用于癌症早期诊断和危险度评价的DNA甲基化指标及其应用的制作方法
本发明涉及一种用于受试者中癌症诊断和/或患癌危险度评价的方法及其试剂盒。
背景技术:
卵巢癌是全球范围内最致命的妇科肿瘤(1)((1)为参考文献编号,以下同)。高致死率主要包含两个原因:缺乏早期检测手段和化疗耐药性(2)。虽然初始化疗对卵巢癌病人具有效果,但是大约25%的病人在六个月之内就会产生化疗耐药性(3)。对肿瘤组织的深入研究发现在卵巢癌发生和进展中存在多样的基因和表观遗传的改变。发现有效的早期检测标志物,尤其在血液中,对改善罹患卵巢癌的预后是迫切需要的。
众所周知,肿瘤细胞溶解释放dna片段(cfdna,游离dna)以及mirna和细胞因子等其他分子到循环系统中(4)。循环系统中肿瘤相关cfdnas的突变是基于晚期肿瘤中已知的突变。原发部位的肿瘤释放到血液中的cfdna是有限的,所以突变检测来检测早期癌症是具有挑战性。另外,早期癌症的驱动事件本质上可能是表观遗传,可能无法基于基因突变进行检测。
最近检查点免疫疗法的成功使我们重新认识到癌症是一项系统性的疾病,可以被免疫系统直接影响(5)。最近的证据显示特殊类型的免疫细胞在卵巢癌细胞增殖和转移中扮演着重要的角色(6-8)。所以,免疫细胞或许能作为窥探肿瘤发病机理和生理平衡的窗口。与免疫系统相关的事件或许可以作为肿瘤理想的标志物,尤其是在早期阶段。可以想象的是,肿瘤细胞的基因或表观遗传改变将会激发免疫监视,并且免疫细胞将会进行表观遗传重新编程来抵抗肿瘤细胞的出现。
最近,外周血的dna甲基化谱已经发现与几种癌症如乳腺癌,肺癌,胃癌,卵巢癌和胰腺具有关联性(9-15)。先前的对卵巢癌风险的基于血液dna甲基化的全表观基因组关联研究,是在一个包含27,000cpgs的低密度平台上实施的(12,16-18)。所以,我们着手现在的研究使用高密度平台去探索白细胞dna甲基化在上皮源性卵巢癌中的潜在价值。
技术实现要素:
本发明人在中国上皮源性卵巢癌病人中实施了一项基于全表观基因组关联性研究的两阶段病例对照研究。在探索阶段,全基因组甲基化实验筛选了450,000个cpg位点,并且对于有显著差异的甲基化cpg位点在验证阶段又进行了验证。
因此,本发明提供以下各项:
1.一种用于受试者中癌症诊断和/或患癌危险度评价的方法,所述方法包括以下步骤:
a)确定在来自受试者的样品中和在来自健康对照组的样品中在至少一个选自由以下组成的组的dna甲基化位点处的dna甲基化状态:
cg11792281,cg08450091,cg14559409,cg13888226,cg17383024,cg09182724,cg22639787,cg07786355,cg04983687,cg21933078,cg26097381,cg07930620,cg05526438,cg22119466,cg01381374,cg02488385,cg06415087,cg00832928,cg19138325,cg09770068,cg14645856,cg10408178,cg16409562,cg05091585,cg09712234,cg25607383,cg21581312,cg20430870,cg06749803,cg21166544,cg19716090,cg12946518,cg20391833,cg17597901,cg06023279,cg24816464,cg15837838,cg15613100,cg07455406,cg09249800,cg24908166,cg22972055,cg03002688,cg26548134,cg15591386,cg10001590,cg26772894,cg12397924,cg06784563,cg00414077,cg26366833,cg16898066,cg00066854,cg20956594,cg15125566,cg05721476,cg00207226,cg23327334,cg03181118,cg25784280,cg15050398,cg17833106,cg01535567,cg20807852,cg20595268,cg07588614,cg24059119,cg18275732,cg12717729,cg08365438,cg12104982,cg01286319,cg04515608,cg22534374,cg14380045,cg16962115,cg22441770,cg12049550,cg02756683,cg01950479,cg26313233,cg13223682,cg14023999,cg23045991,cg12643917,cg16541275,cg22686939,cg21990144,cg11937033,cg02659854,cg17863551,cg27376514,cg20513976,cg27109284,cg11453546,和cg22211672;和
b)将来自受试者的样品中在所述dna甲基化位点处的dna甲基化状态与来自健康对照组的样品中在对应的所述dna甲基化位点处的dna甲基化状态进行比较,如果上述两者存在甲基化状态的显著差异,则表明所述受试者患有癌症或处于患癌的危险中。
2.根据以上1所述的方法,其中所述dna甲基化位点包括至少一个选自由以下组成的组的dna甲基化位点:cg09249800,cg12717729,cg12049550,cg22441770,cg17833106,cg09182724,cg22534374,cg16962115,cg08450091,cg03002688,cg00207226,cg23327334,cg25607383,cg22972055,cg06415087,cg11937033,cg14380045,cg13888226,cg08365438,cg20430870,cg14559409,cg02756683,cg00066854,cg04515608,cg21933078,cg19138325,cg20956594,cg15125566,cg16409562,cg22211672,cg14023999,cg01535567,cg26772894,cg27376514,cg16541275cg06784563,cg01286319,cg12104982,cg24059119,cg17863551,cg00414077,cg22639787,cg02488385,cg11453546,cg19716090,和cg01950479;
更优选选自由以下组成的组:
cg00207226,cg01535567,cg19716090,cg20956594,cg22534374,和cg22639787;或选自由以下组成的组:cg06784563,cg16541275,cg00207226,和cg00066854;或为cg00207226或cg22441770。
3.根据以上1或2所述的方法,其中所述受试者为哺乳动物,优选为人,更优选为亚洲人,更优选为中国人,最优选为汉族人;和/或所述癌症为早期、中期或晚期癌症,例如实体瘤,优选选自乳腺癌、肺癌、胃癌、卵巢癌和胰腺癌等,更优选卵巢癌,例如早期卵巢癌或晚期卵巢癌,或选自浆液性卵巢癌、子宫内膜样卵巢癌和粘液性卵巢癌。
4.根据以上1-3中任一项所述的方法,其中所述受试者和所述健康对照组的样品为基因组dna,优选外周血、尿液、唾液或毛发的基因组dna,最优选为外周血(白细胞)的基因组dna。
5.根据以上1-4中任一项所述的方法,其中所述甲基化状态包括高甲基化(甲基化)和低甲基化(去甲基化)。
6.用于受试者中癌症诊断和/或患癌危险度评价的试剂盒,所述试剂盒包括:
(a)用于确定在来自受试者的样品中和在来自健康对照组的样品中在至少一个选自由以下组成的组的dna甲基化位点处的dna甲基化状态的试剂或反应体系:
cg11792281,cg08450091,cg14559409,cg13888226,cg17383024,cg09182724,cg22639787,cg07786355,cg04983687,cg21933078,cg26097381,cg07930620,cg05526438,cg22119466,cg01381374,cg02488385,cg06415087,cg00832928,cg19138325,cg09770068,cg14645856,cg10408178,cg16409562,cg05091585,cg09712234,cg25607383,cg21581312,cg20430870,cg06749803,cg21166544,cg19716090,cg12946518,cg20391833,cg17597901,cg06023279,cg24816464,cg15837838,cg15613100,cg07455406,cg09249800,cg24908166,cg22972055,cg03002688,cg26548134,cg15591386,cg10001590,cg26772894,cg12397924,cg06784563,cg00414077,cg26366833,cg16898066,cg00066854,cg20956594,cg15125566,cg05721476,cg00207226,cg23327334,cg03181118,cg25784280,cg15050398,cg17833106,cg01535567,cg20807852,cg20595268,cg07588614,cg24059119,cg18275732,cg12717729,cg08365438,cg12104982,cg01286319,cg04515608,cg22534374,cg14380045,cg16962115,cg22441770,cg12049550,cg02756683,cg01950479,cg26313233,cg13223682,cg14023999,cg23045991,cg12643917,cg16541275,cg22686939,cg21990144,cg11937033,cg02659854,cg17863551,cg27376514,cg20513976,cg27109284,cg11453546,和cg22211672;和任选地
(b)说明书,该说明书描述了将来自受试者的样品中在所述dna甲基化位点处的dna甲基化状态与来自健康对照组的样品中在对应的所述dna甲基化位点处的dna甲基化状态进行比较,如果上述两者存在甲基化状态的显著差异,则表明所述受试者患有癌症或处于患癌的危险中。
7.根据以上6所述的试剂盒,其中所述dna甲基化位点包括至少一个选自由以下组成的组的dna甲基化位点:
cg09249800,cg12717729,cg12049550,cg22441770,cg17833106,cg09182724,cg22534374,cg16962115,cg08450091,cg03002688,cg00207226,cg23327334,cg25607383,cg22972055,cg06415087,cg11937033,cg14380045,cg13888226,cg08365438,cg20430870,cg14559409,cg02756683,cg00066854,cg04515608,cg21933078,cg19138325,cg20956594,cg15125566,cg16409562,cg22211672,cg14023999,cg01535567,cg26772894,cg27376514,cg16541275cg06784563,cg01286319,cg12104982,cg24059119,cg17863551,cg00414077,cg22639787,cg02488385,cg11453546,cg19716090,和cg01950479;
更优选选自由以下组成的组:
cg00207226,cg01535567,cg19716090,cg20956594,cg22534374,和cg22639787;或选自由以下组成的组:cg06784563,cg16541275,cg00207226,和cg00066854;或为cg00207226或cg22441770。
8.根据以上6或7所述的试剂盒,其中所述受试者为哺乳动物,优选为人,更优选为亚洲人,更优选为中国人,最优选为汉族人;和/或所述癌症为早期、中期或晚期癌症,例如实体瘤,优选选自乳腺癌、肺癌、胃癌、卵巢癌和胰腺癌等,更优选卵巢癌,例如早期卵巢癌或晚期卵巢癌,或选自浆液性卵巢癌、子宫内膜样卵巢癌和粘液性卵巢癌。
9.根据以上6-8中任一项所述的试剂盒,其中所述受试者和所述健康对照组的样品为基因组dna,优选来自外周血、尿液、唾液或毛发的基因组dna,最优选为来自外周血(白细胞)的基因组dna。
10.根据以上6-9中任一项所述的试剂盒,其中所述试剂或反应体系是用于以下任一种方法来确定dna甲基化状态(高甲基化或低甲基化):
1)甲基化特异性pcr;
2)甲基化特异性限制性内切酶消化;
3)亚硫酸氢盐dna测序;
4)甲基化敏感性单核苷酸引物延伸;
5)限制酶界标基因组扫描;
6)差异性甲基化杂交;
7)基于芯片的甲基化图谱分析;和
8)碱基特异性切割/质谱分析。
附图说明
图1.甲基化生物标志物探索和验证的研究流程图;
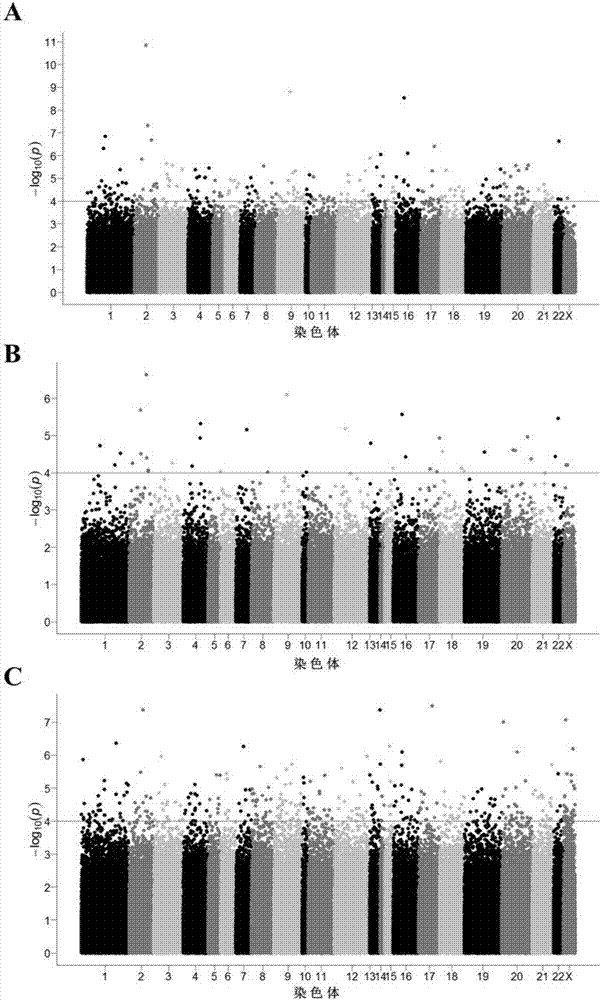
图2.上皮源性卵巢癌全表观基因组关联性研究。曼哈顿图展示了全表观基因组关联性研究的所有病例和对照分析结果(a),早期病例和对照的分析结果(b),晚期病例和对照结果(c),红色长横线代表p<1.0x10-4;
图3.验证阶段具有显著差异的甲基化位点。热点图展示了dna甲基化差异性分析结果,包括所有病例和对照分析结果(总),早期病例和对照(早期),晚期病例和对照(晚期),浆液性卵巢癌和对照(浆液性),子宫内膜样卵巢癌和对照(子宫内膜样),粘液性卵巢癌和对照(粘液性)。差异性甲基化位点的排序是根据所有病例和对照(总)的错误发现率;
图4.6个显著差异甲基化位点的受试者工作特征曲线分析。(a)所有病例和对照(总),早期病例和对照(早期),晚期病例和对照(晚期)的受试者工作特征曲线。(b)浆液性卵巢癌和对照(浆液性),子宫内膜样卵巢癌和对照(子宫内膜样),粘液性卵巢癌和对照(粘液性)的受试者工作特征曲线;
图5.基因本体(go)和差异性甲基化位点的通路分析。(a)文氏图展示了表达差异甲基化位点的重叠部分。(b)与46个表达差异甲基化位点明显相关的go注释(生物进程)。纵轴代表了go分类,横轴代表了具有显著性go注释的富集倍数。(c)在go注释通路中包括的基因;
图6.dna甲基化与凝血因子/血小板参数的相关性。热点图中和卵巢癌风险/生存的相关程度是基于-log10(错误发现率)表示的。对于卵巢癌的风险和生存,每一个热点的的颜色代码已经指定了白色/浅红色作为最低风险,深红色作为最高风险。对于凝血因子和血小板,颜色代码范围从蓝色到白色,再到红色,红色表示正相关,蓝色表示负相关。从白色到红色表示正相关性增强,从白色到深蓝色表示负相关性增强。plcr:血小板大细胞比率,pdw:血小板分布宽度,mpv:平均血小板体积,dd:d-二聚体,plt:血小板计数,pct:血小板比积,fbg:血浆纤维蛋白原,pt:凝血酶原时间,at:抗凝血酶iii;
图7.实施例中40个在所有病例和对照间显著差异甲基化位点的受试者工作特征曲线;
图8.实施例中24个在早期病例和对照间显著差异甲基化位点的受试者工作特征曲线;
图9.实施例中39个在晚期病例和对照间显著差异甲基化位点的受试者工作特征曲线;
图10.实施例中32个在浆液性卵巢癌病例和对照间显著差异甲基化位点的受试者工作特征曲线;
图11.实施例中34个在子宫内膜样卵巢癌病例和对照间显著差异甲基化位点的受试者工作特征曲线;
图12.实施例中11个在粘液性卵巢癌病例和对照间显著差异甲基化位点的受试者工作特征曲线;和
图13.dna甲基化与凝血因子/血小板参数的相关性。热点图中和卵巢癌风险/生存的相关程度是基于-log10(错误发现率)表示的。对于卵巢癌的风险和生存,每一个热点的的颜色代码已经指定了白色/浅红色作为最低风险,深红色作为最高风险。对于凝血因子和血小板,颜色代码范围从蓝色到白色,再到红色,红色表示正相关,蓝色表示负相关。从白色到红色表示正相关性增强,从白色到深蓝色表示负相关性增强。
具体实施方式
实体瘤正逐渐被视为一种系统性的疾病,可以通过dna,rna,蛋白质和血液中代谢产物的变化来识别。虽然许多研究已经报告了循环系统中的基因突变事件,但是很少有研究专注于循环系统中表观遗传dna甲基化标志物。为了识别卵巢癌外周血dna甲基化标志物,本研究实施了一项两个阶段的全表观基因组关联研究。在第一阶段,我们检测了24例上皮源性卵巢癌病例和24例年龄匹配的健康对照的外周血细胞中的485,000多个dna甲基化位点并筛选出96个具有显著差异性的cpg位点。在第二阶段,我们使用illumina’scustomveracode甲基化检测方法在206例上皮源性卵巢癌和205例对照中进行了验证,并确定了46个cpg位点。我们使用46个cpg位点建立预测模型,最终受试者工作特征曲线显示使用其中6个cpg位点所建立的模型的预测准确率为77.3%(95%可信区间:72.9%-81.8%)。我们通过对46个cpg位点的相关基因进行通路分析,发现了免疫系统相关基因的富集,包括lyst(cg16962115,fdr=1.24e-04),cadm1(cg21933078,fdr=1.22e-02),nfatc1(cg06784563,fdr=1.46e-02)。此外,外周血dna甲基化水平和血小板参数/凝血因子水平具有相关性。这项研究揭示了一组表观遗传液体活检标志物与病人整体免疫情况及血小板参数/凝血系统等密切相关,并可以用来检测所有上皮源性卵巢癌的分期和亚型。
因此,在一个方面,本发明提供了一种用于受试者中癌症诊断和/或患癌危险度评价的方法,所述方法包括以下步骤:a)确定在来自受试者的样品中和在来自健康对照组的样品中在至少一个(1至96个,例如,1个、2个、3个、4个、5个、6个、7个、8个、9个、10个、11个、12个、13个、14个、15个、16个、17个、18个、19个、20个、21个、22个、23个、24个、25个、26个、27个、28个、29个、30个、31个、32个、33个、34个、35个、36个、37个、38个、39个、40个、41个、42个、43个、44个、45个、46个、47个、48个、49个、50个、51个、52个、53个、54个、55个、56个、57个、58个、59个、60个、61个、62个、63个、64个、65个、66个、67个、68个、69个、70个、71个、72个、73个、74个、75个、76个、77个、78个、79个、80个、81个、82个、83个、84个、85个、86个、87个、88个、89个、90个、91个、92个、93个、94个、95个、96个)选自由以下组成的组的dna甲基化位点处的dna甲基化状态:cg11792281,cg08450091,cg14559409,cg13888226,cg17383024,cg09182724,cg22639787,cg07786355,cg04983687,cg21933078,cg26097381,cg07930620,cg05526438,cg22119466,cg01381374,cg02488385,cg06415087,cg00832928,cg19138325,cg09770068,cg14645856,cg10408178,cg16409562,cg05091585,cg09712234,cg25607383,cg21581312,cg20430870,cg06749803,cg21166544,cg19716090,cg12946518,cg20391833,cg17597901,cg06023279,cg24816464,cg15837838,cg15613100,cg07455406,cg09249800,cg24908166,cg22972055,cg03002688,cg26548134,cg15591386,cg10001590,cg26772894,cg12397924,cg06784563,cg00414077,cg26366833,cg16898066,cg00066854,cg20956594,cg15125566,cg05721476,cg00207226,cg23327334,cg03181118,cg25784280,cg15050398,cg17833106,cg01535567,cg20807852,cg20595268,cg07588614,cg24059119,cg18275732,cg12717729,cg08365438,cg12104982,cg01286319,cg04515608,cg22534374,cg14380045,cg16962115,cg22441770,cg12049550,cg02756683,cg01950479,cg26313233,cg13223682,cg14023999,cg23045991,cg12643917,cg16541275,cg22686939,cg21990144,cg11937033,cg02659854,cg17863551,cg27376514,cg20513976,cg27109284,cg11453546,cg22211672;和b)将来自受试者的样品中在一个或多个所述dna甲基化位点处的dna甲基化状态与来自健康对照组的样品中在对应的一个或多个所述dna甲基化位点处的dna甲基化状态进行比较,如果上述两者存在甲基化状态的显著差异,则表明所述受试者患有癌症或处于患癌的危险中。
在本发明的一个优选实施方案中,所述dna甲基化位点包括至少一个(1至46个,例如,1个、2个、3个、4个、5个、6个、7个、8个、9个、10个、11个、12个、13个、14个、15个、16个、17个、18个、19个、20个、21个、22个、23个、24个、25个、26个、27个、28个、29个、30个、31个、32个、33个、34个、35个、36个、37个、38个、39个、40个、41个、42个、43个、44个、45个、46个)选自由以下组成的组的dna甲基化位点:cg09249800,cg12717729,cg12049550,cg22441770,cg17833106,cg09182724,cg22534374,cg16962115,cg08450091,cg03002688,cg00207226,cg23327334,cg25607383,cg22972055,cg06415087,cg11937033,cg14380045,cg13888226,cg08365438,cg20430870,cg14559409,cg02756683,cg00066854,cg04515608,cg21933078,cg19138325,cg20956594,cg15125566,cg16409562,cg22211672,cg14023999,cg01535567,cg26772894,cg27376514,cg16541275cg06784563,cg01286319,cg12104982,cg24059119,cg17863551,cg00414077,cg22639787,cg02488385,cg11453546,cg19716090,和cg01950479,这46个cpg位点的甲基化水平在所有类型的卵巢癌中相对于健康受试者对照都具有显著的差异性。
在本发明的一个优选实施方案中,所述dna甲基化位点还优选选自由以下组成的组:cg00207226,cg01535567,cg19716090,cg20956594,cg22534374,和cg22639787。这6个cpg位点在本发明中被用于构建卵巢癌预测模型,成功地预测了卵巢癌风险,并且在卵巢癌的不同分期和组织学亚型的分层分析时诊断效果较好。因此,在本发明的方法中,优选检测1-6个(例如1个、2个、3个、4个、5个或6个)选自以下的dna甲基化位点:cg00207226,cg01535567,cg19716090,cg20956594,cg22534374,和cg22639787。该检测还可以同时检测一个或多个以上列出的其他任何dna甲基化位点。
在本发明的一个优选实施方案中,所述dna甲基化位点选自由以下组成的组:cg06784563,cg16541275,cg00207226,和cg00066854。本发明人首次发现,这4个cpg位点的dna甲基化水平的变化与5个血小板参数(plt:血小板计数,pct:血小板比积,mpv:平均血小板体积,pdw:血小板分布宽度,plcr:大血小板比例)相关。可以推测,这些cpg位点的dna甲基化状态相对于健康对照的变化可以用于确定受试者的血小板状态变化,和/或用于诊断/确定被检测受试者的与血小板相关的疾病(例如炎性疾病)。因此,在本发明的方法中,优选检测1-4个(例如1个、2个、3个或4个)选自以下的dna甲基化位点:cg06784563,cg16541275,cg00207226,和cg00066854。该检测还可以同时检测一个或多个以上列出的其他任何dna甲基化位点。特别优选以上dna甲基化位点为cg00207226,因为该位点的dna甲基化状态相对于健康对照的变化不仅可以用于诊断卵巢癌和/或患卵巢癌危险度评价,而且能够用于诊断/确定被检测受试者的与血小板相关的疾病或病症。
在本发明的一个优选实施方案中,所述dna甲基化位点为cg22441770。本发明人首次发现,该cpg位点的dna甲基化水平的变化与凝血因子参数pt(凝血酶原时间)和atiii(抗凝血酶iii)有相关性。因此,该位点的dna甲基化状态相对于健康对照的变化不仅可以用于诊断卵巢癌和/或患卵巢癌危险度评价,而且能够用于确定凝血因子(特别是与参数pt和atiii)的状态变化,和/或用于诊断/确定与凝血因子(特别是与参数pt和atiii)相关的疾病或病症,例如凝血障碍。
在本发明中,受试者可以是任意人或非人哺乳动物。非人哺乳动物的实例包括灵长类、家畜动物(如马、牛、绵羊、猪、驴)、实验室测试动物(如小鼠、大鼠、兔、豚鼠)、宠物(如狗、猫)和俘获的野生动物(如鹿、狐狸)。优选地,哺乳动物是人。对于人的种群没有特殊限制,但是优选为亚洲人,更优选为中国人,最优选为汉族人。
在本发明中,术语“癌症”不仅包括起源于上皮组织的恶性肿瘤,而且包括起源于间叶组织的恶性肿瘤,还包括其他恶性肿瘤如肾母细胞瘤、恶性畸胎瘤等。对于癌症的类型,没有特别限制,但是在本发明中优选为实体瘤,包括但不限于乳腺癌、肺癌、胃癌、卵巢癌、胰腺癌、肝癌、淋巴瘤、血管瘤等。
在本发明中,术语“诊断”是指判断受试者是否患有某种疾病或其症状、体征等。在本发明中,用于确定本发明dna甲基化位点处的dna甲基化状态相对于健康对照的改变的方法、试剂或反应体系能够单独地或者与一种或多种其他任何癌症诊断方法/标志物组合用于癌症的诊断。在后者的情况下,在某些类型的癌症诊断中,本发明的方法可以是一种诊断癌症的辅助方法。
在本发明中,术语“患癌危险度评价”是指预测受试者患有癌症的可能性或风险的高低程度。
在本发明中,术语“样品”是指含有基因组dna的任何生物材料或基因组dna本身,包括但不限于细胞材料、生物流体(如血液)、粪便、组织活检标本、外科手术标本或引入动物体内并随后移除的流体(例如,从灌肠洗液中重新获得的溶液)或从其中提取的基因组dna。根据本发明方法测试的生物样品可直接测试或者可能在测试之前需要一些形式的处理。例如,活检或外科手术样品可能在测试之前需要均化,或者其可能需要切片以原位测试各个基因的定性表达水平。或者,细胞样品可能在测试之前需要透化。此外,就生物样品不是液体形式(如果需要测试这样的形式)来说,其可能需要添加试剂(例如缓冲剂)以使样品流通。优选地,所述样品为基因组dna,优选来自外周血、尿液、唾液或毛发的基因组dna,最优选为来自外周血(白细胞)的基因组dna。受试者和对照组的样品通常是同种类型的样品。
在本发明中,术语“健康对照组”是指不患有癌症的健康受试者。在本发明的一个实施方案中,该健康受试者没有先前在临床上被诊断患有癌症并且与被检测的受试者在年龄频数上等方面相匹配。健康对照组的确定和选择对于医学领域的技术人员而言是常规的。
在本发明中,术语“dna甲基化”具有在本领域公知的含义。在甲基转移酶的催化下,dna的cg两个核苷酸的胞嘧啶被选择性地添加甲基,形成5-甲基胞嘧啶,这常见于基因的5′-cg-3′序列。大多数脊椎动物基因组dna都有少量的甲基化胞嘧啶,主要集中在基因5′端的非编码区,并成簇存在。dna的甲基化可引起基因的失活,dna甲基化导致某些区域dna构象变化,从而影响了蛋白质与dna的相互作用,导致基因失活。另外,序列特异性甲基化结合蛋白(mbd/mecp)可与启动子区的甲基化cpg结合,阻止转录因子与启动子作用,从而阻抑基因转录过程。所有cpg的70%至80%是甲基化的。cpg可集中成簇,称为“cpg岛”。
在本发明中,术语“dna甲基化位点”是指基因组dna上cpg的位点。具体地,在本发明中,dna甲基化位点的胞嘧啶残基用cgxxxxxxxx表示,其为illunima甲基化芯片的位点号,其对于本领域技术人员而言是清楚的。对于本发明涉及的dna甲基化位点,说明书附图3中还列出了这些位点所在的染色体号和相关基因信息。由于dna的双链互补结构,所以本发明的dna甲基化位点还包括与上述用cgxxxxxxxx表示的胞嘧啶残基对应的在互补dna链上第n+1位处对应的胞嘧啶的甲基化位点。因此,当提及cgxxxxxxxx表示的胞嘧啶残基的dna甲基化位点(n位)时,同时还包括在互补dna链上第n+1位处对应的胞嘧啶的甲基化位点。
在本发明中,术语“dna甲基化状态”应理解为提及dna区域中一个特定核苷酸或多个核苷酸中甲基化的存在、不存在和/或量。在本发明的一个实施方案中,所述dna甲基化位点处的甲基化状态包括但不限于高甲基化和低甲基化。在本发明中,“高甲基化”和“甲基化”可交换使用,意指在某个核苷酸处的cpg存在和高度甲基化;而“低甲基化”和“去甲基化”可交换使用,意指在某个核苷酸处不存在cpg和低度甲基化。如上所述,确定一个以cgxxxxxxxx表示的dna甲基化位点(n位)的胞嘧啶残基的dna甲基化状态时,同时还包括确定在互补dna链上第n+1位处对应的胞嘧啶的甲基化位点的甲基化状态。
用于评估dna甲基化状态的方法是本领域技术人员已知的,包括但不限于以下方法:1)甲基化特异性pcr;2)甲基化特异性限制性内切酶消化;3)亚硫酸氢盐dna测序;4)甲基化敏感性单核苷酸引物延伸;5)限制酶界标基因组扫描;6)差异性甲基化杂交;7)基于芯片的甲基化图谱分析(实例包括,beadarray平台技术(illumina,usa));和8)碱基特异性切割/质谱分析(sequenom,usa)。用于这些方法的试剂或反应体系是本领域普通技术人员熟知的,并且通常以可商购的试剂盒的形式提供。因此,本发明还提供用于评估dna甲基化状态的试剂或反应体系在本发明的方法或制备本发明的试剂盒中的应用。
在本发明中,术语“存在甲基化状态的显著差异”是指与对照组(例如健康对照组)的样品对应的dna甲基化位点处的dna甲基化状态(高甲基化/低甲基化)相比,来自受试者的样品中在所述dna甲基化位点处的dna甲基化状态(高甲基化/低甲基化)存在统计学上的显著性差异,例如p<0.05,优选p<0.01。在本发明的一个特别优选的实施方案中,p<1.0x10-4。
实施例
下文将参考实施例详细描述本发明,所述实施例仅是意图举例说明本发明,而不是意图限制本发明的范围。本发明的范围由后附的权利要求具体限定。
材料和方法
研究人群
卵巢癌病例是2007-2013年天津医科大学肿瘤医院经过组织学诊断确诊的年龄25-85岁原始上皮源性卵巢癌女性(全部为中国汉族人)。病例确诊前6个月有癌症治疗史或者输血史排除在外。病例组的血样在手术室收集,血常规和血凝测试得到血细胞计数、凝血因子值,并且这些都在手术前一周内进行。对照组是天津市社区居民里无肿瘤的受试者(全部为中国汉族人),其先前没有被诊断为癌症并与病例组在年龄频数(5年时间间隔)上匹配。每个受试者完成问卷调查(包括人口统计信息,健康状况,生活方式和饮食习惯)并提供研究用的血液样本。这项研究通过了天津医科大学肿瘤医院和癌症研究所伦理审查委员会的批准,并获得用临床样本进行研究的知情同意书。
在探索阶段,24例上皮性卵巢癌病例组(12例figo分期i-ii和12例figo分期iii-iv)和年龄分布匹配的对照组被随机挑选出来进行全基因组甲基化分析。在验证阶段,206例上皮性卵巢癌病例组和205例年龄分布匹配的对照组用来验证i阶段挑选出来的cpg位点。
dna提取和亚硫酸氢盐转化
收集的样本基因组dna是通过试剂盒qiazendnabloodminikit(qiazen,valencia,ca,usa)从白细胞中提取的,在亚硫酸氢盐处理前储存在-80℃。根据试剂亚硫酸氢盐转化试剂盒ez-96dnamethylation-goldkit(d5008)(zymoresearch,orange,ca)的手册处理基因组dna,其中未甲基化的胞嘧啶转化成尿嘧啶,而甲基化的胞嘧啶则保持原状。亚硫酸氢盐转化的dna储存于-20℃,在转化后一周内使用。
全基因组甲基化表达谱
在探索阶段,全基因组甲基化表达谱是用illuminainfiniumhumanmethylation450(sandiego,ca)芯片处理的。我们用illuminabeadarraytmreader得到芯片图像,用illumina’sgenomestudio软件读取数据。所有样本都经过了质量控制测试,包括染色控制,延伸控制,目标移除控制,杂交控制,亚硫酸氢盐转换控制,特异性控制,阴性控制,非多态性结合控制,每个孔用阴性对照珠进行背景标化。每个cpg位点的dna甲基化值(β值)范围是从0(未甲基化)到1(甲基化),代表减去背景强度的甲基化等位基因信号到总荧光信号的相对比值。
验证筛选的cpg位点
在验证阶段,illunima’scustomveracode甲基化分析法用来确证96个筛选出来的cpg位点。illunima’sbeadxpressreader和illumina’sgenomestudio各自用来扫描芯片和读取数据。分析中实施了包括9项内部控制(特异性等位基因延伸控制,pcr一致性控制,空位延伸控制,性别控制,首次杂交控制,二次杂交控制,阴性控制,污染检测控制)的质控测试。
统计分析:
为比较病例组和对照组的人口统计学特征,对分类变量和连续性变量分别进行卡方检验和独立样本t检验。在初始探索阶段,使用独立样本t检验比较病例组和对照组之间每个cpg位点的中位数β值的差异,并按照上皮性卵巢癌分期(i-ii,iii-iv)进行分层分析。然后我们从全基因组数据中选出96个最有显著差异的甲基化cpg位点进行验证,条件如下:(a)p<1.0×10-4;(b)与对照组相比,i-ii期与iii-iv期上皮性卵巢癌病例之间的变化趋势一致;(c)可以使用illuminacustomveracode甲基化试验测定基因分型。在验证阶段,使用独立样本t检验来评估研究组之间甲基化值的差异。进行pearson相关分析,以评估dna甲基化与血细胞计数/凝血因子的相关性。为了纠正多次测试的误差,利用benjamini和hochberg校正方法计算错误发现率(fdr),fdr<0.05时有意义。
另外,为了评估验证阶段具有差异性的甲基化cpg位点的分类性能,我们利用logistic回归模型建立了受试者工作特征曲线(roc)的预测模型。使用逐步和手动选择的方法选择dna甲基化标志物。
此外,为了确定生物学通路中基因的共同性,我们利用david基因本体工具进行了通路富集分析,该分析用于卵巢癌风险相关的差异甲基化cpg位点附近的基因。所有分析使用sas软件9.3版本(sasinstitute,cary,nc,usa)和r软件3.1.2版本进行。
结果
为了确定卵巢癌外周血白细胞dna甲基化的差异性,我们实施了两阶段的病例-对照全表观基因组关联性研究(ewas,epigenomewideassociationstudy),图1展示了研究设计的流程图。在最开始的探索阶段,全基因组的扫描是在24例上皮性卵巢癌和24例年龄匹配的健康对照中应用illumina’sinfiniumhumanmethylation450k芯片进行的。在病例和对照组,年龄,bmi,吸烟史,癌症家族史和绝经史的差异性均没有统计学意义。所有的病例都是浆液性卵巢癌,12例早期,12例晚期(表1),曼哈顿图展示了全表观基因组分析结果(图2)。在24例上皮性卵巢癌和24例对照组之间有242个cpg位点的甲基化水平显示出明显的统计学差异性(p<1.0×10-4)。当对肿瘤分期进行分层分析,与24例对照组相比,39个cpg位点在12例早期(i&ii期)的上皮性卵巢中具有统计学差异性,375个cpg位点在12例晚期(iii&iv)中具有统计学差异性。我们选择了96个差异最明显的cpg位点在第二期进行了验证(见表2)。
在一个包含206例上皮性卵巢癌和205例对照的独立样本中,我们应用illunima’scustomveracode甲基化检测方法分析了挑选的96个cpg甲基化位点。病例和对照组的年龄,bmi,吸烟史,癌症家族史,绝经史都是一致的。我们发现40个cpg位点和上皮性卵巢癌风险明显相关,其中16个高甲基化,24个低甲基化(见图3,表3)。对肿瘤分期进行分层分析,发现88例早期病例(i&ii期)和对照组相比有24个cpg位点(9个高甲基化和15个低甲基化)甲基化水平具有显著的差异性,115例晚期病例(iii&iv期)和对照相比发现39个cpg位点(21个高甲基化和18个低甲基化)甲基化水平具有显著差异性。本研究也对病例和对照组的甲基化水平进行了组织学分型的分层分析。与205例对照组相比,我们在85例浆液性卵巢癌中找到32个cpg位点,在59例上皮性卵巢癌中找到34个cpg位点,24例粘液性卵巢癌中找到11个cpg位点的甲基化水平具有显著的差异性(图3,表4)。总体来说,我们确定了46个cpg位点的甲基化水平,在所有类型的比较都中有显著的差异性。
单个cpg位点的受试者的代表性工作特征曲线(roc)在图7-12中展示。用受试者工作特征曲线下面积为0.773(0.729-0.818)预测模型筛选出6个cpg位点(cg00207226,cg01535567,cg19716090,cg20956594,cg22534374,cg22639787),表明这6个cpg位点比对照组(77.3%)在卵巢癌风险上有更高的预测能力。这6个cpg位点在不同分期和组织学亚型的分层分析时诊断效果较好:早期肿瘤曲线下面积为0.754(0.693-0.816),晚期肿瘤曲线下面积为0.803(0.754-0.853),浆液性卵巢癌曲线下面积为0.773(0.713-0.833),子宫内膜样卵巢癌曲线下面积为0.799(0.735-0.864),粘液性卵巢癌曲线下面积为0.725(0.609-0.841)(见图4)。
为了确定这46个甲基化cpg位点假定调节的56个基因的共性,我们用david基因本体法进行了通路富集分析。富集分析倍数最高的是白细胞调节的细胞毒性,在这条通路上包括两个基因:lyst(cg16962115,fdr=1.24e-04)和cadm1(cg21933078,fdr=1.22e-02)。大部分的富集分析结果都包含了免疫系统相关的通路;并且我们发现基因nfatc1(cg06784563,fdr=1.46e-02)在reactom通路数据库中也与免疫系统有关(见图5)。另外,我们评估了甲基化位点的差异和血细胞数目/凝血因子之间的相关性(见表5)。在dna甲基化和血细胞数之间,除了血小板数之外其他都没有显著的相关性(图13)。我们发现几个cpg位点和血小板参数及凝血因子具有相关性(图6)。5个血小板参数(plt:血小板计数,pct:血小板比积,mpv:平均血小板体积,pdw:血小板分布宽度,plcr:大血小板比例)和4个凝血因子(pt:凝血酶原时间,d-dimer:d-二聚体,fbg:血纤维蛋白原,atiii:抗凝血酶iii)指标纳入了相关性分析。我们发现四个cpg位点(cg06784563,cg16541275,cg00207226,cg00066854)和所有的血小板参数都有相关性。对于凝血因子,没有一个位点和这四个凝血因子具有相关性。cg22441770位点的dna甲基化水平和pt及atiii有相关性,并且有11个cpg位点和d-二聚体有相关性。
讨论
在这项两阶段的病例对照全表观基因组关联性研究中,我们确定了46个与汉族女性卵巢癌风险显著相关的新的cpg甲基化位点。我们使用6个cpg位点还构建了一个相对高精确性的风险预测模型,尤其适用于早期卵巢癌。对46个具有显著差异的甲基化位点基因进行通路分析,发现它们是免疫系统相关通路的一部分。卵巢癌风险相关的差异性甲基化cpg位点和血细胞计数无关,但却发现与血小板参数/凝血因子具有相关性。据我们所知,这是揭示了外周血白细胞dna甲基化与血小板参数/凝血因子的相关性的首次报道。
外周血dna甲基化标志物在几类癌症中都有报道,包括乳腺癌,头颈癌,卵巢癌,胃癌,结直肠癌和胰腺癌等,但其生物学意义尚不清楚(19)。据悉,dna甲基化与生活方式,日常饮食和环境暴露相关(20)。最近,越来越多研究显示dna甲基化与吸烟和bmi具有相关性(21,22)。先前的研究猜测,外周血dna甲基化的改变或许是由于血细胞成分的转换。为揭示这个机制,koestler等人比较了正常人外周血白细胞亚群(包括b细胞,粒细胞,单核细胞,nk细胞,cd4+t细胞,cd8+t细胞和pan-t细胞)的dna甲基化,确定了50个cpg位点在不同白细胞亚群的甲基化水平具有差异性(11)。然而,这50个cpg位点在我们的研究中均不具有统计学意义,这也和之前在乳腺癌中的报道相一致(9)。在我们的研究中,和卵巢癌相关的甲基化位点与白细胞亚群没有关联性。有趣的是,我们发现外周血dna甲基化与血小板参数和凝血因子具有一定的联系。血液中的血小板据报道在参与凝血,免疫反应,炎症和肿瘤进展/转移中起到关键的作用(8,23)。在卵巢癌中,有报道称血小板数目是和上皮源性卵巢癌预后相关(24)并且血小板能直接促进卵巢癌细胞增殖(6)。在卵巢癌发生上皮间质转化时,dna甲基化的主要改变能被tgfβ1诱导(25),并且血小板分泌的tgfβ1能促进肿瘤细胞的上皮间质转化(26)。然而,外周血中dna甲基化和血小板关系的机制尚不清楚,需要进一步的研究。
在本次研究中,我们确定了与卵巢癌风险相关的差异性cpg位点所在或邻近的基因是参与了免疫系统相关的通路,包括白细胞介导的细胞毒性,细胞杀伤,免疫效应过程。其中有三个基因,nfatc1(cg06784563),lyst(cg16962115),和cadm1(cg21933078)在免疫系统相关通路中。cg06784563是在nfatc1的5’端启动子调控区编码激活t细胞的核转录因子。nfat蛋白已经有报道证明在免疫系统发挥重要的功能(27),而且nfat转录因子在包括肿瘤在内的许多通路中有极其重要的作用。在卵巢癌,nfact1在肿瘤组织相对于配对的正常组织显著的过表达,并通过激活erk1/2/p38/mapk信号通路上调c-myc促进体外肿瘤细胞增殖(29)。cg21933078是在11号染色体cadm1上游15kb处,cadm1是一个细胞黏附分子并认为是一个肿瘤抑制基因(30,31)。cadm1低表达可能是因为启动子区甲基化并因此促进了肿瘤细胞增殖/侵袭(30,31)。在卵巢癌,cadm1下调可以作为预后不好的标志物(32)。cadm1通过使肿瘤细胞对免疫监控机制更敏感发挥抑制肿瘤转移的新功能,并且cadm1丢失是肿瘤免疫编辑的关键一步(33)。cg16962115位于1q42.3编码溶酶体转运调节蛋白lyst上游的35kb处。lyst突变与谢迪亚克—东综合征相关(34)。目前未有lyst与卵巢癌或其他癌症相关的报道。需要进一步的研究去探索这个基因与卵巢癌相关的功能。
先前卵巢癌全表观基因组关联研究已经确定了几个外周血差异性甲基化cpg位点(12,16,17)。然而,没有一个达到我们验证时的选择标准(p<1.0×10-4)。以前研究的探索阶段是基于illumina’s27k甲基化芯片,而我们所用的450k芯片的覆盖范围要比以前大的多。我们研究中挑选出验证的96个cpg位点只有一个是包含在27k芯片中。
总得来说,我们已经确定了一组血源性的dna甲基化标志物与上皮源性卵巢癌风险相关。因此,外周血dna甲基化表达谱可以成为卵巢癌风险评估和早期检测的新工具。大型前瞻性队列研究和深入的生物学机制研究需要来验证这些新的生物标志物,并且为精准医学提供卵巢癌表观基因组治疗策略。
参考文献
1.siegel,r.l.,etal.(2016)cancerstatistics,2016.cacancerjclin,66,7-30.
2.jayson,g.c.,etal.(2014)ovariancancer.thelancet,384,1376-1388.
3.miller,d.s.,etal.(2009)phaseiievaluationofpemetrexedinthetreatmentofrecurrentorpersistentplatinum-resistantovarianorprimaryperitonealcarcinoma:astudyofthegynecologiconcologygroup.journalofclinicaloncology,27,2686-2691.
4.salvi,s.,etal.(2016)cell-freednaasadiagnosticmarkerforcancer:currentinsights.oncotargetsther,9,6549-6559.
5.atkins,m.b.,etal.(2016)immunotherapycombinedorsequencedwithtargetedtherapyinthetreatmentofsolidtumors:currentperspectives.jnatlcancerinst,108.
6.cho,m.s.,etal.(2012)plateletsincreasetheproliferationofovariancancercells.blood,120,4869-4872.
7.davis,a.n.,etal.(2014)plateleteffectsonovariancancer.seminarsinoncology,41,378-384.
8.franco,a.t.,etal.(2015)plateletsattheinterfaceofthrombosis,inflammation,andcancer.blood,126,582-8.
9.xu,z.,etal.(2013)epigenome-wideassociationstudyofbreastcancerusingprospectivelycollectedsisterstudysamples.jnatlcancerinst,105,694-700.
10.brennan,k.,etal.(2012)intragenicatmmethylationinperipheralblooddnaasabiomarkerofbreastcancerrisk.cancerres,72,2304-13.
11.koestler,d.c.,etal.(2012)peripheralbloodimmunecellmethylationprofilesareassociatedwithnonhematopoieticcancers.cancerepidemiolbiomarkersprev,21,1293-302.
12.koestler,d.c.,etal.(2014)integrativegenomicanalysisidentifiesepigeneticmarksthatmediategeneticriskforepithelialovariancancer.bmcmedgenomics,7,8.
13.dauksa,a.,etal.(2014)dnamethylationatselectedcpgsitesinperipheralbloodleukocytesispredictiveofgastriccancer.anticancerres,34,5381-8.
14.pedersen,k.s.,etal.(2011)leukocytednamethylationsignaturedifferentiatespancreaticcancerpatientsfromhealthycontrols.plosone,6,e18223.
15.wang,l.,etal.(2010)methylationmarkersforsmallcelllungcancerinperipheralbloodleukocytedna.jthoraconcol,5,778-85.
16.fridley,b.l.,etal.(2014)methylationofleukocytednaandovariancancer:relationshipswithdiseasestatusandoutcome.bmcmedgenomics,7,21.
17.teschendorff,a.e.,etal.(2009)anepigeneticsignatureinperipheralbloodpredictsactiveovariancancer.plosone,4,e8274.
18.winham,s.j.,etal.(2014)genome-wideinvestigationofregionalblood-baseddnamethylationadjustedforcompletebloodcountsimplicatesbnc2inovariancancer.geneticepidemiology,38,457-466.
19.li,l.,etal.(2012)dnamethylationinperipheralblood:apotentialbiomarkerforcancermolecularepidemiology.jepidemiol,22,384-94.
20.noreen,f.,etal.(2014)modulationofage-andcancer-associateddnamethylationchangeinthehealthycolonbyaspirinandlifestyle.jnatlcancerinst,106.
21.breitling,lutzp.,etal.(2011)tobacco-smoking-relateddifferentialdnamethylation:27kdiscoveryandreplication.theamericanjournalofhumangenetics,88,450-457.
22.dick,k.j.,etal.dnamethylationandbody-massindex:agenome-wideanalysis.thelancet,383,1990-1998.
23.leslie,m.(2010)cellbiology.beyondclotting:thepowersofplatelets.science,328,562-4.
24.stone,r.l.,etal.(2012)paraneoplasticthrombocytosisinovariancancer.nengljmed,366,610-8.
25.cardenas,h.,etal.(2014)tgf-βinducesglobalchangesindnamethylationduringtheepithelial-to-mesenchymaltransitioninovariancancercells.epigenetics,9,1461-1472.
26.labelle,m.,etal.(2011)directsignalingbetweenplateletsandcancercellsinducesanepithelial-mesenchymal-liketransitionandpromotesmetastasis.cancercell,20,576-590.
27.müller,m.r.,etal.(2010)nfat,immunityandcancer:atranscriptionfactorcomesofage.natrevimmunol,10,645-656.
28.shou,j.,etal.(2015)nuclearfactorofactivatedtcellsincancerdevelopmentandtreatment.cancerletters,361,174-184.
29.xu,w.,etal.(2016)nfatc1promotescellgrowthandtumorigenesisinovariancancerup-regulatingc-mycthrougherk1/2/p38mapksignalpathway.tumorbiology,37,4493-4500.
30.woo,h.j.,etal.(2015)hypermethylationofthetumor-suppressorcelladhesionmolecule1inhumanpapillomavirus-transformedcervicalcarcinomacells.intjoncol,46,2656-62.
31.wikman,h.,etal.(2014)lossofcadm1expressionisassociatedwithpoorprognosisandbrainmetastasisinbreastcancerpatients.oncotarget,5,3076-87.
32.yang,g.,etal.(2011)loss/down-regulationoftumorsuppressorinlungcancer1expressionisassociatedwithtumorprogressionandisabiomarkerofpoorprognosisinovariancarcinoma.intjgynecolcancer,21,486-93.
33.faraji,f.,etal.(2012)cadm1isametastasissusceptibilitygenethatsuppressesmetastasisbymodifyingtumorinteractionwiththecell-mediatedimmunity.plosgenet,8,e1002926.
34.al-tamemi,s.,etal.(2014)chediak-higashisyndrome:novelmutationofthechs1/lystgenein3omanipatients.jpediatrhematoloncol,36,e248-50.
表1.全表观基因组检测和验证的研究对象基本情况和临床特征
- 还没有人留言评论。精彩留言会获得点赞!