用于将肽呈递在细胞表面上的系统的制作方法
本申请的主题是一种基于四分子交联体(tetraspanins)的融合蛋白质,其中胞外环完全地或部分地通过其他成分的肽序列替代,以及涉及其制备和作为转运工具的应用。公开的融合蛋白质锚定在细胞的膜中并且能够用于与其融合的外源性肽呈递
背景技术:
细胞的结构设计和其生理机能由基因编码。基因由dna构成并且存在于基因组中或染色体外遗传因子上。所述基因表达初级基因产物和次级基因产物,如rna和蛋白质。所述基因产物能够以多样酶进一步修饰的方式出现。
基因产物主要存在于胞质溶胶中。蛋白质也部分地在区室的细胞内膜和外部细胞膜以及必要时还有细胞壁中复得。在真核生物中,蛋白质和rna前体存在于细胞核中。蛋白质在胞质溶胶中的细胞内膜中分选并且分布在细胞区室上,但也能够通过分泌从细胞中输出。此外,源自内质网(er)的输出蛋白质能够在转运期间在高尔基体中糖基化。
一种特定的蛋白质种类、即膜结合蛋白质被主动地引导到细胞膜上并且在那相对于膜竖直地锚定。但是,水平方向与此相反地,所述膜结合蛋白质由于膜流动性而能够灵活地在膜内部迁移。膜蛋白质与其他异源性膜蛋白质在复合物中部分地复合。因此,多达80%的人类蛋白质在伴生的蛋白质复合体中作用1。一些种类在膜中相互协调地相互作用并且在那形成稳定的结构。
经由膜蛋白质控制大量信号过程,所述信号过程能够使细胞与其环境相互作用。蛋白质经由细胞膜的膜转运连同向外部的分泌过程同样是可行的。
利用细胞上的免疫系统mhc-膜蛋白质来呈递表位,以便识别细胞中潜在的入侵的病原体。该生理机能过程在细胞中大多非常良好地建立,并且所涉及的蛋白质针对该功能高度特定地演化,也就是说,大多非常特定地针对特定的功能。因此,能够期望:附加的、人造的氨基酸序列干扰该功能。
四分子交联体属于哺乳动物中具有33个成员的蛋白质超级家族。四分子交联体的主要特征是其4个跨膜结构域tm1-4,这也产生命名。四分子交联体还包含小的细胞外蛋白质环和大的细胞外蛋白质环(小胞外环和大胞外环(smallandlargeextracellularloop))(也称作sel和lel)。lel包含高度保守的ccg氨基酸模体。此外,包含细胞内的蛋白质环(小胞内环(smallintracellularloop,sil))。n端和c端是细胞内定位34-36。四分子交联体通过四分子交联体中富集的区域(四跨膜蛋白质富集的微结构域(tetraspaninenrichedmicrodomains,tems))中的横向的蛋白质-蛋白质相互作用(ppls)来组织37,38。
需要尤其适合的载体蛋白质(载体),以便将异源性成分的表位和外源性蛋白质以有意的方式例如转运到哺乳动物、例如人类细胞的表面上并且在那呈递。所述载体应当具有足够的电动势并且能够:利用膜电位,以便以经过膜至细胞表面的易位的高的效率确保蛋白质转运2-8。过去已知所谓的“展示”系统,一般地是膜结合蛋白质,,所述膜结合蛋白质通过其在基因方面修饰的方式能够用作用于呈递外源性蛋白质的载体蛋白质。最著名的蛋白质展示系统之一是噬菌体展示。其应用于将蛋白质组分呈递在细菌系统中的丝状噬菌体的表面上9-12。然而,在革兰氏阳性细菌以及革兰氏阴性细菌的情况下,过去也能够在不应用噬菌体展示的情况下成功地呈递最外部的膜上呈递异源性蛋白质。在此,利用细菌细胞内部的不同的转运机制,以便将蛋白质引导至细菌细胞的最外部的膜13-20。展示系统的进一步发展向真核生物系统、如酵母和昆虫细胞移动,以便确保在重组蛋白质上修饰,例如n糖基化,以及蛋白质折叠21-29。也已经开发第一展示系统用于在哺乳动物系统中使用30-33。
技术实现要素:
本发明的目的是基于现有技术提供将任意异源性肽锚定在细胞质膜中并且呈递在细胞膜上的新的可行性。令人惊讶地,,现在已发现:四分子交联体卓越地适合作为工具并且对于与其融合的外源性肽具有高效的转运能力以进行锚定并且呈递在细胞的表面上。所述四分子交联体能够将外源性肽经由内质网和高尔基体高效地引导至哺乳动物的细胞质膜。
四分子交联体家族的蛋白质在野生型中的跨膜结构域tm3和tm4之间具有蛋白质环(大胞外环(largeextracellularloop),lel)。令人惊讶地,在本发明的范围中能够发现:这些胞外环能够完全地或部分地通过其他成分的肽序列替代,进而可行的是:嵌入的异源性肽经由四分子交联体-载体蛋白质转运穿过膜并且在膜中锚定。
本发明基于用于模块化的蛋白质表达系统中的灵活的且可变的序列调节的统一的分子结构的简单性结合经由细胞膜的特定的转运工具和其在那锚定的功能。
提供的载系统统借助更简单的克隆策略实现灵活地交换或补充蛋白质组分(例如,添加单个表位并且还有编制完整的蛋白质数据库)。
在根据本发明的四分子交联体融合构建体(暂时地或稳定地整合到基因组中)表达之后,融合蛋白质在膜中的定位几乎是定量的。四分子交联体蛋白质非常有效地转移到膜上。这在已经已知的这种形式的系统(例如egfr受体)中远未实现。
根据本发明的四分子交联体融合蛋白质的表达相对快速地进行(通常已经整整一夜),反之,在其他转运系统中的表达通常在两天之内完成。
另一方面,与具有七个跨膜结构域的已知的蛋白质对比,融合构造的稳定性和其寿命也相对更好,因为未发现胞质溶胶的分解产物的积聚(已知gpcr受体重组为是不稳定的)。
借助仅具有四个跨膜结构域的根据本发明的系统能够将一个至多个蛋白质稳定地并且毫无问题地转运到细胞的表面上并且在那例如作为固定配偶子在具有小分子、另外的蛋白质、如抗体,,以及其他的蛋白质(例如,催化酶,dna,rna和糖等)的相互作用实验中固着或固定。
在膜上的融合蛋白质的反应配对物现在能够灵活地出自周围的溶液中与膜上的蛋白质或糖基进行特异性反应。
这些反应的前提在于蛋白质的功能化的构象。在根据本发明的融合蛋白质中完全确保所述构象。
因此,本发明的第一主题是一种融合蛋白质,所述融合蛋白质包括第一结构域(i)、第二结构域(ii)和第三结构域(iii),其中第二结构域设置在第一结构域和第三结构域之间,其中
(i)包括四分子交联体的子序列或具有在总长度上至少70%的序列一致性的与其同源的序列,所述子序列包括跨膜结构域1(tm1)、小胞外环(sel)、跨膜结构域2(tm2)、小胞内环(sil)和跨膜结构域3(tm3),
(ii)包括具有预设的氨基酸序列的肽,所述肽与四分子交联体的大胞外环(lel)具有在总长度上小于70%的序列一致性并且
(iii)包括四分子交联体的子序列,所述子序列包括跨膜结构域4(tm4)或其具有在总长度上至少70%的序列一致性的与其同源的序列。
在本发明中发现:序列tm1至tm3和tm4负责融合蛋白质的膜结合性。因此,根据本发明的融合蛋白质一方面包括第一结构域(i)和另一方面包括结构域(iii),所述第一结构域包括具有跨膜结构域tm1至tm3的四分子交联体的子序列,所述结构域(iii)包括具有跨膜结构域tm4的四分子交联体的子序列或其与其同源的序列。
就本发明而言,术语“四分子交联体”表示四分子交联体家族的任意蛋白质,尤其人类四分子交联体33。四分子交联体能够彼此在膜中发生特异性相互作用。由此,有助于经由膜的转运和在那的锚定。在根据本发明的融合蛋白质中能够利用该效应,以便结构域(iii)的外源性肽也有效地经由膜转运并且锚定。就本发明而言,四分子交联体的优选的实例是cd63、cd9、cd82、cd81、cd151和cd53(dna序列和蛋白质序列,见序列表)。尤其优选地,包含在根据本发明的融合蛋白质中的子序列源自四分子交联体cd63(dna序列和蛋白质序列,见序列表)。
原则上可行的是:为结构域(i)选择第一四分子交联体的子序列,并且为结构域(iii)选择另一四分子交联体的子序列。然而,优选的是:出自相同的四分子交联体,例如cd63、cd9、cd82、cd81、cd151或cd53,优选cd63的全部子序列(dna序列和蛋白质序列,见序列表)。
结构域(i)优选包括四分子交联体的跨膜结构域tm1、小胞外环(sel)、跨膜结构域tm2、小胞内环(sil)和跨膜结构域tm3,其中不同的跨膜结构域和跨膜结构域环彼此相随,而没有在所述跨膜结构域和所述跨膜结构域环之间嵌入附加的异源性序列片段。尤其优选地,子序列对应于四分子交联体的野生型,而没有呈例如个别或多个氨基酸的置换、插入或缺失形式的任何偏离。然而,原则上可行的是:只要按野生型四分子交联体计,所述子序列具有在序列的总长度上至少70%的序列一致性,那么对于结构域(i)应用四分子交联体的修饰的子序列。更优选地,序列一致性在四分子交联体的包含在结构域(i)中的子序列的总长度上为至少80%,85%,90%,95%或99%。
在本发明的尤其优选的实施方式中,第一结构域(i)由四分子交联体的上述子序列或具有在总长度上至少70%(更优选至少80%,85%,90%,95%或99%)的序列一致性的与其同源的序列构成。
结构域(iii)包括四分子交联体的子序列,所述子序列包括四跨膜蛋白质的跨膜结构域tm4或具有在总长度上至少70%的序列一致性的与其同源的序列。更优选地,四分子交联体野生型的子序列的序列一致性为在总长度上至少80%,优选至少85%,90%,95%或99%。在尤其优选的实施方式中,结构域(iii)由四分子交联体的上述子序列或其与其同源的序列构成。
结构域(ii)包括具有预设的氨基酸序列的肽。所述肽在此也称为“外源性肽”或“外源性蛋白质”。外源性肽的氨基酸序列不同于野生型四分子交联体的、尤其结构域(i)和结构域(iii)的(一个或多个)四分子交联体的大胞外环(lel)的序列。与野生型四分子交联体的lel对比,序列一致性占在总长度上小于70%,优选小于50%,更优选小于40%或小于30%。
原则上,能够任意选择结构域(ii)中的氨基酸顺序。与外源性肽或结构域(ii)的序列无关,根据本发明的融合蛋白质能够在细胞、尤其哺乳动物细胞(例如hek293t-智人(homosapiens))的表面上锚定并且呈递。
就本发明而言,术语“肽”应理解为由至少两个氨基酸构成的化合物,所述氨基酸彼此经由肽键连接。在此,原则上,肽的长度不受限制。其包括具有直至10个氨基酸的寡肽、具有超过10个氨基酸的多肽和具有大于100个氨基酸的巨肽。在尤其优选的实施方式中,肽的大小为小于100kda,优选小于50kda或小于30kda,例如10kda至30kda。
就本发明而言,术语“氨基酸”优选应理解为存在于二十个天然存在的肽、多肽和蛋白质中的氨基酸,所述氨基酸分别是左旋异构体。但是此外,根据本发明,该术语包括氨基酸的类似物和氨基酸的右旋异构体和其类似物。
就本发明而言,术语“肽”、“多肽”和“蛋白质”应分别理解为可交换的。在优选的实施方式中,肽是具有至少50个氨基酸的蛋白质。在此,优选的是:蛋白质具有预设的折叠,所述折叠例如对于蛋白质的期望的功能是必需的。
在结构域(ii)中能够包括具有预设的氨基酸序列、例如抗原或抗原的表位的肽。在此,术语“肽”应理解为抗原的如下区域,抗体或t细胞受体特异性结合到所述区域上。单个抗原能够包括一个或多个表位。在本发明的实施方式中,具有预设的氨基酸序列的肽包括多个表位,所述表位通过连接肽彼此连接。例如是抗原的多个表位。表位的实例是肿瘤表位,hiv表位(例如,hiv-1的gp41)。对此的实例是v5-6xhis(seqidnos:35,36)或2f5-4e10(seqidnos:37,38)。
在本发明的另一实施方式中,具有结构域(ii)的预设的氨基酸序列的肽包括具有期望功能的蛋白质、例如酶,例如蛋白质酶。
除具有预设的氨基酸序列之外,第二结构域(ii)还能够包括四分子交联体的lel的一个或多个子序列或具有在总长度上至少70%的序列一致性的与其同源的序列。优选地,序列一致性为至少80%,至少85%,至少90%,至少95%或至少99%。四分子交联体的lel的子序列能够相对于具有预设的氨基酸序列的肽设置在c端和/或n端。优选地,结构域(ii)中的lel的子序列源自与结构域(i)和结构域(iii)的序列片段相同的四分子交联体中。
如在此使用的“同源”描述在两个多肽、分子之间的或在两个核酸之间的序列相似性。如果在两个对比序列的两个中的位置都通过相同的碱基单体子单元或氨基酸单体亚组占据,那么在该位置上的相应的分子同源。在两个序列之间按百分比表示的同源是共同具有这两个序列的一致性
同源的序列共同具有相同或相似的氨基酸残基,其中相似的氨基酸残基表示在对齐的参考序列中的相应的氨基酸残基的允许的点突变或相应的氨基酸残基的保守的置换。在这方面,参考序列中的残基的“保守的置换”表示如下置换,所述置换与相应的参考序列在物理上或功能上是相似的,例如所述置换具有相似的大小、形状、电荷、化学性能等,所述化学性能包括形成共价键或氢键的能力。尤其优选的保守的置换是满足如下标准的置换,所述标准针对dayhoff等“atlasofproteinsequenceandstructure”,5:suppl.3,kap.22:354-352,,nat.biomed.res.foundation,washington,d.c.,1978中限定的可接受的点突变来限定。
根据本发明的融合蛋白质的结构域分别按(i)-(ii)-(iii)的顺序设置。在此,术语“融合”涉及结构域经由其各个肽骨架的共线性的耦联。
结构域(i)、结构域(ii)和结构域(iii)的连接可选地能够直接地或经由柔性的连接肽进行。在第一实施方式中,结构域(ii)直接地连接到结构域(i)上。替选地或附加地,结构域(iii)直接地连接到结构域(ii)上。
在另一实施方式中,柔性的连接肽设置在第一结构域和第二结构域之间和/或设置在第二结构域和第三结构域之间。所存在的连接肽能够是相同的或不同的。用于在根据本发明的融合蛋白质中应用的示例性的连接肽例如包括甘氨酸聚合物(g)n、甘氨酸-丙氨酸聚合物和其他本领域技术人员已知的柔性的连接肽,其中n是最小为1的整数。优选的主要是不含丝氨酸并且不含精氨酸的连接肽,尤其优选的是不含丝氨酸并且不含精氨酸的连接肽。更尤其优选的是,连接肽lqefdigggg(对应于seqidnos:41,42)。在图2c中描述其他连接肽(也见seqidnos:39,40)。
结构域(ii)还能够包括另一个或多个识别序列,,所述识别序列能够被酶裂解。实例是用于特定的蛋白质酶、核酸酶或内切糖苷酶的识别位点。替选地,识别位点能够包括用于特定的水解酶、例如磷酸酶、糖苷酶、酰胺酶或酯酶的底物识别位点。术语“蛋白质酶识别序列”在此概括地用于所有能够酶裂解的识别序列。所述识别序列能够相对于具有预设的氨基酸序列的肽设置在c端和/或n端。在尤其优选的实施方式中,蛋白质酶识别序列直接地置于具有预设的氨基酸序列的肽的两侧。原则上,任意蛋白质酶识别序列适用于此。如果第二结构域(ii)除具有预设的氨基酸序列的肽之外还包括四分子交联体的lel的一个或多个子序列,那么优选的是:蛋白质酶识别序列分别嵌入在lel子序列和具有预设的氨基酸序列的肽的c端或n端之间。
在另一实施方式中,根据本发明的融合蛋白质能够附加地包括在融合蛋白质的n端上的第四结构域(iv)和/或在融合蛋白质的c端上的第五结构域(v)。第四结构域(iv)和第五结构域(v)分别能够直接地或经由如上述限定的柔性的连接肽连接。例如,第四结构域(iv)能够直接地或经由柔性的连接肽连接在第一结构域(i)的n端上。第五结构域(v)能够直接地或经由柔性的连接肽连接在结构域(iii)的c端上。此外,可行的是:如上述限定的蛋白质酶识别序列设置在结构域(iv)和结构域(i)之间和/或在结构域(iii)和结构域(v)之间,或只要存在,就设置在连接肽和结构域(i)、结构域(iii)、结构域(iv)和结构域(v)中的一个或多个之间。
在四分子交联体锚定在细胞的细胞质膜中时,四分子交联体的c端和n端位于细胞质侧上。相应的情况也适用于根据本发明的融合蛋白质的膜锚定中。
根据本发明的融合蛋白质的第四结构域(iv)和第五结构域(v)例如能够选择成,使得所述结构域包括标记群或所谓的标签。在此,术语“标签(tag)”应理解为短的肽序列,例如用于标签和识别的蛋白质标签,例如flash-tag或荧光报告蛋白质mcherry。
已发现:根据本发明的融合蛋白质类似于野生型四分子交联体,所述融合蛋白质与野生型四分子交联体至少通过结构域(ii)区分,但正确地折叠。这能够在结构域(ii)中的荧光蛋白质cfp和yfp的实例显示出(见图3b)。在跨膜结构域tm3和tm4之间的表位能够借助于利用扫描电子显微镜在细胞表面上的细胞外的定向来证实(图4)。对于实例cd63,借助扫描电子显微镜示出表位在四分子交联体的tm3和tm4之间的引入并且转运至具有细胞外定向的细胞表面以及在n端和c端上的表位(图5a至图5d)。荧光蛋白质的成功的蛋白质表达附加地能够通过蛋白质印迹分析证实。在此,对于相应的融合蛋白质可能会检测到关于flag以及mcherry表位的特定的光谱带(图6)。同样地,显示出其他四分子交联体(cd9,cd81,cd82和cd151)在细胞表面上的定位(图8)。此外,能够显示出:应用低丝氨酸的和低精氨酸的连接肽提高在细胞表面上定位的效率(图10)。
本发明的另一主题是一种核酸分子,所述核酸分子用于编码根据本发明的融合蛋白质。根据本发明的核酸分子包括用于编码上述融合蛋白质的序列片段或由所述序列片段构成。该序列片段在此也称作“融合基因”。尤其优选地,根据本发明的核酸分子以隔离的形式存在。
就本发明而言的术语“核酸分子”涉及一系列的天然存在的核酸碱基、碱基类似物、碱基衍生物或其混合物形式。术语尤其包括dna和rna以及其派生的核酸,例如cdna和mrna。就本发明而言,核酸类似物、如pna(肽核酸),lna(锁核酸),psna(硫代磷酸核酸)也属于该核酸。这些核酸类似物原则上能够具有天然存在的核酸碱基,当然,所述核酸碱基与例如在dna或rna中不同的方法和方式彼此连接。具有核酸衍生物、例如次黄嘌呤、2,5-二氨基嘌呤和/或甲基胞嘧啶的核酸和被修饰、尤其被标计的核酸也属于该核酸。除碱基衍生物之外,这些核酸衍生物原则上也能够具有天然存在的核酸碱基。标签物能够引入到碱基和/或骨架上。连接能够对应于dna或rna或核酸类似物。核酸能够是双链的或单链的,直链的,支链的或环状的。
本发明还涉及一种载体,所述载体包括如上述的核酸分子。优选地,核酸分子与表达调控序列处于操作性的连接(operatiververknüpfung)。
就本发明而言的术语“表达调控序列”包括在转录水平上调节表达的元件,例如增强子或沉默子,和在后转录水平(例如,剪切、核输出或mrna稳定性)上调节表达的元件。该术语也包括多个元件,所述元件在转录水平或后转录水平上调节表达,或包括转录的和后转录的调节元件的组合。用于识别产生提高的表达的表达调控序列和用于所述表达调控序列分离的技术和辅助手段是本领域技术人员熟知的常规技术手段。表达调控序列尤其能够是启动子,优选真核生物的启动子,例如cmv启动子或sv40启动子;原核生物的启动子或噬菌体特定的启动子,例如sp6启动子或t7启动子。尤其优选地,启动子是cmv启动子。载体能够附加地包括用于核酸分子的终止和/或聚腺苷酸化的序列片段。
此外,根据本发明的载体优选包含选择性标记基因,也就是说,用于编码选择性标记的序列片段。选择性标记的实例是抗生素抗性标记或缺陷型标记。优选使用能扩增的选择性标计基因,例如二氢叶酸还原酶。可选地,根据本发明的载体还能够包括复制起点。
就本申请而言,术语“处于操作性的连接”描述两个或更多个氨基酸序列或子序列的连接,所述氨基酸序列或所述子序列定位成,使得所述子序列能够发挥其预期的功能。例如,当所述启动子/增强子在顺势位置上能够调控或调整连接的基因序列的转录时,启动子/增强子与编码的基因序列功能性连接。一般地,然而并非必需的是,功能性连接的序列紧密相邻,并且只要两个编码的基因序列连接或在分泌信号序列的情况下,就处于相同的阅读框中。尽管功能性连接的启动子一般位于编码的基因序列的上游,但所述启动子不必需是紧密相邻的。只要增强子有利于基因序列的转录,该增强子就同样不一定以紧密相邻的方式存在。为了该目的,所述增强子能够存在于基因序列的上游和下游,必要时,也以一定间距的方式存在。当聚腺苷酸化位点定位在基因序列的3’端上,使得转录经由编码的序列进展呈聚腺苷酸化信号时,聚腺苷酸化位点与基因序列功能性连接。连接能够根据通常的重组方法,例如借助pcr技术通过在适合的限制性酶切位点上连接或通过剪切进行。当不存在适合的限制性酶切位点时,能够以已知的方式使用合成的寡核苷酸连接肽或衔接子。优选地,不经由内含子序列进行功能性连接。
根据本发明的载体优选适合于至少一个(宿主)细胞,尤其用于在该细胞中载体的复制和/或用于在该细胞中蛋白质或肽的表达。复制能够在原核生物细胞和/或真核生物细胞中进行。表达优选在真核生物细胞中进行。载体能够是质粒。
根据现有技术的真核生物表达的载体构建通常具有强烈的、尽可能细胞类型非特异性的真核生物启动子(例如cmv启动子或sv40启动子)和负责转录终止和聚腺苷酸化的序列部分(例如sv40-termination-polya)。在这两个元件之间存在待表达的cds(编码序列),所述cds被或多或少良好突出的、围绕起始密码子的kozak序列引用。在根据本发明的载体构建中,待表达的cds是出自四分子交联体的超级家族的基因。在此,全部四分子交联体基因出自全部生物物种,所述生物物种具有基因超级家族的基因,根据本发明,所述基因产生经由膜的期望的转运性能。当然,根据本发明的融合基因的特征在于:所述融合基因能够具有至少一个人造引入的异源性dna序列作为内部并且必要时具有终止融合体。终止的cds融合体是异源性dna序列,所述dna序列对于融合蛋白质或肽的四分子交联体以5’近端方式(5’-proximal)和/或3’远端方式(3’-distal)编码。根据本发明,在第三跨膜结构域(tm3)和第四跨膜结构域(tm4)之间引入内部的、编码的异源性dna序列。此外,四分子交联体的跨膜结构域负责将异源性编码的序列转运至膜,所述序列负责通过膜和在那的锚定进行易位。
受过分子生物学培训的平均水平的本领域技术人员能够:必要时借助引用文献39来制备功能性的dna融合构建体。
在此,尤其会强调指出其他根据本发明的载体构建体。代替通常的真核生物启动子,所述载体构建体也具有细菌和噬菌体特异性的、但还有真核生物细胞类型特异性或组织特异性的启动子,所述启动子实现与细胞分化无关的表达。能够使用如下启动子类型,以便在任何应用中相同地使用根据本发明的基因构建体。载体的其他根据本发明的实施方式例如也能够具有噬菌体的rna聚合酶启动子,以便在体外或必要时在体内高效地产生mrna分子。实例是噬菌体启动子,,如用于在体外制备mrna分子的sp6特异性启动子或t7特异性启动子,以便必要时能够为治疗目的和应用的输送融合基因mrna。这些变体通常视为调节器的剂型,因为所述变体应当防止核酸重组成基因组。
在细菌或酵母中的四分子交联体基因衍生物的表达能够用于分离融合蛋白质并且在人造膜中使用。但也能够利用粘膜细胞或肌细胞中真核生物的分化特异性或细胞特异性的表达,以便细胞特异性表达dna疫苗(例如见ep1390490b1)。
在用于治疗的批准程序中建立监管规定,所述监管规定强制地要求这种高度特异性的表达特性。
此外,根据本发明的载系统统具有特定的设计,所述设计允许借助唯一的载体构建体在同一细胞中表达多个融合蛋白质(见专利申请de102013006487a9)。借助该设计能够简化地添加附加的元件。
本发明的主题也是如下核酸和载体,所述核酸和所述载体如上述限定那样具有用于编码融合蛋白质的片段,然而其中仅仅存在多克隆位点代替用于编码具有预设的氨基酸序列的肽的序列,所述克隆位点经由限制性核酸内切酶的识别序列实现用于编码具有预设的氨基酸序列的任意的肽的基因的克隆。附加地,多克隆位点也能够存在于结构域(iv)或结构域(v)的n端和/或c端的5’位置和/或3’位置中。
因此,本发明的一个方面涉及核酸分子,所述核酸分子包括第一序列片段(i)、第二序列片段(ii)和第三序列片段(iii),其中第二序列片段设置在第一序列片段和第三序列片段之间,
其中
(i)用于编码四分子交联体的子序列或具有在总长度上至少70%的序列一致性的与其同源的序列,所述子序列包括跨膜结构域1(tm1),小胞外环(sel),跨膜结构域2(tm2),小胞内环(sil)和跨膜结构域3(tm3),
(ii)包括多克隆位点并且
(iii)用于编码四分子交联体的子序列,所述子序列包括跨膜结构域4(tm4)或具有在总长度上至少70%的序列一致性的与其同源的序列。
在此,序列片段(i)至(iii)优选从5’向3’设置。“多克隆位点”或“multiplecloningsite”包括用于限制性核酸内切酶的多个相继紧随的限制性酶切位点。多克隆位点尤其适合于整合核酸分子,其中整合的核酸分子在具有第一序列片段(i)和第三序列片段(iii)的阅读框中用于编码待呈递的肽。肽优选包括预设的氨基酸序列,所述氨基酸序列具有与四分子交联体的大胞外环(lel)在总长度上小于70%的序列一致性。
本发明还涉及一种载体,所述载体包括优选与表达调控序列处于操作性连接的上述核酸分子。
在现有技术中已知用于极其不同的限制性核酸内切酶以及在此所属的限制性核酸内切酶的许多识别序列。优选地,使用由至少七个核苷酸作为识别序列构成的序列。适合的识别序列的列表例如存在于sambrook等(1989)。
具有多克隆位点的根据本发明的载体的特定的序列元件涉及将异源性dna序列引入到四分子交联体基因(例如cd63)的相应末端和内部的、功能允许的位置上本身。因此,总是能够相加地在循环反应中在现有的载体构建体中再次将新的表位-cds或不同的基因和基因片段加入到“独特的”、即单个的限制性酶切位点中。在图2a中的根据本发明的载体pae21(seqidno:1)是示例性的。限制性酶切位点在跨膜结构域tm3和tm4之间的位置上定位。对于该位置以实验的方式示出:所述位置尤其适合于与用于编码异源性肽的异源性dna序列融合,因为所述位置不干扰膜转运的功能。在这些位点上存在蛋白质环,所述蛋白质环突出于膜,其序列能够是可变的,而在细胞培养时基本上不干扰蛋白质转运至四分子交联体蛋白质的细胞质膜。
用于产生n端融合蛋白质的5末端cds融合体能够借助两个ncol通过引入到cd63基因的5’区域中,使得kozak序列在起始区域中再生。这是独特的。
引起c端蛋白质融合的可行的3’末端cds融合元件借助ii型s限制性核酸内切酶bsal插入,所述限制性核酸内切酶bsal在构建体中产生用于ndel和bc/i的特异性的端部。这些位点必要时也能够用于嵌入3’末端基因融合元件。
载系统统的上述实现方案是示例性的,并且除所描述的限制性连接方法之外载体的修饰的任何其他方法或技术同样是根据本发明的,例如是借助适合的重组系统在体内以及在体外系统进行的系统的序列调整。
本发明的另一主题是一种细胞,所述细胞包括根据本发明的核酸分子或根据本发明的载体。
在根据本发明的实施方式中,包括用于编码融合蛋白质的序列并且适合遗传控制功能的核酸分子或载体是基因稳定整合的或存在于细胞中染色体外,并且根据本发明的融合蛋白质在那本构地或可诱导地或可抑制地表达。
就本发明而言的细胞优选是真核动物细胞,优选哺乳动物细胞如、尤其人类细胞。适合的哺乳动物细胞的其他实例是啮齿动物的细胞系,例如小鼠细胞、大鼠细胞和仓鼠细胞或猿猴细胞。根据本发明的核酸分子或根据本发明的载体能够借助已知的技术引入到细胞、尤其哺乳动物细胞中。成功的转染产生转化的、基因修饰的、重组的或转基因的细胞中。
具有根据本发明的核酸分子或根据本发明的载体的真核宿主细胞的转染根据通常的方法(sambrook等,1989;ausubel等,1994)来进行。合适的转染方法例如是脂质体介导的转染,磷酸钙共沉淀,电穿孔,聚阳离子,(例如deae-葡聚糖)介导的转染,原生质体融合,显微注射和病毒感染。根据本发明,细胞优选经由稳定的转染获得,其中构建体整合到宿主细胞的基因组或人造的染色体/微型染色体中或以稳定的方式游离地包含在宿主细胞中。在此,在相应的宿主细胞中实现最佳的转染频率和异源性基因的表达的转染方法是优选的。根据定义,引入到宿主细胞中的每个序列或每个基因相对于宿主细胞称作“异源性序列”或“异源性基因”,即使待引入的序列或待引入的基因与宿主的内源性序列或内源性基因一致也如此。
在根据本发明的实施方式中,其能够是不同细胞类型的转基因的细胞群的细胞,在所述细胞中也在不同的细胞分化状态下表达根据本发明的融合基因。这些细胞能够离体产生并且必要时紧接着在体内治疗使用。
在根据本发明的另一实施方式中,其为转基因组织的细胞群的细胞,其细胞已经过生殖细胞并且发育成多细胞系统,优选发育成生命组织。
在优选的实施方式中,根据本发明的细胞还与至少另一核酸分子或至少另一表达载体转化或转染。另一核酸分子或另一表达载体优选包含用于编码另一预设的蛋白质的片段。根据一个实施方式,另一核酸分子或另一表达载体同样能够是根据本发明的核酸分子或根据本该发明的表达载体,其中在这种情况下为结构域(ii)选择具有其他预设的氨基酸序列的其他肽。在另一实施方式中,另一核酸分子或另一表达载体包含用于编码酶、例如蛋白质酶的片段。当根据本发明的核酸分子和另一核酸分子在这些细胞中表达时,具有预设的氨基酸序列的结构域(ii)的肽和另一蛋白质呈递在膜表面上。如果例如蛋白质酶识别序列置于结构域(ii)的肽两侧,那么经由另一核酸分子表达的蛋白质酶能够在该识别位点上切割进而释放肽。
根据本发明,宿主细胞优选在不含血清条件下,必要时在不含动物蛋白质/肽的培养基中建立、适应并且培养。
本发明的另一主题是膜配制品,所述膜配制品从根据本发明的细胞中获得。
就本发明而言的膜配制品包括至少一个分离的膜,更优选分离的、细胞膜,还更优选分离的、细胞的细胞质膜和/或其他细胞器的分离的、细胞膜。就本发明而言的术语“膜”描述在包括至少一个脂质和可选地至少一个蛋白质的自身闭合的系统。膜的脂质通常形成脂质双分子层。膜配制品优选包括膜颗粒,更优选膜泡。
膜配制品能够从根据本发明的细胞中通过本领域技术人员已知的方法,优选通过细胞裂解(细胞裂解液),更优选通过细胞裂解和随后的分离并且可选地纯化细胞膜(分离的并且可选的纯化的膜)来获得。可选地,表面活性剂、例如tritonx-100能够用于溶解膜。该方法能够附加地包含用于移除细胞碎片的步骤。膜颗粒的直径能够在1nm至1000nm,优选50nm至500nm,更优选75nm至200nm的范围中。至少80%,至少90%,至少95%或至少98%的膜颗粒能够具有在此处描述的范围中的直径。
根据本发明的膜配制品由根据本发明的细胞获得,,所述细胞包括根据本发明的核酸分子或根据本发明的载体。根据本发明的融合蛋白质通过核酸分子或载体在细胞中表达。因此,根据本发明的膜配制品优选包括至少一个分离的膜和至少一个根据本发明的融合蛋白质。在此,根据本发明的融合蛋白质优选锚定在膜中,更优选在膜的表面上,尤其在膜的细胞外侧上。根据本发明的融合蛋白质呈递在膜上,优选在膜的表面上,尤其在膜的细胞外侧上。膜配制品可选地能够附加地包括一个或多个野生型四分子交联体。
在一个实施方式中,根据本发明的膜系统是例如在体外细胞培养的细胞的外膜,在所述细胞中瞬时地表达至少一个根据本发明的融合蛋白质。
本发明还涉及合成的膜系统,所述膜系统包括如上描述的膜和融合蛋白质、核酸分子或载体。
膜能够是细胞的(天然的)或合成的(人造的)膜。细胞膜尤其是细胞的细胞质膜,所述细胞质膜典型地存在于细胞的表面(外侧)上。就本发明而言的细胞膜能够是细胞的一部分,或从细胞中分离出来,也就是说,是分离的细胞膜。细胞膜典型地是非对称的并且具有朝向细胞质的侧(细胞质侧或细胞内侧,内侧)和背离细胞质的侧(外细胞质侧或细胞外侧,外侧)。
合成的膜能够包括合成的和/或分离的、细胞的膜。尤其,在合成的膜中脂质成分和/或蛋白质成分能够与细胞膜的天然存在的成分不同。合成的膜优选不是细胞的一部分并且尤其作为分离的、合成的膜存在。本领域技术人员能够通过选择脂质和/或蛋白质影响合成的膜的特性。
在一个实施方式中,包括根据本发明的核酸分子或根据本发明的载体的合成的膜系统附加地包含用于表达核酸分子或载体的系统,由此能够表达融合蛋白质。用于表达蛋白质的系统在现有技术中是已知的并且优选是不含细胞的。用于表达的系统优选包括用于转译核酸分子,尤其rna或mrna的系统;可选地包括用于转录核酸分子或载体的系统,尤其以便获得rna或mrna。
在替选的实施方式中,融合蛋白质能够直接添加至膜,以便获得根据本发明的膜系统。
在膜系统中,表达的或添加的融合蛋白质锚定在膜中,优选锚定在膜的表面上,尤其锚定在膜的外侧上。根据本发明的融合蛋白质呈递在膜上,优选呈递在膜的表面上,尤其呈递在膜的外侧上。
已发现:在合成的膜系统中,根据本发明的融合蛋白质也能够带引至膜表面,易位并且在那锚定。在这种情况下,可行的是:在体内或在体外人造的、非典范的氨基酸装入到根据本发明的融合蛋白质中。借助不含细胞的蛋白质合成能够获得融合蛋白质,所述融合蛋白质也适合于合成的膜定居并且在那以锚定方式存在。
本发明的另一方面是用于将具有预设的氨基酸序列的肽呈递在细胞的表面上的方法,所述方法包括如下步骤
(a)提供如上述的根据本发明的细胞,并且
(b)在如下条件下培养细胞,其中表达根据本发明的融合蛋白质。
本发明还涉及一种用于将具有预设的氨基酸序列的肽在膜上锚定的方法,所述方法包括如下步骤
(a)提供膜,并且
(b)在如下条件下使膜与如上述的融合蛋白质形成接触,其中将融合蛋白质锚定在膜中,或在如下条件下使膜与如上述的核酸或如上述的载体形成接触,其中表达融合蛋白质并且在膜中锚定。
本发明的另一主题是将上述细胞、膜配制品或合成的膜系统在用于根据肽的相互反应配偶体筛选的方法中的应用,用于研究肽与相互反应配偶体(例如抗体)的相互作用的应用,用于在用于制备肽的方法中的应用,用作为在用于抗体制备的方法中的免疫试剂的应用和/或用作为检测试剂的应用。
本发明还涉及如上述的用作疫苗和/或药物的细胞、膜配制品或合成的膜系统。
在根据本发明的实施方式中,融合蛋白质从转染的、瞬时或稳定表达的细胞、转基因的组织或合成的膜系统中获得,以便应用在研发过程中,如有必要还成为方法的组成部分或者成为可商业化应用的成套试剂的组成部分。实例是生物分析-诊断系统和治疗剂的制造。
在根据本发明的另一实施方式中,获得包含融合蛋白质的膜、细胞、转染的或合成的膜系统,以便在研究和发展中应用,如必要时也作为方法的组成部分或可商购的应用的成套试剂的组成部分。实例是生物分析-诊断系统和治疗剂的制造。
本发明的表达的融合蛋白质在细胞表面上示出转染的细胞作为固定的反应配对物,并且能够用于分析与其他分子的特定的相互作用。一方面,在此感兴趣的是蛋白质-蛋白质相互作用(例如,单克隆抗体或多克隆抗体-互补位-表位的相互作用,螺旋-螺旋的相互作用以及药物-医学、尤其还有分子-机械意义的特定的相互作用)。权利要求中的四分子交联体融合分子也能够用于分析与其他分子,如单糖和多糖,rna,dna并且尤其与小的化学分子如天然物质和化学物质库的相互作用。
多个四分子交联体融合分子或蛋白质种类的蛋白质变体在细胞中的表达对于确定的实验方案与以组合方式使用不同的相互作用的蛋白质是同样令人感兴趣的。
在本发明的一个实施方式中,根据本发明的细胞表达蛋白质结构域(a),蛋白质结构域能够与其他蛋白质结构域(b)进行高度特异性的相互作用。在此,蛋白质结构域(a)对应于具有根据本发明的融合蛋白质的结构域(ii)的预设的氨基酸序列的肽。
然后,这些外部的蛋白质结构域能够与不同功能的其他蛋白质结构域连接。对于全部这些蛋白质结构域模块能够确保:如果所述蛋白质结构域模块是外源性的,那么这些蛋白质结构域模块特异性地结合到用于表达的细胞的配对结构域(a)上,从而能够交换蛋白质配体,所述蛋白质配体就其而言能够再次出现结合,从而能够在细胞表面上证实通过特异性相互反应介导的不同的蛋白质模块的共同存在。
下面,应当更详细描述根据本发明的融合蛋白质、载体、细胞和膜系统的应用的优选的实例。
蛋白质-蛋白质相互作用(ppi)的分析
可供使用地固定在表面上的蛋白质能够借助特别的结构在相应的蛋白质的表面上建立与其他蛋白质的特异性接触。这些其他蛋白质能够是同源蛋白质复合物或异源蛋白质复合物的蛋白质结构组,所述蛋白质结构组能够相互间识别或但也能够识别特定的蛋白质,如抗体和单链,所述特定的蛋白质能够特异性地并且有针对性地结合到确定的蛋白质表面结构上。与其他蛋白质发生特异性相互作用的、编码基因的天然存在的蛋白质发挥许多细胞功能(从在细胞核中的功能直至多种胞质中的功能和膜功能)。这些特异性和功能性的蛋白质-蛋白质相互作用(ppi)在进化过程中形成,并且决定性地负责细胞功能的完整性。有针对性的ppi通过细胞过程和选择蛋白质变种来介导,所述蛋白质变种具有与表面的特异性相互作用。免疫系统针对相对于生物体内的表面的变化的敏感反应设计,并且专门针对于在非常短的时间段内优化抗体的表面相互作用和细胞毒性。在体液免疫中短时间内产生大量表位特异性的、多克隆的抗体。也已知的是骆驼型的(单链(singlechains))和其他天然存在的免疫系统的特定的单链抗体,所述系统发展成高度特异性的蛋白质相互作用。
在医学研究中,这些过程技术上借助细胞培养技术来反应,以产生单克隆抗体,从而实现在体外产生特异性的ppi。
抗体工程设计利用人造的、借助展示技术(核糖体展示,噬菌体展示,酵母展示)制成的、能够特异性识别表位的互补位,以便将所述互补位嵌入到抗体分子中。在此描述的tanchor系统的蛋白质单元的有效的输出功能和其在细胞中的锚定高度适用于ppi分析。此外,借助糖特异性修饰的蛋白质也能借助tanchor技术呈递在细胞表面上。
如下检测系统在贸易中作为细胞分选系统(facs),基于荧光发光或基于生物发光的成像系统,但也作为fret光子的整套设备提供,其中具有平均专业知识的本领域技术人员能够借助所述检测系统识别特异性ppi。在此,质谱分析以杰出的方式也考虑作为分析工具。
单独所使用的细胞系统觉得产生的数据的质量和决定性的区别。与光学装置中的分辨率类似地,其是决定全部其他工艺步骤的参数,所述参数在工艺链结束时获得的数据的质量。
tanchor系统以特定的方法和方式适合于提供用于生物分析诊断应用以及治疗的展示功能。借助所述tanchor系统,蛋白质结构的完整性能够在获得例如结构上的表位条件下保持结构上完好,但还有由结合结构域的完整性在细胞调节蛋白质彼此间的ppi(蛋白质-蛋白质相互作用)时保持结构上的完好。
具有tanchor构建体的小分子的分析
借助结构上完好的蛋白质结构附加地可以进行结合的相互作用研究,当然也可以进行置换的相互作用研究,所述结合是通过具有呈递在细胞上的蛋白质的小分子的ppi结合。小分子的这种相互作用是令人感兴趣的,所述小分子干扰ppi并且其中识别出确定的蛋白质附聚物,所述蛋白质附聚物在小分子的影响下能够再次溶解。ppi分析尤其良好地适合于发现并且开发小分子活性物质。
tanchor技术为此提供杰出的工具,所述工具能够提供蛋白质表达与蛋白质在细胞表面上的同时锚定。结合出自其他表达源的适合的蛋白质对或必要时也通过不同的tanchor分子的共表达能够从中建立用于分析小分子与蛋白质复合物的相互作用的测试系统。
此外,根据本发明的融合蛋白质的在细胞表面上定位的片段能够借助高度特异性裂解的分泌型蛋白质酶(如tev蛋白质酶(烟草蚀纹病毒蛋白质酶))的必要时能诱导的共表达在预设的位点上特异性裂解进而置于溶液中。缺陷型融合蛋白质能够在再合成循环中在膜中持续地被取代,从而也有助于持久的过量表达。
(例如从表达次级代谢产物的组织中提取的)小分子能够以该方式结合到根据本发明的蛋白质上并且所述小分子识别为靶。
在由小分子和根据本发明呈递的蛋白质片段构成的所产生的复合物中,小分子能够特异性地结合到固定在细胞表面上的靶肽上或结合到已经分裂的、作为载体肽处于溶液中的靶肽上。
随后,借助质谱分析能够识别产生的复合物从而识别小分子和其结合对。例如,在非肽的小分子情况下,所述小分子能够在分离处于溶液中的复合物并且例如借助非特异性酶的蛋白质分解等破坏肽之后借助质谱分析识别。
免疫诊断物的开发和生产
根据本发明的实施方式,例如如下应用是令人感兴趣的,其中应当测试对确定模板、例如标准化的96孔模板的抗体测试与暴露tanchor的表位的结合。借助根据本发明的系统,例如将细胞播种在具有96孔模板的微量滴定板中,并且在瞬时转染状态下表达融合构建体。这借助稳定的表达融合蛋白质的细胞系同样是可行的。
示例性地示出tanchor系统在elisa实验中用于检测抗体的应用(图9)。
融合蛋白质存在于细胞表面上并且在那呈递。膜的这些人造的构型能够直接反应开发和生产分析应用和治疗应用。
以该方式能够制备分析系统,在所述分析系统中例如测试患者队列的适合于临床研究的、对应于分别待开发的应用的血清。为了开发免疫诊断和治疗、尤其还有为了治疗伴随的诊断,借助这种系统能够定性以及定量地对患者血清检查抗体的存在和其结合特性。
此外,可行的是:固定、干燥微量滴定板和其他模板,以便借此大量制造生物分析的测试系统和诊断并且大量生产所述生物分析的测试系统和诊断,其中转基因的细胞在所述微量滴定板和其他模板中生长。
此外,也可行的是:利用表达四分子交联体-蛋白质-融合产物的转基因的组织的膜并且纯化所述膜。包含根据本发明的融合产物的人造的膜同样能够提供给如其上述的这些应用。
在此,成本节约是极大的,因为取消用肽或表达的并且纯化的蛋白质相对昂贵的覆层(coating)。
获得纯的蛋白质
在研究和开发中,以及也在较高规模的生产中,,尤其当希望蛋白质呈功能性折叠状态并且必要时以修饰的方式纯化时,借助其表达获得具有高度特异性亲和的足够量的靶蛋白质总仍是具有挑战性的。为此,使用根据本发明的系统是有利的。这能够是非常有利的,因为在培养基中将期望的靶蛋白质在细胞外部的定位与在细胞内部胞质溶胶的定位相比为复杂度低得多的环境。与此相应地,可容易地富集并且分离。通常,表达的蛋白质存在于细胞内并且从胞质溶胶中纯化。
在该应用的根据本发明的实施方式中,用tanchor表达的膜结合蛋白质的分裂能够借助氨基酸-序列模体-特异性的蛋白质酶从膜分裂。特异性蛋白质酶能够一方面外生地输送给培养基或内生地与靶蛋白质共表达。这些途径可能需要序列特异性的内切蛋白质酶的原形式,所述内切蛋白质酶在培养基中活化。在以分离蛋白质为目的的时间持续的培养中,特定的靶蛋白质最后富集在培养基中,因为由tanchor释放的靶蛋白质通过蛋白质的完整性损失持续不断地补充到膜中。
作为治疗应用的dna疫苗接种
结合分化特异性的和/物种特异性的启动子(还在兽医领域中),tanchor系统由于有效的膜转运能够也在生物体的完好的细胞中表达异源性的基因融合元件,其中所述启动子在完全确定的靶细胞中存在其表达。所述基因融合元件能够外生地引入到细胞中。为了接种dna分子或还有rna分子,四分子交联体融合构建体能够直接借助所谓的“基因枪”或还有“粒子枪”以高的速度轰击细胞,并且击中的转染的细胞促使瞬时表达融合蛋白质。这些蛋白质能够是先前已测试其能力的抗原或亚特异性的抗原决定簇、表位。tanchor能够用于提供适合于该目的的诊断生物分析以及也用于治疗性的疫苗接种。
进行如下讨论:dna能够整合到用基因颗粒击中的细胞的基因组中并且在那由于整合例如在肿瘤阻遏物基因中可能会造成损害。但实践教导:非常扩展的同源性基因区域置于根据本发明的基因构建体两侧是必需的,以便真正产生较高的且相关的整合速率。在根据本发明的构建体中不设有这种元件。当然,由于这种缔合链关于在敏感的调节剂方面的dna疫苗接种的危险性,mrna疫苗不断增加。因此,根据本发明,也可行的是:例如借助体外转录来产生mrna,所述mrna用于优化在真核生物细胞中的表达,稳定所述mrna并且将所述mrna固定在纳米珠上或在适合的微米区室或纳米区室(病毒样颗粒,vlp)中包装并且mrna以该形式提供给治疗应用。
由于四分子交联体融合体的特定的特性,在此表达的蛋白质转运到用于表达的细胞的细胞膜上并且在那向外显示。
特定的氨基酸序列的暴露能够被免疫系统识别并且在那引起相应的、期望的免疫反应、体液免疫反应或细胞毒性免疫反应。
利用分离的细胞或细胞膜的治疗应用
附加地,可行的是:理想地,由于hla的不兼容组群和与其相关的可能的不期望的免疫反应,本体的细胞在离体提取之后,必要时在中间培养阶段之后或在中间培养阶段期间在体外转染。受试者(例如人但还有动物)的本体细胞用基于tanchor系统的dna构建体转染实现:在自体或自生的疫苗接种中应用。在此,使用完好的且检查质量的自体的细胞以及使用从这些细胞中分离的细胞膜是可行的。这例如能够通过密度梯度超离心法获得。在此,能够改进表位与总蛋白质的比例,从而有助于提高免疫反应的特异活性。减少非特异性伴随反应和自身免疫反应。
表达佐剂的化合物是如下变体,所述变体同样在根据本发明的tanchor系统的应用中处于改进可能性的范围中,所述表达佐剂必要时能够与多个tanchor构建体在细胞中共表达。
改进物理-化学特性的、例如改进膜的溶解性的不同的蛋白质的共表达,为了疫苗接种而能够显著改进从细胞中分离的膜。在与cd63-抗原/表位融合体的共表达中,天然的佐剂也能够辅助改进疫苗接种反应。
本发明的另一主题是成套试剂,所述成套试剂包括
(i)如上述的核酸分子或载体和
(ii)细胞或膜。
附加地,根据本发明的成套试剂根据预期的应用能够包含不同的组分,例如对照质粒、对照蛋白质等和用于不同应用的特定的质粒。
应当通过下面的附图和实例更详细描述本发明。
附图说明
附图和表格
图1示出根据本发明的融合体和融合蛋白质的定位的示意图。
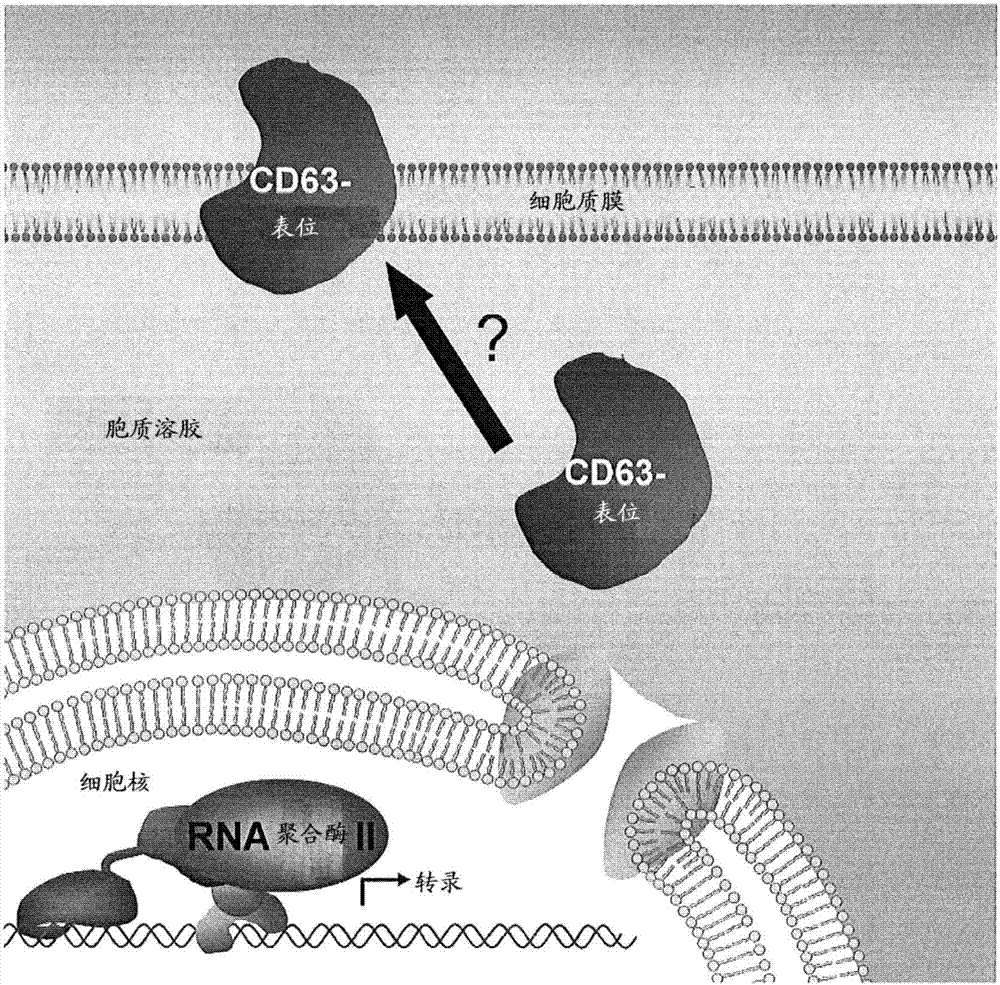
(a)位置,所述位置能够根据具有融合和插入的现有技术用于不同的可适用的解决方案。根据本发明的系统、也称作tanchor系统由四个跨膜结构域(4个tm)构成。在tm3和tm4之间能够克隆用于编码表位或蛋白质的dna序列。在cd63的n末端上示例性地为表位flag-tag,并且在cd63的c末端上例如将荧光蛋白质mcherry附加地在tanchor系统中通过基因融合实施为转染对照(也见图2)。(b)借助为statusquo开发的tanchor系统能够将由人类细胞表达的表位或抗原清楚地定位在细胞质膜上。tanchor系统能够以高的效率将融合蛋白质引至细胞质膜并且在那锚定。
图2示出示例性的tanchor载系统统的示意构造。示例性的第一tanchor载系统统构建在模块化的多表达载体pa4e211(seqidno:1)和该载体的功能性衍生物上。附加地示出cd63δlel组成部分的序列设置的概览(图2b)以及示出荧光报告蛋白质、例如源自pontellinaplumata(桡足类)的mcherry或gfp的flag表位的n端融合以及c端融合。
图2a的表1:tanchor基础系统pa4e211的序列标签的列表:
图2b示出从cd63的编码的野生型(wt)的序列(seqidno:17)中派生cd63δlel序列(seqidno:3)。
野生型cd63蛋白质序列由238个氨基酸构成。为了生成cd63δlel使用跨膜结构域1至3以及跨膜结构域4(标出的序列cd63tm1至tm3,cd63tm4)的编码的dna序列。不包含用于cd63的lel的序列(δlel)的连接肽安置在片段cd63tm1至tm3和cd63tm4之间(seqidno:42)。附加地,gggg结构模体嵌入在连接肽中。在基因序列cd63δlel和用于荧光蛋白质的基因序列之间能够包括用于具有氨基酸序列klidtvdlek(seqidno:46)的连接肽的基因序列(seqidno:45)。
图2a的表2:i型系列的模块化构建的tanchor-成套试剂-载系统统的线性组织:pa4e211
(0)absl-ascl-|flag|cdxx:epitop|reporter|-maubi-sgrdi-selektoren-asrsi-pacl
(1)absl-ascl-|flag|----:------|---------|-maubi-sgrdi-puromycinr-asisi-pacl
(2)absl-ascl-|flag|cd63:-------|---------|-maubi-sgrdi-puromycinr-asisi-pacl
(3)absl-ascl-|flag|cd63:gp41|---------|-maubi-sgrdi-puromycinr-asisi-pacl
(4)absl-ascl-|flag|cd63:eyfp|egfp-gen|-maubl-sgrdi-puromycinr-asisi-pacl
(5)absl-ascl-|flag|cd63:biopepid|egfp-gen|-maubi-sgrdi-puromycinr-asisi-pacl
(6)absl-ascl-|flag|cd63:biopoiid|yfp-gen|-maubi-sgrdi-puromycinr-asisi-pacl
(7)absl-ascl-|flag|cd63:biopoiid|poi-fusion|-maubi·sgrdi-puromycinr-asisi-pacl
用于tanchor-cd63蛋白质呈递系统(展示系统)的阳性对照载体和阴性对照载体。(0)tanchor技术的功能性元件的公式表示:cdxx-(=不同的四分子交联体蛋白质种类)cds(=编码序列)(1)不具有cd63基因、内部的cd63插入和终端报告基因的tanchor载体(2)具有cd63基因的、不具有内部的cd63插入的且不具有如下tanchor载体的终端报告基因的tanchor载体:所述tanchor载体具有cd63基因并且具有例如flag-标签-表位作为内部的cd63插入并且没有终端报告基因(3)具有cd63基因并且gp41表位作为内部的cd63插入以及具有cd63基因的tanchor载体的终端报告基因的tanchor载体(4)具有例如cd63基因和yfp表位作为内部的cd63插入并且具有c端报告基因gfp(e绿色荧光蛋白质基因(egreen-fluorescent-protein-gen))的tanchor载体。(5)具有例如用于分析所使用的poi-biopepid表位(感兴趣的蛋白质/肽)作为内部的cd63插入并且具有c端报告基因egfp(e绿色荧光蛋白质基因(egreen-fluorescent-protein-gen))的tanchor载体,以及(6)具有附加的报告基因,如yfp(黄色荧光蛋白质基因)并且具有相应的实验感兴趣的蛋白质的融合体(poi融合体)。(7)具有根据实验设计的poi融合体等的融合。biopepid是pepid型的生物肽数据库(ep09000893.9)。所述数据库能够在tanchor系统中使用。absl-sgrdi和ascl-maubi以及asisl-pacl是具有用于多个基因的表达的多工复用的八个合并的识别序列的交织的且彼此兼容的限制性核酸内切酶。根据本发明的应用的任何其他的实现方案同样由本专利申请覆盖。
图2c示出不同的根据本发明的连接肽变体与示例性载体pae211_cmv-p-cd63δlel中的cd63δlel组合的实例。限制性酶切位点ecorl、ecorv、hindlll、bamhl和pstl对于根据克隆标准方法以及用于无缝克隆的克隆方插入多个依次相随的表位单元是可行的。(1)seq1,仅根据本发明的且测试功能的限制性酶切位点(r位点)ecorl和ecorv并且n*n=间隔序列(2)seq(1)加上his6-标记加上c-mys(5)tev蛋白质分解位点,所述tev蛋白质分解位点包括中央克隆位点,以便选择性收割表达的外源蛋白质。(6)全部在(8)中提及的转译的和非转译的分子功能元件统一在构建体中(seqidno:39,40)。(9)用于转运至细胞表面的最小连接肽(seqidno:41,42)。
图3示出借助clsm将融合蛋白质定位在hek293t细胞的表面上。
(a)hek293t细胞借助相应的载体共同转染。载体pcmv-cd63-yfp在定位研究中用作用于表达野生型cd63-yfp蛋白质的对照。对于tanchor技术(cd63δlel,图2b)包含的全部蛋白质,通过融合的报告蛋白质mcherry能够检测在细胞表面上的占优势的定位。没有tanchor融合体的mcherry蛋白质细胞质方面均匀地分布。未显示出在细胞表面上的优势。附加地,其他蛋白质、例如具有跨膜结构域和不具有跨膜结构域的hiv-1的糖蛋白质gp41(gp41δtm)能够呈递在表面上。(b)通过cfp和yfp在表面上定位检查蛋白质折叠。能够通过处于对于荧光特定的为405nm(cfp)或514nm(yfp)发射波长中的二极管/氩激光器激励之后检测两种荧光蛋白质。(c)通常分布式地出现在胞质溶胶中的表达的蛋白质mcherry通过tanchor系统以高的效率引导到细胞质膜中。具有实施tanchor技术的mcherry由此甚至能在hek293t细胞的丝状伪足(白色箭头)中如在野生型cd63-yfp中那样检测到。比例尺的长度在图3a,图3b中=10μm或在图3c中=1μm。
图4示出表位的细胞外定向检查的示意图
借助扫描电子显微镜(rem)在转染的hek293t的表面上执行表位定向检测。蛋白质借助初级抗体结合并且借助共轭的次级抗体用黄金颗粒(10nm)目视化。在模型表位yfp,cfp,gp41和gp41δtm中,结合的抗体能够在表面上通过免疫黄金颗粒显像(图5)。
图5a至图5d示出黄金颗粒(10nm)在hek293t细胞的表面上的rem照片。
转染的hek293t细胞分别研究表位定向。在此,相应的表位与特异性的初级抗体结合,然后所述初级抗体借助黄金共轭的次级抗体来证实。对于具有tanchor技术的全部表位而言,细胞外定向能够归因于表位。比例尺的长度在全部附图中为300nm。
图6示出具有生成的tanchor载体的转染的hek293t细胞的蛋白质印迹分析。
转染的hek293t细胞经受蛋白质印迹分析。借助初级抗体以指向c端的报告蛋白质mcherry的方式并且指向n端的表位flag的方式来确定在拆散的细胞裂解液中表达的蛋白质。用载体pmcherry-n1的转染未显示出在与抗flag抗体孵育时的光谱线,因为该载体不包含用于flag表位的序列并且用作用于表达的蛋白质的特定的证明的对照。
图7示出用于检查在具有含v5-6xhis连接肽的内部融合位点的蛋白质转运上其他四分子交联体的构建体的示意图。
为了与四分子交联体超级家族内部的大胞外环无关地检查蛋白质转运的有效性,生成表达载体,所述表达载体包含在根据基于cd63δlel的tanchor原型构建体的第三跨膜结构域和第四跨膜结构域之间的内部的融合体v5-6xhis。内部的融合体v5-6xhis包含在ecorl和ecorv(seqidno:35,36)之间的连接肽。在n端整合flag融合体,在c端在野生型四分子交联体构建体中的荧光蛋白质yfp或在δlel-v5-6xhis构建体中的mcherry融合为报道蛋白质。
图8示出融合蛋白质在hek293t细胞的表面上的定位。
hek293t细胞与生成的载体(a)pcmv-cd9-yfp/pcmv-cd9δlel-v5-6xhis-mcherry,(b)pcmv-cd81-yfp/pcmv-cd81δlel-v5-6xhis-mcherry,(c)pcmv-cd82-yfp/pcmv-cd82δlel-v5-6xhis-mcherry和(d)pcmv-cd151-yfp/pcmv-cd151δlel-v5-6xhis-mcherry共同转染。全部表达的蛋白质能够借助报告蛋白质mcherry的荧光在细胞表面上显性地检测到。如下构建体与具有荧光蛋白质yfp的融合体的相应表达的野生型的四分子交联体共同定位,所述构建体包含具有在四分子交联体cd9,cd81,cd82和cd151的第三跨膜结构域和第四跨膜结构域(图7)之间的连接肽v5-6xhis的内部的融合体。比例尺的长度在全部照片中为10μm。
图9示出用于检测抗体的tanchor系统的应用
(a)借助tanchor系统在96孔板模板中得到elisa实验53的结果。用hrp共轭的抗v5抗体检测在hek293t细胞的细胞表面上的v5表位。tanchor蛋白质的蛋白质表达能够与浓度无关地并且显著地通过抗v5-hrp蛋白质检测。(b)在cd63的第三跨膜结构域和第四跨膜结构域(seqidno:37,38)之间的连接肽2f5-4e10de示意性图。连接肽引入到载体pcmv-cd63δlel中。(c)中和hiv-1的抗体anti-2f5和anti-4e10在每孔0.6μg的恒定的dna量转染hela细胞时引物结合到2f5表位或4e10表位上。能够显著地检测到结合的anti-2f5抗体或anti-4e10抗体,(e)表达的融合蛋白质cd63δlel-v5-6xhis以及cd63δlel-2f5-4e10能够在hek293t细胞的表面上显性地定位用来对照。比例尺的长度在全部照片中为10μm。
图10示出通过使用低丝氨酸和低精氨酸的连接肽提高效率。
(a)在dna序列和氨基酸序列水平上示出所使用的连接肽。具有两个不同的连接肽(seqidnos:41,42或seqidnos:43,44)的融合蛋白质的亚细胞定位的分析明显显示出:使用两侧置有ecorl和ecorv的限制性酶切位点产生蛋白质转运到hek293t细胞的表面的高的效率并且在细胞的所构成的丝状伪足(白色尖头)处能检测到融合蛋白质(b)。比例尺的长度在全部照片中为10μm。
具体实施方式
1.生成用于执行定位表达的基于cd63的模型载体
基于cd63的模型载体的生成基于载体pcmv-tag2b(stratagene)的使用。在该载体中,借助常规的克隆技术将相应的dna序列经由限制性酶切位点引入39。使用包含cd63的全长序列(氨基酸1-238,基因库登录号kf998086)的对照载体pcmv-cd63-yfp或称作pcmv-cd63-yfp-flag,以便借助引物ρα1-01/ρα1-02和基质载体pmcherry-n1(clontech)经由限制性酶切位点xhol和apal将所使用的红色荧光的报告蛋白质mcherry的dna序列引入。借助引物ρα1-03/ρα1-04将具有跨膜结构域tm1至tm3(氨基酸1-110,基因库登录号:kf998086)的子序列经由限制性酶切位点bamhl和pstl引入,并且紧接着借助引物ρα1-05/ρα1-06以及基质载体ppr3-n-cd6349经由ecorv和hindlll将tm4(氨基酸201-238,基因库登录号:kf998086)引入到载体pcmv-tag2b中。所产生的载体包含没有lel的cd63基因序列(pcmv-cd63δlel)(图2b)。
为了简化蛋白质的检测,经由限制性酶切位点xhol和apal,将载体pcmv-cd63-mcherry的限制性的mcherry-dna片段连接到限制性的载体pcmv-cd63δlel中。载体pcmv-cd63δlel-cfp和pcmv-cd63-yfp经由限制性酶切位点ecorl和ecorv借助引物ρα1-07/ρα1-08以及基质载体pscfp3a-c1或psyfp2-c150生成。以相同方式,载体pcmv-gp41δtm和pcmv-gp41借助包含hiv-1的gp41蛋白质的基因序列的基质载体pnl4-351和引物ρα1-09/ρα1-10或ρα1-09/ρα1-11生成。
2.转染和共聚焦激光扫描显微镜(clsm)
为了检查表达的融合蛋白质的定位,hek293t细胞中的在实例1中生成的质粒借助转染试剂转染。在此,hek293t细胞(人胚肾细胞293t)播种在ibitreat的8孔板中并且在补充有10%的胎牛血清、l-谷氨酰胺和青霉素/链霉素的dulbecco’s(杜氏)修饰的eagle培养基(伊格尔氏培养基)中培养。在大约含50%的混合液中,根据制造商说明书,借助
3.借助扫描电子显微镜对hek293t细胞的表面上的表位进行定向分析
表达的蛋白质借助扫描电子显微镜由在hek293t细胞的细胞质膜上的黄金免疫的蛋白质来证实(图4)。为此,转染的并且固定在含2%的多聚甲醛的pbs中的细胞在pbs中洗涤三次并且用具有0.5%的bsa/0.1%的明胶的pbs中阻断。蛋白质cd63δlel-cfp和cd63δlel-yfp用兔的anti-gfp(ab6556,abcam)孵育,洗涤并且用具有带有10nm的共轭的黄金颗粒的山羊抗兔的初级抗体来检测。免疫标记紧随于用戊二醛(0.05m的含2.5%戊二醛的hepes)再固定。接着制作显微标本。为此,将样本以(30%,50%,70%,90%,96%)乙醇梯度分别逐步脱水各15分钟,并在无水乙醇中放置30分钟,转移到hmds(六甲基二硅氮烷)中,从hmds中干燥并且样本表面用炭蒸镀。黄金颗粒用leo1530gemini扫描电子显微镜借助反向散射检测器(centaurus)显像。以相同的方式,gp41蛋白质借助人类anti-2f5抗体(国立卫生研究院,nih)以及利用具有共轭的iggh&l的10nm的黄金颗粒(bbinternational)的山羊抗人类抗体来进行检测(图5a至图5d)。
4.表达的荧光蛋白质的蛋白质印迹分析
为了检查蛋白质表达,对转染的hek293t细胞的细胞裂解液进行蛋白质印迹分析。在此,根据制造商说明书,具有载体的hek293t细胞在6孔模板孔板中借助
5.源自四分子交联体超级家族的基于四分子交联体的杂交蛋白质的亚细胞定位的检查
为了检查在四分子交联体超级家族内的细胞表面上的肽呈递,借助基因合成54(atg:biosynthetics)以及标准克隆方法生成根据示意图的表达载体(图7)。cd9(seqidno:19)、cd81(seqidno:21)、cd82(seqidno:23)和cd151(seqidno:25)的基因序列在载体pcmv-cd63-yfp中,或cd9δlel-v5-6xhis(seqidno:27)的、cd81δlel-v5-6xhis(seqidno:29)的、cd82δlel-v5-6xhis(seqidno:31)的和cd151δlel-v5-6xhis(seqidno:33)的基因序列在载体pcmv-cd63-mcherry中(见1:生成用于执行定位表达的基于cd63的模型载体)经由限制性酶切位点notl/xhol用cd63的四分子交联体序列来交换,并且如在段2中描述的转染和共聚焦激光扫描显微镜(clsm)中描述那样的那样,将野生型基因序列pcmv-cd9-yfp、pcmv-cd81-yfp、pcmv-cd82-yfp、pcmv-cd151-yfp的下面的表达载体以及具有内部连接肽v5-6xhispcmv-cd9δlel-v5-6xhis-mcherry、pcmv-cd81δlel-v5-6xhis-mcherry、pcmv-cd82δlel-v5-6xhis-mcherry和pcmv-cd151δlel-v5-6xhis-mcherry的大胞外环(δlel)的缺失突变转染,并且借助于clsm研究亚细胞定位(图8)。
6.将tanchor系统借助elisa方法53检测抗体的应用
借助tanchor系统融合蛋白质的高效的表面表达适合于检测在呈递的肽上的结合的抗体。作为模型,连接肽v5-6xhis经由ecorl和ecorv从构建体pcmv-cd9δlel-v5-6xhis-mcherry中分离,并且经由标准的克隆方法如在段落1中用于执行定位表达的基于cd63的模型载体的生成中描述的那样嵌入到载体pcmv-cd63δlel-mcherry中。同样地,表位2f5和4e10(hiv-1)借助引物ρα1-12/ρα1-13和基质载体pnl4-3引入到载体pcmv-cd63δlel-mcherry中(图9b)。为了检测在hek293t(图9a)的表面上的结合的抗体,,为此,hek293t细胞或(图9c)hela细胞以播种在96孔板模板中的方式,用质粒pcmv-cd63δlel-v5-6xhis-mcherry(图9a)或pcmv-cd63δlel-2f5-4e10-mcherry(图9c)借助
7.通过在tanchor系统中使用两侧置有ecorl和ecorv的限制性酶切位点来提高蛋白质转运到人类细胞的表面的效率
在tanchor系统中使用两侧置有ecorl和ecorv的限制性酶切位点提高至细胞表面的转运效率。将其他限制性酶切位点引入最小连接肽(lqefdigggg)中引起在cd63的第三跨膜结构域和第四跨膜结构域之间的连接肽(gssgrrslqgggg)中的丝氨酸和精氨酸的转译(图10a)。具有其他酶切位点的载体通过基因合成54(atg:biosynthetics)产生并且在hek293t细胞中,,如在段落2转染和共聚焦激光扫描显微镜(clsm)描述的那样,引起表达并且研究亚细胞定位(图10b)。
序列表
seqidno:1:pa4e211(cd63δlel)的dna序列
seqidno:2:cd63δlel的dna序列
seqidno:3:cd63δlel的蛋白质序列
seqidno:4:引物pa1-01
seqidno:5:引物pa-02
seqidno:6:引物pa1-03
seqidno:7:引物pa1-04
seqidno:8:引物pa1-05
seqidno:9:引物pa1-06
seqidno:10:引物pa1-07
seqidno:11:引物pa1-08
seqidno:12:引物pa1-09
seqidno:13:引物pa1-10
seqidno:14:引物pa1-11
seqidno:15:引物pa1-12
seqidno:16:引物pa1-13
seqidno:17:cd63,基因库登录号kf998086
seqidno:18:cd63的蛋白质序列
seqidno:19:cd9的dna序列(基因库登录号nm_001769.3)
seqidno:20:cd9的蛋白质序列
seqidno:21:cd81的dna序列(基因库登录号nm_004356.3)
seqidno:22:cd81的蛋白质序列
seqidno:23:cd82的dna序列(基因库登录号nm_002231.3)
seqidno:24:cd82的蛋白质序列
seqidno:25:cd151的dna序列(基因库登录号bt007397.1)
seqidno:26:cd151的蛋白质序列
seqidno:27:cd9δlel-v5-6xhis的dna序列
seqidno:28:cd9δlel-v5-6xhis的蛋白质序列
seqidno:29:cd81δlel-v5-6xhis的dna序列
seqidno:30:cd81δlel-v5-6xhis的蛋白质序列
seqidno:31:cd82δlel-v5-6xhis的dna序列
seqidno:32:cd82δlel-v5-6xhis的蛋白质序列
seqidno:33:cd151δlel-v5-6xhis的dna序列
seqidno:34:cd151δlel-v5-6xhis的蛋白质序列
seqidno:35:v5-6xhis连接肽的dna序列
seqidno:36:v5-6xhis连接肽的蛋白质序列
seqidno:37:2f5-4e10-连接肽的dna序列
seqidno:38:2f5-4e10-连接肽的蛋白质序列
seqidno:39:连接肽版本(8)的dna序列
seqidno:40:连接肽版本(8)的蛋白质序列
seqidno:41:在载体pcmv-cd63δlel-mcherry中的连接肽(最小连接肽(9))的dna序列
seqidno:42:在载体pcmv-cd63δlel-mcherry中的连接肽(最小连接肽(9))的蛋白质序列
seqidno:43:在载体pcmv-cd63δlel-v2-mcherry中的连接肽的dna序列
seqidno:44:在载体pcmv-cd63δlel-v2-mcherry中的连接肽的蛋白质序列
seqidno:45:在cd63δlel和荧光蛋白质之间的连接肽的dna序列
seqidno:46:在cd63δlel和荧光蛋白质之间的连接肽的蛋白质序列
在下文中,总结本发明的优选的方面:
1.一种用于使用具有预期的人造的氨基酸顺序的方法,所述氨基酸顺序间接地、至少部分地由基质分子编码或完全是合成的,所述氨基酸顺序
a.能够至少部分地由四分子交联体的蛋白质种类的成员的序列顺序,例如人类基因cd63,cd9,cd82,cd81,cd151、cd53构成
b.但也能够至少部分地由其他生物体的序列同源的四分子交联体构成并且
c.根据a.和b.,其融合产物产生分子,所述分子能够将与其连接的天然的或合成的肽单元或蛋白质单元在适合的系统中
i.指引至一个或多个表面,所述肽单元或所述蛋白质单元
ii.在表面之上转运,并且所述肽单元或所述蛋白质单元
iii.牢固地锚定在表面中
iv.所述肽单元或所述蛋白质单元持续地呈递在表面上。
2.具有根据1.的天然的和/或人造的氨基酸成分的嵌合的蛋白质分子,所述蛋白质分子间接地用于生成蛋白质单元,所述蛋白质单元
a.在表面上修饰,例如糖基化,和/或
b.能够在表面上占据功能构型。
3.具有根据1.和2.的分子,所述分子能够间接地经由编码的基质产生并且所述分子能够从编码的基质中的至少一个的至少子单元的融合体中产生,并且所述分子能够由不同的序列的组合中
a.在体内
b.在体外
产生并且必要时富集并且分离,必要时也在此期间能够被工艺处理。
4.具有根据1.和2.和3.的分子,所述分子是编码的核酸分子,所述核酸分子根据本发明在体内和在内外
i.在体内重组,内生的基因组,(t7系统)或
ii.在体内借助染色体外遗传元件(载体)或
iii.在体内借助天然存在的或非天然存在的核酸分子(rna等)借助环状的或线状的核酸分子或
iv.在体外在耦联的转录或转译系统中
v.在体内在构建人造的氨基酸条件下
表达。
5.用于锚定根据1.至3.的分子的表面,所述表面是天然细胞的天然存在的细胞膜或人造的膜系统。
6.根据1.至4.的分子,所述分子、其基因产物、转染的细胞或细胞的一部分用于:解决关于不同的分子种类(小分子(活性物质数据库,天然材料))、rna、蛋白质和dna)的特异性结合和/或催化作用方面在生物测定法中的结构、生物分析和(兽医)诊断方面提出的问题,并且在该结果基础上建立生物分析系统(bioassay)、诊断(必要时伴随治疗的)和或治疗。
7.根据1.至4.的分子,所述分子、其基因产物、转染的细胞或细胞的一部分用于解决在免疫分析和免疫诊断领域中提出的问题,用于解决免疫分析和诊断提出的问题并且必要时在此基础上建立治疗。
8.根据1.至4.的分子,所述分子、其基因产物、转染的细胞或细胞的一部分用于开发高通量分析系统或运行所述高通量分析系统。
9.根据1.至4.的分子,所述分子、其基因产物、转染的细胞或细胞的一部分直接地或间接地用于开发免疫治疗,所述免疫治疗直接地使用(多克隆抗体或单克隆抗体或具有类似利用可能性的人造的分子)。
10.根据1.至4.的分子,所述分子、其基因产物、转染的细胞或细胞的一部分大量地从发酵/生物生产中提供给第三方来利用和使用。
参考文献
1berggard,t.,linse,s.&james,p.methodsforthedetectionandanalysisofprotein-proteininteractions.proteomics7,,2833-2842,sdoi:10.1002/pmic.200700131(2007)。
2glick,b.s.&malhotra,v.thecuriousstatusofthegolgiapparatus.cell95,883-889(1998)。
3call,m.e.&wucherpfennig,k.w.thetcellreceptor:criticalroleofthemembraneenvironmentinreceptorassemblyandfunction.annulorevimmunol23,101-125,doi:10.1146/annurev.immunol.23.021704.115625(2005)。
4sachs,j.n.&engelman,d.m.introductiontothemembraneproteinreviews:theinterplayofstructure,dynamics,andenvironmentinmembraneproteinfunction.annurevbiochem75,707-712,i5doi:10.1146/annurev.biochem.75.110105.142336(2006)。
5macher,b.a.¥,t.-y.proteinsatmembranesurfaces-areviewofapproaches.molecularbiosystems3,705-713,doi:10.1039/b708581h(2007)。
6lodish,h.f.molecularcellbiology.(w.h.freeman,,2013)。
7nicolson,g.l.thefluid-mosaicmodelofmembranestructure:stillrelevanttounderstandingthestructure,functionanddynamicsofbiologicalmembranesaftermorethan40years.biochimbiophysacta1838,1451-1466,doi:10.1016/j.bbamem.2013.10.019(2014)。
8whitelegge,j.p.integralmembraneproteinsandbilayerproteomics.5analchem85,2558-2568,doi:10.1021/ac303064a(2013)。9smith,g.p.filamentousfusionphage:novelexpressionvectorsthatdisplayclonedantigensonthevirionsurface.science228,1315-1317(1985)。
10crameri,r.,jaussi,r.,menz,g.&blaser,k.displayofexpression0productsofcdnalibrariesonphagesurfaces.aversatilescreeningsystemforselectiveisolationofgenesbyspecificgene-product/ligandinteraction.eurjbiochem226,53-58(1994)。
11gu,h.etal.aphagedisplaysystemforstudyingthesequencedeterminantsofproteinfolding.proteinsei4,1108-1117,doi:10.1002/pro.5560040609(1995)。
12kehoe,j.w.&kay,b.k.filamentousphagedisplayinthenewmillennium.chemrev105,4056-4072,doi:10.1021/cr000261r(2005)。
13chen,w.&georgiou,g.cell-surfacedisplayofheterologousproteins:fromhigh-throughputscreeningtoenvironmentalapplications.biotechnolbioeng79,496-503,doi:10.1002/bit.10407(2002)。
14hoischen,c.etal.novelbacterialmembranesurfacedisplaysystemusingcellwall-lessl-formsofproteusmirabilisandescherichiacoli.applenvironmicrobiol68,525-531(2002)。
15lee,s.y.,choi,j.h.&xu,z.microbialcell-surfacedisplay.trendsbiotechnol21,45-52(2003)。
16rutherford,n.&mourez,m.surfacedisplayofproteinsbygramnegativebacterialautotransporters.microbcellfact5,22,doi:10.1186/1475-2859-5-22(2006)。
17yang,z.,liu,q.,wang,q.&zhang,y.novelbacterialsurfacedisplaysystemsbasedonoutermembraneanchoringelementsfromthemarinebacteriumvibrioanguillarum.applenvironmicrobiol74,4359-4365,doi:10.1128/aem.02499-07(2008)。
18vanbloois,e.,winter,r.t.,kolmar,h.&fraaije,m.w.decoratingmicrobes:surfacedisplayofproteinsonescherichiacoli.trendsbiotechnol29,79-86,doi:10.1016/j.tibtech.2010.11.003(2011)。
19nicolay,t.,vanderleyden,j.&spaepen,s.autotransporter-basedcellsurfacedisplayingram-negativebacteria.critrevmicrobiol,doi:10.3109/1040841x.2013.804032(2013)。
20han,m.-j.&lee,s.h.anefficientbactenalsurfacedisplaysystembasedonanoveloutermembraneanchoringelementfromtheescherichiacoliproteinyiat.(2014)。
21boublik,y.,dibonito,p.&jones,i.m.eukaryoticvirusdisplay:engineeringthemajorsurfaceglycoproteinoftheautographacalifornicanuclearpolyhedrosisvirus(acnpv)forthepresentationofforeignproteinsonthevirussurface.biotechnology(ny)13,1079-1084(1995)。
22grabherr,r.,ernst,w.,doblhoff-dier,o.,sara,m.&katinger,h.expressionofforeignproteinsonthesurfaceofautographacalifornicanuclearpolyhedrosisvirus.biotechniques22,730-735(1997)。
23kim,s.y.,sohn,j.h.,pyun,y.r.&choi,e.s.acellsurfacedisplaysystemusingnovelgpi-anchoredproteinsinhansenulapolymorpha.yeast19,1153-1163,doi:10.1002/yea.911(2002)。
24rahman,m.m.&gopinathan,k.p.bombyxmoriionucleopolyhedrovirus-basedsurfacedisplaysystemforrecombinantproteins.jgenvirol84,2023-2031(2003)。
25baneyx,f.&mujacic,m.recombinantproteinfoldingandmisfoldinginescherichiacoli.natbiotechnol22,1399-1408,doi:10.1038/nbt1029(2004)。
26raty,j.k.etal.enhancedgenedeliverybyavidin-displayingbaculovirus.molther9,282-291,doi:10.1016/j.ymthe.2003.11.004(2004)。
27mao,h.,song,j.,liang,c,yu,z.&chen,x.constructionofeukaryoticsurfacedisplaybasedonthebaculoviralfprotein.obiotechniques41,266,268,270passim(2006)。
28weerapana,e.&imperiali,b.asparagine-iinkedproteinglycosylation:fromeukaryotictoprokaryoticsystems.glycobiology16,91r-101r,doi:10.1093/glycob/cwj099(2006)。
29wang,q.,li,l,chen,m.,qi,q.&wang,p.g.constructionofa5novelsystemforcellsurfacedisplayofheterologousproteinsonpichiapastoris.biotechnollett29,1561-1566,doi:10.1007/s10529-007-9430-6(2007)。
30chesnut,j.d.etal.selectiveisolationoftransientlytransfectedcellsfromamammaliancellpopulationwithvectorsexpressingamembraneoanchoredsingle-chainantibody.jimmunolmethods193,17-27(1996)。
31ellmark,p.,ohlin,m.,borrebaeck,c.a.&furebring,c.anovelmammaliandisplaysystemfortheselectionofprotein-proteininteractionsbydecoyreceptorengagement.jmolrecognit17,316-322,doi:10.1002/jmr.678(2004)。
32zhou,c,jacobsen,f.w.,cai,l.,chen,q.&shen,w.d.developmentofanovelmammaliancellsurfaceantibodydisplayplatform.mabs2,508-518,doi:10.4161/mabs.2.5.12970(2010)。
33tomimatsu,k.etal.arapidscreeningandproductionmethodusinganovelmammaliancelldisplaytoisolatehumanmonoclonalantibodies.biochembiophysrescommuri441,59-64,doi:10.1016/j.bbrc.2013.10.007(2013)。
34stipp,c.s.,kolesnikova,t.v.&hemler,m.e.functionaldomainsintetraspaninproteins.trendsbiochemsci28,106-112(2003)。
35kovalenko,o.v.,metealf,d.g.,degrado,w.f..&hemler,m.e.structuralorganizationandinteractionsoftransmembranedomainsintetraspaninproteins.bmcstructbiol5,11,doi:10.1186/1472-6807-5-1(2005)。
36hemler,m.e.targetingoftetraspaninproteins-potentialbenefitsandstrategies.natrevdrugdiscov7,747-758,doi:10.1038/nrd2659(2008)。
37levy,s.&shoham,t.thetetraspaninwebmodulatesimmune-signallingcomplexes.natrevimmunol5,136-148,doi:10.1038/nri1548(2005)。
38rubinstein,e.etal.cd9,cd63,cd81,andcd82arecomponentsofasurfacetetraspannetworkconnectedtohla-drandvlaintegrins.eurjimmunol26,2657-2665,doi:10.1002/eji.1830261117(1996)。
39green,m.r.&sambrook,j.molecularcloning:alaboratorymanual.1.(coldspringharborlaboratorypress,2012)。
40rognan,d.rationaldesignofprotein-proteininteractioninhibitors.medchemcomm6,51-60,doi:10.1039/c4md00328d(2015)。
41jin,l,wang,w.&fang,g.targetingprotein-proteininteractionbysmallmolecules.annurevpharmacoltoxicol54,435-456,doi:10.1146/annurev-pharmtox-011613-140028(2014)。
42milroy,l.g.,grossmann,t.n.,hennig,s.,brunsveld,l.&ottmann,c.modulatorsofprotein-proteininteractions.chemrev114,4695-4748,doi:10.1021/cr400698c(2014)。
43rao,v.s.,srinivas,k.,sujini,g.n.&kumar,g.n.protein-proteininteractiondetection:methodsandanalysis.intjproteomics2014,147648,doi:10.1155/2014/147648(2014)。
44arkin,m.r.&wells,j.a.small-moleculeinhibitorsofprotein-proteininteractions:progressingtowardsthedream.natrevdrugdiscov3,301-317,doi:10.1038/nrd1343(2004)。
45jones,s.þton,j.m.principlesofprotein-proteininteractions.procnatlacadsciusa93,13-20(1996)。
46jones,s.þton,j.m.protein-proteininteractions:areviewofproteindimerstruetures.progbiophysmolbiol63,31-65(1995)。
47sudhof,t.c.thesynapticvesiclecycle:acascadeofprotein-proteininteractions.nature375,645-653,doi:10.1038/375645a0(1995)。
48mullard,a.protein-proteininteractioninhibitorsgetintothegroove.natrevdrugdiscov11,173-175,doi:10.1038/nrd3680(2012)。
49ivanusic,d.,eschricht,m.&denner,j.investigationofmembraneprotein-proteininteractionsusingcorrelativefret-pla.biotechniques57,188-198,doi:10.2144/000114215(2014)。
50kremers,g.j.,goedhart,j.,vanmunster,e.b.&gadella,t.w.,jr.2ocyanandyellowsuperfluorescentproteinswithimprovedbrightness,proteinfolding,andfretforsterradius.biochemistry45,6570-6580,doi:10.1021/bi0516273(2006)。
51adachi,a.etal.productionofacquiredimmunodeficiencysyndrome-associatedretrovirusinhumanandnonhumancellstransfectedwithan5infectiousmolecularclone.jvirol59,284-291(1986)。
52stephaniecharrin,frangoislenaour,oliviersilvie,pierre-emmanuelmilhiet,claudeboucheix,ericrubinstein,lateralorganizationofmembraneproteins:tetraspaninsspintheirweb,bioch.j.(2009),420(2)133-154;doi:10.1042/bj20082422。
53avoller,abartlett,debidwell,enzymeimmunoassayswithspecialreferencetoelisatechniques,jclinpathoi,1978june,31(6):507-520。
54hughesra,miklosae,ellingtonad,genesynthesis:methodsandapplications,methodsenzymol.2011;498::277-309,doi:10.2016/b978-0-12-385120-8.00012-7。
sequencelisting
<110>发展生物工程与基因技术的彼得和特劳德尔·恩格尔霍恩基金会
<120>用于将肽呈递在细胞表面上的系统
<130>61317pwo
<150>de102015002851
<151>2015-03-05
<160>46
<170>patentinversion3.5
<210>1
<211>4832
<212>dna
<213>artificialsequence
<220>
<223>pa4e211(cd63dellel);kuenstlichesequenzalsshuttlevektorvone.
colinacheukaryotischenzellen(hier:saeugetierzellen)
<400>1
cctcgaggaaggctactacgcctaagctaccgacaggttggctgataagtccccggtctc60
aaggcgcgcctagttattaatagtaatcaattacggggtcattagttcatagcccatata120
tggagttccgcgttacataacttacggtaaatggcccgcctggctgaccgcccaacgacc180
cccgcccattgacgtcaataatgacgtatgttcccatagtaacgccaatagggactttcc240
attgacgtcaatgggtggagtatttacggtaaactgcccacttggcagtacatcaagtgt300
atcatctgccaagtacgccccctattgacgtcaatgacggtaaatggcccgcctggcatt360
atgcccagtacatgaccttatgggactttcctacttggcagtacatctacgtattagtca420
tcgctattaccacggtgatgcggttttggcagtacatcaatgggcgtggatagcggtttg480
actcacggggatttccaagtctccaccccattgacgtcaatgggagtttgttttggcacc540
aaaatcaacgggactttccaaaatgtcgtaacaactccgccccattgacgcaaatgggcg600
gtaggcgtgtacggtgggaggtctatataagcagagctctctggctaactagagaaccca660
ctgcttactggcttatcgaaattaatacgactcactataggaagccgccacctgctagca720
atttatagccaccatggattacaaggatgacgacgataagcccatggcggtggaaggagg780
aatgaaatgtgtgaagttcttgctctacgtcctcctgctggccttttgcgcctgtgcagt840
gggactgattgccgtgggtgtcggggcacagcttgtcctgagtcagaccataatccaggg900
ggctacccctggctctctgttgccagtggtcatcatcgcagtgggtgtcttcctcttcct960
ggtggcttttgtgggctgctgcggggcctgcaaggagaactattgtcttatgattacgtt1020
tgccatctttctgtctcttatcatgttggtggaggtggccgcagccattgctggctatgt1080
gtttagagataagctgcaagaattcgagaatctgtactttcagtcgaagcttggatccct1140
gcagcatcaccatcaccatcacgaaaacctctatttccagagcgaacagaaactgatatc1200
cgaagaagatctggatatcggaggcggaggcaaaaatgtgctggtggtagctgccgcagc1260
ccttggaattgcttttgtcgaggttttgggaattgtctttgcctgctgcctcgtgaagag1320
tatcagaagtggctacgaggtgatgaaacttatcgataccgtcgatctccatatgccagc1380
catgaagatagaatgcaggattacaggcaccctgaatggcgtggagttcgagctggtcgg1440
gggtggagaaggaacacctgagcagggccgaatgacgaacaagatgaagagcaccaaagg1500
agccctgacatttagcccctatctcctctctcatgttatgggctacggcttttatcactt1560
cgggacttacccaagcggatatgaaaaccctttccttcacgccataaacaatgggggtta1620
caccaacacccggattgagaaatatgaggacggtggcgtgttgcatgtctccttcagcta1680
cagatatgaggctggtcgcgtgatcggagattttaaggttgtgggcacaggctttccaga1740
agactccgtaatcttcaccgacaagatcattcggtctaatgccacggtggagcatcttca1800
ccctatgggcgataatgtcctcgtcgggagctttgccagaactttttcactgagggatgg1860
gggatactattcattcgtcgtagactcccacatgcactttaaatccgcaatccacccatc1920
aatcctgcaaaacggagggcctatgtttgcgttcagacgtgtagaggagctgcattctaa1980
cactgaactcggaatcgttgaataccagcacgctttcaaaactcccattgcattcgctgg2040
gccctgatcatgagagtttaaacataaagcctcgactgtgccttctagttgccagccatc2100
tgttgtttgcccctcccccgtgccttccttgaccctggaaggtgccactcccactgtcct2160
ttcctaataaaatgaggaaattgcatcgcattgtctgagtaggtgtcattctattctggg2220
gggtggggtggggcaggacagcaagggggaggattgggaagacaatagcaggcatgctgg2280
ggatgcggtgggctctatggaaaaaacgcgcgcggatataatataacttcgtataatgta2340
tgctatacgaagttatatataatgcgtcgacgcactcaaccctatctcggtctattcttt2400
tgatttataagggattttgccgatttcggcctattggttaaaaaatgagctgatttaaca2460
aaaatttaacgcgaattttaacaaaatattaacgcttacaatttaggtggcacttttcgg2520
ggaaatgtgcgcggaacccctatttgtttatttttctaaatacattcaaatatgtatccg2580
ctcatgagacaataaccctgataaatgcttcaataatattgaaaaaggaagagtatgagt2640
attcaacatttccgtgtcgcccttattcccttttttgcggcattttgccttcctgttttt2700
gctcacccagaaacgctggtgaaagtaaaagatgctgaagatcagttgggtgcccgagtg2760
ggttacatcgaactggatctcaacagcggtaagatccttgagagttttcgccccgaagaa2820
cgttttccaatgatgagcacttttaaagttctgctatgtggcgcggtattatcccgtatt2880
gacgccgggcaagagcaactcggtcgccgcatacactattctcagaatgacttggttgag2940
tattcaccagtcacagaaaagcatcttacggatggcatgacagtaagagaattatgcagt3000
gctgccataaccatgagtgataacactgcggccaacttacttctgacaacgattggagga3060
ccgaaggagctaaccgcttttttgcacaacatgggggatcatgtaactcgccttgatcgt3120
tgggaaccggagctgaatgaagccataccaaacgacgagcgtgacaccacgatgcctgta3180
gcaatggcaacaacgttgcgtaaactattaactggcgaactacttactctagcttcccgg3240
caacaattgatagactggatggaggcggataaagttgcaggaccacttctgcgctcggcc3300
cttccggctggctggtttattgctgataaatctggagccggtgagcgtggttctcgcggt3360
atcattgcagcactggggccagatggtaagccctcccgtatcgtagttatctacacgacg3420
gggagtcaggcaactatggatgaacgaaatagacagatcgctgagataggtgcctcactg3480
attaagcattggtaactgtcagaccaagtttactcatatatactttagattgatttaaaa3540
cttcatttttaatttaaaaggatctaggtgaagatcctttttgataatcttatgaccaaa3600
atcccttaacgtgagttttcgttccactgagcgtcagaccgcgatcgcagcagaccgggg3660
acttatcagccaacctgtcgttaattaaccatgtcagccgttaagtgttcctgtgtcact3720
caaaattgctttgagaggctctaagggcttctcagtgcgttacatccctggcttgttgtc3780
cacaaccgttaaaccttaaaggctttaaaagccttatatattcttttttttcttataaaa3840
cttaaaaccttagaggctatttaagttgctgatttatattaattgaagagcattatacca3900
aaatcccttaacgtgagttttcgttccactgagcgtcagaccccgtagaaaagatcaaag3960
gatcttcttgagatcctttttttctgcgcgtaatatgctgcttgcaaacaaaaaaaccac4020
cgctaccagcggtggtttgtttgccggatcaagagctaccaactctttttccgaaggtaa4080
ctggcttcagcagagcgcagataccaaatactgtccttctagtgtagccgtagttaggcc4140
accacttcaagaactctgtagcaccgcctacatacctcgctctgctaatcctgttaccag4200
tggctgctgccagtggcgataagtcgtgtcttaccgggttggactcaagacgatagttac4260
cggataaggcgcagcggtcgggctgaacggggggttcgtgcacacagcccagcttggagc4320
gaacgacctacaccgaactgagatacctacagcgtgagctatgagaaagcgccacgcttc4380
ccgaagggagaaaggcggacaggtatccggtaagcggcagggtcggaacaggagagcgca4440
cgagggagcttccagggggaaacgcctggtatctttatagtcctgtcgggtttcgccacc4500
tctgacttgagcgtcgatttttgtgatgctcgtcaggggggcggagcctatggaaaaacg4560
ccagcaacgcggcctttttacggttcctggccttttgctggccttttgctcatattagct4620
cttcaaattttattgttcaaacatgagagcttagtacgtgaaacatgagagcttagtacg4680
ttagccatgagagcttagtacgttagccatgagggtttagttcgttaaacatgagagctt4740
agtacgttaaacatgagagcttagtacgtgaaacatgagagcttagtacgtactatcaac4800
aggttgaactgctgatcttcagatcatataat4832
<210>2
<211>474
<212>dna
<213>artificialsequence
<220>
<223>synthetischednasequenzdeshumanencd63gens(cd63dellel);
eukaryotischezellen,hier:s鋟getierzellen);kodierendesequenz
vonseqidno:1;synthetischescd63-codonen
<220>
<221>misc_feature
<222>(1)..(330)
<223>tm1-tm3
<220>
<221>misc_feature
<222>(331)..(360)
<223>minimallinker
<220>
<221>misc_feature
<222>(361)..(474)
<223>tm4
<400>2
atggcggtggaaggaggaatgaaatgtgtgaagttcttgctctacgtcctcctgctggcc60
ttttgcgcctgtgcagtgggactgattgccgtgggtgtcggggcacagcttgtcctgagt120
cagaccataatccagggggctacccctggctctctgttgccagtggtcatcatcgcagtg180
ggtgtcttcctcttcctggtggcttttgtgggctgctgcggggcctgcaaggagaactat240
tgtcttatgatcacgtttgccatctttctgtctcttatcatgttggtggaggtggccgca300
gccattgctggctatgtgtttagagataagctgcaggaattcgatatcggaggaggagga360
aaaaatgtgctggtggtagctgcagcagcccttggaattgcttttgtcgaggttttggga420
attgtctttgcctgctgcctcgtgaagagtatcagaagtggctacgaggtgatg474
<210>3
<211>158
<212>prt
<213>artificialsequence
<220>
<223>cd63dellel;k黱stlichesequenzimshuttlevektor-e.coli-
baculovirus/insektenzellen;translatiertesequenzvonseqid
no:2
<220>
<221>misc_feature
<222>(1)..(110)
<223>tm1-tm3
<220>
<221>misc_feature
<222>(111)..(120)
<223>minimallinker
<220>
<221>misc_feature
<222>(121)..(158)
<223>tm4
<400>3
metalavalgluglyglymetlyscysvallyspheleuleutyrval
151015
leuleuleualaphecysalacysalavalglyleuilealavalgly
202530
valglyalaglnleuvalleuserglnthrileileglnglyalathr
354045
proglyserleuleuprovalvalileilealavalglyvalpheleu
505560
pheleuvalalaphevalglycyscysglyalacyslysgluasntyr
65707580
cysleumetilethrphealailepheleuserleuilemetleuval
859095
gluvalalaalaalailealaglytyrvalpheargasplysleugln
100105110
glupheaspileglyglyglyglylysasnvalleuvalvalalaala
115120125
alaalaleuglyilealaphevalgluvalleuglyilevalpheala
130135140
cyscysleuvallysserileargserglytyrgluvalmet
145150155
<210>4
<211>30
<212>dna
<213>artificialsequence
<220>
<223>pa1-01;konstruktivs.patenttext
<400>4
tttttctcgagatggtgagcaagggcgagg30
<210>5
<211>32
<212>dna
<213>artificialsequence
<220>
<223>pa1-02;konstruktivs.patenttext
<400>5
tttttgggcccatcttgtacagctcgtccatg32
<210>6
<211>33
<212>dna
<213>artificialsequence
<220>
<223>pa1-03;konstruktivs.patenttext
<400>6
ttttttggatccatggcggtggaaggaggaatg33
<210>7
<211>27
<212>dna
<213>artificialsequence
<220>
<223>pa1-04;konstruktivs.patenttext
<400>7
tttttctgcagcttatctctaaacaca27
<210>8
<211>36
<212>dna
<213>artificialsequence
<220>
<223>pa1-05;konstruktivs.patenttext
<400>8
tttttgatatcggaggaggaggaaaaaatgtgctgg36
<210>9
<211>33
<212>dna
<213>artificialsequence
<220>
<223>pa1-06;konstruktivs.patenttext
<400>9
ttttttaagcttcatcacctcgtagccacttct33
<210>10
<211>31
<212>dna
<213>artificialsequence
<220>
<223>pa1-07;konstruktivs.patenttext
<400>10
tttttgaattcatggtgagcaagggcgagga31
<210>11
<211>31
<212>dna
<213>artificialsequence
<220>
<223>pa1-08;konstruktivs.patenttext
<400>11
tttttgatatcctttctgagtccggacttgt31
<210>12
<211>30
<212>dna
<213>artificialsequence
<220>
<223>pa1-09;konstruktivs.patenttext
<400>12
tttttgaattcctgacggtacaggccagac30
<210>13
<211>31
<212>dna
<213>artificialsequence
<220>
<223>pa1-10;konstruktivs.patenttext
<400>13
tttttgatatcgttaaaccaattccacaaac31
<210>14
<211>30
<212>dna
<213>artificialsequence
<220>
<223>pa1-11;konstruktivs.patenttext
<400>14
tttttgatatcctgcctaactctattcact30
<210>15
<211>30
<212>dna
<213>artificialsequence
<220>
<223>pa1-12;konstruktivs.patenttext
<400>15
tttttgaattcattgaagaatcgcaaaacc30
<210>16
<211>31
<212>dna
<213>artificialsequence
<220>
<223>pa1-13;konstruktivs.patenttext
<400>16
tttttgatatcttttatataccacagccaat31
<210>17
<211>714
<212>dna
<213>homosapiens
<220>
<221>misc_feature
<222>(1)..(3)
<223>startkodon
<400>17
atggcggtggaaggaggaatgaaatgtgtgaagttcttgctctacgtcctcctgctggcc60
ttttgcgcctgtgcagtgggactgattgccgtgggtgtcggggcacagcttgtcctgagt120
cagaccataatccagggggctacccctggctctctgttgccagtggtcatcatcgcagtg180
ggtgtcttcctcttcctggtggcttttgtgggctgctgcggggcctgcaaggagaactat240
tgtcttatgatcacgtttgccatctttctgtctcttatcatgttggtggaggtggccgca300
gccattgctggctatgtgtttagagataaggtgatgtcagagtttaataacaacttccgg360
cagcagatggagaattacccgaaaaacaaccacactgcttcgatcctggacaggatgcag420
gcagattttaagtgctgtggggctgctaactacacagattgggagaaaatcccttccatg480
tcgaagaaccgagtccccgactcctgctgcattgatgttactgtgggctgtgggattaat540
ttcaacgagaaggcgatccataaggagggctgtgtggagaagattgggggctggctgagg600
aaaaatgtgctggtggtagctgcagcagcccttggaattgcttttgtcgaggttttggga660
attgtctttgcctgctgcctcgtgaagagtatcagaagtggctacgaggtgatg714
<210>18
<211>238
<212>prt
<213>homosapiens
<220>
<221>misc_feature
<222>(1)..(238)
<223>cd63;translatiertesequenzzuseqidno:17(genbankaccession
no:kf998086)
<400>18
metalavalgluglyglymetlyscysvallyspheleuleutyrval
151015
leuleuleualaphecysalacysalavalglyleuilealavalgly
202530
valglyalaglnleuvalleuserglnthrileileglnglyalathr
354045
proglyserleuleuprovalvalileilealavalglyvalpheleu
505560
pheleuvalalaphevalglycyscysglyalacyslysgluasntyr
65707580
cysleumetilethrphealailepheleuserleuilemetleuval
859095
gluvalalaalaalailealaglytyrvalpheargasplysvalmet
100105110
serglupheasnasnasnpheargglnglnmetgluasntyrprolys
115120125
asnasnhisthralaserileleuaspargmetglnalaaspphelys
130135140
cyscysglyalaalaasntyrthrasptrpglulysileprosermet
145150155160
serlysasnargvalproaspsercyscysileaspvalthrvalgly
165170175
cysglyileasnpheasnglulysalailehislysgluglycysval
180185190
glulysileglyglytrpleuarglysasnvalleuvalvalalaala
195200205
alaalaleuglyilealaphevalgluvalleuglyilevalpheala
210215220
cyscysleuvallysserileargserglytyrgluvalmet
225230235
<210>19
<211>771
<212>dna
<213>homosapiens
<220>
<221>misc_feature
<222>(18)..(21)
<223>startkodon
<400>19
gctagcgcggccgccaccatggattacaaggatgacgacgataagagcccgggcggatcc60
atgccggtcaaaggaggcaccaagtgcatcaaatacctgctgttcggatttaacttcatc120
ttctggcttgccgggattgctgtccttgccattggactatggctccgattcgactctcag180
accaagagcatcttcgagcaagaaactaataataataattccagcttctacacaggagtc240
tatattctgatcggagccggcgccctcatgatgctggtgggcttcctgggctgctgcggg300
gctgtgcaggagtcccagtgcatgctgggactgttcttcggcttcctcttggtgatattc360
gccattgaaatagctgcggccatctggggatattcccacaaggatgaggtgattaaggaa420
gtccaggagttttacaaggacacctacaacaagctgaaaaccaaggatgagccccagcgg480
gaaacgctgaaagccatccactatgcgttgaactgctgtggtttggctgggggcgtggaa540
cagtttatctcagacatctgccccaagaaggacgtactcgaaaccttcaccgtgaagtcc600
tgtcctgatgccatcaaagaggtcttcgacaataaattccacatcatcggcgcagtgggc660
atcggcattgccgtggtcatgatatttggcatgatcttcagtatgatcttgtgctgtgct720
atccgcaggaaccgcgagatggtcaagcttatcgataccgtcgacctcgag771
<210>20
<211>257
<212>prt
<213>homosapiens
<220>
<221>misc_feature
<222>(7)..(7)
<223>startmethionin
<220>
<221>misc_feature
<222>(7)..(257)
<223>cd9;translatiertesequenzzuseqidno:19(genbankaccessionno:
nm_001769.3)
<400>20
alaseralaalaalathrmetasptyrlysaspaspaspasplysser
151015
proglyglysermetprovallysglyglythrlyscysilelystyr
202530
leuleupheglypheasnpheilephetrpleualaglyilealaval
354045
leualaileglyleutrpleuargpheaspserglnthrlysserile
505560
phegluglngluthrasnasnasnasnserserphetyrthrglyval
65707580
tyrileleuileglyalaglyalaleumetmetleuvalglypheleu
859095
glycyscysglyalavalglngluserglncysmetleuglyleuphe
100105110
pheglypheleuleuvalilephealailegluilealaalaalaile
115120125
trpglytyrserhislysaspgluvalilelysgluvalglngluphe
130135140
tyrlysaspthrtyrasnlysleulysthrlysaspgluproglnarg
145150155160
gluthrleulysalailehistyralaleuasncyscysglyleuala
165170175
glyglyvalgluglnpheileseraspilecysprolyslysaspval
180185190
leugluthrphethrvallyssercysproaspalailelysgluval
195200205
pheaspasnlysphehisileileglyalavalglyileglyileala
210215220
valvalmetilepheglymetilephesermetileleucyscysala
225230235240
ileargargasnargglumetvallysleuileaspthrvalaspleu
245250255
glu
<210>21
<211>795
<212>dna
<213>homosapiens
<220>
<221>misc_feature
<222>(18)..(21)
<223>startkodon
<400>21
gctagcgcggccgccaccatggattacaaggatgacgacgataagagcccgggcggatcc60
atgggagtggagggctgcaccaagtgcatcaagtacctgctcttcgtcttcaatttcgtc120
ttctggctggctggaggcgtgatcctgggtgtggccctgtggctccgccatgacccgcag180
accaccaacctcctgtatctggagctgggagacaagcccgcgcccaacaccttctatgta240
ggcatctacatcctcatcgctgtgggcgctgtcatgatgttcgttggcttcctgggctgc300
tacggggccatccaggaatcccagtgcctgctggggacgttcttcacctgcctggtcatc360
ctgtttgcctgtgaggtggccgccggcatctggggctttgtcaacaaggaccagatcgcc420
aaggatgtgaagcagttctatgaccaggccctacagcaggccgtggtggatgatgacgcc480
aacaacgcgaaggctgtggtgaagaccttccacgagacgcttgactgctgtggctccagc540
acactgactgctttgaccacctcagtgctcaagaacaatttgtgtccctcgggcagcaac600
atcatcagcaacctcttcaaggaggactgccaccagaagatcgacgacctcttctccggg660
aagctgtacctcatcggcattgctgccatcgtggtcgctgtgatcatgatcttcgagatg720
atcctgagcatggtgctgtgctgtggcatccggaacagctccgtgtacaagcttatcgat780
accgtcgacctcgag795
<210>22
<211>265
<212>prt
<213>homosapiens
<220>
<221>misc_feature
<222>(7)..(7)
<223>startmethionin
<220>
<221>misc_feature
<222>(7)..(265)
<223>cd81;translatiertesequenzzuseqidno:21(genbankaccession
no:nm_004356.3)
<400>22
alaseralaalaalathrmetasptyrlysaspaspaspasplysser
151015
proglyglysermetglyvalgluglycysthrlyscysilelystyr
202530
leuleuphevalpheasnphevalphetrpleualaglyglyvalile
354045
leuglyvalalaleutrpleuarghisaspproglnthrthrasnleu
505560
leutyrleugluleuglyasplysproalaproasnthrphetyrval
65707580
glyiletyrileleuilealavalglyalavalmetmetphevalgly
859095
pheleuglycystyrglyalaileglngluserglncysleuleugly
100105110
thrphephethrcysleuvalileleuphealacysgluvalalaala
115120125
glyiletrpglyphevalasnlysaspglnilealalysaspvallys
130135140
glnphetyraspglnalaleuglnglnalavalvalaspaspaspala
145150155160
asnasnalalysalavalvallysthrphehisgluthrleuaspcys
165170175
cysglyserserthrleuthralaleuthrthrservalleulysasn
180185190
asnleucysproserglyserasnileileserasnleuphelysglu
195200205
aspcyshisglnlysileaspaspleupheserglylysleutyrleu
210215220
ileglyilealaalailevalvalalavalilemetilepheglumet
225230235240
ileleusermetvalleucyscysglyileargasnserservaltyr
245250255
lysleuileaspthrvalaspleuglu
260265
<210>23
<211>888
<212>dna
<213>homosapiens
<220>
<221>misc_feature
<222>(18)..(21)
<223>startkodon
<400>23
gctagcgcggccgccaccatggattacaaggatgacgacgataagagcccgggcggatcc60
atgggctcagcctgtatcaaagtcaccaaatactttctcttcctcttcaacttgatcttc120
tttatcctgggcgcagtgatcctgggcttcggggtgtggatcctggccgacaagagcagt180
ttcatctctgtcctgcaaacctcctccagctcgcttaggatgggggcctatgtcttcatc240
ggcgtgggggcagtcactatgctcatgggcttcctgggctgcatcggcgccgtcaacgag300
gtccgctgcctgctggggctgtactttgctttcctgctcctgatcctcattgcccaggtg360
acggccggggcactcttctacttcaacatgggcaagctgaagcaggagatgggtggcatc420
gtgactgagctcattcgagactacaacagcagtcgcgaggacagcctgcaggatgcctgg480
gactacgtgcaggctcaggtgaagtgctgcggctgggtcagcttctacaactggacagac540
aacgctgagctcatgaatcgccctgaggtcacctacccctgttcctgcgaagtcaagggg600
gaagaggacaacagcctttctgtgaggaagggcttctgcgaggcccccggcaacaggacc660
cagagtggcaaccaccctgaggactggcctgtgtaccaggagggctgcatggagaaggtg720
caggcgtggctgcaggagaacctgggcatcatcctcggcgtgggcgtgggtgtggccatg780
gtcgagctcctggggatggtcctgtccatctgcttgtgccggcacgtgcattccgaagac840
tacagcaaggtccccaagtacaagcttatcgataccgtcgacctcgag888
<210>24
<211>296
<212>prt
<213>homosapiens
<220>
<221>misc_feature
<222>(7)..(7)
<223>startmethionin
<220>
<221>misc_feature
<222>(7)..(296)
<223>cd82;translatiertesequenzzuseqidno:23(genbankaccession
no:nm_002231.3)
<400>24
alaseralaalaalathrmetasptyrlysaspaspaspasplysser
151015
proglyglysermetglyseralacysilelysvalthrlystyrphe
202530
leupheleupheasnleuilephepheileleuglyalavalileleu
354045
glypheglyvaltrpileleualaasplysserserpheileserval
505560
leuglnthrserserserserleuargmetglyalatyrvalpheile
65707580
glyvalglyalavalthrmetleumetglypheleuglycysilegly
859095
alavalasngluvalargcysleuleuglyleutyrphealapheleu
100105110
leuleuileleuilealaglnvalthralaglyalaleuphetyrphe
115120125
asnmetglylysleulysglnglumetglyglyilevalthrgluleu
130135140
ileargasptyrasnserserarggluaspserleuglnaspalatrp
145150155160
asptyrvalglnalaglnvallyscyscysglytrpvalserphetyr
165170175
asntrpthraspasnalagluleumetasnargprogluvalthrtyr
180185190
procyssercysgluvallysglyglugluaspasnserleuserval
195200205
arglysglyphecysglualaproglyasnargthrglnserglyasn
210215220
hisprogluasptrpprovaltyrglngluglycysmetglulysval
225230235240
glnalatrpleuglngluasnleuglyileileleuglyvalglyval
245250255
glyvalalametvalgluleuleuglymetvalleuserilecysleu
260265270
cysarghisvalhissergluasptyrserlysvalprolystyrlys
275280285
leuileaspthrvalaspleuglu
290295
<210>25
<211>846
<212>dna
<213>homosapiens
<220>
<221>misc_feature
<222>(18)..(21)
<223>startkodon
<400>25
gctagcgcggccgccaccatggattacaaggatgacgacgataagagcccgggcggatcc60
atgggtgagttcaacgagaagaagacaacatgtggcaccgtttgcctcaagtacctgctg120
tttacctacaattgctgcttctggctggctggcctggctgtcatggcagtgggcatctgg180
acgctggccctcaagagtgactacatcagcctgctggcctcaggcacctacctggccaca240
gcctacatcctggtggtggcgggcactgtcgtcatggtgactggggtcttgggctgctgc300
gccaccttcaaggagcgtcggaacctgctgcgcctgtacttcatcctgctcctcatcatc360
tttctgctggagatcatcgctggtatcctcgcctacgcctactaccagcagctgaacacg420
gagctcaaggagaacctgaaggacaccatgaccaagcgctaccaccagccgggccatgag480
gctgtgaccagcgctgtggaccagctgcagcaggagttccactgctgtggcagcaacaac540
tcacaggactggcgagacagtgagtggatccgctcacaggaggccggtggccgtgtggtc600
ccagacagctgctgcaagacggtggtggctctttgtggacagcgagaccatgcctccaac660
atctacaaggtggagggcggctgcatcaccaagttggagaccttcatccaggagcacctg720
agggtcattggggctgtggggatcggcattgcctgtgtgcaggtctttggcatgatcttc780
acgtgctgcctgtacaggagtctcaagctggagcactacaagcttatcgataccgtcgac840
ctcgag846
<210>26
<211>282
<212>prt
<213>homosapiens
<220>
<221>misc_feature
<222>(7)..(7)
<223>startkodon
<220>
<221>misc_feature
<222>(7)..(7)
<223>startmethionin
<220>
<221>misc_feature
<222>(8)..(282)
<223>cd151;translatiertesequenzzuseqidno:25(genbankaccession
no:bt007397.1)
<400>26
alaseralaalaalathrmetasptyrlysaspaspaspasplysser
151015
proglyglysermetglyglupheasnglulyslysthrthrcysgly
202530
thrvalcysleulystyrleuleuphethrtyrasncyscysphetrp
354045
leualaglyleualavalmetalavalglyiletrpthrleualaleu
505560
lysserasptyrileserleuleualaserglythrtyrleualathr
65707580
alatyrileleuvalvalalaglythrvalvalmetvalthrglyval
859095
leuglycyscysalathrphelysgluargargasnleuleuargleu
100105110
tyrpheileleuleuleuileilepheleuleugluileilealagly
115120125
ileleualatyralatyrtyrglnglnleuasnthrgluleulysglu
130135140
asnleulysaspthrmetthrlysargtyrhisglnproglyhisglu
145150155160
alavalthrseralavalaspglnleuglnglngluphehiscyscys
165170175
glyserasnasnserglnasptrpargaspserglutrpileargser
180185190
glnglualaglyglyargvalvalproaspsercyscyslysthrval
195200205
valalaleucysglyglnargasphisalaserasniletyrlysval
210215220
gluglyglycysilethrlysleugluthrpheileglngluhisleu
225230235240
argvalileglyalavalglyileglyilealacysvalglnvalphe
245250255
glymetilephethrcyscysleutyrargserleulysleugluhis
260265270
tyrlysleuileaspthrvalaspleuglu
275280
<210>27
<211>648
<212>dna
<213>artificialsequence
<220>
<223>cd9dellel-v5-6xhis;basierendaufgenbankaccessionno.:
nm_001769.3
<220>
<221>misc_feature
<222>(18)..(405)
<223>tm1-tm3
<220>
<221>misc_feature
<222>(18)..(21)
<223>startkodon
<220>
<221>misc_feature
<222>(406)..(507)
<223>v5-6xhis-linker
<220>
<221>misc_feature
<222>(508)..(648)
<223>tm4
<400>27
gctagcgcggccgccaccatggattacaaggatgacgacgataagagcccgggcggatcc60
atgccggtcaaaggaggcaccaagtgcatcaaatacctgctgttcggatttaacttcatc120
ttctggcttgccgggattgctgtccttgccattggactatggctccgattcgactctcag180
accaagagcatcttcgagcaagaaactaataataataattccagcttctacacaggagtc240
tatattctgatcggagccggcgccctcatgatgctggtgggcttcctgggctgctgcggg300
gctgtgcaggagtcccagtgcatgctgggactgttcttcggcttcctcttggtgatattc360
gccattgaaatagctgcggccatctggggatattcccacaaggatgaattcggaaagggc420
ccgcggttcgaaggtaagcctatccctaaccctctcctcggtctcgattctacgcgtacc480
ggtcatcatcaccatcaccatgatatcggaggaggaggaatcatcggcgcagtgggcatc540
ggcattgccgtggtcatgatatttggcatgatcttcagtatgatcttgtgctgtgctatc600
cgcaggaaccgcgagatggtcaagcttatcgataccgtcgacctcgag648
<210>28
<211>216
<212>prt
<213>artificialsequence
<220>
<223>cd9dellel-v5-6xhis;translatiertesequenz
<220>
<221>misc_feature
<222>(7)..(7)
<223>startmethionin
<220>
<221>misc_feature
<222>(7)..(135)
<223>tm1-tm3
<220>
<221>misc_feature
<222>(7)..(216)
<223>cd9dellel-v5-6xhis
<220>
<221>misc_feature
<222>(136)..(169)
<223>v5-6xhis-linker
<220>
<221>misc_feature
<222>(170)..(216)
<223>tm4
<400>28
alaseralaalaalathrmetasptyrlysaspaspaspasplysser
151015
proglyglysermetprovallysglyglythrlyscysilelystyr
202530
leuleupheglypheasnpheilephetrpleualaglyilealaval
354045
leualaileglyleutrpleuargpheaspserglnthrlysserile
505560
phegluglngluthrasnasnasnasnserserphetyrthrglyval
65707580
tyrileleuileglyalaglyalaleumetmetleuvalglypheleu
859095
glycyscysglyalavalglngluserglncysmetleuglyleuphe
100105110
pheglypheleuleuvalilephealailegluilealaalaalaile
115120125
trpglytyrserhislysaspglupheglylysglyproargpheglu
130135140
glylysproileproasnproleuleuglyleuaspserthrargthr
145150155160
glyhishishishishishisaspileglyglyglyglyileilegly
165170175
alavalglyileglyilealavalvalmetilepheglymetilephe
180185190
sermetileleucyscysalaileargargasnargglumetvallys
195200205
leuileaspthrvalaspleuglu
210215
<210>29
<211>663
<212>dna
<213>artificialsequence
<220>
<223>cd81dellel-v5-6xhis;basierendaufgenbankaccessionno.:
nm_004356.3
<220>
<221>misc_feature
<222>(18)..(413)
<223>tm1-tm3
<220>
<221>misc_feature
<222>(18)..(21)
<223>startkodon
<220>
<221>misc_feature
<222>(414)..(516)
<223>v5-6xhis-linker
<220>
<221>misc_feature
<222>(517)..(663)
<223>tm4
<400>29
gctagcgcggccgccaccatggattacaaggatgacgacgataagagcccgggcggatcc60
atgggagtggagggctgcaccaagtgcatcaagtacctgctcttcgtcttcaatttcgtc120
ttctggctggctggaggcgtgatcctgggtgtggccctgtggctccgccatgacccgcag180
accaccaacctcctgtatctggagctgggagacaagcccgcgcccaacaccttctatgta240
ggcatctacatcctcatcgctgtgggcgctgtcatgatgttcgttggcttcctgggctgc300
tacggggccatccaggaatcccagtgcctgctggggacgttcttcacctgcctggtcatc360
ctgtttgcctgtgaggtggccgccggcatctggggctttgtcaacaaggaccaggaattc420
ggaaagggcccgcggttcgaaggtaagcctatccctaaccctctcctcggtctcgattct480
acgcgtaccggtcatcatcaccatcaccatgatatcggaggaggaggaaagctgtacctc540
atcggcattgctgccatcgtggtcgctgtgatcatgatcttcgagatgatcctgagcatg600
gtgctgtgctgtggcatccggaacagctccgtgtacaagcttatcgataccgtcgacctc660
gag663
<210>30
<211>221
<212>prt
<213>artificialsequence
<220>
<223>cd81dellel-v5-6xhis;translatiertesequenz
<220>
<221>misc_feature
<222>(7)..(7)
<223>startkodon
<220>
<221>misc_feature
<222>(7)..(7)
<223>startmethionin
<220>
<221>misc_feature
<222>(7)..(138)
<223>tm1-tm3
<220>
<221>misc_feature
<222>(7)..(221)
<223>cd81dellel-v5-6xhis
<220>
<221>misc_feature
<222>(139)..(172)
<223>v5-6xhis-linker
<220>
<221>misc_feature
<222>(173)..(221)
<223>tm4
<400>30
alaseralaalaalathrmetasptyrlysaspaspaspasplysser
151015
proglyglysermetglyvalgluglycysthrlyscysilelystyr
202530
leuleuphevalpheasnphevalphetrpleualaglyglyvalile
354045
leuglyvalalaleutrpleuarghisaspproglnthrthrasnleu
505560
leutyrleugluleuglyasplysproalaproasnthrphetyrval
65707580
glyiletyrileleuilealavalglyalavalmetmetphevalgly
859095
pheleuglycystyrglyalaileglngluserglncysleuleugly
100105110
thrphephethrcysleuvalileleuphealacysgluvalalaala
115120125
glyiletrpglyphevalasnlysaspglnglupheglylysglypro
130135140
argphegluglylysproileproasnproleuleuglyleuaspser
145150155160
thrargthrglyhishishishishishisaspileglyglyglygly
165170175
lysleutyrleuileglyilealaalailevalvalalavalilemet
180185190
ilepheglumetileleusermetvalleucyscysglyileargasn
195200205
serservaltyrlysleuileaspthrvalaspleuglu
210215220
<210>31
<211>690
<212>dna
<213>artificialsequence
<220>
<223>cd82dellel-v5-6xhis;basierendaufgenbankaccessionno.:
nm_002231.3
<220>
<221>misc_feature
<222>(18)..(21)
<223>startkodon
<220>
<221>misc_feature
<222>(18)..(405)
<223>tm1-tm3
<220>
<221>misc_feature
<222>(406)..(507)
<223>v5-6xhis-linker
<220>
<221>misc_feature
<222>(508)..(690)
<223>tm4
<400>31
gctagcgcggccgccaccatggattacaaggatgacgacgataagagcccgggcggatcc60
atgggctcagcctgtatcaaagtcaccaaatactttctcttcctcttcaacttgatcttc120
tttatcctgggcgcagtgatcctgggcttcggggtgtggatcctggccgacaagagcagt180
ttcatctctgtcctgcaaacctcctccagctcgcttaggatgggggcctatgtcttcatc240
ggcgtgggggcagtcactatgctcatgggcttcctgggctgcatcggcgccgtcaacgag300
gtccgctgcctgctggggctgtactttgctttcctgctcctgatcctcattgcccaggtg360
acggccggggcactcttctacttcaacatgggcaagctgaagcaggaattcggaaagggc420
ccgcggttcgaaggtaagcctatccctaaccctctcctcggtctcgattctacgcgtacc480
ggtcatcatcaccatcaccatgatatcggaggaggaggagtgcaggcgtggctgcaggag540
aacctgggcatcatcctcggcgtgggcgtgggtgtggccatggtcgagctcctggggatg600
gtcctgtccatctgcttgtgccggcacgtgcattccgaagactacagcaaggtccccaag660
tacaagcttatcgataccgtcgacctcgag690
<210>32
<211>230
<212>prt
<213>artificialsequence
<220>
<223>cd82dellel-v5-6xhis;translatiertesequenz
<220>
<221>misc_feature
<222>(7)..(7)
<223>startmethionin
<220>
<221>misc_feature
<222>(7)..(135)
<223>tm1-tm3
<220>
<221>misc_feature
<222>(7)..(230)
<223>cd82dellel-v5-6xhis
<220>
<221>misc_feature
<222>(136)..(169)
<223>v5-6xhis-linker
<220>
<221>misc_feature
<222>(170)..(230)
<223>tm4
<400>32
alaseralaalaalathrmetasptyrlysaspaspaspasplysser
151015
proglyglysermetglyseralacysilelysvalthrlystyrphe
202530
leupheleupheasnleuilephepheileleuglyalavalileleu
354045
glypheglyvaltrpileleualaasplysserserpheileserval
505560
leuglnthrserserserserleuargmetglyalatyrvalpheile
65707580
glyvalglyalavalthrmetleumetglypheleuglycysilegly
859095
alavalasngluvalargcysleuleuglyleutyrphealapheleu
100105110
leuleuileleuilealaglnvalthralaglyalaleuphetyrphe
115120125
asnmetglylysleulysglnglupheglylysglyproargpheglu
130135140
glylysproileproasnproleuleuglyleuaspserthrargthr
145150155160
glyhishishishishishisaspileglyglyglyglyvalglnala
165170175
trpleuglngluasnleuglyileileleuglyvalglyvalglyval
180185190
alametvalgluleuleuglymetvalleuserilecysleucysarg
195200205
hisvalhissergluasptyrserlysvalprolystyrlysleuile
210215220
aspthrvalaspleuglu
225230
<210>33
<211>663
<212>dna
<213>artificialsequence
<220>
<223>cd151dellel-v5-6xhis;basierendaufgenbankaccessionno.:
bt001769.3
<220>
<221>misc_feature
<222>(18)..(21)
<223>startkodon
<220>
<221>misc_feature
<222>(18)..(420)
<223>tm1-tm3
<220>
<221>misc_feature
<222>(421)..(522)
<223>v5-6xhis-linker
<220>
<221>misc_feature
<222>(523)..(663)
<223>tm4
<400>33
gctagcgcggccgccaccatggattacaaggatgacgacgataagagcccgggcggatcc60
atgggtgagttcaacgagaagaagacaacatgtggcaccgtttgcctcaagtacctgctg120
tttacctacaattgctgcttctggctggctggcctggctgtcatggcagtgggcatctgg180
acgctggccctcaagagtgactacatcagcctgctggcctcaggcacctacctggccaca240
gcctacatcctggtggtggcgggcactgtcgtcatggtgactggggtcttgggctgctgc300
gccaccttcaaggagcgtcggaacctgctgcgcctgtacttcatcctgctcctcatcatc360
tttctgctggagatcatcgctggtatcctcgcctacgcctactaccagcagctgaacacg420
gaattcggaaagggcccgcggttcgaaggtaagcctatccctaaccctctcctcggtctc480
gattctacgcgtaccggtcatcatcaccatcaccatgatatcggaggaggaggactgagg540
gtcattggggctgtggggatcggcattgcctgtgtgcaggtctttggcatgatcttcacg600
tgctgcctgtacaggagtctcaagctggagcactacaagcttatcgataccgtcgacctc660
gag663
<210>34
<211>221
<212>prt
<213>artificialsequence
<220>
<223>cd151dellel-v5-6xhis;translatiertesequenz
<220>
<221>misc_feature
<222>(7)..(7)
<223>startmethionin
<220>
<221>misc_feature
<222>(7)..(140)
<223>tm1-tm3
<220>
<221>misc_feature
<222>(7)..(221)
<223>cd151dellel-v5-6xhis
<220>
<221>misc_feature
<222>(141)..(174)
<223>v5-6xhis-linker
<220>
<221>misc_feature
<222>(175)..(221)
<223>tm4
<400>34
alaseralaalaalathrmetasptyrlysaspaspaspasplysser
151015
proglyglysermetglyglupheasnglulyslysthrthrcysgly
202530
thrvalcysleulystyrleuleuphethrtyrasncyscysphetrp
354045
leualaglyleualavalmetalavalglyiletrpthrleualaleu
505560
lysserasptyrileserleuleualaserglythrtyrleualathr
65707580
alatyrileleuvalvalalaglythrvalvalmetvalthrglyval
859095
leuglycyscysalathrphelysgluargargasnleuleuargleu
100105110
tyrpheileleuleuleuileilepheleuleugluileilealagly
115120125
ileleualatyralatyrtyrglnglnleuasnthrglupheglylys
130135140
glyproargphegluglylysproileproasnproleuleuglyleu
145150155160
aspserthrargthrglyhishishishishishisaspileglygly
165170175
glyglyleuargvalileglyalavalglyileglyilealacysval
180185190
glnvalpheglymetilephethrcyscysleutyrargserleulys
195200205
leugluhistyrlysleuileaspthrvalaspleuglu
210215220
<210>35
<211>102
<212>dna
<213>artificialsequence
<220>
<223>v5-6xhis-linkerintetraspanindellel-v5-6xhiskonstrukten
<220>
<221>misc_feature
<222>(1)..(6)
<223>ecorischnittstelle
<220>
<221>misc_feature
<222>(97)..(102)
<223>ecorvschnittstelle
<400>35
gaattcggaaagggcccgcggttcgaaggtaagcctatccctaaccctctcctcggtctc60
gattctacgcgtaccggtcatcatcaccatcaccatgatatc102
<210>36
<211>34
<212>prt
<213>artificialsequence
<220>
<223>v5-6xhis-linkerintetraspanindellel-v5-6xhiskonstrukten:
translatiertesequenz
<220>
<221>misc_feature
<222>(3)..(23)
<223>v5-epitop
<220>
<221>misc_feature
<222>(27)..(32)
<223>6xhis-epitop
<400>36
glupheglylysglyproargphegluglylysproileproasnpro
151015
leuleuglyleuaspserthrargthrglyhishishishishishis
202530
aspile
<210>37
<211>126
<212>dna
<213>artificialsequence
<220>
<223>2f5-4e10-linker
<220>
<221>misc_feature
<222>(1)..(6)
<223>ecorischnittstelle
<220>
<221>misc_feature
<222>(121)..(126)
<223>ecorvschnittstelle
<400>37
gaattcattgaagaatcgcaaaaccagcaagaaaagaatgaacaagaattattggaatta60
gataaatgggcaagtttgtggaattggtttaacataacaaattggctgtggtatataaaa120
gatatc126
<210>38
<211>42
<212>prt
<213>artificialsequence
<220>
<223>2f5-4e10-linker:translatiertesequenz
<220>
<221>misc_feature
<222>(19)..(24)
<223>2f5-epitop
<220>
<221>misc_feature
<222>(28)..(33)
<223>4e10-epitop
<400>38
glupheileglugluserglnasnglnglnglulysasngluglnglu
151015
leuleuleugluasplystrpalaserleutrpasntrppheasnile
202530
thrasntrpleutrptyrilelysaspile
3540
<210>39
<211>120
<212>dna
<213>artificialsequence
<220>
<223>linkerversion(8)
<220>
<221>misc_feature
<222>(1)..(6)
<223>ecorischnittstelle
<220>
<221>misc_feature
<222>(7)..(26)
<223>tev-proteolyse-i
<220>
<221>misc_feature
<222>(27)..(33)
<223>hindiiischnittstelle
<220>
<221>misc_feature
<222>(34)..(39)
<223>bamhischnittstelle
<220>
<221>misc_feature
<222>(40)..(46)
<223>pstischnittstelle
<220>
<221>misc_feature
<222>(46)..(63)
<223>his6-tag
<220>
<221>misc_feature
<222>(64)..(84)
<223>tev-proteolyse-ii
<220>
<221>misc_feature
<222>(85)..(97)
<223>c-myc-epitope1
<220>
<221>misc_feature
<222>(103)..(114)
<223>c-myc-epitope2
<220>
<221>misc_feature
<222>(115)..(120)
<223>ecorvschnittstelle
<400>39
gaattcgagaatctgtactttcagtcgaagcttggatccctgcagcatcaccatcaccat60
cacgaaaacctctatttccagagcgaacagaaactgatatccgaagaagatctggatatc120
<210>40
<211>40
<212>prt
<213>artificialsequence
<220>
<223>linkerversion(8),translatiertesequenz
<220>
<221>misc_feature
<222>(3)..(8)
<223>tev-proteolysis-i
<220>
<221>misc_feature
<222>(16)..(21)
<223>his6-tag
<220>
<221>misc_feature
<222>(22)..(28)
<223>tev-proteolysis-i
<220>
<221>misc_feature
<222>(29)..(33)
<223>c-myc-epitope1
<220>
<221>misc_feature
<222>(34)..(38)
<223>c-myc-epitope2
<400>40
gluphegluasnleutyrpheglnserlysleuglyserleuglnhis
151015
hishishishishisgluasnleutyrpheglnsergluglnlysleu
202530
ileserglugluaspleuaspile
3540
<210>41
<211>27
<212>dna
<213>artificialsequence
<220>
<223>linkerimvektorpcmv-cd63dellel-mcherry(minimallinker(9))
<220>
<221>misc_feature
<222>(1)..(6)
<223>pstischnittstelle
<220>
<221>misc_feature
<222>(7)..(12)
<223>ecorischnittstelle
<220>
<221>misc_feature
<222>(13)..(18)
<223>ecorvschnittstelle
<220>
<221>misc_feature
<222>(19)..(27)
<223>4xglycin
<400>41
ctgcaggaattcgatatcggaggagga27
<210>42
<211>10
<212>prt
<213>artificialsequence
<220>
<223>linkerimvektorpcmv-cd63dellel-mcherry:translatiertesequenz
(minimallinker(9))
<400>42
leuglnglupheaspileglyglyglygly
1510
<210>43
<211>41
<212>dna
<213>artificialsequence
<220>
<223>linkerimvektorpcmv-cd63dellel-v2-mcherry
<220>
<221>misc_feature
<222>(3)..(8)
<223>bamhischnittstelle
<220>
<221>misc_feature
<222>(18)..(23)
<223>bgliiischnittstelle
<220>
<221>misc_feature
<222>(24)..(29)
<223>pstischnittstelle
<400>43
ttggatccagcggccgcagatctctgcagggaggcggaggc41
<210>44
<211>13
<212>prt
<213>artificialsequence
<220>
<223>linkerimvektorpcmv-cd63dellel-v2-mcherry:translatierte
sequenz(linker)
<400>44
glyserserglyargargserleuglnglyglyglygly
1510
<210>45
<211>30
<212>dna
<213>artificialsequence
<220>
<223>linkerzwischencd63dellelundfluoreszenzprotein
<400>45
aagcttatcgataccgtcgacctcgagaaa30
<210>46
<211>10
<212>prt
<213>artificialsequence
<220>
<223>linkerzwischencd63dellelundfluoreszenzprotein;translatierte
sequenz
<400>46
lysleuileaspthrvalaspleuglulys
1510
- 还没有人留言评论。精彩留言会获得点赞!