一种棕色棉纤维原花青素转运相关的GST蛋白基因及其应用的制作方法
本发明涉及的是植物分子生物学的技术领域,尤其涉及的是一种棕色棉GhTT19基因及其应用。
背景技术:
棉花是锦葵科棉属植物,它既是重要的纤维作物,又是重要的油料作物,还是纺织、精细化工原料和重要的战略物资。棕色棉是一种纤维呈现天然棕色的彩色棉,由于其本身带有天然的颜色,在棉花纺织生产过程中,可以免去漂白、消毒、染色等过程,污染少、环保健康。随着环保理念深入人心,棕色棉迎合了市场的需求,具有广阔的市场前景。
但是,棕色棉与白色棉相比,其品质不够理想,存在纤维颜色单一、着色不均匀、色牢度和色饱和度低等问题,严重制约了棕色棉的生产与应用。因此,必须解析棕色棉纤维色素合成的分子机理,为培育纤维色素稳定的棕色棉新品种奠定基础,同时能增加棉农收入和满足国民经济发展多方面的需要。前人从棕色棉纤维中克隆了原花色素合途径中的结构基因(F3H,DFR,CHI等),结合棕色棉纤维色素化学性质的鉴定结果,初步推断棕色棉纤维色素的合成前体为缩合单宁,即原花色素(proanthocyanidins,PA)。
TRANSPARENT TESTA19是一种参与原花青素、花青素转运的游离蛋白,归属于GST家族。拟南芥TT19基因(AtTT19)编码一种类黄酮代谢途径必需的GST家族的Phi类转运蛋白,其生物学功能是将胞浆内合成的花青素、原花青素等多酚类色素的单体物转运到液泡等亚细胞器官中,然后花青素在酸性条件下显色或原花青素进一步聚合后形成有色色素。
技术实现要素:
本发明的目的在于克服现有技术的不足,提供了一种棕色棉纤维原花青素转运相关的GST蛋白基因及其应用,以提供一种新的与色素转运相关的棉花基因,为培育纤维色素稳定的棕色棉新品种奠定基础。
本发明是通过以下技术方案实现的:
本发明提供了一种棕色棉纤维原花青素转运相关的GST蛋白基因,所述基因具有如SEQ ID NO.1所示的核苷酸序列,命名为GhTT19,所述蛋白的氨基酸序列如SEQ ID NO.2所示。
本发明还提供了上述棕色棉纤维原花青素转运相关的GST蛋白基因在培育棕色棉新品种中的应用。
本发明还提供了一种植物表达载体,所述植物表达载体含有上述棕色棉纤维原花青素转运相关的GST蛋白基因。
进一步地,所述植物表达载体为含有上述基因的pCambia1301a载体。
本发明还提供了一种遗传工程化的宿主细胞,所述宿主细胞含有上述植物表达载体,或其基因组中整合有外源的所述棕色棉纤维原花青素转运相关的GST蛋白基因。
进一步地,所述宿主细胞为含有上述植物表达载体或整合有所述基因的农杆菌细胞。
本发明还提供了一种制备转基因植物的方法,所述植物为拟南芥,所述方法包括以下步骤:
(1)制备侵染液:将所述宿主细胞活化后,用缓冲液重悬,获得侵染液,所述缓冲液为含有质量比为5%的蔗糖和0.02%的Silwet L-77的1/2MS液体培养基,所述侵染液的OD600值为0.8;
(2)将预转化的拟南芥苗的果荚和已开放的花朵全部剪掉,只留花蕾,将全部花蕾倒置浸在步骤(1)的侵染液中45s,再置于黑暗处培养一天,然后置于光照培养箱中继续培养,并控制光照时间为16h/天;
(3)每隔1周重复步骤(2)一次,直至种子成熟,获得T0代种子;
(4)将T0代种子消毒后接种指含有50mg/L潮霉素的MS固体筛选培养基中,置于光照培养箱中培养,并控制光照时间为16h/天,至幼苗长出4-6片莲座后,移入营养土中继续培养并收获T1代种子,只有含有所述植物表达载体的种子能够在含有50mg/L潮霉素的MS固体筛选培养基中生长,由此可筛选出阳性植株;
(5)重复步骤(4)获得稳定遗传的转基因植株及其后代。
本发明相比现有技术具有以下优点:本发明提供了一种棕色棉纤维原花青素转运相关的GST蛋白基因及其应用,通过对该基因编码序列的分析和遗传转化拟南芥,结果表明,该基因能够控制纤维色素的形成,提高植物色泽,为利用基因工程技术对棕色棉色彩性状进行遗传改良提供了参考依据,为培育纤维色素稳定的棕色棉新品种提供了新的途径。
附图说明
图1为pCambia1301a-GhTT19载体构建图谱;
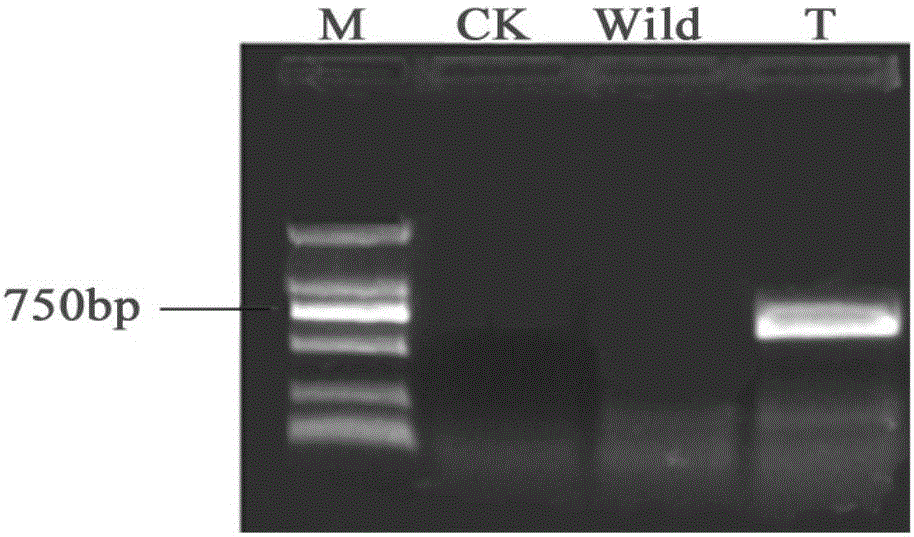
图2为拟南芥阳性苗PCR验证结果图;
图3为转GhTT19基因拟南芥T2代植株组织化学染色结果图;
图4为拟南芥各组织中GhTT19相对表达量柱状图,其中:r:转基因拟南芥根;s:转基因拟南芥茎;l:转基因拟南芥叶;t:转基因拟南芥种皮;w:野生型拟南芥种皮;
图5为拟南芥种皮原花青素含量检测柱状图。
具体实施方式
实施例1
1、材料
下述实施例中所用方法如无特别说明均为常规方法,所用引物均由上海生工生物工程技术服务有限公司合成,测序由上海生工生物工程技术服务有限公司进行,PMD-18T、限制性内切酶BamHI和XbaI、T4连接酶。RNAprep Pure Plant Kit、FastQuant RT Kit均购自TIANGEN公司。DNA胶回收试剂盒、质粒提取试剂盒购、X-Gluc均购自上海生工生物工程技术服务有限公司,方法均照说明书进行。
2、方法
2.1GhTT19的克隆及真核表达载体的构建
2.1.1GhTT19基因克隆
提取棕色棉叶片总RNA,并反转录成cDNA,设计特异性扩增引物GhTT19-F和GhTT19-R,以cDNA为模板进行PCR扩增,PCR产物的回收,连接PMD-18T载体。
所述特异性扩增引物GhTT19-F和GhTT19-R为:
SEQ ID NO.3:GhTT19-F:ATGGTAGTGAAAGTGTATGG
SEQ ID NO.4:GhTT19-R:TCAATAATTAGCGAGCTTCA
具体反应步骤包括:取1μg(2μL)的总RNA,加入2μL 5×gDNA Buffer,2μL 10×Fast RT Buffer,1μL RT Enzyme Mix,2μL FQ-RT Primer Mix,11μL RNase-Free ddH2O,小心混匀,42℃孵育15min,然后95℃孵育3min,冰上放置5min,即获得相应的反转录产物cDNA。以反转录合成的cDNA为模板,利用引物GhTT19F和GhTT19R进行PCR扩增。反应程序为:94℃5min;94℃45s,54℃45s,72℃1min,35个循环;72℃10min。PCR产物经1%琼脂糖凝胶电泳检测后,对目的片段进行回收,并连接到载体PMD-18T上,然后转化到大肠杆菌DH5α感受态细胞,阳性克隆鉴定后委托上海生工生物工程技术服务有限公司进行测序。
测序完成后利用DNAMAN软件进行比对,确保所获序列为目的序列。结果表明,所获基因大小为645bp,其核苷酸序列及其编码的蛋白质的氨基酸序列如SEQ ID NO.1、SEQ ID NO.2所示。
2.1.2植物表达载体的构建
根据目的基因GhTT19的序列设计带有BamHI和XbaI酶切位点的引物GhTT19-BamHI-F和GhTT19-XbaI-R,以cDNA为模板扩增GhTT19基因的ORF,PCR产物经1%琼脂糖凝胶电泳检测后,对目的片段进行回收,并连接到载体PMD-18T上,获得PMD-18T-GhTT19质粒。
所述带有BamHI和XbaI酶切位点的引物GhTT19-BamHI-F和GhTT19-XbaI-R为:
SEQ ID NO.5:GhTT19-BamHI-F:ATGGTAGTGAAAGTGTATGG
SEQ ID NO.6:GhTT19-XbaI-R:TCAATAATTAGCGAGCTTCA
如图1所示,用限制性内切酶BamHI和Xba I分别对PMD-18T-GhTT19质粒和植物表达载体pCambia1301a质粒进行双酶切。酶切产物用1%的琼脂糖凝胶电泳,回收目的片段GhTT19和植物表达载体pCambia1301a。用T4连接酶将2个酶切后的目的片段连接并转化到大肠杆菌DH5α感受态细胞,重组质粒进行酶切验证后并进行测序验证,获得植物表达载体pCambia1301a-GhTT19。
2.1.3宿主细胞的构建
将构建好的植物表达载体pCambia1301a-GhTT19通过电转化到农杆菌LBA4404感受态细胞中,通过卡那霉素和链霉素筛选出阳性菌株,获得宿主细胞。
同时,将未连接GhTT19基因的空载体pCambia1301a按上述同样的方法转化到农杆菌LBA4404感受态细胞中,通过卡那霉素和链霉素筛选出阳性菌株,获得对照细胞。
2.2拟南芥的遗传转化、筛选和鉴定
2.2.1制备侵染液:将所述宿主细胞活化后,用缓冲液重悬,获得侵染液,所述缓冲液为含有质量比为5%的蔗糖和0.02%的Silwet L-77的1/2MS液体培养基,所述侵染液的OD600值为0.8。
2.2.2将预转化的拟南芥苗的果荚和已开放的花朵全部剪掉,只留花蕾,将全部花蕾倒置浸在步骤2.2.1的侵染液中45s,再置于黑暗处培养一天,然后置于光照培养箱中继续培养,并控制光照时间为16h/天。
2.2.3每隔1周重复步骤2.2.2一次,直至种子成熟,获得T0代种子,共1200粒。
2.2.4将1200粒T0代种子消毒后接种至含有50mg/L潮霉素的MS固体筛选培养基中,每个培养基接种200粒,置于光照培养箱中培养,并控制光照时间为16h/天,结果如下表1所示,大部分植株黄化并停止生长,只有6株长出真叶,为阳性转基因植株,转化率为0.50%。
表1拟南芥抗性植株的筛选
待6株幼苗长出2-3片真叶后,取其中一株进行PCR检测,具体为:取少部分叶片,利用EasyPure Plant Genomic DNA Kit提取叶片的DNA,PCR鉴定时,利用目的基因的引物SEQ ID NO.3、SEQ ID NO.4进行检测。反应体系为DNA 2μL,Easytaq buffer 2.5μL,dNTPs2.0μL,上下游引物各1.5μL,Easytaq 0.25μL,水补齐至25μL。反应条件为94℃5min,94℃30s,55℃30s,72℃45s,30个循环,72℃10min。
结果如图2所示,图中,在阳性转基因拟南芥中扩增出预期大小645bp的产物。
待这6株阳性株长出4-6片莲座后,移入营养土中继续培养并收获T1代种子。
2.2.5将T1代种子重复步骤2.2.4进行复筛,获得遗传稳定的拟南芥阳性株。
2.3转基因拟南芥植株的GUS染色
取拟南芥阳性株T2代植株的幼苗,按照GUS染色试剂盒对其进行GUS染色,观察GhTT19的组织表达情况,步骤如下:
(1)预处理:将拟南芥放于1.5mL离心管中,加入预冷的90%丙酮完全覆盖材料,常温处理20-30min。
(2)染色:用蒸馏水将材料漂洗干净后置于1.5mL离心管中,加入适量配置好的GUS染色工作液至完全覆盖材料,锡箔纸包好37℃过夜放置。
(3)洗脱:用95%乙醇进行洗脱,摇床轻摇。
(4)观察:肉眼或显微镜下观察,白色背景上的蓝色部分即为GUS表达位点。
结果发现:转化空载体pCambia1301a的拟南芥细胞中的各个部位都未有蓝色,而转化pCambia1301a-GhTT19的拟南芥幼苗的根、茎、叶都出现蓝色,表明GhTT19在转基因拟南芥的各部位都表达。拟南芥阳性株T2代植株GUS染色结果如图3所示,图中可看出:在拟南芥的根、茎和叶等部位都出现蓝色,表明GhTT19在转基因拟南芥的各部位都表达。
2.4转GhTT19基因拟南芥的表型观察、qRT-PCR分析和原花青素含量检测
2.4.1表型观察
分别取转GhTT19基因拟南芥阳性株和野生型拟南芥种子若干,利用DMACA染色法进行染色处理,结果发现,转GhTT19基因拟南芥阳性株的种皮染色较野生型拟南芥的种皮染色深,说明转GhTT19能适当增加拟南芥种皮中的原花青素含量。
2.4.2qRT-PCR分析
分别从转GhTT19基因拟南芥T2的种皮及其植株组织器官、野生型拟南芥的种皮中提取RNA并进行qRT-PCR分析,结果如图4所示。结果表明转基因拟南芥中,GhTT19在种皮中表达量更高,且转基因拟南芥种皮中GhTT19的表达量高于野生型拟南芥。
2.4.3种皮原花青素含量检测
取转GhTT19基因拟南芥T2种子和野生型拟南芥种子的种皮,根据Ikegami等人提供的方法(Ikegami A,Akagi T,Potter D,et al.Molecular identification of 1-Cys peroxiredoxin and anthocyanidin/flavonol 3-O-galactosyltransferase from proanthocyanidin-rich young fruits of persimmon(Diospyros kaki Thunb.)[J].Planta,2009,230(4):841-855.)提取拟南芥种皮原花青素(PA),获得PAs提取液,利用正丁醇-盐酸法,取400μL PAs提取液,加入1.5mL正丁醇(含5%盐酸),沸水浴20min,在550nm处测定吸光度,对照标准曲线,获得转GhTT19基因拟南芥T2种子和野生型拟南芥种子的种皮中PA含量。结果如图4所示,图中可以看出,转GhTT19基因拟南芥种皮中的总PA含量是野生型的1.32倍。这些结果说明GhTT19在原花青素的转运或积累中起着一定的作用。
以上为本发明一种详细的实施方式和具体的操作过程,是以本发明技术方案为前提下进行实施,但本发明的保护范围不限于上述的实施例。
SEQUENCE LISTING
<110> 安徽农业大学
<120> 一种棕色棉纤维原花青素转运相关的GST蛋白基因及其应用
<130> /
<160> 4
<170> PatentIn version 3.3
<210> 1
<211> 645
<212> DNA
<213> 棕色棉
<400> 1
atggtagtga aagtgtatgg tccaatcaag gcagcttgcc ctcaaagggt attggcatgc 60
cttcttgaga aagaggttga atttcagatc gtcgacgtcg atctcgaagc cggcgatcat 120
aaaaaacccg atttcctcct ccgtcaaccg tttggacaag tcccagctat agaggatggc 180
gacttcaaac tttttgagtc tagggcaatc ataaggtact atgcagccaa atatgaaaag 240
caaggtacaa acctacttgg aaactcattg gaagaacgag caatggtgga tcaatggcta 300
gaagtagaag cccacaactt caacgatttg gcctacactt tggtgtttca actgttgatc 360
ctcccacgaa tgggcaagca gggtgatacg gccttagtgc tcagctgcca acaaaagctg 420
gaaaaagtgt tggacatcta cgagcaacgc ttgtccacca ccgcctatct tgctggagat 480
tcattcacct tggccgacct tagccatcta cccgctcttc gatacttggt cgacgatgtt 540
gggatgtggc acatggtgtc tcaacggaag catgtaaatg catggcggga gaccatttct 600
aaccgagctg cttggaagaa actcatgaag ctcgctaatt attga 645
<210> 2
<211> 214
<212> PRT
<213> 棕色棉
<400> 2
Met Val Val Lys Val Tyr Gly Pro Ile Lys Ala Ala Cys Pro Gln Arg
1 5 10 15
Val Leu Ala Cys Leu Leu Glu Lys Glu Val Glu Phe Gln Ile Val Asp
20 25 30
Val Asp Leu Glu Ala Gly Asp His Lys Lys Pro Asp Phe Leu Leu Arg
35 40 45
Gln Pro Phe Gly Gln Val Pro Ala Ile Glu Asp Gly Asp Phe Lys Leu
50 55 60
Phe Glu Ser Arg Ala Ile Ile Arg Tyr Tyr Ala Ala Lys Tyr Glu Lys
65 70 75 80
Gln Gly Thr Asn Leu Leu Gly Asn Ser Leu Glu Glu Arg Ala Met Val
85 90 95
Asp Gln Trp Leu Glu Val Glu Ala His Asn Phe Asn Asp Leu Ala Tyr
100 105 110
Thr Leu Val Phe Gln Leu Leu Ile Leu Pro Arg Met Gly Lys Gln Gly
115 120 125
Asp Thr Ala Leu Val Leu Ser Cys Gln Gln Lys Leu Glu Lys Val Leu
130 135 140
Asp Ile Tyr Glu Gln Arg Leu Ser Thr Thr Ala Tyr Leu Ala Gly Asp
145 150 155 160
Ser Phe Thr Leu Ala Asp Leu Ser His Leu Pro Ala Leu Arg Tyr Leu
165 170 175
Val Asp Asp Val Gly Met Trp His Met Val Ser Gln Arg Lys His Val
180 185 190
Asn Ala Trp Arg Glu Thr Ile Ser Asn Arg Ala Ala Trp Lys Lys Leu
195 200 205
Met Lys Leu Ala Asn Tyr
210
<210> 3
<211> 20
<212> DNA
<213> 人工序列
<400> 3
atggtagtga aagtgtatgg 20
<210> 4
<211> 20
<212> DNA
<213> 人工序列
<400> 4
tcaataatta gcgagcttca 20
<210> 5
<211> 35
<212> DNA
<213> 人工序列
<400> 5
cgggatccat ggtagtgaaa gtgtatggtc caatc 35
<210> 6
<211> 35
<212> DNA
<213> 人工序列
<400> 6
gctctagaat aattagcgag cttcatgagt ttctt 35
- 还没有人留言评论。精彩留言会获得点赞!