一种检测遗传性耳聋基因的建库试剂盒和应用的制作方法
本发明涉及生物检测
技术领域:
,具体涉及一种建库试剂盒及其制备方法和应用,尤其涉及一种检测遗传性耳聋基因的建库试剂盒和应用。
背景技术:
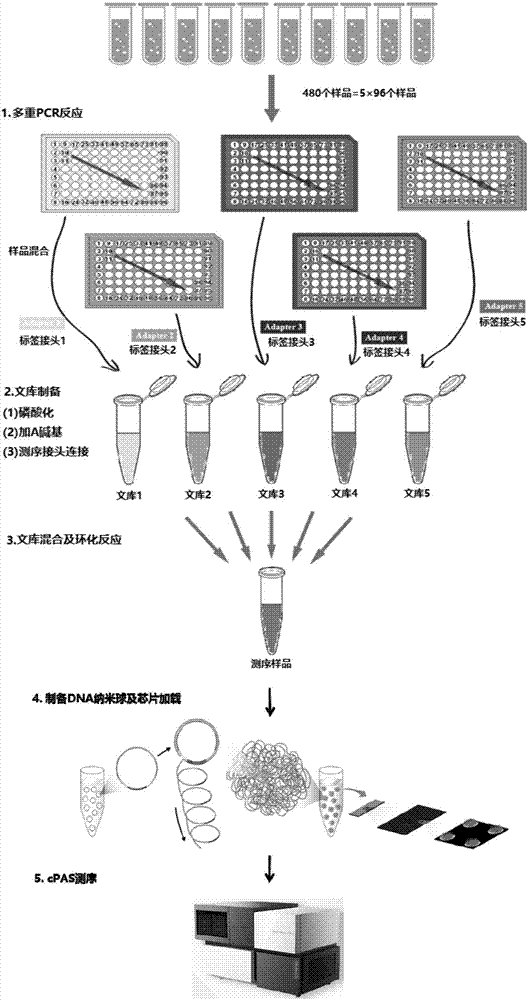
:耳聋是一项非常常见的严重影响人类生活的感觉障碍性疾病,其病因复杂,单一的或者多重的基因突变极可能会导致重度耳聋,而受环境因素影响,诸如药物、创伤、极端环境暴露等,也会突发性致聋。who2013年数据估计全球听力障碍人数达到3.6亿,而据2006年12月公布的全国第二次残疾人抽样调查主要数据公报结果显示,我国的语言听力障碍人数已高达2780万,新生儿中每年新增2万~3万聋儿,发病率高达1‰~3‰。遗传性耳聋具有较高的异质性,即不同耳聋致病基因可导致相同表型的听觉功能障碍,而同一个基因的不同的突变可以引起不同的临床表型的耳聋,由此造成耳聋的病因非常复杂。因此,对致聋的遗传因素的研究成为了探究耳聋病因及有效治疗手段的突破口,而有效检测耳聋相关基因对耳聋的早期防范与治疗起到重要作用。目前已定位的致聋基因有150多种,但分子流行病学数据显示,大多数遗传性耳聋是由几个较为常见的热点突变引起的,中国人群常见耳聋突变主要包括gjb2、gjb3、slc26a4以及线粒体的12srrna基因突变,在人群中的突变比例高达40%。在常染色体隐性遗传的非综合征性耳聋中,大约有一半是由于gjb2基因突变引起的。gjb2目前报道最多的突变位点是167delt、235delc、35delg和176_191del16。大量文献显示,gjb2基因在不同种族中导致耳聋突变的位点上存在很大的差异。在欧美,特别是在北欧、南欧及美国的高加索人中,主要的突变形式是35delg,占所有gjb2突变的70%;另一个突变热点是167delt,在犹太耳聋人群中多见,占所有病理性突变的40%,35delg突变只占18%;而在亚洲人群中,则主要为235delc突变,在日本和中国,235delc位点突变在人群中的携带率为1.0%-1.3%,占所有gjb2病理性等位基因的80%。与gjb2相关的非综合征性耳聋患者一般情况多为双耳同时受累,呈对称性耳聋,多数表现为先天性耳聋,听力损失程度从轻度到极重度不等,大多数为重度和极重度耳聋,损失程度与突变的类型和位点有关,相同基因型的个体听力受损程度也存在差异,临床表现具有多态性,表明基因的表达可能受到修饰基因或者环境的影响。gjb3基因突变可以引起常染色体显性或者隐性遗传非综合征性聋。夏家辉院士等于1998年首次在世界上成功克隆了该基因,应用荧光原位杂交技术发现gjb3定位于1p33-p35,并最早报道了两个gjb3突变位点547g>a以及538c>t:他们筛查了42个遗传性耳聋的家系,在浙江的一个耳聋家系中发现了一个错义突变,cx31基因编码区的第547位碱基由g突变成a,使得连接蛋白cx31的第183号氨基酸由谷氨酸变成了赖氨酸;同时他们还在湖南一个家系中发现了另一个突变,gjb3的第538位碱基由c变成t,导致第180号氨基酸变为终止密码子。slc26a4基因编码蛋白质pendrin。pendrin属于溶质蛋白slc26,通过其转运功能可实现细胞内外氯/碘离子互换。slc26a4基因突变和大前庭水管综合症以及pendred氏综合征有关,临床表现为先天神经性耳聋伴有不同程度的语言障碍,甲状腺肿大,ct或mri显示耳蜗发育不良,前庭导水管扩大,在中国人群的遗传性耳聋患者中突变率仅次于gjb2。到目前为止已发现了150多种slc26a4突变类型,不同人群中突变类型和发生频率存在很大差异,ivs7-2a>g突变类型在中国大前庭水管综合症患者中最常见,有研究证明ivs7-2a>g亦为始祖效应。线粒体dna(mtdna)是唯一存在于细胞质中的dna,由于子代的线粒体由卵细胞提供,故线粒体遗传属于母系遗传。氨基糖苷类抗生素致聋患者大部分是1555a>g、1095t>c、1494c>t突变的携带者,这些突变增加了个体对氨基糖苷类药物耳毒性的敏感性,常出现“一针致聋”的情况,通过早期基因检测,可给出临床用药建议,同时由于该突变属母系遗传,可对家系所有母系成员均起到警戒作用。目前对于遗传性耳聋基因诊断所应用的检测方法主要有sanger测序法、arms-pcr法、荧光pcr法、pcr导流杂交法、dna微阵列技术、飞行时间质谱法等。sanger测序法即双脱氧核苷酸末端终止测序法,是基因突变检测的金标准方法,其原理是利用一种dna聚合酶来延伸结合在待测序列模板上的引物,直到掺入一种链终止核苷酸为止。每一次序列测定由一套四个单独的反应构成,每个反应含有所有四种脱氧核苷酸三磷酸(dntp),并混入限量的四种不同的双脱氧核苷三磷酸(ddntp),该ddntp上偶联上了4种不同颜色的荧光基团。由于ddntp缺乏延伸所需要的3-oh基团,使延长的寡聚核苷酸选择性地在g、a、t或c处终止。终止点由反应中相应的ddntp而定,并发出相应的荧光。每一种dntps和ddntps的相对浓度可以调整,使反应得到一组长几百至几千碱基的链终止产物。它们具有共同的起始点,但终止在不同的的核苷酸上,可通过毛细管电泳分离大小不同的片段,并采集每种链终止产物的荧光信号,从而读出序列模板的碱基序列。但sanger直接测序法的每个反应只能检测一个位点,通量低,检测位点少,导致检测成本高,且数据分析依赖人工阅读,速度慢。突变阻滞扩增系统即arms-pcr法(amplificationrefractorymutationsystem)是一种改进的pcr方法,其基本原理是,如果引物的3’端碱基与模板碱基不互补,则用一般taqdna聚合酶无法延伸,该方法使用4条引物进行碱基突变检测,先在待检测突变位点外侧设计上下游引物(2条外引物),再在紧邻待检测突变位点的上下游设计两条内引物,一条引物的3’端碱基为突变型碱基,另一条引物的3’端碱基为野生型碱基,若存在突变,则产生突变型碱基的产物,若无突变,则产生野生型碱基的产物,通过琼脂糖凝胶电泳检测有无扩增片段及片段长度判定是否有点突变,或在内引物上标记2个不同荧光素,通过对产物荧光分析即可了解到模板中是否存在点突变。arms-pcr法的每个反应只能检测一个或两个位点,检测位点较少,导致检测成本偏高。荧光pcr法是在常规pcr基础上加入荧光标记探针实现碱基突变检测。该方法针对同一待测位点的不同基因型设计两条不同的taqman探针,把它们同时加入到pcr反应体系中,只有与靶序列完全匹配的探针才能被水解并释放出荧光信号。两条探针分别标记不同的荧光基团,分别用实时荧光定量pcr仪中对应的荧光检测通道检测荧光信号。如果只有一条探针能释放荧光信号,则检测的位点是纯合子,如果两条探针都有荧光信号,则检测的位点是杂合子。但荧光pcr法的每个反应只能检测一个或两个位点,检测位点较少,导致检测成本偏高。pcr导流杂交法与dna微阵列技术原理类似,都是根据固相杂交原则设计的检测方法,pcr导流杂交法是将靶dna用带有生物素的引物进行pcr扩增,带生物素的pcr产物与膜固定的捕获探针杂交,再加入交联碱性磷酸酶的链霉亲和素,洗去未结合的链霉亲和素,加入底物显色,由此来检测靶dna是否变异,而dna微阵列技术将生物素换为荧光基团,通过识别不同位置的荧光信号而确定是否有dna变异。但是pcr导流杂交法与dna微阵列技术均依赖探针特异性,探针位置出现其他碱基变异时易出现错误结果。飞行时间质谱法(maldi-tof-ms)首先通过设计出特异性引物,扩增出待测位点的核苷酸片段,再针对待测位点,设计单条特异引物,退火时此引物3’端的碱基刚好与待测位点的前一个碱基结合。在反应体系中加入ddntp和dna聚合酶,当某一ddntp与待测位点碱基互补并结合时,链延伸反应终止,得到延伸一个碱基的延伸产物。由于4种碱基的分子量不同,采用样品分子在电场中的飞行时间与分子的荷质比成正比的原理,通过检测延伸产物的飞行时间,测得延伸产物分子量,从而确定待测位点的碱基类型。但是飞行时间质谱法依赖延伸引物的特异性,探针结合错误或探针位置出现突变易出现假阴性结果,同时易受干扰物质影响出现错误结果。技术实现要素:本发明的目的在于提供一种检测遗传性耳聋基因的建库试剂盒和应用,所述建库试剂盒通过多重pcr方法及高通量测序方法检测遗传性耳聋基因突变。为达到此发明的目的,本发明采用以下技术方案:第一方面,本发明提供了一种检测遗传性耳聋基因的建库试剂盒,所述建库试剂盒包括用于扩增遗传性耳聋基因的扩增引物组;所述扩增引物组包括根据gjb2、gjb3、slc26a4基因和12srrna基因的突变位点的多态性设计的引物;其中,所述gjb2基因包括35delg、167delt、176-191del16、299_300delat和235delc基因;所述gjb3基因包括538c>t和547g>a;所述slc26a4基因包括281c>t、589g>a、ivs7-2a>g、1174a>t、1226g>a、1229c>t、1975g>c、2027t>a、2162c>t、2168a>g和ivs15+5g>a;所述12srrna基因包括1494c>t、1555a>g和1095t>c。根据本发明,所述扩增引物组如seqidno.1-24所示。所述seqidno.1-24所示的引物如下:根据本发明,所述扩增引物组的每条引物的5’端加入样品标签序列。根据本发明,所述样品标签序列的长度为7-10bp,优选为7bp。根据本发明,所述样品标签序列如seqidno.25-120所示。所述seqidno.25-120所示的标签序列如下:本发明中,所述标签本领域技术人员可以根据需要进行调整,只要能够对样品进行区分都是可行的,本申请优选采用上述seqidno.25-120所示的标签序列。第二方面,本发明提供一种遗传性耳聋相关基因的文库构建方法,包括如下步骤:采用第一方面所述的建库试剂盒,在所述引物组的5’端加入建库试剂盒中所述的样品标签序列,加入反应液中对样品进行多重pcr反应,将带有不同样本标签序列的多个样本的pcr产物等比例混合成文一个文库样本,得到所述遗传性耳聋相关基因的文库。根据本发明,所述文库构建方法还包括如下步骤:(1)pcr产物磷酸化;(2)末端修复;(3)连接测序接头;(4)pcr扩增文库;(5)文库环化;(6)纳米球制备;(7)测序分析。根据本发明,所述多重pcr反应的体系为无核酸酶水5.5μl、pcr扩增液12.5μl、扩增引物组2μl和dna样品5μl。根据本发明,所述多重pcr反应的条件为94℃2min;94℃20s,56℃30s,60℃20s,35个循环;72℃5min;pcr扩增结束保存在12℃。本发明中,所述文库的构建还包括磷酸化的步骤、加a碱基的步骤和测序接头连接的步骤,所述磷酸化的步骤、加a碱基的步骤和测序接头连接的步骤为本领域的常规步骤,本领域技术人员可以根据实际需要进行选择。第三方面,本发明提供一种如第二方面所述的方法构建得到的遗传性耳聋相关基因的文库。第四方面,本发明提供一种用于非诊断目的的遗传性耳聋相关基因的检测方法,将第三方面所述遗传性耳聋相关基因的文库采用联合探针锚定聚合测序法(cpas)进行测序。本发明中,所述测序采用bgiseq-500测序仪。本发明中,所述扩增引物组不仅仅可以用于所述基于cpas技术的测序仪,其他构建文库测序的方法也是可行的。本发明中,所述测序之前还包括文库混合和环化反应的步骤及制备dna纳米球的步骤,所述上述步骤为了进一步提高待测片段的浓度,从而提高测序通量和测序准确度。第五方面,本发明提供第一方面所述的检测遗传性耳聋基因的建库试剂盒、如第三方面所述的文库或如第四方面所述的检测方法用于非诊断目的的人遗传性耳聋基因的检测。与现有技术相比,本发明具有的有益效果:(1)本发明采用多重pcr技术,单次反应可检测20个以上耳聋基因突变位点;(2)本发明采用“双标签”系统,使每个样品的dna扩增产物上均带有两套独立的标签序列,因此可将所有样品的扩增产物混合后同时测序,实现高通量检测,从而大幅降低了单个样品的检测成本;(3)本发明采用先进的cpas高通量测序技术,直接读取待测位点碱基信息,不受上下游碱基突变影响,检测结果准确可靠。附图说明图1为本发明基于cpas测序技术的遗传性耳聋基因检测原理图;图2为本发明基于cpas测序的遗传性耳聋基因检测流程图;图3为本发明cpas测序得到的dna序列结构图;图4为本发明文库扩增后的dna片段大小及浓度。具体实施方式为更进一步阐述本发明所采取的技术手段及其效果,以下结合本发明的优选实施例来进一步说明本发明的技术方案,但本发明并非局限在实施例范围内。实施例中未注明具体技术或条件者,按照本领域内的文献所描述的技术或条件,或者按照产品说明书进行。所用试剂或仪器未注明生产厂商者,均为可通过正规渠道商购获得的常规产品。实施例1:引物设计1、引物设计具体包括如下步骤:选择遗传性耳聋相关的gjb2、gjb3、slc26a4基因和12srrna基因的突变位点21个作为检测位点,分别是:gjb2基因的35delg、167delt、176-191del16、299_300delat与235delc,gjb3基因的538c>t与547g>a,slc26a4基因的281c>t、589g>a、ivs7-2a>g、1174a>t、1226g>a、1229c>t、1975g>c、2027t>a、2162c>t、2168a>g与ivs15+5g>a,线粒体基因的1494c>t,1555a>g,1095t>c。使用引物设计软件设计上述基因位点的pcr扩增引物,扩增引物的长度在20个碱基左右。利用软件设计评估样品标签序列,标签序列包含7个碱基,设计标签序列时应避免出现与测序的引物相似度高的序列和添加上标签的pcr扩增引物形成发卡结构或二聚体等二级结构。针对上述4个耳聋基因21个突变位点的pcr扩增引物参见表1,样品标签序列参见表2。表1遗传性耳聋基因21个位点的pcr扩增引物表2样品标签序列实施例2构建文库并测序在本实施例中,检测24份已知上述待检测位点信息(sanger测序检测)的临床dna样品,编号为1~24,其中1~21号样品为突变阳性样品,22~24号样品为突变阴性样品。所述检测的原理如图1所示,检测的流程如图2所示。具体包括如下步骤:2、pcr扩增反应2.1根据表3的比例在适合的离心管中配制pcr扩增反应混合液,混匀后在冰上按18μl每反应孔分装到96孔pcr反应板中;表3pcr扩增反应混合液配方2.2使用八道移液器按顺序吸取2μl储存于96孔pcr反应板中的pcr反应引物加入到步骤(1)的pcr反应板的对应孔中,贴上封口膜,置于板式离心机中4000rmp离心1分钟;2.3分别向步骤(2)的96孔pcr反应板中加入24个待检测dna样品,每个样品加入5μl。完成加样后在pcr反应板上贴上封口膜,置于板式离心机中4000rmp离心1分钟;2.4将pcr反应板置于pcr仪中,运行表4的pcr反应程序;表4pcr反应程序2.5.pcr反应结束后将pcr反应板取出,置于板式离心机中4000rpm离心1分钟。2.6.取步骤2.5的pcr反应板的96个pcr反应产物各10μl至1.5ml离心管中,振荡混匀后置于掌式离心机离心5秒。3、磁珠纯化3.1.将室温平衡30分钟的磁珠振荡混匀,取180μl至1.5ml离心管中,加入100μl步骤2.6的混合样本,混匀,室温静置10分钟,磁力架上放置10分钟至澄清,弃上清。3.2.向离心管加入350μl70%乙醇,保证磁珠完全浸入70%乙醇中,水平180度快速转动离心管,静置1分钟,使管壁上的磁珠迁移至对面管壁,再次水平180度快速转动离心管,静置1分钟,弃上清。3.3.重复步骤3.2,打开离心管管盖,室温放置5分钟,使乙醇充分挥发。3.4.取下离心管,加入25μldna溶解液,与磁珠充分混匀,室温静置5分钟,置于磁力架上至澄清,取上清液至新的1.5ml离心管中。3.5.使用qubit3.0荧光定量检测仪对纯化后样本进行定量检测,测定质量浓度为42.5ng/μl。4、文库制备4.1.磷酸化反应4.1.1.根据表5的比例在合适的离心管中配制磷酸化反应混合液,混匀后在冰上按6μl每反应分装到200μlpcr反应管中。表5磷酸化反应混合液配方试剂名称一个反应标准量磷酸化缓冲液4μl磷酸化酶2μl总体积6μl4.1.2.根据步骤3.5的定量检测的质量浓度(m),按照公式(v=200/m)计算文库制备样本使用体积为4.7μl,取4.7μl步骤3.5的纯化后样本加入到步骤4.1.1中200μlpcr反应管,再加入29.3μl的无核酸酶水混匀,使末端修复反应体系终体积为40μl。4.1.3.将反应管放置pcr仪上37℃孵育30分钟,结束后置于掌式离心机离心5秒,获得经磷酸化反应后的磷酸化文库dna。4.2.接头连接反应4.2.1.根据表6的比例在合适的离心管中配制连接反应混合液,混匀后在冰上待用。表6连接反应混合液配方试剂一个反应标准量连接酶缓冲液27.2μl100mm腺嘌呤核苷三磷酸0.8μl连接酶10μl总体积38μl4.2.2.如表7所示,取38μl步骤4.2.1的连接反应混合液至步骤4.1.3的200μlpcr反应管中,再取2μl标签接头加入到离心管中。测序接头序列如下,高亮标注的碱基为文库标签序列:1链:ttgtcttcctaaggaacgacatggctacgatccgactt2链:/phos/agtcggaggccaagcggtcttaggaagacaataggtccgatcaactccttggctcaca表7连接反应体系4.2.3.将步骤4.2.2的离心管振荡混匀,置于掌式离心机中离心5秒,并置于pcr仪上关闭热盖,25℃孵育20分钟。4.2.4.反应结束后,将pcr反应管置于掌式离心机上离心5秒,并将全部反应液转至新的1.5ml离心管中。4.3.磁珠纯化4.3.1.将室温平衡30分钟的磁珠振荡混匀,取72μl至步骤4.2.4的离心管中,混匀,室温静置10分钟,磁力架上放置10分钟至澄清,弃上清;4.3.2.向离心管加入180μl70%乙醇,保证磁珠完全浸入70%乙醇中,水平180度快速转动离心管,静置1分钟,使管壁上的磁珠迁移至对面管壁,再次水平180度快速转动离心管,静置1分钟,弃上清。4.3.3.重复步骤4.3.2,打开离心管管盖,室温放置5分钟,使乙醇充分挥发;4.3.4.取出离心管,加入25μldna溶解液,与磁珠充分混匀,室温静置5分钟,置于磁力架上至澄清,取7μl上清液至新的200μlpcr反应管中。4.4.文库扩增4.4.1.根据表8的比例在适合的离心管中配制pcr扩增反应混合液,混匀后在冰上按18μl每反应孔分装到4.3.4反应产物的200μlpcr反应管中,混匀。表8pcr扩增反应混合液配方4.4.2.将pcr反应管置于掌式离心机中离心5秒钟。将pcr反应管置于pcr仪中,运行表9的pcr反应程序。表9文库扩增反应程序4.4.3.pcr反应结束后将反应管取出,置于掌式离心机中离心5秒钟。4.5.磁珠纯化4.5.1.将室温平衡30分钟的磁珠振荡混匀,取100μl步骤4.4.3的混合产物到1.5ml离心管中,加入90μl磁珠混匀。室温静置10分钟,磁力架上放置10分钟至澄清,弃上清;4.5.2.向离心管加入350μl70%乙醇,保证磁珠完全浸入70%乙醇中,水平180度快速转动离心管,静置1分钟,使管壁上的磁珠迁移至对面管壁,再次水平180度快速转动离心管,静置1分钟,弃上清。4.5.3.重复步骤4.5.2,打开离心管管盖,室温放置5分钟,使乙醇充分挥发。4.5.4.取出离心管,加入25μldna溶解液,与磁珠充分混匀,室温静置5分钟,置于磁力架上至澄清,取上清液至新的1.5ml离心管中。4.5.5.用安捷伦生物分析仪2100检测步骤4.5.4中文库扩增产物dna片段大小和dna浓度,结果如图4所示,扩增产物dna片段主峰范围为240bp~310bp,文库浓度为35.8ng/μl。5、环化反应5.1根据步骤4.5.5的文库浓度,取4.5μl(约160ng)文库扩增产物,用分子级水将体积补充至48μl,充分混匀后短暂离心,置于pcr仪上95℃孵育5min,孵育完毕即刻取出pcr管放置在冰上冷却。5.2在上述pcr管加入11.6μl环化反应缓冲液、0.5μl连接酶,充分混匀,短暂离心,37℃孵育30min。6、dna纳米球制备6.1取步骤5.2的环化dna文库20μl到0.2mlpcr管内。加入20μldnb制备缓冲液,漩涡振荡器震荡混匀,离心5秒后置于pcr仪中反应。反应条件为:95℃1min,65℃1min,40℃1min,4℃保持。6.2反应完成后取出pcr管,离心5秒,加入40μldnb聚合酶混合液i和4μldnb聚合酶混合液ii,漩涡振荡器震荡混匀,离心5秒后即刻置于pcr仪中开始反应,反应条件:30℃10min,4℃保持。6.3反应完成后取出pcr管至于冰盒上,即刻加入20μldnb终止缓冲液,用移液器和阔口吸头缓慢地吹打混匀,切勿震荡及剧烈吹打,置于4℃保存备用。7、dna纳米球加载参照bgidl-50全自动样品加载系统产品说明书进行操作,将步骤6.3的dna纳米球溶液加载到测序芯片上。8、dna纳米球上机测序参照基因测序仪(bgiseq-500)产品说明书进行操作,将加载了dna纳米球的测序芯片置于测序仪中进行cpas测序,测序得到的dna序列结构图如图3所示。9、数据分析测序完成后,使用“遗传性耳聋基因分析软件”对测序数据进行分析,结果如表10-11所示:表1024例样品cpas测序检测结果表1124例样品cpas测序检测结果与sanger测序结果比较表从表10-11可以看出,24例样品的21个位点的检测结果均与sanger测序检测结果一致,21个位点检测准确性为100%。申请人声明,本发明通过上述实施例来说明本发明的详细方法,但本发明并不局限于上述详细方法,即不意味着本发明必须依赖上述详细方法才能实施。所属
技术领域:
的技术人员应该明了,对本发明的任何改进,对本发明产品各原料的等效替换及辅助成分的添加、具体方式的选择等,均落在本发明的保护范围和公开范围之内。sequencelisting<110>华大基因科技<120>一种检测遗传性耳聋基因的建库试剂盒和应用<130>2017<160>120<170>patentinversion3.3<210>1<211>19<212>dna<213>人工合成序列<400>1cgcccagagtagaagatgg19<210>2<211>20<212>dna<213>人工合成序列<400>2gttgcagacaaagtcggcct20<210>3<211>20<212>dna<213>人工合成序列<400>3ggtgtggggagatgagcagg20<210>4<211>20<212>dna<213>人工合成序列<400>4gatgaacttcctcttcttct20<210>5<211>21<212>dna<213>人工合成序列<400>5ccaacatcgtggactgctaca21<210>6<211>19<212>dna<213>人工合成序列<400>6caggtggaagctcggctgg19<210>7<211>19<212>dna<213>人工合成序列<400>7ctgagattggattgaaaac19<210>8<211>20<212>dna<213>人工合成序列<400>8ttgcagcgtggccactagcc20<210>9<211>20<212>dna<213>人工合成序列<400>9agtgccctgactctgctggt20<210>10<211>20<212>dna<213>人工合成序列<400>10acgtaatttagaaaataatt20<210>11<211>21<212>dna<213>人工合成序列<400>11gtctgtatggcagttaaggaa21<210>12<211>20<212>dna<213>人工合成序列<400>12aatggcagtagcaattatcg20<210>13<211>20<212>dna<213>人工合成序列<400>13aggaattcattgcctttggg20<210>14<211>19<212>dna<213>人工合成序列<400>14attcctcgacttgttctct19<210>15<211>22<212>dna<213>人工合成序列<400>15tttctatggcaatgtcgatggt22<210>16<211>21<212>dna<213>人工合成序列<400>16cacaaagggaagagggtctag21<210>17<211>22<212>dna<213>人工合成序列<400>17aactctgagcttccagtcaaag22<210>18<211>21<212>dna<213>人工合成序列<400>18tactattgccaaagctccaaa21<210>19<211>20<212>dna<213>人工合成序列<400>19tgacgacaacattagaaagg20<210>20<211>20<212>dna<213>人工合成序列<400>20tttgccatttatagaaatca20<210>21<211>20<212>dna<213>人工合成序列<400>21atttagcagtaaactaagag20<210>22<211>21<212>dna<213>人工合成序列<400>22cgtccaagtgcactttccagt21<210>23<211>20<212>dna<213>人工合成序列<400>23ggattagataccccactatg20<210>24<211>19<212>dna<213>人工合成序列<400>24caagaggtggtgaggttga19<210>25<211>7<212>dna<213>人工合成序列<400>25cacatac7<210>26<211>7<212>dna<213>人工合成序列<400>26tgtagtc7<210>27<211>7<212>dna<213>人工合成序列<400>27aacgttg7<210>28<211>7<212>dna<213>人工合成序列<400>28accttct7<210>29<211>7<212>dna<213>人工合成序列<400>29catcatg7<210>30<211>7<212>dna<213>人工合成序列<400>30tcgtctc7<210>31<211>7<212>dna<213>人工合成序列<400>31cgagtga7<210>32<211>7<212>dna<213>人工合成序列<400>32atgtaga7<210>33<211>7<212>dna<213>人工合成序列<400>33aggttag7<210>34<211>7<212>dna<213>人工合成序列<400>34atgaact7<210>35<211>7<212>dna<213>人工合成序列<400>35cactctg7<210>36<211>7<212>dna<213>人工合成序列<400>36caaggat7<210>37<211>7<212>dna<213>人工合成序列<400>37cctacct7<210>38<211>7<212>dna<213>人工合成序列<400>38tgtgaga7<210>39<211>7<212>dna<213>人工合成序列<400>39agcatga7<210>40<211>7<212>dna<213>人工合成序列<400>40agtgcta7<210>41<211>7<212>dna<213>人工合成序列<400>41tcgcaga7<210>42<211>7<212>dna<213>人工合成序列<400>42gaaggta7<210>43<211>7<212>dna<213>人工合成序列<400>43gcgaagt7<210>44<211>7<212>dna<213>人工合成序列<400>44ggtggac7<210>45<211>7<212>dna<213>人工合成序列<400>45gttattg7<210>46<211>7<212>dna<213>人工合成序列<400>46cgatcta7<210>47<211>7<212>dna<213>人工合成序列<400>47taatcgg7<210>48<211>7<212>dna<213>人工合成序列<400>48tataaga7<210>49<211>7<212>dna<213>人工合成序列<400>49ctcgctg7<210>50<211>7<212>dna<213>人工合成序列<400>50agtctga7<210>51<211>7<212>dna<213>人工合成序列<400>51ctgtatg7<210>52<211>7<212>dna<213>人工合成序列<400>52cagtata7<210>53<211>7<212>dna<213>人工合成序列<400>53tccagag7<210>54<211>7<212>dna<213>人工合成序列<400>54ctacgta7<210>55<211>7<212>dna<213>人工合成序列<400>55tgccagt7<210>56<211>7<212>dna<213>人工合成序列<400>56tacgtga7<210>57<211>7<212>dna<213>人工合成序列<400>57tgctcac7<210>58<211>7<212>dna<213>人工合成序列<400>58cagacac7<210>59<211>7<212>dna<213>人工合成序列<400>59tgctaga7<210>60<211>7<212>dna<213>人工合成序列<400>60catgata7<210>61<211>7<212>dna<213>人工合成序列<400>61atatcga7<210>62<211>7<212>dna<213>人工合成序列<400>62cacgcta7<210>63<211>7<212>dna<213>人工合成序列<400>63cacgatg7<210>64<211>7<212>dna<213>人工合成序列<400>64cacagta7<210>65<211>7<212>dna<213>人工合成序列<400>65cgactac7<210>66<211>7<212>dna<213>人工合成序列<400>66tgatatg7<210>67<211>7<212>dna<213>人工合成序列<400>67ctgtgta7<210>68<211>7<212>dna<213>人工合成序列<400>68ctctata7<210>69<211>7<212>dna<213>人工合成序列<400>69tgtctac7<210>70<211>7<212>dna<213>人工合成序列<400>70cagacta7<210>71<211>7<212>dna<213>人工合成序列<400>71cgtagta7<210>72<211>7<212>dna<213>人工合成序列<400>72tcgacac7<210>73<211>7<212>dna<213>人工合成序列<400>73gagcatc7<210>74<211>7<212>dna<213>人工合成序列<400>74gtacgtc7<210>75<211>7<212>dna<213>人工合成序列<400>75agagtac7<210>76<211>7<212>dna<213>人工合成序列<400>76agcgaga7<210>77<211>7<212>dna<213>人工合成序列<400>77cgtatga7<210>78<211>7<212>dna<213>人工合成序列<400>78ctcatga7<210>79<211>7<212>dna<213>人工合成序列<400>79cgagata7<210>80<211>7<212>dna<213>人工合成序列<400>80agtgatc7<210>81<211>7<212>dna<213>人工合成序列<400>81agtacac7<210>82<211>7<212>dna<213>人工合成序列<400>82gctcgta7<210>83<211>7<212>dna<213>人工合成序列<400>83agctatc7<210>84<211>7<212>dna<213>人工合成序列<400>84actcata7<210>85<211>7<212>dna<213>人工合成序列<400>85ctagcga7<210>86<211>7<212>dna<213>人工合成序列<400>86catactg7<210>87<211>7<212>dna<213>人工合成序列<400>87tactatg7<210>88<211>7<212>dna<213>人工合成序列<400>88tcagctc7<210>89<211>7<212>dna<213>人工合成序列<400>89tatgatg7<210>90<211>7<212>dna<213>人工合成序列<400>90tcgagtc7<210>91<211>7<212>dna<213>人工合成序列<400>91cgatatc7<210>92<211>7<212>dna<213>人工合成序列<400>92cactatc7<210>93<211>7<212>dna<213>人工合成序列<400>93atcgtga7<210>94<211>7<212>dna<213>人工合成序列<400>94gtcacac7<210>95<211>7<212>dna<213>人工合成序列<400>95gatcgtc7<210>96<211>7<212>dna<213>人工合成序列<400>96atctgta7<210>97<211>7<212>dna<213>人工合成序列<400>97cgtgatg7<210>98<211>7<212>dna<213>人工合成序列<400>98agacaga7<210>99<211>7<212>dna<213>人工合成序列<400>99tagatga7<210>100<211>7<212>dna<213>人工合成序列<400>100atactga7<210>101<211>7<212>dna<213>人工合成序列<400>101cgtcgtc7<210>102<211>7<212>dna<213>人工合成序列<400>102atgtctc7<210>103<211>7<212>dna<213>人工合成序列<400>103tgcagta7<210>104<211>7<212>dna<213>人工合成序列<400>104tacactg7<210>105<211>7<212>dna<213>人工合成序列<400>105acagatc7<210>106<211>7<212>dna<213>人工合成序列<400>106gatatac7<210>107<211>7<212>dna<213>人工合成序列<400>107tagagta7<210>108<211>7<212>dna<213>人工合成序列<400>108ctctcac7<210>109<211>7<212>dna<213>人工合成序列<400>109tcacgtc7<210>110<211>7<212>dna<213>人工合成序列<400>110cagctac7<210>111<211>7<212>dna<213>人工合成序列<400>111tatctga7<210>112<211>7<212>dna<213>人工合成序列<400>112gtctaga7<210>113<211>7<212>dna<213>人工合成序列<400>113ctagatg7<210>114<211>7<212>dna<213>人工合成序列<400>114ctcacta7<210>115<211>7<212>dna<213>人工合成序列<400>115gctgtga7<210>116<211>7<212>dna<213>人工合成序列<400>116gacgctg7<210>117<211>7<212>dna<213>人工合成序列<400>117gtgcgta7<210>118<211>7<212>dna<213>人工合成序列<400>118ctgatac7<210>119<211>7<212>dna<213>人工合成序列<400>119tgagatc7<210>120<211>7<212>dna<213>人工合成序列<400>120gtgatga7当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1