一种构建果蝇miRNA测序文库进行高通量测序的方法与流程
本发明属于分子生物学技术领域。更具体地,涉及一种构建果蝇mirna测序文库进行高通量测序的方法。
背景技术:
2006年以来,核酸测序技术取得了革命性的突破,第二代测序技术(nextgenerationsequencing,ngs)能够快速测定数百万个序列,在技术上实现了对一个物种的转录组和基因组进行全貌分析。特别是高通量的转录组测序技术(rna-seq)的产生。转录组测序(rna-seq)是利用ngs技术进行测序,全面快速地获取某一物种或特定器官、组织在某一状态下的几乎所有转录本,分析个体、组织或细胞中的全转录组、小rna含量或基因表达谱,是目前深入研究转录组复杂性的强大工具。
将基于微流控技术的超高重pcr(mmpcr)与高通量测序技术相结合而产生超高重pcr测序(mmpcr-seq)是发明人于2014年在斯坦福大学发展的测序新技术,该技术能够同时扩增多达48个样本的960个位点,使这些位点的基因表达水平均一化,甚至对低含量的rna样本都能准确定量其等位基因比例。mmpcr-seq填补了传统rna-seq的靶向测序技术空白,该技术可用于研究转录组的等位基因变化、rna修饰和rna表观遗传组学研究,这一技术的发展通过降低成本而极大的扩展rna-seq的应用领域。
但是目前的mmpcr-seq主要适用于有效构建mrna文库测序,对于mirna文库的构建效果不太好。
技术实现要素:
本发明要解决的技术问题是克服上述现有技术的缺陷和不足,提供一种构建果蝇mirna测序文库进行高通量测序的方法。所述方法能够高效并完整地构建果蝇pri-mirna上的mirna前体区域转录组文库,极大的提高了前体mirna上rna编辑和修饰的检测敏感性,以及rna靶向测序文库的均一性,缩短了构建文库和测序周期,降低了成本。
本发明的目的是提供一种构建果蝇mirna测序文库进行高通量测序的方法。
本发明的上述目的是通过以下技术方案予以实现的:
一种靶向扩增果蝇mirna前体区域的探针,所述探针共247对,其序列依次如seqidno:1~seqidno:494所示。
本发明自主合成的特异性探针可对mirna进行靶向扩增,扩增区域包括pri-mirna上覆盖mirna前体(pre-mirna)的区域,是下一步结合mmpcr-seq技术发展新技术的前提。
因此,上述探针在果蝇mirna测序中的应用亦在本发明保护范围内。
具体地,所述应用为上述探针在构建果蝇mirna高通量测序文库中的应用。
一种构建果蝇mirna测序文库进行高通量测序的方法,包括如下步骤:
s1.提取果蝇总rna,反转成cdna;
s2.以步骤s1所述cdna为模板,将上述247对探针混合在一起成为1个pool,进行超高重pcr预扩增;
s3.再以预扩增产物为模板,将上述247对探针随机分为40个pool,每个pool包含5~7对探针,进行超高重pcr扩增样品中的mirna,同时在扩增产物两端分别接上3'接头和5'接头;
s4.将步骤s3的mmpcr产物连接标签序列;
s5.将加上标签序列的样品进行琼脂糖凝胶电泳,回收纯化电泳产物,即构建好mirna文库;
s6.将得到的多个mirna文库混合,利用二代测序技术进行高通量测序。
本发明以自主设计的特异性靶向果蝇mirna前体区域的探针为引物,先通过一轮超高重pcr预扩增,再以预扩增的产物为模板进行超高重pcr扩增,使rna表达水平均一化,从而保证rna靶向测序文库的均一性。极大的提高了前体mirna上rna编辑和修饰的检测敏感性,缩短了构建文库和测序周期,降低了成本。
优选地,步骤s2所述超高重pcr预扩增的反应体系为kapa2g(2×)5μl,cdna模板>100ng,50μm探针1.25μl,超纯水补充至10μl;反应程序为95℃变性10min,95℃15sec,65℃4min循环两次,95℃15sec,72℃4min循环13次。
优选地,步骤s3所述超高重pcr扩增反应体系为芯片的左侧三列中每个孔中加2×kapa2g2.5μl,20×accessarrayloadingreagent0.25μl,预扩增产物100ng,超纯水补充至5μl;芯片的右侧三列中加同等数量孔,每个孔加4μl的primersolutions;反应程序分为7步:第1步,50℃2min,70℃20min,95℃10min;第2步,95℃15sec,60℃30sec,72℃1min,循环15次;第3步,95℃15sec,80℃30sec,60℃30sec,72℃1min,循环2次;第4步,95℃15sec,60℃30sec,72℃1min,循环8次;第5步,95℃15sec,80℃30sec,60℃30sec,72℃1min,循环2次;第6步,95℃15sec,60℃30sec,72℃1min,循环8次;第7步,95℃15sec,80℃30sec,60℃30sec,72℃1min,循环5次。
优选地,步骤s2所述mmpcr预扩增与步骤s3所述mmpcr分别在特定区段富集捕获系统的两个不同部分中完成。
优选地,步骤s1所述rna在提取过程中及逆转录之前,分别使用dnasei酶处理rna样品。
优选地,步骤s1所述rna样品于65℃变性,使用随机引物将rna逆转录合成cdna,并使用磁珠纯化cdna。
优选地,步骤s2所述超高重pcr预扩增产物在进行下一轮超高重pcr之前进行纯化,优选为磁珠纯化。
优选地,步骤s5所述回收纯化为割胶回收纯化200bp~500bp的电泳产物。
优选地,步骤s6所述高通量测序包括但不限于illumina测序技术,优选为illuminanextseq500测序仪、illuminamiseq测序仪或illuminahiseq2500测序仪。
更优选地,所述测序仪为illuminanextseq500测序仪。
上述高通量测序获得的数据可基于标签序列区分不同的mirna样品,通过生物信息学分析将单个样品测序获得的序列片段比对靶基因,得到mirna的修饰和编辑信息,定位和定量rna修饰信息。
因此,上述方法在定位和定量rna修饰信息方面的应用亦在本发明保护范围内。
同时,本发明还提供一种用于构建果蝇mirna高通量测序文库的试剂盒,所述试剂盒包括权利要求要求所述的247对探针,rna抽提试剂,逆转录试剂,mmpcr所需试剂,accessarrayloadingreagent,高保真聚合酶,磁珠纯化试剂盒以及凝胶回收试剂盒。
具体的,所述试剂盒的使用方法包括如下步骤:
(1)果蝇总rna提取
提取果蝇总rna,rna浓度和纯度用nanodrop2500光度计进行分析;在提取总rna的过程中及下一步逆转录之前,分别使用dnasei酶处理rna样品以消除残留的基因组dna。
(2)rna逆转录成cdna
将步骤(1)的rna样品于65℃变性,按照逆转录试剂盒说明书,使用随机引物,将总rna逆转录成cdna,按照磁珠纯化试剂盒说明书纯化cdna。
(3)mmpcr预扩增
把果蝇的247对特异性扩增探针混合在一起成为1个pool,对cdna进行超高重pcr预扩增,预扩增反应体系为:2×kapa2g5μl,cdna模板>100ng,50μm探针1.25μl,超纯水补充至10μl;反应程序为:95℃变性10min,95℃15sec,65℃4min循环两次,95℃15sec,72℃4min共13个循环;磁珠纯化mmpcr预扩增产物。
(4)mmpcr
以预扩增产物为模板,将247对特异性扩增探针分为40个pool,每个pool包含5~7对探针,使用所有探针pool对mirna进行超高重pcr(mmpcr);芯片的左侧三列中每个孔中加2×kapa2g2.5μl,20×accessarrayloadingreagent0.25μl,预扩增产物100ng,超纯水补充至5μl;芯片的右侧三列中加同等数量孔,每个孔加4μl的primersolutions;反应程序分为7步:第1步,50℃2min,70℃20min,95℃10min;第2步,95℃15sec,60℃30sec,72℃1min,循环15次;第3步,95℃15sec,80℃30sec,60℃30sec,72℃1min,循环2次;第4步,95℃15sec,60℃30sec,72℃1min,循环8次;第5步,95℃15sec,80℃30sec,60℃30sec,72℃1min,循环2次;第6步,95℃15sec,60℃30sec,72℃1min,循环8次;第7步,95℃15sec,80℃30sec,60℃30sec,72℃1min,循环5次;反应的同时在扩增产物两端分别接上3'接头和5'接头。
(5)barcodingpcr
将超高重pcr产物稀释100倍,对mmpcr产物进行barcodingpcr使每一个样品加上特有的标签序列(barcode)。
(6)mirna文库产物混合与纯化
将barcodingpcr产物按比例混合进行2.5%琼脂糖凝胶电泳,割胶回收纯化200bp~500bp长度的产物,即构建好mirna文库,测定mirna文库浓度,琼脂糖凝胶电泳检测文库质量。
(7)illumina测序
将得到的多个mirna文库混合,进行高通量测序。
在本发明采用了超高重pcr(mmpcr)扩增法结合illuminanextseq500高通量测序的建库测序方法,该方法以mirna转录组为模板,使用自主合成的mirna特异性探针pool,通过mmpcr原理对mirna进行靶向扩增,扩增区域包括pri-mirna上覆盖mirna前体(pre-mirna)的区域。扩增产物连接标签序列,进行illumina测序文库制备和测序。
与现有技术相比,本发明具有以下有益效果:
(1)本发明基于mmpcr扩增技术,利用果蝇mirna的序列信息特异性地设计出大量探针,能够特异性地靶向pri-mirna上mirna前体区域。
(2)本发明将自主设计的探针结合最新发展的mmpcr-seq高通量测序技术,发展出适用于构建mirna文库的mir-mmrna-seq高通量测序技术,能够高效并完整地构建果蝇pri-mirna上的mirna前体区域的转录组文库,极大的提高了前体mirna上rna编辑和修饰的检测敏感性,以及rna靶向测序文库的均一性,缩短了构建文库和测序周期,降低了成本。
附图说明
图1为随机挑选的16对mirna探针pcr测试结果电泳示意图。琼脂糖凝胶电泳显示pcr产物为一系列片段大小为100bp~180bp的一条带,其中泳道m为分子量标记物,泳道1~16分别为16个mirna探针的pcr产物电泳结果。电泳结果探针测试通过,可以用于mmpcr预扩增和mmpcr扩增。
图2为提取果蝇总rna后检测rna质量的电泳示意图。琼脂糖凝胶电泳显示6个样本来源的总rna,分别为片段大小1500bp~4700bp的两条带,其中泳道m为分子量标记物,泳道1~6分别为提取自6个样本的总rna电泳结果。rna质量合格即可进行下一步逆转录及之后的mmpcr-seq文库构建。
图3为agilentbioanalyzer2100分析mmpcr扩增产物的结果示意图,显示文库片段大小为200bp~350bp。
图4为barcodingpcr扩增产物电泳示意图。琼脂糖凝胶电泳显示pcr产物为一系列片段大小300bp~400bp的单一条带,其中泳道m为分子量标记物,泳道1~16为16个随机挑选的mmpcr产物通过barcodingpcr分别连接了特定barcode后的电泳结果。之后通过磁珠纯化即基本完成了mmpcr-seq测序文库制备。
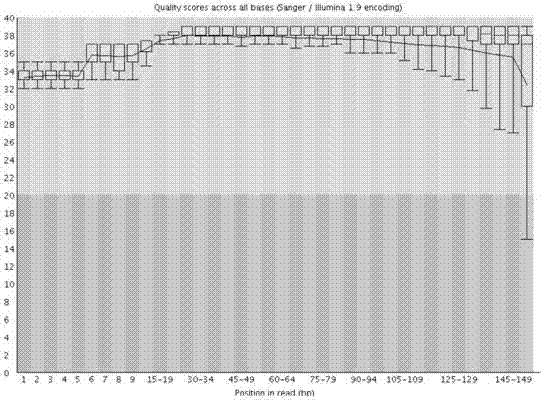
图5为mirna文库illuminanextseq500高通量测序结果示意图。y轴为测序质量quality,测序质量高低以≥q30的比例来衡量,越高质量越好。结果显示测序结果质量≥q30的比例为100%。
图6为测序的覆盖深度和覆盖率示意图。x轴为测到的mirna的数目,y轴为每一个mirna测到的read数。显示所有mirna平均覆盖深度达到300×,所有mirna覆盖率为90%。
具体实施方式
以下结合说明书附图和具体实施例来进一步说明本发明,但实施例并不对本发明做任何形式的限定。除非特别说明,本发明采用的试剂、方法和设备为本技术领域常规试剂、方法和设备。
除非特别说明,以下实施例所用试剂和材料均为市购。
实施例1特定靶向果蝇中microrna(mirna)的探针及使用此批探针构建mirna测序文库进行高通量测序的方法
1、设计探针
设计特异性扩增mirna的超高重pcr探针,所述探针特异性靶向扩增果蝇pri-mirna上覆盖的mirna前体(pre-mirna)的区域,所述探针共247对,其引物序列如表1所示,大部分探针扩增产物长度都在160~300bp。
表1靶向扩增果蝇mirna前体区域的探针序列表
2、总rna提取
提取果蝇总rna,rna浓度和纯度用nanodrop2500光度计进行分析;在提取总rna的过程中及下一步逆转录之前,分别使用dnasei酶处理rna样品以消除残留的基因组dna,提取的总rna作为mir-mmpcr-seq建库的起始模板。
3、rna逆转录成cdna
将rna样品于65℃变性,按照superscriptiii试剂盒说明书、使用随机引物,将总rna逆转录成cdna。逆转录反应在veriti96-wellthermalcyclerpcr仪(美国abi公司)上完成。按照axyprepmagpcrclean-μp(axygen)试剂盒说明书使用磁珠纯化逆转录产物cdna。
4、mmpcr预扩增
把所有的mirna特异性扩增探针混合在一起成为1个pool,按照kapa2gpcrkits及accessarrayloadingreagent(fluidigm)试剂盒说明书,对cdna进行超高重pcr预扩增,预扩增反应体系为2×kapa2g5μl,cdna模板>100ng,50μmpre-primer1.25μl,超纯水补充至10μl;反应参数为95℃变性10min,95℃15sec,65℃4min循环两次,95℃15sec,72℃4min共13个循环;mmpcr扩增反应在fludigmaccessarray特定区段富集捕获系统(fluidigm)上完成。按照axyprepmagpcrclean-μp(axygen)试剂盒说明书使用磁珠纯化mmpcr预扩增产物。
5、mmpcr
把果蝇的特异性扩增探针随机分为40个pool每个pool包含5~7对探针,按照kapa2gpcrkits及accessarrayloadingreagent(fluidigm)试剂盒说明书,使用所有探针pool对mirna进行超高重pcr(mmpcr),反应的体系为芯片的左侧三列中每个孔中加2×kapa2g2.5μl,20×accessarrayloadingreagent0.25μl,预扩增产物100ng,超纯水补充至5μl;芯片的右侧三列中加同等数量孔,每个孔加4μl的primersolutions;反应程序分为7步:第1步,50℃2min,70℃20min,95℃10min;第2步,95℃15sec,60℃30sec,72℃1min,循环15次;第3步,95℃15sec,80℃30sec,60℃30sec,72℃1min,循环2次;第4步,95℃15sec,60℃30sec,72℃1min,循环8次;第5步,95℃15sec,80℃30sec,60℃30sec,72℃1min,循环2次;第6步,95℃15sec,60℃30sec,72℃1min,循环8次;第7步,95℃15sec,80℃30sec,60℃30sec,72℃1min,循环5次。mmpcr反应在fludigmaccessarray特定区段富集捕获系统(美国fluidigm公司)上完成。反应的同时在扩增产物两端分别接上3'接头和5'接头。
7、barcodingpcr
将超高重pcr产物稀释100倍,按照phusion试剂盒说明书,对mmpcr产物进行barcodingpcr使每一个样品加上特有的标签序列(barcode),pcr反应在veriti96-wellthermalcyclerpcr仪(美国abi公司)上完成。
8、mirna文库产物混合与纯化
将barcodingpcr产物按比例混合进行2.5%琼脂糖凝胶电泳,按照zymocleangeldnarecoverykit(zymo)试剂盒说明书割胶回收纯化200bp~500bp长度的产物,即构建好mirna文库。使用qubit3.0测定mirna文库浓度,琼脂糖凝胶电泳检测文库质量。
9、illumina测序
将得到的多个mirna文库混合,使用illumina测序试剂盒,按照试剂盒说明书,进行高通量测序。测序反应在illuminanextseq500高通量测序仪(美国illumina公司)上完成,输出数据。
10、数据分析
采用illuminabcl2fastq软件进行测序原始数据提取。通过bwa软件将原始序列比对到基因组,通过samtools软件进行rna编辑位点的提取。
结果如图6所示,显示5例样本测序的覆盖深度和覆盖率,显示所有mirna平均覆盖深度达到300×,所有mirna覆盖率为90%。表1显示每个样本能测到的mirna种类达到80%。
通过本发明检测了5例果蝇样本,发现和定量了多个编辑位点得到的编辑位点与sanger测序的结果是一致的,说明本发明的方法可行。
- 还没有人留言评论。精彩留言会获得点赞!
- 一种构建Hi‑C高通量测序文库的方法与制造工艺
- 桑葚病原菌高通量鉴定及种属分类方法及其应用与制造工艺
- 基于高通量测序技术检测乳腺癌/卵巢癌易感基因的多重PCR特异性引物、试剂盒及方法与制造工艺
- 基于高通量测序检测人EGFR基因变异的质控方法及试剂盒与制造工艺
- 基于高通量测序技术的小麦病原微生物检测方法及其应用与制造工艺
- 一种基于二代高通量测序技术的地中海贫血症的基因检测试剂盒的制造方法与工艺
- 一种基于集群的高通量数据分析方法与制造工艺
- 高通量双折射干涉成像光谱仪装置及其成像方法与制造工艺
- 高通量测序检测人循环肿瘤DNA EGFR基因的捕获探针及试剂盒的制造方法与工艺
- 一种高通量测序检测无创亲子鉴定的方法与制造工艺