一种检测基因突变的方法、试剂盒及其制备方法与流程
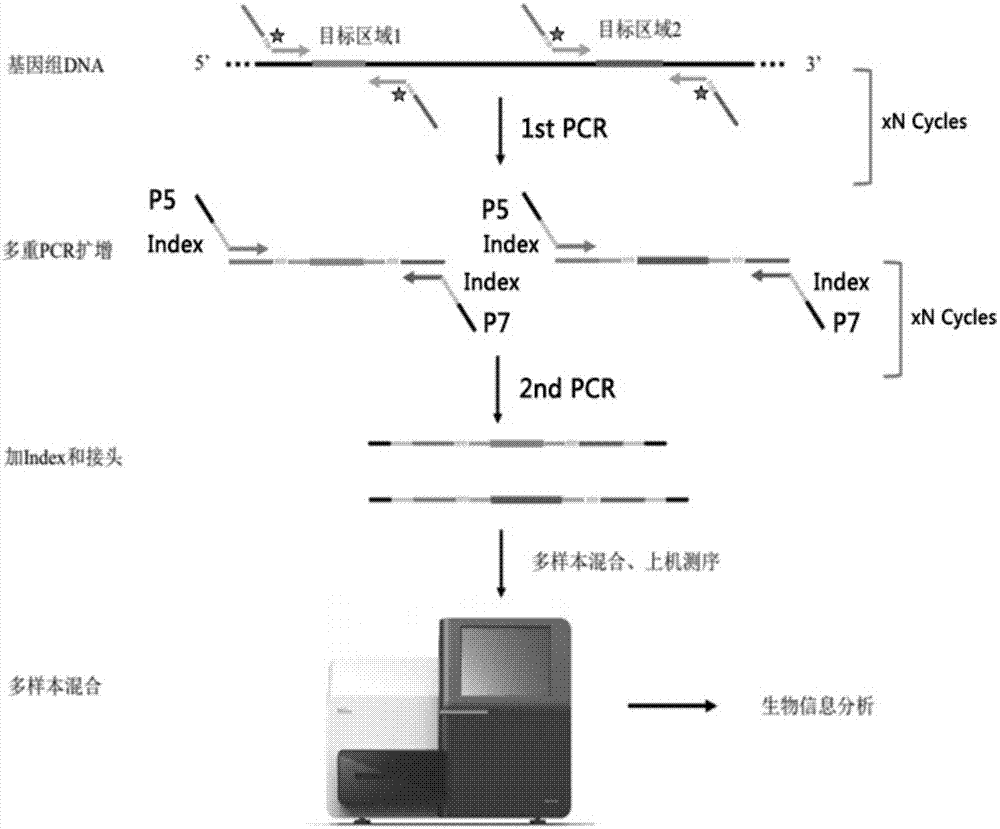
本发明涉及基因诊断领域,尤其涉及一种检测基因突变的方法,试剂盒及其制备方法。
背景技术:
基因突变的检测对于可用于鉴别待测样本的基因型是野生型还是突变型。也可以用于检测待测个体的基因状态。比如是否含有导致某些疾病的基因突变。许多种肿瘤检测,胎儿遗传状态检测都具有重要的价值。目前针对胎儿的基因突变筛查,主要基于snparray等多个平台系统,对胎儿及父母进行全基因组snp分析,成本昂贵;而常规依赖聚合酶的边合成边测序难以保证碱基准确性,发生的碱基错误造成测序噪声,因此常规文库捕获和高通量测序不能解决由扩增和测序造成的变异检测偏差;而较新的循环单分子扩增和重测序技术(csmart)技术受其环化文库效率和背靠背引物位点的设计的影响。孕妇外周血中胎儿来源的游离dna的发现,为无创性检测胎儿遗传状态提供了可能。孕妇外周血cfdna源于母亲和胎儿,胎儿游离dna(cffdna)来源于胎盘滋养层细胞,约占总cfdna的10-20%,片段长度为140-180bp左右。将cffdna从母亲来源的dna中鉴别出来仍然是目前的技术难点,特别是如何检测母血血浆中cffdna所导致的等位基因比例不平衡。目前国际上对cffdna的研究主要通过位点特异性pcr,maldi-tof质谱法,数字pcr和高通量测序等。目前主流的无创产前检测方法均基于胎儿变异位点检测和单体型检测:通过对母血血浆中cfdna进行位点检测,然后利用相对变异剂量(rmd)和相对单体型剂量(rhdo)分析孕妇血浆中胎儿变异位点和单体型的相对含量。通过仅存在父亲中的snp推导了父源部分的胎儿基因组,母亲来源的胎儿基因组部分则使用父亲纯合但母亲杂合的snp进行推导。papasavva等利用该方法对母血cfdna进行分析,成功对其cffdna基因型和变异位点进行分析。因此利用对cffdna中变异位点和单体型的检测,可有效地提高胎儿致病基因的诊断准确度。对于遗传性耳聋无创产前基因诊断的方法学研究,目前国际上仅有极少数报道。由于在耳聋热点变异中存在多个indel变异位点,如gjb2(c.299delat、c.235delc、c.176_191del16),不同的平台系统对其cfdna检测仍然存在多种问题。有必要针对开发一种更便捷和可靠的适合无创检测低拷贝数基因突变的技术。技术实现要素:因此上述领域存在问题和需求,发明人研发了一种快速准确的单分子扩增技术,在常规的多重扩增基础上加入了umi(uniquemolecularidentifier)分子标签,利用umi对二代测序数据进行降噪,从而准确地检测低拷贝数变异:具体方案如下:一种检测基因突变的方法,包括步骤如下:(1)以受试者血浆dna为模板;(2)进行pcr预扩增;(3)对预扩增产物进行indexpcr二次扩增;(4)对pcr二次扩增产物产物进行文库质控,然后在illuminanextseq进行150bp双端测序;(5)将测序的序列中连接接头去掉,然后拼接为一条序列得到原始模板序列;将原始模板序列与人类基因组比对,通过比较原始模板序列的umi分子标签序列,统计出唯一的模板序列集合;利用唯一的模板序列,计算基因组覆盖,用于评估文库特异性;通过计算突变序列与参照序列的比例统计出检测区域体细胞基因突变率;其特征在于:预扩增中采用的引物是多对引物的混合物,所述多对引物用于特异性扩增待测区域中的不同亚目标区域;所有的正向引物从5’端到3’端依次为含有接头序列a、umi分子标签序列和亚目标区域特异性引物序列;所有的反向引物从5’端到3’端依次为含有接头序列b、umi分子标签序列和亚目标区域特异性引物序列;所有引物对中的接头序列a彼此相同,接头序列b彼此相同,接头序列a与接头序列b之间不相同;所述待测区域包含1个或几个特定的基因序列区域,所述亚目标区域指一个特定基因中的不同区域的dna片段。每一对引物扩增基因中的一个亚目标区域,引物对之间包含的umi分子标签序列不同,用于保证来源于同一亚目标区域的dna扩增片段中的umi分子标签序列相同;而来源于不同亚目标区域的dna扩增片段之间的umi分子标签序列彼此不相同;在所述indexpcr二次扩增中,所述二次扩增引物的从3’端到5’端依次为接头序列、通用测序接头序列,其中一侧引物在接头序列、通用测序接头序列之间还包含一段index序;所述二次扩增引物的正向引物和反向引物的3’端分别为所述接头序列a和所述接头序列b的全部或部分;所述index序列用于标示不同样本,不同的样本采用不同的index序列,用于同一样本的二次扩增引物采用同一index序列。上述方法,其特征在于:所述接头序列a、接头序列a的长度为8-15bp,umi分子标签序列长度为5-8bp上述方法,其特征在于:在进行所述indexpcr扩增之前,对pcr预扩增所得dna进行磁珠纯化;上述方法,其特征在于:在进行所述indexpcr扩增之后,对扩增所得dna进行磁珠纯化。用于检测基因突变的试剂盒,其特征在于,包含用于特异性预扩增待测区域中的不同亚目标区域的多个引物对;所有的正向引物从5’端到3’端依次为含有接头序列a、umi分子标签序列和亚目标区域特异性引物序列;所有的反向引物从5’端到3’端依次为含有接头序列b、umi分子标签序列和亚目标区域特异性引物序列;所有引物对中的接头序列a彼此相同,接头序列b彼此相同,接头序列a与接头序列b之间不相同;同一来源的dna片段中的umi分子标签序列相同;而不同来源的dna片段之间的umi分子标签序列彼此不相同;所述待测区域包含1个或几个特定的基因序列区域,所述亚目标区域指一个特定基因中的不同位置区域。上述试剂盒,其特征在于,还包含用于对所述预扩增产物进行扩增的二次扩增引物,所述二次扩增引物的从3’端到5’端依次为接头序列、通用测序接头序列,其中一侧引物在接头序列、通用测序接头序列之间还包含一段index序;所述二次扩增引物的正向引物和反向引物的3’端分别为所述接头序列a和所述接头序列b的全部或部分;所述index序列用于标示不同样本,不同的样本采用不同的index序列,用于同一样本的二次扩增引物采用同一index序列。上述试剂盒,其特征在于:所述接头序列a、接头序列a的长度为8-15bp,umi分子标签序列长度为5-7bp。用于检测基因突变的试剂盒的制备方法,其特征在于,包括合成和/或组装具有以下序列特征的多个引物对,所有的正向引物从5’端到3’端依次为含有接头序列a、umi分子标签序列和亚目标区域特异性引物序列;所有的反向引物从5’端到3’端依次为含有接头序列b、umi分子标签序列和亚目标区域特异性引物序列;所有引物对中的接头序列a彼此相同,接头序列b彼此相同,接头序列a与接头序列b之间不相同;同一来源的dna片段中的umi分子标签序列相同;而不同来源的dna片段之间的umi分子标签序列彼此不相同;所述多个引物对用于特异性预扩增待测区域中的不同亚目标区域;所述待测区域包含1个或几个特定的基因序列区域,所述亚目标区域指一个特定基因中的不同位置区域。上述制备方法,其特征在于,还包含合成和/或组装用于对所述预扩增产物进行扩增的二次扩增引物,所述二次扩增引物的从3’端到5’端依次为接头序列、通用测序接头序列,其中一侧引物在接头序列、通用测序接头序列之间还包含一段index序;所述二次扩增引物的正向引物和反向引物的3’端分别为所述接头序列a和所述接头序列b的全部或部分;所述index序列用于标示不同样本,不同的样本采用不同的index序列,用于同一样本的二次扩增引物采用同一index序列。上述制备方法,其特征在于:所述接头序列a、接头序列a的长度为8-15bp,umi分子标签序列长度为5-7bp。上述方法在gjb2基因突变和slc26a4基因突变检测中的应用衍生出以下技术方案一种无创检测gjb2和/或slc26a4基因突变的方法,包括步骤如下:(1)以受试者血浆dna为模板;(2)进行pcr预扩增;(3)对预扩增产物进行indexpcr二次扩增;(4)对pcr二次扩增产物产物进行文库质控,然后在illuminanextseq进行150bp双端测序;(5)将测序的序列中连接接头去掉,然后拼接为一条序列得到原始模板序列,将原始模板序列与人类基因组比对,通过比较原始模板序列的umi分子标签序列,统计出唯一的模板序列集合;利用唯一的模板序列,计算基因组覆盖,用于评估文库特异性;通过计算突变序列与参照序列的比例统计出检测区域体细胞基因突变率;其特征在于:预扩增中采用seqidno.1-46,89-91,94-96所示的引物构成的26对引物的混合物用于特异性扩增gjb2基因的不同亚目标区域;预扩增中采用seqidno.47-88,92-93,97-98所示的引物构成的23对引物的混合物用于特异性扩增slc26a4基因的不同亚目标区域;或者,预扩增中采用seqidno.1-98所示的引物构成的49对引物的混合物用于同时特异性扩增gjb2基因和slc26a4基因的不同亚目标区域;从而同时检测gjb2基因和slc26a4基因的变异;所述待测区域包含1个或几个特定的基因序列区域,所述亚目标区域指一个特定基因中的不同区域的dna片段;每一对引物扩增基因中的一个亚目标区域,引物对之间包含的umi分子标签序列不同,用于保证来源于同一亚目标区域的dna扩增片段中的umi分子标签序列相同;而来源于不同亚目标区域的dna扩增片段之间的umi分子标签序列彼此不相同;在所述indexpcr二次扩增中,采用seqidno.99和100所示的正反向引物。上述方法,其特征在于:在进行所述indexpcr扩增之前,对pcr预扩增所得dna进行磁珠纯化;上述方法,其特征在于:在进行所述indexpcr扩增之后,对扩增所得dna进行磁珠纯化。用于无创检测gjb2和/或slc26a4基因突变的试剂盒,其特征在于,包括seqidno.1-46,89-91,94-96所示的引物构成的26对引物的混合物,用于特异性扩增gjb2基因的不同亚目标区域;包括seqidno.47-88,92-93,97-98所示的引物构成的23对引物的混合物用于特异性扩增slc26a4基因的不同亚目标区域;或者,包括seqidno.1-98所示的引物构成的49对引物的混合物用于同时特异性扩增gjb2基因和slc26a4基因的不同亚目标区域;从而同时检测gjb2gjb2基因和slc26a4基因的变异。上述试剂盒,其特征在于,还包含用于对所述预扩增产物进行扩增的二次扩增引物,所述二次扩增引物的正反向引物如seqidno.99和100所示。用于检测gjb2基因突变的试剂盒的制备方法,其特征在于,包括合成和/或组装具有seqidno.1-46,89-91,94-96所示核苷酸序列的引物。或用于检测slc26a4基因突变的试剂盒的制备方法,其特征在于,包括合成和/或组装具有seqidno.47-88,92-93,97-98所示核苷酸序列的引物。或用于检测gjb2基因和slc26a4基因突变的试剂盒的制备方法,其特征在于,包括合成和/或组装具有seqidno.1-98所示核苷酸序列的引物。上述制备方法,其特征在于,还包含合成和/或组装用于对所述预扩增产物进行扩增的二次扩增引物,所述二次扩增引物的正反向引物如seqidno.99和100所示。根据本发明提供的基因突变检测方法,针对具体的待测基因,本领域技术人员可以根据选择的各个亚目标区域设计筛选扩增针对不同亚目标区域的特异性引物序列,使得这些引物对之间在同一反应中能够互相干扰最小,保证检测准确性和灵敏性。再根据引物的一般设计规则设计或选择适合所有引物对的接头序列,为不同引物对设计umi分子标签序列,使得扩增同一来源的dna片段的引物采用相同的umi分子标签序列,而扩增不同来源的dna片段的引物之间的umi分子,即可得到针对具体待测基因的预扩增引物组。可用于孕妇外周血cfdna检测胎儿的遗传状态,例如耳聋基因等单基因病,以及通过肿瘤基因游离dna基因癌症检测。本发明的基因突变方法,通过引入umi分子标签,通过生物信息分析将带有相同umi序列的扩增测序产物进行合并,极大地降低了由dna聚合酶扩增过程和二代测序过程所引入的错误和假阳性率,通过降噪实现对低拷贝数变异进行准确检测。实验例通过检测已知gjb2和slc26a4突变位点,验证方法的可靠性。附图说明图1是本发明的原理示意图:首先采用混合引物对血浆dna进行预扩增,目的是扩增所需位点产物;然后将预扩增产物dna纯化,目的是去除体系中的酶和离子;针对预扩增产物进行indexpcr二次扩增,制作成可用于高通量测序的文库;最后对文库进行生物信息学分析得出基因突变数据。图2.生物信息学分析过程示例对带有相同分子标签的序列(即同一样本来源)进行组装成唯一的模板序列具体实施方式本发明的实施例中,提供了一种检测基因突变的方法,包括步骤如下:一种检测基因突变的方法,包括步骤如下:(1)以受试者血浆dna为模板;(2)进行pcr预扩增;(3)对预扩增产物进行indexpcr二次扩增;(4)对pcr二次扩增产物产物进行文库质控,然后在illuminanextseq进行150bp双端测序;(5)将测序的序列中连接接头去掉,然后拼接为一条序列得到原始模板序列;将原始模板序列与人类基因组比对,通过比较原始模板序列的umi分子标签序列,统计出唯一的模板序列集合;利用唯一的模板序列,计算基因组覆盖,用于评估文库特异性;通过计算突变序列与参照序列的比例统计出检测区域体细胞基因突变率;其特征在于:预扩增中采用的引物是多对引物的混合物,所述多对引物用于特异性扩增待测区域中的不同亚目标区域;所有的正向引物从5’端到3’端依次为含有接头序列a、umi分子标签序列和亚目标区域特异性引物序列;所有的反向引物从5’端到3’端依次为含有接头序列b、umi分子标签序列和亚目标区域特异性引物序列;所有引物对中的接头序列a彼此相同,接头序列b彼此相同,接头序列a与接头序列b之间不相同;所述待测区域包含1个或几个特定的基因序列区域,所述亚目标区域指一个特定基因中的不同区域的dna片段。每一对引物扩增基因中的一个亚目标区域,引物对之间包含的umi分子标签序列不同,用于保证来源于同一亚目标区域的dna扩增片段中的umi分子标签序列相同;而来源于不同亚目标区域的dna扩增片段之间的umi分子标签序列彼此不相同;在所述indexpcr二次扩增中,所采用二次扩增引物的从3’端到5’端依次为接头序列、通用测序接头序列,其中一侧引物在接头序列、通用测序接头序列之间还包含一段index序;所述二次扩增引物的正向引物和反向引物的3’端分别为所述接头序列a和所述接头序列b;所述index序列用于标示不同样本,不同的样本采用不同的index序列,用于同一样本的二次扩增引物采用同一index序列。针对具体的待测基因,本领域技术人员可以根据选择的亚目标区域设计扩增该区域的亚目标区域特异性引物序列,根据引物的一般设计规则设计或选择适合的接头序列,并为不同的引物对随机设计umi分子标签序列,即可得到针对具体待测基因的预扩增引物组。例如可用于孕妇外周血cfdna检测胎儿的遗传状态,例如耳聋基因等单基因病,以及通过肿瘤基因游离dna基因癌症检测。一些实施例中,所述接头序列a、接头序列a的长度为8-15bp,umi分子标签序列长度为5-8bp一些实施例中,在进行所述indexpcr二次扩增之前,对pcr预扩增所得dna进行磁珠纯化;一些实施例中,在进行所述indexpcr扩增之后,对扩增所得dna进行磁珠纯化。本发明的实施,还包括提供用于检测基因突变的试剂盒,其特征在于,包含用于特异性预扩增待测区域中的不同亚目标区域的多个引物对;所有的正向引物从5’端到3’端依次为含有接头序列a、umi分子标签序列和亚目标区域特异性引物序列;所有的反向引物从5’端到3’端依次为含有接头序列b、umi分子标签序列和亚目标区域特异性引物序列;所有引物对中的接头序列a彼此相同,接头序列b彼此相同,接头序列a与接头序列b之间不相同;所述待测区域包含1个或几个特定的基因序列区域,所述亚目标区域指一个特定基因中的不同位置区域;每一对引物扩增基因中的一个亚目标区域,引物对之间包含的umi分子标签序列不同,用于保证来源于同一亚目标区域的dna扩增片段中的umi分子标签序列相同;而来源于不同亚目标区域的dna扩增片段之间的umi分子标签序列彼此不相同。一些试剂盒中,还包含用于对所述预扩增产物进行扩增的二次扩增引物,所述二次扩增引物从3’端到5’端依次为接头序列、通用测序接头序列,其中一侧引物在接头序列、通用测序接头序列之间还包含一段index序;所述二次扩增引物的正向引物和反向引物的3’端分别为所述接头序列a和所述接头序列b;所述index序列用于标示不同样本,不同的样本采用不同的index序列,用于同一样本的二次扩增引物采用同一index序列。正反向引物3’序列分别为接头序列a和接头序列b(二次扩增引物的5’端序列针对illumina测序仪的文库要求进行设计)一些试剂盒中,优选所述接头序列a、接头序列a的长度为8-15bp,umi分子标签序列长度为5-7bp。本发明的实施,还包括提供用于检测基因突变的试剂盒的制备方法,其特征在于,包括合成和/或组装具有以下序列特征的多个引物对,所有的正向引物从5’端到3’端依次为含有接头序列a、umi分子标签序列和亚目标区域特异性引物序列;所有的反向引物从5’端到3’端依次为含有接头序列b、umi分子标签序列和亚目标区域特异性引物序列;所有引物对中的接头序列a彼此相同,接头序列b彼此相同,接头序列a与接头序列b之间不相同;所述待测区域包含1个或几个特定的基因序列区域,所述亚目标区域指一个特定基因中的不同位置区域;每一对引物扩增基因中的一个亚目标区域,引物对之间包含的umi分子标签序列不同,用于保证来源于同一亚目标区域的dna扩增片段中的umi分子标签序列相同;而来源于不同亚目标区域的dna扩增片段之间的umi分子标签序列彼此不相同。在一些制备方案中,还包含合成和/或组装用于对所述预扩增产物进行扩增的二次扩增引物,所采用二次扩增引物的从3’端到5’端依次为接头序列、通用测序接头序列,其中一侧引物在接头序列、通用测序接头序列之间还包含一段index序;所述二次扩增引物的正向引物和反向引物的3’端分别为所述接头序列a和所述接头序列b;所述index序列用于标示不同样本,不同的样本采用不同的index序列,用于同一样本的二次扩增引物采用同一index序列。在一些制备方案中,所述接头序列a、接头序列a的长度为8-15bp,umi分子标签序列长度为5-7bp。应用实施例检测gjb2和slc26a4基因突变gjb2基因编码缝隙连接蛋白26,全长678bp,含两个外显子。其蛋白表达于内耳,对维持耳蜗内正常电位具有重要作用。其基因突变可导致常染色体隐性遗传性耳聋和常染色体显性遗传性耳聋,突变所致耳聋约占全部常染色体隐性遗传性耳聋的一半,占散发病例中的60%。slc26a4基因定位于染色体7q22.3上,全长2343bp。其编码蛋白pendrin主要在内耳、甲状腺和肾脏中表达,其功能与氯离子和碳酸氢离子转运相关。slc26a4基因突变可导致pendred综合征和dfnb4型非综合征性耳。gaoz利用snpscan技术对695个中国耳聋患者筛查发现38.7%的耳聋患者是由于gjb2和slc26a4基因变异所导致,并且发现在这些基因中突变呈明显的奠基者效应,因此对这两个基因导致的遗传性耳聋研究具有重要的临床意义。因此,在临床上迫切需要一种快速,高效的检测gjb2和slc26a4基因突变的方法。本发明人在研究片段dna的检测时,发现了一种新的dna片段的检测方法,即通过对扩增引物添加分子标签,然后对dna片段进行扩增。在此基础上,结合第二代高通量测序技术,特别根据gjb2和slc26a4基因对该方法进行了改进,并特别根据gjb2和slc26a4设计了优选的引物,开发了一种通过血浆dna对gjb2和slc26a4基因的突变进行测序和分析的方法以及其试剂盒。对于遗传性耳聋无创产前基因诊断的方法学研究,目前国际上仅有极少数报道。由于在耳聋热点变异中存在多个indel变异位点,如gjb2(c.299delat、c.235delc、c.176_191del16),不同的平台系统对其cfdna检测仍然存在多种问题。如基于snparray等多个平台系统,对胎儿及父母进行全基因组snp分析,成本昂贵;常规依赖聚合酶的边合成边测序难以保证碱基准确性,将产生碱基错误的测序噪声,导致常规文库捕获和高通量测序仍不能解决在变异中由扩增和测序造成偏差;较新的循环单分子扩增和重测序技术(csmart)技术受其环化文库效率和背靠背引物位点的设计的影响。本发明的方法很好地解决了这一问题,在常规的多重扩增基础上加入了umi(uniquemolecularidentifier)分子标签,利用umi对二代测序数据进行降噪,从而对低拷贝数变异进行检测。通过检测gjb2和slc26a4突变位点已知的模板验证方法的可靠性细胞系dna:将含有gjb2和slc26a4基因热点变异的多个dna样本(均来源于陆军军医大学第一附属医院dna样本库)超声破碎,回收166±10bp片断按预设比例混入正常人血浆dna得到突变比例已知的待测样品(根据阳性dna浓度计算)):该样品的gjb2基因热点突变比例预期如下突变位点预期突变比例c.235delc5.50%c.299_300delat6.20%c.176_191del169.30%该样品的slc26a4基因热点突变比例预期如下:突变位点预期突变比例nm_000441:c.919-2a>g2.70%nm_000441:c.2168a>g4.50%测试本发明方法的灵敏性及稳定性。预扩增引物mix:将表1中的第一组中的引物或第二组中的引物,或第一和第二组全部的引物序列加入1.5mlep管中表1预扩增引物组seqidno.1-46,89-91,94-96所示的引物构成的26对引物的混合物,用于特异性扩增gjb2基因的不同亚目标区域的snp位点和热点变异;seqidno.47-88,92-93,97-98所示的引物构成的23对引物的混合物用于特异性扩增slc26a4基因的不同亚目标区域的snp位点和热点变异;每条引物中的“nnnnnn”是一种合成的a/g/c/t随机序列,也就是umi“uniquemolecularidentifier”。每对引物中的umi彼此不同。附注:正向引物中的5’端序列“cctacacgacgctcttccgatct”为接头序列a;反向引物5’端序列“tcagacgtgtgctcttccgatct”为接头序列b.3.pcr预扩增制备如下的反应混合液dna24.8ulbuffer10x3uldntp(2.5mm)1ulprimermix1ultaqpolymerase0.2ul30ulpcr仪上反应:在beckman磁珠上纯化回收dna样品,并在25μ1的无菌dh20或洗脱缓冲液中洗脱。4.indexpcr扩增表2index引物99primerfor:5-aatgatacggcgaccaccgagatctacacacactctttccctacacgacgc-3100indexprimerrev:5-caagcagaagacggcatacgagatctcttaattgactggagttcagacgtgtgct-3primerf中5’端和indexprimerrev5’端序列为illumina公司的公开通用测序接头;反向引物中“ctcttaat”为index序列,用于区分不同样本,3’端序列“cctacacgacgc”,indexprimerrev3’端“tcagacgtgtgct”为表1中引物的接头a和接头b的5’端序列。pcr仪上反应:在beckman磁珠上纯化回收dna样品,并在25μ1的无菌dh20或洗脱缓冲液中洗脱得到文库。将制作好的文库通过qubit检测浓度质控后,illuminanextseq进行150bp双端测序(操作步骤依据illumina公司操作说明进行操作)。将高通量测序的序列依据重复区域拼接为一条序列,去除连接接头,将序列还原回原始模板序列,将模板比对到人类基因组(hg19),通过比较模板序列的分子标签序列,统计出唯一的模板序列集合,如图2:对带有相同分子标签的序列进行组装成唯一的模板序列。利用唯一的模板序列,计算基因组覆盖,用于评估文库特异性,通过计算突变序列与参照序列的比例统计检测区域体细胞突变。传统不带分子标签的引物扩增后,测序并不能区分其相同来源的不同序列。我们通过引入umi分子标签,通过生物信息分析将带有相同来源umi的序列进行合并,极大地降低了由dna聚合酶扩增过程和二代测序过程所引入的错误,通过降噪我们可实现对低拷贝数的变异进行准确检测。针对gjb2基因热点突变位点检测结果如下:突变位点突变比例检测结果c.235delc5.59%检出c.299_300delat6.14%检出c.176_191del169.44%检出针对slc26a4基因热点突变位点检测结果如下:突变位点突变比例检测结果nm_000441:c.919-2a>g2.87%检出nm_000441:c.2168a>g4.56%检出上表是两个基因低比例热点突变的检测结果,与预期结果相符。同时,本发明的方法的检测结果与其他方法的检测结果也高度一致。说明本发明的方法能够准确检测低拷贝突变。针对同样的样本,使用ddpcr法对其中的一个位点突变比例进行了检测,结果与本发明方法检测结果一致。但是ddpcr只能针对单个位点突变进行检测。在一定条件下,针对不同的已知突变,设计适当的引物可以通过pcr扩增方法结合sanger测序直接达到区分突变型和野生型基因的目的。该方法主要针对外周血样本。采用pcr扩增结合针对已知位点检测外周血样本突变类型,然后用本发明方法进行血浆模拟dna检测。最后说明的是,以上实施例仅用以说明本发明的技术方案而非限制,尽管参照较佳实施例对本发明进行了详细说明,本领域的普通技术人员应当理解,可以对本发明的技术方案进行修改或者等同替换,而不脱离本发明技术方案的宗旨和范围,其均应涵盖在本发明的权利要求范围当中。sequencelisting<110>中国人民解放军陆军军医大学第一附属医院<120>一种检测基因突变的方法、试剂盒及其制备方法<130>p1830152cn-cn-ljy-cq-lsh<160>100<170>patentinversion3.3<210>1<211>51<212>dna<213>artificialsequence<220><223>rs7999318_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>1cctacacgacgctcttccgatctnnnnnnaagtgacccgctgtgcatgtat51<210>2<211>51<212>dna<213>artificialsequence<220><223>rs4769954_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>2cctacacgacgctcttccgatctnnnnnnacaggagatgcaggcagtcaac51<210>3<211>51<212>dna<213>artificialsequence<220><223>rs9509170_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>3cctacacgacgctcttccgatctnnnnnngatcccatcagaaggaccctgt51<210>4<211>54<212>dna<213>artificialsequence<220><223>rs1537788_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>4cctacacgacgctcttccgatctnnnnnngagatgcttcaaaatacaggaatgg54<210>5<211>54<212>dna<213>artificialsequence<220><223>rs9552134_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>5cctacacgacgctcttccgatctnnnnnntgcagagatgaaattgtacttgcag54<210>6<211>50<212>dna<213>artificialsequence<220><223>rs12585595_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>6cctacacgacgctcttccgatctnnnnnnccagccattctctcccatgtt50<210>7<211>55<212>dna<213>artificialsequence<220><223>rs9509191_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>7cctacacgacgctcttccgatctnnnnnntgagctacaatcgaactcatctttcc55<210>8<211>51<212>dna<213>artificialsequence<220><223>rs11147616_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>8cctacacgacgctcttccgatctnnnnnnctctgcttcaggggcttgttct51<210>9<211>52<212>dna<213>artificialsequence<220><223>rs9552150_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>9cctacacgacgctcttccgatctnnnnnncaggtaggacccaagatttgtgc52<210>10<211>51<212>dna<213>artificialsequence<220><223>rs9509098_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>10cctacacgacgctcttccgatctnnnnnnggagaagatgcctcctccattc51<210>11<211>49<212>dna<213>artificialsequence<220><223>rs870728_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>11cctacacgacgctcttccgatctnnnnnngactgcctgacccctccaac49<210>12<211>51<212>dna<213>artificialsequence<220><223>rs35967187_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>12cctacacgacgctcttccgatctnnnnnngagtgaccaccctggcacataa51<210>13<211>52<212>dna<213>artificialsequence<220><223>rs2313485_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>13cctacacgacgctcttccgatctnnnnnnccatcagatgtggcattcagaaa52<210>14<211>51<212>dna<213>artificialsequence<220><223>rs7327952_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>14cctacacgacgctcttccgatctnnnnnnaccatcacccatcacccctaat51<210>15<211>52<212>dna<213>artificialsequence<220><223>rs8000719_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>15cctacacgacgctcttccgatctnnnnnnggttgtgacataaacaggggaca52<210>16<211>48<212>dna<213>artificialsequence<220><223>rs9550644_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>16cctacacgacgctcttccgatctnnnnnntgctgggcaggatggagac48<210>17<211>50<212>dna<213>artificialsequence<220><223>rs945368_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>17cctacacgacgctcttccgatctnnnnnngatttttctgggtccccaagc50<210>18<211>50<212>dna<213>artificialsequence<220><223>rs9506430_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>18cctacacgacgctcttccgatctnnnnnncagactgctcaagccctctcc50<210>19<211>51<212>dna<213>artificialsequence<220><223>rs6490533_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>19cctacacgacgctcttccgatctnnnnnnactctctgcgtgaggtgagtgg51<210>20<211>50<212>dna<213>artificialsequence<220><223>rs2872371_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>20cctacacgacgctcttccgatctnnnnnncggctctgtgctaggaaccag50<210>21<211>51<212>dna<213>artificialsequence<220><223>rs9552160_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>21cctacacgacgctcttccgatctnnnnnnttggcacagatgcagacctctt51<210>22<211>52<212>dna<213>artificialsequence<220><223>rs9550642_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>22cctacacgacgctcttccgatctnnnnnnggaaccttgagtactgggctgaa52<210>23<211>51<212>dna<213>artificialsequence<220><223>rs7988514_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>23cctacacgacgctcttccgatctnnnnnntccctcaggtcccatttagctc51<210>24<211>51<212>dna<213>artificialsequence<220><223>rs7999318_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>24tcagacgtgtgctcttccgatctnnnnnngagagtggagagggagctgagg51<210>25<211>51<212>dna<213>artificialsequence<220><223>rs4769954_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>25tcagacgtgtgctcttccgatctnnnnnngggacaggagaccctgtgtttt51<210>26<211>51<212>dna<213>artificialsequence<220><223>rs9509170_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>26tcagacgtgtgctcttccgatctnnnnnngagtccccttctccctgaggtt51<210>27<211>58<212>dna<213>artificialsequence<220><223>rs1537788_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>27tcagacgtgtgctcttccgatctnnnnnnaaaacaaaatataagttttaggggctgaa58<210>28<211>51<212>dna<213>artificialsequence<220><223>rs9552134_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>28tcagacgtgtgctcttccgatctnnnnnnggtcttttgtgggggttgagac51<210>29<211>53<212>dna<213>artificialsequence<220><223>rs12585595_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>29tcagacgtgtgctcttccgatctnnnnnncacagcagacctcaacagacagaa53<210>30<211>51<212>dna<213>artificialsequence<220><223>rs9509191_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>30tcagacgtgtgctcttccgatctnnnnnngccctacacttccattcccatc51<210>31<211>47<212>dna<213>artificialsequence<220><223>rs11147616_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>31tcagacgtgtgctcttccgatctnnnnnncctggccaacaaccacca47<210>32<211>51<212>dna<213>artificialsequence<220><223>rs9552150_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>32tcagacgtgtgctcttccgatctnnnnnntttacaaatgccagccaattcc51<210>33<211>51<212>dna<213>artificialsequence<220><223>rs9509098_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>33tcagacgtgtgctcttccgatctnnnnnntggttgtacgggtattgcatga51<210>34<211>59<212>dna<213>artificialsequence<220><223>rs870728_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>34tcagacgtgtgctcttccgatctnnnnnntgtaatcactatagacagacccttttcagc59<210>35<211>50<212>dna<213>artificialsequence<220><223>rs35967187_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>35tcagacgtgtgctcttccgatctnnnnnngcgagggaggtctcggataag50<210>36<211>51<212>dna<213>artificialsequence<220><223>rs2313485_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>36tcagacgtgtgctcttccgatctnnnnnntttccccaactattccacagca51<210>37<211>52<212>dna<213>artificialsequence<220><223>rs7327952_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>37tcagacgtgtgctcttccgatctnnnnnngatcagcagttgtcagaggctgt52<210>38<211>51<212>dna<213>artificialsequence<220><223>rs8000719_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>38tcagacgtgtgctcttccgatctnnnnnnaaggaaaatggtgcatgcctac51<210>39<211>48<212>dna<213>artificialsequence<220><223>rs9550644_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>39tcagacgtgtgctcttccgatctnnnnnngcgccacttccctgttgaa48<210>40<211>51<212>dna<213>artificialsequence<220><223>rs945368_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>40tcagacgtgtgctcttccgatctnnnnnnggcataggtggattgccaattt51<210>41<211>52<212>dna<213>artificialsequence<220><223>rs9506430_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>41tcagacgtgtgctcttccgatctnnnnnngaggttgctctaatcccacctga52<210>42<211>49<212>dna<213>artificialsequence<220><223>rs6490533_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>42tcagacgtgtgctcttccgatctnnnnnnctgcgtcaacggctcctatg49<210>43<211>49<212>dna<213>artificialsequence<220><223>rs2872371_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>43tcagacgtgtgctcttccgatctnnnnnncactggggaaagggcaaatc49<210>44<211>51<212>dna<213>artificialsequence<220><223>rs9552160_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>44tcagacgtgtgctcttccgatctnnnnnntccaccaagaggaagaaaagga51<210>45<211>51<212>dna<213>artificialsequence<220><223>rs9550642_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>45tcagacgtgtgctcttccgatctnnnnnntcagaggacagttccctgttgg51<210>46<211>51<212>dna<213>artificialsequence<220><223>rs7988514_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>46tcagacgtgtgctcttccgatctnnnnnnccttttcttttgggcacgtttt51<210>47<211>57<212>dna<213>artificialsequence<220><223>rs28690459_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>47cctacacgacgctcttccgatctnnnnnnacaaaagccttcttaagtttaggaaaca57<210>48<211>56<212>dna<213>artificialsequence<220><223>rs73421853_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>48cctacacgacgctcttccgatctnnnnnntggtttaaaggagtatctggagtcaaa56<210>49<211>54<212>dna<213>artificialsequence<220><223>rs17154507_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>49cctacacgacgctcttccgatctnnnnnntgacgttatcaggatacggctatct54<210>50<211>51<212>dna<213>artificialsequence<220><223>rs6944998_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>50cctacacgacgctcttccgatctnnnnnncctgggctcacttgacacaaac51<210>51<211>59<212>dna<213>artificialsequence<220><223>rs10276013_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>51cctacacgacgctcttccgatctnnnnnnccacctattagcttctaaaacaatgtaagg59<210>52<211>51<212>dna<213>artificialsequence<220><223>rs7805114_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>52cctacacgacgctcttccgatctnnnnnnaaaccagtcatgggccttagga51<210>53<211>56<212>dna<213>artificialsequence<220><223>rs10242548_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>53cctacacgacgctcttccgatctnnnnnntggttacaaacataaccctgttattgc56<210>54<211>51<212>dna<213>artificialsequence<220><223>rs6949189_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>54cctacacgacgctcttccgatctnnnnnntgcagacgggtttgaaattttg51<210>55<211>59<212>dna<213>artificialsequence<220><223>rs1008707_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>55cctacacgacgctcttccgatctnnnnnntcactcaactcttaaaagaatcctacttga59<210>56<211>55<212>dna<213>artificialsequence<220><223>rs78502829_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>56cctacacgacgctcttccgatctnnnnnncatgacagccattctacagtttatgc55<210>57<211>51<212>dna<213>artificialsequence<220><223>rs6466197_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>57cctacacgacgctcttccgatctnnnnnnattgtccccaggccaataaact51<210>58<211>52<212>dna<213>artificialsequence<220><223>rs10487275_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>58cctacacgacgctcttccgatctnnnnnnagggacaggatccagtagaaagg52<210>59<211>51<212>dna<213>artificialsequence<220><223>rs3823958_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>59cctacacgacgctcttccgatctnnnnnncaaggtagccagagaagggtga51<210>60<211>54<212>dna<213>artificialsequence<220><223>rs6980029_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>60cctacacgacgctcttccgatctnnnnnntgcacttaaaggtagaggctcagaa54<210>61<211>51<212>dna<213>artificialsequence<220><223>rs4730261_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>61cctacacgacgctcttccgatctnnnnnnctctgaggatggctatggctgt51<210>62<211>51<212>dna<213>artificialsequence<220><223>rs57592817_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>62cctacacgacgctcttccgatctnnnnnnaggctgccgtagatttggtctg51<210>63<211>48<212>dna<213>artificialsequence<220><223>rs2253856_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>63cctacacgacgctcttccgatctnnnnnngtgagcccagtcccctgaa48<210>64<211>51<212>dna<213>artificialsequence<220><223>rs982663_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>64cctacacgacgctcttccgatctnnnnnnagcattggctgagatccattgt51<210>65<211>53<212>dna<213>artificialsequence<220><223>rs6965933_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>65cctacacgacgctcttccgatctnnnnnngctgttatctgttccaaccaagga53<210>66<211>52<212>dna<213>artificialsequence<220><223>rs2712208_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>66cctacacgacgctcttccgatctnnnnnngcaggtcaagacaccaacaatca52<210>67<211>55<212>dna<213>artificialsequence<220><223>rs12534598_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>67cctacacgacgctcttccgatctnnnnnntcaacaacttaaatgtacacgcacag55<210>68<211>53<212>dna<213>artificialsequence<220><223>rs28690459_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>68tcagacgtgtgctcttccgatctnnnnnnttgcctccagttcactcctaaaag53<210>69<211>53<212>dna<213>artificialsequence<220><223>rs73421853_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>69tcagacgtgtgctcttccgatctnnnnnngggggaaaatagacaagaagaagg53<210>70<211>57<212>dna<213>artificialsequence<220><223>rs17154507_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>70tcagacgtgtgctcttccgatctnnnnnnaacatttatcagtagttccaagccaact57<210>71<211>49<212>dna<213>artificialsequence<220><223>rs6944998_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>71tcagacgtgtgctcttccgatctnnnnnncccacagatgcacccaagat49<210>72<211>58<212>dna<213>artificialsequence<220><223>rs10276013_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>72tcagacgtgtgctcttccgatctnnnnnnggatggagtgtaatagtatgggattatgg58<210>73<211>52<212>dna<213>artificialsequence<220><223>rs7805114_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>73tcagacgtgtgctcttccgatctnnnnnngctagtccatcaacttgccttca52<210>74<211>52<212>dna<213>artificialsequence<220><223>rs10242548_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>74tcagacgtgtgctcttccgatctnnnnnncaatgggtcgcattagtcaaaga52<210>75<211>51<212>dna<213>artificialsequence<220><223>rs6949189_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>75tcagacgtgtgctcttccgatctnnnnnncccttctccaccagactcaaag51<210>76<211>53<212>dna<213>artificialsequence<220><223>rs1008707_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>76tcagacgtgtgctcttccgatctnnnnnnttgagcttgagcacattaccaaaa53<210>77<211>51<212>dna<213>artificialsequence<220><223>rs78502829_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>77tcagacgtgtgctcttccgatctnnnnnnagggtccaggctggctactcta51<210>78<211>55<212>dna<213>artificialsequence<220><223>rs6466197_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>78tcagacgtgtgctcttccgatctnnnnnntgagtgttcaggttcaccaaattaca55<210>79<211>51<212>dna<213>artificialsequence<220><223>rs10487275_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>79tcagacgtgtgctcttccgatctnnnnnnggggagctttttagggagagga51<210>80<211>51<212>dna<213>artificialsequence<220><223>rs3823958_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>80tcagacgtgtgctcttccgatctnnnnnnaaggggtggtcttgctgacaat51<210>81<211>53<212>dna<213>artificialsequence<220><223>rs6980029_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>81tcagacgtgtgctcttccgatctnnnnnngcctttgttcagcttgtatccatc53<210>82<211>50<212>dna<213>artificialsequence<220><223>rs4730261_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>82tcagacgtgtgctcttccgatctnnnnnnaggcagtgcttgggtatgagg50<210>83<211>52<212>dna<213>artificialsequence<220><223>rs57592817_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>83tcagacgtgtgctcttccgatctnnnnnntccctaacactcagcacagttgc52<210>84<211>54<212>dna<213>artificialsequence<220><223>rs2253856_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>84tcagacgtgtgctcttccgatctnnnnnntcatttttatccactcctgcagtca54<210>85<211>52<212>dna<213>artificialsequence<220><223>rs982663_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>85tcagacgtgtgctcttccgatctnnnnnntcgttgtgtgggaaatattctgg52<210>86<211>51<212>dna<213>artificialsequence<220><223>rs6965933_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>86tcagacgtgtgctcttccgatctnnnnnnaacacccccaaaccctaagaca51<210>87<211>52<212>dna<213>artificialsequence<220><223>rs2712208_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>87tcagacgtgtgctcttccgatctnnnnnnccacatcctggaatcatttcctc52<210>88<211>51<212>dna<213>artificialsequence<220><223>rs12534598_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>88tcagacgtgtgctcttccgatctnnnnnntttggggactggtacgaaaggt51<210>89<211>49<212>dna<213>artificialsequence<220><223>chr13:20763486_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>89cctacacgacgctcttccgatctnnnnnnctacttccccatctcccaca49<210>90<211>49<212>dna<213>artificialsequence<220><223>chr13:20763421_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>90cctacacgacgctcttccgatctnnnnnnctcccccttgatgaacttcc49<210>91<211>49<212>dna<213>artificialsequence<220><223>chr13:20763530_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>91cctacacgacgctcttccgatctnnnnnntgggagatggggaagtagtg49<210>92<211>53<212>dna<213>artificialsequence<220><223>chr7:107323898_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>92cctacacgacgctcttccgatctnnnnnncaaggaattattaaaaccaatgga53<210>93<211>51<212>dna<213>artificialsequence<220><223>chr7:107350577_f<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>93cctacacgacgctcttccgatctnnnnnntgtgatagaaaagctggagcaa51<210>94<211>49<212>dna<213>artificialsequence<220><223>chr13:20763486_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>94tcagacgtgtgctcttccgatctnnnnnnctacttccccatctcccaca49<210>95<211>48<212>dna<213>artificialsequence<220><223>chr13:20763421_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>95tcagacgtgtgctcttccgatctnnnnnngctcctagtggccatgcac48<210>96<211>48<212>dna<213>artificialsequence<220><223>chr13:20763530_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>96tcagacgtgtgctcttccgatctnnnnnnagatgagcaggccgacttt48<210>97<211>49<212>dna<213>artificialsequence<220><223>chr7:107323898_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>97tcagacgtgtgctcttccgatctnnnnnnttttccaggttggctccata49<210>98<211>55<212>dna<213>artificialsequence<220><223>chr7:107350577_r<220><221>misc_feature<222>(24)..(29)<223>nisa,c,g,ort<400>98tcagacgtgtgctcttccgatctnnnnnnggttgaatatttaccgtttctaaaat55<210>99<211>51<212>dna<213>artificialsequence<220><223>primerfor<400>99aatgatacggcgaccaccgagatctacacacactctttccctacacgacgc51<210>100<211>55<212>dna<213>artificialsequence<220><223>indexprimerrev<400>100caagcagaagacggcatacgagatctcttaattgactggagttcagacgtgtgct55当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1