一种基因变异成因分析方法及系统与流程
本发明涉及基因组学及生物信息学技术,尤其涉及一种基因变异成因分析方法及系统。
背景技术:
随着测序成本的下降,使得在全基因组水平研究结构变异变得可能。在全基因水平识别结构变异,有助于揭示癌症的遗传学因素与致病机理,同时也加快了精准医疗的步伐。
双末端测序的广泛应用使得在全基因组水平检测结构变异及形成机制变得更加灵敏,这是因为双末端测序数据不仅有读片信息,而且还包含了插入距离信息。针对双末端测序数据,目前对于结构变异的识别主要有四种方法:覆盖率分析方法,基于双末端映射的方法,拆分测序片段对映射以及序列拼接方法。覆盖率分析方法只适用于对非平衡性结构变异类型进行检测,基于覆盖率来识别结构变异的算法主要有CNVnator,SegSeq,ReadDepth和CNV-seq等,这种方法的适用范围有限。
技术实现要素:
本发明提供一种基因变异成因分析方法及系统,解决现有技术中基因结构变异的识别方法适用范围有限,操作复杂的技术问题。
本发明的目的是通过以下技术方案实现的:
一种基因变异成因分析方法,包括:
将读片与参考序列进行比对,并根据比对距离以及比对方向,判断出异常读片;
对异常读片进行聚类处理;
根据动态规划算法将每个聚类中的拆分读片与参考序列进行比对,以进行成因分析。
一种基因变异成因分析系统,包括:
异常读片判断模块,用于将读片与参考序列进行比对,并根据比对距离以及比对方向,判断出异常读片;
聚类处理模块,用于对异常读片进行聚类处理;
成因分析模块,用于根据动态规划算法将每个聚类中的拆分读片与参考序列进行比对,以进行成因分析。
本发明提供一种基因变异成因分析方法及系统,通过将读片与参考序列进行比对,并根据比对距离以及比对方向,判断出异常读片;对异常读片进行聚类处理;根据动态规划算法将每个聚类中的拆分读片与参考序列进行比对,以进行成因分析。本发明简化了结构变异成因分析的方法,提高了适用范围。
附图说明
为了更清楚地说明本发明实施例或现有技术中的技术方案,下面将对实施例中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动性的前提下,还可根据这些附图获得其他的附图。
图1为本发明实施例的一种基因变异成因分析方法流程图;
图2为本发明实施例的一种基因变异成因分析系统的结构图。
具体实施方式
为使本发明的上述目的、特征和优点能够更加明显易懂,下面结合附图和具体实施方式对本发明作进一步详细的说明。
本发明实施例提供了一种基因变异成因分析方法,如图1所示,包括:
步骤101、将读片与参考序列进行比对,并根据比对距离以及比对方向,判断出异常读片;
其中,步骤101具体可以包括:
将读片与参考序列进行比对,当至少满足以下一项时,确定为异常读片:
映射方向异常;
成对读片比对到不同染色体上;
插入距离异常。
其中,对于插入距离异常读片的判断,首先算出所有读片的平均插入距离D和标准差σ,当读片的插入距离大于D+3σ或者小于D-3σ时视为异常。具体计算过程为:在SAM文件中可知道读片比对位置的信息,POS表示读片比对到参考序列第一个碱基的位置,PNEXT表示成对读片中另一读片比对到参考序列上第一个碱基的位置,所以每个pair-end成对读片的插入距离l为y-x-L+1,其中:L代表读片的长度;y表示PNEXT;x表示POS。
步骤102、对异常读片进行聚类处理;
其中,步骤102具体可以包括:
首先将异常测序片段对i的比对位置(POS,PNEXT)记为(xi,yi),并计算异常测序片段对i的比对中间值M(xi,yi)=(xi+yi)/2;
同一类的异常读片满足M(,)-M(,)<Dmax,其中,Dmax=lmax,lmax=max(l1,l2,l3…ln),l为插入距离;
同一类中的异常读片之间的插入距离还需满足maxij((,)-(,))≤2lmax。
步骤103、根据动态规划算法将每个聚类中的拆分读片与参考序列进行比对,以进行成因分析。
步骤103具体可以包括:
根据动态规划算法将每个聚类中的拆分读片与参考序列进行比对,识别出该结构变异的间断点;
基于所述间断点处序列的同源性判断结构变异对应的成因。
本发明提供一种基因变异成因分析方法,通过将读片与参考序列进行比对,并根据比对距离以及比对方向,判断出异常读片;对异常读片进行聚类处理;根据动态规划算法将每个聚类中的拆分读片与参考序列进行比对,以进行成因分析。本发明简化了结构变异成因分析的方法,提高了适用范围。
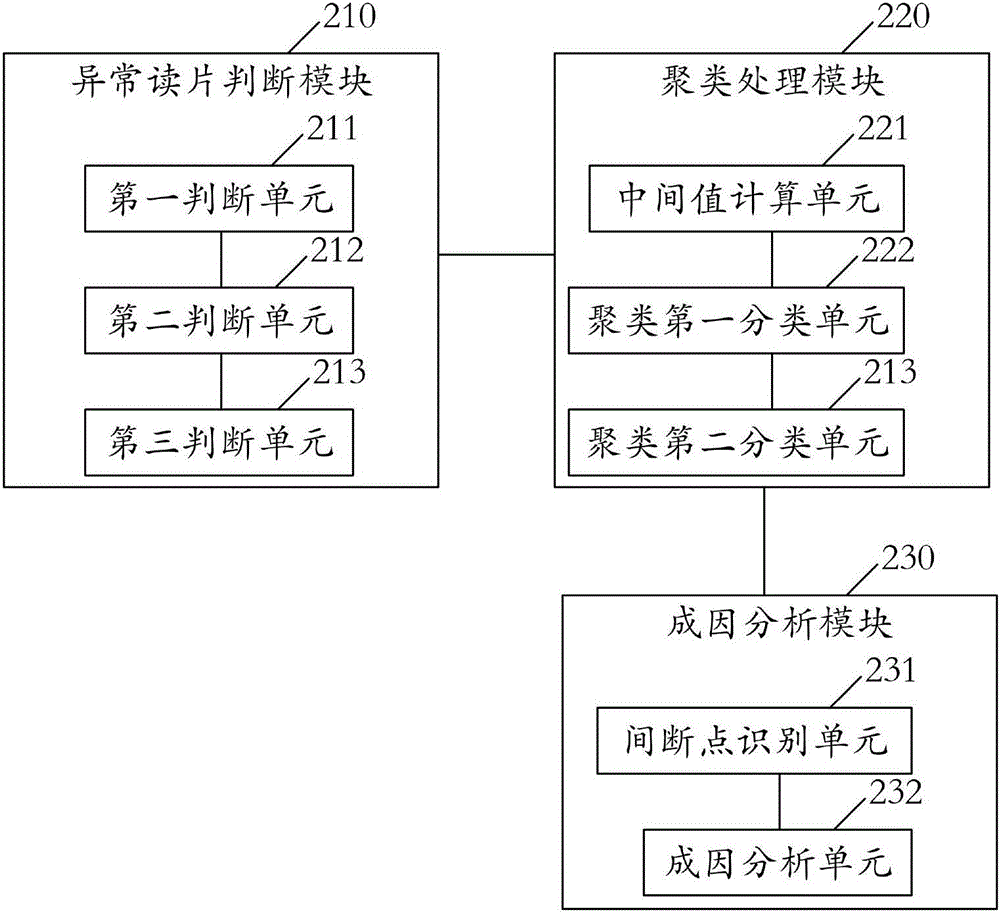
本发明实施例还提供了一种基因变异成因分析系统,如图2所示,包括:
异常读片判断模块210,用于将读片与参考序列进行比对,并根据比对距离以及比对方向,判断出异常读片;
聚类处理模块220,用于对异常读片进行聚类处理;
成因分析模块230,用于根据动态规划算法将每个聚类中的拆分读片与参考序列进行比对,以进行成因分析。
其中,所述成因分析模块230包括:
间断点识别单元231,用于根据动态规划算法将每个聚类中的拆分读片与参考序列进行比对,识别出该结构变异的间断点;
成因分析单元232,用于基于所述间断点处序列的同源性判断结构变异对应的成因。
所述异常读片判断模块210包括:
第一判断单元211,用于判断读片与参考序列的映射方向异常;
第二判断单元212,用于判断是否成对读片比对到不同染色体上;
第三判断单元213,用于判断插入距离异常。
所述聚类处理模块220包括:
中间值计算单元221,用于将异常测序片段对i的比对位置(POS,PNEXT)记为(xi,yi),并计算异常测序片段对i的比对中间值M(xi,yi)=(xi+yi)/2;
聚类第一分类单元222,用将异常读片满足M(,)-M(,)<Dmax的读片归为一类,其中,Dmax=lmax,lmax=max(l1,l2,l3…ln),ln为第n个读片的插入距离,n为自然数;
聚类第二分类单元223,用于在聚类第一分类单元判断的聚类中,通过是否满足maxij((,)-(,))≤2lmax筛选出最终聚类的异常读片。
通过以上的实施方式的描述,本领域的技术人员可以清楚地了解到本发明可借助软件加必需的硬件平台的方式来实现,当然也可以全部通过硬件来实施,但很多情况下前者是更佳的实施方式。基于这样的理解,本发明的技术方案对背景技术做出贡献的全部或者部分可以以软件产品的形式体现出来,该计算机软件产品可以存储在存储介质中,如ROM/RAM、磁碟、光盘等,包括若干指令用以使得一台计算机设备(可以是个人计算机,服务器,或者网络设备等)执行本发明各个实施例或者实施例的某些部分所述的方法。
以上对本发明进行了详细介绍,本文中应用了具体个例对本发明的原理及实施方式进行了阐述,以上实施例的说明只是用于帮助理解本发明的方法及其核心思想;同时,对于本领域的一般技术人员,依据本发明的思想,在具体实施方式及应用范围上均会有改变之处,综上所述,本说明书内容不应理解为对本发明的限制。
- 还没有人留言评论。精彩留言会获得点赞!