核酸酶介导的DNA组装的制作方法
本申请要求2014年6月23日提交的美国临时申请No.62/015,809、2014年6月24日提交的美国临时申请No.62/016,400以及2014年8月13日提交的美国临时申请No.62/036,983的权益,这些美国临时申请中的每一篇均据此全文以引用方式并入本文。
作为通过EFS WEB提交的文本文件
通过EFS-Web以电子方式将序列表的正式文本作为ASCII格式的序列表提交,该文件名称为461002SEQLIST.TXT,创建日期为2015年6月23日,文件大小为66KB,并且该文件与本说明书同时提交。该ASCII格式文档中所含的序列表是本说明书的一部分,并且全文以引用的方式并入本文。
背景技术:
以往,重叠延伸可用作从重叠合成寡核苷酸合成较大双链DNA分子(特别是基因)的一种手段。然而,这些方法不能有效地以快速的方式组合大DNA分子。此外,使用重叠序列对大核酸进行的位点特异性组合通常受限于重叠序列在待组合的核酸中的所需位置处的可用性。被设计成靶向特异性DNA序列的经工程改造的核酸酶作为遗传操作的强大工具已经引起人们关注,用这些酶可以进行定向的基因缺失、替换和修复以及外源序列插入。然而,现有技术的缺点在于精确度有限,这可导致不可预知的脱靶效应和耗时的多步反应。
技术实现要素:
本文提供了用于组装具有重叠序列的核酸的方法。此类方法包括用于组装至少两个核酸的方法,该方法包括:(a)使第一核酸与第一核酸酶试剂接触,其中第一核酸酶试剂在第一靶位点处切割第一核酸,以产生第一经酶切的核酸,在第一经酶切的核酸与第二核酸之间具有重叠末端序列;(b)使第一经酶切的核酸和第二核酸与核酸外切酶接触,以暴露第一经酶切的核酸与第二核酸之间的互补序列;以及(c)组装由步骤(b)生成的两个核酸片段。在一些此类方法中,步骤(c)还包括:(i)使暴露的互补序列退火;(ii)延伸经退火的互补序列的3’端;以及(iii)连接第一核酸和第二核酸。
在一些方法中,步骤(a)还包括使第二核酸与第二核酸酶试剂接触,其中第二核酸不包含重叠末端序列,并且第二核酸酶试剂在第二靶位点处切割第二核酸,以产生第二经酶切的核酸,在第一经酶切的核酸与第二经酶切的核酸之间具有重叠末端序列,并且其中步骤(b)的第二核酸是第二经酶切的核酸。在一些方法中,重叠末端序列的长度在20bp至200bp的范围内。
在一些方法中,第一核酸酶试剂或第二核酸酶试剂中的至少一者包含靶向第一靶位点或第二靶位点的Cas蛋白和向导RNA(gRNA)(gRNA-Cas复合物)。例如,Cas蛋白可为Cas9蛋白。Cas9蛋白可包含RuvC结构域和HNH结构域,这两个结构域中的至少一者缺少核酸内切酶活性。在一些实施例中,gRNA包含编码成簇的规律间隔的短回文重复序列(CRISPR)RNA(crRNA)和反式激活CRISPR RNA(tracrRNA)的核酸序列。第一靶位点和/或第二靶位点可被前间区序列邻近基序(PAM)序列侧接。在一些方法中,核酸酶试剂包括锌指核酸酶或转录激活因子样效应物核酸酶(TALEN)。
在一些方法中,第一核酸、第二核酸或这两个核酸来自细菌人工染色体。细菌人工染色体可包含人DNA、啮齿动物DNA、合成DNA或它们的组合。细菌人工染色体可包含人序列。
本文所公开的方法包括用于组装至少两个核酸的方法,该方法包括:(a)使第一核酸与第一核酸酶试剂和第二核酸酶试剂接触以产生第一经酶切的核酸,其中第一核酸酶试剂在第一核酸的第一链上的第一靶位点处生成切口,并且第二核酸酶试剂在第一核酸的第二链上的第二靶位点处生成切口,以产生在其末端之一处包含5’或3’悬垂序列的第一经酶切的核酸;(b)使第一经酶切的核酸和包含与5’或3’悬垂序列互补的序列的第二核酸退火;以及(c)连接第一经酶切的核酸和第二核酸。在一些方法中,步骤(b)还包括使用第二链作为模板来延伸第一链的3’端,并且使用第一链作为模板来延伸第二链的3’端。在一些方法中,第一靶位点与第二靶位点相隔至少4bp。
在一些方法中,第一核酸酶试剂或第二核酸酶试剂中的至少一者包含靶向第一靶位点或第二靶位点的Cas9蛋白和向导RNA(gRNA)(gRNA-Cas复合物)。gRNA可包含编码成簇的规律间隔的短回文重复序列(CRISPR)RNA(crRNA)和反式激活CRISPR RNA(tracrRNA)的核酸序列。在一些方法中,第一靶位点和第二靶位点中的至少一者被前间区序列邻近基序(PAM)序列侧接。Cas9蛋白可包含RuvC结构域和HNH结构域,这两个结构域中的一者缺少核酸内切酶活性。
在一些方法中,第二核酸不包含与第一经酶切的核酸的5’或3’悬垂序列互补的序列,并且步骤(a)还包括使第一经酶切的核酸和第二经酶切的核酸与接合寡核苷酸接触,其中接合寡核苷酸包含:(i)与第一经酶切的核酸的5’或3’悬垂序列互补的第一互补序列;以及(ii)与第二经酶切的核酸的5’或3’悬垂序列互补的第二互补序列。在一些方法中,第一核酸、第二核酸或这两个核酸来源于细菌人工染色体。细菌人工染色体可包含人DNA、啮齿动物DNA、合成DNA或它们的组合。细菌人工染色体可包含人多核苷酸序列。在一些方法中,第二核酸包含细菌人工染色体。
本文所提供的方法还包括用于组装两个或更多个核酸片段的方法,该方法包括:(a)使第一核酸与至少一种核酸酶试剂接触以生成第一经酶切的核酸;(b)使第一经酶切的核酸与第二核酸、接合寡核苷酸和核酸外切酶接触,其中接合寡核苷酸包含:(i)与第一经酶切的核酸互补的第一互补序列;(ii)间区序列;以及(iii)与第二核酸互补的第二互补序列;其中核酸外切酶使第一互补序列和第二互补序列暴露;以及(c)将接合寡核苷酸与第一经酶切的核酸和第二核酸组装在一起。在一些此类方法中,步骤(c)中的组装包括:(i)使接合寡核苷酸的第一互补序列退火到第一经酶切的核酸上,并使接合寡核苷酸的第二互补序列退火到第二核酸上;以及(ii)将接合寡核苷酸连接到第一经酶切的核酸和第二核酸。
在一些方法中,接合寡核苷酸的第一互补序列和第二互补序列包含15至120个互补碱基。在一些方法中,接合寡核苷酸的间区序列包含非互补核酸。在一些实施例中,第一经酶切的核酸被无缝地组装到第二核酸。
在一些方法中,核酸酶试剂被设计成从将要进行无缝组装的第一核酸末端切割至少20bp片段,其中,接合寡核苷酸的间区序列包含与所述至少20bp片段相同的序列,其中在第一互补序列与所述至少20bp片段之间不存在核酸碱基,并且在第二互补序列与所述至少20bp片段之间不存在核酸碱基,使得所述第一核酸与所述接合寡核苷酸和所述第二核酸的组装重建所述至少20bp片段并无缝地组装第一核酸和第二核酸。在一些方法中,使用来自第二核酸的至少20bp片段作为间区序列来执行相同方法。在一些方法中,间区序列包含约20bp至约120bp。在一些方法中,第二核酸与第二核酸酶试剂和核酸外切酶接触,其中第二核酸酶试剂切割第二核酸,以产生包含与接合寡核苷酸的第二互补序列互补的核苷酸序列的第二经酶切的核酸,其中第一经酶切的核酸被组装到第二经酶切的核酸。在一些方法中,使第二核酸与限制性内切酶或大范围核酸酶和核酸外切酶接触,其中限制性内切酶或大范围核酸酶切割第二核酸,以产生包含与接合寡核苷酸中的第二互补序列互补的核苷酸序列的第二经酶切的核酸,其中第一经酶切的核酸被组装到第二经酶切的核酸。在一些方法中,在步骤(b)中延伸第一经酶切的核酸和/或第二经酶切的核酸的3’端。接合寡核苷酸可在同一反应中或依次地组装到所述第一核酸和所述第二核酸上。在一些方法中,第一核酸、第二核酸或这两个核酸来源于细菌人工染色体,长度为至少10kb,和/或包含人DNA、啮齿动物DNA、合成DNA或它们的组合。
在一些方法中,所述至少一种核酸酶试剂或第二核酸酶试剂包含靶向第一靶位点或第二靶位点的Cas蛋白和向导RNA(gRNA)(gRNA-Cas复合物)。例如,Cas蛋白可为Cas9蛋白。Cas9蛋白可包含RuvC结构域和HNH结构域,这两个结构域中的至少一者缺少核酸内切酶活性。在一些实施例中,gRNA包含编码成簇的规律间隔的短回文重复序列(CRISPR)RNA(crRNA)和反式激活CRISPR RNA(tracrRNA)的核酸序列。第一靶位点和/或第二靶位点可被前间区序列邻近基序(PAM)序列侧接。在一些方法中,所述至少一种核酸酶试剂和/或第二核酸酶试剂包括锌指核酸酶或转录激活因子样效应物核酸酶(TALEN)。
在一些实施例中,接合寡核苷酸包含gBlock。在一些此类方法中,gBlock不包含选择盒。
本文还提供了用于组装两个或更多个核酸的方法,该方法包括:(a)使第一核酸与至少一种核酸酶试剂接触以生成第一经酶切的核酸;(b)使第二核酸与第二核酸酶试剂接触以生成第二经酶切的核酸;(c)使第一经酶切的核酸和第二经酶切的核酸与接合寡核苷酸和核酸外切酶接触,其中接合寡核苷酸包含:(i)与第一经酶切的核酸互补的第一互补序列;(ii)间区序列;以及(iii)与第二经酶切的核酸互补的第二互补序列;其中核酸外切酶使第一互补序列和第二互补序列暴露;以及(d)将接合寡核苷酸与第一经酶切的核酸和第二核酸组装在一起。
本文提供了用于组装具有重叠序列的核酸的方法。此类方法包括用于组装至少两个核酸片段的方法,该方法包括(a)使包含重叠序列的第一核酸和第二核酸与至少一种gRNA-Cas复合物和核酸外切酶接触,从而生成在其末端之一处包含互补序列的两个经酶切的核酸片段;(b)组装由步骤(a)生成的两个核酸片段。在一些方法中,所述至少一种gRNA-Cas复合物在第一靶位点处切割第一核酸,以产生第一经酶切的核酸,在第一经酶切的核酸与第二核酸之间包含互补末端序列。在某些方法中,步骤(b)还包括:(i)使暴露的互补序列退火;(ii)延伸经退火的互补序列的3’端;以及(iii)连接第一核酸和第二核酸。在一些方法中,步骤(a)还包括使第二核酸与第二gRNA-Cas复合物接触,其中第二核酸不包含重叠末端序列,并且第二gRNA-Cas复合物切割第二核酸,以产生第二经酶切的核酸,在第一经酶切的核酸与第二经酶切的核酸之间包含重叠末端序列。例如,gRNA-Cas复合物包含Cas9蛋白。Cas9蛋白可包含RuvC结构域和HNH结构域,这两个结构域中的至少一者缺少核酸内切酶活性。在一些方法中,重叠序列的长度在20bp至200bp的范围内。根据权利要求1至7中任一项所述的方法,其中所述第一核酸、所述第二核酸或这两个核酸来自细菌人工染色体。在一些方法中,细菌人工染色体包含人DNA、啮齿动物DNA、合成DNA或它们的组合。细菌人工染色体可包含人序列。
本文所提供的方法还包括用于组装两个或更多个核酸片段的方法,该方法包括:(a)使第一核酸和第二核酸暴露于至少一种gRNA-Cas复合物,以生成在其末端之一处包含5’或3’悬垂序列的第一经酶切的核酸和第二经酶切的核酸;(b)组装由步骤(a)生成的两个核酸片段。在一些方法中,组装步骤(b)包括:(i)使5’和3’悬垂序列退火;以及(ii)连接第一经酶切的核酸和第二经酶切的核酸。在一些方法中,5’和/或3’悬垂序列包含至少4个互补碱基。在一些方法中,步骤(b)还包括延伸第一经酶切的核酸和第二经酶切的核酸的3’端。在一些方法中,第二核酸不包含与第一经酶切的核酸的5’或3’悬垂序列互补的序列,并且步骤(a)还包括使第一经酶切的核酸和第二经酶切的核酸与接合寡核苷酸接触,其中接合寡核苷酸包含:(i)与第一经酶切的核酸的5’或3’悬垂序列互补的第一互补序列;以及(ii)与第二经酶切的核酸的5’或3’悬垂序列互补的第二互补序列。在一些方法中,gRNA-Cas蛋白复合物包含Cas9蛋白,该Cas9蛋白包含RuvC结构域和HNH结构域,这两个结构域中的一者缺少核酸内切酶活性。在一些方法中,gRNA-Cas复合物作为crRNA、tracrRNA和Cas蛋白单独地提供。在一些方法中,第一核酸和第二核酸包含前间区序列邻近基序(PAM)序列。在一些方法中,第一核酸、第二核酸或这两个核酸来源于细菌人工染色体。在一些方法中,细菌人工染色体包含人DNA、啮齿动物DNA、合成DNA或它们的组合。例如,细菌人工染色体可包含人多核苷酸序列。
本文还提供了用于组装两个或更多个核酸的方法,该方法包括:(a)使第一核酸与至少一种gRNA-Cas复合物接触以生成第一经酶切的核酸;以及(b)使第一经酶切的核酸与第二核酸、接合寡核苷酸和核酸外切酶接触,其中接合寡核苷酸包含:(i)与第一经酶切的核酸互补的第一互补序列;(ii)间区序列;以及(iii)与第二核酸互补的第二互补序列;其中核酸外切酶使第一互补序列和第二互补序列暴露;以及(c)将接合寡核苷酸与第一经酶切的核酸和第二核酸组装在一起。在一些方法中,组装步骤(c)包括(i)使接合寡核苷酸的第一互补序列退火到第一经酶切的核酸上,并使接合寡核苷酸的第二互补序列退火到第二核酸上;以及(ii)将接合寡核苷酸连接到第一经酶切的核酸和第二核酸。在一些方法中,接合寡核苷酸的第一互补序列和第二互补序列包含15至120个互补碱基。在一些方法中,接合寡核苷酸的间区序列包含非互补核酸。
使用接合寡核苷酸,可将第一经酶切的核酸无缝地组装到第二核酸。在一些方法中,gRNA-Cas复合物被设计成从将要进行无缝组装的第一核酸末端切割至少20bp片段,其中,接合寡核苷酸的间区序列包含与所述至少20bp片段相同的序列,其中在第一互补序列与所述至少20bp片段之间不存在核酸碱基,并且在第二互补序列与所述至少20bp片段之间不存在核酸碱基,使得所述第一核酸与所述接合寡核苷酸和所述第二核酸的组装重建所述至少20bp片段并无缝地组装所述第一核酸和第二核酸。在一些方法中,使用来自第二核酸的至少20bp片段作为间区序列来执行相同方法。在一些方法中,间区序列包含约20bp至约120bp。在一些方法中,使第二核酸与第二gRNA-Cas复合物和核酸外切酶接触,其中第二gRNA-Cas复合物切割第二核酸,以产生包含与接合寡核苷酸的第二互补序列互补的核苷酸序列的第二经酶切的核酸,其中第一经酶切的核酸被组装到第二经酶切的核酸。在一些方法中,使第二核酸与限制性内切酶或大范围核酸酶和核酸外切酶接触,其中限制性内切酶或大范围核酸酶切割第二核酸,以产生包含与接合寡核苷酸中的第二互补序列互补的核苷酸序列的第二经酶切的核酸,其中第一经酶切的核酸被组装到第二经酶切的核酸。在一些方法中,在步骤(b)中延伸第一经酶切的核酸和/或第二经酶切的核酸的3’端。接合寡核苷酸可在同一反应中或依次地组装到所述第一核酸和所述第二核酸。在一些方法中,gRNA-Cas复合物包含Cas9蛋白。在一些方法中,第一核酸、第二核酸或这两个核酸来源于细菌人工染色体,长度为至少10kb,和/或包含人DNA、啮齿动物DNA、合成DNA或它们的组合。
附图说明
图1示出了BAC与PCR产物的组装,该PCR产物具有被设计成对BAC具有特异性的重叠区。50bp重叠区通过PCR加到HYG盒。
图2示出了利用每个BAC上的两个Cas9靶位点对具有重叠序列的两个BAC进行组装。使用本文所公开的方法进行组装的过程耗时2天。
图3示出了使用传统方法对具有重叠序列的两个BAC进行组装。使用传统方法进行组装的过程耗时4周。
图4示出了Cas9/等温组装方法的克隆效率以及BAC克隆步骤所需的时间。
图5示出了使用CRISPR/Cas9系统和等温组装来构建大靶向载体(LTVEC)。使用一个或多个接合寡核苷酸和等温组装对用CRISPR/Cas9切割的DNA片段进行了无缝组装。
图6示出了使用接头(接合寡核苷酸)对Cas9切割之后的核酸进行无缝组装的策略。gRNA/Cas9复合物被设计成切割位于目标区域的5’上游的靶位点(箭头),以生成第一经Cas9酶切的DNA片段(5’DNA)。然后将5’DNA的缺失部分(斜线框)用作接合寡核苷酸中的5’和3’重叠序列之间的间区序列。在等温组装反应中组装三种组分:(a)第一经Cas9酶切的DNA片段(5’DNA);(b)接合寡核苷酸;以及(c)第二DNA片段(3’DNA)。接合寡核苷酸从5’至3’包含:(1)与5’DNA重叠的序列,(2)包含第一经酶切的片段的缺失部分的间区序列,以及(3)与3’DNA重叠的序列。在组装步骤期间重建5’DNA的缺失部分。
图7示出了使用CRISPR/Cas9系统和等温组装来构建DNA载体。
图8示出了使用CRISPR/Cas9系统和等温组装来构建大靶向载体。
图9示出了靶向载体的构建,其使用等温组装和两个接头(接合寡核苷酸)将BAC载体的一部分替换为盒。各种比率的mBAC与片段或接头的结果在分图#1、#2、#3和#4中示出。
图10示出了经序列确认,使用两个接头在mBAC(BAC ID:RP23-399M19)与盒之间的组装反应实现了跨两个接合部的无缝组装。
图11示出了使用Cas9和等温组装对两个mBAC进行组装。bMQ50f19载体与包含潮霉素抗性基因泛素启动子的之间的组装是无缝的。
图12示出了在接头1处的无缝组装的序列确认,以及在接头2和接头3处的特意不无缝的组装的序列确认。
图13示出了使用四个接头和等温组装在mBAC上插入大的人基因片段。Cas9从hBAC1切割hGene片段A,从hBAC2切割hGene片段B,并且切割mBAC以去除mGene片段。
图14示出了使用Cas9和等温组装在BAC载体中插入人序列。
图15示出了使用Cas9和等温组装来插入包含大范围核酸酶位点的gBlock。图15A示出了包含PI-SceI位点的gBlock的插入;图15B示出了包含MauBI位点的gBlock的插入。
图16示出了使用三个接合寡核苷酸、Cas9和等温组装对靶向载体进行直接人源化的示例。
图17示出了使用具有上游和下游接合寡核苷酸的供体、Cas9以及等温组装对靶向载体进行间接人源化的示例。
图18示出了使用Cas9和等温组装来引入点突变的示例。
图19示出了通过Cas9和等温组装进行BAC修剪的示例。在该示例中,该修剪去除了Ori序列。使用两个接合寡核苷酸和等温组装将Ori序列重新插入载体中。
具体实施方式
I.定义
在本文中可互换使用的术语“蛋白”、“多肽”和“肽”包括任何长度的氨基酸聚合形式,包括编码氨基酸和非编码氨基酸以及以化学方式或生化方式修饰或衍生的氨基酸。这些术语还包括经过修饰的聚合物,诸如具有经过修饰的肽骨架的多肽。
在本文中可互换使用的术语“核酸”和“多核苷酸”包括任何长度的核苷酸聚合形式,包括核糖核苷酸、脱氧核糖核苷酸或它们的类似物或修饰形式。这些术语包括单链、双链和多链DNA或RNA、基因组DNA、cDNA、DNA-RNA杂交体、以及包含嘌呤碱基、嘧啶碱基、或其他天然的、化学修饰的、生物化学修饰的、非天然的或衍生的核苷酸碱基的聚合物。
“密码子优化”一般包括通过以下方式修饰核酸序列以增强在特定宿主细胞中的表达的过程:将天然序列的至少一个密码子替换为在宿主细胞的基因中更频繁或最频繁使用的密码子,同时保持天然氨基酸序列。例如,可对编码Cas蛋白的核酸进行修饰,以替换成与天然存在的核酸序列相比在给定的原核细胞或真核细胞(包括细菌细胞、酵母细胞、人细胞、非人细胞、哺乳动物细胞、啮齿动物细胞、小鼠细胞、大鼠细胞、仓鼠细胞或任何其他宿主细胞)中具有更高使用频率的密码子。密码子使用表是现成的,例如在“密码子使用数据库(Codon Usage Database)”处提供。这些表格可按多种方式进行改编。参见Nakamura et al.(2000)Nucleic Acids Research 28:292(Nakamura等人,2000年,《核酸研究》,第28卷,第292页)。为实现在特定宿主中的表达而对特定序列进行密码子优化的计算机算法也是现成的(参见例如Gene Forge)。
“有效连接”或“有效连接的”包括两个或更多个组分(例如,启动子和另一个序列元件)的并置,使得这两个组分正常发挥功能并使这些组分中的至少一者有可能介导被施加在其他组分中的至少一者上的功能。例如,如果启动子响应于一个或多个转录调控因子的存在或不存在而对编码序列的转录水平进行控制,则启动子可以是有效连接至编码序列。
核酸的“互补性”意指核酸的一条链中的核苷酸序列因其核碱基基团的取向而与相对核酸链上的另一个序列形成氢键。DNA中的互补碱基通常是A与T及C与G。在RNA中,它们通常是C与G及U与A。互补性可以是完全的或实质的/充分的。两个核酸之间的完全互补性意指这两个核酸可以形成双链体,其中双链体中的每个碱基按照沃森-克里克配对原则与互补碱基结合。“实质”或“充分”互补意指一条链中的序列不与相对链中的序列彻底和/或完全互补,但在一组杂交条件(例如,盐浓度和温度)中这两条链上的碱基之间发生充分键合而形成稳定的杂交复合物。可通过以下方式预测此类条件:使用序列和标准数学计算来预测杂交链的Tm,或使用常规方法凭经验确定Tm。Tm包括在两条核酸链之间形成的一群杂交复合物发生50%变性时的温度。在低于Tm的温度下,有利于杂交复合物的形成,而在高于Tm的温度下,有利于杂交复合物中的两条链的解链或分离。可在1M NaCl水溶液中对具有已知G+C含量的核酸估计Tm,例如使用Tm=81.5+0.41(%G+C),而其他已知的Tm计算法考虑了核酸结构特征。
“杂交条件”包括累积环境,其中一条核酸链通过互补链相互作用和氢键方式键合于第二核酸链,从而产生杂交复合物。此类条件包括含核酸的水溶液或有机溶液的化学组分及其浓度(例如,盐、螯合剂、甲酰胺)以及该混合物的温度。其他因素(例如,温育时间的长度或反应室尺寸)可对环境有影响。参见例如Sambrook et al.,Molecular Cloning,A Laboratory Manual,2.sup.nd ed.,pp.1.90-1.91,9.47-9.51,1 1.47-11.57(Cold Spring Harbor Laboratory Press,Cold Spring Harbor,N.Y.,1989)(Sambrook等人,《分子克隆实验指南》,第2版,第1.90-1.91、9.47-9.51、1 1.47-11.57节,冷泉港实验室出版社,美国纽约州冷泉港,1989年)。
杂交要求两个核酸包含互补序列,但允许碱基之间出现错配。适于两个核酸之间的杂交的条件取决于核酸的长度和互补程度,这些变量是本领域众所周知的。两个核苷酸序列之间的互补程度越大,具有这些序列的核酸的杂交体的解链温度(Tm)值就越大。对于具有短序列段互补性(例如,在35个或更少、30个或更少、25个或更少、22个或更少、20个或更少、或18个或更少核苷酸内的互补性)的核酸之间的杂交,错配的位置变得重要(参见Sambrook等人,出处同上,11.7-11.8)。通常,可杂交核酸的长度为至少约10个核苷酸。可杂交核酸的示例性最小长度包括至少约15个核苷酸、至少约20个核苷酸、至少约22个核苷酸、至少约25个核苷酸以及至少约30个核苷酸。此外,可视需要根据诸如互补区域的长度和互补程度等因素来调节温度和洗涤溶液盐浓度。
多核苷酸的序列不必与其靶核酸的序列100%互补,也能实现特异性杂交。此外,多核苷酸可在一个或多个区段内杂交,使得间插或相邻区段不参与杂交事件(例如,环结构或发夹结构)。多核苷酸(例如,gRNA)可与其所靶向的靶核酸序列内的靶区域具有至少70%、至少80%、至少90%、至少95%、至少99%或100%序列互补性。例如,其中20个核苷酸中有18个与靶区域互补并因此特异性杂交的gRNA将具有90%互补性。在该示例中,剩余的非互补核苷酸可以成簇或散布在互补核苷酸内并且无需彼此邻接或与互补核苷酸邻接。
通常可使用以下程序来确定核酸内的核酸序列的特定序列段之间的互补性百分比:使用本领域已知的BLAST程序(基本局部比对搜索工具)和PowerBLAST程序(Altschul et al.(1990)J.Mol.Biol.215:403-410(Altschul等人,1990年,《分子生物学杂志》,第215卷,第403-410页);Zhang and Madden(1997)Genome Res.7:649-656(Zhang和Madden,1997年,《基因组研究》,第7卷,第649-656页))或使用Gap程序(威斯康星序列分析软件包,适用于Unix的版本8,遗传学计算机组,美国威斯康星州麦迪逊的大学研究园(Wisconsin Sequence Analysis Package,Version 8 for Unix,Genetics Computer Group,University Research Park,Madison Wis.)),这些程序使用默认设置,这使用Smith和Waterman的算法(Adv.Appl.Math.,1981,2,482-489(《应用数学进展》,1981年,第2卷,第482-489页))。
本文所提供的方法和组合物采用多种不同组分。在本说明书通篇中已经确认,一些组分可具有活性变体和片段。此类组分包括例如Cas蛋白、CRISPR RNA、tracrRNA和向导RNA。这些组分中的每一者的生物活性在本文别处描述。
在两个多核苷酸或多肽序列的语境中,“序列同一性”或“同一性”是指在指定比较窗内对齐以实现最大对应性时这两个序列中相同的残基。当使用序列同一性百分比指涉蛋白质时,应认识到,不相同的残基位置通常差别在于保守氨基酸置换,其中氨基酸残基被置换为具有类似化学特性(例如,电荷或疏水性)的其他氨基酸残基且因此不改变分子的功能特性。当序列差别在于保守置换时,可上调序列同一性百分比以校正置换的保守性质。差别在于此类保守置换的序列被称为具有“序列相似性”或“相似性”。进行这种调节的方式是本领域技术人员众所周知的。通常,这涉及将保守置换作为部分错配而非完全错配来评分,从而增加序列同一性百分比。因此,例如,若一个相同氨基酸被给定1的分数且一个非保守置换被给定0的分数,则一个保守置换被给定0至1之间的分数。保守置换的分数例如在程序PC/GENE(美国加利福尼亚州山景城的Intelligenetics公司(Intelligenetics,Mountain View,California))中所执行的那样来计算。
“序列同一性百分比”包括通过在比较窗内比较两个最佳对齐的序列而确定的值,其中与参考序列(其不包含添加或缺失)相比较,多核苷酸序列在比较窗中的部分可包含添加或缺失(即,空位),以便保证这两个序列的最佳对齐。该百分比通过以下方式计算:确定其中相同的核酸碱基或氨基酸残基在两个序列中出现的位置的数目以产生匹配位置的数目,将匹配位置的数目除以在比较窗口中的位置总数,并且将结果乘以100以得到序列同一性百分比。
除非另作说明,否则序列同一性/相似性值包括使用GAP版本10采用以下参数获得的值:核苷酸序列的同一性%和相似性%使用空位权重(GAP Weight)50和长度权重3及nwsgapdna.cmp评分矩阵;氨基酸序列的同一性%或相似性%使用空位权重8和长度权重2及BLOSUM62评分矩阵;或其任何等同程序。“等同程序”包括任何序列比较程序,其为所考虑的任何两个序列产生这样的比对,当与由GAP版本10产生的对应比对相比较时,该比对具有相同的核苷酸或氨基酸残基匹配和相同的序列同一性百分比。
“包含”或“包括”一个或多个所述及的要素的组合物或方法可包括未具体述及的其他要素。例如,“包含”或“包括”某种蛋白质的组合物可包含单独的该蛋白质或与其他成分组合的该蛋白质。
值的范围的指定包括该范围内的或限定该范围的所有整数,以及由该范围内的整数所限定的所有子范围。
除非从上下文明显看出,否则术语“约”涵盖落在规定值的标准测量误差容限(例如,SEM)内的那些值。
除非上下文另外明确指出,否则单数形式的量词“一个”、“一种”和“该”包括复数指代物。例如,术语“Cas蛋白”或“至少一种Cas蛋白”可包括多种Cas蛋白,包括它们的混合物。
II.概述
传统的核酸组装方法采用以下耗时的步骤:用限制性内切酶进行的常规酶切、核酸的克隆以及将核酸连接在一起(有关传统方法和时间线的图解,参见图3和图4)。当要将大片段或载体组装在一起时,这些方法变得更为困难。本文所提供的方法利用核酸酶(例如,向导RNA和Cas9核酸酶)的可塑的靶特异性将核酸转变为适于在快速组装反应中使用的形式。
本文提供了使用诸如通过向导RNA(gRNA)被引导至特定靶位点的核酸酶试剂(例如,通过向导RNA(gRNA)被引导至特定靶位点的Cas蛋白)来组装至少两个核酸的方法。定点核酸酶试剂(例如,向导RNA引导的Cas蛋白)通过选择和操纵由其核酸内切酶活性生成的末端序列,可以实现核酸的快速且有效的组合。本文所提供的方法将第一多核苷酸与对所需靶位点具有特异性的核酸酶试剂(例如,gRNA-Cas复合物)和核酸外切酶组合在一起。可对靶位点进行选择,使得当核酸酶切割核酸时,通过切割产生的所得末端具有与第二核酸的末端互补的区域(例如,重叠末端)。然后可组装这些互补末端,得到单个组装的核酸。由于核酸酶试剂(例如,gRNA-Cas复合物)对单个靶位点具有特异性,本发明的方法使得可以以精确的定点方式修饰核酸。本发明的方法通过使用专门被设计用于组合由核酸酶切割所生成的重叠核酸末端或者被设计并合成用于组装反应的快速且有效的组装方法,而进一步利用了核酸酶试剂(例如,gRNA-Cas复合物)特异性。例如,通过选择对靶位点具有特异性的核酸酶试剂(例如,gRNA-Cas复合物)使得在切割时产生与第二核酸的末端序列互补的末端序列,可以使用等温组装来组装所得的经酶切的核酸。因此,通过选择会产生重叠末端序列的核酸和核酸酶试剂(例如,gRNA-Cas复合物),可以采取快速组合方法来组装核酸,从而以快速且有效的方式产生最终组装的核酸。作为另一种选择,可以将不具有互补末端的核酸和被设计成具有与每个核酸互补的末端的接合寡核苷酸组装在一起。通过使用接合寡核苷酸,可以无缝地组装两个或更多个核酸,从而减少所得组装的核酸中不必要的序列。
III.核酸酶试剂
本发明的方法采用核酸酶试剂对多核苷酸进行定点切割。具体地讲,在所鉴定的靶位点处对多核苷酸进行核酸内切酶切割会产生具有这样的末端的经酶切的多核苷酸,所述末端随后可接合到第二多核苷酸,从而以位点特异性方式组装两个或更多个多核苷酸。
“核酸酶试剂”包含具有DNA切割活性的分子。用于本文所公开的方法中的核酸酶试剂的具体示例包括RNA引导的CRISPR-Cas9系统、锌指蛋白、大范围核酸酶、TAL结构域、TALEN、酵母组装、重组酶、亮氨酸拉链、CRISPR/Cas、核酸内切酶以及本领域技术人员已知的其他核酸酶试剂。可对核酸酶试剂进行选择或设计以实现在给定靶位点处切割的特异性。例如,可对核酸酶试剂进行选择以实现在靶位点处的切割,从而在经切割的多核苷酸与另一不同多核苷酸之间形成重叠末端。如CRISPR-Cas9中那样具有蛋白质和RNA元件两者的核酸酶试剂可作为已复合成核酸酶试剂的试剂提供,或可作为单独的蛋白质和RNA元件提供,在这种情况下,它们在本文所述的反应混合物中复合形成核酸酶试剂。
术语“核酸酶试剂的识别位点”包括核酸酶试剂在其处诱导切口或双链断裂的DNA序列。核酸酶试剂的识别位点对于细胞可为内源的(或天然的),或识别位点对于细胞可为外源的。在具体实施例中,识别位点对于细胞为外源的,从而在细胞基因组中不是天然存在的。在更进一步的实施例中,识别位点对于细胞为外源的,并且对于希望被定位在靶基因座处的目标多核苷酸为外源的。在进一步的实施例中,外源或内源识别位点在宿主细胞的基因组中仅出现一次。在具体实施例中,鉴定了在基因组内仅出现一次的内源或天然位点。然后可使用这种位点来设计将在内源识别位点处产生切口或双链断裂的核酸酶试剂。
识别位点的长度可变,并且包括例如对于锌指核酸酶(ZFN)对为约30-36bp(即,对于每个ZFN为约15-18bp)、对于转录激活因子样效应物核酸酶(TALEN)为约36bp、或对于CRISPR/Cas9向导RNA为约20bp的识别位点。
本文还提供了示例性识别位点的活性变体和片段。此类活性变体可与给定识别位点具有至少65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或更高的序列同一性,其中所述活性变体保留生物活性,从而能够被核酸酶试剂以序列特异性方式识别并切割。测量核酸酶试剂对识别位点造成的双链断裂的测定法是本领域已知的(例如, qPCR测定法,Frendewey D.et al.,Methods in Enzymology,2010,476:295-307(Frendewey D.等人,《酶学方法》,2010年,第476卷,第295-307页),该文献全文以引用的方式并入本文)。
在具体实施例中,识别位点被定位在编码选择标记的多核苷酸内。这种位置可位于选择标记的编码区内或者位于影响选择标记的表达的调控区内。因此,核酸酶试剂的识别位点可位于选择标记的内含子、编码选择标记的多核苷酸的启动子、增强子、调控区或任何非蛋白编码区中。在具体实施例中,识别位点处的切口或双链断裂会破坏选择标记的活性。测定功能选择标记存在与否的方法是已知的。
可在本文所公开的方法和组合物中使用任何会在所需识别位点中诱导切口或双链断裂的核酸酶试剂。可采用天然存在的或天然的核酸酶试剂,只要该核酸酶试剂在所需识别位点中诱导切口或双链断裂即可。作为另一种选择,可采用经修饰或经改造的核酸酶试剂。“经改造的核酸酶试剂”包括由其天然形式改造(修饰或衍生)成会特异性识别所需识别位点并在所需识别位点中诱导切口或双链断裂的核酸酶。因此,经改造的核酸酶试剂可衍生自天然的或天然存在的核酸酶试剂,或其可人工生成或合成。核酸酶试剂的修饰在蛋白切割剂中可少至一个氨基酸,或在核酸切割剂中可少至一个核苷酸。在一些实施例中,经改造的核酸酶在识别位点中诱导切口或双链断裂,其中所述识别位点不是会被天然(未经改造的或未经修饰的)核酸酶试剂识别的序列。在识别位点或其他DNA中产生切口或双链断裂在本文中可称为“切开”或“切割”识别位点或其他DNA。
随后细胞可通过以下两种方式之一修复这些断裂:非同源性末端接合和同源性指导的修复(同源重组)。在非同源性末端接合(NHEJ)中,通过使断裂末端彼此直接连接来修复双链断裂。因此,未在该位点中插入新的核酸物质,但一些核酸物质可能丢失,从而导致缺失。在同源性指导的修复中,与经切割的靶DNA序列具有同源性的供体多核苷酸可用作修复经切割的靶DNA序列的模板,使得遗传信息从供体多核苷酸传递到靶DNA。因此,可在该位点中插入/复制新的核酸物质。因NHEJ和/或同源性指导的修复所引起的靶DNA的修饰,可用于基因修正、基因替换、基因标记、转基因插入、核苷酸缺失、基因破坏、基因突变等。
在一个实施例中,核酸酶试剂为转录激活因子样效应物核酸酶(TALEN)。TAL效应物核酸酶是一类序列特异性核酸酶,其可用于在原核或真核生物基因组中的特定靶序列处产生双链断裂。可通过将天然的或经改造的转录激活因子样(TAL)效应物或其功能部分融合到内切核酸酶如FokI的催化结构域,来生成TAL效应物核酸酶。独特的模块化TAL效应物DNA结合结构域使得可以设计潜在地具有任何给定DNA识别特异性的蛋白质。因此,TAL效应物核酸酶的DNA结合结构域可被改造成识别特定DNA靶位点,故可用于在所需靶序列处产生双链断裂。参见WO 2010/079430;Morbitzer et al.(2010)PNAS 10.1073/pnas.1013133107(Morbitzer等人,2010年,《美国国家科学院院刊》,10.1073/pnas.1013133107);Scholze&Boch(2010)Virulence 1:428-432(Scholze和Boch,2010年,《毒力》,第1卷,第428-432页);Christian et al.Genetics(2010)186:757-761(Christian等人,《遗传学》,2010年,第186卷,第757-761页);Li et al.(2010)Nuc.Acids Res.(2010)doi:10.1093/nar/gkq704(Li等人,2010年,《核酸研究》,2010年,doi:10.1093/nar/gkq704);以及Miller et al.(2011)Nature Biotechnology 29:143–148(Miller等人,2011年,《自然生物技术》,第29卷,第143–148页);所有这些文献均以引用的方式并入本文。
合适的TAL核酸酶的示例以及用于制备合适的TAL核酸酶的方法公开于例如美国专利申请No.2011/0239315 A1、2011/0269234 A1、2011/0145940 A1、2003/0232410 A1、2005/0208489 A1、2005/0026157 A1、2005/0064474 A1、2006/0188987 A1、以及2006/0063231 A1中(每一份专利申请均据此以引用的方式并入)。在各种实施例中,TAL效应物核酸酶被改造成在例如目标基因组位点中的靶核酸序列之中或附近进行切开,其中所述靶核酸序列位于靶向载体将要修饰的序列之处或附近。适合与本文所提供的各种方法和组合物一起使用的TAL核酸酶,包括被专门设计成在本文所述的靶向载体将要修饰的靶核酸序列之处或附近进行结合的那些TAL核酸酶。
在一个实施例中,TALEN的每个单体包含经由两个高变残基识别单碱基对的33-35个TAL重复序列。在一个实施例中,核酸酶试剂为嵌合蛋白,其包含有效连接至独立核酸酶的基于TAL重复序列的DNA结合结构域。在一个实施例中,独立核酸酶为FokI内切核酸酶。在一个实施例中,核酸酶试剂包含第一基于TAL重复序列的DNA结合结构域和第二基于TAL重复序列的DNA结合结构域,其中所述第一基于TAL重复序列的DNA结合结构域和第二基于TAL重复序列的DNA结合结构域中的每一者均有效连接至FokI核酸酶亚基,其中所述第一基于TAL重复序列的DNA结合结构域和第二基于TAL重复序列的DNA结合结构域识别每条DNA靶序列中被可变长度(12-20bp)的间区序列隔开的两条邻接DNA靶序列,并且其中所述FokI核酸酶亚基发生二聚化,从而生成能在靶序列处产生双链断裂的活性核酸酶。
在本文所公开的各种方法和组合物中采用的核酸酶试剂还可包括锌指核酸酶(ZFN)。在一个实施例中,ZFN的每个单体包含3个或更多个基于锌指的DNA结合结构域,其中每个基于锌指的DNA结合结构域结合于3bp亚位点。在其他实施例中,ZFN为包含有效连接至独立核酸酶的、基于锌指的DNA结合结构域的嵌合蛋白。在一个实施例中,独立内切核酸酶为FokI内切核酸酶。在一个实施例中,核酸酶试剂包含第一ZFN和第二ZFN,其中所述第一ZFN和第二ZFN中的每一者均有效连接至FokI核酸酶亚基,其中所述第一ZFN和第二ZFN识别每条DNA靶序列中被约5-7bp间区序列隔开的两条邻接DNA靶序列,并且其中所述FokI核酸酶亚基发生二聚化,从而生成能产生双链断裂的活性核酸酶。参见例如US20060246567;US20080182332;US20020081614;US20030021776;WO/2002/057308A2;US20130123484;US20100291048;WO/2011/017293A2;以及Gaj et al.(2013)Trends in Biotechnology,31(7):397-405(Gaj等人,2013年,《生物技术趋势》,第31卷,第7期,第397-405页);这些文献中的每一篇均以引用的方式并入本文。
在本文所提供的方法的一个实施例中,核酸酶试剂包括(a)包含融合至FokI核酸内切酶的、基于锌指的DNA结合结构域的嵌合蛋白;或(b)包含融合至FokI核酸内切酶的转录激活因子样效应物核酸酶(TALEN)的嵌合蛋白。
在又一个实施例中,核酸酶试剂为大范围核酸酶。已基于保守序列基序将大范围核酸酶分类为四个家族,这些家族是LAGLIDADG(SEQ ID NO:16)、GIY-YIG、H-N-H和His-Cys框家族。这些基序参与金属离子的配位和磷酸二酯键的水解。归巢内切酶以其长识别位点及耐受其DNA底物中的一些序列多态性而著称。大范围核酸酶结构域、结构和功能是已知的,参见例如,Guhan and Muniyappa(2003)Crit Rev Biochem Mol Biol38:199-248(Guhan和Muniyappa,2003年,《生物化学与分子生物学评论》,第38卷,第199-248页);Lucas et al.,(2001)Nucleic Acids Res 29:960-9(Lucas等人,2001年,《核酸研究》,第29卷,第960-969页);Jurica and Stoddard,(1999)Cell Mol Life Sci 55:1304-26(Jurica和Stoddard,1999年,《细胞和分子生命科学》,第55卷,第1304-1326页);Stoddard,(2006)Q Rev Biophys 38:49-95(Stoddard,2006年,《生物物理学季评》,第38卷,第49-95页);以及Moure et al.,(2002)Nat Struct Biol 9:764(Moure等人,2002年,《自然结构生物学》,第9卷,第764页)。在一些示例中,使用天然存在的变体和/或经改造的衍生大范围核酸酶。用于调整动力学、辅因子相互作用、表达、最适条件和/或识别位点特异性及活性筛选的方法是已知的,参见例如,Epinat et al.,(2003)Nucleic Acids Res 31:2952-62(Epinat等人,2003年,《核酸研究》,第31卷,第2952-2962页);Chevalier et al.,(2002)Mol Cell 10:895-905(Chevalier等人,2002年,《分子细胞》,第10卷,第895-905页);Gimble et al.,(2003)Mol Biol 334:993-1008(Gimble等人,2003年,《分子生物学》,第334卷,第993-1008页);Seligman et al.,(2002)Nucleic Acids Res 30:3870-9(Seligman等人,2002年,《核酸研究》,第30卷,第3870-3879页);Sussman et al.,(2004)J Mol Biol 342:31-41(Sussman等人,2004年,《分子生物学杂志》,第342卷,第31-41页);Rosen et al.,(2006)Nucleic Acids Res 34:4791-800(Rosen等人,2006年,《核酸研究》,第34卷,第4791-4800页);Chames et al.,(2005)Nucleic Acids Res 33:e178(Chames等人,2005年,《核酸研究》,第33卷,第e178页);Smith et al.,(2006)Nucleic Acids Res 34:e149(Smith等人,2006年,《核酸研究》,第34卷,第e149页);Gruen et al.,(2002)Nucleic Acids Res 30:e29(Gruen等人,2002年,《核酸研究》,第30卷,第e29页);Chen and Zhao,(2005)Nucleic Acids Res 33:e154(Chen和Zhao,2005年,《核酸研究》,第33卷,第e154页);WO2005105989;WO2003078619;WO2006097854;WO2006097853;WO2006097784;以及WO2004031346。
可在本发明中使用任何大范围核酸酶,包括但不限于I-SceI、I-SceII、I-SceIII、I-SceIV、I-SceV、I-SceVI、I-SceVII、I-CeuI、I-CeuAIIP、I-CreI、I-CrepsbIP、I-CrepsbIIP、I-CrepsbIIIP、I-CrepsbIVP、I-TliI、I-PpoI、PI-PspI、F-SceI、F-SceII、F-SuvI、F-TevI、F-TevII、I-AmaI、I-AniI、I-ChuI、I-CmoeI、I-CpaI、I-CpaII、I-CsmI、I-CvuI、I-CvuAIP、I-DdiI、I-DdiII、I-DirI、I-DmoI、I-HmuI、I-HmuII、I-HsNIP、I-LlaI、I-MsoI、I-NaaI、I-NanI、I-NcIIP、I-NgrIP、I-NitI、I-NjaI、I-Nsp236IP、I-PakI、I-PboIP、I-PcuIP、I-PcuAI、I-PcuVI、I-PgrIP、I-PobIP、I-PorI、I-PorIIP、I-PbpIP、I-SpBetaIP、I-ScaI、I-SexIP、I-SneIP、I-SpomI、I-SpomCP、I-SpomIP、I-SpomIIP、I-SquIP、I-Ssp6803I、I-SthPhiJP、I-SthPhiST3P、I-SthPhiSTe3bP、I-TdeIP、I-TevI、I-TevII、I-TevIII、I-UarAP、I-UarHGPAIP、I-UarHGPA13P、I-VinIP、I-ZbiIP、PI-MtuI、PI-MtuHIP、PI-MtuHIIP、PI-PfuI、PI-PfuII、PI-PkoI、PI-PkoII、PI-Rma43812IP、PI-SpBetaIP、PI-SceI、PI-TfuI、PI-TfuII、PI-ThyI、PI-TliI、PI-TliII、或其任何活性变体或片段。
在一个实施例中,所述大范围核酸酶识别12至40个碱基对的双链DNA序列。在一个实施例中,所述大范围核酸酶识别基因组中的一个完全匹配的靶序列。在一个实施例中,所述大范围核酸酶为归巢核酸酶。在一个实施例中,所述归巢核酸酶为归巢核酸酶的LAGLIDADG(SEQ ID NO:16)家族。在一个实施例中,归巢核酸酶的LAGLIDADG(SEQ ID NO:16)家族选自I-SceI、I-CreI和I-Dmol。
核酸酶试剂还可包括限制性内切核酸酶(限制性内切酶),其包括I型、II型、III型和IV型内切核酸酶。I型和III型限制性内切核酸酶识别特定识别位点,但通常在离核酸酶结合位点的可变位置处切割,该核酸酶结合位点离切割位点(识别位点)可达数百个碱基对。在II型系统中,酶切活性独立于任何甲基化酶活性,并且通常在结合位点之内或附近的特定位点处发生切割。大多数II型酶切开回文序列,但是IIa型酶识别非回文识别位点并在识别位点之外切割,IIb型酶在识别位点之外的两个位点处切开序列两次,并且IIs型酶识别非对称识别位点并在一侧且离识别位点约1-20个核苷酸的限定距离处切割。IV型限制性内切酶靶向甲基化DNA。限制性内切酶进一步在例如REBASE数据库中进行说明和分类(地址为rebase.neb.com的网页;Roberts et al.,(2003)Nucleic Acids Res 31:418-20(Roberts等人,2003年,《核酸研究》,第31卷,第418-420页),Roberts et al.,(2003)Nucleic Acids Res 31:1805-12(Roberts等人,2003年,《核酸研究》,第31卷,第1805-1812页),以及Belfort et al.,(2002)in Mobile DNA II,pp.761-783,Eds.Craigie et al.,(ASM Press,Washington,DC)(Belfort等人,2002年,载于《可移动的DNA II》,第761-783页,Craigie等人编辑,美国华盛顿特区ASM出版社))。在具体实施例中,可将至少两种核酸内切酶选择为核酸酶试剂,其中所述酶产生相容或互补的粘性末端。
在各种方法和组合物中采用的核酸酶试剂还可包括CRISPR/Cas系统。此类系统可采用Cas9核酸酶,其在一些情况下针对要用来表达其的所需细胞类型进行了密码子优化。该系统还采用融合的crRNA-tracrRNA构建体,该构建体与经密码子优化的Cas9一起发挥作用。该单一RNA通常称为向导RNA或gRNA。在gRNA内,crRNA部分被确定为给定识别位点的“靶序列”,并且tracrRNA通常称为“支架”。已证实该系统可在多种真核细胞和原核细胞中发挥作用。简而言之,包含靶序列的短DNA片段被插入到向导RNA表达质粒中。gRNA表达质粒包含靶序列(在一些实施例中约20个核苷酸)、一种形式的tracrRNA序列(支架)以及在细胞中有活性的合适启动子及用于在真核细胞中正确加工的必要元件。这些系统中的多种系统依赖于定制的互补寡核苷酸,这些寡核苷酸退火而形成双链DNA,接着被克隆到gRNA表达质粒中。然后将gRNA表达盒和Cas9表达盒引入到细胞中。参见例如Mali P et al.(2013)Science 2013Feb 15;339(6121):823-6(Mali P等人,2013年,《科学》,2013年2月15日,第339卷,第6121期,第823-826页);Jinek M et al.Science 2012Aug 17;337(6096):816-21(Jinek M等人,《科学》,2012年8月17日,第337卷,第6096期,第816-821页);Hwang WY et al.Nat Biotechnol 2013Mar;31(3):227-9(Hwang WY等人,《自然生物技术》,2013年3月,第31卷,第3期,第227-229页);Jiang W et al.Nat Biotechnol 2013 Mar;31(3):233-9(Jiang W等人,《自然生物技术》,2013年3月,第31卷,第3期,第233-239页);以及Cong L et al.Science 2013Feb 15;339(6121):819-23(Cong L等人,《科学》,2013年2月15日,第339卷,第6121期,第819-823页),这些文献中的每一篇均以引用的方式并入本文。
本文所公开的方法和组合物可利用成簇的规律间隔的短回文重复序列(CRISPR)/CRISPR相关(Cas)系统或此类系统的组分来修饰细胞内的基因组。CRISPR/Cas系统包括参与Cas基因的表达或指导Cas基因的活性的转录物和其他元件。CRISPR/Cas系统可为I型、II型或III型系统。本文所公开的方法和组合物通过利用CRISPR复合物(包含与Cas蛋白复合的向导RNA(gRNA))来采用CRISPR/Cas系统对核酸进行定点切割。
用于本文所公开的方法中的一些CRISPR/Cas系统为非天然存在的。“非天然存在的”系统包括任何表明受到人工干预的系统,诸如该系统的一个或多个组分从其天然存在的状态改变或突变,至少基本上不含其在自然界中与其天然关联的至少一个其他组分,或和不与其天然关联的至少一个其他组分相关联。例如,一些CRISPR/Cas系统采用非天然存在的CRISPR复合物,这些复合物包含在天然情况下不会同时存在的gRNA和Cas蛋白。
本发明还提供了核酸酶试剂的活性变体和片段(即,经改造的核酸酶试剂)。此类活性变体可与天然核酸酶试剂具有至少65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或更高的序列同一性,其中所述活性变体保留在所需识别位点处切开的能力,从而保留了切口或双链断裂诱导活性。例如,本文所述的任何核酸酶试剂可由天然内切核酸酶序列修饰而成,并且可被设计成在不被天然核酸酶试剂识别的识别位点处识别并诱导切口或双链断裂。因此,在一些实施例中,经改造的核酸酶具有在与对应天然核酸酶试剂识别位点不同的识别位点处诱导切口或双链断裂的特异性。针对切口或双链断裂诱导活性的测定法是已知的,并且一般测量内切核酸酶对包含识别位点的DNA底物的总体活性和特异性。
IV.CRISPR/Cas系统(gRNA-Cas复合物)
本发明的方法可采用CRISPR/Cas系统(例如,gRNA-Cas复合物)对核酸进行定点切割。具体地讲,由gRNA引导至所鉴定的靶位点处对核酸进行Cas切割会产生具有这样的末端的经酶切的核酸,所述末端随后可接合到第二核酸,从而以位点特异性方式组装两个或更多个核酸。
“gRNA-Cas复合物”包含Cas蛋白与gRNA的复合物。gRNA可被设计或选择成将Cas切割引导至靶位点,从而在经切割的核酸与另一不同核酸之间形成重叠末端。gRNA-Cas复合物可作为已复合的试剂提供,或可作为单独的蛋白质和RNA元件提供,在这种情况下,它们在本文所述的方法和反应混合物中复合形成gRNA-Cas复合物。
A.Cas RNA引导的核酸内切酶
Cas蛋白一般包含至少一个RNA识别或结合结构域。此类结构域可与向导RNA(gRNA,下文更详细地说明)相互作用。Cas蛋白还可包含核酸酶结构域(例如,DNA酶或RNA酶结构域)、DNA结合结构域、解旋酶结构域、蛋白-蛋白相互作用结构域、二聚化结构域以及其他结构域。核酸酶结构域具有用于核酸切割的催化活性。切割包括核酸分子共价键的断裂。切割可产生平头末端或交错末端,并且其可为单链或双链的。
Cas蛋白的示例包括Cas1、Cas1B、Cas2、Cas3、Cas4、Cas5、Cas5e(CasD)、Cas6、Cas6e、Cas6f、Cas7、Cas8a1、Cas8a2、Cas8b、Cas8c、Cas9(Csn1或Csx12)、Cas10、Casl0d、CasF、CasG、CasH、Csy1、Csy2、Csy3、Cse1(CasA)、Cse2(CasB)、Cse3(CasE)、Cse4(CasC)、Csc1、Csc2、Csa5、Csn2、Csm2、Csm3、Csm4、Csm5、Csm6、Cmr1、Cmr3、Cmr4、Cmr5、Cmr6、Csb1、Csb2、Csb3、Csx17、Csx14、Csx10、Csx16、CsaX、Csx3、Csx1、Csx15、Csf1、Csf2、Csf3、Csf4和Cu1966,以及它们的同源物或修饰形式。
可在本文所公开的方法和组合物中使用任何在所需识别位点中诱导切口或双链断裂的Cas蛋白。可采用天然存在的或天然的Cas蛋白,只要该Cas蛋白在所需识别位点处诱导双链断裂即可。作为另一种选择,可采用经修饰或经改造的Cas蛋白。“经改造的Cas蛋白”包括由其天然形式改造(修饰或衍生)成在所需识别位点中特异性识别并诱导切口或双链断裂的Cas蛋白。因此,经改造的Cas蛋白可衍生自天然的或天然存在的Cas蛋白,或其可人工生成或合成。
在特定实施例中,Cas蛋白为Cas9。这些Cas9蛋白通常共用具有保守架构的四个关键基序。基序1、2和4为RuvC样基序,并且基序3为HNH基序。Cas9的核酸酶活性切割靶DNA,产生双链断裂。随后细胞可通过以下两种方式之一修复这些断裂:非同源性末端接合和同源性指导的修复(同源重组)。在非同源性末端接合(NHEJ)中,通过使断裂末端彼此直接连接来修复双链断裂。因此,未在该位点中插入新的核酸物质,但一些核酸物质可能丢失,从而导致缺失。在同源性指导的修复中,与经切割的靶DNA序列具有同源性的供体多核苷酸可用作修复经切割的靶DNA序列的模板,使得遗传信息从供体多核苷酸传递到靶DNA。因此,可在该位点中插入/复制新的核酸物质。因NHEJ和/或同源性指导的修复所引起的靶DNA的修饰,可用于基因修正、基因替换、基因标记、转基因插入、核苷酸缺失、基因破坏、基因突变等。
Cas蛋白可来自II型CRISPR/Cas系统。例如,Cas蛋白可为Cas9蛋白或衍生自Cas9蛋白。这些Cas9蛋白通常共用具有保守架构的四个关键基序。基序1、2和4为RuvC样基序,并且基序3为HNH基序。Cas9蛋白可来自例如化脓性链球菌(Streptococcus pyogenes)、嗜热链球菌(Streptococcus thermophilus)、链球菌属物种(Streptococcus sp.)、金黄色葡萄球菌(Staphylococcus aureus)、达氏拟诺卡氏菌(Nocardiopsis dassonvillei)、始旋链霉菌(Streptomyces pristinaespiralis)、绿色产色链霉菌(Streptomyces viridochromogenes)、绿色产色链霉菌(Streptomyces viridochromogenes)、粉红链孢囊菌(Streptosporangium roseum)、粉红链孢囊菌(Streptosporangium roseum)、酸热脂环酸芽孢杆菌(AlicyclobacHlus acidocaldarius)、假蕈状芽孢杆菌(Bacillus pseudomycoides)、还原硒酸盐芽孢杆菌(Bacillus selenitireducens)、西伯利亚微小杆菌(Exiguobacterium sibiricum)、德氏乳杆菌(Lactobacillus delbrueckii)、唾液乳杆菌(Lactobacillus salivarius)、海洋微颤菌(Microscilla marina)、伯克氏菌(Burkholderiales bacterium)、萘降解极地单胞菌(Polaromonas naphthalenivorans)、极地单胞菌属物种(Polaromonas sp.)、瓦氏鳄球藻(Crocosphaera watsonii)、蓝杆藻属物种(Cyanothece sp.)、铜绿微囊藻(Microcystis aeruginosa)、聚球藻属物种(Synechococcus sp.)、阿拉伯糖醋盐杆菌(Acetohalobium arabaticum)、制氨菌(Ammonifex degensii)、热解纤维素菌(Caldicelulosiruptor becscii)、Candidatus Desulforudis、肉毒梭菌(Clostridium botulinum)、艰难梭菌(Clostridium difficile)、大芬戈尔德菌(Finegoldia magna)、嗜热盐碱厌氧菌(Natranaerobius thermophilus)、丙酸互营细菌(Pelotomaculum thermopropionicum)、喜温嗜酸硫杆菌(Acidithiobacillus caldus)、嗜酸氧化亚铁硫杆菌(Acidithiobacillus ferrooxidans)、紫色硫细菌(Allochromatium vinosum)、海杆菌属物种(Marinobacter sp.)、嗜盐亚硝化球菌(Nitrosococcus halophilus)、瓦氏亚硝化球菌(Nitrosococcus watsoni)、游海假交替单胞菌(Pseudoalteromonas haloplanktis)、纤线杆菌(Ktedonobacter racemifer)、甲烷盐菌(Methanohalobium evestigatum)、多变鱼腥藻Anabaena variabilis)、泡沫节球藻(Nodularia spumigena)、念珠藻属物种(Nostoc sp.)、极大节螺藻(Arthrospira maxima)、钝顶节螺藻(Arthrospira platensis)、节螺藻属物种(Arthrospira sp.)、鞘丝藻属物种(Lyngbya sp.)、原型微鞘藻(Microcoleus chthonoplastes)、颤藻属物种(Oscillatoria sp.)、运动石袍菌(Petrotoga mobilis)、非洲栖热腔菌(Thermosipho africanus)、或深海单细胞蓝细菌(Acaryochloris marina)。Cas9家族成员的附加示例在WO 2014/131833中有描述,该专利全文以引用的方式并入本文。来自化脓性链球菌(S.pyogenes)或从其衍生的Cas9蛋白是优选的酶。为来自化脓性链球菌的Cas9蛋白分配了SwissProt登录号Q99ZW2。
Cas蛋白可为野生型蛋白(即,自然界存在的蛋白)、经修饰的Cas蛋白(即,Cas蛋白变体)、或者野生型或经修饰的Cas蛋白的片段。Cas蛋白也可以是野生型或经修饰的Cas蛋白的活性变体或片段。活性变体或片段可与野生型或经修饰的Cas蛋白或者其一部分具有至少80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或更高的序列同一性,其中所述活性变体保留了在所需切割位点处切开的能力,从而保留了切口诱导活性或双链断裂诱导活性。针对切口诱导活性或双链断裂诱导活性的测定法是已知的,并且一般测量Cas蛋白对包含切割位点的DNA底物的总体活性和特异性。
可修饰Cas蛋白以提高或降低核酸结合亲和力、核酸结合特异性和/或酶活性。还可修饰Cas蛋白以改变蛋白的任何其他活性或特性,诸如稳定性。例如,Cas蛋白的一个或多个核酸酶结构域可以被修饰、缺失或失活,或者Cas蛋白可以被截短以去除对于蛋白质的功能并非必要的结构域,或优化(例如,增强或降低)Cas蛋白的活性。
一些Cas蛋白包含至少两个核酸酶结构域,诸如DNA酶结构域。例如,Cas9蛋白可包含RuvC样核酸酶结构域和HNH样核酸酶结构域。RuvC结构域和HNH结构域各自可切开双链DNA的不同链,从而在DNA中产生双链断裂。参见例如Jinek et al.(2012)Science 337:816-821(Jinek等人,2012年,《科学》,第337卷,第816-821页),该文献全文据此以引用的方式并入。
这些核酸酶结构域中的一者或两者可以被缺失或突变,使得它们不再有功能或具有降低的核酸酶活性。如果核酸酶结构域之一被缺失或突变,则所得的Cas蛋白(例如,Cas9)可称为切口酶,并且可在双链DNA内的CRISPR RNA识别序列处生成单链断裂,但不会生成双链断裂(即,其可切割互补链或非互补链,但无法同时切割两者)。如果这两个核酸酶结构域都被缺失或突变,则所得的Cas蛋白(例如,Cas9)将具有降低的切割双链DNA两条链的能力。将Cas9转变为切口酶的突变的示例是来自化脓性链球菌的Cas9的RuvC结构域中的D10A(Cas9的第10位处天冬氨酸至丙氨酸)突变。同样,来自化脓性链球菌的Cas9的HNH结构域中的H939A(氨基酸位置839处组氨酸至丙氨酸)或H840A(氨基酸位置840处组氨酸至丙氨酸)可将Cas9转变为切口酶。将Cas9转变为切口酶的突变的其他示例包括来自嗜热链球菌(S.thermophilus)的Cas9的对应突变。参见例如Sapranauskas et al.(2011)Nucleic Acids Research 39:9275-9282(Sapranauskas等人,2011年,《核酸研究》,第39卷,第9275-9282页)和WO 2013/141680,这些文献中的每一篇全文均以引用的方式并入本文。此类突变可使用诸如定点诱变、PCR介导的诱变或全基因合成的方法来生成。其他形成切口酶的突变的示例可见于例如WO/2013/176772A1和WO/2013/142578A1中,这些专利中的每一篇均以引用的方式并入本文。
Cas蛋白也可为融合蛋白。例如,Cas蛋白可融合到切割结构域、表观遗传修饰结构域、转录激活结构域或转录阻遏物结构域。参见WO 2014/089290,该专利全文以引用的方式并入本文。Cas蛋白也可融合到异源多肽,从而提供增强或减弱的稳定性。融合的结构域或异源多肽可位于N端、C端或Cas蛋白的内部。
Cas蛋白可融合到有助于亚细胞定位的异源多肽。此类异源肽包括例如用于靶向细胞核的核定位信号(NLS)如SV40NLS、用于靶向线粒体的线粒体定位信号、ER滞留信号等。参见例如Lange et al.(2007)J.Biol.Chem.282:5101-5105(Lange等人,2007年,《生物化学杂志》,第282卷,第5101-5105页)。此类亚细胞定位信号可位于N端、C端或Cas蛋白内的任何位置处。NLS可包含一段碱性氨基酸,并且可为单分型(monopartite)序列或双分型(bipartite)序列。
Cas蛋白也可连接至细胞穿透结构域。例如,细胞穿透结构域可衍生自HIV-1TAT蛋白、来自人乙肝病毒的TLM细胞穿透基序、MPG、Pep-1、VP22、来自单纯性疱疹病毒的细胞穿透肽、或多聚精氨酸肽序列。参见例如WO 2014/089290,该专利全文以引用的方式并入本文。细胞穿透结构域可位于N端、C端或Cas蛋白内的任何位置处。
Cas蛋白还可包含便于示踪或纯化的异源多肽,诸如荧光蛋白、纯化标签或表位标签。荧光蛋白的示例包括绿色荧光蛋白(例如,GFP、GFP-2、tagGFP、turboGFP、eGFP、Emerald、Azami Green、Monomeric Azami Green、CopGFP、AceGFP、ZsGreenl)、黄色荧光蛋白(例如,YFP、eYFP、Citrine、Venus、YPet、PhiYFP、ZsYellowl)、蓝色荧光蛋白(例如,eBFP、eBFP2、Azurite、mKalamal、GFPuv、Sapphire、T-sapphire)、青色荧光蛋白(例如,eCFP、Cerulean、CyPet、AmCyanl、Midoriishi-Cyan)、红色荧光蛋白(mKate、mKate2、mPlum、DsRed monomer、mCherry、mRFP1、DsRed-Express、DsRed2、DsRed-Monomer、HcRed-Tandem、HcRedl、AsRed2、eqFP611、mRaspberry、mStrawberry、Jred)、橙色荧光蛋白(mOrange、mKO、Kusabira-Orange、Monomeric Kusabira-Orange、mTangerine、tdTomato)以及任何其他合适的荧光蛋白。标签的示例包括谷胱甘肽-S-转移酶(GST)、几丁质结合蛋白(CBP)、麦芽糖结合蛋白、硫氧还蛋白(TRX)、多聚(NANP)、串联亲和纯化(TAP)标签、myc、AcV5、AU1、AU5、E、ECS、E2、FLAG、血凝素(HA)、nus、Softag 1、Softag 3、Strep、SBP、Glu-Glu、HSV、KT3、S、S1、T7、V5、VSV-G、组氨酸(His)、生物素羧基载体蛋白(BCCP)以及钙调蛋白。
在一些实施例中,Cas蛋白可被修饰成使所得核酸酶活性被改变。Cas中的某些突变可降低核酸酶切割靶DNA的互补链和非互补链两者的能力。例如,Cas蛋白可在已知位置中突变,使得核酸酶活性局限于互补链或非互补链的切割。具体地讲,具有D10A(Cas9的氨基酸位置10处天冬氨酸至丙氨酸)突变的Cas9可切割靶DNA的互补链,但切割靶DNA的非互补链的能力降低。在一些实施例中,具有H840A(氨基酸位置840处组氨酸至丙氨酸)突变的Cas9可切割靶DNA的非互补链,但切割靶DNA的互补链的能力降低。具有D10A或H840A突变的Cas9的核酸酶活性将导致单链断裂(SSB)而非DSB。可使其他残基突变以实现相同效果(即,使一个或另一个核酸酶部分失活)。作为非限制性示例,残基D10、G12、G17、E762、H840、N854、N863、H982、H983、A984、D986和/或A987(即,置换)。此外,除丙氨酸之外的置换氨基酸可为合适的。在一些实施例中,当核酸酶具有降低的活性时(例如,当Cas9蛋白具有D10、G12、G17、E762、H840、N854、N863、H982、H983、A984、D986和/或A987突变,诸如D10A、G12A、G17A、E762A、H840A、N854A、N863A、H982A、H983A、A984A和/或D986A时),核酸酶只要保留与gRNA相互作用的能力,就仍可以位点特异性方式结合于靶DNA,因为gRNA仍会将其引导至靶DNA序列。
在一些实施例中,Cas被改变成使得核酸酶不切割靶DNA的互补链或非互补链。例如,具有D10A突变和H840A突变的Cas9切割靶DNA的互补链和非互补链两者的能力降低。可使其他残基突变以实现相同效果(即,使一个或另一个核酸酶部分发生失活)。作为非限制性示例,残基D10、G12、G17、E762、H840、N854、N863、H982、H983、A984、D986和/或可被置换,以基本上消除核酸酶活性。此外,除丙氨酸置换之外的突变可为合适的。
术语“靶位点”或“靶序列”可互换使用,并且包括靶DNA中存在的这样的核酸序列,只要存在充分的结合条件,gRNA的DNA靶向区段就将与该核酸序列结合。例如,靶DNA内的靶位点(或靶序列)被Cas蛋白或gRNA所靶向(或与之结合、或与之杂交、或与之互补)。合适的DNA/RNA结合条件包括通常存在于细胞中的生理条件。其他合适的DNA/RNA结合条件(例如,无细胞系统中的条件)是本领域已知的(参见例如Molecular Cloning:A Laboratory Manual,3rd Ed.(Sambrook et al.,Harbor Laboratory Press 2001)(《分子克隆实验指南》,第3版,Sambrook等人,冷泉港实验室出版社,2001年))。靶DNA的与Cas蛋白或gRNA互补并杂交的链称为“互补链”,并且靶DNA的与“互补链”互补(并因此不与Cas蛋白或gRNA互补)的链称为“非互补链”或“模板链”。
Cas蛋白可在靶序列之内或靶序列之外的位点处切割核酸。“切割位点”包括Cas蛋白在其中产生单链断裂或双链断裂的核酸位置。如果Cas蛋白产生双链断裂,则切割位点可位于核酸的两条链上的相同位置处(产生平头末端),或可位于每条链上的不同位点处(产生粘性或粘着末端)。还可通过使用在每条链上的切割位点处产生单链断裂的两种Cas蛋白来产生粘性末端。Cas9对靶DNA的位点特异性切割可在由以下两者决定的位置处发生:(i)向导RNA与靶DNA之间的碱基配对互补性,以及(ii)靶DNA中的短基序,称为前间区序列邻近基序(PAM)。例如,Cas9的切割位点可为PAM序列上游的约1至约10或者约2至约5个碱基对(例如,3个碱基对)。在一些实施例中(例如,当使用来自化脓性链球菌的Cas9或密切相关的Cas9时),非互补链的PAM序列可为5'-XGG-3',其中X为任何DNA核苷酸,并且X紧邻靶DNA的非互补链的靶序列的3'。因此,互补链的PAM序列将为5'-CCY-3',其中Y为任何DNA核苷酸并且Y紧邻靶DNA的互补链的靶序列的5'。在一些此类实施例中,X和Y可为互补的,并且X-Y碱基对可为任何碱基对(例如,X=C且Y=G;X=G且Y=C;X=A且Y=T,X=T且Y=A)。
Cas蛋白可以任何形式提供。例如,Cas蛋白可以蛋白的形式提供,诸如与gRNA复合的Cas蛋白。作为另一种选择,Cas蛋白可以编码Cas蛋白的核酸的形式提供,诸如RNA(例如,信使RNA(mRNA))或DNA。任选地,编码Cas蛋白的核酸可进行密码子优化,以在特定细胞或生物体中有效翻译成蛋白。例如,可对编码Cas蛋白的核酸进行修饰,以替换成与天然存在的多核苷酸序列相比在细菌细胞、酵母细胞、人细胞、非人细胞、哺乳动物细胞、啮齿动物细胞、小鼠细胞、大鼠细胞或任何其他目标宿主细胞中具有更高使用频率的密码子。当将编码Cas蛋白的核酸引入细胞中时,Cas蛋白可在细胞中瞬时地、条件性地或组成型地表达。
编码Cas蛋白的核酸可稳定整合在细胞的基因组中,并有效连接至细胞中有活性的启动子。作为另一种选择,编码Cas蛋白的核酸可有效连接至表达构建体中的启动子。表达构建体包括任何能够指导目标基因或其他核酸序列(例如,Cas基因)的表达并可将这种目标核酸序列转移到靶细胞中的核酸构建体。例如,编码Cas蛋白的核酸可位于包含核酸插入序列的靶向载体和/或包含编码gRNA的DNA的载体中,或其可位于相对于包含核酸插入序列的靶向载体而言单独的和/或相对于包含编码gRNA的DNA的载体而言单独的载体或质粒中。可用于表达构建体的启动子包括例如在大鼠、真核生物、哺乳动物、非人哺乳动物、人、啮齿动物、小鼠或仓鼠多能细胞中有活性的启动子。此类启动子可为例如条件启动子、诱导型启动子、组成型启动子或组织特异性启动子。其他启动子的示例在本文别处有描述。
B.向导RNA(gRNA)
“向导RNA”或“gRNA”包括结合于Cas蛋白并使Cas蛋白靶向靶DNA内的特定位置的RNA分子。向导RNA(gRNA)可包含两个区段:“DNA靶向区段”和“蛋白结合区段”。“区段”包括分子的区段、部分或区域,诸如RNA中的一个邻接核苷酸段。一些gRNA包含两个单独的RNA分子:“激活因子-RNA”和“靶向因子-RNA”。其他gRNA为单个RNA分子(单个RNA多核苷酸),其也可称为“单分子gRNA”、“单向导RNA”或“sgRNA”。参见例如WO/2013/176772A1、WO/2014/065596A1、WO/2014/089290A1、WO/2014/093622A2、WO/2014/099750A2、WO/2013142578A1以及WO 2014/131833A1,这些专利中的每一篇均以引用的方式并入本文。术语“向导RNA”和“gRNA”包括双分子gRNA和单分子gRNA两者。
示例性的双分子gRNA包含crRNA样(“CRISPR RNA”或“靶向因子-RNA”或“crRNA”或“crRNA重复序列”)分子以及对应的tracrRNA样(“反式作用CRISPR RNA”或“激活因子-RNA”或“tracrRNA”或“支架”)分子。crRNA包含gRNA的DNA靶向区段(单链)和一段核苷酸,该段核苷酸形成gRNA的蛋白结合区段的dsRNA双链体的一半。对应的tracrRNA(激活因子-RNA)包含一段核苷酸,该段核苷酸形成gRNA的蛋白结合区段的dsRNA双链体的另一半。crRNA的一段核苷酸与tracrRNA的一段核苷酸互补并杂交,从而形成gRNA的蛋白结合结构域的dsRNA双链体。因此,每个crRNA可以说成具有对应的tracrRNA。crRNA另外提供了单链DNA靶向区段。因此,gRNA包含与靶序列杂交的序列和tracrRNA。
crRNA和对应的tracrRNA(作为对应的对)杂交而形成gRNA。crRNA另外提供了与CRISPR RNA识别序列杂交的单链DNA靶向区段。如果用于细胞内的修饰,则给定crRNA或tracrRNA分子的确切序列可被设计成对于将在其中使用这些RNA分子的物种具有特异性。参见例如Mali P et al.(2013)Science 2013Feb 15;339(6121):823-6(Mali P等人,2013年,《科学》,2013年2月15日,第339卷,第6121期,第823-826页);Jinek M et al.Science 2012Aug 17;337(6096):816-21(Jinek M等人,《科学》,2012年8月17日,第337卷,第6096期,第816-821页);Hwang WY et al.Nat Biotechnol 2013Mar;31(3):227-9(Hwang WY等人,《自然生物技术》,2013年3月,第31卷,第3期,第227-229页);Jiang W et al.Nat Biotechnol 2013 Mar;31(3):233-9(Jiang W等人,《自然生物技术》,2013年3月,第31卷,第3期,第233-239页);以及Cong L et al.Science 2013 Feb 15;339(6121):819-23(Cong L等人,《科学》,2013年2月15日,第339卷,第6121期,第819-823页),这些文献中的每一篇均以引用的方式并入本文。
给定gRNA的DNA靶向区段(crRNA)包含与靶DNA中的序列互补的核苷酸序列。gRNA的DNA靶向区段通过杂交(即,碱基配对)以序列特异性方式与靶DNA相互作用。因此,DNA靶向区段的核苷酸序列可变化,并且决定将与gRNA和靶DNA相互作用的靶DNA内的位置。可修饰个体gRNA的DNA靶向区段,以与靶DNA内的任何所需序列杂交。天然存在的crRNA随Cas9系统和生物体不同而不同,但通常包含21至72个核苷酸长的靶向区段,该靶向区段被21至46个核苷酸长的两个正向重复序列(DR)侧接(参见例如WO2014/131833)。就化脓性链球菌而言,DR为36个核苷酸长,并且靶向区段为30个核苷酸长。位于3’的DR与对应的tracrRNA互补并杂交,继而结合于Cas9蛋白。
DNA靶向区段的长度可为约12个核苷酸至约100个核苷酸。例如,DNA靶向区段的长度可为约12个核苷酸(nt)至约80nt、约12nt至约50nt、约12nt至约40nt、约12nt至约30nt、约12nt至约25nt、约12nt至约20nt、或约12nt至约19nt。作为另一种选择,DNA靶向区段的长度可为约19nt至约20nt、约19nt至约25nt、约19nt至约30nt、约19nt至约35nt、约19nt至约40nt、约19nt至约45nt、约19nt至约50nt、约19nt至约60nt、约19nt至约70nt、约19nt至约80nt、约19nt至约90nt、约19nt至约100nt、约20nt至约25nt、约20nt至约30nt、约20nt至约35nt、约20nt至约40nt、约20nt至约45nt、约20nt至约50nt、约20nt至约60nt、约20nt至约70nt、约20nt至约80nt、约20nt至约90nt、或约20nt至约100nt。
与靶DNA的核苷酸序列(CRISPR RNA识别序列)互补的DNA靶向区段的核苷酸序列的长度可为至少约12nt。例如,DNA靶向序列(例如,与靶DNA内的CRISPR RNA识别序列互补的DNA靶向区段内的序列)的长度可为至少约12nt、至少约15nt、至少约18nt、至少约19nt、至少约20nt、至少约25nt、至少约30nt、至少约35nt、或至少约40nt。作为另一种选择,与靶DNA的靶序列互补的DNA靶向区段的DNA靶向序列的长度可为约12个核苷酸(nt)至约80nt、约12nt至约50nt、约12nt至约45nt、约12nt至约40nt、约12nt至约35nt、约12nt至约30nt、约12nt至约25nt、约12nt至约20nt、约12nt至约19nt、约19nt至约20nt、约19nt至约25nt、约19nt至约30nt、约19nt至约35nt、约19nt至约40nt、约19nt至约45nt、约19nt至约50nt、约19nt至约60nt、约20nt至约25nt、约20nt至约30nt、约20nt至约35nt、约20nt至约40nt、约20nt至约45nt、约20nt至约50nt、或约20nt至约60nt。与靶DNA的核苷酸序列(靶序列)互补的DNA靶向区段的核苷酸序列(DNA靶向序列)的长度可为至少约12nt。在一些情况下,DNA靶向序列的长度可为至少约20nt。
TracrRNA可为任何形式(例如,全长tracrRNA或有活性的部分tracrRNA)并具有不同长度。它们可包括初级转录物或加工形式。例如,tracrRNA(作为单向导RNA的一部分或作为属于双分子gRNA的一部分的单独分子)可包含以下部分或由以下部分组成:野生型tracrRNA序列的全部或一部分(例如,野生型tracrRNA序列的约或大于约20、26、32、45、48、54、63、67、85个或更多个核苷酸)。来自化脓性链球菌的野生型tracrRNA序列的示例包括171个核苷酸、89个核苷酸、75个核苷酸以及65个核苷酸的形式。参见例如Deltcheva et al.(2011)Nature 471:602-607(Deltcheva等人,2011年,《自然》,第471卷,第602-607页);WO 2014/093661,这些文献中的每一篇全文均以引用的方式并入本文。单向导RNA(sgRNA)内的tracrRNA的示例包括存在于+48、+54、+67和+85形式的sgRNA内的tracrRNA区段,其中“+n”表示野生型tracrRNA的至多+n核苷酸包含在sgRNA中。参见US 8,697,359,该专利全文以引用的方式并入本文。
DNA靶向序列与靶DNA内的CRISPR RNA识别序列之间的互补性百分比可为至少60%(例如,至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少97%、至少98%、至少99%或100%)。DNA靶向序列与靶DNA内的CRISPR RNA识别序列之间的互补性百分比,在靶DNA互补链的靶序列的七个邻接的5'最末端核苷酸内为100%。在某些实施例中,DNA靶向序列与靶DNA内的CRISPR RNA识别序列之间的互补性百分比在约20个邻接核苷酸内可为至少60%。例如,DNA靶向序列与靶DNA内的CRISPR RNA识别序列之间的互补性百分比,在靶DNA的互补链内的CRISPR RNA识别序列的5’最末端的十四个邻接核苷酸内为100%,并且在其余邻接核苷酸内低至0%。在这种情况下,DNA靶向序列可被视为14个核苷酸长。又如,DNA靶向序列与靶DNA内的CRISPR RNA识别序列之间的互补性百分比,在靶DNA的互补链内的CRISPR RNA识别序列的5’最末端的七个邻接核苷酸内为100%,并且在其余邻接核苷酸内低至0%。在这种情况下,DNA靶向序列可被视为7个核苷酸长。
核酸的互补性意指核酸的一条链中的核苷酸序列因其核碱基基团的取向而以氢键方式结合于相对核酸链上的另一个序列。互补碱基通常为,在DNA中:A与T及C与G,在RNA中:C与G及U与A。互补性可以是完全的或实质的/充分的。两个核酸之间的完全互补性意指这两个核酸可以形成双链体,其中双链体中的每个碱基按照沃森-克里克配对原则与互补碱基结合。“实质”或“充分”互补意指一条链中的序列不与相对链中的序列彻底和/或完全互补,但在一组杂交条件(例如,盐浓度和温度)中这两条链上的碱基之间发生充分键合而形成稳定的杂交复合物。可通过以下方式预测此类条件:使用序列和标准数学计算来预测杂交链的Tm,或使用常规方法凭经验确定Tm。Tm是指在两条核酸链之间形成的一群杂交复合物发生50%变性时的温度。在低于Tm的温度下,有利于杂交复合物的形成,而在高于Tm的温度下,有利于杂交复合物中的两条链的解链或分离。可在1M NaCl水溶液中对具有已知G+C含量的核酸估计Tm,例如使用Tm=81.5+0.41(%G+C),而其他已知的Tm计算法考虑了核酸结构特征。
“杂交条件”是指累积环境,其中一条核酸链通过互补链相互作用和氢键方式键合于第二核酸链,从而产生杂交复合物。此类条件包括含核酸的水溶液或有机溶液的化学组分及其浓度(例如,盐、螯合剂、甲酰胺)以及该混合物的温度。其他因素(例如,温育时间的长度或反应室尺寸)可对环境有影响(例如Sambrook et al.,Molecular Cloning,A Laboratory Manual,2.sup.nd ed.,pp.1.90-1.91,9.47-9.51,1 1.47-11.57(Cold Spring Harbor Laboratory Press,Cold Spring Harbor,N.Y.,1989)(Sambrook等人,《分子克隆实验指南》,第2版,第1.90-1.91、9.47-9.51、1 1.47-11.57节,冷泉港实验室出版社,美国纽约州冷泉港,1989年))。
杂交要求两个核酸包含互补序列,但允许碱基之间出现错配。适于两个核酸之间的杂交的条件取决于核酸的长度和互补程度,这些变量是本领域众所周知的。两个核苷酸序列之间的互补程度越大,具有这些序列的核酸的杂交体的解链温度(Tm)值就越大。对于具有短序列段互补性(例如,在35个或更少、30个或更少、25个或更少、22个或更少、20个或更少、或18个或更少核苷酸内的互补性)的核酸之间的杂交,错配的位置变得重要(参见Sambrook等人,出处同上,11.7-11.8)。通常,可杂交核酸的长度为至少约10个核苷酸。可杂交核酸的示例性最小长度为:至少约15个核苷酸、至少约20个核苷酸、至少约22个核苷酸、至少约25个核苷酸以及至少约30个核苷酸。此外,可视需要根据诸如互补区域的长度和互补程度等因素来调节温度和洗涤溶液盐浓度。
多核苷酸的序列不必与其靶核酸的序列100%互补,也能实现特异性杂交。此外,多核苷酸可在一个或多个区段内杂交,使得间插或相邻区段不参与杂交事件(例如,环结构或发夹结构)。多核苷酸(例如,gRNA)可与其靶向的靶核酸序列内的靶区域具有至少70%、至少80%、至少90%、至少95%、至少99%或100%序列互补性。例如,其中gRNA的20个核苷酸中有18个与靶区域互补并因此特异性杂交的gRNA将表示90%互补性。在该示例中,剩余的非互补核苷酸可以成簇或散布在互补核苷酸内并且无需彼此邻接或与互补核苷酸邻接。通常可使用以下程序来确定核酸内的核酸序列的特定序列段之间的互补性百分比:使用本领域已知的BLAST程序(基本局部比对搜索工具)和PowerBLAST程序(Altschul et al.,J.Mol.Biol.,1990,215,403-410(Altschul等人,《分子生物学杂志》,1990年,第215卷,第403-410页);Zhang and Madden,Genome Res.,1997,7,649-656(Zhang和Madden,《基因组研究》,1997年,第7卷,第649-656页))或使用Gap程序(威斯康星序列分析软件包,适用于Unix的版本8,遗传学计算机组,美国威斯康星州麦迪逊的大学研究园(Wisconsin Sequence Analysis Package,Version 8for Unix,Genetics Computer Group,University Research Park,Madison Wis.)),这些程序使用默认设置,这使用Smith和Waterman的算法(Adv.Appl.Math.,1981,2,482-489(《应用数学进展》,1981年,第2卷,第482-489页))。
个体gRNA的蛋白结合区段与Cas蛋白相互作用。个体gRNA经由DNA靶向区段将结合的多肽引导至靶DNA内的特异性核苷酸序列。个体gRNA的蛋白结合区段可包含彼此互补的两段核苷酸。蛋白结合区段的互补核苷酸杂交而形成双链RNA双链体(dsRNA)。个体gRNA的蛋白结合区段与Cas蛋白相互作用,并且gRNA经由DNA靶向区段将结合的Cas蛋白引导至靶DNA内的特异性核苷酸序列。
在某些实施例中,如本文所述的gRNA包含两个单独的RNA分子。个体gRNA的两个RNA分子各自包含一段核苷酸,它们彼此互补,使得这两个RNA分子的互补核苷酸杂交而形成蛋白结合区段的双链RNA双链体(例如,发夹)。个体gRNA可包含任何对应的crRNA和tracrRNA对。在本文所述的方法中,gRNA可用作crRNA和tracrRNA的复合物(例如,gRNA-Cas复合物),或crRNA和对应的tracrRNA可分别进行递送。例如,如果多个gRNA用于切割反应,则可将对每个靶位点具有特异性的单独crRNA和可与每个crRNA复合的标准tracrRNA分别递送。在这种方法中,crRNA可与标准tracrRNA复合以将Cas蛋白引导至靶位点。
向导RNA可包括提供额外所需特征(例如,经修饰或调控的稳定性;亚细胞靶向;用荧光标记物示踪;蛋白或蛋白复合物的结合位点;等等)的修饰或序列。此类修饰的非限制性示例包括例如5'帽(例如,7-甲基鸟苷酸帽(m7G));3'多聚腺苷酸化尾(即,3'多聚(A)尾);核糖开关序列(例如,以实现经调控的稳定性和/或经调控的蛋白和/或蛋白复合物可及性);稳定性控制序列;形成dsRNA双链体(即,发夹)的序列;使RNA靶向亚细胞位置(例如,细胞核、线粒体、叶绿体等)的修饰或序列;提供示踪的修饰或序列(例如,与荧光分子的直接缀合、与有利于荧光检测的部分的缀合、允许荧光检测的序列等);为蛋白质(例如,作用于DNA的蛋白质,包括转录激活因子、转录阻遏物、DNA甲基转移酶、DNA去甲基化酶、组蛋白乙酰转移酶、组蛋白去乙酰化酶等)提供结合位点的修饰或序列;以及它们的组合。
向导RNA可以任何形式提供。例如,gRNA可以RNA的形式(作为两分子(单独的crRNA和tracrRNA)或作为一分子(sgRNA))提供,并任选地以与Cas蛋白的复合物形式提供。gRNA也可以编码RNA的DNA的形式提供。编码gRNA的DNA可编码单个RNA分子(sgRNA)或单独的RNA分子(例如,单独的crRNA和tracrRNA)。在后一情况下,编码gRNA的DNA可作为分别编码crRNA和tracrRNA的单独DNA分子提供。
编码gRNA的DNA可稳定整合在细胞的基因组中,并有效连接至在细胞中有活性的启动子。作为另一种选择,编码gRNA的DNA可有效连接至表达构建体中的启动子。例如,编码gRNA的DNA可位于包含核酸插入序列的靶向载体和/或包含编码Cas蛋白的核酸的载体中,或其可位于相对于包含核酸插入序列的靶向载体而言单独的和/或相对于包含编码Cas蛋白的核酸的载体而言单独的载体或质粒中。此类启动子可例如在大鼠、真核生物、哺乳动物、非人哺乳动物、人、啮齿动物、小鼠或仓鼠多能细胞中有活性。此类启动子可为例如条件启动子、诱导型启动子、组成型启动子或组织特异性启动子。在一些情况下,所述启动子为RNA聚合酶III启动子,诸如人U6启动子、大鼠U6聚合酶III启动子、或小鼠U6聚合酶III启动子。其他启动子的示例在本文别处有描述。当将编码gRNA的DNA引入细胞中时,gRNA可在细胞中瞬时地、条件性地或组成型地表达。
作为另一种选择,可通过各种其他方法制备gRNA。例如,可通过采用例如T7RNA聚合酶的体外转录来制备gRNA(参见例如WO 2014/089290和WO 2014/065596)。向导RNA也可为通过化学合成制备的合成产生的分子。
C.CRISPR RNA识别序列
术语“CRISPR RNA识别序列”包括靶DNA中存在的这样的核酸序列,只要存在充分的结合条件,gRNA的DNA靶向区段就将与该核酸序列结合。例如,CRISPR RNA识别序列包括向导RNA被设计成与之具有互补性的序列,其中CRISPR RNA识别序列与DNA靶向序列之间的杂交促进CRISPR复合物的形成。不必要求完全互补性,只要存在足以引起杂交并促进CRISPR复合物形成的互补性即可。CRISPR RNA识别序列还包括下文更详细说明的Cas蛋白的切割位点。CRISPR RNA识别序列可包含任何多核苷酸,所述多核苷酸可位于例如细胞的细胞核或细胞质中,或位于细胞的细胞器如线粒体或叶绿体内。
靶DNA内的CRISPR RNA识别序列可被Cas蛋白或gRNA所靶向(即,与之结合、或与之杂交、或与之互补)。合适的DNA/RNA结合条件包括通常存在于细胞中的生理条件。其他合适的DNA/RNA结合条件(例如,无细胞系统中的条件)是本领域已知的(参见例如Molecular Cloning:A Laboratory Manual,3rd Ed.(Sambrook et al.,Harbor Laboratory Press 2001)(《分子克隆实验指南》,第3版,Sambrook等人,冷泉港实验室出版社,2001年))。与Cas蛋白或gRNA互补并杂交的靶DNA链可称为“互补链”,并且与“互补链”互补(并因此不与Cas蛋白或gRNA互补)的靶DNA链可称为“非互补链”或“模板链”。
Cas蛋白可在将与gRNA的DNA靶向区段结合的靶DNA中存在的核酸序列之内或之外的位点处切割核酸。“切割位点”包括Cas蛋白产生单链断裂或双链断裂的核酸位置。例如,CRISPR复合物(包含与CRISPR RNA识别序列杂交并与Cas蛋白复合的gRNA)的形成可导致将与gRNA的DNA靶向区段结合的靶DNA中存在的核酸序列之中或附近(例如,在相距1、2、3、4、5、6、7、8、9、10、20、50个或更多个碱基对内)的一条或两条链切割。如果切割位点位于将与gRNA的DNA靶向区段结合的核酸序列之外,则切割位点仍被视为在“CRISPR RNA识别序列”内。切割位点可位于核酸的仅一条链上或两条链上。切割位点可位于核酸的两条链上的相同位置处(产生平头末端),或可位于每条链上的不同位点处(产生交错末端)。可例如通过使用两种Cas蛋白来产生交错末端,每种Cas蛋白在每条链上的不同切割位点处产生单链断裂,从而产生双链断裂。例如,第一切口酶可在双链DNA(dsDNA)的第一链上形成单链断裂,并且第二切口酶可在dsDNA的第二链上形成单链断裂,使得形成悬垂序列。在一些情况下,第一链上的切口酶的CRISPR RNA识别序列与第二链上的切口酶的CRISPR RNA识别序列相隔至少2、3、4、5、6、7、8、9、10、15、20、25、30、40、50、75、100、250、500或1,000个碱基对。
Cas9对靶DNA的位点特异性切割可在由以下两者决定的位置处发生:(i)gRNA与靶DNA之间的碱基配对互补性,以及(ii)靶DNA中的短基序,称为前间区序列邻近基序(PAM)。PAM可侧接CRISPR RNA识别序列。任选地,CRISPR RNA识别序列可被PAM侧接。例如,Cas9的切割位点可为PAM序列上游或下游的约1至约10或者约2至约5个碱基对(例如,3个碱基对)。在一些情况下(例如,当使用来自化脓性链球菌的Cas9或密切相关的Cas9时),非互补链的PAM序列可为5'-N1GG-3',其中N1为任何DNA核苷酸并且紧邻靶DNA的非互补链的CRISPR RNA识别序列的3'。因此,互补链的PAM序列将为5'-CC N2-3',其中N2为任何DNA核苷酸并且紧邻靶DNA的互补链的CRISPR RNA识别序列的5'。在一些此类情况下,N1和N2可为互补的,并且N1-N2碱基对可为任何碱基对(例如,N1=C且N2=G;N1=G且N2=C;N1=A且N2=T,N1=T且N2=A)。
CRISPR RNA识别序列的示例包括与gRNA的DNA靶向区段互补的DNA序列、或除PAM序列之外的这种DNA序列。例如,靶基序可为紧接在Cas蛋白所识别的NGG基序前面的20个核苷酸的DNA序列,诸如GN19NGG(SEQ ID NO:8)或N20NGG(SEQ ID NO:24)(参见例如WO 2014/165825)。5’端的鸟嘌呤可有利于RNA聚合酶在细胞中进行转录。CRISPR RNA识别序列的其他示例可包括5’端的两个鸟嘌呤核苷酸(例如,GGN20NGG;SEQ ID NO:25),以有利于T7聚合酶在体外进行有效转录。参见例如WO 2014/065596。其他CRISPR RNA识别序列可具有4-22个核苷酸长的SEQ ID NO:8、24和25,包括5’G或GG和3’GG或NGG。另外一些CRISPR RNA识别序列可具有14至20个核苷酸长的SEQ ID NO:8、24和25。
CRISPR RNA识别序列可为细胞内源或外源的任何核酸序列。CRISPR RNA识别序列可为编码基因产物(例如,蛋白)的序列或非编码序列(例如,调控序列)或者可包括两者。
在一个实施例中,Cas蛋白为I型Cas蛋白。在一个实施例中,Cas蛋白为II型Cas蛋白。在一个实施例中,II型Cas蛋白为Cas9。在一个实施例中,第一核酸序列编码人密码子优化的Cas蛋白。
在一个实施例中,gRNA包含编码crRNA和tracrRNA的核酸序列。在具体实施例中,Cas蛋白为Cas9。在一些实施例中,gRNA包含(a)核酸序列5’-GUUUUAGAGCUAGAAAUAGCAAGUUAAAAUAAGGCUAGU CCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGCUUUU-3’(SEQ ID NO:1)的嵌合RNA;或(b)核酸序列5’-GUUUUAGAGCUAGAAAU AGCAAGUUAAAAUAAGGCUAGUCCG-3’(SEQ ID NO:2)的嵌合RNA。在另一个实施例中,crRNA包含5’-GUUUUAGAGCUAGAAAUAGCAA GUUAAAAU-3’(SEQ ID NO:3);5’-GUUUUAGAGCUAGAAAU AGCAAGUUAAAAUAAG(SEQ ID NO:4);或5’-GAGUCCGAGCAGA AGAAGAAGUUUUA-3’(SEQ ID NO:5)。在另外其他实施例中,tracrRNA包含5’-AAGGCUAGUCCG-3’(SEQ ID NO:6)或5’-AAGGCUAGUCCGU UAUCAACUUGAAAAAGUGGCACCGAGUCGGUGCUUUU-3’(SEQ ID NO:7)。
V.多核苷酸的组装
本文所公开的方法可在一定条件下组装至少两个核酸,这些条件能有效接合DNA分子,从而形成基本上完整或无缝的双链DNA分子。可根据本文所公开的方法组装具有重叠序列的任何目标核酸。例如,可组装具有重叠序列的任何目标DNA分子,包括天然存在的DNA、克隆的DNA分子、合成产生的DNA等。可根据需要,使用本发明的方法将接合的DNA分子克隆(例如,插入)到载体中。组装两个核酸包括任何接合两个核酸的链的方法。例如,组装包括接合经酶切的核酸,使得来自每个核酸的链退火到另一条链上;以及延伸,其中每条链充当另一条链延伸的模板。
在一些实施例中,将核酸与接合寡核苷酸组装在一起,使得每个核酸组装到接合寡核苷酸,而非直接组装在一起。利用接合寡核苷酸进行的组装可将核酸碱基定位在正在组装的核酸之间,这些核酸不是待组装的核酸的一部分,而是接合寡核苷酸的一部分。因此,即使在核酸之间留下额外碱基,也可成功组装这些核酸。作为另一种选择,可使用接合寡核苷酸进行无缝组装,其中在待组装的核酸之间没有留下额外碱基。
在一些实施例中,可通过用Cas蛋白、限制性内切酶(限制性核酸内切酶)(例如,本文别处提供的各种限制性核酸内切酶中的任一种)、大范围核酸酶(例如,本文别处提供的各种大范围核酸酶中的任一种)或它们的任何组合进行切割,而使这些核酸作好组装的准备。例如,可用Cas蛋白切割待组装的核酸之一,并且可用Cas蛋白、限制性内切酶、大范围核酸酶或它们的任何组合切割待组装的另一个核酸。在用核酸酶切割后,可将经酶切的核酸直接组装到具有重叠末端序列的另一个经酶切的核酸,或组装到未经酶切但具有重叠末端序列的核酸。也可通过使用接合寡核苷酸将经酶切的核酸组装到另一个核酸。
在采用核酸酶试剂(例如,Cas蛋白)在两个核酸分子之间产生重叠末端序列的实施例中,可使用快速组合方法来组装经酶切的核酸。例如,可将具有重叠末端的第一核酸和第二核酸与连接酶、核酸外切酶、DNA聚合酶和核苷酸混合,并在恒定温度下(诸如在50℃下)温育。具体地讲,可使用T5核酸外切酶从dsDNA的5’端去除核苷酸,从而产生互补悬垂序列。然后在50℃下,使互补单链DNA悬垂序列退火,使用DNA聚合酶进行缺口填补,并且使用Taq DNA连接酶密封所得的切口。因此,可在一步等温反应中将共用重叠末端序列的两个核酸接合成共价密封的分子。参见例如Gibson,et al.(2009)Nature Methods 6(5):343-345(Gibson等人,2009年,《自然方法》,第6卷,第5期,第343-345页),该文献全文以引用的方式并入本文。在一些实施例中,使用蛋白酶K或苯酚/氯仿/异戊醇(PCI)纯化,从反应混合物中去除核酸酶试剂(例如,Cas蛋白)。在一些实施例中,可通过基于硅胶的柱纯化,从反应混合物中去除核酸酶试剂(例如,Cas蛋白)。
在某些实施例中,本文所公开的方法将载体与线性多核苷酸组装在一起。在其他实施例中,本文所公开的方法组装至少两个载体,诸如两个BAC载体。术语“BAC载体”包括任何细菌人工染色体。在具体实施例中,对BAC进行修饰以包含具有这样的核苷酸序列的区域,该核苷酸序列与线性核酸或另一个载体(例如,另一个BAC)的区域的核苷酸序列重叠。
当相应末端彼此互补时,第一单链核酸和第二单链核酸具有重叠末端。当第一核酸的一条链的5’端与第二核酸的一条链的3’端互补时,第一双链核酸和第二双链核酸具有重叠末端,反之亦然。例如,对于双链重叠末端序列而言,一个核酸的链可与另一个核酸的对应链具有至少80%、至少85%、至少90%、至少95%、至少96%、至少97%、至少98%、至少99%或100%同一性。在本文所公开的方法中,待组装的dsDNA分子的一条链的5’端与另一个dsDNA分子的一条链的3’端共用重叠末端序列。术语“重叠末端序列”包括dsDNA分子的两条链。因此,当重叠序列的互补区域存在于来自待组装的两个多核苷酸的5’和3’端的单链悬垂序列中时,来自重叠区域的一条链可与其互补链特异性杂交。在一些实施例中,使用核酸外切酶从5’或3’端去除核苷酸而形成悬垂末端序列。在一些实施例中,直到用Cas蛋白进行酶切后,第一核酸和/或第二核酸的重叠区域才存在于5’或3’端上。也就是说,重叠区域可以是内部区域,在用Cas蛋白酶切包含内部重叠区域的核酸后,该内部区域后续被转变为重叠末端序列。Cas蛋白可在重叠区域之内或重叠区域之外的靶位点(例如,切割位点)处切割。
重叠区域的长度优选地足够长,使得该区域在正组装的任一核酸内仅出现一次。这样,可防止其他多核苷酸与末端序列一起退火,并且该组装对靶核酸可具有特异性。重叠区域的长度可从最少约10个碱基对(bp)至约300bp或更长。一般来讲,优选的是,重叠区的长度小于或等于待组合的多核苷酸的大致大小,但不少于约10bp且不超过约1000bp。对于2个或3个多核苷酸的接合而言,约20-30bp重叠区可为足够的。对于超过10个片段而言,优选的重叠区为约80bp至约300bp。在一个实施例中,重叠区域具有使其易于通过合成方法生成的长度,例如约40bp。在具体实施例中,重叠区域的长度可为约20-200bp。重叠区可为约10、20、30、40、50、60、70、80、90、100、150、200、250、300、350、400、450、500、550、600、650、700、750、800、850、900、950或1,000bp长。在一些实施例中,重叠区域的长度为20–200bp。在本文所公开的方法的具体实施例中,可组装至少两个多核苷酸,其中所述多核苷酸中的至少一者上的重叠区域通过与核酸酶试剂(例如,gRNA-Cas复合物)接触而生成。例如,第一多核苷酸的核酸内切酶酶切可形成与第二多核苷酸的末端序列重叠的序列,其中重叠末端序列随后进行组装。
在本文所公开的方法中,可使重叠序列与核酸外切酶接触,以暴露重叠序列之间的互补序列(例如,互补单链序列)。在一定条件下执行核酸外切酶酶切,这些条件能有效去除(“回噬(chew back)”)足够数量的核苷酸,从而使暴露的互补性单链区域可以进行特异性退火。一般来讲,重叠区域的一部分或整个重叠区域被回噬,留下包含重叠区域的一部分或整个重叠区域的悬垂序列。在一些方法中,可在不存在dNTP的情况下由聚合酶(例如,T5DNA聚合酶)执行核酸外切酶酶切,而在其他方法中,可在存在dNTP的情况下由缺乏聚合酶活性的核酸外切酶(例如,核酸外切酶III)执行核酸外切酶酶切。
在本文所公开的方法中,可使用多种5'至3'双链特异性脱氧核糖核酸外切酶中的任一种对核酸的末端进行回噬。术语“5'核酸外切酶”在本文中有时用来指5'至3'脱氧核糖核酸外切酶。如本文所用,“非进行性”核酸外切酶是在每个DNA结合事件期间降解有限数量的(例如,仅一些)核苷酸的核酸外切酶。用5'核酸外切酶进行的酶切在DNA分子中产生了3'单链悬垂序列。所希望的5'核酸外切酶的其他特性包括其缺少3'核酸外切酶活性,其生成5'磷酸末端,以及其引发从5'-磷酸化和非磷酸化末端的降解。还希望该酶可引发从分子5'端的酶切,而不论其是平末端,还是具有较小的5'或3'凹缺末端。合适的核酸外切酶对于技术人员将是显而易见的。这些酶包括例如噬菌体T5核酸外切酶(噬菌体T5基因D15产物)、噬菌体λ核酸外切酶、Rac原噬菌体的RecE、来自大肠杆菌(E.coli)的核酸外切酶VIII、噬菌体T7核酸外切酶(噬菌体T7基因6产物)或参与同源重组反应的多种5'核酸外切酶中的任一种。在本发明的一个实施例中,核酸外切酶是T5核酸外切酶或λ核酸外切酶。在另一个实施例中,核酸外切酶是T5核酸外切酶。在另一个实施例中,核酸外切酶不是噬菌体T7核酸外切酶。用于制备和使用在本发明方法中所采用的核酸外切酶和其他酶的方法是常规的;并且许多方法可得自商业来源,诸如USB Corporation,26111 Miles Road,Cleveland,Ohio 44128(美国俄亥俄州克里夫兰英里路26111号的USB公司,邮编44128),或New England Biolabs,Inc.(NEB),240County Road,Ipswich,Mass.01938-2723(美国马萨诸塞州伊普斯威奇县公路240号的新英格兰生物实验室公司(NEB),邮编01938-2723)。
具体而言,在重叠区域非常长的实施例中,可能仅需要回噬该区域的一部分(例如,超过该重叠区域的一半),前提条件是由此生成的单链悬垂序列具有足够的长度和碱基含量,可以在反应条件下特异性地退火。术语“特异性地退火”包括这样的情况,其中特定的一对单链悬垂序列将优先地(或排他地)彼此退火到一起,而非退火到反应混合物中存在的其他单链悬垂序列(例如,非互补悬垂序列)。所谓“优先地”意指至少约95%的悬垂序列将退火到互补悬垂序列。技术人员可易于确定用于在一组给定的反应条件下实现目标序列的特异性退火的最佳长度。一般来讲,同源重叠区域(单链悬垂序列或其互补序列)包含相同序列。然而,可使用部分相同的序列,前提条件是单链悬垂序列可在反应条件下特异性地退火。
在某些实施例中,核酸酶试剂(例如,Cas蛋白)可在靶位点处形成单链断裂(即,“切口”)而不切割dsDNA的两条链。“切口酶”包括在dsDNA中形成切口的核酸酶试剂(例如,Cas蛋白)。这样,对dsDNA每条链上的靶位点具有特异性的两种单独核酸酶试剂(例如,Cas蛋白)(例如,切口酶)可形成与另一个核酸上的悬垂序列或相同核酸上的单独区域互补的悬垂序列。通过使核酸与对dsDNA两条链上的靶位点具有特异性的两种切口酶接触而形成的悬垂末端可以是5’或3’悬垂末端。例如,第一切口酶可在dsDNA的第一链上形成单链断裂,而第二切口酶可在dsDNA的第二链上形成单链断裂,使得形成悬垂序列。可选择每种形成单链断裂的切口酶的靶位点,使得所形成的悬垂末端序列与第二核酸上的悬垂末端序列互补。因此,可通过本文所公开的方法使第一核酸和第二核酸的互补悬垂末端退火。在一些实施例中,切口酶在第一链上的靶位点不同于切口酶在第二链上的靶位点。dsDNA的单个链上的不同靶位点得到由至少2、3、4、5、6、7、8、9、10、15、20、25、30、40、50、75、100、250、500或1,000个碱基对隔开的单链断裂。
在某些实施例中,还使第二核酸与在第二核酸上的第一靶位点处形成切口的第一切口酶及在第二核酸分子上的第二靶位点处形成切口的切口酶接触。由第二核酸上的两个不同位点处的切口形成的悬垂末端序列,可与由第一核酸上的两个不同位点处的切口形成的悬垂末端序列互补,以使得互补悬垂末端序列发生退火。
在一些实施例中,目标基因的核酸序列跨越两个或更多个BAC。在此类情况下,使用本文所提供的方法时,专门设计的核酸酶试剂可在所需位置处切开所述两个或更多个BAC,所得的核酸片段接合在一起而形成目标基因的序列。
在一些实施例中,由第一核酸的两条链上的不同靶位点处的切口形成的悬垂末端,不与由第二核酸的两条链上的不同靶位点处的切口形成的悬垂末端互补。在其他实施例中,待组装的核酸不具有互补末端,使得需要另外的核酸来组装非互补末端。可使用接合寡核苷酸来接合两个核酸的非互补末端。“接合寡核苷酸”包括互补臂,所述互补臂所包含的多核苷酸或核酸具有与另一不同的多核苷酸或核酸的末端互补的序列。在一些实施例中,接合寡核苷酸具有在5’端与第一核酸互补的臂、中心部分(间区序列)以及在3’端与第二核酸互补的臂。因此,可通过以下方式组装具有彼此不互补的末端序列的核酸:在核酸外切酶处理后,使每个核酸退火到相同的接合寡核苷酸。在具体实施例中,接合寡核苷酸具有与第一经酶切的核酸的5’或3’末端序列互补的第一臂,以及与第二经酶切的核酸的5’或3’序列互补的第二臂。接合寡核苷酸可接合平头的或具有5’或3’悬垂序列的非互补末端序列。
经核酸外切酶处理后,接合寡核苷酸的互补臂序列的长度应当足以退火到待组装的核酸。例如,接合寡核苷酸的互补臂序列的长度可为至少约10、20、30、35、40、45、50、55、60、65、70、75、80、90、100、110、120、130、140、150bp或更长。在具体实施例中,互补臂为15-120bp、20-100bp、30-90bp、30-60bp或20-80bp。在一个具体实施例中,接合寡核苷酸的互补臂序列的长度为40bp。接合寡核苷酸的每个互补臂可具有不同长度。位于与待组装的核酸互补的末端序列之间的、接合寡核苷酸的间区序列可为至少约20bp、30bp、35bp、40bp、45bp、50bp、55bp、60bp、65bp、70bp、75bp、80bp、90bp、100bp、250bp、500bp、750bp、1000bp、2000bp、3000bp、4000bp、5000bp、8000bp、10kb、15kb、20kb或更长。例如,接合寡核苷酸的间区序列可包括BAC载体或LTVEC。在一些实施例中,接合寡核苷酸的间区序列可被设计成具有专用于检测的序列或适用于PCR的序列,以确认成功组装。在一些实施例中,接合寡核苷酸的间区序列可被设计成引入一个或多个限制性内切酶位点。在一些实施例中,接合寡核苷酸的间区序列可被设计成引入药物抗性基因或报告基因。在其他实施例中,间区序列可包含来自待组装的核酸的末端部分的至少20bp,以无缝地组装核酸。例如,为了实现无缝组装,间区序列可为约45bp。
在一些实施例中,核酸与接合寡核苷酸的摩尔比可为约1:1至约1:200。在一些实施例中,核酸与接合寡核苷酸的摩尔比为约1:1、1:2、1:3、1:4、1:5、1:6、1:7、1:8、1:9、1:10、1:11、1:12、1:13、1:14、1:15、1:16、1:17、1:18、1:19、1:20、1:30、1:40、1:50、1:60、1:70、1:80、1:90、1:100、1:120、1:140、1:160、1:180或1:200。在具体实施例中,核酸与接合寡核苷酸的摩尔比可为约1:6至约1:20。在一个实施例中,摩尔比为约1:6。在另一个实施例中,摩尔比为约1:20。
在具体实施例中,使用接合寡核苷酸无缝地组装至少两个核酸。“无缝”组装是指两个核酸的组装,其中待组装的核酸的相邻末端之间不存在间插核酸碱基。例如,无缝组装的核酸不存在不属于待组装的核酸的一部分的核酸碱基。为了无缝地组装两个核酸,接合寡核苷酸的间区序列应包括与待组装的第一核酸或第二核酸的末端部分相同的核酸序列。该末端部分应当先从核酸去除,再与接合寡核苷酸组装在一起。例如,可通过核酸酶试剂(例如,gRNA-Cas复合物)从核酸的末端切割至少20bp的末端部分,诸如从核酸的末端切割至少40bp或至少45bp的末端部分。作为另一种选择,可通过核酸酶试剂(例如,gRNA-Cas复合物)从待组装的核酸的末端切割至少2、至少4、至少6、至少8、至少10、至少12、至少15、至少20、至少25、至少30、至少35、至少37、至少40、至少42、至少45、至少48、至少50、至少55、至少60、至少65、至少70、至少80、至少100、至少110、至少120、至少130、至少140、至少150bp的末端部分。
在一个实施例中,接合寡核苷酸从5’端到3’端可包含:5’核酸的约15-120bp重叠区、5’核酸的3’端区域的约20-50bp以及3’核酸的约15-120bp重叠区。在一个实施例中,接合寡核苷酸从5’端到3’端可包含:5’核酸的约15-120bp重叠区、3’核酸的5’端区域的约20-50bp以及3’核酸的约15-120bp重叠区。因此,当将接合寡核苷酸组装到第一核酸和第二核酸时,来自接合寡核苷酸的间区序列在组装之前重建从核酸去除的部分。参见图5和图6。术语“重建”包括核酸的被切割的末端部分的替换,从而在组装到接合寡核苷酸时提供完全组装的核酸。例如,重建经切割的核酸可将核酸的经切割的部分替换为接合寡核苷酸的间区序列中所含的、具有与经切割的部分相同的序列的核酸。
接合寡核苷酸可以同时地或依次地组装到第一核酸分子和第二核酸分子。当同时组装时,可使接合寡核苷酸在同一反应混合物中与第一核酸和第二核酸接触,使得所得经组装的核酸包含第一核酸、接合寡核苷酸和第二核酸。当依次组装时,使接合寡核苷酸在组装反应中与第一核酸接触,所产生经组装的核酸包含组装到接合寡核苷酸但没有组装到第二核酸的第一核酸。然后使这样的经组装的核酸在单独的组装反应中与第二核酸接触,所产生经组装的核酸包含第一核酸、接合寡核苷酸和第二核酸。在其他实施例中,使接合寡核苷酸在组装反应中与第二核酸接触,所产生经组装的核酸包含组装到接合寡核苷酸但没有组装到第一核酸的第二核酸。然后使这样的经组装的核酸在单独的组装反应中与第一核酸接触,所产生经组装的核酸包含第一核酸、接合寡核苷酸和第二核酸。
可在本文方法中使用任何数量的接合寡核苷酸来组装核酸分子。例如,可使用1个接合寡核苷酸来组装2个核酸分子,可使用2个接合寡核苷酸来组装3个核酸分子,可使用3个接合寡核苷酸来组装4个核酸分子,可使用4个接合寡核苷酸来组装5个核酸分子,或可使用5个接合寡核苷酸来组装6个核酸分子。接合寡核苷酸的数量可为1、2、3、4、5、6、7、8、9、10个或更多个,具体取决于待组装的核酸分子的数量。
在一些实施例中,接合寡核苷酸包含gBlock DNA。“gBlock”为线性双链DNA片段。gBlock可为约50bp至约2000bp。gBlock可为约50bp至约100bp、约100bp至约200bp、约200bp至约300bp、约300bp至约400bp、约400bp至约500bp、约500bp至约600bp、约600bp至约800bp、约800bp至约1000bp、约1000bp至约1250bp、约1250bp至约1500bp、约1500bp至约1750bp、或约1750bp至约2000bp。
可例如通过本文别处所述的PCR测定法(例如,实例10),筛选用gBlock对两个或更多个核酸进行的组装。在一些情况下,gBlock不包含选择盒。这种方法使两个或更多个核酸分子可以快速接合,可通过简单的PCR测定法对该接合进行筛选。gBlock可包含任何目标核酸序列。在一些情况下,gBlock可包含核酸酶试剂的靶位点,或本文所提供的各种大范围核酸酶或限制性内切酶中的任一者的靶位点。在其他实施例中,gBlock可包含选择盒。在一些实施例中,gBlock包含目标DNA序列。在一个实施例中,gBlock包含人DNA序列。
待组装的核酸,或各种接合寡核苷酸中的任一者,还可包含选择盒或报告基因。选择盒可包含编码选择标记的核酸序列,其中所述核酸序列有效连接至启动子。所述启动子可在目标原核细胞中有活性和/或在目标真核细胞中有活性。此类启动子可为诱导型启动子、对于报告基因或细胞为内源的启动子、对于报告基因或细胞为异源的启动子、细胞特异性启动子、组织特异性启动子或发育阶段特异性启动子。在一个实施例中,选择标记选自新霉素磷酸转移酶(neor)、潮霉素B磷酸转移酶(hygr)、嘌呤霉素-N-乙酰转移酶(puror)、杀稻瘟菌素S脱氨酶(bsrr)、黄嘌呤/鸟嘌呤磷酸核糖转移酶(gpt)和单纯性疱疹病毒胸苷激酶(HSV-k)、以及它们的组合。靶向载体的选择标记可被上游同源臂和下游同源臂侧接,或可存在于同源臂的5’或3’。
在一个实施例中,待组装的核酸,或各种接合寡核苷酸中的任一者,包含有效连接至启动子的报告基因,其中所述报告基因编码报告蛋白,所述报告蛋白选自LacZ、mPlum、mCherry、tdTomato、mStrawberry、J-Red、DsRed、mOrange、mKO、mCitrine、Venus、YPet、增强型黄色荧光蛋白(eYFP)、Emerald、增强型绿色荧光蛋白(EGFP)、CyPet、青色荧光蛋白(CFP)、Cerulean、T-Sapphire、荧光素酶、碱性磷酸酶以及它们的组合。此类报告基因可有效连接至在细胞中有活性的启动子。此类启动子可为诱导型启动子、对于报告基因或细胞为内源的启动子、对于报告基因或细胞为异源的启动子、细胞特异性启动子、组织特异性启动子或发育阶段特异性启动子。
在使单链DNA(例如,当待接合的DNA分子是dsDNA时通过核酸外切酶的作用而产生的悬垂序列,或通过在每条链上的不同靶位点处形成切口而产生的悬垂序列)退火后,用合适的非链置换DNA聚合酶填补核酸外切酶所留下的单链缺口,并且用连接酶密封由此形成的切口。如本文所用,“非链置换DNA聚合酶”是这样的DNA聚合酶,其在复制dsDNA分子过程中若遇到位于其路径中的DNA链则会终止DNA的合成,或在复制dsDNA分子过程中会降解遇到的DNA链,与此同时填补由此形成的缺口,从而生成“移动切口”(切口平移)。
在一些实施例中,重叠末端序列在重叠区域之间具有足够的互补性,以使每个多核苷酸的单链互补末端退火。在第一多核苷酸的单链退火到第二多核苷酸的互补链后,可基于第二多核苷酸链的模板来延伸第一多核苷酸的3’端,并且可基于第一多核苷酸链的模板来延伸第二多核苷酸链的3’端。通过延伸每个多核苷酸的互补3’端,可以组装多核苷酸。在组装后,可通过连接反应,密封来自一个片段的一条链的经延伸的3’端与来自另一个片段的一条链的相邻5’端之间的切口。更具体地讲,将第一多核苷酸的经延伸的3’端的羟基连接到第二多核苷酸的5’端的磷酸基团,并且将第二多核苷酸的经延伸的3’端的羟基连接到第一多核苷酸的5’端的磷酸基团。
该连接反应可使用多种合适的耐热DNA连接酶中的任一种执行。合适的连接酶包括例如Taq连接酶、Ampligase耐热DNA连接酶(Epicentre生物技术公司(Epicentre Biotechnologies))、美国专利No.6,576,453中所公开的耐热连接酶、得自百奥尼公司(Bioneer,Inc.)的耐热Tfi DNA连接酶。
反应混合物中合适量的群集剂(诸如PEG)促使、增强或有利于分子群集。不希望受任何特定机制的约束,已表明群集剂促使分子群集并结合和束缚溶液中的水,从而使溶液的组分彼此更紧密接触。例如,待重组的DNA分子可更紧密靠近;从而有利于单链悬垂序列的退火。另外,已表明酶可与其DNA底物更紧密接触,并且可通过去除水分子加以稳定。多种合适的群集剂对于技术人员将是显而易见的。这些群集剂包括多种熟知的大分子,诸如聚合物如聚乙二醇(PEG);聚蔗糖(Ficoll),诸如聚蔗糖70;葡聚糖,诸如葡聚糖70;等等。本申请的许多讨论涉及的是PEG。然而,该讨论意在也适用于其他合适的群集剂。技术人员会了解如何在该方法中实施常规变化以适应其他群集剂的使用。
反应混合物中合适量的群集剂(诸如PEG)促使、增强或有利于分子群集。例如,群集剂可帮助待重组的DNA分子更紧密靠近;这因此有利于单链悬垂序列的退火。另外,已表明酶可与其DNA底物更紧密接触,并且可通过去除水分子加以稳定。多种合适的群集剂对于技术人员将是显而易见的。这些群集剂包括多种熟知的大分子,诸如聚合物如聚乙二醇(PEG);聚蔗糖,诸如聚蔗糖70;葡聚糖,诸如葡聚糖70;等等。一般来讲,当使用PEG时,约5%(重量/体积)的浓度是最佳的。然而,PEG的量可例如在约3至约7%的范围内。可使用任何合适大小的PEG,例如在约PEG-200(例如,PEG-4000、PEG-6000或PEG-8000)至约PEG-20,000或甚至更高的范围内。在本文的实例中,使用了PEG-8000。群集剂除了增强退火反应之外,还增强连接反应。
组装反应混合物中存在的反应组分(诸如盐、缓冲液、合适的能量源(诸如ATP或NAD)、反应混合物的pH等)可能对于单种酶(核酸外切酶、聚合酶和连接酶)并非最佳;相反,它们充当对于整组反应有效的折衷方案。例如,由本发明人确定的一种合适缓冲体系(本文有时称为ISO(等温)缓冲液)通常包含0.1M Tris-Cl pH 7.5;10mM MgCl.sub.2;各0.2mM的dGTP、dATP、dTTP和dCTP;10mM DTT;5%PEG-8000;以及1mM NAD。
在本文所公开的方法中,使至少两个核酸在一定条件下与Cas蛋白和其他酶接触,这些条件能有效组装核酸,以形成其中保留了单个拷贝的重叠区域的经组装的双链DNA分子。可使用所述方法接合任何目标DNA分子,包括天然存在的DNA、克隆的DNA分子、合成产生的DNA等。可根据需要,将接合的DNA分子克隆到载体中(例如,使用本发明的方法)。在一些实施例中,可对待组装的核酸进行密码子优化,以便在目标细胞(例如,啮齿动物细胞、小鼠细胞、大鼠细胞、人细胞、哺乳动物细胞、微生物细胞、酵母细胞等)中引入和表达。
可通过本文所公开的方法接合任何长度的DNA分子。例如,可接合约100bp至约750bp或1,000bp或更长的核酸。可根据本文所述的方法在一个或若干个组装阶段中组装的核酸的数量可为至少约2、3、4、6、8、10、15、20、25、50、100、200、500、1,000、5,000或10,000个DNA分子,例如在约2至约30个核酸的范围内。组装阶段的数量可为约2、4、6、8、10个或更多个。在单个阶段中组装的分子的数量可在约2至约10个分子的范围内。可使用本发明的方法将DNA分子或盒接合在一起,每个所述DNA分子或盒的起始大小为至少或不大于约40bp、60bp、80bp、100bp、500bp、1kb、3kb、5kb、6kb、10kb、18kb、20kb、25kb、32kb、50kb、65kb、75kb、150kb、300kb、500kb、600kb、1Mb或更大。经组装的终产物可为至少约500bp、1kb、3kb、5kb、6kb、10kb、18kb、20kb、25kb、32kb、50kb、65kb、75kb、150kb、300kb、500kb、600kb、1Mb或更大,例如在30kb至1Mb的范围内。
在一些实施例中,经组装的核酸形成环和/或连接到载体而形成环。dsDNA环化的大小下限为约200个碱基对。因此,经接合的片段的总长度(在一些情况下包括载体的长度)为至少约200bp长。没有确切的大小上限,并且可通过本文所公开的方法生成数百千碱基对或更大的经接合的DNA。经接合的核酸可采取环状或线性分子的形式。
可使用本文所述的方法将线性片段与另一个线性片段组装在一起,将线性片段与环状核酸分子组装在一起,将环状核酸分子与另一个环状核酸分子组装在一起,或将线性核酸和环状核酸的任何组合组装在一起。“载体”包括任何环状核酸分子。在某些实施例中,通过本文所公开的方法组装的载体是细菌人工染色体(BAC)。载体(例如,BAC)可包含人DNA、啮齿动物DNA、合成DNA或它们的任何组合。例如,BAC可包含人多核苷酸序列。当接合DNA分子的混合物时,优选的是DNA以适当的等摩尔量存在。
用于由本文所公开的方法进行组装的核酸可为大靶向载体。术语“大靶向载体”或“LTVEC”包括这样的载体,其包含对应于且衍生自用于在细胞中进行同源靶向的核酸序列的同源臂,和/或包含具有意欲在细胞中执行同源重组靶向的核酸序列的插入核酸。例如,LTVEC使得对大基因座的修饰成为可能,而传统的基于质粒的靶向载体由于有大小限制而无法实现这一点。在具体实施例中,LTVEC的同源臂和/或插入核酸包含真核细胞的基因组序列。LTVEC过大而无法通过例如Southern印迹和长片段(例如,1kb-5kb)PCR的常规测定法来筛选靶向事件。LTVEC的示例包括但不限于衍生自细菌人工染色体(BAC)、人类人工染色体或酵母人工染色体(YAC)的载体。LTVEC及其制备方法的非限制性示例描述于例如美国专利No.6,586,251、6,596,541、7,105,348和WO 2002/036789(PCT/US01/45375)及US 2013/0137101中,这些专利每一者均以引用的方式并入本文。
在一些实施例中,可将盒插入载体中,随后可去除这些盒。可构建各种形式的盒,以便可以在特定细胞或组织类型中、在特定发育阶段或在诱导时缺失。此类盒可采用重组酶系统,其中所述盒在两侧上侧接重组酶识别位点,并且可使用在所需细胞类型中表达的、在所需发育阶段表达的、或在诱导时表达或激活的重组酶来去除。可进一步构建此类盒,以包括一系列成对的不同重组酶识别位点,这些位点被布置成使得可生成无效等位基因、条件等位基因或条件/无效组合等位基因,如US 2011/0104799中所述,该专利全文以引用的方式并入。重组酶基因的调控可按各种方式控制,诸如将重组酶基因有效连接至细胞特异性、组织特异性或受发育调控的启动子(或其他调控元件),或将重组酶基因有效连接至3’-UTR,该3’-UTR包含仅在特定细胞类型、组织类型或发育阶段中转录的miRNA的识别位点。重组酶还可例如通过以下方式调控:采用使重组酶处于效应物或代谢物的控制之下的融合蛋白(例如,其活性受到它莫西芬控制的CreERT2),或使重组酶基因处于诱导型启动子(例如,其活性受到强力霉素和TetR或TetR变体控制的诱导型启动子)的控制之下。例如在US 8,518,392、US 8,354,389和US 8,697,851中提供了各种形式的盒及调控重组酶基因的方式的示例,这些专利中的每一篇全文均以引用的方式并入。
如本文所公开的用于组装的载体(例如,LTVEC)可具有任何长度,包括但不限于约20kb至约400kb、约20kb至约30kb、约30kb至40kb、约40kb至约50kb、约50kb至约75kb、约75kb至约100kb、约100kb至125kb、约125kb至约150kb、约150kb至约175kb、约175kb至约200kb、约200kb至约225kb、约225kb至约250kb、约250kb至约275kb或约275kb至约300kb、约200kb至约300kb、约300kb至约350kb、约350kb至约400kb、约350kb至约550kb。在一个实施例中,LTVEC为约100kb。
本文所提供的用于组装核酸的方法可被设计成使得可以缺失约5kb至约10kb、约10kb至约20kb、约20kb至约40kb、约40kb至约60kb、约60kb至约80kb、约80kb至约100kb、约100kb至约150kb、或约150kb至约200kb、约200kb至约300kb、约300kb至约400kb、约400kb至约500kb、约500kb至约1Mb、约1Mb至约1.5Mb、约1.5Mb至约2Mb、约2Mb至约2.5Mb、或约2.5Mb至约3Mb。
在其他情况下,本文所提供的方法被设计成使得可以插入以下范围内的外源核酸序列:约5kb至约10kb、约10kb至约20kb、约20kb至约40kb、约40kb至约60kb、约60kb至约80kb、约80kb至约100kb、约100kb至约150kb、约150kb至约200kb、约200kb至约250kb、约250kb至约300kb、约300kb至约350kb、或约350kb至约400kb。在一个实施例中,插入多核苷酸为约130kb或约155kb。
可通过本文所公开的方法将线性核酸彼此组装在一起或组装到载体。线性分子可以是经过核酸内切酶(例如,Cas蛋白)酶切的载体,或任何合成的、人工的或天然存在的线性核酸。在某些实施例中,线性核酸被形成为使得末端序列与另一个核酸的区域重叠。可通过本领域已知的任何方法引入线性核酸的重叠末端序列,以便生成定制的核酸序列。例如,末端序列可以是合成产生的分子的一部分,可以通过PCR引入,或可以通过传统克隆技术引入。
实例
给出以下实例是为了给本领域的普通技术人员提供如何实施并使用本发明的完整公开和描述,而并非旨在限制本发明人视作其发明的范围,也不旨在表示下文的实验是所进行过的所有或仅有的实验。已尽量确保所使用的数字(例如量、温度等)的准确性,但应考虑到一些实验误差和偏差。除非另外指明,否则份数是重量份数,分子量是重均分子量,温度以摄氏度计,并且压力是大气压或接近大气压。
实例1:用CAS9进行BAC酶切,然后与选择盒组装在一起
人工crRNA和人工tracrRNA被设计成靶向MAID 6177(116kb LTVEC)中的特异性序列,以便与3kb PCR产物(UB-HYG)组装在一起。该PCR产物包含50bp与载体重叠的区域。首先将crRNA和tracrRNA溶解于双链体缓冲液(30mM HEPES,pH 7.5,100mM乙酸钾)中达到100μM。为了使RNA退火,将10μL的100μM crRNA和10μL的100μM tracrRNA添加到80μL的退火缓冲液中。在90℃加热块中加热RNA,然后从加热器中取出加热块并置于工作台上冷却。RNA的最终浓度为约10μM。
为了对BAC进行酶切,使用清洁的maxiprep BAC DNA,并根据以下混合物酶切BAC。
在37℃下酶切1小时,然后脱盐30分钟。最终反应缓冲液包含:20mM Tris 7.5;100-150mM NaCl;10mM MgCl2;1mM DTT;0.1mM EDTA;100μg/mL BSA;最终体积达到15μL。
为了组装BAC和插入序列,酶切质粒或执行PCR以形成插入序列。对于PCR反应,在凝胶上运行少量等分试样并寻找单个产物,如果该产物具有单个条带,则进行PCR清洁而非凝胶提取。BAC:插入序列的1:1-1:6摩尔比是所期望的。通常,50ng的经纯化的插入序列将会有效。可使用以下反应混合物:
BAC酶切产物 4μL
插入序列 1μL
组装混合物 15μL
在冰上添加DNA和混合物,或直接在50℃的PCR仪中添加DNA和混合物。在50℃下温育1小时。添加0.5μL的蛋白酶K(20mg/mL),并且在50℃下温育1小时。脱盐30分钟,并且通过电穿孔法将8μL的反应体系导入DH10B细胞中。可在脉冲场凝胶上运行10μL的BAC酶切产物,以检查酶切效率。使用无RNA酶的水和缓冲液。
按照如下方式执行组装反应:等温缓冲液:3mL 1M Tris-HCL(pH 7.5);150μL 2M MgCl2;以下各60μL 100mM:dGTP、dATP、dTTP、dCTP;300μL 1M DTT;1.5g PEG 8000;300μL 100mM NAD。将该等温缓冲液以320μL等分试样储存在-20℃下。按照如下方式制备主混合物:将320μL等温缓冲液、0.64μL T5核酸外切酶(储液浓度=10U/μL)、20μL Phusion DNA聚合酶(储液浓度=2U/μL)、160μL Taq DNA连接酶(储液浓度=40U/μL)、699.36μL H2O混合在一起,等分为15μL或30μL并且储存在–20℃下。在总体积20μL的反应体系中使用15μL主混合物(MM)。
本实例中所用的tracr RNA序列为:
CAAAACAGCAUAGCAAGUUAAAAUAAGGCUAGUCCGUUAUC(SEQ ID NO:9)。该CRISPR RNA(crRNA)包含:(1)与靶序列互补的约20个核苷酸的RNA,以及(2)将与tracrRNA退火的尾部序列(GUUUUAGAGCUAUGCUGUUUUG(SEQ ID NO:10))。
这些步骤在图1中概述。
实例2:将以下两个重叠BAC拼接在一起:小鼠MHC II基因座(H2-A/H2-E)中的人源化HLA-DQ+人源化HLA-DR
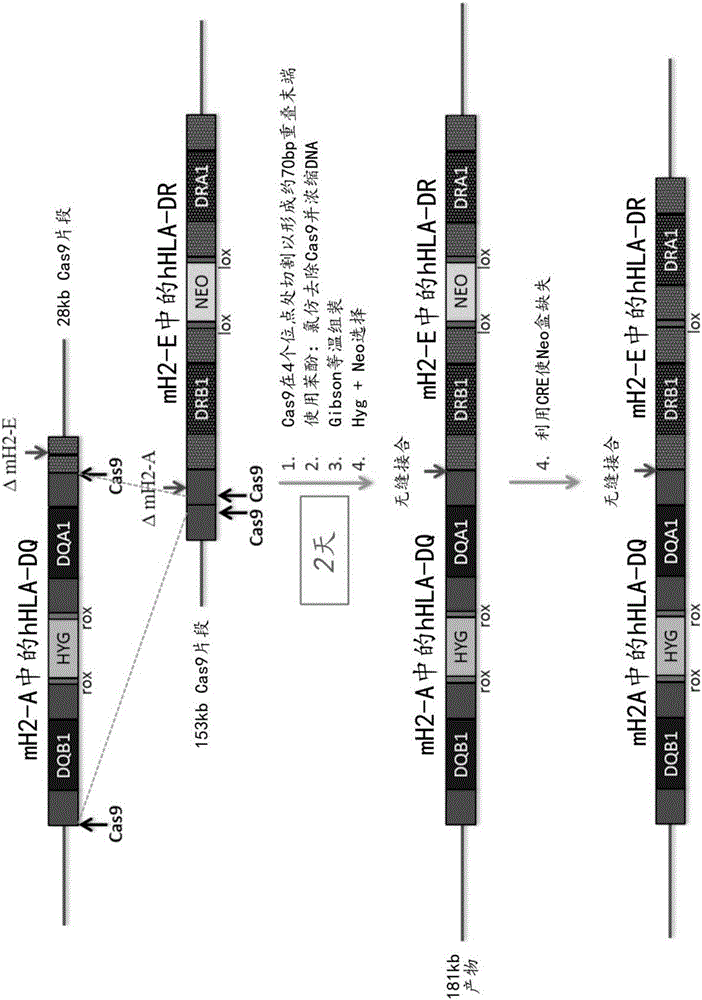
人工crRNA和人工tracrRNA被设计成靶向人源化HLA-DQ BAC中的特定序列,以便与人源化HLA-DR BAC组装在一起。这些载体包含通过Cas9在每个载体上的两个位点处切割而形成的约70bp彼此重叠的区域(参见图2)。将crRNA和tracrRNA溶解于Hybe缓冲液中达到100μM。为了使RNA退火,将10μL的100μM crRNA和10μL的100μM tracrRNA添加到80μL的退火缓冲液中。将RNA放入90℃加热块中,然后从加热器中取出加热块并置于工作台上冷却。RNA的最终浓度为约10μM。
为了对BAC进行酶切,可使用清洁的maxiprep BAC DNA。根据以下混合物单独地酶切每个BAC:
BAC载体应在37℃下酶切1小时,然后在65℃下热灭活20分钟。脱盐30分钟。经由苯酚/氯仿/异戊醇(PCI)提取来纯化经酶切的DNA,然后重悬于35μL TE缓冲液中。
为了组装载体,按照如下方式将2.5μL的BAC用于组装反应:
经酶切的BAC 5μL(总量)
组装混合物 15μL
在冰上添加DNA和混合物,或直接在50℃的PCR仪中添加DNA和混合物。在50℃下温育1小时。脱盐30分钟,并且通过电穿孔法将8μL经组装的DNA导入DH10B细胞中。使用无RNA酶的水和缓冲液。
本实例中所用的tracr RNA序列为:CAAAACAGCAUAGCAAGUUAAAAUAAGGCUAGUCCGUUAUC(SEQ ID NO:9)。该CRISPR RNA(crRNA)包含:(1)与靶序列互补的约20个核苷酸的RNA,以及(2)将与tracrRNA退火的尾部序列(GUUUUAGAGCUAUGCUGUUUUG(SEQ ID NO:10))。
这些步骤在图2中概述。
实例3:使用接头组装由Cas9从2个不同质粒切割的2个片段
为了构建靶向载体,用2个相同的crRNA切割pMJ8502x,使400bp片段和2283bp Amp骨架脱落。(图7)。使用Qiagen柱纯化整个反应。然后用2个不同的crRNA切割R6KZenUbiNeo,以分离成Neo抗性片段(1086bp)和骨架(5390bp)。使用Qiagen柱纯化整个反应。(图7)。切割反应体系:1170ng DNA、30μL缓冲液、4μL经退火的RNA(为100μM)、1.7μL Cas9(为0.89ng/μL),用H2O定容至60μL。将混合物在37℃下温育1小时,并且在Qiagen柱上纯化,之后在30μL洗脱缓冲液中洗脱。
然后根据以下反应混合物,使用两个接头组装经切割的片段,从而实现无缝组装:0.5μL接头1(5ng)、0.5μL接头2(5ng)、2μL Neo切割物(约60ng)、2μL Amp切割物(约60ng)、15μL组装主混合物。将混合物在50℃下温育1小时,并且用H2O透析反应体系。通过电穿孔法将10μL反应体系导入电感受态Pir细胞中,之后接种到Carb/Kan平板上。跨接合部的PCR显示,8个经选择的菌落中有6个是正确的,并得到测序确认。
实例4:使用接头将BAC的一部分替换为盒
为了构建敲除小鼠靶向载体,将40kb的BAC靶向载体替换为被重组识别位点侧接的选择盒。(图8)2个接头被设计成从mBAC缺失目标区域并插入选择盒,一个接头用于5’,另一个接头用于3’。这些接头具有40bp与mBAC重叠的区域以及40bp与选择盒重叠的区域。首先,根据以下反应体系切割206kb靶向载体(mBAC)中的39.5kb:500μL反应体系(用H2O定容):添加1μL Cas9(为0.89μg/μL)、2μL每个RNA双链体(为50μM)、250μL缓冲液、220μL(12.5ng)BAC maxi prep,并且在37℃下温育1小时。经由苯酚/氯仿/异戊醇(PCI)提取来纯化经酶切的DNA,然后重悬于55μL TE缓冲液中。在mBAC切割物的PCI清洁之后,在50℃下进行1小时组装,并且通过电穿孔法将10μL的反应体系导入DH10B细胞中。(图9)。跨接合部的测序确认组装正确。(图10)。接头1(接合寡核苷酸1)从mBAC序列无缝地连接到盒序列(SEQ ID NO:12)。接头2(接合寡核苷酸2)从盒序列无缝地连接到mBAC序列(SEQ ID NO:13)。
实例5:使用接头(接合寡核苷酸)组装两个BAC载体
利用由Cas9/等温组装对2个mBAC进行的拼接法来制备靶向载体,该靶向载体包含对于小鼠基因组区域而言的同源臂,以及用于通过BAC连接反应插入人基因的酶切位点。在BAC连接反应中使用该靶向载体制备人源化靶向载体。根据以下反应体系切割mBAC:12.5μg DNA、2μL每个经退火的RNA(为50μM)、10μL Cas9(为0.89μg/μL)、250μL缓冲液,用H2O定容至500μL。将该混合物在37℃下温育一小时;通过苯酚/氯仿/异戊醇(PCI)提取来清洁;然后重悬于20μL TE中。然后根据以下反应体系用接头将这两个小鼠BAC组装在一起(图11):6μL(2μg)bMQ-208A16切割物、5.6μL(2μg)bMQ-50F19切割物、0.25μL每个接头(为50μM)、4.3μL(100ng)选择盒(Ubi-Hyg)盒、12μL高浓度组装主混合物、11.35μL H2O。将反应混合物在50℃下温育1小时,并且在30℃下用H2O透析。使用10μL或30μL经透析的反应体系来转化DH10B细胞。Sanger测序确认了所有接合部。Illumina测序再次确认了所有接合部(图12和SEQ ID NO:17)。接头1从mBAC无缝地连接到盒(SEQ ID NO:14)。接头2从盒非无缝地连接到mBAC。它按照项目设计掺入人间区序列。接头3从mB2非无缝地连接到mB3。它掺入用于PCR验证的独特序列。当发生线性化以用于ES电穿孔时,该区域被去除(SEQ ID NO:15)。
图13示出了使用4个接合寡核苷酸(接头)并使用四个接头和等温组装在mBAC上插入较大的人基因片段的示例。
实例6:用于切割和组装的试剂和反应混合物
Crispr RNA(crRNA)(作为ssRNA订购)包含:(1)与待切割的靶区域互补的20个核苷酸的RNA;(2)以及将退火到tracr RNA的尾部:<20nt crisprRNA>GUUUUAGAGCUAUGCUGUUUUG(SEQ ID NO:10)。
Tracr RNA(作为ssRNA订购):GUUGGAACCAUUCAA AACAGCAUAGCAAGUUAAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGCUUUUUUU(SEQ ID NO:11)。
将所有RNA重悬于H2O中达到100μM。将crRNA和tracrRNA各2.5μL与5μL的退火缓冲液(最终浓度:10mM Tris pH 7.5-8.0,50mM NaCl,1mM EDTA)组合。然后将该混合物在95℃下温育5分钟,并在1小时内缓慢冷却至室温。Cas9 2X切割缓冲液包含40mM HEPES pH7.5(最终浓度=20mM);300mM KCl(最终浓度=150mM);1mM DTT(最终浓度=0.5mM);0.2mM EDTA(最终浓度=0.1mM);20mM MgCl2(最终浓度=10mM)。
大规模Cas9切割反应:在室温下依次添加:250μL 2x切割缓冲液、12.5μg DNA、各2μL的每种RNA(50μM浓度)、10μL Cas9(0.89mg/mL浓度),用H2O定容至500μL,并且在37℃下温育1小时。
可根据需要缩放该反应体系,例如:25μL缓冲液、125ng DNA、各2μL的每种RNA(5μM浓度)、1μL Cas9(0.89mg/mL浓度),用H2O定容至50μL,并且在37℃下温育1小时。
按照如下方式执行组装反应:等温缓冲液:3mL 1M Tris-HCL(pH 7.5);150μL 2M MgCl2;以下各60μL 100mM:dGTP、dATP、dTTP、dCTP;300μL 1M DTT;1.5g PEG 8000;300μL 100mM NAD。将该等温缓冲液以320μL等分试样储存在-20℃下。按照如下方式制备主混合物:将320μL等温缓冲液、0.64μL T5核酸外切酶(储液浓度=10U/μL)、20μL Phusion DNA聚合酶(储液浓度=2U/μL)、160μL Taq DNA连接酶(储液浓度=40U/μL)、699.36μL H2O混合在一起,等分为15μL或30μL并且储存在–20℃下。在总体积20μL的反应体系中使用15μL主混合物(MM)。
作为另一种选择,可按照如下方式制备高浓度主混合物(GA MM HC):将320μL等温缓冲液、0.64μL T5核酸外切酶(储液浓度=10U/μL)、20μL Phusion DNA聚合酶(储液浓度=2U/μL)、160μL Taq DNA连接酶(储液浓度=40U/μL)混合在一起,等分为6μL或12μL并且储存在–20℃下。在总体积20μL的反应体系中使用6μL的主混合物。
对于所有组装反应而言,应确定DNA的浓度(例如,通过Nano Drop确定)并使用1:6摩尔比(载体与插入序列)。对于标准浓度而言,使用15μL的组装主混合物。将DNA和水添加到200μL PCR管中,达到20μL的最终体积。在50℃热循环仪中反应1小时。然后可将该反应体系储存在-20℃下。对于高浓度而言,使用6μL的高浓度组装主混合物。将DNA和水添加到200μL PCR管中,达到20μL的最终体积。在50℃热循环仪中反应1小时。然后可将该反应体系储存在-20℃下。在反应完成时,用水透析10μL反应体系30分钟,接着通过电穿孔法导入适当的电感受态细胞(例如,DH10B或Pir+细胞)中。
Cas9/等温组装反应:对于Cas9酶切,将2.5μg的每种DNA(例如,BAC DNA)、各4μL的10μM向导RNA/tracr RNA和5μL的Cas9蛋白(0.89mg/mL)在37℃下酶切2小时。将反应体系在65℃下热灭活20分钟,用苯酚氯仿提取(例如,以去除Cas9蛋白),用70%乙醇洗涤一次,接着将DNA重悬于35μL水中。将5μL的DNA与15μL的本文别处所述的主混合物(MM)混合在一起并在50℃下温育1小时,以此进行等温组装。将该反应体系脱盐30分钟,并且可通过电穿孔法将8μL的该反应体系导入细胞中。
实例7:通过Cas9/等温组装将人序列插入BAC载体中
为了构建人源化靶向载体,用gRNA-Cas复合物切割MAID 6236,以生成具有重叠序列的经切割的片段。还用gRNA-Cas复合物切割VI568,以生成与MAID6236的片段重叠的序列。如上所述那样执行Cas9/等温组装,使人源化基因座插入载体(VI599)中。该过程在图14中概述。
实例8:在不进行选择的情况下使用gBlock进行Cas9/等温组装
Cas9酶切和组装还可在不进行选择的情况下例如通过利用gBlock DNA片段来执行。为了测试在没有选择盒的情况下将双链DNA添加到基因座中的可能性,合成了gBlock DNA片段并将这些片段插入构建体中。如图15A和15B中所概述,Cas9/gRNA被设计成靶向TCRβ基因座内的两个位点以缺失4.4kb片段。gBlock被设计成将大范围核酸酶识别位点引入构建体中。gBlock能够在不使用选择标记的情况下插入构建体中。图15A示出了PISceI gBlock的插入,而图15B展示了MauBI gBlock的插入。
使用表1中示出的引物,通过PCR接合部筛选,确认了最终构建体中成功插入了每个gBlock。用于接合部筛选的方案如下:PCR反应体系包含:1μL DNA、0.5μL引物1、0.5μL引物2、1μL DMSO、4μL dNTPs、2.5μL 10x缓冲液、0.5μL Ex-Taq和15μL水。按照以下方式在热循环仪中执行反应:在95℃下持续3分钟,在95℃下持续30秒,在55℃下持续30秒,执行25个循环,然后在72℃持续30秒及在72℃下持续5分钟。通过测序确认了接合序列。
表1:用于对具有PI-SceI gBlock或MauBI gBlock的MAID1715进行接合部筛选的引物
实例9:使用接合寡核苷酸将人序列插入BAC载体中的Cas9/等温组装
图16提供了使用Cas9/等温组装和接合寡核苷酸进行的直接人源化的示例。通过Cas9使人片段和小鼠缺失片段脱落(每个BAC使用2个crispr RNA)。在Gibson组装反应中使用3个接头(接合寡核苷酸)和选择盒将人片段和小鼠骨架连接在一起。
图17提供了使用Cas9/等温组装和接合寡核苷酸组装成大靶向载体(LTVEC)进行的间接人源化的示例。通过采用2个crispr RNA的Cas9切割掉hBAC上的人片段。供体包含上游和下游接合寡核苷酸及选择盒。在通过Cas9进行hBAC切割之后,使用掺有互补悬垂序列的合成供体,通过Gisbon组装来“捕获”片段。通过Gibson组装或BHR来完成靶向载体构建。
实例10:通过Cas9/等温组装来引入点突变
图18提供了利用Cas9/等温组装来引入点突变的示例。通过传统克隆来制备供体。将选择盒插入包含接头重叠区和点突变的合成DNA片段中。用Cas9切割mBAC,从mBAC中去除该序列,并且通过Gibson组装法将mBAC组装到供体,得到包含点突变和选择盒的构建体(LTVEC)。
实例11:通过Cas9/等温组装进行BAC修剪
图19提供了使用Cas9/等温组装方法进行的BAC修剪的示例。使用Cas9来修剪需从LTVEC去除的区域。在本示例中,BAC修剪去除了Ori序列。在Gibson组装反应中使用2个接头(接合寡核苷酸)替换Ori。
实例12:用CAS9酶切BAC后再进行组装的其他方法
可在本文所提供的方法中使用其他方法,包括以下步骤:在反应之前,通过加热到95℃并缓慢冷却到室温,使合成的或体外转录的tracrRNA和crRNA预先退火。在37℃下将天然或线性化的质粒DNA(300ng(约8nM))与经纯化的Cas9蛋白(50-500nM)和tracrRNA:crRNA双链体(50-500nM,1:1)在含或不含10mM MgCl2的Cas9质粒切割缓冲液(20mM HEPES pH 7.5,150mM KCl,0.5mM DTT,0.1mM EDTA)中温育60分钟。用含有250mM EDTA的5X DNA上样缓冲液终止反应,通过0.8%或1%琼脂糖凝胶电泳进行分离,并通过溴化乙锭染色来显色。对于Cas9突变体切割测定法而言,先用5X SDS上样缓冲液(30%甘油,1.2%SDS,250mM EDTA)终止反应,再上样于琼脂糖凝胶中。
人工crRNA和人工tracrRNA被设计成靶向MAID 6177(116kb LTVEC)中的特定序列,以便与3kb PCR产物(UB-HYG)组装在一起。该PCR产物包含50bp与载体重叠的区域。基于分离的缺少3'核酸外切酶活性的不耐热5'至3'核酸外切酶的使用,按照如下方式使用等温一步组装。设定反应体系,其包含以下物质:各100fmol的每种dsDNA底物、16μL 5X ISO缓冲液、16μL T5核酸外切酶(0.2U/μL,Epicentre)、8.0μL Taq DNA连接酶(40U/μL,NEB)、1.0μL PhusionTMDNA聚合酶(2U/μL,NEB),并且用水定容至80μL。5×ISO(等温)缓冲液为25%PEG-8000、500mM Tris-Cl、50mM MgCl2、50mM DTT、5mM NAD和各1000μM的每种dNTP(pH 7.5)。
这得到最终浓度为1.25fmol/μL的每个待组装的dsDNA(或45fmol/μL每个ssDNA)、5%PEG-8000、100mM Tris-Cl pH 7.5、10mM MgCl2、10mM DTT、200MM每种dNTP、1mM NAD、0.02U/μL T5核酸外切酶、4U/μL Taq DNA连接酶以及0.03U/μL PHUSION DNA聚合酶。
对于重叠20-80bp的底物而言,方法使用1.64μL(0.2U/μL)的T5核酸外切酶,而对于具有更大重叠区(例如,200bp)的底物而言,则使用1.6μL(1U/μL)的T5核酸外切酶。T5核酸外切酶作为来自10U/μL T5核酸外切酶(Epicentre)浓缩酶储液的1:50稀释液(稀释于T5核酸外切酶储存缓冲液中)使用。然后将该反应体系在50℃下温育15分钟。
实例13:用于将两个重叠BAC拼接在一起的其他方法
可在本文所提供的方法中使用其他方法,包括以下步骤:在反应之前,通过加热到95℃并缓慢冷却到室温,使合成的或体外转录的tracrRNA和crRNA预先退火。在37℃下将天然或线性化的质粒DNA(300ng(约8nM))与经纯化的Cas9蛋白(50-500nM)和tracrRNA:crRNA双链体(50-500nM,1:1)在含或不含10mM MgCl2的Cas9质粒切割缓冲液(20mM HEPES pH 7.5,150mM KCl,0.5mM DTT,0.1mM EDTA)中温育60分钟。用包含250mM EDTA的5X DNA上样缓冲液终止反应,通过0.8%或1%琼脂糖凝胶电泳进行分离,并通过溴化乙锭染色来显色。对于Cas9突变体切割测定法而言,先用5X SDS上样缓冲液(30%甘油,1.2%SDS,250mM EDTA)终止反应,再上样于琼脂糖凝胶中。
人工crRNA和人工tracrRNA被设计成靶向人源化HLA-DQ BAC中的特定序列,以便与人源化HLA-DR BAC组装在一起。这些载体包含通过Cas9在每个载体上的两个位点处切割而形成的约70bp彼此重叠的区域(参见图2)。基于分离的缺少3'核酸外切酶活性的不耐热5'至3'核酸外切酶的使用,按照如下方式使用等温一步组装。设定反应体系,其大致包含以下物质:各100fmol的每种dsDNA底物、16μL 5X ISO缓冲液、16μt T5核酸外切酶(0.2U/μL,Epicentre)、8.0μL Taq DNA连接酶(40U/μL,NEB)、1.0μL PhusionTMDNA聚合酶(2U/μL,NEB),并且用水定容至80μL。5×ISO(等温)缓冲液为25%PEG-8000、500mM Tris-Cl、50mM MgCl2、50mM DTT、5mM NAD和各1000μM的每种dNTP(pH 7.5)。
这得到最终浓度为约1.25fmol/μL的每个待组装的dsDNA(或45fmol/μL每个ssDNA)、5%PEG-8000、100mM Tris-Cl pH 7.5、10mM MgCl2、10mM DTT、200MM每种dNTP、1mM NAD、0.02U/μL T5核酸外切酶、4U/μL Taq DNA连接酶以及0.03U/μL PHUSION DNA聚合酶。
对于重叠20-80bp的底物而言,方法使用1.64μL 0.2U/μL的T5核酸外切酶,而对于具有更大重叠区(例如,200bp)的底物而言,则使用1.6μL1U/μL的T5核酸外切酶。T5核酸外切酶作为来自10U/μL T5核酸外切酶(Epicentre)浓缩酶储液的1:50稀释液(稀释于T5核酸外切酶储存缓冲液中)使用。然后将该反应体系在50℃下温育15分钟。
实例14:用于将插入序列与BAC载体组装在一起的其他方法
可在本文所提供的方法中使用其他方法,包括以下步骤:将crRNA和tracrRNA溶解于Hybe缓冲液(10X缓冲液:20mM Tris 7.5、100-150mM NaCl、10mM MgCl2、1mM DTT、0.1mM EDTA、100μg/mL BSA)中达到100μM。为了使RNA退火,将10μL的100μM crRNA和10μL的100μM tracrRNA添加到80μL的退火缓冲液中。在90℃加热块中加热RNA,然后从加热器中取出加热块并置于工作台上冷却。RNA的最终浓度为约10μM。
为了对BAC进行酶切,使用清洁的maxiprep BAC DNA,并根据以下混合物酶切BAC。
在37°下酶切1小时,然后脱盐30分钟。
为了组装BAC和插入序列,酶切质粒或执行PCR以形成插入序列。对于PCR反应,在凝胶上运行少量等分试样并寻找洁净产物,如果该产物不洁净,则进行PCR清洁而非凝胶提取。BAC:插入序列的1:1-1:6摩尔比是所期望的。通常,50ng经纯化的插入序列将会有效。可使用以下反应混合物:
BAC酶切产物 4μL
插入序列 1μL
组装混合物 15μL
在冰上添加DNA和混合物,或直接在50℃的PCR仪中添加DNA和混合物。在50℃下温育1小时。添加0.5μL的蛋白酶K(20mg/mL),并且在50℃下温育1小时。脱盐30分钟,并且通过电穿孔法将8μL的反应体系导入DH10B细胞中。可在脉冲场凝胶上运行10μL的BAC酶切产物,以检查酶切效率。使用无RNA酶的水和缓冲液。最终反应缓冲液包含:20mM Tris 7.5;100-150mM NaCl;10mM MgCl2;1mM DTT;0.1mM EDTA;100μg/mL BSA;最终体积达到15μL。
本实例中所用的tracr RNA序列为:
CAAAACAGCAUAGCAAGUUAAAAUAAGGCUAGUCCGUUAUC(SEQ ID NO:9)。该CRISPR RNA(crRNA)包含:(1)与靶序列互补的约20个核苷酸的RNA,以及(2)将与tracrRNA退火的尾部序列(GUUUUAGAGCUAUGCUGUUUUG(SEQ ID NO:10))。
序列表
<110> 克里斯·舒恩赫
约翰·麦克沃特
科里·莫蒙
林恩·麦克唐纳
安德鲁·J.·墨菲
格雷格·S.·沃肖
约瑟·F.·罗哈斯
卡曼·维纳斯·莱
大卫·M.·巴伦苏埃拉
凯特琳·蒙塔尼亚
<120>核酸酶介导的DNA组装
<130> 057766-461002
<140> PCT/US2015/037199
<141> 2015-06-23
<150> US 62/036,983
<151> 2014-08-13
<150> US 62/016,400
<151> 2014-06-24
<150> US 62/015,809
<151> 2014-06-23
<160> 25
<170>适用于Windows的FastSEQ 4.0版
<210> 1
<211> 80
<212> RNA
<213>人工序列
<220>
<223>合成嵌合gRNA
<400> 1
guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc cguuaucaac uugaaaaagu 60
ggcaccgagu cggugcuuuu 80
<210> 2
<211> 42
<212> RNA
<213>人工序列
<220>
<223>合成嵌合gRNA
<400> 2
guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc cg 42
<210> 3
<211> 30
<212> RNA
<213>人工序列
<220>
<223>合成crRNA
<400> 3
guuuuagagc uagaaauagc aaguuaaaau 30
<210> 4
<211> 33
<212> RNA
<213>人工序列
<220>
<223>合成crRNA
<400> 4
guuuuagagc uagaaauagc aaguuaaaau aag 33
<210> 5
<211> 26
<212> RNA
<213>人工序列
<220>
<223>合成crRNA
<400> 5
gaguccgagc agaagaagaa guuuua 26
<210> 6
<211> 12
<212> RNA
<213>人工序列
<220>
<223>合成tracrRNA
<400> 6
aaggcuaguc cg 12
<210> 7
<211> 50
<212> RNA
<213>人工序列
<220>
<223>合成tracrRNA
<400> 7
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugcuuuu 50
<210> 8
<211> 23
<212> DNA
<213>人工序列
<220>
<223> 连接至向导RNA (gRNA)的靶
基因座
<220>
<221> misc_feature
<222> 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18,
19, 20, 21
<223> n = A、T、C或G
<400> 8
gnnnnnnnnn nnnnnnnnnn ngg 23
<210> 9
<211> 41
<212> RNA
<213>人工序列
<220>
<223>合成tracrRNA
<400> 9
caaaacagca uagcaaguua aaauaaggcu aguccguuau c 41
<210> 10
<211> 22
<212> RNA
<213>人工序列
<220>
<223>与tracrRNA互补的合成crRNA区域
<400> 10
guuuuagagc uaugcuguuu ug 22
<210> 11
<211> 89
<212> RNA
<213>人工序列
<220>
<223>合成tracrRNA
<400> 11
guuggaacca uucaaaacag cauagcaagu uaaaauaagg cuaguccguu aucaacuuga 60
aaaaguggca ccgagucggu gcuuuuuuu 89
<210> 12
<211> 145
<212> DNA
<213>人工序列
<220>
<223> 合成-从mBAC到盒的无缝组装
的确认
<400> 12
ttgtgtgaat ataataatat cagtgcttct ttacttccaa aactggacag cgcatcaaac 60
atcagaaaca acagtatcag ctcctgtccc aactaccatg ggtaccgatt taaatgatcc 120
agtggtcctg cagaggagag attgg 145
<210> 13
<211> 205
<212> DNA
<213>人工序列
<220>
<223> 合成-从盒到mBAC的无缝组装
的确认
<400> 13
cagcccctag ataacttcgt ataatgtatg ctatacgaag ttatgctagc tcggtcacac 60
tgtcagcttc ctgtgtttcc taggccatga taagatgcag caaagtttct gcaatgcaca 120
atgaggcagc cgtcggaata gatttgagaa agtcatgatg atgcaatgtg cacactcttc 180
ctttgtattt atctctatcc accat 205
<210> 14
<211> 138
<212> DNA
<213>人工序列
<220>
<223> 合成-从mBAC到盒的无缝组装
的确认
<400> 14
actttagggt ttggttggtt tttaaagccc tatttccagt atgtggaaat gagccaaccc 60
aggacagctt ccgctggatc gtggacagct tctatggccg tcgacgtgta cactcgagat 120
aacttcgtat aatgtatg 138
<210> 15
<211> 147
<212> DNA
<213>人工序列
<220>
<223>合成
<400> 15
tccaaacgac agcagaacta actgagagga gagcacagta gcggccgcaa attgctttga 60
gaggctctat aaaaccttag aggctattta aatttaaatg gccggcccga cggccaggcg 120
gccgccaggc ctacccacta gtcaatt 147
<210> 16
<211> 9
<212> PRT
<213>未知
<220>
<223>合成
<400> 16
Leu Ala Gly Leu Ile Asp Ala Asp Gly
1 5
<210> 17
<211> 49631
<212> DNA
<213>人工序列
<220>
<223>合成
<220>
<221> misc_feature
<222> (22396)...(22533)
<223>接头1
<220>
<221> misc_feature
<222> (22494)...(25426)
<223>盒序列
<220>
<221> misc_feature
<222> (25427)...(25595)
<223>人间区序列
<220>
<221> misc_feature
<222> (25596)...(40791)
<223> BMQ-208A16序列
<220>
<221> misc_feature
<222> (25387)...(25672)
<223>接头2
<220>
<221> misc_feature
<222> (40792)...(40858)
<223>接头3的独特附加序列
<220>
<221> misc_feature
<222> (40752)...(40898)
<223>接头3
<220>
<221> misc_feature
<222> (1)...(22395)
<223> bmq-50F19
<220>
<221> misc_feature
<222> (40899)...(49631)
<223> bmq-50F19
<400> 17
gctggagtgt ggtcaggcaa catccccaaa gggatggaga tgccgggacg acacctttag 60
ggaggcagtg gctctggtcc gggattccgg tgctggccat ccctcaccag ccacagcggt 120
tggcgcagga gggatcgccg cgcgcctggg gctagggggc gaactggacc gacttttcct 180
agttcgccta gctgctccga ccgctgccgc gccgagatgt tgaaagcaca ggcgagttct 240
aacttgcgcg ctcattcttt cagcgcgggg gaatcggtcg agggccctgc gtggcgctgg 300
cttccaccct cgcggccagg gggcaggcgc gggaggccgg cttcggctcc gtgcccctgc 360
aaacttccca agaccttcct tctccccccc acctcacccc ccagttcaat aaaatctacc 420
cttaaaggca gacttgcttt caaatccacg gcacccatta tgtgtttggt gtgaaacgct 480
atcaacattt aaaactctat tgtcccaagc gtcccaaatc cctgtaaatc ttccaccagc 540
ctggactcat tttcatctga aaagcctgtt tagtttgaat agaaaagcaa tcaggcgccc 600
ctctcgctct cgttggaatg tcaattaaaa tgcagatttc tcagagctct ttagcgcccc 660
aagaagtggg acaaaacagg atatttcagg ctgacaaatg aaagaaatgc tacaatgaag 720
tggggtggcg atgtgcaccc caaactgctt ggagtaccca ctgaaagagt aggtcaggga 780
ttatggtctt acttacgaca gcttatattt ttggggtttc gttgtgttta gggccccccc 840
ttggtgtccc ccccccccat gagcccatga cagctccctt ccctattcag ccccgtggag 900
aagtaaggga gccttgaacc agggtagaga ggctacattt agtattaacc tgggagtgtt 960
gacttctccc aggagtaatc cacttgagaa caaaatgcca attgctctgc ccgctgaggt 1020
atcctggaac taccctttaa ggtagcagta cccgtcgcac cgcccccctc ccccaagggc 1080
ttgccttaaa ttaacctgcc ttcttgcagg acaggggaga gtgtgtaaac gtgtataaca 1140
ctgcgcaagc tcaccagccg ggccctttcg gccgggtccc tttgcctgtc tttggaggca 1200
gacttgtgtg gagatgaccc caaggggcgg gtggccgtga agagccatcc gtcagagtga 1260
gggtgaggac tcctccctcg taggctgaga agagagtatc ctttcagggg gaaaaataaa 1320
cacgctgggg ctttctctgg ggttcagcct ccaggaagga ttatggtatt gaaggcagga 1380
agctgggatt gtggccgcca gcagcatgct gggcctgtgt tcccaacacg gagccttggg 1440
acctaattat cctgcctagg aggtcgctca gcacttttgt ccactccggt gaggagctgt 1500
gcagacctgc tgccgtcact tctcgcctta cagaggtttg aggagggggc tcctgtgggg 1560
gctgggactt cgaagaacga acgttcaagt tgagtcagcc tggggcactg gccatcttcc 1620
tcattcagct ggagctgagg tactcctggg tagtggctag tagagacagt gggcccagca 1680
ctctgcttca agacctactg ggacctgaga ttgcaaagtt gctggagagg ggagtttacc 1740
tgcattctga aagttcttag gaaatcaacg agaatgtttg tgcactttcc tttgactggt 1800
atgtagaaat agacaaggaa ttatcttttg tgactcttgg ctttaagaag aaagaagact 1860
tgggggaaca aaaatccttc cagccaacta aaaacactgg gcacctaact gctcatatac 1920
ccctggcttt tgttgttagc tataccattc tacctgtgct taaaaaaaca accaaacagc 1980
agcttcctat tcccctcttg gagatggtac gtcctctctg ccttagtctc agtgaaggct 2040
gaaaggaaca gattttagga cggaggttct ggcagtgtcg aaatcctgtg tcataattga 2100
aagcatcaaa agcgcacggg attagaattc tttttctctt tctctctttt tcattaaaac 2160
gctcacccat ccccagtctc ataaaatggg catcccagca tccaaagccc atggttttgt 2220
gcgatccttt cctgccattg gtttcagcag attctctaaa gctcgtgcat tctgactcaa 2280
agattagtca ctgaagacac tgaacaaaca taaagttatt tgtactgtgg taagcttttt 2340
tttttgggaa attctctgct ttggatctag taaattgagt gcccccttgt aactgatact 2400
tgggaggttt agccaatagg ttagcgtatt gaaagttccc aggccaatca cataccaggg 2460
cagcttgtac gtatcatcac cattactaat aaaatcttga attattcatc aagggttgta 2520
tctttacccc tttgacgtcg gttgcagata tttagttagt atgcctgtac actgccttgt 2580
agtcagtgga agggaattca ggctttgaat cccccggttg gattaaactc actctttgta 2640
agtggctgct tggcggaaga ttgaaataca cgcctgcatt cgaaaatgaa ttctgacaag 2700
tgtaaactgg tgggaatgtt tttgaagcct tcctgagatt ctttgattct gttggtctcc 2760
tttctttctg agaaccgttc tgaagcgagg acgtgccgct cagctcagct gaaatgcggt 2820
tctcagagca gacccttcct ccagtcagcg tcttaaaggc cagctggaat aagagacgtt 2880
aatgaggctg gccatgccaa gcccagcgtt ttaaactcag gtttttctgc agttgccctt 2940
gaaaggaatg aaggtcaagt tgcttcagca accttgcagc tttgatagtg gacggaaggg 3000
cacgctgcag agctgggtgg ctgggtccta cagtgatggt ttatcttgcg tctcttaaaa 3060
gtaagcttaa aaaaaaaaag attagcctac tgcagcttgt ggactagcct ggaaacacct 3120
gggacgctga ggtgaggatg gaaggctttt ccgataatga gaaagaatgt gtttgcgaat 3180
gtattgagag gctgagaaat ggttttatcc catctgggtt taagcaagtt ggcactgggg 3240
gaaaaaactg aatctggctg aatctctctc tttcagtggc agccacagca gcagcagcag 3300
cagcagtggg agcaggaaca gcagtaacaa cagcaacagc agcacagccg cctcagagct 3360
ttggctcctg agccccctgt gggctgaagg cattgcaggt agcccatggt ctcagaagaa 3420
gtgtgcagat gggattaccg tccacgtgga gatatggaag aggaccaggg attggcactg 3480
tgaccatggt cagctggggg cgcttcatct gcctggtctt ggtcaccatg gcaaccttgt 3540
ccctggcccg gccctccttc agtttagttg aggataccac tttagaacca gaaggtaagt 3600
tcatgcgtgc cattttaagg gtaccaagtc gttttgggga tgtgtctggg ggaagtggtc 3660
tttaagtggg aggcctgttt cagccggctg ccatatgagt agtctctctc cgcatcatat 3720
cggagcttag aagggagggt cttgtctccc aggcatgagt ggagtggttt ggtttgctct 3780
gttctttgtg cttgggtgag ggaagcagtg gcagttcttg tttagccagt gccttacagc 3840
actctggagg ggacgtacct tggcagggtg actgtggcct tctgcagttg ctctctagat 3900
tgagggaaaa gccttgaatc acactatctt ttggctaaag gaaataggca gcctctgaaa 3960
gctgactttt tttttctttt tccgcattgt ttaagagaaa agaaggttct gaagttgagc 4020
atggagagcc gtgccatgct ggatcggttt ttaagctggt gtaagctctt tgtgctttca 4080
cccggcatca cagagtgggc aggtttcatg ttgggaagat tggaaagtga atttgccaag 4140
agtcttcccc catctgggga aaagccagat ttcactagtg tgtttggctt tgcacacttg 4200
gttgcaaatg tgagaagcta gttgtgagga ggacgtggct gaaatccgga gctgggcaaa 4260
gcgctggtcc ttctcccagg tccttcagag acgtggtctg tggccaagcc tctctccttg 4320
gtgccgcacg ggaatctgtc atcaggaggg aatattggta ggcgagttat tttttgagtg 4380
gtaatccgag cgtgacactg cagatcgcag cactcatcgc cacttaatga acgtgtttgc 4440
tgagggccca cctggtgccg gctggctttg gagtccgtca cggtcctgag tgctggcagg 4500
tcagctgagt tgctgtggct atgcacactg aatcagggtc ctgattcatc cagatcatcc 4560
agagggggat tgtaggaggg acaggacccc tcccccaagg gtgacctcaa ggagggctat 4620
gtacccatct gagaggaggg cttgagaaat gggtccccag taagatccac ccagacagac 4680
actctccctg gctttgtgtg tatgtcgggc cacacagatg cctggaaatg ttataaatta 4740
ccaggtatct ttggaaagga aatgaggtag gagttttgtg catgaggtgt gttcaacata 4800
cagcctcacg tccttttccg gaaccacctc tctgtgactt atcctgtgac gtcagggaga 4860
gtgtaatctg caacagtgac atgttttcaa agggcttaat gtgaggggga aaggattggg 4920
tttctgaaag tctggtctgc acttctttaa ttttgttaat aattaaaatg gatgtccccc 4980
taattgccgg ttgtccctgg agtgtgtggc tcagcactaa ctaaggaagc tgagctagga 5040
tttcctacag cgtgggcttc agaaacagcc ccggttagga aagaattgtc atttttcatt 5100
tggactctcg gggcagtgtt gctgtgagtt gatttcagtt gcagagtata aaatggtcct 5160
ggagggtttc ctggactgca tctaattacc tcagaaaggt tacaagatgt ttgtactcgc 5220
aaggaggagg caggtggggg agaggaagga cagtgggctg gagtccccca aatggctctt 5280
tgtgtaagaa ccgatatcca acaatgctca cattgttgaa agcagatccc accacctggg 5340
gacctgtagg tacatgtaag gttagggagg gaggctgaga agtctccgaa gttgtaggtc 5400
acactttgcc aatgcccctg ggtacacttt gctaggctca gagtttgcat gaggttcgaa 5460
tcacatatag agttgggaga cgctaagaaa aagaaaagaa aaagaaaaaa ggaaaaaagg 5520
aaaatgtctc aaggtgtgga gtttcaccag agcaagcttg ggaaatgcag agaaacccca 5580
gagccttgat tggtgggatc tctttatcaa tagtcactga acagtagtac catccccaga 5640
tgccttctga ggaccagctc aagagattta gttttcacca gtgacctgga cagaaagcag 5700
aaagcacagc tcctggcatt gatggtggcc ttggccatcc ccatccccag caagctgggg 5760
acaagggggt gcacagttct cagtgcagca aacacggtac cctgagatga atgttgcttt 5820
tggatggagg aggtggtgat gctggatttc ggcagggtct gtgctcactc tccttgtctg 5880
ttagaccaac attgccactg acatccaggc catcaagcta gaggctaggc tccatgctag 5940
gctctggtgt ctaatgtgtg catatgtgca tctctccagc cgccatattt gatgcagcca 6000
ggacttcagc taacactgag ttcagcttct gtctcctgaa gctttaccat ggaaggcatc 6060
cgtttgctaa tttagaagct cagtttagat aatgtctatt gggccggaca aatatgtaat 6120
caggaagttc ctagaaagag cctgtgcctc actactaagg agcccttttg accctctagg 6180
gagatgttat gttcagtcat gtagttctgt gcagtgtatg tagccatgca atgtatgtcc 6240
tcaccccgaa tcctatcctg tccgtgtgtc tctggacact ttctcaagtg gcagcagcag 6300
gattgggtca agtcagttga cctaagaagg cagtcatctc tgtaagattt tcctcggtat 6360
ttcagaatag aaatgattgt atccagctgg tcatccctgt gacaaaggac aacagtatca 6420
acagttgggg acttcggagg ggtggtcccg attctaagta ctgttctgtt gattcaaatc 6480
ctgaatgttc ccagtgtagt caagcttgat tactgccagt ctcggctctt actttcagat 6540
tccccctgac gttgtcacct gctctggtta attaagtcat tgttgacatt aagggaatct 6600
gtttacccca gcccagtagg agctaaaata aaagggcttt cccaaaccca aacccttaat 6660
tactttccca ccctctgcta agtgcaaagg gacggcctgg gggtggggtg ggggtgggag 6720
tgagggagta atttacatgc cttaaaaaac acccaccatt tcttgggcag tcttctgggt 6780
tgatgctgtt ccggattgaa gtgagccagc gaaaacctcg tgagtgtgag gtctacgtgg 6840
agacctgctg aagggttccc cccccctcac caccaccacc acagggtagt tcaagtcctt 6900
tgtcagagag tatcctacat gctgtggtct cagcccccca catttaattg ccagttagaa 6960
gaagagaaaa gaaatatctg cgtggtgcaa gtggatgatt taacaggagt cttgtgtttc 7020
ctattatctg cattttttgt tcctcagtgt gagtgtgaat atttaagagt tgactgtaac 7080
ttgatagttt cgctgaggaa caagggctta ttcttggtca attaaaccaa atgcaggcgc 7140
atgctgttaa acacacaatg aagtacacat tctttattag aatatagtgt atttcacaat 7200
tcatgggcaa ggaactgtgt ttaatattac ttctagagca aaaatctggc cagcccagaa 7260
aattggcatt tatataactc tttcttgctg gcttccactg atctgaatag agcaagtttg 7320
tgtgtgtgtg tgtgtgtgtg tgtgtgtgtg tgtgtgtacc tgtctgtcta tgttgcctgt 7380
ttatttcaat cagttaaaag actaaataac ttacttaaaa aaaaatagcc accccttctc 7440
agatagcctc taaagacttt atgctgtttt taagccttat ttttaaatta ttttaaattg 7500
gggtctgtgt aggcctgtgc acatgagtgc aggtccccct ggaggccaga ggtttttgat 7560
gctagcccgg gagttggttc tggaagtgaa tgtggatcct ctgcaagaac aatatagact 7620
tacaaaaact gagctatctc ttctgctcca atacagttca acattttttc ttttttcttt 7680
ttcttttttt ctgttgaggg tgagaattca aacaaccaag cattccaata tgagatgttt 7740
ccaatatctg atttaatgaa taatcacatg gttatgaaat aactggggta gtgtttaaac 7800
aggaagggga tgtttaatgt tcacattctc tgtggagtgc gtgcagtagc cccgtgcctg 7860
cagtagccct gtgccacact tatagacagt ttggctactt acatagttag ggtggtcatg 7920
aaaagacaac taagtccctt tcatcaggct ccgtcttaac ttttccattt ctgattgaat 7980
ccctgggatc gatccagcag ggtgtcttgt cttggtcagc agctaggagt tattttgggg 8040
gaagggatgc tgcaggctat tttacagata attatgggtt tcctgtgcag aactgtccct 8100
gtaggggctg gagcaagtga tgattctgtg attaagagca cttcctcctt ttgcagagga 8160
ccagtgttgg gtccccaaca cccacaggga agtactcctg attgtcttta tctccaggtg 8220
ctgggggcca gcgcctctga cctactgttg cctgcaggtg cccttctcat gtgggctgcc 8280
cttgccctta ctgtctttgt ggcttcgtag agaatgatgg gaaaaaattc caagatggta 8340
gtcccactgg tgactaaagg tgtttagtag tagctttttc ttaaaataca gttggtgagc 8400
aagatagtgg tgcacacctt taatcccaac actagggagg cagaagcggg tggttctttg 8460
agattgaggc tagcctggtc tacagagtga gtgccaggac tacacacaca cacacacgca 8520
cacacacacc ccagaaagct tgaagttgta gttttacgaa agtgtattta accgtcagga 8580
ctaactatga tctttctttt gggctggtag ctgatggttt ggtttttttt tttttagatg 8640
ggcatctccc acagcctggc ttgggatttg ccttgtagct caggtcggtc tagaactttc 8700
aatcccccta cctcaacttc cactcctaat tgtccggcat ccttgaagag catgtgtctt 8760
gattttctgt aattttgaaa aacttggcct cggattttat ggcttactta tctttatgtg 8820
tatctttatg tgttttgcct gcatgtatgt atgtgtacca tgtatatgtt tagtgcttgc 8880
agagaccaga agaggacttg cggtcccctg gaaatagagt tatctacgtg gttttgagct 8940
agcacccagt ggtcttcact tcccccgtgg ctctccagcc cttggattaa atgtggaatg 9000
tgctgtttgc ttgcttgaag ccaccatagg cagtgacagg ctttgtggac tttctacact 9060
ctgagaataa atgaaagtcc acttgcttgc tcttggctgg gtcaagtcag ggagctaaac 9120
tatcacatac ctcctcttta acttcttgtc caactaaaga atcatgaatc ccaagccgtt 9180
tctggacaga gagaattcca ggttatggtg accatgtttt atgaggatgt taaaaatagc 9240
tcctaaggag gatgctgaca gattcaggaa ggagaacccg gcctcatgtt tatttgggtg 9300
ttatttatgt agcatgttcc tgagacatct caatcctgag cactaaggaa gtcaacacat 9360
tgtttcctaa ccctggaact tgtttttcac ttcttatacc tgacagttta caaatactgg 9420
ttcccccccc cccccatgtg tggccaagtg ttttaaaggt atctaacacc gaaaatggcc 9480
aatttggtgt gctgttatag atcaaaagga gatctttgag actagagatc tctgtcaagt 9540
ttattctctt tggaaaccct tcaagttcac attgagagct gacagttggc tagccctaga 9600
gtcatgtggc ttgcttcaag ccgcctctcc cccattccac ctcaacccct tggactgcca 9660
ctaagactgt tgcttagctg attgtagcag gtaccttgct gaatgtgtaa cctgtataga 9720
ttatgtgttt cagatttaaa accactcagg tctttaaaga ctaagggatc tgatccgaca 9780
tttgacttaa aattttaagt agaaactaag taaagttgtt ttgaatagta tgtgttgtgt 9840
tttctggagg tacagtctca taggaaatcg cccttgggtg ctgagtttga atgtgcctac 9900
tatctacttg accttagtca agtgagataa cctggttgaa attccaagat aatatctgtc 9960
taaattgcac agattgaata cacactggac tgtagttcct ggcccagtgt aggcgcaggg 10020
taagtgctga tttcctccca ccccacacct ttgtcaaact aaataaaacc cacatctcaa 10080
agacctaata tgatgcttgc cttgtaatct ataatgataa atgtcagatt ttcagacctt 10140
aggccttcct ttatccaact ctttttttgc cctcgggttt ttgcaagccc cctggtgttt 10200
agacatgtga ccctttatct gcttacagtc taggtgttca aggttgactt tttttttttt 10260
tcttctgtta aggaagtcaa ccgtagccac ccagcacata gtgagaatat gtcatggtca 10320
tgggtatatt ttggcaggag agtcctctgt ttgaggtttt caaataatcg atgtaggcca 10380
gtgaagggtg gtagagaggt tggtgtgagc ttggttgggt gtgttgggtg tgagcttggt 10440
tgggtgtgtt ggatgtgtta tagtgtgctg tgccctgtcc aagccagtga agaacccatc 10500
caccattgca ggtgttgctt gtcttttgcc atcttctcct gtaatgccac catccatttg 10560
cctgccaggg gagctaggtg ctgggcttcc ggtgggctgt atggcaggga gttcacagaa 10620
ctgtgctggg gtccagacta gtggaagagc tggacattca tgtgcatggt tcctctaaga 10680
ggggcttgtg atggcagagg ctcagggtga gatcgtgtcc ttcaactcag tccttgggct 10740
aatgatggtt tccatgaaga caccttagct cctgctcttt gctccgtgcc ttgtgataag 10800
atgctgaagg tgcagatgct gagagcgcca ggcctttatt aagtgcctgt aagcggctca 10860
catgtgctag ggatgttgac aaattgcccc ttcccaacaa acaggcagat cccaggatcc 10920
catttcatga ataaaatttt tgcaattctt agagatgctg tggtttccgg acaccttcac 10980
agtgaccaca cacccaccct ttaggtgaac taattggtgg aagatggatt tcacagctca 11040
ttcctccttc ctcagcaaga ggatagatat ttgatggagt gttaggcacc cctcttgttt 11100
tttttttttt tttcacccct actttggact ttaaacttca gaggacaggc tggttggttc 11160
tgtttctcct tcacctcccc acacccactt ccttaagtcc tttgaagaag agtttcaggc 11220
aataaaaatt ttctagcact tatattctgt agttctggtg cgatgtaggg agttggtcca 11280
taccctctgc taccgtgggg accagtggga cacagcacag agtcctagac gctgacttat 11340
gctgagtcac tggagaaaag ctcagaacaa gaagggccac cttgctcctg cctgactgtt 11400
cctcatcgtt aggtcttcct ttcctcggat cctccagacc ttagcttcat tgagttgctg 11460
ttttgatagc atttcaagct tctcctttca gcatttcttc ctttttgcaa caaggtggga 11520
aatcaaaggc cacctggact ggactacctg gaactccttc aggctgtggt catgaaaagg 11580
acaggtgtgg aggcctttcc gggaactttt ttctccagag attagggact cacctatctt 11640
ctctccatct ctatctcctc ccctctcccc caggaggaaa aagaaaagaa aaaaattcca 11700
agaacgagaa gtgtggccct aggggcaaaa gaagccagga aatgaagccg ttttaaaagc 11760
cagcaaagct cactttggtg actttaaaaa aaaaaaaagt gacctctggt cgcagctggg 11820
tatggaggtg acagtgactg actaggatac tgatctttgt agaggtcatt tgtgaaatgg 11880
gtggggatgc tcagagacag caggtatgaa gtaaggcaag gtcactgctg aagggaaaga 11940
cccacccaca tcagcttccc tcagagctgt acagcctttg catataacga ccacttccca 12000
ggctggtaga gagaagatgg catctctaga tgtgcttttc tagtctcagg gtaattagtt 12060
ccctttgagt cagtttccca acttattggc tgattggttg acctagagtc tcatgtagcc 12120
cttgaacgtc caattcttct gtctctacct cctgagtgct ggaattacag gcaggcacca 12180
ctttagccag ttcccatctc atctttgttg tagaaaagtg ttcacccttg aaggggtggc 12240
cagtctgagg aagctgcacc gcgctgtagc ttccccttga cgtctctttc ctggcacttc 12300
actctgatgg ctttcttgcc tagccatcat ggaggcaagg aaatggccag ggctgagagg 12360
ccagaaaacc cctgcttctc ttgggcagag taatgatgac ttccctgcct ggcacagtga 12420
cacaccttgt cctcgggaag ccacaatgtt tgggcacctc gcctggatct tcctagactc 12480
agtggctgag tctgaaggga gccacttttc agatttgctt gctttctgaa agccttccct 12540
ccaggcaaag ctgaggtctg tggggcagga gaggaaggtt aaccatggtg ctgccatctt 12600
aatttggaac ttccaagcag atgtggcttt cagtcctcct ggatggcatc cccaggcaga 12660
ggcagagagt cctgtgtcca tccgtccgtc cgtccccccc aacgcaaaac actacagaaa 12720
agtgatcctt ctggctctgg cctacctttc taggtcctgt ggtgttaacc agctgggatg 12780
gtgtggcccc ccgctccaga acgatccctg ccctctcctg aaagcagctt tctgtgaggt 12840
cattgctgtc cagcaacttg cggaccattc ctccagcaga gattcccttc cagcttccat 12900
gcaggcctga gctaactgag ccccagcaac aggatcaaac ccattccaag aggaaggcca 12960
tctgttcctc agcctccagc tgctggccct tcatttgcaa ttggctggga agctttggag 13020
gggtcaggtc ctggggacac atctgcagtc tctgaatggt gttataactg gggtcctgct 13080
gagcagagaa aggccaagcc ctttaaataa acttgctgaa caataccccc ccaaaggtgt 13140
agagtcagag aagcaggagg cagctttgcc ctttagctaa ctcttaacct tggttttgta 13200
gccagggcac ttgaaaacta tttctttatt cagaaagtac ttaccaagcg gagaagggag 13260
gggctgctct gaacaaggaa aatgtcatat aggatttggg catcgatctg ccccttaagg 13320
gaattagagg gcaaatatct ccacttgagt gtatgccatt tattgaatat ttacctcagt 13380
gtcaaagagg tgagcttgtt ccagatgcag cttgtaaaga gccacaggca gcatgaagtc 13440
ctctcgaact tgcctctgga atgcagttca gccttgggaa caccagccaa tctccctagt 13500
tcattgcaag caggtcccca agctgtagct gctttaggtc ctgtggttct tgggcctgtc 13560
tgtagtttgg tgttagggcc cttatttcct gcatccgggg gcctatgata acttagccta 13620
atgctctagg gactttctat agggaccagg ctgtaatcgg gcgtgtgact tcattgagtg 13680
gatgaatggc agttatgcag gtgttgtcca ccttggtttt attgacaggg tctctccctg 13740
actgggaact taccacatag gttaagctgg ctggtgagca ggctccctgg agatgtctct 13800
gtctcattca ctacatctag gtttttttgt ttgtttgttt ttgttttttt ggtttttttt 13860
gtttttgttt ttttgttttt tttttttttt ggtgggttct gaggaattaa agtgatgctt 13920
gcaaggcaag aactttatgg actgagctat ctgtcagcta tctgtcagcc cagcccccag 13980
aggtacaata acctgtgggc cctttggctc actggtttct tgaagcaggt attaggcctg 14040
gtctgtatga gacggagcct tcaggacctg cagatgttta gttccacttg agaactttgc 14100
aggaatcctc gctcagggaa ggcgtgtata taagatgtga cagatttatt cacttgaaag 14160
aaagccctgg tttggagtca gaggcatgca agtggatatt ctcatggggc catcttaacc 14220
ctctgctgac tcatctactg acctgttaga atcaggctgt gacccataaa accaagcccc 14280
aggtggctcc cggctgggtg aatatgtctg cagagcttca ggtagagcat ttgccctact 14340
gtgcacagag tgtttcctct cagtgtgctc ctcacatcag ggtcagtgag ggacttaaca 14400
gaaagccttg ggttccctct ttgtgccacc gtttgccagt agctggcctt tctggtgtct 14460
cagggacaga gggggccgtt cagtacgacc acgttcattt tggacagcag caagccttaa 14520
gctttggtct ttggacaaag ggtttctgag ctggcggtgc catcctcagc tgggagccca 14580
ggagcaccca gccagagcac tcaggccatt caggaggctg accctgggtg gaggtcctta 14640
tgcacgataa acctcggtca ttgcgttcat tttccttcct cccaccttct cagaatgtct 14700
ccacgagaca gttgggtgag aatgaatatg tctgcgtgtt ctacgtggat aaaacatagg 14760
ctgtgacatc atggggatgg ggtgacggca tgtgtcataa tgggaaactg gaaatcttat 14820
agaagagaca tttaggtttt gaaaactgca caggagcctc tcaggtagag aaacagttta 14880
ggtacaggga acagggacag gggacagagg acagacatac cgtctggcta ggcaagccac 14940
catgtgaatg aacgggggga agaggggaaa ctgggggaat gtggtactcg gtaatgatgt 15000
aaagatttcc tagagagaca ctcattatag gttgggtaca ttccattcag gcctttgcct 15060
ctttaggagc ccctatagca ttccttgatg ttgtagctac gaggagcagc aacctggccc 15120
caaaagagat tcaacagact ttcccagtgg cttttgtctg cctgtggatc cagccctaga 15180
tggcaaggtt tgggactagt gtgtcctaag gagtcctgca gaccttgggg agcctgtgct 15240
ttctcttgca agtgcgcctt caggacgcag gaggcctggg cctggctggc cagacctcgg 15300
atacagacgc ctctttgtgc ctctgagcca cgagtgctgg gtactttgac ataacttgta 15360
atgccagttt ctacttcctg ggtgctatgg aatctaatgg ctgagttctc tgggacatgc 15420
tctctcagaa caaaaggttc cattttccag ttcttgctca agcaaagcat caacagctag 15480
gggatttgtg tagctgcgca gatttgatct ctcctcgcgt cttggtggcc cagtgggaat 15540
ttcagtcttg ggagtgtatg aattgagtgc gtatgttgtg accaggcgcc tctgtcattt 15600
ggacactatc gtcgcatgac aggattgggg gggagagagg tgcgggtggg taaggagcta 15660
agctgccgcc gctttgagtc taggtaccgg gtgacacaat gattcttagg cccttttgcc 15720
ttttctgcat ttttattttc tcctgggctc aggcataatt tgtttcaaac tggagggctg 15780
tccaccctgt ttctcaaagc caaacctaaa ttacgagggg tgtgcctaaa tatgaaatat 15840
gtaatggttc ccatattgaa acatttgcta ccttctagtc ctctccgatg ggcggcttga 15900
gccagcccag agtttctggg gctgtccgac tactgcagct gaggtagcta ttggtggggg 15960
tgatgctaac aggaacgtgt ctgaagagat gctccagcta ttggttgtaa acaaagagcc 16020
tgggcagcct gctcacctct ctcctctccc tagcctcacc atcctgccct cccccacccc 16080
ctttttttat gcagccgtat ttcttgaggt tgaaaacttc catctttgtc ctgtatgggt 16140
gttggccccc tcctctcttt caggatgagt tgtacagagg ccttataagg atgctatcag 16200
gatgtgcaag ttggcacact ggtaaagggg aaactttgaa agagtaggag ctgcagcagc 16260
cagctctggg atgtcgtctt tgtgtctggg gacaaggcta gctaggccgc tcttcttcct 16320
gactccacca aaggacccca ttgtccttaa tatcttttat actgaactct ggtgccagct 16380
ccatgctgac agtgccatgc aaaaatatgt acaggagagg ctcttccaag gtcccagtct 16440
tgccaggtgt caccggtttc taaaagccta ggtggacatt ccagtaccat gtgccctgca 16500
ttctgggtgt ccttgatttg aagttacaaa gaacctttca agttctgtac cctgttctat 16560
ggccagtgac cacagctcac caggcccatg gagtggcagg gcatctttat ggctcggagg 16620
gcagagtggg tcaacccttt gccactcacc tgttatgaac ccagtgtcct gtgactttgc 16680
agtgacattt ggcagctcga tccccattct ccgtcaagac ttttggcagt cctgtggctt 16740
tgctgtttat ttgtcttgta ttagatggca ctgtctggga gaacgccggg ccatggtatt 16800
gtcctcgtcc cagggttcct gtgcagtcct actgggctag aggagtgctg ggaggtgggg 16860
acagcttagc tgggcagccc cgtcccttga caggacatgc ctgctgaagc tgtgccttct 16920
cctccaccct cctcctcccc tttccctcct gcctcctctc tcctcttctc tcctcatcgt 16980
cccctttctt ccttgtacgt ccctcttctg ggtgaatcta ctctgattct gctttgtcct 17040
ttccagaaga atgtgttttg ggatctgatt gtgccctgtg gggagccccc ctaagtgggg 17100
ctgtttgagg taccccactg tatctttaac tcagatcctt tagacgctga ctaaagaagt 17160
cattctgggg acaccctaga agtggcttgg tgtggtgcga ggtgatttgt tgccccagag 17220
gtggttggca gaagtggctc cttctccctg cgatggtggg aagctgccat gtgatctgtg 17280
ggagacgatt ggccagggca ggacttggac gcccatctgt tctctgtttg cagttgggcg 17340
ccatttcaga aaccacaggg gaaaagttta taggcaaaca tgataaaaag tgacagtctg 17400
aagtgctgct atcgctggct tggcaactta aagcattacc tgaagcagct tctaacttcc 17460
agacgctcta gctgcaacgg gaaccccaag atggccatcc tgtgggcgct ggggaagatt 17520
tcgtttgtgc gcagtgaggt gtcttagtct cggccccatc tacttcttga aggctccctt 17580
tctagggtga cttcacgaat agcaaggtgt catacccctc ccccctagct tacaggaagg 17640
taaatacaag ctgtcactag tgacatcagg tgaggtccca cccagaggtt gtgacctact 17700
tggatctgta gaaggacttg gagaagggtc aggaagattc tgcctcagtt tccctttcgc 17760
ctgggtctga agcccctctc atttctaaat ccctattacc tcccagggaa tagtggcttg 17820
aggaatcttt gggaagaaag agggctcatg gcagggtaac agtcagccac gtgtgcggaa 17880
ttttaaagac agaatctcac tacatagccc aggctggctt tgactgccct cactcagtag 17940
cttagtaggc ggtaaactct gaagccgatg caggctttga acttatgatc ttcctaaccc 18000
accatgtgcc accatacccc accactgttg atgttttcat tattggattt gatgctgtga 18060
aggaacccct ttatcttttg gtttgtttgt tttctgagta tcagagtagt cagctcactg 18120
aaaatatgac cagtatatag gaaactgctg gcatgtctca agggtttgta acctgtgggt 18180
agaaacacag ctaagcctcc acacaggaga gcctctggcc actgttgtgt ttgtcgcagg 18240
tagaaacagc tgagcagagc cttcccagaa agtaaacatg tcgccttgtt tgttcagaga 18300
gtttaggtaa caatgacagt gtatggccca gctcccatgc atctttccaa gtttccattt 18360
aattatgaaa aatgtatgag aacagacttt ctgtctgcgg aaacccctga aagagcattt 18420
ggtgcctctg ctcgtagctt ctggaacttt ctccccactg tgctgtgcag agtgcagagg 18480
gtggaacttg gaagcgtgtg ctccggtaag ccacggcatc agaaatgtta aatccaggaa 18540
atgttgatat tgctataaaa gagactgttt ggatttccca gggagttcct tgtcctgtgt 18600
caattgtcac gtgttacaca gagcagcttg gcagagtcgg gcaaggagtg gcctgtgtgg 18660
agaggccatc tgagtgggag agacaggtgg ggtgtggcga gcacagtcct tggtgccttg 18720
gccccttata ggacactatg aggtggttac aatatggagt tgtaacacca caggactctt 18780
aagagcaggc agtgattggg aggagccagt cccgaagcct ggtgaaggat ttaggcacag 18840
aagagaagcc tttagctctc aagtctccag ggctaggcgg gagcaggatg gcatcttttc 18900
agcatgccac ttgggttcca tgttcttagt gccctggtcc gtgatgtatc tcatgtgtga 18960
tccatttgca gggagctacc aactgcatct gtgtcctggg atgctgttgg gttggctttt 19020
tcttctcacc cccttattat aatcctgctc tctcctgttt cttccccctc tacggtattt 19080
gaccttctcc tttctttctg ccctttcttt tcctgtattc acccaatctc cctactccct 19140
aggatcacca aggaggaggt aacattgctt tctgctgacg ctgctgaccc ctaagtgggg 19200
cctcttgaga gaaggtcact agggagttgt gcattctgcc tatccaaggc agataccttg 19260
gaggaggcct tggcgttagg atggcttgat ttcatagata cttatctttc tgacgtgctt 19320
gcagatgata ctctatactg tccccaaagc cagtcgtctt cctgggaaac tagagagttt 19380
cccattttgc ccatgccaac ctggcctcac cattgactga gtgagatggg agcccatcag 19440
tgaaagtctt gagattaaaa atccagttgt ttctgaagac agtggagcac cacagttata 19500
gcttgagaac aacggcggat gactgacatt ggttgtggct ggaagatcaa gtatacagcc 19560
ggtggctccc aggcacctcc cgtataatgc cttcttgtat gttggtggtt ggggatcttg 19620
tggctgagag gctatgcagg gcagagagga aatgagccca gtgtccctgt acccagggca 19680
gtgtcccttt accaaacatc cagtgtcctg tcctacctga gacccctctt cttctgtgtt 19740
cctcacagca tggtgataca gtatggtaga attggtccag catggtccag tagtgcagct 19800
aaatttcaat gagtcttggt cctttgttga tgttgggtgg aggaagggtt tctccgtgga 19860
tggtgtagac tttaaggctc catcattctt aacattgtac gaatctttgg tttaaagatg 19920
ttaagaccag actggcagat ggtatgagac ttaggttcaa atggaacccc cctttcccct 19980
ccttatttct cttcctcatc cttaaaaata tgaacccttt gttttacttg ttgttgctgt 20040
tgttcattat tctcagtgtt agtgtgtgtg tgtgtgtgtg tgtgtgtgtg tgtgtgatgt 20100
gtgagtgcat gtagacaaat cagccatgct gtgtaaaggt cagagtgcag tctggtagag 20160
tcaattcctt cttcctcttc taccttttta agggtttctg ggaaccaaac ttggctttag 20220
gcaagcatgc cttcctcctc ggagccatca ttccagaact gcctctccct cattcactta 20280
gccactcagg tcagctgcct cttggtttaa atgggcaggg aaaggcctga gctgagaccc 20340
ttccaactga attctcaatg tctttcaaac ttggttctgt gtagtgccac agggtgtctg 20400
ctacttcttg gaggagactc ctatcccctc ctgcaacaga agctgaaaca ccttcggtga 20460
ggggccacgc tatcagtgtt tggggcttgt agaccatgag attttttttt ttttttcaat 20520
gactctggtc tgcccgtata acacaaaagc agccctagac aatacatacc caagtatgta 20580
ttgagtatgg cactgctcca agaagtcttt gtttacaaaa gcaagtggct gacttgtccc 20640
tcaggccatg ctttgctggc tcctgctgcc cacggggcct tcgcccaccg tttccacatg 20700
aacggctacc tacctgcctc acccttaaga ctcccttaca cacttcctat tttctctgag 20760
gtttttcttc actttcattt gccccactgc aatggagggt ccaccagggc agggatatgg 20820
ctaccctcct gttgcttcct gagtgtacag aacaaagctt ggcctgtggt aggtatgcaa 20880
taaacagagg gcacatgaga taaacaagcc cttgaaacct tacctggctg tcagttgggt 20940
ttgctttctg cccctgcttt gtgtgtgtgt gtgtgtgtgt gtgtgtgtgt gtgtgtgtgt 21000
gtgtggttgt ggtggggttt gtgttccatc aacttctgtt ttcttcccta tgtgggtttt 21060
acttttgtgt tcctgtactg ttaacatctg tgcccctctt ggctgtgtgc atttgaagtg 21120
ggggtcccct gtgagaagcc tcaggcccct tgtgttggct gctgctgcgc ttcttggacc 21180
agatgtttat taaatagcag gactgaaaca tgaattgact gtattctagt cgtgagagaa 21240
tttgttcttt ggagtgggct ctgggcagaa taatcgcctt gtgatgctgc tgcccagatc 21300
tggaacctgc ccagtgtggg gaaggaagca ttgtgttttc caggcttggg actctgggta 21360
ccattcacag ctctcactgt gggatgaaag cttatttcat gagccctcgt ggccacctct 21420
ggccctgagc aaggtcagga gcttccttcc tctcactttt tttgggagaa gctgggaggt 21480
tggatcatag ttggtttcat tctgccctgt ctttagagga aggcaatgtc tgccttctct 21540
gtgtacagca aagatatcca gtgtagggat gggcgtgggc acaatgacct atcagaactg 21600
agctttctga tgtgaaggtt tcctctggaa gtcaggacac ccataggcaa tgtgtctatt 21660
tcagtgtttg gaggtatagg gtaggcagat ggactttaga gtgggagaga cccctttagt 21720
ttccagccag gtgactgatg cagagtgatg gatcatggag ggccatggtt gacctgggca 21780
tcagaggagg aactgggcta aacgggagtg agagggagga ccttgtgttc ataaagaaga 21840
gcaggatgct tgacggagat cagggactct ggggtagtgg tgggttggtg ggcaggatgg 21900
atctggctcc accagtggaa tgctgggtag tagtacatgc tacttatcca gtacatgtag 21960
tctatgtgta tacatggctg gtttatggta tagggccatt aagtgccagt aattccttac 22020
ttttctttct ttggacgtta aaggaccccc agcatctgtc attttgagga agatggaatg 22080
tcccagctcg cccagaacag atctagctca gtcctgatcg ggccccaaga gcacataaaa 22140
acaatcaagc caatagctgc ctcttcccaa gtggtgaaga gtaattttgt agatgggtct 22200
gtttgcccct tgaatttgag acattttatt tatattgaaa agcttggttc tgtgagaaca 22260
ggcaaagtga aatatgaata agtagctaag tcagtgtgag aacgtgtatg tacgtgtgca 22320
tgtatcacat atacagtcat gctggatggc tagcttggaa atcaacttta cagttttctt 22380
gtggattttt cttccacttt agggtttggt tggtttttaa agccctattt ccagtatgtg 22440
gaaatgagcc aacccaggac agcttccgct ggatcgtgga cagcttctat ggccgtcgac 22500
gtgtacactc gagataactt cgtataatgt atgctatacg aagttatatg catggcctcc 22560
gcgccgggtt ttggcgcctc ccgcgggcgc ccccctcctc acggcgagcg ctgccacgtc 22620
agacgaaggg cgcagcgagc gtcctgatcc ttccgcccgg acgctcagga cagcggcccg 22680
ctgctcataa gactcggcct tagaacccca gtatcagcag aaggacattt taggacggga 22740
cttgggtgac tctagggcac tggttttctt tccagagagc ggaacaggcg aggaaaagta 22800
gtcccttctc ggcgattctg cggagggatc tccgtggggc ggtgaacgcc gatgattata 22860
taaggacgcg ccgggtgtgg cacagctagt tccgtcgcag ccgggatttg ggtcgcggtt 22920
cttgtttgtg gatcgctgtg atcgtcactt ggtgagtagc gggctgctgg gctggccggg 22980
gctttcgtgg ccgccgggcc gctcggtggg acggaagcgt gtggagagac cgccaagggc 23040
tgtagtctgg gtccgcgagc aaggttgccc tgaactgggg gttgggggga gcgcagcaaa 23100
atggcggctg ttcccgagtc ttgaatggaa gacgcttgtg aggcgggctg tgaggtcgtt 23160
gaaacaaggt ggggggcatg gtgggcggca agaacccaag gtcttgaggc cttcgctaat 23220
gcgggaaagc tcttattcgg gtgagatggg ctggggcacc atctggggac cctgacgtga 23280
agtttgtcac tgactggaga actcggtttg tcgtctgttg cgggggcggc agttatggcg 23340
gtgccgttgg gcagtgcacc cgtacctttg ggagcgcgcg ccctcgtcgt gtcgtgacgt 23400
cacccgttct gttggcttat aatgcagggt ggggccacct gccggtaggt gtgcggtagg 23460
cttttctccg tcgcaggacg cagggttcgg gcctagggta ggctctcctg aatcgacagg 23520
cgccggacct ctggtgaggg gagggataag tgaggcgtca gtttctttgg tcggttttat 23580
gtacctatct tcttaagtag ctgaagctcc ggttttgaac tatgcgctcg gggttggcga 23640
gtgtgttttg tgaagttttt taggcacctt ttgaaatgta atcatttggg tcaatatgta 23700
attttcagtg ttagactagt aaattgtccg ctaaattctg gccgtttttg gcttttttgt 23760
tagacgtgtt gacaattaat catcggcata gtatatcggc atagtataat acgacaaggt 23820
gaggaactaa accatgaaaa agcctgaact caccgcgacg tctgtcgaga agtttctgat 23880
cgaaaagttc gacagcgtgt ccgacctgat gcagctctcg gagggcgaag aatctcgtgc 23940
tttcagcttc gatgtaggag ggcgtggata tgtcctgcgg gtaaatagct gcgccgatgg 24000
tttctacaaa gatcgttatg tttatcggca ctttgcatcg gccgcgctcc cgattccgga 24060
agtgcttgac attggggaat tcagcgagag cctgacctat tgcatctccc gccgtgcaca 24120
gggtgtcacg ttgcaagacc tgcctgaaac cgaactgccc gctgttctgc agccggtcgc 24180
ggaggccatg gatgcgattg ctgcggccga tcttagccag acgagcgggt tcggcccatt 24240
cggaccgcaa ggaatcggtc aatacactac atggcgtgat ttcatatgcg cgattgctga 24300
tccccatgtg tatcactggc aaactgtgat ggacgacacc gtcagtgcgt ccgtcgcgca 24360
ggctctcgat gagctgatgc tttgggccga ggactgcccc gaagtccggc acctcgtgca 24420
cgcggatttc ggctccaaca atgtcctgac ggacaatggc cgcataacag cggtcattga 24480
ctggagcgag gcgatgttcg gggattccca atacgaggtc gccaacatct tcttctggag 24540
gccgtggttg gcttgtatgg agcagcagac gcgctacttc gagcggaggc atccggagct 24600
tgcaggatcg ccgcggctcc gggcgtatat gctccgcatt ggtcttgacc aactctatca 24660
gagcttggtt gacggcaatt tcgatgatgc agcttgggcg cagggtcgat gcgacgcaat 24720
cgtccgatcc ggagccggga ctgtcgggcg tacacaaatc gcccgcagaa gcgcggccgt 24780
ctggaccgat ggctgtgtag aagtactcgc cgatagtgga aaccgacgcc ccagcactcg 24840
tccgagggca aaggaatagg gggatccgct gtaagtctgc agaaattgat gatctattaa 24900
acaataaaga tgtccactaa aatggaagtt tttcctgtca tactttgtta agaagggtga 24960
gaacagagta cctacatttt gaatggaagg attggagcta cgggggtggg ggtggggtgg 25020
gattagataa atgcctgctc tttactgaag gctctttact attgctttat gataatgttt 25080
catagttgga tatcataatt taaacaagca aaaccaaatt aagggccagc tcattcctcc 25140
cactcatgat ctatagatct atagatctct cgtgggatca ttgtttttct cttgattccc 25200
actttgtggt tctaagtact gtggtttcca aatgtgtcag tttcatagcc tgaagaacga 25260
gatcagcagc ctctgttcca catacacttc attctcagta ttgttttgcc aagttctaat 25320
tccatcagac ctcgacctgc agcccctagc ccgggataac ttcgtataat gtatgctata 25380
cgaagttatg ctagtaacta taacggtcct aaggtagcga gctagcccac cttgccttga 25440
gaatggtcgt cgccttttgg ttcctttggt tgtgctatga tgcgtcagtc tggtgtgcta 25500
actctatggc ctgcttatct gttcctcctc ctgtgatctg caatctagcg cctggaagag 25560
aaaaggagat tacagcttcc ccagactacc tggagatagc tatttactgc ataggggtct 25620
tcttaatcgc ctgcatggtg gtgacagtca tcttttgccg aatgaagacc acgaccaaga 25680
agccagactt cagcagccag ccagctgtgc acaagctgac caagcgcatc cccctgcgga 25740
gacaggtaac agaaagtaga taaagagttt gaagaaattt actcccctcc cccacccagc 25800
cagctcttgg atcttcttcc ctctgatttt ccccctaact tctgtgagct ccagaactgc 25860
aggcaattct aatctgccac tgtgtggagg ttcagtcagc ggttgggact aaagagcatt 25920
aagtcacaat gctgctctgg gcttggtagg ctggctctgg ttttaaagga caagagtgtg 25980
aagactggag ctgcccagtg ggatgggcag aggaggccat gccctctgcg cccctcaagc 26040
tcacggctcc tttgggagaa caagcatttg gtctggctcc attgcttctg tatgaggcca 26100
gatgttcggg ttcaagtttt acccttcata ggaaagagag tttaattttc tttgatttac 26160
tattttaagt agagatcaga aacagaggat ggaggtatac ctgaactaat gcttgcataa 26220
aagtggtctg tgatgtcttc taaactgggt tttggctgat tttgtctggt ttttaaaacg 26280
ctgtatgcgt atagtttatt gttacaggtt tggctaggga ttcagtgata ggatgattgt 26340
gtgtgtgtgt gtgtgtgtgt gtgtgtgtgt gtgtgtgtgt gtgtgtgtgt gtgtgtttgt 26400
atttaggtta taagtacatg tgtgcaggtg cctgtggaga ccaaaagaga gtgtcaggtc 26460
cactagtgct agagtttatc agcataggtg gtaggaattg aactctggtc ttctagaagt 26520
gcagcaagca ctctttaacc actgagccag ttctccagcc cccagatacg atgattgcta 26580
tgtagaacag ggagaaaatt acctttaacc ttgagcttga tctttgatgg ctggctttgg 26640
gggaggtaag gcaatagaac cttccctgtg ccataaaaca aagcccttca aaggtggata 26700
aggaaaaaat gcttgacttc tgtacttgct cctggattcc aagagccagg catgtgtggg 26760
tgtaaatctt tatgataaga ttcggaactt gattctgata agattgtcac tatttttttt 26820
aaattagcaa tggaaatgaa caacctggcc tgtgctatgg ggaggtgcat cttagtgttt 26880
gttaaaactg catattcatt agtttcaacc ctagaaattc ttatttagta cttcttgaat 26940
ggatctgtaa gagtctgcat tttaaacact ttctcgggtg atactgtgta ataccttaag 27000
aatctctggg ttcaacccaa ccctgccttt cctgggccct ttctgtggac aaggtgggaa 27060
ctagcaggtc agtagtggct tggacacagg gccttggctg ttctcaacct agcttcacac 27120
tacaggctga gcaggtcctt gtcaacgtcc ttgaagcctc gtttccaaca ggtgtttctt 27180
gcagagggtt aagatataat ttggagcata tgtcagatgc agcctttggc cagctgttga 27240
atgtggagtc aaaaaggctc agttgggttc cttttaatcc tgagaatgct gtgctacttt 27300
gagtgacacc actgtcattt gtgggaccat agaagctaga tggtgctgaa gttaaagttg 27360
gttgcctgaa tgagttgctg gaagagtcct taataaaact cttacctggc tagatagtgt 27420
taaggcttca ggctgaatgg ccactccttt ggccactcct ttggctactc cttcacagcc 27480
tctcctgatc tcttagccct gggccattct taacatctgg actctggtct agggagttta 27540
agtaaaggga gcaatgtcct gtctcatttg tttttataat agagaaaaga agtaaaatcc 27600
ataagttgag gtagataggc cattgaccct taattatttc atcatttaaa aaactgatgt 27660
gtgtgtgtgt gtgtttatac atgtgtgctg gagctcttgg aaagctggaa gagagcactg 27720
gatccctcag agctggaggg ccggtagttg tgagctctct gatgtggtgc tgggactaga 27780
actcgggtcc tctgcaagag cagcaagcgc tcctaatcac tgagccatct atctctccag 27840
cctgcatcac ttttaaagaa aactctttct atttctccat tttccatttc catttccatc 27900
tttttacatt tatatattac atttatatat ttatgtagct tgggcacgtg tgcgtttgtg 27960
tgggggcatg cacatggcat agcaaggtta gtgaaggtcc ttttgaaatg gtggccaggg 28020
gacaacttac atgggagcca gcacctttct ctaccacgtg attcccagga atcaaacagg 28080
tcaggttcag tcaggtcatc gtatctgatg accgattttg ttgactccat cgcttttaaa 28140
gaaaaaaaga attaacacct attacagcgc tcttcctttt gcttcatgtg aaaaagacag 28200
aggccctgga gttcccaggc acatggattc agcatgtctt cctttctgtt tgtccaactg 28260
agtttcttca ttttctgtcc acctaagctg tccattttgt ttgtttttat attccctgtg 28320
tgaccggagg gaaaagttgt tttttttttt tttcatttac ctccctttct tcttgtattc 28380
attgttattt actgagtgca cagtttcttt tagtgcatgg gcctaaatca ggactcttgg 28440
gctgggagtg tggctcagtg ctggcatgct cgcctagctt gtccaagcct cagtatcacc 28500
aaagaaaata attaagccag tttcgtgtca gagaaagcct gcccatttgc cactggctgc 28560
aaggttagtg aaggtccttt tgaaatgttt cttcatgctg acgctggata acaaatgtgt 28620
gaggcccagg ctctctgcat gaggaagcct ctgggagata aatgggttga aaaggtactg 28680
ataatacccc agcatttcct agaagtcatg gggaagtatg gtactaactg cctcttccca 28740
aaggatttcc caaagcttag gccactggga ggaggaggag gaggaaaagg aggagaagga 28800
ggaggggaga tgcttatcat gagtctggat aaagagggtt ttgggctgta gctggaggcc 28860
tgcagatagg ttaatgacag agtgaattcc tcagggatgc caagcatgcc ttacctggcg 28920
acagatgagc ctgtaatcag atgtctggag gacgggtgct cccaggcact aagaggctag 28980
gctttatttt gtgtaggccc aagcttctat atgatgcagc atccatgccc tggcccttgc 29040
ccaggacggc gaggaggcgc atagcctctc tccattcact ccatctttgt ctttgtttta 29100
gaacgagaaa agttggtttg tttattcatg ctgttttttt ccatgtgcac aagcgcgtgc 29160
tcggaaagtg tgtaggtgtg tacagaaagt gtgctgaggc caaatgataa ccttgggtgt 29220
cattcctcag gtgccgtcca ctccttactc tttgtgtgtg tatgtgtgtg tgtgtgtgtg 29280
tgtgtgtgtg ctaagtttct cacctgcttg cctgaactag ccaagtaagc taggttatct 29340
ggtctgtgag ccccagggtc ccaattgctc ctccttctcc tcctctctgt taggattcca 29400
agtgtcggct cccaagcctg actcttcttt ttttttctga gacagggttc ctctgtgtag 29460
ccctggctgt cctggaactc actctgtaga ccaggctggc cttgaactca gaaatccgcc 29520
tgcctctgcc tccaagtgct gggattaaag gcatggacca ccaccgcccg gctttttttt 29580
tttttttttt tttttttttt taatatagct cctagggatt gctctcaggt caaggaaggc 29640
aggcgttttg atccttcttt ctccttgagg tttgcttccc tgccctgaac ttgtttaaaa 29700
caggcatttc actttaaaaa ggtagggtct ttttttttgt gggtggaggg ggtggggggt 29760
gttttctgta tcaaataaat tctttatagt ctttctagta aacattaatt ttgggagaca 29820
ttgtgcttgg agtaagatac gcaacttttt ggtgggacag cctggtaggt agcctgtggg 29880
atctctaagg aggagtcatc tctctcaccc aaggctagga ctgggcactt tgtaagcgct 29940
tgcgcacttg cctctacttc ttggtaccta gtgttaaatg gcaatagtca gtctagagaa 30000
gggcaccttg tgacccaact ggaccatcag tggtcactgg gcagtggtct ttgtgtactt 30060
ctgagtccaa gtggaaagat ttgccttctg tgatttccac aagtcccttg ttggggaggt 30120
gggccgtatg tgagtgcaga gcggggtgga ggaagccttg ttctgtggag tgcttgtttg 30180
tggaggagct ttcctgggta ggttcagctt ctttctggag ccagaagttt gcttagggca 30240
agatggagat ccatctgtct gtgtccagat gagtgcatag cctacccgat cccccagtct 30300
cacacaggac tgtagtgagt ttgttcccag cctcagccat tgacatgggt agctgagaaa 30360
accagagagc aatttcataa tgcgtttgag acccatggtt attcagggtg ggctgggggg 30420
aacacttaat tccagagctg ttctcagggc aatgtattcg tggtcttaga gtatatgaaa 30480
ctcagtgaaa gtgagtgctg actgcttagc atcccagcac cgtgacctgg aatctccatc 30540
gtacgaggtg tagtcgatcc agagttgcag tgtaccggtt ctgggaaaca tttgggcagc 30600
tggatagttg tggatgaccc gagtggagtc ttcgctttcc tagggatcga tcgctttcct 30660
tgtccccagt ttggcctgtc ttttctctca gcccctgaaa gacatgctgc cttggctgag 30720
atccacccta gacttttgct gatgagctat aagtaggttc agaacacctg agtcaggtac 30780
ttttactgtt gtgacagggc attcaagagt ccagaggagg tagaagctgt ctaaggggca 30840
gtgtgagcaa ttacctagat tttgttatgg aaggaaaaac aaaacaaaac aaaacaaaac 30900
caaaaaactc cactcccaga aactctctga agcttggtgt ggtgcaggtt tttctgttgt 30960
ccatagaggt gtgtggggct agacttaaga tagaacacac tggccctctg ttctgatgtg 31020
gaaggctcca tctgctgcct gggagtcgga gggtgtctca agtctgctgt agtccaaggg 31080
catgtgtcaa ttctcaggaa taaagacaaa cttgactcac ccttccccgt actgtctttg 31140
cttccgcctg cgctgttgtc tgtgaggtcc cctctgaatg ttcagcttca tccagcataa 31200
agggagacgg ctatgacttg gtggctcttt aaaaagaaaa ggggagaaaa cccacttcct 31260
ccgttaatct cccatatgta ccgtggaaat atatgaaaag cacatttagt taaaagcttg 31320
atttatggca cgtggtaaag agatcccggc atgtaaggct gccgaattgg agactgtgaa 31380
gagtgtgcgg ctttctaaaa accgcctgcc aagatttggg gtggggaatg ggggtggggc 31440
ggagcaacag tttactacag tgttagcgtt tattgtttat aagtgaactt ctaacagtgg 31500
gatgttttta agtgcgttga aagggaaact ccaaaatgga agtttctaga ttaaggattg 31560
agaactatct gaggagggaa gttataagta caagagaaag agaagaaagg aagtctgtaa 31620
tacagtggtg tgaggaacct tccaaggtgg gcggtggggc cacaattcag agggaaggag 31680
cccctgaaaa gccaggctcc tccagggacc cctgctgggg attttgccaa gccctccaga 31740
caggttgcct ttctgaggag aggcgagtga agagaaagcc agtcatgctt tatagcccca 31800
gagaggattt taaaagtata gtaaaacgca tggaggtaga attaagatgg acctctgtag 31860
acagggagag cagagtgtat gctcagagac tttgcgtatt ttcttaccct ttcccactct 31920
gggtgttttt tacaaaggta ttttccaggc ttgtacattg aacctgaatc tgcactgtgt 31980
attgaacaaa attcccacac atgaaggcag ttttacattt tgataccaat gtgcagcaac 32040
gactgccaag gttttttttt tttttcttcc gtattagttt agtttttttt tttttttttt 32100
tctccccgtt ttccattttg aaaatgttgc ccttaaaacc ttgtggaggt gctctgttgt 32160
ggggtgggta tgcgtatggg aaacttgcac cccaggcctg tgctgtgcat tctgtttggg 32220
tcaaaggtcc tccacagagt agttgatgtc agactggatg gtaaatctct ctgttttgag 32280
gtaaccccta agtcatggtc accagcggga ccttgctgct ctatggtttt cttcttctcc 32340
tctaattcct acattaaaaa tatatatagt cttgcttact ggaactccag gctatcctgg 32400
ctggcagttt agggtcccat tttgtaaatc agactcgcaa ttcaggtgta tgccatctaa 32460
aatcagaaca aactcacctt gtagagcaga ctggtgagct atggctgtcc cagctcagca 32520
ataagcactt gatgctgtct tcattctgtc ctgctaactc tgagaccacc tgagactcac 32580
atagaccccc ggaatctgac cttgacttca cggtaccatt gaccaggatg tagcctgcca 32640
gggcatcttg gccctgggtg atcaccaggt cacacattga aggatgcgga aacatcacaa 32700
aacagcctgg ggtggggggg acaaaaaaga agtgccatcg ggcgtcttgc tagtttctaa 32760
actgaagtct gcataattca accctgtgcc ttcttttcct gctgttcata tttattttat 32820
tccaaatgct attttggcta aagaaagaat gtctactaaa acacaaagga aacacaagac 32880
cagggtaata aaatctatat gatgtagaaa gttctagaat aagacctgtt tcctaccttg 32940
ctccctattc ttgatctctc actctctctc cgaaggtgac cactgctaaa tccttagata 33000
tctttccaga aaacatttcc tgctttgctt cccaagtctt gatctctctc cccaaagggg 33060
accatggcta aacccttaga tatccttcca gaaaatgcct gtggtcacaa cccatcctgt 33120
aagcctctat gtgctgagta ctgactccca aggacaggcc acagaagctg cgatgtgcca 33180
ctagcctctg gccattacca tcattcagaa ctgtggtctt ctgagatttc tcagcatccc 33240
ctcctcactg gtcttagcac acagtgggtc ctaacaacta agctaggaac tttagggtcc 33300
agtgatgcag aggcaagctg atgatggccc tataaagagt atcctggcta cacacagtct 33360
ctgttggctc tttgctccct ggggtctgtg ttgtctcatt actgggcaga cttttacttg 33420
tttggctgta gcttcttgcc tctgattatc tggtgtgaat ttttactata tttctactgg 33480
gagatgattt ttgcctattt gtgtggaaag actgccagaa agatcttaaa aattaaaaaa 33540
aattacatgc cttttgcaag cataacttgt gagcctgatt cagaatgagt caggtgggtg 33600
gttccacaga agcactatgg accagctcca ttccagaatc ttctgagtcc cttgtctgta 33660
gatggagctc acgatgtttt tgtggccagt ggaaaatgga catcttgatg ttgtcaggaa 33720
acttctggtt tctgatgcag cctgctcacc acagttaggc tggacaccat gcggacagtg 33780
gaaggggctt gggagttatc ttttgtcctg ctgggatgga atgcctattc tggaacaagg 33840
caagtgggtt ctagaggcac tcgcgtgttc cctgctcacc ttcccctgct tgcttctgcg 33900
tttgccttag agattgggat ccttgaatgt atggctctct attacagaat taccaggttc 33960
cttcttcttc ttcttctttt tttttaatta aaaaaaaagc atcaattttt gttgtggcac 34020
aaggagtaaa tgtcctgtct gcatagtata atgtatatac agcttcttct tgggtacggg 34080
tgagatggct caatggacaa aggcacttac gctgatgacc aagcctgacg ctctgtagtc 34140
aatcttcaga gcctatgtgg taggaaaaga gaactgaccc tcagaagttg tcctctgacc 34200
tccacactga tatgcacaca aacacatgca cacagataca ttttttttca tttaaaaaga 34260
aaatcacctt tctccttccc aaaagatact tagaaggttc agaaaagtcc ttatgtgtat 34320
tttaaataat aagatttcat atcaaaattt gcttactgat tttaacattt ctttgtgggg 34380
ttttttttct tttgaggggg ggaggatagg gtctctggga ttgagctcag tgggtagcca 34440
aggataacca tcactgactt taatactgca aacacttttc ttcaattcta ttaagggtag 34500
ttgggtttcc aaagagcaga agggcttgcc aatgggacag tcagtcctgg gaacaacata 34560
ggaccttggg ttcctctgat gagagtctag gatccacatg ggagagttcc tttggcttta 34620
tctttgccag ctggattgag gagtttgtat actcagcagg ggattgtcac ccatgtggga 34680
gctggaagcc tggtgtgctt gctgagtggc tcttgtctaa cctcacaccc atgtctccgg 34740
gaccaaagcc tccgttgtgg tctgagttga aagcagtatc cagcagccca ccatcacacc 34800
aagattgtgt agtcataccc aggcacaggc tttgtgtggg ctctgggtat attttctttc 34860
gcagaaatca gccaaggaga gacggtgtgt ttcagagata gacactgggt ctgacacagt 34920
ctgctataca tcaaggcaaa cttggtgaag ccctgtgtgc tgctgggtga gagaggaccc 34980
ttcccgtgtg gctctgagtg aaagtatctt ttccttaacc cttggtctcc tgtattcact 35040
gctctgcttt ctgaagctaa agtgacaaga gtcagcccat tttcactata tggtctgggc 35100
atcatcaagt ttcagaagga ctggggagag atggagaata gcctccccgt gcctggaact 35160
ctggatttct tgaataaaag acctttgagt taccagaatg ccctttccct gtgtcttagt 35220
taggatttta ttgctgcaaa gagacaacac aatgtaactt aaaaaaatta tttatttgtt 35280
ttatgtatat gagtgcacca tgactctctt cagagacact agaagagggc atcagatccc 35340
attacagatg gttgtgagcc accatgtggt tgctgggaat tgaactgagg acttctcaaa 35400
gagcagttgg tgctcttaac tactgagtca tctctccagc ccccagtgca actcttataa 35460
agaaacacac ttaattgggg cttgcttaca gtttcagagg tttagttcat tattgtcatg 35520
gtggaaagca tggcagcttc ctggcagaca cagtgctgga gagagaagaa gctgagagtt 35580
ctacatcttg atccacaggc agcagaaggg gattgtgtgc catactcttt gaggtttgag 35640
caaaggaaac ctcaaagccc gcccccacag tgagaaactc cctccaacaa ggccacatgt 35700
tctctagcaa ggccacacct cctaatagcg cctatgggcc aggtattcaa accaccacac 35760
catacatatc ttacagctct ttccttgaga tctttcttta tactttggag gcaatggcag 35820
cacggatgac ctcacttgtt agatgtttgt gaatccctcc ctgctgactt gattttggat 35880
gtgtttttat tttatggtgc tggacattgt acatgagaca agcatcctgt aattgagccc 35940
agcctttgag ttagtgatct ataggctgag caaaaaacta taatgaagtc agtagagtct 36000
gtctgcacat tcttaagtgg ctgtcttaaa acaattaagg taaggggctg gagagatgct 36060
tcctcggcta agagcactgg ctgctcttcc agaggacctg ggttcagttc ccagcaccca 36120
tatggcagct cacaactgtc tatacctcca gttccagtct gacatcctca catagacata 36180
catgcaggca aaacaccaat gtacattaaa aaaaacacct aatttttaaa aagttcagat 36240
gaaaagaaga aatactatga ttaaacttct agaaacattt ctatttgtaa acttgacctc 36300
ccaaggtcaa ggatcctgtg acttctcatt tttgcccctg tattttgttg ttgttgttgt 36360
ttttgtttgt ttgttttgtg ttttgtttgt tgtttagttt agtttctcgt tgtttgtttt 36420
gtcctttcct ggttccttcc cctttctttg taagcactcc tgctctggct gggtcccagc 36480
tcacttccag cctcctctga tggagccagc attacatctg ctgttttgca ttttgtatac 36540
aggtttcggc cgagtccagc tcctccatga actccaacac cccgctggtg aggataacaa 36600
cgcgtctgtc ctcaacagcg gacaccccga tgctagcagg ggtctccgag tatgagttgc 36660
cagaggatcc aaagtgggaa ttccccagag ataagtaagt actctccctc tgggagggtc 36720
gttgtctgca cctcctggga ctgagcgcag gtcttggttg tgggagtctc cacctgtgtc 36780
ttggtaatca gggacctgtg tcttggtaat cagggacctt cgaactgtaa actgtaaact 36840
gtaaactgca gcaagatggt gcaattaaca gagctgctgg tgcacagggt aggctaccag 36900
cctgtgccct tgaggtggaa gaccaacctt agctctggga agtgaggatc ctggaaggct 36960
ggcagcttcc ttcttgtagg attagcgtct aaacagcttg agagtaacag aaggtggaaa 37020
aatgggctct ttctgcatca aagacacagg aatacgctcc cagcttgctt gaagacaact 37080
cgtctgccta tcttgacatt ttttcagtgt cttcctaaga ttgttagtga tatgtttaac 37140
acacacagcg tgtgtgtgtg tgtgtgtgtg tgtgtgtgtg tgtgtgtgtg tatacagaga 37200
ttcagaaaca caaagacaag ctttcccaag tctgggtatg gccagctcta gttgaaagtt 37260
gaaggaagct taggccccct tggagcttcg tgtcacacta ttccaggtat tttggttccc 37320
aacttagaga tctttacata tatcctaatc cagggccgag gaactgtctg tgtagtcatc 37380
ttatgtcagt ggaaagaatt tccagtttct ttatatactg cagtgaaaag agcattcatt 37440
cattccttca ttcattccgg gaatgtttag tggacatttt tggggtgagg tatgaaagaa 37500
aacacaaaga atggcttctt tacctataga attttgagaa aggaaggtat gtttctcttg 37560
acccttgcca gcctcctagc tgggtgtgat tatagaaata gctgggttcg tgtgcacatt 37620
cctatgtcta caaggctgga ctggggagtt gtgctcatat gctaaaaact tgcagctttc 37680
gggttgagcc tgtgctttgg gccacctgtg acaccacagt caacagtgtg agtctgtgtt 37740
gcccagcagc tcccacccgg gggtacccaa gtgtggaaaa tctgagcgct atgcatttcc 37800
aagcagtgtt tagcacaaat gtaggtggag cacattccca atgaatgaag gctatttaga 37860
aatggtttat taggttggag gcaggagtta cacaaagaga ggtttgttat ggttgtagta 37920
acaggggcaa taggacataa attagccatt ttccaacgca aatgtttatt ttctgccaag 37980
atgttcaatt taattttagt tcttgactgg aaagggggcg ccttcagcag agcaagtgct 38040
ggtatccatt ttcttttcct tttttgagct tttaaaagcg ttgcgtgctt actgcaaatg 38100
ctgtttactg aggacagctc aatacttgcc tgtactgggc ttatggtttt ttggtttttg 38160
ttttttttaa aacaaaacaa aacaaaacaa aacaaaacac cccgaaaagc atactcaggc 38220
tggagaggta tctctctggt ctaaagcaga cactgccctg taaaggacca gagttcggtt 38280
cccagcaccc atgatgtgta gctcacaacc tctgtaacta cagatctggg gaatctgtct 38340
gggctcctta ggtgcctgtg ctcacatgcc cataccctct ccccagaaac acacatacac 38400
ataatcaaga ataaattgaa aaaaaataaa aataaaaaac acactcctaa gtattaattc 38460
caaagacttc cctgttcctt tggcttctgg aacatctaaa ataatgtcag gtcatttgtc 38520
tgttgtgtat aaaacttaca tgcttagaaa tgtaacttgt gctgttttct attttttttt 38580
ttccttgttt actttgggta gtgataagga atcctaaact tatgtcaaaa aggtatcgtg 38640
cctgatttct agaagttttt cttaatgaga cacgataaat tatttgaaac gtgctgaaga 38700
ttcctaccct gcaactgggc aatcgatgta accataaaat ctaccggtat tgaataatag 38760
tgatttgaga gttgccactt tacagggaca gaaaataaga acagactttc actttttttt 38820
tcacctctgc gacattttaa attataaata tttaattggc tcaagaccaa aagctcccta 38880
tggctggcat gcagggagcc tgaccaccgc gctagcaagg acaccttcca taaagaaaaa 38940
gaaaataaat cgagaggaca aatgtgaaat ttaatagtcc ctccaacagt aattgacgtt 39000
ctggaaaaac atcactaaga aaatagcctg cgtgtgtatc ggaggctcat tggttccata 39060
tgcatgcctc tggaagattt ttatatttag ttctggaatt tccctccctg tgcccctcgg 39120
ccagactcgc ggtgtgctaa tcccgtattt acacatttag gctgacgctg ggcaaacccc 39180
tgggggaagg ttgcttcggg caagtagtca tggctgaagc agtgggaatc gataaagaca 39240
aacccaagga ggcggtcacc gtggcagtga agatgttgaa aggtgagtgg gcggatgggc 39300
ggtcggggag gagagggtct tatcaggagc gagcgttcct tttgtgacat gtgaactctg 39360
cagggacgtg gggtcagaga gcacatactt gacctggcgg ttgagggggt tttcaggata 39420
aatgagcaaa tgagatggag gatttacctt gagctgtgtg tacttaaaaa gaaaagccag 39480
tttagcagca agttgtagct tgctgggctg aaccggtctc taactcctta gaaaagggtc 39540
ccgattctct tcttttctgt gtgttcatgg gtttagaaag tttagggggt ttatttagct 39600
ggttaaattt tggacccaga cttttaacat acaaataagg agaggtaggt gttggagtgg 39660
caactggaga cagaatgtca aaatgtggat tcaaagagtc gcttagaagc caaaaaggag 39720
caaacaattg gaactgatgc agaatcccag ggacatgtaa acaataatgc cacgctataa 39780
atgcccgctt tgttcttttc ttttcttttc ttttcttttc tttttttttt ttttagggga 39840
ggggagggga gggggtctgg gaatttatcc acaacctttc taacacagct tgatgatgac 39900
gcccaaggag cttaaattgc tttcaactat taacttatcc ttgcatgggt attcttttat 39960
cgaagagata aagggaaagg tcacattata aatcctgttg ttggggaatc tcagaaagga 40020
gaaaggagcc atgttcaatg tttccctggc ttgtgggcag agaagtctgt cccgggcctg 40080
tgggatgtgg catgttctca ggagtccgac cttttctctc tttgatagga cacttaccac 40140
atccctccct gatgcagaca acaaagggcc aggacatggt tcattttgtc agttttagtt 40200
attgacctga gactcccagt gaaatctggg atgttccttt ctttggagac tgataccagg 40260
aaggagatag caagtatcgg ggcaccaggg cagaggcagc ccttggtacc tactggaagc 40320
tgtgggttgg gaaggatcag gcatcatact gctttccaca gaacctctgg ttttgagatc 40380
cctggagcta gtgcaaaagg gaggtttagg ggttggccct tccctttaag caagatcacc 40440
caccatcctt ttcatcgtgg tcagaggaca tgccttttca acattctttg tgacagccag 40500
aggatggctg aggtgtaagg aagacaagtg tactgagcca tgtgtctgtc catagtcctc 40560
tcttccctct tctctgtatt ggtcaggata gatttttgga tacctgtgcc tctatttcat 40620
ttttaaccct tttgcttttc ttttagctca gatttttctt ttctaagtat ttctgtattg 40680
aattagctta gtgacagaac acttgcgtgg tgtgcacatg gtactgggtt tgcatcctag 40740
cattacaaga atccaaacga cagcagaact aactgagagg agagcacagt agcggccgca 40800
aattgctttg agaggctcta taaaacctta gaggctattt aaatttaaat ggccggcccg 40860
acggccaggc ggccgccagg cctacccact agtcaattcg ggaggatcga aacggcagat 40920
cgcaaaaaac agtacataca gaaggagaca tgaacatgaa catcaaaaaa attgtaaaac 40980
aagccacagt tctgactttt acgactgcac ttctggcagg aggagcgact caagccttcg 41040
cgaaagaaaa taaccaaaaa gcatacaaag aaacgtacgg cgtctctcat attacacgcc 41100
atgatatgct gcagatccct aaacagcagc aaaacgaaaa ataccaagtg cctcaattcg 41160
atcaatcaac gattaaaaat attgagtctg caaaaggact tgatgtgtgg gacagctggc 41220
cgctgcaaaa cgctgacgga acagtagctg aatacaacgg ctatcacgtt gtgtttgctc 41280
ttgcgggaag cccgaaagac gctgatgaca catcaatcta catgttttat caaaaggtcg 41340
gcgacaactc aatcgacagc tggaaaaacg cgggccgtgt ctttaaagac agcgataagt 41400
tcgacgccaa cgatccgatc ctgaaagatc agacgcaaga atggtccggt tctgcaacct 41460
ttacatctga cggaaaaatc cgtttattct acactgacta ttccggtaaa cattacggca 41520
aacaaagcct gacaacagcg caggtaaatg tgtcaaaatc tgatgacaca ctcaaaatca 41580
acggagtgga agatcacaaa acgatttttg acggagacgg aaaaacatat cagaacgttc 41640
agcagtttat cgatgaaggc aattatacat ccggcgacaa ccatacgctg agagaccctc 41700
actacgttga agacaaaggc cataaatacc ttgtattcga agccaacacg ggaacagaaa 41760
acggatacca aggcgaagaa tctttattta acaaagcgta ctacggcggc ggcacgaact 41820
tcttccgtaa agaaagccag aagcttcagc agagcgctaa aaaacgcgat gctgagttag 41880
cgaacggcgc cctcggtatc atagagttaa ataatgatta cacattgaaa aaagtaatga 41940
agccgctgat cacttcaaac acggtaactg atgaaatcga gcgcgcgaat gttttcaaaa 42000
tgaacggcaa atggtacttg ttcactgatt cacgcggttc aaaaatgacg atcgatggta 42060
ttaactcaaa cgatatttac atgcttggtt atgtatcaaa ctctttaacc ggcccttaca 42120
agccgctgaa caaaacaggg cttgtgctgc aaatgggtct tgatccaaac gatgtgacat 42180
tcacttactc tcacttcgca gtgccgcaag ccaaaggcaa caatgtggtt atcacaagct 42240
acatgacaaa cagaggcttc ttcgaggata aaaaggcaac atttgcgcca agcttcttaa 42300
tgaacatcaa aggcaataaa acatccgttg tcaaaaacag catcctggag caaggacagc 42360
tgacagtcaa ctaataacag caaaaagaaa atgccgatac ttcattggca ttttctttta 42420
tttctcaaca agatggtgaa ttgactagtg ggtagatcca caggacgggt gtggtcgcca 42480
tgatcgcgta gtcgatagtg gctccaagta gcgaagcgag caggactggg cggcggccaa 42540
agcggtcgga cagtgctccg agaacgggtg cgcatagaaa ttgcatcaac gcatatagcg 42600
ctagcagcac gccatagtga ctggcgatgc tgtcggaatg gacgatatcc cgcaagaggc 42660
ccggcagtac cggcataacc aagcctatgc ctacagcatc cagggtgacg gtgccgagga 42720
tgacgatgag cgcattgtta gatttcatac acggtgcctg actgcgttag caatttaact 42780
gtgataaact accgcattaa agcttatcga tgataagctg tcaaacatga gaattgatcc 42840
ggaaccctta atataacttc gtataatgta tgctatacga agttattagg tccctcgact 42900
atagggtcac cgtcgacagc gacacacttg catcggatgc agcccggtta acgtgccggc 42960
acggcctggg taaccaggta ttttgtccac ataaccgtgc gcaaaatgtt gtggataagc 43020
aggacacagc agcaatccac agcaggcata caaccgcaca ccgaggttac tccgttctac 43080
aggttacgac gacatgtcaa tacttgccct tgacaggcat tgatggaatc gtagtctcac 43140
gctgatagtc tgatcgacaa tacaagtggg accgtggtcc cagaccgata atcagaccga 43200
caacacgagt gggatcgtgg tcccagacta ataatcagac cgacgatacg agtgggaccg 43260
tggtcccaga ctaataatca gaccgacgat acgagtggga ccgtggttcc agactaataa 43320
tcagaccgac gatacgagtg ggaccgtggt cccagactaa taatcagacc gacgatacga 43380
gtgggaccat ggtcccagac taataatcag accgacgata cgagtgggac cgtggtccca 43440
gtctgattat cagaccgacg atacgagtgg gaccgtggtc ccagactaat aatcagaccg 43500
acgatacgag tgggaccgtg gtcccagact aataatcaga ccgacgatac gagtgggacc 43560
gtggtcccag tctgattatc agaccgacga tacaagtgga acagtgggcc cagagagaat 43620
attcaggcca gttatgcttt ctggcctgta acaaaggaca ttaagtaaag acagataaac 43680
gtagactaaa acgtggtcgc atcagggtgc tggcttttca agttccttaa gaatggcctc 43740
aattttctct atacactcag ttggaacacg agacctgtcc aggttaagca ccattttatc 43800
gcccttatac aatactgtcg ctccaggagc aaactgatgt cgtgagctta aactagttct 43860
tgatgcagat gacgttttaa gcacagaagt taaaagagtg ataacttctt cagcttcaaa 43920
tatcacccca gcttttttct gctcatgaag gttagatgcc tgctgcttaa gtaattcctc 43980
tttatctgta aaggcttttt gaagtgcatc acctgaccgg gcagatagtt caccggggtg 44040
agaaaaaaga gcaacaactg atttaggcaa tttggcggtg ttgatacagc gggtaataat 44100
cttacgtgaa atattttccg catcagccag cgcagaaata tttccagcaa attcattctg 44160
caatcggctt gcataacgct gaccacgttc ataagcactt gttgggcgat aatcgttacc 44220
caatctggat aatgcagcca tctgctcatc atccagctcg ccaaccagaa cacgataatc 44280
actttcggta agtgcagcag ctttacgacg gcgactccca tcggcaattt ctatgacacc 44340
agatactctt cgaccgaacg ccggtgtctg ttgaccagtc agtagaaaag aagggatgag 44400
atcatccagt gcgtcctcag taagcagctc ctggtcacgt tcattacctg accatacccg 44460
agaggtcttc tcaacactat caccccggag cacttcaaga gtaaacttca catcccgacc 44520
acatacaggc aaagtaatgg cattaccgcg agccattact cctacgcgcg caattaacga 44580
atccaccatc ggggcagctg gtgtcgataa cgaagtatct tcaaccggtt gagtattgag 44640
cgtatgtttt ggaataacag gcgcacgctt cattatctaa tctcccagcg tggtttaatc 44700
agacgatcga aaatttcatt gcagacaggt tcccaaatag aaagagcatt tctccaggca 44760
ccagttgaag agcgttgatc aatggcctgt tcaaaaacag ttctcatccg gatctgacct 44820
ttaccaactt catccgtttc acgtacaaca ttttttagaa ccatgcttcc ccaggcatcc 44880
cgaatttgct cctccatcca cggggactga gagccattac tattgctgta tttggtaagc 44940
aaaatacgta catcaggctc gaacccttta agatcaacgt tcttgagcag atcacgaagc 45000
atatcgaaaa actgcagtgc ggaggtgtag tcaaacaact cagcaggcgt gggaacaatc 45060
agcacatcag cagcacatac gacattaatc gtgccgatac ccaggttagg cgcgctgtca 45120
ataactatga catcatagtc atgagcaaca gtttcaatgg ccagtcggag catcaggtgt 45180
ggatcggtgg gcagtttacc ttcatcaaat ttgcccatta actcagtttc aatacggtgc 45240
agagccagac aggaaggaat aatgtcaagc cccggccagc aagtgggctt tattgcataa 45300
gtgacatcgt ccttttcccc aagatagaaa ggcaggagag tgtcttctgc atgaatatga 45360
agatctggta cccatccgtg atacattgag gctgttccct gggggtcgtt accttccacg 45420
agcaaaacac gtagcccctt cagagccaga tcctgagcaa gatgaacaga aactgaggtt 45480
ttgtaaacgc cacctttatg ggcagcaacc ccgatcaccg gtggaaatac gtcttcagca 45540
cgtcgcaatc gcgtaccaaa cacatcacgc atatgattaa tttgttcaat tgtataacca 45600
acacgttgct caacccgtcc tcgaatttcc atatccgggt gcggtagtcg ccctgctttc 45660
tcggcatctc tgatagcctg agaagaaacc ccaactaaat ccgctgcttc acctattctc 45720
cagcgccggg ttattttcct cgcttccggg ctgtcatcat taaactgtgc aatggcgata 45780
gccttcgtca tttcatgacc agcgtttatg cactggttaa gtgtttccat gagtttcatt 45840
ctgaacatcc tttaatcatt gctttgcgtt tttttattaa atcttgcaat ttactgcaaa 45900
gcaacaacaa aatcgcaaag tcatcaaaaa accgcaaagt tgtttaaaat aagagcaaca 45960
ctacaaaagg agataagaag agcacatacc tcagtcactt attatcacta gcgctcgccg 46020
cagccgtgta accgagcata gcgagcgaac tggcgaggaa gcaaagaaga actgttctgt 46080
cagatagctc ttacgctcag cgcaagaaga aatatccacc gtgggaaaaa ctccaggtag 46140
aggtacacac gcggatagcc aattcagagt aataaactgt gataatcaac cctcatcaat 46200
gatgacgaac taacccccga tatcaggtca catgacgaag ggaaagagaa ggaaatcaac 46260
tgtgacaaac tgccctcaaa tttggcttcc ttaaaaatta cagttcaaaa agtatgagaa 46320
aatccatgca ggctgaagga aacagcaaaa ctgtgacaaa ttaccctcag taggtcagaa 46380
caaatgtgac gaaccaccct caaatctgtg acagataacc ctcagactat cctgtcgtca 46440
tggaagtgat atcgcggaag gaaaatacga tatgagtcgt ctggcggcct ttctttttct 46500
caatgtatga gaggcgcatt ggagttctgc tgttgatctc attaacacag acctgcagga 46560
agcggcggcg gaagtcaggc atacgctggt aactttgagg cagctggtaa cgctctatga 46620
tccagtcgat tttcagagag acgatgcctg agccatccgg cttacgatac tgacacaggg 46680
attcgtataa acgcatggca tacggattgg tgatttcttt tgtttcacta agccgaaact 46740
gcgtaaaccg gttctgtaac ccgataaaga agggaatgag atatgggttg atatgtacac 46800
tgtaaagccc tctggatgga ctgtgcgcac gtttgataaa ccaaggaaaa gattcatagc 46860
ctttttcatc gccggcatcc tcttcagggc gataaaaaac cacttccttc cccgcgaaac 46920
tcttcaatgc ctgccgtata tccttactgg cttccgcaga ggtcaatccg aatatttcag 46980
catatttagc aacatggatc tcgcagatac cgtcatgttc ctgtagggtg ccatcagatt 47040
ttctgatctg gtcaacgaac agatacagca tacgtttttg atcccgggag agactatatg 47100
ccgcctcagt gaggtcgttt gactggacga ttcgcgggct atttttacgt ttcttgtgat 47160
tgataaccgc tgtttccgcc atgacagatc catgtgaagt gtgacaagtt tttagattgt 47220
cacactaaat aaaaaagagt caataagcag ggataacttt gtgaaaaaac agcttcttct 47280
gagggcaatt tgtcacaggg ttaagggcaa tttgtcacag acaggactgt catttgaggg 47340
tgatttgtca cactgaaagg gcaatttgtc acaacacctt ctctagaacc agcatggata 47400
aaggcctaca aggcgctcta aaaaagaaga tctaaaaact ataaaaaaaa taattataaa 47460
aatatccccg tggataagtg gataacccca agggaagttt tttcaggcat cgtgtgtaag 47520
cagaatatat aagtgctgtt ccctggtgct tcctcgctca ctcgagggct tcgccctgtc 47580
gctcaactgc ggcgagcact actggctgta aaaggacaga ccacatcatg gttctgtgtt 47640
cattaggttg ttctgtccat tgctgacata atccgctcca cttcaacgta acaccgcacg 47700
aagatttcta ttgttcctga aggcatattc aaatcgtttt cgttaccgct tgcaggcatc 47760
atgacagaac actacttcct ataaacgcta cacaggctcc tgagattaat aatgcggatc 47820
tctacgataa tgggagattt tcccgactgt ttcgttcgct tctcagtgga taacagccag 47880
cttctctgtt taacagacaa aaacagcata tccactcagt tccacatttc catataaagg 47940
ccaaggcatt tattctcagg ataattgttt cagcatcgca accgcatcag actccggcat 48000
cgcaaactgc acccggtgcc gggcagccac atccagcgca aaaaccttcg tgtagacttc 48060
cgttgaactg atggacttat gtcccatcag gctttgcaga actttcagcg gtataccggc 48120
atacagcatg tgcatcgcat aggaatggcg gaacgtatgt ggtgtgaccg gaacagagaa 48180
cgtcacaccg tcagcagcag cggcggcaac cgcctcccca atccaggtcc tgaccgttct 48240
gtccgtcact tcccagatcc gcgctttctc tgtccttcct gtgcgacggt tacgccgctc 48300
catgagctta tcgcgaataa atacctgtga cggaagatca cttcgcagaa taaataaatc 48360
ctggtgtccc tgttgatacc gggaagccct gggccaactt ttggcgaaaa tgagacgttg 48420
atcggcacgt aagaggttcc aactttcacc ataatgaaat aagatcacta ccgggcgtat 48480
tttttgagtt atcgagattt tcaggagcta aggaagctaa aatggagaaa aaaatcactg 48540
gatataccac cgttgatata tcccaatggc atcgtaaaga acattttgag gcatttcagt 48600
cagttgctca atgtacctat aaccagaccg ttcagctgga tattacggcc tttttaaaga 48660
ccgtaaagaa aaataagcac aagttttatc cggcctttat tcacattctt gcccgcctga 48720
tgaatgctca tccggagttc cgtatggcaa tgaaagacgg tgagctggtg atatgggata 48780
gtgttcaccc ttgttacacc gttttccatg agcaaactga aacgttttca tcgctctgga 48840
gtgaatacca cgacgatttc cggcagtttc tacacatata ttcgcaagat gtggcgtgtt 48900
acggtgaaaa cctggcctat ttccctaaag ggtttattga gaatatgttt ttcgtctcag 48960
ccaatccctg ggtgagtttc accagttttg atttaaacgt ggccaatatg gacaacttct 49020
tcgcccccgt tttcaccatg ggcaaatatt atacgcaagg cgacaaggtg ctgatgccgc 49080
tggcgattca ggttcatcat gccgtttgtg atggcttcca tgtcggcaga atgcttaatg 49140
aattacaaca gtactgcgat gagtggcagg gcggggcgta atttttttaa ggcagttatt 49200
ggtgccctta aacgcctggt tgctacgcct gaataagtga taataagcgg atgaatggca 49260
gaaattcgat gataagctgt caaacatgag aattggtcga cggcgcgcca aagcttgcat 49320
gcctgcagcc gcgtaacctg gcaaaatcgg ttacggttga gtaataaatg gatgccctgc 49380
gtaagcgggg cacatttcat tacctctttc tccgcacccg acatagataa taacttcgta 49440
tagtatacat tatacgaagt tatctagtag acttaattaa ggatcgatcc ggcgcgccaa 49500
tagtcatgcc ccgcgcccac cggaaggagc tgactgggtt gaaggctctc aagggcatcg 49560
gtcgagcttg acattgtagg actatattgc tctaataaat ttgcggccgc taatacgact 49620
cactataggg a 49631
<210> 18
<211> 20
<212> DNA
<213>人工序列
<220>
<223>合成
<400> 18
ggaaagccac cctgtatgct 20
<210> 19
<211> 19
<212> DNA
<213>人工序列
<220>
<223>合成
<400> 19
cttggccaac agtggatgg 19
<210> 20
<211> 42
<212> RNA
<213>人工序列
<220>
<223>合成
<400> 20
cuaaaaugau ucucaucugc guuuuagagc uaugcuguuu ug 42
<210> 21
<211> 42
<212> RNA
<213>人工序列
<220>
<223>合成
<400> 21
gcucucaacu ucacccuuuc guuuuagagc uaugcuguuu ug 42
<210> 22
<211> 23
<212> DNA
<213>人工序列
<220>
<223>合成
<400> 22
ctaaaatgat tctcatctgc agg 23
<210> 23
<211> 23
<212> DNA
<213>人工序列
<220>
<223>合成
<400> 23
gctctcaact tcaccctttc tgg 23
<210> 24
<211> 23
<212> DNA
<213>人工序列
<220>
<223>合成-连接至向导RNA (gRNA)
的靶基因座
<220>
<221> misc_feature
<222> 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17,
18, 19, 20, 21
<223> n = A、T、C或G
<400> 24
nnnnnnnnnn nnnnnnnnnn ngg 23
<210> 25
<211> 25
<212> DNA
<213>人工序列
<220>
<223> 合成-连接至向导RNA (gRNA)
的靶基因座
<220>
<221> misc_feature
<222> 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18,
19, 20, 21, 22, 23
<223> n = A、T、C或G
<400> 25
ggnnnnnnnn nnnnnnnnnn nnngg 25
权利要求书(按照条约第19条的修改)
1.一种用于组装至少两个核酸的方法,包括:
(a)使第一核酸与第一核酸酶试剂接触,其中所述第一核酸酶试剂在第一靶位点处切割所述第一核酸,以产生第一经酶切的核酸,在所述第一经酶切的核酸与第二核酸之间具有重叠末端序列;
(b)使所述第一经酶切的核酸和所述第二核酸与核酸外切酶接触,以暴露所述第一经酶切的核酸与所述第二核酸之间的互补序列;以及
(c)组装由步骤(b)生成的所述两个核酸片段。
2.根据权利要求1所述的方法,其中步骤(c)还包括:
(i)使所述暴露的互补序列退火;
(ii)延伸所述经退火的互补序列的3’端;以及
(iii)连接所述第一核酸和所述第二核酸。
3.根据权利要求1或2所述的方法,其中步骤(a)还包括使所述第二核酸与第二核酸酶试剂接触,其中所述第二核酸不包含所述重叠末端序列,并且所述第二核酸酶试剂在第二靶位点处切割所述第二核酸,以产生第二经酶切的核酸,在所述第一经酶切的核酸与所述第二经酶切的核酸之间具有所述重叠末端序列,并且,
其中步骤(b)的所述第二核酸是所述第二经酶切的核酸。
4.根据权利要求3所述的方法,其中所述第一核酸酶试剂和所述第二核酸酶试剂中的至少一者包含靶向所述第一靶位点或所述第二靶位点的Cas9蛋白和向导RNA(gRNA)(gRNA-Cas复合物)、锌指核酸酶或转录激活因子样效应物核酸酶(TALEN)。
5.根据权利要求4所述的方法,其中所述第一核酸酶试剂和所述第二核酸酶试剂中的至少一者包含所述Cas蛋白和所述向导RNA(gRNA)(gRNA-Cas复合物),
其中所述Cas蛋白是Cas9蛋白,所述gRNA包含编码成簇的规律间隔的短回文重复序列(CRISPR)RNA(crRNA)和反式激活CRISPR RNA(tracrRNA)的核酸序列,并且所述第一靶位点和所述第二靶位点中的至少一者被前间区序列邻近基序(PAM)序列紧邻地侧接。
6.根据权利要求5所述的方法,其中所述Cas9蛋白包含RuvC结构域和HNH结构域,所述两个结构域中的至少一者缺少核酸内切酶活性。
7.根据权利要求1至6中任一项所述的方法,其中所述重叠末端序列的长度在20bp至200bp的范围内。
8.根据权利要求1至7中任一项所述的方法,其中所述第一核酸、所述第二核酸或这两个核酸衍生自细菌人工染色体。
9.根据权利要求8所述的方法,其中所述细菌人工染色体包含人DNA、啮齿动物DNA、合成DNA、人多核苷酸序列或它们的组合。
10.一种用于组装至少两个核酸的方法,包括:
(a)使第一核酸与第一核酸酶试剂和第二核酸酶试剂接触以产生第一经酶切的核酸,其中所述第一核酸酶试剂在所述第一核酸的第一链上的第一靶位点处生成切口,并且所述第二核酸酶试剂在所述第一核酸的第二链上的第二靶位点处生成切口,以产生在其末端之一处包含5′或3′悬垂序列的所述第一经酶切的核酸;
(b)使所述第一经酶切的核酸和包含与所述5′或3′悬垂序列互补的序列的第二核酸退火;以及
(c)连接所述第一经酶切的核酸和所述第二核酸。
11.根据权利要求10所述的方法,其中步骤(b)还包括使用所述第二链作为模板来延伸所述第一链的3’端,并且使用所述第一链作为模板来延伸所述第二链的3’端。
12.根据权利要求10或11所述的方法,其中所述第一核酸酶试剂和所述第二核酸酶试剂中的至少一者包含靶向所述第一靶位点或所述第二靶位点的Cas9蛋白和向导RNA(gRNA)(gRNA-Cas复合物)。
13.根据权利要求12所述的方法,其中所述Cas9蛋白包含RuvC结构域和HNH结构域,所述两个结构域中的一者缺少核酸内切酶活性。
14.根据权利要求10至13中任一项所述的方法,其中所述第一靶位点与所述第二靶位点相隔至少4bp。
15.根据权利要求10至14中任一项所述的方法,其中所述gRNA包含编码成簇的规律间隔的短回文重复序列(CRISPR)RNA(crRNA)和反式激活CRISPR RNA(tracrRNA)的核酸序列,并且其中所述第一靶位点和所述第二靶位点中的至少一者被前间区序列邻近基序(PAM)序列紧邻地侧接。
16.一种用于组装两个或更多个核酸的方法,包括:
(a)使第一核酸与至少一种核酸酶试剂接触以生成第一经酶切的核酸;
(b)使所述第一经酶切的核酸与第二核酸、接合寡核苷酸和核酸外切酶接触,
其中所述接合寡核苷酸包含:
(i)与所述第一经酶切的核酸互补的第一互补序列;
(ii)间区序列;以及
(iii)与所述第二核酸互补的第二互补序列;
其中所述核酸外切酶使所述第一互补序列和所述第二互补序列暴露;以及
(c)将所述接合寡核苷酸与所述第一经酶切的核酸和所述第二核酸组装在一起。
17.根据权利要求16所述的方法,其中步骤(c)中的组装包括:
(i)使所述接合寡核苷酸的所述第一互补序列退火到所述第一经酶切的核酸上,并使所述接合寡核苷酸的所述第二互补序列退火到所述第二核酸上;以及
(ii)将所述接合寡核苷酸连接到所述第一经酶切的核酸和所述第二核酸。
18.根据权利要求16或17所述的方法,其中所述接合寡核苷酸的所述第一互补序列和所述第二互补序列包含15至120个互补碱基。
19.根据权利要求16至18中任一项所述的方法,其中所述接合寡核苷酸的所述间区序列包含非互补核酸。
20.根据权利要求16至18中任一项所述的方法,其中所述第一经酶切的核酸被无缝地组装到所述第二核酸。
21.根据权利要求20所述的方法,其中所述至少一种核酸酶试剂被设计成从将要进行所述无缝组装的所述第一核酸的末端切割至少20bp片段,
其中所述接合寡核苷酸的所述间区序列包含与所述至少20bp片段相同的序列,其中在所述第一互补序列与所述至少20bp片段之间不存在核酸碱基,并且在所述第二互补序列与所述至少20bp片段之间不存在核酸碱基,
使得所述第一核酸与所述接合寡核苷酸和所述第二核酸的组装重建所述至少20bp片段并无缝地组装所述第一核酸和所述第二核酸。
22.根据权利要求20所述的方法,其中所述至少一种核酸酶试剂被设计成从将要进行所述无缝组装的所述第二核酸的末端切割至少20bp片段,
其中所述接合寡核苷酸的所述间区序列包含与所述至少20bp片段相同的序列,其中在所述第一互补序列与所述至少20bp片段之间不存在核酸碱基,并且在所述第二互补序列与所述至少20bp片段之间不存在核酸碱基,
使得所述第一核酸与所述接合寡核苷酸和所述第二核酸的组装重建所述至少20bp片段并无缝地组装所述第一核酸和所述第二核酸。
23.根据权利要求21或22所述的方法,其中所述间区序列包含约20bp至约120bp。
24.根据权利要求16至23中任一项所述的方法,其中步骤(a)还包括使所述第二核酸与第二核酸酶试剂和核酸外切酶接触,其中所述第二核酸酶试剂切割所述第二核酸,以产生包含与所述接合寡核苷酸的所述第二互补序列互补的核苷酸序列的第二经酶切的核酸,其中所述第一经酶切的核酸被组装到所述第二经酶切的核酸。
25.根据权利要求16至23中任一项所述的方法,其中步骤(a)还包括使所述第二核酸与限制性内切酶或大范围核酸酶和核酸外切酶接触,其中所述限制性内切酶或大范围核酸酶切割所述第二核酸,以产生包含与所述接合寡核苷酸中的所述第二互补序列互补的核苷酸序列的第二经酶切的核酸,其中所述第一经酶切的核酸被组装到所述第二经酶切的核酸。
26.根据权利要求24或25所述的方法,其中步骤(b)还包括延伸所述第一经酶切的核酸和/或所述第二经酶切的核酸的3’端。
27.根据权利要求16至26中任一项所述的方法,其中所述接合寡核苷酸在同一反应中被组装到所述第一核酸和所述第二核酸。
28.根据权利要求16至26中任一项所述的方法,其中所述接合寡核苷酸依次被组装到所述第一核酸和所述第二核酸。
29.根据权利要求24至28中任一项所述的方法,其中所述至少一种核酸酶试剂和/或所述第二核酸酶试剂包含靶向所述第一靶位点或所述第二靶位点的Cas蛋白和向导RNA(gRNA)(gRNA-Cas复合物)、锌指核酸酶或转录激活因子样效应物核酸酶(TALEN)。
30.根据权利要求29所述的方法,其中所述第一核酸酶试剂和所述第二核酸酶试剂中的至少一者包含所述Cas蛋白和所述向导RNA(gRNA)(gRNA-Cas复合物),
其中所述Cas蛋白是Cas9蛋白,所述gRNA包含编码成簇的规律间隔的短回文重复序列(CRISPR)RNA(crRNA)和反式激活CRISPR RNA(tracrRNA)的核酸序列,并且所述第一靶位点和所述第二靶位点中的至少一者被前间区序列邻近基序(PAM)序列紧邻地侧接。
31.根据权利要求30所述的方法,其中所述Cas9蛋白包含RuvC结构域和HNH结构域,所述两个结构域中的至少一者缺少核酸内切酶活性。
32.根据权利要求10至31中任一项所述的方法,其中所述第一核酸、所述第二核酸或这两个核酸衍生自细菌人工染色体。
33.根据权利要求10至32中任一项所述的方法,其中所述第一核酸、所述第二核酸或这两个核酸包含人DNA、啮齿动物DNA、合成DNA或它们的组合。
34.根据权利要求10至33中任一项所述的方法,其中所述第一核酸、所述第二核酸或这两个核酸为至少10kb。
35.根据权利要求16至34中任一项所述的方法,其中所述接合寡核苷酸包含线性双链DNA片段。
36.根据权利要求35所述的方法,其中所述线性双链DNA片段不包含选择盒。
37.一种用于组装两个或更多个核酸的方法,包括:
(a)使第一核酸与至少一种核酸酶试剂接触以生成第一经酶切的核酸;
(b)使第二核酸与第二核酸酶试剂接触以生成第二经酶切的核酸;
(c)使所述第一经酶切的核酸和所述第二经酶切的核酸与接合寡核苷酸和核酸外切酶接触,
其中所述接合寡核苷酸包含:
(i)与所述第一经酶切的核酸互补的第一互补序列;
(ii)间区序列;以及
(iii)与所述第二经酶切的核酸互补的第二互补序列;
其中所述核酸外切酶使所述第一互补序列和所述第二互补序列暴露;以及
(d)将所述接合寡核苷酸与所述第一经酶切的核酸和所述第二经酶切的核酸组装在一起。
- 还没有人留言评论。精彩留言会获得点赞!
- 吡啶胺基嘧啶衍生物甲磺酸盐的结晶形式及其制备和应用的制造方法与工艺
- 吡啶胺基嘧啶衍生物的盐及其制备方法和应用与流程
- 用于识别超氧阴离子的荧光探针及其制备方法和应用、光纤探头及其制备方法与流程
- 磁性复合吸附剂去除废水中四环素的方法与流程
- MIIP pS303阻断剂在制备抗肿瘤药物中的应用的制造方法与工艺
- 阻断UBE2M介导Neddylation修饰在肾细胞癌治疗中的应用的制造方法与工艺
- 一种治疗过敏性鼻炎的药物组合物的制造方法与工艺
- 一种皮蛋蛋白源性抗炎肽饮料及其制备方法与流程
- 活性丙烯酰胺类化合物的制造方法与工艺
- 2‑(4‑(4‑乙氧基‑6‑氧代‑1,6‑二氢吡啶‑3‑基)‑2‑氟苯基)‑N‑(5‑(1,1,1‑三氟‑2‑甲基丙‑2‑基)异噁唑‑3‑基)乙酰胺的晶型的制造方法与工艺