氨基酸转运基因OsANT1在水稻选育中的应用的制作方法
技术领域
本发明属于植物基因工程领域,具体涉及氨基酸转运基因OsANT1在水稻选育中的应用。
背景技术:
植物通过吸收土壤中的氨、硝酸根、氨基酸、可溶性肽等来获得氮素;氮的吸收和转运主要依靠铵根运输蛋白(AMT)、硝酸根运输蛋白(NRT)、氨基酸运输蛋白(AAT)、肽运输蛋白(PTR)等运输蛋白来完成(Williams L, Miller A. Transporters responsible for the uptake and partitioning of nitrogenous solutes. Annu Rev Plant Biol and Plant Mol Biol, 2001, 52: 659-688.)。铵通过植物AMT吸收后再通过谷氨酰胺合成酶(GS)和谷氨酸合酶(GOGAT)合成谷氨酰胺和谷氨酸,后者再进一步形成其它氨基酸(Sonoda Y, Ikeda A, Saiki S, et al. Feedback regulation of the ammonium transporter gene family AMT1 by glutamine in rice. Plant Cell Physiol, 2003, 44: 1396-1402.)。植物可通过高亲和转运系统(HATS)的NRT2和低亲和转运系统(LATS)的NRT1吸收环境中的硝酸盐,由硝酸还原酶(NR)和亚硝酸还原酶(NiR)还原形成铵,再进一步形成氨基酸(Paungfoo-Lonhienne C, Lonhienne T G, Rentsch D, et al. Plants can use protein as a nitrogen source without assistance from other organisms. PNAS, 2008, 105: 4524-4529.)。
在高等植物中,AAT是一类跨膜蛋白,将氨基酸从胞外运至胞内,同时还在氨基酸远距离运输、病原反应和非生物胁迫等方面发挥着重要作用(Tegeder M. Transporters for amino acids in plant cells: some functions and many unknowns. Curr Opin Plant Biol, 2012, 15: 1-7.)。AAT基因被分为两个超家族:APC(氨基酸、多胺和胆碱转运)超家族和AAAP(氨基酸/生长素透性酶)超家族。APC超家族分为三个亚家族:CATs(阳离子氨基酸转运蛋白)家族、ACTs(氨基酸/胆碱转运蛋白)家族和PHSs(多胺、H+协同转运蛋白)家族。AAAP超家族分为六个亚家族:AAPs(氨基酸透性酶)家族、LHTs(赖氨酸和组氨酸转运蛋白)家族、ProTs(脯氨酸转运蛋白)家族、GATs(γ-氨基酸丁酸,GABA)家族、AUXs(生长素转运蛋白)家族和ANTs(芳香族和中性氨基酸转运蛋白)家族(Fischer WN, Andre B, Rentsch D, et al. Amino acid transport in plants. Trends Plant Sci, 1998, 3: 188-195.)。
水稻基因组中共找到85个AAT家族成员(Zhao H, Ma H, Yu L, et al. Genome-wide survey and expression analysis of amino acid transporter gene family in rice (Oryza sativa L.). PLoS ONE, 2012, 7: e49210.)。研究发现OsAAT5、OsAAT7、OsAAT24、OsAAT49和OsAAT60的T-DNA插入突变体的稻米产量及植物体干重均下降,证明AAT对水稻的氮素积累及碳氮分配有着重要作用(Lu Y, Song Z, Lu K, et al. Molecular characterization, expression and functional analysis of the amino acid transporter gene family (OsAATs) in rice. Acta physiol Plant, 2012, 34: 1943-1962.)。研究发现超量表达OsAAP6会增加水稻籽粒中储藏性蛋白和氨基酸含量,从而改善稻米营养和风味(Peng B, Kong H, Li Y, et al. OsAAP6 functions as an important regulator of grain protein content and nutritional quality in rice. Nat Commun, 2014, 5: doi:10.1038.)。氨基酸转运蛋白对水稻等各种植物体的氨基酸吸收、转运和储藏具有重要的作用。目前关于水稻AAT家族成员研究的报道很少,ANT基因表达的蛋白主要运输中性氨基酸,目前水稻氨基酸转运家族OsANT1基因的蛋白对水稻的生长发育目前未有任何研究。本发明发现OsANT1基因对水稻生物量影响有较为重要的作用,可应用于植物株型改良。
技术实现要素:
本发明的目的在于解决现有技术中存在的问题,提供氨基酸转运基因OsANT1在水稻选育中的应用。
本发明的目的通过下述技术方案实现:
本发明以水稻的氨基酸转运基因OsANT1为对象,从水稻中花11中克隆了OsANT1的cDNA序列。通过构建OsANT1基因的超表达载体,采用农杆菌EHA105介导的遗传转化方法,将超表达载体导入正常粳稻品种中花11中,得到OsANT1基因表达量上升的超表达植株,超表达植株的根长、根数、株高和鲜重与对照野生型中花11相比显著提高。同时构建了OsANT1基因的干扰载体,将干扰载体导入中花11中,得到OsANT1基因的干扰植株,其根长、根数和鲜重与中花11相比显著降低。这些结果表明,通过提高OsANT1基因表达,可以促进水稻生长,增加生物量;OsANT1基因在阐述氨基酸运输影响植物生长及发育过程方面具有重要的应用价值。
基于本发明发现的OsANT1基因的功能,其可用于水稻选育中。所述的水稻选育为提高水稻根长、根数、株高和鲜重,从而促进水稻生长,提高水稻物量。具体可通过超表达提高OsANT1基因的表达,使水稻根长、根数、株高和鲜重增加,达到提高水稻生物量的目的。所述的OsANT1基因编码的OsANT1蛋白的氨基酸序列如SEQ ID NO.1所示;所述的OsANT1基因的cDNA序列优选如SEQ ID NO.2所示。
应该理解为,在不影响OsANT1蛋白活性的前提下(即不在蛋白的活性中心),本领域技术人员可对SEQ ID NO.1所示的氨基酸序列进行各种取代、添加和/或缺失一个或几个氨基酸获得具有同等功能的氨基酸序列。因此,OsANT1蛋白还包括SEQ ID NO.1所示氨基酸序列经取代、替换和/或增加一个或几个氨基酸获得的具有同等活性的蛋白质。此外,应理解,考虑到密码子的简并性以及不同物种密码子的偏爱性,本领域技术人员可以根据需要使用适合特定物种表达的密码子。
本发明的优点和效果:
(1)本发明克隆的OsANT1基因提高表达后使水稻根长、根数、株高和鲜重增强,说明OsANT1基因对提高水稻生物量较明显,因此,通过基因工程技术提高OsANT1基因的表达能够提高植物生物量。这不仅有助于通过正常施氮条件下培育高产水稻,还可以通过结合分子育种进行植物的品种改良。
(2)OsANT1基因的成功克隆,进一步证实了氨基酸转运家族在氮吸收过程中的重要作用,对阐明氨基酸转运家族的生物学功能有重要的意义,另外对进一步了解植物氮代谢途径,提高氮吸收效率有极大的推动作用。
(3)尽管目前已克隆到了一些氮营养途径中影响植物生长的基因,但对植物生长发育的分子机制仍不清楚。而本发明克隆的OsANT1基因能够提高水稻的生物量,对确定植物增产的关键因素有极大的推动作用。
附图说明
图1是对照中花11、OsANT1基因超表达植株2个株系和OsANT1基因干扰植株2个株系的整株表型图。
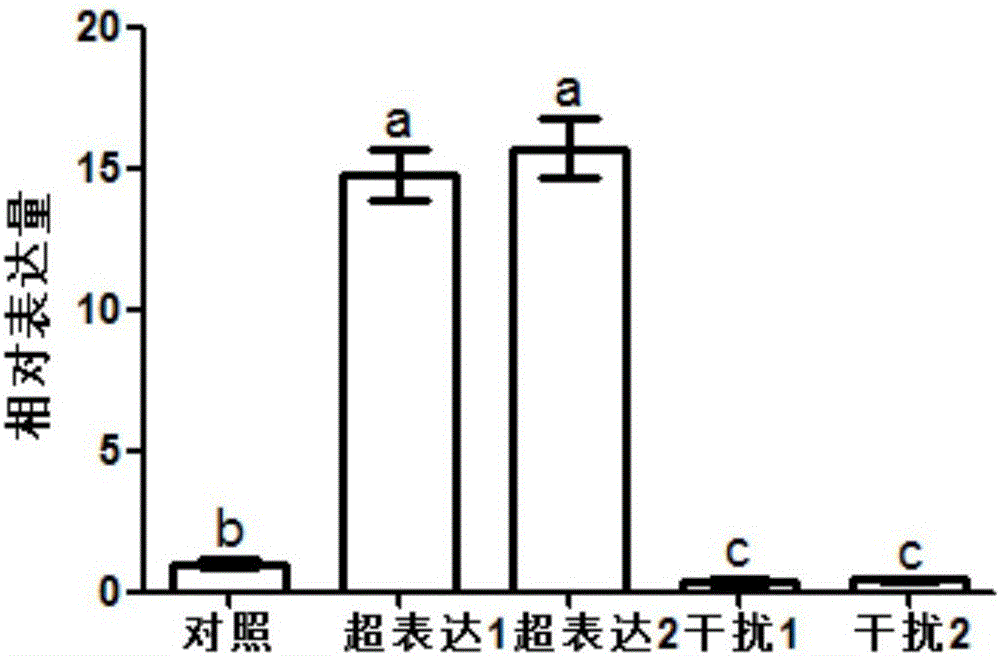
图2是对照中花11、OsANT1基因超表达植株2个株系和OsANT1基因干扰植株2个株系OsANT1基因表达量的统计柱状图,数据采用SPSS软件进行变量分析(ANOVA),使用Duncan’s在0.05水平上进行差异显著性分析,不同组别小写字母(a,b,c)表示之间具有差异显著。
图3是对照中花11和OsANT1基因超表达植株2个株系和OsANT1基因干扰植株2个株系根长的统计柱状图,数据采用SPSS软件进行变量分析(ANOVA),使用Duncan’s在0.05水平上进行差异显著性分析,不同组别小写字母(a,b,c)表示之间具有差异显著。
图4是对照中花11、OsANT1基因超表达植株2个株系和OsANT1基因干扰植株2个株系根数的统计柱状图,数据采用SPSS软件进行变量分析(ANOVA),使用Duncan’s在0.05水平上进行差异显著性分析,不同组别小写字母(a,b,c)表示之间具有差异显著。
图5是对照中花11、OsANT1基因超表达植株2个株系和OsANT1基因干扰植株2个株系株高的统计柱状图,数据采用SPSS软件进行变量分析(ANOVA),使用Duncan’s在0.05水平上进行差异显著性分析,不同组别小写字母(a,b)表示之间具有差异显著。
图6是对照中花11、OsANT1基因超表达植株2个株系和OsANT1基因干扰植株2个株系鲜重的统计柱状图,数据采用SPSS软件进行变量分析(ANOVA),使用Duncan’s在0.05水平上进行差异显著性分析,不同组别小写字母(a,b,c)表示之间具有差异显著。
具体实施方式
下面结合实施例对本发明做进一步详细的说明,但本发明的实施方式不限于此。若未特别指明,下述实施例所用的技术手段为本领域技术人员所熟知的常规手段;所用的实验方法均为常规方法,并可按照已描述的重组技术(参见分子克隆,实验室手册,第2版,冷泉港实验室出版社,冷泉港,纽约)完成;所用的材料、试剂等,均可从商业途径得到。
实施例1 OsANT1基因超表达植株的构建
提取水稻中花11的RNA,并将其反转录成cDNA,利用引物对:
F1:5'-AGATCTATGGAGGGGGCGGCGCCGCTTCT-3'(BglII),
R1:5'-CTTAAGCTGCAGTTGAATCCTGCATTGTGG-3'(AflII);
通过PCR扩增OsANT1基因的cDNA后,通过BglII和AflII酶切后连入pCAMBIA-1301载体(pCAMBIA-1301载体购自Cambia公司),构建出OsANT1基因的超表达载体OsANT1-p1301。采用农杆菌EHA105介导的遗传转化方法,将超表达载体导入正常水稻品种中花11中。
将得到的所有转基因小苗移栽于带泥土的筐中,定期浇水,施肥,待小苗长高约10cm时,将50株水稻转基因小苗浸泡于蒸馏水制备的含浓度为50mg/L的500mL潮霉素溶液中48小时,之后叶子为绿色并舒张、生长状态良好的植株为阳性转基因植株;而叶子枯黄并卷曲的植株为阴性植株,随即死亡。阳性植株单株种植并收种,直至T2代鉴定出在上述潮霉素溶液中无任何叶子枯黄并卷曲的为纯合的转基因植株,即得到OsANT1基因超表达植株。OsANT1基因超表达植株比对照中花11植株根长、根数、株高和鲜重增加,并达到差异显著,如图1、图3-6中所示。
取OsANT1基因超表达植株叶片,提取RNA并将其反转录成cDNA,通过实时荧光定量PCR检测OsANT1基因在超表达植株的表达量,结果显示(图2)超表达植株中OsANT1基因的表达量显著高于对照中花11。实时荧光定量PCR所用引物对为:
F2:5'-GGATGCGCCACGCTCTACTG-3',
R2:5'-GCTACAGATCCACCTGCTTGGGAA-3'。
上述结果表明,OsANT1基因可以通过表达量的提高,增加水稻的根长、根数、株高和鲜重,最终提高水稻生物量。
实施例2 OsANT1基因干扰植株的构建
提取水稻中花11的RNA,并将其反转录成cDNA,利用引物对:
F3:5'-GGTACCGAGTGACTCCGGAAGCCAAGC-3'(KpnI),
R3:5'-GGATCCCAGTAGAGCGTGGCGCATCC-3'(BamH I);
F4:5'-ACTAGTGAGTGACTCCGGAAGCCAAGC-3'(SpeI),
R4:5'-GAGCTCCAGTAGAGCGTGGCGCATCC-3'(Sac I);
各自PCR扩增出OsANT1基因的cDNA片段,通过上述相应的限制性内切酶酶切后连入pTCK303载体,构建出OsANT1基因的干扰表达载体OsANT1-pTCK303。采用农杆菌EHA105介导的遗传转化方法,将干扰表达载体导入正常粳稻品种中花11中。
将得到的所有转基因小苗移栽于带泥土的筐中,定期浇水,施肥,待小苗长高约10cm时,将50株水稻转基因小苗浸泡于蒸馏水制备的含浓度为50mg/L的500mL潮霉素溶液中48小时,之后叶子为绿色并舒张、生长状态良好的植株为阳性转基因植株;而叶子枯黄并卷曲的植株为阴性植株,随即死亡。阳性植株单株种植并收种,直至T2代鉴定出在上述潮霉素溶液中无任何叶子枯黄并卷曲的为纯合的转基因植株,即得到OsANT1基因干扰植株。OsANT1基因干扰植株比对照中花11植株根长、根数和鲜重显著减少,如图1、图3-6中所示。
取OsANT1基因干扰植株叶片,提取RNA并将其反转录成cDNA,通过实时荧光定量PCR检测OsANT1基因在干扰植株的表达量,结果显示(图2)干扰植株中OsANT1基因的表达量比对照中花11降低。实时荧光定量PCR所用引物对同超表达植株的检测。
上述实施例为本发明较佳的实施方式,但本发明的实施方式并不受上述实施例的限制,其他的任何未背离本发明的精神实质与原理下所作的改变、修饰、替代、组合、简化,均应为等效的置换方式,都包含在本发明的保护范围之内。
SEQUENCE LISTING
<110> 武汉生物工程学院
<120> 氨基酸转运基因OsANT1在水稻选育中的应用
<130> 1
<160> 10
<170> PatentIn version 3.3
<210> 1
<211> 424
<212> PRT
<213> Oryza sativa
<400> 1
Met Glu Gly Ala Ala Pro Leu Leu Ala Ala Arg Gly Glu Glu Glu Val
1 5 10 15
Val Glu Gly Arg Arg Arg Gly Gly Gly Gly Gly Ala Thr Ser Ala Gln
20 25 30
Thr Leu Gly Asn Val Val Val Ser Ile Val Gly Thr Gly Val Leu Gly
35 40 45
Leu Pro Tyr Ala Phe Arg Thr Ala Gly Trp Val Ala Gly Ser Leu Gly
50 55 60
Val Ala Ala Ala Gly Cys Ala Thr Leu Tyr Cys Met Leu Leu Leu Val
65 70 75 80
Asp Cys Arg Asp Lys Leu Glu Glu Lys Glu Ser Glu Glu Thr Tyr His
85 90 95
Gly His Tyr Thr Tyr Gly Asp Leu Gly Glu Lys Cys Phe Gly Thr Ile
100 105 110
Gly Arg Cys Leu Thr Glu Ile Leu Ile Leu Val Ser Gln Ala Gly Gly
115 120 125
Ser Val Ala Tyr Leu Ile Phe Ile Gly Gln Asn Leu His Ser Val Phe
130 135 140
Ser Gln Leu Met Ser Pro Ala Ala Phe Ile Phe Ala Ile Leu Leu Pro
145 150 155 160
Met Gln Ile Ala Leu Ser Phe Ile Arg Ser Leu Ser Ser Leu Ser Pro
165 170 175
Phe Ser Ile Phe Ala Asp Val Cys Asn Val Leu Ala Met Ala Ile Val
180 185 190
Ile Lys Glu Asp Leu Gln Leu Phe Asp His Pro Phe Ala Asn Arg Ser
195 200 205
Ala Phe Asn Gly Leu Trp Ala Ile Pro Phe Thr Phe Gly Val Ala Val
210 215 220
Phe Cys Phe Glu Gly Phe Ser Met Thr Leu Ala Leu Glu Ser Ser Met
225 230 235 240
Ala Glu Arg Arg Lys Phe Arg Trp Val Leu Ser Gln Ala Val Val Gly
245 250 255
Ile Ile Ile Val Tyr Ala Cys Phe Gly Val Cys Gly Tyr Leu Ala Tyr
260 265 270
Gly Glu Ala Thr Lys Asp Ile Ile Thr Leu Asn Leu Pro Asn Ser Trp
275 280 285
Ser Ser Ala Ala Val Lys Val Gly Leu Cys Ile Ala Leu Val Phe Thr
290 295 300
Phe Pro Val Met Met His Pro Ile His Glu Ile Val Glu Glu Arg Phe
305 310 315 320
Gln Ser Ser Gly Cys Phe Pro Lys Leu Ser His Lys Val Arg Gly Ala
325 330 335
Glu Trp Val Gly Leu His Ser Ser Arg Ile Val Met Val Thr Ile Leu
340 345 350
Ser Val Val Ala Ser Phe Ile Pro Ala Phe Gly Ser Phe Ile Ser Phe
355 360 365
Val Gly Ser Thr Val Cys Ala Leu Leu Ser Phe Val Leu Pro Thr Ile
370 375 380
Phe His Leu Ser Ile Val Gly Ser Ser Met Ser Pro Trp Arg Arg Trp
385 390 395 400
Gly Asp Tyr Gly Phe Leu Leu Phe Gly Leu Gly Phe Ala Gly Tyr Gly
405 410 415
Leu Ile Arg Ala Leu Phe Ser His
420
<210> 2
<211> 1275
<212> DNA
<213> Oryza sativa
<400> 2
atggaggggg cggcgccgct tctggcggcg cgtggggagg aggaggtggt ggaggggagg 60
cggcgcgggg gagggggcgg ggcgacgtcg gcgcagacgc tggggaacgt ggtggtgtcc 120
atcgtgggga ccggggtgct cggcctcccc tacgcgttcc gcaccgccgg ctgggtcgcc 180
ggctcgctcg gcgtcgccgc cgccggatgc gccacgctct actgcatgct cctcctcgtt 240
gactgcagag ataaattgga agagaaagaa tctgaagaaa cctaccatgg tcactataca 300
tatggtgatt tgggtgaaaa gtgttttggc actataggtc gatgcttgac agaaatcctc 360
atccttgttt cccaagcagg tggatctgta gcttatctaa tattcattgg ccaaaatctt 420
cattctgtct tcagccagtt gatgtcacca gctgccttca tctttgccat tctcctgcct 480
atgcaaatcg ctctatcatt catccgttca ctatcttctc tctccccatt cagcatattt 540
gcagacgtct gcaatgttct tgctatggca attgttatca aagaggatct tcagcttttt 600
gatcacccat ttgcgaatag aagtgctttc aatggacttt gggcgatacc attcactttt 660
ggagttgcgg tcttctgctt cgaaggattc agtatgactt tggcactaga atcatcaatg 720
gcagaacgga gaaaattccg ctgggtactt tctcaagcag ttgtcggtat cataattgtc 780
tatgcatgtt ttggagtttg tggttactta gcctatgggg aggccactaa agacatcata 840
acacttaatc ttcccaatag ttggtcttct gctgccgtta aggttggtct ttgcatagca 900
ctggtattca catttccagt catgatgcac ccaatccacg agattgtcga ggaaagattc 960
caatcaagtg gatgtttccc gaagctctcg cacaaggttc gcggggctga atgggtgggc 1020
ctgcactcaa gccgcatcgt catggtaact atcctgtctg tggtggcatc cttcatacct 1080
gcgttcgggt ccttcatctc ctttgtcggg agcacggttt gcgcgctgct ctccttcgtg 1140
ctgcctacga tcttccatct gagcattgtg ggttcctcga tgagtccgtg gcgacgctgg 1200
ggggactacg gtttccttct cttcggcctt ggttttgcgg gctacgggct cattagggct 1260
ctcttctcac attga 1275
<210> 3
<211> 29
<212> DNA
<213> Artificial
<220>
<223> 引物F1
<400> 3
agatctatgg agggggcggc gccgcttct 29
<210> 4
<211> 30
<212> DNA
<213> Artificial
<220>
<223> 引物R1
<400> 4
cttaagctgc agttgaatcc tgcattgtgg 30
<210> 5
<211> 20
<212> DNA
<213> Artificial
<220>
<223> 引物F2
<400> 5
ggatgcgcca cgctctactg 20
<210> 6
<211> 24
<212> DNA
<213> Artificial
<220>
<223> 引物R2
<400> 6
gctacagatc cacctgcttg ggaa 24
<210> 7
<211> 27
<212> DNA
<213> Artificial
<220>
<223> 引物F3
<400> 7
ggtaccgagt gactccggaa gccaagc 27
<210> 8
<211> 26
<212> DNA
<213> Artificial
<220>
<223> 引物R3
<400> 8
ggatcccagt agagcgtggc gcatcc 26
<210> 9
<211> 27
<212> DNA
<213> Artificial
<220>
<223> 引物F4
<400> 9
actagtgagt gactccggaa gccaagc 27
<210> 10
<211> 26
<212> DNA
<213> Artificial
<220>
<223> 引物R4
<400> 10
gagctccagt agagcgtggc gcatcc 26
- 还没有人留言评论。精彩留言会获得点赞!