一种脯氨酸蛋白酶突变体及其应用的制作方法
本发明属于基因工程技术领域,具体内容涉及一种脯氨酸蛋白酶突变体应用。
技术背景
脯氨酸蛋白酶,也称脯氨酰内肽酶(简称PEP)或脯氨酰寡肽酶,属于丝氨酸蛋白酶中一种新的家族,该家族能专一性地水解多肽中脯氨酸残基的羧基端肽键,含有典型的丝氨酸蛋白酶家族的G-X-S-X-G/A高度保守序列,但是活性位点Ser周围的氨基酸残基和其他典型的丝氨酸蛋白不同,而且与已知的丝氨酸蛋白酶家族(包括胰蛋白酶,枯草杆菌蛋白酶,羧肽酶Y)相比,在一级结构上同源性很低。
脯氨酸蛋白酶不仅能降解催产素,还可以降解许多催产素类似物,抗利尿激素的降解也是由于脯氨酸羧基端肽键的断裂而形成的。此外,很多肽类激素的也是由于脯氨酸残基的羧基端肽键以相同的方式被断裂而灭活的。相反,人体的胃泌素,促肾上腺皮质激素和豚鼠的胶原蛋白却不能被这类酶所降解,脯氨酸蛋白酶也不能够降解很多分子量比较大的蛋白质和多肽,例如血清蛋白、IgG、弹力蛋白、明胶、酪蛋白,促肾上腺激素释放激素,泛素和蛋白酶抑制剂等。根据对脯氨酸蛋白酶降解的多种肽类激素和人工合成多肽的分析,发现不同来源的脯氨酸蛋白酶可以专一性降解肽链内Y-Pro-Y'中的顺式结构Pro-Y'键,Y'可以是除脯氨酸之外的氨基酸、多肽、酰胺、芳香胺或者醇类化合物。
在食品与发酵工业,脯氨酰蛋白酶是一种有价值的酶类,如能通过水解啤酒中富含脯氨酸的蛋白多肽,阻止其与多酚类物质形成大聚合物浑浊沉淀,从而提高啤酒的非生物稳定性。已有报道证明脯氨酸蛋白酶在蛋白水解物中的去苦味作用。此外,鉴于脯氨酸蛋白酶的特异性,该酶还有可能作为一种分子生物学的工具酶,用于蛋白质序列测定、特异位点的酶切、肽段的修饰和加工等。
作为一种非常重要的工业用酶,脯氨酸蛋白酶目前主要应用在食品加工业和啤酒酿酒业,尤其在啤酒生产和面包制造业应用很突出。脯氨酸蛋白酶可以降解啤酒中的浑浊敏感蛋白的脯氨酸而使其失活,在不影响发酵性能的前提下,显著性的提高了啤酒的非生物稳定性。
在工业生产中,追求微生物最大化表达量的酶,提高酶的特定性质的意义重大,不仅可以显著降低生产成本,并减少能源消耗和环境污染。一般情况下,除了通过传统菌种诱变方法提高微生物表达量,还可以通过基因工程手段如突变、利用强启动子、多拷贝基因、合适的信号肽等改造其性能及表达量。因此,通过筛选或改造的方法获得性能优良的脯氨酸蛋白酶成为本领域内的研究热点。
技术实现要素:
本发明为解决现有技术问题,提供了一种新型的脯氨酸蛋白酶突变体及其应用。所述突变体是申请人通过定向进化技术筛选获得的,其耐热性显著高于野生型,有利于脯氨酸蛋白酶的广泛应用。
本发明一方面提供了一种脯氨酸蛋白酶,其氨基酸序列为SEQ ID NO:1。
所述脯氨酸蛋白酶的编码基因的核苷酸序列为SEQ ID NO:2。
本发明另一方面提供了一种脯氨酸蛋白酶突变体,是氨基酸序列为SEQ ID NO:1的脯氨酸蛋白酶的第101位氨基酸由Gly变为Glu,第146位氨基酸由Val变为Ile,第441位氨基酸由Arg变为Trp。
所述脯氨酸蛋白酶突变体的氨基酸序列为SEQ ID NO:3,其编码基因的核酸序列为SEQ ID NO:4。
本发明另一方面提供了一种重组质粒,其携带有编码序列为SEQ ID NO:4的脯氨酸蛋白酶突变体的基因。
本发明还提供了一种重组菌株,是通过将上述重组质粒转化入黑曲霉(Aspergillus niger)中获得的。
本发明还提供了所述脯氨酸蛋白酶突变体在啤酒或饮料生产中的应用。
本发明通过定向进化技术筛选获得的脯氨酸蛋白酶突变体,其最适反应pH值为4.0,最适反应温度为55℃,且在45-60℃范围内均能保持80%以上的酶活,耐热性显著高于野生型。所述突变体可广泛应用于啤酒生产中,能显著降低啤酒的浊度。所述突变体可在麦汁制备过程中添加,其麦汁浊度减少量比添加相同用量的野生型脯氨酸蛋白酶的实验组1提高了29.4%-48.3%;所述突变体也可在啤酒发酵过程中添加,其啤酒的浊度减少量比添加相同用量的野生型脯氨酸蛋白酶的实验组1提高了8.9%-15.4%。从而说明本发明提供的脯氨酸蛋白酶突变体更能耐受麦汁糖化过程中的高温,并且对发酵过程中敏感蛋白的酶解效果明显优于野生型脯氨酸蛋白酶,因而更有利于减少敏感蛋白而引起的浑浊,取得了意料不到的效果。
附图说明
图1为质粒pSU图谱;
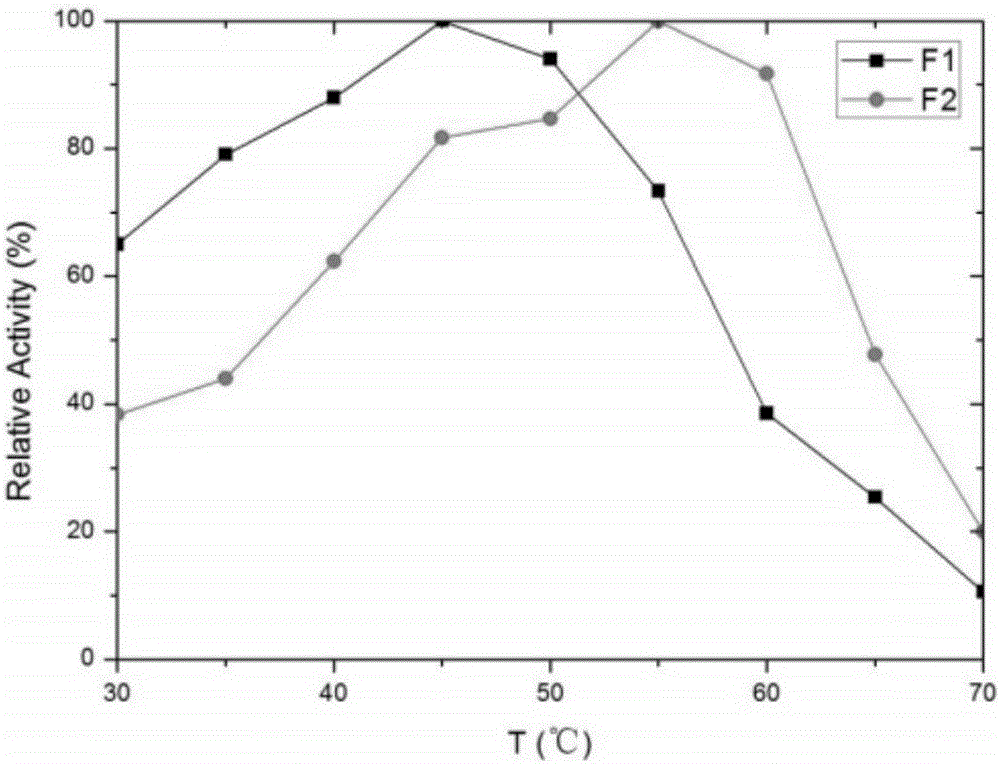
图2为脯氨酸蛋白酶F1及其突变体F2最适反应温度对比;
图3为脯氨酸蛋白酶F1及其突变体F2最适反应pH对比。
具体实施方式
本发明用到了遗传工程和分子生物学领域使用的常规技术和方法,例如MOLECULAR CLONING:ALABORATORY MANUAL,3nd Ed.(Sambrook,2001)和CURRENT PROTOCOLS IN MOLECULAR BIOLOGY(Ausubel,2003)中所记载的方法。这些一般性参考文献提供了本领域技术人员已知的定义和方法。但是,本领域的技术人员可以在本发明所记载的技术方案的基础上,采用本领域其它常规的方法、实验方案和试剂,而不限于本发明具体实施例的限定。
下面结合具体实施方式对本发明进行详细描述。
实施例1脯氨酸蛋白酶基因的获得
提取本实验室保存的一株黑曲霉(Aspergillus niger)Su2-1的基因组。利用下述引物F和引物R进行PCR扩增。反应条件为:94℃变性5min;然后94℃变性30s,56℃复性30s,72℃延伸120s,30个循环后,72℃保温10min。将扩增产物进行琼脂糖电泳,结果显示扩增获得的基因片段大小为2100bp。
引物F:ATGCGTTCCTTCTCCGCTGTCG
引物R:TCAAGCATAATACTCCTCCACC
将获得的基因片段送上海生工生物工程股份有限公司完成测序。测序结果显示该基因片段的核苷酸序列为SEQ ID NO:2,其编码的氨基酸序列为SEQ ID NO:1。通过NCBI BLAST比对发现,该基因编码的蛋白为脯氨酸蛋白酶,命名为F1。
实施例2脯氨酸蛋白酶突变体的获得
申请人为了提高上述脯氨酸蛋白酶F1的耐热性,通过定向进化技术对该酶进行了大量突变的筛选。
以F1基因为模板,以引物F和引物R用GeneMorph II随机突变PCR试剂盒(Stratagene)进行PCR扩增,胶回收PCR产物,EcoRI、NotI进行酶切处理后与经同样酶切后的pET21a载体连接,转化至大肠杆菌BL21(DE3)中,涂布于LB+Amp平板,37℃倒置培养,待转化子出现后,用牙签逐个挑至96孔板,每个孔中加入150ul含有0.1mM IPTG的LB+Amp培养基,37℃220rpm培养6h左右,离心弃上清,将菌体用pH5.5的缓冲液重悬,反复冻融破壁,获得含有脯氨酸蛋白酶F1的大肠杆菌细胞裂解液。
分别取出30μl裂解液至两块新的96孔板,其中一块于50℃处理10min后,稀释到pH5.5的缓冲液中,另一块不处理,稀释到pH5.5的缓冲液中,分别测定其酶活。
结果发现,不同的突变子经过高温处理后保持的酶活性不同,有些突变对脯氨酸蛋白酶的活性没有影响,有些突变甚至使其活性降低了,对依然保持高活性的突变子进行DNA测序。最终,申请人获得了能显著提高脯氨酸蛋白酶F1耐热性的三点突变体:G101E,V146I和R441W。
将上述含G101E,V146I和R441W三点突变的脯氨酸蛋白酶突变体命名为F2,其氨基酸序列为SEQ ID NO:3,参照该序列得到一个编码核苷酸序列为SEQ ID NO:4。
将上述脯氨酸蛋白酶突变体基因分别用引物F、R进行PCR扩增,引物两端引入EcoR I、Not I位点。PCR反应条件为:94℃变性5min;然后94℃变性30s,56℃复性30s,72℃延伸120s,30个循环后,72℃保温10min。琼脂糖凝胶电泳结果显示,上述脯氨酸蛋白酶突变体F2的基因大小约为2100bp。
实施例3重组表达载体的构建
将脯氨酸蛋白酶F1及其突变体F2基因分别通过XbaI连接到表达载体pSU(图1)中,构建得到表达载体pSU-F1、pSU-F2。
原生质体制备:接种宿主菌黑曲霉Su2-1于PDA+U平板,30℃培养5-7d。割取2cm×2cm大小的菌块,接种100ml液体PDA+U培养基中,30℃培养24h生长菌丝体,用于转化。将长好的菌丝体过滤后,用20ml 1.2M硫酸镁溶液重悬,加入0.2g溶菌酶。30℃、100rpm培养2-3h。将裂解好的菌丝用2层擦镜纸过滤,3000rpm离心10min获得原生质体。
转化:原生质体用1.2M山梨醇溶液清洗2遍,再用适量的山梨醇溶液重悬,使原生质体浓度达到108。200ul原生质体中加入10ul准备好的质粒,加入50ul 25%的PEG6000,冰浴20min,再加入2ml 25%的PEG6000室温放置5min,加入4ml山梨醇溶液颠倒混匀。倒入50ml转化上层培养基后,倾倒入4个转化下层平板中,待上层培养基凝固后,于30℃培养箱中倒置培养5d。
转化子筛选:培养5d后,挑取长出的菌落,点种到转化下层平板进行复筛,30℃培养2d。将正常生长的转化子分别接种到新鲜的PDA平板,30℃培养5-7d。每个转化子割取2cm×2cm大小的菌块,分别接种50ml液体摇瓶培养基中发酵,32℃培养5d,每天加入适量氨水,控制pH在4.5左右。培养5d后,离心菌体获得上清液即为粗酶液。通过对粗酶液进行蛋白电泳检测,筛选出有明显蛋白条带表达的转化子。
申请人将筛选到的其中一个重组表达脯氨酸蛋白酶F1的阳性转化子命名为黑曲霉Su-F1(Aspergillus niger Su-F1),一个重组表达脯氨酸蛋白酶突变体F2的阳性转化子命名为黑曲霉Su-F2(Aspergillus niger Su-F2)。
将黑曲霉Su-F1和黑曲霉Su-F2的发酵上清液进行脯氨酸蛋白酶酶活检测,同时以黑曲霉宿主菌发酵上清液做对照。结果表明:黑曲霉宿主菌的发酵上清液酶活仅为3.2U/ml,而黑曲霉Su-F1和黑曲霉Su-F2的发酵上清液酶活分别为30.5u/ml、35.4u/ml。从而进一步说明,本发明构建的重组菌黑曲霉Su-F1和黑曲霉Su-F2能分别重组表达脯氨酸蛋白酶F1和及其突变体F2。
酶活测定方法
(1)脯氨酸蛋白酶酶活单位的定义
在37℃、pH值为5.0的条件下,每分钟分解Z-GLY-Pro-pNA释放1μmolpNA的酶量,定义为一个酶活力单位U。
(2)酶活测定方法
试剂:磷酸氢二钠(0.2mol/L)-柠檬酸(0.1mol/L)缓冲液pH5.0
Z-GLY-Pro-pNA溶液(5mmol/L)
取1ml浓度为5mmol/L的Z-GLY-Pro-pNA溶液于10ml磷酸-柠檬酸缓冲液中混匀,测样前10min配制,可使用4h。
在试管中加入1.1ml上述底物混合液;
将试管在37℃的水浴锅中预热15min;
将比色皿(0.5cm)放置在比色皿架上,比色皿架的温控调整到37℃,分光光度计调节至410nm测定吸光度;
吸取0.1ml稀释的酶液,加入至含底物混合液的试管中,加入酶液时开始计时,涡旋混匀后倒于比色皿中;
将比色皿置于比色皿架上后,读取30s的数据;
酶液反应时如出现吸光值下降后并上升的趋势,则记录下降至谷底的吸光值及时间,并延续记录10min后数据;
如不出现吸光值下降的情况,可直接记录反应前后410nm处的吸光值,反应时间为10min。
通过ΔA410计算脯氨酸蛋白酶酶活:
U=(ΔA410×V×1000×1000×N)/(T×γ×ν×β)
其中:ΔA410:吸光值变化;
T:反应时间(10min);
V:反应体系体积(1.2ml);
1000×1000:将mol换算成μmol;
γ:摩尔消光系数(cm2/mol)8800;
β:比色杯光程(0.5cm);
ν:样品量(0.1ml);
N:稀释倍数;
实施例4酶学性质分析
1、最适反应温度测定
分别在30℃、35℃、40℃、45℃、50℃、55℃、60℃、65℃、70℃,pH5.0条件下测定实施例3获得的重组菌株黑曲霉Su-F1和黑曲霉Su-F2发酵粗酶液的脯氨酸蛋白酶活力,以最高酶活为100%,计算相对酶活,做温度-相对酶活曲线。结果如图2所示,脯氨酸蛋白酶F1的最适反应温度为45℃,而脯氨酸蛋白酶突变体F2的最适反应温度为55℃,且在45-60℃范围内均能保持80%以上的酶活,耐温性得到显著提高。
2、最适反应pH测定
采用pH值分别为3.0、3.5、4.0、4.5、5.0、5.5、6.0、6.5、7.0的缓冲液,将实施例3获得的重组菌株黑曲霉Su-F1和黑曲霉Su-F2发酵粗酶液进行稀释测定,在37℃下进行脯氨酸蛋白酶活力测定,计算酶活,以最高酶活为100%,计算相对酶活,做pH-相对酶活曲线。结果如图3所示,脯氨酸蛋白酶F1的最适反应pH值为4.0,而脯氨酸蛋白酶突变体F2的最适反应pH值也为4.0,没有明显变化。
实施例5脯氨酸蛋白酶在啤酒生产中的应用
应用一:在麦汁制备过程中添加脯氨酸蛋白酶
1、准备10个相同的糖化杯,分别命名为1-10号;向每个糖化杯中各加入250ml、55℃的水,和70g粉碎的麦芽粉,混匀溶解;
2、1号糖化杯作为空白组,不再添加其他物质;
2-4号糖化杯作为实验组1,分别按10u/l、20u/l、30u/l的比例添加本发明提供的野生型脯氨酸蛋白酶;
5-7号糖化杯为实验组2,分别按10u/l、20u/l、30u/l的比例添加本发明提供的脯氨酸蛋白酶突变体;
8-10号糖化杯为实验组3,暂时不添加其他物质。
3、糖化过程:将1-10号糖化杯55℃保温30分钟,以1℃/min的升温速度加热到65℃,保温55分钟,以1℃/min的升温速度到72℃,保温10分钟,77℃保温2分钟后快速降至室温。向各糖化杯中补充水分至450g,过滤得到麦汁。
4、分别测定1-7号麦汁的初始浊度,具体结果见表1。
5、向8-10号麦汁中分别按0.3g/l、0.6g/l、0.9g/l的比例添加PPVP(交联聚乙烯吡咯烷酮,目前啤酒厂普遍使用的用于降低啤酒浊度的化合物),30分钟后,过滤麦汁,测定其初始浊度,具体结果见表1。
6、麦汁浑浊测试:
(1)原理:
啤酒在低温贮藏过程中容易产生浑浊,主要是因为啤酒中的敏感蛋白(富含脯氨酸的蛋白)容易与多酚类物质结合产生絮凝。根据单宁仪的原理,通过添加单宁(属于多酚类),加速啤酒的浑浊,然后通过浊度计测定其浊度的变化趋势,进而确定脯氨酸蛋白酶或PVPP对啤酒浑浊的减少量。
(2)具体步骤:
分别向1-10号麦汁中添加浓度为0.1g/l的单宁,使麦汁中的单宁含量达到7.5mg/l,充分混匀5分钟后,倒入玻璃瓶中,用校正后的浊度计分别测定麦汁浑浊测试后的浊度,计算麦汁浊度减少量,具体结果见表1。
计算公式:
浊度减少量(%)=1-(实验组浑浊测试后浊度-实验组初始浊度)/(空白组浑浊测试后浊度-空白组初始浊度)*100%
表1麦汁浊度表
由表1的结果可知,与空白组相比,实验组通过在麦汁糖化过程中添加本发明提供的脯氨酸蛋白酶以及在麦汁糖化结束后添加PVPP均能显著降低麦汁在浑浊测试后的浊度,说明脯氨酸蛋白酶和PVPP能有效减少麦汁中的敏感蛋白含量,进而减少测试过程中浑浊的产生。其中,添加脯氨酸蛋白酶的实验组1和2制备得到的麦汁,其浊度减少量要高于添加对应剂量PVPP的实验组3,从而说明本发明提供的脯氨酸蛋白酶在降低麦汁浑浊方面的效果要明显优于啤酒厂目前常用的PVPP,能有效减少敏感蛋白而引起的浑浊,提高稳定性。
进一步地,添加脯氨酸蛋白酶突变体的实验组2,其麦汁浊度减少量比添加相同用量的野生型脯氨酸蛋白酶的实验组1提高了29.4%-48.3%,从而说明本发明提供的脯氨酸蛋白酶突变体更能耐受麦芽汁糖化过程中的高温,对敏感蛋白的酶解效果明显优于野生型脯氨酸蛋白酶,因而更有利于减少敏感蛋白而引起的浑浊,取得了意料不到的效果。
应用二:在啤酒发酵过程中添加脯氨酸蛋白酶
1、将纯麦芽经过糖化、过滤、煮沸(添加啤酒花)、旋沉,制备得到麦汁。
2、首先按表2中所述的添加量分别将本发明提供的脯氨酸蛋白酶与酵母液加入到发酵罐中,同时,以不加脯氨酸蛋白酶的发酵罐作为空白对照组;再将麦汁经换热泵降温充氧后灌入发酵罐;9~10℃发酵5天,后升温至12℃,糖度降至3.8~4.20Bix,封罐发酵5天,然后降温至0℃,发酵结束,即得啤酒溶液。将啤酒过滤后,分别测定其浊度,计算浊度减少量,具体结果见表2。
3、向空白对照组制得的啤酒中分别按0.3g/l、0.6g/l、0.9g/l的比例添加PPVP,30分钟后,过滤啤酒,测定其浊度,计算浊度减少量,具体结果见表2。
计算公式:
浊度减少量(%)=(空白组啤酒浊度-实验组啤酒浊度)/空白组啤酒浊度*100%
表2啤酒浊度表
从表2的数据可以知道,与空白对照组相比,实验组通过在发酵过程中添加本发明提供的脯氨酸蛋白酶以及在发酵结束后添加PVPP均能显著降低啤酒的浊度,从而说明说明脯氨酸蛋白酶和PVPP能有效减少啤酒中的敏感蛋白含量,进而减少浑浊的产生。其中,添加脯氨酸蛋白酶的实验组1和2制备得到的啤酒,其浊度减少量要明显高于添加对应剂量PVPP的实验组3,从而说明本发明提供的脯氨酸蛋白酶在降低啤酒浑浊方面的效果要明显优于啤酒厂目前常用的PVPP,能有效减少敏感蛋白而引起的浑浊,提高啤酒的稳定性。
进一步地,添加脯氨酸蛋白酶突变体的实验组2,其啤酒的浊度减少量比添加相同用量的野生型脯氨酸蛋白酶的实验组1提高了8.9%-15.4%,从而说明本发明提供的脯氨酸蛋白酶突变体在发酵过程中对敏感蛋白的酶解效果明显优于野生型脯氨酸蛋白酶,因而更有利于减少敏感蛋白而引起的浑浊,取得了意料不到的效果。
SEQUENCE LISTING
<110> 青岛蔚蓝生物集团有限公司
潍坊康地恩生物科技有限公司
<120> 一种脯氨酸蛋白酶突变体及其应用
<130>
<160> 4
<170> PatentIn version 3.5
<210> 1
<211> 526
<212> PRT
<213> 1
<400> 1
Met Arg Ser Phe Ser Ala Val Ala Ala Ala Ala Leu Ala Leu Ser Trp
1 5 10 15
Ala Ser Leu Ala Gln Ala Ala Arg Pro Arg Leu Val Pro Lys Pro Val
20 25 30
Pro Arg Pro Ala Ser Ser Lys Ser Ala Ala Thr Thr Gly Glu Ala Asn
35 40 45
Phe Glu Gln Leu Leu Asp His His Asn Pro Asp Lys Gly Thr Phe Ser
50 55 60
Gln Arg Tyr Trp Trp Ser Thr Glu Tyr Trp Gly Gly Pro Gly Ser Pro
65 70 75 80
Val Val Leu Phe Thr Pro Gly Glu Val Ser Ala Asp Gly Tyr Glu Gly
85 90 95
Tyr Leu Thr Asn Gly Thr Leu Thr Gly Val Tyr Ala Gln Glu Ile Gln
100 105 110
Gly Ala Val Ile Ile Ile Glu His Arg Tyr Trp Gly Asp Ser Ser Pro
115 120 125
Tyr Glu Val Leu Asn Ala Glu Thr Leu Gln Tyr Leu Thr Leu Asp Gln
130 135 140
Ala Val Leu Asp Leu Thr Tyr Phe Ala Glu Thr Val Lys Leu Gln Phe
145 150 155 160
Asp Asn Ser Thr Arg Ser Asn Ala Gln Asn Ala Pro Trp Val Met Val
165 170 175
Gly Gly Ser Tyr Ser Gly Ala Leu Thr Ala Trp Thr Glu Ser Val Ala
180 185 190
Pro Gly Thr Phe Trp Ala Tyr His Ala Thr Ser Ala Pro Val Glu Ala
195 200 205
Ile Tyr Asp Phe Trp Gln Tyr Phe Tyr Pro Ile Gln Gln Gly Met Ala
210 215 220
Gln Asn Cys Ser Lys Asp Val Ser Leu Val Ala Glu Tyr Val Asp Lys
225 230 235 240
Val Gly Lys Asn Gly Thr Ala Lys Glu Gln Gln Ala Leu Lys Glu Leu
245 250 255
Phe Gly Leu Gly Ala Val Glu His Phe Asp Asp Phe Ala Ala Val Leu
260 265 270
Pro Asn Gly Pro Tyr Leu Trp Gln Asp Asn Asp Phe Ala Thr Gly Tyr
275 280 285
Ser Ser Phe Phe Gln Phe Cys Asp Ala Val Glu Gly Val Glu Ala Gly
290 295 300
Ala Ala Val Thr Pro Gly Pro Glu Gly Val Gly Leu Glu Lys Ala Leu
305 310 315 320
Ala Asn Tyr Ala Asn Trp Phe Asn Ser Thr Ile Leu Pro Asp Tyr Cys
325 330 335
Ala Gly Tyr Gly Tyr Trp Thr Asp Glu Trp Ser Val Ala Cys Phe Asp
340 345 350
Ser Tyr Asn Ala Ser Ser Pro Ile Tyr Thr Asp Thr Ser Val Gly Asn
355 360 365
Pro Val Asp Arg Gln Trp Glu Trp Phe Leu Cys Asn Glu Pro Phe Phe
370 375 380
Tyr Trp Gln Asp Gly Ala Pro Glu Gly Thr Ser Thr Ile Val Pro Arg
385 390 395 400
Leu Val Ser Ala Ser Tyr Trp Gln Arg Gln Cys Ser Leu Tyr Phe Pro
405 410 415
Glu Thr Asn Gly Tyr Thr Tyr Gly Ser Ala Lys Gly Lys Asn Ser Ala
420 425 430
Thr Val Asn Ser Trp Thr Gly Gly Arg Asp Met Thr Arg Asn Thr Thr
435 440 445
Arg Leu Ile Trp Thr Asn Gly Gln Tyr Asp Pro Trp Arg Asp Ser Gly
450 455 460
Val Ser Ser Thr Phe Arg Pro Gly Gly Pro Leu Ala Ser Thr Ala Asn
465 470 475 480
Glu Pro Val Gln Val Ile Pro Gly Gly Phe His Cys Ser Asp Leu Tyr
485 490 495
Met Ala Asp Tyr Tyr Ala Asn Glu Gly Val Lys Lys Val Val Asp Asn
500 505 510
Glu Val Lys Gln Ile Lys Glu Trp Val Glu Glu Tyr Tyr Ala
515 520 525
<210> 2
<211> 1581
<212> DNA
<213> 2
<400> 2
atgcgttcct tctccgctgt cgctgccgca gccctggcgc tctcttgggc gtctctggct 60
caggctgctc gccctcgtct tgtgcccaag cctgtccctc ggccagcttc gagtaaatcg 120
gctgcgacca caggcgaggc taactttgag cagttgctgg accatcataa tccagacaag 180
ggaacgtttt cccagcggta ctggtggagt actgaatact ggggtggtcc tgggtcaccg 240
gttgtcctct ttactcctgg agaggtctct gccgatggct atgaggggta tctcaccaat 300
gggactctca ctggtgttta tgcgcaggag atccagggtg ccgtcattat cattgagcac 360
cgctactggg gtgattcttc gccttatgag gtactcaatg ccgaaactct tcagtatctt 420
acattggacc aagccgttct ggacctgacc tacttcgccg agacggtgaa actgcaattc 480
gataacagca cccgcagcaa tgcgcagaat gctccctggg tcatggtcgg tggatcatac 540
agcggtgcct tgacggcttg gaccgagtct gtcgcgcctg gaacgttctg ggcttaccat 600
gccactagtg ctcctgtgga ggctatctat gacttttggc aatacttcta ccccatccag 660
caaggtatgg cacagaactg cagcaaggac gtgtctctgg tagccgagta tgtcgacaag 720
gttggaaaga acggaactgc caaggagcag caggcactca aggaattgtt tggcttggga 780
gctgttgagc atttcgatga ctttgccgct gtcctcccca acggaccgta cctctggcag 840
gacaacgact ttgccacagg atactcttcc ttcttccagt tctgtgacgc cgtcgagggt 900
gtcgaagccg gcgcggcagt aacccccggc cccgagggtg tcggcctcga aaaggccctg 960
gccaactacg caaactggtt caattcaacc attctccctg attactgcgc aggctacggc 1020
tactggaccg acgaatggag cgtcgcctgc ttcgacagct acaacgcctc gagccctatc 1080
tacaccgata catccgtcgg caatcccgtc gaccgccaat gggaatggtt cctttgcaac 1140
gagcctttct tctactggca agacggtgct cccgagggta cctccaccat tgtgccccgg 1200
ctcgtcagcg cctcctactg gcaacgccaa tgctcgctct acttccccga aacgaacggc 1260
tacacgtacg gtagcgcgaa gggtaagaac tctgccacgg tgaacagctg gaccggtggg 1320
cgggacatga cccgcaacac gacgcggttg atctggacga atgggcaata cgacccctgg 1380
cgcgactccg gtgtgtcgag cactttccgg cctggtggac cgctggcgag cacggcgaat 1440
gaacccgtgc aggttattcc gggcggattc cattgctccg atttgtatat ggcagattat 1500
tatgcgaacg agggggtaaa aaaggtggta gataatgagg tgaagcagat taaggagtgg 1560
gtggaggagt attatgcttg a 1581
<210> 3
<211> 526
<212> PRT
<213> 3
<400> 3
Met Arg Ser Phe Ser Ala Val Ala Ala Ala Ala Leu Ala Leu Ser Trp
1 5 10 15
Ala Ser Leu Ala Gln Ala Ala Arg Pro Arg Leu Val Pro Lys Pro Val
20 25 30
Pro Arg Pro Ala Ser Ser Lys Ser Ala Ala Thr Thr Gly Glu Ala Asn
35 40 45
Phe Glu Gln Leu Leu Asp His His Asn Pro Asp Lys Gly Thr Phe Ser
50 55 60
Gln Arg Tyr Trp Trp Ser Thr Glu Tyr Trp Gly Gly Pro Gly Ser Pro
65 70 75 80
Val Val Leu Phe Thr Pro Gly Glu Val Ser Ala Asp Gly Tyr Glu Gly
85 90 95
Tyr Leu Thr Asn Glu Thr Leu Thr Gly Val Tyr Ala Gln Glu Ile Gln
100 105 110
Gly Ala Val Ile Ile Ile Glu His Arg Tyr Trp Gly Asp Ser Ser Pro
115 120 125
Tyr Glu Val Leu Asn Ala Glu Thr Leu Gln Tyr Leu Thr Leu Asp Gln
130 135 140
Ala Ile Leu Asp Leu Thr Tyr Phe Ala Glu Thr Val Lys Leu Gln Phe
145 150 155 160
Asp Asn Ser Thr Arg Ser Asn Ala Gln Asn Ala Pro Trp Val Met Val
165 170 175
Gly Gly Ser Tyr Ser Gly Ala Leu Thr Ala Trp Thr Glu Ser Val Ala
180 185 190
Pro Gly Thr Phe Trp Ala Tyr His Ala Thr Ser Ala Pro Val Glu Ala
195 200 205
Ile Tyr Asp Phe Trp Gln Tyr Phe Tyr Pro Ile Gln Gln Gly Met Ala
210 215 220
Gln Asn Cys Ser Lys Asp Val Ser Leu Val Ala Glu Tyr Val Asp Lys
225 230 235 240
Val Gly Lys Asn Gly Thr Ala Lys Glu Gln Gln Ala Leu Lys Glu Leu
245 250 255
Phe Gly Leu Gly Ala Val Glu His Phe Asp Asp Phe Ala Ala Val Leu
260 265 270
Pro Asn Gly Pro Tyr Leu Trp Gln Asp Asn Asp Phe Ala Thr Gly Tyr
275 280 285
Ser Ser Phe Phe Gln Phe Cys Asp Ala Val Glu Gly Val Glu Ala Gly
290 295 300
Ala Ala Val Thr Pro Gly Pro Glu Gly Val Gly Leu Glu Lys Ala Leu
305 310 315 320
Ala Asn Tyr Ala Asn Trp Phe Asn Ser Thr Ile Leu Pro Asp Tyr Cys
325 330 335
Ala Gly Tyr Gly Tyr Trp Thr Asp Glu Trp Ser Val Ala Cys Phe Asp
340 345 350
Ser Tyr Asn Ala Ser Ser Pro Ile Tyr Thr Asp Thr Ser Val Gly Asn
355 360 365
Pro Val Asp Arg Gln Trp Glu Trp Phe Leu Cys Asn Glu Pro Phe Phe
370 375 380
Tyr Trp Gln Asp Gly Ala Pro Glu Gly Thr Ser Thr Ile Val Pro Arg
385 390 395 400
Leu Val Ser Ala Ser Tyr Trp Gln Arg Gln Cys Ser Leu Tyr Phe Pro
405 410 415
Glu Thr Asn Gly Tyr Thr Tyr Gly Ser Ala Lys Gly Lys Asn Ser Ala
420 425 430
Thr Val Asn Ser Trp Thr Gly Gly Trp Asp Met Thr Arg Asn Thr Thr
435 440 445
Arg Leu Ile Trp Thr Asn Gly Gln Tyr Asp Pro Trp Arg Asp Ser Gly
450 455 460
Val Ser Ser Thr Phe Arg Pro Gly Gly Pro Leu Ala Ser Thr Ala Asn
465 470 475 480
Glu Pro Val Gln Val Ile Pro Gly Gly Phe His Cys Ser Asp Leu Tyr
485 490 495
Met Ala Asp Tyr Tyr Ala Asn Glu Gly Val Lys Lys Val Val Asp Asn
500 505 510
Glu Val Lys Gln Ile Lys Glu Trp Val Glu Glu Tyr Tyr Ala
515 520 525
<210> 4
<211> 1581
<212> DNA
<213> 4
<400> 4
atgcgttcct tctccgctgt cgctgccgca gccctggcgc tctcttgggc gtctctggct 60
caggctgctc gccctcgtct tgtgcccaag cctgtccctc ggccagcttc gagtaaatcg 120
gctgcgacca caggcgaggc taactttgag cagttgctgg accatcataa tccagacaag 180
ggaacgtttt cccagcggta ctggtggagt actgaatact ggggtggtcc tgggtcaccg 240
gttgtcctct ttactcctgg agaggtctct gccgatggct atgaggggta tctcaccaat 300
gagactctca ctggtgttta tgcgcaggag atccagggtg ccgtcattat cattgagcac 360
cgctactggg gtgattcttc gccttatgag gtactcaatg ccgaaactct tcagtatctt 420
acattggacc aagccattct ggacctgacc tacttcgccg agacggtgaa actgcaattc 480
gataacagca cccgcagcaa tgcgcagaat gctccctggg tcatggtcgg tggatcatac 540
agcggtgcct tgacggcttg gaccgagtct gtcgcgcctg gaacgttctg ggcttaccat 600
gccactagtg ctcctgtgga ggctatctat gacttttggc aatacttcta ccccatccag 660
caaggtatgg cacagaactg cagcaaggac gtgtctctgg tagccgagta tgtcgacaag 720
gttggaaaga acggaactgc caaggagcag caggcactca aggaattgtt tggcttggga 780
gctgttgagc atttcgatga ctttgccgct gtcctcccca acggaccgta cctctggcag 840
gacaacgact ttgccacagg atactcttcc ttcttccagt tctgtgacgc cgtcgagggt 900
gtcgaagccg gcgcggcagt aacccccggc cccgagggtg tcggcctcga aaaggccctg 960
gccaactacg caaactggtt caattcaacc attctccctg attactgcgc aggctacggc 1020
tactggaccg acgaatggag cgtcgcctgc ttcgacagct acaacgcctc gagccctatc 1080
tacaccgata catccgtcgg caatcccgtc gaccgccaat gggaatggtt cctttgcaac 1140
gagcctttct tctactggca agacggtgct cccgagggta cctccaccat tgtgccccgg 1200
ctcgtcagcg cctcctactg gcaacgccaa tgctcgctct acttccccga aacgaacggc 1260
tacacgtacg gtagcgcgaa gggtaagaac tctgccacgg tgaacagctg gaccggtggg 1320
tgggacatga cccgcaacac gacgcggttg atctggacga atgggcaata cgacccctgg 1380
cgcgactccg gtgtgtcgag cactttccgg cctggtggac cgctggcgag cacggcgaat 1440
gaacccgtgc aggttattcc gggcggattc cattgctccg atttgtatat ggcagattat 1500
tatgcgaacg agggggtaaa aaaggtggta gataatgagg tgaagcagat taaggagtgg 1560
gtggaggagt attatgcttg a 1581
- 还没有人留言评论。精彩留言会获得点赞!