一种水稻雄性育性调控基因OsSTRL2及其应用的制作方法
本发明属于植物基因工程和植物育种领域,具体地说,涉及一种水稻雄性育性调控基因OsSTRL2及其应用。
背景技术:
水稻(Rice)作为禾本科植物的一种,是关于人类营养和热量摄入的最重要的作物,提供超过人类全世界消耗的卡路里的五分之一。水稻也是世界上大部分人口中最广泛消费的主食,特别是在亚洲。根据2012年粮农组织统计数据,水稻是世界上第三大产量的农产品,仅次于甘蔗和玉米。作物育种是指改良作物的遗传特性,以培育高产优质品种的技术,又称作物品种改良。它以遗传学为理论基础,并综合应用植物生态、植物生理、生物化学、植物病理和生物统计等多种学科知识,是一项投资少而效益高的生物技术,对发展种植业生产具有十分重要的意义。
杂交育种是培育水稻新品种的主要手段,也是提高水稻产量与品质的主要途径。因为水稻具有明显的杂种优势现象,主要表现在生长旺盛,根系发达,穗大粒多,抗逆性强等方面,因此,利用水稻的杂种优势以大幅度提高水稻产量一直是育种家梦寐以求的愿望。但是,水稻属自花授粉植物,雌雄蕊着生在同一朵颖花里,由于颖花很小,而且每朵花只结一粒种子,因此很难用人工去雄杂交的方法来生产大量的第一代杂交种子,所以长期以来水稻的杂种优势未能得到应用。
植物雄性不育被定义为花粉粒缺失或不起作用,还包括植物不能产生或释放功能性花粉粒。在杂交水稻种子生产中,雄性不育材料的使用具有非常重要的意义,因为它消除了机械去雄的过程。也就是说,借助这种雄性不育水稻母本作为遗传工具,通过人工辅助授粉的办法,就能大量生产杂交种子。但是长久以来,杂交水稻的发展,受到杂交品种资源的局限性的影响,正逐步到达瓶颈。因此,科学家一直在通过分离克隆新的不育基因,创制新的不育材料,来扩展杂交水稻育种的细胞及分子背景。
技术实现要素:
有鉴于此,本发明针对上述的问题,提供了一种水稻基因OsSTRL2的DNA序列、其编码蛋白的序列、启动子的序列及其应用。OsSTRL2基因具有调控水稻育性的功能,对该基因进行突变或抑制其表达导致其功能缺失或表达量下调,这会产生新的水稻雄性不育株系,在未来杂交水稻育种实践中的具有非常重要的应用价值。
本发明的提供的基因DNA序列或相关DNA序列编码的蛋白的用途,用于:
本发明提供的基因OsSTRL2,是一个水稻雄性不育相关基因,OsSTRL2的碱基序列如SEQ ID NO:1所示,氨基酸序列如SEQ ID NO:2所示,在水稻中该基因位于第3号染色体上。
本发明还包括下列a)-c)中任一所述的DNA序列:a)与上述OsSTRL2基因序列具有至少90%(优选为至少95%)序列相似性,且具有相同功能的DNA序列;b)在严格条件下能够与(a)所述序列的DNA杂交的DNA序列;c)与上述任一所述序列互补的DNA序列。
本领域技术人员应该知晓,本发明所述的育性恢复基因包括与OsSTRL2基因高度同源,并且具有同样的育性调控功能的高度同源的功能等价体序列。所述高度同源的功能等价体序列包括在严谨条件下能够与本发明所公开的OsSTRL2基因的核苷酸序列杂交的DNA序列。本发明中所使用的“严谨条件”是公知的,包括诸如在含400mM NaCl、40mM PIPES(pH6.4)和1mM EDTA的杂交液中于60℃杂交12-16小时,然后在65℃下用含0.1SDS、和0.1%SSC的洗涤液洗涤15-60分钟。
功能等价体序列还包括与本发明所公开的OsSTRL2基因所示的序列有至少90%、95%、96%、97%、98%、或99%序列相似性,且具有育性调控功能的DNA序列,可以从任何植物中分离获得。其中,序列相似性的百分比可以通过公知的生物信息学算法来获得,包括Myers和Miller算法(Bioinformatics,4(1):11-17,1988)、Needleman-Wunsch全局比对法(J.Mol.Biol.,48(3):443-53,1970)、Smith-Waterman局部比对法(J.Mol.Biol.,147:195-197,1981)、Pearson和Lipman相似性搜索法(PNAS,85(8):2444-2448,1988)、Karlin和Altschul的算法(Altschul等,J.Mol.Biol.,215(3):403-410,1990;PNAS,90:5873-5877,1993)。这对于本领域技术人员来说是熟悉的。
本发明所述的基因序列可以从任何植物中分离获得,包括但不限于芸苔属、玉米、小麦、高粱、两节荠属、白芥、蓖麻子、芝麻、棉籽、亚麻子、大豆、拟南芥属、菜豆属、花生、苜蓿、燕麦、油菜籽、大麦、燕麦、黑麦(Rye)、粟、蜀黍、小黑麦、单粒小麦、斯佩尔特小麦(Spelt)、双粒小麦、亚麻、格兰马草(Gramma grass)、摩擦禾、假蜀黍、羊茅、多年生麦草、甘蔗、红莓苔子、番木瓜、香蕉、红花、油棕、香瓜、苹果、黄瓜、石斛、剑兰、菊花、百合科、棉花、桉、向日葵、芸苔、甜菜、咖啡、观赏植物和松类等。优选地,植物包括玉米、大豆、红花、芥菜、小麦、大麦、黑麦、稻、棉花和高粱。
本发明所述的应用是:本发明提供了通过影响OsSTRL2的核苷酸序列或者通过调控OsSTRL2基因的转录表达从而影响植株育性的方法,从而获得新的水稻雄性不育系可以用来生产杂交种子。所述影响植株育性是指通过调控OsSTRL2基因的表达,从而使所述植株的育性发生改变,如导致植株雄性不育。具体地,取决于具体应用需求,可以通过多种方法来影响OsSTRL2基因在植物体内的表达,从而达到调控植株雄性育性的效果。更具体地,调控OsSTRL2基因的表达可以使用许多本领域普通技术人员可获得的工具进行,例如,通过突变、诱变、反义基因的转入、共抑制或发夹结构的引入等,都可以用于破坏OsSTRL2基因的正常表达,从而获得雄性不育的植株。另一方面,本发明还涉及一种恢复水稻雄性不育株系的雄性不育性状的方法,包括如下步骤:采用常规遗传手段将所述OsSTRL2基因,转入如前述所述应用获得的水稻雄性不育株系,进而使得突变体恢复野生型表型。
本发明还提供了一种OsSTRL2基因的不育突变体序列及其雄性不育突变体材料。更具体地,所述雄性不育突变体材料是通过突变水稻内源的OsSTRL2基因,或突变与其高度同源的基因的核苷酸序列,使该植物体丧失雄性育性的过程。所述“突变”包括但不限于以下方法,如用物理或化学的方法导致的基因突变,化学方法包括用EMS等诱变剂处理所导致的诱变,所述突变还可以是点突变,也可以是DNA缺失或插入突变,还可以是通过RNAi、定点突变等基因沉默手段产生。
具体地,本发明还提供了一种水稻雄性不育突变体,其含有突变后的雄性不育基因,所述突变后的雄性不育基因的核苷酸序列如SEQ ID NO:15所示,氨基酸序列如SEQ ID NO:16所示。与野生型相比,在不育突变体中,该基因第一个外显子的序列由如SEQ ID NO:17所示突变为如SEQ ID NO:18所示,导致该基因的提前终止。
本发明还提供了一种OsSTRL2基因的启动子,所述启动子具有在花粉中特异表达的功能,相应启动子的序列为OsSTRL2基因从ATG到上游2000bp左右的核苷酸序列。更具体地,在水稻中,所述OsSTRL2基因启动子的核苷酸序列如SEQ ID NO:6所示。将SEQ ID NO:6与报告基因GUS相连,转化植物,检测分析转基因植株中的GUS表达活性和表达模式,通过在转基因植株的根、茎、叶和花中都进行GUS染色分析,结果发现OsSTRL2启动子驱动GUS基因主要在水稻花药中表达,更具体地在花药发育的P9期特异性的高表达。说明本发明所提供的SEQ ID NO:6启动子是一个花药特异性表达的启动子。
本发明所提供的植物花粉特异表达启动子,含有序列表中SEQ ID NO:6所示的核苷酸序列,或包含与SEQ ID NO:6中所列核苷酸序列具有90%以上相似性的核苷酸序列,或包含来源于SEQ ID NO:6序列上的100个及100以上连续的核苷酸片段,并且可以驱动与该启动子操作性连接的核苷酸序列在植物花粉中的表达。含有上述序列的表达载体、转基因细胞系以及宿主菌等均属于本发明的保护范围。扩增本发明所公开的SEQ ID NO:6启动子的任一核苷酸片段的引物对也在本发明的保护范围之内。
本发明所提供的启动子核苷酸序列还可用于从水稻以外的其它植物中分离相应序列,尤其是从其他单子叶植物中进行同源克隆。根据这些相应序列与本文所列启动子序列间的序列同源性,或与本启动子基因的同源性,使用如PCR、杂交等技术来鉴别分离这些相应序列。因此,根据它们与本发明所列的SEQ ID NO:6启动子序列(或其片段)间的序列相似性而分离的相应片段,也包括在实施方案中。
本发明所述的“启动子”是指一种DNA调控区域,其通常包含能指导RNA聚合酶II在特定编码序列的合适转录起始位点起始RNA合成的TATA盒。启动子还可包含其它识别序列,这些识别序列通常位于TATA盒的上游或5’端,通常被称为上游启动子元件,起调控转录效率的作用。本领域技术人员应该知晓,虽然已经鉴定了针对本发明公开的启动子区域的核苷酸序列,但是分离和鉴定处于本发明鉴定的特定启动子区域的TATA盒上游区域的其它调控元件也在本发明的范围内。因此,本文公开的启动子区域通常被进一步界定为包含上游调控元件,例如用于调控编码序列的组织表达性和时间表达功能的那些元件、增强子等。以相同的方式,可以鉴定、分离出使得能在目标组织(例如雄性组织)中进行表达的启动子元件,将其与其它核心启动子一起使用,以验证雄性组织优先的表达。核心启动子指起始转录所需的最小限度的序列,例如被称为TATA盒的序列,这是编码蛋白质的基因的启动子通常都具有的。因此,可选地,OsSTRL2基因的上游启动子可与其自身的或来自其它来源的核心启动子关联使用。
核心启动子可以是任何一种已知的核心启动子,例如花椰菜花叶病毒35S或19S启动子(美国专利No.5,352,605)、泛素启动子(美国专利No.5,510,474)、IN2核心启动子(美国专利No.5,364,780)或玄参花叶病毒启动子。
所述基因启动子的功能可以通过以下方法进行分析:将启动子序列与报告基因可操作性连接,形成可转化的构建体,再将该构建体转入植株中,在获得转基因后代中,通过观察报告基因在植物各个组织器官中的表达情况来确认其表达特性;或者将上述构建体亚克隆进用于瞬时表达实验的表达载体,通过瞬时表达实验来检测启动子或其调控区的功能用来测试启动子或调控区域功能的适当表达载体的选择将取决于宿主和将该表达载体引入宿主的方法,这类方法是本领域普通技术人员所熟知的。对于真核生物,在载体中的区域包括控制转录起始和控制加工的区域。这些区域被可操作地连接到报告基因,所述报告基因包括YFP、UidA、GUS基因或荧光素酶。包含位于基因组片段中的推定调控区的表达载体可以被引入完整的组织,例如阶段性花粉,或引入愈伤组织,以进行功能验证。
此外,本发明的启动子可与并非OsSTRL2基因的核苷酸序列相连,以表达其它异源核苷酸序列。本发明的启动子核苷酸序列及其片段和变体可与异源核苷酸序列一起组装在一个表达盒中,用于在目的植株中表达,更具体地,在该植株的雄性器官中表达。所述表达盒有合适的限制性酶切位点,用于插入所述启动子和异源核苷酸序列。这些表达盒可用于对任何植株进行遗传操作,以获得想要的相应表型。
本发明所公开的OsSTRL2启动子,更具体地为水稻中的OsSTRL2启动子,可用于驱动下列异源核苷酸序列的表达,以使转化的植株获得雄性不育的表型。所述异源核苷酸序列可编码促使碳水化合物降解的酶或修饰酶、淀粉酶、脱支酶和果胶酶,更具体的如α淀粉酶基因、生长素(auxin),rot B、细胞毒素基因、白喉毒素、DAM甲基化酶、亲和素,或者可选自原核调控系统,还可以是显性的雄性不育基因。
在某些实施方式中,本发明中所提到的可操作地连接在本发明启动子下游的核酸,其中所述的“核酸”可以是操作性连接于本文所公开的启动子之上的结构基因、调节基因、结构基因的反义基因、调节基因的反义基因或者能够干扰内源基因表达的小RNA。
更具体地,可以将本发明所提供的育性调控基因SEQ ID NO:1构建到启动子SEQ ID NO:6的下游,从而驱动该育性调控基因在花粉中的特异表达,或是通过RNAi的技术原理,构建由SEQ ID NO:6启动的可以沉默SEQ ID NO:1基因的RNAi载体,从而获得SEQ ID NO:1基因的雄性不育突变体。
本发明所提供的启动子序列可分离自任何植物,包括但不限于芸苔属、玉米、小麦、高粱、两节荠属、白芥、蓖麻子、芝麻、棉籽、亚麻子、大豆、拟南芥属、菜豆属、花生、苜蓿、燕麦、油菜籽、大麦、燕麦、黑麦(Rye)、粟、蜀黍、小黑麦、单粒小麦、斯佩尔特小麦(Spelt)、双粒小麦、亚麻、格兰马草(Gramma grass)、摩擦禾、假蜀黍、羊茅、多年生麦草、甘蔗、红莓苔子、番木瓜、香蕉、红花、油棕、香瓜、苹果、黄瓜、石斛、剑兰、菊花、百合科、棉花、桉、向日葵、芸苔、甜菜、咖啡、观赏植物和松类等。优选地,植物包括玉米、大豆、红花、芥菜、小麦、大麦、黑麦、稻、棉花和高粱。
组织特异表达启动子可用于靶向特定的植物组织中增强转录和/或表达。启动子可在目标组织中表达也在其它植物组织中表达,可在目标组织中强烈表达以及比其它组织程度低得多的表达,或者可高度优选在目标组织中表达。在一种实施方式中,启动子是偏好在植物的雄性或雌性组织中特异表达的类型。本发明不必须在方法中使用任何特定的雄性组织优先型启动子,本领域技术人员已知的很多此类启动子中的任何都可以使用。本文描述的天然的OsSTRL2启动子是可使用的启动子的一个例子。另一种此类启动子是5126启动子、MS45启动子、MS26启动子、BS92-7启动子、SGB6调控元件和TA29启动子等,其偏好于指导其连接的基因在植物雄性组织中的表达。某些构建体中还可以包括配子组织优先表达启动子。雄性配子优先表达启动子包括PG47启动子以及ZM13启动子。
上述构建体中还可包括其它组分,这主要取决于载体构建的目的和用途,例如可进一步包括选择标记基因、靶向或调控序列、稳定序列或引导序列、内含子等。表达盒还将在目标异源核苷酸序列的3’端包括在植物中具有功能的转录和翻译终止子。终止子可以是本发明所提供基因的终止子,也可以是来自外源的终止子。更具体地,上述终止子可以是胭脂氨酸合酶或章鱼碱合酶终止区域。
在希望将异源核苷酸序列的表达产物引向特定细胞器,例如质体、造粉体,或者引向内质网,或在细胞表面或细胞外分泌的情况下,表达盒还可包含用于编码转运肽的核苷酸序列。此类转运肽是本领域所公知的,其包括但不限于Rubisco的小亚基、植物EPSP合酶、玉米Brittle-1叶绿体转运肽等。
在制备表达盒的过程中,可对多种DNA片段加以操作,以提供处于合适方向,或是处于正确读码框中的DNA序列。为达到此目的,可使用衔接子或接头,将DNA片段连起来,或者进一步包括其它操作,以提供方便的限制性酶切位点等。
进一步地,本发明所提供的构建体中还可包括选择标记基因,用于选择经转化的细胞或组织。所述选择标记基因包括赋予抗生素抗性或对除草剂抗性的基因。合适的选择标记基因包括但不限于:氯霉素抗性基因,潮霉素抗性基因,链霉素抗性基因,奇霉素抗性基因,磺胺类抗性基因,草甘磷抗性基因,草丁嶙抗性基因。所述选择标记基因还可以是红色荧光基因、青色荧光蛋白基因、黄色荧光蛋白基因、荧光素酶基因、绿色荧光蛋白基因、花青甙p1等基因。
本发明所提供的表达盒或载体可被插入质粒、粘粒、酵母人工染色体、细菌人工染色体或其他适合转化进宿主细胞中的任何载体中。优选的宿主细胞是细菌细胞,尤其是用于克隆或储存多核苷酸、或用于转化植物细胞的细菌细胞,例如大肠杆菌、根瘤土壤杆菌和毛根土壤杆菌。当宿主细胞是植物细胞时,表达盒或载体可被插入被转化的植物细胞的基因组中。插入可以是定位的或随机的插入。优选地,插入通过诸如同源重组来实现。另外,表达盒或载体可保持在染色体外。本发明的表达盒或载体可存在于植物细胞的核、叶绿体、线粒体和/或质体中。优选地,本发明的表达盒或载体被插入植物细胞核的染色体DNA中。
本发明还涉及一种水稻不育株系在水稻制种中的用途,以如前述所述应用获得的水稻雄性不育株系作为母本,进行杂交育种。具体地,在某些应用的实施方式中,可以应用本发明所提供的OsSTRL2基因或其启动子来实现OsSTRL2或其他类似育性相关基因突变所获得的雄性不育系的繁殖和保持。
更具体地,上述雄性不育系的繁殖和保持,是指以纯合隐性核雄性不育突变体为转化受体材料,将紧密连锁的3个目标基因转化至该不育突变体受体植株中。所述3个目标基因分别是育性恢复基因、花粉失活基因和颜色标记筛选基因。其中,育性恢复基因可使不育的转化受体育性恢复,花粉失活基因可使含有转化的外源基因的花粉失活,即失去授精能力,筛选基因可以用于转基因种子和非转基因种子的分拣,分拣出的非转基因种子用作不育系生产杂交种,转基因种子用作保持系来源源不断地、稳定地生产不育系。
本发明的所提供的花粉特异表达启动子可用于外源基因在花粉中的特异性表达,从而避免该外源基因在植物其他组织中持续表达所带来的不利影响,还可以用于植物花粉生长发育相关基因的功能分析和鉴定;可用于雄性不育系和恢复系的创建;并可应用于花粉败育实验中,从而避免由植物转基因漂移或花粉逃逸所带来的生物安全问题,对植物雄性不育系和恢复系的创造具有重要意义。
本发明的转基因植物使用植物生物技术领域技术人员已知的转化方法制备。任何方法可被用于将重组表达载体转化进植物细胞中,以产生本发明的转基因植物。转化方法可包括直接和间接的转化方法。合适的直接方法包括聚乙二醇诱导的DNA摄入、脂质体介导的转化、使用基因枪导入、电穿孔、以及显微注射,等。在本发明的具体实施方式中,本发明使用了基于土壤杆菌的转化技术(可参见Horsch RB等(1985)Science 225:1229;White FF,Vectors for Gene Transfer in Higher Plants,Transgenic Plants,第1卷,Engineering and Utilization,Academic Press,1993,pp.15-38;Jenes B等.Techniques for Gene Transfer,Transgenic Plants,第1卷,Engineering and Utilization,Academic Press,1993,pp.128-143,等)。土壤杆菌菌株(例如根瘤土壤杆菌或毛根土壤杆菌)包含质粒(Ti或Ri质粒)和T-DNA元件,所述质粒和元件在用土壤杆菌转染后被转移至植物,而T-DNA被整合进植物细胞的基因组中。T-DNA可位于Ri-质粒或Ti-质粒上,或独立地包含在所谓的双元载体中。土壤杆菌介导的转化方法描述于例如中。土壤杆菌介导的转化最适合双子叶植物,但是也适合单子叶植物。土壤杆菌对植物的转化描述于例如中。转化可导致瞬时或稳定的转化和表达。尽管本发明的核苷酸序列可被插入落入这些广泛种类中的任何植物和植物细胞中,但是其尤其适用于作物植物细胞。
与现有技术相比,本发明可以获得包括以下技术效果:
1)本发明通过控制水稻雄性育性OsSTRL2基因及其编码蛋白获得水稻雄性生殖发育的变异株,实现控制水稻生殖过程。
2)本发明获得的水稻突变体在营养期与来源亲本无明显差异,进入生殖生长阶段后雄性生殖发育异常,花粉败育,得到完全雄性不育的不育系,该不育系的育性稳定、不受环境条件影响、能够被野生型转基因恢复。
3)该基因以及该基因突变产生的不育系为构建第三代杂交育种体系提供了必要的元件,该基因突变产生的雄性不育系,用来生产杂交种子,对于突破并改良现有的“三系”和“两系”杂交技术有重要意义,并在杂交水稻构建和农业生产上具有十分重要的应用价值。
当然,实施本发明的任一产品并不一定需要同时达到以上所述的所有技术效果。
附图说明
此处所说明的附图用来提供对本发明的进一步理解,构成本发明的一部分,本发明的示意性实施例及其说明用于解释本发明,并不构成对本发明的不当限定。在附图中:
图1是本发明OsSTRL2-YFP在烟草叶表皮细胞中的亚细胞定位,显示了感染72小时后烟草叶表皮细胞的共焦图像。其中(A)2x35S::YFP的YFP信号作为对照;(B)2x35S::OsSTRL2-YFP的YFP信号;(C)(B)和明场的合并图像;(D和E)共表达的2x35S::OsSTRL2-YFP和ER-标记的YFP和RFP信号;(F)(D和E)和明场的合并信号。标尺=10μm(A,D,E和F);25μm(B和C);
图2是本发明转OsSTRL2pro::GUS阳性植株各组织GUS染色分析;其中(A、B、C、D、E、F和H)分别为根,茎,叶,小花,小花(去除了外敷),小穗(去除了外敷和内敷))和GUS染色花药部分的横切面;标尺=3mm(A-F);30μm(H);
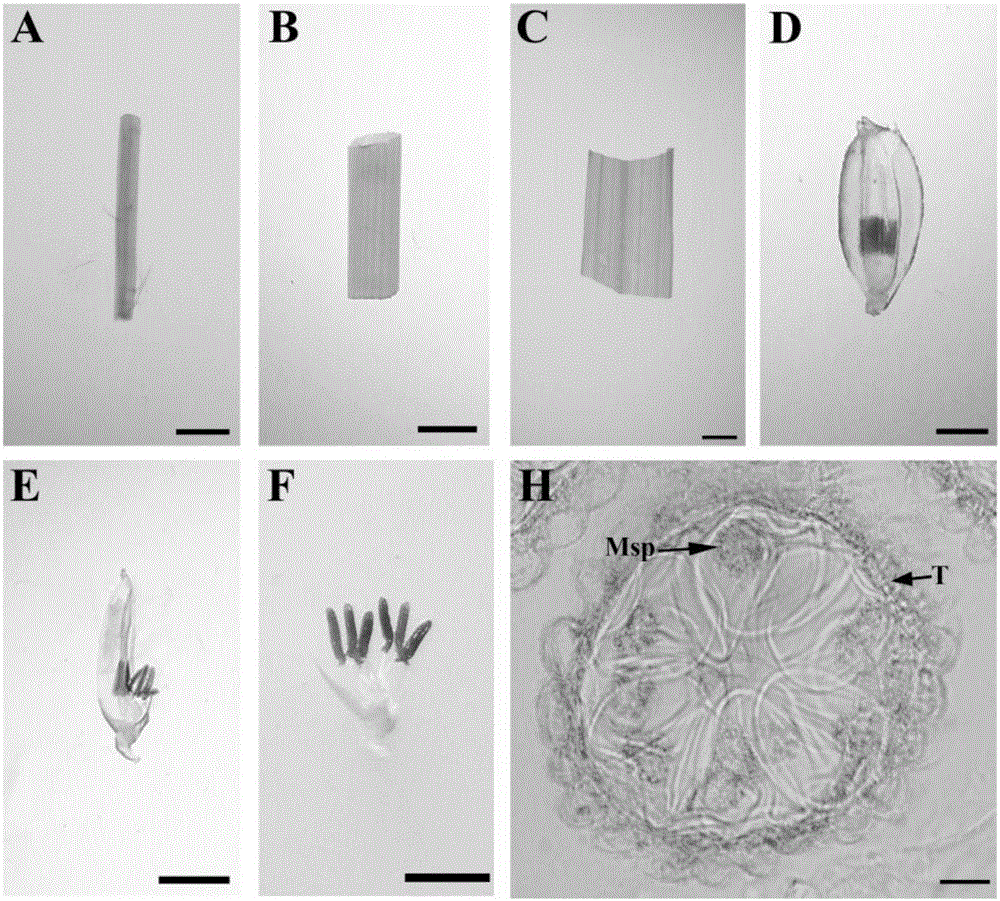
图3是本发明CRISPR/Cas9介导的雄性不育突变体osstrl2的表型观察。其中,(A)野生型(左)和osstrl2(右)在成熟期的表型比较;(B)野生型(左)和osstrl2(右)在抽穗阶段的小花的比较;(C)在抽穗阶段野生型(左)和osstrl2(右)(去除内敷和外敷)小花的比较;(D和E)分别为野生型和osstrl2的I2/KI花粉染色;标尺=0.20mm(B和C);30μm(D和E);
图4是本发明育性得到恢复的osstrl2(互补osstrl2突变体)表型示意图;其中,(A)为野生型小花内部结构图;(B)为野生型成熟花粉I2/KI染色结果;(C)为互补osstrl2突变体后代的小花内部结构图;(D)为互补osstrl2突变体后代的成熟花粉I2/KI染色结果。
具体实施方式
以下将配合实施例来详细说明本发明的实施方式,藉此对本发明如何应用技术手段来解决技术问题并达成技术功效的实现过程能充分理解并据以实施。
实施例1、水稻育性控制蛋白编码基因OsSTRL2的克隆和功能分析
1、OsSTRL2基因全长cDNA克隆、表达载体构建及亚细胞定位
发明人利用本领域技术人员所熟知的生物信息学技术,从水稻基因组中确定一个含完整水稻雄性生殖发育控制蛋白OsSTRL2。经软件分析和cDNA克隆,其CDS为1284bp,如SEQ ID NO:1所示,编码全长427个氨基酸的水稻雄性生殖发育控制蛋白,其序列如SEQ ID NO:2所示。经全核苷酸序列分析结果表明:OsSTRL2基因全长2083bp(SEQ ID NO:3,包含调控区和内含子)。
以正常野生型水稻中花9号品种的RNA为模板,合成第一链cDNA,将OsSTRL2的全长cDNA经引物对OsSTRL2-YFP-F(SEQ ID NO:4)和OsSTRL2-YFP-R(SEQ ID NO:5)扩增后克隆到pA7-YFP载体中以产生2x35S::OsSTRL2-YFP表达盒,即将OsSTRL2的C末端与YFP融合并在花叶病毒双35S启动子的控制下;然后将整个2x35S::OsSTRL2-YFP表达盒导入pCAMBIA1300。同样,将内质网滞留信号KDEL融合与RFP的C端,并置于花叶病毒双35S启动子的控制下作为内质网(ER)定位标记。这些质粒通过农杆菌介导的浸润而在烟草叶表皮细胞中单独表达或共表达。YFP和RFP信号在渗透72小时后用共聚焦扫描显微镜(Nikon A1,Kanagawa,Japan)显像。亚细胞定位结果(见图1)显示OsSRTL2蛋白在细胞中主要定位于内质网上。
2、OsSTRL2基因的启动子克隆及其表达模式分析
以正常野生型水稻中花9号品种的DNA为模板,以所述OsSTRL2基因起始密码子前面约1.7K片段(SEQ ID NO:6)经引物对OsSTRL2-promoter-F(SEQ ID NO:7)和OsSTRL2-promoter-R(SEQ ID NO:8)扩增克隆到载体pCMBIYA1300上产生OsSTRL2pro::GUS表达盒,其中GUS基因由天然的OsSTRL2启动子驱动。将含有OsSTRL2pro::GUS的构建体导入根癌农杆菌菌株EHA105中并通过转基因转移到野生型水稻中花9号中。具体地,将载体通过冻融法导入农杆菌EHA105(四川农业大学水稻所生物技术实验室保存)。农杆菌EHA105平板(4℃保存)挑取单菌落于YEP液体培养基(Km和Rif各50mg/L)中,28℃振荡培养12~18h,然后取1~5mL菌液接到100mL YEP液体培养基(含乙酰丁香酮100μmol/L)中,振荡培养4h后测OD值稀至相应浓度(OD=0.5)。将新鲜菌液于8000rpm、4℃、5min收集菌体,并重悬于三分之一提及的AAM培养基中。加入放有生长旺盛的胚性愈伤组织的灭菌三角瓶中浸泡25min,再吹干表面菌液,将愈伤组织转接于共培养基上,28℃暗培养2~3d。共培养愈伤组织经无菌水和含Cef 500mg/L的无菌水漂洗后,在工作台上吹4h左右,转接于预培养基中28℃暗培养5~7d。将预培养的愈伤组织转接于含Hyg和Cef的筛选培养基上继续培养3~4周,再将抗性愈伤组织转接于仅含Hyg的筛选培养基上,筛选1~2次。取抗性愈伤组织转接于分化培养基上,28℃光照分化。分化小苗转接于生根培养基,28℃光照培养3~4周,开放培养炼苗7d左右,最后移栽到大田。获得的再生植株移栽成活后用潮霉素筛选转化植株。对阳性转基因植株根、茎、叶和花进行GUS染色(B-半乳糖苷酶的活性分析)的结果(图2)显示OsSTRL2启动子驱动GUS主要在水稻小花花药的绒毡层和小孢子中表达,这表明OsSTRL2启动子是一个花药特异型的启动子。
实施例2、水稻雄性不育系的创制
通过基因工程或其他手段创制osstrl2水稻雄性不育株系
本实施例中OsSTRL2基因的编码区序列如SEQ ID NO:2所示。本实施例的osstrl2突变材料是由常规粳稻品种中花9号,经过对OsSTRL2基因的序列变异获得。
具体地,利用CRISPR/Cas9技术首先设计OsSTRL2基因的靶位点(SEQ ID NO:9),然后构建VK005-01-OsSTRL2敲除载体,对重组载体进行鉴定,其中靶位点的表达由OsU3启动子驱动。通过实施例1中的水稻转基因方法将敲除载体导入野生型水稻中花9号基因组中。获得的再生植株移栽成活后用潮霉素筛选转化植株;阳性植株提取叶片总DNA,对中花9号基因组中对应的OsSTRL2基因的靶位点经引物对OsSTRL2-gRNA-seq-F(SEQ ID NO:10)和OsSTRL2-gRNA-seq-R(SEQ ID NO:11)扩增后进行测序验证,同时对转基因植株调查育性表型,从而验证OsSTRL2基因功能。结果如图3和表1,OsSTRL2基因被成功敲除的植株表现为花药白色短小,花粉急剧减少并无活力,进而得到水稻雄性不育株系osstrl2。从上述分析可以看出,OsSTRL2是水稻维持正常雄性育性所必须的基因,缺失该基因会导致植株雄性不育,下调该基因表达则降低植株的雄性育性。
表1 OsSTRL2转基因敲除T0代植株的表型和基因型连锁分析结果:
表中数字表示不同基因型植株所展现的不同的育性表型
实施例3、水稻雄性不育株系osstrl2育性的恢复即转基因功能互补
以正常野生型水稻中花9号品种的DNA为模板,经引物对OsSTRL2-COM-F(SEQ ID NO:12)和OsSTRL2-COM-R(SEQ ID NO:13)扩增后得到片段SEQ ID NO:14,将该片段克隆到载体pCMBIYA1300上,成功构建P1300-OsSTRL2-COM。同样地,通过实施例1中的水稻转基因方法将载体P1300-OsSTRL2-COM导入由水稻雄性不育株系osstrl2的BC1F2后代中不具有潮霉素抗性但为雄性不育表型的幼穗培养的愈伤组织中。获得的再生植株移栽成活后用潮霉素筛选转化植株。对阳性植株的转基因T0代植株观察水稻育性,调查结果(图4)显示转OsSTRL2互补表达基因的植株的育性得到了恢复,变成正常可育,表明OsSTRL2基因的表达是水稻维持正常雄性育性所必须的。
综上所述,本发明通过控制水稻雄性生殖发育相关基因OsSTRL2及其编码蛋白获得水稻雄性生殖发育异常的变异株,实现控制水稻雄性生殖发育和育性;本发明获得的水稻突变体在营养生长时期与来源亲本无明显差异,进入生殖生长阶段后,雄性生殖器官发育异常、花粉败育引起植株不育,在农业生产上具有十分重要的应用。
上述说明示出并描述了发明的若干优选实施例,但如前所述,应当理解发明并非局限于本文所披露的形式,不应看作是对其他实施例的排除,而可用于各种其他组合、修改和环境,并能够在本文所述发明构想范围内,通过上述教导或相关领域的技术或知识进行改动。而本领域人员所进行的改动和变化不脱离发明的精神和范围,则都应在发明所附权利要求的保护范围内。
SEQUENCE LISTING
<110> 四川农业大学
<120> 一种水稻雄性育性调控基因OsSTRL2及其应用
<130> 2017
<160> 18
<170> PatentIn version 3.3
<210> 1
<211> 1293
<212> DNA
<213> OsSTRL2基因
<400> 1
atggaagaga agaagcagca gcagcagcgt ccacagagag ggcgcgatgg catcctgcag 60
tatccgcacc ttttcttcgc ggcgctggcg ctggccctgc tcctcaccga cccgttccac 120
ctcggcccgc tcgccggggt ggactaccgg ccggtgaggc acgagctggc gccgtaccgc 180
gaggtgatgg cgcggtggcc gcgggacaac ggcagccggc tcaggcacgg caggctggag 240
ttcgtcggac aggtgttcgg gccggagtcc atcgagttcg accgccacgg ccgcggcccc 300
tacgccggcc tcgccgacgg ccgcgtcgtg cggtggatgg gggaggacgc cgggtgggag 360
acgttcgccg tcatgagccc tgactggtcg gagaaagttt gtgccaatgg ggtggagtcg 420
acgacgaaga agcagcacga gatggagcga cggtgcggcc ggcctctcgg gctgaggttt 480
cacggcgaga ccggcgagct ctacgtcgcc gacgcatact acgggctcat gtccgtcggt 540
ccgaacggcg gggtggcgac ctctctcgcg agagaagtcg gcgggagccc ggtcaacttc 600
gcgaacgacc tcgacatcca ccgcaacggc tccgtgttct tcaccgacac gagcacgaga 660
tacaacagaa aggatcatct gaacgttctg ctagaaggtg aaggcacagg gaggctgctc 720
agatatgacc cagaaaccaa agctgcccat gtcgtgctga gcgggctggt cttcccgaat 780
ggcgtgcaga tttctgacga ccagcagttc ctcctcttct ccgaaacaac aaactgcagg 840
ataatgcggt actggctgga agggccaaga gccgggcagg tggaggtgtt cgccgacctg 900
ccggggttcc cggacaacgt gcgactgagc agcggcggcg gcggcggacg gttctgggtg 960
gcgatcgact gctgcaggac ggcggcgcag gaggtgttcg ccaagcggcc gtggctgcga 1020
acgctctact tcaagctgcc cctgacgatg cggacgctgg ggaagatggt cagcatgcgg 1080
atgcacaccc tcgtcgcgct cctcgacggc gaaggggacg tcgtcgaggt gctcgaggac 1140
cggggcggcg aggtgatgcg gctggtgagc gaggtgaggg aggtggggcg caagctgtgg 1200
atcggcaccg tggctcataa ccacatcgcc acgatccctt acccgttgga agagcagagt 1260
agcagcagca gcagcaacgt gcttggtgat tga 1293
<210> 2
<211> 430
<212> PRT
<213> OsSTRL2基因的编码蛋白
<400> 2
Met Glu Glu Lys Lys Gln Gln Gln Gln Arg Pro Gln Arg Gly Arg Asp
1 5 10 15
Gly Ile Leu Gln Tyr Pro His Leu Phe Phe Ala Ala Leu Ala Leu Ala
20 25 30
Leu Leu Leu Thr Asp Pro Phe His Leu Gly Pro Leu Ala Gly Val Asp
35 40 45
Tyr Arg Pro Val Arg His Glu Leu Ala Pro Tyr Arg Glu Val Met Ala
50 55 60
Arg Trp Pro Arg Asp Asn Gly Ser Arg Leu Arg His Gly Arg Leu Glu
65 70 75 80
Phe Val Gly Gln Val Phe Gly Pro Glu Ser Ile Glu Phe Asp Arg His
85 90 95
Gly Arg Gly Pro Tyr Ala Gly Leu Ala Asp Gly Arg Val Val Arg Trp
100 105 110
Met Gly Glu Asp Ala Gly Trp Glu Thr Phe Ala Val Met Ser Pro Asp
115 120 125
Trp Ser Glu Lys Val Cys Ala Asn Gly Val Glu Ser Thr Thr Lys Lys
130 135 140
Gln His Glu Met Glu Arg Arg Cys Gly Arg Pro Leu Gly Leu Arg Phe
145 150 155 160
His Gly Glu Thr Gly Glu Leu Tyr Val Ala Asp Ala Tyr Tyr Gly Leu
165 170 175
Met Ser Val Gly Pro Asn Gly Gly Val Ala Thr Ser Leu Ala Arg Glu
180 185 190
Val Gly Gly Ser Pro Val Asn Phe Ala Asn Asp Leu Asp Ile His Arg
195 200 205
Asn Gly Ser Val Phe Phe Thr Asp Thr Ser Thr Arg Tyr Asn Arg Lys
210 215 220
Asp His Leu Asn Val Leu Leu Glu Gly Glu Gly Thr Gly Arg Leu Leu
225 230 235 240
Arg Tyr Asp Pro Glu Thr Lys Ala Ala His Val Val Leu Ser Gly Leu
245 250 255
Val Phe Pro Asn Gly Val Gln Ile Ser Asp Asp Gln Gln Phe Leu Leu
260 265 270
Phe Ser Glu Thr Thr Asn Cys Arg Ile Met Arg Tyr Trp Leu Glu Gly
275 280 285
Pro Arg Ala Gly Gln Val Glu Val Phe Ala Asp Leu Pro Gly Phe Pro
290 295 300
Asp Asn Val Arg Leu Ser Ser Gly Gly Gly Gly Gly Arg Phe Trp Val
305 310 315 320
Ala Ile Asp Cys Cys Arg Thr Ala Ala Gln Glu Val Phe Ala Lys Arg
325 330 335
Pro Trp Leu Arg Thr Leu Tyr Phe Lys Leu Pro Leu Thr Met Arg Thr
340 345 350
Leu Gly Lys Met Val Ser Met Arg Met His Thr Leu Val Ala Leu Leu
355 360 365
Asp Gly Glu Gly Asp Val Val Glu Val Leu Glu Asp Arg Gly Gly Glu
370 375 380
Val Met Arg Leu Val Ser Glu Val Arg Glu Val Gly Arg Lys Leu Trp
385 390 395 400
Ile Gly Thr Val Ala His Asn His Ile Ala Thr Ile Pro Tyr Pro Leu
405 410 415
Glu Glu Gln Ser Ser Ser Ser Ser Ser Asn Val Leu Gly Asp
420 425 430
<210> 3
<211> 2091
<212> DNA
<213> OsSTRL2基因
<400> 3
aagttaagat caccgtctcc accaattccg tctttcctct gcgtgcaaat tccgtcttcc 60
ctcgctcctg atctccatgg aagagaagaa gcagcagcag cagcgtccac agagagggcg 120
cgatggcatc ctgcagtatc cgcacctttt cttcgcggcg ctggcgctgg ccctgctcct 180
caccgacccg ttccacctcg gcccgctcgc cggggtggac taccggccgg tgaggcacga 240
gctggcgccg taccgcgagg tgatggcgcg gtggccgcgg gacaacggca gccggctcag 300
gcacggcagg ctggagttcg tcggacaggt gttcgggccg gagtccatcg agttcgaccg 360
ccacggccgc ggcccctacg ccggcctcgc cgacggccgc gtcgtgcggt ggatggggga 420
ggacgccggg tgggagacgt tcgccgtcat gagccctgac tggtaacgaa cacctcgcct 480
gcattttgct ctcgccctcc acgaaaacac ctctcgtagc agtgtacaat tacgtgttct 540
tatattgcaa aaaaaggtcg gagaaagttt gtgccaatgg ggtggagtcg acgacgaaga 600
agcagcacga gatggagcga cggtgcggcc ggcctctcgg gctgaggttt cacggcgaga 660
ccggcgagct ctacgtcgcc gacgcatact acgggctcat gtccgtcggt ccgaacggcg 720
gggtggcgac ctctctcgcg agagaagtcg gcgggagccc ggtcaacttc gcgaacgacc 780
tcgacatcca ccgcaacggc tccgtgttct tcaccgacac gagcacgaga tacaacagaa 840
agtgtgcagc tgcagtatca ctctcttcag ttgtatcgat tctctatttc cttctatcgt 900
tcaagatttt ctgattagaa tcagttgtgc agggatcatc tgaacgttct gctagaaggt 960
gaaggcacag ggaggctgct cagatatgac ccagaaacca aagctgccca tgtcgtgctg 1020
agcgggctgg tcttcccgaa tggcgtgcag atttctgacg accagcagtt cctcctcttc 1080
tccgaaacaa caaactgcag gtgaaatggc acaagctttc acaggttctg aaaatactaa 1140
aggttaaaca agattcagaa ttgattaaca ttgcacgcat atgctgttct aggataatgc 1200
ggtactggct ggaagggcca agagccgggc aggtggaggt gttcgccgac ctgccggggt 1260
tcccggacaa cgtgcgactg agcagcggcg gcggcggcgg acggttctgg gtggcgatcg 1320
actgctgcag gacggcggcg caggaggtgt tcgccaagcg gccgtggctg cgaacgctct 1380
acttcaagct gcccctgacg atgcggacgc tggggaagat ggtcagcatg cggatgcaca 1440
ccctcgtcgc gctcctcgac ggcgaagggg acgtcgtcga ggtgctcgag gaccggggcg 1500
gcgaggtgat gcggctggtg agcgaggtga gggaggtggg gcgcaagctg tggatcggca 1560
ccgtggctca taaccacatc gccacgatcc cttacccgtt ggaagagcag agtagcagca 1620
gcagcagcaa cgtgcttggt gattgatact ttgataggct ggttttagca gcaacaaagg 1680
tgtactagtt gatgtattgt ttgtgtttgc cgggccatca tagaaagtgc ctggtgatct 1740
ctgggacttg atggcaaatg ttgggcaaat tgtgatcgaa taagattagt actagagtta 1800
tcgtgtaata aggacatgca tggactacca tgtatttcat gttatgacgc tcctaagagc 1860
cacagaccac agcgatggta ttagacccct tctcagaatg gttctgctca ttttcggctt 1920
cgatcgtggt acgcgttcgt gtcttcgtgt gatcggaaaa aaaatatttg ccgtttacaa 1980
gtgatagttt ttcagtggat gtaatttgta cgaaatacca tcgtacaaac gtttgtactt 2040
tttcatcata gtcattagcc tttcatgaat agtactcaca tttgtaaggc c 2091
<210> 4
<211> 43
<212> DNA
<213> 人工序列
<400> 4
accagtctct ctctcaagct tatggaagag aagaagcagc agc 43
<210> 5
<211> 41
<212> DNA
<213> 人工序列
<400> 5
gctcaccata ctagtggatc catcaccaag cacgttgctg c 41
<210> 6
<211> 2141
<212> DNA
<213> OsSTRL2基因
<400> 6
ttacctctct cgacgtaagc catggtcgcg cccctcggat cgccctagcc caccctctgc 60
gatggaaaga actacaatga taagattttc gctcgaggcg agaagaatta agaaggggaa 120
accgaaggag aagggattcg cgacattctt ccttgcgcgt ggagagacta agagaccggt 180
gagagggtcg cgggccttgc gtcttttatt ggccattgac tcgtagaccc gaagttttcg 240
ttgaggtatg gggcccaatg tgtcagtgac acgaattgtt ggcctgcatg ccggtgagga 300
aagagcatcg gcaggaagaa ggtgccacgt caattgtgct tttgtgagcc gttggattaa 360
ggatgtgggt tcgagatgaa ttcaaaaggt caaataatct tccggctgtg gcgctctcta 420
agtctcacct ttctatggtc aaataatgca ttagatagag aagatagtat aaggtttagg 480
gcatgttcac tttgataccg ttttcaacct taccaaattt tggtaaaatt gttaaaaaaa 540
agttgctaca tttagtttga tgccaaattt tagtaactat ataagaaatc attaactata 600
taagaaaaag gttctttttg gcatcaaaat gaataggccc ttatagtata tggtggtcat 660
caattctaat tttatccact cgtctactta ttaattaaga gataacatat ataatatatt 720
gcttttaata tacttactct gatatgctat actaattttt ctctatttgt tttcacttaa 780
tttattggtc tcttctctcg gattcctata agtttgtagt tttgtgtaga gattgtcctt 840
tcgtctcttc ctctctctct ctctctctct ctttctttct ctattccatc ttatcacatg 900
atcctacatg acatcttata gctaagagat ccatagtagg ttgagttcgg gaggcaaaca 960
aagaaagcga ttagagcgat attaatcaag tatcagtcta aaaaagcttg gaaaatggat 1020
taatatgatt tttaaagttt ttttatatag aaaacttttg taaaaaaacg tactttagca 1080
gtttgaaaag cgtgtgcgaa aaaaagatga aggtgggtag gaaaagaagg gagaataaca 1140
cagcataagc atgttttacc cctcgactat taactcttga ttaaggaaca tggtaaaatt 1200
tataccacta agttatcgaa accatatata aatgactttt aagataatct ccggtttttt 1260
ttaatatgaa acgtcattga ttttttaaga tatatttaac cattttcaaa cctaagaggt 1320
ggtttatttt ccctcaagag aacgcgccgt tattttttta cgtcacctaa aaaacataat 1380
tttttggaaa aaaatatatg aaaagacaga tgaatatatg atttttttaa aatacagatt 1440
caaactttac ttacacaaat agaaacaaaa aacaataaat ttgcttatga atggtcaaat 1500
tcattatttt tatttgtacc catgtaagtc aaatttaaac ttcatggtca ttgatatatt 1560
tataaaaaat agttttttag ttgaatttta aagtaacaaa aaaaacaaag ggacaaatag 1620
acttccctat aacccaacgc atgctcaacc acagtaaatg ttttagctta gacttagagt 1680
tagttttctt gcaacatcga cacaccattg cctgtgtctt gtgatttttc ctgcacgttt 1740
agaagggtgg catcgattga acttctggac acttggagtt cttccttctt cgtgatgcac 1800
cttttgcttt acagcgctag caatggccat ggtcagggcc ctcagccctg gccgggttcg 1860
gttacatgca tcgtgatatg cttgttgacc tgtgcatctt gcaccgtcat ccaagcaatg 1920
caaaacatgc aaatccccac ttcgaaagca caagattcct tggctattcc gaaccaacag 1980
aacacctact cccaaacaat cacgctgact catgcaacct ccatgcatcg aagatatatt 2040
ttcgctgcaa aggcattaag aatttaagtt aagatcaccg tctccaccaa ttccgtcttt 2100
cctctgcgtg caaattccgt cttccctcgc tcctgatctc c 2141
<210> 7
<211> 41
<212> DNA
<213> 人工序列
<400> 7
gagatctaca gcgctaagct tttacctctc tcgacgtaag c 41
<210> 8
<211> 40
<212> DNA
<213> 人工序列
<400> 8
ggactgacca cccggggatc cggagatcag gagcgaggga 40
<210> 9
<211> 23
<212> DNA
<213> OsSTRL2基因
<400> 9
ggaacgggtc ggtgaggagc agg 23
<210> 10
<211> 18
<212> DNA
<213> 人工序列
<400> 10
tcttccctcg ctcctgat 18
<210> 11
<211> 18
<212> DNA
<213> 人工序列
<400> 11
tggcacaaac tttctccg 18
<210> 12
<211> 41
<212> DNA
<213> 人工序列
<400> 12
acgacggcca gtgccaagct tttacctctc tcgacgtaag c 41
<210> 13
<211> 42
<212> DNA
<213> 人工序列
<400> 13
tatgaccatg attacgaatt ctgacttact cggttagctc gt 42
<210> 14
<211> 4561
<212> DNA
<213> OsSTRL2基因
<400> 14
ttacctctct cgacgtaagc catggtcgcg cccctcggat cgccctagcc caccctctgc 60
gatggaaaga actacaatga taagattttc gctcgaggcg agaagaatta agaaggggaa 120
accgaaggag aagggattcg cgacattctt ccttgcgcgt ggagagacta agagaccggt 180
gagagggtcg cgggccttgc gtcttttatt ggccattgac tcgtagaccc gaagttttcg 240
ttgaggtatg gggcccaatg tgtcagtgac acgaattgtt ggcctgcatg ccggtgagga 300
aagagcatcg gcaggaagaa ggtgccacgt caattgtgct tttgtgagcc gttggattaa 360
ggatgtgggt tcgagatgaa ttcaaaaggt caaataatct tccggctgtg gcgctctcta 420
agtctcacct ttctatggtc aaataatgca ttagatagag aagatagtat aaggtttagg 480
gcatgttcac tttgataccg ttttcaacct taccaaattt tggtaaaatt gttaaaaaaa 540
agttgctaca tttagtttga tgccaaattt tagtaactat ataagaaatc attaactata 600
taagaaaaag gttctttttg gcatcaaaat gaataggccc ttatagtata tggtggtcat 660
caattctaat tttatccact cgtctactta ttaattaaga gataacatat ataatatatt 720
gcttttaata tacttactct gatatgctat actaattttt ctctatttgt tttcacttaa 780
tttattggtc tcttctctcg gattcctata agtttgtagt tttgtgtaga gattgtcctt 840
tcgtctcttc ctctctctct ctctctctct ctttctttct ctattccatc ttatcacatg 900
atcctacatg acatcttata gctaagagat ccatagtagg ttgagttcgg gaggcaaaca 960
aagaaagcga ttagagcgat attaatcaag tatcagtcta aaaaagcttg gaaaatggat 1020
taatatgatt tttaaagttt ttttatatag aaaacttttg taaaaaaacg tactttagca 1080
gtttgaaaag cgtgtgcgaa aaaaagatga aggtgggtag gaaaagaagg gagaataaca 1140
cagcataagc atgttttacc cctcgactat taactcttga ttaaggaaca tggtaaaatt 1200
tataccacta agttatcgaa accatatata aatgactttt aagataatct ccggtttttt 1260
ttaatatgaa acgtcattga ttttttaaga tatatttaac cattttcaaa cctaagaggt 1320
ggtttatttt ccctcaagag aacgcgccgt tattttttta cgtcacctaa aaaacataat 1380
tttttggaaa aaaatatatg aaaagacaga tgaatatatg atttttttaa aatacagatt 1440
caaactttac ttacacaaat agaaacaaaa aacaataaat ttgcttatga atggtcaaat 1500
tcattatttt tatttgtacc catgtaagtc aaatttaaac ttcatggtca ttgatatatt 1560
tataaaaaat agttttttag ttgaatttta aagtaacaaa aaaaacaaag ggacaaatag 1620
acttccctat aacccaacgc atgctcaacc acagtaaatg ttttagctta gacttagagt 1680
tagttttctt gcaacatcga cacaccattg cctgtgtctt gtgatttttc ctgcacgttt 1740
agaagggtgg catcgattga acttctggac acttggagtt cttccttctt cgtgatgcac 1800
cttttgcttt acagcgctag caatggccat ggtcagggcc ctcagccctg gccgggttcg 1860
gttacatgca tcgtgatatg cttgttgacc tgtgcatctt gcaccgtcat ccaagcaatg 1920
caaaacatgc aaatccccac ttcgaaagca caagattcct tggctattcc gaaccaacag 1980
aacacctact cccaaacaat cacgctgact catgcaacct ccatgcatcg aagatatatt 2040
ttcgctgcaa aggcattaag aatttaagtt aagatcaccg tctccaccaa ttccgtcttt 2100
cctctgcgtg caaattccgt cttccctcgc tcctgatctc catggaagag aagaagcagc 2160
agcagcagcg tccacagaga gggcgcgatg gcatcctgca gtatccgcac cttttcttcg 2220
cggcgctggc gctggccctg ctcctcaccg acccgttcca cctcggcccg ctcgccgggg 2280
tggactaccg gccggtgagg cacgagctgg cgccgtaccg cgaggtgatg gcgcggtggc 2340
cgcgggacaa cggcagccgg ctcaggcacg gcaggctgga gttcgtcgga caggtgttcg 2400
ggccggagtc catcgagttc gaccgccacg gccgcggccc ctacgccggc ctcgccgacg 2460
gccgcgtcgt gcggtggatg ggggaggacg ccgggtggga gacgttcgcc gtcatgagcc 2520
ctgactggta acgaacacct cgcctgcatt ttgctctcgc cctccacgaa aacacctctc 2580
gtagcagtgt acaattacgt gttcttatat tgcaaaaaaa ggtcggagaa agtttgtgcc 2640
aatggggtgg agtcgacgac gaagaagcag cacgagatgg agcgacggtg cggccggcct 2700
ctcgggctga ggtttcacgg cgagaccggc gagctctacg tcgccgacgc atactacggg 2760
ctcatgtccg tcggtccgaa cggcggggtg gcgacctctc tcgcgagaga agtcggcggg 2820
agcccggtca acttcgcgaa cgacctcgac atccaccgca acggctccgt gttcttcacc 2880
gacacgagca cgagatacaa cagaaagtgt gcagctgcag tatcactctc ttcagttgta 2940
tcgattctct atttccttct atcgttcaag attttctgat tagaatcagt tgtgcaggga 3000
tcatctgaac gttctgctag aaggtgaagg cacagggagg ctgctcagat atgacccaga 3060
aaccaaagct gcccatgtcg tgctgagcgg gctggtcttc ccgaatggcg tgcagatttc 3120
tgacgaccag cagttcctcc tcttctccga aacaacaaac tgcaggtgaa atggcacaag 3180
ctttcacagg ttctgaaaat actaaaggtt aaacaagatt cagaattgat taacattgca 3240
cgcatatgct gttctaggat aatgcggtac tggctggaag ggccaagagc cgggcaggtg 3300
gaggtgttcg ccgacctgcc ggggttcccg gacaacgtgc gactgagcag cggcggcggc 3360
ggcggacggt tctgggtggc gatcgactgc tgcaggacgg cggcgcagga ggtgttcgcc 3420
aagcggccgt ggctgcgaac gctctacttc aagctgcccc tgacgatgcg gacgctgggg 3480
aagatggtca gcatgcggat gcacaccctc gtcgcgctcc tcgacggcga aggggacgtc 3540
gtcgaggtgc tcgaggaccg gggcggcgag gtgatgcggc tggtgagcga ggtgagggag 3600
gtggggcgca agctgtggat cggcaccgtg gctcataacc acatcgccac gatcccttac 3660
ccgttggaag agcagagtag cagcagcagc agcaacgtgc ttggtgattg atactttgat 3720
aggctggttt tagcagcaac aaaggtgtac tagttgatgt attgtttgtg tttgccgggc 3780
catcatagaa agtgcctggt gatctctggg acttgatggc aaatgttggg caaattgtga 3840
tcgaataaga ttagtactag agttatcgtg taataaggac atgcatggac taccatgtat 3900
ttcatgttat gacgctccta agagccacag accacagcga tggtattaga ccccttctca 3960
gaatggttct gctcattttc ggcttcgatc gtggtacgcg ttcgtgtctt cgtgtgatcg 4020
gaaaaaaaat atttgccgtt tacaagtgat agtttttcag tggatgtaat ttgtacgaaa 4080
taccatcgta caaacgtttg tactttttca tcatagtcat tagcctttca tgaatagtac 4140
tcacatttgt aaggccgaag gtgttgtcct ataaagaaaa aaaaatgtac cagtaagtag 4200
ggtggctaat gagccaactc ggctcggctc gtaaaaactc gttaaattaa caagctagct 4260
tggcttgact cattaacata ataagtaaaa aactgaactt gactcgactc gttaaaaagt 4320
ctgagttggc tcattaaact cactaaactc gttgcaccaa aatacatgat ttaaaattca 4380
tatacaatat aatttgttgg cctatgacca aagattacga ttgacatgtt atgcatcatg 4440
actcataaga ttgtgctaca atggataatt atgaatttac gacctttaca cctgtaaata 4500
tatagacaat taacttctat tttaatgcat cacgagctca acgagctaac cgagtaagtc 4560
a 4561
<210> 15
<211> 1285
<212> DNA
<213> 水稻雄性不育突变体
<400> 15
atggaagaga agaagcagca gcagcagcgt ccacagagag ggcgcgatgg catcctgcag 60
tatccgcacc ttttcttcgc ggcgctggcg ctggccctgc tacctcaccg acccgttcca 120
cctcggcccg ctcgccgggg tggactaccg gccggtgagg cacgagctgg cgccgtaccg 180
cgaggtgatg gcgcggtggc cgcgggacaa cggcagccgg ctcaggcacg gcaggctgga 240
gttcgtcgga gaggtgttcg ggccggagtc catcgagttc gaccgccacg gccgcggccc 300
ctacgccggc ctcgccgacg gccgcgtcgt gcggtggatg ggggaggacg ccgggtggga 360
gacgttcgcc gtcatgagcc ctgactggtc ggagaaagtt tgtgccaatg gggtggagtc 420
gacgacgaag aagcagcacg agatggagcg acggtgcggc cggcctctcg ggctgaggtt 480
tcacggcgag accggcgagc tctacgtcgc cgacgcgtac tacgggctca tgtccgtcgg 540
tccgaacggc ggggtggcga cctctctcgc gagagaagtc ggcgggagcc cggtcaactt 600
cgcgaacgac ctcgacatcc accgcaacgg ctccgtgttc ttcaccgaca cgagcacgag 660
atacaacaga aaggatcatc tgaacgttct gctagaaggt gaaggcacag ggaggctgct 720
cagatatgac ccagaaacca aagctgccca tgtcgtgctg agcgggctgg tcttcccgaa 780
tggcgtgcag atttctgacg accagcagtt cctcctcttc tccgaaacaa caaactgcag 840
gataatgcgg tactggctgg aagggccaag agccgggcag gtggaggtgt tcgccgacct 900
gccggggttc ccggacaacg tgcgactgag cagcggcggc ggcggcggac ggttctgggt 960
ggcgatcgac tgctgcagga cggcggcgca ggaggtgttc gccaagcggc cgtggctgcg 1020
aacgctctac ttcaagctgc ccctgacgat gcggacgctg gggaagatgg tcagcatgcg 1080
gatgcacacc ctcgtcgcgc tcctcgacgg cgaaggggac gtcgtcgagg tgctcgagga 1140
ccggggcggc gaggtgatgc ggctggtgag cgaggtgagg gaggtggggc gcaagctgtg 1200
gatcggcacc gtggctcata accacatcgc cacgatccct tacccgttgg aagagcagag 1260
tagcagcaac gtgcttggtg attga 1285
<210> 16
<211> 127
<212> PRT
<213> 水稻雄性不育突变体
<400> 16
Met Glu Glu Lys Lys Gln Gln Gln Gln Arg Pro Gln Arg Gly Arg Asp
1 5 10 15
Gly Ile Leu Gln Tyr Pro His Leu Phe Phe Ala Ala Leu Ala Leu Ala
20 25 30
Leu Leu Pro His Arg Pro Val Pro Pro Arg Pro Ala Arg Arg Gly Gly
35 40 45
Leu Pro Ala Gly Glu Ala Arg Ala Gly Ala Val Pro Arg Gly Asp Gly
50 55 60
Ala Val Ala Ala Gly Gln Arg Gln Pro Ala Gln Ala Arg Gln Ala Gly
65 70 75 80
Val Arg Arg Arg Gly Val Arg Ala Gly Val His Arg Val Arg Pro Pro
85 90 95
Arg Pro Arg Pro Leu Arg Arg Pro Arg Arg Arg Pro Arg Arg Ala Val
100 105 110
Asp Gly Gly Gly Arg Arg Val Gly Asp Val Arg Arg His Glu Pro
115 120 125
<210> 17
<211> 386
<212> DNA
<213> 野生水稻
<400> 17
atggaagaga agaagcagca gcagcagcgt ccacagagag ggcgcgatgg catcctgcag 60
tatccgcacc ttttcttcgc ggcgctggcg ctggccctgc tcctcaccga cccgttccac 120
ctcggcccgc tcgccggggt ggactaccgg ccggtgaggc acgagctggc gccgtaccgc 180
gaggtgatgg cgcggtggcc gcgggacaac ggcagccggc tcaggcacgg caggctggag 240
ttcgtcggac aggtgttcgg gccggagtcc atcgagttcg accgccacgg ccgcggcccc 300
tacgccggcc tcgccgacgg ccgcgtcgtg cggtggatgg gggaggacgc cgggtgggag 360
acgttcgccg tcatgagccc tgactg 386
<210> 18
<211> 387
<212> DNA
<213> 水稻雄性不育突变体
<400> 18
atggaagaga agaagcagca gcagcagcgt ccacagagag ggcgcgatgg catcctgcag 60
tatccgcacc ttttcttcgc ggcgctggcg ctggccctgc tacctcaccg acccgttcca 120
cctcggcccg ctcgccgggg tggactaccg gccggtgagg cacgagctgg cgccgtaccg 180
cgaggtgatg gcgcggtggc cgcgggacaa cggcagccgg ctcaggcacg gcaggctgga 240
gttcgtcgga caggtgttcg ggccggagtc catcgagttc gaccgccacg gccgcggccc 300
ctacgccggc ctcgccgacg gccgcgtcgt gcggtggatg ggggaggacg ccgggtggga 360
gacgttcgcc gtcatgagcc ctgactg 387
- 还没有人留言评论。精彩留言会获得点赞!