评价生物分子网络中药物对模块间关系的影响的方法与流程
本发明属于生物信息
技术领域:
。具体而言,本发明涉及对于疾病状态下复杂生物分子网络如蛋白质相互作用网络、基因表达调控网络等,药物干预对该网络中模块间关系的影响的评价方法。
背景技术:
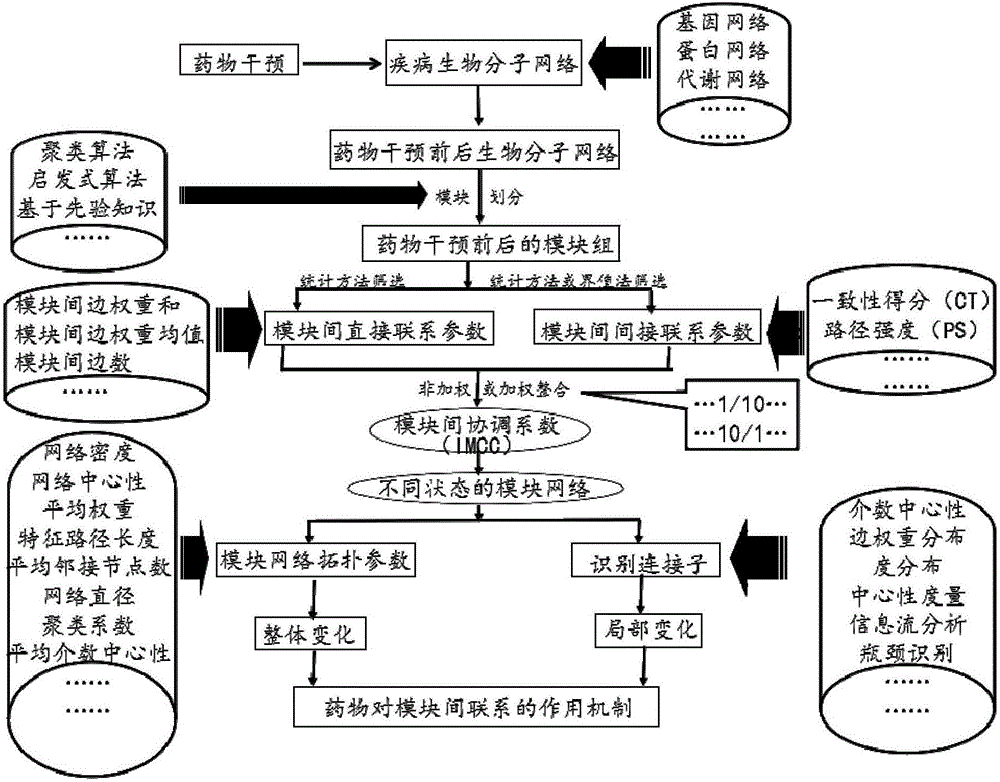
:基因、蛋白、代谢产物等之间存在多种相互作用。使用图论的方法表示,可以将这些基因、蛋白、代谢产物等分子各自视为单独的点,它们之间的相互作用视为边,形成了以点和边为元素的生物分子网络,而网络中连接比较紧密的分子构成的小集团称为模块。随着药物的多靶点性得到越来越多的证据支持,将模块作为药物靶点的研究正逐渐用于药物的研发及药理机制阐发等多个领域。然而,生物分子网络中的模块并不是独立存在的,而是互相联系、协调发展的。将目光集中于局部,单独研究每个模块的功能,不适用于网络全局的研究。特别是表型的变化不仅仅来源于生物分子网络中关键模块的变化,也可能由模块间关系的协调性变化带来。在系统科学的概念中,多个要素相互作用、相互补充、相互制约组成系统后,会出现系统组成前单个要素所不具有的性质,称为“整体涌现性”,即“整体大于部分之和”。这种涌现的现象,也体现在生物分子网络中,因此模块间协调性的变化也是疾病和药物作用的重要靶点。然而,对于将模块之间的关系作为药物靶点进行定量评价的研究方法仍较少。技术实现要素:针对上述技术问题,本发明基于复杂网络及模块的拓扑结构特点,提出一种评价生物分子网络中药物对模块间关系的影响的方法。如本文所用,术语“生物分子网络”是指在生物系统中以不同组织形式存在的网络,其由代表各种生物分子的节点与代表生物分子之间的相互作用关系的边组成。常见的生物分子网络包括基因转录调控网络、基因共表达网络、生物代谢网络、表观遗传网络、表型网络、信号传导网络、蛋白质相互作用网络等。如本文所用,术语“模块”是指生物分子网络中由紧密连接的数个节点构成的亚网络结构。如本文所用,术语“模块网络”是指由模块之间相互连接形成的网络,该网络由代表模块的节点与代表模块间相互作用关系的边构成,反映模块间联系的整体布局。如本文所用,术语“网络拓扑结构”是指网络中由节点和边构成的布局和构型,所述布局和构型的各种定量指标由拓扑参数反映。如本文所用,术语“拓扑参数”是指反映网络拓扑结构的布局和构型的定量指标,如网络密度、平均邻接节点数等。如本文所用,术语“模块间直接联系”是指网络拓扑结构中,两个不同模块(形成一个模块对)直接通过彼此的一个或多个节点之间的边连接,即两个不同模块之间的连接路径为来自不同模块的节点之间的边,如图1中的1A所示。如本文所用,模块间直接联系的紧密程度由术语“模块间直接联系参数”表示。“模块间直接联系参数”基于模块间的边的数量和权重计算并经超几何分布检验和/或介值法筛选得到。如本文所用,术语“模块间间接联系”是指网络拓扑结构中,两个不同模块(形成一个模块对)通过除该模块内节点之外的节点连接,即两个不同模块之间的连接路径为来自不同模块的节点与另外的节点(即中介节点)之间的边,如图1中的1B所示。如本文所用,模块间间接联系的紧密程度由术语“模块间间接联系参数”表示。“模块间间接联系参数”基于由中介节点构成的模块间的路径计算并经超几何分布检验和/或介值法筛选得到。如本文所用,术语“模块间协调系数”(Inter-modulecoordinatecoefficient,下文简称“IMCC”)是用于综合定量评价模块间关系的参数,以反应两个不同模块(形成一个模块对)之间的关系紧密程度。“模块间协调系数”由“模块间直接联系参数”和“模块间间接联系参数”经由加权求和或不加权求和得到。两个模块(形成一个模块对)之间IMCC越大,则该模块对中两个模块之间的关系越紧密,反之亦然。如本文所用,术语“连接子”是指在模块网络中,对整个模块网络拓扑结构的构型具有重要作用的模块,其中重要作用可以表现为介导其它模块之间的连接、与其它模块的连接程度紧密、对整个模块网络连接具有重要作用、构成其它模块之间连接的路径等。如本文所用,术语“连接子离解率”(下文简称“DR”)是指反映连接子(模块)在经过药物干预后的离散程度的参数。本发明提供了一种评价生物分子网络中药物对模块间关系的影响的方法。本发明的方法通过针对药物干预前后的生物分子网络分别进行模块的识别,对模块间直接和间接联系参数进行计算、筛选、整合,获得每组模块中任意两个模块的模块间协调系数,构建模块网络,用于对比药物干预前后的模块网络拓扑参数,或用于识别药物干预前后的模块网络连接子,计算连接子离解率,以综合、定量地比较与分析在疾病状态下药物干预前后模块间关系的整体和局部变化。本发明的实现流程见图2,且可描述为:步骤1,对于疾病相关的药物干预前后的复杂网络进行模块的识别,模块要求节点数目≥3;步骤2,对于获得的药物干预前后的两组模块,分别获得每组模块中任意两个模块的模块间协调系数;步骤3,根据模块间协调系数,构建以模块为单位的模块网络;步骤4.1,根据模块网络,计算疾病相关的药物干预前后的模块网络的拓扑结构变化综合值,作为药物对模块间关系的整体影响;或者步骤4.2,针对模块网络,识别对模块网络拓扑结构具有主要影响的连接子(模块),计算连接子离解率,识别药物对模块间关系的局部影响。具体地,本发明提供一种评价生物分子网络中药物对模块间关系的影响的方法,所述方法包括以下步骤:(1)以节点数目≥3为标准,分别对于疾病相关的药物干预前的生物分子网络和药物干预后的生物分子网络进行模块的识别,以获得药物干预前的生物分子网络的一组模块和药物干预后的生物分子网络的一组模块;(2)对于步骤(1)获得的两组模块,分别获得每组模块中任意两个模块的模块间协调系数;(3),基于步骤(2)获得的模块间协调系数,分别构建以模块为单位的模块网络,以获得药物干预前的模块网络和药物干预后的模块网络;和(4-1)针对步骤(3)获得的两个模块网络,以一个或多个拓扑参数计算药物干预前后模块网络的拓扑结构变化;或者(4-2)针对步骤(3)获得的两个模块网络,通过度量模块重要性的方法识别连接子,计算连接子离解率。在本发明方法的步骤(1)中,优选地,生物分子网络为蛋白质相互作用网络、基因共表达网络、基因转录调控网络、生物代谢网络、表观遗传网络、表型网络、信号传导网络等。更优选地,所述生物分子网络中的生物分子为基因和/或蛋白;优选地,所述生物分子网络为蛋白质相互作用网络、基因共表达网络、基因转录调控网络或信号传导网络。更优选地,所述生物分子网络为蛋白质相互作用网络或基因共表达网络。优选地,所述生物分子网络由50-10000个节点和100-100000条边组成;优选由100-8000个节点和500-80000条边组成;更优选由300-5000个节点和1000-50000条边组成。优选地,通过聚类、启发式算法或基于先验知识的方法进行模块的识别。在本发明方法的步骤(2)中,优选地,所述模块间协调系数通过模块间直接联系参数和模块间间接联系参数的加权求和计算得到,计算公式为:IMCC=α.DC+β.IDC(I)上式(I)中,DC代表模块间直接联系参数,IDC代表模块间间接联系参数,α和β分别为DC和IDC的加权系数。其中,DC基于模块间的边的数量和权重计算并经超几何分布检验和/或介值法筛选得到;优选地,通过计算模块间边的权重和、模块间边的数目或模块间边的权重的平均值并经超几何分布检验和/或介值法筛选得到DC。IDC基于由中介节点构成的模块间的路径计算并经超几何分布检验和/或介值法筛选得到;优选地,通过计算一致性得分(CT)或路径强度(SP)或模块间重叠节点数量并经超几何分布检验和/或介值法筛选得到IDC。根据本发明的具体实施方式,一致性得分(CT)采用Jeh-TingHsu,Chien-HuaPeng,Wen-PingHsieh等人的公式(Anovelmethodtoidentifycooperativefunctionalmodules:studyofmodulecoordinationintheSaccharomycescerevisiaecellcycle.BMCBioinformatics2011,12:281)进行计算:上式(II)中,Mx和My是每组模块中任意两个模块,i是节点,G是包含整个网络内节点的集合,C是G中所有的节点的数量,CLi是G中任一节点i的总度数,Wi是节点i的所有加权度,S和T分别是模块Mx和My内节点的数量,CMx,i和CMy,i分别是节点i和模块Mx和My连接的数量。根据本发明的具体实施方式,路径强度(SP)采用以下公式进行计算:上式(III)中,Mx和My是每组模块中任意两个模块,模块Mx和My之间的路径强度定义为由中介节点m(mediation)介导而形成路径的加权概率。此路径由模块Mx中的节点o(outset)和模块My中的节点e(end),以及中介节点m(mediation)构成,即o-m-e。Wm,o和Wm,e分别是节点m到节点o和节点e的权重,Wm是节点m加权度。路径强度是经这一路径形成的概率校正过的路径的权重。优选地,加权系数α和β通过如下方式确定:以α+β=1为标准,以二者分别5-20个不同取值计算相应的IMCC值(即得到5-20个IMCC值),而后分别将这5-20个IMCC值与生物学数据库(KEGG或GeneOntology)中的模块分类进行数据拟合,选择拟合度最高的α和β值。或者,在步骤(2)中,所述模块间协调系数通过模块间直接联系参数和模块间间接联系参数的不加权求和计算得到,计算公式为:IMCC=DC+IDC(IV)上式(IV)中,DC代表模块间直接联系参数,IDC代表模块间间接联系参数;其中,DC基于模块间的边的数量和权重计算并经超几何分布检验和/或介值法筛选得到;优选地,通过计算模块间边的权重和、模块间边的数目或模块间边的权重的平均值并经超几何分布检验和/或介值法筛选得到DC;其中IDC基于由中介节点构成的模块间的路径计算并经超几何分布检验和/或介值法筛选得到;优选地,通过计算一致性得分(CT)或路径强度(SP)或模块间重叠节点数量并经超几何分布检验和/或介值法筛选得到IDC。在本发明方法的步骤(3)中,优选地,通过以下方式构建模块网络:以每个模块为节点,以任意两个模块之间的IMCC值作为两个模块之间的边,构建模块网络。在本发明方法的步骤(4-1)中,优选地,所述拓扑参数为基于边权重的拓扑参数、基于拓扑构型的拓扑参数和基于边数量的拓扑参数中的至少一种;优选地,所述拓扑参数为选自表1中的一种或多种;更优选地,采用包括网络中心性和平均权重的至少3种拓扑参数。表1拓扑参数拓扑参数名称英文分类网络密度Networkdensity基于边数量网络中心性Networkcentrality基于拓扑构型平均权重Averageweight基于边权重特征路径长度Characteristicpathlength基于边数量平均邻接节点数Averageneighborhood基于边数量网络直径Networkdiameter基于边数量聚类系数Clustercoefficient基于边数量平均介数中心性Averagebetweenness基于边数量根据本发明的具体实施方式,可以采用网络密度、网络中心性和平均权重的组合计算药物干预前后模块网络的拓扑结构变化。在本发明方法的步骤(4-2)中,优选地,所述度量模块重要性的方法为选自表2中的一种或多种。表2度量模块重要性的方法在表2中,介数中心性参见YuH,KimPM,SprecherE,TrifonovV,GersteinM(2007)Theimportanceofbottlenecksinproteinnetworks:Correlationwithgeneessentialityandexpressiondynamics.PLoSComputBiol3(4):e59.。基于边权重分布识别连接子,是指通过将所有模块间边根据权重进行频数分布,识别边权重最大的模块对,将其视为连接子。度分布参见YuH,KimPM,SprecherE,TrifonovV,GersteinM(2007)Theimportanceofbottlenecksinproteinnetworks:Correlationwithgeneessentialityandexpressiondynamics.PLoSComputBiol3(4):e59。中心性度量参见WangY,DiZ,FanY(2011)IdentifyingandCharacterizingNodesImportanttoCommunityStructureUsingtheSpectrumoftheGraph.PLoSONE6(11):e27418。信息流分析参见MissiuroPV,LiuK,ZouL,RossBC,ZhaoG,etal.(2009)InformationFlowAnalysisofInteractomeNetworks.PLoSComputBiol5(4):e1000350。瓶颈识别参见YuH,KimPM,SprecherE,TrifonovV,GersteinM(2007)Theimportanceofbottlenecksinproteinnetworks:Correlationwithgeneessentialityandexpressiondynamics.PLoSComputBiol3(4):e59。优选地,所述步骤(4-2)中的连接子离解率通过以下公式计算:上式(V)中,nA是药物干预前的模块对内模块的数量(nA=2),nB是药物干预后所述模块对离解后的模块数量,NA和NB分别是药物干预前(A状态)和药物干预后(B状态)的网络内总模块数。本发明的优势在于基于疾病和药物干预的复杂网络的拓扑结构特点,基于拓扑结构和统计检验综合定量评价比较疾病或药物干预前后生物网络的模块间关系的整体和局部变化,作为疾病或药物靶向的模块间关系变化。与现存方法相比更具有综合整合、定量的特征。附图说明以下,结合附图来详细说明本发明的实施方案,其中:图1显示了模块间直接联系和间接联系示意图。图2显示了本发明所述评价药物对模块间关系的作用的方法的流程图。图3显示了基因共表达网络中基于IMCC值的Vehicle、BA、JA、UA、CM组的模块网络。其中,黑色边框模块为识别的连接子模块。图4显示了蛋白相互作用网络中基于IMCC值的Vehicle和BA模块网络。图5显示了蛋白相互作用网络中基于介数中心性分别识别的连接子构成亚网络。图6显示了蛋白相互作用网络中Vehicle组连接子模块对(module2和module3)在BA组的离解情况。具体实施方式以下参照具体的实施例来说明本发明。本领域技术人员能够理解,这些实施例仅用于说明本发明,其不以任何方式限制本发明的范围。下述实施例中的实验方法,如无特殊说明,均为常规方法。下述实施例中所用的药材原料、试剂材料等,如无特殊说明,均为市售购买产品。本发明的目的是从药物干预前后的复杂网络中,对比评价模块间关系的整体和局部变化,从而为指导疾病治疗和药物研发提供依据。以下的实施证明了本发明方法的有效性和可行性。这些实施是非限制性的,本发明的方法还可以应用其他类型的网络。实施例1本实施例采用以清开灵有效组分干预小鼠脑缺血模型的基因芯片实验数据为例,(该数据已上传至ArrayExpress数据库,Http://www.ebi.ac.uk/arrayexpress/),构建加权基因共表达网络,具体描述本发明方法:基于清开灵各组分干预脑缺血小鼠模型的加权基因共表达网络及模块划分,脑缺血模型组(Vehicle)、黄芩苷(BA)组、栀子苷(JA)组、胆酸(UA)组、珍珠母(CM)组的模块划分情况及如表3所示。表3基因共表达网络的模块划分情况步骤1,用加权共表达网络分析(WGCNA)工具(一种基于聚类算法的模块划分方法,来源PeterLangfelder,SteveHorvath.WGCNA:anRpackageforweightedcorrelationnetworkanalysis.BMCBioinformatics2008,9:559.)分别对药物干预前后的数据构建基因共表达网络并以节点数=3划分模块,由此分别得到药物干预前(Vehicle组)和药物干预后(BA组、JA组、UA组、CM组)的5个基因共表达网络和相应的5组模块。步骤2,通过计算每一组模块(5个状态,即5组)内任意两两模块之间的直接联系参数(本实施例选择模块间边的权重和,SW)和间接联系参数(本实施例选择模块间一致性得分,CT),而后对SW进行超几何分布检验,筛选出p<0.05的SW;对CT进行介值法筛选,筛选出CT>10的CT值,采用加权求和整合的方式,形成模块间协调系数(IMCC)即α.SW+β.CT=IMCC其中,直接联系参数SW是指模块间边的权重和;间接联系参数CT的采用Jeh-TingHsu,Chien-HuaPeng,Wen-PingHsieh等人的公式(Anovelmethodtoidentifycooperativefunctionalmodules:studyofmodulecoordinationintheSaccharomycescerevisiaecellcycle.BMCBioinformatics2011,12:281)进行计算,公式如下:令α+β=1,α/β分别取1/10、1/8、1/4、1/2、1/1、2/1、4/1、8/1、10/1分别获得9个IMCC值,将每个模块对之间的IMCC值和该模块对富集的KEGG通路的分类的jaccard相似系数对应起来,分别进行曲线拟合,如表4所示,当α/β=1/1时拟合度最佳,R2=0.616,公式即为:0.5SW+0.5CT=IMCCIMCC值分别作为5组模块中的任意两两模块之间关系的定量值。表4α/β不同值时,各模型拟合R2步骤3,基于IMCC值,构建模块网络,如图3所示。步骤4-1,计算干预前后模块网络的拓扑结构变化(从多个拓扑参数中选择8个),如表5所示,作为药物对模块间关系的整体影响。表5基因共表达模块网络拓扑参数注:↑代表与Vehicle组相比升高,↓代表与Vehicle组相比降低由表5结果可知,BA、JA、UA、CM的中心性分别下降至0.156、0.271、0.22、0.314,提示疾病状态下模块网络的呈明显的放射性结构,组分干预之后,这种放射性的结构发生了变化;在BA和UA干预后,网络密度、聚类系数、平均度升高,特征路径长度、平均介数中心性降低,提示模块间的连接更加密集,模块间信息传递路径缩短。JA干预之后,网络密度、模块平均度、聚类系数降低;网络特征路径长度、介数中心性增加,提示模块间连接的中断,形成更加稀疏的网络。步骤4-2,基于模块网络,从多种度量模块重要性的方法中选用基于介数中心性、边权重分布这两种方法识别连接子,两种方法识别的连接子的结果分别见表6和表7。在模型组(Vehicle)的模块网络中,选择两种方法均识别的两个连接子(blue模块和brown模块),计算该两个连接子在不同药物干预后的离解率,计算公式,如下:其中,nA是药物干预前的模块对内模块的数量(nA=2),nB是药物干预后所述模块对离解后的模块数量,NA和NB分别是药物干预前(A状态)和药物干预后(B状态)的网络内总模块数。表6基于介数中心性识别的连接子表7基于边权重分布识别的连接子连接子离解情况如表8所示,以识别不同药物对模块间关系的局部影响。表8药物干预后的连接子模块Blue和模块Brown的离解率由表8结果可知,BA,JA,UA的连接子离解率较CM高,提示BA,JA,UA对要病理状态的逆转程度优于CM。实施例2以清开灵有效组分干预小鼠脑缺血模型的蛋白质相互作用网络为例,具体描述本发明方法:基于清开灵各组分干预脑缺血小鼠模型的蛋白质相互作用网络及模块划分,脑缺血模型组、黄芩苷(BA)组的模块划分情况(参见张莹莹,清开灵多组分干预脑缺血模型蛋白质网络主要模块的识别与比较[D],中国中医科学院,2014),如表9所示。表9蛋白相互作用网络的模块划分情况步骤1,通过将药物干预前后的差异基因映射至string蛋白数据库中,构建清开灵各组分干预前后的蛋白质相互作用网络并以节点数=3划分模块。步骤2,通过对模块之间的直接联系参数——模块间边数(SE)和间接联系参数——路径强度(SP)进行计算,并分别对两个参数进行超几何分布检验筛选,而后将两个参数非加权整合为模块间协调系数(IMCC),该实例中采用的求和的非加权整合方式,公式如下:IMCC=SE+SP其中模块间直接联系参数采用模块间边数(SE),间接联系参数采用路径强度(SP),SP公式如下:其中,模块Mx和My之间的路径强度定义为由中介节点m(mediation)介导而形成路径的加权概率。此路径由模块Mx中的节点o(outset)和模块My中的节点e(end),以及中介节点m(mediation)构成,即o-m-e。Wm,o和Wm,e分别是节点m到节点o和节点e的权重,Wm是节点m加权度。IMCC值分别作为2组模块中的任意两两模块之间间关系的定量值。步骤3,基于IMCC值,构建模块网络,如图4所示。步骤4-1,计算干预前后模块网络的拓扑结构变化(从多个拓扑参数中选择8个),如表10所示,作为药物对模块间关系的整体影响。表10蛋白相互作用网络拓扑参数与模型组(Vehicle)相比,BA干预后,模块网络的中心性下降,提示模型组的放射性结构在BA干预后发生改变,平均介数中心性下降,提示模块间连接经过的节点数减少,及模块间连接更加直接与紧密。步骤4-2,基于模块网络,采用基于介数中心性的方法识别连接子,如图5所示。选取其中连接子模块2和模块3,计算该两个连接子在BA干预后的离解率,公式如下:其中,nA是药物干预前的模块对内模块的数量(nA=2),nB是药物干预后所述模块对离解后的模块数量,NA和NB分别是药物干预前(A状态)和药物干预后(B状态)的网络内总模块数。BA干预后的离解率如表11,图6所示,识别药物对模块间关系的局部影响。表11蛋白相互作用网络连接子模块2和模块3的离解率通过上述分析实例,本发明针对差异基因共表达网络采用两个参数加权整合的方法定量评价,各药物干预组前后的模块间关系,并使用8个拓扑参数评价模块间关系的整体变化,2种方法识别模块网络连接子,评价模块间关系的局部变化。同时本发明针对药物干预前后的蛋白相互作用网络采用两个参数非加权整合的方法定量评价模块间的关系,两个实例分别为生物网络中具有代表性的两类网络,说明评价生物网络中模块间关系的方法具有有效性和可行性。以上是对本发明的描述,而非限定,基于本发明的思想的其他实施方式,均在本发明的保护之中。以上对本发明具体实施方式的描述并不限制本发明,本领域技术人员可以根据本发明作出各种改变或变形,只要不脱离本发明的精神,均应属于本发明所附权利要求的范围。当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1