检测体细胞突变的方法和装置与流程
本发明涉及生物信息领域,具体的,本发明涉及一种检测体细胞突变的方法和一种检测体细胞突变的装置。
背景技术:
目前,临床上肿瘤诊断以组织细胞学检查作为金标准,生物化学检查方法及物理学检查(X光,B超,CT,MRI,PET-CT等)作为辅助手段,但这些肿瘤诊断方法存在现实局限性,即仅能发现0.5cm以上的肿块。当肿块无症状而渐渐长到为自身所觉察的大小时,部分肿瘤已经处于中晚期,有的肿瘤已经发生了转移,很多病人已经丧失了最佳的治疗时期。由于基因编码区的变异是导致疾病的主要原因,因此将个体基因组的某些特定基因编码区即特定外显子区域提取出来进行测序就可以很好的了解该个体基因组特定基因的变异信息,进而评估该个体的患病风险。该技术的基本原理是使用寡核苷酸探针捕获技术或PCR多重扩增的方式来获取基因组上的目标序列,对目标序列产物进行高通量测序,从而识别DNA样品中的碱基序列及变异信息。
随着基于目标区域捕获(target capture)的新一代测序(next generation sequencing,NGS)方法的出现,市面上已出现不少基于该技术、针对特定疾病的基因检测产品,如Life Technologies公司的Ion AmpliSeq Cancer panel,Illumina公司的TruSight One NGS panel及QIAGEN公司的GeneRead DNAseq V2panels等。
技术实现要素:
依据本发明的一方面,本发明提供一种检测体细胞突变的方法,该方法包括:获取第一测序数据,所述第一测序数据包括多个第一读段,所述第一测序数据为待测样本的核酸序列的测定数据;将所述第一测序数据与参考序列比对,获得第一比对结果;基于所述第一比对结果,识别出突变位点,所述突变位点包括SNP和INDEL中的至少之一;比较所述突变位点与正常对照样本的相应突变位点的支持读段数的差异,获得差异显著的突变位点,所述差异显著的突变位点包括差异具有统计意义的突变位点。
所说的测定数据通过对核酸序列进行测序文库制备、上机测序获得,在本发明的一个实施例中,获取所述测定数据,包括:获取待测样本中的核酸,制备所述核酸的测序 文库,对所述测序文库进行测序。测序文库的制备方法根据所选择的测序方法的要求进行,测序方法依据所选的测序平台的不同,可选择但不限于Illumina公司的Hisq2000/2500测序平台、Life Technologies公司的Ion Torrent平台和单分子测序平台,测序方式可以选择单端测序,也可以选择双末端测序,获得的下机数据是测读出来的片段,称为读段(reads)。在本发明的一个实施例中,所称第一测序数据中的读段的长度不相同,例如测序数据是利用Life Technologies公司的Ion Torrent系列中的Proton测序平台进行测序获得的。
所称的比对可以利用已知比对软件进行,例如SOAP、BWA和TeraMap等。
所称的参考序列指预先确定的序列,可以是预先获得的待测样本所属生物类别的任意参考模板,例如,若待测样本来源的为人类个体,参考序列可选择NCBI数据库提供的HG19,进一步地,也可以预先配置包含更多参考序列的资源库,例如依据待测样本来源个体的状态、地域等因素选择或是测定组装出更接近的序列作为参考序列。
在本发明的一个实施例中,所称的突变位点包括SNP(SNV)和INDEL。SNP的识别可以通过运行已知软件进行,可使用的SNP检测软件包括但不限于SomaticSniper、CaVEMan、SAMtools和MuTect。INDEL的识别可以通过运行已知软件进行,INDEL检测可选择的软件包括但不限于Dindel、Pindel、TVC和SOAPindel。
在本发明的一个实施例中,去除所述第一比对结果中的重复的第一读段,例如去除由于测序文库构建过程中的扩增而带来的重复片段。减小后续处理依据的数据量,利于基于比对结果进行快速检测。
所称的正常对照样本的相应突变位点为与待测样本的突变位点相同的位点。正常对照样本的突变位点,例如SNP和/或INDEL,可以在进行目标样本/个体/群体检测时同时测序检测获得,也可以预先确定保存备用。在本发明的一个实施例中,正常对照样本的SNP与待测样本的同时确定,确定所述正常对照样本的SNP包括:获取第二测序数据,所述第二测序数据包括多个第二读段,所述第二测序数据为正常对照样本的核酸序列的测定数据,所述第二测序数据与所述第一测序数据等量,任选的,所述第一测序数据和所述第二测序数据都大于100X;将所述第二测序数据与参考序列比对,获得第二比对结果;基于所述第二比对结果,识别出所述正常对照样本的相应SNP位点。
正常对照样本的INDEL可以同待检样本的同时获得,也可以预先检测确定保存备用。在本发明的一个实施例中,正常对照样本的INDEL所述获取正常对照样本的INDEL包括:获取第二测序数据,所述第二测序数据包括多个第二读段,所述第二测序数据为正常对照样本的核酸序列的测定数据,所述第二测序数据与所述第一测序数据等量,任 选的,所述第一测序数据和所述第二测序数据都大于100X;将所述第二测序数据与参考序列比对,获得第二比对结果;基于所述第二比对结果,检测正常对照样本的INDEL。同样的,INDEL检测可选择的软件包括但不限于Dindel、Pindel、TVC和/或SOAPindel。
所称的差异显著包括统计学上的差异具有显著性和临床或实际中的差异很大,对于前者,例如在本发明的一个实施例中,所述比较突变位点与正常对照样本的相应突变位点的支持读段数的差异,获得差异显著的突变位点,包括过滤掉符合以下(a)-(d)任意之一的SNP:(a)在正常对照样本中没有第二读段比对上,或者比对上的第二读段数目少于10条,(b)在正常对照样本中的支持读段数不少于5,(c)在待测样本中的支持读段数少于5,(d)在正常对照样本中的支持读段数为1-4条,并且与在待测样本中的支持读段数的差异不具统计意义(P>0.005)。在比对过程中,一般对比对参数进行设置,设置一条reads最多允许有s个碱基错配(mismatch),s优选为1或2,若reads中有超过s个碱基发生错配,则视为该reads无法比对到(比对上)参考序列。所称的支持读段,即支持某个SNP的读段指比对上该SNP的读段的相应位置的碱基与该SNP位点一致的读段。
对于后者,例如在本发明的一个实施例中,所述比较突变位点与正常对照样本的相应突变位点的支持读段数的差异,获得差异显著的突变位点,还包括过滤符合以下(e)-(h)任意之一的SNP:(e)两个相邻SNP之间的距离不超过5bp,(f)其支持读段中的低比对质量读段的比例超过50%,任选的,低比对质量读段为比对质量小于30的读段,(g)其位于其支持读段的末端的读段的比例超过50%,任选的,所述读段的末端为从读段的一端的起始核苷酸开始到不大于该读段长度5%的核苷酸的范围,(h)在距其不大于5bp的范围内其支持读段中包含的同时支持其它类型突变或者存在错配的读段的比例超过50%。这里,所称的两个SNP或者两个位点之间的距离指着两位点在参考序列上的距离。所称的比对质量是一般对比软件都有的参数,该实施例是利用BWA软件进行比对,本领域技术人员能够理解,利用不同的比对软件,可能由于计分方式的不同,需要调整设置以达到同该实施例的目的,即该实施例的满足(f)条件的读段。
在本发明的一个实施例中,所述比较突变位点与正常对照样本的相应突变位点的支持读段数的差异,获得差异显著的突变位点,包括过滤掉检出的INDEL中符合以下(i)-(k)任意之一的INDEL:(i)其支持读段同时支持其它一种或多种变异类型,(j)其支持读段中的低比对质量的读段的比例超过50%,任选的,所述低比对质量读段为比对质量小于30的读段,(k)其位于其支持读段的末端的读段的比例超过50%,任选的,读段的末端为从该读段的一端的起始核苷酸开始到不大于该读段长度10%的核苷酸的 范围。
在本发明的一个实施例中,所述比较突变位点与正常对照样本的相应突变位点的支持读段数的差异,获得差异显著的突变位点,包括对位于非同聚体并且符合以下(l)-(o)任意之一的INDEL进行过滤:(l)在正常对照样本中没有第二读段比对上,或者比对上的第二读段数目少于10,(m)在正常对照样本中的支持读段数不少于5,(n)在待测样本中的支持读段数少于5,(o)在正常对照样本中的支持读段数为1-4,并且与在待测样本中的支持读段数的差异不具统计意义(P>0.005)。INDEL的支持读段指比对上该INDEL的且相应位置与该INDEL的相同的读段。同聚体或者同聚物(homopolymer)一般指一段DNA中含有多个连续相同的碱基,在本发明的一个实施例中,定义相同连续的碱基不少于5个的那段序列为同聚物。
在本发明的一个实施例中,所述比较突变位点与正常对照样本的相应突变位点的支持读段数的差异,获得差异显著的突变位点,包括比较待测样本和正常对照样本中的位于同聚体的INDEL的频率的差异,保留待测样本中的频率显著大于正常对照样本的频率的INDEL。在本发明的一个实施例中,要求P<=0.005。
依据本发明的另一方面,提供一种检测体细胞突变的装置,该装置能够实施本发明一方面或者上述任一实施例中的检测突变体细胞的全部或部分步骤,该装置包括:数据输入单元,用以输入数据;数据输出单元,用以输出数据;处理器,用以执行可执行程序,执行所述可执行程序包括完成本发明一方面或者任一实施例中的检测体细胞突变的方法;以及存储单元,与所述数据输入单元、数据输出单元和处理器连接,用以存储数据,其中包括所述可执行程序。本领域技术人员能够理解,所说的计算机可执行程序可以保存在存储介质中,所称存储介质可以包括:只读存储器、随机存储器、磁盘或光盘等。在本发明的一个实施例中,将能够实现本发明检测方法整合成一软件,命名为Oseq-T,能够全过程半自动化实现本发明的检测方法,对计算机I/O资源,内存资源有较好控制,而且该流程用perl、C、Python和/或Java等语言编译,可移植性强;该软件可独立部署、高效运行,不影响以后加入其它类型突变检测分析操作。
本发明一方面提供的体细胞突变(Somatic mutation)检测方法算法和/或装置,旨在解决针对somatic mutation检测方法和工具不完善的问题。本发明的检测算法同时能够整合其他分析模块以形成更完整的肿瘤个体化检测技术方案。该方法和/或装置的检测结果作为中间数据或者辅助结果能够应用于肿瘤个体化检测(Cancer Panel)。该方法算法或者装置包含的计算机可执行程序严格,能够保证Somatic SNV及Indel检测的准确性。可检测突变频率≥3.5%的Somatic SNV突变,测试数据的准确性和敏感性均高于 95%;可检测突变频率≥10%的Somatic indel变异,测试数据显示极高的准确性(100%),同时homopolymer区域有较好的敏感性(56%)。该somatic mutation检测算法适用于各种测序平台,包括BGISEQ-100测序平台,该平台具有灵活、操作简便、省时、成本低、可扩展等优势,方便在移植操作,可以用于临床诊断和指导治疗的基因检测平台。本检测方法/装置系统能够应用于肿瘤个体化检测Cancer Panel,例如用以一次性检测多个肿瘤相关基因,解读注释药物与基因的关系等,利于辅助为个体化用药提供全面解决方案,利于辅助指导临床尽可能多的找到个体化用药依据,利于辅助指导临床正确选择个体化用药,利于辅助指导临床尽可能多的找到靶向药物,根据个体基因的个体差异性辅助或协助医生选择合适的药物和治疗方案,真正实现个体化医疗。
附图说明
本发明的上述和/或附加的方面和优点从结合下面附图对实施方式的描述中将变得明显和容易理解,其中:
图1是本发明的一个实施例中的体细胞突变检测方法的步骤示意图。
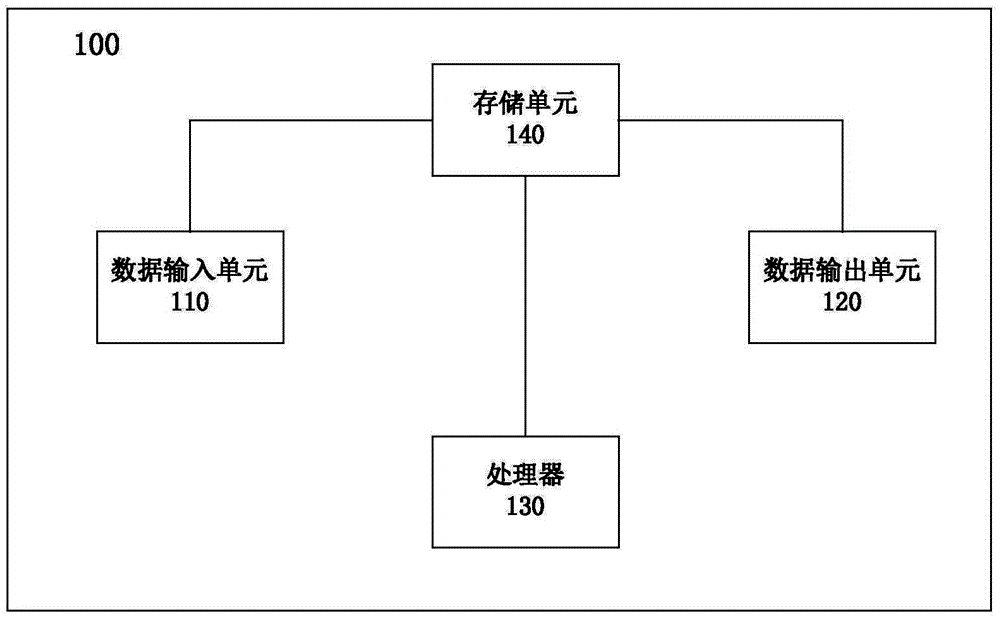
图2是本发明的一个实施例中的体细胞突变检测装置的结构示意图。
图3是本发明的一个实施例中的肿瘤个体化检测软件(Oseq-T)的流程图。
图4是本发明的一个实施例中的目标区域深度分布直方图。
图5是本发明的一个实施例中的目标区域深度累积分布图。
具体实施方式
如图1所示,根据本发明的一个实施例,提供一种检测体细胞突变的方法,该方法包括步骤:S10获取第一测序数据,所述第一测序数据包括多个第一读段,所述第一测序数据为待测样本的核酸序列的测定数据;S20将所述第一测序数据与参考序列比对,获得第一比对结果;S30基于所述第一比对结果,识别出突变位点,所述突变位点包括SNP和INDEL中的至少之一;S40比较所述突变位点与正常对照样本的相应突变位点的支持读段数的差异,获得差异显著的突变位点,所述差异显著的突变位点包括差异具有统计意义的突变位点。所说的测定数据通过对核酸序列进行测序文库制备、上机测序获得,在本发明的一个实施例中,获取所述测定数据,包括:获取待测样本中的核酸,制备所述核酸的测序文库,对所述测序文库进行测序。测序文库的制备方法根据所选择的测序方法的要求进行,测序方法依据所选的测序平台的不同,可选择但不限于Illumina公司的Hisq2000/2500测序平台、Life Technologies公司的Ion Torrent平台和单分子测序平台,测序方式可以选择单端测序,也可以选择双末端测序,获得的下机数据是测读出 来的片段,称为读段(reads)。在本发明的一个实施例中,所称第一测序数据中的读段的长度不相同,例如测序数据是利用Life Technologies公司的Ion Torrent系列中的Proton测序平台进行测序获得的。所称的比对可以利用已知比对软件进行,例如SOAP、BWA和TeraMap等。所称的参考序列指预先确定的序列,可以是预先获得的待测样本所属生物类别的任意参考模板,例如,若待测样本来源的为人类个体,参考序列可选择NCBI数据库提供的HG19,进一步地,也可以预先配置包含更多参考序列的资源库,例如依据待测样本来源个体的状态、地域等因素选择或是测定组装出更接近的序列作为参考序列。SNP的识别可以通过运行已知软件进行,可使用的SNP检测软件包括但不限于SomaticSniper、CaVEMan、SAMtools和MuTect。INDEL的识别可以通过运行已知软件进行,INDEL检测可选择的软件包括但不限于Dindel、Pindel、TVC和/或SOAPindel。
根据本发明的一个实施例中,去除所述第一比对结果中的重复的第一读段,例如去除由于测序文库构建过程中的扩增而带来的重复片段。减小后续处理依据的数据量,利于基于比对结果进行快速检测。
所称的正常对照样本的相应突变位点为与待测样本的突变位点相同的位点。正常对照样本的突变位点,例如SNP和/或INDEL,可以在进行目标样本/个体/群体检测时同时测序检测获得,也可以预先确定保存备用。在本发明的一个实施例中,正常对照样本的SNP与待测样本的同时确定,确定所述正常对照样本的SNP包括:获取第二测序数据,所述第二测序数据包括多个第二读段,所述第二测序数据为正常对照样本的核酸序列的测定数据,所述第二测序数据与所述第一测序数据等量,任选的,所述第一测序数据和所述第二测序数据都大于100X;将所述第二测序数据与参考序列比对,获得第二比对结果;基于所述第二比对结果,识别出所述正常对照样本的SNP。
正常对照样本的INDEL可以同待检样本的同时获得,也可以预先检测确定保存备用。在本发明的一个实施例中,正常对照样本的INDEL所述获取正常对照样本的INDEL包括:获取第二测序数据,所述第二测序数据包括多个第二读段,所述第二测序数据为正常对照样本的核酸序列的测定数据,所述第二测序数据与所述第一测序数据等量,任选的,所述第一测序数据和所述第二测序数据都大于100X;将所述第二测序数据与参考序列比对,获得第二比对结果;基于所述第二比对结果,检测正常对照样本的INDEL。同样的,INDEL检测可选择的软件包括但不限于Dindel、Pindel、TVC和/或SOAPindel。
所称的差异显著可以指统计学上的差异具有显著性,也可以指临床或实际中的差异很大。在本发明的一个实施例中,S40包括过滤掉符合以下(a)-(d)任意之一的SNP:(a)在正常对照样本中没有第二读段比对上,或者比对上的第二读段数目少于10条, (b)在正常对照样本中的支持读段数不少于5,(c)在待测样本中的支持读段数少于5,(d)在正常对照样本中的支持读段数为1-4条,并且与在待测样本中的支持读段数的差异不具统计意义(P>0.005)。在比对过程中,一般对比对参数进行设置,设置一条reads最多允许有s个碱基错配(mismatch),s优选为1或2,若reads中有超过s个碱基发生错配,则视为该reads无法比对到(比对上)参考序列。所称的支持读段,即支持某个SNP的读段指比对上该SNP的读段的相应位置的碱基与该SNP位点一致的读段。将符合(a)-(d)任意之一的SNP过滤掉,利于获得有意义的SNP,且能够使获得的体细胞SNP突变准确性和敏感性高。其中的(d)条件即属于统计学上的差异无显著性。
而在本发明的一个实施例中,S40包括过滤符合以下(e)-(h)任意之一的SNP:(e)两个相邻SNP之间的距离不超过5bp,(f)其支持读段中的低比对质量读段的比例超过50%,任选的,低比对质量读段为比对质量小于30的读段,(g)其位于其支持读段的末端的读段的比例超过50%,任选的,所述读段的末端为从读段的一端的起始核苷酸开始到不大于该读段长度5%的核苷酸的范围,(h)在距其不大于5bp的范围内其支持读段中包含的同时支持其它类型突变或者存在错配的读段的比例超过50%。这里,所称的两个SNP或者两个位点之间的距离指着两位点在参考序列上的距离。所称的比对质量是一般对比软件都有的参数,该实施例是利用BWA软件进行比对,本领域技术人员能够理解,利用不同的比对软件,可能由于计分方式的不同,需要调整设置以达到同该实施例的目的,即该实施例的满足(f)条件的读段。以上(e)-(h)任一条件去除掉的SNP都使得剩余的SNP差异显著,符合(e)-(h)任一条件的SNP都属于临床或实际中的差异不大的情形。将符合(e)-(h)任意之一的SNP过滤掉,有利于获得有意义的体细胞SNP突变,且准确性和敏感性高。
根据本发明的一个实施例,S40包括过滤掉检出的INDEL中符合以下(i)-(k)任意之一的INDEL:(i)其支持读段同时支持其它一种或多种变异类型,(j)其支持读段中的低比对质量的读段的比例超过50%,任选的,所述低比对质量读段为比对质量小于30的读段,(k)其位于其支持读段的末端的读段的比例超过50%,任选的,读段的末端为从该读段的一端的起始核苷酸开始到不大于该读段长度10%的核苷酸的范围。将符合以上(i)-(k)任意之一的INDEL过滤掉,有利于获得有意义的体细胞INDEL突变,且准确性和敏感性高。
根据本发明的一个实施例,S40包括对位于非同聚体并且符合以下(l)-(o)任意之一的INDEL进行过滤:(l)在正常对照样本中没有第二读段比对上,或者比对上的第二读段数目少于10,(m)在正常对照样本中的支持读段数不少于5,(n)在待测样本中 的支持读段数少于5,(o)在正常对照样本中的支持读段数为1-4,并且与在待测样本中的支持读段数的差异不具统计意义(P>0.005)。INDEL的支持读段指比对上该INDEL的且相应位置与该INDEL的相同的读段。同聚体或者同聚物(homopolymer)一般指一段DNA中含有多个连续相同的碱基,在本发明的一个实施例中,定义相同连续的碱基不少于5个为同聚物。
在本发明的一个实施例中,S40包括比较待测样本和正常对照样本中的位于同聚体的INDEL的频率的差异,保留待测样本中的频率显著大于正常对照样本的频率的INDEL。在本发明的一个实施例中,要求P<=0.005。这样,对位于homopolymer和非homopolymer中的INDEL进行不同条件的筛选过滤,使体细胞INDEL检测的准确度和灵敏度提高。
如图2,根据本发明的一个实施例,提供一种检测体细胞突变的装置100,该装置100能够实施本发明一方面或者上述任一实施例中的检测突变体细胞的全部或部分步骤,该装置100包括:数据输入单元110,用以输入数据;数据输出单元120,用以输出数据;处理器130,用以执行可执行程序,执行所述可执行程序包括完成本发明一方面或者任一实施例中的检测体细胞突变的方法;以及存储单元140,与所述数据输入单元110、数据输出单元120和处理器130连接,用以存储数据,其中包括所述可执行程序。本领域技术人员能够理解,所说的计算机可执行程序可以保存在存储介质中,所称存储介质可以包括:只读存储器、随机存储器、磁盘或光盘等。在本发明的一个实施例中,将能够实现本发明检测方法整合成一软件,命名为Oseq-T,能够全过程半自动化实现本发明的检测方法,对计算机I/O资源,内存资源有较好控制,而且该流程用perl、C、Python和/或Java等语言编译,可移植性强;该软件可独立部署、高效运行,不影响以后加入其它类型突变检测分析操作。
本发明一方面提供的体细胞突变(Somatic mutation)检测方法算法和/或装置,旨在解决针对somatic mutation检测方法和工具不完善的问题,该方法和/或装置的检测结果作为中间数据或者辅助结果能够应用于肿瘤个体化检测(Cancer Panel)。该方法算法或者装置包含的计算机可执行程序严格,能够保证Somatic SNV及Indel检测的准确性。可检测突变频率≥3.5%的Somatic SNV突变,测试数据的准确性和敏感性均高于95%;可检测突变频率≥10%的Somatic indel变异,测试数据显示极高的准确性(100%),同时homopolymer区域有较好的敏感性(56%)。该somatic mutation检测算法适用于各种测序平台,包括BGISEQ-100测序平台,该平台具有灵活、操作简便、省时、成本低、可扩展等优势,方便在移植操作,可以用于临床诊断和指导治疗的基因检测平台。本检 测方法/装置系统能够应用于肿瘤个体化检测Cancer Panel,例如用以一次性检测多个肿瘤相关基因,解读注释药物与基因的关系等,利于辅助为个体化用药提供全面解决方案,利于辅助指导临床尽可能多的找到个体化用药依据,利于辅助指导临床正确选择个体化用药,利于辅助指导临床尽可能多的找到靶向药物,根据个体基因的个体差异性辅助或协助医生选择合适的药物和治疗方案,真正实现个体化医疗。
以下结合附图和具体实施例对本发明的体细胞突变检测方法和/或装置进行详细的描述。下面示例,仅用于解释本发明,而不能理解为对本发明的限制。在本发明的描述中,除非另有说明,“多个”的含义是两个或两个以上。
除另有交待,以下实施例中涉及的未特别交待的试剂、序列(接头、标签和引物)、软件及仪器,都是常规市售产品或者开源的,比如购自Life Technologies等。
实施例一
在获得BGISEQ-100测序下机数据后,如图3所示,一般包括如下步骤:
1.与参考基因组比对
对测序数据使用tmap工具比对到参考基因组上,得到精确的比对结果。其中tmap工具源自:https://github.com/iontorrent/TS/tree/master/Analysis/TMAP
2.去除比对结果中的PCR重复片段
对tmap工具比对后的结果(bam格式)使用BamDuplicates工具去除PCR重复片段。其中,BamDuplicates工具源自Ion Torrent Systems,Inc.
3.统计及质量控制
统计目标区域数据量占总数据量的比例、目标区域的平均测序深度、目标区域的覆盖率等,生成一系列质控指标用于判断测序数据的质量情况。
4.Somatic变异检测,主要包括Somatic SNV及indel的检测
1)使用TVC工具进行Somatic SNV检测使用TVC工具
使用TVC工具
(http://ioncommunity.lifetechnologies.com/community/products/torrent-variant-caller),使用其默认参数json文件检测肿瘤相关的SNP,对正常组织突变位点进行堆积(Pile Up),采用统计学的显著性检验方法-P值检验检测肿瘤显著高于正常对照的somatic SNV,满足以下A-D任何一个条件,将被从肿瘤SNV中过滤掉:
A.若SNV位点在正常组织样本中没有reads覆盖或有reads覆盖但深度<10,
B.正常组织样本变异位点支持数>=5,
C.肿瘤组织样本变异位点支持数<5,
D.在正常组织样本变异位点支持数为1至4,且P值>0.005。
进一步针对BGISEQ-100数据特征设计了假阳性过滤方法,满足以下任何一个条件,将被从肿瘤SNV中过滤掉:
A.过滤掉两个相邻5bp之内的SNV,
B.变异位点支持reads中低质量(比对质量<30)比例>50%,
C.变异位点支持reads中突变位点位于reads首末端(5%read长度)比例>50%,
D.变异位点支持reads中突变位点相邻5bp内有突变或mismatch比例>50%。
2)使用TVC工具检测Somatic indel
使用TVC工具,调整其参数json文件,参数data_quality_stringency由8.5改为6,参数filter_unusual_predictions由0.25改为0.3,检测肿瘤组织indel,对检测到的Indel进行假阳性过滤。满足以下任何一个条件TVC检出的肿瘤组织Indel,将被过滤掉:
A.该处有多种变异类型,
B.变异区域支持reads中低质量(比对质量<30)比例>50%,
C.变异区域支持reads中突变位点位于reads首末端(10%read长度)比例>50%。
标记位于homopolymer(长度>=5)的Indel。对正常组织突变位点进行Pile Up,对满足条件的Somatic indel采用统计学的显著性检验方法-P值检验检测肿瘤显著高于正常对照的somatic Indel。对非homopolymer区域(长度>=5)和homopolymer区域的indel差别处理:非homopolymer区域严格筛选以保证准确性——满足以下任何一个条件,将被过滤掉肿瘤组织样品TVC检测的Indel:
A.Indel位点在正常组织样本中没有reads覆盖或有reads覆盖但深度<10,
B.正常组织样本变异位点读段支持数>=5,
C.肿瘤组织样本变异位点读段支持数<5,
D.在正常组织样本变异位点读段支持数为1至4,且P值>0.005;
homopolymer区域适当放松标准——要求肿瘤组织Indel频率大于正常组织Indel频率,并进行统计学的显著性检验-P值检验(满足P值<=0.005),保证准确性的同时很好地提升了敏感性。
5.可选择的,进行Somatic变异注释
对Somatic SNV及Indel进行注释,对预设置的质控位点进行检测,进行Annovar、Cosmic等数据库注释,同时鉴定样本的疾病来源或可能疾病来源,解读相关化疗药物和靶向药物与基因的关系等。
6.可选择的,生成Excel报告
对质控(QC),21个高频寡核酸多态性位点质控(QC21),疾病来源(Signatures),变异列表及变异注释(Variations),FDA临床药物(FDA),其他临床化疗药物(CtDrug)等进行汇总生成一张Excel表,数据解读人员在此Excel表格基础上对数据进行解读。例如包括解读88种肿瘤药物(42种FDA批准靶药,29种临床三、四期药物及17种化疗药物)与基因的关系,为解决个体化用药辅助提供全面解决方案,指导临床尽可能多的找到个体化用药依据,指导临床正确选择个体化用药,避免肿瘤患者选错药,指导临床尽可能多的找到靶向药物,根据患者基因的个体差异性协助医生选择合适的药物和治疗方案,助于真正实现个体化医疗。
实施例二
从医院获得1名结直肠癌女性患者的癌组织样本及血细胞样本(样品名:TJ0002)分别进行目标区域捕获及BGISEQ-100平台测序,按照上述实施例一,对测序有效数据通过tmap比对、BamDuplicates去重、质量控制(QC)、Somatic变异检测、变异注释、报告生成步骤,最终获得该患者的肿瘤个体化检测报告。
将实施例一包含的流程方法整合为软件Oseq-T,该软件的运行环境为Unix/Linux操作系统,通过Unix/Linux命令行运行。
具体操作步骤如下:
在LINUX操作系统计算机终端中输入以下命令以调用该软件:
perl Oseq_chip_proton.pl–c cancer.list–n normol.list,
Oseq-T命令行参数见表1参数说明。
表1
以下为分析结果:
其中,表2为部分的统计及质控分析结果,图4和图5分别为目标区域深度分布直方图和目标区域深度累积分布图。图4中的横坐标为测序深度,纵坐标为目标区域碱基 数比例,理论上应呈正态分布。图5中的横坐标为累计测序深度,纵坐标为目标区域碱基数比例。表3显示21个质控位点,21个高频寡核酸多态性列表,理论上第三、四列SNP类型应相同,与顺序无关(比如,20号染色体(chr20)位置(50238545)GA与AG的SNP类型相同,都表示A/G或G/A类型),可评估该批次测序质量及可行性。
表2
表3
表4显示判断疾病来源(Signatures)的部分结果,第一列为疾病来源标记(Signature ID),第二列表示协方差,第三列表示疾病来源信息。表5显示部分Somatic变异及其注释结果。表6显示部分FDA药物注释结果。表7显示部分其他临床化疗药物注释结果,药物包括铂类化合物Cisplatin(顺铂),Antineoplastic agents(抗肿瘤药)、cisplatin(顺铂)、cyclophosphamide(环磷酰胺)、fluorouracil(氟二氧嘧啶)和paclitaxel(紫杉醇)。
表4
表5
表6
表7
- 还没有人留言评论。精彩留言会获得点赞!