PKD1基因的STR位点及其应用的制作方法
本发明涉及临床分子诊断
技术领域:
,具体涉及一种PKD1基因的STR位点及其应用。
背景技术:
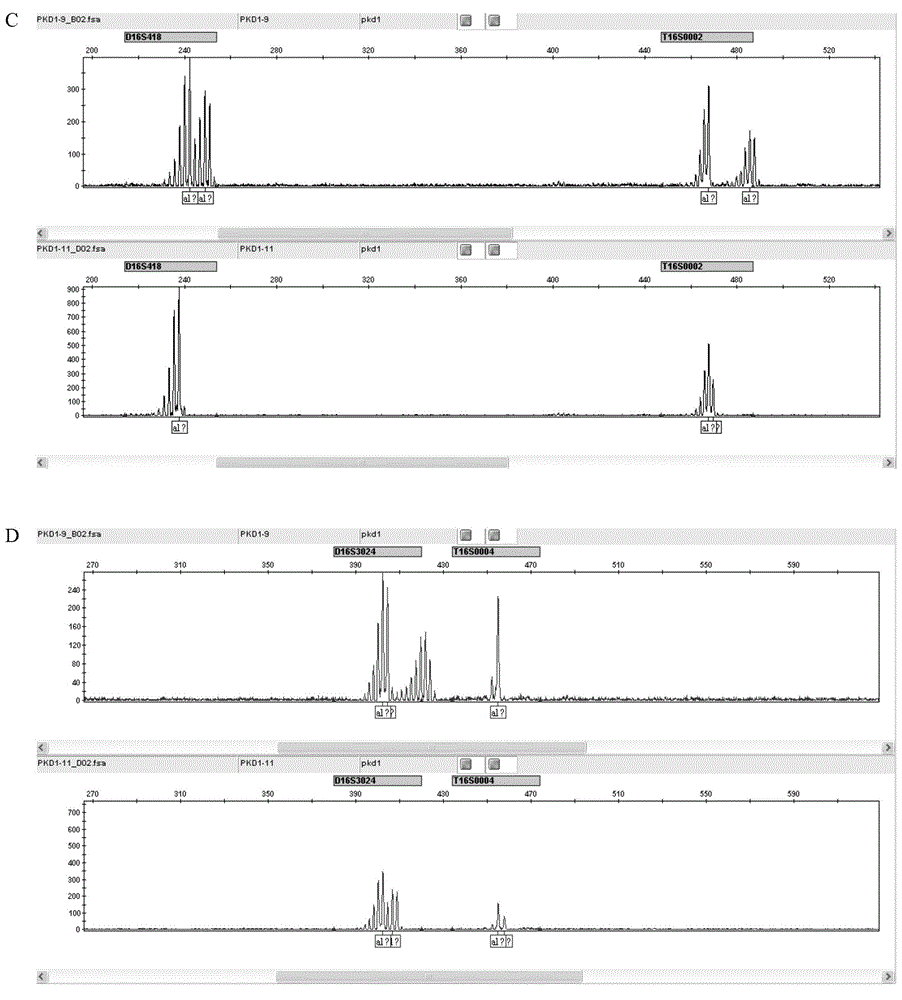
:常染色体显性遗传性多囊肾病(Autosomaldominantpolycystickidneydisease,ADPKD)是人类最常见的单基因遗传病之一,发病率约为0.1%。其主要临床特征是双侧肾脏形成多个液性囊泡,囊肿进行性生长,导致肾脏结构和功能的损害,至60岁时约50%患者发展为终末期肾功能衰竭,约占终末期肾功能衰竭病因的10%。多囊肾病患者除肾脏改变外,还可累及全身多个器官,如肝、胰、脾囊肿、颅内动脉瘤、高血压等。另有学者认为与胰腺癌发生有关。ADPKD属延迟显性遗传,患者一般在40岁左右才出现临床症状,此时多已将致病基因传给下一代。由于ADPKD目前尚无有效治疗方法,因此及时对ADPKD家系中有发病风险的成员进行症状前基因诊断,尤其是对高风险胎儿或胚胎进行产前诊断及植入前遗传学诊断,是控制其发生和发展的关键。ADPKD存在遗传异质性,目前已知引起该病的突变基因可能有3个,分别为PKD1,PKD2和PKD3。其中约85%的患者由PKD1突变所致。PKD1基因长度约52kb,包含46个外显子,转录出14.1kbmRNA,编码4302个氨基酸残基的跨膜蛋白,其中该基因的3’端(34~46外显子)约1/3部分为单拷贝区,另2/3部分(1~33外显子)由于存在至少3个同源序列区,给基因测序及突变检测带来了极大困难。目前PKD1的基因检测主要有以下几种方法:一、限制性片段长度多态性分析(RFLP)RFLP(RestrictionFragmentLengthPolymorphism,限制性内切酶片段长度多态性)作为第一代分子生物学标记自问世以来已广泛运用于多门生物学科研究中。RFLP是根据不同品种(个体)基因组的限制性内切酶的酶切位点碱基发生突变,或酶切位点之间发生了碱基的插入、缺失,导致酶切片段大小发生了变化,这种变化可以通过特定探针杂交进行检测,从而可比较不同品种(个体)的DNA水平的差异(即多态性)。1985年,Reeders等通过RFLP分析方法将PKD1基因定位于16p13,并利用a-珠蛋白基因的3’HVR探针,成功地在世界上首次对一个ADPKD妊娠妇女的胎儿进行了产前基因诊断。由于该技术对DNA质量要求高,需要量大,操作复杂,现已逐渐被其他技术所取代。二、单链构象多态性分析(SSCP)SSCP是利用组成碱基不同导致DNA单链构象的差异,在进行非变性的聚丙烯酰胺凝胶电泳时,迁移速度出现改变的检测方法。SSCP技术由于成本低、简便、快捷,不受家系连锁分析的限制,能够对单个患者进行突变的检测,且对单个位点的变异检出率较高。不过该技术存在5%~30%的漏检率,且只能检测突变的存在而不能确定突变的位置,这些缺陷严重限制了该技术在临床上的推广应用。三、变性高效液相层析(DHPLC)DHPLC是对扩增区域进行PCR扩增,PCR产物加热变性成单链状态,通过高压液相层析分析仪检测其中碱基含量,若发生改变则会出现峰值的移动,从而检测突变是否发生。Mizoguchi等首先采用此技术从PKD1部分编码区(第23~34外显子)和PKD2全编码区检测出8个突变位点。之后Rossetti等应用DHPLC技术分别对45个ADPKD患者进行了PKD1和PKD2全编码区突变检测。DHPLC技术虽然快速、简便,可判断有否突变,但不能确定SNP的位置和类型,需用标准样品或结合测序验证。只能检测杂合突变是DHPLC的主要不足之处,在点突变筛查的检测通量、灵敏度上也与其他检测技术存在差距。四、荧光原位杂交(FISH)荧光原位杂交(fluorescenceinsituhybridization,FISH)是在20世纪80年代末在放射性原位杂交技术的基础上发展起来的一种非放射性分子细胞遗传技术,以荧光标记取代同位素标记而形成的一种新的原位杂交方法,探针首先与某种介导分子(reportermolecule)结合,杂交后再通过免疫细胞化学过程连接上荧光染料.FISH的基本原理是将DNA(或RNA)探针用特殊的核苷酸分子标记,然后将探针直接杂交到染色体或DNA纤维切片上,再用与荧光素分子偶联的单克隆抗体与探针分子特异性结合来检测DNA序列在染色体或DNA纤维切片上的定性、定位、相对定量分析。由于PKD1基因存在同源基因及仅有3%的大片段缺失突变,FISH对于ADPKD诊断价值有限,应用于ADPKD的基因诊断受到极大限制。五、变性梯度凝胶电泳(DGGE)变性梯度凝胶电泳(Denaturinggradientgelelectrophoresis)是根据DNA在不同浓度的变性剂中解链行为的不同而导致电泳迁移率发生变化,从而将片段大小相同而碱基组成不同的DNA片段分开。具体而言,就是将特定的双链DNA片段在含有从低到高的线性变性剂梯度的聚丙烯酰胺凝胶中电泳,随着电泳的进行,DNA片段向高浓度变性剂方向迁移,当它到达其变性要求的最低浓度变性剂处,双链DNA形成部分解链状态,这就导致其迁移速率变慢,由于这种变性具有序列特异性,因此DGGE能区分存在低至单个碱基差异的DNA片段,被用于ADPKD的基因突变检测领域。该技术的缺陷在于检测通量较低,无法确定突变的具体类型,限制了其在临床检测中的应用。六、短串联重复序列(STR)STR在基因组中广泛存在,是以2~6个碱基为核心单位串联重复而成的一类序列,又称为微卫星DNA(microsatelliteDNA)。由于核心序列重复数目的变化,而在群体中呈现出遗传多态性,但在同一家系内具有高度的遗传保守性,以孟德尔方式遗传。因此利用微卫星DNA多态性连锁分析,可在同一家系中筛选出ADPKD患者。目前用于ADPKD连锁分析的STR基本为(CA)n重复,与PKD1连锁的微卫星DNA有SM6,KG8,Cw2和CW4等。由于ADPKD基因的复杂性和突变位点的难确定性,连锁分析仍为目前ADPKD基因诊断的主要手段。但是目前所用的这些STR基因座中并非都是最优选择,有些STR基因座难于进行基因型判定。例如:二核苷酸重复PCR扩增时出现滑行,检测不稳定,容易发生错判;SW6基因座的扩增片段容易产生杂带,并与其他基因座不同的扩增片段由于构象的影响可能会导致它们具有相同的电泳迁移率,变得难以区分;CW2离PKD1基因较远,可能会由于基因重组而导致误诊;实际检测中可能会出现一些STR基因座扩增失败的情况影响检测结果的判读,等等。为了解决这些问题,我们需要筛选出更多离PKD1基因距离近,遗传距离小,重组率低,杂合度高的有信息的STR位点作为遗传标记来进行常染色体显性遗传性多囊肾病的基因检测,以提高检测效率,为该疾病的临床诊断和胚胎植入前诊断提供依据。技术实现要素:本发明要解决目前PKD1基因的STR位点由于存在离PKD1基因距离较远、重组率高、杂合度低等缺点致使检测效率不高的技术问题,提供具有更高分辨率的PKD1基因的STR新位点,该STR新位点用于PKD1基因的连锁遗传分析,能有效提高分型识别效率和准确性。此外,还需要提供一种用于PKD1基因连锁遗传分析的检测试剂盒。为了解决上述技术问题,本发明通过如下技术方案实现:在本发明的一个方面,提供了一种PKD1基因的STR位点,该STR位点具有SEQIDNO.1或SEQIDNO.2所示的核酸序列。在本发明的另一方面,还提供了一种上述PKD1基因的STR位点的应用,用于常染色体显性遗传性多囊肾病的胚胎植入前诊断或产前诊断。在本发明的另一方面,还提供了一种诊断常染色体显性遗传性多囊肾病的检测试剂盒,包含分别针对SEQIDNO.1和SEQIDNO.2所示STR位点的特异性引物对。优选的,还包含针对SEQIDNO.3~SEQIDNO.10中任意一个以上STR位点的特异性引物对。更优选的,还包含分别针对SEQIDNO.3~SEQIDNO.9所示STR位点的特异性引物对。在本发明的另一方面,还提供了一种用于PKD1基因STR位点连锁遗传分析的复合扩增系 统,包含:分别针对SEQIDNO.1~SEQIDNO.10中任意三个以上STR位点的特异性引物对。本发明SEQIDNO.1、SEQIDNO.2所示的两个新STR位点,其所在区域不存在CNV(拷贝数变异)情况,且该两个位点兼容性好,有利于与其它STR基因座位点组成高效的复合扩增系统。本发明的主要优点如下:1.新筛出的STR位点是利用现有最新的人类全基因组序列信息进行筛选获得,排除兼容性差、存在CNV或位于高度重复序列区等影响因素。2.筛选出的2个有关PKD1基因STR新位点,丰富了产前诊断或胚胎植入前诊断等相关应用领域STR基因座的选择范围。3.提供了分辨率更高的STR新位点,该STR新位点与已知的STR位点组成同一个体系检测相关遗传疾病,能更加高效、准确地得到分型数据。附图说明下面结合附图和具体实施方式对本发明作进一步详细的说明。图1是本发明实施例2多个STR位点的原始峰图。具体实施方式本发明筛选出PKD1基因中具有高分辨度的新STR基因座位点,该新STR位点结合一些现有STR基因座应用于常染色体显性遗传性多囊肾病(ADPKD)的分子研究和分子诊断领域,为该疾病的临床诊断和胚胎植入前诊断提供依据。实施例1筛选出2个PKD1基因的STR新位点1.STR的预测(1)使用生物公共数据库查阅整理STR信息,(数据库参照截止2011年11月之前NCBIhttp://www.ncbi.nlm.nih.gov/数据库),获得NCBI有报道STR信息;(2)生物信息学预测STR,得到重复单元2bp重复次数10次以上的基因座若干个;(3)比较这些STR位点在NCBI数据库中的三个基因组(refercese、HuRef、Celera)中的重复次数,获得重复数差异大的STR位点信息;(4)结合文献、生物数据库及生物信息学预测,根据实际STR的序列特征,筛选出2个生物信息学预测的STR,并将其命名为T16S0002、T16S0004。T16S0002的STR位点序列如SEQIDNO.1所示,T16S0004的STR位点序列如SEQIDNO.2所示。2.STR的检测利用荧光标记扩增产物长度多态分析方法检测所有预测的STR位点,引物设计采用primer3软件,设计时尽量避开目前数据库中已知的SNP多态位点和高度重复序列区域等特 殊序列。PCR产物稀释后取少量与内标标记混匀后直接上ABI3130xl进行毛细管电泳,数据文件用GeneMapper4.0(Appliedbiosystems)来分析;获得所筛选到的新的STR基因座的多态信息量(PIC)等信息。实施例2STR新位点样本验证将新筛选出来的2个STR基因座(T16S0002,T16S0004)与7个常用基因座位点D16S521(SEQIDNO.3),D16S3024(SEQIDNO.4),D16S3395(SEQIDNO.5),D16S291(SEQIDNO.6),D16S664(SEQIDNO.7),D16S418(SEQIDNO.8),Amelogenin(AmeloX:SEQIDNO.9;AmeloY:SEQIDNO.10)组合,采用两步法多重荧光PCR技术检测2个个体样本。具体实验操作步骤如下:(1)DNA样本准备在医院中对2个选取的样本个体采集血液;通过DNA提取试剂盒抽提得到DNA样本并各取1μl1%agarose电泳对其样本进行质量检查以及浓度估计,然后根据估计的浓度将样本稀释到工作浓度5-10ng/μl。(2)PCR反应各取1ul样本,进行PCR反应。PCR反应体系总体积为10微升(1μl10×PCR缓冲液(Qiagen公司),0.2μl25mmol氯化镁、1μl检测多个STR基因座的混合引物(引物序列见下表1)、0.8μl2.5mmoldNTP、0.04μlHotStarplusTaq酶(Qiagen公司),5.96μlddH2O。PCR反应条件设置如下:95℃10分钟;7个循环的Touchdown程序(94℃20秒,65℃-1℃/循环40秒和72℃2min),28个循环扩增(94℃20秒,65℃30秒和72℃2min),在72℃延伸2分钟,在60℃延伸60分钟,4℃保存。(3)加荧光反应各取1ul样本,进行PCR反应。PCR反应体系总体积为10微升(1μl10×PCR缓冲液(Qiagen公司),0.2μl25mmol氯化镁、1μl检测多个STR基因座的混合引物、0.8μl2.5mmoldNTP、0.04μlHotStarplusTaq酶(Qiagen公司),5.96μlddH2O。PCR反应条件设置如下:95℃5分钟;30个循环(94℃20秒,56℃40秒和72℃2min),在72℃延伸2分钟,4℃保存。表1PKD1基因9个基因座引物序列STR基因座引物序列荧光标记D16S664F:5’-CCATGGTGCCCGGTCATAAAT-3’(SEQIDNO.11)FAMR:5’-TTGCCATCCGATACTCATCGTTA-3’(SEQIDNO.12)AmeloF:5’-GCCCTGGGCTCTGTAAAGAATAGTG-3’(SEQIDNO.13)FAMR:5’-GTTTCTTGAGGCCAACCATCAGAGCTTA-3’(SEQIDNO.14)D16S291F:5’-GTTAGAGGCACTGAGGGGAGCA-3’(SEQIDNO.15)FAMR:5’-CTCCAAGTGTGGCCTGACAATG-3’(SEQIDNO.16)D16S3395F:5-CCAGCCAGAAGCCATAGTTTCTAAC-3’(SEQIDNO.17)VICR:5-GCCTCAAATCTTCCCTGGCAGT-3’(SEQIDNO.18)D16S521F:5-ATTTCTGCAAAGGCTAAAGGAAGGT-3’(SEQIDNO.19)VICR:5-TTTATTTGCACCCTGGAGAACCTCT-3’(SEQIDNO.20)D16S418F:5-GCTGGACAGACAGCCAGGAAAT-3’(SEQIDNO.21)NEDR:5-CACTGCGTCCATCCCTTAAGTACA-3’(SEQIDNO.22)T16S0002R:5-CAAGCCAGCACCGAGTACAGTG—3’(SEQIDNO.23)NEDR:5-CCCGACGCATTTCCAAAATATG-3’(SEQIDNO.24)D16S3024R:5-GGCTCCTGCAAGGGAGAATCTA-3’(SEQIDNO.25)PETR:5-CCTTGATGCTGACACAGCCTTG-3’(SEQIDNO.26)T16S0004R:5-TCTGTAATGGGCCCCAGATTGT-3’(SEQIDNO.27)PETR:5-CATCACGCCTGGCCTAAAACAT-3’(SEQIDNO.28)PCR反应完成后,将PCR扩增产物稀释20倍,取出1ul加入8.9ulHIDI和0.1ulLIZ,混匀后95℃5min,毛细管电泳上样;因STR位点检测引物带有荧光,通过毛细管电泳检测不同颜色荧光及不同大小的扩增片断,用荧光检测系统分析结果得出STR分型信息(如表格2所示)。结果如图1和表2-4所示。表2是PKD1基因STR位点分布情况。表2PKD1基因STR位点分布情况图1是两个样本多个STR位点检测的原始峰图,分别为样本1(上图)和样本2(下图)。图1中,“A”为FAM峰图,“B”为VIC峰图,“C”为NED峰图,“D”为PET峰图。图1中各STR位点信息说明见下表3。表3PKD1基因各STR位点PCR扩增片段大小由图1得到的STR检测结果见下表4。表49个STR基因座的检测结果由图1和表4可知,新筛选得到的2个STR位点在结果上表现出了较高的区分度,应用到胚胎植入前诊断、STR分型、亲权鉴定、个体识别等生物医学检测中,能有效提高分型识别效率和准确性,并且这一体系具有很高的个体识别率,对PKD1基因的连锁遗传分析有很强的实用性。以上所述实施例仅表达了本发明的实施方式,其描述较为具体和详细,但并不能因此而理解为对本发明专利范围的限制。应当指出的是,对于本领域的普通技术人员来说,在不脱离本发明构思的前提下,还可以做出若干变形和改进,这些都属于本发明的保护范围。因此,本发明专利的保护范围应以所附权利要求为准。当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1