增强发酵单胞菌细胞中D‑木糖和L‑阿拉伯糖的利用的制作方法
本申请涉及并要求2014年6月24日提交的美国专利申请序列号14/312725的权益,其全文以引用方式并入本文。
技术领域
本发明涉及微生物学和基因工程领域。更具体地,发酵单胞菌(Zymomonas)基因组中基因的失活改善了经工程化以利用D-木糖的发酵单胞菌菌株中D-木糖的利用率,并且改善了经工程化以利用D-木糖和L-阿拉伯糖两者的发酵单胞菌菌株中D-木糖和L-阿拉伯糖的利用率。
背景技术:
微生物的乙醇生产提供了化石燃料的替代性能源且因此成为当前研究的重要领域。希望生产乙醇以及其它产品的微生物以能够利用木糖和阿拉伯糖作为碳源,因为这些糖是水解木质纤维素材料中的主要戊糖,它们能够提供丰富可利用的碳底物源用于在发酵中的生物催化利用。
天然不利用D-木糖或L-阿拉伯糖的运动发酵单胞菌(Zymomonas mobilis)和其它产乙醇生物可经基因工程化以利用这些糖。为了提高D-木糖的利用,菌株经工程化以表达编码以下蛋白质的基因:1)D-木糖异构酶,其催化D-木糖转化成D-木酮糖;2)木酮糖激酶,其将D-木酮糖磷酸化以形成D-木酮糖5-磷酸;3)转酮醇酶;和4)转醛醇酶(US 5,514,583,US 6,566,107;Zhang等人(1995)Science 267:240-243)。为了提高阿拉伯糖的利用,已经引入了编码以下蛋白质的基因:1)使L-阿拉伯糖转化成L-核酮糖的L-阿拉伯糖异构酶,2)使L-核酮糖转化成L-核酮糖5-磷酸的L-核酮糖激酶,和3)使L-核酮糖-5-磷酸转化成D-木酮糖5-磷酸的L-核酮糖5-磷酸4-差向异构酶(US 5,843,760)。
在引入D-木糖利用途径基因之后,D-木糖的利用通常并非最佳的。经基因工程化以利用D-木糖的发酵单胞菌菌株适于在包含D-木糖的培养基上的生长,以获得D-木糖耗用改善的菌株(US 7,223,575和US 7,741,119)。改善D-木糖利用的发酵单胞菌基因组的另外基因修饰公开于US 2013-0157331和US 2013-0157332中。
仍然需要这样一种发酵单胞菌菌株:其特别是在包含水解的木质纤维素生物质的培养基中有效利用D-木糖和/或L-阿拉伯糖,并且也利用D-木糖和L-阿拉伯糖两者,以增强乙醇产量。
技术实现要素:
本发明提供经工程化以利用D-木糖的重组发酵单胞菌细胞,以及经工程化以利用D-木糖和L-阿拉伯糖两者并且另外使被鉴定为ZMO0353的发酵单胞菌基因组的基因座失活的重组发酵单胞菌细胞。
因此,本发明提供了一种利用D-木糖的重组发酵单胞菌细胞,所述重组发酵单胞菌细胞包含内源基因的至少一种基因修饰,所述内源基因包含与SEQ ID NO:1具有至少95%的核苷酸序列同一性的编码区,其中基因对功能性蛋白质的表达被破坏。
在一个实施方案中,利用D-木糖的重组发酵单胞菌细胞也是利用L-阿拉伯糖的细胞。
在另一个实施方案中,利用D-木糖或利用D-木糖和L-阿拉伯糖的细胞还包含以下项中的至少一项:
a)降低的葡萄糖-果糖氧化还原酶活性;
b)提高的核糖-5-磷酸异构酶(具有分类EC 5.3.1.6)表达活性;以及
c)编码多核苷酸磷酸化酶的内源基因序列中的至少一种基因修饰,所述基因修饰使编码区缩短,从而导致C-末端截短的蛋白质的表达。
在另一个实施方案中,本发明提供了一种用于生产乙醇的方法,所述方法包括:
a)提供上述重组细胞中的任一种;以及
b)在包含D-木糖和L-阿拉伯糖中至少一种的培养基中培养(a)的细胞,由此将D-木糖和L-阿拉伯糖中的至少一种转化成乙醇。
附图说明和序列描述
图1示出了D-木糖和L-阿拉伯糖利用的代谢途径和产生乙醇的图表,其中glf是指D-葡萄糖易化扩散转运蛋白。
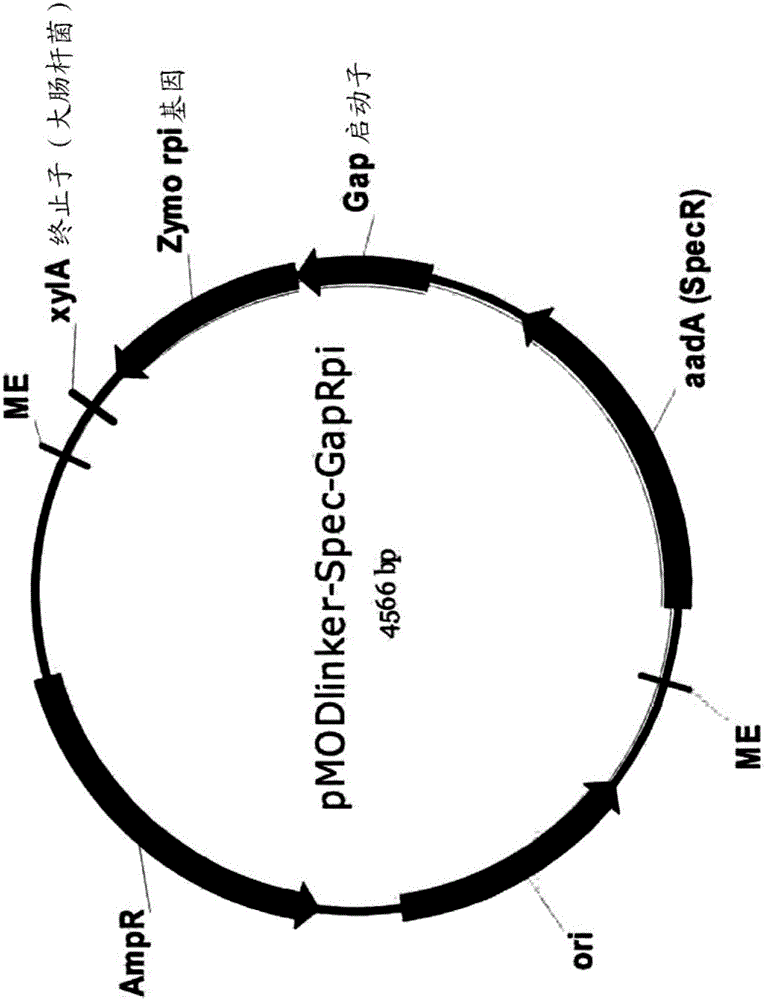
图2示出了pMODlinker-Spec-GapRpi的质粒图谱。
图3示出了p323del的质粒图谱。
图4示出了pARA3003的质粒图谱。
图5示出了pZBpnpIBAD的质粒图谱。
根据下面的详细描述以及构成本申请一部分的附带的序列描述可更充分地理解本发明。
下列序列符合37C.F.R.1.821-1.825(“对含有核酸序列和/或氨基酸序列公开的专利申请的要求-序列规则”)并且符合世界知识产权组织(WIPO)标准ST.25(2009)和EPO和PCT的序列表要求(规则5.2和49.5(a-bis),以及行政性指示的208节和附录C)。用于核苷酸和氨基酸序列数据的符号和格式遵循在37C.F.R.§1.822中示出的规定。
SEQ ID NO:1是运动发酵单胞菌ZM4的ZMO0353 ORF的核苷酸序列。
SEQ ID NO:2是运动发酵单胞菌运动亚种NCIMB11163的ZMO0353 ORF的核苷酸序列。
SEQ ID NO:3是运动发酵单胞菌运动亚种菌株CP4(也称为NRRL B-14023)的ZMO0353 ORF的核苷酸序列。
SEQ ID NO:4是运动发酵单胞菌运动亚种菌株NRRL B-12526的ZMO0353 ORF的核苷酸序列。
SEQ ID NO:5是运动发酵单胞菌运动亚种ATCC 29191的ZMO0353 ORF的核苷酸序列。
SEQ ID NO:6是运动发酵单胞菌运动亚种ATCC 10988的ZMO0353 ORF的核苷酸序列。
SEQ ID NO:7是来自运动发酵单胞菌的野生型GFOR编码区的完整核苷酸序列。
SEQ ID NO:8是来自运动发酵单胞菌ZM4的RPI的氨基酸序列。
SEQ ID NO:9是来自大肠杆菌(E.coli)的RPI的氨基酸序列。
SEQ ID NO:10是来自大肠杆菌的编码RPI的核苷酸序列。
SEQ ID NO:11是运动发酵单胞菌ZM4的ZMO0976编码区的核苷酸序列。
SEQ ID NO:12是由运动发酵单胞菌ZM4的ZMO0976编码区编码的蛋白质的氨基酸序列。
SEQ ID NO:13是来自运动发酵单胞菌菌株ZM4的pnp编码区的核苷酸序列。
SEQ ID NO:14是来自运动发酵单胞菌菌株ZM4的pnp编码的多核苷酸磷酸化酶的氨基酸序列。
SEQ ID NO:15是来自运动发酵单胞菌菌株NCIMB 11163的pnp编码区的核苷酸序列。
SEQ ID NO:16是来自运动发酵单胞菌菌株NCIMB 11163的pnp编码的多核苷酸磷酸化酶的氨基酸序列。
SEQ ID NO:17是来自运动发酵单胞菌菌株ATCC 10988的pnp编码区的核苷酸序列。
SEQ ID NO:18是来自运动发酵单胞菌菌株ATCC 10988的pnp编码的多核苷酸磷酸化酶的氨基酸序列。
SEQ ID NO:19是来自运动发酵单胞菌pomaceae ATCC 29192的pnp编码区的核苷酸序列。
SEQ ID NO:20是来自运动发酵单胞菌菌株ATCC 29192的pnp编码的多核苷酸磷酸化酶的氨基酸序列。
SEQ ID NO:21是菌株I的经修饰的pnp编码的融合蛋白的氨基酸序列,其具有709个天然的N-末端氨基酸和14个附加的C-末端氨基酸。
SEQ ID NO:22是具有695个天然的N-末端氨基酸和2个附加的C-末端氨基酸的经修饰的pnp编码的融合蛋白的氨基酸序列。
SEQ ID NO:23是具有368个天然的N-末端氨基酸和10个附加的C-末端氨基酸的经修饰的pnp编码的融合蛋白的氨基酸序列。
SEQ ID NO:24是具有32个天然的N-末端氨基酸和17个附加的C-末端氨基酸的经修饰的pnp编码的融合蛋白的氨基酸序列。
SEQ ID NO:25和26分别是大肠杆菌araA基因的氨基酸序列和编码区。
SEQ ID NO:27和28分别是大肠杆菌araB基因的氨基酸序列和编码区。
SEQ ID NO:29和30分别是大肠杆菌araD基因的氨基酸序列和编码区。
SEQ ID NO:31是超级GAP启动子(也称为PgapS)的核苷酸序列。
SEQ ID NO:32是RPI表达盒的核苷酸序列。
SEQ ID NO:33是命名为p323del的质粒的核苷酸序列。
SEQ ID NO:34-36和40-43是引物。
SEQ ID NO:37是1,318bp PNP-L片段的核苷酸序列。
SEQ ID NO:38是1,225bp PNP-R片段的核苷酸序列。
SEQ ID NO:39是包含嵌合Pgap-araBAD操纵子的SpeI-FseI DNA片段。
SEQ ID NO:44是来自菌株I的2340bp PCR-扩增的嵌合DNA分子的核苷酸序列,所述菌株I包含菌株I基因组的转座子间断的pnp基因区的一部分(包括ME,其导致融合到Pgap-Rpi表达盒的pnp基因的3’端附近的移框,这在菌株I中导致截短的pnp蛋白质)。
具体实施方式
可使用下列定义阐释权利要求书和说明书:
如本文所使用,术语“包含”、“包含的”、“包括”、“包括的”、“具有”、“具有的”、“含有”或“含有的”,或者它们的任何其它变型旨在涵盖非排他性的包括。例如,包含要素列表的组合物、混合物、工艺、方法、制品或装置不必仅限于那些要素,而可以包括其它未明确列出的要素,或此类组合物、混合物、工艺、方法、制品或装置固有的要素。此外,除非明确指明相反,“或”是指包含性的“或”而非排他性的“或”。例如,条件A或B满足下列中的任一项:A为真实的(或存在的)且B为虚假的(或不存在的),A为虚假的(或不存在的)且B为真实的(或存在的),以及A和B均为真实的(或存在的)。
如本文所用,在本发明的要素或组分之前的不定冠词“一个”、“一种”无意于限制该要素或组分的示例(即出现)的数量。因此,“一个”、“一种”应理解为包括一个/种或至少一个/种,并且要素或组分的单数词语形式还包括复数,除非该数值明显意指单数。
如本文所用,术语“发明”或“本发明”是非限制性术语,并且不旨在意指本发明的任何单独实施方案,而是涵盖如本说明书和权利要求书所述的所有可能的实施方案。
如本文所用,用术语“约”修饰本发明的成分或反应物的量时是指数值量的变化,它们可能发生在例如,典型的测量和用于制备浓缩液或实际使用溶液的液体处理程序中;这些程序中的偶然误差中;制造、来源、或用于制备组合物或实施方法的成分的纯度的差异中等。术语“约”还涵盖因特定初始混合物所得的组合物的不同平衡条件而不同的量。无论是否由术语“约”来修饰,权利要求书包括量的等同量。在一个实施方案中,术语“约”指在报告数值的10%范围内,优选地在报告数值5%范围内。
“基因”指表达特定蛋白质或功能性RNA分子的核酸片段,其可任选地包括位于编码序列之前的调控序列(5′非编码序列)和之后的调控序列(3′非编码序列)。“天然基因”或“野生型基因”指具有其自身调控序列的天然存在的基因。“嵌合基因”指不是天然基因的任何基因,包含在天然情况下不是一起存在的调控序列和编码序列。因此,嵌合基因可包括源于不同来源的调控序列和编码序列,或者包括源于同一来源但以不同于天然存在的方式排列的调控序列和编码序列。“内源基因”是指在生物基因组中处于其天然位置的天然基因。“外来基因”指正常情况下不存在于宿主生物中的基因,但是它通过基因转移引入到宿主生物中。外来基因可包含插入到非天然生物内的天然基因,或嵌合基因。
“启动子”或“启动控制区”指能够控制编码序列或功能RNA的表达的DNA序列。一般来讲,编码序列位于启动子序列的3′端。启动子可整个源于天然基因,或者由源于天然存在的不同启动子的不同元件构成,或者甚至包含合成的DNA片段。本领域内的技术人员应当理解,不同的启动子可以在不同的组织或细胞类型中,或者在不同的发育阶段,或者响应不同的环境条件而引导基因的表达。通常将在一种细胞类型中、在大多数情况下引起基因表达的启动子称为“组成型启动子”。
如本文所使用,术语“表达”指衍生自基因的编码(mRNA)或功能性RNA的转录和稳定积聚。表达也可指将mRNA翻译成多肽。“过表达”指在转基因生物中产生的基因产物超出在正常生物或未转化生物中产生的基因产物的水平。
如本文所用,术语“转化”指将核酸片段转移至宿主生物内,导致在基因上稳定遗传。所转移的核酸可以是宿主细胞中保留的质粒形式,或者某些转移的核酸可以被整合到所述宿主细胞基因组中。含有转移的核酸片段的宿主生物被称为“转基因”或“重组”或“转化的”生物或“转化体”。
如本文所使用,术语“质粒”和“载体”是指通常携带有不属于细胞中心代谢的部分的基因的染色体外元件,并且通常是环状双链DNA分子的形式。这类元件可以是源自任何来源的自主复制序列、基因组整合序列、噬菌体或单链或双链DNA或RNA的核苷酸序列(线性或环状),其中多个核苷酸序列已连接或重组进入独特构建体中,该独特构建体能够将所选基因产物的启动子片段和DNA序列与适当的3′非翻译序列一起引入细胞中。
术语“可操作地连接”指单核酸片段上的核酸序列的关联,使得其中一个核酸序列的功能受到另一个核酸序列的影响。例如,当启动子能够影响编码序列的表达(即,所述编码序列受到启动子的转录控制)时,则该启动子与该编码序列可操作地连接。编码序列可以按有义或反义的取向可操作地连接至调控序列。
术语“选择性标记”指一种鉴定因子,通常是抗生素或化学药品抗性基因,该因子能基于标记基因的效应,即,对抗生素的抗性进行选择,其中所述效应用于追踪遗传的受关注核酸和/或用于鉴定遗传了受关注核酸的细胞或生物。
如本文所用,术语“密码子简并性”指允许核苷酸序列在不影响所编码的多肽的氨基酸序列的情况下发生变化的遗传密码的性质。技术人员将充分意识到:特定宿主细胞在使用核苷酸密码子以确定给定氨基酸时所表现出的“密码子偏好性”。因此,当合成基因用以改善在宿主细胞中的表达时,可能希望对基因进行设计,使得其密码子使用频率接近该宿主细胞优选的密码子使用频率。
术语“密码子优化的”在其涉及用于转化不同宿主的核酸分子的基因或编码区时,是指在不改变多肽序列的情况下,改变核酸分子的基因或编码区中的密码子以使DNA编码的多肽的产生最优化。
术语“在D-木糖上生长适应的”是指在包含D-木糖的培养基中长时间生长之后分离的细胞或菌株。适应可包括在包含D-木糖和D-葡萄糖的培养基中生长一段时间,然后在仅包含D-木糖的培养基中生长一段时间,每种培养基为含D-木糖的培养基。通常,所述长时间的生长是至少约四天。
术语“适应菌株”指为了改善其利用特定碳源生产产品的能力已经被选择在该碳源上生长的微生物。例如“阿拉伯糖适应菌株”是已经选择在作为碳源的阿拉伯糖上生长的微生物菌株。
术语“木糖代谢途径”或“木糖利用代谢途径”是指将D-木糖代谢为D-果糖-6-磷酸和/或甘油醛-3-磷酸的一系列(由基因编码的)酶,并且包括1)D-木糖异构酶,其催化D-木糖至D-木酮糖的转化;2)木酮糖激酶,其使D-木酮糖磷酸化以形成D-木酮糖-5-磷酸;3)转酮醇酶;和4)转醛醇酶。
术语“D-木糖异构酶”或“木糖异构酶”是指催化D-木糖和D-木酮糖的互变的酶。已知分类为EC 5.3.1.5的酶为木糖异构酶(XI)。一些具有木糖异构酶活性的酶可催化除D-木糖和D-木酮糖的互变之外的其它反应,并且可基于它们的其它活性分类。
术语“木糖”是指D-木糖。
术语“阿拉伯糖”是指L-阿拉伯糖。
术语“核糖-5-磷酸异构酶”或“RPI”是指催化D-核酮糖5-磷酸和D-核糖5-磷酸的互变的酶。已知分类为EC 5.3.1.6的酶为核糖-5-磷酸异构酶。一些具有核糖-5-磷酸异构酶活性的酶可催化除D-核酮糖5-磷酸和D-核糖5-磷酸的互变之外的其它反应,并且可基于它们的其它活性分类。
术语“L-阿拉伯糖异构酶”是指催化L-阿拉伯糖和L-核酮糖互变的酶。已知分类为EC 5.3.1.4的酶为L-阿拉伯糖异构酶。一些具有L-阿拉伯糖异构酶活性的酶可催化除L-阿拉伯糖和L-核酮糖的互变之外的其它反应,并且可基于它们的其它活性分类。
术语“L-核酮糖激酶”是指催化L-核酮糖转化成L-核酮糖5-磷酸的酶。已知分类为EC 2.7.1.16的酶为L-核酮糖激酶。一些具有L-核酮糖激酶活性的酶可催化除L-核酮糖转化成L-L-核酮糖5-磷酸之外的其它反应,并且可基于它们的其它活性分类。
术语“L-核酮糖-5-磷酸4-差向异构酶”是指催化L-核酮糖5-磷酸和D-木酮糖5-磷酸互变的酶。已知分类为EC 5.1.3.22的酶为L-核酮糖-5磷酸4-差向异构酶。一些具有L-核酮糖-5磷酸4-差向异构酶活性的酶可催化除L-核酮糖5-磷酸和D-木酮糖5-磷酸的互变之外的其它反应,并且可基于它们的其它活性分类。
术语“碳底物”或“可发酵碳底物”指能够被微生物代谢的碳源。碳底物的一种类型是“可发酵糖”,它是指能够在发酵过程中被微生物用作碳源的寡糖和单糖。
术语“木质纤维素”是指包含木质素和纤维素两者的组合物。木质纤维素材料也可包含半纤维素。
术语“纤维素”是指包含纤维素和附加组分(包括半纤维素)的组合物。
术语“糖化”是指由多糖产生可发酵糖。
术语“经预处理的生物质”是指已经经受过热、物理和/或化学处理,以提高糖化酶对该生物质中的多糖的可用性的生物质。
“生物质”指任何纤维素或木质纤维素材料,包括包含纤维素的材料,并且任选地还包含半纤维素、木质素、淀粉、低聚糖和/或单糖的材料。生物质也可包含附加组分诸如蛋白质和/或脂质。生物质可源自单一来源,或者生物质可包括源自多于一种来源的混合物;例如,生物质可包括玉米棒和玉米秸秆的混合物,或草和叶片的混合物。生物质包括但不限于:生物能作物、农业残余物、市政固体垃圾、工业固体垃圾、来自造纸业的淤渣、庭院垃圾、木材和林业垃圾。生物质的示例包括但不限于玉米棒、作物残余物如玉米壳、玉米秸秆、草、小麦、小麦秸秆、大麦秸秆、干草、稻秆、柳枝稷、废纸、甘蔗渣、高粱、得自谷物研磨物的组分、树、枝、根、叶、木屑、锯末、灌木及灌丛、蔬菜、水果、花和动物粪肥。
“生物质水解产物”指来源于生物质糖化的产物。生物质也可在糖化前进行预处理或预加工。
术语“异源”指非天然存在于受关注的位置。例如异源基因指宿主生物中非天然存在的但是通过转基因被引入到宿主生物中的基因。例如,存在于嵌合基因中的异源核酸分子是与其它嵌合基因片段相关的非天然存在核酸分子,诸如具有彼此非天然相关的编码区和启动子片段的核酸分子。
如本文所用,“分离的核酸分子”是RNA或DNA的聚合物,它是单链或双链的,任选地包含合成的、非天然的或改变的核苷酸碱基。DNA聚合物形式的分离的核酸分子可由cDNA、基因组DNA或合成DNA的一个或多个区段构成。
如本领域所熟知的,术语“百分比同一性”是两种或更多种多肽序列之间或两种或更多种多核苷酸序列之间的关系,该关系通过对序列进行比较而确定。在本领域中,“同一性”还表示多肽或多核苷酸序列之间序列关联的程度,根据具体情况,它由这些序列的序列串之间的匹配程度确定。“同一性”和“相似性”可容易地通过已知方法计算出来,所述的方法包括但不限于以下文献中所描述的那些:1.)Computational Molecular Biology(Lesk,A.M.编辑)Oxford Universitv:NY(1988);2.)Biocomputing:Informatics and Genome Proiects(Smith,D.W.编辑)Academic:NY(1993);3.)Computer Analysis of Sequence Data,部分I(Griffin,A.M.和Griffin,H.G.编辑)Humania:NJ(1994);4.)Sequence Analysis in Molecular Biology(von Heinje,G.编辑)Academic(1987);和5.)Sequence Analysis Primer(Gribskov,M.和Devereux,J.编辑)Stockton:NY(1991)。
设计确定同一性的优选方法来给出待测试序列之间的最佳匹配。确定同一性和相似性的方法在可公开获得的计算机程序中被编成了代码。序列比对和百分比同一性计算可以用LASERGENE生物信息学计算软件包(DNASTAR Inc.,Madison,WI)中的MegAlign程序进行。
序列的多重比对使用“Clustal比对方法”进行,该方法涵盖若干个不同的算法,包括对应于称为Clustal V比对方法的“Clustal V比对方法”(在Higgins和Sharp,CABIOS.5:151-153(1989);Higgins,D.G.等人,Comput.Appl.Bioscl.,8:189-191(1992)中有所描述)并可见于LASERGENE生物信息学计算软件包(DNASTAR Inc.)的MegAlign v8.0程序中。对于多重比对,默认值对应于GAP PENALTY=10,GAP LENGTH PENALTY=10。使用Clustal方法进行蛋白质序列的逐对比对和百分比同一性计算的默认参数是KTUPLE=1,GAP PENALTY=3,WINDOW=5,以及DIAGONALS SAVED=5。对于核酸,这些参数是KTUPLE=2,GAP PENALTY=5,WINDOW=4,以及DIAGONALS SAVED=4。在用Clustal V程序进行序列比对后,可以通过观察同一程序中的“序列距离”表来获得“百分比同一性”。
另外“Clustal W比对方法”是可用的并对应于称为Clustal W的比对方法(在Higgins和Sharp,CABIOS.5:151-153(1989);Higgins,D.G.等人,Comput.Appl.Biosci.8:189-191(1992);Thompson,J.D.等人,Nucleic Acid Research,22(22):4673-4680,1994中有所描述)并可见于LASERGENE生物信息学计算软件包(DNASTAR Inc.)的MegAlign v8.0程序中。用于多重比对的默认参数(规定为蛋白质/核酸(GAP PENALTY=10/15,GAP LENGTH PENALTY=0.2/6.66,Delay Divergen Seqs(%)=30/30,DNA Transition Weight=0.5,Protein Weight Matrix=Gonnet系列,DNA Weight Matrix=IUB)。在用Clustal W程序进行序列比对后,可以通过观察同一程序中的“序列距离”表来获得“百分比同一性”。
本领域的技术人员非常清楚,多种程度的序列同一性用于从其它物种中鉴定多肽,其中这类多肽具有相同或相似的功能或活性。百分比同一性的有用的示例包括但不限于:50%、55%、60%、65%、70%、75%、80%、85%、90%、或95%,或可被用于鉴定所关注的多肽的从50%至100%的任何整数百分比,例如50%、51%、52%、53%、54%、55%、56%、57%、58%、59%、60%、61%、62%、63%、64%、65%、66%、67%、68%、69%、70%、71%、72%、73%、74%、75%、76%、77%、78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%。合适的核酸片段还具有任何上述同一性,并且通常编码具有至少50个氨基酸,优选至少100个氨基酸,更优选至少125个氨基酸的多肽。
术语“序列分析软件”是指可用于分析核苷酸或氨基酸序列的任何计算机算法或软件程序。“序列分析软件”可商购获得或独立开发。典型的序列分析软件包括但不限于:1)GCG程序包(Wisconsin Package Version 9.0,Genetics Computer Group(GCG),Madison,WI);2)BLASTP,BLASTN,BLASTX(Altschul等人,J.Mol.Biol.,215:403-410(1990));3)DNASTAR(DNASTAR,Inc.Madison,WI);4)Vector(Life Technologies);5)Sequencher(Gene Codes Corporation,Ann Arbor,MI);和6)整合了Smith-Waterman算法的FASTA程序(W.R.Pearson,Comput.Methods Genome Res.,[Proc.Int.Symp.](1994),开会日期1992,111-20。编辑:Suhai、Sandor.Plenum:New York,NY)。在本申请的上下文中,应理解,在使用序列分析软件进行分析的情况中,除非另有指明,否则分析的结果将基于所提到的程序的“默认值”。如本文所用,“默认值”将意指软件第一次初始化时原始装载在该软件中的任何数值或者参数组。
本文所用的标准的重组DNA和分子克隆技术为本领域所熟知,并且描述干Sambrook,J.和Russell,D.,Molecular Cloning:A Laboratory Manual,第三版,Cold Spring Harbor Laboratory Press,Cold Spring Harbor,NY(2001);以及Silhavy,T.J.,Bennan,M.L.和Enquist,L.W.,Experiments with Gene Fusions,Cold Spring Harbor Laboratory Press,Cold Spring Harbor,NY(1984);以及Ausubel,F.M.等人,Short Protocols in Molecular Biology,第5版,Current Protocols,John Wilev and Sons,Inc.,N.Y.,2002。
本发明涉及D-木糖利用增强的发酵单胞菌细胞。所述细胞还可具有增强的L-阿拉伯糖利用。本发明的发酵单胞菌细胞可用于产生乙醇。
内源ZMO0353基因修饰
本发明涉及经工程化的发酵单胞菌菌株的细胞,其为利用D-木糖和L-阿拉伯糖中的至少一种并且具有包含与SEQ ID NO:1具有至少95%的核苷酸序列同一性的编码区的经修饰内源基因的重组细胞。SEQ ID NO:1的序列被标记为运动发酵单胞菌ZM4菌株(菌株ATCC 31821;基因组序列GenBank登录号AE008692(Seo等人,Nat.Biotechnol.23(1),63-68(2005))和NCBI参考:NC_006526.2)的基因组序列中的ZMO0353 ORF(开放阅读框)。本文发现,破坏功能性蛋白质表达的ZMO0353 ORF修饰改善了利用D-木糖、或D-木糖和L-阿拉伯糖的工程化发酵单胞菌菌株细胞中的D-木糖利用率。此外,在利用D-木糖和L-阿拉伯糖的工程化的发酵单胞菌菌株细胞中,阿拉伯糖的耗用也得到了改善。
ZMO0353编码区被注释为编码4-二磷酸胞苷-2C-甲基-D-赤藓醇合酶。然而,Follens等人((1999)J.of Bacteriology 181:2001-2007)发现与ZMO0353编码的蛋白质同源的酶具有核酮糖-5-磷酸还原酶和CDP-核糖醇焦磷酸化酶活性两者。该酶的特征还在于具有D-核酮糖磷酸还原活性(Zolli等人(2001)Biochemistry 40:5041-5048)。
运动发酵单胞菌的其它菌株的基因组各自具有等同于ZMO0535 ORF的内源基因。例如,运动发酵单胞菌运动亚种NCIMB11163具有与SEQ ID NO:1有99%同一性的ORF(SEQ ID NO:2),运动发酵单胞菌运动亚种菌株CP4(也称为NRRL B-14023)具有与SEQ ID NO:1有98%同一性的ORF(SEQ ID NO:3),运动发酵单胞菌运动亚种菌株NRRL B-12526具有与SEQ ID NO:1有98%同一性的ORF(SEQ ID NO:4),运动发酵单胞菌运动亚种ATCC 29191具有与SEQ ID NO:1有98%同一性的ORF(SEQ ID NO:5),并且运动发酵单胞菌运动亚种ATCC 10988具有与SEQ ID NO:1有97%同一性的ORF(SEQ ID NO:6)。因此,期望每种运动发酵单胞菌菌株具有与SEQ ID NO:1有至少约95%、96%、97%、98%、99%或100%核苷酸序列同一性的编码区,并且当本文提及到ZMO0353时包括这些编码区。在本发明细胞中,包含鉴定为ZMO0353的编码区的基因对功能性蛋白质的表达被破坏。
包含鉴定为ZMO0353的编码区的基因对功能性蛋白表达的破坏可通过本领域的技术人员已知的任何方法,例如影响其mRNA或蛋白质的表达、或所编码的蛋白质的功能或稳定性的方法来实现。遗传修饰可以是,例如,编码区或基因的其它区如启动子中的插入、缺失、或突变。方法包括但不限于:整个基因或其部分的缺失;向基因中(在启动子或编码区)插入DNA片段,使得所编码的蛋白质不能表达;向编码区中引入突变,突变的引入导致终止密码子的加入或移框,从而使功能性蛋白质不表达;以及向编码区中引入一个或多个突变以改变氨基酸,使得无功能的蛋白质被表达。本领域的技术人员利用已知的靶ZMO0353编码序列(例如SEQ ID NO:1)以及围绕该靶序列的运动发酵单胞菌DNA序列,诸如在运动发酵单胞菌全基因组序列中可用的序列(例如对于ZM4,GenBank登录AE008692)能轻易地实施所有这些方法。
特别适用于在包含靶基因的ZMO0353中创建基因修饰的方法为使基因组的编码序列缺失,如在本文实施例1和2中所举例说明的那样。构建包含位于邻近靶编码区处的基因组序列区的质粒。用质粒转化发酵单胞菌细胞,并且发生两个重组事件:第一个是整合整个质粒,并且第二个是缺失质粒序列和靶编码区。
运动发酵单胞菌中ZMO0353序列的缺失显示于本文实施例3中,以使利用D-木糖的运动发酵单胞菌细胞的D-木糖利用率相比于没有ZMO0353缺失且其它方面相同的细胞有所提高。当在以96.1g/L D-木糖起始的培养基中生长时,该培养基示出,在23小时之后D-木糖利用率提高13.2%,并且在46小时之后提高19.6%。在144小时实验的整个过程中,缺失菌株的D-木糖利用率保持大于未缺失的菌株,D-木糖利用率在144小时时提高3.3%。第二培养基示出木糖利用率提高的更多。
当使以0.05的OD600起始的细胞培养物(即具有ZMO0353缺失的利用D-木糖的运动发酵单胞菌细胞)在初始包含约96g/L的D-木糖的培养基中于33℃下摇动生长46小时时,所述细胞培养物耗用至少约56g/L的D-木糖。在各种实施方案中,具有内源基因的至少一种基因修饰的细胞培养物在这些条件下生长时耗用至少约56,58,60,62,64,66,68,70或更多的g/L D-木糖,所述内源基因包含与SEQ ID NO:1具有至少95%的核苷酸序列同一性的编码区,其中该基因对功能性蛋白质的表达被破坏。
运动发酵单胞菌中ZMO0353序列的缺失显示于本文实施例6中,以使利用D-木糖和L-阿拉伯糖的运动发酵单胞菌细胞中的D-木糖利用率和L-阿拉伯糖利用率提高。当在以约96.8g/L的D-木糖起始的培养基中生长时,与无缺失但其它方面相同的菌株的培养物相比,缺失菌株培养物中的D-木糖利用率在24小时之后平均高七倍以上并且在67小时之后高四倍以上。在48.8g/L L-阿拉伯糖中生长的培养物中,与无缺失但其它方面相同的菌株的培养物相比,培养物或缺失菌株中的L-阿拉伯糖利用率在24小时之后平均大两倍以上并且在67小时之后仍大50%以上。
在利用D-木糖或者利用D-木糖和L-阿拉伯糖的细胞中,D-木糖或L-阿拉伯糖的利用率的提高将根据诸如以下因素而变化:细胞中的其它基因修饰,生长培养基,培养条件,以及分析时间。与除无ZMO0353修饰之外相同的细胞相比,本发明细胞的木糖利用率提高至少约1%、5%、8%、10%、12%、20%、25%、50%、75%、100%、或更大。与除无ZMO0353修饰之外相同的细胞相比,本发明细胞的L-阿拉伯糖利用率提高至少约1%、5%、8%、10%、12%、20%、25%、50%、75%、100%、或更大。
当使以0.06的OD600起始的细胞培养物(即具有ZMO0353缺失的利用D-木糖和L-阿拉伯糖的运动发酵单胞菌细胞)在初始包含约49g/L的L-阿拉伯糖的培养基中于33℃下摇动生长43小时时,所述细胞培养物耗用至少约28g/L的L-阿拉伯糖。在各种实施方案中,具有内源基因的至少一种基因修饰的细胞培养物在这些条件下生长时耗用至少约28,30,32,34,36,38,40或更多的g/L L-阿拉伯糖,所述内源基因包含与SEQ ID NO:1具有至少95%的核苷酸序列同一性的编码区,其中该基因对功能性蛋白质的表达被破坏。
当使以0.06的OD600起始的细胞培养物(即具有ZMO0353缺失的利用D-木糖和L-阿拉伯糖的运动发酵单胞菌细胞)在初始包含约97g/L的D-木糖的培养基中于33℃下摇动生长43小时时,所述细胞培养物耗用至少约20g/L的D-木糖。在各种实施方案中,具有内源基因的至少一种基因修饰的细胞培养物在这些条件下生长时耗用至少约20,25,30,35,40,45,50,55,60,65,70,75,80,85或更多的g/L D-木糖,所述内源基因包含与SEQ ID NO:1具有至少95%的核苷酸序列同一性的编码区,其中该基因对功能性蛋白质的表达被破坏。
在各种实施方案中,细胞以上文给出的各自使用量的任意组合利用L-阿拉伯糖和D-木糖,诸如在以上给出的培养条件下至少约20g/L的D-木糖和至少约28g/L的L-阿拉伯糖。
此外,如本文实施例6中所示,ZMO0353缺失菌株中的乙醇产量也得到改善。在96.8g/L D-木糖中生长的情况下,与来自无缺失但其它方面相同的菌株的那些相比,乙醇产量与缺失菌株的培养物中所利用的木糖量的提高成比例地提高。乙醇产量的提高在2.4倍至6倍,以及更大之间变化。在48.8g/L L-阿拉伯糖中生长的情况下,与无缺失但其它方面相同的菌株的那些相比,乙醇产量也与缺失菌株的培养物中阿拉伯糖利用率的提高成比例地提高。乙醇产量的提高在18%至58%,以及更大之间变化。
本发明细胞的乙醇产量的提高将根据诸如以下因素而变化:细胞中的其它基因修饰,生长培养基,培养条件,以及分析的时间。与除无ZMO0353修饰之外相同的细胞相比,本发明细胞的乙醇产量提高至少约1%、5%、10%、15%、20%、25%、50%、75%、100%、200%、或更大。
利用D-木糖的发酵单胞菌
赋予利用D-木糖的能力的任何基因修饰可存在于本发明的利用D-木糖的重组发酵单胞菌细胞中。发酵单胞菌细胞天然地利用D-葡萄糖、D-果糖和/或蔗糖作为发酵底物生产乙醇,但D-木糖不被代谢。产乙醇的发酵单胞菌菌株,例如运动发酵单胞菌,已经工程化以将D-木糖发酵为乙醇。通常,四个编码区已被引入运动发酵单胞菌,用于表达参与D-木糖代谢的四种酶,以创建D-木糖利用代谢途径(参见图1),如US 5,514,583、US 5,712,133、US 6,566,107、WO 95/28476、Feldmann等人((1992)Appl Microbiol Biotechnol 38:354-361)和Zhang等人((1995)Science 267:240-243)中所述。这些酶包括D-木糖异构酶,该酶催化D-木糖转化成D-木酮糖;以及木酮糖激酶,该酶磷酸化D-木酮糖以形成D-木酮糖5-磷酸。另外还表达了戊糖磷酸途径中的两种酶,转酮醇酶和转醛醇酶,它们将5-磷酸木酮糖转化成联接戊糖代谢与Entner-Douderoff糖酵解途径的中间体,该途径使D-木糖代谢成乙醇(参见图1)。编码这些酶的DNA序列可从能够代谢D-木糖的多种微生物中的任何一种获得,诸如肠细菌和一些酵母以及真菌。编码区的来源可包括黄单胞菌(Xanthomonas)、克雷伯氏菌(Klebsiella)、埃希氏菌(Escherichia)、红细菌(Rhodobacter)、黄杆菌(Flavobacterium)、醋杆菌(Acetobacter)、葡糖杆菌(Gluconobacter)、根瘤菌(Rhizobium)、农杆菌(Agrobacterium)、沙门氏菌(Salmonella)、假单胞菌(Pseudomonads)和发酵单胞菌(Zymomonas)。
因为这些蛋白质及其编码区的序列为人们所熟知,可通过本领域的技术人员使用生物信息学基于酶活性、E.C.编号、和/或序列相似性,并且还可通过如下所述的用于L-阿拉伯糖利用途径基因的实验方法,在利用D-木糖的微生物(例如以上所列的那些)中容易地鉴定合适的蛋白质和编码区。
将编码DNA序列可操作地连接至导致在发酵单胞菌细胞中表达的启动子,诸如运动发酵单胞菌甘油醛-3-磷酸脱氢酶的启动子(GAP启动子)、运动发酵单胞菌烯醇酶的启动子(ENO启动子)、以及密苏里游动放线菌(Actinoplanes missouriensis)木糖异构酶编码基因的启动子(GI启动子,Pgi)。如US 7,989,206(该文献以引用方式并入本文)中所公开的具有提高的表达的突变体GAP启动子,对于在发酵单胞菌中表达也是有用的。编码区可从启动子开始单个表达,或者两个或更多个编码区可接合到一个操纵子中,从相同启动子开始一起表达。可将所得的嵌合基因引入发酵单胞菌细胞并保持在质粒上,或者使用例如同源重组、定点整合、或随机整合而整合进基因组中。
载体是本领域熟知的。对于在发酵单胞菌中的表达而言尤其有用的是兼而能在大肠杆菌和发酵单胞菌中复制的载体,例如在美国专利5,514,583中描述的pZB188。载体可包括在细胞中自主复制的质粒和携带载体整合进细胞基因组的质粒。用于DNA整合的质粒可包括转座子、与靶细胞基因组同源的核酸序列区、定点整合序列、或其它支持整合的序列。在同源重组中,侧翼于靶整合位点的DNA序列位于期望的嵌合基因和任选的选择性标记的边界,从而导致嵌合基因插入到靶基因组位点中。
经工程化以表达D-木糖利用代谢途径的菌株的示例包括CP4(pZB5)(US 5,514,583)、ATCC31821/pZB5(US 6,566,107)、8b(US 20030162271;Mohagheghi等人,(2004)Biotechnol.Lett.25;321-325)以及ZW658(ATTCC#PTA-7858),其具有衍生物ZW800、ZW801-4(US 7,741,119)和ZW705(US 8,247,208)。经工程化以表达D-木糖利用代谢途径的发酵单胞菌细胞还可在包含D-木糖的培养基适应,因为具有D-木糖利用代谢途径的工程化细胞可在包含D-木糖的培养基中适应一段时间之后展示出改善的D-木糖利用率。在含D-木糖的培养基上的适应描述于US 7,223,575和US 7,741,119中,上述文献以引用方式并入本文。
在一个实施方案中,利用D-木糖的发酵单胞菌细胞如上所述。在其它实施方案中,改善D-木糖利用率的一种或多种附加修饰存在于细胞中。可存在于利用D-木糖的发酵单胞菌细胞中的改善D-木糖利用率的附加修饰包括如下所述的那些。基因修饰可存在于降低葡萄糖-果糖氧化还原酶(GFOR)活性的细胞中,如US 7,741,119中所公开(该文献以引用方式并入本文)。降低GFOR的表达可通过本领域的技术人员已知的任何方法进行,例如在US 7,741,119中所述的那些和以下所述破坏包含ZMO0353编码区的基因的那些。运动发酵单胞菌的ZM4菌株中的GFOR编码区序列为SEQ ID NO:7。95%、96%、97%、98%或99%序列同一性的等价序列可存在于其它发酵单胞菌菌株中。围绕GFOR编码序列的DNA序列也可用于一些修饰方法(以下所述),例如可用于运动发酵单胞菌全基因组序列(GenBank登录#AE008692)的那些。
基因修饰可存在于提高核糖-5-磷酸异构酶(RPI)活性的细胞中,如美国专利公布2012-0156746所公开的(该文献以引用方式并入本文)。RPI表达的提高可通过本领域的技术人员已知的任何方法完成实现。例如,可进行修饰以提高内源RPI编码基因的表达,诸如利用活性高于天然启动子的启动子,或通过表达编码在发酵单胞菌中具有核糖-5-磷酸异构酶活性的任何蛋白质或多肽的异源基因。存在各自可被表达的两类核糖-5-磷酸异构酶,称为RPI-A和RPI-B,如在美国专利公布2012-0156746中所述。内源和异源的RPI的示例分别来自:具有SEQ ID NO:8的氨基酸序列的运动发酵单胞菌菌株ZM4,以及氨基酸和编码序列分别为SEQ ID NO:9和10的大肠杆菌。可被表达以提高RPI活性的附加序列包括在美国专利公布2012-0156746中鉴定的那些和本领域的技术人员通过BLAST分析所鉴定的那些。
基因修饰可存在于降低醛糖还原酶活性的细胞中,特别是在辅因子诸如NADPH(其被转化成NADP)或NADH(其被转化成NAD)存在下将木糖转化成木糖醇的酶。在该反应中利用NADPH的酶被称为NADPH依赖性的木糖还原酶并且可属于EC 1.1.1.21,如美国专利申请公布2013-0157332所公开的(该文献以引用方式并入本文)。该修饰允许包含木糖利用途径基因的细胞在仅包含D-木糖(作为碳源)的培养基中即时生长。此类基因修饰可在ZMO0976编码区(SEQ ID NO:11;编码运动发酵单胞菌ZM4菌株的SEQ ID NO:12的蛋白质(GenBank登录#AE008692))和/或其它推定的醛/酮基还原酶中的一个或多个编码区中进行,所述编码区被称为ZMO0976、ZMO1344、ZMO1673和ZMO1773。
另外,使用US 7,989,206和US 7,998,722(这些文献以引用方式并入本文)中所公开的突变的、高活性的启动子,可表达作为D-木糖利用代谢途径的部分被表达的D-木糖异构酶。在其中所公开的突变体启动子是运动发酵单胞菌甘油醛-3-磷酸脱氢酶基因的启动子。此外,作为D-木糖利用代谢途径的部分表达的D-木糖异构酶可为包括在按照EC 5.3.1.5鉴定的酶分类中的组I D-木糖异构酶,如US 8,623,623中所公开的。在利用D-木糖的细胞中表达的D-木糖异构酶可为组1 D-木糖异构酶,其在发酵单胞菌中具有优于组2 D-木糖异构酶的活性,如US 8,623,623中所公开的(该文献以引用方式并入本文)。
此外,本发明的细胞可具有编码多核苷酸磷酸化酶(pnp)的内源基因序列中的至少一种基因修饰,所述基因修饰使编码区缩短,从而导致C-末端截短的蛋白质的表达,如在美国专利申请2013-0157331中所公开的。该修饰可改善利用木糖的细胞中的木糖利用率。被鉴定为编码具有多核苷酸磷酸化酶或多核糖核苷酸核苷酸转移酶活性的蛋白质的发酵单胞菌的任何基因均可提供用于修饰的靶标内源pnp基因。运动发酵单胞菌菌株ZM4的pnp编码区具有SEQ ID NO:13的序列。来自其它发酵单胞菌菌株的已知内源pnp编码区具有与SEQ ID NO:1有99%同一性的序列(发酵单胞菌NCIMB 11163;SEQ ID NO:15),98%同一性的序列(发酵单胞菌ATCC 10988;SEQ ID NO:17),以及83%同一性的序列(运动发酵单胞菌pomaceae ATCC 29192;SEQ ID NO:19)。任何这些序列,或与这些序列中的任何一种具有至少约95%、96%、97%、98%、或99%的同一性,并且被鉴定为编码多核苷酸磷酸化酶或多核糖核苷酸核苷酸转移酶的任何序列,可被用作修饰的靶标。使用BLAST分析或其本领域的技术人员所熟知的其它序列比较分析,可鉴定另外的靶标内源pnp基因序列。
pnp编码区可被修饰,以缩短3′端的编码区,导致与天然地编码的蛋白质相比C-末端截短的蛋白质的表达。运动发酵单胞菌的天然编码的多核苷酸磷酸化酶是约748个氨基酸的蛋白质,其为任何的SEQ ID NO:14、16、18、20或与这些序列中的任何一种有至少约95%、96%、97%、98%、或99%的同一性,并被鉴定为多核苷酸磷酸化酶或多核糖核苷酸核苷酸转移酶的任何序列。从经修饰的pnp编码区表达的截短的蛋白质可保留由编码多核苷酸磷酸化酶的内源基因编码的N-末端氨基酸序列的至少约350并多至约710个氨基酸,它们是天然的N-末端氨基酸。可加入相邻于并在具有截短的天然编码区的框内的非天然氨基酸的附加编码序列,使得产生融合蛋白,如在美国专利申请2013-0157331中所公开的。融合蛋白的示例为SEQ ID NO:21-24。可通过本领域的技术人员已知的任何方法来进行修饰。通常实施靶向整合事件。包含基因修饰的内源pnp基因的发酵单胞菌菌株的示例为菌株I,其描述于本文实施例章节中。
利用阿拉伯糖的发酵单胞菌
赋予利用阿拉伯糖的能力的任何基因修饰可存在于本发明的利用D-木糖和L-阿拉伯糖的发酵单胞菌细胞中。发酵单胞菌细胞不能自然代谢阿拉伯糖。产乙醇的发酵单胞菌的菌株,例如运动发酵单胞菌,已经工程化以将阿拉伯糖发酵为乙醇。通常,三个异源编码区已被引入运动发酵单胞菌,用于表达参与阿拉伯糖代谢的酶,以创建阿拉伯糖利用代谢途径(参见图1),如US 5,843,760中所述,其以引用方式并入本文。所述酶包括使L-阿拉伯糖转化成L-核酮糖的L-阿拉伯糖异构酶、使L-核酮糖转化成L-核酮糖5-磷酸的L-核酮糖激酶、以及使L-核酮糖5-磷酸转化成D-木酮糖5-磷酸的L-核酮糖-5-磷酸4-差向异构酶。
编码这些酶的DNA序列可获取自能够代谢阿拉伯糖的任何微生物。编码区的来源包括克雷伯氏菌(Klebsiella)、埃希氏菌(Escherichia)、根瘤菌(Rhizobium)、农杆菌(Agrobacterium)、和沙门氏菌(Salmonella)。尤其有用的是L-阿拉伯糖异构酶的大肠杆菌编码区:araA编码区(编码区SEQ ID NO:25;蛋白质SEQ ID NO:26)、L-核酮糖激酶的大肠杆菌编码区:araB编码区(编码区SEQ ID NO:27;蛋白质SEQ ID NO:28)、和L-5-磷酸核酮糖-4-差向异构酶的大肠杆菌编码区:araD编码区(编码区SEQ ID NO:29;蛋白质SEQ ID NO:30)。因为这些蛋白质及其编码区的序列为人们所熟知,如在以上给出的序列中举例说明的,可通过本领域的技术人员使用生物信息学基于序列相似性,并且还可通过实验方法,在利用其它阿拉伯糖的微生物(例如以上所列的那些)中容易地鉴定另外合适的蛋白质和编码区。典型地,利用已知的L-阿拉伯糖异构酶、L-核酮糖激酶或L-核酮糖-5磷酸4-差向异构酶氨基酸序列,例如本文所提供的那些,对可公开获得的数据库的BLAST(参见上文)检索被用于鉴定可在本文的菌株中使用的具有相同功能的另外蛋白质和它们的编码序列。这些蛋白质可与SEQ ID NO:26、28或30的任意L-阿拉伯糖异构酶、L-核酮糖激酶、或L-核酮糖-5磷酸4-差向异构酶氨基酸序列分别有至少约80-85%、85%-90%、90%-95%或95%-99%序列同一性,同时具有L-阿拉伯糖异构酶、L-核酮糖激酶、或L-核酮糖-5磷酸4-差向异构酶的活性。同一性基于Clustal W比对方法,所述方法采用GAP PENALTY=10、GAP LENGTH PENALTY=0.1、以及Gonnet 250系列的蛋白质权重矩阵的默认参数。
除了使用蛋白或编码区序列以及生物信息学方法鉴定具有相同活性的附加蛋白质之外,本文所述序列或本领域所述的那些序列还可用于实验鉴定天然的其它同源物。例如本文所述的每个编码核酸片段可用于分离编码同源蛋白的基因。使用序列依赖性规程分离同源基因是本领域熟知的。序列依赖性规程的示例包括但不限于:1.)核酸杂交的方法;2.)DNA和RNA扩增方法,这可通过核酸扩增技术的多种用法来举例说明[如聚合酶链反应(PCR),Mullis等人,美国专利4,683,202;连接酶链反应(LCR),Tabor,S.等人,Proc.Acad.Sci.USA 82:1074(1985);或链置换扩增反应(SDA),Walker等人,Proc.Natl.Acad.Sci.U.S.A.,89:392(1992)];和3.)文库构建和互补筛选方法。
例如,与本文所述的已知L-阿拉伯糖异构酶、L-核酮糖激酶、或L-核酮糖-5磷酸4-差向异构酶编码序列相似的蛋白质或多肽的编码区,可以用本领域的技术人员所熟知的方法,通过将本发明的核酸片段的全部或部分用作DNA杂交探针来筛选来自任何目标生物的文库而被直接分离。基于所公开的核酸序列的特异性寡核苷酸探针,可通过本领域已知的方法(Maniatis,同上)设计和合成。而且,整个序列可用于通过技术人员已知的方法(如随机引物DNA标记、切口平移或末端标记技术)直接合成DNA探针,或使用可用的体外转录体系合成RNA探针。另外,可设计特异性引物并将其用于扩增部分(或全长)的本发明序列。所得的扩增产物可在扩增反应过程中直接标记或在扩增反应后标记,并被用作探针,以通过在适当严格度的条件下的杂交分离全长的DNA片段。
通常,在PCR类型的扩增技术中,引物具有不同的序列而且彼此之间不互补。取决于所需的检测条件,应该设计引物序列以提供既有效又可靠的靶核酸的复制。PCR引物设计方法是本领域常见且熟知的(Thein和Wallace,“The use of as specific hybridization probes in the Diagnosis of Genetic Disorders”,载于Human Genetic Diseases:A Practical Approach,K.E.Davis编辑,(1986),第33-50页,IRL:Herndon,VA;和Rychlik,W.,载于Methods in Molecular Biology,White,B.A.编辑,(1993)第15卷,第31-39页,PCR Protocols:Current Methods and Applications.Humania:Totowa,NJ)。
通常,可以将本文所述序列的两个短片段在聚合酶链反应规程中用于从DNA或RNA扩增编码同源基因的更长的核酸片段。聚合酶链反应也可以用克隆的核酸片段的文库进行,其中一个引物的序列源自本文所述核酸片段,并且另一个引物的序列利用编码微生物基因的mRNA前体的3′端的多腺苷酸片的存在。
另选地,第二个引物序列可以基于来源于克隆载体的序列。例如,技术人员能够按照RACE规程(Frohman等人,PNAS USA 85:8998(1988)),通过利用PCR扩增转录物中的一个单独位点和3′或5′端之间的区域的拷贝,生成cDNA。以3′和5′方向取向的引物可用本发明的序列设计。使用可商购获得的3′RACE或5′RACE体系(例如BRL,Gaithersburg,MD),能够分离特异性的3′或5′cDNA片段(Ohara等人,PNAS USA 86:5673(1989);Loh等人,Science 243:217(1989))。
另选地,可使用所关注编码序列作为杂交试剂用于鉴定同源物。核酸杂交试验的基本组成包括探针、怀疑含有所关注基因或基因片段的样本及特定的杂交方法。探针通常是与待检测核酸序列互补的单链核酸序列。探针与待检测的核酸序列是“可杂交的”。探针长度可从5个碱基至数万个碱基不等,这将取决于具体待完成的测试。通常约15个碱基至约30个碱基的探针长度是合适的。只需要探针分子的部分与待检测的核酸序列互补。另外,探针和靶序列之间不需要完全互补。杂交确实可以在并不完全互补的分子之间发生,结果是杂交区内的一定比率的碱基没有与正确的互补碱基配对。
阿拉伯糖利用途径编码区可操作地连接至启动子以在载体中制备嵌合基因,并且可用于转化发酵单胞菌细胞(全部均如上对木糖利用途径编码区所述的那样)。
此外,在从阿拉伯糖到木糖的生物合成途径中利用转酮醇酶和转醛醇酶活性(参见图1),这对于上述D-木糖利用途径是常见的。
经工程化以表达阿拉伯糖利用代谢途径的发酵单胞菌细胞还可在包含阿拉伯糖的培养基中适应,因为在包含阿拉伯糖的培养基中适应可改善一些具有阿拉伯糖利用代谢途径的工程化细胞的阿拉伯糖利用率。
利用阿拉伯糖的细胞还可例如通过表达araE基因的编码区来表达阿拉伯糖-质子同向转运体,以改善阿拉伯糖利用率,其公开于US 2011/0143408(该文献以引用方式并入本文)。
附加发酵单胞菌细胞修饰
在包含生物质水解产物的培养基中,改善生长和乙醇产量的附加修饰可存在于本发明的利用D-木糖、或利用D-木糖和L-阿拉伯糖的发酵单胞菌细胞中。发酵单胞菌细胞可能已适于在包含乙醇和乙酸铵的胁迫培养物中生长,如US 8,247,208中所公开的,该文献以引用方式并入本文。当使用包含纤维素类生物质水解产物的发酵培养基(其包含乙酸盐)时,具有改善的乙酸盐耐受性的这些发酵单胞菌菌株是尤其有用的。发酵单胞菌细胞可能已适于在水解产物培养基中连续流动培养,如US 8,476,048中所公开的,该文献以引用方式并入本文。发酵单胞菌细胞可具有ZMO1432开放阅读框(NCBI参考:NC_006526.2)的基因修饰,如公开于US 8,476,048,其赋予对生物质水解产物更高的耐受性。
用于乙醇制备的发酵
经工程化的发酵单胞菌细胞可用于发酵以产生乙醇,所述经工程化的发酵单胞菌细胞具有D-木糖利用途径或者具有D-木糖利用途径和L-阿拉伯糖利用途径,以及内源基因的至少一种基因修饰,所述内源基因包含与SEQ ID NO:1具有至少95%的核苷酸序列同一性的编码区,其中该基因对功能性蛋白质的表达被破坏。运动发酵单胞菌为天然产乙醇生物,并且通过发酵单胞菌细胞生产乙醇示于图1中。
例如,描述了通过本发明的运动发酵单胞菌细胞生产乙醇。为了产生乙醇,使重组运动发酵单胞菌细胞与包含糖混合物、或者作为唯一糖的D-木糖或L-阿拉伯糖中的任一种的培养基相接触。通常所述培养基包含包括L-阿拉伯糖、D-木糖、和D-葡萄糖的糖混合物。所述培养基也包含包括这些糖的生物质水解产物,它们来源于经处理的纤维素或木质纤维素生物质。
当混合的糖的浓度高至足以抑制生长时,所述培养基可包括山梨醇、甘露糖醇、或它们的混合物,如US 7,629,156中所公开的。半乳糖醇或核糖醇可代替或与山梨醇或甘露糖醇组合。运动发酵单胞菌细胞在培养基中生长,在培养基中发生发酵并且产生了乙醇。发酵在不补充空气、氧气、或其它气体(这可包括各种条件如厌氧、微氧、或微需氧发酵)的情况下运行至少约24小时,并且可运行30小时或更长时间。达到最大乙醇产量的时间是不确定的,取决于发酵条件。如果抑制剂存在于培养基中,通常需要更长的发酵时间。发酵可在介于约30℃和约37℃之间的温度和约4.5至约7.5的pH下进行。
可以在实验室规模的发酵罐中以及放大发酵中(在其中生产了商业规模量的乙醇),在包含包括D-木糖在内的混合糖培养基中培养本发明的运动发酵单胞菌细胞。当需要商业生产乙醇时,可使用多种培养方法。例如,从本发明的运动发酵单胞菌菌株中的大规模生产可以通过分批培养方法和连续培养方法进行。经典的分批培养方法是封闭系统,其中培养介质的组成在培养开始时设定并且在培养过程期间不进行人工改变。因此,在培养过程开始时,用所需的生物接种培养基,并且不向系统中添加任何物质可以允许发生生长或代谢活性。然而,通常来说,“分批”培养是指碳源的添加是成批的,但经常试图控制诸如pH和氧浓度之类的因素。在分批系统中,代谢产物和生物质组成持续改变直至培养结束时。在分批培养物内,细胞缓慢通过静态延滞期到达高速生长对数期,并最后达到稳定期,此时生长速率减缓或终止。如果不加以处理,稳定期中的细胞将最终死亡。在某些系统中对数期中的细胞通常负责最终产物或中间产物的批量生成。在其它系统中可以获得稳定或指数后期生成。
标准分批系统的一种变型是补料分批系统。分批补料培养方法也适用于本发明的运动发酵单胞菌细胞的培养,并且其包括典型的分批系统,不同的是底物随着培养进行以递增方式添加。在代谢产物往往抑制细胞的代谢作用以及其中期望培养基中具有有限量的底物时,补料分批系统是有用的。补料分批系统中实际底物浓度的测量是困难的,并因此根据可测量因素例如pH和废气如CO2的分压的改变来评估。分批和分批补料培养方法在本领域内是常用的且熟知的,并且示例可见于Biotechnology:A Textbook of Industrial Microbiology,Crueger,Crueger和Brock,第二版(1989)Sinauer Associates,Inc.,Sunderland,MA,或Deshpande,Mukund V.,Appl.Biochem.Biotechnol.,36,227,(1992),将其以引用方式并入本文。
乙醇的商业生产也可通过连续培养进行。连续培养是一种开放式系统,其中将设定好的培养基连续加入生物反应器里,并同时移出等量条件培养基用于加工。连续培养一般使细胞维持在其中细胞主要处于对数生长期的恒定高液相密度。另选地,连续培养可以用固定化细胞来进行,其中连续添加碳和营养素,且连续从细胞团块中取出有价值的产物、副产物或废弃物。细胞固定可使用范围广泛的固体载体进行,所述固体载体由本领域技术人员已知的天然材料和/或合成材料组成。
连续或半连续培养允许调节影响细胞生长或最终产物浓度的一种因素或任何数目的因素。例如,一种方法将维持限制性营养物质例如碳源或氮水平处于固定速率并允许所有其它参数适度。在其它系统中,影响生长的多个因素能够连续改变,而通过培养基浊度测量的细胞浓度保持恒定。连续系统努力维持稳态生长条件,且因此由于培养基取出导致的细胞丧失必须针对培养基中的细胞生长速度进行平衡。对于连续培养过程调节营养素和生长因子的方法以及用于使产物形成速度达到最大的技术是工业微生物学领域众所周知的,并且各种方法由Brock,同上所述。
尤其适用于乙醇生产的发酵方法如下所述。在不摇动的情况下,使期望的本发明运动发酵单胞菌细胞在约30℃至约37℃下再生生长,然后转移至具有半复合培养基的摇瓶中,在约30℃至约37℃下,在回旋摇动器中以约150rpm的转速摇动培养,然后将其转移到包含类似培养基的种子发酵罐中。如果需要,可能需要一系列种子发酵罐以产生期望的生物的量。使种子培养物在种子发酵罐中厌氧生长至期望的生物密度,然后将其转移到生产发酵罐中,其中的发酵参数经最优化用于乙醇生产。从种子罐转移到生产罐的典型种菌体积在约2%至约20%v/v的范围内。典型的发酵培养基包含大于50%培养基体积的生物质水解产物。培养基中可存在终浓度约10mM的山梨醇或甘露糖醇。使用苛性碱溶液(诸如氢氧化铵、氢氧化钾、或氢氧化钠)和硫酸或磷酸将发酵控制在pH 5.0-6.0。发酵罐的温度被控制在30℃-35℃。为了最小化泡沫,按需加入消泡剂(任何种类-硅氧烷基的、有机基的等)到罐中。
上述任何条件组和这些条件中在本领域熟知的附加变化是本发明重组发酵单胞菌细胞产生乙醇的合适条件。
实施例
本发明将在以下实施例中进一步阐述。应该理解,这些实施例尽管说明了本发明的优选实施方案,但仅是以例证的方式给出的。通过上述论述和这些实施例,本领域的技术人员可确定本发明的必要特征,并且在不脱离本发明的实质和范围的前提下,可对本发明进行各种改变和修改以适应多种用途和条件。
一般方法
缩写的含义如下:“kb”指千碱基,“bp”指碱基对,“nt”指核苷酸,“hr”指小时,“min”指分钟,“sec”指秒钟,“d”指天,“L”指升,“mL”指毫升,“μL”指微升,“μg”指微克,“ng”指纳克,“mM”指毫摩尔,“μM”指微摩尔,“nm”指纳米,“μmol”指微摩尔,“pmol”指皮摩尔,“Cm”指氯霉素,“Cmr”或“Cm-R”指氯霉素抗性,“Cms”指氯霉素敏感,“Spr”指奇放线菌素抗性,“Sps”指奇放线菌素敏感,“DCO”指双交换,“UTR”指非翻译区,“~”指约,“OD600”指600nm处的光密度。
除非另行指出,引物由Sigma(St.Luis,MO)合成。
运动发酵单胞菌的转化
首先通过从运动发酵单胞菌的冻存小瓶接种5mL MRMG5培养基的培养物来产生运动发酵单胞菌的感受态细胞。在33℃和125rpm摇动下,使5mL培养物生长约18h。同时,将5mL培养物稀释到100mL的MRM3G5培养基中。使该100mL培养物在33℃和125rpm摇动下生长直至OD600达到约0.4。同时,将培养物置于冰上30分钟,然后通过离心收获得到细胞。除去上清液,并且使所得的细胞沉淀物重悬于0℃的含10%(v/v)甘油的无菌水溶液中至20mL的终体积。通过离心再次收获得到细胞,并且使所得的细胞沉淀物重悬于0℃的含10%甘油的无菌水溶液中至5mL的终体积。通过离心再次收获得到细胞,并且使所得的细胞沉淀物重悬于0℃的含10%甘油的无菌水溶液中至0.5mL的终体积。在-80℃下单独地冷冻0.05mL的等分试样直至需要。
采用电穿孔将质粒DNA引入了运动发酵单胞菌细胞中,基本上如US 5,514,583中所述。简而言之,50-ul的转化反应包含在10%(v/v)甘油中的约1010个细胞/mL和1-2μg的从转化的大肠杆菌SCS110细胞分离的未甲基化质粒DNA。对照反应进行相同处理,但是不接受任何质粒DNA。电穿孔仪的设置是1.6kv/cm、200Ω和25μF,电击杯的间隙宽度是0.1cm。电穿孔之后,用MMG培养基稀释了转化反应,并使细胞在30℃恢复,然后将它们铺板在如所指示的有或没有抗生素的包含1.5%琼脂的MMG培养基(MMG琼脂平板)上。在厌氧腔室中于30-33℃孵育平板,直到菌落出现。更多的细节描述于实施例中。
培养基
MMG培养基:50g/L D-葡萄糖、10g/L酵母提取物、5g/L的胰蛋白胨、2.5g/L的(NH4)2SO4、0.2g/L K2HPO4和1mM MgSO4
MRM3:10g/L酵母提取物、2g/L KH2PO4、1g/L MgSO4.7H2O)
MRM3X10:具有100g/LD-木糖的MRM3
MRM3G5:具有50g/L D-葡萄糖的MRM3
MRM3G10:具有100g/L D-葡萄糖的MRM3
MRM3A10:具有100g/L L-阿拉伯糖的MRM3
MRM3A5:具有50g/L L-阿拉伯糖的MRM3
HPLC分析
以定时的间隔取出发酵样品,并且使用Waters HPLC系统(Alliance system,Waters Corp.,Milford,MA)分析乙醇和残余的糖;条件=0.6mL/min的0.01N H2SO4,注射体积=10μL,柱温=65℃,运行时间=30min,用折射率检测(维持于40℃)。HPLC柱购自BioRad(Aminex HPX-87H,BioRad Inc.,Hercules,CA)。通过折射率检测定量分析物并与已知的标准品进行比较。
运动发酵单胞菌菌株ZM4
运动发酵单胞菌菌株ZM4为ATCC#31821;ZW1为ZM4菌株的另一个名称。
GenBank登录号AE008692(Seo等人,Nat.Biotechnol.23(1),63-68(2005))和NCBI参考:NC_006526.2两者均是ZM4菌株的基因序列的参考。
运动发酵单胞菌菌株的构建
从野生型亲本菌株ZW1开始,构建利用D-木糖的重组菌株ZW801-4的详细描述,提供在US 7,741,119中(该文献以引用方式并入本文)。菌株ZW801-4来源于菌株ZW800,后者来源于菌株ZW658,全部如US 7,741,084中所述,该文献以引用方式并入本文。通过将包含编码区来自大肠杆菌基因的编码D-木糖异构酶(xylA)、木酮糖激酶(xylB)、转醛醇酶(tal)、和转酮醇酶(tkt)的四种利用D-木糖的基因的两个操纵子PgapxylAB和Pgaptaltkt,通过顺序转座事件整合进ZW1(菌株ZM4的重命名;ATCC#31821)的基因组,然后在含D-木糖的选择性培养基上适应以产生菌株X13L3(其被重命名为ZW641),构建了ZW658。ZW641在包含D-木糖的生长培养基上的进一步适应产生了ZW658,其在D-木糖中更好生长并且根据布达佩斯条约(Budapest Treaty)保藏为ATCC PTA-7858。如在共同拥有的US 7,989,206(其以引用方式并入本文)中所公开的,ZW658因表达xylA编码区的启动子(Pgap)中的点突变而具有更大的D-木糖异构酶活性。与ZW641中天然的Pgap(641GAP启动子)相比,该启动子(SEQ ID NO:31),本文中称为801GAP启动子或超级GAP启动子或PgapS,在SEQ ID NO:31的位置116具有“T”而不是“G”。PgapS具有的表达强度是运动发酵单胞菌中Pgap的3至4倍。
在ZW658中,使用宿主介导的双交换同源重组和作为选择性标记的奇放线菌素抗性使编码葡萄糖-果糖氧化还原酶的基因插入失活以产生菌株ZW800。使用Cre重组酶,通过位点特异性重组移除以loxP位点为边界的奇放线菌素抗性标记以产生菌株ZW801-4。
通过针对在包含乙酸铵的培养介质中于胁迫条件下的生长进行适应,由ZW804-1产生菌株ZW705,如US 8,247,208中所描述的,该专利以引用方式并入本文。
菌株I由菌株ZW801-4产生,如US 2013/0157331的实施例1和2中所述,该文献以引用方式并入本文。菌株I由包括运动发酵单胞菌RPI表达盒(Pgap-RPI;SEQ ID NO:32)和Specr-盒的pMODlinker-Spec-GapRpi产生的转座体(图2)的随机整合产生。通过DNA测序将RPI表达转座子在菌株I中的插入位点确定为在运动发酵单胞菌(基因组(GenBank登录号AE008692;Seo等人,Nat.Biotechnol.23(1),63-68(2005))的核苷酸543506和543507之间。插入区的测序显示,转座子的整合导致了编码多核糖核苷酸核苷酸转移酶的pnp基因的开放阅读框3’端的移框。所得的开放阅读框编码突变型蛋白质,该蛋白质缺失了天然蛋白质的最后39个氨基酸残基(保留了从N-末端起的709个氨基酸)并且在其C-末端具有14个新的氨基酸(SEQ ID NO:21)。发现菌株I在D-木糖培养基中比亲本菌株的生长更好。
实施例1
ZMO0353开放阅读框缺失的构建
将命名为p323del的质粒(SEQ ID NO:33)构建成有利于从所公布运动发酵单胞菌菌株ZM4基因序列(GenBank登录号AE008692;Seo等人,Nat.Biotechnol.23(1),63-68(2005))中运动发酵单胞菌基因组的命名为ZMO0353的开放阅读框(SEQ ID NO:1)的缺失。
p323del来源于pMODlinker-CM,其构建描述于美国专利申请公布20130157332的实施例3中,该文献以引用方式并入本文。将赋予氯霉素抗性的DNA片段(Cmr;Cmr编码区具有其相关启动子,得自可商购获得的质粒pACYC184(Boca Scientific,Boca Raton,FL))插入到pMOD-Linker-Spec质粒的NotI和PacI位点之间,其详述于US 7,989,206,置换赋予奇放线菌素抗性的DNA片段(Specr),以产生pMODlinker-CM。pMODlinker-CM具有位于两个与Tn5转座酶相互作用以形成转座体的嵌合端(ME)之间的侧接loxP的Cmr-盒。
质粒p323del的示意性显示于图3。标记为323-L的区(从355位至13580位)与位于邻近ZMO0353的5’端处的ZM4基因组序列的核苷酸350392-351395(GenBank登录号AE008692;Seo等人,Nat.Biotechnol.23(1),63-68(2005))相关联。标记为323-R的区(从1367位至2338位)与位于邻近ZMO0353的3’端处的ZM4基因组序列中的核苷酸352740-353711相关联。
p323del的2412至3625位核苷酸(命名为ZMO0976和CDS571)与ZM4基因组序列的992143-993230位核苷酸相关联。该序列包含ZM4基因组序列中的醛糖还原酶(命名为ZMO0976)的编码区以及包含推定的启动子的上游区。
3690至309位序列得自pMODlinker-CM质粒。用于构建质粒的限制性酶示于图3。
实施例2
ZMO0353开放阅读框缺失菌株的构建
利用D-木糖的运动发酵单胞菌菌株AR3 7-31采用p323del转化(实施例1)。菌株AR3 7-31来源于菌株ZW705(参见一般方法)。在菌株ZW705在恒浊器中生长之后,分离菌株AR3 7-31,如US 8,476,048中所述,该专利以引用方式并入本文;该菌株在其中也称为适应型7-31。在该连续流动培养装置中,乙酸铵和乙醇在水解产物培养基中的浓度随时间推移而增大。将AR3 7-31的整个基因组测序并且与ZW705基因组的序列相比较。发现菌株AR3 7-31在运动发酵单胞菌基因组(NCBI参考:NC_006526.2)的ZMO1432开放阅读框中具有基因修饰,其中ZMO1432注释为编码“镰孢菌酸抗性蛋白”。
菌株AR3 7-31的感受态细胞如一般方法中所述制备。采用实施例1所述的1μg的p323del质粒通过电穿孔转化感受态细胞。使转化的细胞重悬于1mL MMG培养基中并在33℃下孵育三小时。然后通过离心收获得到细胞,除去上清液,并且将细胞重悬于200μL MMG培养基中。将重悬的细胞铺到包含120μg/mL氯霉素(Biomyx)的两个MMG琼脂平板上(每个平板100μL)上。在用于维持厌氧性的包含Gas Pak(Mitsubishi chemicals)的密封箱中,将琼脂平板在33℃下孵育三天。挑取所观察的在平板上生长的三个氯霉素抗性菌落并在培养基相同的平板上再划线接种。在相同条件下将这些新平板再孵育三天。在氯霉素选择下生长的期间,在第一重组事件中,将整个质粒整合进基因组中。
在孵育三天之后,从这些平板挑取三个单菌落并在MMG平板上划线接种,并且在相同条件下孵育一天。在孵育一天之后,挑取三个划线中每者的单菌落并再次再划线接种到MMG平板上并且再孵育一天。同时,挑取三个划线中每者的单菌落并划线接种到单独的MRM3X10平板上。在孵育四天之后,从这些平板挑取细胞并划线接种到单独的MMG平板上,以得到完全分离的单菌落。在孵育一天之后,挑取单菌落并通过PCR进行筛选以确认ZMO0353开放阅读框的缺失。引物323-F1和323-R6(分别为SEQ ID NO:34和35)用于PCR反应。确认具有期望缺失的菌株被命名为AR3 D323。在缺乏氯霉素的培养基上的生长时期允许第二重组发生,即缺失整个质粒和ZMO0353序列。在包含作为碳源的D-木糖的培养基上生长利用初始质粒上醛糖还原酶基因的表达,以创建第二重组事件的选择。先前发现了醛糖还原酶对D-木糖利用率具有有害的影响,如公开于美国专利申请公布2013-0157332中。
实施例3
在包含D-葡萄糖和D-木糖的培养基中ZMO0353开放阅读框缺失菌株对D-木糖利用的改善
将实施例2中所述的AR3 D323菌株的碳水化合物消耗能力与亲本菌株AR3 7-31进行比较。从每种菌株(对于每种菌株标记为a和b)的冻存小瓶接种MRM3G5培养基的两个2mL培养物。在摇动(125rpm)和33℃下,将全部四个培养物孵育约20h。同时,将3mL的新鲜MRM3G5培养基添加至各个培养物中,并且将培养物在相同条件下再孵育5h。同时,测量每种培养物的OD600,向10mL的MRM3X10培养基中添加足够体积的每一者,以给出0.05的计算OD600。在33℃和摇动(125rpm)下,将10mL培养物孵育144h。同时移除样品(示于表1和2中),并且各个培养物中的D-木糖和乙醇的浓度通过如一般方法中所述的HPLC测定。
表1:MRM3X10培养物中的D-木糖利用率
表2:MRM3X10培养物中的乙醇产量
如表1和2所指出的那样,在这些条件下,AR3 D323菌株消耗D-木糖和产生乙醇比AR3 7-31菌株更快,这表明开放阅读框ZMO0353的缺失带来了优势。
实施例4
用于将利用阿拉伯糖的基因插入到pnp基因座中的质粒的构建
命名为pZBpnpIBAD的质粒基于质粒pZX6(SEQ ID NO:36)进行构建,其构建描述于美国专利申请公布20130157332的实施例2中(其以引用方式并入本文)。pZX6是双交换发酵单胞菌-大肠杆菌穿梭载体,其指导整合进运动发酵单胞菌基因组中编码多核苷酸磷酸化酶的内源pnp基因(命名为ZMO0549,GenBank登录号AE008692)中邻近pnp编码序列末端处,并且置换运动发酵单胞菌基因组中的pnp编码序列的片段(从nt-2,084至nt-2,188)。确保用你使用了相同的序列编码标记位置。从nt-767至nt-2,084的1,318bp PNP-L片段(SEQ ID NO:37)为pnp编码序列(SEQ ID NO:13)的片段,同时1,225bp PNP-R片段(SEQ ID NO:38)包括最后59bp(从nt-2189至nt-2247)的pnp编码序列和1,166bp的下游基因序列。pZX6包括:包含使得载体能够在发酵单胞菌细胞中复制的复制起点的2,582bp运动发酵单胞菌基因组DNA片段(Zymo DNA,在图4中),用于选择大肠杆菌或运动发酵单胞菌转化体的911bp氯霉素抗性标记(Cm-R),以及909-bp大肠杆菌复制起点(Ori)。
为了包括工程化的运动发酵单胞菌的基因以利用阿拉伯糖,将包含嵌合Pgap-araBAD操纵子的SpeI-FseI DNA片段(SEQ ID NO:39)插入SpeI和FseI位点之间的pZX6,以置换PgapT-tal-tkt操纵子和Peno-rpi-rpe操纵子,导致命名为pARA3003的12,311-bp DCO穿梭载体并且示于图4中。Pgap-araBAD操纵子描述于US 2011-0143408的实施例1中(其以引用方式并入本文)。SpeI-FseI片段与pARA354的SpeI-EcoRI片段(US 2011-0143408的图3中)相同,操纵子中增加了包括FseI位点3’的序列。SpeI-FseI DNA片段包含:305-bp运动发酵单胞菌Pgap(运动发酵单胞菌甘油醛-3-磷酸脱氢酶编码基因的启动子),大肠杆菌的1,701-bp araB编码区(编码L-核酮糖激酶;ECaraB),大肠杆菌的1,503-bp araA编码区(编码L-阿拉伯糖异构酶;ECaraA),大肠杆菌的696-bp araD编码区(编码L-核酮糖-5磷酸4-差向异构酶;ECaraD),和ECaraD基因的166-bp 3’UTR(ECaraD 3’UTR)。
为了产生pZBpnpIBAD(图5中所示),pARA3003中的SacI/SpeI片段用由重叠PCR产生的新SacI/SpeI片段置换。引物IRpi-F(SEQ ID NO:40)为正向引物,其具有SacI位点,其后为与用于运动发酵单胞菌菌株I基因组中所存在的侧接loxP的Specr-盒和Pgap-RPI表达盒的转座子插入位点(ME)的pnp基因上游结合的24nt(参见一般方法)。引物IRpi-o-R(SEQ ID NO:41)为反向引物。其最后20nt杂交相同ME位点的下游,存在于菌株I中的侧接lox的Specr-盒的仅上游,同时其16nt对应于驱动RPI在菌株I中表达的Pgap启动子的前16nt的反向互补序列。引物IRpi-F和IRpi-o-R用于从菌株I基因组DNA中PCR-扩增pnp基因区,以产生用于重叠PCR反应的一个DNA片段。
用于重叠PCR反应的另一个片段如下产生。引物IRpi-o-F(SEQ ID NO:42)为正向引物并且为引物IRpi-o-R的反向互补。引物IRpi-R(SEQ ID NO:43)为反向引物:其在其5’端包含SpeI位点,其后为仅杂交XylA终止子下游的24nt,该XylA终止子处于存在于菌株I的pnp基因座中的Pgap-RPI表达盒的3’端。IRpi-o-F和IRpi-R用于从菌株IPCR-扩增Pgap-RPI基因区。然后将上述两种PCR产物混合以仅使用引物IRpi-F和IRpi-R进行重叠PCR反应。所得的重叠PCR片段包含菌株I基因组的转座子-间断的pnp基因区的一部分(包括ME,其导致融合到Pgap-Rpi表达盒的pnp基因3’端附近移框,其导致菌株I中截短的pnp蛋白质(参见一般方法))。单独使用引物IRpi-F和IRpi-R从菌株I扩增的上述重叠PCR片段和初始片段之间唯一的显著差异在于重叠PCR产物中不存在侧接loxP的Specr-盒。然后将这种新的2340bp PCR-扩增的嵌合DNA分子(SEQ ID NO:44)用于构建如下所述的质粒pZBpnpIBAD。
pZBpnpIBAD构建的最终步骤是将上述重叠PCR反应产物的2.3kb SacI/SpeI片段与pARA3003的11kb SacI/SpeI片段连接。连接产物用于转化大肠杆菌菌株DH10B,并且在含25μg/mL氯霉素的LB琼脂平板上选择转化体。从Cmr菌落分离质粒DNA,并且通过测序验证该质粒的序列。
实施例5
利用D-木糖和阿拉伯糖、表达Rpi、具有pnp插入和ZMO0353缺失的运动发酵单胞菌菌株的构建
菌株AR3 7-31和AR3 D323 1的感受态细胞如一般方法中所述制备,并且采用实施例4所述的1μg的pZBpnpIBAD质粒通过电穿孔进行转化。通过在包含120μg/mL氯霉素的MMG琼脂板上铺板来选择转化体。AR3 7-31的转化体命名为“AR3BAD”并且AR3 D323 1的那些命名为“AR3D323BAD”。选择单菌落并在MMG氯霉素平板上再划线接种。从该平板选择八个单菌落并再划线接种到MRM3A10平板上。然后挑取菌落并再划线接种到MRM3A10平板上并孵育两天。这种在MRM3A10上重铺平板重复进行另外三次。在MRM3A10琼脂上铺板四次之后,AR3BAD和AR3D323BAD各自挑取四个菌落并划线接种到MRM3X10琼脂和MMG琼脂平板上。全部四个划线接种的AR3BAD细胞在MRM3X10琼脂上表现出极差的生长,而全部四个划线接种的AR3D323BAD细胞在24小时内表现出可见的生长。将得自MMG琼脂平板(在其上两者菌株均生长良好)的单菌落划线接种到包含120μg/mL氯霉素的MMG琼脂上,以证实pZBpnpIBAD质粒已与染色体重组,导致氯霉素抗性标记丧失。保存已确认氯霉素敏感的AR3BAD和AR3D323BAD的各自四个菌落以用于进一步评估。
实施例6
ZMO0353缺失菌株中L-阿拉伯糖和D-木糖消耗以及乙醇产量的提高
对实施例5中所述的AR3BAD和AR3D323BAD各自四个单菌落消耗不同碳水化合物的能力进行评估。在从冻存小瓶接种的MRM3G5培养基的2mL培养物中,使所有八个菌落生长约18小时。在33℃和125rpm摇动下,在15mL管中孵育培养物。在18h之后,测量OD600,并且转移足够的体积以接种MRM3A5或MRM3X10的10mL培养物,至计算的0.06最终OD600。在125rpm摇动和33℃下,孵育这些10mL培养物,并且在表3、4、5和6中所示的时间移除样品。各个培养物中主要碳水化合物(D-木糖或L-阿拉伯糖)和乙醇的浓度通过如一般方法中所述的HPLC进行测定。
表3:MRM3X10培养物中的D-木糖利用率
表4:MRM3X10培养物中的乙醇产量
表5:MRM3A5培养物中的阿拉伯糖利用率
表6:MRM3A5培养物中的乙醇产量
如表中所指出的那样,在这些条件下,AR3D323BAD菌株消耗L-阿拉伯糖或D-木糖比AR3BAD菌株更快,这表明在5-碳单糖的消耗速率上开放阅读框ZMO0353的缺失带来了优势。在D-木糖和L-阿拉伯糖培养基两者中,ΔZMO0353菌株中的乙醇产生也更快。
- 还没有人留言评论。精彩留言会获得点赞!