用于通过使用已缺失BST2基因功能的细胞系来促进病毒感染和增加病毒产生的方法与流程
本发明涉及通过使用缺失Bst2基因功能的细胞系来促进病毒感染和病毒产生的方法且更具体地涉及从能产生病毒的细胞去除Bst2基因由此促进病毒从细胞的释放、抑制细胞的凋亡、促进对细胞的病毒感染和增加细胞中的病毒产生的方法。
背景技术:
当病毒感染身体时,在受感染的细胞中发生病毒增殖且所增殖的病毒颗粒经细胞膜出芽以感染其它周围的细胞。在细胞针对病毒感染的一些防御机制中,Bst2(tetherin)蛋白在细胞膜中表达。Bst2蛋白在N末端和C末端均具有膜结合位点且由此在细胞膜中以中间抬起(像一座桥)的形式表达。Bst2的C末端位于细胞膜的脂筏区即其中发生受体激活的特定区域且N末端位于非脂筏区。当病毒从脂筏区出芽至细胞外时,Bst2蛋白通过其固定于非脂筏区的区域来抑制病毒颗粒通过细胞膜。对于哺乳动物细胞的这种防御机制,一些病毒产生促进Bst2蛋白降解的蛋白从而逃避宿主细胞的此种机制。这反过来表明Bst2基因的功能强效地促进对病毒产生的抑制。
迄今,为了产生病毒以生产疫苗,已使用将病毒种接种至受精的鸡蛋中并培养接种的病毒的方法(KR 2012-0103737A)。然而,这种方法由于一些问题而非常低效,所述问题包括引起过敏、受精的鸡蛋的供应安全性和病毒传播。在尝试克服此类问题的过程中已使用通过动物细胞培养来生产病毒疫苗的方法(KR 2012-0033334A)。这些方法包括通过在无菌条件下培养大量动物细胞并用病毒感染所培养的动物细胞来生产疫苗的方法、通过遗传工程方法仅产生使针对病原体的抗体得以产生的抗原的方法等。通过动物细胞培养来生产疫苗的方法的最大优点是可扩大生产规模。具体地,生产规模可根据用作疫苗生产原料的动物细胞的培养规模来按需扩大。
然而,尽管有这么多优点,但是通过动物细胞培养来生产疫苗不是容易实现的。这是因为每单位体积的产量是较低的。为了在使用动物细胞来生产疫苗时克服每单位体积的低产量,尝试了使用遗传工程技术来开发优异的宿主动物细胞系(Jang J.等人,Appl.Microbiol.Biotechnol 85:1509-1520,2010),但是此类尝试仍然处于不足的水平。因此,为了优化病毒产生,需要鉴定病毒产生细胞系、改善培养条件和改善感染条件。
在先前研究中,本发明人发现当能产生病毒的细胞系中的Bst2基因的功能丧失时,病毒从细胞系的释放得以促进,使得产生病毒的能力增加且宿主细胞的凋亡得以抑制,使得病毒产生细胞系的稳定性增加(WO 2014142433)。
因此,本发明人已进行了大量努力以找到用于通过提高用于使用动物细胞系生产疫苗的靶病毒的产量来产生病毒和产生病毒抗原性蛋白的最佳方法且由此已发现若去除动物细胞系中的Bst2蛋白,则会促进病毒从动物细胞系的释放,会抑制宿主细胞的凋亡,会显著促进宿主细胞的病毒感染并会增加靶病毒的产生,从而完成本发明。
技术实现要素:
技术问题
本发明的目的是提供用于促进靶病毒感染的方法,该方法包括用靶病毒感染缺失Bst2基因功能且能产生病毒的细胞系。
本发明的另一个目的是提供用于增加靶病毒产生的方法,该方法包括:用靶病毒感染缺失Bst2基因功能且能产生病毒的细胞系;及培养所感染的细胞系。
本发明的另一个目的是提供用于产生抗原性蛋白的方法,该方法包括:用靶病毒感染缺失Bst2基因功能且能产生病毒的细胞系;及培养所感染的细胞系。
本发明的另一个目的是提供用于生产针对病毒性疾病的疫苗的方法,该方法包括使用通过上述方法产生的病毒抗原性蛋白。
技术方案
为了实现上述目的,本发明提供用于促进靶病毒感染的方法,该方法包括用靶病毒感染缺失Bst2基因功能且能产生病毒的细胞系。
本发明还提供用于增加靶病毒产生的方法,该方法包括:用靶病毒感染缺失Bst2基因功能且能产生病毒的细胞系;及培养所感染的细胞系。
本发明还提供用于产生抗原性蛋白的方法,该方法包括以下步骤:用靶病毒感染缺失Bst2基因功能且能产生病毒的细胞系;及培养所感染的细胞系。
本发明还提供用于生产针对病毒性疾病的疫苗的方法,该方法包括使用通过上述方法产生的病毒抗原性蛋白。
附图说明
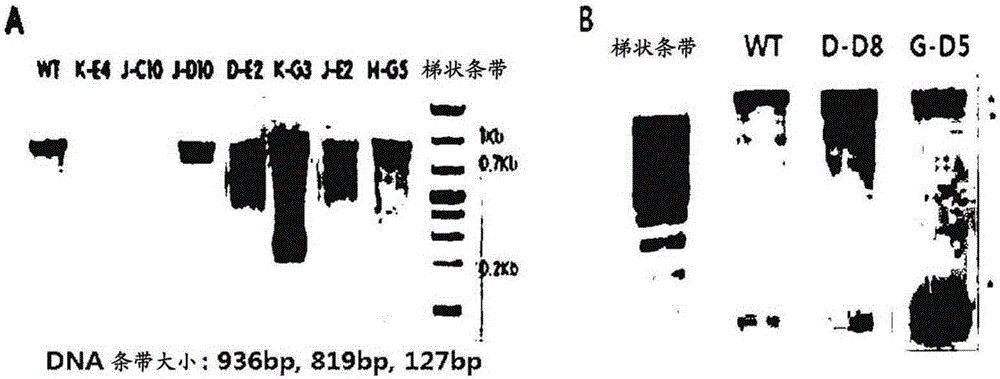
图1显示了先后通过PCR和电泳来鉴定缺失Bst2基因功能的细胞系(A:MDCK细胞系;B:Vero细胞系)中的突变的结果。
图2显示了在野生型细胞系和缺失Bst2基因功能的细胞系之间对感染性进行比较的结果,其以MOI的函数表示。
图3显示了在野生型细胞系和缺失Bst2基因功能的细胞系之间对感染性进行比较的结果,其以感染时间的函数表示。
图4显示了在野生型Vero细胞系和缺失Bst2基因功能的G-D5细胞系之间对胞内病毒抗原性蛋白在总细胞裂解物中的增加进行比较的结果。
图5显示了在野生型Vero细胞系和缺失Bst2基因功能的G-D5细胞系之间对每个培养孔和每个细胞的胞内病毒抗原性蛋白的增加进行比较的结果(A:每个培养孔的病毒抗原性蛋白;B:每个细胞的病毒抗原性蛋白)。
图6显示了在野生型MDCK细胞系和缺失Bst2基因功能的J-D10细胞系之间对胞内病毒抗原性蛋白在总细胞裂解物中和在每个细胞中的增加进行比较的结果(A:总细胞裂解物中的病毒抗原性蛋白;B:每个细胞的胞内病毒抗原性蛋白)。
图7显示了在缺失Bst2基因功能的人293T细胞系中对病毒抗原性蛋白的增加进行比较的结果。
具体实施方式
除另有定义外,本文使用的所有技术和科学术语具有与本发明所属领域技术人员通常理解相同的含义。通常,本文使用的命名法和下述实验方法是在本领域中公知和常用的那些。
本发明已发现当去除动物细胞系中的Bst2蛋白时,促进病毒从动物细胞系的释放,抑制宿主细胞的凋亡,显著促进宿主细胞的病毒感染且增加靶病毒和抗原性蛋白的产生。
根据本发明的方法产生的细胞系的特征在于与野生型宿主细胞相比,即使使用较少量的病毒和较短的感染时间,其也容易被病毒感染且其产量比野生型宿主细胞大的感染病毒的蛋白质。另外,根据本发明的方法产生的细胞系的特征在于与野生型细胞系相比,感染后在细胞系中产生的病毒较容易从细胞释放且感染病毒的细胞是针对凋亡具有抗性的可在延长的时段内产生病毒的稳定的细胞。因此,根据本发明用于促进靶病毒感染和增加靶蛋白产生的方法能增加病毒和抗原性蛋白的产量且由此可用于生产用于预防或治疗病毒性疾病的疫苗。
在一个方面,本发明涉及用于促进靶病毒感染的方法,该方法包括用靶病毒感染缺失Bst2基因功能且能产生病毒的细胞系。
在本发明中,用于促进病毒感染的方法增加用靶病毒感染的比例且由此能在不存在适于培养病毒的细胞系时用于开发针对病毒性疾病的新疫苗。
在一个方面,本发明涉及用于增加靶病毒产生的方法,该方法包括:用靶病毒感染缺失Bst2基因功能且能产生病毒的细胞系;及培养所感染的细胞系。
在本发明中,“增加靶蛋白的产生”是指增加病毒蛋白即抗原性蛋白的绝对产生。
在另一个方面,本发明涉及用于产生抗原性蛋白的方法,该方法包括:用靶病毒感染缺失Bst2基因功能且能产生病毒的细胞系;及培养所感染的细胞系。
在本发明中,用于促进靶病毒感染的方法和用于增加感染病毒产生的方法所具有的优点在于它们能增加抗原性蛋白的产生和使纯化抗原性蛋白的效率最大化。另外,根据本发明,通过病毒感染来抑制凋亡,从而可使抗原性蛋白产生中的细胞导入周期最小化且促进病毒颗粒释放到培养基中,从而还可增加抗原性蛋白的绝对产生。
本文使用的术语“能产生”是指病毒感染宿主细胞,然后在细胞中增殖,使得病毒释放到细胞外培养基中。
本文使用的术语“缺失功能”是指使细胞中Bst2基因的一个或多个核苷酸发生突变,由此促进病毒感染到宿主细胞中,增加所需病毒的产生,促进病毒释放到细胞外培养基中并抑制宿主细胞的凋亡。
本文使用的术语“促进感染”是指使细胞中Bst2基因的一个或多个核苷酸发生突变,由此使少量病毒能在短时间内浸润宿主细胞。
本文使用的术语“促进产生”是指使细胞中Bst2基因的一个或多个核苷酸发生突变,由此增加每个宿主细胞的病毒蛋白在细胞中的量。
本文使用的“抗原性蛋白”是指由感染病毒产生的抗原的总称。这些蛋白质参与病毒的毒力或抗原性且宿主细胞可识别该情况,这显示出对病毒感染的保护作用。流感病毒颗粒具有表面抗原,其被定义为16种(H1至H16)血凝素(HA)和9种(N1至N9)神经氨酸酶(NA)和S抗原(即主要被定义为核糖核蛋白NP抗原的抗原)并通过S抗原的血清学特异性分为A型、B型和C型。
本文使用的术语“凋亡”是指其中细胞主动消耗生物能ATP导致死亡的过程。凋亡包括细胞的收缩、DNA的规则切割和细胞膜的破碎,其在细胞由于异常细胞分裂、辐射、紫外线、细菌感染或病毒感染而不能维持其正常功能时被诱导。
本文使用的术语“病毒”是指可在人或动物中引起疾病的病毒。优选地,该术语是指流感病毒和疱疹病毒。更优选地,其是指选自P/H1N1、A/H1N1、A/H3N2和MHV68的一种病毒,但是不限于此。
在本发明中,病毒可为有包膜的病毒或裸病毒。在本文中,术语“裸病毒”是指仅由核酸、蛋白质或衣壳构成的病毒,而术语“有包膜的病毒”是指具有覆盖衣壳的包膜的病毒。
在本发明中,病毒可为DNA病毒或RNA病毒。在本文中,DNA病毒是具有DNA作为其遗传物质的病毒且其实例包括ssDNA、dsDNA(疱疹病毒)及环状DNA病毒。RNA病毒是具有RNA作为其遗传物质的病毒且其实例包括(-)ssRNA(流感病毒)、(+)ssRNA和dsRNA病毒。
在本发明中,能产生病毒的细胞系可不管其种类而使用,只要其用于开发用于预防或治疗病毒性疾病的疫苗。优选地,可使用动物细胞系。更优选地,细胞系可为选自以下的任何一种:293T人细胞系、MDCK、Vero和小鼠成纤维细胞,但是不限于此。
用于本发明的MDCK(Madin-Darby犬肾)细胞是来源于犬肾的上皮样细胞,而用于本发明的Vero细胞是来源于猴肾的细胞。这两种类型的细胞系对于肝炎病毒、牛痘病毒和流感病毒是易感的且由此是在病毒产生和研究中最常用的。
在本发明中,缺失Bst2基因功能且能产生病毒的细胞系优选如下获得:从Bst2基因的外显子1区删除一个或多个核苷酸或向Bst2基因的外显子1区插入一个或多个核苷酸,所述Bst2基因的外显子1区编码Bst2蛋白的胞质域,但是不限于此。
在本发明中,突变细胞系如下产生:从Bst2基因的外显子1区删除或向Bst2基因的外显子1区插入1-15个核苷酸,其如SEQ ID NO:1或2表示。
在本发明的一个实施例中,通过从MDCK细胞系的Bst2基因的外显子1区删除或向MDCK细胞系的Bst2基因的外显子1区插入一个或多个核苷酸来产生10个突变细胞系,其包含突变的等位基因且具有增加的产生病毒的能力,其如SEQ ID NO:1表示。
[SEQ ID NO:1]
ATGGCACCTACGCTTTACCACTACTACTGGCCTGTGCCCATAACTGACGAGTCAGAGTCAATGTCATCAAGTCAGAAGCTGAGCTGGCTGGAGTGGCTGGGCATCTTGGGGATCCCAGTGGTGATGGGTCTGTCTGTGGCTCTGATCATCTTTGTTGTCAAGACCAACAGCAAAGCCTGCGGGGATGGCCTCCTAGTAGAGCAGGAGTGTCACAATGTCACCAGCCTCCTGGAGCGCCAACTAACCCAAACCCGGCAAGCGTTACAGGGGACCATGGACCAGGCTACCACCTGCAACAAGACTGTG
在本发明的一个实施例中,通过从SEQ ID NO:1的位置45-54删除或向SEQ ID NO:1的位置45-54插入1-10个核苷酸来产生缺失Bst2基因功能的突变细胞系。这些突变细胞系可包括具有SEQ ID NO:3-8和SEQ ID NO:17-21中任何一个核苷酸序列的细胞系并优选地可为K-E4、J-C10、J-D10、D-E2、K-G3、J-E2、H-G5、8-D8、14-F11和12-B8。
[SEQ ID NO:3]
ATGGCACCTACGCTTTACCACTACTACTGGCCTGTGCCCATAACTGA--AGTCAGAGTCAATGTCATCAAGTCAGAAGCTGAGCTGGCTGGAGTGGCTGGGCATCTTGGGGATCCCAGTGGTGATGGGTCTGTCTGTGGCTCTGATCATCTTTGTTGTCAAGACCAACAGCAAAGCCTGCGGGGATGGCCTCCTAGTAGAGCAGGAGTGTCACAATGTCACCAGCCTCCTGGAGCGCCAACTAACCCAAACCCGGCAAGCGTTACAGGGGACCATGGACCAGGCTACCACCTGCAACAAGACTGTG
[SEQ ID NO:4]
ATGGCACCTACGCTTTACCACTACTACTGGCCTGTGCCCATAAC--ACGAGTCAGAGTCAATGTCATCAAGTCAGAAGCTGAGCTGGCTGGAGTGGCTGGGCATCTTGGGGATCCCAGTGGTGATGGGTCTGTCTGTGGCTCTGATCATCTTTGTTGTCAAGACCAACAGCAAAGCCTGCGGGGATGGCCTCCTAGTAGAGCAGGAGTGTCACAATGTCACCAGCCTCCTGGAGCGCCAACTAACCCAAACCCGGCAAGCGTTACAGGGGACCATGGACCAGGCTACCACCTGCAACAAGACTGTG
[SEQ ID NO:5]
ATGGCACCTACGCTTTACCACTACTACTGGCCTGTGCCCATAACT----AGTCAGAGTCAATGTCATCAAGTCAGAAGCTGAGCTGGCTGGAGTGGCTGGGCATCTTGGGGATCCCAGTGGTGATGGGTCTGTCTGTGGCTCTGATCATCTTTGTTGTCAAGACCAACAGCAAAGCCTGCGGGGATGGCCTCCTAGTAGAGCAGGAGTGTCACAATGTCACCAGCCTCCTGGAGCGCCAACTAACCCAAACCCGGCAAGCGTTACAGGGGACCATGGACCAGGCTACCACCTGCAACAAGACTGTG
[SEQ ID NO:6]
ATGGCACCTACGCTTTACCACTACTACTGGCCTGTGCCCATAACT---GAGTCAGAGTCAATGTCATCAAGTCAGAAGCTGAGCTGGCTGGAGTGGCTGGGCATCTTGGGGATCCCAGTGGTGATGGGTCTGTCTGTGGCTCTGATCATCTTTGTTGTCAAGACCAACAGCAAAGCCTGCGGGGATGGCCTCCTAGTAGAGCAGGAGTGTCACAATGTCACCAGCCTCCTGGAGCGCCAACTAACCCAAACCCGGCAAGCGTTACAGGGGACCATGGACCAGGCTACCACCTGCAACAAGACTGTG-3′
[SEQ ID NO:7]
ATGGCACCTACGCTTTACCACTACTACTGGCCTGTGCCCATAAC----GAGTCAGAGTCAATGTCATCAAGTCAGAAGCTGAGCTGGCTGGAGTGGCTGGGCATCTTGGGGATCCCAGTGGTGATGGGTCTGTCTGTGGCTCTGATCATCTTTGTTGTCAAGACCAACAGCAAAGCCTGCGGGGATGGCCTCCTAGTAGAGCAGGAGTGTCACAATGTCACCAGCCTCCTGGAGCGCCAACTAACCCAAACCCGGCAAGCGTTACAGGGGACCATGGACCAGGCTACCACCTGCAACAAGACTGTG
[SEQ ID NO:8]
ATGGCACCTACGCTTTACCACTACTACTGGCCTGTGCCCATAACTGACGACGAGTCAGAGTCAATGTCATCAAGTCAGAAGCTGAGCTGGCTGGAGTGGCTGGGCATCTTGGGGATCCCAGTGGTGATGGGTCTGTCTGTGGCTCTGATCATCTTTGTTGTCAAGACCAACAGCAAAGCCTGCGGGGATGGCCTCCTAGTAGAGCAGGAGTGTCACAATGTCACCAGCCTCCTGGAGCGCCAACTAACCCAAACCCGGCAAGCGTTACAGGGGACCATGGACCAGGCTACCACCTGCAACAAGACTGTG
[SEQ ID NO:17]
atggcacctacgctttaccactactactggcctgtgcccataactgacctatgagagtcagagtcaatgtcatcaagtcagaagctgagctggctggagtggctgggcatcttggggatcccagtggtgatgggtctgtctgtggctctgatcatctttgttgtcaagaccaacagcaaagcctgcggggatggcctcctagtagagcaggagtgtcacaatgtcaccagcctcctggagcgccaactaacccaaacccggcaagcgttacaggggaccatggaccaggctaccacctgcaacaagactgtggtgaccctgtcagcttccctggtgaaggagaaggcctggggtcaggagcagctcacccgaggagagaaacttcagggagagatcgagacattgaagcagcagctgcaggccgctttggaggaggtgaagcagctaagagaagggaaggaggcctcaagcaaggaaagggaaaccagctccgtcagctccttgaaggcaccccccggctccgtggtggtccccgtgtacctgctcctaggccttagggctctgctggcctga
[SEQ ID NO:18]
Atggcacctacgctttaccactactactggcctgtgcccataactga---gtcagagtcaatgtcatcaagtcagaagctgagctggctggagtggctgggcatcttggggatcccagtggtgatgggtctgtctgtggctctgatcatctttgttgtcaagaccaacagcaaagcctgcggggatggcctcctagtagagcaggagtgtcacaatgtcaccagcctcctggagcgccaactaacccaaacccggcaagcgttacaggggaccatggaccaggctaccacctgcaacaagactgtggtgaccctgtcagcttccctggtgaaggagaaggcctggggtcaggagcagctcacccgaggagagaaacttcagggagagatcgagacattgaagcagcagctgcaggccgctttggaggaggtgaagcagctaagagaagggaaggaggcctcaagcaaggaaagggaaaccagctccgtcagctccttgaaggcaccccccggctccgtggtggtccccgtgtacctgctcctaggccttagggctctgctggcctga
[SEQ ID NO:19]
Atggcacctacgctttaccactactactggcctgtgcccataactga-gagtcagagtcaatgtcatcaagtcagaagctgagctggctggagtggctgggcatcttggggatcccagtggtgatgggtctgtctgtggctctgatcatctttgttgtcaagaccaacagcaaagcctgcggggatggcctcctagtagagcaggagtgtcacaatgtcaccagcctcctggagcgccaactaacccaaacccggcaagcgttacaggggaccatggaccaggctaccacctgcaacaagactgtggtgaccctgtcagcttccctggtgaaggagaaggcctggggtcaggagcagctcacccgaggagagaaacttcagggagagatcgagacattgaagcagcagctgcaggccgctttggaggaggtgaagcagctaagagaagggaaggaggcctcaagcaaggaaagggaaaccagctccgtcagctccttgaaggcaccccccggctccgtggtggtccccgtgtacctgctcctaggccttagggctctgctggcctga
[SEQ ID NO:20]
Atggcacctacgctttaccactactactggcctgtgcccataa----------agagtcaatgtcatcaagtcagaagctgagctggctggagtggctgggcatcttggggatcccagtggtgatgggtctgtctgtggctctgatcatctttgttgtcaagaccaacagcaaagcctgcggggatggcctcctagtagagcaggagtgtcacaatgtcaccagcctcctggagcgccaactaacccaaacccggcaagcgttacaggggaccatggaccaggctaccacctgcaacaagactgtggtgaccctgtcagcttccctggtgaaggagaaggcctggggtcaggagcagctcacccgaggagagaaacttcagggagagatcgagacattgaagcagcagctgcaggccgctttggaggaggtgaagcagctaagagaagggaaggaggcctcaagcaaggaaagggaaaccagctccgtcagctccttgaaggcaccccccggctccgtggtggtccccgtgtacctgctcctaggccttagggctctgctggcctga
[SEQ ID NO:21]
Atggcacctacgctttaccactactactggcctgtgcccata---gacga---agagtcaatgtcatcaagtcagaagctgagctggctggagtggctgggcatcttggggatcccagtggtgatgggtctgtctgtggctctgatcatctttgttgtcaagaccaacagcaaagcctgcggggatggcctcctagtagagcaggagtgtcacaatgtcaccagcctcctggagcgccaactaacccaaacccggcaagcgttacaggggaccatggaccaggctaccacctgcaacaagactgtggtgaccctgtcagcttccctggtgaaggagaaggcctggggtcaggagcagctcacccgaggagagaaacttcagggagagatcgagacattgaagcagcagctgcaggccgctttggaggaggtgaagcagctaagagaagggaaggaggcctcaagcaaggaaagggaaaccagctccgtcagctccttgaaggcaccccccggctccgtggtggtccccgtgtacctgctcctaggccttagggctctgctggcctga
在本发明的一个实施例中,分析了所产生的突变细胞系的核苷酸序列且由此在来自MDCK细胞的K-E4、J-C10、J-D10和14-F11细胞的等位基因中可观察到移码突变的发生。
本文使用的术语“移码突变”是指删除或插入1、2或更多个核苷酸(除3的倍数外),其使能读取3核苷酸基因密码的移码发生变化。
在突变细胞系中,J-D10以登录号KCLRF-BP-00285保藏。
在本发明的另一个实施例中,通过从Vero细胞系的Bst2基因的外显子1区删除一个或多个核苷酸来产生2个突变细胞系,其包含突变的等位基因且具有增加的产生病毒的能力,其如SEQ ID NO:2表示。
[SEQ ID NO:2]
ATGGCACCTATTTTGTATGACTATTGCAAAATGCCCATGGATGACATTTGCAAGGAAGACAGGGACAAGTGCTGTAAACTGGCCGTAGGAATTCTGGGGCTCCTGGTCATAGTGCTTCTGGGGGTGCCCCTGATTTTCTTCATCATCAAGGCCAACAGCGAGGCCTGCCAGGATGGCCTCCGGGCAGTGATGGAGTGTCACAATGTCACCTATCTCCTGCAACAAGAGCTGGCCGAGGCCCAGCGGGGCTTTCGGGACGCAGAGGCCCAGGCTGTCACCTGCAACCAGACTGTG
在本发明的一个实施例中,通过从SEQ ID NO:2的位置116-130删除1-12个核苷酸来产生缺失Bst2基因功能的突变细胞系。这些突变细胞系可包括具有SEQ ID NO:9和10中任何一个核苷酸序列的细胞系且优选地可为D-D8和G-D5。
[SEQ ID NO:9]
ATGGCACCTATTTTGTATGACTATTGCAAAATGCCCATGGATGACATTTGCAAGGAAGACAGGGACAAGTGCTGTAAACTGGCCGTAGGAATTCTGGGGCTCCTGGTCATAGTGC------------CCCTGATTTTCTTCATCATCAAGGCCAACAGCGAGGCCTGCCAGGATGGCCTCCGGGCAGTGATGGAGTGTCACAATGTCACCTATCTCCTGCAACAAGAGCTGGCCGAGGCCCAGCGGGGCTTTCGGGACGCAGAGGCCCAGGCTGTCACCTGCAACCAGACTGTG
[SEQ ID NO:10]
ATGGCACCTATTTTGTATGACTATTGCAAAATGCCCATGGATGACATTTGCAAGGAAGACAGGGACAAGTGCTGTAAACTGGCCGTAGGAATTCTGGGGCTCCTGGTCATAGTGCTTCTGGGGGTGCCCTGATTTTCTTCATCATCAAGGCCAACAGCGAGGCCTGCCAGGATGGCCTCCGGGCAGTGATGGAGTGTCACAATGTCACCTATCTCCTGCAACAAGAGCTGGCCGAGGCCCAGCGGGGCTTTCGGGACGCAGAGGCCCAGGCTGTCACCTGCAACCAGACTGTG
在本发明的另一个实施例中,通过从293T人细胞系的Bst2基因的外显子1区删除或向293T人细胞系的Bst2基因的外显子1区插入一个或多个核苷酸来产生3个突变细胞系,其包含突变的等位基因且具有增加的产生病毒的能力,其如SEQ ID NO:24表示。
[SEQ ID NO:24]
CCTGAAAGGGGTTTGCATTTAAGCCCCTCTGTCCCCAGGACCTAGGGAGGAGGCCCAGGTCCCAGGGGCAGCAGCCAAACTCCCCAGGCCAAAACCCCAGATTCTAACTCTTCTTAGATGGCCCTAATGGCTTCCCTGGATGCAGAGAAGGCCCAAGGACAAAAGAAAGTGGAGGAGCTTGAGGGTGAGAAAGGGAGAAGGGAGAGGGCCGGGGAGGGGTGAGTCAGGTATGGAAGAGGGGGTGGGGGCAGGGAGACCAGGGCTGGAGGTTGGGGTAAGGGGGAGGTTCTGTCCCAGAGTGGAGCAGGGCCCCAGCATGGCCACATGCTGACCCGCCCCCTGTTTCTGTCCTCCCACCCTACCAGGAGAGATCACTACATTAAACCATAAGCTTCAGGACGCGTCTGCAGAGGTGGAGCGACTGAGGTCAGAGATAGCCTTCCCCCGCTACCCTCCACCTGCCACATTCCTCTCACCCCCACATCCCTAGCCCAAGACCCAGGATCTCCTTTGCTCCCAAAATCCCCATTGCCCCAAGGGATAAAGTTTGAGTCCCACAAAAGGATAACTTAGCCCCTAGGGTCACAGAGCCATGGGTGGCCGCTGTCCATTCCCTCCCCGGTGACTTGGATTGGGGCGGTGCGGGGGGAACTCCCGGGGGCGGTGGGCTTACAGGGAGGGCGGCAGGAGCCAGGACGAGCAGATGCCTGATTTGCCATCTGTACCGCAGAAGAGAAAACCAGGTCTTAAGCGTGAGAATCGCGGACAAGAAGTACTACCCCAGCTCCCAGGACTCCAGCTCCGCTGCGGCGCCCCAGCTGCTGATTGTGCTGCTGGGCCTCAGCGCTCTGCTGCAGTGA
在本发明的一个实施例中,通过从SEQ ID NO:24的位置51-100删除或向SEQ ID NO:24的位置51-100插入3-27个核苷酸来产生缺失Bst2基因功能的突变细胞系。这些突变细胞系可包括具有SEQ ID NO:25-30中任何一个核苷酸序列的细胞系且优选地可为D-19、B4和B24。
[SEQ ID NO:25]
ATGGCATCTACTTCGTATGACTATTGCAGAGTGCCCATGGAAGACGGGGA----------------------------GATAGGAATTC
[SEQ ID NO:26]
ATGGCATCTACTTCGTATGACTATTGCAGAGTGCCCATGGAAGACGGGGATAAGCGCTGTAAGCTTCTGCTGGGGATAGGAATTC------
[SEQ ID NO:27]
ATGGCATCTACTTCGTATGACTATTGCAGAGTGCCCATGGAAGACGGGGATAAGCGCTGTAAGCTTCtTGCTGGGGATAGGAATTC
[SEQ ID NO:28]
ATGGCATCTACTTCGTATGACTATTGCAGAGTGCCCATGGAAGACGGGGATAAGCGCTGTAAGC---------------------
[SEQ ID NO:29]
ATGGCATCTACTTCGTATGACTATTGCAGAGTGCCCATGGAAGACGGGGATAAGCGCTGTAAGCTTCTGCTGATAGGAATTC---
[SEQ ID NO:30]
ATGGCATCTACTTCGTATGACTATTGCAGAGTGCCCATGGAAGACGGGGATAAGCGCTGTAA--------TGGGGATAGGAATTC
在根据本发明使用缺失Bst2基因功能的细胞系来促进病毒感染和增加病毒产生的方法中,所使用的细胞数目可为1×104至1×107个,优选为5×104至5×106个,更优选为1×105至1×106个。
接种至细胞中的病毒优选在37℃在5%CO2温育箱中以0.0001-0.1的MOI进行感染,但是不限于此。可在新鲜培养基中将感染病毒培养36-72小时并可去除培养基。可在适当的条件下将剩余的细胞离心并可收集上清液并在4℃-15℃以1500-5000rpm离心15分钟,然后收集上清液。
在本发明的一个实施例中,病毒抗原性蛋白如下产生:用靶病毒感染缺失Bst2基因功能且能产生病毒的细胞系,然后培养以增加靶病毒的产生。病毒抗原性蛋白可用于生产用于预防或治疗病毒性疾病的疫苗。
因此,在另一个方面,本发明涉及用于生产针对病毒性疾病的疫苗的方法,该方法包括使用通过上述方法产生的病毒抗原性蛋白。
疫苗组合物还可含有药用辅料或赋形剂。可使用任何辅料,只要其能用于在将辅料注射到体内时促进抗体形成以实现本发明的目的。具体地,可用于本发明的辅料优选为选自铝(Al(OH)3或AlPO4)、角鲨烯、脱水山梨醇、聚山梨醇酯80、CpG、脂质体、胆固醇、单磷酰脂质(MPL)A和吡喃葡萄糖基脂A(GLA)中的至少一种,但是不限于此。
实施例
在下文中,将参考实施例进一步详细地描述本发明。对于本领域技术人员来说显而易见的是,这些实施例仅用于说明性目的,而不应该被解释为限制本发明的范围。
实施例1:缺失Bst2基因功能的细胞系
1-1:MDCK细胞系
将得自Moon-Jung Song教授(Korea University,Korea)的5×105个野生型MDCK细胞在DMEM完全(GIBCO)培养基(含有10%FBS、2mM L-谷氨酰胺、5Mβ-巯基乙醇、10μg/ml庆大霉素、50U/ml青霉素和50μg/ml链霉素)中培养24小时。然后向所培养的细胞添加能结合至Bst2基因外显子1区的一对TALEN载体(ToolGen,Korea)、一个报告载体(ToolGen,Korea)和Turbofect试剂的15μg混合物,然后将其在37℃和5%CO2的条件下温育48小时。通过FACS Aria(BD Bioscience)对所培养的细胞进行打分以选择500个对GFP和RFP均为阳性的细胞并将所选择的细胞接种于96孔板中,由此获得单克隆细胞。
为了确认所获得的单克隆细胞中的突变,提取基因组DNA并使用如SEQ ID NO:11表示的dBst2-F引物和如SEQ ID NO:12表示的dBst2-R引物在以下条件下进行聚合酶链式反应(PCR):95℃5分钟,然后25个循环,每个循环由94℃30秒、60℃30秒和72℃1分钟构成,然后72℃10分钟。将反应产物稀释至10-3并使用如SEQ ID NO:13表示的NdBst2-F引物和如SEQ ID NO:14表示的NdBst2-F引物在以下条件下进行PCR:95℃5分钟,然后25个循环,每个循环由94℃30秒、60℃30秒和72℃1分钟构成,然后72℃10分钟。
[SEQ ID NO:11]
5′-GGTCAGGATGGCTCCTATGC-3′
[SEQ ID NO:12]
5′-AACCTGACAGGGTCACCTGG-3′
[SEQ ID NO:13]
5′-GTAGCCCCAGCCAAAGGTTTC-3′
[SEQ ID NO:14]
5′-AGGCCTCCCCATGCCCAAAC-3′
反应产物的大小通过电泳来分析。结果显示在外显子1区中具有如SEQ ID NO:1表示的推定突变的细胞系具有正常的PCR反应产物大小及由于核酸内切酶切割而产生的两个额外条带,这说明在外显子1区中发生了突变(图1A)。
为了对被确认具有突变的细胞系的核苷酸序列进行分析,用T7E1酶(ToolGen,Korea)处理的PCR反应产物用Bgl-Xba进行消化,与pUC18载体连接,然后在含有氨苄西林和X-gal-IPTG的培养基中培养,由此获得白色群落。在所获得的白色群落中,对于每个克隆选择6个群落并测序。
结果显示在Bst2基因的外显子1区中具有核苷酸的删除或插入的7个突变细胞系(K-E4、J-C10、J-D10、D-E2、K-G3、J-E2和H-G5)中,3个突变细胞系(K-E4、J-C10和J-D10)具有发生在等位基因对中的移码突变。
1-2:Vero细胞系
将5×105个野生型VERO细胞(University of Southern California,Chengyu Liang)在DMEM完全(GIBCO)培养基(含有10%FBS、2mM L-谷氨酰胺、5Mβ-巯基乙醇、10μg/ml庆大霉素、50U/ml青霉素和50μg/ml链霉素)中培养24小时。然后向所培养的细胞添加能结合至Bst2基因外显子1区的一对TALEN载体(ToolGen,Korea)、一个报告载体(ToolGen,Korea)和Turbofect试剂(Thermo,USA)的15μg混合物,然后将其在37℃和5%CO2的条件下温育48小时。通过FACS Aria(BD Bioscience)对所培养的细胞进行打分以选择500个对GFP和RFP均为阳性的细胞并将所选择的细胞接种于96孔板中,由此获得单克隆细胞。
为了确认所获得的单克隆细胞中的突变,提取基因组DNA并使用如SEQ ID NO:11表示的dBst2-F引物和如SEQ ID NO:16表示的AGM-R引物在以下条件下进行聚合酶链式反应(PCR):95℃5分钟,然后25个循环,每个循环由94℃30秒、60℃30秒和72℃1分钟构成,然后72℃10分钟。将反应产物稀释至10-3并使用如SEQ ID NO:15表示的AGM-F引物和如SEQ ID NO:16表示的AGM-R引物在以下条件下进行PCR:95℃5分钟,然后25个循环,每个循环由94℃30秒、60℃30秒和72℃1分钟构成,然后72℃10分钟。
[SEQ ID NO:15]
5′-GAGGGGGAGATCTGGATGG-3′
[SEQ ID NO:16]
5′-CTTCTCTGCATCCAGGGAAG-3′
反应产物的大小通过电泳来分析。结果显示在外显子1区中具有推定突变的两个细胞系具有正常的PCR反应产物大小及由于核酸内切酶切割而产生的两个额外条带,这说明在外显子1区中发生了突变(图1B)。
对被确认具有突变的细胞系的核苷酸序列进行了分析且由此鉴定了在Bst2的外显子1区中具有核苷酸的删除或插入的2个突变细胞系(D-D8:杂合;及G-D5:敲除)。
1-3:成纤维细胞细胞系B2K
为了产生缺失Bst2的小鼠成纤维细胞细胞系(B2K),将100μg MCA(3-甲基胆蒽)注射至24周龄Bst2敲除小鼠(ISU Abxis,Korea)的大腿中。约4个月后,分离在大腿中形成的肿瘤并用70%乙醇洗涤以去除细菌,然后将其转移到含有10%FBS和RPMI培养基的皿中并使用外科剪刀切成细小碎块。将肿瘤碎块和13ml消化溶液(500单位/ml胶原酶IV、150单位/ml DNA酶I)置于50ml锥形管中并在振荡器中以250rpm和37℃振荡1小时30分钟。将经振荡的溶液用10ml移液管吹吸3-5次以使碎块进一步分散,然后通过尼龙网过滤并置于50ml新鲜锥形管中。然后将所得溶液以1250rpm离心5分钟且去除上清液,然后将团块混悬在新鲜培养基中,通过尼龙网过滤并在上述条件下离心。去除上清液后,对团块进行解离,以2.4×107个细胞/10cm皿的密度接种并培养以获得敲除肿瘤细胞系(B2K)。
同时,为了产生野生型肿瘤细胞系(MB19),将100μg MCA(3-甲基胆蒽)注射到24周龄B6野生型小鼠(Orient Bio,Korea)的大腿中。约4个月后,分离在大腿中形成的肿瘤并用70%乙醇洗涤以去除细菌,然后将其转移到含有10%FBS和RPMI培养基的皿中并使用外科剪刀切成细小碎块。将肿瘤碎块和13ml消化溶液(500单位/ml胶原酶IV、150单位/ml DNA酶I)置于50ml锥形管中并在振荡器中以250rpm和37℃振荡1小时30分钟。将经振荡的溶液用10ml移液管吹吸3-5次以使碎块进一步分散,然后通过尼龙网过滤并置于50ml新鲜锥形管中。然后将所得溶液以1250rpm离心5分钟且去除上清液,然后将团块混悬在新鲜培养基中,通过尼龙网过滤并在上述条件下离心。去除上清液后,对团块进行解离,以2.4×107个细胞/10cm皿的密度接种并培养以获得野生型肿瘤细胞系(MB19)。
将Bst2敲除肿瘤细胞系(B2K)和Bst2野生型肿瘤细胞系(MB19)各自的MHC类别(KbDb(R1.21.2)-APC、CD1d(1B1)-PE)、LFA-1(207)-Cyc和Bst2(e.Bio927)生物素+链霉亲和素-PE进行荧光染色并比较其表达水平。
结果显示MB19正常表达Bst2,而B2K肿瘤细胞系不表达Bst2。因此,在以下实施例中使用B2K肿瘤细胞系。
1-4:人293T细胞系
将5×105个人293T细胞在DMEM完全(GIBCO)培养基(含有10%FBS、2mM L-谷氨酰胺、5Mβ-巯基乙醇、10μg/ml庆大霉素、50U/ml青霉素和50μg/ml链霉素)中培养24小时。然后向所培养的细胞添加能结合至Bst2基因外显子1区的一个CRISPR载体(ToolGen,Korea)、一个报告载体(ToolGen,Korea)、Cas9载体和Fugene(Promega,USA)的18μg混合物,然后将其在37℃和5%CO2的条件下温育48小时。通过FACS Aria(BD Bioscience)对所培养的细胞进行打分以选择500个对GFP和RFP均为阳性的细胞并将所选择的细胞接种于96孔板中,由此获得单克隆细胞。
为了确认所获得的单克隆细胞中的突变,提取基因组DNA并使用如SEQ ID NO:22表示的HBst2-F1引物和如SEQ ID NO:23表示的HBst2-R1引物在以下条件下进行聚合酶链式反应(PCR):95℃3分钟,然后10个循环,每个循环由94℃30秒、72℃30秒和72℃45秒组成,然后25个循环,每个循环由95℃30秒、62℃30秒和72℃45秒组成。
[SEQ ID NO:22]
5’-TGG CAC GGC CTA GGC ACT CA-3’
[SEQ ID NO:23]
5’-GGA TAC TGA GGT CCC CGA TT-3’
反应产物的大小通过电泳来分析,然后分析了被确认具有突变的细胞系的核苷酸序列。由此鉴定了在Bst2的外显子1区中具有核苷酸的删除或插入的3个突变细胞系(D19等位基因1:删除27个核苷酸,D19等位基因2:删除6个核苷酸,B4等位基因1:插入1个核苷酸,B4等位基因2:删除21个核苷酸,B24等位基因1:删除3个核苷酸,B24等位基因2:删除8个核苷酸)。
实施例2:促进对缺失Bst2基因的细胞系的感染
2-1:用低MOI的病毒进行感染
为了证实促进对缺失Bst2基因功能的细胞系的感染的作用,比较了病毒对缺失Bst2功能的细胞系的感染与病毒对野生型MDCK细胞系的感染。在该实施例中,使用各自包含突变等位基因的8-D8、14-F11和12-B8,其是具有发生在Bst2的外显子1区中的移码突变从而以与在实施例1中获得的J-D10(登录号KCLRF-BP-00285)细胞系相同的方式使Bst2基因的表达完全沉默的细胞系。8-D8、14-F11和12-B8的突变序列如SEQ ID NO:17-21表示。具体地,8-D8包含SEQ ID NO:17和18的突变等位基因;14-F11包含SEQ ID NO:19和20的突变等位基因;且12-B8包含SEQ ID NO:18和21的突变等位基因。
每种类型的野生型MDCK细胞和缺失Bst2功能的细胞(5×104个细胞/孔)用已从0.001的MOD连续稀释2倍的A/H3N2流感病毒感染30分钟。然后去除培养基且用琼脂糖覆盖细胞层,然后使感染病毒生长48小时。
结果显示缺失Bst2功能的细胞系以与野生型细胞系相同的效率感染,即使感染缺失Bst2功能的细胞系的病毒颗粒的数目为感染野生型细胞系的病毒颗粒的数目的约1/5(图2)。这表明Bst2蛋白从宿主细胞的去除促进了病毒对宿主细胞的感染。
2-2:病毒感染时间的降低
每种类型的野生型MDCK细胞和缺失Bst2功能的细胞(5×104个细胞/孔)用MOI为0.0001的A/H3N2流感病毒感染1分钟、5分钟、10分钟、20分钟、30分钟和1小时。然后去除培养基且用琼脂糖覆盖细胞层,然后使感染病毒生长48小时。
结果显示缺失Bst2功能的细胞系中的斑块形成在1分钟时为3-6pfu/孔,这比野生型细胞系高1.8倍,在5分钟时为6-10pfu/孔,这比野生型细胞系高1.7倍且在10分钟时为9-14pfu/孔,这比野生型细胞系高约1.5倍(图3)。在足够的感染时间即20分钟或更长时,斑块形成在野生型MDCK细胞和缺失Bst2功能的细胞之间没有差异。
这表明病毒可在较短的时间内较容易地穿透缺失Bst2功能的细胞且病毒对缺失Bst2功能的细胞系的感染性显著高于野生型细胞系。因此,发现Bst2蛋白不但如先前报道的那样抑制病毒的细胞外释放,而且抑制病毒穿透进入宿主细胞。也就是说,Bst2的功能从细胞的去除使具有靶病毒的细胞的比例得以增加,由此促进靶病毒对细胞的感染。
实施例3:缺失Bst2基因功能的细胞系中胞内病毒蛋白量的增加
3-1:Vero和MDCK细胞系
为了证实缺失Bst2基因功能的细胞系中病毒抗原性蛋白产生的增加,在野生型Vero和MDCK细胞系及缺失Bst2功能的细胞系之间比较了病毒抗原性蛋白的产生。在该实施例中,使用在实施例1中得到的G-D5和J-D10(登录号KCLRF-BP-00285)细胞系。
野生型Vero和MDCK细胞及缺失Bst2功能的细胞(5×104个细胞/孔)用MOI为0.1的A/H3N2流感病毒感染30分钟,然后将培养基更换为新鲜培养基,然后培养36-72小时。在每个时间点,去除释放到培养基中的病毒且用PBS洗涤留在培养瓶底部的细胞,然后收获,由此获得细胞裂解物。将所获得的细胞裂解物通过SDS-PAGE进行电泳,然后通过Western印迹用识别流感病毒HA蛋白的大片段的抗HA单克隆抗体进行分析。
结果显示缺失Bst2基因功能的G-D5细胞中流感病毒蛋白的量在感染后36小时比野生型细胞大约5倍且在感染后72小时比野生型细胞大约15倍(图5A)。还显示缺失Bst2基因功能的J-D10细胞中流感病毒蛋白的量在感染后36小时大约57倍(图6B)。
3-2:人293T细胞系
为了确认通过从人细胞去除Bst2基因的功能使病毒抗原性蛋白的产生得以增加的作用,在24孔板中用MOI为0.01的H3N2(A/Brisbane/10/2007)流感病毒感染Bst2基因敲除293T细胞(B4、B24和D19)(2.5×105个细胞/孔)30分钟,然后在新鲜培养基中培养2天。
Western印迹分析显示Bst2基因敲除293T细胞中病毒蛋白的量显著增加,如在Vero和MDCK细胞系中那样(图7)。
有效地实现了病毒从缺失Bst2基因功能的细胞系的释放。因此,若病毒在缺失Bst2基因功能的细胞中的生长速率与在野生型细胞中的生长速率相同且若病毒从缺失Bst2基因功能的细胞的释放比从野生型细胞的释放快得多,则预期未从缺失Bst2基因功能的细胞释放的胞内病毒的量比未从野生型细胞释放的胞内病毒的量大得多。然而,从缺失Bst2基因功能的细胞释放的病毒的量和剩余在所述细胞中的病毒的量也显著增加。
因此,病毒从缺失Bst2基因功能的细胞的释放和病毒在所述细胞中的生长都得以促进,这表明Bst2蛋白能抑制病毒在宿主细胞中的生长。也就是说,可促进靶病毒在缺失Bst2功能的细胞中的生长,从而能提高病毒和抗原性蛋白在其中的产生。
实施例4:每个缺失Bst2基因功能的细胞的胞内病毒蛋白的量的增加
在该实施例中,分析了每个缺失Bst2基因功能的细胞的胞内病毒抗原性蛋白的量。因为缺失Bst2基因功能的细胞的凋亡被有效地抑制,所以即使这些细胞被病毒感染(实施例3的结果),在野生型细胞和缺失Bst2基因功能的细胞之间可存在细胞数目的差异,且因此分析了每个细胞的胞内病毒抗原性蛋白的量。
具体地,用MOI为0.1的A/H3N2流感病毒感染野生型Vero和MDCK细胞及缺失Bst2功能的突变细胞(5×104个细胞/孔)30分钟,然后将培养基更换为新鲜培养基,然后培养36-72小时。在每个时间点,去除释放到培养基中的病毒且用PBS洗涤留在培养瓶底部的细胞,然后收获,然后细胞裂解物的量为每个细胞(1×106个细胞/ml)。对应于104个细胞的细胞裂解物通过SDS-PAGE进行电泳,然后通过Western印迹用识别流感病毒HA蛋白的大片段的抗HA单克隆抗体进行分析。
结果显示缺失Bst2基因功能的G-D5细胞中每个细胞的胞内流感病毒蛋白的量在感染后36小时是野生型细胞的约两倍且在感染后72小时是野生型细胞的约17倍(图5B)。还显示缺失Bst2基因功能的J-D10细胞中每个细胞的胞内流感病毒蛋白的量在感染后36小时是野生型细胞的约7倍(图6B)。
这表明包含在相同数目的细胞中的流感病毒的量在缺失Bst2基因功能的细胞中比在野生型细胞中大。因此,发现病毒在缺失Bst2基因功能的细胞系中的生长得以促进,这表明Bst2蛋白能抑制病毒在宿主细胞中的生长。也就是说,缺失Bst2功能的细胞能通过促进靶病毒在其中的生长而产量得以增加的病毒和蛋白质。
工业应用性
如上所述,通过使用缺失Bst2基因功能的动物细胞系来促进靶病毒感染和增加靶蛋白产生的本发明方法能增加靶病毒和抗原性蛋白的产量且由此可用于生产用于预防或治疗病毒性疾病的疫苗。
虽然已参照具体特征而详细描述了本发明,但是对于本领域技术人员来说显而易见的是,该描述仅针对优选的实施方案且不限制本发明的范围。因此,本发明的实质范围将由所附权利要求书及其等同形式所定义。
序列表
<110> 易木农麦克斯有限公司(IMMUNOMAX CO., LTD.)
<120> 用于通过使用已缺失BST2基因功能的细胞系来促进病毒感染和增加病毒产生的方法
<130> PF-B1808
<140> PCT/KR2015/005493
<141> 2016-06-02
<150> 10-2014-0074027
<151> 2014-06-18
<150> 10-2014-0116245
<151> 2014-09-02
<160> 30
<170> PatentIn版本3.2
<210> 1
<211> 306
<212> DNA
<213> 狗
<400> 1
atggcaccta cgctttacca ctactactgg cctgtgccca taactgacga gtcagagtca 60
atgtcatcaa gtcagaagct gagctggctg gagtggctgg gcatcttggg gatcccagtg 120
gtgatgggtc tgtctgtggc tctgatcatc tttgttgtca agaccaacag caaagcctgc 180
ggggatggcc tcctagtaga gcaggagtgt cacaatgtca ccagcctcct ggagcgccaa 240
ctaacccaaa cccggcaagc gttacagggg accatggacc aggctaccac ctgcaacaag 300
actgtg 306
<210> 2
<211> 294
<212> DNA
<213> 猴
<400> 2
atggcaccta ttttgtatga ctattgcaaa atgcccatgg atgacatttg caaggaagac 60
agggacaagt gctgtaaact ggccgtagga attctggggc tcctggtcat agtgcttctg 120
ggggtgcccc tgattttctt catcatcaag gccaacagcg aggcctgcca ggatggcctc 180
cgggcagtga tggagtgtca caatgtcacc tatctcctgc aacaagagct ggccgaggcc 240
cagcggggct ttcgggacgc agaggcccag gctgtcacct gcaaccagac tgtg 294
<210> 3
<211> 304
<212> DNA
<213> 狗
<400> 3
atggcaccta cgctttacca ctactactgg cctgtgccca taactgaagt cagagtcaat 60
gtcatcaagt cagaagctga gctggctgga gtggctgggc atcttgggga tcccagtggt 120
gatgggtctg tctgtggctc tgatcatctt tgttgtcaag accaacagca aagcctgcgg 180
ggatggcctc ctagtagagc aggagtgtca caatgtcacc agcctcctgg agcgccaact 240
aacccaaacc cggcaagcgt tacaggggac catggaccag gctaccacct gcaacaagac 300
tgtg 304
<210> 4
<211> 304
<212> DNA
<213> 狗
<400> 4
atggcaccta cgctttacca ctactactgg cctgtgccca taacacgagt cagagtcaat 60
gtcatcaagt cagaagctga gctggctgga gtggctgggc atcttgggga tcccagtggt 120
gatgggtctg tctgtggctc tgatcatctt tgttgtcaag accaacagca aagcctgcgg 180
ggatggcctc ctagtagagc aggagtgtca caatgtcacc agcctcctgg agcgccaact 240
aacccaaacc cggcaagcgt tacaggggac catggaccag gctaccacct gcaacaagac 300
tgtg 304
<210> 5
<211> 302
<212> DNA
<213> 狗
<400> 5
atggcaccta cgctttacca ctactactgg cctgtgccca taactagtca gagtcaatgt 60
catcaagtca gaagctgagc tggctggagt ggctgggcat cttggggatc ccagtggtga 120
tgggtctgtc tgtggctctg atcatctttg ttgtcaagac caacagcaaa gcctgcgggg 180
atggcctcct agtagagcag gagtgtcaca atgtcaccag cctcctggag cgccaactaa 240
cccaaacccg gcaagcgtta caggggacca tggaccaggc taccacctgc aacaagactg 300
tg 302
<210> 6
<211> 303
<212> DNA
<213> 狗
<400> 6
atggcaccta cgctttacca ctactactgg cctgtgccca taactgagtc agagtcaatg 60
tcatcaagtc agaagctgag ctggctggag tggctgggca tcttggggat cccagtggtg 120
atgggtctgt ctgtggctct gatcatcttt gttgtcaaga ccaacagcaa agcctgcggg 180
gatggcctcc tagtagagca ggagtgtcac aatgtcacca gcctcctgga gcgccaacta 240
acccaaaccc ggcaagcgtt acaggggacc atggaccagg ctaccacctg caacaagact 300
gtg 303
<210> 7
<211> 302
<212> DNA
<213> 狗
<400> 7
atggcaccta cgctttacca ctactactgg cctgtgccca taacgagtca gagtcaatgt 60
catcaagtca gaagctgagc tggctggagt ggctgggcat cttggggatc ccagtggtga 120
tgggtctgtc tgtggctctg atcatctttg ttgtcaagac caacagcaaa gcctgcgggg 180
atggcctcct agtagagcag gagtgtcaca atgtcaccag cctcctggag cgccaactaa 240
cccaaacccg gcaagcgtta caggggacca tggaccaggc taccacctgc aacaagactg 300
tg 302
<210> 8
<211> 309
<212> DNA
<213> 狗
<400> 8
atggcaccta cgctttacca ctactactgg cctgtgccca taactgacga cgagtcagag 60
tcaatgtcat caagtcagaa gctgagctgg ctggagtggc tgggcatctt ggggatccca 120
gtggtgatgg gtctgtctgt ggctctgatc atctttgttg tcaagaccaa cagcaaagcc 180
tgcggggatg gcctcctagt agagcaggag tgtcacaatg tcaccagcct cctggagcgc 240
caactaaccc aaacccggca agcgttacag gggaccatgg accaggctac cacctgcaac 300
aagactgtg 309
<210> 9
<211> 282
<212> DNA
<213> 猴
<400> 9
atggcaccta ttttgtatga ctattgcaaa atgcccatgg atgacatttg caaggaagac 60
agggacaagt gctgtaaact ggccgtagga attctggggc tcctggtcat agtgcccctg 120
attttcttca tcatcaaggc caacagcgag gcctgccagg atggcctccg ggcagtgatg 180
gagtgtcaca atgtcaccta tctcctgcaa caagagctgg ccgaggccca gcggggcttt 240
cgggacgcag aggcccaggc tgtcacctgc aaccagactg tg 282
<210> 10
<211> 293
<212> DNA
<213> 猴
<400> 10
atggcaccta ttttgtatga ctattgcaaa atgcccatgg atgacatttg caaggaagac 60
agggacaagt gctgtaaact ggccgtagga attctggggc tcctggtcat agtgcttctg 120
ggggtgccct gattttcttc atcatcaagg ccaacagcga ggcctgccag gatggcctcc 180
gggcagtgat ggagtgtcac aatgtcacct atctcctgca acaagagctg gccgaggccc 240
agcggggctt tcgggacgca gaggcccagg ctgtcacctg caaccagact gtg 293
<210> 11
<211> 20
<212> DNA
<213> 人工
<220>
<223> dBst2-F引物
<400> 11
ggtcaggatg gctcctatgc 20
<210> 12
<211> 20
<212> DNA
<213> 人工
<220>
<223> dBst2-R引物
<400> 12
aacctgacag ggtcacctgg 20
<210> 13
<211> 21
<212> DNA
<213> 人工
<220>
<223> NdBst2-F引物
<400> 13
gtagccccag ccaaaggttt c 21
<210> 14
<211> 20
<212> DNA
<213> 人工
<220>
<223> NdBst2-R引物
<400> 14
aggcctcccc atgcccaaac 20
<210> 15
<211> 19
<212> DNA
<213> 人工
<220>
<223> AGM-F引物
<400> 15
gagggggaga tctggatgg 19
<210> 16
<211> 20
<212> DNA
<213> 人工
<220>
<223> AGM-R引物
<400> 16
aggcctcccc atgcccaaac 20
<210> 17
<211> 573
<212> DNA
<213> 狗
<400> 17
atggcaccta cgctttacca ctactactgg cctgtgccca taactgacct atgagagtca 60
gagtcaatgt catcaagtca gaagctgagc tggctggagt ggctgggcat cttggggatc 120
ccagtggtga tgggtctgtc tgtggctctg atcatctttg ttgtcaagac caacagcaaa 180
gcctgcgggg atggcctcct agtagagcag gagtgtcaca atgtcaccag cctcctggag 240
cgccaactaa cccaaacccg gcaagcgtta caggggacca tggaccaggc taccacctgc 300
aacaagactg tggtgaccct gtcagcttcc ctggtgaagg agaaggcctg gggtcaggag 360
cagctcaccc gaggagagaa acttcaggga gagatcgaga cattgaagca gcagctgcag 420
gccgctttgg aggaggtgaa gcagctaaga gaagggaagg aggcctcaag caaggaaagg 480
gaaaccagct ccgtcagctc cttgaaggca ccccccggct ccgtggtggt ccccgtgtac 540
ctgctcctag gccttagggc tctgctggcc tga 573
<210> 18
<211> 564
<212> DNA
<213> 狗
<400> 18
atggcaccta cgctttacca ctactactgg cctgtgccca taactgagtc agagtcaatg 60
tcatcaagtc agaagctgag ctggctggag tggctgggca tcttggggat cccagtggtg 120
atgggtctgt ctgtggctct gatcatcttt gttgtcaaga ccaacagcaa agcctgcggg 180
gatggcctcc tagtagagca ggagtgtcac aatgtcacca gcctcctgga gcgccaacta 240
acccaaaccc ggcaagcgtt acaggggacc atggaccagg ctaccacctg caacaagact 300
gtggtgaccc tgtcagcttc cctggtgaag gagaaggcct ggggtcagga gcagctcacc 360
cgaggagaga aacttcaggg agagatcgag acattgaagc agcagctgca ggccgctttg 420
gaggaggtga agcagctaag agaagggaag gaggcctcaa gcaaggaaag ggaaaccagc 480
tccgtcagct ccttgaaggc accccccggc tccgtggtgg tccccgtgta cctgctccta 540
ggccttaggg ctctgctggc ctga 564
<210> 19
<211> 566
<212> DNA
<213> 狗
<400> 19
atggcaccta cgctttacca ctactactgg cctgtgccca taactgagag tcagagtcaa 60
tgtcatcaag tcagaagctg agctggctgg agtggctggg catcttgggg atcccagtgg 120
tgatgggtct gtctgtggct ctgatcatct ttgttgtcaa gaccaacagc aaagcctgcg 180
gggatggcct cctagtagag caggagtgtc acaatgtcac cagcctcctg gagcgccaac 240
taacccaaac ccggcaagcg ttacagggga ccatggacca ggctaccacc tgcaacaaga 300
ctgtggtgac cctgtcagct tccctggtga aggagaaggc ctggggtcag gagcagctca 360
cccgaggaga gaaacttcag ggagagatcg agacattgaa gcagcagctg caggccgctt 420
tggaggaggt gaagcagcta agagaaggga aggaggcctc aagcaaggaa agggaaacca 480
gctccgtcag ctccttgaag gcaccccccg gctccgtggt ggtccccgtg tacctgctcc 540
taggccttag ggctctgctg gcctga 566
<210> 20
<211> 557
<212> DNA
<213> 狗
<400> 20
atggcaccta cgctttacca ctactactgg cctgtgccca taaagagtca atgtcatcaa 60
gtcagaagct gagctggctg gagtggctgg gcatcttggg gatcccagtg gtgatgggtc 120
tgtctgtggc tctgatcatc tttgttgtca agaccaacag caaagcctgc ggggatggcc 180
tcctagtaga gcaggagtgt cacaatgtca ccagcctcct ggagcgccaa ctaacccaaa 240
cccggcaagc gttacagggg accatggacc aggctaccac ctgcaacaag actgtggtga 300
ccctgtcagc ttccctggtg aaggagaagg cctggggtca ggagcagctc acccgaggag 360
agaaacttca gggagagatc gagacattga agcagcagct gcaggccgct ttggaggagg 420
tgaagcagct aagagaaggg aaggaggcct caagcaagga aagggaaacc agctccgtca 480
gctccttgaa ggcacccccc ggctccgtgg tggtccccgt gtacctgctc ctaggcctta 540
gggctctgct ggcctga 557
<210> 21
<211> 561
<212> DNA
<213> 狗
<400> 21
atggcaccta cgctttacca ctactactgg cctgtgccca tagacgaaga gtcaatgtca 60
tcaagtcaga agctgagctg gctggagtgg ctgggcatct tggggatccc agtggtgatg 120
ggtctgtctg tggctctgat catctttgtt gtcaagacca acagcaaagc ctgcggggat 180
ggcctcctag tagagcagga gtgtcacaat gtcaccagcc tcctggagcg ccaactaacc 240
caaacccggc aagcgttaca ggggaccatg gaccaggcta ccacctgcaa caagactgtg 300
gtgaccctgt cagcttccct ggtgaaggag aaggcctggg gtcaggagca gctcacccga 360
ggagagaaac ttcagggaga gatcgagaca ttgaagcagc agctgcaggc cgctttggag 420
gaggtgaagc agctaagaga agggaaggag gcctcaagca aggaaaggga aaccagctcc 480
gtcagctcct tgaaggcacc ccccggctcc gtggtggtcc ccgtgtacct gctcctaggc 540
cttagggctc tgctggcctg a 561
<210> 22
<211> 20
<212> DNA
<213> 人工
<220>
<223> HBst2-F1引物
<400> 22
tggcacggcc taggcactca 20
<210> 23
<211> 20
<212> DNA
<213> 人工
<220>
<223> HBst2-R1引物
<400> 23
ggatactgag gtccccgatt 20
<210> 24
<211> 1881
<212> DNA
<213> 人
<400> 24
atggcatcta cttcgtatga ctattgcaga gtgcccatgg aagacgggga taagcgctgt 60
aagcttctgc tggggatagg aattctggtg ctcctgatca tcgtgattct gggggtgccc 120
ttgattatct tcaccatcaa ggccaacagc gaggcctgcc gggacggcct tcgggcagtg 180
atggagtgtc gcaatgtcac ccatctcctg caacaagagc tgaccgaggc ccagaagggc 240
tttcaggatg tggaggccca ggccgccacc tgcaaccaca ctgtggtaag ctcctcaact 300
cctttggatg gcctagtact aggcggtggg agggacaaga atctctcccc agaaatctga 360
cccagggtgg gtctccaggg agatgcaggg gaggtcctga aactgctcct gggcccccac 420
atcaagggac ctaggttccc ctaccagggt ttgtgggccc ctaacccagt ccagggcact 480
ggtgtagggg cagggtgtta aaactctcca gatcccccaa atcggggacc tcagtatccc 540
cctgggactt aggtgaattt ataaattctt tccagggcac tggtgtcggg ggccttgaaa 600
ctcctcgtgg gcaccagtcc tgggggagta gaaatcccta ttcagggttg aagggggacc 660
tcaccagacc ctgaaaaagg gggcttttga aattttcact tcatccctaa gaaactgaaa 720
tattcacctg ggtcctgata tgggggatct tgaaactctc gctgggcatg tcacttgggc 780
ggggaaatcc cactgcattc tggatttggt agggccctct aacttttctt gggccattgc 840
tcaggcaatc tggaaatgtc cactaaactt tggttatcga tagcctccaa gtttccacgt 900
ggggtggcct caaaactccc attttgagga cccacatgct tatgggtggc cctgggagag 960
tgtgtggttg tggctgttct ttaaggttgg agaccatggt gcagagaggg ttggaagaaa 1020
acctgaaagg ggtttgcatt taagcccctc tgtccccagg acctagggag gaggcccagg 1080
tcccaggggc agcagccaaa ctccccaggc caaaacccca gattctaact cttcttagat 1140
ggccctaatg gcttccctgg atgcagagaa ggcccaagga caaaagaaag tggaggagct 1200
tgagggtgag aaagggagaa gggagagggc cggggagggg tgagtcaggt atggaagagg 1260
gggtgggggc agggagacca gggctggagg ttggggtaag ggggaggttc tgtcccagag 1320
tggagcaggg ccccagcatg gccacatgct gacccgcccc ctgtttctgt cctcccaccc 1380
taccaggaga gatcactaca ttaaaccata agcttcagga cgcgtctgca gaggtggagc 1440
gactgaggtc agagatagcc ttcccccgct accctccacc tgccacattc ctctcacccc 1500
cacatcccta gcccaagacc caggatctcc tttgctccca aaatccccat tgccccaagg 1560
gataaagttt gagtcccaca aaaggataac ttagccccta gggtcacaga gccatgggtg 1620
gccgctgtcc attccctccc cggtgacttg gattggggcg gtgcgggggg aactcccggg 1680
ggcggtgggc ttacagggag ggcggcagga gccaggacga gcagatgcct gatttgccat 1740
ctgtaccgca gaagagaaaa ccaggtctta agcgtgagaa tcgcggacaa gaagtactac 1800
cccagctccc aggactccag ctccgctgcg gcgccccagc tgctgattgt gctgctgggc 1860
ctcagcgctc tgctgcagtg a 1881
<210> 25
<211> 61
<212> DNA
<213> 人
<400> 25
atggcatcta cttcgtatga ctattgcaga gtgcccatgg aagacgggga gataggaatt 60
c 61
<210> 26
<211> 85
<212> DNA
<213> 人
<400> 26
atggcatcta cttcgtatga ctattgcaga gtgcccatgg aagacgggga taagcgctgt 60
aagcttctgc tggggatagg aattc 85
<210> 27
<211> 86
<212> DNA
<213> 人
<400> 27
atggcatcta cttcgtatga ctattgcaga gtgcccatgg aagacgggga taagcgctgt 60
aagcttcttg ctggggatag gaattc 86
<210> 28
<211> 64
<212> DNA
<213> 人
<400> 28
atggcatcta cttcgtatga ctattgcaga gtgcccatgg aagacgggga taagcgctgt 60
aagc 64
<210> 29
<211> 82
<212> DNA
<213> 人
<400> 29
atggcatcta cttcgtatga ctattgcaga gtgcccatgg aagacgggga taagcgctgt 60
aagcttctgc tgataggaat tc 82
<210> 30
<211> 77
<212> DNA
<213> 人
<400> 30
atggcatcta cttcgtatga ctattgcaga gtgcccatgg aagacgggga taagcgctgt 60
aatggggata ggaattc 77
- 还没有人留言评论。精彩留言会获得点赞!