为提高的产率而改造的微生物的制作方法
依据U.S.C.§119(e),本申请要求于2015年9月4日提交的第62/214,780号美国临时专利申请的优先权,该美国临时专利申请的全部内容通过引用并入本文。
对序列表的引用
所附序列表中的材料在此通过引用并入本申请中。命名为SGII1960_1WO_序列表.txt的所附序列表文本文件创建于2016年9月1日,大小为246kb。可以在使用Windows操作系统的计算机上利用Microsoft Word对该文件进行评价。
技术领域
本申请大体上涉及分子生物学和遗传学领域。具体而言,本申请涉及调节微生物(例如,微藻)的生物质产率所涉及的方法和材料。本申请还提供了具有提高的产率的重组微生物,例如微藻。
背景技术:
微藻近来吸引了相当大的关注,因为从这些有机体可以产生许多消费品和应用。基于微藻的产品组合从食品和动物饲料的生物质生产延伸到从微藻生物质提取的有价值的产品,包括可转化为生物柴油的甘油三酯。随着先进的培养和筛选技术的发展,微藻生物技术可有助于满足食品、制药和能源行业的高要求。低生物质产量导致藻类生物燃料及其它产品的相对较高的成本。因此,科学家们正不断努力提高藻类生物质产率。
真核细胞中细胞周期的进展是通过细胞周期调节蛋白的合成/降解和磷酸化/去磷酸化来调节的。包含Skp、Cullin、F-box的复合物(SCF复合物)是泛素连接酶复合物,其通过标记用于蛋白酶体降解的关键蛋白质来控制G1/S期和G2/M期之间的转变(Cordozo和Pagano(2004)Nature Rev Mol Cell Biol 5:739-751;Vodermaier(2014)Curr Biol 14:R787-R796;Wei等(2004)Nature 428:194-198)。除了泛素化细胞周期蛋白之外,SCF复合物还标记用于降解的各种其它细胞蛋白。SCF复合物包含三个核心亚基和一些不那么关键的组分。核心组分包括SKP1、Cullin(CUL1)和F-box蛋白(SKP2,Cdc4)。SKP1充当在Cullin和F-box蛋白之间形成连接的桥连蛋白(Schulman等(2000)Nature 408:381-386)。
技术实现要素:
本申请描述了如下发现:与SCF复合物及相关途径相关的特定基因在微生物例如藻类或异鞭毛微生物中过表达时为微生物赋予提高的产率。所述基因编码生长调节物例如SKP1多肽或衍生自CHORD多肽的多肽。
一方面,本发明提供了包含非天然核酸分子的重组微生物,所述非天然核酸分子包含编码SKP1多肽的核酸序列。所述非天然核酸分子可以包含与编码SKP1多肽的核酸序列并列的一个或多个核酸序列,所述一个或多个核酸序列天然地不与SKP1基因并列。在一些实施方案中,非天然核酸分子包含编码SKP1多肽的核酸序列,所述核酸序列与调控序列(例如,启动子)可操作地连接,所述调控序列天然地不与SKP1编码序列可操作地连接。可选地或另外地,所述非天然核酸分子可以包含用于介导核酸序列整合到宿主基因组中的序列、一种或多种可选择的标记基因和/或一种或多种可检测的标记基因。包含如本文所提供的非天然核酸分子的重组微生物可具有相对于不包含所述非天然核酸分子的对照微生物提高的产率,例如提高的生物质产率(例如,AFDW或TOC产率)和/或提高的脂质产率(例如,提高的FAME产率)。
在各种实施方案中,所述非天然核酸分子可以包含编码SKP1多肽的核酸序列,所述核酸序列与天然SKP1多肽具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%同一性或95%至100%同一性(序列同源性)。编码的SKP1多肽可以包括Pfam PF03931的SKP家族四聚化结构域和Pfam PF01466的SKP家族二聚化结构域中的一者或两者。在一些实施例中,氨基酸序列包含至少一个Pfam03931结构域和至少一个Pfam01466结构域。在一些实施例中,非天然核酸分子包含如下所述的多肽:所述多肽具有与SEQ ID NO:101具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%同一性或95%至100%同一性(序列同源性)的SKP1家族结构域和/或与SEQ ID NO:102具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%同一性或95%至100%同一性(序列同源性)的SKP1家族结构域。
在一些实施方案中,编码SKP1多肽的核酸序列是cDNA,和/或缺少衍生出SKP1编码序列的天然基因的一个或多个内含子。可选地或另外地,编码SKP1多肽的核酸序列可以包含不存在于衍生出SKP1编码序列的天然基因中的一个或多个内含子。例如,包含在SKP1转基因中的一个或多个内含子可以是衍生出该转基因的SKP1基因的天然内含子,或者可以是衍生自不同的天然基因的内含子,和/或一个或多个内含子可以是经完全或部分改造的序列。在各种实施方案中,作为任意上述的替代或补充,编码SKP1多肽的核酸序列可针对宿主微生物进行密码子优化,和/或可编码SKP1多肽,所述SKP1多肽相对于其所衍生自的天然SKP1基因包含一个或多个氨基酸改变、添加或缺失。在各种实施方案中,可以对本文提供的重组微生物进行遗传改造以包含非天然核酸分子,所述非天然核酸分子包含与天然SKP1多肽具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%或95%至100%同一性的编码SKP1多肽的序列,包括但不限于衍生自与重组宿主微生物同种或同属的SKP1多肽。在各种实施例中,如本文所提供的重组微生物包含非天然核酸分子,所述非天然核酸分子包含与选自以下的SKP1多肽具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%或95%至100%同一性的编码SKP1多肽的核酸序列:SEQ ID NO:28、SEQ ID NO:65、SEQ ID NO:66、SEQ ID NO:67、SEQ ID NO:68、SEQ ID NO:69、SEQ ID NO:70、SEQ ID NO:71、SEQ ID NO:72、SEQ ID NO:73、SEQ ID NO:74、SEQ ID NO:75、SEQ ID NO:76、SEQ ID NO:77、SEQ ID NO:78、SEQ ID NO:79、SEQ ID NO:80、SEQ ID NO:81、SEQ ID NO:82、SEQ ID NO:83、SEQ ID NO:84、SEQ ID NO:85、SEQ ID NO:86、SEQ ID NO:87、SEQ ID NO:88、SEQ ID NO:89和SEQ ID NO:90。
在一些实施例中,如本文所提供的重组微生物包含非天然核酸分子,该非天然核酸分子包含与宿主微生物内源的SKP1多肽具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%或95%至100%同一性的编码SKP1多肽的核酸分子。各种实施方案中的非天然核酸分子可以进一步包含与SKP1编码序列可操作地连接的启动子,其中所述启动子可以是SKP1编码序列并非天然地与之可操作地连接的启动子。与SKP1编码核酸序列可操作地连接的启动子可以源自与宿主微生物相同的种属,或者可以源自不同的种属。在示例性实施方案中,根据本发明的重组微生物包含非天然核酸分子,所述非天然核酸分子包含编码SKP1多肽的核酸序列,其中SKP1多肽具有与源自宿主微生物的天然SKP1多肽的氨基酸序列具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、或95%至100%同一性的氨基酸序列,并且编码SKP1多肽的核酸序列可操作地连接至不与SKP1编码序列天然地可操作地连接的启动子。在微生物中表达所述非天然核酸分子以导致存在于重组微生物中的SKP1转录物的水平高于在未用所述非天然核酸分子转化的对照微生物中存在的水平,并且与不包含编码SKP1多肽的非天然核酸分子的对照微生物相比表现出更高的产率,例如更高的生物质或脂质产率。
在替选实施方案中,转化到宿主微生物中的非天然核酸分子不包含与编码SKP1多肽的核酸序列可操作地连接的启动子。在一些这样的实施方案中,在将非天然核酸分子转化到宿主细胞中后,编码SKP1多肽的核酸序列可整合到宿主基因组中,使得其可操作地连接至调控序列,例如引导SKP1编码序列表达的宿主基因组的启动子。在各种实施例中,非天然核酸分子可以包含可选择的标记基因或可检测的标记基因,例如编码荧光蛋白的基因。
在一些实施方案中,如本文所提供的重组微生物可以是异鞭毛例如网粘菌纲(labyrinthulomycete)种属,并且可以包含编码SKP1多肽的非天然基因,所述非天然基因与异鞭毛种属的SKP1多肽具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、或95%至100%同一性序列同源性),所述异鞭毛种属的SKP1多肽例如但不限于网粘菌纲SKP1多肽,例如但不限于SEQ ID NO:73或SEQ ID NO:74。
在另一些实施方案中,如本文所提供的重组微生物可以是异鞭毛藻类例如硅藻种属,并且可以包含与硅藻种属的SKP1多肽具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、或95%至100%同一性(序列同源性)的编码SKP1多肽的非天然基因,所述硅藻种属的SKP1多肽例如但不限于SEQ ID NO:65、SEQ ID NO:70、SEQ ID NO:71、SEQ ID NO:72、SEQ ID NO:75、SEQ ID NO:76、SEQ ID NO:77、SEQ ID NO:78、SEQ ID NO:79、SEQ ID NO:80和SEQ ID NO:81中的任一个。作为非限制性实例,重组宿主微生物可以是双眉藻属(Amphora)、角毛藻属(Chaetoceros)、小环藻属(Cyclotella)、拟脆杆藻属(Fragilaropsis)、舟形藻属(Navicula)、菱形藻属(Nitzschia)、褐指藻属(Phaeodactylum)、海链藻属(Thalassiosira)或菱板藻属(Hantzschia)。
在另一些实施方案中,如本文所提供的重组微生物在一些实施例中可以是异鞭毛藻类,例如黄绿藻(eustigmatophyte)种属,并且可以包含编码SKP1多肽的非天然基因,所述非天然基因与黄绿藻种属的SKP1多肽具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%或95%至100%同一性(序列同源性),所述黄绿藻种属的SKP1多肽例如但不限于SEQ ID NO:28、SEQ ID NO:68和SEQ ID NO:69中的任一个。例如,重组宿主微生物可以是如下种属:真眼点藻属(Eustigmatos)、蒜头藻属(Monodus)、拟角星鼓藻属(Pseudostaurastrum)、魏氏藻属(Vischeria)或拟微球藻属(Nannochloropsis)。
在又一些实施方案中,如本文所提供的重组微生物可以是绿藻例如绿藻纲植物(chlorophyte)种属,并且可以包含编码SKP1多肽的非天然基因,所述非天然基因与绿藻纲植物种属的SKP1多肽具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%或95%至100%同一性(序列同源性),所述绿藻纲植物种属的SKP1多肽例如但不限于SEQ ID NO:66、SEQ ID NO:67、SEQ ID NO:82、SEQ ID NO:83、SEQ ID NO:84、SEQ ID NO:85、SEQ ID NO:86、SEQ ID NO:87、SEQ ID NO:88、SEQ ID NO:89和SEQ ID NO:90中的任一个。作为非限制性实例,重组宿主微生物可以是四鞭藻属(Carteria)、衣藻属(Chlamydomonas)、小球藻属(Chlorella)、拟小球藻属(Parachlorella)、拟绿球藻属(Pseudochlorella)、四球藻属(Tetrachlorella)、链带藻属(Desmodesmus)、栅列藻属(Scenedesmus)、杜氏藻属(Dunaliella)、红球藻属(Haematococcus)、微球藻属(Nannochloris)、新绿藻属(Neochloris)、虫毛球藻属(Ostreococcus)、混营性微藻属(Picochlorum)、四爿藻属(Tetraselmis)和团藻属(Volvox)。
前述序列仅是示例性的。在各种实施例中,重组微生物可以是异鞭毛或藻类微生物,例如网粘菌纲、硅藻(例如,硅藻门(Bacillariophyte))、黄绿藻或绿藻(例如,绿藻门分支的成员,例如但不限于绿藻纲(Chlorophyceae)、四爿藻纲(Chlorodendrophyceae)或共球藻纲(Trebouxiophyceae)的成员),并且非天然核酸分子可以包含编码SKP1多肽的核酸序列,所述核酸序列与宿主微生物的SKP1多肽具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%同一性或95%至100%同一性(序列同源性)。编码SKP1的核酸序列可以与转化到经遗传改造的微生物中的核酸分子上的启动子可操作地连接,或者可以将编码SKP1的核酸序列引入宿主微生物中并指导插入到基因组中使该核酸序列可操作地连接至宿主微生物内源的启动子的位点,所述启动子并非天然地与SKP1基因可操作地连接。
在本发明的另一个方面,提供了相对于对照微生物具有提高的产率的重组微生物,所述产率例如生物质产率,例如AFDW或TOC产率,和/或脂质产率,例如FAME产率,其中重组微生物包含编码包含至少60%的CHORD结构域的多肽的非天然核酸分子。例如,重组微生物可以包含非天然核酸分子,所述非天然核酸分子包含编码CHORD衍生多肽的核酸序列,所述CHORD衍生多肽包含天然CHORD多肽的CHORD结构域的至少60%的氨基酸序列,或与天然CHORD多肽的CHORD结构域具有至少80%同一性的氨基酸序列。在各种实施方案中,重组微生物可以包含编码包含单一CHORD结构域或其部分的CHORD衍生多肽的非天然核酸分子。单一CHORD结构域或其部分可以衍生自宿主微生物的CHORD多肽的序列,并且可以是其中CHORD结构域序列与非CHORD多肽的氨基酸序列融合的嵌合蛋白的一部分。
在各种实施方案中,如本文所提供的重组微生物可以包含非天然核酸分子,所述非天然核酸分子包含编码CHORD衍生多肽的序列,所述序列包含CHORD多肽的单一CHORD结构域或其部分,所述非天然核酸分子与SEQ ID NO:22、SEQ ID NO:91、SEQ ID NO:92、SEQ ID NO:93、SEQ ID NO:94、SEQ ID NO:95和SEQ ID NO:96中的任一个具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%或95%至100%同一性(序列同源性)。衍生出所述序列的CHORD多肽可以是任意CHORD多肽,并且可以任选地衍生自与重组微生物相同的种属的CHORD多肽,或者可以不衍生自与重组微生物相同的种属的CHORD多肽。被引入到经改造的微生物中的非天然核酸分子可以包含可操作地连接到CHORD多肽的启动子。与编码CHORD衍生多肽的核酸序列可操作地连接的启动子可以是不与CHORD基因天然可操作地连接的启动子,并且可以任选地源自宿主微生物。
在替选实施方案中,转化到宿主微生物中的非天然核酸分子不包含与编码SKP1多肽的核酸序列可操作地连接的启动子。在一些这样的实施方案中,在将非天然核酸分子转化到宿主细胞中后,编码SKP1多肽的核酸序列可以整合到宿主基因组中,使得其能可操作地连接至调控序列,例如指导SKP1编码序列表达的宿主基因组的启动子。在各种实施例中,非天然核酸分子可以包含可选择标记基因或可检测标记基因,例如编码荧光蛋白的基因。
在一些实施例中,由引入微生物中的非天然核酸分子的核酸序列编码的CHORD衍生多肽的CHORD结构域可以与SEQ ID NO:4具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%同一性或95%至100%同一性。在其他实施例中,所述多肽包含与SEQ ID NO:99或其至少60%的连续氨基酸具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%同一性或95%至100%同一性的氨基酸序列。在另外的实施例中,CHORD衍生多肽可以与SEQ ID NO:100具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%或95%至100%同一性。
根据任意上述实施例的重组微生物,例如包含如本文所述的编码CHORD衍生多肽或SKP1多肽的非天然基因的重组微生物,可具有相对于对照微生物提高的产率。任意所述重组微生物的提高的产率可以是提高的生物质(例如,AFDW或TOC)产率。相对于对照细胞,生物质产率可以提高至少5%。例如,相对于对照细胞,生物质产率可以提高约5%至约500%、或约10%至约300%、或约10%至约200%、或约10%至约100%。在一些实施例中,相对于对照细胞,生物质(例如,AFDW或TOC)产率可以为约5%至约500%、约5%至约300%、约10%至约200%、约15%至约200%、约20%至约200%、约25%至约200%、约30%至约200%、约40%至约200%、约50%至约200%、约5至约100%、约10%至约100%、约15%至约100%、约20%至约100%、约25%至约100%、约30%至约100%、约40%至约100%、或约50%至约100%。在各种实施例中,可以在半连续生长1、2、3、4、5、6、7、8、9、10、11、12、13、14或更多天后测定生物质产率的提高。或者,可以在分批生长至少1、2、3、4、5、6或7天后证实生物质产率的提高。
本发明的另一个方面是根据任意前述实施例的重组微生物,例如包含如本文所述的编码CHORD衍生多肽或SKP1多肽的非天然基因的重组微生物,其中所述重组微生物示出提高的脂质产率,例如提高的FAME产率。相对于对照微生物,FAME产率可以提高至少5%,例如提高约5%至约500%、或10%至约300%、或约10%至约200%、或约10%至约100%、或约15%至约90%。在一些实施例中,脂质(例如,FAME)产率可以相对于对照细胞为约5%至约500%、约5%至约300%、约10%至约200%、约15%至约200%、约20%至约200%、约25%至约200%、约30%至约200%、约40%至约200%、约50%至约200%、约5%至约100%、约10%至约100%、约15%至约100%、约20%至约100%、约25%至约100%、约30%至约100%、约40%至约100%、或者约50%至约100%。在一些实施例中,可以在半连续生长1、2、3、4、5、6、7、8、9、10、11、12、13、14或更多天后证明提高的FAME产率的提高。可选地或另外地,可以在分批生长至少1、2、3、4、5、6或7天后证明FAME产率的提高。
待使用根据本发明的材料和方法进行修饰的合适的宿主微生物包括但不限于藻类细胞、异鞭毛细胞或真菌细胞。考虑用于本发明的异鞭毛种属包括但不限于硅藻、黄绿藻和网粘菌纲。网粘菌纲包括例如如下种属:网粘菌属(Labryinthula)、类网粘菌属(Labryinthuloides)、破囊壶菌属(Thraustochytrium)、裂殖弧菌(Schizochytrium)、不动壶菌属(Aplanochytrium)、橙黄壶菌属(Aurantiochytrium)、椭圆壶菌属(Oblongichytrium)、日本壶菌属(Japonochytrium)、戴普罗弗菌属(Diplophrys)和吾肯氏壶菌属(Ulkenia)。
适用于本发明方法的藻类种属包括微藻,例如如下种属:曲壳藻属(Achnanthes)、茧形藻属(Amphiprora)、双眉藻属、纤维藻属(Ankistrodesmus)、星胞藻属(Asteromonas)、黄金色藻属(Boekelovia)、波利氏藻属(Bolidomonas)、包特氏藻属(Borodinella)、气球藻属(Botrydium)、葡萄藻属(Botryococcus)、片球藻属(Bracteococcus)、角毛藻属、四鞭藻属、衣藻属、绿球藻属(Chlorococcum)、绿梭藻属(Chlorogonium)、小球藻属、蓝隐藻属(Chroomonas)、金球藻属(Chrysosphaera)、球钙板藻属(Cricosphaera)、隐甲藻属(Crypthecodinium)、隐藻属(Cryptomonas)、小环藻属、链带藻属、杜氏藻属、后棘藻属(Elipsoidon)、圆石藻属(Emiliania)、独球藻属(Eremosphaera)、衣迪斯藻属(Ernodesmius)、眼虫藻属(Euglena)、真眼点藻属、披刺藻属(Franceia)、脆杆藻属(Fragilaria)、拟脆杆藻属、丽丝藻属(Gloeothamnion)、红球藻属、菱板藻属、异弯藻属(Heterosigma)、膜胞藻属(Hymenomonas)、等鞭金藻属(Isochrysis)、鳞孔藻属(Lepocinclis)、微芒藻属(Micractinium)、蒜头藻属、单针藻属(Monoraphidium)、微球藻属、微拟球藻属、舟形藻属、新绿藻属、肾鞭藻属(Nephrochloris)、肾藻属(Nephroselmis)、菱形藻属、棕鞭藻属(Ochromonas)、鞘藻属(Oedogonium)、卵囊藻属(Oocystis)、虫毛球藻属、拟小球藻属、雪衣藻属(Parietochloris)、杜氏亚属盐藻属(Pascheria)、巴夫藻属(Pavlova)、普莱格门(Pelagomonas)、褐指藻属、扁裸藻属(Phagus)、混营性微藻属、扁藻属(Platymonas)、颗石藻属(Pleurochrysis)、肋球藻属(Pleurococcus)、原壁菌属(Prototheca)、拟绿球藻属、拟新绿藻属(Pseudoneochloris)、拟角星鼓藻属、塔胞藻属(Pyramimonas)、桑椹藻属(Pyrobotrys)、栅列藻属、裂衣藻属(Schizochlamydella)、骨条藻属(Skeletonema)、水绵藻属(Spyrogyra)、裂丝藻属(Stichococcus)、四球藻属、四爿藻属、海链藻属、黄丝藻属(Tribonema)、无隔藻属(Vaucheria)、鲜绿球藻属(Viridiella)、魏氏藻属以及团藻属。示例性种属的非限制性实例包括例如硅藻,例如双眉藻属、角毛藻属、小环藻属、脆杆藻属、舟形藻属、菱形藻属、褐指藻属或海链藻属中的任一种的种属,或者黄绿藻,例如真眼点藻属、蒜头藻属、拟角星鼓藻属或魏氏藻属中的任意一种的种属。
在本发明的各个方面,当与不包含非天然基因的对照微生物相比时,包含如本文所提供的非天然基因的微生物可具有提高的产率。例如,可以通过测量生长速率或总有机碳(TOC)或无灰于重累积,或者通过对由重组微生物产生的各种生物分子(例如,一种或多种脂质、聚合物、蛋白质、色素、碳水化合物等)进行定量来证明较高产率。
本文还提供了通过培养具有提高的产率的重组微生物(例如,本文公开的任意重组微生物)来生产生物质或至少一种生物产物的方法。该方法包括在合适的培养基中培养如本文所公开的包含本文公开的编码CHORD衍生多肽或SKP1多肽的非天然核酸的重组微生物以提供微生物培养物,并从所述培养物中回收生物质或至少一种生物产物。该方法可以任选地包括诱导非天然基因的表达。所述微生物可以是异鞭毛种属,例如网粘菌属、类网粘菌属、破囊壶菌属、裂殖弧菌属、不动壶菌属、橙黄壶菌属、椭圆壶菌属、日本壶菌属、戴普罗弗菌属和吾肯氏壶菌属中任意属的网粘菌纲。在一些实施例中,微生物可以是微藻,例如但不限于本文公开的任意属的种属。在一些实施例中,藻类培养物可以是光合自养培养物。可以使用该方法制备的产物的非限制性实例包括生物质、脂质、聚酮化合物、萜类化合物、色素、抗氧化剂、维生素、核苷酸、核酸、氨基酸、碳水化合物、醇、激素、细胞因子、肽、蛋白质或聚合物。生物产物可以进一步定义为食品、饲料、生物燃料、生物化学品、药品或医疗产品。
例如,生产生物质或至少一种生物产物的方法可包括培养如本文所公开的包含编码CHORD衍生多肽或SKP1多肽的非天然核酸分子的重组藻类微生物,其中所述藻类微生物属于选自以下的属:曲壳藻属、茧形藻属、双眉藻属、纤维藻属、星胞藻属、黄金色藻属、波利氏藻属、包特氏藻属、气球藻属、葡萄藻属、片球藻属、角毛藻属、四鞭藻属、衣藻属、绿球藻属、绿梭藻属、小球藻属、蓝隐藻属、金球藻属、球钙板藻属、隐甲藻属、隐藻属、小环藻属、链带藻属、杜氏藻属、后棘藻属、圆石藻属、独球藻属、衣迪斯藻属、眼虫藻属、真眼点藻属、披刺藻属、脆杆藻属、拟脆杆藻属、丽丝藻属、红球藻属、菱板藻属、异弯藻属、膜胞藻属、等鞭金藻属、鳞孔藻属、微芒藻属、蒜头藻属、单针藻属、微球藻属、微拟球藻属、舟形藻属、新绿藻属、肾鞭藻属、肾藻属、菱形藻属、棕鞭藻属、鞘藻属、卵囊藻属、虫毛球藻属、拟小球藻属、雪衣藻属、杜氏亚属盐藻属、巴夫藻属、普莱格门、褐指藻属、扁裸藻属、混营性微藻属、扁藻属、颗石藻属、肋球藻属、原壁菌属、拟绿球藻属、拟新绿藻属、拟角星鼓藻属、塔胞藻属、桑椹藻属、栅列藻属、裂衣藻属、骨条藻属、水绵藻属、裂丝藻属、四球藻属、四爿藻属、海链藻属、黄丝藻属、无隔藻属、鲜绿球藻属、魏氏藻属以及团藻属,以产生生物质或至少一种生物产物。该方法可以任选地进一步包括从培养物中回收生物质或至少一种生物产物,例如从培养基、全部培养物或细胞中回收。例如,藻类细胞可以属于选自以下的属:小球藻属、小环藻属、真眼点藻属、蒜头藻属、微拟球藻属、拟小球藻属、褐指藻属、拟绿球藻属、拟角星鼓藻属、魏氏藻属和四爿藻属。在一些情况下,培养在光合自养条件下进行。可以使用所述方法制备的产物的非限制性实例包括生物质、脂质、聚酮化合物、萜类化合物、色素、抗氧化剂、维生素、核苷酸、核酸、氨基酸、碳水化合物、醇、激素、细胞因子、肽、蛋白质或聚合物。生物产物可以进一步定义为食品、饲料、生物燃料、生物化学、药物或医药产品。
另一方面,本发明提供了分离的或重组的核酸分子,其包含编码如下多肽的核酸序列,所述多肽包含与选自以下的多肽序列具有至少60%、至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%或约100%同一性的氨基酸序列:SEQ ID NO:28、SEQ ID NO:65、SEQ ID NO:66、SEQ ID NO:67、SEQ ID NO:68、SEQ ID NO:69、SEQ ID NO:70、SEQ ID NO:71、SEQ ID NO:72、SEQ ID NO:73、SEQ ID NO:74、SEQ ID NO:75、SEQ ID NO:76、SEQ ID NO:77、SEQ ID NO:78、SEQ ID NO:79、SEQ ID NO:80、SEQ ID NO:81、SEQ ID NO:82、SEQ ID NO:83、SEQ ID NO:84、SEQ ID NO:85、SEQ ID NO:86、SEQ ID NO:87、SEQ ID NO:88、SEQ ID NO:89和SEQ ID NO:90。
在一些实施例中,如本文所提供的分离的或重组的核酸分子可编码包含编码CHORD结构域的至少一部分的氨基酸序列的多肽,并且在一些实施例中可包含单一CHORD结构域或CHORD结构域的一部分,其可以是例如与SEQ ID NO:4或SEQ ID NO:4的至少60%(例如,SEQ ID NO:4的至少36、37、38、39或40个连续氨基酸)具有至少80%同一性的CHORD结构域。例如,分离的或重组的核酸分子可以编码与SEQ ID NO:22、SEQ ID NO:99或SEQ ID NO:100具有至少60%、65%、70%、75%、80%、85%、90%、95%或99%同一性的多肽。
在本发明的另一个方面,提供了分离的或重组的核酸分子,其编码如下多肽,所述多肽包含编码至少一个SKP1家族蛋白结构域的氨基酸序列,所述至少一个SKP1家族蛋白结构域选自与SEQ ID NO:101具有至少80%同一性的至少一个Pfam3931结构域和与SEQ ID NO:102具有至少80%同一性的至少一个Pfam1466结构域。
分离的或重组的核酸分子可以编码作为SKP1蛋白或与植物或微生物种属的SKP1多肽具有至少80%同一性的多肽。可选地或另外地,所述多肽可以与微藻或异鞭毛种属的SKP1多肽具有至少50%、至少55%、至少60%、至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%或约100%同一性。
例如,分离的或重组的核酸分子可以编码如下多肽,所述多肽包含与选自以下的多肽序列具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%或约100%同一性的氨基酸序列:SEQ ID NO:28、SEQ ID NO:65、SEQ ID NO:66、SEQ ID NO:67、SEQ ID NO:68、SEQ ID NO:69、SEQ ID NO:70、SEQ ID NO:71、SEQ ID NO:72、SEQ ID NO:73、SEQ ID NO:74、SEQ ID NO:75、SEQ ID NO:76、SEQ ID NO:77、SEQ ID NO:78、SEQ ID NO:79、SEQ ID NO:80、SEQ ID NO:81、SEQ ID NO:82、SEQ ID NO:83、SEQ ID NO:84、SEQ ID NO:85、SEQ ID NO:86、SEQ ID NO:87、SEQ ID NO:88、SEQ ID NO:89和SEQ ID NO:90。
在多个实施例中,编码所述多肽的重组核酸序列相对于天然基因具有至少一个突变或缺少存在于天然基因中的至少一个内含子。可选地或另外地,所公开的重组核酸序列包含cDNA。进一步可选地或另外地,编码所述多肽的核酸序列可以可操作地连接至异源启动子和/或可以是载体。
在另一个实施例中,核酸分子编码CRISPR/Cas9系统的指导RNA,其中指导RNA靶向天然微生物基因的至少一部分,所述天然微生物基因编码具有与SEQ ID NO:22或SEQ ID NO:92有至少55%、至少60%、至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%或约100%同一性的氨基酸序列的多肽。
在一些实施例中,如上文所公开的重组核酸分子在微生物中的表达导致微生物的提高的产率,例如增强的繁殖、生物质累积或生物分子的产生。
附图说明
图1提供了描绘八种不同的加的斯微拟球藻(Nannochloropsis gaditana)菌株在分批生长的10天内的脂肪酸甲酯(FAME)值的图。所有随机整合突变菌株(GE-5870、GE-5871、GE-5873、GE-5874、GE-5875、GE-5876和GE-5877)优于野生型菌株WE-3730。
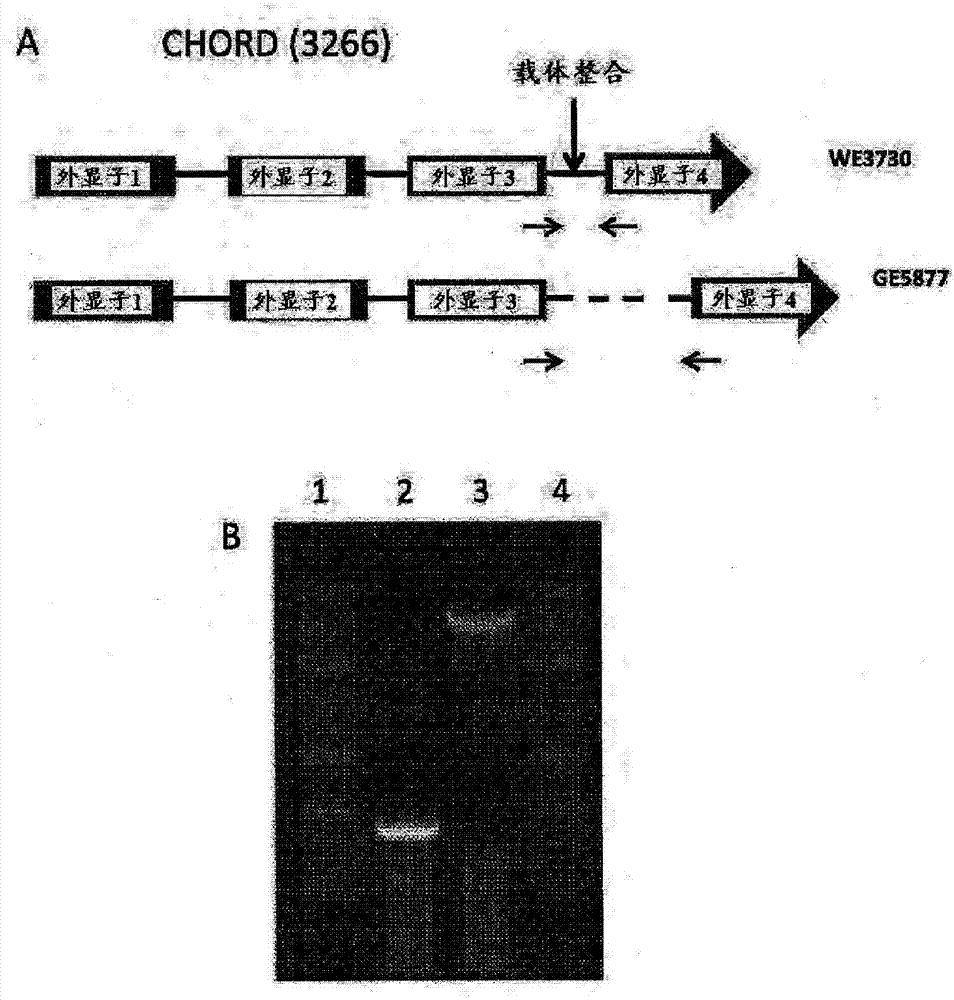
图2A-2G提供了菌株GE-5877的表征信息。A)描绘如通过MiSeq分析确定的载体整合入菌株GE-5877中CHORD基因3266的第三和第四外显子之间的内含子的位点的草图。用于插入的PCR验证的引物的位置描绘为载体整合位点侧翼的细黑色箭头。B)与野生型WE-3730相比,分离插入突变体GE-5877中的CHORD基因组区域的PCR产物后的凝胶图:1和4:分子量标记;2:WE3730(野生型)DNA;3:GE-5877(CHORD插入)DNA。野生型和GE-5877的预期大小分别为386个碱基对和4505个碱基对。C)示出如通过CHORD-3266转录物的两个不同区域的qRT-PCR测定的转录物水平的条形图。使用外显子2和外显子4的序列特异性引物组来扩增来自野生型和GE-5877样品的特定区域。外显子2在载体整合位点的上游,而外显子4在载体整合位点的下游。对在图C所描绘的生长测定的第4天分离的RNA样品进行了qRT-PCR。D)示出光密度的线图和E)经过六天分批生长的野生型和GE-5877的细胞计数。F)描绘FAME的条形图,和G)图C中描绘的生长测定的第4天和第6天总生物有机碳(TOC)产率。
图3A-3B提供了转录组学数据。A)描绘CHORD突变体GE-5877转录组相对于野生型WE-3730的全局基因表达分析的散点图。淡色斑点代表生物学重复的统计学显著倍数变化,而水平条表示1.5倍截止。B)对在CHORD突变菌株GE-5877中富含表达改变的基因的GO类别的基因本体论(GO)分析。示出了具有统计显著性的前10个类别。缩写:CPM,每百万次读数;BP,生物过程;CC,细胞成分;MF,分子功能。
图4A-4C描绘了菌株GE-5877的进一步表征。A)野生型和突变CHORD-3266基因座的图。标记出了外显子,并且介入的细线是内含子。CHORD结构域1和CHORD结构域2分别由外显子2和3以及外显子3和4侧翼的白框来描绘。通过MiSeq分析检测的转录物在基因图下面用细黑箭头表示。野生型菌株WE-3730仅含有天然CHORD-3266转录物,而菌株GE-5877分别表达两种非天然融合转录物,分别标记为5′融合转录物和3′融合转录物。B)用于概括GE-5877表型的方法图。不成功的方法包括敲除外显子1、敲除外显子4和过表达外显子1至3,产率结果用白色向下箭头或等号表示。过表达包含整合载体末端、内含子3和外显子4的3′融合转录物导致与野生型相比提高的产率,这由向上实心箭头描绘。C)描绘了与原始CHORD突变体GE-5877和野生型菌株WE-3730相比,表达3′融合转录物的菌株GE899在分批生长的5天过程中的吸光度的线图。
图5提供了描绘全长CHORD-3266蛋白质(CRD1-336)和用作Y2H筛选中的诱饵的不同肽(CRD1-117、CRD117-179、CRD179-251和CRD179-336)的模块化结构及每个筛选结果的草图。
图6描绘了SKP1-8611的基因图。标记出了外显子,并且介入空间为内含子。标记编码SKP1家族四聚化和二聚化结构域的序列并分别由跨越外显子1至3和外显子4至7的白框描绘。
图7提供了描绘如通过qRT-PCR测定的与野生型菌株WE-3730相比,SKP1-8611过表达菌株GE-8119和GE-8120中的SKP1-8611转录物的稳态mRNA水平的条形图。
图8A-8D提供了描绘GE-8119和WE-3730以30%每日稀释率的两个独立半连续产率测定中的FAME和TOC值的线图。在测定1中培养物产生的A)FAME和B)TOC。在测定2中培养物产生的C)FAME和D)TOC。对于每个半连续测定,各菌株一式三份进行。数据点表示三次重复的平均值且误差条描绘标准偏差。
发明详述
本申请涉及用于改变微生物特征,特别是与提高的产率相关的那些的组合物、方法和相关材料。在各个方面,本申请公开了重组微生物,例如表达影响产率例如生物质或脂质产率的非天然基因的微藻和异鞭毛。
在本公开通篇中,各种信息源被提及和/或通过引用并入。信息源包括例如科学期刊文章、专利文献、教科书和万维网浏览器非活动页面地址。虽然对这些信息源的引用清楚地表明它们可以被本领域技术人员使用,但是本文引用的各个及每一个信息源都明确地整体引入,应注意无论是否具体提及“通过引用并入”。还应当注意的是,对这样的信息源的引用仅仅是为了在递交时提供现有技术的一般性指示的目的。虽然各个及每个信息源的内容和教导内容都可以被本领域技术人员信赖和使用以进行和使用本发明的实施方案,但是具体信息源中的任何讨论和评论决不应被视为承认这样的评论被广泛接受为该领域的一般意见。
一些定义
除非另外定义,否则本文中使用的所有本领域术语、符号和其它科学术语或用辞旨在具有本发明所属领域的技术人员通常理解的含义。在一些情况下,为了清楚和/或为了便于参考起见,在本文中对具有通常理解的含义的术语进行了定义,并且在本文中包括这样的定义不应必须被解释为表示与本领域中通常理解的定义有实质差异。本领域技术人员可以很好地理解本文所描述或引用的许多技术和规程,并且通常采用常规方法来使用。
除非上下文另有明确规定,否则单数形式“一个/种(a/an)”和“该”包括复数指称。例如,术语“细胞”包括一个或多个细胞,包括其混合物。“A和/或B”在本文中用于包括以下所有备选物:“A”、“B”以及“A和B”。
“约”可以表示所提供的值的正或负10%。在提供范围的情况下,它们包括边界值。“约”可以另外地或者替代地指在所述值的10%以内,或者在所述值的5%以内,或者在一些情况下在所述值的2.5%以内,或者“约”可以意指四舍五入到最近的有效数位。
提及“基本上相同”或“基本上等同”的性质而不进一步解释预期含义,旨在意指性质在参考值的10%以内、优选在5%以内、并且可以在2.5%以内。在未阐述在特定上下文中的“基本上”的预期含义的情况下,该术语用于包括较小或不相关的偏差,不认为这些偏差对于在本发明的上下文中被认为重要的特征是实质性的。
如本文所使用的“氨基酸”是指天然和合成的氨基酸,以及以与天然氨基酸类似的方式起作用的氨基酸类似物和氨基酸模拟物。天然氨基酸是由遗传密码编码的氨基酸,包括D/L旋光异构体,以及后来经修饰的那些氨基酸,例如羟基脯氨酸、γ-羧基谷氨酸和O-磷酸丝氨酸。氨基酸类似物是指具有与天然氨基酸相同的基本化学结构(即与氢、羧基、氨基和R基结合的碳)的化合物,例如高丝氨酸、正亮氨酸、甲硫氨酸亚砜、甲硫氨酸甲基锍。这样的类似物具有经修饰的R基(例如,正亮氨酸)或经修饰的肽主链,但保留与天然氨基酸相同的基本化学结构。如本文使用的氨基酸模拟物是指具有与氨基酸的一般化学结构不同的结构,但以与天然氨基酸类似的方式起作用的化合物。
如本文所使用的“减弱”意指数量、程度、强度(intensity)或强度(strength)的降低。减弱的基因表达可以指所述基因的转录的量和/或速率显著降低,或编码的蛋白质的翻译、折叠或装配的显著减少。作为非限制性实例,减弱的基因可以是不编码完整功能性开放阅读框或具有因基因调控序列的突变或破坏所致的降低的表达的突变的或被破坏的基因(例如,通过部分或全部缺失、截短、移码或插入突变破坏的基因)。减弱的基因也可以是由减少基因表达的构建体靶向的基因,例如反义RNA、微小RNA、RNAi分子或核酶。减弱的基因表达可以是被消除的基因表达,例如减少到不显著或检测不到的量。减弱的基因表达还可以是产生不完全功能或无功能的RNA或蛋白质的基因表达,例如减弱的基因表达可以是产生截短的RNA和/或多肽的基因表达。
如本文所使用的“生物燃料”是指来自活生物体(例如高等植物、真菌、藻类或微生物)的可再生能源。因此,生物燃料可以是来源于藻类、真菌、微生物或植物材料的固体、液体或气体燃料、生物质、糖或淀粉,例如来源于植物油或藻油的乙醇或生物柴油等。生物燃料本身就是一种燃料,但可以与石油基燃料共混以生成成品燃料。生物燃料可以用作石化来源的汽油、柴油燃料或喷气燃料的替代品。
“cDNA”是包含mRNA分子的核苷酸序列的至少一部分的DNA分子,不同之处在于DNA分子用核碱基胸腺嘧啶或T代替存在于mRNA序列中的尿苷或U。cDNA可以是单链的或双链的,并且可以是mRNA序列的互补序列。在优选的实施例中,cDNA不包括存在于cDNA所对应的天然基因(在生物体的基因组中)中的一个或多个内含子序列。例如,cDNA可以具有与天然基因的内含子下游的序列并列的天然基因的内含子上游的序列,其中上游序列和下游序列并不是天然地在DNA分子中并列(即,序列在天然基因中不是并列的,而是被内含子分开)。cDNA可以通过mRNA分子的逆转录来产生,或者可以例如通过化学合成和/或通过使用一种或多种限制酶、一种或多种连接酶、一种或多种聚合酶(包括但不限于,可用于聚合酶链式反应(PCR)的耐高温聚合酶)、一种或多种重组酶等基于cDNA序列的知识来合成,其中cDNA序列的知识可任选地基于来自基因组序列的编码区的鉴定和/或从多个部分cDNA的序列编译。
如本发明中使用的“对照微生物”、“对照生物体”或“对照细胞”为测量对象微生物、生物体或细胞的表型变化提供了参考点。对照微生物、生物体或细胞可以包括例如(a)野生型微生物、生物体或细胞,即具有与经历遗传改变而产生对象微生物、生物体或细胞的起始材料相同的基因型;(b)与起始材料具有相同的基因型但已经用无效构建体(即,对目标性状没有已知作用的构建体,例如缺少编码目标多肽的基因、例如缺少编码CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样多肽的基因的构建体)转化的微生物、生物体或细胞;(c)微生物、生物体或细胞,其是对象微生物、生物体或细胞的子代中的非转化分离子;或(d)在目标基因不表达的条件下,对象微生物、生物体或细胞本身。在一些情况下,“对照微生物”在一些情况下可以指不含存在于目标转基因微生物中的外源核酸但在其它方面具有与这样的转基因(“改造的”或“重组”)的微生物相同的或相似的遗传背景的微生物。
“结构域”是可用于表征蛋白质家族和/或蛋白质部分的多肽中的基本上连续的氨基酸组。这样的结构域可具有“指纹”、“基序”或“签名”,其可包括保守的一级序列、二级结构和/或三维构型。一般而言,结构域与特定的体外和/或体内活性相关。结构域可以具有任意大小,举例来说,结构域可以具有4个氨基酸至约400个氨基酸的长度,例如4个至约200个氨基酸、或者8个至约150个氨基酸、或者4个至约10个氨基酸、或者约10个至约100个氨基酸、或者约15个至约65个氨基酸或约20个至约100个氨基酸或约25个至120个氨基酸或约100个至约200个氨基酸或约300个至约500个氨基酸。
“下调”是指相对于基础或天然状态降低表达产物(mRNA、多肽、生物活性或其任意组合)的产生的调节。
在本公开的上下文中,术语“内源的”是指作为细胞的天然部分的任意多核苷酸、多肽或蛋白质序列。
就核酸或基因而言的“外源”指示已经通过人为干预将核酸或基因引入(“转化”)到生物体、微生物或细胞中。通常,通过重组核酸构建体将这样的外源核酸引入细胞或生物体中。外源核酸可以是被引入到另一个种属中的来自一个种属的序列,即异源核酸。外源核酸还可以是与生物体同源的序列(即,核酸序列在该种属中天然存在或编码天然存在于宿主种属中的多肽),该序列已被重新引入该生物体的细胞中。包含同源序列的外源核酸通常可以通过与外源核酸连接的非天然序列(例如重组核酸构建体中同源基因序列侧翼的非天然调控序列)的存在而区别于天然序列。可选地或另外地,稳定地转化的外源核酸可以通过其与已整合的基因组中的序列的并列来检测和/或与天然基因相区分。如果核酸被引入所考虑的细胞、生物体或菌株的祖细胞中,则认为该核酸是外源的。
就多核苷酸而言的“片段”是指多核苷酸分子的克隆或任意部分,特别是保留可用的功能特征的多核苷酸部分。有用的片段包括可用于杂交或扩增技术或复制、转录或翻译的调节的寡核苷酸和多核苷酸。“多核苷酸片段”是指多核苷酸的任意子序列,通常具有本文提供的任意序列的至少约9个连续核苷酸、例如至少约30个核苷酸或至少约50个核苷酸。示例性多核苷酸片段是序列表中列出的多核苷酸的前六十个连续核苷酸。示例性片段可以另外地或可选地包括包含编码多肽的保守CHORD或SKP1家族结构域的区域、基本上由该区域组成或由该区域组成的片段。示例性片段可以另外地或可选地包括包含多肽的保守结构域的片段。
片段可以另外地或可选地包括多肽和蛋白质分子的子序列或多肽的子序列。片段可能因其具有抗原性潜力而有用。在一些情况下,片段或结构域是多肽的子序列,其以与完整多肽基本上相同的方式或相似的程度执行完整多肽的至少一种生物学功能。例如,多肽片段可以包含可识别的结构基序或功能结构域,例如结合DNA启动子区域、激活结构域或蛋白质-蛋白质相互作用结构域的DNA结合位点或结构域,并且可以启动转录。片段的大小可以从少至3个氨基酸残基到完整多肽的全长不等,例如长度为至少约20个氨基酸残基,例如长度为至少约30个氨基酸残基。优选地,片段是具有其所衍生自的多肽的至少一种性质或活性的功能片段,例如,该片段可以包括多肽的功能结构域或保守结构域。结构域可以例如通过Pfam或保守结构域数据库(CDD)指定来表征。
如本文所使用的术语“CHORD衍生多肽”是指包含CHORD结构域的至少60%或包含与天然多肽的CHORD结构域的至少60%具有至少80%同一性的氨基酸序列的多肽,例如所述多肽包含与天然CHORD结构域的至少35个、至少36个、至少37个、至少38个、至少39个或至少40个连续氨基酸具有至少80%同一性的氨基酸序列。在一些具体实施例中,CHORD结构域与SEQ ID NO:4的至少36个连续氨基酸具有至少80%同一性,或者与SEQ ID NO:92的CHORD结构域(第273至338位氨基酸)的至少36个连续氨基酸具有至少80%同一性。在另外的实施例中,所述多肽包含与SEQ ID NO:99的氨基酸序列或其至少35、40、45、50或100个连续氨基酸具有至少80%同一性的氨基酸序列。例如,所述多肽可以与SEQ ID NO:100具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%或95%至100%同一性。
如本文所使用的术语“功能同源物”描述了具有序列相似性并且还共有至少一个功能特征(例如,生物化学活性)的那些分子。功能同源物通常会产生相似但不一定相同的程度的相同特征。功能上同源同源的蛋白质具有相同的特征,其中由一个同源物产生的定量测量值是另一个同源物的至少10%;更通常地,由原始分子产生的定量测量值的至少20%、约30%至约40%;例如,约50%至约60%;约70%至约80%;或约90%至约95%;约98%至约100%,或者大于100%。因此,在分子具有酶活性的情况下,与原始酶相比,功能同源物将具有上述的酶活性百分比。在分子是DNA结合分子(例如,多肽)的情况下,与原始分子相比,同源物将具有如通过结合分子的重量所测量的上述结合亲和力百分比。
功能同源物和参考多肽可以是天然多肽,并且序列相似性可以归因于会聚或发散的进化事件。功能同源物有时被称为直向同源物,其中“直向同源物”是指同源基因或蛋白质,其是另一种属中参考基因或蛋白质的功能等效物。
天然功能同源物的变体(例如,由突变体或野生型编码序列编码的多肽)本身可以是功能同源物。如本文所使用的功能同源物也可以通过对产率调节多肽(例如,CHORD多肽、CHORD衍生多肽或SKP1多肽)的编码序列进行定点诱变或通过组合来自不同天然CHORD多肽、CHORD衍生多肽或SKP1多肽的编码序列的结构域来产生。术语“功能同源物”有时应用于编码功能上同源的多肽的核酸。
功能同源物可以通过分析核苷酸和多肽序列比对来鉴定。例如,对核苷酸或多肽序列的数据库进行查询可以鉴定生物质调节多肽的同源物。序列分析可涉及使用生物质调控多肽的氨基酸序列作为参考序列的非冗余数据库的BLAST、互补BLAST或PSI-BLAST分析。在一些情况下,氨基酸序列是从核苷酸序列推导出来的。通常,数据库中具有大于40%序列同一性的那些多肽是进一步评定作为生物质调节多肽的适合性的候选物。氨基酸序列相似性允许保守的氨基酸替换,例如将一个疏水性残基替换成另一个疏水性残基或将一个极性残基替换成另一个极性残基。如果需要的话,可以对这些候选物进行人工检查,以缩小有待进一步评价的候选物的数量。可以通过选择那些似乎具有存在于产率调节多肽中的结构域(例如,保守的功能结构域)的候选物来进行人工检查。
术语“基因”广义上用于指编码多肽或表达的RNA的核酸分子(通常是DNA,但任选地为RNA)的任意片段。因此,基因包括编码表达的RNA(其可以包括多肽编码序列或例如功能性RNA,例如核糖体RNA、tRNA、反义RNA、微小RNA、短发夹RNA、核酶等)的序列。基因可以进一步包含其表达所需要的或影响其表达的调控序列,以及与其天然状态的蛋白质或RNA编码序列相关的序列,例如内含子序列、5′或3′非翻译序列等。在一些实施例中,“基因”可能仅指DNA或RNA分子的蛋白质编码部分,其可能包含或可能不包含内含子。基因的长度优选为大于50个核苷酸,更优选地长度大于100个核苷酸,并且可以是例如长度为50个核苷酸至500,000个核苷酸,例如长度为100个核苷酸至100,000个核苷酸、或长度为约200个核苷酸至约50,000个核苷酸、或长度为约200个核苷酸至约20,000个核苷酸。基因可以获自多种来源,包括从目标来源克隆或从已知或预测的序列信息合成。
当用于指多核苷酸、基因、核酸、多肽或酶时,术语“异源的”是指不是来源于宿主的种属(例如,来源于相对于宿主细胞而言不同的种属)的多核苷酸、基因、核酸、多肽或酶。例如,用异源去饱和酶基因转化用来自四爿藻属微生物或来自植物的脂肪酸去饱和酶的编码序列转化的转基因微拟球藻属微生物。当提及在核酸构建体或分子中可操作地连接或以其它方式彼此连接的核酸序列时,如本文所使用的“异源序列”是天然地不彼此可操作地连接或彼此不连续的那些。例如,认为来自四爿藻属某种的启动子对于微拟球藻属编码区序列而言是异源的。而且,认为来自编码来自微拟球藻属的CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样基因的基因的启动子对于编码微拟球藻属脂肪酸去饱和酶的序列而言是异源的。类似地,当提及用于维持或操纵基因序列的基因调控序列或辅助核酸序列(例如,启动子、增强子、5′非翻译区、3′非翻译区、Kozak序列、polyA附加序列、内含子序列、剪接位点、核糖体结合位点、内部核糖体进入序列、基因组同源区、重组位点等)时,“异源的”意指调控序列或辅助序列与调节或辅助核酸序列在构建体、基因组、染色体或附加体中与之并列的基因来自不同的来源(例如,不同的基因,无论来自与宿主生物体相同或不同的种属)。当提及蛋白质功能结构域例如定位序列或受体结合位点时,“异源的”还可以意味着蛋白质功能结构域与在经改造的蛋白质中与之并列的蛋白质区的其余部分来自不同的来源(例如,蛋白质)。类似地,当提及经改造基因的启动子序列时,“异源的”意味着启动子源自与通过基因改造与之连接的基因不同的基因。
此外,当提及多核苷酸、基因、核酸、多肽或酶时使用的术语“异源的”是指来自于或来源于与宿主生物体种属不同的来源的多核苷酸、基因、核酸、多肽或酶。相反,“同源的”多核苷酸、基因、核酸、多肽或酶在本文中用于指源自宿主生物体种属的多核苷酸、基因、核酸、多肽或酶。当提及用于维持或操纵基因序列的基因调控序列或辅助核酸序列(例如,启动子、5′非翻译区、3′非翻译区、polyA附加序列、内含子序列、剪接位点、核糖体结合位点、内部核糖体进入序列、基因组同源区、重组位点等),“异源的”意味着调控序列或辅助序列并非与在构建体、基因组、染色体或附加体中与调节或辅助核酸序列并列的基因天然地相关联。因此,可操作地连接于与不在其天然状态下(即,在未经遗传改造的生物体的基因组中)可操作地连接的基因的启动子在本文中称为“异源启动子”,即使启动子可以来源于与其所连接的基因相同的种属(或在某些情况下,相同的生物体)。
当用于指多核苷酸、基因、核酸、多肽或酶时,术语“同源的”是指来源于宿主种属的多核苷酸、基因、核酸、多肽或酶,例如来自相对于宿主细胞而言相同的种属,无论同源多核苷酸、基因、核酸、多肽或酶是否已经被引入到宿主细胞(外源的)中,或者相对于宿主而言是内源的。
如本文所使用的“分离的”核酸或蛋白质是从其天然环境或天然存在核酸或蛋白质的环境中移出。例如,分离的蛋白质或核酸分子是从其自然或天然环境中与其相关的细胞或生物体中移出。在一些情况下,分离的核酸或蛋白质可以是部分或基本上纯化的,而对分离不要求特定的纯化水平。因此,例如,分离的核酸分子可以是从染色体、基因组或天然地整合到其中的附加体切除的核酸序列。
“纯化的”核酸分子或核苷酸序列或蛋白质或多肽序列基本上不含细胞物质和细胞组分。例如,纯化的核酸分子或蛋白质可以不合缓冲剂或溶剂以外的化学试剂。“基本上不含”无意于指检测不到除新核酸分子以外的其它组分。在一些情况下,“基本上不含”可以指核酸分子或核苷酸序列不含至少95%(w/w)的细胞物质和组分。
术语“天然的”在本文中用于指在宿主中天然存在的核酸序列或氨基酸序列。术语“非天然的”在本文中用于指不在宿主中天然存在或不以其在宿主中天然构造的方式构造的核酸序列或氨基酸序列。已经从宿主细胞中移出、经历实验室操纵且被引入或重新引入到宿主细胞中使得其在基因组中的序列或位置相对于其在未经操纵的生物体中的位置不同(即,与在未转化的生物体中未并列或可操作地连接的序列并列或可操作地连接)的核酸序列或氨基酸序列被视为“非天然的”。引入宿主细胞中的合成的或部分合成的基因是“非天然的”。非天然基因还包括可操作地连接到已经重组到宿主基因组中的一个或多个异源调控序列的宿主微生物的内源基因,或者位于基因组中不同于其天然基因座的基因座的宿主生物体的内源基因。
术语“天然存在”和“野生型”是指在自然界中发现的形式。例如,天然存在或野生型核酸分子、核苷酸序列或蛋白质可以存在于天然来源中并且从天然来源分离得到,而不是通过人工操纵有意修饰的。
术语“核酸”或“核酸分子”是指DNA或RNA(例如,mRNA)的区段,并且还包括具有经修饰的骨架(例如,肽核酸、锁核酸)或经修饰的或非天然核碱基的核酸。核酸分子可以是双链的或单链的;包含基因或其部分的单链核酸分子可以是编码(有义)链或非编码(反义)链。
核酸分子或序列可以“来源于”指定的来源,其包括从指定来源分离(全部或部分)核酸区段。核酸分子或序列还可以来自指定的来源,例如通过直接克隆、PCR扩增或从指定的多核苷酸来源或基于与指定的多核苷酸来源相关的序列人工合成,其可以是例如生物体种属。来源于特定来源或种属的基因或核酸分子或序列(例如,启动子)也包括相对于源核酸分子具有序列修饰的基因或核酸分子或序列。例如,源自来源(例如,特定的参考基因)的基因或核酸分子或序列可以包括相对于源基因或核酸分子的非有意的或有意的引入的一个或多个突变,并且如果有意引入一个或多个突变(包括替换、缺失或插入),则可以通过细胞或核酸的随机或定向突变、通过扩增或其它基因合成或分子生物学技术、或通过化学合成、或其任意组合来引入序列突变。在一些实施例中,所述序列可以相对于其衍生自的核酸序列被截短或内部缺失,例如,启动子可以相对于其所衍生自的天然启动子被缩短或内部缺失。源自参考基因或核酸分子或编码功能性RNA或多肽的序列的基因或核酸分子或序列可以编码与参考或源功能性RNA或多肽或者其功能性片段具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%或至少95%序列同一性的功能性RNA或多肽。例如,源自参考基因或核酸分子或编码功能性RNA或多肽的序列的基因或核酸分子或序列可以编码与参考或源功能性RNA或多肽或其功能性片段具有至少85%、至少90%、至少95%、至少96%、至少97%、至少98%或至少99%的序列同一性的功能性RNA或多肽。
“外源核酸分子”或“外源基因”是指已被引入(“转化”)到细胞中的核酸分子或基因。可以将转化的细胞称为重组细胞,可以向该转化的细胞中引入其他外源基因。如果核酸分子转化的细胞的后代继承了外源核酸分子,则也将其称为“转化的”。相对于被转化的细胞,外源基因可以来自不同的种属(因此是“异源的”),或来自相同的种属(因此是“同源的”)。“内源性”核酸分子、基因或蛋白质是天然核酸分子、基因或蛋白质,因为其存在于宿主中或由宿主天然产生。
如本文所使用的“分离的”核酸或蛋白质是从其天然环境或天然存在核酸或蛋白质的环境中移出。例如,分离的蛋白质或核酸分子是从其自然或天然环境中与其相关的细胞或生物体中移出。在一些情况下,分离的核酸或蛋白质可以是部分或基本上纯化的,而对分离不要求特定的纯化水平。因此,例如,分离的核酸分子可以是从染色体、基因组或天然地整合到其中的附加体切除的核酸序列。
“纯化的”核酸分子或核苷酸序列或蛋白质或多肽序列基本上不含细胞物质和细胞组分。例如,纯化的核酸分子或蛋白质可以基本上不含缓冲剂或溶剂以外的化学试剂。“基本上不含”无意于指检测不到除新核酸分子以外的其它组分。
如本文所使用的“可操作地连接”旨在表示两个或更多个序列之间的功能性连接,使得一个序列处或一个序列的活性影响其它序列处或其它序列的活性。例如,目标多核苷酸和调控序列(例如,启动子)之间的可操作的连接是允许目标多核苷酸表达的功能性连接。在这个意义上,术语“可操作地连接”是指调控区和待转录的编码序列的定位,使得调控区对于调控目标编码序列的转录或翻译是有效的。例如,为了可操作地连接编码序列和调控区,编码序列的翻译阅读框的翻译起始位点通常位于调控区的下游约1至约50个核苷酸之间。然而,调控区可以位于翻译起始位点上游约5000个核苷酸处,或者在转录起始位点上游约2000个核苷酸处。可操作地连接的元件可以是连续的或不连续的。当用于指两个蛋白质编码区的连接时,“有效连接”意指编码区处于相同的阅读框中。当用于指增强子的作用时,“可操作地连接”表示增强子增加特定多肽或目的多核苷酸的表达。
如本文所使用的“序列同一性百分比”或“百分比(%)[序列]同一性”是通过在比较窗口中比较两个最佳局部比对的序列来确定的,该比较窗口由这两个序列之间的局部比对长度来定义。(也可以认为其是同源性百分比或“同源性百分数(%)”)。关于两个序列的最佳比对,比较窗口中的氨基酸序列相较于参考序列(其不包含添加或缺失)可以包含添加或缺失(例如,空位或突出端)。两个序列之间的局部比对仅包括根据取决于用于执行比对的算法(例如,BLAST)的标准被认为是足够相似的每个序列的区段。通过确定在两个序列中出现相同核酸碱基或氨基酸残基的位置的数目来计算同一性百分比,以产生相匹配位置的数目,将相匹配位置的数目除以所比较的窗口中的总位置数目并将结果乘以100。用于比较的序列的最佳比对可以通过Smith和Waterman的局部同源性算法(Add.APL.Math.2:482,1981)、Needleman和Wunsch的全局同源性比对算法(J.Biol.Biol.48:443,1970)、Pearson和Lipman的相似性方法搜索(Proc.Natl.Acad.Sci.USA 85:2444,1988)、这些算法的启发式实行方案(NCBI BLAST、WU-BLAST、BLAT、SIM、BLASTZ)或通过检查来进行。例如,可以使用GAP和BESTFIT来确定已被鉴定以用于比较的两个序列的最佳比对。通常,使用如下缺省值:空位权重5.00和空位权重长度0.30。术语多核苷酸或多肽序列之间的“基本序列同一性”是指使用程序与参考序列相比包含具有至少50%的序列同一性的序列的多核苷酸或多肽,例如至少70%、至少80%、至少85%、至少90%、至少95%或至少96%、97%、98%或99%的序列同一性。另外,所使用的配对序列同源性或序列相似性是指两个所比对序列之间相似的残基所占的百分比。具有相似侧链的氨基酸残基家族在本领域中已经被很好地定义。这些家族包括具有碱性侧链的氨基酸(例如赖氨酸、精氨酸、组氨酸)、具有酸性侧链的氨基酸(例如天冬氨酸、谷氨酸)、具有不带电的极性侧链的氨基酸(例如甘氨酸、天冬酰胺、谷氨酰胺、丝氨酸、苏氨酸、酪氨酸、半胱氨酸)、具有非极性侧链的氨基酸(例如丙氨酸、缬氨酸、亮氨酸、异亮氨酸、脯氨酸、苯丙氨酸、甲硫氨酸、色氨酸)、具有β-分支侧链的氨基酸(例如苏氨酸、缬氨酸、异亮氨酸)和具有芳族侧链的氨基酸(例如酪氨酸、苯丙氨酸、色氨酸、组氨酸)。
查询核酸和氨基酸序列是针对存在于公共或专有数据库中的对象核酸或氨基酸序列进行搜索。这样的搜索使用国家生物技术信息基本局部比对搜索工具(NCBI BLAST v 2.18)程序完成。NCBI BLAST程序可以在网上从国家生物技术信息中心(blast.ncbi.nlm.nih.gov/Blast.cgi)获得。通常,使用NCBI BLAST的以下参数:过滤选项设置为“默认”;比较矩阵设置为“BLOSUM62”;空位成本设置为“存在:11,扩展:1”;字体大小设置为3;Expect(E阈值)设置为le-3,以及局部比对的最小长度设置为查询序列长度的50%。序列同一性和相似性还可以使用GENOMEQUESTTM软件(Gene-IT,Worcester,MA,USA)来确定。
“启动子”是指能够在宿主细胞中启动转录并且能够驱动或促进本发明的核苷酸序列或其片段的转录的转录控制序列。这样的启动子不一定是天然序列。另外,应当理解,这样的启动子不一定来源于靶宿主细胞或宿主生物体。术语“启动子”是指能够结合细胞中的RNA聚合酶并启动下游(3′方向)编码序列的转录的核酸序列。启动子包括以高于背景的可检测水平启动转录所必需的最小数目的碱基或元件。启动子可以包括转录起始位点以及负责RNA聚合酶结合的蛋白质结合结构域(共有序列)。真核生物启动子通常但不总是包含“TATA”盒和“CAT”盒。原核生物启动子可含有-10和-35原核生物启动子共有序列。包括来自各种不同来源的组成型、诱导型和抑制型启动子的大量启动子在本领域是公知的。代表性的来源包括例如藻类、病毒、哺乳动物、昆虫、植物、酵母和细菌细胞类型,并且来自这些来源的合适的启动子容易获得,或者可以基于在线公开可得的序列合成制备,或者例如,来自例如ATCC等保藏机构以及其它商业或个人来源。启动子可以是单向的(沿一个方向启动转录)或双向的(沿任一方向启动转录)。启动子可以是组成型启动子、阻抑型启动子或诱导型启动子。除了RNA聚合酶结合以启动转录的基因近端启动子以外,启动子区域可以包括基因上游的其它序列,所述其它序列可以在基因的转录起始位点的1kb、2kb、3kb、4kb、5kb或更多内,其中所述其它序列可以影响下游基因的转录速率,并且可选地影响启动子对发育、环境或生物化学(例如,代谢)状况的反应性。
“多肽”和“蛋白质”在本文中可互换使用,并且指两个或更多个亚基氨基酸、氨基酸类似物或其它肽模拟物的化合物,而无论翻译后修饰例如磷酸化或糖基化为何。亚基可以通过肽键连接,或者在肽模拟物的情况下可以通过诸如酯键或醚键的其它键连接。本定义涵盖全长多肽、截短的多肽、点突变体、插入突变体、剪接变体、嵌合蛋白及其片段。如本文所使用的术语“蛋白质”或“多肽”旨在涵盖单数“多肽”以及复数“多肽”,并且是指由通过酰胺键(也称为肽键)线性连接的单体(氨基酸)构成的分子。术语“多肽”是指两个或更多个氨基酸的任一个或多个链,并且不指产物的特定长度。因此,“多肽”的定义中包括肽、二肽、三肽、寡肽、“蛋白质”、“氨基酸链”或用于指两个或更多个氨基酸的一个或多个链的任意其它术语,可以使用术语“多肽”来代替这些术语中的任一个或与之互换。
如本文所使用的表述“基本上保守的氨基酸序列”是指来自不同来源的相同类型或家族的多肽之间的氨基酸同源性区域。在本发明中,基本上保守的氨基酸序列的实例包括图4和图5中指定为CHORD结构域的那些,以及如图6中突出显示的SKP1家族四聚化和二聚化结构域。本领域技术人员可以对来自不同来源的CHORD样或SKP1样多肽的氨基酸序列与本文所述的CHORD和SKP1多肽序列进行比对,以鉴定其中作为本文所定义的基本上保守的氨基酸序列的区段。然后,本领域技术人员可以确定所鉴定出的区段是否具有在本发明中公开和要求保护的特征。
如本文所使用的“子代”是指生物体的后代、子孙或衍生物。例如,来自转基因藻类的子细胞是转基因藻类的子代。因为某些修饰可能由于突变或环境影响而出现在后代中,所以这样的子代、后代或衍生物实际上可能不与亲本细胞相同,但仍包括在本文所使用的术语的范围内。
如本文在提及核酸分子时所使用的术语“重组”或“改造的”是指通过人为干预已被改变的核酸分子。作为非限制性实例,cDNA是重组DNA分子,如为通过体外聚合酶反应已经产生的或已经连接了接头或已经整合到载体(例如克隆载体或表达载体)中的任意核酸分子。作为非限制性实例,重组核酸分子:1)已经在体外合成或修饰,例如使用核酸分子的化学或酶促技术(例如,采用化学核酸合成,或采用酶进行复制、聚合、核酸外切消化、核酸内切消化、连接、逆转录、转录、碱基修饰(包括例如甲基化)或重组(包括同源和位点特异性重组));2)包括连接在一起的天然不连接的核苷酸序列;3)已经使用分子克隆技术进行改造,使得其相对于天然核酸分子序列缺少一个或多个核苷酸;和/或4)已经使用分子克隆技术操纵,使得其相对于天然核酸序列具有一个或多个序列改变或重排。作为非限制性实例,cDNA是重组DNA分子,如通过体外聚合酶反应已经产生的或已经连接了接头或已经整合到载体(例如克隆载体或表达载体)中的任意核酸分子。
如本文所使用的术语“重组蛋白质”是指通过遗传改造产生的蛋白质,例如通过在细胞中表达经遗传改造的核酸分子。如本文所使用的术语“重组蛋白质”是指通过遗传改造产生的蛋白质,不管氨基酸是否与野生型蛋白质的氨基酸不同。
如本发明中使用的术语“调控区”、“调控序列”、“调控元件”或“调控元件序列”是指影响转录或翻译起始或速率以及转录或翻译产物的稳定性和/或移动性的核苷酸序列。这样的调控区不一定是天然序列。调控序列包括但不限于启动子序列、增强子序列、应答元件、蛋白质识别位点、诱导元件、蛋白质结合序列、5′和3′非翻译区(UTR)、转录起始位点、终止序列、聚腺苷酸化序列、内含子及其组合。调控区通常包含至少一个核心(基础)启动子。调控区还可以包括至少一个控制元件(例如,增强子序列)、上游元件或上游激活区(UAR)。
如本文所使用的“转基因生物体”是指包含异源多核苷酸的生物体,所述异源多核苷酸即已经通过非天然手段(人为干预)引入生物体中的多核苷酸。当应用于生物体时,本文中可互换使用的术语“转基因的”或“重组的”或“经改造的”或“经遗传改造的”是指通过将外源或重组核酸序列引入生物体中而被操纵的生物体。通常,异源多核苷酸被稳定地整合到基因组中,使得多核苷酸被传递到后代,但其也可以存在于附加体上,并且可以存在于转基因生物体的合成染色体上。非天然多核苷酸可以单独整合到基因组中或作为重组表达盒的一部分。在另一些实施例中,转基因微生物可以包括可操作地连接至转基因微生物的内源基因的引入的外源调控序列。这样的操纵的非限制性实例包括基因敲除、靶向突变和基因置换、启动子置换、缺失或插入,以及将转基因引入生物体中。重组或经遗传改造的生物体也可以是引入了用于基因“敲除”的构建体的生物体。这样的构建体包括但不限于RNAi、微小RNA、shRNA、反义和核酶构建体。还包括其基因组已经被大范围核酸酶、锌指核酸酶、TALEN或Crisper核酸酶的活性改变的生物体。如本文所使用的“重组微生物”或“重组宿主细胞”包括本发明重组微生物的后代或衍生物。因为某些修饰可能因突变或环境影响而出现在后代中,事实上,这样的子代或衍生物可能与亲本细胞不同,但仍包括在本文所使用的术语的范围内。
对于核酸和多肽,术语“变体”在本文中用于表示如下多肽、蛋白质或多核苷酸分子,其与参考多肽或多核苷酸相比在其碱基或氨基酸序列中具有合成或天然地产生的一些差异,使得该变体与参考多肽或多核苷酸具有至少70%的序列同一性。在其它实施方案中,变体可以与参考多肽或多核苷酸具有至少80%、至少95%、至少90%或至少95%、至少97%、至少98%或至少99%的序列同一性。例如,这些差异包括参考多肽或多肽中的替换、插入、缺失或这样的变化的任意期望的组合。多肽和蛋白质变体还可以由电荷和/或翻译后修饰(例如,糖基化、甲基化、磷酸化等)的变化组成。
如本文所使用的“载体”是指包含可选择的标记基因或允许载体在宿主细胞中复制的复制起点或自主复制序列(ARS)中的至少一个的核酸分子,并且在一些实施例中包括可选择的标记基因和至少一个复制起点或ARS两者。各种实施例中的载体包含一个或多个表达序列和/或可以包含用于介导重组的至少一个序列。
通常在基因或种属名后的括号中提供的基因和蛋白质登录号是在由美国国立卫生研究院提供的国家生物技术信息中心(NCBI)网站(ncbi.nlm.nih.gov)公开可获得的序列记录的唯一标识符。“GenInfo标识符”(GI)序列标识号对核苷酸或氨基酸序列而言是特定的。如果序列以任意方式改变,则分配新的GI号。序列修订历史工具可用于跟踪出现在特定GenBank记录中的序列的各种GI号、版本号和更新日期。基于登录号和GI号搜索和获得核酸或基因序列或蛋白质序列在例如细胞生物学、生物化学、分子生物学和分子遗传学领域是公知的。
如本文所使用的就核酸或多肽序列而言的术语“同一性百分比”或“同源性”定义为在为了最大同一性百分比和需要时引入空位以获得最大同源性百分比而对序列进行比对之后,候选序列中与已知多肽相同的核苷酸或氨基酸残基所占的百分比。N末端或C末端的插入或缺失不应当被解释为影响同源性,并且多肽序列的内部缺失和/或插入少于约100个、少于约80个、少于约50个、少于约30个、少于约20个、或少于约10个氨基酸残基不应当被解释为影响同源性。核苷酸或氨基酸序列水平的同源性或同一性可以通过BLAST(Basic Local Alignment Search Tool)分析使用程序blastp、blastn、blastx、tblastn和tblastx(Altschul(1997),Nucleic Acids Res.25,3398-3402和Karlin(1990),Proc.Natl.Acad.Sci.USA 87,2264-2268)所采用的算法来确定,上述程序专供序列相似性搜索。BLAST程序所使用的方法是首先考虑查询序列和数据库序列之间在有和无空位的情况下的相似片段,然后评定所有被识别的匹配的统计显著性,最后只总结那些满足预先选定的重要阈值的匹配。对于序列数据库相似性检索中的基本问题的讨论参见Altschul(1994),Nature Genetics 6,119-129。用于直方图、描述、比对、预期(即用于报告与数据库序列匹配的统计显著性阈值)、截止、矩阵和过滤(低复杂度)的搜索参数可以是默认设置。blastp、blastx、tblastn和tblastx使用的默认评分矩阵是BLOSUM62矩阵(Henikoff(1992),Proc.Natl.Acad.Sci.USA 89,10915-10919),推荐用于长度超过85的查询序列(核苷酸碱基或氨基酸)。
对于设计用于比较核苷酸序列的blastn,评分矩阵由M(即,匹配残基对的奖励得分)与N(即,错配残基的罚分)的比率设定,其中M和N的默认值分别为+5和-4。四个blastn参数可以如下调整:Q=10(空位产生罚分);R=10(空位延伸罚分);wink=1(在查询的每个winkth位置产生词条匹配);和gapw=16(设置在其中产生空位对齐的窗口宽度)。用于比较氨基酸序列的等效Blastp参数设置可以是:Q=9;R=2;wink=1;和gapw=32。在GCG软件包10.0版本中可用的序列之间的Bestfit比较可以使用DNA参数GAP=50(空位产生罚分)和LEN=3(空位延伸罚分),并且蛋白质比较中的等效设置可以是GAP=8和LEN=2。
因此,当提及本发明的多肽或核酸序列时,包括与全长多肽或核酸序列或与其包含整个蛋白质的至少50、至少75、至少100、至少125、至少150或更多个氨基酸残基的保持序列的片段具有如下序列同一性:至少40%、至少45%、至少50%、至少55%、至少70%、至少65%、至少70%、至少75%、至少80%、或至少85%、例如至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、至少99%或约100%序列同一性;这样的序列的变体,例如其中至少一个氨基酸残基已被插入所公开的包含插入和替换的序列的N末端和/或C末端和/或其内。所涵盖的变体可以另外地或可选地包括通过例如同源重组或定点诱变或PCR诱变含有预定突变的变体,以及其它种属的相应多肽或核酸,包括但不限于本文所述的那些,等位基因或包含插入和替换的多肽或核酸家族的其它天然变体;和/或衍生物,其中所述多肽已通过用含有插入和替换的天然氨基酸以外的部分(例如,可检测部分例如酶)的替换、化学、酶促或其它适当方式共价地修饰。
如本文所使用的短语“保守的氨基酸替换”或“保守的突变”是指将一种氨基酸用另一种具有共同性质的氨基酸置换。定义单个氨基酸之间共同性质的功能性方式是分析同源生物体的相应蛋白质之间氨基酸变化的归一化频率(Schulz(1979),Principles of Protein Structure,Springer-Verlag)。根据这样的分析,可以定义氨基酸组,其中组内的氨基酸彼此优先交换,并且因此在其对整体蛋白质结构的影响方面彼此最相似(Schulz(1979),Principles of Protein Structure,Springer-Verlag)。以这种方式定义的氨基酸基团的实例可以包括:包括“带电组/极性组”,包括Glu、Asp、Asn、Gln、Lys、Arg和His;“芳族或环状组”,包括Pro、Phe、Tyr和Trp;以及“脂族组”,包括Gly、Ala、Val、Leu、Ile、Met、Ser、Thr和Cys。在每个组中,还可以鉴定子组。例如,可以将带电/极性氨基酸组细分为子组,包括:“带正电的子组”,包括Lys、Arg和His;“带负电的子组”,包括Glu和Asp;和“极性组”,包括Asn和Gln。在另一个实施例中,可以将芳族或环状组细分为子组,包括:氮环子组”,包括Pro、His和Trp;“苯基子组”包括Phe和Tyr。在另一个实施例中,可以将脂族基团细分成子组,包括:“大脂族非极性子组”,包括Val、Leu和Ile;“脂族微极性子组”,包括Met、Ser、Thr和Cys;和“小残基子组”,包括Gly和Ala。保守突变的实例包括上述子组内的氨基酸的氨基酸替换,例如但不限于:Lys替换成Arg,或反之亦然,使得可以保持正电荷;Glu代替成Asp,或反之亦然,使得可以保持负电荷;Ser替换成Thr,或反之亦然,使得保持游离的-OH;以及Gln替换成Asn,或者反之亦然,使得可以保持游离的-NH2。“保守变体”是这样的多肽,其包括已经被替换以置换具有共同性质的氨基酸的参考多肽的一个或多个氨基酸的一个或多个氨基酸(例如,其序列公开于公布或序列数据库中的多肽,或者其序列已经通过核酸测序来确定的多肽),例如,所述参考序列的一个或多个氨基酸属于与上文描述相同的氨基酸组或子组。
如本文所使用的“表达”包括至少在RNA产生水平的基因的表达,并且“表达产物”包括所得产物,例如多肽或功能性RNA(例如,核糖体RNA、tRNA、反义RNA、微小RNA、shRNA、核酶等)。术语“增加的表达”包括为了促进增加的mRNA产生和/或增加的多肽表达的基因表达的改变。与多肽的天然产生或酶活性相比,[基因产物]的“产量增加”包括多肽表达量、多肽酶活性水平或两者的组合的增加。
本发明的一些方面包括特定多核苷酸序列的表达的部分地、大量地、或完全地缺失、沉默、失活或下调。基因可以部分地、大量地或完全地缺失、沉默、失活、或者它们的表达可以被下调以影响由它们所编码的多肽所执行的活性(例如,酶的活性)。基因可以通过插入破坏基因功能和/或表达的核酸序列(例如病毒插入、转座子诱变、大范围核酸酶工程、同源重组或本领域中已知的其它方法)来部分地、大量地或完全地缺失、沉默、失活或下调。术语“消除”、“消减”和“敲除”可以与术语“缺失”、“部分缺失”、“大量缺失”或“完全缺失”互换使用。在某些实施方案中,目标微生物可以通过定点同源重组改造以敲除特定目标基因。在另一些实施方案中,可以使用RNAi或反义DNA(asDNA)构建体来部分地、大量地或完全地沉默、失活或下调特定目标基因。
可以将某些核酸分子或特定多核苷酸序列的这些插入、缺失或其它修饰理解为涵盖“遗传修饰”或“转化”,使得可以将所得到的微生物菌株或宿主细胞理解为“经遗传修饰的”、“经遗传改造的”或“转化的”。
如本文所使用的“上调的”或“上调”包括目标基因或核酸分子的表达或酶的活性的增加,例如与尚未上调的其它相同的基因或酶的表达或活性相比,基因表达或酶活性的增加。
如本文所使用的“下调的”或“下调”包括目标基因或核酸分子的表达或酶的活性的降低,例如与尚未下调的其它相同的基因或酶的表达或活性相比,基因表达或酶活性的降低。
如本文中所用,“突变体”是指在基因中具有突变的生物体,所述突变是传统诱变的结果,例如,使用γ辐射、UV或化学诱变剂。如本文中所用的“突变体”还指由于基因工程改造而具有改变的基因结构或表达的重组细胞,作为非限制性实例,基因工程改造许多包括过表达,包括在不同的暂时性、生物或环境调控下和/或达到不同于自然发生的程度的基因表达和/在重组细胞中不自然表达的基因的表达;同源重组,包括敲除和敲入(例如,用编码比野生型多肽具有更高或更低的活性的多肽和/或显性负性多肽的基因置换基因);经由RNAi、反义RNA或核酶等减弱基因;及使用大范围核酸酶、TALEN和/或CRISPR技术等进行基因组工程改造。因此突变体不是自然存在的生物体。目标突变生物体通常将具有不同于缺乏突变的相应野生型或祖代菌株的表型,其中所述表型可通过生长测定、产物分析、光合特性、生物化学测定等进行评估。当提及基因时“突变体”意指相对于原生或野生型基因而言,该基因具有至少一个碱基(核苷酸)变化、缺失或插入。突变(一个或多个核苷酸的变化、缺失和/或插入)可在基因的编码区内或可在内含子、3’UTR、5’UTR或启动子区内,例如在转录起始位点的2kb以内或在翻译起始位点的3kb以内。作为非限制性实例,突变基因可以是在启动子区内具有可以增加或降低基因表达的插入的基因;可以是具有导致生成非功能性蛋白、截短蛋白、显性负性蛋白或无蛋白质生成的缺失的基因;可以是具有导致所编码的蛋白质的氨基酸变化或导致基因转录物异常剪接等的一个或多个点突变的基因。
术语“Pfam”是指由Pfam联盟维持并可在若干被支持的世界范围的网站获得的蛋白质结构域和蛋白质家族的大集合,包括:pfam.sanger.ac.uk/(Welcome Trust,Sanger Institute);pfam.sbc.su.se(斯德哥尔摩生物信息学中心);pfam.janelia/org/(Janelia农场,霍华德休斯医学研究所);pfam.jouy.inra.fr/(国家农业研究所);和pfam.ccbb.re.kr。Pfam的最新发布版本是基于UniProt蛋白质数据库发布版本2012_06的Pfam 28.0(2015年5月)。使用多重序列比对和隐马尔可夫模型(HMM)来鉴定Pfam结构域和家族。Pfam-A家族或结构域分配是通过使用蛋白质家族的代表成员的策划种子比对和基于种子比对的分布隐马尔可夫模型产生的高质量分配。(除非另有说明,否则所查询的蛋白质与Pfam结构域或家族的匹配是Pfam-A匹配)。然后使用属于该家族的所有鉴定的序列自动产生家族的完全比对(Sonnhammer(1998),Nucleic Acids Research 26,320-322;Bateman(2000)Nucleic Acids Research 26,263-266;Bateman(2004)Nucleic Acids Research 32,Database Issue,D138-D141;Finn(2006)Nucleic Acids Research Database Issue 34,D247-251;Finn(2010)NucleicAcids Research Database Issue 38,D211-222)。通过访问Pfam数据库,例如使用任意上述参考网站,可以使用HMMER同源性搜索软件(例如,HMMER2、HMMER3或更高版本,hmmer.janelia.org/)来针对HMM查询蛋白质序列。将查询的蛋白质鉴定为pfam家族(或具有特定的Pfam结构域)的重要匹配是比特分数大于或等于Pfam结构域的收集阈值的那些匹配。期望值(e值)也可用作在Pfam中包含查询蛋白质的标准或用于确定查询蛋白质是否具有特定Pfam结构域,其中低e值(远小于1.0,例如小于0.1、或小于或等于0.01)表示匹配是由于偶然性的低概率。
如本文所使用的“相同的条件”或“相同的培养条件”是指基本上相同的条件,即,所提及的条件之间的任意差异是微小的并且与对本发明具有实质性的微生物的功能或性质无关,例如不影响脂质生产或生物质生产。
就特定细胞类型而言的“充氮”条件是细胞不经历由于氮不足而造成的生长缺陷的条件。
本文使用的“脂质”或“多种脂质”是指脂肪、蜡、脂肪酸、脂肪酸衍生物例如脂肪醇、蜡酯、烷烃和烯烃、甾醇、甘油单酯、甘油二酯、甘油三酯、磷脂、鞘脂、糖脂和甘油脂。“FAME脂质”或“FAME”是指具有可被衍生为脂肪酸甲酯的酰基部分的脂质,例如单酰甘油酯、二酰甘油酯、三酰甘油酯、蜡酯和膜脂质例如磷脂、半乳糖脂等。可以将脂质产率评定为按毫克每升(mg/L)计的FAME产率,对于藻类,可以报告为克/平方米/天(g/m2/天)。在本文提供的半连续测定中,通过考虑入射辐照的面积(“1 1/2×33/8”的SCPA瓶架开口或0.003145m2)和培养物的体积(550ml),将mg/L值转换成g/m2/天。为了获得按g/m2/天的产率值,将mg/L值乘以每日稀释率(30%)和换算系数0.175。当脂质或其子类别(例如,TAG或FAME)以百分比提及时,该百分比是重量百分比,另有说明除外。
“生物质”是指细胞质量,无论是活细胞还是死细胞,并且可以使用本领域中已知的方法被评定为例如抽吸的沉淀的重量,但更优选为干重(例如,培养样品或沉淀的细胞的冻干物)、无灰干重(AFDW)或总有机碳(TOC)。例如,在生长允许条件下培养物的生长期间生物质增加,并且在分批培养物中可以被称为“生物质积累”。在进行稳定或常规稀释的连续或半连续培养物中,在培养物稀释过程中除去否则将在培养物中积累的所产生的生物质。因此,也可以将这些培养物的日生物质产率(生物质的增加)称为“生物质积累”。生物质产率可以被评定为按毫克/升(mg/L)计的TOC产率,并且对于藻类,可以报告为可/每平方米/天(g/m2/天)。在本文提供的半连续测定中,通过考虑入射辐照的面积(“1 1/2×33/8”的SCPA瓶架开口或0.003145m2)和培养物的体积(550ml),将mg/L值转换成g/m2/天。为了获得按g/m2/天的产率值,将mg/L值乘以每日稀释率(30%)和换算系数0.175。当将生物质表示为百分比时,该百分比是重量百分比,另有说明除外。
在本发明的上下文中,“氮源”是可被主题微生物摄取和代谢并掺入到生物分子中用于生长的氮源。例如,在本发明的上下文中,认为包括不能被微生物摄取和/或代谢的氮的化合物(例如,含氮的生物缓冲剂例如Hepes,Tris等)不是氮源。
本文公开了用于操纵、测定、培养和分析微生物的方法。本文提出的发明还利用本领域中已知的用于细胞培养、微生物转化、遗传改造和生物化学分析的标准方法、技术和试剂。
所有标题都是为了方便读者,并不以任何方式限制本发明。
本说明书中提及的所有出版物和专利申请都通过引用并入本文,其程度如同每个单独的出版物或专利申请被具体地和单独地指出通过引用并入。
不承认任何参考文献构成现有技术。对参考文献的讨论陈述了他们的作者所声称的内容,申请人保留质疑所引用文献的准确性和针对性的权利。将清楚地理解的是,尽管在此引用了许多现有技术出版物,该引用并不构成承认这些文件中的任何一个构成本领域公知常识的一部分。
本文给出的对一般方法的讨论仅仅是为了说明的目的,而无意于是限制性的。在阅读本公开内容之后,其它替代方法和实施方案对于本领域技术人员将是显而易见的。
具有提高的产率的突变体微生物
本发明提供了当突变体微生物和对照微生物在相同条件下培养时,具有与对照微生物相比提高至少5%的生物质产率(例如,AFDW或TOC)和/或相对于对照微生物提高至少5%的脂质产率(例如,FAME)的突变体微生物(例如,每天更高的产率,优选地在整个培养期平均)。可以使用本领域公知的方法将生物质产率评定为例如无灰干重(AFDW)生产或产率(例如,每天产生的量)或总有机碳(TOC)生产或产率。如本文所提供的突变微生物可表现出相对于对照微生物的至少5%的生物质产率提高。例如,生物质产率(例如,AFDW或TOC)可以相对于对照微生物增加约5%至约500%、或约10%至约300%、或约10%至约200%、或约10%至约100%。在各种实施例中,生物质(例如,AFDW或TOC)产率提高可以在1、2、3、4、5、6、7、8、9、10、11、12、13、14或更多天的半连续或持续生长之后测定。可选地或另外地,可以在分批生长至少1、2、3、4、5、6或7天后证明生物质(例如,AFDW或TOC)产率提高。可选地或另外地,在本文中经过可以多达例如1年、180天、90天、30天、14天、7天或5天的一段时间确定产率或生物质产率或生物产物产率。
在一些实施例中,如本文所提供的突变体微生物相对于对照微生物产生更高量的脂质,例如在其中突变体微生物和对照微生物均产生生物质的培养条件下。相对于对照微生物,脂质或FAME产率可以增加至少5%,例如相对于对照微生物增加约5%至约500%、或者10%至约300%、或者约10%至约200%、或约10%至约100%、或约15%至约90%。在一些实施例中,可以在半连续或连续生长1、2、3、4、5、6、7、8、9、10、11、12、13、14或更多天后证实增加的FAME产率增加。可选地或另外地,可以在分批生长至少1、2、3、4、5、6或7天后证明FAME产率增加。在一些实施例中,如本文所提供的突变体微生物在其中突变体微生物和对照微生物均产生生物质并积极分裂的培养条件下相对于对照微生物产生更高量的脂质。
测量由微生物产生的脂质的量的方法在本领域中是公知的,并且提供于本文的实施例中。总可提取脂质可以根据例如Folch等.(1957)J.Biol.Chem.226:497-509;Bligh&Dyer(1959)Can.J.Biochem.Physiol.37:911-917;或Matyash等(2008)J.Lipid Res.49:1137-1146来确定,并且也可以使用傅立叶变换红外光谱(FT-IR)(Pistorius等(208)Biotechnol&Bioengin.103:123-129)来评定作为脂质存在的生物质的百分比。FAME和TAG的重量分析的其它参考例如在US 8,207,363和WO 2011127118中提供,其中每个均通过引用整体并入本文。
生物质可通过测量总有机碳(TOC)或通过其它方法(例如,测量无灰干重(AFDW))来评定。用于测量TOC的方法是本领域已知的(例如,US 8,835,149)并且提供于本文中。测量AFDW的方法也是公知的,并且可以在例如US 8,940,508中找到,其通过引用整体并入本文。
将如本文所提供的具有提高的脂质产率或生物质产率的重组微生物的性质与对照微生物的同一性质进行比较,所述对照微生物可以是与突变体相同种属的野生型生物体,并且优选为过量生产脂质的突变体的祖细胞株。或者,对照微生物可以是与经遗传改造的微生物基本上相同的微生物,除了对照微生物不包含如本文所公开的非天然核酸分子,该非天然核酸分子在重组宿主中的表达导致更高的生物质或脂质产率。例如,对照微生物可以是经遗传改造的微生物或经典地突变的生物体,其已被进一步改造以产生如本文所公开的重组微生物,该重组微生物包含具有增加的生物质(例如,TOC)产率和/或如本文所公开的增加的脂质产率的SKP1或CHORD衍生多肽。
在一些实施例中,对照微生物可以是与包含编码SKP1或CHORD衍生多肽的非天然基因的重组微生物基本上相同的微生物,除了对照微生物不包含如本文所公开的导致增强的生长表型的非天然核酸分子(即,与对照微生物相比,其表达导致提高的生物质(例如AFDW或TOC)产率或提高的脂质(例如,FAME)产率的基因或基因片段)。还将包含本文公开的非天然核酸分子(导致增加的脂质,例如FAME,或生物质,例如AFDW或TOC产率)的增强的产率突变体的性质与不包含所述非天然核酸分子的对照微生物的相同性质(不管所述细胞或微生物是否为“野生型”)进行比较。例如,对照微生物可以是不包含如本文所公开的所述非天然核酸分子的重组微生物,对其效果进行评定等。
本发明的多核苷酸和多肽
在本发明的一个方面,本公开内容提供了分离的或重组的核酸分子、干扰这些核酸分子的核酸分子以及与这些核酸分子杂交的核酸分子。本申请的另外的方面包括由本发明的分离的或重组的核酸分子编码的多肽。
CHORD蛋白质和CHORD衍生多肽
可以通过CHORD结构域的序列特征来鉴定CHORD蛋白。CHORD结构域是长度为约60个氨基酸的结合两个锌离子并且通常串联排列的模块,即典型的CHORD蛋白质包括至少两个CHORD结构域。在CHORD结构域内,六个半胱氨酸残基和两个组氨酸残基是不变的。其它三个残基也是不变的且一些位置被限制为阳性氨基酸、阴性氨基酸或芳族氨基酸。在一些情况下,CHORD结构域具有共有序列C-x(4)-C-x(12-13)-C-x(2)-H-x(14)-CC-x(15-16)-C-x(4)-H,其中“C”表示半胱氨酸;“H”表示组氨酸;“x(n)”表示一串“n”个氨基酸残基,其中氨基酸残基“x”是任意氨基酸残基。CHORD多肽可以通过本领域已知的方法鉴定,例如通过PCR(例如,使用与保守序列例如CHORD结构域同源的简并引物)通过杂交进行计算机同源性搜索(例如,BLAST搜索)、基因组测序和生物信息学分析等。在公共在线数据库中可以获得大量的基因组序列,包括可以搜索SKP1和CHORD基因的NCBI(国家生物技术信息中心)。
如本文所提供的分离的或重组的核酸分子具有编码多肽的序列,所述多肽具有与选自以下的富含半胱氨酸和组氨酸的结构域(CHORD)蛋白具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少96%、至少97%、至少98%或至少99%同一性的氨基酸序列:SEQ ID NO:22、SEQ ID NO:91、SEQ ID NO:92、SEQ ID NO:93、SEQ ID NO:94、SEQ ID NO:95、SEQ ID NO:96及其片段。在各种实施例中,如本文所提供的分离的或重组的核酸分子编码“CHORD衍生多肽”,其包含天然CHORD蛋白的CHORD结构域的至少一部分或与之具有至少80%、至少85%、至少90%、至少95%、至少96%、至少97%、至少98%或至少99%同一性的氨基酸序列。例如,CHORD衍生多肽可以具有不完整的CHORD结构域,例如CHORD结构域的至少60%但不是100%,并且可以具有例如天然CHORD结构域的至少35、至少36、至少37、至少38、至少39或至少40个连续氨基酸或与之具有至少80%、至少85%、至少90%、至少95%、至少96%、至少97%、至少98%或至少99%同一性的氨基酸序列。CHORD衍生多肽可以包含源自天然CHORD蛋白的其他氨基酸序列和/或可以包括并非来源于天然CHORD蛋白的氨基酸序列。例如,CHORD衍生多肽可以包含与非CHORD蛋白的氨基酸序列融合的CHORD蛋白的一部分,使得CHORD衍生多肽包含源自天然CHORD蛋白的氨基酸序列(其可以包括CHORD结构域的至少一部分)以及并非来源于CHORD蛋白的另外的氨基酸序列。一些实施例中的CHORD蛋白片段可以包含SEQ ID NO:4、SEQ ID NO:99或SEQ ID NO:100的氨基酸序列。
本文提供的分离的或重组的核酸分子或如本文所公开的重组微生物的非天然核酸分子可以编码与植物或微生物种属的CHORD多肽(例如植物、微藻或异鞭毛种属的CHORD多肽)的至少一部分具有至少50%、至少55%、至少60%、至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少96%、至少97%、至少98%或至少99%同一性的CHORD衍生多肽。在各种实施例中,本文公开的核酸分子包含编码与藻类或异鞭毛种属的天然多肽的至少一部分具有至少65%的序列同一性的CHORD衍生多肽的核酸序列,例如,与藻类或异鞭毛种属的天然多肽的CHORD结构域具有至少85%的序列同一性。可选地或另外地,核酸序列可以编码与SEQ ID NO:22、SEQ ID NO:91、SEQ ID NO:92、SEQ ID NO:93、SEQ ID NO:94、SEQ ID NO:95或SEQ ID NO:96的CHORD结构域具有至少50%、至少55%、至少60%、至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少96%、至少97%、至少98%或至少99%同一性的CHORD衍生多肽。在一些实施例中,CHORD衍生多肽可以包含SEQ ID NO:4、SEQ ID NO:99或SEQ ID NO:100的氨基酸序列或与SEQ ID NO:4、SEQ ID NO:99或SEQ ID NO:100具有至少50%、至少55%、至少60%、至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少96%、至少97%、至少98%或至少99%同一性的氨基酸序列。
SKP1多肽
本文提供的分离的或重组的核酸分子具有编码多肽的序列,所述多肽具有与S期激酶相关蛋白1(SKP1)(例如,天然SKP1蛋白例如经改造的宿主微生物的SKP1蛋白)具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少96%、至少97%、至少98%或至少99%同一性的氨基酸序列。
例如,如本文所提供的非天然核酸分子可包括编码SKP1多肽的核酸序列,其与选自以下的SKP1多肽具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少96%、至少97%、至少98%或至少99%同一性:SEQ ID NO:28、SEQ ID NO:65、SEQ ID NO:66、SEQ ID NO:67、SEQ ID NO:68、SEQ ID NO:69、SEQ ID NO:70、SEQ ID NO:71、SEQ ID NO:72、SEQ ID NO:73、SEQ ID NO:74、SEQ ID NO:75、SEQ ID NO:76、SEQ ID NO:77、SEQ ID NO:78、SEQ ID NO:79、SEQ ID NO:80、SEQ ID NO:81、SEQ ID NO:82、SEQ ID NO:83、SEQ ID NO:84、SEQ ID NO:85、SEQ ID NO:86、SEQ ID NO:87、SEQ ID NO:88、SEQ ID NO:89和SEQ ID NO:90。
SKP1是SCF复合物的成员,与包含F-box的蛋白质相结合,并且参与泛素蛋白降解途径。如本文所提供的,可以通过SKP1家族四聚化和二聚化结构域的序列特征来鉴定SKP1蛋白。当针对Pfam数据库查询时,包含SKP1家族四聚化结构域的多肽可募集至pfam PF03931,例如具有大于收集截留值(21.9)的比特分数和小于1.00E-2或小于1.00E-10的E值。当针对Pfam数据库查询时,包含SKP1家族二聚化结构域的多肽可募集至pfam PF01466,例如具有比聚集截止值(21.2)更大的比特分数,以及小于1.00E-2或小于1.00E-10的E值。示例性SKP1多肽包含SKP1家族四聚化结构域(pfam PF03931)和SKP1家族二聚化结构域(pfam PF01466)两者。可通过本领域已知的方法鉴定SKP1多肽,例如通过PCR(例如,使用与保守序列同源的简并引物例如SKP1家族二聚化结构域或SKPl家族四聚化结构域)通过杂交等进行计算机同源性搜索(例如,BLAST搜索)、基因组测序和生物信息学分析。
如本文所提供的分离的或重组的核酸分子可编码与植物或微生物种属的SKP1多肽(例如,植物、微藻或异鞭毛种属的SKP1多肽)具有至少50%、至少55%、至少60%、至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少96%、至少97%、至少98%或至少99%同一性的多肽。在各种实施例中,本文公开的核酸分子包含编码与藻类或异鞭毛种属的天然多肽具有至少65%的序列同一性的SKP1多肽的核酸序列,例如与藻类或异鞭毛种属的天然多肽具有至少85%的序列同一性的SKP1多肽的核酸序列。可选地或另外地,核酸序列可以编码与下列各者具有至少50%、至少55%、至少60%、至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少96%、至少97%、至少98%或至少99%同一性的多肽:SEQ ID NO:28、SEQ ID NO:65、SEQ ID NO:66、SEQ ID NO:67、SEQ ID NO:68、SEQ ID NO:69、SEQ ID NO:70、SEQ ID NO:71、SEQ ID NO:72、SEQ ID NO:73、SEQ ID NO:74、SEQ ID NO:75、SEQ ID NO:76、SEQ ID NO:77、SEQ ID NO:78、SEQ ID NO:79、SEQ ID NO:80、SEQ ID NO:81、SEQ ID NO:82、SEQ ID NO:83、SEQ ID NO:84、SEQ ID NO:85、SEQ ID NO:86、SEQ ID NO:87、SEQ ID NO:88、SEQ ID NO:89和SEQ ID NO:90及其片段。
在一些实施例中,本文所提供的分离的或重组的核酸分子或本文所公开的重组微生物的非天然核酸分子可以具有不同于(即,不是100%相同)天然基因的核苷酸序列,和/或分离的或重组的核酸分子可以是cDNA。例如,本文所提供的分离的或重组的核酸分子可以包括缺失在包含所述基因的生物体的基因组中发现的一个或多个插入的非编码序列(内含子)的蛋白质编码区,并且可以包括所述基因的两个或更多个连续的蛋白质编码序列,其中所述两个或更多个序列被生物体的未改变的基因组中的内含子分开。例如,核酸分子可以包含cDNA,其中cDNA包含与在天然生物体的基因组中发现的序列不同的序列。可选地或另外地,核酸分子可以包含编码蛋白质的基因,其包含与未经遗传修饰的生物体的基因组中的核酸分子的蛋白质编码部分不连续的5′非翻译区。可选地或任意上述另外地,核酸分子可以具有相对于生物体基因组中的天然基因的序列具有一个或多个核苷碱基变化的序列。例如,核酸分子可以具有相对于生物体基因组中的天然基因的序列具有一个或多个核碱基替换、缺失或添加的序列。
另外,当在微生物宿主细胞中表达时,如本文所提供的分离的或重组的核酸分子(例如,如本文中公开的非天然核酸分子)可为微生物宿主细胞赋予更高的产率,尤其是脂质例如FAME,和生物质例如AFDW或TOC。在一些实施例中,微藻或异鞭毛细胞中如本文公开的核酸分子的表达可以导致微藻或异鞭毛细胞具有比不表达核酸分子的对照细胞更高的产率,特别是脂质例如FAME和生物质例如AFDW或TOC,例如,微生物宿主细胞可表现出更高的生长速率、更高的生物质产率或更高的生物分子(例如脂质、蛋白质、色素或碳水化合物,包括醇)生产速率或水平。例如,相对于对照细胞,宿主细胞可表现出更高的产率,特别是脂质例如FAME和生物质例如AFDW或TOC,宿主细胞的经改造以合成一种或多种产物。
可以使用重组DNA技术(例如,逆转录、限制性、连接、聚合酶反应,包括聚合酶链式反应(PCR)扩增、克隆、体外或体内重组等中的任意一种或任意组合)或化学合成来产生本发明的分离的核酸分子。分离的核酸分子包括天然核酸分子及其同源物,包括但不限于天然等位基因变体和经修饰的核酸分子,在经修饰的核酸分子中,核苷酸已被插入、缺失和/或替换,以使得这样的修饰为如本文所述的多肽的生物学活性提供期望的效果。
可以使用本领域技术人员已知的多种方法来产生核酸分子变体(参见例如Sambrook等,Molecular Cloning:A Laboratory Manual.第2版.N.Y.,Cold Spring Harbor Laboratory,Cold Spring Harbor Laboratory Press,1989)。例如,可以使用多种技术来修饰核酸分子,所述技术包括但不限于经典诱变技术和重组DNA技术,例如定点诱变、对核酸分子进行化学处理以诱导突变、核酸片段的限制性酶切、核酸片段的连接、核酸序列的选择区域的PCR扩增和/或诱变、寡核苷酸混合物的合成和混合基团的连接以“构建”核酸分子的混合物及其组合。核酸分子同源物可以通过筛选由核酸编码的蛋白质的功能和/或通过与野生型基因杂交而从经修饰的核酸的混合物中选择。
根据本申请的一些实施方案,本发明的核酸分子将包括那些在高度严格条件下与编码多肽的核酸分子特异性杂交或杂交的核酸分子,所述多肽在中度严格或高度严格的条件下与下列各者具有至少50%、至少55%、至少60%、至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少96%、至少97%、至少98%或至少99%同一性:SEQ ID NO:4、SEQ ID NO:99、SEQ ID NO:100、SEQ ID NO:22、SEQ ID NO:91、SEQ ID NO:92、SEQ ID NO:93、SEQ ID NO:94、SEQ ID NO:95、SEQ ID NO:96或SEQ ID NO:101、SEQ ID NO:102、SEQ ID NO:28、SEQ ID NO:65、SEQ ID NO:66、SEQ ID NO:67、SEQ ID NO:68、SEQ ID NO:69、SEQ ID NO:70、SEQ ID NO:71、SEQ ID NO:72、SEQ ID NO:73、SEQ ID NO:74、SEQ ID NO:75、SEQ ID NO:76、SEQ ID NO:77、SEQ ID NO:78、SEQ ID NO:79、SEQ ID NO:80、SEQ ID NO:81、SEQ ID NO:82、SEQ ID NO:83、SEQ ID NO:84、SEQ ID NO:85、SEQ ID NO:86、SEQ ID NO:87、SEQ ID NO:88、SEQ ID NO:89和SEQ ID NO:90及其片段和其互补序列及其片段。
如本文所使用,如果两个分子能够形成反向平行的双链核酸结构,则称这两个分子能够彼此特异性杂交。如果两个分子能够在至少常规的低严格条件下以足够的稳定性彼此杂交以使它们保持彼此退火,则称这两个分子最低限度互补。类似地,如果分子能够在常规的高度严格条件下以足够的稳定性彼此杂交以使它们保持相互退火,则称分子是互补的。如果核酸分子表现出完全的互补性,则称其为另一核酸分子的互补序列。如本文所使用,当分子之一的每个核苷酸与另一个分子的核苷酸互补时,则称分子表现出完全的互补性。只要这样的偏离不能完全排除分子形成双链结构的能力,则允许背离完全互补。因此,为了使本发明的核酸分子或其片段充当引物或探针,它只需要在序列上足够互补以便能够在所采用的特定溶剂和盐浓度下形成稳定的双链结构。
常规的严格条件描述于Sambrook等(同上)和Haymes等,Nucleic Acid Hybridization,A Practical Approach,IRL Press,Washington,D.C.(1985)中。例如,促进DNA杂交的合适的严格条件包括例如在约45℃下6.0×氯化钠/柠檬酸钠(SSC),随后在50℃下以2.0×SSC洗涤。另外,洗涤步骤的温度可以从约22℃的室温下的低严格条件增加到约65℃的高严格条件。温度和盐两者都可以变化,或者温度或盐浓度中的任一者可以保持恒定,而另一变量被改变。这些和其它条件是本领域技术人员已知的,或者可见于Current Protocols in Molecular Biology,John Wiley&Sons,N.Y.(1989),6.3.1-6.3.6。低严格条件可用于选择与靶核酸序列具有较低序列同一性的核酸序列。人们可能希望采用如下条件:在约20℃至约55℃范围内的温度下例如约0.15M至约0.9M氯化钠。可以采用高度严格的条件来选择与所公开的核酸序列具有更高程度的同一性的核酸序列(Sambrook等,1989,同上)。高度严格的条件通常包括在约2×至约10×SSC中(从含有3M氯化钠和0.3M柠檬酸钠的20×SSC储液(pH 7.0于蒸馏水中)稀释)、约2.5×至约5×Denhardt氏溶液(从含有1%(w/v)牛血清白蛋白、1%(w/v)聚蔗糖和1%(w/v)聚乙烯吡咯烷酮在蒸馏水中的50×储液稀释)、约10mg/mL至约100mg/mL鱼精子DNA和约0.02%(w/v)至约0.1%(w/v)SDS的核酸杂交,并在约50℃至约70℃孵育数小时至过夜。高度严格条件优选由6×SSC、5×Denhardt氏溶液、100mg/mL鱼精子DNA和0.1%(w/v)SDS提供,并在55℃孵育数小时。通常在杂交之后进行数个洗涤步骤。洗涤组合物一般包含0.5×至约10×SSC和0.01%(w/v)至约0.5%(w/v)SDS,在约20℃至约70℃孵育15分钟。优选地,核酸片段在65℃于0.1×SSC中洗涤至少一次后保持杂交。
本发明的核酸分子的子集包括由至少12个、至少15个、例如至少16个或17个、或例如至少18个或19个、例如至少20个或更多个保守核苷酸组成的所公开的多核苷酸的片段。这样的寡核苷酸是具有选自序列表中多核苷酸序列的序列的较大分子的片段,并且可用作例如干扰分子、用于检测本发明的多核苷酸的探针和引物。
本发明的核酸分子的最小大小是足以形成探针或寡核苷酸引物的大小,所述探针或寡核苷酸引物能够与可用于本发明的核酸分子的互补序列形成稳定的杂交体(例如,在中度、高度或极高的严格条件下),或其大小足以编码具有根据本发明的多肽,例如本文公开的CHORD、CHORD衍生的、SKP1和其它CHORD样及其它SKP1样多肽的至少一个结构域的生物学活性的氨基酸序列。如此,编码这样的蛋白质的核酸分子的大小可取决于核酸组成和核酸分子与互补序列之间的同源性或同一性百分比以及杂交条件本身(例如温度、盐浓度和甲酰胺浓度)。用作寡核苷酸引物或探针的核酸分子的最小大小通常为:如果核酸分子富含GC,则长度为至少约12至约15个核苷酸,如果核酸分子富含AT,则长度为至少约15至约18个碱基。除了实际限制以外,对本发明核酸分子的最大大小没有限制,因为核酸分子可以包括足以编码CHORD、CHORD衍生的或SKP1多肽的结构域的生物活性片段、整个CHORD、CHORD衍生的或SKP1多肽或编码CHORD、CHORD衍生的或SKP1多肽的开放阅读框中的数个结构域的序列。
在多个实施例中,本发明提供了包含编码多肽的区的核苷酸序列,所述多肽可以是由所述多核苷酸表示的基因编码的完整蛋白质,或者可以是编码的蛋白质的片段。例如,本文提供的多核苷酸可编码构成完整蛋白质的主要部分或其一个结构域的多肽,例如构成完整蛋白质的足够部分以提供相关的生物学活性,例如CHORD结构域或其部分的活性。特定目标是编码CHORD结构域的至少35个连续氨基酸的本发明的多核苷酸,其可以任选地以与其它非CHORD序列的融合蛋白形式提供。这样的多核苷酸可以在转基因细胞或转基因生物体中表达以产生具有较高产率的细胞和生物体,例如较高生物质例如AFDW或TOC或脂质例如FAME产率。
此外,如本文所提供的核酸分子,包括包含编码CHORD、CHORD衍生的或SKP1多肽的序列或其片段的核酸分子,可以在重组宿主细胞中表达,并且可以测定核酸分子的表达对生物体产率特别是脂质例如FAME和生物质例如AFDW或TOC的影响进行评定。可以例如通过生长测定(例如通过细胞计数或光密度监测生殖)、通过测定随时间积累的无灰干重的总有机碳(TOC)或者通过使用本领域中所用的方法(包括但不限于气相色谱(GC)、HPLC、免疫学检测、生物化学和/或酶检测等)评定任何目标产物(例如蛋白质、碳水化合物、脂质、色素等)的量来测量产率。
本发明中还关注的是本文提供的多核苷酸的变体。这样的变体可以是天然的、包括来自相同或不同种属的同源多核苷酸,或者可以是非天然变体,例如使用化学合成方法合成的、或使用重组DNA技术产生的多核苷酸。就核苷酸序列而言,遗传密码的简并性提供了用不同碱基替换基因的蛋白质编码序列的至少一个碱基的可能性,而不引起由基因产生的多肽的氨基酸序列发生改变。因此,本发明的DNA也可以具有通过根据遗传密码的简并进行替换而从序列表中的任何多核苷酸序列改变的任何碱基序列。描述密码子使用的参考资料易于得到。
此外,本领域技术人员将进一步理解,可通过本发明的核苷酸序列的突变来引入改变,从而导致编码的CHORD、CHORD衍生的或SKP1蛋白的氨基酸序列的变化,而不改变蛋白质的生物活性。因此,可以通过将一个或多个核苷酸替换、添加或缺失引入本文公开的相应核苷酸序列而产生变体分离的核酸分子,使得将一个或多个氨基酸替换、添加或缺失引入编码的蛋白质中。可以通过标准技术引入突变,例如定点诱变和PCR介导的诱变。这样的变体核苷酸序列也涵盖在本发明中。
例如,可以对一个或多个预测的非必需氨基酸残基进行保守氨基酸替换。如本文所使用的“非必需”氨基酸残基是可以从目前公开的CHORD、CHORD衍生蛋白、CHORD样蛋白、SKP1或SKP1样蛋白的野生型序列改变的残基,而不改变生物活性,而“必需”氨基酸残基为生物活性所需。“保守氨基酸替换”是其中氨基酸残基被具有相似侧链的氨基酸残基取代的替换。具有相似侧链的氨基酸残基家族在本领域中已经被很好地定义。这些家族包括具有碱性侧链的氨基酸(例如赖氨酸、精氨酸、组氨酸)、具有酸性侧链的氨基酸(例如天冬氨酸、谷氨酸)、具有不带电的极性侧链的氨基酸(例如甘氨酸、天冬酰胺、谷氨酰胺、丝氨酸、苏氨酸、酪氨酸、半胱氨酸)、具有非极性侧链的氨基酸(例如丙氨酸、缬氨酸、亮氨酸、异亮氨酸、脯氨酸、苯丙氨酸、甲硫氨酸、色氨酸)、具有β-分支侧链的氨基酸(例如苏氨酸、缬氨酸、异亮氨酸)和具有芳族侧链的氨基酸(例如酪氨酸、苯丙氨酸、色氨酸、组氨酸)。
在一个特定的非限制性示例中,所公开的CHORD蛋白的保守残基、结构域和基序示于图4中,并且可以在序列表的序列中被识别。在一个特定的非限制性示例中,所公开的SKP1蛋白的保守残基、结构域和基序示于图6中,并且可以在序列表的序列中被识别。如上所讨论的,本领域技术人员将理解,可以在保留多肽功能的非保守区域中进行氨基酸替换。一般而言,对保守氨基酸残基或保守基序内的氨基酸残基不进行这样的替换,其中这样的残基对于蛋白质活性可能是必需的。对于蛋白质活性而言保守的和可能是必不可少的残基的实例包括例如在本发明的氨基酸序列与已知的CHORD、CHORD衍生的或SKP1蛋白质序列的比对中所含的所有蛋白质之间相同的残基。保守的但可能允许保守的氨基酸替换并仍保留活性的残基的实例包括例如在本发明的氨基酸序列与已知的CHORD、CHORD衍生的或SKP1序列的比对中所含的所有蛋白质之间仅具有保守替换的残基。然而,本领域技术人员会理解,功能变体可能在保守残基中具有较小的保守或非保守的改变。
CHORD变体包括具有如下所述氨基酸序列的蛋白质,该氨基酸序列与SEQ ID NO:4、SEQ ID NO:99、SEQ ID NO:22、SEQ ID NO:91、SEQ ID NO:92、SEQ ID NO:93、SEQ ID NO:94、SEQ ID NO:95和SEQ ID NO:96中的多肽中的任一个的不同之处为在相应于本文所述的保守氨基酸残基的一个或多个位置处的至少一个氨基酸缺失、插入或替换及其任意组合。在一些优选的实施方案中,这样的CHORD变体包括具有与序列表中任意一种多肽不同之处在于如下所述的氨基酸序列的蛋白质:如此前所鉴定的在对应于保守氨基酸残基的一个或多个位置处的氨基酸缺失、插入或替换及其任意组合。
SKP1变体包括具有与下列各者任意一种多肽不同的氨基酸序列的蛋白质:SEQ ID NO:28、SEQ ID NO:65、SEQ ID NO:66、SEQ ID NO:67、SEQ ID NO:68、SEQ ID NO:69、SEQ ID NO:70、SEQ ID NO:71、SEQ ID NO:72、SEQ ID NO:73、SEQ ID NO:74、SEQ ID NO:75、SEQ ID NO:76、SEQ ID NO:77、SEQ ID NO:78、SEQ ID NO:79、SEQ ID NO:80、SEQ ID NO:81、SEQ ID NO:82、SEQ ID NO:83、SEQ ID NO:84、SEQ ID NO:85、SEQ ID NO:86、SEQ ID NO:87、SEQ ID NO:88、SEQ ID NO:89和SEQ ID NO:90,不同之处在于在对应于本文所述的保守氨基酸残基的一个或多个位置处的至少一个氨基酸缺失、插入或替换及其任意组合。在一些优选的实施方案中,这样的SKP1变体包括具有与序列表中任意一种多肽不同的氨基酸序列的蛋白质,不同之处在于在对应于此前所鉴定的保守氨基酸残基的一个或多个位置处的氨基酸缺失、插入或替换及其任意组合。
可选地或另外地,可以通过如下方式来制备变体核苷酸序列:沿全部或部分编码序列随机引入突变,例如通过饱和诱变;随后可以筛选所得突变体赋予CHORD衍生蛋白、CHORD样蛋白或SKP1样蛋白的活性的能力,以分别鉴定保留CHORD或SKP1蛋白活性的突变体。例如,在诱变后,可以重组表达编码的蛋白质,并且可以使用如上所公开的标准测定技术来测定蛋白质的活性。
这样的操作的方法在本领域中是已知的。例如,CHORD或SKP1蛋白的氨基酸序列变体可以通过DNA的突变来制备。这也可以通过几种形式的诱变和/或定向进化来完成。在一些方面,氨基酸序列中编码的变化不会实质上影响蛋白质的功能。这样的变体将具有期望的CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样活性。然而,应当理解,CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样赋予产率提高的能力,特别是脂质例如FAME和生物质例如AFDW或TOC,可由在本发明的组合物上采用这种技术来改进。例如,可以在DNA复制过程中表现出高碱基错误整合率的宿主细胞中表达CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样多肽,所述宿主细胞例如Stratagene XL-1 Red细胞(Fischer Scientific)。在这些菌株或细胞中繁殖后,可以分离CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样蛋白或CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样编码DNA(例如通过制备质粒DNA,或通过PCR扩增并将得到的PCR片段克隆到载体中),接着在非突变菌株或细胞中培养突变的CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样蛋白或CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样基因,并例如通过进行测定以测试CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样的体内和体外活性来鉴定具有提高宿主细胞产率、尤其是脂质例如FAME和生物质例如AFDW或TOC产率的能力的突变的CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样蛋白或CHORD、CHORD衍生的、CHORD样、SKPQ或SKP1样基因。
可选地或另外地,可以对许多蛋白质的氨基或羧基末端的蛋白质序列进行改变,而基本上不影响活性。这可以包括通过现代分子方法例如PCR引入的插入、缺失或改变,包括借助于在PCR扩增中使用的编码寡核苷酸中的序列的氨基酸来改变或延伸蛋白质编码序列的PCR扩增。或者,所添加的蛋白质序列可以包括完整的蛋白质编码序列,例如本领域常用于产生蛋白质融合体的那些。这样的融合蛋白通常用于(1)增加目标蛋白质的表达;(2)引入结合结构域、酶活性或表位以促进蛋白质纯化、蛋白质检测或本领域已知的其它实验用途;(3)靶向蛋白质向亚细胞器例如革兰氏阴性菌的周质间隙或真核细胞的内质网的分泌或翻译,后者通常导致蛋白质的糖基化。
结构域交换或改组是产生改变的CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样蛋白质的另一种机制。保守的结构域可以在CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样蛋白质之间交换,导致具有提高的生物质(例如AFDW或TOC)产率的杂交或嵌合CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样多肽。用于产生重组蛋白质并测试其提高的生物质(例如AFDW或TOC)产率的方法在本领域中是已知的。因此,本发明的分子还包括两种或更多种CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样基因或多肽之间的融合物。不同基因或多肽的不同结构域可以融合。CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样基因融合体可以直接连接,或者可以通过连接两个或多个融合伴侣的附加氨基酸连接。
基因融合可以通过碱性重组DNA技术产生,其实例在下文中有描述。基因融合的选择将取决于由基因融合引起的期望的表型。例如,如果与一个CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样蛋白的A结构域相关的表型期望地具有与第二CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样蛋白的B结构域相关的表型,则将产生第一CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样蛋白的A结构域与第二CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样蛋白的B结构域的融合体。随后可以在体外或体内测试融合体的期望的表型。
CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样多肽也涵盖在本发明中。在该方面的一个实施方案中,“CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样多肽”欲指具有包含序列表中的任一个氨基酸序列(例如,SEQ ID NO:4、SEQ ID NO:99或SEQ ID NO:100、SEQ ID NO:22、SEQ ID NO:91、SEQ ID NO:92、SEQ ID NO:93、SEQ ID NO:94、SEQ ID NO:95、SEQ ID NO:96、SEQ ID NO:28、SEQ ID NO:65、SEQ ID NO:66、SEQ ID NO:67、SEQ ID NO:68、SEQ ID NO:69、SEQ ID NO:70、SEQ ID NO:71、SEQ ID NO:72、SEQ ID NO:73、SEQ ID NO:74、SEQ ID NO:75、SEQ ID NO:76、SEQ ID NO:77、SEQ ID NO:78、SEQ ID NO:79、SEQ ID NO:80、SEQ ID NO:81、SEQ ID NO:82、SEQ ID NO:83、SEQ ID NO:84、SEQ ID NO:85、SEQ ID NO:86、SEQ ID NO:87、SEQ ID NO:88、SEQ ID NO:89和SEQ ID NO:90)或其片段或变体的氨基酸序列的多肽。还提供了其片段、生物活性部分及变体,并且可以用于实践本发明的方法。
改变或改进的变体:预期可以通过多种方法改变CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样的DNA序列及各同源物,并且这些改变可以产生编码具有不同于由本发明的CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样基因编码的氨基酸序列的蛋白质的DNA序列。CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样蛋白质可以以多种方式改变,包括序列表中给出的多肽序列的一个或更多个氨基酸的氨基酸替换、缺失、截短和插入,包括多达约2、约3、约4、约5、约6、约7、约8、约9、约10、约15、约20、约25、约30、约35、约40、约45、约50、约55、约60、约65、约70、约75、约80、约85、约90、约100、约105、约110、约115、约120、约125、约130个或更多个氨基酸替换、缺失或插入。
还考虑的是与SEQ ID NO:4、SEQ ID NO:99或SEQ ID NO:100、SEQ ID NO:22、SEQ ID NO:91、SEQ ID NO:92、SEQ ID NO:93、SEQ ID NO:94、SEQ ID NO:95、SEQ ID NO:96、SEQ ID NO:28、SEQ ID NO:65、SEQ ID NO:66、SEQ ID NO:67、SEQ ID NO:68、SEQ ID NO:69、SEQ ID NO:70、SEQ D NO:71、SEQ ID NO:72、SEQ ID NO:73、SEQ ID NO:74、SEQ ID NO:75、SEQ ID NO:76、SEQ ID NO:77、SEQ ID NO:78、SEQ ID NO:79、SEQ ID NO:80、SEQ ID NO:81、SEQ ID NO:82、SEQ ID NO:83、SEQ ID NO:84、SEQ ID NO:85、SEQ ID NO:86、SEQ ID NO:87、SEQ ID NO:88、SEQ ID NO:89和SEQ ID NO:90或其片段或保守结构域例如CHORD结构域、SKP1家族四聚化结构域或SKP1家族二聚化结构域具有至少约50%、至少55%、至少60%、至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少96%、至少97%、至少98%或至少99%同一性的多肽。就结构属性而言,所述多肽优选是具生物活性,例如多肽与抗体结合的能力或结合靶核苷酸序列(或与另一分子竞争这样的结合)的能力。可选地或另外地,这样的属性可以是具催化性并且因此涉及分子介导化学反应(对于酶蛋白质)或转录调节反应(对于转录因子)或结构(对于较大复合物的蛋白质亚单位)的能力。本发明的多肽和多肽也可以是重组的。
一般而言,蛋白质或结构域的生物活性或生物作用是指如由体内(即,在蛋白质的天然生理学环境中)或体外(即,在实验室条件下)测量或观察到的该蛋白质或结构域所呈现或执行的归因于该蛋白质的天然形式的任意功能。如本文所使用的CHORD、CHORD衍生的或SKP1多肽的功能结构域是能够执行CHORD、CHORD衍生的或SKP1多肽的生物学功能的结构域。例如,如本文所分别讨论的,CHORD、CHORD衍生的或SKP1多肽和构成CHORD、CHORD衍生的或SKP1多肽的单个结构域的生物学活性包括CHORD结构域或SKP1家族四聚化结构域或SKP1家族二聚化。还考虑的是CHORD、CHORD衍生的或CHORD样多肽片段,其充当阻断天然全长CHORD或CHORD样多肽的功能的显性阴性多肽。
可以使用本领域公知的多种方法中的任一种来制造或获得一种或多种上述多肽。本发明的多肽可以是化学合成的,或者可以使用标准重组技术在异源表达系统例如大肠杆菌、酵母、昆虫等中制备多肽。本发明多肽的抗体或其变体或片段也涵盖在内。用于生产抗体的多种技术和方法在本领域中是公知的(参见例如Harlow和Lane(1988)Antibodies:A Laboratory Manual,Cold Spring Harbor Laboratory,Cold Spring Harbor,NY;第4,196,265号美国专利),并且可以用于制备根据本文公开的本发明的抗体。
核酸构建体
本发明的另一个方面涉及重组核酸分子,所述重组核酸分子包含编码如下氨基酸序列的核酸序列,所述氨基酸序列具有如本文所述的CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样多肽的至少一个结构域的生物学活性。通常,这样的重组核酸分子包括与一个或多个转录控制序列可操作地连接的至少一个本发明的核酸分子。如本文所使用的短语“重组分子”或“重组核酸分子”主要指与转录控制序列可操作地连接的核酸分子或核酸序列,但这样的核酸分子是本文所讨论的重组分子时,其可与短语“核酸分子”互换使用。
本发明提供了包含如本文提供的核酸序列的核酸构建体,所述核酸序列可操作地连接至一个或多个可调节或介导核苷酸序列转录、翻译或整合至宿主基因组中的序列。例如,本发明提供了表达构建体,其包含一个或多个“表达控制元件”或调控可操作地连接的基因的表达转录或转录的RNA的翻译的序列。例如,表达控制元件可以是可以在表达构建体或“表达盒”中可操作地连接至目标基因或反义序列的启动子。多种藻类启动子是已知的并且可以使用,包括在第US 2013/0023035号美国专利申请公开、2012年6月1日提交的美国专利申请13/486,930、2012年12月4日提交的美国专利申请13/693,585以及2013年6月11日提交的美国专利申请13/915,522中公开的那些。构建体中使用的启动子在一些情况下可以是可调控的,例如诱导型的。
诱导型启动子可以响应于例如光强度或高或低的温度,和/或可以响应于特定化合物。诱导型启动子可以是例如激素响应型启动子(例如,蜕皮激素响应型启动子,例如第6,379,945号美国专利中所描述的)、金属硫蛋白启动子(例如第6,410,828号美国专利)、可响应于诸如水杨酸、乙烯、硫胺素和/或BTH的化学品的病原体相关(PR)启动子(第5,689,044号美国专利)等或它们的一些组合。诱导型启动子也可以响应于光或暗(例如第8,318,482号美国专利;第5,750,385号美国专利;第5,639,952号美国专利)、金属(Eukaryotic Cell 2:995-1002(2003))或温度(第5,447,858号美国专利;Abe等,Plant Cell Physiol.49:625-632(2008);Shroda等,Plant J.21:121-131(2000)。就可以使用的启动子类型或特定启动子而言,前述实例是非限制性的。启动子序列可以来自任意生物体,只要它在宿主生物体中具有功能性即可。在某些实施方案中,通过将来自已知的诱导型启动子的一个或多个部分或结构域与可以在宿主细胞中起作用的不同启动子的至少一部分融合来形成诱导型启动子,例如以为在宿主种中起作用的启动子赋予诱导性。
在核酸构建体不含有与编码目标基因(例如,CHORD衍生的或SKP1基因)的核酸序列可操作地连接的启动子的方面,可以将核酸序列转化到细胞中,以使其通过例如同源重组、位点特异性整合和/或载体整合可操作地连接至内源性启动子。在一些情况下,包含于核酸构建体中用以介导同源重组入宿主基因组中的宿主基因组序列可以包含基因调控序列,例如启动子序列,其可以调控基因或核酸构建体的反义或RNAi序列的表达。在这样的实例中,构建体的转基因可以可操作地连接至对于宿主微生物内源的启动子。内源启动子可以是可调控的,例如诱导型的。
本发明的重组核酸分子还可以含有其他调控序列,例如翻译调控序列、复制起点以及与重组细胞相容的其它调控序列。在一个实施方案中,本发明的重组分子(包括整合到宿主细胞染色体中的那些分子)还包含分泌信号(即,信号区段核酸序列),以使得能够从产生蛋白质的细胞分泌表达的蛋白质。合适的信号区段包含与待表达的蛋白质天然相关联的信号区段或根据本发明能够指导蛋白质分泌的任意异源信号区段。在另一个实施方案中,本发明的重组分子包含前导序列以使表达的蛋白质能够递送至并插入宿主细胞的膜中。
合适的前导序列包括与蛋白质天然相关联的前导序列,或任何能够指导蛋白质递送和插入细胞膜中的异源前导序列。在另一些实施方案中,本发明的重组分子包含细胞器靶向信号以使表达的蛋白质能够被转运并递送至靶细胞器。本领域技术人员将会理解,可以使用多种细胞器靶向信号,包括但不限于核定位信号(NLS)、叶绿体靶向信号和线粒体靶向序列。
如本文所述的核酸分子可以被克隆到合适的载体中并且可以用于转化或转染任何合适的宿主。载体和构建它们的方法的选择是本领域公知的并且在一般技术参考文献(参见例如Sambrook和Russell,Molecular Cloning,A Laboratory Manual,Cold Spring Harbor LaboratoryPress,2001)中有所描述。因此,在本发明的一些实施方案中,重组核酸分子是重组载体。根据本发明,重组载体是经改造的(即人工产生的)核酸分子,其被用作操纵选择的核酸序列和将此核酸序列引入宿主细胞的工具。所述重组载体因此适合用于克隆、测序和/或以其它方式操纵选择的核酸序列,例如通过将选择的核酸序列表达和/或递送到宿主细胞中以形成重组细胞。这样的载体通常含有异源核酸序列。载体可以是原核生物或真核生物的RNA或DNA,并且通常是质粒。载体可以保持为染色体外元件(例如质粒),或者可以将其整合到重组生物体(例如,微生物或植物)的染色体中。整个载体可原位保留在宿主细胞内,或在某些条件下,质粒DNA可被缺失,留下本发明的核酸分子。整合的核酸分子可以在染色体启动子控制下、在天然或质粒启动子控制下、或在几个启动子控制的组合下。核酸分子的一个或多个拷贝可整合到染色体中。本发明的重组载体可以含有一种或多种可选择的遗传标记。
在另一个实施方案中,用于本发明的重组核酸分子中的重组载体是靶向载体。如本文所使用的短语“靶向载体”用于指用于将特定核酸分子递送到重组宿主细胞中的载体,其中所述核酸分子用于缺失或灭活宿主细胞或微生物内的内源基因(即用于靶向基因破坏、修饰或敲除技术)。这样的载体也可以在本领域中被称为“敲除”载体。在该实施方案的一个方面,通常是插入载体中的核酸分子(即插入片段)的载体的一部分具有与宿主细胞中的靶基因(即靶向被修饰、缺失或失活的基因)的核酸序列同源的核酸序列。载体插入物的核酸序列被设计为与靶基因相结合,使得靶基因和插入物经历同源重组,由此内源靶基因被修饰、缺失、失活或减弱(即,使至少部分内源性靶基因突变或缺失)。
用于同源重组进入藻类或异鞭毛基因组(例如用于调控基因的破坏或基因置换)的构建体可以包括CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样基因或直向同源物的核苷酸序列同源,例如本文中提供的任一个,或与宿主生物体中的CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样基因相邻的来自藻类或异鞭毛基因组的序列。例如,用于同源重组的构建体可包括靶向敲除或基因替代的基因的至少50、至少100、至少200、至少300、至少400、至少500、至少600、至少700、至少800、至少900、至少1,000、至少1,200、至少1,500、至少1,750或至少2,000个核苷酸,所述基因例如CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样基因或直向同源物,例如本文公开的任一个同源,和/或与其相邻的基因组DNA。例如,用于介导构建体中的同源重组的序列可以包括来自编码CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样多肽的天然藻类或异鞭毛基因或与之相邻的一个或多个核苷酸序列,其中所述CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样多肽包含与序列表中的氨基酸序列中的任一个具有至少40%、例如至少45%、至少50%、至少55%、至少60%、至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%或至少99%同一性的氨基酸序列。在示例性实施方案中,构建体可以包含序列表中的任一个核酸序列的至少50、至少100、至少200、至少300、至少400、至少500、至少600、至少700、至少800、至少900、至少1,000、至少1,200、至少1,500、至少1,750或至少2,000个核苷酸,和/或相应基因组的相邻区域。
例如,用于介导构建体中的同源重组的序列可以包括来自编码CHORD、CHORD-衍生的、CHORD-样、SKP1或SKP1样多肽的天然藻类或异鞭毛基因或与之相邻的一个或多个核苷酸序列,其中CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样多肽包含与下列各者中的任一个具有至少80%、例如至少85%、至少90%、至少95%或至少99%同一性的氨基酸序列:SEQ ID NO:4、SEQ ID NO:99或SEQ ID NO:100、SEQ ID NO:22、SEQ ID NO:91、SEQ ID NO:92、SEQ ID NO:93、SEQ ID NO:94、SEQ ID NO:95、SEQ ID NO:96、SEQ ID NO:28、SEQ ID NO:65、SEQ ID NO:66、SEQ ID NO:67、SEQ ID NO:68、SEQ ID NO:69、SEQ ID NO:70、SEQ ID NO:71、SEQ ID NO:72、SEQ ID NO:73、SEQ ID NO:74、SEQ ID NO:75、SEQ ID NO:76、SEQ ID NO:77、SEQ ID NO:78、SEQ ID NO:79、SEQ ID NO:80、SEQ ID NO:81、SEQ ID NO:82、SEQ ID NO:83、SEQ ID NO:84、SEQ ID NO:85、SEQ ID NO:86、SEQ ID NO:87、SEQ ID NO:88、SEQ ID NO:89和SEQ ID NO:90。例如,用于同源重组的构建体可以包括例如本文中公开的任意编码CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样多肽的调控基因的至少50、至少100、至少200、至少300、至少400、至少500、至少600、至少700、至少800、至少900、至少1,000、至少1,200、至少1,500、至少1,750或至少2,000个核苷酸,和/或与其相邻的基因组DNA。例如,用于介导构建体中的同源重组的序列可以包括来自编码CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样蛋白质的天然藻类或异鞭毛基因或与之相邻的一个或多个核苷酸序列,其中,CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样蛋白质包含与以下中的任一个具有至少40%、例如至少45%、至少50%、至少55%、至少60%、至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%或至少99%的同一性的氨基酸序列:SEQ ID NO:4、SEQ ID NO:99或SEQ ID NO:100、SEQ ID NO:22、SEQ ID NO:91、SEQ ID NO:92、SEQ ID NO:93、SEQ ID NO:94、SEQ ID NO:95、SEQ ID NO:96、SEQ ID NO:28、SEQ ID NO:65、SEQ ID NO:66、SEQ ID NO:67、SEQ ID NO:68、SEQ ID NO:69、SEQ ID NO:70、SEQ ID NO:71、SEQ ID NO:72、SEQ ID NO:73、SEQ ID NO:74、SEQ ID NO:75、SEQ ID NO:76、SEQ ID NO:77、SEQ ID NO:78、SEQ ID NO:79、SEQ ID NO:80、SEQ ID NO:81、SEQ ID NO:82、SEQ ID NO:83、SEQ ID NO:84、SEQ ID NO:85、SEQ ID NO:86、SEQ ID NO:87、SEQ ID NO:88、SEQ ID NO:89和SEQ ID NO:90。在示例性实施方案中,构建体可以包含序列表中示出的CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样和/或相应基因组的相邻区域的核酸序列中的任一个的至少50、至少100、至少200、至少300、至少400、至少500、至少600、至少700、至少800、至少900、至少1,000、至少1,200、至少1,500、至少1,750或至少2,000个核苷酸。
以上关于重组核酸分子和宿主细胞转化的一般性讨论旨在应用于本文讨论的任意重组核酸分子,包括编码具有来自CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样多肽的至少一个结构域的生物活性的任意氨基酸序列的那些,编码来自其它CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样多肽的氨基酸序列的那些和编码其它蛋白质或结构域的那些。
序列表中的信息
对序列表中提供的氨基酸序列进行了注释以指示各序列的一个或数个已知的同源物。一些序列包含指示特定功能和/或应用的“Pfam”结构域。特定的Pfam结构域通过各种来源更详细地描述,例如“sanger.ac.uk”或“pfam.janelia.org”。因此,基于与已知序列的相似性,序列表中的氨基酸序列的各种实际应用对于本领域技术人员而言是显而易见的。
还对序列表中提供的氨基酸序列进行注释以指示各序列的一个或数个已知的同源物。一些氨基酸序列含有保守结构域,例如CHORD结构域,其募集到pfam PF04968。申请人已经在本文所述的多肽中鉴定的指示SKP1家族成员的保守结构域包括募集至pfam PF03931的SKP1家族四聚化结构域和募集至Pfam PF01466的SKP1家族二聚化结构域。
序列应用的附加信息来自与公共数据库中序列的相似性。序列表中标记为“NCBI GI:”和“NCBI Desc”的“杂项特征”部分的条目提供了关于各同源序列的附加信息。在一些情况下,可从www.ncbi.nlm.nih.gov检索到的相应的公共记录引用具有指示注释序列使用的数据的出版物。本文所附的序列描述和序列表符合37C.F.R.§1.182-1.185中所给出的支配专利申请中的核苷酸和/或氨基酸序列公开内容的规定。
从序列表的公开内容可以看出,根据相应的各序列,本发明的核苷酸和多肽可用于制备具有一种或多种改变的生长和表型特征的转基因生物体,所述特征例如提高的产率,例如提高的生物质(例如AFDW或TOC)产率或提高的脂质(例如,FAME)产率。本发明进一步涵盖编码上述多肽的核苷酸,例如包括在序列表中的那些,以及其互补序列和/或片段,并且包括基于遗传密码简并性的其替代方案。
重组微生物
本发明还提供了包含非天然核酸分子的重组微生物,所述非天然核酸分子包含编码CHORD、CHORD衍生的或SKP1蛋白的核酸序列,其中所述重组微生物具有比对照微生物更高的产率,例如,更高的生物产率,例如AFDW或TOC产率,和/或脂质产率,例如FAME产率,对照微生物与重组微生物基本上相同,不同之处在于对照微生物不包含含有编码CHORD、CHORD衍生的或SKP1蛋白的核酸序列的非天然核酸分子。CHORD、CHORD衍生的或SKP1蛋白可以是任意CHORD、CHORD衍生的或SKP1蛋白,例如,其序列可得自基因、蛋白质或基因组数据库或科学文献者,或其变体。在一些实施例中,如本文所提供的重组微生物可包括如本文所提供的编码SKP1蛋白的非天然核酸分子,例如,可包含编码与重组微生物的内源性SKP1具有至少95%同一性的多肽的核酸序列。在一些实施例中,如本文所提供的重组微生物可以包含如本文所提供的编码CHORD衍生蛋白的非天然核酸分子,例如,可以包含编码包含如下氨基酸序列的多肽的核酸序列,所述氨基酸序列与CHORD结构域或重组微生物的内源CHORD多肽的CHORD结构域的至少60%具有至少95%同一性。
在各种实施例中,如本文所提供的重组微生物包含非天然基因,所述非天然基因编码具有如下氨基酸序列的多肽,所述氨基酸序列与选自以下的CHORD、CHORD衍生的或SKP1多肽具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少96%、至少97%、至少98%或至少99%同一性:SEQ ID NO:4、SEQ ID NO:99、SEQ ID NO:100、SEQ ID NO:22、SEQ ID NO:91、SEQ ID NO:92、SEQ ID NO:93、SEQ ID NO:94、SEQ ID NO:95、SEQ ID NO:96、SEQ D NO:28、SEQ ID NO:65、SEQ ID NO:66、SEQ ID NO:67、SEQ ID NO:68、SEQ ID NO:69、SEQ ID NO:70、SEQ ID NO:71、SEQ ID NO:72、SEQ ID NO:73、SEQ ID NO:74、SEQ ID NO:75、SEQ ID NO:76、SEQ ID NO:77、SEQ ID NO:78、SEQ ID NO:79、SEQ ID NO:80、SEQ ID NO:81、SEQ ID NO:82、SEQ ID NO:83、SEQ ID NO:84、SEQ ID NO:85、SEQ ID NO:86、SEQ ID NO:87、SEQ ID NO:88、SEQ ID NO:89和SEQ ID NO:90。在一些实施例中,所述非天然基因编码具有CHORD、CHORD衍生的或SKP1多肽或其功能结构域的多肽,其中所述多肽与微藻或异鞭毛种属的CHORD、CHORD衍生的或SKP1多肽具有至少50%、至少55%、至少60%、至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少96%、至少97%、至少98%或至少99%同一性。重组微生物可表现出更高的产率,例如更高的生物质产率,例如AFDW或TOC产率,并且还可表现出比对照微生物更高的脂质产率,例如更高的FAME产率,所述对照微生物与包含编码具有CHORD、CHORD衍生的或SKP1多肽的非天然基因的重组微生物基本上相同,不同之处在于对照微生物不包含编码具有CHORD、CHORD衍生的或SKP1多肽或其片段的非天然基因。例如,非天然基因在藻类或异鞭毛细胞中的表达可导致藻类或异鞭毛细胞产生更大量的生物质或更大量的一种或多种生物分子,例如但不限于脂质、萜类化合物、聚酮化合物、蛋白质、肽、一种或多种氨基酸、碳水化合物、醇、核酸、一种或多种核苷酸、核苷或核碱基、维生素、辅因子、激素、抗氧化剂或者色素或着色剂。
具有编码具有CHORD、CHORD衍生的或SKP1多肽的多肽的非天然基因的重组微生物可包含例如本文所述的任意编码多肽的核酸分子,所述多肽包含CHORD、CHORD衍生的或SKP1多肽或其功能结构域。此外,重组宿主细胞可以包含本文所述的任意构建体或载体。在一些方面,编码多肽的核酸序列可以对于重组宿主细胞是异源的,并且可以是编码源自任意种属的CHORD、CHORD衍生的或SKP1多肽的基因,所述种属包括植物、动物或微生物种属,或其变体。或者,编码CHORD、CHORD衍生的或SKP1多肽的基因可以与宿主生物体同源。例如,非天然CHORD、CHORD衍生的或SKP1基因可以是与宿主微生物相同种属的CHORD、CHORD衍生的或SKP1基因,并且在允许调控的表达或过表达同源CHORD、CHORD衍生的或SKP1基因的表达盒中引入重组微生物中。或者,CHORD、CHORD衍生的或SKP1非天然基因对于微生物而言可以是内源性的,并且可以将异源启动子引入宿主微生物中,使得其与内源性CHORD、CHORD衍生的或SKP1基因并列且可操作地连接以实现过表达和/或受调控的表达。
本领域技术人员将会理解,许多转化方法可以用于微生物的遗传转化,因此可以用于本发明的方法。“稳定转化”意指引入生物体中的核酸构建体整合到生物体的基因组中,或者是稳定的游离型构建体的一部分,并且能够被其子代遗传。“瞬时转化”意指多核苷酸被引入生物体中并且不整合到基因组中或以其它方式被建立和由后代稳定地遗传。
遗传转化可导致来自细胞核或质体的转基因的稳定插入和/或表达,并且在一些情况下可导致转基因的瞬时表达。例如,据报道,已有30种以上不同的微藻菌株成功地进行了微藻的遗传转化,所述微藻菌株属于至少约22种绿藻、红藻和褐藻、硅藻、裸藻(euglenid)和双鞭毛藻(dianoflagellate)(参见例如Radakovits等,Eukaryotic Cell,2010;和Gong等,J.Ind.Microbiol.Biotechnol.,2011)。这种有用的转化方法的非限制性实例包括在玻璃珠或碳化硅晶须存在下对细胞进行搅动,如由例如Dunahay,Biotechniques,15(3):452-460,1993;Kindle,Proc.Natl.Acad.Sci.USA,1990;Michael和Miller,Plant J.,13,427-435,1998中所报道。电穿孔技术已经成功地用于数种微藻种属的遗传转化,包括微拟球藻属某种(参见,例如,Chen等,J.Phycol.,44:768-76,2008)、小球藻属某种(参见,例如,Chen等,Curr.Genet.,39:365-370,2001;Chow和Tung,Plant Cell Rep.第18卷,第9期,778-780,1999);衣藻属(Shimogawara等,Genetics,148:1821-1828,1998);杜氏藻属(Sun等,Mol.Biotechnol.,30(3):185-192,2005)。基因枪轰击(也称为微粒轰击、基因枪转化或生物弹道轰击)已经成功地用于数种藻类种属,包括例如硅藻种属,例如褐指藻属(Apt等,Mol.Gen.Genet.,252:572-579,1996)、小环藻属和舟形藻属(Dunahay等,J.Pycol.,31:1004-1012,1995)、筒柱藻属(Cylindrotheca)(Fischer等,J.Pycol.,35:113-120,1999)和角毛藻属某种(Chaetoceros sp.)(Miyagawa-Yamaguchi等,Phycol.Res.59:113-119,2011)以及绿藻种属例如小球藻属(El-Sheekh,Biologia Plantarum,第42卷,第2期:209-216,1999)和团藻属(Jakobiak等,Protist,155:381-93,2004)。此外,农杆菌介导的基因转移技术也可用于微藻的遗传转化,例如Kumar,PlantSci.,166(3):731-738,2004和Cheney等,J.Phycol.,第37卷,增刊11,2001中所报道。
如本文所述的转化载体将通常包含赋予靶宿主细胞(例如藻类细胞)可选择的或可分化的表型的标记基因。已经成功地开发出了许多可选择的标记以有效地分离藻类的遗传转化体。常见的可选择的标记包括抗生素抗性、荧光标记和生化标记。已经成功地使用了数种不同的抗生素抗性基因来选择微藻转化体,包括灭瘟素(blastocydin)、博来霉素(参见例如Apt等,1996,同上;Fischer等,1999,同上;Fuhrmann等,Plant J,19,353-61,1999,Lumbreras等,Plant J.,14(4):441-447,1998;Zaslavskaia等,J.Pycol.,36:379-386,2000)、壮观霉素(Cerutti等,Genetics,145:97-110,1997;Doetsch等,Curr.Genet.,39,49-60,2001;Fargo,Mol.Cell.Biol.,19:6980-90,1999)、链霉素(Berthold等,Protist,153:401-412,2002)、巴龙霉素(Jakobiak等,Protist,同上;Sizova等,Gene,277:221-229,2001)、诺尔斯菌素(nourseothricin)(zaslavskaia等,2000,同上)、G418(Dunahay等,1995,同上;Poulsen和Kroger,FEBS Lett.,272:3413-3423,2005;Zaslavskaia等,2000,同上)、潮霉素(Berthold等,2002,同上)、氯霉素(Poulsen和Kroger,2005,同上)以及许多其它。用于微藻例如衣藻属的另外的可选择的标记可以是为如下提供抗性的标记:卡那霉素和丁胺卡那霉素抗性(Bateman,Mol.Gen.Genet.263:404-10,2000)、希奥霉素(zeomycin)和腐草霉素抗性(例如,ZEOCINTM腐草霉素D1)抗性(Stevens,Mol.Gen.Genet.251:23-30,1996)和巴龙霉素和新霉素抗性(Sizova等,2001年,同上)。已经使用的其它荧光或显色标记包括荧光素酶(Falciatore等,J.Mar.Biotechnol.1:239-251,1999;Fuhrmann等,Plant Mol.Biol.,2004;Jarvis和Brown,Curr.Genet.,19:317-322,1991)、β-葡萄糖醛酸酶(Chen等,2001,同上;Cheney等,2001,同上;Chow和Tung,1999,同上;El-Sheekh,1999,同上;Falciatore等,1999,同上;Kubler等,J.Mar.Biotechnol.1:165-169,1994)、β-半乳糖苷酶(Gan等,J.Appl.Phycol.,15:345-349,2003;Jiang等,Plant Cell Rep.,21:1211-1216,2003;Qin等,High Technol.Lett.,13:87-89,2003)和绿色荧光蛋白(GFP)(Cheney等,2001,同上;Ender等,Plant Cell,2002,Franklin等,Plant J.,2002;56,148,210)。
本领域技术人员将容易理解,根据本发明,多种已知的启动子序列可以有效地用于微藻种属的转化系统。例如,通常用于驱动微藻中的转基因表达的启动子包括花椰菜花叶病毒启动子35S(CaMV35S)的各种形式,其已经用于双鞭毛藻和绿藻门(Chow等,Plant Cell Rep.,18:778-780,1999;Jarvis和Brown,Curr.Genet.,317-321,1991;Lohuis和Miller,Plant J.,13:427-435,1998)。据报道,猿猴病毒的SV40启动子在数种藻类中也具有活性(Gan等人,J.Appl.Phycol.,151345-349,2003;Qin等,Hydrobiologia 398-399,469-472,1999)。来自衣藻属的RBCS2(核酮糖二磷酸羧化酶,小亚基)(Fuhrmann等,Plant J.,19:353-361,1999)和PsaD(丰富的光合系统I复合物蛋白质;Fischer和Rochaix,FEBS Lett.581:5555-5560,2001)的启动子也可以是有用的。HSP70A/RBCS2和HSP70A/β2TUB(微管蛋白)(Schroda等,Plant J.,21:121-131,2000)的融合启动子也可用于转基因的改进的表达,其中HSP70A启动子可在置于其它启动子的上游时作为转录激活子。在编码硅藻岩藻黄素-叶绿素a/b结合蛋白的fcp基因(Falciatore等,Mar.Biotechnol,1:239-251,1999;Zaslavskaia等,J.Pycol.36:379-386,2000)或编码黄绿藻紫黄素-叶绿素a/b结合蛋白的vcp基因(参见第8,318,482号美国专利)的启动子的控制下,在例如硅藻种属中也可以实现目标基因的高水平表达。如果期望的话,诱导型启动子可以在转基因微藻中提供基因的快速和严密受控的表达。例如,编码硝酸还原酶的NR基因的启动子区域可以用作这样的诱导型启动子。NR启动子活性通常被铵抑制,并且当铵被硝酸盐替代时被诱导(Poulsen和Kroger,FEBS Lett 272:3413-3423,2005),因此当微藻类细胞在铵/硝酸盐的存在下生长时可以关闭或开启基因表达。可用于本文提供的构建体和转化系统中的另外的藻类启动子包括在第US 2013/0023035号美国专利申请公开、2012年6月1日提交的第US 2013/0323780号美国专利申请公开、2012年12月4日提交的第US 2014/0154806号美国专利申请公开;和2013年6月11日提交的第US 2014/0363892号美国专利申请公开中公开的那些。
宿主微生物或细胞可以是未转化的细胞或已经用至少一种核酸分子转染的细胞。例如,包含如本文所提供的编码CHORD、CHORD衍生的或SKP1基因、同源物或变体的非天然基因的宿主细胞可以进一步包含一个或多个可赋予任意期望的性状的基因,所述性状包括但不限于目标生物分子(例如一种或多种蛋白质、色素、醇或脂质)的产量增加。例如,为了产生脂质,宿主细胞(例如但不限于藻类或异鞭毛宿主细胞)可以任选地包含编码在脂质生物合成中起作用的多肽的一种或多种非天然基因,包括但不限于,编码用于生产脂肪酸、脂肪酸衍生物和/或甘油脂质的酶的多肽,包括但不限于二酰甘油酰基转移酶(DGAT)基因、甘油磷酸酰基转移酶(GPAT)基因、溶血磷脂酸酰基转移酶(脱氢酶)(LPAAT)基因、磷脂酸磷酸酶(PAP)基因和/或单酰甘油酰基转移酶(MGAT)基因。
使用根据本发明的材料和方法进行修饰的合适的宿主细胞包括但不限于细菌、原生生物、微藻、浮游植物、异鞭毛体、真菌和原生动物。例如,该过程可以用于对水产养殖重要或引起关注的藻类属种,或用于在生产液体燃料分子和其它化学品中使用的生物质的生产。
考虑用于本发明的异鞭毛种属包括但不限于芽孢杆菌属、黄绿藻属、拉普利门(Labrinthulids)和破囊壶菌属。在一些实施例中,菌株可以是如下种属:网粘菌属、类网粘菌属、破囊壶菌、裂殖弧菌、不动壶菌属、橙黄壶菌属、日本壶菌属、戴普罗弗菌属或吾肯氏壶菌属。
适用于本发明方法的藻类种属包括微藻,例如如下种属:曲壳藻属、茧形藻属、双眉藻属、纤维藻属、星胞藻属、黄金色藻属、波利氏藻属、包特氏藻属、气球藻属、葡萄藻属、片球藻属、角毛藻属、四鞭藻属、衣藻属、绿球藻属、绿梭藻属、小球藻属、蓝隐藻属、金球藻属、球钙板藻属、隐甲藻属、隐藻属、小环藻属、链带藻属、杜氏藻属、后棘藻属、圆石藻属、独球藻属、衣迪斯藻属、眼虫藻属、真眼点藻属、披刺藻属、脆杆藻属、拟脆杆藻属、丽丝藻属、红球藻属、菱板藻属、异弯藻属、膜胞藻属、等鞭金藻属、鳞孔藻属、微芒藻属、蒜头藻属、单针藻属、微球藻属、微拟球藻属、舟形藻属、新绿藻属、肾鞭藻属、肾藻属、菱形藻属、棕鞭藻属、鞘藻属、卵囊藻属、虫毛球藻属、拟小球藻属、雪衣藻属、杜氏亚属盐藻属、巴夫藻属、普莱格门、褐指藻属、扁裸藻属、混营性微藻属、扁藻属、颗石藻属、肋球藻属、原壁菌属、拟绿球藻属、拟新绿藻属、拟角星鼓藻属、塔胞藻属、桑椹藻属、栅列藻属、裂衣藻属、骨条藻属、水绵藻属、裂丝藻属、四球藻属、四爿藻属、海链藻属、黄丝藻属、无隔藻属、鲜绿球藻属、魏氏藻属以及团藻属。
在本申请的一些实施方案中,待遗传改造的优选微生物包括但不限于光合生物体例如蓝细菌、藻类、硅藻等。示例性种属的非限制性实例包括例如黄绿藻或硅藻,例如如下种属:双眉藻属、角毛藻属、小环藻属、真眼点藻属、脆杆藻属、拟脆杆藻属、蒜头藻属、微拟球藻属、舟形藻属、菱形藻属、巴夫藻属、褐指藻属、海链藻属或魏氏藻属。在一些实施方案中,微拟球藻属成员,例如但不限于加的斯微拟球藻、颗粒微拟球藻(N.granulata)、湖泊微拟球藻(N.limnetica)、海洋微拟球藻(N.oceanica)、眼点微拟球藻(N.oculata)和盐生微拟球藻(N.salina),被转化或过表达如本文所提供的编码CHORD、CHORD衍生的、CHORD样、SKP1或SKP1样多肽的核酸分子。
包含如本文所提供的包含编码SKP1多肽的核酸序列的非天然核酸分子的微生物可以证明SKP1多肽的过表达,例如可以证明相较于不包含非天然核酸分子的对照微生物更高水平的SKP1转录物。包含如本文所提供的包含编码CHORD衍生多肽的核酸序列的非天然核酸分子的微生物可以证明CHORD衍生多肽的过表达,例如可以证明相较于不包含非天然核酸分子的对照微生物更高水平的CHORD转录物。
与不包含编码CHORD、CHORD衍生的或SKP1多肽的非天然基因的对照微生物相比,包含如本文所提供的非天然基因的微生物可具有提高的产产率,尤其是脂质例如FAME和生物质例如AFDW或TOC。通过例如使用流式细胞仪测量生长速率,或者通过以高于700nm、例如730或750nm的波长测量光密度可以证明更高的产率。如本文的实施例中所提供的,也可以测量无灰干重。可以通过提取藻类生物质、部分或大量纯化目标生物分子产物以及通过本领域已知的任意方法(例如但不限于化学或生物化学分析、光谱学或免疫学检测和/或活性测定)对产物进行定量来评定各种生物分子的产生。
生产藻类产物的方法
本文还提供了通过培养具有调节的生长特征的微生物细胞(例如,本文公开的宿主细胞)来生产生物质或至少一种生物产物的方法。所述方法包括在合适的培养基中培养如本文所公开的包含编码CHORD、CHORD衍生的或SKP1蛋白的非天然基因,例如本文公开的编码CHORD、CHORD衍生的或SKP1多肽的核酸分子的微生物细胞以提供藻类培养物和从培养物中回收生物质或至少一种生物产物。
异养或混养培养基可以包含还原性碳源,其可以是例如糖、有机酸、碳水化合物、醇、醛、酮、氨基酸、肽等。可以使用多种单糖例如葡萄糖、寡糖、多糖、纤维素材料、木糖和阿拉伯糖、二糖例如蔗糖、饱和或不饱和脂肪酸、琥珀酸盐、乳酸盐、乙酸盐、乙醇等或其混合物。
在一些实施例中,微生物可以是微藻。藻类培养物可以任选地为光合自养培养物,其中培养基优选地不包含大量的还原性碳,即培养物不包含处于可以为藻类用于生长的形式或水平的还原性碳。
可以在任意合适的容器中培养藻类,包括烧瓶或生物反应器,其中可以将藻类暴露于人造光或自然光。可以在可以是例如自然或程序化亮/暗循环的亮/暗循环中培养包含具有调节的生长特征的藻类细胞的培养物,作为说明性实例,可以提供12小时的光照到12小时的黑暗、十四个小时的光照到十个小时的黑暗、十六个小时的光照到八个小时的黑暗等。
培养是指通过使用选择的和/或受控的条件对一种或多种细胞的生长(例如细胞大小、细胞内容物和/或细胞活性的增加)和/或繁殖(例如,细胞数量通过有丝分裂的增加)进行有意的培育。可以将生长和繁殖两者的组合称为增殖。如本文实施例中所证实,本文提供的具有调节的生长特征的宿主细胞可实现增强的生长,如例如通过培养物经过例如一周或更多的时间相对于生长特征未调节的相同菌株的培养物野生型藻类细胞的更高细胞密度所证实。例如,可以将本文所述的本发明宿主细胞培养至少五天、至少六天、至少七天、至少八天、至少九天、至少十天、至少十一天、至少十二天、至少十三天、至少十四天、或至少十五天、或者至少一周、两周、三周、四周、五周、六周、七周、八周、九周或十周或更长时间。
可用于培养重组微生物的选择的和/或受控的条件的非限制性实例可以包括使用确定的培养基(具有已知特征例如pH、离子强度和/或碳源)、特定的温度、氧气张力、二氧化碳水平、生物反应器中的生长等,或其组合。在一些实施方案中,微生物或宿主细胞可以使用光和还原性碳源两者混养生长。或者,可以光合自养培养微生物或宿主细胞。当光合自养生长时,藻类菌株可以有利地使用光作为能源。无机碳源例如CO2或碳酸氢盐可用于微生物合成生物分子。如本文所使用的“无机碳”包括不能被生物体用作可持续能源的含碳化合物或分子。通常,“无机碳”可以呈CO2(二氧化碳)、碳酸、碳酸氢盐、碳酸盐、碳酸氢盐等或其组合的形式,其不能被进一步氧化以用于可持续能源,也不用作生物体减少能量的来源。可以使光合自养的微生物在培养基上生长,其中无机碳基本上是碳的唯一来源。例如,在其中无机碳基本上是碳的唯一来源的培养物中,可以在培养基中提供的任意有机(还原性)碳分子或有机碳化合物不能被细胞摄取和/或代谢供能和/或并非以足以为细胞培养物的生长和增殖提供可持续的能量的量存在。
根据本发明的方法可以使用的微生物和宿主细胞可以在世界各地的不同位置和环境中找到。用于脂质和/或其它产物的最佳繁殖和产生的特定生长培养基可以不同,并且可以被优化以促进诸如脂质、蛋白质、色素、抗氧化剂等的产物的生长、繁殖或生产。在一些情况下,某些微生物菌株可能不能在特定的生长培养基中生长,原因是存在一些抑制成分或不存在微生物或宿主细胞的特定菌株的一些基本营养需求。
固体和液体生长培养基一般可以从多种来源获得,如制备适用于各种微生物菌株的特定培养基的说明亦是如此。例如,各种淡水和盐水介质可以包括在Barsanti(2005)Algae:Anatomy,Biochemistry&Biotechnology,CRC Press中关于培养藻类的培养基和方法所描述的那些。藻类培养基配方也可以在各个藻类培养物保藏中心的网站上找到,作为非限制性实例包括UTEX藻类培养物保藏中心(www.sbs.utexas.edu/utex/media.aspx);藻类和原生动物的培养物保藏中心(www.ccap.ac.uk);和Katedra Botaniky(botany.natur.cuni.cz/algo/caup-media.html)。
培养方法可以任选地包括诱导一种或多种基因的表达以产生产物,例如但不限于参与脂质、一种或多种蛋白质、抗氧化剂或色素的产生和/或调节微生物代谢途径的蛋白质。诱导表达可以包括向培养物添加营养物或化合物、从培养基移除一种或多种组分、增加或减少光和/或温度、和/或促进目的基因表达的其它操作。这样的操作在很大程度上取决于与目的基因可操作地连接的(异源)启动子的性质。
在本发明的一些实施方案中,可以在发酵罐或生物反应器中培养如本文所述的具有调节的生长特征的微生物,其中生物反应器可以任选地为装备有人造光源的“光生物反应器”,和/或具有一个或多个足够透光(包括阳光)的壁以能够促进和/或维持光合微生物的生长和繁殖的生物反应器。为了产生脂肪酸产物或甘油三酯,可另外地或可选地在摇瓶、试管、小瓶、微量滴定盘、培养皿等或其组合中培养光合微生物或宿主细胞。
另外地或可选地,重组光合微生物或宿主细胞可以在池塘、运河、海基生长容器、沟槽、沟道、通道等或其组合中生长。与标准的生物反应器一样,无机碳源(例如但不限于CO2、碳酸氢盐、碳酸盐等)包括但不限于空气、富含CO2的空气、烟道气等或其组合,可以被供应给培养物。当供应除了CO2之外可能含有CO的烟道气和/或其它无机源时,可能需要预处理此类来源,使得引入(光)生物反应器中的CO水平相对于微生物的生长、繁殖和/或存活不构成危险和/或致死剂量。
可以通过从培养基中收获微生物来回收微生物培养物的生物质,例如通过过滤、沉降、离心或其组合。在根据本发明的生物质生产实施方案中,通过本文所述的方法产生和/或回收的、以无灰干重(AFDW)测量的生物质的量可以有利地为至少约0.05g/升培养物,例如至少约0.1g、至少约0.2g、至少约0.3g、至少约0.4g、至少约0.5g、至少约0.6g、至少约0.7g/升培养物、至少约1g/升培养物、至少约1.5g/升培养物、至少约2g/升培养物、至少约2.5g/升培养物或至少约5g/升培养物。虽然目标可以多次生产和/或回收尽可能多的生物质,但是在一些情况下,通过本文所述的方法生产和/或回收的以无灰干重(AFDW)测量的生物质的量可以限于约15g或更少/升培养物,例如约12g或更少/升培养物、约10g或更少/升培养物、约5g或更少/升培养物、约2g或更少/升培养物、约1g或更少/升培养物、或约0。5g或更少/升培养物。
生物质可以通过例如离心或过滤来收获。生物质可以被干燥和/或冷冻。其它产物可以从生物质中分离,例如脂质或一种或多种蛋白质。因此,本发明的一个方面还提供了包含具有调节的生长和/或表型特征的藻类宿主细胞的藻类生物质,例如包含本发明的核酸分子的藻类宿主细胞,其中核酸的提高的表达导致更高的生物质(例如AFDW或TOC)产率。
生物质可以以多种方式中的任意一种使用,例如,其可以通过从生物质产生合成气而被加工用作生物燃料,可以被供应到厌氧消化器用以生产一种或多种醇,或者可提取生物质以提供藻类脂质,例如但不限于甘油单酯、甘油二酯或甘油三酯、脂肪酸烷基酯、脂肪酸和/或脂肪酸衍生物。
如本文所述的宿主藻类细胞可以包括编码用于产生产物的多肽的一种或多种非天然基因,所述产物例如但不限于脂质、着色剂或色素、抗氧化剂、维生素、核苷酸、核酸、氨基酸、激素、细胞因子、肽、蛋白质或聚合物。例如,非天然基因可以编码酶、代谢调节物、辅因子、载体蛋白或转运蛋白。
在一些实施方案中,产物例如脂肪酸和脂肪酸衍生物可以通过本领域普通技术人员已知的回收手段从培养物中回收,例如通过全培养提取,例如使用有机溶剂。在一些情况下,如本文的实施例中所提供,可以通过使细胞均质化来增强脂肪酸或脂肪酸衍生物(例如,脂肪酸酯)的回收。当脂肪酸从微生物充分释放到培养基中时,回收方法可以适用于有效地仅回收释放的脂肪酸、仅回收微生物内产生和储存的脂肪酸或者产生和释放的脂肪酸两者。
在其它实施方案中,可以以各种方式回收由上述重组微生物分泌/释放到培养基中的产物,例如但不限于游离脂肪酸和脂肪酸衍生物。可以使用简单的分离方法,例如通过使用不混溶溶剂进行分配。另外地或可选地,可以使用颗粒吸附剂。这些可以包括亲脂性颗粒和/或离子交换树脂,这取决于回收方法的设计。它们可以在分离的介质中循环,然后收集,和/或介质可以通过含有这些颗粒的固定床柱,例如色谱柱。然后可以从颗粒吸附剂洗脱脂肪酸,例如通过使用适当的溶剂。在这种情况下,一种分离方法可以包括进行溶剂的蒸发,然后进一步处理分离的脂肪酸和脂质,以产生可以用于各种商业目的的化学品和/或燃料。
本发明的一些实施方案涉及包括培养如本文所述的藻类宿主细胞以产生生物质或至少一种藻类产物的方法,所述宿主细胞进一步包括编码参与产生产物的多肽的至少一种非天然基因。可以通过本领域普通技术人员已知的回收手段从培养物中回收诸如脂质和蛋白质的产物,例如通过全培养提取,例如使用有机溶剂。在某些情况下,可以通过细胞的均质化来加强脂肪酸产物的回收。例如,脂质例如脂肪酸、脂肪酸衍生物和/或甘油三酯可以通过在升高的温度和/或压力下用溶剂萃取藻类而从藻类中分离,如在2012年2月29日提交的名称为“Solvent Extraction of Products from Algae”的共同待决的美国专利申请公开2013/中所描述,该申请公开的全部内容通过引用并入本文。
对本文给出的一般方法的讨论仅用于说明的目的。在阅读本公开内容后,其它替代方法和实施方案对于本领域技术人员将是显而易见的,并且将被包括在本申请的精神和范围内。
除任一个上述实施方案之外或作为另外一种选择,本发明涵盖以下实施方案:
实施方案1是包含非天然核酸分子的重组微生物,所述非天然核酸分子包含编码SKP1多肽的核酸序列,其中编码SKP1多肽的核酸序列可操作地连接至异源启动子,其中所述重组微生物具有比不包含非天然核酸分子的对照微生物更高的生物质产率和/或更大的脂质产率。
实施方案2是根据实施方案1的重组微生物,其中
任选地为源自宿主微生物种属的启动子的异源启动子在转化到宿主中的核酸分子构建体上与编码SKP1多肽的核酸序列可操作地连接;或者
异源启动子是宿主基因组的内源启动子,其中编码SKP1多肽的核酸序列被转化到宿主微生物中,使得其整合到基因组中而变得可操作地连接至内源宿主启动子。
实施方案3是根据实施方案1或2的重组微生物,其中满足以下中的任一个或多个:
微生物是异鞭毛或藻类;
SKP1多肽的氨基酸序列与选自以下的SKP1多肽具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%或95%至100%同一性:SEQ ID NO:28、SEQ ID NO:65、SEQ ID NO:66、SEQ ID NO:67、SEQ ID NO:68、SEQ ID NO:69、SEQ ID NO:70、SEQ ID NO:71、SEQ ID NO:72、SEQ ID NO:73、SEQ ID NO:74、SEQ ID NO:75、SEQ ID NO:76、SEQ ID NO:77、SEQ ID NO:78、SEQ ID NO:79、SEQ ID NO:80、SEQ ID NO:81、SEQ ID NO:82、SEQ ID NO:83、SEQ ID NO:84、SEQ ID NO:85、SEQ ID NO:86、SEQ ID NO:87、SEQ ID NO:88、SEQ ID NO:89和SEQ ID NO:90;
SKP1多肽包括SKP1家族二聚化结构域和SKP1家族二聚化结构域中的一个或两个;和
SKP1多肽的氨基酸序列与宿主微生物的SKP1多肽具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、或95%至100%同一性。
实施方案4是根据实施方案1至3中任一项的重组微生物,其中:
所述重组微生物是网粘菌纲,并且SKP1多肽与网粘菌纲种属的SKP1多肽具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%或95%至100%同一性,任选地,其中SKP1多肽与SEQ ID NO:73或SEQ ID NO:74具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、或95%至100%同一性;
所述重组微生物是硅藻,并且SKP1多肽与硅藻种属的SKP1多肽具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%或95%至100%同一性,任选地其中所述SKP1多肽与SEQ ID NO:65、SEQ ID NO:70、SEQ ID NO:71、SEQ ID NO:72、SEQ ID NO:75、SEQ ID NO:76、SEQ ID NO:77、SEQ ID NO:78、SEQ ID NO:79、SEQ ID NO:80和SEQ ID NO:81具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%或95%至100%同一性;
所述重组微生物是黄绿藻种属,并且SKP1多肽与黄绿藻种属的SKP1多肽具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%或95%至100%同一性,任选地,所述SKP1多肽与SEQ ID NO:28、SEQ ID NO:68和SEQ ID NO:69具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、或95%至100%同一性;或者
所述重组微生物是绿藻纲植物藻类并且SKP1多肽与绿藻纲植物种属的SKP1多肽具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%或95%至100%同一性,任选地,其中所述SKP1多肽与SEQ ID NO:66、SEQ ID NO:67、SEQ ID NO:82、SEQ ID NO:83、SEQ ID NO:84、SEQ ID NO:85、SEQ ID NO:86、SEQ ID NO:87、SEQ ID NO:88、SEQ ID NO:89或SEQ ID NO:90具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%或95%至100%同一性。
实施方案5是包含非天然核酸分子的重组微生物,所述非天然核酸分子包含编码包含CHORD结构域的至少一部分的CHORD衍生多肽的核酸序列,其中编码CHORD衍生多肽的核酸序列与异源启动子可操作地连接,其中所述重组微生物具有比不包含所述非天然核酸分子的对照微生物更大的生物质产率和/或更大的脂质产率。
实施方案6是根据实施方案5的重组微生物,其中
异源启动子是宿主基因组的内源启动子,其中编码CHORD衍生多肽的核酸序列被转化到宿主微生物中,使得其整合到基因组中而变得可操作地连接至内源启动子;或者
异源启动子在转化入宿主微生物的核酸分子构建体上可操作地连接至编码CHORD衍生多肽的核酸序列。
实施方案7是根据实施方案6的重组微生物,其中CHORD衍生多肽包含天然CHORD多肽的CHORD结构域的一部分或与其具有至少80%同一性的氨基酸序列,任选地其中与其具有至少80%同一性的CHORD结构域或氨基酸序列的一部分是天然CHORD多肽的CHORD结构域的连续氨基酸的至少60%,或者是天然CHORD多肽的CHORD结构域的至少36、37、38、39、40个氨基酸,任选地,其中CHORD结构域与SEQ ID NO:4具有至少80%、至少85%、至少90%、至少95%、至少96%、至少97%、至少98%、至少99%或100%同一性。
实施方案8是根据实施方案6或实施方案7的重组微生物,其中CHORD衍生多肽包含CHORD结构域或其部分或与其具有至少80%同一性的氨基酸序列,并且进一步其中CHORD结构域或其部分或与其具有至少80%同一性的氨基酸序列与异源氨基酸序列融合,任选地,其中所述CHORD衍生多肽包含与SEQ ID NO:4、SEQ ID NO:99或SEQ ID NO:100具有至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少96%、至少97%、至少98%或至少99%同一性的氨基酸序列。
实施方案9是根据实施方案1至8中任一项的重组微生物,其中相对于在相同条件下培养的对照微生物,所述重组微生物具有提高的生物质产率,其中所述条件是分批、半连续或连续培养。
实施方案10是根据实施方案9的重组微生物,其中所述重组微生物是藻类,并且显示出提高的产率的培养条件是光合自养的。
实施方案11是一种生产生物质或至少一种生物产物的方法,其包括在合适的培养基中培养权利要求1至10中任一项的微生物以产生生物质或至少一种生物产物,任选地进一步包括从培养物中回收生物质或至少一种生物产物。
实施方案12是根据实施例11的方法,其中培养条件是分批、半连续或连续培养。
实施方案13是根据实施方案11或12的方法,其中所述重组微生物是藻类并且培养条件是光合自养的。
实施方案14是包含根据实施方案1至9中任一项的重组微生物的生物质。
实施例
申请人已鉴定并分离了藻类菌株加的斯微拟球藻,其两个基因改变的表达赋予微生物提高的产率,例如提高的生物质和脂质产率。这些发现是通过如下所述进行:鉴定编码藻类菌株微拟球藻WT-3730的基因组中的细胞周期调控家族成员的基因,构建被设计为改变基因表达的载体,将其转化入微拟球藻,并分析所得藻系的提高的产率,特别是提高的脂质例如FAME和生物质例如AFDW或TOC的产率。
实施例中使用的培养基
以下培养基用于实施例中。
PM066培养基包含硝酸盐作为唯一的氮源。PM066培养基包含10mM硝酸盐(NO3)和0.417mM磷酸盐(PO4)以及即溶海洋盐中的痕量金属和维生素。通过将5.71ml的1.75M NaNO3储液(148.7g/L)和5.41ml的77mM K2HPO4·3H2O储液(17.57g/L)加入到981ml的即溶海洋盐溶液(35g/L)以及4m1螯合金属储液和4ml维生素储液中来制备PM066培养基。如下制备螯合金属储液:向400ml水中加入2.18g Na2EDTA-2H2O;1.575g FeCl3·6H2O;500μl的39.2mM储液(0.98g/100ml)CuSO4-5H2O;500μl 77.5mM储液(2.23g/100ml);ZnSO4·7H2O;500μl的42.0mM储液(1.00g/100ml);CoCl2·6H2O;500μl的910.0mM储液(18.0/100ml)MnCl2·4H2O;500μl的26.0mM储液(0.63g/100ml)Na2MoO4·2H2O;补足至500ml最终体积,并过滤消毒。如下制备维生素储液:向400毫升水中加入0.05克硫胺素HCl;500μl的0.37mM储液(0.05g/100ml)氰钴胺素;和2.5ml的0.41mM储液(0.01g/100ml)生物素,补足至500ml最终体积,并过滤灭菌。
PM074是作为10X F/2的充氮培养基,其如下制备:添加1.3ml F/2藻类饲料部分A(Aquatic Eco-Systems)和1.3ml F/2藻类饲料部分B(Aquatic Eco-Systems)至即溶海洋盐溶液(35g/L)(Aquatic Eco Systems,Apopka,FL)中,最终体积为1升。脯氨酸A和脯氨酸B一起包括8.8mM NaNO3、0.361mM NaH2PO4·H2O、10X F/2痕量金属和10X F/2维生素(Guillard(1975)Culture of phytoplankton for feeding marine invertebrates,“Culture of Marine Invertebrate Animals.”(编著:Smith W.L.和Chanley M.H.),Plenum Press,New York,USA,第26至60页)。
实施例1:加的斯微拟球藻的插入诱变
野生型加的斯微拟球藻菌株WT-3730是从Provasoli-Guillard国家海洋藻类和微生物中心(NCMA,Maine,USA)(原海洋浮游植物培养物保藏中心(CCMP))获得的加的斯微拟球藻菌株CCMP1894的传代分离株,WT-3730被用作插入诱变和遗传操纵的野生型背景。将包含抗性基因的构建体转化到加的斯微拟球藻细胞中。构建体(SEQ ID NO:57)包括针对微拟球藻属表达进行密码子优化的芽孢杆菌属杀稻瘟菌素抗性基因(SEQ ID NO:98),其可操作地连接至微拟球藻属TCTP启动子(SEQ ID NO:97),如2013年12月6日提交的第20140220638号美国专利公开中所描述,该美国专利公开的全部内容通过引用并入本文。基本上如第20140220638号美国专利公开中所描述通过电穿孔转化加的斯微拟球藻细胞。
实施例2:筛选和分离增加的脂质突变体
首先,使用与荧光中性脂质特异性染料BODIPY 505/515(Life Technologies)偶联的荧光激活细胞分选(FACS)技术针对提高的脂质含量筛选转化的微拟球藻的随机插入标签文库。使出现在转化平板上的抗生素抗性集落重悬于液体培养物中并适应低光强度,用BODIPY染色,并使用BD FACSAria II流式细胞仪(BD Biosciences,San Jose,CA)通过流式细胞术进行分选,以便选择高BOPDIPY荧光细胞。通过处理1ml具有12.5%甘油和0.1μg/ml BODIPY的终浓度的细胞进行BODIPY染色。一般而言,总细胞群体的约0.5%至2%被选为具有来自该第一筛选程序的最高BODIPY荧光。在一些情况下,基于增加的BODIPY荧光进行两轮连续的分选。将分选的细胞合并并在充氮培养基中的25m1分批培养物中培养,通过细胞数量归一化,并评定TOC和FAME水平。用分选的细胞接种含有大约30ml PM066充氮培养基的烧瓶(25cm2),并在Adaptis生长室中培养,在含有1%富CO2空气的环境中以大约130rpm振荡并以16小时光照(在30℃):8小时黑暗(在25℃)周期暴露于大约274μE-m-2s-1光照。
对2mL样品进行FAME分析,该样品使用GeneVac HT-4X干燥。向经干燥的沉淀中加入以下物质:500μL含500mM KOH的甲醇、200μL含有0.05%丁基化羟基甲苯的四氢呋喃、40μL的2mg/ml C11:0游离脂肪酸/C13:0甘油三酯/C23:0脂肪酸甲酯内标混合物和500μL的玻璃珠(425-600μm直径)。用开口顶部PTFE隔垫内衬帽盖上小瓶,并以1.65krpm放置在SPEX GenoGrinder中7.5分钟。然后将样品在80℃加热5分钟并使其冷却。为了衍生化,向样品中加入500μL的含10%三氟化硼的甲醇,然后在80℃下加热30分钟。使管冷却,然后加入2mL庚烷和500μL的5M NaCl。将样品以2krpm涡旋5分钟,最后以1krpm离心3分钟。使用Gerstel MPS自动取样器对庚烷层进行取样。定量使用了80μg C23:0FAME内标。
通过用去离子水稀释2mL细胞培养物至总体积20mL来测定总有机碳(TOC)。将每次测量的三次注射液注入到Shimadzu TOC-Vcsj分析仪中以测定总碳(TC)和总无机碳(TIC)。燃烧炉的温度设定为720℃,从TC减去TIC,以确定TOC。四点校准范围从2ppm到200ppm,对于未稀释培养物,对应于20-2000ppm,相关系数r2>0.999。
将具有与野生型相比提高的TOC和FAME的批次涂铺到含有100μg/mL杀稻瘟菌素的PM066平板上并在恒定光下(约80pmol光子m-2sec-1)孵育直至出现单个集落(约2至3周)。进一步评定所回收单个集落的产率提高,如在以下实施例中所描述。
实施例3:具有增加的脂质产生的克隆的脂质产率评定
在分批生长测定中评定七种高产脂质分离物以测试脂质产率。在该测定中,用藻类接种每种菌株的一式三份的225cm2烧瓶,以在总体积为500mL的包含8.8mM硝酸盐作为氮源的PM066培养基中提供0.15OD 730nm的培养物密度。将搅拌棒添加到每个烧瓶中,并且对烧瓶安装具有用于在100ml/min的速率下空气/CO2递送的注射器式过滤器的塞子和用于取样的可来福接头,将烧瓶沿着16烧瓶架分配随机位置。架下方的搅拌板以450rpm操作。LED灯库提供16∶8的灯光方案,该灯光方案被设计为16小时提供1800μE-m-2-s-1,然后是8个小时的黑暗。温度从25℃变化到34℃。对于TOC和FAME分析,在第3、5、6、7、8、9和10天取样(通常2mL)。第5天之后,所有7种含有高脂质的分离株在FAME产率上明显优于野生型(图1)。到第10天,与野生型WE-3730(图1和表1)相比,菌株GE-5877的FAME体积产率提高62%。
表1.在分批生长评定的第10天,FAME产率值和相对于WE-3730的提高百分比。
实施例4:增加的脂质突变体的基因分型
对具有确认的增加的脂质表型的7种菌株进行测序以鉴定诱因突变。根据推荐的规程(Illumina Inc,San Diego,CA),将加的斯微拟球藻突变体的全基因组DNA用于Nextera DNA文库制备。在Illumina MiSeq仪器上通过配对末端测序对文库进行测序。在七种情况中的每一种中,在编码CHORD(富含半胱氨酸和组氨酸的结构域)蛋白质产物的基因(称为基因3266或“CHORD-3266”(SEQ ID NO:1))的第三和第四外显子之间发生了单个载体整合事件。由于除了非编码区中的SNP外,所有七个突变分离物基本上相同,因此挑选菌株GE-5877进行更详细的表征(中断的3266基因座的序列提供为SEQ ID NO:56)。
实施例5:GE-5877的生理学评定
在摇瓶中将GE5877和野生型菌株WE3730以16∶8昼夜循环中用约100μE光生长六天。在整个培养期间取样用于细胞计数、OD730、FAME、TOC和多核苷酸提取。基本上如第20150191515号和第20150183838号美国专利公开中所公开来进行多核苷酸提取、聚合酶链式反应(PCR)和定量实时PCR(qRT-PCR),这两篇专利均通过引用整体并入。最初披露诱变载体的测序结果插在CHORD-3266基因座的外显子3和外显子4之间(实施例4)。使用位于插入位点侧翼的引物(SEQ ID NO:58和SEQ ID NO:59)的PCR确认了载体的存在(图2A)。qRT-PCR表明由载体整合引起的障碍干扰了全长CHORD基因的转录(图2A和2B)。使用外显子2(SEQ ID NO:60和SEQ ID NO:61)和外显子4(SEQ ID NO:62和SEQ ID NO:63)的序列特异性引物进行qRT-PCR扩增。由扩增外显子2的引物相对于扩增外显子4的引物对得到的qRT-PCR产物披露对应于破坏的CHORD-3266基因的这两个区域的不同转录水平(图2C)。这些结果表明,两个单独的转录物正在产生。在通过光密度(图2D)和细胞计数(图2E)两者测量的细胞密度方面,GE5877优于WE3730,并且在生物质和FAME产率(图2E和2F;表2)方面优于野生型菌株,通过时间过程中的细胞计数所见生成时间缩短(图2E)。
表2.WE-3730和GE-5877分批生长培养物的FAME μg/mL和总有机碳(TOC)μg/mL含量。
实施例6:GE-5877的转录组学分析
如实施例5中所描述,使GE5877和野生型菌株WE3730在具有大约100μE 16∶8的昼夜循环的摇瓶中生长六天。经过一周的适应后,将野生型和CHORD敲除的GE5877两者接种到生物学一式三份重复的OD730 0.25(t=0),在接种3小时后收获以进行RNA提取,此时两种菌株的密度基本相同,适应状态相似。
为了分离总RNA,将10mL的藻类细胞培养物以4000x×g离心5分钟,并倾倒出上清液。将沉淀重悬于1.8mL缓冲液A(5mL TLE研磨缓冲液,5mL苯酚,1mL 1-溴-3-氯丙烷和20μL巯基乙醇,其中TLE研磨缓冲液包含9mL的1M Tris pH 8、5mL 10%SDS、0.6mL的7.5M LiCl和0.45M EDTA,终体积为50mL)中并转移至含有约0.5mL的200μm锆珠粒的2mL微量离心管中。将管在4℃剧烈涡旋5分钟,然后以11.8xg离心2分钟。然后取出水层并移液至新的2mL管中,向其中加入1mL 25∶24∶1苯酚提取缓冲液(25mL苯酚pH 8或5.1;24mL 1-溴-3-氯丙烷和1mL异戊基醇),剧烈摇动管,以11.8×g离心2分钟。离心后,除去水层,移液到新的2mL离心管中,向其中加入1ml 1-溴-3-氯丙烷。摇动试管,再次以11.8xg离心2分钟。将水层移至新管中并加入0.356体积的7.5M LiCl。将管倒置10至12次并在-20℃下储存过夜。次日,使样品不经混合而达到室温,并以16,000xg离心30分钟。除去上清液,用1mL冰冷的80%乙醇洗涤沉淀。将管以16,000xg离心30分钟,并在除去上清液后使其风干。最后,将RNA沉淀重悬于50μl超纯水中。根据制造商的说明,使用Agilent 2100生物分析仪和RNA6000LabChip通过片上凝胶电泳评定RNA质量。
使用TruSeq链mRNA样品制备试剂盒(Illumina)按照生产商的说明从分离的RNA制备下一代测序文库。使用mRNA-Seq程序(在Mortazavi等(2008)Nature Methods 5:621-628中描述)使用合成测序法(Illumina MiSeq)对TruSeq文库进行测序以产生100bp的配对末端读数。使用TopHat(tophat.cbcb.umd.edu/)将可映射的读数与加的斯微拟球藻参考基因组序列进行比对。使用Cufflinks软件(cufflinks.cbcb.umd.edu)的Cuffdiff组件为每个注释计算表达水平。使用R软件包edgeR进行差异表达分析(McCarthy等(2012)Nucl.Acids Res.40:doi:10/1093/nar/gks042))。使用标准参数针对每个样品中的每个基因报告以片段/千碱基转录物/百万匹配读数(FPKM)为单位的表达水平。FPKM是相对转录水平的量度,其归一化转录物长度的差异。
CHORD敲除菌株GE-5877针对野生型(WE-3730)转录组的全局分析披露大部分差异调控的基因相对于野生型在菌株中下调(图3A)。在所有重复中鉴定出具有统计学显著表达水平的大约3,000个基因中,与野生型相比,突变体中仅3.5%上调超过2倍,而20%下调不到2倍。对基因本体论类别的分析(提供了描述基因产物特征及其功能的术语的受控词汇,参见:geneontology.org)披露了相对较小的上调基因子集富含与细胞周期进展和有丝分裂有关的基因(图3B)。事实上,诸如“有丝分裂细胞周期的M/G1转变”和在细胞分裂中起关键作用的细胞组分例如“动粒”和“蛋白酶体复合物”之类的过程是富含上调基因的前10个基因本体类别之一。这些表达谱与CHORD敲除所观察到的增长速率一致,并表明细胞周期控制在该突变体中失调。
实施例7:GE-5877突变的概况
实施例6中获得的转录组学数据披露了菌株GE-5877表达两个突变CHORD转录物-第一个跨越外显子1-3(SEQ ID NO:2),第二个起始于插入的杀稻瘟菌素基因的3'区并且跨越内含子4和外显子4的其余部分(SEQ ID NO:3)(图4A)。为了概括GE-5877表型,在加的斯微拟球藻WE-3730背景菌株中均使用基因敲除和过表达方法两者。将抗生素抗性标记插入外显子1或外显子4并不能概括增长的生长速率表型(图4B)。类似地,跨越外显子1-3的过表达基因组DNA不导致期望的表型(图4B)。出乎意料的是,如在菌株GE-8999中那样,载体的3′末端的转录产物的过表达和跨越内含子4和外显子4(表达构建体如SEQ ID NO:64所提供)引起了与原始的GE-5877突变体表型相似的提高的生长速率(图4B和4C;表3)。该截短和突变的转录物(SEQ ID NO:3)编码包含由外显子4(SEQ ID NO:99)编码的氨基酸序列的融合多肽(SEQ ID NO:100),所述外显子4包含第二CHORD结构域的65个氨基酸中的最后40个(CHORD结构域2的61%,即,SEQ ID NO:4的61%)。这些结果支持与表达未改变的CHORD-3266基因转录物的野生型菌株相比,与异源序列融合的外显子4(其包括约60%的CHORD结构域2)的过表达足以增加生物质。
表3.分批生长四天后菌株的光密度(OD)。
实施例8:酵母双杂交实验
在四种不同条件下从生长的培养物独立地分离总微拟球菌RNA,所述条件随后汇集:标准的充氮条件、氮剥夺、缺磷和高光强度。为了在酵母双杂交分析中筛选cDNA,通过从藻类细胞培养物中除去10mL来分离RNA,然后以4000xg离心5分钟,倾倒出上清液。将沉淀重悬于1.8mL缓冲液A(5mL TLE研磨缓冲液、5mL苯酚、1mL 1-溴-3-氯丙烷和20μL巯基乙醇,其中TLE研磨缓冲液包含9mL的1M Tris,pH8,5mL的10%SDS、0.6mL 7.5M LiCl和0.45M EDTA,终体积为50mL)中并转移至含有约0.5mL的200μm锆珠粒的2mL微量离心管中。将管在4℃剧烈涡旋5分钟,然后以11.8xg离心2分钟。然后移出水层并移入新的2mL管中,向其中加入1mL 25∶24∶1苯酚提取缓冲液(25mL苯酚pH 8或5.1;24mL 1-溴-3-氯丙烷和1mL异戊基醇),剧烈摇动管,以11.8xg离心2分钟。离心后,除去水层,移液到新的2mL离心管中,向其中加入1ml 1-溴-3-氯丙烷。摇动试管,再次以11.Sxg离心2分钟。将水层移至新管中并加入0.356体积的7.5M LiCl。将管倒置10至12次并在-20℃下储存过夜。次日,使样品不经混合而达到室温,并以16,000xg离心30分钟。除去上清液,用1mL冰冷的80%乙醇洗涤沉淀。将管以16,000xg离心30分钟,并在除去上清液后使其风干。最后,将RNA沉淀重悬于50μl超纯水中。根据制造商的说明,使用Agilent 2100生物分析仪和RNA6000 LabChip通过片上凝胶电泳评定RNA质量。
使用Make Your Own“Mate&Plate TM”实验室系统使用者手册作为指导(Clontech,Mountain View,CA)合成cDNA文库。然而,代替使用试剂盒提供的SMART III Oligo,使用经修饰的5′引物进行第一链(SEQ ID NO:5)合成,经修饰的5′引物利用在微拟球藻中鉴定的先前描述的剪接前导序列(参见于2013年12月5日提交的美国专利申请公开2014/0186842,“Nannochloropsis Spliced Leader Sequences and Uses ThereFor”,其在此整体并入本文)。还将修饰的3′引物用于第二链合成(SEQ ID NO:6)。经修饰的5′和3′引物两者均含有序列延伸,其添加了与酵母表达载体pGADT7-rec(Clontech)相容的核苷酸序列以允许随后通过环化聚合酶延伸克隆进行克隆(cpec;参见例如Quan&Tijan(2009)PLoS One 4(7):e6441)。在将第二链cDNA克隆到pGAD-T7-rec中后,将所得文库转化到大肠杆菌中。获得了大约75万个集落,其代表了微拟球藻属转录组的至少25倍的覆盖度。通过测序验证文库的低冗余度,并将文库转化到酵母菌株Y2HGold(Clontech)中。最终的酵母表达文库由超过200多万个克隆组成。
为了鉴定潜在的蛋白结合配偶体,将CHORD蛋白片段用于酵母双杂交(Y2H)筛选。将编码全长CHORD蛋白(SEQ ID NO:22)的CHORD-3266的编码序列分成不同的结构域:CRD1-117(SEQ ID NO:23)、CRD117-179(SEQ ID NO:24)、CRD179-251(SEQ ID NO:25)和CRD179-336(SEQ ID NO:26)。在Y2H筛选中,将每个结构域用作诱饵(图5A)。(参见例如Chien等(1991)Proc.Natl.Acad.Sci.88:9578-9582;Guarente(1993)Proc.Natl.Acad.Sci.90∶1639-1641;Rutisjmu&Golemis(2008)Biotechniques 44:655-662)。使用含有悬挂序列的序列特异性引物扩增全长序列和单独的结构域,以克隆到酵母双杂交质粒中:CRD1-336(SEQ ID NO:15和SEQ ID NO:16)、CRD1-117(SEQ ID NO:15和SEQ ID NO:17)、CRD117-179(SEQ ID NO:18和SEQ ID NO:19)、CRD179-251(SEQ ID NO:20和SEQ ID NO:21)和CRD179-336(SEQ ID NO:20和SEQ ID NO:16)。
如前段所述,使用正向引物和反向引物从cDNA扩增CHORD-3266的编码序列片段。通过循环聚合酶延伸克隆将单个片段克隆到诱饵载体pGBKT7(Clontech)中,并转化到大肠杆菌中。在确认序列后,将其转化到酵母菌株Y187(Clontech)中,并筛选针对克隆到捕食载体中的微拟球藻cDNA文库的相互作用,如上所述。
根据MatchmakerTM Gold Yeast双杂交系统使用者手册(Clontech),通过将含菌株的文库(猎物)的菌株与每个单独的诱饵菌株(即,表达CRD1-117的菌株)配对来筛选猎物文库。CRD1-117筛选的配对效率达到约4.5%(良好的匹配效率通常在3%至5%之间)。根据这些数字,预计有超过一千万的相互作用被测试。
一些CHORD结构域导致多重假阳性,但是一个结构域(CRD1-117)具有更高的选择性,并且基于它们在选择性培养基上的生长和蓝色,只有少数阳性克隆被获取,这是由于含有CRD 1-117的表达蛋白的相对作用以及随后的营养缺陷型标记和报道基因的激活。在从其它结构域命中减去假阳性之后,保留对该结构域而言独特的基因:编码在加的斯微拟球藻基因组基因座Naga_100120gl2编码的SGT1(SEQ ID NO:27)的基因;Genbank登录号AZIL01000354.1。该基因通常被称为“skp1的G2等位基因的抑制子”,因为它是在旨在抑制skp1-4的遗传抑制筛选中发现的,该突变体在酵母动粒功能中具有缺陷(Hieter等,1999Nature 402:362-363)。SGT1是酵母动粒组装途径的重要组成部分,并且已经与不同生物体中的许多不同生物学作用相关联。此外,已知哺乳动物SGT1与CHORD-3266的人类同源物Hsp90分子伴侣和melusin相互作用。类似地,拟南芥CHORD-3266同源物RAR1也已知结合拟南芥SGT1(Takahashi等(2003)PNAS USA 100:11777-11782)。鉴于CHORD-3266同源物与SGT1蛋白结合的文献中有先例,Y2H观察到的相互作用可能是体内发生的相互作用。
为了确定SGT1(SEQ ID NO:27)和Skp1是否是相同途径的成员,我们对微拟球藻SGT1是否能够通过Y2H与微拟球藻属Skp1结合进行了测试。在微拟球藻、SKP1-8611(多核苷酸SEQ ID NO:9和多肽SEQ ID NO:28)和Skp1-7479(在加的斯微拟球藻基因组的基因座Naga_100005g56处编码的SEQ ID NO:29,Genbank登录号AZIL01000936)中鉴定了两种不同的Skp1蛋白。使用SGT1(SEQ ID NO:30和SEQ ID NO:31)、SKP-8611(SEQ ID NO:32和SEQ ID NO:33)和SKP-7479(SEQ ID NO:34和SEQ ID NO:35)的序列特异性引物扩增蛋白质编码序列,并如上所述克隆到猎物和诱饵载体中。这些构建体用于直接酵母双杂交测定,并且均通过Y2H与SGT1产生阳性相互作用(图5B)。
实施例9:在加的斯微拟球藻中SKP1的过表达
在转录组学和Y2H数据的指导下,对SKP-Cullin-F-box E3泛素连接酶复合物(泛素-蛋白酶体系统的关键组分)进行靶向操纵。基于其对细胞周期的正调控作用,过表达SKP1-8611(SEQ ID NO:9,编码SEQ ID NO:28;图6)。使用引物(SEQ ID NO:7和SEQ ID NO:8)对SKP1-8611(SEQ ID NO:9)进行PCR扩增,然后将其克隆到含有5901启动子(SEQ ID NO:103)和T9终止子(SEQ ID NO:104)的载体中以产生SKP1-8611过表达载体(SEQ ID NO:10)。通过电穿孔将SKP1-8611过表达载体(SEQ ID NO:10)转化到微拟球藻中。在含有500μg/mL潮霉素抗生素的PM74琼脂平板上回收集落并进行PCR筛选以确认SKP1-8611过表达构建体的存在。对阳性菌株GE-8119和GE-8120进行qRT-PCR分析以确认SKP1转录物的过表达。
与野生型对照系相比,进行qRT-PCR实验以评定拟微球菌转基因系GE-8119和GE-8120中SKP1-8611转基因的稳态mRNA水平。基本上如实施例8中所描述分离总RNA。根据制造商的方案使用商业逆转录酶将分离的RNA转化为cDNA。对于PCR,将Ssofast EvaGreen Supermix(Bio-Rad,Hercules,CA)与基因特异性引物一起使用。PCR反应在与CFX实时系统(BioRad)结合的C1000热循环仪上进行。引物和cDNA浓度按照制造商的推荐。使用序列特异性引物(SEQ ID NO:11和SEQ ID NO:12)对SKP1-8611转录物进行PCR扩增。将每个样品的转录物水平相对于在不同培养条件下具有一致表达水平的管家基因进行归一化,具体是使用序列特异性引物(SEQ ID NO:13和SEQ ID NO:14)扩增的T5001704。使用BioRad的CFX Manager软件采用ddCT方法计算相对表达水平。图7显示相对于野生型(WT)绘制在y轴上的归一化表达值,其中WE-3730的SKP1-8611的表达等于1。误差线表示三次技术重复的标准误差。发现菌株GE-8119和GE-8120过表达SKP1-8611转基因,其水平接近野生型亲本的4至5倍(图7)。
实施例10:菌株GE-8119的产率评定
测试确认的SKP1过表达株系GE-8119的FAME和TOC水平,以确定编码SKP1的cDNA序列的过表达是否也导致产率提高。在该半连续培养评定中,用藻类接种每种菌株的一式三份的225cm2烧瓶以提供总体积为500mL的PM074培养基中的0.15OD 730nm的培养物密度。将搅拌棒添加到每个烧瓶中,并且对烧瓶安装具有用于在100ml/min的速率下空气/CO2递送的注射器式过滤器的塞子和用于取样的可来福接头,将烧瓶沿着16烧瓶架分配随机位置。架下方的搅拌板以450rpm操作。LED灯库提供了程序化正弦16∶8的光方案,该光方案被设计为经16小时稳定地上升到2000μE-m-2s-1峰值和回落到0μE-m-2s-1,然后是8小时黑暗。温度从25℃变化到34℃。将培养物每天稀释30%以实现半连续生长,并且一旦培养物达到稳定的生长状态,为了TOC和FAME分析,经5至6天每天移出样品(通常2mL)。图7总结了评定GE-8119和野生型WE-3730的脂肪酸甲酯(FAME,图8)和总有机碳(TOC,图8)值的产率水平的实验结果。发现过表达SKP1的转基因系GE-8119的FAME和TOC产率都优于野生型(图8,表4)。
表4.在半连续培养中与野生型相比SKP1过表达株系GE-8119的FAME和TOC产率。
实施例11:其他细胞周期基因
基于来自实施例6和8的转录组学和酵母双杂交数据,SKP-Cullin-Fbox E3泛素连接酶复合物和其它细胞周期调节基因的其它成员被靶向敲除或过表达(表5)。这些其他靶标是SGT1(多肽SEQ ID NO:27)、SKP1-7479(多肽SEQ ID NO:29)、SKP2-6789(多肽SEQ ID NO:36)、CDC25(多肽SEQ ID NO:37)、FBW7-1(多肽SEQ ID NO:38)、FBW7-2(多肽SEQ ID NO:39)、FBW7-3(多肽SEQ ID NO:40)、FBW7-4(多肽SEQ ID NO:41)、FBW7-5(多肽SEQ ID NO:51)、Weel-1(多肽SEQ ID NO:43)、Weel-2(多肽SEQ ID NO:44)、Weel-3(多肽SEQ ID NO:45)、Weel-4(多肽SEQ ID NO:46)、Weel-5(多肽SEQ ID NO:47)、细胞周期蛋白-6855(SEQ ID NO:48)、细胞周期蛋白-3560(多肽SEQ ID NO:49)、细胞周期蛋白-9008(多肽SEQ ID NO:50)、细胞周期蛋白-4163(多肽SEQ ID NO:51)、CDKA1-3735(多肽SEQ ID NO:52)、CDKA1-864(多肽SEQ ID NO:53)、CDKA1-9049(多肽SEQ ID NO:54)和CDKA1-8325(多肽SEQ ID NO:55)。如实施例5中所描述的,评定各菌株在分批生长测定中提高的FAME产率。在测试的突变体中,只有过表达株系GE-8119和GE-8120(其包括编码多肽SEQ ID NO:28的基因SEQ ID NO:9)的SKP-8611与WE-3730相比表现出提高的FAME产率(表5)。由于在这些分批测定中,GE-8119和GE-8120相对于野生型细胞的生物质产率提高反映在更高的FAME体积产率中,所以在任意其他突变体中无法检测到更高的FAME产率也被认为是缺乏提高的整体生物质产率。
表5.拟微球菌中SKP-Cullin-F box E3泛素连接酶复合物和细胞周期调节基因的基因表达变化
已经对本发明的多个实施方案进行了描述。然而,应当理解的是,可以组合本文描述的实施方案的要素来形成另外的实施方案,并且在不背离本发明的精神和范围的情况下可以进行各种修改。因此,其它实施方案、替代方案和等效方案也在本文所描述和要求保护的本发明的范围内。
本申请中的标题仅仅是为了方便读者,而不以任意方式限制本发明或其实施方案的范围。
本说明书中提到的所有出版物和专利申请均通过引用并入本文,其程度如同每个单独的出版物或专利申请被具体地和单独地指明通过引用并入。
序列表
<110> 合成基因组股份有限公司
N•鲍曼
I•阿加维
<120> 为提高的产率而改造的微生物
<130> SGI1960-1WO
<150> US 62/214,780
<151> 2015-09-04
<160> 104
<170> PatentIn版本3.5
<210> 1
<211> 1327
<212> DNA
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> CHORD-3266编码序列
<400> 1
atgaaactct atgtgcacta cgaggaggct ggtcaggatg aaaaggcatt gacgcttaag 60
ttgactctgc ccaaaagctg ggcggagcag ccgttgcttc aagtactgga gctgttcatc 120
gaatcctaca acaagaaaaa gaccggtcta cctcccttgg acaaagactt tgtccacatg 180
gaaaaagctg ggtaagtcct tactcgtgac agcgttccct ttctccagac tagacgccta 240
atagtgttct aatgtaccac tgggacacgc ctcgctgcct gtgcaccatg ctccatactc 300
aacgctgcta caggggcgta atccttccag tcggcaacat tgtgagcgac atgttgagcg 360
atagagatga tttgtatatc agatccgggc cagggcctgc tcgtgggaag attgcccatc 420
tcagttcgcc cccaaacgcg cacgcttcga gtgagtcgag cacaggattg ttgcgctgca 480
aaaactatgg atgcaatcag tcattttcgg aagaaaacaa ttcagaagag gcgtgccgct 540
ttcacaaggc accccccgtc tttcatgata cgaagaaagg gtggtcgtgc tgcgcgaagc 600
gagtatatga ctgggacgag ttccatacgg taagcgtgga agtgttcgtt ctcggcccca 660
ggactttgtt ttgaggcaat tggtgtactt taattggcgg ataaagggag gactcacaac 720
tttcgatatt caccgtctcc agatcgaggg gtgcaccaca ggacggcaca gtctcatcga 780
tccgaaggaa attttcgcgc cgtcccccac cctggctgca gccgcgcagg ccgagagggg 840
agattgcagc aatacgtcaa gcgctgctac agtcatcaag agcattgatg aattcaatca 900
gtcgaatcca aatgccgccg ctgcatgcaa aacagcagcc tcgatgacgc tggcgggcac 960
gcgctgcacc gtcaaaccgg acgggtctgc cacctgtttg aacaaaggct gccaaaagga 1020
ctacttgctc aaggagaatc acccctctgc atgtcggtaa ggacaccgcg ctcgatggaa 1080
tcgtgagctt tacgttccca cgccaacact tcgccatttc tcctcccttc ctttctttag 1140
ctaccacgca gccggccccg tcttccacga cgcgggtaaa tactggtcat gttgccctgg 1200
aacggtcaag tacgacttcg acgactttct caagatccct ggatgcatgc tcagtagtca 1260
ttacgacgga agccaggaga gcctggaggc gttcactaga cacgccaaaa cgtctgaggg 1320
cacatga 1327
<210> 2
<211> 821
<212> DNA
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> CHORD-3266 5'融合转录物
<400> 2
atgaaactct atgtgcacta cgaggaggct ggtcaggatg aaaaggcatt gacgcttaag 60
ttgactctgc ccaaaagctg ggcggagcag ccgttgcttc aagtactgga gctgttcatc 120
gaatcctaca acaagaaaaa gaccggtcta cctcccttgg acaaagactt tgtccacatg 180
gaaaaagctg ggggcgtaat ccttccagtc ggcaacattg tgagcgacat gttgagcgat 240
agagatgatt tgtatatcag atccgggcca gggcctgctc gtgggaagat tgcccatctc 300
agttcgcccc caaacgcgca cgcttcgagt gagtcgagca caggattgtt gcgctgcaaa 360
aactatggat gcaatcagtc attttcggaa gaaaacaatt cagaagaggc gtgccgcttt 420
cacaaggcac cccccgtctt tcatgatacg aagaaagggt ggtcgtgctg cgcgaagcga 480
gtatatgact gggacgagtt ccatacgatc gaggggtgca ccacaggacg gcacagtctc 540
atcgatccga aggaaatttt cgcgccgtcc cccaccctgg ctgcagccgc gcaggccgag 600
aggggagatt gcagcaatac gtcaagcgct gctacagtca tcaagagcat tgatgaattc 660
aatcagtcga atccaaatgc cgccgctgca tgcaaaacag cagcctcgat gacgctggcg 720
ggcacgcgct gcaccgtcaa accggacggg tctgccacct gtttgaacaa aggctgccaa 780
aaggactact tgctcaagga gaatcacccc tctgcatgtc g 821
<210> 3
<211> 420
<212> DNA
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> CHORD-3266 3'融合转录物
<400> 3
atgggcagcc cacagcggtt ggcatcaggg agttgcttcc ctctggctac gtctgggagg 60
gttgaaattc actggccgtc gttttacaac gtcgtaactg ggaaaaccct ggcgttaccc 120
aacttaatcg ccttgcagca catccccctt tcgccagtgg caaacagcta taatcgtgag 180
ctttacgttc ccacgccaac acttcgccat ttctcctccc ttcctttctt tagctaccac 240
gcagccggcc ccgtcttcca cgacgcgggt aaatactggt catgttgccc tggaacggtc 300
aagtacgact tcgacgactt tctcaagatc cctggatgca tgctcagtag tcattacgac 360
ggaagccagg agagcctgga ggcgttcact agacacgcca aaacgtctga gggcacatga 420
<210> 4
<211> 65
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> CHORD-3266结构域2
<400> 4
Gly Ser Ala Thr Cys Leu Asn Lys Gly Cys Gln Lys Asp Tyr Leu Leu
1 5 10 15
Lys Glu Asn His Pro Ser Ala Cys Arg Tyr His Ala Ala Gly Pro Val
20 25 30
Phe His Asp Ala Gly Lys Tyr Trp Ser Cys Cys Pro Gly Thr Val Lys
35 40 45
Tyr Asp Phe Asp Asp Phe Leu Lys Ile Pro Gly Cys Met Leu Ser Ser
50 55 60
His
65
<210> 5
<211> 51
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<220>
<221> misc_feature
<223> 5'引物MCA-1185
<400> 5
ttccacccaa gcagtggtat caacgcagag tggcctaagg gaaaacaaca g 51
<210> 6
<211> 60
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<220>
<221> misc_feature
<223> 用于Y2H文库第二链合成的引物
<400> 6
gtatcgatgc ccaccctcta gaggccgagg cggccgacac ggtacccgct tttttttttt 60
<210> 7
<211> 44
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<220>
<221> misc_feature
<223> 引物JLC-P6317-5901PRO-8611-F
<400> 7
gtttttcttt ggcaggcaaa catgcaagca tcccaaaaga cgtc 44
<210> 8
<211> 43
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<220>
<221> misc_feature
<223> 引物JLC-P6317-8611-T9-R
<400> 8
ctatgcacct tcccccttga ctcttagctt gagttgctgt tgc 43
<210> 9
<211> 1918
<212> DNA
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> SKP-8611编码序列
<220>
<221> misc_feature
<222> (1136)..(1136)
<223> n是a、c、g或t
<400> 9
atgcaagcat cccaaaagac gtccgatgtg caagaggagg atggtccgcc caaatacatt 60
acattacaag cccgagatgg cacgctcgac gagccagtgg acgcccgcat tctcttgccg 120
tccgacctcc tgcgaagcat gctccccgaa aagtgtaagc tgcgaagtga atgcacaaaa 180
gagataggag ccatgttcta ttttggtgtc gtttttgggg ttgtgtgact gattttgtcc 240
cgtacgaagg gcgtgtctgt ctctcctttt tgaggatgtg tcgatatacg tctcgtcctt 300
tttatcaatt cttatcgggt tcgtccactc ccaaacggct tgcatttcag tgtccgaaat 360
tgaggacgat ttccagatac cgctgcaggg agttgacaag gccgtactgg agaaggtagg 420
gtggaaggga aattaggatg gggagaggaa gggagatgga taggagagga taacaatgcc 480
gtatttcatt ttgtttcctt gtcttatcat ggtaggtggt agagtattta catctttatc 540
gcgaagagcc gatgtacaag atcgagaagg taatcgaggg agggagggag ggaggggggg 600
agggagaggg gagggaaggg agaggggaaa ggaggaggga tcaattcctc attctgggcg 660
tgtacctcac tctctctcct tctccttcat tttcaagccc ttgaacaaag tgaagctgta 720
cgacctggtt caaccccaat acgatcagtt catcaatgcc cttcattaca agacgatatt 780
ccagatcatc gatgcggtgc gcttcctccc ttccttcatt tctccctccc ttcctctctt 840
ctccctccct tcttccctcc caccctccct cccttctccc tcccctcttc cttcccacct 900
ccctttcttc ctctttccct ttcttcctcc ctttctcccc cttgccccat tctctctttg 960
ttcttttcct gttttcccct cgcctccttc cctgacttcc tttcgagttt ttgtgttaat 1020
atgtatatat atatatacaa tttttaacta tatatatata atcttaacct caaagcgtat 1080
ccctagcggt cttagctgac atgtgcaaat cacagaatcc cttcttaccc ctcctnccct 1140
cgacccatcc ctcgcccgtt cttctcgcgg ccttcttgca aaccactcgt ggtagacatg 1200
gtccaatcct ttccgcctcc cccttccccc ctccctctgt cccttcctcc tccccctgcg 1260
tcttgctcct tgactgtcta ctccaggcga atttcctggg aatcgagccc ctcctttccc 1320
tctcgttatc gtgggtggcc ttcgtcctta aaggtaccac tgggaagaag gagtggaggg 1380
aggagtggag gagtcaaagg agggagatga aacacatgaa gcaatactcg aacaagaatt 1440
aagacctccc tggcatcgaa tttttggcgc ccgtgcctcg gccggtctca acaccacctc 1500
ctcctcccgc cctcccgccc atatatccct cttgataatc cttgtttccc gatagggcct 1560
actgtggaag agttcaagaa actgttcacg atccataacg attttacgcc ggaggtgagg 1620
gaaaaacgag tggattgagg gggagagggg agaaggaaga gagcagggat cgctaggagg 1680
tagcacacgt cggcgggtgt ggtgtggcat ttccccgttc ccctccctca tgtggtcgag 1740
tttcattcga tttcataatg tcttttgacc ccccgttctc cttcctcgca cggtcgcggc 1800
cttctcctta caggaggagg ctatcttcag gcgcgagtac ctccttccgc gccggaatcg 1860
ggcgacaagc gagggcggga gggagagagg cgcgtctggc aacagcaact caagctaa 1918
<210> 10
<211> 19193
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<220>
<221> misc_feature
<223> SKP-8611过表达载体
<400> 10
gctagtgata ataagtgact gaggtatgtg ctcttcttat ctccttttgt agtgttgctc 60
ttattttaaa caactttgcg gttttttgat gactttgcga ttttgttgtt gctttgcagt 120
aaattgcaag atttaataaa aaaacgcaaa gcaatgatta aaggatgttc agaatgaaac 180
tcatggaaac acttaaccag tgcataaacg ctggtcatga aatgacgaag gctatcgcca 240
ttgcacagtt taatgatgac agcccggaag cgaggaaaat aacccggcgc tggagaatag 300
gtgaagcagc ggatttagtt ggggtttctt ctcaggctat cagagatgcc gagaaagcag 360
ggcgactacc gcacccggat atggaaattc gaggacgggt tgagcaacgt gttggttata 420
caattgaaca aattaatcat atgcgtgatg tgtttggtac gcgattgcga cgtgctgaag 480
acgtatttcc accggtgatc ggggttgctg cccataaagg tggcgtttac aaaacctcag 540
tttctgttca tcttgctcag gatctggctc tgaaggggct acgtgttttg ctcgtggaag 600
gtaacgaccc ccagggaaca gcctcaatgt atcacggatg ggtaccagat cttcatattc 660
atgcagaaga cactctcctg cctttctatc ttggggaaaa ggacgatgtc acttatgcaa 720
taaagcccac ttgctggccg gggcttgaca ttattccttc ctgtctggct ctgcaccgta 780
ttgaaactga gttaatgggc aaatttgatg aaggtaaact gcccaccgat ccacacctga 840
tgctccgact ggccattgaa actgttgctc atgactatga tgtcatagtt attgacagcg 900
cgcctaacct gggtatcggc acgattaatg tcgtatgtgc tgctgatgtg ctgattgttc 960
ccacgcctgc tgagttgttt gactacacct ccgcactgca gtttttcgat atgcttcgtg 1020
atctgctcaa gaacgttgat cttaaagggt tcgagcctga tgtacgtatt ttgcttacca 1080
aatacagcaa tagcaatggc tctcagtccc cgtggatgga ggagcaaatt cgggatgcct 1140
ggggaagcat ggttctaaaa aatgttgtac gtgaaacgga tgaagttggt aaaggtcaga 1200
tccggatgag aactgttttt gaacaggcca ttgatcaacg ctcttcaact ggtgcctgga 1260
gaaatgctct ttctatttgg gaacctgtct gcaatgaaat tttcgatcgt ctgattaaac 1320
cacgctggga gattagataa tgaagcgtgc gcctgttatt ccaaaacata cgctcaatac 1380
tcaaccggtt gaagatactt cgttatcgac accagctgcc ccgatggtgg attcgttaat 1440
tgcgcgcgta ggagtaatgg ctcgcggtaa tgccattact ttgcctgtat gtggtcggga 1500
tgtgaagttt actcttgaag tgctccgggg tgatagtgtt gagaagacct ctcgggtatg 1560
gtcaggtaat gaacgtgacc aggagctgct tactgaggac gcactggatg atctcatccc 1620
ttcttttcta ctgactggtc aacagacacc ggcgttcggt cgaagagtat ctggtgtcat 1680
agaaattgcc gatgggagtc gccgtcgtaa agctgctgca cttaccgaaa gtgattatcg 1740
tgttctggtt ggcgagctgg atgatgagca gatggctgca ttatccagat tgggtaacga 1800
ttatcgccca acaagtgctt atgaacgtgg tcagcgttat gcaagccgat tgcagaatga 1860
atttgctgga aatatttctg cgctggctga tgcggaaaat atttcacgta agattattac 1920
ccgctgtatc aacaccgcca aattgcctaa atcagttgtt gctctttttt ctcaccccgg 1980
tgaactatct gcccggtcag gtgatgcact tcaaaaagcc tttacagata aagaggaatt 2040
acttaagcag caggcatcta accttcatga gcagaaaaaa gctggggtga tatttgaagc 2100
tgaagaagtt atcactcttt taacttctgt gcttaaaacg tcatctgcat caagaactag 2160
tttaagctca cgacatcagt ttgctcctgg agcgacagta ttgtataagg gcgataaaat 2220
ggtgcttaac ctggacaggt ctcgtgttcc aactgagtgt atagagaaaa ttgaggccat 2280
tcttaaggaa cttgaaaagc cagcaccctg atgcgaccac gttttagtct acgtttatct 2340
gtctttactt aatgtccttt gttacaggcc agaaagcata actggcctga atattctctc 2400
tgggcccact gttccacttg tatcgtcggt ctgataatca gactgggacc acggtcccac 2460
tcgtatcgtc ggtctgatta ttagtctggg accacggtcc cactcgtatc gtcggtctga 2520
ttattagtct gggaccacgg tcccactcgt atcgtcggtc tgataatcag actgggacca 2580
cggtcccact cgtatcgtcg gtctgattat tagtctggga ccatggtccc actcgtatcg 2640
tcggtctgat tattagtctg ggaccacggt cccactcgta tcgtcggtct gattattagt 2700
ctggaaccac ggtcccactc gtatcgtcgg tctgattatt agtctgggac cacggtccca 2760
ctcgtatcgt cggtctgatt attagtctgg gaccacgatc ccactcgtgt tgtcggtctg 2820
attatcggtc tgggaccacg gtcccacttg tattgtcgat cagactatca gcgtgagact 2880
acgattccat caatgcctgt caagggcaag tattgacatg tcgtcgtaac ctgtagaacg 2940
gagtaacctc ggtgtgcggt tgtatgcctg ctgtggattg ctgctgtgtc ctgcttatcc 3000
acaacatttt gcgcacggtt atgtggacaa aatacctggt tacccaggcc gtgccggcac 3060
gttaaccggg ctgcatccga tgcaagtgtg tcgctgtcga cgagctcgcg agctcggaca 3120
tgaggttgcc ccgtattcag tgtcgctgat ttgtattgtc tgaagttgtt tttacgttaa 3180
gttgatgcag atcaattaat acgatacctg cgtcataatt gattatttga cgtggtttga 3240
tggcctccac gcacgttgtg atatgtagat gataatcatt atcactttac gggtcctttc 3300
cggtgatccg acaggttacg gggcggcgac ctcgcgggtt ttcgctattt atgaaaattt 3360
tccggtttaa ggcgtttccg ttcttcttcg tcataactta atgtttttat ttaaaatacc 3420
ctctgaaaag aaaggaaacg acaggtgctg aaagcgagct ttttggcctc tgtcgtttcc 3480
tttctctgtt tttgtccgtg gaatgaacaa tggaagtccg agctcatcgc taataacttc 3540
gtatagcata cattatacga agttatattc gatgcgcccg ggcgcctatt tccgagtgca 3600
tgtcccagtc gaaggacgcc tggggggtga tgcaaggata catcgatcat ttggccgtgc 3660
attttgtcaa accgggtaag tggattacgg agaatcttcg tttctttcct cctcttctct 3720
tagtggcctt ccctcctttt tttttcattt gtttcctttc ccgtgcctgt ttgcacggca 3780
aaattttcct ctttccacct taacgtcctt cctcttctcc tttcttcctg ccctcccccc 3840
gattcctctc cccccttcct cccctccctc cctccccagt cgtggaaacc gacgtcatcc 3900
ggagcaacgg cggcctggtc gtgacccgtt cggtccactc ctggctcacg ggccgccggg 3960
agcccatcct cgccctctcc ctcgactcct cagaccctcg agtcctcttc tccttcctcg 4020
gcgccgactt gagcctgagc ccggagggcg gggtggaggg cgggcacggc acctgccgta 4080
tgtccaacga cgtggaaaac accgtcaccg cctgtgcccc cgtcctggac gaggcgtcgg 4140
gggaaatccg ggaataccga aaaaccccga gcaccttcca caccggcaag catttcccgc 4200
accggatcgg acagttggcc gcccaccatg gagacgagaa aatttggggc gtgtggcctc 4260
gaaagacgat cgttatccag gcggggtccc gtgagggagg cagcaatgtg caatacggac 4320
attccgacgg catgcagttc ccccccttgc accgcctcga agaagcccgc ccggtcgcct 4380
cggaggatac cggcgtgtcc tcggtattaa acggcacact cgggctgaag gaactggcgg 4440
ccttgtcctt cttggacgcg ggggaggcgg agacctcgac ggggatggaa agagaggagg 4500
ttgtcatggc ggatccggcg tcggtatcgg aggatcaact ttcggtgttt ttgagcaagt 4560
tcgtcctccc cctctggttc cgcttccagg aaacggtctc cttgcgaggc atccgccttt 4620
tgaaattcaa cctctcgccg ttgactttct ccccgcaacg gccggacgcc tcgggctttt 4680
ccatgcttgg cctgggggag ggcgggcggg agggtctatt caatctgacc gaccttcaaa 4740
cgatgccatc ttttctgagc acgccacaat ttcacggtga gaaccaggga gggacgcgtg 4800
gggagggtga tggatgggga agaaacaggt gggagggtaa ggtaacgcga gagcgtaggg 4860
ttccacgtca tctctctctc tacattcttg ccccttcaat ccttcacacc gtgggacatg 4920
agagaacaag cgtgttcctt gtgcccactc ccggcccctc cccgccccgc ccttccctcc 4980
ccatcgcctt cttccaggat acccctccct gcgaaagctt ccctgtctgg ccagcctcag 5040
gttgtcggag atcgggaaga ggcaggccgc ccgaggaagc tacttggcgg tggagcccta 5100
caccggtacg gacaagacag aggaatgcac aggcgggagg acggggggat taaagggatg 5160
gggcaagtta aggaagcttc aatatcctgg atcccggggg gcgggcgtgg gcgagtcggg 5220
gagggctcca gacgaggttt ggtcttctcg gtcgccgaaa ttcggcccct tcctgccgca 5280
tcgcggaatt cccacgtctt ccaccctcat cacatgacgc ctccccaatc ttcctttcct 5340
ttcctctcca cctcctatca ttgccttgtc cctccgtcct cctcacgctc ttcttccctc 5400
ctcatcactg tccttccttc cctcctcatc ccaacacacc tcttttccct tcctcctcgg 5460
ccccttcctt tgccctcctg acctccacct tcctcccctc ctcatccctc cttacctcca 5520
ccttcctccc ctcctcatcc cttcttacct cgaccttcct tccctcctca tccctcctta 5580
cctcgacctt ccttccctcc tcatccctcc ttccctccac cctccctcca cctcaccgct 5640
cttcaccttc cttcttccct cctcctcctc cctccttcct tccatctccc ctcctccttc 5700
ccgccccctc acccgtaggc aagacaatga gcgcccacat tgccatgcaa gtctctggcg 5760
ccatcaaaac cgcctccggg aacggaccca gctatgacat ctggtaaggg agggagggag 5820
ggagggaggg aggaaggaag ggagggaggg agggagggag ggagggaggg aggtagaatg 5880
acacaacgtc gagtgcgaag gtcgccctcc ccctcgcaac gagccgtgcc aggaacagga 5940
ccttaccttc tcctccccct tgtccttccc atcccatcac ccccgcctcg ctccttccct 6000
ccttatcccc tcctccctcc ttcccacctc atcaggcacc atcaggtata ccccgccgag 6060
atcgtcccca ccttgtggat ggctcaatct gccgaggttg gccctgcgga tgcccgtacc 6120
ttccacgata aaatctatcg ctctgccagc atcttggccg atatcaggcg ctactccttc 6180
atcgggggaa taacggcctt ggccctgggg ctcttgcttg tgcttgggag ctgttggtgg 6240
gcgaggaagg gagggaggga gggagggagg gagggaggga gggagggagg gaggaggcag 6300
gacgtggaag cggcggtgga gatgcaggag agaggggagg gaggaggagg agggaggaag 6360
ccggggaaga aggatctctt cggagcgaga cgtccagtgc cggccgttta agcgccgtcc 6420
gttacttcgc aacggctgta tttttaaatt tgaaatttaa aagggggagt ctggaggcgt 6480
agagaccctg gggcctgggc gttcggctct gtcattgttc gcgtgcgtgg ccgtgccact 6540
ggttctttcc agctttaggg ggaagaaggg agggagggag ggagggaggg agggaagtag 6600
gaaaggaagg agcggggaag ttcatgggac tgagagcgtg agaagaagag agggaaggcg 6660
gggagttcag tcataccatg tctcgtctct tggggaagga gaggtggagg gacgttggga 6720
tggcggggtg gagccacagt gggaggagaa agacgtttat ggaaagatct agaaccttcg 6780
gcgaaagtgt aggaaaaact ccagggctgg gtgcctctca cagcaagcct tcaaccgggg 6840
gagaaagatt tcaaatttca aaacagaaga ggaagacttc gcagtcgtaa cggctcccca 6900
acggttgaaa cgccctgggt cccttccagc ctcatcacgt gcattttctc tttctttcct 6960
ccctccttcc tcctcctccc cgctctcccc ttccctcttc ctcaagcttt tctttggcat 7020
cctaccgccc ctaccttcac ctcttcacgc actcacacct tccagatcca tgcgatcgca 7080
acaagttttg atttttgtgt tgtcgtattt ttacgcagga aaatgtcaga gccgatacat 7140
tcgagctacc tggggttgtc tttttaagag tcccagcatt gtgttcacct acgatagtgc 7200
atggctgcaa gtaaattcat tttgcgataa ccacaaaact agccaattta cgaatctgtg 7260
aagaagtttt tcgcgttcta cggtcctctt gtagtaaccc atcgatgttg cttcaagata 7320
tgggcgctgg tcgatggaaa aaaccacagg actagttaaa atcatagatt ccacgaaatt 7380
attaaaatat tatttttcat gccctgcggt ccgtgaagac tgcgagcacg cgccttagga 7440
gatgagagcc tctcgacatc tcctgcatac atctgcagac gacaagagag catcaggggc 7500
taagggaggc atggaatagc gtcgacacat cgacagctca ttccatattc tctctaagtt 7560
cccctgtgga gcgattgctt cgatcagtga agatcacctg gccctttcct cgttgaaagt 7620
gaggctcagg acggtcggaa ggtattgtat tggtacgctt tgacgtagcc ttctcgttga 7680
cactttgact tgcatacgac ccttgtggcg acggctgcat ttgtgcgctg ttgctccaag 7740
aactatgcat cgaaggcaca cgccctggaa gccccgacac ctctgtttga gacactgctt 7800
caaatcactt gagcaggact cacaatgaga gtacatcgta ttgggtttgg cggccgccgt 7860
atggtcgacg gttgctcgga tggggggggc ggggagcgat ggagggagga agatcaggta 7920
aggtctcgac agactagaga agcacgagtg caggtataag aaacagcaaa aaaaagtaat 7980
gggcccaggc ctggagaggg tatttgtctt gtttttcttt ggccaggaac ttgttctcct 8040
ttcttcgttt ctaggacccc gatccccgct cgcatttctc tcttcctcag ccgaagcgca 8100
gcggtaaagc atccatttta tcccaccgaa agggcgctcc cagccttcgt cgagcggaac 8160
cggggttaca gtgcctcact cctttgcacg cggtcgggtg ctcggcctac ggttgcccga 8220
gtccgcaagc acctcaacac agccgtctgt ccacaccgca gccgaccggc gtgcgatttg 8280
ggtccgaccc accgtcccag ccccgctgcg gactatcgcg tcgcagcggc cctgcgccca 8340
cgcggcgtcg tcgaagttgc cgtcgacgag agactggtaa agctgatcga gtccgatacg 8400
caacatatag gcgcggagtc gtggggagcc ggccagctcc gggtgcctcc gttcaaagta 8460
gcgtgtctgc tgctccatgc acgccaacca gggacgccag aagaatatgt tcgccacttc 8520
gtattggcta tcaccaaaca tcgcttcgga ccagtcgatg acagcagtaa tccgaccatt 8580
gtctgtaagt acgttattgc tgccgaaatc cgcgtgcacc aggtgcctga cctcagggca 8640
atcctcggcc cacaacatga gttcgtccag tgcttgggcc acggatgcag acacggtgtc 8700
atccatgact gtctgccaat gatagacgtg aggatcggca atggcgcaga tgaagtctcg 8760
ccaggtcgtg tactgcccga tgccctgggg cccaaaaggt ccaaagccgg acgtctgaga 8820
cagatctgcg gcagcgatcg cgtccatggc ctcggccacg ggttgcaaaa cggcaggcaa 8880
ttcagtttcg ggcagatctt gcaacgtcac tccctgggct cggcgcgaga tgcagtacgt 8940
gagagattcg ctaaactccc caatgtccag tacctctggt atggggagag cggcggaggc 9000
gaaatgacgg tagacatacc gatccttgta gaacccgtcc gcacaactat taaccctcaa 9060
cacgtatccc cgacccccta cgtcaaacga gaacgcccta ctctcctctc cctcgctcag 9120
ttgcatcaag tcggagacag agtcgaactt ctcaataagg aatttctcca cggacgtagc 9180
ggtcagttcc ggtttcttcc ccatcgagct cggtacccgg ggatccatga ttgttgtatt 9240
atgtacctat gtttgtgatg agacaataaa tatgagaaga gaacgttgcg gccacttttt 9300
tctccttcct tcgcgtgctc atgttggtgg tttgggaggc agaagatgca tggagcgcca 9360
cacattcggt aggacgaaac agcctccccc acaaagggac catgggtagc taggatgacg 9420
cacaagcgag ttcccgctct cgaagggaaa cccaggcatt tccttcctct tttcaagcca 9480
cttgttcacg tgtcaacaca attttggact aaaatgcccc tcggaactcg gcaggcctcc 9540
ctctgctccg ttgtcctggt cgccgagaac gcgagaccgt gccgcatgcc atcgatctgc 9600
tcgtctgtac tactaatcgt gtgcgtgttc gtgcttgttt cgcacgaaat tgtcctcgtt 9660
cggccctcac aacggtggaa atcggtgcta gaataaagtg aggtggctta tttcaatggc 9720
ggccgtcatc atgcgggatc aactgaagta cggcgggttc tcgagatttc atcgtgctcg 9780
tccagagcag gtgttttgcc tgcagctctt catgtttagg ggtcatgatt tcatctgata 9840
tgccgtaaga aaaccaatat tcacttctca attttccatg gaaaggtgaa ggcctaggtt 9900
gtgtgcgagg caacgactgg ggagggatcg caacattctt gctaacctcc cctctatctt 9960
ggccgctgtg aatcggcata tttaccgggc tgaattgaga aagtgttttg agggaattaa 10020
aaggtggctg tcttgcaagc ttggcttcag tgcctgctta attcgaaccg atccagcttg 10080
tgatgaggcc ttcctaagcc tggtagtcag aagcgacatg gcgctataaa tttcgtctca 10140
gttggagagt agaaaagcat gattcgaaca cggttttcaa ctgccaaaga tatctccatt 10200
gtttccttca atctgtacac ctgcacggtg caccagttgg tacggcatat tatggtttaa 10260
taagcataca tcatatgaat acaattcagc ttaaatttat catacaaaga tgtaagtgca 10320
gcgtgggtct gtaacgatcg ggcgtaattt aagataatgc gagggaccgg gggaggtttt 10380
ggaacggaat gaggaatggg tcatggccca taataataat atgggtttgg tcgcctcgca 10440
cagcaaccgt acgtgcgaaa aaggaacaga tccatttaat aagttgaacg ttattctttc 10500
ctatgcaatg cgtgtatcgg aggcgagagc aagtcatagg tggctgcgca caataattga 10560
gtctcagctg agcgccgtcc gcgggtggtg tgagtggtca tcctcctccc ggcctatcgc 10620
tcacatcgcc tctcaatggt ggtggtgggg cctgatatga cctcaatgcc gacccatatt 10680
aaaacccagt aaagcattca ccaacgaacg aggggctctt ttgtgtgtgt tttgagtatg 10740
attttacacc tctttgtgca tctctctggt cttccttggt tcccgtagtt tgggcatcat 10800
cactcacgct tccctcgacc ttcgttcttc ctttacaacc ccgacacagg tcagagttgg 10860
agtaatcaaa aaaggggtgc acgaatgaga tacattagat tttgacagat atccttttac 10920
tggagagggt tcaagggatc aaatgaacag cgggcgttgg caatctaggg agggatcgga 10980
ggttggcagc gagcgaaagc gtgtccatcc ttttggctgt cacacctcac gaaccaactg 11040
ttagcaggcc agcacagatg acatacgaga atctttatta tatcgtagac cttatgtgga 11100
tgacctttgg tgctgtgtgt ctggcaatga acctgaaggc ttgataggga ggtggctccc 11160
gtaaaccctt tgtcctttcc acgctgagtc tcccccgcac tgtcctttat acaaattgtt 11220
acagtcatct gcaggcggtt tttctttggc aggcaaacat gcaagcatcc caaaagacgt 11280
ccgatgtgca agaggaggat ggtccgccca aatacattac attacaagcc cgagatggca 11340
cgctcgacga gccagtggac gcccgcattc tcttgccgtc cgacctcctg cgaagcatgc 11400
tccccgaaaa gttgtccgaa attgaggacg atttccagat accgctgcag ggagttgaca 11460
aggccgtact ggagaaggtg gtagagtatt tacatcttta tcgcgaagag ccgatgtaca 11520
agatcgagaa gcccttgaac aaagtgaagc tgtacgacct ggttcaaccc caatacgatc 11580
agttcatcaa tgcccttcat tacaagacga tattccagat catcgatgcg gcgaatttcc 11640
tgggaatcga gcccctcctt tccctctcgt tatcgtgggt ggccttcgtc cttaaagggc 11700
ctactgtgga agagttcaag aaactgttca cgacccataa cgattttacg ccggaggagg 11760
aggctatctt caggcgcgag tacctccttc cgcgccggaa tcgggcgaca agcgagggcg 11820
ggagggagag aggcgggtct ggcaacagca actcaagcta agagtcaagg gggaaggtgc 11880
atagtgtgca acaacagcat taacgtcaaa gaaaactgca cgttcaagcc cgcgtgaacc 11940
tgccggtctt ctgatcgcct acatatagca gatactagtt gtactttttt ttccaaaggg 12000
aacattcatg tatcaatttg aaataaacat ctatcctcca gatcaccagg gccagtgagg 12060
ccggcataaa ggacggcaag gaaagaaaag aaagaaagaa aaggacactt atagcatagt 12120
ttgaagttat aagtagtcgc aatctgtgtg cagccgacag atgctttttt tttccgtttg 12180
gcaggaggtg tagggatgtc gaagaccagt ccagctagta tctatcctac aagtcaatca 12240
tgctgcgaca aaaatttctc gcacgaggcc tctcgataaa caaaacttta aaagcacact 12300
tcattgtcat gcagagtaat aactcttccg cgtcgatcaa tttatcaatc tctatcattt 12360
ccgccccttt ccttgcatag agcaagaaaa gcgacccgga tgaggataac atgtcctgcg 12420
ccagtagtgt ggcattgcct gtctctcatt tacacgtact gaaagcataa tgcacgcgca 12480
taccaatatt tttcgtgtac ggagatgaag agacgcgaca cgtaagatca cgagaaggcg 12540
agcacggttg ccaatggcag acgcgctagt ctccattatc gcgttgttcg gtagcttgct 12600
gcatgtcttc agtggcacta tatccactct gcctcgtctt ctacacgagg gccacatcgg 12660
tgcaagttcg aaaaatcata tctcaatctt cagatccttt ccagaaacgg tgctcaggcg 12720
ggaaagtgaa ggttttctac tctagtggct accccaattc tctccgactg tcgcagacgg 12780
tccttcgttg cgcacgcacc gcgcactacc tctgaaattc gacaaccgaa gttcaatttt 12840
acatctaact tctttcccat tctctcacca aaagcctagc ttacatgttg gagagcgacg 12900
agagcggcct gcccgccatg gagatcgagt gccgcatcac cggcaccctg aacggcgtgg 12960
agttcgagct ggtgggcggc ggagagggca cccccgagca gggccgcatg accaacaaga 13020
tgaagagcac caaaggcgcc ctgaccttca gcccctacct gctgagccac gtgatgggct 13080
acggcttcta ccacttcggc acctacccca gcggctacga gaaccccttc ctgcacgcca 13140
tcaacaacgg cggctacacc aacacccgca tcgagaagta cgaggacggc ggcgtgctgc 13200
acgtgagctt cagctaccgc tacgaggccg gccgcgtgat cggcgacttc aaggtgatgg 13260
gcaccggctt ccccgaggac agcgtgatct tcaccgacaa gatcatccgc agcaacgcca 13320
ccgtggagca cctgcacccc atgggcgata acgatctgga tggcagcttc acccgcacct 13380
tcagcctgcg cgacggcggc tactacagct ccgtggtgga cagccacatg cacttcaaga 13440
gcgccatcca ccccagcatc ctgcagaacg ggggccccat gttcgccttc cgccgcgtgg 13500
aggaggatca cagcaacacc gagctgggca tcgtggagta ccagcacgcc ttcaagaccc 13560
cggatgcaga tgccggtgaa gaataagggt gggaaggagt cggggagggt cctggcagag 13620
cggcgtcctc atgatgtgtt ggagacctgg agagtcgaga gcttcctcgt cacctgattg 13680
tcatgtgtgt ataggttaag ggggcccact caaagccata aagacgaaca caaacactaa 13740
tctcaacaaa gtctactagc atgccgtctg tccatcttta tttcctggcg cgcctatgct 13800
tgtaaaccgt tttgtgaaaa aatttttaaa ataaaaaagg ggacctctag ggtccccaat 13860
taattagtaa tataatctat taaaggtcat tcaaaaggtc atccagtcga ccaattctca 13920
tgtttgacag cttatcatcg aatttctgcc attcatccgc ttattatcac ttattcaggc 13980
gtagcaacca ggcgtttaag ggcaccaata actgccttaa aaaaattacg ccccgccctg 14040
ccactcatcg cagtactgtt gtaattcatt aagcattctg ccgacatgga agccatcaca 14100
aacggcatga tgaacctgaa tcgccagcgg catcagcacc ttgtcgcctt gcgtataata 14160
tttgcccatg gtgaaaacgg gggcgaagaa gttgtccata ttggccacgt ttaaatcaaa 14220
actggtgaaa ctcacccagg gattggctga gacgaaaaac atattctcaa taaacccttt 14280
agggaaatag gccaggtttt caccgtaaca cgccacatct tgcgaatata tgtgtagaaa 14340
ctgccggaaa tcgtcgtggt attcactcca gagcgatgaa aacgtttcag tttgctcatg 14400
gaaaacggtg taacaagggt gaacactatc ccatatcacc agctcaccgt ctttcattgc 14460
catacgaaat tccggatgag cattcatcag gcgggcaaga atgtgaataa aggccggata 14520
aaacttgtgc ttatttttct ttacggtctt taaaaaggcc gtaatatcca gctgaacggt 14580
ctggttatag gtacattgag caactgactg aaatgcctca aaatgttctt tacgatgcca 14640
ttgggatata tcaacggtgg tatatccagt gatttttttc tccattttag cttccttagc 14700
tcctgaaaat ctcgataact caaaaaatac gcccggtagt gatcttattt cattatggtg 14760
aaagttggaa cctcttacgt gccgatcaac gtctcatttt cgccaaaagt tggcccaggg 14820
cttcccggta tcaacaggga caccaggatt tatttattct gcgaagtgat cttccgtcac 14880
aggtatttat tcgcgataag ctcatggagc ggcgtaaccg tcgcacagga aggacagaga 14940
aagcgcggat ctgggaagtg acggacagaa cggtcaggac ctggattggg gaggcggttg 15000
ccgccgctgc tgctgacggt gtgacgttct ctgttccggt cacaccacat acgttccgcc 15060
attcctatgc gatgcacatg ctgtatgccg gtataccgct gaaagttctg caaagcctga 15120
tgggacataa gtccatcagt tcaacggaag tctacacgaa ggtttttgcg ctggatgtgg 15180
ctgcccggca ccgggtgcag tttgcgatgc cggagtctga tgcggttgcg atgctgaaac 15240
aattatcctg agaataaatg ccttggcctt tatatggaaa tgtggaactg agtggatatg 15300
ctgtttttgt ctgttaaaca gagaagctgg ctgttatcca ctgagaagcg aacgaaacag 15360
tcgggaaaat ctcccattat cgtagagatc cgcattatta atctcaggag cctgtgtagc 15420
gtttatagga agtagtgttc tgtcatgatg cctgcaagcg gtaacgaaaa cgatttgaat 15480
atgccttcag gaacaataga aatcttcgtg cggtgttacg ttgaagtgga gcggattatg 15540
tcagcaatgg acagaacaac ctaatgaaca cagaaccatg atgtggtctg tccttttaca 15600
gccagtagtg ctcgccgcag tcgagcgaca gggcgaagcc ctcgagctgg ttgccctcgc 15660
cgctgggctg gcggccgtct atggccctgc aaacgcgcca gaaacgccgt cgaagccgtg 15720
tgcgagacac cgcggccggc cgccggcgtt gtggatacct cgcggaaaac ttggccctca 15780
ctgacagatg aggggcggac gttgacactt gaggggccga ctcacccggc gcggcgttga 15840
cagatgaggg gcaggctcga tttcggccgg cgacgtggag ctggccagcc tcgcaaatcg 15900
gcgaaaacgc ctgattttac gcgagtttcc cacagatgat gtggacaagc ctggggataa 15960
gtgccctgcg gtattgacac ttgaggggcg cgactactga cagatgaggg gcgcgatcct 16020
tgacacttga ggggcagagt gctgacagat gaggggcgca cctattgaca tttgaggggc 16080
tgtccacagg cagaaaatcc agcatttgca agggtttccg cccgtttttc ggccaccgct 16140
aacctgtctt ttaacctgct tttaaaccaa tatttataaa ccttgttttt aaccagggct 16200
gcgccctgtg cgcgtgaccg cgcacgccga aggggggtgc ccccccttct cgaaccctcc 16260
cggtcgagtg agcgaggaag caccagggaa cagcacttat atattctgct tacacacgat 16320
gcctgaaaaa acttcccttg gggttatcca cttatccacg gggatatttt tataattatt 16380
ttttttatag tttttagatc ttctttttta gagcgccttg taggccttta tccatgctgg 16440
ttctagagaa ggtgttgtga caaattgccc tttcagtgtg acaaatcacc ctcaaatgac 16500
agtcctgtct gtgacaaatt gcccttaacc ctgtgacaaa ttgccctcag aagaagctgt 16560
tttttcacaa agttatccct gcttattgac tcttttttat ttagtgtgac aatctaaaaa 16620
cttgtcacac ttcacatgga tctgtcatgg cggaaacagc ggttatcaat cacaagaaac 16680
gtaaaaatag cccgcgaatc gtccagtcaa acgacctcac tgaggcggca tatagtctct 16740
cccgggatca aaaacgtatg ctgtatctgt tcgttgacca gatcagaaaa tctgatggca 16800
ccctacagga acatgacggt atctgcgaga tccatgttgc taaatatgct gaaatattcg 16860
gattgacctc tgcggaagcc agtaaggata tacggcaggc attgaagagt ttcgcgggga 16920
aggaagtggt tttttatcgc cctgaagagg atgccggcga tgaaaaaggc tatgaatctt 16980
ttccttggtt tatcaaacgt gcgcacagtc catccagagg gctttacagt gtacatatca 17040
acccatatct cattcccttc tttatcgggt tacagaaccg gtttacgcag tttcggctta 17100
gtgaaacaaa agaaatcacc aatccgtatg ccatgcgttt atacgaatcc ctgtgtcagt 17160
atcgtaagcc ggatggctca ggcatcgtct ctctgaaaat cgactggatc atagagcgtt 17220
accagctgcc tcaaagttac cagcgtatgc ctgacttccg ccgccgcttc ctgcaggtct 17280
gtgttaatga gatcaacagc agaactccaa tgcgcctctc atacattgag aaaaagaaag 17340
gccgccagac gactcatatc gtattttcct tccgcgatat cacttccatg acgacaggat 17400
agtctgaggg ttatctgtca cagatttgag ggtggttcgt cacatttgtt ctgacctact 17460
gagggtaatt tgtcacagtt ttgctgtttc cttcagcctg catggatttt ctcatacttt 17520
ttgaactgta atttttaagg aagccaaatt tgagggcagt ttgtcacagt tgatttcctt 17580
ctctttccct tcgtcatgtg acctgatatc gggggttagt tcgtcatcat tgatgagggt 17640
tgattatcac agtttattac tctgaattgg ctatccgcgt gtgtacctct acctggagtt 17700
tttcccacgg tggatatttc ttcttgcgct gagcgtaaga gctatctgac agaacagttc 17760
ttctttgctt cctcgccagt tcgctcgcta tgctcggtta cacggctgcg gcgagcatca 17820
cgtgctataa aaataattat aatttaaatt ttttaatata aatatataaa ttaaaaatag 17880
aaagtaaaaa aagaaattaa agaaaaaata gtttttgttt tccgaagatg taaaagactc 17940
tagggggatc gccaacaaat actacctttt atcttgctct tcctgctctc aggtattaat 18000
gccgaattgt ttcatcttgt ctgtgtagaa gaccacacac gaaaatcctg tgattttaca 18060
ttttacttat cgttaatcga atgtatatct atttaatctg cttttcttgt ctaataaata 18120
tatatgtaaa gtacgctttt tgttgaaatt ttttaaacct ttgtttattt ttttttcttc 18180
attccgtaac tcttctacct tctttattta ctttctaaaa tccaaataca aaacataaaa 18240
ataaataaac acagagtaaa ttcccaaatt attccatcat taaaagatac gaggcgcgtg 18300
taagttacag gcaagcgatc ctagtacact ctatattttt ttatgcctcg gtaatgattt 18360
tcattttttt ttttccacct agcggatgac tctttttttt tcttagcgat tggcattatc 18420
acataatgaa ttatacatta tataaagtaa tgtgatttct tcgaagaata tactaaaaaa 18480
tgagcaggca agataaacga aggcaaagat gacagagcag aaagccctag taaagcgtat 18540
tacaaatgaa accaagattc agattgcgat ctctttaaag ggtggtcccc tagcgataga 18600
gcactcgatc ttcccagaaa aagaggcaga agcagtagca gaacaggcca cacaatcgca 18660
agtgattaac gtccacacag gtatagggtt tctggaccat atgatacatg ctctggccaa 18720
gcattccggc tggtcgctaa tcgttgagtg cattggtgac ttacacatag acgaccatca 18780
caccactgaa gactgcggga ttgctctcgg tcaagctttt aaagaggccc tactggcgcg 18840
tggagtaaaa aggtttggat caggatttgc gcctttggat gaggcacttt ccagagcggt 18900
ggtagatctt tcgaacaggc cgtacgcagt tgtcgaactt ggtttgcaaa gggagaaagt 18960
aggagatctc tcttgcgaga tgatcccgca ttttcttgaa agctttgcag aggctagcag 19020
aattaccctc cacgttgatt gtctgcgagg caagaatgat catcaccgta gtgagagtgc 19080
gttcaaggct cttgcggttg ccataagaga agccacctcg cccaatggta ccaacgatgt 19140
tccctccacc aaaggtgttc ttatgtagtt ttacacagga gtctggactt gac 19193
<210> 11
<211> 22
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<220>
<221> misc_feature
<223> 引物JLC-SKP-8611-RT-F SKP qRT
<400> 11
accccaatac gatcagttca tc 22
<210> 12
<211> 22
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<220>
<221> misc_feature
<223> 引物JLC-SKP-8611-RT-R SKP qRT
<400> 12
cttccacagt aggcccttta ag 22
<210> 13
<211> 19
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<220>
<221> misc_feature
<223> 引物JLC-RT-1704-F管家qRT
<400> 13
gaggaagcgg aagaggatg 19
<210> 14
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<220>
<221> misc_feature
<223> 引物JLC-RT-1704-R管家qRT
<400> 14
tcaagtacca gttccacacg 20
<210> 15
<211> 37
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<220>
<221> misc_feature
<223> 用于FL CHORD Y2H的引物JLC-Chord-pGB-F
<400> 15
catggaggcc gaattcatga aactctatgt gcactac 37
<210> 16
<211> 38
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<220>
<221> misc_feature
<223> 用于FL CHORD Y2H的引物JLC-Chord-pGB-R
<400> 16
gcaggtcgac ggatcctcat gtgccctcag acgttttg 38
<210> 17
<211> 41
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<220>
<221> misc_feature
<223> 用于Y2H的引物JLC-pGB-ChDom1-R
<400> 17
gcaggtcgac ggatcctcac aacaatcctg tgctcgactc a 41
<210> 18
<211> 38
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<220>
<221> misc_feature
<223> 用于Y2H的引物JLC-pGB-CRD117-179
<400> 18
catggaggcc gaattcatgt tgcgctgcaa aaactatg 38
<210> 19
<211> 41
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<220>
<221> misc_feature
<223> 用于Y2H的引物JLC-pGB-ChDom1-R
<400> 19
gctgcaggtc gacggatcct cagtgccgtc ctgtggtgca c 41
<210> 20
<211> 40
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<400> 20
catggaggcc gaattcatga gtctcatcga tccgaaggaa 40
<210> 21
<211> 38
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<400> 21
gcaggtcgac ggatcctcag tccggtttga cggtgcag 38
<210> 22
<211> 335
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> 全长CHORD T3266 CRD1-336
<400> 22
Met Lys Leu Tyr Val His Tyr Glu Glu Ala Gly Gln Asp Glu Lys Ala
1 5 10 15
Leu Thr Leu Lys Leu Thr Leu Pro Lys Ser Trp Ala Glu Gln Pro Leu
20 25 30
Leu Gln Val Leu Glu Leu Phe Ile Glu Ser Tyr Asn Lys Lys Lys Thr
35 40 45
Gly Leu Pro Pro Leu Asp Lys Asp Phe Val His Met Glu Lys Ala Gly
50 55 60
Gly Val Ile Leu Pro Val Gly Asn Ile Val Ser Asp Met Leu Ser Asp
65 70 75 80
Arg Asp Asp Leu Tyr Ile Arg Ser Gly Pro Gly Pro Ala Arg Gly Lys
85 90 95
Ile Ala His Leu Ser Ser Pro Pro Asn Ala His Ala Ser Ser Glu Ser
100 105 110
Ser Thr Gly Leu Leu Arg Cys Lys Asn Tyr Gly Cys Asn Gln Ser Phe
115 120 125
Ser Glu Glu Asn Asn Ser Glu Glu Ala Cys Arg Phe His Lys Ala Pro
130 135 140
Pro Val Phe His Asp Thr Lys Lys Gly Trp Ser Cys Cys Ala Lys Arg
145 150 155 160
Val Tyr Asp Trp Asp Glu Phe His Thr Ile Glu Gly Cys Thr Thr Gly
165 170 175
Arg His Ser Leu Ile Asp Pro Lys Glu Ile Phe Ala Pro Ser Pro Thr
180 185 190
Leu Ala Ala Ala Ala Gln Ala Glu Arg Gly Asp Cys Ser Asn Thr Ser
195 200 205
Ser Ala Ala Thr Val Ile Lys Ser Ile Asp Glu Phe Asn Gln Ser Asn
210 215 220
Pro Asn Ala Ala Ala Ala Cys Lys Thr Ala Ala Ser Met Thr Leu Ala
225 230 235 240
Gly Thr Arg Cys Thr Val Lys Pro Asp Gly Ser Ala Thr Cys Leu Asn
245 250 255
Lys Gly Cys Gln Lys Asp Tyr Leu Leu Lys Glu Asn His Pro Ser Ala
260 265 270
Cys Arg Tyr His Ala Ala Gly Pro Val Phe His Asp Ala Gly Lys Tyr
275 280 285
Trp Ser Cys Cys Pro Gly Thr Val Lys Tyr Asp Phe Asp Asp Phe Leu
290 295 300
Lys Ile Pro Gly Cys Met Leu Ser Ser His Tyr Asp Gly Ser Gln Glu
305 310 315 320
Ser Leu Glu Ala Phe Thr Arg His Ala Lys Thr Ser Glu Gly Thr
325 330 335
<210> 23
<211> 117
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> CHORD蛋白质氨基酸1-117
<400> 23
Met Lys Leu Tyr Val His Tyr Glu Glu Ala Gly Gln Asp Glu Lys Ala
1 5 10 15
Leu Thr Leu Lys Leu Thr Leu Pro Lys Ser Trp Ala Glu Gln Pro Leu
20 25 30
Leu Gln Val Leu Glu Leu Phe Ile Glu Ser Tyr Asn Lys Lys Lys Thr
35 40 45
Gly Leu Pro Pro Leu Asp Lys Asp Phe Val His Met Glu Lys Ala Gly
50 55 60
Gly Val Ile Leu Pro Val Gly Asn Ile Val Ser Asp Met Leu Ser Asp
65 70 75 80
Arg Asp Asp Leu Tyr Ile Arg Ser Gly Pro Gly Pro Ala Arg Gly Lys
85 90 95
Ile Ala His Leu Ser Ser Pro Pro Asn Ala His Ala Ser Ser Glu Ser
100 105 110
Ser Thr Gly Leu Leu
115
<210> 24
<211> 63
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> CHORD蛋白质氨基酸117-179
<400> 24
Leu Arg Cys Lys Asn Tyr Gly Cys Asn Gln Ser Phe Ser Glu Glu Asn
1 5 10 15
Asn Ser Glu Glu Ala Cys Arg Phe His Lys Ala Pro Pro Val Phe His
20 25 30
Asp Thr Lys Lys Gly Trp Ser Cys Cys Ala Lys Arg Val Tyr Asp Trp
35 40 45
Asp Glu Phe His Thr Ile Glu Gly Cys Thr Thr Gly Arg His Ser
50 55 60
<210> 25
<211> 71
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> CHORD蛋白质氨基酸179-251
<400> 25
Leu Ile Asp Pro Lys Glu Ile Phe Ala Pro Ser Pro Thr Leu Ala Ala
1 5 10 15
Ala Ala Gln Ala Glu Arg Gly Asp Cys Ser Asn Thr Ser Ser Ala Ala
20 25 30
Thr Val Ile Lys Ser Ile Asp Glu Phe Asn Gln Ser Asn Pro Asn Ala
35 40 45
Ala Ala Ala Cys Lys Thr Ala Ala Ser Met Thr Leu Ala Gly Thr Arg
50 55 60
Cys Thr Val Lys Pro Asp Gly
65 70
<210> 26
<211> 156
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> CHORD蛋白质氨基酸179-336
<400> 26
Leu Ile Asp Pro Lys Glu Ile Phe Ala Pro Ser Pro Thr Leu Ala Ala
1 5 10 15
Ala Ala Gln Ala Glu Arg Gly Asp Cys Ser Asn Thr Ser Ser Ala Ala
20 25 30
Thr Val Ile Lys Ser Ile Asp Glu Phe Asn Gln Ser Asn Pro Asn Ala
35 40 45
Ala Ala Ala Cys Lys Thr Ala Ala Ser Met Thr Leu Ala Gly Thr Arg
50 55 60
Cys Thr Val Lys Pro Asp Gly Ser Ala Thr Cys Leu Asn Lys Gly Cys
65 70 75 80
Gln Lys Asp Tyr Leu Leu Lys Glu Asn His Pro Ser Ala Cys Arg Tyr
85 90 95
His Ala Ala Gly Pro Val Phe His Asp Ala Gly Lys Tyr Trp Ser Cys
100 105 110
Cys Pro Gly Thr Val Lys Tyr Asp Phe Asp Asp Phe Leu Lys Ile Pro
115 120 125
Gly Cys Met Leu Ser Ser His Tyr Asp Gly Ser Gln Glu Ser Leu Glu
130 135 140
Ala Phe Thr Arg His Ala Lys Thr Ser Glu Gly Thr
145 150 155
<210> 27
<211> 390
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> SGT1 1852多肽
<400> 27
Met Glu Gln Ala Gln Leu Ala Phe Met Asp Glu Asp Tyr Ala Thr Ala
1 5 10 15
Asp Glu Leu Phe Ser Ala Tyr Ile Ala Thr His Pro Asp Asp Ala Thr
20 25 30
Ala Leu Val Ser Arg Ala Ala Val His Leu Lys Leu Asn Gln Pro Ser
35 40 45
Gln Ala Leu His Asp Ala Glu Ala Ala Ala Ser Leu Lys Ser Asp Phe
50 55 60
Glu Val Ala Trp Tyr Arg Gln Gly Val Ala Ala Phe Ala Leu Glu Asp
65 70 75 80
Phe Ser Arg Ala Lys Glu Ala Phe Met Arg Gly Arg Ala Leu Leu Gly
85 90 95
Asp Arg Pro Glu Pro Gln Gly Arg Arg Tyr Asp Val Trp Leu Arg Lys
100 105 110
Thr Thr Leu Glu Leu Gly Glu His Asp Glu Gln Gly Met Thr Val Val
115 120 125
Ala Glu Gly Gly Arg Lys Glu Leu Arg Gln Glu Glu Gly Ala Lys Gly
130 135 140
Ile Glu Ala Thr Val Pro Arg Ser Gln Pro Ser Pro Thr His Pro Pro
145 150 155 160
Ser Ser Ala Pro Ala Ile Pro Ala Gly His Val Lys Phe Gln Phe Tyr
165 170 175
Gln Thr Thr Ser Tyr Val Thr Val Thr Ile Leu Tyr Lys Gly Leu Arg
180 185 190
Glu Glu Asp Ala Asn Val Ser Val Gln Pro Arg His Leu Lys Val Ser
195 200 205
Val Gly Ala Asp Gly Asp Val Leu Phe Asp Arg Ala Leu Phe Glu Thr
210 215 220
Val Val Pro Glu Glu Ser Thr Ser Lys Ile Phe Ala Thr Lys Ile Glu
225 230 235 240
Val Lys Leu Arg Lys Ala Met Glu Gly Leu Thr Trp Pro Glu Leu Cys
245 250 255
Ala Leu Ser Pro Gly Ala Ala Ala Ser Val His Thr Val Ser Ala Pro
260 265 270
Thr Pro Asp Ser His Ala Thr Ala Leu Pro Ser Leu Ala Gln Gly Pro
275 280 285
Ala Thr Ala Pro Ser Ser Lys Pro Leu Arg Pro Tyr Ala Ser Thr Lys
290 295 300
Asp Trp Gly Ala Val Glu Lys Glu Ile Ser Lys Glu Leu Glu Ser Glu
305 310 315 320
Lys Pro Glu Gly Asp Glu Ala Leu Asn Thr Leu Phe Arg Asp Ile Tyr
325 330 335
Ala Lys Gly Ser Asp Glu Thr Arg Arg Ala Met Val Lys Ser Phe Gln
340 345 350
Thr Ser Gly Gly Thr Val Leu Ser Thr Asn Trp Glu Glu Val Gly Lys
355 360 365
Lys Asp Tyr Glu Lys Glu Glu Glu Arg Lys Ala Pro Glu Gly Met Glu
370 375 380
Trp Arg Lys Trp Gly Val
385 390
<210> 28
<211> 200
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> SKP-8611多肽
<400> 28
Met Gln Ala Ser Gln Lys Thr Ser Asp Val Gln Glu Glu Asp Gly Pro
1 5 10 15
Pro Lys Tyr Ile Thr Leu Gln Ala Arg Asp Gly Thr Leu Asp Glu Pro
20 25 30
Val Asp Ala Arg Ile Leu Leu Pro Ser Asp Leu Leu Arg Ser Met Leu
35 40 45
Pro Glu Lys Leu Ser Glu Ile Glu Asp Asp Phe Gln Ile Pro Leu Gln
50 55 60
Gly Val Asp Lys Ala Val Leu Glu Lys Val Val Glu Tyr Leu His Leu
65 70 75 80
Tyr Arg Glu Glu Pro Met Tyr Lys Ile Glu Lys Pro Leu Asn Lys Val
85 90 95
Lys Leu Tyr Asp Leu Val Gln Pro Gln Tyr Asp Gln Phe Ile Asn Ala
100 105 110
Leu His Tyr Lys Thr Ile Phe Gln Ile Ile Asp Ala Ala Asn Phe Leu
115 120 125
Gly Ile Glu Pro Leu Leu Ser Leu Ser Leu Ser Trp Val Ala Phe Val
130 135 140
Leu Lys Gly Pro Thr Val Glu Glu Phe Lys Lys Leu Phe Thr Ile His
145 150 155 160
Asn Asp Phe Thr Pro Glu Glu Glu Ala Ile Phe Arg Arg Glu Tyr Leu
165 170 175
Leu Pro Arg Arg Asn Arg Ala Thr Ser Glu Gly Gly Arg Glu Arg Gly
180 185 190
Ala Ser Gly Asn Ser Asn Ser Ser
195 200
<210> 29
<211> 172
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> SKP-7479多肽
<400> 29
Met Gly Asp Lys Ala Lys Gly Ala Glu Ser Gly Lys Val Val His Leu
1 5 10 15
Val Ser Gln Glu Gly Asp Gln Tyr Glu Val Glu Val Ser Val Cys Lys
20 25 30
Met Ser Glu Leu Val Lys Thr Met Leu Pro Asp Asp Asp Asp Ser Ser
35 40 45
Glu Thr Gln Glu Ile Pro Leu Pro Asn Val Lys Asn Asn Val Leu Ala
50 55 60
Lys Val Ile Glu Phe Cys Lys His His Lys Glu Asp Pro Met Asn Asp
65 70 75 80
Ile Glu Lys Pro Leu Lys Ser Ala Asn Met His Glu Val Val Gln Asp
85 90 95
Trp Tyr Ala Asn Phe Val Asn Val Asp Gln Glu Leu Leu Phe Glu Leu
100 105 110
Ile Leu Ala Ala Asn Tyr Met Asp Ile Lys Pro Leu Leu Asp Leu Thr
115 120 125
Cys Ala Thr Val Ala Ser Met Ile Lys Gly Lys Thr Pro Glu Glu Ile
130 135 140
Arg Arg Thr Phe Asn Ile Thr Asn Asp Phe Thr Pro Glu Glu Glu Ala
145 150 155 160
Gln Val Arg Glu Glu Asn Lys Trp Cys Glu Glu Val
165 170
<210> 30
<211> 48
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<220>
<221> misc_feature
<223> 引物SGTI Y2H
<400> 30
catatggcca tggaggccga attcatggag caggcgcagc ttgcattc 48
<210> 31
<211> 45
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<220>
<221> misc_feature
<223> SGTI Y2H
<400> 31
gctgcaggtc gacggatccc cctacacccc ccacttccgc cactc 45
<210> 32
<211> 46
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<220>
<221> misc_feature
<223> SKP-8611 Y2H
<400> 32
catggaggcc agtgaattcc acatgcaagc atcccaaaag acgtcc 46
<210> 33
<211> 46
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<220>
<221> misc_feature
<223> SKP-8611 Y2H
<400> 33
gatcccgtat cgatgcccac ccgttagctt gagttgctgt tgccag 46
<210> 34
<211> 44
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<220>
<221> misc_feature
<223> SKP-7479 Y2H
<400> 34
catggaggcc agtgaattcc acatgggtga caaagcgaag ggag 44
<210> 35
<211> 45
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<220>
<221> misc_feature
<223> SKP-7479 Y2H
<400> 35
gatcccgtat cgatgcccac ccgtcacacc tcctcacacc acttg 45
<210> 36
<211> 646
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> SKP2-6789多肽
<400> 36
Met Gly Gly Ile Phe Phe Lys Gly Leu Glu Glu Gly Gly Arg Asp Glu
1 5 10 15
Gly Gly Val Gly Gly Arg Ser Ser Arg His Tyr Glu Asp Ala Ala Cys
20 25 30
Pro Ser Arg Ser Arg Leu Ala Val Asp Glu Glu Asp Glu Ser Glu Ala
35 40 45
Thr Ser Ile Asn Leu Ser Ser Glu Ala Ser Val His Arg Phe Leu Gln
50 55 60
Glu Gln Tyr Ser Leu Ala Arg Gly Gly Arg Cys Arg Arg Cys Arg Pro
65 70 75 80
Ala Glu Gly Asn Gly Glu Ile Met Ser Leu Ile Asp Leu Cys Ile Asp
85 90 95
Ser Ile Cys Gln Asn Ile Leu Cys Tyr Glu Val Pro Pro Ser Phe Asp
100 105 110
Val Leu Pro Pro Asp Leu Val Arg Arg Ile Phe Asp Ser Leu Thr Ser
115 120 125
His Lys Ala Leu Thr Lys Leu Thr Leu Leu Pro Leu Arg Tyr Cys Asp
130 135 140
Val Thr Arg Leu Asp Leu Ser Pro Cys Lys Gly Val Thr Asp Gln Trp
145 150 155 160
Leu Arg Pro Phe Thr Asp Lys Thr Leu Thr His Leu Cys Leu Ala Phe
165 170 175
Cys Asn Ala Ile Thr Asp Ala Gly Leu Leu Asn Leu Thr Glu Leu Gly
180 185 190
Gly Leu Gln Val Ala Asp Leu Thr Ala Cys Ala Gly Leu Arg Thr Gly
195 200 205
Gly Leu Ala Ala Phe Arg Glu Ser Trp Ser Met Thr Ser Leu Thr Leu
210 215 220
Asn Gly Cys Arg Asn Leu Thr Asp Glu Ala Val Lys Pro Leu Ala Tyr
225 230 235 240
Leu Ser Ala Leu Gln His Leu Arg Leu Arg Gly Cys Trp Gln Leu Ser
245 250 255
Asp Ala Ala Leu Asn Ala Leu Ala Pro Leu Met Pro Gln Leu Lys Thr
260 265 270
Leu Asp Val Asn Ala Cys Arg Gln Phe Ser Ser Thr Ser Ile Ser Phe
275 280 285
Cys Val Gly Lys Met Arg Glu Ile Arg His Leu Asp Leu Gly Tyr Cys
290 295 300
Pro Arg Gly Val Gly Gly Gly Ala Leu Glu Val Leu Thr Arg Gly Pro
305 310 315 320
Ala Pro Thr Thr Leu Gln Val Leu Ile Leu Asp Ser Ser Arg Ser Leu
325 330 335
Ser Asn Ala Asp Leu Phe Ser Leu Gly Lys Phe Arg Cys Leu Gln Arg
340 345 350
Leu Ser Leu Arg Asn Cys Thr Thr Ile Ser Asp Ala Gly Leu Cys Gln
355 360 365
Leu Pro Ser Ser Leu Glu Thr Leu Asp Ala Ser His Cys Arg Gly Val
370 375 380
Arg Arg Leu Pro Ala Gln Ser Leu Pro Cys Leu Arg Ser Val Asn Phe
385 390 395 400
Ala His Ser Gly Leu Arg Asp Ser Asp Val Gly Ser Leu Ala Val Phe
405 410 415
Pro Ala Leu Val Asn Ile Asn Leu Asp Ser Cys Ser Ile Gly Asn Ser
420 425 430
Gly Leu Val Ser Leu Leu Pro Leu Lys Lys Leu Lys Arg Leu Asn Leu
435 440 445
Ala Asp Thr Gly Val Ser Asn Gly Gly Met Asp Val Leu Ala Lys Leu
450 455 460
Ser Ser Leu Ser Val Ile Ser Leu Phe Tyr Thr Ser Ile Ser Asp Ser
465 470 475 480
Gly Val Arg Gln Leu Ser Val Leu Gln Asn Leu Thr Asp Leu Asn Leu
485 490 495
Asp Asn Arg Asp Leu Thr Asp Thr Ser Leu Leu His Leu Ser Ser Leu
500 505 510
Thr Lys Leu Arg Arg Leu Asp Met Phe Ser Ser Arg Val Ser Asp Val
515 520 525
Gly Leu Cys Phe Ile Thr Ser Leu Val Glu Leu Val Asp Leu Glu Ile
530 535 540
Cys Gly Gly Arg Ile Thr Asp Lys Gly Leu Glu Tyr Ile Ser Gln Leu
545 550 555 560
Pro Ser Leu Gln Arg Leu Asn Val Ser Gln Asn Val Gln Ile Ser Asn
565 570 575
Ser Gly Leu His His Leu Ala Ser Leu Lys Asp Leu Glu Ala Leu Asn
580 585 590
Val Ser His Ser Gln Val Ala Ala Pro Gly Ala Leu Lys Pro Leu Cys
595 600 605
Arg Leu Arg Gly Leu Lys Ile Leu Ala Val Asn Gly Cys Arg Gly Met
610 615 620
Asp Asp Thr Thr Leu His Gln Ile Gln Ser Ser Leu Pro Asn Leu Arg
625 630 635 640
Thr Val Arg Ala Val Ser
645
<210> 37
<211> 303
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> CDC25-9451多肽
<400> 37
Met Val Tyr Pro Pro Val His Ser Val Thr Gly Leu Tyr Thr Pro Leu
1 5 10 15
Trp Lys Thr Phe Gly Ala Trp Cys Met Pro Gly His Pro Ser Ser Ser
20 25 30
Leu Arg Val Arg Pro Asp Ser Cys Asn Phe Phe Arg Gly Ala Phe Pro
35 40 45
Arg Pro Phe Leu Phe Ala Arg Tyr Phe Arg Ser Ser Ser Ala Leu Leu
50 55 60
Leu Phe Leu Thr Ile Pro Leu Ser Tyr Cys Phe Val Arg Pro Pro Gly
65 70 75 80
Pro Phe Ser Ile Ala Ser Leu Cys Pro Ser Trp Gly Thr Arg Ser Ile
85 90 95
Val Ser Asn Phe Pro Arg Pro Asp Ala Arg Arg Ala Phe Thr Thr Phe
100 105 110
Ser Gly Thr Asp Val His Leu Ala Met Ser Ser Ser Ser Val Ser His
115 120 125
Ala Leu Ser Gly Pro Ala Tyr Ile Glu Gln Glu Glu Leu Leu Ala Ile
130 135 140
Leu His Ala Arg Arg Arg Gly Glu Arg Ser Leu Lys Leu Gln Ile Leu
145 150 155 160
Asp Val Arg Asp Asp Asp Tyr Thr Gly Gly Glu Arg Thr Gly Gly Leu
165 170 175
Ala Lys Leu Pro Gly Ala Ile Asn Val Pro Ser Glu Asp Trp Arg Asp
180 185 190
Glu Glu Arg Val Val Ala Leu Ala Glu Ser Leu Lys Asp His Asp Met
195 200 205
Ile Val Leu His Cys Met Leu Ser Gln Val Arg Gly Pro Phe Cys Ser
210 215 220
Ala Arg Leu Met Ala His Phe Ser Cys Ala Val Gly Glu Asp Gly Gly
225 230 235 240
Ala Ala Thr Lys Gln Glu Arg Asn Gln Ser Ser Gly Arg Asn Asp Gly
245 250 255
Glu Glu Thr Leu Pro Ala Asn Glu Ser Lys Lys Glu Arg Arg Arg Glu
260 265 270
Gln Gly Val Pro Gln Val Leu Val Leu Arg Gly Gly Phe Gln Gly Trp
275 280 285
Tyr Ala Arg Tyr Arg Glu Glu Ile Gly Met Val Glu Pro Cys Glu
290 295 300
<210> 38
<211> 2071
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> FBW7.1-2293多肽
<400> 38
Met Ala His Ser Ile Leu Ala Lys Gly Cys Asp Thr Met Glu Glu Gln
1 5 10 15
Ser Gln Glu Gly Asp His Arg Ala Gly Pro Gly Val Lys Glu Ser Ile
20 25 30
Gly Ser Ser Leu Arg Ala Ser Asn Gly Ala Ala Asp Ala Asn Gly Val
35 40 45
Leu Glu Val Arg Val Ile Glu Ala Thr Asp Leu Ile Glu Arg Pro Arg
50 55 60
Glu Arg Lys Gln Arg Ser Phe Phe Ala Gly Met Ala Glu Ser Ser Gly
65 70 75 80
Val Met Ser Arg Ala Glu Tyr Gly Trp Pro Tyr Val Thr Ile Arg Ala
85 90 95
Gly Arg Asp Ala Pro Arg Arg Thr Met Pro Gly Ile Met Ser Met Gln
100 105 110
Ala Glu Glu Asp Arg Arg Gly Ser His Asn Asn Ala Gly Leu Ser Ser
115 120 125
Gly Met Val Ala Trp His Glu Asp Phe Val Phe Gly Pro Val Ser Ser
130 135 140
Arg Glu Glu Val Val Val Thr Cys Tyr Met His Arg Arg Ser Ala Gly
145 150 155 160
Pro Asp Gly Arg Ser Ala Gln Cys Ser Val Val Gly Asp Val His Ile
165 170 175
Pro Val Asn Arg Leu Pro Glu Gly His Ala Ile Glu Gln Trp Tyr Gln
180 185 190
Leu Leu Pro Gln Gln Glu Thr Pro Arg Glu Asp Glu Leu Gly Arg Ser
195 200 205
Arg Pro Arg Pro Ser Arg Lys Gly Val Val Ser Lys Ala Ala Ile Lys
210 215 220
Leu Arg Leu Tyr Tyr Gln Val Arg Asp Pro Ala Phe Ala Pro Arg Gly
225 230 235 240
Glu Gly Ala Val Ala Phe Val Gly Pro Glu Thr Met Gly Arg Thr Gln
245 250 255
Arg Ile Gly Ala Glu Gly Val Ser Ala Thr Tyr Pro Ser Pro Ser Ser
260 265 270
Thr Thr Gly Ala Ser Gly Leu Asp Thr Pro Arg Ser Ala Gln Glu Ser
275 280 285
Gln Ser Gln Asp Glu Gln Gln Arg Gln Val Met Leu Ser Ser Ala Gly
290 295 300
Gly Gly Leu Leu Ser Ser Ala Pro Gly Thr Glu Gly Gly Thr Gly Asp
305 310 315 320
Pro Val Glu Gly Ala Ser Pro Arg Arg Leu Pro Thr Pro Ser Ser Met
325 330 335
Ala Glu Leu Ala Gln Glu Glu Leu Pro Thr Gly Leu Val Asp Tyr Phe
340 345 350
Cys Ile Met Gly Pro Arg Leu Asp Glu Ala Thr Gly Leu Pro Ser Leu
355 360 365
His Asn Gly Ala Ile Leu Leu Arg His Pro Val Glu Asp Lys Ala Gly
370 375 380
Gln Pro Leu Pro Asp Ser Pro Gln Phe Phe Cys Phe Pro Ala Gly Met
385 390 395 400
Ala Leu Ala Tyr Gly Pro Ser Pro Pro Lys Pro Ala Pro Leu Ala Tyr
405 410 415
Thr Phe Val Ile Lys His Ser Gly Val Ser Ser Tyr Gly Val Cys Leu
420 425 430
His Phe His Arg Arg Trp Glu Gln Leu Ser Lys Asn Val Leu Ser Pro
435 440 445
Ser Val Gly Leu Leu Gly Val Thr Gly Asp Thr Ser Gln Glu His Asp
450 455 460
Arg Pro Ser Gly Gln Glu Asp Pro Lys Gln Gln Gly Thr Thr Val Trp
465 470 475 480
Ala Pro Val Cys Phe Cys Leu Leu Thr Arg Val Pro Val Val Gln Pro
485 490 495
Leu Leu Asn Trp Leu Val His Ala Tyr Asp His Met Asp Arg Leu Leu
500 505 510
Pro Pro Thr Tyr Asp Ala Leu Leu Gly Asp Pro Leu Asp Pro Ala Ser
515 520 525
Val Pro Gly Ala His Leu Thr Asp Leu Leu Arg Thr His Ile Val Gln
530 535 540
Leu Thr Leu Glu Val Pro Leu Pro Ile Pro Gly Ala Leu Gly Val Gln
545 550 555 560
Phe Asp Phe Leu Gly Arg Pro Ile Thr Cys Arg Leu Ala Gly Pro Gly
565 570 575
Ala Leu Pro Ser Leu Cys Tyr Pro Leu Ser Pro Phe Leu Arg Thr Phe
580 585 590
Ser Ala Arg Asn Val Leu Ala Leu Val Ala Ala Ala Leu Thr Glu Ser
595 600 605
Lys Val Leu Leu His Ser His Asp Leu Ser Val Leu Pro Val Met Ala
610 615 620
Glu Ser Leu Leu Ser Leu Ile Tyr Pro Leu Gln Trp Gln His Pro Tyr
625 630 635 640
Leu Val Pro Leu Pro Arg Glu Leu Leu Val Val Val Glu Thr Pro Thr
645 650 655
Asn Tyr Ile Leu Gly Val His Thr Glu Trp Leu Ala Asp Val Pro Arg
660 665 670
Asp Ser Leu Lys Asp Val Val Cys Val Asp Cys Asp Ser Gly Ala Val
675 680 685
Arg Leu Pro Pro Arg Pro Phe Asn Pro Pro Ser Phe Pro Pro Ser Val
690 695 700
Leu Tyr Pro Leu Leu Arg Arg Leu Arg Gly Thr Val Tyr Pro Met Leu
705 710 715 720
Thr Arg Leu Asp Ser Ala Ser Ser Arg Val Ser Leu Asp Asp Leu Pro
725 730 735
Ile Ile Ser Thr Ser Ala Pro Ser Glu Ile Ser Pro Ala Ser Glu Glu
740 745 750
Glu Leu Arg Arg Leu Phe Leu Arg Cys Leu Ala Tyr Leu Leu Ser Gly
755 760 765
Tyr His Asp Cys Val Phe Tyr Ile Asp Pro Asn Ser Pro Ile Phe Asn
770 775 780
Arg Ala Arg Phe Leu Ala Glu Tyr Ala Pro Ala Glu Asp His Ala Phe
785 790 795 800
Leu Ser Arg Leu Leu Asp Thr Gln Ser Phe Gln Ala Phe Leu Glu Asn
805 810 815
Gln Asp Gly Pro Ser Ile Asn Leu Phe Arg Arg Thr Leu Phe Gln Ala
820 825 830
Phe Ser Arg Ala Thr Ser Arg Pro Pro Ser Pro Leu Pro Gly Glu Thr
835 840 845
Thr Ser Ser Pro Ala Ala Ser Thr Ser Phe Pro Ser Leu Lys Lys Cys
850 855 860
Pro Ala Asp Phe Arg Ile Leu Met Glu Glu Glu Glu Ser Gly Asn Glu
865 870 875 880
Trp Leu Glu Thr Pro Gly Ala Gly His Arg Glu Thr His Gly Glu Ala
885 890 895
Trp Glu Gln Arg Pro Ser Ala Gly Arg Gly Gly Glu Glu Cys Glu Val
900 905 910
Lys Leu Met Leu Lys Ile Pro Pro Pro Pro Leu Tyr Gly Glu Thr Glu
915 920 925
Glu Asp Glu Ser Glu Ser Asp Gly Ser Val Asp Glu Asp Cys Asp Pro
930 935 940
Ala Thr Thr Arg Gly Glu Glu Ala Val Pro Gly Gly Arg Arg Glu Phe
945 950 955 960
Arg Phe Asp Gly Asp Gln Asp Gln Lys Ala Glu Gly Trp Glu Gly Val
965 970 975
Ser Glu Glu Arg Asp Val Ser Gly Arg Gln Arg Thr Ser Val Arg Phe
980 985 990
Asn Leu Gly Glu Leu Glu Asn Pro Gly Glu Ser Lys Gly Asp Arg Gly
995 1000 1005
Asp Ser Glu Val Met Ser Ser Asp Arg Met Ala Asn Thr Ile Thr
1010 1015 1020
Ala Ser Glu Gly Leu Ser Thr Val Val Ser Ala Glu Ser Asn Arg
1025 1030 1035
His Lys Glu Lys Arg Ala Lys Ala Lys Lys Ala Arg Arg Pro Leu
1040 1045 1050
Asn Leu Pro Gly Leu Ala Val Gly Ser Pro Pro Thr Arg Ser Ser
1055 1060 1065
Leu Glu Gly Thr Phe Gly Glu Ala Asp Leu Asp Ala Leu Met Arg
1070 1075 1080
Lys Thr Met Asn Leu Thr Gly Ala Thr Tyr Gly Arg Asn Val Pro
1085 1090 1095
Ala Ala Ser His Thr Gly Gly Tyr Thr Cys Gly Asp Glu Gly Asp
1100 1105 1110
Ser Met Asp Ser Ser Val Val Pro Ala Arg Thr Val Asp Leu Asp
1115 1120 1125
Ser Gly Tyr Gln Gly Gly Ala Ala Glu Ala Lys Gly Pro Thr Asn
1130 1135 1140
Ala Ala Thr Gly Val Ala Trp Arg Lys Ala Arg Arg Trp Ser Val
1145 1150 1155
Glu Ala Ala Ala Gly Met Met Asp Val Thr Val Arg Asp Val Ala
1160 1165 1170
Arg Ala Phe Gly Leu Asn Phe Asp Leu Gln Arg Val Met Thr Arg
1175 1180 1185
Gly His Thr Ile Phe Gly Phe Phe Asp Asp Val Pro Pro Ser Trp
1190 1195 1200
Gly Arg Glu Gln Gly Ser Lys Gly Gly Glu Gly Cys Arg Glu Gly
1205 1210 1215
Arg Ala Thr Glu Gly Ser Trp Leu Gly Lys Glu Asp Asp Gly Leu
1220 1225 1230
Glu Lys Asp Gly Ala Gly Ala Gln Thr Gly Val Leu Ser Gly Asp
1235 1240 1245
Met Ala Lys Leu Asn Arg Cys Leu Ala Glu Ala Phe Ser Thr Glu
1250 1255 1260
Lys Pro Ser Lys Glu Leu Phe Arg Glu Val Glu Ile Ala Phe Arg
1265 1270 1275
Ala Arg Gly Val Arg Thr Arg Phe Leu Ala Ile Leu Ser Gln Pro
1280 1285 1290
Arg Ser Lys Arg Met Ile Gln Arg His Gln Phe Leu Ile Gly Gly
1295 1300 1305
Gly Thr Ser Gly His Phe Arg Val His Ser Thr Gly Phe Glu Ala
1310 1315 1320
Leu Met Gln Leu Ala Ser Ala Val Cys Asp Ala Cys Val Val Asp
1325 1330 1335
Arg Asp Phe Pro Thr Ala His Ala Leu Leu Gln Leu Met Gly Lys
1340 1345 1350
Tyr Tyr Arg Val Leu Glu Gly Gly Gly Gly Gly Asn His Ala Trp
1355 1360 1365
Ala Ala Ala Thr Leu Arg Gln Gly Glu Gly Gly Ala Gly Ala Gln
1370 1375 1380
Gln Gln His Lys Glu Phe Leu Ser Ser Arg Leu Arg His His Gln
1385 1390 1395
Ile Tyr Gln Cys Val Glu Leu Trp Met His Val Leu Glu Glu Gln
1400 1405 1410
Leu Gly Ala Gly Lys Gly Pro Thr Ala Thr Arg Arg Asp Ser Thr
1415 1420 1425
Asn Val Asn Ser Val Asn Gly Phe Lys Leu Ala Lys Ala Pro Gly
1430 1435 1440
Ala Lys Thr Thr Asn Arg Asp Leu Pro Ser Val Ala Glu Ala Asp
1445 1450 1455
Asp Pro Ser Ser Val Gly Thr Val Asp Val Lys Ala Gly Gly Lys
1460 1465 1470
Gly Lys Gly Ser Ala Leu Gly Gln Gly Glu Ala Ala Ala Glu Glu
1475 1480 1485
Thr Pro Gly Ser Glu Met Asp Asp Ala Glu Val Asp Pro Arg Lys
1490 1495 1500
Phe Ile Leu Arg Val Lys Ser Ile Leu Ala Glu Met His Gly Val
1505 1510 1515
Gly Met Pro Asp His Arg Ala Leu Ala Phe Val Gly Arg Ile Cys
1520 1525 1530
Glu Val His Glu Ala Gly Met Glu Ser Lys Gln Ala Leu Val Arg
1535 1540 1545
Leu Val Gln Lys Ile Trp Gly Ile Ser Pro Ala Pro Thr Pro Gln
1550 1555 1560
Gln Met Phe Ile Ser Gln Glu Pro Val Asn Arg Gly Glu Ser Val
1565 1570 1575
Gly Ser Asp Ala Pro Arg Pro Ala Ala Ser Ser Ser Ser Ser Ser
1580 1585 1590
Thr Ser Pro Ala Trp Thr Ser Pro Glu Gly Ile Ala Pro Met Val
1595 1600 1605
Pro Pro Arg Arg Val Ser Phe Gln Gly Val Gly Ser Glu Ile Gly
1610 1615 1620
Arg Arg Ser Ser Asp Ser Ile Gly Asp Ser Val Arg Lys Asn Ser
1625 1630 1635
Thr Leu Leu Asn Asp Leu Arg Gly Pro Ser Pro Ser Pro Ser Phe
1640 1645 1650
Ser Ser Thr Val Ser Ser Ser Ser Ser Gly Ala Arg Leu His Arg
1655 1660 1665
Ser Thr Ser Pro Ile Ser His Leu Met Gly Gly Met Gly Leu Gly
1670 1675 1680
Leu Gly Met Gly Ser Ala Ala Pro Pro Glu Ala Ser Met His Arg
1685 1690 1695
Gly Pro Val Leu Ser Val Asp Val Asp Ala Ala Gly Ser Val Gly
1700 1705 1710
Val Ser Gly Gly Ala Asp Lys Leu Leu Ile Val Tyr Ser Leu Gln
1715 1720 1725
Gln Arg Ser Arg Ile Thr Ser Phe Ser Gly His Thr Gly Pro Val
1730 1735 1740
Thr Cys Val Lys Ile Phe Arg Asp His Ala Asn Asp Pro Leu Val
1745 1750 1755
Ala Ser Ala Ser Met Asp Ser Thr Leu Arg Ile Trp Lys Leu Gly
1760 1765 1770
Gly Gly Gly Gly Gly Asp Pro Gly Ala Phe Thr Gly Ala Arg Leu
1775 1780 1785
Leu Ser Ser Phe Thr Thr Ala Lys Asp Val Arg His Ile Leu Thr
1790 1795 1800
Gly His Ala Lys Gly Ile Val Cys Leu Asp Lys Cys Glu Asp Leu
1805 1810 1815
Gln Leu Leu Ala Thr Gly Ala Met Asp Arg Ala Val Lys Leu Trp
1820 1825 1830
Asn Val Ser Gln Gly Arg Asn Thr Ala Thr Leu Ile Gly His Thr
1835 1840 1845
Arg Thr Val Asn Ser Leu Arg Phe Met Asp Gln Ser Gln Gly Tyr
1850 1855 1860
Arg Ile Leu Ser Ala Gly Gln Asp Arg Thr Met Ile Leu Trp Asp
1865 1870 1875
Ser Gly Arg Gly Ser Cys Ile Arg Val Phe Lys Gly His Glu Ser
1880 1885 1890
Trp Ile Arg Gln Val Glu Ala Trp Gly Arg Asp Leu Ala Val Thr
1895 1900 1905
Ala Ser Asn Asp Arg Thr Leu Arg Val Trp Asp Leu Arg Val His
1910 1915 1920
Asn Cys Val Gln Lys Leu Ala Glu His Lys Gly Ala Val Thr Cys
1925 1930 1935
Met Gln Val Ser Lys Glu Gln Asp Ala Pro Val Val Tyr Ser Gly
1940 1945 1950
Ser Thr Asp Ser Thr Val Lys Ile Trp Asp Leu Arg Gly Gly Gly
1955 1960 1965
Gly Arg Cys Thr Ala Thr Leu Glu Gly His Ala Glu Ala Val Thr
1970 1975 1980
Gly Leu Ala Leu Glu Ser Pro Met Ala Ala Gly Ile Gly Lys Ser
1985 1990 1995
Lys Asn Gly Gly Gly Ser Lys Leu Val His Gln Lys Leu Val Ser
2000 2005 2010
Val Gly Glu Asp Lys Arg Val Val Glu Trp Asp Thr Arg Thr Gly
2015 2020 2025
Ala Leu Leu Gln Ser Arg Met Gly His Ser Asp Gly Ile Ser Cys
2030 2035 2040
Val Gln Val Ser Lys Tyr Gly Val Ile Val Thr Gly Ser Trp Asp
2045 2050 2055
Ala Ser Val Arg Leu Trp Glu Gly Ile Gln Ile Ser Val
2060 2065 2070
<210> 39
<211> 544
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> FBW7.2-284多肽
<400> 39
Met Gly Arg Ser Leu Leu Pro His Pro Ser Ser Leu Met Leu Ser His
1 5 10 15
Pro Ser Ser Arg Leu Leu Ser Ile Met Thr Thr Ser Leu Ala Asp Met
20 25 30
Ile Leu Gly Asn Arg Lys Arg Thr Cys Asp Leu Tyr Thr Ala Glu Met
35 40 45
Thr Ser Gly Thr Thr Pro Pro Phe Leu Pro Ala Gln Lys Leu Lys Val
50 55 60
Ala Val Lys Val Asp Gln Glu Tyr Lys Ile Pro Cys Ser Ser Pro Thr
65 70 75 80
Val His Thr Ser Ser Thr Lys Gln Glu Ser Glu Ala Thr Ser Ala Pro
85 90 95
Gly Asn Glu Glu Thr Arg His Arg Gly Pro Ser Val Leu Ala Gly Gln
100 105 110
Gly Lys Ala Lys Gly Glu Ala Thr Asp Thr Leu Asn Ile Leu Asn Ser
115 120 125
Leu Arg Asn Glu Glu Ala Glu Arg Glu Leu Arg Gln Tyr Arg Gly Lys
130 135 140
Tyr Gly Gly Ser Arg Ala Ala Ser Arg Gly Ala Leu Val Ala Tyr Lys
145 150 155 160
Gly Asn Asp Ala Gly Gly Ala Met Val Ser Arg Ser Asn Lys Arg Asp
165 170 175
Gly Arg Thr Pro Pro Gly Ala Ala Ala Ala Ala Ala Ala Gly Ile Gly
180 185 190
Asn Ser Ser Ala Leu Ala Leu His Arg Thr Pro Ala Lys Val Pro Thr
195 200 205
Pro Thr Trp His Ala Pro Trp Lys Leu His Ala Val Val Ala Gly His
210 215 220
Leu Gly Trp Val Arg Ala Val Ala Phe Asp Pro Ala Asn Glu Trp Phe
225 230 235 240
Val Thr Gly Ser Ala Asp Arg Thr Ile Lys Val Trp Asp Leu Ala Lys
245 250 255
Cys Ala Ala Gly Ala Glu Gly Gly Leu Arg Leu Thr Leu Thr Gly His
260 265 270
Ile Ser Ala Val Arg Ala Leu Ala Val Ser Asn Arg His Pro Tyr Leu
275 280 285
Phe Ser Val Ala Glu Asp Lys Thr Val Lys Cys Trp Asp Leu Glu Gln
290 295 300
Asn Lys Val Ile Arg His Tyr His Gly His Leu Ser Gly Val Tyr Ser
305 310 315 320
Leu Ala Leu His Pro Thr Leu Asp Val Leu Val Thr Gly Gly Arg Asp
325 330 335
Ser Val Ala Arg Val Trp Asp Met Arg Thr Lys Met Gln Val His Val
340 345 350
Leu Gly Gly His Thr Asn Thr Val Gly Ala Leu Ala Thr Asn Ser Val
355 360 365
Asp Pro Gln Ile Ile Thr Gly Ser Tyr Asp Ser Thr Ile Lys Leu Trp
370 375 380
Asp Ile Val Ala Gly Lys Ser Met Ala Thr Leu Thr Asn His Lys Lys
385 390 395 400
Ala Val Arg Asp Leu Lys Val His Pro Lys Glu Leu Ser Phe Val Ser
405 410 415
Gly Ala Gln Asp Asn Leu Lys Arg Trp Gln Val Arg Asp Gly Lys Phe
420 425 430
Leu Lys Asn Leu Ser Gly His Asn Ala Val Ile Asn Thr Leu Ala Ile
435 440 445
Asn Glu Asp Asn Val Leu Val Ser Cys Gly Asp Asn Gly Ser Leu Arg
450 455 460
Phe Trp Asp Tyr Gln Thr Gly Tyr Cys Phe Gln Arg Leu Glu Thr Ile
465 470 475 480
Val Gln Pro Gly Ser Leu Asp Cys Glu Ala Gly Ile Tyr Ala Ser Ala
485 490 495
Phe Asp Met Ser Gly Thr Arg Phe Leu Thr Cys Glu Ala Asp Lys Thr
500 505 510
Val Lys Ile Trp Lys Glu Asp Ala Glu Ala Ser Pro Glu Thr His Pro
515 520 525
Ile Asp Met Glu Ala Trp Lys Gln Glu Cys Leu Ser His Lys Arg Trp
530 535 540
<210> 40
<211> 437
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> FBW7.3-4601多肽
<400> 40
Met Leu Thr Glu Arg Gln Lys Ala Asp Leu His Val Ala Ile Leu Glu
1 5 10 15
Tyr Leu Arg Asp Glu Gly Asp Ser Leu Ala Lys Thr Ala Glu Leu Phe
20 25 30
Ala Leu Glu Thr Gly Leu Glu Ala Ala Ala Val Pro Lys Leu Thr Gly
35 40 45
Thr Leu Glu Lys Lys Trp Ser Ala Val Val Arg Leu Gln Lys Lys Leu
50 55 60
Met Glu Leu Glu Glu Arg Met Leu Val Ala Glu Gln Glu Leu Lys Ala
65 70 75 80
Tyr Arg Gly His Ser His Met Gly Gly Thr Arg Ala Pro Ala Ala Gly
85 90 95
Asp Asp Arg Asn Leu Pro Arg Ala Pro Ala Ile Arg Ser Phe Leu Gly
100 105 110
His Arg Gly Gly Val Thr Cys Leu Ala Met His Pro Ile Phe Ala Leu
115 120 125
Leu Val Ser Gly Ser Asp Asp Ala Thr Ile Lys Thr Trp Asp Leu Glu
130 135 140
Ser Gly Ala His Glu Leu Thr Leu Lys Gly His Thr Asn Gly Val Gln
145 150 155 160
Ala Val Val Phe Asn Arg Ala Gly Thr Leu Leu Ala Ser Cys Ser Ser
165 170 175
Asp Leu Ser Ile Lys Leu Trp Asn Phe Gln Ser Pro Ser Thr Ala Pro
180 185 190
Glu Cys Val Arg Thr Leu Arg Gly His Asp His Thr Ile Ser Gly Leu
195 200 205
Ala Phe Ile Gly Pro Thr Asp Ala Gln Leu Ala Ser Cys Ser Arg Asp
210 215 220
Thr Thr Val Arg Leu Trp Glu Val Ser Thr Gly Phe Cys Gln Arg Ser
225 230 235 240
Leu Val Gly Ala His Thr Asp Trp Val Arg Cys Ile Ala Thr Ser Ala
245 250 255
Asp Gly Ala Leu Leu Ala Ser Gly Gly Ser Asp Arg Leu Val Ala Val
260 265 270
Trp Ala Leu Asp Thr Cys Ala Pro Val Ala Val Leu Arg Glu His Ser
275 280 285
His Val Val Glu Ala Val Ala Phe Pro Pro Pro Gly Val Ala Val Lys
290 295 300
Ile Asp Gly Asn Lys Gly Gly Ser Thr Gly Leu Gly Ser Glu Asn Gly
305 310 315 320
Glu Ala Ser Ala Gly Leu Gln Ser Gln Gly Ser Ala Glu Glu Ala Tyr
325 330 335
Leu Val Ser Gly Ser Arg Asp Lys Thr Ile Met Leu Trp Asn Ala Arg
340 345 350
Thr Gly Gln Cys Leu Leu Arg Leu Ala Asp His Glu Asn Trp Val Arg
355 360 365
Ser Val Arg Phe His Pro Ser Gly Gln Phe Leu Leu Ser Val Ser Asp
370 375 380
Asp Arg Ser Leu Arg Val Phe Asp Ile Ala Lys Ala Arg Cys Ile Arg
385 390 395 400
Ser Leu Pro Asp Ala His Glu Gln Phe Val Ser Ala Leu Ala Gln His
405 410 415
Pro Thr Leu Pro Tyr Leu Ala Thr Gly Ser Val Asn Arg Glu Ile Lys
420 425 430
Leu Trp Glu Cys Arg
435
<210> 41
<211> 544
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> FBW7.4-3015多肽
<400> 41
Met Gly Phe Phe Tyr Phe Phe Tyr Leu Thr Leu Gln Arg Thr Arg Arg
1 5 10 15
Phe Leu Glu Thr Thr Ser Pro Val Thr Leu Leu Arg Thr Pro Ser Met
20 25 30
Thr Ser Glu Ala Leu Pro Pro Pro Ala Ser Glu Leu His Val Thr Lys
35 40 45
Lys Ala Lys Lys Leu Pro Thr Leu Pro Gly Ser Met Val Val Gln Phe
50 55 60
Gln Asp Ala Thr Gly Lys Gln Thr Gly Pro Arg Ile Asp Leu Pro Thr
65 70 75 80
Asp Ser Thr Pro Ala Gln Leu Glu Leu Leu Ile Asn Glu Leu Arg Lys
85 90 95
Ala Thr Asp Pro Thr Glu Glu Ser Gln Gly Lys Val Pro Tyr Ser Cys
100 105 110
Tyr Ile Asn Asp Val Glu Val Leu Asp Ser Leu Arg Asp Thr Leu Glu
115 120 125
Ser Gln Gly Ile Val Asn Gly Glu Ala Val Ile Asn Ile Asn Tyr Gln
130 135 140
Pro Leu Ala Val Phe Arg Val Arg Pro Val Val Arg Cys Thr Asp Thr
145 150 155 160
Met Pro Gly His Thr Glu Ala Val Ile His Val Ser Phe Ser Pro Asp
165 170 175
Gly Arg Arg Leu Ala Ser Gly Gly Gly Asp Thr Thr Val Arg Phe Trp
180 185 190
Asp Thr Gly Thr Ser Leu Pro Lys Phe Thr Cys Arg Gly His Arg His
195 200 205
His Val Leu Cys Thr Ala Trp Ser Pro Asp Gly Ser Arg Phe Ala Ser
210 215 220
Ala Asp Lys Ala Gly Glu Ile Arg Leu Trp Asp Pro Ala Thr Gly Leu
225 230 235 240
Ala Val Gly Gln Pro Leu Gln Gly His Lys Gln His Ile Thr Ser Leu
245 250 255
Ala Trp Glu Pro Leu His Leu Asn Arg Gly Lys Gly Glu Arg Leu Ala
260 265 270
Ser Ser Ser Lys Asp Gly Thr Val Arg Val Trp Asn Val Arg Thr Gly
275 280 285
Ala Cys Leu Thr Thr Leu Ala Gln His Thr Asn Ser Val Glu Cys Cys
290 295 300
Lys Trp Gly Gly Gln Gly Val Leu Tyr Thr Gly Ser Arg Asp Arg Thr
305 310 315 320
Val Lys Ile Trp Ala Leu Gln Gly Arg Asp Gly Glu Ala Gly Phe Gly
325 330 335
Lys Leu Val Lys Thr Leu Val Gly His Gly His Arg Ile Asn Thr Leu
340 345 350
Ala Leu Asn Thr Asp Tyr Val Leu Arg Thr Gly Pro Phe Asp His Thr
355 360 365
Gly Ser Leu Ala Leu Asp Ala Ala Ser Pro Met Glu Ala Ala Glu Ala
370 375 380
Lys Tyr Arg Lys Phe Leu Glu Gly Ser Glu Gly Arg Glu Arg Leu Val
385 390 395 400
Ser Gly Ser Asp Asp Phe Thr Leu Phe Leu Trp Asp Pro Leu Gly Glu
405 410 415
Glu Gly Gly Lys Lys Pro Leu Ala Arg Met Thr Gly His Gln Gln Ala
420 425 430
Val Asn His Ile Ser Phe Ser Pro Asp Gly Arg Tyr Val Ala Ser Ala
435 440 445
Ser Phe Asp Lys Lys Val Lys Thr Trp Asp Gly Arg Thr Gly Arg Phe
450 455 460
Leu Ser Thr Leu Val Gly His Val Gly Ala Val Tyr Met Val Ala Trp
465 470 475 480
Ser Pro Asp Ser Arg Leu Leu Val Ser Ala Ser Lys Asp Ser Thr Leu
485 490 495
Lys Leu Trp Asp Val Ala Lys Gly Ala Lys Ala Lys Glu Thr Leu Pro
500 505 510
Gly His Met Asp Glu Val Tyr Ala Leu Asp Trp Ala Pro Asn Gly Ala
515 520 525
Ser Val Ala Ser Gly Ser Lys Asp Arg Thr Ile Lys Ile Trp Arg Ala
530 535 540
<210> 42
<211> 601
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> FBW7.5-4195多肽
<400> 42
Met Thr Gly Pro Ser Thr Arg Asn Ser Ser Ser Leu Thr Ala Ala His
1 5 10 15
Trp Arg Thr Ser Val Leu Asn Ala Leu Gln Gln Gln Asn Asp Val Glu
20 25 30
Val Glu Pro Phe Arg Gly Ile Ile Leu Ser Tyr Thr Asp Leu Ala Arg
35 40 45
Gln Asn Gln Val Leu Lys Ala His Val Asp His Gln Glu Lys Glu Leu
50 55 60
Val Thr Leu Arg His Glu Ala Leu Glu Gln Ser Asp Ser Arg Gly Gln
65 70 75 80
Gly Gly Ala Gly Ala Cys Gly Ala Lys Asp Glu Gln Thr Arg Lys Leu
85 90 95
Gln Ser Lys Val Gln Arg Leu Gln Glu Glu Leu Thr Asp Lys Leu Arg
100 105 110
Leu Glu Val Gln Gly Thr Thr Ser Gln Leu Asn Met Ser Lys Glu Ile
115 120 125
Gln Asp Leu Phe Gln Lys Trp Gln Leu Ser Arg Ala Glu Ala Glu Lys
130 135 140
Leu Arg Thr Glu Val Asp Gly Phe Arg Ala Arg Glu Ala Thr Leu Glu
145 150 155 160
Ala Gln Ala Gly Met Ala Thr Arg Asp Leu Glu Ile Val Gln Glu Glu
165 170 175
Leu Lys Arg Val Arg Ala Arg Leu Asn Thr Val Glu Lys Glu Tyr Leu
180 185 190
Asp Val Arg Arg Gln Asn Glu Glu Leu Val Arg Arg Met Val Gly Glu
195 200 205
Lys Ser His Asn Ala Glu Glu Met Asn Arg Met Thr Glu Leu Val Glu
210 215 220
Arg Met Arg Ala Gln Leu Lys Ala Arg Asn Leu Glu Ile Glu Ser Thr
225 230 235 240
Gln Ala Val Ala Ala Asp Ile Glu Ile Glu Ala Ala Pro Ser Ser Gly
245 250 255
Leu Arg Thr Asp Glu Leu Pro Ser His Cys Asn Gln Gln Phe Arg Ala
260 265 270
His Ala Ala Asp Val Asn Asp Leu Val Tyr Cys Asp Thr Gly Gln Trp
275 280 285
Leu Ala Thr Ala Gly Gly Asp Gly Lys Val Arg Val Trp Glu Ala Gly
290 295 300
Ser Gly Arg Leu Lys Ala Thr Leu His Gly Gln Asp Val Met Leu Gly
305 310 315 320
Leu Asp Phe Arg Gly Asp Phe Val Val Gly Gly Ser Ser Asp His Thr
325 330 335
Cys Lys Leu Trp Ser Leu Ala Ser Gly Arg Leu His Arg Thr Phe Val
340 345 350
Gly His Ser Gly Asn Val Tyr Ala Val Lys Leu Ile Ala Gly Asp Leu
355 360 365
Arg Ala Val Leu Thr Gly Gly Ala Asp Arg Thr Ile Arg Leu Trp Asp
370 375 380
Val Gly Arg Ala Ser Cys Arg Gln Val Leu Arg Ser Gly Ser Thr Cys
385 390 395 400
Asn Gly Leu Asp Ile Gly Leu Asp Gly His Ala Pro Val Ser Ala His
405 410 415
Gln Asp Gly Gly Leu Arg Phe Trp Asp Leu Arg Ala Gly Asn Pro Thr
420 425 430
Met Ile Val Arg Ala Phe Glu Thr Gln Ala Thr Ser Val Gln Tyr Gly
435 440 445
Gln Asn Phe Thr Ala Leu Ala Asn Ser Arg Asp Asn Ala Leu Lys Ile
450 455 460
Ile Asp Thr Arg Thr Phe Glu Thr Leu His Val Leu Arg His Glu Asp
465 470 475 480
Tyr Arg Thr Phe Leu Asn Trp Ser Arg Ala Cys Phe Ser Pro Ser Ser
485 490 495
Ser Tyr Val Ala Ala Gly Ser Ala Thr Gly Gln Leu Phe Val Trp Glu
500 505 510
Thr Ala Ser Gly Glu Met Lys Ser Ile Leu Ser His Ala Pro Asp Ser
515 520 525
Arg His Ser Phe Ser Ser Ser Pro Glu His Asp Glu Leu Arg Cys Gln
530 535 540
Gly Met Ser Leu Ser Ala Ser Gly Asp Arg Ile Arg Thr Ser Ser Glu
545 550 555 560
Ala Leu Glu Gly Ser Thr Ser Gly Ala Gly Gln Arg Gly Gly Gly Gly
565 570 575
Gly Ile Ile Ser Cys Ala Trp Lys Ala Ser Arg Leu Ser Ala Cys Thr
580 585 590
Arg Ser Gly Asn Val Cys Ile Trp Ser
595 600
<210> 43
<211> 578
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> Wee1.1-6397多肽
<400> 43
Met Thr His Ser Leu Pro Leu Tyr Leu Phe Glu Lys Val Met Ser Leu
1 5 10 15
Val Leu Asp Pro Arg Asn Leu Ile Ser Pro Cys Pro Phe Leu Ser Thr
20 25 30
Ser His Thr Ala Thr Gln Met Ser Ser Met Met Met Asp Leu Asp Asp
35 40 45
Asp Asp Gly Pro Leu Lys Met Glu Asp Gly Gly Gly Ser Gly Gly Ser
50 55 60
Pro Gly Gly Ser Asp Asp Tyr Ile Leu Pro Arg Pro Pro Ser Pro Gly
65 70 75 80
Gly Leu Gln Arg Thr Phe Thr Pro Val Asp Asp Tyr Glu Thr Thr Cys
85 90 95
Ser Pro Leu Pro Pro Leu Pro Pro Ser Ser Ser Ser Thr Ser Ser Asp
100 105 110
Tyr Lys Arg Asn Gly Arg Ala Asn Cys Asp Phe His Leu Ser Arg Pro
115 120 125
Ile Ala Ser Pro Phe Ser Gly Arg Asn Ser Gln His Gln Pro His Ser
130 135 140
Ala Pro Met Gly Pro Pro Gln Ala His Leu Arg Glu Asn Ile Asn Arg
145 150 155 160
Asp Leu Lys Arg Gly Tyr Ala Met Gln Asp Glu Leu Gly Lys Asn Tyr
165 170 175
Phe Leu Asp Glu Ser Pro Ser Asp Val Met Ala Phe Pro Pro Pro Thr
180 185 190
Pro Tyr Lys Pro Ala His Pro Gly Tyr Ala Pro Ala Ser Ala Thr Arg
195 200 205
Thr Pro Ser Ser Leu His Arg His Pro His Glu Pro His Ser His His
210 215 220
His Pro Thr His Ser Ser Lys Arg Asn Gly Arg Gly Ser Asn Pro Cys
225 230 235 240
Ser Thr Asp Lys His Arg Gly Ser Gly Ala Gly Pro Gly Ser Ser Gly
245 250 255
Arg Arg Thr Ser Leu His Glu Gly His Gly Pro Thr Thr Pro Val Ser
260 265 270
Arg Phe Gln Thr Asp Phe Asp Val Val Arg Val Ile Gly Ser Gly Cys
275 280 285
Phe Gly Glu Val Tyr Arg Val Arg Ser Arg Val Asp Gly Val Glu Tyr
290 295 300
Ala Val Lys Cys Thr Arg Arg Arg Phe Arg Gly Pro Ala Asp Arg Asn
305 310 315 320
Arg Tyr Leu Gln Glu Val Lys Ala Leu Ala Lys Val Cys Ala Ala Asp
325 330 335
Ser Ser Glu Glu Val Leu His Val Val Arg Tyr His Gln Ala Trp Ile
340 345 350
Glu Asp Glu Arg Leu Phe Met Gln Thr Glu Leu Cys Glu Glu Ser Leu
355 360 365
Asp Gly Ala Leu Arg Ala Gly Glu Lys Met Gly Phe Glu Glu Val Phe
370 375 380
Asp Phe Met Arg Gln Met Leu Leu Ala Leu Asp Val Leu His Arg His
385 390 395 400
Gly Leu Val His Leu Asp Val Lys Pro Gly Asn Ile Phe Ile Lys Ala
405 410 415
Gly Val Tyr Lys Leu Gly Asp Phe Gly Leu Val Ala Ser Val Asn Ser
420 425 430
Ser Asp Gly Leu Gly Asp Ser Leu Val Glu Gly Asp Ser Arg Tyr Met
435 440 445
Ser Ala Glu Leu Leu Gln Asp Gly Pro Lys Asp Leu Thr Lys Cys Asp
450 455 460
Ile Phe Ser Leu Gly Ala Thr Val Tyr Glu Met Gly Arg Gly Arg Ala
465 470 475 480
Leu Pro Pro Asn Gly Glu Glu Trp His Ala Leu Arg Ser Gly His Pro
485 490 495
Pro Ser Leu Lys Gly Glu Pro Ala Val Leu Val Ser Asp Leu Met Arg
500 505 510
Val Leu Ala Gln Met Met Ala Arg Glu Pro Ser Gln Arg Pro Ser Ala
515 520 525
Ala Val Leu Leu Thr His Pro Arg Leu Arg Ser Lys Leu Glu Arg Glu
530 535 540
Leu Leu Gln Glu Lys Met Lys Ser Lys Lys Leu Val Lys Ala Leu Val
545 550 555 560
Gln Asn Gln Gln Gln Gln Ser Lys Gly Arg Lys Leu Glu Arg Thr Gln
565 570 575
Thr Tyr
<210> 44
<211> 596
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> Wee1.2-4623多肽
<400> 44
Met Gly Leu Asp Asp Phe Glu Val Ile Gln Gly Leu Gly Lys Gly Ala
1 5 10 15
Phe Ala Arg Val Asp Lys Val Lys Arg Lys Ile Asp Gly Lys Val Tyr
20 25 30
Ala Leu Lys Arg Val Asn Ile Ser Thr Ile Pro Pro Lys Asp Leu Glu
35 40 45
Asp Ser Leu Asn Glu Ile Arg Ile Leu Ala Ser Phe Lys His Pro Arg
50 55 60
Leu Ile Arg Trp Tyr Glu Thr Phe Val Glu Asn Ala Lys Glu Glu Leu
65 70 75 80
Cys Ile Val Met Glu Leu Cys Pro Tyr Gly Asp Leu Glu Gln Lys Ile
85 90 95
Lys Arg His Lys His Arg Lys Gln Tyr Ile Asp Glu Arg Glu Ile Trp
100 105 110
Val Tyr Ala Val Asn Leu Leu Glu Gly Leu Ala Ala Leu His Ser Lys
115 120 125
Gly Val Val His Arg Asp Leu Lys Pro Ala Asn Cys Leu Ile Asp Ser
130 135 140
Gln Gly Cys Val Lys Ile Ala Asp Met Asn Ile Ser Lys Val Ser Lys
145 150 155 160
Gly Gly Asn Met Gln Thr Gln Val Gly Thr Pro Tyr Phe Ile Cys Pro
165 170 175
Glu Ile Tyr Leu Lys Arg Pro Tyr Thr Ser Thr Ser Asp Ile Trp Ser
180 185 190
Leu Gly Gly Val Leu Tyr Asn Leu Ala Ala Leu Arg Pro Pro Phe Leu
195 200 205
Ala Asp Asn Ile Gln Asn Leu Arg Arg Val Val Ile Arg Gly Ser Phe
210 215 220
Asp Pro Leu Pro Ser Val Phe Gly Gln Ser Leu Thr Thr Leu Ile Gly
225 230 235 240
Gln Leu Leu Gln Ile Asn Pro Ser Asp Arg Pro Glu Ala Lys Glu Ile
245 250 255
Leu Lys Asp Pro Leu Val Glu Arg His Lys Tyr Leu Leu Leu His Pro
260 265 270
Leu Pro Ala Gly Gln Glu Glu Ala Glu Gly Glu Met Leu Pro Thr Ile
275 280 285
Arg Val Ala Ser Asp Lys Glu Gly Thr Lys Thr Ile Arg Leu Pro Gly
290 295 300
Pro Ala Tyr Glu Glu Glu Glu Thr Gly Ala Gly Thr Arg Ala Gly Lys
305 310 315 320
Glu Arg Gly Ala Arg Asp Arg Pro Pro Ser Pro Arg Ser Pro Thr Ser
325 330 335
Val Phe Val Val Ser Ser Pro Arg Ala Lys Asp Lys Ala Asn Ser Pro
340 345 350
Ala Ser His Pro Ala Arg Glu Ser Thr Ser Pro Ser Asn Ser Ser Ala
355 360 365
Gly Ser Ala Ala Arg Thr Glu Pro Ala Pro Arg Val Gln Ser Ser Pro
370 375 380
Pro Ala Asp Ala Pro Arg Arg Pro Pro Arg Ser Ser Pro Pro Pro Ser
385 390 395 400
Ser Pro Pro Thr Ser Pro Ser Gln Leu Pro Met Gly Gly Arg Leu Ser
405 410 415
Pro Leu Lys Thr Pro Ser Pro Thr Gln Met Phe Pro Ser Phe Pro Phe
420 425 430
Ala Ser Pro Lys Val Gly Gly Ser Lys Pro Ser Ser Pro Ser Asn Gly
435 440 445
Thr Ser Thr Ala Pro Thr Ala Ala Pro Gly Ser Pro Pro Arg Ser Gly
450 455 460
Ser Pro Leu Ser Ser Leu Leu Pro Ala Leu Gln Asp Met Gly Arg Asp
465 470 475 480
Met Val Arg His Leu Pro Val Arg Pro Pro Gly Met Lys Val Ser Lys
485 490 495
Glu Glu Lys Ala Arg Leu Ala Ala Ile Ala Ala Gly Glu Tyr Gly Gly
500 505 510
Gly Ala Val Phe Ile Asp Thr Gly Glu Tyr Asp Asp Val Gly Asp Val
515 520 525
Ala Gly Ser Pro Ala Thr Ser Thr Ser Val Gly Gly Lys Ala Lys Arg
530 535 540
Ser Ser Ile Gln Arg Val Phe Asp Gly Glu Gly Leu Pro Ser Phe Pro
545 550 555 560
Thr Leu Glu Ala Phe Pro Val Ala Glu Val Lys Asn Phe Phe Ala Asn
565 570 575
Ala Asn Gly Arg Arg Thr Pro Pro Pro Gly Glu Lys Pro Asp Met Glu
580 585 590
Glu Trp Arg Leu
595
<210> 45
<211> 505
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> Wee1.3-8521多肽
<400> 45
Met Gly Ile Glu Gln Phe Glu Ile Leu Lys Ser Leu Gly Glu Gly Ala
1 5 10 15
Phe Ala Ser Val His Lys Val Thr Arg Leu Val Asp Gly Lys Thr Tyr
20 25 30
Ala Leu Lys Lys Val Asp Val Ser Ser Leu Asp Asp Lys Glu Leu Leu
35 40 45
Ser Ala Leu Asn Glu Ile Arg Leu Leu Ala Ser Phe Gly His Pro Arg
50 55 60
Ile Val Arg Leu His Glu Thr Phe Met Asp Gly Asn Asn Leu Cys Ile
65 70 75 80
Val Met Glu Tyr Cys Gly Trp Gly Asp Leu Ala Met Lys Ile Lys Arg
85 90 95
Tyr Val Lys Arg Arg Glu Tyr Ile Asp Glu Arg Val Ile Trp Val Tyr
100 105 110
Met Ile Gln Ile Leu Glu Gly Leu Lys Ala Leu His Glu Arg Asn Val
115 120 125
Leu His Arg Asp Leu Lys Pro Ala Asn Cys Phe Leu Ala Glu Asp Gly
130 135 140
Ser Ile Lys Ile Gly Asp Met Asn Val Ser Lys Val Met Lys Asp Gly
145 150 155 160
Asn Ala Lys Thr Gln Ile Gly Thr Pro Tyr Tyr Met Ser Pro Glu Ile
165 170 175
Trp Ala Arg Arg Pro Tyr Asn His Ala Thr Asp Ile Trp Ser Leu Gly
180 185 190
Cys Leu Ile Tyr Glu Leu Cys Ala Leu Arg Pro Pro Phe Leu Gly Asn
195 200 205
Asn Met Ser Glu Leu Lys Thr Ala Val Leu Gly Gly Asn Phe Asn Pro
210 215 220
Val Pro Ser Val Tyr Ser Lys Asp Leu Gly Ser Val Ile Ala Arg Met
225 230 235 240
Leu Leu Ala Ala Ala Arg Asp Arg Pro Ser Ala Ala Glu Ala Leu Ala
245 250 255
Tyr Pro Glu Val Asn Ala Arg Lys Cys Leu Val Lys Asn Val Leu Arg
260 265 270
Glu Glu Glu Leu Tyr Thr Lys Gly Gly Lys Gly Gly Tyr Gly Ala Glu
275 280 285
Asp Ala Leu Met Pro Thr Ile His Ile Gly Ser Leu Arg Glu Leu Gly
290 295 300
Ile Lys Leu Pro Gly Pro Ser Tyr Pro Ser Asp Gly Pro Pro Thr Pro
305 310 315 320
Thr Thr Pro Ile Met Leu Ala His Asp Ala Ser Pro Val Asn Glu Lys
325 330 335
Ala Thr Leu Ala Ser Pro Ile His His Arg Gln Lys His Glu Gln Glu
340 345 350
Phe Pro Ala Ser Gly Ser Pro Ser His His Arg Arg Ala Gly Leu Pro
355 360 365
Gly Ala Lys Ser Glu Ser His Ser Pro Leu Ser Pro Ser Ile Pro Pro
370 375 380
Ile Asp Lys Gly Arg Gln Gln Ser Pro Gly Val Gly Ser Leu Asp Arg
385 390 395 400
Asp Leu Ser Gly Ala Lys Glu Arg Gly Ser Ala Ser Tyr Pro Gln Asp
405 410 415
Arg Arg Arg Val Pro Thr Pro Pro Pro Asn Gly Ala Pro Ala Lys Pro
420 425 430
Leu Pro Ile Gly Arg Ile Pro Ser His Gly Lys Gly Ser Ser Ala Ala
435 440 445
Ser Ser Ser Leu Pro Val Leu Lys Ile Lys Thr Ser Ser Ser Gly Ser
450 455 460
Gly His Val Thr Pro Val Gly Gly Val Gly Asp Glu Tyr Ser Met Leu
465 470 475 480
Pro Arg Thr Glu Gln Lys Asn Ser Val Lys Asn Val Leu Gln Ala Val
485 490 495
Gln Lys Glu Ile Ser Arg Pro Pro Tyr
500 505
<210> 46
<211> 289
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> Wee1.4-7374多肽
<400> 46
Met Asn Gly Gly Lys Arg Gly Glu Gly Gly Gly Tyr Asp Gly Ser Glu
1 5 10 15
Ala Cys Leu Leu Arg Val Ala Arg Asp Val Gly Gly Ala Leu Asp Phe
20 25 30
Met His Ser Arg Gly Ile Val His Met Asp Val Lys Pro Gly Asn Ile
35 40 45
Phe Ile Ala Ala Asp Gly Ser Phe Lys Leu Gly Asp Leu Gly His Ala
50 55 60
Ile Lys Ala Asp Gly Ser Met His Val Leu Glu Gly Asp Glu Arg Tyr
65 70 75 80
Leu Ser Met Glu Val Leu Lys Gly Leu Asp Leu Phe Lys Leu Ser Ala
85 90 95
Gly Lys Glu Val Pro Pro Tyr Arg Thr Ile Leu Glu Pro Asn Asp Ile
100 105 110
Phe Gly Leu Gly Ala Ser Leu Tyr Glu Ala Trp Ser Arg Val Pro Leu
115 120 125
Ala Gly Ala Gly Pro Glu Phe Leu Ala Val Arg Glu Gly Arg Leu Thr
130 135 140
His Leu Pro Ser Asn Gly Glu Lys Ser Val Ser Glu Gly Phe Glu Arg
145 150 155 160
Phe Leu Arg Asn Leu Leu Ala Pro Arg Gly Glu Asp Arg Pro Thr Ala
165 170 175
Ala Glu Val Val Gly Arg Ala Met Gly Leu Leu Gly Ala Gly Ser Gly
180 185 190
Gly Arg His His Ala His Gly Trp Glu Gly Gly Trp Gln Thr Gln Ala
195 200 205
Ala Glu Asp Thr Asp Asn Gly Ser Pro Arg Ala Ser Arg Arg Gln Glu
210 215 220
Glu Asp His His Phe Ser Asp Ala Lys Gly Arg Ala Ala Ala Ala Val
225 230 235 240
Ala Ala Ala Ala Ala Gly Ala Gln Ser Glu Leu Asn Arg Asp Ala Pro
245 250 255
Leu Arg Val Pro Phe Ala Cys Glu Leu Pro Glu Cys Gln Ala Arg Gln
260 265 270
Gln Arg Ile Arg Ala Leu Glu Thr Met Ile Leu Ser Leu Thr Gly Glu
275 280 285
Gly
<210> 47
<211> 540
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> Wee1.5-9810多肽
<400> 47
Met Tyr Ser Gln Ala Ser Ser Gln Phe Ser Gln Glu Asp Tyr Leu Thr
1 5 10 15
Pro Ala Thr Gln Asp His Thr Ser Ala Pro Phe Leu Pro Pro His Tyr
20 25 30
Ala Ile Ala Ser Pro Ser Pro Ile Lys Lys Lys Ser Arg Pro Ser Arg
35 40 45
Arg Gly Ser Asn Gly Ala Ala Thr Arg His Gln Ala Met Met Met Ser
50 55 60
Val Leu Pro Glu Gly Ile Pro Leu Pro Asn Gly Gly His Ala Ile Asn
65 70 75 80
His Lys Ser Pro Arg Asp Ala Leu Pro Pro Met Gln Val His Pro Gly
85 90 95
Pro Ile Leu His Ala Gly Asp Thr His Arg Ser Leu Gly Lys Ala Gln
100 105 110
Gly Ser His Ala Ser Asn Ser Thr Leu Pro Ser Ser Leu Gly Arg Glu
115 120 125
Glu Gly Asn Pro Ser Pro Pro Thr Pro Ser Lys Leu Arg Glu Arg Val
130 135 140
Asp Arg Arg Leu Asp Pro Arg His Lys Pro Leu Asn Arg Asp Leu His
145 150 155 160
Gly Leu Pro Ala Ser Pro Leu His Ser Arg Ala Ser Leu Gly Ser Ala
165 170 175
Ser Val Leu Arg Ala Arg Asp Ala Ser Leu Gly Leu Thr Ser Ser Ser
180 185 190
Ser Ser Ser Ala Ser Ser Ser Ala Ser Pro Pro Pro Ser Ala Ser Phe
195 200 205
Leu Pro Pro Arg Gly Pro Gly Pro Asp Ala Leu Gly Pro Gly Leu Asp
210 215 220
Leu Pro His Pro His Arg Ser Asp Glu Lys Arg Gly Ser Ser Phe Gln
225 230 235 240
Ser Leu Ser Gln Ser Val Val Asp Cys Phe Gly Ser Leu Arg Val His
245 250 255
Gly Pro Ser Pro Glu Glu Val Pro Asn Glu Ser Lys Gly Lys Gly Ala
260 265 270
Leu Gly Phe Gln Gly Ala Arg Gly Glu Glu Gly Glu Glu Glu Gly Gly
275 280 285
Glu Gly Glu Gly Trp Ala Met Gln Ser Ser Gln Asp Met Ala Gly Gly
290 295 300
Phe Arg Leu Lys Ser Val Gly Ala Val Trp Asp Glu Glu Glu Gly Ala
305 310 315 320
Glu Met Glu Ala Gly Gly Gly Leu Arg Gly Glu Gly Glu Gly Glu Arg
325 330 335
Gly Arg Gln Arg Ala Met Thr Gly Gly Pro Ile Ser Leu Asp Ala Trp
340 345 350
Gly Gly Arg Gly Gly Gly Arg Glu Asp Glu Ala Gly Gly Glu Val Val
355 360 365
His Val Val Ser Ile Pro Ala Cys Asn Pro Asn Pro Phe Gly Pro Pro
370 375 380
Ser Ser Arg Ala Ser Glu Asp Leu Pro Glu Gly Arg Lys Arg Thr Arg
385 390 395 400
Gln Arg Lys Ile Lys Ile Pro Cys Ser Arg Gly Thr Gly Glu Gly Gly
405 410 415
Arg Glu Gly Ala Gly Gly Gly Gly Glu Gly Glu Val Ser Arg Tyr Leu
420 425 430
Leu Glu Phe Glu Gln Leu Glu Thr Ile Gly Ala Gly Arg Phe Ser Leu
435 440 445
Val His Lys Val Arg Lys Arg Leu Asp Gly Trp Val Tyr Ala Ile Lys
450 455 460
Arg Ser Arg His Ser Leu Val Ser Glu Gly Glu Lys Glu Gly Ala Met
465 470 475 480
Arg Glu Val Tyr Ala Leu Ala Ala Leu Gln Gly Cys Pro His Leu Val
485 490 495
Arg Tyr Met Ser Ala Trp Met Glu Ala Ser Tyr Leu Phe Ile Gln Thr
500 505 510
Glu Tyr Cys Pro Gly Gly Cys Leu Glu Arg Ala Val Phe Asp Ser Val
515 520 525
Arg Ala Arg Gly Gln Gly Ala Gly Glu Gly Pro Gly
530 535 540
<210> 48
<211> 518
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> Cyclin-6855多肽
<400> 48
Met Val His Ser Lys Arg Lys Ala Leu Glu Asp Ile Thr Ala Val Ala
1 5 10 15
Leu Ser Gly Ser Gln Pro Phe Arg Ala Ala Tyr Asp Thr Gly Gly Lys
20 25 30
His Lys Arg Ala Cys Ile Asp Thr Val Asn Gln Thr Ser Lys Arg Arg
35 40 45
Val Pro Asp Ala Pro Cys Arg Val Thr Arg Arg Ser Ser Ala Leu Ala
50 55 60
Ala Phe Ser Leu Thr Thr Asp Gly Pro Ala Gln Asp Gly Lys Ala Thr
65 70 75 80
Phe Arg Arg Leu Tyr Ser Thr Arg Ser Cys Pro Asn Val Glu Ile Asn
85 90 95
Cys Pro Val Pro Ala Ser Ser Lys Val Lys Asp Asp Lys Asp Ser Asp
100 105 110
Ala Asp Leu His Leu Ala Cys Lys Glu Ala Leu Ala Met Glu Glu Ile
115 120 125
Ser Gly Ser Pro Asp Arg Arg Pro Thr Asp Glu Pro Val Ala Leu Pro
130 135 140
Ser Ser Ala Ser Gln Ser Ser Asp Ser Glu Val Asn Thr Gly Leu Glu
145 150 155 160
His His Leu Ser Asp Ala Thr Ser Phe Pro Phe Ser Ser Ser Leu Ser
165 170 175
Ser Ser Thr Leu Pro Val Gly Ile Ile Asp Ile Asp Glu Thr Gln Gly
180 185 190
Ser Ser Gln His Pro Arg Ser His Leu Leu Pro His Gln Val Gly Lys
195 200 205
Ala Ser His Lys Gln Ser Asp Asn Val Ile Val Pro Arg Ser Ser Asn
210 215 220
Leu Ala Leu Thr Asp Gly Thr Pro Gln Arg Leu Ala Ser Tyr Ser Arg
225 230 235 240
Asp Tyr Phe Ala Tyr Leu Arg Glu Arg Glu Arg Glu Gln Gln Asp Arg
245 250 255
Pro Leu Pro Asp Tyr Met Ser Arg Ile Gln Gly Gly Ala Leu Thr Gln
260 265 270
Asp Met Arg Ala Leu Leu Val Asp Trp Leu Val Thr Val Cys Glu Glu
275 280 285
Cys Glu Leu Met Pro Ser Thr Leu Tyr His Cys Val Glu Leu Ile Asp
290 295 300
Arg Ala Leu Ser Lys Leu Gln Val Pro Lys Glu Gln Leu Gln Cys Met
305 310 315 320
Gly Cys Ala Cys Leu Phe Ile Ala Cys Lys Phe Glu Glu Thr Thr Val
325 330 335
Pro Ser Leu Glu Glu Phe Thr Tyr Met Ala Ala Glu Ser Phe Ser Lys
340 345 350
Lys Gln Leu Thr Asp Leu Glu Leu Arg Val Val Glu Ala Leu Ser Phe
355 360 365
Arg Leu Ser Thr Val Thr Ala Tyr Asn Phe Leu Ser Arg Phe Thr Leu
370 375 380
Ala Ala Gly Ser Gly Pro Arg Glu Thr Ala Leu Val Tyr Tyr Phe Ser
385 390 395 400
Glu Leu Ser Leu Leu His Tyr Glu Phe Leu Gly Phe Ser Pro Ser Ile
405 410 415
Arg Ala Ala Ala Ala Leu Tyr Leu Ala Arg Gln Thr Leu Ala Val Ser
420 425 430
Asn Thr Asp Ser Ala Arg Arg Ala Ala Glu Glu Arg Asn Ile Trp Thr
435 440 445
Pro Thr Ile Ala Phe Tyr Thr Gly Tyr Ser Pro Ala Thr Thr Leu Pro
450 455 460
Leu Gln Glu Cys Val Arg Leu Leu Arg Arg Ala His Ala Gly Val Glu
465 470 475 480
Tyr Ser Pro Tyr Glu Ala Leu Lys Leu Lys Tyr Gly Ala Pro Ala Leu
485 490 495
Leu His Val Gly Asp Ile Ala Cys Val Glu Glu Lys Asp Leu Gly Ala
500 505 510
Thr Ile Leu Asn Leu Asn
515
<210> 49
<211> 688
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> Cyclin-3560多肽
<400> 49
Met Arg Lys Arg Arg His Asp Asp Ser Ser Gln Asn Ser Glu Val Ala
1 5 10 15
Leu Ser Ser Gln Val Lys Arg Ala Val Asn Pro Arg Ser Thr Arg Ser
20 25 30
His Ser Arg Ser Thr Asp Lys Gly Ile Ser Asp Glu Ala Lys Lys Asn
35 40 45
Gln Phe Ser Ala Pro Asp Ser Lys Ser Ala Ala Ser Arg Pro Ser Arg
50 55 60
Gly Gln Phe Ser Leu Lys His Ala Arg Glu Glu Pro Ala Thr Glu Gln
65 70 75 80
Asn Leu Val Asn Gln Ser Arg Asp Ser Pro Arg Pro Pro Met Gln Pro
85 90 95
Cys Pro Val His Cys His Gly Ala Met Glu Asp Met Asp Ser Ile Ser
100 105 110
Ser Thr Asn Thr Thr Lys Arg Asn Val Glu Gly Arg Pro Ser Leu Ile
115 120 125
Ile Leu Gln Thr Gln Cys Leu Glu Ala Ile Leu Gln Asn Leu Thr Pro
130 135 140
Ile Ser Arg Ser Pro Ser Ala Pro Pro Val Leu Leu Ala Glu Glu Arg
145 150 155 160
Leu Ala Leu Arg Lys Leu Gly Leu Gln Leu Met Ala Leu Thr Ala Pro
165 170 175
Asp Asp Gly Glu Asn Ser Gly Phe Pro Leu Ser Tyr Ala Gly Ala Glu
180 185 190
His Ala Gly Val Asp Met Val Ser Ala Ser Glu Glu Glu Arg Glu Glu
195 200 205
Arg Arg Gly Glu Ala Ser Lys Ala Phe Ala Pro Leu Cys Gly Arg Gly
210 215 220
Ile Ala Ile Asp Glu Ser Ser Thr Tyr Asn Gly Leu Ser Ser Thr Asp
225 230 235 240
Asn Gly Ala Gln Arg Ser Gly Asn Arg Pro Ser Arg Leu Ser Ser Ser
245 250 255
Ser Ser Ile Pro Ala Ala Ser Leu Ser Ser Pro Ser Leu Ser Leu Lys
260 265 270
Arg Pro Ser Ser Val Met Arg Ala Phe Arg Tyr Lys Cys Gly Pro Cys
275 280 285
Pro Lys His Ala Ser Tyr Asp Leu Pro Arg Thr Ile Lys Gly Asn Pro
290 295 300
Gly Gly Ile Thr Arg Ala Gly Lys Pro Pro Asp Asp Gly Leu Leu Val
305 310 315 320
Leu Pro Glu Ser Val Trp Gln His Ala Phe Ser Phe Val Pro Ala His
325 330 335
Asp Leu Leu Ser Val Met Leu Thr Ala Arg Pro Phe Cys Ser Met Ala
340 345 350
Glu Pro Phe Lys Gly Phe Tyr Phe His Trp Asn Leu Arg Ala Ala Glu
355 360 365
Ser Leu Gln Ser Leu Arg Pro Tyr Ile Ser Lys Leu Arg His Leu Asn
370 375 380
Ala Lys Met Arg Ala Ile Leu Leu Asp Trp Val Thr Asp Val His Gln
385 390 395 400
Ser Leu Ser Phe Ala Pro Ala Thr Leu Tyr Arg Thr Ala Gln Val Leu
405 410 415
Asp Gln Phe Leu Ser Arg Thr Glu Asn Val Thr Arg Glu Lys Leu Gln
420 425 430
Leu Val Gly Val Thr Ala Phe Met Val Ala Ala Lys Gly Val Glu His
435 440 445
Thr Pro Pro Asp Pro Asp Asp Cys Ala Tyr Trp Thr Asp Asn Ala Tyr
450 455 460
Ser Gly Leu Glu Val Ser Ser Met Glu Ala Arg Leu Leu Lys Val Leu
465 470 475 480
Ser Gln Ser Pro Phe Arg Pro Pro Ser Leu Pro Pro Thr Ala Gln Asp
485 490 495
Phe Leu Thr Leu Tyr Leu Lys Glu Val Gly Ala Gly Lys Leu Ala Ser
500 505 510
Cys Arg Ala Gln Tyr Tyr Cys Glu Arg Thr Leu Gln Glu His Asp Met
515 520 525
Leu Ser Phe Pro Pro Ser Leu Ile Ala Ala Ala Ser Val Ile Leu Ala
530 535 540
Leu Lys Ser Ser Pro Ile Pro Val Val Val His Pro Cys Ser Thr Gln
545 550 555 560
Lys Pro Lys Ser Trp Thr Glu Ala Val Ala His Tyr Ser Gly Tyr Ser
565 570 575
Asp Thr Lys Val Ala Ala Cys Ala Arg Arg Ile Cys Gln His Val Arg
580 585 590
His Thr Val Thr Thr Leu Ser Gly Arg Lys Leu Asp Ala Val Lys Arg
595 600 605
Lys Tyr Ala Asn Gly Leu Phe Leu Ala Val Ser Arg Met Glu Pro Pro
610 615 620
Thr Trp Gly Gly Gly Pro Gly Glu Asp Ala Glu Lys Ala Glu Gly Gly
625 630 635 640
Gly Gly Gln Glu Glu Gly Gly Glu Ile Ser Asn Ala Glu Gly Gly Asn
645 650 655
Ala Pro Gly Gly Asp Glu Val Asp Val Glu Glu Ala Gly Glu Glu Gly
660 665 670
Asn Gly Asp Gly Gly Met Gly Gly Asp Val Glu Gly Gly Leu Glu Val
675 680 685
<210> 50
<211> 729
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> Cyclin-9008多肽
<400> 50
Met Tyr Gly Leu Asn Lys Ala Cys Glu Asp Ser Val Leu Ala Ala Ala
1 5 10 15
Cys His Glu Thr Leu Glu Met Glu Asp Leu Glu Ala Leu Ala Thr Cys
20 25 30
Ala Lys Ser Thr Tyr Ser Gly Asp Val Gly Gly Asp Thr Trp Ile Gln
35 40 45
Ser Arg Arg Pro Ser Arg Ser Ser Phe Tyr Leu Lys Thr Gly Leu Phe
50 55 60
Leu Pro Pro Asp Val Thr Leu His Asn Met Lys His Tyr Gly Arg Arg
65 70 75 80
His Phe Leu Lys Met Arg Glu Ser Glu Met His Ile Tyr Ala Cys Asp
85 90 95
Pro Cys Tyr Glu Glu Arg Gln Ile Glu Leu Arg Pro Asp Met Arg Ala
100 105 110
Gln Leu Val Asp Trp Leu Met Glu Val Cys Ala Asn Phe Ser Val His
115 120 125
Arg Arg Thr Phe Gln Ala Ala Val Asn His Cys Asp Arg Tyr Leu Ser
130 135 140
Leu Cys Ala Arg Gly Phe Pro Lys Gln Arg Leu Gln Leu Leu Ala Ile
145 150 155 160
Thr Ala Leu Phe Val Ala Ala Lys Met Asp Glu Val Tyr Pro Pro Lys
165 170 175
Ala His Asp Leu Ala Glu Ala Thr Ala Gly Ala Phe His Ala Arg Asp
180 185 190
Leu Val Val Phe Glu Gln Asp Leu Leu Thr Thr Leu Ser Trp Asn Leu
195 200 205
Thr Pro Pro Thr Pro Asp Asp Trp Ala Glu Trp Tyr Phe Leu Ala Phe
210 215 220
Leu Glu Arg Gly Leu Pro Ala Ala Ala Thr Glu Met Lys Ala Ala Ser
225 230 235 240
Ala Pro Ser Leu Ser Gln Gln Asp Leu Pro Ser Leu Ala Leu Leu Gln
245 250 255
His Leu Pro Thr Gly Ile Ala Gln Lys Val Gln Val Leu Leu Asp Leu
260 265 270
Ala Leu Leu Asp Val Thr Ser Ile His Phe Phe Pro Ser Met Leu Ala
275 280 285
Ala Ala Gly Leu Tyr Val Leu Leu Pro Pro Val Phe Tyr Pro Ala Leu
290 295 300
Ala Leu Ala Thr Gly Tyr Pro Pro Asn Asp Lys Ala Leu Glu His Cys
305 310 315 320
Lys Ala Tyr Leu Thr Phe Leu Ala Thr Gly Leu Phe Asp Ser Ser Leu
325 330 335
Pro Leu Gln Ala Leu Pro Leu Ser Arg Arg His Leu Gln His Gly Gln
340 345 350
Pro Gly Gln Arg Ala Gly Gly Gly Phe Gly Trp Thr Pro Glu Ala Ser
355 360 365
Glu Ala Ala Gln Ser Gly Val Pro Leu Trp Asp Lys His Ser Leu Gln
370 375 380
Ser His Pro Ser Arg Leu Leu Pro His Leu Leu Gly Arg Ile Ser Ala
385 390 395 400
Ile Ser Asp His Cys Asp Asp Leu Pro Pro Pro Leu Pro His Ala Pro
405 410 415
Lys Thr Gln Gly Ala Gln Ala Asp Asn Pro Ala Gly Asn Ala Ala Ala
420 425 430
Leu Ala Thr Ser Ser Leu Ser Pro Pro Cys Ser Ser Ala Ser Pro Ser
435 440 445
Ala Ser Thr Leu Pro Ser Ala Thr Arg Ala Pro Ala Ala Leu Ala Asp
450 455 460
Leu Val Thr Pro Ile His Ala Lys Ala Arg Leu Leu Ser Pro Leu Pro
465 470 475 480
Ser Phe Leu Gly Tyr Arg Ser Val Ser Ser Lys Asn Leu Ile Ala Gly
485 490 495
Arg Lys Gly Arg Cys Asp Glu Trp Glu Asp Gly Glu Ala Met Phe Met
500 505 510
Glu Thr Leu Glu Met Trp Pro Met Asp Glu Ser Glu Asn Gly Glu Glu
515 520 525
Thr Glu Glu Glu Glu Glu Glu Glu Glu Glu Gly Gly Ala Glu Glu Glu
530 535 540
Glu Glu Glu Lys Gly Gly Ala Glu Glu Glu Glu Glu Gly Arg Lys Gly
545 550 555 560
Met Asp Glu Asp Ile Phe Gly His Ser Leu Glu Leu His Glu Glu Trp
565 570 575
Glu Ser Gly Arg Arg Gly Arg Glu Arg Lys Ala Gly Arg Cys Gly Leu
580 585 590
Gly Gln Asp Lys Gly Arg Gly Ala Gly Pro Gln Ser Leu Arg Leu Glu
595 600 605
Ala Leu Asp Gly Glu Ala Ser Glu Asn Glu Leu Gly Trp Ser Met Ala
610 615 620
Thr Thr Val Asp Pro Gln Thr Thr Arg Asp Ser Val Phe Ser Phe Phe
625 630 635 640
Ser Leu Glu Gly Thr Glu Gly Gly Gln Asp Gly Gly His Gly Leu Gly
645 650 655
Ser Gly Arg Gly Ser Glu Glu Met Leu Gly Trp Gln His Glu Val Arg
660 665 670
Glu Arg Phe Gly Glu Asp Cys Thr Asn Thr Gln Glu Ser Cys Gly Glu
675 680 685
Gly Glu Glu Leu Glu Glu Met Arg Asn Val Ala Leu Ser Ser Pro Cys
690 695 700
Leu Thr Pro Ile Phe Cys Pro Asp Glu Ala Lys Val Ala Ser Gly Leu
705 710 715 720
Gly Ala Ser Ala Thr Thr Thr Ala Pro
725
<210> 51
<211> 463
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> Cyclin-4163多肽
<400> 51
Met His Ala Thr Arg Ser Glu Ala Ala Thr Phe His Gly Lys His Ser
1 5 10 15
Ser Ala His Glu His Glu Ala Leu Ala Thr Asp Asp Gly Pro Gln Gly
20 25 30
Pro Asp Cys Leu Gly Pro Leu Ile Lys Pro Gly Val Ser Thr Arg Ser
35 40 45
Gly Leu Ala Ser Gly Ala Ser Arg Arg Ala Leu Gly Asp Ile Thr Asn
50 55 60
Asn Arg Gly Ala Pro Ser Gly Lys Pro Gly Gln His Asn Thr Ser Lys
65 70 75 80
Pro Met Thr Arg Ala Met Trp Ala Thr Ala Ala Ala Gly Glu Gly Pro
85 90 95
Leu Pro Gly Ala Thr Ser Ser Val Gly Leu Ala Ser Val Ala Pro Pro
100 105 110
Pro Val Leu Tyr Glu Gln Pro Leu Pro Ala Leu Gln Lys Ser Asp Met
115 120 125
Asp Gly Lys Arg Arg Glu Glu Ala Val Asp Asp Met Asp Leu Ile Gln
130 135 140
Glu Val Glu Glu Ile Asp Met His Ile Glu Glu Ala Ser Glu Met Pro
145 150 155 160
Gln Glu Ala Ile Gln Ala Glu Ser Gln Glu Ala Leu Gly Ala Ile Ile
165 170 175
Glu Asp Leu Gln Gly Met Thr Leu Lys Tyr Ser Thr Ala Arg Pro Val
180 185 190
Leu Gly Leu Gly Val Asp Asp Ile Asp Ala Leu Asp Ala Ser Asn Pro
195 200 205
Leu Ala Cys Val Asp Tyr Val Glu Ser Gln Tyr Ser His Tyr Arg Glu
210 215 220
Lys Glu Cys Arg Pro Gly Tyr Asp Pro Gly Tyr Met Lys Lys Gln Pro
225 230 235 240
Tyr Ile Asn Val Arg Met Arg Ala Ile Leu Val Asp Trp Leu Val Glu
245 250 255
Val His Tyr Lys Phe Lys Cys Cys Pro Glu Thr Leu Tyr Leu Thr Val
260 265 270
Asn Leu Ile Asp Arg Phe Leu Asp Arg Lys Gln Val Pro Arg Pro Lys
275 280 285
Leu Gln Leu Val Gly Val Thr Ala Phe Leu Ile Ala Cys Lys Tyr Glu
290 295 300
Glu Ile Tyr Pro Pro Glu Val Lys Glu Leu Val Tyr Met Thr Asp Ala
305 310 315 320
Ala Tyr Thr Arg Lys Gln Ile Ile Asp Met Glu Ala Phe Met Leu Ala
325 330 335
Thr Leu Lys Phe Gln Val Thr Val Cys Thr Thr His Cys Phe Leu Val
340 345 350
Arg Phe Leu Lys Ala Gly His Ala Asp Asn Lys Leu Tyr Phe Leu Ala
355 360 365
Ser Tyr Ile Ala Glu Arg Thr Leu Gln Glu Val Asp Val Leu Cys Phe
370 375 380
Leu Pro Ser Met Val Ala Ala Ala Ala Val Tyr Leu Ala Arg Lys Asn
385 390 395 400
Cys Gly Met Arg Ser Trp Ser Pro Thr Leu Asn His Tyr Thr Lys Tyr
405 410 415
Ser Glu Glu Ala Leu Leu Pro Cys Leu Arg Val Leu Ser Pro Trp Leu
420 425 430
Asn Ser Arg Ser Gln Thr Leu Gln Ala Ile Arg Lys Lys Tyr Gly Ala
435 440 445
Ala Lys Phe Met Met Val Ser Ser Leu Glu Leu Thr Gly Val Val
450 455 460
<210> 52
<211> 397
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> CDKA1-3735多肽
<400> 52
Met Glu Arg Tyr Gln Lys Leu Glu Lys Ile Gly Glu Gly Thr Tyr Gly
1 5 10 15
Val Val Phe Lys Ala Lys Asp Arg Val Thr Asn Glu Ile Leu Ala Leu
20 25 30
Lys Lys Ile Arg Leu Glu Ala Glu Asp Glu Gly Ile Pro Ser Thr Ala
35 40 45
Ile Arg Glu Ile Ser Leu Leu Lys Glu Leu Gln His Pro Asn Ile Val
50 55 60
Arg Leu Tyr Asp Val Val His Thr Glu Arg Lys Leu Thr Leu Val Phe
65 70 75 80
Glu Tyr Leu Asp Gln Asp Leu Lys Lys Tyr Leu Asp Thr Cys Glu Ser
85 90 95
Gly Leu Asp Leu Pro Val Leu Gln Ser Phe Leu Tyr Gln Leu Leu His
100 105 110
Gly Val Ala Phe Cys His Asp His Arg Val Leu His Arg Asp Leu Lys
115 120 125
Pro Gln Asn Leu Leu Ile Asn Arg Glu Gly Glu Leu Lys Leu Ala Asp
130 135 140
Phe Gly Leu Ala Arg Ala Phe Gly Ile Pro Val Arg Ser Tyr Thr His
145 150 155 160
Glu Val Val Thr Leu Trp Tyr Arg Ala Pro Asp Val Leu Met Gly Ser
165 170 175
Arg Lys Tyr Ser Thr Pro Val Asp Ile Trp Ser Ile Gly Cys Ile Phe
180 185 190
Ala Glu Met Ala Asn Gly Arg Pro Leu Phe Ala Gly Ser Ser Glu Ser
195 200 205
Asp Gln Leu Asp Arg Ile Phe Arg Ala Leu Gly Thr Pro Thr Glu Gly
210 215 220
Met Tyr Pro Gly Ile Val Glu Leu Pro Glu Phe Gln Lys Val Lys Asn
225 230 235 240
Gln Phe Pro Arg Tyr Ser Pro Leu Glu Ser Trp Ala Pro Leu Val Pro
245 250 255
Thr Leu Pro Pro Val Gly Ile Asp Leu Leu Gly Lys Met Leu Thr Phe
260 265 270
Asp Pro Ala Lys Arg Val Ser Ala Arg Asp Ala Leu Ser His Pro Phe
275 280 285
Phe Gly Asp Ile His Ala His Gly His Ala His Pro Ala Gln Ile Glu
290 295 300
Pro Gly Met Thr Ala Gly Gly Arg Gly Gly Ile Pro Pro Tyr Ser Ile
305 310 315 320
His His Pro Pro His Gln His His Ile His Gln Gln Pro Ser Gln Gln
325 330 335
Gln His Phe Gln His Gly Pro Met His Ser Val His Pro Gln Gly Ser
340 345 350
His His Pro Thr Gln Ala His Gly Ala Ser Leu Ala Leu Gly Pro Pro
355 360 365
His Ala Gly His Pro Ala Gln Gln Pro Ser Ser Met Ala Pro Ala Ala
370 375 380
Val Gly Asp Val Gly Leu Ala Gly Trp Glu Ala Arg Gly
385 390 395
<210> 53
<211> 334
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> CDKA1-864多肽
<400> 53
Met Asp Arg Phe Thr Asn Ile Gln Gln Ile Gly Glu Gly Thr Tyr Gly
1 5 10 15
Ile Val Tyr Lys Gly Tyr Met Lys Gly Pro Gly Ser Ala Thr Thr Ser
20 25 30
Ser Asn Gly Glu Leu Ile Ala Leu Lys Gln Ile Arg Leu Ala Asp Glu
35 40 45
Asp Glu Gly Val Pro Ser Thr Ala Ile Arg Glu Ile Ser Leu Leu Lys
50 55 60
Glu Leu Ser His Pro Asn Val Val Thr Leu Lys Asp Val Ile Tyr Ala
65 70 75 80
Asp Asn Arg Leu Tyr Leu Val Phe Glu Tyr Leu Asp Gln Asp Leu Lys
85 90 95
Arg Tyr Met Asp Gly Cys Lys Thr Gly Leu Asp Ser Thr Leu Val Lys
100 105 110
Ser Tyr Leu His Gln Met Ile Gln Gly Val Ala Phe Cys His Ser His
115 120 125
Arg Val Leu His Arg Asp Leu Lys Pro Gln Asn Leu Leu Ile Asp Arg
130 135 140
Gln Gly Arg Leu Lys Leu Ala Asp Phe Gly Leu Ala Arg Ala Phe Asn
145 150 155 160
Val Pro Leu Arg Gln Tyr Thr Arg Glu Val Val Thr Leu Trp Tyr Arg
165 170 175
Ala Pro Glu Ile Leu Leu Gly Ala Glu His Tyr Ser Thr Pro Val Asp
180 185 190
Thr Trp Ser Ile Gly Cys Ile Phe Ala Glu Met Leu Asn Lys Glu Pro
195 200 205
Leu Phe Pro Gly Asp Ser Glu Ile Asp Glu Leu Phe Arg Ile Phe Arg
210 215 220
Val Leu Gly Thr Pro Asn Asp Glu Ile Trp Pro Lys Val Thr Glu Leu
225 230 235 240
Pro Asn Tyr Lys Thr Gln Phe Pro Lys Trp Lys Arg Gln Ser Leu Asp
245 250 255
Arg Asn Val Ser Arg Leu Cys Ala Asp Gly Leu Asp Leu Leu Ser Val
260 265 270
Cys Ala Thr Ser Lys Lys Leu Ser Trp Arg Ala Lys Glu Val Val Ile
275 280 285
Asn Thr Leu Pro His Phe Phe Leu Ser Tyr Leu Pro Lys Arg Leu Leu
290 295 300
Thr Tyr Glu Pro Thr Ala Arg Ile Thr Cys Arg Glu Ala Gln Asp His
305 310 315 320
Ala Tyr Phe Ala Gly Leu Gly Ala Cys Lys Asp Arg Met Val
325 330
<210> 54
<211> 303
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> CDKA1-9049多肽
<400> 54
Met His Ile Tyr Leu Arg Tyr Gln Lys Ile Glu Lys Asn Gly Gly Gly
1 5 10 15
Asn Leu Gly Glu Gly Thr Tyr Gly Val Val Tyr Lys Ala Arg Asp Lys
20 25 30
Gln Thr Asn Asp Ile Val Ala Leu Lys Arg Ile Arg Leu Glu Met Glu
35 40 45
Asp Glu Gly Ile Pro Ser Thr Ala Leu Arg Glu Ile Ser Leu Leu Gln
50 55 60
Glu Leu Arg His Pro Cys Ile Val Glu Leu Leu Asp Cys Val Gln Ser
65 70 75 80
Glu Gly Arg Leu Tyr Leu Val Phe Glu Phe Val Asp Arg Asp Leu Lys
85 90 95
Lys Tyr Met Glu Ala Gln Pro Gly Thr Leu Ser Arg Gly Val Val Lys
100 105 110
Ser Phe Leu Phe Gln Ile Phe His Gly Leu Ala Phe Cys His Ala Arg
115 120 125
Gly Ile Met His Arg Asp Leu Lys Pro Gln Asn Leu Leu Val Ser Lys
130 135 140
Glu Gly Arg Leu Lys Ile Ala Asp Phe Gly Leu Ala Arg Ala Phe Val
145 150 155 160
Pro Pro Ile Arg Pro Leu Thr His Glu Val Val Thr Leu Trp Tyr Arg
165 170 175
Pro Pro Glu Ile Leu Leu Gly Ser Gln Thr Tyr Ala Pro Pro Val Asp
180 185 190
Val Trp Ala Cys Gly Ala Ile Phe Val Glu Leu Leu Cys Lys Arg Ala
195 200 205
Met Phe Gln Gly Asp Ser Glu Val Asp Gln Leu Phe Lys Ile Phe Arg
210 215 220
Ser Leu Gly Thr Pro Ser Glu Glu Thr Trp Pro Gly Val Thr Ala Leu
225 230 235 240
Gln Asp Trp Asn Pro Ala Phe Pro Val Trp Pro Pro Val Lys Leu Thr
245 250 255
Lys Tyr Cys Pro Ser Ile Asp Glu Ala Gly Leu Asp Leu Leu Glu Lys
260 265 270
Leu Val Val Leu Asp Pro Lys Ser Arg Ile Ser Ala Lys Thr Ala Leu
275 280 285
Tyr His Arg Tyr Phe Asp Asp Leu Asp Lys Gln Gln Phe Arg Gln
290 295 300
<210> 55
<211> 528
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> CDKA1-8325多肽
<400> 55
Met Thr Ser Ile Cys Glu Glu Cys Glu Glu Met Pro Ala Ala Met Arg
1 5 10 15
Cys Glu Asp Cys Asp Leu Ile Tyr Cys Leu Pro Cys Ala Lys His Phe
20 25 30
His Ala Lys Gly Asn Arg Ala Lys His Val Leu Asp Phe Phe Pro Ser
35 40 45
Gln Asn Cys Gly Asn Leu Ala Gln Asp Asp Gly Thr Glu Ser Met Leu
50 55 60
Ser Thr Ser Ser Ser Leu Thr Arg Arg Arg Ser Gly Ser Gly Ala Leu
65 70 75 80
Leu Gly Pro Pro Leu Arg Gln Thr Thr Pro Gln Ser Gln Gln Lys Arg
85 90 95
Arg Ser Arg Leu Pro Leu Gly Ile Pro Thr Ala Asn Ile Met Ala Glu
100 105 110
Asn Ala Thr Thr Gln Thr Ala Val Val Lys Ser Val Thr Gly Pro Gln
115 120 125
Tyr Pro Gln Thr Thr Val Leu Gly Val Asn Gln Asn His Asn Gly Gly
130 135 140
Ser Gly Arg Arg Ser Ser Thr Leu Lys Thr Pro Thr Thr Thr Ala Thr
145 150 155 160
Glu Ser His Ala Cys Arg Met Ser Gly Ser Asn Leu Gly Gln Gly Pro
165 170 175
Asp Ser Ser Ile Ser Ser Met Lys Trp Ser Ala Ser Asp Phe Val Val
180 185 190
Gly Arg Pro Leu Gly Arg Gly Lys Phe Gly Asn Val Tyr Leu Ala Arg
195 200 205
Glu Ser Arg Thr Ser Lys Ser Val Ala Leu Lys Val Ile Phe Lys Asn
210 215 220
Ser Leu Thr Gly Gly Lys Ser Phe Asn Leu Leu Arg Arg Glu Val Glu
225 230 235 240
Ile Gln Ile Arg Leu Arg His Pro His Ile Leu Arg Leu Tyr Gly Tyr
245 250 255
Phe His Asp Pro Lys Ser Cys Phe Leu Val Leu Glu Trp Ala Ser Gly
260 265 270
Gly Glu Leu Tyr Lys His Leu Lys Ala Leu Pro Glu Asn Arg Leu Glu
275 280 285
Glu Ser Leu Ala Ala Gln Tyr Met Arg Gln Val Ala Leu Ala Val Gln
290 295 300
Tyr Leu His Ala Cys His Val Ile His Arg Asp Ile Lys Pro Glu Asn
305 310 315 320
Leu Leu Leu Ala Gly Asp Leu Pro Pro Ser Ala Ala Pro Ala Thr Ala
325 330 335
Gly Ser His Gly Arg Arg Ser Ser Gly Ser Thr Gly Val Gly Val Ala
340 345 350
Gly Val Leu Pro Ala Asp His Val Leu Lys Leu Cys Asp Phe Gly Trp
355 360 365
Ala Val His Ala Pro Pro Pro Asp Gln His Trp Arg Gln Thr Leu Cys
370 375 380
Gly Thr Ala Glu Tyr Leu Ser Pro Glu Met Val Ala Gly Lys Pro Tyr
385 390 395 400
Asp Tyr Thr Val Asp Val Trp Ala Leu Gly Ile Leu Ala Tyr Glu Leu
405 410 415
Leu Gln Gly Lys Thr Pro Phe Tyr Val Pro Glu Gly Glu Asn Arg Gly
420 425 430
Ala Val Ser Met Asp Arg Gly Glu Gly Arg Glu Gly Gly Gly Gly Gly
435 440 445
Lys Gly Val Gly Val Ala Ala Ala Ala Val Ala Ala Ala Thr Glu Asp
450 455 460
Gly Asp Met Lys Lys Gly Arg Glu Glu Val Tyr Ala Arg Ile Ala Ala
465 470 475 480
Tyr Glu Gly Ala Leu Thr Phe Pro His Pro Val Ser Val Glu Ala Arg
485 490 495
Ser Phe Val Asn Ala Leu Leu Val Pro Asp Pro Gln Asp Arg Val Ser
500 505 510
Leu Asp Ala Val Leu Arg His Pro Trp Leu Met Glu Glu Glu Asp Val
515 520 525
<210> 56
<211> 5457
<212> DNA
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> 菌株GE-5877中破坏的CHORD-3266基因座
<400> 56
atgaaactct atgtgcacta cgaggaggct ggtcaggatg aaaaggcatt gacgcttaag 60
ttgactctgc ccaaaagctg ggcggagcag ccgttgcttc aagtactgga gctgttcatc 120
gaatcctaca acaagaaaaa gaccggtcta cctcccttgg acaaagactt tgtccacatg 180
gaaaaagctg ggtaagtcct tactcgtgac agcgttccct ttctccagac tagacgccta 240
atagtgttct aatgtaccac tgggacacgc ctcgctgcct gtgcaccatg ctccatactc 300
aacgctgcta caggggcgta atccttccag tcggcaacat tgtgagcgac atgttgagcg 360
atagagatga tttgtatatc agatccgggc cagggcctgc tcgtgggaag attgcccatc 420
tcagttcgcc cccaaacgcg cacgcttcga gtgagtcgag cacaggattg ttgcgctgca 480
aaaactatgg atgcaatcag tcattttcgg aagaaaacaa ttcagaagag gcgtgccgct 540
ttcacaaggc accccccgtc tttcatgata cgaagaaagg gtggtcgtgc tgcgcgaagc 600
gagtatatga ctgggacgag ttccatacgg taagcgtgga agtgttcgtt ctcggcccca 660
ggactttgtt ttgaggcaat tggtgtactt taattggcgg ataaagggag gactcacaac 720
tttcgatatt caccgtctcc agatcgaggg gtgcaccaca ggacggcaca gtctcatcga 780
tccgaaggaa attttcgcgc cgtcccccac cctggctgca gccgcgcagg ccgagagggg 840
agattgcagc aatacgtcaa gcgctgctac agtcatcaag agcattgatg aattcaatca 900
gtcgaatcca aatgccgccg ctgcatgcaa aacagcagcc tcgatgacgc tggcgggcac 960
gcgctgcacc gtcaaaccgg acgggtctgc cacctgtttg aacaaaggct gccaaaagga 1020
ctacttgctc aaggagaatc acccctctgc atgtcggtaa ggacaccgcg ctcgatggat 1080
atcataatac gatgggtatt atgggtctgg cgtaatagcg aagaggcccg caccgatcgc 1140
ccttcccaac agttgcgcag cctgaatggc gaatggcgcc tgatgcggta ttttctcctt 1200
acgcatctgt gcggtatttc acaccgcata tggtgcactc tcagtacaat ctgctctgat 1260
gccgcatagt taagccagcc ccgacacccg ccaacacccg ctgacgcgcc ctgacgggct 1320
tgtctgctcc cggcatccgc ttacagacaa gctgtgaccg tctccgggag ctgcatgtgt 1380
cagaggtttt caccgtcatc accgaaacgc gcgagacgaa agggcctcgt gatacgccta 1440
tttttatagg ttaatgtcat gataataatg gtttcttaga cgtcaggtgg cacttttcgg 1500
ggaaatgtgc gcggaacccc tatttgttta tttttctaaa tacattcaaa tatgtatccg 1560
ctcatgagac aataaccctg ataaatgctt caataatatt gaaaaaggaa gagtatgagt 1620
attcaacatt tccgtgtcgc ccttattccc ttttttgcgg cattttgcct tcctgttttt 1680
gctcacccag aaacgctggt gaaagtaaaa gatgctgaag atcagttggg tgcacgagtg 1740
ggttacatcg aactggatct caacagcggt aagatccttg agagttttcg ccccgaagaa 1800
cgttttccaa tgatgagcac ttttaaagtt ctgctatgtg gcgcggtatt atcccgtatt 1860
gacgccgggc aagagcaact cggtcgccgc atacactatt ctcagaatga cttggttgag 1920
tactcaccag tcacagaaaa gcatcttacg gatggcatga cagtaagaga attatgcagt 1980
gctgccataa ccatgagtga taacactgcg gccaacttac ttctgacaac gatcggagga 2040
ccgaaggagc taaccgcttt tttgcacaac atgggggatc atgtaactcg ccttgatcgt 2100
tgggaaccgg agctgaatga agccatacca aacgacgagc gtgacaccac gatgcctgta 2160
gcaatggcaa caacgttgcg caaactatta actggcgaac tacttactct agcttcccgg 2220
caacaattaa tagactggat ggaggcggat aaagttgcag gaccacttct gcgctcggcc 2280
cttccggctg gctggtttat tgctgataaa tctggagccg gtgagcgtgg gtctcgcggt 2340
atcattgcag cactggggcc agatggtaag ccctcccgta tcgtagttat ctacacgacg 2400
gggagtcagg caactatgga tgaacgaaat agacagatcg ctgagatagg tgcctcactg 2460
attaagcatt ggtaactgtc agaccaagtt tactcatata tactttagat tgatttaaaa 2520
cttcattttt aatttaaaag gatctaggtg aagatccttt ttgataatct catgaccaaa 2580
atcccttaac gtgagttttc gttccactga gcgtcagacc ccgtagaaaa gatcaaagga 2640
tcttcttgag atcctttttt tctgcgcgta atctgctgct tgcaaacaaa aaaaccaccg 2700
ctaccagcgg tggtttgttt gccggatcaa gagctaccaa ctctttttcc gaaggtaact 2760
ggcttcagca gagcgcagat accaaatact gttcttctag tgtagccgta gttaggccac 2820
cacttcaaga actctgtagc accgcctaca tacctcgctc tgctaatcct gttaccagtg 2880
gctgctgcca gtggcgataa gtcgtgtctt accgggttgg actcaagacg atagttaccg 2940
gataaggcgc agcggtcggg ctgaacgggg ggttcgtgca cacagcccag cttggagcga 3000
acgacctaca ccgaactgag atacctacag cgtgagctat gagaaagcgc cacgcttccc 3060
gaagggagaa aggcggacag gtatccggta agcggcaggg tcggaacagg agagcgcacg 3120
agggagcttc cagggggaaa cgcctggtat ctttatagtc ctgtcgggtt tcgccacctc 3180
tgacttgagc gtcgattttt gtgatgctcg tcaggggggc ggagcctatg gaaaaacgcc 3240
agcaacgcgg cctttttacg gttcctggcc ttttgctggc cttttgctca catgttcttt 3300
cctgcgttat cccctgattc tgtggataac cgtattaccg cctttgagtg agctgatacc 3360
gctcgccgca gccgaacgac cgagcgcagc gagtcagtga gcgaggaagc ggaagagcgc 3420
ccaatacgca aaccgcctct ccccgcgcgt tggccgattc attaatgcag ctggcacgac 3480
aggtttcccg actggaaagc gggcagtgag cgcaacgcaa ttaatgtgag ttagctcact 3540
cattaggcac cccaggcttt acactttatg cttccggctc gtatgttgtg tggaattgtg 3600
agcggataac aatttcacac aggaaacagc tatgaccatg attacgccaa gcttgcatgc 3660
ctgcaggtcg actctagagc gtgcaggtgt acagattgaa ggaaacaatg gagatatctt 3720
tggcagttga aaaccgtgtt cgaatcatgc tcttctactc tccaactgag acgaaattta 3780
tagcgccatc tcgcttctga ctaccaggct taggaaggcc tcatcacaag ctggatcggt 3840
tcgaattaag caggcactga agccaagctt gcaagacagc caccttttaa ttctctcaaa 3900
acactttctc aattcagccc ggtaaatatg ccgattcaca gcggccaaga tagaggggag 3960
gttagcaaga atgttgcgat ccctccccag tcgttgcctc gcacacaacc taggacttca 4020
cctttccatg gaaaattgag aagtgaatat tggttttctt acggcatatc agatgaaatc 4080
atgaccccta aacatgaaga gctgcaggca aaacacctgc tctggacgag cacgatgaaa 4140
tctcgagaac ccgccgtact tcagttgatc ccgcatgatg acggccgcca ttgaaataag 4200
ccacctcact ttattctagc accgatttcc accgttgtga gggccgaacg aggacaattt 4260
cgtgcgaaac aagcacgaac gcgcacacga ttagtaggac agacgagcag atcgatggca 4320
tgcggcacgg tctcgcgttc tcggcgacca ggacaacgga gcagagggag gcctgccgag 4380
ttccgagggg cattttagtc ccaaaattgt gttgacacgt gaacaagtgg cttgaaaaga 4440
ggaaggaaat gcctgggttt cccttcgaga gcgggaactc gcttgtgcgt catcctagct 4500
acccatggtc cctttgtggg ggaggctgtt tcgtcctacc gaatgtgtgg cgctccatgc 4560
atcttctgcc tcccaaacca ccaacatgag cacgcgaagg aaggagaaaa aagtggccgc 4620
aacgttctct tctcatattt attgtctcat cacaaacata ggtacataat acaacaatca 4680
tggatccccg ggtaccgagc tcgatggcca agcctttgtc ccaagaggaa tccacgctga 4740
tcgaacgtgc aactgcgacc atcaacagca tacctattag cgaggactac tcggtggcca 4800
gtgcagccct ctcgtccgac ggtcggatct ttaccggcgt gaatgtatat catttcaccg 4860
gagggccatg cgcggagctc gtggtcctcg gaacggccgc tgcggctgct gccggaaatc 4920
tgacgtgcat agtggccatc gggaacgaaa accgcggcat tctgtctccg tgcgggcgat 4980
gtcggcaggt gctgcttgac ttgcacccgg ggatcaaggc aattgtcaaa gattccgatg 5040
ggcagcccac agcggttggc atcagggagt tgcttccctc tggctacgtc tgggagggtt 5100
gaaattcact ggccgtcgtt ttacaacgtc gtgactggga aaaccctggc gttacccaac 5160
ttaatcgcct tgcagcacat ccccctttcg ccagtggcaa acagctataa tcgtgagctt 5220
tacgttccca cgccaacact tcgccatttc tcctcccttc ctttctttag ctaccacgca 5280
gccggccccg tcttccacga cgcgggtaaa tactggtcat gttgccctgg aacggtcaag 5340
tacgacttcg acgactttct caagatccct ggatgcatgc tcagtagtca ttacgacgga 5400
agccaggaga gcctggaggc gttcactaga cacgccaaaa cgtctgaggg cacatga 5457
<210> 57
<211> 4118
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<220>
<221> misc_feature
<223> 插入诱变中所使用的载体
<400> 57
gcgcccaata cgcaaaccgc ctctccccgc gcgttggccg attcattaat gcagctggca 60
cgacaggttt cccgactgga aagcgggcag tgagcgcaac gcaattaatg tgagttagct 120
cactcattag gcaccccagg ctttacactt tatgcttccg gctcgtatgt tgtgtggaat 180
tgtgagcgga taacaatttc acacaggaaa cagctatgac catgattacg ccaagcttgc 240
atgcctgcag gtcgactcta gagcgtgcag gtgtacagat tgaaggaaac aatggagata 300
tctttggcag ttgaaaaccg tgttcgaatc atgctcttct actctccaac tgagacgaaa 360
tttatagcgc catctcgctt ctgactacca ggcttaggaa ggcctcatca caagctggat 420
cggttcgaat taagcaggca ctgaagccaa gcttgcaaga cagccacctt ttaattctct 480
caaaacactt tctcaattca gcccggtaaa tatgccgatt cacagcggcc aagatagagg 540
ggaggttagc aagaatgttg cgatccctcc ccagtcgttg cctcgcacac aacctaggac 600
ttcacctttc catggaaaat tgagaagtga atattggttt tcttacggca tatcagatga 660
aatcatgacc cctaaacatg aagagctgca ggcaaaacac ctgctctgga cgagcacgat 720
gaaatctcga gaacccgccg tacttcagtt gatcccgcat gatgacggcc gccattgaaa 780
taagccacct cactttattc tagcaccgat ttccaccgtt gtgagggccg aacgaggaca 840
atttcgtgcg aaacaagcac gaacgcgcac acgattagta ggacagacga gcagatcgat 900
ggcatgcggc acggtctcgc gttctcggcg accaggacaa cggagcagag ggaggcctgc 960
cgagttccga ggggcatttt agtcccaaaa ttgtgttgac acgtgaacaa gtggcttgaa 1020
aagaggaagg aaatgcctgg gtttcccttc gagagcggga actcgcttgt gcgtcatcct 1080
agctacccat ggtccctttg tgggggaggc tgtttcgtcc taccgaatgt gtggcgctcc 1140
atgcatcttc tgcctcccaa accaccaaca tgagcacgcg aaggaaggag aaaaaagtgg 1200
ccgcaacgtt ctcttctcat atttattgtc tcatcacaaa cataggtaca taatacaaca 1260
atcatggatc cccgggtacc gagctcgatg gccaagcctt tgtcccaaga ggaatccacg 1320
ctgatcgaac gtgcaactgc gaccatcaac agcataccta ttagcgagga ctactcggtg 1380
gccagtgcag ccctctcgtc cgacggtcgg atctttaccg gcgtgaatgt atatcatttc 1440
accggagggc catgcgcgga gctcgtggtc ctcggaacgg ccgctgcggc tgctgccgga 1500
aatctgacgt gcatagtggc catcgggaac gaaaaccgcg gcattctgtc tccgtgcggg 1560
cgatgtcggc aggtgctgct tgacttgcac ccggggatca aggcaattgt caaagattcc 1620
gatgggcagc ccacagcggt tggcatcagg gagttgcttc cctctggcta cgtctgggag 1680
ggttgaaatt cactggccgt cgttttacaa cgtcgtgact gggaaaaccc tggcgttacc 1740
caacttaatc gccttgcagc acatccccct ttcgccagac ccataatacc cataatagct 1800
gtttgccact ggcgtaatag cgaagaggcc cgcaccgatc gcccttccca acagttgcgc 1860
agcctgaatg gcgaatggcg cctgatgcgg tattttctcc ttacgcatct gtgcggtatt 1920
tcacaccgca tatggtgcac tctcagtaca atctgctctg atgccgcata gttaagccag 1980
ccccgacacc cgccaacacc cgctgacgcg ccctgacggg cttgtctgct cccggcatcc 2040
gcttacagac aagctgtgac cgtctccggg agctgcatgt gtcagaggtt ttcaccgtca 2100
tcaccgaaac gcgcgagacg aaagggcctc gtgatacgcc tatttttata ggttaatgtc 2160
atgataataa tggtttctta gacgtcaggt ggcacttttc ggggaaatgt gcgcggaacc 2220
cctatttgtt tatttttcta aatacattca aatatgtatc cgctcatgag acaataaccc 2280
tgataaatgc ttcaataata ttgaaaaagg aagagtatga gtattcaaca tttccgtgtc 2340
gcccttattc ccttttttgc ggcattttgc cttcctgttt ttgctcaccc agaaacgctg 2400
gtgaaagtaa aagatgctga agatcagttg ggtgcacgag tgggttacat cgaactggat 2460
ctcaacagcg gtaagatcct tgagagtttt cgccccgaag aacgttttcc aatgatgagc 2520
acttttaaag ttctgctatg tggcgcggta ttatcccgta ttgacgccgg gcaagagcaa 2580
ctcggtcgcc gcatacacta ttctcagaat gacttggttg agtactcacc agtcacagaa 2640
aagcatctta cggatggcat gacagtaaga gaattatgca gtgctgccat aaccatgagt 2700
gataacactg cggccaactt acttctgaca acgatcggag gaccgaagga gctaaccgct 2760
tttttgcaca acatggggga tcatgtaact cgccttgatc gttgggaacc ggagctgaat 2820
gaagccatac caaacgacga gcgtgacacc acgatgcctg tagcaatggc aacaacgttg 2880
cgcaaactat taactggcga actacttact ctagcttccc ggcaacaatt aatagactgg 2940
atggaggcgg ataaagttgc aggaccactt ctgcgctcgg cccttccggc tggctggttt 3000
attgctgata aatctggagc cggtgagcgt gggtctcgcg gtatcattgc agcactgggg 3060
ccagatggta agccctcccg tatcgtagtt atctacacga cggggagtca ggcaactatg 3120
gatgaacgaa atagacagat cgctgagata ggtgcctcac tgattaagca ttggtaactg 3180
tcagaccaag tttactcata tatactttag attgatttaa aacttcattt ttaatttaaa 3240
aggatctagg tgaagatcct ttttgataat ctcatgacca aaatccctta acgtgagttt 3300
tcgttccact gagcgtcaga ccccgtagaa aagatcaaag gatcttcttg agatcctttt 3360
tttctgcgcg taatctgctg cttgcaaaca aaaaaaccac cgctaccagc ggtggtttgt 3420
ttgccggatc aagagctacc aactcttttt ccgaaggtaa ctggcttcag cagagcgcag 3480
ataccaaata ctgttcttct agtgtagccg tagttaggcc accacttcaa gaactctgta 3540
gcaccgccta catacctcgc tctgctaatc ctgttaccag tggctgctgc cagtggcgat 3600
aagtcgtgtc ttaccgggtt ggactcaaga cgatagttac cggataaggc gcagcggtcg 3660
ggctgaacgg ggggttcgtg cacacagccc agcttggagc gaacgaccta caccgaactg 3720
agatacctac agcgtgagct atgagaaagc gccacgcttc ccgaagggag aaaggcggac 3780
aggtatccgg taagcggcag ggtcggaaca ggagagcgca cgagggagct tccaggggga 3840
aacgcctggt atctttatag tcctgtcggg tttcgccacc tctgacttga gcgtcgattt 3900
ttgtgatgct cgtcaggggg gcggagccta tggaaaaacg ccagcaacgc ggccttttta 3960
cggttcctgg ccttttgctg gccttttgct cacatgttct ttcctgcgtt atcccctgat 4020
tctgtggata accgtattac cgcctttgag tgagctgata ccgctcgccg cagccgaacg 4080
accgagcgca gcgagtcagt gagcgaggaa gcggaaga 4118
<210> 58
<211> 28
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<220>
<221> misc_feature
<223> CHORD插入侧翼的PCR引物
<400> 58
gtcaagcgct gctacagtca tcaagagc 28
<210> 59
<211> 29
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<220>
<221> misc_feature
<223> CHORD插入侧翼的PCR引物
<400> 59
cagggatctt gagaaagtcg tcgaagtcg 29
<210> 60
<211> 22
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<220>
<221> misc_feature
<223> qRT引物CHORD外显子2
<400> 60
ggatgcaatc agtcattttc gg 22
<210> 61
<211> 21
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<220>
<221> misc_feature
<223> qRT引物CHORD外显子2
<400> 61
cccagtcata tactcgcttc g 21
<210> 62
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<220>
<221> misc_feature
<223> qRT引物CHORD外显子4
<400> 62
atactggtca tgttgccctg 20
<210> 63
<211> 21
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<220>
<221> misc_feature
<223> qRT CHORD外显子4
<400> 63
gttttggcgt gtctagtgaa c 21
<210> 64
<211> 7822
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<220>
<221> misc_feature
<223> CHORD外显子4融合表达载体
<400> 64
gcggccgccg tatggtcgac ggttgctcgg atgggggggg cggggagcga tggagggagg 60
aagatcaggt aaggtctcga cagactagag aagcacgagt gcaggtataa gaaacagcaa 120
aaaaaagtaa tgggcccagg cctggagagg gtatttgtct tgtttttctt tggccaggaa 180
cttgttctcc tttcttcgtt tctaggaccc cgatccccgc tcgcatttct ctcttcctca 240
gccgaagcgc agcggtaaag catccatttt atcccaccga aagggcgctc ccagccttcg 300
tcgagcggaa ccggggttac agtgcctcac tcctttgcac gcggtcgggt gctcggccta 360
cggttgcccg agtccgcaag cacctcaaca cagccgtctg tccacaccgc agccgaccgg 420
cgtgcgattt gggtccgacc caccgtccca gccccgctgc ggactatcgc gtcgcagcgg 480
ccctgcgccc acgcggcgtc gtcgaagttg ccgtcgacga gagactggta aagctgatcg 540
agtccgatac gcaacatata ggcgcggagt cgtggggagc cggccagctc cgggtgcctc 600
cgttcaaagt agcgtgtctg ctgctccatg cacgccaacc agggacgcca gaagaatatg 660
ttcgccactt cgtattggct atcaccaaac atcgcttcgg accagtcgat gacagcagta 720
atccgaccat tgtctgtaag tacgttattg ctgccgaaat ccgcgtgcac caggtgcctg 780
acctcagggc aatcctcggc ccacaacatg agttcgtcca gtgcttgggc cacggatgca 840
gacacggtgt catccatgac tgtctgccaa tgatagacgt gaggatcggc aatggcgcag 900
atgaagtctc gccaggtcgt gtactgcccg atgccctggg gcccaaaagg tccaaagccg 960
gacgtctgag acagatctgc ggcagcgatc gcgtccatgg cctcggccac gggttgcaaa 1020
acggcaggca attcagtttc gggcagatct tgcaacgtca ctccctgggc tcggcgcgag 1080
atgcagtacg tgagagattc gctaaactcc ccaatgtcca gtacctctgg tatggggaga 1140
gcggcggagg cgaaatgacg gtagacatac cgatccttgt agaacccgtc cgcacaacta 1200
ttaaccctca acacgtatcc ccgaccccct acgtcaaacg agaacgccct actctcctct 1260
ccctcgctca gttgcatcaa gtcggagaca gagtcgaact tctcaataag gaatttctcc 1320
acggacgtag cggtcagttc cggtttcttc cccatcgagc tcggtacccg gggatccatg 1380
attgttgtat tatgtaccta tgtttgtgat gagacaataa atatgagaag agaacgttgc 1440
ggccactttt ttctccttcc ttcgcgtgct catgttggtg gtttgggagg cagaagatgc 1500
atggagcgcc acacattcgg taggacgaaa cagcctcccc cacaaaggga ccatgggtag 1560
ctaggatgac gcacaagcga gttcccgctc tcgaagggaa acccaggcat ttccttcctc 1620
ttttcaagcc acttgttcac gtgtcaacac aattttggac taaaatgccc ctcggaactc 1680
ggcaggcctc cctctgctcc gttgtcctgg tcgccgagaa cgcgagaccg tgccgcatgc 1740
catcgatctg ctcgtctgta ctactaatcg tgtgcgtgtt cgtgcttgtt tcgcacgaaa 1800
ttgtcctcgt tcggccctca caacggtgga aatcggtgct agaataaagt gaggtggctt 1860
atttcaatgg cggccgtcat catgcgggat caactgaagt acggcgggtt ctcgagattt 1920
catcgtgctc gtccagagca ggtgttttgc ctgcagctct tcatgtttag gggtcatgat 1980
ttcatctgat atgccgtaag aaaaccaata ttcacttctc aattttccat ggaaaggtga 2040
aggcctaggt tgtgtgcgag gcaacgactg gggagggatc gcaacattct tgctaacctc 2100
ccctctatct tggccgctgt gaatcggcat atttaccggg ctgaattgag aaagtgtttt 2160
gagggaatta aaaggtggct gtcttgcaag cttggcttca gtgcctgctt aattcgaacc 2220
gatccagctt gtgatgaggc cttcctaagc ctggtagtca gaagcgacat ggcgctataa 2280
atttcgtctc agttggagag tagaaaagca tgattcgaac acggttttca actgccaaag 2340
atatctccat tgtttccttc aatctgtaca cctgcacggt gcaccagttg gtacggcata 2400
ttatggttta aagttgaaaa tgctaacagt gaagtgatat ccttttttaa tggagtgttg 2460
aggtgaagtc tagcatcgta ggggaaaaca ggattctgtg tcttccattc tactccttga 2520
taaagcgaag aaatccgaca aaaccaaaga gattgttcaa gtttaagatt tgtaagcgta 2580
caactatgaa cttcttctct ttgtaggcct gagtggtcgt atgcatacga ttcatgaagt 2640
gaatcagtat cgctggattt tgcttaggag taaagcacaa ctaagaaaat atgctgcctg 2700
gcaggcatcc tgagacatga ggcaagcgac gtagcaattg aatcctaatt taagccaggg 2760
catctgtatg actctgttag ttaattgatg aaccaatgag ctttaaaaaa aaatcgttgc 2820
gcgtaatgta gttttaattc tccgccttga ggtgcggggc catttcggac aaggttcttt 2880
ggacggagat ggcagcatgt gtcccttctc caaattggtc cgtgtggtag ttgagatgct 2940
gccttaaaat tctgctcggt catcctgcct tcgcattcac tcctttcgag ctgtcgggtt 3000
cctcacgagg cctccgggag cggattgcgc agaaaggcga cccggagaca cagagaccat 3060
acaccgacta aattgcactg gacgatacgg catggcgacg acgatggcca agcattgcta 3120
cgtgattatt cgccttgtca ttcagggaga aatgatgaca tgtgtgggac ggtctttata 3180
tgggaagagg gcatgaaaat aacatggcct ggcgggatgg agcgtcacac ctgtgtatgc 3240
gttcgatcca caagcaactc accatttgcg tcggggcctg tctccaatct gctttaggct 3300
acttttctct aatttagcct attctataca gacagagaca cacagggatc taatgggcag 3360
cccacagcgg ttggcatcag ggagttgctt ccctctggct acgtctggga gggttgaaat 3420
tcactggccg tcgttttaca acgtcgtaac tgggaaaacc ctggcgttac ccaacttaat 3480
cgccttgcag cacatccccc tttcgccagt ggcaaacagc tataatcgtg agctttacgt 3540
tcccacgcca acacttcgcc atttctcctc ccttcctttc tttagctacc acgcagccgg 3600
ccccgtcttc cacgacgcgg gtaaatactg gtcatgttgc cctggaacgg tcaagtacga 3660
cttcgacgac tttctcaaga tccctggatg catgctcagt agtcattacg acggaagcca 3720
ggagagcctg gaggcgttca ctagacacgc caaaacgtct gagggcacat gacgtagagt 3780
caagggggaa ggtgcatagt gtgcaacaac agcattaacg tcaaagaaaa ctgcacgttc 3840
aagcccgcgt gaacctgccg gtcttctgat cgcctacata tagcagatac tagttgtact 3900
tttttttcca aagggaacat tcatgtatca atttgaaata aacatctatc ctccagatca 3960
ccagggccag tgaggccggc ataaaggacg gcaaggaaag aaaagaaaga aagaaaagga 4020
cacttatagc atagtttgaa gttataagta gtcgcaatct gtgtgcagcc gacagatgct 4080
ttttttttcc gtttggcagg aggtgtaggg atgtcgaaga ccagtccagc tagtatctat 4140
cctacaagtc aatcatgctg cgacaaaaat ttctcgcacg aggcctctcg ataaacaaaa 4200
ctttaaaagc acacttcatt gtcatgcaga gtaataactc ttccgcgtcg atcaatttat 4260
caatctctat catttccgcc cctttccttg catagagcaa gaaaagcgac ccggatgagg 4320
ataacatgtc ctgcgccagt agtgtggcat tgcctgtctc tcatttacac gtactgaaag 4380
cataatgcac gcgcatacca atatttttcg tgtacggaga tgaagagacg cgacacgtaa 4440
gatcacgaga aggcgagcac ggttgccaat ggcagacgcg ctagtctcca ttatcgcgtt 4500
gttcggtagc ttgctgcatg tcttcagtgg cactatatcc actctgcctc gtcttctaca 4560
cgagggccac atcggtgcaa gttcgaaaaa tcatatctca atcttcagat cctttccaga 4620
aacggtgctc aggcgggaaa gtgaaggttt tctactctag tggctacccc aattctctcc 4680
gactgtcgca gacggtcctt cgttgcgcac gcaccgcgca ctacctctga aattcgacaa 4740
ccgaagttca attttacatc taacttcttt cccattctct caccaaaagc ctagcttaca 4800
tgttggagag cgacgagagc ggcctgcccg ccatggagat cgagtgccgc atcaccggca 4860
ccctgaacgg cgtggagttc gagctggtgg gcggcggaga gggcaccccc gagcagggcc 4920
gcatgaccaa caagatgaag agcaccaaag gcgccctgac cttcagcccc tacctgctga 4980
gccacgtgat gggctacggc ttctaccact tcggcaccta ccccagcggc tacgagaacc 5040
ccttcctgca cgccatcaac aacggcggct acaccaacac ccgcatcgag aagtacgagg 5100
acggcggcgt gctgcacgtg agcttcagct accgctacga ggccggccgc gtgatcggcg 5160
acttcaaggt gatgggcacc ggcttccccg aggacagcgt gatcttcacc gacaagatca 5220
tccgcagcaa cgccaccgtg gagcacctgc accccatggg cgataacgat ctggatggca 5280
gcttcacccg caccttcagc ctgcgcgacg gcggctacta cagctccgtg gtggacagcc 5340
acatgcactt caagagcgcc atccacccca gcatcctgca gaacgggggc cccatgttcg 5400
ccttccgccg cgtggaggag gatcacagca acaccgagct gggcatcgtg gagtaccagc 5460
acgccttcaa gaccccggat gcagatgccg gtgaagaata agggtgggaa ggagtcgggg 5520
agggtcctgg cagagcggcg tcctcatgat gtgttggaga cctggagagt cgagagcttc 5580
ctcgtcacct gattgtcatg tgtgtatagg ttaagggggc ccactcaaag ccataaagac 5640
gaacacaaac actaatctca acaaagtcta ctagcatgcc gtctgtccat ctttatttcc 5700
tggcgcgcct atgcttgtaa accgttttgt gaaaaaattt ttaaaataaa aaaggggacc 5760
tctagggtcc ccaattaatt agtaatataa tctattaaag gtcattcaaa aggtcatcca 5820
gacgaaaggg cctcgtgata cgcctatttt tataggttaa tgtcatgata ataatggttt 5880
cttagacgtc aggtggcact tttcggggaa atgtgcgcgg aacccctatt tgtttatttt 5940
tctaaataca ttcaaatatg tatccgctca tgagacaata accctgataa atgcttcaat 6000
aatattgaaa aaggaagagt atgagtattc aacatttccg tgtcgccctt attccctttt 6060
ttgcggcatt ttgccttcct gtttttgctc acccagaaac gctggtgaaa gtaaaagatg 6120
ctgaagatca gttgggtgca cgagtgggtt acatcgaact ggatctcaac agcggtaaga 6180
tccttgagag ttttcgcccc gaagaacgtt ttccaatgat gagcactttt aaagttctgc 6240
tatgtggcgc ggtattatcc cgtattgacg ccgggcaaga gcaactcggt cgccgcatac 6300
actattctca gaatgacttg gttgagtact caccagtcac agaaaagcat cttacggatg 6360
gcatgacagt aagagaatta tgcagtgctg ccataaccat gagtgataac actgcggcca 6420
acttacttct gacaacgatc ggaggaccga aggagctaac cgcttttttg cacaacatgg 6480
gggatcatgt aactcgcctt gatcgttggg aaccggagct gaatgaagcc ataccaaacg 6540
acgagcgtga caccacgatg cctgtagcaa tggcaacaac gttgcgcaaa ctattaactg 6600
gcgaactact tactctagct tcccggcaac aattaataga ctggatggag gcggataaag 6660
ttgcaggacc acttctgcgc tcggcccttc cggctggctg gtttattgct gataaatctg 6720
gagccggtga gcgtgggtct cgcggtatca ttgcagcact ggggccagat ggtaagccct 6780
cccgtatcgt agttatctac acgacgggga gtcaggcaac tatggatgaa cgaaatagac 6840
agatcgctga gataggtgcc tcactgatta agcattggta actgtcagac caagtttact 6900
catatatact ttagattgat ttaaaacttc atttttaatt taaaaggatc taggtgaaga 6960
tcctttttga taatctcatg accaaaatcc cttaacgtga gttttcgttc cactgagcgt 7020
cagaccccgt agaaaagatc aaaggatctt cttgagatcc tttttttctg cgcgtaatct 7080
gctgcttgca aacaaaaaaa ccaccgctac cagcggtggt ttgtttgccg gatcaagagc 7140
taccaactct ttttccgaag gtaactggct tcagcagagc gcagatacca aatactgtcc 7200
ttctagtgta gccgtagtta ggccaccact tcaagaactc tgtagcaccg cctacatacc 7260
tcgctctgct aatcctgtta ccagtggctg ctgccagtgg cgataagtcg tgtcttaccg 7320
ggttggactc aagacgatag ttaccggata aggcgcagcg gtcgggctga acggggggtt 7380
cgtgcacaca gcccagcttg gagcgaacga cctacaccga actgagatac ctacagcgtg 7440
agctatgaga aagcgccacg cttcccgaag ggagaaaggc ggacaggtat ccggtaagcg 7500
gcagggtcgg aacaggagag cgcacgaggg agcttccagg gggaaacgcc tggtatcttt 7560
atagtcctgt cgggtttcgc cacctctgac ttgagcgtcg atttttgtga tgctcgtcag 7620
gggggcggag cctatggaaa aacgccagca acgcggcctt tttacggttc ctggcctttt 7680
gctggccttt tgctcacatg ttctttcctg cgttatcccc tgattctgtg gataaccgta 7740
ttaccgcctt tgagtgagct gataccgctc gccgcagccg aacgaccgag cgcagcgagt 7800
cagtgagcga ggaagcggaa ga 7822
<210> 65
<211> 171
<212> PRT
<213> 小环藻属某种
<220>
<221> misc_feature
<223> SKP1-0244多肽
<400> 65
Met Trp Ala Tyr Asn Gly Ala Asp Thr Gln Ile Ser Arg Glu Gly Val
1 5 10 15
Arg Tyr Gln Val Pro Ser Asn Ala Ala Lys Leu Ser Phe Phe Val Ala
20 25 30
Leu Cys Val Asp Asn Glu Asp Ile Glu Asp Gly Gly Asp Thr Asp Gln
35 40 45
Asn Asn Gly Asn Gly Ser Gly Val Val His Glu Ile Pro Leu His Ala
50 55 60
Ile Asn Ser Val Val Leu Ala Lys Ile Val Lys Tyr Met Glu Tyr Tyr
65 70 75 80
Lys Gln Asp Pro Met Gln Lys Ile Ile Thr Leu Phe Thr Ser Ser Arg
85 90 95
Asn Cys Asn Phe Phe Gln Glu Glu Tyr Ala Ile Val Ile Asp Val Asp
100 105 110
Arg Asn Thr Leu Phe Glu Leu His Ala Ala Ala Asp Phe Leu Gly Ile
115 120 125
Gln Pro Leu Leu Glu Leu Thr Ser Leu Ala Val Leu Leu Ser Tyr Met
130 135 140
Trp Val Gly Val Leu Ile Tyr Leu Thr Leu Asp Gly Ile Val Thr Ser
145 150 155 160
Cys Asn Ser Ile His Pro Ala Leu Leu Asp Asn
165 170
<210> 66
<211> 174
<212> PRT
<213> 四爿藻属某种
<220>
<221> misc_feature
<223> SKP1-4935多肽
<400> 66
Met Ala Ala Pro Ser Gln Pro Ser Leu Gly Ser Asp Val Val Arg Leu
1 5 10 15
Arg Ser Ala Asp Gly Val Thr His Glu Val Asn Arg Glu Ala Leu Thr
20 25 30
Arg Leu Ser Pro Val Leu Glu Gly Leu Leu Gly Glu Cys Gly Ala Glu
35 40 45
Glu Ala Val Pro Leu Pro Ser Val Gln Gly Arg Glu Leu Gly Arg Val
50 55 60
Ala Glu Phe Cys Arg Gln Trp Asp Ala His Glu Ala Ala Ala Arg Gly
65 70 75 80
Ala Asp Gly Ser Gly Ala Ala Ala Ala Ala Ala Ala Ala Lys Glu Arg
85 90 95
Ala Ala Trp Glu Ala Ala Tyr Leu Ala Pro Leu Asn Ala Asp Asp Leu
100 105 110
His Asp Leu Leu Leu Ala Ala Asn Phe Leu His Ala Glu Pro Leu Met
115 120 125
Thr Leu Cys Phe Arg Ala Val Gly Asp Ala Met Arg Gly Lys Ala Pro
130 135 140
Ala Glu Ile Arg Ala His Phe Gly Ile Pro Asn Asp Phe Glu Pro Gly
145 150 155 160
Glu Glu Asp Ala Ile Arg Arg Glu Asn Gln Arg Tyr Phe Ser
165 170
<210> 67
<211> 165
<212> PRT
<213> 普通小球藻
<220>
<221> misc_feature
<223> SKP1-9804多肽
<400> 67
Met Arg Ser Ile Ile Leu Arg Ser Cys Asp Gly Ala Asp His Val Val
1 5 10 15
Ala Gln Glu Ala Ala Cys Leu Ser Lys Thr Val Gln Ser Leu Leu Glu
20 25 30
Glu Leu Glu Glu Ser Thr Leu Val Val Pro Leu Pro Asn Val Cys Asp
35 40 45
Cys Thr Leu Arg Lys Val Leu Gln Tyr Cys Thr Gln His Thr Ala Leu
50 55 60
Gln Arg Arg Val Thr Asp Ile Ser Asp Glu Leu Arg Thr Arg Glu Met
65 70 75 80
Glu Ala Trp Asp Lys Arg Tyr Ile Met Val Ser Thr Asp Glu Leu Tyr
85 90 95
His Leu Val Met Ala Ala His Tyr Leu Asn Val Pro Gly Leu Leu Glu
100 105 110
Leu Cys Cys Glu Gly Ile Ala Asn Leu Ile Arg Gly Lys Ser Pro Glu
115 120 125
His Val Arg Gln Cys Phe Gly Leu Val Lys Asn Phe Glu Ala Pro Glu
130 135 140
Glu Glu Asn Ile Arg Arg Thr Asn Leu Trp Ala Leu Val Asp Glu Lys
145 150 155 160
Glu Gln Ala Lys Lys
165
<210> 68
<211> 187
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> SKP1-9734多肽
<400> 68
Met Ala Gly Trp Thr Ser Val Pro Asp Pro Phe Gln Ala Ala Lys Met
1 5 10 15
Ser Asp Glu Ala Lys Gly Thr Glu Ser Gly Lys Val Val His Leu Val
20 25 30
Ser Gln Glu Gly Asp Gln Tyr Glu Val Glu Val Ala Val Cys Lys Met
35 40 45
Ser Glu Leu Val Lys Thr Met Leu Pro Asp Asp Asp Asp Asn Thr Asp
50 55 60
Thr Gln Glu Ile Pro Leu Pro Asn Val Lys Asn Ser Val Leu Ala Lys
65 70 75 80
Val Ile Glu Phe Cys Lys His His Lys Asp Asp Pro Met Asn Asp Ile
85 90 95
Glu Lys Pro Leu Lys Ser Ala Asn Met His Glu Val Val Gln Asp Trp
100 105 110
Tyr Ala Asn Phe Val Asn Val Asp Gln Glu Leu Leu Phe Glu Leu Ile
115 120 125
Leu Ala Ala Asn Tyr Met Asp Ile Lys Pro Leu Leu Asp Leu Thr Cys
130 135 140
Ala Thr Val Ala Ser Met Ile Lys Gly Lys Thr Pro Glu Glu Ile Arg
145 150 155 160
Arg Thr Phe Asn Ile Thr Asn Asp Phe Thr Pro Glu Glu Glu Ala Gln
165 170 175
Val Arg Glu Glu Asn Lys Trp Cys Glu Glu Val
180 185
<210> 69
<211> 172
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> SKP1-5078多肽
<400> 69
Met Ser Asp Glu Ala Lys Gly Thr Glu Ser Gly Lys Val Val His Leu
1 5 10 15
Val Ser Gln Glu Gly Asp Gln Tyr Glu Val Glu Val Ala Val Cys Lys
20 25 30
Met Ser Glu Leu Val Lys Thr Met Leu Pro Asp Asp Asp Asp Asn Thr
35 40 45
Asp Thr Gln Glu Ile Pro Leu Pro Asn Val Lys Asn Ser Val Leu Ala
50 55 60
Lys Val Ile Glu Phe Cys Lys His His Lys Asp Asp Pro Met Asn Asp
65 70 75 80
Ile Glu Lys Pro Leu Lys Ser Ala Asn Met His Glu Val Val Gln Asp
85 90 95
Trp Tyr Ala Asn Phe Val Asn Val Asp Gln Glu Leu Leu Phe Glu Leu
100 105 110
Ile Leu Ala Ala Asn Tyr Met Asp Ile Lys Pro Leu Leu Asp Leu Thr
115 120 125
Cys Ala Thr Val Ala Ser Met Ile Lys Gly Lys Thr Pro Glu Glu Ile
130 135 140
Arg Arg Thr Phe Asn Ile Thr Asn Asp Phe Thr Pro Glu Glu Glu Ala
145 150 155 160
Gln Val Arg Glu Glu Asn Lys Trp Cys Glu Glu Val
165 170
<210> 70
<211> 169
<212> PRT
<213> 三角褐指藻
<400> 70
Met Met Asp Val Glu Gln Glu Thr Thr Val Asn Leu Ile Ser Lys Asp
1 5 10 15
Gly Asp Ser Phe Ser Val Pro Leu Ala Val Ala Lys Met Ser Glu Leu
20 25 30
Val Lys Gly Met Ile Asp Glu Asp Ala Glu Asp Glu Gly Asp Lys Ile
35 40 45
Glu Ile Pro Leu Pro Asn Val Lys Ser Gln Val Leu Asn Lys Val Ile
50 55 60
Glu Phe Cys Glu His His Leu Gln Glu Pro Met Thr Glu Ile Glu Lys
65 70 75 80
Pro Leu Lys Ser Gln Val Met Ala Asp Val Val Gln Lys Trp Tyr Ala
85 90 95
Asp Phe Val Asp Val Glu Gln Val Leu Leu Phe Glu Leu Ile Leu Ala
100 105 110
Ala Asn Tyr Met Asp Ile Lys Pro Leu Leu Asp Leu Thr Cys Ala Thr
115 120 125
Val Ala Gly Met Ile Lys Gly Lys Thr Pro Glu Asp Ile Arg Gln Thr
130 135 140
Phe Gly Ile Gln Asn Asp Phe Ser Pro Glu Glu Glu Ala Gln Val Arg
145 150 155 160
Glu Glu Asn Lys Trp Cys Glu Glu Ala
165
<210> 71
<211> 193
<212> PRT
<213> 圆柱拟脆杆藻
<220>
<221> misc_feature
<223> SKP1-6453多肽
<400> 71
Met Asp Val Glu Asp Ser Lys Glu Thr Ala Ala Ser Thr Ala Asp Thr
1 5 10 15
Ser Ser Ser Gly Val Thr Ile Gln Leu Ile Ser Lys Glu Gly Asp Lys
20 25 30
Phe Pro Val Pro Ile Thr Val Ser Asn Met Ser Glu Leu Val Lys Ser
35 40 45
Met Met Asp Asp Lys Asp Asp Asp Asp Asp Asp Asp Asp Asp Glu Asp
50 55 60
Asp Asp Gly Lys Glu Lys Ile Thr Glu Ile Pro Leu Pro Asn Val Lys
65 70 75 80
Ser Glu Val Leu Lys Lys Val Ile Glu Phe Cys Glu His His Leu Ala
85 90 95
Glu Pro Met Thr Glu Ile Glu Lys Pro Leu Lys Ser Gln Asn Met Ala
100 105 110
Asp Val Val Gln Lys Trp Tyr Ala Asp Phe Val Asp Leu Glu Gln Val
115 120 125
Leu Leu Phe Glu Leu Ile Leu Ala Ala Asn Tyr Met Asp Ile Lys Pro
130 135 140
Leu Leu Asp Leu Thr Cys Ala Thr Val Ala Ser Met Ile Lys Gly Lys
145 150 155 160
Thr Pro Asp Glu Ile Arg Ala Thr Phe Asn Ile Thr Asn Asp Phe Ser
165 170 175
Pro Glu Glu Glu Ala Gln Val Arg Glu Glu Asn Lys Trp Cys Glu Glu
180 185 190
Ala
<210> 72
<211> 156
<212> PRT
<213> 假微型海链藻
<220>
<221> misc_feature
<223> SKP1-7124多肽
<400> 72
Ser Lys Glu Gly Asp Thr Phe Pro Val Asp Ile Glu Val Ala Arg Met
1 5 10 15
Ser Glu Leu Val Lys Gly Met Leu Glu Asp Asp Ala Asp Asp Asp Glu
20 25 30
Glu Ala Thr Glu Ile Pro Leu Pro Asn Val Lys Ser Thr Val Leu Lys
35 40 45
Lys Val Ile Glu Phe Cys Lys His His Arg Ser Glu Pro Met Thr Glu
50 55 60
Ile Glu Lys Pro Leu Lys Ser Ala Ala Met Ala Glu Val Val Gln Lys
65 70 75 80
Trp Tyr Ala Asp Phe Val Asn Val Glu Gln Val Leu Leu Phe Glu Leu
85 90 95
Ile Leu Ala Ala Asn Tyr Met Asp Ile Lys Pro Leu Leu Asp Leu Thr
100 105 110
Cys Ala Thr Val Ala Ser Met Ile Lys Gly Lys Thr Pro Glu Glu Ile
115 120 125
Arg Lys Thr Phe Asn Ile Ala Asn Asp Phe Ser Pro Glu Glu Glu Ala
130 135 140
Gln Val Arg Glu Glu Asn Lys Trp Cys Glu Glu Pro
145 150 155
<210> 73
<211> 167
<212> PRT
<213> 克革伦不动壶菌
<220>
<221> misc_feature
<223> SKP1-5159多肽
<400> 73
Met Glu Thr Glu Ser Leu Ala Ser Arg Ile Tyr Asn Leu Val Ser Gln
1 5 10 15
Glu Lys Glu Lys Phe Pro Val Ser Lys Glu Val Ala Glu Met Ser Asn
20 25 30
Leu Val Lys Glu Met Thr Glu Asp Glu Glu Asp Glu Asp Ser Asp Ile
35 40 45
Pro Leu Pro Asn Val Ser Ser Arg Val Leu Lys Lys Val Ile Glu Phe
50 55 60
Cys Gln His His Glu Lys Glu Lys Met Pro Glu Ile Glu Lys Pro Leu
65 70 75 80
Lys Ser Ser Asn Met Ala Glu Val Val Ser Asp Trp Asp Ala Lys Phe
85 90 95
Val Glu Val Glu Gln Glu Leu Leu Phe Gln Leu Ile Leu Ala Ala Asn
100 105 110
Tyr Met Asp Ile Lys Ser Leu Leu Asp Leu Ser Cys Ala Lys Val Ala
115 120 125
Ser Met Ile Lys Gly Lys Thr Pro Glu Glu Ile Arg Lys Thr Phe Asn
130 135 140
Ile Thr Asn Asp Phe Thr Pro Glu Glu Glu Cys Thr Val Arg Glu Glu
145 150 155 160
Asn Lys Trp Ser Glu Glu Ser
165
<210> 74
<211> 172
<212> PRT
<213> 裂壶藻
<220>
<221> misc_feature
<223> SKP1-0889多肽
<400> 74
Met Gly Asp Ala Pro Glu Val Gly Ala Thr Lys Gly Met Cys Ala Leu
1 5 10 15
Val Ser Gln Glu Gly Lys Arg Tyr Glu Val Ser Arg Glu Thr Ala Met
20 25 30
Gln Ser Met Leu Val Lys Glu Met Val Asp Glu Glu Glu Gly Glu Asp
35 40 45
Ser Val Leu Asp Ile Pro Leu Pro Asn Val Lys Ser Ser Val Leu Glu
50 55 60
Lys Val Ile Glu Phe Cys Ser His His Val Lys Glu Pro Met Pro Asp
65 70 75 80
Ile Glu Lys Pro Leu Lys Ser Ala Asn Met Asn Glu Val Val Ser Glu
85 90 95
Trp Asp Ala Asn Phe Val Asp Leu Asp Gln Glu Ala Leu Phe Glu Leu
100 105 110
Ile Leu Ala Ala Asn Tyr Met Asp Ile Lys Ser Leu Leu Asp Leu Thr
115 120 125
Cys Ala Lys Val Ala Ser Met Ile Lys Gly Lys Ser Pro Glu Glu Ile
130 135 140
Arg Glu Thr Phe Asn Ile Thr Asn Asp Phe Thr Pro Glu Glu Glu Ala
145 150 155 160
Arg Val Arg Glu Glu Asn Lys Trp Cys Glu Arg Ser
165 170
<210> 75
<211> 166
<212> PRT
<213> 小环藻属某种
<220>
<221> misc_feature
<223> SKP1-3944多肽
<400> 75
Met Glu Asp Thr Ala Arg Thr Ile Asn Leu Val Ser Lys Thr Gly Asp
1 5 10 15
Lys Tyr Glu Leu Ser Tyr Lys Ala Ala Lys Leu Ser Gln Leu Val Phe
20 25 30
Asp Ala Ser Glu Asn Lys Glu Asp Glu Glu Cys Ser Asp Val Pro Ile
35 40 45
Leu Lys Val Glu Ser Glu Cys Leu Glu Lys Val Val Glu Phe Leu Lys
50 55 60
His Tyr Glu Gln Glu Pro Leu Lys Glu Ile Lys Ser Pro Leu Glu Asp
65 70 75 80
Asn Thr Phe Glu Gly Val Val Lys Gln Glu Trp Tyr Arg Asn Phe Val
85 90 95
Gln Glu Val Asp Ser Pro Met Leu Phe Asp Leu Val Thr Ala Ala Asn
100 105 110
Phe Met Ala Ile Gln Pro Leu Leu Asp Leu Ala Cys Leu Lys Val Ser
115 120 125
Cys Leu Leu Met Gly Lys Ser Ser Glu Glu Ile Arg Ile Ile Leu Asn
130 135 140
Ile Pro Gln Met Thr Pro Gln Glu Glu Glu His Ala Arg Arg Glu His
145 150 155 160
Arg Trp Ile Phe Asp Asp
165
<210> 76
<211> 158
<212> PRT
<213> 假微型海链藻
<220>
<221> misc_feature
<223> SKP1-8843多肽
<400> 76
Ile Lys Leu Ile Ser Arg Ala Gly Asp Ser Phe Glu Leu Pro Tyr Ala
1 5 10 15
Ala Ala Ile Leu Ser Gln Thr Val Lys Asp Ala Gln Ser Cys Glu Asp
20 25 30
Asp Glu Glu Asn Glu Asn Pro Asp Asp Val Glu Ile Val Lys Val Glu
35 40 45
Ser Arg Cys Leu Glu Lys Val Val Glu Phe Leu Val His His Leu Glu
50 55 60
Glu Pro Leu Ala Glu Ile Lys Thr Pro Leu Glu Asp Asn Thr Phe Asp
65 70 75 80
Gly Val Val Lys Gln Gln Phe Tyr Arg Asp Phe Val Lys Gly Val Asp
85 90 95
Gln Pro Met Leu Phe Asp Leu Val Thr Ala Ala Asn Phe Met Ala Ile
100 105 110
Gln Pro Leu Leu Asp Leu Thr Cys Leu Gln Val Ser Cys Gln Leu Met
115 120 125
Gly Lys Ser Ala Asp Glu Ile Arg Thr Ile Leu Asn Ile Pro Gln Met
130 135 140
Thr Pro Glu Glu Glu Ala Lys Ala Arg Gln Glu His Arg Trp
145 150 155
<210> 77
<211> 175
<212> PRT
<213> 三角褐指藻
<220>
<221> misc_feature
<223> SKP1-0332多肽
<400> 77
Met Gly Ser Glu Thr Ile Thr Phe Ile Ser Asn Asp Lys Gln Glu Phe
1 5 10 15
Asp Leu Pro Phe Glu Ala Ala Lys Thr Ala Gly Leu Val Glu Asp Phe
20 25 30
Phe Glu Ser Ser Asp Asp Asn Glu Glu Asn Asn Asp Gly Gly Lys Pro
35 40 45
Asn Arg Arg Glu Met Glu Phe Pro Arg Val Glu Gly Arg Ile Leu Ser
50 55 60
Leu Ile Val Asp Phe Leu Lys His His Asn Glu Glu Gln Met Lys Glu
65 70 75 80
Ile Pro Val Pro Leu Gly Gly Ser Thr Phe Asp Glu Val Met Asp Gln
85 90 95
Glu Trp Tyr Lys Glu Phe Ala His Ala Leu Ser Gln Asn Lys Thr Leu
100 105 110
Phe Glu Val Leu Thr Ala Ala Asn Tyr Met Asn Ile Lys Pro Leu Leu
115 120 125
Asp Leu Ala Cys Leu Glu Ile Thr Phe Lys Leu Thr Gly Met Ser Ala
130 135 140
Glu Gln Val Arg Val Tyr Leu Asn Leu Pro Gln Leu Thr Ala Glu Gln
145 150 155 160
Glu Ala Glu Ala Arg Glu Arg His Pro Trp Ile Phe Glu Ser His
165 170 175
<210> 78
<211> 158
<212> PRT
<213> 三角褐指藻
<220>
<221> misc_feature
<223> SKP1-8957多肽
<400> 78
Ser Lys Glu Gly Asp Ala Tyr Glu Val Pro Met Ala Val Ala Lys Met
1 5 10 15
Ser Val Leu Val Ala Asp Thr Phe Asp Ala Asp Glu Asp Asp Asp Glu
20 25 30
Ala Glu Pro Val Lys Asp Phe Pro Leu Pro Asn Val Thr Ser Gly Val
35 40 45
Leu Glu Lys Val Ile Glu Phe Cys Lys His Phe Gln Glu Glu Pro Met
50 55 60
Thr Thr Ile Gln Thr Pro Leu Lys Ser Ser Lys Leu Glu Asp Leu Val
65 70 75 80
Gln Gln Trp Tyr Ala Asp Phe Val Lys Val Pro Lys Thr Leu Leu Phe
85 90 95
Asp Leu Val Ala Ala Ala Asn Tyr Met Asp Ile Lys Pro Leu Leu Asp
100 105 110
Leu Thr Cys Leu Ala Val Ser Ile Leu Ile Lys Gly Lys Ser Ala Ala
115 120 125
Glu Leu Arg Ser Met Phe Asn Leu Ser Asp Glu Leu Ser His Glu Glu
130 135 140
Glu Ala Gln Met Ala Gln Gly Asn Gln Gln Phe Ala Asp Arg
145 150 155
<210> 79
<211> 194
<212> PRT
<213> 圆柱拟脆杆藻
<220>
<221> misc_feature
<223> SKP1-9785多肽
<400> 79
Met Asp Glu Glu Asn Gly Ile Val Lys Leu Val Ser Lys Glu Gly Asp
1 5 10 15
Val His Glu Val Pro Ile Asn Val Ala Lys Met Ser Ser Leu Ile Leu
20 25 30
Ser Thr Leu Asp Asp Asp Glu Glu Asp Asp Asp Asp Asp Glu Glu Thr
35 40 45
Lys Lys His Leu Glu Ile Pro Leu Pro Asn Val Lys Asn Ala Val Leu
50 55 60
Thr Lys Val Ile Glu Tyr Cys Lys His Tyr Thr Asn Asp Glu Ala Met
65 70 75 80
Thr Gln Ile Gln Thr Pro Leu Lys Ser Ser Lys Ile Glu Asp Leu Val
85 90 95
Gln Thr Trp Tyr Ala Gly Phe Val Asp Val Glu Gln Thr Leu Leu Phe
100 105 110
Glu Leu Val Thr Ala Ala Asn Phe Met Asp Ile Lys Pro Leu Leu Asp
115 120 125
Leu Thr Cys Leu Ala Val Ser Ile Ser Ile Lys Gly Lys Thr Ala Pro
130 135 140
Gln Leu Arg Glu Ile Phe Asn Ile Ser Asn Asp Phe Ser Gln Glu Glu
145 150 155 160
Glu Ala Gln Val Arg Glu Glu Ser Gln Trp Ser Gln Glu Asp Thr Pro
165 170 175
Ala Val Pro Ala Ala Ala Ala Ala Ala Ala Ala Asn Glu Glu Glu Lys
180 185 190
Glu Glu
<210> 80
<211> 207
<212> PRT
<213> 小环藻属某种
<220>
<221> misc_feature
<223> SKP1-0115多肽
<400> 80
Met Leu Leu Trp Val Cys Ala Cys Lys Arg Pro Pro Pro Gln Arg Leu
1 5 10 15
Thr Lys Pro Val Leu Asn Tyr Gln Thr Ile Met Glu Gln Asp Asp Ser
20 25 30
Val Arg Thr Val Lys Leu Ile Ser Asn Glu Gly Glu Ile Phe Lys Val
35 40 45
Pro Leu Glu Ile Ala Lys Leu Ser Asn Leu Val Val Thr Thr Leu Gly
50 55 60
Glu Glu Glu His Asp Asp Asp His Asp Asp Asn Gly Asp Asp Asp Asp
65 70 75 80
Ala Val Glu Ile Pro Leu Pro Asn Val Lys Ala Ser Val Leu Ala Lys
85 90 95
Val Val Asp Phe Cys Thr His Tyr Lys Glu Glu Pro Met Lys Pro Ile
100 105 110
Thr Thr Pro Leu Glu Ser Met Val Ile Asp Glu Ile Val Gln Thr Phe
115 120 125
Tyr Ala Arg Phe Val Glu Val Asp Gln Val Met Leu Phe Glu Leu Val
130 135 140
Thr Ala Ala Asn Phe Met Asp Ile Lys Pro Leu Leu Asp Leu Thr Cys
145 150 155 160
Leu Ala Val Ser Ile Tyr Ile Lys Gly Lys Ser Pro Asp Glu Ile Arg
165 170 175
Arg Ile Phe Asn Ile Ser Asn Asp Met Ser Gln Gly Asp Gly Gly Gln
180 185 190
Val Gly Asp Glu Ser Lys Met Asn Pro Asn Gly Ser Ala Ser His
195 200 205
<210> 81
<211> 176
<212> PRT
<213> 假微型海链藻
<220>
<221> misc_feature
<223> SKP1-5712多肽
<400> 81
Met Asp Gly Asp Asp Ala His Gly Thr Val Lys Leu Val Ser Lys Glu
1 5 10 15
Gly Asp Thr Phe Glu Val Pro Ile Glu Val Ala Lys Leu Ser Asn Leu
20 25 30
Val Val Thr Thr Leu Gly Glu Glu Asp Asp Tyr Asp Asp Asp Asp Asp
35 40 45
Asn Met Val Glu Ile Pro Leu Pro Asn Val Lys Ser Ser Val Leu Ala
50 55 60
Lys Val Ile Glu Tyr Cys Thr His Tyr Asn Gln Asp Pro Met Thr Pro
65 70 75 80
Ile Thr Thr Pro Leu Lys Ser Asn Arg Ile Glu Glu Ile Val Gln Glu
85 90 95
Trp Tyr Ala His Phe Val Asp Val Glu Gln Ile Leu Leu Phe Glu Leu
100 105 110
Val Thr Ala Ala Asn Phe Met Asp Ile Lys Ala Leu Leu Asp Leu Thr
115 120 125
Cys Leu Ala Val Ser Val Leu Ile Lys Gly Lys Ser Ala Glu Glu Ile
130 135 140
Arg Arg Ile Phe Asn Ile Ser Asn Asp Phe Ser Pro Glu Glu Glu Ala
145 150 155 160
Gln Val Ser Lys Glu Asn Gln Phe Thr Asp Gly Thr Ser Ser Ser Ser
165 170 175
<210> 82
<211> 158
<212> PRT
<213> 强壮团藻
<220>
<221> misc_feature
<223> SKP1-3608多肽
<400> 82
Met Ser Gly Ser Lys Val Lys Leu Met Ser Ser Asp Thr Gln Met Phe
1 5 10 15
Glu Val Asp Glu Glu Ala Ala Phe Gln Ser Gln Thr Val Lys Asn Leu
20 25 30
Val Glu Asp Ala Gly Thr Asp Asp Ala Ile Pro Leu Pro Asn Val Ser
35 40 45
Gly Arg Ile Leu Ala Lys Val Ile Glu Tyr Cys Lys Tyr His Val Glu
50 55 60
Ala Glu Lys Lys Gly Ala Asp Asp Lys Pro Met Lys Thr Glu Asp Glu
65 70 75 80
Val Lys Arg Trp Asp Glu Glu Phe Val Lys Val Asp Gln Ala Thr Leu
85 90 95
Phe Asp Leu Ile Leu Ala Ala Asn Tyr Leu Asn Ile Lys Gly Leu Leu
100 105 110
Asp Leu Thr Cys Gln Thr Val Ala Gln Met Ile Lys Gly Lys Thr Pro
115 120 125
Glu Glu Ile Arg Lys Thr Phe Asn Ile Lys Asn Asp Phe Thr Pro Glu
130 135 140
Glu Glu Glu Glu Val Arg Arg Glu Asn Gln Trp Ala Phe Asp
145 150 155
<210> 83
<211> 157
<212> PRT
<213> 莱茵衣藻
<220>
<221> misc_feature
<223> SKP1-6936多肽
<400> 83
Met Ala Thr Lys Val Lys Leu Met Ser Ser Asp Ala Gln Met Phe Glu
1 5 10 15
Val Asp Glu Asp Val Ala Phe Gln Ser Gln Thr Val Lys Asn Leu Val
20 25 30
Glu Asp Ala Gly Thr Glu Asp Ala Ile Pro Leu Pro Asn Val Ser Gly
35 40 45
Arg Ile Leu Ala Lys Val Ile Glu Tyr Ser Lys Tyr His Val Glu Ala
50 55 60
Glu Lys Lys Gly Ala Asp Asp Lys Pro Thr Lys Thr Glu Asp Asp Val
65 70 75 80
Lys Arg Trp Asp Asp Glu Phe Val Lys Val Asp Gln Ala Thr Leu Phe
85 90 95
Asp Leu Ile Leu Ala Ala Asn Tyr Leu Asn Ile Lys Gly Leu Leu Asp
100 105 110
Leu Thr Cys Gln Thr Val Ala Gln Met Ile Lys Gly Lys Thr Pro Glu
115 120 125
Glu Ile Arg Lys Thr Phe Asn Ile Lys Asn Asp Phe Thr Pro Glu Glu
130 135 140
Glu Glu Glu Val Arg Arg Glu Asn Gln Trp Ala Phe Asp
145 150 155
<210> 84
<211> 160
<212> PRT
<213> 四爿藻属某种
<220>
<221> misc_feature
<223> SKP1-5885多肽
<400> 84
Met Ser Glu Lys Val Lys Leu Arg Ser Ala Asp Gly Glu Met Phe Glu
1 5 10 15
Val Asp Ala Asp Val Ala Phe Ser Ser Leu Thr Val Lys Asn Met Ile
20 25 30
Glu Asp Thr Gly Ala Ser Ala Pro Val Pro Val Pro Asn Val Asn Ser
35 40 45
Lys Val Leu Ser Lys Ile Ile Glu Tyr Cys Ser Tyr His Val Asp Gln
50 55 60
Glu Arg Arg Ser Lys Asp Ala Asp Asp His Thr Arg Arg Gln Ile Glu
65 70 75 80
Asp Glu Thr Ser Lys Trp Asp Lys Asp Tyr Ile Cys Val Asp Gln Ala
85 90 95
Val Leu Tyr Glu Leu Ile Leu Ala Ala Asn Phe Leu Asn Ile Lys Gly
100 105 110
Leu Leu Asp Leu Cys Cys Gln Thr Val Ala Asp Ile Ile Lys Gly Lys
115 120 125
Thr Pro Glu Gln Ile Arg Gln Tyr Phe His Ile Lys Asn Asp Phe Thr
130 135 140
Pro Glu Glu Glu Glu Glu Val Arg Lys Glu Asn Gln Trp Ala Phe Glu
145 150 155 160
<210> 85
<211> 158
<212> PRT
<213> 普通小球藻
<220>
<221> misc_feature
<223> SKP1-2686多肽
<400> 85
Met Ala Gln Lys Val Thr Leu Val Ser Ser Asp Ser Gln Asp Tyr Thr
1 5 10 15
Val Thr Glu Glu Val Ala Phe Met Ser Glu Thr Val Lys Asn Thr Leu
20 25 30
Glu Glu Thr Gly Gly Glu Asp Thr Lys Val Pro Leu Pro Asn Val His
35 40 45
Ser Lys Ile Leu Ser Lys Val Leu Glu Tyr Cys Asn Phe His Val Asp
50 55 60
Ala Ser Lys Lys Asn Thr Asp Asp Lys Pro Ala Lys Thr Glu Glu Glu
65 70 75 80
Val Lys Thr Trp Asp Ser Asp Phe Val Lys Val Asp Gln Ala Thr Leu
85 90 95
Phe Glu Leu Ile Leu Ala Ala Asn Tyr Leu Asn Ile Lys Ser Leu Leu
100 105 110
Asp Leu Gly Cys Leu Thr Val Ala Asn Met Ile Lys Gly Lys Thr Pro
115 120 125
Glu Glu Ile Arg Lys Thr Phe Asn Ile Pro Asn Asp Phe Thr Pro Glu
130 135 140
Glu Glu Glu Glu Val Arg Arg Glu Asn Gln Trp Ala Phe Glu
145 150 155
<210> 86
<211> 159
<212> PRT
<213> 拟小球藻属某种
<220>
<221> misc_feature
<223> SKP1-2323多肽
<400> 86
Met Ala Glu Gln Lys Val Lys Leu Val Ser Ser Asp Gly Gln Val Phe
1 5 10 15
Glu Val Glu Glu Asp Val Ala Lys Gln Ser Val Thr Leu Gln Asn Thr
20 25 30
Met Asp Glu Ile Asp Ala Ala Asp Glu Gln Ile Pro Leu Pro Asn Val
35 40 45
Ser Gly Lys Ile Leu Ala Lys Val Val Glu Tyr Cys Lys Tyr His Val
50 55 60
Glu Ala Glu Gln Lys Asp Glu His Gly Lys Ala Ala Lys Ser Glu Asp
65 70 75 80
Glu Val Lys Thr Trp Asp Thr Glu Phe Cys Lys Val Asp Gln Gly Thr
85 90 95
Leu Phe Glu Leu Ile Leu Ala Ala Asn Tyr Leu Asn Ile Lys Thr Leu
100 105 110
Leu Asp Leu Thr Cys Leu Thr Val Ala Asn Met Ile Lys Gly Lys Thr
115 120 125
Pro Glu Glu Ile Arg Lys Thr Phe Asn Ile Glu Asn Asp Phe Thr Pro
130 135 140
Glu Glu Glu Glu Glu Val Arg Arg Glu Asn Gln Trp Ala Phe Glu
145 150 155
<210> 87
<211> 168
<212> PRT
<213> 拟小球藻属某种
<220>
<221> misc_feature
<223> SKP1-2322多肽
<400> 87
Met Tyr Gln Met Gln Lys Val Cys Ser Arg Arg Val Val Gln Val Lys
1 5 10 15
Leu Val Ser Ser Asp Gly Gln Val Phe Glu Val Glu Glu Asp Val Ala
20 25 30
Lys Gln Ser Val Thr Leu Gln Asn Thr Met Asp Glu Ile Asp Ala Ala
35 40 45
Asp Glu Gln Ile Pro Leu Pro Asn Val Ser Gly Lys Ile Leu Ala Lys
50 55 60
Val Val Glu Tyr Cys Lys Tyr His Val Glu Ala Glu Gln Lys Asp Glu
65 70 75 80
His Gly Lys Ala Ala Lys Ser Glu Asp Glu Val Lys Thr Trp Asp Thr
85 90 95
Glu Phe Cys Lys Val Asp Gln Gly Thr Leu Phe Glu Leu Ile Leu Ala
100 105 110
Ala Asn Tyr Leu Asn Ile Lys Thr Leu Leu Asp Leu Thr Cys Leu Thr
115 120 125
Val Ala Asn Met Ile Lys Gly Lys Thr Pro Glu Glu Ile Arg Lys Thr
130 135 140
Phe Asn Ile Glu Asn Asp Phe Thr Pro Glu Glu Glu Glu Glu Val Arg
145 150 155 160
Arg Glu Asn Gln Trp Ala Phe Glu
165
<210> 88
<211> 182
<212> PRT
<213> 拟小球藻属某种
<220>
<221> misc_feature
<223> SKP1-0087多肽
<400> 88
Met Phe Trp Trp Thr Thr Phe Ser His Leu Ser Leu Arg Cys Arg Tyr
1 5 10 15
Pro Arg Gln Arg Tyr Gly Arg Thr Lys Val Val Gln Val Lys Leu Val
20 25 30
Ser Ser Asp Gly Gln Val Phe Glu Val Glu Glu Asp Val Ala Lys Gln
35 40 45
Ser Val Thr Leu Gln Asn Thr Met Asp Glu Ile Asp Ala Ala Asp Glu
50 55 60
Gln Ile Pro Leu Pro Asn Val Ser Gly Lys Ile Leu Ala Lys Val Val
65 70 75 80
Glu Tyr Cys Lys Tyr His Val Glu Ala Glu Gln Lys Asp Glu His Gly
85 90 95
Lys Ala Ala Lys Ser Glu Asp Glu Val Lys Thr Trp Asp Thr Glu Phe
100 105 110
Cys Lys Val Asp Gln Gly Thr Leu Phe Glu Leu Ile Leu Ala Ala Asn
115 120 125
Tyr Leu Asn Ile Lys Thr Leu Leu Asp Leu Thr Cys Leu Thr Val Ala
130 135 140
Asn Met Ile Lys Gly Lys Thr Pro Glu Glu Ile Arg Lys Thr Phe Asn
145 150 155 160
Ile Glu Asn Asp Phe Thr Pro Glu Glu Glu Glu Glu Val Arg Arg Glu
165 170 175
Asn Gln Trp Ala Phe Glu
180
<210> 89
<211> 157
<212> PRT
<213> 小球藻属某种
<220>
<221> misc_feature
<223> SKP1-0684多肽
<400> 89
Met Thr Glu Lys Val Lys Leu Leu Ser Ser Asp Thr Gln His Phe Glu
1 5 10 15
Val Asp Ala Glu Val Ala Lys Gln Ser Val Thr Ile Leu Asn Thr Ile
20 25 30
Glu Glu Ile Gly Ser Asp Glu Val Ile Pro Val Pro Asn Val Asn Ser
35 40 45
Lys Ile Leu Ser Lys Val Ile Glu Tyr Cys Ser Phe His Val Ala Ala
50 55 60
Glu Lys Lys Asp Glu His Gly Lys Thr Gly Lys Thr Glu Asp Glu Ile
65 70 75 80
Lys Ala Phe Asp Ala Glu Phe Thr Lys Val Asp Gln Gly Val Leu Phe
85 90 95
Glu Leu Ile Leu Ala Ala Asn Tyr Leu Asn Ile Lys Ser Leu Leu Asp
100 105 110
Leu Thr Cys Leu Thr Val Ala Asn Met Ile Lys Gly Lys Thr Pro Glu
115 120 125
Glu Ile Arg Lys Thr Phe Asn Ile Glu Asn Asp Phe Thr Pro Glu Glu
130 135 140
Glu Glu Glu Val Arg Arg Glu Asn Gln Trp Ala Phe Glu
145 150 155
<210> 90
<211> 175
<212> PRT
<213> 四爿藻属某种
<220>
<221> misc_feature
<223> SKP1-9377多肽
<400> 90
Val Ala Ala Pro Glu Val Arg Asn Ser Ser Phe Thr Gln Arg Thr Tyr
1 5 10 15
Thr Met Ala Glu Thr Lys Val Lys Leu Arg Ser Ser Asp Glu Gln Met
20 25 30
Phe Glu Val Glu Glu Asp Val Ala Phe Glu Ser Leu Thr Val Lys Asn
35 40 45
Met Ile Glu Asp Thr Gly Thr Glu Ala Pro Ile Pro Leu Pro Asn Val
50 55 60
Ser Ser Lys Ile Leu Ala Lys Val Ile Glu Tyr Cys Lys Tyr His Val
65 70 75 80
Asp Ala Arg Lys Lys Thr Asp Ala Asp Lys Pro Ser Lys Leu Asp Asp
85 90 95
Asp Val Lys Ala Trp Asp Met Glu Phe Val Lys Val Asp Gln Gly Thr
100 105 110
Leu Phe Glu Leu Ile Leu Ala Ala Asn Tyr Leu Asn Ile Lys Thr Leu
115 120 125
Leu Asp Leu Thr Cys Leu Thr Val Ala Asn Met Ile Lys Gly Lys Thr
130 135 140
Pro Glu Glu Ile Arg Lys Thr Phe Asn Ile Lys Asn Asp Phe Thr Pro
145 150 155 160
Glu Glu Glu Glu Glu Val Arg Lys Glu Asn Gln Trp Ala Phe Glu
165 170 175
<210> 91
<211> 252
<212> PRT
<213> 四爿藻属某种
<220>
<221> misc_feature
<223> CHORD多肽
<400> 91
Met Ala Glu Asp Ala Ala Asn Gly Ala Ala Ala Leu Ala Ala Ala Leu
1 5 10 15
Ser Pro Gly Val Gly Lys Leu Ser Leu Gly Gly Leu Gly Thr Gln Ser
20 25 30
Pro Ala Gln Ala Gly Pro His Thr Cys His Arg Met Gly Cys Gly Glu
35 40 45
Lys Phe Asp Pro Ala Ala Asn Ser Asp Ser Ser Cys Arg Tyr His Pro
50 55 60
Asn Pro Pro Tyr Phe His Asp Gly Met Lys Glu Trp Thr Cys Cys Lys
65 70 75 80
Lys Lys Ser His Asp Phe Gly Glu Phe Met Ala Ile Pro Gly Cys Thr
85 90 95
Thr Gly Arg His Ser Ser Glu Lys Pro Glu Lys Pro Ala Ala Lys Pro
100 105 110
Val Pro Ala Ala Ala Ala Pro Pro Val Ala Ser Thr Pro Ala Ala Ala
115 120 125
Ser Thr Cys Leu Arg Cys Ala Gln Gly Phe Phe Cys Ser Asp His Ala
130 135 140
Gly Val Pro Ala Val Val Pro Val Ser Val Ala Ala Ala Ala Pro Val
145 150 155 160
His Pro Gln Val Glu Pro Ala Pro Lys Val Ala Arg Pro Ala Pro Val
165 170 175
Pro Asp Ala Asp Gly Asn Leu Val Cys Arg His Phe Ala Cys Gly Asn
180 185 190
Lys Tyr Lys Glu Gly Glu Asn His Gly Glu Ala Cys His His His Pro
195 200 205
Gly Pro Ala Val Phe His Asp Arg Gln Lys Gly Trp Gly Cys Cys Asn
210 215 220
Lys Phe Glu Arg Asp Phe Asp Ala Phe Leu Ala Ile Pro Pro Cys Ala
225 230 235 240
Tyr Gly Glu His Asp Ala Ala Phe Glu Gly Thr Phe
245 250
<210> 92
<211> 358
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> CHORD多肽
<400> 92
Met Lys Leu Tyr Phe His Tyr Glu Glu Thr Asp Asp Ala Gly Asp Val
1 5 10 15
Glu Glu Ala Lys Ala Leu Thr Leu Lys Leu Thr Leu Pro Lys Ser Trp
20 25 30
Val Gly Gln Pro Leu Leu Gln Val Leu Glu Leu Phe Leu Glu Asn Tyr
35 40 45
Asn Asn Lys Lys Ala Arg Leu Glu Pro Leu Asp Ile Ser Gly Val His
50 55 60
Leu Glu Lys Ala Asp Gly Met Lys Ile His Thr Thr Asp Ile Val Met
65 70 75 80
Asp Ile Leu Ser Asp Arg Asp Asp Val Tyr Val Lys His Gly Ala Glu
85 90 95
Gln Pro Ala Lys Ala Lys Arg Thr Pro Pro Ser Ser Ser Ser Ser Ser
100 105 110
Ser Ser Ser Ser Ser Ser Ser Ser Ser Ser Ser Ser Ser Ile Pro Gly
115 120 125
Ala Thr Thr Glu Gly Ser Thr Gly Leu Leu Arg Cys Lys Asn Tyr Gly
130 135 140
Cys Asn Gln Ser Phe Ala Gln Glu Thr Asn Thr Glu Ser Ala Cys Arg
145 150 155 160
Phe His Arg Ala Pro Pro Val Phe His Asp Thr Lys Lys Gly Trp Ala
165 170 175
Cys Cys Thr Lys Arg Val Tyr Asp Trp Asp Glu Phe His Thr Ile Glu
180 185 190
Gly Cys Ala Thr Gly Arg His Ser Thr Leu Asp Pro Lys Glu Val Phe
195 200 205
Ala Pro Ser Pro Thr Leu Ala Ala Ala Asn Gln Ala Gly Ala Asn Gly
210 215 220
Gly Ser Asp Ala Pro Gly Ala Ser Ser Thr Ala Leu Lys Ser Ile Glu
225 230 235 240
Asp Tyr Asn Gln Ala Asn Pro Asp Ala Ala Thr Ala Ala Lys Ser Ala
245 250 255
Ala Ser Ser Val Thr Lys Pro Gln Ala Arg Cys Thr Val Lys Ala Asp
260 265 270
Gly Ser Ala Thr Cys Leu Asn Lys Gly Cys Gln Lys Glu Phe Gln Val
275 280 285
Lys Glu Asn His Pro Thr Ala Cys Cys Tyr His Ala Ser Gly Pro Val
290 295 300
Phe His Asp Ala Gly Lys Phe Trp Ser Cys Cys Pro Gly Val Ile Lys
305 310 315 320
Tyr Asp Phe Glu Glu Phe Leu Lys Ile Pro Gly Cys Met Val Ser Ser
325 330 335
His Leu Asp Gly Ser Glu Glu Ser Ser Arg Phe Phe Glu Ser His Ala
340 345 350
Arg Lys Arg Glu Asp Arg
355
<210> 93
<211> 301
<212> PRT
<213> 三角褐指藻
<220>
<221> misc_feature
<223> CHORD多肽
<400> 93
Met Lys Val Leu Leu His Tyr Glu Asp Asn Glu Asp Ser Ser Leu His
1 5 10 15
Lys Ser Leu Lys Ile Thr Leu Pro Lys Ser Trp Lys Thr Gly Pro Thr
20 25 30
Ser Arg Leu Leu Thr Gln Phe Leu Glu Ser Tyr Asn Ala Asn Glu Ser
35 40 45
Phe Arg Ser Asn Pro Leu Thr Glu Ala Thr Met His Leu Glu Thr Arg
50 55 60
Ser Ile Ser Thr Glu Ser Gly Pro Thr Val Ser Gly Arg Val Ala Leu
65 70 75 80
Ala Ser Asp Ala Val Val Val Asp Val Ile Ala Asp Arg Ala Asp Ile
85 90 95
Tyr Ile Val His Gly Pro Ser Arg Thr Leu Gln Asp Met Ala Asp Glu
100 105 110
Val Ala Glu Ala Lys Arg Gln Lys Ala Glu Arg Leu Lys Gly Ser Val
115 120 125
Ala Cys Leu His Phe Gly Cys Gln Asn Arg Phe Pro Lys Gly Gly Pro
130 135 140
Tyr Pro Asp Cys Arg Tyr His Lys Ala Pro Pro Val Phe His Glu Thr
145 150 155 160
Ala Lys Phe Trp Ser Cys Cys Pro Asn Lys Lys Ala Tyr Asp Trp Glu
165 170 175
Thr Phe Gln Ala Ile Pro Gly Cys Glu Thr Gly Thr Cys Thr Asp Val
180 185 190
Arg Glu Glu Gly Asp Asp Gly Lys Gln Phe Leu Gly Gly Ser Asp Leu
195 200 205
Arg Glu Lys Thr Glu Ala Val Pro Leu Lys Ser Ile Asp Asp Phe Asn
210 215 220
Lys Ala Gln Thr Ser Gly Glu Ala Ala Pro Ile Leu Glu Arg Leu Glu
225 230 235 240
Thr Val Leu Leu Gln Leu Gly Val Glu Lys Glu Leu Phe Gln Gln Val
245 250 255
Val His Gly Met Lys Val Asn Leu Glu Ala Gln Thr Ala Asn Glu Ala
260 265 270
Glu Leu Met Glu Ala Val Lys Asn Glu Leu Gly Gly Lys Leu Lys Ala
275 280 285
Ala Ile Lys Ala Val Ala Val Glu Gln Leu Arg Ile Lys
290 295 300
<210> 94
<211> 297
<212> PRT
<213> 舟形藻属某种
<220>
<221> misc_feature
<223> CHORD多肽
<400> 94
Met Lys Val Leu Leu His Tyr Glu Asp Asn Glu Asn Thr Ala Leu His
1 5 10 15
Lys Ser Leu Lys Ile Thr Leu Pro Lys Ser Trp Lys Thr Gly Pro Ser
20 25 30
Ser Lys Leu Leu Asp Gln Phe Val Glu Ser Tyr Asn Gly Asn Glu Thr
35 40 45
Leu Gly Ala Asn Asn Pro Leu Asp Ala Ser Arg Leu His Leu Ala Leu
50 55 60
Lys Gln Pro Asp Asn Ser Phe Arg Leu Ile Ala Ser Asp Ala Thr Ala
65 70 75 80
Val Asp Asp Ile Pro Asp Arg Ala Asp Val Tyr Ile Arg His Gly Ala
85 90 95
Ser Lys Thr Lys Gln Asp Ile Ala Thr Glu Gln Arg Arg Ala Gln Glu
100 105 110
Ala Leu Glu Leu Ala Arg Lys Asp Ser Val Ala Cys Thr His Phe Gly
115 120 125
Cys Arg Asn Arg Phe Pro Lys Asn Gly Pro Phe Pro Glu Cys Arg Tyr
130 135 140
His Lys Ala Pro Pro Val Phe His Glu Thr Ala Lys Phe Trp Ser Cys
145 150 155 160
Cys Pro Gln Lys Lys Ala Tyr Asp Trp Glu Asp Phe Gln Asn Ile Pro
165 170 175
Gly Cys Met Thr Gly Ile Cys Thr Ala Val Lys Glu Thr Glu Gly Lys
180 185 190
Gln Phe Leu Gly Gly Thr Asp Leu Arg Glu Asn Ala Glu Val Ala Thr
195 200 205
Leu Lys Ser Ile Asp Asp Phe Asn Lys Ser Gln Ala Ala Gly Gly Ser
210 215 220
Ala Ala Ala Pro Val Leu Glu Arg Leu Ala Gly Val Leu Glu Glu Leu
225 230 235 240
Gly Ile Glu Lys Glu Leu Phe Gln Gln Val Thr Asn Gly Ile Arg Asp
245 250 255
Glu Lys Arg Lys Ser Gly Ile Ile Thr Ser Glu Ala Glu Leu Leu Asp
260 265 270
Gln Val Lys Glu Glu Leu Gly Ala Lys Leu Lys Ala Ala Val Lys Ala
275 280 285
Ile Ala Val Glu Gln Leu Arg Ile Lys
290 295
<210> 95
<211> 287
<212> PRT
<213> 假微型海链藻
<220>
<221> misc_feature
<223> CHORD多肽
<400> 95
Met Lys Val Phe Leu Arg Tyr Glu Glu Asn Asp Asp Glu Ser Thr His
1 5 10 15
Lys Thr Leu Lys Ile Thr Leu Pro Lys Ser Trp Lys Thr Gly Pro Thr
20 25 30
Ser Arg Leu Leu Asp Gln Phe Val Glu Ser Tyr Asn Gly Gly Lys Glu
35 40 45
Gly Glu Ala Asn Pro Leu Asp Ala Ser Thr Leu His Leu Ser Ile Arg
50 55 60
Arg Pro Ala Ser Thr Thr Val Arg Thr Ser Ser Ala Ser Ala Asp Asp
65 70 75 80
Gly Ala Thr Val Leu Lys Glu Leu Pro Ser Asp Gly Ile Ile Val Glu
85 90 95
Thr Ile Glu Asp Arg Asp Asp Val Tyr Val Cys His Gly Pro Ser Leu
100 105 110
Thr Ser Thr Glu Met Asn Ala Glu Arg Gln Ala Lys Ile Asp Lys Glu
115 120 125
Lys Glu Glu Lys Lys Asn Leu Ser Gln Cys Val His Phe Gly Cys Asn
130 135 140
Asn Arg Phe Pro Lys Gly Gly Pro Tyr Pro Asp Cys Lys Tyr His Ser
145 150 155 160
Gly Pro Pro Val Phe His Glu Thr Ala Lys Phe Trp Ser Cys Cys Pro
165 170 175
Asp Lys Lys Ala Tyr Asp Trp Glu Gly Phe Gln Cys Leu Pro Thr Cys
180 185 190
Gln Ser Gly Pro Lys Leu Lys Ser Ile Asp Asp Phe Asn Ala Ser Ile
195 200 205
Ala Ala Gly Gly Ser Glu Gly Ala Pro Val Leu Glu Arg Leu Arg Ser
210 215 220
Val Leu Gly Glu Leu Gly Val Glu Asn Glu Leu Phe Asp Gln Val Phe
225 230 235 240
Glu Gly Val Lys Lys Glu Val Arg Glu Lys Asn Gly Val Asp Cys Glu
245 250 255
Asp Ala Lys Val Leu Asp Glu Ala Ala Gln Met Leu Gly Gly Lys Leu
260 265 270
Lys Ser Ala Met Lys Ala Ile Ala Val Glu Gln Leu Arg Ile Ser
275 280 285
<210> 96
<211> 313
<212> PRT
<213> 小环藻属某种
<220>
<221> misc_feature
<223> CHORD多肽
<400> 96
Met Lys Val Phe Leu Arg Tyr Glu Glu Asn Asp Asp Glu Ser Thr His
1 5 10 15
Lys Thr Leu Lys Ile Thr Leu Pro Lys Ser Trp Lys Thr Gly Pro Thr
20 25 30
Ser Arg Leu Leu Asp Gln Phe Ile Glu Ser Tyr Asn Ala Gly Lys Glu
35 40 45
Gly Glu Ala Asn Pro Leu Glu Ala Asn Ala Met His Leu Ser Val Arg
50 55 60
Arg Arg Ile Ser Thr Asn Gly Ser Asn Ser Asp Asp Asp Thr Ile Leu
65 70 75 80
Lys Asp Leu Pro Ser Asp Gly Ile Val Val Glu Leu Ile Ser Asp Arg
85 90 95
Asp Asp Val Tyr Val Cys His Gly Pro Ser Arg Thr Ser Ser Glu Ile
100 105 110
Asn Ser Glu Arg Glu Ala Gln Leu Lys Lys Glu Lys Glu Glu Lys Lys
115 120 125
Asn Gln Ser Gln Cys Val His Phe Gly Cys Asn Lys Arg Phe Pro Lys
130 135 140
Gly Gly Pro Tyr Pro Glu Cys His Tyr His Ser Gly Pro Pro Val Phe
145 150 155 160
His Glu Thr Ala Lys Phe Trp Ser Cys Cys Pro Asp Lys Lys Ala Tyr
165 170 175
Asp Trp Glu Ser Phe Gln Ser Leu Pro Thr Cys Gln Ser Gly Thr Cys
180 185 190
Thr Asp Val Arg Glu Glu Ser Asp Ala Pro Arg Lys Glu Phe Leu Gly
195 200 205
Gly Cys Asp Leu Arg Glu Gln Ile Ser Ala Gly Pro Lys Leu Arg Ser
210 215 220
Ile Asp Asp Phe Asn Ala Ser Val Ala Ala Gly Gly Ser Glu Arg Ala
225 230 235 240
Pro Val Ala Val Arg Leu Arg Ser Val Leu Glu Glu Leu Gly Val Glu
245 250 255
Asn Glu Leu Phe Asp Gln Val Phe Asp Gly Ile Lys Lys Gln Val Lys
260 265 270
Glu Lys Asn Gly Asp Thr Ala Asp Gly Asp Asp Ala Arg Val Val Asp
275 280 285
Glu Ala Val Lys Ile Leu Gly Thr Lys Leu Lys Ser Ala Met Lys Ser
290 295 300
Ile Ala Val Glu Gln Leu Arg Ile Arg
305 310
<210> 97
<211> 1003
<212> DNA
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> TCTP引物
<400> 97
cgtgcaggtg tacagattga aggaaacaat ggagatatct ttggcagttg aaaaccgtgt 60
tcgaatcatg ctcttctact ctccaactga gacgaaattt atagcgccat ctcgcttctg 120
actaccaggc ttaggaaggc ctcatcacaa gctggatcgg ttcgaattaa gcaggcactg 180
aagccaagct tgcaagacag ccacctttta attctctcaa aacactttct caattcagcc 240
cggtaaatat gccgattcac agcggccaag atagagggga ggttagcaag aatgttgcga 300
tccctcccca gtcgttgcct cgcacacaac ctaggacttc acctttccat ggaaaattga 360
gaagtgaata ttggttttct tacggcatat cagatgaaat catgacccct aaacatgaag 420
agctgcaggc aaaacacctg ctctggacga gcacgatgaa atctcgagaa cccgccgtac 480
ttcagttgat cccgcatgat gacggccgcc attgaaataa gccacctcac tttattctag 540
caccgatttc caccgttgtg agggccgaac gaggacaatt tcgtgcgaaa caagcacgaa 600
cgcgcacacg attagtagga cagacgagca gatcgatggc atgcggcacg gtctcgcgtt 660
ctcggcgacc aggacaacgg agcagaggga ggcctgccga gttccgaggg gcattttagt 720
cccaaaattg tgttgacacg tgaacaagtg gcttgaaaag aggaaggaaa tgcctgggtt 780
tcccttcgag agcgggaact cgcttgtgcg tcatcctagc tacccatggt ccctttgtgg 840
gggaggctgt ttcgtcctac cgaatgtgtg gcgctccatg catcttctgc ctcccaaacc 900
accaacatga gcacgcgaag gaaggagaaa aaagtggccg caacgttctc ttctcatatt 960
tattgtctca tcacaaacat aggtacataa tacaacaatc atg 1003
<210> 98
<211> 399
<212> DNA
<213> 人工序列
<220>
<223> 合成的
<220>
<221> misc_feature
<223> 针对加的斯微拟球藻密码子优化的曲霉菌属杀稻瘟菌素抗性基因
<400> 98
atggccaagc ctttgtccca agaggaatcc acgctgatcg aacgtgcaac tgcgaccatc 60
aacagcatac ctattagcga ggactactcg gtggccagtg cagccctctc gtccgacggt 120
cggatcttta ccggcgtgaa tgtatatcat ttcaccggag ggccatgcgc ggagctcgtg 180
gtcctcggaa cggccgctgc ggctgctgcc ggaaatctga cgtgcatagt ggccatcggg 240
aacgaaaacc gcggcattct gtctccgtgc gggcgatgtc ggcaggtgct gcttgacttg 300
cacccgggga tcaaggcaat tgtcaaagat tccgatgggc agcccacagc ggttggcatc 360
agggagttgc ttccctctgg ctacgtctgg gagggttga 399
<210> 99
<211> 61
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> CHORD外显子4的氨基酸序列
<400> 99
Tyr His Ala Ala Gly Pro Val Phe His Asp Ala Gly Lys Tyr Trp Ser
1 5 10 15
Cys Cys Pro Gly Thr Val Lys Tyr Asp Phe Asp Asp Phe Leu Lys Ile
20 25 30
Pro Gly Cys Met Leu Ser Ser His Tyr Asp Gly Ser Gln Glu Ser Leu
35 40 45
Glu Ala Phe Thr Arg His Ala Lys Thr Ser Glu Gly Thr
50 55 60
<210> 100
<211> 139
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> CHORD 3'融合载体-内含子-外显子4氨基酸序列
<400> 100
Met Gly Ser Pro Gln Arg Leu Ala Ser Gly Ser Cys Phe Pro Leu Ala
1 5 10 15
Thr Ser Gly Arg Val Glu Ile His Trp Pro Ser Phe Tyr Asn Val Val
20 25 30
Thr Gly Lys Thr Leu Ala Leu Pro Asn Leu Ile Ala Leu Gln His Ile
35 40 45
Pro Leu Ser Pro Val Ala Asn Ser Tyr Asn Arg Glu Leu Tyr Val Pro
50 55 60
Thr Pro Thr Leu Arg His Phe Ser Ser Leu Pro Phe Phe Ser Tyr His
65 70 75 80
Ala Ala Gly Pro Val Phe His Asp Ala Gly Lys Tyr Trp Ser Cys Cys
85 90 95
Pro Gly Thr Val Lys Tyr Asp Phe Asp Asp Phe Leu Lys Ile Pro Gly
100 105 110
Cys Met Leu Ser Ser His Tyr Asp Gly Ser Gln Glu Ser Leu Glu Ala
115 120 125
Phe Thr Arg His Ala Lys Thr Ser Glu Gly Thr
130 135
<210> 101
<211> 68
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> SKP四聚化结构域
<400> 101
Lys Tyr Ile Thr Leu Gln Ala Arg Asp Gly Thr Leu Asp Glu Pro Val
1 5 10 15
Asp Ala Arg Ile Leu Leu Pro Ser Asp Leu Leu Arg Ser Met Leu Pro
20 25 30
Glu Lys Leu Ser Glu Ile Glu Asp Asp Phe Gln Ile Pro Leu Gln Gly
35 40 45
Val Asp Lys Ala Val Leu Glu Lys Val Val Glu Tyr Leu His Leu Tyr
50 55 60
Arg Glu Glu Pro
65
<210> 102
<211> 68
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> SKP二聚化结构域
<400> 102
Ile Asn Ala Leu His Tyr Lys Thr Ile Phe Gln Ile Ile Asp Ala Ala
1 5 10 15
Asn Phe Leu Gly Ile Glu Pro Leu Leu Ser Leu Ser Leu Ser Trp Val
20 25 30
Ala Phe Val Leu Lys Gly Pro Thr Val Glu Glu Phe Lys Lys Leu Phe
35 40 45
Thr Ile His Asn Asp Phe Thr Pro Glu Glu Glu Ala Ile Phe Arg Arg
50 55 60
Glu Tyr Leu Leu
65
<210> 103
<211> 1000
<212> PRT
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> 5901启动子序列
<400> 103
Ala Ala Thr Ala Ala Gly Cys Ala Thr Ala Cys Ala Thr Cys Ala Thr
1 5 10 15
Ala Thr Gly Ala Ala Thr Ala Cys Ala Ala Thr Thr Cys Ala Gly Cys
20 25 30
Thr Thr Ala Ala Ala Thr Thr Thr Ala Thr Cys Ala Thr Ala Cys Ala
35 40 45
Ala Ala Gly Ala Thr Gly Thr Ala Ala Gly Thr Gly Cys Ala Gly Cys
50 55 60
Gly Thr Gly Gly Gly Thr Cys Thr Gly Thr Ala Ala Cys Gly Ala Thr
65 70 75 80
Cys Gly Gly Gly Cys Gly Thr Ala Ala Thr Thr Thr Ala Ala Gly Ala
85 90 95
Thr Ala Ala Thr Gly Cys Gly Ala Gly Gly Gly Ala Cys Cys Gly Gly
100 105 110
Gly Gly Gly Ala Gly Gly Thr Thr Thr Thr Gly Gly Ala Ala Cys Gly
115 120 125
Gly Ala Ala Thr Gly Ala Gly Gly Ala Ala Thr Gly Gly Gly Thr Cys
130 135 140
Ala Thr Gly Gly Cys Cys Cys Ala Thr Ala Ala Thr Ala Ala Thr Ala
145 150 155 160
Ala Thr Ala Thr Gly Gly Gly Thr Thr Thr Gly Gly Thr Cys Gly Cys
165 170 175
Cys Thr Cys Gly Cys Ala Cys Ala Gly Cys Ala Ala Cys Cys Gly Thr
180 185 190
Ala Cys Gly Thr Gly Cys Gly Ala Ala Ala Ala Ala Gly Gly Ala Ala
195 200 205
Cys Ala Gly Ala Thr Cys Cys Ala Thr Thr Thr Ala Ala Thr Ala Ala
210 215 220
Gly Thr Thr Gly Ala Ala Cys Gly Thr Thr Ala Thr Thr Cys Thr Thr
225 230 235 240
Thr Cys Cys Thr Ala Thr Gly Cys Ala Ala Thr Gly Cys Gly Thr Gly
245 250 255
Thr Ala Thr Cys Gly Gly Ala Gly Gly Cys Gly Ala Gly Ala Gly Cys
260 265 270
Ala Ala Gly Thr Cys Ala Thr Ala Gly Gly Thr Gly Gly Cys Thr Gly
275 280 285
Cys Gly Cys Ala Cys Ala Ala Thr Ala Ala Thr Thr Gly Ala Gly Thr
290 295 300
Cys Thr Cys Ala Gly Cys Thr Gly Ala Gly Cys Gly Cys Cys Gly Thr
305 310 315 320
Cys Cys Gly Cys Gly Gly Gly Thr Gly Gly Thr Gly Thr Gly Ala Gly
325 330 335
Thr Gly Gly Thr Cys Ala Thr Cys Cys Thr Cys Cys Thr Cys Cys Cys
340 345 350
Gly Gly Cys Cys Thr Ala Thr Cys Gly Cys Thr Cys Ala Cys Ala Thr
355 360 365
Cys Gly Cys Cys Thr Cys Thr Cys Ala Ala Thr Gly Gly Thr Gly Gly
370 375 380
Thr Gly Gly Thr Gly Gly Gly Gly Cys Cys Thr Gly Ala Thr Ala Thr
385 390 395 400
Gly Ala Cys Cys Thr Cys Ala Ala Thr Gly Cys Cys Gly Ala Cys Cys
405 410 415
Cys Ala Thr Ala Thr Thr Ala Ala Ala Ala Cys Cys Cys Ala Gly Thr
420 425 430
Ala Ala Ala Gly Cys Ala Thr Thr Cys Ala Cys Cys Ala Ala Cys Gly
435 440 445
Ala Ala Cys Gly Ala Gly Gly Gly Gly Cys Thr Cys Thr Thr Thr Thr
450 455 460
Gly Thr Gly Thr Gly Thr Gly Thr Thr Thr Thr Gly Ala Gly Thr Ala
465 470 475 480
Thr Gly Ala Thr Thr Thr Thr Ala Cys Ala Cys Cys Thr Cys Thr Thr
485 490 495
Thr Gly Thr Gly Cys Ala Thr Cys Thr Cys Thr Cys Thr Gly Gly Thr
500 505 510
Cys Thr Thr Cys Cys Thr Thr Gly Gly Thr Thr Cys Cys Cys Gly Thr
515 520 525
Ala Gly Thr Thr Thr Gly Gly Gly Cys Ala Thr Cys Ala Thr Cys Ala
530 535 540
Cys Thr Cys Ala Cys Gly Cys Thr Thr Cys Cys Cys Thr Cys Gly Ala
545 550 555 560
Cys Cys Thr Thr Cys Gly Thr Thr Cys Thr Thr Cys Cys Thr Thr Thr
565 570 575
Ala Cys Ala Ala Cys Cys Cys Cys Gly Ala Cys Ala Cys Ala Gly Gly
580 585 590
Thr Cys Ala Gly Ala Gly Thr Thr Gly Gly Ala Gly Thr Ala Ala Thr
595 600 605
Cys Ala Ala Ala Ala Ala Ala Gly Gly Gly Gly Thr Gly Cys Ala Cys
610 615 620
Gly Ala Ala Thr Gly Ala Gly Ala Thr Ala Cys Ala Thr Thr Ala Gly
625 630 635 640
Ala Thr Thr Thr Thr Gly Ala Cys Ala Gly Ala Thr Ala Thr Cys Cys
645 650 655
Thr Thr Thr Thr Ala Cys Thr Gly Gly Ala Gly Ala Gly Gly Gly Thr
660 665 670
Thr Cys Ala Ala Gly Gly Gly Ala Thr Cys Ala Ala Ala Thr Gly Ala
675 680 685
Ala Cys Ala Gly Cys Gly Gly Gly Cys Gly Thr Thr Gly Gly Cys Ala
690 695 700
Ala Thr Cys Thr Ala Gly Gly Gly Ala Gly Gly Gly Ala Thr Cys Gly
705 710 715 720
Gly Ala Gly Gly Thr Thr Gly Gly Cys Ala Gly Cys Gly Ala Gly Cys
725 730 735
Gly Ala Ala Ala Gly Cys Gly Thr Gly Thr Cys Cys Ala Thr Cys Cys
740 745 750
Thr Thr Thr Thr Gly Gly Cys Thr Gly Thr Cys Ala Cys Ala Cys Cys
755 760 765
Thr Cys Ala Cys Gly Ala Ala Cys Cys Ala Ala Cys Thr Gly Thr Thr
770 775 780
Ala Gly Cys Ala Gly Gly Cys Cys Ala Gly Cys Ala Cys Ala Gly Ala
785 790 795 800
Thr Gly Ala Cys Ala Thr Ala Cys Gly Ala Gly Ala Ala Thr Cys Thr
805 810 815
Thr Thr Ala Thr Thr Ala Thr Ala Thr Cys Gly Thr Ala Gly Ala Cys
820 825 830
Cys Thr Thr Ala Thr Gly Thr Gly Gly Ala Thr Gly Ala Cys Cys Thr
835 840 845
Thr Thr Gly Gly Thr Gly Cys Thr Gly Thr Gly Thr Gly Thr Cys Thr
850 855 860
Gly Gly Cys Ala Ala Thr Gly Ala Ala Cys Cys Thr Gly Ala Ala Gly
865 870 875 880
Gly Cys Thr Thr Gly Ala Thr Ala Gly Gly Gly Ala Gly Gly Thr Gly
885 890 895
Gly Cys Thr Cys Cys Cys Gly Thr Ala Ala Ala Cys Cys Cys Thr Thr
900 905 910
Thr Gly Thr Cys Cys Thr Thr Thr Cys Cys Ala Cys Gly Cys Thr Gly
915 920 925
Ala Gly Thr Cys Thr Cys Cys Cys Cys Cys Gly Cys Ala Cys Thr Gly
930 935 940
Thr Cys Cys Thr Thr Thr Ala Thr Ala Cys Ala Ala Ala Thr Thr Gly
945 950 955 960
Thr Thr Ala Cys Ala Gly Thr Cys Ala Thr Cys Thr Gly Cys Ala Gly
965 970 975
Gly Cys Gly Gly Thr Thr Thr Thr Thr Cys Thr Thr Thr Gly Gly Cys
980 985 990
Ala Gly Gly Cys Ala Ala Ala Cys
995 1000
<210> 104
<211> 163
<212> DNA
<213> 加的斯微拟球藻
<220>
<221> misc_feature
<223> T9终止子序列
<400> 104
gagtcaaggg ggaaggtgca tagtgtgcaa caacagcatt aacgtcaaag aaaactgcac 60
gttcaagccc gcgtgaacct gccggtcttc tgatcgccta catatagcag atactagttg 120
tacttttttt tccaaaggga acattcatgt atcaatttga aat 163
- 还没有人留言评论。精彩留言会获得点赞!