外排泵SMeDEF与Ⅰ类整合酶基因联合介导嗜麦芽寡养单胞菌多重耐药基因定位的方法与流程
本发明属于医学技术领域,具体涉及一种外排泵SMeDEF与Ⅰ类整合酶基因联合介导嗜麦芽寡养单胞菌多重耐药基因定位的方法。
背景技术:
近年来,随着临床上大量使用抗菌药物,特别是第三代头孢菌素的广泛应用,出现了对临床使用的三类或三类以上抗菌药物同时呈现耐药的细菌即多重耐药菌(Multidrug-Resistant Organism,MDRO),细菌耐药性问题给临床感染性疾病的治疗带来严峻挑战。嗜麦芽寡养(窄食)单胞菌(Stenotrophomonasmaltophilia)是一种非发酵需氧革兰阴性杆菌,该菌在自然界分布极为广泛,同时也是人和动物体内常见的定植菌。作为条件致病菌的嗜麦芽寡养单胞菌获得了大量繁殖的机遇,成为了一种医院感染重要的新兴多重耐药病原菌,几乎对复方新诺明以外的全部抗菌药耐药,呈现泛耐药性,给临床带来了严重的感染问题,患病率及死亡率呈上升趋势。由此,嗜麦芽寡养单胞菌耐药机制、耐药基因的传播与转移成为近年来国内外研究的热点。
技术实现要素:
本发明的目的是提供一种外排泵SMeDEF与Ⅰ类整合酶基因联合介导嗜麦芽寡养单胞菌多重耐药基因定位的方法。
为了实现上述目的,本发明的技术方案具体如下:
一种外排泵SMeDEF与Ⅰ类整合酶基因联合介导嗜麦芽寡养单胞菌多重耐药基因定位的方法,包括以下步骤:
(1)对收集到的嗜麦芽寡养单胞菌重新鉴定和药敏检测,确立标准菌株;
(2)将标准菌株分别接种于仅含不同浓度环丙沙星的琼脂平板、同时含碳酰氰基-对-氯苯腙(CCCP)和不同浓度环丙沙星的琼脂平板、以及仅含CCCP的琼脂平板上,进行外排表型鉴定,筛选出外排泵基因阳性菌株;
(3)选用外排泵基因阳性菌株提取并纯化细菌基因组DNA作为扩增模版,铜绿假单胞ATCC25783作为阴性菌株,采用RT-PCR进行嗜麦芽寡养单胞菌的外排泵编码基因SMeDEF检测;
(4)采用RT-PCR和菌落杂交实验检测嗜麦芽寡养单胞菌的外排泵编码基因SMeDEF阳性菌株的Ⅰ类整合酶基因;
(5)利用质粒图谱、核酸内切酶图谱进行外排泵编码基因SMeDEF、Ⅰ类整合酶基因比对分析,确定外排泵SMeDEF与Ⅰ类整合酶基因联合介导耐药基因;
(6)将质粒DNA和酶切的染色体DNA与经过地高辛标记的Ⅰ类整合酶基因及外排泵编码基因SMeDEF进行分子杂交实验,完成嗜麦芽寡养单胞菌多重耐药基因定位。
在上述技术方案中,步骤(1)具体为:采用法国梅里埃公司VITEK-2-COMPACT全自动细菌分析系统和药敏测试系统对收集到的嗜麦芽寡养单胞菌进行重新鉴定和药敏检测,确立标准菌株。
在上述技术方案中,步骤(3)中:各种靶基因PCR扩增体系均为:P1、P2引物各0.5μL,KCl 10mmol/L,(NH4)2SO4 8mmol/L,MgCl2 2mmol/L,pH为9.0的Tris-HCl 10mmol/L,BSA质量体积比为0.02%,Taq DNA聚合酶1U;总反应体积20μL,其中模板液5μL;热循环参数:93℃2min,然后93℃30s、55℃30s、72℃60s,循环35周期,最后72℃延伸5min;产物经2%琼脂糖凝胶电泳,EB染色,最后凝胶成像仪观察结果并摄像保存;阳性PCR产物纯化后进行测序,所测序列与GenBank登录的AMEs基因进行序列比较及同源性分析。
在上述技术方案中,步骤(4)具体步骤为:
1)RT-PCR检测
DNA模板的制备:用加热裂解法,将过夜培养的细菌置于60L pH 8.0的10mmol/LTris·Cl和1mmol EDTA的TE缓冲液中,配成一定浓度的菌悬液,并振荡混匀,将菌悬液放入97℃水浴中10min,-20℃放置1min,12000r/min离心5min后取上清,于-20℃放置备用;
基因扩增:采用PCR法设计int1、int2、int3基因引物,反应体系:反应体积为5μL,PremixTaq PCR反应液25μL,基因模板2μL,基因引物各1μL,灭菌蒸馏水2μL;整合子基因扩增条件:预变性94℃5min,变性94℃30s,退火55℃30s,延伸72℃60s,再延伸72℃2min,取扩增产物10μL与缓冲液混匀,点样于2%琼脂糖凝胶中,以100V电压电泳30min,紫外检测仪中观察结果,凝胶成像系统采集图像并保存所测序列与GenBank登录的AMEs基因进行序列比较及同源性分析,int N1引物序列P1:ACGAGCGCAAGGT T TCGGT 565;P2:GAAAGGTCT GGTCAT ACATG;
2)菌落杂交检测
在用RT-PCR方法鉴定Ⅰ类整合子的同时,采用菌落杂交的方法检测73株嗜麦芽寡养单胞菌携带整合子的情况,将73株实验菌株过夜培养后,中间间隔一定距离,并按一定次序接种到一张尼龙膜上,培养过夜后将所有菌株的DNA释放出来,并固定在尼龙膜上;以地高辛标记的Ⅰ类整合酶基因intI1为探针,与73株待测的嗜麦芽寡养单胞菌释放出来的总DNA进行杂交,出现阳性信号的为Ⅰ类整合酶阳性菌株。
在上述技术方案中,步骤(5)具体步骤为:
先将细菌用EDTA处理,破坏细胞膜,然后用十二烷基硫酸钠(SDS)和NaOH溶液裂解细胞;轻混溶液以免染色体DNA切成小片段,进而在抽提过程中混于质粒DNA中,然后加入乙酸中和pH值,使变性的单链DNA复性;用酚/氯仿混合分离后,复性的DNA仍留在溶液中,而变性的染色体DNA、RNA和蛋白质则形成大块凝集沉淀,最后用乙醇将质粒DNA沉淀下来;在营养丰富培养基上生长到对数生长期后加入氯霉素,培养过夜,将细菌培养物在4℃贮存1-2天,再抽提质粒,质粒中是否含有Ⅰ类整合子及整合子的拷贝数,通过质粒接合转移和Southern杂交实验进行分析。
在上述技术方案中,步骤(6)具体步骤为:
1)用碱裂解法提取阳性菌株携带质粒,确定其质粒图谱;
2)Ⅰ整合子阳性菌株作为供体菌,EcoliNK5409F-△为受体菌,在含有链霉素和利福平两种抗生素的培养基中,重复培养实验;
3)用CTAB法分别提取Ⅰ类整合子和外排泵阳性菌株,提取的染色体DNA用EcorI和EcorV进行限制性酶切,酶切后的染色体DNA和质粒DNA分别凝胶电泳,然后分别将DNA转印到尼龙膜上;用地高辛标记的Ⅰ类整合酶基因和外排泵编码基因SMeDEF作为探针进行杂交。
本发明的有益效果是:
(1)本发明提供的外排泵SMeDEF与Ⅰ类整合酶基因联合介导嗜麦芽寡养单胞菌多重耐药基因定位的方法,对嗜麦芽寡养单胞菌多药耐药菌株进行整合子及外排泵筛选及其特性分析;
(2)采用实时PCR(RT-PCR)、间隔重复序列(ERIC)、Southern方法对嗜麦芽寡养单胞菌的整合子和外排泵阳性菌株进行基因指纹图谱分析,确定其Ⅰ类整合子和外排泵特性与基因型的关系,确定基因定位,实现了嗜麦芽寡养单胞菌多重耐药基因定位,将对开发能够控制细菌耐药基因盒地捕获和表达的抗生素佐剂,寻找有效耐药逆转策,提供有力证据。
附图说明
下面结合附图和具体实施方式对本发明作进一步详细说明。
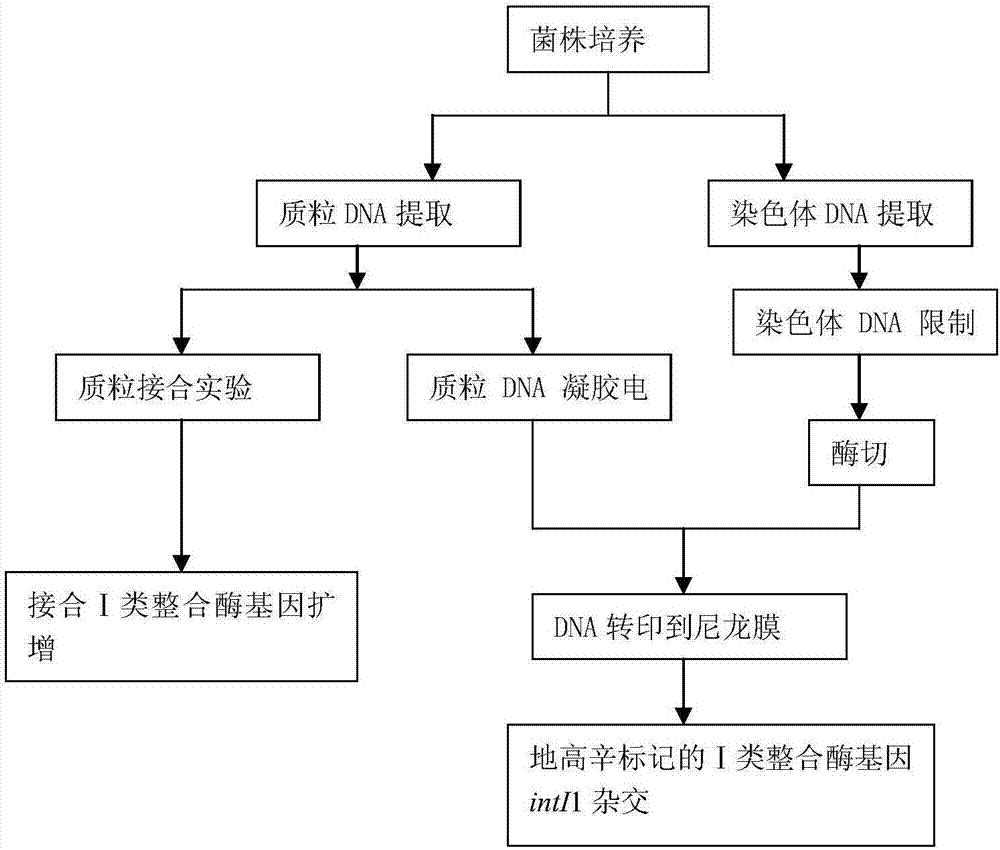
图1为SMeDEF外排泵与Ⅰ类整合酶基因联合介导嗜麦芽寡养单胞菌多重耐药基因确定流程图。
图2为SMeDEF外排泵与Ⅰ类整合酶基因联合介导嗜麦芽寡养单胞菌多重耐药基因定位与拷贝数分析流程图。
图3为Ⅰ类整合酶阳性菌株的质粒表达图谱。
图4为嗜麦芽寡养单胞菌整合酶基因PCR检测结果图。
具体实施方式
下面结合附图对本发明做以详细说明。
实施例
结合图1-2具体说明本发明提供的SMeDEF外排泵与Ⅰ类整合酶基因联合介导嗜麦芽寡养单胞菌多重耐药基因定位的方法的具体步骤:
(1)嗜麦芽寡养单胞菌菌株的筛选:采用法国梅里埃公司VITEK-2-COMPACT全自动细菌分析系统和药敏测试系统对收集到的嗜麦芽寡养单胞菌进行重新鉴定和药敏检测,确立标准菌株。
(2)将标准菌株分别接种于仅含不同浓度环丙沙星的琼脂平板、同时含碳酰氰基-对-氯苯腙(CCCP)和不同浓度环丙沙星的琼脂平板、以及仅含CCCP的琼脂平板上,进行外排表型鉴定,筛选出外排泵基因阳性菌株;
(3)选用外排泵基因阳性菌株提取并纯化细菌基因组DNA作为扩增模版,铜绿假单胞ATCC25783作为阴性菌株,采用RT-PCR进行嗜麦芽寡养单胞菌的外排泵编码基因SMeDEF检测;
根据GenBank中已发布的各型基因序列由博亚生物技术有限公司合成引物。各种靶基因PCR扩增体系均为:P1、P2引物各0.5μL,KCl 10mmol/L,(NH4)2SO4 8mmol/L,MgCl22mmol/L,Tris-HCl(pH9.0)10mmol/L,BSA0.02%(w/v),Taq DNA聚合酶1U。总反应体积20μL(其中模板液5μL)。热循环参数:93℃2min,然后93℃30s、55℃30s、72℃60s,循环35周期,最后72℃延伸5min。产物经2%琼脂糖凝胶电泳,EB染色,最后凝胶成像仪观察结果并摄像保存。阳性PCR产物纯化后进行测序,所测序列与GenBank登录的AMEs基因进行序列比较及同源性分析。
(4)采用RT-PCR和菌落杂交实验检测嗜麦芽寡养单胞菌的外排泵编码基因SMeDEF阳性菌株的Ⅰ类整合酶基因;
1)RT-PCR
DNA模板的制备:用加热裂解法,将过夜培养的细菌置于60L pH 8.0的TE缓冲液(10mmol/LTris·Cl和1mmol EDTA)中,配成一定浓度的菌悬液,并振荡混匀,将菌悬液放入97℃水浴中10min,-20℃放置1min,12000r/min离心5min后取上清,于-20℃放置备用。
基因扩增:采用PCR法设计int1、int2、int3基因引物,反应体系:反应体积为5μL,PremixTaq PCR反应液25μL,基因模板2μL,引物各1μL,灭菌蒸馏水2μL整合子基因扩增条件:预变性94℃5min,变性94℃30s,退火55℃30s,延伸72℃60s,再延伸72℃2min,取扩增产物10μL与缓冲液混匀,点样于2%琼脂糖凝胶中,以100V电压电泳30min,紫外检测仪中观察结果,凝胶成像系统采集图像并保存所测序列与GenBank登录的AMEs基因进行序列比较及同源性分析。int N1引物序列P1:ACGAGCGCAAGGT T TCGGT 565;P2:GAAAGGTCT GGTCAT ACATG。
2)菌落杂交
在用PCR方法鉴定Ⅰ类整合子的同时,采用菌落杂交的方法检测73株嗜麦芽寡养单胞菌携带整合子的情况。将73株实验菌株过夜培养后,中间间隔一定距离,并按一定次序接种到一张尼龙膜上,培养过夜后将所有菌株的DNA释放出来,并固定在尼龙膜上。以地高辛标记的Ⅰ类整合酶基因intI1为探针,与73株待测的嗜麦芽寡养单胞菌释放出来的总DNA进行杂交,出现阳性信号的为Ⅰ类整合酶阳性菌株,与PCR反应检测到的结果相符合,证实PCR方法检测到Ⅰ类整合酶阳性菌株的结果。
(5)利用质粒图谱、核酸内切酶图谱进行外排泵编码基因SMeDEF、Ⅰ类整合酶基因比对分析,确定外排泵SMeDEF与Ⅰ类整合酶基因联合介导耐药基因;
对革兰氏阴性细菌的质粒DNA提取,最常用的是碱裂解法。先将细菌用EDTA(螯合二价阳离子)处理,破坏细胞膜,然后用十二烷基硫酸钠(SDS)和NaOH溶液裂解细胞。轻混溶液以免染色体DNA切成小片段,进而在抽提过程中混于质粒DNA中,然后加入乙酸中和PH值,使变性的单链DNA复性。用酚/氯仿混合分离后,复性的DNA仍留在溶液中,而变性的染色体DNA、RNA和蛋白质则形成大块凝集沉淀,最后用乙醇将质粒DNA沉淀下来。为了增进质粒DNA相对于染色体DNA的产量,在营养丰富培养基上生长到对数生长期后加入氯霉素,培养过夜,将细菌培养物在4℃贮存1-2天,再抽提质粒,质粒中是否含有Ⅰ类整合子及整合子的拷贝数,通过质粒接合转移和Southern杂交实验进行分析。
(6)基因定位分析:分别采用质粒接合转移和质粒DNA与染色体Southern杂交试验进行,即将质粒DNA和酶切的染色体DNA与经过地高辛标记的Ⅰ类整合酶基因及外排泵编码基因SMeDEF进行分子杂交实验,完成嗜麦芽寡养单胞菌多重耐药基因定位。
1)用碱裂解法提取阳性菌株携带质粒,确定其质粒图谱。
2)在质粒接合转移实验中,Ⅰ类整合子阳性菌株作为供体菌,EcoliNK5409F-△为受体菌,在含有链霉素和利福平两种抗生素的培养基中,经过3次重复培养实验后,实验结果均阴性,推测Ⅰ类整合子不在质粒DNA上。
3)用CTAB法分别提取整合子和外排泵阳性菌株,提取的染色体DNA用EcorI和EcorV进行限制性酶切,酶切后的染色体DNA和质粒DNA分别凝胶电泳,然后分别将DNA转印到尼龙膜上。用地高辛标记的Ⅰ类整合酶和SMeDEF基因做为探针进行杂交。
图3为Ⅰ类整合酶阳性菌株的质粒表达图谱,M.整合酶ⅠDNA标志物,相对分子量由下至上分别为:50、100、150、200、300、500、600、800、900、1000、1100、1200、1500Bp;1~5.Ⅰ类整合酶阳性菌株。
图4为嗜麦芽寡养单胞菌整合酶基因PCR检测结果图,在Ⅰ类整合酶基因intI1均无酶切点,Ⅲ:整合酶Ⅲ;Ⅱ:整合酶Ⅱ;Ⅰ:整合酶Ⅰ;M:整合酶ⅠDNA Marker,500Bp;1-10:整合酶Ⅰ临床阳性菌株。
显然,上述实施例仅仅是为清楚地说明所作的举例,而并非对实施方式的限定。对于所属领域的普通技术人员来说,在上述说明的基础上还可以做出其它不同形式的变化或变动。这里无需也无法对所有的实施方式予以穷举。而由此所引伸出的显而易见的变化或变动仍处于本发明创造的保护范围之中。
- 还没有人留言评论。精彩留言会获得点赞!