DNA连接酶介导的DNA扩增技术的制作方法
相关申请
本申请要求于2016年4月1日提交的申请号为201610206799.9的中国专利申请的优先权,其全部内容通过引用并入本申请。
本发明涉及dna连接酶介导的扩增技术,特别地涉及一种dna连接酶介导的扩增低含量dna的技术。该扩增技术可用于snp或者基因拷贝数检测。
背景技术:
在人类和其他哺乳动物中,很多疾病都与基因的突变密切相关。突变有多种形式,比如在单个个体或细胞中最为常见类型的突变往往会发生在dna序列的单一位点上(singlenucleotidepolymorphism,snp);而另一种常见的突变则为拷贝数变异(copynumbervariation,cnv)。
snp是指主要是指在基因组水平上由单个核苷酸的变异所引起的dna序列多态性。snp所表现的多态性只涉及到单个碱基的变异,这种变异可由单个碱基的转换(transition)或颠换(transversion)所引起,也可由碱基的插入或缺失所致,但通常所说的snp并不包括后两种情况。在人类基因组中单核苷酸变异的频率约为0.1%,即大概每1000个碱基就有一个snp。研究表明可以通过snp发现疾病相关基因突变,虽然有些snp并不直接导致疾病,但由于其所处位置与某些疾病基因相邻,因而也成为某些疾病的重要标记物。
cnv一般指长度为1kb以上的基因组大片段的拷贝数增加或者减少,其主要表现为亚显微水平的缺失和重复。cnv是基因组结构变异(structuralvariation,sv)的重要组成部分。cnv位点的突变率远高于snp,是人类疾病的重要致病因素之一。cnv可以导致呈孟德尔遗传的单基因病与罕见疾病,同时与复杂疾病也相关,例如大量研究表明一些不同类型的肿瘤中均存在cnv。cnv致病的可能机制有基因剂量效应、基因断裂、基因融合和位置效应等。对cnv的深入研究,可以使我们对人类基因组的构成、个体间的遗传差异、以及遗传致病因素有新的认识。对cnv的检测也有可能有助于准确判断个体的疾病类型、亚型或抗药性等,从而可以有针对性地制定特殊的治疗方案。
当前用于探究snp和cnv的强大工具就是单细胞测序。单细胞测序是指在单细胞水平(并不限于一个细胞,而是指相对少量的细胞(如少于103个细胞),或等同单细胞水平的量的dna(如单染色体或0.5-3pg的基因组dna))上进行基因测序的技术。由于单个细胞里的dna或rna仅仅处在皮克(picograms,pg)级的水平,其远远达不到现有测序仪的最低上样需求,因此必须先对单细胞内的微量核酸分子进行扩增以便用于测序。在单细胞水平上进行基因扩增会带来一些问题:1.前对于单细胞转录组的扩增技术,很难得到全长转录本。2.扩增存在偏性,即,有些转录本或者某些区域容易被扩增一些,使得最终的基因的表达量之间的数量关系并没有真实的反应细胞里的真实情况。3.在扩增过程中会引入一些错误,由于模板量非常低,上述错误会经由扩增被放大很多倍。4.扩增过程3’和5’端的偏性会导致表达量计算过程中出现问题。而对于snp和cnv来说,扩增过程中的偏性会大大影响snp和cnv识别的准确度和分辨率。
因此,目前急需一种能够尽量降低扩增中引入的误差和/或有效去除扩增偏性的方法。
技术实现要素:
本发明的一个方面提供了一种在dna水平上扩增目标区域的方法,特别是在低含量的dna中扩增目标区域的方法。
在一些实施方式中,所述在dna水平上扩增目标区域的方法包括:重复n个循环的第一轮扩增,其中n为整数且1≤n≤40,所述第一轮扩增包括以下步骤:i)变性dna模板以获得单条目标dna链;ii)将用于扩增目标区域的第一轮扩增引物对杂交至所述目标dna链上,每对所述第一轮扩增引物对包括一个上游引物和一个下游引物,所述上游引物杂交至目标区域的第一核酸序列,所述下游引物杂交至目标区域的第二核酸序列,其中第一核酸序列和第二核酸序列在同一条目标dna链上,并且所述第一核酸序列和第二核酸序列之间间隔m个核苷酸,其中m为≥0的整数,其中第一核酸序列在所述目标dna链上位于第二核酸序列的下游,并且所述下游引物包含磷酸化的5’末端;iii)可选地,在所述上游引物和/或一个下游引物3’末端以所述反义链作为模板进行延伸;iv)通过将上述上游引物或其延伸产物与上述下游引物或其延伸产物连接得到包含目标区域的半扩增产物,其中所述半扩增产物为所述单条目标dna链的反义链。在另一些实施方式中,所述初始dna模板为双链dna。在一些实施方式中,所述初始dna模板为单链dna。
在一些实施方式中,第一核酸序列和第二核酸序列的长度范围为不低于6bp、不低于7bp、不低于8bp、不低于9bp、不低于10bp、不高于50bp、不高于40bp、不高于30bp、6-50bp、6-40bp、6-30bp、6-20bp。在一些实施方式中,第一核酸序列和第二核酸序列的长度范围为6-30bp。
在一些实施方式中,m=0,即第一核酸序列与第二核酸序列紧邻。在一些实施方式中,m≤100,即第一核酸序列与第二核酸序列之间间隔不高于100bp。在一些实施方式中,第一核酸序列与第二核酸序列之间间隔不高于90bp、不高于80bp、不高于70bp、不高于60bp、不高于50bp、不高于40bp、不高于30bp、不高于20bp、不高于10bp。在另一些实施方式中,第一核酸序列与第二核酸序列之间间隔不低于90bp、不低于80bp、不低于70bp、不低于60bp、不低于50bp、不低于40bp、不低于30bp、不低于20bp、不低于10bp。
在一些实施方式中,所述上游引物包含接头序列。在一些实施方式中,所述上游引物和所述下游引物均包含接头序列。在一些实施方式中,所述上游引物和所述下游引物包含杂交序列。在一些特定的实施方式中,所述杂交序列中进一步包含随机序列。在一些实施方式中,所述上游引物包括的杂交序列包括或为随机序列,所述下游引物包含的杂交序列为特异性识别所述目标区域的杂交序列。在一些实施方式中,所述上游引物和所述下游引物包含的杂交序列均为特异性识别所述目标区域的杂交序列。
在一些实施方式中,所述上游引物具有5’末端接头序列。在一些实施方式中,所述上游引物具有5’末端接头序列,所述下游引物具有3’末端接头序列。
在一些实施方式中,第一核酸序列与第二核酸序列紧邻,通过使用耐高温连接酶将上游引物3’端与下游引物或下游引物的延伸产物的5’端直接连接得到半扩增产物。
在一些实施方式中,第一核酸序列与第二核酸序列之间间隔m个核苷酸,通过dna聚合酶在上游引物3’端延伸m个核苷酸至与下游引物5’端紧邻的位置,获得上游引物的延伸产物。在一些实施方式中,通过使用耐高温连接酶将上游引物的延伸产物与下游引物或其延伸产物连接得到半扩增产物。
在一些实施方式中,第一轮扩增为线性扩增,所述半扩增产物即为第一轮扩增产物,在dna水平上扩增目标区域的方法进一步包括:对所述半扩增产物进行测序以确定所述目标区域的序列。
在一些实施方式中,所述上游引物包括的杂交序列为随机序列,所述第一轮扩增进一步包括:v)将所述上游引物杂交至所述半扩增产物,并且在所述上游引物的3’末端以所述半扩增产物作为模板进行延伸,获得第一轮扩增产物。
在一些实施方式中,在dna水平上扩增目标区域的方法进一步包括:使用所述第一轮扩增产物为模板通过聚合酶链式反应pcr扩增所述目标区域以生成指数扩增产物。在一些实施方式中,在dna水平上扩增目标区域的方法进一步包括:对所述指数扩增产物进行测序以确定所述目标区域的序列。
在一些实施方式中,在dna水平上扩增目标区域的方法进一步包括:将所述目标序列的测序结果与参考样本相比较以确定所述目标序列的拷贝数。在一些实施方式中,所述参考样本序列具有两个拷贝。
本发明的另一个方面提供了一种用于在dna水平上扩增目标区域的试剂盒。在一些实施方式中,所述试剂盒包括:第一轮扩增引物对,其中每对所述引物对包括一个上游引物和一个下游引物,所述上游引物和下游引物杂交至目标dna链上,其中所述上游引物杂交至所述目标区域的第一核酸序列,所述下游引物杂交至所述目标区域的第二核酸序列,其中所述第一核酸序列和所述第二核酸序列之间间隔m个核苷酸,其中m为≥0的整数,其中所述第一核酸序列在所述目标dna链上位于第二核酸序列的下游,并且所述下游引物包含磷酸化的5’末端;以及连接试剂,其中所述连接试剂用于将所述上游引物或其延伸产物与所述下游引物或其延伸产物连接得到半扩增产物。
在一些实施方式中,所述连接试剂包括连接酶及连接酶反应溶液。在一些实施方式中,所述连接酶为耐高温连接酶。
在一些实施方式中,用于在dna水平上扩增目标区域的试剂盒进一步包括:延伸试剂,所述延伸试剂用于在所述上游引物和/或所述下游引物3’末端以所述反义链作为模板进行延伸,得到所述上游引物和/或所述下游引物的延伸产物。在一些实施方式中,延伸试剂包括dna聚合酶、反应试剂以及dntp,所述dntp由datp、dttp、dgtp、dctp中的任意一种或多种组成。在一些实施方式中,所述延伸试剂还可用于当所述上游引物杂交至所述半扩增产物上时,在所述上游引物的3’末端以所述半扩增产物作为模板进行延伸,获得第一轮扩增产物。
在一些实施方式中,所述上游引物具有5’末端接头序列。在一些实施方式中,所述上游引物具有5’末端接头序列,所述下游引物具有3’末端接头序列。在一些实施方式中,所述上游引物和所述下游引物包含杂交序列。在一些特定的实施方式中,所述杂交序列中进一步包含随机序列。在一些实施方式中,所述上游引物包括的杂交序列包括或为随机序列,所述下游引物包含的杂交序列为特异性识别所述目标区域的杂交序列。在一些实施方式中,所述上游引物和所述下游引物包含的杂交序列均为特异性识别所述目标区域的杂交序列。在一些实施方式中,用于在dna水平上扩增目标区域的试剂盒进一步包括:指数扩增试剂,所述指数扩增试剂用于以所述第一轮扩增产物为模板扩增所述目标区域得到指数扩增产物。在一些实施方式中,指数扩增试剂包括dna聚合酶、反应试剂以及dntp。
在一些实施方式中,指数扩增试剂进一步包括指数扩增通用引物,其中所述指数扩增通用引物包括与第一轮扩增中所述上游引物的5’末端接头序列相同或反向互补的序列和/或第一轮所述下游引物的3’末端接头序列相同或反向互补的序列。
在一些实施方式中,用于在dna水平上扩增目标区域的试剂盒进一步包括:测序试剂,所述测序试剂用于对所述第一轮扩增产物或所述指数扩增产物进行测序。
附图说明
通过下面说明书和所附的权利要求书并与附图结合,将会更加充分地描述本申请内容的上述和其他特征。可以理解,这些附图仅描绘了本申请内容的若干实施方式,因此不应认为是对本申请内容范围的限定。通过采用附图,本申请内容将会得到更加明确和详细的说明。
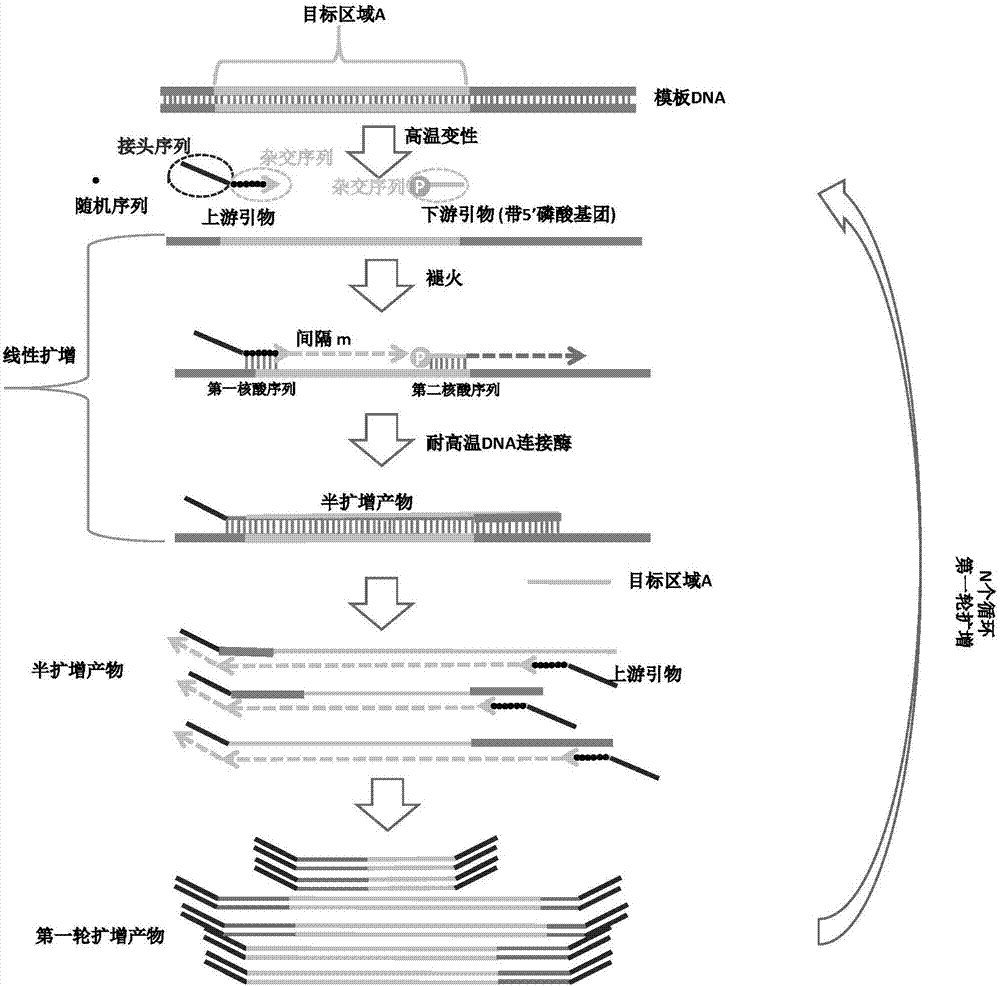
图1为本申请扩增方法中第一轮扩增为线性扩增时的基本原理示意图;
图2为本申请扩增方法中第一轮扩增不为线性扩增时的基本原理示意图;
图3为本申请扩增方法中指数扩增的基本原理示意图;
图4为本申请一种示例性的扩增方法用于dna拷贝数差异(cnv)检测的基本原理示意图;
图5为本申请中的扩增方法的一种具体实施方式。
具体实施方式
本发明的一个方面提供了一种在dna水平上扩增目标区域的方法,特别是在低含量dna中扩增目标区域的方法。本发明至少部分基于以下发现,即,dna连接酶介导的dna扩增方法可以实现:1)降低在扩增过程中引入的误差;2)有效去除扩增偏性,这两个效果中的一个或两个。
在一些实施方式中,本申请提供的在dna水平上扩增目标区域的方法包括:重复n个循环的第一轮扩增,其中n为整数且1≤n≤40,所述第一轮扩增包括以下步骤:i)变性dna模板以获得单条目标dna链;ii)将用于扩增目标区域的引物对杂交至所述目标dna单链上,每对所述引物对包括一个上游引物和一个下游引物,所述上游引物杂交至目标区域的第一核酸序列,所述下游引物杂交至目标区域的第二核酸序列,其中第一核酸序列和第二核酸序列之间间隔m个核苷酸,其中m为≥0的整数,其中第一核酸序列在所述目标dna链上位于第二核酸序列的下游,并且所述下游引物包含磷酸化的5’末端;iii)可选地,在所述上游引物和/或一个下游引物3’末端以所述反义链作为模板进行延伸;iv)通过将上述上游引物或其延伸产物与上述下游引物或其延伸产物连接得到半扩增产物。
本申请所述的“dna”是指由脱氧核苷酸组成的遗传指令的长链聚合物生物大分子。“dna水平”是指核苷酸水平。dna中的每个核苷酸由含氮碱基、五碳糖(2-脱氧核糖)和磷酸基团组成。相邻的核苷酸通过脱氧核糖和磷酸形成酯键相连从而组成长链骨架组成。核苷酸分子两端并不对称,其分别含有磷酸基团和羟基基团,dna链中相邻的核苷酸分子彼此间形成磷酸二酯键,而dna链两端的分子分别保留了一个磷酸基团和一个羟基基团,其中含有磷酸基团的一端被称为5’末端,而含有羟基基团的一端被称为3’末端。在同一dna链上的某一dna片段/碱基a相对于另一片段/碱基b的位置可以用上下游来表示,上下游是一个相对概念,当描述dna片段/碱基a位于片段/碱基b的上游时,是指该dna片段/碱基a相对于片段/碱基b而言更接近于其所处dna链的5’末端,反之当描述dna片段/碱基a位于片段/碱基b的下游时,是指该dna片段/碱基a相对于片段/碱基b而言更接近于其所处dna链的3’末端。
dna的核苷酸中的含氮碱基一般有四种,分别为腺嘌呤(a)、鸟嘌呤(g)和胞嘧啶(c)、胸腺嘧啶(t)。两条dna长链上的碱基通过氢键配对,其中腺嘌呤(a)和胸腺嘧啶(t)配对,鸟嘌呤(g)和胞嘧啶(c)配对,从而使得大部分dna以双链双螺旋结构存在,而该双链结构在经热或碱处理时会发生氢链的断裂,从而使得双链dna分子变性分离为两条单链dna。
本申请中所述的“目标区域”泛指一切目标检测位置。在一些优选的实施方式中,所述目标区域是已知的与某些疾病(例如肿瘤、炎症、出生缺陷等)有关的cnv或snp区域。在一些优选的实施方式中,所述目标区域是与染色体微重复和微缺失综合征(mms)相关的基因组区域。
在进行第1个循环的第一轮扩增时,本文所述的“dna模板”是指初始dna模板;而在n>1的第n次循环时,本文所述的“dna模板”是指第n-1次循环中步骤iii)中获得的扩增产物(半扩增产物和/或第一轮扩增产物)中所包含的双链模板。
本申请所述的“初始dna模板”是指用于本申请的扩增方法的最初的作为模板的dna样本。本申请中用于扩增的初始dna模板可以为单链dna,也可以为双链dna。
初始dna模板可来源于任意形式的生物样本。本申请所述的“生物样本”包括但不限于细胞(包括但不限于:细菌、病毒或动植物细胞)、组织(包括但不限于:正常、坏死、癌、癌旁组织等)、体液(包括但不限于:血液、血浆、血清、唾液、羊水穿刺液、胸腔积液、腹腔积液)等。本申请涉及的生物样本可以来自任何物种或生物种类,包括但不限于,人类、哺乳动物、牛、猪、羊、马、啮齿动物、禽类、鱼类、斑马鱼、虾、植物、酵母、病毒或细菌。本领域技术人员可以通过任何公知的方法获取该生物样本,包括但不限于通过细胞培养、手术、解剖、抽血、擦拭、灌洗等取样方式。生物样品可以以任何适当的形式提供,例如可以以新鲜分离、石蜡包埋、冷藏、冷冻等形式提供。
在一些实施方式中,生物样本为细胞,初始dna模板为基因组dna。在一些实施方式中,初始dna模板来源于单细胞。在一些实施方式中,初始dna模板来源于多个细胞,例如来源于两个或多个同类细胞。本申请所述的“多个细胞”是指不超过103、不超过102、不超过10个或者103以上的细胞。上述单个细胞或多个同类细胞可以来自,例如,植入前的胚胎、孕妇外周血中的胚胎细胞、单精子、卵细胞、受精卵、癌细胞、细菌细胞、肿瘤循环细胞、肿瘤组织细胞、或者从任意组织获得的单个或多个同类细胞。可以通过本领域公知的方法获得单细胞,包括但不限于,流式细胞分选、荧光激活细胞分选法、磁珠分离、半自动细胞挑取仪等。在一些实施方式中,可以根据单个细胞不同的性质来进行挑选特定类型的细胞,例如表达某种特定的生物标记的细胞。
在另一些实施方式中,生物样本为体液。在一些实施方式中,初始dna模板来源于血液、血清、血浆或羊水。在一些实施方式中,初始dna模板为细胞外的游离dna。“细胞外的游离dna”是指在循环系统(例如,血液)中发现的游离于细胞外的dna。其来源一般认为是由于细胞凋亡过程中释放的基因组dna。研究发现,人体内绝大部分的细胞外的游离dna的大小在160bp左右(参见fanetal.,(2010)analysisofthesizedistributionsoffetalandmaternalcell-freednabypaired-endsequencing,clinchem56:81279-86)。在一些实施方式中,在所述细胞外的游离dna中包含循环肿瘤dna。“循环肿瘤dna”是指来源于肿瘤细胞的细胞外游离dna。肿瘤细胞在人体内会因为细胞凋亡、免疫反应等原因释放其基因组dna到血液中。由于正常细胞也同样会将其基因组dna释放到血液中,因此,一般情况下,循环肿瘤dna只占细胞外游离dna的很小一部分。在一些实施方式中,初始dna模板为源于怀孕母体的细胞外游离dna,其中包含胎儿游离dna。“胎儿游离dna”是指在母体血液中包含的来源于胎儿的细胞外游离dna片段。
本领域技术人员可以通过公知的任何方法从生物样品中获得初始dna模板。在一些实施方式中,可以通过对组织或细胞进行裂解(例如可以通过热裂解、碱裂解、酶裂解、机械裂解等方式)并释放细胞内的核酸物质,之后经过纯化等处理步骤最终获得初始dna模板。在一些特定的实施方式中,裂解后的核酸物质无需进行纯化即可作为初始dna模板用于后续扩增。在一些实施方式中,可以通过从血液或血清中分离或富集其中包含的细胞外游离dna,从而获得初始dna模板。
如前所述本申请的方法可以用于扩增一些宝贵的样本例如低含量的初始dna模板,如来源于人类的卵细胞、生殖细胞、体外受精的胚胎细胞等的初始dna模板,或者如肿瘤循环dna、胎儿游离dna等。本申请中所述的“低含量的初始dna模板”是指来源于单细胞的dna模板、来源于不超过103、不超过102、不超过10个或者103以上细胞的dna模板、或者指等同单细胞水平的量的初始dna模板,例如≤0.5pg、≤3pg、≤5pg、≤10pg、≤50pg、≤100pg、≤0.5ng、≤1ng、≤3ng、>3ng的初始dna模板。
本申请中所述的“变性dna模板”是指通过本领域技术人员公知的任何方法,包括但不限于通过热变性(例如高于90℃)、碱(例如naoh)处理等方法分离双链dna的两条链。当dna模板为双链时,通过上述变性方法获得单条目标dna链。当初始dna模板为单链dna时,在第一个循环的扩增中,可以不包括变性手段,初始的单链dna即为所述单条目标dna链;又或者在第一个循环的扩增中,也可以包括变性手段(例如加热至高于90℃或者加热碱溶液等),但对初始的单链dna模板不产生实质性的影响,因此即便经过变性步骤,初始的单链dna还是所述单条目标dna链。本申请中所述的“目标dna链”是指在扩增中步骤ii)内用于扩增目标区域的“引物对”能够杂交至的那条链。
本申请中所述的“引物”是指一段短的单链dna片段,其可杂交至dna或rna链上与之互补的区域并作为dna聚合作用的起始点,dna聚合酶可在其3’末端依次添加与dna模板链互补的核苷酸从而合成新的dna链。
本申请中所述的“互补”是指核酸能够通过传统的watson-crick碱基配对方式或其他非传统方式与另一核酸之间形成氢键的能力。互补百分比被用来表示在一条核酸链分子中能与第二核酸序列形成氢键(例如,watson-crick碱基对)的残基数量百分比,例如一条10个核酸组成的核酸链中5、6、7、8、9或10个核酸能够和第二核酸序列通过氢键互补,则相应的互补百分比为50%、60%、70%、80%、90%或100%。“完全互补”是指核酸序列的全部的连续残基与第二核酸序列上相同数目的全部连续残基依次形成氢键。“基本互补”是指在8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、30、35、40、45、50个或更多的核酸区域内具有与第二核酸序列至少60%、65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%的互补百分比,或者指在严格条件下能够杂交的两个核酸分子。
第一轮扩增中的“引物对”包括一个上游引物和一个下游引物,其中上游引物能够杂交至单链dna上目标区域的第一核酸序列,下游引物能够杂交至单链dna上目标区域的第二核酸序列,其中第一核酸序列在包含目标区域的单链dna上位于第二核酸序列的下游。“杂交”是指包含互补碱基序列的两条dna链,可以通过在互补的碱基序列处彼此形成氢键配对,从而生成稳定的双链区。即,上游引物能够与目标区域的第一核酸序列形成氢键配对从而生成稳定的双链区,下游引物能够与目标区域的第二核酸序列形成氢键配对从而生成稳定的双链区。上游引物和下游引物可以包含任意的可与天然核酸进行碱基配对的核苷酸,包括但不限于a、t、g、和c四种天然碱基以及本领域技术人员公知的其他核苷酸类似物、经修饰的核苷酸等,只要能够与目标区域上的第一或第二核酸序列配对并实现扩增反应即可。
在一些实施方式中,上游引物和/或下游引物分别包含接头序列。接头序列在本申请中是指位于上游引物5’端、下游引物3’端的特定序列,其长度可以在8-40bp、8-32bp、10-30bp、12-28bp、15-25bp、18-22bp、20-24bp之间。上下游引物包含的接头序列可以是相同的或者是不同的。在本申请中,选择适当的接头序列,从而使得所述接头序列不与目标区域结合,并且能够避免上下游引物自身或者上下游引物之间的聚合。在一些实施方式中,后续的指数扩增中所用的扩增引物包括部分或全部与上、下游接头序列互补的序列。在一些实施方式中,选择接头序列以使得半扩增产物/第一轮扩增产物/指数扩增产物能够直接被用于测序。
在一些实施方式中,上游引物和下游引物包含杂交序列。杂交序列在本申请中是指位于上游引物3’端、下游引物5’端的特定序列,其长度可以在10-40bp、15-35bp、18-32bp、20-30bp、22-28bp、24-26bp之间。在本申请的一些实施方式中,“杂交序列”由随机序列组成。在本申请的一些实施方式中,“杂交序列”由至少4个、至少5个、至少6个、至少7个、至少8个连续的随机序列组成。在本申请的一些实施方式中,“杂交序列”由固定序列组成。在本申请的一些实施方式中,“杂交序列”基本由固定序列组成,但在该固定序列的一个或多个碱基位置引入随机序列,上述一个或多个碱基位置可以位于杂交序列的3’或5’端也可以位于杂交序列的中间部分,并且上述一个或多个碱基位置可以是连续的也可以是不连续的。当杂交序列由或基本由固定序列组成时,可以根据目标区域选择适当的固定序列,例如选择与已知目标区域序列上相邻或间隔的两个区域序列互补的序列作为引物中的固定序列。由于基因组中很多位点存在多种突变或者存在单核苷酸多态性(snp),因此当目标区域的第一核酸序列和/或第二核酸序列区域内包括此等位点时,可以在引物的固定序列中引入随机序列从而使得包括不同的位点突变和snp的模板均被扩增,并且使得上述不同的位点突变和snp能够在后续可能存在的测序步骤中被检测出。在本申请的一些实施方式中,上游引物包含由连续的随机序列组成的杂交序列,下游引物包含由特异性结合目标区域的固定序列组成的杂交序列。
“随机序列”中的核苷酸序列可以有多种变化的可能,在引物的某一个特定碱基位置引入随机序列的意思是指,该引物实际上为一个引物混合物,其中包含了在上述特定碱基位置包含不同的核苷酸序列的引物的集合。随机序列中每个碱基位点可以仅包括a、t、g、c中的任意两种、三种或四种核苷酸。可以通过简并标识方法表示该碱基位置的核苷酸种类,例如,在随机序列中的某一碱基位置仅包含a、g两种核苷酸,即可表示为该位点序列为r(即r=a/g),其他的简并标识包括:y=c/t、m=a/c、k=g/t、s=c/g、w=a/t、h=a/c/t、b=c/g/t、v=a/c/g、d=a/g/t、n=a/c/g/t。随机序列的长度可以为1bp、2bp、3bp、4bp、5bp、6bp、7bp、8bp、9bp、10bp或10bp以上。假设随机序列的长度为1bp,该1bp的位置包含的随机核苷酸标识为n,则包含该随机序列的引物为4种引物的混合物。或者假设,随机序列为3bp,其中每个位置包含的随机核苷酸标识为h(3种核苷酸a/c/t),则包含该随机序列的引物为3x3x3=27种引物的混合物。在一些实施方式中,为了排除一些不希望的情况或者增加与目标dna区域的匹配程度,在最大程度的随机性(即包含全部a、t、g、c四种可能性)的基础上进一步增加了某些限定条件,从而选择随机序列上或随机序列中的某个位点上所包含的核苷酸的种类。例如,在某些实施方式中,接头序列包含大量g、t,因而为减少杂交序列与接头序列产生互补配对的可能性,可选择在随机序列中排除包含c、a,随机序列的每个位点以k标识。
此外,本申请中所述的下游引物包含磷酸化的5’末端,即下游引物杂交序列的5’末端包含磷酸基团,而上游引物杂交至的第一核酸序列和下游引物杂交至的第二核酸序列之间间隔了m个核苷酸,其中m为≥0的整数;当m=0时,第一核酸序列与第二核酸序列紧邻,则直接利用dna连接酶连接上游引物的3’端与下游引物的5’获得半扩增产物;或者当m>0时,第一核酸序列与第二核酸序列之间存在间隔,在这种情况下所述第一轮扩增在步骤ii)之后的步骤iii)延伸步骤中,通过dna聚合酶在上游引物3’端延伸m个核苷酸至与下游引物5’端紧邻的位置,获得上游引物的延伸产物,之后再利用dna连接酶将上游引物的延伸产物和下游引物或下游引物的延伸产物相连得到半扩增产物。
本领域技术人员可以根据实际情况选择利用任何公知的dna聚合酶并且采用任何公知的方法对上游引物进行延伸,其中所述dna聚合酶包括但不限于适合的核酸聚合酶包括,但不限于:taq聚合酶、pfudna聚合酶、
本申请所述的“dna连接酶”是指具有可以通过在两个dna分子的借由形成磷酸二酯键将dna在3′端的尾端与5′端的前端连在一起。
在一些实施方式中,所述dna连接酶是t4dna连接酶,其可以催化具有粘端或平端的双链dna或rna的5’-p末端和3’-oh末端之间以磷酸二酯键结合,该催化反应需atp作为辅助因子,但其最佳反应温度在约6℃左右,高于65℃则失去酶活性。t4dna连接酶可以修补双链dna、双链rna或dna/rna杂合物上的单一条链上面具有的缺口。
在一些所述方式中,所述dna连接酶为耐高温的dna连接酶。
在一些实施方式中,所述dna连接酶是耐高温的双链dna连接酶(例如但不限于
在另一些实施方式中,所述dna连接酶是耐高温的单链dna连接酶(例如但不限于circligasetmssdnaligase,epicentre科技公司),其是一种热稳定的、atp依赖的连接酶,其能够催化单链dna的5’-磷酸和3’-羟基基团的连接,从而使单链dna环化。circligasetmssdnaligase与t4dnaligase和不同,t4dnaligase和
当上下游引物均包含接头序列时,一轮线性扩增后得到的扩增产物为一条两端具有接头序列中间为目标区域的反向互补序列的dna链(半扩增产物)与dna模板链之间通过氢键连接形成双链区域的产物分子(见图1)。可以理解的是,当上下游引物的杂交序列由或基本由与目标序列的特定区域反向互补的序列组成时,由于下游引物5’端包含接头序列,3’端包括磷酸基团,在扩增过程中下游引物不能延伸,上游引物延伸至紧邻下游引物3’端时,利用dna连接酶将该上游引物延伸产物与下游引物连接得到半扩增产物与dna模板链之间形成的双链分子,其中在这两条链中,半扩增产物不能作为下一个循环扩增的模板,每个循环的扩增实际只能利用初始的目标dna链作为模板,因此该扩增方式被称为线性扩增。在这种情况下,在第n轮线性扩增后(其中n>1),所述线性扩增产物包括n-1条单链半扩增产物,以及一条半扩增产物与dna模板链之间形成的双链分子。在一些实施方式中,线性扩增重复不少于5个、不少于10个、不少于15个、不少于20个、不少于30个循环。在一些实施方式中,线性扩增重复不高于100个、不高于90个、不高于80个、不高于70个、不高于60个循环、不高于50个循环。
在一些实施方式中,上游引物由5’端至3’端分别包括接头序列和由连续的随机序列组成的杂交序列,下游引物包括由或基本由与目标序列的特定区域反向互补的序列组成的杂交序列且不包含接头序列,在第一轮扩增的第一个循环中得到的半扩增产物为一条5’端具有接头序列、中间为目标区域的反向互补序列的部分或全部的dna链,其与dna模板链之间通过氢键连接形成双链区域的产物分子(见图3)。可以理解的是,与下游引物包含3’端接头序列的情况相反,当上游引物的杂交序列由随机序列组成,下游引物的杂交序列由或基本由与目标序列的特定区域反向互补的序列组成时,上游引物杂交至dna模板链的多个任意位置并向下游延伸,由于下游引物5’端不包含接头序列,在扩增过程中下游引物也向下延伸,当上游引物杂交至下游引物识别区域的上游时,其可继续向下游延伸至紧邻下游引物3’端,随后利用dna连接酶将该上游引物延伸产物与下游引物或下游引物的延伸产物连接得到半扩增产物与dna模板链之间形成的双链分子,由于上游引物的杂交序列由随机序列组成,其也能识别半扩增产物上的位置,因此此时能够以上游引物作为引物进一步将半扩增产物作为下一轮扩增的模板进行扩增。在这种情况下,在第一轮扩增的多个循环后,所述第一轮扩增产物包括多条单链半扩增产物,以及两端分别包含上游引物的接头序列和与上游引物接头序列互补的序列的双链分子。
在一些实施方式中,本申请提供的在初始dna模板上扩增目标区域的方法进一步包括:在n个循环的第一轮扩增之后,以第一轮扩增片段为模板,通过聚合酶链式反应(pcr)扩增所述目标区域以生成指数扩增产物。所述聚合酶链式反应pcr包括但不限于常规pcr、滚环pcr、反向pcr、巢式pcr等。本领域技术人员可以根据实际情况决定pcr的反应温度、反应程序、反应cycle数等反应条件。在指数扩增中可以采用指数扩增通用引物,其包括与第一轮扩增中上游引物的接头序列(如有)相同或反向互补的序列和/或与线性扩增中下游引物的接头序列(如有)相同或反向互补的序列。
在一些实施方式中,本申请提供的在初始dna模板上扩增目标区域的方法包括测序步骤,其中所述测序步骤可以在第一轮扩增之后,即用于测序的序列是第一轮扩增产物;或者其中所述测序步骤可以在指数扩增之后,即用于测序的序列是指数扩增产物。在一些特定的实施方式中,为了使得指数扩增产物能够成为可直接用于测序的dna文库,可以选择第一轮扩增的上下游引物中的接头序列使其包括一段与测序用引物的部分或全部相同或反向互补的特定序列,从而使得第一轮扩增产物能够成为可直接用于测序的dna文库。在一些特定的实施方式中,为了使得指数扩增产物能够成为可直接用于测序的dna文库,指数扩增的上下游引物在其5’端还可以进一步包括一段测序所需的配接序列(adapter),例如但不限于,与测序用引物的部分或全部相同或反向互补的特定序列、与测序用平台的测序板上捕获序列的部分或全部相同或反向互补的特定序列。在一些特定的实施方式中,为了使得指数扩增产物能够成为可直接用于测序的dna文库,可以选择第一轮扩增的上下游引物中的接头序列使其包括一段与测序用引物的部分或全部相同或反向互补的特定序列,相应地指数扩增的引物中包含与线性扩增的上下游引物中的接头序列相同或反向互补的序列,也即指数扩增的引物中包含一段与测序用引物的部分或全部相同或反向互补的特定序列。
本申请中所述的“dna测序文库”是指丰度达到可测序的程度的dna片段集合,其中所述dna片段集合中每条片段的一端或两端包含与测序用引物部分或全部反向互补的特定序列,从而可以直接用于后续的测序上机。
以下将根据附图简要说明本申请所述的dna酶介导的扩增方法的两个示例以及本申请所述的dna酶介导的扩增扩增方法的一种应用。
第一轮扩增
图1是第一轮扩增的一种示例性实施方式,其中所述第一轮扩增为线性扩增。如图1所示,初始dna模板为一条包括目标区域a的双链dna模板,第一轮扩增的引物对中包括一条上游引物和一条下游引物,其中上游引物从5’端到3’端依次为接头序列和杂交序列,并且杂交序列中包含两个不连续碱基位置的随机序列n,其中下游引物从5’端到3’端依次为杂交序列和接头序列,并且下游引物的5’端包含磷酸基团。选择接头序列使其不与dna模板的任何位置发生杂交反应。选择上游引物的杂交序列使其除了在两个随机序列所处的碱基位置以外其余位置与目标区域a内的第一核酸序列完全互补配对。选择下游引物的杂交序列使其与目标区域a内的第二核酸序列完全互补配对。其中在所述目标dna链上所述第一核酸序列位于所述第二核酸序列的下游,并且两个区域之间间隔m个核苷酸。
重复n个循环的扩增步骤(其中n为>1的整数):1)通过高温变性步骤从双链dna模板中分离出目标dna链(例如反义链);2)通过退火步骤将上游引物和下游引物分别杂交至所述目标区域a内的第一核酸序列和第二核酸序列处;3)通过dna聚合酶在上游引物的3’末端进行延伸直至紧邻下游引物5’末端位置,得到上游引物的延伸产物;4)通过耐高温dna连接酶(例如
图2是第一轮扩增的另一种示例性实施方式,如图2所示,初始dna模板为一条包括目标区域a的双链dna模板,第一轮扩增的引物对中包括一条上游引物和一条下游引物,其中上游引物从5’端到3’端依次为接头序列和杂交序列,其中杂交序列中包含多个连续碱基位置的随机序列n,其中下游引物由或基本由与目标序列的特定区域反向互补的序列组成,并且下游引物的5’端包含磷酸基团。选择接头序列使其不与dna模板的任何位置发生杂交反应。选择下游引物的杂交序列使其与目标区域a内的第二核酸序列完全或基本完全互补配对,上游引物的杂交序列与与目标区域a内的第一核酸序列完全或基本完全互补配对,其中在所述目标dna链上所述第一核酸序列位于所述第二核酸序列的下游,并且两个区域之间间隔m个核苷酸。
重复n个循环的线性扩增步骤(其中n为>1的整数):1)通过高温变性步骤从双链dna模板中分离出目标dna链(例如反义链);2)通过退火步骤将上游引物和下游引物分别杂交至所述目标区域a内的第一核酸序列和第二核酸序列处;3)通过dna聚合酶在上游引物和/或下游引物的3’末端进行延伸,上游引物被延伸至紧邻下游引物5’末端位置,得到上游引物的延伸产物;4)通过耐高温dna连接酶(例如
指数扩增
如图3所示,在第一轮扩增后,通过引入指数扩增引物对实现指数扩增,其中所述指数扩增引物对包括指数扩增上游引物和指数扩增下游产物。其中指数扩增上游引物从5’端到3’端依次包括与拟使用的测序平台(例如ngs测序)中的测序上游引物的部分或全部反向互补或相同的序列、与第一轮扩增上下游引物的接头序列(如有)相同或互补的序列。指数扩增的下游引物从5’端到3’端依次包括与拟使用的测序平台(例如ngs测序)中的测序下游引物的部分或全部反向互补或相同的序列、与第一轮扩增上下游引物的接头序列(如有)相同或反向互补的序列。此处所述上下游引物与线性扩增中所述的上下游引物的概念不同,第一轮扩增中的步骤ii中上下游引物杂交至同一条目标dna链上,而指数扩增的上下游引物能够分别杂交至dna双链的两条链上。在一些实施方式中,所述指数扩增上下游引物序列相同。
在指数扩增中的首轮扩增为以第一轮扩增中获得的半扩增产物或第一轮扩增产物为模板进行扩增,在扩增过程中形成的dna双链分子每条链均可再次作为模板进行下一轮的扩增。在经数轮指数扩增后得到的产物为两端均具有与ngs测序平台中的测序引物的部分或全部反向互补或相同的序列,即生成的扩增产物为可以直接用于ngs测序的dna文库。
dna连接酶介导的扩增方法在cnv检测中的应用
如图4所示为上述包括线性扩增和指数扩增步骤的dna连接酶介导的扩增方法在cnv检测中的应用。其中假设目标dna序列为模板dna2,期望测定该dna序列在其来源于的生物样本中的拷贝数。已知参考样本(模板dna1)的拷贝数为2,在平行操作的dna连接酶介导的扩增实验中扩增两种模板dna,如图所示,若模板dna2的拷贝数为3,则经数轮线性扩增再经指数扩增放大后,模板dna2的指数扩增产物的丰度相对于模板dna1的指数扩增产物的丰度为3∶2,由此丰度比值可以反推知模板dna2的拷贝数为3。
应当理解,此处仅为本申请所述扩增方法的一个示例性应用,并不旨在限制本申请所述的扩增方法仅能用于上述检测。本领域技术人员可以根据实际需要灵活选择本申请所述的扩增方法适用的范围。
本申请的另一个方面提供了,一种用于在dna水平上扩增目标区域的试剂盒。在一些实施方式中,所述试剂盒包括:第一轮扩增引物对,其中每对所述引物对包括一个上游引物和一个下游引物,所述上游引物和下游引物杂交至目标dna链上,其中所述上游引物杂交至所述目标区域的第一核酸序列,所述下游引物杂交至所述目标区域的第二核酸序列,其中所述第一核酸序列和所述第二核酸序列之间间隔m个核苷酸,其中m为≥0的整数,其中所述第一核酸序列在所述目标dna链上位于第二核酸序列的下游,并且所述下游引物包含磷酸化的5’末端;以及连接试剂,其中所述连接试剂用于将所述上游引物或其延伸产物与所述下游引物或其延伸产物连接得到半扩增产物。
在一些实施方式中,所述连接试剂包括连接酶及连接酶反应溶液。在一些实施方式中,所述连接酶为耐高温连接酶。在一些实施方式中,所述耐高温连接酶为
在一些实施方式中,用于在dna水平上扩增目标区域的试剂盒进一步包括:延伸试剂,所述延伸试剂用于在所述上游引物和/或下游引物的3’末端以所述反义链作为模板进行延伸,得到上游引物和/或下游引物的的延伸产物。在一些实施方式中,延伸试剂包括dna聚合酶、反应试剂以及dntp,所述dntp由datp、dttp、dgtp、dctp中的任意一种或多种组成。在一些实施方式中,所述上游引物具有5’末端接头序列。在一些实施方式中,所述上游引物具有5’末端接头序列,所述下游引物具有3’末端接头序列。在一些实施方式中,所述上游引物具有5’末端接头序列,所述下游引物不具有接头序列。在一些实施方式中,所述第一轮扩增为线性扩增,所述半扩增产物即为第一轮扩增产物。在一些实施方式中,所述第一轮扩增不为线性扩增,所述上游引物包括的杂交序列包括或为随机序列,所述下游引物包含的杂交序列为特异性识别所述目标区域的杂交序列,所述延伸试剂还可用于当所述上游引物杂交至所述半扩增产物上时,在所述上游引物的3’末端以所述半扩增产物作为模板进行延伸,获得第一轮扩增产物。
在一些实施方式中,用于在dna水平上扩增目标区域的试剂盒进一步包括:指数扩增试剂,所述指数扩增试剂用于以所述第一轮扩增产物为模板扩增所述目标区域得到指数扩增产物。
在一些实施方式中,指数扩增试剂包括dna聚合酶、反应试剂以及dntp。在一些实施方式中,指数扩增试剂进一步包括指数扩增通用引物,其中所述指数扩增通用引物包括与第一轮扩增中所述上游引物的5’末端接头序列相同或反向互补的序列和/或第一轮所述下游引物的3’末端接头序列相同或反向互补的序列。。
在一些实施方式中,用于在dna水平上扩增目标区域的试剂盒进一步包括:测序试剂,所述测序试剂用于对所述半扩增产物或所述第一轮扩增产物或所述指数扩增产物进行测序。在一些实施方式中,指数扩增试剂包括指数扩增通用引物。在一些实施方式中,指数扩增试剂进一步包括dna聚合酶、反应试剂以及dntp。
实施例
以下将通过一些非限制性实施例进一步描述本发明。需要说明的是这些实施例仅用于进一步说明本发明的技术特征,并非旨在也不能够被解释为对本发明的限制。所述实验例不包含本领域一般技术人员所公知的传统方法(不同种类样本中dna的提取、纯化等)的详细描述。
实施例1:连接酶介导的dna扩增方法用于在早期胚胎中检测染色体拷贝数异常
目前试管婴儿的成功率在20-30%之间,染色体非整倍体(染色体数目异常)是试管婴儿失败、流产,以及极少数情况下的异常妊娠和活产的主要原因。提高试管婴儿的成功率,关键在于选择优质胚胎。有数据显示第3天的胚胎约60%存在染色体异常,即只有约40%的胚胎是正常的,因此胚胎植入着床之前,可以对早期胚胎的染色体数目进行检测筛查具有遗传物质异常的胚胎,从而挑选正常的胚胎植入子宫,以期获得正常的妊娠,提高试管婴儿的成功率。
本申请提供的连接酶介导的dna扩增方法可用于准确快速地扩增目标区域,并可通过后续对目标区域的测序来检测这种拷贝数的变异。
初始dna模板
受精卵在体外培养,可以在卵裂期(例如,体外培养24小时内)取单个卵裂球细胞或者在囊胚期(例如,体外培养第3天)取外胚滋养层的多个细胞(1-8个细胞)进行染色体拷贝数异常的检测。采集卵裂细胞或者囊胚外胚滋养层细胞的方法可以为任何本领域技术人员公知的方法,例如但不限于wangl,cramdsetal.validationofcopynumbervariationsequencingfordetectingchromosomeimbalancesinhumanpreimplantationembryos.biolreprod,2014,91(2):37中所述的方法。使用pbs将经分离的卵裂球细胞或滋养层细胞洗涤3次,并用25μl的pbs溶液重悬,作为包含基因组dna的样品溶液直接应用于第一反应溶液体系中。起始的样品溶液中包含约3-24pg总基因组dna。
目标区域
针对全部23条染色体进行检测,其中一个示例性的目标区域为x染色体上的lamp2基因(位于x染色体上从碱基对120442594-120442644)区域,以下所述的引物也是相应地针对该示例性的目标区域的。
参考样本
以已知染色体拷贝数正常的血液样本作为参照进行平行试验。当第一轮扩增为线性扩增,上下游引物均包含接头序列。
线性引物
上游引物(seqidno.3)从5’到3’为接头序列+杂交序列(包含随机序列)。
接头序列为(5’到3’)seqidno.1:5’-cctacacgacgctcttccgatct-3’。
杂交序列为(5’到3’)seqidno.2:5’-cttaccrgagccattaaccaaatac-3’。
下游引物(seqidno.6)从5’到3’为杂交序列(不包含随机序列)+接头序列,并且包含5’磷酸基团。
杂交序列为(5’到3’)seqidno.4:5’-atctgaaggaagtgaacatcagcat-3’。
接头序列为(5’到3’)seqidno.5:5’-gtgactggagttcagacgtgtgc-3’。
线性扩增
在线性扩增中使用耐高温连接酶
指数扩增
线性扩增结束后,可以利用dna纯化试剂盒纯化线性扩增产物后用于指数扩增,或者直接在线性扩增体系中加入指数扩增通用引物对进行指数扩增。
指数扩增上游引物从5’到3’为测序所必要的配接序列+线性扩增上游引物的接头序列的相同序列。
测序所必要的配接序列:
seqidno.7:5’-aatgatacggcgaccaccgagatctacacacactctttc-3’。
线性扩增上游引物的接头序列的相同序列seqidno.8:
5’-cctacacgacgctcttccgatct-3’。
指数扩增上游引物seqidno.9:
5’-aatgatacggcgaccaccgagatctacacacactctttccctacacgacgctcttccgatct-3’。
指数扩增下游引物从5’到3’为测序所必要的配接序列+线性扩增下游引物的接头引物的反向互补序列
测序所必要的配接序列seqidno.10:
5’-caagcagaagacggcatacgagatgatcggaaga-3’。
线性扩增下游引物的接头引物的反向互补序列seqidno.11:
5’-gcacacgtctgaactccagtcac-3’。
指数扩增下游引物seqidno.12:
5’-caagcagaagacggcatacgagatgatcggaagagcacacgtctgaactccagtcac-3’。
在指数扩增中使用的反应条件与经典的聚合酶链反应所用条件的相似,例如使用taq聚合酶,先对dna进行94℃的2分钟初始变性,之后重复30个循环94℃的30秒变性,58℃引物退火20秒和65℃延伸30秒的步骤。
dna测序
由于上述指数扩增步骤中产生的指数扩增产物两端已包含部分或全部与测序用引物互补的序列,因此该指数扩增产物可以被认为是可直接进行测序的dna文库。使用高通量dna测序的方法对dna文库进行测序。测序前可以通过利用寡核苷酸探针对目标测序dna进行富集。
分析测序结果得出参考样本与待检测样本各自的相对丰度,并且通过将参考样本和待检测样本的相对丰度进行比对得出待检测早期胚胎样本中是否存在染色体拷贝数异常。
实施例2:引物的设计及使用
本申请的一种实施方式中,第一轮线性扩增使用一个包含随机序列和接头序列的上游引物a和一个特异性识别目标序列的下游引物b。以下为下游引物b的设计及评估的一个示例。设计特异性识别染色体相关区域而不识别或较少识别基因组的其他位置的引物b。在人基因组数据库,例如hg19数据库中blast设计好的引物b,获得hg19频率,其代表引物b在hg19基因组中的完美匹配数,同时在与疾病对应的目标区域内blast引物,获得mms频率,其代表微重复和微缺失综合征(mms)目标区域内的完美匹配数。当“hg19频率”和“mms频率”一致时,则意味着引物b针对对该mms区域有特异性。在引物具有特异性的前提下,尽量选择在目标mms区域有更多完美匹配数目的引物。本申请中利用上述引物a和引物b对目标区域进行扩增的一种实施方法如下:在第一轮pcr使用一个由5’端到3’端包括接头序列、内切酶识别位点以及随机六聚体组成的上游引物和一个特异性针对目标区域(微重复和缺失综合征(mms)相关的目标区域)的下游引物,通过将上游引物或其延伸产物与下游引物连接得到扩增产物,它包含特定区域(引物b序列)两端分别包含接头序列和接头序列的互补序列的片段集合(第一轮扩增产物)。在第二轮pcr中,使用指数扩增引物c来放大所有的信号获得指数扩增产物,所述指数扩增引物c包含与第一轮扩增上游引物的全部接头序列和部分内切酶识别位点序列。经过两轮pcr,可以使用限制性内切酶消化指数扩增产物,并在凝胶电泳中检验各样本中目标区域的拷贝数,或者可以通过第二代测序检验样本中目标区域的拷贝数情况。
以猫眼综合征(ces)为例,其与第22号染色体上部分区域的拷贝数异常相关。根据上述设计评估原则,能够用于检测猫眼综合征的引物b如表1所示,而相应的引物b在染色体上识别的位置定位如表2所示。
表1:特异性针对猫眼综合征的引物b的评估
表2:特异性针对猫眼综合征的引物b在染色体上识别位置的定位
利用
在第一轮pcr中使用如下的上下游引物:
上游引物a(seqidno.15):5’-gttctacacgagtcactgcagnnnnnnn-3’
下游引物b(seqidno.13):5’-cttcgatcacacg-3’。
在第一轮扩增中使用耐高温连接酶
使用
测序结果中包含引物b的序列的读数量将被记录以表征所述mms区域对应的拷贝数,在测序过程中非mms区域的正常双拷贝区域被用作内参。使用标准z检测来评估mms区域读数量与正常双拷贝区域的读数量的比值以便确认mms区域的拷贝数情况(比值>1代表拷贝数增加,比值<1代表拷贝数减少)。
根据本实施例描述的设计及评估方法,针对不同的cnv相关疾病挑选出了一系列特异性识别相关疾病相关区域的下游引物b,具体如下表3所示。
表3:针对不同cnv相关疾病设计选择的特异性下游引物
应当理解,上述的实验操作仅为示例性的,本领域技术人员可以采用任何市售的试剂盒完成上述任一步骤,其中在这些步骤中任选地使用的试剂、反应条件、反应时间等各不相同但基本原理基本一致。
尽管在上文的实施例中详细地描述了本发明公开的方法的一些具体步骤的操作方法,但是这种描述仅仅是示例性的而并非是对本发明的限制。实际上,根据本发明的实施例,本技术领域的一般技术人员可以通过研究说明书、公开的内容及附图和所附的权利要求书,理解和实施对披露的实施方式的其他改变。在权利要求中,措词“包括”不排除其他的元素和步骤,并且措辞“一”、“一个”不排除复数。说明书中“基本上”是指大于80%、大于85%、大于90%、大于95%、大于98%、或者大于99%的程度。
序列表
<110>广州市基准医疗有限责任公司
<120>dna连接酶介导的dna扩增技术
<130>064938-8003cn02
<150>cn201610206799.9
<151>2016-04-01
<160>139
<170>patentinversion3.5
<210>1
<211>23
<212>dna
<213>人工序列
<220>
<223>合成的
<400>1
cctacacgacgctcttccgatct23
<210>2
<211>25
<212>dna
<213>人工序列
<220>
<223>合成的
<220>
<221>variation
<222>(7)..(7)
<223>r=aorg
<400>2
cttaccrgagccattaaccaaatac25
<210>3
<211>48
<212>dna
<213>人工序列
<220>
<223>合成的
<220>
<221>variation
<222>(30)..(30)
<223>r=aorg
<400>3
cctacacgacgctcttccgatctcttaccrgagccattaaccaaatac48
<210>4
<211>25
<212>dna
<213>人工序列
<220>
<223>合成的
<400>4
atctgaaggaagtgaacatcagcat25
<210>5
<211>23
<212>dna
<213>人工序列
<220>
<223>合成的
<400>5
gtgactggagttcagacgtgtgc23
<210>6
<211>48
<212>dna
<213>人工序列
<220>
<223>合成的
<400>6
atctgaaggaagtgaacatcagcatgtgactggagttcagacgtgtgc48
<210>7
<211>39
<212>dna
<213>人工序列
<220>
<223>合成的
<400>7
aatgatacggcgaccaccgagatctacacacactctttc39
<210>8
<211>23
<212>dna
<213>人工序列
<220>
<223>合成的
<400>8
cctacacgacgctcttccgatct23
<210>9
<211>62
<212>dna
<213>人工序列
<220>
<223>合成的
<400>9
aatgatacggcgaccaccgagatctacacacactctttccctacacgacgctcttccgat60
ct62
<210>10
<211>34
<212>dna
<213>人工序列
<220>
<223>合成的
<400>10
caagcagaagacggcatacgagatgatcggaaga34
<210>11
<211>23
<212>dna
<213>人工序列
<220>
<223>合成的
<400>11
gcacacgtctgaactccagtcac23
<210>12
<211>57
<212>dna
<213>人工序列
<220>
<223>合成的
<400>12
caagcagaagacggcatacgagatgatcggaagagcacacgtctgaactccagtcac57
<210>13
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>13
cttcgatcacacg13
<210>14
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>14
atcgcacacgccc13
<210>15
<211>28
<212>dna
<213>人工序列
<220>
<223>合成的
<220>
<221>misc_feature
<222>(16)..(21)
<223>pstienzymedigestionsite
<220>
<221>variation
<222>(22)..(28)
<223>n=aortorcorg
<220>
<221>misc_feature
<222>(22)..(28)
<223>nisa,c,g,ort
<400>15
gttctacacgagtcactgcagnnnnnnn28
<210>16
<211>19
<212>dna
<213>人工序列
<220>
<223>合成的
<400>16
gttctacacgagtcactgc19
<210>17
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>17
ccgtatcggttcc13
<210>18
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>18
ccaagacccgtac13
<210>19
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>19
taataggaacgcg13
<210>20
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>20
atgtagtcgccgt13
<210>21
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>21
tttcgtagcgtgc13
<210>22
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>22
atactggcgagta13
<210>23
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>23
cggcgccggacaa13
<210>24
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>24
ctcgtcgacccac13
<210>25
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>25
cgcgcggttagca13
<210>26
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>26
cgcggttagcatg13
<210>27
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>27
taagagcgcgttc13
<210>28
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>28
atcgtagtgtacc13
<210>29
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>29
ccgatgtgcgcag13
<210>30
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>30
cgtgcccgcgtca13
<210>31
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>31
cgcctgcgattat13
<210>32
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>32
atctcgatacgat13
<210>33
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>33
cgaatcggacgag13
<210>34
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>34
aatcggacgagac13
<210>35
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>35
actatggtatccg13
<210>36
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>36
cgctatacggact13
<210>37
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>37
tcgccgccggttc13
<210>38
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>38
gccgccggttcta13
<210>39
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>39
taaatcacggcgg13
<210>40
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>40
gcgcgcttagcta13
<210>41
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>41
gcgcactatcgat13
<210>42
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>42
cacgatacggcca13
<210>43
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>43
agcgcactatcga13
<210>44
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>44
agtccaattcgtg13
<210>45
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>45
gtcggcgaccctt13
<210>46
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>46
aggacgacgctac13
<210>47
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>47
tcgtctggtacga13
<210>48
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>48
taaagggacgcgc13
<210>49
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>49
cgtggttcgcggc13
<210>50
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>50
tcctaacgcgccg13
<210>51
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>51
cagtcgctcggtt13
<210>52
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>52
ccgtggttcgcgg13
<210>53
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>53
atgcgcgcatgtc13
<210>54
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>54
cgggtccacgact13
<210>55
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>55
ctcgcgagtgtac13
<210>56
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>56
tcgcgagtgtaca13
<210>57
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>57
ttgcgtgacgcag13
<210>58
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>58
caacggcgttgct13
<210>59
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>59
aacggcgttgcta13
<210>60
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>60
gcgttgctacacg13
<210>61
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>61
acgagtcgtcgat13
<210>62
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>62
cggctaaacccgc13
<210>63
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>63
gcggctcgtgcgt13
<210>64
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>64
acgagtcgtcgat13
<210>65
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>65
cggctaaacccgc13
<210>66
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>66
gcggctcgtgcgt13
<210>67
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>67
cactaactcgcgc13
<210>68
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>68
cactaactcgcgc13
<210>69
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>69
actaactcgcgcc13
<210>70
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>70
actaactcgcgcc13
<210>71
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>71
tcacgtacaccgc13
<210>72
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>72
acgtacaccgcag13
<210>73
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>73
cggctacggagat13
<210>74
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>74
acgcaaaggcgac13
<210>75
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>75
ctcttagccggtt13
<210>76
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>76
tagccggttaggg13
<210>77
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>77
cccgccgacggtc13
<210>78
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>78
ccgacggtctcgc13
<210>79
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>79
cagtgaaacgacg13
<210>80
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>80
cagcggcgatcgg13
<210>81
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>81
gaacggagcgcat13
<210>82
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>82
gcttaggcgctta13
<210>83
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>83
gtcgagcctcgag13
<210>84
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>84
tcgagcctcgagt13
<210>85
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>85
cggacggcatttc13
<210>86
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>86
tataacgtacgaa13
<210>87
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>87
aacgtacgaagtc13
<210>88
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>88
gtactatcgaacg13
<210>89
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>89
ttagtcggcttcg13
<210>90
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>90
cacgcgatgcaac13
<210>91
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>91
tttgcggcgtttc13
<210>92
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>92
acacggtcggtaa13
<210>93
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>93
cgttcgaacgtgg13
<210>94
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>94
tcgaacgtggcga13
<210>95
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>95
accgccctcgcat13
<210>96
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>96
cgaataccgccct13
<210>97
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>97
cgaccatgcggct13
<210>98
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>98
acgcaacgcctcc13
<210>99
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>99
cgagcttcgtgcg13
<210>100
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>100
cgtgtagctcgat13
<210>101
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>101
gagtgatacgcgg13
<210>102
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>102
gtgatacgcggac13
<210>103
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>103
tgatacgcggaca13
<210>104
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>104
gatacgcggacaa13
<210>105
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>105
tcgcacagacgtt13
<210>106
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>106
cggcggcgatctt13
<210>107
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>107
atcgcacacgccc13
<210>108
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>108
cttcgatcacacg13
<210>109
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>109
gccgaacccgaga13
<210>110
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>110
cacgagtcggtgc13
<210>111
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>111
tacgcccaggtat13
<210>112
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>112
atcgggacccgat13
<210>113
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>113
atcgagggttacc13
<210>114
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>114
tcgccgtccacga13
<210>115
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>115
ctgtcgtgtgcgg13
<210>116
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>116
atgcgtgaagtcg13
<210>117
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>117
atgactccgacgt13
<210>118
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>118
cggttgaataagc13
<210>119
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>119
acgctgaaatcgg13
<210>120
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>120
gtacgcgggctta13
<210>121
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>121
tatcggacaaggc13
<210>122
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>122
atcatgaacgacg13
<210>123
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>123
attgtcaacgacc13
<210>124
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>124
tagggttaccgcc13
<210>125
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>125
aggtagtcgccta13
<210>126
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>126
tcgacaacgtttc13
<210>127
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>127
tcgaacactggta13
<210>128
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>128
gctcgttatagat13
<210>129
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>129
ttcgaattgaccg13
<210>130
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>130
cgaattgaccgta13
<210>131
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>131
tcgtgcttcgggc13
<210>132
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>132
cggagtctacggg13
<210>133
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>133
gcggttaactagt13
<210>134
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>134
cgattgccaaacg13
<210>135
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>135
gatgtcgataacc13
<210>136
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>136
aatcgtctatgcg13
<210>137
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>137
atcgtctatgcgc13
<210>138
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>138
cgccgtatgcaac13
<210>139
<211>13
<212>dna
<213>人工序列
<220>
<223>合成的
<400>139
gccttaatccgct13
- 还没有人留言评论。精彩留言会获得点赞!
- 一种霍乱弧菌环介导等温扩增试剂盒及其应用的制造方法与工艺
- 用于检测番茄晚疫病菌的LAMP引物组合物和应用的制造方法与工艺
- 基于颜色判定的环介导等温扩增(LAMP)技术检测雪松疫霉根腐病菌的制造方法与工艺
- Sortase A介导的(S)‑羰基还原酶寡聚体高效催化(S)‑苯基乙二醇的合成的制造方法与工艺
- 用于旋转机器中的超导线圈的等温支承部和真空容器的制造方法与工艺
- 基于环介导等温扩增消光技术的转基因玉米MIR604检测方法与制造工艺
- 基于环介导等温扩增消光技术的玉米检测方法与制造工艺
- 一套鉴定甘蔗上三种短体线虫的环介导等温扩增引物及其试剂盒的制造方法与工艺
- RNA扩增方法与制造工艺
- 用于扩增核酸样品的等温方法与制造工艺
- 吡啶胺基嘧啶衍生物甲磺酸盐的结晶形式及其制备和应用的制造方法与工艺
- 吡啶胺基嘧啶衍生物的盐及其制备方法和应用与流程
- 用于识别超氧阴离子的荧光探针及其制备方法和应用、光纤探头及其制备方法与流程
- 磁性复合吸附剂去除废水中四环素的方法与流程
- MIIP pS303阻断剂在制备抗肿瘤药物中的应用的制造方法与工艺
- 阻断UBE2M介导Neddylation修饰在肾细胞癌治疗中的应用的制造方法与工艺
- 一种治疗过敏性鼻炎的药物组合物的制造方法与工艺
- 一种皮蛋蛋白源性抗炎肽饮料及其制备方法与流程
- 活性丙烯酰胺类化合物的制造方法与工艺
- 2‑(4‑(4‑乙氧基‑6‑氧代‑1,6‑二氢吡啶‑3‑基)‑2‑氟苯基)‑N‑(5‑(1,1,1‑三氟‑2‑甲基丙‑2‑基)异噁唑‑3‑基)乙酰胺的晶型的制造方法与工艺