酵母中RAS/CAMP/PKA信号通路的调节的制作方法
本公开涉及酵母细胞的选择以及重组酵母宿主细胞的基因工程,以在保持其繁殖效率的同时提高其发酵效率。
背景技术
在需氧、葡萄糖受限、补料分批酵母生产(aerobic,glucose-limited,fedbatchyeastproduction)期间,繁殖的酵母必须优选保持初始接种物中存在的相同(或更好)的所需品质。在采用重组酵母生产燃料乙醇时,已经确定的多种菌株,它们在酵母繁殖后具有非所需的表型(较差的乙醇耐受性,高糖残留)。
提供一种展现出对培养基发酵能力改进而同时保持其在高细胞密度下的繁殖效率的酵母菌株,将是高度合乎需要的。
技术实现要素:
本公开涉及基于在其ras/camp/pka途径中表现出信号活性增加而选择的或经过基因工程化以在其ras/camp/pka途径中表现出信号活性增加的酵母细胞。在一些实施方式中,当与相应野生型ras蛋白相比时,这些酵母细胞表达具有增加的活性的突变ras1蛋白和/或突变ras2蛋白。在其他实施方式中,这些酵母细胞表达具有对所述野生型ras1或所述野生型ira蛋白降低的抑制活性的突变ira1蛋白和/或突变ira2蛋白。在所述酵母的发酵期间优选观察到这些表达/活性差异。
根据第一方面,本公开提供了对发酵培养基进行发酵以获得发酵产物的方法。广义上而言,所述方法包括(i)可选地在繁殖培养基中繁殖第一酵母细胞和/或第二重组酵母宿主细胞从而获得酵母细胞的繁殖群体;和(ii)使所述第一酵母细胞,所述第二重组酵母宿主细胞和/或所述繁殖群体与所述发酵培养基在允许产生所述发酵产物的条件下接触。在所述方法中,所述第一酵母细胞由于能够在ras/camp/pka途径中表现出增加的信号(并且在一些实施方式中,这种特征在发酵期间观察到而在繁殖期间未观察到)而被选出。此外,所述第二重组酵母宿主细胞包含对能够增加所述ras/camp/pka途径中的信号(并且在一些实施方式中,在发酵过程中观察到这种特征,而在繁殖期间未观察到这种特征)的异源蛋白进行编码的异源核酸分子。而且,所述繁殖群体包含所述第一酵母细胞或所述第二重组酵母宿主细胞中的至少之一。在一个实施方式中,所述方法包括使所述第一酵母细胞或包含所述第一酵母细胞的所述繁殖群体与所述繁殖培养基接触。在另一个实施方式中,所述方法进一步包括,在繁殖步骤和/或接触步骤之前或期间,从所述酵母细胞群体中选择出所述第一酵母细胞。在还有的另一个实施方式中,所述方法进一步包括,在繁殖步骤和/或接触步骤之前或期间,从所述繁殖培养基和/或发酵培养基中排除或去除缺乏在所述ras/camp/pka途径中表现出信号增加的能力的酵母细胞。在另一个实施方式中,所述第一酵母细胞由于能够表达与野生型ras2蛋白相比具有增加的活性的突变ras2蛋白而被选出或已经被选出。在另一个实施方式中,与野生型ras2蛋白相比,所述突变ras2蛋白包含至少一个氨基酸残基变异。在还有的另一个实施方式中,所述突变ras2蛋白具有seqidno:2的氨基酸序列或由具有seqidno:3的序列的核酸分子进行编码。在另一个实施方式中,所述第一酵母细胞因为能够表达具有与野生型ira2蛋白相比对野生型ras1蛋白和/或野生型ras2蛋白降低的抑制活性的突变ira2蛋白而被选出或已经被选出。在另一个实施方式中,所述突变ira2蛋白是野生型ira2蛋白的片段。在还有的另一个实施方式中,所述突变ira2蛋白具有seqidno:9的氨基酸序列或由具有seqidno:7的序列的核酸分子进行编码。在一个实施方式中,所述方法包括使所述第二重组酵母宿主细胞或包含所述第二重组酵母宿主细胞的繁殖群体与所述繁殖培养基接触。在还有的一个实施方式中,所述异源核酸分子包含对与野生型ras2蛋白相比具有增加的活性的突变ras2蛋白进行编码的第一核酸。在还有的另一个实施方式中,与野生型ras2蛋白相比,所述突变ras2蛋白包含至少一个氨基酸残基变异,并且在还有的进一步的实施方式中,具有seqidno:2的氨基酸序列或由具有seqidno:3的序列的核酸分子进行编码。在还有的另一个实施方式中,所述异源核酸分子包含对与野生型ira2蛋白相比对野生型ras1蛋白和/或野生型ras2蛋白具有降低的抑制活性的突变ira2蛋白进行编码的第二核酸分子。例如,所述突变ira2蛋白能够是所述野生型ira2蛋白的片段和/或具有seqidno:9的氨基酸序列或由具有seqidno:7的序列的核酸分子进行编码。在一个实施方式中,所述异源核酸分子进一步包含与所述第一核酸分子和/或所述第二核酸分子可操作地连接的启动子,并且与在繁殖期间所述第一核酸分子和/或所述第二核酸分子的表达相比,所述启动子能够提高在发酵期间所述第一核酸分子和/或所述第二核酸分子的表达。在一些实施方式中,所述启动子不是来自ras2基因的ras2启动子或来自ras1基因的ras1启动子。在一个进一步的实施方式中,与所述第二重组酵母宿主细胞处于需氧条件下时获得的所述第一核酸分子和/或所述第二核酸分子的表达水平相比,当所述第二重组酵母宿主细胞处于至少部分厌氧条件下时,所述启动子能够提高所述第一核酸分子和/或所述第二核酸分子的表达(例如,所述启动子可以是来自dan1基因的dan1启动子或来自anb1基因的anb1启动子)。在还有的另一个实施方式中,所述启动子是葡萄糖抑制型启动子(例如,所述启动子能够是来自hxk1基因的hxk1启动子)。在还有的另一个实施方式中,所述第一酵母细胞和/或所述第二重组酵母宿主细胞包含编码野生型ras2蛋白的ras2基因的至少一个拷贝。在一个实施方式中,所述第一酵母细胞或所述第二重组酵母宿主细胞能够利用木糖和/或阿拉伯糖。在一个实施方式中,所述发酵产物是乙醇。在另一个实施方式中,所述第一酵母细胞和/或所述第二重组酵母宿主细胞来自酵母属(saccharomyces),克鲁维酵母属(kluyveromyces),阿氏酵母属(arxula),德巴利酵母属(debaryomyces),假丝酵母属(candida),毕赤酵母属(pichia),法夫酵母属(phaffia),裂殖酵母属(schizosaccharomyces),汉逊氏酵母属(hansenula),克勒克氏酵母属(kloeckera),许旺氏酵母属(schwanniomyces)或耶氏酵母属(yarrowia)。在一个实施方式中,所述第一酵母细胞和/或所述第二重组酵母宿主细胞来自酵母属(saccharomyces),在另一个实施方式中,能够来自酿酒酵母种(saccharomycescerevisiae)。
根据第二方面,本公开提供了包含具有旨在与编码突变ras2蛋白的第一核酸分子可操作地连接的启动子的第一异源核酸分子的重组酵母宿主细胞。在这种重组酵母宿主细胞中,所述启动子能够与所述第一核酸分子可操作地连接。在所述重组酵母宿主细胞中,与在繁殖期间所述第一核酸分子和/或所述第二核酸分子的表达相比,所述启动子能够提高在发酵期间所述第一核酸分子和/或所述第二核酸分子的表达。还有在所述重组酵母宿主细胞中,与相应野生型ras2蛋白相比,所述突变ras2蛋白表现出增加的活性。在一个实施方式中,所述重组酵母宿主细胞进一步包含在所述ras2启动子控制之下编码所述野生型ras2蛋白的ras2基因的至少一个拷贝。在还有的另一个实施方式中,所述第一异源核酸分子进一步包含所述第一核酸分子。在另一个实施方式中,所述启动子不是来自ras2基因的ras2启动子。在还有的另一个实施方式中,相比于在所述重组酵母宿主细胞置于需氧条件下时获得的所述第一核酸分子的表达水平,所述启动子能够提高在所述重组酵母宿主细胞处于至少部分厌氧条件时所述第一核酸分子的表达(例如,所述启动子可以是来自dan1基因的dan1启动子或来自anb1基因的anb1启动子)。在还有的另一个实施方式中,所述启动子是葡萄糖抑制型的(例如,所述启动子能够是来自hxk1基因的hxk1启动子)。在一些实施方式中,所述野生型ras2蛋白具有seqidno:1的氨基酸。在另一个实施方式中,相比于所述野生型ras2蛋白,所述突变ras2蛋白具有至少一个氨基酸取代,在一个进一步的实施方式中,能够具有seqidno:2的氨基酸序列或由具有seqidno:3的序列的核酸分子进行编码。在另一个实施方式中,所述重组酵母宿主细胞能够包含含有旨在可操作地连接至对与野生型ira2蛋白相比对于野生型ras2蛋白具有降低的抑制活性的突变ira2蛋白进行编码的第二核酸分子的所述启动子的第二异源核酸分子。在一个实施方式中,所述突变ira2蛋白是所述野生型ira2蛋白的片段,并且在一个进一步的实施方式中,具有seqidno:9的氨基酸序列或由具有seqidno:7的序列的核酸分子进行编码。在一个实施方式中,所述第二异源核酸分子包含所述第二核酸分子。在还有的另一个实施方式中,所述重组酵母宿主细胞能够利用木糖和/或阿拉伯糖。在还有的一个进一步的实施方式中,所述重组酵母宿主细胞能够包含对异源木糖异构酶进行编码的第三异源核酸。在还有的另一个实施方式中,所述重组酵母宿主细胞能够来自酵母属,克鲁维酵母属,阿氏酵母属,德巴利酵母属,假丝酵母属,毕赤酵母属,法夫酵母属,裂殖酵母属,汉逊氏酵母属,克勒克氏酵母属,许旺氏酵母或耶氏酵母属,例如,来自酵母属。在还有的另一个实施方式中,所述重组酵母宿主细胞能够来自酿酒酵母种。
根据第三方面,本公开提供了在繁殖培养基中繁殖酵母细胞的方法,所述方法包括在允许所述重组酵母宿主细胞繁殖的条件下使本文描述的所述重组酵母宿主细胞与所述繁殖培养基接触。在一个实施方式中,所述繁殖培养基包含糖蜜(molasses)。
根据第四方面,本发明提供了使发酵培养基发酵从而获得发酵产物的方法。所述方法包括(i)可选地繁殖本文描述的所述重组酵母宿主细胞从而获得重组酵母宿主细胞繁殖群体;和(ii)在允许产生发酵产物的条件下使所述重组酵母宿主细胞和/或所述繁殖群体与所述发酵培养基接触。在一个实施方式中,所述发酵培养基包含木质纤维素材料,木糖,阿拉伯糖和/或麦芽糖。在另一个实施方式中,所述发酵产物是乙醇。在还有的另一个实施方式中,所述繁殖培养基包含糖蜜。
附图说明
因此经过对本发明本质进行一般性描述,现在将参考附图,它们通过图示说明方式揭示其优选实施方式,并且其中:
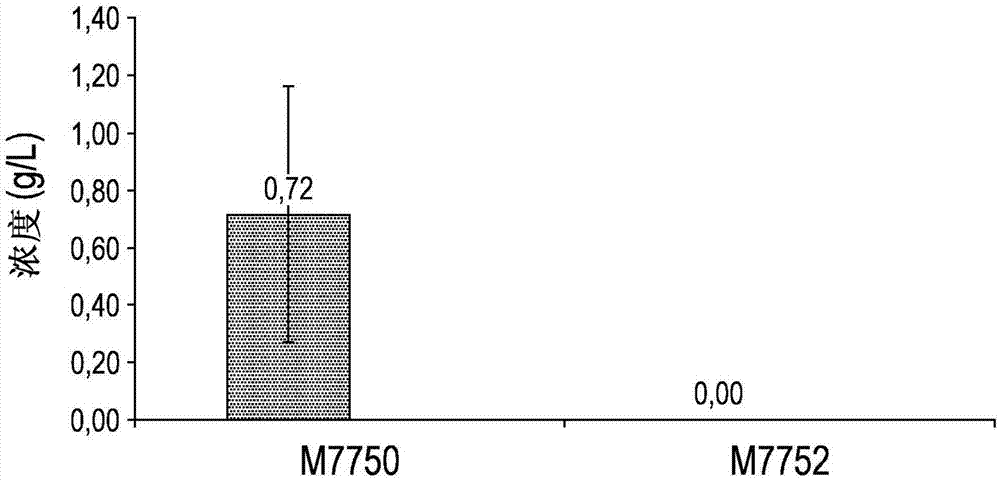
图1图示说明了作为所使用的酵母菌株的函数在葡萄糖/麦芽糖培养基发酵期间的葡萄糖消耗。结果显示为对于所测试的各种酿酒酵母菌株(m7750或m7752)在发酵1.5h(灰色条)或4h(白色条)的培养基中的残余葡萄糖浓度(以g/l计)。
图2图示说明了作为所使用的酵母菌株的函数在葡萄糖/麦芽糖培养基发酵期间的麦芽糖消耗。结果显示为对于所使用的各种酿酒酵母菌株(m7750或m7752)在发酵1.5h(灰色条)或4h(白色条)的培养基中残余麦芽糖浓度(以g/l计)。
图3图示说明了作为所使用的酵母菌株的函数在葡萄糖/麦芽糖培养基发酵期间的乙醇产量。结果显示为对于所使用的各种酿酒酵母菌株(m7750或m7752),在发酵1.5h(灰色条)或4h(白色条)的培养基中产生的乙醇浓度(以g/l计)。
图4图示说明了作为所使用的酵母菌株的函数在葡萄糖/麦芽糖培养基发酵期间的甘油产量。结果显示为对于所使用的各种酿酒酵母菌株(m7750或m7752)在发酵1.5h(灰色条)或4h(白色条)的培养基中产生的甘油浓度(以g/l计)。
图5图示说明了作为所使用的酵母菌株的函数在仅有葡萄糖的培养基中培养期间的camp水平。结果显示为对于所使用的所述各种酿酒酵母菌株(m7750或m7752)在细胞收集之前在存在(白色条)或不存在(灰色条)100mm葡萄糖尖峰(glucosespike)时在含有葡萄糖作为碳源的酵母提取物蛋白胨培养基中发酵8小时后的camp水平(pm/g)。
图6图示说明了对于所使用的所述各种酿酒酵母菌株(m7750或m7752)在图5中所示的所述葡萄糖尖峰与基部camp水平的比率(ratiosoftheglucosespike-to-basalcamplevels)。
图7比较了对于厌氧生长的酿酒酵母菌株m8349(实线(regularline))或m12076(虚线)作为发酵小时数的函数的co2产量(以ml计)。
图8比较了作为厌氧生长的所用酿酒酵母菌株(m8349或m12076)的函数在培养基中的残留葡萄糖(g/l,白色条),甘油产量(g/l,灰色条)和乙醇产量(g/l,▲)。
图9a和图9b比较了作为酿酒酵母菌株m12196(实线)和m12543(虚线)中发酵小时数的函数的co2产量(以ml计)。发酵在ypd10x70培养基(a)或sp3培养基(b)中进行。
图10a和图10b比较了在各种酿酒酵母菌株(m11321,m12557,m12621(例如,在所述ras2启动子控制下表达突变ras2a66t蛋白的m12557),m12624(例如,在所述dan1启动子控制下表达突变ras2a66t蛋白的m12557)或m12625(例如,在anb1启动子控制下表达突变ras2a66t蛋白的m12557))发酵的sp3培养基中的(图10a)co2产量(以ml计),(图10b)残留木糖(g/l,灰色条)和乙醇产量(g/l,▲)。
图11图示说明了在由各种酿酒酵母菌株(m11321,m12557,m12621和m12624)发酵的sp4培养基中的残留木糖(以g/l计,灰色条)和乙醇产量(以g/l计,▲表示)。
图12图示说明了对于各种酿酒酵母菌株(m2390,m11321,m12557,m12621,m12624,m12625或m12627)在两种不同糖蜜培养基上有氧繁殖期间的干细胞重量(以g/l计)产量:csl/dap(上图,白色条)或脲/mgso4(下图,灰色条)。
图13图示说明了对于各种酿酒酵母菌株(m12359,m13414,m13570或m13578)在ypd5a35培养基中的发酵动力学。结果提供为作为发酵小时数的函数的每平方英寸尺度的累积磅数(cumulativepoundspersquareinchgauge)(作为co2产量的代表(proxy))。
图14提供了对于各种酿酒酵母菌株(m12359,m13414,m13570或m13578)发酵70小时后的终点代谢物的高效液相色谱(hplc)分析。结果提供为作为所使用的酿酒酵母菌株的函数以g/l计的残留葡萄糖(黑色条),阿拉伯糖(白色条)或乙醇(阴影条)。
具体实施方式
根据本公开,提供了在发酵期间具有在ras/camp/pka途径中增加的信号活性的酵母细胞。在一些实施方式中,这种信号活性的增加限于发酵(例如,在厌氧条件下)并且在繁殖期间(例如,如在葡萄糖受限的需氧条件下)并未观察到。所述酵母细胞能够从酵母细胞群体中(基于其在ras/camp/pka途径中表现出增加的信号活性的能力)进行选择,并且在一个进一步的实施方式中,能够是天然存在的。所述酵母细胞能够包括一种或多种遗传修饰以增加其在ras/camp/pka途径中的信号活性。
具有在ras/camp/pka途径中增加的信号活性的酵母菌株
本公开提供了在发酵期间具有在ras/camp/pka途径中增加的信号活性的酵母细胞。正如在本公开的上下文中所用,具有“在ras/camp/pka途径中增加的信号活性”的酵母细胞或菌株表现出在所述ras/camp/pka途径中的一种或多种蛋白中生物活性增加,最终导致与相应的对照酵母细胞相比,camp产量提高。正如本领域已知的是,由这种生物途径引起的camp增加会导致pka蛋白解离成bcy1蛋白和tpk1-3蛋白。然后,所述解离的tpk1-3蛋白有利于发酵和海藻糖迁移。ras/camp/pka信号的这种增加优选在发酵期间(例如,在厌氧条件下)观察到,并且在一些实施方式中,在繁殖期间(例如,在葡萄糖受限的需氧条件下)并未观察到。
为了实现ras/camp/pka信号的这种增加,能够提高所述ras/camp/pka途径的一种或多种蛋白的表达和/或活性(与相应的对照酵母细胞或菌株相比时)。能够提高其表达或生物活性的一种或多种蛋白包括,但不限于,cdc25蛋白(能够激活ras1蛋白和/或ras2蛋白的膜结合鸟嘌呤核苷酸交换因子),sdc25蛋白(能够激活ras1蛋白和/或ras2蛋白的ras鸟嘌呤核苷酸交换因子),ras1蛋白(其活性会提高所述cyr1蛋白活性的gtp酶)和/或ras2蛋白(其活性会提高所述cyr1的活性的gtp酶))。
在一个实施方式中,提高所述ras2蛋白表达或其生物学活性以引起所述ras/camp/pka途径的所述信号活性的增加。在这种实施方式中,所述酵母细胞能够在所述ras2蛋白中表达突变(本文称为突变ras2蛋白),这会增加其生物活性。例如,所述突变ras2蛋白能够是所述野生型ras2蛋白的变体或片段,这导致所述ras2蛋白的生物活性增加。所述ras2蛋白是gtp酶,因此其生物活性包括与gtp结合和将gtp水解成gdp。因此,在本公开的上下文中,与所述野生型ras2蛋白相比,具有增加的(生物学)活性的突变ras2蛋白能够表现出对gtp的更高结合亲和力,更高的gtp水解活性,或这两者。在一个实施方式中,所述突变ras2蛋白能够具有一个或多个氨基酸取代。例如,所述突变ras2蛋白能够在对应于seqidno:1的位置66的残基(或另一种野生型ras2蛋白中的相应残基)处具有氨基酸取代。在一个实施方式中,所述突变ras2蛋白的氨基酸取代限于位于seqidno:1的66位的残基(或另一种野生型ras2蛋白中的相应残基)。在酿酒酵母的所述野生型ras2蛋白(seqidno:1)中,66位的所述氨基酸残基是丙氨酸残基。在一个实施方式中,所述突变ras2蛋白不具有位于seqidno:1的66位(或在另一种野生型ras2蛋白的相应位置)上的丙氨酸残基,而相反具有组氨酸,异亮氨酸,精氨酸,亮氨酸,天冬酰胺,赖氨酸,天冬氨酸,蛋氨酸,半胱氨酸,苯丙氨酸,谷氨酸,苏氨酸,谷氨酰胺,色氨酸,甘氨酸,缬氨酸,脯氨酸,丝氨酸或酪氨酸残基。在一个实施方式中,所述突变ras2蛋白在seqidno:1的66位(或在另一野生型ras2蛋白的相应位置)不具有脂肪族氨基酸残基如,例如,甘氨酸,缬氨酸,亮氨酸或异亮氨酸残基。在还有的另一个实施方式中,所述突变ras2蛋白在seqidno:1的66位(或在另一种野生型ras2蛋白相应位置)上具有含羟基或硫/硒的氨基酸如,例如,丝氨酸,半胱氨酸,苏氨酸或甲硫氨酸残基。在还有的另一个实施方式中,所述突变ras2蛋白在seqidno:1的66位(或在另一种野生型ras2蛋白的相应位置)上具有苏氨酸残基。在还有的一个进一步的实施方式中,所述突变ras2蛋白具有seqidno:2的所述氨基酸序列,并能够由具有含有seqidno:3或seqidno:5的序列的核酸分子的核酸分子进行编码。
在一个实施方式中,提高所述ras1蛋白的表达或生物活性以引起所述ras/camp/pka途径的信号活性增加。在这种实施方式中,所述酵母细胞能够在所述ras1蛋白中表达突变(本文称为突变的ras1蛋白),这会提高其生物活性。例如,所述突变ras1蛋白能够是所述野生型ras1蛋白的变体或片段,这会导致ras1蛋白的生物活性增加。所述ras1蛋白是gtp酶,因此其生物活性包括与gtp结合和将gtp水解成gdp。因此,在本公开的上下文中,与所述野生型ras1蛋白相比,具有增加的(生物学)活性的所述突变ras1蛋白能够表现出对gtp的更高结合亲和力,更高的gtp水解活性,或这两者。在一个实施方式中,所述突变ras1蛋白能够具有氨基酸取代。例如,正如temelesetal.,1985中所述,所述突变ras1蛋白能够在对应于seqidno:10的位置66的残基处(或在另一种野生型ras1蛋白的相应残基处)具有氨基酸取代。在一个实施方式中,所述突变ras1蛋白的所述氨基酸取代限于位于seqidno:10的66位处的所述残基(或另一种野生型ras1蛋白中的相应残基)。在酿酒酵母的所述野生型ras1蛋白(seqidno:10)中,66位处的所述氨基酸残基是丙氨酸残基。在一个实施方式中,所述突变ras1蛋白不具有位于seqidno:10的66位处(或另一种野生型ras1蛋白的相应位置上)的丙氨酸残基,而相反却具有组氨酸,异亮氨酸,精氨酸,亮氨酸,天冬酰胺,赖氨酸,天冬氨酸,蛋氨酸,半胱氨酸,苯丙氨酸,谷氨酸,苏氨酸,谷氨酰胺,色氨酸,甘氨酸,缬氨酸,脯氨酸,丝氨酸或酪氨酸残基。在一个实施方式中,所述突变ras1蛋白在seqidno:10的66位上(或在另一种野生型ras1蛋白的相应位置上)不具有脂族氨基酸残基如,例如,甘氨酸,缬氨酸,亮氨酸或异亮氨酸残基。在还有的另一个实施方式中,所述突变ras1蛋白在seqidno:1的66位上(或在另一种野生型ras1蛋白的相应位置上)具有含羟基或硫/硒的氨基酸如,例如,丝氨酸,半胱氨酸,苏氨酸或甲硫氨酸残基。在还有的另一个实施方式中,所述突变ras1蛋白在seqidno:1的66位上(或在另一种野生型ras1蛋白的相应位置上)具有苏氨酸残基。在还有的一个进一步的实施方式中,所述突变ras1蛋白具有seqidno:11的所述氨基酸序列。
在另一个实例中,可以降低所述ras/camp/pka途径的一种或多种蛋白的表达和/或活性(与相应的对照酵母细胞或菌株相比时)以实现所述信号活性ras/camp/pka途径中的增加。能够降低其表达或生物活性的一种或多种蛋白包括,但不限于,ira1蛋白(gtp酶活化蛋白,其活性会降低所述野生型ras1蛋白和/或所述野生型ras2蛋白的活性)和/或ira2蛋白(gtp酶活化蛋白,其活性会降低所述野生型ras1蛋白和/或所述野生型ras2蛋白的活性)。
正如上所述,在一个实施方式中,能够降低所述ira2蛋白的表达和/或活性以在所述酵母细胞中实现所述ras/camp/pka途径中的信号活性的增加。在一个实施方式中,所述酵母细胞会表达所述ira2蛋白中的突变(本文称为突变ira2蛋白),这会降低其生物活性。例如,所述突变ira2蛋白能够是野生型ira2蛋白的变体或片段,会导致所述野生型ras1蛋白和/或所述野生型ras2蛋白的生物活性增加。正如本领域已知的是,所述ira2蛋白会将所述野生型ras1蛋白或所述野生型ras2蛋白从其gtp结合形式转化成其gdp结合失活形式。所述ira2蛋白的所述生物活性包括与所述野生型ras1蛋白结合以及与所述野生型ras2蛋白结合。因此,在本公开的上下文中,与所述野生型ira2相比,具有降低的(生物学)活性的突变ira2蛋白能够表现出对所述野生型ras1蛋白,所述野生型ras2蛋白或这两者具有较低的结合亲和力。在一个具体实施方式中,所述突变ira2蛋白能够是由核酸分子或基因(包括移码(frame-shift)突变)编码的截短ira2蛋白。所述突变ira2蛋白能够具有seqidno:9的所述氨基酸序列或由具有seqidno:7的所述序列的核酸分子进行编码。
在一个实施方式中,能够降低所述ira1蛋白的表达和/或活性以在所述酵母细胞或菌株中实现所述ras/camp/pka途径中的信号活性的增加。在一个实施方式中,所述酵母细胞会表达所述ira1蛋白中的突变(本文称为突变ira1蛋白),这会降低其生物活性。例如,所述突变ira1蛋白能够是所述野生型ira1蛋白的变体或片段,这会导致所述野生型ras1蛋白和/或所述野生型ras2蛋白的生物活性增加。所述ira1蛋白将所述野生型ras1蛋白或所述野生型ras2蛋白从其gtp结合形式转化成其gdp结合失活形式。所述ira1蛋白的生物活性包括与所述野生型ras1蛋白结合和/或与所述野生型ras2蛋白结合。因此,在本公开的上下文中,具有降低的(生物活性)的突变ira1蛋白能够表现出对所述野生型ras1蛋白,所述野生型ras2蛋白或这两者具有较低的结合亲和力。在一个具体实施方式中,所述突变ira1蛋白能够是由核酸分子或基因(包括移码突变)编码的截短ira1蛋白。
在还有的另一个实例中,可以提高所述ras/camp/pka途径的一种或多种蛋白的表达和/或活性以及可以降低所述ras/camp/pka途径的一种或多种蛋白的表达和/或活性(均与相应的对照酵母细胞或菌株相比)以实现所述信号活性ras/camp/pka途径中的增加。
为了实现ras/camp/pka信号中的这种增加,还有可能在所述转录后的水平下调节所述ras/camp/pka信号途径的一种或多种蛋白的活性。例如,有可能对所述重组酵母宿主细胞进行遗传修饰以允许所述ras/camp/pka信号途径中的一种或多种蛋白(例如,所述ira1蛋白和/或所述ira2蛋白)的葡萄糖诱导蛋白转换(glucose-inducedproteinturnover)。
所述ras/camp/pka途径的突变蛋白(也称为变体)能够具有与所述相应野生型蛋白至少70%,80%,85%,90%,95%,96%,97%,98%或99%的一致性,条件是所述突变蛋白允许所述ras/camp/pka途径的增加的信号。当与所述相应野生型蛋白的所述氨基酸序列相比时,变体包含至少一个氨基酸差异。
正如本领域已知的是,所述术语“百分比一致性”是两个或更多个多肽序列或两个或更多个多核苷酸序列之间的关系(通过序列比对进行确定)。一致性水平通常使用已知的计算机程序能够确定。用于确定百分比一致性方法,正如下文关于多核苷酸一致性的更详细讨论,也与评价多肽序列一致性相关。“一致性”能够通过已知的方法很容易计算,包括但不限于以下所述的那些:计算分子生物学(computationalmolecularbiology)(lesk,a.m.,主编)牛津大学出版社(oxforduniversitypress),ny(1988);生物计算:信息学和基因组计划(biocomputing:informaticsandgenomeprojects)(smith,d.w.主编)学术出版社(academicpress),ny(1993);序列数据的计算机分析(computeranalysisofsequencedata),第一部分(griffin,a.m.和griffin,h.g.主编)胡马纳出版社(humanapress),nj(1994);分子生物学中的序列分析(sequenceanalysisinmolecularbiology)(vonheinje,g.主编)学术出版社(1987);和序列分析引物(sequenceanalysisprimer)(gribskov,m.和devereux,j.,主编)斯托克顿出版社(stocktonpress),ny(1991)。设计确定一致性的优选方法以给出所述测试的序列之间的最佳匹配。确定一致性和相似性的方法编纂于公开可用的计算机程序中。使用lasergene生物信息学计算套件(dnastarinc.,madison,wis.)的megalign程序能够完成序列比对和百分比一致性计算。使用比对的clustal方法(higgins和sharp(1989)cabios.5:151-153)采用默认参数(gappenalty=10,gaplengthpenalty=10)完成本文公开的所述序列的多重比对。使用所述clustal方法进行成对比对的默认参数是ktuplb1,gappenalty=3,window=5和diagonalssaved=5。
本文描述的所述变体或突变蛋白可以是(i)其中一个或多个所述氨基酸残基被保守或非保守氨基酸残基(优选保守氨基酸残基)取代并且这种取代氨基酸残基可以是或可以不是由遗传密码编码的情况,或(ii)其中一个或多个所述氨基酸残基包含取代基的情况,或(iii)其中所述成熟多肽与另一个化合物,如用于增加所述多肽半衰期的化合物(例如,聚乙二醇)融合的情况,或(iv)其中所述额外的氨基酸与所述成熟多肽融合以纯化所述多肽的情况。
所述ras/camp/pka途径的所述突变蛋白也能够是所述相应野生型蛋白的片段。所述“片段”具有所述相应野生型蛋白的至少10,20,30,40,50,60,70,80,90,100,200,300,400或500或更多个连续氨基酸。当与所述相应野生型蛋白的所述氨基酸序列相比时,片段包含至少一个更少的氨基酸残基。
在一个实施方式中,所述酵母细胞不需要进行基因工程化以增加所述ras/camp/pka途径中的信号活性。例如,所述酵母细胞能够选自与所述群体中的其他酵母细胞相比其能够在所述ras/camp/pka途径中表现出高的信号活性的酵母细胞群体(其可以是天然存在的)。所述选择能够在核酸水平上(通过测定编码所述ras/camp/pka途径中的一种或多种蛋白的一种或多种基因的所述核酸序列或编码所述ras/camp/pka途径中的一种或多种蛋白的一种或多种基因的表达水平),在所述蛋白水平上(通过测定所述ras/camp/pka途径中的一种或多种蛋白的所述氨基酸序列,所述ras/camp/pka途径中的一种或多种蛋白的表达水平或所述ras/camp/pka途径中的一种或多种蛋白的所述活性)或在所述中间体水平上(例如,通过测定gtp,gdp或camp的量)作出。所述选择能够在所述繁殖步骤之前,在繁殖步骤期间,在发酵步骤之前和/或在发酵步骤期间进行。在一个实施方式中,所述选择在所述发酵步骤之前进行。在另一个实施方式中,所述选择在所述繁殖步骤之前进行。
在另一个实例中,为了实现由于与群体中的其他酵母细胞相比,其在所述ras/camp/pka途径中表现出增加的信号活性的能力来选择所述酵母细胞,有可能排除表现出所述ras/camp/pka途径中低信号活性的酵母细胞。这种排除能够在所述核酸水平上(通过测定编码所述ras/camp/pka途径中的一种或多种蛋白的一种或多种基因的所述核酸序列或编码所述ras/camp/pka途径中的一种或多种蛋白的一种或多种基因的表达水平),在所述蛋白水平上(通过测定所述ras/camp/pka途径中的一种或多种蛋白的所述氨基酸序列,所述ras/camp/pka途径中的一种或多种蛋白的表达水平或所述ras/camp/pka途径中的一种或多种蛋白的所述活性)或在所述中间体水平上(例如,通过测定gtp,gdp或camp的量)作出。所述排除能够在所述繁殖步骤之前,在繁殖步骤期间,在发酵步骤之前和/或在发酵步骤期间进行。在一个实施方式中,所述排除在所述发酵步骤之前进行。在另一个实施方式中,所述排除在所述繁殖步骤之前进行。
在一些实施方式中,推荐对重组酵母宿主细胞进行遗传工程化以(当与不包括此类遗传修饰的相应酵母细胞相比时)提高其在所述ras/camp/pka途径中的信号活性。所述重组酵母宿主细胞能够包括所述ras/camp/pka途径的一种或多种蛋白中的遗传修饰。例如,所述遗传修饰能够包括(例如,在中性整合位点)添加所述ras/camp/pka途径的一种或多种蛋白的表达盒(expressioncassette)或替换所述ras/camp/pka途径的一种或多种天然蛋白。
所述遗传修饰能够是引入编码异源启动子和/或异源核酸分子的异源核酸分子。当参考核酸分子(如启动子或编码序列)或蛋白(如所述ras/camp/pka途径中的蛋白)而使用时,所述术语“异源”是指并非天然存在于宿主生物或细胞中的所述核酸分子或蛋白。“异源”还包括从所述源生物体中移出并随后以不同于相应天然基因的形式,例如,不处于其在所述生物体基因组中的天然位置内而重新引入所述源生物体的天然编码区,或其部分。所述异源核酸分子有目的地引入所述宿主细胞中。“异源”核酸分子或蛋白可以源自任何来源,例如,真核生物,原核生物,病毒等。在一个实施方式中,所述异源核酸分子可以源自真核生物(如,例如,另一种酵母)或原核生物(如,例如,细菌)。正如本文所用的所述术语“异源的”也是指源自除内源性来源之外的来源的元件(核酸或蛋白)。因此,例如,异源元件能够来自不同的宿主细胞株,或来自不同分类群(taxonomicgroup)的生物(例如,不同的界(kingdom),门(phylum),纲(class),目(order),科(family),属(genus)或种(species),或这些分类之一中的任何亚组)。所述术语“异源”在本文中也与所述术语“外源”同义使用。
在一个实例中,所述重组酵母宿主细胞能够包括包含编码如本文所述的异源和突变ras2蛋白的第一核酸分子的异源核酸分子。当与所述野生型ras2蛋白相比时,所述突变ras2蛋白能够在一个或多个位置上包括氨基酸取代。在一个实施方式中,所述重组酵母宿主细胞还能够表达野生型ras2蛋白(其能够是所述重组酵母宿主细胞天然的或经过基因工程化的)。野生型ras2蛋白已经表征于酿酒酵母(genbank登录号caa95974或amqb00000000.1),解脂耶氏酵母(yarrowialipolytica)(genbank登录号aak52675)和假丝酵母(candidaorthopsilosis)(genbank登录号ccg25689)中。在一个实施方式中,所述野生型ras2蛋白具有seqidno:1的所述氨基酸序列或由具有seqidno:4的所述序列的核酸分子进行编码。在另一个实施方式中,所述突变ras2蛋白具有seqidno:2的所述氨基酸序列或由具有seqidno:3的所述序列的所述核酸分子进行编码。
在另一个实例中,所述重组酵母宿主细胞能够包括包含编码如本文所述的异源和突变ras1蛋白的第二核酸分子(单独或与其他异源核酸分子组合)的异源核酸分子。当与所述野生型ras1蛋白相比时,所述突变ras1蛋白能够在一个或多个位置上包括氨基酸取代。在一个实施方式中,所述重组酵母宿主细胞还能够表达野生型ras1蛋白(其可以是所述重组酵母宿主细胞天然的或经过基因工程化的)。
在还有的另一个实例中,所述重组酵母宿主细胞能够包括包含编码如本文所述的异源和突变ira2蛋白的第三核酸分子(单独或与其他异源核酸分子组合)的异源核酸。所述突变ira2蛋白可以是野生型ira2蛋白的片段。所述野生型ira2蛋白已经表征于酿酒酵母(genbank登录号caa99093或amqb00000000.1),马克斯克鲁维酵母(kluyveromycesmarxianus)(genbank登录号bao41432),毕赤酵母(scheffersomycesstipites)(genbank登录号xp_001386919),假丝酵母(candidaorthopsilosis)(genbank登录号ccg24772)和威克汉姆西弗酵母(wickerhamomycesciferrii)(genbank登录号xp_011275426)中。在一个实施方式中,所述突变ira2蛋白具有seqidno:8的所述氨基酸序列或由具有seqidno:6的所述序列的核酸分子进行编码。
在还有的另一个实例中,所述重组酵母宿主细胞能够包括包含编码如本文所述的异源和突变ira1蛋白的第四核酸分子(单独或与其他异源核酸分子组合)的异源核酸。所述突变ira1蛋白可以是所述野生型ira1蛋白的片段。
当制备和使用重组酵母宿主细胞时,其包括的所述异源核酸分子能够进一步包含用于控制所述核酸分子表达的启动子(编码所述ras/camp/pka信号途径的蛋白)。“启动子”是指能够控制编码序列或功能性rna(在本公开中,所述第一核酸分子)的表达的dna片段。正如本文所用,所述术语“表达”是指来自本文描述的所述异源核酸分子的有义(mrna)的转录和稳定积累。表达还可以指mrna翻译成多肽。启动子可以整体源自天然基因,或由源自天然存在的不同启动子的不同元件组成,或甚至包含合成dna片段。应该进一步认识到的是,由于在大多数情况下调控序列的确切边界尚未完全定义,不同长度的dna片段可以具有相同的启动子活性。启动子通常在其3'末端被所述转录起始位点结合并向上游(5'方向)延伸从而包括在高于背景的可检测水平下启动转录所必需的最少数量的碱基或元件。在所述启动子内将发现转录起始位点(例如,通过用核酸酶s1作图(mapping)很方便地定义),以及负责所述聚合酶结合的蛋白结合结构域(共有序列)。在本公开的上下文中,一种或多种启动子可以用于表达所述异源核酸分子。
在本公开内容的上下文中,所述启动子旨在允许所述核酸分子在发酵期间(例如,厌氧条件)表达,但在酵母生产/繁殖期间(例如,葡萄糖受限和需氧条件)不表达。正如在本公开的上下文中所用,所述表述“繁殖”是指商业过程的扩增阶段,其中所述酵母在需氧条件下繁殖从而最大化所述底物向生物质的转化。还正如在本公开的上下文中所用,所述表述“发酵”是指使用所述繁殖的生物质从而最大化从培养基中产生一种或多种所需的代谢物的生产。在一些实施方式中,所述启动子能够允许所述异源核酸分子在所述酵母的静止阶段(stationaryphase)中(例如,在繁殖之后但在发酵之前,例如,所述ygp1基因的所述启动子)表达。发酵诱导型启动子包括,但不限于,所述pdc1基因的启动子,所述tdh1基因的启动子,所述tdh3基因的启动子,所述eno2基因的启动子,所述cdc19基因的启动子,所述hxt3基因的启动子和/或所述hor7基因的启动子。不严格厌氧的示例性发酵诱导型启动子是所述pdc1基因的启动子。在还有的另一个实施方式中,所述启动子不是需氧启动子(例如,在氧存在下有活性,比如所述icl1启动子,所述tdh1基因的启动子,所述tdh2基因的启动子,所述tdh3基因的启动子,所述eno2基因的启动子,所述cdc19基因的启动子,所述hxt3基因的启动子和所述hor7基因的启动子),组成型启动子(例如,在所有条件下都有活性,如所述adh1基因的启动子或所述tef2基因的启动子),静止阶段启动子(例如,仅在细胞生长停止后有活性,比如所述ygp1基因的启动子)或渗透应力诱导型启动子(例如,响应于渗透应力而有活性,比如所述stl1基因的启动子)。
在一个实施方式中,所述启动子允许或有利于所述核酸分子在部分或完全厌氧条件下的表达(例如,厌氧调节的或缺氧调节的启动子)。因此,当与环境空气中的氧水平(例如,氧通常以约21体积%存在于环境空气中的有氧条件)相比,所使用的所述启动子有利于在氧水平降低(例如,厌氧条件或缺氧)的环境中所述核酸分子的表达。当所述重组酵母宿主细胞置于需氧条件下时,所述启动子能够允许所述核酸分子的表达,然而,在启动子的控制下,与置于(部分或全部)厌氧条件下的相同重组酵母宿主细胞的表达水平相比,在置于需氧条件的所述重组酵母宿主细胞中所述核酸分子的表达水平更低。因此,当所述重组酵母宿主细胞置于至少部分厌氧或缺氧的条件下时,所述启动子允许所述核酸分子优先表达。正如在本公开的上下文中所用,所述术语“厌氧条件”是指空气中氧水平(按体积计)低于21%(例如,低于或等于约20,19,18,17,16,15,14,13,12,11,10,9,8,7,6,5,4,3,2,1或0)的情况。因为所述酵母细胞通常在液体中培养,所述表述“厌氧条件”或“缺氧”是指与在与环境空气平衡的培养基中的所述饱和溶解氧水平(例如,需氧条件,在环境空气中具有约21体积%的典型氧水平)相比,所述氧水平降低的环境条件(例如,厌氧条件)。因此,所述术语“厌氧条件”是指所述溶解氧水平低于100%饱和(例如,低于或等于约90,80,70,60,50,40,30,20,10,5,4,3,2,1,0.5,0.1,0.05,0.01或0%饱和)的条件。
能够包括于本公开的所述异源核酸分子中的所述厌氧调节型启动子能够包括,但不限于,所述dan1基因的启动子,所述pau5基因的启动子,所述gpd2基因的启动子,所述pdc1基因的启动子,所述tpi1基因的启动子,所述anb1基因的启动子,所述acs2基因的启动子以及kwastetal.,2002,terlindetal.,1999和taietal.,2002中所列的所述基因的所述厌氧调节型启动子。
在还有的另一个实施方式中,所述启动子允许或有利于在不存在葡萄糖的情况下表达所述异源核酸分子(例如,葡萄糖抑制型启动子)。因此,能够使用的一些所述启动子有利于在不存在葡萄糖的环境中表达所述异源核酸分子。当所述重组酵母宿主细胞置于存在一些葡萄糖的情况下时,所述启动子能够允许所述核酸分子的表达,然而,在所述葡萄糖可抑制型启动子的控制下,当与所述核酸分子在不存在葡萄糖的情况下相应重组酵母宿主细胞中的表达水平相比时,在存在葡萄糖的情况下所述重组酵母宿主细胞中所述核酸分子表达水平更低。因此,所述启动子允许在不存在葡萄糖的情况下优先表达所述异源核酸分子。示例性的葡萄糖可抑制型启动子包括,但不限于,所述hxt7启动子和所述hxk1启动子。
在所述异源核酸分子中,所述启动子(或启动子的组合)和编码所述异源蛋白的所述核酸分子彼此可操作地连接。在本公开的上下文中,所述表述“可操作地连接”或“可操作地结合”是指所述启动子与编码所述异源蛋白的核苷酸分子以在某些条件下允许表达来自所述核酸分子的所述异源蛋白的方式进行物理结合的事实。在一个实施方式中,所述启动子能够位于编码所述异源蛋白的核酸序列的上游(5')。在还有的另一个实施方式中,所述启动子能够位于编码所述异源蛋白的所述核酸序列的下游(3')。在本公开的上下文中,所述异源核酸分子中能够包含一个或一个以上的启动子。当一个以上的启动子与所述异源核酸分子可操作地连接时,每种所述启动子与编码所述异源蛋白的所述核酸序列可操作地连接。对于编码所述异源蛋白的所述核酸分子,所述启动子能够位于上游,下游以及上游和下游两者。
所述启动子能够与编码所述ras/camp/pka信号途径的所述蛋白的所述核酸分子异源。所述启动子能够异源于或衍生于来自与所述重组酵母宿主细胞相同的属或种的菌株。在一个实施方式中,所述启动子衍生自所述酵母宿主细胞的相同属或种,并且所述异源蛋白源自所述酵母宿主细胞的不同属。
在本公开的上下文中,所述异源核酸分子能够整合到所述重组酵母宿主细胞的基因组中。正如本文所用的所述术语“整合的”是指通过分子生物学技术置于宿主细胞基因组中的遗传元件。例如,遗传元件能够置于所述酵母宿主细胞的染色体中,而不是载体如宿主细胞携带的质粒中。将遗传元件整合到酵母宿主细胞基因组中的方法是本领域熟知的,包括,例如,同源重组。所述异源核酸分子能够存在于所述酵母宿主细胞基因组中的一个或多个拷贝中。
可替代地,所述异源核酸分子能够独立地从所述酵母基因组中复制。在这样的实施方式中,所述核酸分子能够是稳定的且自我复制的。
使用载体能够将所述异源核酸分子引入所述酵母宿主细胞中。“载体”,例如,“质粒”,“粘粒(cosmid)”或“yac”(酵母人工染色体)是指额外的染色体元件并通常是以环状双链dna分子形式。此类载体可以是源自任何来源的单链或双链dna或rna的线性的、环状或超螺旋的自主复制序列、基因组整合序列、噬菌体或核苷酸序列,其中许多核苷酸序列已经连接或重组成能够将所选择的基因产物的启动子片段和dna序列连同合适的3'非翻译序列一起引入细胞中的独特构建体。
本公开的酵母细胞和重组酵母宿主细胞能够经过选择或设计从而能够利用木糖和/或阿拉伯糖。正如在本发明的上下文中所用,所述表述“能够利用木糖和/或阿拉伯糖”是指所述酵母细胞或重组酵母宿主细胞代谢木糖和/或阿拉伯糖的能力。
在所述细胞能够利用木糖的实施方式中。在这样的实施方式中,所述重组酵母宿主细胞能够经过修饰从而表达木糖还原酶,木糖醇脱氢酶,木糖异构酶,木酮糖激酶,木糖还原酶,木糖脱氢酶,木糖酸脱水酶,木糖转酮酶和/或木糖转醛酶蛋白中的至少之一(或其组合)。示例性的木糖异构酶包括,但不限于,源自狄氏副拟杆菌(parabacteroidesdistasonis),阿氏枝梗鞭菌(cyllamycesaberensis),软弱贫养菌(abiotrophiadefectiva),chitinophagapinensis,栖瘤普雷沃菌(prevotellaruminicola),单鞭毛球菌(piromycesequi),lachnoclostridiumphytofermentans,clostridiumphytofermentans,疾病卡托菌(catonellamorbi)和/或多形拟杆菌(bacteroidesthetaiotaomicron)的那些以及描述于wo/2016/024215以及美国专利8986948中的那些。在一些实施方式中,所述异源木酮糖激酶(xks1)能够由所述xks1基因进行编码。
在还有的另一个实施方式中,所述细胞具有“木聚糖分解活性(xylanolyticactivity)”,例如,具有所述酵母细胞和所述重组酵母宿主细胞水解寡聚戊糖和聚戊糖中的糖苷键的能力。所述术语“木聚糖酶”是给予一类酶的名称,其将所述线性多糖β-1,4-木聚糖降解为木糖,从而分解植物细胞壁的主要组分之一的半纤维素。木聚糖酶包括与酶学委员会编号3.2.1.8相对应的那些酶。在一个实施方式中,所述重组酵母宿主细胞能够包括一种或多种提供木聚糖水解活性的遗传修饰(编码一种或多种异源蛋白)。
经遗传工程化以发酵木糖的示例性重组酵母宿主细胞公开于wo/2016/024215和美国专利申请14/821,955中,并能够如本文所述进一步修饰从而提高其ras/camp/pka信号途径。
在所述细胞能够利用阿拉伯糖的实施方式中,本公开提供了表达编码阿拉伯糖异构酶(ai,如,例如,来自多形拟杆菌(bacteroidesthetaiotaomicron))的所述araa蛋白)的核酸分子、编码核酮糖激酶(rk,如,例如,来自多形拟杆菌的所述arab蛋白)的异源多核苷酸、编码核酮糖5-磷酸差向异构酶(r5pe,如,例如,来自多形拟杆菌的arad蛋白)的异源多核苷酸和/或编码阿拉伯糖转运蛋白(arat)的异源多核苷酸的酵母宿主细胞或包含编码阿拉伯糖异构酶(ai,如,例如,来自多形拟杆菌的所述araa蛋白)的核酸分子、编码核酮糖激酶(rk,如,例如,来自多形拟杆菌的所述arab蛋白)的异源多核苷酸、编码核酮糖5-磷酸差向异构酶(r5pe,如,例如,来自多形拟杆菌的arad蛋白)的异源多核苷酸和/或编码阿拉伯糖转运蛋白(arat)的异源多核苷酸的重组酵母宿主细胞。wo2013/071112中描述了能够利用阿拉伯糖的重组酵母宿主细胞的实施方式。所述异源阿拉伯糖异构酶(araa)能够,例如,源自大肠杆菌(araa),植物乳杆菌(lactobacillusplantarum)(araa),金黄节杆菌(arthrobacteraurescens)(araa),密执克杆菌(clavibactermichiganensis)(araa),gramellaforsetii(araa),地衣芽孢杆菌(bacilluslicheniformis),丙酮丁醇梭菌(clostridiumacetobutylicum),枯草芽孢杆菌(bacillussubtilis)或耻垢分枝杆菌(mycobacteriumsmegmatis)或描述于美国专利9,206,444;美国专利9,303,253;美国专利8,993,301;美国专利申请2010/0304454或欧洲专利1499708中。所述异源核酮糖激酶(arad)能够,例如,源自多形拟杆菌(arab),大肠杆菌(arab),植物乳杆菌(arab),金黄节杆菌(arab),密执克杆菌(arab)或gramellaforsetii(arab)或描述于美国专利9,206,444;美国专利9,303,253;美国专利申请2010/0304454或欧洲专利1499708中。所述异源核酮糖-差向异构酶(arad)能够源自多形拟杆菌(arad),大肠杆菌(arad),植物乳杆菌(arad),金黄节杆菌(arad),密执克杆菌(arad)或gramellaforsetii(arad)或描述于美国专利9,206,444;美国专利9,303,253;美国专利申请2010/0304454或欧洲专利1499708中。所述异源阿拉伯糖转运蛋白能够源自乳酸克鲁维酵母(kluveromyceslactis),耐热克鲁维酵母(kluveromycesthermotolerans),鲁氏接合酵母(zygosaccharomycesrouxii),范式酵母(vanderwaltozymapolyspora),汉逊德巴利酵母(debaryomyceshanseii),黑曲霉(aspergillusniger),产黄青霉(penicilliumchrysogenum),圭勒蒙迪毕赤酵母(pichiaguilermondii),黄曲霉(aspergillusflavus),葡萄牙假丝酵母(candidalusitaniae),白色念珠菌(candidaalbicans),马克斯克鲁维酵母(kluveromycesmarxianus),毕赤酵母(pichiastipites)或阿拉伯发酵假丝酵母(candidaarabinofermentans)。为了促进阿拉伯糖的转运,所述重组酵母宿主细胞能够表达所述gal2基因和/或gal2基因。wo2013/071112中描述了能够转运阿拉伯糖的示例性重组酵母宿主细胞。
在一些实施方式中,所述重组酵母宿主细胞还能够表达来自所述戊糖磷酸途径(例如,tal1转醛酶,tkl1转酮酶,rpe1d-核酮糖-5-磷酸3-差向异构酶和/或rki1核糖-5-磷酸酮醇异构酶)的一个或多个基因和/或铁硫簇基因突变体(如,例如,yfh1t163p,isu1d71n,isu1d71g,isu1s98f,nfs1l115w,nfs1e458d,全部描述于wo2016/024215中)。在一些实施方式中,所述重组酵母宿主细胞的所述醛糖还原酶基因(如,例如,编码所述ypr1蛋白的所述ypr1基因和/或编码所述gre3蛋白的所述gre3基因)的表达能够被下调或删除。
在一些实施方式中,所述异源核酸分子相对于所述预期的受体酵母宿主细胞进行密码子优化。正如本文所用,所述术语“密码子优化的编码区”是指通过用一个或多个更常用于该生物体的所述基因中的密码子取代至少一个或一个以上的密码子从而适应于给定生物的所述细胞中的表达的核酸编码区。通常而言,生物体中高度表达的基因偏向于被该生物体中最丰富的trna物质识别的密码子。这种偏向的一个衡量标准是“密码子适应指数(codonadaptationindex)”或“cai”,其度量用于编码特定基因中每个氨基酸的密码子在多大程度上是最经常出现于来自生物体的高度表达基因的参照集合中那些密码子。本文所述的密码子优化序列的所述cai对应于约0.8~1.0,约0.8~0.9,或约1.0。
密码子优化的序列可以进一步经过修饰从而在特定生物中表达,这取决于受体的生物学限制。例如,如果已知这些序列会对转录产生负面影响,能够从所述序列中移除大量的“as”或“ts”序列(run)(例如,大于4,5,6,7,8,9或10个连续碱基的序列(run)。此外,特异性限制酶位点出于分子克隆目的可以移除。此外,能够检查所述核酸分子的长度为10个碱基或更长的直接重复,反向重复和镜像重复,这能够通过用“第二最佳”密码子,即以第二个最高频率出现于优化所述序列的所述特定生物体内的密码子,替换密码子而手动修改。
在本公开的上下文中,所述酵母细胞或重组酵母宿主细胞能够来自酵母属,克鲁维酵母属,阿氏酵母属,德巴利酵母属,假丝酵母属,毕赤酵母属,法夫酵母属,裂殖酵母属,汉逊氏酵母属,克勒克氏酵母属,许旺氏酵母属或耶氏酵母属。合适的酵母种可以包括,例如,酿酒酵母,博伊丁酵母(s.bulderi),巴尼特酵母(s.barnetti),少孢酵母(s.exiguus),葡萄汁酵母(s.uvarum),糖化酵母(s.diastaticus),乳酸克鲁维酵母(k.lactis),马克斯克鲁维酵母(k.marxianus)或脆弱克鲁维酵母(k.fragilis)。在一些实施方式中,所述酵母选自由酿酒酵母,粟酒裂殖酵母(schizzosaccharomycespombe),白色念珠菌(candidaalbicans),巴斯德毕赤酵母(pichiapastoris),树干毕赤酵母(pichiastipitis),解脂耶氏酵母(yarrowialipolytica),多形汉逊酵母(hansenulapolymorpha),红法夫酵母(phaffiarhodozyma),产朊假丝酵母(candidautilis),解嘌呤阿氏酵母(arxulaadeninivorans),汉逊德巴利酵母(debaryomyceshansenii),多形汉逊酵母(debaryomycespolymorphus),裂殖酵母(schizosaccharomycespombe)和西方许旺氏酵母(schwanniomycesoccidentalis)组成的组。在一个具体实施方式中,所述酵母是酿酒酵母(saccharomycescerevisiae)。在一些实施方式中,所述酵母细胞能够是产油酵母细胞。例如,所述产油酵母宿主细胞能够来自布拉属(generablakeslea),念珠菌属,隐球酵母(cryptococcus)属,小克银汉属(cunninghamella),油脂酵母属(lipomyces),被孢霉属(mortierella),毛霉属(mucor),须霉属(phycomyces),腐霉属(pythium),红冬孢酵母属(rhodosporidum),红酵母属(rhodotorula),皮状丝孢酵母属(trichosporon)或耶氏酵母属(yarrowia)。在一些备选实施方式中,所述宿主细胞能够是产油性微藻宿主细胞(例如,来自破囊壶菌属(thraustochytrium)或裂殖壶菌属(schizochytrium))。
正如本文所述,所述重组酵母宿主细胞采用编码所述异源蛋白的所述异源核酸分子进行基因工程化(转导或转化或转染)。所述核酸分子能够在载体上引入所述宿主细胞中,所述载体可以是,例如,克隆载体或包含编码异源蛋白的序列的表达载体。所述宿主细胞能够包含一种或多种异源核酸分子,每种都以整合拷贝或独立复制拷贝的形式存在。
如上所述的重组宿主细胞或细胞培养物能够对于纤维素水解,或淀粉,或戊糖利用(例如,通过糖检测分析测试),对于特定类型的糖解酶活性(例如,通过测量各个内切葡聚糖酶,葡糖苷酶,纤维二糖水解酶,木聚糖酶,葡聚糖酶,木糖苷酶,木聚糖酯酶,阿拉伯呋喃糖苷酶,半乳糖苷酶,纤维二糖磷酸化酶,纤维糊精磷酸化酶,甘露聚糖酶,甘露糖苷酶,木葡聚糖酶,木聚糖内切酶,葡糖醛酸酶,乙酰木聚糖酯酶,阿拉伯呋喃糖水解酶,膨胀素,葡萄糖醛酸酯,棒曲霉素(expansin),果胶酶(pectinase),阿魏酸酯酶(feruoylesterase),α-淀粉酶,β-淀粉酶,葡糖淀粉酶,支链淀粉酶(pullulanase),异支链淀粉酶(isopullulanase),α-葡糖苷酶,β-葡糖苷酶,半乳糖苷酶,阿拉伯糖酶,阿拉伯木聚糖酶,阿拉伯糖苷酶,阿拉伯呋喃糖苷酶,阿拉伯木聚糖酶,阿拉伯糖苷酶,阿拉伯呋喃糖苷酶,阿拉伯糖异构酶,核酮糖5-磷酸4-差向异构酶,木糖异构酶,木酮糖激酶,木糖还原酶,木糖脱氢酶,木糖醇脱氢酶,木糖酸酯脱水酶,木糖转酮酶和/或木糖转醛酶)或对于总纤维素酶活性进行进一步分析。内切葡聚糖酶活性能够通过,例如,测量内切葡聚糖酶特异性cmc或羟乙基纤维素(hec)底物中还原末端的增加而进行测定。纤维二糖水解酶活性能够,例如,通过使用不溶性纤维素底物如所述无定形底物磷酸膨胀纤维素(pasc)或微晶纤维素(aviceltm)和测定所述底物的水解程度进行测量。β-葡糖苷酶活性能够通过各种测试法,例如,使用纤维二糖进行测量。其他糖解酶类型的活性的测试法在本领域内是已知的,并将在下面举例说明。
总糖解酶的活性(其能够包括内切葡聚糖酶,葡糖苷酶,纤维二糖水解酶,木聚糖酶,葡聚糖酶,木糖苷酶,木聚糖酯酶,阿拉伯呋喃糖苷酶,半乳糖苷酶,纤维二糖磷酸化酶,纤维糊精磷酸化酶,甘露聚糖酶,甘露糖苷酶,木葡聚糖酶,木聚糖内切酶,葡糖醛酸酶,乙酰木聚糖酯酶,阿拉伯呋喃糖糖水解酶,膨胀素,葡萄糖醛酸酯酶,棒曲霉素,果胶酶,阿魏酸酯酶蛋白,α-淀粉酶,β-淀粉酶,葡糖淀粉酶,α-葡糖苷酶,β-葡糖苷酶,半乳糖苷酶,阿拉伯糖酶,阿拉伯木聚糖酶,阿拉伯糖苷酶,阿拉伯呋喃糖苷酶,阿拉伯木聚糖酶,阿拉伯糖苷酶,支链淀粉酶,异支链淀粉酶,阿拉伯糖异构酶,核酮糖5-磷酸4-差向异构酶,木糖异构酶,木酮糖激酶,木糖还原酶,木糖脱氢酶,木糖醇脱氢酶,木糖酸酯脱水酶,木糖转酮酶和/或木糖转醛酶的活性)能够协同地水解生物质原料。因此可以例如使用不溶性底物,包括纯纤维素底物如whatman1号滤纸,棉绒,微晶纤维素,细菌纤维素,藻类纤维素和含纤维素底物如染色纤维素,α-纤维素或预处理的木质纤维素来测量总纤维素酶的活性。纤维素酶的比活性也能够通过本领域普通技术人员已知的方法进行测量,例如,通过(如上所述)将通过针对样品测量的蛋白(纤维素酶)浓度进行标准化的avicel测试法。总糖分解活性还能够使用含有淀粉、纤维素和半纤维素如玉米醪(cornmash)的复合底物通过测量释放的单体糖而进行测量。
在另外的实施方式中,所述酵母宿主细胞和所述重组宿主细胞或细胞培养物能够对其产生乙醇的能力进行测试。乙醇产量能够通过本领域普通技术人员已知的技术进行测定,例如,通过标准hplc折射率方法。
酵母细胞的繁殖
在另一方面中,本发明提供了能够在发酵前以与未遗传修饰的相应酵母宿主细胞相似的水平繁殖的重组酵母宿主细胞。在一个实施方式中,本公开提供了发酵前在繁殖培养基中繁殖重组酵母宿主细胞的方法。所述方法优选在葡萄糖受限且需氧条件下进行。通常为液体形式的所述繁殖培养基包含允许进行繁殖的营养物。在一些实施方式中,所述繁殖培养基经过选择从而实现快速生长。例如,所述培养基能够包含碳源(如,例如,糖蜜,蔗糖,葡萄糖,右旋糖糖浆,乙醇和/或玉米浆(cornsteepliquor)),氮源(如,例如,氨)和磷源(如,例如,磷酸)。
所述方法能够进一步包括在繁殖后用于储存的所述繁殖的重组酵母宿主细胞的处理。例如,所述方法能够进一步包括制备所述繁殖的重组酵母宿主细胞的高细胞密度制剂。这能够通过浓缩在液体培养基中的所述繁殖的重组酵母宿主细胞(通过,例如,离心或过滤)而实现。所述浓缩的繁殖重组酵母宿主细胞也能够混合于储存介质(其能够含有甘油)中。所述高细胞密度制剂还能够包括冻干所述繁殖的重组酵母宿主细胞。所述冻干的繁殖重组酵母宿主细胞也能够掺混于储存介质(优选干燥的储存介质)中。
使用所述酵母细胞进行发酵的方法
本公开提供了使发酵培养基发酵的方法,包括使本文所述的酵母细胞或所述重组酵母宿主细胞与所述发酵培养基接触。在一个实施方式中,所述发酵培养基包含葡萄糖和至少一种另外的可发酵碳源(如,例如,c5碳源,单糖(例如,木糖或阿拉伯糖)或二糖(例如,麦芽糖或纤维二糖))。在还有的另一个实施方式中,所述发酵培养基包含木质纤维素材料如,例如甘蔗渣,木材或玉米纤维。在还有的另一个实施方式中,所述发酵在低乙醇应力的条件下进行(例如,所述获得的发酵不超过高于130g/l,120g/l,110g/l或100g/l的乙醇浓度)。
本公开的所述酵母细胞(在一个实施方式中,其未经遗传修饰)能够有利地用于食品生产,例如,面包制作。在这样的实施方式中,所述酵母宿主细胞置于包含葡萄糖和麦芽糖的培养基中。在这样的实施方式中,所述发酵培养基能够包含小麦。
本公开的所述重组酵母宿主细胞能够用于发酵许多生物质原料。在一些实施方式中,基于其在水中的溶解度,所述底物(或发酵培养基)能够分为两类,可溶的和不可溶的。可溶性底物包括α-糊精,纤维糊精或衍生物,羧甲基纤维素(cmc)或羟乙基纤维素(hec)。不溶性底物包括不溶性淀粉(原生的(raw)或凝胶化的),结晶纤维素,微晶纤维素(aviceltm),无定形纤维素,如磷酸溶胀纤维素(pasc),染色或荧光纤维素和木质纤维素生物质。这些底物通常是高度有序的纤维素材料,因此仅微溶。应当理解的是,合适的木质纤维素材料可以是含有可溶性和/或不溶性纤维素和半纤维素的任何原料,其中所述不溶性纤维素和半纤维素可以是结晶或非结晶形式。在各种实施方式中,所述木质纤维素生物质包括,例如,木材,玉米,玉米秸秆,锯末,树皮,叶子,农业和林业残余物,草如柳枝稷,反刍动物消化产物,市政废物,造纸厂废水,再生纸类产品(如,例如,报纸,纸板)或其组合。这种方法能够容许产生一种或多种发酵产物如,例如,乙醇,丁醇,乙酸盐,氨基酸和维生素。
在一些实施方式中,本文描述的所述方法能够用于以特定速率生产乙醇。例如,在一些实施方式中,乙醇以至少约0.1mg/h/l,至少约0.25mg/h/l,至少约0.5mg/h/l,至少约0.75mg/h/l,约1.0mg/h/l,至少约2.0mg/h/l,至少约5.0mg/h/l,至少约10mg/h/l,至少约15mg/h/l,至少约20.0mg/h/l,至少约25mg/h/l,至少约30mg/h/l,至少约50mg/h/l,至少约100mg/h/l,至少约200mg/h/l,或至少约500mg/h/l的速率进行生产。
乙醇产量能够使用本领域已知的任何方法进行测量。例如,发酵样品中乙醇的量能够使用hplc分析进行评价。许多乙醇测试试剂盒能够商购获得,其使用例如,基于醇氧化酶的测试法。
通过参考以下实施例将更容易理解本发明,这些实施例用于举例说明本发明而不是限制其范围。
实施例i—ras2突变的表征
表1中提供了该实施例中使用的酿酒酵母菌株的描述。
表1.实施例i中表征的所述菌株的描述
在设计用于模拟面包制作条件的条件下,在高细胞密度下进行发酵。具体而言,在含有10%麦芽糖,2%葡萄糖,4%氯化钠,0.1%柠檬酸钠,0.1%酵母提取物,ph5.5的培养基中,用2%干酵母剂量当量进行发酵。使用hplc测量葡萄糖,麦芽糖,乙醇和甘油浓度。用elisa测定法测量camp的水平。
据发现,在含有与“贫”(无添加糖)面团相似浓度的葡萄糖和麦芽糖混合物的液体培养基中,携带所述ras2a66t突变的菌株更快地消耗葡萄糖和麦芽糖(图1和图2)并产生更多乙醇(和因此产生的更多二氧化碳),表明与其野生型对应物相比具有更高的发酵能力(图3)。此外,携带所述ras2a66t突变的菌株具有改进的甘油产量,这是面团发酵的重要特征(图4)。
还发现,携带ras2a66t突变的菌株对葡萄糖尖峰具有异常的camp反应,表明camp信号失调(dysregulated)。这些效应类似于ras2val19过度活性突变体对葡萄糖尖峰的已知反应(mbonyietal.,1988)(图5和图6)。实施例ii—木糖发酵上ras2突变的表征
多个酿酒酵母菌株被基因工程化,容许通过表达异源木糖异构酶,上调所述磷酸戊糖途径(rki1,rpe1,tkl1,tki1)和xks1,并缺失醛糖还原酶活性(例如,gre3)进行木糖发酵。对在这些菌株中引入异源突变ras2蛋白(带有a66t突变)的效应进行了表征。
表2.实施例ii的所述酿酒酵母菌株的描述
m8349和m12076菌株在35℃下在ypd40(含有40g/l右旋糖)中繁殖过夜。将含有30mlypd10x70培养基(含有10g/l右旋糖和70g/l木糖)的血清瓶用30μl所述过夜培养物接种,密封并在32℃下以150rpm振荡生长。监测co2废气的流速作为乙醇产量的代表进行监测,并在生长90小时后采集最终时间点hplc样品(用于葡萄糖,甘油和乙醇测定)。将含有50ml含有300g/l麦芽糖糊精的yp培养基的第二组血清瓶用50μl所述过夜ypd40培养物接种,并计量给予0.48agu/g
比较了m8349和m12076菌株在ypd10x70培养基中的所述厌氧生长。如图7和图8所示,当与缺乏这种突变ras2蛋白的亲本菌株m8349相比时,所述突变ras2蛋白(a66t)在所述m12076菌株中的表达提高了co2产量,糖消耗,甘油产量和乙醇产量。
m12196和m12543的过夜培养物在35℃下的ypd40培养基中繁殖。将30μl每种培养物的等分试样作为接种物用于30mlypd10x70培养基以及基于ynb的合成培养基“sp3”(6.700g/l具有氨基酸的ynb,5.000g/l柠檬酸三钠,60.000g/l葡萄糖,45.000g/l木糖,5.000g/l半乳糖,110.000g/l可发酵糖,49.5g/l理论乙醇(0.45g/g),0.006g/lfeso4,8.000g/l乙酸钾,1.000g/l乳酸,0.500g/lhmf,0.500g/l糠醇,0.200g/l山梨酸,ph5.5)。将所述血清瓶密封并在32℃下以150rpm振荡孵育。监测co2废气作为乙醇生成的代表。如图9a和图9b所示,当与缺乏这种突变ras2蛋白的相应亲本菌株m12196相比时,所述突变ras2蛋白(a66t)在m12543中的表达提高了ypd10x70和sp3培养基两者中的co2产量。
m11321,m12557,m12621,m12624,m12625和m12627在ypd40培养基中于35℃繁殖过夜。具有30mlsp3培养基的血清瓶用60μl每种所述过夜培养物接种,密封并在32℃下以150rpm振荡孵育。m12557,m12621和m12624也用作“sp4”培养基(6.700g/l含氨基酸的ynb,5.000g/l柠檬酸三钠脱水物,30.000g/l葡萄糖,75.000g/l木糖,5.000g/l半乳糖,110.000g/l可发酵糖,49.5g/l理论乙醇(0.45g/g),0.006g/lfeso4·7h2o,9.000g/l乙酸钾,1.000g/l乳酸,0.500g/l香草醛,0.500g/l糠醛,0.200g/l山梨酸,ph5.5)中的接种物。将所述sp4样品在33℃下以150rpm振荡孵育。测量所述sp3瓶的co2废气作为乙醇产量的代表,并对sp3和sp4瓶采集终点hplc(用于葡萄糖,甘油和乙醇测定)。此外,为了评价生产的可行性,测试了m12557,m12621,m12624,m12625和m12627在糖蜜上的生长。使用两种独立的糖蜜条件:含有6g/lcsl和4g/ldap或10g/l尿素和2g/lmgso4的80%糖蜜。将50ml糖蜜的等分试样从每个菌株的ypd40过夜培养物接种至起始od600为0.05,并在需氧摇瓶中于30℃以200rpm振荡孵育。生长24h后,测量每种培养物的干细胞重量。
在测试的实验条件下,表达所述突变的ras2a66t蛋白的菌株m12621,m12624和m12625以比其相应的亲本菌株m12557更快的速率产生co2,并产生相同或更多的乙醇(图10a和图10b)。这些结果表明,所述突变ras2a66t蛋白的表达确实提高了木糖的乙醇发酵的速率。
当在sp4培养基中生长时,菌株m12624(在所述dan1启动子控制下表达所述ras2a66t蛋白)表现出比菌株m12621(在所述ras2启动子控制下表达ras2a66t蛋白)或亲本菌株m12557更高的木糖消耗和乙醇产量(图11)。
当在糖蜜培养基中生长时,菌株m12624,m12625和m12627(在所述dan1,anb1或hxk1启动子的控制下表达所述ras2a66t蛋白)表现出比菌株m12621(在所述ras2启动子控制下表达ras2a66t蛋白)更高的产量(图12)。因此,用于表达ras2a66t蛋白的启动子的选择能够同时实现良好的繁殖和木糖发酵。
实施例iii—阿拉伯糖发酵上ras2突变的表征
表达异源多形拟杆菌araa,arab,arad并且gal80缺失的所述阿拉伯糖技术被整合到两种利用木糖的菌株m12359和m13414中。除了在所述dan1启动子和天然ras2终止子控制下在m13414的中性整合位点处存在ras2a66t等位基因外,所述两个亲本菌株是相同的。两种菌株仍含有天然野生型ras2基因座。
表3.实施例iii的酿酒酵母菌株的描述
菌株m12359,m13414,m13570和m13578在35℃下在5ml含有40g/l葡萄糖的yp培养基中生长过夜。将300μl每种所述过夜培养物接种到装有30ml含有5g/l葡萄糖和35g/l阿拉伯糖的yp培养基的60ml血清瓶中,不调节ph(约ph6.5)。将所述血清瓶在32℃下以170rpm振荡孵育。在整个发酵过程中监测压力并用作co2和乙醇产量的估计。
在70h发酵后,通过hplc在h-柱上测量终点代谢物浓度。简而言之,从每个血清瓶中取样1ml,并通过离心沉淀细胞。将400μl上清液与10μl10%h2so4混合,并离心通过0.2μm过滤器。将所获得的样品储存于4℃直至通过hplc进行分析。
虽然阿拉伯糖发酵速率在所述两种菌株之间看起来相同(数据未显示),但表达所述ras2突变体等位基因的m13578似乎产生更大量的co2(图13)。该结果与hplc分析中的较高最终乙醇滴定度相关(图14)。所述ras2等位基因将来自阿拉伯糖的乙醇产量从m13570中的0.44g乙醇/g阿拉伯糖提高至m13578中的0.46g乙醇/g阿拉伯糖,这导致阿拉伯糖衍生乙醇的产率提高了4.5%(图14)。
虽然已经结合本发明的具体实施方式描述了本发明,但应该理解的是,所述权利要求的范围不应该受到实施例中阐述的优选实施方式的限制,而应该做出与作为整体的描述相一致的最广泛的解释。
参考文献
kwastke,lailc,mendan,jamesdt3rd,arefs,burkepv.genomicanalysesofanaerobicallyinducedgenesinsaccharomycescerevisiae:functionalrolesofrox1andotherfactorsinmediatingtheanoxicresponse.jbacteriol.2002jan;184(1):250-65.
mbonyi,k.,beullens,m.,detremerie,k.,geerts,l.,andthevelein,j.m.(1988).requirementofonefunctionalrasgeneandinabilityofanoncogenicrasvarianttomediatetheglucose-inducedcyclicampsignalintheyeastsaccharomycescerevisiae.mol.cell.biol.8,3051-3057.
taisl,boervm,daran-lapujadep,walshmc,dewindejh,daranjm,pronkjt.two-dimensionaltranscriptomeanalysisinchemostatcultures.combinatorialeffectsofoxygenavailabilityandmacronutrientlimitationinsaccharomycescerevisiae.jbiolchem.2005jan7;280(1):437-47.
temeles,g.l.,gibbs,j.b.,d’alonzo,j.s.,sigal,i.s.,andscolnick,e.m.(1985).yeastandmammalianrasproteinshaveconservedbiochemicalproperties.nature313,700-703.
terlindejj,liangh,davisrw,steensmahy,vandijkenjp,pronkjt.genome-widetranscriptionalanalysisofaerobicandanaerobicchemostatculturesofsaccharomycescerevisiae.jbacteriol.1999dec;181(24):7409-13.
序列表
<110>拉勒曼德匈牙利流动性管理有限责任公司(lallemandhungaryliquiditymanagementllc)
<120>酵母中ras/camp/pka信号通路的调节(regulationoftheras/camp/pkasignalingpathwayinyeasts)
<130>n410440wo
<150>62/310251
<151>2016-03-18
<160>11
<170>patentinversion3.5
<210>1
<211>322
<212>prt
<213>酿酒酵母种(saccharomycescerevisiae)
<400>1
metproleuasnlysserasnileargglutyrlysleuvalvalval
151015
glyglyglyglyvalglylysseralaleuthrileglnleuthrgln
202530
serhisphevalaspglutyraspprothrilegluaspsertyrarg
354045
lysglnvalvalileaspaspgluvalserileleuaspileleuasp
505560
thralaglyglngluglutyrseralametarggluglntyrmetarg
65707580
asnglygluglypheleuleuvaltyrserilethrserlysserser
859095
leuaspgluleumetthrtyrtyrglnglnileleuargvallysasp
100105110
thrasptyrvalproilevalvalvalglyasnlysseraspleuglu
115120125
asnglulysglnvalsertyrglnaspglyleuasnmetalalysgln
130135140
metasnalapropheleugluthrseralalysglnalaileasnval
145150155160
gluglualaphetyrthrleualaargleuvalargaspgluglygly
165170175
lystyrasnlysthrleuthrgluasnaspasnserlysglnthrser
180185190
glnaspthrlysglyserglyalaasnservalproargasnsergly
195200205
glyhisarglysmetserasnalaalaasnglylysasnvalasnser
210215220
serthrthrvalvalasnalaargasnalaserilegluserlysthr
225230235240
glyleualaglyasnglnalathrasnglylysthrglnthrasparg
245250255
thrasnileaspasnserthrglyglnalaglyglnalaasnalagln
260265270
seralaasnthrvalasnasnargvalasnasnasnserlysalagly
275280285
glnvalserasnalalysglnalaarglysglnglnalaalaprogly
290295300
glyasnthrserglualaserlysserglyserglyglycyscysile
305310315320
ileser
<210>2
<211>322
<212>prt
<213>酿酒酵母种(saccharomycescerevisiae)
<400>2
metproleuasnlysserasnileargglutyrlysleuvalvalval
151015
glyglyglyglyvalglylysseralaleuthrileglnleuthrgln
202530
serhisphevalaspglutyraspprothrilegluaspsertyrarg
354045
lysglnvalvalileaspaspgluvalserileleuaspileleuasp
505560
thrthrglyglngluglutyrseralametarggluglntyrmetarg
65707580
asnglygluglypheleuleuvaltyrserilethrserlysserser
859095
leuaspgluleumetthrtyrtyrglnglnileleuargvallysasp
100105110
thrasptyrvalproilevalvalvalglyasnlysseraspleuglu
115120125
asnglulysglnvalsertyrglnaspglyleuasnmetalalysgln
130135140
metasnalapropheleugluthrseralalysglnalaileasnval
145150155160
gluglualaphetyrthrleualaargleuvalargaspgluglygly
165170175
lystyrasnlysthrleuthrgluasnaspasnserlysglnthrser
180185190
glnaspthrlysglyserglyalaasnservalproargasnsergly
195200205
glyhisarglysmetserasnalaalaasnglylysasnvalasnser
210215220
serthrthrvalvalasnalaargasnalaserilegluserlysthr
225230235240
glyleualaglyasnglnalathrasnglylysthrglnthrasparg
245250255
thrasnileaspasnserthrglyglnalaglyglnalaasnalagln
260265270
seralaasnthrvalasnasnargvalasnasnasnserlysalagly
275280285
glnvalserasnalalysglnalaarglysglnglnalaalaprogly
290295300
glyasnthrserglualaserlysserglyserglyglycyscysile
305310315320
ileser
<210>3
<211>969
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>variation
<223>编码突变ras2p的核酸序列(nucleicacidsequencecodingforamutatedras2p)
<220>
<221>variation
<222>(198)..(198)
<223>n=a,c,t或g
<220>
<221>misc_feature
<222>(198)..(198)
<223>n是a,c,g,或t
<400>3
atgcctttgaacaagtcgaacataagagagtacaagctagtcgtcgttggtggtggtggt60
gttggtaaatctgctttgaccatacaattgacccaatcgcactttgtagatgaatacgat120
cccacaattgaggattcatacaggaagcaagtggtgattgatgatgaagtgtctatattg180
gacattttggatactacngggcaggaagaatactctgctatgagggaacaatacatgcgc240
aacggcgaaggattcctattggtttactctataacatccaagtcgtctcttgatgagctt300
atgacttactatcaacagatattgagagtcaaagataccgactatgttccaattgtggtt360
gttggtaacaaatctgatttagaaaacgaaaaacaggtctcttaccaggacgggttgaac420
atggcaaagcaaatgaacgctcctttcttggagacatctgctaagcaagcaatcaacgtg480
gaagaggcgttttacactctagcacgtttagttagagacgaaggcggcaagtacaacaag540
actttgacggaaaatgacaactccaagcaaacttctcaagatacaaaagggagcggtgcc600
aactctgtgcctagaaatagcggtggccacaggaagatgagcaatgctgccaacggtaaa660
aatgtgaacagtagcacaactgtcgtgaatgccaggaatgcaagcatagagagtaagaca720
gggttggcaggcaaccaggcgacaaatggtaagacacaaactgatcgcaccaatatagac780
aattccacgggccaagctggtcaggccaacgctcaaagcgctaatacggttaataatcgt840
gtaaataataatagtaaggccggtcaagtttcaaatgctaaacaggctaggaagcagcaa900
gctgcacccggcggtaacaccagtgaagcctccaagagcggatcgggtggctgttgtatt960
ataagttaa969
<210>4
<211>969
<212>dna
<213>酿酒酵母种(saccharomycescerevisiae)
<400>4
atgcctttgaacaagtcgaacataagagagtacaagctagtcgtcgttggtggtggtggt60
gttggtaaatctgctttgaccatacaattgacccaatcgcactttgtagatgaatacgat120
cccacaattgaggattcatacaggaagcaagtggtgattgatgatgaagtgtctatattg180
gacattttggatactgcagggcaggaagaatactctgctatgagggaacaatacatgcgc240
aacggcgaaggattcctattggtttactctataacatccaagtcgtctcttgatgagctt300
atgacttactatcaacagatattgagagtcaaagataccgactatgttccaattgtggtt360
gttggtaacaaatctgatttagaaaacgaaaaacaggtctcttaccaggacgggttgaac420
atggcaaagcaaatgaacgctcctttcttggagacatctgctaagcaagcaatcaacgtg480
gaagaggcgttttacactctagcacgtttagttagagacgaaggcggcaagtacaacaag540
actttgacggaaaatgacaactccaagcaaacttctcaagatacaaaagggagcggtgcc600
aactctgtgcctagaaatagcggtggccacaggaagatgagcaatgctgccaacggtaaa660
aatgtgaacagtagcacaactgtcgtgaatgccaggaatgcaagcatagagagtaagaca720
gggttggcaggcaaccaggcgacaaatggtaagacacaaactgatcgcaccaatatagac780
aattccacgggccaagctggtcaggccaacgctcaaagcgctaatacggttaataatcgt840
gtaaataataatagtaaggccggtcaagtttcaaatgctaaacaggctaggaagcagcaa900
gctgcacccggcggtaacaccagtgaagcctccaagagcggatcgggtggctgttgtatt960
ataagttaa969
<210>5
<211>969
<212>dna
<213>酿酒酵母种(saccharomycescerevisiae)
<400>5
atgcctttgaacaagtcgaacataagagagtacaagctagtcgtcgttggtggtggtggt60
gttggtaaatctgctttgaccatacaattgacccaatcgcactttgtagatgaatacgat120
cccacaattgaggattcatacaggaagcaagtggtgattgatgatgaagtgtctatattg180
gacattttggatactacagggcaggaagaatactctgctatgagggaacaatacatgcgc240
aacggcgaaggattcctattggtttactctataacatccaagtcgtctcttgatgagctt300
atgacttactatcaacagatattgagagtcaaagataccgactatgttccaattgtggtt360
gttggtaacaaatctgatttagaaaacgaaaaacaggtctcttaccaggacgggttgaac420
atggcaaagcaaatgaacgctcctttcttggagacatctgctaagcaagcaatcaacgtg480
gaagaggcgttttacactctagcacgtttagttagagacgaaggcggcaagtacaacaag540
actttgacggaaaatgacaactccaagcaaacttctcaagatacaaaagggagcggtgcc600
aactctgtgcctagaaatagcggtggccacaggaagatgagcaatgctgccaacggtaaa660
aatgtgaacagtagcacaactgtcgtgaatgccaggaatgcaagcatagagagtaagaca720
gggttggcaggcaaccaggcgacaaatggtaagacacaaactgatcgcaccaatatagac780
aattccacgggccaagctggtcaggccaacgctcaaagcgctaatacggttaataatcgt840
gtaaataataatagtaaggccggtcaagtttcaaatgctaaacaggctaggaagcagcaa900
gctgcacccggcggtaacaccagtgaagcctccaagagcggatcgggtggctgttgtatt960
ataagttaa969
<210>6
<211>9246
<212>dna
<213>酿酒酵母种(saccharomycescerevisiae)
<400>6
atgtcccagcccactaagaataagaagaaagaacacgggaccgattccaagtcatcccgc60
atgactcggacgttggttaatcatattctttttgaaagaattctcccgatccttccggtg120
gagtctaatctaagtacctattcggaagtggaagagtattcctcattcatttcatgcaga180
tctgtgctcattaacgttaccgtttcccaagatgcaaacgctatggtggaaggcaccttg240
gagttgatagaatcgcttcttcaagggcacgaaatcatttcagataagtgtagcagtgac300
gttattgaatcaatactgattatactaagattgttaagtgatgcgctagagtataattgg360
caaaatcaagaaagccttcattacaacgacatttcgactcacgtagaacatgaccaagaa420
cagaagtacagaccaaagcttaacagtattctgcccgactactcgtcgactcattccaat480
ggcaacaaacactttttccaccagagcaaacctcaggcactgataccggaactggcatcg540
aaattgcttgagagttgcgcgaagttgaagttcaatacaagaactttgcaaattttacaa600
aatatgatcagtcatgttcatggaaacattctaacgactttgagttcctcgattcttccc660
cgccacaaatcctatctgacaaggcacaaccatccttctcattgtaaaatgattgactct720
actctaggccatattctccgatttgtagcggcttccaatccgtccgagtattttgaattt780
atcagaaagagtgtgcaagtgcccgtaacacagacacacacacacacgcattcacactcc840
cattcacactctttgccatcttccgtttataacagcatagtgccccactttgatcttttc900
agcttcatctatttaagcaagcataattttaagaaatacttggaactcatcaaaaactta960
tcggtgacgttaaggaaaacgatttatcattgcctacttttgcattacagcgccaaagca1020
ataatgttttggataatggctaggcctgcggaatattatgaactcttcaacttattaaaa1080
gataataacaatgaacactcgaaatccttaaacacgttaaaccatacacttttcgaggag1140
atccattcgacttttaatgtgaatagcatgataaccaccaatcaaaatgctcatcaaggc1200
tcatcttccccttcgtcctcctcgccatcgtcaccacctagctcatcatcatcggataac1260
aacaatcaaaacataatagcaaaatccttaagtcgtcagctttctcaccaccagtcatac1320
attcaacagcagtctgaaagaaaactacattcttcatggactacaaactctcaatcctct1380
acttcactgtcatcttcaacgtctaattcaacaacaactgatttctctactcacactcaa1440
ccaggagaatacgacccttccttaccagatactcccacgatgtctaacatcactattagt1500
gcatcttcattattatctcaaactccaactccaacaacacaattgcaacagcggttgaac1560
tcagcagctgcagccgccgccgcagctgcttcaccatcgaattccaccccaactggatac1620
acagcagagcaacaaagtcgcgcttcatacgatgcacacaaaactggccatactggtaag1680
gattatgacgaacattttttgtctgtcactcgtttggataatgttttggagttatacacg1740
cactttgatgatactgaggtactaccacacacatccgtactgaagtttttaactactttg1800
acaatgttcgatattgacctttttaatgaattaaacgctacatcattcaaatatattcct1860
gattgtactatgcatcgtccaaaagaaagaacaagttctttcaataatactgcacacgag1920
acaggttccgaaaagacttcgggtataaaacatattacacaaggcttaaagaaattaact1980
tctttaccttcctcaaccaaaaaaactgtaaaatttatgaagatgttgctaagaaattta2040
aatgggaatcaagctgtatcagatgttgccctcttagatacaatgagggccttactatca2100
ttctttacaatgacttctgcggtctttctcgtggatagaaacttaccctcagtacttttt2160
gccaagagactcatccccataatggggacaaatttaagcgtcggtcaagactggaattca2220
aaaataaataacagtttgatggtttgtttgaaaaaaaactccaccacgtttgttcaatta2280
caattaatattcttctcttcagctattcaattcgatcatgaattattactggcacgtctg2340
agcatcgatacaatggccaacaatttaaacatgcagaagctatgcctttatactgaagga2400
ttcaggatattcttcgacataccaagtaagaaggaattgcggaaggcaattgcggttaaa2460
atttctaaatttttcaaaacattattctccattatagcagatattcttttacaagaattt2520
ccgtattttgatgagcaaatcaccgacatagttgcttccattcttgacggtacaattatc2580
aatgagtatggtacgaagaaacatttcaaggggagctcaccctctttatgttcgacaacc2640
cggtcaagatcaggatctacatctcaaagttcaatgacaccagtttctccgctgggactg2700
gatactgatatacgtccaatgaacaccctgtctttagttggttcaagtacttcaagaaat2760
tctgacaacgttaattcattaaacagttcaccaaagaacttgtcttctgatccatacttg2820
tcacatcttgtggccccaagagcacgtcatgctttaggtgggccatctagtattataagg2880
aataaaataccgactacattgacttcacctccaggaacggaaaaatcttcaccagtacaa2940
cgtccgcaaacggaaagcatcagtgccacaccaatggccataacaaattctactccatta3000
tcgtcggcagcattcggaattcgatcgcctttgcagaaaataagaacgaggcgttattcc3060
gatgaaagtttaggaaaattcatgaaatcaacaaataattacattcaagaacatttgata3120
ccaaaagatttgaatgaagcaactcttcaagatgctagaagaataatgattaatattttc3180
agtatttttaagagaccgaatagttacttcatcattcctcacaatataaattcgaattta3240
caatgggtttcgcaggattttagaaatattatgaaaccgattttcgtcgccatcgtaagt3300
ccggatgtagatttacagaatactgctcaatcattcatggataccttattatcgaatgtt3360
attacttatggtgaatcagatgagaatatcagtattgaagggtatcatcttctttgcagt3420
tacactgtaacattatttgcaatgggccttttcgatttgaaaattaataatgaaaagcgt3480
caaattctcttggatataactgtcaagtttatgaaggttagatcacatttagcagggatc3540
gcggaggcctcacaccacatggaatacataagtgattctgaaaaactcacctttccgctg3600
attatggggactgttggtagggccctatttgtttcattatactctagtcaacaaaaaatt3660
gaaaagactttaaatattgcttacacagagtatctttctgcaatcaattttcatgagagg3720
aatattgatgatgctgataaaacttgggttcataatattgagtttgtagaagcgatgtgt3780
catgacaactacacaacttctggttcaattgctttccaaaggaggacaagaaataatatt3840
ttacgatttgctactattcctaacgctatcttacttgattctatgaggatgatctataag3900
aagtggcatacttacacacacagtaaaagtttagaaaaacaagaacggaacgacttcaga3960
aatttcgcgggtattttagcctctttgtcgggtatcctattcatcaataaaaagatattg4020
caagaaatgtatccatacctactcgacaccgtttcagaattgaaaaaaaatatagactct4080
tttatctcaaaacaatgccaatggttaaactatccggatttattaacgagagaaaattca4140
agagatattctaagtgtagaactgcatcctttgtcttttaacttactttttaataatttg4200
aggctcaagttaaaagaacttgcttgttcagacttatcaataccagaaaatgaaagttcc4260
tatgttttattagaacaaataatcaaaatgctgcggacaatcctaggtcgtgatgatgac4320
aattatgtaatgatgcttttttccacagagattgtagatcttattgatttattgacagat4380
gaaataaaaaaaataccagcctattgtccaaaatatctcaaggcaattattcaaatgacc4440
aaaatgttcagtgccttgcagcactcagaggttaatttaggtgtcaaaaatcattttcac4500
gttaaaaataaatggttgaggcaaatcactgattggtttcaagtgagtattgcgagagag4560
tacgatttcgaaaacttgtcaaaacctctaaaagaaatggatttggtaaaaagagacatg4620
gatattctatacatagatacggcaatcgaagcttcaaccgctattgcgtacctcacgaga4680
catactttcttagagattccacctgccgcgtcagatcccgaactatctcgatctaggtct4740
gtgatatttgggttttatttcaacatcttaatgaaaggccttgaaaaaagtagtgatcgt4800
gacaattacccagtattcttgaggcacaaaatgagtgtcctcaacgacaatgtaatactt4860
tcattaacaaatctttcaaacaccaatgttgatgcgagtttgcagttcaccttaccgatg4920
ggctattcgggaaatcgaaacattaggaatgcatttttggaggtcttcattaatatcgtt4980
acgaactatcggacatacacggctaaaactgaccttggaaaattagaggcagcagacaaa5040
tttttgcgatatacgattgaacatccccagctatcgtcctttggagcagcggtttgtccc5100
gctagcgatattgatgcttatgctgctggcttaataaatgcatttgaaacgaggaatgcc5160
acccacattgtagtggcacagttgattaaaaatgaaattgaaaaatcttccagacctacg5220
gatatccttagaagaaatagctgtgctacgagatcattatctatgctagccaggtccaag5280
ggtaacgaatatttgattcgcactttgcaaccattactaaaaaaaattatccagaacaga5340
gatttttttgaaattgagaaactaaaaccggaagattcagatgctgaacgtcaaatagag5400
ctcttcgttaaatacatgaatgaattattggaatccatatccaactccgtatcttatttt5460
ccccctcctttattttatatttgccaaaacatttataaagttgcgtgtgaaaaatttccg5520
gatcacgcaattatcgccgctgggtctttcgtgtttttacggtttttttgtcctgcttta5580
gtcagccctgattctgaaaatatcatagatatttctcacttgagcgaaaagcgtaccttc5640
atcagcttggctaaagttatccaaaatattgccaatggctcagaaaatttctccagatgg5700
ccagctttgtgttcccaaaaggattttcttaaagaatgtagcgatagaattttcagattc5760
ctagctgaactttgtagaacagatcgcacgatagacatccaagtgagaacagacccaacg5820
ccaattgcatttgactatcaattccttcattcctttgtttacctttacggtcttgaggtg5880
agaaggaatgtgctaaatgaagcaaaacatgatgatggtgacattgatggtgacgatttc5940
tataagaccacatttttacttattgatgatgttcttggccaattaggccaacctaaaatg6000
gaattttccaatgaaataccaatatacataagagaacatatggacgactatccggaactg6060
tatgagttcatgaataggcacgcgttcagaaacattgagacttcaacagcgtacagccca6120
agcgttcacgagtccacctcaagtgaaggcattccaattattacgttaacaatgtcaaat6180
ttctcagacagacatgtggacattgatacagttgcttacaagttcttgcaaatttatgct6240
cgaatctggaccaccaaacactgtttaataatcgactgtacagaatttgacgagggaggg6300
cttgatatgaggaaatttatttctttggttatgggactattaccagaagttgcacccaaa6360
aattgtataggctgttactactttaacgtaaacgagacatttatggataattatggaaaa6420
tgtttggacaaagacaacgtatatgtttcctcgaaaattcctcattatttcattaatagt6480
aactctgatgaaggacttatgaaatctgtgggtataactggacaagggttgaaggttctg6540
caagatattcgtgtctctctgcatgatatcacgctttatgacgaaaaaagaaatagattt6600
acgccggtatcgttgaaaataggcgatatttactttcaagtcttgcatgaaactcctagg6660
caatataaaataagagacatgggtactttattcgacgtaaaattcaatgatgtctacgaa6720
attagccgaatatttgaagtacatgtttcgtcaataactggagtggcagctgaatttaca6780
gtaacttttcaggacgagagaaggttgatttttagtagtccgaaataccttgaaattgtg6840
aagatgttctattacgcacagatccggttagaaagtgaatatgaaatggataataattcg6900
agtacctcctccccaaattcaaacaacaaggacaaacagcagaaagagagaacaaaatta6960
ttgtgccacctactgttagtatctcttattggtctgtttgatgagagtaaaaaaatgaaa7020
aacagttcgtataacctaatagctgccactgaggcgtcatttggcttgaactttggctcc7080
cattttcatcgctctcccgaggtgtacgtccccgaatatactacaacatttttaggtgtt7140
attggaaagtctcttgcagagtctaatccagaactcacagcctatatgtttatctatgtt7200
ttggaggcattgaagaacaacgtaattcctcacgtttacatccctcataccatttgcggt7260
ttgtcttattggatccctaatttataccaacatgtgtatttggctgatgatgaagaaggc7320
cccgaaaacatatctcacattttccgaattcttatcaggctctctgtgagagagactgac7380
tttaaagccgtatacatgcaatatgtttggttgctacttttagatgatggccgcttaact7440
gacattatcgttgatgaagttattaatcatgcgttagaaagagactccgaaaaccgcgat7500
tggaagaaaacaatatcgttactgactgtcctacccactactgaggttgctaataacatt7560
attcaaaaaatattggcaaaaattagatcatttttaccgtcattgaagttagaagctatg7620
acccaaagttggtctgaactaacaatattagttaagataagcatccacgttttttttgaa7680
acttctttgctggtacagatgtacttaccagagatcctgtttatcgtatccttattaatt7740
gatgttggtccaagggaactcagatcatcactacaccagctattaatgaatgtatgccat7800
tccttggctattgactcagctttatcacaagatcatagaaataatctagatgaaataagt7860
gatatatttgcacatcaaaaggtgaagtttatgtttgggttcagcgaggacaaaggacga7920
attttacagatttttagcgcttcttcttttgcaagcaagtttaatattctggatttcttc7980
atcaataatatattattgctgatggaatattcttcaacgtacgaagcaaacgtgtggaag8040
acaagatacaagaaatatgtcttggaatctgtgtttacaagtaattcttttctttcggca8100
cgttcaatcatgattgttggtataatgggtaaatcttacataactgaagggttatgcaag8160
gctatgttaattgaaaccatgaaagttatcgccgaaccaaagattactgacgagcatctt8220
ttcttagccatatctcatatttttacttattccaaaattgttgaaggtttggatcccaac8280
cttgacttaatgaagcacttattttggttttcaacactcttccttgaatcacgtcacccg8340
ataatttttgagggtgcccttctctttgtgtcaaactgtataaggcgcctatacatggcc8400
cagtttgaaaatgaaagcgaaacatcattgataagtactttacttaaggggagaaagttt8460
gctcatacctttttaagcaagatagagaatcttagtggtattgtttggaatgaagataat8520
tttacacacattctgattttcatcattaataaaggactatccaatcctttcattaagagt8580
acggcttttgatttcttgaagatgatgtttagaaactcctactttgagcatcaaatcaat8640
cagaaatctgatcattatttgtgctatatgttcctattgtattttgttttaaactgcaat8700
caatttgaggaacttttaggtgacgttgattttgaaggagaaatggttaacattgaaaac8760
aagaacaccattcctaaaattttgttagagtggttgagttcggataacgaaaatgcaaac8820
attaccctctatcaaggtgcgatactgttcaaatgttcagttacggatgaaccaagtaaa8880
tttaggtttgcgttgattattaggcatctattgacaaagaaacccatttgtgcattgcgt8940
ttttacagtgttattcgtaacgaaataagaaaaatatcagcatttgagcaaaattcggat9000
tgtgttccacttgctttcgatattttaaacttattagtgacgcattcagagtctaattcg9060
ttagaaaaacttcacgaagaatccattgaacgtctaaccaaaagaggtttatcgattgtg9120
acttcttctggtatatttgcgaagaattccgacatgatgatacctttagatgtaaaacct9180
gaagatatctatgaacgtaagagaataatgacaatgattttatcaaggatgtcatgttct9240
gcttag9246
<210>7
<211>9248
<212>dna
<213>酿酒酵母种(saccharomycescerevisiae)
<400>7
atgtcccagcccactaagaataagaagaaagaacacgggaccgattccaagtcatcccgc60
atgactcggacgttggttaatcatattctttttgaaagaattctcccgatccttccggtg120
gagtctaatctaagtacctattcggaagtggaagagtattcctcattcatttcatgcaga180
tctgtgctcattaacgttaccgtttcccaagatgcaaacgctatggtggaaggcaccttg240
gagttgatagaatcgcttcttcaagggcacgaaatcatttcagataagtgtagcagtgac300
gttattgaatcaatactgattatactaagattgttaagtgatgcgctagagtataattgg360
caaaatcaagaaagccttcattacaacgacatttcgactcacgtagaacatgaccaagaa420
cagaagtacagaccaaagcttaacagtattctgcccgactactcgtcgactcattccaat480
ggcaacaaacactttttccaccagagcaaacctcaggcactgataccggaactggcatcg540
aaattgcttgagagttgcgcgaagttgaagttcaatacaagaactttgcaaattttacaa600
aatatgatcagtcatgttcatggaaacattctaacgactttgagttcctcgattcttccc660
cgccacaaatcctatctgacaaggcacaaccatccttctcattgtaaaatgattgactct720
actctaggccatattctccgatttgtagcggcttccaatccgtccgagtattttgaattt780
atcagaaagagtgtgcaagtgcccgtaacacagacacacacacacacacgcattcacact840
cccattcacactctttgccatcttccgtttataacagcatagtgccccactttgatcttt900
tcagcttcatctatttaagcaagcataattttaagaaatacttggaactcatcaaaaact960
tatcggtgacgttaaggaaaacgatttatcattgcctacttttgcattacagcgccaaag1020
caataatgttttggataatggctaggcctgcggaatattatgaactcttcaacttattaa1080
aagataataacaatgaacactcgaaatccttaaacacgttaaaccatacacttttcgagg1140
agatccattcgacttttaatgtgaatagcatgataaccaccaatcaaaatgctcatcaag1200
gctcatcttccccttcgtcctcctcgccatcgtcaccacctagctcatcatcatcggata1260
acaacaatcaaaacataatagcaaaatccttaagtcgtcagctttctcaccaccagtcat1320
acattcaacagcagtctgaaagaaaactacattcttcatggactacaaactctcaatcct1380
ctacttcactgtcatcttcaacgtctaattcaacaacaactgatttctctactcacactc1440
aaccaggagaatacgacccttccttaccagatactcccacgatgtctaacatcactatta1500
gtgcatcttcattattatctcaaactccaactccaacaacacaattgcaacagcggttga1560
actcagcagctgcagccgccgccgcagctgcttcaccatcgaattccaccccaactggat1620
acacagcagagcaacaaagtcgcgcttcatacgatgcacacaaaactggccatactggta1680
aggattatgacgaacattttttgtctgtcactcgtttggataatgttttggagttataca1740
cgcactttgatgatactgaggtactaccacacacatccgtactgaagtttttaactactt1800
tgacaatgttcgatattgacctttttaatgaattaaacgctacatcattcaaatatattc1860
ctgattgtactatgcatcgtccaaaagaaagaacaagttctttcaataatactgcacacg1920
agacaggttccgaaaagacttcgggtataaaacatattacacaaggcttaaagaaattaa1980
cttctttaccttcctcaaccaaaaaaactgtaaaatttatgaagatgttgctaagaaatt2040
taaatgggaatcaagctgtatcagatgttgccctcttagatacaatgagggccttactat2100
cattctttacaatgacttctgcggtctttctcgtggatagaaacttaccctcagtacttt2160
ttgccaagagactcatccccataatggggacaaatttaagcgtcggtcaagactggaatt2220
caaaaataaataacagtttgatggtttgtttgaaaaaaaactccaccacgtttgttcaat2280
tacaattaatattcttctcttcagctattcaattcgatcatgaattattactggcacgtc2340
tgagcatcgatacaatggccaacaatttaaacatgcagaagctatgcctttatactgaag2400
gattcaggatattcttcgacataccaagtaagaaggaattgcggaaggcaattgcggtta2460
aaatttctaaatttttcaaaacattattctccattatagcagatattcttttacaagaat2520
ttccgtattttgatgagcaaatcaccgacatagttgcttccattcttgacggtacaatta2580
tcaatgagtatggtacgaagaaacatttcaaggggagctcaccctctttatgttcgacaa2640
cccggtcaagatcaggatctacatctcaaagttcaatgacaccagtttctccgctgggac2700
tggatactgatatacgtccaatgaacaccctgtctttagttggttcaagtacttcaagaa2760
attctgacaacgttaattcattaaacagttcaccaaagaacttgtcttctgatccatact2820
tgtcacatcttgtggccccaagagcacgtcatgctttaggtgggccatctagtattataa2880
ggaataaaataccgactacattgacttcacctccaggaacggaaaaatcttcaccagtac2940
aacgtccgcaaacggaaagcatcagtgccacaccaatggccataacaaattctactccat3000
tatcgtcggcagcattcggaattcgatcgcctttgcagaaaataagaacgaggcgttatt3060
ccgatgaaagtttaggaaaattcatgaaatcaacaaataattacattcaagaacatttga3120
taccaaaagatttgaatgaagcaactcttcaagatgctagaagaataatgattaatattt3180
tcagtatttttaagagaccgaatagttacttcatcattcctcacaatataaattcgaatt3240
tacaatgggtttcgcaggattttagaaatattatgaaaccgattttcgtcgccatcgtaa3300
gtccggatgtagatttacagaatactgctcaatcattcatggataccttattatcgaatg3360
ttattacttatggtgaatcagatgagaatatcagtattgaagggtatcatcttctttgca3420
gttacactgtaacattatttgcaatgggccttttcgatttgaaaattaataatgaaaagc3480
gtcaaattctcttggatataactgtcaagtttatgaaggttagatcacatttagcaggga3540
tcgcggaggcctcacaccacatggaatacataagtgattctgaaaaactcacctttccgc3600
tgattatggggactgttggtagggccctatttgtttcattatactctagtcaacaaaaaa3660
ttgaaaagactttaaatattgcttacacagagtatctttctgcaatcaattttcatgaga3720
ggaatattgatgatgctgataaaacttgggttcataatattgagtttgtagaagcgatgt3780
gtcatgacaactacacaacttctggttcaattgctttccaaaggaggacaagaaataata3840
ttttacgatttgctactattcctaacgctatcttacttgattctatgaggatgatctata3900
agaagtggcatacttacacacacagtaaaagtttagaaaaacaagaacggaacgacttca3960
gaaatttcgcgggtattttagcctctttgtcgggtatcctattcatcaataaaaagatat4020
tgcaagaaatgtatccatacctactcgacaccgtttcagaattgaaaaaaaatatagact4080
cttttatctcaaaacaatgccaatggttaaactatccggatttattaacgagagaaaatt4140
caagagatattctaagtgtagaactgcatcctttgtcttttaacttactttttaataatt4200
tgaggctcaagttaaaagaacttgcttgttcagacttatcaataccagaaaatgaaagtt4260
cctatgttttattagaacaaataatcaaaatgctgcggacaatcctaggtcgtgatgatg4320
acaattatgtaatgatgcttttttccacagagattgtagatcttattgatttattgacag4380
atgaaataaaaaaaataccagcctattgtccaaaatatctcaaggcaattattcaaatga4440
ccaaaatgttcagtgccttgcagcactcagaggttaatttaggtgtcaaaaatcattttc4500
acgttaaaaataaatggttgaggcaaatcactgattggtttcaagtgagtattgcgagag4560
agtacgatttcgaaaacttgtcaaaacctctaaaagaaatggatttggtaaaaagagaca4620
tggatattctatacatagatacggcaatcgaagcttcaaccgctattgcgtacctcacga4680
gacatactttcttagagattccacctgccgcgtcagatcccgaactatctcgatctaggt4740
ctgtgatatttgggttttatttcaacatcttaatgaaaggccttgaaaaaagtagtgatc4800
gtgacaattacccagtattcttgaggcacaaaatgagtgtcctcaacgacaatgtaatac4860
tttcattaacaaatctttcaaacaccaatgttgatgcgagtttgcagttcaccttaccga4920
tgggctattcgggaaatcgaaacattaggaatgcatttttggaggtcttcattaatatcg4980
ttacgaactatcggacatacacggctaaaactgaccttggaaaattagaggcagcagaca5040
aatttttgcgatatacgattgaacatccccagctatcgtcctttggagcagcggtttgtc5100
ccgctagcgatattgatgcttatgctgctggcttaataaatgcatttgaaacgaggaatg5160
ccacccacattgtagtggcacagttgattaaaaatgaaattgaaaaatcttccagaccta5220
cggatatccttagaagaaatagctgtgctacgagatcattatctatgctagccaggtcca5280
agggtaacgaatatttgattcgcactttgcaaccattactaaaaaaaattatccagaaca5340
gagatttttttgaaattgagaaactaaaaccggaagattcagatgctgaacgtcaaatag5400
agctcttcgttaaatacatgaatgaattattggaatccatatccaactccgtatcttatt5460
ttccccctcctttattttatatttgccaaaacatttataaagttgcgtgtgaaaaatttc5520
cggatcacgcaattatcgccgctgggtctttcgtgtttttacggtttttttgtcctgctt5580
tagtcagccctgattctgaaaatatcatagatatttctcacttgagcgaaaagcgtacct5640
tcatcagcttggctaaagttatccaaaatattgccaatggctcagaaaatttctccagat5700
ggccagctttgtgttcccaaaaggattttcttaaagaatgtagcgatagaattttcagat5760
tcctagctgaactttgtagaacagatcgcacgatagacatccaagtgagaacagacccaa5820
cgccaattgcatttgactatcaattccttcattcctttgtttacctttacggtcttgagg5880
tgagaaggaatgtgctaaatgaagcaaaacatgatgatggtgacattgatggtgacgatt5940
tctataagaccacatttttacttattgatgatgttcttggccaattaggccaacctaaaa6000
tggaattttccaatgaaataccaatatacataagagaacatatggacgactatccggaac6060
tgtatgagttcatgaataggcacgcgttcagaaacattgagacttcaacagcgtacagcc6120
caagcgttcacgagtccacctcaagtgaaggcattccaattattacgttaacaatgtcaa6180
atttctcagacagacatgtggacattgatacagttgcttacaagttcttgcaaatttatg6240
ctcgaatctggaccaccaaacactgtttaataatcgactgtacagaatttgacgagggag6300
ggcttgatatgaggaaatttatttctttggttatgggactattaccagaagttgcaccca6360
aaaattgtataggctgttactactttaacgtaaacgagacatttatggataattatggaa6420
aatgtttggacaaagacaacgtatatgtttcctcgaaaattcctcattatttcattaata6480
gtaactctgatgaaggacttatgaaatctgtgggtataactggacaagggttgaaggttc6540
tgcaagatattcgtgtctctctgcatgatatcacgctttatgacgaaaaaagaaatagat6600
ttacgccggtatcgttgaaaataggcgatatttactttcaagtcttgcatgaaactccta6660
ggcaatataaaataagagacatgggtactttattcgacgtaaaattcaatgatgtctacg6720
aaattagccgaatatttgaagtacatgtttcgtcaataactggagtggcagctgaattta6780
cagtaacttttcaggacgagagaaggttgatttttagtagtccgaaataccttgaaattg6840
tgaagatgttctattacgcacagatccggttagaaagtgaatatgaaatggataataatt6900
cgagtacctcctccccaaattcaaacaacaaggacaaacagcagaaagagagaacaaaat6960
tattgtgccacctactgttagtatctcttattggtctgtttgatgagagtaaaaaaatga7020
aaaacagttcgtataacctaatagctgccactgaggcgtcatttggcttgaactttggct7080
cccattttcatcgctctcccgaggtgtacgtccccgaatatactacaacatttttaggtg7140
ttattggaaagtctcttgcagagtctaatccagaactcacagcctatatgtttatctatg7200
ttttggaggcattgaagaacaacgtaattcctcacgtttacatccctcataccatttgcg7260
gtttgtcttattggatccctaatttataccaacatgtgtatttggctgatgatgaagaag7320
gccccgaaaacatatctcacattttccgaattcttatcaggctctctgtgagagagactg7380
actttaaagccgtatacatgcaatatgtttggttgctacttttagatgatggccgcttaa7440
ctgacattatcgttgatgaagttattaatcatgcgttagaaagagactccgaaaaccgcg7500
attggaagaaaacaatatcgttactgactgtcctacccactactgaggttgctaataaca7560
ttattcaaaaaatattggcaaaaattagatcatttttaccgtcattgaagttagaagcta7620
tgacccaaagttggtctgaactaacaatattagttaagataagcatccacgttttttttg7680
aaacttctttgctggtacagatgtacttaccagagatcctgtttatcgtatccttattaa7740
ttgatgttggtccaagggaactcagatcatcactacaccagctattaatgaatgtatgcc7800
attccttggctattgactcagctttatcacaagatcatagaaataatctagatgaaataa7860
gtgatatatttgcacatcaaaaggtgaagtttatgtttgggttcagcgaggacaaaggac7920
gaattttacagatttttagcgcttcttcttttgcaagcaagtttaatattctggatttct7980
tcatcaataatatattattgctgatggaatattcttcaacgtacgaagcaaacgtgtgga8040
agacaagatacaagaaatatgtcttggaatctgtgtttacaagtaattcttttctttcgg8100
cacgttcaatcatgattgttggtataatgggtaaatcttacataactgaagggttatgca8160
aggctatgttaattgaaaccatgaaagttatcgccgaaccaaagattactgacgagcatc8220
ttttcttagccatatctcatatttttacttattccaaaattgttgaaggtttggatccca8280
accttgacttaatgaagcacttattttggttttcaacactcttccttgaatcacgtcacc8340
cgataatttttgagggtgcccttctctttgtgtcaaactgtataaggcgcctatacatgg8400
cccagtttgaaaatgaaagcgaaacatcattgataagtactttacttaaggggagaaagt8460
ttgctcatacctttttaagcaagatagagaatcttagtggtattgtttggaatgaagata8520
attttacacacattctgattttcatcattaataaaggactatccaatcctttcattaaga8580
gtacggcttttgatttcttgaagatgatgtttagaaactcctactttgagcatcaaatca8640
atcagaaatctgatcattatttgtgctatatgttcctattgtattttgttttaaactgca8700
atcaatttgaggaacttttaggtgacgttgattttgaaggagaaatggttaacattgaaa8760
acaagaacaccattcctaaaattttgttagagtggttgagttcggataacgaaaatgcaa8820
acattaccctctatcaaggtgcgatactgttcaaatgttcagttacggatgaaccaagta8880
aatttaggtttgcgttgattattaggcatctattgacaaagaaacccatttgtgcattgc8940
gtttttacagtgttattcgtaacgaaataagaaaaatatcagcatttgagcaaaattcgg9000
attgtgttccacttgctttcgatattttaaacttattagtgacgcattcagagtctaatt9060
cgttagaaaaacttcacgaagaatccattgaacgtctaaccaaaagaggtttatcgattg9120
tgacttcttctggtatatttgcgaagaattccgacatgatgatacctttagatgtaaaac9180
ctgaagatatctatgaacgtaagagaataatgacaatgattttatcaaggatgtcatgtt9240
ctgcttag9248
<210>8
<211>3081
<212>prt
<213>酿酒酵母种(saccharomycescerevisiae)
<400>8
metserglnprothrlysasnlyslyslysgluhisglythraspser
151015
lysserserargmetthrargthrleuvalasnhisileleupheglu
202530
argileleuproileleuprovalgluserasnleuserthrtyrser
354045
gluvalgluglutyrserserpheilesercysargservalleuile
505560
asnvalthrvalserglnaspalaasnalametvalgluglythrleu
65707580
gluleuilegluserleuleuglnglyhisgluileileserasplys
859095
cysserseraspvalilegluserileleuileileleuargleuleu
100105110
seraspalaleuglutyrasntrpglnasnglngluserleuhistyr
115120125
asnaspileserthrhisvalgluhisaspglngluglnlystyrarg
130135140
prolysleuasnserileleuproasptyrserserthrhisserasn
145150155160
glyasnlyshisphephehisglnserlysproglnalaleuilepro
165170175
gluleualaserlysleuleuglusercysalalysleulyspheasn
180185190
thrargthrleuglnileleuglnasnmetileserhisvalhisgly
195200205
asnileleuthrthrleuserserserileleuproarghislysser
210215220
tyrleuthrarghisasnhisproserhiscyslysmetileaspser
225230235240
thrleuglyhisileleuargphevalalaalaserasnproserglu
245250255
tyrpheglupheilearglysservalglnvalprovalthrglnthr
260265270
histhrhisthrhisserhisserhisserhisserleuproserser
275280285
valtyrasnserilevalprohispheaspleupheserpheiletyr
290295300
leuserlyshisasnphelyslystyrleugluleuilelysasnleu
305310315320
servalthrleuarglysthriletyrhiscysleuleuleuhistyr
325330335
seralalysalailemetphetrpilemetalaargproalaglutyr
340345350
tyrgluleupheasnleuleulysaspasnasnasngluhisserlys
355360365
serleuasnthrleuasnhisthrleupheglugluilehisserthr
370375380
pheasnvalasnsermetilethrthrasnglnasnalahisglngly
385390395400
serserserproserserserserproserserproproserserser
405410415
serseraspasnasnasnglnasnileilealalysserleuserarg
420425430
glnleuserhishisglnsertyrileglnglnglnsergluarglys
435440445
leuhissersertrpthrthrasnserglnserserthrserleuser
450455460
serserthrserasnserthrthrthrasppheserthrhisthrgln
465470475480
proglyglutyraspproserleuproaspthrprothrmetserasn
485490495
ilethrileseralaserserleuleuserglnthrprothrprothr
500505510
thrglnleuglnglnargleuasnseralaalaalaalaalaalaala
515520525
alaalaserproserasnserthrprothrglytyrthralaglugln
530535540
glnserargalasertyraspalahislysthrglyhisthrglylys
545550555560
asptyraspgluhispheleuservalthrargleuaspasnvalleu
565570575
gluleutyrthrhispheaspaspthrgluvalleuprohisthrser
580585590
valleulyspheleuthrthrleuthrmetpheaspileaspleuphe
595600605
asngluleuasnalathrserphelystyrileproaspcysthrmet
610615620
hisargprolysgluargthrserserpheasnasnthralahisglu
625630635640
thrglyserglulysthrserglyilelyshisilethrglnglyleu
645650655
lyslysleuthrserleuproserserthrlyslysthrvallysphe
660665670
metlysmetleuleuargasnleuasnglyasnglnalavalserasp
675680685
valalaleuleuaspthrmetargalaleuleuserphephethrmet
690695700
thrseralavalpheleuvalaspargasnleuproservalleuphe
705710715720
alalysargleuileproilemetglythrasnleuservalglygln
725730735
asptrpasnserlysileasnasnserleumetvalcysleulyslys
740745750
asnserthrthrphevalglnleuglnleuilephepheserserala
755760765
ileglnpheasphisgluleuleuleualaargleuserileaspthr
770775780
metalaasnasnleuasnmetglnlysleucysleutyrthrglugly
785790795800
pheargilephepheaspileproserlyslysgluleuarglysala
805810815
ilealavallysileserlysphephelysthrleupheserileile
820825830
alaaspileleuleuglnglupheprotyrpheaspgluglnilethr
835840845
aspilevalalaserileleuaspglythrileileasnglutyrgly
850855860
thrlyslyshisphelysglyserserproserleucysserthrthr
865870875880
argserargserglyserthrserglnsersermetthrprovalser
885890895
proleuglyleuaspthraspileargprometasnthrleuserleu
900905910
valglyserserthrserargasnseraspasnvalasnserleuasn
915920925
serserprolysasnleuserseraspprotyrleuserhisleuval
930935940
alaproargalaarghisalaleuglyglyproserserileilearg
945950955960
asnlysileprothrthrleuthrserproproglythrglulysser
965970975
serprovalglnargproglnthrgluserileseralathrpromet
980985990
alailethrasnserthrproleuserseralaalapheglyilearg
99510001005
serproleuglnlysileargthrargargtyrseraspgluser
101010151020
leuglylysphemetlysserthrasnasntyrileglngluhis
102510301035
leuileprolysaspleuasnglualathrleuglnaspalaarg
104010451050
argilemetileasnilepheserilephelysargproasnser
105510601065
tyrpheileileprohisasnileasnserasnleuglntrpval
107010751080
serglnasppheargasnilemetlysproilephevalalaile
108510901095
valserproaspvalaspleuglnasnthralaglnserphemet
110011051110
aspthrleuleuserasnvalilethrtyrglygluseraspglu
111511201125
asnileserilegluglytyrhisleuleucyssertyrthrval
113011351140
thrleuphealametglyleupheaspleulysileasnasnglu
114511501155
lysargglnileleuleuaspilethrvallysphemetlysval
116011651170
argserhisleualaglyilealaglualaserhishismetglu
117511801185
tyrileseraspserglulysleuthrpheproleuilemetgly
119011951200
thrvalglyargalaleuphevalserleutyrserserglngln
120512101215
lysileglulysthrleuasnilealatyrthrglutyrleuser
122012251230
alaileasnphehisgluargasnileaspaspalaasplysthr
123512401245
trpvalhisasnilegluphevalglualametcyshisaspasn
125012551260
tyrthrthrserglyserilealapheglnargargthrargasn
126512701275
asnileleuargphealathrileproasnalaileleuleuasp
128012851290
sermetargmetiletyrlyslystrphisthrtyrthrhisser
129513001305
lysserleuglulysglngluargasnasppheargasnpheala
131013151320
glyileleualaserleuserglyileleupheileasnlyslys
132513301335
ileleuglnglumettyrprotyrleuleuaspthrvalserglu
134013451350
leulyslysasnileaspserpheileserlysglncysglntrp
135513601365
leuasntyrproaspleuleuthrarggluasnserargaspile
137013751380
leuservalgluleuhisproleuserpheasnleuleupheasn
138513901395
asnleuargleulysleulysgluleualacysseraspleuser
140014051410
ileprogluasnglusersertyrvalleuleugluglnileile
141514201425
lysmetleuargthrileleuglyargaspaspaspasntyrval
143014351440
metmetleupheserthrgluilevalaspleuileaspleuleu
144514501455
thraspgluilelyslysileproalatyrcysprolystyrleu
146014651470
lysalaileileglnmetthrlysmetpheseralaleuglnhis
147514801485
sergluvalasnleuglyvallysasnhisphehisvallysasn
149014951500
lystrpleuargglnilethrasptrppheglnvalserileala
150515101515
argglutyraspphegluasnleuserlysproleulysglumet
152015251530
aspleuvallysargaspmetaspileleutyrileaspthrala
153515401545
ileglualaserthralailealatyrleuthrarghisthrphe
155015551560
leugluileproproalaalaseraspprogluleuserargser
156515701575
argservalilepheglyphetyrpheasnileleumetlysgly
158015851590
leuglulysserseraspargaspasntyrprovalpheleuarg
159516001605
hislysmetservalleuasnaspasnvalileleuserleuthr
161016151620
asnleuserasnthrasnvalaspalaserleuglnphethrleu
162516301635
prometglytyrserglyasnargasnileargasnalapheleu
164016451650
gluvalpheileasnilevalthrasntyrargthrtyrthrala
165516601665
lysthraspleuglylysleuglualaalaasplyspheleuarg
167016751680
tyrthrilegluhisproglnleuserserpheglyalaalaval
168516901695
cysproalaseraspileaspalatyralaalaglyleuileasn
170017051710
alaphegluthrargasnalathrhisilevalvalalaglnleu
171517201725
ilelysasngluileglulysserserargprothraspileleu
173017351740
argargasnsercysalathrargserleusermetleualaarg
174517501755
serlysglyasnglutyrleuileargthrleuglnproleuleu
176017651770
lyslysileileglnasnargaspphephegluileglulysleu
177517801785
lysprogluaspseraspalagluargglnilegluleupheval
179017951800
lystyrmetasngluleuleugluserileserasnservalser
180518101815
tyrpheproproproleuphetyrilecysglnasniletyrlys
182018251830
valalacysglulyspheproasphisalaileilealaalagly
183518401845
serphevalpheleuargphephecysproalaleuvalserpro
185018551860
aspsergluasnileileaspileserhisleuserglulysarg
186518701875
thrpheileserleualalysvalileglnasnilealaasngly
188018851890
sergluasnpheserargtrpproalaleucysserglnlysasp
189519001905
pheleulysglucysseraspargilepheargpheleualaglu
191019151920
leucysargthraspargthrileaspileglnvalargthrasp
192519301935
prothrproilealapheasptyrglnpheleuhisserpheval
194019451950
tyrleutyrglyleugluvalargargasnvalleuasngluala
195519601965
lyshisaspaspglyaspileaspglyaspaspphetyrlysthr
197019751980
thrpheleuleuileaspaspvalleuglyglnleuglyglnpro
198519901995
lysmetglupheserasngluileproiletyrilearggluhis
200020052010
metaspasptyrprogluleutyrgluphemetasnarghisala
201520202025
pheargasnilegluthrserthralatyrserproservalhis
203020352040
gluserthrsersergluglyileproileilethrleuthrmet
204520502055
serasnpheserasparghisvalaspileaspthrvalalatyr
206020652070
lyspheleuglniletyralaargiletrpthrthrlyshiscys
207520802085
leuileileaspcysthrglupheaspgluglyglyleuaspmet
209020952100
arglyspheileserleuvalmetglyleuleuprogluvalala
210521102115
prolysasncysileglycystyrtyrpheasnvalasngluthr
212021252130
phemetaspasntyrglylyscysleuasplysaspasnvaltyr
213521402145
valserserlysileprohistyrpheileasnserasnserasp
215021552160
gluglyleumetlysservalglyilethrglyglnglyleulys
216521702175
valleuglnaspileargvalserleuhisaspilethrleutyr
218021852190
aspglulysargasnargphethrprovalserleulysilegly
219522002205
aspiletyrpheglnvalleuhisgluthrproargglntyrlys
221022152220
ileargaspmetglythrleupheaspvallyspheasnaspval
222522302235
tyrgluileserargilephegluvalhisvalserserilethr
224022452250
glyvalalaalagluphethrvalthrpheglnaspgluargarg
225522602265
leuilepheserserprolystyrleugluilevallysmetphe
227022752280
tyrtyralaglnileargleugluserglutyrglumetaspasn
228522902295
asnserserthrserserproasnserasnasnlysasplysgln
230023052310
glnlysgluargthrlysleuleucyshisleuleuleuvalser
231523202325
leuileglyleupheaspgluserlyslysmetlysasnserser
233023352340
tyrasnleuilealaalathrglualaserpheglyleuasnphe
234523502355
glyserhisphehisargserprogluvaltyrvalproglutyr
236023652370
thrthrthrpheleuglyvalileglylysserleualagluser
237523802385
asnprogluleuthralatyrmetpheiletyrvalleugluala
239023952400
leulysasnasnvalileprohisvaltyrileprohisthrile
240524102415
cysglyleusertyrtrpileproasnleutyrglnhisvaltyr
242024252430
leualaaspaspglugluglyprogluasnileserhisilephe
243524402445
argileleuileargleuservalarggluthraspphelysala
245024552460
valtyrmetglntyrvaltrpleuleuleuleuaspaspglyarg
246524702475
leuthraspileilevalaspgluvalileasnhisalaleuglu
248024852490
argaspsergluasnargasptrplyslysthrileserleuleu
249525002505
thrvalleuprothrthrgluvalalaasnasnileileglnlys
251025152520
ileleualalysileargserpheleuproserleulysleuglu
252525302535
alametthrglnsertrpsergluleuthrileleuvallysile
254025452550
serilehisvalphephegluthrserleuleuvalglnmettyr
255525602565
leuprogluileleupheilevalserleuleuileaspvalgly
257025752580
proarggluleuargserserleuhisglnleuleumetasnval
258525902595
cyshisserleualaileaspseralaleuserglnasphisarg
260026052610
asnasnleuaspgluileseraspilephealahisglnlysval
261526202625
lysphemetpheglyphesergluasplysglyargileleugln
263026352640
ilepheseralaserserphealaserlyspheasnileleuasp
264526502655
phepheileasnasnileleuleuleumetglutyrserserthr
266026652670
tyrglualaasnvaltrplysthrargtyrlyslystyrvalleu
267526802685
gluservalphethrserasnserpheleuseralaargserile
269026952700
metilevalglyilemetglylyssertyrilethrgluglyleu
270527102715
cyslysalametleuilegluthrmetlysvalilealaglupro
272027252730
lysilethraspgluhisleupheleualaileserhisilephe
273527402745
thrtyrserlysilevalgluglyleuaspproasnleuaspleu
275027552760
metlyshisleuphetrppheserthrleupheleugluserarg
276527702775
hisproileilephegluglyalaleuleuphevalserasncys
278027852790
ileargargleutyrmetalaglnphegluasnglusergluthr
279528002805
serleuileserthrleuleulysglyarglysphealahisthr
281028152820
pheleuserlysilegluasnleuserglyilevaltrpasnglu
282528302835
aspasnphethrhisileleuilepheileileasnlysglyleu
284028452850
serasnpropheilelysserthralapheasppheleulysmet
285528602865
metpheargasnsertyrphegluhisglnileasnglnlysser
287028752880
asphistyrleucystyrmetpheleuleutyrphevalleuasn
288528902895
cysasnglnpheglugluleuleuglyaspvalasppheglugly
290029052910
glumetvalasnilegluasnlysasnthrileprolysileleu
291529202925
leuglutrpleuserseraspasngluasnalaasnilethrleu
293029352940
tyrglnglyalaileleuphelyscysservalthraspglupro
294529502955
serlyspheargphealaleuileilearghisleuleuthrlys
296029652970
lysproilecysalaleuargphetyrservalileargasnglu
297529802985
ilearglysileseralaphegluglnasnseraspcysvalpro
299029953000
leualapheaspileleuasnleuleuvalthrhissergluser
300530103015
asnserleuglulysleuhisglugluserilegluargleuthr
302030253030
lysargglyleuserilevalthrserserglyilephealalys
303530403045
asnseraspmetmetileproleuaspvallysprogluaspile
305030553060
tyrgluarglysargilemetthrmetileleuserargmetser
306530703075
cysserala
3080
<210>9
<211>293
<212>prt
<213>酿酒酵母种(saccharomycescerevisiae)
<400>9
metserglnprothrlysasnlyslyslysgluhisglythraspser
151015
lysserserargmetthrargthrleuvalasnhisileleupheglu
202530
argileleuproileleuprovalgluserasnleuserthrtyrser
354045
gluvalgluglutyrserserpheilesercysargservalleuile
505560
asnvalthrvalserglnaspalaasnalametvalgluglythrleu
65707580
gluleuilegluserleuleuglnglyhisgluileileserasplys
859095
cysserseraspvalilegluserileleuileileleuargleuleu
100105110
seraspalaleuglutyrasntrpglnasnglngluserleuhistyr
115120125
asnaspileserthrhisvalgluhisaspglngluglnlystyrarg
130135140
prolysleuasnserileleuproasptyrserserthrhisserasn
145150155160
glyasnlyshisphephehisglnserlysproglnalaleuilepro
165170175
gluleualaserlysleuleuglusercysalalysleulyspheasn
180185190
thrargthrleuglnileleuglnasnmetileserhisvalhisgly
195200205
asnileleuthrthrleuserserserileleuproarghislysser
210215220
tyrleuthrarghisasnhisproserhiscyslysmetileaspser
225230235240
thrleuglyhisileleuargphevalalaalaserasnproserglu
245250255
tyrpheglupheilearglysservalglnvalprovalthrglnthr
260265270
histhrhisthrargilehisthrproilehisthrleucyshisleu
275280285
propheilethrala
290
<210>10
<211>309
<212>prt
<213>酿酒酵母种(saccharomycescerevisiae)
<400>10
metglnglyasnlysserthrileargglutyrlysilevalvalval
151015
glyglyglyglyvalglylysseralaleuthrileglnpheilegln
202530
sertyrphevalaspglutyraspprothrilegluaspsertyrarg
354045
lysglnvalvalileaspasplysvalserileleuaspileleuasp
505560
thralaglyglngluglutyrseralametarggluglntyrmetarg
65707580
thrglygluglypheleuleuvaltyrservalthrserargasnser
859095
pheaspgluleuleusertyrtyrglnglnileglnargvallysasp
100105110
serasptyrileprovalvalvalvalglyasnlysleuaspleuglu
115120125
asngluargglnvalsertyrgluaspglyleuargleualalysgln
130135140
leuasnalapropheleugluthrseralalysglnalaileasnval
145150155160
aspglualaphetyrserleuileargleuvalargaspaspglygly
165170175
lystyrasnsermetasnargglnleuaspasnthrasngluilearg
180185190
aspsergluleuthrserseralathralaaspargglulyslysasn
195200205
asnglysertyrvalleuaspasnserleuthrasnalaglythrgly
210215220
serserserlysseralavalasnhisasnglygluthrthrlysarg
225230235240
thraspglulysasntyrvalasnglnasnasnasnasngluglyasn
245250255
thrlystyrserserasnglyasnglyasnargseraspileserarg
260265270
glyasnglnasnasnalaleuasnserargserlysglnseralaglu
275280285
proglnlysasnserseralaasnalaarglysgluserserglygly
290295300
cyscysileilecys
305
<210>11
<211>309
<212>prt
<213>酿酒酵母种(saccharomycescerevisiae)
<400>11
metglnglyasnlysserthrileargglutyrlysilevalvalval
151015
glyglyglyglyvalglylysseralaleuthrileglnpheilegln
202530
sertyrphevalaspglutyraspprothrilegluaspsertyrarg
354045
lysglnvalvalileaspasplysvalserileleuaspileleuasp
505560
thrthrglyglngluglutyrseralametarggluglntyrmetarg
65707580
thrglygluglypheleuleuvaltyrservalthrserargasnser
859095
pheaspgluleuleusertyrtyrglnglnileglnargvallysasp
100105110
serasptyrileprovalvalvalvalglyasnlysleuaspleuglu
115120125
asngluargglnvalsertyrgluaspglyleuargleualalysgln
130135140
leuasnalapropheleugluthrseralalysglnalaileasnval
145150155160
aspglualaphetyrserleuileargleuvalargaspaspglygly
165170175
lystyrasnsermetasnargglnleuaspasnthrasngluilearg
180185190
aspsergluleuthrserseralathralaaspargglulyslysasn
195200205
asnglysertyrvalleuaspasnserleuthrasnalaglythrgly
210215220
serserserlysseralavalasnhisasnglygluthrthrlysarg
225230235240
thraspglulysasntyrvalasnglnasnasnasnasngluglyasn
245250255
thrlystyrserserasnglyasnglyasnargseraspileserarg
260265270
glyasnglnasnasnalaleuasnserargserlysglnseralaglu
275280285
proglnlysasnserseralaasnalaarglysgluserserglygly
290295300
cyscysileilecys
305
- 还没有人留言评论。精彩留言会获得点赞!