碳水化合物传感器的制作方法
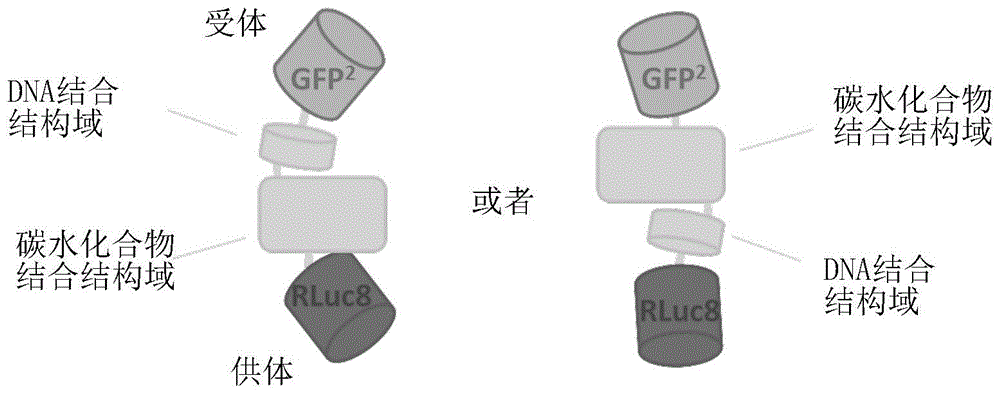
本发明涉及用于检测样品中诸如乳糖的碳水化合物的传感器和方法。传感器和方法也可用于测定样品中碳水化合物的量。
背景技术:
:用于检测包括糖和糖衍生物的碳水化合物的测定法广泛用于食品和医疗工业。食品工业特别感兴趣的是检测乳制品中诸如乳糖的碳水化合物的测定法。目前,乳制品中的常规乳糖分析用高效液相色谱(hplc)实现。该方法产生对乳糖的准确、灵敏和选择性测量,但是分析需要将样品运输到实验室设备,该实验室设备具有由训练有素的人员操作的昂贵设备(euber和brunner,1979;indyk等,1996;xinmin等人,2008;erich等人,2012)。用于检测/测量乳糖的其他技术使用酶级联来间接测量溶液中乳糖的浓度。这些测定通常通过用β-半乳糖苷酶将乳糖酶水解成半乳糖和葡萄糖,然后氧化任一种单糖来进行(kleyn,1985;ansari等人,2012;gia等人,2014)。乳糖的定量通过使用分光光度法、安培法或比色法测量氧化步骤的化学计量副产物nadh或h2o2来实现。这些测定需要许多试剂和许多步骤,使得它们对于加工厂中的常规使用来说太冗长和麻烦。它们具有固有的低选择性。或者,高水平的乳糖(例如3.9-4.8%(w/v))可以通过近红外光谱测定(tsenkova等人,1999)。然而,对于测量较低水平的乳糖,如低于1%(w/v)乳糖,该方法是不准确的,并且需要昂贵的设备。因此,需要另外的检测和定量样品中碳水化合物的量的方法,优选可以实时进行的方法,具有增加的灵敏度和/或不必将样品送到非现场进行分析。技术实现要素:本发明人已经鉴定了可用于检测样品中碳水化合物的传感器。本发明人还鉴定了使用这些传感器检测样品中碳水化合物存在的改进方法。在一些实施方案中,这些传感器和方法可用于测量样品中碳水化合物的浓度。他们还鉴定了使用这些传感器检测乳制品乳糖的改进方法。在一些实施方案中,传感器和方法可用于测量乳制品的乳糖含量。在一些实施方案中,传感器和方法可用于基于乳制品的乳糖含量对乳制品进行分类。在一个方面,提供了用于检测碳水化合物的传感器分子,所述传感器包括:i)螺旋-转角-螺旋转录因子的碳水化合物结合结构域,或所述碳水化合物结合结构域的变体;ii)化学发光供体结构域;以及iii)受体结构域;其中当所述碳水化合物结合至所述碳水化合物结合结构域时,所述化学发光供体结构域相对于所述受体结构域的空间位置和/或偶极取向改变。在一些实施方案中,螺旋-转角-螺旋转录因子是细菌螺旋-转角-螺旋转录因子或其变体。在一些实施方案中,细菌螺旋-转角-螺旋转录因子是gntr转录因子或其变体。在一些实施方案中,细菌螺旋-转角-螺旋转录因子或其变体具有与seqidno:1提供的氨基酸序列至少60%相同的氨基酸序列。在另一个实施方案中,结合结构域具有与seqidno:9提供的氨基酸序列至少60%相同的氨基酸序列。在另一个实施方案中,结合结构域具有与seqidno:9和56-74中任何一个或多个提供的氨基酸序列至少30%相同的氨基酸序列。在一些实施方案中,碳水化合物是糖或糖衍生物。在一些实施方案中,碳水化合物是糖。在一些实施方案中,糖是二糖。在优选的实施方案中,二糖是乳糖。在另一个实施方案中,二糖是乳果糖。在一些实施方案中,碳水化合物是糖衍生物。在一些实施方案中,糖衍生物选自由以下组成的组:氨基糖、酸性糖、脱氧糖、糖醇、葡基胺和磷酸糖。在一些实施方案中,化学发光供体结构域是生物发光蛋白。在一些实施方案中,生物发光蛋白是荧光素酶、β-半乳糖苷酶、内酰胺酶、辣根过氧化物酶、碱性磷酸酶、β-葡糖醛酸糖苷酶或β-葡糖苷酶。在一些实施方案中,生物发光蛋白是荧光素酶。在一些实施方案中,所述萤光素酶是海肾萤光素酶、萤火虫萤光素酶、腔肠动物(coelenterate)萤光素酶、北美萤火虫(northamericanglowworm)萤光素酶、叩头虫(clickbeetle)萤光素酶、铁路蠕虫(railroadworm)萤光素酶、细菌萤光素酶、gaussia萤光素酶、水母(aequorin)发光蛋白、蕈蚊(arachnocampa)萤光素酶、oplophorusgracilirostris萤光素酶或其任何一种的生物活性变体或片段,或其两种或更多种的嵌合体。在一些实施方案中,化学发光供体结构域能够修饰底物。在一些实施方案中,所述底物是荧光素,钙,腔肠素,furimazine,或腔肠素、荧光素或furimazine的衍生物、类似物或稳定衍生物。在一些实施方案中,受体结构域是荧光受体结构域。在一些实施方案中,其中所述荧光受体结构域选自由以下组成的组:绿色荧光蛋白(gfp)、gfp的蓝色荧光变体(bfp)、gfp的青色荧光变体(cfp)、gfp的黄色荧光变体(yfp)、增强的gfp(egfp)、增强的cfp(ecfp)、增强的yfp(eyfp)、gfps65t、emerald、venus、morange、topaz、gfpuv、去稳定egfp(degfp)、去稳定ecfp(decfp)、去稳定eyfp(deyfp)、hc红(hcred)、t-hc红(t-hcred)、ds红(dsred)、ds红2(dsred2)、t-二聚体2、t二聚体2(12)、mrfp1、杯型珊瑚色(pocilloporin)、海肾gfp(renillagfp)、monstergfp、pagfp、kaede蛋白、tdtomato、mcherry、tagrfp、turbofb和藻胆蛋白,和其任何一个的生物活性变体或片段。在一些实施方案中,在存在和/或不存在碳水化合物的情况下,化学发光供体结构域和受体结构域的分离和相对取向在费尔斯特距离的±50%内。在一些实施方案中,化学发光供体结构域和受体结构域的费尔斯特距离至少为5.6nm。在一些实施方案中,化学发光供体结构域和受体结构域之间费尔斯特距离在约5.6nm和约10nm之间。另一方面,还提供了检测样品中碳水化合物的方法,该方法包括:i)使样品与本文定义的传感器分子接触;以及ii)确定化学发光供体结构域相对于受体结构域的空间位置和/或偶极取向在样品存在下是否已经改变,其中所述化学发光供体结构域相对于所述受体结构域的空间位置和/或偶极取向的改变表明所述样品中存在碳水化合物。在一些实施方案中,所述方法还包括测定样品中碳水化合物的浓度。在一些实施方案中,所述方法在微流体装置上进行。在一些实施方案中,样品是空气、液体、生物材料或土壤。在一些实施方案中,样品包含乳制品。在优选的实施方案中,样品是乳品。另一方面提供了一种用于检测乳糖的传感器分子,其包含共价连接至共振能量转移供体结构域和共振能量转移受体结构域的细菌bgar转录因子或其变体,其中当乳糖结合至转录因子时,所述供体结构域相对于所述受体结构域的空间位置和/或偶极取向改变。乳糖与传感器分子的结合产生共振能量转移(ret)的变化,例如bret的变化或fret的变化。因此,传感器可用于检测样品中乳糖的存在和/或测定样品中乳糖的浓度。另一方面提供了一种用于检测乳果糖的传感器分子,其包含共价连接至共振能量转移供体结构域和共振能量转移受体结构域的细菌bgar转录因子或其变体,其中当乳果糖结合至转录因子时,所述供体结构域相对于所述受体结构域的空间位置和/或偶极取向改变。乳果糖与传感器分子的结合产生共振能量转移(ret)的变化,例如bret的变化或fret的变化。因此,传感器可用于检测样品中乳果糖的存在和/或测定样品中乳糖的浓度。在一些实施方案中,所述转录因子或其变体具有与seqidno:1中提供的氨基酸序列至少60%、70%、80%、85%、90%、95%、98%或100%相同的氨基酸序列。在一些实施方案中,转录因子或其变体具有seqidno:1提供的氨基酸序列。传感器分子可以是基于bret或fret的传感器。在一些实施方案中,传感器分子是基于bret的传感器,使得乳糖与传感器分子的结合产生bret的变化。因此,在一些实施方案中,共振能量转移供体结构域是生物发光蛋白。合适的生物发光蛋白包括荧光素酶、β-半乳糖苷酶、内酰胺酶、辣根过氧化物酶、碱性磷酸酶、β-葡糖醛酸糖苷酶或β-葡糖苷酶。在一些实施方案中,生物发光蛋白是荧光素酶。在一些实施方案中,所述萤光素酶是海肾萤光素酶、萤火虫萤光素酶、腔肠动物(coelenterate)萤光素酶、北美萤火虫(northamericanglowworm)萤光素酶、叩头虫(clickbeetle)萤光素酶、铁路蠕虫(railroadworm)萤光素酶、细菌萤光素酶、gaussia萤光素酶、水母(aequorin)发光蛋白、蕈蚊(arachnocampa)萤光素酶、oplophorusgracilirostris萤光素酶或其任何一种的生物活性变体或片段,或其两种或更多种的嵌合体。在一些实施方案中,供体结构域能够修饰底物。合适的底物包括荧光素,钙,腔肠素,furimazine,或腔肠素、荧光素或furimazine的衍生物、类似物或稳定衍生物。在一些实施方案中,传感器分子是基于fret的传感器,使得乳糖与传感器分子的结合产生fret的变化。因此,在一些实施方案中,共振能量转移供体结构域是荧光蛋白。在一些实施方案中,其中所述荧光蛋白选自由以下组成的组:绿色荧光蛋白(gfp)、gfp的蓝色荧光变体(bfp)、gfp的青色荧光变体(cfp)、gfp的黄色荧光变体(yfp)、增强的gfp(egfp)、增强的cfp(ecfp)、增强的yfp(eyfp)、gfps65t、emerald、venus、morange、topaz、gfpuv、去稳定egfp(degfp)、去稳定ecfp(decfp)、去稳定eyfp(deyfp)、hc红(hcred)、t-hc红(t-hcred)、ds红(dsred)、ds红2(dsred2)、t-二聚体2、t二聚体2(12)、mrfp1、杯型珊瑚色(pocilloporin)、海肾gfp(renillagfp)、monstergfp、pagfp、kaede蛋白、tdtomato、mcherry、tagrfp、turbofb和藻胆蛋白,和其任何一个的生物活性变体或片段。对于基于bret和fret的乳糖传感器,共振能量转移受体结构域可以是荧光受体结构域。在一些实施方案中,荧光受体结构域是荧光蛋白。在一些实施方案中,其中所述荧光受体结构域选自由以下组成的组:绿色荧光蛋白(gfp)、gfp的蓝色荧光变体(bfp)、gfp的青色荧光变体(cfp)、gfp的黄色荧光变体(yfp)、增强的gfp(egfp)、增强的cfp(ecfp)、增强的yfp(eyfp)、gfps65t、emerald、venus、morange、topaz、gfpuv、去稳定egfp(degfp)、去稳定ecfp(decfp)、去稳定eyfp(deyfp)、hc红(hcred)、t-hc红(t-hcred)、ds红(dsred)、ds红2(dsred2)、t-二聚体2、t二聚体2(12)、mrfp1、杯型珊瑚色(pocilloporin)、海肾gfp(renillagfp)、monstergfp、pagfp、kaede蛋白、tdtomato、mcherry、tagrfp、turbofb和藻胆蛋白,和其任何一个的生物活性变体或片段。在一些实施方案中,共振能量转移供体结构域是海肾荧光素酶或其变体,且共振能量转移受体结构域是gfp或其变体。在一些实施例中,本发明提供了一种传感器分子,该传感器分子与选自seqidno:15、seqidno:16、seqidno:17和seqidno:18组成的组的多肽序列具有至少60%、70%、80%、85%、90%、95%、98%、99%或100%的序列一致性。在这些实施方案中,乳糖与传感器分子的结合产生bret的变化。在一些实施方案中,共振能量转移供体结构域是cfp或其变体,且共振能量转移受体结构域是yfp或其变体。一些实施方案中,本发明提供了一种传感器分子,其与seqidno:23提供的多肽具有至少60%、70%、80%、85%、90%、95%、98%、99%或100%的序列一致性。在这些实施方案中,乳糖与传感器分子的结合产生fret的变化。在一些实施方案中,在存在和/或不存在乳糖的情况下,供体结构域和受体结构域的分离和相对取向在费尔斯特距离的±50%内。在一些实施方案中,供体结构域和受体结构域的费尔斯特距离为至少5.6nm。在一些实施方案中,供体结构域和受体结构域的费尔斯特距离为约5.6nm-约10nm。另一方面,还提供了检测样品中乳糖的方法,所述方法包括:i)使样品与如本文所定义的用于检测乳糖的传感器分子接触;以及ii)确定发光供体结构域相对于受体结构域的空间位置和/或偶极取向在所述样品存在下是否已经改变,其中所述供体结构域相对于所述受体结构域的空间位置和/或偶极取向的改变表明所述样品中存在乳糖。在一些实施方案中,所述方法还包括测定样品中乳糖的浓度。在一些实施方案中,所述方法在微流体装置上进行。在一些实施方案中,样品是空气、液体、生物材料或土壤。在一些实施方案中,样品包含乳制品。在一些实施方案中,乳制品是乳品。另一方面,还提供了一种检测样品中乳果糖的方法,所述方法包括:i)使样品与本文定义的传感器分子接触;以及ii)确定发光供体结构域相对于受体结构域的空间位置和/或偶极取向在所述样品存在下是否已经改变,其中供体结构域相对于受体结构域的空间位置和/或偶极取向的改变表明样品中存在乳果糖。在一些实施方案中,所述方法还包括测定样品中乳果糖的浓度。在一些实施方案中,所述方法在微流体装置上进行。在一些实施方案中,样品是空气、液体、生物材料或土壤。在一些实施方案中,样品包含乳制品。在一些实施方案中,乳制品是乳品。在一些实施方案中,传感器分子是单个多肽。在一些实施方案中,化学发光供体结构域位于n-末端,且受体结构域位于c-末端。在另一个实施方案中,受体结构域位于n-末端,且化学发光供体结构域位于c-末端。在一些实施方案中,共振能量转移供体结构域位于n-末端,且共振能量转移受体结构域位于c-末端。在可选择的实施方案中,共振能量转移受体结构域位于n-末端,且共振能量转移供体结构域位于c-末端。在一些实施方案中,传感器分子包含至少一个肽接头。另一方面,还提供了编码本文定义的传感器分子的多核苷酸。另一方面,还提供了包含编码本文定义的传感器分子的多核苷酸的载体。另一方面,还提供了包含本文定义的多核苷酸和/或载体的宿主细胞。另一方面,还提供了生产传感器分子的方法,该方法包括在允许编码多肽的多核苷酸表达的条件下培养本文定义的宿主细胞或载体,并回收表达的多肽。除非另外具体说明,否则本文中的任何实施例应被理解为比照适用于任何其他实施例。本发明不限于这里描述的特定实施例的范围,这些实施例仅用于示例的目的。如本文所述,功能等同的产品、组合物和方法显然在本发明的范围内。在整个说明书中,除非另有特别说明或上下文另有要求,提及单个步骤、物质组成、步骤组或物质组成组应被认为包括一个和多个(即一个或多个)那些步骤、物质组成、步骤组或物质组成组。以下通过下述非限制性实施例并参考附图描述本发明。附图说明图1-本发明的实施方案的示意图。所示传感器分子lacb1包含bgar转录因子,其n-和c-末端分别侧接gfp2和rluc8。图2-水(深灰色条)或1mm乳糖(浅灰色条)中1μm的lacb1和lacf1传感器的ret比率(平均值±标准偏差(sd),n=3)。添加17μm腔肠素400a底物后记录bret2扫描。*p<0.0001。图3-lacb1对0.000034w/v%-0.34%w/v乳糖的bret2响应。图4-本发明的实施方案的示意图。lacb1包含bgar转录因子(浅灰色),在n-和c-末端分别侧接gfp2(中灰色)和rluc8(深灰色)。lacb2包含lacb1,其氨基酸接头-ggtggg-插入到bgar和gfp2以及bgar和rluc8之间。lacb3包含lacb1,其接头-ggtggg-插入bgar和gfp2之间。lacb4包含lacb1,其接头-ggtggg-插入bgar和rluc8之间。接头的位置由连接bgar和gfp2和/或bgar和rluc8的黑色部分表示。图5-lacb1(1)、lacb2(2)、lacb3(3)和lacb4(4)对1mm乳糖存在的bret2响应。图6-在二糖乳糖、麦芽糖和乳果糖以及单糖半乳糖和葡萄糖存在下,lacb1传感器的bret2比率的变化。在与指定的糖在30℃下温育30分钟后,向1μm的lacb1中添加17μm腔肠素400a后,记录bret2的比率(平均值±sd,n=3)。将bret2的比例标准化为水响应,并表示为bret2的变化百分比。图7-lacb1对一系列二糖和单糖的响应,所有浓度均为1mm。在与指定的糖在30℃下温育5分钟后,向1μm的lacb1中添加17μm腔肠素400a后,记录bret2的比率(平均值±sd,n=3)。将bret2的比例标准化为水响应,并表示为bret2的变化百分比。图8-在pbs(a)中半乳糖和葡萄糖以及透析乳(c、e)中半乳糖和葡萄糖存在下通过lacb1传感器检测乳糖(平均值±s.d.,n=3)。在pbs(b)中半乳糖和葡萄糖以及透析乳(d)中半乳糖和葡萄糖存在下,通过lacf1传感器检测乳糖(平均值±s.d.,n=3)。图9-在pbs和补充有乳糖、半乳糖和葡萄糖的10%(v/v)透析全脂乳中,通过lacb1传感器检测乳糖和乳果糖。实线:lacb1在pbs中的乳糖浓度依赖性。ec50=12±1μm,lod=1μm。虚线:加入的乳糖、半乳糖和葡萄糖使得([乳糖]+[半乳糖+葡萄糖]/2=13.9mm)的10%透析全脂乳中lacb1的乳糖浓度依赖性。ec50=21±2μm,lod=0.2μm。点画线:lacb1在pbs中的乳糖浓度依赖性。ec50=2.4±0.2mm,lod=0.1mm。图10-(a)使用装置测定的在测定缓冲液(0.45%明胶的磷酸盐缓冲液中)中存在3mm乳糖的情况下,lacb1传感器的bret2比值的示例迹线。(b)在测定缓冲液中存在3mm乳糖的情况下,使用装置为lacb1记录的供体发射(上窗口,深灰色)和受体发射(上窗口,浅灰色)强度的示例迹线。图11–用装置确定的lacb1的bret2比值的变化。用装置记录在30μm和3mm乳糖存在下lacb1的bret2比率。将bret2比率标准化为测定缓冲液响应,并表示为bret2变化的百分比(平均值±s.d.,n=2或3)。序列表的关键字seqidno:1-bgarhth转录因子的氨基酸序列。seqidno:2-8-接头序列。seqidno:9-bgarhth碳水化合物结合结构域。seqidno:10-14-引物序列。seqidno:15-lacb1的氨基酸序列(gfp2-bgar-rluc8融合蛋白)。seqidno:16-lacb2的氨基酸序列(gfp2-bgar-rluc8融合蛋白)。seqidno:17-lacb3的氨基酸序列(gfp2-bgar-rluc8融合蛋白)。seqidno:18-lacb4的氨基酸序列(gfp2-bgar-rluc8融合蛋白)。seqidno:19-编码lacb1的核苷酸序列(gfp2-bgar-rluc8融合蛋白)。seqidno:20-编码lacb2的核苷酸序列(gfp2-bgar-rluc8融合蛋白)。seqidno:21-编码lacb3的核苷酸序列(gfp2-bgar-rluc8融合蛋白)。seqidno:22-编码lacb4的核苷酸序列(gfp2-bgar-rluc8融合蛋白)。seqidno:23-lacf1的氨基酸序列(his6-cfp-bgar-yfp融合蛋白)。seqidno:24-编码lacf1的核苷酸序列(his6-cfp-bgar-yfp融合蛋白)。seqidno:25-lacb1的氨基酸序列(his6-gfp2-bgar-rluc8融合蛋白)。seqidno:26-lacb2的氨基酸序列(his6-gfp2-bgar-rluc8融合蛋白)。seqidno:27-lacb3的氨基酸序列(his6-gfp2-bgar-rluc8融合蛋白)。seqidno:28-lacb4的氨基酸序列(his6-gfp2-bgar-rluc8融合蛋白)。seqidno:29-编码lacb1的核苷酸序列(his6-gfp2-bgar-rluc8融合蛋白)。seqidno:30-编码lacb2的核苷酸序列(his6-gfp2-bgar-rluc8融合蛋白)。seqidno:31-编码lacb3的核苷酸序列(his6-gfp2-bgar-rluc8融合蛋白)。seqidno:32-编码lacb4的核苷酸序列(his6-gfp2-bgar-rluc8融合蛋白)。seqidno:33-lacb11-171的氨基酸序列(gfp2-bgar1-171-rluc8融合蛋白)。根据本发明的实施方案的传感器分子,其包含侧接gfp2和rluc8的bgar的残基1-171。seqidno:34-lacb11-150的氨基酸序列(gfp2-bgar1-150-rluc8融合蛋白)。根据本发明的实施方案的传感器分子,其包含侧接gfp2和rluc8的bgar的残基1-150。seqidno:35-lacb112-171的氨基酸序列(gfp2-bgar12-171-rluc8融合蛋白)。根据本发明的实施方案的传感器分子,其包含侧接gfp2和rluc8的bgar的残基12-171。seqidno:36-lacb112-150的氨基酸序列(gfp2-bgar12-150-rluc8融合蛋白)。根据本发明的实施方案的传感器分子,其包含侧接gfp2和rluc8的bgar的残基12-150。seqidno:37-uniprot登录号:a0a133mux6的氨基酸序列。seqidno:38-uniprot登录号:b1v7n0的氨基酸序列。seqidno:39-uniprot登录号:a0a127egd8的氨基酸序列。seqidno:40-uniprot登录号:a0a1c6jub7的氨基酸序列。seqidno:41-uniprot登录号:a0a174hyb7的氨基酸序列。seqidno:42-uniprot登录号:a0a1c6ky47的氨基酸序列。seqidno:43-uniprot登录号:a0a174lzq7的氨基酸序列。seqidno:44-uniprot登录号:n9yr91的氨基酸序列。seqidno:45-uniprot登录号:a0a174i591的氨基酸序列。seqidno:46-uniprot登录号:a0a2a7me67的氨基酸序列。seqidno:47-uniprot登录号:a0a2k4azl9的氨基酸序列。seqidno:48-uniprot登录号:a0a166ppm9的氨基酸序列。seqidno:49-uniprot登录号:a0a2t4r7g1的氨基酸序列。seqidno:50-uniprot登录号:a0a2a4hcu9的氨基酸序列。seqidno:51-uniprot登录号:a0a2t4ms83的氨基酸序列。seqidno:52-uniprot登录号:o33813的氨基酸序列。seqidno:53-uniprot登录号:a0a1d4lkb2的氨基酸序列。seqidno:54-uniprot登录号:a0a133qvv5的氨基酸序列。seqidno:55-uniprot登录号:a9qsr3的氨基酸序列。seqidno:56-uniprot登录号:a0a133mux6的假定碳水化合物结合结构域(cbd)的氨基酸序列。seqidno:57-uniprot登录号:b1v7n0的假定cbd的氨基酸序列。seqidno:58-uniprot登录号:a0a127egd8的假定cbd的氨基酸序列。seqidno:59-uniprot登录号:a0a1c6jub7的假定cbd的氨基酸序列。seqidno:60-uniprot登录号:a0a174hyb7的假定cbd的氨基酸序列。seqidno:61-uniprot登录号:a0a1c6ky47的假定cbd的氨基酸序列。seqidno:62-uniprot登录号:a0a174lzq7的假定cbd的氨基酸序列。seqidno:63-uniprot登录号:n9yr91的假定cbd的氨基酸序列。seqidno:64-uniprot登录号:a0a174i591的假定cbd的氨基酸序列。seqidno:65-uniprot登录号:a0a2a7me67的假定cbd的氨基酸序列。seqidno:66-uniprot登录号:a0a2k4azl9的假定cbd的氨基酸序列。seqidno:67-uniprot登录号:a0a166ppm9的假定cbd的氨基酸序列。seqidno:68-uniprot登录号:a0a2t4r7g1的假定cbd的氨基酸序列。seqidno:69-uniprot登录号:a0a2a4hcu9的假定cbd的氨基酸序列。seqidno:70-uniprot登录号:a0a2t4ms83的假定cbd的氨基酸序列。seqidno:71-uniprot登录号:o33813的假定cbd的氨基酸序列。seqidno:72-uniprot登录号:a0a1d4lkb2的假定cbd的氨基酸序列。seqidno:73-uniprot登录号:a0a133qvv5的假定cbd的氨基酸序列。seqidno:74-uniprot登录号:a9qsr3的假定cbd的氨基酸序列。具体实施方式一般技术和定义除非另外具体定义,否则本文所用的所有技术和科学术语应认为具有与本领域(例如,在传感器技术、分子生物学、蛋白质化学、乳品科学、乳品技术、生物化学等中)普通技术人员通常理解的相同含义。除非另有说明,用于本发明的重组蛋白、细胞培养物和免疫学技术是本领域技术人员熟知的标准方法。在以下来源的整个文献中描述了这种技术,诸如:j.perbal,《分子克隆实用指南》,johnwileyandsons(1984),j。sambrook等,《分子克隆:实验室手册》,冷泉港实验室出版社(1989),tabrown(编辑),基本分子生物学:实用方法,第1卷和第2卷,irl出版社(1991),dmglover和bdhames(编辑),dna克隆:实用方法,第1-4卷,irl出版社(1995和1996),以及fmausubel等(编者),《分子生物学的最新方案》,greene.associates和wiley-interscience(1988,包括到目前为止的所有更新),edharlow和davidlane(编者)抗体:实验室手册,冷泉港实验室,(1988)和jecoligan等人,(编辑)《免疫学最新方案》,约翰·威利父子出版社(johnwiley&sons)(j.perbal,apracticalguidetomolecularcloning,johnwileyandsons(1984),j.sambrooketal.,molecularcloning:alaboratorymanual,coldspringharbourlaboratorypress(1989),t.a.brown(editor),essentialmolecularbiology:apracticalapproach,volumes1and2,irlpress(1991),d.m.gloverandb.d.hames(editors),dnacloning:apracticalapproach,volumes1-4,irlpress(1995and1996),andf.m.ausubeletal.,(editors),currentprotocolsinmolecularbiology,greenepub.associatesandwiley-interscience(1988,includingallupdatesuntilpresent),edharlowanddavidlane(editors)antibodies:alaboratorymanual,coldspringharbourlaboratory,(1988),和j.e.coliganetal.,(editors)currentprotocolsinimmunology,johnwiley&sons)(包括到目前为止的所有更新)。适用于本发明方法的多肽可通过其氨基酸序列与参考氨基酸序列的一致性程度(%一致性)、或通过与一个参考氨基酸序列比与另一个参考氨基酸序列具有更大的%一致性来定义。多肽与参考氨基酸序列的%一致性通常通过gap分析确定(needleman和wunsch,1970;gcg程序),其中参数的缺口产生罚分=5,缺口延伸罚分=0.3。查询序列长度为至少100个氨基酸,且gap分析在至少100个氨基酸的区域上比对两个序列。甚至更优选地,查询序列长度为至少250个氨基酸,且gap分析在至少250个氨基酸的区域上比对两个序列。甚至更优选地,查询序列长度为至少450个氨基酸,且gap分析在至少450个氨基酸的区域上比对两个序列。甚至更优选地,gap分析在其全长上比对两个序列。术语“和/或”,例如“x和/或y”应理解为意指“x和y”或“x或y”,并且应理解为提供对两种含义或两种含义的明确支持。除非上下文另有说明,否则以单数形式提及的术语(例如传感器和底物)显然也意指复数形式。例如,逻辑上许多单独的传感器分子将流过该装置或包含在孔中而不是单个分子中。如本文所用,除非另有相反说明,术语“约”是指指定值的+/-10%、更优选+/-5%、甚至更优选+/-1%。在整个说明书中,词语“包含(comprise)”或诸如“包含(comprises)”或“包含(comprising)”的变化将被理解为暗示包含所述元件、整体或步骤,或元件、整体或步骤的组,但不排除任何其他元件、整体或步骤,或元件、整体或步骤的组。除非另有说明或上下文另有说明,%浓度是重量/体积(%w/v)。传感器在整个说明书中,“传感器”和“传感器分子”可互换使用。在一个方面,提供了用于检测碳水化合物的传感器分子,所述传感器包括:i)螺旋-转角-螺旋转录因子的碳水化合物结合结构域,或所述碳水化合物结合结构域的变体;ii)化学发光供体结构域;以及iii)受体结构域;其中当所述碳水化合物结合至所述碳水化合物结合结构域时,所述化学发光供体结构域相对于所述受体结构域的空间位置和/或偶极取向改变。在一些实施方案中,传感器是氨基酸的连续片段(换句话说,传感器是单个多肽)。例如,碳水化合物结合结构域、化学发光供体蛋白结构域和受体结构域是氨基酸的单个片段,例如但不限于,共价连接至碳水化合物结合结构域n末端的化学发光供体蛋白结构域和与碳水化合物结合结构域的c-末端共价连接的受体蛋白结构域,或与碳水化合物结合结构域的n-末端共价连接的受体蛋白结构域和与碳水化合物结合结构域的c-末端共价连接的化学发光供体蛋白结构域。在图1中提供了实施例。例如,在一些实施方案中,多肽具有选自由seqidno:15、seqidno:16、seqidno:17、seqidno:18、seqidno:25、seqidno:26、seqidno:27和seqidno:28组成的组的序列,或其片段或变体。在一些实施方案中,多肽可具有与seqidno:15、seqidno:16、seqidno:17、seqidno:18、seqidno:25、seqidno:26、seqidno:27和seqidno:28中的任何一个所示的序列至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列,或与其一部分至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列。如本文所用,多肽的“部分”保留多肽的相关活性,例如,多肽的部分保留结合碳水化合物的能力。例如,在一些实施方案中,多肽具有选自由seqidno:15、seqidno:16、seqidno:17、和seqidno:18组成的组的序列,或其片段或变体。在一些实施方案中,多肽可具有与seqidno:15、seqidno:16、seqidno:17、和seqidno:18中的任何一个所示的序列至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列,或与其一部分至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列。例如,多肽具有选自由seqidno:25、seqidno:26、seqidno:27和seqidno:28组成的组的序列,或其片段或变体。在一些实施方案中,多肽可具有与seqidno:25、seqidno:26、seqidno:27和seqidno:28中的任何一个所示的序列至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列,或与其一部分至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列。在一些实施方案中,还提供了包含编码本文定义的传感器的多核苷酸序列的核酸。在一些实施方案中,核酸是分离的核酸。例如,在一些实施方案中,核酸分子包含编码多肽序列的序列,所述多肽序列选自由seqidno:15、seqidno:16、seqidno:17、seqidno:18、seqidno:25、seqidno:26、seqidno:27和seqidno:28组成的组。在一些实施方案中,核酸分子包含编码多肽序列的序列,所述多肽序列选自由seqidno:15、seqidno:16、seqidno:17和seqidno:18组成的组。在一些实施方案中,核酸分子包含编码多肽序列的序列,所述多肽序列选自由seqidno:25、seqidno:26、seqidno:27和seqidno:28组成的组。在一些实施方案中,核酸分子包含编码seqidno:15或seqidno:25的多肽序列的序列。在一些实施方案中,所述核酸分子包含编码seqidno:15的多肽序列的序列。在一些实施方案中,所述核酸分子包含编码seqidno:25的多肽序列的序列。在一些实施方案中,核酸分子包含编码多肽的序列,所述多肽具有与seqidno:15、seqidno:16、seqidno:17、seqidno:18、seqidno:25、seqidno:26、seqidno:27和seqidno:28中的任何一个所示的序列至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列,或与其一部分至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列。在一些实施方案中,核酸分子包含编码多肽的序列,所述多肽具有与seqidno:15、seqidno:16、seqidno:17、和seqidno:18中的任何一个所示的序列至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列,或与其一部分至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列。在一些实施方案中,核酸分子包含编码多肽的序列,所述多肽具有与seqidno:25、seqidno:26、seqidno:27、和seqidno:28中的任何一个所示的序列至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列,或与其一部分至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列。在一些实施方案中,核酸分子包含选自由seqidno:19、seqidno:20、seqidno:21、seqidno:22、seqidno:29、seqidno30、seqidno:31、和seqidno:32组成的组的序列,或与seqidno:19、seqidno:20、seqidno:21、seqidno:22、seqidno:29、seqidno30、seqidno:31、和seqidno:32中的任何一个的一部分至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列。在一些实施方案中,核酸分子包含选自由seqidno:19、seqidno:20、seqidno:21、和seqidno:22组成的组的序列,或与seqidno:19、seqidno:20、seqidno:21、和seqidno:22中的任何一个的一部分至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列。在一些实施方案中,核酸分子包含选自由seqidno:19、seqidno:20、seqidno:21、和seqidno:22组成的组的序列,或与seqidno:29、seqidno30、seqidno:31、和seqidno:32中的任何一个的一部分至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列。在另一方面,本发明提供了一种用于检测乳糖或乳果糖的传感器分子,其包含共价连接至共振能量转移供体结构域和共振能量转移受体结构域的细菌bgar转录因子或其变体,其中当乳糖结合至转录因子时,所述供体结构域相对于所述受体结构域的空间位置和/或偶极取向改变。乳糖或乳果糖与该方面的传感器分子的结合产生共振能量转移的变化,例如bret的变化或fret的变化。在一些实施方案中,本发明提供用于检测乳糖的传感器分子。在一些实施方案中,本发明提供了用于检测乳果糖的传感器分子。在一些实施方案中,传感器是氨基酸的连续片段(换句话说,传感器是单个多肽)。例如,细菌bgar转录因子或其变体,共振能量转移供体结构域和共振能量转移受体结构域是氨基酸的单个片段,例如但不限于,共价连接至细菌bgar转录因子的n末端的供体蛋白结构域和共价连接至细菌bgar转录因子的c末端的受体蛋白结构域,或共价连接至细菌bgar转录因子的n末端的受体蛋白结构域和共价连接至细菌bgar转录因子的c末端的供体蛋白结构域。例如,在一些实施方案中,该多肽具有seqidno:23或其片段或变体提供的序列。在一些实施方案中,多肽可具有与seqidno:23中所示的序列至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列,或与其一部分至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列。在一些实施方案中,还提供了包含编码本文定义的传感器的多核苷酸序列的核酸分子。在一些实施方案中,核酸分子是分离的核酸分子。在一些实施方案中,核酸分子包含编码seqidno:23或其片段或变体提供的多肽序列的序列。在一些实施方案中,核酸分子包含编码多肽的序列,所述多肽可具有与seqidno:23中所示的序列至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列,或与其一部分至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列。在一些实施方案中,核酸分子包含seqidno:24提供的序列,或与其一部分至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列。本发明的传感器、组合物、试剂盒、方法和用途包括具有指定序列或与其基本上相同或相似的序列的多肽和核酸,例如,与指定序列至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列。在氨基酸序列的上下文中,术语“基本上相同”在本文中用于指,第一氨基酸,其含有足够或最小数目的第二氨基酸序列中比对的氨基酸残基的i)相同的氨基酸残基,或ii)保守取代,使得第一和第二氨基酸序列可以具有共同的结构域和/或共同的功能活性。例如,含有与指定序列具有至少约85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%一致性的共同结构域的氨基酸序列被称为基本上相同的。在核苷酸序列的上下文中,术语“基本上相同”在本文中用于指,第一核酸序列,其含有足够或最小数目的与第二核酸序列中的比对的核苷酸相同的核苷酸,使得第一和第二核苷酸序列编码具有共同功能活性的多肽,或编码共同结构多肽结构域或共同功能多肽活性。例如,与指定序列具有至少约85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%一致性的核苷酸序列被称为基本上相同的。序列之间同源性或序列一致性的计算(术语在本文中可互换使用)使用本领域技术人员已知的技术进行。例如,为了确定两个氨基酸序列或两个核酸序列的百分比一致性,为了最佳比较的目的对序列进行比对(例如,为了最佳比对可以在第一和第二氨基酸或核酸序列中的一个或两个中引入缺口,并且为了比较的目的可以忽略非同源序列)。在优选的实施方案中,为了比较目的而比对的参考序列的长度是参考序列长度的至少30%,优选至少40%,更优选至少50%、60%,甚至更优选至少70%、80%、90%、100%。然后比较相应氨基酸位置或核苷酸位置处的氨基酸残基或核苷酸。当第一序列中的位置被与第二序列中相应位置相同的氨基酸残基或核苷酸占据时,则分子在该位置是相同的(如本文所用,氨基酸或核酸“一致性”相当于氨基酸或核酸“同源性”)。考虑到缺口的数目和每个缺口的长度,两个序列之间的一致性百分比是序列共有的相同位置的数目的函数,为了两个序列的最佳比对需要引入缺口的数目和每个缺口的长度。序列的比较和两个序列之间一致性百分比的确定可以使用数学算法完成。在一些实施例中,使用已并入gcg软件包中的gap程序中的needleman和wunsch(1970)算法,使用blossum62矩阵或pam250矩阵,且缺口重量为16、14、12、10、8、6或4且长度重量为1、2、3、4、5或6,来确定两个氨基酸序列之间的百分比一致性。在其他实施方案中,使用gcg软件包中的gap程序,使用nwsgapdna.cmp矩阵,且缺口重量为40、50、60、70、或80且长度重量为1、2、3、4、5或6,来测定两个核苷酸序列之间的百分比一致性。两个氨基酸或核苷酸序列之间的百分比一致性可以使用meyers和miller(1989)的算法测定,该算法已经被引入align程序(2.0版),使用例如pam120重量残基表,缺口长度罚分12和缺口罚分4。本文所述的核酸和蛋白质序列可用作“查询序列”,以对公共数据库进行搜索,例如鉴定其他族成员或相关序列。这种搜索可以使用例如altschul等,(1990)的nblast和xblast程序(2.0版)以及blastp进行。blast核苷酸搜索可以用nblast程序进行,得分=100,字长=12,以获得与本发明一些实施方案的核酸分子同源的核苷酸序列。blast蛋白质搜索可以用xblast程序进行,得分=50,字长=3,以获得与本发明一些实施方案的蛋白质分子同源的氨基酸序列。为了获得用于比较目的的缺口比对,可以使用如altschul等,(1997)所述的缺口blast。当使用blast和gappedblast程序时,可以使用各个程序(例如blastp、xblast和nblast)的默认参数。参见http:www.ncbi.nlm.nih.gov/blast。对应于本文所述序列的天然等位基因变体、同源物、直系同源物或其他相关序列(例如,旁系同源物)的核酸分子可以基于它们与编码本文公开的氨基酸序列的核酸的同源性而分离,例如通过使用所有或部分已知序列作为探针进行标准或严格杂交反应。这种核酸杂交和克隆方法是本领域熟知的。本文提供的肽的同系物通常与这些肽具有结构相似性。多肽的同系物包括一个或多个保守氨基酸取代,其可以选自氨基酸所属类别的相同或不同成员。在一些实施方案中,序列还可以具有氨基酸残基的缺失、插入或取代,其产生沉默变化并产生功能等同的物质。可以基于残基的极性、电荷、溶解性、疏水性、亲水性和/或两亲性的相似性进行有意的氨基酸取代,只要保留物质的二级结合活性即可。例如,带负电荷的氨基酸包括天冬氨酸和谷氨酸;带正电荷的氨基酸包括赖氨酸和精氨酸;具有相似亲水性值的不带电荷的极性头基的氨基酸包括:亮氨酸、异亮氨酸、缬氨酸、甘氨酸、丙氨酸、天冬酰胺、谷氨酰胺、丝氨酸、苏氨酸、苯丙氨酸和酪氨酸。本发明的一些实施方案还包括保守取代(取代和置换均在本文中用于指现有氨基酸残基与替代残基的互换),其可以发生例如同类取代,诸如碱性取代碱性、酸性取代酸性、极性取代极性等。非保守取代也可以例如从一类残基到另一类残基发生,或者涉及内含非天然氨基酸,诸如鸟氨酸(下文称为z)、二氨基丁酸鸟氨酸(下文称为b)、正亮氨酸鸟氨酸(下文称为o)、吡啶基丙氨酸、噻吩基丙氨酸、萘基丙氨酸和苯基甘氨酸。可以进行的保守取代是,例如,在碱性氨基酸(精氨酸、赖氨酸和组氨酸),酸性氨基酸(谷氨酸和天冬氨酸),脂族氨基酸(丙氨酸、缬氨酸、亮氨酸、异亮氨酸),极性氨基酸(谷氨酰胺、天冬酰胺、丝氨酸、苏氨酸),芳族氨基酸(苯丙氨酸、色氨酸和酪氨酸),羟基氨基酸(丝氨酸、苏氨酸),大氨基酸(苯丙氨酸和色氨酸)和小氨基酸(甘氨酸、丙氨酸)。除了编码本发明传感器分子的序列之外,核酸分子还可以含有其他序列,例如引物位点、转录因子结合位点、载体插入位点和抗核酸分解降解的序列(例如聚腺苷尾)。核酸分子可以是dna或rna,并且可以包括合成的核苷酸,条件是多核苷酸仍然能够被翻译以合成本发明的蛋白质。在一些实施方案中,核酸形成诸如质粒的载体的一部分。除上述核酸序列外,质粒还包含其他元件,例如原核复制起点(例如,大肠杆菌or1复制起点)、自主复制序列、着丝粒序列;等等。能够在宿主细胞中表达该核酸的启动子序列,该启动子序列可操作地连接至该核酸、位于该核酸序列下游的终止子序列、抗生素抗性基因和/或分泌信号序列。包含自主复制序列的载体也是酵母人工染色体。在一些可选择的实施方案中,载体是病毒,例如噬菌体,并且除了本发明的核酸序列之外,还包含用于噬菌体复制的核酸序列,例如结构蛋白、启动子、转录激活因子等。本发明的核酸或载体可用于转染或转化宿主细胞以合成本发明的传感器分子。合适的宿主细胞包括诸如大肠杆菌的原核细胞和诸如酵母细胞或哺乳动物或植物细胞系的真核细胞。使用本领域已知的技术转染或转化宿主细胞,如电穿孔;磷酸钙碱法;基因枪技术或使用病毒载体。转染/转化后,根据需要转录和翻译本发明的核酸或载体。在一些实施方案中,合成的蛋白质从宿主细胞中提取,或者由于其从细胞中分泌,例如由于载体中分泌信号的存在,或者通过宿主细胞的裂解和从中纯化蛋白质。在一些实施方案中,还提供了生产本文定义的传感器分子的方法,该方法包括在允许编码多肽的多核苷酸表达的条件下培养本文定义的宿主细胞或载体,并回收表达的多肽。在一些实施方案中,传感器作为无细胞组合物提供。如本文所用,术语“无细胞组合物”是指包含如果有的话则很少的完整细胞并包含传感器的分离的组合物。无细胞组合物的实例包括细胞(诸如酵母细胞)提取物和含有分离的和/或重组的传感器分子(诸如蛋白质)的组合物。由细胞制备无细胞组合物的方法是本领域熟知的。传感器分子任选地包含至少一个接头。例如,传感器可包含将碳水化合物结合结构域(或包含碳水化合物结合结构域的螺旋-转角-螺旋转录因子)连接至化学发光供体结构域和/或受体结构域的接头。在另一个实例中,传感器分子可以在传感器分子的n-和/或c-末端包含接头。在一些实施方案中,传感器分子包含至少一个肽接头。在一些实施方案中,接头可以位于碳水化合物结合结构域(或包含碳水化合物结合结构域的螺旋-转角-螺旋转录因子)的n-和/或c-末端。优选地,接头是肽或多肽。在一些实施方案中,接头包含一个或多个甘氨酸、丝氨酸和/或苏氨酸残基。例如,在一些实施方案中,接头包含选自以下的氨基酸序列:gssggs(seqidno:2)、ggsggs(seqidno:3)、ggtggg(seqidno:4)、gggggt(seqidno:5)lqggtggg(seqidno:6)、feggtggg(seqidno:7)和ggsggsl(seqidno:8)。在一些实施方案中,接头是25个氨基酸或更少、20个氨基酸或更少、15个氨基酸或更少、10个氨基酸或更少、8个氨基酸或更少、6个氨基酸或更少、4个氨基酸或更少、或3个氨基酸或更少。在一些实施方案中,接头的长度为1-10个氨基酸、2-9个氨基酸或4-8个氨基酸。接头序列可以位于碳水化合物结合结构域的n-末端、碳水化合物结合结构域的c-末端或两者。当接头位于n-和c-末端时,接头序列可以相同或不同。不希望被理论所束缚,接头可用于以下目的中的一个或多个:(i)帮助确保碳水化合物结合位点处于优选的结合构象;(ii)改善碳水化合物结合位点的可及性;(iii)增加化学发光供体结构域相对于受体结构域的空间位置和/或偶极取向变化的幅度(例如,接头序列可起到增加bret比的作用);和/或(iv)优化化学发光供体结构域相对于受体结构域的空间位置和/或偶极取向。在一些实施方案中,传感器还包含蛋白酶切割位点和/或纯化标签。在一些实施方案中,接头包含可用于纯化和/或连接化学发光供体结构域和/或受体结构域的氨基酸或一系列氨基酸。例如,接头可包含组氨酸标签,用于纯化或与化学发光供体结构域和/或受体结构域自组装。在另一个实例中,接头可以包含用于添加化学发光供体结构域和/或受体结构域的反应基团(例如半胱氨酸或赖氨酸)。在一些实施方案中,传感器包含蛋白酶切割位点。蛋白酶切割位点可用于除去纯化标签。本发明的多肽可以多种方式生产,包括天然多肽的生产和回收,重组多肽的生产和回收,以及多肽的化学合成。在一个实施方案中,通过在有效产生多肽的条件下培养能够表达多肽的细胞并回收多肽来产生分离的多肽。有效培养条件可由本领域技术人员确定,包括但不限于允许多肽产生的有效培养基、生物反应器、温度、ph和氧条件。有效培养基是指培养细胞以产生多肽的任何培养基。这种培养基通常包括水性培养基,水性培养基具有可同化的碳,氮和磷酸盐源,以及合适的盐、矿物质、金属和诸如维生素的其他营养物。细胞可以在常规发酵生物反应器、摇瓶、试管、微量滴定皿和陪替氏平皿中培养。培养可以在适合于重组细胞的温度、ph和氧含量下进行。这些培养条件在本领域普通技术人员的专业知识范围内。本发明的多肽可以通过本领域技术人员已知的方法从诸如植物、细菌或酵母细胞的重组细胞中提取和纯化,产生所述多肽。在一个实施方案中,该方法包括:通过匀浆细胞/组织/植物来提取总可溶性蛋白质,并使用ni-nta或talon分离六组氨酸多肽。可以用常规凝胶或亲和色谱法实现另外的纯化。碳水化合物本发明的传感器能够结合碳水化合物。如本文所用的术语“碳水化合物”被广义地定义并且是指单糖、寡糖和多糖以及衍生自单糖的物质,例如通过还原羰基(形成糖醇),通过将一个或多个端基氧化成羧酸,或通过用氢原子、氨基、硫醇基或类似基团置换一个或多个羟基(形成衍生物)。它还包括这些化合物的衍生物(参见iupac(国际理论和应用化学联合会).compendiumofchemicalterminology(化学术语总目录)第二版.(金本),a.d.mcnaught和a.wilkinson编辑.blackwellscientificpublications(布莱克韦尔科学出版物),牛津(1997))。如本领域技术人员所知,碳水化合物可以含有不对称中心,因此具有立体异构体。可用于本发明的传感器、方法、试剂盒和组合物中的碳水化合物可以是d-立体异构形式和/或l-形式(对映异构体)形式。开链和闭环结构都属于碳水化合物的定义。在一些实施方案中,碳水化合物是糖或糖衍生物。在一些实施方案中,碳水化合物是糖。如本文所用,术语“糖”包括包含至少一个羟基和至少一个醛基或酮基的多羟基醛和多羟基酮。在一些实施方案中,糖是单糖、寡糖或多糖。合适的单糖可以包括丙糖(例如甘油醛),丁糖(例如赤藓糖和苏糖),戊糖(例如核酮糖、阿拉伯糖、木糖、核糖和来苏糖),己糖(例如葡萄糖、甘露糖、半乳糖、艾杜糖、古洛糖、果糖、阿卓糖、阿洛糖、岩藻糖和塔罗糖),庚糖(例如景天庚酮糖),辛糖(例如丙三氧基-d-甘露-辛酮糖)和戊糖环糖(例如呋喃核糖和吡喃核糖)。在一些实施方案中,单糖选自由以下组成的组:核糖、葡萄糖、甘露糖、半乳糖和果糖。本文所用的“寡糖”是含有二到十个通过糖苷键连接的糖的糖类聚合物。在一些实施方案中,寡糖包含二、三、四、五、六、七、八、九、或十种糖类。例如,寡糖包括但不限于二糖或三糖。合适的寡糖包括但不限于蔗糖、乳糖、乳果糖、海藻糖、龙胆二糖、麦芽糖、异麦芽糖、纤维二糖、松三糖、蜜三糖、水苏糖、纤维三糖、蜜二糖和毛蕊花糖。合适的寡糖包括但不限于蔗糖、乳糖、海藻糖、龙胆二糖、麦芽糖、异麦芽糖、纤维二糖、松三糖、蜜三糖、水苏糖、纤维三糖、蜜二糖和毛蕊花糖。在一些实施方案中,碳水化合物是二糖。在一些实施方案中,碳水化合物是乳糖或乳果糖。在一些实施方案中,碳水化合物是乳糖。在一些实施方案中,碳水化合物是乳果糖。在优选的实施方案中,碳水化合物是乳糖。多糖是其中单糖或寡糖通过糖苷键化学连接在一起的糖。合适的多糖包括但不限于直链淀粉、支链淀粉和糖原。在一些实施方案中,碳水化合物是糖衍生物。如本文所用,“糖衍生物”是指已被改性以用不同取代基取代一个或多个羟基的糖,或通过糖的氧化还原反应获得的糖变体。合适的取代基包括但不限于烷基、取代的烷基、环烷基、取代的环烷基、烯基、取代的烯基、环烯基、取代的环烯基、炔基、取代的炔基、烷氧基、取代的烷氧基、碳环基团、取代的碳环基团、杂环基团、取代的杂环基团、卤素、羟基、取代的羟基、硫醇、取代的硫醇、氰基、二氧磷基、取代的二氧磷基、硝基、氨基、取代的氨基、羧基、取代的羧基、酰基、取代的酰基、硫羧基、取代的硫羧基、酰胺、取代的酰胺、取代的羰基、取代的硫羰基、取代的磺酰基和取代的亚磺酰基。在一些实施方案中,糖衍生物选自由以下组成的组:糖醇(也称为糖醛或醛糖醇)、酮糖、氨基糖(或糖胺)、脱氧糖、磷酸糖、酸性糖、糖苷和内酯。合适的糖醇包括但不限于赤藓糖醇、葡萄糖醇、山梨糖醇或甘露糖醇。合适的酮糖包括但不限于二羟基丙酮、赤藓酮糖、核酮糖、木酮糖、假果糖、果糖、山梨糖和塔格糖。合适的氨基糖包括但不限于葡糖胺、半乳糖胺、n-乙酰葡糖胺、n-乙酰半乳糖胺、胞壁酸、n-乙酰胞壁酸和n-乙酰神经氨酸(唾液酸)。合适的糖苷包括但不限于吡喃葡萄糖和甲基吡喃葡萄糖。合适的内酯包括但不限于葡糖酸内酯。合适的脱氧糖包括但不限于脱氧核糖和鼠李糖。合适的磷酸糖包括但不限于6-磷酸葡萄糖、6-磷酸果糖、4-磷酸赤藓糖、5-磷酸核糖、1,6-二磷酸果糖和5-磷酸木酮糖。在一些实施方案中,碳水化合物是衍生自单糖的物质。在一些实施方案中,碳水化合物是通过还原羰基或通过将一个或多个端基氧化成羧酸而衍生自单糖的物质。例如,衍生自单糖的物质可以是但不限于糖醇(如赤藓糖醇、葡萄糖醇、山梨糖醇或甘露糖醇)和糖酸(如葡糖酸、甘露糖酸、苏糖酸和甘油酸)。在一些实施方案中,衍生自单糖的物质选自由乳酸盐和丙酮酸盐组成的组。碳水化合物结合结构域如本文所用,“碳水化合物结合结构域”是能够结合碳水化合物的多肽。碳水化合物结合结构域包含至少一个结合碳水化合物的结合位点。术语“与碳水化合物结合”是指碳水化合物与碳水化合物结合结构域的非共价结合。这种结合可以涉及碳水化合物和碳水化合物结合结构域结合结构域之间的非共价相互作用,例如盐桥、氢键、范德华力、堆积力、络合物形成或其组合。它还可以包括在结合位点与水分子的相互作用。合适的碳水化合物结合结构域可以存在于仅由结合结构域氨基酸序列组成的多肽链上,或者可以存在于更大的多肽分子(即,包含除结合结构域之外的氨基酸的多肽分子)的上下文中。因此,碳水化合物结合结构域可以是全长蛋白质(例如,全长螺旋-转角-螺旋转录因子)或其片段(例如,包含碳水化合物结合结构域的螺旋-转角-螺旋转录因子的片段)或其变体。碳水化合物结合结构域可包含天然或非天然氨基酸序列。保持与碳水化合物结合并经历构象变化的碳水化合物结合结构域的最小长度可以由本领域技术人员确定,所述构象变化足以并适于本文所述的碳水化合物检测。在一些实施方案中,碳水化合物结合结构域是天然存在的多肽。在一些实施方案中,碳水化合物结合结构域是天然存在的多肽的变体。例如,在一些实施方案中,碳水化合物结合结构域是改变的氨基酸(即,根据具体情况,通过至少一个氨基酸或核苷酸的插入、缺失或取代),使得碳水化合物结合结构域序列不再是天然发现的。在一些实施方案中,变异的位置在形成碳水化合物结合结构域的残基内。变体可以包含天然或非天然氨基酸序列。在一些实施方案中,变体碳水化合物结合结构域包含与螺旋-转角-螺旋转录因子的天然存在的碳水化合物结合结构域具有至少30%一致性的氨基酸序列。例如,在一些实施方案中,变体碳水化合物结合结构域包含与螺旋-转角-螺旋转录因子的天然存在的碳水化合物结合结构域至少30%相同至少35%相同至少40%相同至少45%相同至少50%相同至少55%相同至少60%相同至少65%相同至少70%相同至少75%相同至少80%相同至少85%相同至少90%相同至少95%相同至少98%相同至少99%相同或至少99.5%相同的氨基酸序列。在一些实施方案中,碳水化合物结合结构域是糖或糖衍生物结合结构域。在一些实施方案中,碳水化合物结合结构域是糖结合结构域。在一些实施方案中,碳水化合物结合结构域是糖衍生物结合结构域。在一些实施方案中,碳水化合物结合结构域以高亲和力结合碳水化合物。在一些实施方案中,碳水化合物结合结构域以下述的碳水化合物浓度下发生的半数最大结合与碳水化合物结合:在1nm或以下、10nm或以下、50nm或以下、100nm或以下、500nm或以下、1μm或以下、10μm或以下、50μm或以下、100μm或以下、500μm或以下、1mm以下或10mm以下。例如,在一些实施例中,ec50在约0.1μm与150μm之间、在约1μm与100μm之间或在约5μm与50μm之间。在一些实施例中,ec50在约10μm与25μm之间。或者,在一些实施方案中,ec50为约0.1mm-150mm、约1mm-100mm、约2mm-50mm或约2mm-5mm。当碳水化合物与碳水化合物结合结构域结合时,合适的碳水化合物结合结构域经历构象变化,该构象变化足以并适于本文所述的碳水化合物检测。可用于本发明传感器的碳水化合物结合结构域衍生自含有螺旋-转角-螺旋(hth)结构域(也称为螺旋-转角-螺旋基序)的转录因子。即,传感器包含螺旋-转角-螺旋转录因子的碳水化合物结合结构域,或碳水化合物结构域的变体。在一些实施方案中,传感器分子包含螺旋-转角-螺旋转录因子的碳水化合物结合结构域和存在于螺旋-转角-螺旋转录因子中的一个或多个另外的氨基酸。例如,传感器还可以包含一个或多个也存在于螺旋-转角-螺旋转录因子中的功能域(例如dna结合域)。在一个实施方案中,多肽缺乏转录因子的螺旋-转角-螺旋结构域。在另一个实施方案中,多肽具有转录因子的螺旋-转角-螺旋结构域。可用于本文所述传感器的任何变体、部分或片段保留结合碳水化合物的能力。在一些实施方案中,碳水化合物结合结构域包含公开的结合至碳水化合物的蛋白质折叠。结合至碳水化合物的蛋白质折叠的非限制性实例包括nudix水解酶折叠、碳水化合物结合模块或arac碳水化合物识别结构域。在一些实施方案中,hth转录因子是细菌hth转录因子。在一些实施方案中,hth转录因子可以来源于革兰氏阴性菌或革兰氏阳性菌。这种hth转录因子的实例示于表1。除了本文所讨论的变体或其片段之外,还可以使用表1中列出的hth转录因子的天然存在的物种变体。表1中所列hth转录因子的同源物(例如源自相关细菌物种的直系同源物)也可用于本文所述的传感器分子中。此外,预期术语“hth转录因子”包括任何天然存在的hth转录因子的变体、部分、片段或衍生物,只要变体、部分、片段或衍生物保留结合碳水化合物的能力即可。还应当理解,本领域技术人员能够通过本领域已知的合适技术修饰和优化天然存在的hth转录因子,例如体外或体内诱变、pcr改组诱变、化学修饰等。表1.示例性螺旋-转角-螺旋转录因子在一些实施方案中,hth转录因子是转录因子gntr家族的成员。gntr家族,命名为枯草芽孢杆菌中的葡糖酸操纵子阻遏物,是hth转录因子的最普遍的超家族之一(haydon和guest,1991;zheng等人,2009)。该hth蛋白家族通常具有dna结合结构域和效应子结合结构域(aravind和anantharman,2003;rigali等人,2002;rigali等,2004)。dna结合结构域在该超家族的成员中相对保守,具有中心β-片层簇和三个α-螺旋,其中两个与连接环一起构成hth基序(zheng等,2009;rigali等人,2002;kong等,2009)。相反,效应子结构域在gntr超家族中是不同的,并且它们的结构差异导致gntr转录因子的六个亚家族:四个主要亚家族(fadr、hutc、mocr、ytra)和两个次要亚家族(arar和plma)(zheng等,2009;rigali等人,2002;rigali等人,2004;wiethaus等人,2008;lee等人,2003;zhang等人,2012;franco等人,2006;franco等人,2007)。效应子结构域通常结合分子,例如碳水化合物。通常,gntr家族成员具有n-末端dna结合结构域和c-末端效应子结合。然而,在arar亚家族中,dna结合结构域通常位于c-末端,而效应子结合结构域位于n-末端。合适的细菌hth转录因子的一个实例是bgar。bgar是来自产气荚膜梭菌(clostridiumperfringins)菌株13(cpe0770;uniprot登录号:q8xmb9)的转录因子,并且是arar亚家族的假定成员(hartman等,2011)。bgar结合至乳糖并形成乳糖诱导型调节系统的一部分。在一些实施方案中,传感器包含螺旋-转角-螺旋转录因子bgar或其片段或变体。在一些实施方案中,碳水化合物结合结构域包含seqidno:1提供的氨基酸序列或其片段或变体。在一些实施方案中,碳水化合物结合结构域具有与seqidno:1提供的氨基酸序列至少30%相同、至少35%相同、至少40%相同、至少45%相同、至少50%相同、至少55%相同、至少60%相同、至少65%相同、至少70%相同、至少75%相同、至少80%相同、至少85%相同、至少90%相同、至少95%相同、至少98%相同、至少99%相同或至少99.5%相同的氨基酸序列。在一些实施方案中,碳水化合物结合结构域具有与其一部分(例如,包含seqidno:1的氨基酸1-179、氨基酸1-171、1-157、氨基酸1-150、氨基酸12-179、氨基酸12-171、氨基酸12-150、氨基酸16-179、氨基酸16-171、氨基酸16-151或氨基酸16-129)至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的氨基酸序列。在一些实施方案中,碳水化合物结合结构域包含seqidno:9提供的氨基酸序列或是其保留碳水化合物结合活性的片段或变体。在一些实施方案中,碳水化合物结合结构域具有与seqidno:9至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的氨基酸序列。bgar(或另一种hth转录因子)的最小碳水化合物结合结构域可以使用本领域技术人员已知的技术测定。例如,本文所述的蛋白质序列可用作“查询序列”,以对保守结构域数据库进行搜索,以例如鉴定假定的碳水化合物结合结构域和/或hth结构域(marchler-bauer等,2017;marchler-bauer等,2015;marchler-bauer等,2011;marchler-bauer和bryant,2004)。当使用保守结构域数据库搜索保守结构域时,可以使用默认参数。参见https:www.ncbi.nlm.nih.gov/structure/cdd/wrpsb.cgi。预测的碳水化合物结合结构域结合碳水化合物的能力可以使用本领域技术人员已知的技术证实。在一些实施方案中,bgar的碳水化合物结合结构域包含seqidno:1的氨基酸1-179、氨基酸1-171、氨基酸1-157、氨基酸1-150、氨基酸12-179、氨基酸12-171、氨基酸12-157、氨基酸12-150、氨基酸16-179、氨基酸16-171、氨基酸16-157、氨基酸16-151或氨基酸16-129。在一些实施方案中,bgar的碳水化合物结合结构域包含seqidno:1的氨基酸1-150、氨基酸1-171、氨基酸1-179、氨基酸12-171或氨基酸12-150。在一些实施方案中,碳水化合物结合结构域具有与seqidno:1的氨基酸1-157、氨基酸16-151、氨基酸16-129至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的氨基酸序列。在一些实施方案中,碳水化合物结合结构域具有与seqidno:1的氨基酸1-150、氨基酸1-171、氨基酸12-150或氨基酸12-171至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的氨基酸序列。在一些实施方案中,碳水化合物结合结构域包含seqidno:9提供的氨基酸序列。在一些实施方案中,碳水化合物结合结构域具有与seqidno:9提供的氨基酸序列至少30%相同、至少35%相同、至少40%相同、至少45%相同、至少50%相同、至少55%相同、至少60%相同、至少65%相同、至少70%相同、至少75%相同、至少80%相同、至少85%相同、至少90%相同、至少95%相同、至少98%相同、至少99%相同、至少99.5%相同或100%相同的氨基酸序列。在一些实施方案中,碳水化合物结合结构域具有与seqidno:9的一部分具有至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的氨基酸序列,或与其一部分至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的氨基酸序列。其他合适的转录因子可由本领域技术人员使用工具如blastp鉴定。例如,在一些实施方案中,转录因子包含碳水化合物结合结构域,该碳水化合物结合结构域包含氨基酸序列,该氨基酸序列与bgar(例如,seqidno:9中的氨基酸,或与氨基酸1-179、氨基酸1-171、氨基酸1-150、氨基酸12-179、氨基酸12-171或氨基酸12-150)的碳水化合物结合结构域至少30%相同、至少35%相同、至少40%相同、至少45%相同、至少50%相同、至少55%相同、至少60%相同、至少65%相同、至少70%相同、至少75%相同、至少80%相同、至少85%相同、至少90%相同、至少95%相同、至少98%相同、至少99%相同、或至少99.5%相同。合适的转录因子包括但不限于,来自产气荚膜梭菌(clostridiumperfringens)(uniprot登录号:a0a133mux6)的假定的乳糖操纵子转录激活剂、arac家族转录调节因子(产气荚膜梭菌dstr.(clostridiumperfringensdstr.)jgs1721)(uniprot登录号:b1v7n0)、arac家族转录调节因子(产气荚膜梭菌(clostridiumperfringens))(uniprot登录号:a0a127egd8)、阿拉伯糖操纵子调节蛋白(未培养的梭菌属物种)(uniprot登录号:a0a1c6jub7)、转录调节因子(甘草梭菌(clostridiumdisporicum))(uniprot登录号:a0a174hyb7)、阿拉伯糖操纵子调节蛋白(未培养的梭菌属物种)(uniprot登录号:a0a1c6ky47)、转录调节因子(甘草梭菌(clostridiumdisporicum))(uniprot登录号:a0a174lzq7)、arac家族转录调节因子(类腐败梭菌(clostridiumparaputrificum))(uniprot登录号:a0a174i591)、未表征的蛋白(丁酸梭菌(clostridiumbutyricum)60e.3)(uniprot登录号:n9yr91)、arac家族转录调节因子(新生梭状芽孢杆菌(clostridiumneonatale))(uniprot登录号:a0a2a7me67)、arac家族转录调节因子(中间葡萄球菌(staphylococcusintermedius)nctc11048)(uniprot登录号:a0a2k4azl9)。arac家族转录调节因子(伪中间型葡萄球菌(staphylococcuspseudintermedius))(uniprot登录号:a0a166ppm9)、arac家族转录调节因子(猪葡萄球菌(staphylococcushyicus))(uniprot登录号:a0a2t4r7g1)、arac家族转录调节因子(海豚葡萄球菌(staphylococcusdelphini))(uniprot登录号:a0a2a44hcu9),arac家族转录调节因子(磁葡萄球菌(staphylococcusmagnetis))(uniprot登录号:a0a2t4ms83)、乳糖操纵子转录激活子(木糖葡萄球菌(staphylococcusxylosus))(uniprot登录号:o333813)、乳糖操纵子转录激活子(腐生葡萄球菌(staphylococcussaprophyticus)(uniprot登录号:a0a1d4lkb2)、假定的乳糖操纵子转录激活子(模仿葡萄球菌(staphylococcussimulans))(uniprot登录号:a0a133qvv5)、转录调节因子arac家族(乳酸乳球菌乳酸亚种(lactococcuslactissubsp.lactis)菌株kf147)(uniprot登录号:a9qsr3。在一些实施方案中,传感器包含具有氨基酸序列的转录因子,该氨基酸序列选自由seqidno:37、seqidno:38、seqidno:39、seqidno:40、seqidno:41、seqidno:42、seqidno:43、seqidno:44、seqidno:45、seqidno:46、seqidno:48、seqidno:49、seqidno:50、seqidno:51、seqidno:52、seqidno:53、seqidno:54和seqidno:55提供的氨基酸序列组成的组,或者是其片段或变体。在一些实施方案中,传感器包含具有氨基酸序列的转录因子,该氨基酸序列与由seqidno:37、seqidno:38、seqidno:39、seqidno:40、seqidno:41、seqidno:42、seqidno:43、seqidno:44、seqidno:45、seqidno:46、seqidno:48、seqidno:49、seqidno:50、seqidno:51、seqidno:52、seqidno:53、seqidno:54和seqidno:55提供的氨基酸序列至少30%相同、至少35%相同、至少40%相同、至少45%相同、至少50%相同、至少55%相同、至少60%相同、至少65%相同、至少70%相同、至少75%相同、至少80%相同、至少85%相同、至少90%相同、至少95%相同,至少98%相同、至少99%相同、至少99.5%相同,或者是其片段或变体。在一些实施方案中,碳水化合物结合结构域包含氨基酸序列,该氨基酸序列选自由seqidno:56、seqidno:58、seqidno:59、seqidno:60、seqidno:61、seqidno:62、seqidno:63、seqidno:64、seqidno:65、seqidno:66、seqidno:67、seqidno:68、seqidno:69、seqidno:70、seqidno:71、seqidno:72、seqidno:54和seqidno:74提供的氨基酸序列组成的组,或者是其片段或变体。在一些实施方案中,碳水化合物结合结构域具有氨基酸序列,该氨基酸序列与seqidno:56,seqidno:57、seqidno:58、seqidno:59、seqidno:60、seqidno:61、seqidno:62、seqidno:63、seqidno:64、seqidno:65、seqidno:66、seqidno:67、seqidno:68、seqidno:69、seqidno:70、seqidno:71、seqidno:72、seqidno:73或seqidno:74提供的氨基酸序列至少30%相同、至少35%相同、至少40%相同、至少45%相同、至少50%相同、至少55%相同、至少60%相同、至少65%相同、至少70%相同、至少75%相同、至少80%相同、至少85%相同、至少90%相同、至少95%相同、至少98%相同、至少99%相同或至少99.5%相同。在一些实施方案中,核酸分子包含编码多肽的序列,所述多肽具有与seqidno:15、seqidno:16、seqidno:17、seqidno:18、seqidno:25、seqidno:26、seqidno:27或seqidno:28中的任何一个示出的序列至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列,或与其一部分(例如,一部分包含seqidno:15、seqidno:16、seqidno:17、seqidno:18、seqidno:25、seqidno:26、seqidno:27或seqidno:28中的任何一个序列的氨基酸11-349、氨基酸18-349、氨基酸28-349、氨基酸38-349或氨基酸39-349)至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列。在一些实施方案中,核酸分子包含编码多肽的序列,所述多肽具有与seqidno:15、seqidno:16、seqidno:17、和seqidno:18中的任何一个所示的序列至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列,或与其一部分(例如seqidno:15、seqidno:16、seqidno:17、或seqidno:18中的任何一个的氨基酸11-349、氨基酸18-349、氨基酸28-349、氨基酸38-349或氨基酸39-349)至少30%、35%、40%、45%、50%、55%、60%、65%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列。在一些实施方案中,核酸分子包含编码多肽的序列,所述多肽具有与seqidno:25、seqidno:26、seqidno:27、和seqidno:28中的任何一个所示的序列至少50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列,或与其一部分(例如seqidno:25、seqidno:26、seqidno:27、和seqidno:28中的任何一个的氨基酸11-349、氨基酸18-349、氨基酸28-349、氨基酸38-349或氨基酸39-349)至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列。例如,核酸分子包括选自由seqidno:19、seqidno:20、seqidno:21和seqidno:22组成的组的序列,或其片段或变体。在一些实施方案中,核酸分子包含编码多肽的序列,所述多肽具有与seqidno:19、seqidno:20、seqidno:21、和seqidno:22中的任何一个所示的序列至少50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列,或与seqidno:19、seqidno:20、seqidno:21、和seqidno:22中的任何一个的一部分至少50%、55%、60%、65%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列。在一些实施方案中,核酸分子包含选自由seqidno:19、seqidno:20、seqidno:21、seqidno:22、seqidno:29、seqidno:30、seqidno:31和seqidno:32组成的组的序列,或其片段或变体。在一些实施方案中,核酸分子包含编码多肽的序列,所述多肽具有与seqidno:19、seqidno:20、seqidno:21、和seqidno:22中的任何一个序列至少50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列;或与seqidno:19、seqidno:20、seqidno:21、seqidno:22、seqidno:29、seqidno30、seqidno:31、和seqidno:32中的任何一个的一部分至少50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列。例如,核酸分子包括选自由seqidno:29、seqidno:30、seqidno:31和seqidno:32组成的组的序列,或其片段或变体。在一些实施方案中,核酸分子包含编码多肽的序列,所述多肽具有与seqidno:19、seqidno:20、seqidno:21、和seqidno:22中的任何一个所示的序列至少50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列,或与seqidno:29、seqidno:30、seqidno:31、和seqidno:32中的任何一个所示的序列的一部分至少50%、55%、60%、65%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列。本发明的传感器、组合物、方法和用途包括具有指定序列或与其基本上相同或相似的序列的多肽和核酸,例如,与指定序列至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列。在氨基酸序列的上下文中,术语“基本上相同”在本文中用于指,第一氨基酸,其含有足够或最小数目的第二氨基酸序列中比对的氨基酸残基的i)相同的氨基酸残基,或ii)保守取代,使得第一和第二氨基酸序列可以具有共同的结构域和/或共同的功能活性。例如,含有与指定序列具有至少约85%、90%、91%、92%、93%、94%、95%、96%、97%、99%或98%或99%或100%一致性的共同结构域的氨基酸序列被称为基本上相同的。共振能量转移诸如乳糖的碳水化合物与本发明的传感器的结合可导致共振能量转移(ret)的变化,包括但不限于生物发光共振能量转移(“bret”)和荧光共振能量转移(“fret”)。如本文所用,“bret”是基于生物发光蛋白供体和受体分子之间能量的非辐射转移的邻近测定法。“生物发光共振能量转移”和“bret”可互换使用。如本文所用,“fret”是基于例如两种荧光团的两种生色团之间的能量的非辐射转移的邻近测定。“fret”和“荧光共振能量转移”可互换使用。一方面,传感器分子包含共价连接至转录因子或其片段或变体的供体结构域和受体结构域。在一些实施方案中,供体结构域是化学发光供体结构域。在可选择的实施方案中,供体结构域是荧光团。在一些实施方案中,受体结构域是荧光受体结构域。在一些实施方案中,供体结构域共价连接至转录因子或其片段或变体的n末端,受体结构域共价连接至转录因子或其片段或变体的c末端。在可选择的实施方案中,供体结构域共价连接至转录因子或其片段或变体的c-末端,且受体结构域共价连接至转录因子或其片段或变体的n-末端。a.供体结构域本发明的传感器分子包含供体结构域。供体结构域能够用作共振能量转移对(例如bret对或fret对)中的供体结构域,并且根据上下文,也称为“共振能量转移供体结构域”。如本文所用,术语“供体”是指发射光的分子,例如当用某一波长的光照射时发射光的分子或由于化学反应引起光发射的分子。合适的供体结构域包括化学发光结构域和荧光结构域。在一些优选的实施方案中,供体结构域能够用作bret对中的供体结构域。例如,供体结构域可以是化学发光供体结构域。化学发光是由于化学反应而具有有限的热发射(发冷光)的能量发射。本文所用的术语“化学发光”包括依赖于酶活性的生物发光。非酶化学发光是在催化剂存在下有机染料和氧化剂之间的化学反应的结果。当化学诱导的有机染料激发态的能量衰减到基态时,发生化学发光发射。化学发光发射的持续时间和强度主要取决于反应溶液中存在的化学试剂的程度。在优选实施方案中,化学发光供体结构域是生物发光蛋白。如本文所用,术语“生物发光蛋白”是指能够作用于合适底物以产生发光的任何蛋白。本领域应当理解,生物发光蛋白是将底物转化为活化产物,然后当其松弛时释放能量的酶。活化产物(由底物上生物发光蛋白的活性产生)是转移到受体分子上的生物发光蛋白产生的发冷光的来源。有许多不同的生物发光蛋白可用于本发明(参见,例如,表2)。发光系统已知并分离自许多发光生物体,包括细菌、原生动物、腔肠动物、软体动物、鱼、多足类、苍蝇、真菌、蠕虫,甲壳类和甲虫,特别是火甲虫属(pyrophorus)的叩头虫和北美萤火虫(photinus)、妖扫萤属(photuris)和熠萤(luciola)属的萤火虫。显示生物发光的其他生物体列于wo00/024878,wo99/049019和viviani(2002)。表2.示例性生物发光蛋白。任何合适的生物发光蛋白可用于本发明的传感器中。一个非常熟知的例子是称为荧光素酶的一类蛋白质,其催化产生能量的化学反应,其中特定的生化物质荧光素(天然存在的荧光团)被具有荧光素酶活性的酶氧化(hastings,1996)。原核生物和真核生物(包括细菌、藻类、真菌、昆虫、鱼和其他海洋形式的物种)的巨大多样性可以以这种方式发射光能,并且各自具有与其他生物的荧光素酶活性和荧光素化学上不同的特异性荧光素酶活性和荧光素。萤光素/萤光素酶系统在形式、化学和功能上非常多样。因此,具有荧光素酶活性的生物发光蛋白可从多种来源或通过多种方法获得。具有荧光素酶活性的生物发光蛋白的实例可以在us5,229,285、5,219,737、5,843,746、5,196,524、和5,670,356中找到。两种最广泛使用的荧光素酶是:(i)海肾荧光素酶(来自r.reniformis),一种35kda的蛋白质,它使用腔肠素作为底物并在480nm处发光(lorenz等,1991);和(ii)萤火虫荧光素酶(来自photinuspyralis),一种61kda的蛋白质,其以荧光素为底物并在560nm处发光(dewet等,1987)。gaussia荧光素酶(来自gaussiaprincps)已经用于生物化学测定(verhaegen等,2002)。gaussia荧光素酶是20kda蛋白质,其以快速反应氧化腔肠素,导致470nm处的明亮光发射。用于本发明的荧光素酶的特征还在于anachnocampasp(wo2007/019634)。这些酶的大小约为59kda,并且是atp依赖性荧光素酶,其催化发射光谱在光谱的蓝色部分内的发光反应。天然存在的生物发光蛋白的生物活性变体或片段可由本领域技术人员容易地制备。可用于本发明的这些变体的三个实例是rluc2(loening等,2006)、rluc8(loening等,2006)和rluc8.6-535(loening等,2007),它们各自是海肾(renilla)荧光素酶的变体。在另一个优选的实施方案中,选择bret化学发光供体的序列以具有比掺入天然海肾(renilla)荧光素酶传感器的传感器分子更大的热稳定性。rluc2或rluc8是合适选择的方便实例,其因此显示比掺入天然海肾(renilla)荧光素酶序列的传感器高≥5x或≥10x的亮度。这种增强的亮度具有显著的益处,因为它允许对于任何给定的时间分辨率更经济地使用试剂。或者,可用于本发明的非荧光素酶生物发光蛋白是可作用于合适底物以产生发光信号的任何酶。这些酶的具体实例是β-半乳糖苷酶,内酰胺酶,辣根过氧化物酶,碱性磷酸酶,β-葡糖醛酸糖苷酶和β-葡糖苷酶。用于这些酶的合成发光底物是本领域公知的,并且可从公司例如tropix公司(美国马萨诸塞州贝德福德)商购获得。hushpulian等,(2007)描述了可用于本发明的过氧化物酶的实例。在一些实施方案中,生物发光蛋白是荧光素酶、β-半乳糖苷酶、内酰胺酶、辣根过氧化物酶、碱性磷酸酶、β-葡糖醛酸糖苷酶或β-葡糖苷酶。在一些实施方案中,生物发光蛋白是荧光素酶。合适的萤光素酶包括但不限于,海肾萤光素酶、萤火虫萤光素酶、腔肠动物(coelenterate)萤光素酶、北美萤火虫(northamericanglowworm)萤光素酶、叩头虫(clickbeetle)萤光素酶、铁路蠕虫(railroadworm)萤光素酶、细菌萤光素酶、gaussia萤光素酶、水母(aequorin)发光蛋白、蕈蚊(arachnocampa)萤光素酶、oplophorusgracilirostris萤光素酶或其任何一种的生物活性变体或片段,或其两种或更多种的嵌合体。在一个实例中,优选的荧光素酶是rluc8或其变体。如本文所用,“生物活性片段”是如本文所述的多肽的一部分,其保持全长多肽的限定的活性。如本文所用,“生物活性变体”是通过一个或多个氨基酸与天然存在的和/或确定的分子不同但保持限定的活性的分子,如以上对于生物活性片段所定义的。生物活性变体通常与天然存在和/或限定的分子具有至少50%、更优选至少80%、更优选至少90%、更优选至少95%、更优选至少97%、甚至更优选至少99%的一致性。在一个优选的实施方案中,具有小分子量的生物发光蛋白用于防止由于空间位阻而抑制相互作用。生物发光蛋白优选由单个多肽链组成。此外,生物发光蛋白优选不形成低聚物或聚集体。生物发光蛋白renilla荧光素酶,gaussia荧光素酶和萤火虫(firefly)荧光素酶满足所有或大部分这些标准。在一些实施方案中,化学发光供体结构域能够修饰底物。如本文所用,术语“底物”是指可与化学发光供体结合使用以产生或吸收发光的任何分子。底物的选择可以影响由化学发光供体产生的光的波长和强度。在一些实施方案中,生物发光蛋白具有底物,所述底物选自荧光素,钙,腔肠素,furimazine,或腔肠素、荧光素或furimazine的衍生物、类似物或稳定衍生物。腔肠素(coelenterazine)是广泛已知的底物,其存在于刺细胞动物(cnidarians)、桡足类(coppods)、毛颚类(chaetognaths)、栉水母类(ctenophores)、十足虾(decapodshrinps)、糠虾(mysideshrinps),放射虫(radiolaris)和一些鱼类类群中(greenr和szalay,2002)。例如,对于海肾荧光素酶,可获得腔肠素类似物/衍生物,其导致418和547nm之间的光发射(inouye等,1997,loening等,2007)。已经描述了腔肠素类似物/衍生物(400a,deepbluec)与海肾荧光素酶一起发射400nm的光(wo01/46691)。腔肠素类似物/衍生物的其他实例是栉水母类、prolume紫、prolume紫ii、prolume紫iii、viviren和furimazine。腔肠素类似物/衍生物的其他实例包括但不限于pct/us2013057660和us20140302539中公开的化合物。如本文所用,术语“荧光素”被广义地定义,并且是指在能够生物发光的生物体中发现的一类发光生物颜料以及合成类似物或功能等同的化学品,其在酶荧光素酶的存在下被氧化以产生光形式的氧化荧光素和能量。首先从萤火虫photinuspyralis中分离出d-荧光素或2-(6-羟基苯并噻唑-2-基)-2-噻唑啉-4-甲酸。从那时起,已经从各种不同的生物体,主要从海洋,例如鱼和鱿鱼中,发现和研究了荧光素的各种化学上不同的形式,然而,已经在陆地居住生物体中鉴定了许多荧光素,例如蠕虫、甲虫和各种其他昆虫(day等,2004;vivivani,2002)。如本文所用,荧光素还包括荧光素的衍生物或类似物。除了完全合成的荧光素,例如环状烷基氨基荧光素(cycluc1)之外,还存在至少五种一般类型的生物演化的荧光素,它们各自在化学上不同并且由化学和结构上不同的荧光素酶催化,所述荧光素酶使用广泛的不同辅因子。首先,萤火虫萤光素是萤火虫萤光素酶的底物,其需要atp用于催化(ec1.13.12.7)。第二,是细菌荧光素,也在一些鱿鱼和鱼中发现,其由长链醛和还原的核黄素磷酸酯组成。细菌荧光素酶是fmnh依赖性的。第三,是甲藻萤光素,一种在甲藻(海洋浮游生物)中发现的四吡咯叶绿素衍生物,该生物体造成夜间海洋磷光。甲藻萤光素酶催化甲藻萤光素的氧化,并且由三个相同的催化活性结构域组成。第四,是咪唑并吡嗪介形虫荧光素(vargulin),其被发现于某些介形类和深海鱼类中,例如光蟾鱼(porichthys)。最后,是腔肠素(coelenterazine)(一种咪唑并吡嗪),一种蛋白质水母(aequorin)发光蛋白的光发射体,发现于放射虫、栉水母类、刺细胞动物、鱿鱼、桡足类、毛颚类、鱼和虾中。在一些实施方案中,生物发光蛋白需要辅因子。辅因子的实例包括但不一定限于atp、镁、氧、fmnh2、钙或其任何两种或多种的组合。在另一个实施方案中,共振能量转移供体结构域是荧光供体结构域。荧光供体结构域可以是荧光蛋白或非蛋白。在一些实施方案中,荧光供体结构域是非蛋白质。适合用作供体结构域的荧光团的非限制性实例包括但不限于alexafluor染料(例如af680、af750)、bodipy染料、cy染料、荧光素、丹磺酰基、伞形酮、荧光微球、发光微球、荧光纳米晶体、marina蓝、cascade蓝、cascade黄、pacific蓝、oregon绿、四甲基若丹明、若丹明、德克萨斯红、稀土元素螯合物或其任何组合或衍生物。在一些实施方案中,供体结构域是荧光蛋白。非限制性实例包括诸如以下的蛋白:绿色荧光蛋白(gfp)、gfp的蓝色荧光变体(bfp)、gfp的青色荧光变体(cfp)、gfp的黄色荧光变体(yfp)、增强的gfp(egfp)、增强的cfp(ecfp)、增强的yfp(eyfp)、gfps65t、emerald、venus、morange、topaz、gfpuv、去稳定egfp(degfp)、去稳定ecfp(decfp)、去稳定eyfp(deyfp)、hc红(hcred)、t-hc红(t-hcred)、ds红(dsred)、ds红2(dsred2)、t-二聚体2、t二聚体2(12)、mrfp1、杯型珊瑚色(pocilloporin)、海肾gfp(renillagfp)、monstergfp、pagfp、tdtomato、mcherry、kaede蛋白、tagrfp、turbofb或藻胆(色素)蛋白,或其任何一个的生物活性变体或片段。在一些实施方案中,优选的荧光供体结构域是cfp。b.受体结构域本发明的传感器分子还包含受体结构域。受体结构域能够用作共振能量转移对(例如bret对或fret对)中的受体结构域,并且根据上下文,也称为“共振能量转移受体结构域”。如本文所用,“受体结构域”是能够接受由于供体结构域的活性而发射的能量的任何分子。在一些实施方案中,受体结构域(本文也称为“受体分子”)是荧光受体结构域。如本文所用,术语“荧光受体结构域”(本文也称为“荧光受体分子”)是指能够接受由于供体结构域的活性而发射的能量并将其作为光能再发射的任何化合物。有许多不同的受体结构域可用于本发明。合适的受体结构域可以是蛋白质或非蛋白质的。在一些实施方案中,荧光受体结构域是荧光蛋白。一个众所周知的例子是荧光团组,其包括:来自水母维多利亚多管发光水母(aequoreavictoria)的绿色荧光蛋白,和由例如诱变和嵌合蛋白技术的分子生物学应用产生的许多其他变体(gfps)(tsien,1998)。gfps基于其发色团的独特组分进行分类,每一类具有不同的激发和发射波长:第1类,中性酚和阴离子酚盐的野生型混合物:第2类,酚盐阴离子:第3类,中性酚:第4类,具有堆叠s-电子系统的酚盐阴离子:第5类,吲哚:第6类,咪唑:和第7类,苯基。已经突变(变体)的天然存在的受体分子也可用于本发明。适用于bret的工程化系统的一个实例是海肾荧光素酶和gfp配对的增强黄色突变体(eyfp),其在不存在介导蛋白(在这种情况下,g蛋白偶联受体)的情况下彼此不直接显著程度地相互作用(xu等,1999)。实例包括但不限于:绿色荧光蛋白(gfp)、gfp的蓝色荧光变体(bfp)、gfp的青色荧光变体(cfp)、gfp的黄色荧光变体(yfp)、增强的gfp(egfp)、增强的gfp(egfp)、增强的cfp(ecfp)、增强的yfp(eyfp)、gfps65t、emerald、venus、morange、topaz、gfpuv、去稳定egfp(degfp)、去稳定ecfp(decfp)、去稳定eyfp(deyfp)、hc红(hcred)、t-hc红(t-hcred)、ds红(dsred)、ds红2(dsred2)、t-二聚体2、t二聚体2(12)、mrfp1、杯型珊瑚色(pocilloporin)、海肾gfp(renillagfp)、monstergfp、pagfp、kaede蛋白、tdtomato、mcherry、tagrfp、turbofb和藻胆蛋白,或其任何一个的生物活性变体或片段。在一些实施方案中,优选的荧光受体结构域是gfp2。在其他实施方案中,优选的荧光受体结构域是yfp。在一些实施方案中,荧光受体结构域是非蛋白质。非蛋白质的受体分子的实例包括但不限于alexafluor染料(例如af680、af750)、bodipy染料、cy染料、荧光素、丹磺酰基、伞形酮、荧光微球、发光微球、荧光纳米晶体、marina蓝、cascade蓝、cascade黄、pacific蓝、oregon绿、四甲基若丹明、若丹明、德克萨斯红、稀土元素螯合物或其任何组合或衍生物。c.供体结构域和受体结构域对在本发明的传感器中可以使用任何数量的供体-受体组合。供体-受体组合应该能够用作bret对或fret对。本领域技术人员能够选择允许有效能量转移的供体和受体对。在优选的实施方案中,在存在和/或不存在碳水化合物的情况下,供体结构域和受体结构域的分离和相对取向在费尔斯特距离的±50%之内。如本文所用,术语“供体结构域和受体结构域在存在和/或不存在碳水化合物的情况下的分离和相对取向在费尔斯特距离的±50%内”是指可以在r0的±50%的范围内进行的稳态ret测量。该短语包括从供体结构域到受体结构域的发光能量转移效率为10-90%。在一些实施方案中,化学发光供体结构域和受体结构域的费尔斯特距离为至少4nm、为至少5.6nm、或为至少6nm。在一些实施方式中,该费尔斯特距离小于12nm、小于11nm、小于10nm或小于9nm。在一些实施方案中,供体结构域和受体结构域的费尔斯特距离是约4nm-约11nm、约5.6nm-约11nm或约7nm-约11nm。不希望受理论的束缚,本发明人相信供体结构域和受体结构域的费尔斯特距离与可用于本申请传感器的碳水化合物结合结构域的大小相匹配。碳水化合物结合结构域可以是:例如全长hth转录因子,其保留碳水化合物结合活性的片段,hth转录因子的碳水化合物结合结构域或其变体。在确定合适配对时应考虑的标准是受体分子与供体分子相比的相对发射/荧光光谱。供体的发射光谱应当与受体分子的吸收光谱重叠,使得来自供体发射的光能的波长能够激发受体分子,从而当两个分子彼此适当接近和取向时促进受体分子荧光。例如,基于可观察到的发射光谱峰,已经证明海肾荧光素酶/egfp配对不如海肾荧光素酶/eyef配对(xu等,1999;wang等,(1997)在生物发光和化学发光:molecularreportingwithphotons(光子分子报告),hastings等编辑(纽约威利),第419-422页)。为了研究可能的配对,制备含有选择的供体和受体结构域的蛋白质融合体(例如),并且如果需要,在合适的底物存在下进行测试。还应当确认供体和受体结构域彼此不虚假地缔合。例如,这可以通过在相同细胞中分别共表达生物发光蛋白和受体分子,然后监测发光光谱以确定bret是否发生来实现。这可以例如使用xu等(1999)的方法来实现。如果观察到几乎没有或没有bret,则所选择的生物发光蛋白和受体分子形成合适的bret对。可以对fret对进行类似的实验。在一些实施方案中,本发明的传感器分子包含化学发光供体结构域和荧光受体结构域。在一些实施例中,可通过对底物的修改来操纵供体发射。在海肾(renilla)荧光素酶的情况下,底物是腔肠素。改变供体发射的基本原理是提高供体发射和受体发射之间的分辨率。原始bret系统使用海肾(renilla)荧光素酶作为供体,eyfp(或topaz)作为受体,腔肠素h衍生物作为底物。当在bret测定中组合时,这些组分对于生物发光蛋白产生475-480nm范围内的光,对于受体分子产生525-530nm范围内的光,得到45-55nm的光谱分辨率。不幸的是,海肾(renilla)荧光素酶产生基本上与gfp发射重叠的宽发射峰,这又有助于降低系统的信噪比。用于本发明的一种bret系统具有coe1400a作为海肾(renilla)荧光素酶底物并提供供体和受体发射波长(-105nm)之间的宽光谱分辨率。具有coe1400a的海肾(renilla)荧光素酶产生390-400nm之间的光,制备gfp衍生物(gfp2),其吸收该范围内的光并重新发射505-508nm的光。由于海肾(renilla)荧光素酶和gfp发射之间光谱分辨率的增加,该bret系统提供了监测碳水化合物与本申请传感器结合的极好的生物工具。然而,较小的斯托克斯(stokes)位移bret系统也将允许碳水化合物的灵敏测量。本领域已知各种腔肠素衍生物,包括coel400a,其由于海肾荧光素酶活性而产生各种波长的光(不同于野生型腔肠素产生的光)。本领域技术人员将理解,因为供体的发光峰已经改变,所以必须选择将吸收该波长的光并由此允许有效能量转移的受体分子。这可以例如通过改变第4类gfp以使其变成第3类或第1类gfp来完成。供体的光发射和受体的光吸收峰之间的光谱重叠是有效能量转移的一个条件。已知第3类或第1类gfp吸收400nm的光并在505-511nm之间重新发射。这导致供体和受体发射之间的波长差约为111nm。其他生物发光蛋白和受体分子对的实例提供于表3中。表3.示例性bret生物发光蛋白和受体分子对。在一些实施方案中,优选的生物发光蛋白和受体结构域对是rluc8和gfp2。在一些实施方案中,本发明的传感器分子包含荧光供体结构域和荧光受体结构域。任何适当选择的荧光团都可以用作供体和/或受体,条件是供体的发射光谱与受体的激发光谱充分重叠即可。在确定合适配对时应考虑的标准是受体分子与供体的激发光谱相比的激发光谱。如本领域技术人员将理解的,在供体结构域的激发最大值处应该存在受体结构域的最小直接激发。其他荧光供体和受体结构域对的实例提供于表4中。荧光供体和受体结构域对的其他实例在bajar等(2016)中讨论。表4.示例性fret荧光供体和受体结构域对。荧光供体受体波长(em)荧光受体分子受体波长(exc)fitc520nmtritc550nmcy3566nmcy5649nmegfp508nmcy3554nmcfp477nmyfp514nmegfp508nmyfp514nmgfp2yfp514nmecfp475nmeyfp513nmmturquioise2474nmmcitrine516nmmclover3518nmmruby3558nmeqfp650650nmirfp690nmmametrine526nmtdtomato554nm在一些实施方案中,优选的供体结构域和受体结构域对是cfp和yfp。碳水化合物结合碳水化合物与本发明传感器的碳水化合物结合结构域的结合改变供体结构域相对于受体结构域的空间位置和/或偶极取向。在一些实施方案中,空间位置和/或偶极取向的改变导致bret的改变。在一些实施方案中,空间位置和/或偶极取向的改变导致fret的改变。如本文所用,术语“空间位置”是指供体相对于受体分子的三维定位,其由于分析物结合或从传感器分子释放而改变。如本文所用,术语“偶极取向”是指与供体和/或受体分子相关的偶极矩在三维空间中相对于它们在三维空间中的取向的方向。偶极矩是分子上电荷变化的结果。以bret为例,在一个实施方案中,将生物发光蛋白和受体分子之间发生的能量转移表示为使用选择特定波长的滤光片(一个用于受体分子发射,另一个用于生物发光蛋白发射)测量的发射的计算比率(参见等式1)。ea/ed=bret比率(1)其中ea定义为受体分子发射强度(使用适合于受体发射的特定滤光器选择发射光),且ed定义为生物发光蛋白发射强度(使用适合于生物发光蛋白发射的特定滤光器选择发射光)。本领域技术人员应当容易理解,滤光器可以是允许适用于bret的波长区分的任何类型的滤光器。例如,根据本发明使用的滤光器可以是干涉滤光器、长通滤光器、短通滤光器等。通过滤光器的波长的强度(通常以每秒计数(cps)或相对发光单位(rlu))可以使用光电倍增管(pmt)、包括级联光电二极管的光电二极管、光电二极管阵列或诸如电荷耦合器件(ccd)照相机的敏感照相机来量化。量化的信号随后用于计算bret比率并表示能量转移效率。bret比率随着受体发射强度的增加而增加。通常,确定受体发射强度与供体发射强度的比率(参见等式1),其是反映能量转移效率的以任意单位表示的数。该比率随着能量转移效率的增加而增加(参见xu等人,1999)。能量转移效率也可以使用供体发射强度与受体发射强度的反比来表示(参见等式2)。在这种情况下,比率随着能量传递效率的增加而减小。在进行该计算之前,针对背景光的存在和底物的自发光来校正发光强度。这种校正通常通过从含有底物但不含本发明的生物发光蛋白、受体分子或多肽的对照样品中减去在适当波长下测量的发射强度来进行。ed/ea=bret比率(2)其中ea和ed如上所定义。生物发光蛋白和受体分子发射的光强度也可以使用诸如分光荧光计的基于单色器的仪器、电荷耦合器件(ccd)照相机或二极管阵列检测器来定量。使用分光荧光计,进行发射扫描,使得在加入底物时检测生物发光蛋白和受体分子发射峰。峰值下的面积代表相对光强度并用于计算比率,如上所述。能够测量来自同一样品的生物发光蛋白和受体分子的光的任何仪器都可用于监测本发明的bret系统。在另一个实施方案中,受体分子发射单独适用于bret的有效检测和/或定量。在这种情况下,仅使用受体发射强度来表示能量转移效率。对于本领域技术人员显而易见的是,为了测量能量转移,可以使用受体发射强度而不进行任何比率计算。这是由于理想情况下受体分子仅在吸收从生物发光蛋白转移的光时才发光。在这种情况下,只需要一个滤光器。在相关的实施方案中,单独的生物发光蛋白发射适于bret的有效检测和/或定量。在这种情况下,仅使用生物发光蛋白发射强度计算能量转移效率。对于本领域技术人员显而易见的是,为了测量能量转移,可以使用供体发射强度而不进行任何比率计算。这是由于当受体分子吸收从生物发光蛋白转移的光时,生物发光蛋白的可检测发射相应减少。在这种情况下,只需要一个滤光器。在替代实施例中,使用仅需要一个滤光器来进行测量的比率测量来表示能量转移效率。在这种情况下,使用适当的滤光器确定供体或受体的光强度,并且在不使用任何滤光器的情况下进行样品的另一测量(开放光谱的强度)。在后一测量中,量化总光输出(对于所有波长)。然后使用等式3或4进行比率计算。对于等式3,只需要用于受体的滤光器。对于等式4,只需要用于供体的滤光器。ea/eo-ea=bret比率或=eo-ea/ea(3)eo-ed/ed=bret比率或=ed/eo-ed(4)其中ea和ed如上所定义,eo定义为所有组合波长(开放光谱)的发射强度。对于本领域技术人员显而易见的是,可以从等式1到4导出另外的等式。例如,一种这样的衍生物包括校正存在于生物发光蛋白和/或受体分子的发射波长处的背景光。在进行bret测定时,可以使用bretcount测定每个孔的光发射。bretcount仪器是改进的topcount,其中topcount是由帕卡德仪器(packardinstrument)(康涅狄格州梅里登市)销售的微量滴定板闪烁和发光计数器。与利用两个光电倍增管(pmt)同时消除背景噪声的传统计数器不同,topcount采用单pmt技术和时间分辨脉冲计数以降低噪声,从而允许在标准不透明微量滴定板中计数。不透明微滴定板的使用可以将光学串扰降低到可忽略的水平。topcount有各种格式,包括1、2、6和12个检测器(pmt),它们分别允许同时读取1、2、6或12个样本。除了bretcount之外,其他市售仪器能够进行bret:victor2(芬兰瓦拉克(珀金埃尔默生命科学))和fusion(梅里登市帕卡德仪器)。bret可以使用能够检测至少受体分子发射并且优选两个波长(对于受体分子和生物发光蛋白)或更多波长的读取器来进行。bret是一种比率测定技术,其可以消除由于测定体积、测定条件和穿过板中不同孔的信号衰减的变化引起的光输出波动引起的数据可变性。基于ret的反应是均相的,通常在没有固相附着的溶液中发生。这允许在不分离的情况下检测不同形式的分析物,例如液体、气体甚至微粒。乳糖传感器分子本文定义的传感器分子的一个非限制性实例是可用于检测和/或测量乳糖浓度的传感器分子。因此,在一些实施方案中,本发明提供了用于检测乳糖的传感器分子,其包含能够结合乳糖或其变体的细菌转录因子,该细菌转录因子共价连接至共振能量转移供体结构域和共振能量转移受体结构域,其中当乳糖结合至转录因子时,供体结构域相对于受体结构域的空间位置和/或偶极取向改变。在一些实施方案中,本发明提供了一种用于检测乳糖的传感器分子,其包含共价连接至共振能量转移供体结构域和共振能量转移受体结构域的细菌bgar转录因子或其变体,其中当乳糖结合至转录因子时,所述供体结构域相对于所述受体结构域的空间位置和/或偶极取向改变。乳糖与传感器分子的结合产生共振能量转移(ret)的变化,例如bret的变化或fret的变化。根据选择的供体结构域和受体结构域,ret的变化可以是bret的变化或fret的变化。在一些实施方案中,bgar转录因子或其变体具有氨基酸序列,所述氨基酸序列与seqidno:1提供的序列至少50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同,或具有与其一部分至少30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%相同的序列。在一些实施方案中,bgar转录因子与seqidno:1提供的100%相同。在一些实施方案中,所述bgar转录因子或其变体具有与seqidno:9中提供的氨基酸序列至少50%、60%、70%、80%、85%、90%、95%、98%或100%相同的氨基酸序列。在一些实施方案中,bgar转录因子或其变体与seqidno:9提供的100%相同。因此,在一些实施方案中,共振能量转移供体结构域是生物发光蛋白。合适的生物发光蛋白的非限制性实例如上所述,且包括荧光素酶、β-半乳糖苷酶、内酰胺酶、辣根过氧化物酶、碱性磷酸酶、β-葡糖醛酸糖苷酶或β-葡糖苷酶。在一些实施方案中,生物发光蛋白是荧光素酶。萤光素酶可以选自海肾萤光素酶、萤火虫萤光素酶、腔肠动物(coelenterate)萤光素酶、北美萤火虫(northamericanglowworm)萤光素酶、叩头虫(clickbeetle)萤光素酶、铁路蠕虫(railroadworm)萤光素酶、细菌萤光素酶、gaussia萤光素酶、水母(aequorin)发光蛋白、蕈蚊(arachnocampa)萤光素酶、深海虾(oplophorusgracilirostris)萤光素酶或其任何一种的生物活性变体或片段,或其两种或更多种的嵌合体。在一些实施方案中,共振能量转移供体结构域是海肾荧光素酶。在一些实施方案中,共振能量转移供体域是rluc8。在一些实施方案中,共振能量转移供体结构域能够修饰底物。底物的非限制性实例包括荧光素,钙,腔肠素,furimazine,或腔肠素、荧光素或furimazine的衍生物、类似物或稳定衍生物。在这些实施方案中,乳糖与传感器分子的结合产生bret的变化。因此,在一些实施方案中,共振能量转移供体结构域是荧光蛋白。合适的荧光蛋白的非限制性实例包括:绿色荧光蛋白(gfp)、gfp的蓝色荧光变体(bfp)、gfp的青色荧光变体(cfp)、gfp的黄色荧光变体(yfp)、增强的gfp(egfp)、增强的cfp(ecfp)、增强的yfp(eyfp)、gfps65t、emerald、venus、morange、topaz、gfpuv、去稳定egfp(degfp)、去稳定ecfp(decfp)、去稳定eyfp(deyfp)、hc红(hcred)、t-hc红(t-hcred)、ds红(dsred)、ds红2(dsred2)、t-二聚体2、t二聚体2(12)、mrfp1、杯型珊瑚色(pocilloporin)、海肾gfp(renillagfp)、monstergfp、pagfp、kaede蛋白、tdtomato、mcherry、tagrfp、turbofb和藻胆蛋白,和其任何一个的生物活性变体或片段。在一些实施方案中,供体结构域是cfp。在这些实施方案中,乳糖与传感器分子的结合产生fret的变化。在一些实施方案中,共振能量转移受体结构域是荧光受体结构域。在一些实施方案中,荧光受体结构域是荧光蛋白。合适的荧光蛋白的非限制性实例包括:绿色荧光蛋白(gfp)、gfp的蓝色荧光变体(bfp)、gfp的青色荧光变体(cfp)、gfp的黄色荧光变体(yfp)、增强的gfp(egfp)、增强的cfp(ecfp)、增强的yfp(eyfp)、gfps65t、emerald、venus、morange、topaz、gfpuv、去稳定egfp(degfp)、去稳定ecfp(decfp)、去稳定eyfp(deyfp)、hc红(hcred)、t-hc红(t-hcred)、ds红(dsred)、ds红2(dsred2)、t-二聚体2、t二聚体2(12)、mrfp1、杯型珊瑚色(pocilloporin)、海肾gfp(renillagfp)、monstergfp、pagfp、kaede蛋白、tdtomato、mcherry、tagrfp、turbofb和藻胆蛋白,和其任何一个的生物活性变体或片段。在一些实施方案中,受体结构域是yfp。在其他实施方案中,受体结构域是gfp,优选gfp2。在优选的实施方案中,供体结构域是cfp或其变体,且受体结构域是yfp或其变体。在一些实施方案中,传感器还包含yfp和bgar之间和/或cfp和bgar之间的接头。在一些实施方案中,传感器还包含蛋白酶切割位点和/或纯化标签。在一些实施方案中,传感器包含与seqidno:23提供的氨基酸序列至少30%相同、至少35%相同、至少40%相同、至少45%相同、至少50%相同、至少55%相同、至少60%相同、至少65%相同、至少70%相同、至少75%相同、至少80%相同、至少85%相同、至少90%相同、至少95%相同、至少98%相同、至少99%相同、至少99.5%相同的氨基酸序列。在一些实施方案中,传感器与seqidno:23提供的100%相同。在这些实施方案中,乳糖与传感器分子的结合产生fret的变化。在优选的实施方案中,供体结构域是海肾(renilla)荧光素酶或其变体,且受体结构域是gfp或其变体。例如,供体结构域可以是rluc8,且受体结构域可以是gfp2。在一些实施方案中,传感器分子是包含rluc8-bgar-gfp2的单个多肽。在一些实施方案中,传感器分子是是包含gfp2-bgar-rluc8的单个多肽。在一些实施方案中,传感器还包含gfp2和bgar之间和/或rluc8和bgar之间的接头。在一些实施方案中,传感器还包含蛋白酶切割位点和/或纯化标签。在一些实施方案中,传感器包含氨基酸序列,该氨基酸序列具有与选自由seqidno:15、seqidno:16、seqidno:17、seqidno:18、seqidno:25、seqidno:26、seqidno:27、seqidno:28组成的组的氨基酸序列至少30%相同、至少35%相同、至少40%相同、至少45%相同、至少50%相同、至少55%相同、至少60%相同、至少65%相同、至少70%相同、至少75%相同、至少80%相同、至少85%相同、至少90%相同、至少95%相同、至少98%相同、至少99%相同或至少99.5%相同。在一些实施方案中,传感器包含氨基酸序列,该氨基酸序列与选自由seqidno:33、seqidno:34、seqidno:35和seqidno:36组成的组的氨基酸序列至少30%相同、至少35%相同、至少40%相同、至少45%相同、至少50%相同、至少55%相同、至少60%相同、至少65%相同、至少70%相同、至少75%相同、至少80%相同、至少85%相同、至少90%相同、至少95%相同、至少98%相同、至少99%相同、至少99.5%相同。在一些实施方案中,传感器包含氨基酸序列,该氨基酸序列与选自由seqidno:15、seqidno:16、seqidno:17和seqidno:18组成的组的氨基酸序列至少30%相同、至少35%相同、至少40%相同、至少45%相同、至少50%相同、至少55%相同、至少60%相同、至少65%相同、至少70%相同、至少75%相同、至少80%相同、至少85%相同、至少90%相同、至少95%相同、至少98%相同、至少99%相同、至少99.5%相同。在一些实施方案中,传感器包含氨基酸序列,该氨基酸序列具有与选自由seqidno:25、seqidno:26、seqidno:27、和seqidno:28组成的组的氨基酸序列至少30%相同、至少35%相同、至少40%相同、至少45%相同、至少50%相同、至少55%相同、至少60%相同、至少65%相同、至少70%相同、至少75%相同、至少80%相同、至少85%相同、至少90%相同、至少95%相同、至少98%相同、至少99%相同或至少99.5%相同。在一些实施方案中,传感器包含氨基酸序列,该氨基酸序列与选自由seqidno:15、seqidno:16、seqidno:17、seqidno:18、seqidno:25、seqidno:26、seqidno:27、和seqidno:28组成的组的氨基酸序列100%相同。在一些实施方案中,传感器包含氨基酸序列,该氨基酸序列与选自由seqidno:15、seqidno:16、seqidno:17、和seqidno:18组成的组的氨基酸序列100%相同。在一些实施方案中,传感器包含氨基酸序列,该氨基酸序列与选自由seqidno:25、seqidno:26、seqidno:27、和seqidno:28组成的组的氨基酸序列100%相同。在一些实施方案中,传感器包含氨基酸序列,该氨基酸序列与选自由seqidno:33、seqidno:34、seqidno:35、和seqidno:36组成的组的氨基酸序列100%相同。在一些实施方案中,传感器包含氨基酸序列,该氨基酸序列与选自seqidno:15、seqidno:16、seqidno:17、seqidno:18、seqidno:25、seqidno:26、seqidno:27、和seqidno:28组成的组的氨基酸序列100%相同,或是其片段或变体。在一些实施方案中,传感器包含氨基酸序列,该氨基酸序列与选自seqidno:15、seqidno:16、seqidno:17、和seqidno:18组成的组的氨基酸序列100%相同,或是其片段或变体。在一些实施方案中,传感器包含氨基酸序列,该氨基酸序列与选自seqidno:25、seqidno:26、seqidno:27、和seqidno:28组成的组的氨基酸序列100%相同,或是其片段或变体。在这些实施方案中,乳糖与传感器分子的结合产生bret的变化。可用于检测和/或测量乳糖浓度的传感器分子特别适用于测定无乳糖产品中的残留乳糖。在乳品和乳制品(奶油、黄油、冰淇淋、干酪、奶粉)中发现高水平的乳糖(5%)(fernandessilveira等,2015)。无乳糖和减少乳糖的产品的市场正在增长,但是并没有便宜、快速、灵敏的方法来测量在处理以除去或减少乳糖的量之后乳制品中乳糖的残留量。根据澳大利亚和新西兰食品标准局(fsanz),乳糖减少的乳制品必须含有不超过0.3%的乳糖。不含乳糖的产品被定义为具有不可检测水平的二糖,这是经过解释的定义。然而,欧洲和中国市场需要低于0.01%的乳糖水平。尽管已经很好地建立了导致乳中乳糖减少/消除的酶促方法,但是对于那些进行酶促方法以验证在加工时乳糖减少的程度的人来说,还没有已建立的方法。目前,将乳糖减少和不含乳糖的乳样品从加工厂送至专门实验室进行分析。这导致额外的成本,是由于物流的原因,需要专门的实验室设备和专业知识,以及需要额外地持有被评估的货物。除了与当前分析方法和储存相关的成本之外,并非所有不含乳糖的乳都被测试,导致乳的不均匀处理和产品对消费者的潜在不可靠性。因此,需要用于测量例如乳糖减少的和无乳糖的乳制品的食品中乳糖浓度的替代方法和传感器。优选地,所述方法和传感器将为乳制品加工者提供快速、灵敏、选择性、在线的方法,用于在加工厂测量乳中的低水平乳糖。乳中残留乳糖的测量在至少两个水平上代表了一个挑战:i)经过酶处理降解乳糖后,乳中乳糖的含量约为0.01%w/v;ii)由于存在高浓度的乳糖衍生的单糖,可能会干扰乳糖的测量,因此具有选择性。优选地,在一些实施方案中描述的方法和传感器将能够检测浓度为约0约.0001%w/v或更高、约0.0003%w/v或更高、约0.0005%w/v或更高、约0.0007%w/v或更高、约0.001%w/v或更高、约0.003%w/v或更高、约0.005%w/v或更高、约0.007%w/v或更高、约0.01%w/v或更高、约0.03%w/v或更高、约0.05%w/v或更高、约0.07%w/v或更高、或约0.1%w/v或更高的乳糖。优选地,在一些实施方案中描述的方法和传感器将能够在例如乳糖衍生的单糖和/或乳果糖的其他碳水化合物存在下检测乳糖。在一些实施方案中,所述方法和传感器可在至少0.1mm、至少1mm、至少10mm、至少20mm、至少50mm、至少100mm、至少130mm、至少200mm、至少260mm碳水化合物、至少300mm或至少350mm总碳水化合物存在下检测乳糖。如本领域技术人员将理解的,总碳水化合物浓度不包括乳糖(例如,如果样品包含乳糖、半乳糖和葡萄糖,则该浓度是指葡萄糖和半乳糖的浓度)。在一些实施方案中,所述方法和传感器可在至少0.1mm、至少1mm、至少10mm、至少20mm、至少50mm、至少100mm、至少130mm、至少200mm、至少260mm碳水化合物、至少300mm或至少350mm葡萄糖和半乳糖存在下检测乳糖。在一些实施方案中,提供了用于检测乳糖的传感器分子,所述传感器包含i)螺旋-转角-螺旋转录因子的乳糖结合结构域,或碳水化合物结合结构域的变体;ii)化学发光供体结构域;以及iii)受体结构域;其中当所述碳水化合物与所述乳糖结合结构域结合时,所述化学发光供体结构域相对于所述受体结构域的空间位置和/或偶极取向改变。在一些实施方案中,提供了用于检测乳糖的传感器分子,所述传感器包含i)细菌bgar转录因子;ii)共振能量转移供体结构域;以及iii)共振能量转移受体结构域;其中当乳糖与转录因子结合时,化学发光供体结构域相对于受体结构域的空间位置和/或偶极取向改变。共振能量转移供体结构域和共振能量转移受体结构域如本文所定义。乳果糖传感器分子本文定义的传感器分子的另一非限制性实例是可用于检测和/或测量乳果糖浓度的传感器分子。因此,在一些实施方案中,本发明提供了如本文所定义的用于检测乳果糖的传感器分子。在一些实施方案中,本文所述的方法和传感器将能够检测浓度为约0.05mm或更高、约0.1mm或更高、约0.5mm或更高、约1mm或更高、约1.5mm或更高、约1.8mm或更高或约2mm或更高的乳果糖。在一些实施方案中,在一些实施方案中描述的方法和传感器将能够检测浓度为约0.1mm或更高的乳果糖。组合物,试剂盒,方法和用途本文所述的传感器可被包括在用于检测碳水化合物的组合物中。例如,本文所述的传感器可包括在用于检测糖或糖衍生物的组合物中。在一个实施方案中,本文所述的传感器可被包括在用于检测乳糖的组合物中。在一个实施方案中,本文所述的传感器可被包括在用于检测乳果糖的组合物中。在一些实施方案中,提供了包含根据本发明的传感器和可接受的载体的组合物。如本文所用,术语“可接受的载体”包括与本发明的方法和用途相容的任何和所有固体或溶剂(例如磷酸盐缓冲盐水缓冲液、水、盐水)分散介质,包衣等。在与组合物的其他成分相容的意义上,可接受的载体必须是“可接受的”,不损害被测试的碳水化合物并且不抑制碳水化合物与碳水化合物结合结构域的结合。通常,合适的可接受的载体是本领域已知的并且是基于最终用途应用来选择的。如本领域技术人员将理解的,本申请的传感器可用于检测样品中碳水化合物的存在或不存在,并且如果存在,也可用于测定样品中碳水化合物的存在量。因此,在一些实施方式中,提供了一种检测样品中的碳水化合物的方法,该方法包括:i)使样品与本发明的传感器分子接触;以及ii)确定在样品存在下化学发光供体结构域相对于受体结构域的空间位置和/或偶极方向是否已改变,其中化学发光供体结构域的空间位置和/或偶极方向相对于受体结构域的的改变表示样品中存在碳水化合物。在一些实施方案中,确定化学发光供体结构域相对于受体结构域的空间位置和/或偶极取向在样品存在下是否已经改变包括在加入样品之前和之后测量bret比率。在一些实施方案中,所述方法还包括测定样品中碳水化合物的浓度。在一些实施方案中,碳水化合物选自由乳糖和乳果糖组成的组。在一些实施方案中,碳水化合物是乳糖。在一些实施方案中,碳水化合物是乳果糖。传感器可用于检测和定量样品中的碳水化合物。“样品”可以是可能含有碳水化合物的任何物质或组合物。在一些实施方案中,样品是空气、液体、生物材料或土壤。在一些实施方案中,样品选自由以下组成的组:乳制品或其提取物,土壤或其提取物,生物材料或其提取物等。样品可以直接从环境或来源获得,或者可以在进行本发明的方法之前通过合适的程序提取和/或至少部分纯化。在一些实例中,样品包含生物材料。如本文所用,“生物材料”广义地定义且包括全部或部分衍生自生物体的任何材料。生物材料包括但不限于体液、细胞、软组织(如结缔组织和非结缔组织)和硬组织(如骨和软骨)。在一些实施方案中,体液是血液、血清、痰、粘液、脓、腹膜液、尿或其他体液。在一些实施方案中,这样的材料可以从活生物体中收获,然后进行进一步的加工和/或化学处理。在一个实施例中,传感器不用于检测活细胞内的碳水化合物。在一些实施例中,传感器离体使用。在一些实施方案中,样品包含乳制品。如本文所用,术语“乳制品”包括乳品和部分或全部衍生自乳品的产品。乳品可以获自任何哺乳动物,例如牛、绵羊、山羊、马、骆驼、水牛、人等。乳制品包括但不限于,原料乳、低脂乳、脱脂乳、巴氏灭菌乳、延长保质期乳、uht乳、乳糖改性uht乳、强化uht乳、调味uht乳和这些产品的组合,以及uht婴儿配方、乳酪、酸奶、乳清、酪乳、奶油、奶粉、粉状婴儿配方、冰淇淋和奶油等。在一些实例中,样品是乳或稀释乳。乳制品还可以是乳制品的提取物,例如部分纯化的部分,其包含或疑似包含感兴趣的碳水化合物。在一些实施方案中,本发明的传感器可用于检测乳制品中的乳糖。因此,提供了一种检测乳制品中乳糖的方法,该方法包括:i)使样品与本发明的传感器分子接触;以及ii)确定在样品存在下化学发光供体结构域相对于受体结构域的空间位置和/或偶极方向是否已改变,其中化学发光供体结构域的空间位置和/或偶极方向相对于受体结构域的的改变表示样品中存在乳糖。本发明的传感器还可用于监测样品中碳水化合物的浓度。在一些实施方案中,本发明的传感器可用于检测乳制品中的乳果糖。因此,提供了一种检测乳制品中乳果糖的方法,该方法包括:i)使样品与本发明的传感器分子接触;以及ii)确定在样品存在下化学发光供体结构域相对于受体结构域的空间位置和/或偶极方向是否已改变,其中化学发光供体结构域的空间位置和/或偶极方向相对于受体结构域的的改变表示样品中乳果糖。在一些实施方案中,所述方法还包括测定样品中乳果糖的浓度。乳果糖已经由国际乳业联盟和欧洲联盟提出作为热处理引起的乳损害的指标并且作为区分巴氏灭菌乳([乳果糖]<10μm)、超高温(uht)处理乳([乳果糖]<1.8mm)和容器内消毒乳([乳果糖]>1.8mm)的标准(marconi等,2004;montilla等,1996)。在一些实施方案中,本发明的传感器可用于监测样品中乳果糖的浓度。在一些实施例中,本发明的传感器可用于提供由热处理引起的乳损坏的指示。在一些实施例中,本发明的传感器可用于区分各种形式的乳,例如巴氏杀菌乳、超高温(uht)处理乳和容器内消毒乳。在一些实施方案中,还提供了传感器分子用于检测碳水化合物的用途,所述传感器分子包含:i)螺旋-转角-螺旋转录因子的碳水化合物结合结构域,或所述碳水化合物结合结构域的变体;ii)化学发光供体结构域;以及iii)受体结构域;其中当所述碳水化合物结合至所述碳水化合物结合结构域时,所述化学发光供体结构域相对于所述受体结构域的空间位置和/或偶极取向改变。在一些实施方案中,所述用途还包括测定样品中碳水化合物的浓度。另一方面提供了一种用于检测乳糖的传感器分子,该传感器分子包含共价连接至共振能量转移供体结构域和共振能量转移受体结构域的细菌bgar转录因子或其变体,其中当乳糖结合至转录因子时,所述供体结构域相对于所述受体结构域的空间位置和/或偶极取向改变。在一些实施方案中,所述用途还包括测定样品中乳糖的浓度。在一些实施方案中,还提供了一种用于检测乳果糖的传感器分子,该传感器分子包含共价连接至共振能量转移供体结构域和共振能量转移受体结构域的细菌bgar转录因子或其变体,其中当乳果糖结合至转录因子时,所述供体结构域相对于所述受体结构域的空间位置和/或偶极取向改变。在一些实施方案中,所述方法还包括测定样品中乳果糖的浓度。如本领域技术人员所知,本发明的传感器也可以是多路复用的。在该系统中,提供了检测不同碳水化合物的两种或多种不同的传感器分子。例如,检测乳糖的本发明的传感器分子可以与检测诸如乳果糖、半乳糖和/或葡萄糖的其他碳水化合物的传感器复用。在一些实施方案中,每个不同的传感器分子可以包括不同的供体和/或受体分子,使得它们以不同的波长发射,从而能够检测和定量不同的靶化合物。在一些实施方案中,每个不同的传感器分子可以包括相同的供体和/或受体分子。在一些实施方案中,使用单个流体检测室。在一些实施方案中,可以使用多通道检测设备。在一些实施方案中,样品是水性液体。例如,样品包括但不限于乳、果汁、其他饮料和包括血清的体液。本发明的方法可以在适于测量可检测的变化的任何系统上进行。如本领域技术人员将理解的,本发明的方法可以分批(例如使用平板读数器的分批形式)或流动形式进行。例如,本发明的方法可以使用装备有适当过滤器的微孔板读数器以微孔板形式进行。本发明的方法还可以在微流体装置上进行,例如wo2013/155553中所述。另一方面,本发明提供一种包括本文所述传感器的试剂盒。在一些实施方案中,试剂盒还包含标准品(例如乳糖和/或乳果糖标准品)。实施例实施例1-lacb1和lacf1传感器的构建通过genscript(美国)合成编码bgar(为在大肠杆菌中表达而优化的密码子)的dna构建体。如果需要,使用genscript合成的bgar作为模板和适当的引物,通过pcr将接头序列添加到dna构建体中(表5)。引物包括接头序列(如果需要)和psti或bstbi的限制性位点。扩增的pcr产物用psti和bstbi消化并克隆到prset载体(澳大利亚biolabs)中,使用psti/bstbi限制性位点预先用gfp2和rluc8(对于lacb传感器而言)或cfp和yfp(对于lacf传感器而言)克隆,使得表达的融合蛋白具有n-末端组氨酸标签。表5.用于制备乳糖传感器的寡核苷酸取向序列p1正向aaaaaactgcagatgcagattctgtg(seqidno:10)p2反向acacacttcgaaaatgctcggtttat(seqidno:11)p3正向aaaaaactgcagggtggtaccggaggcggcatgcagattctgtggaaaaa(seqidno:12)p4反向aaaaaattcgaagccgcctccggtaccaccaatgctcggtttattaactt(seqidno:13)p5反向aaaaaactgcagaatgctcggtttat(seqidno:14)用编码传感器的prset载体转化大肠杆菌菌株bl21(de3)(新英格兰生物实验室)的细胞。使用本领域技术人员已知的协议在大肠杆菌菌株bl21(de3)中表达传感器。为了表达传感器,将补充有2%(v/v)葡萄糖和100μg/ml氨苄青霉素的50mllb(10g胰蛋白胨,5g酵母提取物,5gnacl/l水(ph7.4))用单个菌落接种,并在37℃,200rpm下温育,直到其达到0.8的abs600nm。将补充有100μg/ml氨苄青霉素的250mllb使用起始培养物接种至0.05的abs600nm,并且在28℃,200rpm下温育16小时并且收获细胞。或者,将含有100μg/ml氨苄青霉素的200mllb用单个菌落接种,并将培养物在28℃温育48小时,同时以200rpm振荡,并收获细胞。通过在5000×g(4℃)离心10分钟收获细胞,用磷酸盐缓冲盐水(pbs;137mmnacl,2.7mmkcl,10mmna2hpo4,1.8mmkh2po4,ph7.4)并重悬于磷酸钠缓冲液(50mmna2hpo4,300mmnacl,ph7.0)中。使细胞悬浮液在≈20,000psi的压力下通过均化器(microfluidicsm-110p(美国马萨诸塞州牛顿市)),并通过在15,000×g(4℃)下离心15分钟分离可溶性蛋白质级分。根据制造商的说明书,使用钴亲和层析(超低金属亲和树脂(澳大利亚的takara出品公司))纯化蛋白质。用150mm咪唑洗脱纯化的蛋白质后,使用透析单元(gehealthcare,vivaspin6,10kdamwco)用三羟甲基氨基甲烷(tris)缓冲液(50mmtris,100mmnacl,0.1mmedta,ph8.0)透析样品。将500μl的纯化蛋白质的等分试样在液氮中速冻并且在-80℃储存。通过280nm处的吸光度测定蛋白质浓度。纯化的lacb1传感器是具有序列seqidno:15的多肽。lacb1传感器含有gfp2-bgar-rluc8。纯化的lacf1传感器是具有序列seqidno:23的多肽。lacf1传感器含有cfp-bgar-yfp。组氨酸(his)标记的lacb1和lacf1传感器的示意图如图2所示。实施例2-通过lacb1和lacf1的乳糖结合材料和方法bret测定在最终体积为100μl的96孔板中进行。使用pbs(58mmna2h2po4、17mmnah2po4、68mmnacl,ph7.4)将纯化的传感器和乳糖稀释至所需浓度。lacb1传感器用1mm乳糖或水在30℃下温育5分钟。在温育时间结束时,添加5μl的在etoh中的腔肠素400a(至17μm的最终腔肠素400a浓度),并且立即记录光谱扫描。fret测量以类似于bret2测定的方式进行,具有以下修改。lacf1传感器与1mm乳糖在30℃下温育5分钟。以荧光模式记录光谱扫描(λex=435nm,455nm截止,20nm增量)。用spectramaxm3读板分光荧光计(美谷分子仪器(moleculardevices))以发光模式(20nm增量)在白色96孔板(opti-platetm-96,珀金埃尔默)中记录光谱扫描。数据分析bret2比率计算为500nm处的受体发射强度与420nm处的供体发射强度的比率。fret比率计算为520nm处的受体发射强度与480nm处的供体发射强度的比率。结果在水或1mm乳糖存在下测量lacb1传感器和lacf1传感器的ret比率(图2)。对于两种传感器,1mm乳糖的存在导致ret比率降低。对于lacb1,变化为从1.09±0.01至0.800±0.003。对于lacf1,变化为从1.216±0.004变为1.089±0.004。因此,1mm乳糖引起lacb1传感器的ret比下降27%,而lacf1传感器下降10%。实施例3-通过lacb1检测乳糖结合的bret测定材料和方法bret测定在最终体积为100μl的96孔板中进行。使用pbs(58mmna2h2po4、17mmnah2po4、68mmnacl,ph7.4)将纯化的传感器和乳糖稀释至所需浓度。将纯化的传感器(1μm)用不同量的乳糖(0.000036%-0.36%w/v)或其他碳水化合物在30℃温育30分钟。对于bret测量,在温育期后添加5μl腔肠素400a底物(最终[coel400a]=16.7μm))。加入底物后立即记录光谱扫描。用spectramaxm2读板分光荧光计(美谷分子仪器)记录光谱扫描。数据分析bret2比率计算为最大受体发射强度(500nm)与最大供体发射强度(420nm)的比率。结果在乳糖量增加的情况下,lacb1传感器的bret2比率如图3所示。实施例4-接头长度对乳糖结合的影响材料和方法为了研究改变连接碳水化合物结合结构域与化学发光供体结构域和/或受体结构域的接头长度的影响,构建了乳糖传感器,其中接头序列-ggtggg-被包含在bgar前后(lacb2;seqidno:16),bgar前(lacb3;seqidno:17)和bgar后(lacb4;seqidno:18)。lacb2传感器包含rluc8-ggtggg-bgar-ggtggg-gfp2。lacb3传感器包含rluc8-ggtggg-bgar-gfp2。lacb4传感器包含rluc8-bgar-ggtggg-gfp2。乳糖传感器的示意图如图4所示。与lacb1的相同区域相比,接头位置用黑色部分表示。使用实施例3中描述的bret测定评估这些传感器与1mm乳糖的结合。结果在1mm乳糖存在和不存在下lacb2、lacb3和lacb4传感器的bret2比率如图5所示。包括在存在和不存在1mm乳糖的情况下对于lacb1传感器的bret2比率作为比较。尽管所有传感器在1mm乳糖存在下bret2比率都发生了变化,但在1mm乳糖存在下lacb1(无接头)给出了最显着的bret2比值变化。实施例5-lacb1和lacf1传感器的灵敏度用于产生无乳糖和乳糖减少的乳的方法使用β半乳糖苷酶(也称为乳糖酶)将二糖乳糖分解成其两组分单糖,半乳糖和葡萄糖。半乳糖和葡萄糖以高浓度保留在最终乳制品中。由于它们与乳糖的结构相似性,半乳糖和葡萄糖具有竞争性结合乳糖生物传感器的潜力,干扰痕量乳糖的测量。此外,对乳进行热处理以产生长寿命产品(例如uht或奶粉,但不是巴氏灭菌的“新鲜”乳)导致二糖乳糖部分异构化为乳果糖。在uht乳中乳果糖的水平通常达到0.32-2.16mm(0.011-0.074%(w/v))(morales等,2000;marconi等,2004)。乳果糖不通过β-半乳糖苷酶处理水解成其组分单糖。与单糖半乳糖和葡萄糖类似,热处理乳中乳果糖的存在可能干扰乳制品中低水平乳糖的测定。材料和方法为了确定lacb1传感器是否具有足够的特异性和灵敏度以避免例如半乳糖、葡萄糖和乳果糖的糖的干扰,使用实施例2和3中描述的bret测定评估lacb1传感器结合一系列二糖以及单糖半乳糖和葡萄糖的能力,所述二糖在结构上与乳糖(β-d-半乳糖基-(1→4)-d-葡萄糖)相关的二糖,即乳果糖(4-o-β-d-半乳糖基-d-果糖)、蜜二糖(d-半乳糖基-α(1→6)-d-葡萄糖)、麦芽糖(4-o-α-d-葡萄糖基-d-葡萄糖)、纤维二糖(4-o-β-d-葡萄糖基-d-葡萄糖)、海藻糖(α-d-葡萄糖基-(1→1)-α-d-葡萄糖)和蔗糖(β-d-果糖基α-d-葡萄糖)简言之,lacb1传感器分别与0.1mm或1mm乳糖、1mm乳果糖、蜜二糖、麦芽糖、纤维二糖、海藻糖或蔗糖或1mm或10mm半乳糖或葡萄糖温育。接着,按照实施例3中详述的方案产生在葡萄糖和半乳糖存在下lacb1和lacf1传感器的“校正”校准曲线,然而,为了模拟在磷酸盐缓冲液(pbs)中1比10稀释的处理过的奶,包括葡萄糖和半乳糖,其浓度使得[乳糖]+0.5×[半乳糖]+0.5×[葡萄糖]=13.9mm且[半乳糖]=[葡萄糖]。例如,如果在乳糖酶处理之前在乳中发现139mm乳糖,在乳糖酶处理至1mm的残余[乳糖]之后,半乳糖和葡萄糖将以138mm的浓度存在。在这种1比10稀释在pbs中的乳糖酶处理的乳样品情况下,稀释的样品将含有0.1mm残余乳糖和13.8mm半乳糖和葡萄糖。因此,当反应混合物含有0.1mm乳糖时,也加入13.8mm葡萄糖和13.8mm半乳糖。还构建了lacb1和lacf1传感器在葡萄糖和半乳糖存在下的“校正”校准曲线,使用无乳糖全脂乳作为稀释剂,该无乳糖全脂乳经透析除去低分子量组分,特别是乳糖、半乳糖和葡萄糖。为了除去,根据以下方案透析全奶油无乳糖乳。在4℃下,将10ml的无乳糖全脂乳用1000mlpbs透析两次,持续24h,以除去存在于无乳糖乳中的任何残留乳糖。将1ml透析的乳的等分试样冷冻在干冰上并且在-80℃储存。透析后的乳用作“乳基质”,在pbs中以1比10稀释。结果与讨论在乳糖、乳果糖、蜜二糖、麦芽糖、纤维二糖、海藻糖、蔗糖、半乳糖和葡萄糖存在30分钟时,lacb1传感器bret2比率的变化如图6所示。lacb1传感器与1mm(0.034%w/v)二糖乳果糖温育导致bret2比率变化约21%,而其他二糖导致bret2比率变化3-10%。相比之下,添加1mm(0.034%)乳糖时bret2比率的变化为约35%。这表明lacb1传感器对乳糖的选择性优于其他糖类。lacb1传感器与1和10mm(0.018%和0.18%)半乳糖或葡萄糖温育导致bret2比率随半乳糖变化3%和13%,随葡萄糖变化9%和6%。当在添加腔肠素400a之前在乳糖、乳果糖、蜜二糖、麦芽糖、纤维二糖、海藻糖、蔗糖、半乳糖和葡萄糖的存在下温育lacb1传感器5分钟时,获得类似的结果(图7)。图8显示了pbs中lacb1和lacf1传感器和pbs中10%透析乳的校正校准曲线。图8a和8b显示pbs中校正的校准曲线。图8c、8d和8e显示了在pbs中的10%透析乳中的校正校准曲线。校正的校准曲线在至少2log单位上是线性的以允许乳糖定量。具有较高乳糖含量的样品可以使用相同的方法通过用缓冲液10:90稀释样品来分析(参见图8)。实施例6-lacb1与乳糖和乳果糖的结合在实施例5中测试的糖中,乳糖引起bret2比率的最大变化,随后是乳果糖。由于lacb1对乳糖和乳果糖表现出最大的响应,进一步研究了生物传感器对每种糖的亲和力。材料和方法用spectramaxm3读板分光荧光计(美谷分子仪器(moleculardevices))以发光模式(20nm增量)在白色96孔板(opti-platetm-96,珀金埃尔默)中记录光谱扫描。将1μm的纯化蛋白质用于bret测定,最终体积为100μl,其中蛋白质和分析物在磷酸盐缓冲盐水(pbs;10mm磷酸盐,137mmnacl,2.7mmkcl,ph7.3)或10%(v/v)透析的无乳糖全奶油乳在pbs中的溶液。纯化的蛋白质与乳糖或乳果糖在30℃温育5分钟℃。在温育时间结束时,添加5μl的在etoh中的腔肠素400a(至17μm的最终腔肠素400a浓度),并且立即记录光谱扫描。数据分析bret2比率计算为500nm处的受体发射强度与420nm处的供体发射强度的比率。结果与讨论在乳糖或乳果糖存在下,lacb1传感器bret2比率的变化如图9所示。lacb1对乳糖和乳果糖(在pbs中)的响应是浓度依赖性的。lacb1对乳糖的响应在几乎3个对数单位上是准线性的,ec50为12±1μm,检测极限为1μm。lacb1对乳果糖的亲和力大约弱150倍,ec50为2.4±0.2mm。乳果糖的检测极限为0.1mm,即比乳糖高100倍。乳果糖响应在几乎2个对数单位上是准线性的。lacb1对乳果糖(0.1mm)的检测极限比巴氏灭菌乳(10μm)中发现的乳果糖水平高10倍,这意味着其可用于测定乳糖酶处理的巴氏灭菌乳中乳糖的任何相关水平。lacb1传感器对10%(v/v)透析乳中乳糖的响应是浓度依赖性的,ec50为21±2μm,线性超过几乎3个对数单位,检测极限为1μm。在10%(v/v)透析乳和饱和浓度的半乳糖和葡萄糖中lacb1对乳糖的敏感性在统计学上不同于仅在pbs中观察到的敏感性(11-14μm和18-23μm)。然而,由于存在10%(v/v)透析的奶或高浓度的葡萄糖和半乳糖,lacb1对乳糖的亲和力没有显著降低。不希望受理论束缚,认为这是由于传感器对乳糖的高选择性和/或由于bret2转导机制在复杂培养基中的效率。lacb1与乳糖和乳果糖结合的特征突出了使用结合蛋白作为生物传感的分析物识别元件的内在能力,特别是当与bret2转导机制偶联以检测变化时。用于构建lacb1的乳糖结合转录调节因子bgar在低微摩尔范围内产生敏感性,具有区分结构相关二糖的能力,如乳糖和测试的第二种最有效的糖乳果糖之间观察到的ec50的200倍差异所证明的。实施例7-在模拟乳系统中lacb1与乳糖的结合为了研究在模拟乳系统中测量乳糖浓度的效果,使用透析的乳样品,其中当乳糖浓度从13.9mm(相当于未改性的10%(v/v)全脂乳)降低到零时,通过添加补偿量的葡萄糖和半乳糖来保持糖的总浓度恒定。材料和方法将全奶油乳用水透析以除去小分子。简言之,将20ml全奶油无乳糖乳在d-tubetm透析器(merck,3.5kda截流分子量(mwco))中在4℃下用1l水透析90分钟两次。将1ml透析的乳的等分试样冷冻在干冰上并且在-80℃储存。透析的乳用于以精确限定水平的乳糖、半乳糖和葡萄糖的范围重建10%(v/v)乳基质,其中([乳糖]+[半乳糖+葡萄糖]/2=13.9mm。当测量样品中的乳糖处于或低于300μm‘无乳糖'阈值时,即在乳糖酶处理之后,选择10倍稀释因子以精确地模拟测定条件。如实施例6所述进行bret测定。结果与讨论模拟乳系统中lacb1传感器bret2比率的变化如图9所示。在这些与乳样品中的情况非常相似的条件下,乳糖的lod为0.2μm(0.00003%w/v)。在这些条件下,乳糖的ec50从12μm变化至21μm,但差异没有统计学差异。乳糖的ec50比“无乳糖”乳制品的最严格客观调节标准(0.01%w/v)低约15倍。在存在或不存在10%(w/v)全奶油乳的情况下对数浓度-响应函数的相似性是显著的,因为在后一种情况下,在较低浓度的乳糖下,在13.9mm葡萄糖和半乳糖的存在下进行测量。不希望受理论的束缚,认为生物传感器“忽略”潜在干扰物质的强能力源自传感器的选择性,bret2转导机制的稳健的比率测定性质和/或不存在外部照明源,这将导致光散射并增加例如乳的混浊介质中的噪声,即使稀释十倍时也是如此。实施例8–乳糖的测定材料和方法将lacb1在测定缓冲液(磷酸盐缓冲液中的0.45%明胶:淡水鱼皮中的0.45%(w/v)明胶(sigmaaldrich)),58mmna2h2po4、17mmnah2po4,68mmnacl,ph7.4中)稀释至1200μm。将35μl分析物(在测定缓冲液中的30μm或3mm乳糖或单独的测定缓冲液),lacb1(1200μm)和腔肠素400a(在15%etoh/测定缓冲液中的30μm)放置在微流控芯片的三个入口中的每一个中,并且以1200μl/h的流速进行测定100秒。使用装置以1200μl/h的流速记录bret比率并且供体和受体发光强度平均在80秒-100秒。bret2比率由装置软件程序计算为最大受体发射强度(绿色滤光片)与最大供体发射强度(蓝色滤光片)的比率。结果图10显示了具有3mm乳糖的测定缓冲液的示例装置迹线。如图11所示,测定可以用于检测30μm和3mm两者下的乳糖。添加30μm和3mm乳糖分别导致bret2比率的大约22%和41%的变化。实施例9-全脂乳中乳糖的估计设计传感器以在受控实验室条件下定量确定的缓冲液中的分析物本身是一个挑战,但是在真实世界条件下精确定量分析物甚至更具有挑战性。特别地,复杂和干扰的样品基质,例如乳、乳品和其他生物样品,可能使生物传感器性能复杂化和劣化。本文所述的传感器的一种用途是定量未改性乳中乳糖水平,取决于种类,其范围为约4.5-7.0%(w/v)。本发明人比较了使用本文所述的传感器获得的乳糖估计值,相对于pbs中已知的乳糖量或10%的透析乳基质已通过目前商业上使用的两种方法进行了校正,这两种方法是偶联酶乳糖测定试剂盒(biovision)和在nata认可的分析实验室中进行的带折射率检测器的hplc。材料和方法使用市售试剂盒(biovision乳糖比色/荧光测定试剂盒(美国旧金山,#k624-100)),通过具有折射率(ri)检测的hplc和使用本文所述的lacb1传感器来估计全脂乳中乳糖的浓度。用于这些测定的样品是购自超市的全巴氏消毒牛乳。牛乳纸盒上的营养板表明乳糖的代表值为137mm。根据制造商的说明书使用biovision乳糖比色/荧光测定试剂盒来估计乳糖和半乳糖浓度。简言之,使用0、2、4、6、8或10μl的乳糖标准物(在所提供的乳糖测定缓冲液中1mm)制备标准曲线。将所需体积吸移到透明96孔板(格瑞纳(greiner)的uv-starmicroplate)的微量孔中。将购买的全巴氏消毒牛乳在水中稀释约104倍并且将10μl用于该测定。此外,向各孔中加入2μl的显色探针,2μl的酶混合物和2μl的辣根氧化酶(hrp),用乳糖测定缓冲液将体积补足至100μl并充分混合。将反应混合物在37℃温育60分钟并避光。abs570用spectramaxm3读板分光荧光计(moleculardevices)以吸收模式记录abs570nm(终点测量abs570nm)。测定一式三份进行。通过商业实验室进行乳糖浓度的hplc估计。简言之,将200ml全脂乳冷冻并储存在-80℃下并在干冰上运送至商业nata(澳大利亚国家检测机构协会)-认可的测试实验室(澳大利亚墨尔本的dts/asurequality)。根据实验室的标准商业方案,使用hplc和ri检测进行分析(chaves-services等,2004;southgate,1969)。结果以g糖/100ml乳报告,使用1.033g/ml作为全奶油全脂乳的密度。没有报告误差值。使用lacb1传感器进行乳糖浓度的估计,如本文所述,相对于pbs中已知量的乳糖或10%透析的乳基质进行校准。lacb1对乳糖的ec50为12μm,即比在未改性牛乳中发现的乳糖浓度低约104倍。因此,在乳糖估计之前将全乳样品在水中稀释3200倍。结果使用lacb1传感器,biovision试剂盒和hplc测定的巴氏灭菌全牛乳的乳糖浓度列于表6中。使用biovision偶联酶试剂盒并按照制造商的方案,发明人估计全乳样品的乳糖浓度为129±1mm。将相同乳的样品置于nata认可的实验室中,通过hplc/折射率(ri)分析进行乳糖估计。实验室报道乳糖浓度为134mm。在这种情况下,没有报告误差值。使用本文所述的lacb1传感器,发明人估计全乳样品中的乳糖浓度为157±6mm。表6.用lacb1传感器和两种独立方法测定巴氏灭菌全牛乳乳糖浓度的比较。[乳糖](mm)[乳糖](%w/v)lacb1传感器157±65.4±0.2偶联酶分析(biovision)129±14.4±0.03hplc用ri检测*134*4.6实施例10-乳糖酶处理乳中乳糖的估计乳糖生物传感器的另一用途是测量不同等级的乳糖酶处理的乳中的乳糖,其特征为“减少的乳糖”或“不含乳糖”。乳糖酶处理的乳中乳糖的估计是有挑战性的,因为分析物的水平低,乳培养基的复杂性,以及可能干扰乳糖本身测量的高水平葡萄糖和半乳糖的存在。澳大利亚和新西兰食品标准(fsanz)规定乳糖减少的乳制品含有不超过0.3%(8.8mm)的乳糖,而不含乳糖的产品应当含有“不可检测的乳糖”,这是主观的方法依赖性定义。欧洲当局规定无乳糖食品的目标阈值为0.01%(w/v)(0.3mm)。乳是一种复合基质,其包含各自浓度为约3%(w/v)的蛋白质和脂质(kailasopathy,2009)。为了最小化干扰,分析实验室通常在通过hplc或比色偶联酶测定分析糖含量之前从乳样品中沉淀脂肪和蛋白质。除了耗时和导致额外成本之外,在分析之前对样品进行处理增加了由于产率变化和样品体积的改变而导致的误差风险。需要一种改进的测定样品中例如乳糖的碳水化合物浓度的方法,该方法避免了与hplc或比色偶联酶测定有关的至少一些缺点。材料和方法使用市售试剂盒(biovision乳糖比色/荧光测定试剂盒(美国旧金山,#k624-100)),通过具有折射率(ri)检测的hplc和使用如实施例9所述的lacb1传感器,估计商业获得的全奶油“无乳糖”牛乳中乳糖的浓度。在这些试验中使用的样品是商业上获得的全奶油“无乳糖”牛乳。“无乳糖”牛乳的10倍稀释液用于lacb1测定。还使用市售试剂盒(biovision乳糖比色/荧光测定试剂盒(美国旧金山,#k624-100))和使用标准方案通过具有折射率(ri)检测的hplc测定样品中半乳糖的浓度。结果lacb1传感器用于估计市售全奶油“无乳糖”牛乳的10倍稀释液中的乳糖浓度。bret2比率降低16%,相当于2.7±0.1m的浓度,对应于原始乳样品中27±1μm乳糖(表7)。使用实施例9中描述的biovision乳糖比色/荧光测定试剂盒估计全奶油“无乳糖”牛乳的乳糖浓度的尝试是不成功的。认为这是“无乳糖”牛乳中存在高浓度半乳糖的结果。样品中半乳糖的浓度估计为163±2mm。将相同的全奶油无乳糖牛乳的样品置于nata认可的分析实验室,通过hplc-折射率检测进行分析。没有检测到乳糖,检测极限为0.1%(w/v)或约3mm(表7)。表7.用lacb1传感器和两种独立方法比较新鲜“无乳糖”全奶油牛乳中乳糖的浓度。*没有引用错误因此,lacb1传感器似乎适于直接测定无乳糖市售乳制品中残留乳糖的浓度。本申请要求2017年8月8日提交的澳大利亚申请号2017903148的优先权,其全部内容通过引用并入本文。本领域技术人员应当理解,在不脱离广泛描述的本发明的精神或范围的情况下,可以对具体实施例中所示的本发明进行多种变化和/或修改。因此,本实施例在所有方面都被认为是说明性的而非限制性的。本文讨论和/或引用的所有出版物均全文引入本文。包括在本说明书中的文件、动作、材料、装置、物品等的任何讨论仅仅是为了提供本发明的上下文。不应视为承认这些内容中的任何或全部形成现有技术基础的一部分或者是与本发明相关的领域中的公知常识,因为其存在于本申请的每个权利要求的优先权日之前。参考文献ansarietal.(2012)processbiochem.47:2427–2433.aravindandanantharaman(2003)femsmicrobiol.rev.222:17-23.aravindetal.(2005)femsmicrobiol.rev.29:231-262.altschuletal.(1990)j.mol.biol.215:403-10.altschuletal.(1997)nucleicacidsres.25:3389-402.bajaretal.(2016)sensors.16:1488-1512.chávez-servi′netal.(2004)j.ofchromatogr.a.1043:211-215.dacresetal.(2009a)anal.biochem.385:194-202.dacresetal.(2009b)biosensorsandbioelectronics24:1164-1170.dacresetal.(2010)anal.chem.82:432-435.dacresetal.(2011)biosens.bioelectron.29:119-124.dacresetal.(2012)biochem.biophys.res.commun.425:625-629.dacresetal.(2014)posterpresentation.biosensors2014,may27-30th,melbourne.dayetal.(2004)luminescence19:8-20.dewetetal.(1987)mol.cell.biol.2987:725-737.euberandbrunner(1979)j.dairysci.62:685–690.erichetal.(2012)foodchem.135:2393–2396.fernandessilveiraetal.(2015)j.chem.id185967,6pages.(1948)ann.physik.2:55.(1959)discuss.faradaysoc.27:7–17.(1960)rad.res.suppl.2:326.francoetal.(2006)j.bacteriol.188:3024–3036.francoetal.(2007)nucleicacidsres.35:4755–4766.greerandszalay(2002)luminescence17:43-74.hartmanetal.(2011)appl.environ.microbiol.77:471-8.haydonandguest(1991)femsmicrobiol.lett.63:291-295.hastings(1996)gene173:5-11.hushpulianetal.(2007)biotransformation25:2–4.indyketal.(1996)foodchem.57:575–580.inouyeetal.(1997)biochem.j.233:349–353.jiaetal.(2014)biotechnol.bioeng.111:209–222.kailasapathy,k.(2009)“chemicalcomposition,physicalandfunctionalpropertiesofmilkandmilkingredients;dairyprocessing&qualityassurance”,wiley-blackwell,chapter4.kimandkim(2012)theranostics2:127-138.kleyn(1985)j.dairy.sci.68:2791–2798.kongetal.(2009)nucleicacidsres.37:1915-1924.leeetal.(2003)j.bacteriol.185:4315–4325.loeningetal.(2006)proteineng.des.sel.19:391-400.loeningetal.(2007)naturemethods4:641-643.lorenzetal.(1991)proc.natl.acad.sci.usa88:4438-4442.marchler-baueretal.(2017),nucleicacidsres.45:d200-3.marchler-baueretal.(2015),nucleicacidsres.43:d222-6.marchler-baueretal.(2011),nucleicacidsres.39:d225-9.marchler-bauerandbryant(2004),nucleicacidsres.32:w327-331.marconietal.(2004)foodchem.84:447-450.mcsweeneyetal.(1993)foodbiotechnology.7:143-158.milkandmilkproducts–determinationoflactosecontentbyhighperformanceliquidchromatography(referencemethod)–iso22622:2007.montillaetal.(1996)j.foodprot.59:1061-1064.moralesetal.(2000)int.j.foodsci.tech.35:193-200.myersandmiller(1988),comput.appl.biosci.4:11-7.needlemanandwunsch(1970)j.mol.biol.48:443–53.paboandsauer(1992)annu.rev.biochem.61:1053-95.pflegerandeidne(2006)naturemethods3:165-174.rigalietal.(2002)j.biol.chem.15:12507-12515.rigalietal.(2004)nuc.acidsres.32:3418-3426.southgate(1969)j.sci.food.agric.20:326.tsenkovaetal.(1999).j.dairysci.82:2344-2351.tsien(1998)ann.rev.biochem.63:509-544.verhaegenetal.(2002)anal.chem.74:4378-4385.viviani(2002)cell.mol.lifesci.59:1833-1850.wangetal.(1997)“bioluminescenceandchemiluminescence:molecularreportingwithphotons",wiley,419–422.wiethausetal.(2008)j.bacteriol.190:487–93.xinminetal.(2008)j.foodcompos.anal.21:255–258.xuetal.(1999)proc.natl.acad.sci.usa.96:151-156.zhangetal.(2012)j.bacteriol.194:1055–64.zhengetal.(2009)actacrystallogr.dbiol.crystallogr.,d65:356-365.序列表<110>联邦科学技术研究组织<120>碳水化合物传感器<130>524959<150>au2017903148<151>2017-08-08<160>74<170>patentin3.5版<210>1<211>279<212>prt<213>人工序列<220><223>bgar<400>1metglnileleutrplyslystyrvallysgluasnpheglumetasn151015valaspglucysglyilegluglnglyileproglyleuglytyrasn202530tyrgluvalleulysasnalavalilehistyrvalthrlysglytyr354045glythrphelyspheasnglylysvaltyrasnleulysglnglyasp505560ilepheileleuleulysglymetglnvalglutyrvalalaserile65707580aspaspprotrpglutyrtyrtrpileglypheserglyserasnala859095asnglutyrleuasnargthrserilethrasnsercysvalalaasn100105110cysglugluasnserlysileproglnileileleuasnmetcysglu115120125ileserlysthrtyrasnproserargseraspaspileleuleuleu130135140lysgluleutyrserleuleutyralaleuilegluglupheprolys145150155160propheglutyrlysasplysgluleuhisthrtyrileglnaspala165170175leuasnpheileasnserasntyrmethisserilethrvalglnglu180185190ilealaasptyrvalasnleuserargsertyrleutyrlysmetphe195200205ilelysasnleuglyileserproglnargtyrleuileasnleuarg210215220mettyrlysalathrleuleuleulysserthrlysleuproilegly225230235240gluvalalaserservalglytyrseraspserleuleupheserlys245250255thrpheserlyshisphesermetserproleuasntyrargasnasn260265270glnvalasnlysproserile275<210>2<211>6<212>prt<213>人工序列<220><223>接头序列<400>2glyserserglyglyser15<210>3<211>6<212>prt<213>人工序列<220><223>接头序列<400>3glyglyserglyglyser15<210>4<211>6<212>prt<213>人工序列<220><223>接头序列<400>4glyglythrglyglygly15<210>5<211>6<212>prt<213>人工序列<220><223>接头序列<400>5glyglyglyglyglythr15<210>6<211>8<212>prt<213>人工序列<220><223>接头序列<400>6leuglnglyglythrglyglygly15<210>7<211>8<212>prt<213>人工序列<220><223>接头序列<400>7phegluglyglythrglyglygly15<210>8<211>7<212>prt<213>人工序列<220><223>接头序列<400>8glyglyserglyglyserleu15<210>9<211>157<212>prt<213>人工序列<220><223>bgar假定cbd<400>9metglnileleutrplyslystyrvallysgluasnpheglumetasn151015valaspglucysglyilegluglnglyileproglyleuglytyrasn202530tyrgluvalleulysasnalavalilehistyrvalthrlysglytyr354045glythrphelyspheasnglylysvaltyrasnleulysglnglyasp505560ilepheileleuleulysglymetglnvalglutyrvalalaserile65707580aspaspprotrpglutyrtyrtrpileglypheserglyserasnala859095asnglutyrleuasnargthrserilethrasnsercysvalalaasn100105110cysglugluasnserlysileproglnileileleuasnmetcysglu115120125ileserlysthrtyrasnproserargseraspaspileleuleuleu130135140lysgluleutyrserleuleutyralaleuilegluglu145150155<210>10<211>26<212>dna<213>人工序列<220><223>引物<400>10aaaaaactgcagatgcagattctgtg26<210>11<211>26<212>dna<213>人工序列<220><223>引物<400>11acacacttcgaaaatgctcggtttat26<210>12<211>50<212>dna<213>人工序列<220><223>引物<400>12aaaaaactgcagggtggtaccggaggcggcatgcagattctgtggaaaaa50<210>13<211>50<212>dna<213>人工序列<220><223>引物<400>13aaaaaattcgaagccgcctccggtaccaccaatgctcggtttattaactt50<210>14<211>26<212>dna<213>人工序列<220><223>引物<400>14aaaaaactgcagaatgctcggtttat26<210>15<211>833<212>prt<213>人工序列<220><223>lacb1<400>15metvalserlysglyglugluleuphethrglyvalvalproileleu151015valgluleuaspglyaspvalasnglyhislyspheservalsergly202530gluglygluglyaspalathrtyrglylysleuthrleulyspheile354045cysthrthrglylysleuprovalprotrpprothrleuvalthrthr505560leusertyrglyvalglncyspheserargtyrproasphismetlys65707580glnhisaspphephelysseralametprogluglytyrvalglnglu859095argthrilephephelysaspaspglyasntyrlysthrargalaglu100105110vallysphegluglyaspthrleuvalasnargilegluleulysgly115120125ileaspphelysgluaspglyasnileleuglyhislysleuglutyr130135140asntyrasnserhisasnvaltyrilemetalaasplysglnlysasn145150155160glyilelysvalasnphelysilearghisasnilegluaspglyser165170175valglnleualaasphistyrglnglnasnthrproileglyaspgly180185190provalleuleuproaspasnhistyrleuserthrglnseralaleu195200205serlysaspproasnglulysargasphismetvalleuleugluphe210215220valthralaalaglyilethrleuglymetaspgluleutyrlysleu225230235240glnmetglnileleutrplyslystyrvallysgluasnpheglumet245250255asnvalaspglucysglyilegluglnglyileproglyleuglytyr260265270asntyrgluvalleulysasnalavalilehistyrvalthrlysgly275280285tyrglythrphelyspheasnglylysvaltyrasnleulysglngly290295300aspilepheileleuleulysglymetglnvalglutyrvalalaser305310315320ileaspaspprotrpglutyrtyrtrpileglypheserglyserasn325330335alaasnglutyrleuasnargthrserilethrasnsercysvalala340345350asncysglugluasnserlysileproglnileileleuasnmetcys355360365gluileserlysthrtyrasnproserargseraspaspileleuleu370375380leulysgluleutyrserleuleutyralaleuileglugluphepro385390395400lyspropheglutyrlysasplysgluleuhisthrtyrileglnasp405410415alaleuasnpheileasnserasntyrmethisserilethrvalgln420425430gluilealaasptyrvalasnleuserargsertyrleutyrlysmet435440445pheilelysasnleuglyileserproglnargtyrleuileasnleu450455460argmettyrlysalathrleuleuleulysserthrlysleuproile465470475480glygluvalalaserservalglytyrseraspserleuleupheser485490495lysthrpheserlyshisphesermetserproleuasntyrargasn500505510asnglnvalasnlysproserilepheglumetalaserlysvaltyr515520525aspprogluglnarglysargmetilethrglyproglntrptrpala530535540argcyslysglnmetasnvalleuaspserpheileasntyrtyrasp545550555560serglulyshisalagluasnalavalilepheleuhisglyasnala565570575thrsersertyrleutrparghisvalvalprohisilegluproval580585590alaargcysileileproaspleuileglymetglylysserglylys595600605serglyasnglysertyrargleuleuasphistyrlystyrleuthr610615620alatrpphegluleuleuasnleuprolyslysileilephevalgly625630635640hisasptrpglyalaalaleualaphehistyralatyrgluhisgln645650655aspargilelysalailevalhismetgluservalvalaspvalile660665670glusertrpaspglutrpproaspileglugluaspilealaleuile675680685lysserglugluglyglulysmetvalleugluasnasnphepheval690695700gluthrvalleuproserlysilemetarglysleugluprogluglu705710715720phealaalatyrleugluprophelysglulysglygluvalargarg725730735prothrleusertrpproarggluileproleuvallysglyglylys740745750proaspvalvalglnilevalargasntyrasnalatyrleuargala755760765seraspaspleuprolysleupheilegluseraspproglyphephe770775780serasnalailevalgluglyalalyslyspheproasnthrgluphe785790795800vallysvallysglyleuhispheleuglngluaspalaproaspglu805810815metglylystyrilelysserphevalgluargvalleulysasnglu820825830gln<210>16<211>845<212>prt<213>人工序列<220><223>lacb2<400>16metvalserlysglyglugluleuphethrglyvalvalproileleu151015valgluleuaspglyaspvalasnglyhislyspheservalsergly202530gluglygluglyaspalathrtyrglylysleuthrleulyspheile354045cysthrthrglylysleuprovalprotrpprothrleuvalthrthr505560leusertyrglyvalglncyspheserargtyrproasphismetlys65707580glnhisaspphephelysseralametprogluglytyrvalglnglu859095argthrilephephelysaspaspglyasntyrlysthrargalaglu100105110vallysphegluglyaspthrleuvalasnargilegluleulysgly115120125ileaspphelysgluaspglyasnileleuglyhislysleuglutyr130135140asntyrasnserhisasnvaltyrilemetalaasplysglnlysasn145150155160glyilelysvalasnphelysilearghisasnilegluaspglyser165170175valglnleualaasphistyrglnglnasnthrproileglyaspgly180185190provalleuleuproaspasnhistyrleuserthrglnseralaleu195200205serlysaspproasnglulysargasphismetvalleuleugluphe210215220valthralaalaglyilethrleuglymetaspgluleutyrlysleu225230235240glnglyglythrglyglyglymetglnileleutrplyslystyrval245250255lysgluasnpheglumetasnvalaspglucysglyilegluglngly260265270ileproglyleuglytyrasntyrgluvalleulysasnalavalile275280285histyrvalthrlysglytyrglythrphelyspheasnglylysval290295300tyrasnleulysglnglyaspilepheileleuleulysglymetgln305310315320valglutyrvalalaserileaspaspprotrpglutyrtyrtrpile325330335glypheserglyserasnalaasnglutyrleuasnargthrserile340345350thrasnsercysvalalaasncysglugluasnserlysileprogln355360365ileileleuasnmetcysgluileserlysthrtyrasnproserarg370375380seraspaspileleuleuleulysgluleutyrserleuleutyrala385390395400leuilegluglupheprolyspropheglutyrlysasplysgluleu405410415histhrtyrileglnaspalaleuasnpheileasnserasntyrmet420425430hisserilethrvalglngluilealaasptyrvalasnleuserarg435440445sertyrleutyrlysmetpheilelysasnleuglyileserprogln450455460argtyrleuileasnleuargmettyrlysalathrleuleuleulys465470475480serthrlysleuproileglygluvalalaserservalglytyrser485490495aspserleuleupheserlysthrpheserlyshisphesermetser500505510proleuasntyrargasnasnglnvalasnlysproserilepheglu515520525glyglythrglyglyglymetalaserlysvaltyraspproglugln530535540arglysargmetilethrglyproglntrptrpalaargcyslysgln545550555560metasnvalleuaspserpheileasntyrtyraspserglulyshis565570575alagluasnalavalilepheleuhisglyasnalathrsersertyr580585590leutrparghisvalvalprohisilegluprovalalaargcysile595600605ileproaspleuileglymetglylysserglylysserglyasngly610615620sertyrargleuleuasphistyrlystyrleuthralatrppheglu625630635640leuleuasnleuprolyslysileilephevalglyhisasptrpgly645650655alaalaleualaphehistyralatyrgluhisglnaspargilelys660665670alailevalhismetgluservalvalaspvalileglusertrpasp675680685glutrpproaspileglugluaspilealaleuilelyssergluglu690695700glyglulysmetvalleugluasnasnphephevalgluthrvalleu705710715720proserlysilemetarglysleugluproglugluphealaalatyr725730735leugluprophelysglulysglygluvalargargprothrleuser740745750trpproarggluileproleuvallysglyglylysproaspvalval755760765glnilevalargasntyrasnalatyrleuargalaseraspaspleu770775780prolysleupheilegluseraspproglyphepheserasnalaile785790795800valgluglyalalyslyspheproasnthrgluphevallysvallys805810815glyleuhispheleuglngluaspalaproaspglumetglylystyr820825830ilelysserphevalgluargvalleulysasnglugln835840845<210>17<211>839<212>prt<213>人工序列<220><223>lacb3<400>17metvalserlysglyglugluleuphethrglyvalvalproileleu151015valgluleuaspglyaspvalasnglyhislyspheservalsergly202530gluglygluglyaspalathrtyrglylysleuthrleulyspheile354045cysthrthrglylysleuprovalprotrpprothrleuvalthrthr505560leusertyrglyvalglncyspheserargtyrproasphismetlys65707580glnhisaspphephelysseralametprogluglytyrvalglnglu859095argthrilephephelysaspaspglyasntyrlysthrargalaglu100105110vallysphegluglyaspthrleuvalasnargilegluleulysgly115120125ileaspphelysgluaspglyasnileleuglyhislysleuglutyr130135140asntyrasnserhisasnvaltyrilemetalaasplysglnlysasn145150155160glyilelysvalasnphelysilearghisasnilegluaspglyser165170175valglnleualaasphistyrglnglnasnthrproileglyaspgly180185190provalleuleuproaspasnhistyrleuserthrglnseralaleu195200205serlysaspproasnglulysargasphismetvalleuleugluphe210215220valthralaalaglyilethrleuglymetaspgluleutyrlysleu225230235240glnglyglythrglyglyglymetglnileleutrplyslystyrval245250255lysgluasnpheglumetasnvalaspglucysglyilegluglngly260265270ileproglyleuglytyrasntyrgluvalleulysasnalavalile275280285histyrvalthrlysglytyrglythrphelyspheasnglylysval290295300tyrasnleulysglnglyaspilepheileleuleulysglymetgln305310315320valglutyrvalalaserileaspaspprotrpglutyrtyrtrpile325330335glypheserglyserasnalaasnglutyrleuasnargthrserile340345350thrasnsercysvalalaasncysglugluasnserlysileprogln355360365ileileleuasnmetcysgluileserlysthrtyrasnproserarg370375380seraspaspileleuleuleulysgluleutyrserleuleutyrala385390395400leuilegluglupheprolyspropheglutyrlysasplysgluleu405410415histhrtyrileglnaspalaleuasnpheileasnserasntyrmet420425430hisserilethrvalglngluilealaasptyrvalasnleuserarg435440445sertyrleutyrlysmetpheilelysasnleuglyileserprogln450455460argtyrleuileasnleuargmettyrlysalathrleuleuleulys465470475480serthrlysleuproileglygluvalalaserservalglytyrser485490495aspserleuleupheserlysthrpheserlyshisphesermetser500505510proleuasntyrargasnasnglnvalasnlysproserilepheglu515520525metalaserlysvaltyraspprogluglnarglysargmetilethr530535540glyproglntrptrpalaargcyslysglnmetasnvalleuaspser545550555560pheileasntyrtyraspserglulyshisalagluasnalavalile565570575pheleuhisglyasnalathrsersertyrleutrparghisvalval580585590prohisilegluprovalalaargcysileileproaspleuilegly595600605metglylysserglylysserglyasnglysertyrargleuleuasp610615620histyrlystyrleuthralatrpphegluleuleuasnleuprolys625630635640lysileilephevalglyhisasptrpglyalaalaleualaphehis645650655tyralatyrgluhisglnaspargilelysalailevalhismetglu660665670servalvalaspvalileglusertrpaspglutrpproaspileglu675680685gluaspilealaleuilelysserglugluglyglulysmetvalleu690695700gluasnasnphephevalgluthrvalleuproserlysilemetarg705710715720lysleugluproglugluphealaalatyrleugluprophelysglu725730735lysglygluvalargargprothrleusertrpproarggluilepro740745750leuvallysglyglylysproaspvalvalglnilevalargasntyr755760765asnalatyrleuargalaseraspaspleuprolysleupheileglu770775780seraspproglyphepheserasnalailevalgluglyalalyslys785790795800pheproasnthrgluphevallysvallysglyleuhispheleugln805810815gluaspalaproaspglumetglylystyrilelysserphevalglu820825830argvalleulysasnglugln835<210>18<211>839<212>prt<213>人工序列<220><223>lacb4<400>18metvalserlysglyglugluleuphethrglyvalvalproileleu151015valgluleuaspglyaspvalasnglyhislyspheservalsergly202530gluglygluglyaspalathrtyrglylysleuthrleulyspheile354045cysthrthrglylysleuprovalprotrpprothrleuvalthrthr505560leusertyrglyvalglncyspheserargtyrproasphismetlys65707580glnhisaspphephelysseralametprogluglytyrvalglnglu859095argthrilephephelysaspaspglyasntyrlysthrargalaglu100105110vallysphegluglyaspthrleuvalasnargilegluleulysgly115120125ileaspphelysgluaspglyasnileleuglyhislysleuglutyr130135140asntyrasnserhisasnvaltyrilemetalaasplysglnlysasn145150155160glyilelysvalasnphelysilearghisasnilegluaspglyser165170175valglnleualaasphistyrglnglnasnthrproileglyaspgly180185190provalleuleuproaspasnhistyrleuserthrglnseralaleu195200205serlysaspproasnglulysargasphismetvalleuleugluphe210215220valthralaalaglyilethrleuglymetaspgluleutyrlysleu225230235240glnmetglnileleutrplyslystyrvallysgluasnpheglumet245250255asnvalaspglucysglyilegluglnglyileproglyleuglytyr260265270asntyrgluvalleulysasnalavalilehistyrvalthrlysgly275280285tyrglythrphelyspheasnglylysvaltyrasnleulysglngly290295300aspilepheileleuleulysglymetglnvalglutyrvalalaser305310315320ileaspaspprotrpglutyrtyrtrpileglypheserglyserasn325330335alaasnglutyrleuasnargthrserilethrasnsercysvalala340345350asncysglugluasnserlysileproglnileileleuasnmetcys355360365gluileserlysthrtyrasnproserargseraspaspileleuleu370375380leulysgluleutyrserleuleutyralaleuileglugluphepro385390395400lyspropheglutyrlysasplysgluleuhisthrtyrileglnasp405410415alaleuasnpheileasnserasntyrmethisserilethrvalgln420425430gluilealaasptyrvalasnleuserargsertyrleutyrlysmet435440445pheilelysasnleuglyileserproglnargtyrleuileasnleu450455460argmettyrlysalathrleuleuleulysserthrlysleuproile465470475480glygluvalalaserservalglytyrseraspserleuleupheser485490495lysthrpheserlyshisphesermetserproleuasntyrargasn500505510asnglnvalasnlysproserilephegluglyglythrglyglygly515520525metalaserlysvaltyraspprogluglnarglysargmetilethr530535540glyproglntrptrpalaargcyslysglnmetasnvalleuaspser545550555560pheileasntyrtyraspserglulyshisalagluasnalavalile565570575pheleuhisglyasnalathrsersertyrleutrparghisvalval580585590prohisilegluprovalalaargcysileileproaspleuilegly595600605metglylysserglylysserglyasnglysertyrargleuleuasp610615620histyrlystyrleuthralatrpphegluleuleuasnleuprolys625630635640lysileilephevalglyhisasptrpglyalaalaleualaphehis645650655tyralatyrgluhisglnaspargilelysalailevalhismetglu660665670servalvalaspvalileglusertrpaspglutrpproaspileglu675680685gluaspilealaleuilelysserglugluglyglulysmetvalleu690695700gluasnasnphephevalgluthrvalleuproserlysilemetarg705710715720lysleugluproglugluphealaalatyrleugluprophelysglu725730735lysglygluvalargargprothrleusertrpproarggluilepro740745750leuvallysglyglylysproaspvalvalglnilevalargasntyr755760765asnalatyrleuargalaseraspaspleuprolysleupheileglu770775780seraspproglyphepheserasnalailevalgluglyalalyslys785790795800pheproasnthrgluphevallysvallysglyleuhispheleugln805810815gluaspalaproaspglumetglylystyrilelysserphevalglu820825830argvalleulysasnglugln835<210>19<211>2502<212>dna<213>人工序列<220><223>核苷酸序列编码lacb1<400>19atggtgagcaagggcgaggagctgttcaccggggtggtgcccatcctggtcgagctggac60ggcgacgtaaacggccacaagttcagcgtgtccggcgagggcgagggcgatgccacctac120ggcaagctgaccctgaagttcatctgcaccaccggcaagctgcccgtgccctggcccacc180ctcgtgaccaccctgagctacggcgtgcagtgcttcagccgctaccccgaccacatgaag240cagcacgacttcttcaagtccgccatgcccgaaggctacgtccaggagcgcaccatcttc300ttcaaggacgacggcaactacaagacccgcgccgaggtgaagttcgagggcgacaccctg360gtgaaccgcatcgagctgaagggcatcgacttcaaggaggacggcaacatcctggggcac420aagctggagtacaactacaacagccacaacgtctatatcatggccgacaagcagaagaac480ggcatcaaggtgaacttcaagatccgccacaacatcgaggacggcagcgtgcagctcgcc540gaccactaccagcagaacacccccatcggcgacggccccgtgctgctgcccgacaaccac600tacctgagcacccagtccgccctgagcaaagaccccaacgagaagcgcgatcacatggtc660ctgctggagttcgtgaccgccgccgggatcactctcggcatggacgagctgtacaagctg720cagatgcagattctgtggaaaaaatacgtcaaagaaaactttgaaatgaacgtggatgaa780tgcggcattgaacaaggcattccgggcctgggttataactacgaagttctgaaaaatgca840gtcatccattatgtgaccaaaggctatggtacgtttaaattcaacggcaaagtctataat900ctgaaacagggtgacattttcatcctgctgaaaggcatgcaagtggaatacgttgcgagc960attgatgacccgtgggaatattactggatcggctttagtggttccaacgcgaatgaatat1020ctgaaccgtaccagcattaccaacagctgcgtggccaactgtgaagaaaatagcaaaatt1080ccgcagattatcctgaacatgtgtgaaatctctaaaacctacaacccgtcacgctcggat1140gacattctgctgctgaaagaactgtattccctgctgtacgcactgatcgaagaatttccg1200aaaccgtttgaatacaaagataaagaactgcatacctacattcaggacgcgctgaacttc1260atcaactcaaattatatgcactcgattacggtgcaagaaatcgccgattacgttaatctg1320agccgttcttacctgtacaaaatgttcatcaaaaacctgggtatcagtccgcagcgttat1380ctgattaatctgcgcatgtacaaagcaaccctgctgctgaaatctacgaaactgccgatc1440ggcgaagttgcgagcagcgtgggttatagtgattccctgctgtttagtaaaaccttctcc1500aaacacttttcaatgtcgccgctgaactaccgcaacaatcaagttaataaaccgagcatt1560ttcgaaatggcttccaaggtgtacgaccccgagcaacgcaaacgcatgatcactgggcct1620cagtggtgggctcgctgcaagcaaatgaacgtgctggactccttcatcaactactatgat1680tccgagaagcacgccgagaacgccgtgatttttctgcatggtaacgctacctccagctac1740ctgtggaggcacgtcgtgcctcacatcgagcccgtggctagatgcatcatccctgatctg1800atcggaatgggtaagtccggcaagagcgggaatggctcatatcgcctcctggatcactac1860aagtacctcaccgcttggttcgagctgctgaaccttccaaagaaaatcatctttgtgggc1920cacgactggggggctgctctggcctttcactacgcctacgagcaccaagacaggatcaag1980gccatcgtccatatggagagtgtcgtggacgtgatcgagtcctgggacgagtggcctgac2040atcgaggaggatatcgccctgatcaagagcgaagagggcgagaaaatggtgcttgagaat2100aacttcttcgtcgagaccgtgctcccaagcaagatcatgcggaaactggagcctgaggag2160ttcgctgcctacctggagccattcaaggagaagggcgaggttagacggcctaccctctcc2220tggcctcgcgagatccctctcgttaagggaggcaagcccgacgtcgtccagattgtccgc2280aactacaacgcctaccttcgggccagcgacgatctgcctaagctgttcatcgagtccgac2340cctgggttcttttccaacgctattgtcgagggagctaagaagttccctaacaccgagttc2400gtgaaggtgaagggcctccacttcctccaggaggacgctccagatgaaatgggtaagtac2460atcaagagcttcgtggagcgcgtgctgaagaacgagcagtaa2502<210>20<211>2538<212>dna<213>人工序列<220><223>核苷酸序列编码lacb2<400>20atggtgagcaagggcgaggagctgttcaccggggtggtgcccatcctggtcgagctggac60ggcgacgtaaacggccacaagttcagcgtgtccggcgagggcgagggcgatgccacctac120ggcaagctgaccctgaagttcatctgcaccaccggcaagctgcccgtgccctggcccacc180ctcgtgaccaccctgagctacggcgtgcagtgcttcagccgctaccccgaccacatgaag240cagcacgacttcttcaagtccgccatgcccgaaggctacgtccaggagcgcaccatcttc300ttcaaggacgacggcaactacaagacccgcgccgaggtgaagttcgagggcgacaccctg360gtgaaccgcatcgagctgaagggcatcgacttcaaggaggacggcaacatcctggggcac420aagctggagtacaactacaacagccacaacgtctatatcatggccgacaagcagaagaac480ggcatcaaggtgaacttcaagatccgccacaacatcgaggacggcagcgtgcagctcgcc540gaccactaccagcagaacacccccatcggcgacggccccgtgctgctgcccgacaaccac600tacctgagcacccagtccgccctgagcaaagaccccaacgagaagcgcgatcacatggtc660ctgctggagttcgtgaccgccgccgggatcactctcggcatggacgagctgtacaagctg720cagggtggtaccggaggcggcatgcagattctgtggaaaaaatacgtcaaagaaaacttt780gaaatgaacgtggatgaatgcggcattgaacaaggcattccgggcctgggttataactac840gaagttctgaaaaatgcagtcatccattatgtgaccaaaggctatggtacgtttaaattc900aacggcaaagtctataatctgaaacagggtgacattttcatcctgctgaaaggcatgcaa960gtggaatacgttgcgagcattgatgacccgtgggaatattactggatcggctttagtggt1020tccaacgcgaatgaatatctgaaccgtaccagcattaccaacagctgcgtggccaactgt1080gaagaaaatagcaaaattccgcagattatcctgaacatgtgtgaaatctctaaaacctac1140aacccgtcacgctcggatgacattctgctgctgaaagaactgtattccctgctgtacgca1200ctgatcgaagaatttccgaaaccgtttgaatacaaagataaagaactgcatacctacatt1260caggacgcgctgaacttcatcaactcaaattatatgcactcgattacggtgcaagaaatc1320gccgattacgttaatctgagccgttcttacctgtacaaaatgttcatcaaaaacctgggt1380atcagtccgcagcgttatctgattaatctgcgcatgtacaaagcaaccctgctgctgaaa1440tctacgaaactgccgatcggcgaagttgcgagcagcgtgggttatagtgattccctgctg1500tttagtaaaaccttctccaaacacttttcaatgtcgccgctgaactaccgcaacaatcaa1560gttaataaaccgagcattttcgaaggtggtaccggaggcggcatggcttccaaggtgtac1620gaccccgagcaacgcaaacgcatgatcactgggcctcagtggtgggctcgctgcaagcaa1680atgaacgtgctggactccttcatcaactactatgattccgagaagcacgccgagaacgcc1740gtgatttttctgcatggtaacgctacctccagctacctgtggaggcacgtcgtgcctcac1800atcgagcccgtggctagatgcatcatccctgatctgatcggaatgggtaagtccggcaag1860agcgggaatggctcatatcgcctcctggatcactacaagtacctcaccgcttggttcgag1920ctgctgaaccttccaaagaaaatcatctttgtgggccacgactggggggctgctctggcc1980tttcactacgcctacgagcaccaagacaggatcaaggccatcgtccatatggagagtgtc2040gtggacgtgatcgagtcctgggacgagtggcctgacatcgaggaggatatcgccctgatc2100aagagcgaagagggcgagaaaatggtgcttgagaataacttcttcgtcgagaccgtgctc2160ccaagcaagatcatgcggaaactggagcctgaggagttcgctgcctacctggagccattc2220aaggagaagggcgaggttagacggcctaccctctcctggcctcgcgagatccctctcgtt2280aagggaggcaagcccgacgtcgtccagattgtccgcaactacaacgcctaccttcgggcc2340agcgacgatctgcctaagctgttcatcgagtccgaccctgggttcttttccaacgctatt2400gtcgagggagctaagaagttccctaacaccgagttcgtgaaggtgaagggcctccacttc2460ctccaggaggacgctccagatgaaatgggtaagtacatcaagagcttcgtggagcgcgtg2520ctgaagaacgagcagtaa2538<210>21<211>2520<212>dna<213>人工序列<220><223>核苷酸序列编码lacb3<400>21atggtgagcaagggcgaggagctgttcaccggggtggtgcccatcctggtcgagctggac60ggcgacgtaaacggccacaagttcagcgtgtccggcgagggcgagggcgatgccacctac120ggcaagctgaccctgaagttcatctgcaccaccggcaagctgcccgtgccctggcccacc180ctcgtgaccaccctgagctacggcgtgcagtgcttcagccgctaccccgaccacatgaag240cagcacgacttcttcaagtccgccatgcccgaaggctacgtccaggagcgcaccatcttc300ttcaaggacgacggcaactacaagacccgcgccgaggtgaagttcgagggcgacaccctg360gtgaaccgcatcgagctgaagggcatcgacttcaaggaggacggcaacatcctggggcac420aagctggagtacaactacaacagccacaacgtctatatcatggccgacaagcagaagaac480ggcatcaaggtgaacttcaagatccgccacaacatcgaggacggcagcgtgcagctcgcc540gaccactaccagcagaacacccccatcggcgacggccccgtgctgctgcccgacaaccac600tacctgagcacccagtccgccctgagcaaagaccccaacgagaagcgcgatcacatggtc660ctgctggagttcgtgaccgccgccgggatcactctcggcatggacgagctgtacaagctg720cagggtggtaccggaggcggcatgcagattctgtggaaaaaatacgtcaaagaaaacttt780gaaatgaacgtggatgaatgcggcattgaacaaggcattccgggcctgggttataactac840gaagttctgaaaaatgcagtcatccattatgtgaccaaaggctatggtacgtttaaattc900aacggcaaagtctataatctgaaacagggtgacattttcatcctgctgaaaggcatgcaa960gtggaatacgttgcgagcattgatgacccgtgggaatattactggatcggctttagtggt1020tccaacgcgaatgaatatctgaaccgtaccagcattaccaacagctgcgtggccaactgt1080gaagaaaatagcaaaattccgcagattatcctgaacatgtgtgaaatctctaaaacctac1140aacccgtcacgctcggatgacattctgctgctgaaagaactgtattccctgctgtacgca1200ctgatcgaagaatttccgaaaccgtttgaatacaaagataaagaactgcatacctacatt1260caggacgcgctgaacttcatcaactcaaattatatgcactcgattacggtgcaagaaatc1320gccgattacgttaatctgagccgttcttacctgtacaaaatgttcatcaaaaacctgggt1380atcagtccgcagcgttatctgattaatctgcgcatgtacaaagcaaccctgctgctgaaa1440tctacgaaactgccgatcggcgaagttgcgagcagcgtgggttatagtgattccctgctg1500tttagtaaaaccttctccaaacacttttcaatgtcgccgctgaactaccgcaacaatcaa1560gttaataaaccgagcattttcgaaatggcttccaaggtgtacgaccccgagcaacgcaaa1620cgcatgatcactgggcctcagtggtgggctcgctgcaagcaaatgaacgtgctggactcc1680ttcatcaactactatgattccgagaagcacgccgagaacgccgtgatttttctgcatggt1740aacgctacctccagctacctgtggaggcacgtcgtgcctcacatcgagcccgtggctaga1800tgcatcatccctgatctgatcggaatgggtaagtccggcaagagcgggaatggctcatat1860cgcctcctggatcactacaagtacctcaccgcttggttcgagctgctgaaccttccaaag1920aaaatcatctttgtgggccacgactggggggctgctctggcctttcactacgcctacgag1980caccaagacaggatcaaggccatcgtccatatggagagtgtcgtggacgtgatcgagtcc2040tgggacgagtggcctgacatcgaggaggatatcgccctgatcaagagcgaagagggcgag2100aaaatggtgcttgagaataacttcttcgtcgagaccgtgctcccaagcaagatcatgcgg2160aaactggagcctgaggagttcgctgcctacctggagccattcaaggagaagggcgaggtt2220agacggcctaccctctcctggcctcgcgagatccctctcgttaagggaggcaagcccgac2280gtcgtccagattgtccgcaactacaacgcctaccttcgggccagcgacgatctgcctaag2340ctgttcatcgagtccgaccctgggttcttttccaacgctattgtcgagggagctaagaag2400ttccctaacaccgagttcgtgaaggtgaagggcctccacttcctccaggaggacgctcca2460gatgaaatgggtaagtacatcaagagcttcgtggagcgcgtgctgaagaacgagcagtaa2520<210>22<211>2520<212>dna<213>人工序列<220><223>核苷酸序列编码lacb4<400>22atggtgagcaagggcgaggagctgttcaccggggtggtgcccatcctggtcgagctggac60ggcgacgtaaacggccacaagttcagcgtgtccggcgagggcgagggcgatgccacctac120ggcaagctgaccctgaagttcatctgcaccaccggcaagctgcccgtgccctggcccacc180ctcgtgaccaccctgagctacggcgtgcagtgcttcagccgctaccccgaccacatgaag240cagcacgacttcttcaagtccgccatgcccgaaggctacgtccaggagcgcaccatcttc300ttcaaggacgacggcaactacaagacccgcgccgaggtgaagttcgagggcgacaccctg360gtgaaccgcatcgagctgaagggcatcgacttcaaggaggacggcaacatcctggggcac420aagctggagtacaactacaacagccacaacgtctatatcatggccgacaagcagaagaac480ggcatcaaggtgaacttcaagatccgccacaacatcgaggacggcagcgtgcagctcgcc540gaccactaccagcagaacacccccatcggcgacggccccgtgctgctgcccgacaaccac600tacctgagcacccagtccgccctgagcaaagaccccaacgagaagcgcgatcacatggtc660ctgctggagttcgtgaccgccgccgggatcactctcggcatggacgagctgtacaagctg720cagatgcagattctgtggaaaaaatacgtcaaagaaaactttgaaatgaacgtggatgaa780tgcggcattgaacaaggcattccgggcctgggttataactacgaagttctgaaaaatgca840gtcatccattatgtgaccaaaggctatggtacgtttaaattcaacggcaaagtctataat900ctgaaacagggtgacattttcatcctgctgaaaggcatgcaagtggaatacgttgcgagc960attgatgacccgtgggaatattactggatcggctttagtggttccaacgcgaatgaatat1020ctgaaccgtaccagcattaccaacagctgcgtggccaactgtgaagaaaatagcaaaatt1080ccgcagattatcctgaacatgtgtgaaatctctaaaacctacaacccgtcacgctcggat1140gacattctgctgctgaaagaactgtattccctgctgtacgcactgatcgaagaatttccg1200aaaccgtttgaatacaaagataaagaactgcatacctacattcaggacgcgctgaacttc1260atcaactcaaattatatgcactcgattacggtgcaagaaatcgccgattacgttaatctg1320agccgttcttacctgtacaaaatgttcatcaaaaacctgggtatcagtccgcagcgttat1380ctgattaatctgcgcatgtacaaagcaaccctgctgctgaaatctacgaaactgccgatc1440ggcgaagttgcgagcagcgtgggttatagtgattccctgctgtttagtaaaaccttctcc1500aaacacttttcaatgtcgccgctgaactaccgcaacaatcaagttaataaaccgagcatt1560ttcgaaggtggtaccggaggcggcatggcttccaaggtgtacgaccccgagcaacgcaaa1620cgcatgatcactgggcctcagtggtgggctcgctgcaagcaaatgaacgtgctggactcc1680ttcatcaactactatgattccgagaagcacgccgagaacgccgtgatttttctgcatggt1740aacgctacctccagctacctgtggaggcacgtcgtgcctcacatcgagcccgtggctaga1800tgcatcatccctgatctgatcggaatgggtaagtccggcaagagcgggaatggctcatat1860cgcctcctggatcactacaagtacctcaccgcttggttcgagctgctgaaccttccaaag1920aaaatcatctttgtgggccacgactggggggctgctctggcctttcactacgcctacgag1980caccaagacaggatcaaggccatcgtccatatggagagtgtcgtggacgtgatcgagtcc2040tgggacgagtggcctgacatcgaggaggatatcgccctgatcaagagcgaagagggcgag2100aaaatggtgcttgagaataacttcttcgtcgagaccgtgctcccaagcaagatcatgcgg2160aaactggagcctgaggagttcgctgcctacctggagccattcaaggagaagggcgaggtt2220agacggcctaccctctcctggcctcgcgagatccctctcgttaagggaggcaagcccgac2280gtcgtccagattgtccgcaactacaacgcctaccttcgggccagcgacgatctgcctaag2340ctgttcatcgagtccgaccctgggttcttttccaacgctattgtcgagggagctaagaag2400ttccctaacaccgagttcgtgaaggtgaagggcctccacttcctccaggaggacgctcca2460gatgaaatgggtaagtacatcaagagcttcgtggagcgcgtgctgaagaacgagcagtaa2520<210>23<211>799<212>prt<213>人工序列<220><223>lacf1<400>23metargglyserhishishishishishisglymetalasermetthr151015glyglyglnglnmetglyargaspleutyraspaspaspasplysasp202530argtrpglysergluphemetvalserlysglyglugluleuphethr354045glyvalvalproileleuvalgluleuaspglyaspvalasnglyhis505560argpheservalserglygluglygluglyaspalathrtyrglylys65707580leuthrleulyspheilecysthrthrglylysleuprovalprotrp859095prothrleuvalthrthrleuthrtrpglyvalglncyspheserarg100105110tyrproasphismetlysglnhisaspphephelysseralametpro115120125gluglytyrvalglngluargthrilephephelysaspaspglyasn130135140tyrlysthrargalagluvallysphegluglyaspthrleuvalasn145150155160argilegluleulysglyileaspphelysgluaspglyasnileleu165170175glyhislysleuglutyrasntyrileserhisasnvaltyrilethr180185190alaasplysglnlysasnglyilelysalahisphelysilearghis195200205asnilegluaspglyservalglnleualaasphistyrglnglnasn210215220thrproileglyaspglyprovalleuleuproaspasnhistyrleu225230235240serthrglnseralaleuserlysaspproasnglulysargasphis245250255metvalleuleugluphevalthralaalaglyilethrleuglymet260265270aspgluleutyrlysleuglnmetglnileleutrplyslystyrval275280285lysgluasnpheglumetasnvalaspglucysglyilegluglngly290295300ileproglyleuglytyrasntyrgluvalleulysasnalavalile305310315320histyrvalthrlysglytyrglythrphelyspheasnglylysval325330335tyrasnleulysglnglyaspilepheileleuleulysglymetgln340345350valglutyrvalalaserileaspaspprotrpglutyrtyrtrpile355360365glypheserglyserasnalaasnglutyrleuasnargthrserile370375380thrasnsercysvalalaasncysglugluasnserlysileprogln385390395400ileileleuasnmetcysgluileserlysthrtyrasnproserarg405410415seraspaspileleuleuleulysgluleutyrserleuleutyrala420425430leuilegluglupheprolyspropheglutyrlysasplysgluleu435440445histhrtyrileglnaspalaleuasnpheileasnserasntyrmet450455460hisserilethrvalglngluilealaasptyrvalasnleuserarg465470475480sertyrleutyrlysmetpheilelysasnleuglyileserprogln485490495argtyrleuileasnleuargmettyrlysalathrleuleuleulys500505510serthrlysleuproileglygluvalalaserservalglytyrser515520525aspserleuleupheserlysthrpheserlyshisphesermetser530535540proleuasntyrargasnasnglnvalasnlysproserilepheglu545550555560metvalserlysglyglugluleuphethrglyvalvalproileleu565570575valgluleuaspglyaspvalasnglyhislyspheservalsergly580585590gluglygluglyaspalathrtyrglylysleuthrleulyspheile595600605cysthrthrglylysleuprovalprotrpprothrleuvalthrthr610615620pheglytyrglyvalglncysphealaargtyrproasphismetarg625630635640glnhisaspphephelysseralametprogluglytyrvalglnglu645650655argthrilephephelysaspaspglyasntyrlysthrargalaglu660665670vallysphegluglyaspthrleuvalasnargilegluleulysgly675680685ileaspphelysgluaspglyasnileleuglyhislysleuglutyr690695700asntyrasnserhisasnvaltyrilemetalaasplysglnlysasn705710715720glyilelysvalasnphelysilearghisasnilegluaspglyser725730735valglnleualaasphistyrglnglnasnthrproileglyaspgly740745750provalleuleuproaspasnhistyrleusertyrglnseralaleu755760765serlysaspproasnglulysargasphismetvalleuleugluphe770775780valthralaalaglyilethrleuglymetaspgluleutyrlys785790795<210>24<211>2397<212>dna<213>人工序列<220><223>核苷酸序列编码lacf1<400>24atgcggggttctcatcatcatcatcatcatggtatggctagcatgactggtggacagcaa60atgggtcgggatctgtacgacgatgacgataaggatcgatggggatccgaattcatggtg120agcaagggcgaggagctgttcaccggggtggtgcccatcctggtcgagctggacggcgac180gtaaacggccacaggttcagcgtgtccggcgagggcgagggcgatgccacctacggcaag240ctgaccctgaagttcatctgcaccaccggcaagctgcccgtgccctggcccaccctcgtg300accaccctgacctggggcgtgcagtgcttcagccgctaccccgaccacatgaagcagcac360gacttcttcaagtccgccatgcccgaaggctacgtccaggagcgtaccatcttcttcaag420gacgacggcaactacaagacccgcgccgaggtgaagttcgagggcgacaccctggtgaac480cgcatcgagctgaagggcatcgacttcaaggaggacggcaacatcctggggcacaagctg540gagtacaactacatcagccacaacgtctatatcaccgccgacaagcagaagaacggcatc600aaggcccacttcaagatccgccacaacatcgaggacggcagcgtgcagctcgccgaccac660taccagcagaacacccccatcggcgacggccccgtgctgctgcccgacaaccactacctg720agcacccagtccgccctgagcaaagaccccaacgagaagcgcgatcacatggtcctgctg780gagttcgtgaccgccgccgggatcactctcggcatggacgagctgtacaagctgcagatg840cagattctgtggaaaaaatacgtcaaagaaaactttgaaatgaacgtggatgaatgcggc900attgaacaaggcattccgggcctgggttataactacgaagttctgaaaaatgcagtcatc960cattatgtgaccaaaggctatggtacgtttaaattcaacggcaaagtctataatctgaaa1020cagggtgacattttcatcctgctgaaaggcatgcaagtggaatacgttgcgagcattgat1080gacccgtgggaatattactggatcggctttagtggttccaacgcgaatgaatatctgaac1140cgtaccagcattaccaacagctgcgtggccaactgtgaagaaaatagcaaaattccgcag1200attatcctgaacatgtgtgaaatctctaaaacctacaacccgtcacgctcggatgacatt1260ctgctgctgaaagaactgtattccctgctgtacgcactgatcgaagaatttccgaaaccg1320tttgaatacaaagataaagaactgcatacctacattcaggacgcgctgaacttcatcaac1380tcaaattatatgcactcgattacggtgcaagaaatcgccgattacgttaatctgagccgt1440tcttacctgtacaaaatgttcatcaaaaacctgggtatcagtccgcagcgttatctgatt1500aatctgcgcatgtacaaagcaaccctgctgctgaaatctacgaaactgccgatcggcgaa1560gttgcgagcagcgtgggttatagtgattccctgctgtttagtaaaaccttctccaaacac1620ttttcaatgtcgccgctgaactaccgcaacaatcaagttaataaaccgagcattctgcag1680atggtgagcaagggcgaggagctgttcaccggggtggtgcccatcctggtcgagctggac1740ggcgacgtaaacggccacaagttcagcgtgtccggcgagggcgagggcgatgccacctac1800ggcaagctgaccctgaagttcatctgcaccaccggcaagctgcccgtgccctggcccacc1860ctcgtgaccaccttcggctacggcgtgcagtgcttcgcccgctaccccgaccacatgcgc1920cagcacgacttcttcaagtccgccatgcccgaaggctacgtccaggagcgcaccatcttc1980ttcaaggacgacggcaactacaagacccgcgccgaggtgaagttcgagggcgacaccctg2040gtgaaccgcatcgagctgaagggcatcgacttcaaggaggacggcaacatcctggggcac2100aagctggagtacaactacaacagccacaacgtctatatcatggccgacaagcagaagaac2160ggcatcaaggtgaacttcaagatccgccacaacatcgaggacggcagcgtgcagctcgcc2220gaccactaccagcagaacacccccatcggcgacggccccgtgctgctgcccgacaaccac2280tacctgagctaccagtccgccctgagcaaagaccccaacgagaagcgcgatcacatggtc2340ctgctggagttcgtgaccgccgccgggatcactctcggcatggacgagctgtacaag2397<210>25<211>873<212>prt<213>人工序列<220><223>lacb1<400>25metargglyserhishishishishishisglymetalasermetthr151015glyglyglnglnmetglyargaspleutyraspaspaspasplysasp202530argtrpglysergluphemetvalserlysglyglugluleuphethr354045glyvalvalproileleuvalgluleuaspglyaspvalasnglyhis505560lyspheservalserglygluglygluglyaspalathrtyrglylys65707580leuthrleulyspheilecysthrthrglylysleuprovalprotrp859095prothrleuvalthrthrleusertyrglyvalglncyspheserarg100105110tyrproasphismetlysglnhisaspphephelysseralametpro115120125gluglytyrvalglngluargthrilephephelysaspaspglyasn130135140tyrlysthrargalagluvallysphegluglyaspthrleuvalasn145150155160argilegluleulysglyileaspphelysgluaspglyasnileleu165170175glyhislysleuglutyrasntyrasnserhisasnvaltyrilemet180185190alaasplysglnlysasnglyilelysvalasnphelysilearghis195200205asnilegluaspglyservalglnleualaasphistyrglnglnasn210215220thrproileglyaspglyprovalleuleuproaspasnhistyrleu225230235240serthrglnseralaleuserlysaspproasnglulysargasphis245250255metvalleuleugluphevalthralaalaglyilethrleuglymet260265270aspgluleutyrlysleuglnmetglnileleutrplyslystyrval275280285lysgluasnpheglumetasnvalaspglucysglyilegluglngly290295300ileproglyleuglytyrasntyrgluvalleulysasnalavalile305310315320histyrvalthrlysglytyrglythrphelyspheasnglylysval325330335tyrasnleulysglnglyaspilepheileleuleulysglymetgln340345350valglutyrvalalaserileaspaspprotrpglutyrtyrtrpile355360365glypheserglyserasnalaasnglutyrleuasnargthrserile370375380thrasnsercysvalalaasncysglugluasnserlysileprogln385390395400ileileleuasnmetcysgluileserlysthrtyrasnproserarg405410415seraspaspileleuleuleulysgluleutyrserleuleutyrala420425430leuilegluglupheprolyspropheglutyrlysasplysgluleu435440445histhrtyrileglnaspalaleuasnpheileasnserasntyrmet450455460hisserilethrvalglngluilealaasptyrvalasnleuserarg465470475480sertyrleutyrlysmetpheilelysasnleuglyileserprogln485490495argtyrleuileasnleuargmettyrlysalathrleuleuleulys500505510serthrlysleuproileglygluvalalaserservalglytyrser515520525aspserleuleupheserlysthrpheserlyshisphesermetser530535540proleuasntyrargasnasnglnvalasnlysproserilepheglu545550555560leuglnmetalaserlysvaltyraspprogluglnarglysargmet565570575ilethrglyproglntrptrpalaargcyslysglnmetasnvalleu580585590aspserpheileasntyrtyraspserglulyshisalagluasnala595600605valilepheleuhisglyasnalathrsersertyrleutrparghis610615620valvalprohisilegluprovalalaargcysileileproaspleu625630635640ileglymetglylysserglylysserglyasnglysertyrargleu645650655leuasphistyrlystyrleuthralatrpphegluleuleuasnleu660665670prolyslysileilephevalglyhisasptrpglyalaalaleuala675680685phehistyralatyrgluhisglnaspargilelysalailevalhis690695700metgluservalvalaspvalileglusertrpaspglutrpproasp705710715720ileglugluaspilealaleuilelysserglugluglyglulysmet725730735valleugluasnasnphephevalgluthrvalleuproserlysile740745750metarglysleugluproglugluphealaalatyrleugluprophe755760765lysglulysglygluvalargargprothrleusertrpproargglu770775780ileproleuvallysglyglylysproaspvalvalglnilevalarg785790795800asntyrasnalatyrleuargalaseraspaspleuprolysleuphe805810815ilegluseraspproglyphepheserasnalailevalgluglyala820825830lyslyspheproasnthrgluphevallysvallysglyleuhisphe835840845leuglngluaspalaproaspglumetglylystyrilelysserphe850855860valgluargvalleulysasnglugln865870<210>26<211>885<212>prt<213>人工序列<220><223>lacb2<400>26metargglyserhishishishishishisglymetalasermetthr151015glyglyglnglnmetglyargaspleutyraspaspaspasplysasp202530argtrpglysergluphemetvalserlysglyglugluleuphethr354045glyvalvalproileleuvalgluleuaspglyaspvalasnglyhis505560lyspheservalserglygluglygluglyaspalathrtyrglylys65707580leuthrleulyspheilecysthrthrglylysleuprovalprotrp859095prothrleuvalthrthrleusertyrglyvalglncyspheserarg100105110tyrproasphismetlysglnhisaspphephelysseralametpro115120125gluglytyrvalglngluargthrilephephelysaspaspglyasn130135140tyrlysthrargalagluvallysphegluglyaspthrleuvalasn145150155160argilegluleulysglyileaspphelysgluaspglyasnileleu165170175glyhislysleuglutyrasntyrasnserhisasnvaltyrilemet180185190alaasplysglnlysasnglyilelysvalasnphelysilearghis195200205asnilegluaspglyservalglnleualaasphistyrglnglnasn210215220thrproileglyaspglyprovalleuleuproaspasnhistyrleu225230235240serthrglnseralaleuserlysaspproasnglulysargasphis245250255metvalleuleugluphevalthralaalaglyilethrleuglymet260265270aspgluleutyrlysleuglnglyglythrglyglyglymetglnile275280285leutrplyslystyrvallysgluasnpheglumetasnvalaspglu290295300cysglyilegluglnglyileproglyleuglytyrasntyrgluval305310315320leulysasnalavalilehistyrvalthrlysglytyrglythrphe325330335lyspheasnglylysvaltyrasnleulysglnglyaspilepheile340345350leuleulysglymetglnvalglutyrvalalaserileaspasppro355360365trpglutyrtyrtrpileglypheserglyserasnalaasnglutyr370375380leuasnargthrserilethrasnsercysvalalaasncysgluglu385390395400asnserlysileproglnileileleuasnmetcysgluileserlys405410415thrtyrasnproserargseraspaspileleuleuleulysgluleu420425430tyrserleuleutyralaleuilegluglupheprolyspropheglu435440445tyrlysasplysgluleuhisthrtyrileglnaspalaleuasnphe450455460ileasnserasntyrmethisserilethrvalglngluilealaasp465470475480tyrvalasnleuserargsertyrleutyrlysmetpheilelysasn485490495leuglyileserproglnargtyrleuileasnleuargmettyrlys500505510alathrleuleuleulysserthrlysleuproileglygluvalala515520525serservalglytyrseraspserleuleupheserlysthrpheser530535540lyshisphesermetserproleuasntyrargasnasnglnvalasn545550555560lysproserileglyglythrglyglyglyphegluleuglnmetala565570575serlysvaltyraspprogluglnarglysargmetilethrglypro580585590glntrptrpalaargcyslysglnmetasnvalleuaspserpheile595600605asntyrtyraspserglulyshisalagluasnalavalilepheleu610615620hisglyasnalathrsersertyrleutrparghisvalvalprohis625630635640ilegluprovalalaargcysileileproaspleuileglymetgly645650655lysserglylysserglyasnglysertyrargleuleuasphistyr660665670lystyrleuthralatrpphegluleuleuasnleuprolyslysile675680685ilephevalglyhisasptrpglyalaalaleualaphehistyrala690695700tyrgluhisglnaspargilelysalailevalhismetgluserval705710715720valaspvalileglusertrpaspglutrpproaspileglugluasp725730735ilealaleuilelysserglugluglyglulysmetvalleugluasn740745750asnphephevalgluthrvalleuproserlysilemetarglysleu755760765gluproglugluphealaalatyrleugluprophelysglulysgly770775780gluvalargargprothrleusertrpproarggluileproleuval785790795800lysglyglylysproaspvalvalglnilevalargasntyrasnala805810815tyrleuargalaseraspaspleuprolysleupheilegluserasp820825830proglyphepheserasnalailevalgluglyalalyslysphepro835840845asnthrgluphevallysvallysglyleuhispheleuglngluasp850855860alaproaspglumetglylystyrilelysserphevalgluargval865870875880leulysasnglugln885<210>27<211>879<212>prt<213>人工序列<220><223>lacb3<400>27metargglyserhishishishishishisglymetalasermetthr151015glyglyglnglnmetglyargaspleutyraspaspaspasplysasp202530argtrpglysergluphemetvalserlysglyglugluleuphethr354045glyvalvalproileleuvalgluleuaspglyaspvalasnglyhis505560lyspheservalserglygluglygluglyaspalathrtyrglylys65707580leuthrleulyspheilecysthrthrglylysleuprovalprotrp859095prothrleuvalthrthrleusertyrglyvalglncyspheserarg100105110tyrproasphismetlysglnhisaspphephelysseralametpro115120125gluglytyrvalglngluargthrilephephelysaspaspglyasn130135140tyrlysthrargalagluvallysphegluglyaspthrleuvalasn145150155160argilegluleulysglyileaspphelysgluaspglyasnileleu165170175glyhislysleuglutyrasntyrasnserhisasnvaltyrilemet180185190alaasplysglnlysasnglyilelysvalasnphelysilearghis195200205asnilegluaspglyservalglnleualaasphistyrglnglnasn210215220thrproileglyaspglyprovalleuleuproaspasnhistyrleu225230235240serthrglnseralaleuserlysaspproasnglulysargasphis245250255metvalleuleugluphevalthralaalaglyilethrleuglymet260265270aspgluleutyrlysleuglnglyglythrglyglyglymetglnile275280285leutrplyslystyrvallysgluasnpheglumetasnvalaspglu290295300cysglyilegluglnglyileproglyleuglytyrasntyrgluval305310315320leulysasnalavalilehistyrvalthrlysglytyrglythrphe325330335lyspheasnglylysvaltyrasnleulysglnglyaspilepheile340345350leuleulysglymetglnvalglutyrvalalaserileaspasppro355360365trpglutyrtyrtrpileglypheserglyserasnalaasnglutyr370375380leuasnargthrserilethrasnsercysvalalaasncysgluglu385390395400asnserlysileproglnileileleuasnmetcysgluileserlys405410415thrtyrasnproserargseraspaspileleuleuleulysgluleu420425430tyrserleuleutyralaleuilegluglupheprolyspropheglu435440445tyrlysasplysgluleuhisthrtyrileglnaspalaleuasnphe450455460ileasnserasntyrmethisserilethrvalglngluilealaasp465470475480tyrvalasnleuserargsertyrleutyrlysmetpheilelysasn485490495leuglyileserproglnargtyrleuileasnleuargmettyrlys500505510alathrleuleuleulysserthrlysleuproileglygluvalala515520525serservalglytyrseraspserleuleupheserlysthrpheser530535540lyshisphesermetserproleuasntyrargasnasnglnvalasn545550555560lysproserilephegluleuglnmetalaserlysvaltyrasppro565570575gluglnarglysargmetilethrglyproglntrptrpalaargcys580585590lysglnmetasnvalleuaspserpheileasntyrtyraspserglu595600605lyshisalagluasnalavalilepheleuhisglyasnalathrser610615620sertyrleutrparghisvalvalprohisilegluprovalalaarg625630635640cysileileproaspleuileglymetglylysserglylyssergly645650655asnglysertyrargleuleuasphistyrlystyrleuthralatrp660665670phegluleuleuasnleuprolyslysileilephevalglyhisasp675680685trpglyalaalaleualaphehistyralatyrgluhisglnasparg690695700ilelysalailevalhismetgluservalvalaspvalilegluser705710715720trpaspglutrpproaspileglugluaspilealaleuilelysser725730735glugluglyglulysmetvalleugluasnasnphephevalgluthr740745750valleuproserlysilemetarglysleugluprogluglupheala755760765alatyrleugluprophelysglulysglygluvalargargprothr770775780leusertrpproarggluileproleuvallysglyglylysproasp785790795800valvalglnilevalargasntyrasnalatyrleuargalaserasp805810815aspleuprolysleupheilegluseraspproglyphepheserasn820825830alailevalgluglyalalyslyspheproasnthrgluphevallys835840845vallysglyleuhispheleuglngluaspalaproaspglumetgly850855860lystyrilelysserphevalgluargvalleulysasnglugln865870875<210>28<211>879<212>prt<213>人工序列<220><223>lacb4<400>28metargglyserhishishishishishisglymetalasermetthr151015glyglyglnglnmetglyargaspleutyraspaspaspasplysasp202530argtrpglysergluphemetvalserlysglyglugluleuphethr354045glyvalvalproileleuvalgluleuaspglyaspvalasnglyhis505560lyspheservalserglygluglygluglyaspalathrtyrglylys65707580leuthrleulyspheilecysthrthrglylysleuprovalprotrp859095prothrleuvalthrthrleusertyrglyvalglncyspheserarg100105110tyrproasphismetlysglnhisaspphephelysseralametpro115120125gluglytyrvalglngluargthrilephephelysaspaspglyasn130135140tyrlysthrargalagluvallysphegluglyaspthrleuvalasn145150155160argilegluleulysglyileaspphelysgluaspglyasnileleu165170175glyhislysleuglutyrasntyrasnserhisasnvaltyrilemet180185190alaasplysglnlysasnglyilelysvalasnphelysilearghis195200205asnilegluaspglyservalglnleualaasphistyrglnglnasn210215220thrproileglyaspglyprovalleuleuproaspasnhistyrleu225230235240serthrglnseralaleuserlysaspproasnglulysargasphis245250255metvalleuleugluphevalthralaalaglyilethrleuglymet260265270aspgluleutyrlysleuglnmetglnileleutrplyslystyrval275280285lysgluasnpheglumetasnvalaspglucysglyilegluglngly290295300ileproglyleuglytyrasntyrgluvalleulysasnalavalile305310315320histyrvalthrlysglytyrglythrphelyspheasnglylysval325330335tyrasnleulysglnglyaspilepheileleuleulysglymetgln340345350valglutyrvalalaserileaspaspprotrpglutyrtyrtrpile355360365glypheserglyserasnalaasnglutyrleuasnargthrserile370375380thrasnsercysvalalaasncysglugluasnserlysileprogln385390395400ileileleuasnmetcysgluileserlysthrtyrasnproserarg405410415seraspaspileleuleuleulysgluleutyrserleuleutyrala420425430leuilegluglupheprolyspropheglutyrlysasplysgluleu435440445histhrtyrileglnaspalaleuasnpheileasnserasntyrmet450455460hisserilethrvalglngluilealaasptyrvalasnleuserarg465470475480sertyrleutyrlysmetpheilelysasnleuglyileserprogln485490495argtyrleuileasnleuargmettyrlysalathrleuleuleulys500505510serthrlysleuproileglygluvalalaserservalglytyrser515520525aspserleuleupheserlysthrpheserlyshisphesermetser530535540proleuasntyrargasnasnglnvalasnlysproserileglygly545550555560thrglyglyglyphegluleuglnmetalaserlysvaltyrasppro565570575gluglnarglysargmetilethrglyproglntrptrpalaargcys580585590lysglnmetasnvalleuaspserpheileasntyrtyraspserglu595600605lyshisalagluasnalavalilepheleuhisglyasnalathrser610615620sertyrleutrparghisvalvalprohisilegluprovalalaarg625630635640cysileileproaspleuileglymetglylysserglylyssergly645650655asnglysertyrargleuleuasphistyrlystyrleuthralatrp660665670phegluleuleuasnleuprolyslysileilephevalglyhisasp675680685trpglyalaalaleualaphehistyralatyrgluhisglnasparg690695700ilelysalailevalhismetgluservalvalaspvalilegluser705710715720trpaspglutrpproaspileglugluaspilealaleuilelysser725730735glugluglyglulysmetvalleugluasnasnphephevalgluthr740745750valleuproserlysilemetarglysleugluprogluglupheala755760765alatyrleugluprophelysglulysglygluvalargargprothr770775780leusertrpproarggluileproleuvallysglyglylysproasp785790795800valvalglnilevalargasntyrasnalatyrleuargalaserasp805810815aspleuprolysleupheilegluseraspproglyphepheserasn820825830alailevalgluglyalalyslyspheproasnthrgluphevallys835840845vallysglyleuhispheleuglngluaspalaproaspglumetgly850855860lystyrilelysserphevalgluargvalleulysasnglugln865870875<210>29<211>2622<212>dna<213>人工序列<220><223>核苷酸序列编码lacb1<400>29atgcggggttctcatcatcatcatcatcatggtatggctagcatgactggtggacagcaa60atgggtcgggatctgtacgacgatgacgataaggatcgatggggatccgaattcatggtg120agcaagggcgaggagctgttcaccggggtggtgcccatcctggtcgagctggacggcgac180gtaaacggccacaagttcagcgtgtccggcgagggcgagggcgatgccacctacggcaag240ctgaccctgaagttcatctgcaccaccggcaagctgcccgtgccctggcccaccctcgtg300accaccctgagctacggcgtgcagtgcttcagccgctaccccgaccacatgaagcagcac360gacttcttcaagtccgccatgcccgaaggctacgtccaggagcgcaccatcttcttcaag420gacgacggcaactacaagacccgcgccgaggtgaagttcgagggcgacaccctggtgaac480cgcatcgagctgaagggcatcgacttcaaggaggacggcaacatcctggggcacaagctg540gagtacaactacaacagccacaacgtctatatcatggccgacaagcagaagaacggcatc600aaggtgaacttcaagatccgccacaacatcgaggacggcagcgtgcagctcgccgaccac660taccagcagaacacccccatcggcgacggccccgtgctgctgcccgacaaccactacctg720agcacccagtccgccctgagcaaagaccccaacgagaagcgcgatcacatggtcctgctg780gagttcgtgaccgccgccgggatcactctcggcatggacgagctgtacaagctgcagatg840cagattctgtggaaaaaatacgtcaaagaaaactttgaaatgaacgtggatgaatgcggc900attgaacaaggcattccgggcctgggttataactacgaagttctgaaaaatgcagtcatc960cattatgtgaccaaaggctatggtacgtttaaattcaacggcaaagtctataatctgaaa1020cagggtgacattttcatcctgctgaaaggcatgcaagtggaatacgttgcgagcattgat1080gacccgtgggaatattactggatcggctttagtggttccaacgcgaatgaatatctgaac1140cgtaccagcattaccaacagctgcgtggccaactgtgaagaaaatagcaaaattccgcag1200attatcctgaacatgtgtgaaatctctaaaacctacaacccgtcacgctcggatgacatt1260ctgctgctgaaagaactgtattccctgctgtacgcactgatcgaagaatttccgaaaccg1320tttgaatacaaagataaagaactgcatacctacattcaggacgcgctgaacttcatcaac1380tcaaattatatgcactcgattacggtgcaagaaatcgccgattacgttaatctgagccgt1440tcttacctgtacaaaatgttcatcaaaaacctgggtatcagtccgcagcgttatctgatt1500aatctgcgcatgtacaaagcaaccctgctgctgaaatctacgaaactgccgatcggcgaa1560gttgcgagcagcgtgggttatagtgattccctgctgtttagtaaaaccttctccaaacac1620ttttcaatgtcgccgctgaactaccgcaacaatcaagttaataaaccgagcattttcgaa1680cttcagatggcttccaaggtgtacgaccccgagcaacgcaaacgcatgatcactgggcct1740cagtggtgggctcgctgcaagcaaatgaacgtgctggactccttcatcaactactatgat1800tccgagaagcacgccgagaacgccgtgatttttctgcatggtaacgctacctccagctac1860ctgtggaggcacgtcgtgcctcacatcgagcccgtggctagatgcatcatccctgatctg1920atcggaatgggtaagtccggcaagagcgggaatggctcatatcgcctcctggatcactac1980aagtacctcaccgcttggttcgagctgctgaaccttccaaagaaaatcatctttgtgggc2040cacgactggggggctgctctggcctttcactacgcctacgagcaccaagacaggatcaag2100gccatcgtccatatggagagtgtcgtggacgtgatcgagtcctgggacgagtggcctgac2160atcgaggaggatatcgccctgatcaagagcgaagagggcgagaaaatggtgcttgagaat2220aacttcttcgtcgagaccgtgctcccaagcaagatcatgcggaaactggagcctgaggag2280ttcgctgcctacctggagccattcaaggagaagggcgaggttagacggcctaccctctcc2340tggcctcgcgagatccctctcgttaagggaggcaagcccgacgtcgtccagattgtccgc2400aactacaacgcctaccttcgggccagcgacgatctgcctaagctgttcatcgagtccgac2460cctgggttcttttccaacgctattgtcgagggagctaagaagttccctaacaccgagttc2520gtgaaggtgaagggcctccacttcctccaggaggacgctccagatgaaatgggtaagtac2580atcaagagcttcgtggagcgcgtgctgaagaacgagcagtaa2622<210>30<211>2658<212>dna<213>人工序列<220><223>核苷酸序列编码lacb2<400>30atgcggggttctcatcatcatcatcatcatggtatggctagcatgactggtggacagcaa60atgggtcgggatctgtacgacgatgacgataaggatcgatggggatccgaattcatggtg120agcaagggcgaggagctgttcaccggggtggtgcccatcctggtcgagctggacggcgac180gtaaacggccacaagttcagcgtgtccggcgagggcgagggcgatgccacctacggcaag240ctgaccctgaagttcatctgcaccaccggcaagctgcccgtgccctggcccaccctcgtg300accaccctgagctacggcgtgcagtgcttcagccgctaccccgaccacatgaagcagcac360gacttcttcaagtccgccatgcccgaaggctacgtccaggagcgcaccatcttcttcaag420gacgacggcaactacaagacccgcgccgaggtgaagttcgagggcgacaccctggtgaac480cgcatcgagctgaagggcatcgacttcaaggaggacggcaacatcctggggcacaagctg540gagtacaactacaacagccacaacgtctatatcatggccgacaagcagaagaacggcatc600aaggtgaacttcaagatccgccacaacatcgaggacggcagcgtgcagctcgccgaccac660taccagcagaacacccccatcggcgacggccccgtgctgctgcccgacaaccactacctg720agcacccagtccgccctgagcaaagaccccaacgagaagcgcgatcacatggtcctgctg780gagttcgtgaccgccgccgggatcactctcggcatggacgagctgtacaagctgcagggt840ggtaccggaggcggcatgcagattctgtggaaaaaatacgtcaaagaaaactttgaaatg900aacgtggatgaatgcggcattgaacaaggcattccgggcctgggttataactacgaagtt960ctgaaaaatgcagtcatccattatgtgaccaaaggctatggtacgtttaaattcaacggc1020aaagtctataatctgaaacagggtgacattttcatcctgctgaaaggcatgcaagtggaa1080tacgttgcgagcattgatgacccgtgggaatattactggatcggctttagtggttccaac1140gcgaatgaatatctgaaccgtaccagcattaccaacagctgcgtggccaactgtgaagaa1200aatagcaaaattccgcagattatcctgaacatgtgtgaaatctctaaaacctacaacccg1260tcacgctcggatgacattctgctgctgaaagaactgtattccctgctgtacgcactgatc1320gaagaatttccgaaaccgtttgaatacaaagataaagaactgcatacctacattcaggac1380gcgctgaacttcatcaactcaaattatatgcactcgattacggtgcaagaaatcgccgat1440tacgttaatctgagccgttcttacctgtacaaaatgttcatcaaaaacctgggtatcagt1500ccgcagcgttatctgattaatctgcgcatgtacaaagcaaccctgctgctgaaatctacg1560aaactgccgatcggcgaagttgcgagcagcgtgggttatagtgattccctgctgtttagt1620aaaaccttctccaaacacttttcaatgtcgccgctgaactaccgcaacaatcaagttaat1680aaaccgagcattggtggtaccggaggcggcttcgaacttcagatggcttccaaggtgtac1740gaccccgagcaacgcaaacgcatgatcactgggcctcagtggtgggctcgctgcaagcaa1800atgaacgtgctggactccttcatcaactactatgattccgagaagcacgccgagaacgcc1860gtgatttttctgcatggtaacgctacctccagctacctgtggaggcacgtcgtgcctcac1920atcgagcccgtggctagatgcatcatccctgatctgatcggaatgggtaagtccggcaag1980agcgggaatggctcatatcgcctcctggatcactacaagtacctcaccgcttggttcgag2040ctgctgaaccttccaaagaaaatcatctttgtgggccacgactggggggctgctctggcc2100tttcactacgcctacgagcaccaagacaggatcaaggccatcgtccatatggagagtgtc2160gtggacgtgatcgagtcctgggacgagtggcctgacatcgaggaggatatcgccctgatc2220aagagcgaagagggcgagaaaatggtgcttgagaataacttcttcgtcgagaccgtgctc2280ccaagcaagatcatgcggaaactggagcctgaggagttcgctgcctacctggagccattc2340aaggagaagggcgaggttagacggcctaccctctcctggcctcgcgagatccctctcgtt2400aagggaggcaagcccgacgtcgtccagattgtccgcaactacaacgcctaccttcgggcc2460agcgacgatctgcctaagctgttcatcgagtccgaccctgggttcttttccaacgctatt2520gtcgagggagctaagaagttccctaacaccgagttcgtgaaggtgaagggcctccacttc2580ctccaggaggacgctccagatgaaatgggtaagtacatcaagagcttcgtggagcgcgtg2640ctgaagaacgagcagtaa2658<210>31<211>2640<212>dna<213>人工序列<220><223>核苷酸序列编码lacb3<400>31atgcggggttctcatcatcatcatcatcatggtatggctagcatgactggtggacagcaa60atgggtcgggatctgtacgacgatgacgataaggatcgatggggatccgaattcatggtg120agcaagggcgaggagctgttcaccggggtggtgcccatcctggtcgagctggacggcgac180gtaaacggccacaagttcagcgtgtccggcgagggcgagggcgatgccacctacggcaag240ctgaccctgaagttcatctgcaccaccggcaagctgcccgtgccctggcccaccctcgtg300accaccctgagctacggcgtgcagtgcttcagccgctaccccgaccacatgaagcagcac360gacttcttcaagtccgccatgcccgaaggctacgtccaggagcgcaccatcttcttcaag420gacgacggcaactacaagacccgcgccgaggtgaagttcgagggcgacaccctggtgaac480cgcatcgagctgaagggcatcgacttcaaggaggacggcaacatcctggggcacaagctg540gagtacaactacaacagccacaacgtctatatcatggccgacaagcagaagaacggcatc600aaggtgaacttcaagatccgccacaacatcgaggacggcagcgtgcagctcgccgaccac660taccagcagaacacccccatcggcgacggccccgtgctgctgcccgacaaccactacctg720agcacccagtccgccctgagcaaagaccccaacgagaagcgcgatcacatggtcctgctg780gagttcgtgaccgccgccgggatcactctcggcatggacgagctgtacaagctgcagggt840ggtaccggaggcggcatgcagattctgtggaaaaaatacgtcaaagaaaactttgaaatg900aacgtggatgaatgcggcattgaacaaggcattccgggcctgggttataactacgaagtt960ctgaaaaatgcagtcatccattatgtgaccaaaggctatggtacgtttaaattcaacggc1020aaagtctataatctgaaacagggtgacattttcatcctgctgaaaggcatgcaagtggaa1080tacgttgcgagcattgatgacccgtgggaatattactggatcggctttagtggttccaac1140gcgaatgaatatctgaaccgtaccagcattaccaacagctgcgtggccaactgtgaagaa1200aatagcaaaattccgcagattatcctgaacatgtgtgaaatctctaaaacctacaacccg1260tcacgctcggatgacattctgctgctgaaagaactgtattccctgctgtacgcactgatc1320gaagaatttccgaaaccgtttgaatacaaagataaagaactgcatacctacattcaggac1380gcgctgaacttcatcaactcaaattatatgcactcgattacggtgcaagaaatcgccgat1440tacgttaatctgagccgttcttacctgtacaaaatgttcatcaaaaacctgggtatcagt1500ccgcagcgttatctgattaatctgcgcatgtacaaagcaaccctgctgctgaaatctacg1560aaactgccgatcggcgaagttgcgagcagcgtgggttatagtgattccctgctgtttagt1620aaaaccttctccaaacacttttcaatgtcgccgctgaactaccgcaacaatcaagttaat1680aaaccgagcattttcgaacttcagatggcttccaaggtgtacgaccccgagcaacgcaaa1740cgcatgatcactgggcctcagtggtgggctcgctgcaagcaaatgaacgtgctggactcc1800ttcatcaactactatgattccgagaagcacgccgagaacgccgtgatttttctgcatggt1860aacgctacctccagctacctgtggaggcacgtcgtgcctcacatcgagcccgtggctaga1920tgcatcatccctgatctgatcggaatgggtaagtccggcaagagcgggaatggctcatat1980cgcctcctggatcactacaagtacctcaccgcttggttcgagctgctgaaccttccaaag2040aaaatcatctttgtgggccacgactggggggctgctctggcctttcactacgcctacgag2100caccaagacaggatcaaggccatcgtccatatggagagtgtcgtggacgtgatcgagtcc2160tgggacgagtggcctgacatcgaggaggatatcgccctgatcaagagcgaagagggcgag2220aaaatggtgcttgagaataacttcttcgtcgagaccgtgctcccaagcaagatcatgcgg2280aaactggagcctgaggagttcgctgcctacctggagccattcaaggagaagggcgaggtt2340agacggcctaccctctcctggcctcgcgagatccctctcgttaagggaggcaagcccgac2400gtcgtccagattgtccgcaactacaacgcctaccttcgggccagcgacgatctgcctaag2460ctgttcatcgagtccgaccctgggttcttttccaacgctattgtcgagggagctaagaag2520ttccctaacaccgagttcgtgaaggtgaagggcctccacttcctccaggaggacgctcca2580gatgaaatgggtaagtacatcaagagcttcgtggagcgcgtgctgaagaacgagcagtaa2640<210>32<211>2640<212>dna<213>人工序列<220><223>核苷酸序列编码lacb4<400>32atgcggggttctcatcatcatcatcatcatggtatggctagcatgactggtggacagcaa60atgggtcgggatctgtacgacgatgacgataaggatcgatggggatccgaattcatggtg120agcaagggcgaggagctgttcaccggggtggtgcccatcctggtcgagctggacggcgac180gtaaacggccacaagttcagcgtgtccggcgagggcgagggcgatgccacctacggcaag240ctgaccctgaagttcatctgcaccaccggcaagctgcccgtgccctggcccaccctcgtg300accaccctgagctacggcgtgcagtgcttcagccgctaccccgaccacatgaagcagcac360gacttcttcaagtccgccatgcccgaaggctacgtccaggagcgcaccatcttcttcaag420gacgacggcaactacaagacccgcgccgaggtgaagttcgagggcgacaccctggtgaac480cgcatcgagctgaagggcatcgacttcaaggaggacggcaacatcctggggcacaagctg540gagtacaactacaacagccacaacgtctatatcatggccgacaagcagaagaacggcatc600aaggtgaacttcaagatccgccacaacatcgaggacggcagcgtgcagctcgccgaccac660taccagcagaacacccccatcggcgacggccccgtgctgctgcccgacaaccactacctg720agcacccagtccgccctgagcaaagaccccaacgagaagcgcgatcacatggtcctgctg780gagttcgtgaccgccgccgggatcactctcggcatggacgagctgtacaagctgcagatg840cagattctgtggaaaaaatacgtcaaagaaaactttgaaatgaacgtggatgaatgcggc900attgaacaaggcattccgggcctgggttataactacgaagttctgaaaaatgcagtcatc960cattatgtgaccaaaggctatggtacgtttaaattcaacggcaaagtctataatctgaaa1020cagggtgacattttcatcctgctgaaaggcatgcaagtggaatacgttgcgagcattgat1080gacccgtgggaatattactggatcggctttagtggttccaacgcgaatgaatatctgaac1140cgtaccagcattaccaacagctgcgtggccaactgtgaagaaaatagcaaaattccgcag1200attatcctgaacatgtgtgaaatctctaaaacctacaacccgtcacgctcggatgacatt1260ctgctgctgaaagaactgtattccctgctgtacgcactgatcgaagaatttccgaaaccg1320tttgaatacaaagataaagaactgcatacctacattcaggacgcgctgaacttcatcaac1380tcaaattatatgcactcgattacggtgcaagaaatcgccgattacgttaatctgagccgt1440tcttacctgtacaaaatgttcatcaaaaacctgggtatcagtccgcagcgttatctgatt1500aatctgcgcatgtacaaagcaaccctgctgctgaaatctacgaaactgccgatcggcgaa1560gttgcgagcagcgtgggttatagtgattccctgctgtttagtaaaaccttctccaaacac1620ttttcaatgtcgccgctgaactaccgcaacaatcaagttaataaaccgagcattggtggt1680accggaggcggcttcgaacttcagatggcttccaaggtgtacgaccccgagcaacgcaaa1740cgcatgatcactgggcctcagtggtgggctcgctgcaagcaaatgaacgtgctggactcc1800ttcatcaactactatgattccgagaagcacgccgagaacgccgtgatttttctgcatggt1860aacgctacctccagctacctgtggaggcacgtcgtgcctcacatcgagcccgtggctaga1920tgcatcatccctgatctgatcggaatgggtaagtccggcaagagcgggaatggctcatat1980cgcctcctggatcactacaagtacctcaccgcttggttcgagctgctgaaccttccaaag2040aaaatcatctttgtgggccacgactggggggctgctctggcctttcactacgcctacgag2100caccaagacaggatcaaggccatcgtccatatggagagtgtcgtggacgtgatcgagtcc2160tgggacgagtggcctgacatcgaggaggatatcgccctgatcaagagcgaagagggcgag2220aaaatggtgcttgagaataacttcttcgtcgagaccgtgctcccaagcaagatcatgcgg2280aaactggagcctgaggagttcgctgcctacctggagccattcaaggagaagggcgaggtt2340agacggcctaccctctcctggcctcgcgagatccctctcgttaagggaggcaagcccgac2400gtcgtccagattgtccgcaactacaacgcctaccttcgggccagcgacgatctgcctaag2460ctgttcatcgagtccgaccctgggttcttttccaacgctattgtcgagggagctaagaag2520ttccctaacaccgagttcgtgaaggtgaagggcctccacttcctccaggaggacgctcca2580gatgaaatgggtaagtacatcaagagcttcgtggagcgcgtgctgaagaacgagcagtaa2640<210>33<211>765<212>prt<213>人工序列<220><223>gfp2-bgar1-171-rluc8<400>33metargglyserhishishishishishisglymetalasermetthr151015glyglyglnglnmetglyargaspleutyraspaspaspasplysasp202530argtrpglysergluphemetvalserlysglyglugluleuphethr354045glyvalvalproileleuvalgluleuaspglyaspvalasnglyhis505560lyspheservalserglygluglygluglyaspalathrtyrglylys65707580leuthrleulyspheilecysthrthrglylysleuprovalprotrp859095prothrleuvalthrthrleusertyrglyvalglncyspheserarg100105110tyrproasphismetlysglnhisaspphephelysseralametpro115120125gluglytyrvalglngluargthrilephephelysaspaspglyasn130135140tyrlysthrargalagluvallysphegluglyaspthrleuvalasn145150155160argilegluleulysglyileaspphelysgluaspglyasnileleu165170175glyhislysleuglutyrasntyrasnserhisasnvaltyrilemet180185190alaasplysglnlysasnglyilelysvalasnphelysilearghis195200205asnilegluaspglyservalglnleualaasphistyrglnglnasn210215220thrproileglyaspglyprovalleuleuproaspasnhistyrleu225230235240serthrglnseralaleuserlysaspproasnglulysargasphis245250255metvalleuleugluphevalthralaalaglyilethrleuglymet260265270aspgluleutyrlysleuglnmetglnileleutrplyslystyrval275280285lysgluasnpheglumetasnvalaspglucysglyilegluglngly290295300ileproglyleuglytyrasntyrgluvalleulysasnalavalile305310315320histyrvalthrlysglytyrglythrphelyspheasnglylysval325330335tyrasnleulysglnglyaspilepheileleuleulysglymetgln340345350valglutyrvalalaserileaspaspprotrpglutyrtyrtrpile355360365glypheserglyserasnalaasnglutyrleuasnargthrserile370375380thrasnsercysvalalaasncysglugluasnserlysileprogln385390395400ileileleuasnmetcysgluileserlysthrtyrasnproserarg405410415seraspaspileleuleuleulysgluleutyrserleuleutyrala420425430leuilegluglupheprolyspropheglutyrlysasplysgluleu435440445histhrphegluleuglnmetalaserlysvaltyraspproglugln450455460arglysargmetilethrglyproglntrptrpalaargcyslysgln465470475480metasnvalleuaspserpheileasntyrtyraspserglulyshis485490495alagluasnalavalilepheleuhisglyasnalathrsersertyr500505510leutrparghisvalvalprohisilegluprovalalaargcysile515520525ileproaspleuileglymetglylysserglylysserglyasngly530535540sertyrargleuleuasphistyrlystyrleuthralatrppheglu545550555560leuleuasnleuprolyslysileilephevalglyhisasptrpgly565570575alaalaleualaphehistyralatyrgluhisglnaspargilelys580585590alailevalhismetgluservalvalaspvalileglusertrpasp595600605glutrpproaspileglugluaspilealaleuilelyssergluglu610615620glyglulysmetvalleugluasnasnphephevalgluthrvalleu625630635640proserlysilemetarglysleugluproglugluphealaalatyr645650655leugluprophelysglulysglygluvalargargprothrleuser660665670trpproarggluileproleuvallysglyglylysproaspvalval675680685glnilevalargasntyrasnalatyrleuargalaseraspaspleu690695700prolysleupheilegluseraspproglyphepheserasnalaile705710715720valgluglyalalyslyspheproasnthrgluphevallysvallys725730735glyleuhispheleuglngluaspalaproaspglumetglylystyr740745750ilelysserphevalgluargvalleulysasnglugln755760765<210>34<211>744<212>prt<213>人工序列<220><223>gfp2-bgar1-150-rluc8<400>34metargglyserhishishishishishisglymetalasermetthr151015glyglyglnglnmetglyargaspleutyraspaspaspasplysasp202530argtrpglysergluphemetvalserlysglyglugluleuphethr354045glyvalvalproileleuvalgluleuaspglyaspvalasnglyhis505560lyspheservalserglygluglygluglyaspalathrtyrglylys65707580leuthrleulyspheilecysthrthrglylysleuprovalprotrp859095prothrleuvalthrthrleusertyrglyvalglncyspheserarg100105110tyrproasphismetlysglnhisaspphephelysseralametpro115120125gluglytyrvalglngluargthrilephephelysaspaspglyasn130135140tyrlysthrargalagluvallysphegluglyaspthrleuvalasn145150155160argilegluleulysglyileaspphelysgluaspglyasnileleu165170175glyhislysleuglutyrasntyrasnserhisasnvaltyrilemet180185190alaasplysglnlysasnglyilelysvalasnphelysilearghis195200205asnilegluaspglyservalglnleualaasphistyrglnglnasn210215220thrproileglyaspglyprovalleuleuproaspasnhistyrleu225230235240serthrglnseralaleuserlysaspproasnglulysargasphis245250255metvalleuleugluphevalthralaalaglyilethrleuglymet260265270aspgluleutyrlysleuglnmetglnileleutrplyslystyrval275280285lysgluasnpheglumetasnvalaspglucysglyilegluglngly290295300ileproglyleuglytyrasntyrgluvalleulysasnalavalile305310315320histyrvalthrlysglytyrglythrphelyspheasnglylysval325330335tyrasnleulysglnglyaspilepheileleuleulysglymetgln340345350valglutyrvalalaserileaspaspprotrpglutyrtyrtrpile355360365glypheserglyserasnalaasnglutyrleuasnargthrserile370375380thrasnsercysvalalaasncysglugluasnserlysileprogln385390395400ileileleuasnmetcysgluileserlysthrtyrasnproserarg405410415seraspaspileleuleuleulysgluleutyrserleuphegluleu420425430glnmetalaserlysvaltyraspprogluglnarglysargmetile435440445thrglyproglntrptrpalaargcyslysglnmetasnvalleuasp450455460serpheileasntyrtyraspserglulyshisalagluasnalaval465470475480ilepheleuhisglyasnalathrsersertyrleutrparghisval485490495valprohisilegluprovalalaargcysileileproaspleuile500505510glymetglylysserglylysserglyasnglysertyrargleuleu515520525asphistyrlystyrleuthralatrpphegluleuleuasnleupro530535540lyslysileilephevalglyhisasptrpglyalaalaleualaphe545550555560histyralatyrgluhisglnaspargilelysalailevalhismet565570575gluservalvalaspvalileglusertrpaspglutrpproaspile580585590glugluaspilealaleuilelysserglugluglyglulysmetval595600605leugluasnasnphephevalgluthrvalleuproserlysilemet610615620arglysleugluproglugluphealaalatyrleugluprophelys625630635640glulysglygluvalargargprothrleusertrpproarggluile645650655proleuvallysglyglylysproaspvalvalglnilevalargasn660665670tyrasnalatyrleuargalaseraspaspleuprolysleupheile675680685gluseraspproglyphepheserasnalailevalgluglyalalys690695700lyspheproasnthrgluphevallysvallysglyleuhispheleu705710715720glngluaspalaproaspglumetglylystyrilelysserpheval725730735gluargvalleulysasnglugln740<210>35<211>754<212>prt<213>人工序列<220><223>gfp2-bgar12-171-rluc8<400>35metargglyserhishishishishishisglymetalasermetthr151015glyglyglnglnmetglyargaspleutyraspaspaspasplysasp202530argtrpglysergluphemetvalserlysglyglugluleuphethr354045glyvalvalproileleuvalgluleuaspglyaspvalasnglyhis505560lyspheservalserglygluglygluglyaspalathrtyrglylys65707580leuthrleulyspheilecysthrthrglylysleuprovalprotrp859095prothrleuvalthrthrleusertyrglyvalglncyspheserarg100105110tyrproasphismetlysglnhisaspphephelysseralametpro115120125gluglytyrvalglngluargthrilephephelysaspaspglyasn130135140tyrlysthrargalagluvallysphegluglyaspthrleuvalasn145150155160argilegluleulysglyileaspphelysgluaspglyasnileleu165170175glyhislysleuglutyrasntyrasnserhisasnvaltyrilemet180185190alaasplysglnlysasnglyilelysvalasnphelysilearghis195200205asnilegluaspglyservalglnleualaasphistyrglnglnasn210215220thrproileglyaspglyprovalleuleuproaspasnhistyrleu225230235240serthrglnseralaleuserlysaspproasnglulysargasphis245250255metvalleuleugluphevalthralaalaglyilethrleuglymet260265270aspgluleutyrlysleuglnasnpheglumetasnvalaspglucys275280285glyilegluglnglyileproglyleuglytyrasntyrgluvalleu290295300lysasnalavalilehistyrvalthrlysglytyrglythrphelys305310315320pheasnglylysvaltyrasnleulysglnglyaspilepheileleu325330335leulysglymetglnvalglutyrvalalaserileaspaspprotrp340345350glutyrtyrtrpileglypheserglyserasnalaasnglutyrleu355360365asnargthrserilethrasnsercysvalalaasncysglugluasn370375380serlysileproglnileileleuasnmetcysgluileserlysthr385390395400tyrasnproserargseraspaspileleuleuleulysgluleutyr405410415serleuleutyralaleuilegluglupheprolyspropheglutyr420425430lysasplysgluleuhisthrphegluleuglnmetalaserlysval435440445tyraspprogluglnarglysargmetilethrglyproglntrptrp450455460alaargcyslysglnmetasnvalleuaspserpheileasntyrtyr465470475480aspserglulyshisalagluasnalavalilepheleuhisglyasn485490495alathrsersertyrleutrparghisvalvalprohisileglupro500505510valalaargcysileileproaspleuileglymetglylyssergly515520525lysserglyasnglysertyrargleuleuasphistyrlystyrleu530535540thralatrpphegluleuleuasnleuprolyslysileilepheval545550555560glyhisasptrpglyalaalaleualaphehistyralatyrgluhis565570575glnaspargilelysalailevalhismetgluservalvalaspval580585590ileglusertrpaspglutrpproaspileglugluaspilealaleu595600605ilelysserglugluglyglulysmetvalleugluasnasnphephe610615620valgluthrvalleuproserlysilemetarglysleugluproglu625630635640gluphealaalatyrleugluprophelysglulysglygluvalarg645650655argprothrleusertrpproarggluileproleuvallysglygly660665670lysproaspvalvalglnilevalargasntyrasnalatyrleuarg675680685alaseraspaspleuprolysleupheilegluseraspproglyphe690695700pheserasnalailevalgluglyalalyslyspheproasnthrglu705710715720phevallysvallysglyleuhispheleuglngluaspalaproasp725730735glumetglylystyrilelysserphevalgluargvalleulysasn740745750glugln<210>36<211>733<212>prt<213>人工序列<220><223>gfp2-bgar12-150-rluc8<400>36metargglyserhishishishishishisglymetalasermetthr151015glyglyglnglnmetglyargaspleutyraspaspaspasplysasp202530argtrpglysergluphemetvalserlysglyglugluleuphethr354045glyvalvalproileleuvalgluleuaspglyaspvalasnglyhis505560lyspheservalserglygluglygluglyaspalathrtyrglylys65707580leuthrleulyspheilecysthrthrglylysleuprovalprotrp859095prothrleuvalthrthrleusertyrglyvalglncyspheserarg100105110tyrproasphismetlysglnhisaspphephelysseralametpro115120125gluglytyrvalglngluargthrilephephelysaspaspglyasn130135140tyrlysthrargalagluvallysphegluglyaspthrleuvalasn145150155160argilegluleulysglyileaspphelysgluaspglyasnileleu165170175glyhislysleuglutyrasntyrasnserhisasnvaltyrilemet180185190alaasplysglnlysasnglyilelysvalasnphelysilearghis195200205asnilegluaspglyservalglnleualaasphistyrglnglnasn210215220thrproileglyaspglyprovalleuleuproaspasnhistyrleu225230235240serthrglnseralaleuserlysaspproasnglulysargasphis245250255metvalleuleugluphevalthralaalaglyilethrleuglymet260265270aspgluleutyrlysleuglnasnpheglumetasnvalaspglucys275280285glyilegluglnglyileproglyleuglytyrasntyrgluvalleu290295300lysasnalavalilehistyrvalthrlysglytyrglythrphelys305310315320pheasnglylysvaltyrasnleulysglnglyaspilepheileleu325330335leulysglymetglnvalglutyrvalalaserileaspaspprotrp340345350glutyrtyrtrpileglypheserglyserasnalaasnglutyrleu355360365asnargthrserilethrasnsercysvalalaasncysglugluasn370375380serlysileproglnileileleuasnmetcysgluileserlysthr385390395400tyrasnproserargseraspaspileleuleuleulysgluleutyr405410415serleuphegluleuglnmetalaserlysvaltyraspproglugln420425430arglysargmetilethrglyproglntrptrpalaargcyslysgln435440445metasnvalleuaspserpheileasntyrtyraspserglulyshis450455460alagluasnalavalilepheleuhisglyasnalathrsersertyr465470475480leutrparghisvalvalprohisilegluprovalalaargcysile485490495ileproaspleuileglymetglylysserglylysserglyasngly500505510sertyrargleuleuasphistyrlystyrleuthralatrppheglu515520525leuleuasnleuprolyslysileilephevalglyhisasptrpgly530535540alaalaleualaphehistyralatyrgluhisglnaspargilelys545550555560alailevalhismetgluservalvalaspvalileglusertrpasp565570575glutrpproaspileglugluaspilealaleuilelyssergluglu580585590glyglulysmetvalleugluasnasnphephevalgluthrvalleu595600605proserlysilemetarglysleugluproglugluphealaalatyr610615620leugluprophelysglulysglygluvalargargprothrleuser625630635640trpproarggluileproleuvallysglyglylysproaspvalval645650655glnilevalargasntyrasnalatyrleuargalaseraspaspleu660665670prolysleupheilegluseraspproglyphepheserasnalaile675680685valgluglyalalyslyspheproasnthrgluphevallysvallys690695700glyleuhispheleuglngluaspalaproaspglumetglylystyr705710715720ilelysserphevalgluargvalleulysasnglugln725730<210>37<211>279<212>prt<213>人工序列<220><223>a0a133mux6<400>37metglnileleutrplyslystyrvallysgluasnpheglumetasn151015valaspglucysglyilegluglnglyileproglyleuglytyrasn202530tyrgluvalleulysasnalavalilehistyrvalthrlysglytyr354045glythrphelyspheasnglylysvaltyrasnleulysglnglyasp505560ilepheileleuleulysglymetglnvalglutyrvalalaserile65707580aspaspprotrpglutyrtyrtrpileglypheserglyserasnala859095asnglutyrleuasnargthrserilethraspsercysvalalaasn100105110cysglugluasnserlysileproglnileileleuasnmetcysglu115120125ileserlysthrtyrasnproserargseraspaspileleuleuleu130135140lysgluleutyrserleuleutyralaleuilegluglupheprolys145150155160propheglutyrlysasplysgluleuhisthrtyrileglnaspala165170175leuasnpheileasnserasntyrmethisserilethrvalglnglu180185190ilealaasptyrvalasnleuserargsertyrleutyrlysmetphe195200205ilelysasnleuglyileserproglnargtyrleuileasnleuarg210215220mettyrlysalathrleuleuleulysglythrlysleuproilegly225230235240gluvalalaserservalglytyrseraspserleuleupheserlys245250255thrpheserlyshisphesermetserproleuasntyrargasnasn260265270glnvalasnlysproasnile275<210>38<211>279<212>prt<213>人工序列<220><223>b1v7n0<400>38metglnileleutrplyslystyrvallysgluasnpheglumetasn151015valaspglucysglyilegluglnglyileproglyleuglytyrlys202530tyrgluvalleulysasnalavalilehistyrvalthrlysglytyr354045glythrphelyspheasnglylysvaltyrthrleulysglnglyasp505560ilepheileleuleulysglymetglnvalasptyrvalalaserile65707580aspaspprotrpglutyrtyrtrpileglypheserglyserasnala859095asnglutyrleuasnargthrserilethrasnsercysvalalaasn100105110cysglugluasnserlysileproglnileileleuasnmetcysglu115120125ileserlysthrtyrasnproserargseraspaspileleuleuleu130135140lysgluleutyrserleuleutyralaleuilegluglupheprolys145150155160propheglutyrlysasplysgluleuhisthrtyrileglnaspala165170175leuasnpheileasnserasntyrmethisserilethrvalglnglu180185190ilealaasptyrvalasnleuserargsertyrleutyrlysmetphe195200205ilelysasnleuglyileserproglnargtyrleuileasnleuarg210215220mettyrlysalathrleuleuleulysglythrlysleuproilegly225230235240gluvalalaserservalglytyrseraspserleuleupheserlys245250255thrpheserlyshisphesermetserproleuasntyrargasnasn260265270glnvaltyrlysserserile275<210>39<211>279<212>prt<213>人工序列<220><223>a0a127egd8<400>39metglnileleutrplyslystyrilelysgluasnpheglumetasn151015valaspglucysglyilegluglnglyileproglyleuglytyrlys202530tyrgluvalleulysasnalavalilehistyrvalthrlysglytyr354045glythrphelyspheasnglylysvaltyrthrleulysglnglyasp505560ilepheileleuleulysglymetlysvalglutyrvalalaserile65707580aspaspprotrpglutyrtyrtrpileglypheserglyserasnala859095asnglutyrleuasnargthrserilethrasnsertyrvalalaasn100105110cysglulysasnserlysileproglnileileleuasnmetcysglu115120125ileserlysthrtyrasnproserserseraspaspileleuleuleu130135140lysgluleutyrserleuleutyrthrleuilegluglupheprolys145150155160propheasptyrlysasplysgluleuhisthrtyrileglnaspala165170175leuasnpheileasnserasntyrmetasnserilethrvalglnglu180185190ilealaasptyrvalasnleuserargsertyrleutyrlysmetphe195200205ilelysasnleuglyileserproglnargtyrleuileasnleuarg210215220mettyrlysalathrleuleuleulysglythrlysleuproilegly225230235240gluvalalaserargileglytyrseraspserleuleupheserlys245250255thrpheserlyshisphesermetserproleuasntyrargasnasn260265270glnvalasnlysproserile275<210>40<211>278<212>prt<213>人工序列<220><223>a0a1c6jub7<400>40metglnileleutrplyslystyrthrlysgluasnpheglumetasn151015valaspglucysglyilegluglnglyileproglypheglytyrlys202530tyrgluvalleulysasnalavalilehistyrileserlysglyasn354045glythrphelyspheasnasplysvaltyrasnleulysglnglyasp505560valpheileleuleulysglymetlysvalglutyrilealaserile65707580aspaspprotrpglutyrtyrtrpileglypheserglyserasnala859095asnglutyrleuasnargthrserileileaspsertyralaalaasn100105110cyslysgluaspserlysileproaspileileserasnmetcysglu115120125ileserlysthrtyrasnproserargseraspaspileleuleuleu130135140lysgluleutyrserleuleutyralapheilegluglupheprolys145150155160alapheglutyrlysasplysgluleuhisthrtyrileglngluala165170175ileasppheileasnserasntyrmetasnserilethrvalasnasp180185190ilealagluhisvalasnleuserargsertyrleutyrlysmetphe195200205metlysasnleulysvalserproglnlystyrleuileasnleuarg210215220mettyrlysalathrleuleuleulysasnthrargileproilegly225230235240gluvalalaserlysvalglytyrseraspserleuleupheserlys245250255thrpheserlystyrphesermetserproleuasntyrargasnasn260265270glnmetasnglyasnlys275<210>41<211>278<212>prt<213>人工序列<220><223>a0a174hyb7<400>41metglnileleutrplyslystyrthrlysgluasnpheglumetasn151015valaspglucysglyilegluglnglyileproglypheglytyrlys202530tyrgluvalleulysasnalavalilehistyrileserlysglyasn354045glythrphelyspheasnasplysvaltyrasnleulysglnglyasp505560valpheileleuleulysglymetlysvalglutyrilealaserile65707580aspaspprotrpglutyrtyrtrpileglypheserglyserasnala859095asnglutyrleuasnargthrserileileaspsertyralaalalys100105110cyslysglyaspserlysileproaspileileserasnmetcysglu115120125ileserlysthrtyrasnproserargseraspaspileleuleuleu130135140lysgluleutyrserleuleutyralapheilegluglupheprolys145150155160alapheglutyrlysasplysgluleuhisthrtyrileglngluala165170175ileasppheileasnserasntyrmetasnserilethrvalasnasp180185190ilealagluhisileasnleuserargsertyrleutyrlysmetphe195200205metlysasnleulysvalserproglnlystyrleuileasnleuarg210215220mettyrlysalathrleuleuleulysglythrargileproilegly225230235240gluvalalaserlysvalglytyrseraspserleuleupheserlys245250255thrpheserlystyrphesermetserproleuasntyrargasnasn260265270glnthrseraspasnlys275<210>42<211>278<212>prt<213>人工序列<220><223>a0a1c6ky47<400>42metglnileleutrplyslystyrilelysgluasnpheglumetasn151015valaspglucysglyilegluglnglyileproglypheglytyrlys202530tyrgluvalleulysasnservalilehistyrilethrlysglyhis354045glythrphelysileasnasplysleutyrasnleuglyglnglyasp505560valpheileleuleulysglymetlysvalglutyrmetalaserile65707580aspaspprotrpglutyrtyrtrpileglypheserglyserasnala859095asnglutyrleuasnargthrserileileaspsertyralaalathr100105110cyslysgluaspserlysileprocysileileserasnmetcysglu115120125ileserlysthrtyrasnprosercyscysaspaspileleuleuleu130135140lysgluleutyrserleuleutyralapheilegluglupheprolys145150155160alapheglutyrlysasplysgluleuhisthrtyrileglngluala165170175ileasnpheileasnserasntyrmetlysserilethrvalasnasp180185190ilealagluhisvalasnleuserargsertyrleutyrlysmetphe195200205metlysasnleulysvalserproglnlystyrleuileasnleuarg210215220mettyrlysalathrleuleuleulysglythrargileproilegly225230235240gluvalalaserlysvalglytyrseraspserleuleupheserlys245250255thrpheserlystyrphesermetserproleuasntyrargasnasn260265270glnthrseraspasnlys275<210>43<211>277<212>prt<213>人工序列<220><223>a0a174lzq7<400>43metglnileleutrparglystyrlyslysgluasnpheaspileasn151015leuaspglucysglyilegluhisglyileproglypheglytyrarg202530tyrlysvalleulysasnservalilehistyrvalileargglytyr354045glythrphelysvalasnasplysvaltyrasnleulysgluglyasp505560ilepheileleuleulysglymetaspvalglutyrmetalasermet65707580aspasnprotrpglutyrcystrpileglypheserglyserlysala859095aspglutyrleuasnargthrserileileaspserhisvalalaasn100105110cysasngluasnserlysileprocysileileleuasnilecysglu115120125ileserlysasntyrasnproserasnseraspaspileleuleuleu130135140asngluleutyrserleuleutyrgluleuileglyglupheprolys145150155160propheglutyrlysasplysgluilehislystyrileglnaspthr165170175ileasnpheileasnserasntyrmetasnasnilethrvalasnglu180185190ilealagluhisvalasnleuserargsertyrleutyrlysmetphe195200205ilelysasnleulysileserproglnlystyrleuileasnleuarg210215220mettyrlysalathrleuleuleulysasnthrlysleuproilegly225230235240gluilealaasnlysvalglytyralaaspserleuleupheserlys245250255thrpheserlystyrpheservalserproleuasntyrargasnasn260265270lysvalasnlysglu275<210>44<211>279<212>prt<213>人工序列<220><223>n9yr91<400>44metglnileleutrplyslystyrlysasnileasnpheaspserasn151015leuaspglucysglyilegluglnglythrproglythrglytyrlys202530tyrgluvalvallysasnalavalilehistyrileserlysglyser354045glyilephelysileasnasplysiletyrasnleulysargglyasp505560glypheileleuleulysglymethisvalglutyrileserserile65707580aspaspprotrplystyrtyrtrpvalglypheserglylysasnala859095asnglutyrleulysargthrserileileaspthrcysileileasn100105110pheserlyslysserlysvalproasnthrileileaspmetcysasn115120125ileserlyslystyrasnglnthrserseraspaspileleuleuleu130135140serlysleuhisleuleuleutyrtyrileserserglupheprolys145150155160serphelystyrtyrasnasnleualahisthrtyrileglngluala165170175valasppheileasnasnasntyrmetlysserilethrvalglnglu180185190valalaasnhisvalasnleuserargsertyrleutyrlysmetphe195200205ilelystyrleuglyglnserthrglnsertyrleuileasnilearg210215220mettyrlysserserleuleuleulysgluthrasnleuserileleu225230235240gluilealaasnlysvalglytyraspaspproglyleupheserlys245250255thrpheserlyshisphesermetseralaserlystyrarglysile260265270tyrglnlysasnlysthrasn275<210>45<211>279<212>prt<213>人工序列<220><223>a0a174i591<400>45metglnileleutrpasnlystyrlysserasnasnpheglualaasn151015leuaspglucysglyilegluglnglythrproglyalaglytyrasn202530tyrlysvalglulysasnalavalilehistyrileserlysglyser354045glythrphelysileasnasplysiletyrthrleulyslysglyasp505560glypheileleuleulysaspmetasnvalglutyrileproserile65707580aspaspprotrplystyrtyrtrpileglypheserglyglnserleu859095asnglutyrleulysargthrserileileaspsercysvalileasn100105110pheserlyslysserlysileproasnleuileileaspmetcysasn115120125ileserlyslystyraspglnthrserseraspaspileleuleuleu130135140serlysleuhisleuleuleutyrtyrileserserglupheprolys145150155160alaphelystyrasnasnasnleuthrhisthrtyrileglngluala165170175thrthrpheileasnasnasntyrmetasnproilethrvalglnglu180185190valalaasphisvalasnleuserargsertyrleutyrlysmetphe195200205ilelyshisleuglygluserthrglnsertyrleuileasnilearg210215220mettyrlysserserileleuleulysgluthrserleuserileala225230235240gluilealaasnlysvalglytyrseraspproleuleupheserlys245250255ilepheserlyshisphesermetseralaserlystyrarglysser260265270hisglnlysasnlyslyscys275<210>46<211>275<212>prt<213>人工序列<220><223>a0a2a7me67<400>46metglnileleutrplyslystyrlysilethrasnpheglumetasn151015leuaspglucysglyilegluglncysthrproglyilelystyrasn202530tyrgluvalvallysasnservalilehistyrilesergluglyglu354045glythrphelysileasnasnglnilepheaspleulyslysglyasp505560glypheileleuphelysglymetasnvalglutyrthralaserile65707580aspasnprotrplystyrtyrtrpvalglypheserglythrasnala859095asnglutyrleuhisargserserilepheaspasntyrileileasn100105110tyrglnserasnserlysileproserileilelysasnmetcysala115120125leuserlysthrtyraspglnasnserseraspaspileleuleuleu130135140asnlysleutyrtyrleuleutyrthrilethrglnglupheprolys145150155160propheglnleuvalasnasnleuthrhisthrtyrileglnglnser165170175ileasppheileasnserlystyralaglulysilethrvalglngln180185190ilealaaspasnvalasnleuserargsertyrleutyrlysleuphe195200205ilelystyrleuglygluserproglnlystyrleuleuasnleuarg210215220mettyrlysalathrleuleuleulysgluthraspleuserileser225230235240glnileserserasnileglytyraspaspproleuphepheserlys245250255thrpheserlyshispheserileseralaserglntyrarglysleu260265270tyrlyslys275<210>47<211>274<212>prt<213>人工序列<220><223>a0a2k4azl9<400>47metglnleuleutrplysmetphelyslysasnglnpheglualaasn151015ileaspglucysglyilegluileglythrproglnglyglytyrgln202530tyrgluvalthrlysproalavalleuhisvalvalmetserglythr354045glythrleuthrtyrasnglnlyslystyrthrleulysproglyasp505560leupheleuleucysargglymetlysvalhistyrgluserthrleu65707580aspgluprotrpthrtyrtyrtrpvalglypheserglylysleuala859095metasptyrleuasnargthrthrleutyrgluthrargvalilegln100105110asnglnglnthrserthrileargglnileiletyrglnmetcyshis115120125argserileasptyrasnprogluhisseraspaspileglnhismet130135140argaspleutyraspleuleutyralaleuhisglnhispheprolys145150155160prophehisvalvallysasnglulystyrserasnvalarggluala165170175ileargtyrileasnaspasntyrmethisglyileserilehisasp180185190valalalyshisvalasnvalserargsertyrleutyrlysmetphe195200205lyslyshisileaspglnserproglnhistyrleuilehisilearg210215220mettyrhisalaserglnleuphelysaspthraspleuglnsergln225230235240gluilealaaspargvalglytyrlysaspproleuleupheserarg245250255alaphelyslyshispheglyilethralathrglntyrarggluthr260265270hisgln<210>48<211>286<212>prt<213>人工序列<220><223>a0a166ppm9<400>48metglnleuleutrplysmetphelyslysasnglnpheglualaasn151015ileaspglucysglyilegluileglythrprohisglyglytyrgln202530tyrgluvalthrlysproalavalleuhisilevalmetserglythr354045glythrleuthrtyrasnglnlyslystyrthrleulysproglyasp505560leupheleuleucysargglymetasnvalhistyrgluserthrleu65707580aspgluprotrpthrtyrtyrtrpvalglypheserglylysleuval859095pheasptyrleuasnargthrserleutyrgluthrargvalilegln100105110asnglnprothrasnthrileargglnileiletyrargmetcysgln115120125argserileglutyralathrgluasnseraspaspileglnhismet130135140argaspleutyrgluleuleutyrgluleuhisglnhispheprolys145150155160prophehisvalvallysasngluargtyrserasnvalarggluala165170175ileargtyrileasnaspasntyrmethisalaileserileasnasp180185190valalalyshisvalasnvalserargsertyrleutyrlysmetphe195200205lyslyshisileaspglnserproglnhistyrleuilehisilearg210215220mettyrhisalaserglnleuphelysgluthraspleuglnsergln225230235240gluilealaaspargvalglytyrlysaspproleuleupheserarg245250255alaphelyslyshispheglyvalthralathrglntyrarggluglu260265270glnglnleuargilegluserthrleuaspasnglnlysarg275280285<210>49<211>274<212>prt<213>人工序列<220><223>a0a2t4r7g1<400>49metglnleuleutrplysleuphelysargasnhispheglualaasn151015ileaspglucysglyilegluileglythrproasnvalsertyrgln202530tyrthrvalvallysproalavalleuhisileilevalalaglythr354045glyserphethrtyrglnglnserthrtyrglnleulysserglyasp505560metpheleuleuglngluglymethisvalhistyrglualaserala65707580aspaspprotrpthrtyrhistrpvalglypheserglyasnleuala859095ileasptyrleulysargthrserleuileaspcysprovalvalmet100105110asnlysaspthrseraspileserlysvalmettyrglnilecysglu115120125argalailethrtyrgluthralathrseraspaspilehishisleu130135140seraspleutyrlysleuleupheleuilethrglncysalaprolys145150155160propheglulysgluhisasngluiletyrserservalglnaspala165170175valasptyrmetasnglnasntyrmettyralailethrileaspasp180185190ilealaglntyralalysvalserargsertyrleutyrlysleuphe195200205ilelystrpmetaspglnserproglnglntyrleuiletyrleuarg210215220leutyrhisalasersermetleulysthrthrserlysproilegln225230235240aspilealaglnalavalglytyrseraspproleuleupheserlys245250255alaphearglyshispheaspmetproproserthrtyrarglysval260265270tyrlys<210>50<211>274<212>prt<213>人工序列<220><223>a0a2a4hcu9<400>50metglnleuleutrplysmetphelyslysasnglnpheglualaasn151015ileaspglucysglyilegluileglythrproglnargglytyrgln202530tyrgluvalthrlysproalavalleuhisvalvalmetserglythr354045glythrleuthrtyrasnglnlyslystyrthrleulysproglyasp505560leupheleuleucysargglymetasnvalhistyrgluserthrleu65707580aspgluprotrpthrtyrtyrtrpvalglypheserglylysleuval859095pheasptyrleuasnargthrserleutyrgluthrargvalilegln100105110asnglnprothrasnalaileargglnileiletyrargmetcyshis115120125argserileglutyralathrgluasnseraspaspileglnhismet130135140argaspleutyrgluleuleutyrgluleuhisglnhispheprolys145150155160prophehisvalvallysasnglulystyrserasnvalarggluala165170175ileargtyrmetasnaspasntyrmethisalaileserileasnasp180185190valalalyshisvalasnvalserargsertyrleutyrlysmetphe195200205lyslyshisileaspglnserproglnhistyrleuilehisilearg210215220mettyrhisalaserglnleuphelysgluthraspleuglnsergln225230235240gluilealaaspargvalglytyrlysaspproleuleupheserarg245250255alaphelyslyshispheglyvalthralathrglntyrarggluglu260265270hisgln<210>51<211>271<212>prt<213>人工序列<220><223>a0a2t4ms83<400>51metglnleuleutrplysleuphelyslysasnhispheglualaasn151015ileaspglucysglyilegluileglythrproasnvalsertyrgln202530tyrthrvalvallysproalavalleuhisileilemetserglythr354045glythrphethrhisglnhisthrsertyrgluleulysalaglyasp505560metpheleuleuarggluglymetargvalhistyrglualaserthr65707580aspaspprotrpthrtyrhistrpvalglypheserglyasnleuala859095metasptyrleulysargthrthrleuileaspcysprovalvalleu100105110asnglnaspthrserlysleuserlysleumettyrglnilecysglu115120125argalailethrtyrgluthrthralaseraspaspilehishisleu130135140seraspleutyrlysleuleupheleuleuthrglnleuserprolys145150155160prophegluserhisproasngluiletyrserservalglnalaala165170175valasntyrmetasnglnhistyrmethisthrileserileaspasp180185190valalaglnhisalalysvalserargsertyrleutyrlysleuphe195200205metlystrpmetaspglnserproglnglntyrleuvaltyrleuarg210215220leutyrhisalasersermetleulysthrthrserlysproilegln225230235240gluilealaglnasnvalglytyrasnaspproleuleupheserlys245250255alaphearglyshispheaspmetproproserthrtyrarglys260265270<210>52<211>279<212>prt<213>人工序列<220><223>o33813<400>52metglnvalleutrplyslyspheglnlyslysleuileaspalaasn151015leualaglucysglyilegluileglyvalproasnvalglytyrasn202530tyrthrvalpheglnlysservalleuhisilevalthrglnglyglu354045glythrphesertyralaglygluthrtyrhisleuthralaglyasp505560ilepheleuleugluargglymetgluvalglutyrlysproserphe65707580serasnprotrpthrtyrtyrtrpvalglymetasnglylysglnile859095leuasntyrleuserargcysserilevalaspserhisvalileleu100105110glyglnaspthrthraspilelysasnileileglnleuilecyslys115120125leuserglnserilegluserasnasnserasnaspileleuilemet130135140glntyrleutyrglnleuvaltyrthrleuglnglulyspheprolys145150155160ilepheservalglnvalaspilevalasngluaspileglnhisala165170175valasppheileasnthrasntyrglnlyshisilethrvalgluasp180185190valalalysservalasnilethrargserhisleutyrlysleuphe195200205lyslysasnleuglycysserprolysglutyrleuthrtyrilearg210215220mettyrhisalaserglnleuleuilehisthrserthrleuileser225230235240aspileserargglnvalglytyrlysaspproleuleupheserlys245250255asnphethrlyshisphegluileseralaserglutyrarghishis260265270pheserileasnasnlysgln275<210>53<211>277<212>prt<213>人工序列<220><223>a0a1d4lkb2<400>53metglnileleutrplyslyspheglnlyslysleuvalaspalaasn151015leualaglucysglyilegluileglyileproasnvalglytyrasp202530tyrthrvalleuglnglnservalleuhisilevalthraspglyglu354045glyvalphelystyrasnasngluiletyrhisleulyslysglyasp505560ilepheleuleugluargglymetservallystyrmetproserphe65707580serasnprotrpthrtyrtyrtrpvalglyileasnglylysglnleu859095leuasntyrleumetargsertyrilevalaspthrhisvalileile100105110glylysaspthrglnaspilelysvalileileglnlysleucyslys115120125leualalysaspileglnserthrasnserasnaspileleuilemet130135140glntyrleutyrlysleuvaltyrthrpheglnasplyspheprolys145150155160thrphethrvalproleuaspilevalasngluaspileglnhisala165170175ileglupheileasnthrhistyrglnasnglyilethrilethrasp180185190valthrasnservalasnmetserargsertyrleutyrlysleuphe195200205lyslyshisleuasncysserprolyssertyrleuthrtyrilearg210215220mettyrhisalaserglnleuleuileasnserasnleuleuvalser225230235240gluileserglnargvalglytyrseraspproleuleupheserlys245250255asnphethrlyshisphegluileseralaseralatyrargphehis260265270pheglnglnasnlys275<210>54<211>314<212>prt<213>人工序列<220><223>a0a133qvv5<400>54metthrleuleuserilepheglnlysleutyrasnphevallystyr151015thrcystyrasnglyserileilegluarglysargvalargglnval202530glnvalphetrpthrlysleulyslysthrsertyrglualaglnval354045aspglucysglylysgluasnleutyrvalglyasnglytyrglutyr505560gluvalthrlysproalavalleuhisilevalthrglnglythrgly65707580thrphethrvalasnaspthrthrtyrhisleulyslysglyaspval859095pheleuleuleulysglymethisvallystyrhisalathrglyglu100105110thrprotrphistyrmettrpvalglypheserglythrhisalaile115120125serpheilethrargthrserleuseraspgluphevalleuleuasn130135140glnasnthrgluthrleuphelysleuilephelysilecysileleu145150155160alaasnserhisthrprogluaspthrhisaspileleuleulysile165170175argleuphegluleuleutyrpheleuthrglnglnasnglnlysglu180185190ilevalileproaspglnargglualathraspleulysaspalaleu195200205glutyrpheasnaspasnphelysserlysaspthrthrvalaspasn210215220alaalaglnvalalaasnmetserargserglnleutyrlysargphe225230235240lyslysglnpheglyalaserproserargtyrleuthraspleuarg245250255metalaphealaalagluglnleulysphethrasnlysthrvalgln260265270alailealaaspgluleuasntyraspvalserleualapheserlys275280285alapheserlystyrpheasncysproprothrglntyrarglysasn290295300tyrlysalaarglysalaleuglnserglu305310<210>55<211>289<212>prt<213>人工序列<220><223>a9qsr3<400>55metglutyrlysgluphehisglnasnpheleuaspileasnleuasp151015phevalglyasnglualathrileproasnpheserpheglyproala202530ilearggluasntyrvalilehistyrilethrserglyserglyarg354045tyrmetiletyrglyphegluhisglnleulysalaglyaspcysphe505560ileileproalaaspvalgluthrphetyrglnseraspalaleuthr65707580protrpalatyrtyrtrpleuglyleuserglyhisvalvalasnasp859095leuphealaargthralaleuaspasplysglytrpileleugluasn100105110valserlysthrglupheilegluhispheserlysileglnasnleu115120125ileseraspaspasplysthrvalaspleuaspileglnvalgluleu130135140phealaleumetlysserleuilethrleupheprolysserilethr145150155160gluhislysasnglnserasptyrtyralaglulysalatyrthrphe165170175ileasnglnasntyrserglnserilelysilelysaspvalleuala180185190hisvalmetileserargalatyrleuphethrilephelyshislys195200205tyrglyleuserproglnlystyrleuileaspleuargmetalalys210215220alaalametleuleuilehisseraspasnleuvalserglnileser225230235240glualavalglypheseraspserleuserpheserseralaphelys245250255lysargtyrglyvalserprothrlysphelysthrglnlyshisasp260265270asnleumetleugluthrleuasnasnmetasnleuasnargleulys275280285lys<210>56<211>139<212>prt<213>人工序列<220><223>a0a133mux6cbd<400>56asnpheglumetasnvalaspglucysglyilegluglnglyilepro151015glyleuglytyrasntyrgluvalleulysasnalavalilehistyr202530valthrlysglytyrglythrphelyspheasnglylysvaltyrasn354045leulysglnglyaspilepheileleuleulysglymetglnvalglu505560tyrvalalaserileaspaspprotrpglutyrtyrtrpileglyphe65707580serglyserasnalaasnglutyrleuasnargthrserilethrasp859095sercysvalalaasncysglugluasnserlysileproglnileile100105110leuasnmetcysgluileserlysthrtyrasnproserargserasp115120125aspileleuleuleulysgluleutyrserleu130135<210>57<211>139<212>prt<213>人工序列<220><223>b1v7n0cbd<400>57asnpheglumetasnvalaspglucysglyilegluglnglyilepro151015glyleuglytyrlystyrgluvalleulysasnalavalilehistyr202530valthrlysglytyrglythrphelyspheasnglylysvaltyrthr354045leulysglnglyaspilepheileleuleulysglymetglnvalasp505560tyrvalalaserileaspaspprotrpglutyrtyrtrpileglyphe65707580serglyserasnalaasnglutyrleuasnargthrserilethrasn859095sercysvalalaasncysglugluasnserlysileproglnileile100105110leuasnmetcysgluileserlysthrtyrasnproserargserasp115120125aspileleuleuleulysgluleutyrserleu130135<210>58<211>139<212>prt<213>人工序列<220><223>a0a127egd8cbd<400>58asnpheglumetasnvalaspglucysglyilegluglnglyilepro151015glyleuglytyrlystyrgluvalleulysasnalavalilehistyr202530valthrlysglytyrglythrphelyspheasnglylysvaltyrthr354045leulysglnglyaspilepheileleuleulysglymetlysvalglu505560tyrvalalaserileaspaspprotrpglutyrtyrtrpileglyphe65707580serglyserasnalaasnglutyrleuasnargthrserilethrasn859095sertyrvalalaasncysglulysasnserlysileproglnileile100105110leuasnmetcysgluileserlysthrtyrasnproserserserasp115120125aspileleuleuleulysgluleutyrserleu130135<210>59<211>139<212>prt<213>人工序列<220><223>a0a1c6jub7cbd<400>59asnpheglumetasnvalaspglucysglyilegluglnglyilepro151015glypheglytyrlystyrgluvalleulysasnalavalilehistyr202530ileserlysglyasnglythrphelyspheasnasplysvaltyrasn354045leulysglnglyaspvalpheileleuleulysglymetlysvalglu505560tyrilealaserileaspaspprotrpglutyrtyrtrpileglyphe65707580serglyserasnalaasnglutyrleuasnargthrserileileasp859095sertyralaalaasncyslysgluaspserlysileproaspileile100105110serasnmetcysgluileserlysthrtyrasnproserargserasp115120125aspileleuleuleulysgluleutyrserleu130135<210>60<211>139<212>prt<213>人工序列<220><223>a0a174hyb7cbd<400>60asnpheglumetasnvalaspglucysglyilegluglnglyilepro151015glypheglytyrlystyrgluvalleulysasnalavalilehistyr202530ileserlysglyasnglythrphelyspheasnasplysvaltyrasn354045leulysglnglyaspvalpheileleuleulysglymetlysvalglu505560tyrilealaserileaspaspprotrpglutyrtyrtrpileglyphe65707580serglyserasnalaasnglutyrleuasnargthrserileileasp859095sertyralaalalyscyslysglyaspserlysileproaspileile100105110serasnmetcysgluileserlysthrtyrasnproserargserasp115120125aspileleuleuleulysgluleutyrserleu130135<210>61<211>139<212>prt<213>人工序列<220><223>a0a1c6ky47cbd<400>61asnpheglumetasnvalaspglucysglyilegluglnglyilepro151015glypheglytyrlystyrgluvalleulysasnservalilehistyr202530ilethrlysglyhisglythrphelysileasnasplysleutyrasn354045leuglyglnglyaspvalpheileleuleulysglymetlysvalglu505560tyrmetalaserileaspaspprotrpglutyrtyrtrpileglyphe65707580serglyserasnalaasnglutyrleuasnargthrserileileasp859095sertyralaalathrcyslysgluaspserlysileprocysileile100105110serasnmetcysgluileserlysthrtyrasnprosercyscysasp115120125aspileleuleuleulysgluleutyrserleu130135<210>62<211>139<212>prt<213>人工序列<220><223>a0a174lzq7cbd<400>62asnpheaspileasnleuaspglucysglyilegluhisglyilepro151015glypheglytyrargtyrlysvalleulysasnservalilehistyr202530valileargglytyrglythrphelysvalasnasplysvaltyrasn354045leulysgluglyaspilepheileleuleulysglymetaspvalglu505560tyrmetalasermetaspasnprotrpglutyrcystrpileglyphe65707580serglyserlysalaaspglutyrleuasnargthrserileileasp859095serhisvalalaasncysasngluasnserlysileprocysileile100105110leuasnilecysgluileserlysasntyrasnproserasnserasp115120125aspileleuleuleuasngluleutyrserleu130135<210>63<211>139<212>prt<213>人工序列<220><223>n9yr91cbd<400>63asnpheaspserasnleuaspglucysglyilegluglnglythrpro151015glythrglytyrlystyrgluvalvallysasnalavalilehistyr202530ileserlysglyserglyilephelysileasnasplysiletyrasn354045leulysargglyaspglypheileleuleulysglymethisvalglu505560tyrileserserileaspaspprotrplystyrtyrtrpvalglyphe65707580serglylysasnalaasnglutyrleulysargthrserileileasp859095thrcysileileasnpheserlyslysserlysvalproasnthrile100105110ileaspmetcysasnileserlyslystyrasnglnthrserserasp115120125aspileleuleuleuserlysleuhisleuleu130135<210>64<211>139<212>prt<213>人工序列<220><223>a0a174i591cbd<400>64asnpheglualaasnleuaspglucysglyilegluglnglythrpro151015glyalaglytyrasntyrlysvalglulysasnalavalilehistyr202530ileserlysglyserglythrphelysileasnasplysiletyrthr354045leulyslysglyaspglypheileleuleulysaspmetasnvalglu505560tyrileproserileaspaspprotrplystyrtyrtrpileglyphe65707580serglyglnserleuasnglutyrleulysargthrserileileasp859095sercysvalileasnpheserlyslysserlysileproasnleuile100105110ileaspmetcysasnileserlyslystyraspglnthrserserasp115120125aspileleuleuleuserlysleuhisleuleu130135<210>65<211>139<212>prt<213>人工序列<220><223>a0a2a7me67cbd<400>65asnpheglumetasnleuaspglucysglyilegluglncysthrpro151015glyilelystyrasntyrgluvalvallysasnservalilehistyr202530ilesergluglygluglythrphelysileasnasnglnilepheasp354045leulyslysglyaspglypheileleuphelysglymetasnvalglu505560tyrthralaserileaspasnprotrplystyrtyrtrpvalglyphe65707580serglythrasnalaasnglutyrleuhisargserserilepheasp859095asntyrileileasntyrglnserasnserlysileproserileile100105110lysasnmetcysalaleuserlysthrtyraspglnasnserserasp115120125aspileleuleuleuasnlysleutyrtyrleu130135<210>66<211>139<212>prt<213>人工序列<220><223>a0a2k4azl9cbd<400>66glnpheglualaasnileaspglucysglyilegluileglythrpro151015glnglyglytyrglntyrgluvalthrlysproalavalleuhisval202530valmetserglythrglythrleuthrtyrasnglnlyslystyrthr354045leulysproglyaspleupheleuleucysargglymetlysvalhis505560tyrgluserthrleuaspgluprotrpthrtyrtyrtrpvalglyphe65707580serglylysleualametasptyrleuasnargthrthrleutyrglu859095thrargvalileglnasnglnglnthrserthrileargglnileile100105110tyrglnmetcyshisargserileasptyrasnprogluhisserasp115120125aspileglnhismetargaspleutyraspleu130135<210>67<211>139<212>prt<213>人工序列<220><223>a0a166ppm9cbd<400>67glnpheglualaasnileaspglucysglyilegluileglythrpro151015hisglyglytyrglntyrgluvalthrlysproalavalleuhisile202530valmetserglythrglythrleuthrtyrasnglnlyslystyrthr354045leulysproglyaspleupheleuleucysargglymetasnvalhis505560tyrgluserthrleuaspgluprotrpthrtyrtyrtrpvalglyphe65707580serglylysleuvalpheasptyrleuasnargthrserleutyrglu859095thrargvalileglnasnglnprothrasnthrileargglnileile100105110tyrargmetcysglnargserileglutyralathrgluasnserasp115120125aspileglnhismetargaspleutyrgluleu130135<210>68<211>139<212>prt<213>人工序列<220><223>a0a2t4r7g1cbd<400>68hispheglualaasnileaspglucysglyilegluileglythrpro151015asnvalsertyrglntyrthrvalvallysproalavalleuhisile202530ilevalalaglythrglyserphethrtyrglnglnserthrtyrgln354045leulysserglyaspmetpheleuleuglngluglymethisvalhis505560tyrglualaseralaaspaspprotrpthrtyrhistrpvalglyphe65707580serglyasnleualaileasptyrleulysargthrserleuileasp859095cysprovalvalmetasnlysaspthrseraspileserlysvalmet100105110tyrglnilecysgluargalailethrtyrgluthralathrserasp115120125aspilehishisleuseraspleutyrlysleu130135<210>69<211>139<212>prt<213>人工序列<220><223>a0a2a4hcu9cbd<400>69glnpheglualaasnileaspglucysglyilegluileglythrpro151015glnargglytyrglntyrgluvalthrlysproalavalleuhisval202530valmetserglythrglythrleuthrtyrasnglnlyslystyrthr354045leulysproglyaspleupheleuleucysargglymetasnvalhis505560tyrgluserthrleuaspgluprotrpthrtyrtyrtrpvalglyphe65707580serglylysleuvalpheasptyrleuasnargthrserleutyrglu859095thrargvalileglnasnglnprothrasnalaileargglnileile100105110tyrargmetcyshisargserileglutyralathrgluasnserasp115120125aspileglnhismetargaspleutyrgluleu130135<210>70<211>139<212>prt<213>人工序列<220><223>a0a2t4ms83cbd<400>70hispheglualaasnileaspglucysglyilegluileglythrpro151015asnvalsertyrglntyrthrvalvallysproalavalleuhisile202530ilemetserglythrglythrphethrhisglnhisthrsertyrglu354045leulysalaglyaspmetpheleuleuarggluglymetargvalhis505560tyrglualaserthraspaspprotrpthrtyrhistrpvalglyphe65707580serglyasnleualametasptyrleulysargthrthrleuileasp859095cysprovalvalleuasnglnaspthrserlysleuserlysleumet100105110tyrglnilecysgluargalailethrtyrgluthrthralaserasp115120125aspilehishisleuseraspleutyrlysleu130135<210>71<211>139<212>prt<213>人工序列<220><223>o33813cbd<400>71leuileaspalaasnleualaglucysglyilegluileglyvalpro151015asnvalglytyrasntyrthrvalpheglnlysservalleuhisile202530valthrglnglygluglythrphesertyralaglygluthrtyrhis354045leuthralaglyaspilepheleuleugluargglymetgluvalglu505560tyrlysproserpheserasnprotrpthrtyrtyrtrpvalglymet65707580asnglylysglnileleuasntyrleuserargcysserilevalasp859095serhisvalileleuglyglnaspthrthraspilelysasnileile100105110glnleuilecyslysleuserglnserilegluserasnasnserasn115120125aspileleuilemetglntyrleutyrglnleu130135<210>72<211>139<212>prt<213>人工序列<220><223>a0a1d4lkb2cbd<400>72leuvalaspalaasnleualaglucysglyilegluileglyilepro151015asnvalglytyrasptyrthrvalleuglnglnservalleuhisile202530valthraspglygluglyvalphelystyrasnasngluiletyrhis354045leulyslysglyaspilepheleuleugluargglymetservallys505560tyrmetproserpheserasnprotrpthrtyrtyrtrpvalglyile65707580asnglylysglnleuleuasntyrleumetargsertyrilevalasp859095thrhisvalileileglylysaspthrglnaspilelysvalileile100105110glnlysleucyslysleualalysaspileglnserthrasnserasn115120125aspileleuilemetglntyrleutyrlysleu130135<210>73<211>139<212>prt<213>人工序列<220><223>a0a133qvv5cbd<400>73asnphevallystyrthrcystyrasnglyserileilegluarglys151015argvalargglnvalglnvalphetrpthrlysleulyslysthrser202530tyrglualaglnvalaspglucysglylysgluasnleutyrvalgly354045asnglytyrglutyrgluvalthrlysproalavalleuhisileval505560thrglnglythrglythrphethrvalasnaspthrthrtyrhisleu65707580lyslysglyaspvalpheleuleuleulysglymethisvallystyr859095hisalathrglygluthrprotrphistyrmettrpvalglypheser100105110glythrhisalaileserpheilethrargthrserleuseraspglu115120125phevalleuleuasnglnasnthrgluthrleu130135<210>74<211>139<212>prt<213>人工序列<220><223>a9qsr3<400>74aspileasnleuaspphevalglyasnglualathrileproasnphe151015serpheglyproalailearggluasntyrvalilehistyrilethr202530serglyserglyargtyrmetiletyrglyphegluhisglnleulys354045alaglyaspcyspheileileproalaaspvalgluthrphetyrgln505560seraspalaleuthrprotrpalatyrtyrtrpleuglyleusergly65707580hisvalvalasnaspleuphealaargthralaleuaspasplysgly859095trpileleugluasnvalserlysthrglupheilegluhispheser100105110lysileglnasnleuileseraspaspasplysthrvalaspleuasp115120125ileglnvalgluleuphealaleumetlysser130135当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1