一种羰基还原酶突变体mut-AcCR(E144A/G152L)及其应用与编码基因的制作方法
本发明属于酶分子改造领域,具体涉及一种羰基还原酶突变体mut-accr(e144a/g152l)及其应用与编码该突变体的基因。
背景技术:
光学纯的手性醇及其衍生物是合成手性药物、液晶材料、香精香料、农药的重要手性中间体,在医药及其他化工领域占有重要位置。手性醇可以通过化学法和和生物法合成。化学合成法一般需要高温高压等严苛的条件;采用大量的有机试剂,环境污染严重;制备过程复杂,往往存在多次保护和去保护步骤;更重要的是化学方法得到的产物的对映体选择性低。相比于化学法,生物法一般在常温常压下反应,条件温和,设备要求低,环境污染小,底物专一性强,具有较高的立体选择性和区域选择性,产物对映体纯度高等优点。生物催化合成手性醇正在以强劲的势头在制备药物中间体中发挥越来越重要的作用。基于绿色化学和可持续发展的原则,生物催化制备手性醇是既绿色又可持续的合成途径。
羰基还原酶作为一种高效、高选择性的可用于不对称还原反应的生物催化剂,常用于制备具有光学活性的手性化合物。羰基还原酶在催化不对称还原反应时存在两个极具挑战性的问题,并且限制其进一步应用于不对称合成反应。第一,很多高价值手性醇的潜手性底物的溶解性很低或不可溶于酶的天然介质。这个缺点可以通过采用非水介质提高底物溶解度的方法得以解决,如采用有机溶剂、离子液体、超临界二氧化碳等单或双相反应介质进行生物催化反应。一部分羰基还原酶以这些溶剂为介质,在高底物浓度时亦表现出较高的酶活。但是,一些羰基还原酶的活性、选择性和稳定性在非水介质中往往会出现不同程度的降低。因此,在选择羰基还原酶非水反应介质时需要对反应介质进行筛选。第二,由于羰基还原酶具有严格的底物特异性、辅酶依赖性以及生理环境的不适应等特点,使羰基还原酶并非总能有效地完成一个特定的立体专一性合成反应,如在高浓度底物、高温、极端ph等环境下,羰基还原酶往往表现出活性低、选择性和稳定性差等不足。
随着基因组学和蛋白质组学的发展,科学家将更多的目光投入到蛋白质工程上。以定向进化和理性设计等为基础,对蛋白序列进行修饰,改变其结构,进而影响其催化性能,使其具有更好的区域和立体选择性、更高的稳定性和底物耐受性,为手性醇的合成提供更有效的方式。将蛋白质工程更好地应用于酶分子改造,可以使以酶或细胞为主导的生物催化在底物、介质、反应器等其他方面得到进一步的发展,使蛋白质工程取得的成果得到更有效、更持续性地应用。
本发明前期研究中,从醋酸杆菌acetobactersp.cctccm209061中克隆表达一种羰基还原酶。该羰基还原酶能够催化多种底物进行不对称还原反应,具有良好的立体选择性。但是,该羰基还原酶对于4’-氯苯乙酮等芳香族羰基化合物的酶活较低,对于这类底物的耐受性比较差,只有在底物浓度较低时才能达到良好的催化效果。
技术实现要素:
为了克服该酶活力低及底物耐受性差的缺点,本发明的首要目的在于提供一种活性和底物耐受性有明显提高的遵循反-prelog规则的羰基还原酶突变体mut-accr(e144a/g152l)。
本发明的另一个目的在于提供上述羰基还原酶突变体mut-accr(e144a/g152l)的基因及氨基酸序列。
本发明的再一个目的在于提供上述羰基还原酶突变体mut-accr(e144a/g152l)的应用。
本发明的目的通过以下技术方案实现。
一种羰基还原酶突变体mut-accr(e144a/g152l),其氨基酸序列如seqidno.1所示。
本发明还提供了编码上述羰基还原酶突变体mut-accr(e144a/g152l)的基因,其基因序列如seqidno.2所示。
进一步的,上述一种羰基还原酶突变体mut-accr(e144a/g152l)是将羰基化还原酶accr通过酶分子突变的手段获得羰基还原酶突变体mut-accr(e144a/g152l)。
进一步的,将accr的基因序列通过标准方法翻译出其氨基酸序列,以该序列在pdb数据库中搜索,选取同源性分别为53%、51%、51%和51%的4rf2、1zjy、1nxq和1zk3三级结构为模板,进行同源建模,并进行能量最小化,获得了羰基还原酶accr的三级结构模型。再采用拉氏图(ramachandranplot)和profile-3d对同源建模结果中的每个氨基酸残基结构合理性以及所建蛋白模型与蛋白氨基酸序列的匹配度进行评估。确定所建模型合理,可用于后续实验分析。
进一步的,将羰基还原酶accr三级结构与辅酶nadh进行对接,通过hotspot2.0预测羰基还原酶的突变热点,选取144e和152g位点作为突变位点。
进一步的,通过分子对接分析突变前后羰基还原酶与4’-氯苯乙酮之间的变化,分别将羰基还原酶accr、突变体mut-accr(e144a/g152l)与4’-氯苯乙酮进行分子对接,分析酶活性位点ser142、tyr155和辅酶nadh烟酰胺环c4位于底物4’-氯苯乙酮之间距离及作用力的变化。
进一步的,测定羰基还原酶突变体mut-accr(e144a/g152l)的活性,测定羰基还原酶突变体mut-accr(e144a/g152l)的酶学性质,通过酶活测定的方式研究突变体mut-accr(e144a/g152l)的最适温度和ph以及稳定性。
本发明还提供了上述一种羰基还原酶突变体mut-accr(e144a/g152l)在催化羰基化合物的不对称还原中的应用。
与现有技术相比,本发明具有如下优点和有益效果:本发明提供的一种羰基还原酶突变体mut-accr(e144a/g152l),克服了原有羰基还原酶酶活力低及底物耐受性差的缺点,具有较高的酶活力和底物耐受性。
附图说明
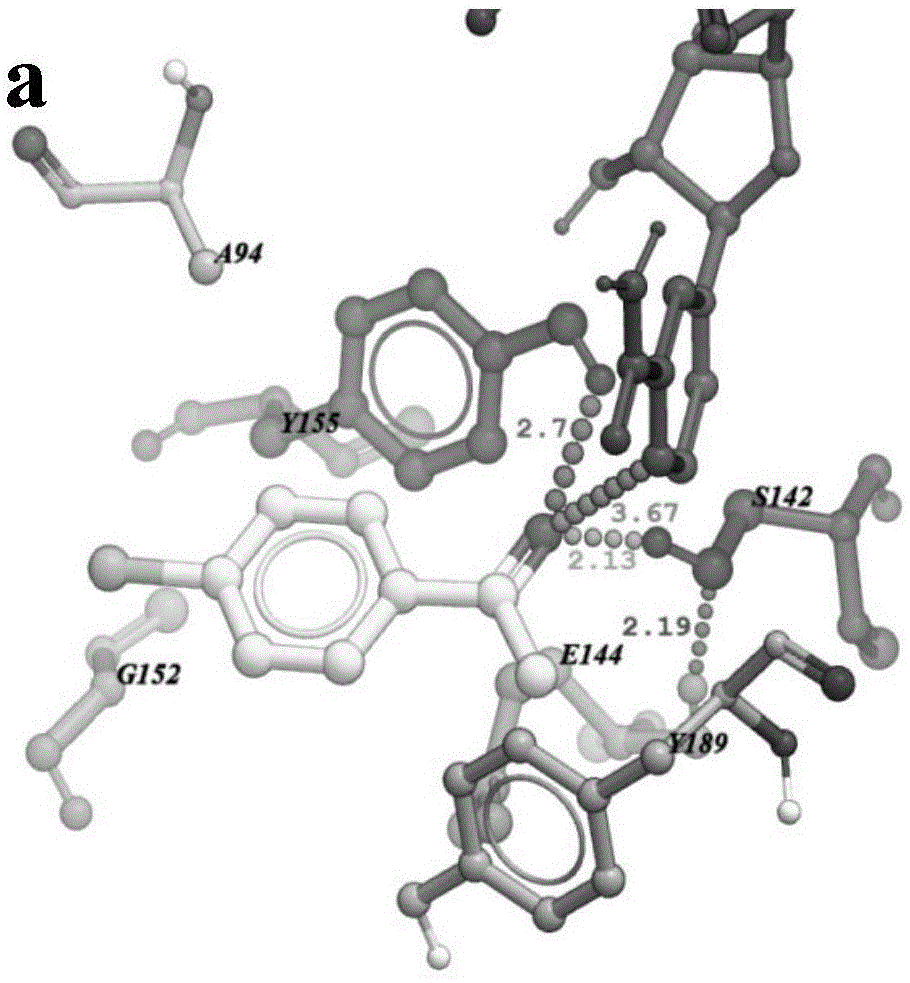
图1a、图1b为accr及mut-e144a/g152l与4’-氯苯乙酮的对接结果图。
图2a、图2b为温度对突变体mut-e144a/g152l活性及稳定性的影响对比图;
图3a、图3b为缓冲液ph对突变体mut-e144a/g152l活性及稳定性的影响对比图。
具体实施方式
下面结合实施例和附图对本发明作进一步详细的描述,但本发明的实施方式不限于此。需指出的是以下若有未特别详细说明之处,均是本领域技术人员可参照现有技术实现或理解的。
实施例1
将accr的基因序列通过标准方法翻译出其氨基酸序列,以该序列在pdb数据库中搜索,选取同源性分别为53%、51%、51%和51%的4rf2、1zjy、1nxq和1zk3(数据库中蛋白质序列的名称)三级结构为模板,进行同源建模,并进行能量最小化,获得了羰基还原酶accr的三级结构模型。进一步采用拉氏图(ramachandranplot)和profile-3d对同源建模结果中的每个氨基酸残基结构合理性以及所建蛋白模型与蛋白氨基酸序列的匹配度进行评估。确定所建模型合理,可用于后续实验分析。将羰基还原酶accr三级结构与辅酶nadh进行对接,通过hotspot2.0预测羰基还原酶的突变热点,选取144e和152g位点作为突变位点。
实施例2
通过分子对接分析突变前后羰基还原酶与4’-氯苯乙酮之间的变化;分别将羰基还原酶accr、突变体mut-accr(e144a/g152l)与4’-氯苯乙酮进行对接,分析酶活性位点ser142、tyr155和辅酶nadh烟酰胺环c4位于底物4’-苯乙酮之间距离及作用力的变化。结果见图1a、图1b所示accr及mut-e144a/g152l与4’-氯苯乙酮的对接结果图。从图1a、图1b可以看出突变后催化位点s142、y155及nadh烟酰胺环4位上的氢原子与4’-氯苯乙酮的羰基氧原子之间的距离减小明显,分别缩短
实施例3
采用primestarmaxdnapolymerase,通过进行pgex-accr全质粒扩增的方法获得含有突变体mut-accr(e144a/g152l)基因的质粒pgex-mut-e144a/g152l。
定点突变所用引物:位点e144a的突变引物为primer1:5'-atctgtcttccattgccggactgat-3';
primer2:5'-atcagtccggcaatggaagacagat-3';位点g152l的突变引物为primer3:5'-acccaatgttggccgcctataac-3';
primer4:5'-gttataggcggccaacattgggt-3'。
定点突变所采用的pcr扩增体系及反应条件如下:
聚合酶链式反应(pcr)扩增体系
pcr反应条件:
反应结束后,将反应产物用限制性内切酶dpni进行处理,作用于gm6a^tc位点,消化体系中的模板质粒。反应体系为:
将配制好的酶切反应体系置于37℃下孵育15min,即完成模板质粒的酶切过程。将酶切产物直接转化大肠杆菌dh5α。采用菌落pcr验证阳性转化子,提取质粒,进行测序。
本发明中从重组菌dh5α(pgex-mut-e144a/g152l)提取测序正确的重组质粒pgex-mut-e144a/g152l,并将其转化入大肠杆菌表达菌株bl21(de3)中,得到重组表达菌株bl21(de3)(pgex-mut-e144a/g152l)。
实施例4
挑取阳性转化子于4ml含100μg/ml氨苄的lb培养基中,于37℃、180r/min条件下过夜培养,取1ml培养液转接至50ml含100μg/ml氨苄的lb培养基中,37℃、180r/min培养至od600至约1.2,将培养温度将至20℃后加入终浓度0.4mmol/l的iptg,培养15-18h,4℃、8000r/min离心5min收集菌体。4℃、8000r/min离心5min,去除上清液,将菌体用0.85%生理盐水洗涤三次后称重,均匀分散于磷酸盐缓冲液(50mmol/l,ph6.5)中,配制成30mg/ml细胞悬浮液,置于4℃备用。采用超声破碎仪破碎细胞,超声破碎仪设置为功率350w,工作3s,间歇5s,超声时间设置为20min,整个破碎过程细胞悬液始终处于冰水浴中,保持低温环境,以免温度过高,使酶变性失活。超声破碎结束后,将破碎悬液置于离心机中,4℃、8000r/min离心30min,所得上清液即为重组羰基还原酶的粗酶提取液。
采用bio-radngcquesttm10中高压层析系统对重组羰基还原酶粗酶液进行纯化。将提取的粗酶液用0.22μm滤膜过滤后置于4℃备用。纯化的一般步骤为:用5倍柱体积的buffera(4.3mmna2hpo4,1.47mmkh2po4,137mmnacl,2.7mmkcl,ph7.3)预平衡bio-scaleminiprofinitygst预装柱(5ml,蛋白负载量500mg),流速5ml/min;预平衡结束后开始上样,采用泵上样,流速1ml/min,上样量约500mg蛋白;上样结束后,继续用buffera平衡柱子至基线与预平衡时基线相当;然后,用bufferb(4.3mmol/lna2hpo4,1.47mmkh2po4,637mmnacl,2.7mmkcl,ph7.3)继续冲洗柱子,去除比较牢固的杂蛋白;最后,用洗脱缓冲液bufferc(50mmtris-hcl,2.5g/lglutathione,ph8.0)洗脱重组蛋白accr,洗脱得到的溶液即为分离纯化后的单一重组蛋白accr。将还有重组蛋白accr的洗脱液置于透析袋中,用聚乙二醇20000,于4℃环境中浓缩,浓缩后的重组酶溶液于4℃保存。
重组羰基还原酶accr还原活性的测定条件:0.25mmol/lnadh,2ml磷酸盐缓冲液(50mmol/l,ph6.5),20mmol/l4’-氯苯乙酮,35℃孵育5min,加入适量酶液,检测反应体系在340nm紫外波长下,3min内吸光值的变化。
酶活定义:在上述条件下,每分钟催化氧化1μmolnadh的酶活力为1个酶活力单位,用u表示。
酶活力的计算公式如下:
酶活(u)=ew×v×103/(6220×1)
ew:1min内340nm处吸光度值的变化量;v:反应液的总体积,ml;6220:摩尔消光系数,l/mol/cm;1:光程距离,cm。
酶活测定结果见表1关于突变体mut-accr(e144a/g152l)对4’-氯苯乙酮的活性部分。
实施例5
通过酶活测定的方式研究突变体mut-accr(e144a/g152l)的最适温度和ph以及稳定性。重组羰基还原酶accr的最适反应温度通过测定不同温度时该酶的酶活力确定,即分别在25、30、35、40、45℃下,参考标准方法测定重组羰基还原酶accr的酶活。该酶的热稳定性测定,即将该酶置于不同的温度(25-45℃)孵育36h,定时取样,按照标准方法测定该酶不同时间点的酶活力。设定孵育前该酶的活力为100%相对酶活。每个温度的反应设置两个平行。结果见图2a温度对突变体mut-e144a/g152l活性及稳定性(图2b)的影响。由图2a、图2b可知,该突变体的最适反应温度为35℃,与突变前的重组accr的最适温度一致;在所研究的温度范围(25-45℃)内,该突变体的相对活性均保持在78%以上,特别是当温度高于35℃时,当温度为40-45℃时,该酶的相对活性在80%-92%之间,明显优于未突变accr(70-90%)。研究该突变酶的热稳定性发现在所考察的温度范围内,孵育相同时间,温度越低,突变体mut-e144a/g152l的相对酶活越好,热稳定性越好。
当孵育温度在40-45℃时,随着温度升高,以及孵育时间延长,突变体mute144a/g152l蛋白变性速度加快,残余活性下降明显,稳定性降低。当孵育温度在25-35℃范围内,孵育12h,相对酶活均能在80%以上;在25℃孵育24h,相对酶活仍有75%,36h后相对活性仍在60%左右。与未突变accr比较,该突变体在40-45℃相对酶活以及热稳定性在一定程度上有所提高。结合分子对接结果,底物与酶分子氢键作用的增强可能是其在所研究的温度范围内相对酶活增加的原因。另外,酶分子中的glu突变为leu,leu的疏水侧链使酶分子内残基的疏水结合增加,酶分子内部疏水作用增强,从而使蛋白结构更稳定。
重组羰基还原酶accr的最适反应ph通过测定不同ph时该酶的酶活力确定,即分别在ph5.0至8.0时,参考标准方法测定重组羰基还原酶accr的酶活。该酶的ph稳定性测定,即将该酶置于4℃,不同的ph范围内(6.0-8.0)孵育96h,定时取样,按照标准方法测定该酶不同时间点的酶活力。设定孵育前该酶的相对酶活力为100%。不同ph范围所用缓冲液分别为柠檬酸-磷酸盐缓冲液(ph4.5-8.0)、tris-hcl缓冲液(ph8.0-8.5)、甘氨酸-氢氧化钠缓冲液(ph8.5-9.5)。每个ph值设置两个平行。结果缓冲液ph对突变体mut-e144a/g152l活性(图3a)及稳定性(图3b)的影响。由图3a、图3b可知,突变前后,accr的最适ph并未改变,仍为6.5,缓冲液ph在6.0-8.0范围内,突变体mut-e144a/g152l的相对活性均在80%以上,具有较高的催化活性。研究其相对酶活大于80%的ph范围内的稳定性,发现其在ph6.0-8.0范围内比较稳定,特别是ph在6.5-7.5之间,96h仍残留约70%的相对酶活,具有良好的ph稳定性。
实施例7
羰基还原酶突变体mut-accr(e144a/g152l)催化不同底物的活性测定:将2ml含有不同底物(20mmol/l)的磷酸盐缓冲液(100mmol/l,ph6.5)分别加入到已做标记的相应的10ml具塞三角瓶中,一式两份,35℃孵育10min,再一定量纯化后的accr突变体,然后,参照标准方法测定该酶对不同底物的酶活力,将该酶对4’-氯苯乙酮的酶活力作为该酶的100%相对酶活。结果见表1所示羰基还原酶突变前后对于不同底物活性的比较。突变体mut-e144a/g152l除了对4’-氯苯乙酮的酶活有所提高外,对4’位被取代的苯乙酮的活性均有不同程度的提高。
表1
表2
e.e:对映体过量值
羰基还原酶突变体mut-accr(e144a/g152l)催化不同底物的产率及对映体选择性测定:将2ml含有不同底物(200mmol/l)、0.1mmol/lnadh和400mmol/l异丙醇的磷酸盐缓冲液(100mmol/l,ph6.5)中,分别加入到已做标记的相应的10ml具塞三角瓶中,一式两份。未突变羰基还原酶accr催化不同底物的产率测定,2ml含不同底物(50mmol/l)、0.1mmol/lnadh和150mmol/l异丙醇的磷酸盐缓冲液(100mmol/l,ph6.5)中,分别加入到已做标记的相应的10ml具塞三角瓶中,一式两份。35℃孵育10min后,一定量accr突变体。将反应瓶置于35℃气浴恒温振荡器中进行反应(200rpm),定时取样25μl,供gc或hplc分析。结果见表2羰基还原酶突变体mut-accr(e144a/g152l)催化潜手性羰基化合物的不对称还原所示。结果表明,突变酶对于底物的耐受性和催化反应的具有显著提高。在200mmol/l的底物浓度下(r)-3’-甲氧基苯乙酮的产率由未突变前61.6%(50mmol/l底物)提高至(200mmol/l底物)87.05%,提高了近26%;(r)-4’-甲氧基苯乙醇的产率提高至79.5%,与为突变酶催化该反应的产率相比提高了29%。
序列表
<110>华南理工大学
<120>一种羰基还原酶突变体mut-accr(e144a/g152l)及其应用与编码基因
<160>2
<170>siposequencelisting1.0
<210>1
<211>253
<212>prt
<213>羰基还原酶突变体(mut-accr(e144a/g152l))
<400>1
metthrargvalalaglylysvalalailevalserglyalaalaasn
151015
glyileglylysalathralaglnleuleualalysgluglyalalys
202530
valvalileglyaspleulysglugluaspglyglnlysalavalala
354045
gluilelysalaalaglyglyglualaalaphevallysleuasnval
505560
thraspglualaalatrplysalaalailegluglnthrleulysleu
65707580
tyrglyargleuaspilealavalasnasnalaglyilealatyrser
859095
glyservalgluserthrserleugluasptrpargargvalglnser
100105110
ileasnleuaspglyvalpheleuglythrglnvalalailegluala
115120125
metlyslysserglyglyglyserilevalasnleuserserileala
130135140
glyleuileglyaspprometleualaalatyrasnalaserlysgly
145150155160
glyvalargleuphethrlysseralaalaleuhiscysalalysser
165170175
glytyrlysileargvalasnservalhisproglytyriletrpthr
180185190
prometvalalaglyleuthrlysgluaspalaalaalaargglnlys
195200205
leuvalaspleuhisproileglyhisleuglygluproasnaspile
210215220
alatyrglyileleutyrleualaseraspgluserlysphevalthr
225230235240
glysergluleuvalileaspglyglytyrthralagln
245250
<210>2
<211>762
<212>dna
<213>羰基还原酶突变体(mut-accr(e144a/g152l))
<400>2
atgacacgtgtagcaggcaaggttgccattgtttctggggccgctaatggcattggcaag60
gcaaccgcacagcttttggccaaggaaggcgcaaaagttgttattggtgatttaaaagaa120
gaagatgggcagaaagctgttgcagaaattaaggcagcaggtggtgaagccgcatttgtc180
aaactgaatgtaacagatgaggctgcatggaaagccgctattgagcaaacgcttaagctt240
tatgggcggctggatattgcagtgaacaatgcaggcattgcgtattctggcagtgtagaa300
agcacatctctggaagattggcggcgcgttcagtctatcaatctggatggcgtgtttttg360
ggcacacaggtggctattgaggccatgaagaagtcgggcggtggatccattgtcaatctg420
tcttccattgccggactgattggggacccaatgttggccgcctataacgccagtaaaggt480
ggggtaaggctgtttacaaaatctgcggccctacattgcgccaaatctggatacaaaatt540
cgggtaaactcagtgcatcccggctatatctggacacctatggtggccggtttaacaaag600
gaagatgctgctgcacgccaaaagctggtggatctgcaccccattggccacttgggtgag660
cccaacgatattgcttacggtattttgtatcttgcctctgatgaatccaagtttgttaca720
gggagcgaactggtcattgatggtgggtacaccgcgcaataa762
- 还没有人留言评论。精彩留言会获得点赞!