突变孔的制作方法
本发明涉及Msp的突变形式。本发明还涉及使用Msp进行的多核苷酸表征。
背景技术:
目前需要涉及广泛应用范围的快速且廉价的多核苷酸(例如DNA或RNA)测序和识别技术。现有技术缓慢和昂贵,主要是因为它们依靠扩增技术来产生大量的多核苷酸,并且需要大量的专门的荧光化学物质用于信号检测。
跨膜孔(纳米孔)具有作为用于聚合物和各种小分子的直接的电生物传感器的巨大潜力。特别地,作为潜在DNA测序技术的纳米孔已成为最近焦点。
当跨越纳米孔施加电势时,当分析物(例如核苷酸)在桶状体中短暂驻留一段时间后,电流发生变化。核苷酸的纳米孔检测给出了已知标记和持续时间的电流变化。在链测序方法中,使单个多核苷酸链穿过孔,得到对核苷酸的识别。链测序可以包括使用多核苷酸结合蛋白来控制多核苷酸穿过孔的运动。
Msp的不同形式是来自耻垢分枝杆菌(Mycobacterium smegmatis)的孔蛋白。MspA为来自耻垢分枝杆菌的157kDa八聚体孔蛋白。野生型MspA不以允许DNA被表征或被测序的方式与DNA相互作用。MspA的结构以及其与DNA相互作用并表征DNA所需的修饰已有大量文献记载(Butler,2007,核酸的纳米孔分析,哲学博士论文,华盛顿大学;Gundlach,Proc Natl Acad Sci USA.2010Sep 14;107(37):16060-5.Epub 2010Aug 26;以及国际申请No.PCT/GB2012/050301(公开号为WO/2012/107778)。一些关键残基已经被鉴定和修饰以提供可观察到的DNA和MspA之间的相互作用,这对DNA表征或测序是必需的。特别地,前文的Butler教导了,需要从90、91和93的所有这三个位置去除负电荷,以提供可观察到的DNA和MspA之间的相互作用。
技术实现要素:
令人惊讶地,本发明人已证明,可使用在位置93保留有负电荷的MspA的新突变体来表征多核苷酸,如DNA,只要它们在帽状体形成区和/或桶状体形成区的内衬中具有减少的净负电荷即可。在位置93的负电荷不影响突变体捕获多核苷酸、与多核苷酸相互作用并区分多核苷酸中各核苷酸的能力。
因此,本发明提供了一种包含SEQ ID NO:2中所示序列的变体的Msp突变单体,其中所述变体:
(a)在位置90不包含天冬氨酸(D);
(b)在位置91不包含天冬氨酸(D);
(c)在位置93包含天冬氨酸(D)或谷氨酸(E);和
(d)包含一个或多个修饰,所述修饰降低单体的帽状体形成区和/或桶状体形成区中面向内的氨基酸的净负电荷。
本发明还提供:
-包含两个或更多个共价连接的MspA单体的构建体,其中至少一个所述单体是本发明的突变体单体。
-编码本发明的突变单体或本发明的构建体的多核苷酸。
-衍生自Msp的同源寡聚孔,包含本发明的相同突变体单体或本发明的相同构建体。
-衍生自Msp的异源寡聚孔,所述Msp包含至少一种本发明的突变单体或至少一种本发明的构建体。
-表征目标多核苷酸的方法,包括:
a)使多核苷酸与本发明的孔接触,使得多核苷酸移动通过孔;和
b)随着所述多核苷酸相对于所述孔移动,获取一个或多个测量值,其中所述测量值代表所述多核苷酸的一个或多个特征,由此表征该目标多核苷酸。
-用于表征目标多核苷酸的试剂盒,其包含(a)本发明的孔,和(b)膜的组分。
-用于表征样品中目标多核苷酸的设备,包括(a)多个本发明的孔,和(b)多个膜。
-表征目标多核苷酸的方法,包括:
a)使多核苷酸与本发明的孔、聚合酶和标记的核苷酸接触,使得当通过聚合酶将核苷酸加成至第一多核苷酸分析物时,磷酸盐标记的物质被顺序释放,其中所述磷酸盐物质含有对每个核苷酸特异性的标记;和
b)使用所述孔检测磷酸盐标记的物质,由此表征多核苷酸。
附图说明
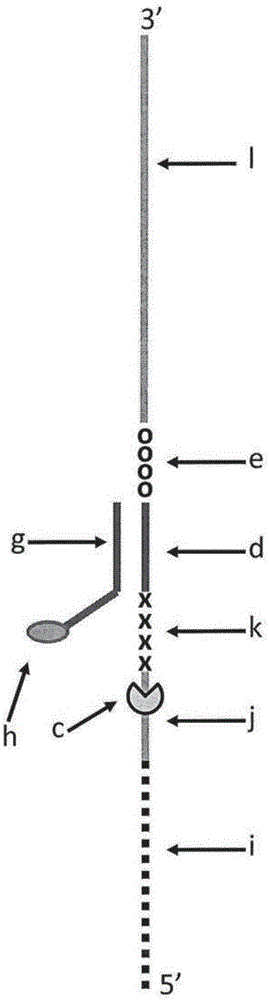
图1示出了实施例1中使用的DNA构建体X。DNA构建体X的区段a对应于SEQ ID NO:26。区段b对应于四个iSpC3间隔区。C对应于解旋酶T4Dda-E94C/C109A/C136A/A360C(具有突变E94C/C109A/C136A/A360C的SEQ ID NO:24),所述解旋酶可以结合到标记为a的区段。区段d对应于SEQ ID NO:27。区段e对应于四个5′-硝基吲哚。区段f对应于SEQ ID NO:28。区段g对应于SEQ ID NO:29,其3′端连接到6个iSp18间隔区,所述间隔区的另一端连接到两个胸腺嘧啶和3′胆固醇TEG(标记的h)。
图2示出了实施例1中使用的DNA构建体Y。DNA构建体X的区段i对应于连接到SEQ ID NO:3(标记的j)的5′端的25个iSpC3间隔区。区段j是解旋酶T4Dda-E94C/C109A/C136A/A360C(标记的c)可结合到的构建体Y的区域,区域k对应于四个iSp18间隔区。区段d对应于SEQ ID NO:27。区域e对应于四个5′-硝基吲哚。区域I对应于SEQ ID NO:4。区域g对应于SEQ ID NO:29,其3′端连接到6个iSp18间隔区,所述间隔区的另一端连接到两个胸腺嘧啶和3′胆固醇TEG(标记的h)。
图3示出了当解旋酶(T4Dda-E94C/C109A/C136A/A360C)控制DNA构建体X穿过MspA纳米孔MspA-(G75S/G77S/L88N/D90N/D91N/D118R/Q126R/D134R/E139K)8移位时的示例电流轨迹(所有三条轨迹的y轴坐标=电流(pA),x轴坐标=时间(s))。B和C部分显示了电流轨迹A的放大区域。
图4示出了当解旋酶(T4Dda-E94C/C109A/C136A/A360C)控制DNA构建体Y穿过MspA纳米孔MspA-(G75S/G77S/L88N/D90N/D91N/A96D/D118R/Q126R/D134R/E139K)8移位时的示例电流轨迹(这两条轨迹的y轴坐标=电流(pA),x轴坐标=时间(s))。B部分显示了电流轨迹A的放大区域。
图5示出了当解旋酶(T4Dda-E94C/C109A/C136A/A360C)控制DNA构建体X穿过MspA纳米孔MspA-(G75S/G77S/L88N/D90N/D91N/N102G/D118R/Q126R/D134R/E139K)8移位时的示例性电流轨迹(所有三条轨迹的y轴坐标=电流(pA),x轴坐标=时间(s))。B和C部分显示了电流轨迹A的放大区域。
图6示出了当解旋酶(T4Dda-E94C/C109A/C136A/A360C)控制DNA构建体X穿过MspA纳米孔MspA-(G75S/G77S/L88N/D90N/D91N/S103A/D118R/Q126R/D134R/E139K)8移位时的示例性电流轨迹(所有三条轨迹的y轴坐标=电流(pA),x轴坐标=时间(s))。B和C部分显示了电流轨迹A的放大区域。
图7示出了当解旋酶(T4Dda-E94C/C109A/C136A/A360C)控制DNA构建体X穿过MspA纳米孔MspA-(G75S/G77S/L88N/D90N/D91N/N108S/D118R/Q126R/D134R/E139K)8移位时的示例性电流轨迹(所有三条轨迹的y轴坐标=电流(pA),x轴坐标=时间(s))。B和C部分显示了电流轨迹A的放大区域。
图8示出了当解旋酶(T4Dda-E94C/C109A/C136A/A360C)控制DNA构建体X穿过MspA纳米孔MspA-(G75S/G77S/L88N/D90N/D91N/N108P/D118R/Q126R/D134R/E139K)8移位时的示例性电流轨迹(所有三条轨迹的y轴坐标=电流(pA),x轴坐标=时间(s))。B和C部分显示了电流轨迹A的放大区域。
图9示出了当解旋酶(T4Dda-E94C/C109A/C136A/A360C)控制DNA构建体X穿过MspA纳米孔MspA-(G75S/G77S/L88N/D90N/D91N/A96D/N108P/D118R/Q126R/D134R/E139K)8移位时的示例性电流轨迹(所有三条轨迹的y轴坐标=电流(pA),x轴坐标=时间(s))。B和C部分显示了电流轨迹A的放大区域。
图10示出了当解旋酶(T4Dda-E94C/C109A/C136A/A360C)控制DNA构建体X穿过MspA纳米孔MspA-(G75S/G77S/L88N/D90N/D91N/A96D/N108A/D118R/Q126R/D134R/E139K)8移位时的示例性电流轨迹(所有三条轨迹的y轴坐标=电流(pA),x轴坐标=时间(s))。B和C部分显示了电流轨迹A的放大区域。
图11示出了当解旋酶(T4Dda-E94C/C109A/C136A/A360C)控制DNA构建体X穿过MspA纳米孔MspA-(G75S/G77S/L88N/I89F/D90N/D91N/D118R/Q126R/D134R/E139K)8移位时的示例性电流轨迹(这两条轨迹的y轴坐标=电流(pA),x轴坐标=时间(s))。B部分显示了电流轨迹A的放大区域。
图12示出了当解旋酶(T4Dda-E94C/C109A/C136A/A360C)控制DNA构建体X穿过MspA纳米孔MspA-(G75S/G77S/D90N/D91N/D118R/Q126R/D134R/E139K)8移位时的示例性电流轨迹(所有三条轨迹的y轴坐标=电流(pA),x轴坐标=时间(s))。B和C部分显示了电流轨迹A的放大区域。
图13示出了当解旋酶(T4Dda-E94C/C109A/C136A/A360C)控制DNA构建体X穿过MspA纳米孔MspA-(G75S/G77S/L88K/D90N/D91N/I105E/D118R/Q126R/D134R/E139K)8移位时的示例性电流轨迹(这两条轨迹的y轴坐标=电流(pA),x轴坐标=时间(s))。B部分显示了电流轨迹A的放大区域。
图14示出了当解旋酶(T4Dda-E94C/C109A/C136A/A360C)控制DNA构建体X穿过MspA纳米孔MspA-(G75S/G77S/L88N/D90N/D91N/D118G/Q126R/D134R/E139K)8移位时的示例性电流轨迹(所有三条轨迹的y轴坐标=电流(pA),x轴坐标=时间(s))。B和C部分显示了电流轨迹A的放大区域。
图15示出了当解旋酶(T4Dda-E94C/C109A/C136A/A360C)控制DNA构建体X穿过MspA纳米孔MspA-(G75S/G77S/L88N/D90N/D91N/D118N/Q126R/D134R/E139K)8移位时的示例性电流轨迹(所有三条轨迹的y轴坐标=电流(pA),x轴坐标=时间(s))。B和C部分显示了电流轨迹A的放大区域。
图16示出了当解旋酶(T4Dda-E94C/C109A/C136A/A360C)控制DNA构建体X穿过MspA纳米孔MspA-(G75S/G77S/L88N/D90N/D91N/D118R/D134R/E139K)8移位时的示例性电流轨迹(所有三条轨迹的y轴坐标=电流(pA),x轴坐标=时间(s))。B和C部分显示了电流轨迹A的放大区域。
图17示出了当解旋酶(T4Dda-E94C/C109A/C136A/A360C)控制DNA构建体X通过MspA纳米孔MspA-(G75S/G77S/L88K/D90N/D91N/N108E/D118R/Q126R/D134R/E139K)8移位时的示例性电流轨迹(所有三条轨迹的y轴坐标=电流(pA),x轴坐标=时间(s))。B和C部分显示了电流轨迹A的放大区域。
图18示出了当解旋酶(T4Dda-E94C/C109A/C136A/A360C)控制DNA构建体Y穿过MspA纳米孔MspA-(G75S/G77S/L88N/D90N/D91N/T95E/P98K/D118R/Q126R/D134R/E139K)8移位时的示例性电流轨迹(所有三条轨迹的y轴坐标=电流(pA),x轴坐标=时间(s))。B和C部分显示了电流轨迹A的放大区域。
图19示出了当解旋酶(T4Dda-E94C/C109A/C136A/A360C)控制DNA构建体X穿过MspA纳米孔MspA-(G75S/G77S/L88N/D90N/D91N/D93N/D118R/Q126R/D134R/E139K)8移位时的示例性电流轨迹(所有三条轨迹的y轴坐标=电流(pA),x轴坐标=时间(s))。区段B和C部分显示了电流轨迹A的放大区域。
图20示出了野生型MspA纳米孔的卡通示意图。区域1对应于帽状体形成区并且包括1-72和122-184位残基。区域2对应于桶状体形成区域并且包括73-82和112-121位残基。区域3对应于收缩和环形(constriction and loops)区域并且包括83-111位残基。
序列表说明
SEQ ID NO:1示出了编码野生型MspA单体的密码子优化的多核苷酸序列。该突变体缺少信号序列。
SEQ ID NO:2示出了野生型MspA单体的成熟形式的氨基酸序列。该突变体缺少信号序列。
SEQ ID NO:3和4示出了实施例1中使用的多核苷酸序列。
SEQ ID NO:5至7示出了MspB、C和D的氨基酸序列。
SEQ ID NO:8示出了编码Phi29DNA聚合酶的多核苷酸序列。
SEQ ID NO:9示出了Phi29DNA聚合酶的氨基酸序列。
SEQ ID NO:10示出了来自大肠杆菌的sbcB基因的密码子优化的多核苷酸序列。它编码来自大肠杆菌的核酸外切酶I(EcoExo I)。
SEQ ID NO:11示出了来自大肠杆菌的核酸外切酶I(EcoExo I)的氨基酸序列。
SEQ ID NO:12示出了来自大肠杆菌的xthA基因的密码子优化的多核苷酸序列。它编码来自大肠杆菌的核酸外切酶III。
SEQ ID NO:13示出了来自大肠杆菌的核酸外切酶III的氨基酸序列。该酶从双链DNA(dsDNA)的一条链以3′-5′方向进行5′单磷酸核苷的分布消化。链上的酶的引发需要约4个核苷酸的5′突出。
SEQ ID NO:14示出了来自嗜热栖热菌(T.thermophilus)的recJ基因的密码子优化的多核苷酸序列。它编码来自嗜热栖热菌的RecJ酶(TthRecJ-cd)。
SEQ ID NO:15示出了来自嗜热栖热菌的RecJ酶的氨基酸序列(TthRecJ-cd)。该酶从ssDNA以5′-3′方向进行5′单磷酸核苷的进行性(processive)消化。链上的酶引发需要至少4个核苷酸。
SEQ ID NO:16示出了衍生自噬菌体λexo(redX)基因的密码子优化的多核苷酸序列。它编码噬菌体λ核酸外切酶。
SEQ ID NO:17示出了噬菌体λ核酸外切酶的氨基酸序列。该序列是组装成三聚体的三个相同亚基之一。该酶从dsDNA的一条链以5′-3′方向进行核苷酸的高度进行性消化(http://www.neb.com/nebecomm/products/productM0262.asp)。链上的酶引发优先需要具有5′磷酸的约4个核苷酸的5′突出。
SEQ ID NO:18示出了Hel308Mbu的氨基酸序列。
SEQ ID NO:19示出了Hel308Csy的氨基酸序列。
SEQ ID NO:20示出了Hel308Tga的的氨基酸序列。
SEQ ID NO:21示出了Hel308Mhu的氨基酸序列。
SEQ ID NO:22示出了TraI Eco的氨基酸序列。
SEQ ID NO:23示出了XPD Mbu的氨基酸序列。
SEQ ID NO:24示出了Dda 1993的氨基酸序列。
SEQ ID NO:25示出了Trwc Cba的氨基酸序列。
SEQ ID NO:26-29示出了实施例1中使用的多核苷酸序列。
具体实施方式
应该理解的是,所公开的产品和方法的不同应用可以适用于本领域中的特定需要。也应理解的是,这里使用的术语仅用于描述本发明的具体实施方案,并且不意在限制。
此外,除非内容另外明确指出,否则用于本说明书和所附权利要求书中的单数形式“一”,“一个”,和“所述”包括复数指代。因此,例如,涉及“多核苷酸”时包括两个或更多个多核苷酸,涉及“解旋酶”时包括两个或更多个解旋酶,涉及“单体”时包括两个或更多个单体,涉及“孔”时包括两个或更多个孔,等。
文本无论在上文还是下文中引用的所有出版物、专利和专利申请,以全文引用的方式纳入本文。
Msp突变单体
本发明提供Msp突变单体。Msp突变单体可用于形成本发明的孔。Msp突变单体是其序列从野生型Msp单体的序列变化而来并且保留形成孔的能力的单体。用于证实突变体单体形成孔的能力的方法是本领域公知的,并且在下面更详细地论述。
突变单体具有改进的多核苷酸读取性质,即显示改进的多核苷酸捕获和核苷酸鉴别。特别地,由突变单体构建的孔比野生型更容易捕获核苷酸和多核苷酸。此外,由突变单体构建的孔显示增加的电流范围,这使得更容易区分不同的核苷酸,以及减少的状态的差异,这增加了信噪比。此外,当多核苷酸移动通过由突变体构建的孔时,有助于形成电流的核苷酸的数目减少。这使得更容易鉴定当多核苷酸移动通过孔时观察到的电流与多核苷酸序列之间的直接关系。
本发明的突变体单体包含SEQ ID NO:2中所示序列的变体。SEQ ID NO:2是野生型MspA单体。SEQ ID NO:2的变体是具有这样的氨基酸序列的多肽:从SEQ ID NO:2的氨基酸序列变化而来并且保留其形成孔的能力。变体形成孔的能力可以使用本领域已知的任何方法测定。例如,变体可以与其它合适的亚基一起插入两亲性层中,并且可以确定其寡聚以形成孔的能力。用于将亚基插入膜例如两亲性层中的方法是本领域已知的。例如,亚基可以以纯化形式悬浮在含有三嵌段共聚物膜的溶液中,使得它扩散到膜并通过结合到膜并组装成功能状态而插入。或者,亚基可以使用M.A.Holden,H.Bayley.J.Am.Chem.Soc.2005,127,6502-6503和国际申请No.PCT/GB2006/001057(公开号WO 2006/100484)中所述的“拾取和放置”方法直接插入膜中。
位置90和91
在野生型MspA中,氨基酸90和91都是天冬氨酸(D)。每个单体中的这些氨基酸形成孔的内部收缩的一部分(图20)。变体在位置90不包含天冬氨酸(D)。变体优选在位置90不包含天冬氨酸(D)或谷氨酸(E)。变体在位置90优选不具有带负电荷的氨基酸。
变体在位置91不包含天冬氨酸(D)。变体优选在位置91不包含天冬氨酸(D)或谷氨酸(E)。变体在位置91优选不具有带负电荷的氨基酸。
变体在位置90和/或91优选包含丝氨酸(S)、谷氨酰胺(Q)、亮氨酸(L)、甲硫氨酸(M)、异亮氨酸(I)、丙氨酸(A)、缬氨酸(V)、甘氨酸(G)、苯丙氨酸(F)、色氨酸(W)、酪氨酸(Y)、组氨酸(H)、苏氨酸(T)、精氨酸(R)、赖氨酸(K)、天冬酰胺(N)或半胱氨酸。本发明可以想到在位置90和91这些氨基酸的任意组合。所述变体在位置90和/或位置91优选包含天冬酰胺(N)。所述变体在位置90和位置91更优选包含天冬酰胺(N)。这些氨基酸优选通过取代在位置90和/或91插入。
位置93
在野生型MspA中,氨基酸93是天冬氨酸(D)。每个单体中的这一氨基酸也形成孔的内部收缩的一部分(图20)。
变体在位置93包含天冬氨酸(D)或谷氨酸(E)。因此,变体在位置93具有负电荷。谷氨酸(E)优选通过取代引入。
帽状体形成区
在野生型MspA中,氨基酸1至72和122至184形成孔的帽状体(图20)。在这些氨基酸中,V9,Q12,D13,R14,T15,W40,I49,P53,G54,D56,E57,E59,T61,E63,Y66,Q67,I68,F70,P123,I125,Q126,E127,V128,A129,T130,F131,S132,V133,D134,S136,G137,E139,V144,H148,T150,V151,T152,F163,R165,I167,S169,T170和S173面向内进入孔的通道中。
桶状体形成区
在野生型MspA中,氨基酸73至82和112至121形成孔的桶状体(图20)。在这些氨基酸中,S73,G75,G77,N79,S81,G112,S114,S116,D118和G120面向内进入孔的通道中。S73,G75,G77,N79,S81面向内进入下游的链中,并且G112,S114,S116,D118和G120面向内进入上游的链中。
减少的净负电荷
变体包含一个或多个修饰,所述修饰减少单体的帽状体形成区和/或桶状体形成区中的面向内的氨基酸的净负电荷。变体优选包含两个或更多个修饰,所述修饰减少单体的帽状体形成区和桶状体形成区中的面向内的氨基酸的净负电荷。
变体可以包含任何数量的修饰,例如1个或多个,2个或多个,3个或多个,4个或多个,5个或多个,6个或多个,7个或多个,8个或多个,9个或多个,10个或更多,15个或更多个,20个或更多个,30个或更多个,或40个或更多个修饰。
净负电荷可以通过本领域已知的任何方式减少。净负电荷以不干扰突变单体形成孔的能力的方式减少。这可以如上所述测量。
帽状体形成区和/或桶状体形成区中面向内的氨基酸的净负电荷减少。这意味着帽状体形成区和/或桶状体形成区中面向内的氨基酸包含比SEQ ID NO:2中更少的带负电荷的氨基酸和/或包含比SEQ ID NO:2中更多的带正电荷的氨基酸。所述一个或多个修饰可导致帽状体形成区和/或桶状体形成区中面向内的氨基酸中产生净正电荷。
净电荷可以使用本领域已知的方法测量。例如,等电点可以用于限定帽状体形成区和/或桶状体形成区中的面向内的氨基酸的净电荷。
所述一个或多个修饰优选是带负电荷的氨基酸的一个或多个缺失。去除一个或多个带负电荷的氨基酸减少帽状体形成区和/或桶状体形成区中面向内的氨基酸的净负电荷。带负电荷的氨基酸是具有净负电荷的氨基酸。带负电荷的氨基酸包括但不限于天冬氨酸(D)和谷氨酸(E)。从蛋白质例如MspA单体缺失氨基酸的方法是本领域熟知的。
所述一个或多个修饰优选是用一个或多个带正电荷的、不带电荷的、非极性的和/或芳香族的氨基酸取代带负电荷的氨基酸中的一个或多个。带正电荷的氨基酸是具有净正电荷的氨基酸。带正电荷的氨基酸(一个或多个)可以是天然存在的或非天然存在的。带正电荷的氨基酸(一个或多个)可以是合成的或修饰的。例如,具有净正电荷的修饰的氨基酸可以特别设计而用于本发明。对氨基酸的许多不同类型的修饰是本领域公知的。
优选的天然存在的带正电荷的氨基酸包括但不限于组氨酸(H),赖氨酸(K)和精氨酸(R)。H,K和/或R的任何数目和组合可以取代为帽状体形成区和/或桶状体形成区中面向内的氨基酸。
不带电荷的氨基酸、非极性氨基酸和/或芳香族氨基酸可以是天然存在的或非天然存在的。它们可以是合成的或修饰的。未带电荷的氨基酸没有净电荷。合适的不带电荷的氨基酸包括但不限于半胱氨酸(C)、丝氨酸(S)、苏氨酸(T)、甲硫氨酸(M)、天冬酰胺(N)和谷氨酰胺(Q)。非极性氨基酸具有非极性侧链。合适的非极性氨基酸包括但不限于甘氨酸(G)、丙氨酸(A)、脯氨酸(P)、异亮氨酸(I)、亮氨酸(L)和缬氨酸(V)。芳香族氨基酸具有芳香族侧链。合适的芳族氨基酸包括但不限于组氨酸(H)、苯丙氨酸(F)、色氨酸(W)和酪氨酸(Y)。任何数目和组合的这些氨基酸可以取代成帽状体形成区和/或桶状体形成区中的面向内的氨基酸。
所述一个或多个带负电荷的氨基酸优选用丙氨酸(A)、缬氨酸(V)、天冬酰胺(N)或甘氨酸(G)取代。优选的取代包括但不限于D用A取代,D用V取代,D用N取代和D用G取代。
所述一个或多个修饰优选是引入一个或多个带正电荷的氨基酸。正电荷的引入降低了净负电荷。所述一个或多个带正电荷的氨基酸可以通过添加或取代而引入。任何氨基酸可以被带正电荷的氨基酸取代。一个或多个不带电荷的氨基酸、非极性氨基酸和/或芳香族氨基酸可以用一个或多个带正电荷的氨基酸取代。可以引入任何数量的带正电荷的氨基酸。
野生型MspA在位置126包含极性谷氨酰胺(Q)。所述一个或多个修饰优选减少位置126处的净负电荷。所述一个或多个修饰优选增加位置126的净正电荷。这可以通过用带正电荷的氨基酸替换位置126处的极性氨基酸或者相邻或附近的面向内的氨基酸来实现。变体优选地在位置126包含带正电荷的氨基酸。所述变体优选地在位置123,125,127和128中的一个或多个处包含带正电荷的氨基酸。所述变体在位置123,125,127和128可以包含任何数量和组合的带正电荷的氨基酸。带正电荷的氨基酸(一个或多个)可以通过添加或取代引入。
所述一个或多个修饰优选是引入一个或多个带正电荷的氨基酸,该氨基酸中和一个或多个带负电荷的氨基酸。对负电荷的中和减少了净负电荷。所述一个或多个带正电荷的氨基酸可以通过添加或取代而引入。任何氨基酸可以用带正电荷的氨基酸取代。一个或多个不带电荷的氨基酸、非极性氨基酸和/或芳香族氨基酸可以被一个或多个带正电荷的氨基酸取代。可以引入任何数目的带正电荷的氨基酸。该数目通常与帽状体形成区和/或桶状体形成区中面向内的氨基酸中的带负电荷的氨基酸的数目相同。
所述一个或多个带正电荷的氨基酸可以在帽状体形成区和/或桶状体形成区中的任何位置引入,只要它们中和所述一个或多个带负电荷的氨基酸的负电荷即可。为了有效中和负电荷,在变体中引入的每个带正电荷的氨基酸和其要中和的带负电荷的氨基酸之间通常有5个或更少的氨基酸。在变体中引入的每个带正电荷的氨基酸和其要中和的带负电荷的氨基酸之间优选具有4个或更少、3个或更少或者2个或更少的氨基酸。在变体中引入的每个带正电荷的氨基酸和其要中和的带负电荷的氨基酸之间更优选具有两个氨基酸。在变体中每个带正电荷的氨基酸最优选在邻近其要中和的带负电荷的氨基酸位置引入。
所述一个或多个带正电荷的氨基酸可以在帽状体形成区和/或桶状体形成区中的面向内的氨基酸中的任何位置处引入,只要它们中和一个或多个带负电荷的氨基酸的负电荷即可。为了有效中和负电荷,在引入的每个带正电荷的氨基酸和其要中和的带负电荷的氨基酸之间通常有5个或更少的面内向的氨基酸。在引入的每个带正电荷的氨基酸和其要中和的带负电荷的氨基酸之间优选有4个或更少、3个或更少或者2个或更少的面内向的氨基酸。在引入的每个带正电荷的氨基酸和其要中和的带负电荷的氨基酸之间更优选具有一个面内向的氨基酸。每个带正电荷的氨基酸最优选在邻近其要中和的带负电荷的氨基酸的面向内的位置处引入。
野生型MspA在位置118和134包含天冬氨酸(D),在位置139包含谷氨酸(E)。每个单体中的氨基酸118存在于孔的桶状体内(图20)。变体优选在位置114,116,120,123,70,73,75,77和79中的一个或多个位置包含带正电荷的氨基酸。在一个或多个这些位置的正电荷中和位置118处的负电荷。带正电荷的氨基酸可以存在于114,116,120,123,70,73,75,77和79中任何数目的位置和位置组合处。氨基酸可以通过添加或取代而引入。
每个单体中的氨基酸134和139是帽状体的一部分(图20)。变体在位置129,132,136,137,59,61和63中的一个或多个处包含带正电荷的氨基酸。在这些位置中的一个或多个处的正电荷中和位置134处的负电荷。带正电荷的氨基酸可以存在于位置129,132,136,137,59,61和63中任何数目的位置和位置组合处。氨基酸可以通过添加或取代而引入。
所述变体优选在位置137,138,141,143,45,47,49和51中的一个或多个处包含带正电荷的氨基酸。在这些位置的一个或多个处的正电荷中和位置139处的负电荷。带正电荷的氨基酸可以存在于位置137,138,141,143,45,47,49和51中任何数目的位置和位置组合处。氨基酸可以通过添加或取代而引入。
位置118,126,134和139
所述一个或多个修饰优选减少位置118,126,134和139中的一个或多个处的净负电荷。所述一个或多个修饰优选减少118;126;134;139;118和126;118和134;118和139;126和134;126和139;134和139;118,126和134;118,126和139;118,134和139;126,134和139;或118,126,134和139处的净负电荷。
所述变体优选在位置118,126,134和139中的一个或多个处不包含天冬氨酸(D)或谷氨酸(E)。所述变体优选在上文公开的位置118,126,134和139的任何组合处不包含天冬氨酸(D)或谷氨酸(E)。所述变体更优选在位置118,126,134和139中的一个或多个位置,例如上文公开的位置118,126,134和139的任何组合,包含精氨酸(R)、甘氨酸(G)或天冬酰胺(N)。所述变体最优选包含D118R,Q126R,D134R和E139K。
用于引入或取代天然存在的氨基酸的方法是本领域熟知的。例如,可以通过在编码突变单体的多核苷酸中的相关位置处,用精氨酸(CGT)的密码子替换甲硫氨酸(ATG)的密码子,用精氨酸(R)取代甲硫氨酸(M)。然后可以如下所述表达所述多核苷酸。
引入或取代非天然存在的氨基酸的方法也是本领域熟知的。例如,非天然存在的氨基酸可以通过在用于表达突变单体的IVTT系统中包括合成的氨酰基-tRNA来引入。或者,它们可以通过在大肠杆菌中表达突变单体而引入,所述大肠杆菌在那些特定氨基酸的合成(即非天然存在的)类似物的存在下对特定氨基酸是营养缺陷型的。如果使用部分肽合成制备突变单体,它们也可以通过裸连接(naked ligation)产生。
所述一个或多个修饰优选是一个或多个带负电荷的氨基酸的一个或多个化学修饰,所述化学修饰中和它们的负电荷。例如,所述一个或多个带负电荷的氨基酸可以与碳二亚胺反应。
其它修饰
所述变体优选包含以下中的一个或多个:
(a)位置75处的丝氨酸(S);
(b)位置77处的丝氨酸(S);和
(c)位置88处的天冬酰胺(N)或赖氨酸(K)。
所述变体可以包括(a)至(c)中的任意数目及组合,包括(a),(b),(c),(a)和(b),(b)和(c),(a)和(c),以及(a),(b)和(c)。所述变体优选包含G75S,G77S和L88N。
所述变体最优选包含G75S,G77S,L88N,D90N,D91N,D118R,Q126R,D134R和E139K。
所述变体优选还包含以下中的一种或多种:
(d)位置89处的苯丙氨酸(F);
(e)位置95处的谷氨酸(E)和位置98处的赖氨酸(K);
(f)位置96处的天冬氨酸(D);
(g)位置102处的甘氨酸(G);
(h)位置103处的丙氨酸(A);和
(i)位置108处的丙氨酸(A)、丝氨酸(S)或脯氨酸(P)。
所述变体可以包括(d)至(i)中的任何数目及其组合。
变体
除了上文讨论的特定突变之外,所述变体可以包括其他突变。在SEQ ID NO:2的氨基酸序列的整个长度上,基于氨基酸同一性,变体将优选与该序列至少50%同源。更优选地,基于氨基酸同一性,所述变体可以是至少55%,至少60%,至少65%,至少70%,至少75%,至少80%,至少85%,至少90%,且更优选至少95%,97%或99%在整个序列上与SEQ ID NO:2的氨基酸序列同源。在100个或更多个,例如125,150,175或200个或更多个连续氨基酸的片段(stretch)上,可以具有至少80%,例如至少85%,90%或95%的氨基酸同一性(“严格同源性(hard homology)”)。
可用本领域的标准方法测定同源性。例如,UWGCG包提供了用于计算同源性的BESTFIT程序,例如使用其默认设置(Devereux等人(1984)Nucleic Acids Research 12,p387-395)。可用PILEUP和BLAST算法计算同源性或比对序列(line up sequence)(例如鉴定等同残基或相应序列(通常基于其默认设置)),例如Altschul S.F.(1993)J Mol Evol36:290-300;Altschul,S.F等人(1990)JMol Biol 215:403-10中所述。用于执行BLAST分析的软件可通过国家生物技术信息中心(http://www.ncbi.nlm.nih.gov/)公开获得。
SEQ ID NO:2是野生型MspA单体的成熟形式。与MspA相比,变体可以包含MspB、C或D单体中的任何突变。MspB、C和D的成熟形式示于SEQ ID NO:5至7中。特别地,所述变体可以包含存在于MspB:A138P中的以下取代。所述变体可以包含存在于MspC中的一个或多个下列取代:A96G,N102E和A138P。所述变体可包含存在于MspD中的一个或多个以下突变:G1,L2V,E5Q,L8V,D13G,W21A,D22E,K47T,I49H,I68V,D91G,A96Q,N102D,S103T,V104I,S136K和G141A的缺失。所述变体可以包含来自Msp B、C和D中的一个或多个突变和取代的组合。
除了上文论述的那些之外,可以对SEQ ID NO:2的氨基酸序列进行氨基酸取代,例如高达1,2,3,4,5,10,20或30个取代。保守取代用具有相似化学结构、相似化学性质或相似侧链体积的其他氨基酸替换氨基酸。引入的氨基酸可以具有与它们替代的氨基酸相似的极性、亲水性、疏水性、碱性、酸性、中性或电荷。或者,保守取代可以引入另一个芳香族或脂肪族氨基酸代替预先存在的芳香族或脂肪族氨基酸。保守氨基酸变化是本领域公知的,并且可以依据下表1中限定的20种主要氨基酸的性质进行选择。当氨基酸具有相似的极性时,这也可以通过参考表2中氨基酸侧链的亲水性大小(hydropathy scale)来确定。
表1-氨基酸的化学性质
表2-亲水性大小
SEQ ID NO:2的氨基酸序列的一个或多个氨基酸残基可以另外从上述多肽中缺失。可以缺失多至1,2,3,4,5,10,20或30个残基或更多个残基。
变体可以包括SEQ ID NO:2的片段。这类片段保留成孔活性。片段的长度可以为至少50,100,150或200个氨基酸。这样的片段可以用于产生孔。片段优选包含SEQ ID NO:2的孔形成结构域。片段必须包括SEQ ID NO:2的残基88,90,91,105,118和134中的一个。通常,片段包括SEQ ID NO:2的所有残基88,90,91,105,118和134。
一个或多个氨基酸可以替代地或另外地添加到上述多肽。可以在SEQ ID NO:2或其片段的多肽变体的氨基酸序列的氨基末端或羧基末端提供延伸。延伸可以非常短,例如1至10个氨基酸长度。或者,延伸可以较长,例如高达50或100个氨基酸。载体蛋白可以与本发明的氨基酸序列融合。其它融合蛋白在下面更详细地论述。
如上所述,变体是具有下述氨基酸序列的多肽:从SEQ ID NO:2的氨基酸序列变化而来并且保留其形成孔的能力。变体通常包含负责形成孔的SEQ ID NO:2的区域。含有β桶状体的Msp的孔形成能力由每个亚基中的β片体提供。SEQ ID NO:2的变体通常包含SEQ ID NO:2中形成β片体的区域。可以对SEQ ID NO:2的形成β片体的区域进行一个或多个修饰,只要所得变体保留其形成孔的能力即可。SEQ ID NO:2的变体优选包括在其α螺旋和/或环区域内的一个或多个修饰,例如取代、添加或缺失。
衍生自Msp的单体可例如通过添加链霉亲和素标签或通过添加信号序列以促进其从细胞分泌而进行修饰以有助于对其的鉴定或纯化,其中所述单体不天然含有这样的序列。下面更详细地论述其他合适的标签。单体可以用显示性标记物进行标记。显示性标记物可以是允许对单体检测的任何合适标记物。合适的标记物如下所述。
衍生自Msp的单体也可以使用D-氨基酸产生。例如,衍生自Msp的单体可包含L-氨基酸和D-氨基酸的混合物。这在生产这种蛋白质或肽的领域是常规的。
衍生自Msp的单体含有一种或多种特异性修饰以便于对核苷酸的鉴别。衍生自Msp的单体也可以含有其它非特异性修饰,只要它们不干扰孔形成即可。许多非特异性侧链修饰是本领域已知的,并且可以对衍生自Msp的单体的侧链进行修饰。这样的修饰包括例如通过与醛反应、然后用NaBH4还原而进行的氨基酸的还原性烷基化,用甲基乙酰亚胺酯(methylacetimidate)进行的脒基化,或用乙酸酐进行的酰化。
衍生自Msp的单体可以使用本领域中已知的标准方法来制备。衍生自Msp的单体可以通过合成或重组方式制成。例如,所述单体可通过体外翻译和转录(IVTT)合成。制造孔和单体的合适的方法在国际申请No.PCT/GB09/001690(公开号为WO 2010/004273),PCT/GB09/001679(公开号为WO 2010/004265)or PCT/GB10/000133(公开号为WO2010/086603)中有论述。论述了将孔插入膜的方法。
在一些实施方案中,突变单体是经化学修饰的。所述突变单体可以任何方式在任何位点进行化学修饰。所述突变单体优选通过下述进行化学修饰:将分子连接到一个或多个半胱氨酸(半胱氨酸连接),将分子连接到一个或多个赖氨酸,将分子连接到一个或多个非天然氨基酸,表位的酶修饰或末端的修饰。适用于进行这些修饰的方法是本领域已知的。所述突变单体可通过连接任何分子被化学修饰。例如,所述突变单体可以通过连接染料或荧光团被化学修饰。
在一些实施方案中,突变单体用促进包含单体的孔与目标核苷酸或目标多核苷酸序列之间的相互作用的分子适配器(adaptor)进行化学修饰。适配器的存在改善了孔和核苷酸或多核苷酸序列的主-客体化学,从而提高了由突变单体形成的孔的测序能力。主客体化学的原理是本领域公知的。适配器对孔的改善其与核苷酸或多核苷酸序列的相互作用的物理或化学性质具有影响。适配器可改变孔的桶状体或通道的电荷,或与核苷酸或多核苷酸序列特异性相互作用或结合,从而促进其与孔的相互作用。
分子适配器优选是环状分子、环糊精、能够杂交的物质、DNA结合剂或相互螯合剂(interchelator)、肽或肽类似物、合成聚合物、芳香族平面分子、带正电荷小分子,或能够氢键合的小分子。
所述适配器可以是环状的。环状适配器优选具有与孔相同的对称性。适配器优选具有八重对称性,因为Msp通常具有围绕中心轴的八个亚基。这将在下面更详细地论述。
适配器通常通过主-客体化学与核苷酸或多核苷酸序列相互作用。适配器通常能够与核苷酸或多核苷酸序列相互作用。适配器包含一个或多个能够与核苷酸或多核苷酸序列相互作用的化学基团。所述一个或多个化学基团优选通过非共价相互作用(例如疏水性相互作用、氢键合、范德华力、π-阳离子相互作用和/或静电力)与核苷酸或多核苷酸序列相互作用。能够与核苷酸或多核苷酸序列相互作用的一个或多个化学基团优选是带正电荷的。能够与核苷酸或多核苷酸序列相互作用的一个或多个化学基团更优选包含氨基。氨基可以连接到伯、仲或叔碳原子。适配器甚至更优选包含氨基环,例如6个、7个或8个氨基的环。适配器最优选包含八个氨基的环。质子化氨基的环可以与核苷酸或多核苷酸序列中的带负电荷的磷酸基团相互作用。
适配器在孔内的正确定位可以通过适配器和包含突变单体的孔之间的主-客体化学来促进。适配器优选包含一个或多个能够与孔中的一个或多个氨基酸相互作用的化学基团。适配器更优选包含能够通过非共价相互作用,例如疏水性相互作用、氢键合、范德华力、π-阳离子相互作用和/或静电力,与孔中的一个或多个氨基酸相互作用的一个或多个化学基团。能够与孔中的一个或多个氨基酸相互作用的化学基团通常是羟基或胺。所述羟基可以连接到伯、仲或叔碳原子上。所述羟基可以与孔中不带电荷的氨基酸形成氢键。可以使用促进孔和核苷酸或多核苷酸序列之间的相互作用的任何适配器。
合适的适配器包括但不限于环糊精、环肽和葫芦脲。适配器优选是环糊精或其衍生物。环糊精或其衍生物可以是Eliseev,A.V.和Schneider,H-J.(1994)J.Am.Chem.Soc.116,6081-6088中公开的任何环糊精或其衍生物。适配器更优选是七-6-氨基-β-环糊精(am7-βCD)、6-单脱氧-6-单氨基-β-环糊精(am1-βCD)或七-(6-脱氧-6-胍基)环糊精(gu7-βCD)。gu7-βCD中的胍基具有比am7-βCD中的伯胺高得多的pKa,因此其带更多正电荷。该gu7-βCD适配器可以用于增加核苷酸在孔中的停留时间,以提高测量的残余电流的精确度,以及增加高温或低数据采集速率下的碱基检测率。
如下文更详细论述的,如果使用3-(2-吡啶基二硫代)丙酸琥珀酰亚胺酯(SPDP)交联剂,适配器优选是七(6-脱氧-6-氨基)-6-N-单(2-吡啶基)二硫代丙酰基-β-环糊精(am6amPDP1-βCD)。
更合适的适配器包括γ-环糊精,其包含8个糖单元(因此具有8重对称性)。所述γ-环糊精可以含有连接分子或可以被修饰以包含用于上述β-环糊精实例中的所有或更多的修饰的糖单元。
分子适配器优选共价连接到突变单体。适配器可以使用本领域已知的任何方法共价连接到孔。适配器通常通过化学键连接。如果分子适配器通过半胱氨酸键连接,则优选通过取代将一个或多个半胱氨酸引入突变体。当然,本发明的突变单体可以在位置88,90,91,103和105中的一个或多个位置包含半胱氨酸残基。突变单体可以通过将分子适配器连接到这些半胱氨酸中一个或多个,例如2,3,4或5个进行化学修饰。或者,突变单体可以通过将分子连接到在其它位置引入的一个或多个半胱氨酸来进行化学修饰。分子适配器优选连接至SEQ ID NO:2的位置90,91和103中的一个或多个位置。
半胱氨酸残基的反应性可以通过修饰相邻残基来增强。例如,侧翼精氨酸、组氨酸或赖氨酸残基的碱基将半胱氨酸硫醇基团的pKa变为更具反应性的S-基团的pKa。半胱氨酸残基的反应性可以用硫醇保护基团如dTNB保护。所述保护基团可以在接头连接之前与突变单体的一个或多个半胱氨酸残基反应。
所述分子可以直接连接到突变单体。所述分子优选使用接头例如化学交联剂或肽接头连接到突变单体。
合适的化学交联剂是本领域公知的。优选的交联剂包括3-(吡啶-2-基二硫烷基)丙酸2,5-二氧代吡咯烷-1-基酯、4-(吡啶-2-基二硫烷基)丁酸2,5-二氧代吡咯烷-1-基酯和8-(吡啶-2-基二硫烷基)辛酸2,5-二氧代吡咯烷-1-基酯。最优选的交联剂是3-(2-吡啶基二硫代)丙酸琥珀酰亚胺酯(SPDP)。通常,在分子/交联剂复合物共价连接到突变单体之前,所述分子共价连接到双官能交联剂,但是在双官能交联剂/单体复合物连接到所述分子之前,也可以将双官能交联剂共价连接到所述单体。
所述接头优选对二硫苏糖醇(DTT)具有抗性。合适的接头包括但不限于基于碘乙酰胺和基于马来酰亚胺的接头。
在其它实施方案中,所述单体可以连接到多核苷酸结合蛋白。这形成可用于本发明的测序方法中的模块化测序系统。多核苷酸结合蛋白在下文论述。
多核苷酸结合蛋白优选共价连接到突变单体。蛋白质可以使用本领域已知的任何方法共价连接到所述单体。所述单体和蛋白质可以是化学融合或基因融合的。如果整个构建体从单个多核苷酸序列表达,则所述单体和蛋白质是基因融合的。国际申请No.PCT/GB09/001679(公开号WO 2010/004265)中论述了单体与多核苷酸结合蛋白的基因融合。
如果多核苷酸结合蛋白通过半胱氨酸键连接,则优选通过取代将一个或多个半胱氨酸引入突变体。当然,本发明的突变单体可以在位置10至15,51至60,136至139和168至172中的一个或多个上包含半胱氨酸残基。这些位置存在于同系物内具有低保守性的环区域中,代表可以耐受突变或插入。因此它们适于连接多核苷酸结合蛋白。半胱氨酸残基的反应性可以通过如上所述的修饰来增强。
多核苷酸结合蛋白可以直接地或通过一个或多个接头连接到突变单体。所述分子可以使用国际申请No.PCT/GB10/000132(公开号WO 2010/086602)中描述的杂交接头连接到突变单体。或者,可以使用肽接头。肽接头是氨基酸序列。肽接头的长度、柔性和亲水性通常设计为,使得其不干扰所述单体和分子的功能。优选的柔性肽接头是2至20个,例如4,6,8,10或16个丝氨酸和/或甘氨酸氨基酸的片段。更优选的柔性接头包括(SG)1,(SG)2,(SG)3,(SG)4,(SG)5和(SG)8,其中S是丝氨酸,G是甘氨酸。优选的刚性接头是2至30个,例如4,6,8,16或24个脯氨酸氨基酸的片段。更优选的刚性接头包括(P)12,其中P是脯氨酸。
突变单体可以用分子适配器和多核苷酸结合蛋白进行化学修饰。
半胱氨酸残基的反应性可以通过修饰相邻残基来增强。例如,侧翼精氨酸、组氨酸或赖氨酸残基的碱基将使半胱氨酸硫醇基团的pKa变为更具反应性的S-基团的pKa。半胱氨酸残基的反应性可以通过硫醇保护基团如dTNB保护。所述保护基团可以在连接接头之前与单体的一个或多个半胱氨酸残基反应。
所述分子(用其对所述单体进行化学修饰)可直接地连接或通过国际申请No.PCT/GB09/001690(公开号WO 2010/004273),PCT/GB09/001679(公开号WO 2010/004265)或PCT/GB10/000133(公开号WO 2010/086603)中公开的接头连接到所述单体。
本文所述的任何蛋白质,例如突变单体和本发明的孔,可以例如通过添加组氨酸残基(his标签)、天冬氨酸残基(asp标签)、链霉亲和素标签、flag标签、SUMO标签、GST标签或MBP标签,或通过添加信号序列促进它们从不天然含有这类序列的多肽的细胞进行分泌,而进行修饰,以有助于对其进行鉴定或纯化。引入遗传标签的替代方案是使标签通过化学反应而连接到蛋白质上的天然或工程位置。这样的一个实例是使凝胶移位试剂与工程改造到蛋白质外部上的半胱氨酸反应。这已经被证明是分离溶血素异源寡聚体的方法(ChemBiol.1997Jul;4(7):497-505)。
本文所述的任何蛋白质,例如本发明的突变单体和孔,可以用显示性标记物标记。显示性标记物可以是允许检测蛋白质的任何合适的标记物。合适的标记物质包括但不限于荧光分子、放射性同位素例如125I、35S、酶、抗体、抗原、多核苷酸和配体如生物素。
本文所述的任何蛋白质,例如本发明的单体或孔,可通过合成或通过重组方法制备。例如,蛋白质可以通过体外翻译和转录(IVTT)来合成。蛋白质的氨基酸序列可以被修饰为包括非天然存在的氨基酸或增加蛋白质的稳定性。当通过合成方法产生蛋白质时,可以在制备过程引入这类氨基酸。蛋白质也可以在合成或重组制备后改变。
蛋白质也可以使用D-氨基酸制备。例如,蛋白质可以包含L-氨基酸和D-氨基酸的混合物。这在制备这种蛋白质或肽的领域是常规的。
蛋白质也可以含有其它非特异性修饰,只要它们不干扰蛋白质的功能即可。许多非特异性侧链修饰是本领域已知的,并且可以对蛋白质(一个或多个)的侧链进行修饰。这样的修饰包括例如通过与醛反应、然后用NaBH4还原而进行的氨基酸的还原性烷基化,用甲基乙酰亚胺酯进行的脒基化作用,或用乙酸酐进行的酰化。
本文所述的任何蛋白质,包括本发明的单体和孔,可以使用本领域已知的标准方法制备。编码蛋白质的多核苷酸序列可以使用本领域的标准方法衍生和复制。编码蛋白质的多核苷酸序列可以使用本领域标准技术在细菌宿主细胞中表达。蛋白质可以通过在细胞中由重组表达载体原位表达多肽而制备。表达载体任选地携带诱导型启动子以控制多肽的表达。这些方法在Sambrook,J.和Russell,D.(2001).Molecular Cloning:A Laboratory Manual,第3版,Cold Spring Harbor Laboratory Press,Cold Spring Harbor,NY中有描述。
蛋白质可以从产生蛋白质的生物体或在重组表达后通过任何蛋白质液相色谱系统纯化后大规模生产。典型的蛋白质液相色谱系统包括FPLC系统、AKTA系统、Bio-Cad系统、Bio-Rad BioLogic系统和Gilson HPLC系统。
构建体
本发明还提供了包含两个或更多个共价连接的MspA单体的构建体,其中至少一个单体是本发明的突变单体。本发明的构建体保持其形成孔的能力。这可以按如上所述确定。本发明的一种或多种构建体可用于形成用于表征例如测序多核苷酸的孔。所述构建体可以包含至少2个、至少3个、至少4个、至少5个、至少6个、至少7个、至少8个、至少9个或至少10个单体。所述构建体优选包含两个单体。所述两个或更多个单体可以相同或不同。
所述构建体可以包含一种或多种不是本发明突变单体的单体。为本发明非突变单体的MspA突变单体包含SEQ ID NO:2的比较变体。构建体中的至少一种单体可包含SEQ ID NO:2中所示序列的比较变体,其包含D90N,D91N,D93N,D118R,D134R和E139K。构建体中的至少一种单体可以是国际申请No.PCT/GB2012/050301(公开号WO/2012/107778)中公开的任何单体,包括包含SEQ ID NO:2中所示序列的比较变体的那些,所述比较变体包含G75S,G77S,L88N,D90N,D91N,D93N,D118R,Q126R,D134R和E139K。SEQ ID NO:2的比较变体基于氨基酸同一性与SEQ ID NO:2的整个序列至少50%同源。更优选地,所述比较变体基于氨基酸同一性可与SEQ ID NO:2的氨基酸序列在整个序列上至少55%,至少60%,至少65%,至少70%,至少75%,至少80%,至少85%,至少90%,且更优选至少95%,97%或99%同源。
构建体中的至少一种单体是本发明的突变单体。构建体中的所有单体可以是本发明的突变单体。突变单体可以相同或不同。在更优选的实施方案中,构建体包含本发明的两种单体。
构建体中的单体优选为基因融合的。如果整个构建体从单个多核苷酸序列表达,则单体为基因融合的。单体的编码序列可以以任何方式组合以形成编码该构建体的单个多核苷酸序列。
单体可以以任何构型基因融合。各单体可以通过它们的末端氨基酸融合。例如,一个单体的氨基末端可以与另一个单体的羧基末端融合。构建体中的第二和随后的单体(在氨基至羧基方向)可以在它们的氨基末端(其中每一个与在前单体的羧基末端融合)包含甲硫氨酸。例如,如果M是单体(没有氨基末端甲硫氨酸)并且mM是具有氨基末端甲硫氨酸的单体,则构建体可以包含序列M-mM,M-mM-mM或M-mM-mM-mM。这些甲硫氨酸的存在通常源于在编码整个构建体的多核苷酸内编码第二或随后单体的多核苷酸的5′端的起始密码子(即ATG)的表达。构建体中的第一单体(在氨基至羧基方向上)还可以包含甲硫氨酸(例如mM-mM,mM-mM-mM或mM-mM-mM-mM)。
两个或更多个单体可以直接基因融合在一起。单体优选使用接头进行基因融合。接头可以设计成限制单体的移动性。优选的接头是氨基酸序列(即肽接头)。可以使用上述任何肽接头。
在另一个优选的实施方案中,单体是化学融合的。如果两个部分化学连接,例如通过化学交联剂,则两个单体是化学融合的。可以使用上述任何化学交联剂。接头可以连接到引入本发明的突变单体中的一个或多个半胱氨酸残基。或者,接头可以连接到构建体中单体之一的末端。
如果构建体包含不同的单体,则可以通过将单体中接头的浓度保持大量过量来防止单体自身的交联。或者,使用两个接头的情况下可以使用“锁和钥匙”布置。每个接头仅一端可以一起反应以形成更长的接头,并且接头的另一端各自与不同的单体反应。这类接头在国际申请No.PCT/GB10/000132(公开号WO 2010/086602)中有描述。
多核苷酸
本发明还提供编码本发明突变单体的多核苷酸序列。突变单体可以是上面论述的任何一种。所述多核苷酸序列优选包含基于核苷酸同一性与SEQ ID NO:1的序列在整个序列上至少50%,60%,70%,80%,90%或95%同源的序列。可以在300或更多个,例如375,450,525或600个或更多连续核苷酸的片段上具有至少80%,例如至少85%,90%或95%的核苷酸同源性(“严格核苷酸”)。同源性可如上所述计算。在遗传密码的简并性的基础上,多核苷酸序列可以包含不同于SEQ ID NO:1的序列。
本发明还提供编码本发明的任何基因融合构建体的多核苷酸序列。多核苷酸优选包含SEQ ID NO:1中所示序列的两个或更多个变体。基于核苷酸同一性,多核苷酸序列优选包含两个或更多个与SEQ ID NO:1在整个序列上具有至少50%,60%,70%,80%,90%或95%同源性的序列。在600或更多,例如750,900,1050或1200或更多个连续核苷酸的片段上,可以具有至少80%,例如至少85%,90%或95%核苷酸同一性(“严格同源性”)。同源性可按上述计算。
多核苷酸序列可以使用本领域的标准方法衍生和复制。编码野生型Msp的染色体DNA可以从产生孔的生物体例如耻垢分枝杆菌提取。可以使用包括特异性引物的PCR扩增编码孔亚基的基因。然后扩增的序列可以进行定点诱变。定点诱变的合适方法是本领域已知的,包括例如接合链反应。编码本发明的构建体的多核苷酸可以使用熟知的技术来制备,例如Sambrook,J.和Russell,D.(2001).Molecular Cloning:A Laboratory Manual,3rd Edition.Cold Spring Harbor Laboratory Press,Cold Spring Harbor,NY中描述的技术。
然后可将所得的多核苷酸序列纳入重组可复制载体如克隆载体中。载体可以用于在相容的宿主细胞中复制多核苷酸。因此,可以通过将多核苷酸引入可复制载体中、将载体引入相容的宿主细胞中,然后在能产生载体复制的条件下使宿主细胞生长来制备多核苷酸序列。载体可以从宿主细胞中回收。用于克隆多核苷酸的合适的宿主细胞是本领域已知的,并在下文更详细地描述。
多核苷酸序列可以克隆到合适的表达载体中。在表达载体中,多核苷酸序列通常可操作地连接到能够提供宿主细胞表达编码序列的控制序列。这种表达载体可用于表达孔亚基。
术语“可操作地连接”是指并置,其中所述组分处于允许它们以它们预期的方式起作用的关系中。控制序列“可操作地连接”到编码序列时以这样的方式连接:在与控制序列相容的条件下实现编码序列的表达。可以将多个拷贝的相同或不同多核苷酸序列引入载体中。
然后可以将表达载体引入合适的宿主细胞中。因此,本发明的突变单体或构建体可以通过将多核苷酸序列插入表达载体中、将载体引入相容的细菌宿主细胞中,然后在能产生多核苷酸序列的表达的条件下使宿主细胞生长而制备。重组表达的单体或构建体可以自组装成宿主细胞膜中的孔。或者,以这种方式产生的重组孔可以从宿主细胞中移出并插入另一个膜中。当产生包含至少两种不同单体或构建体的孔时,不同的单体或构建体可以如上所述在不同的宿主细胞中单独表达,从宿主细胞中移出并组装到单独的膜例如兔细胞膜中的孔中。
载体可以是例如提供有复制起点,可选的用于表达所述多核苷序列的启动子和可选的启动子的调节子的质粒、病毒或噬菌体载体。载体可以含有一个或多个可选择标记基因,例如四环素抗性基因。启动子和其它表达调节信号可以选择为与设计表达载体所针对的宿主细胞相容。通常使用T7,trc,lac,ara或λL启动子。
宿主细胞通常以高水平表达单体或构建体。选择用多核苷酸序列转化的宿主细胞,以与用于转化细胞的表达载体相容。宿主细胞通常是细菌,优选大肠杆菌。具有λDE3溶素原的任何细胞,例如C41(DE3),BL21(DE3),JM109(DE3),B834(DE3),TUNER,Origami和Origami B,可以表达包含T7启动子的载体。除了上面列出的条件之外,Proc Natl Acad Sci USA.2008Dec 30;105(52):20647-52中引用的任何方法可以用于表达Msp蛋白。
本发明还包括制备本发明的突变单体或本发明的构建体的方法。该方法包括在合适的宿主细胞中表达本发明的多核苷酸。多核苷酸优选是载体的一部分,并且优选可操作地连接至启动子。
孔
本发明还提供各种孔。本发明的孔对于表征例如测序多核苷酸序列是理想的,因为它们可以以高灵敏度区分不同核苷酸。令人惊奇地,孔可以区分DNA和RNA中的四个核苷酸。本发明的孔甚至可以区分甲基化和非甲基化核苷酸。本发明的孔的碱基分辨率令人惊讶地高。所述孔显示,所有四种DNA核苷酸几乎完全分离。基于在孔中的停留时间和流经孔的电流,所述孔进一步区分脱氧胞苷单磷酸酯(dCMP)和甲基-dCMP。
本发明的孔也可以在一系列条件下区分不同的核苷酸。特别地,所述孔将在有利于表征(例如测序)核酸的条件下区分核苷酸。本发明的孔可以区分不同核苷酸的程度可以通过改变施加的电位、盐浓度、缓冲液、温度和添加剂如脲、甜菜碱和DTT的存在来控制。这允许对孔的功能进行微调,特别是当测序时。这将在下面更详细地论述。本发明的孔也可用于从与一种或多种单体的相互作用中,而不是通过核苷酸基础在核苷酸上鉴别多核苷酸聚合物。
本发明的孔可以是分离的、基本分离的、纯化的或基本纯化的。如果本发明的孔完全不含任何其它组分,例如脂质或其它孔,则其是分离的或纯化的。如果孔与不会干扰其预期用途的载体或稀释剂混合,则孔是基本上被分离的。例如,如果孔以包含小于10%,小于5%,小于2%或小于1%的其它组分的形式存在,则孔基本被分离或基本纯化,所述其它组分例如三嵌段共聚物、脂质或其他孔。或者,本发明的孔可存在于膜中。合适的膜在下面论述。
本发明的孔可以作为单独或单个孔存在。或者,本发明的孔可以存在于两个或更多个孔的同源或异源群体中。
同源寡聚孔
本发明还提供了衍生自Msp的同源寡聚孔,包含相同的本发明突变单体。同源寡聚孔可包含本发明的任何突变体。本发明的同寡聚孔对于表征例如测序多核苷酸是理想的。本发明的同源寡聚孔可具有上述任何优点。
同源寡聚孔可以包含任何数量的突变单体。所述孔通常包含至少7个,至少8个,至少9个或至少10个相同突变单体,例如7,8,9或10个突变单体。所述孔优选地包括八个相同的突变单体。一个或多个,例如2,3,4,5,6,7,8,9或10个突变单体,优选如上所述进行化学修饰。
下面更详细地论述制备孔的方法。
异源寡聚孔
本发明还提供了一种源自Msp的异源寡聚孔,包含至少一种本发明突变单体。本发明的异源寡聚孔对表征例如测序多核苷酸而言是理想的。异源寡聚孔可以使用本领域中已知的方法制备(例如Protein Sci.2002Jul;11(7):1813-24)。
所述异源寡聚孔含有足够的用于形成孔的单体。单体可以是任何类型的。孔通常包含至少7个、至少8、至少9或至少10个单体,如7、8、9或10个单体。所述孔优选包括8个单体。
在一个优选的实施方案中,所有单体(如10、9、8或7个单体)为本发明的突变单体,并且它们中的至少一个与其它不同。在一个更优选的实施方案中,孔包括8个本发明的突变单体,并且它们中的至少一个与其它不同。它们可以全部彼此不同。
在另一个优选的实施方案中,至少一个突变单体不是本发明的突变单体。在本实施方案中,其余单体优选是本发明的突变单体。因此,所述孔可以包括9,8,7,6,5,4,3,2或1个本发明的突变单体。孔中任何数目的单体可以不是本发明的突变单体。所述孔优选包括7个本发明的突变单体和不为本发明单体的单体,例如含如上所述的比较变体的单体。本发明的突变单体可以相同或不同。
在上面论述的所有实施方案中,优选对一个或多个,例如2,3,4,5,6,7,8,9或10个突变单体如上所述进行化学修饰。
下文更详细论述制造所述孔的方法。
含构建体的孔
本发明还提供包含本发明至少一个构建体的孔。本发明构建体包含源自Msp的两个或更多个共价连接的单体,其中至少一个所述单体是本发明的突变单体。换言之,构建体必须包含多于一个的单体。所述孔含有足够的构建体和——如果需要——单体,用以形成所述孔。例如,一个八聚体的孔可包括:(a)四个构建体,每一个包含两个构建体,(b)两个构建体,每一个包含四个单体,或(b)一个构建体,其包含两个单体和六个不构成的构建体的一部分的单体。构建体和单体的其他组合是本领域技术人员可以想到的。
孔中至少两个单体是本发明构建体的形式。所述构建体并因此所述孔,包含至少一个本发明突变单体。所述孔通常共包含至少7,至少8,至少9或至少10个单体,如7,8,9或10个单体,(其中至少两个必须是在构建体中)。所述孔优选包含8个单体(其中至少两个必须在构建体中)。
含孔的构建体可以是同源寡聚物(即包括相同构建体)或异源寡聚物(即,其中至少一个构建体与其他的不同)。
孔通常包含(a)包含两个单体的一个构建体,和(b)5,6,7或8个单体。所述构建体可以是上文论述的任何构建体。所述单体可以是上文论述的任何单体,包括本发明的突变单体和包含上文所述的SEQ ID NO:2的比较变体的突变单体。
另一典型的孔包含多于一个的本发明构建体,如两个、三个或四个本发明构建体。如果需要,这类孔可进一步包含足够的另外的单体或构建体,用以形成所述孔。所述另外的单体(一个或多个)可以是上文论述的任何单体,包括本发明的突变单体和包含上文所述的SEQ ID NO:2的比较变体的突变单体。所述另外的构建体(一个或多个)可以是上文论述的任何构建体,或者可以是含有两个或更多个共价连接的MspA单体的构建体,每一个所述MspA单体含有上文所述的SEQ ID NO:2的比较变体。
本发明的再一个孔仅包括含2个单体的构建体,例如孔可以包括含2个单体的4,5,6,7或8个构建体。至少一个构建体是本发明的构建体,即至少一个构建体中的至少一个单体,优选至少一个构建体中的每一个单体,是本发明的突变单体。含有2单体的所有构建体都可以是本发明的构建体。
本发明的特定孔包含四个本发明的构建体,每个构建体包含两个单体,其中每个构建体中的至少一个单体,并且优选每个构建体中的每个单体,是本发明的突变单体。所述构建体可以寡聚到孔中形成这样的结构:使得每个构建体中只有一个单体有助于孔的通道。通常,所述构建体中的其他单体将在所述孔的通道的外部上。例如,本发明的孔可包括含有2个单体的5,6,7或8个构建体,其中所述通道包含8个单体。
如上所述,可在构建体中引入突变。所述突变可以是交替的,即,对于一个两单体构建体中的每个单体,突变是不同的,并且所述构建体组装成同源寡聚物,导致交替的修饰。换句话说,包含MutA和MutB的单体相融合并组装以形成A-B:A-B:A-B:A-B孔。替代地,所述突变可以是相邻的,即,相同的突变被引入到构建体中的两个单体中,然后其与不同突变单体或构建体进行寡聚。换句话说,包含MutA的单体相融合,之后与含MutB的单体寡聚,形成A-A:B:B:B:B:B:B。
在含构建体的孔中的一个或多个本发明单体可以如上所述进行化学修饰。
多核苷酸的表征
本发明提供了表征目标多核苷酸的方法。该方法包括测量所述目标多核苷酸的一个或多个特征。所述目标多核苷酸也可被称为模板多核苷酸或感兴趣的多核苷酸。
多核苷酸
多核苷酸,如核酸,是含有两个或更多个核苷酸的大分子。所述多核苷酸或核酸可以包含任何核苷酸的任意组合。所述核苷酸可以是天然存在的或人造的。所述多核苷酸中的一个或多个核苷酸可以被氧化或甲基化。所述多核苷酸中的一个或多个核苷酸可以被破坏。例如,所述多核苷酸可以包含嘧啶二聚体。此类二聚体通常与紫外光的破坏相关,并且是皮肤黑素瘤的首要原因。所述多核苷酸中的一个或多个核苷酸可以被修饰,例如用标记物或标签修饰。合适的标记物在下面描述。所述多核苷酸可包含一个或多个间隔区。
核苷酸通常包含核碱基、糖和至少一个磷酸基团。核碱基和糖形成核苷。
核碱基通常为杂环。核碱基包括但不限于:嘌呤和嘧啶,更具体是腺嘌呤(A)、鸟嘌呤(G)、胸腺嘧啶(T)、尿嘧啶(U)和胞嘧啶(C)。
所述糖通常为戊糖。核苷酸的糖包括,但不限于,核糖和脱氧核糖。所述糖优选是脱氧核糖。
所述多核苷酸优选包括下列核苷:脱氧腺苷(dA)、脱氧尿苷(dU)和/或胸苷(dT)、脱氧鸟苷(dG)和脱氧胞苷(dC)。
所述核苷酸一般是核糖核苷酸或脱氧核糖核苷酸。所述核苷酸一般包含单磷酸、二磷酸或三磷酸。所述核苷酸可包含多于三个的磷酸酯,如4或5个磷酸酯。磷酸酯可以连接在核苷酸的5′或3′侧。核苷酸包括但不限于,腺苷一磷酸(AMP)、鸟苷一磷酸(GMP)、胸苷一磷酸(TMP)、尿苷一磷酸(UMP)、5-甲基胞苷一磷酸、5-羟甲基胞苷一磷酸、胞苷一磷酸(CMP)、环状腺苷一磷酸(cAMP)、环状鸟苷一磷酸(cGMP)、脱氧腺苷一磷酸(dAMP)、脱氧鸟苷一磷酸(dGMP)、脱氧胸苷一磷酸(dTMP)、脱氧尿苷一磷酸(dUMP)、脱氧胞苷一磷酸(dCMP的)和脱氧甲基胞苷一磷酸。所述核苷酸优选选自AMP,TMP,GMP,CMP,UMP,dAMP,dTMP,dGMP,dCMP和dUMP。
核苷酸可以是无碱基的(即缺乏核碱基)。核苷酸也可以缺乏核碱基和糖(即,是C3间隔区)。
所述多核苷酸中的核苷酸可以以任何方式彼此连接。所述核苷酸一般通过它们的糖和磷酸基团进行连接,同在核酸中一样。所述核苷酸可以通过它们的核碱基进行连接,同在嘧啶二聚体中一样。
所述多核苷酸可以是单链或双链的。优选至少所述多核苷酸的一部分是双链的。
所述多核苷酸可以是核酸,例如脱氧核糖核酸(DNA)或核糖核酸(RNA)。所述多核苷酸可包含杂交到DNA的一条链的RNA的一条链。所述多核苷酸可以是本领域中已知的任何合成的核酸,例如肽核酸(PNA)、甘油核酸(GNA)、苏糖核酸(TNA)、锁定核酸(LNA),或其它具有核苷酸侧链的合成聚合物。PNA骨架由通过肽键连接的N-(2-氨基乙基)-甘氨酸重复单元组成。GNA骨架由通过磷酸二酯键连接的乙二醇重复单元组成。TNA骨架由通过磷酸二酯键连接在一起的苏糖重复单元组成。LNA由如上所述的核糖核苷酸形成,具有额外的桥连接核糖部分中的2′氧和4′碳。
所述多核苷酸最优选为核糖核酸(RNA)或脱氧核糖核酸(DNA)。
所述多核苷酸可以是任何长度。例如,所述多核苷酸可以是至少10个,至少50个,至少100,至少150,至少200,至少250,至少300,至少400或至少500个核苷酸或核苷酸对长度。所述多核苷酸可以是1000或更多个核苷酸或核苷酸对、5000或更多个核苷酸或核苷酸对长度,或者100000或更多个核苷酸或核苷酸对长度。
对任何数量的多核苷酸进行研究。例如,本发明的方法可以涉及表征2,3,4,5,6,7,8,9,10,20,30,50,100或更多个多核苷酸。如果对两个或更多个多核苷酸进行表征,则它们可以是不同的多核苷酸或同一多核苷酸的两种情形。
所述多核苷酸可以是天然存在的或人造的。例如,该方法可以用来验证制造的寡核苷酸的序列。该方法通常在体外进行。
样本
每个分析物通常存在于任何合适的样本中。本发明通常针对已知含有或怀疑含有分析物的两个或更多个样本实施。替换地,本发明可以对两个或更多个样本实施本发明,以确认对已知或预期存在于样本中的两个或更多个分析物的身份。
第一样本和/或第二样本可以是生物样本。本发明可以使用获取自或提取自任何有机体或微生物的至少一个样本在体外进行。所述第一样本和/或第二样本可以是非生物样本。非生物样本优选为的流体样品。非生物样本的实例包括外科流体;水,如饮用水、海水或河水;和实验室测试用试剂。
第一样本和/或第二样本通常在用于本发明之前进行处理,例如通过离心或通过膜过滤掉不需要的分子或细胞,例如红细胞。所述第一样本和/或第二样本可以在获取后立即进行测量。所述第一样本和/或第二样本通常也可在测试之前进行存储,优选在低于-70℃下进行存储。
表征
所述方法可以包括测量所述多核苷酸的两个、三个、四个或五个或更多个特征。所述的一个或多个特征优选选自(i)多核苷酸的长度,(ii)多核苷酸的同一性,(iii)多核苷酸的序列,(iv)多核苷酸的二级结构,以及(v)多核苷酸是否被修饰。根据本发明可以测量(i)到(v)的任意组合,例如{i},{ii},{iii},{iv},{v},{i,ii},{i,iii},{i,iv},{i,v},{ii,iii},{ii,iv},{ii,v},{iii,iv},{iii,v},{iv,v},{i,ii,iii},{i,ii,iv},{i,ii,v},{i,iii,iv},{i,iii,v},{i,iv,v},{ii,iii,iv},{ii,iii,v},{ii,iv,v},{iii,iv,v},{i,ii,iii,iv},{i,ii,iii,v},{i,ii,iv,v},{i,iii,iv,v},{ii,iii,iv,v}或{i,ii,iii,iv,v}。可以测量第一多核苷酸的与第二多核苷酸相比不同的(i)到(v)的组合,包括上面所列的任意组合。
对于(i),多核苷酸的长度可以例如通过确定所述多核苷酸和所述孔之间相互作用的次数或所述多核苷酸和所述孔之间相互作用的持续时间进行测量。
对于(ii),对多核苷酸的鉴别可以通过多种方式进行测定。对多核苷酸的识别可以联合或不联合多核苷酸序列的测定而进行测定。前者是直接测定;对所述多核苷酸进行测序,并由此进行识别。后者可以多种方式完成。例如,可以测定多核苷酸中特定模序的存在(不需要测定多核苷酸的其余序列)。替代地,在所述方法中特定电和/或光信号的测定可以鉴别来自特定来源的多核苷酸。
对于(iii),多核苷酸的序列可以如前文所述进行测定。适合的测序方法,特别是那些使用电测量的方法,在Stoddart D等人,Proc Natl Acad Sci,12;106(19):7702-7,Lieberman KR等人,JAm Chem Soc.2010;132(50):17961-72,以及国际申请WO 2000/28312中有描述。
对于(iv),二级结构可以多种方式测定。例如,如果所述方法涉及电测量,则二级结构可以利用流经孔的停留时间的变化或电流的变化进行测定。这使得能够对单链和双链多核苷酸区域进行辨别。
对于(v),可以测定任何修饰的存在与否。所述方法优选包括确定所述多核苷酸是否通过甲基化、氧化、损伤、用一个或多个蛋白质或一个或多个标记物、标签或间隔区进行了修饰。特定的修饰将导致与孔的特定的相互作用,这可使用下述方法测定。例如,可以基于孔与每个核苷酸的相互作用过程中流经所述孔的电流,来区分甲基胞嘧啶和胞嘧啶。
使目标多核苷酸与本发明的孔接触。所述孔通常存在于膜中。合适的膜在下文论述。该方法可以使用适于研究膜/孔系统——其中孔存在于膜中——的任何设备进行。该方法可以使用适合于跨膜孔传感的任何设备进行。例如,所述设备包括含水溶液的腔室,以及将所述腔室分成两个区段的屏障。所述屏障典型地具有开口,含所述孔的膜形成于所述开口中。替代地,所述屏障形成其中存在孔的膜。
该方法可使用国际申请No.PCT/GB08/000562(WO 2008/102120)中描述的设备进行。
可进行多种不同类型的测量。其包括但不限于:电测量和光学测量。可能的电测量包括:电流测量、阻抗测量、隧道测量(Ivanov AP等人,Nano Lett.2011Jan 12;11(1):279-85),以及FET测量(国际申请WO 2005/124888)。光学测量可以与电测量结合(Soni GV等人,Rev Sci Instrum.2010 Jan;81(1):014301)。所述测量可以是跨膜电流测量,例如流经所述孔的离子电流的测量。
电测量可以使用标准单通道记录设备进行,如Stoddart D等人,Proc Natll Acad Sci,12;106(19):7702-7,Lieberman KR等人,J Am Chem Soc.2010;132(50):17961-72,和国际申请WO 2000/28312中描述的。替代地,电测量可以使用多通道系统进行,例如国际申请WO2009/077734和国际申请WO 2011/067559中所描述的。
所述方法优选利用跨所述膜而施加的电势实施。所施加的电势可以是电压电势。替代地,所施加的电势可以是化学电势。其一个例子是使用跨所述膜如两亲性层的盐梯度。盐梯度在Holden人,J Am Chem Soc.2007 Jul 11;129(27):8650-5中有公开。在一些情况下,使用当多核苷酸相对于所述孔移动时穿过所述孔的电流来估计或确定多核苷酸的序列。这就是链测序。
所述方法可以包括测量所述多核苷酸相对于所述孔移动时穿过所述孔的电流。因此,在本方法中使用的设备也可以包括能够施加电势并测量穿过所述膜和孔的电信号的电路。所述方法可以使用膜片钳或电压钳进行。所述方法优选涉及使用电压钳。
本发明方法可包括,当多核苷酸相对于孔移动时,测量穿过该孔的电流。用于测量穿过跨膜蛋白孔的离子电流的适宜条件是本领域已知的并在实施例中公开。所述方法通常通过跨膜和孔施加电压来实施。使用的电压通常为+5V到-5V,例如+4V到-4V,+3V到-3V或+2V到-2V。使用的电压通常为-600mV到+600mV或-400mV到+400mV。使用的电压优选在具有以下下限和上限的范围内,所述下限选自-400mV,-300mV,-200mV,-150mV,-100mV,-50mV,-20mV和0mV,所述上限独立地选自+10mV,+20mV,+50mV,+100mV,+150mV,+200mV,+300mV和+400mV。使用的电压更优选在100mV到240mV范围内,最优选在120mV到220mV范围内。可通过对孔施加提高的电势来增加对不同核苷酸的鉴别力。
所述方法通常在任何载荷子诸如金属盐(如碱金属盐)、卤盐(如氯化物盐(如碱金属氯化物盐))的存在下实施。载荷子可包括离子液体或有机盐,例如四甲基氯化铵、三甲基苯基氯化铵、苯基三甲基氯化铵或1-乙基-3-甲基氯化咪唑鎓。在上述示例性设备中,所述盐以水溶液存在于室中。通常使用氯化钾(KCl)、氯化钠(NaCl)、氯化铯(CsCl),或亚铁氰化钾和铁氰化钾的混合物。优选KCl、NaCl和亚铁氰化钾和铁氰化钾的混合物。载荷子可以是跨所述膜不对称的。例如,所述载荷子的类型和/或浓度在膜的每一侧上可以不同。
所述盐浓度可以是饱和的。所述盐浓度可以是3M或更低并且通常为0.1-2.5M,0.3-1.9M,0.5-1.8M,0.7-1.7M,0.9-1.6M或1M-1.4M。所述盐浓度优选为150mM-1M。所述方法优选使用至少0.3M,例如至少0.4M,至少0.5M,至少0.6M,至少0.8M,至少1.0M,至少1.5 M,至少2.0M,至少2.5M或至少3.0M的盐浓度而实施。高的盐浓度提供高的信噪比并使得能在正常电流波动的背景下识别代表核苷酸存在的电流。
所述方法通常在缓冲剂的存在下实施。在上述示例性设备中,所述缓冲剂以水溶液存在于所述室中。任何缓冲剂均可用在本发明方法中。通常,所述缓冲剂为磷酸盐缓冲剂。另一个适宜的缓冲剂为HEPES和三羟甲基氨基甲烷-HCl(Tris-HCl)缓冲剂。所述方法通常在4.0-12.0,4.5-10.0,5.0-9.0,5.5-8.8,6.0-8.7或7.0-8.8或7.5-8.5的pH下实施。所用pH优选约为7.5。
所述方法可在0℃-100℃、15℃-95℃、16℃-90℃、17℃-85℃、18℃-80℃、19℃-70℃或20℃-60℃实施。所述方法通常在室温实施。所述方法可选地在能支持酶功能的温度下,例如约37℃实施。
多核苷酸结合蛋白
步骤(a)优选包括使多核苷酸与多核苷酸结合蛋白接触,使得所述蛋白控制多核苷酸穿过所述孔的移动。
更优选地,所述方法包括(a)使多核苷酸与本发明的孔和多核苷酸结合蛋白接触,使得所述蛋白控制所述多核苷酸穿过所述孔的移动,和(b)当多核苷酸相对于所述孔移动时,获取一个或多个测量值,其中所述测量值代表所述多核苷酸的一个或多个特征,由此表征所述多核苷酸。
更优选地,所述方法包括(a)使多核苷酸与本发明的孔和多核苷酸结合蛋白接触,使得所述蛋白控制所述多核苷酸穿过所述孔的移动,和(b)测量多核苷酸相对于所述孔移动时穿过所述孔的电流,其中所述电流代表所述多核苷酸的一个或多个特征,由此表征所述多核苷酸。
所述多核苷酸结合蛋白可以是能够结合到多核苷酸并控制其移动穿过所述孔的任何蛋白质。现有技术中可直接确定蛋白质是否结合到多核苷酸。该蛋白质通常与多核苷酸相互作用并修改其至少一种性质。该蛋白质可通过使多核苷酸裂解形成各单个核苷酸或短链核苷酸如二核苷酸或三核苷酸而修饰该多核苷酸。该蛋白质可以通过将多核苷酸定向或将其移动到特定位置,即控制其移动,而修饰所述多核苷酸。
所述多核苷酸结合蛋白优选地衍生自多核苷酸处理酶。多核苷酸处理酶是能够与多核苷酸相互作用并修饰其至少一种性质的多肽。该酶可以通过使多核苷酸裂解形成各单个核苷酸或短链核苷酸如二核苷酸或三核苷酸而修饰该多核苷酸。该酶可以通过使其定向或将其移动到特定位置而修饰所述多核苷酸。所述多核苷酸处理酶不需要显示酶活性,只要它能够结合多核苷酸并控制其移动穿过所述孔即可。例如,该酶可以被修饰,以除去其酶活性,或者可以在防止其作为酶发挥作用的条件下使用。这样的条件在下面更详细地论述。
所述多核苷酸处理酶优选衍生自溶核酶。在酶的构建体中使用的所述多核苷酸处理酶更优选衍生自酶分类(EC)组3.1.11,3.1.13,3.1.14,3.1.15,3.1.16,3.1.21,3.1.22,3.1.25,3.1.26,3.1.27,3.1.30和3.1.31中的任意成员。所述酶可以是国际申请PCT/GB10/000133(公开号为WO 2010/086603)中公开的任意酶。
优选的酶为聚合酶、核酸外切酶、解旋酶和拓扑异构酶,例如促旋酶。合适的酶包括,但不限于,来自大肠杆菌的核酸外切酶I(SEQ ID NO:11)、来自大肠杆菌的核酸外切酶III(SEQ ID NO:13)、来自嗜热栖热菌(T.thermophilus)的RecJ(SEQ ID NO:15),和噬菌体λ核酸外切酶(SEQ ID NO:17)及其变体。包含SEQ ID NO:15或其变体所示序列的三个亚基相互作用形成三聚体核酸外切酶。所述酶优选是Phi29DNA聚合酶(SEQ ID NO:9)或其变体。拓扑异构酶优选是部分分类(EC)组5.99.1.2和5.99.1.3中的任意成员。
所述酶最优选衍生自解旋酶,如Hel308Mbu(SEQ ID NO:18),Hel308Csy(SEQ ID NO:19),Hel308Mhu(SEQ ID NO:21),TraI Eco(SEQ ID NO:22),XPD Mbu(SEQ ID NO:23)或其变体。本发明可以使用任何解旋酶。所述解旋酶可以是或者衍生自Hel308解旋酶、RecD解旋酶,例如TraI解旋酶或TrwC解旋酶、XPD解旋酶或Dda解旋酶。所述解旋酶可以是国际申请No.PCT/GB2012/052579(公开号WO 2013/057495);PCT/GB2012/053274(公开号WO2013/098562);PCT/GB2012/053273(公开号WO2013098561);PCT/GB2013/051925(公开号WO 2014/013260);PCT/GB2013/051924(公开号WO 2014/013259)和PCT/GB2013/051928(公开号WO 2014/013262);以及PCT/GB2014/052736(公开号WO/2015/055981)中公开的解旋酶、经修饰的解旋酶或解旋酶构建体中的任意解旋酶。
所述解旋酶优选包含SEQ ID NO:25(Trwc Cba)或其变体中所示序列,SEQ ID NO:18(Hel308Mbu)或其变体中所示序列,或者SEQ ID NO:24(Dda)或其变体中所示序列。各变体可以以下文针对跨膜孔所述任意方式不同于天然序列。SEQ ID NO:8的优选变体包含E94C/A360C和之后的(ΔM1)G1G2(即M1缺失,然后添加G1和G2)。
本发明可以使用任何数目的解旋酶。例如,可以使用1,2,3,4,5,6,7,8,9,10或更多个解旋酶。在一些实施方案中,可使用不同数目的解旋酶。
本发明方法优选包括使多核苷酸与两个或更多个解旋酶接触。所述两个或更多个解旋酶通常为相同解旋酶。所述两个或更多个解旋酶可以是不同解旋酶。
所述两个或更多个解旋酶可以是上文提及的解旋酶的任意组合。所述两个或更多个解旋酶可以是两个或更多个Dda解旋酶。所述两个或更多个解旋酶可以是一个或多个Dda解旋酶和一个或多个TrwC解旋酶。所述两个或更多个解旋酶可以是相同解旋酶的不同变体。
所述两个或更多个解旋酶优选彼此连接。所述两个或更多个解旋酶更优选彼此共价连接。所述解旋酶可以以任何顺序使用任何方法连接。用于本发明的优选的解旋酶构建体在国际申请PCT/GB2013/051925(公开号WO 2014/013260);PCT/GB2013/051924(公开号WO 2014/013259)和PCT/GB2013/051928(公开号WO 2014/013262);以及在PCT/GB2014/052736(公开号WO/2015/055981)中有描述。
SEQ ID NO:9,11,13,15,17,18,19,20,21,22,23,24或25的变体是具有下述氨基酸序列的酶:所述氨基酸序列从SEQ ID NO:9,11,13,15,17,18,19,20,21,22,23,24或25变化而来并保留多核苷酸结合能力。这可以使用本领域已知的任何方法测定。例如,使所述变体与多核苷酸接触,然后测量其结合到多核苷酸并沿多核苷酸移动的能力。所述变体可以包括能促进对多核苷酸的结合和/或促进其在高盐浓度和/或室温下活性的修饰。各变体可以被修饰为,使其结合多核苷酸(即保留多核苷酸结合能力),但不具有解旋酶功能(即当提供了有助于移动的所有必要组分例如ATP和Mg2+时仍不能沿多核苷酸移动)。这种修饰是本领域公知的。例如,对解旋酶中Mg2+结合结构域的修饰通常导致不具有解旋酶功能的变体。这些类型的变体可用作分子制动器(见下文)。
基于氨基酸同一性,在SEQ ID NO:9,11,13,15,17,18,19,20,21,22,23,24或25的氨基酸序列的整个长度上,变体将优选与该序列至少50%同源。更优选,基于氨基酸同一性,所述变体多肽可以与SEQ ID NO:9,11,13,15,17,18,19,20,21,22,23,24或25的氨基酸序列在整个长度上,至少55%,至少60%,至少65%,至少70%,至少75%,至少80%,至少85%,至少90%且更优选至少95%,97%或99%同源。在200或更多,例如230,250,270,280,300,400,500,600,700,800,900或1000或更多的连续氨基酸片段上,可以具有至少为80%,例如至少85%,90%或95%的氨基酸同源性(“严格同源性”)。同源性可如上文所述进行确定。所述变体可以上文针对SEQ ID NO:2和4论述的任意方式不同于野生型序列。所述酶可以共价连接到孔。可使用任何方法将所述酶共价连接到孔。
优选的分子制动器是TrwC Cba-Q594A(SEQ ID NO:25,具有突变Q594A)。该变异不具有解旋酶的功能(即能结合多核苷酸,但当提供了有助于移动的所有必要组分例如ATP和Mg2+时仍不能沿多核苷酸移动)。
在链测序中,所述多核苷酸顺着或逆着所施加电势移位穿过所述孔。逐渐地或进行性地作用于双链多核苷酸上的核酸外切酶可用于在施加的电势下在所述孔的顺侧上使剩余的单链穿过,或者在反向电势下在所述孔的反侧上使剩余的单链穿过。同样地,解开双链DNA的解旋酶也可以类似的方式使用。也可以使用聚合酶。还有可能有这样的测序应用,其需要使链逆着施加电势移位,但是DNA必须在反向电势或没有电势的情况下首先被所述酶“捕获”。然后随着在结合之后电势被翻转,所述链将从顺侧到反侧而穿过所述孔并通过电流保持延伸的构造。单链DNA核酸外切酶或单链DNA依赖性聚合酶可用作分子马达,用于以受控和逐步的方式将最近移位的单链逆着施加的电势从反侧到顺侧穿过所述孔而拉回。
所述方法中可以使用任何解旋酶。解旋酶可以两种模式相对于所述孔起作用。首先,所述方法优选使用解旋酶实施,使得所述解旋酶顺着由施加电压产生的场而移动所述多核苷酸穿过所述孔。在这种模式下,多核苷酸的5′端首先在孔中被捕获,然后解旋酶将多核苷酸移动到孔中,使得多核苷酸顺着所述场穿过所述孔,直到其最终移位穿至膜的反侧。替代地,所述方法优选以下述方式实施:使得解旋酶将多核苷酸逆着由施加电压产生的场移动穿过所述孔。在这种模式下,多核苷酸的3′端首先在孔中被捕获,然后解旋酶使多核苷酸移动穿过所述孔,使得多核苷酸逆着施加的场被拉出所述孔,直到其最终返回至膜的顺侧。
所述方法也可以按相反的方向实施。使多核苷酸的3′端首先在孔中被捕获,然后解旋酶可以将多核苷酸移动到所述孔中,使得多核苷酸顺着所述场穿过所述孔,直到其最终移位穿至膜的反侧。
当解旋酶不具有有助于移动的必要组分或被修饰为阻碍或阻止其移动时,所述解旋酶可以结合到多核苷酸并作为制动器在多核苷酸被所施加场拉入孔中时减缓多核苷酸的移动。在非活性模式下,多核苷酸是在3′或5′端被捕获并不重要,是所施加的场用起制动器作用的酶朝着反侧将多核苷酸拉入孔中。当在非活性模式时,解旋酶对多核苷酸的移动控制可以多种方式描述,包括齿合、滑动和制动。缺少解旋酶活性的解旋酶变体也可以这种方式使用。
所述多核苷酸可与多核苷酸结合蛋白和孔以任何顺序接触。优选的是,当多核苷酸与多核苷酸结合蛋白如解旋酶和孔接触时,多核苷酸首先与蛋白质形成复合物。然后当跨越所述孔施加电压时,多核苷酸/蛋白质复合物与所述孔形成复合物,并控制多核苷酸移动穿过所述孔。
使用多核苷酸结合蛋白的方法中的任何步骤通常在游离核苷酸或游离核苷酸类似物以及促进多核苷酸结合蛋白起作用的酶辅因子存在下实施。所述游离核苷酸可以是上述各多核苷酸中的任意一个或多个。所述游离核苷酸包括但不限于,腺苷单磷酸(AMP),腺苷二磷酸(ADP),腺苷三磷酸(ATP),鸟苷单磷酸(GMP),鸟苷二磷酸(GDP),鸟苷三磷酸(GTP),胸苷单磷酸(TMP),胸苷二磷酸(TDP),胸苷三磷酸(TTP),尿苷单磷酸(UMP),尿苷二磷酸(UDP),尿苷三磷酸(UTP),胞苷单磷酸(CMP),胞苷二磷酸(CDP),胞苷三磷酸(CTP),环状腺苷单磷酸(cAMP),环状鸟苷单磷酸(cGMP),脱氧腺苷单磷酸(dAMP),脱氧腺苷二磷酸(dADP),脱氧腺苷三磷酸(dATP),脱氧鸟苷单磷酸(dGMP),脱氧鸟苷二磷酸(dGDP),脱氧鸟苷三磷酸(dGTP),脱氧胸苷单磷酸(dTMP),脱氧胸苷二磷酸(dTDP),脱氧胸苷三磷酸(dTTP),脱氧尿苷单磷酸(dUMP),脱氧尿苷二磷酸(dUDP),脱氧尿苷三磷酸(dUTP),脱氧胞苷单磷酸(dCMP),脱氧胞苷二磷酸(dCDP)和脱氧胞苷三磷酸(dCTP)。所述游离核苷酸优选选自AMP,TMP,GMP,CMP,UMP,dAMP,dTMP,dGMP或dCMP。所述游离核苷酸优选为腺苷三磷酸(ATP)。所述酶辅因子为使构建体发挥功能的因子。所述酶辅因子优选为二价金属阳离子。所述二价金属阳离子优选为Mg2+,Mn2+,Ca2+或Co2+。所述酶辅因子最优选为Mg2+。
解旋酶(一个或多个)和分子制动器(一个或多个)
在优选的实施方案中,所述方法包括:
(a)提供所述多核苷酸,具有一个或多个解旋酶和一个或多个分子制动器连接到所述多核苷酸;
(b)使所述多核苷酸与本发明的孔接触并跨所述孔施加电势,使得所述一个或多个解旋酶和所述一个或多个分子制动器联合在一起来控制所述多核苷酸移动穿过所述孔;
(c)当所述多核苷酸相对于所述孔移动时,获取一个或多个测量值,其中所述测量值代表所述多核苷酸的一个或多个特征,由此表征所述多核苷酸。这类方法在国际申请PCT/GB2014/052737中有详细论述。
所述一个或多个解旋酶可以是上文论述的任何解旋酶。所述一个或多个分子制动器可以是能结合到所述多核苷酸并减缓所述多核苷酸穿过所述孔的移动的任何化合物或分子。所述一个或多个分子制动器优选包括能结合到所述多核苷酸的一种或多种化合物。所述一种或多种化合物优选为一种或多种大环化合物。合适的大环化合物包括,但不限于,环糊精、杯芳烃、环肽、冠醚、葫芦脲、柱芳烃、其衍生物或其组合。所述环糊精或其衍生物可以是任何在Eliseev,A.V.,and Schneider,H-J.(1994)J..Am.Chem.Soc.116,6081-6088中公开的那些。该试剂更优选为七-6-氨基-β-环糊精(am7-βCD)、6-单脱氧-6-单氨基-β-环糊精(am1-βCD)或七-(6-脱氧-6-胍基)-环糊精(gu7-βCD)。
所述一个或多个分子制动器优选不为一个或多个单链结合蛋白(SSB)。所述一个或多个分子制动器更优选不为包含不具有净负电荷的羧基末端(C-末端)区域的单链结合蛋白(SSB),或不为(ii)C-末端区域中包含能减少C-末端区域的净负电荷的一个或多个修饰的经修饰SSB。所述一个或多个分子制动器最优选不为在国际申请PCT/GB2013/051924(公开号WO 2014/013259)中公开的任何SSB。
所述一个或多个分子制动器优选为一个或多个多核苷酸结合蛋白。所述多核苷酸结合蛋白可以是能够结合到多核苷酸并控制其移动穿过所述孔的任何蛋白质。现有技术中可直接确定蛋白质是否结合到多核苷酸。所述蛋白通常与多核苷酸相互作用并修饰所述多核苷酸的至少一种特性。所述蛋白可通过使多核苷酸裂解形成各单个核苷酸或短链核苷酸,如二核苷酸或三核苷酸,来修饰所述多核苷酸。该部分可以通过将多核苷酸定向或将其移动到特定位置,即控制其移动,来修饰所述多核苷酸。
所述多核苷酸结合蛋白优选衍生自多核苷酸处理酶。所述一个或多个分子制动器可以衍生自上述的任何多核苷酸处理酶。用作分子制动器的Phi29聚合酶的经修饰的版本(SEQ ID NO:9)在美国专利No.5576204中公开。充当分子制动器的Phi29聚合酶的经修饰的变体(SEQ ID NO:8)在美国专利5,576,204中有公开。所述一个或多个分子制动器优选衍生自解旋酶。
可以使用任何数量的衍生自解旋酶的分子制动器。例如,可以使用1、2、3、4、5、6、7、8、9、10或更多个解旋酶作为分子制动器。如果使用两个或更多个解旋酶作为分子制动器,所述两个或更多个解旋酶通常是相同的解旋酶。所述两个或更多个解旋酶可以是不同的解旋酶。
所述两个或更多个解旋酶可以是上文提到的解旋酶的任意组合。所述两个或更多个解旋酶可以是两个或更多个Dda解旋酶。所述两个或更多个解旋酶可以是一个或多个Dda解旋酶和一个或多个TrwC解旋酶。所述两个或更多个解旋酶可以是相同解旋酶的不同变体。
所述两个或更多个解螺旋酶优选彼此连接。所述两个或更多个解旋酶更优选彼此共价连接。所述解旋酶可以以任何顺序使用任何方法连接。从解旋酶衍生的所述一个或多个分子制动器优选被修饰以减少多核苷酸结合结构域域中开口的大小,所述多核苷酸可在至少一个构象状态下通过所述开口从解旋酶解绑。这在WO 2014/013260中公开。
用于本发明的优选的解旋酶构建体在国际申请No.PCT/GB2013/051925(公开号WO2014/013260);PCT/GB2013/051924(公开号WO 2014/013259)和PCT/GB2013/051928(公开号WO 2014/013262);和在PCT/GB2014/052736(公开号WO/2015/055981)中有描述。
间隔区
所述一个或多个解旋酶可以在一个或多个间隔区停滞,如在国际申请No.PCT/GB2014/050175中论述的。本发明中可以使用在该国际申请中公开的一个或多个解旋酶和一个或多个间隔区的任意构造。
膜
本发明的孔可以存在于膜中。在本发明方法中,多核苷酸通常与本发明的孔在膜中接触。任何膜可以按照本发明使用。合适的膜是本领域公知的。所述膜优选为两亲层。两亲层是由具有亲水性和亲脂性的两亲分子形成的层,如磷脂。所述两亲分子可以是合成的或天然存在的。非天然存在的两亲物和形成单层的两亲物在本领域中是已知的,且包括,例如,嵌段共聚物(Gonzalez-Perez等人,Langmuir,2009,25,10447-10450)。嵌段共聚物是其中两个或更多个单体亚基一起聚合以生成单个聚合物链的聚合物材料。嵌段共聚物通常具有由每个单体亚基贡献的特性。然而,嵌段共聚物可具有由各个亚基形成的聚合物不具有的独特的特性。嵌段共聚物可以被加工,使得单体亚基中的一个是疏水性的(即亲脂的),而其他亚基在含水介质中是亲水性的。在这种情况下,该嵌段共聚物可具有两亲特性,并且可以形成模仿生物膜的结构。嵌段共聚物可以是二嵌段(由两个单体亚基组成),但也可以由多于两个的单体亚基构成以形成充当两亲物的更复杂的排列。所述共聚物可以是三嵌段、四嵌段或五嵌段共聚物。膜优选为三嵌段共聚物膜。
古细菌双极四醚脂质是天然存在的脂质,其被构造为使得所述脂质形成单层膜。这些脂质通常在恶劣的生物环境生存的极端微生物、嗜热菌、嗜盐菌和嗜酸菌中被发现。它们的稳定性被认为是从最终双层的融合性质中衍生的。比较简单的是,通过生成具有亲水性-疏水性-亲水性一般基序的三嵌段聚合物构造模仿这些生物实体的嵌段共聚物材料。所述材料可以形成行为类似于脂质双层且包含从囊泡到层状膜的一系列相似行为的单体膜。由这些三嵌段共聚物形成的膜与生物脂质膜相比持有一些优点。因为三嵌段共聚物是合成的,因此可以仔细控制其确切构造以提供用于形成膜和与孔及其它蛋白相互作用所需的正确链长度和特性。
嵌段共聚物也可以从不被分类为脂质辅料(lipid sub-materials)的亚基构建;例如疏水性聚合物可从硅氧烷或其它非基于烃的单体制成。嵌段共聚物的亲水性子段也可具有低的蛋白质结合特性,这使得当暴露于未处理的生物样品时,其可生成高抗性的膜。该头部基团单元也可以从非经典的脂质头部基团衍生。
三嵌段共聚物膜与生物脂质膜相比,还具有增强的机械稳定性和环境稳定性,例如高得多的操作温度或pH范围。嵌段共聚物的合成性质为定制用于广泛应用范围的基于聚合物的膜提供了平台(platform)。
所述膜最优选为在国际申请No.PCT/GB2013/052766或PCT/GB2013/052767中公开的膜之一。
所述两亲分子可以是经化学修饰的或官能化的以有助于多核苷酸的耦合。
该两亲层可以是单层或双层。两亲层通常是平面的。两亲层可以是弯曲的。两亲层可以被支撑。
两亲膜通常天然是移动的,本质上充当具有约10-8cm s-1的脂质扩散速率的二维流体。这意味着孔和耦合的多核苷酸通常在两亲膜内移动。
膜可以是脂质双层。脂质双层是细胞膜的模型,并充当用于一系列实验研究的优秀的平台。例如,脂质双层可通过单通道记录用于膜蛋白的体外研究。或者,脂质双层可用作生物传感器以检测一系列物质的存在。脂质双层可以是任何脂质双层。合适的脂质双层包括,但不限于,平面脂双层、支撑双层或脂质体。脂质双层优选平面脂质双层。合适的脂质双层在国际申请No.PCT/GB08/000563(公开号WO 2008/102121)、国际申请No.PCT/GB08/004127(公开号WO2009/077734)和国际申请No.PCT GB2006/001057(公开号WO 2006/100484)中公开。
耦合
所述多核苷酸优选耦合到包含本发明孔的膜。该方法可以包括将多核苷酸耦合到包含本发明孔的膜。所述多核苷酸优选地使用一个或多个锚耦合到膜。所述多核苷酸可以使用任何已知方法耦合到膜。
每个锚包括一个耦合(或结合)到多核苷酸的基团和耦合(或结合)到膜的基团。每个锚可以共价耦合(或结合)到多核苷酸和/或膜。如果使用Y适配器和/或发夹环适配器,则多核苷酸优选地使用适配器(一个或多个)耦合到膜。
多核苷酸可以使用任何数目的锚耦合到膜,如2、3、4各或更多锚。例如,多核苷酸可以使用两个锚耦合到膜,每个锚分别耦合(或结合)到多核苷酸和膜。
所述一个或多个锚可以包括一个或多个解旋酶和/或一个或多个分子制动器。
如果膜是两亲性层,如共聚物膜或脂质双层,则所述一个或多个锚优选地包括在所述膜中存在的多肽锚和/或在所述膜中存在的疏水锚。所述疏水锚优选是脂质、脂肪酸、甾醇、碳纳米管、多肽、蛋白质或氨基酸,例如胆固醇、棕榈酸酯或生育酚。在优选的实施方案中,所述一个或多个锚不是孔。
膜的组分,如两亲分子、共聚物或脂质,可以是化学修饰的或官能化的,以形成一个或多个锚。合适的化学修饰和合适的官能化所述膜的组分的方式的实例在下面更详细地讨论。任何比例的膜组分可以被官能化,例如至少0.01%、至少0.1%、至少1%、至少10%、至少25%、至少50%或100%。
多核苷酸可以直接耦合到膜。用于将多核苷酸耦合到膜的一个或多个锚优选包含接头。所述一个或多个锚可包含一个或多个接头,例如2、3、4个或更多。一个接头可用于耦合多于1个,例如2、3、4或更多个多核苷酸到膜上。
优选的接头包括,但不限于,聚合物,如多核苷酸、聚乙二醇(PEG)、多糖和多肽。这些接头可以是直链的、支链的或环状的。例如,所述连接体可以是环形的多核苷酸。所述多核苷酸可杂交到环状多核苷酸接头上的互补序列。
所述耦合可以是永久的或稳定的。换言之,耦合可以是,使得当多核苷酸与孔相互作用时,多核苷酸保持耦合到膜上。
该耦合可以是短暂的。换言之,耦合可以是,使得当多核苷酸与孔相互作用时,多核苷酸可以从膜上解耦合。
将多核苷酸耦合到接头或官能化的膜上还可以通过许多其他手段实现,条件是互补的反应性基团或锚定基团被添加到所述多核苷酸。反应性基团添加到多核苷酸的任一端之前已经有报道。所述一个或多个锚优选将多核苷酸通过杂交耦合到膜。在一个或多个锚中的杂交使得耦合以瞬时方式进行,如上所述。所述一个或多个锚可包括单链或双链多核苷酸。锚的一部分可以接合到单链或双链多核苷酸。使用T4RNA连接酶I连接ssDNA的短片段已经被报道(Troutt,A.B.,M.G.McHeyzer-Williams等人(1992).″连接-锚定PCR:具有单侧特异性的简单扩增技术.″Proc Natl Acad Sci USA 89(20):9823-5)。或者,单链或双链多核苷酸可以连接到双链多核苷酸,然后通过热变性或化学变性分离两条链。如果多核苷酸是合成的链,所述一个或多个锚可以在多核苷酸的化学合成过程中引入。例如,多核苷酸可以使用连接有反应性基团的引物来合成。
理想地,多核苷酸耦合到膜,而无需必须将所述多核苷酸官能化。这可以通过将所述一个或多个锚,如多核苷酸结合蛋白或化学基团,耦合到膜,并使得所述一个或多个锚与所述多核苷酸相互作用或通过官能化所述膜来实现。所述一个或多个锚可以通过本文所述的任何方法耦合到膜。特别地,所述一个或多个锚可以包含一个或多个接头,如马来酰亚胺官能化的接头。
在本实施方案中,多核苷酸通常是RNA,DNA,PNA,TNA或LNA,并且可以是双链或单链的。这一实施方案特别适合于基因组DNA多核苷酸。
如果所述一个或多个锚包含蛋白,则它们无需进一步官能化,能够直接锚定到膜中,例如,如果所述蛋白已经具有一个与膜相容的外部疏水区时。这类蛋白的实例包括,但不限于,跨膜蛋白、膜内蛋白和膜蛋白。
根据优选的实施方案,当多核苷酸结合到优先旋入孔中的前导序列时,所述一个或多个锚可用于将多核苷酸耦合到膜上。前导序列在下面更详细地论述。优选地,该多核苷酸被结合到(例如连接到)优先旋入孔中的前导序列。这种前导序列可以包含均聚多核苷酸或脱碱基区。前导序列通常被设计为,直接杂交到或通过一个或多个间体多核苷酸(或夹板体)杂交到所述一个或多个锚。在这种情况下,所述一个或多个锚通常包含与前导序列中的序列或所述一个或多个中间体多核苷酸(或夹板体)中的序列互补的多核苷酸序列。在这种情况下,所述一个或多个夹板体通常包含与前导序列中的序列互补的多核苷酸序列。
双链多核苷酸
所述多核苷酸可以是双链的。如果多核苷酸是双链的,则在接触步骤之前,所述方法优选进一步包括将发夹适配器接合到所述多核苷酸的一端。然后所述多核苷酸的两条链可在所述多核苷酸与本发明的孔接触时或接触之前分离。当多核苷酸穿过所述孔的移动是由多核苷酸结合蛋白,如解旋酶或分子制动器控制时,所述两条链可以被分离。这在国际申请No.PCT/GB2012/051786(公开号WO 2013/014451)中有描述。将两条链以这种方式连接或相互接合到双链构建体上提高了表征的效率和准确性。
拐角处测序(Round the corner sequencing)
在一优选的实施方案中,目标双链多核苷酸的一端设有一个发夹环适配器,并且所述方法包括使多核苷酸与本发明的孔接触,使得多核苷酸的两条链移动穿过所述孔,以及当多核苷酸的两条链相对于所述孔移动时,获取一个或多个测量值,其中所述测量值代表多个核苷酸的链的一个或多个特征,由此表征该目标双链多核苷酸。上面论述的任何实施方案也同样适用于本实施方案。
前导序列
在接触步骤之前,所述方法优选包括将优先旋入所述孔的前导序列连接到所述多核苷酸。所述前导序列有利于本发明方法。所述前导序列被设计为优先旋入本发明的孔中,从而便于所述多核苷酸移动穿过所述孔。所述前导序列还可以用于如以上所论述的将所述多核苷酸连接到一个或多个锚。
双耦合
本发明方法可以包括双链多核苷酸的双耦合。在一优选的实施方案中,本发明方法包括:
(a)提供双链多核苷酸,其一端具有Y适配器且另一端具有发夹环适配器,其中,Y适配器包含用于将所述多核苷酸耦合到膜的一个或多个第一锚,所述发夹环适配器包含用于将所述多核苷酸耦合到所述膜的一个或多个第二锚,且其中,所述发夹环适配器耦合到所述膜的强度大于所述Y适配器耦合到所述膜的强度;
(b)使步骤(a)中提供的所述多核苷酸与本发明的孔接触,使得所述多核苷酸移动穿过所述孔;和
(c)当所述多核苷酸相对于所述孔移动时获取一个或多个测量值,其中所述测量值代表所述多核苷酸的一个或多个特征,由此表征该目标多核苷酸。这类方法在国际申请PCT/GB2015/050991中有详细论述。
添加发夹环和前导序列
提供之前,双链多核苷酸可以与MuA转座酶和一组双链MuA底物接触,其中所述组中一部分底物为包含前导序列的Y适配器,并且其中所述组中一部分底物为发夹环适配器。转座酶使双链多核苷酸分析物片段化并将MuA底物接合到片段的一端或两端。这产生多个经修饰的双链多核苷酸,所述经修饰的双链多核苷酸一端含有前导序列且另一端含有发夹环。然后,使用本发明方法研究所述经修饰的双链多核苷酸。这些基于MuA的方法在国际申请PCT/GB2014/052505(公开号WO/2015/022544)中公开。它们也在国际申请PCT/GB2015/050991中被详细论述。
一个或多个解旋酶可以在与所述双链多核苷酸和MuA转座酶接触之前,连接到MuA底物Y适配器。替代地,一个或多个解旋酶可以在与所述双链多核苷酸和MuA转座酶接触之前,结合到MuA底物发夹环适配器。
一个或多个分子制动器可以在与所述双链多核苷酸和MuA转座酶接触之前,连接到MuA底物发夹环适配器。替代地,一个或多个分子制动器可以在与所述双链多核苷酸和MuA转座酶接触之前,连接到MuA底物Y适配器。
解耦合
本发明方法可以包括表征多个目标多核苷酸和解耦合至少第一目标多核苷酸。
在一优选实施方案中,本发明包括表征两个或更多个目标多核苷酸。所述方法包括:
(a)提供第一样本中的第一多核苷酸;
(b)提供第二样本中的第二多核苷酸;
(c)将第一样本中的第一多核苷酸使用一个或多个锚耦合到膜;
(d)使第一多核苷酸与本发明的孔接触,使得第一多核苷酸移动穿过所述孔;
(e)当第一多核苷酸相对于所述孔移动时获取一个或多个测量值,其中所述测量值代表第一多核苷酸的一个或多个特征,由此表征第一多核苷酸。
(f)将第一多核苷酸从所述膜上解耦合;
(g)将第二样本中的第二多核苷酸使用一个或多个锚耦合到所述膜;
(h)使第二多核苷酸与本发明的孔接触,使得第二多核苷酸移动穿过所述孔;和
(i)当第二多核苷酸相对于所述孔移动时获取一个或多个测量值,其中所述测量值代表第二多核苷酸的一个或多个特征,由此表征第二多核苷酸。
这类方法在国际申请PCT/GB2015/050992中被详细论述。
如果一个或多个锚包含疏水性锚,例如胆固醇,则该试剂优选是环糊精或其衍生物或脂质。所述环糊精或其衍生物可以是任何在Eliseev,A.V.,and Schneider,H-J.(1994)J.Am.Chem.Soc.116,6081-6088中公开的那些。该试剂更优选为七-6-氨基-β-环糊精(am7-βCD)、6-单脱氧-6-单氨基-β-环糊精(am1-βCD)或七-(6-脱氧-6-胍基)-环糊精(gu7-βCD)。本文公开的任何脂质都可以使用。
修饰的多核苷酸
表征前,在所述聚合酶使用所述目标多核苷酸作为模板形成经修饰多核苷酸的条件下,目标多核苷酸可通过使该多核苷酸与聚合酶和一组游离核苷酸接触而进行修饰,其中所述聚合酶在形成所述经修饰多核苷酸时用不同核苷酸物质替代目标多核苷酸中的一个或多个核苷酸物质。然后,可对经修饰多核苷酸提供一个或多个连接到该多核苷酸的解旋酶和一个或多个连接到该多核苷酸的分子制动器。这类修饰在国际申请PCT/GB2015/050483中进行了描述。上面论述的任何聚合酶均可以使用。聚合酶优选为Klenow或9o North。
在所述聚合酶使用模板多核苷酸作为模板形成经修饰多核苷酸的条件下,使所述模板多核苷酸与聚合酶接触。这种条件是本领域已知的。例如,通常使该多核苷酸与所述聚合酶在市售聚合酶缓冲液中接触,如购自New England的缓冲液。温度优选为,Klenow时为20-37℃,9o North时为60-75℃。通常使用引物或3′发夹作为聚合酶延伸的成核点。
使用跨膜孔表征,如测序,多核苷酸通常包括,分析由k个核苷酸构成的聚合物单元,其中k是正整数(即,‘k聚体’)。这在国际申请PCT/GB2012/052343(公开号WO2013/041878)中进行了论述。虽然希望的是,不同k聚体的电流测量值之间具有明确的分离,但是通常这些测量值中的一些是重叠的。尤其是k聚体中具有高数目的聚合物单元,即高k值时,变得难以分辨由不同k聚体产生的测量值,难以确定关于多核苷酸的导出信息,例如评估该多核苷酸的潜在序列(underlying sequence)。
通过将目标多核苷酸中的一个或多个核苷酸物质用经修饰多核苷酸中的不同核苷酸物质替代,所述经修饰多核苷酸含有与目标多核苷酸中的k聚体不同的k聚体。所述经修饰多核苷酸中的所述不同的k聚体能够产生与目标多核苷酸中的k聚体不同的电流测量值,由此所述经修饰多核苷酸提供来自目标多核苷酸的不同信息。来自所述经修饰多核苷酸的这些额外信息使得更容易表征所述目标多核苷酸。在一些情况下,所述经修饰多核苷酸本身可以更容易地表征。例如,可将所述经修饰多核苷酸设计为包括其电流测量值之间具有增强的分离或明确的分离的k聚体,或者包括具有降低的噪声的k聚体。
所述聚合酶优选将目标多核苷酸中的两个或更多个核苷酸物质在形成经修饰多核苷酸时用不同核苷酸物质替代。所述聚合酶可以用不同核苷酸物质替代目标多核苷酸中所述两种或更多种核苷酸物质中的每一个。所述聚合酶可以用相同核苷酸物质替代目标多核苷酸中所述两种或更多种核苷酸物质中的每一个。
如果目标多核苷酸为DNA,则所述经修饰多核苷酸中的不同核苷酸物质通常含有不同于腺嘌呤、鸟嘌呤、胸腺嘧啶、胞嘧啶或甲基胞嘧啶的核碱基,和/或含有不同于脱氧腺苷、脱氧鸟苷、胸苷、脱氧胞苷或脱氧甲基胞苷的核苷。如果目标多核苷酸是RNA,则所述经修饰多核苷酸中的不同核苷酸物质通常含有不同于腺嘌呤、鸟嘌呤、尿嘧啶、胞嘧啶或甲基胞嘧啶的核碱基,和/或含有不同于腺苷、鸟苷、尿苷、胞苷或甲基胞苷的核苷。所述不同核苷酸物质可以是任何在上文论述的一般核苷酸。
所述聚合酶可以用不同核苷酸物质替代所述一个或多个核苷酸物质,所述不同核苷酸物质包含所述一个或多个核苷酸物质中不存在的化学基团或原子。所述化学基团可以是丙炔基、硫基、氧基、甲基、羟甲基、甲酰基、羧基、羰基、苄基、炔丙基或炔丙胺基。
所述聚合酶可以用缺少所述一个或多个核苷酸物质中的化学基团或原子的不同核苷酸物质替代所述一个或多个核苷酸物质。所述聚合酶可以用具有改变的电负性的不同核苷酸物质替代所述一个或多个核苷酸物质。所述具有改变的电负性的不同核苷酸物质优选包括卤素原子。
所述方法优选进一步包括,选择性地移除所述经修饰多核苷酸中的所述一个或多个不同核苷酸物质中的核碱基。
其他表征方法
在另一个实施方案中,通过检测被标记的且当聚合酶将核苷酸插入多核苷酸时释放的物质来表征所述多核苷酸。所述聚合酶使用所述多核苷酸作为模板。各个标记的物质对每个核苷酸具有特异性。使所述多核苷酸与本发明的孔、聚合酶和标记的核苷酸接触,使得在核苷酸通过聚合酶添加到所述多核苷酸(一个或多个)时,磷酸盐标记的物质依次释放,其中磷酸盐物质包含对每个核苷酸具有特异性的标记物。所述聚合酶可以是任何上文论述的聚合酶。使用所述孔检测磷酸盐标记的物质,由此表征所述多核苷酸。这类方法在欧洲专利申请13187149.3(公开号EP 2682460)中有公开。上文论述的任何实施方案同样适用于该方法。
试剂盒
本发明还提供了用于表征目标多核苷酸的试剂盒。所述试剂盒包含本发明的孔和膜的组分。所述膜优选由所述组分形成。所述孔优选存在于所述膜中。所述试剂盒可包括上文公开的任何膜(例如两亲性层或三嵌段共聚物膜)的组分。
所述试剂盒还可以包括多核苷酸结合蛋白。
所述试剂盒还可以包括用于将所述多核苷酸耦合到所述膜的一个或多个锚。
所述试剂盒优选用于表征双链多核苷酸并优选包括Y适配器和发夹环适配器。Y适配器优选连接有一个或多个解旋酶,发夹环适配器优选连接有一个或多个分子制动器。Y适配器优选包括一个或多个第一锚,用于将所述多核苷酸耦合到所述膜,所述发夹环适配器优选包括一个或多个第二锚,用于将所述多核苷酸耦合到所述膜,所述发夹环适配器耦合到所述膜的强度大于所述Y适配器耦合到所述膜的强度。
本发明的试剂盒可另外包括一种或多种能实施上述任何实施方案的其他试剂或仪器。所述试剂或仪器包括以下中的一种或多种:合适的缓冲剂(水溶液)、用于从受体获得样本的工具(如含有针的导管或仪器)、用于扩增和/或表达多核苷酸的工具,或者电压钳或膜片钳设备。试剂可以干态存在于所述试剂盒中,使得液体样本使该试剂重悬(resuspend)。可选地,所述试剂盒还可包括使该试剂盒能在本发明方法中使用的说明书或关于该方法可以用于哪种有机体的详细资料。
设备
本发明还提供用于表征目标多核苷酸的设备。该设备包括多个本发明的孔和多个膜。所述多个孔优选存在于所述多个膜中。孔和膜的数量优选相等。优选地,每个膜中存在单个孔。
所述设备优选进一步包括用于实施本发明方法的说明书。所述设备可以是任何用于多核苷酸分析的常规设备,例如阵列或芯片。上面论述的关于本发明方法的任何实施方案同样适用于本发明的设备。所述设备可以进一步包括存在于本发明试剂盒中的任何特征。
所述设备优选被设置成用于实施本发明方法。
所述设备优选包括:
传感器装置,其能够支持所述多个孔和膜并且为可操作的以使用所述孔和膜来实施多核苷酸表征;和
至少一个端口,其用于传输实施表征所用的材料。
或者,所述设备优选包括:
传感器装置,其能够支持所述多个孔和膜为可操作的以使用所述孔和膜来实施多核苷酸表征;和
至少一个存储器,用于容纳实施表征所用的材料。
所述设备更优选包括:
传感器装置,其能够支持所述膜和多个孔和膜为可操作的以使用所述孔和膜来实施多核苷酸表征;
至少一个存储器,用于容纳实施表征所用的材料;
流体系统,其配置成将材料从至少一个所述存储器可控地供应到所述传感器装置;和
一个或多个容器,其用于接收各个样本,所述流体系统被配置成将所述样本选择性地从一个或多个容器供应到所述传感器装置。
所述设备可以是那些在国际申请No.PCT/GB08/004127(公开号WO 2009/077734)、PCT/GB10/000789(公开号WO 2010/122293)、国际申请No.PCT/GB10/002206(公开号WO 2011/067559)或国际申请No.PCT/US99/25679(公开号WO 00/28312)中描述的任何设备。
以下实施例示意说明本发明。
实施例1
该实施例描述了使用解旋酶-T4Dda-E94C/C109A/C136A/A360C(SEQ ID NO:24具有突变E94C/C109A/C136A/A360C)来控制DNA构建体X移动(图1中示出)通过多个不同MspA纳米孔。测试的所有纳米孔显示了当DNA移位通过纳米孔时的电流变化。
材料和方法
建立实验之前,用T4Dda-E94C/C109A/C136A/A360C(加入到纳米孔系统的终浓度为10nM,具有突变E94C/A360C的SEQ ID NO:24在缓冲液(253mM KCl,50mM磷酸钾,pH8.0,2mM EDTA)中提供)将DNA构建体X或Y(参照图1和2,分别为构建体X和Y中使用的图解和序列,加入到纳米孔系统的终浓度为0.1nM)在室温下预培育5分钟。5分钟后,向预混物中添加TMAD(加入到纳米孔体系的终浓度为100μM),并将该混合物再培育5分钟。最后,向预混物中添加MgCl2(加入到纳米孔体系的终浓度为1或2mM)、ATP(加入到纳米孔体系的终浓度为2mM)和KCl(加入到纳米孔体系的终浓度为500mM)。
从在缓冲液(500mM KCl,25mM磷酸钾pH 8.0)中插入嵌段共聚物的单MspA纳米孔获得电测量值(测试的测量值请参阅下表5)。在将单个孔插入所述嵌段共聚物之后,使缓冲液(1mL,500mM KCl,25mM磷酸钾)流经该系统,以除去任何过量的MspA纳米孔。然后将酶(T4Dda-E94C/C109A/C136A/A360C,10nM终浓度)、DNA构建体X或Y(0.1nM终浓度)、燃料((MgCl2,1或2mM终浓度,ATP 2mM终浓度)预混物(共150μL)流入所述单纳米孔实验系统中,并使用以下实验方案运行该实验:120mV下900秒,-180mV下2秒,0mV下2秒,然后返回到120mV下900秒(该翻转实验方案重复8次),监测解旋酶控制的DNA移动。
结果
所测试的MspA纳米孔示于下表3中。使用T4Dda-E94C/C109A/C136A/A360C,对DNA构建X和Y观察解旋酶控制的DNA移动(分别为图1和2)(见表5,其中各图对应于所测试的各纳米孔突变体)。
表3
进入位22(图19)示出了MspA突变体MspA-(G75S/G77S/L88N/D90N/D91N/D93N/D118R/Q126R/D134R/E139K)8,其位置93处的天冬氨酸被甘氨酸取代。当DNA移位穿过该突变体时,观察到解旋酶控制的DNA移动。但是进入1(图3)的位置93变回天冬氨酸MspA-(G75S/G77S/L88N/D90N/D91N/D118R/Q126R/D134R/E139K)8(其为同一氨基酸,存在于野生型MspA中),并且,其也表现出解旋酶控制的DNA移动。因此,可以将天冬氨酸放回纳米孔的氨基酸序列,并且仍能观察到解旋酶控制的DNA移动。
序列表
<110> 牛津纳米孔技术公司
<120> 突变孔
<130> N402816WO
<150> GB 1407809.1
<151> 2014-05-02
<150> GB 1417712.5
<151> 2014-10-07
<150> GB 1417708.3
<151> 2014-10-07
<160> 29
<170> PatentIn version 3.5
<210> 1
<211> 552
<212> DNA
<213> 耻垢分枝杆菌(Mycobacterium smegmatis)
<400> 1
ggcctggata acgaacttag cctggtggac ggccaagatc gcacgctgac ggtgcaacaa 60
tgggatacct tcctgaatgg tgtgtttccg ctggatcgta accgcctgac ccgtgaatgg 120
tttcattccg gtcgcgcaaa atatatcgtc gcaggcccgg gtgctgacga attcgaaggc 180
acgctggaac tgggttatca gattggcttt ccgtggtcac tgggcgttgg tatcaacttc 240
tcgtacacca cgccgaatat tctgatcgat gacggtgata ttaccgcacc gccgtttggc 300
ctgaacagcg tgattacgcc gaacctgttt ccgggtgtta gcatctctgc cgatctgggc 360
aacggtccgg gcattcaaga agtggcaacc tttagtgtgg acgtttccgg cgctgaaggc 420
ggtgtcgcgg tgtctaatgc ccacggtacc gttacgggcg cggccggcgg tgtcctgctg 480
cgtccgttcg cgcgcctgat tgcgagcacc ggcgactctg ttacgaccta tggcgaaccg 540
tggaatatga ac 552
<210> 2
<211> 184
<212> PRT
<213> 耻垢分枝杆菌
<400> 2
Gly Leu Asp Asn Glu Leu Ser Leu Val Asp Gly Gln Asp Arg Thr Leu
1 5 10 15
Thr Val Gln Gln Trp Asp Thr Phe Leu Asn Gly Val Phe Pro Leu Asp
20 25 30
Arg Asn Arg Leu Thr Arg Glu Trp Phe His Ser Gly Arg Ala Lys Tyr
35 40 45
Ile Val Ala Gly Pro Gly Ala Asp Glu Phe Glu Gly Thr Leu Glu Leu
50 55 60
Gly Tyr Gln Ile Gly Phe Pro Trp Ser Leu Gly Val Gly Ile Asn Phe
65 70 75 80
Ser Tyr Thr Thr Pro Asn Ile Leu Ile Asp Asp Gly Asp Ile Thr Ala
85 90 95
Pro Pro Phe Gly Leu Asn Ser Val Ile Thr Pro Asn Leu Phe Pro Gly
100 105 110
Val Ser Ile Ser Ala Asp Leu Gly Asn Gly Pro Gly Ile Gln Glu Val
115 120 125
Ala Thr Phe Ser Val Asp Val Ser Gly Ala Glu Gly Gly Val Ala Val
130 135 140
Ser Asn Ala His Gly Thr Val Thr Gly Ala Ala Gly Gly Val Leu Leu
145 150 155 160
Arg Pro Phe Ala Arg Leu Ile Ala Ser Thr Gly Asp Ser Val Thr Thr
165 170 175
Tyr Gly Glu Pro Trp Asn Met Asn
180
<210> 3
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> 实施例1中使用的多核苷酸序列
<400> 3
tttttttttt tttttttttt 20
<210> 4
<211> 3595
<212> DNA
<213> 人工序列
<220>
<223> 实施例1中使用的多核苷酸序列
<400> 4
gccatcagat tgtgtttgtt agtcgctgcc atcagattgt gtttgttagt cgcttttttt 60
ttttggaatt ttttttttgg aatttttttt ttgcgctaac aacctcctgc cgttttgccc 120
gtgcatatcg gtcacgaaca aatctgatta ctaaacacag tagcctggat ttgttctatc 180
agtaatcgac cttattccta attaaataga gcaaatcccc ttattggggg taagacatga 240
agatgccaga aaaacatgac ctgttggccg ccattctcgc ggcaaaggaa caaggcatcg 300
gggcaatcct tgcgtttgca atggcgtacc ttcgcggcag atataatggc ggtgcgttta 360
caaaaacagt aatcgacgca acgatgtgcg ccattatcgc ctagttcatt cgtgaccttc 420
tcgacttcgc cggactaagt agcaatctcg cttatataac gagcgtgttt atcggctaca 480
tcggtactga ctcgattggt tcgcttatca aacgcttcgc tgctaaaaaa gccggagtag 540
aagatggtag aaatcaataa tcaacgtaag gcgttcctcg atatgctggc gtggtcggag 600
ggaactgata acggacgtca gaaaaccaga aatcatggtt atgacgtcat tgtaggcgga 660
gagctattta ctgattactc cgatcaccct cgcaaacttg tcacgctaaa cccaaaactc 720
aaatcaacag gcgccggacg ctaccagctt ctttcccgtt ggtgggatgc ctaccgcaag 780
cagcttggcc tgaaagactt ctctccgaaa agtcaggacg ctgtggcatt gcagcagatt 840
aaggagcgtg gcgctttacc tatgattgat cgtggtgata tccgtcaggc aatcgaccgt 900
tgcagcaata tctgggcttc actgccgggc gctggttatg gtcagttcga gcataaggct 960
gacagcctga ttgcaaaatt caaagaagcg ggcggaacgg tcagagagat tgatgtatga 1020
gcagagtcac cgcgattatc tccgctctgg ttatctgcat catcgtctgc ctgtcatggg 1080
ctgttaatca ttaccgtgat aacgccatta cctacaaagc ccagcgcgac aaaaatgcca 1140
gagaactgaa gctggcgaac gcggcaatta ctgacatgca gatgcgtcag cgtgatgttg 1200
ctgcgctcga tgcaaaatac acgaaggagt tagctgatgc taaagctgaa aatgatgctc 1260
tgcgtgatga tgttgccgct ggtcgtcgtc ggttgcacat caaagcagtc tgtcagtcag 1320
tgcgtgaagc caccaccgcc tccggcgtgg ataatgcagc ctccccccga ctggcagaca 1380
ccgctgaacg ggattatttc accctcagag agaggctgat cactatgcaa aaacaactgg 1440
aaggaaccca gaagtatatt aatgagcagt gcagatagag ttgcccatat cgatgggcaa 1500
ctcatgcaat tattgtgagc aatacacacg cgcttccagc ggagtataaa tgcctaaagt 1560
aataaaaccg agcaatccat ttacgaatgt ttgctgggtt tctgttttaa caacattttc 1620
tgcgccgcca caaattttgg ctgcatcgac agttttcttc tgcccaattc cagaaacgaa 1680
gaaatgatgg gtgatggttt cctttggtgc tactgctgcc ggtttgtttt gaacagtaaa 1740
cgtctgttga gcacatcctg taataagcag ggccagcgca gtagcgagta gcattttttt 1800
catggtgtta ttcccgatgc tttttgaagt tcgcagaatc gtatgtgtag aaaattaaac 1860
aaaccctaaa caatgagttg aaatttcata ttgttaatat ttattaatgt atgtcaggtg 1920
cgatgaatcg tcattgtatt cccggattaa ctatgtccac agccctgacg gggaacttct 1980
ctgcgggagt gtccgggaat aattaaaacg atgcacacag ggtttagcgc gtacacgtat 2040
tgcattatgc caacgccccg gtgctgacac ggaagaaacc ggacgttatg atttagcgtg 2100
gaaagatttg tgtagtgttc tgaatgctct cagtaaatag taatgaatta tcaaaggtat 2160
agtaatatct tttatgttca tggatatttg taacccatcg gaaaactcct gctttagcaa 2220
gattttccct gtattgctga aatgtgattt ctcttgattt caacctatca taggacgttt 2280
ctataagatg cgtgtttctt gagaatttaa catttacaac ctttttaagt ccttttatta 2340
acacggtgtt atcgttttct aacacgatgt gaatattatc tgtggctaga tagtaaatat 2400
aatgtgagac gttgtgacgt tttagttcag aataaaacaa ttcacagtct aaatcttttc 2460
gcacttgatc gaatatttct ttaaaaatgg caacctgagc cattggtaaa accttccatg 2520
tgatacgagg gcgcgtagtt tgcattatcg tttttatcgt ttcaatctgg tctgacctcc 2580
ttgtgttttg ttgatgattt atgtcaaata ttaggaatgt tttcacttaa tagtattggt 2640
tgcgtaacaa agtgcggtcc tgctggcatt ctggagggaa atacaaccga cagatgtatg 2700
taaggccaac gtgctcaaat cttcatacag aaagatttga agtaatattt taaccgctag 2760
atgaagagca agcgcatgga gcgacaaaat gaataaagaa caatctgctg atgatccctc 2820
cgtggatctg attcgtgtaa aaaatatgct taatagcacc atttctatga gttaccctga 2880
tgttgtaatt gcatgtatag aacataaggt gtctctggaa gcattcagag caattgaggc 2940
agcgttggtg aagcacgata ataatatgaa ggattattcc ctggtggttg actgatcacc 3000
ataactgcta atcattcaaa ctatttagtc tgtgacagag ccaacacgca gtctgtcact 3060
gtcaggaaag tggtaaaact gcaactcaat tactgcaatg ccctcgtaat taagtgaatt 3120
tacaatatcg tcctgttcgg agggaagaac gcgggatgtt cattcttcat cacttttaat 3180
tgatgtatat gctctctttt ctgacgttag tctccgacgg caggcttcaa tgacccaggc 3240
tgagaaattc ccggaccctt tttgctcaag agcgatgtta atttgttcaa tcatttggtt 3300
aggaaagcgg atgttgcggg ttgttgttct gcgggttctg ttcttcgttg acatgaggtt 3360
gccccgtatt cagtgtcgct gatttgtatt gtctgaagtt gtttttacgt taagttgatg 3420
cagatcaatt aatacgatac ctgcgtcata attgattatt tgacgtggtt tgatggcctc 3480
cacgcacgtt gtgatatgta gatgataatc attatcactt tacgggtcct ttccggtgaa 3540
aaaaaaggta ccaaaaaaaa catcgtcgtg agtagtgaac cgtaagcagc gacgg 3595
<210> 5
<211> 184
<212> PRT
<213> 耻垢分枝杆菌
<400> 5
Gly Leu Asp Asn Glu Leu Ser Leu Val Asp Gly Gln Asp Arg Thr Leu
1 5 10 15
Thr Val Gln Gln Trp Asp Thr Phe Leu Asn Gly Val Phe Pro Leu Asp
20 25 30
Arg Asn Arg Leu Thr Arg Glu Trp Phe His Ser Gly Arg Ala Lys Tyr
35 40 45
Ile Val Ala Gly Pro Gly Ala Asp Glu Phe Glu Gly Thr Leu Glu Leu
50 55 60
Gly Tyr Gln Ile Gly Phe Pro Trp Ser Leu Gly Val Gly Ile Asn Phe
65 70 75 80
Ser Tyr Thr Thr Pro Asn Ile Leu Ile Asp Asp Gly Asp Ile Thr Ala
85 90 95
Pro Pro Phe Gly Leu Asn Ser Val Ile Thr Pro Asn Leu Phe Pro Gly
100 105 110
Val Ser Ile Ser Ala Asp Leu Gly Asn Gly Pro Gly Ile Gln Glu Val
115 120 125
Ala Thr Phe Ser Val Asp Val Ser Gly Pro Ala Gly Gly Val Ala Val
130 135 140
Ser Asn Ala His Gly Thr Val Thr Gly Ala Ala Gly Gly Val Leu Leu
145 150 155 160
Arg Pro Phe Ala Arg Leu Ile Ala Ser Thr Gly Asp Ser Val Thr Thr
165 170 175
Tyr Gly Glu Pro Trp Asn Met Asn
180
<210> 6
<211> 184
<212> PRT
<213> 耻垢分枝杆菌
<400> 6
Gly Leu Asp Asn Glu Leu Ser Leu Val Asp Gly Gln Asp Arg Thr Leu
1 5 10 15
Thr Val Gln Gln Trp Asp Thr Phe Leu Asn Gly Val Phe Pro Leu Asp
20 25 30
Arg Asn Arg Leu Thr Arg Glu Trp Phe His Ser Gly Arg Ala Lys Tyr
35 40 45
Ile Val Ala Gly Pro Gly Ala Asp Glu Phe Glu Gly Thr Leu Glu Leu
50 55 60
Gly Tyr Gln Ile Gly Phe Pro Trp Ser Leu Gly Val Gly Ile Asn Phe
65 70 75 80
Ser Tyr Thr Thr Pro Asn Ile Leu Ile Asp Asp Gly Asp Ile Thr Gly
85 90 95
Pro Pro Phe Gly Leu Glu Ser Val Ile Thr Pro Asn Leu Phe Pro Gly
100 105 110
Val Ser Ile Ser Ala Asp Leu Gly Asn Gly Pro Gly Ile Gln Glu Val
115 120 125
Ala Thr Phe Ser Val Asp Val Ser Gly Pro Ala Gly Gly Val Ala Val
130 135 140
Ser Asn Ala His Gly Thr Val Thr Gly Ala Ala Gly Gly Val Leu Leu
145 150 155 160
Arg Pro Phe Ala Arg Leu Ile Ala Ser Thr Gly Asp Ser Val Thr Thr
165 170 175
Tyr Gly Glu Pro Trp Asn Met Asn
180
<210> 7
<211> 183
<212> PRT
<213> 耻垢分枝杆菌
<400> 7
Val Asp Asn Gln Leu Ser Val Val Asp Gly Gln Gly Arg Thr Leu Thr
1 5 10 15
Val Gln Gln Ala Glu Thr Phe Leu Asn Gly Val Phe Pro Leu Asp Arg
20 25 30
Asn Arg Leu Thr Arg Glu Trp Phe His Ser Gly Arg Ala Thr Tyr His
35 40 45
Val Ala Gly Pro Gly Ala Asp Glu Phe Glu Gly Thr Leu Glu Leu Gly
50 55 60
Tyr Gln Val Gly Phe Pro Trp Ser Leu Gly Val Gly Ile Asn Phe Ser
65 70 75 80
Tyr Thr Thr Pro Asn Ile Leu Ile Asp Gly Gly Asp Ile Thr Gln Pro
85 90 95
Pro Phe Gly Leu Asp Thr Ile Ile Thr Pro Asn Leu Phe Pro Gly Val
100 105 110
Ser Ile Ser Ala Asp Leu Gly Asn Gly Pro Gly Ile Gln Glu Val Ala
115 120 125
Thr Phe Ser Val Asp Val Lys Gly Ala Lys Gly Ala Val Ala Val Ser
130 135 140
Asn Ala His Gly Thr Val Thr Gly Ala Ala Gly Gly Val Leu Leu Arg
145 150 155 160
Pro Phe Ala Arg Leu Ile Ala Ser Thr Gly Asp Ser Val Thr Thr Tyr
165 170 175
Gly Glu Pro Trp Asn Met Asn
180
<210> 8
<211> 1830
<212> DNA
<213> 噬菌体 phi-29
<400> 8
atgaaacaca tgccgcgtaa aatgtatagc tgcgcgtttg aaaccacgac caaagtggaa 60
gattgtcgcg tttgggccta tggctacatg aacatcgaag atcattctga atacaaaatc 120
ggtaacagtc tggatgaatt tatggcatgg gtgctgaaag ttcaggcgga tctgtacttc 180
cacaacctga aatttgatgg cgcattcatt atcaactggc tggaacgtaa tggctttaaa 240
tggagcgcgg atggtctgcc gaacacgtat aataccatta tctctcgtat gggccagtgg 300
tatatgattg atatctgcct gggctacaaa ggtaaacgca aaattcatac cgtgatctat 360
gatagcctga aaaaactgcc gtttccggtg aagaaaattg cgaaagattt caaactgacg 420
gttctgaaag gcgatattga ttatcacaaa gaacgtccgg ttggttacaa aatcaccccg 480
gaagaatacg catacatcaa aaacgatatc cagatcatcg cagaagcgct gctgattcag 540
tttaaacagg gcctggatcg catgaccgcg ggcagtgata gcctgaaagg tttcaaagat 600
atcatcacga ccaaaaaatt caaaaaagtg ttcccgacgc tgagcctggg tctggataaa 660
gaagttcgtt atgcctaccg cggcggtttt acctggctga acgatcgttt caaagaaaaa 720
gaaattggcg agggtatggt gtttgatgtt aatagtctgt atccggcaca gatgtacagc 780
cgcctgctgc cgtatggcga accgatcgtg ttcgagggta aatatgtttg ggatgaagat 840
tacccgctgc atattcagca catccgttgt gaatttgaac tgaaagaagg ctatattccg 900
accattcaga tcaaacgtag tcgcttctat aagggtaacg aatacctgaa aagctctggc 960
ggtgaaatcg cggatctgtg gctgagtaac gtggatctgg aactgatgaa agaacactac 1020
gatctgtaca acgttgaata catcagcggc ctgaaattta aagccacgac cggtctgttc 1080
aaagatttca tcgataaatg gacctacatc aaaacgacct ctgaaggcgc gattaaacag 1140
ctggccaaac tgatgctgaa cagcctgtat ggcaaattcg cctctaatcc ggatgtgacc 1200
ggtaaagttc cgtacctgaa agaaaatggc gcactgggtt ttcgcctggg cgaagaagaa 1260
acgaaagatc cggtgtatac cccgatgggt gttttcatta cggcctgggc acgttacacg 1320
accatcaccg cggcccaggc atgctatgat cgcattatct actgtgatac cgattctatt 1380
catctgacgg gcaccgaaat cccggatgtg attaaagata tcgttgatcc gaaaaaactg 1440
ggttattggg cccacgaaag tacgtttaaa cgtgcaaaat acctgcgcca gaaaacctac 1500
atccaggata tctacatgaa agaagtggat ggcaaactgg ttgaaggttc tccggatgat 1560
tacaccgata tcaaattcag tgtgaaatgc gccggcatga cggataaaat caaaaaagaa 1620
gtgaccttcg aaaacttcaa agttggtttc agccgcaaaa tgaaaccgaa accggtgcag 1680
gttccgggcg gtgtggttct ggtggatgat acgtttacca ttaaatctgg cggtagtgcg 1740
tggagccatc cgcagttcga aaaaggcggt ggctctggtg gcggttctgg cggtagtgcc 1800
tggagccacc cgcagtttga aaaataataa 1830
<210> 9
<211> 608
<212> PRT
<213> 噬菌体 phi-29
<400> 9
Met Lys His Met Pro Arg Lys Met Tyr Ser Cys Ala Phe Glu Thr Thr
1 5 10 15
Thr Lys Val Glu Asp Cys Arg Val Trp Ala Tyr Gly Tyr Met Asn Ile
20 25 30
Glu Asp His Ser Glu Tyr Lys Ile Gly Asn Ser Leu Asp Glu Phe Met
35 40 45
Ala Trp Val Leu Lys Val Gln Ala Asp Leu Tyr Phe His Asn Leu Lys
50 55 60
Phe Asp Gly Ala Phe Ile Ile Asn Trp Leu Glu Arg Asn Gly Phe Lys
65 70 75 80
Trp Ser Ala Asp Gly Leu Pro Asn Thr Tyr Asn Thr Ile Ile Ser Arg
85 90 95
Met Gly Gln Trp Tyr Met Ile Asp Ile Cys Leu Gly Tyr Lys Gly Lys
100 105 110
Arg Lys Ile His Thr Val Ile Tyr Asp Ser Leu Lys Lys Leu Pro Phe
115 120 125
Pro Val Lys Lys Ile Ala Lys Asp Phe Lys Leu Thr Val Leu Lys Gly
130 135 140
Asp Ile Asp Tyr His Lys Glu Arg Pro Val Gly Tyr Lys Ile Thr Pro
145 150 155 160
Glu Glu Tyr Ala Tyr Ile Lys Asn Asp Ile Gln Ile Ile Ala Glu Ala
165 170 175
Leu Leu Ile Gln Phe Lys Gln Gly Leu Asp Arg Met Thr Ala Gly Ser
180 185 190
Asp Ser Leu Lys Gly Phe Lys Asp Ile Ile Thr Thr Lys Lys Phe Lys
195 200 205
Lys Val Phe Pro Thr Leu Ser Leu Gly Leu Asp Lys Glu Val Arg Tyr
210 215 220
Ala Tyr Arg Gly Gly Phe Thr Trp Leu Asn Asp Arg Phe Lys Glu Lys
225 230 235 240
Glu Ile Gly Glu Gly Met Val Phe Asp Val Asn Ser Leu Tyr Pro Ala
245 250 255
Gln Met Tyr Ser Arg Leu Leu Pro Tyr Gly Glu Pro Ile Val Phe Glu
260 265 270
Gly Lys Tyr Val Trp Asp Glu Asp Tyr Pro Leu His Ile Gln His Ile
275 280 285
Arg Cys Glu Phe Glu Leu Lys Glu Gly Tyr Ile Pro Thr Ile Gln Ile
290 295 300
Lys Arg Ser Arg Phe Tyr Lys Gly Asn Glu Tyr Leu Lys Ser Ser Gly
305 310 315 320
Gly Glu Ile Ala Asp Leu Trp Leu Ser Asn Val Asp Leu Glu Leu Met
325 330 335
Lys Glu His Tyr Asp Leu Tyr Asn Val Glu Tyr Ile Ser Gly Leu Lys
340 345 350
Phe Lys Ala Thr Thr Gly Leu Phe Lys Asp Phe Ile Asp Lys Trp Thr
355 360 365
Tyr Ile Lys Thr Thr Ser Glu Gly Ala Ile Lys Gln Leu Ala Lys Leu
370 375 380
Met Leu Asn Ser Leu Tyr Gly Lys Phe Ala Ser Asn Pro Asp Val Thr
385 390 395 400
Gly Lys Val Pro Tyr Leu Lys Glu Asn Gly Ala Leu Gly Phe Arg Leu
405 410 415
Gly Glu Glu Glu Thr Lys Asp Pro Val Tyr Thr Pro Met Gly Val Phe
420 425 430
Ile Thr Ala Trp Ala Arg Tyr Thr Thr Ile Thr Ala Ala Gln Ala Cys
435 440 445
Tyr Asp Arg Ile Ile Tyr Cys Asp Thr Asp Ser Ile His Leu Thr Gly
450 455 460
Thr Glu Ile Pro Asp Val Ile Lys Asp Ile Val Asp Pro Lys Lys Leu
465 470 475 480
Gly Tyr Trp Ala His Glu Ser Thr Phe Lys Arg Ala Lys Tyr Leu Arg
485 490 495
Gln Lys Thr Tyr Ile Gln Asp Ile Tyr Met Lys Glu Val Asp Gly Lys
500 505 510
Leu Val Glu Gly Ser Pro Asp Asp Tyr Thr Asp Ile Lys Phe Ser Val
515 520 525
Lys Cys Ala Gly Met Thr Asp Lys Ile Lys Lys Glu Val Thr Phe Glu
530 535 540
Asn Phe Lys Val Gly Phe Ser Arg Lys Met Lys Pro Lys Pro Val Gln
545 550 555 560
Val Pro Gly Gly Val Val Leu Val Asp Asp Thr Phe Thr Ile Lys Ser
565 570 575
Gly Gly Ser Ala Trp Ser His Pro Gln Phe Glu Lys Gly Gly Gly Ser
580 585 590
Gly Gly Gly Ser Gly Gly Ser Ala Trp Ser His Pro Gln Phe Glu Lys
595 600 605
<210> 10
<211> 1390
<212> DNA
<213> 大肠杆菌
<400> 10
atgatgaacg atggcaaaca gcagagcacc ttcctgtttc atgattatga aaccttcggt 60
acccatccgg ccctggatcg tccggcgcag tttgcggcca ttcgcaccga tagcgaattc 120
aatgtgattg gcgaaccgga agtgttttat tgcaaaccgg ccgatgatta tctgccgcag 180
ccgggtgcgg tgctgattac cggtattacc ccgcaggaag cgcgcgcgaa aggtgaaaac 240
gaagcggcgt ttgccgcgcg cattcatagc ctgtttaccg tgccgaaaac ctgcattctg 300
ggctataaca atgtgcgctt cgatgatgaa gttacccgta atatctttta tcgtaacttt 360
tatgatccgt atgcgtggag ctggcagcat gataacagcc gttgggatct gctggatgtg 420
atgcgcgcgt gctatgcgct gcgcccggaa ggcattaatt ggccggaaaa cgatgatggc 480
ctgccgagct ttcgtctgga acatctgacc aaagccaacg gcattgaaca tagcaatgcc 540
catgatgcga tggccgatgt ttatgcgacc attgcgatgg cgaaactggt taaaacccgt 600
cagccgcgcc tgtttgatta tctgtttacc caccgtaaca aacacaaact gatggcgctg 660
attgatgttc cgcagatgaa accgctggtg catgtgagcg gcatgtttgg cgcctggcgc 720
ggcaacacca gctgggtggc cccgctggcc tggcacccgg aaaatcgtaa cgccgtgatt 780
atggttgatc tggccggtga tattagcccg ctgctggaac tggatagcga taccctgcgt 840
gaacgcctgt ataccgccaa aaccgatctg ggcgataatg ccgccgtgcc ggtgaaactg 900
gttcacatta acaaatgccc ggtgctggcc caggcgaaca ccctgcgccc ggaagatgcg 960
gatcgtctgg gtattaatcg ccagcattgt ctggataatc tgaaaatcct gcgtgaaaac 1020
ccgcaggtgc gtgaaaaagt ggtggcgatc ttcgcggaag cggaaccgtt caccccgagc 1080
gataacgtgg atgcgcagct gtataacggc ttctttagcg atgccgatcg cgcggcgatg 1140
aaaatcgttc tggaaaccga accgcgcaat ctgccggcgc tggatattac ctttgttgat 1200
aaacgtattg aaaaactgct gtttaattat cgtgcgcgca attttccggg taccctggat 1260
tatgccgaac agcagcgttg gctggaacat cgtcgtcagg ttttcacccc ggaatttctg 1320
cagggttatg cggatgaact gcagatgctg gttcagcagt atgccgatga taaagaaaaa 1380
gtggcgctgc 1390
<210> 11
<211> 485
<212> PRT
<213> 大肠杆菌
<400> 11
Met Met Asn Asp Gly Lys Gln Gln Ser Thr Phe Leu Phe His Asp Tyr
1 5 10 15
Glu Thr Phe Gly Thr His Pro Ala Leu Asp Arg Pro Ala Gln Phe Ala
20 25 30
Ala Ile Arg Thr Asp Ser Glu Phe Asn Val Ile Gly Glu Pro Glu Val
35 40 45
Phe Tyr Cys Lys Pro Ala Asp Asp Tyr Leu Pro Gln Pro Gly Ala Val
50 55 60
Leu Ile Thr Gly Ile Thr Pro Gln Glu Ala Arg Ala Lys Gly Glu Asn
65 70 75 80
Glu Ala Ala Phe Ala Ala Arg Ile His Ser Leu Phe Thr Val Pro Lys
85 90 95
Thr Cys Ile Leu Gly Tyr Asn Asn Val Arg Phe Asp Asp Glu Val Thr
100 105 110
Arg Asn Ile Phe Tyr Arg Asn Phe Tyr Asp Pro Tyr Ala Trp Ser Trp
115 120 125
Gln His Asp Asn Ser Arg Trp Asp Leu Leu Asp Val Met Arg Ala Cys
130 135 140
Tyr Ala Leu Arg Pro Glu Gly Ile Asn Trp Pro Glu Asn Asp Asp Gly
145 150 155 160
Leu Pro Ser Phe Arg Leu Glu His Leu Thr Lys Ala Asn Gly Ile Glu
165 170 175
His Ser Asn Ala His Asp Ala Met Ala Asp Val Tyr Ala Thr Ile Ala
180 185 190
Met Ala Lys Leu Val Lys Thr Arg Gln Pro Arg Leu Phe Asp Tyr Leu
195 200 205
Phe Thr His Arg Asn Lys His Lys Leu Met Ala Leu Ile Asp Val Pro
210 215 220
Gln Met Lys Pro Leu Val His Val Ser Gly Met Phe Gly Ala Trp Arg
225 230 235 240
Gly Asn Thr Ser Trp Val Ala Pro Leu Ala Trp His Pro Glu Asn Arg
245 250 255
Asn Ala Val Ile Met Val Asp Leu Ala Gly Asp Ile Ser Pro Leu Leu
260 265 270
Glu Leu Asp Ser Asp Thr Leu Arg Glu Arg Leu Tyr Thr Ala Lys Thr
275 280 285
Asp Leu Gly Asp Asn Ala Ala Val Pro Val Lys Leu Val His Ile Asn
290 295 300
Lys Cys Pro Val Leu Ala Gln Ala Asn Thr Leu Arg Pro Glu Asp Ala
305 310 315 320
Asp Arg Leu Gly Ile Asn Arg Gln His Cys Leu Asp Asn Leu Lys Ile
325 330 335
Leu Arg Glu Asn Pro Gln Val Arg Glu Lys Val Val Ala Ile Phe Ala
340 345 350
Glu Ala Glu Pro Phe Thr Pro Ser Asp Asn Val Asp Ala Gln Leu Tyr
355 360 365
Asn Gly Phe Phe Ser Asp Ala Asp Arg Ala Ala Met Lys Ile Val Leu
370 375 380
Glu Thr Glu Pro Arg Asn Leu Pro Ala Leu Asp Ile Thr Phe Val Asp
385 390 395 400
Lys Arg Ile Glu Lys Leu Leu Phe Asn Tyr Arg Ala Arg Asn Phe Pro
405 410 415
Gly Thr Leu Asp Tyr Ala Glu Gln Gln Arg Trp Leu Glu His Arg Arg
420 425 430
Gln Val Phe Thr Pro Glu Phe Leu Gln Gly Tyr Ala Asp Glu Leu Gln
435 440 445
Met Leu Val Gln Gln Tyr Ala Asp Asp Lys Glu Lys Val Ala Leu Leu
450 455 460
Lys Ala Leu Trp Gln Tyr Ala Glu Glu Ile Val Ser Gly Ser Gly His
465 470 475 480
His His His His His
485
<210> 12
<211> 804
<212> DNA
<213> 大肠杆菌
<400> 12
atgaaatttg tctcttttaa tatcaacggc ctgcgcgcca gacctcacca gcttgaagcc 60
atcgtcgaaa agcaccaacc ggatgtgatt ggcctgcagg agacaaaagt tcatgacgat 120
atgtttccgc tcgaagaggt ggcgaagctc ggctacaacg tgttttatca cgggcagaaa 180
ggccattatg gcgtggcgct gctgaccaaa gagacgccga ttgccgtgcg tcgcggcttt 240
cccggtgacg acgaagaggc gcagcggcgg attattatgg cggaaatccc ctcactgctg 300
ggtaatgtca ccgtgatcaa cggttacttc ccgcagggtg aaagccgcga ccatccgata 360
aaattcccgg caaaagcgca gttttatcag aatctgcaaa actacctgga aaccgaactc 420
aaacgtgata atccggtact gattatgggc gatatgaata tcagccctac agatctggat 480
atcggcattg gcgaagaaaa ccgtaagcgc tggctgcgta ccggtaaatg ctctttcctg 540
ccggaagagc gcgaatggat ggacaggctg atgagctggg ggttggtcga taccttccgc 600
catgcgaatc cgcaaacagc agatcgtttc tcatggtttg attaccgctc aaaaggtttt 660
gacgataacc gtggtctgcg catcgacctg ctgctcgcca gccaaccgct ggcagaatgt 720
tgcgtagaaa ccggcatcga ctatgaaatc cgcagcatgg aaaaaccgtc cgatcacgcc 780
cccgtctggg cgaccttccg ccgc 804
<210> 13
<211> 268
<212> PRT
<213> 大肠杆菌
<400> 13
Met Lys Phe Val Ser Phe Asn Ile Asn Gly Leu Arg Ala Arg Pro His
1 5 10 15
Gln Leu Glu Ala Ile Val Glu Lys His Gln Pro Asp Val Ile Gly Leu
20 25 30
Gln Glu Thr Lys Val His Asp Asp Met Phe Pro Leu Glu Glu Val Ala
35 40 45
Lys Leu Gly Tyr Asn Val Phe Tyr His Gly Gln Lys Gly His Tyr Gly
50 55 60
Val Ala Leu Leu Thr Lys Glu Thr Pro Ile Ala Val Arg Arg Gly Phe
65 70 75 80
Pro Gly Asp Asp Glu Glu Ala Gln Arg Arg Ile Ile Met Ala Glu Ile
85 90 95
Pro Ser Leu Leu Gly Asn Val Thr Val Ile Asn Gly Tyr Phe Pro Gln
100 105 110
Gly Glu Ser Arg Asp His Pro Ile Lys Phe Pro Ala Lys Ala Gln Phe
115 120 125
Tyr Gln Asn Leu Gln Asn Tyr Leu Glu Thr Glu Leu Lys Arg Asp Asn
130 135 140
Pro Val Leu Ile Met Gly Asp Met Asn Ile Ser Pro Thr Asp Leu Asp
145 150 155 160
Ile Gly Ile Gly Glu Glu Asn Arg Lys Arg Trp Leu Arg Thr Gly Lys
165 170 175
Cys Ser Phe Leu Pro Glu Glu Arg Glu Trp Met Asp Arg Leu Met Ser
180 185 190
Trp Gly Leu Val Asp Thr Phe Arg His Ala Asn Pro Gln Thr Ala Asp
195 200 205
Arg Phe Ser Trp Phe Asp Tyr Arg Ser Lys Gly Phe Asp Asp Asn Arg
210 215 220
Gly Leu Arg Ile Asp Leu Leu Leu Ala Ser Gln Pro Leu Ala Glu Cys
225 230 235 240
Cys Val Glu Thr Gly Ile Asp Tyr Glu Ile Arg Ser Met Glu Lys Pro
245 250 255
Ser Asp His Ala Pro Val Trp Ala Thr Phe Arg Arg
260 265
<210> 14
<211> 1275
<212> DNA
<213> 嗜热栖热菌(Thermus thermophilus)
<400> 14
atgtttcgtc gtaaagaaga tctggatccg ccgctggcac tgctgccgct gaaaggcctg 60
cgcgaagccg ccgcactgct ggaagaagcg ctgcgtcaag gtaaacgcat tcgtgttcac 120
ggcgactatg atgcggatgg cctgaccggc accgcgatcc tggttcgtgg tctggccgcc 180
ctgggtgcgg atgttcatcc gtttatcccg caccgcctgg aagaaggcta tggtgtcctg 240
atggaacgcg tcccggaaca tctggaagcc tcggacctgt ttctgaccgt tgactgcggc 300
attaccaacc atgcggaact gcgcgaactg ctggaaaatg gcgtggaagt cattgttacc 360
gatcatcata cgccgggcaa aacgccgccg ccgggtctgg tcgtgcatcc ggcgctgacg 420
ccggatctga aagaaaaacc gaccggcgca ggcgtggcgt ttctgctgct gtgggcactg 480
catgaacgcc tgggcctgcc gccgccgctg gaatacgcgg acctggcagc cgttggcacc 540
attgccgacg ttgccccgct gtggggttgg aatcgtgcac tggtgaaaga aggtctggca 600
cgcatcccgg cttcatcttg ggtgggcctg cgtctgctgg ctgaagccgt gggctatacc 660
ggcaaagcgg tcgaagtcgc tttccgcatc gcgccgcgca tcaatgcggc ttcccgcctg 720
ggcgaagcgg aaaaagccct gcgcctgctg ctgacggatg atgcggcaga agctcaggcg 780
ctggtcggcg aactgcaccg tctgaacgcc cgtcgtcaga ccctggaaga agcgatgctg 840
cgcaaactgc tgccgcaggc cgacccggaa gcgaaagcca tcgttctgct ggacccggaa 900
ggccatccgg gtgttatggg tattgtggcc tctcgcatcc tggaagcgac cctgcgcccg 960
gtctttctgg tggcccaggg caaaggcacc gtgcgttcgc tggctccgat ttccgccgtc 1020
gaagcactgc gcagcgcgga agatctgctg ctgcgttatg gtggtcataa agaagcggcg 1080
ggtttcgcaa tggatgaagc gctgtttccg gcgttcaaag cacgcgttga agcgtatgcc 1140
gcacgtttcc cggatccggt tcgtgaagtg gcactgctgg atctgctgcc ggaaccgggc 1200
ctgctgccgc aggtgttccg tgaactggca ctgctggaac cgtatggtga aggtaacccg 1260
gaaccgctgt tcctg 1275
<210> 15
<211> 425
<212> PRT
<213> 嗜热栖热菌
<400> 15
Met Phe Arg Arg Lys Glu Asp Leu Asp Pro Pro Leu Ala Leu Leu Pro
1 5 10 15
Leu Lys Gly Leu Arg Glu Ala Ala Ala Leu Leu Glu Glu Ala Leu Arg
20 25 30
Gln Gly Lys Arg Ile Arg Val His Gly Asp Tyr Asp Ala Asp Gly Leu
35 40 45
Thr Gly Thr Ala Ile Leu Val Arg Gly Leu Ala Ala Leu Gly Ala Asp
50 55 60
Val His Pro Phe Ile Pro His Arg Leu Glu Glu Gly Tyr Gly Val Leu
65 70 75 80
Met Glu Arg Val Pro Glu His Leu Glu Ala Ser Asp Leu Phe Leu Thr
85 90 95
Val Asp Cys Gly Ile Thr Asn His Ala Glu Leu Arg Glu Leu Leu Glu
100 105 110
Asn Gly Val Glu Val Ile Val Thr Asp His His Thr Pro Gly Lys Thr
115 120 125
Pro Pro Pro Gly Leu Val Val His Pro Ala Leu Thr Pro Asp Leu Lys
130 135 140
Glu Lys Pro Thr Gly Ala Gly Val Ala Phe Leu Leu Leu Trp Ala Leu
145 150 155 160
His Glu Arg Leu Gly Leu Pro Pro Pro Leu Glu Tyr Ala Asp Leu Ala
165 170 175
Ala Val Gly Thr Ile Ala Asp Val Ala Pro Leu Trp Gly Trp Asn Arg
180 185 190
Ala Leu Val Lys Glu Gly Leu Ala Arg Ile Pro Ala Ser Ser Trp Val
195 200 205
Gly Leu Arg Leu Leu Ala Glu Ala Val Gly Tyr Thr Gly Lys Ala Val
210 215 220
Glu Val Ala Phe Arg Ile Ala Pro Arg Ile Asn Ala Ala Ser Arg Leu
225 230 235 240
Gly Glu Ala Glu Lys Ala Leu Arg Leu Leu Leu Thr Asp Asp Ala Ala
245 250 255
Glu Ala Gln Ala Leu Val Gly Glu Leu His Arg Leu Asn Ala Arg Arg
260 265 270
Gln Thr Leu Glu Glu Ala Met Leu Arg Lys Leu Leu Pro Gln Ala Asp
275 280 285
Pro Glu Ala Lys Ala Ile Val Leu Leu Asp Pro Glu Gly His Pro Gly
290 295 300
Val Met Gly Ile Val Ala Ser Arg Ile Leu Glu Ala Thr Leu Arg Pro
305 310 315 320
Val Phe Leu Val Ala Gln Gly Lys Gly Thr Val Arg Ser Leu Ala Pro
325 330 335
Ile Ser Ala Val Glu Ala Leu Arg Ser Ala Glu Asp Leu Leu Leu Arg
340 345 350
Tyr Gly Gly His Lys Glu Ala Ala Gly Phe Ala Met Asp Glu Ala Leu
355 360 365
Phe Pro Ala Phe Lys Ala Arg Val Glu Ala Tyr Ala Ala Arg Phe Pro
370 375 380
Asp Pro Val Arg Glu Val Ala Leu Leu Asp Leu Leu Pro Glu Pro Gly
385 390 395 400
Leu Leu Pro Gln Val Phe Arg Glu Leu Ala Leu Leu Glu Pro Tyr Gly
405 410 415
Glu Gly Asn Pro Glu Pro Leu Phe Leu
420 425
<210> 16
<211> 738
<212> DNA
<213> 噬菌体λ
<400> 16
tccggaagcg gctctggtag tggttctggc atgacaccgg acattatcct gcagcgtacc 60
gggatcgatg tgagagctgt cgaacagggg gatgatgcgt ggcacaaatt acggctcggc 120
gtcatcaccg cttcagaagt tcacaacgtg atagcaaaac cccgctccgg aaagaagtgg 180
cctgacatga aaatgtccta cttccacacc ctgcttgctg aggtttgcac cggtgtggct 240
ccggaagtta acgctaaagc actggcctgg ggaaaacagt acgagaacga cgccagaacc 300
ctgtttgaat tcacttccgg cgtgaatgtt actgaatccc cgatcatcta tcgcgacgaa 360
agtatgcgta ccgcctgctc tcccgatggt ttatgcagtg acggcaacgg ccttgaactg 420
aaatgcccgt ttacctcccg ggatttcatg aagttccggc tcggtggttt cgaggccata 480
aagtcagctt acatggccca ggtgcagtac agcatgtggg tgacgcgaaa aaatgcctgg 540
tactttgcca actatgaccc gcgtatgaag cgtgaaggcc tgcattatgt cgtgattgag 600
cgggatgaaa agtacatggc gagttttgac gagatcgtgc cggagttcat cgaaaaaatg 660
gacgaggcac tggctgaaat tggttttgta tttggggagc aatggcgatc tggctctggt 720
tccggcagcg gttccgga 738
<210> 17
<211> 226
<212> PRT
<213> 噬菌体λ
<400> 17
Met Thr Pro Asp Ile Ile Leu Gln Arg Thr Gly Ile Asp Val Arg Ala
1 5 10 15
Val Glu Gln Gly Asp Asp Ala Trp His Lys Leu Arg Leu Gly Val Ile
20 25 30
Thr Ala Ser Glu Val His Asn Val Ile Ala Lys Pro Arg Ser Gly Lys
35 40 45
Lys Trp Pro Asp Met Lys Met Ser Tyr Phe His Thr Leu Leu Ala Glu
50 55 60
Val Cys Thr Gly Val Ala Pro Glu Val Asn Ala Lys Ala Leu Ala Trp
65 70 75 80
Gly Lys Gln Tyr Glu Asn Asp Ala Arg Thr Leu Phe Glu Phe Thr Ser
85 90 95
Gly Val Asn Val Thr Glu Ser Pro Ile Ile Tyr Arg Asp Glu Ser Met
100 105 110
Arg Thr Ala Cys Ser Pro Asp Gly Leu Cys Ser Asp Gly Asn Gly Leu
115 120 125
Glu Leu Lys Cys Pro Phe Thr Ser Arg Asp Phe Met Lys Phe Arg Leu
130 135 140
Gly Gly Phe Glu Ala Ile Lys Ser Ala Tyr Met Ala Gln Val Gln Tyr
145 150 155 160
Ser Met Trp Val Thr Arg Lys Asn Ala Trp Tyr Phe Ala Asn Tyr Asp
165 170 175
Pro Arg Met Lys Arg Glu Gly Leu His Tyr Val Val Ile Glu Arg Asp
180 185 190
Glu Lys Tyr Met Ala Ser Phe Asp Glu Ile Val Pro Glu Phe Ile Glu
195 200 205
Lys Met Asp Glu Ala Leu Ala Glu Ile Gly Phe Val Phe Gly Glu Gln
210 215 220
Trp Arg
225
<210> 18
<211> 760
<212> PRT
<213> 伯顿拟甲烷球菌(Methanococcoides burtonii)
<400> 18
Met Met Ile Arg Glu Leu Asp Ile Pro Arg Asp Ile Ile Gly Phe Tyr
1 5 10 15
Glu Asp Ser Gly Ile Lys Glu Leu Tyr Pro Pro Gln Ala Glu Ala Ile
20 25 30
Glu Met Gly Leu Leu Glu Lys Lys Asn Leu Leu Ala Ala Ile Pro Thr
35 40 45
Ala Ser Gly Lys Thr Leu Leu Ala Glu Leu Ala Met Ile Lys Ala Ile
50 55 60
Arg Glu Gly Gly Lys Ala Leu Tyr Ile Val Pro Leu Arg Ala Leu Ala
65 70 75 80
Ser Glu Lys Phe Glu Arg Phe Lys Glu Leu Ala Pro Phe Gly Ile Lys
85 90 95
Val Gly Ile Ser Thr Gly Asp Leu Asp Ser Arg Ala Asp Trp Leu Gly
100 105 110
Val Asn Asp Ile Ile Val Ala Thr Ser Glu Lys Thr Asp Ser Leu Leu
115 120 125
Arg Asn Gly Thr Ser Trp Met Asp Glu Ile Thr Thr Val Val Val Asp
130 135 140
Glu Ile His Leu Leu Asp Ser Lys Asn Arg Gly Pro Thr Leu Glu Val
145 150 155 160
Thr Ile Thr Lys Leu Met Arg Leu Asn Pro Asp Val Gln Val Val Ala
165 170 175
Leu Ser Ala Thr Val Gly Asn Ala Arg Glu Met Ala Asp Trp Leu Gly
180 185 190
Ala Ala Leu Val Leu Ser Glu Trp Arg Pro Thr Asp Leu His Glu Gly
195 200 205
Val Leu Phe Gly Asp Ala Ile Asn Phe Pro Gly Ser Gln Lys Lys Ile
210 215 220
Asp Arg Leu Glu Lys Asp Asp Ala Val Asn Leu Val Leu Asp Thr Ile
225 230 235 240
Lys Ala Glu Gly Gln Cys Leu Val Phe Glu Ser Ser Arg Arg Asn Cys
245 250 255
Ala Gly Phe Ala Lys Thr Ala Ser Ser Lys Val Ala Lys Ile Leu Asp
260 265 270
Asn Asp Ile Met Ile Lys Leu Ala Gly Ile Ala Glu Glu Val Glu Ser
275 280 285
Thr Gly Glu Thr Asp Thr Ala Ile Val Leu Ala Asn Cys Ile Arg Lys
290 295 300
Gly Val Ala Phe His His Ala Gly Leu Asn Ser Asn His Arg Lys Leu
305 310 315 320
Val Glu Asn Gly Phe Arg Gln Asn Leu Ile Lys Val Ile Ser Ser Thr
325 330 335
Pro Thr Leu Ala Ala Gly Leu Asn Leu Pro Ala Arg Arg Val Ile Ile
340 345 350
Arg Ser Tyr Arg Arg Phe Asp Ser Asn Phe Gly Met Gln Pro Ile Pro
355 360 365
Val Leu Glu Tyr Lys Gln Met Ala Gly Arg Ala Gly Arg Pro His Leu
370 375 380
Asp Pro Tyr Gly Glu Ser Val Leu Leu Ala Lys Thr Tyr Asp Glu Phe
385 390 395 400
Ala Gln Leu Met Glu Asn Tyr Val Glu Ala Asp Ala Glu Asp Ile Trp
405 410 415
Ser Lys Leu Gly Thr Glu Asn Ala Leu Arg Thr His Val Leu Ser Thr
420 425 430
Ile Val Asn Gly Phe Ala Ser Thr Arg Gln Glu Leu Phe Asp Phe Phe
435 440 445
Gly Ala Thr Phe Phe Ala Tyr Gln Gln Asp Lys Trp Met Leu Glu Glu
450 455 460
Val Ile Asn Asp Cys Leu Glu Phe Leu Ile Asp Lys Ala Met Val Ser
465 470 475 480
Glu Thr Glu Asp Ile Glu Asp Ala Ser Lys Leu Phe Leu Arg Gly Thr
485 490 495
Arg Leu Gly Ser Leu Val Ser Met Leu Tyr Ile Asp Pro Leu Ser Gly
500 505 510
Ser Lys Ile Val Asp Gly Phe Lys Asp Ile Gly Lys Ser Thr Gly Gly
515 520 525
Asn Met Gly Ser Leu Glu Asp Asp Lys Gly Asp Asp Ile Thr Val Thr
530 535 540
Asp Met Thr Leu Leu His Leu Val Cys Ser Thr Pro Asp Met Arg Gln
545 550 555 560
Leu Tyr Leu Arg Asn Thr Asp Tyr Thr Ile Val Asn Glu Tyr Ile Val
565 570 575
Ala His Ser Asp Glu Phe His Glu Ile Pro Asp Lys Leu Lys Glu Thr
580 585 590
Asp Tyr Glu Trp Phe Met Gly Glu Val Lys Thr Ala Met Leu Leu Glu
595 600 605
Glu Trp Val Thr Glu Val Ser Ala Glu Asp Ile Thr Arg His Phe Asn
610 615 620
Val Gly Glu Gly Asp Ile His Ala Leu Ala Asp Thr Ser Glu Trp Leu
625 630 635 640
Met His Ala Ala Ala Lys Leu Ala Glu Leu Leu Gly Val Glu Tyr Ser
645 650 655
Ser His Ala Tyr Ser Leu Glu Lys Arg Ile Arg Tyr Gly Ser Gly Leu
660 665 670
Asp Leu Met Glu Leu Val Gly Ile Arg Gly Val Gly Arg Val Arg Ala
675 680 685
Arg Lys Leu Tyr Asn Ala Gly Phe Val Ser Val Ala Lys Leu Lys Gly
690 695 700
Ala Asp Ile Ser Val Leu Ser Lys Leu Val Gly Pro Lys Val Ala Tyr
705 710 715 720
Asn Ile Leu Ser Gly Ile Gly Val Arg Val Asn Asp Lys His Phe Asn
725 730 735
Ser Ala Pro Ile Ser Ser Asn Thr Leu Asp Thr Leu Leu Asp Lys Asn
740 745 750
Gln Lys Thr Phe Asn Asp Phe Gln
755 760
<210> 19
<211> 707
<212> PRT
<213> 泉古菌(Cenarchaeum symbiosum )
<400> 19
Met Arg Ile Ser Glu Leu Asp Ile Pro Arg Pro Ala Ile Glu Phe Leu
1 5 10 15
Glu Gly Glu Gly Tyr Lys Lys Leu Tyr Pro Pro Gln Ala Ala Ala Ala
20 25 30
Lys Ala Gly Leu Thr Asp Gly Lys Ser Val Leu Val Ser Ala Pro Thr
35 40 45
Ala Ser Gly Lys Thr Leu Ile Ala Ala Ile Ala Met Ile Ser His Leu
50 55 60
Ser Arg Asn Arg Gly Lys Ala Val Tyr Leu Ser Pro Leu Arg Ala Leu
65 70 75 80
Ala Ala Glu Lys Phe Ala Glu Phe Gly Lys Ile Gly Gly Ile Pro Leu
85 90 95
Gly Arg Pro Val Arg Val Gly Val Ser Thr Gly Asp Phe Glu Lys Ala
100 105 110
Gly Arg Ser Leu Gly Asn Asn Asp Ile Leu Val Leu Thr Asn Glu Arg
115 120 125
Met Asp Ser Leu Ile Arg Arg Arg Pro Asp Trp Met Asp Glu Val Gly
130 135 140
Leu Val Ile Ala Asp Glu Ile His Leu Ile Gly Asp Arg Ser Arg Gly
145 150 155 160
Pro Thr Leu Glu Met Val Leu Thr Lys Leu Arg Gly Leu Arg Ser Ser
165 170 175
Pro Gln Val Val Ala Leu Ser Ala Thr Ile Ser Asn Ala Asp Glu Ile
180 185 190
Ala Gly Trp Leu Asp Cys Thr Leu Val His Ser Thr Trp Arg Pro Val
195 200 205
Pro Leu Ser Glu Gly Val Tyr Gln Asp Gly Glu Val Ala Met Gly Asp
210 215 220
Gly Ser Arg His Glu Val Ala Ala Thr Gly Gly Gly Pro Ala Val Asp
225 230 235 240
Leu Ala Ala Glu Ser Val Ala Glu Gly Gly Gln Ser Leu Ile Phe Ala
245 250 255
Asp Thr Arg Ala Arg Ser Ala Ser Leu Ala Ala Lys Ala Ser Ala Val
260 265 270
Ile Pro Glu Ala Lys Gly Ala Asp Ala Ala Lys Leu Ala Ala Ala Ala
275 280 285
Lys Lys Ile Ile Ser Ser Gly Gly Glu Thr Lys Leu Ala Lys Thr Leu
290 295 300
Ala Glu Leu Val Glu Lys Gly Ala Ala Phe His His Ala Gly Leu Asn
305 310 315 320
Gln Asp Cys Arg Ser Val Val Glu Glu Glu Phe Arg Ser Gly Arg Ile
325 330 335
Arg Leu Leu Ala Ser Thr Pro Thr Leu Ala Ala Gly Val Asn Leu Pro
340 345 350
Ala Arg Arg Val Val Ile Ser Ser Val Met Arg Tyr Asn Ser Ser Ser
355 360 365
Gly Met Ser Glu Pro Ile Ser Ile Leu Glu Tyr Lys Gln Leu Cys Gly
370 375 380
Arg Ala Gly Arg Pro Gln Tyr Asp Lys Ser Gly Glu Ala Ile Val Val
385 390 395 400
Gly Gly Val Asn Ala Asp Glu Ile Phe Asp Arg Tyr Ile Gly Gly Glu
405 410 415
Pro Glu Pro Ile Arg Ser Ala Met Val Asp Asp Arg Ala Leu Arg Ile
420 425 430
His Val Leu Ser Leu Val Thr Thr Ser Pro Gly Ile Lys Glu Asp Asp
435 440 445
Val Thr Glu Phe Phe Leu Gly Thr Leu Gly Gly Gln Gln Ser Gly Glu
450 455 460
Ser Thr Val Lys Phe Ser Val Ala Val Ala Leu Arg Phe Leu Gln Glu
465 470 475 480
Glu Gly Met Leu Gly Arg Arg Gly Gly Arg Leu Ala Ala Thr Lys Met
485 490 495
Gly Arg Leu Val Ser Arg Leu Tyr Met Asp Pro Met Thr Ala Val Thr
500 505 510
Leu Arg Asp Ala Val Gly Glu Ala Ser Pro Gly Arg Met His Thr Leu
515 520 525
Gly Phe Leu His Leu Val Ser Glu Cys Ser Glu Phe Met Pro Arg Phe
530 535 540
Ala Leu Arg Gln Lys Asp His Glu Val Ala Glu Met Met Leu Glu Ala
545 550 555 560
Gly Arg Gly Glu Leu Leu Arg Pro Val Tyr Ser Tyr Glu Cys Gly Arg
565 570 575
Gly Leu Leu Ala Leu His Arg Trp Ile Gly Glu Ser Pro Glu Ala Lys
580 585 590
Leu Ala Glu Asp Leu Lys Phe Glu Ser Gly Asp Val His Arg Met Val
595 600 605
Glu Ser Ser Gly Trp Leu Leu Arg Cys Ile Trp Glu Ile Ser Lys His
610 615 620
Gln Glu Arg Pro Asp Leu Leu Gly Glu Leu Asp Val Leu Arg Ser Arg
625 630 635 640
Val Ala Tyr Gly Ile Lys Ala Glu Leu Val Pro Leu Val Ser Ile Lys
645 650 655
Gly Ile Gly Arg Val Arg Ser Arg Arg Leu Phe Arg Gly Gly Ile Lys
660 665 670
Gly Pro Gly Asp Leu Ala Ala Val Pro Val Glu Arg Leu Ser Arg Val
675 680 685
Glu Gly Ile Gly Ala Thr Leu Ala Asn Asn Ile Lys Ser Gln Leu Arg
690 695 700
Lys Gly Gly
705
<210> 20
<211> 720
<212> PRT
<213> 伽马超嗜热古菌(Thermococcus gammatolerans)
<400> 20
Met Lys Val Asp Glu Leu Pro Val Asp Glu Arg Leu Lys Ala Val Leu
1 5 10 15
Lys Glu Arg Gly Ile Glu Glu Leu Tyr Pro Pro Gln Ala Glu Ala Leu
20 25 30
Lys Ser Gly Ala Leu Glu Gly Arg Asn Leu Val Leu Ala Ile Pro Thr
35 40 45
Ala Ser Gly Lys Thr Leu Val Ser Glu Ile Val Met Val Asn Lys Leu
50 55 60
Ile Gln Glu Gly Gly Lys Ala Val Tyr Leu Val Pro Leu Lys Ala Leu
65 70 75 80
Ala Glu Glu Lys Tyr Arg Glu Phe Lys Glu Trp Glu Lys Leu Gly Leu
85 90 95
Lys Val Ala Ala Thr Thr Gly Asp Tyr Asp Ser Thr Asp Asp Trp Leu
100 105 110
Gly Arg Tyr Asp Ile Ile Val Ala Thr Ala Glu Lys Phe Asp Ser Leu
115 120 125
Leu Arg His Gly Ala Arg Trp Ile Asn Asp Val Lys Leu Val Val Ala
130 135 140
Asp Glu Val His Leu Ile Gly Ser Tyr Asp Arg Gly Ala Thr Leu Glu
145 150 155 160
Met Ile Leu Thr His Met Leu Gly Arg Ala Gln Ile Leu Ala Leu Ser
165 170 175
Ala Thr Val Gly Asn Ala Glu Glu Leu Ala Glu Trp Leu Asp Ala Ser
180 185 190
Leu Val Val Ser Asp Trp Arg Pro Val Gln Leu Arg Arg Gly Val Phe
195 200 205
His Leu Gly Thr Leu Ile Trp Glu Asp Gly Lys Val Glu Ser Tyr Pro
210 215 220
Glu Asn Trp Tyr Ser Leu Val Val Asp Ala Val Lys Arg Gly Lys Gly
225 230 235 240
Ala Leu Val Phe Val Asn Thr Arg Arg Ser Ala Glu Lys Glu Ala Leu
245 250 255
Ala Leu Ser Lys Leu Val Ser Ser His Leu Thr Lys Pro Glu Lys Arg
260 265 270
Ala Leu Glu Ser Leu Ala Ser Gln Leu Glu Asp Asn Pro Thr Ser Glu
275 280 285
Lys Leu Lys Arg Ala Leu Arg Gly Gly Val Ala Phe His His Ala Gly
290 295 300
Leu Ser Arg Val Glu Arg Thr Leu Ile Glu Asp Ala Phe Arg Glu Gly
305 310 315 320
Leu Ile Lys Val Ile Thr Ala Thr Pro Thr Leu Ser Ala Gly Val Asn
325 330 335
Leu Pro Ser Phe Arg Val Ile Ile Arg Asp Thr Lys Arg Tyr Ala Gly
340 345 350
Phe Gly Trp Thr Asp Ile Pro Val Leu Glu Ile Gln Gln Met Met Gly
355 360 365
Arg Ala Gly Arg Pro Arg Tyr Asp Lys Tyr Gly Glu Ala Ile Ile Val
370 375 380
Ala Arg Thr Asp Glu Pro Gly Lys Leu Met Glu Arg Tyr Ile Arg Gly
385 390 395 400
Lys Pro Glu Lys Leu Phe Ser Met Leu Ala Asn Glu Gln Ala Phe Arg
405 410 415
Ser Gln Val Leu Ala Leu Ile Thr Asn Phe Gly Ile Arg Ser Phe Pro
420 425 430
Glu Leu Val Arg Phe Leu Glu Arg Thr Phe Tyr Ala His Gln Arg Lys
435 440 445
Asp Leu Ser Ser Leu Glu Tyr Lys Ala Lys Glu Val Val Tyr Phe Leu
450 455 460
Ile Glu Asn Glu Phe Ile Asp Leu Asp Leu Glu Asp Arg Phe Ile Pro
465 470 475 480
Leu Pro Phe Gly Lys Arg Thr Ser Gln Leu Tyr Ile Asp Pro Leu Thr
485 490 495
Ala Lys Lys Phe Lys Asp Ala Phe Pro Ala Ile Glu Arg Asn Pro Asn
500 505 510
Pro Phe Gly Ile Phe Gln Leu Ile Ala Ser Thr Pro Asp Met Ala Thr
515 520 525
Leu Thr Ala Arg Arg Arg Glu Met Glu Asp Tyr Leu Asp Leu Ala Tyr
530 535 540
Glu Leu Glu Asp Lys Leu Tyr Ala Ser Ile Pro Tyr Tyr Glu Asp Ser
545 550 555 560
Arg Phe Gln Gly Phe Leu Gly Gln Val Lys Thr Ala Lys Val Leu Leu
565 570 575
Asp Trp Ile Asn Glu Val Pro Glu Ala Arg Ile Tyr Glu Thr Tyr Ser
580 585 590
Ile Asp Pro Gly Asp Leu Tyr Arg Leu Leu Glu Leu Ala Asp Trp Leu
595 600 605
Met Tyr Ser Leu Ile Glu Leu Tyr Lys Leu Phe Glu Pro Lys Glu Glu
610 615 620
Ile Leu Asn Tyr Leu Arg Asp Leu His Leu Arg Leu Arg His Gly Val
625 630 635 640
Arg Glu Glu Leu Leu Glu Leu Val Arg Leu Pro Asn Ile Gly Arg Lys
645 650 655
Arg Ala Arg Ala Leu Tyr Asn Ala Gly Phe Arg Ser Val Glu Ala Ile
660 665 670
Ala Asn Ala Lys Pro Ala Glu Leu Leu Ala Val Glu Gly Ile Gly Ala
675 680 685
Lys Ile Leu Asp Gly Ile Tyr Arg His Leu Gly Ile Glu Lys Arg Val
690 695 700
Thr Glu Glu Lys Pro Lys Arg Lys Gly Thr Leu Glu Asp Phe Leu Arg
705 710 715 720
<210> 21
<211> 799
<212> PRT
<213> 亨氏产甲烷螺菌(Methanospirillum hungatei)
<400> 21
Met Glu Ile Ala Ser Leu Pro Leu Pro Asp Ser Phe Ile Arg Ala Cys
1 5 10 15
His Ala Lys Gly Ile Arg Ser Leu Tyr Pro Pro Gln Ala Glu Cys Ile
20 25 30
Glu Lys Gly Leu Leu Glu Gly Lys Asn Leu Leu Ile Ser Ile Pro Thr
35 40 45
Ala Ser Gly Lys Thr Leu Leu Ala Glu Met Ala Met Trp Ser Arg Ile
50 55 60
Ala Ala Gly Gly Lys Cys Leu Tyr Ile Val Pro Leu Arg Ala Leu Ala
65 70 75 80
Ser Glu Lys Tyr Asp Glu Phe Ser Lys Lys Gly Val Ile Arg Val Gly
85 90 95
Ile Ala Thr Gly Asp Leu Asp Arg Thr Asp Ala Tyr Leu Gly Glu Asn
100 105 110
Asp Ile Ile Val Ala Thr Ser Glu Lys Thr Asp Ser Leu Leu Arg Asn
115 120 125
Arg Thr Pro Trp Leu Ser Gln Ile Thr Cys Ile Val Leu Asp Glu Val
130 135 140
His Leu Ile Gly Ser Glu Asn Arg Gly Ala Thr Leu Glu Met Val Ile
145 150 155 160
Thr Lys Leu Arg Tyr Thr Asn Pro Val Met Gln Ile Ile Gly Leu Ser
165 170 175
Ala Thr Ile Gly Asn Pro Ala Gln Leu Ala Glu Trp Leu Asp Ala Thr
180 185 190
Leu Ile Thr Ser Thr Trp Arg Pro Val Asp Leu Arg Gln Gly Val Tyr
195 200 205
Tyr Asn Gly Lys Ile Arg Phe Ser Asp Ser Glu Arg Pro Ile Gln Gly
210 215 220
Lys Thr Lys His Asp Asp Leu Asn Leu Cys Leu Asp Thr Ile Glu Glu
225 230 235 240
Gly Gly Gln Cys Leu Val Phe Val Ser Ser Arg Arg Asn Ala Glu Gly
245 250 255
Phe Ala Lys Lys Ala Ala Gly Ala Leu Lys Ala Gly Ser Pro Asp Ser
260 265 270
Lys Ala Leu Ala Gln Glu Leu Arg Arg Leu Arg Asp Arg Asp Glu Gly
275 280 285
Asn Val Leu Ala Asp Cys Val Glu Arg Gly Ala Ala Phe His His Ala
290 295 300
Gly Leu Ile Arg Gln Glu Arg Thr Ile Ile Glu Glu Gly Phe Arg Asn
305 310 315 320
Gly Tyr Ile Glu Val Ile Ala Ala Thr Pro Thr Leu Ala Ala Gly Leu
325 330 335
Asn Leu Pro Ala Arg Arg Val Ile Ile Arg Asp Tyr Asn Arg Phe Ala
340 345 350
Ser Gly Leu Gly Met Val Pro Ile Pro Val Gly Glu Tyr His Gln Met
355 360 365
Ala Gly Arg Ala Gly Arg Pro His Leu Asp Pro Tyr Gly Glu Ala Val
370 375 380
Leu Leu Ala Lys Asp Ala Pro Ser Val Glu Arg Leu Phe Glu Thr Phe
385 390 395 400
Ile Asp Ala Glu Ala Glu Arg Val Asp Ser Gln Cys Val Asp Asp Ala
405 410 415
Ser Leu Cys Ala His Ile Leu Ser Leu Ile Ala Thr Gly Phe Ala His
420 425 430
Asp Gln Glu Ala Leu Ser Ser Phe Met Glu Arg Thr Phe Tyr Phe Phe
435 440 445
Gln His Pro Lys Thr Arg Ser Leu Pro Arg Leu Val Ala Asp Ala Ile
450 455 460
Arg Phe Leu Thr Thr Ala Gly Met Val Glu Glu Arg Glu Asn Thr Leu
465 470 475 480
Ser Ala Thr Arg Leu Gly Ser Leu Val Ser Arg Leu Tyr Leu Asn Pro
485 490 495
Cys Thr Ala Arg Leu Ile Leu Asp Ser Leu Lys Ser Cys Lys Thr Pro
500 505 510
Thr Leu Ile Gly Leu Leu His Val Ile Cys Val Ser Pro Asp Met Gln
515 520 525
Arg Leu Tyr Leu Lys Ala Ala Asp Thr Gln Leu Leu Arg Thr Phe Leu
530 535 540
Phe Lys His Lys Asp Asp Leu Ile Leu Pro Leu Pro Phe Glu Gln Glu
545 550 555 560
Glu Glu Glu Leu Trp Leu Ser Gly Leu Lys Thr Ala Leu Val Leu Thr
565 570 575
Asp Trp Ala Asp Glu Phe Ser Glu Gly Met Ile Glu Glu Arg Tyr Gly
580 585 590
Ile Gly Ala Gly Asp Leu Tyr Asn Ile Val Asp Ser Gly Lys Trp Leu
595 600 605
Leu His Gly Thr Glu Arg Leu Val Ser Val Glu Met Pro Glu Met Ser
610 615 620
Gln Val Val Lys Thr Leu Ser Val Arg Val His His Gly Val Lys Ser
625 630 635 640
Glu Leu Leu Pro Leu Val Ala Leu Arg Asn Ile Gly Arg Val Arg Ala
645 650 655
Arg Thr Leu Tyr Asn Ala Gly Tyr Pro Asp Pro Glu Ala Val Ala Arg
660 665 670
Ala Gly Leu Ser Thr Ile Ala Arg Ile Ile Gly Glu Gly Ile Ala Arg
675 680 685
Gln Val Ile Asp Glu Ile Thr Gly Val Lys Arg Ser Gly Ile His Ser
690 695 700
Ser Asp Asp Asp Tyr Gln Gln Lys Thr Pro Glu Leu Leu Thr Asp Ile
705 710 715 720
Pro Gly Ile Gly Lys Lys Met Ala Glu Lys Leu Gln Asn Ala Gly Ile
725 730 735
Ile Thr Val Ser Asp Leu Leu Thr Ala Asp Glu Val Leu Leu Ser Asp
740 745 750
Val Leu Gly Ala Ala Arg Ala Arg Lys Val Leu Ala Phe Leu Ser Asn
755 760 765
Ser Glu Lys Glu Asn Ser Ser Ser Asp Lys Thr Glu Glu Ile Pro Asp
770 775 780
Thr Gln Lys Ile Arg Gly Gln Ser Ser Trp Glu Asp Phe Gly Cys
785 790 795
<210> 22
<211> 1756
<212> PRT
<213> 大肠杆菌
<400> 22
Met Met Ser Ile Ala Gln Val Arg Ser Ala Gly Ser Ala Gly Asn Tyr
1 5 10 15
Tyr Thr Asp Lys Asp Asn Tyr Tyr Val Leu Gly Ser Met Gly Glu Arg
20 25 30
Trp Ala Gly Lys Gly Ala Glu Gln Leu Gly Leu Gln Gly Ser Val Asp
35 40 45
Lys Asp Val Phe Thr Arg Leu Leu Glu Gly Arg Leu Pro Asp Gly Ala
50 55 60
Asp Leu Ser Arg Met Gln Asp Gly Ser Asn Lys His Arg Pro Gly Tyr
65 70 75 80
Asp Leu Thr Phe Ser Ala Pro Lys Ser Val Ser Met Met Ala Met Leu
85 90 95
Gly Gly Asp Lys Arg Leu Ile Asp Ala His Asn Gln Ala Val Asp Phe
100 105 110
Ala Val Arg Gln Val Glu Ala Leu Ala Ser Thr Arg Val Met Thr Asp
115 120 125
Gly Gln Ser Glu Thr Val Leu Thr Gly Asn Leu Val Met Ala Leu Phe
130 135 140
Asn His Asp Thr Ser Arg Asp Gln Glu Pro Gln Leu His Thr His Ala
145 150 155 160
Val Val Ala Asn Val Thr Gln His Asn Gly Glu Trp Lys Thr Leu Ser
165 170 175
Ser Asp Lys Val Gly Lys Thr Gly Phe Ile Glu Asn Val Tyr Ala Asn
180 185 190
Gln Ile Ala Phe Gly Arg Leu Tyr Arg Glu Lys Leu Lys Glu Gln Val
195 200 205
Glu Ala Leu Gly Tyr Glu Thr Glu Val Val Gly Lys His Gly Met Trp
210 215 220
Glu Met Pro Gly Val Pro Val Glu Ala Phe Ser Gly Arg Ser Gln Ala
225 230 235 240
Ile Arg Glu Ala Val Gly Glu Asp Ala Ser Leu Lys Ser Arg Asp Val
245 250 255
Ala Ala Leu Asp Thr Arg Lys Ser Lys Gln His Val Asp Pro Glu Ile
260 265 270
Arg Met Ala Glu Trp Met Gln Thr Leu Lys Glu Thr Gly Phe Asp Ile
275 280 285
Arg Ala Tyr Arg Asp Ala Ala Asp Gln Arg Thr Glu Ile Arg Thr Gln
290 295 300
Ala Pro Gly Pro Ala Ser Gln Asp Gly Pro Asp Val Gln Gln Ala Val
305 310 315 320
Thr Gln Ala Ile Ala Gly Leu Ser Glu Arg Lys Val Gln Phe Thr Tyr
325 330 335
Thr Asp Val Leu Ala Arg Thr Val Gly Ile Leu Pro Pro Glu Asn Gly
340 345 350
Val Ile Glu Arg Ala Arg Ala Gly Ile Asp Glu Ala Ile Ser Arg Glu
355 360 365
Gln Leu Ile Pro Leu Asp Arg Glu Lys Gly Leu Phe Thr Ser Gly Ile
370 375 380
His Val Leu Asp Glu Leu Ser Val Arg Ala Leu Ser Arg Asp Ile Met
385 390 395 400
Lys Gln Asn Arg Val Thr Val His Pro Glu Lys Ser Val Pro Arg Thr
405 410 415
Ala Gly Tyr Ser Asp Ala Val Ser Val Leu Ala Gln Asp Arg Pro Ser
420 425 430
Leu Ala Ile Val Ser Gly Gln Gly Gly Ala Ala Gly Gln Arg Glu Arg
435 440 445
Val Ala Glu Leu Val Met Met Ala Arg Glu Gln Gly Arg Glu Val Gln
450 455 460
Ile Ile Ala Ala Asp Arg Arg Ser Gln Met Asn Leu Lys Gln Asp Glu
465 470 475 480
Arg Leu Ser Gly Glu Leu Ile Thr Gly Arg Arg Gln Leu Leu Glu Gly
485 490 495
Met Ala Phe Thr Pro Gly Ser Thr Val Ile Val Asp Gln Gly Glu Lys
500 505 510
Leu Ser Leu Lys Glu Thr Leu Thr Leu Leu Asp Gly Ala Ala Arg His
515 520 525
Asn Val Gln Val Leu Ile Thr Asp Ser Gly Gln Arg Thr Gly Thr Gly
530 535 540
Ser Ala Leu Met Ala Met Lys Asp Ala Gly Val Asn Thr Tyr Arg Trp
545 550 555 560
Gln Gly Gly Glu Gln Arg Pro Ala Thr Ile Ile Ser Glu Pro Asp Arg
565 570 575
Asn Val Arg Tyr Ala Arg Leu Ala Gly Asp Phe Ala Ala Ser Val Lys
580 585 590
Ala Gly Glu Glu Ser Val Ala Gln Val Ser Gly Val Arg Glu Gln Ala
595 600 605
Ile Leu Thr Gln Ala Ile Arg Ser Glu Leu Lys Thr Gln Gly Val Leu
610 615 620
Gly His Pro Glu Val Thr Met Thr Ala Leu Ser Pro Val Trp Leu Asp
625 630 635 640
Ser Arg Ser Arg Tyr Leu Arg Asp Met Tyr Arg Pro Gly Met Val Met
645 650 655
Glu Gln Trp Asn Pro Glu Thr Arg Ser His Asp Arg Tyr Val Ile Asp
660 665 670
Arg Val Thr Ala Gln Ser His Ser Leu Thr Leu Arg Asp Ala Gln Gly
675 680 685
Glu Thr Gln Val Val Arg Ile Ser Ser Leu Asp Ser Ser Trp Ser Leu
690 695 700
Phe Arg Pro Glu Lys Met Pro Val Ala Asp Gly Glu Arg Leu Arg Val
705 710 715 720
Thr Gly Lys Ile Pro Gly Leu Arg Val Ser Gly Gly Asp Arg Leu Gln
725 730 735
Val Ala Ser Val Ser Glu Asp Ala Met Thr Val Val Val Pro Gly Arg
740 745 750
Ala Glu Pro Ala Ser Leu Pro Val Ser Asp Ser Pro Phe Thr Ala Leu
755 760 765
Lys Leu Glu Asn Gly Trp Val Glu Thr Pro Gly His Ser Val Ser Asp
770 775 780
Ser Ala Thr Val Phe Ala Ser Val Thr Gln Met Ala Met Asp Asn Ala
785 790 795 800
Thr Leu Asn Gly Leu Ala Arg Ser Gly Arg Asp Val Arg Leu Tyr Ser
805 810 815
Ser Leu Asp Glu Thr Arg Thr Ala Glu Lys Leu Ala Arg His Pro Ser
820 825 830
Phe Thr Val Val Ser Glu Gln Ile Lys Ala Arg Ala Gly Glu Thr Leu
835 840 845
Leu Glu Thr Ala Ile Ser Leu Gln Lys Ala Gly Leu His Thr Pro Ala
850 855 860
Gln Gln Ala Ile His Leu Ala Leu Pro Val Leu Glu Ser Lys Asn Leu
865 870 875 880
Ala Phe Ser Met Val Asp Leu Leu Thr Glu Ala Lys Ser Phe Ala Ala
885 890 895
Glu Gly Thr Gly Phe Thr Glu Leu Gly Gly Glu Ile Asn Ala Gln Ile
900 905 910
Lys Arg Gly Asp Leu Leu Tyr Val Asp Val Ala Lys Gly Tyr Gly Thr
915 920 925
Gly Leu Leu Val Ser Arg Ala Ser Tyr Glu Ala Glu Lys Ser Ile Leu
930 935 940
Arg His Ile Leu Glu Gly Lys Glu Ala Val Thr Pro Leu Met Glu Arg
945 950 955 960
Val Pro Gly Glu Leu Met Glu Thr Leu Thr Ser Gly Gln Arg Ala Ala
965 970 975
Thr Arg Met Ile Leu Glu Thr Ser Asp Arg Phe Thr Val Val Gln Gly
980 985 990
Tyr Ala Gly Val Gly Lys Thr Thr Gln Phe Arg Ala Val Met Ser Ala
995 1000 1005
Val Asn Met Leu Pro Ala Ser Glu Arg Pro Arg Val Val Gly Leu
1010 1015 1020
Gly Pro Thr His Arg Ala Val Gly Glu Met Arg Ser Ala Gly Val
1025 1030 1035
Asp Ala Gln Thr Leu Ala Ser Phe Leu His Asp Thr Gln Leu Gln
1040 1045 1050
Gln Arg Ser Gly Glu Thr Pro Asp Phe Ser Asn Thr Leu Phe Leu
1055 1060 1065
Leu Asp Glu Ser Ser Met Val Gly Asn Thr Glu Met Ala Arg Ala
1070 1075 1080
Tyr Ala Leu Ile Ala Ala Gly Gly Gly Arg Ala Val Ala Ser Gly
1085 1090 1095
Asp Thr Asp Gln Leu Gln Ala Ile Ala Pro Gly Gln Ser Phe Arg
1100 1105 1110
Leu Gln Gln Thr Arg Ser Ala Ala Asp Val Val Ile Met Lys Glu
1115 1120 1125
Ile Val Arg Gln Thr Pro Glu Leu Arg Glu Ala Val Tyr Ser Leu
1130 1135 1140
Ile Asn Arg Asp Val Glu Arg Ala Leu Ser Gly Leu Glu Ser Val
1145 1150 1155
Lys Pro Ser Gln Val Pro Arg Leu Glu Gly Ala Trp Ala Pro Glu
1160 1165 1170
His Ser Val Thr Glu Phe Ser His Ser Gln Glu Ala Lys Leu Ala
1175 1180 1185
Glu Ala Gln Gln Lys Ala Met Leu Lys Gly Glu Ala Phe Pro Asp
1190 1195 1200
Ile Pro Met Thr Leu Tyr Glu Ala Ile Val Arg Asp Tyr Thr Gly
1205 1210 1215
Arg Thr Pro Glu Ala Arg Glu Gln Thr Leu Ile Val Thr His Leu
1220 1225 1230
Asn Glu Asp Arg Arg Val Leu Asn Ser Met Ile His Asp Ala Arg
1235 1240 1245
Glu Lys Ala Gly Glu Leu Gly Lys Glu Gln Val Met Val Pro Val
1250 1255 1260
Leu Asn Thr Ala Asn Ile Arg Asp Gly Glu Leu Arg Arg Leu Ser
1265 1270 1275
Thr Trp Glu Lys Asn Pro Asp Ala Leu Ala Leu Val Asp Asn Val
1280 1285 1290
Tyr His Arg Ile Ala Gly Ile Ser Lys Asp Asp Gly Leu Ile Thr
1295 1300 1305
Leu Gln Asp Ala Glu Gly Asn Thr Arg Leu Ile Ser Pro Arg Glu
1310 1315 1320
Ala Val Ala Glu Gly Val Thr Leu Tyr Thr Pro Asp Lys Ile Arg
1325 1330 1335
Val Gly Thr Gly Asp Arg Met Arg Phe Thr Lys Ser Asp Arg Glu
1340 1345 1350
Arg Gly Tyr Val Ala Asn Ser Val Trp Thr Val Thr Ala Val Ser
1355 1360 1365
Gly Asp Ser Val Thr Leu Ser Asp Gly Gln Gln Thr Arg Val Ile
1370 1375 1380
Arg Pro Gly Gln Glu Arg Ala Glu Gln His Ile Asp Leu Ala Tyr
1385 1390 1395
Ala Ile Thr Ala His Gly Ala Gln Gly Ala Ser Glu Thr Phe Ala
1400 1405 1410
Ile Ala Leu Glu Gly Thr Glu Gly Asn Arg Lys Leu Met Ala Gly
1415 1420 1425
Phe Glu Ser Ala Tyr Val Ala Leu Ser Arg Met Lys Gln His Val
1430 1435 1440
Gln Val Tyr Thr Asp Asn Arg Gln Gly Trp Thr Asp Ala Ile Asn
1445 1450 1455
Asn Ala Val Gln Lys Gly Thr Ala His Asp Val Leu Glu Pro Lys
1460 1465 1470
Pro Asp Arg Glu Val Met Asn Ala Gln Arg Leu Phe Ser Thr Ala
1475 1480 1485
Arg Glu Leu Arg Asp Val Ala Ala Gly Arg Ala Val Leu Arg Gln
1490 1495 1500
Ala Gly Leu Ala Gly Gly Asp Ser Pro Ala Arg Phe Ile Ala Pro
1505 1510 1515
Gly Arg Lys Tyr Pro Gln Pro Tyr Val Ala Leu Pro Ala Phe Asp
1520 1525 1530
Arg Asn Gly Lys Ser Ala Gly Ile Trp Leu Asn Pro Leu Thr Thr
1535 1540 1545
Asp Asp Gly Asn Gly Leu Arg Gly Phe Ser Gly Glu Gly Arg Val
1550 1555 1560
Lys Gly Ser Gly Asp Ala Gln Phe Val Ala Leu Gln Gly Ser Arg
1565 1570 1575
Asn Gly Glu Ser Leu Leu Ala Asp Asn Met Gln Asp Gly Val Arg
1580 1585 1590
Ile Ala Arg Asp Asn Pro Asp Ser Gly Val Val Val Arg Ile Ala
1595 1600 1605
Gly Glu Gly Arg Pro Trp Asn Pro Gly Ala Ile Thr Gly Gly Arg
1610 1615 1620
Val Trp Gly Asp Ile Pro Asp Asn Ser Val Gln Pro Gly Ala Gly
1625 1630 1635
Asn Gly Glu Pro Val Thr Ala Glu Val Leu Ala Gln Arg Gln Ala
1640 1645 1650
Glu Glu Ala Ile Arg Arg Glu Thr Glu Arg Arg Ala Asp Glu Ile
1655 1660 1665
Val Arg Lys Met Ala Glu Asn Lys Pro Asp Leu Pro Asp Gly Lys
1670 1675 1680
Thr Glu Leu Ala Val Arg Asp Ile Ala Gly Gln Glu Arg Asp Arg
1685 1690 1695
Ser Ala Ile Ser Glu Arg Glu Thr Ala Leu Pro Glu Ser Val Leu
1700 1705 1710
Arg Glu Ser Gln Arg Glu Arg Glu Ala Val Arg Glu Val Ala Arg
1715 1720 1725
Glu Asn Leu Leu Gln Glu Arg Leu Gln Gln Met Glu Arg Asp Met
1730 1735 1740
Val Arg Asp Leu Gln Lys Glu Lys Thr Leu Gly Gly Asp
1745 1750 1755
<210> 23
<211> 726
<212> PRT
<213> 伯顿拟甲烷球菌(Methanococcoides burtonii)
<400> 23
Met Ser Asp Lys Pro Ala Phe Met Lys Tyr Phe Thr Gln Ser Ser Cys
1 5 10 15
Tyr Pro Asn Gln Gln Glu Ala Met Asp Arg Ile His Ser Ala Leu Met
20 25 30
Gln Gln Gln Leu Val Leu Phe Glu Gly Ala Cys Gly Thr Gly Lys Thr
35 40 45
Leu Ser Ala Leu Val Pro Ala Leu His Val Gly Lys Met Leu Gly Lys
50 55 60
Thr Val Ile Ile Ala Thr Asn Val His Gln Gln Met Val Gln Phe Ile
65 70 75 80
Asn Glu Ala Arg Asp Ile Lys Lys Val Gln Asp Val Lys Val Ala Val
85 90 95
Ile Lys Gly Lys Thr Ala Met Cys Pro Gln Glu Ala Asp Tyr Glu Glu
100 105 110
Cys Ser Val Lys Arg Glu Asn Thr Phe Glu Leu Met Glu Thr Glu Arg
115 120 125
Glu Ile Tyr Leu Lys Arg Gln Glu Leu Asn Ser Ala Arg Asp Ser Tyr
130 135 140
Lys Lys Ser His Asp Pro Ala Phe Val Thr Leu Arg Asp Glu Leu Ser
145 150 155 160
Lys Glu Ile Asp Ala Val Glu Glu Lys Ala Arg Gly Leu Arg Asp Arg
165 170 175
Ala Cys Asn Asp Leu Tyr Glu Val Leu Arg Ser Asp Ser Glu Lys Phe
180 185 190
Arg Glu Trp Leu Tyr Lys Glu Val Arg Ser Pro Glu Glu Ile Asn Asp
195 200 205
His Ala Ile Lys Asp Gly Met Cys Gly Tyr Glu Leu Val Lys Arg Glu
210 215 220
Leu Lys His Ala Asp Leu Leu Ile Cys Asn Tyr His His Val Leu Asn
225 230 235 240
Pro Asp Ile Phe Ser Thr Val Leu Gly Trp Ile Glu Lys Glu Pro Gln
245 250 255
Glu Thr Ile Val Ile Phe Asp Glu Ala His Asn Leu Glu Ser Ala Ala
260 265 270
Arg Ser His Ser Ser Leu Ser Leu Thr Glu His Ser Ile Glu Lys Ala
275 280 285
Ile Thr Glu Leu Glu Ala Asn Leu Asp Leu Leu Ala Asp Asp Asn Ile
290 295 300
His Asn Leu Phe Asn Ile Phe Leu Glu Val Ile Ser Asp Thr Tyr Asn
305 310 315 320
Ser Arg Phe Lys Phe Gly Glu Arg Glu Arg Val Arg Lys Asn Trp Tyr
325 330 335
Asp Ile Arg Ile Ser Asp Pro Tyr Glu Arg Asn Asp Ile Val Arg Gly
340 345 350
Lys Phe Leu Arg Gln Ala Lys Gly Asp Phe Gly Glu Lys Asp Asp Ile
355 360 365
Gln Ile Leu Leu Ser Glu Ala Ser Glu Leu Gly Ala Lys Leu Asp Glu
370 375 380
Thr Tyr Arg Asp Gln Tyr Lys Lys Gly Leu Ser Ser Val Met Lys Arg
385 390 395 400
Ser His Ile Arg Tyr Val Ala Asp Phe Met Ser Ala Tyr Ile Glu Leu
405 410 415
Ser His Asn Leu Asn Tyr Tyr Pro Ile Leu Asn Val Arg Arg Asp Met
420 425 430
Asn Asp Glu Ile Tyr Gly Arg Val Glu Leu Phe Thr Cys Ile Pro Lys
435 440 445
Asn Val Thr Glu Pro Leu Phe Asn Ser Leu Phe Ser Val Ile Leu Met
450 455 460
Ser Ala Thr Leu His Pro Phe Glu Met Val Lys Lys Thr Leu Gly Ile
465 470 475 480
Thr Arg Asp Thr Cys Glu Met Ser Tyr Gly Thr Ser Phe Pro Glu Glu
485 490 495
Lys Arg Leu Ser Ile Ala Val Ser Ile Pro Pro Leu Phe Ala Lys Asn
500 505 510
Arg Asp Asp Arg His Val Thr Glu Leu Leu Glu Gln Val Leu Leu Asp
515 520 525
Ser Ile Glu Asn Ser Lys Gly Asn Val Ile Leu Phe Phe Gln Ser Ala
530 535 540
Phe Glu Ala Lys Arg Tyr Tyr Ser Lys Ile Glu Pro Leu Val Asn Val
545 550 555 560
Pro Val Phe Leu Asp Glu Val Gly Ile Ser Ser Gln Asp Val Arg Glu
565 570 575
Glu Phe Phe Ser Ile Gly Glu Glu Asn Gly Lys Ala Val Leu Leu Ser
580 585 590
Tyr Leu Trp Gly Thr Leu Ser Glu Gly Ile Asp Tyr Arg Asp Gly Arg
595 600 605
Gly Arg Thr Val Ile Ile Ile Gly Val Gly Tyr Pro Ala Leu Asn Asp
610 615 620
Arg Met Asn Ala Val Glu Ser Ala Tyr Asp His Val Phe Gly Tyr Gly
625 630 635 640
Ala Gly Trp Glu Phe Ala Ile Gln Val Pro Thr Ile Arg Lys Ile Arg
645 650 655
Gln Ala Met Gly Arg Val Val Arg Ser Pro Thr Asp Tyr Gly Ala Arg
660 665 670
Ile Leu Leu Asp Gly Arg Phe Leu Thr Asp Ser Lys Lys Arg Phe Gly
675 680 685
Lys Phe Ser Val Phe Glu Val Phe Pro Pro Ala Glu Arg Ser Glu Phe
690 695 700
Val Asp Val Asp Pro Glu Lys Val Lys Tyr Ser Leu Met Asn Phe Phe
705 710 715 720
Met Asp Asn Asp Glu Gln
725
<210> 24
<211> 439
<212> PRT
<213> 菊欧文氏菌(Dickeya dadantii)
<400> 24
Met Thr Phe Asp Asp Leu Thr Glu Gly Gln Lys Asn Ala Phe Asn Ile
1 5 10 15
Val Met Lys Ala Ile Lys Glu Lys Lys His His Val Thr Ile Asn Gly
20 25 30
Pro Ala Gly Thr Gly Lys Thr Thr Leu Thr Lys Phe Ile Ile Glu Ala
35 40 45
Leu Ile Ser Thr Gly Glu Thr Gly Ile Ile Leu Ala Ala Pro Thr His
50 55 60
Ala Ala Lys Lys Ile Leu Ser Lys Leu Ser Gly Lys Glu Ala Ser Thr
65 70 75 80
Ile His Ser Ile Leu Lys Ile Asn Pro Val Thr Tyr Glu Glu Asn Val
85 90 95
Leu Phe Glu Gln Lys Glu Val Pro Asp Leu Ala Lys Cys Arg Val Leu
100 105 110
Ile Cys Asp Glu Val Ser Met Tyr Asp Arg Lys Leu Phe Lys Ile Leu
115 120 125
Leu Ser Thr Ile Pro Pro Trp Cys Thr Ile Ile Gly Ile Gly Asp Asn
130 135 140
Lys Gln Ile Arg Pro Val Asp Pro Gly Glu Asn Thr Ala Tyr Ile Ser
145 150 155 160
Pro Phe Phe Thr His Lys Asp Phe Tyr Gln Cys Glu Leu Thr Glu Val
165 170 175
Lys Arg Ser Asn Ala Pro Ile Ile Asp Val Ala Thr Asp Val Arg Asn
180 185 190
Gly Lys Trp Ile Tyr Asp Lys Val Val Asp Gly His Gly Val Arg Gly
195 200 205
Phe Thr Gly Asp Thr Ala Leu Arg Asp Phe Met Val Asn Tyr Phe Ser
210 215 220
Ile Val Lys Ser Leu Asp Asp Leu Phe Glu Asn Arg Val Met Ala Phe
225 230 235 240
Thr Asn Lys Ser Val Asp Lys Leu Asn Ser Ile Ile Arg Lys Lys Ile
245 250 255
Phe Glu Thr Asp Lys Asp Phe Ile Val Gly Glu Ile Ile Val Met Gln
260 265 270
Glu Pro Leu Phe Lys Thr Tyr Lys Ile Asp Gly Lys Pro Val Ser Glu
275 280 285
Ile Ile Phe Asn Asn Gly Gln Leu Val Arg Ile Ile Glu Ala Glu Tyr
290 295 300
Thr Ser Thr Phe Val Lys Ala Arg Gly Val Pro Gly Glu Tyr Leu Ile
305 310 315 320
Arg His Trp Asp Leu Thr Val Glu Thr Tyr Gly Asp Asp Glu Tyr Tyr
325 330 335
Arg Glu Lys Ile Lys Ile Ile Ser Ser Asp Glu Glu Leu Tyr Lys Phe
340 345 350
Asn Leu Phe Leu Gly Lys Thr Ala Glu Thr Tyr Lys Asn Trp Asn Lys
355 360 365
Gly Gly Lys Ala Pro Trp Ser Asp Phe Trp Asp Ala Lys Ser Gln Phe
370 375 380
Ser Lys Val Lys Ala Leu Pro Ala Ser Thr Phe His Lys Ala Gln Gly
385 390 395 400
Met Ser Val Asp Arg Ala Phe Ile Tyr Thr Pro Cys Ile His Tyr Ala
405 410 415
Asp Val Glu Leu Ala Gln Gln Leu Leu Tyr Val Gly Val Thr Arg Gly
420 425 430
Arg Tyr Asp Val Phe Tyr Val
435
<210> 25
<211> 970
<212> PRT
<213> 肉毒梭菌(Clostridium botulinum)
<400> 25
Met Leu Ser Val Ala Asn Val Arg Ser Pro Ser Ala Ala Ala Ser Tyr
1 5 10 15
Phe Ala Ser Asp Asn Tyr Tyr Ala Ser Ala Asp Ala Asp Arg Ser Gly
20 25 30
Gln Trp Ile Gly Asp Gly Ala Lys Arg Leu Gly Leu Glu Gly Lys Val
35 40 45
Glu Ala Arg Ala Phe Asp Ala Leu Leu Arg Gly Glu Leu Pro Asp Gly
50 55 60
Ser Ser Val Gly Asn Pro Gly Gln Ala His Arg Pro Gly Thr Asp Leu
65 70 75 80
Thr Phe Ser Val Pro Lys Ser Trp Ser Leu Leu Ala Leu Val Gly Lys
85 90 95
Asp Glu Arg Ile Ile Ala Ala Tyr Arg Glu Ala Val Val Glu Ala Leu
100 105 110
His Trp Ala Glu Lys Asn Ala Ala Glu Thr Arg Val Val Glu Lys Gly
115 120 125
Met Val Val Thr Gln Ala Thr Gly Asn Leu Ala Ile Gly Leu Phe Gln
130 135 140
His Asp Thr Asn Arg Asn Gln Glu Pro Asn Leu His Phe His Ala Val
145 150 155 160
Ile Ala Asn Val Thr Gln Gly Lys Asp Gly Lys Trp Arg Thr Leu Lys
165 170 175
Asn Asp Arg Leu Trp Gln Leu Asn Thr Thr Leu Asn Ser Ile Ala Met
180 185 190
Ala Arg Phe Arg Val Ala Val Glu Lys Leu Gly Tyr Glu Pro Gly Pro
195 200 205
Val Leu Lys His Gly Asn Phe Glu Ala Arg Gly Ile Ser Arg Glu Gln
210 215 220
Val Met Ala Phe Ser Thr Arg Arg Lys Glu Val Leu Glu Ala Arg Arg
225 230 235 240
Gly Pro Gly Leu Asp Ala Gly Arg Ile Ala Ala Leu Asp Thr Arg Ala
245 250 255
Ser Lys Glu Gly Ile Glu Asp Arg Ala Thr Leu Ser Lys Gln Trp Ser
260 265 270
Glu Ala Ala Gln Ser Ile Gly Leu Asp Leu Lys Pro Leu Val Asp Arg
275 280 285
Ala Arg Thr Lys Ala Leu Gly Gln Gly Met Glu Ala Thr Arg Ile Gly
290 295 300
Ser Leu Val Glu Arg Gly Arg Ala Trp Leu Ser Arg Phe Ala Ala His
305 310 315 320
Val Arg Gly Asp Pro Ala Asp Pro Leu Val Pro Pro Ser Val Leu Lys
325 330 335
Gln Asp Arg Gln Thr Ile Ala Ala Ala Gln Ala Val Ala Ser Ala Val
340 345 350
Arg His Leu Ser Gln Arg Glu Ala Ala Phe Glu Arg Thr Ala Leu Tyr
355 360 365
Lys Ala Ala Leu Asp Phe Gly Leu Pro Thr Thr Ile Ala Asp Val Glu
370 375 380
Lys Arg Thr Arg Ala Leu Val Arg Ser Gly Asp Leu Ile Ala Gly Lys
385 390 395 400
Gly Glu His Lys Gly Trp Leu Ala Ser Arg Asp Ala Val Val Thr Glu
405 410 415
Gln Arg Ile Leu Ser Glu Val Ala Ala Gly Lys Gly Asp Ser Ser Pro
420 425 430
Ala Ile Thr Pro Gln Lys Ala Ala Ala Ser Val Gln Ala Ala Ala Leu
435 440 445
Thr Gly Gln Gly Phe Arg Leu Asn Glu Gly Gln Leu Ala Ala Ala Arg
450 455 460
Leu Ile Leu Ile Ser Lys Asp Arg Thr Ile Ala Val Gln Gly Ile Ala
465 470 475 480
Gly Ala Gly Lys Ser Ser Val Leu Lys Pro Val Ala Glu Val Leu Arg
485 490 495
Asp Glu Gly His Pro Val Ile Gly Leu Ala Ile Gln Asn Thr Leu Val
500 505 510
Gln Met Leu Glu Arg Asp Thr Gly Ile Gly Ser Gln Thr Leu Ala Arg
515 520 525
Phe Leu Gly Gly Trp Asn Lys Leu Leu Asp Asp Pro Gly Asn Val Ala
530 535 540
Leu Arg Ala Glu Ala Gln Ala Ser Leu Lys Asp His Val Leu Val Leu
545 550 555 560
Asp Glu Ala Ser Met Val Ser Asn Glu Asp Lys Glu Lys Leu Val Arg
565 570 575
Leu Ala Asn Leu Ala Gly Val His Arg Leu Val Leu Ile Gly Asp Arg
580 585 590
Lys Gln Leu Gly Ala Val Asp Ala Gly Lys Pro Phe Ala Leu Leu Gln
595 600 605
Arg Ala Gly Ile Ala Arg Ala Glu Met Ala Thr Asn Leu Arg Ala Arg
610 615 620
Asp Pro Val Val Arg Glu Ala Gln Ala Ala Ala Gln Ala Gly Asp Val
625 630 635 640
Arg Lys Ala Leu Arg His Leu Lys Ser His Thr Val Glu Ala Arg Gly
645 650 655
Asp Gly Ala Gln Val Ala Ala Glu Thr Trp Leu Ala Leu Asp Lys Glu
660 665 670
Thr Arg Ala Arg Thr Ser Ile Tyr Ala Ser Gly Arg Ala Ile Arg Ser
675 680 685
Ala Val Asn Ala Ala Val Gln Gln Gly Leu Leu Ala Ser Arg Glu Ile
690 695 700
Gly Pro Ala Lys Met Lys Leu Glu Val Leu Asp Arg Val Asn Thr Thr
705 710 715 720
Arg Glu Glu Leu Arg His Leu Pro Ala Tyr Arg Ala Gly Arg Val Leu
725 730 735
Glu Val Ser Arg Lys Gln Gln Ala Leu Gly Leu Phe Ile Gly Glu Tyr
740 745 750
Arg Val Ile Gly Gln Asp Arg Lys Gly Lys Leu Val Glu Val Glu Asp
755 760 765
Lys Arg Gly Lys Arg Phe Arg Phe Asp Pro Ala Arg Ile Arg Ala Gly
770 775 780
Lys Gly Asp Asp Asn Leu Thr Leu Leu Glu Pro Arg Lys Leu Glu Ile
785 790 795 800
His Glu Gly Asp Arg Ile Arg Trp Thr Arg Asn Asp His Arg Arg Gly
805 810 815
Leu Phe Asn Ala Asp Gln Ala Arg Val Val Glu Ile Ala Asn Gly Lys
820 825 830
Val Thr Phe Glu Thr Ser Lys Gly Asp Leu Val Glu Leu Lys Lys Asp
835 840 845
Asp Pro Met Leu Lys Arg Ile Asp Leu Ala Tyr Ala Leu Asn Val His
850 855 860
Met Ala Gln Gly Leu Thr Ser Asp Arg Gly Ile Ala Val Met Asp Ser
865 870 875 880
Arg Glu Arg Asn Leu Ser Asn Gln Lys Thr Phe Leu Val Thr Val Thr
885 890 895
Arg Leu Arg Asp His Leu Thr Leu Val Val Asp Ser Ala Asp Lys Leu
900 905 910
Gly Ala Ala Val Ala Arg Asn Lys Gly Glu Lys Ala Ser Ala Ile Glu
915 920 925
Val Thr Gly Ser Val Lys Pro Thr Ala Thr Lys Gly Ser Gly Val Asp
930 935 940
Gln Pro Lys Ser Val Glu Ala Asn Lys Ala Glu Lys Glu Leu Thr Arg
945 950 955 960
Ser Lys Ser Lys Thr Leu Asp Phe Gly Ile
965 970
<210> 26
<211> 46
<212> DNA
<213> 人工序列
<220>
<223> 实施例1中使用的多核苷酸序列
<400> 26
tttttttttt tttttttttt tttttttttt tttttttttt tttttt 46
<210> 27
<211> 27
<212> DNA
<213> 人工序列
<220>
<223> 实施例1中使用的多核苷酸序列
<400> 27
ggttgtttct gttggtgctg atattgc 27
<210> 28
<211> 3588
<212> DNA
<213> 人工序列
<220>
<223> 实施例1中使用的多核苷酸序列
<400> 28
gccatcagat tgtgtttgtt agtcgctgcc atcagattgt gtttgttagt cgcttttttt 60
ttttggaatt ttttttttgg aatttttttt ttgcgctaac aacctcctgc cgttttgccc 120
gtgcatatcg gtcacgaaca aatctgatta ctaaacacag tagcctggat ttgttctatc 180
agtaatcgac cttattccta attaaataga gcaaatcccc ttattggggg taagacatga 240
agatgccaga aaaacatgac ctgttggccg ccattctcgc ggcaaaggaa caaggcatcg 300
gggcaatcct tgcgtttgca atggcgtacc ttcgcggcag atataatggc ggtgcgttta 360
caaaaacagt aatcgacgca acgatgtgcg ccattatcgc ctagttcatt cgtgaccttc 420
tcgacttcgc cggactaagt agcaatctcg cttatataac gagcgtgttt atcggctaca 480
tcggtactga ctcgattggt tcgcttatca aacgcttcgc tgctaaaaaa gccggagtag 540
aagatggtag aaatcaataa tcaacgtaag gcgttcctcg atatgctggc gtggtcggag 600
ggaactgata acggacgtca gaaaaccaga aatcatggtt atgacgtcat tgtaggcgga 660
gagctattta ctgattactc cgatcaccct cgcaaacttg tcacgctaaa cccaaaactc 720
aaatcaacag gcgccggacg ctaccagctt ctttcccgtt ggtgggatgc ctaccgcaag 780
cagcttggcc tgaaagactt ctctccgaaa agtcaggacg ctgtggcatt gcagcagatt 840
aaggagcgtg gcgctttacc tatgattgat cgtggtgata tccgtcaggc aatcgaccgt 900
tgcagcaata tctgggcttc actgccgggc gctggttatg gtcagttcga gcataaggct 960
gacagcctga ttgcaaaatt caaagaagcg ggcggaacgg tcagagagat tgatgtatga 1020
gcagagtcac cgcgattatc tccgctctgg ttatctgcat catcgtctgc ctgtcatggg 1080
ctgttaatca ttaccgtgat aacgccatta cctacaaagc ccagcgcgac aaaaatgcca 1140
gagaactgaa gctggcgaac gcggcaatta ctgacatgca gatgcgtcag cgtgatgttg 1200
ctgcgctcga tgcaaaatac acgaaggagt tagctgatgc taaagctgaa aatgatgctc 1260
tgcgtgatga tgttgccgct ggtcgtcgtc ggttgcacat caaagcagtc tgtcagtcag 1320
tgcgtgaagc caccaccgcc tccggcgtgg ataatgcagc ctccccccga ctggcagaca 1380
ccgctgaacg ggattatttc accctcagag agaggctgat cactatgcaa aaacaactgg 1440
aaggaaccca gaagtatatt aatgagcagt gcagatagag ttgcccatat cgatgggcaa 1500
ctcatgcaat tattgtgagc aatacacacg cgcttccagc ggagtataaa tgcctaaagt 1560
aataaaaccg agcaatccat ttacgaatgt ttgctgggtt tctgttttaa caacattttc 1620
tgcgccgcca caaattttgg ctgcatcgac agttttcttc tgcccaattc cagaaacgaa 1680
gaaatgatgg gtgatggttt cctttggtgc tactgctgcc ggtttgtttt gaacagtaaa 1740
cgtctgttga gcacatcctg taataagcag ggccagcgca gtagcgagta gcattttttt 1800
catggtgtta ttcccgatgc tttttgaagt tcgcagaatc gtatgtgtag aaaattaaac 1860
aaaccctaaa caatgagttg aaatttcata ttgttaatat ttattaatgt atgtcaggtg 1920
cgatgaatcg tcattgtatt cccggattaa ctatgtccac agccctgacg gggaacttct 1980
ctgcgggagt gtccgggaat aattaaaacg atgcacacag ggtttagcgc gtacacgtat 2040
tgcattatgc caacgccccg gtgctgacac ggaagaaacc ggacgttatg atttagcgtg 2100
gaaagatttg tgtagtgttc tgaatgctct cagtaaatag taatgaatta tcaaaggtat 2160
agtaatatct tttatgttca tggatatttg taacccatcg gaaaactcct gctttagcaa 2220
gattttccct gtattgctga aatgtgattt ctcttgattt caacctatca taggacgttt 2280
ctataagatg cgtgtttctt gagaatttaa catttacaac ctttttaagt ccttttatta 2340
acacggtgtt atcgttttct aacacgatgt gaatattatc tgtggctaga tagtaaatat 2400
aatgtgagac gttgtgacgt tttagttcag aataaaacaa ttcacagtct aaatcttttc 2460
gcacttgatc gaatatttct ttaaaaatgg caacctgagc cattggtaaa accttccatg 2520
tgatacgagg gcgcgtagtt tgcattatcg tttttatcgt ttcaatctgg tctgacctcc 2580
ttgtgttttg ttgatgattt atgtcaaata ttaggaatgt tttcacttaa tagtattggt 2640
tgcgtaacaa agtgcggtcc tgctggcatt ctggagggaa atacaaccga cagatgtatg 2700
taaggccaac gtgctcaaat cttcatacag aaagatttga agtaatattt taaccgctag 2760
atgaagagca agcgcatgga gcgacaaaat gaataaagaa caatctgctg atgatccctc 2820
cgtggatctg attcgtgtaa aaaatatgct taatagcacc atttctatga gttaccctga 2880
tgttgtaatt gcatgtatag aacataaggt gtctctggaa gcattcagag caattgaggc 2940
agcgttggtg aagcacgata ataatatgaa ggattattcc ctggtggttg actgatcacc 3000
ataactgcta atcattcaaa ctatttagtc tgtgacagag ccaacacgca gtctgtcact 3060
gtcaggaaag tggtaaaact gcaactcaat tactgcaatg ccctcgtaat taagtgaatt 3120
tacaatatcg tcctgttcgg agggaagaac gcgggatgtt cattcttcat cacttttaat 3180
tgatgtatat gctctctttt ctgacgttag tctccgacgg caggcttcaa tgacccaggc 3240
tgagaaattc ccggaccctt tttgctcaag agcgatgtta atttgttcaa tcatttggtt 3300
aggaaagcgg atgttgcggg ttgttgttct gcgggttctg ttcttcgttg acatgaggtt 3360
gccccgtatt cagtgtcgct gatttgtatt gtctgaagtt gtttttacgt taagttgatg 3420
cagatcaatt aatacgatac ctgcgtcata attgattatt tgacgtggtt tgatggcctc 3480
cacgcacgtt gtgatatgta gatgataatc attatcactt tacgggtcct ttccggtgaa 3540
aaaaaaggta ccaaaaaaaa catcgtcgtg agtagtgaac cgtaagca 3588
<210> 29
<211> 28
<212> DNA
<213> 人工序列
<220>
<223> 实施例1中使用的多核苷酸序列
<400> 29
gcaatatcag caccaacaga aacaacct 28
- 还没有人留言评论。精彩留言会获得点赞!