通过实时PCR从细胞裂解物定量HBVcccDNA的高通量新方法与流程
发明领域
本发明涉及(1)可以用于乙型肝炎病毒(hbv)共价闭合型环状dna(cccdna)的高度高效实时qpcr的新dna引物组和(2)使用这个新dna引物组通过实时qpcr从细胞定量hbvcccdna的高通量新方法。它还涉及这种方法在实验感染的肝细胞中监测cccdna水平和评价对hbv的治疗效果中的用途。
发明背景
由于cccdna在hbv生物学中发挥重要作用,所以已经建立许多实验系统以研究靶向cccdna的潜在候选药物。为了提供cccdna的可控细胞模型,ladner等人(ladner,s.k.,otto,m.j.,barker,c.s.,zaifert,k.,wang,g.h.,guo,j.t.,seeger,c.,king,r.w.,antimicrobagentschemother.1997年8月;41(8):1715-20)已经插入了在tet启动子系统的转录性控制下的hbv病毒基因组。另外,插入物的长度超过hbv全基因组的长度,这提供了在nt(核苷酸)1805-1900的序列重叠(cai等人,antimicrobagentschemother.2012年8月;56(8):4277-88)。这种方案允许利用eag产生作为cccdna水平和功能的替代标志物。最后,guo等人(j.virol.2007,81(22):12472)提出,在以上遗传背景下进一步突变sag基因时,病毒dna的形成不能用来形成成熟病毒粒子,因此在名为hepdes19的细胞系中遵循一个待循环入胞核的途径以为cccdna形成提供更多前体。因为这些变化导致受控的cccdna产生、cccdna的替代测量和消除有功能的病毒粒子,所以基于hepdes19的细胞模型大大促进了cccdna研究并且导致建立利用eag作为cccdna的替代标志物的高通量筛选(hts)测定法(antimicrobagentschemother.2012年8月;56(8):4277-88)。
但是,cccdna是治疗hbv疾病的直接目的靶。直接测量细胞系中、尤其hepdes19细胞中的cccdna因此在针对cccdna产生和稳定性的活性评价方面重要。
不能利用借助常规技术如dna印迹杂交对cccdna的测量作为高通量方法,原因在于其灵敏度或通量低。he(bbrc295(2002)1102-1107,美国专利申请:20040058314)、shao等人(cn101503740b)和lu等人(cn101285105b)先前已经报告了基于qpcr的cccdna定量测定法。尽管详细的qpcr方法可能不同,但全部方法均需要在分析之前提取hbvdna。dna提取是复杂的并且除去对qpcr重要的系统关键组分。因此,当涉及这类dna提取时,通常通过使用等量的起始材料(如细胞数目)设置对照。尽管这种对照管提供了如何尝试补偿结果的指示,但严格地说,它们不包含真实的实验对照。
因为包括了dna纯化步骤,所以通过qpcr直接测量cccdna不能放大成高通量筛选模式(hirt,b,j.mol.biol.1967;26,365-9)。为了提供可以在全部样品源、尤其来自hepdes19细胞模型系统的那些样品源中实现高通量测量cccdna的更好方法,需要稳健的新筛查方案。
许多人已经描述了在不预先纯化dna的情况下直接测量细胞裂解物样品中特定形式的某种核酸的方法(yangk.等人,gene.2013年12月1日,531(2):199-204;kumarjs等人,molcellprobes.2014年4月13日;pangz等人,plosone.2014年4月21日,9(4):e95635)。这些方案显著地增加测定效率。但是,这些方案不能适用于he、shao和lu描述的cccdnaqpcr方法。基于来自他们的参比方法的引物序列信息,他们的反向引物全部位于hbv的nt-1990上游的序列范围内,因为它们可能轻易地用于q-pcr测定法中。因为这一点,那些基于qpcr的方法全部从1.1×hbv基因组的染色体插入物扩增dna信号(guo等人,jvirol.2007年11月,81(22):12472-84),因此未能在hepdes19细胞中选择性地扩增cccdna。另外,直接裂解,然后qpcr需要所涉及引物的更高特异性,以将cccdna与其他形式的hbvdna(如细胞中插入的松弛型环状dna(rcdna)和hbv基因组)区分。
总之,不存在不预提取hbvdna的情况下可用于定量hbv感染的细胞、尤其hepdes19中cccdna水平的敏感和特异性hts直接测定法。因此,需要以高灵敏度、高通量和高特异性定量cccdna的方法。如本文中所述的本发明的目的将满足这些需求。
发明简述
本发明计划开发一种在不事先提取和纯化dna的情况下,通过从hbv感染的细胞、尤其hepdes19细胞直接实时pcr来一步cccdna定量hts(高通量筛选)方法。
我们已经设计了一组cccdna特异性引物。已经证明这种新引物组在从hbvcccdna而不从基因组dna或染色体插入的hbvdna扩增dna片段方面具有cccdna特异性。这些引物的cccdna特异性消除了dna提取和cccdna纯化步骤的需求并且允许我们以微量滴定板测定样式按hts模式定量cccdna。
本发明的一个实施方案是一种反向引物,所述反向引物在其3’最末端包含识别乙型肝炎病毒(hbv)基因组(seqidno:1)的核苷酸中至少16个连续核苷酸的序列。
本发明的另一个实施方案是一个由具有如上文所述的意思的反向引物和通用正向引物组成的引物对,其中通用正向引物的长度是16至200个核苷酸、17至35个核苷酸、18至30个核苷酸、21至30个核苷酸、24至30个核苷酸或21至24个核苷酸并且识别hbv基因组的dr2的上游。
本发明的另一个实施方案是一种用于检测提取的dna或一步qpcr测定法的裂解物的探针,其中探针的长度是16至200个核苷酸、17至35个核苷酸、18至30个核苷酸、21至30个核苷酸、24至30个核苷酸或21至24个核苷酸。
本发明涉及包含反向引物和探针的组合,其中反向引物和探针具有如上文所述的重要性。
本发明涉及通过pcr检测hbvcccdna的方法,所述方法包括使用hbv基因组(seqidno:1)作为模板、使用反向引物和通用正向引物的引物对或使用包含反向引物和探针的组合,其中反向引物、通用正向引物和探针具有如上文所述的意思。
本发明涉及一种在评价对hbv的治疗效果中使用的方法,其中方法具有如上文所述的意思。
本发明涉及反向引物,反向引物和通用正向引物的引物对,或包含反向引物和探针的组合用于评价对hbv的治疗效果的用途,其中反向引物、通用正向引物和探针具有如上文所述的意思。
本发明涉及反向引物,反向引物和通用正向引物的引物对,或包含反向引物和探针的组合在制备检测剂中的用途,所述检测剂用于评价对hbv的治疗效果,其中反向引物、通用正向引物和探针具有如上文所述的意思。
本发明涉及内参基因作为归一化对照的用途。它包括使用针对基因组dna上基因的引物作为qpcr定量对照。
在本发明中保留全部dna组分的比例允许使用这种定量对照。
本发明涉及用于评价对hbv的治疗效果的试剂盒,所述试剂盒包含反向引物、引物对或组合,其中反向引物、引物对和组合具有如上文所述的意思。
本发明还涉及用于评价对hbv的治疗效果的多重试剂盒,所述多重试剂盒包含反向引物、引物对、参比对照基因引物、以及针对hbvdna和针对参比基因的一对探针,其中反向引物及引物对具有如上文所述的意思。
定义
除非另外规定,否则本文中所用的全部术语及科学术语具有与本发明相关领域的技术人员通常理解的相同意义。尽管基本上与本文所述的那些方法和材料相似的任意方法和材料可以用于本发明的实施或检测,然而仅描述示例性的方法和材料。为了本发明的目的,下文定义以下术语。
除非上下文另外清楚地指明,否则术语“核苷酸”应当在本文中理解为除了指天然存在的核糖核苷酸单体或脱氧核糖核苷酸单体之外,还指相对于正在使用的核苷酸的特定上下文(例如,杂交至互补碱基),在功能上等同的其相关的结构变体,包括衍生物和类似物。
在两个或更多个核酸序列或多肽序列的情境下,术语“同一性”指相同的两个或更多个序列或子序列。如果在比较窗口或指定区域范围内为最大对应性而进行比较和比对时,如使用以下序列比较算法之一或通过手工比对和目视审查所测量,各序列具有指定百分数的相同核苷酸或氨基酸残基(例如,在指定区域范围内至少20%、至少25%、至少30%、至少35%、至少40%、至少45%、至少50%、至少55%、至少60%、至少65%、至少70%、至少75%、至少80%、至少85%、至少90%或至少95%同一性),则它们是彼此“基本上同一的”。这些定义还指测试序列的互补序列。任选地,同一性存在于具有至少约50个核苷酸长度的区域内,或更一般地存在于具有100个至500个或1000或更多个核苷酸长度的区域内。
如本文所用的术语“pcr”指聚合酶链反应。
术语“qpcr”通常指称作实时定量聚合酶链反应、定量聚合酶链反应或动力学聚合酶链反应的pcr技术。这项技术利用pcr同时地扩增并定量靶核酸,其中借助嵌入性荧光染料或序列特异性探针定量,所述序列特异性探针含有仅与靶核酸杂交才可检出的荧光报道分子。
如本文所用的术语“引物”指能够在qpcr反应中充当dna复制反应起始子的寡核苷酸dna。
如本文所用的术语“探针”指具有荧光标签的寡核苷酸,所述荧光标签能够改变荧光强度,其中荧光强度的变化与反应中存在的互补dna的水平增加相关。
术语“cp”指允许定量输入靶核酸的值。cp值可以根据二阶导数最大法确定(vanluu-the等人,“improvedreal-timert-pcrmethodforhigh-throughputmeasurementsusingsecondderivativecalculationanddoublecorrection,”biotechniques,vol.38,no.2,2005年2月,第287-293页)。在二阶导数法中,cp对应于二阶导数曲线的第一峰。这个峰对应于对数线性阶段的起点。二阶导数法计算实时荧光强度曲线的二阶导数值,并且仅获得一个值。原始cp法基于强度值的局部定义的可微分逼近,例如,通过多项式函数逼近。随后计算三阶导数。cp值是三阶导数的最小根。也可以使用拟合点法确定cp,其中通过对数线性区域中平行于阈值线的交叉,确定cp(vanluu-the等人,biotechniques,vol.38,no.2,2005年2月,第287-293页)。这些计算容易地由本领域的任何技术人员实施。
如本文所用的术语“提取”指根据改良hirt程序的方法纯化染色体外dna(aradu,biotechniques.1998may;24(5):760-2;hirt,b,j.mol.biol.1967;26,365-9)。
术语“常规”指基于本领域充分认可的已出版参考文献的实验方法和材料。为了避免含混性,附图中的“常规组”和“常规”具有不同意含义,所述含义在下文缩写部分中说明。
术语“一步qpcr”指其中将细胞材料裂解并直接进行qpcr分析的实验程序。
术语“z因子”是测定法统计重现性的量度。已经提出它用于高通量筛选(其中它又称作z-prime,并且常书写为z'),以判定具体测定法中的反应是否大到足以保证进一步注意。z-因子根据四个参数定义:阳性对照(p)和阴性对照(n)的均数和标准差(μp、σp、μn和σn)。给定这些值,将z-因子定义为:
术语“tamra”指作为染料的5-羧基-四甲基罗丹明n-琥珀酰亚胺酯。染料代用品:当寻求染料备选物时,以下标准是重要的:1)激发波长和检测波长与仪器光源和检测系统相容。2)对于探针,猝灭剂在报道分子的发射波长处有效地吸收光。3)消光系数越高,染料越明亮,这有助于灵敏检测。
术语“bhq2”指作为猝灭剂的染料猝灭剂blackholequencher-2。猝灭剂:猝灭分子一般置于单个分子探针的3’末端。猝灭剂可以是荧光分子(tamratm)或非荧光分子(dabcyl、blackhole
附图简述
图1:图1描述了hepdes19稳定细胞中的整合hbvdna和设计原理示意图。hepdes19细胞中的整合hbvdna由1.1倍超长hbv基因组组成,其置于tetcmv启动子下游,始于核苷酸1805。hbvnt位置依据galibert命名法(galibert,f等人,nature281:646-650)。如图中所示,正向引物(f)序列的位置处于双链松弛型环状dna(rcdna)的单链部分内部。因为这一点,rcdna中的逆反应产物不能在下一个pcr循环中由正向引物用作模板。另外,qpcr设计中的探针在rcdna中切口的下游。物理断口的存在阻止正向引物反应移入rcdna中的探针位置。在cccdna中,不存在切口结构,这引起聚合酶切割探针,造成荧光增加。这两种因素解释了qpcr方案对cccdna的选择性高于rcdna。但是,在hepdes19细胞中,还存在染色体1.1倍hbv基因组插入物。这个1.1倍超长hbv基因组的3’端冗余性序列是从nt1805至1990。
我们首先发现如果反向引物从1990的上游设计,则染色体hbvdna插入物将在qpcr测定法中扩增,这令提取病毒dna对于cccdna定量是必需的。这解释了当使用常规反向引物时为何需要提取病毒dna(takkenberg等人,methodsinmolecularbiology,vol.903,doi10.1007/978-1-61779-937-2_7)。我们令人惊讶地发现,通过在1990的下游设计反向引物,这种新的反向引物位于图中正向引物的左侧(彼此指向相反),在qpcr测定法中不扩增基因组hbvdna插入物,并且新的反向引物仍指向cccdna上的正向引物,导致生产性qpcr反应,原因在于cccdna的环形本质。
图2:图2描述了使用针对已提取病毒基因组dna的常规cccdna引物对和使用一步qpcr测定法的qpcr测定法。
图3:图3描述了用于检测来自hepdes19的已提取dna或无提取的直接裂解物(一步qpcr测定法)的新cccdna引物的设计和检测。
图4:图4描述了用于检测来自hepdes19的已提取dna或一步测定法的直接裂解物(一步qpcr测定法)的新cccdna探针的设计和检测。
图5:图5提供通过dna琼脂糖凝胶,来自phbv1.3阳性对照质粒(关于phbv1.3阳性对照质粒,请参考j.virol.2007年9月;81(18):10072-80epub2007年7月3日)和一步qpcr测定法的pcr产物的大小比较。
图6:图6提供使用phbv1.3阳性质粒和hbvdna对照,通过qpcr测定法确认cccdna引物的特异性。
图7:图7提供在采用或不采用plasmidsafeatp-依赖性dna酶(psad,其可以切割非环dna,购自epicentre,madison,wi,usa,目录号:e3101k)预处理,接着qpcr测定法确认cccdna引物对的特异性。
图8:图8提供使用hepg2.2.15细胞、hepde19细胞和hepdes19细胞的上清液,通过qpcr测定法确认cccdna引物的特异性。
图9:图9描述96孔微量滴定板(corning3599)中检测cccdna水平。
图10:图10描述新反向引物的设计及其检测hbvcccdna的能力的比较。
本发明的具体描述
本发明提供通过实时pcr从细胞裂解物定量hbvcccdna的高通量新方法及其在感染的肝细胞中监测cccdna水平和评价对hbv的治疗效果中的用途。
本发明的一个实施方案是(i)一种反向引物,所述反向引物在其3’最末端包含识别乙型肝炎病毒(hbv)基因组(seqidno:1)的核苷酸中至少16个连续核苷酸的序列。
本发明的又一个实施方案是(ii)一种反向引物,所述反向引物在其3’最末端包含识别hbv基因组的核苷酸中从seqidno:1的核苷酸1996至核苷酸2278的至少16个连续核苷酸的序列。
本发明的另一个实施方案是(iii)一种具有在实施方案(i)或(ii)中所给出的意思的反向引物,其中反向引物的长度是16至200个核苷酸、17至35个核苷酸、18至30个核苷酸、21至30个核苷酸、24至30个核苷酸或21至24个核苷酸。
本发明的另一个实施方案是(iv)一种反向引物,所述反向引物优选地在其3’最末端处核苷酸在5’至3’方向包含选自以下的序列:
seqidno:10:r02005-2025(反向互补物)
5’-aaggcttcccgatacagagct-3’;
seqidno:11:r11996-2016(反向互补物)
5’-cgatacagagctgaggcggta-3’;
seqidno:12:r22001-2021(反向互补物)
5’-cttcccgatacagagctgagg-3’;
seqidno:13:r32046-2067(反向互补物)
5’-ctgagtgcagtatggtgaggtg-3’;
seqidno:14:r42097-2118(反向互补物)
5’-cccacccaggtagctagagtca-3’;
seqidno:15:r52129-2152(反向互补物)
5’-taggtctctagacgctggatcttc-3’;
seqidno:16:r62183-2206(反向互补物)
5’-ccacaagagttgcctgaactttag-3’;
seqidno:17:r72188-2210(反向互补物)
5’-gaaaccacaagagttgcctgaac-3’;
seqidno:18:r82210-2233(反向互补物)
5’-tccaaaagtgagacaagaaatgtg-3’;
seqidno:19:r92249-2272(反向互补物)
5’-cactccgaaagacaccaaatactc-3’;
seqidno:20:r102251-2274(反向互补物)
5’-cacactccgaaagacaccaaatac-3’;
seqidno:21:r112257-2278(反向互补物)
5’-aatccacactccgaaagacacc-3’;和
seqidno:24:r4(30bp)2097-2126(反向互补物)
5’-aattaacacccacccaggtagctagagtca-3’。
本发明的另一个实施方案是(v)一个由反向引物和通用正向引物组成的引物对,其中反向引物具有如前文定义的意思,通用正向引物的长度是16至200个核苷酸、17至35个核苷酸、18至30个核苷酸、21至30个核苷酸、24至30个核苷酸或21至24个核苷酸,并且通用正向引物识别hbv基因组的dr2的上游。
本发明的又一个实施方案是(vi)一个由反向引物和通用正向引物组成的引物对,其中反向引物具有如前文定义的意思,通用正向引物的长度是16至200个核苷酸、17至35个核苷酸、18至30个核苷酸、21至30个核苷酸、24至30个核苷酸或21至24个核苷酸,并且通用正向引物与从核苷酸1528至核苷酸1548的核苷酸序列seqidno:5具有至少80%序列同一性。
本发明的又一个实施方案是(vii)一个由反向引物和通用正向引物组成的引物对,其中反向引物具有如前文定义的意思,通用正向引物的长度是16至200个核苷酸、17至35个核苷酸、18至30个核苷酸、21至30个核苷酸、24至30个核苷酸或21至24个核苷酸,并且通用正向引物识别hbv基因组的dr2的上游并且与从核苷酸1528至核苷酸1548的核苷酸序列seqidno:5具有至少80%序列同一性。
本发明的又一个实施方案是(viii)一个由反向引物和通用正向引物组成的引物对,其中反向引物具有如上文定义的意思;通用正向引物的长度是16至200个核苷酸、17至35个核苷酸、18至30个核苷酸、21至30个核苷酸、24至30个核苷酸或21至24个核苷酸,并且通用正向引物在5’至3’方向包含seqidno:5。
本发明的另一个实施方案是(ix)一种用于检测提取的dna或一步qpcr测定法的裂解物的探针,其中探针的长度是16至200个核苷酸、17至35个核苷酸、18至30个核苷酸、21至30个核苷酸、24至30个核苷酸或21至24个核苷酸。特别地,本发明还涉及一种用于检测来自hepdes19的已提取dna或一步qpcr测定法的裂解物的探针,其中探针的长度是16至200个核苷酸、17至35个核苷酸、18至30个核苷酸、21至30个核苷酸、24至30个核苷酸或21至24个核苷酸。
本发明的又一个实施方案是(x)一种用于检测提取的dna或一步qpcr测定法的裂解物的探针,其中探针与包含选自seqidno:6、seqidno:7、seqidno:8和seqidno:9的序列的一部分的序列具有至少80%序列同一性。
本发明的其它实施方案是(xi)探针与seqidno:6、seqidno:7、seqidno:8和seqidno:9具有至少80%序列同一性,并且其他技术特征具有如上文定义的意思。
本发明的其它实施方案是(xii)探针在5’至3’方向具有选自seqidno:6、seqidno:7、seqidno:8和seqidno:9的序列,并且其他技术特征具有如上文定义的意思。
本发明的又一个实施方案是(xiii)p0、p1、p2或p3的探针,其中
p0是5’-染料+seqidno:6+猝灭剂-3’;
p1是5’-染料+seqidno:7+猝灭剂-3’;
p2是5’-染料+seqidno:8+猝灭剂-3’;
p3是5’-染料+seqidno:9+猝灭剂-3’。
本发明的又一个实施方案是(xiv)与包含选自seqidno:6、seqidno:7、seqidno:8和seqidno:9的序列的一部分的序列具有至少80%序列同一性的探针,其中染料是6-fam、joe或tet,而猝灭剂是bhq-1或tamra,或者染料是tamra,而猝灭剂是bhq-2。
本发明的另一个实施方案是(xv)一种包含反向引物和探针的组合,其中反向引物是r0、r1、r2、r3、r4、r5、r6、r7、r8、r9、r10、r11或r4(30bp);并且探针是p0、p1、p2或p3。
本发明的又一个实施方案是(xvi)一种包含反向引物和探针的组合,其中反向引物是r3、r4、r5或r4(30bp)并且探针是tamra+seqidno:6+bhq2或tamra+seqidno:8+bhq2。
本发明的另一个实施方案是(xvii)一种通过pcr检测hbvcccdna的方法,所述方法包括使用hbv基因组(seqidno:1)作为模板,以及使用反向引物和通用正向引物的引物对,或包含反向引物和探针的组合;所述反向引物、通用正向引物和探针具有如上文定义的意思。
本发明的另一个实施方案是(xviii)一种通过pcr检测hbvcccdna的方法,所述方法包括使用hbv基因组(seqidno:1)作为模板,以及使用反向引物和通用正向引物的引物对,或包含反向引物和探针的组合;其中pcr是实时pcr;反向引物、通用正向引物和探针具有如上文定义的意思。
本发明的另一个实施方案是(xix)一种通过pcr检测hbvcccdna的方法,所述方法包括使用hbv基因组(seqidno:1)作为模板,以及使用反向引物和通用正向引物的引物对,或包含反向引物和探针的组合;其中该方法是高通量方法;反向引物、通用正向引物和探针具有如上文定义的意思。
本发明的另一个实施方案是(xx)一种在无dna预先提取及纯化的情况下通过实时pcr的一步cccdna检测方法,其中该方法包括以下操作:
(1)用待测试的化合物处理细胞;
(2)处理后收获细胞;
(3)向收获的细胞添加裂解缓冲液;和
(4)实施具有如上文定义的意思的实时pcr。
本发明的又一个实施方案是(xxi)一种根据实施方案(xx)在无dna预先提取及纯化的情况下,通过实时pcr的一步cccdna检测方法,其中裂解缓冲液选自市售裂解缓冲液如ripa缓冲液:50mmtris-hcl(ph7.5)、150mmnacl、1%tritonx-100、0.5%脱氧胆酸钠、0.1%sds和5mmedta;np-40缓冲液:50mmtris-hcl(ph8.0)、150mmnacl、0.5%np-40替代品和5mmedta;和dsp缓冲液:40mmhepes(ph7.5)、120mmnacl、1%tritonx-100、1mmedta、10mmβ-甘油磷酸盐和50mmnaf;并且其他技术特征具有如上文定义的意思。
本发明的又一个实施方案是(xxii)一种根据实施方案(xx)在无dna预先提取及纯化的情况下,通过实时pcr的一步cccdna检测方法,其中裂解缓冲液来自invitrogen并且目录号是am8723;并且其他技术特征具有如上文定义的意思。
本发明的又一个实施方案是(xxiii)一种根据实施方案(xx)在无dna预先提取及纯化的情况下,通过实时pcr的一步cccdna检测方法,其中将细胞在烘箱中在50℃和75℃之间的温度处理30分钟至2小时,在96孔平板中每孔添加20-100μl裂解缓冲液,并且随后在0℃和25℃之间的温度实施实时pcr,并且其他技术特征具有如上文定义的意思。
本发明的另一个实施方案是(xxiv)一种在评价对hbv的治疗效果中使用的方法,所述方法包括使用具有如上文定义的意思的方法。
本发明的另一个实施方案是(xxv)反向引物,反向引物和通用正向引物的引物对,或包含反向引物和探针的组合用于评价对hbv的治疗效果的用途,其中反向引物、通用正向引物和探针具有如上文定义的意思。
本发明的另一个实施方案是(xxvi)反向引物、反向引物和通用正向引物的引物对,或包含反向引物和探针的组合在制备检测剂中的用途,所述检测剂用于评价对hbv的治疗效果,其中反向引物、通用正向引物和探针具有如上文定义的意思。
本发明的另一个实施方案是(xxvii)用于评价对hbv的治疗效果的试剂盒,所述试剂盒包含反向引物、包含反向引物和通用正向引物的引物对,或者包含含有反向引物和探针的组合,其中反向引物、通用正向引物和探针具有如上文定义的意思。
本发明的另一个实施方案是(xxviii)新引物和探针通过实时pcr检测hbvcccdna的原理:选择性引物和qpcr设计的一般原理如图1中显示。通过在1.1×hbv整合的基因组的至关重要位置精心设计qpcr引物,可能在使用cccdna作为pcrdna来源时产生特定大小的qpcr产物,而当使用整合的基因组hbvdna作为pcr来源时将不产生生产性pcr产物。这种引物对提供了足够的特异性和选择性,从而不需要使用纯化的病毒dna制备物作为qpcr分析的来源。
材料和方法
细胞培养:
将hepg2.2.15细胞在37℃在补充有10%(v/v)胎牛血清(clontech,tet系统批准的fbs,目录编号:631106)和300μg/mlg418(invitrogen,目录编号:10131-027)的dmem/f12培养基(invitrogen,目录编号:10565-018)中培养。
将hepde19细胞和hepdes19细胞在37℃在补充有10%(v/v)胎牛血清、300μg/mlg418和1μg/ml四环素(sigma,目录编号:t0600000)的dmem/f12培养基中培养。为了获得hbv病毒dna,将hepdes19细胞在60mm培养皿中接种于无四环素的补充有10%(v/v)胎牛血清、300μg/mlg418的dmem/f12培养基中。
dna提取方法(改良hirtdna提取法,1998,biotechniques24:760-762):
将细胞在具有或没有50μg/mlrna酶a(sigma,目录号:r6148)的情况下重悬于250μl50mmtris-hclph7.5和10mmedta中。随后将样品与250μl1.2%sds(sigma,目录编号:74255)涡旋混合并且随后静置5分钟。通过添加350μl3mcscl、1m乙酸钾和0.67m乙酸沉淀细胞残片和染色体dna。将混合物转移至1.5ml管,并且随后将该管立即但温和地通过移液器混合并置于冰上15分钟。将样品在14000g(rcf)在4℃离心15分钟后,将上清液加载于柱(qiagen离心柱;qiagengmbh,希尔登,德国,来自qiaampdna微量试剂盒,目录号:51306)上。通过14000g(rcf)在4℃离心,将柱用750μl洗涤缓冲液(80mm乙酸钾,10mmtris,ph7.5,40μmedta和60%乙醇v/v)洗涤。随后通过14000g(rcf)在4℃离心,将dna用50μl水或te缓冲液(10mmtris-hcl,ph8.0,1mmedta)洗脱。
一步qpcr测定法:
将96孔平板中的40,000个细胞/孔在37℃温育6天后,将细胞通过无ca2+和mg2+的磷酸盐缓冲盐水(pbs)洗涤并且通过50μl特定裂解缓冲液(invitrogen,目录号:am8723)裂解。将所产生的样品在烘箱中在75℃温育2小时。保持裂解物处在4℃直至进行实时pcr。为了进行qpcr,取2μl裂解物用于分析。对于不同样品,通过lightcycler480ii仪器(rochediagnositic,inc)以含有750nm每种引物、400nm探针和10μllightcycler480probesmaster(rd04887301001-01,roche)的终体积20μl进行每个反应。pcr循环程序如下进行:在50℃启动2分钟,随后在95℃的温度变性10分钟,随后45个扩增循环:在95℃变性温度10秒,在58℃复性温度持续5秒,在63℃持续10秒,并且在延伸温度72℃持续40秒,并且随后在72℃的温度终末延伸10分钟。在完成pcr循环程序后,获得cp值以确定输入靶核酸的定量。
实施例
提供以下实施例旨在说明,但不限制要求保护的本发明。
本文所用的缩略语如下:
常规组:意指与常规反向引物(seqidno:4)配对的常规正向引物(seqidno:3),和常规探针(5’-tamra+seqidno:2+bhq2-3’),“常规组”在全部图中均缩写成“常规的”。
hbvdna组:意指与hbv-r(seqidno:26)配对的hbv-f(seqidno:25),和hbvdna探针(5’-tamra+seqidno:27+bhq2-3’)
p0:5’-染料+seqidno:6+猝灭剂-3’
p1:5’-染料+seqidno:7+猝灭剂-3’
p2:5’-染料+seqidno:8+猝灭剂-3’
p3:5’-染料+seqidno:9+猝灭剂-3’
pair0:与r0(seqidno:10)配对的cccdna新正向引物(seqidno:5)
pair1:与r1(seqidno:11)配对的cccdna新正向引物(seqidno:5)
pair2:与r2(seqidno:12)配对的cccdna新正向引物(seqidno:5)
pair3:与r3(seqidno:13)配对的cccdna新正向引物(seqidno:5)
pair4:与r4(seqidno:14)配对的cccdna新正向引物(seqidno:5)
实施例1:使用针对已提取病毒基因组dna的常规cccdna引物对和使用一步qpcr测定法的qpcr测定法的比较
细胞材料源来自六天培养后无诱导cccdna(背景,因存在1μg/ml四环素所致)或有诱导(cccdna表达,因四环素未存在)的hepdes19。测试常规组。dna提取方法和一步qpcr测定法遵循上文描述的程序实施。
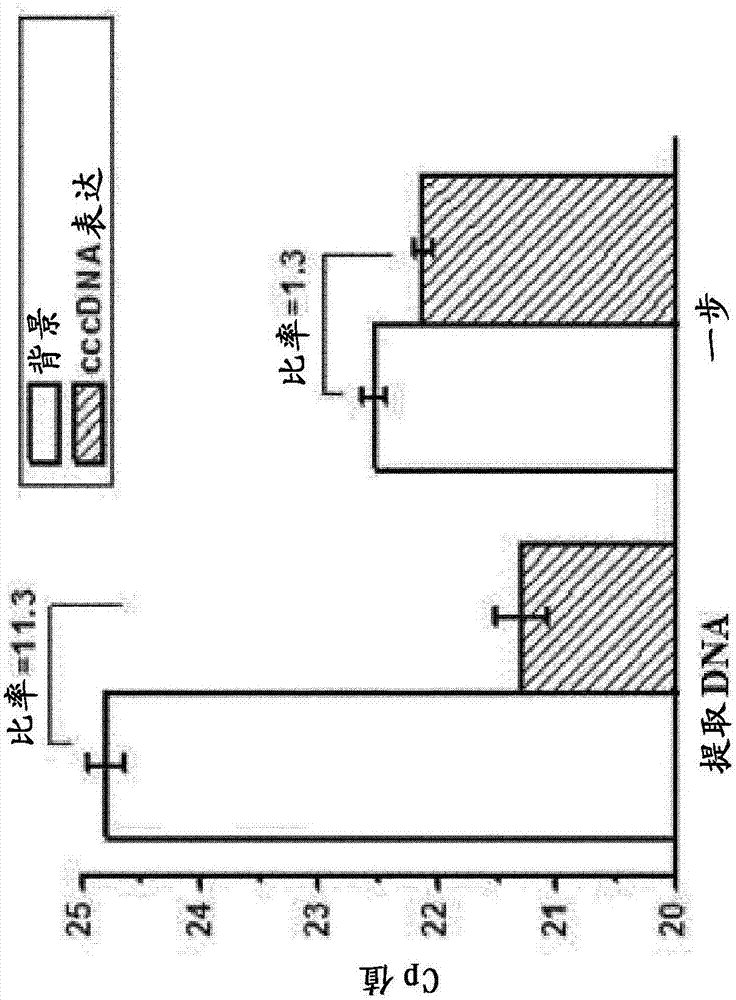
图2中所示的结果描述了来自tet-on(背景)条件的dna和来自tet-off(cccdna表达)条件dna之间的范围。对于提取dna法,常规组在qpcr测定法中显示11.3倍差异。对于一步qpcr测定法,仅存在1.3倍差异。通过比较提取dna法和一步qpcr测定法的结果,常规组不能用来建立一步cccdna定量测定法。
实施例2:用于检测来自hepdes19的已提取dna或无提取的直接裂解物(一步qpcr测定法)的新cccdna引物的试验
细胞材料源来自六天培养后无诱导cccdna(背景,因存在1μg/ml四环素所致)或有诱导(cccdna表达,因四环素未存在)的hepdes19。该实施例中使用以下材料:常规组、pair0-p0、pair1-p0、pair2-p0、pair3-p0和pair4-p0。p0中使用的染料是tamra并且p0中使用的猝灭剂是bhq2。遵循上文描述的程序实施一步qpcr测定法。
图3a中所示的结果显示,hepdes19细胞中来自背景的提取dna和来自cccdna表达的提取dna之间的范围从常规组中的11.3倍改善至pair0-p0、pair1-p0、pair2-p0、pair3-p0和pair4-p0中的50-80倍。这表明,所述的新引物对和探针,即pair0-p0、pair1-p0、pair2-p0、pair3-p0和pair4-p0,具有以低背景检测cccdna的更好灵敏度。图3b中所示的结果表明,对于一步qpcr测定法,背景和cccdna表达之间存在几乎6.8倍差异。值得指出的是,与常规组不同,所述的新cccdna引物对和探针,即pair0-p0、pair1-p0、pair2-p0、pair3-p0和pair4-p0,不能检出hepdes19细胞中的hbv基因组dna并且它可用于一步qpcr测定法。这解释了与所述新引物对和探针相关的改善。
实施例3:用于检测来自hepdes19的已提取dna或无提取的一步qpcr测定法的直接裂解物(一步qpcr测定法)的新cccdna探针的试验。
细胞材料源来自六天培养后无诱导cccdna(背景,因存在1μg/ml四环素所致)或有诱导(cccdna表达,因不存在四环素)的hepdes19。
对于dna提取研究,使用的探针是p0、p1、p2和p3,其中在每种探针中,使用的染料是tamra并且使用的猝灭剂bhq2。用于全部反应的引物是pair4。
对于一步qpcr测定法,使用的探针是p2,并且p2中使用的染料是tamra并且p2中使用的猝灭剂是bhq2。使用的引物分别是pair1和pair4。也使用常规组用于对照。
图4a中所示的结果描述了新探针的设计和它们在提取的病毒dna中检出hbvcccdna的能力的比较。在qpcr测定法中固定pair4,p0和p2显示良好效率并且它们在背景和cccdna表达之间具有几乎90倍差异。对于一步qpcr测定法,图4b中所示的结果显示,pair1-p2和pair4-p2具有超过8倍差异,而对于常规组,仅存在1.3倍差异。从这个结果得出如下结论,所述的新引物和探针对可用于开发高通量测定法。
实施例4:通过dna琼脂糖凝胶比较来自phbv1.3阳性对照质粒和一步qpcr测定法的pcr产物的大小
细胞材料源来自六天培养后有诱导(cccdna表达,无四环素)的hepdes19。
在dna提取法和一步qpcr测定法中使用pair1-p2、pair4-p2和常规组。使用hbvdna组和常规组作为对照。p2中使用的染料是tamra并且p2中使用的猝灭剂是bhq2。
图5的左部分是来自使用phbv1.3作为模板的qpcr的反应产物的1%琼脂糖凝胶电泳结果。图5的右部分给出了使用相同引物和探针记号的一步qpcr测定法的结果。
图5中所示的结果显示,来自phbv1.3阳性对照质粒和来自一步qpcr测定法的pcr产物的大小相同,与来自一步法一致的pcr产物是正确产物。来自pair4-p2的一步qpcr产物比来自常规引物对的pcr产物具有更特异的条带。
实施例5:确认cccdna引物和探针的特异性。
细胞材料源来自六天培养后有诱导(cccdna表达,无四环素)的hepdes19。使用phbv1.3质粒作为阳性对照并且通过dna提取方法提取hbvdna。这项研究中使用hbvdna组、pair1-p2和pair4-p2。对于p2,使用的染料是tamra并且使用的猝灭剂是bhq2。阳性对照phbv1.3质粒用来校正各种dna引物对的水平。在校正后,引物对生成等量的pcr产物(白色柱)。但是,当相同量的引物对施加至提取的hepdes19dna时,检出qpcr产物的不同cp值。如图6中所示,在hepdes19细胞中,使用hbvdna组的实验中检出的hbvdna水平高于使用pair1-p2和pair4-p2的实验中检出的cccdna水平20倍。
实施例6:确认cccdna引物对的特异性
遵循dna提取法的程序,这项研究中使用hbvdna组、pair1-p2和pair4-p2。反应混合物含有6.8μl提取的dna,4upsad、1mmatp和2μl反应缓冲液,以水补足至终体积20μl。在30℃实施消化16小时。细胞材料源来自六天培养后无诱导cccdna(背景,因存在1μg/ml四环素所致)或有诱导(cccdna表达,因不存在四环素)的hepdes19。dna源是从hepdes19细胞提取的dna。psad可以切割非环形hbvdna,包括双链松弛型环状dna(rcdna)。图7中显示结果。使用通常的hbvdna组时,在psad处理或psad未处理的dna样品之间存在2.4倍差异。但是,使用pair1-p2和pair4-p2时,不存在差异,表明用pair1-p2和pair4-p2的qpcr模板是环形dna源(cccdna)。
实施例7:使用hepg2.2.15细胞、hepde19细胞和hepdes19细胞的上清液,通过qpcr测定法确认cccdna引物的特异性。
该实施例中使用pair4-p2。阳性对照质粒phbv1.3用来校正hbvdna引物和cccdna引物的效率。将hepg2.2.15细胞在37℃在补充有10%(v/v)胎牛血清和300μg/mlg418的dmem/f12培养基中培养。将hepde19细胞和hepdes19细胞在37℃在补充有10%(v/v)胎牛血清、300μg/mlg418和1μg/ml四环素的dmem/f12培养基中培养。在hepg2.2.15细胞(稳定细胞系,持续表达hbv病毒)的上清液中未检出cccdna。在hepde19细胞(撤除四环素后六天)和hepdes19细胞(撤除四环素后六天)的上清液中,cccdna仅以背景水平检出,但是在这些细胞系中检出rcdna。在温育细胞6天后,为了进行qpcr,2μl上清液用于分析。对于不同样品,通过使用lightcycler480ii仪器(roche)以含有750nm每种引物、400nm探针和10μllightcycler480probesmaster(rd04887301001-01,roche)的终体积20μl进行每个反应。pcr循环程序如下进行:在50℃启动2分钟,随后使用95℃的温度变性10分钟,随后45个扩增循环:在95℃变性温度10秒,在58℃复性温度持续5秒,在63℃持续10秒,并且在延伸温度72℃持续40秒,并且随后在72℃的温度终末延伸10分钟。图8中显示结果。
实施例8:通过实时pcr检测hbvcccdna的测定法开发
这个实施例中,pair4-p2用于一步qpcr测定法中。接种40,000个细胞/孔。细胞材料源来自六天培养后无诱导cccdna(背景,因存在1μg/ml四环素所致)或有诱导(cccdna表达,因不存在四环素)的hepdes19。从孔1至孔48的样品是cccdna表达并且从孔49-96的样品是背景。
图9中所示的结果描述了优化的条件。带细胞的裂解的最佳温育是约半小时至一小时。96孔微量滴定板上表达cccdna的细胞数是40,000个细胞/孔。与teton(cccdna表达)相比时,cp差别是无tet(背景)后约3.0个循环,并且z因子是约0.53,这适于高通量筛选或高通量筛选以筛选阻断cccdna在感染的活细胞中堆积的化合物。
实施例9:通过一步qpcr测定法比较新反向引物检测hbvcccdna的能力
通过一步qpcr测定法在96孔微量滴定板(corning3599)中检测cccdna的水平。在全部情况下,固定cccdna新正向引物(seqidno:5)和p2,其中p2中使用的染料是tamra并且p2中使用的猝灭剂是bhq2。还检测了多个反向引物,其中反向引物是r5(seqidno:15);r6(seqidno:16);r7(seqidno:17);r8(seqidno:18);r9(seqidno:19);r10(seqidno:20);r11(seqidno:21);或r4(30bp)(seqidno:24)。接种40,000个细胞/孔。细胞材料源来自六天培养后无诱导cccdna(背景,因存在1μg/ml四环素所致)或有诱导(cccdna表达,因不存在四环素)的hepdes19。从孔1至孔48的样品是cccdna表达并且从孔49-96的样品是背景。这个测定法中使用pair4-p2。
图10中显示结果。反向引物r5至r10显示良好效率并且它们在背景和cccdna表达之间具有几乎8倍差异。还检测了r4(30bp)(seqidno:24)。图10中的结果显示与r4(seqidno:14)相比,r4(30bp)(seqidno:24)类似地有效。
可以理解本文所述的实施例和实施方案的目的仅在于说明,并且所述教导会启发本领域技术人员进行多种修改或改变。
序列表信息
seqidno:1:hepdes19基因组的hbvdna插入物:
ttccacaaccttccaccaagctcctgcagatcccagagtgagaggcctgtatttccctgctggtggctccagttcaggaacagtaaaccctgttctgactactgcctctcccttatcgtcaatcttctcgaggattggggaccctgcgctgaacatggagaacatcacatcaggattcctaggaccccttctcgtgttacaggcggggtttttcttgttgacaagaatcctcacaataccgcagagtctagactcgtggtggacttctctcaattttctagggggaactaccgtgtgtcttggccaaaattcgcagtccccaacctccaatcactcaccaacctcttgtcctccaacttgtcctggttatcgctggatgtgtctgcggcgttttatcatcttcctcttcatcctgctgctatgcctcatcttcttgttggttcttctggactatcaaggtatgttgcccgtttgtcctctaattccaggatcctcaacaaccagcacgggaccatgccggacctgcatgactactgctcaaggaacctctatgtatccctcctgttgctgtaccaaaccttcggacggaaattgcacctgtattcccatcccatcatcctgggctttcggaaaattcctatgggagtgggcctcagcccgtttctcctggctcagtttactagtgccatttgttcagtggttcgtagggctttcccccactgtttggctttcagttatatggatgatgtggtattgggggccaagtctgtacagcatcttgagtccctttttaccgctgttaccaattttcttttgtctttgggtatacatttaaaccctaacaaaacaaagagatggggttactctctaaattttatgggttatgtcattggatgttatgggtccttgccacaagaacacatcatacaaaaaatcaaagaatgttttagaaaacttcctattaacaggcctattgattggaaagtatgtcaacgaattgtgggtcttttgggttttgctgccccttttacacaatgtggttatcctgcgttgatgcctttgtatgcatgtattcaatctaagcaggctttcactttctcgccaacttacaaggcctttctgtgtaaacaatacctgaacctttaccccgttgcccggcaacggccaggtctgtgccaagtgtttgctgacgcaacccccactggctggggcttggtcatgggccatcagcgcatgcgtggaaccttttcggctcctctgccgatccatactgcggaactcctagccgcttgttttgctcgcagcaggtctggagcaaacattatcgggactgataactctgttgtcctatcccgcaaatatacatcgtttccatggctgctaggctgtgctgccaactggatcctgcgcgggacgtcctttgtttacgtcccgtcggcgctgaatcctgcggacgacccttctcggggtcgcttgggactctctcgtccccttctccgtctgccgttccgaccgaccacggggcgcacctctctttacgcggactccccgtctgtgccttctcatctgccggaccgtgtgcacttcgcttcacctctgcacgtcgcatggagaccaccgtgaacgcccaccaaatattgcccaaggtcttacataagaggactcttggactctcagcaatgtcaacgaccgaccttgaggcatacttcaaagactgtttgtttaaagactgggaggagttgggggaggagattaggttaaaggtctttgtactaggaggctgtaggcataaattggtctgcgcaccagcaccatgcaactttttcacctctgcctaatcatctcttgttcatgtcctactgttcaagcctccaagctgtgccttgggtggctttggggcatggacatcgacccttataaagaatttggagctactgtggagttactctcgtttttgccttctgacttctttccttcagtacgagatcttctagataccgcctcagctctgtatcgggaagccttagagtctcctgagcattgttcacctcaccatactgcactcaggcaagcaattctttgctggggggaactaatgactctagctacctgggtgggtgttaatttggaagatccagcgtctagagacctagtagtcagttatgtcaacactaatatgggcctaaagttcaggcaactcttgtggtttcacatttcttgtctcacttttggaagagaaacagttatagagtatttggtgtctttcggagtgtggattcgcactcctccagcttatagaccaccaaatgcccctatcctatcaacacttccggagactactgttgttagacgacgaggcaggtcccctagaagaagaactccctcgcctcgcagacgaaggtctcaatcgccgcgtcgcagaagatctcaatctcgggaatctcaatgttagtattccttggactcataaggtggggaactttactgggctttattcttctactgtacctgtctttaatcctcattggaaaacaccatcttttcctaatatacatttacaccaagacattatcaaaaaatgtgaacagtttgtaggcccactcacagttaatgagaaaagaagattgcaattgattatgcctgccaggttttatccaaaggttaccaaatatttaccattggataagggtattaaaccttattatccagaacatctagttaatcattacttccaaactagacactatttacacactctatggaaggcgggtatattatataagagagaaacaacacatagcgcctcattttgtgggtcaccatattcttgggaacaagatctacagcatggggcagaatctttccaccagcaatcctctgggattctttcccgaccaccagttggatccagccttcagagcaaacaccgcaaatccagattgggacttcaatcccaacaaggacacctggccagacgccaacaaggtaggagctggagcattcgggctgggtttcaccccaccgcacggaggccttttggggtggagccctcaggctcagggcatactacaaactttgccagcaaatccgcctcctgcctccaccaatc
gccagtcaggaaggcagcctaccccgctgtctccacctttgagaaacactcatcctcaggccatgcagtggaa
seqidno:2:5'-cgtcgcatggaraccaccgtgaacgcc-3’
seqidno:3:常规正向引物1545-1563”
5'-ctccccgtctgtgccttct-3'
seqidno:4:常规反向引物1883-1900(反向互补物)
5'-gccccaaagccacccaag-3'
seqidno:5:cccdna新正向引物1528-1548
5’-acctctctttacgcggactcc-3’
seqidno:6:1617-1641
5’-accgtgaacgcccaccaaatattgc-3’
seqidno:7:1813-1841
5’-catgcaactttttcacctctgcctaatca-3’
seqidno:8:1793-1814
5’-attggtctgcgcaccagcacca-3’
seqidno:9:1855-1881
5’-tcctactgttcaagcctccaagctgtg-3’
seqidno:10:r02005-2025(反向互补物)
5’-aaggcttcccgatacagagct-3’
seqidno:11:r11996-2016(反向互补物)
5’-cgatacagagctgaggcggta-3’
seqidno:12:r22001-2021(反向互补物)
5’-cttcccgatacagagctgagg-3’
seqidno:13:r32046-2067(反向互补物)
5’-ctgagtgcagtatggtgaggtg-3’
seqidno:14:r42097-2118(反向互补物)
5’-cccacccaggtagctagagtca-3’
seqidno:15:r52129-2152(反向互补物)
5’-taggtctctagacgctggatcttc-3’
seqidno:16:r62183-2206(反向互补物)
5’-ccacaagagttgcctgaactttag-3’
seqidno:17:r72188-2210(反向互补物)
5’-gaaaccacaagagttgcctgaac-3’
seqidno:18:r82210-2233(反向互补物)
5’-tccaaaagtgagacaagaaatgtg-3’
seqidno:19:r92249-2272(反向互补物)
5’-cactccgaaagacaccaaatactc
seqidno:20:r102251-2274(反向互补物)
5’-cacactccgaaagacaccaaatac-3’
seqidno:21:r112257-2278(反向互补物)
5’-aatccacactccgaaagacacc-3’
seqidno:24:r4(30bp)2097-2126(反向互补物)
5’-aattaacacccacccaggtagctagagtca-3’
seqidno:25:hbv-f:5’-aagaaaaaccccgcctgtaa-3’
seqidno:26:hbv-r:5’-cctgttctgactactgcctctcc-3’
seqidno:27:5'-cctgatgtgatgttctccatgttcagc-3'
- 还没有人留言评论。精彩留言会获得点赞!