一种双基因缺失肠炎沙门氏菌、其构建方法及含有该双基因缺失肠炎沙门氏菌的疫苗与流程
本发明属于基因敲除技术领域,具体涉及一种双基因缺失肠炎沙门氏菌、其构建方法及含有该双基因缺失肠炎沙门氏菌的疫苗。
背景技术:
肠炎沙门氏菌是一种人兽共患的重要病原菌,感染沙门氏菌的家禽是沙门氏菌最为常见的储存库,人类通常是由于摄入被污染的蛋类或未完全煮熟的禽肉而感染沙门菌的。世界范围内,因食入肠炎沙门菌污染的禽肉蛋制品引起的食物中毒,各国都有发生且数量不断增加。因此,肠炎沙门氏菌不但严重阻碍了养禽业的发展,而且还威胁着人畜健康,已成为医学、兽医学和公共卫生领域的一个重要问题。目前,主要是使用抗生素类药物来预防和治疗沙门氏菌,但随着抗生素的广泛使用,导致了沙门氏菌的耐药性和耐药谱的不断变化以及多重耐药现象。
疫苗是预防肠炎沙门氏菌感染的另一种有效措施,如今使用的疫苗有灭活苗和减毒活疫苗,灭活苗需要经皮下多次接种,使用不方便,且不能诱导产生消化道局部保护力和细胞免疫,因而无法激发强有力的免疫力以抵御野生致病菌株的感染等不足。而活疫苗因能够模拟自然感染状况,具有诱导细胞、体液和黏膜免疫的特点,因此国际上提倡用活疫苗控制家禽SE感染,活疫苗的研发已经得到世界卫生组织的认可,并作为控制沙门氏菌病完整策略的一部分。活疫苗主要是由基因突变或敲除构建的减毒突变株,目前在鸡广泛使用的减毒活疫苗,虽然都有一定的免疫保护效力,然而在多数实验中,对野生株很难产生较强的定殖抑制效应,还可诱发野生株的全身性传播;此外,也不能有效诱导对其它非宿主适应性血清型沙门菌的交叉保护。因此,目前还没有一种非常有效的减毒活疫苗来控制家禽SE感染。随着人感染肠炎沙门菌的持续流行,急需开发更加有效的疫苗以控制家禽SE细菌的传播。
技术实现要素:
针对现有技术存在的上述不足,本发明解决的技术问题是:针对现有技术中肠炎沙门 氏菌活疫苗对野生株很难产生较强的定殖抑制效应,容易诱发野生株的全身性传播,且也不能有效诱导对其他非宿主适应性血清型沙门菌交叉保护的技术问题,而提供一种在宿主中清除效果好,且免疫效果优良的双基因缺失肠炎沙门氏菌、其构建方法及含有该双基因缺失肠炎沙门氏菌的疫苗。
为了解决上述技术问题,本发明采用如下技术方案:一种双基因缺失肠炎沙门氏菌,为rfaH基因和sopB基因都被敲除的肠炎沙门氏菌。
一种双基因缺失肠炎沙门氏菌的构建方法,采用同源重组的方法对所述肠炎沙门氏菌中的rfaH基因和sopB基因进行敲除。具体包括如下步骤:
1)根据肠炎沙门氏菌的基因组序列,设计出rfaH基因的上游同源臂序列rU的上游引物rfaH-1F和下游引物rfaH-1R,rfaH基因的下游同源臂序列rD的上游引物rfaH-2F和下游引物rfaH-2R,所述rfaH-1F的核苷酸序列如SEQ ID NO.3所示,所述rfaH-1R的核苷酸序列如SEQ ID NO.4所示,所述rfaH-2F的核苷酸序列如SEQ ID NO.5所示,所述rfaH-2R的核苷酸序列如SEQ ID NO.6所示;
2)采用PCR扩增方法,以肠炎沙门菌的基因组DNA为模板,以rfaH-1F和rfaH-1R为引物扩增得到rfaH的上游同源臂rU,以rfaH-2F和rfaH-2R为引物扩增得到rfaH的下游同源臂rD;
3)以步骤2)扩增得到的rU和rD为模板,rfaH-1F和rfaH-2R为引物,进行交叠PCR反应,获得交叠PCR产物rUD;
4)将步骤3)得到的交叠PCR产物rUD与pMD19-T载体进行连接,将连接产物转化感受态大肠杆菌DH5α,采用蓝白筛选和氨苄青霉素抗性筛选挑选阳性克隆,采用LB培养基培养挑选出的阳性克隆,提取质粒,获得重组pMD-ΔrfaH质粒;
5)用限制性内切酶BglⅡ分别酶切自杀质粒pYG4和重组pMD-ΔrfaH质粒,回收自杀质粒pYG4酶切后获得的5797bp片段和重组pMD-ΔrfaH质粒酶切后获得的1885bp片段,将获得的5797bp片段和1885bp片段于金属浴中连接过夜;
6)将步骤5)连接后的产物热激转化大肠杆菌S17-l/λpir感受态细胞,对转化后的细胞进行培养,将培养菌液涂布于卡拉霉素抗性LB平板上,挑取单菌落,增菌后提取质粒,获得重组自杀质粒pYG4-ΔrfaH;
7)在含有萘啶酸的LB培养基中培养受体菌肠炎沙门氏菌,在含有卡拉霉素的LB培养基中培养供体菌含重组自杀质粒pYG4-ΔrfaH的大肠杆菌S17-l/λpir,对所述受体菌和供体菌培养后分别收集菌体并重悬,将重悬后的供体菌和受体菌混合并于无抗生素的LB培养基上培养,将培养得到的菌液涂布于含卡那霉素和萘啶酸的双抗平板上,筛选阳性克隆单整合菌株,将筛选得到的单整合菌株于无抗生素的LB培养基中培养后,用蔗糖平板筛选,得到发生染色体内重组的肠炎沙门氏菌株;以序列如SEQ ID NO.7所示的rfaH-1和序列如SEQ ID NO.8所示的rfaH-2为引物,采用PCR方法对发生染色体内重组的肠炎沙门氏菌株进行筛选鉴定,挑选PCR扩增出631bp的菌株,得到敲除rfaH基因的肠炎沙门氏菌株;
8)根据肠炎沙门氏菌的基因组序列,设计出sopB基因的上游同源臂序列sU的上游引物sopB-1F和下游引物sopB-1R,sopB基因的下游同源臂序列sD的上游引物sopB-2F和下游引物sopB-2R,所述sopB-1F的核苷酸序列如SEQ ID NO.9所示,所述sopB-1R的核苷酸序列如SEQ ID NO.10所示,所述sopB-2F的核苷酸序列如SEQ ID NO.11所示,所述sopB-2R的核苷酸序列如SEQ ID NO.12所示;
9)采用PCR扩增方法,以肠炎沙门菌的基因组DNA为模板,以sopB-1F和sopB-1R为引物扩增得到sopB的上游同源臂sU,以sopB-2F和sopB-2R为引物扩增得到sopB的下游同源臂sD;
10)以步骤9)扩增得到的sU和sD为模板,sopB-1F和sopB-2R为引物进行交叠PCR反应,获得交叠PCR产物sUD;
11)将步骤10)得到的交叠PCR产物sUD与pMD19-T载体进行连接,将连接产物转化感受态大肠杆菌DH5α,采用蓝白筛选和氨苄青霉素抗性筛选挑选阳性克隆,采用LB培养基培养挑选出的阳性克隆,提取质粒,获得重组pMD-ΔsopB质粒;
12)用限制性内切酶BglⅡ分别酶切自杀质粒pYG4和重组pMD-ΔsopB质粒,回 收自杀质粒pYG4酶切后获得的5797bp片段和重组pMD-ΔsopB质粒酶切后获得的1917bp片段,将获得的5797bp片段和1917bp片段于金属浴中连接过夜;
13)将步骤12)连接后的产物热激转化大肠杆菌S17-l/λpir感受态细胞,对转化后的细胞进行培养,将培养菌液涂布于卡拉霉素抗性LB平板上,挑取单菌落,增菌后提取质粒,获得重组自杀质粒pYG4-ΔsopB;
14)在含有萘啶酸的LB培养基中培养受体菌步骤7)得到的敲除rfaH基因的肠炎沙门氏菌株,在含有卡拉霉素的LB培养基中培养供体菌含重组自杀质粒pYG4-ΔsopB的大肠杆菌S17-l/λpir,对所述受体菌和供体菌培养后分别收集菌体并重悬,将重悬后的供体菌和受体菌混合并于无抗生素的LB培养基上培养,将培养得到的菌液涂布于含卡那霉素和萘啶酸的双抗平板上,筛选阳性克隆单整合菌株,将筛选得到的单整合菌株于无抗生素的LB培养基中培养后,用蔗糖平板筛选,得到发生染色体内重组的肠炎沙门氏菌株;以序列如SEQ ID NO.13所示的sopB-1和序列如SEQ ID NO.14所示的sopB-2为引物,采用PCR方法对发生染色体内重组的肠炎沙门氏菌株进行筛选鉴定,挑选PCR扩增出379bp的菌株,得到缺失rfaH基因和sopB基因的双基因缺失肠炎沙门氏菌菌株。
一种肠炎沙门氏菌疫苗,其组分包括采用上述方法制得的双基因缺失肠炎沙门氏菌。
相比现有技术,本发明具有如下优点:
1、本发明通过研究发现由rfaH基因编码的RfaH蛋白是一种抗转录终止蛋白,它可以在细菌转录过程中协助RNA聚合酶越过ρ非依赖型转录终止子结构,实现下游基因的通读,从而消除长转录子的极性。在大肠杆菌中,rfaH蛋白是一种毒力调控蛋白,它影响脂多糖(LPS)层、溶血素和荚膜等的合成;在鼠伤寒沙门氏菌中,这个蛋白也可影响LPS核心区和0抗原的表达及其他毒力基因的表达,rfaH基因缺失鼠伤寒沙门氏菌,侵入上皮细胞和抗原递呈细胞的能力也都得到增强,而抵抗宿主细胞内抗菌肽等物质的抵抗力却减弱,因而在宿主细胞内净增殖数量较野生株大大降低,损失引起系统性疾病的能力,但却能有效保护野毒株的攻击。肠炎沙门氏菌亦存在rfaH基因,敲除肠炎沙门氏菌rfaH基因同样可以实现LPS及其它沙门氏菌毒力基因的减毒效果,同时能够激发有效的免疫保护反应,是构建SE疫苗候选株的理想的敲除目标基因。Sop蛋白是沙门氏菌的另一毒力因子,有肌醇磷酸酯磷酸酶活性,转移到宿主细胞后,能诱导细胞骨架重排,可引起腹泻症状。SopB突变株除能增强体液和细胞免疫外,还能增强粘膜免疫,此外还减少 液体分泌从而减轻腹泻。本发明发明人在上述研究发现的基础上,采用自杀质粒同源重组系统,通过基因同源重组和蔗糖敏感基因的反向筛选技术,首先构建了rfaH基因(1-489)缺失的肠炎沙门氏菌-ΔrfaH缺失株(D315),在此基础上,又利用同样的原理和技术,敲除了与肠炎沙门氏菌毒力相关的另一个基因sopB(1-1686),获得了遗传稳定的肠炎沙门氏菌氏菌ΔrfaH-ΔsopB双基因缺失突变株(D316)。
2、本发明敲除双基因的肠炎沙门氏菌细胞免疫和黏膜免疫得到增强,SE属于肠道病原菌,局部的体液免疫对肠道的保护和菌体的清除效率高于全身免疫,胃肠炎的恢复与肠道局部产生sIgA密切有关,因此能增强粘膜免疫将有助于SE的清除;同时SE是胞内菌,强的细胞免疫对消除SE感染将发挥重要作用;经过试验验证,采用本发明敲除双基因的肠炎沙门氏菌攻毒保护效果效果明显。
3、本发明敲除双基因的肠炎沙门氏菌对宿主和环境的安全性得到提高,因所得突变株在宿主细胞内的抵抗力减弱,致使存活时间和排毒量都大大减少,在体内3~4周内就可以被定殖清除去,安全性能优异。
4、本发明敲除双基因的肠炎沙门氏菌对其他血清型的免疫交叉保护得到提高,原因是该双基因缺失株的核心LPS(O-LPS)链的减短,增强了对其他表面蛋白的免疫反应,而这些蛋白在不同沙门氏菌中是相对保守的,因而能提高交叉免疫保护性,提高对其他血清型沙门氏菌的预防效果。
5、本发明敲除双基因的肠炎沙门氏菌双突变能降低返主的风险,更安全;且本发明敲除双基因的肠炎沙门氏菌rfaH基因突变后突变菌株会变为半粗糙型,而野生菌株为光滑型,为区分SE的自然感染和疫苗免疫株提供方便。
附图说明
图1为rfaH上下游同源臂rU\rD的PCR扩增片段凝胶电泳图。(1,DL-5000Marker DNA Marker(DL-5000);2,ddH2O阴性对照;3,rU片段;4,rD片段)
图2为rU\rD交叠PCR扩增片段rUD凝胶电泳图。(1,DL-5000Marker;2rU D片段)
图3为缺失菌株构建图。
图4为缺失rfaH基因菌株的PCR鉴定图。(1,DL-5000Marker;2缺失菌株;3,亲本菌株)
图5为sopB上下游同源臂sU\sD的PCR扩增片段凝胶电泳图。(1,DL-5000Marker DNA Marker(DL-5000);2,sU片段;3,sD片;4,ddH2O阴性对照段)
图6为sU\sD交叠PCR扩增片段sUD凝胶电泳图。(1,DL-5000Marker;2sUD片段)
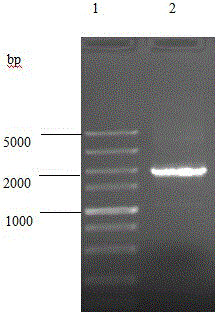
图7为缺失rfaH基因和sopB基因菌株的PCR鉴定图。(1,DL-5000Marker;2缺失菌株;3,亲本菌株)
具体实施方式
下面结合具体实施例和说明书附图对本发明作进一步详细说明。本实施案例在以本发明技术为前提下进行实施,现给出详细的实施方式和具体的操作过程来说明本发明具有创造性,但本发明的保护范围不限于以下的实施例。
下述实施例中使用的肠炎沙门氏菌D1203(由重庆理工大学药学与生物工程学院中心实验室分离、鉴定并保存);下述步骤为敲除rfaH基因和sopB基因的具体步骤,所述rfaH基因的核苷酸序列如SEQ ID NO.1所示,所述sopB基因的核苷酸序列如SEQ ID NO.2所示
实施例1肠炎沙门菌的增殖及基因组的提取
将肠炎沙门菌D1203接种于液体LB培养基中,37℃培养过夜,取1.5mL培养菌液,放置于Eppendorf离心管中,10000g离心2min,弃上清液,加入1.5mLPBS缓冲液重悬沉淀,10000g离心2min,弃上清液,向沉淀中加入240μLTE重悬菌体,并加入15μL溶菌酶,0℃冰浴30min,加入质量浓度10%SDS 15μL,混合均匀后加入蛋白酶K,65℃水浴30min,加入5M NaCl溶液200μL,震荡,加入500μL酚/氯仿/异戊醇(体积比1:24:25)溶液,混合均匀,12000g离心10min,取上清液至离心管中,记录其体积,加入等体积的冷冻无水乙醇,放置于-80℃冰箱10min,12000g离心10min,弃上清液,自然风干,加入30μL ddH2O溶解,放置-20℃冰箱保存,用于PCR反应的模板。
实施例2rfaH基因敲除突变株的构建
2.1交叠PCR
根据GeneBank上已发表的肠炎沙门菌基因组序列,设计出rfaH基因的上游同源臂序列rU的上游引物rfaH-1F和下游引物rfaH-1R,rfaH基因的下游同源臂序列rD的上游引物rfaH-2F和下游rfaH-2R,所述rfaH-1F的核苷酸序列如SEQ ID NO.3所示,所述rfaH-1R的核苷酸序列如SEQ ID NO.4所示,所述rfaH-2F的核苷酸序列如SEQ ID NO.5所示,所 述rfaH-2F的核苷酸序列如SEQ ID NO.6所示。以上述提取的肠炎沙门菌D1203基因组DNA为模板,PCR方法扩增rfaH上、下游同源臂序列rU、rD。rU、rD的50μLPCR扩增体系如表1:
表1PCR扩增体系
PCR反应条件:94℃预变性5min;94℃变性30s,55℃退火40s,72℃延伸1min,共25个循环;最后72℃延伸5min。
将采用上述PCR反应体系和反应条件扩增得到的上、下游同源臂扩增产物rU和rD经1.0%琼脂糖凝胶检测,结果可见得到与预期大小(919bP和933bp)相符的两条带(图1),分别切取两条带,按照Omega公司的凝胶回收试剂盒的使用说明进行回收纯化。
由于rfaH-1R、rfaH-2F相互有部分互补序列,混合纯化后的rU和rD,并以其为模板,rfaH-1F/rfaH-2R为引物进行交叠PCR。交叠PCR 50μLPCR扩增体系同前,PCR反应条件:94℃预变性5min;94℃变性30s,55℃退火45s,72℃延伸2min,共25个循环;最后72℃延伸5min。
2.2中间转移质粒pMD-ΔrfaH的构建
交叠PCR产物用1.0%琼脂糖凝胶检测,可见约2000bP条带rUD(图2),切取该条带,按照Omega公司的胶回收试剂盒的使用说明进行回收纯化,得到交叠PCR产物rUD。将回收目的片段rUD与pMD19-T载体连接。连接体系(10μL):10×连接酶缓冲液1μL,T4DNA连接酶1μL,pMD19-T载体1μL,rUD 5μL,灭菌ddH2O 2μL,16℃金属浴过夜。
将上述得到的连接产物转化感受态大肠杆菌DH5α,挑选(基于蓝白斑和氨苄青霉素 抗性)阳性克隆,并于LB培养基培养阳性克隆,提取质粒,进行酶切、PCR鉴定和序列测定。将测定正确的重组质粒命名为pMD-ΔrfaH。
重组质粒转化大肠杆菌DH5α具体步骤如下:
取大肠杆菌DH5α感受态细胞,置于冰上融化,吸取重组连接产物10μL于已融化的大肠杆菌DH5α感受态细胞中,混匀后冰浴30min,42℃热激90s,冰浴3min,加入1ml LB液体培养基,37℃摇床培养1h,10000g离心2min,弃上清,向沉淀中加入100μL LB重悬菌体,将菌液涂布于100μg/mL氨苄青霉素抗性LB平板,37℃培养过夜。
2.3重组自杀质粒pYG4-ΔrfaH的构建
小量提取自杀质粒pYG4和重组质粒pMD-ΔrfaH,分别用限制性内切酶BglⅡ酶切,酶切体系(50μL):10×H Buffer缓冲液5μL,质粒(pMD-ΔrfaH或PYG4)30μL,BglⅡ2μL,灭菌ddH2O 13μL,37℃水浴酶切2h。酶切产物经1%琼脂糖凝胶电泳,质粒pYG4酶切成1个片段(5797bp),质粒pMD-ΔrfaH酶切成2个片段(2707bp和1885bp)。DNA纯化试剂盒回收纯化pYG4片段和pMD-ΔrfaH的1885bp片段,上述两纯化片段在6℃金属浴连接过夜。金属浴连接体系(10μl):10×连接酶缓冲液1μL,T4DNA连接酶1μL,pYG4片段4μL,ΔrfaH片段3μL,灭菌ddH2O 1μL。
将金属浴连接产物热激转化大肠杆菌S17-l/λpir感受态细胞,涂布卡拉霉素(100μg/ml)LB平板,挑取单菌落,增菌后提取质粒,进行酶切、PCR鉴定和序列测定。将鉴定正确 的重组自杀质粒命名为pYG4-ΔrfaH。
热激转化感受态细胞大肠杆菌S17-l/λpir具体步骤如下:
取大肠杆菌S17-l/λpir感受态细胞,置于冰上融化,吸取重组连接产物20μL于已融化的大肠杆菌S17-l/λpir感受态细胞中,混匀后冰浴30min,42℃热激90s,冰浴3min,加入1ml LB液体培养基,37℃摇床培养1h,10000g离心2min,弃上清,向沉淀中加入100μL LB重悬菌体,将菌液涂布于100μg/mL卡拉霉素抗性LB平板,37℃培养过夜。
2.4接合转移及肠炎沙门菌ΔrfaH单缺失株的筛选
ΔrfaH缺失株的构建路线如图3,通过二步法筛选出基因突变株。具体步骤:在含有奈碇酸(NaL,40μg/ml)的LB培养基中培养受体菌肠炎沙门氏菌D1203(NalR),在含有卡拉霉素(Kan,100μg/mL)的LB培养基中培养含pYG4-ΔrfaH自杀质粒的大肠杆菌S17-l/λpir供体菌,37℃培养过夜后分别收集菌体,LB培养基重悬菌体,各取100μL细菌悬液混合,于无抗生素的LB平板上过夜培养,收集菌体,系列稀释后,取适量菌液涂布于含卡那霉素(Kan,100μg/mL)和萘啶酸(NaL,40μg/ml)的双抗平板上,筛选阳性克隆单整合菌株。
将获得的整合菌株置于无抗生素的LB培养基中培养过夜,培养后于5%蔗糖平板筛选发生染色体内重组的菌株。由于sacB片段的反向选择作用,带有sacB片段的整合株无法在含有5%蔗糖的平板进行上生长,而长出来的菌落都是发生了染色体内重组,从而去掉了载体序列(包括sacB片段)的重组菌株。重组有两种情况,其中50%的可能性产生敲除株,50%的可能性产生野生亲本株。根据GeneBank上已发表的肠炎沙门菌基因组序列,设计一对引物rfaH-1/rfaH-2,rfaH-1序列如SEQ ID NO.7所示,rfaH-2序列如SEQ ID NO.8所示,通过PCR方式筛选鉴定敲除株。PCR反应体系同前,PCR反应条件:94℃预变性5min;94℃变性30s,55℃退火40s,72℃延伸1.5min,共25个循环;最后72℃延伸5min。PCR反应结束,1.0%琼脂糖凝胶检测,如图4,敲除株扩增出一个条带,位于631bp处,野生株扩增出一个条带,位于1120bp左右。将敲除株的PCR扩增产物送至大连宝生工生物工程有限公司进行序列测定。测序结果表明成功敲除掉rfaH基因489bp序列,将该敲除菌株命名为D315。
实施例3肠炎沙门菌ΔrfaHΔsopB双缺失株的构建
3.1交叠PCR
根据GeneBank上已发表的肠炎沙门菌基因组序列,设计出sopB基因的上游同源臂序列sU的上游引物sopB-1F和下游引物sopB-1R,sopB基因的下游同源臂序列sD的上游引物sopB-2F和下游引物sopB-2R,所述sopB-1F的核苷酸序列如SEQ ID NO.9所示,所述sopB-1R的核苷酸序列如SEQ ID NO.10所示,所述sopB-2F的核苷酸序列如SEQ ID NO.11所示,所述sopB-2R的核苷酸序列如SEQ ID NO.12所示。以肠炎沙门菌D1203基因组DNA为模板,PCR方法扩增sopB上、下游同源臂序列sU、sD。sU、sD的50μL PCR扩增体系及PCR反应条件同rU和rD的扩增。
将采用上述方法扩增得到的上、下游同源臂扩增产物sU、sD经1.0%琼脂糖凝胶检测,结果可见得到与预期大小(920bP和964bp)相符条带(图5),分别切取两条带,按照Omega公司的凝胶回收试剂盒的使用说明进行回收纯化。
由于sopB-1R、sopB-2F相互有部分互补序列,混合纯化后的sU和sD,并以其为模板,sopB-1F/sopB-2R为引物进行交叠PCR。交叠PCR 50μL扩增体系同前。PCR反应条件:94℃预变性5min;94℃变性30s,55℃退火45s,72℃延伸2min,共25个循环;最后72℃延伸5min,得到交叠PCR反应产物sUD。
3.2中间转移质粒pMD-ΔsopB的构建
交叠PCR产物经1.0%琼脂糖凝胶电泳检测,可见得到与预期大小(1917bP)相符条带sUD(图6),切取该条带,按照Omega公司的胶回收试剂盒的使用说明进行回收纯化。将回收目的片段sUD与pMD19-T载体连接,连接体系(10μL):10×连接酶缓冲液1μL,T4DNA连接酶1μL,pMD19-T载体1μL,rUD 5μL,灭菌ddH2O 2μL,16℃金属浴过夜。
将连接产物转化感受态大肠杆菌DH5α,挑选(基于蓝白斑和氨苄青霉素抗性)阳性克隆,并于LB培养基培养阳性克隆,提取质粒,进行酶切、PCR鉴定和序列测定。将测定正确的重组质粒命名为pMD-ΔsopB。
3.3重组自杀质粒pYG4-ΔsopB的构建
小量提取自杀质粒pYG4和重组质粒pMD-ΔsopB,分别用限制性内切酶BglⅡ酶切,酶切体系(50μL):10×H Buffer缓冲液5μL,质粒(pMD-ΔrfaH或PYG4)30μL,Bgl 2μL,灭菌ddH2O 13μL,37℃水浴酶切2h。酶切产物经1%琼脂糖凝胶电泳,质粒pYG4酶切成1个片段(5797bp),质粒pMD-ΔrfaH酶切成2个片段(2707bp和1917bp),DNA纯化试剂盒回收纯化pYG4片段和pMD-ΔrfaH的1917bp片段,将回收的上述两纯化片段在6℃金属浴连接过夜。连接体系(10ul):10×连接酶缓冲液1μL,T4DNA连接酶1μL,pYG4片段4μL,ΔsopB片段3μL,灭菌ddH2O 1μL。
将金属浴连接产物热激转化大肠杆菌S17-l/λpir感受态细胞,涂布卡拉霉素(100μg/ml)LB平板,挑取单菌落,增菌后提取质粒,进行酶切、PCR鉴定和序列测定。将鉴定正确的重组自杀质粒命名为pYG4-ΔsopB。
3.4接合转移及肠炎沙门菌ΔrfaHΔsopB双缺失株的筛选
以含pYG4-ΔsopB自杀质粒的大肠杆菌S17-l/λpir为供体菌,前述2.4中所获得的缺失rfaH基因的菌株D315为受体菌进行接合转移。供体菌和受体菌分别接种于含有卡拉霉素(Kan,100μg/mL)和奈碇酸(NaL,40μg/ml)的LB培养基中,37℃培养过夜后收集菌体,并用LB培养基重悬菌体,各取100μL细菌悬液混合,于无抗生素的LB平板上过夜培养。收集菌体系列稀释后,取适量菌液涂布于含卡那霉素(Kan,100μg/mL)和萘啶酸 (NaL,40μg/ml)的双抗平板上,筛选阳性克隆单整合菌株。将整合菌株置于含有5%蔗糖的平板进行筛选,带有sacB片段的整合株无法在该平板上生长,而长出来的菌落都是发生了染色体内重组,从而去掉了载体序列(包括sacB片段)的重组菌株。
重组有两种情况,其中50%的可能性产生敲除株,50%的可能性产生野生亲本株。根据GeneBank上已发表的肠炎沙门菌基因组序列,设计一对引物sopB-1/sopB-2,sopB-1的序列如SEQ ID NO.13所示,sopB-2序列如SEQ ID NO.14所示,通过PCR方式筛选鉴定敲除株。PCR反应体系同前,PCR反应条件:94℃预变性5min;94℃变性30s,55℃退火40s,72℃延伸2.5min,共25个循环;最后72℃延伸5min。PCR反应结束,1.0%琼脂糖凝胶检测,如图7,敲除株扩增出一个条带,位于379bp左右,野生株扩增出一个条带,位于2065bp左右。将敲除株的PCR扩增产物送至大连宝生工生物工程有限公司进行序列测定,测序结果表明又成功敲除掉sopB基因的1686bp序列,将该双基因敲除菌株命名为D316。
实施例4细菌在体内定殖清除
双基因敲除突变菌株D316和野生菌株分别接种LB液体培养基,37℃振荡培养过夜,2%接种新鲜LB培养基,37℃振荡培养至OD600=0.7,将菌液于10000g离心5min,重悬于灭菌PBS,将细菌浓度调节到108CFU/mL。
1日龄海兰白雏鸡随机分成3组,每组20只。其中1组口服1×107CFU/0.1mL突变菌株D316,1组口服1×107CFU/0.1mL野生菌株,1组口服0.1mLPBS作为对照组。分别于免疫后1周、2周、3周、4周随机剖杀各免疫组和对照组鸡各5只,无菌采取肝脏、脾脏和盲肠,称重,无菌PBS研磨后分2份,其中1份适当稀释后涂布沙门氏菌显色培养基平板,37℃培养后计数;另一份做增菌培养后,涂布沙门氏菌显色培养基平板,PCR确定。
细菌在体内定殖清除情况见表2。接种后的第1、2、3、4周,对照组鸡肝脏、脾脏和盲肠均未检出沙门氏菌,突变菌在肝脏、脾脏和盲肠中都最终被完全清除,野生株在在肝脏、脾脏和盲肠中的数量显著高于对照组,第四周时宿主仍不能完全清除。
表2接种D316后各内脏器官的菌株分布(mean±SEM log10cfu/g)
实施例5免疫保护性
40只雌性1日海兰白雏鸡,随机分为2组,每组20只。其中1组在1日龄和5周龄时分别口服1×107CFU/0.1mL突变菌株D316,另1组在1日龄和5周龄时分别口服0.1mLPBS作为对照组。在9周龄时,两组鸡都口服1×109CFU/0.2mL野生菌株进行攻毒。分别于攻毒后1周、2周、随机剖杀各免疫组和对照组鸡各10只,无菌采取肝脏、脾脏和盲肠,称重,无菌PBS研磨后分2份,其中1份适当稀释后涂布沙门氏菌显色培养基平板,37℃培养后计数;另一份做增菌培养后,涂布沙门氏菌显色培养基平板,PCR确定。
保护率及攻毒菌株在体内定殖清除情况见表3。接种后的第1、2周,免疫组内脏器官携带攻毒菌株的鸡只数都低于对照组,说明缺失株免疫后能降低鸡只肠炎沙门菌的带菌率。同时,免疫组在肝脏、脾脏和盲肠的带菌量也低于对照组,说明所构建的缺失株能提供良好的免疫保护效果。
表3攻毒保护率及各内脏器官的菌株分布(mean±SEM log10cfu/g)
实施例6肠炎沙门氏菌疫苗
根据上述实施例验证结果,本发明敲除双基因的缺失肠炎沙门氏菌具有优异的免疫保护效果,且在体内能够定殖清除掉,可以作为疫苗使用,进一步,本实施例提供一种肠炎沙门氏菌口服疫苗,其组分包括本发明敲除rfaH基因和sopB基因的双基因缺失沙门氏菌,所述双基因缺失沙门氏菌在所述口服疫苗中的浓度可以为1×107CFU/0.1mL。
最后说明的是,以上实施例仅用以说明本发明的技术方案而非限制,尽管参照较佳实施例对本发明进行了详细说明,本领域的普通技术人员应当理解,可以对本发明的技术方案进行修改或者等同替换,而不脱离本发明技术方案的宗旨和范围,其均应涵盖在本发明的权利要求范围当中。
<110> 重庆理工大学;
<120> 一种双基因缺失肠炎沙门氏菌、其构建方法及含有该双基因缺失肠炎沙门氏菌的疫苗;
<160> 14
<170> PatentIn version 3.5
<210> 1
<211> 489
<212> DNA
<213> 人工序列
<220>
<223> rfaH基因的核苷酸序列
<400> 1
atgcaatcct ggtatttact gtactgcaaa cgcgggcaac ttcagcgtgc tcaggaacac 60
ctcgaaagac aagcggtaag ttgcctgaca ccgatgatca ccctggaaaa aatggtacgc 120
ggaaaacgta cctccgtcag cgaaccgctc tttcctaatt atctgttcgt tgaatttgat 180
ccggaagtga tacataccac tacaatcaac gccacgcgcg gcgtcagcca ttttgtgcgc 240
tttggcgcgc atcctgcgat cgtgccttcc agcgttattc atcagctttc tatctacaag 300
cccgaaggcg ttgtcgatcc tgaaaccccc tatcccggcg atagcgtcat catcacggaa 360
ggcgcatttg aagggctgaa agcgattttt accgaaccgg atggcgaaac gcgttcgatg 420
ttactgctta atttactcaa taaagaagtg aagcagagcg taaaaaacac cggttttcgc 480
aagatttag 489
<210> 2
<211> 1686
<212> DNA
<213> 人工序列
<220>
<223> sopB基因的核苷酸序列
<400> 2
atgcaaatac agagcttcta tcactcagct tcactaaaaa cccaggaggc ttttaaaagc 60
ctacaaaaaa ccttatacaa cggaatgcag attctctcag gccagggcaa agcgccggct 120
aaagcgcccg acgctcgccc ggaaattatt gtcctgcgag aacctggcgc gacatggggg 180
aattatctac agcatcagaa gacgtctaac cactcgctgc ataacctcta taacttacag 240
cgcgatcttc ttaccgtcgc ggcaaccgtt ctgggtaaac aagacccggt tctaacgtca 300
atggcaaacc aaatggagtt agccaaagtt aaagcggacc ggccagcaac aaaacaagaa 360
gaagccgcgg caaaagcatt gaagaaaaat cttatcgaac ttattgcagc acgcactcag 420
cagcaggatg gcttacctgc aaaagaagct catcgctttg cggcagtagc gtttagagat 480
gctcaggtca agcagcttaa taaccagccc tggcaaacca taaaaaatac actcacgcat 540
aacgggcatc actataccaa cacgcagctc cctgcagcag agatgaaaat cggcgcaaaa 600
gatatctttc ccagtgctta tgagggaaag ggcgtatgca gttgggatac caagaatatt 660
catcacgcca ataatttgtg gatgtccacg gtgagtgtgc atgaggacgg taaagataaa 720
acgctttttt gcgggatacg tcatggcgtg ctttccccct atcatgaaaa agatccgctt 780
ctgcgtcacg tcggcgctga aaacaaagcc aaagaagtat taactgcggc actttttagt 840
aaacctgagt tgcttaacaa agccttagcg ggcgaggcgg taagcctgaa actggtatcc 900
gtcgggttac tcaccgcgtc gaatattttc ggcaaagagg gaacgatggt cgaggaccaa 960
atgcgcgcat ggcaatcgtt gacccagccg ggaaaaatga ttcatttaaa aatccgcaat 1020
aaagatggcg atctacagac ggtaaaaata aaaccggacg tcgccgcatt taatgtgggt 1080
gttaatgagc tggcgctcaa gctcggcttt ggccttaagg catcggatag ctataatgcc 1140
gaggcgctac atcagttatt aggcaatgat ttacgccctg aagccagacc aggtggctgg 1200
gttggcgaat ggctggcgca atacccggat aattatgagg tcgtcaatac attagcgcgc 1260
cagattaagg atatatggaa aaataaccaa catcataaag atggcggcga accctataaa 1320
ctcgcacaac gccttgccat gttagcccat gaaattgacg cggtacccgc ctggaattgt 1380
aaaagcggca aagatcgtac aggaatgatg gattcagaaa tcaagcgaga gcacatttct 1440
ttacatcaga cccatatgtt aagtgcgcct ggtagtcttc cggatagcgg tggacagaaa 1500
attttccaaa aagtattact gaatagcggt aacctggaga ttcagaaaca aaatacgggc 1560
ggggcgggaa acaaagtaat gaaaaattta tcgccagagg tgctcaatct ttcctatcaa 1620
aaacgagttg gggatgaaaa tatttggcag tcagtaaaag gcatttcttc attaatcaca 1680
tcttga 1686
<210> 3
<211> 32
<212> DNA
<213> 人工序列
<220>
<223> rfaH-1F的核苷酸序列
<400> 3
gaagatctgc aaaggcatac tccgacagag ta 32
<210> 4
<211> 57
<212> DNA
<213> 人工序列
<220>
<223> rfaH-1R的核苷酸序列
<400> 4
gttataaatt tggagtgtga aggttattgc gtgaatgact cttatccgct tgttcgg 57
<210> 5
<211> 59
<212> DNA
<213> 人工序列
<220>
<223> rfaH-2F的核苷酸序列
<400> 5
cacgcaataa ccttcacact ccaaatttat aacctatcgt tcagaatacg acctcaaat 59
<210> 6
<211> 32
<212> DNA
<213> 人工序列
<220>
<223> rfaH-2R的核苷酸序列
<400> 6
gaagatctga aattggcgtt ttctgctcac gc 32
<210> 7
<211> 21
<212> DNA
<213> 人工序列
<220>
<223> rfaH-1的核苷酸序列
<400> 7
ttgcacagca ccggcatggc g 21
<210> 8
<211> 22
<212> DNA
<213> 人工序列
<220>
<223> rfaH-2的核苷酸序列
<400> 8
aacgacgcgg gcttgagcta cg 22
<210> 9
<211> 32
<212> DNA
<213> 人工序列
<220>
<223> sopB-1F的核苷酸序列
<400> 9
gaagatctgc agcagtataa gatggagcag ag 32
<210> 10
<211> 59
<212> DNA
<213> 人工序列
<220>
<223> sopB-1R的核苷酸序列
<400> 10
gttataaatt tggagtgtga aggttattgc gtgagcgttt ttaatattcc tgaataggg 59
<210> 11
<211> 59
<212> DNA
<213> 人工序列
<220>
<223> sopB-2F的核苷酸序列
<400> 11
cacgcaataa ccttcacact ccaaatttat aacgtcttga ggtaactata tggaaagtc 59
<210> 12
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> sopB-2R的核苷酸序列
<400> 12
gaagatctgt accggccgca tgcaaatatc 30
<210> 13
<211> 24
<212> DNA
<213> 人工序列
<220>
<223> sopB-1的核苷酸序列
<400> 13
atgctgcaaa gtcaggatgt cgtc 24
<210> 14
<211> 23
<212> DNA
<213> 人工序列
<220>
<223> sopB-2的核苷酸序列
<400> 14
tgtaccgatc tcccccatga tcg 23
- 还没有人留言评论。精彩留言会获得点赞!