一种高分辨率核酸结合/熔解荧光的检测方法与流程
本发明涉及分子生物学技术领域,尤其涉及一种高分辨率核酸结合/熔解荧光的检测方法。
背景技术:
荧光定量PCR(Realtime quantitative PCR,qPCR)是一种基因表达定量技术。qPCR技术的基础是聚合酶链式反应(Polymerase Chain Reaction,PCR),其以单链模板与引物链按照碱基配对原则正确配对为前提。所以,在包含碱基差异的区域,设计一组只有单碱基差异的qPCR引物,利用常见的qPCR扩增仪,进行扩增,进而通过荧光染料或荧光标记的特异性探针,对PCR产物进行标记跟踪,实时在线监控反应过程,结合相应软件可以对产物进行分析,计算待测样品模板碱基差异区域的初始浓度。利用双链核酸在高温下变性的特性,在70-94℃间记录熔解曲线作为检测核酸特异性的参数。核酸的熔解曲线完全取决于核酸序列特征,序列中由于单个碱基的改变造成核酸的解链温度发生改变。互补的双链核酸在经过高温变性后,随着温度的下降分开的两条链会重新缔合,这个过程称为复性。利用核酸复性的特征,则可以判断两条互补的寡核苷酸是否可以发生相互作用。
饱和荧光染料是一种小分子的非序列特异荧光染料,可以和核酸双链的小沟充分结合,在解链中不会发生重排,荧光信号强度可以准确地反映解链难易程度。常用于检测单碱基突变、小片段插入或缺失(Wittwer et al.,2003;Liew et al.,2004)。目前,可以用来检测RNAs序列多态性干扰RNA-RNA分子相互作用的方法为表面等离子共振技术(Surface Plasmon Resonance,SPR),该方法价格比较昂贵,实验操作环境要求严格,必须以相关的大型仪器平台作为支撑,实验运行灵活性差。
技术实现要素:
本发明的目的在于提供一种高分辨率核酸结合/熔解荧光的检测方法。本发明提供的检测方法检测效率高、灵活性好,且配套条件简单,实验成本低。
本发明提供了一种高分辨率核酸结合/熔解荧光检测方法,包括以下步骤:
1)预测RNA-RNA相互作用区域;
所述RNA-RNA包括miRNA及其靶基因或lncRNAs及其靶基因;
所述miRNAs及其靶基因相互作用区域的预测方法包括:利用psRNATarget在线预测工具进行预测,所述预测方法的规则包括:最大期望值:0~1.0;互补区域的长度:18~19bp;未匹配靶位点的最大能量:15~20;靶位点的侧翼长度:上游17bp/下游13bp;导致延伸抑制的中央错配范围:9~11nt;
所述lncRNAs及其靶基因相互作用区域的预测方法包括:利用Blast进行序列互补计算,所述序列互补计算时的参数设置为E value=1e-10,identity=90%;将所述互补计算得到的序列再利用RNAplex进行热力学上的互补计算,参数设置为e=-40;
2)对所述步骤1)得到的RNA-RNA相互作用区域内序列进行多态性分析,筛选得到多态性位点作为靶位点;
所述多态性分析的方法包括:
当分析对象为模式物种时,通过物种重测序数据库进行相互作用区域内的SNPs、InDels位点的发现,所述位点在群体中的频率均大于5%;
当分析对象为非模式物种时,通过克隆相互作用区域cDNA全长进行相互作用区域内的SNPs位点的发现,至少利用20个没有亲缘关系的个体进行序列多态性分析;
3)以所述步骤2)得到的相互作用区域内的含1~5个靶位点的序列为模板,合成相互作用区域内的寡核苷酸片段;
4)将所述步骤3)得到的任意两条相互作用区域内的寡核苷酸片段与饱和荧光染料混合,利用Realtime-PCR进行升温处理,通过相互作用的寡核苷酸片段产生的荧光强度检测寡核苷酸序列的熔解;利用Realtime-PCR进行降温处理,通过相互作用的寡核苷酸片段产生的荧光强度检测寡核苷酸序列的结合;
5)根据所述步骤4)得到的荧光强度绘制荧光随温度的变化曲线,曲线的峰值对应的温度代表寡核苷酸结合或熔解一半时所需要的温度;在降温处理中,所述峰值对应的温度越高,则代表RNA-RNA结合能力越强;在升温处理中,所述峰值的温度越高,则代表RNA-RNA形成的互补双链结构稳定性越强。
优选的是,所述步骤3)寡核苷酸片段的长度为20~180bp。
优选的是,所述步骤4)中Realtime-PCR的反应体系为:每25μl体系包括11.25μl去离子水;0.625μl 10μM的寡核苷酸片段;12.50μl饱和荧光染料。
优选的是,所述饱和荧光染料为LC Green。
优选的是,所述步骤4)Realtime-PCR的反应条件为:
RNA-RNA寡核苷酸序列升温处理体系:95℃,30s;95℃,30s,每个循环降低0.5℃,共40个循环;每个循环记录荧光强度;
RNA-RNA寡核苷酸序列降温处理体系:95℃,30s;自然冷却至室温(25℃);75℃,30s,每个循环提高0.5℃,共40个循环;每个循环记录荧光强度。
本发明提供了一种高分辨率核酸结合/熔解荧光的检测方法。利用Realtime-PCR,通过监测升温/降温过程中相互作用的寡核苷酸片段产生的荧光强度来分析核酸相互作用能力。本发明在Realtime-PCR技术的灵活性与高通量特性的基础上结合高分辨率荧光染料的灵敏性为核酸相互作用检测提供了一种检测效率高、灵活性好,且配套条件简单,实验成本低的方法。试验结果表明,通过观测荧光强度变化曲线,就能得出RNA-RNA寡核苷酸序列结合或熔解的具体情况,本发明提供的检测方法灵敏度高,结果观测简单,为核酸相互作用的研究奠定了基础。
附图说明
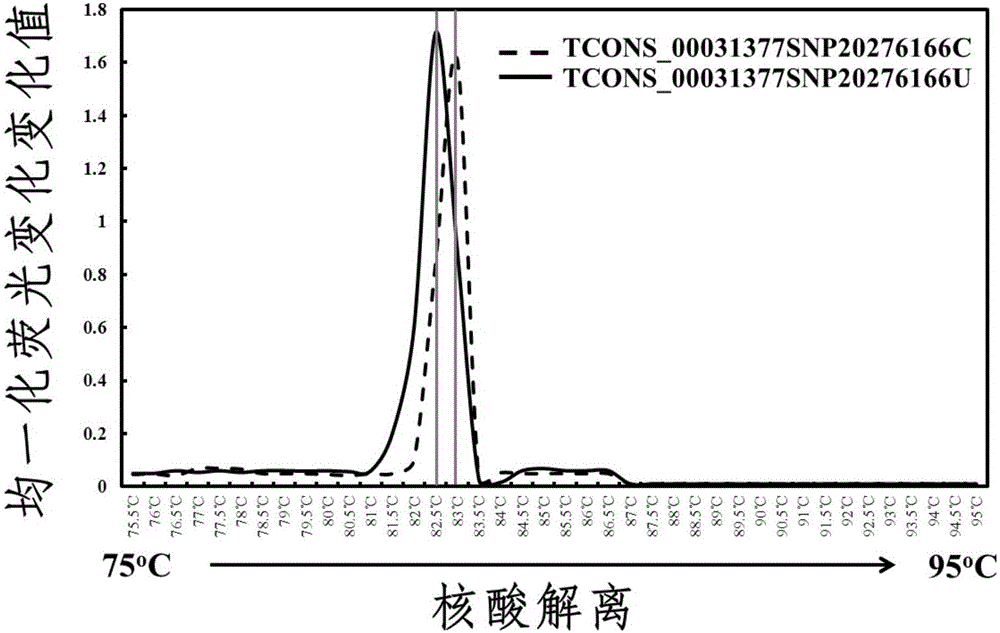
图1是本发明实施例1提供的毛白杨lncRNAs与靶基因结合荧光曲线;
图2是本发明实施例1提供的毛白杨lncRNAs与靶基因熔解荧光曲线。
具体实施方式
本发明提供了一种高分辨率核酸结合/熔解荧光检测方法,包括以下步骤:
1)预测RNA-RNA相互作用区域;
所述RNA-RNA包括miRNA及其靶基因或lncRNAs及其靶基因;
所述miRNAs及其靶基因相互作用区域的预测方法包括:利用psRNATarget在线预测工具进行预测,所述预测方法的规则包括:最大期望值:0~1.0;互补区域的长度:18~19bp;未匹配靶位点的最大能量:15~20;靶位点的侧翼长度:上游17bp/下游13bp;导致延伸抑制的中央错配范围:9~11nt;
所述lncRNAs及其靶基因相互作用区域的预测方法包括:利用Blast进行序列互补计算,所述序列互补计算时的参数设置为E value=1e-10,identity=90%;将所述互补计算得到的序列再利用RNAplex进行热力学上的互补计算,参数设置为e=-40;
2)对所述步骤1)得到的RNA-RNA相互作用区域内序列进行多态性分析,筛选得到多态性位点作为靶位点;
所述多态性分析的方法包括:
当分析对象为模式物种时,通过物种重测序数据库进行相互作用区域内的SNPs、InDels位点的发现,所述位点在群体中的频率均大于5%;
当分析对象为非模式物种时,通过克隆相互作用区域cDNA全长进行相互作用区域内的SNPs位点的发现,至少利用20个没有亲缘关系的个体进行序列多态性分析;
3)以所述步骤2)得到的相互作用区域内的含1~5个靶位点的序列为模板,合成相互作用区域内的寡核苷酸片段;
4)将所述步骤3)得到的任意两条相互作用区域内的寡核苷酸片段与饱和荧光染料混合,利用Realtime-PCR进行升温处理,通过相互作用的寡核苷酸片段产生的荧光强度检测寡核苷酸序列的熔解;利用Realtime-PCR进行降温处理,通过相互作用的寡核苷酸片段产生的荧光强度检测寡核苷酸序列的结合;
5)根据所述步骤4)得到的荧光强度绘制荧光随温度的变化曲线,曲线的峰值对应的温度代表寡核苷酸结合或熔解一半时所需要的温度;在降温处理中,所述峰值对应的温度越高,则代表RNA-RNA结合能力越强;在升温处理中,所述峰值的温度越高,则代表RNA-RNA形成的互补双链结构稳定性越强。
本发明预测RNA-RNA相互作用区域。在本发明中,所述RNA-RNA包括miRNA及其靶基因或lncRNAs及其靶基因;在本发明中,所述miRNAs及其靶基因相互作用区域的预测方法包括:利用psRNATarget在线预测工具进行预测,所述预测方法的规则包括:最大期望值:0~1.0;互补区域的长度:18~19bp;未匹配靶位点的最大能量:15~20;靶位点的侧翼长度:上游17bp/下游13bp;导致延伸抑制的中央错配范围:9~11nt。本发明所述RNA优选为植物RNA。
所述lncRNAs及其靶基因相互作用区域的预测方法包括:利用Blast进行序列互补计算,所述序列互补计算时的参数设置为E value=1e-10,identity=90%;将所述互补计算得到的序列再利用RNAplex进行热力学上的互补计算,参数设置为e=-40,得到lncRNAs及其靶基因相互作用区域。
得到预测的RNA-RNA相互作用区域后,本发明对所述RNA-RNA相互作用区域内序列进行多态性分析,筛选得到多态性位点作为靶位点。在本发明中,所述多态性分析根据是否为模式物种有两种不同的方法,具体的当分析对象为模式物种时,所述多态性分析的方法包括:通过物种重测序数据库进行相互作用区域内的SNPs、InDels位点的发现,所述位点在群体中的频率均大于5%;在本发明中,所述模式物种指的是在Phytozome数据库中存有群体基因组的物种,具体如拟南芥、杨树、桉树等植物。在本发明中,所述物种重测序数据库指的具有储存该物种群体基因组数据的数据库,如Phytozome数据库等。在本发明中,所述SNPs、InDels位点的频率代表该多态性位点在群体中发生的频率,本发明所述位点在群体中的频率优选均大于5%,更优选大于10%。
当分析对象为非模式物种时,所述多态性分析的方法包括:通过克隆相互作用区域cDNA全长进行相互作用区域内的SNPs位点的发现,至少利用20个没有亲缘关系的个体进行序列多态性分析;在本发明中,所述非模式物种指的是在Phytozome数据库中存在群体基因组的物种,所述物种优选为植物,具体优选为柳树、槐树、油松和杨树等。在本发明中,所述没有亲缘关系的个体是指所属同一个物种,但不具有遗传关系的个体。所述序列多态性分析指的是发现用于该区域内的SNPs位点作为后续实验的靶位点。限定个体数下限是为了提高SNPs位点发现的机率。
进行多态分析后,以得到的相互作用区域内的含1~5个靶位点的序列为模板,合成相互作用区域内的寡核苷酸片段。即本发明根据得到的序列多态性分析结果即靶位点,选择miRNA及其靶基因相互作用区域或lncRNAs及其靶基因相互作用区域内存在1~5个靶位点的序列为模板,合成寡核苷酸片段。在本发明中,所述寡核苷酸片段的长度为20~180bp。本发明对所述寡核苷酸片段的合成方法没有特殊的限定,采用本领域技术人员熟知的基因序列合成方法即可,如人工合成等。
得到寡核苷酸片段后,将得到的任意两条相互作用区域内的寡核苷酸片段与饱和荧光染料混合,利用Realtime-PCR进行升温处理,通过相互作用的寡核苷酸片段产生的荧光强度检测寡核苷酸序列的熔解;利用Realtime-PCR进行降温处理,通过相互作用的寡核苷酸片段产生的荧光强度检测寡核苷酸序列的结合。在本发明中,所述饱和荧光染料为LC Green染料,所述饱和荧光染料的作用为均匀的嵌合在核酸双链上,在核酸双链结构改变时提供荧光指示。在本发明中,所述Realtime-PCR的反应体系为:每25μl体系包括11.25μl去离子水;0.625μl 10μM的寡核苷酸片段;12.50μl饱和荧光染料。
在本发明中,所述步骤4)Realtime-PCR的反应条件为:
RNA-RNA寡核苷酸序列升温处理体系:95℃,30s;95℃,30s,每个循环降低0.5℃,共40个循环;每个循环记录荧光强度;
RNA-RNA寡核苷酸序列降温处理体系:95℃,30s;自然冷却至室温(25℃);75℃,30s,每个循环提高0.5℃,共40个循环;每个循环记录荧光强度。
本发明根据所述得到的荧光强度绘制荧光随温度的变化曲线,在绘制曲线前,本发明优选对得到的荧光强度数值进行Z-score值均一化处理,利用均一化后的数据绘制荧光变化曲线;本发明对所述Z-score值均一化的方法没有特殊的限定,采用本领域技术人员熟知的Z-score值均一化处理方法即可。得到的荧光曲线中,曲线的峰值对应的温度代表寡核苷酸结合或熔解一半时所需要的温度;在降温处理中,所述峰值对应的温度越高,则代表RNA-RNA结合能力越强;在升温处理中,所述峰值的温度越高,则代表RNA-RNA形成的互补双链结构稳定性越强。
下面结合实施例,对本发明提供的一种高分辨率核酸结合/熔解荧光检测方法进行详细的描述,但是不能将它们理解为对本申请保护范围的限定。
实施例1
利用高分辨率核酸结合/熔解荧光检测方法对lncRNAs(TCONS_00031377,如SEQUENCE ID NO:1所示)及其靶基因相互作用区域存在的SNPs位点对RNA-RNA相互作用的干扰效应进行检测。
1)预测lncRNAs(TCONS_00031377)靶基因,利用Blast进行序列互补计算,参数设置为E-value=1e-10,identity=90%;利用RNAplex进行热力学上的互补计算,参数设置为e=-40。预测得到其靶基因为Potri.001G204300,在基因组中的位置为Chr01:20271786..20276405,如SEQUENCE ID NO:2所示,位于反义链上。RNA-RNA相互作用区域为Chr01:20276090-20276215,如SEQUENCE ID NO:3所示。
2)利用毛白杨重测序数据对所述相互作用区域内SNPs、InDels位点进行筛选,筛选标准为在群体中的频率需要大于5%,SNP(Chr01:20276166-C/U)位点满足以上要求,该SNP候选区域被筛选为候选区域。
3)根据所述筛选得到的候选区域,确定需要合成的寡核苷酸长度为51bp。合成寡核苷酸片段如下:
SEQUENCE ID NO:4(TCONS_00031377SNP20276166C F:5'-GUUGCGUCGAAAUUUGAAAGATAGAUGCACCCGUUGCUGCCAUUUUAGUUG-3'),
SEQUENCE ID NO:5(TCONS_00031377SNP20276166U F:5'-GUUGUGUCGAAAUUUGAAAGAUAGAUGCACCCGUUGCUGCCAUUUUAGUUG-3'),
SEQUENCE ID NO:6(Potri.001G204300interact region F:5'-CAACUAAAAUGGCAGCAACGGGUGCAUCUAUCUUUCAAAUUUCGACGCAAC-3');
SEQUECE NO:4和SEQUENCE NO:5分别与SEQUENCE NO:6存在相互作用区域。
4)将寡核苷酸片段与荧光染料混合,所述混合体系为:25μl体系包括去离子水,11.25μl;0.625μl 10μM的lncRNA互作区域寡核苷酸序列SEQ ID NO:4或SEQ ID NO:5;0.625μl 10μM的lncRNA互作区域寡核苷酸序列SEQ ID NO:6;LC Green饱和荧光染料,12.50μl。Realtime-PCR检测条件为:①RNA-RNA寡核苷酸序列结合检测体系为:95℃,30s;40个循环95℃,30s(-0.5℃/循环),每个循环记录荧光强度。②RNA-RNA寡核苷酸序列熔解检测体系为:95℃,30s;自然冷却至室温(25℃);40个循环75℃,30s(+0.5℃/循环)。
5)对得到荧光强度数值进行Z-score值均一化处理,利用均一化后的数据绘制荧光变化曲线。曲线的峰值所对应的温度代表寡核苷酸结合或熔解一半时所需要的温度。在结合测试中,TCONS_00031377SNP20276166C荧光变化峰值处温度为81.5℃,TCONS_00031377SNP20276166U荧光变化峰值处温度为81℃,证明TCONS_00031377SNP20276166C与靶基因结合能力较强。在熔解测试中,TCONS_00031377SNP20276166C荧光变化峰值处温度为83℃,TCONS_00031377SNP20276166U荧光变化峰值处温度为82.5℃,证明TCONS_00031377SNP20276166C与靶基因结合后形成的双链结构更为稳定。
毛白杨lncRNAs与靶基因结合荧光曲线如图1所示,虚线代表lncRNAs(TCONS_00031377SNP20276166C)与靶基因结合荧光变化曲线;实线代表lncRNAs(TCONS_00031377SNP20276166U)与靶基因结合荧光变化曲线。虚线代表结合过程中荧光变化峰值处的温度。
毛白杨lncRNAs与靶基因熔解荧光曲线如图2所示,虚线代表lncRNAs(TCONS_00031377SNP20276166C)与靶基因熔解荧光变化曲线;实线代表lncRNAs(TCONS_00031377SNP20276166U)与靶基因熔解荧光变化曲线。虚线代表熔解过程中荧光变化峰值处的温度。
以上所述仅是本发明的优选实施方式,应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以做出若干改进和润饰,这些改进和润饰也应视为本发明的保护范围。
SEQUENCE LISTING
<110> 北京林业大学
<120> 一种高分辨率核酸结合/熔解荧光的检测方法
<130> 2017
<160> 6
<170> PatentIn version 3.3
<210> 1
<211> 212
<212> DNA
<213> 人工序列
<400> 1
tgctcttttc ttcttagtag tgctccgttg caaagaagaa ccgatgaaag aactagcaag 60
agtgttcgag acggatggat tagcattatt aatattagca gtagtaattg gcctcgtgaa 120
gacacgaggt tgcgtcgaaa tttgaaagat agatgcaccc gttgctgcca ttttagttga 180
ttacatatat ctatatctat atctatgatt tt 212
<210> 2
<211> 4620
<212> DNA
<213> 人工序列
<400> 2
ttgcttgctt tttgtctgtt tccatgcact tactttgtgt gtttgtaaag atatttcctt 60
ggaaaattgg tgcttgtggc tgtctctcct tctatatatc tccttatctc atactctgtc 120
tcataactaa taggttcaag ctatttcctc tctttttttt aattctttgc tgtcttgata 180
gttaaagcag aatcaactaa aatggcagca acgggtgcat ctatctttca aatttcgacg 240
caacctcgtg tcttcacgag gccaattact actgctaata ttaataatgc taatccatcc 300
gtctcgaaca ctcttggtga gtgtatcttt aaaaccttgt cactttctta ttttattcat 360
gttagaatcc tagagaattt tgtagtgtga tttctaatat acttgaactc atatcttatt 420
aatgacatgt atgcaaagta attactagga agaaatggca aggtaagaat ggatgaaacc 480
actgacaatt taatttcatc aagaaaacga ttgaaagatg ttctgacaaa caagttgcta 540
ggtacgtagt tccaagagct ctaagttgtt tatcatcact ggagactgct tgttaactgt 600
ttgaatgaga gtatatggtg aataccatga atttcttggg caatcttgta ctttaccatc 660
agacttgaat ggcttgactg gattttggtt tgtaaaaaaa ttggctgttt tcctttttcc 720
ccgattttaa tcttgaatgg ctatggttat ccgtgactgc actgtgtttt catctgtaat 780
attgttttaa ggcgtttttg ctttcaaatt taagtagcaa gttagccaaa cctcgtgcgt 840
tgatcttttt ttactaagct gctccgtctg tgttctatct ccatcacaag tacaagtaca 900
gcaacgagtt ttgaccttaa gcttcaaaaa gcttgacatt tttatgtttc ttattgcagc 960
tagttctttc atcggttctt ctttgcaacg gagcactact aagaagaaaa gagcagttaa 1020
gattggtagg aaagtgatcg ctgcagctgc tgttactgtt gccactcctt caagggagga 1080
agttaaggag tatgaatttc ctcttccttc catttaatgg gattttatct tacttgtgta 1140
aagggctcta tcacaagtcc tctacctcac agttctgttc ataaaggaag tttttcccat 1200
tccattaata cttgcgttct tgtagagcgg gtaaacgaga aacaagcaaa ccagcataaa 1260
aaaggataca gcaaatacta cagtcgtatc atgaggctta attttctatt acaaaagcag 1320
cttttttgtt atcatttcct tttaaaagag cagaatcgta atggtcatct aattctgtaa 1380
tcacttcttt ccagatatgc acgtcctaca tgggccatgt ttgaacttgg aagtgccccc 1440
gtcttctgga aaaccatgaa cggccttcct ccatcttctg taagttagcg ctcacaggat 1500
tcttttctct cttttgaagg tggtgctagt ctttcaatga tgttgaactt gaaatcaatt 1560
gacaggggga aaacctaaag cttttctaca atccagctgc aaacaagctt gttccaaacg 1620
aagaatttgg gattgctttt aatggtaatt tgttattcaa ctcatctcag taagtgtcac 1680
aaaatactcc ttgcaataaa gggtctgtaa aatttccaca agtaataact tgctgccctt 1740
ggaatgtcat agagcatcag attaaagcac tttgaacata acgatatcat gcataatttc 1800
aaataccaga agaatttaca tttcattggt ggtgctctgc tcggcataca aggagtgctc 1860
ggtggttcca ccttcctcaa gatatcaagt attgtttaat caatctccaa ccccttactt 1920
tgcaggaggt tttaatcagc ccatcatgtg tggtggtgag ccaagggcaa tgctgaggaa 1980
agttagaggg aaggctgatc cgccatttta caccatccag atatgcgttc ctaagcatgg 2040
tatggttggt accattttca caattttatt gctttatggg acatttaatt ccttgtaaga 2100
aactctcgta tcctttttgc agctgtgaac ttgatattct cgttcacaaa tggagttgac 2160
tgggatggtc cttacaggct gcagtttcaa gttcacaatg gctggcgaaa caagcccatc 2220
gaattcttca acgaggtact aacacataaa caaagttttg atatatccct tgagaatttc 2280
ctcaagcttt gtaataacta ggtgtcaatc ccaaacgtga caaccttgaa tgataagaaa 2340
agagaaggta tgaactttac tgatacatgc acggactgtt ttattcattt atcacaaagc 2400
atatgcattt tactaattga tgcgaaccca tagaaataat ataaaaagct aatatcataa 2460
aatctccatc caaagattat cttgaattga aaaatccaag ttattgaatg aaaaatccaa 2520
aactttaatt ataaagaatc aagaactgaa tgtataagag aaaatattac aaaaacattt 2580
aacttttttt ataagtatga agaatcaaga atatgcataa cctcaccact cacaaaatat 2640
tattcataaa taataagcaa tttgaaaaaa aaaactaaat acaagctaaa taatcatttt 2700
tatattaggt aaaaacatcc taaataaagc tagaaaaata aaaaaaaaat attaatccaa 2760
ataagaaaaa gaatcttaaa ctaactaaga aaataatgca aatctcgcat agtaacttct 2820
aaatcttatt gtaaggcttt ctcgatatct gatcgttaca acattttaac cctataaaaa 2880
acaaaacctt tagaatcttt aggaaaaatt tcaataaaat tcaatggtca aattaaaagt 2940
tatatttatt agcataaaaa tatgtattgg aaaaaataaa ctcaaaacac cctctaatgt 3000
ccttgaataa taaggattgt tgttgtataa agaatctttc taaaataagg attgttgcct 3060
aattaatctt attggaaagt aagaaatcga tttcacatat aatgaaatcc ttaccttgaa 3120
agaatacaca aatcatgatg aaaataataa ttttccgact ttccttgttt actggcagat 3180
ttcaagtttg acttcaaaca cgtcgttctc tcatatctag atgagttaat aagcctatca 3240
tatttagatg gaaagcttaa gatatatagt ttttatctca actagaatcg caataaaact 3300
ccatttatag ctttaaatct ggttcaaaca ctacatgaag gtctagtcta gtaaatatgc 3360
aatttgttaa cccaaatatc atgaatcaac ttaaataatt tctctttgat ttttttagtt 3420
ttaaacctaa ttagtccact tctaagtact ttaataatgc tccctaattt atttattttg 3480
gaaagttttg gtgcttaaat caattgtaat atctggttgt ttggaatgct acgacatctt 3540
agtgttgttg gtgcaacaaa tccttgggta cttcgcatcc gaaaacatca aatttccagg 3600
tttccttata ccagaatata taagaaagct gggagattat ttgcctaaat ctagaagact 3660
ttgctcattt tgacatggct tataacatgt taaataagtg attcaattct gaccaattta 3720
gccacaaatt tagataaaag gaacaaacaa aagacgtttc cattttgaca gggcctggca 3780
gaagagttga gtaaagaagg tgcgtgcgaa aaagcaattt ttccggatac agatatcatt 3840
gttacgagat gtgctatgat cggcaactta tccattgaag gggtaagagt actcattgtt 3900
tcatctgtct cggtttcctt gcttccattt ttcttgaaat atgacaaatt gacttttaat 3960
ttcacaaatg aaactttctt gcagggtgat cgttgcgatc ttgatctagt ctcaggatgc 4020
atggatccta gctcacattt atataacccc cttgccaatg tggatgatgg aacctgccca 4080
attgagatgg aagattagca gtagcctggc aatacccctt tgatgttgat gtaaatacca 4140
tatacattta ctgtatacct aaggccttcc tcatacataa tggcaaatat actaatgaaa 4200
ttactacttg ctcaagtaaa aatgcttgct ttgagcgagg tctatatgaa acatctccag 4260
ttcccttgat gatgacaaga agtatagtgc acatcctgat agtaatttga aggcaacatt 4320
ccagtttgaa gctatgcttc aaagggaagg tgtggatcga ctttcagttt ggcaaaccta 4380
aaatggaacg agcctctttc agcacagagt ctcaggctgc aagtatgaag taattattct 4440
tacaagacaa agtgaatgga atttttagat tttatttgtg tcagaatagt ggctgtgtca 4500
gctcgcagaa aagcgactcc aaaagaaata ttataaactt catagtcgtt ccgatagaaa 4560
aaaactgacc attttttttt cctctttatt ttgttatatt gcacaggctt agttatttca 4620
<210> 3
<211> 126
<212> DNA
<213> 人工序列
<400> 3
caagagtgtt cgagacggat ggattagcat tattaatatt agcagtagta attggcctcg 60
tgaagacacg aggttgcgtc gaaatttgaa agatagatgc acccgttgct gccattttag 120
ttgatt 126
<210> 4
<211> 51
<212> RNA
<213> 人工序列
<400> 4
guugcgucga aauuugaaag auagaugcac ccguugcugc cauuuuaguu g 51
<210> 5
<211> 51
<212> RNA
<213> 人工序列
<400> 5
guugugucga aauuugaaag auagaugcac ccguugcugc cauuuuaguu g 51
<210> 6
<211> 51
<212> RNA
<213> 人工序列
<400> 6
caacuaaaau ggcagcaacg ggugcaucua ucuuucaaau uucgacgcaa c 51
- 还没有人留言评论。精彩留言会获得点赞!