一种山羊CSN1S1基因插入/缺失的检测方法及其应用与流程
本发明属于生物技术与家畜育种领域,涉及基因插入/缺失(indel)的检测,特别涉及快速、准确检测山羊csn1s1基因nc_030813.1:g.6654-6664位11-bp插入/缺失(indel)多态性的方法及其应用。
背景技术:
动物育种技术主要包括以表型和表型值为基础的常规育种技术和以dna多态为基础的分子育种技术。作为分子育种技术体系的重要组成部分,分子标记辅助选择(marker-assistedselection,mas)育种技术首先检测重要基因的dna多态性,然后分析dna多态与遗传性状之间的相关性,最后再根据与遗传性状显著相关的dna标记进行性状选择。作为现代分子生物学技术发展过程中孕育而生的新技术,mas育种技术可以在dna水平上快速准确地分析个体的遗传组成,通过有性杂交将目的基因转移到需要改良的亲本中,将目标基因型的鉴定与传统育种相结合,从而实现对基因型的直接选择,提高育种目标的定向性,在克服表型鉴定的困难、早期选择、进行无损害的性状评价和选择及提高回交育种效率等方面具有优越性。
在mas育种技术体系中,寻找重要功能基因、筛查重要基因遗传变异位点,并分析重要功能基因遗传变异位点与生长性能的相关性,是标记辅助选择(mas)技术应用的前提和关键。插入/缺失(insertion/deletion,indel)多态性是一种新型分子标记,它是指dna序列上核苷酸片段的插入或缺失而引起dna序列的改变,造成包括人类在内的物种之间染色体基因组的多样性,其片段长度在1-50bp之间。利用indel分析个体基因组可以更好地解释个体的表型差异,因此从dna水平上对小片段核苷酸的差异进行indel检测在动物分子育种的mas体系中具有重要意义。
插入/缺失(indel)是dna或蛋白质序列水平上发生频率仅次于残基替换的进化改变,indel多态性是人类基因组中一种特殊类型的二等位基因遗传标记,表现为基因组中插入或缺失了不同大小的小片段dna。indel大致分成以下5大类:(1)单碱基对的插入/缺失;(2)单一碱基的插入/缺失;(3)重复单元为2~15个碱基的多碱基对插入/缺失;(4)转座子插入/缺失;(5)任意dna序列的插入/缺失多态性。随着比较基因组学的深入研究,indel为理论研究和遗传育种应用研究提供了大量的生物信息,其作为新一代的遗传学鉴定标记,兼具snp的优点。与snp相比,均是源于单突变事件,其突变频率较低,约为10-8,相对比较稳定:在结构上属于二等位基因多态性,等位基因都固定且已知,能够通过很小的扩增子进行扩增(<50bp),提高了扩增被高度降解dna的成功率。作为一种重要的遗传标记,indel的研究最早聚焦在分子生物学及生物医学领域。遍布于整个基因组的indel发生频率仅次于snp位居第二,其中约三分之一位于已知的基因区域内,还有一些位于决定基因功能的关键性区域如启动子区和外显子区。分子生物学家最早将其用于基因表型相关的研究,希望通过将人类性状、疾病症状或是易感性进行联系,从而达到基因诊断和治疗的目的。2005年schnabel等人结合ssr标记和indel标记对控制奶牛产奶量进行了研究,并成功进行了精细定位,同时提出indel和snp的形成机制可能不同,但其进化史是相似的。indel的研究多集中在人类和各种农作物(如水稻和玉米等)的基因组研究中,在畜禽上则集中在对鸡生长性状的研究,在反刍动物上研究和应用甚少。由此,对反刍家畜的功能性基因的indel标记研究亟待开拓和深入。
随着经济发展和人们生活水平的提高,社会对山羊产品的需求不断加强,但由于近年来羊肉、羊奶、羊绒等羊产品严重短缺,所以山羊育种专家期望尽快培育出更早、更好、更快地获得山羊产品的优良品种。在高产、优质和高效的山羊育种目标上,如何提高产羔数一直是关注热点,不仅依靠传统育种手段,还需要依靠分子育种技术。即首先在dna水平上筛查和检测与山羊生长发育性状密切相关的dna标记,然后进行基因多态性的检测,最后是进行基因多态性与产羔性状的关联分析,从而实现mas和早期诊断选择。
哺乳动物泌乳期乳腺合成分泌大量蛋白质,酪蛋白(caseins)占乳蛋白总量80%(mercierandvilotte,1993),包含αs1酪蛋白(csn1s1)、β酪蛋白(csn2)、αs2酪蛋白(csn1s2)以及κ酪蛋白(csn3)(ollieretal.,2008;nilsenetal.,2009)。酪蛋白在乳腺中广泛表达,含有几乎全部必需氨基酸,在肠钙吸收、炎症反应(hatorietal.,2008)和免疫调控(biceretal.,2009)等方面有重要作用,近几年发现,不同基因型的csn1s1基因与酪蛋白含量、脂肪含量、乳凝固时间、凝固硬度以及乳固体含量显著相关(calvoetal.,2013;mastrangeloetal.,2013;mestawetetal.,2013;skeieetal.,2014)。最近又有研究表明其在生长性状上又可影响牛犊的体重(taitetal.,2016)。
目前,对csn1s1基因的研究主要集中在与泌乳及乳成分相关性状的研究上,对csn1s1基因遗传变异与繁殖性状相关性的研究匮乏。
技术实现要素:
本发明的目的在于提供一种山羊csn1s1基因插入/缺失的检测方法及其应用,即利用pcr扩增方法检测山羊csn1s1基因的插入/缺失(indel)多态性,从而加快良种选育速度。
为达到上述目的,本发明采用了以下技术方案:
一种山羊csn1s1基因插入/缺失多态性的检测方法,包括以下步骤:
以待测山羊全基因组dna为模板,以引物对p1为引物,pcr扩增山羊csn1s1基因部分片段(内含子);再对pcr扩增片段进行琼脂糖凝胶电泳;根据琼脂糖凝胶电泳结果鉴定山羊csn1s1基因上的插入/缺失多态性;
所述引物对p1包括用于pcr扩增山羊csn1s1基因上11-bp插入/缺失多态位点的上、下游引物:
上游引物:5’-gctggaagcagttcgtca-3’;
下游引物:5’-gggttgatagccttgtatgtt-3’。
所述山羊csn1s1基因上的插入/缺失多态性是指nc_030813.1:g.6654-6664位11-bp插入/缺失多态性。
所述pcr扩增的反应程序为:95℃预变性5min;94℃变性30s,60.5℃复性30s,72℃延伸25s,35个循环;72℃延伸10min。
所述的琼脂糖凝胶电泳是质量浓度3.0%的琼脂糖凝胶电泳。
所述山羊csn1s1基因上的插入/缺失(indel)多态性的琼脂糖凝胶电泳结果为:插入/插入(ii)基因型表现为170bp的一条带纹;插入/缺失(id)基因型表现为170bp和159bp以及异源双链条带共三条带纹;缺失/缺失(dd)基因型表现为159bp的一条带纹。
一种山羊csn1s1基因插入/缺失多态性的检测试剂盒,包括上述引物对p1。
山羊csn1s1基因nc_030813.1:g.6654-6664位存在的11-bp插入/缺失(indel)多态性位点在山羊分子标记辅助选择育种中应用。
所述插入/缺失(indel)多态性位点的插入/插入(ii)基因型可作为提高山羊母羊第一胎产羔数的dna分子标记。
本发明的有益效果体现在:
本发明根据山羊csn1s1基因nc_030813.1:g.6654-6664位存在11-bp插入/缺失(indel)多态性设计引物,以山羊基因组dna为模板,进行pcr扩增,通过琼脂糖凝胶电泳鉴定,能够简单、快速、低成本、精确地检测山羊csn1s1基因第6654-6664位的插入/缺失多态性。
本发明对山羊csn1s1基因第6654-6664位插入/缺失多态性位点进行基因型和基因频率分析,对上述插入/缺失多态性位点与山羊相关繁殖性状(第一胎产羔数)进行关联分析,结果表明该位点能够作为山羊产羔数(p<0.01)的分子标记,有利于快速建立高产羔数性状的优良山羊种群,加快良种选育速度。
附图说明
图1为引物对p1扩增山羊csn1s1基因产物的3%琼脂糖凝胶电泳结果;m表示marker,a表示异源双链条带。
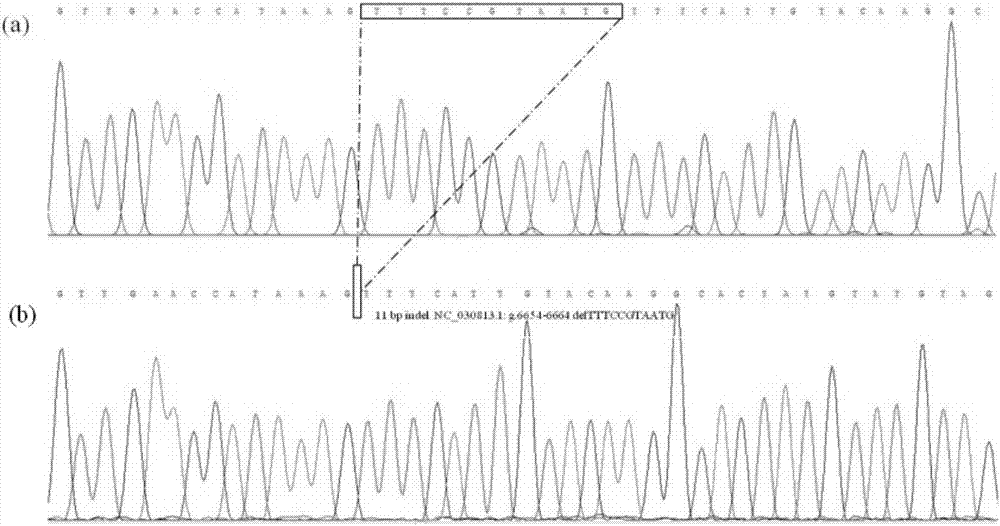
图2为山羊csn1s1基因pcr扩增产物测序图;(a)ii基因型,(b)dd基因型,方框标出的部分为11-bp缺失序列:nc_030813.1:g.6654-6664deltttccgtaatg。
图3为山羊csn1s1基因11-bpindel序列分析图;图中上方序列为插入型,下方序列为缺失型,参考序列为ncbi网站上公布的山羊csn1s1基因序列nc_030813.1。
图4indel基因型(genotypes)与产羔数(littersize)关联分析图。
具体实施方式
下面结合附图和实施例对本发明做详细说明,所述是对本发明的解释而不是限定。
本发明利用pcr方法对山羊csn1s1基因第6654-6664位点突变可能产生的插入/缺失多态性进行检测,并将其与山羊相关繁殖性状(第一胎产羔数)进行关联分析,验证其是否可以作为山羊分子育种中辅助选择的分子标记。
1.实验药品与试剂
1.1生化试剂与生物学试剂:①taqdna聚合酶(购自fermantas即mbi公司);②蛋白酶k(购自华美生物工程公司);③markeri(购自天根生化科技(北京)有限公司)。
1.2普通试剂:普通试剂从华美生物工程公司购买,为进口分装产品:柠檬酸、柠檬酸钠、葡萄糖、tris、edta、nacl、naoh、kcl、na2hpo4、kh2po4、tris饱和酚、氯仿、异戊醇、无水乙醇、醋酸钠、十二烷基磺酸钠(sds)、溴化乙锭(eb)、溴酚蓝、二甲基苯氰ff、乙酸、蔗糖、硼酸、琼脂糖等。
1.3溶液与缓冲液:所有溶液与缓冲液均采用去离子超纯水配制。高压灭菌条件为15bf/in(1.034×105pa),25min。试剂配制方法均参考sambrook等编著的《分子克隆实验指南》。
1)提取组织样dna所用溶液:
除了基因组dna提取时的公用溶液外,还配制以下溶液:①2mol/lnacl:11.688g溶于水,定容至100ml,高压灭菌。②组织dna提取液(100ml):lmol/ltris-cl(ph8.0)lml,0.5mol/ledta(ph8.0)20ml,2mol/lnacl5ml,定容至100ml。
2)琼脂糖电泳分析所用溶液
①0.5×tbe缓冲液:取10×tbe50ml定容至1000ml。②上样缓冲液:0.25%溴酚蓝,0.25%二甲基苯氰ff,以及40.0%(w/v)蔗糖水溶液。
2.设计山羊csn1s1基因indel引物
在ncbi上检索山羊csn1s1基因的序列(山羊csn1s1基因参考序列nc_030813.1),并利用primer5.0设计能够扩增包含山羊csn1s1基因第6654-6664位区域indel位点的pcr引物对p1,其引物序列如下,参见图2、图3:
上游引物:5’-gctggaagcagttcgtca-3’(18bp,seq.id.no.1);
下游引物:5’-gggttgatagccttgtatgtt-3’(21bp,seq.id.no.2)。
上述引物对p1对山羊基因组扩增,能够扩增包含山羊csn1s1基因(nc_030813.1:g.6654-6664序列)的片段。理论上,当6654与6664位之间的tttccgtaatg(seq.id.no.3)缺失时,pcr产物经琼脂糖凝胶电泳检测之后是159bp大小的带纹;6654与6664位之间的tttccgtaatg存在时,pcr产物经琼脂糖凝胶电泳检测之后是170bp大小的一条带纹。6654与6664位之间的tttccgtaatg同时出现插入和缺失时,pcr产物经琼脂糖凝胶电泳检测之后是159bp+170bp两条带纹以及一条异源双链条带。为此,根据理论分析结果,插入/插入(ii)基因型表现为170bp一条带纹;插入/缺失(id)基因型表现为159bp+170bp及异源双链三条带纹;缺失/缺失(dd)基因型表现为159bp一条带纹。
3.pcr扩增待测山羊csn1s1基因片段
3.1山羊耳组织样品的采集
实验所用的动物为陕北白绒山羊共计1835个样本,采自陕西省榆林市狄青塬陕北白绒山羊原种场、佳县方塌镇瑞兴种羊场、神木县聚科农牧发展有限公司、定边县香草园农牧业科技有限公司和榆林学院羊场;横山县横山镇、赵石畔镇、塔湾镇养殖场;榆林市榆阳区芹河镇、金鸡滩镇、麻黄梁镇养殖场。每个个体都具有完整的第一胎产羔数记录。采用随机采样方式采取个体耳组织样品,70%乙醇保存,冰盒低温带回实验室后置于-80℃冻存。
3.2组织样品基因组dna的提取与分离
1)取约10mg耳组织样,放于1.5ml的离心管中,用小剪刀尽量剪碎。
2)加入600μl组织dna提取液,10%sds至终浓度为1%,蛋白酶k至终浓度为100μg/ml,55.0℃消化过夜,最好保证组织样较均匀地分布在组织dna提取液中。
3)将溶液冷却至室温,加入等体积的tris饱和酚,盖紧管盖,缓慢地来回颠倒离心管,至少持续10min以上,12000r/min离心15min。
4)取上清液,加入等体积的tris饱和酚:氯仿(1:1,v/v),盖紧管盖,缓慢地来回颠倒离心管,至少持续10min以上,12000r/min离心15min。
5)取上清液,加入等体积的氯仿:异戊醇(24:1,v/v),盖紧管盖,缓慢地来回颠倒离心管,至少持续10min以上,12000r/min离心15min。
6)取上清液,加入2倍体积的冰冷无水乙醇和1/10体积的3mol/l乙酸钠,盖紧管盖,缓慢地来回颠倒离心管,直至液体清亮,出现白色絮状dna。
7)挑出dna,放进一个1.5ml的离心管中,加入500μl70%乙醇,盖紧管盖,缓慢地来回颠倒离心管,然后12000r/min离心3~5min,小心倒掉乙醇,将管倒置于吸水纸上。
8)再一次向离心管中加入500μl70%乙醇,盖紧管盖,缓慢地来回颠倒离心管,然后12000r/min离心3~5min,小心倒掉乙醇,将管倒置于吸水纸上。
9)待干燥后,加入60μl灭菌超纯水,为使其完全溶解,4℃保存过夜,待检测。
3.3琼脂糖凝胶电泳检测dna
1)将凝胶电泳槽洗干净,用胶带纸将两端封住,插上梳子。
2)称取1.2g的琼脂糖,转入三角瓶中,加入0.5×tbe40ml使其悬浮,微波炉中火加热,待沸腾2次拿出,待其冷却至不烫手时加入终浓度为0.5μg/ml的eb。
3)混匀后(约60℃),立即将琼脂糖溶液到入槽内。如出现气泡,立即用移液器移出。
4)完全冷却凝固(约25~40min)后,拔掉梳子,去掉两端胶带纸。
5)向电泳槽中加入1×tbe缓冲液,使液面高出胶面2~5mm。
6)取基因组dna样品2~4μl,加2μl上样缓冲液后混匀,统一上样(注意枪头的顺序应前后对应),并将dnamarker加在一边。
7)80v电压电泳2h。
8)在紫外分析仪上观察,如果有rna则需要纯化,如果有明显降解,需重新提取相应样品的dna。
3.4dna的纯化
1)500μl的dna溶液中加入10%sds使其终浓度为0.1%,加入蛋白酶k至终浓度达到100μg/ml。
2)55℃保温10h左右。
3)苯酚:氯仿:异戊醇(25:24:1,v/v/v)和等体积氯仿分别抽提一次。
4)12000r/min离心5min分相,吸取上层水相至另一离心管中。
5)加入1/10体积3mol/l醋酸钠和2倍体积冰冷无水乙醇沉淀dna。
6)倒掉液体,70%乙醇洗涤后凉干,加入60μl灭菌超纯水溶解,4℃待检测。
3.5分光光度法检测基因组dna
用紫外光光度计测定dna样品在260nm、280nm处的od值。计算dna含量和od260/od280的比值。如od260/od280比值小于1.6,说明样品中含有较多的蛋白质或酚,则应进行纯化;若比值大于1.8,则应该考虑去除rna。
dna浓度(ng/μl)=50×od260值×稀释倍数
dna检测完毕后,取出一定的量稀释至50ng/μl(模板dna),存于-20℃备用,其余的存放于-80℃。
3.6pcr扩增
pcr反应体系采用混合加样法,即根据每一个反应体系所需的各种组分的数量和1次反应所需的pcr反应的个数,算出各种反应组分的总量,加入到1个1.5ml离心管中,充分混匀后瞬时离心,再分装到每个0.2mlpcr管中,然后加入模板dna,再瞬时离心后进行pcr扩增;pcr反应体系包括2×taqpcrsupermix(包括taqdna聚合酶、dntps和反应缓冲液,浓度为2×)12.5μl;上游引物1.0μl;下游引物1.0μl(上、下游引物浓度为10pmol/μl);基因组dna(浓度为50ng/μl山羊基因组dna)1.0μl;去离子水9.5μl;共25μl体积的pcr扩增体系。
3.7pcr反应的程序
pcr扩增反应程序为:
1)95℃预变性5min;
2)94℃变性30s,60.5℃复性30s,72℃延伸25s,共35个循环;
3)之后,72℃延伸10min;10℃保存扩增产物。
4.扩增pcr产物后琼脂糖凝胶电泳检测分析
琼脂糖凝胶电泳检测分3步:
1)制作3.0%的琼脂糖凝胶,点样后120v电压电泳1.0-1.2h,电泳结束后eb染色;
2)待分子量不同的dna片段分离清晰时,在bio-radgeldoc2000凝胶成像系统成像;
3)根据琼脂糖凝胶电泳结果分析indel多态性;
用bio-radgeldoc2000凝胶成像系统照相分析,判断indel的多态性:
参见图1,山羊基因组的csn1s1基因的第6654-6664位点的indel的多态性的琼脂糖凝胶电泳结果为ii基因型表现为170bp一条带纹;id基因型表现为159bp+170bp及异源双链三条带纹;dd基因型表现为159bp一条带纹。
5.山羊csn1s1基因indel位点的频率统计分析
1)基因和基因型频率
基因型频率是指一个群体中某一性状的某种基因型个体数占总个体数的比率。pnn=nnn/n,其中pnn代表某一位点的nn基因型频率;nnn表示群体中具有nn基因型的个体数;n为检测群体的总数量。
基因频率是指一个群体中某一基因数对其等位基因总数的相对比率。计算的公式可以写成:pn=(2nnn+nna1+nna2+nna3+nna4+……+nnam)/2n
式中,pn表示等位基因n频率,nnn表示群体中具有nn基因型的个体数量,nnai表示群体中具有nai基因型个体数量,a1-am为等位基因n的m个互不相同的复等位基因。
山羊csn1s1基因插入/缺失多态性位点中的等位基因型频率及等位基因频率如表1所示。
表1.山羊csn1s1基因第6654-6664位indel基因频率分布表
6.山羊csn1s1基因indel位点基因效应的关联分析
基因型数据:pcr扩增后琼脂糖凝胶电泳识别的基因型;
生产数据:陕北白绒山羊初产羔数。
关联分析模型:利用spss(18.0)软件来分析品种,不同因素与生长性状相关性。首先要对所得数据描述性的统计分析,来确定是不是存在离群值。然后根据数据的特性,利用方差分析、多元线性模型或者t分析进而来分析基因型的效应。在数据处理的过程中,考虑到个体的效应,基因之间的互作以及基因型的效应,采用固定的模型来进行相关分析。此外,根据实际条件来进行取舍,完整模型如下所示:y=μ+g+e,其中,y:个体表型记录;u:总体均值;g:标记基因型效应;e:随机误差。
结果表明:第一胎产单羔和产双羔的山羊个体csn1s1基因不同基因型频率分布之间存在显著差异(χ2=65.042,p<0.001),表明csn1s1基因不同基因型对山羊第一胎产羔数性状有显著差异;第一胎产单羔和产双羔的山羊个体csn1s1基因不同等位基因频率分布之间存在显著差异(χ2=74.654,p<0.001),表明csn1s1基因不同等位基因对山羊第一胎产羔数性状有显著差异。
由表2和图4可以看出,在对1835只陕北白绒山羊的产羔性状研究中,cs1ns1基因的插入/缺失多态性对其产羔数有极显著影响(p<0.01),ii基因型个体性状优于id和dd基因型个体;id基因型个体性状优于dd基因型个体。结论:ii基因型可以作为山羊繁殖性状(产羔数)的遗传标记。
表2.山羊csn1s1基因11bpindel不同基因型与第一胎产羔数之间的相关性分析
总之,本发明建立了一种山羊csn1s1基因插入/缺失多态性的检测方法,为山羊繁殖性状的标记辅助选择(mas)应用提供理论和实践支撑。
<110>西北农林科技大学
<120>一种山羊csn1s1基因插入/缺失的检测方法及其应用
<160>3
<210>1
<211>18
<212>dna
<213>人工合成
<400>1
gctggaagcagttcgtca18
<210>2
<211>21
<212>dna
<213>人工合成
<400>2
gggttgatagccttgtatgtt21
<210>3
<211>11
<212>dna
<213>capraaegagrushircus
<400>3
tttccgtaatg11
- 还没有人留言评论。精彩留言会获得点赞!