烟草生长素转运蛋白NtPIN4及其应用的制作方法
本发明属于植物基因工程技术领域,具体涉及一个烟草生长素转运蛋白ntpin4及其应用的专利申请。
背景技术:
生长素(auxin,iaa)是植物最重要的激素之一,也是最早发现能够调控植物分枝的激素之一,在调节植物生长发育过程,例如:在细胞分裂、细胞伸长、维管分化、胚形成、根形态构建等众多的生物学过程中,发挥着重要的作用。
植物生长过程中,分枝发育具有顶端优势的现象,即顶芽能够抑制侧芽的发生,去除顶芽(打顶)则会促进侧芽的产生。一直以来,人们认为这种抑制侧芽发育的原因就是生长素。其主要原因是:研究发现,去除顶芽后,植株茎内的iaa含量明显下降,侧芽发生,此时外加iaa并不能完全抑制侧芽的发生;此外,向植物施加生长素极性运输抑制剂降低茎内的生长素含量,也并不能促进侧芽的发生。由此推测,除生长素外还有其他因素参与顶端优势的建立。
研究认为,生长素向茎基部的运输,同时影响了第二信使向上运输进入侧芽。细胞分裂素被认为是生长素第二信使的首选。主要原因是:细胞分裂素对腋芽生长有直接的促进作用,生长素可能是通过阻止细胞分裂素在茎中以及在根中的合成和运输来发挥其抑制作用。另有研究发现,独脚金内酯与生长素在调控分枝发育、构建分枝发育调控方面具有重要的互作关系,独脚金内酯是新发现的能够抑制植物分枝的激素。
就生长素的合成与转运而言,生长素主要在植物地上部分的叶原基、营养芽、嫩叶中合成,然后运输到特定的作用部位。调节生长素极性运输的关键蛋白是一种pin-formed(pin)蛋白,属于植物所特有的一类跨膜蛋白,在细胞膜上调节生长素的定向运输。植物中的第一个pin蛋白基因是在拟南芥中克隆的,研究表明,拟南芥pin-formed1突变体具有异常的花序,将atpin1基因突变后,生长素运输蛋白的缺失导致由茎向根中的生长素运输能力降低。有研究还发现,在新形成的叶原基中,pin1将生长素极性向下运输到表皮下层细胞。
随着各物种基因组测序工作的完成,已在水稻、拟南芥,高粱、玉米和番茄中对pin基因进行了全基因组范围的鉴定和分析。烟草作为一种重要的科研模式作物,也是重要的经济作物和农作物,烟草的分枝发育与烟叶产量及其内在品质具有十分密切的关系,但是现有技术中,尚未见到关于烟草生长素运输蛋白与其分枝发育方面的详细报道。因而,对于烟草中生长素运输蛋白的详细研究,具有十分重要的研究理论意义和实用价值。
技术实现要素:
本申请目的在于提供一个烟草生长素转运蛋白编码基因ntpin4基因,及其所编码烟草生长素转运蛋白ntpin4蛋白,该蛋白与烟草中生长素转运相关,在烟草生长发育、分枝发育中具有十分重要的应用价值。
本申请所采取的技术方案详述如下。
一个烟草生长素转运蛋白编码基因ntpin4基因,该基因的碱基序列如seqidno.1所示。
所述烟草生长素转运蛋白编码基因ntpin4基因所编码烟草生长素转运蛋白ntpin4蛋白,其氨基酸序列如seqidno.2所示。
所述烟草生长素转运蛋白ntpin4蛋白在烟草中的应用,能够转运烟草生长素,具有抑制侧枝及腋芽生长的作用;将ntpin4蛋白的编码基因ntpin4基因沉默后,基因沉默植株中,烟草侧枝明显增多,腋芽的生长数量明显增加;通过对烟草生长素转运蛋白ntpin4蛋白的调控,对于烟草株型的调控和提高烟叶产量有着重要的应用意义。
用于敲除(沉默)烟草中烟草生长素转运蛋白ntpin4基因的重组crispr/cas9表达载体,通过如下步骤构建获得:
设计敲除引物序列pin4-k-f和pin4-k-r如下:
pin4-k-f:5’-gattgcatatggttctgtccgatgg-3’,
pin4-k-r:5’-aaacccatcggacagaaccatatgc-3’;
利用上述引物序列,获得靶位点的dna双链(退火);
将所获得靶位点的dna双链(退火产物)与bsaⅰ酶切后的crispr/cas9载体进行连接,并转化、筛选、鉴定,获得用于敲除烟草中烟草生长素转运蛋白ntpin4基因的重组crispr/cas9表达载体。
所述用于敲除(沉默)烟草中烟草生长素转运蛋白ntpin4基因的重组crispr/cas9表达载体在烟草中应用,将该载体转化烟草后,能够敲除烟草中烟草生长素转运蛋白ntpin4基因,使转化植株中烟草生长素转运蛋白ntpin4蛋白表达量明显降低,甚至不表达。
利用所述crispr/cas9表达载体构建转基因植株的方法,利用农杆菌介导的基因转化方法,将crispr/cas9表达载体转化农杆菌,筛选获得转化用工程菌,结合组培方式、利用工程菌对植物组培组织进行浸染,并经组培、鉴定获得ntpin4基因缺失的植物新品种。
通过设计特异性引物,发明人克隆获得了对烟草生长素转运蛋白ntpin4蛋白的编码基因ntpin4基因,实时定量pcr分析表明,正常烟草植株中,ntpin4基因在茎和腋芽的中表达量较高。通过开展不同激素处理条件下ntpin4基因表达分析,发明人对烟草生长素转运蛋白ntpin4蛋白在烟草生长中的作用有了初步了解。为进一步研究ntpin4基因(ntpin4蛋白)在烟草生长中的作用,发明人构建了crispr/cas9载体,将该载体转化烟草植株后,构建获得了ntpin4基因敲除的突变株系。表型观察结果表明,ntpin4基因敲除的突变株系,其分枝明显增多,腋芽增长活跃。因此,利用ntpin4基因(ntpin4蛋白)在烟草生长中的作用,通过外源施加ntpin4蛋白或者超表达ntpin4基因,进而抑制烟草分枝和腋芽的增长;亦可通过构建ntpin4基因缺失的转基因突变株,从而促进烟草分枝和腋芽增长。总之,通过对ntpin4基因(ntpin4蛋白)的深入研究,对于烟草生长调控、株型调控具有重要的理论意义和实际应用价值。
附图说明
图1为ntpin4基因在不同外源植物激素处理下的表达特征(10μmol/l的生长素iaa、10μmol/l独脚金内酯类似物gr24、10μmol/l的细胞分裂素6-ba、10μmol/l的脱落酸aba);
图2为ntpin4基因在不同组织中的表达特征;
图3为ntpin4基因敲除的靶位点选择示意图(20bp的靶位点后面为pam区域,+显示正义链,星号指示sgrna的相对位置);
图4为t0、t1代转基因系敲除靶位点测序结果;
图5为t0、t1代pin4突变体的表型,其中a图为t0代植株表型观察结果,b图为t1代植株表型观察结果。
具体实施方式
下面结合实施例对本申请做进一步的解释说明,在介绍具体实施例前,就下述实施例中涉及部分生物材料、实验试剂、实验设备等情况简要介绍如下。
生物材料:
烟草品种:红花大金元,一种商品化烟草,实施例中所采用种子由国家烟草基因研究中心保存提供;
载体:peasy-t1simple载体,购自北京全式金生物技术有限公司;
crispr/cas9载体由西南大学家蚕基因组生物学国家重点实验室友情提供;
菌株:
trans1-t1化学感受态细胞,购自北京全式金生物技术有限公司;
lba4404农杆菌菌株,生物实验中常用菌株,可公开获得;
引物的合成和dna测序由北京六合华大基因科技股份有限公司提供完成;
实验试剂:
rna提取试剂盒,superpureplantpolyrnakit;
荧光定量pcr酶(sybrqpcrkit),购自郑州安赛生物科技有限公司;
反转录试剂盒、t4连接酶,购自宝生物工程(大连)有限公司;
限制性内切酶bsai,购自neb公司;
dna扩增酶,购自北京全式金生物技术有限公司;
植物基因组提取试剂盒、dna纯化试剂盒购自qiagen公司;
实验设备:
pcr合成仪tprofessionalthermocycler,biometra公司;
定量pcr仪cfx96,bio-rad公司;
紫外凝胶成像系统biospectrum,uvp公司。
实施例1
本实施例主要就ntpin4基因的克隆获得过程简要介绍如下。
(1)制备cdna作为克隆模板
取旺长期烟草(红花大金元)的茎100mg作为样品,在液氮中充分研磨,参照rna提取试剂盒说明书提取总rna,然后反转录为cdna备用;
(2)设计引物,进行pcr扩增
设计用于扩增烟草生长素转运蛋白基因ntpin4的引物序列如下:
ntpin4-f:5’-atgatcacttggcacgatcta-3’,
ntpin4-r:5’-ttataatccaagaatgatgtagtacac-3’;
以步骤(1)中所制备cdna为模板,利用上述引物进行pcr扩增,pcr扩增条件为:94℃预变性4min;94℃变性30s,56℃退火30s,72℃延伸40s,共25个循环;再72℃终延伸10min;pcr扩增4℃保存备用,或者直接进行电泳检测分析。
参照胶回收试剂盒说明书纯化pcr扩增后产物,纯化产物再连接至peasy-t1载体上,连接体系如下:
dna扩增产物,6μl;
peasy-t1载体,1μl;
混匀后,25℃连接25min。
将连接产物转化至大肠杆菌感受态细胞中,具体转化过程简要介绍如下:
从-80℃冰箱中取出感受态细胞,置于冰上使其溶解,加连接产物于50μl的trans1-t1感受态细胞中,轻弹混匀,冰浴30min;
42℃水浴中热激30s,立即置于冰上2min;加入250μl平衡至室温的lb(不含抗生素),37℃摇荡培养1h;
取8μl的500mm的iptg、40μl的20mg/ml的x-gal,混合后均匀地涂布到lb固体平板(含60μg/μl氨苄青霉素)平板上,倒置培养皿,37℃培养过夜。
挑取白斑扩增培养后,提取各质粒dna,通过质粒pcr扩增对重组质粒进行了鉴定,将对应的阳性克隆送样测序,获得ntpin4基因序列。
测序分析结果表明,ntpin4基因编码区长度为1953bp个核苷酸,具体如seqidno.1所示;对该基因分析后可知,其所编码的ntpin4蛋白的氨基酸序列如seqidno.2所示。
实施例2
本实施例中,发明人通过不同外源植物激素处理和采集不同组织、器官,利用荧光定量pcr对ntpin4基因的表达模式进行了分析,相关实验简要介绍如下。
将烟草种子置于培养皿中,加入hoagland溶液进行培养(光照/黑暗=18/6h,23℃~28℃),培养至发芽2周后,将幼苗移至不同激素的溶液中,并在叶片喷施相应浓度激素,处理时间为5h,采集样品经液氮速冻后置于-80℃冰箱中,保存备用;另外正常培育后,采集现蕾期烟草的根、茎、叶、腋芽和花作为样品,经液氮速冻后置于-80℃冰箱中,保存备用。
实验组具体处理方式如下:
10μmol/l的生长素iaa,
10μmol/l独脚金内酯类似物gr24,
10μmol/l的细胞分裂素6-ba,
10μmol/l的脱落酸aba;
同时设置对照组,对照组烟草幼苗用1%(v/v)的dmso溶液处理。
将处理后烟草幼苗材料分别置于1.5ml离心管中,液氮速冻,-80℃保存备用。
将所保存的材料提取rna,利用反转录试剂盒合成cdna后(参考试剂盒说明书操作即可),以烟草ntl25基因为内参,进行荧光定量pcr检测,检测时,引物序列设计如下:
检测ntpin4基因的荧光定量引物,引物序列为:
ntpin4-q-f:5’-gcagtccctttactttcctttca-3’,
ntpin4-q-r:5’-aagtgtattaggaagtgttgaaagtgag-3’;
检测烟草ntl25基因时,具体引物为:
ntl25-f:5’-caaaagttacattccaccg-3’,
ntl25-f:5’-tttcttcgtcccatcaggc-3’;
荧光定量pcr的条件为:第一步预变性,95℃10s;第二步pcr反应,95℃5s,57-60℃30s,39个循环;第三步溶解曲线。
每个样品进行3次生物学重复,通过2-△△ct方法分析相对基因表达差异。分析结果如图1所示,与对照组相比较,ntpin4基因的相对表达量受生长素的诱导显著上调,但受脱落酸、细胞分裂素以及独脚金内酯的诱导显著下调。
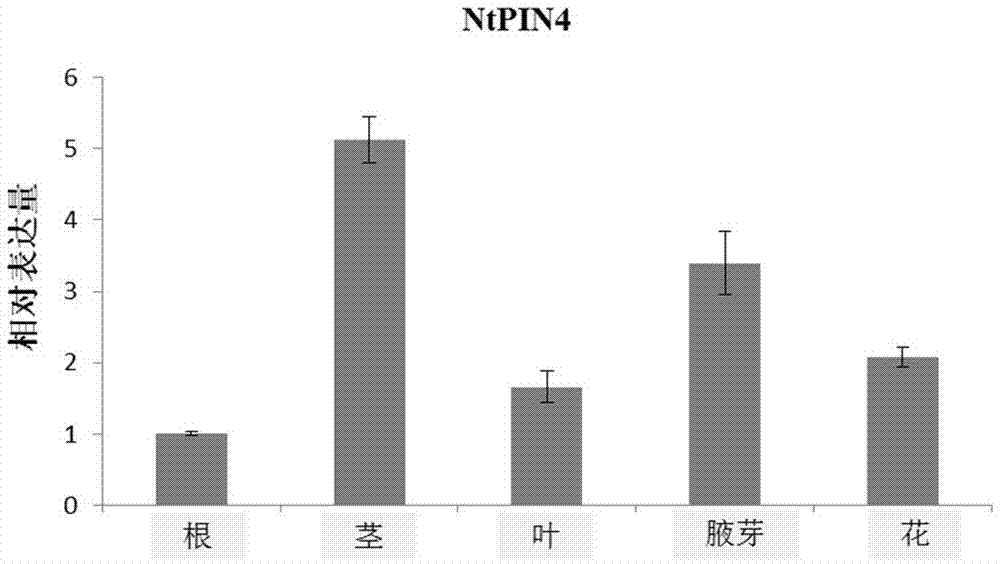
不同组织表达分析结果如图2所示,ntpin4基因在茎中的表达量最高,腋芽中次之,在根和叶片中相对较低。
实施例3
为进一步了解ntpin4基因在植株生长中的调节作用,发明人构建了用于敲除ntpin4基因的crispr/cas9表达载体,下面就构建过程简要介绍如下。
首先根据实施例1所知的ntpin4基因组和编码区序列,根据靶位点设计的标准,在ntpin4基因的第一外显子序列上设计20bp长度的靶位点(示意图如图3所示),设计敲除引物序列pin4-k-f和pin4-k-r如下:
pin4-k-f:5’-gattgcatatggttctgtccgatgg-3’,
pin4-k-r:5’-aaacccatcggacagaaccatatgc-3’;
设计反应体系,以获得靶位点的dna双链(退火),20μl反应体系设计如下:
annealingbufferfordnaoligos(5×),4μl;
上下游引物(pin4-k-f、pin4-k-r),各4μl(50μmol/μl);
nuclease-freewater补充至20μl;
反应程序为:95℃5min,每8s下降0.1℃,降至25℃;反应产物4℃保存备用,或者直接进行后续反应。
将上述将退火产物与bsaⅰ酶切后的crispr/cas9载体进行连接,并筛选获得用于敲除ntpin4基因的crispr/cas9表达载体,20μl连接体系设计如下:
退火产物,6μl;
酶切产物(bsaⅰ酶切后的crispr/cas9载体),3μl;
10×t4dnaligasebuffer,2μl;
t4dnaligase,1μl;
灭菌水补充至20μl,37℃连接3h。
然后将连接产物转化至大肠杆菌感受态细胞中,通过挑取阳性克隆、扩大培养,提取质粒后,经pcr确认载体构建成功后,低温保存,用于农杆菌转化。
实施例4
将实施例3中所构建crispr/cas9表达载体转化农杆菌,并进而转化烟草植株,构建ntpin4基因敲除的转基因植株,具体实验过程简要介绍如下。
(1)农杆菌的转化
从-80℃冰箱中取出农杆菌感受态细胞,在冰上冻融,待即将解冻时加入6μl实施例3所制备crispr/cas9表达载体,轻弹混匀;将混合物置于预冷的电转杯中,冰上放置5min;
将电转仪参数调至:电压2.5kv,电容25μf,电阻200ω;然后用吸水纸将电转杯外壁上的水滴吸除干净后,把电转杯放入电击槽中,电击5ms;
迅速加入800μl的预热至28℃的yeb液体培养基,220rpm、28℃振荡复苏3h;
然后将菌液4500rpm离心1min,弃一半体积的上清,重新悬浮后均匀涂于含有rif(100μg/ml)、str(50μg/ml)和kan(50μg/ml)的yeb固体培养基上,28℃倒置培养约2~3d,直至单菌落形成;
挑取单菌落,扩培后对菌液进行pcr鉴定,鉴定正确的阳性克隆菌株,即为转化正确的工程菌。
(2)转化烟草植株
取生长一个月左右的烟草无菌苗叶片,用打孔器将叶片处理成直径0.5cm大小的叶盘,将处理后叶盘在ms固体培养基上预培养3d;
将上述所制备转化后农杆菌工程菌,培养至od600=0.6左右,4000rpm离心5min收集菌体,再用20ml的ms液体培养基悬浮菌体;
然后将上述预培养后叶盘置于菌液中,侵染10min;
用无菌的滤纸吸干浸染后叶盘周围多余的菌液,在ms+6-ba(2mg/l)+naa(0.5mg/l)的固体培养基上暗培养3d;
用含有cef(400mg/l)的无菌水清洗叶盘,并用无菌滤纸吸去多余的液体,将叶盘转到含有6-ba(2mg/l)、naa(0.5mg/l)、cef(200mg/l)和kan(50mg/l)的ms固体筛选培养基上,28℃光照培养;
待不定芽长到0.5cm时,转移到含cef(200mg/l)和kan(50mg/l)的ms固体培养基上生根。
待生长一个月左右,取少量叶片,参照植物基因组提取试剂盒说明书提取dna,通过pcr扩增、克隆、测序的方法,检测阳性转基因株系和突变形式。具体鉴定方法为:
在ntpin4基因组上,设计一对检测引物,该引物位于敲除靶位点的两侧,具体为:
pin4-j-f:5’-tcagagataagtcagcaccacac-3’,
pin4-j-r:5’-taaagaaacaacatcagattccact-3’;
以t0代转基因株系dna模板,进行pcr扩增;pcr条件为:94℃预变性4min;94℃变性30s,56℃退火30s,72℃延伸40s,共25个循环;再72℃终延伸10min;
将pcr扩增产物纯化之后,连接至peasy-t1载体上,通过转化大肠杆菌感受态细胞,挑取单菌落进行pcr扩增验证,并送测序。
测序结果如图4所示,在9株t0代植株中,检测到ntpin4基因有6中形式的突变,分别是1、2、5、17、20个碱基的删除(delete)和1个碱基的插入(insert),这些突变均发生在敲除的靶位点上,而野生型植株的ntpin4基因未检测的任何突变。这说明在t0代植株中已经成功实现了对ntpin4基因进行了突变(换言之,以碱基删除或碱基插入形式对ntpin4基因进行了沉默)。
转基因株系表型变化情况:
分别将野生型和转基因株系的t0代植株移栽至盆中,温室培养7周左右,表型观察结果显示,与野生型烟草相比较,阳性植株的腋芽生长加快。当生长至12周时(图5a),阳性株的分枝表型更加显著,腋芽生长明显,叶片增加。
为了验证pin4突变体突变位点和分枝表型能否稳定遗传至下一代,研究收集了t0代阳性株的种子,继续种植后获得t1代植株,开展pcr扩增、单克隆测序、突变位点及表型分析。分析表明t0代突变位点能够稳定遗传至t1代,而且获得了只含有5个碱基删除的纯合突变株。同时将野生型与t1代植株在温室中培养4周左右,表型观察结果如图5b所示。从图中可以看出,与野生型烟草相比较,在突变体中消除了腋芽抑制,腋芽增大,分枝表型显著增加。
综上,通过调控ntpin4基因的表达情况可以控制烟草的腋芽及分枝的增生情况,从而为烟草株型调控及最终烟草产量奠定应用基础。
sequencelisting
<110>中国烟草总公司郑州烟草研究院
<120>烟草生长素转运蛋白ntpin4及其应用
<130>none
<160>2
<170>patentinversion3.5
<210>1
<211>1953
<212>dna
<213>nicotianatabacuml.cv.honghuadajinyuan
<400>1
atgatcacttggcacgatctatatgttgtgttaacagcagttgttcctttgtatgttgct60
atgatcttggcatatggttctgtccgatggtggaaaatcttttcccctgatcaatgctct120
ggtataaacagatttgtggctatttttgcagtccctttactttcctttcactttatagcc180
atgaacaacccgtatgaaatgaacttccgtttcattgctgctgattctttacaaaaggtt240
attatgttagtggtgctttcattatgggctaatttgacaaaaaatggtagcctagaatgg300
agcattacaattttctcactttcaacacttcctaatacacttgttatgggaattcctttg360
ttaattgctatgtatggtgagtactctggtagtttaatggtacaagttgtagtgttacag420
tgtattatttggtacacccttttgcttttcttgttcgaatatcgtggggctaagatgctt480
attatggaacaatttcctgaaactgctgcttccattgtttcatttaaagtggaatctgat540
gttgtttctttagatggacatgattttcttgaaactgacgctgaaattggccaagatggt600
aaacttcatgttactgtaagaaaatcaaatgcgtctaggagatcatttgctatggaccat660
agaccatcaaatctaactggagctgaaatttatagcttaagttcttcaagaaatccaact720
cctagaggatctaattttaatcataatgatttttactctatgatgggttttcctggagga780
agattatccaattttggtcctgcggataatatgtattctgttcaatcgtctcggggtcca840
accccgagaccgtctaattttgaagaaaattgtgctccgggaggtctgattcagaactct900
ccgaagtttggatttttcccggcgcagactgctaccccaggttattatcctgcaccgaat960
cccgaaattgcttcgacagtacctaagaatacaaaacctcagcaacaacaaaatgttcaa1020
gtacaaaagcaagatgtacaacaacagctgaatgctaaaggtaataatcatgatgctaag1080
gaacttcacatgtttgtttggagctcgagtaattcgccggtgtcggaagccggcggcctc1140
catgtgttcggcggcaatgatttcagtgctaacgaacaatctggtcggtccgatggtgct1200
aaagaaattaggatgttggtttctgatcatcctcaaaatggggataatactaaagccatt1260
ccgcaaagtggggactttgttagggaagattttagctttggaggtgccaatggtggtgga1320
aaagatggagatgaagaaaaaggagaaaaagagggacccactggactgactaaactgggg1380
tccagttccacatcggagctacacccgaaaaccgccggagttcaagattccggtaccgga1440
aaacagatgccaccggcgagtgttatgactcgtttaatcttaatcatggtttggcgtaag1500
cttatccgtaacccgaacacgtattcgagcctcattggtctgatttggtcactaatctca1560
tttaggtggcatgtgcatatgcccaaaatcatagagaaatcaatctctattctctcagac1620
gctggtcttggaatggctatgtttagcttaggtctgtttatggctttgcaaccaaagatc1680
attgcatgtgggaacacagtggctacatttgcaatggctgtgaggtttttgactggccca1740
gcagttatggctgctgcttccattgccgttggtcttcgtggtaccctcctccatgtagcc1800
attgtacaggctgcattgccacaagggattgtcccatttgtgtttgctaaggagtacaat1860
gttcatccagctattcttagcactgcggttattttcgggatgttgatagcattgccaatc1920
acattagtgtactacatcattcttggattataa1953
<210>2
<211>650
<212>prt
<213>nicotianatabacuml.cv.honghuadajinyuan
<400>2
metilethrtrphisaspleutyrvalvalleuthralavalvalpro
151015
leutyrvalalametileleualatyrglyservalargtrptrplys
202530
ilepheserproaspglncysserglyileasnargphevalalaile
354045
phealavalproleuleuserphehispheilealametasnasnpro
505560
tyrglumetasnpheargpheilealaalaaspserleuglnlysval
65707580
ilemetleuvalvalleuserleutrpalaasnleuthrlysasngly
859095
serleuglutrpserilethrilepheserleuserthrleuproasn
100105110
thrleuvalmetglyileproleuleuilealamettyrglyglutyr
115120125
serglyserleumetvalglnvalvalvalleuglncysileiletrp
130135140
tyrthrleuleuleupheleupheglutyrargglyalalysmetleu
145150155160
ilemetgluglnpheprogluthralaalaserilevalserphelys
165170175
valgluseraspvalvalserleuaspglyhisasppheleugluthr
180185190
aspalagluileglyglnaspglylysleuhisvalthrvalarglys
195200205
serasnalaserargargserphealametasphisargproserasn
210215220
leuthrglyalagluiletyrserleuserserserargasnprothr
225230235240
proargglyserasnpheasnhisasnaspphetyrsermetmetgly
245250255
pheproglyglyargleuserasnpheglyproalaaspasnmettyr
260265270
servalglnserserargglyprothrproargproserasnpheglu
275280285
gluasncysalaproglyglyleuileglnasnserprolysphegly
290295300
phepheproalaglnthralathrproglytyrtyrproalaproasn
305310315320
progluilealaserthrvalprolysasnthrlysproglnglngln
325330335
glnasnvalglnvalglnlysglnaspvalglnglnglnleuasnala
340345350
lysglyasnasnhisaspalalysgluleuhismetphevaltrpser
355360365
serserasnserprovalserglualaglyglyleuhisvalphegly
370375380
glyasnasppheseralaasngluglnserglyargseraspglyala
385390395400
lysgluileargmetleuvalserasphisproglnasnglyaspasn
405410415
thrlysalaileproglnserglyaspphevalarggluasppheser
420425430
pheglyglyalaasnglyglyglylysaspglyaspgluglulysgly
435440445
glulysgluglyprothrglyleuthrlysleuglyserserserthr
450455460
sergluleuhisprolysthralaglyvalglnaspserglythrgly
465470475480
lysglnmetproproalaservalmetthrargleuileleuilemet
485490495
valtrparglysleuileargasnproasnthrtyrserserleuile
500505510
glyleuiletrpserleuileserpheargtrphisvalhismetpro
515520525
lysileileglulysserileserileleuseraspalaglyleugly
530535540
metalametpheserleuglyleuphemetalaleuglnprolysile
545550555560
ilealacysglyasnthrvalalathrphealametalavalargphe
565570575
leuthrglyproalavalmetalaalaalaserilealavalglyleu
580585590
argglythrleuleuhisvalalailevalglnalaalaleuprogln
595600605
glyilevalprophevalphealalysglutyrasnvalhisproala
610615620
ileleuserthralavalilepheglymetleuilealaleuproile
625630635640
thrleuvaltyrtyrileileleuglyleu
645650
- 还没有人留言评论。精彩留言会获得点赞!