一种醇脱氢酶突变体及其应用的制作方法
本发明涉及酶工程技术领域,尤其涉及一种醇脱氢酶突变体及其应用。
背景技术:
(s)-6-氯-5-羟基-3-羰基己酸叔丁酯((s)-choh)是非常重要的手性中间体,主要用于合成hmg-coa还原酶抑制剂,其进一步不对称还原的产物既能经脱卤上氰基、羟基保护等步骤合成阿托伐他汀钙(立普妥),也可由羟基保护、酰化和水解反应等步骤合成瑞舒伐他汀钙(可定),两者均为全球畅销的降血脂药物。
目前,主要通过两种合成路线合成(s)-choh,一种是以4-氯-3-羰基丁酸乙酯为底物,通过化学法或生物法不对称合成(s)-4-氯-3-羟基丁酸乙酯,再通过一步克莱森缩合反应,与醋酸叔丁酯反应生成(s)-6-氯-5-羟基-3-羰基己酸叔丁酯;另一种方法则是从6-氯-3,5-二羰基己酸叔丁酯(cdoh)出发,利用羰基还原酶对其5-位上的羰基进行不对称还原,生成(s)-choh。
其中,路线2不仅在反应步骤上减少了一步化学反应,且酶催化方法反应条件温和,产物易于分离,无论是从经济角度还是环境角度,路线2都比路线1更具潜力。然而,目前工业上大部分采用路线1制备(s)-choh,而对路线2的研究却偏少。其中缺少高效生物催化剂是阻碍该路线走向产业化的瓶颈问题。
为了使酶能更好的应用于工业生产,研究者们采取了不同的分子改造方法来提高酶的催化活性。根据文库构建原理的不同,可分为定向进化、理性设计和半理性设计这三种策略。酶的定向进化技术不需要了解酶的结构与反应机理之间的关系,适用范围广。但在实际操作中由于其构建的突变文库较大,依赖于高通量筛选方法。酶的理性设计与定向进化技术不同,该方法具有目的性强,效率高的优点。理性设计需要在充分了解酶的空间结构和催化机理的基础上,对酶的结构和功能进行全面的分析,然后定位到关键的结构上进行精确的调控,从而获得想要的突变体。而半理性设计则是结合了上述两种酶改造技术的优势的一种方法。半理性设计是借助蛋白质数据库中的信息并通过计算机模拟等手段识别出可供分子改造的关键氨基酸位点,减少突变数目,降低筛选工作量,提高酶分子改造的成功率。
目前,仅发现3种醇脱氢酶能催化cdoh不对称还原得到(s)-choh。第一种是来自于lactobacillusbrevisdsm20054的nadph依赖型醇脱氢酶lbadh;第二种是来源于lactobacilluskefirdsm20587的醇脱氢酶lkadh,两者在氨基酸序列上有88.6%的相似度;而第三种是申请人于2017年授权的专利(申请号cn201410810512.4)中的lkadh的两点突变体a202l/l199h。为提高筛选效率本发明采用基于结构的“半理性设计”的方法对其进行改造,得到了酶活显著提高的醇脱氢酶突变体。
技术实现要素:
本发明提供了一种醇脱氢酶突变体及其应用,该醇脱氢酶突变体具有高酶活的特点,可高效催化6-氯-3,5-二羰基己酸叔丁酯制备(s)-6-氯-5-羟基-3-羰基己酸叔丁酯,对他汀类药物中间体的生产具有重要意义。
具体技术方案如下:
本发明提供了一种醇脱氢酶突变体,所述醇脱氢酶突变体为下列(1)和/或(2);
(1)将seqidno.1所示氨基酸序列的第147位的苯丙氨酸替换为亮氨酸、异亮氨酸、甲硫氨酸或半胱氨酸;
(2)将seqidno.1所示氨基酸序列的第202位的丙氨酸替换为亮氨酸、异亮氨酸或缬氨酸。
作为优选,所述醇脱氢酶突变体为将seqidno.1所示氨基酸序列的第147位的苯丙氨酸替换异亮氨酸,第202位的丙氨酸替换为亮氨酸;
并且,将seqidno.1所示氨基酸序列如(a)~(c)中所述位点的一种或多种突变进行组合;
(a)将第145位的谷氨酸替换为亮氨酸;
(b)将第190位的酪氨酸替换为苯丙氨酸;
(c)将第96位的丝氨酸替换为丙氨酸。
更优选,所述醇脱氢酶突变体为将seqidno.1所示氨基酸序列的第147位的苯丙氨酸替换为异亮氨酸,第202位的丙氨酸替换为亮氨酸,第190位的酪氨酸替换为苯丙氨酸,第96位的丝氨酸替换为丙氨酸,第145位的谷氨酸替换为亮氨酸。
具体的,本发明突变获得的醇脱氢酶突变体为下列中的一种:
(i)将seqidno.1所示氨基酸序列的第147位的苯丙氨酸替换为亮氨酸、异亮氨酸、甲硫氨酸或半胱氨酸;所得单点突变体依次命名为f147l、f147i、f147m、f147c;
(ii)将seqidno.1所示氨基酸序列的第202位的丙氨酸替换为亮氨酸、异亮氨酸或缬氨酸;所得单点突变体依次命名为a202l、a202i、a202v:
(iii)将seqidno.1所示氨基酸序列的第147位的苯丙氨酸替换为亮氨酸,第202位的丙氨酸替换为亮氨酸,命名为f147l/a202l;
或,第147位的苯丙氨酸替换为异亮氨酸,第202位的丙氨酸替换为亮氨酸,命名为f147i/a202l;
或,第147位的苯丙氨酸替换为异亮氨酸,第202位的丙氨酸替换为异亮氨酸,命名为f147i/a202i;
或,第147位的苯丙氨酸替换为甲硫氨酸,第202位的丙氨酸替换为异亮氨酸,命名为f147m/a202i;
或,第147位的苯丙氨酸替换为甲硫氨酸,第202位的丙氨酸替换为缬氨酸,命名为f147m/a202v;
或,第147位的苯丙氨酸替换为异亮氨酸,第202位的丙氨酸替换为缬氨酸,命名为f147i/a202v;
或,第147位的苯丙氨酸替换为半胱氨酸,第202位的丙氨酸替换为缬氨酸,命名为f147c/a202v。
(iv)将seqidno.1所示氨基酸序列的第147位的苯丙氨酸替换异亮氨酸,第202位的丙氨酸替换为亮氨酸,第145位的谷氨酸替换为亮氨酸;所得三点突变体命名为f147i/a202l/e145l;
(v)将seqidno.1所示氨基酸序列的第147位的苯丙氨酸替换为异亮氨酸,第202位的丙氨酸替换为亮氨酸,第190位的酪氨酸替换为苯丙氨酸;所得三点突变体命名为f147i/a202l/y190f;
(vi)将seqidno.1所示氨基酸序列的第147位的苯丙氨酸替换为异亮氨酸,第202位的丙氨酸替换为亮氨酸,第96位的丝氨酸替换为丙氨酸;所得三点突变体命名为f147i/a202l/s96a;
(vii)将seqidno.1所示氨基酸序列的第147位的苯丙氨酸替换为异亮氨酸,第202位的丙氨酸替换为亮氨酸,第190位的酪氨酸替换为苯丙氨酸,第96位的丝氨酸替换为丙氨酸;所得四点突变体命名为f147i/a202l/y190f/s96a;
(viii)将seqidno.1所示氨基酸序列的第147位的苯丙氨酸替换为异亮氨酸,第202位的丙氨酸替换为亮氨酸,第190位的酪氨酸替换为苯丙氨酸,第145位的谷氨酸替换为亮氨酸;所得四点突变体命名为f147i/a202l/y190f/e145l;
(ix)将seqidno.1所示氨基酸序列的第147位的苯丙氨酸替换为异亮氨酸,第202位的丙氨酸替换为亮氨酸,第96位的丝氨酸替换为丙氨酸,第145位的谷氨酸替换为亮氨酸;所得四点突变体命名为f147i/a202l/s96a/e145l;
(x)将seqidno.1所示氨基酸序列的第147位的苯丙氨酸替换为异亮氨酸,第202位的丙氨酸替换为亮氨酸,第190位的酪氨酸替换为苯丙氨酸,第96位的丝氨酸替换为丙氨酸,第145位的谷氨酸替换为亮氨酸;所得五点突变体命名为f147i/a202l/y190f/s96a/e145l。
本发明基于结构的半理性设计方法,对来源于开菲尔乳杆菌(lactobacilluskefirdsm20587),氨基酸序列如seqidno.1所示的野生型醇脱氢酶(ncbi登录号:ay267012.1;基因大小:759bp)进行改造,得到了酶活显著提高的醇脱氢酶突变体。
本发明提供了一种所述醇脱氢酶突变体的编码基因。
本发明还提供了一种包含所述编码基因的表达载体。
本发明还提供了一种包含所述编码基因的基因工程菌。
本发明还提供了所述醇脱氢酶突变体或所述基因工程菌在催化6-氯-3,5-二羰基己酸叔丁酯制备(s)-6-氯-5-羟基-3-羰基己酸叔丁酯中的应用。
具体地,所述应用,包括:在辅因子和氢供体的存在下,以醇脱氢酶突变体或基因工程菌为催化剂,催化底物6-氯-3,5-二羰基己酸叔丁酯,反应生成(s)-6-氯-5-羟基-3-羰基己酸叔丁酯。
作为优选,所述辅因子为nadph;氢供体为异丙醇;所述反应的温度为18~30℃,ph为5.0~7.0。
与现有技术相比,本发明具有以下有益效果:
本发明采用半理性设计对来源于开菲尔乳杆菌的醇脱氢酶lkadh进行改造,获得了多个酶活提高的醇脱氢酶突变体。其中,突变体f147l/a202l比酶活为18u/mg、f147i/a202l比酶活为23u/mg,分别是原始酶lkadh(0.57u/mg)31.6倍和40.3倍。组合突变体f147i/a202l/y190f/s96a,其比酶活为44u/mg,是原始酶lkadh(0.57u/mg)的77倍。
附图说明
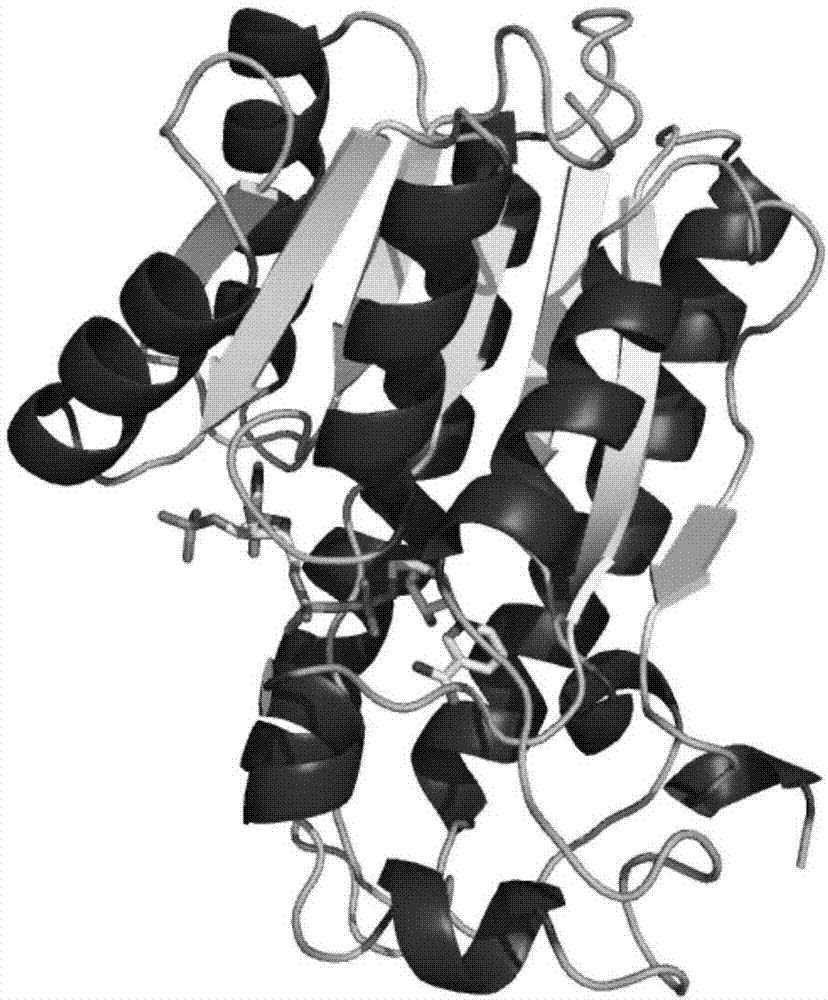
图1为实施例1中醇脱氢酶mf147i-a202l同源性模拟结构示意图。
图2为醇脱氢酶mf147i-a202l催化活性口袋附近的关键氨基酸。
具体实施方式
下面结合具体实施方式对本发明做进一步描述。其中,下列实施例中涉及的工程菌构建、酶的诱导表达与分离纯化以及酶活的测定均采用下列方法,具体如下:
1、野生型醇脱氢酶重组菌的构建
根据醇脱氢酶的基因序列设计引物,用引物f-lkadh/r-lkadh进行pcr扩增来获得目的基因。
引物f-lkadh的序列为:5’-cgcggatccatgactgatcgttaaaag-3’;
引物r-lkadh的序列为:5’-ccgctcgagcttattgagcagtgtatccac-3’。
pcr反应体系为:
pcr程序为:98℃2min;98℃10s,58℃15s,72℃10s(一般5s/kb),30个循环;72℃5min;4℃保温。
pcr产物的大小通过1%琼脂糖凝胶电泳的验证后用pcr纯化试剂盒进行纯化回收。将纯化后的pcr产物和质粒pet30a(+)分别用限制性内切酶bamhi与xhoi进行双酶切。酶切结束后样品经核酸琼脂糖凝胶电泳分离纯化后切胶回收,测定各自的浓度后进行酶连反应。
采用热激法将连接产物转化到大肠杆菌bl21(dh5α)感受态细胞中并送去铂尚生物技术(上海)有限公司-杭州测序部测序。从测序验证正确的阳性转化子中提取重组质粒转化到大肠杆菌bl21(de3)中,最后得到重组菌。
2、突变型醇脱氢酶重组菌的构建
以连接有醇脱氢酶基因的重组质粒pet-30a(+)-lkadh为模板,设计一对包含突变位点的正反向引物(具体见实施例1),进行pcr,得到重组基因。
其中,pcr的反应体系为:
pcr程序:1)95℃预加热2min;2)98℃变性10s;3)58℃退火15s;4)72℃延伸1min;5)循环30次;6)再延伸72℃3min;7)4℃保温2h。
pcr产物经过琼脂糖凝胶电泳的验证后正确后进行纯化回收。用限制性内切酶dpni对纯化后的pcr产物进行消化,除去带有甲基的模板质粒。
消化体系为:dna10μl、buffer2μl、dpni1μl、去离子水7μl。37℃2h,酶切后的混合物用pcr纯化试剂盒进行纯化回收。
最后,采用热激法将纯化产物转入大肠杆菌bl21(de3)感受态细胞中,测序正确的单菌落,即为构建成功的突变子。
3、醇脱氢酶的诱导表达与分离纯化
挑取重组菌(野生型醇脱氢酶基因工程菌和突变型醇脱氢酶基因工程菌)单菌落于5mllb液体培养基中(含50μg/ml卡那霉素),生长8-10h后,按2%接种量将种子发酵液接至含50μg/ml卡那霉素的50mllb培养基中(1%蛋白胨,0.5%酵母提取物,1%氯化钠,ph7.0),转速200rpm,37℃培养约2-3h(od600达到0.6-0.8),加入终浓度为0.5mm的异丙基硫代-β-d-半乳糖苷,在30℃,200rpm下诱导14h。
将菌液离心弃上清,用纯净水洗涤湿菌体后再次离心弃上清,最后加入一定量磷酸钾缓冲液(ph7.5)将湿菌体重悬,将浓缩10倍的菌液置于冰水浴中进行超声破胞。将超声破碎后的破胞液进行离心,取上清液用ni柱进行亲和层析,用洗杂缓冲液(20mm磷酸钾缓冲液,0.5mnacl,50mm咪唑,ph7.5)洗去杂质,用洗脱缓冲液(20mm磷酸钾缓冲液,0.5mnacl,250mm咪唑,ph7.5)洗脱目的蛋白,将分离出来的目标蛋白置于超滤离心管中进行脱盐浓缩,得到电泳纯的目标蛋白。
4、醇脱氢酶重组菌的酶活测定
反应体系为1ml,其中包括1.0mmnadph,10mm底物cdoh(预溶于50%v异丙醇中),ph7的柠檬酸-磷酸氢二钠-naoh缓冲液以及适量的酶,30℃,600rpm金属浴中振荡反应20min,再12,000rpm离心2min除去反应菌体终止全细胞反应(当用破胞液反应时,加入0.5ml乙腈终止反应,过滤后液相检测),取上清过滤后hplc检测产物吸收峰面积的大小。
利用液相色谱法对产物(s)-choh进行定量分析。
色谱柱型号为pntulipstmqs-c18(5μm×4.6mm×250mm)。检测波长225nm,流动相流速为1.0ml/min,乙腈:10mm乙酸钠溶液(ph=7)=55:50(v/v),进样量为20μl,柱温为30℃。酶活反应体系根据实验需求而定,终止反应后,将需要检测的产物的浓度控制在检测范围内即可。
酶活单位(u)定义为每分钟产生1μmol(s)-choh所需的酶量。
实施例1
1、醇脱氢酶lkadh的半理性设计
1.1147位和202位的定点突变
(1)f147l、f147i、f147m、f147c、a202l、a202i、a202v突变体
以连接有醇脱氢酶lkadh基因的重组质粒pet-30a(+)-lkadh为模板,设计一对包含突变位点的正反向引物f-primer/r-primer(表1,2),对147位和202位通过pcr进行定点突变。
表1147位点定点突变引物序列
表2202位点定点突变引物序列
(2)f147l/a202l、f147i/a202l突变体的获得:
以连接有醇脱氢酶lkadh(a202l)基因的重组质粒pet-30a(+)-lkadh(a202l)为模板,设计一对包含突变位点的正反向引物f-primer/r-primer(表3),通过pcr进行定点突变。
表3147、202位点组合突变引物序列
如表4所示,对147位和202位进行定点突变,一系列突变体的酶活分别为醇脱氢酶lkadh的6.40-49.9倍。
表4147、202位点突变体及其相对酶活
1.2基于结构知识的设计与改造
来源于开菲尔乳杆菌的醇脱氢酶lkadh的三维结构已经得到解析,f147i/a202l为lkadh的两点突变体,其他突变体与lkadh相比也只有少量氨基酸的差异,序列同源性高于99%,故选择醇脱氢酶lkadh(pdbid:4rf2)进行建模。
用ds软件进行分子对接。将同源模建所得的f147i/a202l突变体的蛋白结构文件和处理好的底物分子文件分别导入ds中,对蛋白进行去水、加氢处理保留,并保留单亚基用于对接。
采用ds软件中的cdocker方法将cdoh对接进酶的活性中心,以是否与ser143和tyr156形成氢键来筛选产生的pose。若有多个pose满足,选择分数最高的pose做后续优化。
根据分子对接的结果,发现底物处于所附图2所示的疏水口袋中,酶和底物结合口袋部位的氨基酸变化会对催化口袋造成较大的影响,选取催化活性口袋周围的氨基酸进行饱和突变。
由于缺少有效的高通量筛选方法,故选取靠近底物5位碳上的羰基(氯甲基端)的位点i144、e145、l153以及y190、g189进行定点饱和突变。在确定突变位点后,一一构建相应的突变子。
以连接有醇脱氢酶基因f147i/a202l的重组质粒pet-30a(+)-f147i/a202l为模板,进行全质粒pcr。设计一对包含突变位点的正反向引物(表5),通过引物将突变引入目的片段中。
表5i144、e145、l153以及y190、g189定点突变引物序列
每个位点取一对引物为例,突变成其他氨基酸时只需将下划线处密码子替换成对应氨基酸的密码子即可。
pcr产物经过琼脂糖凝胶电泳的验证后正确后进行纯化回收。用限制性内切酶dpni对纯化后的pcr产物进行消化,除去带有甲基的模板质粒。
消化体系为:dna10μl、buffer2μl、dpni1μl、去离子水7μl。37℃2h,酶切后的混合物用pcr纯化试剂盒进行纯化回收。最后采用热激法将纯化产物转入大肠杆菌bl21(de3)感受态细胞中,测序正确的单菌落即为构建成功的突变子。
e145、l153、y190分别突变成另外19种氨基酸的57个突变体,i144突变成另外12种氨基酸的12个突变体、g189突变成另外11种氨基酸的11个突变体(ndt密码子编码的包括f、l、i、v、y、h、n、d、c、r、s、g在内的12种氨基酸。)经过反复筛选验证后,发现f147i/a202l/e145l、f147i/a202l/y190f的酶活有一定程度的提高。酶活分别为醇脱氢酶lkadh的71.2和132倍。
表6145、190、96位点突变体及其相对酶活
由于底物口袋附近及底物通道处氨基酸较多,故利用discoverystudio的calculationmutationenergy(binding)模块计算各个位点突变成丙氨酸的突变能量。选出了6个对亲和力有提高的位点m141a、i191a、s96a、m206a、n90a、p188a进行定点突变。
经测序正确后,对m141a、i191a、s96a、m206a、n90a、p188a这6个突变体进行酶活的测定,经反复筛选验证后发现s96a的酶活有明显提高;酶活为醇脱氢酶lkadh的89.8倍。
3、突变体的组合
为进一步提高突变体的酶活,我们将得到的正向突变体进行组合。引物序列如下:
f-s96a:5’-gggattgccgttgccaaaagcgttgaagac-3’;
r-s96a:5’-gtcttcaacgcttttggcaacggcaatccc-3’;
f-e145l:5’-atgagcagtattctggggattgtaggcgat-3’;
r-e145l:5’-atcgcctacaatccccagaatactgctcat-3’;
通过全质粒pcr将s96a的突变引入突变体f147i/a202l/y190f和突变体f147i/a202l/e145l中,得到四点突变体f147i/a202l/y190f/s96a和f147i/a202l/e145l/s96a;将e145l的突变引入突变体f147i/a202l/y190f中,得到四点突变体f147i/a202l/y190f/e145l;将s96a的突变引入突变体f147i/a202l/y190f/e145l中得到五点突变体f147i/a202l/y190f/e145l/s96a。
表7组合突变体及其酶活
最后,得到酶活最高的组合突变体f147i/a202l/y190f/s96a。f147i/a202l/y190f/s96a在30℃下的比酶活(u/mg)为44,是醇脱氢酶f147l-a202l比酶活的2.44倍,醇脱氢酶lkadh比酶活的77倍。
序列表
<110>浙江大学
<120>一种醇脱氢酶突变体及其应用
<160>36
<170>siposequencelisting1.0
<210>1
<211>252
<212>prt
<213>开菲尔乳杆菌(lactobacilluskefir)
<400>1
metthraspargleulysglylysvalalailevalthrglyglythr
151015
leuglyileglyleualailealaasplysphevalglugluglyala
202530
lysvalvalilethrglyarghisalaaspvalglyglulysalaala
354045
lysserileglyglythraspvalileargphevalglnhisaspala
505560
seraspglualaglytrpthrlysleupheaspthrthrglugluala
65707580
pheglyprovalthrthrvalvalasnasnalaglyilealavalser
859095
lysservalgluaspthrthrthrgluglutrparglysleuleuser
100105110
valasnleuaspglyvalphepheglythrargleuglyileglnarg
115120125
metlysasnlysglyleuglyalaserileileasnmetserserile
130135140
gluglyphevalglyaspprothrleuglyalatyrasnalaserlys
145150155160
glyalavalargilemetserlysseralaalaleuaspcysalaleu
165170175
lysasptyraspvalargvalasnthrvalhisproglytyrilelys
180185190
thrproleuvalaspaspleugluglyalagluglumetmetsergln
195200205
argthrlysthrprometglyhisileglygluproasnaspileala
210215220
trpilecysvaltyrleualaseraspgluserlysphealathrgly
225230235240
alagluphevalvalaspglyglytyrthralagln
245250
<210>2
<211>759
<212>dna
<213>开菲尔乳杆菌(lactobacilluskefir)
<400>2
atgactgatcgtttaaaaggcaaagtagcaattgtaactggcggtaccttgggaattggc60
ttggcaatcgctgataagtttgttgaagaaggcgcaaaggttgttattaccggccgtcac120
gctgatgtaggtgaaaaagctgccaaatcaatcggcggcacagacgttatccgttttgtc180
caacacgatgcttctgatgaagccggctggactaagttgtttgatacgactgaagaagca240
tttggcccagttaccacggttgtcaacaatgccggaattgcggtcagcaagagtgttgaa300
gataccacaactgaagaatggcgcaagctgctctcagttaacttggatggtgtcttcttc360
ggtacccgtcttggaatccaacgtatgaagaataaaggactcggagcatcaatcatcaat420
atgtcatctatcgaaggttttgttggtgatccaactctgggtgcatacaacgcttcaaaa480
ggtgctgtcagaattatgtctaaatcagctgccttggattgcgctttgaaggactacgat540
gttcgggttaacactgttcatccaggttatatcaagacaccattggttgacgatcttgaa600
ggggcagaagaaatgatgtcacagcggaccaagacaccaatgggtcatatcggtgaacct660
aacgatatcgcttggatctgtgtttacctggcatctgacgaatctaaatttgccactggt720
gcagaattcgttgtcgatggtggatacactgctcaataa759
<210>3
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>3
cgcggatccatgactgatcgttaaaag27
<210>4
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>4
ccgctcgagcttattgagcagtgtatccac30
<210>5
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>5
agcagtattgaggggctggtaggcgatccg30
<210>6
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>6
cggatcgcctaccagcccctcaatactgct30
<210>7
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>7
agcagtattgaggggatagtaggcgatccg30
<210>8
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>8
cggatcgcctactatcccctcaatactgct30
<210>9
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>9
agcagtattgaggggatggtaggcgatccg30
<210>10
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>10
cggatcgcctaccatcccctcaatactgct30
<210>11
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>11
agcagtattgaggggtgcgtaggcgatccg30
<210>12
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>12
cggatcgcctacgcacccctcaatactgct30
<210>13
<211>33
<212>dna
<213>人工序列(artificialsequence)
<400>13
gatgatcttgaaggtctcgaggaaatgatgtca33
<210>14
<211>33
<212>dna
<213>人工序列(artificialsequence)
<400>14
tgacatcatttcctcgagaccttcaagatcatc33
<210>15
<211>33
<212>dna
<213>人工序列(artificialsequence)
<400>15
gatgatcttgaaggtatcgaggaaatgatgtca33
<210>16
<211>33
<212>dna
<213>人工序列(artificialsequence)
<400>16
tgacatcatttcctcgataccttcaagatcatc33
<210>17
<211>33
<212>dna
<213>人工序列(artificialsequence)
<400>17
gatgatcttgaaggtgtcgaggaaatgatgtca33
<210>18
<211>33
<212>dna
<213>人工序列(artificialsequence)
<400>18
tgacatcatttcctcgacaccttcaagatcatc33
<210>19
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>19
agcagtattgaggggctggtaggcgatccg30
<210>20
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>20
cggatcgcctaccagcccctcaatactgct30
<210>21
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>21
agcagtattgaggggatagtaggcgatccg30
<210>22
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>22
cggatcgcctactatcccctcaatactgct30
<210>23
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>23
atgagcagtattaacgggattgtaggcgat30
<210>24
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>24
atcgcctacaatcccgttaatactgctcat30
<210>25
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>25
ggcgatccgacgttcggggcatacaacgct30
<210>26
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>26
agcgttgtatgccccgaacgtcggatcgcc30
<210>27
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>27
gtacatccgggcttcatcaagaccccgctg30
<210>28
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>28
cagcggggtcttgatgaagcccggatgtac30
<210>29
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>29
aatatgagcagtgctgaggggattgtaggc30
<210>30
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>30
gcctacaatcccctcagcactgctcatatt30
<210>31
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>31
acagtacatccgctgtatatcaagaccccg30
<210>32
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>32
cggggtcttgatatacagcggatgtactgt30
<210>33
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>33
gggattgccgttgccaaaagcgttgaagac30
<210>34
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>34
gtcttcaacgcttttggcaacggcaatccc30
<210>35
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>35
atgagcagtattctggggattgtaggcgat30
<210>36
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>36
atcgcctacaatccccagaatactgctcat30
- 还没有人留言评论。精彩留言会获得点赞!