与草鱼性状相关的SNP分子标记及其应用的制作方法
本发明属于分子生物学dna标记技术与应用领域,特别涉及与草鱼性状相关的snp分子标记及其应用。
背景技术:
草鱼(ctenopharyngodonidella)属雅罗鱼亚科(leuciscinae)、草鱼属(ctenopharyngodon),具有生长速度快,肉质鲜美,营养价值高等优点,目前是我国淡水养殖鱼类中年产量最大的养殖对象,从2007年到2016年草鱼产量从356万吨逐年增加到590万吨,十年产量增加了65.73%。由此可见,草鱼养殖业的健康良性发展,不仅为我国乃至发展中国家人民提供了大量优质动物蛋白,同时带动了相关产业如饲料加工、运输和销售行业的发展壮大,解决了大批人员的就业问题。至今,虽然水产工作者已经付出了很多的努力,但草鱼仍未有通过国家良种审定委员会鉴定的人工选育的良种,实际生产上多为野生种直接驯化而成,缺少定向选育,生产上经常出现因亲本质量差,造成后代生长速度慢、规格不齐、抗病力差等问题,养殖户常常面临重大的经济损失,因此,选育高产优质的草鱼良种一直是草鱼育种工作的中心目标。
选育出生长速度更快的草鱼新品种可以节约饲料成本,缩短养殖周期进而大幅度降低草鱼养殖成本,因此,开发快速可靠的育种技术,进行草鱼快长品种(系)的培育十分必要。在开展常规选育研究的同时,开发和应用分子标记辅助育种技术可加快草鱼良种选育进程。snp是指基因组dna序列中由于单个核苷酸变异而产生的多态性,位于基因编码区的非同义snp可导致氨基酸的变化,从而影响蛋白质的功能,特别是发生在结构功能区域的snp尤其重要,最终引起生物表型的变化。
全基因组关联分析(genome-wideassociationstudy,gwas)旨在从全基因组范围内筛选与性状关联的snps。gwas分析的成本主要有2个:第一,用于分析的样本数量;第二,基因分型的费用。数量越多,其分型成本、测定性状的成本也越高。为减少成本,只测序极端样品的gwas方法被开发出来了,如bsr-seq(rna-seqbasedbsa)、xp-gwas(extreme-phenotypegenome-wideassociationstudy)等。研究证明xp-gwas可有效降低基因分型的工作量,能够进行低成本高效益的snp筛选。snp分型技术也逐渐由早期阶段的中、低通量,发展到高通量的基因芯片、重测序技术。其中rad-seq(restrictionassociationsitednasequencing)就是一种便宜、高效的snp发掘与分型高通量方法。综合考虑实验成本和效率,可以采用rad-seq技术结合xp-gwas策略在全基因组范围内寻找与草鱼生长性状相关的snp位点,旨在寻找新的遗传标记,为分子标记辅助选育快长草鱼新品种选育提供基础资料。
技术实现要素:
本发明的首要目的在于提供与草鱼体重相关的4个snps标记及应用。
本发明所采取的技术方案是:
草鱼生长相关的基因序列如seqidno.1~seqidno.4所示。
上述基因序列,seqidno.1的第159位y为碱基t或c,seqidno.2的第410位y为碱基c或t,seqidno.3的第370位y为碱基c或t,seqidno.4的第374位y为碱基c或t。
上述所述的基因序列seqidno.1~seqidno.4在判断草鱼生长快慢的应用。
草鱼生长快慢相关的snp位点,其为seqidno.1第159位碱基、seqidno.1第410位碱基、seqidno.1第370位碱基和seqidno.1第374位碱基。
上述所述的snp位点在判断或鉴别草鱼生长快慢中的应用。
上述所述的snp位点在选育生长优异草鱼品种的应用。
一种筛选草鱼生长快慢的方法,包括以下步骤:
检测草鱼基因序列seqidno.1的第159位碱基处的snp位点是否为基因型cc,若是,则为体重优异,生长快的草鱼;
或/和,检测草鱼基因seqidno.2的第410位碱基处的snp位点是否为基因型cc,若是,则为体重优异,生长快的草鱼;
或/和,检测草鱼基因seqidno.3的第370位碱基处的snp位点是否为基因型cc,若是,则为体重优异,生长快的草鱼;
或/和,检测草鱼基因seqidno.1的第374位碱基处的snp位点是否为基因型tt,若是,则为体重优异,生长快的草鱼。
进一步的,包括以下步骤:
(1)提取草鱼的dna;
(2)以提取的dna为模板,进行pcr扩增实验,获取含有snp位点的目标片段,检测草鱼基因seqidno.1第159位碱基、基因seqidno.2第410位碱基、基因seqidno.3第370位碱基的基因型是否为cc,或基因seqidno.4第374位碱基处的基因型是否为tt。
进一步的,所述的步骤(2)pcr扩增:使用snp24-f、snp24-r、snp35-f、snp35-r、snp36-f、snp36-r、snp37-f以及snp37-r引物对进行pcr扩增,得到pcr产物,所述引物的核苷酸序列如下:
snp24-f:5'-cgtagtcacgacaggacacgt-3';(seqidno.5)
snp24-r:5'-cagtgattcttctgttaattct-3';(seqidno.6)
snp35-f:5'-gatatggatgactgtctctcc-3';(seqidno.7)
snp35-r:5'-caatccaggtcagatagtaca-3';(seqidno.8)
snp36-f:5'-tcagggttcaaagtttgagtg-3';(seqidno.9)
snp36-r:5'-gtgcgaggtgattggtatctg-3';(seqidno.10)
snp37-f:5'-gacaatgagttcactattct-3';(seqidno.11)
snp37-r:5'-caaggttctggaatcattcct-3'(seqidno.12);
再将pcr产物使用延伸引物snp24-p、snp35-p、snp36-p和snp37-p进行pcr延伸扩增,确定草鱼基因seqidno.1第159位碱基、基因seqidno.2第410位碱基、基因seqidno.3第370位碱基以及基因seqidno.4第374位碱基处的基因型;所述延伸引物核苷酸序列如下:
snp24-p:5'-tacgctgtgagaaactgtgagg-3';(seqidno.13)
snp35-p:5'-gtgaaagggggaatactataac-3';(seqidno.14)
snp36-p:5'-ttttttttttcggactcggaggttgagagcta-3';(seqidno.15)
snp37-p:5'-agatctcaatccaagcgagcac-3'(seqidno.16)。
进一步的,所述的pcr扩增体系为:
所述的pcr扩增反应条件为:94℃变性15s;54℃退火15s,72℃延伸30s,24个循环。
本发明的有益效果是:
(1)本发明所获得的基因型是依据基因内部产生的碱基突变,因此不存在遗传的交换,也不需要表型的进一步验证。
(2)本发明通过检测草鱼的四个snp标记,snp24、snp35、snp36以及snp37,能够在相同养殖条件下有效的挑选体重更大的草鱼,能够有效用于草鱼的分子标记辅助育种。进而能够根据实际育种需求对草鱼亲本进行基因型鉴定,挑选合适的基因型草鱼亲本进行繁殖,可以获得生长较快的草鱼后代(鱼苗),节约育种时间,成本低廉,准确性高,加快草鱼育种进程。
附图说明
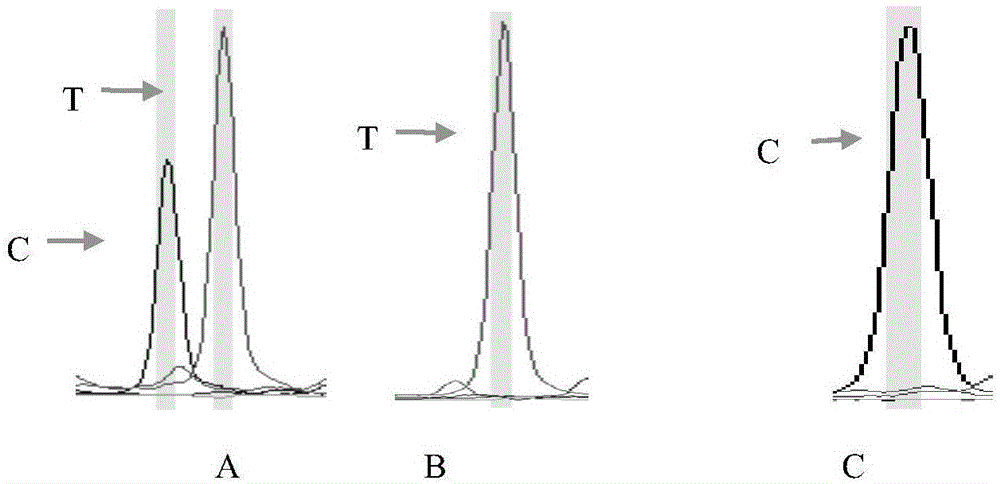
图1为snp延伸反应后的检测峰图,峰图的颜色及对应核苷酸为:绿色—a,红色—t,黑色—c,蓝色—g。
具体实施方式
下面结合实施例对本发明作进一步说明。
实施例1snp标记的获得
1.本发明测序获得的草鱼基因序列如seqidno.1~seqidno.4,在seqidno.1第159位碱基、seqidno.1第410位碱基、seqidno.1第370位碱基和seqidno.1第374位碱基处各发现一个snp位点,seqidno.1第159位碱基、seqidno.1第410位碱基、seqidno.1第370位碱基和seqidno.1第374位碱基处有等位基因c和t,组成cc、tc、tt基因型。
本发明用于生长性状(体重)分析的样本为同批繁殖且同塘饲养的25月龄草鱼群体,随机挑选298尾草鱼用于生长性状(体重)关联分析,选取体重极大的个体20尾(平均体重为2659±126.40g),体重极小的个体20尾(平均体重为744.76±73.35g),分别抽取亲本和后代尾静脉血液各2ml(添加acd抗凝剂抗凝)用于后续实验。
以下序列中的y代表的是突变的碱基。
snp24的基因序列片段如seqidno.1,rad库标签(ctg294657.0),突变位置为t159c:
atcggtccacgtaactctcccgaaagcctttccgcaccgcgcgggccatactgaccaccgtagtcacgacaggacacgtgctgaaacccaagaggacgggatgatggtctttaattcattaacatgacgagtgctgtgtgtgtgtgtgtgtgtttgttycctcacagtttctcacagcgtatgccgctcgtccccgtcggacacacacaggtgttcagagggccgcaggcagcaccatgttggcagggaggaacacacggtgtagtgacagctgcacacacacacacaattacatgatcacacactacatcaaaacaccacagaattaacagaagaatcactgcaaatgaaactgaaactgaaaaaatgaatgtcatatgaatgattcaatgattaatgatgtcataatgaaaggattataat(seqidno.1);
snp35序列片段,rad库标签(ctg355219.0),突变位置为c410t:
attacaacacaccagtggtgaggtcagctggtggtagcatgtgtttcctgatgttgattagtttgattttgtctagtataagtgcattctttttctttggagaacccacatctgcactttgcctcctgcgaaatgccatatttgcatttttcttcactgtctgtatttcctgtttgactgtccgttcctttcaaattgtttgtgtttttaaaatggctgctcagttccctaaggtgcacagcctttgggtaaagcacaatggccagtggctcttcattgcatttacttctgtcattcatttaatttcttgtgtgatatggatgactgtctctcctgtcaaagtcacggctgactggtggacttatacagatcaaattatgctcgtctgtgaaagggggaatactataacyttaaccatagttgtgttcataggttggtttcttggtttcctgtgtctcctgttttcctacatgggaagagatctgccgaaaaattacaatgaggccaaatcaataaccttcagtctaactttgtactatctgacctggattggatatttcacagcatacctctctttcaaaagcaaatacatcatccttttgaatgcactggctcaaatatccagtataaatggaatt(seqidno.2);
snp36序列片段,rad库标签(ctg36503.0),突变位置为c370t:
agtgttccgtgaggaaatggtgcgttgaccacagggctttgattacagaagcggctggtgtaatggagacggaggactctctgttctcctgaggcccccagacgcttcccgcatgtgtttacttaacgcagttagggcttacgccatgcatgtgattaccaagtggctgacacttcatatcgcaaatagacaaactaaacagagaggccacacaacttcaccctaatttttttttggagggaaattctgtggtttggatgtgaaattctgtgaatggtccctgtagagtttaaaatcttcttttttttaatatcttcccccatgtatcagggttcaaagtttgagtgcggactcggaggttgagagctaygcgtccttcatagcccaggctctagaaaagacgcgtggccgtgagtgtgtgccctcttgggaggagatccaggggctgatgggaaggcaggagatactgtgtgctgtgcactaccctggaccgggctgctgccagataccaatcacctcgcacactacggctaatgaggtattgcatcagtcgtttcctctacagcagggattgggaac(seqidno.3);
snp37序列片段,rad库标签(ctg378190.0),突变位置为c374t::
aatcgactctaaagtctggatcggatggacgtgttcgaatgcacacctgtgaccacatataactataataataaaaaaataaaaaaataatttagggcaagtattatgtttttttttttgtttttttggctgttggcaattcagctttgcagttttaaattgcaatatttcacaatatgtctgcttttactgtatttttgatcaaataaatgcagcattggtgagactttaccaaccctaaacttttggccctaaaccagcaggataacgccacaaagcttaaatcatctcaaactggttttttaaacaagacaatgagttcactattctcaaatggcctccacagtctccagatctcaatccaagcgagcacytttgggatgtggtgcaacaggagatttgcataatggatgtgcagctgacaaatctgcagcaactgtgtgatgccatcatgataatatggaccaaactctctgaggaatgattccagaaccttgttgaatttatgccactatgaattaaggcagttctgaaatcaacttggtactagaaagatgtacagtacctaataaagtggccggttagtgtatgtatacacacacacacacacacacacacacacac(seqidno.4).
实施例2生长差异标记的筛选
利用构建的极端大个体20尾和极端小个体20尾样品对筛选出来的84个差异snps标记进行初步验证。在所选择的84个snps位点中,其中40个未检测到snp突变,44个检测到snp突变。将检测到突变型的44个snps在极端大个体组和极端小个体组的差异趋近分析表明,snp24,snp36差异显著(p<0.05),snp37,snp38差异极显著(p<0.01)(见表2)。
表2在极端群体分型的标记
注:“_”表示此位点p<0.05。
实施例3草鱼体重关联的snp标记与体重相关的验证
1.样品dna提取
(1)取待测鱼的血液100ul或剪碎后鳍条组织3mg,加入0.5ml的裂解液(10mmol/ltris-hcl;0.1mol/ledta;0.5%sds;30mg/lrnase;100mg/l蛋白酶k,ph8.0),55℃消化1小时,其间不时轻轻摇动。
(2)加入等体积的酚/氯仿/异戊醇(25:24:1),颠倒混匀,室温下静置5分钟后,12000转/分钟离心10分钟,取上清液,再用氯仿抽提一次,室温下静置5分钟后,12000转/分钟离心10分钟,取上清液。
(3)加入2倍体积的无水乙醇,室温静置10分钟沉淀dna,12000转/分钟离心10分钟。
(4)用70%乙醇洗涤1次,12000转/分钟离心2分钟,吸去上清,室温静置干燥10分钟,加入50μlte(10mmol/ltris-hcl;1mmol/ledta,ph8.0)溶解dna,4℃贮存备用。
2.引物设计和合成
根据草鱼基因片段(seqidno.1~seqidno.4)设计合成了引物对,以及与体重相关的4个snp位点的snapshot引物,引物序列如下:
snp24-f:5'-cgtagtcacgacaggacacgt-3';(seqidno.5)
snp24-r:5'-cagtgattcttctgttaattct-3';(seqidno.6)
snp35-f:5'-gatatgggatgactgtctctcc-3';(seqidno.7)
snp35-r:5'-caatccaggtcagatagtaca-3';(seqidno.8)
snp36-f:5'-tcagggttcaaagtttgagtg-3';(seqidno.9)
snp36-r:5'-gtgcgaggtgattggtatctg-3';(seqidno.10)
snp37-f:5'-gacaatgagttcactattct-3';(seqidno.11)
snp37-r:5'-caaggttctggaatcattcct-3'(seqidno.12)
3.pcr扩增反应体系:
pcr扩增反应条件:
94℃变性15s;54℃退火15s,72℃延伸30s,24个循环;72℃延伸3min。得到pcr扩增反应产物。扩增获得条带大小为423bp、638bp、579bp或624bp的条带。
4.取3ulpcr扩增反应的产物用exoi和sap纯化,去除pcr产物中的引物及dntp。
纯化反应体系:
纯化反应条件:37℃45min,80℃15min,得到纯化的pcr产物。
5.对纯化的pcr产物进行单碱基延伸
反应体系为:
延伸引物序列如下:
snp24-p:5'-tacgctgtgagaaactgtgagg-3';(seqidno.13)
snp35-p:5'-gtgaaagggggaatactataac-3';(seqidno.14)
snp36-p:5'-ttttttttttcggactcggaggttgagagcta-3';(seqidno.15)
snp37-p:5'-agatctcaatccaagcgagcac-3';(seqidno.16)
延伸反应条件:
5.取1μl延伸产物,加8μl上样loading,95℃变性3min,立即冰水浴。
6.草鱼基因型分析
利用测序仪(测序仪型号为abi3730xl)对pcr产物进行snapshot分型,根据测序结果峰图的颜色可判断各个样品草鱼的基因型,草鱼基因seqidno.1第159位碱基处的snp位点是否为cc基因型,如果是cc基因型,则为体重更重、快速增长的草鱼;基因seqidno.2第410位碱基处的snp位点是否为cc基因型,若为cc基因型,则为体重更重、快速增长的草鱼;基因seqidno.3第370位碱基处的snp位点是否为cc基因型,若为cc基因型,则为体重更重、快速增长的草鱼;或/和基因seqidno.4第374位碱基处的snp位点是否为tt基因型,若为tt基因型,则为体重更重、快速增长的草鱼。
利用最小二乘法对筛选到的差异显著的snps与草鱼体重进行关联性分析,在随机群体中与在极端群体验证的结果相同,即这4个snps与生长性状均显著相关(p<0.05)。标记snp24,snp35和snp36标记cc型体重均值均显著高于tt型,而snp37标记tt型体重均值显著高于cc和tc基因型(见表3)。
表34个snps标记与体重性状关联分析
注:相同字母表示两种基因型之间差异不显著(p>0.05),不同字母表示两种基因型差异显著(p<0.05)。
从表3中可知,草鱼基因seqidno.1第159位碱基、基因seqidno.2第410位碱基、基因seqidno.3第370位碱基,或/和基因seqidno.4第374位碱基处的snp位点,与草鱼的体重密切相关。本发明通过检测草鱼的四个snp标记,snp24、snp35、snp36以及snp37,能够在相同养殖条件下有效的挑选体重更大的草鱼。本发明发现snp标记与草鱼的体重能紧密相关,能够有效用于草鱼的分子标记辅助育种。进而能够根据实际育种需求对草鱼亲本进行基因型鉴定,挑选合适的基因型草鱼亲本进行繁殖,可以获得生长较快的草鱼后代(鱼苗),对快长草鱼苗种生产有积极意义,并可以大大节约育种时间,成本低廉,准确性高,加快草鱼育种进程。
上述实施例为本发明较佳的实施方式,但本发明的实施方式并不受上述实施例的限制,其他的任何未背离本发明的精神实质与原理下所作的改变、修饰、替代、组合、简化,均应为等效的置换方式,都包含在本发明的保护范围之内。
sequencelisting
<110>中国水产科学研究院珠江水产研究所
<120>与草鱼性状相关的snp分子标记及其应用
<130>
<160>16
<170>patentinversion3.5
<210>1
<211>423
<212>dna
<213>人工序列
<400>1
atcggtccacgtaactctcccgaaagcctttccgcaccgcgcgggccatactgaccaccg60
tagtcacgacaggacacgtgctgaaacccaagaggacgggatgatggtctttaattcatt120
aacatgacgagtgctgtgtgtgtgtgtgtgtgtttgttycctcacagtttctcacagcgt180
atgccgctcgtccccgtcggacacacacaggtgttcagagggccgcaggcagcaccatgt240
tggcagggaggaacacacggtgtagtgacagctgcacacacacacacaattacatgatca300
cacactacatcaaaacaccacagaattaacagaagaatcactgcaaatgaaactgaaact360
gaaaaaatgaatgtcatatgaatgattcaatgattaatgatgtcataatgaaaggattat420
aat423
<210>2
<211>638
<212>dna
<213>人工序列
<400>2
attacaacacaccagtggtgaggtcagctggtggtagcatgtgtttcctgatgttgatta60
gtttgattttgtctagtataagtgcattctttttctttggagaacccacatctgcacttt120
gcctcctgcgaaatgccatatttgcatttttcttcactgtctgtatttcctgtttgactg180
tccgttcctttcaaattgtttgtgtttttaaaatggctgctcagttccctaaggtgcaca240
gcctttgggtaaagcacaatggccagtggctcttcattgcatttacttctgtcattcatt300
taatttcttgtgtgatatggatgactgtctctcctgtcaaagtcacggctgactggtgga360
cttatacagatcaaattatgctcgtctgtgaaagggggaatactataacyttaaccatag420
ttgtgttcataggttggtttcttggtttcctgtgtctcctgttttcctacatgggaagag480
atctgccgaaaaattacaatgaggccaaatcaataaccttcagtctaactttgtactatc540
tgacctggattggatatttcacagcatacctctctttcaaaagcaaatacatcatccttt600
tgaatgcactggctcaaatatccagtataaatggaatt638
<210>3
<211>579
<212>dna
<213>人工序列
<400>3
agtgttccgtgaggaaatggtgcgttgaccacagggctttgattacagaagcggctggtg60
taatggagacggaggactctctgttctcctgaggcccccagacgcttcccgcatgtgttt120
acttaacgcagttagggcttacgccatgcatgtgattaccaagtggctgacacttcatat180
cgcaaatagacaaactaaacagagaggccacacaacttcaccctaatttttttttggagg240
gaaattctgtggtttggatgtgaaattctgtgaatggtccctgtagagtttaaaatcttc300
ttttttttaatatcttcccccatgtatcagggttcaaagtttgagtgcggactcggaggt360
tgagagctaygcgtccttcatagcccaggctctagaaaagacgcgtggccgtgagtgtgt420
gccctcttgggaggagatccaggggctgatgggaaggcaggagatactgtgtgctgtgca480
ctaccctggaccgggctgctgccagataccaatcacctcgcacactacggctaatgaggt540
attgcatcagtcgtttcctctacagcagggattgggaac579
<210>4
<211>624
<212>dna
<213>人工序列
<400>4
aatcgactctaaagtctggatcggatggacgtgttcgaatgcacacctgtgaccacatat60
aactataataataaaaaaataaaaaaataatttagggcaagtattatgtttttttttttg120
tttttttggctgttggcaattcagctttgcagttttaaattgcaatatttcacaatatgt180
ctgcttttactgtatttttgatcaaataaatgcagcattggtgagactttaccaacccta240
aacttttggccctaaaccagcaggataacgccacaaagcttaaatcatctcaaactggtt300
ttttaaacaagacaatgagttcactattctcaaatggcctccacagtctccagatctcaa360
tccaagcgagcacytttgggatgtggtgcaacaggagatttgcataatggatgtgcagct420
gacaaatctgcagcaactgtgtgatgccatcatgataatatggaccaaactctctgagga480
atgattccagaaccttgttgaatttatgccactatgaattaaggcagttctgaaatcaac540
ttggtactagaaagatgtacagtacctaataaagtggccggttagtgtatgtatacacac600
acacacacacacacacacacacac624
<210>5
<211>21
<212>dna
<213>人工引物
<400>5
cgtagtcacgacaggacacgt21
<210>6
<211>22
<212>dna
<213>人工引物
<400>6
cagtgattcttctgttaattct22
<210>7
<211>22
<212>dna
<213>人工引物
<400>7
gatatgggatgactgtctctcc22
<210>8
<211>21
<212>dna
<213>人工引物
<400>8
caatccaggtcagatagtaca21
<210>9
<211>21
<212>dna
<213>人工引物
<400>9
tcagggttcaaagtttgagtg21
<210>10
<211>21
<212>dna
<213>人工引物
<400>10
gtgcgaggtgattggtatctg21
<210>11
<211>20
<212>dna
<213>人工引物
<400>11
gacaatgagttcactattct20
<210>12
<211>21
<212>dna
<213>人工引物
<400>12
caaggttctggaatcattcct21
<210>13
<211>22
<212>dna
<213>人工引物
<400>13
tacgctgtgagaaactgtgagg22
<210>14
<211>22
<212>dna
<213>人工引物
<400>14
gtgaaagggggaatactataac22
<210>15
<211>32
<212>dna
<213>人工引物
<400>15
ttttttttttcggactcggaggttgagagcta32
<210>16
<211>22
<212>dna
<213>人工引物
<400>16
agatctcaatccaagcgagcac22
- 还没有人留言评论。精彩留言会获得点赞!