用于结核分枝杆菌复合群的CRISPR检测引物组及其用途的制作方法
本发明涉及基于crispr技术的基因检测技术领域,特别是涉及一种用于结核分枝杆菌复合群的crispr检测引物组及其用途。
背景技术:
结核病(tuberculosis,tb)是全世界死亡人口的十大死因之一。据世界卫生组织(worldhealthorganization,who)报告,2016年全球估计新发结核病1040万例,其中仅有630万例得到发现和报告,尚存在410万例的差距(数据源于《2017年全球结核病报告》)。因此,结核病的防控关系到国家的公共卫生安全,而控制结核病传播的关键就是早期准确诊断。
结核病的致病菌是结核分枝杆菌复合群(mycobacteriumtuberculosiscomplex,mtbc),包括结核分枝杆菌(m.tuberculosis)、牛分枝杆菌(m.bovis)、非洲分枝杆菌(m.africanum)、坎纳分支杆菌(m.canettii)和田鼠分枝杆菌(m.microti)等。
目前结核病的诊断主要依赖于对病原体的检查,常用方法为涂片染色镜检、分离培养、免疫学诊断和分子生物学诊断等。其中,分离培养法是目前诊断结核的金标准,但培养需要4~8周的时间,延误临床诊断与治疗。涂片染色镜检法操作简单,快速,但该法灵敏度低、特异性差。免疫学诊断则由于现有的抗原或抗体与其它微生物存在交叉,导致其特异性较差、假阳性率高。分子生物学诊断具有快速、灵敏的优势,且利用特异性的dna片段还可以区分结核分枝杆菌复合体内菌种。who推荐使用的genexpert全自动检测系统采用pcr技术对结核分枝杆菌复合群进行检测,灵敏度特异性均较好,且不需长时间等待结果,但是仪器设备价格昂贵,难以在中低收入地区普及。近年也出现诸如lamp,rpa等多种恒等温扩增技术,可用于现场检测,但均存在缺乏较为有效的扩增产物检测手段等问题。因此,亟需建立一种可应用于现场、简易、快速、且高灵敏度的检测技术。
重组酶扩增技术(rpa,recombinasepolymeraseamplification,重组酶聚合酶等温扩增技术)是近年兴起的一种恒温扩增技术,通过三种酶的混合物,即能结合单链核酸(寡核苷酸引物)的重组酶、单链dna结合蛋白(ssb)和链置换dna聚合酶,在常温下也有活性,最佳反应温度在37℃左右,整个过程一般可在十分钟之内获得可检出水平的扩增产物。但是,从原理上,其单级扩增反应的本质并未改变,相较于目前市场上多数的实时定量pcr(q-pcr)检测方式,并不能有实质性的灵敏度提升。
技术实现要素:
基于此,有必要针对上述问题,提供一种用于结核分枝杆菌复合群的crispr检测引物组,将此引物组用于基于crispr技术的基因检测中,可快速现场检测结核分枝杆菌复合群(结核分枝杆菌、牛分枝杆菌、非洲分枝杆菌、坎纳分支杆菌、田鼠分枝杆菌),具有特异高、灵敏高、简易的优点。
一种用于结核分枝杆菌复合群的crispr检测引物组,包括扩增引物对和crrna;所述扩增引物对用于扩增结核分枝杆菌、牛分枝杆菌、非洲分枝杆菌、坎纳分支杆菌和田鼠分枝杆菌的共有保守序列;所述crrna包括锚定序列和向导序列,所述锚定序列与cas蛋白结合,所述向导序列与所述共有保守序列中的靶向dna片段相匹配。
上述引物组,针对结核分枝杆菌、牛分枝杆菌、非洲分枝杆菌、坎纳分支杆菌和田鼠分枝杆菌的共有保守序列进行设计,且利用crispr(clusteredregularlyinterspacedshortpalindromicrepeats,成簇规律间隔短回文重复序列)技术进行检测,在crisprcas系统中,cas蛋白在crrna(crispr-derivedrna)的引导下,识别目标序列后启动“附带切割”活性。在体系中加入荧光报告分子,借用cas酶附带切割活性,实现待检序列信息向荧光信号的转化。通过rpa与cas蛋白的偶联,能够实现“序列扩增”(rpa完成)加上“酶促级联”(cas酶完成)的两级放大,从而超越q-pcr这种单级扩增的灵敏度。此外,因为rpa扩增方式无需复杂的温度改变,从而摆脱了对q-pcr仪等精密仪器的依赖,使得crispr-cas技术在结核病的现场诊断方面具有广阔的应用前景
在其中一个实施例中,所述共有保守序列如seqidno:1所示。发明人在前期研究基础上,经过多番筛选和比对,发现seqidno:1所示共有保守序列既可特异性的用于结核分枝杆菌复合群的检测,又可以crispr-cas技术高效检出。
在其中一个实施例中,所述向导序列如seqidno:2所示。选取seqidno:2所示序列为向导序列,也即检测的靶向序列,seqidno:2序列为aucagcucggucuuguauag,具有较好的检测效果。
在其中一个实施例中,所述crrna序列如seqidno:3所示。seqidno:3所示序列的构成为:5’-锚定序列-向导序列-3’,以上述向导序列配合lbcas12a的锚定序列5’-uaauuucuacuaaguguagau-3’,即seqidno:3序列为uaauuucuacuaaguguagauaucagcucggucuuguauag,具有较好的检测效果。
在其中一个实施例中,所述扩增引物对如下:
正向扩增引物:5’-ggtcggaagctcctatgacaatgcactagcc-3’(seqidno:4)
反向扩增引物:5’-ttgagcgtagtaggcagcctcgagttcgac-3’(seqidno:5)。
本发明人通过大量实验研究后发现,以上述扩增引物对进行rpa扩增再配合上述crrna,具有较好的扩增效率和灵敏度。
本发明还公开了上述用于结核分枝杆菌复合群的crispr检测引物组在开发和/或制备具有结核诊断和/或预后评估用途的产品中的应用。
可以理解的,上述产品可以是试剂盒,也可以是一体化检测设备等。
本发明还公开了一种用于检测结核分枝杆菌复合群的试剂盒,包括上述的用于结核分枝杆菌复合群的crispr检测引物组。
在其中一个实施例中,该试剂盒还包括cas12a蛋白,信号报告探针。所述cas12a蛋白可来源于lbcas12a、ascas12a和fncas12a等,但需根据不同来源蛋白调整其锚定序列和其它试剂成分。
在其中一个实施例中,所述cas12a蛋白为lbcas12a;所述信号报告探针序列为:5’-6-fam-tttttttttttt-bhq1-3’。以上述引物组配合lbcas12a和特定探针序列,具有较好的检测效果。
本发明还公开了一种非诊断治疗目的结核分枝杆菌复合群检测方法,包括以下步骤:
样本提取:取待测样本,提取其中dna;
rpa扩增:以上述扩增引物,通过rpa方法扩增上述提取得到的待测样本dna,得扩增产物;
crispr反应检测:取上述扩增产物,加入信号报告探针、cas蛋白和上述crrna,进行crispr反应检测,读取检测信号,即得。
上述检测步骤均在恒温条件完成,无需复杂的温度改变,从而摆脱了对q-pcr仪等精密仪器的依赖,具有较为广阔的应用前景。
与现有技术相比,本发明具有以下有益效果:
本发明的一种用于结核分枝杆菌复合群的crispr检测引物组,针对结核分枝杆菌、牛分枝杆菌、非洲分枝杆菌、坎纳分支杆菌和田鼠分枝杆菌的共有保守序列进行设计,且利用crispr技术进行检测,在crisprcas系统中,cas蛋白在向导rna的引导下,识别目标序列后启动“附带切割”活性。在体系中加入荧光报告分子,借用cas酶附带切割活性,实现待检序列信息向荧光信号的转化。通过rpa与cas蛋白的偶联,能够实现“序列扩增”(rpa完成)加上“酶促级联”(cas酶完成)的两级放大,从而超越q-pcr这种单级扩增的灵敏度。此外,因为rpa扩增方式无需复杂的温度改变,从而摆脱了对q-pcr仪等精密仪器的依赖,使得crispr-cas技术在结核病的现场诊断方面具有广阔的应用前景。
并且,本发明人还通过对靶标序列选定、扩增引物对和crrna的设计和筛选,最终得到了具有扩增效率高、灵敏度好且特异性强的可临床实用的引物组,缩短了结核分枝杆菌复合群的检测时间,可在90min内完成检测,并且最低可以检测5cfu/ml的结核分枝杆菌符合群,与非结核分枝杆菌、肺炎链球菌等其他人体常见致病菌均无交叉反应,极大的提高了crispr-cas技术在结核病现场诊断的实操性。
附图说明
图1为实施例1中引物和crrna序列扩增效率筛选结果示意图;
图2为实施例1中引物和crrna序列灵敏度筛选结果示意图;
图3为实施例1中特异性检测结果示意图。
具体实施方式
为了便于理解本发明,下面将参照相关附图对本发明进行更全面的描述。附图中给出了本发明的较佳实施例。但是,本发明可以以许多不同的形式来实现,并不限于本文所描述的实施例。相反地,提供这些实施例的目的是使对本发明的公开内容的理解更加透彻全面。
除非另有定义,本文所使用的所有的技术和科学术语与属于本发明的技术领域的技术人员通常理解的含义相同。本文中在本发明的说明书中所使用的术语只是为了描述具体的实施例的目的,不是旨在于限制本发明。本文所使用的术语“和/或”包括一个或多个相关的所列项目的任意的和所有的组合。
以下实施例所涉及材料及仪器如下:
1、实验材料:
结核分枝杆菌(m.tuberculosish37ra株)、牛分枝杆菌(m.bovisatcc19210)、非洲分枝杆菌(m.africanum)、坎纳分支杆菌(m.canettii)、田鼠分枝杆菌(m.microti)dna提取物由上海华山医院提供。15种非特异性菌株(鸟分枝杆菌、土地分枝杆菌、施氏分枝杆菌、堪萨斯分枝杆菌、亚洲分枝杆菌、瘰疬分枝杆菌、戈登分枝杆菌、龟脓分枝杆菌、偶然分枝杆菌、草分枝杆菌、巴西诺卡氏菌、北京棒杆菌、嗜肺军团菌、肺炎链球菌和百日咳博德特氏菌)购自中国食品药品检定研究院。
引物与信号报告探针由上海生工生物有限公司合成,crrna由金斯瑞生物科技有限公司合成,lbcas12a蛋白由金斯瑞生物科技有限公司表达纯化。其他生化试剂均为进口分装或国产分析纯。
2、实验仪器
金属浴,离心机,涡旋仪,荧光检测仪器等。
实施例1
结核分枝杆菌复合群crispr检测引物序列的设计和筛选。
一、序列的设计。
1、靶标序列选定。
发明人在前期研究基础上,经过多番筛选和比对,选定如seqidno:1所示的序列作为结核分枝杆菌(m.tuberculosis)、牛分枝杆菌(m.bovis)、非洲分枝杆菌(m.africanum)、坎纳分支杆菌(m.canettii)和田鼠分枝杆菌(m.microti)的靶标序列,该序列作为上述菌种的保守序列,可将上述结核致病菌检出。seqidno:1序列如下:
aaagaccgcgtcggctttcttcgcggccgagctcgaccggccagcacgctaattacccggttcatcgccgatcatcagggccaccgcgagggccccgatggtttgcggtggggtgtcgagtcgatctgcacacagctgaccgagctgggtgtgccgatcgccccatcgacctactacgaccacatcaaccgggagcccagccgccgcgagctgcgcgatggcgaactcaaggagcacatcagccgcgtccacgccgccaactacggtgtttacggtgcccgcaaagtgtggctaaccctgaaccgtgagggcatcgaggtggccagatgcaccgtcgaacggctgatgaccaaactcggcctgtccgggaccacccgcggcaaagcccgcaggaccacgatcgctgatccggccacagcccgtcccgccgatctcgtccagcgccgcttcggaccaccagcacctaaccggctgtgggtagcagacctcacctatgtgtcgacctgggcagggttcgcctacgtggcctttgtcaccgacgcctacgctcgcaggatcctgggctggcgggtcgcttccacgatggccacctccatggtcctcgacgcgatcgagcaagccatctggacccgccaacaagaaggcgtactcgacctgaaagacgttatccaccatacggataggggatctcagtacacatcgatccggttcagcgagcggctcgccgaggcaggcatccaaccgtcggtcggagcggtcggaagctcctatgacaatgcactagccgagacgatcaacggcctatacaagaccgagctgatcaaacccggcaagccctggcggtccatcgaggatgtcgagttggccaccgcgcgctgggtcgactggttcaaccatcgccgcctctaccagtactgcggcgacgtcccgccggtcgaactcgaggctgcctactacgctcaacgccagagaccagccgccggctga
2、扩增引物对和crrna的设计。
针对上述共有保守序列,设计多条crrna和相应的扩增引物对,下表列出部分示例性引物序列。
表1.crrna和引物序列
二、序列的筛选。
1、扩增效率筛选
1.1方法
利用平板计数法计算结核分枝杆菌(m.tuberculosis)菌液浓度,分别以10倍倍比稀释成5.0×104-5.0×10-1cfu/ml菌液,取5.0×103cfu/ml菌液1ml,采用细菌基因组dna提取试剂盒(天根生化科技(北京)有限公司,货号dp320)提取菌液基因组dna,用20μlte缓冲液溶解,分别取2μl基因组dna为模板进行rpa扩增,同时以鸟分枝杆菌核酸dna为阴性对照,按照以下操作进行:
a.rpa扩增
扩增体系:
rpa反应体系为50μl:其中包括rpa上游引物0.5-2.5μl(浓度10μm),rpa下游引物0.5-2.5μl(浓度10μm),rpa酶预混液41.5μl(其中含有:磷酸肌酸(浓度20-80mm)、肌酸激酶(浓度50-150mm)、dntps(浓度100-300μm)、atp(浓度20-80mm)、dtt(浓度1-10mm)、醋酸钾(浓度50-200mm)、重组酶uxsx(50-300ng/μl)、uxsy(10-100ng/μl)、单链结合蛋白(200-1000ng/μl)、bsu聚合酶(10-100ng/μl)),醋酸镁0.5-2μl(浓度280mm),待测样本基因组dna2μl。
扩增程序:恒温37℃反应15-60min。
b.基于cas12a的crispr反应
crispr反应体系:
上述rpa扩增产物(22μl),lbcas12a蛋白1μl(浓度1-5μm),crrna1μl(浓度1-5μm),信号报告探针1μl(5’6-fam-tttttttttttt-bhq1-3’,浓度1-10μm)。
反应条件和时间:37℃反应30-60min,每1min读取fam荧光值。
结果分析:使用荧光检测仪得到的累积荧光值作为信号强度,按照以下标准进行分析判定:
阴性判断标准:荧光量小于或等于阴性对照荧光量的2倍。
阳性判断标准:荧光量大于阴性对照荧光量的2倍。
阴性对照组为每个实验组对应设置的以加入非结核分枝杆菌(鸟分枝杆菌)核酸dna作为模板的阴性信号组。
1.2结果
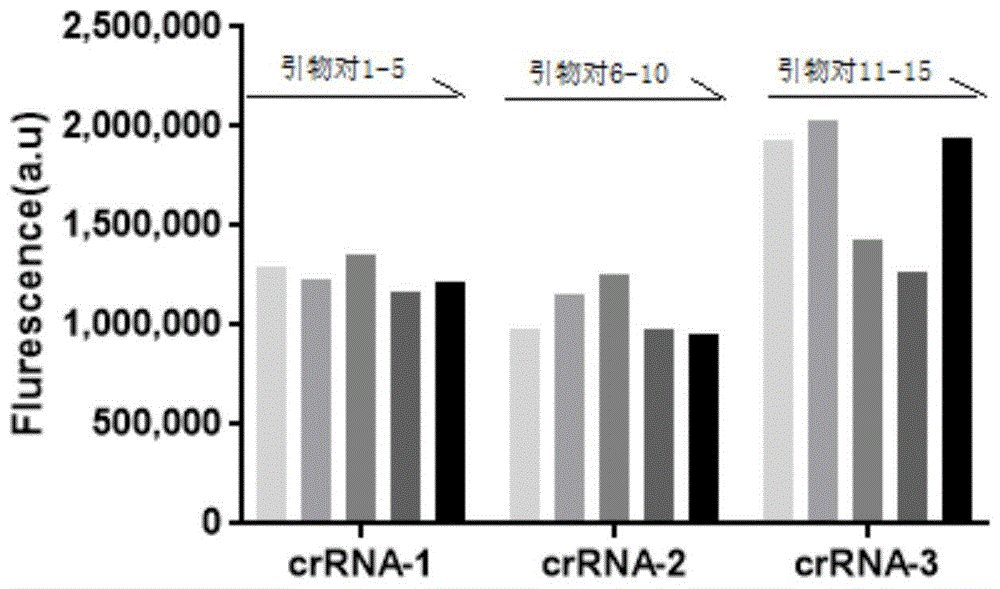
对上表中特异性crrna及其对应的各5对引物对组合进行了检测筛选,最终选出一组扩增效率好的特异性crrna-3序列,筛选结果如图1所示。
结果表示,采用crrna-3配合引物对12具有最佳的扩增效率。
2、灵敏度筛选
1.1方法
利用平板计数法计算结核分枝杆菌菌液浓度,分别以10倍倍比稀释成5.0×104-5.0×10-1cfu/ml菌液,取相应稀释倍数的菌液各1ml,采用细菌基因组dna提取试剂盒(天根生化科技(北京)有限公司)提取各稀释倍数的菌液基因组dna,用20μlte溶解,分别取2μl基因组dna为模板进行rpa扩增,同时以鸟分枝杆菌核酸dna为阴性对照,对筛选到的扩增效率较好的crrna-3序列及相应的引物对11,12和15进行灵敏度筛选,按照上述方法进行检测。
1.2结果
结果如图2所示,以crrna-3序列(seqidno:3)及相应的引物对12(seqidno:4-5),最低可以检测5×100cfu/ml的结核分枝杆菌复合群,具有最佳的灵敏度。
3、特异性考察
1.1方法
选取crrna-3序列(seqidno:3)及相应的引物对12(seqidno:4-5),按照上述方法对结核分枝杆菌(m.tuberculosis)、牛分枝杆菌(m.bovis)、非洲分枝杆菌(m.africanum)、坎纳分支杆菌(m.canettii)、田鼠分枝杆菌(m.microti)基因组dna和15种非特异性菌株(鸟分枝杆菌、土地分枝杆菌、施氏分枝杆菌、堪萨斯分枝杆菌、亚洲分枝杆菌、瘰疬分枝杆菌、戈登分枝杆菌、龟脓分枝杆菌、偶然分枝杆菌、草分枝杆菌、巴西诺卡氏菌、北京棒杆菌、嗜肺军团菌、肺炎链球菌和百日咳博德特氏菌)进行扩增,同时以鸟分枝杆菌核酸dna为阴性对照,检验本方法的特异性。
1.2结果
结果如图3所示,图中nc表示以双蒸水作为模板的阴性对照,本发明方法仅对结核分枝杆菌复合群(人型结核分枝杆菌、牛型结核分枝杆菌、非洲分枝杆菌、坎纳分支杆菌、田鼠分枝杆菌)样品有扩增,而其它的非结核分枝杆菌则无扩增,表明该方法特异性好。
实施例2
一种用于检测结核分枝杆菌复合群的试剂盒,包括:
(1)rpa扩增体系:
rpa扩增引物对:
正向:5’-ggtcggaagctcctatgacaatgcactagcc-3’(seqidno:4),浓度为0.1~1μm
反向:5’-ttgagcgtagtaggcagcctcgagttcgac-3’(seqidno:5),浓度为0.1~1μm。
rpa酶预混液:磷酸肌酸(浓度20-80mm)、肌酸激酶(浓度50-150mm)、dntps(浓度100-300μm)、atp(浓度20-80mm)、dtt(浓度1-10mm)、醋酸钾(浓度50-200mm)、重组酶uxsx(50-300ng/μl)、uxsy(10-100ng/μl)、单链结合蛋白(200-1000ng/μl)、bsu聚合酶(10-100ng/μl)等。
醋酸镁溶液:浓度为10-20mm
(2)crispr反应体系:
crrna:5’-uaauuucuacuaaguguagauaucagcucggucuuguauag-3’(seqidno:3),浓度为20-100nm
lbcas12a蛋白:浓度为20-100nm。
信号报告探针:5’6-fam-tttttttttttt-bhq1-3’,浓度为50-500nm。
采用上述试剂盒进行结核分枝杆菌复合群检测的方法如下:
(1)检测样本的dna提取
检测样本可以是结核分枝杆菌复合群菌株,也可以是临床样本(主要包括组织样本、痰液、脑脊液、胸腹水、尿液、脓液、血液样本等)或是其它科研实验样本。
(2)通过rpa技术对病原体核酸进行扩增
扩增体系:
rpa反应体系为50μl:其中包括rpa上游引物0.5-2.5μl(浓度10μm),rpa下游引物0.5-2.5μl(浓度10μm),rpa酶预混液41.5μl,醋酸镁14mm,待测样本基因组dna2μl。
扩增程序:恒温37℃反应15-60min。
(3)基于cas12a的crispr反应
上述rpa扩增产物(22μl),lbcas12a蛋白1μl(浓度1-5μm),crrna1μl(浓度1-5μm),信号报告探针1μl(5’6-fam-tttttttttttt-bhq1-3’,浓度1-10μm)。
反应条件和时间:37℃反应30-60min,每1min读取fam荧光值。
(4)结果分析:
上述检测过程中,由于在反应体系中加入两端分别连接荧光物质和淬灭剂的信号报告探针,当cas12a蛋白在crrna的帮助下,识别具有靶向序列的靶向dna后,被激活的cas12a酶可以降解该带有信号的信号报告探针,从而释放荧光信号,实现检测。
使用荧光检测仪得到的累积荧光值作为信号强度,按照以下标准进行分析判定:
阴性判断标准:荧光量小于或等于阴性对照荧光量的2倍。
阳性判断标准:荧光量大于阴性对照荧光量的2倍。
其中,阴性对照组为每个实验组对应设置的以加入非结核分枝杆菌(鸟分枝杆菌)核酸dna作为模板的阴性信号组。
实施例3
以上述实施例2的试剂盒进行临床样品的结核分枝杆菌复合群检测。
1.方法
(1)样本来源:上海华山医院感染科。
临床样本共120例,用市场上广泛认可的genexpert检测系统及本发明方法进行盲检测。
(2)检测方法:
参考实施例2中的本发明的相关检测步骤对120例样本进行检测。
参考genexpert检测系统相关操作说明书检测步骤对120例样本进行检测。
2.结果
使用两种方法的检测结果如下表所示:
表2不同方法对临床样品检测结果
经过计算可知:
阳性符合率=97.06%
阴性符合率=100%
总符合率=99.17%
总体预测值95%可信区间为[0.9389~1.0197]
kappa值=0.9793
上述结果说明本发明检测方法与genexpert检测方法具有较高的检测效果一致性。
而上述结果中一例本发明crispr检测结果为阴性,而genexpert检测结果为阳性的案例,经分析,可能是由于使用的样本量不同造成,因genexpert使用原始样本量均多于0.5ml,提取的核酸全部用来扩增检测,但是本发明的crispr方法,在提取样本基因组dna后,只取了2μl进行检测;同时,一个样本的差异,也可能是有一些其它不明原因导致。
而从灵敏度考虑,本发明的方法,最低可以检测5×100cfu/ml的结核分枝杆菌复合群(实施例1结果),而按照genexpert说明书(
以上所述实施例的各技术特征可以进行任意的组合,为使描述简洁,未对上述实施例中的各个技术特征所有可能的组合都进行描述,然而,只要这些技术特征的组合不存在矛盾,都应当认为是本说明书记载的范围。
以上所述实施例仅表达了本发明的几种实施方式,其描述较为具体和详细,但并不能因此而理解为对发明专利范围的限制。应当指出的是,对于本领域的普通技术人员来说,在不脱离本发明构思的前提下,还可以做出若干变形和改进,这些都属于本发明的保护范围。因此,本发明专利的保护范围应以所附权利要求为准。
序列表
<110>广州微远基因科技有限公司
<120>用于结核分枝杆菌复合群的crispr检测引物组及其用途
<160>35
<170>siposequencelisting1.0
<210>1
<211>987
<212>dna
<213>结核分枝杆菌(mycobacteriumtuberculosis)
<400>1
aaagaccgcgtcggctttcttcgcggccgagctcgaccggccagcacgctaattacccgg60
ttcatcgccgatcatcagggccaccgcgagggccccgatggtttgcggtggggtgtcgag120
tcgatctgcacacagctgaccgagctgggtgtgccgatcgccccatcgacctactacgac180
cacatcaaccgggagcccagccgccgcgagctgcgcgatggcgaactcaaggagcacatc240
agccgcgtccacgccgccaactacggtgtttacggtgcccgcaaagtgtggctaaccctg300
aaccgtgagggcatcgaggtggccagatgcaccgtcgaacggctgatgaccaaactcggc360
ctgtccgggaccacccgcggcaaagcccgcaggaccacgatcgctgatccggccacagcc420
cgtcccgccgatctcgtccagcgccgcttcggaccaccagcacctaaccggctgtgggta480
gcagacctcacctatgtgtcgacctgggcagggttcgcctacgtggcctttgtcaccgac540
gcctacgctcgcaggatcctgggctggcgggtcgcttccacgatggccacctccatggtc600
ctcgacgcgatcgagcaagccatctggacccgccaacaagaaggcgtactcgacctgaaa660
gacgttatccaccatacggataggggatctcagtacacatcgatccggttcagcgagcgg720
ctcgccgaggcaggcatccaaccgtcggtcggagcggtcggaagctcctatgacaatgca780
ctagccgagacgatcaacggcctatacaagaccgagctgatcaaacccggcaagccctgg840
cggtccatcgaggatgtcgagttggccaccgcgcgctgggtcgactggttcaaccatcgc900
cgcctctaccagtactgcggcgacgtcccgccggtcgaactcgaggctgcctactacgct960
caacgccagagaccagccgccggctga987
<210>2
<211>20
<212>rna
<213>人工序列(artificialsequence)
<400>2
aucagcucggucuuguauag20
<210>3
<211>41
<212>rna
<213>人工序列(artificialsequence)
<400>3
uaauuucuacuaaguguagauaucagcucggucuuguauag41
<210>4
<211>31
<212>dna
<213>人工序列(artificialsequence)
<400>4
ggtcggaagctcctatgacaatgcactagcc31
<210>5
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>5
ttgagcgtagtaggcagcctcgagttcgac30
<210>6
<211>42
<212>rna
<213>人工序列(artificialsequence)
<400>6
uaauuucuacuaaguguagauucaccgacgccuacgcucgca42
<210>7
<211>39
<212>dna
<213>人工序列(artificialsequence)
<400>7
ctgatccggccacagcccgtcccgccgatctcgtccagc39
<210>8
<211>35
<212>dna
<213>人工序列(artificialsequence)
<400>8
gaggaccatggaggtggccatcgtggaagcgaccc35
<210>9
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>9
ccacagcccgtcccgccgatctcgtccagc30
<210>10
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>10
gccatcgtggaagcgacccgccagcccagg30
<210>11
<211>32
<212>dna
<213>人工序列(artificialsequence)
<400>11
gctgatgaccaaactcggcctgtccgggacca32
<210>12
<211>39
<212>dna
<213>人工序列(artificialsequence)
<400>12
tggtggataacgtctttcaggtcgagtacgccttcttgt39
<210>13
<211>32
<212>dna
<213>人工序列(artificialsequence)
<400>13
tcgaacggctgatgaccaaactcggcctgtcc32
<210>14
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>14
ccgtatggtggataacgtctttcaggtcgagtacgccttc40
<210>15
<211>33
<212>dna
<213>人工序列(artificialsequence)
<400>15
ttcggaccaccagcacctaaccggctgtgggta33
<210>16
<211>38
<212>dna
<213>人工序列(artificialsequence)
<400>16
ggataacgtctttcaggtcgagtacgccttcttgttgg38
<210>17
<211>42
<212>rna
<213>人工序列(artificialsequence)
<400>17
uaauuucuacuaaguguagauuagcagaccucaccuaugugu42
<210>18
<211>32
<212>dna
<213>人工序列(artificialsequence)
<400>18
tgaaccgtgagggcatcgaggtggccagatgc32
<210>19
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>19
gatcctgcgagcgtaggcgtcggtgacaaaggccacgtag40
<210>20
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>20
gccacagcccgtcccgccgatctcgtccag30
<210>21
<211>32
<212>dna
<213>人工序列(artificialsequence)
<400>21
agatggcttgctcgatcgcgtcgaggaccatg32
<210>22
<211>35
<212>dna
<213>人工序列(artificialsequence)
<400>22
gaacggctgatgaccaaactcggcctgtccgggac35
<210>23
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>23
tcctgcgagcgtaggcgtcggtgacaaagg30
<210>24
<211>32
<212>dna
<213>人工序列(artificialsequence)
<400>24
tcgaacggctgatgaccaaactcggcctgtcc32
<210>25
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>25
catcgtggaagcgacccgccagcccaggat30
<210>26
<211>32
<212>dna
<213>人工序列(artificialsequence)
<400>26
cggctgatgaccaaactcggcctgtccgggac32
<210>27
<211>32
<212>dna
<213>人工序列(artificialsequence)
<400>27
atcgcgtcgaggaccatggaggtggccatcgt32
<210>28
<211>32
<212>dna
<213>人工序列(artificialsequence)
<400>28
cctatgacaatgcactagccgagacgatcaac32
<210>29
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>29
ctggtctctggcgttgagcgtagtaggcag30
<210>30
<211>36
<212>dna
<213>人工序列(artificialsequence)
<400>30
gctcctatgacaatgcactagccgagacgatcaacg36
<210>31
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>31
gccaactcgacatcctcgatggaccgccag30
<210>32
<211>35
<212>dna
<213>人工序列(artificialsequence)
<400>32
ggaagctcctatgacaatgcactagccgagacgat35
<210>33
<211>32
<212>dna
<213>人工序列(artificialsequence)
<400>33
tggtagaggcggcgatggttgaaccagtcgac32
<210>34
<211>34
<212>dna
<213>人工序列(artificialsequence)
<400>34
tcctatgacaatgcactagccgagacgatcaacg34
<210>35
<211>35
<212>dna
<213>人工序列(artificialsequence)
<400>35
acgtcgccgcagtactggtagaggcggcgatggtt35
- 还没有人留言评论。精彩留言会获得点赞!
- 一种敲除c‑di‑AMP分解酶的重组耻垢分枝杆菌株及其应用的制造方法与工艺
- 检测结核分枝杆菌感染的抗原多肽池及其应用的制造方法与工艺
- 一种猕猴桃分枝定位器的制造方法与工艺
- 一种xanthone类化合物及其单晶的制备方法与作为抗海分枝杆菌药物的应用与制造工艺
- 一种糖肽类抗生素耐药基因检测试剂盒的制造方法与工艺
- 分枝杆菌快速分离培养方法与制造工艺
- 一种用于检测潜在结核的特异性标志物及其应用的制造方法与工艺
- 结核分枝杆菌抗原蛋白Rv2941及其T细胞表位肽的应用的制造方法与工艺
- 一种微流控芯片上基于荧光量子点磁性富集并分离结核分枝杆菌TB的方法及装置与制造工艺
- 二肽meso‑DAP‑D‑Ala和/或四肽L‑Ala‑D‑Glu‑L‑Lys‑D‑Ala的新用途的制造方法与工艺