一种全人源抗huTIGIT单克隆抗体及其应用的制作方法
本发明涉及基因工程产品,即公开了几株具有高亲和力和阻断活性的全人源抗hutigit特异性单克隆抗体。本发明构建了大容量的全合成人源噬菌体抗体库,从中筛选并优化获得了4株优选的全人源抗人tigit抗体tbb8、tdc8、3tb3和5tb10。本发明的优选抗体d1y1a有较高的亲和力和阻断活性,能够有效地识别人tigit抗原。
背景技术:
近年来,治疗性抗体显示出越来越广阔的应用前景。但由于人抗鼠抗体(humananti-mouseantibody,hama)的产生使其应用受到很大限制。抗体库技术的出现为这一问题的解决提供了一条新的途径,为各种不同免疫原包括自身抗原的抗体获得提供了一条有效途径。抗体库技术是20世纪90年代抗体工程领域的重大进展,它的出现得益于三大技术的发展,即pcr扩增技术、大肠杆菌表达系统的优化及噬菌体展示技术,尤其是噬菌体展示技术是实现抗体库技术的重要前提。
噬菌体展示蛋白质工程或分子进化是蛋白质工程中的一个富有潜力的崭新领域,该技术的显著优点,一是表达的融合蛋白或肽类直接与编码它们的基因相关联,将活的噬菌体繁殖扩增即可鉴定和分析结合肽或蛋白的序列,其二,噬菌体展示肽或蛋白的结构和功能常常与天然肽或蛋白具有一样或极为相似的结构和功能,这一技术已成为基础研究和药物筛选的重要手段。噬菌体抗体库技术避免了传统的从杂交瘤细胞中筛选鼠源单抗,再进行人源化改造的繁琐过程,可在较短时间内,得到完全的人源抗体片段,而且无需经过免疫即可得到大量的针对各种抗原的特异性抗体,使人源性抗体制备技术获得突破性的进展。
全合成人源噬菌体抗体库的设计,需要选择具有代表性的骨架序列以及制定覆盖范围广的cdr序列。抗体库技术的发展和对抗体结构功能认识的深化使人们具备了完全人工设计抗体可变区的能力。随着人类抗体胚系(germline)基因测序完成,人们对抗体结构的认识更深入了一步,使得对人抗体基因的操作更加简单易行。对抗体胚系基因使用频率分析表明,胚系基因的使用具有较强的偏向性,许多胚系基因从不或很少使用(tomlinson,1995),这一特点是构建抗体库的重要基础。为保证骨架区的代表性和保守性,骨架区的选择和设计通常采用人抗体胚系基因作为基础骨架。选用多个骨架序列构建多骨架库能够更全面地涵盖人抗体库基因。cdr区序列的设计是合成抗体库的关键,这使合成抗体库具有可控性强的优点,可根据需要人为设计改造抗体的基因。合成抗体库的组分清楚,能够在一定程度上改善抗体多样性的可控性,抗体库的内容、位点变异性、库的整体多样性均可根据设计加以控制调节。
人为地控制骨架序列和cdr区的设计与合成,可使抗体库的构成和多样性明确化。早期抗体库构建筛选的成功证实了通过全合成技术构建抗体库的可行性,并且为近年来新型合成抗体库的构建积累了丰富的经验。
tigit(tcellimmunoglobulinanditimdomain)是近年新发现的一种i型跨膜蛋白,由免疫球蛋白可变区(igv)、跨膜区和受体酪氨酸抑制基序(immunoreceptortyrosine-basedinhibitormotif,itim)组成,主要表达在调节性t细胞(treg)、消耗性cd8+t细胞、记忆性t细胞以及自然杀伤细胞(nk)表面(stengelkf,eta1.(2012)procnatlacadsciusa.109)。tigit是一种共抑制性受体蛋白,其和共刺激分子cd226与共同的配体脊髓灰质炎病毒受体pvr/cd155结合。cd226参与增强t细胞活化,而tigit参与抑制t细胞应答。cd155表达于未成熟或静止的树突细胞(dcs),tigit与cd155的结合会阻断tigit/cd226成熟的信号传递,刺激并增加其分泌il-10(yux,eta1.(2009)eurjimmunol.39:3160)。正常情况下,与cd226相比,tigit结合到cd155亲和力较强,tigit共抑制信号占主导,这意味着激活的免疫抑制是典型的负调节信号。
高亲和力的tigit/pvr相互作用会产生抑制信号,可阻止对正常表达pvr的(自身)细胞的杀伤,也可通过抑制nk细胞的抗肿瘤反应,保护肿瘤细胞免于免疫细胞介导的杀伤作用(stanietskyetal.(2009)proc.nat'lacad.sei.(usa)106:17858;lozanoeta1.(2012),j.immunol.188:3869)。有研究发现,tigit在肿瘤浸润性treg细胞上高度富集。肿瘤组织中的tigit+treg细胞表现出高度活性和抑制性的treg细胞表型。实验表明,cd155在黑色素瘤细胞上(inozumeetal.(2014)j.invest,dermato1.134:s121-abstract693)和在各种其它肿瘤上过表达。
因此,抗tigit抗体可增强抗肿瘤免疫反应机制,或者总体上阻断tigit/pvr的结合,可用于治疗癌症,例如结肠直肠癌(hampton.(2015)jmn313:1305),黑色素瘤等。
技术实现要素:
本发明的目的是提供一种全人源抗hutigit单克隆抗体及其应用。
为实现上述目的,本发明采用以下技术方案:
本发明从构建的大容量全合成人源抗体库中,筛选获得了4株能特异性结合人tigit,以及阻断hutigit与人pvr/cd155相互作用的全人源抗人tigit抗体。本发明的申请人进行了大量的实验,采用分子进化的方法,对上述一株单克隆抗体3tb3进行亲和力成熟,结合基因工程技术构建了1株优选的具有高亲和力和阻断活性的抗人tigit单克隆抗体,并对其生物学活性进行鉴定。该抗体有较高的亲和力和阻断活性,能够有效地识别人tigit抗原。
本发明的全合成人源抗体库采用以下设计方案。应用生物信息学方法,通过检索absys、vbase2、ncbi、kabat、genbank、pdb等数据库,下载人源和鼠源等抗体基因序列,比较分析大量序列之间的同一性和差异性,对抗体骨架序列在人类抗体库中的出现频率进行排序,以保证抗体库的代表性和保守性为原则,选择了优化的vh/vl框架序列,确定了构建抗体库的germline基因,即两种人源igg型抗体轻链可变区master基因,vκ2和vκ3,以及两种人源igg型抗体重链可变区master基因,vh3和vh4。为了便于文库构建,对抗体骨架序列进行模块化设计,在骨架区和cdr区的边界引入稀有的单一酶切位点,便于cdr区与骨架区的灵活拆卸与组装。对于人源抗体可变区的cdr区域,收集抗体可变区的氨基酸序列(包括所有的人抗体胚系基因和重排序列),进行序列同源性分析和氨基酸使用频率和偏向性分析。选择cdr区域多样性较高的氨基酸位置,按大肠杆菌密码子使用原则将其转换为核苷酸序列,使用简并密码子进行了长度多样性和序列多样性设计,尽量合理地覆盖所有典型的cdr结构。利用成熟的抗体库构建和噬菌体展示技术,筛选特异性结合人tigit抗原的分子。
本发明所使用的抗体库,是大容量的全合成人源scfv噬菌体抗体库。为了提高和改变获得的抗体分子亲和力,采用了亲和力成熟技术,通过分子进化改进tigit单抗分子的结合能力。
本发明的分子进化技术是通过pcr引入突变的,它包括关键氨基酸(ka)扫描和突变引物设计。突变引物设计是这一技术中获得足够数量的有用突变的关键步骤。本发明中所述的目标区域(即拟突变的区域)是抗体3tb3分子的cdr区,优先选择的是轻链和重链的cdr3。
本发明中在分子进化之前或之后通过淘筛获得的抗体分子是scfv形式的。这种形式可以通过本领域的技术人员熟知的基因工程技术方便地进一步转换成fab、全长抗体或其他形式。
本发明中经亲和力成熟获得的高亲和力抗人tigit单克隆抗体d1y1a,其原型分子3tb3是用人tigit作为抗原在上述抗体库进行淘筛获得的。通过spr法分析测定,d1y1a和3tb3分别以2.661×10-10和1.128×10-9或更小的平衡解离常数kd(m)结合hutigit。其特征在于所述抗体与人tigit的亲和力相比3tb3提高了约4倍,这也是通过elisa实验的半数有效浓度ec50体现的。
本发明公开了全人源抗人tigit单抗的氨基酸序列:
4株特异性抗人tigit单抗tbb8、tdc8、3tb3和5tb10,和分子进化后优选的1株高亲和力抗人tigit单抗d1y1a,包含全人源框架区以及全人源轻链和重链可变区,所述轻链可变区(vl)分别为seqidno:5,seqidno:7,seqidno:9,seqidno:11,seqidno:13所示氨基酸,所述重链可变区(vh)分别为seqidno:6,seqidno:8,seqidno:10,seqidno:12,seqidno:14所示氨基酸。
本发明中,所述抗体分子的重链和轻链是全人源的,尽管嵌合的或人源化的也可以使用,但是用于人体时,全人源的可以避免很多副作用,例如hama和haca反应引起的副作用。
本发明的全人源框架和fc区,以及人tigit和cd155序列是直接或间接从人体得到的。所述的直接方法包括但不限于基因组dna克隆或cdna文库。所述的间接方法包括但不限于genbank或其他出版物或网站提供的生物信息为基础部分,合成或完全从头合成完整的dna。dna合成技术包括但不限于以pcr为基础的dna合成方法。
为获得全长轻链基因,可以把编码vl区的dna整合到人轻链恒定区。优先选择的是人κ或λ恒定区,最优先选择的是人κ恒定区。为了获得全长重链基因,可以把编码vh区的dna整合到重链的恒定区。重链恒定区可以从以下几种中选择,igg、iga、ige、igm或igd。优先选择的是iggl~igg4,最优先选择igg1的恒定区。
为了表达本发明所述的抗体或抗体的片段,可以把其相应的轻链和重链编码序列插入到表达载体的转录和翻译控制序列之间。本发明所述的表达载体包含调控序列如启动子、增强子等。所述的表达载体及其控制序列应当与受体细胞兼容。编码轻链和重链的基因可以分别插入到两个独立的载体,也可以插入到同一个载体,所述表达载体可以预置了恒定区,也可以不预置恒定区。
本发明的抗体或抗体片段中,轻链和重链基因在受体细胞中的表达可以通过瞬时表达来实现。该表达策略包括用一种或多种带有编码抗体轻链和重链的dna片段的表达载体共转染哺乳动物细胞,从而使轻链和重链在受体细胞中表达并组装,优选的表达方式是分泌到培养基中,可以用本领域技术人员熟知的层析等方法从中回收抗体。标准的重组dna方法可以有效地用于轻链和重链基因的获得、克隆至表达载体以及引入到受体细胞。
附图说明
图1为pcd-tigit-avi-his质粒构建图解,该质粒经转染进入真核细胞,可获得表达的rhtigit-avi-his蛋白。
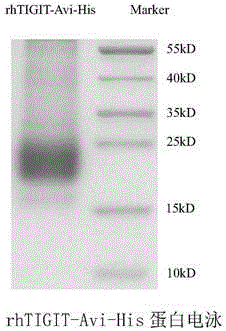
图2为sds-page(聚丙烯酰胺凝胶电泳)检测rhtigit-avi-his蛋白纯度。其中,泳道1为rhtigit-avi-his蛋白,泳道2为蛋白ladder。
图3为elisa法测定4株全人源抗人tigit单抗的结合活性。elisa结果表明,全人源抗人tigit单抗可以与rhtigit-avi-his抗原特异性结合。
图4为facs法测定4株全人源抗人tigit单抗的结合活性。结果表明,全人源抗人tigit单抗能够识别细胞表面的tigit蛋白,且呈现出浓度梯度依赖特性。
图5为facs法测定4株全人源抗人tigit单抗的阻断活性。结果表明,全人源抗人tigit单抗能够阻断细胞表面的tigit蛋白与rhcd155-cfc的相互作用,且呈现出浓度梯度依赖特性。
图6为elisa法测定全人源抗人tigit单抗d1y1a的结合活性。elisa结果表明,d1y1a可以与rhtigit-avi-his抗原特异性结合,且相比3tb3提高了约3倍。
图7为facs法测定全人源抗人tigit单抗d1y1a的阻断活性。结果表明,d1y1a能够阻断细胞表面的tigit蛋白与rhcd155-cfc的相互作用,且呈现出浓度梯度依赖特性。
图8为spr法测定全人源抗人tigit单抗d1y1a和3tb3与rhtigit-avi-his-bio蛋白的结合动力学,测得其平衡解离常数kd(m)值约为2.661×10-10和1.128×10-9。
图9为elisa法测定全人源抗人tigit单抗d1y1a与其他物种tigit分子的结合活性。结果表明,d1y1a能够与小鼠tigit分子(rmtigit)以及食蟹猴/恒河猴tigit分子(rcynotigit)结合。
具体实施方案
以下实施例仅对本发明进行进一步的说明,不应理解为对本发明的限制。实施例不包括对传统方法的详细描述,如那些用于构建载体和质粒的方法,将编码蛋白的基因插入到这样的载体和质粒的方法或将质粒引入宿主细胞的方法。这样的方法对于本领域中具有普通技术的人员是众所周知的。
抗体库构建
本研究对噬菌体展示载体pcomb3进行了改造,获得适用于本实验室建库体系的噬菌体展示载体pc3xfss。参考相关书籍(therapeuticantibodymethodsandprotocols,antonys.dimitrov,2009;phagedisplayinbiotechnologyanddrugdiscovery,sachdevs.sidhu,2005.)中的建库方法,使用qiagen公司的qiaexiigelextractionkit,axygen公司的pcr清洁回收试剂盒,dna凝胶回收试剂盒和质粒小量制备试剂盒,通过重叠拼接延伸pcr的方法获得抗体基因,构建全合成人源抗体库。经过几次电击转化大肠杆菌tg1,构建了库容量分别约为1×109的8类不同骨架类型的全合成人源抗体库。菌落测序及高通量测序结果表明,抗体库质量优良,为筛选人源性治疗抗体奠定了基础。使用适用于本实验室噬菌体展示体系的辅助噬菌体m13eea,参考相关书籍(antibodyphagedisplaymethodsandprotocols,philippam.o’brienandrobertaitken,2002.)中的噬菌体抗体库构建方法,构建了滴度都达到约3×1012的8类不同骨架类型的全合成人源噬菌体抗体库。
膜表达人tigit稳转细胞株制备
下载人tigit核苷酸序列(ncbireferencesequence:nm_173799.3)和氨基酸序列(ncbireferencesequence:np_776160.2),针对核苷酸序列进行去相关酶切位点或减少茎环结构等改造,委托通用生物系统(安徽)有限公司全基因合成抗人tigit全长基因,包括信号肽、胞外段、跨膜区和胞内段,两端都设置酶切位点ecori。预混50ul酶切体系,使用ecori酶(购自thermo)单酶切合成的基因,同样预混50ul酶切体系,用ecori酶切真核表达载体gc-id(改造自pmh3质粒,杭州安普),并去磷酸化(fastap酶,购自thermo),将tigit全长基因构建到真核表达载体gc-id中,构建过程同上,获得gc-id-tigit质粒,插入的片段处在载体的启动子和ires元件之间。用pvui酶(购自thermo)线性化该质粒后,利用脂质体法,转染该质粒到cho-dg44细胞中,从而构建膜表达人tigit稳转细胞株dg44-tigit,用于流式细胞检测tigit单抗的结合活性和阻断活性。
rhtigit-avi-his-bio抗原蛋白制备
针对合成的tigit全长基因,设计合适的引物,正向引物序列为aatggatccactggtgacatgatgacaggcacaataga,反向引物序列为aatggtacctccgcctgagctttctaggacctccaggaaga,pcr体系为4ul2.5mmdntps,5ulfastpfubuffer,1ulforwardprimer,1ulreverseprimer,1ulfastpfudnapolymerase,适量的基因模板,以及ddh2o补齐到50ul,pcr反应程序为,95℃,2min;95℃20s,56℃20s,72℃15s,30cycles;72℃,5min。单一条带产物回收后使用bamhi和kpni酶切,与同样酶切的重组蛋白真核表达的载体pcd-avi-his(改造自pcdna3.1+,invitrogen公司)连接,从而将胞外段的免疫球蛋白可变区(igv,22aa-130aa)样结构域构建到表达载体中,构建过程同上,获得真核表达质粒pcd-tigit-avi-his,如图1所示。使用质粒中抽试剂盒(axygen,货号ap-md-p-25)抽提足量的质粒,将该质粒转染足够量的expi293悬浮细胞,收获上清用3.5kd透析膜(购自bbi,货号f132594)透析过夜,使用ge公司的镍柱纯化系统ni-nta树脂进行纯化,用截留分子量为3kda的超滤离心管(millipore公司)超滤浓缩。sds-page验证纯度和大小(图2)。同样的方法制备rmtigit和rcynotigit蛋白。利用bira生物素化酶将biotin共价连接到含有avi标签肽序列的rhtigit-avi-his蛋白,完成生物素化酶标记,biaa酶和biotinsolution都来自genecopoeia公司,hitrapdesalting除盐柱来自ge公司。采用streptavidin亲和胶(购自thermo/pierce,货号20359)吸附法检测标记效率,得到浓度较高,标记效率较好的rhtigit-avi-his-bio蛋白,用于抗体淘筛实验。
rhcd155-cfc配体蛋白制备
根据文献报道(sakisakat,etal.(2004)curropincellbiol.16:513-521.),人cd155(pvr)在在树突状细胞(dcs)上高表达,因此从人pbmc中提取totalrna,使用rna提取试剂盒axyprepmultisourcetotalrnaminiprepkit(购自axygen,货号ap-mv-ms-rna-50)。下载cd155全长基因核苷酸序列(ncbireferencesequence:nm_006505.4)和氨基酸序列(ncbireferencesequence:np_006496.4),包括信号肽、胞外段、跨膜区和胞内段,设计合适的引物,正向引物序列为ccgtcttccaccaaacgc,反向引物序列为cccgtcgttttgaggtct,以totalrna为模板进行反转录,使用反转录试剂盒primescripttmrt-pcrkit(购自takara,货号rr014a),调取cd155全长基因。设计构建克隆的引物,正向引物序列aatggatccactggtgacgtcgtcgtgcaggcgc,反向引物序列aattgcggccgcgttacgggacatgcctgagt,两端设置bamhi和noti酶切位点,将调取的cd155全长基因构建到用于重组蛋白真核表达的载体pcd-cfc(改造自pcdna3.1+,购自invitrogen公司)中,构建过程同上,获得真核表达质粒pcd-cd155-cfc。经过proteina亲和层析胶纯化大量瞬转expi293悬浮细胞后的上清液,获得rhcd155-cfc纯蛋白,sds-page验证纯度。为了体现tigit单抗在细胞水平上对tigit和cd155分子的阻断活性,使用nh2-reactivehilytefluor647试剂盒(购自同仁化学)标记rhcd155-cfc蛋白,得到hilytefluortm647荧光标记的rhcd155-cfc-647蛋白,用于流式检测tigit单抗的阻断活性。
tigit单抗序列筛选
利用制备好的rhtigit-avi-his-bio抗原蛋白,对8类全合成人源噬菌体抗体库进行多轮液相筛选,筛选过程参考书籍(therapeuticantibodyengineering,williamr.strohlandlilamm.strohl,2012;antibodyengineeringmethodsandprotocols,patrickchames,2012.)。从第二轮及以后的细菌培养板上挑取大量的单克隆,制备重组phage和可溶性scfv抗体。
elisa检测phage上清结合活性
具体操作如下:rhtigit-avi-his蛋白铺板,nahco3缓冲液将其稀释至1ug/ml,铺96孔elisa板(购自nunc公司,货号469957),每孔100ul,于4℃冰箱过夜;tpbs(pbs+0.1%tween20)洗三遍,3%脱脂奶粉溶于tpbs作为封闭剂,加满每孔(约350ul),37℃封闭1小时;tpbs洗三遍,加重组phage上清,100ul/孔,室温震荡温育1小时;tpbs洗三遍,加入1:5000稀释的anti-m13/hrp二抗(购自ge,货号27-9421-01),每孔100ul,室温震荡温育30min;tpbs洗三遍,每孔加入混有0.1%过氧化氢的opd邻苯二胺(购自sigma公司,货号78412)底物工作液100ul进行显色,约1-20分钟后加入100ul1m硫酸终止,酶标仪(购自biotek公司,货号elx800)测定od490值。选择吸光值相对较高的孔对应的克隆测序,分析序列多样性。鉴定出了70个高吸光值且序列唯一的单克隆,进一步检测这些克隆的可溶性抗体结合活性。
elisa检测可溶性抗体结合活性
具体操作如下:rhtigit-avi-his蛋白铺板,nahco3缓冲液将其稀释至1ug/ml,铺96孔elisa板(购自nunc公司,货号469957),每孔100ul,于4℃冰箱过夜;tpbs(pbs+0.1%tween20)洗三遍,3%脱脂奶粉溶于tpbs作为封闭剂,加满每孔(约350ul),37℃封闭1小时;tpbs洗三遍,加可溶性表达上清,100ul/孔,室温震荡温育1小时;tpbs洗三遍,加入1:5000稀释的anti-flag/hrp二抗(购自sigma,货号a8592),每孔100ul,室温震荡温育30min;tpbs洗三遍,每孔加入混有0.1%过氧化氢的opd邻苯二胺(购自sigma公司,货号78412)底物工作液100ul进行显色,约1-20分钟后加入100ul1m硫酸终止,酶标仪(购自biotek公司,货号elx800)测定od490值。根据检测结果,获得了11株结合活性较强的scfv单链抗体。
11株单链抗体的真核表达质粒构建
本实验室用于抗体真核表达的载体为pcd-hukappa和pcd-huigg1,都改造自pcdna3.1+(invitrogen公司)。针对单链抗体轻链序列设计合适的引物,正向引物序列为attggatccactggtgacatcgtgttgacgcagtctccagc,反向引物序列为aagcttggtaccctgaccgaaggt,分别带有bamhi和kpni酶切位点,针对单链抗体轻链序列设计合适的引物,正向引物序列为attggatccactggtgacgtacagctgcaagagtcaggt,反向引物序列为caccaaggtaccctgaccccagta,分别带有bamhi和kpni酶切位点,pcr体系为4ul2.5mmdntps,5ulfastpfubuffer,1ulforwardprimer,1ulreverseprimer,1ulfastpfudnapolymerase,适量的基因模板,以及ddh2o补齐到50ul,pcr反应程序为,95℃,2min;95℃20s,56℃20s,72℃15s,30cycles;72℃,5min。单一条带产物回收后使用bamhi和kpni酶切,预混50ul的酶切体系,取适量的回收产物通过t4dnaligase(购自thermo)将轻链可变区基因构建到同样酶切的pcd-hukappa载体上,重链可变区基因构建到同样酶切的pcd-huigg1载体上,连接体系为10ul,插入的片段处在载体的信号肽和cl或者ch之间。两个载体上的信号肽基因序列一致(atggagacagacacactcctgctatgggtactgctgctctgggttccaggatccactggtgac),来自psectag2a质粒(invitrogen公司)。筛选阳性克隆酶切验证并测序,获得11株单链抗体的轻重链真核表达质粒。将轻重链质粒瞬时共转染到expi293悬浮细胞后,针对fc融合蛋白,使用ge公司的proteina/mabselectsure亲和层析胶纯化大量瞬转后的上清液,得到11株抗体的纯蛋白。
elisa测定抗体相对亲和力
实验步骤如下:首先需要进行抗原铺板,用50mmnahco3将rhtigit-avi-his稀释到0.5ug/ml,铺96孔elisa板,每孔100ul,于4℃冰箱过夜;tpbs洗三遍,3%脱脂奶粉溶于tpbs作为封闭剂,加满每孔(约350ul),37℃封闭1小时;tpbs洗三遍,用封闭剂将单抗稀释到1ug/ml,随后3倍梯度稀释,共稀释8个梯度,每孔100ul,室温震荡温育1小时;tpbs洗三遍,加入1:4000稀释的羊抗人/hrp二抗,每孔100ul,室温震荡温育30min;tpbs洗三遍,每孔加入混有0.1%过氧化氢的opd邻苯二胺(购自sigma公司,货号78412)底物工作液100ul进行显色,约1-20分钟后加入100ul1m硫酸终止,酶标仪测定od490值。该结果可用于初步计算tigit单抗与抗原rhtigit-avi-his的相对亲和力ec50值,即半数有效浓度。
流式细胞术(fluorescence-activatedcellsorting,facs)检测tigit单抗细胞水平结合活性
取对数生长期的dg44-tigit稳转细胞株,计数后分装到每管3×105细胞,500g离心3min,弃上清,用含2%fbs的pbs洗液清洗两遍,将单抗稀释到10ug/ml,随后3倍梯度稀释,共稀释5个梯度,分别100ul加入对应的离心管中,细胞空白对照管和阴性对照管中加入100ul洗液,冰上温育1h(每10min轻弹混匀);用含2%fbs的pbs洗液清洗三遍,羊抗人igg/fitc按照1:200稀释,除细胞对照管外,每管加入100ul,冰上避光温育30min;用含2%fbs的pbs洗液清洗三遍,500ul的pbs悬浮细胞,立即上机检测。该结果可用于计算tigit单抗与人tigit分子细胞水平的结合活性ec50。
流式细胞术(fluorescence-activatedcellsorting,facs)检测tigit单抗细胞水平阻断活性
取对数生长期的dg44-tigit稳转细胞株,计数后分装到每管3×105细胞,500g离心3min,弃上清,用含2%fbs的pbs洗液清洗两遍,取50ul用洗液稀释的待测tigit单抗纯蛋白(浓度梯度10,2,0.4,0.08,0.016ug/ml)与50ulrhcd155-cfc-647(1ug/ml)混合加入实验管中,50ul洗液与50ulrhcd155-cfc-647(1ug/ml)混合加入阴性对照管中,细胞空白对照管加100ul洗液,冰上避光温育1h(每10min轻弹混匀);用洗液清洗三遍,500ul的pbs悬浮细胞,立即上机检测。该结果可用于计算tigit单抗阻断rhcd155-cfc与dg44-tigit细胞的阻断活性ic50值。
经过elisa测定抗体相对亲和力、流式检测其与人tigit分子细胞水平的结合活性,以及其阻断rhcd155-cfc与dg44-tigit细胞的活性后进行排序发现,这11株单克隆抗体中,tbb8、tdc8、3tb3、5tb10四个抗体分子的结合和阻断能力最强。在elisa测定抗体相对亲和力实验中,四个抗体分子的ec50分别为0.2798、0.2929、0.03071和0.7543ug/ml,如图3所示;在facs检测tigit单抗细胞水平结合实验中,四个抗体分子的ec50分别为0.09599、0.05851、0.05887和0.0872ug/ml,如图4所示;在facs检测tigit单抗细胞水平阻断实验中,四个抗体分子的ic50分别为0.5629、0.3085、0.2121和0.4347ug/ml,如图5所示。
从以上4株全人源抗人tigit抗体中,选择以3tb3为亲和力成熟的原型分子。根据dna测序结果推断的3tb3轻链和重链的可变区氨基酸序列分别为seqidno:9和seqidno:10。
3tb3单抗的亲和力成熟。
首先,我们采用生物信息学的方法对3tb3抗体的6个cdr区按kabat数据库进行定义,参照相同类别下已知抗体的序列设计饱和突变,将序列多样性直接针对那些最可能与抗原接触的残基,鉴定出保守性弱的残基,这些残基即为优选的突变位点。这样就保证了突变设计的合理性,避免随机突变导致的抗体结合抗原表位的变化,维持了抗体的稳定性,提高亲和力成熟抗体库的有效库容的同时,大大减少了冗余序列。下表中下划线部分是3tb3中的优选氨基酸突变位点。
表13tb3分子的优选氨基酸突变位点
将设计好的突变序列引物委托苏州金唯智生物技术有限公司合成。进一步,以3tb3质粒为模板,使用transstartkdpluspcrsupermix(购自transgene,货号as301-01)聚合酶反应体系,通过重叠拼接pcr的方法分别扩增突变部分,pcr反应程序为,95℃,2min;95℃20s,60℃20s,72℃15s,10cycles;72℃,5min;95℃20s,56℃20s,72℃15s,20cycles;72℃,5min。用上述全合成人源抗体库同样的载体和辅助噬菌体,快速有效地构建了3类由突变cdr区组合的亲和力成熟抗体文库,此scfv亲和力成熟噬菌体抗体库为一级库。
以本领域熟知的方法,对6个突变cdr区的3类组合文库使用不同浓度梯度的rhtigit-avi-his-bio抗原,进行了多轮液相筛选,淘筛到的克隆用原核表达的可溶性抗体elisa判定相对亲和力。对亲和力高于野生型3tb3的优选克隆进行序列分析,获得每个cdr区对亲和力提高或者维持有意义的突变序列信息,并以上述筛选到的克隆作为模板,分别扩增相应的cdr-l1+cdr-l2,cdr-l3,cdr-h1+cdr-h2和cdr-h3,进行4片段体外重组,获得cdr突变体组成的二级抗体库。针对二级库使用更低浓度的抗原进行了多轮液相筛选和富集,从第二轮及以后的细菌培养板上挑取大量的单克隆,得到若干亲和力提高的多cdr突变的scfv单链抗体。将部分有潜力的克隆如d1y1a,转入真核表达系统。使用elisa(图6)和facs(图7),对上述获得的真核表达的单抗进行相对亲和力和阻断活性检测。
平衡解离常数测定
用表面等离子共振(surfaceplasmonresonance,spr)方法,利用t200分子间相互作用仪测定tigit单抗的亲和力,即平衡解离常数。具体操作如下:使用sa芯片(ge;br-1005-31),用pbs稀释生物素标记的抗原rhtigit-avi-his-bio至100ng/ml,抗原标记至45ru,每个样品测试7梯度,用pbs稀释2个tigit单抗蛋白至:5ug/ml,2.5,1.25,0.625,0.3125,0.15625,0.078125。测试结合、解离和glycine2.0(ge;br-1003-55)再生条件。最后确定结合2分钟,解离10分钟,再生1分钟。计算d1y1a和3tb3的平衡解离常数分别为2.661×10-10和1.128×10-9,如图8。
elisa测定d1y1a单抗与rmtigit和rcynotigit的结合,两种抗原铺板浓度都为1ug/ml,d1y1a单抗从1ug/ml开始3倍梯度稀释,共稀释8个梯度,如图9所示,d1y1a单抗能够与rmtigit和rcynotigit结合。
将3tb3以及d1y1a的单抗可变区序列与已公布的tigit单抗序列比对,均没有重复,因此,3tb3以及d1y1a的单抗轻重链可变区是全新单抗序列。
应该理解,尽管参考其示例性的实施方案,已经对本发明进行具体地显示和描述,但是本领域的普通技术人员应该理解,在不背离由权利要求书所定义的本发明的精神和范围的条件下,可以在其中进行各种形式和细节的变化,可以进行各种实施方案的任意组合。
sequencelisting
<110>安徽安科生物工程(集团)股份有限公司
<120>一种全人源抗hutigit单克隆抗体及其应用
<130>2019
<160>14
<170>patentinversion3.3
<210>1
<211>244
<212>prt
<213>humantigit
<400>1
metargtrpcysleuleuleuiletrpalaglnglyleuargglnala
151015
proleualaserglymetmetthrglythrilegluthrthrglyasn
202530
ileseralaglulysglyglyserileileleuglncyshisleuser
354045
serthrthralaglnvalthrglnvalasntrpgluglnglnaspgln
505560
leuleualailecysasnalaaspleuglytrphisileserproser
65707580
phelysaspargvalalaproglyproglyleuglyleuthrleugln
859095
serleuthrvalasnaspthrglyglutyrphecysiletyrhisthr
100105110
tyrproaspglythrtyrthrglyargilepheleugluvalleuglu
115120125
serservalalagluhisglyalaargpheglnileproleuleugly
130135140
alametalaalathrleuvalvalilecysthralavalilevalval
145150155160
valalaleuthrarglyslyslysalaleuargilehisservalglu
165170175
glyaspleuargarglysseralaglyglngluglutrpserproser
180185190
alaproserproproglysercysvalglnalaglualaalaproala
195200205
glyleucysglygluglnargglygluaspcysalagluleuhisasp
210215220
tyrpheasnvalleusertyrargserleuglyasncysserphephe
225230235240
thrgluthrgly
<210>2
<211>110
<212>prt
<213>cyno/rhesustigit-ecd
<400>2
metmetthrglythrilegluthrthrglyasnileseralalyslys
151015
glyglyservalileleuglncyshisleuserserthrmetalagln
202530
valthrglnvalasntrpgluglnhisasphisserleuleualaile
354045
argasnalagluleuglytrphisiletyrproalaphelysasparg
505560
valalaproglyproglyleuglyleuthrleuglnserleuthrmet
65707580
asnaspthrglyglutyrphecysthrtyrhisthrtyrproaspgly
859095
thrtyrargglyargilepheleugluvalleugluserser
100105110
<210>3
<211>105
<212>prt
<213>mousetigit-ecd
<400>3
thrileaspthrlysargasnileseralaglugluglyglyserval
151015
ileleuglncyshispheserseraspthralagluvalthrglnval
202530
asptrplysglnglnaspglnleuleualailetyrservalaspleu
354045
glytrphisvalalaservalpheseraspargvalvalproglypro
505560
serleuglyleuthrpheglnserleuthrmetasnaspthrglyglu
65707580
tyrphecysthrtyrhisthrtyrproglyglyiletyrlysglyarg
859095
ilepheleulysvalglngluserser
100105
<210>4
<211>417
<212>prt
<213>humancd155
<400>4
metalaargalametalaalaalatrpproleuleuleuvalalaleu
151015
leuvalleusertrpproproproglythrglyaspvalvalvalgln
202530
alaprothrglnvalproglypheleuglyaspservalthrleupro
354045
cystyrleuglnvalproasnmetgluvalthrhisvalserglnleu
505560
thrtrpalaarghisglygluserglysermetalavalphehisgln
65707580
thrglnglyprosertyrsergluserlysargleugluphevalala
859095
alaargleuglyalagluleuargasnalaserleuargmetphegly
100105110
leuargvalgluaspgluglyasntyrthrcysleuphevalthrphe
115120125
proglnglyserargservalaspiletrpleuargvalleualalys
130135140
proglnasnthralagluvalglnlysvalglnleuthrglyglupro
145150155160
valprometalaargcysvalserthrglyglyargproproalagln
165170175
ilethrtrphisseraspleuglyglymetproasnthrserglnval
180185190
proglypheleuserglythrvalthrvalthrserleutrpileleu
195200205
valproserserglnvalaspglylysasnvalthrcyslysvalglu
210215220
hisgluserpheglulysproglnleuleuthrvalasnleuthrval
225230235240
tyrtyrproprogluvalserileserglytyraspasnasntrptyr
245250255
leuglyglnasnglualathrleuthrcysaspalaargserasnpro
260265270
gluprothrglytyrasntrpserthrthrmetglyproleupropro
275280285
phealavalalaglnglyalaglnleuleuileargprovalasplys
290295300
proileasnthrthrleuilecysasnvalthrasnalaleuglyala
305310315320
argglnalagluleuthrvalglnvallysgluglyproproserglu
325330335
hisserglymetserargasnalaileilepheleuvalleuglyile
340345350
leuvalpheleuileleuleuglyileglyiletyrphetyrtrpser
355360365
lyscysserarggluvalleutrphiscyshisleucysproserser
370375380
thrgluhisalaseralaseralaasnglyhisvalsertyrserala
385390395400
valserarggluasnserserserglnaspproglnthrgluglythr
405410415
arg
<210>5
<211>109
<212>prt
<213>tbb8vl
<400>5
aspilevalleuthrglnserproalathrleuserleuserprogly
151015
gluargalathrleusercysargalaserglnaspileglyvalser
202530
leuasntrptyrglnglnlysproglyglnalaproargleuleuile
354045
tyrtrpthrserpheargalathrglyvalprothrargphesergly
505560
serglyserglythraspphethrleuthrileserserleuglupro
65707580
gluaspphealavaltyrtyrcysglnglnalaasnglyvalproleu
859095
phethrpheglyglnglythrlysleugluilelysarg
100105
<210>6
<211>119
<212>prt
<213>tbb8vh
<400>6
aspvalglnleuglngluserglyproglyleuvallysproserglu
151015
thrleuserleuthrcysthrvalserglytyrserileserthrgly
202530
tyralatrpsertrpileargglnproproglylysglyleuglutrp
354045
ileglyserileseralapheglyglyasnthrtyrtyrasnproser
505560
leulysserargvalthrileservalaspthrserlysasnglnphe
65707580
serleulysleuserservalthralaalaaspthrservaltyrtyr
859095
cysalaargthralaargtrpilealapheasptyrtrpglyglngly
100105110
thrleuvalthrvalserser
115
<210>7
<211>109
<212>prt
<213>tdc8vl
<400>7
aspilevalleuthrglnserproalathrleuserleuserprogly
151015
gluargalathrleusercysargalaserglnserilehisalagly
202530
tyrleualatrptyrglnglnlysproglyglnalaproargleuleu
354045
iletyrtrpthrasnleuargalathrglyvalprothrargpheser
505560
glyserglyserglythraspphethrleuthrileserserleuglu
65707580
progluaspphealavaltyrtyrcysleuglnleumetargtyrpro
859095
prothrpheglyglnglythrlysleugluilelysarg
100105
<210>8
<211>122
<212>prt
<213>tdc8vh
<400>8
aspvalglnleuglngluserglyproglyleuvallysproserglu
151015
thrleuserleuthrcysthrvalserglyglyserileserserlys
202530
glyphethrtrpsertrpileargglnproproglylysglyleuglu
354045
trpileglyglnileserproleuaspserserthrsertyrasnpro
505560
serleulysserargvalthrileservalaspthrserlysasngln
65707580
pheserleulysleuserservalthralaalaaspthrservaltyr
859095
tyrcysalaargglyiletrpthrtyrleutyrglypheasptyrtrp
100105110
glyglnglythrleuvalthrvalserser
115120
<210>9
<211>108
<212>prt
<213>3tb3vl
<400>9
aspilevalleuthrglnserproalathrleuserleuserprogly
151015
gluargalathrleusercysargalaserglnglyvalglyilearg
202530
leusertrptyrglnglnlysproglyglnalaproargleuleuile
354045
tyrtyrthralatyrargalathrglyvalprothrargphesergly
505560
serglyserglythraspphethrleuthrileserserleuglupro
65707580
gluaspphealavaltyrtyrcysleuglnthrpheglyaspprotyr
859095
thrpheglyglnglythrlysleugluilelysarg
100105
<210>10
<211>119
<212>prt
<213>3tb3vh
<400>10
aspvalglnleuglngluserglyproglyleuvallysproserglu
151015
thrleuserleuthrcysthrvalserglyglyserileserglygly
202530
tyrsertrpsertrpileargglnproproglylysglyleuglutrp
354045
ileglyserilevalprohisargseralathrsertyrasnproser
505560
leulysserargvalthrileservalaspthrserlysasnglnphe
65707580
serleulysleuserservalthralaalaaspthrservaltyrtyr
859095
cysalaargsertyrglyglyasnargleuasptyrtrpglyglngly
100105110
thrleuvalthrvalserser
115
<210>11
<211>109
<212>prt
<213>5tb10vl
<400>11
aspilevalleuthrglnserproalathrleuserleuserprogly
151015
gluargalathrleusercysargalaserglnaspvallysileleu
202530
leuasntrptyrglnglnlysproglyglnalaproargleuleuile
354045
tyrtrpthrseraspargalathrglyvalprothrargphesergly
505560
serglyserglythraspphethrleuthrileserserleuglupro
65707580
gluaspphealavaltyrtyrcysglnglnhisleuaspargleupro
859095
tyrthrpheglyglnglythrlysleugluilelysarg
100105
<210>12
<211>119
<212>prt
<213>5tb10vh
<400>12
aspvalglnleuglngluserglyproglyleuvallysproserglu
151015
thrleuserleuthrcysthrvalserglyaspservalseraspala
202530
glyvalglytrpasntrpileargglnproproglylysglyleuglu
354045
trpileglythrilevalproargargglyilethrthrtyrasnpro
505560
serleulysserargvalthrileservalaspthrserlysasngln
65707580
pheserleulysleuserservalthralaalaaspthrservaltyr
859095
tyrcysalaargvalthrthralaglypheasptyrtrpglyglngly
100105110
thrleuvalthrvalserser
115
<210>13
<211>100
<212>prt
<213>d1y1avl
<400>13
aspilevalleuthrglnserproalathrleuserleuserprogly
151015
gluargalathrleusercysargalaserglnglyvalx1x2arg
202530
leusertrptyrglnglnlysproglyglnalaproargleuleuile
354045
tyrtyrx3x4tyrargalathrglyvalproalaargphesergly
505560
serglyserglythraspphethrleuthrileserserleuglupro
65707580
gluaspphealavaltyrtyrcysleuglnx5phex6x7prox8
859095
thrpheglyglnglythrlysleugluilelysarg
100105
x1=gly,arg
x2=ile,glu,thr,asp
x3=thr,ala,ser,gly
x4=ala,leu,met,trp,val,ile
x5=thr,met
x6=gly,gln,ser,his,asn
x7=val,ile,asp
x8=tyr,phe,his
<210>14
<211>109
<212>prt
<213>d1y1avh
<400>14
aspvalglnleuglngluserglyproglyleuvallysproserglu
151015
thrleuserleuthrcysthrvalserglyglyserileserx1x2
202530
tyrx3trpsertrpileargglnproproglylysglyleuglutrp
354045
ileglyserilex4x5x6x7x8alathrsertyrasnproser
505560
leulysserargx9thrx10servalaspthrserlysasnglnphe
65707580
serleulysleuserservalthralaalaaspthralavaltyrtyr
859095
cysalaargsertyrglyglyasnargleuasptyrtrpglyglngly
100105110
thrleuvalthrvalserser
115
x1=gly,arg,asn,gln,thr,asp
x2=gly,ala
x3=ser,met,ala
x4=val,phe
x5=pro,his
x6=phe,his
x7=ser,arg
x8=his,ser,arg
x9=val,ala
x10=val,ile
- 还没有人留言评论。精彩留言会获得点赞!