一种产林可霉素的基因工程菌及其构建方法和应用与流程
本发明属于基因工程领域,具体地说,是关于一种产林可霉素的基因工程菌及其构建方法和应用。
背景技术:
林可霉素(lincomycin)是由林肯链霉菌(streptomyceslincolnensis)产生的一种具有广泛临床药用价值的林可酰胺类抗生素。林可霉素及其衍生物(如氯代衍生物克林霉素)对于厌氧型革兰氏阳性菌,以及原生动物具有抑制作用。林可霉素通过与核糖体50s亚基中的23srrna的中心环疏水性结合,抑制蛋白质翻译过程中的多肽链转移,从而发挥抑菌功能。林可霉素与大多数药用抗生素都不存在交叉抗性,临床上广泛用于治疗呼吸道、泌尿系统、皮肤及软组织感染,慢性骨髓炎,脑膜炎,中耳炎和眼科疾病等,特别适用于对内酰胺类抗生素有抗性的细菌感染患者。
过去几十年的生产实践中,林可霉素工业菌株的获取主要依靠物理或化学方法诱变,不但耗时,且随机性较大,无法对育种提供理论性指导。林可霉素的发酵生产水平与其他大宗抗生素相比仍存在较大差距,亟需通过新的技术手段获得优良的高产菌株,以满足工业生产的需求。
tpr(tetratricopeptiderepeat)是一个含34个氨基酸的蛋白重复序列,编码一个α螺旋-转角-α螺旋的二级结构片段,通常以前后串连的形式重复出现在蛋白结构中。前后相连的tpr折叠形成一个介导蛋白之间相互作用的结构域。含tpr结构域的蛋白广泛分布于原核和真核生物中,在细胞中行使各种各样的功能(细胞循环调节、转录调控、蛋白折叠与分泌)。目前已有多个含tpr结构域的蛋白质被发现在链霉菌中发挥抗生素合成调控的功能。目前尚无在林肯链霉菌中发现tpr调控林可霉素合成的案例报道。
rna酶通过在细胞内对rna加工或降解,在转录后水平调控基因表达量,进而广泛影响包括细胞生长、形态分化、核糖体组装及次级代谢等在内的细胞过程。早期研究发现,rnasej是一种同时具有核酸外切和内切活性的rna酶,在天蓝色链霉菌和委内瑞拉链霉菌体内影响多种抗生素的生物合成。但目前尚无rna酶调控林可霉素合成的案例被报道。
xre(xenobioticresponseelement)家族蛋白质是一类于真核生物、细菌和古细菌都广泛存在的转录调控因子。其结构上包含n末端的发挥nda结合功能的hth(螺旋-转角-螺旋)结构域,以及c末端的配体结合结构域。该家族成员已知的生物学功能包括噬菌体的转录调控,细菌质粒拷贝数的控制等。编码xre家族蛋白的基因广泛存在于链霉菌基因组,但生物学功能被解析的只有极少数。目前尚无通过改造xre家族基因提高林可霉素产量的案例。
技术实现要素:
本发明致力于通过基因工程手段定向改造基因,以获得用于林可霉素及其药用衍生物工业生产的高产菌株。为此,本发明通过研究,在林肯链霉菌野生菌株nrrl2936的基因组中发现了多个编码含tpr结构域蛋白质的基因,并从中选取了三个基因(slinc566、slinc4481、slinc6027)进行了基因敲除尝试。其中缺失slinc4481基因后,林肯链霉菌nrrl2936中林可霉素产量提高了18.7倍,说明slinc4481是一个参与林可霉素合成过程的负调控因子。
同时,本发明经过序列同源分析,林肯链霉菌nrrl2936基因组上的slinc6156基因被发现编码rnasej。缺失slinc6156基因后,林可霉素产量较出发菌株提高了22.4倍,说明slinc6156是一个参与林可霉素生物合成过程的负调控因子。
同时,本发明在在林肯链霉菌野生菌株nrrl2936的基因组中选取了两个编码xre家族蛋白质的基因(slinc348与slinc4742)进行了基因敲除尝试。其中缺失slinc348基因后,林肯链霉菌nrrl2936中林可霉素产量检测不到,说明slinc348是一个参与林可霉素合成过程的正调控因子。但缺失slinc4742基因对于林可霉素产量几乎无影响。在林肯链霉菌中过表达slinc348后,林可霉素产量提高7.8倍。
因此,本发明的第一个目的是提供一种产林可霉素的基因工程菌,以解决现有的林肯链霉菌的林可霉素产量低的问题。本发明的第二个目的是提供一种产林可霉素的基因工程菌的构建方法,以构建一种新的可以提高林可霉素产量的基因工程菌。本发明的第三个目的是提供一种产林可霉素的基因工程菌在发酵生产林可霉素中的应用。
为了达到上述目的,本发明提供了如下的技术方案:
作为本发明的第一个方面,一种产林可霉素的基因工程菌,其是将林肯链霉菌的编码含tpr结构域蛋白质的基因敲除和将编码rna酶的基因敲除并过表达林肯链霉菌的编码xre家族蛋白的基因构建获得。
根据本发明,所述编码含tpr结构域蛋白质的基因为slinc4481基因;所述编码rna酶的基因为slinc6156基因;所述编码xre家族蛋白的基因为slinc348基因。
根据本发明,所述林肯链霉菌为林肯链霉菌nrrl2936。
作为本发明的第二个方面,一种上述所述的基因工程菌的构建方法,其是以林肯链霉菌为出发菌株,将林肯链霉菌的编码含tpr结构域蛋白质的基因和编码rna酶的基因敲除,并过表达编码xre家族蛋白,构建获得产林可霉素的基因工程菌。
进一步的,所述的基因工程菌的构建方法,其是以林肯链霉菌为出发菌株,将林肯链霉菌的slinc4481基因和slinc6156基因敲除,并过表达slinc348基因,构建获得产林可霉素的基因工程菌δ4481δ6156::348。
根据本发明,所述的基因工程菌的构建方法,包括如下步骤:
步骤一、构建基因工程菌δ4481
以林肯链霉菌为出发菌株,将林肯链霉菌的slinc4481基因敲除,构建获得产林可霉素的基因工程菌δ4481;
步骤二、构建基因工程菌δ4481δ6156
以基因工程菌δ4481为出发菌株,将基因工程菌δ4481的slinc6156基因敲除,构建获得产林可霉素的基因工程菌δ4481δ6156;
步骤三、构建基因工程菌δ4481δ6156::348
以基因工程菌δ4481δ6156为出发菌株,过表达slinc348基因,构建获得产林可霉素的基因工程菌δ4481δ6156::348。
根据本发明,根据本发明,pcr反应条件为:98℃,10分钟;98℃,40秒;64℃,30秒;72℃,80秒,30个循环;72℃,10分钟;25℃,2分钟。
根据本发明,所述的基因工程菌的构建方法,包括如下步骤:
步骤一、(1)构建slinc4481敲除载体
a、以u4481-f/r和d4481-f/r为引物,林肯链霉菌基因组为模板,进行pcr扩增,获得slinc4481上下游同源臂,所述u4481-f/r和d4481-f/r的引物序列分别如seqidno:9、seqidno:10、seqidno:11、seqidno:12所示;
b、以slinc4481上下游同源臂片段为模板,以sg4481/u4481-r为引物,进行pcr扩增,获得以添加特异性识别slinc4481基因内序列的sgrna序列;sg4481的引物序列如seqidno:8所示;
c、将步骤b的sgrna序列克隆接入经spei/hindiii双酶切pkccas9do线性载体,获得pkccas9d4881敲除载体;
(2)构建δ4481突变株
将pkccas9d4881质粒转入大肠杆菌s17-1,获得pkccas9d4881/s17-1,将培养后的pkccas9d4881/s17-1与林肯链霉菌进行接合转移;通过含阿普拉霉素培养基筛选到转化子,转化子培养获得δ4481突变株;
(3)构建产林可霉素的基因工程菌δ4481
将δ4481突变株通过不含抗生素的培养基和含阿普拉霉素培养基进行培养和筛选进行培养和筛选获得基因工程菌δ4481,该基因工程菌δ4481通过发酵生产可以获得高产量的林可霉素;
步骤二、(1)构建slinc6156敲除载体
a、以u6156-f/r和d6156-f/r为引物,林肯链霉菌基因组为模板,进行pcr扩增,获得slinc6156上下游同源臂,所述u6156-f/r和d6156-f/r的引物序列分别如seqidno:23、seqidno:24、seqidno:25、seqidno:26所示;
b、以slinc6156上下游同源臂片段为模板,以sg6156/u6156-r为引物,进行pcr扩增,获得以添加特异性识别slinc6156基因内序列的sgrna序列;sg6156的引物序列如seqidno:22所示;
c、将步骤b的sgrna序列克隆接入经spei/hindiii双酶切pkccas9do线性载体,获得pkccas9d6156敲除载体;
(2)构建δ4481δ6156突变株
将pkccas9d6156质粒转入大肠杆菌s17-1,获得pkccas9d6156/s17-1,将培养后的pkccas9d6156/s17-1与基因工程菌δ4481进行接合转移;通过含阿普拉霉素培养基筛选到转化子,转化子培养获得δ4481δ6156突变株;
(3)构建基因工程菌δ4481δ6156
δ4481δ6156突变株通过不含抗生素的培养基和含阿普拉霉素培养基进行培养和筛选获得基因工程菌δ4481δ6156,该基因工程菌δ4481δ6156通过发酵生产可以获得高产量的林可霉素;
步骤三、(1)构建pib348重组质粒
分别以348-f/r和348-f/r为引物,林肯链霉菌基因组为模板,pcr扩增slinc348完整基因阅读框;纯化后的pcr产物与pib139质粒分别用限制性内切酶xbai和ecori酶切;将纯化的外源和载体使用t4连接酶链接成环,得到pib348重组质粒;
(2)构建δ4481δ6156::348突变株
将pib348重组质粒转入大肠杆菌s17-1,并培养;接着,与基因工程菌δ4481δ6156进行接合转移;通过含阿普拉霉素培养基筛选到转化子,转化子培养获得δ4481δ6156::348突变株;
(3)构建基因工程菌δ4481δ6156::348
δ4481δ6156::348突变株通过不含抗生素的培养基和含阿普拉霉素培养基进行培养和筛选获得δ4481δ6156::348,该基因工程菌δ4481δ6156::348通过发酵生产可以获得高产量的林可霉素。
根据本发明,步骤一和步骤二的pcr反应条件为:98℃,10分钟;98℃,40秒;64℃,30秒;72℃,80秒,30个循环;72℃,10分钟;25℃,2分钟;
步骤三的pcr反应条件为:所述pcr反应条件为:98℃,10分钟;98℃,40秒;64℃,30秒;72℃,140秒,30个循环;72℃,10分钟;25℃,2分钟。
作为本发明的第三个方面,一种产林可霉素的基因工程菌,其是将林肯链霉菌的编码含tpr结构域蛋白质的基因和编码rna酶的基因敲除构建获得。
根据本发明,所述编码含tpr结构域蛋白质的基因为slinc4481基因;所述编码rna酶的基因为slinc6156基因。
根据本发明,所述林肯链霉菌为林肯链霉菌nrrl2936。
作为本发明的第四个方面,一种上述所述的产林可霉素的基因工程菌的构建方法,其是以林肯链霉菌为出发菌株,将林肯链霉菌的编码含tpr结构域蛋白质的基因和编码rna酶的基因敲除,构建获得产林可霉素的基因工程菌。
进一步的,所述的产林可霉素的基因工程菌的构建方法,其是以林肯链霉菌为出发菌株,将林肯链霉菌的slinc4481基因和slinc6156敲除,构建获得产林可霉素的基因工程菌δ4481δ6156。
根据本发明,所述的产林可霉素的基因工程菌的构建方法,包括如下步骤:
步骤一、构建基因工程菌δ4481
以林肯链霉菌为出发菌株,将林肯链霉菌的slinc4481基因敲除,构建获得产林可霉素的基因工程菌δ4481;
步骤二、构建基因工程菌δ4481δ6156
以基因工程菌δ4481为出发菌株,将基因工程菌δ4481的slinc6156基因敲除,构建获得产林可霉素的基因工程菌δ4481δ6156。
根据本发明,pcr反应条件为:98℃,10分钟;98℃,40秒;64℃,30秒;72℃,80秒,30个循环;72℃,10分钟;25℃,2分钟。
作为本发明的第五个方面,一种产林可霉素的基因工程菌,其是将林肯链霉菌的编码含tpr结构域蛋白质的基因敲除构建获得。
根据本发明,所述编码含tpr结构域蛋白质的基因为slinc4481基因。
根据本发明,所述林肯链霉菌为林肯链霉菌nrrl2936。
作为本发明的第六个方面,一种上述所述的产林可霉素的基因工程菌的构建方法,其是以林肯链霉菌为出发菌株,将林肯链霉菌的编码含tpr结构域蛋白质的基因敲除,构建获得产林可霉素的基因工程菌。
进一步的,所述的产林可霉素的基因工程菌的构建方法,其是以林肯链霉菌为出发菌株,将林肯链霉菌的slinc4481基因敲除,构建获得产林可霉素的基因工程菌δ4481。
作为本发明的第七个方面,一种产林可霉素的基因工程菌,其是将林肯链霉菌的编码rna酶的基因敲除构建获得。
根据本发明,所述编码rna酶的基因为slinc6156基因。
根据本发明,所述林肯链霉菌为林肯链霉菌nrrl2936。
作为本发明的第八个方面,一种上述所述的产林可霉素的基因工程菌的构建方法,其是以林肯链霉菌为出发菌株,将林肯链霉菌的编码rna酶的基因敲除,构建获得产林可霉素的基因工程菌。
进一步的,所述的产林可霉素的基因工程菌的构建方法,其是以林肯链霉菌为出发菌株,将林肯链霉菌的slinc6156基因敲除,构建获得产林可霉素的基因工程菌δ6156。
根据本发明,所述的产林可霉素的基因工程菌的构建方法,包括如下步骤:
步骤一、构建slinc6156敲除载体
a、以u6156-f/r和d6156-f/r为引物,林肯链霉菌基因组为模板,进行pcr扩增,获得slinc6156上下游同源臂,所述u6156-f/r和d6156-f/r的引物序列分别如seqidno:23、seqidno:24、seqidno:25、seqidno:26所示;
b、以slinc6156上下游同源臂片段为模板,以sg6156/u6156-r为引物,进行pcr扩增,获得以添加特异性识别slinc6156基因内序列的sgrna序列;sg6156的引物序列如seqidno:22所示;
c、将步骤b的sgrna序列克隆接入经spei/hindiii双酶切pkccas9do线性载体,获得pkccas9d6156敲除载体;
步骤二、构建δ6156突变株
将pkccas9d6156质粒转入大肠杆菌s17-1,获得pkccas9d6156s17-1,将培养后的pkccas9d6156/s17-1与林肯链霉菌离心并进行接合转移;通过含阿普拉霉素培养基筛选到转化子,转化子培养获得δ6156突变株;
步骤三、构建产林可霉素的基因工程菌δ6156
将δ6156突变株通过不含抗生素的培养基和含阿普拉霉素培养基进行培养和筛选获得基因工程菌δ6156,该基因工程菌δ6156通过发酵生产可以获得高产量的林可霉素。
根据本发明,pcr反应条件为:98℃,10分钟;98℃,40秒;64℃,30秒;72℃,80秒,30个循环;72℃,10分钟;25℃,2分钟。
作为本发明的第九个方面,一种产林可霉素的基因工程菌,其是通过过表达林肯链霉菌的编码xre家族蛋白的基因构建获得。
根据本发明,所述编码xre家族蛋白的基因为slinc348基因。
作为本发明的第十个方面,一种上述所述的产林可霉素的基因工程菌的构建方法,其是以林肯链霉菌为出发菌株,过表达林肯链霉菌的编码xre家族蛋白的基因,构建获得产林可霉素的基因工程菌。
进一步的,所述的产林可霉素的基因工程菌的构建方法,其是以林肯链霉菌为出发菌株,过表达林肯链霉菌的slinc348基因,构建获得产林可霉素的基因工程菌。
作为本发明的第十一个方面,一种上述所述的产林可霉素的基因工程菌在发酵生产林可霉素中的应用。
本发明的产林可霉素的基因工程菌及其构建方法,其有益效果:其构建方法简便,通过敲除菌株nrrl2936的slinc6156基因、slinc6156基因、同时敲除slinc6156基因和slinc6156基因、或者同时敲除slinc6156基因和slinc6156基因并过表达slinc348基因,构建获得的林可霉素的基因工程菌δ4481δ6156::348、基因工程菌δ4481、基因工程菌δ6156以及基因工程菌δ4481δ6156,均可以提高林可霉素的工业产量,具有良好的工业应用价值。
附图说明
图1为slinc566基因敲除质粒的图谱。
图2为slinc4481基因敲除质粒的图谱。
图3为slinc6027基因敲除质粒的图谱。
图4为slinc6156基因敲除质粒的图谱。
图5为slinc348基因敲除质粒的图谱。
图6为slinc4742基因敲除质粒的图谱。
图7为slinc566基因敲除突变株构建示意图。
图8为slinc4481基因敲除突变株构建示意图。
图9为slinc6027基因敲除突变株构建示意图。
图10为slinc6156基因敲除突变株构建示意图。
图11为slin348基因敲除突变株构建示意图。
图12为slinc4742基因敲除突变株构建示意图。
图13为nrrl2936中slinc566、slinc4481、slinc6027、slinc6156、slinc348、slinc4742基因敲除突变株pcr鉴定电泳图。
图14为nrrl2936中slinc4481与slinc6156基因联合敲除突变株pcr鉴定电泳图。
图15为nrrl2936与δ4481δ6156中过表达slinc348突变株pcr鉴定电泳图。
图16为δ566、δ4481、δ6027、δ6156、δ348、δ4742突变株发酵液生物效价图。
图17为δ4481δ6156突变株发酵液生物效价图。
图18为o348突变株发酵液生物效价图。
图19为δ4481δ6156::348基因联合敲除突变株发酵液生物效价图。
具体实施方式
以下结合具体实施例,对本发明做进一步说明。应理解,以下实施例仅用于说明本发明而非用于限制本发明的范围。下列实施例中未注明具体条件的实验方法,按照常规方法和条件,或按照商品说明书选择。
1、本发明所用的引物序列见表1。
表1本发明研究中所用的引物序列
备注:斜体代表sgrna特异性识别靶基因的序列;加粗代表与相应同源臂重叠的序列;加粗并斜体代表与pkccas9do重叠的序列;划横线代表酶切位点。
2、本发明所用的培养基见表2
表2本发明研究中所用的培养基
3、本发明涉及的菌株和质粒:
(1)林肯链霉菌(streptomyceslincolnensis):nrrl2936;
(2)藤黄微球菌(micrococcusluteus):cgmcc28001,林可霉素生物效价指示菌。
(3)大肠杆菌dh5α、大肠杆菌s17-1为市售产品;
(4)质粒pkccas9do购自于addgene(http://www.addgene.org/62552/)。
(5)质粒pib139:链霉菌整合质粒,可购自北京华越洋生物(http://www.huayueyang.com.cn/product/277434631)。
4、本发明的用于质粒克隆的大肠杆菌dh5α和用于接合转移的大肠杆菌s17-1在28℃的lb固体(含1.8%琼脂)培养基或lb液体培养基培养。林肯链霉菌及其衍生株在28℃的ms固体培养基或yeme液体培养基培养。
5、slinc566、slinc4481、slinc6027、slinc6156、slinc348、slinc4742的序列分别如seqidno:47、seqidno:48、seqidno:49、seqidno:50、seqidno:51和seqidno:52所示。
6、pcr反应体系:taq酶mixture,5μl;模板1μl;引物各1μl;水;2μl。
实施例1slinc566、slinc4481、slinc6027、slinc6156、slinc348、slinc4742敲除载体的构建
本实施例中的基因敲除方案是基于crispr/cas9技术。
本实施例的pcr反应条件为:98℃,10分钟;(98℃,40秒;64℃,30秒;72℃,80秒)×30个循环;72℃,10分钟;25℃,2分钟。
1、slinc566敲除载体的构建
步骤一、以u566-f/r和d566-f/r为引物,林肯链霉菌nrrl2936基因组为模板,pcr扩增slinc566上下游同源臂。经1%琼脂糖凝胶电泳检测,将1.1kb处的目标条带切胶回收纯化。
步骤二、将上一步pcr反应得到的上下游同源臂片段作为模板,以sg566/u566-r为引物,pcr扩增以添加特异性识别slinc566基因内序列的sgrna序列。经1%琼脂糖凝胶电泳检测,将1.1kb处的目标条带切胶回收纯化。
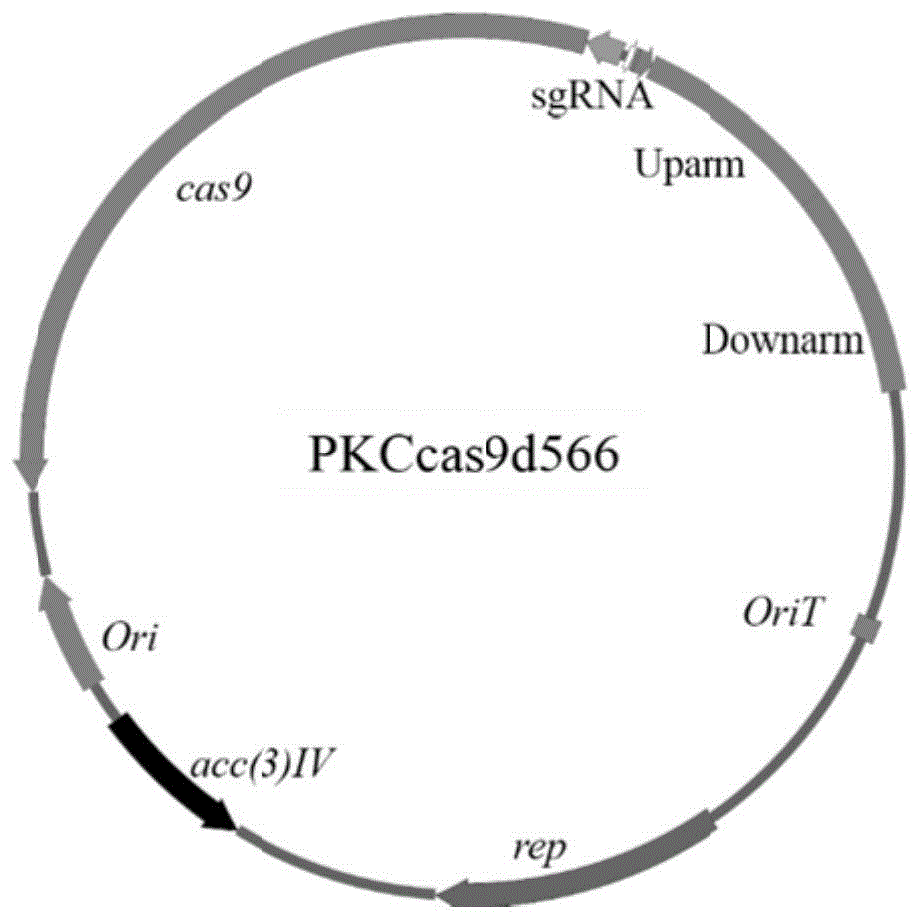
步骤三、将上述得到的带有sgrna序列的上游同源臂、下游同源臂,通过in-fusion克隆连接入经spei/hindiii酶切的pkccas9do线性载体,构建得到pkccas9d566敲除载体。pkccas9d566序列图谱如图1所示。
2、slinc4481敲除载体的构建
slinc4481敲除载体的构建的步骤与slinc566的步骤大致相同,区别是引物序列对不同。区别为:步骤一是以u4481-f/r和d4481-f/r为引物,pcr扩增slinc4481上下游同源臂;步骤二是以sg4481/u4481-r为引物,以slinc4481上下游同源臂片段为模板pcr扩增以添加特异性识别slinc4481基因内序列的sgrna序列。步骤三构建获得的pkccas9d4481的序列图谱如图2所示。
3、slinc6027敲除载体的构建
slinc6027敲除载体的构建的步骤与slinc566的步骤大致相同,区别是引物序列对不同。区别为:步骤一是以u6027-f/r和d6027-f/r为引物,pcr扩增slinc6027上下游同源臂;步骤二是以sg6027/u6027-r为引物,以slinc6027上下游同源臂片段为模板,pcr扩增以添加特异性识别slinc6027基因内序列的sgrna序列。步骤三构建获得的pkccas9d6027的序列图谱如图3所示。
4、slinc6156敲除载体的构建
slinc6156敲除载体的构建的步骤与slinc566的步骤大致相同,区别的地方是引物序列对不同。具体区别为:步骤一是以u6156-f/r和d6156-f/r为引物,pcr扩增slinc6156上下游同源臂;步骤二是以sg6156/u6156-r为引物,以slinc6156上下游同源臂片段为模板,pcr扩增以添加特异性识别slinc6156基因内序列的sgrna序列。步骤三构建获得的pkccas9d6156的序列图谱如图4所示。
5、slinc348敲除载体的构建
slinc348敲除载体的构建的步骤与slinc566的步骤大致相同,区别的地方是引物序列对不同。具体区别为:步骤一是以u348-f/r和d348-f/r为引物,pcr扩增slinc348上下游同源臂;步骤二是以sg348/u348-r为引物,以slinc348上下游同源臂片段为模板,pcr扩增以添加特异性识别slinc348基因内序列的sgrna序列。步骤三构建获得的pkccas9d4742的序列图谱如图5所示。
6、slinc4742敲除载体的构建
slinc4742敲除载体的构建的步骤与slinc566的步骤大致相同,区别的地方是引物序列对不同。具体区别为:步骤一是以u4742-f/r和d4742-f/r为引物,pcr扩增slinc4742上下游同源臂;步骤二是以sg4742/u4742-r为引物,以slinc4742上下游同源臂片段为模板,pcr扩增以添加特异性识别slinc4742基因内序列的sgrna序列。步骤三构建获得的pkccas9d4742的序列图谱如图6所示。
实施例2slinc566、slinc4481、slinc6027、slinc6156、slinc348、slinc4742基因敲除。
本实施例的pcr反应条件为:98℃,10分钟;(98℃,40秒;64℃,30秒;72℃,80秒)×30个循环;72℃,10分钟;25℃,2分钟。
1、本发明的slinc566基因敲除原理如图7所示。
步骤一、将pkccas9d566质粒转入大肠杆菌s17-1。经lb培养基液体培养至od600=0.4的pkccas9d566/s17-1与经yeme培养基液体培养至菌丝均匀浓密的林肯链霉菌nrrl2936,离心收集清洗后,用2×yt培养基重悬,共同涂布于isp4培养基,28℃培养,进行接合转移。涂板18h后,覆盖含阿普拉霉素(apramycin,20μg/ml)与萘啶酮酸(nalidixicacid,25μg/ml)的1ml无菌水溶液进行筛选。28℃继续培养4-7天后,可长出转化子菌落。挑取10个转化子菌丝体转接至含20μg/μl阿普拉霉素的ms培养基,28℃继续培养3-5天后,接种至yeme培养基28℃培养3天,抽提染色体,以jd566-f/r为引物,pcr鉴定阳性克隆,得到δ566突变株。引物验证结果如图13所示。
步骤二、将δ566突变株接种于yeme培养基,37℃培养2天后,按4%比例翻至新的yeme培养基,重复两次后,取菌液稀释1000倍后涂布于不含抗生素的ms培养基,28℃继续培养3-5天待长出单克隆后,挑20个单克隆分别转接至含20μg/μl阿普拉霉素与不含抗生素的ms培养基相同位置。培养3-5天后,若不含抗生素的ms培养基上长出克隆,而含阿普拉霉素的平板对应位置无克隆长出,则质粒顺利消除,得到无痕敲除株δ566(基因工程菌δ566)。
2、本发明的slinc4481基因敲除原理如图8所示。
敲除slinc4481基因的步骤与敲除slinc566基因的步骤大致相同,区别是引物序列对不同。区别为:步骤一将pkccas9d4481质粒转入大肠杆菌s17-1。经lb培养基液体培养至od600=0.4的pkccas9d4481/s17-1与经yeme培养基液体培养至菌丝均匀浓密的林肯链霉菌nrrl2936;另外,步骤一是以jd4481-f/r为引物,pcr鉴定阳性克隆,得到δ4881突变株。引物验证结果如图13所示。
步骤二是将δ4881突变株接种于yeme培养基。
3、本发明的slinc6027基因敲除原理如图9所示。
敲除slinc6027基因的步骤与敲除slinc566基因的步骤大致相同,区别是引物序列对不同。区别为:步骤一将pkccas9d6027质粒转入大肠杆菌s17-1。经lb培养基液体培养至od600=0.4的pkccas9d6027/s17-1与经yeme培养基液体培养至菌丝均匀浓密的林肯链霉菌nrrl2936;另外,步骤一是以jd6027-f/r为引物,pcr鉴定阳性克隆,得到δ6027突变株。引物验证结果如图13所示。
步骤二是将δ6027突变株接种于yeme培养基。
4、本发明的slinc6156基因敲除原理如图10所示。
敲除slinc6156基因的步骤与敲除slinc566基因的步骤大致相同,区别是引物序列对不同。区别为:步骤一将pkccas9d6156质粒转入大肠杆菌s17-1。经lb培养基液体培养至od600=0.4的pkccas9d6156/s17-1与经yeme培养基液体培养至菌丝均匀浓密的林肯链霉菌nrrl2936;另外,步骤一是以jd6156-f/r为引物,pcr鉴定阳性克隆,得到δ6156突变株。引物验证结果如图13所示。
步骤二是将δ6156突变株接种于yeme培养基。
5、本发明的slinc348基因敲除原理如图11所示。
敲除slinc348基因的步骤与敲除slinc566基因的步骤大致相同,区别是引物序列对不同。区别为:步骤一将pkccas9d348质粒转入大肠杆菌s17-1。经lb培养基液体培养至od600=0.4的pkccas9d348/s17-1与经yeme培养基液体培养至菌丝均匀浓密的林肯链霉菌nrrl2936;另外,步骤一是以jd348-f/r为引物,pcr鉴定阳性克隆,得到δ348突变株。引物验证结果如图13所示。
步骤二是将δ348突变株接种于yeme培养基。
6、本发明的slinc4742基因敲除原理如图12所示。
敲除slinc4742基因的步骤与敲除slinc566基因的步骤大致相同,区别是引物序列对不同。区别为:步骤一将pkccas9d4742质粒转入大肠杆菌s17-1。经lb培养基液体培养至od600=0.4的pkccas9d4742/s17-1与经yeme培养基液体培养至菌丝均匀浓密的林肯链霉菌nrrl2936;另外,步骤一是以jd4742-f/r为引物,pcr鉴定阳性克隆,得到δ4742突变株。引物验证结果如图13所示。
步骤二是将δ4742突变株接种于yeme培养基。
实施例3基因工程菌δ4481δ6156的构建
本实施例的pcr反应条件与实施例2相同。
步骤一、将pkccas9d6156质粒转入大肠杆菌s17-1。经lb培养基液体培养至od600=0.4的pkccas9d6156/s17-1与经yeme培养基液体培养至菌丝均匀浓密的基因工程菌株δ4481,离心收集清洗后,用2×yt培养基重悬,共同涂布于isp4培养基,28℃培养,进行接合转移。涂板18h后,覆盖含阿普拉霉素(apramycin,20μg/ml)与萘啶酮酸(nalidixicacid,25μg/ml)的1ml无菌水溶液进行筛选。28℃继续培养4-7天后,可长出转化子菌落。挑取10个转化子菌丝体转接至含20μg/μl阿普拉霉素的ms培养基,28℃继续培养3-5天后,接种至yeme培养基28℃培养3天,抽提染色体,以jd6156-f/r为引物,pcr鉴定阳性克隆,得到δ4481δ6156突变株。引物验证结果如图14所示。
步骤二、将δ4481δ6156联合敲除突变株接种于yeme培养基,37℃培养2天后,按4%比例翻至新的yeme培养基,重复两次后,取菌液稀释1000倍后涂布于不含抗生素的ms培养基,28℃继续培养3-5天待长出单克隆后,挑20个单克隆分别转接至含20μg/μl阿普拉霉素与不含抗生素的ms培养基相同位置。培养3-5天后,若不含抗生素的ms培养基上长出克隆,而含阿普拉霉素的平板对应位置无克隆长出,则质粒顺利消除,得到无痕联合敲除株δ4481δ6156(基因工程菌株δ4481δ6156)。
实施例4slinc348过表达菌株的构建
分别以348-f/r和348-f/r为引物,林肯链霉菌nrrl2936基因组为模板,pcr扩增slinc348完整基因阅读框。pcr反应条件为:98℃,10分钟;(98℃,40秒;64℃,30秒;72℃,140秒)×30个循环;72℃,10分钟;25℃,2分钟。经1%琼脂糖凝胶电泳检测,将2.3kb处的目标条带切胶回收纯化。纯化后的pcr产物与pib139质粒分别用限制性内切酶xbai和ecori酶切(37℃,1小时)。经1%琼脂糖凝胶电泳检测并回收对应大小条带后,将纯化的外源和载体使用t4连接酶链接成环(22℃,1小时),得到pib348重组质粒。
将pib348质粒转入大肠杆菌s17-1后按实施例2中的步骤转入野生株nrrl2936,得到o348突变株,引物对824f/r用于pcr鉴定。引物验证结果如图15所示。
将pib348质粒转入大肠杆菌s17-1后按实施例2中的步骤转入突变株δ4481δ6156,得到δ4481δ6156::348突变株,引物对824f/r用于pcr鉴定。引物验证结果如图15所示。
实施例5基因工程菌δ566、基因工程菌δ4481、基因工程菌δ6027、基因工程菌δ6156、基因工程菌δ348、基因工程菌δ4742、基因工程菌o348、基因工程菌δ4481δ6156、基因工程菌δ4481δ6156::348的摇瓶发酵及效价检测。
分别用无菌牙签挖取约1.5厘米×1.5厘米的ms培养基上生长4-5天的基因工程菌δ566、δ4481、δ6027、δ6156、δ4481δ6156、δ4481δ6156::348,接种于含25mlsfpi培养基的一级摇瓶,28℃培养(200转/秒)4天后,取1ml接种于含25mlsfpii培养基的二级摇瓶,28℃培养(200转/秒)6天。取1ml发酵结束的样品于1.5ml离心管,12000转/秒离心10分钟后将上清转移,保存待测。
本发明中的效价检测采用管碟法,以藤黄微球菌(micrococcusluteus28001)作为指示菌株(涂布于培养基iii培养20小时后,用无菌的0.9%nacl溶液将菌苔洗下备用),利用林可霉素在琼脂培养基中的扩散作用,绘制标准品扩散曲线,并将待测品与之比较,从而测定待测品效价。
将15ml培养基iii下层固体培养基均匀摊铺于培养皿中,待其冷却凝固后,取200μl指示菌悬液与5ml融化且温度适宜的培养基iii上层培养基均匀混合后倒在下层培养基上面,凝固后放置灭菌牛津杯。
向牛津杯中加入200μl浓度为2、4、6、8、10、12mg/l的标准品及待检测发酵上清,于37℃恒温培养箱正置培养20小时后,用游标卡尺准确量取所产生抑菌圈的直径。以标准品浓度值的自然对数为横坐标,抑菌圈直径为纵坐标绘制标准曲线。将发酵液上清抑菌圈直径代入标准曲线,计算可得林可霉素浓度。结果如图16-19所示。
图16-19的结果显示,
(1)突变株δ566、突变株δ6027以及突变株δ4742的林可霉素产量与出发菌株nrrl2936的林可霉素产量几乎一致。说明敲除slinc566基因、敲除slinc6027基因或slinc4742基因获得的突变株的产林可霉素的能力几乎不变。
(2)突变株δ4481的林可霉素产量达到1.55g/l,为出发菌株nrrl2936的18.7倍。说明突变株δ4481产林可霉素的能力大幅度提升,有助于提高林可霉素的产量。
(3)突变株δ6156的林可霉素产量达到1.89g/l,为出发菌株nrrl2936的22.4倍。说明突变株δ6156产林可霉素的能力大幅度提升,有助于提高林可霉素的产量。
(4)突变株δ4481δ6156的林可霉素产量达到2.74g/l,为出发菌株nrrl2936的32.4倍。说明突变株δ4481δ6156产林可霉素的能力大幅度提升,有助于提高林可霉素的产量。
(5)突变株δ348的林可霉素的产量检测不到,说明slinc348基因是一个参与林可霉素合成过程的正调控因子。
(6)突变株o348的林可霉素的产量可达到0.57g/l。说明突变株o348产林可霉素的能力大幅度提升,有助于提高林可霉素的产量。
(7)突变株δ4481δ6156::348的林可霉素产量达到3.58g/l,为出发菌株nrrl2936的42.4倍。说明突变株δ4481δ6156::348产林可霉素的能力大幅度提升,有助于提高林可霉素的产量。
结论:敲除slinc4481基因、敲除slinc6156基因、过表达slinc348基因、同时敲除slinc4481基因与slinc6156基因的方法、或者同时敲除slinc4481与slinc6156并过表达slinc348的方法均可以应用于林可霉素高产菌株的构建方法中,以提高林可霉素的产量。
以上所述,仅为本发明的较佳实施例,并非对本发明作任何形式上和实质上的限制,凡熟悉本专业的技术人员,在不脱离本发明技术方案范围内,当可利用以上所揭示的技术内容,而作出的些许更动、修饰与演变的等同变化,均为本发明的等效实施例;同时,凡依据本发明的实质技术对以上实施例所作的任何等同变化的更动、修饰与演变,均仍属于本发明的技术方案的范围内。
序列表
<110>华东理工大学
<120>一种产林可霉素的基因工程菌及其构建方法和应用
<141>2020-01-16
<160>52
<170>siposequencelisting1.0
<210>1
<211>88
<212>dna
<213>人工序列(artificialsequence)
<400>1
cagtcctaggtataatactaggctgcaacgcaaggcggtcggttttagagctagaaatag60
caagttaaaataaggctagtccgttatc88
<210>2
<211>79
<212>dna
<213>人工序列(artificialsequence)
<400>2
tagcaagttaaaataaggctagtccgttatcaacttgaaaaagtggcaccgagtcggtgc60
cgatgccgtgcagcagttt79
<210>3
<211>17
<212>dna
<213>人工序列(artificialsequence)
<400>3
cgcctgccggttgacca17
<210>4
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>4
cgcgaggctggtcaaccggcaggcgctggcgctgatgtcgatgg44
<210>5
<211>39
<212>dna
<213>人工序列(artificialsequence)
<400>5
acgacggccagtgccaagctggacggtgcacatgatggg39
<210>6
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>6
cgtgtcgtgcatcgaacgc19
<210>7
<211>17
<212>dna
<213>人工序列(artificialsequence)
<400>7
cttcgtcctcggcaggg17
<210>8
<211>88
<212>dna
<213>人工序列(artificialsequence)
<400>8
cagtcctaggtataatactagccacgacctcgcgttactgagttttagagctagaaatag60
caagttaaaataaggctagtccgttatc88
<210>9
<211>78
<212>dna
<213>人工序列(artificialsequence)
<400>9
tagcaagttaaaataaggctagtccgttatcaacttgaaaaagtggcaccgagtcggtgc60
ccgtcctctcctcggctc78
<210>10
<211>17
<212>dna
<213>人工序列(artificialsequence)
<400>10
cgggccacggtgtccat17
<210>11
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>11
cgcggatcatggacaccgtggcccgggcacgctgtctggaatggc45
<210>12
<211>37
<212>dna
<213>人工序列(artificialsequence)
<400>12
acgacggccagtgccaagctcagacgcgacgacgttg37
<210>13
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>13
ggtgtccggtcggtcgaa18
<210>14
<211>17
<212>dna
<213>人工序列(artificialsequence)
<400>14
cgctggtgtccgtctcc17
<210>15
<211>88
<212>dna
<213>人工序列(artificialsequence)
<400>15
cagtcctaggtataatactagcgacggcgatgttccgccaggttttagagctagaaatag60
caagttaaaataaggctagtccgttatc88
<210>16
<211>79
<212>dna
<213>人工序列(artificialsequence)
<400>16
tagcaagttaaaataaggctagtccgttatcaacttgaaaaagtggcaccgagtcggtgc60
cttggtcgccgtgtccagg79
<210>17
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>17
cagctcgcccttggaccag19
<210>18
<211>46
<212>dna
<213>人工序列(artificialsequence)
<400>18
agcggctggtccaagggcgagctgatgcagatcgccaagaaggtcc46
<210>19
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>19
acgacggccagtgccaagctccaggaaccggcgttgaagc40
<210>20
<211>17
<212>dna
<213>人工序列(artificialsequence)
<400>20
gctcggcgtaccggtca17
<210>21
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>21
ccatggcgcacaggacac18
<210>22
<211>88
<212>dna
<213>人工序列(artificialsequence)
<400>22
cagtcctaggtataatactaggaaggtcccaccgggtctgggttttagagctagaaatag60
caagttaaaataaggctagtccgttatc88
<210>23
<211>79
<212>dna
<213>人工序列(artificialsequence)
<400>23
tagcaagttaaaataaggctagtccgttatcaacttgaaaaagtggcaccgagtcggtgc60
gtcctcaccgccatggtca79
<210>24
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>24
gaccgccgtactcgaagacc20
<210>25
<211>48
<212>dna
<213>人工序列(artificialsequence)
<400>25
gaaacatgacggtcttcgagtacggcggtcgtggtcgaaccccaccag48
<210>26
<211>43
<212>dna
<213>人工序列(artificialsequence)
<400>26
acgacggccagtgccaagctccaataattccggacaacgcttg43
<210>27
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>27
agcgcgagatcgagatggt19
<210>28
<211>16
<212>dna
<213>人工序列(artificialsequence)
<400>28
gccccgctccaggttg16
<210>29
<211>88
<212>dna
<213>人工序列(artificialsequence)
<400>29
cagtcctaggtataatactagcacccgcacgccagttgttcgttttagagctagaaatag60
caagttaaaataaggctagtccgttatc88
<210>30
<211>79
<212>dna
<213>人工序列(artificialsequence)
<400>30
tagcaagttaaaataaggctagtccgttatcaacttgaaaaagtggcaccgagtcggtgc60
cttcgctccgggtgtactg79
<210>31
<211>17
<212>dna
<213>人工序列(artificialsequence)
<400>31
gcccagcgcgtccgatt17
<210>32
<211>43
<212>dna
<213>人工序列(artificialsequence)
<400>32
cggtccgcaatcggacgcgctgggccgcgccctggagttcgac43
<210>33
<211>38
<212>dna
<213>人工序列(artificialsequence)
<400>33
acgacggccagtgccaagcttccaggtctcggcggtca38
<210>34
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>34
gaagtcgtggatgatgccct20
<210>35
<211>17
<212>dna
<213>人工序列(artificialsequence)
<400>35
tccctgccccggtgaag17
<210>36
<211>88
<212>dna
<213>人工序列(artificialsequence)
<400>36
cagtcctaggtataatactagaggccaatgtggcgggctgggttttagagctagaaatag60
caagttaaaataaggctagtccgttatc88
<210>37
<211>82
<212>dna
<213>人工序列(artificialsequence)
<400>37
tagcaagttaaaataaggctagtccgttatcaacttgaaaaagtggcaccgagtcggtgc60
aggtcccgcgctcgtctacacc82
<210>38
<211>17
<212>dna
<213>人工序列(artificialsequence)
<400>38
ccgcgcctcacgcagtc17
<210>39
<211>36
<212>dna
<213>人工序列(artificialsequence)
<400>39
ctgcgtgaggcgcggggtgccttcacgatcctgtcc36
<210>40
<211>39
<212>dna
<213>人工序列(artificialsequence)
<400>40
acgacggccagtgccaagcttgttgccgcccagttcgag39
<210>41
<211>17
<212>dna
<213>人工序列(artificialsequence)
<400>41
cgcccgcccaagctgta17
<210>42
<211>17
<212>dna
<213>人工序列(artificialsequence)
<400>42
acctggccctggtggac17
<210>43
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>43
ggaacgtctagacgtggtcggtccgcaatc30
<210>44
<211>31
<212>dna
<213>人工序列(artificialsequence)
<400>44
gtcccggaattctcagtcgtgtgttccgggc31
<210>45
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>45
agtgtccgttcgagtggcggcttg24
<210>46
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>46
gcaccccaggctttacactttatgcttc28
<210>47
<211>1485
<212>dna
<213>人工序列(artificialsequence)
<400>47
gtgagcggtcaacccaacacccgtctgacggacctgttcggcctggccggctggtccaag60
ggcgaactcgcgaggctggtcaaccggcaggcggcggccatgggccaccctcagctggcg120
accgacacctcccgggtgcggcggtggatcgacatgggagagatcccacgcgatccggtg180
ccgcgggtgctggcggccctgttcaccgagcgtctcggccgtgtcgtgaccatcgaggac240
ctcggtctggtccggcacgggcgtacggggaaacggccacgcgacgggagtgcggaacat300
cccgacggtgtgccgtgggcgcccgagcggactgctgcggtcctcaccgaattcacggga360
atggacctcatgctcaaccgacgcggcttggtgggcgcgggtgccgcgctcgccgcggga420
tcagccctcagcagtgccatgtacgactggctgcacaccgatccgaccctgaccgccgac480
gctccccggttcgacgaccccctgcatgccgaccccgctgggttcgaccgctacgaggcc540
gcccccatcgggtcgcaggagatcgaggaactggaacgctccgtcgaagtgttccgggcc600
tgggacgcggcccgcggcggcgggctgcaacgcaaggcggtcgtgggccaactcaacgag660
gtgggcggcatgctcgcctaccgacaccccgaccatctccagcggcgcctgtggggcgtc720
gccgccaacctcgccgtcctcgcgggctggatgtcgcacgacgtcggtctcgaacccacg780
gcccagaagtacttcgtcatcgccgcccatgccgccagagagggcggcgaccggccccgc840
gccggggaggcgctctcccgagcggctcgccagatggtgcaccttggccggcccgacgac900
gcgctcgacctgatgaacctcgcccagtccggctccggcgaacaggtgctgccccgcacc960
cgggcgatgctccacaccatcgaggcctgggcacaggcctcgatgggcaagggccaggcc1020
atgcgccgcaccctcggagtggcggaggacctcttcgtctccgacaagggcgacgtgccg1080
ccgccgagctggatgcagaccttcagggaggaggacctgtacggcatgcaggccctggcc1140
taccgcacgctggccgagttcgagcccggcgcggccgcgcacgcccagcactacgcggag1200
aaggccctggccctgcgggtcgacggacggcagcggtcgaagatcttcgacttcctgtcc1260
atggcgtcggcctgcttcatcgccgacgaccccgaacaggccgaccggtacgcacgcctg1320
gcgctgatgtcgatgggctccaactcctcccaccgcacctgggaccggctgcgtcagatg1380
taccggctcaccgccgagtactccggctatccgaagatcagcgaactgcgggaggagatc1440
aggctggccctgccgaggacgaagggcagctcggtcaggacgtga1485
<210>48
<211>2448
<212>dna
<213>人工序列(artificialsequence)
<400>48
gtgcgtgcttcctccccggacggaccggaggcgttgctgtcggccctggccacctgcggc60
gccgacccggattcccccgcatggcgcgacctgacgcggatcatggacaccgtggcccgc120
gcggatccggcggtgtcccggttctggcccccacatgtgacgggggggcatcccgccgag180
ccgacgcactggcgggaactggcccgggaactccacgtcctggcgtcgcgtgacgcggcg240
gcacgccaggccctgaccgagtgggtgcgtcaccaccccggcgccgcccgtacgccgtcc300
ggtccggcgccctcggcgacggccaatgtcatcagcggcgactccgtgctccacggcccc360
agcgtgcaagcccgggacatccacggcggcatccacttccacccgtcctccccgccacca420
cgggcgggcctccccgtgccccgccaactgcctccggtgaccgcacggttcgtcgaccgg480
gaggacgaccgacgggccctggacgccctgcgtgcccggcgcgcgccgcacgcacctcag540
gtcctggtggtcagcgggctcccgggcgtcggaaagaccaccttcgccacccactggctg600
catgagcacgccgaaagtttccccgacggccagctctacgccgacctcggcggacgggcc660
accgacgagggcgacggacctgtgtcccccgccgccgtactggaagccttcctggtcgca720
ctcggcgcgtcctcggtaccgcccggtagcgctcaacggagcgcgctgtggcgttcgatg780
acctcaggtctacgactggcgctgctcctggacagcgcgttcacggctgcgcaggtacgc840
ccgctcctgcccggcacaccgaccggcctcaccgtggtgaccagcacaagcatgctgacc900
ggactccacatcgacggcgcgtccgtacaccggctggaggggctccccgccgaatcggcc960
gtcgaactcctcgccttcggcggtgggacgcgcgtggcacgggagccgaccgccgcgcgt1020
gaagtggtcagactctgcggccatctgcctctggcggtggctctggcctctgcccagctc1080
gccctgcgtcctcaccgttccgtgtccgccctggtcgacagcctgggtcgtcgaagcccc1140
gtcgagacgctgcacgtcgaaggggaggccgtgatgcgtacggcactcgacaggacgtac1200
gacgttctcccggacaacagccgtgtcctctaccgccggatgggacttctgcccgcggat1260
cgccacgacctcgcgttactgacggccctcgcgggcaacgaggaggacacgcgcgacacc1320
gggcaggctgccgacatcgccgtccatgctctggtggaggcgaacctcctgcaggagacg1380
ggcccggggacctatcgcttccacgacctcgtccagccgcacgcccgtcgactcggcgag1440
gagctggaggacgccgaccggcaggagcgcacactgcgctgcttcgtggactggtgcctg1500
tccaccgcggcttcggcggagaccctcctcacccccagccaccggctgcccgggcacgac1560
ctctccgcgtgcaccgtgcctccgaccccgctcacgggtcccgaggaagccctcgcctgg1620
ctggacacgtaccgcaacggcctgatgggggcggtacggcactcggcccgggtcggctgg1680
gactccttctgctggcgcctggtggacctcatatggccccttttcctgcgattacgtccg1740
tccgagatgtggatcgaggcgcatcgactcggtctcgacgcggcgcgccgatgccgttcg1800
cggcagggcgagggccgcatgctcacctccggcgcgatcggcttgcggaacgcgggccgg1860
tactcggaggcggcggactggtaccggcaggcactggagcaggctgcggccgacgacgac1920
gtacgccaacaggcacaagccgtcagcgggctgggccatgtgagcctcctggcccaccgg1980
ctcgacgaggcacgcggccacttcgaacacgcgctgcgtctgagggaggcgatcggctac2040
cgacgcggagccgccctctcccggcggcgtctcggcgaaaccgctctggccggcggcgac2100
ctggccacggccaccgaacaactgcgccgtgccggcaccgaactggactcactgggcgag2160
acatacgaggcgacccgcgtcctggctcttctcggccatgtcctggaccgcagcggggac2220
ggtgagggaggcgcgcgccgactgcgtgaggctctcgcgcgattccgaacgggcggcacc2280
cggtccgagcactgggaggcacgctgtctggaatggctcggccaggccgcggagtcgctt2340
ggtgatcatgatgaatcgatacgccactacgaggccgcccgggaactcttccgacgcctg2400
aaccccgaggacgcgcaacgcctggacggtcggctgcggcggtcatga2448
<210>49
<211>1494
<212>dna
<213>人工序列(artificialsequence)
<400>49
gtgagcggcaacggcgatggcggtacgaacgcgacgaatgccggcaccacggacaagcgc60
cccaacgagctgctcacttcatggttcgtgcgcagcggctggtccaagggcgagctggcc120
cgccaggtcaaccggcgggcccgccagctgggggccaaccacatctccaccgacacctcg180
cgcgtgcgccgctggctggacggtgagaatccccgcgaacccatccccaggatcctgtcc240
gagctgttctcggagcggttcggctgtgtcgtctccgtcgaggatctcggactgcgcgcc300
gcccgccagtcaccctccgcgaccggtgtcgacctgccctggacgggcccgcagacggtg360
gccctgctcagcgagttctcgcgcagcgacctgatgctggcgcggcgcggcttcctcgga420
acctcgctggtcctgtccgcgggcccgtccctcatcgagcccatgcagcgctggctggtg480
ccggcgccctccgtcccgcggcacgagcccgagccggctccgtcgtcggcccgccgcggc540
cggctctccaagcccgagctggacctcctggagtcgacgacggcgatgttccgccagtgg600
gacgcccagtgcggcggcggtctgcgccgcaaggcggtcgtcggccagctgcacgaggtg660
accgacctgctccaggagccgcagcccgaggccaccacccgcaagctgttcaaggtcgcc720
gccgagctcgccgaactggccggctggatgtcgtacgacgtcgggctccagcccaccgcg780
cagaagtacttcgtcctcgccctgcacgcggccaaggaagccggggacaagccgctcggc840
tcctatgtcctgtccagcatgagccgccagatgatccacctcgggcggcccgaggacgcg900
ctggagctgatccacctcgcgcagtacggcagccgcgactgcgcgagcccgcgcacccag960
tcgatgctgtatgcgatggaggcccgcgcctacgccaacatgggccagcccggtaagtgc1020
aagcgggcggtccggatggccgaggacaccttcggtgaggccgacgagtgggacgagccc1080
gaccccgactggatccgcttcttctccgaggccgagctctacggcgagaactcccactcc1140
taccgcgacctcgcctatgtcgccggtcgcagccccacctacgcctccctggccgagccc1200
ctgatgcagcgggccgtcgagctcttcgccaaggacgacgaacaccagcggtcgtacgca1260
ctgaacctgatcggcatggccacggtgcacctgctgcggcgcgagcccgaacagagcacc1320
gtgctggccgccgaggccatgcagatcgccaagaaggtccgctccgagcgggtcaacact1380
cgtatccgaaagacggtcgacacggccgtacgcgatttcggtgacctcgccgaggtcatc1440
cacctcaccgaccagctcgccatcgagctgcccgagaccgcggaggcggtctga1494
<210>50
<211>1686
<212>dna
<213>人工序列(artificialsequence)
<400>50
ttgagtcatccgcatcctgaactcggcacgcccccgccgctgcccgagggcggcctccgg60
gtcaccccactgggcggcctcggcgaaatcggccgaaacatgacggtcttcgagtacggc120
ggtcgcctgctgatcgtcgactgcggcgtgctcttcccggaggaggagcagcccggaatc180
gatctgatcctgccggacttctcgtccatcagggaccgcctcgacgacatcgagggcatc240
gtcctcacccatggccacgaggaccacatcggcggcgtccccttcctcctgcgcgagaag300
ccggacatcccgctgatcggctccaagctgaccctcgccctgatcgaggccaagctccag360
gagcaccggatccgcccgtacaccctggaggtggcggaggggcaccgcgagcgcgtcggc420
cccttcgactgcgagttcgtcgcggtcaaccactccatcccggacgccctggccgtggcc480
atccgcacccccgccggcatggtggtgcacacgggcgacttcaagatggaccagctcccg540
ctggacaaccgcctcaccgatctgcacgcgttcgcccggctgagcgaggaagggatcgac600
ctcctcctctccgactctacgaacgccgaggtccccgggttcgtcccgccggagcgggac660
atctccaacgtcctgcgtcaggtcttcggcaacgcccgcaagcggatcatcgtggcgagc720
ttcgccagccacgtccaccgcatccagcagatcctggacgccgcccacgagtacggccgc780
agggtcgccttcgtcggccgctcgatggtccgcaacatgggcatcgcgcgcgacctgggc840
tacctgaaggtcccaccgggtctggtggtcgacgtcaagacactcgacgacctccccgac900
cacgaggtggtcctggtctgcacgggctcccagggcgaaccgatggccgccctctcccgc960
atggccaaccgggaccaccagatccggatcgtcaacggcgacacggtgatcctggcgtcg1020
tccctgatccccggcaacgagaacgcggtctaccgcgtgatcaacggcctgacccgctgg1080
ggcgccaacgtcgtccacaagggcaacgccaaggtccatgtctccggccacgcgtccgcg1140
ggcgagttgctgtacttctacaacatctgccgcccgaagaacctgatgccggtccacggc1200
gaatggcgccacctccgggccaacgccgaactgggcgccctcaccggcgtcccgcacgac1260
cggatcgtcatcgcggaggacggcgtcgtcgtcgacctcgtcgagggcaaggccaagatc1320
tccggcaaggtccaggcgggctacgtgtacgtcgacggcctctcggtcggagatgtcggt1380
gagccggcgctcaaggaccgcaagatcctcggcgacgagggcatcatctcggtcttcgtg1440
gtcatcgacgcctccaccggcaagatcacaggtggtccgcatatccaggcccgcggctcg1500
ggcatcgaggactccgccttcgccgacgtcatcccgagggtcaccgaagtcctggagcgt1560
tcggcccaggacggcgtggtcgaaccccaccagctccagcagctggtgcggcgaaccctc1620
ggcaagtgggtctcggacacctatcggcgcaggccgatgatcctgcctgtcgtggtggag1680
gtctga1686
<210>51
<211>2334
<212>dna
<213>人工序列(artificialsequence)
<400>51
gtggtcggtccgcaatcggacgcgctgggccccttgctgcgcggccacagacacgcggcg60
gggatgaccctggaggaactcgccctcgcctccggtgtcagcgaccgcgccatcggcgac120
atggaacgcggccgcagccgaggaccccaggcccgcaccgtccaggccctcgccaccgcc180
ctccgcctgccccgggaggacaccgaccgcctgctcaccgccgcgcgcgacgggcgccgc240
cgaacccgccgaacccggcgccccccgcacggcctgtgcgatcttcccccggccgtaccg300
gacttcgccgggcgcgacgaggagatccgccggctcgacgcggccgggacgccgggaacc360
gccgcgatcccggtgatcgtcagcggcgcccccggcctggggaagacgacactggtcgtc420
caggccgcgcaccggcaggcggcgcgctacgacgacggatgcctgttcctcaacctgcgc480
ggcctggacgaggaacccctcgacccgcacgacgcgctggcccgcctgctcaaggcactc540
ggagtgcccgaacacgacctgcccgcggacctggaggagcgccaggtgctccatcgacgc600
gtcgtccgggacaagaacgtcctggtcatcctcgacaacgccgccgacgaggcccaactg660
cgccccctgctgccgggcgacgggaagtgcgtgttctgggtcaccagccgccgcgcgctg720
accggcatcgagcacgcccgccgactcgtgccgaccccgctcccggcccacgcggccacc780
accctgctggagacgatcaccgccgaccgcacggaccggcaggacaccgcgtccctacga840
cgcatcgccgacctctgcggcggcctgcccctcgccctgcgcatcgcaggcaaccgcctg900
gtgagccgcccggcctggagcgcgaccggcctcgccgaccggctcgcggccgaggacctg960
cgcctggaccggctcaccgccggagacctgaaggtgaaatccgccttcactctctcctac1020
gagcaactcaccgcacccgcacgccagttgttccggcgcctctccctcgtccagggcccc1080
gacttctcgcccgcccacggcgcggcgctcacctcgttcacgccggcacgcgtcgagcgg1140
atgaccgacgaactcatcgaactcggcctgctgaacgccgccggcggcgaccgggtctcc1200
ttccacgacctggtccggctgtacgcccaccagcgcctgcgtgacgaggagacacccgag1260
gaacgcgaggccgcccgagcgcggttggacggctggctcctggacaccgcccgcgccgcc1320
ggacagtggttcgaacccggcgacgacccgcggccggtccccggtccgccggccgacggc1380
cacgacccggcctccgcgcagcgctggctccagacggagagcgcccactggttcgccgcc1440
ctgcgggccgcggcggcggccggccaccaccggcgggtcgtcgacgtcgcggagtccatg1500
cactggttctccgaccgctggtcccactgggggcactggcacactgtcttcgccctgtcg1560
cgggccgccgctcacgcgctcggtgaccccggactcgaagcgacccacgccaactacctg1620
tcctgggcggtcgcccaatgcctcgaccgcccggccgagggcatggcgatcgcgctggag1680
gccgcggaactggcccggcgcgcgcacgacccggttcaggaggcatggtccctgacgtac1740
gccgcctatgccgcccgggattcgggggagttcacgtccgccgtggagcagacccggcgg1800
gcggccgaactgttcgccgccgcgggcgacaaggaggggcacccgcaggcgctgctgggc1860
ctcgccctcaacctgcgcgccgtgggccggctggaggaggcggccgacgccttcgacgcc1920
gccgtggcacgggtgagcgatccgcgtaccgccccggcaccgcacatcgccgacttcacg1980
gcgatgaacgccctcagcggcaaggcgcgagtactcctggacctggaggactgggaggcc2040
gcgcacgcggtctgcgaccgcgccctggagttcgacgcccgggtcggggtcgccctgaac2100
cgcggagtgccgacgatgcgcaaggcccaggccctgtgggcgctgggccggcgaccggag2160
gcactcgcggcgctggaggacgccctgcgctgcttcaccggggcagggagcgaccgatgg2220
ctggacgaggcgaggaacctgcaccagcagtggaccgcgcccgaccacaggccagggccg2280
cctcctgccactcccggggcgccaggccgtacgcggcccggaacacacgactga2334
<210>52
<211>825
<212>dna
<213>人工序列(artificialsequence)
<400>52
atgctgctcggctcgcaactcaggcgactgcgtgaggcgcggggcatcacgcgtgaggcg60
gcgggctactcgatccgcgcgtccgagtcgaagatcagccggatggagttgggccgggtg120
agcttcaagacacgggacgtcgaggatctgctgacgctgtacggcatcacggacgagcag180
gagcgcacgtcactcctctctctcgcccgggaggccaatgtggcgggctggtggcacagt240
tactcggacgtcctgccgagctggttccccacctatgtgggcctggagggcgcggccgcc300
ctgatccgggcctacgaggtgcagttcgtgcacggtctgctccagaccgaggcgtacgcg360
cgcgcggtcgtgcggcgcggtatgaagggcgcgagcgaggccgacgtcgaacgccgggtg420
ggggtgcgcctggagcggcagaagtacctggtcgacgagaacgcgccggacgtccacatc480
gtcctggacgaggccgcgctgcgccggccctacggcgaccgcgaggtgatgcggggtcag540
ctccagcatctgatcgagatctccgagcggcccaatgtgcgccttcagatcatgccgttc600
ggcttcggcgggcactccggcgagagcggtgccttcacgatcctgtccttcccggactcc660
gacctgtcggatgtcgtgtacctggagcagctcaccagcgcgctgtacctcgacaagcgc720
gaggacgtcacgcagtacgagaaggcgctcaaggagctccagcaggacagcccggggccg780
gacgagagccgtgatcttctgcgcggtctcctccaactctcctag825
- 还没有人留言评论。精彩留言会获得点赞!