对修饰后自体表位的抗肿瘤免疫应答的制作方法
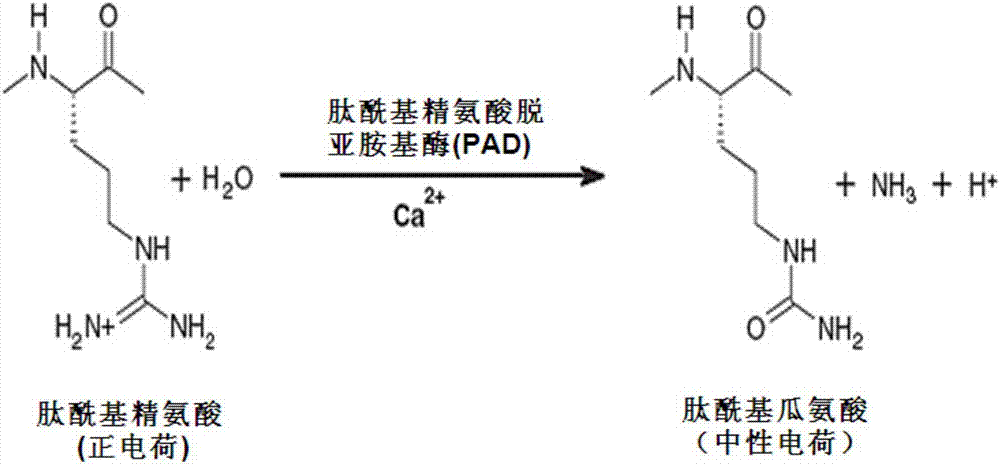
本发明主要涉及可用作癌症免疫疗法标靶的修饰多肽。所述修饰多肽可用作单克隆抗体(mab)疗法的疫苗或标靶。所述疫苗或mab可用于治疗癌症。抗肿瘤免疫应答的焦点主要集中在产生肿瘤特异性cd8应答上,这部分是因为大多数实体瘤缺乏mhcii类表达。因此,这些肿瘤更适合作为cd8t细胞的标靶,而不是cd4t细胞。但是,cd8t细胞参与的疗法仅对患者表现出中等且短效的应答。多年来已知cd4辅助细胞在抗体或cd8介导的表位特异性免疫应答的诱导中均起到关键作用。据报告,若在没有cd4辅助的情况下诱导记忆cd8应答,则该应答被破坏[1、2]。最初以为这是因为cd4辅助细胞分泌il-2[3],但近来认为还因为树突细胞(dcs)的修饰/激活,从而激活了cd8细胞[4-7]。在大多数情况下,该辅助是由源自病原或插入疫苗中的外源cd4表位所提供的。但是,肿瘤位点处也需要肿瘤特异性cd4应答来增强发炎,从而增强对cd8细胞、nk细胞和其他抗肿瘤免疫性炎性介质的招募和滞留[8-11]。使用cd4或mhcii类缺陷小鼠的研究中,肿瘤进程接连发生,这进一步证实了cd4辅助t细胞参与癌症免疫,表明了cd4t细胞在根除肿瘤中的重要性。更新的文献报告暗示了肿瘤反应性cd4t细胞的重要性,它在体内肿瘤根除和肿瘤特异性th17细胞介导的优秀肿瘤破坏作用中的直接作用[12-15]。若细胞库不受制于耐受性,则可产生对肿瘤特异性表位的高频率和亲抗原性的cd4应答[16-18]。若细胞库受制于自身耐受性,则对表位特异性cd4应答的诱导会受到很大限制,且所得应答频率和亲抗原性低。肿瘤中有若干种肿瘤表位可供标靶,包括肿瘤特异性表位和肿瘤相关表位。前者因肿瘤的发生和发病机理中获得的较大dna片段突变或重复或删除而产生。点突变难以被免疫系统靶定,这是由于它们必须创造出新表位,创造出可与mhc相结合的新锚定残基或与t细胞或b细胞受体(tcr或bcrs)反应更强烈的新氨基酸。即使突变真的产生了新表位,也往往仅在少数个别合适的mhc单倍型中被识别。基因重复和删除难以被靶定,因为他们通常是个别癌症中独特存在的。相反,肿瘤相关表位是过度表达的正常表位。所述正常表位通常在胸腺中直接或通过aire酶表达,且能以高亲和性识别这些表位的t细胞被删除或被分化为天然t调节细胞。虽然已发现了对肿瘤相关表位的cd8应答,对肿瘤相关表位的cd4应答却很鲜见,暗示着对这些表位的细胞库中,cd4细胞受到比cd8细胞更重的调控。唯一被发现的cd4应答是针对睾丸癌抗原,其在胚胎发生期间表达,但仅在成人的配子中保持活性,而可能不会受到胸腺耐受,但最新证据表明,aire也可以在胸腺中呈现这些表位。因此,困难在于找到能够识别自体表位并攻击肿瘤的t细胞库。发明人意外地发现了t细胞或b细胞表位经某些修饰能得到与未修饰表位相比引发免疫应答更强的表位。令人吃惊地,发明人发现某些修饰后表位与肿瘤相关,由此可用于产生针对所述肿瘤的免疫应答。在本发明的第一方面中,提供了用于医药的刺激对肿瘤的免疫反应的肿瘤相关表位,所述表位经过选自如下的修饰:精氨酸到瓜氨酸的脱亚氨基反应、酪氨酸硝化反应、色氨酸氧化、谷氨酰胺或天冬酰胺的脱氨基反应。本发明还提供用于癌症治疗方法中的刺激对肿瘤的免疫反应的肿瘤相关表位,和/或含有编码所述表位的序列的核酸,所述表位经过选自如下的修饰:精氨酸到瓜氨酸的脱亚氨基反应、酪氨酸硝化反应、色氨酸氧化、谷氨酰胺或天冬酰胺的脱氨基反应;所述表位,及所述表位和/或核酸在制备用于治疗癌症的药物中的用途。本发明还提供一种治疗癌症的方法,包括将本发明的表位或核酸向需要改治疗的对象给药。所述表位可以是t细胞或b细胞表位。本发明中的表位可单独或组合使用。此外,它们还可以和其他治疗剂共同使用,例如抗癌药剂,包括但不限于检查点阻断药物,如易普利姆玛(ipilimumab)。优选的修饰之一为精氨酸脱亚氨基反应形成瓜氨酸。自由精氨酸可以通过真核细胞中的氧化亚氮合成酶或细菌中的精氨酸脱亚氨酶转化为自由瓜氨酸。瓜氨酸是一种修饰后的氨基酸,但由于其不具有trna,无法直接添加入蛋白中,而是来自由肽酰基精氨酸脱亚胺基酶(pad,存在于各种组织中的酶家族)作用的精氨酸翻译后修饰。术语“瓜氨酸化(citrullination)”或“脱亚氨基(deimination)”指的是精氨酸到瓜氨酸的修饰,可以是由酶pad在多肽序列中进行的翻译后修饰。附图中的图1中展示了更多细节。在精氨酸到瓜氨酸的反应中,精氨酸侧链的末端氮原子之一被氧原子取代。该反应使用了一个h2o分子,生成副产物氨。所述精氨酸到瓜氨酸的转化可以对蛋白的结构和功能有重要影响,因为精氨酸在中性ph下带正电,而瓜氨酸则不带电荷。随着蛋白疏水性的增加,蛋白折叠也可能发生变化。pad酶有5种,称为pad1、2、3、4和6。虽然它们高度同源,在氨基酸水平上具有5-60%的序列一致性,但却具有不同的位点(pad1-表皮和子宫;pad2-大脑,女性生殖道,骨骼肌和造血细胞;pad3-毛囊和上皮;pad4-造血细胞,肺,食道,乳腺和卵巢癌;pad6-卵母细胞和植入前胚胎[19])。虽然与靶蛋白有些重叠,但每个家族成员还似乎能够靶定独有的一套细胞蛋白。这是由精氨酸的表面裸露和聚簇决定的,使它们成为良好标靶和优选的氨基酸侧翼序列。对于pad4,位点-2和+2上优选为非极性小氨基酸,且精氨酸侧翼的脯氨酸防止瓜氨酸化[20,21]。因此,不是蛋白质中的所有精氨酸都瓜氨酸化,不是所有精氨酸都在mhc抗原呈现以供t细胞识别。在本发明中,可用于刺激抗癌免疫的瓜氨酸化表位源自bip、ny-eso-1、mmp7、细胞角蛋白、muc1、cea.cam5、cd59、bcl2、β-连环蛋白、cxcl8、cxcl10、cxcl12、α烯醇酶、髓鞘碱性蛋白、组蛋白、核磷酸蛋白、b23、共活化剂复合体、抗凝血酶、aggregan、延长因子1α、腺苷酸环化酶相关蛋白(cap1)、葡萄糖调节蛋白、aldh2、软骨中间层蛋白(clip)、醛缩酶、磷酸甘油酸激酶1(pgk1)、钙网蛋白、hsp60、hsp90、远端上游元件结合蛋白1和2(fuse-bps)、无孢蛋白、组织蛋白酶d、肝素结合蛋白、β-肌动蛋白、f-肌动蛋白、加帽蛋白α-1亚单位(capzα-1)、白蛋白、组胺受体、蛋白质二硫键异构酶er60前体、线粒体醛脱氢酶(aldh2)和糖原合成酶激酶3β(gsk3β)。所述蛋白的通用蛋白库(uniprot)索引如附图中的图32所示。针对各种瓜氨酸化蛋白(包括丝聚合蛋白、胶原蛋白、α-烯醇酶、纤维蛋白原和波形蛋白)的抗体被发现于风湿性关节炎(ra)病患中,并可用作诊断该疾病的特异性标记[22-24]。最近,若干其他瓜氨酸化蛋白被描述为ra病患中的抗体标靶,包括组蛋白、核磷酸蛋白、b23、共活化剂复合体、抗凝血剂、aggregan、延长因子1α、腺苷酸环化酶相关蛋白(cap1)、葡萄糖调节蛋白、aldh2、软骨中间层蛋白(clip)、醛缩酶、磷酸甘油酸激酶1(pgk1)、钙网蛋白、hsp60、hsp90、bip、远端上游元件结合蛋白1和2(fuse-bps)、无孢蛋白、组织蛋白酶d、肝素结合蛋白、β-肌动蛋白、f-肌动蛋白、加帽蛋白α-1亚单位(capzα-1)、白蛋白、组胺受体、蛋白质二硫键异构酶er60前体、线粒体醛脱氢酶(aldh2)[49]。此外,据假定,瓜氨酸自身不足以生成变异反应,而瓜氨酸周围的氨基酸对确定表位的抗原性则是必要的[25-27]。除瓜氨酸化外,本发明中的数种其他翻译后修饰也在mhc结合的多肽中观察到,包括酪氨酸残基的硝化[28]和色氨酸残基的氧化[29]。巨噬细胞和树突细胞的激活,包括生成氧化亚氮和其他活性氧类。蛋白质的裸露可以带来酪氨酸的硝化和色氨酸的较低程度硝化。据显示,cd4细胞在鸡卵溶菌酶来源的表位上特异识别所述修饰[29]。但是,尚未确定硝化表位在任何疾病中的作用。前列腺肿瘤浸润性淋巴细胞中存在的高水平硝化酪氨酸,暗示着过氧亚硝基的本地生成。精氨酸代谢中的关键酶在恶性前列腺组织中高度表达但在正常前列腺中则否,抑制精氨酸酶和氧化亚氮合成酶活性,降低酪氨酸硝化和肿瘤浸润性淋巴细胞的恢复[30]。谷氨酰胺或天冬酰胺的脱氨基化可能是因为老化,但该修饰的显著部分是因酶机理而产生。脱氨基化形成了主要带负电荷的侧链(谷氨酸盐或天冬氨酸盐),并改变了谷氨酰胺和天冬酰胺形成氢键的能力。天冬酰胺可以通过n-聚糖酶转化为天冬氨酸盐,因此,该修饰与n-糖基化反应紧密相连。谷氨酰胺残基可以通过组织转谷氨酸盐酶来脱氨基。来自饮食小麦蛋白中的谷氨酰胺脱氨基化,是乳糜泻疾病(一种与遗传导致的个体谷蛋白不耐受相关的自免疫疾病)的核心。hla单倍型、dq2和dq8对脱氨基化的醇溶蛋白多肽具有强亲和性,并刺激t细胞对胃肠道内壁的应答[31]。本发明的表位可以包括以下序列、由其组成或主要由其组成:iqklygkrs,优选为sqddikgiqklygkrs(mmp7-247),niltirltaa,优选为pgvllkeftvsgniltirltaadhr(nyeso-1-119),ltirltaa,优选为pgvllkeftvsgniltirltaadhr(nyeso-1-119),eirelqsq,优选为eeeirelqsqisdtsvvls(cytokeratin8229-247),akqdmarqlreyqel,优选为akqdmarqlreyqelmnvkl(cytokeratin8:363-382),akqdmarq,优选为lqrakqdmarqlreyqelm(cytokeratin8:360-378),issssfsrv,优选为pgsrissssfsrvgss(cytokeratin8:29-44),prafssrs,优选为stsgprafssrsytsgpg(cytokeratin8:13-30),eaalqrakq,优选为eleaalqrakqdmarql(cytokeratin8:355-371),levdpniqavrtqe,优选为levdpniqavrtqekeqi(cytokeratin8:78-95),和qkklklvrt,优选为aqkklklvrtspeygmp(ing4158-174),lklvrtspe,优选为aqkklklvrtspeygmp(ing4158-174),kklklvrts,优选为aqkklklvrtspeygmp(ing4158-174),ymssarsls,mssarslss或teymssars,优选为klateymssarslsseek(ing444-58),fdlfenrkk,优选为rapfdlfenrkkknn(hsp90-346-360),ylnfirgvv或firgvvdse,优选为ipeylnfirgvvdse(hsp90-378-392),lryytsasg,llryytsas或lsellryyt,优选为rkklsellryytsasgdemvsl(hsp90-456-477),rrrlsellryhtsqs(hsp90beta456-460),vgvfkngrv或fkngrveii,优选为yscvgvfkngrveii(bip39-53),yfndaqrqa,优选为vpayfndaqrqatkda(bip172-186),vtfeidvng,优选为evtfeidvngilrvt(bip497-511),itndqnrlt,优选为kititndqnrltpee(bip522-536),lqivarlkn或varlknnnr,优选为ncalqivarlknnnr(cxcl12-54-68),veiiatmkk或rveiiatmk,优选为cprveiiatmkkkge(cxcl1057-71)。其中,一个或多个r残基被替代为瓜氨酸。在nyeso-1-119中,优选地,第一个r残基(位点136)被瓜氨酸取代。在细胞角蛋白8:360-378中,优选地,第二个r残基(位点369)被瓜氨酸取代。在细胞角蛋白8:29-44中,优选地,第二个r残基(位点40)被瓜氨酸取代。在hsp90、bip、ing4、cxcl10和cxcl12中,优选地,序列中所有r残基被瓜氨酸取代。本发明还在进一步的方面中提供了所述表位。特别地,本发明提供了含有序列yvttstcittyslgsalcit或由该序列组成或主要由该序列组成,且可选地含有序列citsyvttstcittyslgsalcitpsts(vim28-49)或由该序列组成或主要由该序列组成的多肽,其中cit代表瓜氨酸。本发明的优选表位之一源自波形蛋白,且瓜氨酸化。发明人意外地发现,来自波形蛋白的瓜氨酸化表位可用作产生对肿瘤的免疫应答,包括但不限于,黑色素瘤、乳腺癌、子宫内膜癌、结肠直肠癌和卵巢癌。来自波形蛋白的优选表位含有vttstrtyslgsalr(波形蛋白30-45)、由其组成或主要由其组成,其可以可选地瓜氨酸化。其中两个精氨酸之一或两者均可以瓜氨酸化。该表位可以包括rsyvttstrtyslgsalrpsts(波形蛋白28-49)、由其组成或主要由其组成,其可以可选地瓜氨酸化。其中三个精氨酸中的任一个、任两个或所有三个可以瓜氨酸化。优选地,所有三个r残基皆为瓜氨酸化。第二个r残基(位点36)可以被瓜氨酸取代。源自波形蛋白的可选表位包括至少一个以下序列、由其组成或主要由其组成:vrlrssvpg或rlrssvpgv,优选为savrlrssvpgvr(vim65-77),和fsslnlret,优选为lpnfsslnlretnldslpl(vim415-433),其中一个或多个所述r残基被瓜氨酸取代。在vim65-77中,优选地,第二个r残基(位点70)被瓜氨酸取代。源自波形蛋白的更多可选表位包括至少一个以下序列、由其组成或主要由其组成:rssvpgvrl,savrlrssv,atrssavrl,yatrssavrlrssvpgvrl(vim61-79),rssvpgvrl,gvrllqdsv,rlrssvpgvrllqdsvdfs(vim69-87),qlkgqgksr,ksrlgdlye,eqlkgqgksrlgdlyeeem(vim125-154),elrrqvdql,emrelrrqv,dlyeeemrelrrqvdqltn(vim148-166),qltndkarv,veverdnla,ltndkarve,vdqltndkarveverdnla(vim161-179),everdnlae,ndkarveverdnlaedimr(vim166-184),imrlreklq,dnlaedimrlreklqeeml(vim176-194),qreeaentl,klqeemlqr,eklqeemlqreeaentlqs(vim187-205),和/或frqdvdnas,entlqsfrq,eaentlqsfrqdvdnasla(vim198-216),其中一个或多个所述r残基可以被瓜氨酸取代。在上述序列中,优选被瓜氨酸取代的r残基以下划线标出。核心序列显示在表位之前。本发明的一个方面中,提供了包含序列yvttstrtyslgsalr、由其组成或主要由其组成的表位,且可选地,包含序列rsyvttstrtyslgsalrpsts(vim28-49),和/或编码所述表位的核酸,以用于治疗癌症的方法中。本发明的另一个方面中,提供了包含序列fsslnlret(优选为lpnfsslnlretnldslpl(vim415-433))、由其组成或主要由其组成的表位,和/或编码所述表位的核酸,以用于治疗自免疫疾病的方法中。发明人发现,该表位刺激能抑制整体免疫反应的il10应答。除非另有说明,上述序列中下划线的残基指示的是核心序列。所述核心序列以外的氨基酸可以被其他氨基酸保守取代(优选为以与被取代的原生氨基酸具有相似大小和/或电荷的氨基酸来取代)或可以被删除。此外,或可选地,1、2、3、4、5或所有非核心氨基酸可以被取代或删除。本发明中可用的表位可以具有供hlaii类分子的cd4+t细胞识别的优化长度、疏水性和/或电荷。本发明的表位可以包括至少13、14、15、16或更多氨基酸。本领域技术人员可以领会,本发明中可用的其他表位可以使用如本文实施例10中所述的各种技术来鉴别。发明人还发现,编码全长波形蛋白(人波形蛋白序列参见图2。可以包括或不包括n端蛋氨酸残基)的核酸(例如dna或rna)的给药,会引起强免疫反应,这构成了本发明的又一实施例。波形蛋白(vim)在具有该基因克隆的物种之间高度保守(鸡、鼠、狗、羊、牛、马、猪和人)。相应地,例如如上所述的波形蛋白和源自波形蛋白的表位,及其编码核酸,可用于治疗人类以外的哺乳动物的癌症。申请人此前展示了,在抗体dna结构中包含t细胞表位,能增强t细胞应答的频率和亲抗原性[32,33]。如下文实施例中将会详细描述,在hla转基因小鼠中,发明人向抗体dna结构中克隆了各种外源和自体表位,并筛选t细胞应答。所有外源表位都刺激了强t细胞应答,但刺激了显著应答的自体表位仅有波形蛋白28-49。即使是与原生小鼠表位仅有1个保守氨基酸变化差异的人gp100表位,也在小鼠中刺激了强应答,而完全保守的小鼠表位未能在小鼠中刺激应答。这暗示着对识别小鼠表位的t细胞的完全耐受/删除,而对极为相似的人表位则没有。这暗示着人表位不会在人体内刺激t细胞响应,而且,虽然小鼠表位可能可以,但t细胞却无法识别肿瘤中天然加工的人表位。相反地,自体波形蛋白28-49表位能在正常供体或风湿性关节炎(ra)患者中刺激应答[34]。相反地,发明人展示了癌症患者可对vim28-49做出免疫应答。这暗示着,该表位令人意外地不受制于自体耐受性,人和小鼠中均存在一个能够识别该表位并作出应答的t细胞库。波形蛋白(gi/4507895)是发现于间叶细胞和其他非上皮细胞的中间纤丝(ifs),尤其是iii类ifs中的同源二聚体胞内蛋白。ifs是高等真核细胞的细胞骨架的主要成分,由数种不同结构相关的蛋白所组成。不同的if蛋白基因在不同的组织中表达。人和鼠波形蛋白基因的特性均得到了辨认(参见例如[35,36])。波形蛋白以及其他if蛋白已被用于人类肿瘤的组织学分类(参见[37,38]的综述)和数种肿瘤类型去分化的标记。文献记载了多药物抗药性肿瘤病的波形蛋白指导诊断和治疗(美国专利申请20100260667),以及与波形蛋白和其他ifs共定位肿瘤标记(参见例如wo0127269)。后者描述了用于成神经细胞瘤的新型标记(称为vip54)及其在作为肿瘤发展和进程标记的if检测和细胞成像中的用途。数个其他已知实施例展示了不同人实体瘤和实体瘤细胞系种类中波形蛋白的表达水平和细胞间分布[39,40]。但是,至今为止,还未发现对未修饰波形蛋白的t细胞应答。人波形蛋白为57kda蛋白,包含466个氨基酸(参见图2),也是iii型if蛋白家族最广泛表达且高度保守的蛋白之一。它不存在于细胞液中,但在细胞核中表达,并作为胞外蛋白表达。它参与细胞骨架的动态组织,在细胞器转运、细胞迁移和繁殖中具有重要功能。上皮间叶细胞转换(emt);波形蛋白亦在各种上皮癌症中过度表达,包括前列腺癌、胃肠道癌、乳腺癌肺癌、恶性黑色素瘤和中央神经系统瘤。它还见于宫颈癌[41]、透明细胞肾细胞癌[42]、某些类型的淋巴癌[43]、甲状腺乳头状癌[44]和子宫内膜癌[45]。它在癌症中的过度表达与肿瘤加速生长、侵袭和不良预后具有相关性。在胃癌中,波形蛋白表达最经常与胃癌侵袭表型相关,且有证据暗示它在胃癌的转移中具有重要作用,且可充当预后指标[46、47]。软组织肉瘤和一些表现出上皮细胞向间叶细胞转化(emt)表型的上皮癌症中表达了波形蛋白。癌细胞通过emt从上皮细胞向间叶细胞状转化的表型,被看做是实体瘤转移的相关步骤(参见图3)。此外,所述癌细胞表型转换与降低放射、化学疗法和某些小分子靶向疗法的抗肿瘤作用的癌症抗性机制的获得有关。虽然波形蛋白被当做是emt的标记,它在肿瘤发生中的作用仍不清楚。研究最多的emt诱导物之一为tgfβ,它在多种肿瘤类型中都有发现,与rtk-信号传导通路或其他通路合作,驱使肿瘤细胞成为更接近间叶细胞的转移性表现型。ivaska[48]展示了波形蛋白通过上调书中emt相连基因的表达来协助emt,尤其是促迁移受体酪氨酸激酶axl。若干研究支持波形蛋白功能是emt的积极调控子且其上调是emt诱导的前提的说法[48、49]。上述所有癌症以及卵巢癌和胃肠道癌均可以根据本发明进行治疗,无论其表位为何。本发明还可以涉及但不限于治疗前列腺癌、乳腺癌、肺癌、恶性黑色素瘤、中央神经系统瘤、宫颈癌、透明细胞肾细胞癌、淋巴瘤、甲状腺乳头状癌和子宫内膜癌。本发明的波形蛋白28-49及其他表位可以多肽形式进行体内递送,可选地,以wo02/058728中所公开的多肽形式。发明人意外地发现,本发明中可用的表位以多肽形式给药时,会引起强免疫应答。所述表位可仅以表位的序列给药,或以含有该表位的多肽给药,或以甚至全长蛋白给药。可选地,本发明的表位可以作为编码该表位的核酸、编码含有该表位的多肽的核酸,或甚至编码该全长蛋白的核酸来体内给药。所述核酸可以是小基因形式,即编码前导序列和该表位,或编码前导序列和全长蛋白。可选地,其可以是如wo2008/116937所公开的核酸形式。在另一个方面中,本发明提供了含有至少一个编码免疫球蛋白分子重组重链的序列的核酸,所述重链具有至少一个异源t细胞表位,其中所述t细胞表位是本发明第一方面中的表位,优选为波形蛋白28-49。优选地,该核酸还包括非特异性启动子,且可以是dna。所述免疫球蛋白分子可以是抗体。所述表位可以插入重链的cdr中,优选为该重链的cdr3。本发明中可用表位的编码核酸可以靶向抗原呈递细胞和其他表达pad酶(优选为pad4酶)的细胞。本发明的核酸的靶向,可以通过包括含有靶向剂的核酸,所述靶向剂例如为fc或靶向apcs上不同抗原的单克隆抗体,例如抗dec205mab,或通过皮内注射法,因为皮肤中有大量apcs。此前的研究辨明,在位点28和36处瓜氨酸化的vim26-44、在位点36和45处瓜氨酸化的vim36-54和在位点424处瓜氨酸化的vim415-433,能刺激hla-dr4小鼠和/或ra患者中的t细胞应答[34]。如实施例中的详细记载,要测试dna结构中的vim表位是否刺激对瓜氨酸化波形蛋白的免疫应答,使小鼠对编码波形蛋白表位的dna免疫接种,并筛选对未修饰表位和瓜氨酸化表位的应答。以vim28-49dna或编码全波形蛋白序列的dna所免疫接种的小鼠,对瓜氨酸化(cit)28-49表位的应答强于野生型(wt)表位。这暗示着,dna翻译后,vim表位瓜氨酸化,瓜氨酸化表位与mhc结合的亲和性更高,或以未修饰波形蛋白表位刺激的t细胞识别瓜氨酸化表位的亲抗原性更高。与vim28-49相反,vim415-433dna或编码全波形蛋白序列的dna仅刺激了识别瓜氨酸化表位vim415-433的应答,而非野生型vim415-433,证实了表位呈递细胞(apcs)中的dna被翻译成了被瓜氨酸化的多肽。基于此,近期研究表明,树突细胞表达瓜氨酸化酶pad2和pad4[51]。另一有趣之处在于,编码vim415-433的dna引起强th17应答。编码来自mmp7和ny-eso-1的表位的dna疫苗还刺激能够识别瓜氨酸化和未修饰表位的t细胞。mmp-7应答主要是th17,而nyeso-1应答主要是th1。类似地,癌症患者表现出对mmp7和nyeso-1修饰多肽和未修饰多肽的应答。有数篇报告表明,单核细胞表达pad酶[27,52,53]。近期来,发现骨髓性树突细胞和腹膜巨噬细胞均表达pad2和pad4[51]。瓜氨酸化是发炎过程的一部分,对其研究刚刚开始。刺激具有ifnγ和双链rna的外周血单核细胞,使得趋化因子cxcl8和cxcl10瓜氨酸化,这对其受体使用、蛋白酶解加工和生物活性均有根本影响[54-56]。这可能意味着经由toll受体、damp受体或热休克蛋白的细胞应激介质可能允许pad酶在apcs和目标细胞(例如感染细胞或肿瘤细胞)中的生理激活,从而能够破坏对修饰字体表位的耐受,允许诱导免疫应答。树突细胞中的瓜氨酸化似乎涉及自体吞噬[57],但这仅在鸡卵溶菌酶中发现过。本文的公开中,编码未修饰表位的dna可以刺激瓜氨酸化表位特异性t细胞应答。这意味着,该dna必须在apcs中经过翻译和翻译后瓜氨酸化,才能刺激瓜氨酸化表位特异性t细胞。这是对核酸疫苗刺激瓜氨酸化的首次展示。并无任何报告显示肿瘤对mhc分子上呈现的任何表位进行瓜氨酸化。除了显示抗体dna中的编码表位得到的应答频率较高、亲抗原性更高外,发明人还在此展示了瓜氨酸化多肽可以刺激t细胞应答。瓜氨酸化的vim28-49多肽主要刺激th1应答,识别修饰后表位,但也与野生型多肽有一些交叉反应。野生型vim28-49刺激针对野生型和修饰后表位的th1应答。当癌症患者以两种多肽中的任一种进行刺激时,2/11对未修饰表位做出应答,5/11对瓜氨酸化表位vim28-49做出了应答。hilletal.[50]此前展示了修饰后的瓜氨酸化vim65-77表位,其中位点33处的a被l取代,所述表位与hla-dr4的结合比未修饰表位更强。但是,其为合成抗原,且据发现,dr4转基因小鼠中,即使是瓜氨酸化的天然vim65-77表位也不会引起免疫应答[34]。相反,我们展示了,在hla-dr4小鼠中,瓜氨酸化vim28-49和vim65-77多肽刺激了主要识别修饰后表位的th1应答,而野生型表位则是不良免疫原。进一步的分析证实了所述应答受到cd4t细胞的调控。瓜氨酸化vim65-77还刺激了hla-a2.dr1转基因小鼠中的应答。但是,进一步分析也揭示了该应答为cd8应答,且特异性表位为rlcitssvpgv。这是对瓜氨酸化cd8表位的首次描述。我们展示了,出乎意料地,在癌症病人中,4/9的病人对vim65-77做出应答,4/9对瓜氨酸化vim65-77应答,但仅其中2人对两种多肽均有应答。对于dna免疫接种,以瓜氨酸化vim415-433多肽免疫接种的小鼠刺激了对修饰后表位的特异性应答,而野生型表位未能刺激应答。此外,瓜氨酸化vim415-433多肽刺激了非常强烈的il-17应答。相反,在癌症病人中,8/11对野生型表位应答,而仅5/11对瓜氨酸化vim415-433做出应答。这暗示着,未修饰表位和修饰表位均能刺激患者的t细胞应答,且所述t细胞应答并非总是交叉反应。这也是癌症病人能够对瓜氨酸化表位做出应答的首次展示,此前人们以为该应答仅限于ra病人。修饰后多肽和编码所述多肽的dna能够刺激对修饰后表位的免疫应答,且所述修饰后表位由肿瘤靶细胞表达。瓜氨酸化波形蛋白存在于炎性巨噬细胞[58、59]中,且在钙-离子载体诱导的人和鼠巨噬细胞凋亡过程中被观察到。巨噬细胞在rna和蛋白水平上表达pad[60],且瓜氨酸化波形蛋白可在离子载体诱导的ca2+输入后于巨噬细胞样细胞中体外表达[27,58]。但是,正常情况下,细胞溶质ca2+浓度(约为10-7m)对pad酶活性而言过低。pad活性需要的最低ca2+浓度比正常细胞溶质ca2+浓度高约100倍。由于所述离子载体和随之而来的ca2+输入引起细胞凋亡,这暗示着波形蛋白瓜氨酸化参与了细胞凋亡。的确,瓜氨酸在准备用于凋亡过程中的降解的胞内蛋白上具有重要作用。据发现,波形蛋白在凋亡中的巨噬细胞中被瓜氨酸化,而针对瓜氨酸化波形蛋白的抗体则在凋亡物质处置不当时产生[27]。该研究暗示,翻译后修饰在调控波形蛋白功能时的重要作用,尤其是指定修饰有细胞和组织特异性时。但是,若pads仅在凋亡肿瘤细胞中激活,则瓜氨酸化蛋白便是不良标靶,因为这些细胞已在死亡。但是,根据发明人展示,识别修饰后表位的t细胞做出强烈抗肿瘤应答,暗示着如apcs等活性肿瘤细胞能够在其局部释放高胞内钙水平,导致靶蛋白的瓜氨酸化。此外,由瓜氨酸化vim415-433和vim28-49诱导的t细胞,能诱导cd4t细胞,在mhcii类和表位表达的情况下识别和杀灭癌细胞,但在正常细胞中则不会。虽然据显示,瓜氨酸化vim415-433能够刺激t细胞应答,而未修饰表位不会,这可能与人和鼠vim415-433的两个氨基酸差异有关。因此,筛选小鼠对鼠表位的应答。以瓜氨酸化人波形蛋白免疫接种的小鼠可识别波形蛋白。此外,与人表位相似地,其为th1/th17应答。此前,据发现,th1或th17诱导应答受到为适当应答建立正确细胞因子环境的免疫佐剂的极大影响。ifnγ和il-12触发th1细胞分化。th1细胞产生ifnγ、il-2、tnf和gm-csf,并刺激例如cd8+t细胞的激活。相反,th2细胞因il-4分化。它们分泌il-4、il-5和il-13,并辅助b细胞进行抗体生成,且下调th1细胞诱导的促炎性应答。tgfβ、il-1、il-6、il-21和il-23触发th17细胞的分化。它们分泌il-17和il-6。当无促炎性细胞因子但存在高浓度tgfβ时,cd4+t细胞分化为itreg(参见图4)。所述细胞分泌tgfβ和il-10,且特征在于具有转录因子叉头框p3(foxp3)。它们控制并中和其他效应子t细胞的免疫应答,且参与预防自免疫性[61]。瓜氨酸化vim415-433多肽因此以各种佐剂或无佐剂免疫接种。免疫刺激性佐剂,例如cpg/mpla和gmcsf,均能刺激th1/th17应答,且是必需的,因为在缺乏佐剂的情况下不发生应答。惰性佐剂,例如明矾和弗氏不完全佐剂主要诱导il-10应答,与诱导il-10应答的野生型vim415-433多肽免疫接种相似,暗示着对itreg应答的诱导。相反,瓜氨酸化vim415-433多肽与优化佐剂的组合诱导ifnγ/il-17应答,暗示着th17应答。这是首次展示t细胞表位能确定t辅助细胞分化。由于小鼠对瓜氨酸化vim415-433多肽的单免疫接种做出了强th1/th17应答,这可能反映了对天然treg应答的促进。但是,删除天然tregs后,并未影响瓜氨酸化vim415-433免疫应答的频率或亲抗原性,暗示着此原理不太可能。可能是原位呈现的野生型vim415-433表位刺激了itreg应答,以il-10生成为特征。的确,以瓜氨酸化vim415-433免疫接种的小鼠做出了ifnγ、il-17和il-10应答,而以未修饰表位免疫接种的小鼠仅刺激了il-10应答。未修饰vim415-433多肽还刺激了癌症病人的il-10应答。抗ctla-4mabs能够阻断ctla-4与其同源受体cd80/86的相互作用,由此防止该配体诱导的t细胞抑制。在存在抗ctla-4mab的情况下,小鼠以瓜氨酸化vim415-433多肽免疫接种,显著增加了t细胞应答的亲抗原性,从10-6m到10-8m。在本发明的又一个方面中,提供了一种用于医药用途的刺激对自体抗原的il-10免疫反应的修饰后表位,和/或编码所述表位的核酸,所述表位的修饰选自:精氨酸到瓜氨酸的脱亚氨基反应、酪氨酸硝化反应、色氨酸氧化、谷氨酰胺或天冬酰胺的脱氨基反应。在本发明的又一个方面中,提供了一种调制t辅助细胞分化的方法,包括:令t辅助细胞与本文所述表位和/或核酸以及佐剂(可选)相接触。本发明的又一个方面中,提供本文所述的表位和/或核酸,以及佐剂(可选),用于调制t辅助细胞分化的方法。本发明的又一个方面中,提供本文所述的表位和/或核酸以及佐剂(可选)在制备用于调制t辅助细胞分化的药物中的用途。本发明所述方面中可用的表位可以是如本发明第一方面所述的表位,和/或可以是刺激对自体抗原的il-10免疫反应的修饰后表位,所述表位的修饰选自:精氨酸到瓜氨酸的脱亚氨基反应、酪氨酸硝化反应、色氨酸氧化、谷氨酰胺或天冬酰胺的脱氨基反应。本发明中可用的表位和/或核酸可用于引导t辅助细胞分化,以刺激免疫应答。如此一来,其可以与疫苗联用,以提高所述疫苗的效力。如发明人所示,瓜氨酸化vim415-433多肽诱导了ifnγ/il-17应答,暗示着th17应答。这与使得t辅助细胞分化为itreg细胞的vim415-433和其他自体表位的正常应答大为不同。可选地,本发明中可用的表位和/或核酸可用于引导t辅助细胞分化以抑制免疫应答。瓜氨酸化ing4产生il-10应答,暗示着itreg应答和免疫应答受抑制。所述表位可用于治疗需要免疫抑制的疾病,例如自体免疫性疾病和移植物抗宿主疾病(gvhd)。自体免疫疾病的例子包括:斑秃、强直性脊柱炎、抗磷脂综合征、自身免疫性阿狄森氏病、自身免疫性溶血性贫血、自身免疫性肝炎、自身免疫性内耳疾病、自身免疫性淋巴增生综合征(alps)、自身免疫性血小板减少性紫癜(atp)、白塞氏病(behcet’sdisease)、大疱性类天疱疮、心肌病、口炎性腹泻-皮炎、慢性疲劳综合征免疫缺陷综合症(cfids)、慢性炎性脱髓鞘性多发性神经病、瘢痕性类天疱疮、冷凝集素病、crest综合症、克罗恩病(crohn’sdisease)、恶性萎缩性丘疹病(dego’sdisease)、皮肌炎、幼年型皮肌炎、盘状红斑狼疮、原发性冷球蛋白血症、纤维肌痛-纤维肌炎、格雷夫斯病(grave’sdisease)、吉兰-巴雷综合征(guillain-barre)、桥本氏甲状腺炎(hashimoto’sthyroiditis)、特发性肺纤维化、特发性血小板减少性紫癜(itp)、iga肾病、胰岛素依赖性糖尿病(i型)、幼年型关节炎、狼疮、美尼尔氏症(meniere’sdisease)、混合性结缔组织病、多发性硬化症、重症肌无力、寻常型天疱疮、恶性贫血、结节性多动脉炎、多软骨炎、polyglancular综合征、风湿性多肌痛、多发性肌炎和皮肌炎、原发性丙种球蛋白血症、原发性胆汁性肝硬化、银屑病、雷诺氏现象(raynaud’sphenomenon)、赖特综合征(reiter’ssyndrome)、风湿热、类风湿关节炎、结节病、硬皮病、干燥综合征(sjogren’ssyndrome)、僵人综合征(stiff-mansyndrome)、多发性大动脉炎(takayasuarteritis)、颞动脉炎/巨细胞动脉炎、溃疡性结肠炎、葡萄膜炎、血管炎、白癜风和韦格纳氏肉芽肿(wegener’sgranulomatosis)。在本发明的又一个方面中,提供了一种预测结肠直肠癌生存期的方法,包括测定结肠直肠癌细胞中的肽酰基精氨酸脱亚胺基酶4(pad4)水平。94%的肿瘤表达pad4。肿瘤细胞缺失pad4表达,表明预后不良,且暗示着降低的生存期。因此,若结肠直肠癌表现出的pad4表达极少或没有,则患者具有不良预后。在不受理论约束的情况下,这可能与能被t细胞识别和杀灭的瓜氨酸化表位的缺失有关。因此,肿瘤逃避了免疫监控。相反地,若肿瘤表达pad4,则目标脱亚氨基化,肿瘤生长受到免疫应答的控制,患者的预后较好。pad4水平可以用抗体或测量细胞中的pad4蛋白的其他免疫分析来测定。可选地,pad4mrna水平可以用原位杂交来测定。本领域技术人员熟知用于测定mrna水平的适当可选技术,以及测定蛋白水平的可选技术。90%的结肠直肠瘤表达了波形蛋白,其主要见于基质细胞和浸润性细胞。15%的细胞中,也有些上皮细胞染色,且12%的肿瘤中,所有细胞中有超过75%被染色。结肠直肠瘤中仅6%未显示出pad4特异性mab染色。卡普兰迈耶存活分析显示,pad4强度和存活之间具有相关性。在多变量模型中,tnm分期(p=<0.0001)、血管入侵(p=<0.0001)和pad4表达(p=0.004)是患者生存期的独立指标,暗示着pad4能够成为有用的结肠直肠癌预后指标。pad4表达与bcl2(p=0.01)、β-连环蛋白(p=0.001)、cd8t细胞数量(p=0.006),muc1(p=0.000)、cea(p=0.000)、cd59(p=0.038)和波形蛋白(p=0.000)均具有相关性,暗示着所有这些均可作为修饰后蛋白的标靶。发明人所进行的实验中,89%的卵巢瘤显示出强烈的波形蛋白染色,而仅有4%未显示抗pad4抗体染色。此外,82%还表达了瓜氨酸,暗示着该酶的活性。pad4的表达与应激相关蛋白raet1e和ulbp1具有相关性,暗示着该酶在细胞应激状态下激活。其与波形蛋白之间也存在强烈相关性,暗示着瓜氨酸化波形蛋白是卵巢癌的良好标靶。hmgb1在87%肿瘤中表达,且也与波形蛋白表达相关。在本发明的又一个方面中,提供了一种预测卵巢癌生存期的方法,包括在卵巢癌细胞中测定肽酰基精氨酸脱亚胺基酶2(pad2)和/或高迁移率族蛋白b1(hmgb1)的水平。91%的肿瘤表达pad2。肿瘤细胞缺失pad2表达,说明预后不良,且暗示生存期下降。因此,若卵巢癌表现出极少或没有pad2表达,则患者具有不良预后。在不受理论约束的情况下,这可能与缺乏能够被t细胞识别和杀灭的瓜氨酸化表位有关。因此,肿瘤逃避了免疫监控。相反地,若肿瘤表达pad2,则标靶脱亚氨基化,肿瘤生长受到免疫应答的控制,则患者具有较好预后。发明人进行的实验表明,卵巢肿瘤中有16%表现出强烈的pad2染色。此外,91%仍表达瓜氨酸。pad2的表达与hmgb1表达相关,暗示着与自体吞噬相关;其也与mhc表达相关,暗示着与免疫应答的关联。87%的肿瘤表达hmgb1。缺失hmgb1表达的肿瘤细胞说明预后较好,并暗示着生存期增加。hmgb1水平高的肿瘤具有强自体吞噬性,这是有力的存活机制,并与不良预后相关。因此,若卵巢癌表现出极少或无hmgb1表达,则说明患者预后良好,低水平表达说明预后较好,而高表达说明预后不良。hmgb1水平可以使用抗体或其他免疫分析方法来测定,以测量细胞中的hmgb1蛋白。可选地,hmgb1mrna水平可以通过原位杂交来测量。本领域技术人员熟知用于测定mrna水平的适当可选技术,以及用于测定蛋白水平的可选技术。87%的卵巢肿瘤表达hmgb1,主要发现于细胞核与细胞质。卡普兰迈耶(kaplanmeier)存活分析表明,hmgb1水平与生存期之间存在相关性(p=0.001)。在多变量模型中,tnm分期(p=<0.0001)、肿瘤类型(p=0.031)、对化疗的应答(p=<0.001)和hmgb1缺失(p=0.002)是患者生存期的独立制表,暗示着hmgb1可以是卵巢癌的有用预后指标。在显示出高hmgb1和低pad2表达的患者中,310个患者中有219个(70%)具有最差中位生存期,即50个月,而具有低hmgb1和低pad2的患者的生存期较好,310个患者中有41个(13%)的中位生存期为101个月。在不受理论束缚的情况下,这可能与缺乏瓜氨酸化表位和缺乏自体吞噬性有关,肿瘤逃避了免疫监控,具有不良存活能力。在哺乳动物中,发现了5种pad同种型,各由不同基因编码[60]。所有这些酶的活性均强烈依赖于ca2+的存在。pad的所有同种型均表现出广泛的共有序列同源性。同种型之间最引人瞩目的差异是它们的组织特异性表达。所有同种型均可在体外利用可利用的精氨酸将大多数蛋白瓜氨酸化[62],虽然有些蛋白在个别pads作用下比其他蛋白瓜氨酸化更迅速[63]。对多肽的研究表明,精氨酸残基侧翼的某些氨基酸影响其对瓜氨酸化的敏感性[20]。所有这些酶的活性均强烈依赖ca2+的存在。pad的所有同种型均表现出广泛的共有序列同源性。同种型之间最引人瞩目的差异是它们的组织特异性表达。所有同种型均可在体外利用可用精氨酸将大多数蛋白瓜氨酸化[62],虽然有些蛋白在个别pads作用下比其他蛋白瓜氨酸化更迅速[63]。对多肽的研究表明,精氨酸残基侧翼的某些氨基酸影响其对瓜氨酸化的敏感性[20]。最广泛表达的pad同种型,pad2,存在于许多不同组织中,例如骨骼肌、大脑、脾腺和巨噬细胞。虽然其具有广泛的表达模式,只有中央神经系统中(其中生理性瓜氨酸化对延续髓鞘电绝缘性质十分重要)的髓磷脂结合蛋白(mbp),以及波形蛋白,被鉴定为天然基质。在多发性硬化(一种cns的慢性炎性病症)中,髓鞘被破坏,患者对瓜氨酸化的mbp产生自免疫应答。在健康成人中,18%的mbp瓜氨酸化,而ms中,超过45%瓜氨酸化,导致去折叠和降解的增加。pad1主要在上皮和子宫中表达,而且,在角化细胞的成体终末分化中,角蛋白(k1和k10)和角蛋白相关蛋白丝聚合蛋白均瓜氨酸化。一般而言,瓜氨酸化降低蛋白质上的电荷,导致心房去折叠,降低表皮弹性和角化。银屑病是由pad1在银屑病性表皮中的异常瓜氨酸化引起的。pad3被限制在毛发/毛绒的毛囊中,其天然基质为毛透明蛋白,是毛囊内根鞘的主要结构蛋白。pad6由卵细胞和胚胎表达。炎性白血球包括滑膜t和b细胞、巨噬细胞、中性粒细胞,以及成纤维样滑膜细胞,表达2种pad同种型,pad2和pad4[27,64]。不像其他pads都主要位于细胞的细胞质中,pad4位于细胞核中。pad4的细胞核定位信号见于蛋白n端区域。pad4主要在外周血粒细胞和单核细胞中表达,还由许多肿瘤组织包括腺癌所表达[65]。免疫组织化学法还检测出pad4和细胞角蛋白的共定位,免疫印迹法则在提取自肿瘤的细胞角蛋白中检测到瓜氨酸信号。此外,体外瓜氨酸化的细胞角蛋白8、细胞角蛋白18、细胞角蛋白19抵抗半胱天冬酶的消化。这可能赋予肿瘤生长优势。外周白血球中检测不到含瓜氨酸的蛋白,即使这些细胞显示出强烈的pad4表达。据推测,钙浓度是激活pad4和后续瓜氨酸化的关键因素。此外,风湿性关节炎(ra)滑膜的瓜氨酸化主要发生在其胞外沉淀中,而瓜氨酸化蛋白则被定位于肿瘤细胞内。组蛋白的瓜氨酸化可以改变肿瘤中的基因表达,而ing4的瓜氨酸化抑制其与p53的结合,导致该有力肿瘤抑制基因的失活[66]。本发明的范围内还包括具有如上和/或如表3、8、9、10所列举的氨基酸序列以及与其具有高度一致性(例如与其一致性为70%、80%、85%、80%、95%或99%)的序列的多肽,及其在医药中,尤其是在治疗癌症的方法中的用途。两种氨基酸序列或两种核酸序列的一致性百分比通常由将序列以最优化对照目的(例如,可以在第一序列中引入间隔来得到与第二序列的最佳比对)进行比对,并比较对应位点上的氨基酸残基或核苷酸。“最佳比对”是两条序列之间获得最高相似度的比对。一致性百分比的测定是通过比较序列中的相同氨基酸残基或核苷酸的数目来确定的(即,%一致性=一致位点数目/位点总数目x100)。两条序列之间的一致性百分比的测定,可以通过使用本领域技术人员所知的数学算法来完成。用于比对两条序列的数学算法的例子之一,为karlin和altschul改进算法[67]。altschul,etal的nblast和xblast程序合并了所述算法[68]。可以用xblast程序(score=50,wordlength=3)来执行blast蛋白检索,得到与本发明蛋白质分子同源的氨基酸序列。为了获得用于比对目的的空位比对,可以使用gappedblast,如[69]所述。可选地,可以使用psi-blast来执行重复检索,以检测分子之间的远缘关系。当使用blast、gappedblast和psi-blast程序时,可以使用各程序(e.g.,xblastandnblast)的默认参数。参见http://www.ncbi.nlm.nih.gov。可用于序列比对的数学算法的另一例子为myers和miller的算法[70]。align程序(版本2.0)是gcg序列比对软件包的一部分,合并了所述算法。本领域所知的用于序列分析的其他算法包括如[71]所述的advance和adam,以及如[72]所述的fasta。在fasta中,ktup是一个控制选项,用于设定搜索的敏感性和速度。本文中所称的术语“治疗”包括能够使人类或非人类的动物受益的任何方案。所述多肽或核酸可与药学可接受的一种或多种载体共同使用。所述载体可以包括但不限于,盐水、缓冲盐水、右旋葡萄糖、脂质体、水、甘油、乙醇及其组合。本发明中可用的多肽可以是以fmoc化学法或其他本领域技术人员已知的标准技术来合成。可以预期,本发明的组合物的治疗给药的主要途径是注射,但也可以使用导管或其他外壳用管来递送。一些适当的给药途径包括静脉内、皮下、皮内、腹膜内和肌肉内给药。粉末制剂可以重构为液体制剂后使用。对于静脉内注射或病痛部位注射,活性物质将处于肠胃可接受的水溶液形式,其无热原、ph适当、等渗且保持稳定。本领域技术人员能够使用例如等渗赋形剂例如氯化钠注射液、林格氏注射液(ringer’sinjection)或乳酸林格氏注射液(lactatedringer’sinjection),熟练地制备适当溶液。根据需要,可以包含防腐剂、稳定剂、缓冲液、抗氧化剂和/或其他添加剂。口服给药的药用组合物可以是片剂、胶囊、粉剂或液体形式。片剂可以包括固体载体例如明胶,或佐药。液体药用组合物一般包括液体载体,例如水、石油、动物或植物油、矿物油、或合成油。可以包括生理盐溶液、右旋葡萄糖或其他糖类溶液,或甘油例如乙二醇、丙二醇或聚乙二醇。当制剂为液体时,其可以是例如含有ph6.8-7.6的非磷酸缓冲液的生理盐溶液,或冻干粉末。所述组合物可以在肿瘤部位或其他所需部位进行局部给药,或可以用靶向肿瘤或其他细胞的方式递送给药。所述组合物优选为以“药学有效剂量”对个体给药,所述剂量足以令个体受益。给要得实际剂量、速率和时间进程取决于治疗病症的性质和严重程度。治疗处方,例如剂量的决策等,是全科医生和其他医生的职责范围,且一般需要考虑到所知道的的病症、患者的个别情况、递送位点、给药方法和医师所知的其他因素。本发明的组合物尤其是涉及癌症的治疗,对所述病症在初始治疗或手术后的复发的预防。上述技术和方案的例子可以参见remington’spharmaceuticalsciences[73]。组合物可以单独给药或与其他治疗一起给药,其可以使同时给药或相继给药,取决于所治疗的病症。其他癌症治疗包括本领域所知的其他单克隆抗体、其他化疗药剂、其他放疗技术或其他免疫疗法。本发明所述的组合物的一种具体应用在于作为手术的辅助,即帮助降低肿瘤移除后再次复发癌症的风险。本发明的组合物可以全部或部分通过化学合成来制备。该化合物可以根据本领域熟知的标准液体法,或优选地,固相多肽合成法,来便利地制备,所述方法的一般性描述随处可得(例如参见solidphasepeptidesynthesis,2ndedition[74]、thepracticeofpeptidesynthesis[75]和appliedbiosystems430ausersmanual,abiinc.);或者,其可以在溶液中通过液相法制备,或通过顾想法、液相法和溶液化学法的任意组合来制备,例如首先完成各个多肽部分,然后根据需要和是否适当,在去除存在的任何保护性基团后,通过各碳酸或磺酸或其反应性衍生物的反应来引入残基x。另一种制备本发明组合物的便利方法在于,通过在表达体系中使用编码所述组合物的核酸来对其进行表达。本发明还包括编码本发明组合物的核酸分离物。在一个优选的方面中,本发明提供了编码如上所述的本发明组合物的核酸。本领域技术人员能够确定仍提供本发明的组合物的所述核酸的取代、删除和/或添加。所述核酸可以是dna、cdna或rna,例如通过克隆或化学合成产生的mrna。对于医疗用途,所述核酸形式优选为能够在治疗对象中表达的形式。本发明中可用的多肽或本发明的核酸可以提供为分离物,处于分离和/或纯化形式,或不含或基本不含其天然关联的物质。对于核酸而言,它可以不含或基本不含人类基因组中的基因侧翼核酸,但可能仍含一个或多个表达调控序列。核酸可以全部或部分是合成的,且可以包括基因组dna、cdna或rna。当本发明的核酸包含rna时,对所示序列的提及应当被理解为亦包含其等同rna(u被取代为t)。只要有了所述核酸序列和克隆,本领域技术人员可以很方便地制备编码本发明中可用的多肽的核酸序列,例如使用本文所提供的信息和文献以及本领域已知技术(例如参见[76,77])。所述技术包括(i)使用聚合酶链反应(pcr)来扩增所述核酸样本,例如从基因组来源;(ii)化学合成;或(iii)制备cdna序列。编码所述多肽的dna可以用本领域技术人员已知的任何适当方法来制备和使用,包括,取得编码dna,在待表达部分两端找到适当的限制性酶识别位点,并从所述dna切取所述部分。该部分随后可以通过标准市售表达体系,可操作地连接在适当的启动子上,另一重组途径是,以适当pcr引物扩增该dna的相关部分。可以通过例如使用定点突变来对所述序列进行修饰,以获得修饰后多肽的表达或考虑宿主细胞中用于表达所述核酸的优选密码子。本发明还提供含有至少一个上述核酸的质粒、载体、转录或表达组件形式的结构。本发明还提供包括含有一个或多个上述结构的重组宿主细胞。如上所述,编码本发明组合物的核酸构成本发明的一个方面;制备所述组合物的方法也构成本发明的一个方面,所述方法包括编码核酸的表达。通过在适当条件下培养含有所述核酸的重组宿主细胞,能够便利地使其表达。表达制备后,可以使用任何适当技术来分离和/或纯化组合物,并适当使用。各种不同宿主细胞中的多肽克隆和表达体系是众所周知的。适当的宿主细胞包括细菌、哺乳类细胞、酵母和杆状病毒体系。本领域可用于表达异源多肽的哺乳类细胞系,包括中国仓鼠卵巢细胞、hela细胞、仓鼠崽肾细胞、nso小鼠黑色素瘤细胞和许多其他细胞。常见的优选细菌宿主为e.coli。抗体和抗体片段在原核细胞(如e.coli)中的表达为本领域所熟知。参见文献[78]的综述。本领域技术人员还可选择使用在真核细胞培养物中的表达,来生成特异性结合成分,参见例如文献[79,80]中的近期综述。可以选择或构件含有适当调控序列的适当载体,所述调控序列包括启动子序列、终止序列、多聚腺苷酸化序列、增强子序列、标记基因和其他适当序列。载体可以是质粒、病毒(如噬菌体)或噬菌粒,视需要而定。更多详情参见例如molecularcloning:alaboratorymanual[76]。shortprotocolsinmolecularbiology[77]中详细记载了许多核酸操作的已知技术和实验方案,例如核酸结构的制备、诱变、测序、dna引入细胞和基因表达、蛋白分析。因此,本发明的又一方面中,提供一种含有本文所公开的核酸的宿主细胞,其可以被分离。在又一个方面中,提供一种方法,包括将所述核酸引入宿主细胞。所述引入可以使用任何可用技术。对于真核细胞,适当的技术可以包括磷酸钙转染、deae-葡聚糖、电穿孔、脂质体介导的转染,和使用反转录病毒或其他病毒(例如牛痘,或对昆虫细胞而言,杆状病毒)的转导。对于细菌细胞,适当的技术可以包括氯化钙转化、电穿孔和使用噬菌体进行转染。该引入的后续可以是引发或允许该核酸的表达,例如通过在该基因的表达条件下培养宿主细胞。在一个实施例中,本发明的核酸被整合入宿主细胞的基因组(例如染色体)中。可以根据标准技术,通过包含促进与该基因组重组的序列来促进整合。本发明还提供了一种方法,包括在表达体系中使用上述结构以表达如上所述的多肽。本发明的每个方面中的优选特征,可在必要的细节变更基础上用于其他方面。本文中提到的现有技术文件在法律允许的最大范围内并入本文。实施例下面将引用以下实施例和附图,进一步描述本发明。图1:蛋白质中的精氨酸向瓜氨酸的酶促转化由pad催化。图2:人波形蛋白的氨基酸序列。图3:限定上皮向间叶细胞转化(emt)及其相反过程(间叶细胞向上皮转化)的相关表型变化。本图引自[81]。图4:cd4+t细胞亚种群。cd4+t细胞的主要种群为th1、th2和th17,以及itregs。treg控制免疫效应器细胞的应答。本图引自kaufmann[81]。图5:cd4t细胞对抗体dna中编码的辅助表位的应答。用基因枪以cdrl1或cdrh3中含有辅助表位的抗体dna结构对hla-dr4或hla-dr1转基因小鼠免疫接种。所有小鼠均在第0、7、14天时免疫接种三次。第20天时,采用ifnγ酶联免疫斑点法(elispot)分析对所述辅助表位对相关辅助多肽和无关对照的特异性应答进行间接体内分析。应答测量为斑点/百万脾细胞。图6:对插入huigg1抗体dna双表达载体cdrh3位点的波形蛋白28-49辅助表位进行体内加工、瓜氨酸化和呈递。连续3周每周一次用基因枪以抗体dna结构对hla-dr4转基因小鼠免疫接种。第19天时,通过(i)ifnγ(ii)il-17和(iii)il-2酶联免疫斑点法以浓度为5μm的波形蛋白28-49野生型和瓜氨酸化(cit)多肽对脾细胞进行体外分析图7:对插入huigg1抗体dna双表达载体cdrh3位点的波形蛋白415-433辅助表位进行体内加工、瓜氨酸化和呈递。连续3周每周一次用基因枪以抗体dna结构对hla-dr4转基因小鼠免疫接种。第19天时,通过(i)ifnγ(ii)il-17和(iii)il-2酶联免疫斑点法以浓度为5μm的波形蛋白415-433野生型和瓜氨酸化多肽对脾细胞进行体外分析。图8:以编码mmp-7辅助表位的抗体dna结构免疫接种诱导dr1小鼠的cd4应答。用基因枪以含有mmp-7247辅助表位的抗体dnahuigg1结构对hla-dr1转基因小鼠免疫接种。所有小鼠均在第0、7、14天时免疫接种三次。第20天时,通过(i)ifnγ(n=14)和(ii)il-17(n=14)酶联免疫斑点法对辅助表位对mmp7人247野生型和瓜氨酸化多肽的特异性应答进行体外分析。应答测量为斑点/百万脾细胞。图9:抗体dna中编码的nyeso1119-143cd4表位刺激cd4应答,伴随着抗肿瘤应答。第0天时以2.5x104b16hhdiinyeso-1细胞注入hhdii/dr1转基因小鼠。小鼠在第3、10和17天时用基因枪以在cdrl1位点上含有辅助表位nyeso-1119-143的抗体dnahuigg1dna免疫接种。(i)第20天时,通过ifnγ酶联免疫斑点法对辅助表位对野生型、瓜氨酸化nyeso-1119-143辅助多肽和无关对照的特异性应答进行体外分析。应答测量为斑点/百万脾细胞。(ii)以nyeso-1抗体dna单独免疫接种或与homspera佐剂免疫接种或以homspera佐剂单独免疫接种的动物存活。图10:来自全抗原dna结构的波形蛋白28-49和415-433表位进行体内加工、瓜氨酸化和呈递。连续3周每周1次通过基因枪以编码鼠波形蛋白的dna结构或通过皮下注射以cpg/mpla中的vim28-49和415-433瓜氨酸化多肽,来对hla-dr4转基因小鼠免疫接种。第19天,通过ifnγ酶联免疫斑点实验,以浓度为5μm的28-49和415-433野生型和瓜氨酸化多肽对脾细胞进行体外分析。图11:对比以人野生型和瓜氨酸化波形蛋白28-49多肽免疫接种获得的hla-dr4转基因小鼠的cd4应答。第0天以25μg波形蛋白28-49人野生型或瓜氨酸化多肽对hla-dr4转基因小鼠进行免疫接种。第14天时,对脾细胞进行体外分析,对(a)浓度为5μm的波形蛋白28-49和415-433野生型和瓜氨酸化多肽,通过(i)ifnγ(ii)il-17(iii)il-2(iv)il-10的酶联免疫斑点实验进行分析;(b)瓜氨酸化表位特异性应答的亲抗原性,通过ifnγ和il-17的酶联免疫斑点实验测量对递增多肽浓度的应答进行分析;(c)5μm的波形蛋白28-49三聚、二聚和单体瓜氨酸化多肽,通过ifnγ酶联免疫斑点实验进行分析。应答测定为平均斑点/百万脾细胞。图12:vim28-49、415-433、瓜氨酸化vim28-49、瓜氨酸化415-433多肽与hla-dr0401在竞争结合实验中的结合度。将多肽与源自流行性感冒(ha306-318)的预定浓度的生物素化多肽混合,然后分析与纯化hla-dr0401的结合度。使用未标记的ha306-318多肽作为阳性对照。图13:对比以人野生型和瓜氨酸化波形蛋白415多肽免疫接种获得的hla-dr4转基因小鼠的cd4应答。第0天以25μg波形蛋白人野生型或瓜氨酸化415多肽对hla-dr4转基因小鼠进行免疫接种。第14天时,对脾细胞进行体外分析,对(a)浓度为5μm的波形蛋白415野生型和瓜氨酸化多肽,通过(i)ifnγ(ii)il-17(iii)il-2(iv)il-10的酶联免疫斑点实验进行分析;(b)瓜氨酸化表位特异性应答的亲抗原性,通过ifnγ和il-17的酶联免疫斑点实验测量对递增多肽浓度的应答进行分析。应答测定为平均斑点/百万脾细胞。图14:hla-dr4转基因小鼠的以人瓜氨酸化波形蛋白415多肽免疫接种获得的cd4应答以及与等同的鼠瓜氨酸化多肽的交叉反应性。第0天以25μg波形蛋白人瓜氨酸化415-433多肽对hla-dr4转基因小鼠进行免疫接种。第14天时,通过(i)ifnγ(ii)il-17的酶联免疫斑点实验,对浓度为5μm的人和鼠瓜氨酸化波形蛋白415对脾细胞进行体外分析。应答测定为平均斑点/百万脾细胞。图15:验证hla-dr4转基因小鼠中以(a)人瓜氨酸化波形蛋白415多肽和(b)瓜氨酸化波形蛋白28多肽免疫接种获得的t细胞应答是cd4应答。第0天以25μg波形蛋白人瓜氨酸化415-433多肽对hla-dr4转基因小鼠进行免疫接种。第14天时,通过ifnγ酶联免疫斑点实验,在存在或不存在l243hla-dr阻断抗体的情况下,以浓度为5μm的人瓜氨酸化波形蛋白415或28多肽对全脾细胞或清空cd4的脾细胞进行体外分析。应答测定为平均斑点/百万脾细胞。图16:hla-dr4和hla-a2/dr1转基因小鼠中以瓜氨酸化波形蛋白65多肽免疫接种获得的cd4应答。第0天以25μg波形蛋白65瓜氨酸化多肽对(a)hla-dr4或(b)hla-a2/dr1转基因小鼠进行免疫接种。第14天时,通过ifnγ、il-17或il-10的酶联免疫斑点实验,以浓度为5μm的波形蛋白65野生型和瓜氨酸化多肽,对全脾细胞进行体外分析;(c)第14天时,通过ifnγ酶联免疫斑点实验,在存在或不存在l243hla-dr阻断抗体的情况下,以浓度为5μm的人波形蛋白瓜氨酸化65多肽对全脾细胞进行体外分析;应答测定为平均斑点/百万脾细胞。(d)对脾细胞中cd8、ifnγ和tnfα的免疫荧光染色和facs分析。(e)第14天,以人瓜氨酸化波形蛋白65多肽脉冲lps冲击来对脾细胞进行体外刺激。刺激后6天,以铬释放试验来评估ctl系溶解以瓜氨酸化人波形蛋白65多肽和t2细胞或单独以b16hhd冲击的t2或b16hhd肿瘤细胞的能力。应答测定为%细胞毒性。图中所示的p值是针对目标:效应器比例100:1显示的)通过ifnγ酶联免疫斑点实验,以最低预测值的野生型和瓜氨酸化hla-a2结合短多肽波形蛋白68和65以及浓度为5μm的野生型和瓜氨酸化波形蛋白65多肽,对来自免疫接种小鼠的脾细胞进行体外分析;应答测定为平均斑点/百万脾细胞。(g)通过ifnγ酶联免疫斑点实验,分析免疫接种小鼠的脾细胞对瓜氨酸化和野生型vim65和vim68多肽以及el4hhd和b16肿瘤靶细胞的应答。图17:在存在或不存在天然t调控细胞的存在下,生成的对鼠波形蛋白415-433瓜氨酸化多肽的辅助应答。第0天以25μg鼠形蛋白415-433瓜氨酸化多肽对hla-dr4转基因小鼠进行免疫接种。免疫接种3天前,使用抗cd25单克隆抗体(pc61)耗尽t调控细胞。第14天,通过ifnγ酶联免疫斑点实验,以瓜氨酸化鼠波形蛋白415多肽滴定浓度来分析脾细胞。图18:佐剂影响对瓜氨酸化波形蛋白415和28多肽的应答中的th1/th17平衡。以alum、ifa、gmcsf、mpla和cpg或tmx201为佐剂的人瓜氨酸化波形蛋白415-433和28-49多肽(25μg)进行皮下给药。免疫接种14天后,通过i)ifnγ、(ii)il-17和(iii)il-10酶联免疫斑点实验,以野生型多肽和瓜氨酸化多肽和无关对照,来分析脾细胞对辅助表位的特异性应答。应答测定为斑点/百万脾细胞。图19:抗ctla-4mab提高对瓜氨酸化波形蛋白415多肽的应答的亲抗原性。第0、7、14天时,以25μg人瓜氨酸化波形蛋白415多肽对hla-dr4转基因小鼠免疫接种。半数小鼠还在第0、7、14天时接受了抗ctla-4mab。第21天,对脾细胞进行体外分析,(i)用5μm瓜氨酸化波形蛋白415进行ifnγ酶联免疫斑点实验;(ii)在ifnγ酶联免疫斑点实验中测定对递增多肽浓度的表位特异性应答的亲抗原性。应答测定为斑点/百万脾细胞。图20:从患者分离的外周血单核细胞(pbmc’s)对野生型和瓜氨酸化多肽的增殖应答。以10μg/ml的(i)野生型和瓜氨酸化人波形蛋白415-433多肽、(ii)野生型和瓜氨酸化人波形蛋白28-49多肽、(iii)野生型和瓜氨酸化人波形蛋白65-77多肽和(iv)瓜氨酸化nyeso-1119-143多肽,培养患者的pbmc’s4、7和11天。添加3[h]–胸腺嘧啶核苷以分析淋巴细胞增殖。增殖应答测定为刺激指数(si)。si计算为受刺激多肽cpm平均值与未受刺激培养物cpm平均值之比值。图21:a)肿瘤表达pad2的卵巢癌患者的卡普兰迈耶存活曲线。b)肿瘤表达hmgb1的卵巢癌患者的卡普兰迈耶存活曲线。c)肿瘤表达pad2和hmgb1的卵巢癌患者的卡普兰迈耶存活曲线。d)肿瘤表达pad4的结肠直肠癌患者的卡普兰迈耶存活曲线。图22:第0、7、14天以cpg和mpla为佐剂的瓜氨酸化人波形蛋白415-433多肽(b)或瓜氨酸化vim28-48多肽(c)或两者(a,d&e)对hla-dr4转基因小鼠免疫接种。a:第14天,通过ifnγ酶联免疫斑点实验,在存在或不存在3-ma或ci-脒的情况下,以5μm的人瓜氨酸化或未瓜氨酸化波形蛋白415和28多肽,以及通过血清饥饿诱导自体吞噬的b16dr4肿瘤细胞,分析脾细胞。b-e:通过酶联免疫试验,分析间接体内ifnγ酶联免疫斑点实验的上清液中存在的颗粒酶b。图23:肿瘤细胞的体外杀灭。(ai)第0、7、14天以cpg和mpla为佐剂的瓜氨酸化人波形蛋白415-433多肽对hla-dr4转基因小鼠免疫接种。第19天,以人瓜氨酸化波形蛋白415-433多肽脉冲冲击来体外刺激脾细胞。刺激后6天,使用铬释放试验来评估ctl系对以瓜氨酸化人波形蛋白415-433多肽脉冲的dr4脾细胞、以瓜氨酸化人波形蛋白415-433多肽脉冲的t2dr4肿瘤细胞、单独的t2dr4细胞的溶解能力。应答测定为%细胞毒性。图中所示p值针对的是靶细胞:效应器细胞比例10:1。对20:1和40:1的p值也高度显著p<0.0001。(aii)第0、7和14天用以cpg和mpla为佐剂的瓜氨酸化人波形蛋白28-49多肽对hhd/dr1转基因小鼠免疫接种。第19天,以人瓜氨酸化波形蛋白28-49多肽脉冲冲击对脾细胞进行体外刺激。刺激后6天,使用铬释放试验来评估ctl系对以瓜氨酸化人波形蛋白28-49多肽脉冲的t2dr1肿瘤细胞、单独的t2dr1细胞、单独的t2细胞的溶解能力。应答测定为%细胞毒性。图中所示p值针对的是靶细胞:效应器细胞比例12.5:1且p值显著。对25:1(t2dr1p=0.0017,t2dr1+vim28citp<0.0001)和50:1(t2dr1p=0.0008,t2dr1+vim28citp=0.0005)的p值均高度显著。图24:瓜氨酸化波形蛋白415-433和波形蛋白28-49cd4应答影响抗肿瘤免疫应答。第0天,向hla-dr4转基因小鼠注射2.5x104b16f1-dr4细胞。(a)第4、11和18天,用对照抗体dna,或在cdrh3编码hla-dr4限制性vim415辅助表位的抗体dna疫苗,或以mpla和cpg为佐剂的鼠瓜氨酸化波形蛋白415-433多肽(25μg),使用基因枪对小鼠皮下给药免疫接种。(i)(b)第4、11和18天,用对照抗体dna,或在cdrh3编码hla-dr4限制性vim28辅助表位的抗体dna疫苗,或以mpla和cpg为佐剂的瓜氨酸化或野生型波形蛋白28-49多肽(25μg),使用基因枪对小鼠皮下给药免疫接种。(c)用以mpla和cpg为佐剂的瓜氨酸化波形蛋白415-433多肽(25μg)或波形蛋白28-49或两者(25μg),对小鼠皮下给药免疫接种。(d)用以mpla和cpg为佐剂的瓜氨酸化波形蛋白415-433多肽(25μg)联合抗cd4抗体(克隆gk1.5),对小鼠皮下给药免疫接种。(e)用以mpla和cpg为佐剂的瓜氨酸化波形蛋白28-49多肽(25μg)联合抗cd4抗体(克隆gk1.5),对小鼠皮下给药免疫接种。图25:瓜氨酸化波形蛋白415-433和波形蛋白28-49的抗肿瘤免疫应答部分由ifnγ和il-17介导。第0天,向hla-dr4转基因小鼠注射2.5x104b16f1-dr4细胞。用以mpla和cpg为佐剂的(a和c)瓜氨酸化波形蛋白415-433多肽(25μg)或(b和d)波形蛋白28-49多肽(25μg);在存在或不存在ifnγ中和单克隆抗体(a和b)或il-17中和抗体(c和d)的情况下,对小鼠进行皮下免疫接种。图26:瓜氨酸化波形蛋白415-433抗肿瘤免疫应答,而非波形蛋白28-49抗肿瘤免疫应答,部分受到肿瘤细胞上的hla-dr0401的直接识别的介导。第0天以(a)2.5x104b16f1细胞或(b)b16f1-dr4细胞注射hla-dr4转基因小鼠。以(a)以mpla和cpg为佐剂的瓜氨酸化波形蛋白415-433多肽(25μg)或波形蛋白28-49多肽(25μg)或两者;(b)以mpla和cpg为佐剂的波形蛋白415-433多肽(25μg),在存在或不存在hla-dr0401中和单克隆抗体的情况下,对小鼠进行皮下免疫接种。图27:不同物种间的波形蛋白同源性。图28:筛选波形蛋白中刺激t细胞应答的新型表位。用以mpla和cpg为佐剂的人瓜氨酸化细胞角蛋白多肽(3x10μg)皮下给药。a)hla-a2/dr1和b)c57/blmice小鼠免疫接种14天后,通过对辅助多肽和无关对照的(i)ifnγ和(ii)il-17酶联免疫斑点实验来检测脾细胞对辅助表位的特异性应答。c)用以mpla和cpg为佐剂的瓜氨酸化vim14多肽皮下给药。免疫接种14天后,通过对辅助多肽和无关对照的ifnγ酶联免疫斑点实验来检测脾细胞对辅助表位的特异性应答。应答测定为斑点/百万脾细胞。图29:筛选细胞角蛋白8中刺激t细胞应答的新型表位。用以mpla和cpg为佐剂的人瓜氨酸化细胞角蛋白多肽(3x10μg)皮下给药。a)hla-a2/dr1和b)c57/blmice小鼠免疫接种14天后,通过对辅助多肽和无关对照的(i)ifnγ和(ii)il-17酶联免疫斑点实验来检测脾细胞对辅助表位的特异性应答。应答测定为斑点/百万脾细胞。图30:野生型波形蛋白刺激分泌il-10的itreg细胞应答。用以mpla和cpg为佐剂的人415波形蛋白多肽(25μg)皮下给药。hla-dr4小鼠免疫接种14天后,通过对辅助多肽和无关对照的(i)ifnγ,(ii)il-17和(iii)il-10酶联免疫斑点实验来检测脾细胞对辅助表位的特异性应答。应答测定为斑点/百万脾细胞。图31:瓜氨酸化ing4刺激cd4应答。第0、7、14天,用以mpla和cpg为佐剂的瓜氨酸化ing4多肽158-174多肽(25μg)皮下给药。对hla-a2/dr1最后一次免疫接种后7天,在存在或不存在hla-dr阻断单克隆抗体的情况下,用对辅助多肽的ifnγ酶联免疫斑点实验测试hla-a2/dr1小鼠脾细胞对辅助表位的特异性应答。应答测定为斑点/百万脾细胞。图32:本发明中可用表位的蛋白质在通用蛋白质库中的来源索引。图33:全抗原波形蛋白dna诱导瓜氨酸化波形蛋白特异性t细胞应答。在第0、7、14天,通过基因枪以1μg编码全鼠波形蛋白序列的dna对(a)hla-dr4转基因小鼠或(b)hla-a2/dr1转基因小鼠进行免疫接种。第20天,通过酶联免疫斑点实验,分析脾细胞对一组跨全蛋白的瓜氨酸化波形蛋白多肽的ifnγ应答。应答测定为斑点/百万脾细胞。图34:预测多肽的瓜氨酸特异性免疫应答的筛选。第0、7、14天用以cpg和mpla佐剂的源自bip,hsp90和ing4的预测瓜氨酸化多肽(25ug)对hla-dr4转基因小鼠进行皮下给药。第14天或20天,通过ifng酶联免疫斑点实验,分析脾细胞对相关瓜氨酸化和未修饰多肽(5um)和背景对照的免疫应答。应答测定为平均斑点/百万脾细胞。方法2.1.市售mabs从abcam购入一级兔抗人波形蛋白(克隆epr3776)、兔抗人pad2(克隆pab0197)、兔抗人瓜氨酸(克隆ab6464)。从covalab购得一次兔抗人pad-4(克隆pab0199)并从ebioscience购得抗人hla-drpe-cy7标记抗体(克隆l243)。从bioxcell购得抗-cd25抗体(克隆pc61)、抗ifnγ抗体(克隆xmg1.2)、抗il-17(克隆17f3)抗体和抗cd4(克隆gk1.5)抗体。通过琼脂糖蛋白g亲和层析法从hb304杂交瘤细胞培养物上清液(atcc,usa)中纯化抗ctla4抗体。通过琼脂糖蛋白g亲和层析法从hb-55杂交瘤细胞培养物上清液(atcc,usa)中纯化抗hla-dr抗体。从cellsignalingtechnology公司购入兔mab抗hmgb1(克隆d3e5)。2.2.细胞系t细胞/b细胞杂交细胞系t2[80]以功能ii型dr4(drb1*0401;t2dr4)或dr1(drb1*0101;t2dr1)稳定转染在文献中已有记述[82,83],并由janiceblum博士和lawrencestern教授友情提供。小鼠黑色素瘤b16f1和b16f10细胞系来自atcc。除非另有说明,否则所有细胞系均在补充了10%fcs、l-谷氨酰胺(2mm)和碳酸氢钠缓冲液的rpmi培养基1640(gibco/brl)中培养。hb304杂交瘤细胞在杂交瘤sfm(invitrogen,uk)中进行培养。为了生成呈递瓜氨酸化表位的肿瘤标靶用于体外分析,4℃下以含1%bsa的0.1m柠檬酸(ph3.0)处理细胞2分钟,然后以培养基洗涤细胞,并37℃无血清培养20小时。该20小时无血清培养基培养中,添加了自噬和pad抑制剂,即终浓度分别为10nm和50μg/ml的3-甲基腺嘌呤(sigma)和ci-脒(calbiochem)。2.3.免疫原2.3.1多肽通过多肽合成材料(fareham,uk)合成大于90%纯度的多肽。-80℃冻干贮藏在0.2mg小份中。使用时,以10%二甲基甲酰胺重建适当浓度。2.4.质粒其他文献已详细叙述了抗体dna结构的生成[84]。简单而言,为了生成抗体dna结构,使用标准分析生物学技术将表位并入抗体的轻链可变域和重链可变区的互补决定区中。将来自表位酪氨酸酶448-462(dysylqdsdpdsfqd)、鼠和人hla-dr4gp10044-59表位(wnrqlypewtevqgsn/wnrqlypewteaqrld)和来自hepb核蛋白128-140(tppayrppnapil)的i-ab限制性表位限制性辅助cd4表位插入取代κ链的cdrl1。相似地,来自表位mmp7247-262(sqddikgqklygkrs)、ssx233-48(keewekmkasekify)、nyeso-187-111(llefylampfatpmeaelarrslaq)和119-143(pgvllkeftvsgniltirltaadhr)的hla-dr1限制性辅助cd4表位并入cdrl1,而来自表位磷酸丙糖异构酶23-37(geligilnaakvpad)的修饰后表位被插入取代κ链的cdrl3。hla-dr4限制性cd4波形蛋白415-433(lpnfsslnlretnldslpl)和hla–dr128-49(rsyvttstrtyslgsalrpsts)限制性表位均被并入cdrh3。dr1cd4nyeso-187-111(llefylampfatpmeaelarrslaq)也被编码在克隆入抗体dna双表达载体中的扩展序列83-111中。还生成了含有全部3种波形蛋白表位的人igg1和鼠igg2a免疫体载体。hla–dr128-49(rsyvttstrtyslgsalrpsts)、65-77(savrlrssvpgvr)和hla-dr4限制性人cd4波形蛋白415-433(lpnfsslnlretnldslpl)表位分别并入cdrh1、cdrh2和cdrh3。将来自从b16f1细胞系分离得到的mrna的全长序列作为模版cdna,通过扩增生成质粒pvaxi鼠全长波形蛋白。所用的正反向引物分别设计为并入hindiii和bamhi位点。通过野生型序列的扩增和确认,将全长鼠波形蛋白并入哺乳类表达载体pvaxi(invitrogen)的多克隆位点的hindiii/bamhi位点。为了生成质粒pvitro2,从转基因hla-dr4小鼠的脾细胞中分离得到mrna,并以此生成嵌合hla-dr4cdna。将其用作模版,以分别含有fspi/ecori和bamhi/sali位点的正向和反向引物来分别扩增嵌合α链和β链。在序列确认中,由鼠h2-ea和hla-draα1域组成的人全长嵌合α链结合在载体pvitro2-hygro-mcs(invivogen)的fspi/ecorimcs2位点中。由鼠h2-eb和人drb1*0401β1域组成的β链随后与嵌合α链一同插入载体的bamhi/salimcs1位点。为了生成hhd质粒,从el4-hhd细胞分离全rna并以此合成cdna。将其用作模版以利用正向和反向引物扩增hhd,并亚克隆入pcr2.1中。随后将hhd链插入购自invitrogen的哺乳类表达载体pcdna3.1的ecorv/hindiii位点,所述hhd链包括人hla-a2前导,和通过甘氨酸丝氨酸连接子共价连接至人hla-0201mhc1类分子的α1和2域及小鼠h-2db1类分子的α3跨膜和胞浆区的人b2微球蛋白分子。为了构建编码鼠tap2和nyeso-1全长链的哺乳类双表达质粒,用从geneservice公司购得的image克隆40146393以分别包含bamh1/xhoi位点的正向和反向引物扩增nyeso-1。在序列确认中,将全长nyeso-1连接到抗体dna双表达载体的bamhi/xho1多克隆位点上,取代轻链。用从编码序列移除了hindiii位点并将其插入在起始密码子前的映像克隆6530488来扩增鼠tap2,并使用hindiii/ecorv将其克隆入表达载体porighib。然后,使用hindiii/avrii将鼠tap2与全长nyeso-1一道转移至双表达载体中,取代重链。为了降低鼠b2微球蛋白和鼠mhcii类在细胞系b16f10中的表达,使用了rna干扰。将靶定鼠b2微球蛋白的序列266和鼠mhcii类序列159的互补寡核苷酸退火并分别插入pcdna6.2gwmir(invitrogen)中。使用bamhi/xhoi切离含有mirna266的mirna前体表达组件,并连接到pcdna6.2gwmir159的xhoi/bglii位点上,以将两条mirna链连接在一起,并在相同载体中的初级转录物中表达。使用qiagen无内毒素质粒提取试剂盒(endofreeqiagenmaxiprepkit,qiagen,crawley)生成不含内毒素的质粒dna。2.5.转染使用质粒体转染剂以编码全长ny-eso-1和鼠tap2、hhdii和sirna的表达载体,相继转染b16f10细胞,以降低鼠mhcii类和鼠β2微球蛋白的表达。转染后的细胞分别在博来霉素(300μg/ml)、g418(500μg/ml)和杀稻瘟菌素(4μg/ml)的存在下进行生长选择。通过有限稀释法克隆细胞系,并通过流式细胞仪确认表达。根据厂商说明,使用质粒体转染剂(invitrogen)以4μg编码全长嵌合α和β链的质粒pvitro2嵌合hla-dr401来转染b16f1细胞。在潮霉素b(200μg/ml)的存在下选择转染细胞的生长。通过有限稀释法克隆细胞系,并使用购自ebioscience的hla-drpe-cy7标记抗体(克隆l243)抗人通过流式细胞仪确认表达。2.6hladr0401结合实验简单而言,将浓度递增的实验多肽与预定浓度的生物素化ha306-318对照多肽混合,并添加到固定在板上的hladr0401中。与hladr0401结合的生物素化对照多肽的量,使用链霉亲和素标记酶步骤和随后的显色底物检测来进行量化。将生物素化ha306-318多肽单独达到的数值看作最大结合度。作为阳性对照,未标记ha306-318多肽用于与生物素化ha306-318多肽竞争。2.7.免疫接种2.7.1免疫接种方案使用了8-12周龄的c57bl/6小鼠(charlesriver,uk)、hla-dr4小鼠(taconic,usa)、hhdii小鼠(pasteurinstitute,france)和hhdii/dr1小鼠(pasteurinstitute,france),由诺丁汉伦特大学(nottinghamtrentuniversity)的员工照顾。所有工作均由英国内政部项目许可证授权。将多肽在10%二甲基甲酰胺中制为1mg/ml溶液并以不同佐剂进行乳化(系列稀释):cpg和mpla各为6μg/小鼠(invivogen,uk)、弗氏不完全佐剂50μl/小鼠(sigma,uk),以及gmcsf10μg/小鼠(peprotech,uk)。在尾根处皮下注射多肽(25μg/小鼠)。根据厂商说明将dna(1μg/小鼠)涂布在1.0μm金颗粒(biorad,hemelhempstead,uk)上,并以基因枪(biorad)皮内给药。使用基因枪皮下注射地免疫接种homspera(10nm/小鼠)(peptidesynthetics,uk)。小鼠在第0天免疫接种多肽,或在第0、7、14天进行多肽和基因枪免疫接种。除非另有说明,否则第14天摘除脾脏并分析多肽,第20天则摘除分析多肽或基因枪接种。免疫接种前3天,以含400μg抗cd25抗体(pc61)的盐水腹腔注射(i.p.)给药。免疫接种第7和第14天,用基因枪或多肽免疫接种以含200μg抗ctla-4抗体(uc10-4f10-11)的盐水腹腔注射(i.p.)给药。对于肿瘤激发实验,在基础免疫3天前,以2.5x104b16hhdiinyeso/tap2siβ2m1f10细胞或b16dr42e7细胞对小鼠进行右侧皮下给药,然后如上所述地进行免疫。肿瘤植入后第2、7、11和14天,以含抗ifnγ抗体(300μg/剂)、抗il-17抗体(200μg/剂)和抗hla-dr抗体(300μg/剂)的盐水进行腹腔注射给药。在肿瘤植入后的第2和8天以含抗cd4抗体(500μg/剂)的盐水进行腹腔注射给药。以3-4天为间隔时间监控肿瘤生长,一旦肿瘤直径达到≥10mm,即将小鼠安乐死。2.8.免疫应答分析2.8.1间接体内酶联免疫斑点法分析根据厂商说明(mabtech,sweden),使用ifnγ、il-17和il-10捕获和检测试剂进行酶联免疫分析(elispot)。简单而言,将抗ifnγ、il-17和il-10抗体涂布在96孔immobilin-p板的孔上。将合成多肽(各种浓度)和每孔5x105的脾细胞加入所述板孔中,一式三联。将癌症靶细胞按5x104/孔一式三联地加入相关位点,将板在37℃下培养40小时。培养后,以生物素化抗ifnγ、il-17和il-10抗体来检测捕获的ifnγ、il-2、il-17和il-10,并以链霉亲和素碱性磷酸酶和显色底物进行显影。使用自动分析仪(cellulartechnologiesltd)来分析和计数斑点。使用效应器功能对多肽浓度作图,将功能性亲抗原性计算为调控50%最大效应器功能时的浓度。2.8.2来自脾细胞培养物的cd8和cd4细胞的间接体内清空根据厂商说明,使用抗体涂覆磁珠(miltenyibiotech)令脾细胞完全隔离于cd4或cd8细胞。对于mhcii类阻断实验,向酶联免疫斑点实验中加入20μg/ml抗hla-dr(克隆l243)抗体。2.8.3颗粒酶b的酶联免疫吸附测定脾细胞的间接体内ifnγ酶联免疫斑点实验,40小时后取上清液,并根据厂商说明进行颗粒酶b的酶联免疫吸附测定(r&dsystems公司)。2.8.4luminex多重分析使用三步间接过程进行igg抗体对il-10、il-17、ifnу、tnfα、il-2和il-4的多重luminex分析(invitrogen)。取标样、对照和未知血清,以50%分析稀释液(invitrogen)和50%无血清rpmi进行1:2稀释。每一实验中均包括系列标准稀释。取每一标样、对照和未知血清稀释溶液,与一组偶联luminex微球在96孔滤板(milliporemultiscreen;milliporecorporation,bedford,mass.)中混合,并在室温下摇动培养2小时。真空过滤收集微球,并以pbst洗涤。向每孔中加入生物素化检测抗体,室温下摇动1小时。培养30分钟并洗涤后,将微球重悬在pbst中,并以配备xy平台的bioradbioplexluminex分析仪进行分析。以luminex软件(bioplexsystems)进行数据采集和分析。2.8.5增殖实验用ficol-hypaque(sigma)梯度离心从新鲜抽取的肝素化血液中分离外周单核细胞(pbmc)。在含有5%合并自体人血清、2mm谷氨酰胺、20mmhepes和青霉素-链霉素(1%)且终体积为2ml的rpmi培养基中,以单肽(终浓度为10μg/ml)刺激pbmc(1.5×106细胞/孔)。纯化蛋白衍生物ppd(终浓度10μg/ml)的刺激作为pbmc增殖能力的阳性对照。作为阴性对照,将pbmc与培养基单独培养。将pbmc在37℃下、5%co2的空气中培养4、7、11天。为了检测所述时间点上的增殖,从每一培养物中一式三份地取100μl,移入96孔板的圆底孔中,并添加3h-胸腺嘧啶核苷(0.0185mbq/孔),再在37℃下培养8小时。将培养物收获到unifilter板上,并以β-闪光计数测定3h-胸腺嘧啶核苷的结合。分析结果,计算刺激指数(si)为受刺激表位每分钟计数(cpm)平均值与未受刺激培养物之间的比值。当si>2.5时,增殖实验认为是阳性。2.8.651cr-释放试验在存在或不存在10μg/ml肽的情况下,以1.85mbq络酸钠(51cr)(amersham,essex,uk)标记靶细胞1小时。培养后,以rpmi洗涤3次。建立5x103/孔的96孔尖底板,并与不同密度的效应器细胞共培养,rpmi终体积为200μl,含10%fcs(sigma)、20mmhepes缓冲液、2mm左旋谷氨酰胺、100单位/ml青霉素和100μg/ml链霉素。37℃下培养20小时后,从每孔取50μl上清液,移入lumaplate(packard,rigaweg,thenetherlands)中。以topcount微孔板闪烁记数器(packard)。特异性溶解百分比以下述公式计算:特异性溶解=100x[(实验释放-自发释放)/最大释放–自发释放]。2.9免疫组织化学分析组织阵列切片首先以二甲苯脱蜡,通过梯度酒精再水化,并浸没在含0.3%双氧水的甲醇中20分钟,以阻断内源性过氧化酶活性。为了获得抗原性,将切片浸没在500ml的ph6.0柠檬酸盐缓冲剂中,微波炉设定第六感加热20分钟。使用亲和素/生物素阻断试剂盒(vectorlabs)阻断内源性亲和素/生物素的结合。为了阻断一级抗体的非特异性结合,所有切片随后以100μl的1/5正常马血清(nhs)pbs溶液处理15分钟。以100μl一级抗体pbs稀释溶液22℃下处理测试切片1小时,或4℃下过夜处理。阳性对照组织包括以1/1000pbs(dako)稀释的β2-微球蛋白染色的结肠直肠癌组织的全切片,所有切片均用100μl生物素化羊抗鼠/兔免疫球蛋白(dako)的1:100nhs稀释液培养30分钟。在此用pbs洗涤切片,并以100μl预成型链霉亲和素-生物素/hrp复合物(dako)室温(rt)培养60分钟。随后,使用dab对表位表达进行可视化。最后,以苏木精(dako)对切片轻微对比染色,以酒精脱水,用二甲苯澄清(gentamedica,york,uk)并以联苯乙烯、塑化剂和二甲苯(dpx;bdh)装片。染色评估:为了允许玻片的永久保存,使用nanozoomer2.0玻片成像系统(hamamatsu,higashi-ku,japan)对其进行x20成像。使用nanozoomer数字病理性虚拟玻片查看器(nanozoomerdigitalpathologyvirtualslideviewer)中的图像分析组织上的标记表达(hamamatsu)。两名具有评分经验的研究人员在不知道临床资料的情况下同时对标记表达进行筛选。对于h评分(hscore),简要分析芯样,将阴性、弱、中、强芯样用作全组织微阵列(tma)的参考。对于染色强度,估计了阳性染色的肿瘤细胞百分比。然后,合并两种评分来形成h评分,其中h=染色细胞百分比x强度(范围=0-300)。使用nanozoomer查看器,测量肿瘤和基质的面积,以及每个区域中技术的阳性细胞数目。然后,计算每mm2中的阳性细胞值。2.9.1.结肠直肠肿瘤tma在胃癌tma上筛选抗血清的肿瘤结合。患者研究和设计:研究人群包括一系列在英国诺丁汉的大学医院选择性手术切除了组织学证实的散发性原发性结肠直肠癌的462位连贯病人(表1)。这些病人在1994年1月1日至2000年12月31日间接受治疗;该时间段允许对本研究的预后指标进行有意义的评估。在此时间段中治疗的病人认为是有资格参与研究。若肿瘤体积50%以上由粘液组成,则肿瘤被分类为粘液瘤。表1:结肠直肠tma病人群体(n=462)的临床病理变量临床病理学:仅相关病理材料不可用的病例被排除在本研究以外。跟进期自切除原始肿瘤时起算,在数据分析中,2003年12月31日的所有仍存活病例被截去,产生的所有病例跟进期中位值为37个月(范围:0-116),存活病例的跟进期中位值为75个月(范围:36-116)。使用前瞻性维护数据库来记录相关临床病理学数据,数据来自英国国家统计局(ukofficefornationalstatistics);超过99%的病例中存在可用数据。所采集的信息通过已故病人的病例笔记来独立验证。使用疾病特异性存活来作为主要研究终点;但是,也采集了各种其他相关临床和组织病理学参数数据,总结在表2.3中。对油阳性淋巴结的病患保留了由folfox组成的佐剂化疗,但手术和佐剂治疗由主治医生决定。诺丁汉地方研究与伦理委员会(nottinghamlocalresearchandethicscommittee)对本研究进行了前置伦理审查,并批准了本研究。阵列模块的构建包含了一系列选择性切除的结肠直肠瘤,并被认为是对英国的结肠直肠癌人群具有广泛代表性。266(58%)位病人为男性,196(42%)为女性。手术时的年龄中位值为72岁,与英国结肠直肠癌诊断年龄中位值70-74岁相一致。阵列中69(15%)个肿瘤为淋巴结转移(tnm)第1分期的肿瘤,174(38%)为第2分期,155(34%)为第3分期,54(11%)为第4分期;有3例为原位疾病。这些数字分别与诊断为第1-4分期的全国数据,11、35、26、29%分别一致[85]。大多数肿瘤(392,85%)为腺癌,且最频繁为中度组织学分级(353,77%)。注意到128(28%)个肿瘤有壁外血管入侵的组织学迹象,224(48%)个无血管入侵迹象,且在110(24%)个病例无该信息。对数据分析进行截尾时,228(49%)个病人已因病去世,64(14%)因其他原因去世,169(37%)例存活。该人群的5年疾病特异性中位生存期为58个月,与英国结肠直肠癌[86]的约45%的全国平均5年存活相仿。2.9.2.子宫癌tma在抗血清中筛选与卵巢癌tma的肿瘤结合。卵巢癌tma代表了362位患有原发性卵巢癌并于2000-2007年间在诺丁汉大学医院接受治疗的病人人群。使用国际妇产科联合会(internationalfederationofobstetricsandgynaecology)标准来对癌症进行分期。本研究中包含的所有病例均根据当前标准化疗方案进行治疗,其中65位病人(41.4%)采用单一药剂卡铂,89位病人(56.7%)进行基于铂类的联合化疗,另有3位病人拒绝化疗。若病人在一线铂类化疗治疗期间,病情发展,或在治疗后6个月内复发,则定义为对铂类具有抗性。所有病人均经过手术;超过44%的病例(n=69)被认定为是首次手术后亚最佳减积(剩余肿瘤1cm)。随后,对病人跟进,进行体检、计算机断层扫描和ca-125水平。这些病人的肿瘤的苏木精-伊红染色切片由一位不知晓临床数据和病理诊断的妇科病理学家查看。对每一肿瘤,还以sd查看其类型和分化。从病人的病历或通过医院的电子记录(notis)采集并记录每一病例相关的临床数据。该数据包括:病人诊断时的年龄、figo分期、手术细胞减灭程度和化疗的类型、持续时间和应答。记录所有病人的佐剂治疗的详情、疾病特异性生存期(dss)和总生存期。生存期的计算自手术日期起算,到2008年5月30日止,并截去所有剩余的生存者的数据。跟进期中位值为36个月。采集研究样本和相关数据的伦理批准由诺丁汉郡地方研究伦理委员会批准(nottinghamshirelocalresearchethicscommittee)。2.9.3.正常组织tma正常组织tma含有38个正常器官的59个代表性芯样。根据样本来源对每个芯样进行分类;来自非癌症患者的正常组织、来自癌症涉及其他器官的癌症患者的正常组织、癌症附近的正常组织。组织类型和分类详情如表2所示。表2:正常组织tma和组织类型详情实施例1:野生型小鼠对自体和外源表位的cd4应答对肿瘤相关表位的t细胞应答通常微弱或不存在,这是由于胸腺中的耐受性和t细胞删减。尽管如此,我们仍筛选了各种自体和外源cd4表位的刺激辅助应答的能力。由于此前的研究已表明,抗体dna结构产生的免疫应答最强[84],因此在各结构中并入了各种cd4外源和自体表位,并在野生型小鼠中进行筛选。表3列出了所有表位及其适当的小鼠同源物的序列。表3:cd4表位精氨酸残基以下划线表示非同源残基以斜体表示图5展示了对大多数外源cd4表位的良好cd4应答,但对自体表位则不然。乙肝核蛋白128-140、cd4i-ab辅助表位在c57b1小鼠的elispot检测中表现出50-1,000/百万脾细胞(平均值为382/百万脾细胞)的应答。小鼠睾丸癌表位nyeso-1和ssx2为外源表位,dr1表位nyeso-1(87-111)和ssx2(34-48)在hla-dr1转基因小鼠中刺激了良好应答:nyeso-1(87-111)为250-900/百万脾细胞(平均值567/百万脾细胞),ssx2(34-48)为500-1200/百万脾细胞(平均值765/百万脾细胞)。hla-dr1突变tpi表位与野生型的不同仅在于一个氨基酸,表现出300-1300/百万脾细胞的应答。人gp100hla-dr4酪氨酸酶表位44-59与同源小鼠表位相比具有1个氨基酸改变,并具有263-1521/百万脾细胞(中位值为745/百万脾细胞)的应答。相反,同源鼠自体表位未能在hla-dr4转基因小鼠中刺激应答。人hla-dr4酪氨酸酶表位与同源小鼠表位相比具有6个氨基酸改变,并具有405-832/百万脾细胞(中位值为555/百万脾细胞)的应答。人和小鼠之间的hla-dr1mmp7247-262表位具有4个氨基酸变化,其中一个据预测位于核心mhc结合/tcr识别区域(表3)。该表位未能刺激hla-dr1转基因小鼠中高于背景水平的应答。hla-dr4波形蛋白415-433表位在人与小树之间有2个氨基酸差异,但据预测并不位于核心mhc结合/tcr识别区域(表3)。它未能在野生型hla-dr4转基因小鼠中刺激应答。相反,波形蛋白28-49在人和小鼠中为同源,且是真实自体表位,能在hla-dr4转基因小鼠中刺激200-500/百万脾细胞(平均值390/百万脾细胞)的应答。该应答非常吸引人,并使得我们试图解释是否存在对该表位的删减/耐受。实施例2:dna免疫接种导致对瓜氨酸化的vim28、vim415、mmp7、ny-eso-1的应答。ra病人表现出对瓜氨酸化波形蛋白表位的t细胞应答。由于apcs能够结构性地瓜氨酸化表位,因此可能抗体dna结构正在经历瓜氨酸化,并刺激应答。因此,以编码自体vim28表位的抗体dna结构对hla-dr4转基因小鼠免疫接种。在所述小鼠中刺激所得的t细胞进行对瓜氨酸化和未瓜氨酸化vim28多肽的ifnγ、il-17、il-2的应答的体外筛选。图6显示,虽然在elispot实验中,小鼠对野生型多肽做出了应答,产生了全部三种细胞因子(平均值:ifnγ400/百万脾细胞、il-17120/百万脾细胞和il-2150/百万脾细胞),对瓜氨酸化多肽的ifnγ和il-17应答显著较高(平均值:ifnγ1250/百万脾细胞;p=0.0046,il-17250/百万脾细胞;p=0.0392),但il-2则不然(250/百万脾细胞)。为了确定对vim415表位抗体是否产生相似应答,使用dna结构来对小鼠免疫接种(图7)。与vim28-49相反,且与我们的最初观察相一致,对野生型vim415-433无应答,但对瓜氨酸化多肽强应答,且对il-17的应答与对ifnγ的应答同样强(平均:ifnγ400/百万脾细胞,p=0.0067;il-17350/百万脾细胞,p=0.002;和il-2250/百万脾细胞,p=0.0056)。这证实了抗体dna翻译后被瓜氨酸化。相反,mmp7247-262并入dna疫苗(图8)后,未发现对野生型或瓜氨酸化多肽的ifnγ应答,但却发现了对两种野生型(平均:312/百万脾细胞;p=0.0061)和瓜氨酸化(平均:309/百万脾细胞;p=0.0267)多肽的显著il-17应答。以b16hhdiinyeso-1肿瘤细胞注射小鼠后,然后以附加或不附加homspera佐剂的含nyeso-1119抗体-dna疫苗(图9)免疫接种,仅在发现了对瓜氨酸化多肽的ifnγ应答,而野生型多肽中则不然,且该应答伴随着抗肿瘤应答。由于抗原呈递细胞能够结构性地瓜氨酸化表位,我们想要发现,瓜氨酸化自体表位特异性应答能够由以dna疫苗形式递送的全长抗原来进行诱导。因此,以编码全鼠波形蛋白抗原的dna结构对hla-dr4转基因小鼠进行免疫接种。在这些小鼠中刺激得到的t细胞中,筛选对瓜氨酸化和未瓜氨酸化vim415-433和vim28-49多肽的ifnγ应答。图10显示,以dna结构免疫接种的小鼠对两种瓜氨酸化多肽均作出ifnγ应答,但对野生型则不然。这证实了dna翻译后被瓜氨酸化。这些结果暗示,来自抗体-dna结构的表位被瓜氨酸化,且识别所述修饰后多肽的t细胞并未耗尽/失去对抗原的反应性。实施例3.多肽免疫接种引起对瓜氨酸化vim28、vim415、vim65的应答。为了确定这是否仅限于dna疫苗,或瓜氨酸化多肽是否还能刺激该细胞库,以野生型和瓜氨酸化vim28-49和瓜氨酸化vim415-433多肽联合cpg和mpla佐剂,对小鼠免疫接种。图11展示了瓜氨酸化vim28-49刺激了对瓜氨酸化多肽的强烈ifnγ应答(平均600/百万脾细胞;p=0.0037)和对野生型多肽的弱应答(平均250/百万脾细胞;p=0.0175)。野生型多肽刺激的对未瓜氨酸化多肽(平均700/百万脾细胞;p=0.003)和瓜氨酸化多肽(平均1100/百万脾细胞;p=0.0182)的ifnγ应答相似,以及弱il-17和il-2细胞。未观察到显著il-10应答。对ifnγ和il-17的应答亲抗原性为10-6m。对小鼠中的vim28-49的应答是对自体的应答,因为氨基酸序列在小鼠和人中是一样的,暗示着对该表位的t细胞库未被删除。对瓜氨酸化vim28-49的应答是对修饰自体的应答,但小鼠也可以识别野生型多肽。此前的研究表明,在位点36和45处瓜氨酸化的多肽30-40能够在hla-dr4转基因小鼠中刺激t细胞应答。我们注意到,位点28处还存在又一个精氨酸,因此,我们延长了该多肽,以提供28-49和瓜氨酸化位点28、36、45(图11c)。三瓜氨酸化vim28-49多肽比位点36、45处瓜氨酸化的vim28-49多肽表现出显著更强的应答(分别有p=0.02和p=0.0007)。仅在位点28瓜氨酸化的vim28-49,对三瓜氨酸化多肽和vim45瓜氨酸化多肽均产生应答。vim28和vim36瓜氨酸化多肽也对该三瓜氨酸化多肽做出应答。该数据表明,瓜氨酸位点对该序列产生的免疫应答程度有影响,并暗示着位点28最为重要。但是,所述三瓜氨酸化多肽诱导了对其他瓜氨酸化版本交叉反应性更高的应答。与野生型版本相比,三瓜氨酸化vim28-49多肽对hla-dr0401表现出的结合性更好,这一点可从hla-dr0401结合实验中其与ha306-318标记蛋白竞合更好而看出(图12)。图13表明,瓜氨酸化vim415刺激了对瓜氨酸化多肽的强ifnγ应答(平均1000/百万脾细胞;p=<0.0001),而对野生型多肽无应答。它还刺激了对瓜氨酸化多肽的强烈il-17应答(平均530/百万脾细胞;p=<0.0001)和对野生型多肽的弱应答(平均140/百万脾细胞;p=0.04)。ifnγ和il-17应答的亲抗原性均为10-6m。对瓜氨酸化表位的il-2应答更具有可变性(平均350/百万脾细胞;p=0.046)但对野生型多肽无应答。野生型vim415-433多肽刺激了对瓜氨酸化多肽的弱il-2应答。未观察到显著il-10应答。人vim415-433表位与同源小鼠表位有2个氨基酸不同,该2个氨基酸未预测处于核心mhc结合/tcr识别区域。为了测试这一假设,以瓜氨酸化人vim415多肽对小鼠免疫接种,然后筛选瓜氨酸化小鼠vim415。t细胞表现出对两种多肽的相等应答(图14)。在间接体内酶联免疫斑点实验之前清空cd4细胞,或向酶联免疫斑点实验培养物中添加mhcii类阻断抗体,发现瓜氨酸化vim415-433和vim28-49应答由cd4介导(图15)。使用瓜氨酸化vim28和vim415对c57bl小鼠和hhd1/dr1小鼠免疫接种,但其均未产生应答,暗示着该表位在i-ab或hla-dr0401上均不存在。图16a显示,在hla-dr4小鼠中,瓜氨酸化波形蛋白65多肽刺激了对瓜氨酸化多肽的ifnγ应答(平均值550/百万脾细胞;p=0.0046),但对野生型多肽无应答。它还刺激了il-17应答(平均值550/百万脾细胞),但对野生型多肽无应答。野生型vim65-77多肽刺激了对野生型和瓜氨酸化多肽的弱ifnγ和il-17应答。还测试了hla-a2/dr1小鼠中的vim65-77多肽,也表现出对瓜氨酸化多肽的高频率ifnγ应答,还表现出与野生型多肽的一些交叉反应性(图16b)。还观察到对瓜氨酸化多肽的低频率il-10应答。对nhcii类的阻断未消除瓜氨酸化多肽特异性应答,由此表明vim65-77特异性应答是mhci性限制性(图16c)。这进一步被胞内细胞因子染色所证实,该染色表明,对vim65-77瓜氨酸化多肽应答而产生ifnγ和tnfa的细胞为cd8阳性细胞(图16d)。瓜氨酸化vim65-77特异性应答还表现出肽脉冲hla-a2阳性靶细胞(图16e)的细胞毒性(图16e),表明了hla-a2限制性。使用2个高预测hla-a2结合性的9聚体多肽来绘制vim65-77序列中的最小hla-a2限制性表位,揭示了最佳序列位于vim68-76区域(图16f)。对该瓜氨酸化9聚多肽的特异性应答未与野生型交叉反应。分析对肿瘤靶细胞的应答,发现相对于hla错配b16细胞,瓜氨酸化65-77多肽诱导的间接体内应答对转基因hla-a2改造el4细胞系(el4hhd)具有良好识别(图16g)。实施例4:测定对自体表位的cd4应答是否为初次应答、记忆应答或treg应答小鼠对人瓜氨酸化vim415的单免疫接种产生了强力ifnγ和il-17应答,暗示着其促进了记忆应答或treg应答。为了测定这是否是转化为ifnγ/il-17应答的天然treg应答,用抗cd25mab清空小鼠的天然tregs,并以瓜氨酸化vim415对小鼠进行免疫接种。清空天然treg对该应答的频率或亲抗原性未产生影响,暗示着天然tregs并非应答细胞群(图17)。为了确定该应答是否也可由其他佐剂来诱导,用以明矾、弗氏不完全佐剂(ifa)、gmcsf、mpla、tmx201、mpla/tmx201或cpg/mpla为佐剂的瓜氨酸化vim415-433和瓜氨酸化28-49,对小鼠免疫接种,并筛选ifnγ、il-17或il-10生成(图18)。cpg/mpla、gmcsf和tmx201诱导了强烈的瓜氨酸化vim415-433和瓜氨酸化28-49表位的ifnγ/il-17应答,但是,当以明胶或ifa为佐剂进行多肽给药时,未产生应答。多肽和ifa的免疫接种诱导了对瓜氨酸化多肽的高频率il-10应答。抗ctla-4单克隆抗体可以阻断ctla-4与其同源受体cd80/86的相互作用,由此防止该配体诱导的t细胞抑制。在存在抗ctla-4mab的情况下,以瓜氨酸化vim415免疫接种的小鼠,t细胞应答的亲抗原性从10-6m显著提高为10-8m(图19)。实施例5:癌症患者对自体多肽的应答对9位黑色素瘤病人筛选其对一系列自体多肽的应答(图20)。8个病人中有5人在第4(1)、7(1)或大于11(3)天表现出对瓜氨酸化vim415的应答。11个病人中有6人在>第10天对未修饰vim415做出应答。所述病人中仅有4人对修饰后多肽和未修饰多肽均产生应答。8个病人中有2人在第11天对vim28产生应答,而5人对瓜氨酸化vim28应答。对未修饰多肽产生应答的2人也能识别修饰后的多肽。8个病人中有4人对vim65产生应答,且8人中有3人对瓜氨酸化vim65产生应答。这些病人中仅2人对修饰后多肽和未修饰多肽均产生应答。8个病人还对瓜氨酸化nyeso-1119-143产生应答;所述应答中有6个在第4-7天达到峰值,暗示强烈应答或记忆应答。8个病人还对未修饰nyeso-1119-143产生应答,这些病人具有一系列hla类型(表4)。仅有一个是hla-a2/dr4,这暗示着其他hla单倍型可以对这些多肽产生应答。表4:癌症病人的单倍型pt019a2a3b7b55dr7dr16pt020a2b27b40dr3dr13pt021a3a25b44b35dr1dr13pt023a2a3b7b35dr1dr15pt028a2a24b7b35dr1dr13pt029a2a25b15dr15pt032a1a11b51b18dr15pt033b7dr11dr15pt034a11b7b35dr1dr4pt035a29a30b50dr1dr7表6:pad酶、波形蛋白和瓜氨酸的表白瓜氨酸化由pad酶执行,尤其是pad2和pad4酶。这些酶要求高浓度钙,且通常在死亡细胞或死亡中的细胞中激活。因此,似乎不可能在健康肿瘤细胞中表达瓜氨酸化蛋白。因此,对结肠直肠瘤和子宫瘤以及正常组织进行波形蛋白、pad2和pad4酶的瓜氨酸化和表达的染色。正常组织正常组织的波形蛋白、pad2、pad4和瓜氨酸的表达如表5所示。间充质细胞,例如结缔组织细胞、血细胞和神经元细胞,都表达波形蛋白作为细胞骨骼蛋白。脾脏、甲状腺、睾丸、宫颈、卵巢、扁桃体、子宫、肺、胸腺和乳腺中的大多数细胞均可对波形蛋白强烈染色。胎盘、直肠、结肠、胰腺和十二指肠骨骼肌和平滑肌、胆囊、食道、肾脏、肝脏、膀胱、回肠、空肠、胃中,小于50%的细胞为弱染色。皮肤、脂肪组织、骨骼肌、直肠、大脑、小脑、隔膜或心脏中未观察到染色。大多数肝脏细胞可以抗pad2mab强烈染色。平滑肌细胞和大脑细胞大多数为弱染色。小脑、胰腺和睾丸的过半细胞以pad2强烈染色。胆囊、回肠、空肠、胃和结肠中低于50%细胞对pad2强烈染色。食道、直肠、骨骼肌、膀胱、胸腺、肾脏、胎盘、心脏、隔膜、十二指肠、甲状腺和肺部细胞低于50%为弱染色。皮肤、脂肪组织、脾脏、扁桃体、胸腺、卵巢和子宫细胞无染色。表5波形蛋白、pad2、pad4和瓜氨酸化蛋白在正常组织中的表达肝脏和小脑细胞大多数对抗pad4mab强烈染色。大脑和心脏细胞大多数弱染色。结肠和胃低于50%的细胞对pad4强烈染色,而回肠、隔膜、十二指肠、甲状腺、睾丸、乳腺、胆囊、食道低于50%的细胞弱染色。皮肤、骨骼肌和平滑肌、膀胱、胰脏、空肠、肾脏、肝脏、宫颈、肺部、卵巢、扁桃体、脾脏、子宫、直肠、脂肪组织、胸腺无染色。小脑和肝脏对抗瓜氨酸mab强烈染色。皮肤、心脏、肾脏肺部、胰脏和大脑细胞大多数弱染色。超过50%十二指肠强染色,且超过50%的胸腺细胞和睾丸细胞弱染色。胸腺、结肠和空肠中低于50%细胞弱染色。胸腺中低于25%的细胞强烈染色,且食道、直肠、胆囊、骨骼肌、膀胱、回肠、胸腺、扁桃体、隔膜和子宫中超过25%弱染色。脾脏、皮肤、胃、卵巢、扁桃体、脂肪组织、胎盘和宫颈对瓜氨酸无染色。卵巢瘤卵巢瘤起源于间叶细胞,因此预期可能表达波形蛋白。pad4在正常卵巢中弱表达。219个卵巢瘤被波形蛋白特异性mab染色。仅9/219(4%)肿瘤无染色,还有16/219(7%)弱染色,而194/219(89%)强染色。卡普兰迈耶存活分析显示,波形蛋白表达和存活之间无相关性。波形蛋白表达与应激相关蛋白ulbp1(p=0.017)、pad4(p=0.018)和cea-cam4(p=0.033)之间有弱相关性。219个卵巢瘤以pad4特异性mab染色(图23)。肿瘤中仅9/219(4%)未染色,126/219(58%)弱染色,而84/219(38%)强染色。卡普兰迈耶存活分析表明,pad表达和存活之间无相关性。pad4表达与应激相关蛋白之间raet1e(p=0.036)和ulbp1之间(p=0.016)之间存在弱相关性,而与波形蛋白表达(p=0.001)之间和lewisy(p=0.006)之间存在强相关性。虽然肿瘤表达pad4,但其应仅在死亡中的细胞中激活。为了评估其真实性,卵巢肿瘤对抗瓜氨酸多肽特异性mab染色。仅7/228(3%)肿瘤未染色,34/228(15%)为弱染色,而187/228(82%)为强染色。但是,并非肿瘤中的所有细胞均被染色。在69/228(30%)肿瘤中,低于25%的细胞染色。83/228(36%)的肿瘤中,25-50%的细胞染色,且如前所述,这些细胞主要是来源于基质的细胞。53/228(23%)的肿瘤中,50-75%的细胞染色,包括一些上皮细胞;16/228(7%)肿瘤中,大于75%的细胞染色。卡普兰迈耶存活分析表明,瓜氨酸表达和存活之间不存在相关性。瓜氨酸表达强度和cea-cam5(p=0.037)之间、bcl2(p=0.011)之间、lewisy(p=0.053)之间存在相关性。表达瓜氨酸的细胞百分比和分级(p=0.034)之间、bcl2(p=0.035)之间,cd59(p=0.049)之间和ulbp1(p=0.044)之间存在相关性。360个卵巢瘤对pad2染色。9%因为缺少足量组织芯样或芯样中无可评测的肿瘤细胞(即全为基质)而无法进行评测。329个以pad2特异性mab染色的可评测的卵巢瘤中,所有肿瘤均表达pad2。其中277/329(84%)弱染色,52/329(16%)强染色。卡普兰迈耶分析(图21a)表明,pad2表达和存活之间存在相关性,pad2高表达具有保护性(p=0.033)。pad2表达与mhc表达之间(p=0.038)和hmgb1表达之间(p=0.008)存在相关性。多变量分析表明,pad2是独立的预后因素(p=0.002)。360个卵巢瘤对hmgb1染色。10%因为缺少足量组织芯样或芯样中无可评测的肿瘤细胞(即全为基质)而无法进行评测。316个以hmgb-1特异性mab染色的可评测的卵巢瘤中,仅23/360(7%)肿瘤未染色。42/316(13%)弱染色,52/329(87%)强染色。卡普兰迈耶分析(图21b)表明,hmgb1表达和存活之间存在相关性,hmgb1低表达具有保护性(p=0.002)。hmgb1表达与波形蛋白表达之间(p=0.034)存在弱相关性。多变量分析表明,hmgb1是独立的预后因素(p=0.02)。肿瘤分期、肿瘤类型和对化疗的应答也与病人存活相关。在多变量模型中,tnm分期(p=<0.0001)、肿瘤类型(p=<0.031)、对化疗的应答(p=<0.0001)和hmgb1表达(p=0.002)是独立的病人存活指标。若将高和低pad2细胞表达与高和低hmgb1表达相比(图21c),表现出高hmgb1和低pad2表达的病人中,310人中有219人(70%)具有最差中位生存期50个月,而具有低hmgb1和高pad2的病人则表现出较好存活,310人中有41人(13%)的中位生存期为101个月。结肠直肠瘤结肠直肠瘤是上皮起源,且预期并不表达波形蛋白,除非经历上皮到间叶细胞转化。正常结肠中可见pad2和pad4表达。取282个结肠直肠瘤以波形蛋白特异性mab染色。仅25/282(9%)肿瘤未染色,4/282(1%)弱染色,53/282(90%)强染色。但是,一个肿瘤中并非所有细胞均染色。114/282(40%)个肿瘤中,低于25%细胞染色,且全部为基质细胞。68/282(24%)个肿瘤中,25-50%细胞染色,且也主要是基质起源。42/282(15%)个肿瘤中,50-75%细胞染色,包括一些上皮细胞;33/282(12%)个肿瘤中,大于75%的细胞染色。卡普兰迈耶存活分析表明,波形蛋白强度或染色细胞百分比和存活之间无相关性。波形蛋白表达和trailr2(p=0.003)、肿瘤中的il-17(p=0.021)、muc1(p=0.001)、pad2(p=0.025)和pad4(p=<0.0001)相关。取296个结肠直肠瘤以pad2特异性mab染色(图26)。仅45/296(15%)肿瘤未染色,60/296(20%)弱染色,191/296(65%)强染色。但是,一个肿瘤中并非所有细胞军染色。99/296(33%)个肿瘤中,低于25%细胞染色,且全部为基质细胞。82/296(28%)个肿瘤中,25-50%细胞染色,且也主要是基质起源。55/296(19%)个肿瘤中,50-75%细胞染色;15/296(5%)个肿瘤中,大于75%的细胞染色。卡普兰迈耶存活分析表明,pad2强度或染色细胞百分比和存活之间无相关性。pad2表达和cd59(p=0.006)、β-连环蛋白(p=0.005)、cd8t细胞数量(p=0.009)、muc1(p=0.012)、波形蛋白(p=0.038)和pad4(p=0.000)相关。取291个结肠直肠瘤以pad4特异性mab染色(图26)。仅18/291(6%)肿瘤未染色,158/291(54%)弱染色,115/291(40%)强染色。但是,一个肿瘤中并非所有细胞军染色。65/291(22%)个肿瘤中,低于25%细胞染色,且全部为基质细胞。68/291(23%)个肿瘤中,25-50%细胞染色,且也主要是基质起源。98/291(34%)个肿瘤中,50-75%细胞染色;42/291(14%)个肿瘤中,大于75%的细胞染色。卡普兰迈耶存活分析表明,pad4强度和存活之间无相关性(图21d;表6;p=0.032)。表6:表达pad4的结肠直肠癌病人的生存期平均值a若截尾,则估计值被限制为最大生存期。肿瘤分期和血管入侵也与病人存活相关。在多变量模型tnm分期(p=<0.0001),中,血管入侵(p=<0.0001)和pad4表达(p=0.017)均为病人存活的独立指标。pad4表达和bcl2(p=0.01)、β-catenin(p=0.001)、cd8t细胞数量(p=0.006)、muc1(p=0.000)、cea.cam5(p=0.000)、cd59(p=0.038)波形蛋白(p=0.000)和pad2(p=0.000)之间也存在相关性。虽然肿瘤表达pad4,但这应该仅在死亡中的细胞中表达。为了评估其真实性,结肠直肠肿瘤对抗瓜氨酸多肽特异性mab染色。所有被染色的肿瘤中,41/316(13%)为弱染色,而275/316(87%)为强染色。卡普兰迈耶存活分析表明,瓜氨酸表达和存活之间存在弱相关性(p=0.078)。瓜氨酸表达和放射治疗之间存在相关性(p>0.001)。实施例7:对瓜氨酸化多肽的抗肿瘤应答小鼠和人均对瓜氨酸化自体多肽和dna疫苗表现出应答。但是,若肿瘤中的这些表位并未瓜氨酸化,则t细胞无抗肿瘤活性。由于人肿瘤表达波形蛋白、pad2/4和瓜氨酸,在鼠模型中评估了瓜氨酸化波形蛋白的抗肿瘤应答。对以瓜氨酸化波形蛋白415-433和28-49多肽免疫接种的小鼠,评价其脾细胞对b16肿瘤细胞的体外应答能力。图22a展示了血清饥饿诱导自体吞噬表达hla-dr4(b16dr4)的b16肿瘤细胞特异性的ifnγ释放,与未处理细胞或hla-错配细胞相比较,以说明对肿瘤细胞的识别(p<0.0001)。当以自体吞噬抑制剂3-甲基腺嘌呤(3-ma)(p<0.0001)或pad抑制剂ci脒(p=0.0012)的存在下处理细胞时,对诱导自体吞噬b16dr4细胞的识别显著降低。因此,这表明该识别具有自体吞噬和瓜氨酸化依赖性。以瓜氨酸化vim28-49或vim415-433多肽或两者免疫接种的小鼠的脾细胞显示,当以vim415-433(p<0.0001)和vim28-49(p<0.0001)瓜氨酸化多肽,而非野生型进行刺激时,发生颗粒酶b(一种细胞毒性的标记)释放(图22b-d)。颗粒酶b还在对血清饥饿b16dr4肿瘤靶细胞的应答中释放,暗示着呈递瓜氨酸化表位的肿瘤靶细胞的细胞毒性(p=0.014)(图22e)。测定以瓜氨酸化vim415或瓜氨酸化vim28免疫接种的小鼠体外杀灭以人dr4转染的t2肿瘤细胞的能力。图23a显示,两种瓜氨酸化多肽均能诱导cd细胞杀灭转染靶细胞,无论是否曾以适当多肽进行脉冲冲击。由于t2细胞表达波形蛋白,这暗示着所述多肽由t2细胞内源性呈递。相反地,未杀灭同样表达波形蛋白但不表达pad酶的正常脾细胞。向hla/dr4转基因小鼠置入以dr4转染的b16肿瘤。小鼠以vim415-433瓜氨酸化多肽或编码vim415-433序列的dna疫苗免疫接种,并监控肿瘤生长。以vim415-433瓜氨酸化多肽或编码于dna疫苗中的vim415-433免疫接种的小鼠,刺激了强烈的抗肿瘤应答(图24a)。未免疫接种小鼠中,肿瘤迅速生长,且所有小鼠在第23天时都不得不处死。相反,以vim415瓜氨酸化多肽免疫的小鼠有50%在第35天无肿瘤,且30%肿瘤治愈。以vim28-49瓜氨酸化多肽或以编码vim28-49序列的dna疫苗接种的小鼠,相对于未免疫接种对照或以vim28-49野生型多肽而言,表现出显著提高的生存期(图24b)。以vim28-49野生型多肽免疫接种的小鼠表现出几乎达到显著的抗肿瘤应答。以vim415-433和28-49瓜氨酸化多肽联合免疫接种,相对于对照得到更好的肿瘤保护和总生存期(p<0.0001)(图24c)。这些研究表明,肿瘤表达瓜氨酸化波形蛋白,而后者是细胞毒性cd4杀手t细胞的标靶。为了展示这些抗肿瘤应答由cd4t细胞调控,以抗cd4抗体处理小鼠,以体内清空cd4细胞。免疫接种联合cd4t细胞清空,完全终止了由瓜氨酸化vim415(p=0.0005)和瓜氨酸化vim28多肽(p=0.0001)所调控的抗肿瘤应答(图24d和e)。vim415-433和vim28-49瓜氨酸化多肽均诱导了高频率ifnγ应答。体内阻断ifnγ终止了vim415-433和vim28-49瓜氨酸化多肽特异性抗肿瘤应答(图25a和b)。瓜氨酸化vim415-433特异性应答还表现出il-17应答。对这些进行体内阻断,对体内抗肿瘤作用的影响为低显著性(图25c)。对il-17的阻断还对体内瓜氨酸化vim28-49特异性抗肿瘤应答具有低显著性的影响作用(图25d)。为了确定vim415-433和vim28-49瓜氨酸化特异性应答的直接肿瘤识别的重要性,以缺失hla-dr0401表达的b16肿瘤处理小鼠,随后以vim415-433或vim28-49瓜氨酸化多肽免疫接种。以vim28-49瓜氨酸化多肽免疫接种的小鼠,与对照相比,表现出延迟肿瘤生长和提高的生存期(图26a),暗示着对肿瘤细胞上的hla/dr4和同源多肽的直接识别对抗肿瘤应答并非必需条件,但对表达肿瘤环境中的同源多肽和hla/dr4的抗原呈递细胞产生应答而从旁释放ifnγ,则是该抗肿瘤应答的成因。相反,以vim415-433瓜氨酸化多肽免疫接种的小鼠,在此模型中未显示任何肿瘤应答,暗示着对肿瘤细胞上的hla/dr4和同源多肽的直接识别对抗肿瘤应答是对该表位进行抗肿瘤应答的必需条件。为了确认vim415-433特异性应答依赖于直接肿瘤识别,以表达hla-dr0401的b16肿瘤处理后的小鼠,以vim415-433瓜氨酸化多肽联合抗hla-dr阻断抗体进行免疫接种。对hla-dr的阻断阻止了抗肿瘤应答的发生(图26b)。实施例8:不同物种之间的波形蛋白同源性鸡、鼠、狗、羊、牛、马、猪和人之间的波形蛋白高度保守(图27)。由于疫苗诱导人、鼠的t细胞应答,以及鼠的抗肿瘤应答,可以假定,类似应答也可见于其他物种中。实施例9:通过其他hla单倍型限制的波形蛋白应答hla-dr4转基因小鼠对人vim415瓜氨酸化多肽的单免疫接种产生了有力的ifnγ和il-17应答。为了确定这一或其他瓜氨酸化波形蛋白表位可以在其他单倍型中诱导免疫应答,在hla-dr1和c57/bl小鼠中,对涵盖波形蛋白全长并包含所有被瓜氨酸残基取代的精氨酸的一系列20聚体多肽(表8)进行ifnγ/il-17应答筛选(图28)。以3种瓜氨酸化波形蛋白多肽和cpg和mpla佐剂的组合对小鼠免疫接种,然后对所述组合中的每种多肽单独进行ifnγ和il-17应答的筛选。图28a显示,在hla-dr1转基因小鼠中,瓜氨酸化波形蛋白多肽9、10、14、15和16展示了显著的ifnγ应答(200-350斑点/百万脾细胞p<0.02),且多肽10展示了il-17应答(350斑点/百万脾细胞)。图28b显示,在c57bl/6小鼠中,瓜氨酸化波形蛋白多肽16、17、18、19和20展示了显著ifnγ应答(300-500斑点/百万脾细胞p<0.02),且多肽19和20展示了il-17应答(~400斑点/百万脾细胞p<0.05)。表7展示了所述多肽的序列,瓜氨酸化氨基酸的位点和在波形蛋白中的位点。图28c展示了以波形蛋白14瓜氨酸化多肽进行免疫接种的hla-a2/dr1转基因小鼠中的应答。表7:瓜氨酸化波形蛋白ifnγ/il-17应答瓜氨酸取代的精氨酸残基以下划线表示实施例10如何筛选瓜氨酸化t细胞表位文献中所述的任何瓜氨酸化蛋白都可能是潜在的t细胞目标。但是,首先它必须具有在mhci类和/或ii类mhc抗原上呈递的能力,且它必须被t细胞受体所识别。抗原呈递细胞进行结构性自体吞噬,且其双膜自噬体中能够累积足以激活pad酶(呈递于mhc抗原上的瓜氨酸表位)的胞内ca2+。最后,要成为抗肿瘤标靶,肿瘤细胞必须还在自噬体中诱导瓜氨酸化,并将相同修饰后抗体呈递在mhc抗原上。因此,要鉴别还能刺激抗肿瘤免疫反应的瓜氨酸化表位,需要筛选诱导t细胞应答和肿瘤识别的靶蛋白。a)瓜氨酸化多肽诱导下的人外周血t细胞体外增殖人外周血可以如实施例5所述进行体外刺激。可在跨全蛋白的瓜氨酸化20聚体多肽中筛选t细胞增殖。cd4和cd8t细胞分类,能鉴别cd4和cd8表位,且可以通过hla分型供体来鉴别hla限制性。b)以跨全靶蛋白的20聚体瓜氨酸化多肽体外或体内刺激来自常规或hla转基因小鼠的细胞为了测定瓜氨酸化细胞角蛋白-8表位能否诱导免疫应答,在涵盖细胞角蛋白-8全长且包含预期芯样结合区域中被瓜氨酸残基取代的精氨酸的一系列20聚体多肽(表8)中,筛选hla-dr1和c57bi小鼠中的ifnγ/il-17应答(图29)。以3个瓜氨酸化细胞角蛋白8多肽与cpg和mpla佐剂的组合对小鼠免疫接种,然后筛选对该组合中每个多肽单独的ifnγ和il-17应答。图28a展示了hla-dr1转基因小鼠中表现出显著ifnγ应答(200-300斑点/百万脾细胞p<0.02)的瓜氨酸化细胞角蛋白8多肽1、2、3、13、16和17,以及显示出il-17应答的多肽1、2、3、13和14(250-350斑点/百万脾细胞;p<0.02)。图29b显示,细胞角蛋白8多肽未在c57b1小鼠中刺激应答。表8瓜氨酸化细胞角蛋白的ifnγ/il-17应答瓜氨酸取代的精氨酸残基以下划线表示c)筛选已知瓜氨酸化表位的t细胞应答ing4蛋白在其nls区域的位点133处被pad4酶瓜氨酸化。这防止了它与p53结合,该结合是p53激活所必需的。瓜氨酸化ing4蛋白迅速降解,暗示着它可能在mhcii类上大量表达。ing4多肽aqkklklvrtspeygmp未能在hla-dr1转基因小鼠中刺激任何免疫应答,但aqkklklvcittspeygmp刺激了对瓜氨酸化多肽的ifnγ/il-17应答(平均200斑点/百万脾细胞),但对野生型多肽无应答。对瓜氨酸化多肽的应答被mhcii类阻断mab所阻断(图31a)。这是能够刺激瓜氨酸化/肿瘤特异性cd4应答的蛋白的又一示例。d)筛选任意蛋白的瓜氨酸化t细胞表位如申请人在实施例2中所示,以编码全抗原的dna进行免疫接种,得到对瓜氨酸化多肽的应答。在此,我们展示了,若以编码全抗原的dna进行免疫接种,并对所有可能的瓜氨酸化20聚体多肽进行筛选,仅对hla分子呈递的瓜氨酸化表位的t细胞应答刺激了免疫应答。瓜氨酸化多肽组如表7所示。图33a展示了hla-dr4转基因小鼠以波形蛋白dna免疫接种后产生的应答,图33b展示了hla-dr1转基因小鼠中的应答。hla-dr4转基因小鼠展现出对瓜氨酸化波形蛋白28-49和415-433的高频率特异性应答,以及对波形蛋白19-33、26-44和36-54多肽的低频率应答。hla-a2/dr1转基因小鼠展现出对瓜氨酸化波形蛋白65-77多肽的高频率特异性应答。这证明,编码全抗原的dna可用于诱导瓜氨酸化t细胞应答,且这是一种用于筛选更多瓜氨酸化t细胞表位的极好方法。类似地,可以通过在存在高水平钙的情况下,将蛋白与pad酶共培养,以进行间接体内瓜氨酸化。这些蛋白质可用于对小鼠免疫接种,随后针对所有可能的瓜氨酸化20聚体多肽进行筛选。e)筛选根据mhc结合评分和核心区域中的精氨酸残基选择的预测多肽。本文展示了,可以在已知瓜氨酸化表位中诱导应答,但在此,还展示了可根据预测mhc结合评分和存在于核心mhc结合区域中的精氨酸残基,选择多肽表位。在此实施例中,多肽以syfpeithi预测算法(www.syfpeithi.de),选自对hla-dr4具有高结合预期的bip、hsp90、cxcl10、cxcl12和ing4,然后进一步通过存在于核心mhc结合区域中的精氨酸残基(以iedb预测算法(www.iedb.org)确定)进行限制(表9)。对含有所有转化为瓜氨酸的精氨酸的多肽进行测试。表9:预测hla-dr4多肽以瓜氨酸化多肽对hla-dr4转基因小鼠免疫接种至多3次,然后以ifng酶联免疫斑点法对相关瓜氨酸化多肽和未修饰多肽的应答进行间接体内分析。图34展示了对瓜氨酸化bip39-53、bip172-186、hsp90346-360、hsp90456-477和ing444-58的显著应答,相对于未修饰多肽或背景对照。这提供了又一种选择瓜氨酸化t细胞表位的方法。为了证明所述t细胞识别肿瘤,可以对其进行识别肿瘤靶细胞的筛选,除酸以促进mhc循环,采用无血清以诱导自体吞噬(图22a和22e)。瓜氨酸化和自体吞噬的作用可以使用pad和自体吞噬抑制剂来加以证实(图22a)。体内抗肿瘤应答可以通过触发肿瘤、以瓜氨酸多肽免疫接种、监控肿瘤生长来测量,如图24-26所示。实施例11此前的研究表明,可能对原生cd4细胞受刺激后根据其细胞因子环境分化为不同辅助表型进行测定。相反地,我们在图15中首次公布,某些表位可以无视细胞因子环境地测定t辅助细胞分化。瓜氨酸化vim415刺激了th1/il-17表现型,即使是在th2佐剂完全弗氏佐剂的存在下。这暗示着cd4t细胞受体与mhc多肽的结合强度能够决定t细胞分化。基于此,我们展示了野生型vim415刺激的il-10应答(图30;平均值580斑点/百万脾细胞;p=0.0249),即使是在th1佐剂cpg/mpla的存在下。但是,未能刺激显著ifnγ或il-17应答。参考文献1.gao,f.g.,etal.,antigen-specificcd4+t-cellhelpisrequiredtoactivateamemorycd8+tcelltoafullyfunctionaltumorkillercell.cancerres,2002.62(22):p.6438-41.2.janssen,e.m.,etal.,cd4+tcellsarerequiredforsecondaryexpansionandmemoryincd8+tlymphocytes.nature,2003.421(6925):p.852-6.3.fearon,e.r.,etal.,interleukin-2productionbytumorcellsbypassesthelperfunctioninthegenerationofanantitumorresponse.cell,1990.60(3):p.397-403.4.baxevanis,c.n.,etal.,tumor-specificcd4+tlymphocytesfromcancerpatientsarerequiredforoptimalinductionofcytotoxictcellsagainsttheautologoustumor.jimmunol,2000.164(7):p.3902-12.5.cella,m.,etal.,ligationofcd40ondendriticcellstriggersproductionofhighlevelsofinterleukin-12andenhancestcellstimulatorycapacity:t-thelpviaapcactivation.jexpmed,1996.184(2):p.747-52.6.ridge,j.p.,f.dirosa,andp.matzinger,aconditioneddendriticcellcanbeatemporalbridgebetweenacd4+t-helperandat-killercell.nature,1998.393(6684):p.474-8.7.schoenberger,s.p.,etal.,t-cellhelpforcytotoxictlymphocytesismediatedbycd40-cd40linteractions.nature,1998.393(6684):p.480-3.8.ayyoub,m.,etal.,animmunodominantssx-2-derivedepitoperecognizedbycd4+tcellsinassociationwithhla-dr.jclininvest,2004.113(8):p.1225-33.9.halder,t.,etal.,isolationofnovelhla-drrestrictedpotentialtumor-associatedantigensfromthemelanomacelllinefm3.cancerres,1997.57(15):p.3238-44.10.pardoll,d.m.ands.l.topalian,theroleofcd4+tcellresponsesinantitumorimmunity.curropinimmunol,1998.10(5):p.588-94.11.topalian,s.l.,mhcclassiirestrictedtumorantigensandtheroleofcd4+tcellsincancerimmunotherapy.curropinimmunol,1994.6(5):p.741-5.12.muranski,p.,etal.,tumor-specificth17-polarizedcellseradicatelargeestablishedmelanoma.blood,2008.112(2):p.362-73.13.paludan,c.,etal.,epstein-barrnuclearantigen1-specificcd4(+)th1cellskillburkitt'slymphomacells.jimmunol,2002.169(3):p.1593-603.14.quezada,s.a.,etal.,tumor-reactivecd4(+)tcellsdevelopcytotoxicactivityanderadicatelargeestablishedmelanomaaftertransferintolymphopenichosts.jexpmed,2010.207(3):p.637-50.15.xie,y.,etal.,naivetumor-specificcd4(+)tcellsdifferentiatedinvivoeradicateestablishedmelanoma.journalofexperimentalmedicine,2010.207(3):p.651-667.16.brandmaier,a.g.,etal.,high-avidityautoreactivecd4+tcellsinducehostctl,overcomet(regs)andmediatetumordestruction.jimmunother,2009.32(7):p.677-88.17.lauwen,m.m.,etal.,self-tolerancedoesnotrestrictthecd4+t-helperresponseagainstthep53tumorantigen.cancerres,2008.68(3):p.893-900.18.touloukian,c.e.,etal.,identificationofamhcclassii-restrictedhumangp100epitopeusingdr4-ietransgenicmice.jimmunol,2000.164(7):p.3535-42.19.mohanan,s.,etal.,potentialroleofpeptidylargininedeiminaseenzymesandproteincitrullinationincancerpathogenesis.biochemresint,2012.2012:p.895343.20.nomura,k.,specificityandmodeofactionofthemuscle-typeprotein-argininedeiminase.archbiochembiophys,1992.293(2):p.362-9.21.stensland,m.e.,etal.,primarysequence,togetherwithotherfactors,influencepeptidedeiminationbypeptidylargininedeiminase-4.biolchem,2009.390(2):p.99-107.22.klareskog,l.,etal.,immunitytocitrullinatedproteinsinrheumatoidarthritis.annualreviewofimmunology,2008.26:p.651-675.23.migliorini,p.,etal.,theimmuneresponsetocitrullinatedantigensinautoimmunediseases.autoimmunityreviews,2005.4(8):p.561-564.24.nijenhuis,s.,etal.,autoantibodiestocitrullinatedproteinsinrheumatoidarthritis:clinicalperformanceandbiochemicalaspectsofanra-specificmarker.clinicachimicaacta,2004.350(1-2):p.17-34.25.sebbag,m.,etal.,epitopesofhumanfibrinrecognizedbytherheumatoidarthritis-specificautoantibodiestocitrullinatedproteins.europeanjournalofimmunology,2006.36(8):p.2250-2263.26.vossenaar,e.r.,etal.,rheumatoidarthritisspecificanti-saantibodiestargetcitrullinatedvimentin.arthritisresther,2004.6(2):p.r142-50.27.vossenaar,e.r.,etal.,expressionandactivityofcitrullinatingpeptidylargininedeiminaseenzymesinmonocytesandmacrophages.annalsoftherheumaticdiseases,2004.63(4):p.373-381.28.birnboim,h.c.,etal.,cuttingedge:mhcclassii-restrictedpeptidescontainingtheinflammation-associatedmarker3-nitrotyrosineevadecentraltoleranceandelicitarobustcell-mediatedimmuneresponse.jimmunol,2003.171(2):p.528-532.29.herzog,j.,etal.,activatedantigen-presentingcellsselectandpresentchemicallymodifiedpeptidesrecognizedbyuniquecd4tcells.proceedingsofthenationalacademyofsciencesoftheunitedstatesofamerica,2005.102(22):p.7928-7933.30.bronte,v.,etal.,boostingantitumorresponsesoftlymphocytesinfiltratinghumanprostatecancers.journalofexperimentalmedicine,2005.201(8):p.1257-1268.31.arentz-hansen,h.,etal.,theintestinaltcellresponsetoalpha-gliadininadultceliacdiseaseisfocusedonasingledeamidatedglutaminetargetedbytissuetransglutaminase.jexpmed,2000.191(4):p.603-12.32.pudney,v.a.,etal.,dnavaccinationwitht-cellepitopesencodedwithinabmoleculesinduceshigh-avidityanti-tumorcd8(+)tcells.europeanjournalofimmunology,2010.40(3):p.899-910.33.durrant,l.g.m.,rachaellouise;pudney,victoriaanne;,nucleicacids,2008.34.feitsma,a.l.,etal.,identificationofcitrullinatedvimentinpeptidesastcellepitopesinhla-dr4-positivepatientswithrheumatoidarthritis.arthritisandrheumatism,2010.62(1):p.117-125.35.kryszke,m.h.andp.vicart,regulationoftheexpressionofthehumanvimentingene:applicationtocellularimmortalization.pathologiebiologie,1998.46(1):p.39-45.36.paulin,d.,expressionofthegenes-codingforthehumanintermediatefilamentproteins.pathologiebiologie,1989.37(4):p.277-282.37.ramaekers,f.c.,etal.,useofantibodiestointermediatefilamentsinthecharacterizationofhumantumors.coldspringharbsympquantbiol,1982.46pt1:p.331-9.38.thomas,p.a.,etal.,associationbetweenkeratinandvimentinexpression,malignantphenotype,andsurvivalinpostmenopausalbreastcancerpatients.clinicalcancerresearch,1999.5(10):p.2698-2703.39.conforti,g.,etal.,differentvimentinexpressionin2clonesderivedfromahumancolocarcinomacell-line(lovo)showingdifferentsensitivitytodoxorubicin.britishjournalofcancer,1995.71(3):p.505-511.40.moran,e.,etal.,co-expressionofmdr-associatedmarkers,includingp-170,mrpandlrpandcytoskeletalproteins,inthreeresistantvariantsofthehumanovariancarcinomacellline,oaw42.europeanjournalofcancer,1997.33(4):p.652-660.41.gilles,c.,etal.,vimentinexpressionincervicalcarcinomas:associationwithinvasiveandmigratorypotential.jpathol,1996.180(2):p.175-80.42.williams,a.a.,etal.,cd9andvimentindistinguishclearcellfromchromophoberenalcellcarcinoma.bmcclinpathol,2009.9:p.9.43.gustmann,c.,etal.,cytokeratinexpressionandvimentincontentinlargecellanaplasticlymphomasandothernon-hodgkins-lymphomas.americanjournalofpathology,1991.138(6):p.1413-1422.44.yamamoto,y.,k.izumi,andh.otsuka,animmunohistochemicalstudyofepithelialmembraneantigen,cytokeratin,andvimentininpapillarythyroid-carcinoma-recognitionoflethalandfavorableprognostictypes.cancer,1992.70(9):p.2326-2333.45.coppola,d.,etal.,prognosticsignificanceofp53,bcl-2,vimentin,ands100protein-positivelangerhanscellsinendometrialcarcinoma.humanpathology,1998.29(5):p.455-462.46.fuyuhiro,y.,etal.,clinicalsignificanceofvimentin-positivegastriccancercells.anticancerresearch,2010.30(12):p.5239-5243.47.takemura,k.,etal.,expressionofvimentiningastric-cancer-apossibleindicatorforprognosis.pathobiology,1994.62(3):p.149-154.48.ivaska,j.,vimentin:centralhubinemtinduction?smallgtpases,2011.2(1):p.51-53.49.vuoriluoto,k.,etal.,vimentinregulatesemtinductionbyslugandoncogenich-rasandmigrationbygoverningaxlexpressioninbreastcancer.oncogene,2011.30(12):p.1436-1448.50.hill,j.a.,etal.,cuttingedge:theconversionofargininetocitrullineallowsforahigh-affinitypeptideinteractionwiththerheumatoidarthritis-associatedhla-drb1*0401mhcclassiimolecule.jimmunol,2003.171(2):p.538-541.51.ireland,j.,j.herzog,ande.r.unanue,cuttingedge:uniquetcellsthatrecognizecitrullinatedpeptidesareafeatureofproteinimmunization.jimmunol,2006.177(3):p.1421-1425.52.asaga,h.,etal.,immunocytochemicallocalizationofpeptidylargininedeiminaseinhumaneosinophilsandneutrophils.journalofleukocytebiology,2001.70(1):p.46-51.53.nagata,s.andt.senshu,peptidylargininedeiminaseinratandmousehematopoietic-cells.experientia,1990.46(1):p.72-74.54.loos,t.,etal.,citrullinationofcxcl10andcxcl11bypeptidylargininedeiminase:anaturallyoccurringposttranslationalmodificationofchemokinesandnewdimensionofimmunoregulation.blood,2008.112(7):p.2648-2656.55.proost,p.,etal.,citrullinationofcxcl8bypeptidylargininedeiminasealtersreceptorusage,preventsproteolysis,anddampenstissueinflammation.journalofexperimentalmedicine,2008.205(9):p.2085-2097.56.struyf,s.,etal.,citrullinationofcxcl12differentiallyreducescxcr4andcxcr7bindingwithlossofinflammatoryandanti-hiv-1activityviacxcr4.jimmunol,2009.182(1):p.666-674.57.ireland,j.m.ande.r.unanue,autophagyinantigen-presentingcellsresultsinpresentationofcitrullinatedpeptidestocd4tcells.journalofexperimentalmedicine,2011.208(13):p.2625-2632.58.asaga,h.,m.yamada,andt.senshu,selectivedeiminationofvimentinincalciumionophore-inducedapoptosisofmouseperitonealmacrophages.biochembiophysrescommun,1998.243(3):p.641-6.59.mor-vaknin,n.,etal.,vimentinissecretedbyactivatedmacrophages.naturecellbiology,2003.5(1):p.59-63.60.vossenaar,e.r.,etal.,pad,agrowingfamilyofcitrullinatingenzymes:genes,featuresandinvolvementindisease.bioessays,2003.25(11):p.1106-18.61.kaufmann,s.h.e.,thecontributionofimmunologytotherationaldesignofnovelantibacterialvaccines.naturereviewsmicrobiology,2007.5(7):p.491-504.62.kubilus,j.,r.f.waitkus,andh.p.baden,partial-purificationandspecificityofanarginine-convertingenzymefrombovineepidermis.biochimicaetbiophysicaacta,1980.615(1):p.246-251.63.senshu,t.,etal.,studiesonspecificityofpeptidylargininedeiminasereactionsusinganimmunochemicalprobethatrecognizesanenzymaticallydeiminatedpartialsequenceofmousekeratink1.journalofdermatologicalscience,1999.21(2):p.113-126.64.chang,x.,etal.,localizationofpeptidylargininedeiminase4(padi4)andcitrullinatedproteininsynovialtissueofrheumatoidarthritis.rheumatology,2005.44(1):p.40-50.65.chang,x.andj.han,expressionofpeptidylargininedeiminasetype4(pad4)invarioustumors.molcarcinog,2006.45(3):p.183-96.66.guo,q.andw.fast,citrullinationofinhibitorofgrowth4(ing4)bypeptidylargininedeminase4(pad4)disruptstheinteractionbetweening4andp53.jbiolchem,2011.286(19):p.17069-78.67.karlin,s.ands.f.altschul,applicationsandstatisticsformultiplehigh-scoringsegmentsinmolecularsequences.procnatlacadsciusa,1993.90(12):p.5873-7.68.altschul,s.f.,etal.,basiclocalalignmentsearchtool.jmolbiol,1990.215(3):p.403-10.69.altschul,s.f.,etal.,gappedblastandpsi-blast:anewgenerationofproteindatabasesearchprograms.nucleicacidsres,1997.25(17):p.3389-402.70.myers,e.w.andw.miller,approximatematchingofregularexpressions.bullmathbiol,1989.51(1):p.5-37.71.torelli,a.andc.a.robotti,advanceandadam:twoalgorithmsfortheanalysisofglobalsimilaritybetweenhomologousinformationalsequences.computapplbiosci,1994.10(1):p.3-5.72.pearson,w.r.andd.j.lipman,improvedtoolsforbiologicalsequencecomparison.procnatlacadsciusa,1988.85(8):p.2444-8.73.remington,r.,remington'spharmaceuticalsciences.16thed.1980:mackpub.co.74.stewart,solidphasepeptidesynthesis.2nded.1984,rockford:illinoispiercechemicalcompany.75.bodanzsky,b.,thepracticeofpeptidesynthesis.1984,newyork:springerverlag.76.sambrook,j.,fritschandmaniatis,molecularcloning:alaboratorymanual.2nded.1989:coldspringharborlaboratorypress.77.ausubel,j.,shortprotocolsinmolecularbiology.1992:johnwiley&sons78.pluckthun,a.,antibodyengineering:advancesfromtheuseofescherichiacoliexpressionsystems.biotechnology(ny),1991.9(6):p.545-51.79.reff,m.e.,high-levelproductionofrecombinantimmunoglobulinsinmammaliancells.curropinbiotechnol,1993.4(5):p.573-6.80.trill,j.j.,a.r.shatzman,ands.ganguly,productionofmonoclonalantibodiesincosandchocells.curropinbiotechnol,1995.6(5):p.553-60.81.palena,c.,etal.,strategiestotargetmoleculesthatcontroltheacquisitionofamesenchymal-likephenotypebycarcinomacells.expbiolmed(maywood),2011.236(5):p.537-45.82.denzin,l.k.,etal.,assemblyandintracellulartransportofhla-dmandcorrectionoftheclassiiantigen-processingdefectint2cells.immunity,1994.1(7):p.595-606.83.kovats,s.,etal.,presentationofabundantendogenousclassiidr-restrictedantigensbydm-negativebcelllines.eurjimmunol,1997.27(4):p.1014-21.84.metheringham,r.l.,etal.,antibodiesdesignedaseffectivecancervaccines.mabs,2009.1(1):p.71-85.85.quinnmjbp,b.a.,kirbyea,jonesj,cancertrendsinenglandandwales,1950-1999,inlondon:stationeryoffice2001.p.206-207.86.nice,improvingoutcomesincolorectalcancers:manualupdate,2004:london:nationalinstituteforclinicalexcellence.序列表<110>斯坎塞尔有限公司<120>对修饰后自体表位的抗肿瘤免疫应答<130>p55129wo/njl<140>pct/gb2013/052109<141>2013-08-07<150>gb1214007.5<151>2012-08-07<160>159<170>patentinversion3.5<210>1<211>9<212>prt<213>artificialsequence<220><223>modifiedepitopewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>1ileglnlysleutyrglylysargser15<210>2<211>16<212>prt<213>artificialsequence<220><223>modifiedepitopewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>2serglnaspaspilelysglyileglnlysleutyrglylysargser151015<210>3<211>10<212>prt<213>artificialsequence<220><223>modifiedepitopewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>3asnileleuthrileargleuthralaala1510<210>4<211>25<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>4proglyvalleuleulysgluphethrvalserglyasnileleuthr151015ileargleuthralaalaasphisarg2025<210>5<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>5ileleuthrileargleuthralaala15<210>6<211>8<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>6gluilearggluleuglnsergln15<210>7<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>7gluglugluilearggluleuglnserglnileseraspthrserval151015valleuser<210>8<211>15<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>8alalysglnaspmetalaargglnleuargglutyrglngluleu151015<210>9<211>20<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>9alalysglnaspmetalaargglnleuargglutyrglngluleumet151015asnvallysleu20<210>10<211>8<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>10alalysglnaspmetalaarggln15<210>11<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>11leuglnargalalysglnaspmetalaargglnleuargglutyrgln151015gluleumet<210>12<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>12ileserserserserpheserargval15<210>13<211>16<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>13proglyserargileserserserserpheserargvalglyserser151015<210>14<211>8<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>14proargalapheserserargser15<210>15<211>18<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>15serthrserglyproargalapheserserargsertyrthrsergly151015progly<210>16<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>16glualaalaleuglnargalalysgln15<210>17<211>17<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>17gluleuglualaalaleuglnargalalysglnaspmetalaarggln151015leu<210>18<211>14<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>18leugluvalaspproasnileglnalavalargthrglnglu1510<210>19<211>18<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>19leugluvalaspproasnileglnalavalargthrglnglulysglu151015glnile<210>20<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>20glnlyslysleulysleuvalargthr15<210>21<211>17<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>21alaglnlyslysleulysleuvalargthrserproglutyrglymet151015pro<210>22<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>22leulysleuvalargthrserproglu15<210>23<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>23lyslysleulysleuvalargthrser15<210>24<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>24tyrmetserseralaargserleuser15<210>25<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>25metserseralaargserleuserser15<210>26<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>26thrglutyrmetserseralaargser15<210>27<211>18<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>27lysleualathrglutyrmetserseralaargserleuserserglu151015glulys<210>28<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>28pheaspleuphegluasnarglyslys15<210>29<211>15<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>29argalapropheaspleuphegluasnarglyslyslysasnasn151015<210>30<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>30tyrleuasnpheileargglyvalval15<210>31<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>31pheileargglyvalvalaspserglu15<210>32<211>15<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>32ileproglutyrleuasnpheileargglyvalvalaspserglu151015<210>33<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>33leuargtyrtyrthrseralasergly15<210>34<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>34leuleuargtyrtyrthrseralaser15<210>35<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>35leusergluleuleuargtyrtyrthr15<210>36<211>22<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>36arglyslysleusergluleuleuargtyrtyrthrseralasergly151015aspglumetvalserleu20<210>37<211>15<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>37argargargleusergluleuleuargtyrhisthrserglnser151015<210>38<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>38valglyvalphelysasnglyargval15<210>39<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>39phelysasnglyargvalgluileile15<210>40<211>15<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>40tyrsercysvalglyvalphelysasnglyargvalgluileile151015<210>41<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>41tyrpheasnaspalaglnargglnala15<210>42<211>16<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>42valproalatyrpheasnaspalaglnargglnalathrlysaspala151015<210>43<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>43valthrphegluileaspvalasngly15<210>44<211>15<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>44gluvalthrphegluileaspvalasnglyileleuargvalthr151015<210>45<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>45ilethrasnaspglnasnargleuthr15<210>46<211>15<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>46lysilethrilethrasnaspglnasnargleuthrprogluglu151015<210>47<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>47leuglnilevalalaargleulysasn15<210>48<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>48valalaargleulysasnasnasnarg15<210>49<211>15<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>49asncysalaleuglnilevalalaargleulysasnasnasnarg151015<210>50<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>50valgluileilealathrmetlyslys15<210>51<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>51argvalgluileilealathrmetlys15<210>52<211>15<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>52cysproargvalgluileilealathrmetlyslyslysglyglu151015<210>53<211>16<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinxaaiscitrulline<220><221>mod_res<222>(7)..(7)<223>xaaiscitrulline<220><221>mod_res<222>(16)..(16)<223>xaaiscitrulline<400>53tyrvalthrthrserthrxaathrtyrserleuglyseralaleuxaa151015<210>54<211>22<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinxaaiscitrulline<220><221>mod_res<222>(1)..(1)<223>xaaiscitrulline<220><221>mod_res<222>(9)..(9)<223>xaaiscitrulline<220><221>mod_res<222>(18)..(18)<223>xaaiscitrulline<400>54xaasertyrvalthrthrserthrxaathrtyrserleuglyserala151015leuxaaproserthrser20<210>55<211>16<212>prt<213>artificialsequence<220><223>modifiedpeptidewhichoptionallycanbecitrullinated.eitherorbothargininesmaybecitrullinated.<400>55tyrvalthrthrserthrargthrtyrserleuglyseralaleuarg151015<210>56<211>22<212>prt<213>artificialsequence<220><223>modifiedpeptidewhichoptionallymaybecitrullinated.anyoneortwo,orall,ofthethreeargininespresentmaybecitrullinated.<400>56argsertyrvalthrthrserthrargthrtyrserleuglyserala151015leuargproserthrser20<210>57<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>57valargleuargserservalprogly15<210>58<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>58argleuargserservalproglyval15<210>59<211>13<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>59seralavalargleuargserservalproglyvalarg1510<210>60<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>60pheserserleuasnleuarggluthr15<210>61<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>61leuproasnpheserserleuasnleuarggluthrasnleuaspser151015leuproleu<210>62<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>62argserservalproglyvalargleu15<210>63<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline<400>63seralavalargleuargserserval15<210>64<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>64alathrargserseralavalargleu15<210>65<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>65tyralathrargserseralavalargleuargserservalprogly151015valargleu<210>66<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>66argserservalproglyvalargleu15<210>67<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>67glyvalargleuleuglnaspserval15<210>68<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>68argleuargserservalproglyvalargleuleuglnaspserval151015asppheser<210>69<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>69glnleulysglyglnglylysserarg15<210>70<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>70lysserargleuglyaspleutyrglu15<210>71<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>71gluglnleulysglyglnglylysserargleuglyaspleutyrglu151015gluglumet<210>72<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>72gluleuargargglnvalaspglnleu15<210>73<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>73glumetarggluleuargargglnval15<210>74<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>74aspleutyrglugluglumetarggluleuargargglnvalaspgln151015leuthrasn<210>75<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>75glnleuthrasnasplysalaargval15<210>76<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>76valgluvalgluargaspasnleuala15<210>77<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>77leuthrasnasplysalaargvalglu15<210>78<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>78valaspglnleuthrasnasplysalaargvalgluvalgluargasp151015asnleuala<210>79<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>79gluvalgluargaspasnleualaglu15<210>80<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>80asnasplysalaargvalgluvalgluargaspasnleualagluasp151015ilemetarg<210>81<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>81ilemetargleuargglulysleugln15<210>82<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>82aspasnleualagluaspilemetargleuargglulysleuglnglu151015glumetleu<210>83<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>83glnarggluglualagluasnthrleu15<210>84<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>84lysleuglngluglumetleuglnarg15<210>85<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>85glulysleuglngluglumetleuglnarggluglualagluasnthr151015leuglnser<210>86<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>86pheargglnaspvalaspasnalaser15<210>87<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>87gluasnthrleuglnserphearggln15<210>88<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<400>88glualagluasnthrleuglnserpheargglnaspvalaspasnala151015serleuala<210>89<211>15<212>prt<213>artificialsequence<220><223>epitope<400>89asptyrsertyrleuglnaspseraspproaspserpheglnasp151015<210>90<211>16<212>prt<213>artificialsequence<220><223>epitope<400>90trpasnargglnleutyrproglutrpthrgluvalglnglyserasn151015<210>91<211>16<212>prt<213>artificialsequence<220><223>epitope<400>91trpasnargglnleutyrproglutrpthrglualaglnargleuasp151015<210>92<211>13<212>prt<213>artificialsequence<220><223>epitope<400>92thrproproalatyrargproproasnalaproileleu1510<210>93<211>15<212>prt<213>artificialsequence<220><223>epitope<400>93serglnaspaspilelysglyglnlysleutyrglylysargser151015<210>94<211>15<212>prt<213>artificialsequence<220><223>epitope<400>94lysgluglutrpglulysmetlysalaserglulysilephetyr151015<210>95<211>25<212>prt<213>artificialsequence<220><223>epitope<400>95leuleugluphetyrleualametprophealathrprometgluala151015gluleualaargargserleualagln2025<210>96<211>15<212>prt<213>artificialsequence<220><223>epitope<400>96glygluleuileglyileleuasnalaalalysvalproalaasp151015<210>97<211>19<212>prt<213>artificialsequence<220><223>epitope<400>97leuproasnpheserserleuasnleuarggluthrasnleuaspser151015leuproleu<210>98<211>22<212>prt<213>artificialsequence<220><223>epitope<400>98argsertyrvalthrthrserthrargthrtyrserleuglyserala151015leuargproserthrser20<210>99<211>9<212>prt<213>artificialsequence<220><223>cd4epitope<400>99leutyrproglutrpthrgluvalgln15<210>100<211>9<212>prt<213>artificialsequence<220><223>cd4epitope<400>100thrtyrserleuglyseralaleuarg15<210>101<211>9<212>prt<213>artificialsequence<220><223>cd4epitope<400>101tyrvalthrthrserthrargthrtyr15<210>102<211>9<212>prt<213>artificialsequence<220><223>cd4epitope<400>102leutyrproglutrpthrglualagln15<210>103<211>9<212>prt<213>artificialsequence<220><223>cd4epitope<400>103tyrleuglnaspseraspproaspser15<210>104<211>9<212>prt<213>artificialsequence<220><223>cd4epitope<400>104ileglyileleuasnalaalalysval15<210>105<211>9<212>prt<213>artificialsequence<220><223>cd4epitope<400>105phetyrleualametprophealathr15<210>106<211>9<212>prt<213>artificialsequence<220><223>cd4epitope<400>106asnileleuthrileargleuthrala15<210>107<211>9<212>prt<213>artificialsequence<220><223>cd4epitope<400>107trpglulysmetlysalaserglulys15<210>108<211>9<212>prt<213>artificialsequence<220><223>cd4epitope<400>108tyrargproproasnalaproileleu15<210>109<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(4)..(4)<223>arginineresiduesubstitutedwithcitrulline<220><221>mod_res<222>(12)..(12)<223>arginineresiduesubstitutedwithcitrulline<220><221>mod_res<222>(13)..(13)<223>arginineresiduesubstitutedwithcitrulline<400>109metserthrargservalsersersersertyrargargmetphegly151015glyprogly<210>110<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(1)..(1)<223>arginineresiduesubstitutedwithcitrulline<220><221>mod_res<222>(9)..(9)<223>arginineresiduesubstitutedwithcitrulline<220><221>mod_res<222>(10)..(10)<223>arginineresiduesubstitutedwithcitrulline<400>110argservalsersersersertyrargargmetpheglyglyprogly151015thralaser<210>111<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(10)..(10)<223>arginineresiduesubstitutedwithcitrulline<220><221>mod_res<222>(15)..(15)<223>arginineresiduesubstitutedwithcitrulline<400>111metpheglyglyproglythralaserargproserserserargser151015tyrvalthr<210>112<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(5)..(5)<223>arginineresiduesubstitutedwithcitrulline<220><221>mod_res<222>(10)..(10)<223>arginineresiduesubstitutedwithcitrulline<220><221>mod_res<222>(18)..(18)<223>arginineresiduesubstitutedwithcitrulline<400>112glythralaserargproserserserargsertyrvalthrthrser151015thrargthr<210>113<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(3)..(3)<223>arginineresiduesubstitutedwithcitrulline<220><221>mod_res<222>(11)..(11)<223>arginineresiduesubstitutedwithcitrulline<400>113serserargsertyrvalthrthrserthrargthrtyrserleugly151015seralaleu<210>114<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(1)..(1)<223>arginineresiduesubstitutedwithcitrulline<220><221>mod_res<222>(10)..(10)<223>arginineresiduesubstitutedwithcitrulline<220><221>mod_res<222>(15)..(15)<223>arginineresiduesubstitutedwithcitrulline<400>114argthrtyrserleuglyseralaleuargproserthrserargser151015leutyrala<210>115<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(5)..(5)<223>arginineresiduesubstitutedwithcitrulline<220><221>mod_res<222>(10)..(10)<223>arginineresiduesubstitutedwithcitrulline<400>115glyseralaleuargproserthrserargserleutyralaserser151015proglygly<210>116<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(10)..(10)<223>arginineresiduesubstitutedwithcitrulline<220><221>mod_res<222>(15)..(15)<223>arginineresiduesubstitutedwithcitrulline<220><221>mod_res<222>(17)..(17)<223>arginineresiduesubstitutedwithcitrulline<400>116serserproglyglyvaltyralathrargserseralavalargleu151015argserser<210>117<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(10)..(10)<223>arginineresiduesubstitutedwithcitrulline<400>117alaileasnthrgluphelysasnthrargthrasnglulysvalglu151015leuglnglu<210>118<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(11)..(11)<223>arginineresiduesubstitutedwithcitrulline<400>118glulysvalgluleuglngluleuasnaspargphealaasntyrile151015asplysval<210>119<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(1)..(1)<223>arginineresiduesubstitutedwithcitrulline<220><221>mod_res<222>(10)..(10)<223>arginineresiduesubstitutedwithcitrulline<400>119argphealaasntyrileasplysvalargpheleugluglnglnasn151015lysileleu<210>120<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(7)..(7)<223>arginineresiduesubstitutedwithcitrulline<220><221>mod_res<222>(12)..(12)<223>arginineresiduesubstitutedwithcitrulline<400>120aspasnalaserleualaargleuaspleugluarglysvalgluser151015leuglnglu<210>121<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(9)..(9)<223>arginineresiduesubstitutedwithcitrulline<220><221>mod_res<222>(12)..(12)<223>arginineresiduesubstitutedwithcitrulline<400>121lysproaspleuthralaalaleuargaspvalargglnglntyrglu151015servalala<210>122<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(10)..(10)<223>arginineresiduesubstitutedwithcitrulline<220><221>mod_res<222>(16)..(16)<223>arginineresiduesubstitutedwithcitrulline<400>122phealaaspleuserglualaalaasnargasnasnaspalaleuarg151015glnalalys<210>123<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(4)..(4)<223>arginineresiduesubstitutedwithcitrulline<220><221>mod_res<222>(10)..(10)<223>arginineresiduesubstitutedwithcitrulline<400>123alaalaasnargasnasnaspalaleuargglnalalysglngluser151015thrglutyr<210>124<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(10)..(10)<223>arginineresiduesubstitutedwithcitrulline<220><221>mod_res<222>(11)..(11)<223>arginineresiduesubstitutedwithcitrulline<400>124glnalalysglngluserthrglutyrargargglnvalglnserleu151015thrcysglu<210>125<211>19<212>prt<213>artificialsequence<220><223>modifiedepitopewhereinoneormoreoftherresiduesissubstitutedforcitrulline.<220><221>mod_res<222>(9)..(9)<223>arginineresiduesubstitutedwithcitrulline<220><221>mod_res<222>(12)..(12)<223>arginineresiduesubstitutedwithcitrulline<400>125lysglythrasngluserleugluargglnmetargglumetgluglu151015asnpheala<210>126<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(10)..(10)<223>arginineresiduesubstitutedwithcitrulline<400>126alaalaasntyrglnaspthrileglyargleuglnaspgluilegln151015asnmetlys<210>127<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(9)..(9)<223>arginineresiduesubstitutedwithcitrulline<220><221>mod_res<222>(12)..(12)<223>arginineresiduesubstitutedwithcitrulline<400>127glnasnmetlysgluglumetalaarghisleuargglutyrglnasp151015leuleuasn<210>128<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(10)..(10)<223>arginineresiduesubstitutedwithcitrulline<220><221>mod_res<222>(19)..(19)<223>arginineresiduesubstitutedwithcitrulline<400>128alaleuaspilegluilealathrtyrarglysleuleugluglyglu151015gluserarg<210>129<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(1)..(1)<223>arginineresiduesubstitutedwithcitrulline<220><221>mod_res<222>(10)..(10)<223>arginineresiduesubstitutedwithcitrulline<400>129arglysleuleugluglyglugluserargileserleuproleupro151015asnpheser<210>130<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(10)..(10)<223>arginineresiduesubstitutedwithcitrulline<400>130leuproleuvalaspthrhisserlysargthrleuleuilelysthr151015valgluthr<210>131<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(10)..(10)<223>arginineresiduesubstitutedwithcitrulline<400>131thrleuleuilelysthrvalgluthrargaspglyglnvalileasn151015gluthrser<210>132<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptide<400>132gluilearggluleuglnserglnile15<210>133<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptide<400>133alaargglnleuargglutyrglnglu15<210>134<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptide<400>134alalysglnaspmetalaargglnleu15<210>135<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(5)..(5)<223>arginineresiduesubsitutedwithcitrulline<400>135leulysglyglnargalaserleuglualaalailealaaspalaglu151015glnarggly<210>136<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(14)..(14)<223>arginineresiduesubsitutedwithcitrulline<400>136ileseraspthrservalvalleusermetaspasnserargserleu151015aspmetasp<210>137<211>20<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(13)..(13)<223>arginineresiduesubsitutedwithcitrulline<400>137aspasnmetpheglusertyrileasnasnleuargargglnleuglu151015thrleuglygln20<210>138<211>20<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(14)..(14)<223>arginineresiduesubsitutedwithcitrulline<400>138ilealathrtyrarglysleuleugluglyglugluserargleuglu151015serglymetgln20<210>139<211>18<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(10)..(10)<223>arginineresiduesubsitutedwithcitrulline<400>139lysalaglntyrgluaspilealaasnargserargalaglualaglu151015sermet<210>140<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(10)..(10)<223>arginineresiduesubsitutedwithcitrulline<400>140alavalvalvallyslysilegluthrargaspglylysleuvalser151015gluserser<210>141<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(12)..(12)<223>arginineresiduesubsitutedwithcitrulline<400>141asnasnlysphealaserpheileasplysvalargpheleuglugln151015glnasnlys<210>142<211>20<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(12)..(12)<223>arginineresiduesubsitutedwithcitrulline<400>142glualatyrmetasnlysvalgluleugluserargleugluglyleu151015thraspgluile20<210>143<211>19<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(18)..(18)<223>arginineresiduesubsitutedwithcitrulline<400>143valgluleugluserargleugluglyleuthraspgluileasnphe151015leuarggln<210>144<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptide<400>144glyproargalapheserserargser15<210>145<211>20<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(13)..(13)<223>arginineresiduesubsitutedwithcitrulline<400>145aspphelysasnlystyrgluaspgluileasnlysargthrglumet151015gluasngluphe20<210>146<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptide<400>146asnileglnalavalargthrglnglu15<210>147<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptide<400>147aspproasnileglnalavalargthr15<210>148<211>20<212>prt<213>artificialsequence<220><223>modifiedpeptide<220><221>mod_res<222>(8)..(8)<223>arginineresiduesubsitutedwithcitrulline<400>148lyshisglyaspaspleuargargthrlysthrgluileserglumet151015asnargasnile20<210>149<211>17<212>prt<213>artificialsequence<220><223>modifiedpeptidewhereinxaaiscitrulline<220><221>mod_res<222>(9)..(9)<223>xaaiscitrulline<400>149alaglnlyslysleulysleuvalxaathrserproglutyrglymet151015pro<210>150<211>8<212>prt<213>artificialsequence<220><223>modifiedpeptide<400>150valglyvalphelysasnglyarg15<210>151<211>9<212>prt<213>artificialsequence<220><223>modifiedpeptide<400>151phelysasnglyargvalgluileile15<210>152<211>466<212>prt<213>homosapiens<400>152metserthrargservalsersersersertyrargargmetphegly151015glyproglythralaserargproserserserargsertyrvalthr202530thrserthrargthrtyrserleuglyseralaleuargproserthr354045serargserleutyralaserserproglyglyvaltyralathrarg505560serseralavalargleuargserservalproglyvalargleuleu65707580glnaspservalasppheserleualaaspalaileasnthrgluphe859095lysasnthrargthrasnglulysvalgluleuglngluleuasnasp100105110argphealaasntyrileasplysvalargpheleugluglnglnasn115120125lysileleuleualagluleugluglnleulysglyglnglylysser130135140argleuglyaspleutyrglugluglumetarggluleuargarggln145150155160valaspglnleuthrasnasplysalaargvalgluvalgluargasp165170175asnleualagluaspilemetargleuargglulysleuglngluglu180185190metleuglnarggluglualagluasnthrleuglnserphearggln195200205aspvalaspasnalaserleualaargleuaspleugluarglysval210215220gluserleuglnglugluilealapheleulyslysleuhisgluglu225230235240gluileglngluleuglnalaglnileglngluglnhisvalglnile245250255aspvalaspvalserlysproaspleuthralaalaleuargaspval260265270argglnglntyrgluservalalaalalysasnleuglnglualaglu275280285glutrptyrlysserlysphealaaspleuserglualaalaasnarg290295300asnasnaspalaleuargglnalalysglngluserthrglutyrarg305310315320argglnvalglnserleuthrcysgluvalaspalaleulysglythr325330335asngluserleugluargglnmetargglumetglugluasnpheala340345350valglualaalaasntyrglnaspthrileglyargleuglnaspglu355360365ileglnasnmetlysgluglumetalaarghisleuargglutyrgln370375380aspleuleuasnvallysmetalaleuaspilegluilealathrtyr385390395400arglysleuleugluglyglugluserargileserleuproleupro405410415asnpheserserleuasnleuarggluthrasnleuaspserleupro420425430leuvalaspthrhisserlysargthrleuleuilelysthrvalglu435440445thrargaspglyglnvalileasngluthrserglnhishisaspasp450455460leuglu465<210>153<211>460<212>prt<213>gallusgallus<400>153metserphethrserserlysasnsersertyrargargmetphegly151015glyglyserargproserserglythrargtyrilethrserserthr202530argtyrserleuglyseralaleuargproserseralaargtyrval354045seralaserproglyglyvaltyrargthrlysalathrservalarg505560leuargsersermetproprometargmethisaspalavalaspphe65707580thrleualaaspalaileasnthrgluphelysalaasnargthrasn859095glulysvalgluleuglngluleuasnaspargphealaasntyrile100105110asplysvalargpheleugluglnglnasnlysileleuleualaglu115120125leugluglnleulysglylysglythrserargleuglyaspleutyr130135140glugluglumetargaspvalargargglnvalaspglnleuthrasn145150155160asplysalaargvalgluvalgluargaspasnleualaaspaspile165170175metargleuargglulysleuglngluglumetleuglnarggluglu180185190alagluserthrleuglnserpheargglnaspvalaspasnalaser195200205leualaglyleuaspleugluargprovalgluserleuglngluglu210215220ilevalpheleulyslysleuhisaspglugluilearggluleugln225230235240alaglnleuglngluglnhisileglnileaspmetaspvalserlys245250255proaspleuthralaalaleuargaspvalargglnglntyrgluser260265270valalaalalysasnleuglnglualagluglutrptyrlysserlys275280285phealaaspleuserglualaalaasnargasnasnaspalaleuarg290295300glnalalysglnglualaasnglutyrargargglnileglnserleu305310315320thrcysgluvalaspalaleulysglyserasngluserleugluarg325330335glnmetargglumetglugluasnphealavalglualaalaasntyr340345350glnaspthrileglyargleuglnaspgluileglnasnmetlysglu355360365glumetalaarghisleuargglutyrglnaspleuleuasnvallys370375380metalaleuaspilegluilealathrtyrarglysleuleuglugly385390395400glugluserargileasnmetproileprothrphealaserleuasn405410415leuarggluthrasnilegluserglnproilevalaspthrhisser420425430lysargthrleuleuilelysthrvalgluthrargaspglyglnval435440445ileasngluthrserglnhishisaspaspleuglu450455460<210>154<211>466<212>prt<213>musmusculus<400>154metserthrargservalsersersersertyrargargmetphegly151015glyserglythrserserargproserserasnargsertyrvalthr202530thrserthrargthrtyrserleuglyseralaleuargproserthr354045serargserleutyrserserserproglyglyalatyrvalthrarg505560serseralavalargleuargserservalproglyvalargleuleu65707580glnaspservalasppheserleualaaspalaileasnthrgluphe859095lysasnthrargthrasnglulysvalgluleuglngluleuasnasp100105110argphealaasntyrileasplysvalargpheleugluglnglnasn115120125lysileleuleualagluleugluglnleulysglyglnglylysser130135140argleuglyaspleutyrglugluglumetarggluleuargarggln145150155160valaspglnleuthrasnasplysalaargvalgluvalgluargasp165170175asnleualagluaspilemetargleuargglulysleuglngluglu180185190metleuglnarggluglualagluserthrleuglnserphearggln195200205aspvalaspasnalaserleualaargleuaspleugluarglysval210215220gluserleuglnglugluilealapheleulyslysleuhisaspglu225230235240gluileglngluleuglnalaglnileglngluglnhisvalglnile245250255aspvalaspvalserlysproaspleuthralaalaleuargaspval260265270argglnglntyrgluservalalaalalysasnleuglnglualaglu275280285glutrptyrlysserlysphealaaspleuserglualaalaasnarg290295300asnasnaspalaleuargglnalalysglngluserasnglutyrarg305310315320argglnvalglnserleuthrcysgluvalaspalaleulysglythr325330335asngluserleugluargglnmetargglumetglugluasnpheala340345350leuglualaalaasntyrglnaspthrileglyargleuglnaspglu355360365ileglnasnmetlysgluglumetalaarghisleuargglutyrgln370375380aspleuleuasnvallysmetalaleuaspilegluilealathrtyr385390395400arglysleuleugluglyglugluserargileserleuproleupro405410415thrpheserserleuasnleuarggluthrasnleugluserleupro420425430leuvalaspthrhisserlysargthrleuleuilelysthrvalglu435440445thrargaspglyglnvalileasngluthrserglnhishisaspasp450455460leuglu465<210>155<211>466<212>prt<213>canisfamiliaris<400>155metserthrargservalsersersersertyrargargmetphegly151015glyproglythrglyserargproserserthrargsertyrvalthr202530thrserthrargthrtyrserleuglyseralaleuargproserthr354045serargserleutyralaserserproglyglyalatyralathrarg505560serseralavalargleuargserservalproglyvalargleuleu65707580glnaspservalasppheserleualaaspalaileasnthrgluphe859095lysasnthrargthrasnglulysvalgluleuglngluleuasnasp100105110argphealaasntyrileasplysvalargpheleugluglnglnasn115120125lysileleuleualagluleugluglnleulysglyglnglylysser130135140argleuglyaspleutyrglugluglumetarggluleuargarggln145150155160valaspglnleuthrasnasplysalaargvalgluvalgluargasp165170175asnleualagluaspilemetargleuargglulysleuglngluglu180185190metleuglnarggluglualagluserthrleuglnserphearggln195200205aspvalaspasnalaserleualaargleuaspleugluarglysval210215220gluserleuglnglugluilealapheleulyslysleuhisaspglu225230235240gluileglngluleuglnalaglnileglnaspglnhisvalglnile245250255aspmetaspvalserlysproaspleuthralaalaleuargaspval260265270argglnglntyrgluservalalaalalysasnleuglnglualaglu275280285glutrptyrlysserlysphealaaspleuserglualaalaasnarg290295300asnasnaspalaleuargglnalalysglngluserasnglutyrarg305310315320argglnvalglnserleuthrcysgluvalaspalaleulysglythr325330335asngluserleugluargglnmetargglumetglugluasnpheala340345350valglualaalaasntyrglnaspthrileglyargleuglnaspglu355360365ileglnasnmetlysgluglumetalaarghisleuargglutyrgln370375380aspleuleuasnvallysmetalaleuaspilegluilealathrtyr385390395400arglysleuleugluglyglugluserargilealaleuproleupro405410415asnpheserserleuasnleuarggluthrasnleuaspserleupro420425430leuvalaspthrhisserlysargthrleuleuilelysthrvalglu435440445thrargaspglyglnvalileasngluthrserglnhishisaspasp450455460leuglu465<210>156<211>466<212>prt<213>ovisaries<400>156metserthrargservalsersersersertyrargargmetphegly151015glyproglythralaserargproserserthrargsertyrvalthr202530thrserthrargthrtyrserleuglyseralaleuargproserthr354045serargthrleutyrthrserserproglyglyvaltyralathrarg505560serseralavalargleuargserglyvalproglyvalargleuleu65707580glnaspservalasppheserleualaaspalaileasnthrgluphe859095lysasnthrargthrasnglulysvalgluleuglngluleuasnasp100105110argphealaasntyrileasplysvalargpheleugluglnglnasn115120125lysileleuleualagluleugluglnleulysglyglnglylysser130135140argleuglyaspleutyrglugluglumetarggluleuargarggln145150155160valaspglnleuthrasnasplysalaargvalgluvalgluargasp165170175asnleualagluaspilemetargleuargglulysleuglngluglu180185190metleuglnarggluglualagluserthrleuglnserphearggln195200205aspvalaspasnalaserleualaargleuaspleugluarglysval210215220gluserleuglnglugluilealapheleulyslysleuhisaspglu225230235240gluileglngluleuglnalaglnileglngluglnhisvalglnile245250255aspmetaspvalserglnproaspleuthralaalaleuargaspval260265270argglnglntyrgluservalalaalalysasnleuglnglualaglu275280285glutrptyrlysserlysphealaaspleuserglualaalaasnarg290295300asnasnaspalaleuargglnalalysglngluserasnglutyrarg305310315320argglnvalglnserleuthrcysgluvalaspalaleulysglythr325330335asngluserleugluargglnmetargglumetglugluasnpheser340345350valglualaalaasntyrglnaspthrileglyargleuglnaspglu355360365ileglnasnmetlysgluglumetalaarghisleuargglutyrgln370375380aspleuleuasnvallysmetalaleuaspilegluilealathrtyr385390395400arglysleuleugluglyglugluserargileserleuproleupro405410415asnpheserserleuasnleuarggluthrasnleuaspserleupro420425430leuvalaspthrhisserlysargthrleuleuilelysthrvalglu435440445thrargaspglyglnvalileasngluthrserglnhishisaspasp450455460leuglu465<210>157<211>466<212>prt<213>bostaurus<400>157metserthrargservalsersersersertyrargargmetphegly151015glyproglythralaserargproserserthrargsertyrvalthr202530thrserthrargthrtyrserleuglyseralaleuargprothrthr354045serargthrleutyrthrserserproglyglyvaltyralathrarg505560serseralavalargleuargserglyvalproglyvalargleuleu65707580glnaspservalasppheserleualaaspalaileasnthrgluphe859095lysasnthrargthrasnglulysvalgluleuglngluleuasnasp100105110argphealaasntyrileasplysvalargpheleugluglnglnasn115120125lysileleuleualagluleugluglnleulysglyglnglylysser130135140argleuglyaspleutyrglugluglumetarggluleuargarggln145150155160valaspglnleuthrasnasplysalaargvalgluvalgluargasp165170175asnleualagluaspilemetargleuargglulysleuglngluglu180185190metleuglnarggluglualagluserthrleuglnserphearggln195200205aspvalaspasnalaserleualaargleuaspleugluarglysval210215220gluserleuglnglugluilealapheleulyslysleuhisaspglu225230235240gluileglngluleuglnalaglnileglngluglnhisvalglnile245250255aspmetaspvalserlysproaspleuthralaalaleuargaspval260265270argglnglntyrgluservalalaalalysasnleuglnglualaglu275280285glutrptyrlysserlysphealaaspleuserglualaalaasnarg290295300asnasnaspalaleuargglnalalysglngluserasnglutyrarg305310315320argglnvalglnthrleuthrcysgluvalaspalaleulysglythr325330335asngluserleugluargglnmetargglumetglugluasnpheser340345350valglualaalaasntyrglnaspthrileglyargleuglnaspglu355360365ileglnasnmetlysgluglumetalaarghisleuargglutyrgln370375380aspleuleuasnvallysmetalaleuaspilegluilealathrtyr385390395400arglysleuleugluglyglugluserargileserleuproleupro405410415asnpheserserleuasnleuarggluthrasnleuaspserleupro420425430leuvalaspthrhisserlysargthrleuleuilelysthrvalglu435440445thrargaspglyglnvalileasngluthrserglnhishisaspasp450455460leuglu465<210>158<211>466<212>prt<213>equuscaballus<400>158metserthrargservalsersersersertyrargargmetphegly151015glyproglythralaserargproserserserargsertyrvalthr202530thrserthrargthrtyrserleuglyseralaleuargproserser354045serargthrleutyrserserserproserglyvaltyralathrarg505560serseralavalargleuargserservalproglyvalargleuleu65707580glnaspservalasppheserleualaaspalaileasnthrgluphe859095lysasnthrargthrasnglulysvalgluleuglngluleuasnasp100105110argphealasertyrileasplysvalargpheleugluglnglnasn115120125lysileleuleualagluleugluglnleulysglyglnglylysser130135140argleuglyaspleutyrglugluglumetarggluleuargarggln145150155160valaspglnleuthrasnasplysalaargvalgluvalgluargasp165170175asnleualagluaspilemetargleuargglulysleuglngluglu180185190metvalglnarggluglualagluserthrleuglnserphearggln195200205aspvalaspasnalaserleualaargleuaspleugluarglysval210215220gluserleuglnglugluilealapheleulyslysleuhisgluglu225230235240gluileglngluleuglnalaglnileglngluglnhisvalglnile245250255aspvalaspvalserlysproaspleuthralaalaleuargaspval260265270argglnglntyrgluservalalaalalysasnleuglnglualaglu275280285glutrptyrlysserlysphealaaspleuserglualaalaasnarg290295300asnasnaspalaleuargglnalalysglngluserasnglutyrarg305310315320argglnvalglnserleuthrcysgluvalaspalaleulysglythr325330335asngluserleugluargglnmetargglumetglugluasnpheala340345350valglualaalaasntyrglnaspthrileglyargleuglnaspglu355360365ileglnasnmetlysgluglumetalaarghisleuargglutyrgln370375380aspleuleuasnvallysmetalaleuaspilegluilealathrtyr385390395400arglysleuleugluglyglugluserargileserleuproleupro405410415asnpheserserleuasnleuarggluthrasnleugluserleupro420425430leuvalaspthrhisserlysargthrleuleuilelysthrvalglu435440445thrargaspglyglnvalileasngluthrserglnhishisaspasp450455460leuglu465<210>159<211>275<212>prt<213>susscrofa<400>159serthrargthrvalsersersersertyrargargmetpheglygly151015proglythralaserargproserserserargsertyrvalthrthr202530serthrargthrtyrserleuglyseralaleuargproserthrser354045argserleuserthrserserproglyglyvalglytyrtyralathr505560argserseralavalargleuargserservalproglyvalargleu65707580leuglnaspalavalasppheserleualaaspalaileasnthrglu859095phelystrptyrlysserlysphealaaspleuserglualaalaasn100105110argasnasnaspalaleuargglnalalysglngluserasnglutyr115120125argargglnvalglnserleuthrcysgluvalaspalaleulysgly130135140thrasngluserleugluargglnmetargglumetglugluasnphe145150155160alavalglualaalaasntyrglnaspthrileglyargleuglnasp165170175gluileglnasnmetlysgluglumetalaarghisleuargglutyr180185190glnaspleuleuasnvallysmetalaleuaspilegluilealathr195200205tyrarglysleuleugluglyglugluserargileserleuproleu210215220proasnpheserserleuasnleuarggluthrasnleugluserleu225230235240proleuvalaspthrhisserlysargthrleuleuilelysthrval245250255gluthrargaspglyglnvalileasngluthrserglnhishisasn260265270aspleuglu275当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1
- 抗PD-L1纳米抗体及其编码序列和用途的制造方法与工艺
- 用于HPV‑相关病症的新方法、生物试验和生物标记物与制造工艺
- 用于去除血液中的促炎介质以及粒细胞和单核细胞的系统的制造方法与工艺
- 活化人CD40的抗体和针对人PD‑L1的抗体的组合疗法的制造方法与工艺
- 免疫原性组合产品的制造方法与工艺
- 白介素2的超级激动剂、部分激动剂和拮抗剂的制造方法与工艺
- 表达免疫应答刺激细胞因子以吸引和/或激活免疫细胞的基因修饰间充质干细胞的制造方法与工艺
- 一种研究北虫草多糖对机体的体液免疫应答和细胞免疫应答反应的方法与制造工艺
- 金黄色葡萄球菌肠毒素C2的核酸适配体C203及其筛选方法和应用与制造工艺
- 一种干扰乙肝病毒复制的siRNA、其构建方法及应用与制造工艺