DNA文库的制备方法以及试剂盒与流程
本发明涉及DNA文库的制备及靶向捕获方法以及试剂盒。尤其涉及血浆游离DNA文库、白细胞gDNA文库或组织来源的DNA文库的制备及靶向捕获方法以及试剂盒。
背景技术:
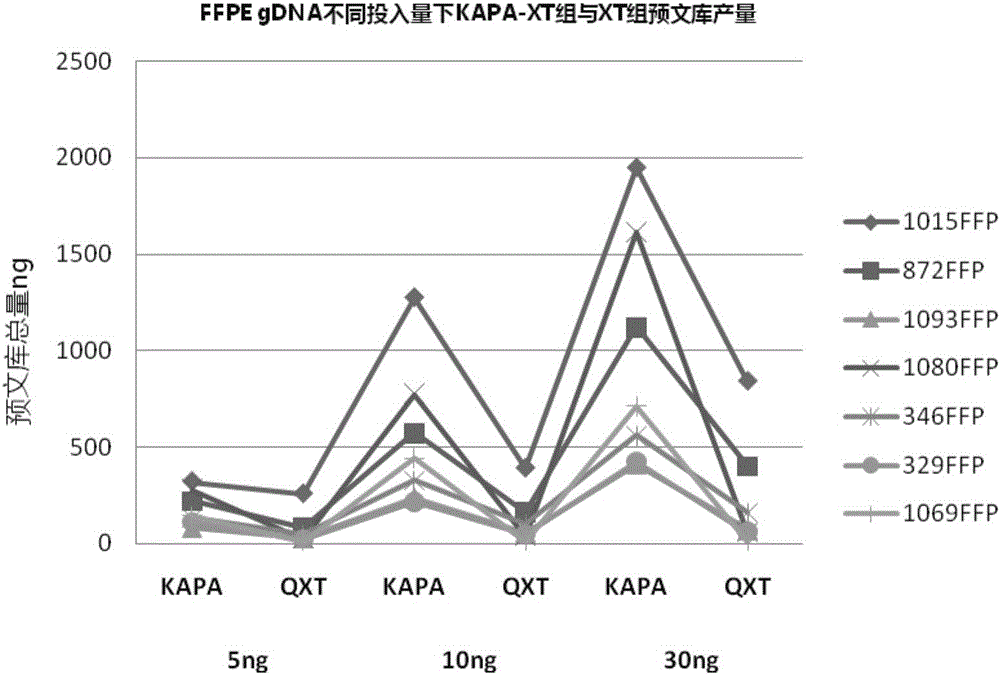
:研究发现,癌症患者血液中存在少量的游离DNA,即血浆游离DNA(cfDNA),而这些游离DNA中有一部分是来自于肿瘤细胞,这部分游离于血液中的肿瘤基因组片段被称为循环肿瘤DNA(ctDNA)。ctDNA作为一种新的肿瘤标志物,在肿瘤的个体化用药检测、复发及疗效监测等方面发挥着重要的作用。通过检测从肿瘤组织释放到血液中的ctDNA以获得癌症相关基因变异信息的方法,即为“液体活检”。近年来基于ctDNA的液体活检概念逐渐被医生们所接受和认可。它不仅可以辅助诊断实体瘤,而且能够实现耐药复发监测,疗效评估,是组织活检的有力补充。基于全基因组建库后探针捕获的二代测序法技术,能实现多基因多靶点的平行检测,且拥有极高的敏感性与特异性,能实现ctDNA的液体活检。因此,基于二代测序技术的ctDNA文库的构建对于实现液体活检以及肿瘤的辅助诊断、临床指导用药等都具有重要的意义。技术实现要素:本发明提供了一种DNA文库的制备方法以及试剂盒,所述方法适用于cfDNA、白细胞gDNA、以及来源于组织样本的DNA文库的构建。本发明的方法能够便于实现靶基因的所有变异类型,如突变、缺失、插入、融合、以及拷贝数扩增的检测,克服了目前在DNA水平上通过PCR的方法富集靶基因序列,但只能检测靶基因的突变和缺失,无法检测融合的缺点。本发明的DNA文库制备方法,是在SureSelectXT的建库方法基础上,对预文库以及终文库构建过程中的细节进行了改进,例如末端修复以及3’端加A在同一个反应体系中进行,改进杂交投入量、并对杂交捕获截断的阻断制剂进行优化,由此获得了更高的捕获效率,更高的文库多样性,和更均一的覆盖度,适合于微量以及低丰度突变的样本文库的构建,并实现了技术流程上的节约。本发明一方面涉及DNA文库的制备方法,其包括预文库和终文库的制备过程,其中所述的预文库制备过程包括DNA的制备、末端修复和3’端加A,接头连接,接头连接产物纯化,预文库扩增,扩增的预文库纯化,所述的终文库制备过程包括预文库杂交,捕获洗脱,终文库制备,以及终文库纯化过程,其中所述DNA文库为cfDNA文库、白细胞gDNA文库或组织来源的DNA文库。根据本发明第一方面任一项的制备方法,其中所述的末端修复和3’端加A是在一个反应体系中进行。根据本发明第一方面任一项的制备方法,其中末端修复和3’端加A的反应体系含有ER&AT缓冲液,ER&AT酶,以及cfDNA,并且ER&AT缓冲液:ER&AT酶:cfDNA的体积比为7:3:50。根据本发明第一方面任一项的制备方法,其中末端修复和3’端加A的反应体系条件为:20℃30分钟(85℃热盖)65℃30分钟(85℃热盖)4℃维持。根据本发明第一方面任一项的制备方法,其中预文库杂交过程中所用的阻断剂为双向短链阻断剂。根据本发明第一方面任一项的制备方法,其中所述的双向短链阻断剂结构如下:根据本发明第一方面任一项的制备方法,其中纯化步骤中所加入的磁珠量较常规减少一半。根据本发明第一方面任一项的制备方法,其中接头连接产物纯化过程中连接反应体系和磁珠体积比为1:0.8。根据本发明第一方面任一项的制备方法,其中扩增的预文库纯化过程中PCR体积和磁珠体积1:1.2。根据本发明第一方面任一项的制备方法,其还适用于白细胞gDNA、以及组织样本DNA的文库构建。根据本发明第一方面任一项的制备方法,其中进行白细胞gDNA、以及组织样本的DNA文库构建时还涉及打断时间、打断稀释液、以及漂洗缓冲液吹打位置的优化。根据本发明第一方面任一项的制备方法,其中所述白细胞gDNA、以及组织样本的DNA文库构建时打断时间为30s到150s。根据本发明第一方面任一项的制备方法,其中所述白细胞gDNA、以及组织样本的DNA文库构建时所用的打断稀释液为IDTE(10mMTris0.1MEDTA,pH8.0)。根据本发明第一方面任一项的制备方法,其中所述白细胞gDNA、以及组织样本的DNA文库构建时漂洗缓冲液吹打位置为PCR仪于为65℃下进行。根据本发明第一方面任一项的制备方法,其中还进一步包括纯化的预文库或终文库的质检、纯化的预文库或终文库的片段大小检测。本发明的第二方面提供了双向短链阻断剂。根据本发明第二方面任一项的双向短链阻断剂,其具体结构如下:所述双向短链阻断剂,能够在杂交过程中产生更好的阻断作用,使得杂交更彻底,结果更准确。根据本发明第二方面任一项的双向短链阻断剂在制备用于构建DNA文库的试剂盒中的应用,其中所述DNA文库为cfDNA文库、白细胞gDNA文库或组织来源的DNA文库。附图说明图1为预文库总量结果图。图1A为cfDNA预文库总量;图1B为WBCgDNA预文库总量,其中样本数为8。图2为FFPEgDNA不同投入量下KAPA-XT组与XT组预文库产量,其中样本数为7。其中KAPA即为如下的KAPA-XT组,QXT即为XT-30组。图3为文库浓度结果图。图3A为cfDNA结果,图3B为WBCgDNA文库结果,其中样本同图1。图4为文库中片段大小结果图。图4A为cfDNA结果,图4B为WBCgDNA文库结果。图5为捕获效率的结果比较图,其中横坐标表示上述8个样本的cfDNA和WBCgDNA的共16个文库。图6为文库复杂度和测序深度的结果图。图6A和图6B为cfDNA文库的复杂度(5000X)和测序深度结果;图6C和图6D为WBCgDNA文库的复杂度(5000X)和测序深度结果。图7为KAPA+XT组实验操作流程图。上述WBCgDNA即为白细胞来源的gDNA,KAPA-30即为如下的KAPA-XT组,XT-30即为如下的XT-30组。具体实施方式下面结合具体实施例进一步阐述本发明,应理解,以下实施例仅用于说明本发明而不用于限制本发明的保护范围。实施例1.实验分组实验分为两组进行,具体分组情况如下:第一组(XT-30组):根据现有的SureSelectXT方法的标准操作流程进行,并作为下面所述不同亚组的对照组,所用的cfDNA或白细胞来源的gDNA与各个亚组所用量保持一致。第二组(KAPA-XT组):该组进一步依据不同的样本来源(血浆来源的cfDNA,白细胞来源的gDNA,FFPE(福尔马林固定石蜡包埋)来源的gDNA)分为三个亚组。三个亚组依据下面所述的具体步骤进行。2.实验步骤2.1DNA提取以及质检依据《cfDNA提取标准流程》提取30ngcfDNA并进行洗脱,洗脱条件为室温,体积为55μL。50μL进入下面所述流程,3μL用来进行质量控制流程。在Qubit3.0HSKit试剂盒标准流程的基础上,进行了如下改进:白细胞的打断时间由90s调整为150s打断,打断稀释液由ddH2O调整为IDTE,漂洗缓冲液的吹打位置由操作台面调整为PCR仪内于65℃下进行,提取200nggDNA,然后进行洗脱,洗脱条件为室温,体积为55μL。50μL进入以下流程,3μL用来进行质量控制流程。提取FFPE来源的gDNA,并将FFPE来源的gDNA超声打断到200-250bp左右,打断时间设置为30s,60s,以及150s,为建库做准备。2.2末端修复,3’端加A1)本流程对接步骤2.1最后一步,打开KAPAHyperPrep96reactionKit(-20℃),取出ER&AT缓冲液以及ER&AT酶试剂置于冰上融化。2)设置PCR仪(BioRadT100或ABVeriti),具体程序(程序名为“ERA”)设置如下:20℃30分钟(85℃热盖)65℃30分钟(85℃热盖)4℃维持。3)依据步骤2.1分别定量准备30ng血浆cfDNA,200ng白细胞来源的gDNA,5ng、10ng、30ngFFPE来源的gDNA,终末体积为50μL。4)冰上配制末端修复和加A反应体系混匀液(表1),手指轻弹3-5次,上下颠倒混匀2-3次,台式微型离心机离心1-3秒。5)用P20移液器吸取10μL混匀液加入上述50μLDNA溶液中,上下轻柔吹打10次混匀,台式微型离心机短暂离心1-3秒。表1末端修复和3’端加A反应体系试剂反应体积ER&AT缓冲液7μLER&AT酶3μLcfDNA或片段化的gDNA50μL总量60μL6)放入PCR仪Bio-RadT100或ABVeriti中,使用程序“ERA”,(85℃热盖,20℃30分钟,65℃30分钟,4℃维持)。2小时内进入下一步。2.3接头连接1)准备如下表2所示的试剂:表2接头连接与纯化试剂2)设置程序:设置(BioRadT100orABVeriti)定义程序名为“LIG”20℃15分钟(85℃热盖)70℃10分钟(85℃热盖)4℃保持。3)冰上配制接头连接反应体系混匀液(表3),手指轻弹3-5次,上下颠倒混匀2-3次,台式微型离心机离心1-3秒。4)用P100移液器吸取50μL混匀液加入上述0.2mL管中,移液器保持50μL上下吹打5次混匀,台式微型离心机离心1-3秒。表3连接反应体系设置试剂预反应体积连接缓冲液30μLDNA连接酶10μLAdaptorOligoMix10μL末端修复混合液60μL总体积110ul5)在PCR仪(T100orABVeriti)上运行程序“LIG”(20℃15分钟,70℃10分钟,4℃维持(85℃热盖))。2.4接头连接产物纯化1)将AMPUREXP磁珠置于室温至少30分钟,上下颠倒2-3次,涡旋混匀5-10秒已经回复室温的AMPUREXP磁珠,使其均一化。2)按照每个文库600μL配制新鲜的75%乙醇。3)取EppendorfLoBind1.5mL离心管,按连接反应体系和磁珠体积1:0.8比例,先后加入88μL均一化的磁珠和110μL加接头的预文库。移液器P200吹打5次混匀,温和旋转孵育5分钟。4)将离心管置于磁力架。等待溶液澄清(约3-5分钟)。5)将离心管置于磁力架上不动,打开管盖,用移液器P200小心吸走澄清上清(约200μL),避免碰到磁珠。6)将离心管仍置于磁力架上,用移液器P1000在每管加入300μL新鲜配制的75%乙醇。7)等待1分钟使磁珠充分沉淀,期间沿水平方向缓慢旋转离心管一圈,吸走乙醇。8)重复6,7步骤一次。9)台式微型离心机离心1-3秒,将离心管重新放回磁力架静置30秒,使用P10移液器除净残留乙醇,保持管盖开启。10)室温2分钟使磁珠干燥,以磁珠表面不反光,磁珠块没有裂纹为准。11)加入28μLEB溶液,充分吹打混匀,室温孵育3分钟。12)将离心管置于磁力架2分钟,直至溶液澄清。13)移取上清27.5μL至新的0.2mLPCR管,冰上备用。2.5预文库扩增1)预文库扩增所需试剂以及反应条件参见表4和表5:表4PCR纯化准备表5预文库扩增PCR反应条件2)为避免交叉污染,在专用的干净区域或紫外线灭菌和带正压气流的PCR台配制PCR(除文库DNA的所有成分)反应液。3)按照表6准备反应体系混匀液(冰上配制),手指轻弹3-5次,上下颠倒混匀2-3次,台式微型离心机离心1-3秒。4)在“接头连接产物纯化”这一步中的PCR管中(含27.5μL接头纯化产物)每管用P100移液器加入22.5μL反应混匀液,上下轻柔吹打10次混匀。5)置于PCR仪上运行“PRE”程序。表6预富集PCR体系试剂预反应体积5xKAPAHiFiBuffer10μL10mMdNTPMix1.5μLSureSelectPrimer5μLPre-CapturePCRReversePrimer5μLKAPAHiFi1μL干净的连接混合液27.5μL总体积50μL2.6扩增的预文库纯化1)将AMPUREXP磁珠置于室温至少30分钟。2)按照每个文库600μL配制新鲜的75%乙醇。3)上下颠倒2-3次,涡旋混匀5-10秒已经回复室温的AMPUREXP磁珠,使其均一化。4)取EppendorfLoBind1.5mL离心管,按PCR体积和磁珠体积1:1.2比例,先后加入60μL均一化的磁珠和50μLPCR预文库。移液器P200调到100μL上下吹打5次,温和旋转孵育5分钟。5)将离心管置于磁力架。等待溶液澄清(约3-5分钟)。6)离心管仍置于磁力架不动,打开管盖,小心吸走上清(约110μL),避免碰到磁珠。7)离心管仍置于磁力架上,用移液器P1000在每管加入300μL新鲜配制的75%乙醇。8)等待1分钟使磁珠充分沉淀,期间沿水平方向缓慢旋转离心管一圈,吸走乙醇。9)重复7,8步骤一次。10)台式微型离心机离心1-3秒,将离心管重新放回磁力架静置30s,使用P10移液器除净残留乙醇,保持管盖开启。11)室温2分钟使磁珠干燥,以磁珠表面不反光,磁珠块没有裂纹为准。12)加入28μLddH2O,充分吹打混匀,室温孵育3分钟。13)将离心管置于磁力架2分钟,直至溶液澄清。14)移取上清27.5μL至新的0.2mLPCR管,冰上备用。2.7纯化的预文库质检将上述步骤获得的纯化预文库稀释。取1μL纯化后的预文库到一个新的1.5mLEP管中,加入10μLddH2O,P20移液器上下吹打混匀10次,其中1μL用作Qubit定量,其余用作下一步的LabchipQC(使用LabchipDNAHSKit试剂盒进行)。2.8预文库杂交1)准备表7中的试剂。2)程序设置:设置(BioRadS1000)定义程序名为“FASTHYB”表7预文库杂交试剂准备其中,预文库杂交过程中所用的阻断剂BLK为双向短链阻断剂,其具体结构如下:3)该杂交反应需要至少300ng预文库,根据Qubit定量结果,判断是否可以进行杂交。如果预文库产量大于1500ng,移取12μL纯化后预文库到48孔板。如果预文库产量大于750ng但小于1500ng,移取相应于750ng纯化后预文库到48孔板,用水补齐体积至12μL。如果预文库产量小于750ng,但大于300ng,可尝试杂交,移取所有预文库到48孔板,用水补齐体积至12μL。4)按表8常温配制预文库封闭体系,按表9常温配置探针体系。表8预文库封闭体系预文库封闭预反应体积SureSelectQXTFastBlockerMix5μLBLK1μL预文库12μL总体积(HybBuffer)18μL表9探针配置体系5)将含有预文库封闭体系的48孔板盖上8连管盖,放入PCR仪。运行程序“FASTHYB”程序95℃5分钟,至第三步65℃开始,暂停PCR程序,打开8连管盖。6)将相应的探针组分按每个样本12μL的体积用P20排枪迅速转移到已含有预文库组分的杂交板孔,总体积30μL,吹打5次混匀,每次取样探针组分需更换枪头。将八连管在板上盖盖紧,再贴上剪切成一半的microsealB贴膜以防止蒸干。运行杂交程序至程序结束(65℃保持)。如需过夜杂交,也可将杂交程序65℃保持过夜至16小时后进行下一步。2.9捕获洗脱1)设置恒温金属浴温度65℃。2)SCB/T1磁珠(为一种亲和链霉磁珠,选取来自于ThermoFisher65601的MyOneTMStreptavidinT1)室温平衡30分钟以上。3)按600μL/样本的用量将SureSelectWashBuffer2置于15ml锥形管内金属加热器65℃孵育。4)取出SCB/T1磁珠,上下颠倒混匀5次,涡旋混匀10秒,室温静置半小时以上,涡旋混匀10秒,按样品数分装入1.5mlLoBind管,每个样品需50μL,每个Lobind管最多放250μL。静置磁力架上3分钟,弃上清。5)每25μL原始磁珠加200μLSureSelectBindingBuffer,涡旋混匀3秒,短暂离心,静置磁力架上3分钟,弃上清。6)重复以上步骤2次,共3次。7)每25μL原始磁珠加入200μLSureSelectBindingBuffer,涡旋混匀3秒,重悬SCB/T1,分装200μL/管于LoBind管中。8)将杂交样本约28μL用P200转移至重悬的SCB/T1磁珠中,置于旋转混匀仪上,室温旋转孵育30分钟。9)将磁珠样品混合液管置于磁力架上静置3分钟,用P1000吸去约230μL上清。加入500μLSureSelectWashBuffer1,涡旋混匀5秒,置于旋转混匀仪上,室温旋转孵育15分钟。10)将磁珠样品混合液管磁力架上静置3分钟,用P1000吸去约500μL上清。用P20吸尽残留液体。11)加入200μL预热至65℃的SureSelectWashBuffer2,涡旋混匀10秒,置于干式混匀仪上,65℃震荡孵育10分钟,转速为300rpm。磁力架上静置3分钟,用P1000吸去约200μL上清,再用P20吸尽残留液体。11)重复步骤10两次,共三次。12)用P20除净样本管中残留液体,P100移液器加入30μLEB,上下吹打10次混匀,放置冰上备用。2.10终文库制备1)按表10准备以下试剂;依据表11设置PCR仪(BioRadS1000)程序“POST”。表10捕获文库扩增及纯化表11PostPCR设置2)取20μLSCB/T1结合文库的磁珠,涡旋混匀加到0.2mLPCR管中。3)按表12PostPCR体系所示的体系,依次加入0.2mlPCR管,用P100设置量程30μL吹打10次混匀,短暂离心,盖上盖子。在PCR仪上运行表“POST”程序。表12PCR体系设定2.11终文库纯化1)将PCR产物置于微型离心机快速离心1s,静置在96孔磁力架上5分钟,吸取50μL上清加入到新的1.5mLEppendorfLoBind管中。2)加入50μLAMPUREXP磁珠,混匀,涡旋混匀仪上室温孵育5分钟。3)将离心管置于磁力架。等待溶液澄清(约2分钟)。4)离心管仍置于磁力架不动,同时小心吸走澄清上清约100μL,不要碰到磁珠。5)离心管仍置于磁力架上,每管加入300μL新鲜配制的75%乙醇。等待1分钟使磁珠充分沉淀,期间沿水平方向缓慢旋转离心管一圈,吸走乙醇。75%乙醇重复洗涤一次。6)将离心管重新放回磁力架静置30s,使用P10移液器除净残留乙醇。7)开盖,室温2分钟使磁珠干燥,立刻进行下一步。过度干燥会严重降低溶解效率。8)加入30μLEB,充分吹打混匀,室温孵育3分钟。9)将离心管置于磁力架2分钟,直至溶液澄清。10)移取约30μL上清至新的1.5ml离心管,弃去磁珠。3实验结果3.1预文库总量以及预文库QC在与XT-30组投入量相同的情况下,KAPA+XT方法下的cfDNA和WBCgDNA预文库得率显著高于XT-30组,具体结果参见图1。选取质量不同的FFPEgDNA样本进行比对测试,KAPA+XT组的方法流程明显优于基于XT组的建库流程,10个PCR循环后的预文库得率明显高于XT组流程,具体结果参见表13以及图2。并且由于XT组预文库产量过低,因此未进行后续的测序比较。表13FFPEgDNA不同投入量下KAPA-XT组与XT组预文库产量3.2文库浓度测定纯化后文库的浓度。浓度过低(<10nM)则预示PCR循环数不够或者纯化丢失过多,过高(>50nM)也可能预示过分扩增或者非特异性捕获组成过多。本发明KAPA+XT组所构建的cfDNA以及白细胞gDNA文库浓度均显著高于XT-30组,具体结果参见图3。3.3纯化后文库片段大小检测通过检测文库的片段大小,判断文库是否正常。结果分析:KAPA+XT组中加接头的cfDNA的文库片段分布在160-170bp之间,白细胞gDNA的文库片段分布在200bp左右,参见图4,均符合文库构建的要求,文库构建结果正常。3.4捕获效率KAPA+XT组的捕获效率平均在67%左右,且比XT-30组更稳定,具体参见图5。3.5复杂度和测序深度cfDNA在相同投入量(30ng),同测序深度(5000×)下,KAPA+XT组和XT-30组在复杂度上保持较好的一致性,具体参见图6,且两个样本中,无论是文库复杂度还是测序深度,KAPA+XT组均优于XT-30组。3.6终文库构建所需要时间根据本发明的构建方法,节省了实验操作流程,时间按8个样本计算,手动操作时间为170分钟,总计实验时间约10小时,参见图7。按照标准的SureSelectXT方法,时间按8个样本计算,手动操作时间为230小时,总计实验时间约26小时。通过上述实验结果可以看出,本发明的方法相比标准方法在文库构建的各个指标方面均产生了更优异的技术效果,而这与上述提及的对标准方法的改进之处,以及所使用的例如双向短链阻断剂直接相关。实施例中所用试剂具体参见表14:当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1