由体细胞突变基因编码的HLA限制性表位的制作方法
技术领域
本发明涉及抗体产生领域。具体地,其涉及在单链或其他类型的抗体分子中含有抗体可变区的构建物。
背景技术:
癌症是癌基因和肿瘤抑制基因相继突变的结果(1)。理论上,体细胞突变是理想的治疗靶点,因为它们几乎未见于任何正常细胞(2)。即使这些突变的蛋白质产物通常仅略微不同于野生型(wt)形式,经常在于单个氨基酸,但这种不同对于有效的靶向是足够的。当蛋白质为酶,例如由BRAF编码的酶时,产生的结构改变可以提供用于结合具体的酶抑制剂的袋(3-5)。抗体是现代药剂最成功的类型之一,并已被显示能够特异性识别不同仅在于单个氨基酸或仅在于单个氨基酸的修饰的蛋白质(5-11)。但是,用于临床的所有抗体直接针对细胞表面或分泌的蛋白质而不是细胞内蛋白质。细胞内蛋白质不能接近大的分子例如抗体,但是不幸地,由突变基因编码的绝大多数异常表位不在细胞表面上(2)。
细胞内抗原,例如病毒组分,可以被免疫系统识别,但是这种识别是基于与细胞表面的人白细胞抗原(HLA)分子复合的蛋白水解加工的肽的识别(12)。实际上,癌症中10%至20%的由突变基因产生的表位(下文称为MANA,突变相关的新抗原)被预测结合常见的HLA型(12)。此外,可以结合这种肽-HLA复合物的T细胞的实例已经见于患者以及实验动物(13-16)。
体内产生的针对MANA的大多数T细胞应答是“专用的(私人的,private)”,即直接针对由存在于个别患者或小鼠的癌症中但不常见于患者且不驱动瘤生长的偶发突变(伴随突变,passenger mutations)编码的突变表位(2)。靶向这种抗原的免疫剂仅用于具有特定MANA的个别患者的治疗(16-20)。
本领域持续地需要鉴定用于包括但不限于癌症的疾病的新的治疗剂、诊断剂和分析剂。
技术实现要素:
根据本发明的一个方面,提供了包括抗体可变区的分离的分子。抗体可变区特异性结合人白细胞抗原(HLA)分子、β-2-微球蛋白分子和作为蛋白质的部分的肽的复合物。肽包括在蛋白质的细胞内表位中的突变残基。当HLA分子不在复合物中时,分子不特异性结合HLA分子。分子也不特异性结合野生型形式的肽。任选地,当不存在于HLA复合物内时分子不特异性结合肽。分离的分子可以用于检测或监测癌细胞或用于治疗癌症。
根据本发明的另一个方面,提供了用于从核酸文库中选择scFv或Fab或TCR的方法,所述scFv或Fab或TCR特异性结合(a)人白细胞抗原(HLA)分子、(b)β-2-微球蛋白分子和(c)蛋白质肽部分的第一形式的复合物。第一形式包括突变残基,并且突变残基在蛋白质的细胞内表位中。当HLA分子不在复合物中时,scFv或Fab或TCR不特异性结合HLA分子。scFv或Fab或TCR不特异性结合处于其野生型形式的肽。方法包括如下步骤:在包括(a)结合(b)HLA和(c)β-2-微球蛋白的肽部分的第二形式的竞争复合物的存在下阳性地选择结合所述复合物的scFv或Fab或TCR。第二形式选自野生型形式和具有与第一形式不同的突变残基的肽。在任选的步骤的连续执行过程中,复合物和竞争复合物的量可被改变以便竞争复合物与相关复合物的比例增加。
根据本发明的另一个方面,提供了用于从核酸文库中选择特异性结合蛋白质的肽部分的第一形式的scFv或Fab或T细胞受体。第一形式包括突变残基,其在蛋白质的细胞内表位中。scFv或Fab或TCR不特异性结合处于其野生型形式的肽。方法包括如下步骤:在肽部分的竞争物第二形式的存在下阳性地选择结合第一形式的scFv或Fab或T细胞受体,其中第二形式选自野生型形式和具有与第一形式不同的突变残基的肽。
本领域技术人员在阅读说明书后将会明白的这些和其他实施方式为本领域提供了用于接近(accessing)通常在细胞内但在疾病状况中展示在特定细胞表面上的表位的剂。
附图说明
图1.MANAbody的产生。利用在中心突出显示的竞争性噬菌体选择概述MANAbody产生的方法。
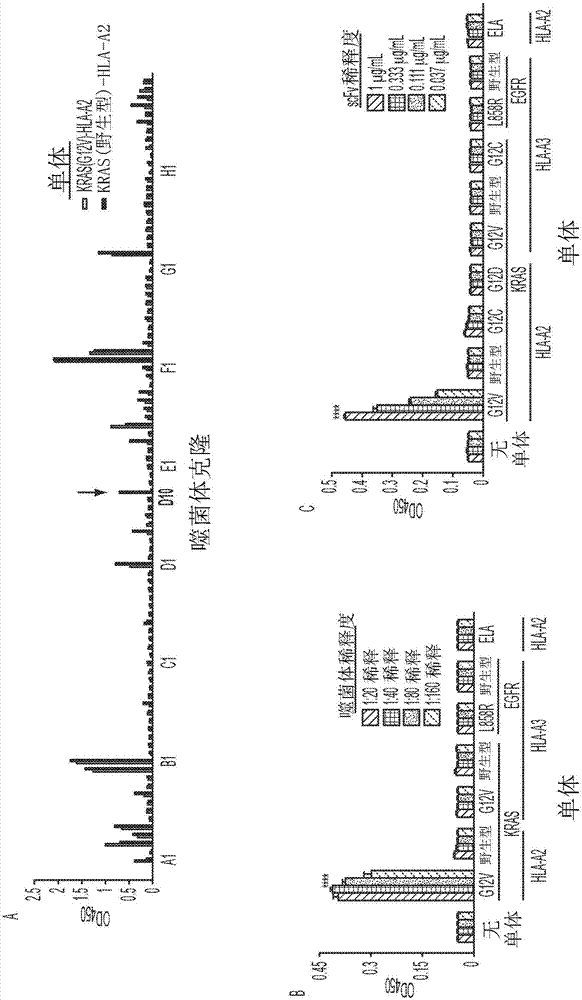
图2A-2E.噬菌体和纯化的scFv与突变单体的选择性结合。将用指示的肽、β-2-微球蛋白和HLA分子折叠的单体与不同稀释度的噬菌体克隆或纯化的scFv温育,然后用抗-M13(对于噬菌体)或抗-Flag标签(对于scFv)抗体进行ELISA。(图2A)在最后选择阶段后收集和扩增的噬菌体克隆对于KRAS(G12V)-HLA-A2结合物(binder)的选择性结合。克隆D10通过红色箭头突出显示。(图2B)噬菌体克隆D10与不同单体的选择性结合。在1:80稀释度时,KRAS(G12V)-HLA-A2与每种其它单体相比,****,P<0.0001。(图2C)纯化的D10scFv与不同单体的选择性结合。在1μg/mL稀释度时,KRAS(G12V)-HLA-A2与每种其它单体相比,****,P<0.0001。(图2D)噬菌体克隆C9与不同单体的选择性结合。在1:900稀释度时,EGFR(L858R)-HLA-A3与每种其它单体相比,****,P<0.0001。(图2E)纯化的C9scFv与不同单体的选择性结合。在1μg/mL稀释度时,EGFR(L858R)-HLA-A3与每种其它单体相比,****,P<0.0001。(B-E),用野生型或指定的突变肽和HLA分子折叠的单体在x轴示出。ELA:阴性对照肽,无单体(No Monomer):孔用链霉亲和素包被而没有单体结合。
图3A-3D.候选噬菌体克隆或纯化的D10scFv与在细胞表面展示突变肽的细胞的选择性结合。T2或T2A3细胞用指示的肽脉冲(pulsed),然后与D10噬菌体(图3A)、纯化的D10scFv(图3B)或C9噬菌体(图3C)温育,然后通过流式细胞术分析染色的细胞。ELA,LLG:阴性对照;对于C9噬菌体,KRAS(野生型)被用作阴性对照肽。(图3D),scFv介导的补体依赖的细胞杀伤。在T2细胞被用指示的肽脉冲或未脉冲或脉冲后,通过将T2细胞与10%兔补体和预缀合至抗-V5抗体的D10scFv或D10-7scFv温育来执行CDC试验。CellTiter-被用于评估细胞的生存能力。在0.66nM(在x轴上-0.18)抗体浓度时,KRAS(G12V)/D10-7与所有其它点相比,***P<0.001。在0.66nM抗体浓度时,KRAS(WT)/D10-7与未脉冲的/D10-7相比,ns,不显著(P=0.488)。
图4A-4B.D10MANAbody的选择性亲和力。(图4A)D10MANAbody与KRAS(G12V)-HLA-A2的选择性结合。将用指示的肽和HLA分子折叠的单体与不同稀释度的D10MANAbody温育,然后用抗-人IgG抗体进行ELISA。在1μg/mL稀释度时,KRAS G12V HLA-A2与每种其它单体相比,***,P<0.0001。(图4B)D10MANAbody与在细胞表面展示突变肽的细胞的选择性结合。T2细胞未脉冲或被用指示的肽脉冲,然后与D10MANAbody或与同种型对照抗体温育,然后通过流式细胞术分析染色的细胞。
图5.在噬菌粒中的scFv/M13pIII开放阅读框的线性呈现。pelB,pelB周质分泌信号;VL和VH,scFv中的轻链和重链;myc,myc标签;TEV,TEV蛋白酶切割识别序列;M13pIII,M13pIII外壳蛋白质。
图6A-6C.竞争选择的流程图。选择方法由10轮选择和扩增组成,其被分为三个阶段:富集阶段(图6A;第1-3轮),竞争阶段(图6B;第4-8轮)和最后选择阶段(图6C;第9-10轮)。在每轮竞争中使用的突变(MUT)单体与野生型(WT)竞争单体的比例被示出。
图7A-7B.在不同选择阶段后噬菌体的结合。将用指示的肽和HLA分子折叠的单体与不同稀释度的噬菌体(全体地)温育,然后用抗-M13抗体进行ELISA。(图7A)在富集阶段后收集的噬菌体的结合。(图7B)在最后选择阶段后收集的噬菌体的结合。KRAS(G12V),具有G12V突变的KRAS肽;KRAS(WT),野生型KRAS肽。
图8.纯化的D10scFv不结合未与HLA分子复合的KRAS肽或变性单体。将单独的生物素化的KRAS肽、天然单体或热变性单体与不同稀释度的纯化的scFv温育,然后用抗-Flag标签抗体进行ELISA。KRAS(G12V),具有G12V突变的KRAS肽;KRAS(WT),野生型KRAS肽;无单体(No Monomer),孔用链霉亲和素包被而没有单体结合。
图9.纯化的D10-7scFv与不同单体的选择性结合。将用指示的肽、β-2-微球蛋白和HLA分子折叠的单体与不同稀释度的D10-7scFv温育,然后用抗-Flag标签抗体进行ELISA。肽在柱状图下的线上示出,结合单体的HLA蛋白质类型在所述肽下的线上示出。在0.037μg/mL稀释度时,KRAS(G12V)-HLA-A2与每种其它单体相比,****,P<0.0001。
图10.产生C9噬菌体的修改的竞争选择的流程图。选择方法由9轮选择和扩增组成,其被分为三个阶段:富集阶段(第1-5轮),竞争阶段(第6-8轮)和最后选择阶段(第9轮)。在每轮竞争中使用的突变(MUT)单体与野生型(WT)竞争单体的比例被示出。
图11A-E.通过W6/32抗体染色评估的肽装载效率。T2或T2A3细胞未脉冲或用指示的肽脉冲,然后与W6/32抗体温育,然后通过流式细胞术分析染色的细胞。ELA:对照肽。图11A:KRAS(G12V),图11B:KRAS(WT),图11C:EGFR(L858R),图11D:EGFR(WT),图11E:ELA对照。
图12.D10噬菌体与在细胞表面展示突变肽的细胞的选择性结合。T2细胞用指示的肽脉冲,然后与D10噬菌体、作为对照的C9噬菌体或无噬菌体温育,然后通过流式细胞术分析染色的细胞。KRAS(G12V),具有G12V突变的KRAS肽;KRAS(WT),野生型KRAS肽;ELA,不相关肽。
图13.C9噬菌体与在细胞表面展示突变肽的细胞的选择性结合。T2A3细胞用指示的肽脉冲,然后与C9噬菌体或无噬菌体温育,然后通过流式细胞术分析染色的细胞。EGFR(L858R),具有L858R突变的EGFR肽;EGFR(WT),野生型EGFR肽;KRAS(WT),野生型KRAS肽。
图14.W6/32抗体介导的补体依赖的细胞杀伤。在T2细胞用指示的肽脉冲或未脉冲后,通过将T2细胞与W6/32抗体和10%兔补体温育来执行CDC试验。CellTiter-被用于评估细胞的生存能力。
图15.D10MANAbody与在细胞表面展示突变肽的细胞的选择性结合。T2细胞用指示的肽脉冲,然后与或不与D10MANAbody温育,然后通过流式细胞术分析染色的细胞。还使用了对照抗体(7A)。
图16显示了使用噬菌体上呈现的β联蛋白S45F特异性scFv的酶联免疫吸附试验。CTNNB1S45F scFv候选物(E10):噬菌体ELISA(归一化的)。图例指示了使用的噬菌体的稀释度。使用的单体和噬菌体克隆在X轴被标记。*指示相同的序列。ELISA使用表达E10或G7的噬菌体。两种scFv显示了比野生型更显著的对突变表位(CTNNB1S45F)的结合。ELISA使用表达E10或G7的噬菌体。两种scFv显示了比野生型更显著的对突变表位(CTNNB1S45F)的结合。当与野生型复合物相比时,两种scFv显示了对突变表位HLA-A3复合物的增加的结合。见实施例11。
图17显示了E10 CTNNB S45F噬菌体染色的流式细胞试验结果。噬菌体克隆对CTNNB S45F肽-脉冲的细胞是特异性的。scFv直接针对CTNNB1(β联蛋白)S45F突变。W6/32数据显示了相对于b2m(阴性对照)的抗体结合的增加,其显示突变肽和野生型肽可以在存在于T2A3细胞系上的HLA-A3复合物上被呈递。E10噬菌体染色显示,相对于对照肽(600-800MFU),scFv特异性结合S45F表位(80,400k MFU)。标记1-5的行证明,突变的和野生型的β联蛋白表位可以在细胞表面HLA-A3复合物上被呈递。标记6-9的行显示与用指示的肽脉冲的T2A3细胞结合的E10噬菌体的平均荧光强度(MFI)。E10噬菌体特异性识别突变肽,并且不与细胞表面呈递的用野生型肽或对照(K3WT)肽脉冲的复合物结合。直方图提供了E10噬菌体的特异性的可选的表示。见实施例11。
图18显示了补体依赖的细胞毒性试验(CDC)结果。在具有E10(CTNNB1S45F HLA-A3)scFv:抗-V5缀合物的T2A3细胞上用CTG(Cell Titer GloTM)进行CDC。在用S45F脉冲的细胞条件下在E10:V5缀合物系列中看到的相对萤光素酶单位的减少证明了对呈现β联蛋白S45F突变的细胞的特异性杀伤。该试验按照Proc Natl Acad Sci U S A.2015,8月11日;112(32):9967-72中所述进行,除了仅使用了单一的抗体浓度(10ug/ml)。T2A3细胞没有用肽进行脉冲,或用野生型或突变的β联蛋白肽进行脉冲。当仅与补体血清温育时,用肽脉冲的细胞经历了CDC依赖的细胞死亡。见实施例11。
图19.组合的EGFR T790M 9-mer和10-mer命中(14个独特的):噬菌体上清和沉淀的噬菌体ELISA。两个实验(噬菌体上清和沉淀的噬菌体ELISA)分别在左侧和右侧示出。图例指示了测试的单体(HLA类型);数据证明噬菌体对于被预测呈现这种突变的9个和10个氨基酸表位的高特异性。如所指示的,该图显示了噬菌体上清或沉淀的噬菌体的ELISA测试的结果。包括D3E6、D2D8和D2D6的某些噬菌体克隆在上清测试后被进一步分析。观察到了对于所有三个候选物的高特异性。EGFR T790对照肽不是生物相关的(野生型)对照,而是与用于竞争淘选的T790M 9-mer高度相似的序列。见实施例12和13。
图20(表)和21(直方图)显示了在表中标记1-7的样品的结果,证明两个突变EGFR T790M表位(9-mer和10-mer)都可以在细胞表面HLA-A2复合物上被呈递。在图20的表中标记8-10的行显示结合用指示的肽脉冲的T2细胞的D3E6噬菌体的平均荧光强度(MFI)。D3E6噬菌体特异性识别突变肽,并且不结合细胞表面呈递的用对照肽脉冲的复合物。直方图(图21)是显示D3E6噬菌体的特异性的可选的表示。D3E6噬菌体使得突变体而不是野生型790肽脉冲的细胞染色,并且没有使ELA(阴性HLA-A2对照肽)染色。见实施例12和13。
图22-25(图22的表和图23-25的流式细胞术)进一步确认D3E6、D2D8和D2D6克隆特异性识别突变肽,并且不结合细胞表面呈递的用对照肽脉冲的复合物。图23:T2细胞的EGFR T790M克隆1(“D3E6”)染色;图24:T2细胞的EGFR T790M克隆2(“D2D8”)染色;图25:T2细胞的EGFR T790M克隆3(“D2D6”)染色。见实施例12和13。
图26显示了测试沉淀的噬菌体的ELISA结果。对于F10候选物,观察到了对HLA-A2中KRAS G12V的高特异性。图例指示了使用的噬菌体的稀释度。使用的单体在X轴上指示。见实施例10。
图27提供了流式细胞术试验结果的直方图,显示了F10亲和力成熟的变体相对于野生型对照可以特异性识别T2细胞上脉冲的突变KRAS表位。见实施例10。
具体实施方式
发明人已经开发了产生和鉴定抗体可变区的方法,所述抗体可变区选择性靶向含有常见HLA类型的复合物,所述HLA类型结合常见突变癌基因的肽产物。由于这些HLA-肽复合物被预期唯一存在于癌细胞或其他疾病相关细胞的表面,靶向它们的抗体原则上可用于治疗或监测目的。这些用于鉴定抗体可变区的相同方法还可以用于鉴定T细胞受体。
癌基因和肿瘤抑制基因中的突变驱动肿瘤发生,它们的蛋白质产物形成正常细胞所没有的治疗靶。但是,几乎所有这样的突变表位都位于细胞的内部,在细胞质或细胞核中,使直接针对突变体的免疫治疗复杂化。本文描述的抗体可变区和T细胞受体通过靶向展示在细胞表面上的形式克服了在细胞内的细胞内靶的屏障。然而,本文描述的方法和方式不仅可用于肿瘤抑制基因和癌基因,还可用于偶发突变(不是致癌作用的驱动者)以及用于作为体细胞突变发生的产物或作为体细胞突变发生的结果在细胞表面表达的其它蛋白质。
可靶向的细胞内蛋白质的实例包括但不限于EGFR、KRAS、NRAS、HRAS、p53、PIK3CA、ABL1、β-联蛋白和IDH1/2。为了具有最广泛的适用性,期望选择癌症群体中普遍存在的突变。这种突变的实例包括在残基EGFR L858、KRAS G12、KRAS G13、HRAS G12、NRAS G12、HRAS Q61、NRAS Q61、IDH1R132、β-联蛋白S45、IDH2R140和IDH2R172中的那些。常见突变包括EGFR L858R、KRAS G12V、KRAS G12C、KRAS G12D、HRAS Q61P、NRAS Q61P、HRAS Q61R、NRAS Q61R、HRAS Q61K、NRAS Q61K、EGFR T790M、IDH1R132H、β-联蛋白S45F、IDH2R140Q和IDH2R172K。但是,即使是编码细胞内表位的私人或个人疾病特异性的突变可为scFv或Fab或T细胞受体的靶。
可以制造和筛选的文库包括产生有用的特异性结合分子例如scFv、Fab和TCR的任何文库。结合分子的所有组分(repertoire)的复杂度优选得非常高。文库可在任何适合的载体系统中制造,包括但不限于M13噬菌体、核糖体和酵母。对于Fab文库参见Lee et al.,J Mol Biol.“High-affinity human antibodies from phage-displayed synthetic Fab libraries with a single framework scaffold,”2004,7月23日;340:1073-93。对于T细胞受体文库参见Kieke et al.,“Selection of functional T cell receptor mutants from a yeast surface-display library,”Proc Natl Acad Sci U S A.1999;96:5651–5656。对于核糖体展示文库参见Stafford et al.,Protein Eng Des Sel.“In vitro Fab display:a cell-free system for IgG discovery.”2014;27:97-109。文库可使用例如合成的寡核苷酸、合成的三聚物或合成的脱氧核糖核苷酸来制造。每种选择都允许混合物的偏倚(bias)以使最终的文库组成偏倚。
在文库中需要的scFv或Fab或TCR的稀少某种程度上是由于期望的靶的性质。期望的靶包括HLA分子、β-2-微球蛋白蛋白质和肽的复合物。但是,在该整个复合物中,期望的scFv或Fab或TCR将仅识别含有突变残基——最可能是一个氨基酸替换另一个氨基酸——的特定表位。此外,其将不特异性识别其中残基为野生型的相同的大分子复合物。由于这种极其窄的聚焦,除了文库中非常大量的多样性之外,还需要强的选择方法。已经设计了针对期望的scFv或Fab或T细胞受体的阳性选择步骤,其在竞争复合物的存在下执行。竞争复合物包括结合HLA和β-2微球蛋白的肽的野生型形式。可选地,竞争复合物可包括具有与突变肽高度相似的序列的肽,例如具有一个或多个另外的突变残基的肽或在与突变肽相同残基处具有替换的非野生型残基的肽。阳性选择剂包括HLA、β-2-微球蛋白和“突变”肽。任选地,竞争选择步骤将被重复执行。随着所述步骤被重复执行,竞争复合物与阳性选择剂的比例可增加。任选地,在竞争淘选之后是使用竞争复合物的阴性选择步骤。任选地,竞争复合物和/或阳性选择剂可展示或表达在用于选择步骤的细胞的表面上。在本发明的可选方面,这种类型的选择方法可用于淘选识别不是HLA/β-2微球蛋白复合物的部分的蛋白质或肽中单个氨基酸差异的结合分子。在另一个选项中,肽不表示细胞内表位。
用于呈递具有突变残基的肽的HLA分子可来自任何HLA基因(A、B、C、E、F和G)和这些基因的等位基因。更普遍的基因和等位基因例如HLA-A2、HLA-A3和HLA-B7将在某些群体的人类患者中有更广泛的用途。可使用的其他HLA基因为HLA DP、DM、DOA、DOB、DQ和DR。
当特异性结合(1)HLA分子、(2)β-2-微球蛋白和(3)包括突变残基(发现于完全的天然蛋白质中的细胞内表位)的肽的复合物的有用分子被鉴定时,它们可被用于多种目的和多种衍生物中。分子可被结合或连接至可检测标记。可检测标记可以是本领域已知的任何标记,包括但不限于放射性核素、生色团、酶和荧光分子。这些分子可被用于例如监测抗肿瘤治疗或检测样品中的癌细胞或诊断癌症。可选地,分子可以被结合、缀合或连接至治疗剂。这些治疗剂可以通过鉴定的scFv或Fab或T细胞受体特异性靶向表达所述蛋白质的细胞。可有效地制造的所述鉴定的分子的另外的衍生物为嵌合抗原受体(CAR)。这种衍生物包括:作为单个蛋白质的部分,包括抗体可变区的所述鉴定的分子、铰链区、跨膜区和细胞内结构域。参见例如Curran et al.,“Chimeric antigen receptors for T cell immunotherapy:current understanding and future directions,”J.Gene Med 2012;14:405-415。如实施例中所述,有用的分子的CDR序列可并入完整抗体以形成MANAbody。可选地,有用的分子不是完整抗体分子的部分。有用的分子还可被包括作为具有另外的scFv/抗体例如抗-CD3scFv的嵌合蛋白质的部分,以形成双特异性靶向剂。这种嵌合蛋白质可用于靶向针对肿瘤的T细胞,诱导抗肿瘤活性。
本领域已知的任何诊断技术,具体地任何免疫诊断技术可以与所述有用的分子一起使用。例如它们可以被用于作为组织样品或组织匀浆的样品。它们可以被用于免疫组织化学、ELISA、免疫沉淀、免疫印迹等。检测将依赖于被连接或使用以鉴定免疫复合物的可检测标记。任何检测技术可被使用。治疗剂施用可以利用适合施用抗体或特异性结合分子的任何已知手段来完成。例如施用可通过注射或输注到外周循环系统或瘤内、脊柱内、小脑内、腹膜内等。
发明人已经建立了用于产生选择性结合嵌入HLA-β-2微球蛋白复合物内的突变肽的scFv的方法。使用该方法,发明人获得了当与两种常见的HLA类型(分别为A2和A3)复合时,针对两种常见突变癌基因(KRAS和EGFR)的产物的scFv。这些scFv结合细胞表面上的肽-HLA复合物并且当补体存在时可以杀伤这些细胞。将scFv转变为含有Fc区的完整二价抗体有时导致亲和力损失(46,47)。但是,发明人成功地使用D10scFv序列产生了完整抗体,并且这种MANAbody保留了scFv的特异性(图4B、图15)。发明人还没有尝试使用针对与HLA-A3复合的突变EGFR肽的C9scFv来产生MANAbody。
过去已经产生了针对肽-HLA复合物的其它抗体,称为TCR模拟物(TCRmimics)(48-49)。发明人研究的第一个重要方面是产生基于抗体的试剂,该试剂差别地识别含有仅有单个氨基酸不同的肽的HLA复合物。发明人研究的第二个重要方面是变体肽通常发现于人类癌症中。
在癌症诊断和治疗中的最大挑战是特异性——开发识别或杀伤癌细胞而不是正常细胞的试剂。目前对于强有力的免疫治疗剂例如嵌合抗原受体和双特异性抗体的更广泛实施来说,特异性的相对缺乏成为主要阻碍(57-60)。在该上下文中,改变癌症驱动基因的编码蛋白质的特异性体细胞突变表现出以无与伦比的方式(以独特地方式,in unparalleled fashion)区分癌细胞与正常细胞的生化特征。本文描述的工作的优势为,其证明了产生在临床相关环境中(细胞表面)识别这些改变的蛋白质的高特异性试剂的可行性。这为进一步探索这种试剂和将其并入到适合的诊断和治疗载体中打好了基础(铺平了道路,set the stage)。
上述公开内容概述了本发明。本文公开的所有参考文献清楚地通过引用被并入。通过参考下文具体实施例可以获得更完全的理解,本文提供的具体实施例仅用于示例目的,不意欲限制本发明的范围。
实施例1
材料和方法
细胞系。T2细胞(ATCC,马纳萨斯,VA)在37℃在5%CO2下被培养在具有10%FBS(GE Hyclone,Logan,Utah,USA)、1%青霉素链霉素(Life Technologies)和20IU/mL重组人IL-2(ProleukinTM,Prometheus Laboratories)的RPMI-1640(ATCC)中。T2A3细胞(由约翰·霍普金斯大学(JHU)的Eric Lutz和Liz Jaffee惠赠)生长在与T2细胞相同的条件下,但还加入500ug/mL遗传霉素(Geneticin)(Life Technologies)和1×非必需氨基酸(Life Technologies)。
噬菌体展示文库构建。使用混合和裂池简并(mixed and split pool degenerate)寡核苷酸合成法在DNA 2.0(Menlo Park,CA)合成了寡核苷酸。寡核苷酸被并入到pADL-10b噬菌粒(Antibody Design Labs,San Diego,CA)。该噬菌粒含有F1起点,和由乳糖操纵基因(operator)和乳糖阻抑蛋白(repressor)组成的转录阻抑蛋白单元以限制非诱导表达。scFv用pelB周质分泌信号合成并被亚克隆到乳糖操纵基因的下游。其后为TEV蛋白酶切割识别序列的myc表位标签被立即放置在重链可变区的下游,之后在框中是全长M13pIII外壳蛋白质序列。(图5)通过对从小部分连接产物的转化获得的45个随机克隆进行Sanger测序确认成功的克隆。所述克隆中的24个含有期望序列或沉默突变,4个含有框架区内的框内突变,17个含有一个或多个碱基对缺失,指示53%的成功合成和克隆分数。这随后如下文讨论的下述文库电穿孔确认。
将10ng的连接产物在冰上与10μL电转感受态SS320细胞(Lucigen,Middleton,WI)和14μL双蒸水(ddH2O)混合。将该混合物使用Gene Pulser电穿孔系统(Bio-Rad,Hercules,CA)进行电穿孔,并在37℃在Recovery Media(Lucigen)中恢复60min。将用60ng连接产物转化的细胞合并然后铺板在含有补充有羧苄青霉素(100μg/mL)和2%葡萄糖的2×YT培养基的24-cm×24-cm板上。细胞在37℃生长6小时,然后在4℃放置过夜。为测定每个电穿孔系列的转化效率,取小份并通过连续稀释滴定。板上生长的细胞被刮到850mL具有羧苄青霉素(100μg/mL)和2%葡萄糖的2×YT培养基中,最终OD600为5-15。取所述850mL培养物中的2mL并~1:200稀释以达到0.05-0.07的最终OD600。向剩余的培养物加入150mL无菌甘油,然后迅速冷冻以产生甘油储存物。将稀释的细菌培养至OD600为0.2-0.4,用M13K07辅助噬菌体(NEB,Ipswich,MA or Antibody Design Labs)以1MOI感染,然后允许其在37℃恢复30min,然后在37℃再摇动30min。将培养物离心,将细胞重悬在具有羧苄青霉素(100μg/mL)和卡那霉素(50μg/mL)的2×YT培养基中,然后在30℃培养过夜用于噬菌体生产。第二天早上,将细菌培养物分装到50mLFalcon管中并离心沉淀(pellet)两次,第一次转速3000g,然后转速12000g,以获得澄清上清。将装载有噬菌体的上清用20%PEG-8000/2.5M NaCl溶液以1:4的PEG/NaCl:上清的比例在冰上沉淀40min。沉淀后,将来自每50mL培养物的噬菌体以12,000g离心40分钟,然后重悬在1mL体积的1×TBS、2mMEDTA中。将来自多个管的噬菌体合并,再次沉淀,然后在15%甘油中重悬至1×1013cfu/mL的平均滴度。获得的转化体总数被测定为5.5×109。将文库分装并在-80℃保存在15%甘油中。
完整噬菌体文库的下一代测序(Next-generation sequencing)。使用在CDR-H3区侧面的如下引物扩增来自文库的DNA(正向:GGATACCGCTGTCTACTACTGTAGCCG,SEQ ID NO:1反向:CTGCTCACCGTCACCAATGTGCC,SEQ ID NO:2)。将另外的分子条形码序列并入在这些引物的5'端以帮助清楚地枚举不同的噬菌体序列。PCR扩增和测序的程序以前公开在参考文献(1)中。使用定制的SQL数据库处理和翻译序列,使用Microsoft Excel分析核苷酸序列和氨基酸翻译物。
肽和HLA-单体。被预测结合HLA-A2的野生型KRAS肽(KLVVVGAGGV;SEQ ID NO:3),被预测结合HLA-A2的突变KRAS(G12V)肽(KLVVVGAVGV;SEQ ID NO:4),被预测结合HLA-A2的突变KRAS(G12C)肽(KLVVVGACGV;SEQ ID NO:5),被预测结合HLA-A2的突变KRAS(G12D)肽(KLVVVGADGV;SEQ ID NO:6),被预测结合HLA-A3的突变KRAS(G12V)肽(VVGAVGVGK;SEQ ID NO:7),被预测结合HLA-A3的突变KRAS(G12C)肽(VVGACGVGK;SEQ ID NO:8),被预测结合HLA-A3的突变EGFR(L858R)肽(KITDFGRAK;SEQ ID NO:9),被预测结合HLA-A3的野生型EGFR肽(KITDFGLAK;SEQ ID NO:10),和被预测结合HLA-A3的野生型KRAS肽(VVGAGGVGK;SEQ ID NO:11),以及HLA-A2阴性对照肽ELA(ELAGIGILTV;SEQ ID NO:12)和LLG(LLGRNSFEV;SEQ ID NO:13)由Peptide 2.0(Chantilly,VA)以>90%的纯度合成。将肽以10mg/mL重悬在DMSO中并保存在-80℃。HLA-A2和HLA-A3单体通过将重组HLA与肽和β-2微球蛋白再折叠合成,通过凝胶过滤纯化,并且生物素化(Fred Hutchinson Immune Monitoring Lab,Seattle,WA)。确认单体被折叠,然后通过使用仅识别折叠HLA的W6/32抗体(BioLegend,San Diego,CA)执行ELISA来选择(59)。识别折叠和未折叠HLA的兔抗-HLA-A抗体EP1395Y(Abcam,Cambridge,MA)被用作非折叠单体结合ELISA板的对照。
选择结合突变KRAS-HLA-A2的噬菌体。将含有HLA和β-2-微球蛋白蛋白质的生物素化单体缀合至MyOne T1链霉亲和素磁珠(Life Technologies,Carlsbad,CA)或链霉亲和素琼脂糖(Novagen,Millipore,Darmstadt,Germany)。将生物素化单体与25μL MyOne T1珠或100μL链霉亲和素琼脂糖在封闭缓冲液(PBS,0.5%BSA,0.1%叠氮化钠)中在室温(RT)温育1小时。在初始温育后,将复合物用1ml封闭缓冲液洗涤3次,然后重悬在100μL封闭缓冲液中。
富集阶段:选择的富集阶段由第1-3轮组成。在第1轮中,将表示文库的250倍覆盖的1.4×1012的噬菌体(140μL)在25μl洗涤的裸MyOne T1珠和1μg(100μl)缀合至MyOne T1珠的热变性HLA-A2的混合物中温育30分钟。应该注意,在热变性后,仅生物素化HLA分子,而不是肽或β-2-微球蛋白,能够结合MyOne T1珠。该步骤称为“阴性选择(负选择,negative selection)”,这对于去除任何识别链霉亲和素或变性单体的噬菌体是必要的,所述变性单体小幅度地出现在生物化单体的每次制备中。在该阴性选择后,将珠用DynaMag-2磁体(magnet)(Life Technologies)固定,然后将含有未结合噬菌体的上清转移用于针对突变KRAS-HLA-A2单体进行阳性选择(正选择,positive selection)。单体的数量从第1轮的1μg减少至第2轮的500ng,第3轮的250ng,然后将噬菌体温育30分钟。在洗脱前,在第1轮至第3轮分别用含有0.05%、0.1%和0.25%吐温(Tween)-20的1ml 1×TBS将珠洗涤10次。通过将珠重悬于1mL pH2.2的0.2M甘氨酸中洗脱噬菌体。在10分钟的温育后,通过加入150μL pH 9.0的1M Tris来中和溶液。使用洗脱的噬菌体感染10mL对数中期SS320的培养物,并加入M13K07辅助噬菌体(MOI为4)和2%葡萄糖。然后将细菌如前所述温育,在第二天早上将噬菌体用PEG/NaCl沉淀。
竞争阶段:在竞争阶段(第4-8轮),阴性选择并入了相同的热变性HLA-A2单体和裸的链霉亲和素-包被的磁珠,也并入了1μg的天然HLA-A3单体。在阴性选择后,将珠用磁体分离,然后将含有未结合噬菌体的上清转移用于竞争选择。这是在缀合至链霉亲和素-包被的琼脂糖珠(Novagen EMD Millipore,Darmstadt,Germany)的野生型KRAS-HLA-A2单体的存在下通过将噬菌体与缀合至磁性链霉亲和素-包被的MyOne T1磁珠的突变KRAS-HLA-A2单体共温育来执行的。突变单体与野生型单体的比例每轮下降2倍,从1:1到1:32,保持野生型单体的量恒定为1μg。在洗脱前,在1ml含有0.5%吐温(Tween)-20的1×TBS中将珠洗涤10次。噬菌体被洗脱并如以上富集阶段所述用于感染对数中期的SS320细胞。
最后选择阶段:在最后选择阶段(第9-10轮),各1μg变性的和天然的KRAS-(WT)-HLA-A2单体被用于阴性选择以去除残留的野生型结合单体的噬菌体。在阴性选择后,如以上富集阶段所述,将珠用磁体固定,然后将含有未结合噬菌体的上清转移用于使用62.5ng突变KRAS-HLA-A2单体的阳性选择。
选择结合突变EGFR-HLA-A3的噬菌体。这是如以上分离结合突变KRAS-HLA-A2的噬菌体所述执行,但有如下修改。选择用2.5×1012的输入噬菌体起始,输入噬菌体的数量在选择过程中下降(见下文)。为在早期的轮中增加scFv-pIII融合蛋白的表达,使用IPTG将乳糖操纵子去阻抑。在第3轮从始至终以10μM的起始浓度加入IPTG,随后在剩余的6轮选择中降低至5μM。另外,在第1至8轮中,用2μg缀合至链霉亲和素磁珠(MyOne T1)的热变性的生物素化HLA-A2和HLA-A3以及25μL裸的链霉亲和素包被的磁珠来执行阴性选择。在竞争阶段,突变单体与野生型单体的比例从1:2逐渐降低至1:64,加入的噬菌体的量从2.5×1012逐渐减少至10×106。
亲和力成熟。D10的亲和力成熟在AxioMx按下文执行。简略地,D10scFv序列(SEQ ID NO:37)被合成并被用作基于易错PCR的诱变的模板。所产生的诱变文库经历三轮选择和扩增,其中将针对KRAS(WT)-HLA-A2单体对噬菌体进行阴性选择,然后针对KRAS(G12V)-HLA-A2单体进行阳性选择,随后进行扩增。选择和扩增后,将可能的噬菌体分离,测序并通过ELISA测试。以鉴定更高亲和力的D10变体。八个克隆被鉴定为对KRAS(G12V)-HLA-A2具有更高亲和力,而不结合KRAS(WT)-HLA-A2,其中一个克隆D10-7(SEQ ID NO:38)被选择用于进一步表征。
ELISA。将链霉亲和素包被的96孔板(Thermo Scientific,Walthan,MA)用封闭缓冲液(具有0.5%BSA、2mM EDTA和0.1%叠氮化钠的PBS)中的200nM生物素化单体溶液在4℃包被过夜。将板简略地用1×TBST(TBS+0.05%吐温-20)洗涤。将噬菌体在1×TBST中连续稀释至指定稀释度,并将100μL加入至每个孔。将噬菌体在室温温育1小时,然后剧烈洗涤(使用喷瓶(Fisher Scientific,Waltham,MA)用1×TBST洗涤6次)。将结合的噬菌体与100μL在1×TBST中1:2000稀释的兔抗M13抗体(Pierce,Rockford,IL)在室温温育1小时,然后额外洗涤6×次,并且与100μL在1×TBST中1:10,000稀释的抗兔IgG-HRP(Jackson Labs,Bar Harbor,Maine)在室温温育45分钟。在最后用1×TBST洗涤6次后,将100μL TMB底物(Biolegend,San Diego,CA)加入到孔中,将反应用1N HCl或2N硫酸进行猝灭。用光吸收酶标仪(SpectraMax Plus 384plate reader)(Molecular Devices,Sunnyvale,CA)或多功能微孔板检测仪(Synergy H1Multi-Mode Reader)(BioTek,Winooski,VT)测量450nm的吸光度。
通过选择用从最后选择阶段获得的噬菌体的有限稀释转导的SS320细胞的单个克隆执行单克隆噬菌体ELISA。将单个克隆接种到200μl的含有100μg/mL羧苄青霉素和2%葡萄糖的2×YT培养基中并在37℃培养3小时。然后将细胞在深孔96孔板中用1.6×107的M13K07辅助噬菌体(Antibody Design Labs,San Diego,CA)以4MOI感染,并在37℃不摇动地温育30分钟,然后摇动30分钟。将细胞离心沉淀,重悬在300μL含有羧苄青霉素(100μg/mL)和卡那霉素(50μg/mL)的2×YT培养基中,并在30℃培养过夜。将细胞离心沉淀,将装载有噬菌体的上清如上述用于ELISA。用纯化的scFv基本如上述执行ELISA,从1μg/mL的起始浓度进行连续稀释,使用1:2000稀释的抗Flag-HRP抗体(Abcam)用于检测。用全长D10MANAbody类似地执行ELISA,从1μg/mL的起始浓度进行连续稀释,1:2000稀释的抗人IgG-HRP抗体作为二抗(Life Technologies)。首先通过将单体稀释到100μL ddH2O中,然后在100℃进行5分钟热封闭温育来执行单体热变性。
scFv生产。设计引物以扩增整个scFv编码区。将GatewayTM定向克隆序列加入到正向引物以帮助亚克隆到GatewayTM入门载体(entry vectors)中,将AviTagTM序列加入到反向引物以允许将来的重组scFv的生物素化。将克隆测序验证并重组到含有C末端V5和His表位标签的pET-DEST42目标载体(Life Technologies)中。
将用重组质粒转化的BL21DE3Gold细胞以1升批量培养至OD600为1.0,冷却至大约20℃并用500μM IPTG诱导。蛋白质在20℃过夜表达。第二天早上将细菌离心沉淀,重悬于周质提取缓冲液(50mM Tris pH 7.4,20%蔗糖,1mM EDTA,5mM MgCl2),并在1/10体积1mg/ml溶菌酶的存在下在冰上孵育30分钟。在将细胞以12,000g离心沉淀30分钟后,将上清通过22μM滤器(Millipore)过滤,然后与1ml Ni-NTA树脂(Qiagen)温育1小时。将上清和珠混合物装载到重力柱上,洗涤,然后用渐增量的咪唑洗脱。使来自每个分装的样品在SDS聚丙烯酰胺凝胶上移动(run),将含有纯的蛋白质的级分在pH 7.4的1×PBS中在4℃透析过夜。使用抗V5HRP抗体(LifeTechnologies)依照标准操作方法执行ELISA以确保scFv结合能力和特异性。
可选地,将scFv序列提供给AxioMx Inc.,亚克隆到含有周质定位序列和N-末端Flag和C末端His标签的载体中。然后将scFv通过镍色谱层析纯化。
抗体生产。将scFv序列移植到曲妥珠单抗(trastuzamab,4D5)序列上用于重组抗体表达。将重链和轻链序列提供给Geneart.用于密码子优化、合成、亚克隆和蛋白质生产(Geneart,Life Technologies,Carlsbad,CA)。将IgG信号序列包括在每条链中用于使用Expi293TM细胞培养系统进行蛋白质表达和抗体分泌。在72小时蛋白质表达后,将来自1升培养物的抗体用柱色谱层析纯化,在17mL PBS中洗脱,以8.25mg/ml分装和运输。
T2和T2A3细胞染色。对于肽的脉冲,将T2和T2A3细胞在50mL PBS中洗涤一次,在50mL无血清RPMI-1640中洗涤一次,然后以每mL 5×105个细胞在含有50μg/mL肽和10μg/mL人β-2微球蛋白(ProSpec,EastBrunswick,NJ)的无血清RPMI-1640中在37℃温育4小时或过夜。将脉冲的细胞离心沉淀,在染色缓冲液(含有0.5%BSA、2mM EDTA和0.1%叠氮化钠的PBS)中洗涤一次,然后在染色缓冲液中重悬。噬菌体染色是在200μl总体积中用~1×109个噬菌体在4℃进行30分钟,然后通过在6℃以500g离心5分钟在染色缓冲液中进行3×4mL漂洗来执行的。将细胞在200μL染色缓冲液中重悬,用1μL兔抗M13抗体(Pierce,Rockford,IL)在冰上染色30分钟,然后用4mL染色缓冲液漂洗三次。然后将细胞重悬于200μL染色缓冲液,并与1μL抗兔Alexa Fluor 488TM(Life Technologies)在冰上孵育30分钟,另外漂洗三次,然后分析。scFv染色是在100μl染色缓冲液中用1μg scFv在冰上30分钟,然后在4℃在染色缓冲液中漂洗3次来执行的。然后将细胞用1μL鼠抗V5-FITC(Life Technologies,Grand Island,NY)在冰上染色30分钟,然后在6℃在染色缓冲液中漂洗三次。抗体染色是通过将细胞重悬于100μL染色缓冲液中,并用1μg山羊抗人抗体(Life Technologies)在冰上封闭30分钟,然后在4℃漂洗三次来执行的。将细胞在200μL染色缓冲液中重悬,用5μg D10抗体(或同种型对照)在冰上染色30分钟,然后漂洗三次。将细胞在200μL染色缓冲液中重悬,用2μL山羊抗人PE抗体(LifeTechnologies)在冰上染色30分钟,然后漂洗三次。肽的脉冲是通过将经脉冲的T2或T2A3细胞与5μL W6/32-PE(Bilegend)在100μL染色缓冲液中温育来评估。将染色的T2和T2A3细胞使用FACSCalibur或LSRII流式细胞仪(Becton Dickinson,Mansfield,MA)分析。
T2细胞补体依赖的细胞毒性。在4℃将scFv与抗V5鼠单克隆抗体(LifeTechnologies,Grand Island,NY)以2:1摩尔比例缀合过夜。将缀合的scFv或对照抗HLA抗体W6/32(Bio-X-Cell)在冰上在无血清RPMI-1640中连续稀释。将用冰冷的ddH2O重悬的幼兔补体(Cedarlane)加入到连续稀释的抗体缀合物中,然后转移60μL至96孔板。将含有20,000个细胞的另外40μL预冷的肽脉冲的T2细胞转移至板并温和混合。在所有情形中,10%的最终补体浓度被用于试验。将板在37℃温育1小时,随后通过CellTiter-发光细胞活力检测(Luminescent Cell Viability Assay)(Promega,)按照制造商说明书读板。通过首先将三种细胞亚型(用不同肽或仅β-2微球蛋白对照脉冲的细胞)的每一种归一化为最大萤光素酶信号(无抗体对照),然后从100%减去来计算细胞死亡。比细胞死亡(specific cell death)被定义为针对给定的三种细胞亚型的每一种在用W6/32抗体处理后在特定抗体浓度的细胞死亡除以观察到的最大细胞死亡。
细胞死亡(%)=100–(100×萤光素酶信号/萤光素酶信号最大)
亲和力和koff测量。亲和力测量在AxioMx Inc.如下所述执行。将生物素化的KRAS(G12V)-HLA-A2单体、未生物化的KRAS(G12V)-HLA-A2单体和Flag标签化的D10scFv同时加入到含有链霉亲和素包被的激发波长为680nm的供体珠和与抗Flag抗体缀合的受体珠的溶液。受体珠的刺激取决于供体珠的接近度(proximity),当两种珠非常接近时导致520-620nm的激发。因此通过改变竞争的未生物素化单体的量并测量所导致的吸光度测定D10scFv与KRAS(G12V)-HLA-A2的EC50和亲和力。
为保存有限量的抗原并加快筛选新的scFv,发明人开发了解离率的基于ELISA的动力学试验以测量koff,从而测量scFv/单体解离的半衰期。将100μL各自的生物素化单体的20nM溶液缀合至链霉亲和素包被的96孔条形板(R&D Systems)。在洗涤后,将100μL的D10scFv、D10-7scFv或C9scFv的37.0nM、9.3nM或2.3nM溶液加入到板,并允许其在22℃在单体包被的孔中平衡2小时。在不同时间点,移除所述板的一条,剧烈洗涤,然后放入22℃的2升1×TBST中同时摇动,直至所有时间点完成。在0小时时间点后,从第一条洗涤的时间开始允许总共16小时的scFv解离,将所有条重新组装到板上,如前所述使用抗Flag-HRP抗体和TMB底物执行ELISA。通过拟合指数函数(At=Aoe-kt)至产生的数据点然后减去背景,并使用一阶反应方程t1/2=ln(2)/k来测定半衰期。对D10MANAbody的解离率估计按上述执行,除了如下例外:将100μL10nM的单体包被在板上使得链霉亲和素复合物与单体比例为1:2,使用了更低浓度(1.8nM)的抗体,试验进行到32小时时间点。
统计。所有统计分析是用Prism 5(GraphPad Software)执行的。除非另有说明,误差条表示三个技术重复的标准差。统计显著性用未配对双尾t检验执行。
实施例2
基于scFv-基的噬菌体展示的scFv文库的设计和构建。发明人开始这些研究企图在小鼠中产生针对突变KRAS肽的抗体(下文叙述这种选择的基础)。使用常规方法在小鼠免疫后获得单克隆抗体,这些努力失败了,因为没有鉴定出与MANA特异性反应的抗体。发明人因此转向噬菌体展示方法用于产生MANAbody(图1)。噬菌体展示文库的设计遵循公开研究中使用的原则(22)并且包括一些特别的特征。文库的框架是基于针对由ERBB2编码的蛋白质产生的人源化4D5抗体(曲妥单抗,Trastuzumab)的scFv序列(23)。该框架由于其在噬菌体上的稳定性以及其易于转化为可溶的scFv、Fab或抗体(22、24)而被选择。人源化4D5的高分辨率晶体结构已经鉴定了在抗原结合中起最重要作用的高度可变的互补决定区(CDR)内的残基(25)。这使得发明人聚焦于用于抗原结合的最重要残基而不是主链残基的可变性。在发明人的文库中,氨基酸置换被限制为限定的四个CDR,CDR-L3、CDR-H1、CDR-H2和CDR-H3中的互补位残基(26)。发明人使文库偏斜以包括以前被证明在抗原结合中起重要作用或在天然存在抗体中富集的CDR内的特异性氨基酸。一个重要的随机化是在CDR-L3处,其含有丝氨酸和酪氨酸的混合,之前显示所述两种氨基酸促进了最低要求(最简的,minimalist)的文库设计方案(26、27)。已经显示重链中的CDR在抗原结合多样性中起更重要的作用(28-30),因此发明人在重链CDR中引入比在轻链CDR中更多的简并性。另外,在其中追求增加的多样性的位置,更常用的IUB核苷酸(指定的NNK和NNS简并密码子)由DMT密码子替代;这排除了不想要的残基。在所有情形中,发明人使用了简并核苷酸混合物意欲使产生半胱氨酸或终止密码子的序列的并入最小化。最终,发明人在CDR-H3中引入了长度多态性,允许7个氨基酸的茎环结合长度多样性。这些改变导致计算出的理论文库复杂度为5×1013。
将合成的寡核苷酸文库克隆到噬菌粒载体用于scFv表达。这种scFv携带myc标签,并通过烟草蚀纹病毒(TEV)切割位点被融合到噬菌体M13pIII外壳蛋白(图5)。这种设计帮助scFv从噬菌体颗粒纯化并提供在随后的噬菌体选择方法中通过TEV切割完成的可选的洗脱方法。在文库合成后,将45个克隆通过Sanger方法测序。测序显示文库成功率为53%,所述成功率由框架区内的突变不存在和CDR内的期望氨基酸的存在所定义。文库多样性基于文库构建期间达到的转化效率计算,产生的估计的多样性为5.5×109。为进一步评估文库的质量,将文库进行大规模平行测序(31)。该分析揭露了3,785,138个独特的克隆(分析的所有克隆的46.5%)。测序区域仅包括CDR-H3,不包括被系统地改变的其他三个CDR(CDR-L3、CDR-H1和CDR-H2)。因此独特克隆部分(46.5%)表示多样性的最小估计。序列的随机亚组的翻译进一步显示了CDR-H3中的期望氨基酸分布以及长度多样性。
实施例3
用于鉴定选择性反应的噬菌体克隆的靶点选择和竞争策略。发明人基于具体突变的频率和其预测的结合HLA等位基因的强度选择了MANAbody靶。KRAS是人类癌症中最常突变的基因之一,其中突变具体在胰腺、结肠直肠和肺腺癌中普遍存在。发明人选择G12V突变作为靶,因为含有它的相关肽被预测以高亲和力结合HLA-A2,HLA-A2是许多人种群中最常见的HLA等位基因(32)。这种生物信息学(in silico)预测是使用NetMHC v3.4算法进行的(33-35)。另外,基于其它肽-HLA复合物的结构研究,关键的突变残基(密码子12处的V)被预期暴露于HLA蛋白质的表面上(36)。肽KLVVVGAVGV(SEQ ID NO:4),其中在第8位的缬氨酸残基(V)表示G12V突变,通过常规方式被化学合成。对应于突变等位基因产物的肽将在此后称为“突变肽”,而表示野生型等位基因产物的肽将被称为“野生型肽”。然后突变的KRAS肽被折叠为HLA-A2和β-2-微球蛋白的复合物(单体)[KRAS(G12V)-HLA-A2]。对应于野生型KRAS序列的两个肽也被合成并与HLA-A2或HLA-A3一起折叠以分别形成KRAS(WT)-HLA-A2和KRAS(WT)-HLA-A3单体。对应于其它密码子12的突变的另外的突变KRAS单体也被组装。在大多数情形中,将单体生物素化以帮助纯化和随后的实验(见材料和方法)。
噬菌体展示选择方法由10轮选择和扩增组成,其被分为三个不同的阶段:富集阶段(第1-3轮),竞争阶段(第4-8轮)和最后选择阶段(第9-10轮)(图6)。这些阶段的总目标是最大化回收比KRAS(WT)-HLA-A2或单独的HLA更好结合KRAS(G12V)-HLA-A2的克隆。在每轮富集阶段,用热变性的生物素化HLA-A2单体进行阴性选择后,用KRAS(G12V)-HLA-A2进行阳性选择(图6A以及材料和方法)。在每个连续轮(第2和3轮)中,减少KRAS(G12V)-HLA-A2单体的量以富集更强的结合物。
本研究中描述的新的竞争阶段意欲富集相对于KRAS(WT)-HLA-A2结合物稀有的突变KRAS-(G12V)--HLA-A2结合物,以及发明人预期在富集阶段后存在于文库中的更频繁出现的全-HLA结合物(泛-HLA结合物,pan-HLAbinder)。每轮竞争阶段使用变性的HLA-A2和天然的HLA-A3单体以阴性选择开始(图6B)。然后,将噬菌体同时地与结合链霉亲和素磁珠的KRAS(G12V)-HLA-A2和结合链霉亲和素琼脂糖珠的KRAS(WT)-HLA-A2温育。KRAS(WT)-HLA-A2充当竞争物,因为与其结合的噬菌体不会在磁珠捕获步骤中被回收(图6B)。此外,在每轮竞争阶段,使用逐渐减少量的KRAS(G12V)-HLA-A2,但使用相同量的KRAS(WT)-HLA-A2,企图富集高亲和力的结合物。在最后选择阶段,每轮使用非常过量的变性和天然的KRAS(WT)-HLA-A2单体以严格的阴性选择起始,并用KRAS(G12V)-HLA-A2单体以阳性选择继续(图6C)。
实施例4
评估选择的噬菌体克隆。发明人使用酶联免疫吸附测定(ELISA)评估噬菌体与肽-HLA复合物的结合。在富集阶段后(图6A),所选择的噬菌体(全体地)相对于与HLA-A2复合的野生型KRAS肽没有显示对突变体的偏好,或者相对于结合HLA-A3的KRAS肽没有显示出对结合HLA-A2的KRAS肽的偏好。(图7A)仅在最后选择阶段(图6C)对结合HLA-A2的突变KRAS的特异性才出现。具体地,这些噬菌体与KRAS(G12V)-HLA-A2结合比于KRAS(WT)-HLA-A2或与KRAS(WT)-HLA-A3结合得更好(图7B)。这些噬菌体通过有限稀释被克隆并以96孔板形式扩增。一个克隆(D10;SEQ ID NO:37)显示与KRAS(G12V)-HLA-A2单体的实质结合(图2A)。该D10克隆对KRAS(G12V)-HLA-A2是高度特异性的,因为其没有结合测试的所有其它单体(图2B)。
为产生与M13pIII分开的D10scFv,纯化了来自D10噬菌体的单链DNA(ssDNA)。将scFv部分通过聚合酶链式反应(PCR)扩增,测序,然后克隆到含有除了6×His标签之外的Flag或V5表位标签的原核表达载体。这帮助D10scFv的高水平表达和亲和纯化。与表达D10scFv:pIII融合蛋白的噬菌体类似,纯化的D10scFv与KRAS(G12V)-HLA-A2以高度特异的方式相互作用(图2C)。重要地,D10scFv没有显示出任何与KRAS(WT)-HLA-A2、KRAS(WT)-HLA-A3或与结合HLA-A2的其它KRAS突变体(KRAS G12C或KRAS G12D)的上述结合的背景。另外,当没有与HLA蛋白质复合时,D10scFv不结合KRAS肽(图8)。这些结果证明成功选择了结合HLA情况下肽的scFv。
亲和力成熟。使用亲和力测量的方法(37),D10scFv对KRAS(G12V)-HLA-A2的亲和力被评估为49nM。发明人接下来继续使D10亲和力成熟。简略地,从原始的D10scFv序列产生通过易错PCR诱变的D10scFv文库,并针对KRAS(G12V)-HLA-A2和KRAS(WT)-HLA-A2单体进行三轮选择。评价克隆得到候选物,D10-7,其显示了新获得的结合KRAS(G12C)-HLA-A2的能力,同时仍然保留区分突变和野生型KRAS表位的能力(图9)。为比较D10-7和D10与KRAS(G12V)-HLA-A2的相对结合,发明人使用基于解离率(off-rate)的试验以测量koff值。不像亲和力测量,这些试验允许多个scFv在同一测试内快速比较,因此提供对多个克隆的相对结合的更直接的比较(38)。解离率测量显示,与D10scFv(5.7×10-6sec-1)相比,亲和力成熟的D10-7scFv的解离率常数(3.2×10-6sec-1)几乎两倍的减少。KRAS(WT)-HLA-A2单体与这些scFv没有出现可测量的结合,证明KRAS(G12V)-HLA-A2的koff比KRAS(WT)-HLA-A2低至少200倍。这些scFv与同HLA-A2复合的突变体和野生型肽的大区分度的结合在下文描述的其他试验中也是明显的。
实施例5
鉴定可结合不同MANA的噬菌体。为确定该方法是否适用于其它MANA,发明人试图鉴定对与不同HLA等位基因复合的突变EGFR肽特异的scFv。EGFR L858R突变在~10%的肺腺癌中被发现并且在这种癌症类型中占所有EGFR突变的~40%(39)。密码子858在EGFR蛋白质的胞质区,而不是胞外或胞膜区,并且通常不在细胞表面上可见(40)。当通过NetMHC v3.4算法分析时,含有该突变的肽(KITDFGRAK;SEQ ID NO:9)被预测以高亲和力结合HLA-A3等位基因。为鉴定对该肽-HLA复合物特异的scFv,发明人采用了修改的选择和扩增方案,其中使用了逐渐降低浓度的异丙基β-D-1-硫代半乳糖苷(IPTG)以减少后面几轮中的scFv表达。另外,每个选择阶段中轮的数量被调整以有利于富集需要的噬菌体而不是消除不需要的噬菌体(图9与图6相比,还见于材料和方法)。通过这些修改,发明人能够获得,与多个对照单体——包括与HLA-A3结合的野生型EGFR——相比,对与HLA-A3复合的突变EGFR肽[EGFR(L858R)-HLA-A3]显示出选择性结合的噬菌体克隆(C9;SEQ ID NO:39)(图2D)。由该克隆产生的C9scFv对EGFR(L858R)-HLA-A3显示出相似的选择性结合(图2E)。与HLA-A3结合的突变EGFR肽的估计koff比野生型肽的koff低一个数量级(值分别为2.6×10-6sec-1和3.0×10-5sec-1)。
实施例6
与在细胞表面展示突变肽的细胞的选择性结合。然后发明人试图确定D10和C9scFv是否结合细胞表面上的突变KRAS和EGFR肽-HLA复合物。T2细胞系衍生自爱-巴病毒(Epstein-Barr virus)转化的人淋巴母细胞系,该人淋巴母细胞系由于缺失涉及TAP1和TAP2肽转运蛋白的基因而在内源性HLA相关肽抗原的呈递中有缺陷(41)。T2A3是稳定表达HLA-A3转基因的修改版本的T2(42、43)。T2和T2A3细胞表达低水平的HLA,其可通过添加外源的HLA结合肽来稳定,因此可充当用于测试与特异性HLA结合肽的相互作用的平台(44、45)。发明人首先用KRAS(G12V)、KRAS(WT)或阴性对照肽脉冲的T2细胞。为评估装载效率,发明人使用了靶向通过任何HLA结合肽稳定的HLA分子的W6/32抗体。由抗W6/32染色可知,野生型和突变肽之间的肽装载效率是相当的(图11)。在与D10噬菌体温育后通过流式细胞术对脉冲的细胞的分析显示D10噬菌体与KRAS(G12V)肽脉冲的细胞的结合是明显的,同时观察到了与KRAS(WT)或对照肽脉冲的细胞的临界(marginal)结合(超过背景)(图3A、图S8)。用纯化的D10scFv而不是D10噬菌体进行的类似实验确认了与在细胞表面呈递的KRAS(G12V)的选择性结合(图3B)。发明人还用突变体EGFR(L858R)、EGFR(WT)或阴性对照肽脉冲T2A3细胞,并通过流式细胞术评估C9噬菌体结合。再次地,C9噬菌体与EGFR(L858R)肽脉冲的细胞的结合是明显的,同时没有观察到与EGFR(WT)或对照肽脉冲的细胞的结合。当噬菌体或scFv没有包括在反应中或细胞没有装载有所述肽时,仅观察到背景荧光(图3A-3C、图12和13)。
然后发明人试图确定用突变KRAS(G12V)肽脉冲的T2细胞是否可被D10scFv靶向并在补体依赖的细胞毒性(CDC)试验中被杀伤。作为阳性对照,发明人用KRAS(G12V)或KRAS(WT)肽脉冲的T2细胞并用W6/32抗体执行CDC试验。如所预期的,该抗体在补体的存在下有效地杀伤肽脉冲的T2细胞(图14)。随后发明人测试了D10scFv和亲和力成熟的D10-7scFv,两者在CDC试验中都缀合至含有补体固定Fc区的抗V5表位标签抗体。两种scFv都以剂量依赖的方式杀伤KRAS(G12V)脉冲的T2细胞,并且亲和力成熟的D10-7scFv在杀伤效率方面显示出显著的提高(D10-7的EC50为0.79nM和D10的EC50为11.2nM,图3D)。相比之下,用KRAS(WT)脉冲的细胞或没有用外源肽脉冲的细胞仅显示出最低限度(marginal)的细胞死亡。
实施例7
全长MANAbody的产生。免疫治疗剂的临床应用通常使用完整抗体,其包括Fc结构域而不是仅包含scFv组分。完整抗体的另一个特性是由于最初单价scFv的二价化而实现的更高的亲和力(avidity)。为从D10scFv产生完整MANAbody,发明人将D10scFv序列移植到临床使用的人源化4D5抗体曲妥珠单抗的恒定区上。D10MANAbody的高水平表达在哺乳动物细胞中实现(每升Expi293细胞培养物139毫克蛋白质)。与D10scFv类似,如通过ELISA所评估的,D10MANAbody与KRAS(G12V)-HLA-A2相互作用(图4A)。没有观察到与KRAS(WT)-HLA-A2单体或测试的任何其它单体的结合。D10MANAbody还显示出与用KRAS(WT)或阴性对照肽脉冲的T2细胞相比,用突变KRAS(G12V)肽脉冲的T2细胞的相对更强的染色(图4B、图15)。最后,当评估单价解离时,观察到的全长D10MANAbody的半衰期与其scFv衍生物的半衰期类似。因此,D10MANAbody保留了与D10scFv所观察到的一致的高特异性和低解离率常数。
实施例8
噬菌体淘选方法的修改。以前描述的噬菌体选择方法的变化被执行以针对另外的HLA-肽复合物(CTNNB1S45F)和(EGFR T790M)鉴定抗体。CTNNB1是编码蛋白质β-联蛋白的基因名称。这些名称在本文中互换使用。
第一个改变是包括来自表达正被筛选的特定HLA等位基因的细胞系的细胞。但是这些细胞不含有感兴趣的相关突变。发明人将这些细胞加到在每轮开始时出现的阴性选择步骤(与其中发明人检查(interrogate)针对变性HLA和裸链霉亲和素珠的噬菌体的步骤相同的步骤)。细胞可以在最初两轮或筛选期间被加入到该步骤。该步骤的目的是去除任何结合与发明人的突变表位类似的HLA-表位序列的噬菌体。
发明人还证明通过改变富集阶段、竞争阶段和最后选择阶段的轮的数量,发明人能够鉴定以前没有被检测到的抗体。具体地,执行富集阶段的0或1轮,然后执行竞争选择的多达5轮。但是,在竞争选择的第二轮后开始,噬菌体小份在每天被转换到阴性选择(这既减少了噬菌体选择的轮的数量又允许发明人鉴定在后面几轮淘选中不存在的scFv候选物)。
在2、3和4轮竞争选择后,使噬菌体进行两轮连续的阴性选择,产生已经经历4-8轮总选择的噬菌体。
这些改变导致鉴定针对β-联蛋白/CTNNB1(S45F)-HLA-A3和EGFRT790M-HLA-A2突变以及可能的p53突变的scFv候选物。
实施例9
KRAS G12V-HLA-A2克隆F10。对于F10克隆的噬菌体选择按照对C9噬菌体选择的描述进行,除了使用了突变[KRAS(G12V)-HLA-A2]和野生型[KRAS(野生型)-HLA-A2]单体。这证明对于给定的HLA复合物可以鉴定多个scFv候选物。
F10克隆:
DIQMTQSPSSLSASVGDRVTITCRASQDVNTAVAWYQQKPGKAPKLLIYSASFLYSGVPSRFSGSRSGTDFTLTISSLQPEDFATYYCQQYYYYPPTFGQGTKVEIKRTGGGSGGGGSGASEVQLVESGGGLVQPGGSLRLSCAASGFNINSNYIHWVRQAPGKGLEWVAYITPETGYYRYADSVKGRFTISADTSKNTAYLQMNSLRAEDTAVYYCSRNYYSAYAMDVWGQGTLVTVSS(SEQ ID NO:21)
实施例10
KRAS F10克隆的亲和力成熟。如同KRAS D10克隆,F10scFv也能够经历有效的亲和力成熟并且变体保留了其相对于KRAS野生型对KRAS突变体的特异性。
KRAS(wt)-HLA-A2信号保持在背景附近,如同F10scFv(这是由于包括N末端表位标签)。F10亲和力成熟的变体展示了显著的结合(平均荧光强度增加多达131倍)。
用于F10亲和力成熟的衍生物的scFv序列:
KRAS G12V_F10AM#1
DIQMTQSPSSLSASVGDRVTITCRASQDVNTAVAWYQQKPGKAPKLLIYSASFLYSGVPSRFSGSRSGTDFTLTISSLQPEDFATYYCQQYYYYPPTFGQGTKVEIKRTGGGSGGGGSGASEVQLVESGGGLVQPGGSLRLSCAASGFNINSNYIHWVRQAPGKGLEWVAYITPETGYYHYADSVKGRFTISADTSKNTAYLQMNSLRAEDTAVYYCSRNYYSAYAMDVWGQGTLVTVSS(SEQ ID NO:22)
KRAS G12V_F10AM#2
DIQMTQSPSSLSASVGDRVTITCRASQDVNTAVAWYQQKPGKAPKLLIYGASFLYSGVPSRFSGSRSGTDFTLTISSLQPEDFATYYCQQYYYYPPTFGQGTKVEIKRTGGGSGGGGSGASEVQLVESGGGLVQPGGSLRLSCAASGFYINSNYIHWVRQAPGKGLEWVAYITPETGYYHYADSVKGRFTISADTSKNTAYLQMNSLRAEDTAVYYCSRNYYSAYAMDVWGQGTLVTVSS(SEQ ID NO:23)
KRAS G12V_F10AM#3
DIQMSQSPSSLSASVGDRVTITCRTSQDANTAVAWYQQKPGKAPKLLFYSASFLFSGVPSRFSGSRSGTDFTLTISSLQPEDFATYYCQQYYYYPPTFGQGTKVEIKRTGGGSGGGGSGASEVQLVESGGGLVQPGGSLRLSCAASGFNINSNYIHWVRQAPGKGLEWVAYITPETGYYRYADSVKGRFTISADTSKNTAYLQMNSLRAEDTAVYYCSRNYYSAYAMDVWGQGTLVTVSS(SEQ ID NO:24)
实施例11
CTNNB1(S45F)-HLA-A3致癌突变(TTAPFLSGK;SEQ ID NO:27)。在CTNNB1(β-联蛋白)的蛋白质产物的第45残基处的突变是致癌基因中第二常见的。(S->F突变是最普遍的氨基酸改变)。针对S45F鉴定抗体还表明针对S45P的抗体衍生物是可能的。另外,最常见的CTNNB1突变,T41A突变也被预测以相同的氨基酸坐标(coordinate)(ATAPSLSGK;SEQ ID NO:36)结合HLA-A3。这也证明了靶向该突变的可行性。
淘选β-联蛋白/CTNNB1S4F5和EGFR T790M突变的改变在下文描述。这证明了基于竞争的筛选中内在的灵活性。
E10scFv序列:
DIQMTQSPSSLSASVGDRVTITCRASQDVNTAVAWYQQKPGKAPKLLIYSASFLYSGVPSRFSGSRSGTDFTLTISSLQPEDFATYYCQQSYYSPPTFGQGTKVEIKRTGGGSGGGASEVQLVESGGGLVQPGGSLRLSCAASGFNINNTYIHWVRQAPGKGLEWVASIYPTDGYTRYADSVKGRFTISADTSKNTAYLQMNSLRAEDTAVYYCSRTYYSYYSAMDVWGQGTLVTVSS(SEQ ID NO:16)
W6/32数据显示相对于b2m(阴性对照)的抗体结合的增加,显示所述肽由HLA-A3复合物呈递。E10噬菌体染色显示相对于对照肽(600-800MFI),scFv特异性结合S45F表位(80,400k MFI)。
噬菌体克隆E10对CTNNB S10F肽-脉冲的细胞是特异的。
实施例12
EGFR T790M。EGFR T790M突变是第二最频繁的EGFR突变(在L858R之后)。这是一个重要的突变,因为其作为抗性突变响应于抗EGFR治疗而频繁地出现。另外,文献中的证据表明T790M突变在肿瘤细胞上由HLA-A2复合物内源性地加工和呈递(作为9个和10个氨基酸的表位)。
所述9个氨基酸的突变表位为:
IMQLMPFGC(SEQ ID NO:28;突变残基T->M有下划线和粗体)
所述10个氨基酸的突变表位为:
LIMQLMPFGC(SEQ ID NO:29;突变残基T->M有下划线和粗体)实施例13
噬菌体选择。噬菌体选择如上文所述完成。鉴定了来自二和三和四天的竞争选择(其后是两天的阴性选择)的可能候选物(具有不同的scFv序列)。这些候选物被称为D2D6、D3E6、D2D8。D3E6在这组中看起来是最有前景的。
D3E6scFv序列为:
DIQMTQSPSSLSASVGDRVTITCRASQDVNTAVAWYQQKPGKAPKLLIYSASFLYSGVPSRFSGSRSGTDFTLTISSLQPEDFATYYCQQYYYYPPTFGQGTKVEIKRTGGGSGGGGSGGGASEVQLVESGGGLVQPGGSLRLSCAASGFNISTSYIHWVRQAPGKGLEWVATIDPNDGYSRYADSVKGRFTISADTSKNTAYLQMNSLRAEDTAVYYCSRTNNTAADAMDVWGQGTLVTVSS(SEQ ID NO:18)
D2D6scFv序列为:
DIQMTQSPSSLSASVGDRVTITCRASQDVNTAVAWYQQKPGKAPKLLIYSASFLYSGVPSRFSGSRSGTDFTLTISSLQPEDFATYYCQQSYYSPPTFGQGTKVEIKRTGGGSGGGGSGGGASEVQLVESGGGLVQPGGSLRLSCAASGFNITSSYIHWVRQAPGKGLEWVAYISPADGYNRYADSVKGRFTISADTSKNTAYLQMNSLRAEDTAVYYCSRTDSTAYTAMDVWGQGTLVTVSS(SEQ ID NO:40)
D2D8scFv序列为:
DIQMTQSPSSLSASVGDRVTITCRASQDVNTAVAWYQQKPGKAPKLLIYSASFLYSGVPSRFSGSRSGTDFTLTISSLQPEDFATYYCQQYYSYPPTFGQGTKVEIKRTGGGSGGGGSGGGASEVQLVESGGGLVQPGGSLRLSCAASGFNINSSYIHWVRQAPGKGLEWVAYISPTDGYYRYADSVKGRFTISADTSKNTAYLQMNSLRAEDTAVYYCSRTSDTSYAAMDVWGQGTLVTVSS(SEQ ID NO:41)
实施例14
p53R248W克隆。TP53是癌症中最经常突变的基因。发明人已经获得了能够结合p53R248W突变的scFv。发明人目前正在测试它们的特异性,但是证明了可获得针对该表位的抗体。另一个常见突变p53R248Q,其序列除了W改变为Q外是相同的,以相似方式结合HLA-A2。
克隆D2F2序列:
DIQMTQSPSSLSASVGDRVTITCRASQDVNTAVAWYQQKPGKAPKLLIYSASFLYSGVPSRFSGSRSGTDFTLTISSLQPEDFATYYCQQSYSSPPTFGQGTKVEIKRTGGGSGGGASEVQLVESGGGLVQPGGSLRLSCAASGFNINDTYIHWVRQAPGKGLEWVAYISPASGNSRYADSVKGRFTISADTSKNTAYLQMNSLRAEDTAVYYCSRSYAAMDVWGQGTLVTVSS(SEQ ID NO:25)
实施例15
ABL1E255K突变(KVYEGVWKK;SEQ ID NO:26)。ABL1在所有CML中的~30%是突变的。E255K突变赋予了对伊马替尼(imatinab)和尼洛替尼(nilotinab)的耐药性。该突变被预测位于表位内的第1位。这使得抗体或TCR非常难于区分突变体和野生型。但是,HLA-A3对于突变表位的预测的亲和力比预测的野生型亲和力高10倍(29nM对比228nM)。另外,具有不同N末端氨基酸的表位的蛋白水解加工可产生不同的分裂产物,从而影响内源性的呈递。这表明突变的和野生型表位(在第1位有突变)的scFv识别可能不妨碍体内突变表位的特异性。
Abl1E255K_命中1
MASDIQMTQSPSSLSASVGDRVTITCRASQDVNTAVAWYQQKPGKAPKLLIYSASFLYSGVPSRFSGSRSGTDFTLTISSLQPEDFATYYCQQYYSSPPTFGQGTKVEIKRTGGGSGGGGSGGGASEVQLVESGGGLVQPGGSLRLSCAASGFNINSSSIHWVRQAPGKGLEWVASIAPARGSTRYADSVKGRFTISADTSKNTAYLQMNSLRAEDTAVYYCSRNYYAYTAMDVWGQGTLVTVSS(SEQ ID NO:14)
Abl1E255K_命中2
DIQMTQSPSSLSASVGDRVTITCRASQDVNTAVAWYQQKPGKAPKLLIYSASFLYSGVPSRFSGSRSGTDFTLTISSLQPEDFATYYCQQYSSSPPTFGQGTKVEIKRTGGGSGGGASEVQLVESGGGLVQPGGSLRLSCAASGFNINTSYIHWVRQAPGKGLEWVASIYPNDGYNRYADSVKGRFTISADTSKNTAYLQMNSLRAEDTAVYYCSRAAYAMDVWGQGTLVTVSS(SEQ ID NO:15)
参考文献
引用的每篇参考文献的公开内容明确地并入本文。
1.Fearon ER&Vogelstein B(1990)A genetic model for colorectal tumorigenesis.Cell 61(5):759-767.
2.Vogelstein B,et al.(2013)Cancer genome landscapes.Science 339(6127):1546-1558.
3.Tsai J,et al.(2008)Discovery of a selective inhibitor of oncogenic B-Raf kinase with potent antimelanoma activity.Proc Natl Acad Sci U S A 105(8):3041-3046.
4.Halilovic E&Solit DB(2008)Therapeutic strategies for inhibiting oncogenic BRAF signaling.Curr Opin Pharmacol 8(4):419-426.
5.Bradbury AR,Sidhu S,Dubel S,&McCafferty J(2011)Beyond natural antibodies:the power of in vitro display technologies.Nat Biotechnol 29(3):245-254.
6.Sliwkowski MX&Mellman I(2013)Antibody therapeutics in cancer.Science 341(6151):1192-1198.
7.Weiner LM,Surana R,&Wang S(2010)Monoclonal antibodies:versatile platforms for cancer immunotherapy.Nature reviews.Immunology 10(5):317-327.
8.Ayriss J,Woods T,Bradbury A,&Pavlik P(2007)High-throughput screening of single-chain antibodies using multiplexed flow cytometry.J Proteome Res 6(3):1072-1082.
9.Kato Y,et al.(2009)A monoclonal antibody IMab-1specifically recognizes IDH1R132H,the most common glioma-derived mutation.Biochem Biophys Res Commun 390(3):547-551.
10.Yu J,et al.(2009)Mutation-specific antibodies for the detection of EGFR mutations in non-small-cell lung cancer.Clin Cancer Res 15(9):3023-3028.
11.Roblek M,et al.(2010)Monoclonal antibodies specific for disease-associated point-mutants:lamin A/C R453W and R482W.PLoS One 5(5):e10604.
12.Segal NH,et al.(2008)Epitope landscape in breast and colorectal cancer.Cancer Res 68(3):889-892.
13.Rahma OE,et al.(2014)The immunological and clinical effects of mutated ras peptide vaccine in combination with IL-2,GM-CSF,or both in patients with solid tumors.Journal of translational medicine 12:55.
14.Abrams SI,Hand PH,Tsang KY,&Schlom J(1996)Mutant ras epitopes as targets for cancer vaccines.Semin Oncol 23(1):118-134.
15.Gubin MM,et al.(2014)Checkpoint blockade cancer immunotherapy targets tumour-specific mutant antigens.Nature 515(7528):577-581.
16.Castle JC,et al.(2012)Exploiting the mutanome for tumor vaccination.Cancer Res 72(5):1081-1091.
17.Vonderheide RH&June CH(2014)Engineering T cells for cancer:our synthetic future.Immunol Rev 257(1):7-13.
18.Schreiber RD,Old LJ,&Smyth MJ(2011)Cancer immunoediting:integrating immunity's roles in cancer suppression and promotion.Science 331(6024):1565-1570.
19.Robbins PF,et al.(2013)Mining exomic sequencing data to identify mutated antigens recognized by adoptively transferred tumor-reactive T cells.Nat Med 19(6):747-752.
20.Yadav M,et al.(2014)Predicting immunogenic tumour mutations by combining mass spectrometry and exome sequencing.Nature 515(7528):572-576.
21.Bondgaard AL,Hogdall E,Mellemgaard A,&Skov BG(2014)High specificity but low sensitivity of mutation-specific antibodies against EGFR mutations in non-small-cell lung cancer.Mod Pathol 27(12):1590-1598.
22.Sidhu SS,et al.(2004)Phage-displayed antibody libraries of synthetic heavy chain complementarity determining regions.J Mol Biol 338(2):299-310.
23.Carter P,et al.(1992)Humanization of an anti-p185HER2 antibody for human cancer therapy.Proc Natl Acad Sci U S A 89(10):4285-4289.
24.Bostrom J&Fuh G(2009)Design and construction of synthetic phage-displayed Fab libraries.Methods in molecular biology 562:17-35.
25.Eigenbrot C,Randal M,Presta L,Carter P,&Kossiakoff AA(1993)X-ray structures of the antigen-binding domains from three variants of humanized anti-p185HER2 antibody 4D5 and comparison with molecular modeling.J Mol Biol 229(4):969-995.
26.Fellouse FA,et al.(2007)High-throughput generation of synthetic antibodies from highly functional minimalist phage-displayed libraries.J Mol Biol 373(4):924-940.
27.Koide S&Sidhu SS(2009)The importance of being tyrosine:lessons in molecular recognition from minimalist synthetic binding proteins.ACS chemical biology 4(5):325-334.
28.Kuroda D,Shirai H,Kobori M,&Nakamura H(2008)Structural classification of CDR-H3 revisited:a lesson in antibody modeling.Proteins 73(3):608-620.
29.Kunik V,Peters B,&Ofran Y(2012)Structural consensus among antibodies defines the antigen binding site.PLoS computational biology 8(2):e1002388.
30.Sela-Culang I,Kunik V,&Ofran Y(2013)The structural basis of antibody-antigen recognition.Frontiers in immunology 4:302.
31.Kinde I,Wu J,Papadopoulos N,Kinzler KW,&Vogelstein B(2011)Detection and quantification of rare mutations with massively parallel sequencing.Proc Natl Acad Sci U S A 108(23):9530-9535.
32.Ellis JM,et al.(2000)Frequencies of HLA-A2 alleles in five U.S.population groups.Predominance Of A*02011 and identification of HLA-A*0231.Human immunology 61(3):334-340.
33.Nielsen M,et al.(2003)Reliable prediction of T-cell epitopes using neural networks with novel sequence representations.Protein science:a publication of the Protein Society 12(5):1007-1017.
34.Lundegaard C,et al.(2008)NetMHC-3.0:accurate web accessible predictions of human,mouse and monkey MHC class I affinities for peptides of length 8-11.Nucleic Acids Res 36(Web Server issue):W509-512.
35.Lundegaard C,Lund O,&Nielsen M(2008)Accurate approximation method for prediction of class I MHC affinities for peptides of length 8,10 and 11 using prediction tools trained on 9mers.Bioinformatics 24(11):1397-1398.
36.Bouvier M,Guo HC,Smith KJ,&Wiley DC(1998)Crystal structures of HLA-A*0201 complexed with antigenic peptides with either the amino-or carboxyl-terminal group substituted by a methyl group.Proteins 33(1):97-106.
37.Miller,M.D.,et al.(2005)."Ahuman monoclonal antibody neutralizes diverse HIV-1 isolates by binding a critical gp41 epitope."Proc Natl Acad Sci U S A102(41):14759-14764.
38.Ylera,F.,et al.(2013)."Off-rate screening for selection of high-affinity anti-drug antibodies."Anal Biochem 441(2):208-213.
39.D'Angelo SP,et al.(2011)Incidence of EGFR exon 19 deletions and L858R in tumor specimens from men and cigarette smokers with lung adenocarcinomas.J Clin Oncol 29(15):2066-2070.
40.Sharma SV,Bell DW,Settleman J,&Haber DA(2007)Epidermal growth factor receptor mutations in lung cancer.Nat Rev Cancer 7(3):169-181.
41.Salter RD,Howell DN,&Cresswell P(1985)Genes regulating HLA class I antigen expression in T-B lymphoblast hybrids.Immunogenetics 21(3):235-246.
42.Anderson KS,Alexander J,Wei M,&Cresswell P(1993)Intracellular transport of class I MHC molecules in antigen processing mutant cell lines.J Immunol 151(7):3407-3419.
43.Thomas AM,et al.(2004)Mesothelin-specific CD8(+)T cell responses provide evidence of in vivo cross-priming by antigen-presenting cells in vaccinated pancreatic cancer patients.J Exp Med 200(3):297-306.
44.Luft T,et al.(2001)Exogenous peptides presented by transporter associated with antigen processing(TAP)-deficient and TAP-competent cells:intracellular loading and kinetics of presentation.J Immunol 167(5):2529-2537.
45.Brickner AG,et al.(2006)The PANE1 gene encodes a novel human minor histocompatibility antigen that is selectively expressed in B-lymphoid cells and B-CLL.Blood 107(9):3779-3786.
46.Steinwand M,et al.(2014)The influence of antibody fragment format on phage display based affinity maturation of IgG.mAbs 6(1):204-218.
47.McConnell AD,et al.(2013)An integrated approach to extreme thermostabilization and affinity maturation of an antibody.Protein engineering,design&selection:PEDS 26(2):151-164.
48.Miller KR,et al.(2012)T cell receptor-like recognition of tumor in vivo by synthetic antibody fragment.PLoS One 7(8):e43746.
49.Verma B,et al.(2010)TCR mimic monoclonal antibody targets a specific peptide/HLA class I complex and significantly impedes tumor growth in vivo using breast cancer models.J Immunol 184(4):2156-2165.
50.Bernal M,et al.(2012)Implication of theβ2-microglobulin gene in the generation of tumor escape phenotypes.Cancer Immunol Immunother 61(9):1359-71.
51.Nolan JP,Lauer S,Prossnitz ER,&Sklar LA(1999)Flow cytometry:a versatile tool for all phases of drug discovery.Drug Discov Today 4(4):173-180.
52.Porgador A,Yewdell JW,Deng Y,Bennink JR,&Germain RN(1997)Localization,quantitation,and in situ detection of specific peptide-MHC class I complexes using a monoclonal antibody.Immunity 6(6):715-726.
53.Hoof I,van Baarle D,Hildebrand WH,&Kesmir C(2012)Proteome sampling by the HLA class I antigen processing pathway.PLoS computational biology 8(5):e1002517.
54.Wang W,et al.(1997)A naturally processed peptide presented by HLA-A*0201 is expressed at low abundance and recognized by an alloreactive CD8+cytotoxic T cell with apparent high affinity.J Immunol 158(12):5797-5804.
55.De Verteuil D,Granados DP,Thibault P,&Perreault C(2012)Origin and plasticity of MHC I-associated self peptides.Autoimmunity reviews 11(9):627-635.
56.Stone JD,Aggen DH,Schietinger A,Schreiber H,&Kranz DM(2012)A sensitivity scale for targeting T cells with chimeric antigen receptors(CARs)and bispecific T-cell Engagers(BiTEs).Oncoimmunology 1(6):863-873.
57.Jena B,Dotti G,&Cooper LJ(2010)Redirecting T-cell specificity by introducing a tumor-specific chimeric antigen receptor.Blood 116(7):1035-1044.
58.Kershaw MH,Westwood JA,Slaney CY,&Darcy PK(2014)Clinical application of genetically modified T cells in cancer therapy.Clinical&translational immunology 3(5):e16.
59.Link BK,et al.(1998)Anti-CD3-based bispecific antibody designed for therapy of human B-cell malignancy can induce T-cell activation by antigen-dependent and antigen-independentmechanisms.Int J Cancer 77(2):251-256.
60.Thakur A&Lum LG(2010)Cancer therapy with bispecific antibodies:Clinical experience.Current opinion in molecular therapeutics 12(3):340-349.
序列表
<110> 约翰霍普金斯大学
<120> 由体细胞突变基因编码的HLA限制性表位
<130> 001107.01140
<150> 62/136,843
<151> 2015-03-23
<150> 62/186,455
<151> 2015-06-30
<160> 41
<170> FastSEQ for Windows Version 4.0
<210> 1
<211> 27
<212> DNA
<213> 人工序列
<220>
<223> 合成引物
<400> 1
ggataccgct gtctactact gtagccg 27
<210> 2
<211> 23
<212> DNA
<213> 人工序列
<220>
<223> 合成引物
<400> 2
ctgctcaccg tcaccaatgt gcc 23
<210> 3
<211> 10
<212> PRT
<213> 人工序列
<220>
<223> 合成肽
<400> 3
Lys Leu Val Val Val Gly Ala Gly Gly Val
1 5 10
<210> 4
<211> 10
<212> PRT
<213> 人工序列
<220>
<223> 合成肽
<400> 4
Lys Leu Val Val Val Gly Ala Val Gly Val
1 5 10
<210> 5
<211> 10
<212> PRT
<213> 人工序列
<220>
<223> 合成肽
<400> 5
Lys Leu Val Val Val Gly Ala Cys Gly Val
1 5 10
<210> 6
<211> 10
<212> PRT
<213> 人工序列
<220>
<223> 合成肽
<400> 6
Lys Leu Val Val Val Gly Ala Asp Gly Val
1 5 10
<210> 7
<211> 9
<212> PRT
<213> 人工序列
<220>
<223> 合成肽
<400> 7
Val Val Gly Ala Val Gly Val Gly Lys
1 5
<210> 8
<211> 9
<212> PRT
<213> 人工序列
<220>
<223> 合成肽
<400> 8
Val Val Gly Ala Cys Gly Val Gly Lys
1 5
<210> 9
<211> 9
<212> PRT
<213> 人工序列
<220>
<223> 合成肽
<400> 9
Lys Ile Thr Asp Phe Gly Arg Ala Lys
1 5
<210> 10
<211> 9
<212> PRT
<213> 人工序列
<220>
<223> 合成肽
<400> 10
Lys Ile Thr Asp Phe Gly Leu Ala Lys
1 5
<210> 11
<211> 9
<212> PRT
<213> 人工序列
<220>
<223> 合成肽
<400> 11
Val Val Gly Ala Gly Gly Val Gly Lys
1 5
<210> 12
<211> 10
<212> PRT
<213> 人工序列
<220>
<223> 合成肽
<400> 12
Glu Leu Ala Gly Ile Gly Ile Leu Thr Val
1 5 10
<210> 13
<211> 9
<212> PRT
<213> 人工序列
<220>
<223> 合成肽
<400> 13
Leu Leu Gly Arg Asn Ser Phe Glu Val
1 5
<210> 14
<211> 245
<212> PRT
<213> 人工序列
<220>
<223> 合成肽
<400> 14
Met Ala Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala
1 5 10 15
Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asp Val
20 25 30
Asn Thr Ala Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys
35 40 45
Leu Leu Ile Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val Pro Ser Arg
50 55 60
Phe Ser Gly Ser Arg Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser
65 70 75 80
Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Ser
85 90 95
Ser Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr
100 105 110
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Ala Ser Glu Val
115 120 125
Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu
130 135 140
Arg Leu Ser Cys Ala Ala Ser Gly Phe Asn Ile Asn Ser Ser Ser Ile
145 150 155 160
His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Ser
165 170 175
Ile Ala Pro Ala Arg Gly Ser Thr Arg Tyr Ala Asp Ser Val Lys Gly
180 185 190
Arg Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn Thr Ala Tyr Leu Gln
195 200 205
Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ser Arg
210 215 220
Asn Tyr Tyr Ala Tyr Thr Ala Met Asp Val Trp Gly Gln Gly Thr Leu
225 230 235 240
Val Thr Val Ser Ser
245
<210> 15
<211> 234
<212> PRT
<213> 人工序列
<220>
<223> 合成肽
<400> 15
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asp Val Asn Thr Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Ser Ser Pro Pro
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Gly Gly Gly
100 105 110
Ser Gly Gly Gly Ala Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly
115 120 125
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
130 135 140
Phe Asn Ile Asn Thr Ser Tyr Ile His Trp Val Arg Gln Ala Pro Gly
145 150 155 160
Lys Gly Leu Glu Trp Val Ala Ser Ile Tyr Pro Asn Asp Gly Tyr Asn
165 170 175
Arg Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Ala Asp Thr
180 185 190
Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
195 200 205
Thr Ala Val Tyr Tyr Cys Ser Arg Ala Ala Tyr Ala Met Asp Val Trp
210 215 220
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
225 230
<210> 16
<211> 238
<212> PRT
<213> 人工序列
<220>
<223> 合成肽
<400> 16
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asp Val Asn Thr Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Tyr Ser Pro Pro
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Gly Gly Gly
100 105 110
Ser Gly Gly Gly Ala Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly
115 120 125
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
130 135 140
Phe Asn Ile Asn Asn Thr Tyr Ile His Trp Val Arg Gln Ala Pro Gly
145 150 155 160
Lys Gly Leu Glu Trp Val Ala Ser Ile Tyr Pro Thr Asp Gly Tyr Thr
165 170 175
Arg Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Ala Asp Thr
180 185 190
Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
195 200 205
Thr Ala Val Tyr Tyr Cys Ser Arg Thr Tyr Tyr Ser Tyr Tyr Ser Ala
210 215 220
Met Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
225 230 235
<210> 17
<211> 243
<212> PRT
<213> 人工序列
<220>
<223> 合成肽
<400> 17
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asp Val Asn Thr Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Ser Tyr Pro Pro
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Gly Gly Gly
100 105 110
Ser Gly Gly Gly Gly Ser Gly Gly Gly Ala Ser Glu Val Gln Leu Val
115 120 125
Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser
130 135 140
Cys Ala Ala Ser Gly Phe Asn Ile Thr Ser Ser Tyr Ile His Trp Val
145 150 155 160
Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Tyr Ile Ser Pro
165 170 175
Glu Asp Gly Tyr Ala Arg His Ala Asp Ser Val Lys Gly Arg Phe Thr
180 185 190
Ile Ser Ala Asp Thr Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Ser
195 200 205
Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ser Arg Asp Asp Thr
210 215 220
Tyr Tyr Tyr Ser Ala Met Asp Val Trp Gly Gln Gly Thr Leu Val Thr
225 230 235 240
Val Ser Ser
<210> 18
<211> 243
<212> PRT
<213> 人工序列
<220>
<223> 合成肽
<400> 18
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asp Val Asn Thr Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Tyr Tyr Pro Pro
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Gly Gly Gly
100 105 110
Ser Gly Gly Gly Gly Ser Gly Gly Gly Ala Ser Glu Val Gln Leu Val
115 120 125
Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser
130 135 140
Cys Ala Ala Ser Gly Phe Asn Ile Ser Thr Ser Tyr Ile His Trp Val
145 150 155 160
Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Thr Ile Asp Pro
165 170 175
Asn Asp Gly Tyr Ser Arg Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr
180 185 190
Ile Ser Ala Asp Thr Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Ser
195 200 205
Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ser Arg Thr Asn Asn
210 215 220
Thr Ala Ala Asp Ala Met Asp Val Trp Gly Gln Gly Thr Leu Val Thr
225 230 235 240
Val Ser Ser
<210> 19
<211> 241
<212> PRT
<213> 人工序列
<220>
<223> 合成肽
<400> 19
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Ala Cys Arg Ala Ser Gln Asp Val Asn Thr Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Tyr Tyr Pro Pro
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Gly Gly Gly
100 105 110
Ser Gly Gly Gly Gly Ser Gly Gly Gly Ala Ser Glu Val Gln Leu Val
115 120 125
Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser
130 135 140
Cys Ala Ala Ser Gly Phe His Ile Asn Gly Ser Tyr Ile His Trp Val
145 150 155 160
Arg Gln Ala Pro Gly Lys Gly Leu Lys Trp Val Ala Tyr Ile Asp Pro
165 170 175
Glu Thr Gly Tyr Ser Arg Tyr Ala Asp Ser Val Lys Gly Arg Phe Ala
180 185 190
Ile Ser Ala Asp Met Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Ser
195 200 205
Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ser Arg Asp Ser Ala
210 215 220
Ser Asp Ala Met Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser
225 230 235 240
Ser
<210> 20
<211> 241
<212> PRT
<213> 人工序列
<220>
<223> 合成肽
<400> 20
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asp Val Asn Thr Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Tyr Tyr Pro Pro
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Gly Gly Gly
100 105 110
Ser Gly Gly Gly Gly Ser Gly Gly Gly Ala Ser Glu Val Gln Leu Val
115 120 125
Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser
130 135 140
Cys Ala Ala Ser Gly Phe Asn Ile Asn Gly Ser Tyr Ile His Trp Val
145 150 155 160
Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Tyr Ile Asp Pro
165 170 175
Glu Thr Gly Tyr Ser Arg Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr
180 185 190
Ile Ser Ala Asp Thr Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Ser
195 200 205
Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ser Arg Asp Ser Ala
210 215 220
Ser Asp Ala Met Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser
225 230 235 240
Ser
<210> 21
<211> 240
<212> PRT
<213> 人工序列
<220>
<223> 合成肽
<400> 21
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asp Val Asn Thr Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Tyr Tyr Pro Pro
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Gly Gly Gly
100 105 110
Ser Gly Gly Gly Gly Ser Gly Ala Ser Glu Val Gln Leu Val Glu Ser
115 120 125
Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala
130 135 140
Ala Ser Gly Phe Asn Ile Asn Ser Asn Tyr Ile His Trp Val Arg Gln
145 150 155 160
Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Tyr Ile Thr Pro Glu Thr
165 170 175
Gly Tyr Tyr Arg Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser
180 185 190
Ala Asp Thr Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Ser Leu Arg
195 200 205
Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ser Arg Asn Tyr Tyr Ser Ala
210 215 220
Tyr Ala Met Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
225 230 235 240
<210> 22
<211> 240
<212> PRT
<213> 人工序列
<220>
<223> 合成肽
<400> 22
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asp Val Asn Thr Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Tyr Tyr Pro Pro
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Gly Gly Gly
100 105 110
Ser Gly Gly Gly Gly Ser Gly Ala Ser Glu Val Gln Leu Val Glu Ser
115 120 125
Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala
130 135 140
Ala Ser Gly Phe Asn Ile Asn Ser Asn Tyr Ile His Trp Val Arg Gln
145 150 155 160
Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Tyr Ile Thr Pro Glu Thr
165 170 175
Gly Tyr Tyr His Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser
180 185 190
Ala Asp Thr Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Ser Leu Arg
195 200 205
Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ser Arg Asn Tyr Tyr Ser Ala
210 215 220
Tyr Ala Met Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
225 230 235 240
<210> 23
<211> 240
<212> PRT
<213> 人工序列
<220>
<223> 合成肽
<400> 23
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asp Val Asn Thr Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Phe Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Tyr Tyr Pro Pro
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Gly Gly Gly
100 105 110
Ser Gly Gly Gly Gly Ser Gly Ala Ser Glu Val Gln Leu Val Glu Ser
115 120 125
Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala
130 135 140
Ala Ser Gly Phe Tyr Ile Asn Ser Asn Tyr Ile His Trp Val Arg Gln
145 150 155 160
Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Tyr Ile Thr Pro Glu Thr
165 170 175
Gly Tyr Tyr His Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser
180 185 190
Ala Asp Thr Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Ser Leu Arg
195 200 205
Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ser Arg Asn Tyr Tyr Ser Ala
210 215 220
Tyr Ala Met Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
225 230 235 240
<210> 24
<211> 240
<212> PRT
<213> 人工序列
<220>
<223> 合成肽
<400> 24
Asp Ile Gln Met Ser Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Thr Ser Gln Asp Ala Asn Thr Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Phe
35 40 45
Tyr Ser Ala Ser Phe Leu Phe Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Tyr Tyr Pro Pro
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Gly Gly Gly
100 105 110
Ser Gly Gly Gly Gly Ser Gly Ala Ser Glu Val Gln Leu Val Glu Ser
115 120 125
Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala
130 135 140
Ala Ser Gly Phe Asn Ile Asn Ser Asn Tyr Ile His Trp Val Arg Gln
145 150 155 160
Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Tyr Ile Thr Pro Glu Thr
165 170 175
Gly Tyr Tyr Arg Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser
180 185 190
Ala Asp Thr Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Ser Leu Arg
195 200 205
Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ser Arg Asn Tyr Tyr Ser Ala
210 215 220
Tyr Ala Met Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
225 230 235 240
<210> 25
<211> 234
<212> PRT
<213> 人工序列
<220>
<223> 合成肽
<400> 25
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asp Val Asn Thr Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Ser Pro Pro
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Gly Gly Gly
100 105 110
Ser Gly Gly Gly Ala Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly
115 120 125
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
130 135 140
Phe Asn Ile Asn Asp Thr Tyr Ile His Trp Val Arg Gln Ala Pro Gly
145 150 155 160
Lys Gly Leu Glu Trp Val Ala Tyr Ile Ser Pro Ala Ser Gly Asn Ser
165 170 175
Arg Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Ala Asp Thr
180 185 190
Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
195 200 205
Thr Ala Val Tyr Tyr Cys Ser Arg Ser Tyr Ala Ala Met Asp Val Trp
210 215 220
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
225 230
<210> 26
<211> 9
<212> PRT
<213> 人工序列
<220>
<223> 合成肽
<400> 26
Lys Val Tyr Glu Gly Val Trp Lys Lys
1 5
<210> 27
<211> 9
<212> PRT
<213> 人工序列
<220>
<223> 合成肽
<400> 27
Thr Thr Ala Pro Phe Leu Ser Gly Lys
1 5
<210> 28
<211> 9
<212> PRT
<213> 人工序列
<220>
<223> 合成肽
<400> 28
Ile Met Gln Leu Met Pro Phe Gly Cys
1 5
<210> 29
<211> 10
<212> PRT
<213> 人工序列
<220>
<223> 合成肽
<400> 29
Leu Ile Met Gln Leu Met Pro Phe Gly Cys
1 5 10
<210> 30
<211> 9
<212> PRT
<213> 人工序列
<220>
<223> 合成肽
<400> 30
Lys Ile Thr Asp Phe Gly Arg Ala Lys
1 5
<210> 31
<211> 10
<212> PRT
<213> 人工序列
<220>
<223> 合成肽
<400> 31
Lys Leu Val Val Val Gly Ala Val Gly Val
1 5 10
<210> 32
<211> 10
<212> PRT
<213> 人工序列
<220>
<223> 合成肽
<400> 32
Gly Met Asn Trp Arg Pro Ile Leu Thr Ile
1 5 10
<210> 33
<211> 241
<212> PRT
<213> 人工序列
<220>
<223> 合成抗体
<400> 33
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asp Val Asn Thr Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Tyr Tyr Pro Pro
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Gly Gly Gly
100 105 110
Ser Gly Gly Gly Gly Ser Gly Gly Gly Ala Ser Glu Val Gln Leu Val
115 120 125
Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser
130 135 140
Cys Ala Ala Ser Gly Phe Asn Ile Asn Gly Ser Tyr Ile His Trp Val
145 150 155 160
Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Tyr Ile Asp Pro
165 170 175
Glu Thr Gly Tyr Ser Arg Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr
180 185 190
Ile Ser Ala Asp Thr Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Ser
195 200 205
Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ser Arg Asp Ser Ala
210 215 220
Ser Asp Ala Met Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser
225 230 235 240
Ser
<210> 34
<211> 241
<212> PRT
<213> 人工序列
<220>
<223> 合成抗体
<400> 34
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Ala Cys Arg Ala Ser Gln Asp Val Asn Thr Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Tyr Tyr Pro Pro
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Gly Gly Gly
100 105 110
Ser Gly Gly Gly Gly Ser Gly Gly Gly Ala Ser Glu Val Gln Leu Val
115 120 125
Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser
130 135 140
Cys Ala Ala Ser Gly Phe His Ile Asn Gly Ser Tyr Ile His Trp Val
145 150 155 160
Arg Gln Ala Pro Gly Lys Gly Leu Lys Trp Val Ala Tyr Ile Asp Pro
165 170 175
Glu Thr Gly Tyr Ser Arg Tyr Ala Asp Ser Val Lys Gly Arg Phe Ala
180 185 190
Ile Ser Ala Asp Met Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Ser
195 200 205
Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ser Arg Asp Ser Ala
210 215 220
Ser Asp Ala Met Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser
225 230 235 240
Ser
<210> 35
<211> 243
<212> PRT
<213> 人工序列
<220>
<223> 合成抗体
<400> 35
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asp Val Asn Thr Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Ser Tyr Pro Pro
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Gly Gly Gly
100 105 110
Ser Gly Gly Gly Gly Ser Gly Gly Gly Ala Ser Glu Val Gln Leu Val
115 120 125
Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser
130 135 140
Cys Ala Ala Ser Gly Phe Asn Ile Thr Ser Ser Tyr Ile His Trp Val
145 150 155 160
Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Tyr Ile Ser Pro
165 170 175
Glu Asp Gly Tyr Ala Arg His Ala Asp Ser Val Lys Gly Arg Phe Thr
180 185 190
Ile Ser Ala Asp Thr Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Ser
195 200 205
Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ser Arg Asp Asp Thr
210 215 220
Tyr Tyr Tyr Ser Ala Met Asp Val Trp Gly Gln Gly Thr Leu Val Thr
225 230 235 240
Val Ser Ser
<210> 36
<211> 9
<212> PRT
<213> 人工序列
<220>
<223> 合成肽
<400> 36
Ala Thr Ala Pro Ser Leu Ser Gly Lys
1 5
<210> 37
<211> 241
<212> PRT
<213> 人工序列
<220>
<223> scFv序列
<400> 37
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asp Val Asn Thr Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Tyr Tyr Pro Pro
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Gly Gly Gly
100 105 110
Ser Gly Gly Gly Gly Ser Gly Gly Gly Ala Ser Glu Val Gln Leu Val
115 120 125
Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser
130 135 140
Cys Ala Ala Ser Gly Phe Asn Ile Asn Gly Ser Tyr Ile His Trp Val
145 150 155 160
Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Tyr Ile Asp Pro
165 170 175
Glu Thr Gly Tyr Ser Arg Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr
180 185 190
Ile Ser Ala Asp Thr Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Ser
195 200 205
Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ser Arg Asp Ser Ala
210 215 220
Ser Asp Ala Met Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser
225 230 235 240
Ser
<210> 38
<211> 241
<212> PRT
<213> 人工序列
<220>
<223> scFv序列
<400> 38
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Ala Cys Arg Ala Ser Gln Asp Val Asn Thr Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Tyr Tyr Pro Pro
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Gly Gly Gly
100 105 110
Ser Gly Gly Gly Gly Ser Gly Gly Gly Ala Ser Glu Val Gln Leu Val
115 120 125
Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser
130 135 140
Cys Ala Ala Ser Gly Phe His Ile Asn Gly Ser Tyr Ile His Trp Val
145 150 155 160
Arg Gln Ala Pro Gly Lys Gly Leu Lys Trp Val Ala Tyr Ile Asp Pro
165 170 175
Glu Thr Gly Tyr Ser Arg Tyr Ala Asp Ser Val Lys Gly Arg Phe Ala
180 185 190
Ile Ser Ala Asp Met Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Ser
195 200 205
Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ser Arg Asp Ser Ala
210 215 220
Ser Asp Ala Met Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser
225 230 235 240
Ser
<210> 39
<211> 243
<212> PRT
<213> 人工序列
<220>
<223> scFv序列
<400> 39
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asp Val Asn Thr Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Ser Tyr Pro Pro
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Gly Gly Gly
100 105 110
Ser Gly Gly Gly Gly Ser Gly Gly Gly Ala Ser Glu Val Gln Leu Val
115 120 125
Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser
130 135 140
Cys Ala Ala Ser Gly Phe Asn Ile Thr Ser Ser Tyr Ile His Trp Val
145 150 155 160
Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Tyr Ile Ser Pro
165 170 175
Glu Asp Gly Tyr Ala Arg His Ala Asp Ser Val Lys Gly Arg Phe Thr
180 185 190
Ile Ser Ala Asp Thr Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Ser
195 200 205
Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ser Arg Asp Asp Thr
210 215 220
Tyr Tyr Tyr Ser Ala Met Asp Val Trp Gly Gln Gly Thr Leu Val Thr
225 230 235 240
Val Ser Ser
<210> 40
<211> 243
<212> PRT
<213> 人工序列
<220>
<223> 单链可变区
<400> 40
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asp Val Asn Thr Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Tyr Ser Pro Pro
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Gly Gly Gly
100 105 110
Ser Gly Gly Gly Gly Ser Gly Gly Gly Ala Ser Glu Val Gln Leu Val
115 120 125
Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser
130 135 140
Cys Ala Ala Ser Gly Phe Asn Ile Thr Ser Ser Tyr Ile His Trp Val
145 150 155 160
Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Tyr Ile Ser Pro
165 170 175
Ala Asp Gly Tyr Asn Arg Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr
180 185 190
Ile Ser Ala Asp Thr Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Ser
195 200 205
Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ser Arg Thr Asp Ser
210 215 220
Thr Ala Tyr Thr Ala Met Asp Val Trp Gly Gln Gly Thr Leu Val Thr
225 230 235 240
Val Ser Ser
<210> 41
<211> 243
<212> PRT
<213> 人工序列
<220>
<223> 单链可变区
<400> 41
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asp Val Asn Thr Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Ser Tyr Pro Pro
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Gly Gly Gly
100 105 110
Ser Gly Gly Gly Gly Ser Gly Gly Gly Ala Ser Glu Val Gln Leu Val
115 120 125
Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser
130 135 140
Cys Ala Ala Ser Gly Phe Asn Ile Asn Ser Ser Tyr Ile His Trp Val
145 150 155 160
Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Tyr Ile Ser Pro
165 170 175
Thr Asp Gly Tyr Tyr Arg Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr
180 185 190
Ile Ser Ala Asp Thr Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Ser
195 200 205
Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ser Arg Thr Ser Asp
210 215 220
Thr Ser Tyr Ala Ala Met Asp Val Trp Gly Gln Gly Thr Leu Val Thr
225 230 235 240
Val Ser Ser
- 还没有人留言评论。精彩留言会获得点赞!