用于产生1,3‑丁二醇的方法和微生物与流程
本申请要求美国专利申请no.62/195,011的优先权。
本申请包括作为其描述的一部分的包括42条序列且以电子形式与本申请一起提交的序列表,并且该序列表以其整体并入本申请中。
本公开涉及用于产生包括1,3-丁二醇(1,3-bdo)在内的有机化合物的生物合成工艺。
技术背景
1,3-丁二醇可用作1,3-丁二烯的前体,1,3-丁二烯是一种常用商品化学品,每年全球市场总值约为220亿美元。1,3-丁二烯是用于生产橡胶、乳胶、树脂和塑料的重要基础材料。目前,1,3-丁二烯是使用石油基工艺生产的。近来,人们的兴趣主要集中在使用生物技术途径生产1,3-丁二醇上,主要原因是有机会从1,3-丁二醇中催化生产1,3-丁二烯。1,3-丁二醇除了作为1,3-丁二烯的前体外,还可应用于化妆品和药品中。1,3-丁二醇的对映纯形式,诸如(r)-1,3-丁二醇,可用于合成诸如信息素、芳香剂和杀虫剂的高价值化合物。仍在继续寻求1,3-丁二醇的生产技术。
概述
一方面,提供了具有1,3-bdo途径的非天然存在的微生物。所述微生物包括以下1,3-bdo途径酶中的至少一种:催化两个乙醛缩合以产生3-羟基丁醛的醛缩酶;和将3-羟基丁醛还原为1,3-bdo的醛酮还原酶、氧化还原酶、醛还原酶或醇脱氢酶。所述微生物具有至少一种编码来自所述1,3-bdo途径的酶的外源性核酸。
在一个实施方案中,非天然存在的微生物包含至少一种对编码来自所述1,3-bdo途径的酶或影响来自所述1,3-bdo途径的酶的表达的内源性核酸的修饰。
在一个实施方案中,微生物具有编码醛缩酶的外源性核酸。在一个实施方案中,外源性核酸包含seqidno:1、2、3、4、5、6、7、8、9或10的核苷酸序列。在一个实施方案中,醛缩酶是脱氧核糖-5-磷酸醛缩酶(dera)。在一个实施方案中,醛缩酶包含seqidno:15、16、17、18、19、20、21、22、23或24的氨基酸序列或其活性片段或同源物。在一个实施方案中,醛缩酶包含在酶的活性位点中的以下保守的氨基酸残基:赖氨酸167、赖氨酸201、天冬氨酸16和天冬氨酸102,其中与每个残基相关的编号是指seqidno.20的大肠埃希氏菌dera的氨基酸序列中的残基编号。
在一个实施方案中,所述微生物包含编码醛酮还原酶、氧化还原酶、醛还原酶或醇脱氢酶的外源性核酸。在一个实施方案中,所述微生物表达包含seqidno:25的氨基酸序列或其活性片段或同源物的醛酮还原酶。在一个实施方案中,醛酮还原酶、氧化还原酶、醛还原酶或醇脱氢酶包含nadp结合袋和活性位点中的以下保守残基:arg214、arg227、arg281、gln285、gly279、arg208,其中第二编号是指seqid25的氨基酸序列中的氨基酸残基。在一个实施方案中,外源性核酸包含seqidno.11的核苷酸序列。
在一个实施方案中,非天然存在的微生物还包含:能够使丙酮酸盐脱羧以产生乙醛和二氧化碳的脱羧酶。在各种实施方案中,脱羧酶包括丙酮酸脱羧酶(pdc),其可包含seqidno:26的氨基酸序列或其活性片段或同源物;苯甲酰甲酸脱羧酶(bfd),其可包含seqidno:27的氨基酸序列或其活性片段或同源物;或α-酮酸脱羧酶(alpha-detoaciddecarboxylase)(kdc),其可包含seqidno:28的氨基酸序列或其活性片段或同源物。所述微生物可可替代地或另外表达表3中鉴定的酶。
在一个实施方案中,相较于野生型,编码利用丙酮酸盐的酶的一种或多种基因从非天然存在的微生物中缺失。在一个实施方案中,相较于野生型,编码醇脱氢酶、乳酸脱氢酶或丙酮酸甲酸裂解酶的一种或多种基因相从非天然存在的微生物中缺失。
还提供了用于产生1,3-bdo的方法,其包括在产生1,3-bdo的条件下培养如本文所述的微生物并持续足够的时间段。在一个实施方案中,所述方法在基本上厌氧的培养基中进行。
还提供了生物合成工艺,其包括:使用来自醛缩酶类别的酶将两个乙醛分子缩合为3-羟基丁醛;和使用属于醛酮还原酶类别、氧化还原酶类别、醛还原酶类别或醇脱氢酶类别的酶将3-羟基丁醛选择性还原为1,3-bdo。在一个实施方案中,所述工艺还包括通过生物合成方法产生乙醛。在一个实施方案中,所述工艺还包括使丙酮酸盐脱羧以获得乙醛。
还提供了产生对映纯(r)-1,3-bdo的方法,其包括培养如本文所述的非天然存在的微生物或进行如本文所述的生物合成工艺。
附图简述
图1显示了根据本发明的一个方面的乙醛转化为1,3-bdo的示意图。
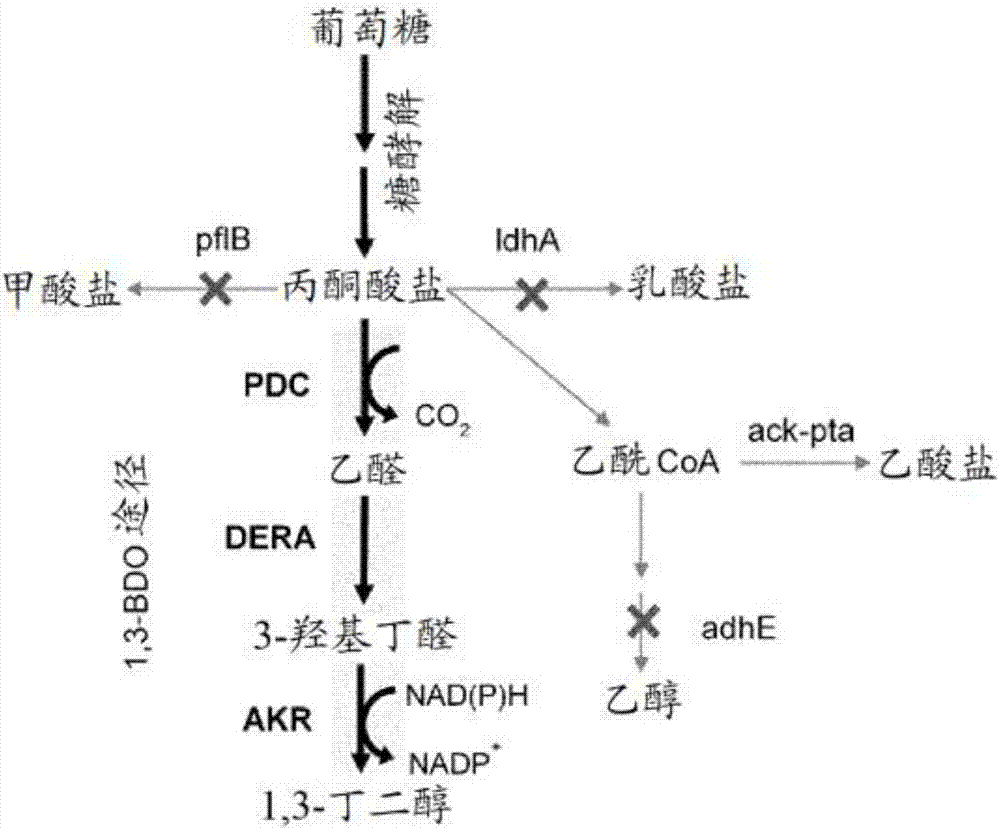
图2显示了可能由经遗传修饰的大肠埃希氏菌实施的使用葡萄糖进行的1,3-丁二醇产生的合成途径。
图3示意性地示出了五个公布的使用发酵产生1,3-bdo的途径。
图4示意性地示出了使用mafft进行的十五种dera(脱氧核糖-5-磷酸醛缩酶)酶的多重序列比对。使用blosum62矩阵。位于dera的磷酸盐-结合袋中的五个残基用框突出显示,且基于与tm1559和ec1535的比较。这些残基对于来自海栖热袍菌(thermotogamaritima)(tm1559)的dera中存在的那些残基是保守的,但不同于来自大肠埃希氏菌(escherichiacoli)(ec1535)的dera。
图5示意性地示出了使用mafft进行的十五种dera酶的多重序列比对。使用blosum62矩阵。位于活性位点中且在使用供体乙醛形成所需的席夫碱中间体(schiff-basedintermediate)方面起作用的四个残基用框突出显示,且基于与tm1559和ec1535的比较。这些残基在所有dera酶中是严格保守的(除了d16(编号是指ec1535序列),其在所有序列中是保守的,除了具有甘氨酸残基的tm1559之外)。
图6示意性地示出了醛酮还原酶的多重序列比对。
图7示意性地示出了三种脱羧酶的多重序列比对。
图8示意性地示出了筛选十种醛缩酶(各自来自不同的物种)用于乙醛羟醛缩合的结果。比活性根据在含有0.8mg每种酶的反应混合物中经5小时乙醛缩合变化确定。经筛选的醛缩酶包括来自大肠埃希氏菌(ec1535)、蜡样芽胞杆菌(bacilluscereus)(bce1975)、嗜碱芽孢杆菌(bacillushalodurans)(bh1352)和枯草芽孢杆菌(bacillussubtilis)(bsu3938)的dera。
详述
本公开描述了非天然存在的微生物,其通过表达编码参与乙醛转化为1,3-丁二醇(1,3-bdo)的新型生物化学途径的酶的基因进行工程化。本公开还描述了可用于改善1,3-bdo生产途径的性能的另外的遗传修饰。遗传修饰可以朝向优化表达系统或针对非天然生物以改善生产指标,包括产率、效价和生产力。另外,遗传修饰可以旨在改善非天然微生物的特征,包括但不限于对原料中发现的抑制剂的耐受性、产物耐受性、耐高渗和有效产物分泌。
定义
如本文所用,术语1,3-bdo用于指代1,3-丁二醇,其也被称为丁二醇(butyleneglycol)、1,3-丁二醇(1,3-butyleneglycol)、丁烷-1,3-二醇、1,3-二羟基丁烷,且以(r)-1,3-丁二醇或(s)-1,3-丁二醇呈对映纯形式。
如本文所用,术语3-hb和3hb可互换使用来指代3-羟基丁醛,其也被称为3-羟基丁醛(3-hydroxybutaraldehyde)、羟基丁醛且以(r)-3-羟基丁醛或(s)-3-羟基丁醛呈对映纯形式。
如本文所用,术语dera是指属于醛缩酶类别的酶脱氧核糖-5-磷酸醛缩酶,术语akr是指醛酮还原酶类别,术语adh是指酶醇脱氢酶,术语pdc是指酶丙酮酸脱羧酶,术语bfd是指酶苯甲酰甲酸脱羧酶且术语kdc是指酶α-酮酸脱羧酶,
如本文所用的“酶”包括由细胞产生的能够催化生物化学反应的蛋白质。此外,除非上下文中另外地指出,否则如本文所用的“酶”包括保留相关催化活性的蛋白质片段,并且可能包括合成的保留相关催化活性的人工酶。
与酶或(多)肽相关的表达“来源于”表示酶或(多)肽从(微)生物分离,或其包括从此类(微)生物分离或表征的酶或(多)肽的氨基酸序列的全部或生物活性部分。
如本文所用,术语“微生物”意在指代任何以微观细胞形式存在的生物,且涵盖原核或真核细胞或具有微观尺寸的生物,并且包括所有物种的细菌、古细菌和真细菌以及真核微生物诸如酵母菌和真菌。该术语还包括任何物种的细胞培养物(包括如植物和哺乳动物细胞),其可以经培养用于生物化学物质产生。
如本文所用,术语“非天然存在的”当用于指微生物时是指微生物具有至少一个在参考物种的天然存在的菌株(包括参考物种的野生型菌株)中不常见的遗传改变。遗传改变包括例如引入编码代谢多肽的可表达的核酸的修饰、其它核酸添加、核酸缺失和/或微生物遗传物质的其它功能性破坏。此类修饰包括例如用于所参考物种的异源性多肽、同源性多肽或异源性多肽和同源性多肽两者的编码区和其功能片段。另外的修饰包括,例如其中修饰改变基因或操纵子的表达的非编码调控区。
术语“内源性”是指源自宿主微生物的参考分子或活性。类似地,该术语当用于指代编码核酸的表达时是指微生物内所含的编码核酸的表达。
如本文所用,术语“外源性”是指引入宿主微生物中的分子或活性。可以例如通过将编码核酸引入宿主遗传物质中,诸如通过整合到宿主染色体中或作为非染色体遗传物质诸如质粒来引入分子。关于编码核酸的表达,该术语是指将编码核酸以可表达的形式引入微生物中。当用于指生物合成活性时,该术语是指被引入参考宿主生物中的活性。来源可以是,例如引入宿主微生物中后表现出该活性的编码核酸。
如本文所用,“同源物”是指功能上等效的蛋白质,即具有与具有指定序列识别号的氨基酸序列的酶相同的酶促活性,但在氨基酸序列中可以具有有限数量的氨基酸取代、缺失、插入或添加。为了维持蛋白质的功能,取代可以是保守取代,从而将一种氨基酸用具有相似性质的氨基酸替代。
在各个方面,每种酶的同源物是指一种与对应于该酶的seqidno的氨基酸序列具有至少25%、至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%或至少99%的同一性并保留酶促活性的蛋白质。用于测定序列同一性的算法是公开可用的,并且包括例如通过美国国家生物技术信息中心(nationalcenterforbiotechnologyinformation)(ncbi)可获得的blast。本领域技术人员可以确定序列是否相似至指示同源性且从而相似或相同的功能的程度。
本领域技术人员可通过使用诸如位点特异性诱变的方法对由本文公开的核苷酸组成的酶的dna适当地引入取代、缺失、插入和/或添加来获得编码每种酶的同源物的多核苷酸(nucleicacidres.10,第6487页(1982),methodsinenzymol.100,第448页(1983),molecularcloning第2版,coldspringharborlaboratorypress(1989),pcrapracticalapproachirlpress第200页(1991))。编码每种酶的同源物的多核苷酸可被引入宿主中并在其中表达,以获得同源物。
本文所述的每种酶可以附接到另外的氨基酸序列上,只要它保留了与该酶的活性功能上等效的活性即可。如上所提及,应理解的是,每种酶或其同源物可以是(多)肽片段,只要其保留与该酶的活性功能等效的活性即可。
术语“异源性”是指源自除所提及的物种以外的的分子或活性,而“同源性”是指来自宿主微生物生物的分子或活性。因此,本发明的编码核酸的外源性表达可以使用异源性或同源性编码核酸中的一者或两者。
如本文所用,术语“可操作地连接”是指一个或多个表达控制序列与待表达的多核苷酸中的编码区之间的键联,以这样的一种方式使得在与表达控制序列相容的条件下实现表达。
1,3-bdo用作聚酯增塑剂制造中的中间体,且也可用作食品业和化妆品业应用中香料的溶剂。此外,光学纯(r)-1,3-bdo用于合成各种光学活性化合物诸如信息素、芳香剂和杀虫剂,并且是用于合成青霉烯类和碳青霉烯类抗生素的中间体[zheng,r.-c.,ge,z.,qiu,z.-k.,wang,y.-s.&zheng,y.-g.asymmetricsynthesisof(r)-1,3-butanediolfrom4-hydroxy-2-butanonebyanewlyisolatedstraincandidakruseizjb-09162.appliedmicrobiologyandbiotechnology94,969–76(2012)]。
可以使用生物技术途径生产1,3-bdo,这主要是由于有机会从1,3-bdo催化产生1,3-丁二烯[burgard,a.p.,burk,m.j.&pharkya,p.methodsandorganismsforconvertingsynthesisgasorothergaseouscarbonsourcesandmethanolto13butanediol.(2011)]。1,3-丁二烯是一种潜在市场价值超过220亿美元的基础材料化学品,且可用于制造橡胶、乳胶和树脂,以及包含其中一种或多种的制品例如轮胎。使用1,3-bdo作为组分或中间体生产的产品的非限制性实例包括有机溶剂、聚氨酯树脂、聚酯树脂和降血糖药。因此,在一些实施方案中,提供了有机溶剂、聚氨酯树脂、聚酯树脂和降血糖药,其具有根据本文所述的工艺制备的1,3-bdo作为组分或使用根据本文所述的工艺制备的1,3-bdo作为中间体产生。
一方面,提供了使用酶促转化使用乙醛作为前体产生1,3-bdo的新型途径。
一方面,公开了包括以下步骤的途径:使用来自醛缩酶类别的酶将两个乙醛分子缩合为3-羟基丁醛;和使用属于醛酮还原酶/氧化还原酶/醛还原酶/醇脱氢酶类别(ec1.1.1.a)的酶将3-羟基丁醛选择性还原为1,3-bdo。图1显示了该途径的示意图。
在另一方面,公开了包括以下步骤的途径:根据下述途径中的一个或多个产生乙醛;使用来自醛缩酶类别的酶将两个乙醛分子缩合为3-羟基丁醛;和使用属于醛酮还原酶/氧化还原酶/醛还原酶/醇脱氢酶类别(ec1.1.1.a)的酶将3-羟基丁醛选择性还原为1,3-bdo。参见图2。
在一些实施方案中,由新型途径产生的中间体3-羟基丁醛呈对映异构体过量。
在一些实施方案中,过量产生的3-羟基丁醛的对映异构体是(r)-3-羟基丁醛。
在一些实施方案中,由新型途径产生的1,3-bdo呈对映异构体过量。
在一些实施方案中,过量产生的1,3-bdo的对映异构体是(r)-1,3-bdo。
与公布的需要至少4-5个步骤,每个步骤通过异源性酶催化以产生1,3-bdo的多步骤途径(图3显示了此类途径的实例)相比,该途径较短,仅需要两个简单的酶催化步骤来合成1,3-bdo。此外,前述大多数途径使用难以合成的前体诸如丙氨酸、4-羟基丁酰-coa等。前体乙醛的优势在于,实现高通量是简单的,因为它已经存在于大肠埃希氏菌和酿酒酵母两者的中心碳代谢中。
一方面,公开的工艺包括使用如本文所述的醛缩酶缩合两个醛分子,由使用如本文所述的醛缩酶两个醛分子组成或基本上由使用如本文所述的醛缩酶缩合两个醛分子组成。
醛缩酶通过将亲核供体立体控制加成到亲电子醛受体上来催化羟醛缩合。由于机理的要求,醛缩酶对亲核供体组分相当专一,但在受体范围方面表现出很大的灵活性。因此,醛缩酶根据其亲核供体分类。不同类别的醛缩酶是1)乙醛依赖性醛缩酶,2)丙酮酸盐/磷酸烯醇丙酮酸盐依赖性醛缩酶,3)二羟丙酮磷酸/二羟丙酮依赖性醛缩酶,和4)甘氨酸依赖性醛缩酶。
在一些实施方案中,醛缩酶可为乙醛依赖性醛缩酶。
在一些实施方案中,醛可为供体或受体。
在一些实施方案中,供体可包括乙醛(acetaldehyde)(乙醛(ethanal))、丙醛、2-甲基丙醛、甲基乙二醛、乳醛、乙醇醛或丙烯醛。
在一些实施方案中,供体可为非醛,包括丙酮酸盐、丙酮(propanone)(丙酮(acetone))、乙醛酸或3-丙烯醇。
在一些实施方案中,受体可包括以下中的一个或多个:乙醛(乙醛)、丙醛、丁醛、异丁醛、2-甲基-1-丁醛、3-甲基-1-丁醛、戊醛、己醛、3-甲基-1-戊醛、4-甲基-1-戊醛、琥珀酸半醛、乳醛、乙二醇醛(glycoldehyde)、甘油醛、2-苯基乙醛、肉桂醛、乙二醛、乙醛酸、甲基乙二醛、丙烯醛、丁二醛、戊二醛、己二醛、丙二醛、丙二酸半醛(3-氧代丙酸)、粘康酸半醛或2-羟基粘康酸半醛。
如上所述,将两个乙醛分子缩合为3-羟基丁醛可使用来自醛缩酶类别的酶进行。在一个实施方案中,来自醛缩酶类别的酶是脱氧核糖-5-磷酸醛缩酶(dera)(ec4.1.2.4.)。
在一些实施方案中,dera酶可以描述为形成共价席夫碱中间体的i类醛缩酶。在所有研究的结构中,dera采用经典的八叶tim桶形折叠。dera的寡聚状态似乎取决于生物的温度。例如,来自大肠埃希氏菌的dera是同型二聚体,而来自海栖热袍菌的dera是同型四聚体。寡聚程度似乎不影响催化,但可能影响各种条件下的稳定性。
一方面,如本文所述的dera源自以下各属的微生物:芽孢杆菌属(bacillus)、埃希氏菌属(escherichia)、热袍菌属(thermotoga)、奇球菌属(deinococcus)、李斯特菌属(listeria)、葡萄球菌属(staphylococcus)、链球菌属(streptococcus)和甲烷嗜热杆菌(methanothermobacter)。在某些实施方案中,dera源自嗜碱芽孢杆菌、蜡样芽胞杆菌、枯草芽孢杆菌、大肠埃希氏菌、海栖热袍菌、耐辐射奇球菌(deinococcusradiodurans)、单核细胞增多性李斯特氏菌(listeriamonocytogenes)、金黄色葡萄球菌(staphylococcusaureus)、肺炎链球菌(streptococcuspneumonia)和热自养甲烷嗜热杆菌(methanothermobacterthermautotrophicus)。一方面,如本文所述的工艺中所用的dera包含seqidno:15、16、17、18、19、20、21、22、23或24的氨基酸序列或其活性片段或同源物。
在一些实施方案中,dera是包含由包含seqidno:1、2、3、4、5、6、7、8、9或10的核苷酸序列的dna编码的氨基酸序列的酶。
一方面,醛缩酶在酶的活性位点中包含以下保守的氨基酸残基:赖氨酸167、赖氨酸201、天冬氨酸16和天冬氨酸102,其中与每个残基相关的编号是指seqidno.20的大肠埃希氏菌dera的氨基酸序列中的残基编号,和在seqidno2的核苷酸序列中相应的密码子。图4显示了各种dera酶的多重序列比对并以框突出显示了上述的关键保守残基,而图5突出显示了当与大肠埃希氏菌dera相比时在一些dera酶中可能赋予最佳乙醛羟醛缩合的保守残基。
一方面,酶属于pfam数据库[finnr.d.等,pfam:theproteinfamiliesdatabasenucl.acidsres.(2014年1月1日)42(d1):d222-d230]组pf01791(deoc/lacd家族醛缩酶)包括脱氧核糖-5-磷酸醛缩酶,其还属于interpro家族ipr002915、ipr013785、ipr011343和ipr028581。本领域技术人员可以获得属于interpro和pfam蛋白质家族的蛋白质序列,使得它们是本文描述的dera的同源物。
使3-羟基丁醛(3-hydroxybutanaldehyde)还原为1,3-bdo可以通过使用适当的醇脱氢酶(adh)、醛酮还原酶、氧化还原酶或醛还原酶,使用还原性等效物作为辅因子(其在一个实施方案中可为nadh或nadph)进行。然而,在一个实施方案中,adh、akr、氧化还原酶或醛还原酶对于3-羟基丁醛基本上是专一的,并且不作用于乙醛,从而基本上避免或消除作为副产物的乙醇的产生。
在一些实施方案中,本文所述的途径酶的编码核酸的来源没有特别的限制,并且可以包括其中编码的基因产物可以催化相关反应的任何物种。酶可来源于但不限于以下物种:根癌土壤杆菌(agrobacteriumtumefaciens)、蜡样芽胞杆菌、嗜碱芽孢杆菌、枯草芽孢杆菌、幽门螺杆菌(helicobacterpylori)、短乳杆菌(lactobacillusbrevis)、绿脓假单胞菌(pseudomonasaeruginosa)、恶臭假单胞菌(pseudomonasputida)、丁香假单胞菌(pseudomonassynringae)、沼泽红假单胞菌(rhodopseudomonaspalustris)、鼠伤寒沙门氏菌(salmonellatyphimurium)、酿酒酵母(saccharomycescerevisiae)、丙酮丁醇梭菌(clostridiumacetobutylicum)。表1包括示例性醛酮还原酶。
表1
一方面,本文所述的酶可以属于interpro超家族家族ipr023210、ipr001395、ipr018170和ipr020471,其描述了具有β-α-β折叠的酶的醛酮还原酶家族,所述折叠包含平行的8个β/α桶,所述β/α桶含有nadp结合基序。
一方面,提供了能够将3-羟基丁醛选择性还原为1,3-bdo的醇脱氢酶、醛酮还原酶(akr)、氧化还原酶或醛还原酶。一方面,该酶的来源不特别受限。
一方面,本文所述的酶包含nadp结合袋和活性位点中的以下保守残基:arg214、arg227、arg281、gln285、gly279、arg208,其中第二编号指seqid25的氨基酸序列中的氨基酸残基。所描述的两个示例性醛酮还原酶中保守的关键残基显示于图6中的多重序列比对中。
一方面,如本文所述的醛酮还原酶(akr)源自属假单胞菌属的微生物。一方面,如本文提供的akr源自绿脓假单胞菌。一方面,本文所述的工艺中使用的akr包含seqidno:25的氨基酸序列或其活性片段或同源物。
在一些实施方案中,akr是包含由包含seqidno.11的核苷酸序列的dna编码的氨基酸序列的酶。
两步途径的起始代谢物是乙醛,其是常见的中心代谢物,或者可以由丙酮酸盐脱羧来产生。
在一些实施方案中,乙醛通过由丙酮酸脱羧酶(pdc)(ec4.1.1.1)使丙酮酸盐脱羧得到乙醛和二氧化碳来产生。来自酿酒酵母的pdc对于脂肪族2-酮酸具有广泛的底物范围。其已在大肠埃希氏菌中已被广泛研究、工程化和表达[candy,j.m.,duggleby,r.g.,&mattick,j.s.(1991).expressionofactiveyeastpyruvatedecarboxylasein.journalofgeneralmicrobiology,(137),5–9;killenberg-jabs,m.,
表2
一方面,如本文所述的pdc源自属发酵单胞菌属(zymomonas)的微生物。一方面,pdc源自运动发酵单胞菌。一方面,如本文所述的工艺中使用的pdc包含seqidno:26的氨基酸序列或其活性片段或同源物。
在一个实施方案中,pdc是包含由包含seqidno:12的核苷酸序列的dna编码的氨基酸序列的酶。
已显示出对丙酮酸盐起作用用于产生乙醛的丙酮酸脱羧酶包括但不限于源自恶臭假单胞菌的苯甲酰甲酸脱羧酶(bfd)(ec4.1.1.7),和源自乳酸乳球菌(lactococcuslactis)的支链α-酮酸脱羧酶(kdc)[gocke,d.,graf,t.,brosi,h.,frindi-wosch,i.,walter,l.,müller,m.,&pohl,m.(2009).comparativecharacterisationofthiaminediphosphate-dependentdecarboxylases.journalofmolecularcatalysisb:enzymatic,61(1-2),30–35]。此外,pdc和bfd的突变体已经通过定点诱变(包括但不限于:pdci472a、pdci476f、pdci472a/i476f、bfda460i、bfdf464i和bfda460i/f464i)产生,也显示出对朝向乙醛形成的丙酮酸盐的活性[siegert,p.,mcleish,m.j.,baumann,m.,iding,h.,kneen,m.m.,kenyon,g.l.,&pohl,m.(2005).exchangingthesubstratespecificitiesofpyruvatedecarboxylasefromzymomonasmobilisandbenzoylformatedecarboxylasefrompseudomonasputida.proteinengineering,design&selection:peds,18(7),345–57]。
在一个实施方案中,本文所述的bfd包含seqidno:27的氨基酸序列或其活性片段或同源物,以及seqidno:28的kdc的氨基酸序列或其活性片段或同源物。
在一个实施方案中,bfd是包含由包含seqidno:13的核苷酸序列的dna编码的氨基酸序列的酶。
在一个实施方案中,kdc是包含由包含seqidno:14的核苷酸序列的dna编码的氨基酸序列的酶。
在其它实施方案中,本文所述的酶的同源物可用于如本文所述的方法中。图7显示了本文所述的pdc、bfd和kdc的多重序列比对,并突出显示了脱羧活性所需的蛋白质结构域。
尽管在某些实施方案中,乙醛是由pdc通过丙酮酸盐的脱羧获得的,但在其它实施方案中,乙醛是(可替代地或另外地)通过下表3中鉴定的一个或多个反应途径获得的。
表3
反应id参考自2015年5月13日起的京都基因和基因组百科全书(kyotoencyclopediaofgenesandgenomes)(kegg)数据库(http://www.genome.jp/kegg/)。
在一个实施方案中,如本文所述的工艺是用活细胞进行的。在其它实施方案中,所述工艺在体外用裂解细胞或用部分或基本完全纯化的酶进行。在一个实施方案中,所述工艺是用透化细胞进行的。在其它实施方案中,方法在体外进行并且酶被固定。用于将酶固定在不同载体上的手段和方法是本领域技术人员公知的。
表达如本文所述的一种或多种酶的微生物可以以各种形式提供,包括活体形式例如在水溶液中或在培养基中,或以“静息”形式,诸如以冻干或片剂形式。
在一个实施方案中,所述方法在培养物中用一种或多种宿主微生物进行,从而产生一种或多种途径酶。
在一个实施方案中,提供了微生物,其经遗传修饰以便含有编码能够缩合两个乙醛分子的如本文所述的缩醛酶的核酸分子。
在一个实施方案中,提供了具有1,3-bdo途径的非天然存在的微生物,其中所述微生物包括以下1,3-bdo途径酶:催化两个乙醛缩合以产生3-羟基丁醛的醛缩酶;以及将3-羟基丁醛脱氢为1,3-bdo的醛酮还原酶、氧化还原酶、醛还原酶或醇脱氢酶;其中所述微生物包含至少一种编码来自所述1,3-bdo途径的酶的外源性核酸。在一个实施方案中,所述微生物还包含用于丙酮酸盐脱羧以产生乙醛和二氧化碳的pdc。在一个实施方案中,所述微生物表达表3中鉴定的酶或其活性片段或同源物用于产生乙醛。在一个实施方案中,多于一种或所有的核酸对宿主微生物是外源性的。
在一个实施方案中,根据一个实施方案的方法中所用的微生物是经遗传修饰以含有编码能够将两个乙醛分子缩合为3-羟基丁醛的醛缩酶的核酸分子的微生物。在一个实施方案中,所述微生物经遗传修饰以含有编码能够将3-羟基丁醛还原为1,3-bdo的酮还原酶的核酸。在一个实施方案中,所述微生物经遗传修饰以含有编码能够使酮酸盐脱羧以产生乙醛和二氧化碳的pdc的核酸。
当提及包含在微生物中的多于一种外源性核酸时,应理解这是指参考的编码核酸或生物化学活性,而不是引入宿主生物中的的单独核酸的数目。如本领域技术人员将理解的,此类外源性核酸可以在单独的核酸分子上、多顺反子核酸分子上或其组合上引入宿主生物中。例如,当将编码不同的酶促活性的两种或更多种外源性核酸引入到宿主生物中时,两种或更多种外源性核酸可作为单个核酸引入,例如,在单个质粒上、在单独的质粒上,可在单个位点或多个位点整合到宿主染色体中,且仍然被认为是两种或更多种外源性核酸。
如本领域技术人员显而易见的,根据所选宿主微生物,可将一些或全部的所述1,3-bdo途径酶的核酸引入宿主生物中。如果宿主微生物内源性表达一种或多种途径基因,则可能没有必要引入这些基因,而只需要微生物缺乏的那些编码该途径中的一种或多种酶的核酸。如对于本领域技术人员显而易见的是,当选择表达一种或多种途径基因的宿主微生物时,可以对微生物进行工程化,使得编码该酶的基因过表达和/或编码竞争途径的酶或蛋白质的基因可能被缺失。
如本领域技术人员显而易见的,宿主微生物可经工程化以增加nadh和/或nadph的辅因子汇集物以改善朝向1,3-bdo的代谢流量。在一个实施方案中,如果大肠埃希氏菌被用作宿主生物,则可以使葡萄糖磷酸异构酶(pgi)基因缺失以将通量转移到磷酸戊糖途径以增加nadph汇集物。其它策略可能涉及将内源性nadh依赖性甘油醛-3-磷酸脱氢酶(gapdh)转向具有源自丙酮丁醇梭菌的外源性nadph依赖性甘油醛-3-磷酸脱氢酶的宿主大肠埃希氏菌菌株。在另一种方法中,可以将nadh激酶(pos5p)从酿酒酵母中引入宿主大肠埃希氏菌菌株中。后者成功地用于增加通过nadph依赖性途径产生的几种产物[lee,w.-h.,kim,m.-d.,jin,y.-s.,&seo,j.-h.(2013).engineeringofnadphregeneratorsinescherichiacoliforenhancedbiotransformation.appliedmicrobiologyandbiotechnology.97(7):2761-72]。
如对于本领域技术人员显而易见的,如果选择大肠埃希氏菌作为宿主生物,则可以通过限制竞争途径来增加nadh汇集物,尽管编码nadh依赖性酶的基因缺失,所述nadh依赖性酶包括但不限于:醇脱氢酶(adhe)、乳酸脱氢酶(ldha)和丙酮酸-甲酸裂解酶(pflb)(参见图2)。
如本领域技术人员显而易见的,可以在宿主菌株中表达细菌微区室(bmc)以增加辅因子汇集物和途径代谢物浓度,或减少来自途径中间体的副产物形成。此外,bmc可以减少中间体醛对细胞的毒性作用,并减少由于其挥发性性质造成的损失[cai,f.,sutter,m.,bernstein,s.l.,kinney,j.n.,&kerfeld,c.a.(2014).engineeringbacterialmicrocompartmentshells:chimericshellproteinsandchimericcarboxysomeshells.acssynthbiol.2015,4(4):444-53]。已经在大肠埃希氏菌中表达的天然存在的bmc的实例包括来自肠炎沙门氏菌(salmonellaenterica)的丙二醇利用bmc(pdu)和乙醇胺利用bmc(eut)。用于这些bmc的蛋白质壳可以在没有将被1,3-1,3-bdo途径酶替代的内部途径的情况下表达。
宿主微生物可以选自例如细菌、酵母、真菌或可用作宿主生物的多种其它微生物中的任一种以及在它们中产生的非天然存在的微生物。
在一些实施方案中,细菌物种可包括:大肠埃希氏菌、枯草芽孢杆菌、地衣芽孢杆菌(bacilluslicheniformis)、蜡样芽胞杆菌、巨大芽胞杆菌(bacillusmegaterium)、短乳杆菌、短小芽胞杆菌(bacilluspumilus)、谷氨酸棒杆菌(corynebacteriumglutamicum)、运动发酵单胞菌、丙酮丁醇梭菌、丁酸梭菌(clostridiumbutylicum)、克氏梭菌(clostridiumkluyveri)、产乙醇梭菌(clostridiumautoethanogenum)、热醋穆尔氏菌(moorellathermoacetica)、醋酸梭菌(clostridiumaceticum)、拜氏梭菌(clostridiumbeijerinckii)、糖乙酸多丁醇梭菌(clostridiumsaccharoperbutylacetonicum)、产气荚膜梭菌(clostridiumperfringens)、艰难梭菌(clostridiumdifficile)、肉毒梭菌(clostridiumbotulinum)、酪丁酸梭菌(clostridiumtyrobutyricum)、假破伤风梭菌(clostridiumtetanomorphum)、破伤风梭菌(clostridiumtetani)、丙酸梭菌(clostridiumpropionicum)、氨基丁酸梭菌(clostridiumaminobutyricum)、近端梭菌(clostridiumsubterminale)、斯氏梭菌(clostridiumsticklandii)、富养罗尔斯通氏菌(ralstoniaeutropha)、牛分枝杆菌(mycobacteriumbovis)、结核分支杆菌(mycobacteriumtuberculosis)、牙龈卟啉单胞菌(porphyromonasgingivalis)、荧光假单胞菌(pseudomonasfluorescens)、恶臭假单胞菌、绿脓假单胞菌、食羧假单胞菌(pseudomonascarboxidovorans)(食羧寡养菌(oligotrophacarboxidovorans))、施氏假单胞菌(pseudomonasstutzeri)、肺炎克雷伯菌(klebsiellapneumonia)、产酸克雷伯菌(klebsiellaoxytoca)、产琥珀酸厌氧螺菌(anaerobiospirillumsucciniciproducens)、产琥珀酸放线杆菌(actinobacillussuccinogenes)、产琥珀酸曼氏杆菌(mannheimiasucciniciproducens)、菜豆根瘤菌(rhizobiumetli)、氧化葡糖杆菌(gluconobacteroxydans)、乳酸乳球菌、植物乳杆菌(lactobacillusplantarum)、天蓝色链霉菌(streptomycescoelicolor)、弗氏柠檬酸杆菌(citrobacterfreundii)、无丙二酸柠檬酸杆菌(citrobacteramalonaticus)、醋酸钙不动杆菌(acinetobactercalcoaceticus)、贝氏不动杆菌(acinetobacterbaylyi)、海栖热袍菌、盐生盐杆菌(halobacteriumsalinarum)、粘质沙雷氏菌(serratiamarcescens)、深红红螺菌(rhodospirillumrubrum)、艾德昂菌属某种(ideonellasp.)、荚膜红细菌(rhodobactercapsulatus)、荚膜甲基球菌(methylococcuscapsulatus)、发孢甲基弯菌(methylosinustrichosporium)、扭脱甲基杆菌(methylobacteriumextorquens)、甲基孢囊菌属gb25(methylocystisgb25)、荚膜食甲基菌(methylotrophuscapsulatus)、甲基单胞菌属某种16a(methylomonassp.16a)、强烈火球菌(pyrococcusfuriosus)。
在一些实施方案中,酵母或真菌可包括:酿酒酵母、粟酒裂殖酵母(schizosaccharomycespombe)、saccharomycopsiscrataegensis、乳酸克鲁维酵母(kluyveromyceslactis)、马克斯克鲁维酵(kluyveromycesmarxianus)、土曲霉(aspergillusterreus)、黑曲霉(aspergillusniger)、树干毕赤酵母(pichiastipitis)、巴斯德毕赤酵母(pichiapastoris)、少根根霉(rhizopusarrhizus)、米根霉(rhizobusoryzae)、解脂耶氏酵母(yarrowialipoiytica)、东方伊萨酵母(issatchenkiaorientalis)、西方伊萨酵母(issatchenkiaoccidentalis)、郎比可假丝酵母(candidalambica)、山梨木糖假丝酵母(candidasorboxylosa)、candidazemplinina、candidageochares、膜璞毕赤酵母(pichiamembranifaciens)、zygosaccharomyceskombuchaensis、candidasorbosivorans、candidavanderwaltii、candidasorbophila、二孢接合酵母(zygosaccharomycesbisporus)、缓慢接合酵母(zygosaccharomyceslentus)、贝酵母(saccharomycesbayanus)、博伊丁酵母(saccharomycesbulderi)、汉氏德巴利氏酵母(debaryomycescastellii)、博伊丁假丝酵母(candidaboidinii)、埃切假丝酵母(candidaetchellsii)、杰丁毕赤酵母(pichiajadinii)、异常毕赤酵母(pichiaanomala)、产黄青霉(penicilliumchrysogenum)
在一些实施方案中,蓝藻细菌可包括:海滨蓝藻菌mbic11017(acaryochlorismarinambic11017)、鱼腥藻属某种pcc7120(anabaenasp.pcc7120)、多变鱼腥藻atcc29413(anabaenavariabilisatcc29413)、阿格门氏藻(agmenellumquadruplicatum)、绿硫细菌tls(chlorobiumtepidumtls)、蓝丝菌属某种atcc51142(cyanothecesp.atcc51142)、无类囊体蓝藻pcc7421(gloeobacterviolaceuspcc7421)、铜绿微囊藻nies-843(microcystisaeruginosanies-843)、点形念珠藻atcc29133(nostocpunctiformeatcc29133)、海洋原绿球藻med4(prochlorococcusmarinusmed4)、海洋原绿球藻mit9313(prochlorococcusmarinusmit9313)、海洋原绿球藻ss120(prochlorococcusmarinusss120)、海洋原绿球藻菌株as9601(prochlorococcusmarinusstr.as9601)、海洋原绿球藻菌株mit9211(prochlorococcusmarinusstr.mit9211)、海洋原绿球藻菌株mit9215(prochlorococcusmarinusstr.mit9215)、海洋原绿球藻菌株mit9301(prochlorococcusmarinusstr.mit9301)、海洋原绿球藻菌株mit9303(prochlorococcusmarinusstr.mit9303)、海洋原绿球藻菌株mit9312(prochlorococcusmarinusstr.mit9312)、海洋原绿球藻菌株mit9515(prochlorococcusmarinusstr.mit9515)、海洋原绿球藻菌株natl1a(prochlorococcusmarinusstr.natl1a)、海洋原绿球藻菌株natl2a(prochlorococcusmarinusstr.natl2a)、沼泽红假单胞菌cga009、细长聚球藻pcc6301(synechococcuselongatuspcc6301)、聚球藻属某种pcc7942(synechococcuselongatuspcc7942)、聚球藻属某种cc9311(synechococcussp.cc9311)、聚球藻属某种cc9605(synechococcussp.cc9605)、聚球藻属某种cc9902(synechococcussp.cc9902)、聚球藻属某种ja-2-3b(synechococcussp.ja-2-3b)、聚球藻属某种ja-3-3ab(synechococcussp.ja-3-3ab)、聚球藻属某种pcc7002(synechococcussp.pcc7002)、聚球藻属某种rcc307(synechococcussp.rcc307)、聚球藻属某种wh7803(synechococcussp.wh7803)、聚球藻属某种wh8102(synechococcussp.wh8102)、集胞藻属某种pcc6803(synechocystissp.pcc6803)、细长嗜热蓝细菌聚球藻bp-1(thermosynechococcuselongatusbp-1)、红海束毛藻ims101(trichodesmiumerythraeumims101)。
在一些实施方案中,藻类可包括:布朗葡萄藻(botryococcusbraunii)、莱茵衣藻(chlamydomonasreinhardii)、小球藻某种(chlorellasp.)、寇氏隐甲藻(crypthecodiniumcohnii)、细柱藻属某种(cylindrothecasp.)、普氏杜氏藻(dunaliellaprimolecta)、等鞭金藻某种(isochrysissp.)、盐生单肠藻(monallanthussalina)、微球藻某种(nannochlorissp.)、微拟球藻某种(nannochloropsissp.)、富油新绿藻(neochlorisoleoabundans)、菱形藻属某种(nitzschiasp.)、三角褐指藻(phaeodactylumtricornutum)、裂殖壶菌属某种(schizochytriumsp.)、干扁藻(tetraselmissueica)。
然而,在其它实施方案中,宿主微生物不受特别限制,并且本领域技术人员可以使用本领域已知和/或如本文所述的方法将酶促活性或活性引入任何合适的宿主生物中。
大肠埃希氏菌和酿酒酵母是尤为适用的宿主生物,因为它们是适合于基因工程的经充分表征的微生物。此外,乙醛是大肠埃希氏菌和酿酒酵母的天然代谢物,存在于两个物种的中央碳代谢中。
编码如本文所述的酶的核酸分子可以单独或作为载体的一部分使用。核酸分子可以包含可操作地连接至核酸分子中所包含的多核苷酸的表达控制序列。这些表达控制序列可能适于确保细菌或真菌中可翻译rna的转录和合成。表达是指异源性dna序列的转录,优选转录成可翻译的mrna。确保在真菌和细菌中表达的调控元件是本领域技术人员已知的并且包括启动子、增强子、终止信号、靶向信号等。与核酸分子结合使用的启动子就其来源和/或待表达的基因而言可以是同源性的或异源性的。适合的启动子是例如使得它们本身组成型表达的启动子。然而,也可以使用仅在由外部影响决定的时间点激活的启动子。可以使用人工和/或化学诱导型启动子。化学诱导型启动子可以包括但不限于:iptg诱导型启动子诸如t7或ptrc,或四环素诱导型启动子诸如plteto-1,其序列是本领域技术人员已知的。
不同表达系统的综述例如包含于bitter等(methodsinenzymology153(1987),516-544)和sawers等(appliedmicrobiologyandbiotechnology46(1996),1-9),billman-jacobe(currentopinioninbiotechnology7(1996),500-4),hockney(trendsinbiotechnology12(1994),456-463),griffiths等,(methodsinmolecularbiology75(1997),427-440)中。酵母菌表达系统的综述例如由hensing等(antonievanleuwenhoek67(1995),261-279),bussineau等(developmentsinbiologicalstandardization83(1994),13-19),gellissen等(antonievanleuwenhoek62(1992),79-93,fleer(currentopinioninbiotechnology3(1992),486-496),vedvick(currentopinioninbiotechnology2(1991),742-745)和buckholz(bio/technology9(1991),1067-1072)给出。
表达载体已经在文献中被广泛描述。通常,它们不仅含有选择标记基因和确保所选宿主中的复制的复制起点,而且还含有细菌或病毒启动子,并且在大多数情况下含有用于转录的终止信号。在启动子和终止信号之间,通常存在至少一个允许插入编码dna序列的限制性位点或多接头。如果在所选宿主生物中具有活性,那么天然控制相应基因转录的dna序列可以用作启动子序列。然而,这个序列也可以换成其它启动子序列。可能使用确保基因组成型表达的启动子和允许对基因表达精细控制的诱导型启动子。文献中详细描述了具有这些性质的细菌和病毒启动子序列。用于在包括大肠埃希氏菌和酿酒酵母的微生物中表达的调控序列描述在本领域技术人员已知的文献中。允许下游序列的特别高表达的启动子是例如t7启动子[studier等,methodsinenzymology185(1990),60-89]、lacuv5、trp、trp-lacuv5[deboer等,rodriguez和chamberlin(编),promoters,structureandfunction;praeger,newyork,(1982),462-481;deboer等,proc.natl.acad.sci.usa(1983),21-25]、ip1、rac[boros等,gene42(1986),97-100]。文献中也描述了用于转录的终止信号。
可使用可提供比组成型启动子更高的多肽产率的诱导型启动子。适合地,在某些实施方案中,可以使用两阶段工艺:首先在最佳条件下培养宿主细胞直至相对高的细胞密度;然后诱导转录。
如本领域技术人员将理解的,当两种或更多种外源性编码核酸被共表达时,两个核酸可被插入例如一个表达载体中或插入单独的表达载体中。对于单个载体表达,编码核酸可以可操作地连接到常见的表达控制序列,或连接到不同的表达控制序列,诸如一种诱导型启动子和一种组成型启动子。
应当理解,在一个实施方案中,产生途径中间体或产物的非天然存在的微生物可以与表达一种或多种下游或上游途径酶的另一种生物(或其它生物)组合使用来生产期望的产物。例如,野生型或经工程化的生物可用于产生和积累丙酮酸盐、乙醛和/或3-羟基丁酸。然后,这些中间体可用作表达1,3-bdo途径基因中的一个或多个的另一种经工程化生物的底物以转化为1,3-bdo。
在其它实施方案中,可以任选地将如本文提供的微生物工程化以缺失一个或多个副产物或可替代途径。在图2中用x表示此类示例性途径。在一个实施方案中,从宿主微生物中缺失一种或多种编码醇脱氢酶、乳酸脱氢酶或丙酮酸甲酸裂解酶的基因。在一个实施方案中,宿主微生物是大肠埃希氏菌,并且缺失了基因adhe、idha和pflb中的一个或所有。也可缺失大肠埃希氏菌原生且与adhe、ldha和pflb中的一个或多个同源的其它基因。例如,本领域技术人员可以通过多重序列比对算法(诸如clustalw)鉴定与催化乙醛还原成乙醇的adhe共有相似功能的醛还原酶和醇脱氢酶。对乙醛显示活性的大肠埃希氏菌原生的醛还原酶或醇脱氢酶包括但不限于下表4中存在的序列数据。
表4.
然而,本领域技术人员还可以测定上述醛还原酶和醇脱氢酶对在1,3-bdo途径中作为中间体的3-羟基丁醛的活性。在一个实施方案中,可以在宿主生物中过表达本文所述的显示底物偏好和对3-羟基丁醛的活性的一种或多种醛还原酶和醇脱氢酶以改善1,3-bdo产生。本领域技术人员还可以进行序列相似性搜索以鉴定源自其它生物的同源物为对3-羟基丁醛显示活性的大肠埃希氏菌中的原生醛还原酶和醇脱氢酶。
在一些实施方案中,1,3-bdo途径也可能产生副产物,其可能包括乙酸盐、乙醇和乙偶姻。在某些实施方案中,用于将这些副产物转化成乙醛前体的途径过表达。以下(表5)是可将副产物乙酸盐、乙醇和乙偶姻转化为乙醛的途径。
表5.
*反应id参考自2015年5月13日起的京都基因和基因组百科全书(kegg)数据库(http://www.genome.jp/kegg/)。
在一个实施方案中,根据本发明的实施方案使用的丙酮酸盐由可再生原料(诸如葡萄糖)产生。在一个实施方案中,给宿主生物提供糖原料。此类来源包括例如糖,诸如葡萄糖、木糖、阿拉伯糖、半乳糖、甘露糖、果糖、淀粉及其组合。葡萄糖可以从各种含碳水化合物的来源获得,包括常规生物可再生资源诸如玉米(corn/maize)、小麦、马铃薯、木薯和水稻,以及可替代的来源诸如能源作物、植物生物质、农业废弃物、林业残余物、糖加工残余物和植物源的家庭废物。
碳水化合物的来源包括可再生的原料和生物质,例如纤维素生物质、半纤维素生物质和木质素原料。其它可再生原料和生物质将是本领域技术人员已知的。
可根据本发明使用的生物可再生原料来源包括包含碳水化合物来源的任何可再生有机物质。这些包括例如草、树(硬木和软木)、植物和作物残余物。其它来源可以包括例如废料(例如废纸、绿色废物、城市废物等)。适合的碳水化合物,包括葡萄糖,可以使用本领域已知的方法从生物可再生材料中分离。参见,例如,centi和vansanten,catalysisforrenewables,wiley-vch,weinheim2007;kamm,gruber和kamm,biorefineries-industrialprocessesandproducts,wiley-vch,weinheim2006;shang-tianyang,bioprocessingforvalue-addedproductsfromrenewableresourcesnewtechnologiesandapplications,elsevierb.v.2007;furia,starchinthefoodindustry,第8章,crchandbookoffoodadditives第2版crcpress,1973。还参见专用于starch,sugarandsyrupswithinkirk-othmerencyclopediaofchemicaltechnology第5版,johnwileyandsons2001的章节。用于将淀粉转化为葡萄糖的方法也是本领域熟知的,参见,例如,schenck,“glucoseandglucosecontainingsyrups”inullmann'sencyclopediaofindustrialchemistry,wiley-vch2009。此外,将纤维素转化为葡萄糖的方法是本领域已知的,参见,例如,centi和vansanten,catalysisforrenewables,wiley-vch,weinheim2007;kamm,gruber和kamm,biorefineries-industrialprocessesandproducts,wiley-vch,weinheim2006;shang-tianyang,bioprocessingforvalue-addedproductsfromrenewableresourcesnewtechnologiesandapplications,elsevierb.v.2007。
可替代碳源可以是从生物柴油生产植物获得的粗甘油,从废聚乳酸降解获得的乳酸,从乳制品业获得的乳糖或乳酪乳清渗透物,从富含几丁质的废物获得的葡糖胺。碳源还可以是从植物或植物产物诸如菜籽油、椰子油、玉米油、橄榄油、棕榈油、红花油、花生油、大豆油、芝麻油、葵花籽油及其组合获得的脂肪酸及其酯(甘油一酯、甘油二酯和甘油三酯)。
另一种碳源可以是合成气体或“合成气”,其主要是h2和co的混合物,可以含有co2并且是基于有机或化石燃料的含碳材料的气化产物。
诸如一氧化碳(co)、二氧化碳(co2)和甲烷(ch4)的c1化合物可作为原料源自来自诸如钢铁制造、炼油、煤炭工业的废气,和天然气、页岩气、沼气和甲烷水合物,以及呈由可持续资源(诸如生物质以及生活废弃物和农业废弃物)气化产生的合成气体(或合成气)形式。
另外的可持续碳源可以通过使用co2的电化学还原为甲酸或草酸并且在发酵中使用这些化合物作为碳源来实现。
目前已知六种不同途径用于碳固定:还原性戊糖磷酸途径(卡尔文)循环、还原性乙酰-coa(wood-ljungdahl)途径、还原性柠檬酸循环、3-羟基丙酸盐双循环,二羧酸盐/4-羟基丁酸盐循环和3-羟基丙酸盐/4-羟基丁酸盐循环[fuchsg,2011alternativepathwaysofcarbondioxidefixation:insightsintotheearlyevolutionoflife?annualreviewsinmicrobiology,65:631-658]。除了这些途径之外,还可以使用属于丙二酰-coa-草酰乙酸-乙醛酸(mog)家族途径的家族的合成碳固定途径[bar-even等,designandanalysisofsyntheticcarbonfixationpathways.proceedingsofnationalacademyofsciencesusa,2010,107:8889-8894]。
在一个实施方案中,所提供的工艺可以在发酵罐中进行。
经工程化的生物可以在各种反应器系统中培养,并且该工艺可以以不同的操作模式进行。最常用的生物反应器是搅拌罐生物反应器或充气发酵罐。发酵罐配备有无菌气源,气泡分散体的混合是通过机械搅动来实现的,并且可以利用使蒸汽或冷却水循环的夹套或盘管来维持温度。对于充气容器,可以选择高度/直径比(>3)来增加气泡和液相之间的接触时间。生物反应器的其它变体是在没有机械搅动的情况下实现混合的气升式生物反应器,以及固定生物催化剂时使用的填充床或流化床生物反应器。
发酵可以以三种不同的模式进行:分批、补料分批和连续模式。标准分批生物反应器被认为是“封闭”系统。在分批模式下,将所有的培养基组分都添加到生物反应器,同时确保无菌。培养基准备好后,将生物反应器接种适当的接种物接种,并且使发酵继续进行直到结束而对培养基没有任何改变,即没有任何其它组分的进料。然而,可以加入诸如酸和/或碱的组分来维持ph,并且可以添加空气/氧气以维持溶解的氧气水平。在分批发酵中,生物质和产物浓度随时间推移变化,直到发酵完成。细胞经历经典的滞后期、指数生长期、固定期生长,然后是死亡期。
批量模式的变化是补料分批模式,其中随着工艺进行,包括碳源的营养物被添加到发酵罐中。
除分批或补料分批模式外,也可使用连续发酵模式。与分批模式相比,连续系统被认为是“开放”系统。在连续模式下,将确定的生产培养基连续添加到生物反应器中,并以相同的速率除去等量的生物反应器内容物。连续操作可以在去除包括细胞在内的容器内容物的恒化器中或者在使用灌注培养的生物反应器中进行,其允许将活细胞再循环回到生物反应器,从而获得高细胞密度。
常用的发酵罐设计和不同的操作模式在文献[biochemicalengineeringfundamentals,第2版,j.e.bailey和d.f.ollis,mcgrawhill,newyork,1986;developmentofsustainablebioprocesses:modelingandassessment,e.heinzle,a.p.biwer和c.l.cooney,johnwiley&sons,ltd.,2006;bioprocessengineering:basicconcepts,第2版,m.l.shuler和f.kargi,prenticehall,2001]中已充分确认。
可以采用分批、补料分批或连续发酵程序。
在一个实施方案中,如本文所提供的工艺在基本上厌氧的条件下进行。“基本上厌氧的”当参考培养或生长条件使用时在一个实施方案中意指氧气的量小于液体培养基中溶解氧气的饱和度的约10%。在一个实施方案中,该术语包括用小于约1%氧气的气氛维持的液体或固体培养基的密封室。在另一个实施方案中,所述工艺在基本上好氧的条件下进行。如本文所用,术语“基本上好氧的”当参考培养或生长条件使用时在一个实施方案中意指氧气的量等于或大于液体培养基中溶解氧气的饱和度的约10%。在一个实施方案中,该术语包括用大于约1%氧气的气氛维持的液体或固体培养基的密封室。在生物反应器内维持好氧或厌氧条件的方法是本领域技术人员已知的。
如本领域技术人员将理解,可将各种组分添加到培养基中以支持本文所述的微生物生长和/或代谢工艺,包括例如营养物质、ph调节剂、渗透保护剂。
生物可以生长在任何适合的生长培养基中,诸如luria-bertani肉汤培养基、terrific肉汤培养基或酵母菌提取物-蛋白胨-右旋糖(ypd)培养基。对于生产,根据宿主的选择,可以使用合成基本培养基诸如m9基本培养基、酵母菌合成基本培养基、酵母菌氮碱、bg-11或其变型。适合的基本培养基必须含有至少一种碳源、至少一种氮源、盐、辅因子、缓冲剂和生长和维持重组微生物所需的其它组分。碳源可以是前述的一种或多种碳源,氮源可以是铵盐或硝酸盐,包括但不限于(nh4)2so4、nh4cl、(nh4)2hpo4、nh4oh、kno3、nano3。培养基可以补充复合或有机氮源,诸如尿素、酵母菌提取物、酪蛋白氨基酸、蛋白胨、胰蛋白胨、大豆粉、玉米浸出液或酪蛋白水解物。此外,可向基本培养基供应痕量金属,包括但不限于h3bo3、mncl2、znso4、na2moo4、cuso4、co(no3)2、cucl2、zncl2、cocl2、fecl3、ki。基本培养基可补充维生素和/或非维生素化合物,包括但不限于生物素、泛酸盐、叶酸、肌醇、烟酸、对氨基苯甲酸、吡哆醇、核黄素、硫胺素、氰钴胺、柠檬酸、乙二胺四乙酸(edta)、柠檬酸铁铵。可以通过直接喷射或者以nahco3或na2co3的形式向培养基供应二氧化碳。
根据所使用的宿主生物,基本培养基可以适合地具有ph2.0–ph10.0之间的ph范围。
发酵可以在范围为25℃至42℃的温度内进行。如果所选宿主生物是嗜热的,其中培养温度可能高达80℃,则可以使用更高的温度。
发酵可以在好氧、微好氧或厌氧条件下进行。它也可以在两个不同的阶段进行,涉及好氧生长阶段和微好氧或厌氧生产阶段。可引入无菌空气或氧气以维持培养基中所需的溶解氧气水平。
可以使用本领域已知的方法诸如高效液相色谱(hplc)、气相色谱(gc)、液相色谱-质谱(lc-ms)、气相色谱-质谱(gc-ms)来测定培养基中产物的量。
测定1,3-丁二醇产生的方法是本领域技术人员已知的,并且在实施例3中进一步举例说明。例如,产品、中间体和副产物形成可以通过方法诸如配备有折射率和/或一个或多个光电二极管阵列检测器的hplc(高效液相色谱)、gc-ms(气相色谱-质谱)、gc-fid(气相色谱-火焰离子化检测器)和lc-ms(液相色谱-质谱)来分析。也可以使用本领域公知的方法测定来自外源性dna序列的个别酶促活性。
在一些实施方案中,如本文所公开的工艺还包括纯化工艺的产物,例如1,3-bdo。此类纯化方法是本领域技术人员已知的,且包括例如通过液体萃取、过滤、蒸馏或蒸发。从发酵液中分离化合物取决于所需化合物的最终纯度。分离技术可以包括:离心、微滤、超滤、纳滤、蒸发、结晶、蒸馏和离子交换。典型的下游加工操作将包括一系列工艺,包括使用离心或微滤分离细胞,使用超滤除去发酵液中的附加固体,使用纳滤、离子交换或蒸发结晶从发酵液除去盐,和最后使用蒸馏纯化1,3-bdo。
如本领域技术人员将理解,在一个实施方案中,可以产生如本文所述的微生物以分泌所得产物,无论是通过选择具有对应于产物的分泌信号的宿主生物,还是通过对宿主生物进行工程化以提供分泌信号。例如,可以在宿主生物中过表达膜结合转运蛋白以改善1,3-bdo向发酵液的分泌,包括但不限于:yhjx基因,其编码丙酮酸盐-诱导的内膜蛋白和属于主要促进因子蛋白质超家族的推定的转运蛋白,并且可以从培养基微生物中回收产物。在其它实施方案中,产物可以从微生物中提取。在一种方法中,可以使微生物破裂,并且可以离心培养基或裂解物以除去颗粒细胞碎片,并且如果有必要可以分离膜和可溶性蛋白质级分。
应理解,本领域技术人员将显而易见对其的诸多修改。因此,上面的描述和附图应被认为是对本发明的说明,而不具有限制意义。将进一步理解的是,旨在覆盖大体上符合本发明的原理并且包括虽然不属于本公开内容但属于本发明所属领域内的已知或习惯做法且可以被应用于前文阐述的必要特征,并且落入所附权利要求的范围内的对本发明的任何变化、使用或改动。
本文引用的所有文档都通过引用并入,然而,应理解,通过引用并入本文的任何专利、出版物或其它公开材料全部或部分内容仅以这样的程度并入,即所并入的材料不与本公开中阐述的定义、陈述或其它公开材料相冲突。因此,并且在必要的程度上,如本文明确阐述的公开内容取代通过引用并入本文的任何冲突的材料。
以上描述的本发明的实施方案仅旨在是示例性的。因此,本发明的范围旨在仅由所附权利要求的范围来限定。
实施例
在以下实施例中进一步描述了本发明。所提供的实施例仅仅是对本发明的说明,而不具有限制意义。
在文档中提供的实施例中构建和使用的菌株和质粒列于下表6中:
表6.
实施例1:选择dera酶用于1,3-丁二醇途径
本实施例描述了用于1,3-bdo途径的酶候选物的筛选、选择和表征。
1,3-bdo生物合成途径中的第一步是由脱氧核糖-5-磷酸醛缩酶缩合两个乙醛分子以产生3-羟基丁醛(3hb)。为了鉴定可缩合两个乙醛分子的醛缩酶,基于与大肠埃希氏菌脱氧核糖-5-磷酸醛缩酶(dera)的序列相似性,鉴定72种i类醛缩酶同源物或推定的醛缩酶的列表。这72种推定的醛缩酶列于表7中。
表7.
此外,在大肠埃希氏菌的活性位点中的三个残基已经显示在使用对于羟醛缩合发生是关键的供体醛形成席夫碱中间体中起关键作用。这些残基是:lys167、lys201和asp102(编号是指大肠埃希氏菌dera中的残基编号)。使用mafft进行多重序列比对,发现上述残基严格保守的72种酶中的34种。表8列出了这34种所选酶的列表。
表8.
为了进一步缩小来自34种酶的潜在醛缩酶的列表,选择先前显示相对于大肠埃希氏菌dera对乙醛展现更高的羟醛缩合活性的来自海栖热袍菌(tm1559)的醛缩酶。tm1559因此代表了比大肠埃希氏菌dera可能更好的dera。因此,tm1559用作筛选之前没有表征但可以展现出更高的乙醛羟醛缩合活性的潜在dera的基础。
我们对来自海栖热袍菌的dera的晶体结构的进一步分析[heine,a.,luz,j.g.,wong,c.-h.&wilson,i.aanalysisoftheclassialdolasebindingsitearchitecturebasedonthecrystalstructureof2-deoxyribose-5-phosphatealdolaseat0.99aresolution.journalofmolecularbiology343,1019–34(2004)]和多重序列比对分析揭示了tm1559的磷酸盐结合袋中令人有兴趣的残基,其可能在针对乙醛的羟醛缩合活性中起作用。这些残基在来自剩余34种酶的一些dera酶中保守,但在大肠埃希氏菌dera中不保守。这些残基是:n21k,y49n,a71c或v,k172f,n176h或g(其中第一个字母和编号是指残基及其在ec1535中的位置,且最后一个字母是指tm1559中相应的残基)。基于这些残基,该列表进一步缩小到具有更高的乙醛羟醛缩合活性的潜力的15种酶(tm1559和ec1535也被列入列表中比较)。经选择用于进一步分析的15种dera的多重序列比对示于图4中,并在表9中列出。
表9.
实施例2:通过体外测定表征用于乙醛羟醛缩合的dera酶.
来自十五种物种的dera(十三种候选物连同来自海栖热袍菌和大肠埃希氏菌的dera)各自在宿主生物中表达,且成功纯化十种(表9)。
使用先前描述的酶促测定法按对乙醛羟醛缩合的活性对十种纯化的dera酶进行筛选[chen,l.,dumas,d.p.&wong,c.deoxyribose-5-phosphatealdolaseasacatalystinasymmetricaldolcondensation.journaloftheamericanchemicalsociety114,741–748(1992)]。简而言之,使每种酶与乙醛反应5小时。取来自每个反应的20μl等分试样并置于冰上。然后将2.5μl样品在水中稀释20倍。为了测定反应中剩余的乙醛的量,将6μl稀释的样品加入到96孔板内含有0.3mmnadh、1mg酵母菌醇脱氢酶于100mm三乙醇胺缓冲液(tea)(ph7.5)中的混合物中,使每孔含有200μl的终体积。使用读板仪在340nm处监测nadh的吸光度(∈340nm=6220m-1·cm-1),其中由nadh消耗引起的吸光度降低与每个反应中剩余的乙醛量成正比。因此,在每个反应中消耗的乙醛的量是在每个羟醛反应中使用的乙醛的初始量与酶测定中消耗的nadh的量之间的差值。比活性是由含有0.8mg每种酶的反应混合物中经5小时消耗的乙醛量确定的。
测定结果显示在图8中。10种酶全部展现出对乙醛羟醛缩合的具有一定活性。来自海栖热袍菌的dera似乎展现出最高活性,其次是来自六种生物的dera,它们展现出比大肠埃希氏菌dera更高的活性。这六种生物与tm1559共有严格保守的残基:n21k,y49n,k172f,n176g,除了具有a71c代替a71v之外(其中第一个字母和编号是指残基及其在ec1535中的位置,且最后的字母是指在tm1559中相应的残基)。
实施例3:来自使用dera进行的乙醛缩合的产物的鉴定
为了鉴定由实施例2中测试的dera进行的由乙醛的羟醛缩合形成的产物,再次制备更大体积的酶促反应混合物并通过高效液相色谱(hplc)分析。
简而言之,通过在100mm三乙醇胺缓冲液(ph7.5)中加入20mm乙醛和600mg嗜碱芽孢杆菌dera制备6ml反应混合物。平行地,与反应类似地制备阴性对照,但省略乙醛。将这两种混合物在室温下孵育。每五分钟从每个取500μl样品,持续一个小时。将200μl浓硫酸的等分试样加入到每个样品中以猝灭反应并置于冰上。然后在配备有折射率检测器的dionexultimate3000系统上通过hplc分析样品。利用以下方法使用hpx-87h柱分离分析物乙醛和3hb:30℃,5mmh2so4,0.8ml/min,20min运行时间。使用ri检测器鉴定峰。3-hb标准物在11.8min洗脱,且乙醛标准物在13.5min洗脱。使用hplc测试的酶包括来自嗜碱芽孢杆菌(bh1352)和大肠埃希氏菌(ec1535)的那些(结果未显示)。使用任一种酶的反应都会导致乙醛消耗和3hb合成。乙醛和3hb的浓度汇总于下表10中:
表10.
实施例4:选择用于1,3-丁二醇途径的醛酮还原酶
本文所教导的产生1,3-丁二醇(1,3-bdo)的生物合成途径中的第二步涉及使用醛酮还原酶将3hb还原成1,3-bdo。通过筛选37种推定的候选醛酮还原酶来进行该反应所需的酶的筛选(表11)。在含有k2hpo4(50mm,ph7.0)、kcl(10mm)、edta(0.5mm)、nadph(1mm)、乙醛和3-羟基丁醛(作为底物)(1mm)以及蛋白质(20μg)的反应混合物(终体积为200μl)中,在96孔板中于37℃下采用分光光度法测量在宽松的条件下对乙醛和3hb的底物专一性,持续30min。通过追踪作为辅因子nadph(∈340nm=6220m-1·cm-1)向nadp+的转化的量度的在340nm处的吸光度的降低来监测反应。
表11.
选择来自绿脓假单胞菌的候选还原酶(pa1127),因为它表现出对3hb的显著活性,但对乙醛没有活性。
使用hplc根据以下方法鉴定pa1127还原酶催化的反应的产物:hpx-87h柱,60℃,0.4ml/min和5mmh2so4。使用ri检测器检测峰。首先运行以下标准:在28.46min洗脱1,3-bdo,在27.46min洗脱乙醛且在23.35min洗脱3hb。反应混合物通过混合55mm的3hb(还含有约38mm乙醛)和还原酶60mg的pa1127在100mmtea缓冲液(ph7.5)中制备。在不同的时间点,使用hplc分析反应混合物的等分试样。在存在酶pa1127的情况下,3hb随着时间推移消耗,同时1,3-bdo浓度增加,而乙醛浓度保持恒定。在阴性对照中,没有检测到3hb转化为1,3-bdo(表12),从而验证了pa1127对3hb的专一性。
表12.
实施例5:1,3-bdo途径的体外验证
为了同时测试醛缩酶和还原酶的活性,使用以下方法进行偶联酶反应:在于100mmtea缓冲液(ph7.5)中的含有25mm乙醛、60mgdera、60mg还原酶和20mmnadph的0.6ml反应混合物中,测试来自嗜碱芽孢杆菌的dera和来自绿脓假单胞菌的还原酶。在不同的时间点,使用与实施例4所述方法类似的hplc分析每个反应混合物的等分试样。结果汇总于表13。
随着时间推移,乙醛浓度降低,同时1,3-bdo增加。在没有酶的阴性对照中没有鉴定到反应产物3hb或1,3-bdo。由于乙醛的高挥发性,有些在反应期间损失。剩余的反应乙醛可能已经转化为双重顺序的羟醛缩合产物(2,4,6-三脱氧-d-赤式-吡喃己糖苷),其由于缺乏可用的标准物进行验证而不能通过hplc鉴定。
表13.
实施例6:野生型大肠埃希氏菌中1,3-bdo途径的体内验证
用于表达1,3-bdo途径基因的载体骨架是具有诱导型laci启动子和氨苄青霉素抗性的ptrc99a。将表达pdc、dera和akr所需的三个基因组装到表达载体中。每个dna部分侧接有独特的核苷酸序列(uns)[orella,j.p.,lienert,f.,boehm,c.r.,chen,j.-h.,way,j.c.,&silver,p.a.(2014).uniquenucleotidesequence-guidedassemblyofrepetitivednapartsforsyntheticbiologyapplications.natureprotocols,9(9),2075–89]。表14列出了用于扩增每个部分的引物。然后,如[kok,s.de,stanton,l.h.,slaby,t.,durot,m.,holmes,v.f.,patel,k.g.,chandran,s.s.(2014).rapidandreliablednaassemblyvialigasecyclingreaction.acs,3,97–106]所述,使用具有与一个部分的3'uns的5'同源区且具有与连续的dna部分的5;uns的3'同源区的ssdna桥接寡核苷酸,通过连接酶循环反应(lcr)组装dna部分。简而言之,首先按照制造商的方案,使用
通过两阶段补料分批发酵来一式三份地表征ecbd-6菌株。通过用单菌落接种5mllb(补充有100μg/ml羧苄青霉素)来制备种菌培养物,然后在设定为250rpm的旋转摇床上在37℃下生长过夜。然后用过夜培养物接种50ml补充有100μg/ml羧苄青霉素的tb,并在30℃下好氧生长,直到在600nm处的光密度达到0.6。然后用1mmiptg诱导蛋白质表达。蛋白质表达5小时后,将细胞沉淀并以高细胞密度重悬于25ml含有3%(w/v)葡萄糖的基本盐培养基(m9盐培养基)中。在37℃下在密封的250ml带挡板烧瓶中进行发酵。在距发酵开始20和50小时,再次以15g/l的浓度供给葡萄糖。在不同的时间点,获取样品以测定600nm的细胞光密度。使用配备有uv和折射率检测器的dionexultimate-3000hplc系统通过hplc分析测定底物和产物的浓度。使用的柱是以5mmh2so4作为洗脱剂且在36℃下操作的aminexhpx-87h。发酵93小时后,ecbd-6产生1.42g/l的1,3-丁二醇。
表14显示了用于组装表达1,3-bdo途径基因的质粒pbd3的引物,包括来自运动发酵单胞菌的pdc、来自嗜碱芽孢杆菌的dera和来自绿脓假单胞菌的akr。载体骨架ptrc99a也用含有uns的引物扩增。然后通过连接酶循环反应组装四个dna部分。
表14
实施例7:丙酮酸盐累积菌株的构建
由于丙酮酸盐是1,3-丁二醇途径中的第一代谢物,因此增加1,3-丁二醇产量需要可以累积丙酮酸盐的宿主生物。后者代表了最常见微生物的中心碳代谢的关键代谢物。在大肠埃希氏菌中,丙酮酸盐是几种原生发酵副产物的主要前体(图2)。为了在大肠埃希氏菌中产生高效价的丙酮酸盐,缺失了将丙酮酸盐排入发酵产物的途径。因此,从大肠埃希氏菌中缺失了三个基因adhe(编码醇脱氢酶)、ldha(编码乳酸脱氢酶)和pflb(编码丙酮酸甲酸裂解酶),以分别消除乙醇、乳酸盐和甲酸盐的形成(图2)。使用p1转导方法将基因缺失从来自keio集合的相应单基因缺失突变体按顺序转移到野生型菌株。使用在序列表中提供的引物使用pcr确认基因缺失:f-adhe-check和r-adhe-check(序列id37和38),f-ldha-check和r-ldha-check(序列id39和40),f-pflb-check和r-pfla-check(序列id41和42)。将具有三个基因缺失的所得菌株命名为lmse-25。
在含有15g/l葡萄糖、3.5g/l(nh4)2hpo4、5g/lk2hpo4和3.5g/lkh2po4、0.25gmgso4·7h2o、15mgcacl2·2h2o、0.5mg硫胺素和1ml痕量金属原料的无机培养基上表征菌株lmse-25。痕量金属原料在0.1mhcl中制备,且每升由以下组成:1.6gfecl3、0.2gcocl2·6h2o、0.1gcucl2、0.2gzncl2·4h2o、0.2gnamoo4和0.05gh3bo3(causey,t.b.,zhou,s.,shanmugam,k.t.&ingram,l.o.(2003)proc.natl.acad.sci.usa100,825–832)。在500ml发酵罐中进行表征,使用4mkoh将ph控制在7.0并鼓吹空气用于溶解氧控制(>70%)。
对照野生型大肠埃希氏菌没有累积可检测量的丙酮酸盐,而突变菌株在5小时内产生了0.67g/l的最大效价,代表了20%的葡萄糖最大理论产率。
实施例8:菌株ecbd-5的构建
菌株ecbd-5是通过将质粒pbd3引入丙酮酸盐产生菌株lmse-25中来构建。通过使用葡萄糖作为碳源并且在半好氧条件下进行的补料分批发酵来表征由此产生的1,3-丁二醇产生大肠埃希氏菌菌株(ecbd-5)。菌株首先在30℃下在250ml带挡板烧瓶内的50ml富培养基(terrificbroth)中培养以表达途径蛋白。蛋白质表达5小时后,将细胞沉淀并以高细胞密度重悬于含有3%(w/v)葡萄糖的25ml基本盐培养基(m9盐培养基)中。在37℃下在密封的250ml带挡板烧瓶中进行发酵。在距发酵开始20和50小时,再次以15g/l的浓度供给葡萄糖。本实验一式三份进行。
在不同的时间点,获取样品以测定600nm处的细胞光密度。使用配备有uv和折射率检测器的dionexultimate-3000hplc系统通过hplc分析测定底物和产物的浓度。使用的柱是以5mmh2so4作为洗脱剂且在36℃下操作的aminexhpx-87h。
发酵93小时后,ecbd-5产生2.1g/l的1,3-bdo,而对照ecbd-6产生1.42g/l的1,3-bdo。
序列表
<110>universityoftoronto
<120>用于产生1,3-丁二醇的方法和微生物
<130>05014925-101pct
<160>42
<170>patentin3.5版
<210>1
<211>672
<212>dna
<213>蜡样芽胞杆菌atcc10987
<300>
<308>ae017194.1
<309>2014-01-31
<313>(1909205)..(1909876)
<400>1
atgaacattgcaaagttaattgaccatacaattttaaaagctaatactactaaagaagat60
gttatgaaagtaatcgaagaagcaaaggaatataaattcgcttctgtttgtattaatcct120
acatgggtaaagctagctgctgaggaattagctggacatgatgtagatgtttgtactgta180
atcggtttcccattaggcgcaagtactactgaaacaaaagctttcgaaacaaaagatgct240
atcgcaaaaggtgcaactgaagttgacatggtaatcaacgtaggcgctttaaaagatggc300
gacgacgaacttgttgaaaaagacatttatgaagtagtacaagcagcaaaaggaaaagct360
cttgtaaaagtaatcattgaaacttgcctattaacagatgaagagaaagtacgcgcttgt420
gaattatcagtaaaagctggggctgatttcgtaaaaacttcaactggattctcaactggc480
ggagcaactgctgaagatatcgcattaatgcgtaaaacagttggaccaaacgttggtgta540
aaagcatctggtggcgttcgtacacgtgaagatgcagaaaaaatggtagctgctggagct600
tctcgcgttggagcaagtgctagtgttgcaatcgtattaaatgatgcaaaaggtgctaca660
gataactactaa672
<210>2
<211>780
<212>dna
<213>大肠埃希氏菌k12
<300>
<308>cp009685
<309>2014-12-15
<313>(3719004)..(3719783)
<400>2
atgactgatctgaaagcaagcagcctgcgtgcactgaaattgatggacctgaccaccctg60
aatgacgacgacaccgacgagaaagtgatcgccctgtgtcatcaggccaaaactccggtc120
ggcaataccgccgctatctgtatctatcctcgctttatcccgattgctcgcaaaactctg180
aaagagcagggcaccccggaaatccgtatcgctacggtaaccaacttcccacacggtaac240
gacgacatcgacatcgcgctggcagaaacccgtgcggcaatcgcctacggtgctgatgaa300
gttgacgttgtgttcccgtaccgcgcgctgatggcgggtaacgagcaggttggttttgac360
ctggtgaaagcctgtaaagaggcttgcgcggcagcgaatgtactgctgaaagtgatcatc420
gaaaccggcgaactgaaagacgaagcgctgatccgtaaagcgtctgaaatctccatcaaa480
gcgggtgcggacttcatcaaaacctctaccggtaaagtggctgtgaacgcgacgccggaa540
agcgcgcgcatcatgatggaagtgatccgtgatatgggcgtagaaaaaaccgttggtttc600
aaaccggcgggcggcgtgcgtactgcggaagatgcgcagaaatatctcgccattgcagat660
gaactgttcggtgctgactgggcagatgcgcgtcactaccgctttggcgcttccagcctg720
ctggcaagcctgctgaaagcgctgggtcacggcgacggtaagagcgccagcagctactaa780
<210>3
<211>675
<212>dna
<213>嗜碱芽孢杆菌c-125
<300>
<308>ba000004
<309>2009-04-16
<313>(1438985)..(1439659)
<400>3
atgtcacgttcgattgcacaaatgattgatcatacgctacttaaaccaaatacaacagaa60
gaccaaattgtaaagctctgtgaggaagcaaaggaatattcatttgcatctgtttgtgtg120
aatcctacttgggtcgctcttgctgcgcagttgctaaaagatgcacctgatgtgaaagta180
tgtacagttatcggctttccgttaggggcaacgactccggaagtgaaagcgtttgaaacg240
actaatgccattgaaaatggagcgacagaagtggacatggtcattaacattggagcgtta300
aaagataaacaatacgagcttgttggacgcgacattcaagcggttgttaaagcagcagaa360
gggaaagcattaacgaaagtaatcattgaaacatcgttattaacggaggaagagaagaag420
gctgcgtgtgagcttgccgtaaaagcaggagccgactttgtcaaaacgtcgactggattc480
tctggcggaggtgctacggctgaggatatcgcgctcatgcgaaaagtggtcggaccaaat540
ttaggagtcaaagcttctggaggtgttagagatctgtccgacgcgaaagcgatgattgat600
gctggtgctactcggattggtgcgagtgctggggtggcgattgttaacggggagcgtagc660
gaagggagttattaa675
<210>4
<211>672
<212>dna
<213>枯草芽孢杆菌枯草亚种菌株168
<300>
<308>al009126.3
<309>2015-02-27
<313>(4051602)..(4052273)
<400>4
atgtcattagccaacataattgatcatacagctttgaaaccgcatacacaaaaagcggac60
attctaaaactaattgaagaagcgaaaacatacaaatttgcttcagtatgtgtcaatccg120
acatgggtggagcttgctgcaaaagagcttaagggaactggagtcgacgtttgtacggtc180
atcggcttcccgctcggtgccaatacaactgaaacaaaagcgttcgaaacaaaagacgcc240
atttcaaaaggcgccactgaagtggatatggtcattaatattgccgctttaaaagacaag300
gaagacgatgtggtggaagctgatatccgcggtgtagtggaagctgtagccggaaaagcg360
cttgtcaaagtcattatcgaaacgtgccttctgactgatgaagaaaaagaacgtgcatgc420
cgtttagcggtgtctgcgggagcggatttcgtaaaaacatcaacaggcttttctacaggc480
ggcgcaacgaaggaagatatcgccttaatgcgcaaaacagtagggcctgatatcggcgtg540
aaagcatctggcggcgtcagaacgaaagaagatgtagacacaatggtagaggccggagca600
agccgaattggcgccagcgcaggcgtttctatcgtaaaaggagaaaatgcatcaggcgga660
gacaactattaa672
<210>5
<211>747
<212>dna
<213>海栖热袍菌
<300>
<308>cp011108
<309>2015-04-24
<313>(1550526)..(1551270)
<400>5
atgatagagtacaggattgaggaggcagtagcgaagtacagagagttctacgaattcaag60
cccgtcagagaaagcgcaggtattgaagatgtgaaaagtgctatagagcacacgaatctg120
aaaccgtttgccacaccagacgatataaaaaaactctgtcttgaagcaagggaaaatcgt180
ttccatggagtctgtgtgaatccgtgttatgtgaaactggctcgtgaagaactcgaagga240
accgatgtgaaagtcgtcaccgttgttggttttccactgggagcgaacgaaactcggacg300
aaagcccatgaggcgattttcgctgttgagagtggagccgatgagatcgatatggtcatc360
aacgttggcatgctcaaggcaaaggagtgggagtacgtttacgaggatataagaagtgtt420
gtcgaatcggtgaaaggaaaagttgtgaaggtgatcatcgaaacgtgctatctggatacg480
gaagagaagatagcggcgtgtgtcatttccaaacttgctggagctcatttcgtgaagact540
tccacgggatttggaacaggaggggcgaccgcagaagacgttcatctcatgaaatggatc600
gtgggagatgagatgggtgtaaaagcttccggagggatcagaaccttcgaggacgctgtt660
aaaatgatcatgtacggtgctgatagaataggaacgagttcgggagttaagatcgttcag720
gggggagaagagagatatggaggttaa747
<210>6
<211>675
<212>dna
<213>热自养甲烷嗜热杆菌
<300>
<308>ap011952
<309>2015-03-03
<313>(720590)..(721263)
<400>6
gtggttaaaatgaatgtggagacaagggaggaacttgcatcacttatagaccacaccaat60
gtgagggctgatgcaacagaaaatgatattgagaggctatgcagggaggcggtcagctac120
ggcttcaggtgcgcggtggtcacacccaccaatgtcaggctggcggctgaactccttgag180
gggaccgatgtgacggtctgctcagttgttggtttcccggcaggcgtcagtacaccccgc240
gttaaggcccttgaagcctctgaggccgttgagaacggggccggtgaggtggacatggtc300
atgaatatcggggccatgaagtcaggcaatagggagctcgtatacagggatatcagcggc360
gttgttgatgccgccggcgtccccgtcaaggttatacttgaaacagcctatctcacagac420
aaggagaaggttgaagcctgccttataagtaaagaggccggtgcggcatttgttaaaaca480
tcaacagcctatggtggactagccggcgccacagttgaggatgtgatgctcatgcggaaa540
acggtgggtgatgagatgggagtcaaggcatctgggggaataagggatcttgaaacagcc600
cttgcgatgatagatgctggggcagacaggatcgggacatcaaccggtgtacagataatc660
gagggatggaggtaa675
<210>7
<211>663
<212>dna
<213>耐辐射奇球菌
<300>
<308>ae000513
<309>2014-01-31
<313>(1215027)..(1215687)
<400>7
atgtcactcgcctcctacatcgaccacacgctgcttaaggccaccgccacgctcgccgac60
atccgcacgctgtgtgaggaagcccgcgagcactcgttctacgcggtgtgcatcaacccg120
gtctttattccccacgcccgcgcctggctcgaaggcagcgacgtgaaggtcgccaccgtc180
tgcggctttcccctcggcgccatcagctccgagcagaaagctctggaagcccgcctgagc240
gccgaaacgggcgccgacgaaatcgatatggtcatccacatcggctcggcgcttgccggc300
gactgggacgcggtggaagccgacgtgcgggcagtgcgccgcgcggtgcccgagcaggtg360
ctcaaggtgattatcgaaacctgctacctgaccgacgagcaaaagcgcttggcgactgag420
gtcgccgtacagggcggcgccgacttcgtgaagacgagcacaggcttcggcaccggcggc480
gccaccgtggacgacgtgcgcctgatggcggaagtgatcgggggccgcgccggactcaag540
gcggcgggcggcgtccgcactcctgccgacgcgcaagccatgatcgaggcgggcgcgacc600
cggctgggcacctcgggcggcgtgggtctggtgtcgggcggcgaaaacggagccggctac660
taa663
<210>8
<211>663
<212>dna
<213>金黄色葡萄球菌金黄色亚种mu50
<300>
<308>ba000017
<309>2009-01-09
<313>(2265358)..(2266020)
<400>8
atgaatagtgcaaaattgattgatcacactttattgaagcctgagtcaacacgtacgcaa60
atcgatcaaatcatcgatgaagcgaaagcataccattttaaatctgtatgtgtgaatcca120
acgcatgttaaatatgcagcagagcgactagctgattcagaggtgttagtttgtacggta180
ataggattcccattaggtgcatcgacaactgcgacgaaagcatttgaaacagaagatgcg240
attcaaaatggtgcagatgaaattgacatggtcatcaacatcggcgcattaaaagatgga300
cgttttgatgatgtacaacaagacattgaagcagtggtgaaagctgcgaaaggtcacaca360
gtaaaagtgattattgagacggtattgttggaccatgacgaaatcgtaaaagcgagtgaa420
ttaacaaaagtggctggtgcggacttcgttaaaacttcaacaggttttgcaggtggcggt480
gcgactgcagaagacgttaaattaatgaaagatacagtaggtgctgatgtagaagtaaaa540
gcatcaggtggcgtacgtaatttagaagatttcaataaaatggttgaagcaggtgcgaca600
cgtattggtgcgagcgcaggcgttcaaattatgcaaggtttagaagcagattcagattac660
taa663
<210>9
<211>672
<212>dna
<213>单核细胞增多性李斯特氏菌egd-e
<300>
<308>hg421741
<309>2015-02-27
<313>(2068136)..(2068807)
<400>9
atgacaattgctaaaatgatcgatcatactgctttaaaaccagacacaacgaaagaacaa60
attttaaccctaacaaaagaagcaagagaatacggctttgcatccgtatgtgtgaaccca120
acttgggtaaaactatccgctgaacaacttgctggagcagaatccgtagtatgtactgtt180
atcggtttcccactaggagcgaatacccctgaagtaaaagcatttgaagtgaaagatgcc240
atccaaaacggcgcgaaagaagtcgatatggttatcaatatcggtgcacttaaagacaag300
gacgacgaattagtagaacgcgatattcgcgctgttgtcgatgttgctaaaggcaaagca360
ttagtaaaagtaattatcgaaacttgcctattaacagacgaagaaaaagtgcgcgcatgc420
gaaatcgctgtaaaagcaggaacagacttcgttaaaacatctacaggattttcaacaggt480
ggcgcaactgccgaagatatcgccttgatgcgtaaaacagttggaccgaacatcggtgta540
aaagcatctggtggggttcgtacgaaagaagacgtagaaaaaatgatcgaagcaggcgca600
actcgtatcggcgcaagtgcaggcgttgcaattgtttccggcgaaaaaccagctaaacct660
gataattactaa672
<210>10
<211>663
<212>dna
<213>肺炎链球菌tigr4
<300>
<308>ae005672
<309>2014-01-31
<313>(790292)..(790952)
<400>10
atgaaattaaataaatatatagatcatacgcttttaaaacaagatgcaaagaaaaaacaa60
attgatagtttgttgtctgaggctagagagtatgactttgccagtgtttgcgttaatccg120
acctgggttgaacatgctaaaaaaggacttgaaggcacagatgttaaggtttgcacagta180
gtaggtttccctttgggagcaacaacttcagccgtgaaagcatttgagacaaaagaagct240
atccaaaatggtgcagatgagattgatatggtgatcaatgttggagctctcaaatcaggt300
aatttagccttggttgagtcagatattcgcgcagtagtggaagcaagtggtgataagtta360
gtgaaagtcattattgaagcttgccttctgacagaccaagaaaaagttgttgtttgccaa420
ttggcccaaaaagctggggctgactttgtcaaaacatctactggcttttcaactggtggt480
gctacgatagcagatgttacattaatgcgtgaaacagttggatctgatatgggtgtcaag540
gccgccggtggagctcgttcttatgcagatgctcttgcctttgtcgaagcaggtgcgacc600
cgtatcggaacgtcagctggggtagctattttaaaaggagaattggcagatggcgactac660
taa663
<210>11
<211>990
<212>dna
<213>绿脓假单胞菌
<300>
<308>ae004091.2
<309>2014-01-31
<313>(1219623)..(1220610)
<400>11
atgagcgttgaaagcattcgcatcgagggtatcgacacgccggtctcgcgcatcggcctc60
ggcacctgggccatcggcggctggatgtggggcggcgctgacgacgcgacgtcggtggaa120
accatccggcgtgcggtggaatccgggatcaacctgatcgacaccgcgccggtctatggc180
ttcggccattccgaagaggtcgtcggcaaggccttgcagggcctgcgcgacaaggcggtg240
atcgccaccaaggcggcgctggagtggagcgacgcgggcatccaccgcaacgcctccgcc300
gcacgcatccgccgggaggtcgaggactcgctgcggcggctgaagaccgatcgtatcgac360
ctgtaccagattcactggccggacccgctggtggcgcacgaggaaaccgccggcgaactg420
gagcgcctgcgccgcgacggcaagatcctcgccatcggcgtgagcaactattcgccggaa480
cagatggacgggttccgccagttcgctccgctggccagcgtgcagccaccctacaacctg540
ttcgagcgcgccatcgacgccgacgtgctgccctacgccgagcgtaacggcatcgtcgtg600
ctggcctacggagcgctgtgccgcggcctgctttccggacggatgaacgccgagacccgc660
ttcgatggcgacgacctgcgcaagtccgacccgaagttccagcagccccgcttcgcccag720
tacctggcagcggtcgcgcaactggaggaactggctcgcgagcgctatggcaagtcggtg780
ctggccctggccatccgctggattctcgatcgcggccctacggtggcgctgtggggggcg840
cgcaagccggagcagttgaacggcatcgccgacgccttcggctggcgcctggacgacgag900
gccatggcccgcatcgagcggatcctcgccgagaccatccaggatccggtcggtcccgag960
ttcatggcgccgcccagccgcaacgcctaa990
<210>12
<211>1707
<212>dna
<213>运动发酵单胞菌
<300>
<308>ae008692.2
<309>2014-01-31
<313>(1373366)..(1375072)
<400>12
atgagttatactgtcggtacctatttagcggagcggcttgtccagattggtctcaagcat60
cacttcgcagtcgcgggcgactacaacctcgtccttcttgacaacctgcttttgaacaaa120
aacatggagcaggtttattgctgtaacgaactgaactgcggtttcagtgcagaaggttat180
gctcgtgccaaaggcgcagcagcagccgtcgttacctacagcgtcggtgcgctttccgca240
tttgatgctatcggtggcgcctatgcagaaaaccttccggttatcctgatctccggtgct300
ccgaacaacaatgatcacgctgctggtcacgtgttgcatcacgctcttggcaaaaccgac360
tatcactatcagttggaaatggccaagaacatcacggccgccgctgaagcgatttacacc420
ccggaagaagctccggctaaaatcgatcacgtgattaaaactgctcttcgtgagaagaag480
ccggtttatctcgaaatcgcttgcaacattgcttccatgccctgcgccgctcctggaccg540
gcaagcgcattgttcaatgacgaagccagcgacgaagcttctttgaatgcagcggttgaa600
gaaaccctgaaattcatcgccaaccgcgacaaagttgccgtcctcgtcggcagcaagctg660
cgcgcagctggtgctgaagaagctgctgtcaaatttgctgatgctctcggtggcgcagtt720
gctaccatggctgctgcaaaaagcttcttcccagaagaaaacccgcattacatcggcacc780
tcatggggtgaagtcagctatccgggcgttgaaaagacgatgaaagaagccgatgcggtt840
atcgctctggctcctgtcttcaacgactactccaccactggttggacggatattcctgat900
cctaagaaactggttctcgctgaaccgcgttctgtcgtcgttaacggcattcgcttcccc960
agcgtccatctgaaagactatctgacccgtttggctcagaaagtttccaagaaaaccggt1020
gcattggacttcttcaaatccctcaatgcaggtgaactgaagaaagccgctccggctgat1080
ccgagtgctccgttggtcaacgcagaaatcgcccgtcaggtcgaagctcttctgaccccg1140
aacacgacggttattgctgaaaccggtgactcttggttcaatgctcagcgcatgaagctc1200
ccgaacggtgctcgcgttgaatatgaaatgcagtggggtcacattggttggtccgttcct1260
gccgccttcggttatgccgtcggtgctccggaacgtcgcaacatcctcatggttggtgat1320
ggttccttccagctgacggctcaggaagtcgctcagatggttcgcctgaaactgccggtt1380
atcatcttcttgatcaataactatggttacaccatcgaagttatgatccatgatggtccg1440
tacaacaacatcaagaactgggattatgccggtctgatggaagtgttcaacggtaacggt1500
ggttatgacagcggtgctggtaaaggcctgaaggctaaaaccggtggcgaactggcagaa1560
gctatcaaggttgctctggcaaacaccgacggcccaaccctgatcgaatgcttcatcggt1620
cgtgaagactgcactgaagaattggtcaaatggggtaagcgcgttgctgccgccaacagc1680
cgtaagcctgttaacaagctcctctag1707
<210>13
<211>1587
<212>dna
<213>恶臭假单胞菌
<300>
<308>ap013070.1
<309>2014-03-01
<313>(5446976)..(5448562)
<400>13
atggcttcggtacacggcaccacatacgaactcttgcgacgtcaaggcatcgatacggtc60
ttcggcaatcctggctcgaacgagctcccgtttttgaaggactttccagaggactttcga120
tacatcctggctttgcaggaagcgtgtgtggtgggcattgcagacggctatgcgcaagcc180
agtcggaagccggctttcattaacctgcattctgctgctggtaccggcaatgctatgggt240
gcactcagtaacgcctggaactcacattccccgctgatcgtcactgccggccagcagacc300
agggcgatgattggcgttgaagctctgctgaccaacgtcgatgccgccaacctgccacga360
ccacttgtcaaatggagctacgagcccgcaagcgcagcagaagtccctcatgcgatgagc420
agggctatccatatggcaagcatggcgccacaaggccctgtctatctttcggtgccatat480
gacgattgggataaggatgctgatcctcagtcccaccacctttttgatcgccatgtcagt540
tcatcagtacgcctgaacgaccaggatctcgatattctggtgaaagctctcaacagcgca600
tccaacccggcgatcgtcctgggcccggacgtcgacgcagcaaatgcgaacgcagactgc660
gtcatgttggccgaacgcctcaaagctccggtttgggttgcgccatccgctccacgctgc720
ccattccctacccgtcatccttgcttccgtggattgatgccagctggcatcgcagcgatt780
tctcagctgctcgaaggtcacgatgtggttttggtaatcggcgctccagtgttccgttac840
caccaatacgacccaggtcaatatctcaaacctggcacgcgattgatttcggtgacctgc900
gacccgctcgaagctgcacgcgcgccaatgggcgatgcgatcgtggcagacattggtgcg960
atggctagcgctcttgccaacttggttgaagagagcagccgccagctcccaactgcagct1020
ccggaacccgcgaaggttgaccaagacgctggccgacttcacccagagacagtgttcgac1080
acactgaacgacatggccccggagaatgcgatttacctgaacgagtcgacttcaacgacc1140
gcccaaatgtggcagcgcctgaacatgcgcaaccctggtagctactacttctgtgcagct1200
ggcggactgggcttcgccctgcctgcagcaattggcgttcaactcgcagaacccgagcga1260
caagtcatcgccgtcattggcgacggatcggcgaactacagcattagtgcgttgtggact1320
gcagctcagtacaacatccccactatcttcgtgatcatgaacaacggcacctacggtgcg1380
ttgcgatggtttgccggcgttctcgaagcagaaaacgttcctgggctggatgtgccaggg1440
atcgacttccgcgcactcgccaagggctatggtgtccaagcgctgaaagccgacaacctt1500
gagcagctcaagggttcgctacaagaagcgctttctgccaaaggcccggtacttatcgaa1560
gtaagcaccgtaagcccggtgaagtga1587
<210>14
<211>1644
<212>dna
<213>乳酸乳球菌
<300>
<308>ay548760.1
<309>2004-12-27
<313>(100)..(1743)
<400>14
atgtatacagtaggagattacctgttagaccgattacacgagttgggaattgaagaaatt60
tttggagttcctggtgactataacttacaatttttagatcaaattatttcacgcgaagat120
atgaaatggattggaaatgctaatgaattaaatgcttcttatatggctgatggttatgct180
cgtactaaaaaagctgccgcatttctcaccacatttggagtcggcgaattgagtgcgatc240
aatggactggcaggaagttatgccgaaaatttaccagtagtagaaattgttggttcacca300
acttcaaaagtacaaaatgacggaaaatttgtccatcatacactagcagatggtgatttt360
aaacactttatgaagatgcatgaacctgttacagcagcgcggactttactgacagcagaa420
aatgccacatatgaaattgaccgagtactttctcaattactaaaagaaagaaaaccagtc480
tatattaacttaccagtcgatgttgctgcagcaaaagcagagaagcctgcattatcttta540
gaaaaagaaagctctacaacaaatacaactgaacaagtgattttgagtaagattgaagaa600
agtttgaaaaatgcccaaaaaccagtagtgattgcaggacacgaagtaattagttttggt660
ttagaaaaaacggtaactcagtttgtttcagaaacaaaactaccgattacgacactaaat720
tttggtaaaagtgctgttgatgaatctttgccctcatttttaggaatatataacgggaaa780
ctttcagaaatcagtcttaaaaattttgtggagtccgcagactttatcctaatgcttgga840
gtgaagcttacggactcctcaacaggtgcattcacacatcatttagatgaaaataaaatg900
atttcactaaacatagatgaaggaataattttcaataaagtggtagaagattttgatttt960
agagcagtggtttcttctttatcagaattaaaaggaatagaatatgaaggacaatatatt1020
gataagcaatatgaagaatttattccatcaagtgctcccttatcacaagaccgtctatgg1080
caggcagttgaaagtttgactcaaagcaatgaaacaatcgttgctgaacaaggaacctca1140
ttttttggagcttcaacaattttcttaaaatcaaatagtcgttttattggacaaccttta1200
tggggttctattggatatacttttccagcggctttaggaagccaaattgcggataaagag1260
agcagacaccttttatttattggtgatggttcacttcaacttaccgtacaagaattagga1320
ctatcaatcagagaaaaactcaatccaatttgttttatcataaataatgatggttataca1380
gttgaaagagaaatccacggacctactcaaagttataacgacattccaatgtggaattac1440
tcgaaattaccagaaacatttggagcaacagaagatcgtgtagtatcaaaaattgttaga1500
acagagaatgaatttgtgtctgtcatgaaagaagcccaagcagatgtcaatagaatgtat1560
tggatagaactagttttggaaaaagaagatgcgccaaaattactgaaaaaaatgggtaaa1620
ttatttgctgagcaaaataaatag1644
<210>15
<211>248
<212>prt
<213>海栖热袍菌
<300>
<308>aad36625.1
<309>2014-01-31
<313>(1)..(248)
<400>15
metileglutyrargilegluglualavalalalystyrarggluphe
151015
tyrgluphelysprovalarggluseralaglyilegluaspvallys
202530
seralailegluhisthrasnleulysprophealathrproaspasp
354045
ilelyslysleucysleuglualaarggluasnargphehisglyval
505560
cysvalasnprocystyrvallysleualaargglugluleuglugly
65707580
thraspvallysvalvalthrvalvalglypheproleuglyalaasn
859095
gluthrargthrlysalahisglualailephealavalglusergly
100105110
alaaspgluileaspmetvalileasnvalglymetleulysalalys
115120125
glutrpglutyrvaltyrgluaspileargservalvalgluserval
130135140
lysglylysvalvallysvalileilegluthrcystyrleuaspthr
145150155160
gluglulysilealaalacysvalileserlysleualaglyalahis
165170175
phevallysthrserthrglypheglythrglyglyalathralaglu
180185190
aspvalhisleumetlystrpilevalglyaspglumetglyvallys
195200205
alaserglyglyileargthrphegluaspalavallysmetilemet
210215220
tyrglyalaaspargileglythrserserglyvallysilevalgln
225230235240
glyglyglugluargtyrglygly
245
<210>16
<211>224
<212>prt
<213>热自养甲烷嗜热杆菌
<300>
<308>aab85318.1
<309>2014-01-30
<313>(1)..(224)
<400>16
metvallysmetasnvalgluthrargglugluleualaserleuile
151015
asphisthrasnvalargalaaspalathrgluasnaspilegluarg
202530
leucysargglualavalsertyrglypheargcysalavalvalthr
354045
prothrasnvalargleualaalagluleuleugluglythraspval
505560
thrvalcysservalvalglypheproalaglyvalserthrproarg
65707580
vallysalaleuglualaserglualavalgluasnglyalaglyglu
859095
valaspmetvalmetasnileglyalametlysserglyasnargglu
100105110
leuvaltyrargaspileserglyvalvalaspalaalaglyvalpro
115120125
vallysvalileleugluthralatyrleuthrasplysglulysval
130135140
glualacysleuileserlysglualaglyalaalaphevallysthr
145150155160
serthralatyrglyglyleualaglyalathrvalgluaspvalmet
165170175
leumetarglysthrvalglyaspglumetglyvallysalasergly
180185190
glyileargaspleugluthralaleualametileaspalaglyala
195200205
aspargileglythrserthrglyvalglnileilegluglytrparg
210215220
<210>17
<211>220
<212>prt
<213>耐辐射奇球菌
<300>
<308>aaf10775.1
<309>2014-01-31
<313>(1)..(220)
<400>17
metserleualasertyrileasphisthrleuleulysalathrala
151015
thrleualaaspileargthrleucysgluglualaarggluhisser
202530
phetyralavalcysileasnprovalpheileprohisalaargala
354045
trpleugluglyseraspvallysvalalathrvalcysglyphepro
505560
leuglyalailesersergluglnlysalaleuglualaargleuser
65707580
alagluthrglyalaaspgluileaspmetvalilehisileglyser
859095
alaleualaglyasptrpaspalavalglualaaspvalargalaval
100105110
argargalavalprogluglnvalleulysvalileilegluthrcys
115120125
tyrleuthraspgluglnlysargleualathrgluvalalavalgln
130135140
glyglyalaaspphevallysthrserthrglypheglythrglygly
145150155160
alathrvalaspaspvalargleumetalagluvalileglyglyarg
165170175
alaglyleulysalaalaglyglyvalargthrproalaaspalagln
180185190
alametileglualaglyalathrargleuglythrserglyglyval
195200205
glyleuvalserglyglygluasnglyalaglytyr
210215220
<210>18
<211>220
<212>prt
<213>金黄色葡萄球菌金黄色亚种mu50
<300>
<308>wp_001083318.1
<309>2015-04-27
<313>(1)..(220)
<400>18
metasnseralalysleuileasphisthrleuleulysprogluser
151015
thrargthrglnileaspglnileileaspglualalysalatyrhis
202530
phelysservalcysvalasnprothrhisvallystyralaalaglu
354045
argleualaaspsergluvalleuvalcysthrvalileglyphepro
505560
leuglyalaserthrthralathrlysalaphegluthrgluaspala
65707580
ileglnasnglyalaaspgluileaspmetvalileasnileglyala
859095
leulysaspglyargpheaspaspvalglnglnaspileglualaval
100105110
vallysalaalalysglyhisthrvallysvalileilegluthrval
115120125
leuleuasphisaspgluilevallysalasergluleuthrlysval
130135140
alaglyalaaspphevallysthrserthrglyphealaglyglygly
145150155160
alathralagluaspvallysleumetlysaspthrvalglyalaasp
165170175
valgluvallysalaserglyglyvalargasnleugluasppheasn
180185190
lysmetvalglualaglyalathrargileglyalaseralaglyval
195200205
glnilemetglnglyleuglualaaspserasptyr
210215220
<210>19
<211>220
<212>prt
<213>肺炎链球菌tigr4
<300>
<308>wp_000773677.1
<309>2013-05-15
<313>(1)..(220)
<400>19
metlysleuasnlystyrileasphisthrleuleulysglnaspala
151015
lyslyslysglnileaspserleuleuserglualaargglutyrasp
202530
phealaservalcysvalasnprothrtrpvalgluhisalalyslys
354045
glyleugluglythraspvallysvalcysthrvalvalglyphepro
505560
leuglyalathrthrseralavallysalaphegluthrlysgluala
65707580
ileglnasnglyalaaspgluileaspmetvalileasnvalglyala
859095
leulysserglyasnleualaleuvalgluseraspileargalaval
100105110
valglualaserglyasplysleuvallysvalileileglualacys
115120125
leuleuthraspglnglulysvalvalvalcysglnleualaglnlys
130135140
alaglyalaaspphevallysthrserthrglypheserthrglygly
145150155160
alathrilealaaspvalthrleumetarggluthrvalglyserasp
165170175
metglyvallysalaalaglyglyalaargsertyralaaspalaleu
180185190
alaphevalglualaglyalathrargileglythrseralaglyval
195200205
alaileleulysglygluleualaaspglyasptyr
210215220
<210>20
<211>259
<212>prt
<213>大肠埃希氏菌k12
<300>
<308>np_418798.1
<309>2014-12-16
<313>(1)..(259)
<400>20
metthraspleulysalaserserleuargalaleulysleumetasp
151015
leuthrthrleuasnaspaspaspthraspglulysvalilealaleu
202530
cyshisglnalalysthrprovalglyasnthralaalailecysile
354045
tyrproargpheileproilealaarglysthrleulysgluglngly
505560
thrprogluileargilealathrvalthrasnpheprohisglyasn
65707580
aspaspileaspilealaleualagluthrargalaalailealatyr
859095
glyalaaspgluvalaspvalvalpheprotyrargalaleumetala
100105110
glyasngluglnvalglypheaspleuvallysalacyslysgluala
115120125
cysalaalaalaasnvalleuleulysvalileilegluthrglyglu
130135140
leulysaspglualaleuilearglysalasergluileserilelys
145150155160
alaglyalaasppheilelysthrserthrglylysvalalavalasn
165170175
alathrprogluseralaargilemetmetgluvalileargaspmet
180185190
glyvalglulysthrvalglyphelysproalaglyglyvalargthr
195200205
alagluaspalaglnlystyrleualailealaaspgluleuphegly
210215220
alaasptrpalaaspalaarghistyrargpheglyalaserserleu
225230235240
leualaserleuleulysalaleuglyhisglyaspglylysserala
245250255
sersertyr
<210>21
<211>223
<212>prt
<213>单核细胞增多性李斯特氏菌egd-e
<300>
<308>np_465519.1
<309>2014-12-17
<313>(1)..(223)
<400>21
metthrilealalysmetileasphisthralaleulysproaspthr
151015
thrlysgluglnileleuthrleuthrlysglualaargglutyrgly
202530
phealaservalcysvalasnprothrtrpvallysleuseralaglu
354045
glnleualaglyalagluservalvalcysthrvalileglyphepro
505560
leuglyalaasnthrprogluvallysalaphegluvallysaspala
65707580
ileglnasnglyalalysgluvalaspmetvalileasnileglyala
859095
leulysasplysaspaspgluleuvalgluargaspileargalaval
100105110
valaspvalalalysglylysalaleuvallysvalileilegluthr
115120125
cysleuleuthraspgluglulysvalargalacysgluilealaval
130135140
lysalaglythraspphevallysthrserthrglypheserthrgly
145150155160
glyalathralagluaspilealaleumetarglysthrvalglypro
165170175
asnileglyvallysalaserglyglyvalargthrlysgluaspval
180185190
glulysmetileglualaglyalathrargileglyalaseralagly
195200205
valalailevalserglyglulysproalalysproaspasntyr
210215220
<210>22
<211>224
<212>prt
<213>嗜碱芽孢杆菌c-125
<300>
<308>wp_010897517.1
<309>2013-05-15
<313>(1)..(224)
<400>22
metserargserilealaglnmetileasphisthrleuleulyspro
151015
asnthrthrgluaspglnilevallysleucysgluglualalysglu
202530
tyrserphealaservalcysvalasnprothrtrpvalalaleuala
354045
alaglnleuleulysaspalaproaspvallysvalcysthrvalile
505560
glypheproleuglyalathrthrprogluvallysalaphegluthr
65707580
thrasnalailegluasnglyalathrgluvalaspmetvalileasn
859095
ileglyalaleulysasplysglntyrgluleuvalglyargaspile
100105110
glnalavalvallysalaalagluglylysalaleuthrlysvalile
115120125
ilegluthrserleuleuthrglugluglulyslysalaalacysglu
130135140
leualavallysalaglyalaaspphevallysthrserthrglyphe
145150155160
serglyglyglyalathralagluaspilealaleumetarglysval
165170175
valglyproasnleuglyvallysalaserglyglyvalargaspleu
180185190
seraspalalysalametileaspalaglyalathrargileglyala
195200205
seralaglyvalalailevalasnglygluargsergluglysertyr
210215220
<210>23
<211>223
<212>prt
<213>蜡样芽胞杆菌atcc10987
<300>
<308>wp_001017443.1
<309>2013-08-28
<313>(1)..(223)
<400>23
metasnilealalysleuileasphisthrileleulysalaasnthr
151015
thrlysgluaspvalmetlysvalilegluglualalysglutyrlys
202530
phealaservalcysileasnprothrtrpvallysleualaalaglu
354045
gluleualaglyhisaspvalaspvalcysthrvalileglyphepro
505560
leuglyalaserthrthrgluthrlysalaphegluthrlysaspala
65707580
ilealalysglyalathrgluvalaspmetvalileasnvalglyala
859095
leulysaspglyaspaspgluleuvalglulysaspiletyrgluval
100105110
valglnalaalalysglylysalaleuvallysvalileilegluthr
115120125
cysleuleuthraspgluglulysvalargalacysgluleuserval
130135140
lysalaglyalaaspphevallysthrserthrglypheserthrgly
145150155160
glyalathralagluaspilealaleumetarglysthrvalglypro
165170175
asnvalglyvallysalaserglyglyvalargthrarggluaspala
180185190
glulysmetvalalaalaglyalaserargvalglyalaseralaser
195200205
valalailevalleuasnaspalalysglyalathraspasntyr
210215220
<210>24
<211>223
<212>prt
<213>枯草芽孢杆菌枯草亚种菌株168
<300>
<308>cab15978.2
<309>2015-02-27
<313>(1)..(223)
<400>24
metserleualaasnileileasphisthralaleulysprohisthr
151015
glnlysalaaspileleulysleuilegluglualalysthrtyrlys
202530
phealaservalcysvalasnprothrtrpvalgluleualaalalys
354045
gluleulysglythrglyvalaspvalcysthrvalileglyphepro
505560
leuglyalaasnthrthrgluthrlysalaphegluthrlysaspala
65707580
ileserlysglyalathrgluvalaspmetvalileasnilealaala
859095
leulysasplysgluaspaspvalvalglualaaspileargglyval
100105110
valglualavalalaglylysalaleuvallysvalileilegluthr
115120125
cysleuleuthraspgluglulysgluargalacysargleualaval
130135140
seralaglyalaaspphevallysthrserthrglypheserthrgly
145150155160
glyalathrlysgluaspilealaleumetarglysthrvalglypro
165170175
aspileglyvallysalaserglyglyvalargthrlysgluaspval
180185190
aspthrmetvalglualaglyalaserargileglyalaseralagly
195200205
valserilevallysglygluasnalaserglyglyaspasntyr
210215220
<210>25
<211>329
<212>prt
<213>绿脓假单胞菌
<300>
<308>np_249818.1
<309>2015-04-20
<313>(1)..(329)
<400>25
metservalgluserileargilegluglyileaspthrprovalser
151015
argileglyleuglythrtrpalaileglyglytrpmettrpglygly
202530
alaaspaspalathrservalgluthrileargargalavalgluser
354045
glyileasnleuileaspthralaprovaltyrglypheglyhisser
505560
glugluvalvalglylysalaleuglnglyleuargasplysalaval
65707580
ilealathrlysalaalaleuglutrpseraspalaglyilehisarg
859095
asnalaseralaalaargileargarggluvalgluaspserleuarg
100105110
argleulysthraspargileaspleutyrglnilehistrpproasp
115120125
proleuvalalahisglugluthralaglygluleugluargleuarg
130135140
argaspglylysileleualaileglyvalserasntyrserproglu
145150155160
glnmetaspglypheargglnphealaproleualaservalglnpro
165170175
protyrasnleuphegluargalaileaspalaaspvalleuprotyr
180185190
alagluargasnglyilevalvalleualatyrglyalaleucysarg
195200205
glyleuleuserglyargmetasnalagluthrargpheaspglyasp
210215220
aspleuarglysseraspprolyspheglnglnproargphealagln
225230235240
tyrleualaalavalalaglnleuglugluleualaarggluargtyr
245250255
glylysservalleualaleualaileargtrpileleuasparggly
260265270
prothrvalalaleutrpglyalaarglysprogluglnleuasngly
275280285
ilealaaspalapheglytrpargleuaspaspglualametalaarg
290295300
ilegluargileleualagluthrileglnaspprovalglyproglu
305310315320
phemetalaproproserargasnala
325
<210>26
<211>568
<212>prt
<213>运动发酵单胞菌
<300>
<308>wp_011241152.1
<309>2015-04-27
<313>(1)..(568)
<400>26
metsertyrthrvalglythrtyrleualagluargleuvalglnile
151015
glyleulyshishisphealavalalaglyasptyrasnleuvalleu
202530
leuaspasnleuleuleuasnlysasnmetgluglnvaltyrcyscys
354045
asngluleuasncysglypheseralagluglytyralaargalalys
505560
glyalaalaalaalavalvalthrtyrservalglyalaleuserala
65707580
pheaspalaileglyglyalatyralagluasnleuprovalileleu
859095
ileserglyalaproasnasnasnasphisalaalaglyhisvalleu
100105110
hishisalaleuglylysthrasptyrhistyrglnleuglumetala
115120125
lysasnilethralaalaalaglualailetyrthrproglugluala
130135140
proalalysileasphisvalilelysthralaleuargglulyslys
145150155160
provaltyrleugluilealacysasnilealasermetprocysala
165170175
alaproglyproalaseralaleupheasnaspglualaseraspglu
180185190
alaserleuasnalaalavalglugluthrleulyspheilealaasn
195200205
argasplysvalalavalleuvalglyserlysleuargalaalagly
210215220
alagluglualaalavallysphealaaspalaleuglyglyalaval
225230235240
alathrmetalaalaalalysserphepheproglugluasnprohis
245250255
tyrileglythrsertrpglygluvalsertyrproglyvalglulys
260265270
thrmetlysglualaaspalavalilealaleualaprovalpheasn
275280285
asptyrserthrthrglytrpthraspileproaspprolyslysleu
290295300
valleualagluproargservalvalvalasnglyileargphepro
305310315320
servalhisleulysasptyrleuthrargleualaglnlysvalser
325330335
lyslysthrglyalaleuaspphephelysserleuasnalaglyglu
340345350
leulyslysalaalaproalaaspproseralaproleuvalasnala
355360365
gluilealaargglnvalglualaleuleuthrproasnthrthrval
370375380
ilealagluthrglyaspsertrppheasnalaglnargmetlysleu
385390395400
proasnglyalaargvalglutyrglumetglntrpglyhisilegly
405410415
trpservalproalaalapheglytyralavalglyalaprogluarg
420425430
argasnileleumetvalglyaspglyserpheglnleuthralagln
435440445
gluvalalaglnmetvalargleulysleuprovalileilepheleu
450455460
ileasnasntyrglytyrthrilegluvalmetilehisaspglypro
465470475480
tyrasnasnilelysasntrpasptyralaglyleumetgluvalphe
485490495
asnglyasnglyglytyraspserglyalaglylysglyleulysala
500505510
lysthrglyglygluleualaglualailelysvalalaleualaasn
515520525
thraspglyprothrleuileglucyspheileglyarggluaspcys
530535540
thrglugluleuvallystrpglylysargvalalaalaalaasnser
545550555560
arglysprovalasnlysleuleu
565
<210>27
<211>528
<212>prt
<213>恶臭假单胞菌
<300>
<308>wp_016501746.1
<309>2015-04-27
<313>(1)..(528)
<400>27
metalaservalhisglythrthrtyrgluleuleuargargglngly
151015
ileaspthrvalpheglyasnproglyserasngluleupropheleu
202530
lysasppheprogluasppheargtyrileleualaleuglngluala
354045
cysvalvalglyilealaaspglytyralaglnalaserarglyspro
505560
alapheileasnleuhisseralaalaglythrglyasnalametgly
65707580
alaleuserasnalatrpasnserhisserproleuilevalthrala
859095
glyglnglnthrargalametileglyvalglualaleuleuthrasn
100105110
valaspalaalaasnleuproargproleuvallystrpsertyrglu
115120125
proalaseralaalagluvalprohisalametserargalailehis
130135140
metalasermetalaproglnglyprovaltyrleuservalprotyr
145150155160
aspasptrpasplysaspalaaspproglnserhishisleupheasp
165170175
arghisvalserserservalargleuasnaspglnaspleuaspile
180185190
leuvallysalaleuasnseralaserasnproalailevalleugly
195200205
proaspvalaspalaalaasnalaasnalaaspcysvalmetleuala
210215220
gluargleulysalaprovaltrpvalalaproseralaproargcys
225230235240
propheprothrarghisprocyspheargglyleumetproalagly
245250255
ilealaalaileserglnleuleugluglyhisaspvalvalleuval
260265270
ileglyalaprovalpheargtyrhisglntyraspproglyglntyr
275280285
leulysproglythrargleuileservalthrcysaspproleuglu
290295300
alaalaargalaprometglyaspalailevalalaaspileglyala
305310315320
metalaseralaleualaasnleuvalglugluserserargglnleu
325330335
prothralaalaprogluproalalysvalaspglnaspalaglyarg
340345350
leuhisprogluthrvalpheaspthrleuasnaspmetalaproglu
355360365
asnalailetyrleuasngluserthrserthrthralaglnmettrp
370375380
glnargleuasnmetargasnproglysertyrtyrphecysalaala
385390395400
glyglyleuglyphealaleuproalaalaileglyvalglnleuala
405410415
gluprogluargglnvalilealavalileglyaspglyseralaasn
420425430
tyrserileseralaleutrpthralaalaglntyrasnileprothr
435440445
ilephevalilemetasnasnglythrtyrglyalaleuargtrpphe
450455460
alaglyvalleuglualagluasnvalproglyleuaspvalprogly
465470475480
ileasppheargalaleualalysglytyrglyvalglnalaleulys
485490495
alaaspasnleugluglnleulysglyserleuglnglualaleuser
500505510
alalysglyprovalleuilegluvalserthrvalserprovallys
515520525
<210>28
<211>547
<212>prt
<213>乳酸乳球菌
<300>
<308>wp_046124870.1
<309>2015-04-08
<313>(1)..(547)
<400>28
mettyrthrvalglyasptyrleuleuaspargleuhisgluleugly
151015
ileglugluilepheglyvalproglyasptyrasnleuglnpheleu
202530
aspglnileileserarggluaspmetlystrpileglyasnalaasn
354045
gluleuasnalasertyrmetalaaspglytyralaargthrlyslys
505560
alaalaalapheleuthrthrpheglyvalglygluleuseralaile
65707580
asnglyleualaglysertyralagluasnleuprovalvalgluile
859095
valglyserprothrserlysvalglnasnaspglylysphevalhis
100105110
histhrleualaaspglyaspphelyshisphemetlysmethisglu
115120125
provalthralaalaargthrleuleuthralagluasnalathrtyr
130135140
gluileaspargvalleuserglnleuleulysgluarglysproval
145150155160
tyrileasnleuprovalaspvalalaalaalalysalaglulyspro
165170175
alaleuserleuglulysgluserserthrthrasnthrthrglugln
180185190
valileleuserlysileglugluserleulysasnalaglnlyspro
195200205
valvalilealaglyhisgluvalileserpheglyleuglulysthr
210215220
valthrglnphevalsergluthrlysleuproilethrthrleuasn
225230235240
pheglylysseralavalaspgluserleuproserpheleuglyile
245250255
tyrasnglylysleusergluileserleulysasnphevalgluser
260265270
alaasppheileleumetleuglyvallysleuthraspserserthr
275280285
glyalaphethrhishisleuaspgluasnlysmetileserleuasn
290295300
ileaspgluglyileilepheasnlysvalvalgluasppheaspphe
305310315320
argalavalvalserserleusergluleulysglyileglutyrglu
325330335
glyglntyrileasplysglntyrgluglupheileproserserala
340345350
proleuserglnaspargleutrpglnalavalgluserleuthrgln
355360365
serasngluthrilevalalagluglnglythrserphepheglyala
370375380
serthrilepheleulysserasnserargpheileglyglnproleu
385390395400
trpglyserileglytyrthrpheproalaalaleuglyserglnile
405410415
alaasplysgluserarghisleuleupheileglyaspglyserleu
420425430
glnleuthrvalglngluleuglyleuserileargglulysleuasn
435440445
proilecyspheileileasnasnaspglytyrthrvalgluargglu
450455460
ilehisglyprothrglnsertyrasnaspilepromettrpasntyr
465470475480
serlysleuprogluthrpheglyalathrgluaspargvalvalser
485490495
lysilevalargthrgluasngluphevalservalmetlysgluala
500505510
glnalaaspvalasnargmettyrtrpilegluleuvalleuglulys
515520525
gluaspalaprolysleuleulyslysmetglylysleuphealaglu
530535540
glnasnlys
545
<210>29
<211>49
<212>dna
<213>引物
<400>29
cctcgtctcaaccaaagcaatcaacccatcagtcgacctgcaggcatgc49
<210>30
<211>53
<212>dna
<213>引物
<400>30
cgagacagcctgagaatggatgcgagtaatgcgagctcgaattccatggtctg53
<210>31
<211>63
<212>dna
<213>引物
<400>31
gctgggagttcgtagacggaaacaaacgcaaggaggtattgcacatgtcacgttcgattg60
cac63
<210>32
<211>51
<212>dna
<213>引物
<400>32
gtttccagtgcgattgaggaccttcagtgcttaataactcccttcgctacg51
<210>33
<211>65
<212>dna
<213>引物
<400>33
ctgacctcctgccagcaatagtaagacaacaggaggtatcgggcatgagcgttgaaagca60
ttcgc65
<210>34
<211>49
<212>dna
<213>引物
<400>34
acttgagtgaggttgtaaagggagttggctcttaggcgttgcggctggg49
<210>35
<211>66
<212>dna
<213>引物
<400>35
ctcgttcgctgccacctaagaatactctacgaggaggtattcgccatgagttatactgtc60
ggtacc66
<210>36
<211>51
<212>dna
<213>引物
<400>36
agtagcggaaatgtcagagccagcgtcttgctagaggagcttgttaacagg51
<210>37
<211>24
<212>dna
<213>引物
<400>37
ggtcaactaatccttaactgatcg24
<210>38
<211>23
<212>dna
<213>引物
<400>38
aagcaaatcatcaccgcactgac23
<210>39
<211>20
<212>dna
<213>引物
<400>39
caggcttagcgcaacaaacg20
<210>40
<211>19
<212>dna
<213>引物
<400>40
ggttgcgcctacactaagc19
<210>41
<211>24
<212>dna
<213>引物
<400>41
tccacttaagaaggtaggtgttac24
<210>42
<211>23
<212>dna
<213>引物
<400>42
aaagttgccgctttacggggaaa23
- 还没有人留言评论。精彩留言会获得点赞!