微量基因组DNA的探针连接扩增检测方法与流程
本发明涉及一种微量基因组DNA的探针连接扩增检测方法。
背景技术:
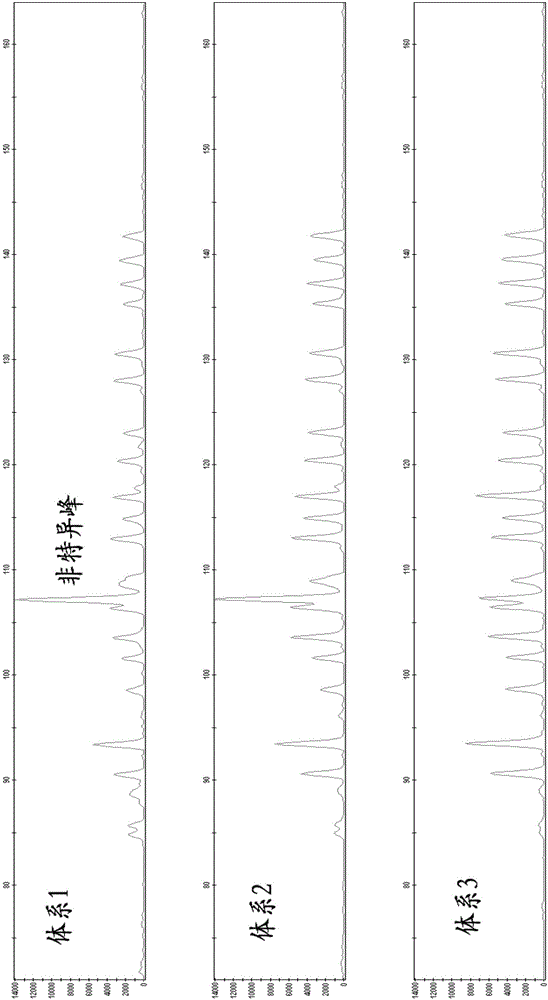
:多重探针连接扩增技术(MLPA)是2002年由荷兰学者Schouten发明的一种对待测核酸中靶序列进行定性和定量分析的技术。利用探针杂交、连接及多重PCR反应,它可以同时检测多个目标核酸序列的存在和拷贝数变化。MLPA以其高特异性、方便快捷等特点,被广泛地应用于基因或染色体片段拷贝数异常的检测。2012年,研究人员在MLPA的技术基础上发明了一种高通量探针连接扩增技术HLPA(high-throughputligation-dependentprobeamplification),大大提升了检测的通量和效率。2015年,Zhang等人使用HLPA成功开展了对先天性心脏病的诊断,体现了这一技术在临床诊断中的应用。此外,作为核型分析的有益补充,探针连接扩增技术可用于临床羊水标本中胎儿染色体非整倍体的产前诊断。虽然MLPA和HLPA在科研和临床中应用广泛,但在技术上仍存在不足,例如在探针结合目标DNA以及后续的酶连接反应中需要使用较多的模板DNA。在常规连接反应体系(http://www.mrc-holland.com)中对模板DNA的需求为50-250ng。这样一来,对于一些微量的DNA样本就可能存在样本量无法满足实验要求的情况,因为研究者可能会从极其微小的组织、甚至是单细胞中提取待检测的核酸。因此,难以检测微量DNA成为了MLPA和HLPA在分子诊断领域中的技术瓶颈。对于起始核酸量较低的情况,研究者通常采取以下方法:(1)选取更多的样本抽提DNA/RNA。这一方法在实际操作中可行性不高,因为样本(组织、血液等)有限,无法大量获得;(2)使用提取量更高的核酸抽提方法。这一手段往往意味着实验成本的增加,不利于在临床中的应用;(3)引物延伸预扩增法(PrimerExtensionPre-amplification,PEP)使用随机引物对含量较低的模板DNA进行预扩增。但这一做法会导致扩增的不均一性和非特异性扩增,影响后续检测的准确性。在目前,尚无任何报道显示探针连接扩增技术可以对50ng以下的DNA进行检测。考虑到这一技术的成本较低且对拷贝数的检测非常精确,如果能够有效地检测微量DNA,将成为临床分子诊断技术的一项新突破。技术实现要素:本发明的目的在于克服现有技术存在的以上问题,提供一种微量基因组DNA的探针连接扩增检测方法,本方法是将起始量低于50ng的DNA进行浓缩或蒸干呈粉末状后,以微量反应体系(反应液总体积不高于20μL,模板DNA加入量不超过50ng)进行连接反应(液体表面覆盖有机试剂,如矿物油以防止挥发),以达到预期的实验效果。为实现上述技术目的,达到上述技术效果,本发明通过以下技术方案实现:一种微量基因组DNA的探针连接扩增检测方法,包括以下步骤:1)微量基因组DNA样本进行浓缩或蒸干呈粉末状;2)以浓缩或蒸干的基因组DNA为模板进行连接反应,连接体系的体积不高于20μL,模板DNA量不高于50ng;3)微量连接体系采取防挥发措施,减少反应体系与空气的接触面积,比如在液面覆盖有机试剂、采用末端封闭的毛细管进行连接反应等。4)以连接反应的产物为模板进行多重PCR扩增;5)得到扩增产物。进一步的,所述扩增产物用于后续实验,包括荧光毛细电泳、高通量二代测序或基因芯片检测中的一种或多种实验。本发明的有益效果是:1、模板需求量少:在本发明的微量连接体系中,DNA模板的总量低于50ng也可达到常规连接体系的检测效果,对DNA含量极少的情况,比如孕妇血浆游离DNA可以进行检测,突破了传统探针连接扩增反应的瓶颈。2、试剂损耗小:本发明的微量连接体系相对常规体系体积减少较多,因此各个成分的试剂损耗也相对减少,节约了试剂成本。3、反应特异性、稳定性高:在微量连接体系中,DNA模板量低于50ng可以达到常规连接体系的特异性和稳定性。上述说明仅是本发明技术方案的概述,为了能够更清楚了解本发明的技术手段,并可依照说明书的内容予以实施,以下以本发明的较佳实施例并配合附图详细说明如后。本发明的具体实施方式由以下实施例及其附图详细给出。附图说明此处所说明的附图用来提供对本发明的进一步理解,构成本申请的一部分,本发明的示意性实施例及其说明用于解释本发明,并不构成对本发明的不当限定。在附图中:图1为微量基因组DNA探针连接反应流程;图2为不同体系检测样本峰图;图3位不同检测体系的变异系数(CV)比较;图4为不同体系所有待测位点的拷贝数平均值比较。具体实施方式下面将参考附图并结合实施例,来详细说明本发明。该实施例对起始量为5ng的人类基因组DNA进行微量DNA体系的探针连接反应,以MLPA技术为例,对21号染色体上的13个位点(待测位点)进行拷贝数检测,选取了5个非21号染色体的位点作为参照位点。所选取的位点信息参见表1。探针序列见表2和表3,其中,序列如序列表SEQIDNO.:1至SEQIDNO.:36所示。在探针与基因组待测位点序列特异性结合后,采用连接酶使用不同体积的反应体系(体系1:体积20μL;体系2:体积12μL;体系3:体积5μL)进行连接反应。连接产物进行多重荧光PCR扩增、通过毛细电泳分离不同大小的片段并对13个待测位点的拷贝数进行计算,每个体系进行5次重复实验。表1待测位点和参照位点信息表25’端探针序列表33’端探针序列一、检测样本样本为从血细胞中提取的健康人基因组DNA。二、检测方法主要包括以下步骤:将待测样本用real-timePCR进行精确定量,然后取总量为5ng的DNA真空抽干作为待测样本。本实验需要以下试剂:ddH2O,10×ligasebuffer(NEB,B0208S),TaqDNA连接酶(NEB,M0208L),连接探针混合液,待测基因组DNA(真空抽干),10×PCRbuffer(Takara,R007WZ),MgCl2,dNTP,TaqDNA聚合酶(Takara,R007WZ),PCR引物混合液。A.连接体系配制:体系1(20μL):体系2(12μL):体系3(5μL):备注:体系3需加入10μL矿物油,防止反应体系挥发。连接反应条件设置如下:95℃2min;4个循环(96℃20s,60℃4h);94℃2min,在72℃保存。反应结束后需加入与连接反应体积等体积的反应终止液(20mMEDTA)停止反应,震荡混匀后离心。B.连接反应的产物用带有荧光标记的通用引物进行多重PCR扩增,引物序列和荧光标记见表4。表4多重荧光PCR反应引物信息引物名称荧光标记序列(5'-3')正向引物VICTATTCGCTCATAACGGGTTCG反向引物无GTTTCTTGCTTCCCTAGAGCGGGTGATTT多重荧光PCR反应体系配制(30μL):PCR反应条件设置如下:95℃2min;5个循环扩增(94℃20s,62℃-1℃每循环40s和72℃90s);28个循环扩增(94℃20s,57℃40s和72℃90s),在68℃延伸60min,4℃保存。C.毛细管电泳分离扩增产物取1μl荧光多重PCR产物用ddH2O稀释10倍,然后取1μl加入到8.9μl的Hi-Di(ABI)和0.1μl的LIZ500分子内标(ABI)中,95℃5min变性后,置于3730XL测序仪上进行毛细管电泳。电泳结果使用GeneMapper4.0软件进行数据分析,读取峰高等数据,参照图2、图3所示。三、数据分析1.位点峰型和峰高原始数据图2和图3显示了不同反应体系阴性对照及样本检测峰图结果。以体系3为例说明拷贝数的计算方法。从13个待测位点中选取一个位点(T015),对应5个参照位点(TREF37,38,39,40,41)。表5为待测位点(T015)和对应的参照位点(TREF)的峰高数据。表5样本位点峰高数据2.样本校正计算待测位点峰高分别与对应的参照位点峰高的比值,取同一体系中所有样本该待测位点峰高比值的中位数即为该位点的校正值。表6待测位点与参照位点的峰高比值3.位点拷贝数计算用每个参照位点计算得到的峰高比值除以对应的校正值,所有对应参照位点计算的相对拷贝数的中位数乘以2即为绝对拷贝数。针对所有参照位点计算得到的绝对拷贝数的平均值即为该位点的最终拷贝数计算值。计算结果见表7。表7位点拷贝数计算结果4.单个样本所有位点的CV值计算表8至表10为单个样本所有位点的CV值计算方法公式:CV=待测位点拷贝数的标准差(SD)/待测位点拷贝数的平均值(Mean)表8体系1所有位点的平均CV值表9体系2所有位点的平均CV值表10体系3所有位点的平均CV值5.不同体系的所有位点拷贝数计算比较图4显示了13个待测位点在体系1,2,3实验中计算得到的平均拷贝数。因为样本为正常基因组DNA,所以理论拷贝数为2。可以明显看出,体系3得到的拷贝数在不同待测位点间的误差最小,CV值在三个体系中最小(图3),计算结果也最接近理论值。四、结论通过对比实验发现,对于低于50ng的基因组DNA样本的检测,通过样本浓缩蒸干,以微量体系进行连接反应,可以达到预期实验效果。在模板DNA固定的情况下,缩小反应体系体积得到的CV更小,结果更精确。在模板DNA加入量一定的条件下,与体系1和体系2相比,体系3具有如下优势:1、阴性对照中的非特异性峰高度相对较低;2、样本检测结果峰图中,非特异性峰高明显降低,每个位点对应的峰高更高,且峰型更均一;3、样本所有位点的波动小,实验稳定性更好。以上所述仅为本发明的优选实施例而已,并不用于限制本发明,对于本领域的技术人员来说,本发明可以有各种更改和变化。凡在本发明的精神和原则之内,所作的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。SEQUENCELISTING<110>天昊生物医药科技(苏州)有限公司<120>微量基因组DNA的探针连接扩增检测方法<160>36<170>PatentInversion3.5<210>1<211>44<212>DNA<213>ArtificialSequence<400>1tattcgctcataacgggttcgtgcaaaactgtggccagttatgc44<210>2<211>42<212>DNA<213>ArtificialSequence<400>2tattcgctcataacgggttcgcagcggttcgccaacaagaag42<210>3<211>48<212>DNA<213>ArtificialSequence<400>3tattcgctcataacgggttcgccctaagagagaagccatgtggaggag48<210>4<211>51<212>DNA<213>ArtificialSequence<400>4tattcgctcataacgggttcgcgatccagagagcagggcaggagatgatac51<210>5<211>48<212>DNA<213>ArtificialSequence<400>5tattcgctcataacgggttcgcgatttctgctggcagacaccatttgt48<210>6<211>50<212>DNA<213>ArtificialSequence<400>6tattcgctcataacgggttcgcgattcattttccatttagggaatgtggg50<210>7<211>52<212>DNA<213>ArtificialSequence<400>7tattcgctcataacgggttcgcgatggtatttccaaatcagacattttgccc52<210>8<211>54<212>DNA<213>ArtificialSequence<400>8tattcgctcataacgggttcgcgatcgatcgtgctctttcctctccaaatggga54<210>9<211>55<212>DNA<213>ArtificialSequence<400>9tattcgctcataacgggttcgcgatcgtctttttaacaactcatctccctgagcc55<210>10<211>54<212>DNA<213>ArtificialSequence<400>10tattcgctcataacgggttcgcgatcgatccctttgactggtttgtggccatct54<210>11<211>57<212>DNA<213>ArtificialSequence<400>11tattcgctcataacgggttcgcgatcgatcgttttaaaaaatgccctactccttccc57<210>12<211>61<212>DNA<213>ArtificialSequence<400>12tattcgctcataacgggttcgcgatcgatcgatcgaatcctgaaccacactctcagagcc60t61<210>13<211>62<212>DNA<213>ArtificialSequence<400>13tattcgctcataacgggttcgcgatcgatcgatcgcatagacatatggaccagcaatcag60gc62<210>14<211>62<212>DNA<213>ArtificialSequence<400>14tattcgctcataacgggttcgcgatcgatcgatcgatcgatggtgcgcgctcctcctaac60tc62<210>15<211>66<212>DNA<213>ArtificialSequence<400>15tattcgctcataacgggttcgcgatcgatcgatcgatcgatcgccaccctccagtagcct60tttcct66<210>16<211>67<212>DNA<213>ArtificialSequence<400>16tattcgctcataacgggttcgcgatcgatcgatcgatcgatgcacacaagctcacccact60caagtta67<210>17<211>66<212>DNA<213>ArtificialSequence<400>17tattcgctcataacgggttcgcgatcgatcgatcgtattttctatcaccttggaaaggca60ctattt66<210>18<211>67<212>DNA<213>ArtificialSequence<400>18tattcgctcataacgggttcgcgatcgatcgatcgattctattatagtttgaccatagcc60acatggg67<210>19<211>41<212>DNA<213>ArtificialSequence<400>19cgcagcacggagtgagtgacaaatcacccgctctagggaag41<210>20<211>46<212>DNA<213>ArtificialSequence<400>20tggtaaagtagcaaaaatggggatgaaatcacccgctctagggaag46<210>21<211>44<212>DNA<213>ArtificialSequence<400>21cgaggcgggtttgatgtgtgcgaaaatcacccgctctagggaag44<210>22<211>45<212>DNA<213>ArtificialSequence<400>22agaaataagagcaggggagaggggaaatcacccgctctagggaag45<210>23<211>49<212>DNA<213>ArtificialSequence<400>23gaatggacagttttccagatggtggcgaaaatcacccgctctagggaag49<210>24<211>50<212>DNA<213>ArtificialSequence<400>24agctctggtcagagacccacagatcgatcaaatcacccgctctagggaag50<210>25<211>51<212>DNA<213>ArtificialSequence<400>25aaaattgctgcccctctctggttcgatcgaaaatcacccgctctagggaag51<210>26<211>53<212>DNA<213>ArtificialSequence<400>26cttccccaaatcaggaaaatgtgacccgatcgaaatcacccgctctagggaag53<210>27<211>55<212>DNA<213>ArtificialSequence<400>27ctgcaaaacagagcacctcctcccgatcgatcgaaaatcacccgctctagggaag55<210>28<211>56<212>DNA<213>ArtificialSequence<400>28gttgtcctctctgttacggcctcatccgatcgatcaaatcacccgctctagggaag56<210>29<211>58<212>DNA<213>ArtificialSequence<400>29accactggaaatttgggcagagtccgatcgatcgatcaaatcacccgctctagggaag58<210>30<211>58<212>DNA<213>ArtificialSequence<400>30aatgcatcacttgtgccagaagaatccgatcgatcgaaaatcacccgctctagggaag58<210>31<211>60<212>DNA<213>ArtificialSequence<400>31gaatacagagccctttggacatcttgccgatcgatcgataaatcacccgctctagggaag60<210>32<211>62<212>DNA<213>ArtificialSequence<400>32ggggtcgcttttagcgagaagtgcgatcgatcgatcgatcgaaatcacccgctctaggga60ag62<210>33<211>63<212>DNA<213>ArtificialSequence<400>33ggtggttctgcattgtacctgagaaacgatcgatcgatcgataaatcacccgctctaggg60aag63<210>34<211>65<212>DNA<213>ArtificialSequence<400>34catgtatacatatgcaggaaggcacaagtaccgatcgatcgatcaaatcacccgctctag60ggaag65<210>35<211>66<212>DNA<213>ArtificialSequence<400>35ctctgagcctggatttgttttctgccgatcgatcgatcgatcgataaatcacccgctcta60gggaag66<210>36<211>67<212>DNA<213>ArtificialSequence<400>36aagttatgccagtggacattcctttgcgatcgatcgatcgatcgataaatcacccgctct60agggaag67当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1
- 带DNA分子探针的石墨烯微电极电化学检测传感器的制造方法与工艺
- 一种小型化时间分辨的激光泵浦X射线探测仪的制造方法与工艺
- 一种气冷高温稳态压力探针的制造方法与工艺
- 基于超磁致伸缩与石英摆线复合机理的隔微振装置的制造方法
- 纳米材料制备装置及纳米材料制备方法与流程
- 一种用于EB病毒荧光定量PCR检测的引物和探针组、及试剂盒的制造方法与工艺
- 一种用于鉴定川贝母及伪品的荧光PCR检测引物、探针组合物、试剂盒及检测方法与应用与流程
- 用于基因甲基化检测的引物和探针、采样方法、试剂盒与流程
- 一种基于荧光PCR检测ESR1基因突变的引物及探针的制造方法与工艺
- 转基因甜菜GTSB77品系特异性实时荧光PCR检测引物、探针、方法和试剂盒与流程