一种双酶偶联全细胞催化马来酸合成L-天冬氨酸的方法与流程
本发明涉及一种双酶偶联全细胞催化马来酸合成L-天冬氨酸的方法,属于生物工程领域。
背景技术:
L-天冬氨酸(L-aspartic acid)是构成蛋白质的20种基础氨基酸之一。其在食品、医药、化工等领域都有着广泛的应用和较大的市场开发潜力。
目前,工业生产L-天冬氨酸的工艺主要是以顺丁烯二酸酐为原料,在无机催化剂、强酸性(pH=1左右)条件下转化成富马酸;分离纯化获得富马酸,然后富马酸与过量的氨在L-天冬氨酸裂解酶催化下转化生成L-天冬氨酸铵,反应液用硫酸中和过量的氨后,分离纯化得到产品L-天冬氨酸。此工艺虽然流程简单,但是其缺点明显:需要在高温、高压、过渡金属催化剂及强酸性条件下进行,对设备的要求严格,造成的环境污染严重,同时需要分离纯化中间产物富马酸,会造成产率的下降等。相比较而言,双酶偶联全细胞催化转化法具有专一性强,转化率高、工艺简洁、设备投资少及环境污染小等优点,因此双酶偶联全细胞催化转化法具有更好的应用前景。
目前,关于双酶偶联全细胞催化马来酸合成L-天冬氨酸的研究还较少,大多数都还是停留在以富马酸为底物的研究基础上,主要原因受限于马来酸顺反异构酶的稳定性差、酶活低、难以异源表达。
技术实现要素:
本发明提供了一种偶联表达马来酸顺反异构酶(粘质沙雷氏菌来源)和L-天冬氨酸裂解酶(大肠杆菌来源)的重组菌株,并对该重组菌株进行改造优化,获得能够全细胞高转化率催化马来酸生成L-天冬氨酸的重组菌。
所述偶联表达马来酸顺反异构酶和L-天冬氨酸裂解酶的重组菌株,是以大肠杆菌为宿主,表达以pRSFDuet-1为表达载体构建的重组表达载体pRSFDuet-1-maiA-aspA。
在本发明的一种实施方式中,所述大肠杆菌是敲除了基因组中fum A-fum C基因的E.coliBL21(DE3)△fumAC。
在本发明的一种实施方式中,编码所述马来酸顺反异构酶的基因的核苷酸序列如SEQ ID NO.2所示。
在本发明的一种实施方式中,编码所述L-天冬氨酸裂解酶的基因的核苷酸序列如SEQ ID NO.4所示。
在本发明的一种实施方式中,所述马来酸顺反异构酶的第27位的甘氨酸突变为丙氨酸并且第171位的甘氨酸突变为丙氨酸;或者第27位的甘氨酸突变为丙氨酸并且第104位的赖氨酸突变为精氨酸。
在本发明的一种实施方式中,还在SEQ ID NO.4基础上将编码L-天冬氨酸裂解酶的基因的RBS序列替换为SEQ ID NO.23或SEQ ID NO.24或SEQ ID NO.25所示的序列。
本发明还提供应用所述重组菌株全细胞转化马来酸生成L-天冬氨酸的方法,是以pH 8.0的2M的马来酸溶液为底物,加入菌体浓度OD600为40的静息细胞悬液,静息细胞悬液与底物溶液的体积比为2:8。
所述方法在pH 8.0的50mM的Na2HPO4-KH2PO4缓冲液中进行,反应温度为37℃。
本发明提供了一株能高效表达马来酸顺反异构酶和L-天冬氨酸裂解酶的重组菌株,能够利用价格相对便宜的马来酸生成L-天冬氨酸,反应40~120min,马来酸反应完全,而且几乎没有中间产物富马酸的积累,转化率到达98%以上。
附图说明
图1MaiA与AspA偶联表达的不同策略
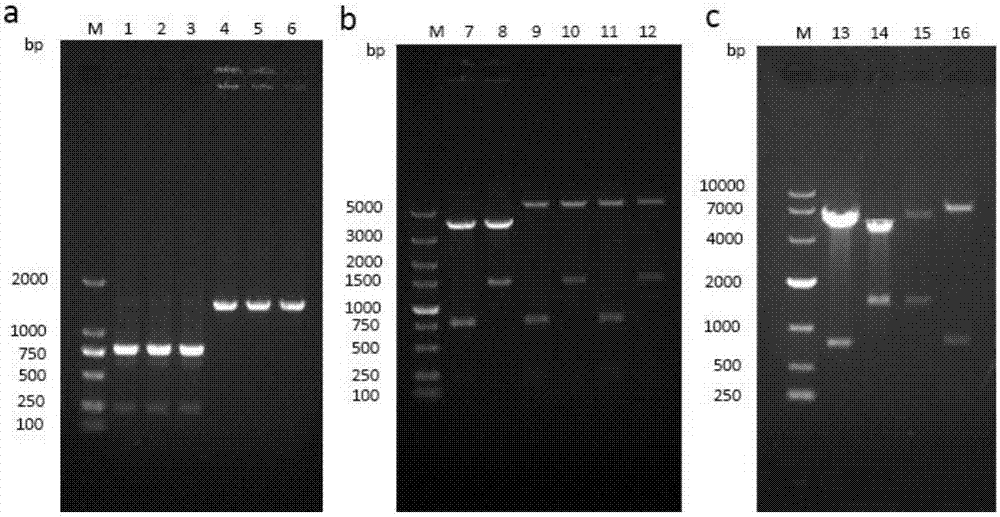
图2PCR扩增maiA和aspA基因及重组质粒的酶切验证结果:M:marker;1BamHI-maiA-HindIII的PCR产物;2NdeI-maiA-XhoI的PCR产物;3HindIII-maiA-XhoI的PCR产物;4BamHI-aspA-HindIII的PCR产物;5NdeI-aspA-XhoI的PCR产物;6HindIII-aspA-XhoI的PCR产物;7pRSFDuet-1-maiA重组质粒的双酶切验证;8pRSFDuet-1-aspA重组质粒的双酶切验证;9pETDuet-1-maiA重组质粒的双酶切验证;10pETDuet-1-aspA重组质粒的双酶切验证;11pET-28a(+)-maiA重组质粒的双酶切验证;12pET-28a(+)-aspA重组质粒的双酶切验证;13pRSFDuet-1-aspA-maiA重组质粒的双酶切验证;14pRSFDuet-1-maiA-aspA重组质粒的双酶切验证;15pET-28a(+)-maiA-aspA重组质粒的双酶切验证;16pET-28a(+)-aspA-maiA重组质粒的双酶切验证
图3不同偶联表达的SDS-PAGE结果:M:marker;1pRSFDuet-1-maiA表达结果;2pRSFDuet-1-aspA表达结果;3pMA 1表达结果;4pAM 1表达结果;5pMA2表达结果;6pAM2表达结果;7pMA 3表达结果;8pAM 3表达结果;
图4pMA2全细胞催化结果
图5pMA2中的MaiA的RBS改造后SDS-PAGE结果:M:marker;1pMA2表达结果;2pMA2-1表达结果;3pMA2-2表达结果;4pMA2-3表达结果;5pMA2-4表达结果;
图6pMA2-4全细胞催化结果
图7pMA2-4(G27A-G171A)全细胞催化结果
图8pMA2-4(G27A-K104R)全细胞催化结果
具体实施方式
马来酸、富马酸和L-天冬氨酸的检测方法:
马来酸、富马酸和L-天冬氨酸的浓度都用高效液相色谱(HPLC)法检测。马来酸、富马酸的HPLC检测条件:色谱柱Prevail Organic Acid(250mm×4.6mm,5m;Grace DavisonDiscovery Sciences),流动相为pH 2.5、浓度25mM的K2HPO4溶液,流速1mL/min,柱温40℃,紫外检测器波长210nm,进样量10μL。L-天冬氨酸的检测要先经过苯基异硫酸酯(PITC)进行衍生,在其氨基端连接一个苯环,便于分离。衍生方法为:取500μL反应液,加入250μL 1M的三乙胺-乙腈溶液和250μL 0.1M的PITC-乙腈溶液,振荡混匀,避光反应1h。待衍生结束后,700μL正己烷,振荡30s萃取出残余的衍生试剂,静置,待反应液有明显分层后吸取下层溶液,用0.22μm针头式有机滤膜过滤。HPLC检测条件:色谱柱La ChromC18(5μm,4.6mm×250mm),用梯度洗脱的方法进行检测。A流动相为80%的乙腈溶液,B流动相为97:3的0.1M乙酸钠-乙腈溶液;梯度洗脱条件为:0~35min,B流动相由95%降至65%;35~40min,B流动相由65%升至95%;40~45min,B流动相浓度不变。检测温度为40℃,检测波长为254nm。
实施例1构建偶联表达马来酸顺反异构酶和L-天冬氨酸裂解酶的重组菌株
1)马来酸顺反异构酶的氨基酸序列如SEQ ID NO.1所示,编码马来酸顺反异构酶的基因的核苷酸序列如SEQ ID NO.2所示;L-天冬氨酸裂解酶的氨基酸序列如SEQ ID NO.3所示,编码L-天冬氨酸裂解酶的基因的核苷酸序列如SEQ ID NO.4所示。根据目的基因和载体,选择酶切位点,设计引物(如表1);
表1酶切连接引物设计
注:下划线标出的为酶切位点
2)分别以pET-24a(+)–maiA和pET-28a(+)–aspA为模板进行PCR,得到如图2a所示的带不同酶切位点的maiA和aspA基因片段:BamHI-maiA-HindIII、NdeI-maiA-XhoI、HindIII-maiA-XhoI、BamHI-aspA-HindIII、NdeI-aspA-XhoI、HindIII-aspA-XhoI;
3)纯化PCR片段,用对应的酶进行双酶切2h,也将对应的质粒载体pRSFDuet-1、pETDuet-1、pET-28a(+)用对应的双酶切2h,然后将酶切产物切胶回收纯化;
4)用核酸定量仪测定回收后的基因和载体片段的浓度,按基因片段:载体片段=3:1的比例混合,再加T4DNA连接酶在16℃过夜连接;
5)将连接产物转化到JM109的感受态细胞里,然后涂布到载体相应的抗生素抗性的LB平板培养基上;
6)先菌落PCR验证,然后挑取单菌落到5mL的抗生素浓度为50μg/mL的LB液体试管培养基(10g蛋白胨、5g酵母膏、10gNaCl定容到1L)中,37℃、200rpm培养8h,提质粒进行双酶切验证(如图2b和2c),将验证正确的菌进行测序;
7)将测序正确的重组质粒转化到表达宿主E.coli BL21(DE3)△fumAC(E.coli BL21(DE3)△fumAC的构建方法参见文章:房月芹,周丽,周哲敏.重组大肠杆菌全细胞转化马来酸高效合成富马酸[J].食品与生物技术学报,2016,35(12):1323-1329.)感受态细胞里,然后涂布到载体相应的抗生素抗性的LB平板培养基上;挑取单菌落得到六种偶联表达体系:pMA 1:pET-28a(+)–maiA-aspA;pAM 1:pET-28a(+)–aspA-maiA;pMA 2:pRSFDuet-1-maiA-aspA;pAM 2:pRSFDuet-1-aspA-maiA;pMA 3:pRSFDuet-1-maiA-pETDuet-1-aspA;pAM 3:pRSFDuet-1-aspA-pETDuet-1-maiA 6。
8)挑取单菌落接种于5mL的抗生素浓度为50μg/mL的LB培养基中,37℃、200rpm培养8小时,然后以2%的接种量转接到装有50mL的2YT培养基(16g蛋白胨、10g酵母膏、5gNaCl定容到1L)的250mL的摇瓶中,抗生素浓度为50μg/mL,37℃、200rpm培养到OD为0.8,然后加终浓度为0.2mmol/L的IPTG在20℃下诱导表达20小时;
9)各取相同量的诱导表达细胞,离心收集菌体,再用pH 8.0的50mM的Na2HPO4-KH2PO4缓冲液重悬细胞,超声破碎得到粗酶液。SDS-PAGE电泳分析目的蛋白的表达情况(如图3),当MaiA和AspA分别单独表达时,其表达量都比较高,而当其串联表达时,其表达量都有相应的降低,而且不同的串联方式,两个酶的表达水平相差也较大。期中pMA 1和pAM 1的串联体系的两个酶的表达量都较低;pMA2的串联体系的MaiA表达较好,而AspA的表达较低;pAM 2的串联体系的两个酶几乎都没有表达;pMA3和pAM3的串联体系的AspA的表达较好,而MaiA的表达较低。取由各重组细胞生产得到的100μL粗酶液,加入到50μL的2M的pH 8.0的马来酸铵溶液,用pH 8.0的50mM的Na2HPO4-KH2PO4缓冲液补充体系到500μL,在40℃反应10min,然后再100℃煮沸10min,离心取上清,检测上清中马来酸、富马酸和L-天冬氨酸的含量。如表2,pMA2的串联体系的催化生成L-天冬氨酸的效果最佳,这是因为AspA的酶活比MaiA要高的多,故此催化体系的限速因素是MaiA的总酶活,因此当MaiA的表达量越高时,催化效果越好。
表2重组菌粗酶催化200mM的马来酸的结果比较
实施例2偶联表达马来酸顺反异构酶和L-天冬氨酸裂解酶的重组菌株全细胞pMA 2转化马来酸生成L-天冬氨酸
首先配置pH 8.0的马来酸溶液,此过程用氨水调节马来酸的pH到8.0。收集实施例1诱导表达的细胞pMA 2,用pH 8.0的50mM的Na2HPO4-KH2PO4缓冲液重悬细胞,稀释到OD600到40,然后按20%(体积比)的静息细胞和80%(体积比)的底物马来酸混合,反应体系为30mL,在200r/min、37℃的摇床中催化反应,每隔20min取样检测反应液中马来酸、富马酸和L-天冬氨酸的含量。如图4,在120min底物马来酸反应完,而且几乎没有中间产物富马酸的积累,转化率到达98%以上。
实施例3 MaiA基因序列中的RBS改造
(1)通过RBS Calculator软件预测不同的起始翻译速率的RBS序列选择四个不同的翻译起始速率的RBS序列,设计引物(如表3);
表3 MaiA的RBS改造引物设计
注:斜体下划线标出的为RBS Calculator预测的RBS序列
(2)以pMA2为模板,通过全质粒PCR的方法替换pRSFDuet-1-maiA-aspA中MaiA的原始RBS序列,用DpnI酶对PCR产物进行过夜消化;
(3)将消化产物转化到JM109的感受态细胞里,然后涂布到带卡那霉素的LB平板培养基上;
(4)挑取单菌落进行测序,将正确突变的突变体重组质粒转化到表达宿主E.coliBL21(DE3)△fumAC感受态细胞里,然后涂布到带卡那霉素的LB平板培养基上,获得四株MaiA的RBS的翻译起始速率不同的菌株:pMA 2-1、pMA 2-2、pMA 2-3、pMA 2-4;
(5)分别挑取单菌落接种于5mL的抗生素浓度为50μg/mL的LB培养基中,37℃、200rpm培养8小时,然后以2%的接种量转接到装有50mL的2YT培养基(16g蛋白胨、10g酵母膏、5g NaCl定容到1L)的250mL的摇瓶中,抗生素浓度为50μg/mL,37℃、200rpm培养到OD为0.8,然后加终浓度为0.2mmol/L的IPTG在20℃下诱导表达20小时;
(6)离心收集等量的菌体,再用pH 8.0的50mM的Na2HPO4-KH2PO4缓冲液重悬细胞,超声破碎得到粗酶液,SDS-PAGE验证MaiA的表达量,如图5,随着RBS起始翻译速率的增加,MaiA的表达量也逐渐增加,pMA 2-4的表达量最高。接下来对pMA 2-4进行全细胞催化。
(7)配置pH 8.0的马来酸溶液,此过程用氨水调节马来酸的pH到8.0。收集步骤(5)诱导表达的pMA 2-4细胞,用pH 8.0的50mM的Na2HPO4-KH2PO4缓冲液重悬细胞,稀释到OD600到40,然后按20%的静息细胞和80%的底物马来酸铵混合,反应体系为30mL,在200r/min、37℃的摇床中催化反应,每隔20min取样检测反应液中马来酸、富马酸和L-天冬氨酸的含量。如图6,相同条件下pMA 2-4完全催化转化底物马来酸只需要80min,且也几乎没有中间产物富马酸的积累,转化率也在98%以上。
实施例4马来酸顺反异构酶的突变改造
(1)分别以pET-24a(+)–maiA(G27A-G171A)和pET-24a(+)–maiA(G27A-K104R)为模板PCR获得突变体maiA基因片段maiA(G27A-G171A)和maiA(G27A-K104R)。
(2)参照实施例1步骤2~9构建并诱导培养重组菌;参照实施例2进行全细胞反应。
G27A-G171A在55℃的半衰期是野生型的2.9倍,酶活是野生型的1.96倍,G27A-K104R在55℃的半衰期是野生型的4.18倍,酶活是野生型的1.59倍。结果如图7,相比实施例3,pMA 2-4(G27A-G171A)完全催化转化底物马来酸只需要40min,且也几乎没有中间产物富马酸的积累,转化率也在98%以上;图8,相同条件下pMA 2-4(G27A-K104R)完全催化转化底物马来酸只需要60min,且也几乎没有中间产物富马酸的积累,转化率也在98%以上。
虽然本发明已以较佳实施例公开如上,但其并非用以限定本发明,任何熟悉此技术的人,在不脱离本发明的精神和范围内,都可做各种的改动与修饰,因此本发明的保护范围应该以权利要求书所界定的为准。
SEQUENCE LISTING
<110> 江南大学
<120> 一种双酶偶联全细胞催化马来酸合成L-天冬氨酸的方法
<160> 25
<170> PatentIn version 3.3
<210> 1
<211> 250
<212> PRT
<213> 粘质沙雷氏菌
<400> 1
Met Ser Asn His Tyr Arg Ile Gly Gln Ile Val Pro Ser Ser Asn Thr
1 5 10 15
Thr Met Glu Thr Glu Ile Pro Ala Met Leu Gly Ala Arg Gln Leu Ile
20 25 30
Arg Pro Glu Arg Phe Thr Phe His Ser Ser Arg Met Arg Met Lys His
35 40 45
Val Asn Lys Glu Glu Leu Ala Ala Met Asp Ala Glu Ser Asp Arg Cys
50 55 60
Ala Leu Glu Leu Ser Asp Ala Arg Val Asp Val Leu Gly Tyr Ala Cys
65 70 75 80
Leu Val Ala Ile Met Ala Met Gly Leu Gly Tyr His Arg Glu Ser Gln
85 90 95
Ala Arg Leu Ala Gln Val Thr Lys Asp Asn Gln Ala Ala Ala Pro Val
100 105 110
Ile Ser Ser Ala Gly Ala Leu Val Asn Gly Leu Lys Val Ile Gly Ala
115 120 125
Lys Arg Ile Ala Leu Val Ala Pro Tyr Met Lys Pro Leu Thr Gln Leu
130 135 140
Val Val Asp Tyr Ile Gln His Glu Gly Ile Glu Val Lys Val Trp Arg
145 150 155 160
Ala Leu Glu Ile Pro Asp Asn Leu Asp Val Gly Arg His Asp Pro Ala
165 170 175
Arg Leu Pro Gly Ile Val Ala Glu Met Asp Leu Arg Glu Val Asp Ala
180 185 190
Ile Val Leu Ser Ala Cys Val Gln Met Pro Ser Leu Pro Ala Val Pro
195 200 205
Thr Val Glu Ala Gln Thr Gly Lys Pro Val Ile Thr Ala Ala Ile Ala
210 215 220
Thr Thr Tyr Ala Met Leu Thr Ala Leu Glu Leu Glu Pro Ile Val Pro
225 230 235 240
Gly Ala Gly Ala Leu Leu Ser Gly Ala Tyr
245 250
<210> 2
<211> 753
<212> DNA
<213> 粘质沙雷氏菌
<400> 2
atgagcaacc actaccgcat cggccagatc gtgcccagct ccaacaccac gatggaaacc 60
gagatcccgg cgatgctggg cgcgcgccag ctgatacgcc cggagcgttt cacctttcac 120
tccagccgca tgcgcatgaa acacgtcaat aaagaagaat tggcggcgat ggacgccgag 180
tccgatcgct gcgcgctgga gctgtccgac gcgcgggtcg acgtgctcgg ctacgcctgc 240
ctggtggcca tcatggcgat ggggctgggc taccaccgcg aatcgcaggc ccggctggcg 300
caggtgacga aagacaatca ggccgccgcg ccggtcatca gcagcgccgg cgcgctggtc 360
aacggcctga aggtgatcgg cgccaaacgc atcgcgctgg tggcgcccta catgaaaccg 420
ctgacccagc tggtggtgga ctacatccag cacgaaggca tcgaggtcaa ggtatggcgc 480
gcgctggaga tcccggacaa cctcgacgtc ggccggcacg atccggccag gctgccgggg 540
atcgtcgccg agatggactt acgcgaggtc gatgctatcg tgctgtccgc ctgcgtgcag 600
atgccttcgc tgccggccgt cccgacggtg gaggcccaaa ccggcaaacc ggtgatcacc 660
gccgccatcg ccaccactta cgcgatgctg accgcgctgg agctggaacc gatcgttccc 720
ggcgccggcg ccctgctgtc cggcgcttat tga 753
<210> 3
<211> 478
<212> PRT
<213> 大肠杆菌
<400> 3
Met Ser Asn Asn Ile Arg Ile Glu Glu Asp Leu Leu Gly Thr Arg Glu
1 5 10 15
Val Pro Ala Asp Ala Tyr Tyr Gly Val His Thr Leu Arg Ala Ile Val
20 25 30
Asn Phe Tyr Ile Ser Asn Asn Lys Ile Ser Asp Ile Pro Glu Phe Val
35 40 45
Arg Gly Met Val Met Val Lys Lys Ala Ala Ala Met Ala Asn Lys Glu
50 55 60
Leu Gln Thr Ile Pro Lys Ser Val Ala Asn Ala Ile Ile Ala Ala Cys
65 70 75 80
Asp Glu Val Leu Asn Asn Gly Lys Cys Met Asp Gln Phe Pro Val Asp
85 90 95
Val Tyr Gln Gly Gly Ala Gly Thr Ser Val Asn Met Asn Thr Asn Glu
100 105 110
Val Leu Ala Asn Ile Gly Leu Glu Leu Met Gly His Gln Lys Gly Glu
115 120 125
Tyr Gln Tyr Leu Asn Pro Asn Asp His Val Asn Lys Cys Gln Ser Thr
130 135 140
Asn Asp Ala Tyr Pro Thr Gly Phe Arg Ile Ala Val Tyr Ser Ser Leu
145 150 155 160
Ile Lys Leu Val Asp Ala Ile Asn Gln Leu Arg Glu Gly Phe Glu Arg
165 170 175
Lys Ala Val Glu Phe Gln Asp Ile Leu Lys Met Gly Arg Thr Gln Leu
180 185 190
Gln Asp Ala Val Pro Met Thr Leu Gly Gln Glu Phe Arg Ala Phe Ser
195 200 205
Ile Leu Leu Lys Glu Glu Val Lys Asn Ile Gln Arg Thr Ala Glu Leu
210 215 220
Leu Leu Glu Val Asn Leu Gly Ala Thr Ala Ile Gly Thr Gly Leu Asn
225 230 235 240
Thr Pro Lys Glu Tyr Ser Pro Leu Ala Val Lys Lys Leu Ala Glu Val
245 250 255
Thr Gly Phe Pro Cys Val Pro Ala Glu Asp Leu Ile Glu Ala Thr Ser
260 265 270
Asp Cys Gly Ala Tyr Val Met Val His Gly Ala Leu Lys Arg Leu Ala
275 280 285
Val Lys Met Ser Lys Ile Cys Asn Asp Leu Arg Leu Leu Ser Ser Gly
290 295 300
Pro Arg Ala Gly Leu Asn Glu Ile Asn Leu Pro Glu Leu Gln Ala Gly
305 310 315 320
Ser Ser Ile Met Pro Ala Lys Val Asn Pro Val Val Pro Glu Val Val
325 330 335
Asn Gln Val Cys Phe Lys Val Ile Gly Asn Asp Thr Thr Val Thr Met
340 345 350
Ala Ala Glu Ala Gly Gln Leu Gln Leu Asn Val Met Glu Pro Val Ile
355 360 365
Gly Gln Ala Met Phe Glu Ser Val His Ile Leu Thr Asn Ala Cys Tyr
370 375 380
Asn Leu Leu Glu Lys Cys Ile Asn Gly Ile Thr Ala Asn Lys Glu Val
385 390 395 400
Cys Glu Gly Tyr Val Tyr Asn Ser Ile Gly Ile Val Thr Tyr Leu Asn
405 410 415
Pro Phe Ile Gly His His Asn Gly Asp Ile Val Gly Lys Ile Cys Ala
420 425 430
Glu Thr Gly Lys Ser Val Arg Glu Val Val Leu Glu Arg Gly Leu Leu
435 440 445
Thr Glu Ala Glu Leu Asp Asp Ile Phe Ser Val Gln Asn Leu Met His
450 455 460
Pro Ala Tyr Lys Ala Lys Arg Tyr Thr Asp Glu Ser Glu Gln
465 470 475
<210> 4
<211> 1437
<212> DNA
<213> 大肠杆菌
<400> 4
atgtcaaaca acattcgtat cgaagaagat ctgttgggta ccagggaagt tccagctgat 60
gcctactatg gtgttcacac tctgagagcg attgtaaact tctatatcag caacaacaaa 120
atcagtgata ttcctgaatt tgttcgcggt atggtaatgg ttaaaaaagc cgcagctatg 180
gcaaacaaag agctgcaaac cattcctaaa agtgtagcga atgccatcat tgccgcatgt 240
gatgaagtcc tgaacaacgg aaaatgcatg gatcagttcc cggtagacgt ctaccagggc 300
ggcgcaggta cttccgtaaa catgaacacc aacgaagtgc tggccaatat cggtctggaa 360
ctgatgggtc accaaaaagg tgaatatcag tacctgaacc cgaacgacca tgttaacaaa 420
tgtcagtcca ctaacgacgc ctacccgacc ggtttccgta tcgcagttta ctcttccctg 480
attaagctgg tagatgcgat taaccaactg cgtgaaggct ttgaacgtaa agctgtcgaa 540
ttccaggaca tcctgaaaat gggtcgtacc cagctgcagg acgcagtacc gatgaccctc 600
ggtcaggaat tccgcgcttt cagcatcctg ctgaaagaag aagtgaaaaa catccaacgt 660
accgctgaac tgctgctgga agttaacctt ggtgcaacag caatcggtac tggtctgaac 720
acgccgaaag agtactctcc gctggcagtg aaaaaactgg ctgaagttac tggcttccca 780
tgcgtaccgg ctgaagacct gatcgaagcg acctctgact gcggcgctta tgttatggtt 840
cacggcgcgc tgaaacgcct ggctgtgaag atgtccaaaa tctgtaacga cctgcgcttg 900
ctctcttcag gcccacgtgc cggcctgaac gagatcaacc tgccggaact gcaggcgggc 960
tcttccatca tgccagctaa agtaaacccg gttgttccgg aagtggttaa ccaggtatgc 1020
ttcaaagtca tcggtaacga caccactgtt accatggcag cagaagcagg tcagctgcag 1080
ttgaacgtta tggagccggt cattggccag gccatgttcg aatccgttca cattctgacc 1140
aacgcttgct acaacctgct ggaaaaatgc attaacggca tcactgctaa caaagaagtg 1200
tgcgaaggtt acgtttacaa ctctatcggt atcgttactt acctgaaccc gttcatcggt 1260
caccacaacg gtgacatcgt gggtaaaatc tgtgccgaaa ccggtaagag tgtacgtgaa 1320
gtcgttctgg aacgcggtct gttgactgaa gcggaacttg acgatatttt ctccgtacag 1380
aatctgatgc acccggctta caaagcaaaa cgctatactg atgaaagcga acagtaa 1437
<210> 5
<211> 32
<212> DNA
<213> 人工序列
<400> 5
tttggatccg atgagcaacc actaccgcat cg 32
<210> 6
<211> 30
<212> DNA
<213> 人工序列
<400> 6
tttaagcttt caataagcgc cggacagcag 30
<210> 7
<211> 28
<212> DNA
<213> 人工序列
<400> 7
tttcatatga gcaaccacta ccgcatcg 28
<210> 8
<211> 30
<212> DNA
<213> 人工序列
<400> 8
tttctcgagt caataagcgc cggacagcag 30
<210> 9
<211> 31
<212> DNA
<213> 人工序列
<400> 9
tttaagctta tgagcaacca ctaccgcatc g 31
<210> 10
<211> 35
<212> DNA
<213> 人工序列
<400> 10
tttggatccg atgtcaaaca acattcgtat cgaag 35
<210> 11
<211> 37
<212> DNA
<213> 人工序列
<400> 11
tttaagcttt tactgttcgc tttcatcagt atagcgt 37
<210> 12
<211> 34
<212> DNA
<213> 人工序列
<400> 12
tttcatatgt caaacaacat tcgtatcgaa gaag 34
<210> 13
<211> 37
<212> DNA
<213> 人工序列
<400> 13
tttctcgagt tactgttcgc tttcatcagt atagcgt 37
<210> 14
<211> 34
<212> DNA
<213> 人工序列
<400> 14
tttaagctta tgtcaaacaa cattcgtatc gaag 34
<210> 15
<211> 52
<212> DNA
<213> 人工序列
<400> 15
cgaaaatccc taaggagctt aagcatgggc agcagccatc accatcatca cc 52
<210> 16
<211> 71
<212> DNA
<213> 人工序列
<400> 16
gcttaagctc cttagggatt ttcgattaaa gttaaacaaa attatttcta caggggaatt 60
gttatccgct c 71
<210> 17
<211> 55
<212> DNA
<213> 人工序列
<400> 17
catcaccgtt agacgaggag gtatcctatg ggcagcagcc atcaccatca tcacc 55
<210> 18
<211> 74
<212> DNA
<213> 人工序列
<400> 18
aggatacctc ctcgtctaac ggtgatgatt aaagttaaac aaaattattt ctacagggga 60
attgttatcc gctc 74
<210> 19
<211> 51
<212> DNA
<213> 人工序列
<400> 19
aataccctac taaggaggta agcatgggca gcagccatca ccatcatcac c 51
<210> 20
<211> 70
<212> DNA
<213> 人工序列
<400> 20
gcttacctcc ttagtagggt attattaaag ttaaacaaaa ttatttctac aggggaattg 60
ttatccgctc 70
<210> 21
<211> 58
<212> DNA
<213> 人工序列
<400> 21
gaactcgaac atagtcttaa ggaggttcaa atgggcagca gccatcacca tcatcacc 58
<210> 22
<211> 77
<212> DNA
<213> 人工序列
<400> 22
ttgaacctcc ttaagactat gttcgagttc attaaagtta aacaaaatta tttctacagg 60
ggaattgtta tccgctc 77
<210> 23
<211> 27
<212> DNA
<213> 人工序列
<400> 23
catcaccgtt agacgaggag gtatcct 27
<210> 24
<211> 23
<212> DNA
<213> 人工序列
<400> 24
aataccctac taaggaggta agc 23
<210> 25
<211> 30
<212> DNA
<213> 人工序列
<400> 25
gaactcgaac atagtcttaa ggaggttcaa 30
- 还没有人留言评论。精彩留言会获得点赞!