一种调控米尔贝霉素合成的基因簇、重组链霉菌及其制备方法和应用与流程
本发明涉及生物工程技术领域,具体涉及一种调控米尔贝霉素合成的基因簇、重组链霉菌及其制备方法和应用。
背景技术
生物药物尤其是大环内酯类正在并且越来越多地应用于兽医学和农业。米尔贝霉素(milbemycin)是其中一个有代表性的对害虫具有广谱抑制同时具有较好的生态友好性的商业化农药及兽药。米尔贝霉素是一类十六元大环内酯类抗生素,化学结构与阿维菌素相似。米尔贝霉素1967年首次在日本被发现,并最先在日本登记为杀螨剂,用于农作物及经济作物等的害虫防治。之后在2006年于澳大利亚被登记用作集中花卉的二斑叶螨的杀螨剂。到现在,米尔贝霉素已在美国、中国、意大利等43个国家和地区被批准用于共24种农作物、经济作物及花卉的害虫防治。还有一些米尔贝霉素的衍生物也表现出非常好的生物活性,并进行了商业化的应用。如米尔贝肟是米尔贝霉素半合成产品,通常被用来预防恶丝虫病以及治疗钩虫和线虫等引起的猫和犬类疾病,同时还可以控制犬类的鞭虫病。此外,还有米尔贝霉素半合成药物乐平霉素(lepimectin)被登记用于蔬菜和水果的鳞翅目、半翅类等害虫的防治,另外还有研究认为乐平霉素的衍生物可以用于治疗人类的皮肤病;半合成拉替菌素(latidectin)也已商业化,用于防治动物寄生虫。
尽管米尔贝霉素系列产品有巨大的商业价值,世界各国也争相研发产米尔贝霉素菌株,但都没有成功,近半个世纪来全球现仅日本sankyo公司垄断生产米尔贝霉素原药。1999年课题组筛选出了产米尔贝霉素新菌株(cn101100651b、cn10046743c、cn100467472c、cn100490648c),被命名为冰城链霉菌。这一菌株为我国工业化生产米尔贝霉素提供了基础。除已知的米尔贝霉素类化合物外,冰城链霉菌还产生12个未报道的米尔贝霉素类化合物、环五肽、一个大环内酯类化合物及聚醚类的杀虫剂南昌霉素。
冰城链霉菌在2010年完成了全基因组测序,genebank登录号为cp002047,其基因组大小为11,936,683bp。基因组中至少有47个与聚酮类、非核糖体肽类或萜类等化合物生物合成相关的基因簇(wangxetal.genomesequenceofthemilbemycin-producingbacteriumstreptomycesbingchenggensis.jbacteriol.2010;192:4526-4527)。在使用冰城链霉菌生产米尔贝霉素的过程中,有一种水溶性黄绿色素总是与米尔贝霉素伴随合成,对米尔贝霉素的分离提纯带来困难。
技术实现要素:
本发明针对目前米尔贝霉素的分离提纯困难的问题,本发明提供了一种调控米尔贝霉素合成的基因簇,所述基因簇包括kelc基因、keld基因和kele基因;所述基因kelc,其核苷酸序列如seqidno.1所示,其编码的蛋白质氨基酸序列如seqidno.4所示;所述基因keld,其核苷酸序列如seqidno.2所示,其编码的蛋白质氨基酸序列如seqidno.5所示;所述基因kele,其核苷酸序列如seqidno.3所示,其编码的蛋白质氨基酸序列如seqidno.6所示。
本发明还提供了一种重组链霉菌,相对于产生该重组链霉菌的出发菌株,所述重组链霉菌基因组中的kelc基因、keld基因和kele基因中的任一个、任两个或全部三个基因失活:
所述基因kelc,其核苷酸序列如seqidno.1所示,其编码的蛋白质氨基酸序列如seqidno.4所示;
所述基因keld,其核苷酸序列如seqidno.2所示,其编码的蛋白质氨基酸序列如seqidno.5所示;
所述基因kele,其核苷酸序列如seqidno.3所示,其编码的蛋白质氨基酸序列如seqidno.6所示;
所述重组链霉菌的出发菌株为冰城链霉菌streptomycesbingchenggensis;
所述基因的失活是指基因核苷酸序列中的一个或多个碱基点突变、缺失、插入或重排。
“失活”包括部分失活和完全失活,是指基因功能部分或全部丧失,不能产生表达其编码的蛋白质、或蛋白质表达水平降低或消除、或表达的蛋白质的相关生物学活性降低或消失,例如基因不能被转录或者转录的rna不能被翻译为具有相应活性的蛋白质、或者产生的蛋白质的量或其活性与未进行所述失活操作的基因翻译的蛋白质的量或活性相比是降低或者消失的。
本领域技术人员应理解,已知的任何适于链霉菌中的基因失活方法均可以用于进行本发明的基因失活,例如包括但不限于基因替换、基因敲除、插入失活、移码突变、定点诱变、基因部分缺失、基因沉默、rnai、反义抑制等。对于通过上述方法失活基因可以参见本领域已知的教科书、技术手册和参考文献(例如kiesert,bibbm.practicalstreptomycesgenetics[m].norwich:thejohninnesfoundation,2000)。
所述冰城链霉菌为streptomycesbingchenggensisbcwt、streptomycesbingchenggensisbc-109-6、streptomycesbingchenggensisbc-101-4、streptomycesbingchenggensisbc-102-26、streptomycesbingchenggensisbc-103-46、streptomycesbingchenggensisbc-104-28、streptomycesbingchenggensisx-4、streptomycesbingchenggensisbc-x-1、streptomycesbingchenggensisbcj13、streptomycesbingchenggensisbcj36、streptomycesbingchenggensisbc-120-4、streptomycesbingchenggensisbcj60、streptomycesbingchenggensisbcj60/pcmf、streptomycesbingchenggensisbc04::milr中的任一种。
所述streptomycesbingchenggensisbcwt记载在高爱丽,王相晶,etal.(2007)."吸水链霉菌新种的筛选和鉴定."东北农业大学学报38(3):361-364.
所述streptomycesbingchenggensisbc-109-6,streptomycesbingchenggensisbc-101-4、streptomycesbingchenggensisbc-102-26、streptomycesbingchenggensisbc-103-46、streptomycesbingchenggensisbc-104-28记载在wang,x.j.,x.c.wang,etal.(2009)."improvementofmilbemycin-producingstreptomycesbingchenggensisbyrationalscreeningofultraviolet-andchemicallyinducedmutants."worldjournalofmicrobiologyandbiotechnology25(6):1051-1056
所述streptomycesbingchenggensisx-4,记载在zhang,b.-x.,h.zhang,etal.(2011)."newmilbemycinsfrommutantstreptomycesbingchenggensisx-4."journalofantibiotics64(11):753-756
所述streptomycesbingchenggensisbc-x-1,记载在zhang,b.,x.wang,etal.(2011)."optimizationoffermentationmediumforenhancedproductionofmilbemycinbyamutantofstreptomycesbingchenggensisbc-x-1usingresponsesurfacemethodology."africanjournalofbiotechnology10(37):7225-7235.
所述streptomycesbingchenggensisbcj13、streptomycesbingchenggensisbcj36,记载在zhang,j.,j.an,etal.(2013)."geneticengineeringofstreptomycesbingchenggensistoproducemilbemycinsa3/a4asmaincomponentsandeliminatethebiosynthesisofnanchangmycin."appliedmicrobiologyandbiotechnology:1-11
所述streptomycesbingchenggensisbc-120-4、streptomycesbingchenggensisbcj60、streptomycesbingchenggensisbcj60/pcmf,记载在wang,h.y.,j.zhang,etal.(2014)."combinedapplicationofplasmamutagenesisandgeneengineeringleadsto5-oxomilbemycinsa3/a4asmaincomponentsfromstreptomycesbingchenggensis."appliedmicrobiologyandbiotechnology98(23):9703-9712
所述streptomycesbingchenggensisbc04::milr,记载在zhang,y.,h.he,etal.(2016)."characterizationofapathway-specificactivatorofmilbemycinbiosynthesisandimprovedmilbemycinproductionbyitsoverexpressioninstreptomycesbingchenggensis."microbcellfact15(1):152
上述重组链霉菌的制备方法,包括如下步骤:
1)目的基因阻断质粒的构建:以冰城链霉菌基因组dna为模板,通过pcr扩增获得下述目的基因的同源重组的左臂片段及右臂片段,将左臂片段与右臂片段连入pkc1139载体骨架中,获得目的基因阻断质粒;
2)将目的基因阻断质粒转化大肠杆菌,通过属间接合转移转入冰城链霉菌,获得目的基因阻断突变株,即为重组链霉菌。
优选地,步骤1)、步骤2)所述的冰城链霉菌为streptomycesbingchenggensisbcwt、streptomycesbingchenggensisbc-109-6、streptomycesbingchenggensisbc-101-4、streptomycesbingchenggensisbc-102-26、streptomycesbingchenggensisbc-103-46、streptomycesbingchenggensisbc-104-28、streptomycesbingchenggensisx-4、streptomycesbingchenggensisbc-x-1、streptomycesbingchenggensisbcj13、streptomycesbingchenggensisbcj36、streptomycesbingchenggensisbc-120-4、streptomycesbingchenggensisbcj60、streptomycesbingchenggensisbcj60/pcmf、streptomycesbingchenggensisbc04::milr中的一种。
所述streptomycesbingchenggensisbcwt记载在高爱丽,王相晶,etal.(2007)."吸水链霉菌新种的筛选和鉴定."东北农业大学学报38(3):361-364.
所述streptomycesbingchenggensisbc-109-6,streptomycesbingchenggensisbc-101-4、streptomycesbingchenggensisbc-102-26、streptomycesbingchenggensisbc-103-46、streptomycesbingchenggensisbc-104-28记载在wang,x.j.,x.c.wang,etal.(2009)."improvementofmilbemycin-producingstreptomycesbingchenggensisbyrationalscreeningofultraviolet-andchemicallyinducedmutants."worldjournalofmicrobiologyandbiotechnology25(6):1051-1056
所述streptomycesbingchenggensisx-4,记载在zhang,b.-x.,h.zhang,etal.(2011)."newmilbemycinsfrommutantstreptomycesbingchenggensisx-4."journalofantibiotics64(11):753-756
所述streptomycesbingchenggensisbc-x-1,记载在zhang,b.,x.wang,etal.(2011)."optimizationoffermentationmediumforenhancedproductionofmilbemycinbyamutantofstreptomycesbingchenggensisbc-x-1usingresponsesurfacemethodology."africanjournalofbiotechnology10(37):7225-7235.
所述streptomycesbingchenggensisbcj13、streptomycesbingchenggensisbcj36,记载在zhang,j.,j.an,etal.(2013)."geneticengineeringofstreptomycesbingchenggensistoproducemilbemycinsa3/a4asmaincomponentsandeliminatethebiosynthesisofnanchangmycin."appliedmicrobiologyandbiotechnology:1-11
所述streptomycesbingchenggensisbc-120-4、streptomycesbingchenggensisbcj60、streptomycesbingchenggensisbcj60/pcmf,记载在wang,h.y.,j.zhang,etal.(2014)."combinedapplicationofplasmamutagenesisandgeneengineeringleadsto5-oxomilbemycinsa3/a4asmaincomponentsfromstreptomycesbingchenggensis."appliedmicrobiologyandbiotechnology98(23):9703-9712
所述streptomycesbingchenggensisbc04::milr,记载在zhang,y.,h.he,etal.(2016)."characterizationofapathway-specificactivatorofmilbemycinbiosynthesisandimprovedmilbemycinproductionbyitsoverexpressioninstreptomycesbingchenggensis."microbcellfact15(1):152
所述目的基因为下述基因基因的任一个、任两个或全部三个基因:
1)基因kelc,其核苷酸序列如seqidno.1所示,其编码的蛋白质氨基酸序列如seqidno.4所示;
2)基因keld,其核苷酸序列如seqidno.2所示,其编码的蛋白质氨基酸序列如seqidno.5所示;
3)基因kele,其核苷酸序列如seqidno.3所示,其编码的蛋白质氨基酸序列如seqidno.6所示。
优选地,目的基因同源重组片段扩增用的引物,为如下所述6种情况之一:
①当失活的目的基因为kelc时,kelc基因同源重组左臂片段pcr扩增所用的引物:
上游引物为:kelcdup-f:5'cccaagcttacgcgatacggcggatatgtgct3'
下游引物为:kelcdup-r:5'gctctagaactcggtgtccaggctggtggtgg3'
kelc基因同源重组右臂片段pcr扩增所用的引物:
上游引物为:kelcdown-f:5'cggaattccaccgccaacctccacgaac3'
下游引物为:kelcdown-r:5'gctctagacgggcgaacctcagataccc3'
获得kelc的左臂片段2199bp和kelc的右臂片段2090bp;
②当失活的目的基因为keld时,keld基因同源重组左臂片段pcr扩增所用的引物:
上游引物为:keldup-f:5'cccaagctttgtcccgcatatcgagaagg3'
下游引物为:keldup-r:5'gctctagaggtgttgacggcgtagaacc3'
keld基因同源重组右臂片段pcr扩增所用的引物:
上游引物为:kelddown-f:5'cggaattccggttacgccgccaccttcg3'
下游引物为:kelddown-r:5'gctctagagcctcggggtcctgctgctc3'
获得keld的左臂片段2369bp和keld的右臂片段1872bp;
③当失活的目的基因为kele时,kele基因同源重组左臂片段pcr扩增所用的引物:
上游引物为:keleup-f:5'cccaagctttgtcctgggcgagggttc3'
下游引物为:keleup-r:5'gctctagacagcgtcatccgggtcatct3'
kele基因同源重组右臂片段pcr扩增所用的引物:
上游引物为:keledown-f:5'cggaattcgctgctggccgacgacacgg3'
下游引物为:keledown-r:5'gctctagatgggcactgacgatcaggatcttctt3'
获得kele的左臂片段1906bp和kele的右臂片段2454bp;
④当失活的目的基因为kelc和keld时,kelc基因同源重组左臂片段pcr扩增所用的引物:
上游引物为:kelcdup-f:5'cccaagcttacgcgatacggcggatatgtgct3'
下游引物为:kelcdup-r:5'gctctagaactcggtgtccaggctggtggtgg3'
keld基因同源重组右臂片段pcr扩增所用的引物:
上游引物为:kelcddown-f:5'cggaattcgagggtcttgaactcgacctggtga3'
下游引物为:kelcddown-r:5'gctctagagtggactactacgtccgcctggaa3'
获得kelc的左臂片段2199bp和keld的右臂片段2443bp;
⑤当失活的目的基因为keld和kele时,keld基因同源重组左臂片段pcr扩增所用的引物:
上游引物为:keldup-f:5'cccaagctttgtcccgcatatcgagaagg3'
下游引物为:keldup-r:5'gctctagaggtgttgacggcgtagaacc3'
kele基因同源重组右臂片段pcr扩增所用的引物:
上游引物为:keledown-f:5'cggaattcgctgctggccgacgacacgg3'
下游引物为:keledown-r:5'gctctagatgggcactgacgatcaggatcttctt3'
获得keld的左臂片段2369bp和kele的右臂片段2454bp;
⑥当失活的目的基因为kelc、keld和kele时,kelc基因同源重组左臂片段pcr扩增所用的引物:
上游引物为:kelcdup-f:5'cccaagcttacgcgatacggcggatatgtgct3'
下游引物为:kelcdup-r:5'gctctagaactcggtgtccaggctggtggtgg3'
kele基因同源重组右臂片段pcr扩增所用的引物:
上游引物为:keledown-f:5'cggaattcgctgctggccgacgacacgg3'
下游引物为:keledown-r:5'gctctagatgggcactgacgatcaggatcttctt3'
获得kelc的左臂片段2199bp和kele的右臂片段2454bp。
优选地,步骤1)所述的同源重组的左臂片段经hindiii和xbai酶切后先连入到经同样酶切的中间载体puc119::neo中,获得带有目的基因左臂片段的重组中间载体pucbcupneo,然后经hindiii和ecori酶切后,回收带有左臂片段和neo基因的大片段upneo;同源重组右臂片段经xbai和ecori酶切后,与upneo连入经hindiii和xbai酶切的pkc1139中,获得目的基因阻断质粒。
优选地,步骤2)所述大肠杆菌为e.coliet12567\puz8002。
本发明所述的重组链霉菌在生产、回收、分离或纯化米尔贝霉素中的应用,是在适于米尔贝霉素产生的条件下培养本发明所述的重组链霉菌用于回收、分离和/或纯化产生的米尔贝霉素,将重组链霉菌依次经种子培养基培养、发酵培养基发酵培养后合成米尔贝霉素。
包括含有seqidno.1-3的任一核苷酸序列的基因或含有选自seqidno.4-6的任一氨基酸序列的多肽均适用于生产米尔贝霉素。
有益效果
1.本发明通过同源重组双交换确定了冰城链霉菌发酵生产米尔贝霉素时,副产物水溶性黄绿色素的生物合成基因簇。在该基因簇中基因kelc、keld、kele分别编码ii型聚酮合酶中的聚酮合酶α、β亚基和酰基载体蛋白(acp),组成了合成芳环类化合物的最小聚酮合酶。通过对水溶性黄绿色素的生物合成基因簇中结构基因kelc、keld、kele中的至少一个进行失活,废除了米尔贝霉素合成过程中杂质组分水溶性黄绿色素的合成,便利了米尔贝霉素的分离提纯,并提高了米尔贝霉素的产量。
2.采用本发明方法生产米尔贝霉素,降低了生产成本,提高了经济效益。
附图说明
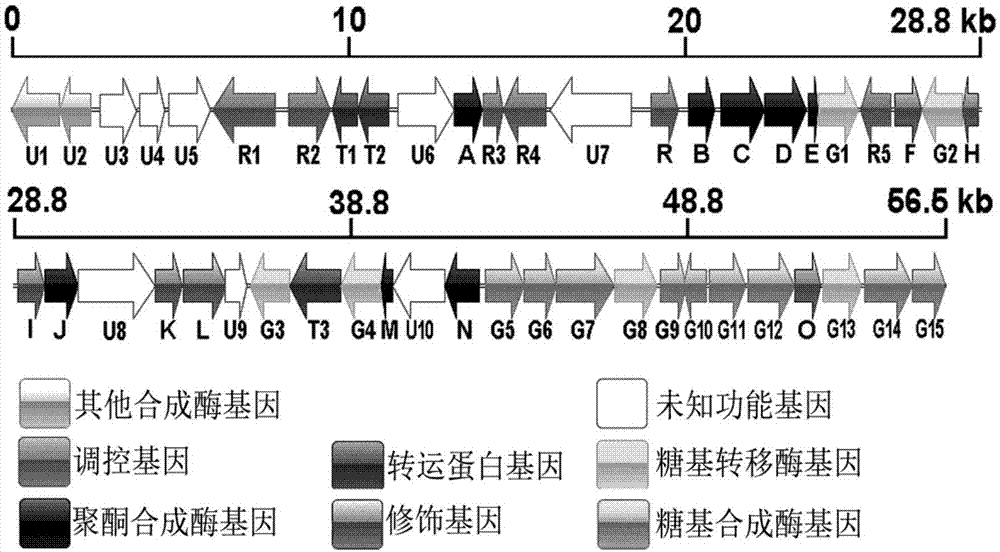
图1为水溶性黄绿色素的生物合成基因簇组成示意图,其中c为kelc,编码ii型聚酮合酶中的聚酮合酶α亚基;d为keld,编码ii型聚酮合酶中的聚酮合酶β亚基;e为kele,编码ii型聚酮合酶中的酰基载体蛋白,三者组成了合成芳环类化合物的最小聚酮合酶。
图2中a为卡那霉素抗性基因质粒puc119::neo的物理谱图;b为基因阻断用质粒pkc1139的物理谱图。
图3为kelc和keld阻断突变株基因重组的构建示意图。
具体实施方式
下面通过实施例对本发明做进一步说明,但本发明并不受其限制。下列实施例中未注明具体条件的实验方法,通常按照常规条件,或按照制造厂商所建议的条件。
实施例1.kelc、keld基因失活重组链霉菌的制备方法。
1)kelc和keld阻断质粒的pkc4445的构建。
分别利用引物kelcdup-f/kelcdup-r和kelcddown-f/kelcddown-r,以冰城链霉菌streptomycesbingchenggensisbcwt基因组为模板,pcr扩增得到kelc基因同源重组左臂片段(即,kelc的上游片段)及keld基因同源重组右臂片段(即keld的下游片段),引物序列由北京六合华大基因科技股份有限公司合成(下同),下划线代表限制性内切酶酶切位点(下同):
kelc的上游片段扩增用的引物对:
kelcdup-f:5'cccaagcttacgcgatacggcggatatgtgct3'(aagctt为hindiii酶切位点);
kelcdup-r:5'gctctagaactcggtgtccaggctggtggtgg3'(tctaga为xbai酶切位点);
keld的下游片段扩增用的引物对:
kelcddown-f:5'cggaattcgagggtcttgaactcgacctggtga3'(gaattc为ecori酶切位点);
kelcddown-r:5'gctctagagtggactactacgtccgcctggaa3'(tctaga为xbai酶切位点);
pcr反应体系:10×kod-plus缓冲液5μl,2mmdntps5μl,25mmmgso42μl,kod-plus(1u/μl)1μl(以上试剂购自toyobo,japan);10μm引物kelcdup-f和kelcdup-r各1.5μl,(或10μm引物kelcddown-f和kelcddown-r各1.5μl),基因组模板1μl(1-50ng),32μl,dmso2.5μl,加ddh2o至50μl。
pcr循环条件:预变性:94℃,2min。变性:94℃,15sec;退火:60℃,30sec。延伸:68℃,1min,30个循环;68℃,2min,4℃,保存。
扩增得到kelc的上游片段2199bp和keld的下游片段2443bp,kelc的上游片段经hindiii和xbai双酶切(限制性内切酶均购自takara,下同),keld的下游片段经xbai和ecori双酶切,利用天根琼脂糖dna回收试剂盒(tiangen,beijing)进行回收。kelc的上游片段连入用hindiii和xbai酶切的质粒puc119::neo骨架(该质粒的物理谱图如图2中a所示),得到的重组质粒为puccdupneo。用hindiii和ecori双酶切puccdupneo,回收3181bp的upneo片段,将upneo片段及keld下游片段连入经hindiii和xbai酶切的pkc1139(该质粒的物理谱图如图2中b所示),得到kelc和keld阻断质粒pkc4445。
酶切体系按照takara产品使用指南手册进行。连接体系使用t4dnaligase,购自thermofisherscientific公司,具体方法按照thermofisherscientific公司提供的t4dnaligase使用说明书进行。
2)kelc及keld基因失活重组链霉菌的获得。
将质粒pkc4445转化e.coliet12567\puz8002感受态细胞,进而通过属间接合转移转入冰城链霉菌bcwt中。筛选具有安普霉素(apramycin,apr)或卡那霉素(kanamycin,kan)抗性的接合子。将得到的接合子转接至含有卡那霉素和安普霉素的液体培养基sspy中培养,所述卡那霉素在sspy培养基中的终浓度为25ug/ml,所述安普霉素在sspy培养基中的终浓度为30ug/ml,本申请中其他涉及卡那霉素和安普霉素的培养基,抗生素浓度均按此浓度添加。经质粒提取和酶切验证的接合子挑取6个转接至含有卡那霉素的sspy平板上,28℃培养7天,制备孢子悬液,刮取成熟孢子至含玻璃珠三角瓶,5只5-15ml的斜面培养基培养的孢子用大约20ml水,250rpm,30min震荡打散孢子;4000rpm离心10min弃上清回收孢子沉淀,等体积2×yt液体培养基洗涤一次后收集孢子沉淀,再重悬浮于5ml2×yt液体培养基中;以每个平皿约104个孢子的浓度涂布在含有卡那霉素的sspy平板上,于37℃培养。
pkc1139具有温度敏感型复制子,在高于34℃条件下培养不能自主复制,重组质粒只有与冰城链霉菌bcwt染色体dna发生同源重组,才能在含有卡那霉素的ms培养基上生长。若发生双交换,则卡那霉素抗性基因正确地插入到靶基因内部,此时菌落表现为kanraprs(卡那霉素抗性,安普霉素敏感)。将表现kanraprs的菌落同时影印到sspy/kan和sspy/apr平板上,于28℃培养。随机挑选突变株,接种在没有任何选择压力的sspy培养基上,28℃培养。转接3代后重新接种到分别含有卡那霉素和安普霉素的sspy培养基上,结果仍然表现kanraprs,说明通过双交换得到了遗传稳定的kelc和keld阻断突变株,命名为δkelcd,其基因重组图谱如图3所示。
实施例2.kelc基因单独失活重组链霉菌的制备方法。
利用相同原理,参照实施例1所述的构建方法,构建单独失活黄绿色素生物合成基因簇(如图1所示)中kelc基因的重组载体,其他步骤参照步骤2),获得kelc基因单独失活的重组链霉菌。
kelc基因同源重组左臂片段pcr扩增所用的引物:
上游引物为:kelcdup-f:5'cccaagcttacgcgatacggcggatatgtgct3'
下游引物为:kelcdup-r:5'gctctagaactcggtgtccaggctggtggtgg3'
kelc基因同源重组右臂片段pcr扩增所用的引物:
上游引物为:kelcdown-f:5'cggaattccaccgccaacctccacgaac3'
下游引物为:kelcdown-r:5'gctctagacgggcgaacctcagataccc3'
获得kelc的左臂片段2199bp和kelc的右臂片段2090bp;
实施例3.keld基因单独失活重组链霉菌的制备方法。
利用相同原理,参照实施例1所述的构建方法,构建单独失活黄绿色素生物合成基因簇(如图1所示)中keld基因的重组载体,其他步骤参照步骤2),获得keld基因单独失活的重组链霉菌。
keld基因同源重组左臂片段pcr扩增所用的引物:
上游引物为:keldup-f:5'cccaagctttgtcccgcatatcgagaagg3'
下游引物为:keldup-r:5'gctctagaggtgttgacggcgtagaacc3'
keld基因同源重组右臂片段pcr扩增所用的引物:
上游引物为:kelddown-f:5'cggaattccggttacgccgccaccttcg3'
下游引物为:kelddown-r:5'gctctagagcctcggggtcctgctgctc3'
获得keld的左臂片段2369bp和keld的右臂片段1872bp;
实施例4.kele基因单独失活重组链霉菌的制备方法。
利用相同原理,参照实施例1所述的构建方法,构建单独失活黄绿色素生物合成基因簇(如图1所示)中kele基因的重组载体,其他步骤参照步骤2),获得kele基因单独失活的重组链霉菌。
kele基因同源重组左臂片段pcr扩增所用的引物:
上游引物为:keleup-f:5'cccaagctttgtcctgggcgagggttc3'
下游引物为:keleup-r:5'gctctagacagcgtcatccgggtcatct3'
kele基因同源重组右臂片段pcr扩增所用的引物:
上游引物为:keledown-f:5'cggaattcgctgctggccgacgacacgg3'
下游引物为:keledown-r:5'gctctagatgggcactgacgatcaggatcttctt3'
获得kele的左臂片段1906bp和kele的右臂片段2454bp;
实施例5.keld和kele基因失活重组链霉菌的制备方法。
利用相同原理,参照实施例1所述的构建方法,构建失活黄绿色素生物合成基因簇(如图1所示)中keld和kele基因阻断质粒,其他步骤参照步骤2),获得keld和kele基因失活的重组链霉菌。
所述keld基因同源重组左臂片段pcr扩增所用的引物:
上游引物为:keldup-f:5'cccaagctttgtcccgcatatcgagaagg3'
下游引物为:keldup-r:5'gctctagaggtgttgacggcgtagaacc3'
kele基因同源重组右臂片段pcr扩增所用的引物:
上游引物为:keledown-f:5'cggaattcgctgctggccgacgacacgg3'
下游引物为:keledown-r:5'gctctagatgggcactgacgatcaggatcttctt3'
获得keld的左臂片段2369bp和kele的右臂片段2454bp;
实施例6.kelc、keld和kele基因失活重组链霉菌的制备方法。
利用相同原理,参照实施例1所述的构建方法,构建失活黄绿色素生物合成基因簇(如图1所示)中kelc、keld和kele基因阻断质粒,其他步骤参照步骤2),获得kelc、keld和kele基因失活的重组链霉菌。
kelc基因同源重组左臂片段pcr扩增所用的引物:
上游引物为:kelcdup-f:5'cccaagcttacgcgatacggcggatatgtgct3'
下游引物为:kelcdup-r:5'gctctagaactcggtgtccaggctggtggtgg3'
kele基因同源重组右臂片段pcr扩增所用的引物:
上游引物为:keledown-f:5'cggaattcgctgctggccgacgacacgg3'
下游引物为:keledown-r:5'gctctagatgggcactgacgatcaggatcttctt3'
获得kelc的左臂片段2199bp和kele的右臂片段2454bp。
上述各实施例中所述的冰城链霉菌streptomycesbingchenggensisbcwt可替换为streptomycesbingchenggensisbc-109-6、streptomycesbingchenggensisbc-101-4、streptomycesbingchenggensisbc-102-26、streptomycesbingchenggensisbc-103-46、streptomycesbingchenggensisbc-104-28、streptomycesbingchenggensisx-4、streptomycesbingchenggensisbc-x-1、streptomycesbingchenggensisbcj13、streptomycesbingchenggensisbcj36、streptomycesbingchenggensisbc-120-4、streptomycesbingchenggensisbcj60、streptomycesbingchenggensisbcj60/pcmf、streptomycesbingchenggensisbc04::milr中的任一种,可同样获得相应的目的基因失活的重组链霉菌。
实施例7.kelc、keld基因失活重组链霉菌的制备方法。重复实施例1,与实施例1的不同在于,将质粒pkc4445转化e.coliet12567\puz8002感受态细胞,进而通过属间接合转移转入冰城链霉菌bc-120-4中。
实施例8.重组链霉菌的米尔贝霉素合成分析。
以实施例1制备的重组链霉菌为例,对其进行米尔贝霉素合成分析。
一、发酵培养采用如下培养基
种子培养基(1l):蔗糖10g,脱脂奶粉1g,蛋白胨3.5g,酵母浸粉5g,k2hpo4·3h2o0.5g,蒸馏水1l,ph7.0。121℃,1.01×105pa灭菌20分钟。
发酵培养基(1l):蔗糖80g,黄豆饼粉20g,脱脂奶粉1g,k2hpo4·3h2o1g,feso4·7h2o0.1g,caco33g,蒸馏水1l,ph7.0-7.2。121℃,1.01×105pa灭菌30分钟。
二、发酵工艺
将预先准备好的冰城链霉菌streptomycesbingchenggensisbcwt、上述构建好的敲除突变株δkelcd的孢子在ms平板分别进行划线活化,铲取约2cm2菌丝块接种到内装25ml上述种子培养基的三角瓶中,28℃,250r/min振荡培养2天后。将所得的种子菌液按5%接种量转接到发酵培养基中,28℃、250r/min振荡培养10天。
三、发酵液中米尔贝霉素的高效液相色谱分析
取0.5ml发酵料液,加入1.5ml无水乙醇,振荡15min,之后4000rpm离心15min。将上清用0.22μm滤膜过滤后滤液保存于-20℃冰箱。
hplc测定条件如下:
色谱柱:agilenttc-c18,5μm,150×4.6mm
流动相:a:mecn-h2o-meoh(350:50:100,v/v/v),b:meoh
流速:1ml/min
检测波长:242nm
进样体积:10μl
分析:0-15min,b相从0%梯度洗脱到100%。
以冰城链霉菌野生型bcwt发酵液为对照,培养发现kelcd阻断突变株水溶性黄绿色素合成终止,kelc和keld基因是所在ii型聚酮合成酶基因簇中的酮基合成酶,kelcd阻断实验证明该基因簇在冰城链霉菌中负责水溶性黄绿色素的合成。
从中挑选8株工程菌,按照实施例7的方法对其进行发酵及米尔贝霉素液相色谱检测,与出发菌株冰城链霉菌streptomycesbingchenggensisbcwt进行比较,结果表明高产米尔贝霉素基因工程菌米尔贝霉素产量平均达到1022mg/l,出发菌株冰城链霉菌streptomycesbingchenggensisbcwt的米尔贝霉素产量为800mg/l,米尔贝霉素产量提高了约27.8%。说明kelcd失活的重组工程菌能够获得比出发菌株更高的米尔贝霉素产量。
采用上述发酵方法,利用实施例2-6所构建的重组链霉菌经发酵后合成米尔贝霉素,产量统计,如下表1所示:
表1不同基因失活情况下,重组链霉素合成米尔贝霉素的产量统计。
综上所述,通过将kelc、keld、keld中至少一个基因失活,构建获得的重组链霉菌,不再生物合成米尔贝霉素发酵过程中伴随产生的水溶性黄绿色素,便利了米尔贝霉素的纯化,且米尔贝霉素产量升高。
sequencelisting
<110>中国农业科学院植物保护研究所
<120>一种调控米尔贝霉素合成的基因簇、重组链霉菌及其制备方法和应用
<130>
<160>20
<170>patentinversion3.5
<210>1
<211>1299
<212>dna
<213>基因kelc
<400>1
gtggcgatcaccggcataggcgtggtggccccgggcggcgtggggaccaaggagttctgg60
tcgctgctgaccgagggccgcaccgccacccgcgccatctccctcttcgacgccacccgc120
ttccgctcccggatcgccgccgaggccgacttcgacccgcaccacgagggcctgaccccg180
caagaggtacgccggatggaccgcgccgcgcagttcgcggtggtcgcggcccgggaggcg240
gtgtccgacagcggtctcgaggccgccggggcgcttggggccgccggggcgcttggggcc300
gccgggcccgacccgtaccgcgtgggcgtgaccgtcggcagcgcggtcggcgccaccacc360
agcctggacaccgagtaccgggtggtcagcgacggcggggccaagtggcatgtcgactac420
acatacgcggtgccgcatctcttcgaccacttcgtgcccagctccttcgccgccgaggtc480
gcctgggccgtgggcgcccagggaccggcgtccgtggtctccaccggctgcacctcgggc540
ctggacgcggtcggccacgccgtcgagctgatccgggagggcagcgcggacatcatggtc600
gcgggggccaccgacgcgcccatctccccgatcaccgtcgcctgcttcgacgccatcaag660
gccacctcgccgcgcaacgacgaccccgaacacgcctcgcgccccttcgaccgcacccgt720
aacggctttgtcctgggcgagggttccgcgctgttcgtcctcgaggagctgtccctggcg780
caccgccggggcgcccatgtctacgcggagatcgtcggcttcgcgtcccgctgcaacgcg840
taccacatgaccgggctgcgccccgacggccgggagatggccgaggccatccgcgtcgcc900
ctcgacgaggcccggctgccggtgacggccgtcgactacatcaacgcccatggctccggc960
accaagcagaacgaccgccacgagaccgccgccttcaaacgcgccctcggcgaacacgcc1020
agggccacccccatcagctcgatcaagtcgatggtcggccactcgctgggcgccatcggc1080
tccctggagatcgccgccagcgccctggccatcgagcacggtgtcgtaccgcccaccgcc1140
aacctccacgaacccgaccccgaatgcgacctcgactacaccccgctggtcgcccgcgag1200
cagcgcgtcgaggtggtgctgagcgtggggagcgggttcggcggtttccagagcgcgatg1260
gtgctggcccgtgaggggctggagaggggtacgtggtga1299
<210>2
<211>1239
<212>dna
<213>基因keld
<400>2
gtggtgagcgccgtcgccgtgttcaccggtatcggcgtcgccgcgcccaacggcctgtcg60
accccggtgtggtggaaggccaccctcaacggcgagagcggcatccgcccgatcagccgc120
ttcgacgcctcccgctacccggcccggctcgcgggcgaggtgcccggcttcgacgccgcc180
gaacacatcccgaaccgtctgctcccgcagaccgaccacatgacccggctcgccctgacc240
gccgcccaggaggcgttcgacgacgcggacaccgaccccgcccagctccccgagtacgcc300
gccggggtggtcaccgccagctcggccggcggtttcgagttcggccagcgcgagctggag360
gcgctgtggagcaagggcgggcagtacgtcagcgcgtatcagtccttcgcctggttctac420
gccgtcaacaccggccagatctccatccggcacgggctgcgcgggcccagcggggtcctc480
gtcaccgagcaggcgggcggcctcgacgcggtggcccaggcccgcagacggctgcgcggc540
ggcagtcggctcatcgtcaccggcggtgtggacggggcgatctgcccatggggctggacg600
gcgcagctcgcgggcggccggctcagcctcgccgacgaccccacacgcgcctatctgccc660
ttcgacgccgcggcccgtggctatgtgccgggcgagggcggggcgatcctgatcctggag720
gaggccgccgcggcgcgcgcccgcggcgcccgcgtgtacggcgaactggccggttacgcc780
gccaccttcgacgcgaagtccgggagcgagcagcacctcagcggcgtccccggtctgcgc840
cgcgccgtggaactcgccctcgccgacgccggcctcgccccccacgacatcgacgtcgtc900
ttcgcagacgcctccggcgtaccccacctcgaccgggccgaggcggaggccatcaccgcc960
gtcttcgggccccggcgggtgccggtcaccgcgcccaagaccatgacggggcggctgtac1020
gcgggaggggccgccctcgacctcgccgccgccctgctggcgatccgcgacgcgctgatc1080
ccgccgacggtgaacatcacccggctcgccgagggtcttgaactcgacctggtgagcacc1140
gagccgcggccctgccccgtacgcgccgccctggtgctggcccgtggccgcggcggcttc1200
aacgcggcggccgtggtcgtcggggcccggccccactga1239
<210>3
<211>285
<212>dna
<213>基因kele
<400>3
atgacccagatgacccggatgacgctggccgagctgaccaccctgctgcgcgaatgcgcc60
ggcgaggacgagaccgtcgacctcgacggagatgtgctggacacccccttcgccgacctc120
aactacgactccctcgcggtcctgcagacggtcggccgtatcgagcgcgaatacggggtg180
ctgctggccgacgacacggtcgccgaggccgccacgccccggctgctgctgcagttcgtc240
aacgcgggcctgaccgaggcgcgcacccgccctgcccaggcttag285
<210>4
<211>432
<212>prt
<213>kelc基因编码的蛋白氨基酸序列
<400>4
valalailethrglyileglyvalvalalaproglyglyvalglythr
151015
lysgluphetrpserleuleuthrgluglyargthralathrargala
202530
ileserleupheaspalathrargpheargserargilealaalaglu
354045
alaasppheaspprohishisgluglyleuthrproglngluvalarg
505560
argmetaspargalaalaglnphealavalvalalaalaarggluala
65707580
valseraspserglyleuglualaalaglyalaleuglyalaalagly
859095
alaleuglyalaalaglyproaspprotyrargvalglyvalthrval
100105110
glyseralavalglyalathrthrserleuaspthrglutyrargval
115120125
valseraspglyglyalalystrphisvalasptyrthrtyralaval
130135140
prohisleupheasphisphevalproserserphealaalagluval
145150155160
alatrpalavalglyalaglnglyproalaservalvalserthrgly
165170175
cysthrserglyleuaspalavalglyhisalavalgluleuilearg
180185190
gluglyseralaaspilemetvalalaglyalathraspalaproile
195200205
serproilethrvalalacyspheaspalailelysalathrserpro
210215220
argasnaspaspprogluhisalaserargpropheaspargthrarg
225230235240
asnglyphevalleuglygluglyseralaleuphevalleugluglu
245250255
leuserleualahisargargglyalahisvaltyralagluileval
260265270
glyphealaserargcysasnalatyrhismetthrglyleuargpro
275280285
aspglyargglumetalaglualaileargvalalaleuaspgluala
290295300
argleuprovalthralavalasptyrileasnalahisglysergly
305310315320
thrlysglnasnasparghisgluthralaalaphelysargalaleu
325330335
glygluhisalaargalathrproileserserilelyssermetval
340345350
glyhisserleuglyalaileglyserleugluilealaalaserala
355360365
leualailegluhisglyvalvalproprothralaasnleuhisglu
370375380
proaspproglucysaspleuasptyrthrproleuvalalaargglu
385390395400
glnargvalgluvalvalleuservalglyserglypheglyglyphe
405410415
glnseralametvalleualaarggluglyleugluargglythrtrp
420425430
<210>5
<211>412
<212>prt
<213>keld基因编码的蛋白氨基酸序列
<400>5
valvalseralavalalavalphethrglyileglyvalalaalapro
151015
asnglyleuserthrprovaltrptrplysalathrleuasnglyglu
202530
serglyileargproileserargpheaspalaserargtyrproala
354045
argleualaglygluvalproglypheaspalaalagluhisilepro
505560
asnargleuleuproglnthrasphismetthrargleualaleuthr
65707580
alaalaglnglualapheaspaspalaaspthraspproalaglnleu
859095
proglutyralaalaglyvalvalthralaserseralaglyglyphe
100105110
glupheglyglnarggluleuglualaleutrpserlysglyglygln
115120125
tyrvalseralatyrglnserphealatrpphetyralavalasnthr
130135140
glyglnileserilearghisglyleuargglyproserglyvalleu
145150155160
valthrgluglnalaglyglyleuaspalavalalaglnalaargarg
165170175
argleuargglyglyserargleuilevalthrglyglyvalaspgly
180185190
alailecysprotrpglytrpthralaglnleualaglyglyargleu
195200205
serleualaaspaspprothrargalatyrleupropheaspalaala
210215220
alaargglytyrvalproglygluglyglyalaileleuileleuglu
225230235240
glualaalaalaalaargalaargglyalaargvaltyrglygluleu
245250255
alaglytyralaalathrpheaspalalysserglysergluglnhis
260265270
leuserglyvalproglyleuargargalavalgluleualaleuala
275280285
aspalaglyleualaprohisaspileaspvalvalphealaaspala
290295300
serglyvalprohisleuaspargalaglualaglualailethrala
305310315320
valpheglyproargargvalprovalthralaprolysthrmetthr
325330335
glyargleutyralaglyglyalaalaleuaspleualaalaalaleu
340345350
leualaileargaspalaleuileproprothrvalasnilethrarg
355360365
leualagluglyleugluleuaspleuvalserthrgluproargpro
370375380
cysprovalargalaalaleuvalleualaargglyargglyglyphe
385390395400
asnalaalaalavalvalvalglyalaargprohis
405410
<210>6
<211>94
<212>prt
<213>kele基因编码的蛋白氨基酸序列
<400>6
metthrglnmetthrargmetthrleualagluleuthrthrleuleu
151015
argglucysalaglygluaspgluthrvalaspleuaspglyaspval
202530
leuaspthrprophealaaspleuasntyraspserleualavalleu
354045
glnthrvalglyargilegluargglutyrglyvalleuleualaasp
505560
aspthrvalalaglualaalathrproargleuleuleuglnpheval
65707580
asnalaglyleuthrglualaargthrargproalaglnala
8590
<210>7
<211>32
<212>dna
<213>kelcdup-f
<400>7
cccaagcttacgcgatacggcggatatgtgct32
<210>8
<211>32
<212>dna
<213>kelcdup-r
<400>8
gctctagaactcggtgtccaggctggtggtgg32
<210>9
<211>28
<212>dna
<213>kelcdown-f
<400>9
cggaattccaccgccaacctccacgaac28
<210>10
<211>28
<212>dna
<213>kelcdown-r
<400>10
gctctagacgggcgaacctcagataccc28
<210>11
<211>29
<212>dna
<213>keldup-f
<400>11
cccaagctttgtcccgcatatcgagaagg29
<210>12
<211>28
<212>dna
<213>keldup-r
<400>12
gctctagaggtgttgacggcgtagaacc28
<210>13
<211>28
<212>dna
<213>kelddown-f
<400>13
cggaattccggttacgccgccaccttcg28
<210>14
<211>28
<212>dna
<213>kelddown-r
<400>14
gctctagagcctcggggtcctgctgctc28
<210>15
<211>27
<212>dna
<213>keleup-f
<400>15
cccaagctttgtcctgggcgagggttc27
<210>16
<211>28
<212>dna
<213>keleup-r
<400>16
gctctagacagcgtcatccgggtcatct28
<210>17
<211>28
<212>dna
<213>keledown-f
<400>17
cggaattcgctgctggccgacgacacgg28
<210>18
<211>34
<212>dna
<213>keledown-r
<400>18
gctctagatgggcactgacgatcaggatcttctt34
<210>19
<211>33
<212>dna
<213>kelcddown-f
<400>19
cggaattcgagggtcttgaactcgacctggtga33
<210>20
<211>32
<212>dna
<213>kelcddown-r
<400>20
gctctagagtggactactacgtccgcctggaa32
- 还没有人留言评论。精彩留言会获得点赞!