以Nicastrin为靶点的药物筛选方法及其应用与流程
本发明属于生物信息学领域,计算生物学范畴,特别涉及一种计算机药物筛选方法及其应用。
技术背景
随着近年来功能性基因组学和结构生物学的飞速发展,越来越多的基因信息被发掘,越来愈多的蛋白结构被解析,传统的非基于受体结构的纯实验性新药研发模式被动摇。2012年的诺贝尔化学奖授予了为解析GPCR晶体结构做出卓越贡献的科学家Lefkowitz(杜克大学)和Kobilka(斯坦福大学)。之后2013年诺贝尔化学奖授予了计算模拟界的三位著名科学家马汀·卡普拉斯(Martin Karplus)、迈克尔·莱维特(Michael Levitt)和亚利耶·瓦谢尔(Arieh Warshel)。
在现代药物发现中使用虚拟筛选发现新的先导化合物将越来越成为一种既高效有精准的手段。基于生物信息的计算药物筛选平台可以有效的节约时间和费用。
类药化合物数目在1062个,即使现行组合化学及高通量筛选能每天筛选数万个化合物,它们仅占类药化合物比例的有效空间,随机试错式筛选有如大海捞针,效率低下且成本极高。随机药物筛选必须与计算机辅助技术有机结合才能发挥其应用的作用。计算机辅助药物设计已发展有数十年历史,近十几年来取得较大的实质性进步,已在许多药物的研究中去得成功。如HIV蛋白酶抑制剂Indinavir的设计(已上市)、HIV蛋白酶环脲类抑制剂的设计(曾进入一期临床)、唾液酸酶抑制剂的设计(以上市)、老年痴呆症药物E2020(donepezil)的开发等等。CADD技术在国外高校及大型制药企业已成为药物设计的常规手段,如加州理工学院、加州大学洛杉矶分校及美国匹兹堡大学、Merck公司、Pfizer公司等等。随着人类基因组学的不断深化发展,大量的与疾病相关的基因被陆续发现,这使得药物靶标分子的数量急剧增加,尤其是大量受体晶体结构的解析及最近几年冷冻电镜技术的发展。这使得人们对药物机理作用的认识、对化合物源的数目及多样性的要求会逐步降低,命中率逐步提高。
阿尔兹海默症是一类神经退行性疾病,临床表现为脑组织切片中出现淀粉样斑块,神经元逐渐死亡,认知和记忆能力受损,大脑功能逐渐丧失,病人逐渐丧失独立生活能力,最后脑功能严重受损直至死亡。美国前总统里根和英国前首相撒切尔夫人都罹患该疾病。据不完全统计,我国目前大约有500万阿尔兹海默症患者,占世界发病总数的四分之一。由于缺乏特效药物,该疾病不但给病人及家属造成极大痛苦,也同时带来沉重的社会负担。
阿尔兹海默病的发生和大脑中淀粉样斑块的形成密切相关。研究证明:淀粉样斑块是由膜整合蛋白酶复合物γ-secretase异常切割淀粉样前体蛋白APP(amyloid precursor protein)而产生过量易聚集的Aβ42肽段所致。γ-secretase是由四个膜整合蛋白组成的包含19次跨膜螺旋的复合体,包括早老素Presenilin(PS1),Aph-1,Pen-2和Nicastrin四个亚基,其中Presenilin是执行酶活功能的膜整合蛋 白酶(intramembrane protease)活性亚基。目前已经在Presenilin上鉴定出一百多个与阿尔兹海默症有关联的氨基酸突变。2014年施一公团队解析了部分γ-secretase的三维结构。其中Nicastrin亚基已被证明作为靶点接收天然配体或者药物。
目前药品专利的保护期限为20年,如果药物上市前研究与开发(R&D)花费的时间为10年,那么药品的有效市场销售时间就仅有10年。如果R&D时间能缩短2~3年,那么不但能节约R&D的经费,而且能为市场赢得宝贵的时间,这将产生巨大的经济效益和社会效益。因此,CADD方法已被国外许多制药公司用于新药的研究与开发,并且近年来已取得了极大的成功。所以,CADD方法与应用的研究不但具有深远的科学意义,而且具有巨大的应用价值。研究者发现通过CADD可有效减少新药研发过程中33%的费用和30%的时间成本,其中虚拟筛选的介入,使新药研发的平均周期缩短了0.9年,直接研发费用平均降低了1.3亿美元。
技术实现要素:
本发明目的在于开发一种以分子对接和虚拟筛选技术为基础的高精准计算机药物筛选方法,能够适用于基于结构的药物设计。
本发明的首要目的是通过虚拟筛选方式得到gama-secretase中的Nicastrin的先导药物。该先导化合物有对Nicastrin有抑制活性的潜力。
本发明第二个目的是通过Discovery Studio精确地选中靶点Nicastrin的绑定口袋。以供分子对接和虚拟筛选使用。
本发明的第三个目的是使用生物信息学分析工具对蛋白的一维及二维信息进行基本分析。
本发明第四个目的是通过同源建模工具对已知序列进行三维建模,供药物设计使用。
本发明第五个目的是通过Autodock进行以Nicastrin为靶点以Zinc数据库为基准数据库的虚拟筛选。
本发明还针对先导化合物与受体残基空间构象及静电作用力的研究,优化先导化合物,并进行IC50测试,以验证虚拟筛选的准确度。
为实现上述发明目标,本发明采用如下技术方案:
一种以Nicastrin为靶点的药物筛选方法,其特征在于:以生物信息学分析、同源建模、分子对接虚拟筛选技术为基础进行计算机药物筛选,所述以Nicastrin为靶点的药物筛选方法包括以下步骤:
确定Nicastrin在Uniprot中的蛋白序列;
Nicastrin蛋白序列的生物信息学一级结构分析;
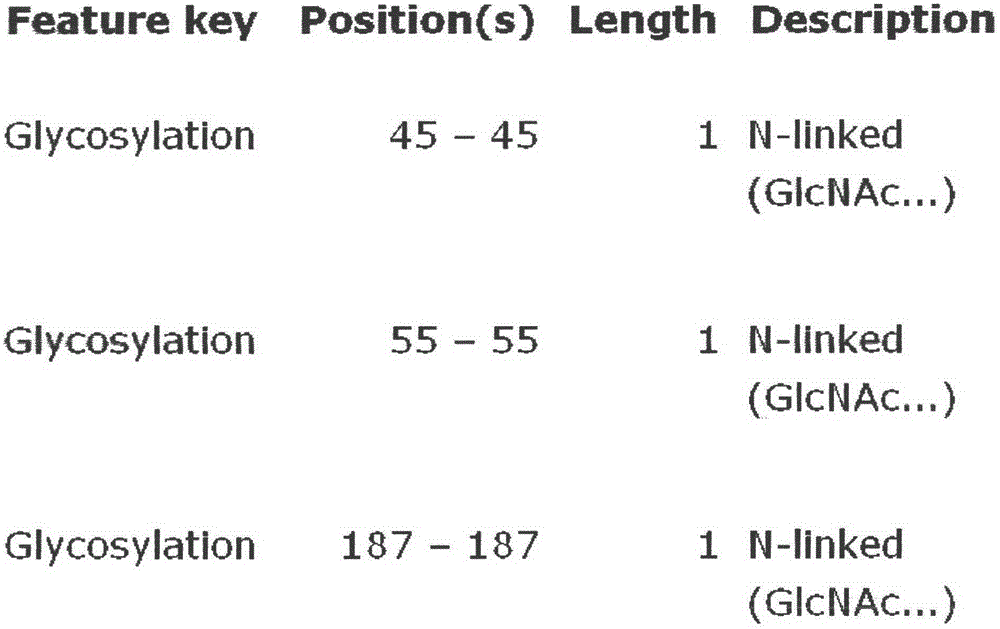
通过Uniprot查询Nicastrin的翻译后修饰发现:45、55、187、200、204、264、387、417、435、464、506、530、562、573、580、612位残基都有N-糖基化报导,1-33位序列是信号肽。在Nicastrin的亚细胞定位部分发现该靶点有一次跨膜,其中34-669处于细胞膜外,而691-709处于细胞膜内,中间肽段670-690横跨磷脂双分子层。因此正常的小分子结合口袋不会处于670-690内。与此同时结合TMHMM工 具分析靶点的跨膜分布发现670-690为跨膜区;
Nicastrin蛋白序列二级结构分析;
采用Lasegene软件对靶点蛋白进行二级结构及相关物理性质的预测。首先打开Lasegene中的Editseq模块,新建Protein窗口并将靶点序列粘帖至该窗口中,并保存文件类型为EditSeq Protein Sequence File类型。然后将该文件用Lasegene中的Protean模块打开,通过Protean预测该靶点的二级结构、柔性区域、表面倾向性等因素。同时结合Functional Sites in Protein工具预测蛋白的功能位点及球状和混乱区域;
Nicastrin同源建模:使用Swiss-modeling进行同源建模。首先打开Swiss-modeling工具网站并点击Modelling,将靶点序列黏贴至序列窗口并点击Built Model,Swiss-modeling工具将搜寻同源模板将输入序列进行同源膜建,待程序大约跑15分钟后查看建模结果。我们选择4upc(分辨率5.4埃)作为模板所建模型GMQE为0.71.我们发现该靶点33-41及666-709区间三维结构缺失,因此我们通过Modelle程序进行片段模建。我们将所建造的3D模型用DS进行能量最小化优化,然后将模板与序列建构进行叠合后发现两者RMSD为1埃,同源建模模型可信,swiss-modeling工具对同源建模有较高的准确度。E333,Y337,R281,S297and T333;
采用Discovery Studio程序,为Nicastrin确定绑定口袋;
采用Glide软件对Nicastrin抑制剂小分子数据库进行分子对接及虚拟筛选;
小分子抑制剂IC50活性测试。
本发明还采用如下技术方案:以Nicastrin为靶点的药物筛选方法在以γ-分泌酶为靶点的先导化合物计算机筛选的应用。
本发明还采用如下技术方案:以Nicastrin为靶点的药物筛选方法在无晶体结构GPCR蛋白但与其他GPCR晶体结构同源相似率达75%以上的受体抑制剂筛选的应用。
本发明还采用如下技术方案:以Nicastrin为靶点的药物筛选方法在分子对接软件Autodock平台的应用。
本发明还采用如下技术方案:以Nicastrin为靶点的药物筛选方法在Autodock-vina平台的应用。
本发明还采用如下技术方案:以Nicastrin为靶点的药物筛选方法在swiss-model同源建模平台上的应用。
本发明还采用如下技术方案:以Nicastrin为靶点的药物筛选方法在PGB蛋白抑制剂计算机筛选的应用。
本发明还采用如下技术方案:以Nicastrin为靶点的药物筛选方法在Zinc数据库的应用
附图说明
图1是本发明所述基于生物信息学的计算机药物筛选方法中的靶点Nicastrin一级结构分析及转录后 修饰情况。其中1-33序列属于信号肽区间。
图2是本发明所述基于生物信息学的计算机药物筛选方法中的靶点Nicastrin二级结构分析及亚细胞定位分析。34-669位氨基酸处于细胞膜外,670-690区间处于跨膜区,691-709区间位于细胞膜内。
图3是本发明所述基于生物信息学的计算机药物筛选方法中的靶点Nicastrin的二级结构分析,基于隐马尔夫模型算法的蛋白拓扑结构预测。
图4是基于Protein软件的蛋白质二级结构及亲水性等因素预测。
图5是本发明所述基于生物信息学的计算机药物筛选方法中的靶点Nicastrin功能位点的预测分析。其中90-270区间基本处于Disorder(混乱区域),而270-600区间处于球状区域。
图6是本发明所述基于生物信息学的计算机药物筛选方法中同源建模的参数设置及分析;其中所选用模板为4UIS,序列一致度达91.15,分辨率4.40埃。GMQE值0.71.
图7是本发明所述基于生物信息学的计算机药物筛选方法中配体作用口袋关键氨基酸残基的分析。关键氨基酸主要是E333,Y337,R281,S297and T333.
图8是本发明所述基于生物信息学的计算机药物筛选方法中配体作用口袋的设定,定义口袋半径为5埃。
图9是通过虚拟筛选得到的小分子先导化合物母核。
图10小分子化合物对细胞中Nicastrin的抑制曲线。
图11是本发明的基于生物信息学的计算机药物筛选方法的流程图。
具体实施方式
具体来说,本发明提供一种通过计算模拟的方法来定义受体绑定口袋并进行基于结构的虚拟药物筛选的方法。步骤如下:
(1)确定Nicastrin在Uniprot中的蛋白序列。
(2)Nicastrin蛋白序列的生物信息学一级结构分析。
(3)Nicastrin蛋白序列二级结构分析。
(4)Nicastrin同源建模。
(5)采用Discovery Studio程序,为Nicastrin确定绑定口袋。
(6)采用Autodock软件对Nicastrin抑制剂小分子数据库进行虚拟筛选。
(7)小分子抑制剂活性测试。
下面我们针对本发明进行具体描述:
确定Nicastrin在Uniprot中的蛋白序列。
将Uniprot ID Q92542输入Uniprot网站。得到Nicastrin的相关注释信息,并得到Nicastrin序列:MATAGGGSGADPGSRGLLRLLSFCVLLAGLCRGNSVERKIYIPLNKTAPCVRLLNATHQIGCQSSISGDTGVIHVVEKEEDLQWVLTDGPN PPYMVLLESKHFTRDLMEKLKGRTSRIAGLAVSLTKPSPASGFSPSVQCPNDGFGVYSNSYGPEFAHCREIQWNSLGNGLAYEDFSFPIFLLEDENETKVIKQCYQDHNLSQNGSAPTFPLCAMQLFSHMHAVISTATCMRRSSIQSTFSINPEIVCDPLSDYNVWSMLKPINTTGTLKPDDRVVVAATRLDSRSFFWNVAPGAESAVASFVTQLAAAEALQKAPDVTTLPRNVMFVFFQGETFDYIGSSRMVYDMEKGKFPVQLENVDSFVELGQVALRTSLELWMHTDPVSQKNESVRNQVEDLLATLEKSGAGVPAVILRRPNQSQPLPPSSLQRFLRARNISGVVLADHSGAFHNKYYQSIYDTAENINVSYPEWLSPEEDLNFVTDTAKALADVATVLGRALYELAGGTNFSDTVQADPQTVTRLLYGFLIKANNSWFQSILRQDLRSYLGDGPLQHYIAVSSPTNTTYVVQYALANLTGTVVNLTREQCQDPSKVPSENKDLYEYSWVQGPLHSNETDRLPRCVRSTARLARALSPAFELSQWSSTEYSTWTESRWKDIRARIFLIASKELELITLTVGFGILIFSLIVTYCINAKADVLFIAPREPGAVSY。
Nicastrin蛋白序列的生物信息学一级结构分析。
通过Uniprot查询Nicastrin的翻译后修饰发现:45、55、187、200、204、264、387、417、435、464、506、530、562、573、580、612位残基都有N-糖基化报导,1-33位序列是信号肽。在Nicastrin的亚细胞定位部分发现该靶点有一次跨膜,其中34-669处于细胞膜外,而691-709处于细胞膜内,中间肽段670-690横跨磷脂双分子层。因此正常的小分子结合口袋不会处于670-690内。与此同时结合TMHMM工具分析靶点的跨膜分布发现670-690为跨膜区。
Nicastrin蛋白序列二级结构分析。
采用Lasegene软件对靶点蛋白进行二级结构及相关物理性质的预测。首先打开Lasegene中的Editseq模块,新建Protein窗口并将靶点序列粘帖至该窗口中,并保存文件类型为EditSeq Protein Sequence File类型。然后将该文件用Lasegene中的Protean模块打开,通过Protean预测该靶点的二级结构、柔性区域、表面倾向性等因素。同时结合Functional Sites in Protein工具预测蛋白的功能位点及球状和混乱区域。
Nicastrin同源建模。
使用Swiss-modeling进行同源建模。首先打开Swiss-modeling工具网站并点击Modelling,将靶点序列黏贴至序列窗口并点击Built Model,Swiss-modeling工具将搜寻同源模板将输入序列进行同源膜建,待程序大约跑15分钟后查看建模结果。我们选择4upc(分辨率5.4埃)作为模板所建模型GMQE为0.71.我们发现该靶点33-41及666-709区间三维结构缺失,因此我们通过Modelle程序进行片段模建。我们将所建造的3D模型用DS进行能量最小化优化,然后将模板与序列建构进行叠合后发现两者RMSD为1埃,同源建模模型可信,swiss-modeling工具对同源建模有较高的准确度。E333,Y337,R281,S297and T333.
采用Discovery Studio程序,为Nicastrin确定绑定口袋。
载入同源建模后的受体PDB结构,使PDB结构以卡通图模式展示。选定E333,Y337,R281,S297残基,定义绑定口袋区域,定义口袋半径为5埃。
采用Autodock软件对Nicastrin抑制剂小分子数据库进行虚拟筛选。将同源建模后的晶体结构用Autodock Tools进行处理,去掉冗余分子,按Autodock Vina运行规则进行分子优化处理,加电荷,并将 受体结构转化为pdbqt格式。所有的化合物结构从ZINC数据库下载,用PyRx处理和转换成pdbqt格式.建立一个处理后的化合物数据库.
根据已经确定的活性位点.其它条件设为默认.选择Lamarckian genetical gorithm(LGA)计算方法,用PyRx运行AutodockVina,首轮筛选从大约4万个化合物库(ZINC数据库)中进行高通量筛选,得到800个类药小分子化合物的库.再从这800个小分子化合物中筛选针对Nicastrin的抑制剂.通过对建立的化合物数据库进行4轮筛选,筛选出活性最高的1个化合物,对这个化合物做进一步分析.
小分子抑制剂的细胞实验活性分析。
细胞接种在96孔板上,使用0.2%DMSO或DAPT(浓度为2.6μM-150μM)处理48小时。使用MTT染料减少检测实验,稍微修正,测定细胞毒性。与DAPT温育后,25μL MTT溶液(5mg/mL,溶于PBS)加到含200μL培养基的每孔中,实验板在37℃下温育6小时,随后每孔加入200μL DMSO,在室温下震荡20分钟混合。通过酶联免疫吸附法在490nm处测定吸光值。使用α-MEM及等量的MTT溶液和溶剂,作为空白对照。使用PROBIT程序在SPSS中计算IC50值。
本发明还包括:以Nicastrin为靶点的药物筛选方法在以γ-分泌酶为靶点的先导化合物计算机筛选的应用。
本发明还包括:以Nicastrin为靶点的药物筛选方法在无晶体结构GPCR蛋白但与其他GPCR晶体结构同源相似率达75%以上的受体抑制剂筛选的应用。
本发明还包括:以Nicastrin为靶点的药物筛选方法在分子对接软件Autodock平台的应用。
本发明还包括:以Nicastrin为靶点的药物筛选方法在Autodock-vina平台的应用。
本发明还包括:以Nicastrin为靶点的药物筛选方法在swiss-model同源建模平台上的应用。
本发明还包括:以Nicastrin为靶点的药物筛选方法在PGB蛋白抑制剂计算机筛选的应用。
本发明还包括:以Nicastrin为靶点的药物筛选方法在Zinc数据库的应用。
本发明还包括:小分子化合物在抑制γ-secretase酶活性的应用。
- 还没有人留言评论。精彩留言会获得点赞!