使植物的生物质增加的新基因及对其的利用的制作方法
本发明涉及使植物的生物质增加的新基因,更详细而言,涉及导入了该基因的植物、使用该基因使植物的生物质增加的方法、以及使用该基因制备增加了生物质的植物的方法等。
背景技术:
生物质通常被理解为以物质的量(质量)的形式表示特定时刻某个空间中存在的生物(bio)的量的概念。生物质在日语中有时使用“生物体量”或“生物量”这样的用语,在生态学中也被称为“现存量(standingcrop)”。生物质的数值化通常以质量或能量来进行,也以每单位面积的相应生物的干重来表示。在多数情况下植物在生物质方面被利用,增加植物的生物质不仅提供生物燃料、可再生能源,而且从使农作物的产量增高的观点考虑对稳定食物供给也是有用的。
关于植物生物质的增加,为了开发在工业上有用的植物新品种,以往应用了使植物彼此杂交并挑选后代的杂交育种法、对植物诱发突变的突变育种法等。另外,近年来也开发了将有用基因导入植物并使其功能表达的基因重组植物。现状是,虽然在这样的新品种的开发中,累积能够赋予优异性质的基因的方法是有效的,但仍希望进一步提高作物的生产性,可利用的基因种类仍然少,特别是希望能够对与高产性状等生物质的增加相关的基因进行鉴定。
在促进植物的繁殖中,多数情况下植物激素会造成影响,已知其中之一为油菜素类固醇。油菜素类固醇是一组具有甾体骨架的化合物,代表性的油菜素类固醇有油菜素内酯。关于植物的繁殖,油菜素类固醇具有以下作用:(i)促进茎、叶及根的伸长生长、(ii)促进细胞分裂、(iii)促进从叶肉细胞向导管或管胞的分化、(iv)促进乙烯合成、(v)促进种子的发芽、以及(vi)赋予对环境胁迫的耐性等。尝试进行了利用这样的油菜素类固醇的生理活性,将油菜素类固醇生物合成基因导入稻中,使其产量增加(非专利文献1)。但是,该效果并不一定足够。另外,虽然鉴定了若干与油菜素类固醇的合成、信息传递相关的基因,但尚未对充分实现植物的生物质增加的基因进行鉴定。
现有技术文献
非专利文献
非专利文献1:chuan-yinwuetal.theplantcell,2008,20(8):2130-2145
发明的内容
发明要解决的课题
如上所述,对使植物的生物质增加的基因、特别是使种子产量提高的基因的探索尚不充分。因此,强烈地要求发现有效增加植物的生物质的新基因,并开发利用该基因的技术,例如,开发利用该基因使生物质增加的植物体等。
本发明是鉴于上述问题而完成的,其目的在于鉴定有效增加植物的生物质的新基因,同时提供该基因及利用该基因的技术。
解决课题的方法
本发明人等将在作为油菜素类固醇生物合成抑制剂的芸苔素唑(brassinazole)(brz)的存在下、且在暗处发芽的胚轴形态作为选择基准,尝试进行了拟南芥(arabidopsisthaliana)突变体的筛选及相关基因的分离。具体而言,使用基于fox追寻系统(huntingsystem)(全长cdna过表达基因追寻系统)的方法进行突变体的筛选,研究了相关基因。其结果是发现了芸苔素唑耐性极强的突变体,在进一步进行基因分析时发现bil7(brz-insensitive-long-hypocotyl7)基因与该突变相关。
本发明人等制备了表达bil7基因的构建体,并将其导入植物,制备了过表达该基因的转化体。然后,在对该转化体的形态进行研究时发现,与野生体相比,在花序长度等各个方面促进了繁殖。基于这些见解,本发明人等完成了本发明。
本发明优选通过以下所述的方式进行,但并不限定于此。
[方式1]
一种导入了编码蛋白质的核酸的植物,其中,
该蛋白质包含:
序列号1所示的氨基酸序列、以及
与序列号7或11所示的氨基酸序列具有25%以上的同一性或75%以上的相似性的氨基酸序列,且
该蛋白质具有使植物的生物质增加的活性。
[方式2]
根据方式1的植物,其中,序列号1所示的氨基酸序列为序列号2所示的氨基酸序列。
[方式3]
根据方式1或2的植物,其中,蛋白质包含序列号3~5中任一项所示的氨基酸序列。
[方式4]
根据方式1~3中的任一种植物,其中,核酸为源自单子叶植物或双子叶植物的核酸,植物为单子叶植物。
[方式5]
一种使植物的生物质增加的方法,该方法包括向植物导入编码蛋白质的核酸的工序,其中,
该蛋白质包含:
序列号1所示的氨基酸序列、以及
与序列号7或11所示的氨基酸序列具有25%以上的同一性或75%以上的相似性的氨基酸序列,且
该蛋白质具有使植物的生物质增加的活性。
[方式6]
根据方式5的方法,其中,序列号1所示的氨基酸序列为序列号2所示的氨基酸序列。
[方式7]
根据方式5或6的方法,其中,蛋白质包含序列号3~5中任一项所示的氨基酸序列。
[方式8]
根据方式5~7中的任一方法,其中,核酸为源自单子叶植物或双子叶植物的核酸,植物为单子叶植物。
[方式9]
一种增加了生物质的植物的制备方法,该方法包括向植物导入编码蛋白质的核酸的工序,其中,
该蛋白质包含:
序列号1所示的氨基酸序列、以及
与序列号7或11所示的氨基酸序列具有25%以上的同一性或75%以上的相似性的氨基酸序列,且
该蛋白质具有使植物的生物质增加的活性。
[方式10]
根据方式9的方法,其中,序列号1所示的氨基酸序列为序列号2所示的氨基酸序列。
[方式11]
根据方式9或10的方法,其中,蛋白质包含序列号3~5中任一项所示的氨基酸序列。
[方式12]
根据方式9~11中的任一方法,其中,核酸为源自单子叶植物或双子叶植物的核酸,植物为单子叶植物。
[方式13]
一种包含编码蛋白质的核酸及启动子的构建体,其中,
该蛋白质包含:
序列号1所示的氨基酸序列、以及
与序列号7或11所示的氨基酸序列具有25%以上的同一性或75%以上的相似性的氨基酸序列,且
该蛋白质具有使植物的生物质增加的活性。
[方式14]
根据方式13的构建体,其中,序列号1所示的氨基酸序列为序列号2所示的氨基酸序列。
[方式15]
根据方式13或14的构建体,其中,蛋白质包含序列号3~5中任一项所示的氨基酸序列。
[方式16]
一种载体,其包含方式13~15中的任一构建体。
[方式17]
一种宿主细胞,其包含方式16的载体。
[方式18]
一种植物,其导入了方式17的载体。
[方式19]
根据方式18的植物,其中,核酸为源自单子叶植物或双子叶植物的核酸,植物为单子叶植物。
[方式20]
一种增加了生物质的植物的筛选方法,该方法包括:
(1)在受试植物及野生型植物中,对蛋白质或编码该蛋白质的核酸的表达量进行测定的工序,该蛋白质包含:
序列号1所示的氨基酸序列、以及
与序列号7或11所示的氨基酸序列具有25%以上的同一性或75%以上的相似性的氨基酸序列,且
该蛋白质具有使植物的生物质增加的活性,
(2)对由工序(1)得到的表达量进行比较的工序,以及
(3)选择比野生型植物中的表达量显示出更高表达量的受试植物的工序。
[方式21]
根据方式20的方法,其中,序列号1所示的氨基酸序列为序列号2所示的氨基酸序列。
[方式22]
根据方式20或21的方法,其中,蛋白质包含序列号3~5中任一项所示的氨基酸序列。
[方式23]
根据方式20~22中的任一方法,其中,核酸为源自单子叶植物或双子叶植物的核酸,植物为单子叶植物。
[方式24]
一种对增加了生物质的植物进行判定的方法,该方法包括:
(1)在受试植物及野生型植物中,对蛋白质或编码该蛋白质的核酸的表达量进行测定的工序,该蛋白质包含:
序列号1所示的氨基酸序列、以及
与序列号7或11所示的氨基酸序列具有25%以上的同一性或75%以上的相似性的氨基酸序列,且
该蛋白质具有使植物的生物质增加的活性,
(2)对由工序(1)得到的表达量进行比较的工序,以及
(3)确认受试植物中的表达量高于野生型植物中的表达量的工序。
[方式25]
根据方式24的方法,其中,序列号1所示的氨基酸序列为序列号2所示的氨基酸序列。
[方式26]
根据方式24或25的方法,其中,蛋白质包含序列号3~5中任一项所示的氨基酸序列。
[方式27]
根据方式24~26中的任一方法,其中,核酸为源自单子叶植物或双子叶植物的核酸,植物为单子叶植物。
发明的效果
根据本发明,能够有效地使植物的生物质增加。另外,通过使用本发明,可以提供有效地增加了生物质的植物及其制备方法,还可以提供对于增加了生物质的植物的筛选有用的方法、以及使植物的生物质增加的物质的筛选方法。
附图说明
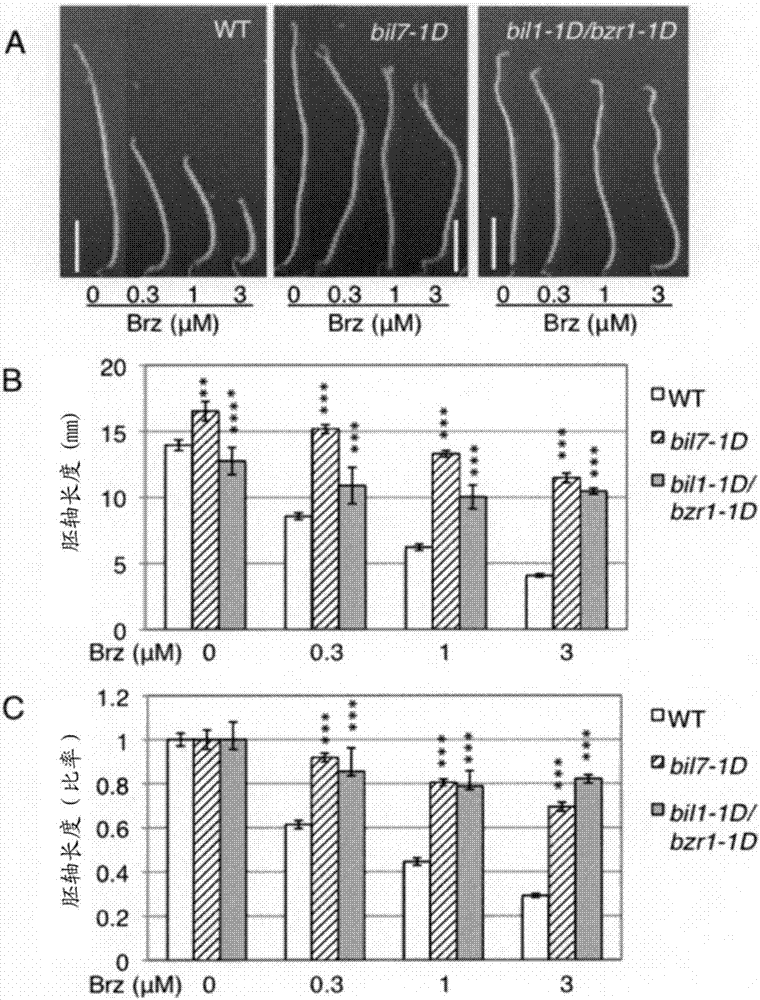
图1是示出在暗条件下bil7-1d显示出芸苔素唑(brz)耐性的图。根据图1可知,bil7-1d在暗处发芽中显示出brz耐性。a表示暗处第7天的brz0、0.3、1、3μm条件下的胚轴伸长的情况。b表示暗处第7天的brz0、0.3、1、3μm条件下的胚轴的伸长长度。c表示暗处第7天的brz0、0.3、1、3μm条件下的胚轴的伸长率。比例尺=3mm、误差棒=s.d.、n=30、***:p<0.001、相对于brz0μm的studentt检验。
图2是示出bil7-1d的形态学特征的图。根据图2可知,bil7-1d显示出促进繁殖的形态。a表示第60天的植物的情况(比例尺=10cm)。b表示第40天的植物体的莲座叶(rosetteleaf)的形态(比例尺=1cm)。c表示第70天的植物体的花葶长度(n=12)。d表示第70天的植物体的花葶数(n=12)。e表示第70天的植物体的二次花葶数(n=12)。误差棒=s.d.、***:p<0.001、相对于wt的studentt检验。
图3是示出bil7-1d的花瓣、长角果(silique)及种子的形态学特征的图。根据图3可知,bil7-1d的种子重量增加。a表示bil7-1d的正常开花(左)和异常开花(右)(比例尺=1mm)。b表示bil7-1d的正常开花的长角果(左)和异常开花的长角果(右)(比例尺=5mm)。c表示野生型的种子(左)和bil7-1d的种子(右)(比例尺=0.5mm)。d表示第70天的植物体的花数(n=12)。e表示第70天的植物体的正常长角果数(n=12)。f表示每1个长角果的种子数(n=12)。g表示100粒种子的重量(n=5)。误差棒=s.d.、*:p<0.1、**:p<0.01、***:p<0.001、相对于wt的studentt检验。
图4是示出bil7-1d的花葶伸长期间及开花期间的图。a表示开花期的莲座叶的片数(n=5)。b表示至开花所需的天数(n=5)。c表示花葶长度的经时变化及开花期间(n=24)。误差棒=s.d.、*:p<0.1、***:p<0.001、相对于wt的studentt检验。
图5是示出暗处第7天的bil7-1d中的bil7候选基因的表达量的图。根据图5可知,能够确认在bil7-1d中bil7候选基因高表达。
图6是示出暗条件下的bil7高表达转化体(bil7-ox)及bil7表达抑制转化体(bil7-rnai)的brz耐性及bil7表达量的图。根据图6可知,bil7表达量越高的品系在暗处brz条件下的发芽中越显示出胚轴伸长。a表示暗处第7天的brz3μm条件下的胚轴(比例尺=3mm)。b表示暗处第7天的brz3μm条件下的胚轴长度(误差棒=s.d.、n=50、***:p<0.001、相对于wt的studentt检验)。c表示第27天的植物体的各品系的bil7表达量(误差棒=s.d.)。wt:野生型、bil7-ox1、2:35s::bil7过表达者(overexpressor)1、2、bil7-rnai:bil7-rnai抑制者(suppressor)。
图7是示出bil7-ox及bil7-rnai的形态学特征的图。根据图7可知,bil7表达量越高的品系在成熟时越显示出花序伸长形态。a表示第63天的植物的情况(比例尺=10cm)。b表示第86天的植物体的花序长度(n=10、误差棒=s.d.、*:p<0.1、***:p<0.001、相对于wt的studentt检验)。wt:野生型、bil7-ox1、2:35s::bil7过表达者1、2、bil7-rnai:bil7-rnai抑制者。
图8是示出bil7-ox及bil7-rnai的形态学特征的图。根据图8可知,bil7-ox显示出二次花序数及种子重量增加的倾向。a表示第72天的植物体的莲座叶的形态(比例尺=5cm)。b表示各品系的种子(比例尺=1mm)。c表示第86天的植物体的花序数(n=10)。d表示第86天的植物体的二次花序长度(n=10)。e表示第86天的植物体的正常长角果数(n=10)。f表示100粒种子的重量(n=5)。误差棒=s.d.、*:p<0.1、**:p<0.01、***:p<0.001、相对于wt的studentt检验、wt:野生型、bil7-ox1、2:35s::bil7过表达者1、2、bil7-rnai:bil7-rnai抑制者。
图9是示出osbil7高表达转化体(osbil7-ox)中的生物质增加的图。a表示t1的结果。根据a可知,在使稻中的bil7同源基因osbil7高表达化的稻-osbil7-ox株(t1)中,能够确认每1株植物体的总重量、分蘖数、稻谷数、结实种子数(150%)的增加。b表示t2的结果。根据b可知,在使稻中的bil7同源基因osbil7高表达化的稻-osbil7-ox株(t2)中,能够确认每1株的分蘖数、稻谷数、结实种子数(140%)的增加。
图10是示出osbil7稻转化体移植到盆中17天后的植物姿态的图。在稻(品种:yukihikari)中,可以观察到转化后的osbil7重组体(左)比对照用载体的重组体(右)略微生长茂盛的情况。
图11是示出osbil7稻转化体的成熟期的植物姿态的图。在稻(品种:yukihikari)中,可以观察到转化后的osbil7重组体(左)比对照用载体的重组体(右)生长茂盛的情况。
图12是示出bil7稻转化体移植到盆中17天后的植物姿态的图。在稻(品种:yukihikari)中,可以观察到转化后的bil7重组体(左)比对照用载体的重组体(右)生长茂盛的情况。
图13是示出bil7稻转化体的成熟期的植物姿态的图。在稻(品种:yukihikari)中,可以观察到转化后的bil7重组体(右)比对照用载体的重组体(左)生长茂盛的情况。
具体实施方式
(1)核酸及蛋白质
在本发明中,使用bil7(brz不敏感性长胚轴7,brz-insensitive-long-hypocotyl7)基因作为有助于植物生物质的增加的核酸。bil7基因是与油菜素类固醇信号转导相关的基因,其是从在作为油菜素类固醇生物合成抑制剂的芸苔素唑(brz)的存在下也能够观察到胚轴徒长的突变体中发现的,能够在各种植物中见到。bil7基因的核苷酸序列及该基因所编码的蛋白质(即,bil7蛋白质)的氨基酸序列随各种植物而有所不同,但本发明的蛋白质具有下述序列号1所示的氨基酸序列的共同基序。
序列号1:
ala-pro-pro-ser-ser-pro-ala-ser-x1-x2-x3-ser-x4-x5-x6-ser-x7-x8-x9-x10-pro-x11-gly-pro-tyr-ala-x12-glu-x13-x14-x15-val-x16-pro-pro-val-phe-ser-x17-x18-x19-thr-x20-pro-ser-x21-ala-pro-x22-thr-pro-pro-x23-pro-ser-ser-pro-x24-val-pro-x25-ala-x26-pro-x27-ser-pro-x28-ser-pro
(式中,x1表示phe或tyr,x2表示phe、leu、thr或ala,x3表示gln、pro、his或asn,x4表示glu、gly、asp、ala或met,x5表示pro、gly、leu或ala,x6表示pro、ala、thr或ser,x7表示ala、ile、val、ser、或thr,x8表示thr、val、ser或ala,x9表示gln或his,x10表示ser或thr,x11表示15~30个氨基酸残基,x12表示his或asn,x13表示thr或pro,x14表示gln或ala,x15表示leu或pro,x16表示ser或thr,x17表示thr或ala,x18表示tyr或phe,x19表示thr、ile或pro,x20表示glu或ala,x21表示ser或thr,x22表示ile、val、tyr或phe,x23表示3~15个氨基酸残基,x24表示glu或asp,x25表示phe或tyr,x26表示20~50个氨基酸残基,x27表示gly、glu或asp,x28表示5或6个氨基酸残基。)
在上述氨基酸序列中,x11优选为17~29个氨基酸残基,更优选为20~26个氨基酸残基,x23优选为3~12个氨基酸残基,更优选为8~12个氨基酸残基,x26优选为23~49个氨基酸残基,更优选为23~30个氨基酸残基,x28优选为5个氨基酸残基。
作为具有包含上述共同基序的bil7蛋白质的植物,没有特别限定,可以列举例如:拟南芥(arabidopsisthaliana)、大豆(glycinemax)、稻(oryzasativa)、玉米(zeamays)、萝卜(raphanussativus)、杨(populustrichocarpa)、葡萄(vitisvinifera)、小立碗藓(physcomitrellapatens)等。各植物的编码bil7蛋白质的mrna(及cdna)的核苷酸序列及bil7蛋白质的氨基酸序列已经鉴定,如下表所述注册了genbank收录号。
表1
另外,在具有上述共同基序的bil7蛋白质中包含各植物的bil7蛋白质的同源物(homolog)。例如,在拟南芥bil7中存在3种同源物蛋白质,对于其mrna(及cdna)的核苷酸序列及氨基酸序列,如下表所述注册了genbank收录号。
表2
另外,本发明的蛋白质的特征在于,除了包含上述共同基序以外,还包含以拟南芥或稻的bil7蛋白质作为基准、与序列号7或11所示的氨基酸序列具有25%以上的同一性或75%以上的相似性的氨基酸序列。在本发明中,上述共同基序(即,序列号1所示的氨基酸序列)包含在与序列号7或11所示的氨基酸序列具有25%以上的同一性或75%以上的相似性的氨基酸序列中(换言之,上述共同基序(即,序列号1所示的氨基酸序列)作为与序列号7或11所示的氨基酸序列具有25%以上的同一性或75%以上的相似性的氨基酸序列的一部分而包含(存在))。
在本说明书中,氨基酸序列的同一性是指成为对象的2个蛋白质之间的氨基酸序列的同一性,其通过氨基酸残基的比例(%)来表示,所述氨基酸残基的比例是在使用该技术领域中公知的数学算法而形成的氨基酸序列的最优排列中一致的氨基酸残基的比例。氨基酸序列的同一性可以通过目测及数学计算来确定,可以使用本领域技术人员公知的同源性检索程序(例如,blast、fasta)、序列排列程序(例如,clustalw))或遗传信息处理软件(例如,genetyx[注册商标])等来计算。具体而言,本说明书中的氨基酸序列的同一性可以使用ddbj(dnadatabankofjapan)的网站上公开的系统分析程序clustalw(http://clustalw.ddbj.nig.ac.jp/index.php?lang=ja)在初始设定的条件(version2.1、alignmenttype:slow、dnaweightmatrix:gonnet、gapopen:10、gapextension:0.1)下求出。
本发明的蛋白质可以包含与序列号7或11所示的氨基酸序列具有25%以上、30%以上、40%以上、50%以上、60%以上、70%以上、80%以上、85%以上、90%以上、95%以上、97%以上、98%以上或99%以上的同一性的氨基酸序列。
拟南芥的bil7蛋白质与其同源物之间的同一性分别为41%(同源物1)、43%(同源物2)及28%(同源物3)。因此,作为优选的一个方式,本发明的蛋白质包含与序列号7所示的氨基酸序列具有25%以上、30%以上或40%以上的同一性的氨基酸序列。拟南芥的bil7蛋白质与大豆、稻及玉米的bil7蛋白质之间的同一性分别为44%(大豆bil7)、41%(稻bil7)及40%(玉米bil7)。因此,作为优选的一个方式,本发明的蛋白质包含与序列号7所示的氨基酸序列具有40%以上的同一性的氨基酸序列。稻的bil7蛋白质与大豆及玉米的bil7蛋白质之间的同一性分别为42%(大豆bil7)及85%(玉米bil7)。因此,作为优选的一个方式,本发明的蛋白质包含与序列号11所示的氨基酸序列具有40%以上、50%以上、60%以上、70%以上、80%以上或85%以上的同一性的氨基酸序列。
本说明书中的氨基酸序列的相似性是指成为对象的2个蛋白质之间的氨基酸序列的相似性,其通过在使用该技术领域中公知的数学算法而形成的氨基酸序列的最优排列中一致的氨基酸残基及表示相似性的氨基酸残基的比例(%)来表示。氨基酸残基的相似性可以通过物理化学性质相互类似的氨基酸残基的关系来表示,例如,在芳香族氨基酸(phe、tyr、trp)、疏水性氨基酸(asp、glu)、脂肪族氨基酸(ala、leu、ile、val)、极性氨基酸(asn、gln)、碱性氨基酸(lys、arg、his)、酸性氨基酸(asp、glu)、含羟基氨基酸(ser、thr)、支链小的氨基酸(gly、ala、ser、thr、met)等的组中,属于同一组的氨基酸之间可以被认为是相互类似的氨基酸残基。可以预测,显示出这样的相似性的氨基酸残基不对蛋白质的表型造成影响。氨基酸序列的相似性可以与同一性同样地通过目测及数学计算来确定,可以使用本领域技术人员公知的序列相似性检索程序(例如,blast、psi-blast、hmmer)、遗传信息处理软件(例如,genetyx[注册商标])等来计算。具体而言,本说明书中的氨基酸序列的相似性可以使用genetyx[注册商标]网络版ver.11.1.3(genetyx公司)在初始设定的条件(将unitsizetocompare设定为2)下求出蛋白质与蛋白质全局同源性。
本发明的蛋白质可以包含与序列号7或11所示的氨基酸序列具有75%以上、80%以上、85%以上、90%以上、91%以上、92%以上、93%以上、94%以上、95%以上、96%以上、97%以上、98%以上或99%以上的相似性的氨基酸序列。
拟南芥的bil7蛋白质与大豆、稻及玉米的bil7蛋白质之间的相似性分别为76%(大豆bil7)、76%(稻bil7)及75%(玉米bil7)。因此,作为优选的一个方式,本发明的蛋白质包含与序列号7所示的氨基酸序列具有75%以上的相似性的氨基酸序列。稻的bil7蛋白质与大豆及玉米的bil7蛋白质之间的相似性分别为83%(大豆bil7)及97%(玉米bil7)。因此,作为优选的一个方式,本发明的蛋白质包含与序列号11所示的氨基酸序列具有75%以上、80%以上、85%以上、90%以上、95%以上、97%以上的相似性的氨基酸序列。
本发明的蛋白质的特征在于,包含上述共同基序,且包含与序列号7或11所示的氨基酸序列具有给定的同一性或相似性的氨基酸序列,并且还具有使植物的生物质增加的活性。
在本说明书中,植物的生物质是指植物个体的整体、一部分、个别器官或其组合的量。作为植物个体的整体、一部分或个别器官,可以列举例如:整体、地上部分、根、茎、叶、果实、种子、胚、胚珠、子房、苗端(shootapex)、花药、花粉或穗等。其中,优选为果实、种子、穗、根、茎、叶或花药。另外,作为量,可以列举例如:数量、大小、长度、宽度、重量、面积或体积等。因此,作为生物质的例子,可以列举:整体重量、地上部分重量(例如,地上部分干重)、产量、茎径、茎数、秆(culm)长、株高、叶面积、叶数、旗叶长度、叶片长度、叶片宽度、稻谷数、稻谷重量(例如,1000粒稻谷重量、全部稻谷重量)、种子数、分蘖数、穗数、单穗粒数、单穗结实粒数、结实率、穗长、最大穗长、单穗重或总穗重等,但并不限定于此。另外,“增加”可以是上述例示出的植物生物质中的任一者单独增加,或是多者组合增加。
对于使植物的生物质增加的活性而言,例如,可以使用对照(亲本植株、非转化体、野生型等)的生物质作为增加的指标,通过在植物的成熟体中与该对照比较来进行评价。在通过与对照的比较能够观察到增加的情况下,可以判定具有使植物的生物质增加的活性,在能够将该生物质数值化时,在比较数值的基础上,例如在能够观察到1%以上、3%以上、5%以上、10%以上、15%以上、20%以上、25%以上、30%以上、40%以上、50%以上、60%以上或70%以上的增加的情况下,可以判定具有使植物的生物质增加的活性。
在本发明中,序列号1所示的氨基酸序列可以为下述序列号2所示的氨基酸序列。作为具有包含序列号2所示的氨基酸序列作为共同基序的蛋白质的植物,可以列举例如:拟南芥、大豆、稻、玉米等。另外,这些植物的bil7蛋白质的同源物(例如,拟南芥bil7的同源物蛋白质)也包含于具有序列号2所示的氨基酸序列的蛋白质中。
序列号2:
ala-pro-pro-ser-ser-pro-ala-ser-phe-x29-x30-ser-x31-x32-x33-ser-x34-x35-x36-x37-pro-x38-ser-x39-x40-x41-x42-gly-pro-tyr-ala-x43-glu-thr-gln-x44-val-x45-pro-pro-val-phe-ser-x46-x47-x48-thr-glu-pro-ser-x49-ala-pro-x50-thr-pro-pro-x51-pro-ser-ser-pro-x52-val-pro-x53-ala-x54-pro-x55-ser-pro-x56-leu-x57-ser-pro
(式中,x29表示phe、leu或thr,x30表示gln、his、pro或asn,x31表示glu、gly、asp或ala,x32表示pro、gly或leu,x33表示pro、ala、thr或ser,x34表示ala、val、ile或thr,x35表示thr、ala、val或ser,x36表示gln或his,x37表示ser或thr,x38表示10~25个氨基酸残基,x39表示ile、val、ala或met,x40表示phe或tyr,x41表示ala或thr,x42表示ile、val或thr,x43表示his或asn,x44表示leu或pro,x45表示ser或thr,x46表示thr或ala,x47表示tyr或phe,x48表示thr或ile,x49表示ser或thr,x50表示ile、phe或tyr,x51表示3~15个氨基酸残基,x52表示glu或asp,x53表示phe或tyr,x54表示20~35个氨基酸残基,x55表示gly、glu或asp,x56表示3~5个氨基酸残基,x57表示ile或arg。)
在上述氨基酸序列中,x38优选为12~21个氨基酸残基,更优选为15~21个氨基酸残基,x51优选为3~12个氨基酸残基,更优选为8~12个氨基酸残基,x54优选为23~35个氨基酸残基,更优选为23~30个氨基酸残基,x56优选为3或4个氨基酸残基,更优选为3个氨基酸残基。
另外,本发明的蛋白质可以包含下述序列号3所示的氨基酸序列。序列号3所示的氨基酸序列中包含上述序列号1或2所示的氨基酸序列(换言之,序列号1或2所示的氨基酸序列作为序列号3所示的氨基酸序列的一部分而包含(存在))。另外,与序列号7或11所示的氨基酸序列具有25%以上的同一性或75%以上的相似性的氨基酸序列中包含序列号3所示的氨基酸序列(换言之,序列号3所示的氨基酸序列作为与序列号7或11所示的氨基酸序列具有25%以上的同一性或75%以上的相似性的氨基酸序列的一部分而包含(存在))。作为包含序列号3所示的氨基酸序列作为共同基序的蛋白质的植物,可以列举例如:拟南芥、稻等。
序列号3:
met-x58-ser-gly-x59-asn-x60-x61-asp-thr-x62-asn-ala-ala-ala-x63-ala-ile-x64-x65-x66-x67-x68-arg-x69-arg-lys-trp-x70-x71-x72-x73-ser-x74-x75-x76-cys-phe-gly-ser-x77-x78-x79-x80-x81-arg-ile-x82-x83-x84-val-leu-val-pro-glu-pro-x85-pro-phe-x86-ala-pro-pro-ser-ser-pro-ala-ser-phe-x87-gln-ser-x88-x89-x90-ser-x91-x92-gln-ser-pro-val-gly-x93-x94-ser-phe-ser-pro-leu-x95-x96-asn-x97-pro-ser-ile-phe-ala-ile-gly-pro-tyr-ala-his-glu-thr-gln-leu-val-ser-pro-pro-val-phe-ser-x98-x99-thr-thr-glu-pro-ser-x100-ala-pro-x101-thr-pro-pro-x102-ser-x103-x104-leu-thr-thr-x105-pro-ser-ser-pro-glu-val-pro-x106-ala-x107-leu-x108-x109-ser-x110-glu-x111-gln-x112-tyr-gln-x113-x114-pro-x115-ser-pro-x116-gly-x117-leu-ile-ser-pro-ser-x118-ser-gly-x119-x120-ser-pro-phe-pro-asp-x121-ser-x122-phe-pro-x123-phe-x124-val-x125-x126-pro-pro-lys-x127-leu-x128-gly-x129-his-x130-val-ser-phe-x131-leu-x132-x133-x134-x135-val-x136-arg-cys-x137-x138-x139-lys-x140-pro-x141-x142-ser-x143-asp-x144-ser-leu-x145-x146-x147-lys-glu-phe-x148-phe-x149-val-x150-x151-x152-x153-x154-ala-x155-x156-lys-x157-trp-ser-phe-phe-pro-val-x158-gln-x159-gly
(式中,x58表示arg或gln,x59表示3~10个氨基酸残基,x60表示val或ser,x61表示phe或val,x62表示ile或val,x63表示ser或val,x64表示ala或val,x65表示ser或thr,x66表示ser或ala,x67表示asp或glu,x68表示asp或ser,x69表示5~10个氨基酸残基,x70表示trp或ala,x71表示asn或asp,x72表示arg或trp,x73表示trp或leu,x74表示leu或val,x75表示leu或tyr,x76表示lys或phe,x77表示ser或gln,x78表示arg或lys,x79表示gln或asn,x80表示arg或gly,x81表示lys或arg,x82表示gly或ser,x83表示asn或his,x84表示ser或ala,x85表示20~25个氨基酸残基,x86表示ile或val,x87表示phe或leu,x88表示glu或gly,x89表示pro或gly,x90表示pro或ala,x91表示ala或ile,x92表示thr或val,x93表示ile或ala,x94表示leu或pro,x95表示pro或ser,x96表示cys或pro,x97表示1~10个氨基酸残基,x98表示thr或ala,x99表示tyr或phe,x100表示ser或thr,x101表示ile或phe,x102表示1~5个氨基酸残基,x103表示ile或val,x104表示tyr或his,x105表示0~5个氨基酸残基,x106表示phe或tyr,x107表示gln或lys,x108表示phe或leu,x109表示asn或thr,x110表示10~20个氨基酸残基,x111表示phe或leu,x112表示phe或ser,x113表示leu或ile,x114表示pro或tyr,x115表示gly或glu,x116表示leu或ile,x117表示gln或arg,x118表示1~5个氨基酸残基,x119表示pro或thr,x120表示thr或cys,x121表示1~10个氨基酸残基,x122表示leu或thr,x123表示his或ser,x124表示gln或pro,x125表示ser或arg,x126表示asp或glu,x127表示leu或ile,x128表示3~20个氨基酸残基,x129表示10~30个氨基酸残基,x130表示1~5个氨基酸残基,x131表示asp或glu,x132表示asp或thr,x133表示ala或val,x134表示asp或glu,x135表示his或asp,x136表示ile或ala,x137表示val或leu,x138表示asp或glu,x139表示gln或lys,x140表示3~25个氨基酸残基,x141表示glu或arg,x142表示ala或glu,x143表示ser或asn,x144表示5~25个氨基酸残基,x145表示gly或arg,x146表示ser或lys,x147表示asn或ala,x148表示asn或lys,x149表示5~15个氨基酸残基,x150表示asp或gly,x151表示glu或ser,x152表示his或asp,x153表示arg或trp,x154表示ser或trp,x155表示ser或asn,x156表示pro或glu,x157表示5~15个氨基酸残基,x158表示met或ala,x159表示ser或pro。)
在上述氨基酸序列中,x59优选为4~7个氨基酸残基,x69优选为8~10个氨基酸残基,x85优选为21~22个氨基酸残基,x97优选为2~7个氨基酸残基,x102优选为2~4个氨基酸残基,x105优选为0~2个氨基酸残基,x110优选为10~17个氨基酸残基,x118优选为2~3个氨基酸残基,x121优选为3~8个氨基酸残基,x128优选为5~18个氨基酸残基,x129优选为13~28个氨基酸残基,x130优选为1~2个氨基酸残基,x140优选为5~24个氨基酸残基,x144优选为7~22个氨基酸残基,x149优选为8~11个氨基酸残基,x157优选为4~11个氨基酸残基。
另外,本发明的蛋白质可以包含下述序列号4所示的氨基酸序列。序列号4所示的氨基酸序列包含上述序列号1或2所示的氨基酸序列(换言之,序列号1或2所示的氨基酸序列作为序列号4所示的氨基酸序列的一部分而包含(存在))。另外,与序列号7或11所示的氨基酸序列具有75%以上的同一性或75%以上的相似性的氨基酸序列中包含序列号4所示的氨基酸序列(换言之,序列号4所示的氨基酸序列作为与序列号7或11所示的氨基酸序列具有25%以上的同一性或75%以上的相似性的氨基酸序列的一部分而包含(存在))。作为包含序列号4所示的氨基酸序列作为共同基序的蛋白质的植物,可以列举例如:拟南芥、大豆等。
序列号4:
met-arg-x160-gly-ala-asn-gly-x161-asn-asn-x162-x163-x164-thr-ile-asn-ala-ala-ala-x165-x166-ile-ala-ser-x167-x168-x169-arg-leu-x170-gln-x171-x172-pro-x173-x174-x175-lys-x176-x177-trp-x178-asn-x179-x180-ser-x181-x182-x183-cys-phe-gly-x184-x185-x186-x187-arg-x188-arg-ile-gly-x189-x190-val-leu-val-pro-glu-x191-x192-x193-x194-x195-x196-x197-asn-x198-thr-x199-ile-x200-x201-x202-x203-phe-x204-ala-pro-pro-ser-ser-pro-ala-ser-phe-x205-x206-ser-glu-pro-pro-ser-x207-x208-gln-ser-pro-x209-x210-ile-leu-ser-x211-x212-pro-x213-ser-ile-phe-ala-ile-gly-pro-tyr-ala-his-glu-thr-gln-leu-val-ser-pro-pro-val-phe-ser-thr-x214-thr-thr-glu-pro-ser-x215-ala-pro-x216-thr-pro-pro-x217-thr-thr-pro-ser-ser-pro-glu-val-pro-phe-ala-gln-leu-x218-x219-x220-asn-x221-x222-x223-x224-x225-x226-x227-x228-x229-phe-x230-tyr-x231-phe-x232-x233-tyr-gln-leu-x234-pro-gly-ser-pro-x235-gly-gln-leu-ile-ser-pro-x236-ser-x237-x238-x239-ser-pro-phe-pro-asp-x240-ser-leu-x241-x242-x243-phe-gln-x244-x245-asp-x246-ser-x247-x248-x249-x250-gly-x251-x252-thr-pro-x253-gln-x254-x255-x256-x257-pro-x258-x259-x260-val-ser-x261-x262-x263-x264-ala-x265-x266-val-x267-x268-cys-val-x269-lys-leu-x270-thr-x271-x272-pro-x273-glu-x274-x275-ser-asp-x276-glu-x277-x278-x279-his-x280-lys-glu-phe-asn-phe-x281-x282-x283-glu-x284-leu-x285-x286-asp-x287-ala-ser-x288-ser-asn-x289-trp-ser-phe-phe-pro-x290-x291-x292-x293-gly
(式中,x160表示0~3个氨基酸残基,x161表示0~5个氨基酸残基,x162表示val或thr,x163表示phe或leu,x164表示asp或glu,x165表示ser或thr,x166表示ala或val,x167表示ser或val,x168表示asp或glu,x169表示asp或asn,x170表示his或asp,x171表示ser或pro,x172表示ser或his,x173表示ile或his,x174表示his或val,x175表示lys或gln,x176表示arg或lys,x177表示lys或ser,x178表示trp或gly,x179表示arg或trp,x180表示trp或leu,x181表示leu或ile,x182表示leu或tyr,x183表示lys或trp,x184表示ser或his,x185表示ser或arg,x186表示arg或lys,x187表示gln或asn,x188表示lys或gln,x189表示asn或his,x190表示ser或ala,x191表示pro或arg,x192表示val或ile,x193表示ser或pro,x194表示met或ser,x195表示ser或gly,x196表示ser或thr,x197表示ser或asp,x198表示ser或ala,x199表示5~15个氨基酸残基,x200表示thr或ile,x201表示thr或pro,x202表示leu或phe,x203表示pro或his,x204表示ile或val,x205表示phe或leu,x206表示gln或his,x207表示ala或val,x208表示thr或ala,x209表示val或ser,x210表示gly或ala,x211表示phe或leu,x212表示ser或thr,x213表示1~10个氨基酸残基,x214表示tyr或phe,x215表示ser或thr,x216表示ile或phe,x217表示3~15个氨基酸残基,x218表示phe或leu,x219表示asn或asp,x220表示ser或pro,x221表示his或asn,x222表示gln或lys,x223表示thr或asn,x224表示gly或ser,x225表示ser或glu,x226表示tyr或thr,x227表示gly或tyr,x228表示tyr或gln,x229表示lys或arg,x230表示3~7个氨基酸残基,x231表示glu或asp,x232表示gln或his,x233表示phe或ser,x234表示pro或his,x235表示leu或val,x236表示1~10个氨基酸残基,x237表示gly或ser,x238表示pro或thr,x239表示thr或ser,x240表示1~10个氨基酸残基,x241表示phe或leu,x242表示pro或leu,x243表示his或asn,x244表示val或thr,x245表示ser或asp,x246表示3~10个氨基酸残基,x247表示pro或his,x248表示lys或gln,x249表示thr或gly,x250表示ala或ser,x251表示val或ser,x252表示thr或leu,x253表示1~10个氨基酸残基,x254表示lys或ala,x255表示ile或ser,x256表示val或phe,x257表示pro或leu,x258表示his或ser,x259表示lys或his,x260表示pro或trp,x261表示phe或ile,x262表示asp或glu,x263表示leu或val,x264表示asp或ser,x265表示asp或gln,x266表示his或glu,x267表示ile或phe,x268表示arg或asn,x269表示1~15个氨基酸残基,x270表示arg或lys,x271表示thr或asp,x272表示phe或ala,x273表示0~15个氨基酸残基,x274表示ala或thr,x275表示ser或pro,x276表示1~15个氨基酸残基,x277表示ser或arg,x278表示met或val,x279表示asn或his,x280表示5~10个氨基酸残基,x281表示gly或asp,x282表示thr或asn,x283表示asp或ala,x284表示1~10个氨基酸残基,x285表示thr或val,x286表示val或ala,x287表示1~10个氨基酸残基,x288表示1~7个氨基酸残基,x289表示asp或asn,x290表示val或met,x291表示met或ile,x292表示gln或arg,x293表示ser或pro。)
在上述氨基酸序列中,x160优选为0~1个氨基酸残基,x161优选为0~3个氨基酸残基,x199优选为7~9个氨基酸残基,x213优选为4~7个氨基酸残基,x217优选为6~10个氨基酸残基,x230优选为4~5个氨基酸残基,x236优选为3~6个氨基酸残基,x240优选为3~9个氨基酸残基,x246优选为5~9个氨基酸残基,x253优选为3~7个氨基酸残基,x269优选为2~12个氨基酸残基,x273优选为0~11个氨基酸残基,x276优选为1~11个氨基酸残基,x280优选为6~10个氨基酸残基,x284优选为2~8个氨基酸残基,x287优选为4~7个氨基酸残基,x288优选为3~5个氨基酸残基。
另外,本发明的蛋白质可以包含下述序列号5所示的氨基酸序列。序列号5所示的氨基酸序列中包含上述序列号1或2所示的氨基酸序列(换言之,序列号1或2所示的氨基酸序列作为序列号5所示的氨基酸序列的一部分而包含(存在))。另外,与序列号7或11所示的氨基酸序列具有25%以上的同一性或75%以上的相似性的氨基酸序列中包含序列号5所示的氨基酸序列(换言之,序列号5所示的氨基酸序列作为与序列号7或11所示的氨基酸序列具有25%以上的同一性或75%以上的相似性的氨基酸序列的一部分而包含(存在))。作为包含序列号5所示的氨基酸序列作为共同基序的蛋白质的植物,可以列举例如:稻、玉米等。
序列号5:
met-gln-ser-gly-x294-x295-met-arg-pro-val-his-asn-ser-val-asp-thr-val-asn-ala-ala-ala-val-ala-ile-val-thr-ala-glu-ser-arg-thr-gln-pro-x296-ala-glu-x297-arg-arg-lys-trp-ala-asp-x298-leu-ser-val-tyr-phe-cys-phe-gly-ser-gln-lys-asn-gly-arg-x299-arg-x300-x301-his-ala-x302-leu-val-pro-glu-pro-x303-pro-x304-arg-thr-asp-ala-pro-x305-x306-glu-ile-pro-x307-his-pro-pro-pro-pro-val-phe-pro-phe-val-ala-pro-pro-ser-ser-pro-ala-ser-phe-leu-gln-ser-x308-x309-x310-ser-ile-val-gln-ser-pro-x311-gly-ala-pro-x312-phe-ser-pro-leu-ser-pro-asn-ser-x313-ser-pro-thr-gly-pro-pro-ser-ile-phe-ala-ile-gly-pro-tyr-ala-his-glu-thr-gln-leu-val-ser-pro-pro-val-phe-ser-ala-phe-thr-thr-glu-pro-ser-thr-ala-pro-phe-thr-pro-pro-pro-glu-ser-val-his-leu-thr-thr-pro-ser-ser-pro-glu-val-pro-tyr-ala-lys-leu-leu-thr-ser-ile-asn-asn-ser-lys-asn-x314-glu-x315-gly-x316-leu-gln-ser-tyr-x317-x318-tyr-pro-x319-ser-pro-ile-gly-arg-leu-ile-ser-pro-ser-ser-x320-cys-ser-gly-thr-x321-ser-pro-phe-pro-asp-pro-glu-x322-gln-x323-ser-ser-arg-ser-x324-x325-x326-x327-phe-pro-val-arg-glu-pro-pro-lys-ile-leu-asp-gly-glu-gly-x328-ala-thr-gln-lys-leu-ile-pro-arg-his-met-arg-asn-gly-gly-ser-leu-leu-asp-gly-x329-ile-ser-ala-ala-val-pro-val-val-asp-phe-ser-ala-arg-leu-gln-x330-asn-x331-his-ala-met-asp-his-arg-val-ser-phe-glu-leu-thr-val-glu-asp-val-ala-arg-cys-leu-glu-lys-lys-thr-x332-ile-x333-gly-x334-ser-x335-x336-ala-ser-phe-x337-leu-x338-pro-thr-gly-x339-gly-asp-x340-his-x341-arg-glu-ser-asn-x342-x343-arg-ala-gly-leu-x344-val-asp-glu-x345-tyr-his-asp-leu-pro-glu-lys-ala-arg-arg-ser-leu-ser-leu-arg-x346-ala-lys-glu-phe-x347-phe-asn-asn-val-asp-x348-x349-ser-val-glu-pro-ser-val-gly-ser-asp-trp-trp-ala-asn-glu-lys-val-ala-gly-x350-thr-x351-glu-pro-x352-lys-x353-trp-ser-phe-x354-pro-val-x355-gln-pro-gly-val-ser
(式中,x294表示ser或gly,x295表示glu或asp,x296表示gln或pro,x297表示pro或gln,x298表示trp或arg,x299表示0~5个氨基酸残基,x300表示ile或val,x301表示ser或asn,x302表示val或ala,x303表示leu或ala,x304表示pro或gln,x305表示met或ala,x306表示pro或ala,x307表示ile或asn,x308表示gly或glu,x309表示gly或pro,x310表示ala或thr,x311表示0~5个氨基酸残基,x312表示ser或ala,x313表示pro或gln,x314表示ala或gly,x315表示thr或ala,x316表示0~5个氨基酸残基,x317表示gln或pro,x318表示ile或asn,x319表示glu或asp,x320表示ala或gly,x321表示cys或ser,x322表示val或met,x323表示thr或ala,x324表示thr或ala,x325表示phe或leu,x326表示pro或arg,x327表示ser或leu,x328表示ile或val,x329表示his或gln,x330表示asn或pro,x331表示asp或glu,x332表示asn或ala,x333表示asn或ser,x334表示glu或asp,x335表示ala或gly,x336表示ala或thr,x337表示arg或his,x338表示val或ala,x339表示asn或ser,x340表示0~5个氨基酸残基,x341表示pro或his,x342表示asp或glu,x343表示thr或ala,x344表示cys或tyr,x345表示thr或ser,x346表示lys或leu,x347表示lys或asn,x348表示ala或val,x349表示pro或gly,x350表示ile或met,x351表示ser或thr,x352表示arg或lys,x353表示ser或asn,x354表示phe或his,x355表示ala或val。)
在上述氨基酸序列中,x299优选为0~2个氨基酸残基,x311优选为1~2个氨基酸残基,x316优选为1~2个氨基酸残基,x340优选为0~2个氨基酸残基。
在本发明中,可以使用编码上述蛋白质的核酸。在本说明书中,核酸是指由磷酸酯键连接核苷酸而形成的高分子,可以与多核苷酸及寡核苷酸这样的用语互换使用。核酸的结构没有特别限定,可以是单链和双链的任一种。核酸中包含脱氧核糖核酸(dna)、核糖核酸(rna)及它们的混合物(例如,dna-rna的杂交双链、dna及rna连接成单链的嵌合核酸(chimericnucleicacid))。核酸的结构单元中主要包含腺嘌呤(a)、鸟嘌呤(g)等嘌呤碱基及胸腺嘧啶(t)、胞嘧啶(c)、尿嘧啶(u)等嘧啶碱基,只要可以进行向对象蛋白质的翻译,也可以包含这些碱基的修饰物。本发明中使用的核酸优选为编码对象蛋白质的mrna或cdna。
本发明中的核酸及蛋白质只要能使植物的生物质增加即可,没有特别限定,优选源自于双子叶植物或单子叶植物。或者可以源自于小立碗藓。作为双子叶植物,可以列举例如:拟南芥、大豆、萝卜、杨、葡萄、棉、油菜、甜菜、烟草、番茄等,其中优选为拟南芥、大豆、萝卜,更优选为拟南芥、大豆,进一步优选为拟南芥。作为单子叶植物,可以列举:稻、玉米、小麦、大麦、高粱、甘蔗、洋葱等,其中优选为稻、玉米、小麦,更优选为稻、玉米。
作为本发明的蛋白质,可以利用源自各种植物的bil7蛋白质,例如可以为源自拟南芥、大豆、稻、玉米、萝卜、杨、葡萄或小立碗藓的bil7蛋白质(即,包含序列号7、9、11、13、15、17、19及21中任一者所示的氨基酸序列的蛋白质)。另外,本发明的蛋白质也可以利用源自各种植物的bil7蛋白质的同源物,例如,可以使用拟南芥bil7的同源物蛋白质(即,包含序列号23、25及27中任一者所示的氨基酸序列的蛋白质)。
另外,在本发明中,作为一个方式,在源自各种植物的bil7蛋白质及其同源物的氨基酸序列(例如,序列号7、9、11、13、15、17、19、21、23、25及27中任一者所示的氨基酸序列)中,也包含由缺失、插入、取代或添加了1个或多个氨基酸的氨基酸序列构成、且具有使植物的生物质增加的活性的蛋白质。这里,作为多个氨基酸,例如是指2~40个、2~30个、2~20个、2~10个、2~7个、2~5个、5个、4个、3个、2个氨基酸的意思。氨基酸的缺失、插入、取代或添加可以使用该技术领域中公知的方法(例如,通过改变核酸)来进行。例如,这样的氨基酸序列优选包含序列号1所示的氨基酸序列。
向核酸导入突变可以通过kunkel法或缺口双链体(gappedduplex)法等、或者以此为基准的方法来进行,例如,可以使用利用了部位特异性突变诱发法的突变导入用试剂盒(例如,transformertmsite-directedmutagenesiskit(clontechlaboratories,inc.)、或quikchangesite-directedmutagenesiskit(agilenttechnologies)。另外,在向核酸导入突变中可以使用ems(甲磺酸乙酯)、5-溴尿嘧啶、2-氨基嘌呤、羟胺、n-甲基-n’-硝基-n-亚硝基胍、其它致癌性化合物这样的化学性突变剂,也可以使用x射线、α射线、β射线、γ射线、离子束等放射线或紫外线。
另外,在本发明中,作为一个方式,在源自各种植物的bil7蛋白质及其同源物的氨基酸序列(例如,序列号7、9、11、13、15、17、19、21、23、25及27中任一者所示的氨基酸序列)中,也包含如下蛋白质,所述蛋白质具有25%以上、30%以上、40%以上、50%以上、60%以上、70%以上、80%以上、85%以上、90%以上、95%以上、96%以上、97%以上、98%以上、99%以上的同一性、或者70%以上、80%以上、85%以上、90%以上、95%以上、96%以上、97%以上、98%以上、99%以上的相似性,且具有使植物的生物质增加的活性。关于氨基酸序列的同一性及相似性,如上述说明所述。另外,这样的氨基酸序列优选包含序列号1所示的氨基酸序列。
作为本发明的蛋白质,在使用了源自拟南芥、大豆、稻、玉米、萝卜、杨、葡萄、或小立碗藓的bil7蛋白质时,作为编码该蛋白质的核酸,主要使用包含序列号6、8、10、12、14、16、18及20中任一者所示的核苷酸序列的核酸。另外,在使用了拟南芥bil7的3种同源物蛋白质时,作为编码该蛋白质的核酸,主要使用包含序列号22、24及26中任一者所示的核苷酸序列的核酸。
作为本发明的一个方式,还包括如下所述的核酸,所述核酸对于包含序列号6、8、10、12、14、16、18、20、22、24及26中任一者所示的核苷酸序列的核酸而言,在严格条件下与由与该核苷酸序列互补的核苷酸序列构成的核酸杂交,而且编码具有使植物的生物质增加的活性的蛋白质。
这里,“严格条件下”是指以中程度或高程度在严格条件下进行杂交。具体而言,对于中程度的严格条件而言,例如,具有通常技术的本领域技术人员可以基于核酸的长度而容易地确定。基本的条件示于sambrook等,molecularcloning:alaboratorymanual,第3版,第6章,coldspringharborlaboratorypress,2001,包含使用例如5×ssc、0.5%sds、1.0mmedta(ph8.0)的预清洗溶液、约42℃下的、约50%甲酰胺、2×~6×ssc、优选为5×~6×ssc、0.5%sds(或约42℃下的约50%甲酰胺中的、施托克溶液(starksolution)等其它同样的杂交溶液)的杂交条件,以及例如约50℃~68℃、0.1×~6×ssc、0.1%sds的清洗条件。中程度严格条件优选包含约50℃、6×ssc、0.5%sds的杂交条件(及清洗条件)。
另外,对于高严格条件而言,例如,也可以由本领域技术人员基于核酸的长度而容易地确定。通常,这样的条件被定义为含有比中程度严格条件更高的温度和/或更低的盐浓度下的杂交(例如,约0.5%sds,约65℃、6×ssc~0.2×ssc、优选为6×ssc、更优选为2×ssc、更优选为0.2×ssc、或0.1×ssc的杂交)和/或清洗,例如,如上所述的杂交条件、以及伴随有约65℃~68℃、0.2×~0.1×ssc、0.1%sds的清洗。在杂交及清洗的缓冲液中,可以使用sspe(1×sspe为0.15mnacl、10mmnah2po4及1.25mmedta、ph7.4)代替ssc(1×ssc为0.15mnacl及15mm柠檬酸钠),清洗在杂交结束后进行15分钟~1小时左右。
另外,也可以使用在探针中不使用放射性物质的市售的杂交试剂盒。具体而言,可以列举使用了ecl直接标记和检测系统(directlabeling&detectionsystem)(amersham公司制造)的杂交等。作为严格的杂交,可以列举如下条件,例如,在试剂盒中的杂交缓冲液中加入封闭试剂5%(w/v),使得nacl为0.5m,在42℃下进行4小时,并在0.4%sds、0.5×ssc中于55℃下进行2次20分钟的清洗,在2×ssc中于室温下进行1次5分钟的清洗。
另外,在本发明中,作为一个方式,还包括如下所述的核酸,所述核酸与序列号6、8、10、12、14、16、18、20、22、24及26中任一者所示的核苷酸序列具有70%以上、80%以上、85%以上、90%以上、95%以上、96%以上、97%以上、98%以上、99%以上的同一性,且编码具有使植物的生物质增加的活性的蛋白质。
2个核苷酸序列的同一性可以通过目测和数学计算来确定,或者更优选通过使用计算机程序比较序列信息来完成该比较。代表性的优选计算机程序是geneticscomputergroup(gcg;威斯康星州麦迪逊(madison))的wisconsinpackage,10.0版程序“gap”(devereux,etal.,1984,nucl.acidsres.,12:387)。通过使用该“gap”程序,除了进行2个核苷酸序列的比较以外,还能够进行2个氨基酸序列的比较、核苷酸序列与氨基酸序列的比较。这里,在“gap”程序的优选的默认参数中包含:(1)关于核苷酸的(关于相同物,含有1的值,关于非相同物含有0的值)一元(unary)比较矩阵的gcg实行、和在schwartz及dayhoff主编的“多肽的序列及结构的图谱(atlasofpolypeptidesequenceandstructure)”国立生物医学研究财团、353-358页、1979所记载的、gribskov及burgess,nucl.acidsres.,14:6745,1986的加权氨基酸比较矩阵;或者其它能够进行比较的比较矩阵;(2)对氨基酸的各间隙进行30的补偿并对各间隙中的各符号追加1的补偿;或者对核苷酸序列的各间隙进行50的补偿并对各间隙中的各符号追加3的补偿;(3)对末端间隙无补偿:以及(4)对长间隙没有最大补偿。对于本领域技术人员使用的其它序列比较程序而言,例如,可以使用通过美国国立医学图书馆的网站:http://www.ncbi.nlm.nih.gov/blast/bl2seq/bls.html可以使用的blastn程序2.2.7版、或uw-blast2.0algorithm。关于uw-blast2.0的标准默认参数的设定,记载于以下的互联网网站:http://blast.wustl.edu。另外,blastalgorithm使用blosum62氨基酸打分矩阵,可使用的选择参数如下所述:(a)包含用于屏蔽具有较低组成复杂性的检索序列的区段(根据wootton及federhen的seg程序(computersandchemistry,1993)来确定;也希望参照wootton及federhen,1996“序列数据库中的组成偏离区域的分析(analysisofcompositionallybiasedregionsinsequencedatabases)”methodsenzymol.,266:544-71)、或者由短周期性的内部重复构成的区段(根据claverie及states(computersandchemistry,1993)的xnu程序来确定)的过滤器;以及(b)用于报告对数据库序列的匹配的有统计学上意义的阈值、或e-评分(根据karlin及altschul,1990)的统计学模型,单纯通过偶然而发现匹配的期望概率;在源于某种匹配的有统计学上意义的差大于e-评分阈值的情况下,不报告该匹配);优选的e-评分阈值的数值为0.5,或者按优选度增加的顺序为0.25、0.1、0.05、0.01、0.001、0.0001、1e-5、1e-10、1e-15、1e-20、1e-25、1e-30、1e-40、1e-50、1e-75或1e-100。
(2)构建体
本发明提供包含上述核酸及启动子的构建体。在本说明书中,构建体是指使多个核酸连接而成的接合物,在本发明中,作为其结构单元,包含上述核酸和启动子。本发明的核酸与启动子不需要直接连接,可以隔着其它种类的核酸间接地连接。启动子优选可发挥作用地连接于本发明的核酸。“可发挥作用地连接”是指启动子能够发挥其功能地连接,即,以能够进行对象核酸的转录的方式进行连接。
启动子只要能够在植物细胞内进行对象核酸的转录即可,没有特别限定。作为这样的启动子,可以列举例如:花椰菜花叶病毒(cauliflowermosaicvirus)35s启动子(camv35s)、各种泛素启动子、各种肌动蛋白启动子、烟草pr1a基因启动子、胭脂碱合成酶基因启动子、油菜籽蛋白(napin)基因启动子、油质蛋白基因启动子等。
另外,在本发明中,也可以使用具有在植物中部位特异性表达的功能的启动子。作为这样的启动子,可以列举:叶部特异性表达核酸的启动子(例如,稻psb0基因启动子(日本特开2010-166924))、茎部特异性表达核酸的启动子(例如,拟南芥fa6启动子(guptaetal.2012plantcellrep.31:839-850.))、根部特异性表达核酸的启动子(例如,rcc3启动子(xuetal.1995plantmolbiol27:237-248))、主要在根、茎、叶等营养器官中表达的启动子(例如,拟南芥as启动子:非专利文献1)等。
另外,在本发明中还可以使用诱导性启动子。作为这样的启动子,可以列举例如:已知因丝状真菌/细菌/病毒的感染或入侵、低温、高温、干燥、紫外线的照射、植物生长素或油菜素类固醇这样的激素等特定化合物的散布等的外因而表达的启动子等。作为这样的启动子,可以列举例如:因丝状真菌/细菌/病毒的感染、入侵而表达的稻壳多糖酶基因的启动子(xuetal.1996plantmol.biol.30:387)、烟草的pr蛋白质基因的启动子(ohshimaetal.1990plantcell2:95),因低温而诱导的稻的“lip19”基因的启动子(aguanetal.1993mol.gen.genet.240:1),因高温而诱导的稻的“hsp80”基因和“hsp72”基因的启动子(vanbreusegemetal.1994planta193:57)、因干燥而诱导的拟南芥的“rab16”基因的启动子(nundyetal.1990proc.natl.acad.sci.usa87:1406)、因紫外线的照射而诱导的欧芹的查耳酮合成酶基因的启动子(schulze-lefertetal.1989emboj.8:651)、厌氧条件下诱导的玉米的醇脱氢酶基因的启动子(walkeretal.1987proc.natl.acad.sci.usa84:6624)、因盐胁迫而诱导的启动子(shinozaki,k.andyamaguchi-shinozaki,k.,curr.opin.plantbiol.3,217-223(2000))等。
(3)载体
本发明提供包含上述构建体的载体。即,在本发明中,可以提供包含能够发挥作用地连接的启动子和本发明的核酸的载体。
载体可以通过利用通常方法将希望的核酸连接于本领域能够获得的重组用载体而简便地制备。在使用本发明的核酸使植物的生物质增加时,植物转化用载体特别有用。本发明中使用的载体只要是为了在植物细胞中达到实现本发明的目的的效果而能够使用的载体即可,没有特别限定,可以使用例如:pbi系的载体、pbluescript系的载体、puc系的载体等。作为pbi系的载体,可以列举例如:pbi121、pbi101、pbi101.2、pbi101.3、pbi221等。从能够经由土壤杆菌向植物导入目标核酸的观点考虑,优选pbi系的载体等双元载体。另外,作为pbluescript系的载体,可以列举例如:pbluescriptsk(+)、pbluescriptsk(-)、pbluescriptiiks(+)、pbluescriptiiks(-)、pbluescriptiisk(+)、pbluescriptiisk(-)等。作为puc系的载体,可以列举例如:puc19、puc119等。从能够将核酸直接导入植物的观点考虑,优选pbluescript系的载体、puc系的载体。另外,还可以优选使用pgreen系列(www.pgreen.ac.uk)、pcambia系列(www.cambia.org)等双元载体、psb11(komarietal,1996,plantj,10:165-174)、psb200(komorietal,2004,plantj,37:315-325)等超级双元载体。
另外,上述载体优选包含转录终止子序列,所述转录终止子序列包含转录产物的稳定化所需要的聚腺苷酸化部位。本领域技术人员可以适当选择转录终止子序列。
转录终止子序列只要具有作为转录终止部位的功能即可,没有特别限定,可以是公知的转录终止子序列。转录终止子序列可以根据使用的启动子来选择,例如可以使用花椰菜花叶病毒35s的转录终止区域(camv35s终止子)、胭脂碱合成酶基因的转录终止区域(nos终止子)等。在上述重组表达载体中,将转录终止子序列设置于适当的位置,由此可以在导入植物细胞后防止不必要地合成长转录物的现象的发生等。
另外,在上述重组表达载体中还可以含有其它核酸区段。该其它核酸区段没有特别限定,可以列举:转化体挑选标记、增强子、用于提高翻译效率的碱基序列等。另外,上述重组表达载体还可以具有t-dna区域。特别是在使用土壤杆菌将上述重组表达载体导入植物体的情况下,t-dna区域能够提高基因导入的效率。
作为转化体挑选标记,例如可以使用耐药性基因。作为所述耐药性基因的具体例子,可以列举例如:对潮霉素、博来霉素、卡那霉素、庆大霉素、氯霉素等的耐药性基因(对抗生素卡那霉素或庆大霉素有耐性的新霉素磷酸转移酶基因、对潮霉素有耐性的潮霉素磷酸转移酶基因)。另外,也可以利用对除草剂膦丝菌素(phosphinothricin)有耐性的膦丝菌素乙酰转移酶基因等。由此,可以通过在含有上述抗生物质、除草剂的培养基中生长繁殖的植物体来容易地挑选转化后的植物体。
作为用于提高翻译效率的核苷酸序列,可以列举例如:源自烟草花叶病毒的omega序列。可以通过使该omega序列设置于启动子的非翻译区域(5’utr)来提高上述融合基因的翻译效率。
另外,作为增强子,可以列举包含camv35s启动子内的上游侧的序列的增强子区域。这样,上述重组表达载体中可以根据其目的而包含各种核酸区段。
对重组表达载体的构建方法没有特别限定,可以按照给定的顺序将本发明的核酸、启动子、及终止子序列、以及根据需要的其它dna区段导入适当选择的作为母体的载体。向作为母体的载体中插入核酸可以按照通常方法进行,可以使用将纯化后的核酸用适当的限制酶切断并插入适当的载体的限制酶部位或多克隆位点的方法等(例如,molecularcloning,5.61-5.63)。
对于本领域技术人员而言,可以根据通常的基因工程技术适当地制备具有希望基因的载体。通常可以通过利用市售的各种载体而容易地制备。
(4)宿主细胞
本发明提供包含上述载体的宿主细胞(换言之,导入了本发明的核酸的宿主细胞)。
本发明的宿主细胞没有特别限定,优选为植物细胞。该植物细胞包括各种形态的植物细胞,例如:悬浮培养细胞、原生质体、植物体中的细胞等。
植物细胞没有特别限定,可以使用源自双子叶植物或源自单子叶植物的细胞。作为双子叶植物,可以列举:拟南芥、大豆、棉、油菜、甜菜、烟草、番茄、萝卜、葡萄、杨等,其中优选为拟南芥、大豆、棉、油菜、烟草、番茄,进一步优选为拟南芥、大豆、棉、油菜。作为单子叶植物,可以列举:稻、玉米、小麦、大麦、高粱、甘蔗、洋葱等,其中优选为稻、玉米、小麦、高粱,更优选为稻、玉米。
本发明的核酸可以使用源自各种植物品种的核酸,在本发明中,核酸的来源植物品种与宿主细胞(植物细胞)的来源植物品种可以相同或不同。即,本发明可以提供:导入了源自与宿主细胞(植物细胞)的植物品种相同的植物品种的核酸或包含该核酸的载体的宿主细胞(植物细胞)、以及导入了源自与宿主细胞(植物细胞)的植物品种不同的植物品种的核酸或包含该核酸的载体的宿主细胞(植物细胞)中的任一者。另外,在本发明中,在核酸的来源植物为双子叶植物的情况下,可以选择双子叶植物的宿主细胞,另外,在核酸的来源植物为单子叶植物的情况下,可以选择单子叶植物的宿主细胞。
作为在宿主细胞内使对象核酸表达的方法,可以列举将该核酸插入适当的载体中,例如:聚乙二醇法、土壤杆菌法、脂质体法、阳离子脂质体法、磷酸钙沉淀法、电脉冲穿孔法(电穿孔)(currentprotocolsinmolecularbiologyedit.ausubeletal.(1987)publish.johnwiley&sons.section9.1-9.9)、脂质转染法(gibco-brl公司制造)、显微注射法、基因枪法等通过本领域技术人员公知的方法导入生体内的方法。在本发明中,可以优选使用土壤杆菌法。在将核酸导入植物细胞内时,可以使用显微注射法、电穿孔法、聚乙二醇法等直接地导入核酸,也可以插入对植物导入基因用的质粒中,将其作为载体,经由具有植物感染能力的病毒或细菌间接地导入植物细胞。作为所述病毒,例如,作为代表性的病毒可以列举:花椰菜花叶病毒、烟草花叶病毒、双粒病毒组等,作为细菌,可以列举土壤杆菌等。在通过土壤杆菌法进行对植物的基因导入时,可以使用市售的质粒。
(5)转化植物
本发明提供导入了上述载体的植物(换言之,导入了本发明的核酸的植物)。在本发明的宿主细胞为植物细胞时,本发明的植物(转化植物)包含该植物细胞。本发明的植物不仅包括植物细胞,还包括植物体整体、植物器官(例如,根、茎、叶、花瓣、种子、果实、成熟胚、未成熟胚、胚珠、子房、苗端、花药、花粉等)、植物组织(例如,表皮、韧皮部、薄壁组织、木质部、维管束等)、它们的切片、愈伤组织、枝条原基(shootprimordia)、幼苗、丛生芽(multipleshoot)、毛状根及培养根等中的任一者。
本发明的植物为双子叶植物或单子叶植物。作为双子叶植物,可以列举:拟南芥、大豆、棉、油菜、甜菜、烟草、番茄、萝卜、葡萄、杨等,其中优选为拟南芥、大豆、棉、油菜、烟草、番茄,进一步优选为拟南芥、大豆、棉、油菜。作为单子叶植物,可以列举:稻、玉米、小麦、大麦、高粱、甘蔗、洋葱等,其中优选为稻、玉米、小麦、高粱,更优选为稻、玉米。
对于本发明的植物而言,其品种与上述宿主细胞同样地可以与导入的核酸的来源植物品种相同或不同。即,本发明可以提供:导入了源自同一品种植物的核酸或包含该核酸的载体的植物(导入了核酸或包含该核酸的载体的植物,且该植物的品种与该核酸的来源植物品种相同)、以及导入了源自不同种植物的核酸或包含该核酸的载体的植物(导入了核酸或包含该核酸的载体的植物,且该植物的品种与该核酸的来源植物品种不同)中的任一者。另外,在本发明中,在核酸的来源植物为双子叶植物时,导入目标植物可以为双子叶植物,另外,在核酸的来源植物为单子叶植物时,导入目标植物可以为单子叶植物。
本发明可以提供以下植物。
(i)导入了源自单子叶植物的核酸或包含该核酸的载体的单子叶植物。
(ii)导入了源自双子叶植物的核酸或包含该核酸的载体的单子叶植物。
(iii)导入了源自双子叶植物的核酸或包含该核酸的载体的双子叶植物。
(iv)导入了源自单子叶植物的核酸或包含该核酸的载体的双子叶植物。
其中,优选为:
(i)导入了源自单子叶植物的核酸或包含该核酸的载体是单子叶植物、
(ii)导入了源自双子叶植物的核酸或包含该核酸的载体的单子叶植物、以及
(iii)导入了源自双子叶植物的核酸或包含该核酸的载体的双子叶植物,
更优选为:
(i)导入了源自单子叶植物的核酸或包含该核酸的载体的单子叶植物、及
(ii)导入了源自双子叶植物的核酸或包含该核酸的载体的单子叶植物。
本发明的植物包括导入了本发明的核酸或包含该核酸的载体的植物细胞生长繁殖而得到的植物体、及该植物体的后代、作为后代或克隆的植物、以及它们的繁殖材料(例如,种子、果实、切穗、块茎、块根、原种(stock)、愈伤组织、原生质体等)。从转化植物细胞进行植物体的再生可以根据植物细胞的种类用本领域技术人员公知的方法来进行。上述技术已经确立,且在本发明的技术领域中广泛使用,在本发明中可以适当地使用上述方法。
使转化后的植物细胞再分化而使植物体再生的方法根据植物细胞的种类而不同,例如,在稻的情况下可以列举fujimura等(planttissueculturelett.2:74(1995))的方法,在玉米的情况下可以列举shillito等(bio/technology7:581(1989))的方法、gorden-kamm等(plantcell2:603(1990))的方法。通过上述方法再生且栽培的转化植物体中导入的外来核酸的存在可以通过公知的pcr法、southern杂交法、或者通过对植物体中的dna碱基序列进行分析来确认。在该情况下,从转化植物体中提取dna可以按照公知的j.sambrook等的方法(molecularcloning、第2版、coldspringharborlaboratorypress,1989)来实施。
例如,在使用pcr法对再生后的植物体中存在的外来核酸进行分析的情况下,如上所述以从再生植物体中提取出的dna作为模板进行扩增反应。另外,可以使用合成的寡核苷酸作为引物,并在混合了它们的反应液中进行扩增反应,所述合成的寡核苷酸具有根据本发明的核酸或修饰后的核酸的核苷酸序列适当选择的核苷酸序列。在扩增反应中,重复数十次dna的变性、退火、延长反应时,可以得到包含对象核酸的核苷酸序列的dna片段的扩增产物。将包含扩增产物的反应液进行例如琼脂糖电泳时,扩增后的各种dna片段被分离,能够确认其dna片段对应于本发明的基因。
一旦获得了在基因组内导入了对象核酸的转化植物体,就能够通过有性生殖或无性生殖从该植物体得到后代。另外,可以从该植物体、其后代或克隆获得繁殖材料,并以此为基础批量制备该植物体。本发明包括导入了本发明的核酸或包含该核酸的重组表达载体的植物细胞、包含该细胞的植物体、该植物体的后代及克隆、以及该植物体、其后代和克隆的繁殖材料。即,本发明包括作为实施了转化处理的再分化初代的“t0代”、作为t0代植物的种子的“t1代”等后代植物、以它们为一个亲体杂交而得到的杂种植物、其后代植物。
本发明的植物(转化植物)包含将天然启动子取代为上述各种启动子的所谓的“部位特异性转化体”,所述天然启动子控制野生型植物(包括未从外部导入本发明的核酸(例如bil7基因)的植物)所具有的bil7基因。这样的植物因取代的启动子而使该植物原本具有的bil7基因的表达增加,从而具有增加生物质这样的特性。本发明也包括从结果上看给定核酸(例如bil7基因)在植物内强表达(过表达)的植物作为一个方式,因此这样的部位特异性转化体也可以作为本发明的对象。作为制备这样的植物的方法,可以使用crispr法、zfn法、tal效应物核酸酶(talen)法等公知的基因组编辑技术。
与普通的植物相比,可以期待这样制备的转化植物具有生物质增加这样的有利特性。本发明中成为转化对象而使用的植物没有特别限定,可以根据本发明的方法制备生物质增加的各种转化植物。
(6)使植物的生物质增加的方法
本发明提供使植物的生物质增加的方法,该方法包括将上述核酸导入植物的工序。更具体而言,本发明的方法为使植物的生物质增加的方法,该方法包括:制备包含本发明的核酸及启动子的载体的工序,所述启动子与该核酸能够发挥作用地连接;将该载体导入宿主细胞(植物细胞)的工序;以及由导入了核酸的植物细胞再生植物体的工序。通过利用本发明的核酸编码的蛋白质所具有的使植物的生物质增加的活性,可以得到本发明的方法。
在本发明的方法中,不仅是上述核酸,还可以将包含该核酸的载体导入植物。将核酸或包含该核酸的载体导入植物的方法如上所述,可以通过导入宿主细胞(植物细胞)来向植物导入该核酸。另外,本发明的方法中成为对象的植物种类、与被导入的核酸的来源植物品种的关系等也如上所述,其它在该方法中应考虑的用语、材料、方法等也按照上述说明及定义等进行解释。
(7)增加了生物质的植物的制备方法
本发明提供增加了生物质的植物的制备方法,该方法包括将上述核酸导入植物的工序。更具体而言,本发明的方法为增加了生物质的植物的制备方法,该方法包括:制备包含本发明的核酸及启动子的载体的工序,所述启动子与该核酸能够发挥作用地连接;将该载体导入宿主细胞(植物细胞)的工序;以及由导入了核酸的植物细胞再生植物体的工序。通过利用本发明的核酸编码的蛋白质所具有的使植物的生物质增加的活性,可以得到本发明的方法。
在本发明的方法中,不仅是上述核酸,还可以将包含该核酸的载体导入植物。将核酸或包含该核酸的载体导入植物的方法如上所述,可以通过导入宿主细胞(植物细胞)来向植物导入该核酸。另外,本发明的方法中成为对象的植物种类、与被导入的核酸的来源植物品种的关系等也如上所述,其它在该方法中应考虑的用语、材料、方法等也按照上述说明及定义等进行解释。
(8)增加了生物质的植物的筛选方法
本发明提供利用了上述说明的蛋白质或核酸的增加了生物质的植物的筛选方法,该方法包括下述工序。
(1)在受试植物及野生型植物中,对本发明的蛋白质或编码该蛋白质的核酸的表达量进行测定的工序,
(2)对通过工序(1)而得到的表达量进行比较的工序,以及
(3)选择比野生型植物中的表达量显示出更高表达量的受试植物的工序。
在本发明的筛选方法中,作为对象植物的形态,与上述相同,不仅包括植物细胞,还包括植物体整体、植物器官(例如,根、茎、叶、花瓣、种子、果实、成熟胚、未成熟胚、胚珠、子房、苗端、花药、花粉等)、植物组织(例如,表皮、韧皮部、薄壁组织、木质部、维管束等)、它们的切片、愈伤组织、枝条原基、幼苗、丛生芽、毛状根及培养根等,从进行筛选的观点考虑,其中特别优选植物为成熟体前的状态或幼小的状态。因此,在本发明的筛选方法中,种子(成熟种子、未成熟种子)、成熟胚、未成熟胚、愈伤组织、枝条、幼苗等是特别优选的植物形态。
在本说明书中,野生型植物是对象植物中最经常发现的表型的系统或个体,主要是指未进行任何基因操作的植物品种。野生型植物的形态可以利用上述形态中的任一种,优选与受试植物的形态相同。
测定蛋白质或核酸的表达量的方法可以利用该技术领域中公知的方法。例如,对于蛋白质而言,从植物中提取蛋白质,制备或获得对本发明的蛋白质(例如,各种植物的bil7蛋白质)的抗体,使用该抗体进行蛋白质印迹法(westernblotting)、免疫测定(elisa等)或基于它们的方法,由此可以测定其表达量。使用的抗体可以是单克隆抗体及多克隆抗体中的任一种,另外,该抗体可以是抗体分子本身,也可以是fab、fab’、或f(ab’)2等的片段。另外,抗体的标签可以使用本身公知的放射性同位素、酶、荧光物质、发光物质等。对于核酸的表达量而言,例如,从植物中提取rna,制备或获得能够基于本发明的核酸(例如,各种植物的bil7基因)的核苷酸序列对该核酸进行特异性扩增的引物或能够特异性检测的探针,使用该引物或探针进行rt-pcr、rna印迹法(northernblotting)或基于它们的方法,由此能够测定其表达量。
蛋白质或核酸的表达量可以是定性的,也可以是定量的,优选以数值(测定值)的形式进行定量化。对由受试植物及野生型植物得到的表达量进行相互比较,在受试植物的表达量大于野生型植物的表达量的情况下,可以判定植物的生物质增加,通过选择这样的受试植物,可以进行增加了植物生物质的植物的筛选。
(9)对增加了生物质的植物进行判定的方法
本发明提供对增加了生物质的植物进行判定的方法,该方法利用了上述说明的蛋白质或核酸,该方法包括下述工序。
(1)在受试植物及野生型植物中,对本发明的蛋白质或编码该蛋白质的核酸的表达量进行测定的工序,
(2)对通过工序(1)而得到的表达量进行比较的工序,以及
(3)确认受试植物中的表达量高于野生型植物中的表达量的工序。
本发明的判定方法与使用本发明的蛋白质或核酸作为标记物(蛋白质标记物或核酸标记物)含义相同。即,在植物中检测出本发明的蛋白质或核酸、且其表达量高于野生型中的表达量的情况下,可以期待该植物的植物生物质增加。
在本发明的判定方法中,对象植物的形态与上述的筛选方法相同,包括上述例示出的所有部位等作为其形态,特别优选植物处于成熟体前的状态或幼小状态的形态。因此,在本发明的判定方法中,种子(成熟种子、未成熟种子)、成熟胚、未成熟胚、愈伤组织、枝条、幼苗等是特别优选的植物形态。
本发明的判定方法中的蛋白质或核酸的表达量测定方法可以利用该技术领域中公知的方法,其具体例子如上述筛选方法中所述。另外,在本发明中,蛋白质或核酸的表达量可以是定性的,也可以是定量的,优选以数值(测定值)的形式进行定量化。将由受试植物及野生型植物得到的表达量进行相互比较,确认受试植物中的表达量大于野生型植物中的表达量,由此可以判定植物的生物质增加。
实施例
以下,通过实施例对本发明具体地进行说明,但这些例子并不用于限定本发明的技术范围。本领域技术人员可以基于本说明书的记载而容易地对本发明进行修改/变更,这些修改/变更也包含在本发明的技术范围内。
实施例1bil7突变体的挑选
fox追寻系统(全长cdna过表达基因追寻系统)是根据通过将全长cdna导入植物并使其高表达所带来的性状变化来确定dna的功能的方法(wo03/018808)。这里,从约8,800种拟南芥(arabidopsis)(拟南芥)fox品系(ichikawaetal.2006)中,在作为油菜素类固醇(br)生物合成抑制剂的芸苔素唑(brz)存在下于暗处进行了品系的挑选,挑选的品系与野生型拟南芥相比,显示出胚轴更加伸长的bil(brz不敏感性长胚轴(brz-insensitive-long-hypocothl))形态。在brz存在下、暗处中显示出bil形态的植物体可以认为具有brz耐性。挑选进行至二次挑选,对于挑选出的品系而言,确认了通过与野生型的回交(backcross)而制备的杂交f1代植物体在brz存在的暗处下显示出bil形态,确认了bil突变为显性性状。由于fox品系为功能获得型突变体,因此可以认为显性突变表示该突变原因来自于fox。
首先,将20~40个fox品系的种子混合,在暗处、brz3μm发芽条件下进行培育,对胚轴伸长品系进行了一次挑选。接着,对一次挑选的个体进行培育,将得到的种子再次在暗处、brz3μm发芽条件下进行培育,对胚轴伸长品系进行了二次挑选。需要说明的是,在二次挑选时,从分离成bil形态强的品系为1/4、bil形态中等程度的品系为2/4、与野生型同等的品系为1/4的情况也可以确认bil突变是显性性状。该挑选的结果是得到了比野生型columbia-0(col-0)明显显示出胚轴伸长的多个品系的候选突变体。其中,将显示出最长的胚轴伸长形态的no.72品系命名为bil7-1d(brz不敏感性长胚轴7-1d,brz-insensitive-long-hypocothl7-1d),进行分析。bil7-1d在brz3μm存在的暗处下的胚轴伸长约为野生型的2.5倍,可以认为与br信息转导的主转录因子(mastertranscriptionfactor)bil1/bzr1的功能获得型突变体bil1-1d/bzr1-1d(显示出耐brz的阳性对照)具有同等强度的耐brz突变性状(图1)。
为了分析bil7-1d的形态上的特征,进行了成熟时的形态观察。对于可以认为bil7-1d突变体基本上停止了生长的第60天的成熟个体进行了形态分析,结果表明,与野生型相比,bil7-1d的花序长度伸长至约1.5倍(图2a、c)。另外可知,与野生型相比,花序数基本上没有变化,但二次花序数增加至约2倍(图2d、e)。另外,与野生型相比,莲座叶具有圆弧,显示出与bil1-1d/bzr1-1d的莲座叶相似的形态(图2b)。
另外,对生殖器官进行了分析。对于bil7-1d而言,在花中未正常地发生花瓣的展开(图3a),因此,虽然没有在全部花中,但在约45%左右的花中观察到了即使存在花也未生长成正常的长角果的现象(图3b)。因此,对于bil7-1d而言,虽然与野生型相比花的数量有所增加,但正常的长角果的数量减少(图3d、e)。而且,由于每1个长角果的种子数也减少,因此总种子数减少(图3f)。种子与野生型相比增大(图3c),种子重量与野生型相比增加至约2倍(图3g)。
对bil7-1d的生长过程进行了分析,结果可以认为,虽然有些开花延迟(图4a)、生长延迟(图4b)的倾向,但考虑到报告了开花延迟型的bil1-1d/bzr1-1d(zhangetal.2013)的值,并没有与野生型的较大差别。另外,在经时观察中,虽然bil7-1d的花序伸长速度与野生型基本没有变化,但与野生型在播种后约第45天开花期停止、且花序伸长也停止的情况相比,bil7-1d在约第60天为止仍继续开花期和花序伸长(图4c)。根据这些结果可知,bil7-1d在花序伸长方面显著地显示出促进生长的形态。
实施例2引起bil7-1d突变的基因的分离和鉴定
根据bil7-1d植物体的形态分析,可以推测引起突变的基因bil7造成花序伸长等促进生长形态、br信息转导的活性化。因此,分离引起bil7-1d突变的基因bil7,进行了同源蛋白质的分析、功能域的研究。接着,制备bil7高表达转化体(bil7-ox),使bil7候选基因高表达,由此确认bil7-1d形态重现的情况,进行了bil7基因的确定。
2-1引起bil7-1d突变的基因的分离
关于bil7-1d突变体的导入cdna,以突变体基因组作为模板,通过使用了对35scamv启动子和nos终止子特异性的引物的pcr法和测序而得到了基因片段。
从bil7-1d突变体上采集莲座叶,使用nucleondnaextractionkit(amersham),提取了基因组dna。接着,对提取出的dna进行了pcr。pcr反应液、反应条件、引物如下所述。
表3
表4
引物集:
对由此得到的pcr产物确定了碱基序列,结果是编码功能未知的新蛋白质的基因表示为bil7候选基因。
利用实时rt-pcr对该bil7候选基因的表达进行了分析。使用rneasyplantminikit(qiagen)从植物体中提取出totalrna。接着,使用takaraprimescriptrtregantkit(perfectrealtime)制备反应液,利用cdna反应(37℃15min、85℃5sec、4℃结束)合成了cdna。以这样合成的cdna作为模板,在以下条件下进行了实时pcr。
表5
表6
rt-pcr引物集:
其结果是,bil7-1d中的bil7候选基因的表达量比野生型增加至约40倍以上(图5)。该结果与bil7-1d为显性突变体的事实一起暗示了bil7候选基因的过表达是bil7-1d的原因。
2-2bil7基因的氨基酸序列的分析
bil7候选基因编码了功能未知的蛋白质,因此,为了推测翻译产物的功能,以bil7的氨基酸序列为基础,进行了基于prosite(http://prosite.expasy.org/)和psort(http://psort.hgc.jp/form.html)数据库的基序检索及基于genetyx-mac的疏水性分析。
其结果是,bil7在其序列中存在n-十四烷酰化预测位点和n-糖基化预测位点。此外不存在能够预测功能的区域。从未发现跨膜区域,但存在核定位信号(nls)的情况可以推测核定位。
bil7同源蛋白质在拟南芥(arabidopsisthaliana)中存在3种。另外已知在其它种植物中,在萝卜(raphanussativus)、大豆(glycinemax)、杨(populustrichocarpa)、葡萄(vitisvinifera)、稻(oryzasativa)、小立碗藓(physcomitellapatens)等广泛的植物品种中存在同源性高的蛋白质。这些均是没有报告例的功能未知的新蛋白质。另外,在这些同源蛋白质组中,确认到了保守性特别高的区域。以该区域为基础进一步进行了显示出同源性的氨基酸的检索,但不存在能够推测功能的蛋白质。另外,可以观察到十四烷酰化预测位点的序列部分在其它基因中比较保守,因此暗示了bil7的功能可能与这些蛋白质中共同的功能相关。
2-3bil7高表达转化体(bil7-ox)的制备及形态观察
将构建体转化至野生型拟南芥中制备bil7高表达转化体(bil7-ox),进行了形态的观察,所述构建体将bil7基因连接于35scamv启动子的下游。
使用rneasyplantminikit(qiagen)从野生型拟南芥的莲座叶提取了rna。接着,使用“superscripttmiiifirst-strandsynthesissystemforrt-pcr”(invitrogen),按照kit的技术方案合成了cdna。以该cdna作为模板,按照使用了以下引物的pcr法对bil7进行了扩增。
表7
向入门载体(pentr)克隆的引物
对于扩增后的bil7而言,使用“penter/dtopocloningkit”(invitrogen)进行了向入门载体(pentr)的克隆。通过gatewaytechonology使用“gatewaylrclonaseiienzymemix(invitrogen)”将制备的pentr载体导入包含35s启动子的pgwb2及包含rnai构建体的pgwb80,得到了插入了bil7的转化用载体pgwb2-bil7及pgwb80-bil7-rnai。
接着,将制备的载体导入土壤杆菌,利用flowerdipping法进行了野生型拟南芥的转化。
相对于土壤杆菌感受态细胞50μl添加2μl制备的载体并混和,在冰上静置30分钟。在液氮中静置1分钟,然后在37℃下融化1分钟。加入yep液体培养基250μl,在28℃下培养1小时,散布于包含卡那霉素和潮霉素50μg/ml、利福霉素(rifampycin)100μg/ml的yep培养基中,利用菌落pcr确认了有无载体导入。
将导入了载体的土壤杆菌的菌落在yep液体培养基中培养过夜,然后将培养液按比例放大至500ml,培养过夜。对培养液进行5000rpm、10分钟的离心,去除上清,然后悬浮于含有5%(w/v)蔗糖的ms培养基。通过浸花(flowerdipping)法对去除了长角果的野生型进行了转化。在含有卡那霉素25μg/ml的ms培养基中对得到的t1种子进行了挑选,得到了bil7高表达转化体(bil7-ox)及bil7表达抑制转化体(bil7-rnai)(图6)。
关于bil7-ox品系,对实施例1中叙述的bil7-1d中观察到的形态特征进行了研究(图7、8)。其结果是,在bil7表达量低的bil7-ox1中未见到bil7-1d中观察到的莲座叶形态,而仅在bil7表达量高的bil7-ox2中确认到bil7-1d中观察到的莲座叶形态(图7a)。在花序长度和二次花序数方面显示出bil7表达量越多的品系越得到促进的倾向(图7a、b、图8d)。另外,对于在bil7-1d中与野生型基本没有变化的花序数而言,观察到一些随bil7表达增强而花序数减少的倾向(图8c)。在种子方面,虽然bil7的表达越高,正常的长角果数越减少,但种子重量与bil7-1d同样地有所增加(图8b、e、f)。
如上所述,在bil7-1d中观察到的成熟形态全部在bil7-ox品系中观察到了。与胚轴的brz耐性形态中的观察结果组合可以认为,因bil7候选基因的高表达重现了bil7-1d的形态。根据这些结果,可以确认该基因为引起bil7-1d的基因。
实施例3稻的bil7同源基因osbil7向稻中的转化
关于稻中的bil7同源基因osbil7,构建了连接于泛素启动子下游的载体,进行了向稻中的转化,所述泛素启动子是稻中的组成性表达启动子(constitutiveexpressionpromoter)。
首先进行了稻的bil7同源基因osbil7的克隆。使用qiagen的rnaeasyplant试剂盒提取了日本晴(nipponbare)稻野生型totalrna。接着,根据其使用invitrogen的superscriptii试剂盒合成了cdna。以该cdna为模板,用以下引物进行pcr扩增了osbil7。
表8
osy72-gw-f1:caccatgcagagtgggagcgagat(序列号44)
osy72-gw-r1:ttagctgacccctggctgt(序列号45)
使用“penter/dtopo克隆试剂盒(cloningkit)”(invitrogen)对扩增后的osbil7进行了克隆。
接下来,使用制备的载体进行了向稻中的转化。稻的品种使用日本晴。使用土壤杆菌eha105株通过高速转化法(tokietal.plantj.47:969-76,2006)对上述中构建的载体进行了转化。对得到的转化t1代、同源t2代进行了产量性状的研究。其结果是,在t1代、同源t2代中均确认到了分蘖数的增加、种子产量的增加等(图9a、b)。
由以上结果可以确认,对于稻而言,bil7使产量提高。
实施例4使osbil7基因过表达的稻品种“yukihikari”的转化体中的生物质评价
将实施例3中构建的载体经由土壤杆菌lba4404株转化至稻(品种:yukihikari)中。同时,以psb4(komarietal.1996plantj10:165-174)作为对象进行转化。转化按照hieietal.(2008plantj6:271-282)的方法进行。将挑选培养基、再分化培养基及生根培养基中的潮霉素的浓度设为30μg/ml。在日本烟草产业株式会社植物创新中心的重组体专用温室中进行了得到的转化体的初代(t0代)的栽培评价。昼长设为14.5小时的长日照条件,温度设为昼温28℃、夜温21℃。从苗木箱移植至盆中18天后,选择生长良好的苗各36株,将每1株移植至塑料盆(直径12cm、容量:830cc)。为了研究有无导入基因,对osbil7基因及潮霉素抗性基因进行了pcr检测。其结果是确认了4个个体中的osbil7基因脱落。另一方面,对照用载体的全部36株中没有潮霉素抗性基因脱落的个体。因此,汇总了32株osbil7重组体及36株对照用载体重组体的数据。测定的性状为秆长、分蘖数、穗数、穗长、单穗粒数、单穗结实粒数、单穗重、总穗重、地上部分干重及全部稻谷重量。需要说明的是,秆长测定了最长的秆的长度。穗长、单穗粒数、单穗结实粒数、单穗重以测定了秆长的秆(=最长的秆)的穗作为测定对象。穗数研究了除去晚穗后的穗的数量。从早熟个体开始依次进行收获。性状研究结束后,汇总数据,进行了统计分析。作为其结果,将各种性状的平均值示于表9。另外,分别将即将移植至塑料盆之前的苗的生长状况示于图10,将成熟期的状况示于图11。
表9
可知,与对照用载体的重组体相比,osbil7重组体具有以下特性。秆长高14cm,穗长长4.6cm,单穗粒数多至23粒。由于种子能育性(fertility)相同,因此单穗结实粒数多至20粒。单穗重增加了0.66g(170%),总穗重增加了2.00g(135%)。最终,相当于种子产量的全部稻谷重量增加了1.80g(135%)。另外,地上部分干重增加了5.00g(140%)。
对以上结果进行总结可知,对于稻品种“yukihikari”而言,在通过泛素启动子使osbil7基因过表达时,可在使稻的地上部分生物质增加的同时也使种子产量增加。
实施例5使拟南芥bil7基因过表达的稻的转化体中的生物质评价
在使拟南芥bil7基因在单子叶植物稻中过表达的情况下,对能否带来使稻产量增加的效果进行了研究。
通过pcr对实施例2中构建的bil7基因cdna克隆(pentr入门载体)的bil7基因编码区域进行扩增,并连接于实施例3中使用的稻转化用双元载体的泛素启动子下游。pcr中使用的引物如下所述。
表10
将得到的构建体ubi-atbil7经由土壤杆菌lba4404株转化至稻(品种:yukihikari)中。同时,以作为pig121hm的修饰型的p121hm载体(在t-dna区域包含“camv35s启动子、潮霉素耐性基因、nos终止子”盒)作为对照进行了转化。转化按照hieietal.(2008plantj6:271-282)的方法进行。将挑选培养基、再分化培养基及生根培养基的潮霉素的浓度设为30μg/ml。在日本烟草产业株式会社植物创新中心的重组体专用温室中进行了得到的转化体的初代(t0代)的栽培评价。昼长设为14.5小时的长日照条件,温度设为昼温28℃、夜温21℃。从苗木箱移植至盆中,选择20天后生长良好的苗各36株,将每1株移植至塑料盆(直径12cm、容量:830cc)。为了研究有无导入基因,对于包括对照在内的全部72个重组体,对bil7基因及潮霉素抗性基因进行了pcr检测。测定的性状为移植至盆中5周后的株高、最长秆的旗叶长度、秆长、穗数、穗长、单穗粒数、单穗结实粒数、单穗结实粒重、单穗重、总穗重、地上部分干重、全部稻谷重量。需要说明的是,秆长测定了最长的秆的长度。穗长、单穗粒数、单穗结实粒数、单穗结实粒重、单穗重以测定了秆长的秆(=最长的秆)的穗作为测定对象。穗数研究了除去晚穗后的穗的数量。1000粒稻谷重量通过将单穗结实稻谷重除以单穗结实粒数而得到的值乘以1000来计算。
对栽培的36个bil7重组体检测了有无bil7基因。其结果是,在bil7重组体中没有bil7基因脱落的个体。另外,在对照用载体的全部36个个体中没有潮霉素抗性基因脱落的个体。因此,汇总了36个bil7重组体及36个对照用载体的重组体的数据。作为其结果,将各种性状的平均值示于表11。另外,分别将即将移植至塑料盆之前的苗的生长状况示于图12,将成熟期的状况示于图13。
表11
可知,与对照用载体的重组体相比,bil7重组体具有以下特性。播种5周后的株高高15cm,旗叶长度长9cm,秆长高13cm。穗长长4.2cm,单穗粒数多至14粒。由于种子能育性相同,单穗结实粒数多至13粒。1000粒稻谷重量重2.6g。单穗重增加了0.48g(146%)。总穗重增加了1.34g(120%),最终,相当于种子产量的全部稻谷重量增加了1.18g(120%)。另外,地上部分干重增加了3.77g(128%)。
对以上结果进行总结可知,对于稻品种“yukihikari”而言,在通过泛素启动子使bil7基因过表达时,可在使稻的地上部分生物质增加的同时也使种子产量增加。
实施例6使osbil7基因过表达的玉米的转化体中的生物质评价
按照ishidaetal.(2007)的方法将实施例3中构建的载体经由土壤杆菌lba4404株转化至玉米(品种:a188)中。在日本烟草产业株式会社植物创新中心的重组体专用温室中进行了得到的转化体的栽培评价。昼长设为14.5小时,温度设为昼温28℃、夜温20℃。抽出的雄穗在开花前进行了切除。将从未转化的玉米(品种:a188)中采集的花粉与从雌穗中充分抽出的玉米穗丝(cornsilk)杂交,得到了t1种子。使用该种子进行了生物质相关性状的评价试验。需要说明的是,在评价试验中,考虑到t0代中的osbil7基因及潮霉素抗性基因的pcr检测结果、植物姿态、种子数及种子重量,将被采集的25个个体中的6个个体供于试验。该评价试验分2次(第1次:3个系统、第2次:3个系统,总计6个系统)进行。在容量570ml的塑料盆中每1个系统播种1粒(1个系统25粒,总计25盆)。在播种后17~18天切下叶的一部分,浸渍于潮霉素溶液中,研究了潮霉素的抗性及敏感性。除去初期生长差的个体,将每1个系统16个个体移植至容量5100cc的塑料盆中继续进行栽培。测定的性状为穗丝抽出天数、总叶数、第9叶至第13叶的叶片长度及株高。需要说明的是,穗丝抽出天数是从播种至穗丝抽出日的天数,株高测定了播种后84天的最终株高。对各系统进行了潮霉素抗性个体(即,视为保有osbil7基因的个体)与潮霉素敏感性个体(即,视为osbil7基因脱落个体)的比较。将其结果示于表12。其结果是,在穗丝抽出天数及总叶数方面,在抗性个体与敏感性个体之间没有确认到差别。另一方面,在株高及第9叶至第13叶的叶片长度方面,抗性个体与敏感性个体相比有所增加(表12)。
对以上结果进行总结可知,对于玉米品种“a188”而言,在使osbil7基因过表达时,可使玉米的地上部分生物质增加。
表12
工业实用性
本发明在能够利用生物质的工业领域是有用的,例如在食品领域、能源领域、环境领域等是有用。通过使用本发明,能够使植物的生物质有效地增加。
序列表
<110>日本烟草产业株式会社(japantobaccoinc.)
国立研究开发法人理化学研究所(riken)
<120>使植物的生物质增加的新基因及对其的利用
<130>fa0392-15166
<150>jp2014-209154
<151>2014-10-10
<160>49
<170>patentinversion3.5
<210>1
<211>70
<212>prt
<213>unknown
<220>
<223>共同基序
<220>
<221>各种特征(misc_feature)
<222>(9)..(9)
<223>xaa为phe或tyr.
<220>
<221>各种特征
<222>(10)..(10)
<223>xaa为phe,leu,thr或ala.
<220>
<221>各种特征
<222>(11)..(11)
<223>xaa为gln,pro,his或asn.
<220>
<221>各种特征
<222>(13)..(13)
<223>xaa为glu,gly,asp,ala或met.
<220>
<221>各种特征
<222>(14)..(14)
<223>xaa为pro,gly,leu或ala.
<220>
<221>各种特征
<222>(15)..(15)
<223>xaa为pro,ala,thr或ser.
<220>
<221>各种特征
<222>(17)..(17)
<223>xaa为ala,ile,val,ser或thr.
<220>
<221>各种特征
<222>(18)..(18)
<223>xaa为thr,val,ser或ala.
<220>
<221>各种特征
<222>(19)..(19)
<223>xaa为gln或his.
<220>
<221>各种特征
<222>(20)..(20)
<223>xaa为ser或thr.
<220>
<221>各种特征
<222>(22)..(22)
<223>xaa表示15-30个任意氨基酸残基。
<220>
<221>各种特征
<222>(27)..(27)
<223>xaa为his或asn.
<220>
<221>各种特征
<222>(29)..(29)
<223>xaa为thr或pro.
<220>
<221>各种特征
<222>(30)..(30)
<223>xaa为gln或ala.
<220>
<221>各种特征
<222>(31)..(31)
<223>xaa为leu或pro.
<220>
<221>各种特征
<222>(33)..(33)
<223>xaa为ser或thr.
<220>
<221>各种特征
<222>(39)..(39)
<223>xaa为thr或ala.
<220>
<221>各种特征
<222>(40)..(40)
<223>xaa为tyr或phe.
<220>
<221>各种特征
<222>(41)..(41)
<223>xaa为thr,ile或pro.
<220>
<221>各种特征
<222>(43)..(43)
<223>xaa为glu或ala.
<220>
<221>各种特征
<222>(46)..(46)
<223>xaa为ser或thr.
<220>
<221>各种特征
<222>(49)..(49)
<223>xaa为ile,val,tyr或phe.
<220>
<221>各种特征
<222>(53)..(53)
<223>xaa表示3-15个任意氨基酸残基。
<220>
<221>各种特征
<222>(58)..(58)
<223>xaa为glu或asp.
<220>
<221>各种特征
<222>(61)..(61)
<223>xaa为phe或tyr.
<220>
<221>各种特征
<222>(63)..(63)
<223>xaa表示20-50个任意氨基酸残基。
<220>
<221>各种特征
<222>(65)..(65)
<223>xaa为gly,glu或asp.
<220>
<221>各种特征
<222>(68)..(68)
<223>xaa表示5或6个任意氨基酸残基。
<400>1
alaproproserserproalaserxaaxaaxaaserxaaxaaxaaser
151015
xaaxaaxaaxaaproxaaglyprotyralaxaagluxaaxaaxaaval
202530
xaaproprovalpheserxaaxaaxaathrxaaproserxaaalapro
354045
xaathrproproxaaproserserproxaavalproxaaalaxaapro
505560
xaaserproxaaserpro
6570
<210>2
<211>77
<212>prt
<213>unknown
<220>
<223>共同基序
<220>
<221>各种特征
<222>(10)..(10)
<223>xaa为phe,leu或thr.
<220>
<221>各种特征
<222>(11)..(11)
<223>xaa为gln,his,pro或asn.
<220>
<221>各种特征
<222>(13)..(13)
<223>xaa为glu,gly,asp或ala.
<220>
<221>各种特征
<222>(14)..(14)
<223>xaa为pro,gly或leu.
<220>
<221>各种特征
<222>(15)..(15)
<223>xaa为pro,ala,thr或ser.
<220>
<221>各种特征
<222>(17)..(17)
<223>xaa为ala,val,ile或thr.
<220>
<221>各种特征
<222>(18)..(18)
<223>xaa为thr,ala,val或ser.
<220>
<221>各种特征
<222>(19)..(19)
<223>xaa为gln或his.
<220>
<221>各种特征
<222>(20)..(20)
<223>xaa为ser或thr.
<220>
<221>各种特征
<222>(22)..(22)
<223>xaa表示10-25个任意氨基酸残基。
<220>
<221>各种特征
<222>(24)..(24)
<223>xaa为ile,val,ala或met.
<220>
<221>各种特征
<222>(25)..(25)
<223>xaa为phe或tyr.
<220>
<221>各种特征
<222>(26)..(26)
<223>xaa为ala或thr.
<220>
<221>各种特征
<222>(27)..(27)
<223>xaa为ile,val或thr.
<220>
<221>各种特征
<222>(32)..(32)
<223>xaa为his或asn.
<220>
<221>各种特征
<222>(36)..(36)
<223>xaa为leu或pro.
<220>
<221>各种特征
<222>(38)..(38)
<223>xaa为ser或thr.
<220>
<221>各种特征
<222>(44)..(44)
<223>xaa为thr或ala.
<220>
<221>各种特征
<222>(45)..(45)
<223>xaa为tyr或phe.
<220>
<221>各种特征
<222>(46)..(46)
<223>xaa为thr或ile.
<220>
<221>各种特征
<222>(51)..(51)
<223>xaa为ser或thr.
<220>
<221>各种特征
<222>(54)..(54)
<223>xaa为ile,phe或tyr.
<220>
<221>各种特征
<222>(58)..(58)
<223>xaa表示3~15个任意氨基酸残基.
<220>
<221>各种特征
<222>(63)..(63)
<223>xaa为glu或asp.
<220>
<221>各种特征
<222>(66)..(66)
<223>xaa为phe或tyr.
<220>
<221>各种特征
<222>(68)..(68)
<223>xaa表示20-35个任意氨基酸残基。
<220>
<221>各种特征
<222>(70)..(70)
<223>xaa为gly,glu或asp.
<220>
<221>各种特征
<222>(73)..(73)
<223>xaa表示3-5个任意氨基酸残基。
<220>
<221>各种特征
<222>(75)..(75)
<223>xaa为ile或arg.
<400>2
alaproproserserproalaserphexaaxaaserxaaxaaxaaser
151015
xaaxaaxaaxaaproxaaserxaaxaaxaaxaaglyprotyralaxaa
202530
gluthrglnxaavalxaaproprovalpheserxaaxaaxaathrglu
354045
proserxaaalaproxaathrproproxaaproserserproxaaval
505560
proxaaalaxaaproxaaserproxaaleuxaaserpro
657075
<210>3
<211>261
<212>prt
<213>unknown
<220>
<223>共同基序
<220>
<221>各种特征
<222>(2)..(2)
<223>xaa为arg或gln.
<220>
<221>各种特征
<222>(5)..(5)
<223>xaa表示3-10个任意氨基酸残基。
<220>
<221>各种特征
<222>(7)..(7)
<223>xaa为val或ser.
<220>
<221>各种特征
<222>(8)..(8)
<223>xaa为phe或val.
<220>
<221>各种特征
<222>(11)..(11)
<223>xaa为ile或val.
<220>
<221>各种特征
<222>(16)..(16)
<223>xaa为ser或val.
<220>
<221>各种特征
<222>(19)..(19)
<223>xaa为ala或val.
<220>
<221>各种特征
<222>(20)..(20)
<223>xaa为ser或thr.
<220>
<221>各种特征
<222>(21)..(21)
<223>xaa为ser或ala.
<220>
<221>各种特征
<222>(22)..(22)
<223>xaa为asp或glu.
<220>
<221>各种特征
<222>(23)..(23)
<223>xaa为asp或ser.
<220>
<221>各种特征
<222>(25)..(25)
<223>xaa表示5-10个任意氨基酸残基。
<220>
<221>各种特征
<222>(29)..(29)
<223>xaa为trp或ala.
<220>
<221>各种特征
<222>(30)..(30)
<223>xaa为asn或asp.
<220>
<221>各种特征
<222>(31)..(31)
<223>xaa为arg或trp.
<220>
<221>各种特征
<222>(32)..(32)
<223>xaa为trp或leu.
<220>
<221>各种特征
<222>(34)..(34)
<223>xaa为leu或val.
<220>
<221>各种特征
<222>(35)..(35)
<223>xaa为leu或tyr.
<220>
<221>各种特征
<222>(36)..(36)
<223>xaa为lys或phe.
<220>
<221>各种特征
<222>(41)..(41)
<223>xaa为ser或gln.
<220>
<221>各种特征
<222>(42)..(42)
<223>xaa为arg或lys.
<220>
<221>各种特征
<222>(43)..(43)
<223>xaa为gln或asn.
<220>
<221>各种特征
<222>(44)..(44)
<223>xaa为arg或gly.
<220>
<221>各种特征
<222>(45)..(45)
<223>xaa为lys或arg.
<220>
<221>各种特征
<222>(48)..(48)
<223>xaa为gly或ser.
<220>
<221>各种特征
<222>(49)..(49)
<223>xaa为asn或his.
<220>
<221>各种特征
<222>(50)..(50)
<223>xaa为ser或ala.
<220>
<221>各种特征
<222>(57)..(57)
<223>xaa表示20-25个任意氨基酸残基。
<220>
<221>各种特征
<222>(60)..(60)
<223>xaa为ile或val.
<220>
<221>各种特征
<222>(70)..(70)
<223>xaa为phe或leu.
<220>
<221>各种特征
<222>(73)..(73)
<223>xaa为glu或gly.
<220>
<221>各种特征
<222>(74)..(74)
<223>xaa为pro或gly.
<220>
<221>各种特征
<222>(75)..(75)
<223>xaa为pro或ala.
<220>
<221>各种特征
<222>(77)..(77)
<223>xaa为ala或ile.
<220>
<221>各种特征
<222>(78)..(78)
<223>xaa为thr或val.
<220>
<221>各种特征
<222>(84)..(84)
<223>xaa为ile或ala.
<220>
<221>各种特征
<222>(85)..(85)
<223>xaa为leu或pro.
<220>
<221>各种特征
<222>(91)..(91)
<223>xaa为pro或ser.
<220>
<221>各种特征
<222>(92)..(92)
<223>xaa为cys或pro.
<220>
<221>各种特征
<222>(94)..(94)
<223>xaa表示1-10个任意氨基酸残基。
<220>
<221>各种特征
<222>(117)..(117)
<223>xaa为thr或ala.
<220>
<221>各种特征
<222>(118)..(118)
<223>xaa为tyr或phe.
<220>
<221>各种特征
<222>(124)..(124)
<223>xaa为ser或thr.
<220>
<221>各种特征
<222>(127)..(127)
<223>xaa为ile或phe.
<220>
<221>各种特征
<222>(131)..(131)
<223>xaa表示1-5个任意氨基酸残基。
<220>
<221>各种特征
<222>(133)..(133)
<223>xaa为ile或val.
<220>
<221>各种特征
<222>(134)..(134)
<223>xaa为tyr或his.
<220>
<221>各种特征
<222>(138)..(138)
<223>xaa表示0-5个任意氨基酸残基。
<220>
<221>各种特征
<222>(146)..(146)
<223>xaa为phe或tyr.
<220>
<221>各种特征
<222>(148)..(148)
<223>xaa为gln或lys.
<220>
<221>各种特征
<222>(150)..(150)
<223>xaa为phe或leu.
<220>
<221>各种特征
<222>(151)..(151)
<223>xaa为asn或thr.
<220>
<221>各种特征
<222>(153)..(153)
<223>xaa表示10-20个任意氨基酸残基。
<220>
<221>各种特征
<222>(155)..(155)
<223>xaa为phe或leu.
<220>
<221>各种特征
<222>(157)..(157)
<223>xaa为phe或ser.
<220>
<221>各种特征
<222>(160)..(160)
<223>xaa为leu或ile.
<220>
<221>各种特征
<222>(161)..(161)
<223>xaa为pro或tyr.
<220>
<221>各种特征
<222>(163)..(163)
<223>xaa为gly或glu.
<220>
<221>各种特征
<222>(166)..(166)
<223>xaa为leu或ile.
<220>
<221>各种特征
<222>(168)..(168)
<223>xaa为gln或arg.
<220>
<221>各种特征
<222>(174)..(174)
<223>xaa表示1-5个任意氨基酸残基。
<220>
<221>各种特征
<222>(177)..(177)
<223>xaa为pro或thr.
<220>
<221>各种特征
<222>(178)..(178)
<223>xaa为thr或cys.
<220>
<221>各种特征
<222>(184)..(184)
<223>xaa表示1-10个任意氨基酸残基。
<220>
<221>各种特征
<222>(186)..(186)
<223>xaa为leu或thr.
<220>
<221>各种特征
<222>(189)..(189)
<223>xaa为his或ser.
<220>
<221>各种特征
<222>(191)..(191)
<223>xaa为gln或pro.
<220>
<221>各种特征
<222>(193)..(193)
<223>xaa为ser或arg.
<220>
<221>各种特征
<222>(194)..(194)
<223>xaa为asp或glu.
<220>
<221>各种特征
<222>(198)..(198)
<223>xaa为leu或ile.
<220>
<221>各种特征
<222>(200)..(200)
<223>xaa表示3-20个任意氨基酸残基。
<220>
<221>各种特征
<222>(202)..(202)
<223>xaa表示10-30个任意氨基酸残基。
<220>
<221>各种特征
<222>(204)..(204)
<223>xaa表示1-5个任意氨基酸残基。
<220>
<221>各种特征
<222>(208)..(208)
<223>xaa为asp或glu.
<220>
<221>各种特征
<222>(210)..(210)
<223>xaa为asp或thr.
<220>
<221>各种特征
<222>(211)..(211)
<223>xaa为ala或val.
<220>
<221>各种特征
<222>(212)..(212)
<223>xaa为asp或glu.
<220>
<221>各种特征
<222>(213)..(213)
<223>xaa为his或asp.
<220>
<221>各种特征
<222>(215)..(215)
<223>xaa为ile或ala.
<220>
<221>各种特征
<222>(218)..(218)
<223>xaa为val或leu.
<220>
<221>各种特征
<222>(219)..(219)
<223>xaa为asp或glu.
<220>
<221>各种特征
<222>(220)..(220)
<223>xaa为gln或lys.
<220>
<221>各种特征
<222>(222)..(222)
<223>xaa表示3-25个任意氨基酸残基。
<220>
<221>各种特征
<222>(224)..(224)
<223>xaa为glu或arg.
<220>
<221>各种特征
<222>(225)..(225)
<223>xaa为ala或glu.
<220>
<221>各种特征
<222>(227)..(227)
<223>xaa为ser或asn.
<220>
<221>各种特征
<222>(229)..(229)
<223>xaa表示5-25个任意氨基酸残基。
<220>
<221>各种特征
<222>(232)..(232)
<223>xaa为gly或arg.
<220>
<221>各种特征
<222>(233)..(233)
<223>xaa为ser或lys.
<220>
<221>各种特征
<222>(234)..(234)
<223>xaa为asn或ala.
<220>
<221>各种特征
<222>(238)..(238)
<223>xaa为asn或lys.
<220>
<221>各种特征
<222>(240)..(240)
<223>xaa表示5-15个任意氨基酸残基。
<220>
<221>各种特征
<222>(242)..(242)
<223>xaa为asp或gly.
<220>
<221>各种特征
<222>(243)..(243)
<223>xaa为glu或ser.
<220>
<221>各种特征
<222>(244)..(244)
<223>xaa为his或asp.
<220>
<221>各种特征
<222>(245)..(245)
<223>xaa为arg或trp.
<220>
<221>各种特征
<222>(246)..(246)
<223>xaa为ser或trp.
<220>
<221>各种特征
<222>(248)..(248)
<223>xaa为ser或asn.
<220>
<221>各种特征
<222>(249)..(249)
<223>xaa为pro或glu.
<220>
<221>各种特征
<222>(251)..(251)
<223>xaa表示5-15个任意氨基酸残基。
<220>
<221>各种特征
<222>(258)..(258)
<223>xaa为met或ala.
<220>
<221>各种特征
<222>(260)..(260)
<223>xaa为ser或pro.
<400>3
metxaaserglyxaaasnxaaxaaaspthrxaaasnalaalaalaxaa
151015
alailexaaxaaxaaxaaxaaargxaaarglystrpxaaxaaxaaxaa
202530
serxaaxaaxaacyspheglyserxaaxaaxaaxaaxaaargilexaa
354045
xaaxaavalleuvalprogluproxaaprophexaaalaproproser
505560
serproalaserphexaaglnserxaaxaaxaaserxaaxaaglnser
65707580
provalglyxaaxaaserpheserproleuxaaxaaasnxaaproser
859095
ilephealaileglyprotyralahisgluthrglnleuvalserpro
100105110
provalpheserxaaxaathrthrgluproserxaaalaproxaathr
115120125
proproxaaserxaaxaaleuthrthrxaaproserserprogluval
130135140
proxaaalaxaaleuxaaxaaserxaagluxaaglnxaatyrglnxaa
145150155160
xaaproxaaserproxaaglyxaaleuileserproserxaasergly
165170175
xaaxaaserpropheproaspxaaserxaapheproxaaphexaaval
180185190
xaaxaaproprolysxaaleuxaaglyxaahisxaavalserphexaa
195200205
leuxaaxaaxaaxaavalxaaargcysxaaxaaxaalysxaaproxaa
210215220
xaaserxaaaspxaaserleuxaaxaaxaalysgluphexaaphexaa
225230235240
valxaaxaaxaaxaaxaaalaxaaxaalysxaatrpserphephepro
245250255
valxaaglnxaagly
260
<210>4
<211>308
<212>prt
<213>未知
<220>
<223>共同基序
<220>
<221>各种特征
<222>(3)..(3)
<223>xaa表示0-3个任意氨基酸残基。
<220>
<221>各种特征
<222>(8)..(8)
<223>xaa表示0-5个任意氨基酸残基。
<220>
<221>各种特征
<222>(11)..(11)
<223>xaa为val或thr.
<220>
<221>各种特征
<222>(12)..(12)
<223>xaa为phe或leu.
<220>
<221>各种特征
<222>(13)..(13)
<223>xaa为asp或glu.
<220>
<221>各种特征
<222>(20)..(20)
<223>xaa为ser或thr.
<220>
<221>各种特征
<222>(21)..(21)
<223>xaa为ala或val.
<220>
<221>各种特征
<222>(25)..(25)
<223>xaa为ser或val.
<220>
<221>各种特征
<222>(26)..(26)
<223>xaa为asp或glu.
<220>
<221>各种特征
<222>(27)..(27)
<223>xaa为asp或asn.
<220>
<221>各种特征
<222>(30)..(30)
<223>xaa为his或asp.
<220>
<221>各种特征
<222>(32)..(32)
<223>xaa为ser或pro.
<220>
<221>各种特征
<222>(33)..(33)
<223>xaa为ser或his.
<220>
<221>各种特征
<222>(35)..(35)
<223>xaa为ile或his.
<220>
<221>各种特征
<222>(36)..(36)
<223>xaa为his或val.
<220>
<221>各种特征
<222>(37)..(37)
<223>xaa为lys或gln.
<220>
<221>各种特征
<222>(39)..(39)
<223>xaa为arg或lys.
<220>
<221>各种特征
<222>(40)..(40)
<223>xaa为lys或ser.
<220>
<221>各种特征
<222>(42)..(42)
<223>xaa为trp或gly.
<220>
<221>各种特征
<222>(44)..(44)
<223>xaa为arg或trp.
<220>
<221>各种特征
<222>(45)..(45)
<223>xaa为trp或leu.
<220>
<221>各种特征
<222>(47)..(47)
<223>xaa为leu或ile.
<220>
<221>各种特征
<222>(48)..(48)
<223>xaa为leu或tyr.
<220>
<221>各种特征
<222>(49)..(49)
<223>xaa为lys或trp.
<220>
<221>各种特征
<222>(53)..(53)
<223>xaa为ser或his.
<220>
<221>各种特征
<222>(54)..(54)
<223>xaa为ser或arg.
<220>
<221>各种特征
<222>(55)..(55)
<223>xaa为arg或lys.
<220>
<221>各种特征
<222>(56)..(56)
<223>xaa为gln或asn.
<220>
<221>各种特征
<222>(58)..(58)
<223>xaa为lys或gln.
<220>
<221>各种特征
<222>(62)..(62)
<223>xaa为asn或his.
<220>
<221>各种特征
<222>(63)..(63)
<223>xaa为ser或ala.
<220>
<221>各种特征
<222>(69)..(69)
<223>xaa为pro或arg.
<220>
<221>各种特征
<222>(70)..(70)
<223>xaa为val或ile.
<220>
<221>各种特征
<222>(71)..(71)
<223>xaa为ser或pro.
<220>
<221>各种特征
<222>(72)..(72)
<223>xaa为met或ser.
<220>
<221>各种特征
<222>(73)..(73)
<223>xaa为ser或gly.
<220>
<221>各种特征
<222>(74)..(74)
<223>xaa为ser或thr.
<220>
<221>各种特征
<222>(75)..(75)
<223>xaa为ser或asp.
<220>
<221>各种特征
<222>(77)..(77)
<223>xaa为ser或ala.
<220>
<221>各种特征
<222>(79)..(79)
<223>xaa表示5-15个任意氨基酸残基。
<220>
<221>各种特征
<222>(81)..(81)
<223>xaa为thr或ile.
<220>
<221>各种特征
<222>(82)..(82)
<223>xaa为thr或pro.
<220>
<221>各种特征
<222>(83)..(83)
<223>xaa为leu或phe.
<220>
<221>各种特征
<222>(84)..(84)
<223>xaa为pro或his.
<220>
<221>各种特征
<222>(86)..(86)
<223>xaa为ile或val.
<220>
<221>各种特征
<222>(96)..(96)
<223>xaa为phe或leu.
<220>
<221>各种特征
<222>(97)..(97)
<223>xaa为gln或his.
<220>
<221>各种特征
<222>(103)..(103)
<223>xaa为ala或val.
<220>
<221>各种特征
<222>(104)..(104)
<223>xaa为thr或ala.
<220>
<221>各种特征
<222>(108)..(108)
<223>xaa为val或ser.
<220>
<221>各种特征
<222>(109)..(109)
<223>xaa为gly或ala.
<220>
<221>各种特征
<222>(113)..(113)
<223>xaa为phe或leu.
<220>
<221>各种特征
<222>(114)..(114)
<223>xaa为ser或thr.
<220>
<221>各种特征
<222>(116)..(116)
<223>xaa表示1-10个任意氨基酸残基。
<220>
<221>各种特征
<222>(139)..(139)
<223>xaa为tyr或phe.
<220>
<221>各种特征
<222>(145)..(145)
<223>xaa为ser或thr.
<220>
<221>各种特征
<222>(148)..(148)
<223>xaa为ile或phe.
<220>
<221>各种特征
<222>(152)..(152)
<223>xaa表示3~15个任意氨基酸残基.
<220>
<221>各种特征
<222>(166)..(166)
<223>xaa为phe或leu.
<220>
<221>各种特征
<222>(167)..(167)
<223>xaa为asn或asp.
<220>
<221>各种特征
<222>(168)..(168)
<223>xaa为ser或pro.
<220>
<221>各种特征
<222>(170)..(170)
<223>xaa为his或asn.
<220>
<221>各种特征
<222>(171)..(171)
<223>xaa为gln或lys.
<220>
<221>各种特征
<222>(172)..(172)
<223>xaa为thr或asn.
<220>
<221>各种特征
<222>(173)..(173)
<223>xaa为gly或ser.
<220>
<221>各种特征
<222>(174)..(174)
<223>xaa为ser或glu.
<220>
<221>各种特征
<222>(175)..(175)
<223>xaa为tyr或thr.
<220>
<221>各种特征
<222>(176)..(176)
<223>xaa为gly或tyr.
<220>
<221>各种特征
<222>(177)..(177)
<223>xaa为tyr或gln.
<220>
<221>各种特征
<222>(178)..(178)
<223>xaa为lys或arg.
<220>
<221>各种特征
<222>(180)..(180)
<223>xaa表示3-7个任意氨基酸残基。
<220>
<221>各种特征
<222>(182)..(182)
<223>xaa为glu或asp.
<220>
<221>各种特征
<222>(184)..(184)
<223>xaa为gln或his.
<220>
<221>各种特征
<222>(185)..(185)
<223>xaa为phe或ser.
<220>
<221>各种特征
<222>(189)..(189)
<223>xaa为pro或his.
<220>
<221>各种特征
<222>(194)..(194)
<223>xaa为leu或val.
<220>
<221>各种特征
<222>(201)..(201)
<223>xaa表示1-10个任意氨基酸残基。
<220>
<221>各种特征
<222>(203)..(203)
<223>xaa为gly或ser.
<220>
<221>各种特征
<222>(204)..(204)
<223>xaa为pro或thr.
<220>
<221>各种特征
<222>(205)..(205)
<223>xaa为thr或ser.
<220>
<221>各种特征
<222>(211)..(211)
<223>xaa表示1-10个任意氨基酸残基。
<220>
<221>各种特征
<222>(214)..(214)
<223>xaa为phe或leu.
<220>
<221>各种特征
<222>(215)..(215)
<223>xaa为pro或leu.
<220>
<221>各种特征
<222>(216)..(216)
<223>xaa为his或asn.
<220>
<221>各种特征
<222>(219)..(219)
<223>xaa为val或thr.
<220>
<221>各种特征
<222>(220)..(220)
<223>xaa为ser或asp.
<220>
<221>各种特征
<222>(222)..(222)
<223>xaa表示3-10个任意氨基酸残基。
<220>
<221>各种特征
<222>(224)..(224)
<223>xaa为pro或his.
<220>
<221>各种特征
<222>(225)..(225)
<223>xaa为lys或gln.
<220>
<221>各种特征
<222>(226)..(226)
<223>xaa为thr或gly.
<220>
<221>各种特征
<222>(227)..(227)
<223>xaa为ala或ser.
<220>
<221>各种特征
<222>(229)..(229)
<223>xaa为val或ser.
<220>
<221>各种特征
<222>(230)..(230)
<223>xaa为thr或leu.
<220>
<221>各种特征
<222>(233)..(233)
<223>xaa表示1-10个任意氨基酸残基。
<220>
<221>各种特征
<222>(235)..(235)
<223>xaa为lys或ala.
<220>
<221>各种特征
<222>(236)..(236)
<223>xaa为ile或ser.
<220>
<221>各种特征
<222>(237)..(237)
<223>xaa为val或phe.
<220>
<221>各种特征
<222>(238)..(238)
<223>xaa为pro或leu.
<220>
<221>各种特征
<222>(240)..(240)
<223>xaa为his或ser.
<220>
<221>各种特征
<222>(241)..(241)
<223>xaa为lys或his.
<220>
<221>各种特征
<222>(242)..(242)
<223>xaa为pro或trp.
<220>
<221>各种特征
<222>(245)..(245)
<223>xaa为phe或ile.
<220>
<221>各种特征
<222>(246)..(246)
<223>xaa为asp或glu.
<220>
<221>各种特征
<222>(247)..(247)
<223>xaa为leu或val.
<220>
<221>各种特征
<222>(248)..(248)
<223>xaa为asp或ser.
<220>
<221>各种特征
<222>(250)..(250)
<223>xaa为asp或gln.
<220>
<221>各种特征
<222>(251)..(251)
<223>xaa为his或glu.
<220>
<221>各种特征
<222>(253)..(253)
<223>xaa为ile或phe.
<220>
<221>各种特征
<222>(254)..(254)
<223>xaa为arg或asn.
<220>
<221>各种特征
<222>(257)..(257)
<223>xaa表示1-15个任意氨基酸残基。
<220>
<221>各种特征
<222>(260)..(260)
<223>xaa为arg或lys.
<220>
<221>各种特征
<222>(262)..(262)
<223>xaa为thr或asp.
<220>
<221>各种特征
<222>(263)..(263)
<223>xaa为phe或ala.
<220>
<221>各种特征
<222>(265)..(265)
<223>xaa表示0-15个任意氨基酸残基。
<220>
<221>各种特征
<222>(267)..(267)
<223>xaa为ala或thr.
<220>
<221>各种特征
<222>(268)..(268)
<223>xaa为ser或pro.
<220>
<221>各种特征
<222>(271)..(271)
<223>xaa表示1-15个任意氨基酸残基。
<220>
<221>各种特征
<222>(273)..(273)
<223>xaa为ser或arg.
<220>
<221>各种特征
<222>(274)..(274)
<223>xaa为met或val.
<220>
<221>各种特征
<222>(275)..(275)
<223>xaa为asn或his.
<220>
<221>各种特征
<222>(277)..(277)
<223>xaa表示5-10个任意氨基酸残基。
<220>
<221>各种特征
<222>(283)..(283)
<223>xaa为gly或asp.
<220>
<221>各种特征
<222>(284)..(284)
<223>xaa为thr或asn.
<220>
<221>各种特征
<222>(285)..(285)
<223>xaa为asp或ala.
<220>
<221>各种特征
<222>(287)..(287)
<223>xaa表示1-10个任意氨基酸残基。
<220>
<221>各种特征
<222>(289)..(289)
<223>xaa为thr或val.
<220>
<221>各种特征
<222>(290)..(290)
<223>xaa为val或ala.
<220>
<221>各种特征
<222>(292)..(292)
<223>xaa表示1-10个任意氨基酸残基。
<220>
<221>各种特征
<222>(295)..(295)
<223>xaa表示1-7个任意氨基酸残基。
<220>
<221>各种特征
<222>(298)..(298)
<223>xaa为asp或asn.
<220>
<221>各种特征
<222>(304)..(304)
<223>xaa为val或met.
<220>
<221>各种特征
<222>(305)..(305)
<223>xaa为met或ile.
<220>
<221>各种特征
<222>(306)..(306)
<223>xaa为gln或arg.
<220>
<221>各种特征
<222>(307)..(307)
<223>xaa为ser或pro.
<400>4
metargxaaglyalaasnglyxaaasnasnxaaxaaxaathrileasn
151015
alaalaalaxaaxaailealaserxaaxaaxaaargleuxaaglnxaa
202530
xaaproxaaxaaxaalysxaaxaatrpxaaasnxaaxaaserxaaxaa
354045
xaacyspheglyxaaxaaxaaxaaargxaaargileglyxaaxaaval
505560
leuvalprogluxaaxaaxaaxaaxaaxaaxaaasnxaathrxaaile
65707580
xaaxaaxaaxaaphexaaalaproproserserproalaserphexaa
859095
xaasergluproproserxaaxaaglnserproxaaxaaileleuser
100105110
xaaxaaproxaaserilephealaileglyprotyralahisgluthr
115120125
glnleuvalserproprovalpheserthrxaathrthrgluproser
130135140
xaaalaproxaathrproproxaathrthrproserserprogluval
145150155160
prophealaglnleuxaaxaaxaaasnxaaxaaxaaxaaxaaxaaxaa
165170175
xaaxaaphexaatyrxaaphexaaxaatyrglnleuxaaproglyser
180185190
proxaaglyglnleuileserproxaaserxaaxaaxaaserprophe
195200205
proaspxaaserleuxaaxaaxaapheglnxaaxaaaspxaaserxaa
210215220
xaaxaaxaaglyxaaxaathrproxaaglnxaaxaaxaaxaaproxaa
225230235240
xaaxaavalserxaaxaaxaaxaaalaxaaxaavalxaaxaacysval
245250255
xaalysleuxaathrxaaxaaproxaagluxaaxaaseraspxaaglu
260265270
xaaxaaxaahisxaalysglupheasnphexaaxaaxaagluxaaleu
275280285
xaaxaaaspxaaalaserxaaserasnxaatrpserphepheproxaa
290295300
xaaxaaxaagly
305
<210>5
<211>428
<212>prt
<213>unknown
<220>
<223>共同基序
<220>
<221>各种特征
<222>(5)..(5)
<223>xaa为ser或gly.
<220>
<221>各种特征
<222>(6)..(6)
<223>xaa为glu或asp.
<220>
<221>各种特征
<222>(34)..(34)
<223>xaa为gln或pro.
<220>
<221>各种特征
<222>(37)..(37)
<223>xaa为pro或gln.
<220>
<221>各种特征
<222>(44)..(44)
<223>xaa为trp或arg.
<220>
<221>各种特征
<222>(59)..(59)
<223>xaa表示0-5个任意氨基酸残基。
<220>
<221>各种特征
<222>(61)..(61)
<223>xaa为ile或val.
<220>
<221>各种特征
<222>(62)..(62)
<223>xaa为ser或asn.
<220>
<221>各种特征
<222>(65)..(65)
<223>xaa为val或ala.
<220>
<221>各种特征
<222>(71)..(71)
<223>xaa为leu或ala.
<220>
<221>各种特征
<222>(73)..(73)
<223>xaa为pro或gln.
<220>
<221>各种特征
<222>(79)..(79)
<223>xaa为met或ala.
<220>
<221>各种特征
<222>(80)..(80)
<223>xaa为pro或ala.
<220>
<221>各种特征
<222>(84)..(84)
<223>xaa为ile或asn.
<220>
<221>各种特征
<222>(107)..(107)
<223>xaa为gly或glu.
<220>
<221>各种特征
<222>(108)..(108)
<223>xaa为gly或pro.
<220>
<221>各种特征
<222>(109)..(109)
<223>xaa为ala或thr.
<220>
<221>各种特征
<222>(116)..(116)
<223>xaa表示0-5个任意氨基酸残基。
<220>
<221>各种特征
<222>(120)..(120)
<223>xaa为ser或ala.
<220>
<221>各种特征
<222>(129)..(129)
<223>xaa为pro或gln.
<220>
<221>各种特征
<222>(199)..(199)
<223>xaa为ala或gly.
<220>
<221>各种特征
<222>(201)..(201)
<223>xaa为thr或ala.
<220>
<221>各种特征
<222>(203)..(203)
<223>xaa表示0-5个任意氨基酸残基。
<220>
<221>各种特征
<222>(208)..(208)
<223>xaa为gln或pro.
<220>
<221>各种特征
<222>(209)..(209)
<223>xaa为ile或asn.
<220>
<221>各种特征
<222>(212)..(212)
<223>xaa为glu或asp.
<220>
<221>各种特征
<222>(224)..(224)
<223>xaa为ala或gly.
<220>
<221>各种特征
<222>(229)..(229)
<223>xaa为cys或ser.
<220>
<221>各种特征
<222>(237)..(237)
<223>xaa为val或met.
<220>
<221>各种特征
<222>(239)..(239)
<223>xaa为thr或ala.
<220>
<221>各种特征
<222>(244)..(244)
<223>xaa为thr或ala.
<220>
<221>各种特征
<222>(245)..(245)
<223>xaa为phe或leu.
<220>
<221>各种特征
<222>(246)..(246)
<223>xaa为pro或arg.
<220>
<221>各种特征
<222>(247)..(247)
<223>xaa为ser或leu.
<220>
<221>各种特征
<222>(262)..(262)
<223>xaa为ile或val.
<220>
<221>各种特征
<222>(282)..(282)
<223>xaa为his或gln.
<220>
<221>各种特征
<222>(298)..(298)
<223>xaa为asn或pro.
<220>
<221>各种特征
<222>(300)..(300)
<223>xaa为asp或glu.
<220>
<221>各种特征
<222>(325)..(325)
<223>xaa为asn或ala.
<220>
<221>各种特征
<222>(327)..(327)
<223>xaa为asn或ser.
<220>
<221>各种特征
<222>(329)..(329)
<223>xaa为glu或asp.
<220>
<221>各种特征
<222>(331)..(331)
<223>xaa为ala或gly.
<220>
<221>各种特征
<222>(332)..(332)
<223>xaa为ala或thr.
<220>
<221>各种特征
<222>(336)..(336)
<223>xaa为arg或his.
<220>
<221>各种特征
<222>(338)..(338)
<223>xaa为val或ala.
<220>
<221>各种特征
<222>(342)..(342)
<223>xaa为asn或ser.
<220>
<221>各种特征
<222>(345)..(345)
<223>xaa表示0-5个任意氨基酸残基。
<220>
<221>各种特征
<222>(347)..(347)
<223>xaa为pro或his.
<220>
<221>各种特征
<222>(352)..(352)
<223>xaa为asp或glu.
<220>
<221>各种特征
<222>(353)..(353)
<223>xaa为thr或ala.
<220>
<221>各种特征
<222>(358)..(358)
<223>xaa为cys或tyr.
<220>
<221>各种特征
<222>(362)..(362)
<223>xaa为thr或ser.
<220>
<221>各种特征
<222>(378)..(378)
<223>xaa为lys或leu.
<220>
<221>各种特征
<222>(383)..(383)
<223>xaa为lys或asn.
<220>
<221>各种特征
<222>(389)..(389)
<223>xaa为ala或val.
<220>
<221>各种特征
<222>(390)..(390)
<223>xaa为pro或gly.
<220>
<221>各种特征
<222>(409)..(409)
<223>xaa为ile或met.
<220>
<221>各种特征
<222>(411)..(411)
<223>xaa为ser或thr.
<220>
<221>各种特征
<222>(414)..(414)
<223>xaa为arg或lys.
<220>
<221>各种特征
<222>(416)..(416)
<223>xaa为ser或asn.
<220>
<221>各种特征
<222>(420)..(420)
<223>xaa为phe或his.
<220>
<221>各种特征
<222>(423)..(423)
<223>xaa为ala或val.
<400>5
metglnserglyxaaxaametargprovalhisasnservalaspthr
151015
valasnalaalaalavalalailevalthralagluserargthrgln
202530
proxaaalagluxaaargarglystrpalaaspxaaleuservaltyr
354045
phecyspheglyserglnlysasnglyargxaaargxaaxaahisala
505560
xaaleuvalprogluproxaaproxaaargthraspalaproxaaxaa
65707580
gluileproxaahisproproproprovalpheprophevalalapro
859095
proserserproalaserpheleuglnserxaaxaaxaaserileval
100105110
glnserproxaaglyalaproxaapheserproleuserproasnser
115120125
xaaserprothrglyproproserilephealaileglyprotyrala
130135140
hisgluthrglnleuvalserproprovalpheseralaphethrthr
145150155160
gluproserthralaprophethrproproprogluservalhisleu
165170175
thrthrproserserprogluvalprotyralalysleuleuthrser
180185190
ileasnasnserlysasnxaagluxaaglyxaaleuglnsertyrxaa
195200205
xaatyrproxaaserproileglyargleuileserproserserxaa
210215220
cysserglythrxaaserpropheproaspprogluxaaglnxaaser
225230235240
serargserxaaxaaxaaxaapheprovalarggluproprolysile
245250255
leuaspglygluglyxaaalathrglnlysleuileproarghismet
260265270
argasnglyglyserleuleuaspglyxaaileseralaalavalpro
275280285
valvalasppheseralaargleuglnxaaasnxaahisalametasp
290295300
hisargvalserphegluleuthrvalgluaspvalalaargcysleu
305310315320
glulyslysthrxaailexaaglyxaaserxaaxaaalaserphexaa
325330335
leuxaaprothrglyxaaglyaspxaahisxaaarggluserasnxaa
340345350
xaaargalaglyleuxaavalaspgluxaatyrhisaspleuproglu
355360365
lysalaargargserleuserleuargxaaalalysgluphexaaphe
370375380
asnasnvalaspxaaxaaservalgluproservalglyserasptrp
385390395400
trpalaasnglulysvalalaglyxaathrxaagluproxaalysxaa
405410415
trpserphexaaprovalxaaglnproglyvalser
420425
<210>6
<211>1524
<212>dna
<213>拟南芥(arabidopsisthaliana)
<220>
<221>cds
<222>(182)..(1258)
<400>6
attcccagaaatttcggggttacgtgtcgctttatttgtaaaacgatttttcagattttg60
attgttaaagaagctttctgtgtattatttttgatttttactagagaaaaactcgaatgc120
tctgttttgttctctcgagaaaattctcagactcagagaaaagtaaaaacagagaagaaa180
gatgagaagcggtgctaatggaaacaacgttttcgatactataaacgca229
metargserglyalaasnglyasnasnvalpheaspthrileasnala
151015
gctgcttctgctattgcttcttctgatgatcgtcttcatcaatcttct277
alaalaseralailealaserseraspaspargleuhisglnserser
202530
ccgattcataagaagagaaaatggtggaatcgatggagtctgttgaaa325
proilehislyslysarglystrptrpasnargtrpserleuleulys
354045
tgtttcggatcttcaagacaaagaaaacgaataggaaactcggttctt373
cyspheglyserserargglnarglysargileglyasnservalleu
505560
gttcctgaaccggtatcaatgtcttcttcaaattcaacaacatcaaat421
valprogluprovalsermetserserserasnserthrthrserasn
65707580
tccggttatcgttcggttatcacaacacttccttttatagctccacct469
serglytyrargservalilethrthrleupropheilealapropro
859095
tcatctccagcttcatttttccaatcagagccaccttctgctacacaa517
serserproalaserphepheglnsergluproproseralathrgln
100105110
tcacctgttggaatcctctcttttagtcctttgccttgtaacaaccgt565
serprovalglyileleuserpheserproleuprocysasnasnarg
115120125
ccttccatcttcgccattggaccttacgctcatgaaactcaattggta613
proserilephealaileglyprotyralahisgluthrglnleuval
130135140
tctcctccggttttctcaacttacactactgaaccatcttcagctcca661
serproprovalpheserthrtyrthrthrgluproserseralapro
145150155160
atcacacctcctcttgatgactcatctatctacttaaccaccacaaca709
ilethrproproleuaspaspserseriletyrleuthrthrthrthr
165170175
ccttcttcacctgaagtgccttttgctcagctctttaactcgaaccat757
proserserprogluvalprophealaglnleupheasnserasnhis
180185190
caaaccggtagctatggttataagttcccaatgtcttctagttatgag805
glnthrglysertyrglytyrlyspheprometsersersertyrglu
195200205
tttcagttttaccaacttcctcctggtagtccacttggtcagcttatt853
pheglnphetyrglnleuproproglyserproleuglyglnleuile
210215220
tccccgagccctggttctggtccaacttctccttttcccgatggagaa901
serproserproglyserglyprothrserpropheproaspglyglu
225230235240
acctcgctgttccctcactttcaagtctctgatcctccaaaactgttg949
thrserleupheprohispheglnvalseraspproprolysleuleu
245250255
agtccaaagactgctggtgttacaactccttgtaaagagcagaagatt997
serprolysthralaglyvalthrthrprocyslysgluglnlysile
260265270
gtaagaccgcataaaccggtttcattcgatcttgatgctgatcatgtc1045
valargprohislysprovalserpheaspleuaspalaasphisval
275280285
attagatgcgtggatcagaagctaagaacaacgttccctgaagcatca1093
ileargcysvalaspglnlysleuargthrthrpheproglualaser
290295300
tcagatcaagaatcaatgaatcattcgtctctcgggtccaataaggaa1141
seraspglnglusermetasnhisserserleuglyserasnlysglu
305310315320
ttcaatttcggcacggatgagaaacatttgaccgttgatgaacataga1189
pheasnpheglythraspglulyshisleuthrvalaspgluhisarg
325330335
tcagcttcgccgaagaacagcaatgattggtcttttttccctgtgatg1237
seralaserprolysasnserasnasptrpserphepheprovalmet
340345350
cagtcaggtacactaagctaaccttcatcagaagaatagaaatctgaaatt1288
glnserglythrleuser
355
tagatatcgattcggacaaatatcttgttcaagattcaagaacaattatagaatttttag1348
atgattctgttcaggatcttaaggatattttcttgtctctctttttggttttgtaataaa1408
tatttggcatcgttagttgttgtatatggctactctttatgtagttttttgtttttgtga1468
atacacatttgatcgcatttgtaaataaaatttaatcagtttcttcggagaaattc1524
<210>7
<211>358
<212>prt
<213>拟南芥(arabidopsisthaliana)
<400>7
metargserglyalaasnglyasnasnvalpheaspthrileasnala
151015
alaalaseralailealaserseraspaspargleuhisglnserser
202530
proilehislyslysarglystrptrpasnargtrpserleuleulys
354045
cyspheglyserserargglnarglysargileglyasnservalleu
505560
valprogluprovalsermetserserserasnserthrthrserasn
65707580
serglytyrargservalilethrthrleupropheilealapropro
859095
serserproalaserphepheglnsergluproproseralathrgln
100105110
serprovalglyileleuserpheserproleuprocysasnasnarg
115120125
proserilephealaileglyprotyralahisgluthrglnleuval
130135140
serproprovalpheserthrtyrthrthrgluproserseralapro
145150155160
ilethrproproleuaspaspserseriletyrleuthrthrthrthr
165170175
proserserprogluvalprophealaglnleupheasnserasnhis
180185190
glnthrglysertyrglytyrlyspheprometsersersertyrglu
195200205
pheglnphetyrglnleuproproglyserproleuglyglnleuile
210215220
serproserproglyserglyprothrserpropheproaspglyglu
225230235240
thrserleupheprohispheglnvalseraspproprolysleuleu
245250255
serprolysthralaglyvalthrthrprocyslysgluglnlysile
260265270
valargprohislysprovalserpheaspleuaspalaasphisval
275280285
ileargcysvalaspglnlysleuargthrthrpheproglualaser
290295300
seraspglnglusermetasnhisserserleuglyserasnlysglu
305310315320
pheasnpheglythraspglulyshisleuthrvalaspgluhisarg
325330335
seralaserprolysasnserasnasptrpserphepheprovalmet
340345350
glnserglythrleuser
355
<210>8
<211>1909
<212>dna
<213>大豆(glycinemax)
<220>
<221>cds
<222>(193)..(1431)
<400>8
tatacacaacaagctcgtaccataggatatacaaactatacagagattgattttattttc60
aattgatctcttagcaaaacaaacatttgtcaagtatcaactttcacgcctctacgattt120
caagcttgtttgtttttgggtatgtagtaaatctagaaaaatctgcttttgtaaagaaaa180
aaggtactagagatgagaggtgctaatggaggcgctgtgaacaacactttg231
metargglyalaasnglyglyalavalasnasnthrleu
1510
gagactatcaacgctgctgcaactgtcatcgcttccgtcgagaatcgc279
gluthrileasnalaalaalathrvalilealaservalgluasnarg
152025
cttgatcaaccccatcctcacgtccagaagaaaagctggggaaactgg327
leuaspglnprohisprohisvalglnlyslyssertrpglyasntrp
30354045
ttaagcatatattggtgttttggacatcgcaaaaaccgacagcgcatt375
leuseriletyrtrpcyspheglyhisarglysasnargglnargile
505560
gggcatgcagtccttgttcccgaaagaataccttctggcacagataat423
glyhisalavalleuvalprogluargileproserglythraspasn
657075
gcaacagtaaattcaacacaagcacctattatcccattccacttcgtt471
alathrvalasnserthrglnalaproileileprophehispheval
808590
gcacctccctcatctcctgcatctttccttcactcagaacctccttca519
alaproproserserproalaserpheleuhissergluproproser
95100105
gttgcacaatccccaagtgctatactatctctcactcccggtggtcct567
valalaglnserproseralaileleuserleuthrproglyglypro
110115120125
ttctctatctttgccattggaccttatgcccacgaaacacaattagtt615
pheserilephealaileglyprotyralahisgluthrglnleuval
130135140
tcaccacctgttttctcaacattcaccacagaaccatcaaccgctcct663
serproprovalpheserthrphethrthrgluproserthralapro
145150155
ttcactccccctcctgaatctaaccacttgactacaccttcttcacct711
phethrproproprogluserasnhisleuthrthrproserserpro
160165170
gaagtgccatttgctcaactgcttgaccccaacaacaaaaatagtgaa759
gluvalprophealaglnleuleuaspproasnasnlysasnserglu
175180185
acctatcagaggttccaaatacctcagtatgatttccactcttatcag807
thrtyrglnargpheglnileproglntyraspphehissertyrgln
190195200205
cttcatcccggaagcccggttggtcaactcatttcaccaagatctgct855
leuhisproglyserprovalglyglnleuileserproargserala
210215220
ttctcagcttctagcacatcatctcctttccctgacactgacactgag903
pheseralaserserthrserserpropheproaspthraspthrglu
225230235
tttaattctcgcgcctccctcctccttaactttcaaacagatgataaa951
pheasnserargalaserleuleuleuasnpheglnthraspasplys
240245250
ctgtctactaatgaaaaccataagtcacatcaaggttctggctctcta999
leuserthrasngluasnhislysserhisglnglyserglyserleu
255260265
accccagattctataagatccacaactcaagctagttttcttccaagt1047
thrproaspserileargserthrthrglnalaserpheleuproser
270275280285
cactgggtctctattgaagtgtctgcccaagaagtattcaattgtgtg1095
histrpvalserilegluvalseralaglngluvalpheasncysval
290295300
gaaaacaaggcagtggcatggactaaaccactttcaaaactcaaaact1143
gluasnlysalavalalatrpthrlysproleuserlysleulysthr
305310315
gatgcaccgggagaagataattcaagagaagtccttgtcatcgaaact1191
aspalaproglygluaspasnserarggluvalleuvalilegluthr
320325330
cccagtgatgcaccacaacagaccgctgacgatggagatgtggaaaga1239
proseraspalaproglnglnthralaaspaspglyaspvalgluarg
335340345
gttcatcataaggatgaatgtataacattttctgctgctaaagaattc1287
valhishislysaspglucysilethrpheseralaalalysgluphe
350355360365
aactttgataatgcagaaggaggggattctcctactcctaatttagtt1335
asnpheaspasnalagluglyglyaspserprothrproasnleuval
370375380
gctgactggtgggctaaggagaaagttgcaagcaaggaaggaggatcc1383
alaasptrptrpalalysglulysvalalaserlysgluglyglyser
385390395
tctaataattggtctttcttcccaatgattcgacccggtgttggttaa1431
serasnasntrpserphepheprometileargproglyvalgly
400405410
tcgccaataaggactcacttagtttagtgactttcacttaacatagccttgctggaattt1491
gacatttttttttttttataaaaaaaattgtgcaattgtgtcacagcagccaacaccagt1551
acaccacccctgctaaattgaatcataggcaactaagaattttacattggtagatctaac1611
aaatagaaaggtggagataattttgcagactagctataaattttgcaggtaaacctactt1671
tggattaggaatggctttacatttataacatctccattctccacacccgttctttatcct1731
ttttgtttgtaagtgtaatacagttgtcatcaacacattggttaccaataaccaatgatg1791
attttgatttttgactatgatgtgtttttttattgccccttgccttgactgtaattgtag1851
gaatgcaattgcagaacttggagaaggggaggaaaccttgtgtgaaacattttagcaa1909
<210>9
<211>412
<212>prt
<213>大豆(glycinemax)
<400>9
metargglyalaasnglyglyalavalasnasnthrleugluthrile
151015
asnalaalaalathrvalilealaservalgluasnargleuaspgln
202530
prohisprohisvalglnlyslyssertrpglyasntrpleuserile
354045
tyrtrpcyspheglyhisarglysasnargglnargileglyhisala
505560
valleuvalprogluargileproserglythraspasnalathrval
65707580
asnserthrglnalaproileileprophehisphevalalapropro
859095
serserproalaserpheleuhissergluproproservalalagln
100105110
serproseralaileleuserleuthrproglyglypropheserile
115120125
phealaileglyprotyralahisgluthrglnleuvalserpropro
130135140
valpheserthrphethrthrgluproserthralaprophethrpro
145150155160
proprogluserasnhisleuthrthrproserserprogluvalpro
165170175
phealaglnleuleuaspproasnasnlysasnsergluthrtyrgln
180185190
argpheglnileproglntyraspphehissertyrglnleuhispro
195200205
glyserprovalglyglnleuileserproargseralapheserala
210215220
serserthrserserpropheproaspthraspthrglupheasnser
225230235240
argalaserleuleuleuasnpheglnthraspasplysleuserthr
245250255
asngluasnhislysserhisglnglyserglyserleuthrproasp
260265270
serileargserthrthrglnalaserpheleuproserhistrpval
275280285
serilegluvalseralaglngluvalpheasncysvalgluasnlys
290295300
alavalalatrpthrlysproleuserlysleulysthraspalapro
305310315320
glygluaspasnserarggluvalleuvalilegluthrproserasp
325330335
alaproglnglnthralaaspaspglyaspvalgluargvalhishis
340345350
lysaspglucysilethrpheseralaalalysglupheasnpheasp
355360365
asnalagluglyglyaspserprothrproasnleuvalalaasptrp
370375380
trpalalysglulysvalalaserlysgluglyglyserserasnasn
385390395400
trpserphepheprometileargproglyvalgly
405410
<210>10
<211>2049
<212>dna
<213>稻(oryzasativa)
<220>
<221>cds
<222>(416)..(1702)
<400>10
gtcttgtgcttctctcttccccggcagcgcgtccatcgaagtagtagcatactactctct60
actcctctcctctcctctcctccgctttggtttgctccgatctgcagcgagagcgggagg120
tcgtcgccggcgaggaggcgcggcggaggaatcctctccttcctcttcttggtctgcgag180
taaagctcggccgccgccggcgacgaactggtttggggagagccacgtacggcgccgcct240
cgaatcgaatcgaattggaggttgatttgactcgttggttggagcgctccatcgattaag300
cagacggagtcatgcttcttctttgggtataaaagtttggatctcggaggctcgattcaa360
tctctggagtgggaacaatcgtttgctcgtggtcgtgggatagctcctctgcgagatg418
met
1
cagagtgggagcgagatgagacccgtgcacaacagtgtcgacacggtg466
glnserglyserglumetargprovalhisasnservalaspthrval
51015
aacgcggctgctgttgccattgttacagccgagagccgcacgcaacct514
asnalaalaalavalalailevalthralagluserargthrglnpro
202530
caggcagagccgcgaagaaaatgggctgattggttgagtgtgtacttc562
glnalagluproargarglystrpalaasptrpleuservaltyrphe
354045
tgctttggatcacagaaaaatggccgacgcatcagccatgctgttctt610
cyspheglyserglnlysasnglyargargileserhisalavalleu
50556065
gtcccagaacctttacctccgaggacagatgcacctatgccagaaatt658
valprogluproleuproproargthraspalaprometprogluile
707580
ccaatccatccgccacccccggtattcccctttgtcgcacctccatcc706
proilehisproproproprovalpheprophevalalaproproser
859095
tctcctgcttcttttctccaatcaggaggtgcatctattgtacaatca754
serproalaserpheleuglnserglyglyalaserilevalglnser
100105110
cctgttggtgctccatctttttcgcctctctcgccaaattctccatcc802
provalglyalaproserpheserproleuserproasnserproser
115120125
cccactgggccaccgtccatctttgctatcggaccatatgcacatgag850
prothrglyproproserilephealaileglyprotyralahisglu
130135140145
acacagctagtctctcctcctgtcttctcagccttcacaactgaacct898
thrglnleuvalserproprovalpheseralaphethrthrglupro
150155160
tcaacagctcctttcactcccccaccagagtctgtgcatctgacaacc946
serthralaprophethrproproprogluservalhisleuthrthr
165170175
ccttcctcaccagaggtgccatatgcaaagctacttacctcaatcaac994
proserserprogluvalprotyralalysleuleuthrserileasn
180185190
aacagcaaaaatgctgaaacaggtgagcttcagtcataccagatttac1042
asnserlysasnalagluthrglygluleuglnsertyrglniletyr
195200205
cctgagagcccaataggacgtctgatatctccaagctcagcttgttca1090
progluserproileglyargleuileserproserseralacysser
210215220225
gggacttgctctccatttcctgaccctgaggtgcagacttcctcacgt1138
glythrcysserpropheproaspprogluvalglnthrserserarg
230235240
tccacattcccctcattcccagttcgtgagcctccaaagatactggat1186
serthrpheproserpheprovalarggluproprolysileleuasp
245250255
ggcgagggaattgcaacacagaagttgatacctcgccatatgcgcaat1234
glygluglyilealathrglnlysleuileproarghismetargasn
260265270
ggcggttctctcttggatggacatatttctgctgctgtaccagtcgta1282
glyglyserleuleuaspglyhisileseralaalavalprovalval
275280285
gacttctctgcccgacttcaaaataatgatcatgctatggatcatcgg1330
asppheseralaargleuglnasnasnasphisalametasphisarg
290295300305
gtttcatttgagttaacagtagaagatgtagctcgctgtcttgagaag1378
valserphegluleuthrvalgluaspvalalaargcysleuglulys
310315320
aaaaccaacattaatggggagtctgctgcagcttcttttcgccttgtg1426
lysthrasnileasnglygluseralaalaalaserpheargleuval
325330335
cccaccggtaacggagatcacattcatcccagagaatcgaatgataca1474
prothrglyasnglyasphisilehisproarggluserasnaspthr
340345350
agagcagggctatgtgttgatgaaacataccatgatctgcctgagaaa1522
argalaglyleucysvalaspgluthrtyrhisaspleuproglulys
355360365
gcacggcgctccttgtcccttcgtaaggctaaagaattcaagttcaac1570
alaargargserleuserleuarglysalalysgluphelyspheasn
370375380385
aatgttgatgctcctagtgtggagccgagcgttggatcagactggtgg1618
asnvalaspalaproservalgluproservalglyserasptrptrp
390395400
gcaaacgagaaagttgctgggatcacatcagagccaaggaaaagctgg1666
alaasnglulysvalalaglyilethrsergluproarglyssertrp
405410415
tccttcttcccagtggcacagccaggggtcagctaacagttgcact1712
serphepheprovalalaglnproglyvalser
420425
gaccataatcttacaagatgaagaaataagcttctgtcatttttgctcctgctgtttctc1772
agttccccagataccatcatcataactgcaggagatgcaaacttatgcaaaatattccag1832
ttttagcggtaagttggagttagttagtaactcagtgtccagctaatatcatcataagat1892
actaaaggtgatgcagattctggaacctgttgttataaaaccataatacttccatggtgt1952
agcgtctgttgtatcctccttaactgactggctgttctgatactgatgctgtttatgtac2012
ataataaactagcgaaaagagtagaactcttcctagc2049
<210>11
<211>428
<212>prt
<213>稻(oryzasativa)
<400>11
metglnserglyserglumetargprovalhisasnservalaspthr
151015
valasnalaalaalavalalailevalthralagluserargthrgln
202530
proglnalagluproargarglystrpalaasptrpleuservaltyr
354045
phecyspheglyserglnlysasnglyargargileserhisalaval
505560
leuvalprogluproleuproproargthraspalaprometproglu
65707580
ileproilehisproproproprovalpheprophevalalapropro
859095
serserproalaserpheleuglnserglyglyalaserilevalgln
100105110
serprovalglyalaproserpheserproleuserproasnserpro
115120125
serprothrglyproproserilephealaileglyprotyralahis
130135140
gluthrglnleuvalserproprovalpheseralaphethrthrglu
145150155160
proserthralaprophethrproproprogluservalhisleuthr
165170175
thrproserserprogluvalprotyralalysleuleuthrserile
180185190
asnasnserlysasnalagluthrglygluleuglnsertyrglnile
195200205
tyrprogluserproileglyargleuileserproserseralacys
210215220
serglythrcysserpropheproaspprogluvalglnthrserser
225230235240
argserthrpheproserpheprovalarggluproprolysileleu
245250255
aspglygluglyilealathrglnlysleuileproarghismetarg
260265270
asnglyglyserleuleuaspglyhisileseralaalavalproval
275280285
valasppheseralaargleuglnasnasnasphisalametasphis
290295300
argvalserphegluleuthrvalgluaspvalalaargcysleuglu
305310315320
lyslysthrasnileasnglygluseralaalaalaserpheargleu
325330335
valprothrglyasnglyasphisilehisproarggluserasnasp
340345350
thrargalaglyleucysvalaspgluthrtyrhisaspleuproglu
355360365
lysalaargargserleuserleuarglysalalysgluphelysphe
370375380
asnasnvalaspalaproservalgluproservalglyserasptrp
385390395400
trpalaasnglulysvalalaglyilethrsergluproarglysser
405410415
trpserphepheprovalalaglnproglyvalser
420425
<210>12
<211>2131
<212>dna
<213>玉米(zeamays)
<220>
<221>cds
<222>(359)..(1651)
<400>12
gcactccctccctgcccctgttctctctctcgtggcgacggcgacggcgacgactgggcg60
gccgttgctgccgccctactcgccaggcgcccaggtattcttcggccccttcgccggcga120
cgagtgtcaccaggttcctcgtctccggaacgcagtgcaaagcagatcgatctcggccca180
gcaggggcctcacgacttgactcccaagggctcccaaccttgcaggctgactcgatctct240
ttggtttcgtggaccgtgatcagactccagggctggatcgtgtttcttccagtgtgcaag300
cttggatctcgtaggtctggtttagaaaggaccgtgcttgctctttgctgccgcaatc358
atgcagagtgggggcgacatgaggcctgtgcacaacagtgtcgataca406
metglnserglyglyaspmetargprovalhisasnservalaspthr
151015
gttaacgctgctgctgttgccatcgtcaccgcggagagccgcacacag454
valasnalaalaalavalalailevalthralagluserargthrgln
202530
cctcctgctgaacagagaaggaaatgggctgaccggttgagcgtgtac502
proproalagluglnargarglystrpalaaspargleuservaltyr
354045
ttctgctttggatcacagaagaatggccggcgcatgcgtgtcaaccat550
phecyspheglyserglnlysasnglyargargmetargvalasnhis
505560
gccgcacttgtcccagaacctgcacctcaaaggacagatgcgcctgca598
alaalaleuvalprogluproalaproglnargthraspalaproala
65707580
gcagaaattccaaaccacccaccgcctcctgtgttcccctttgtggca646
alagluileproasnhisproproproprovalpheprophevalala
859095
ccgccatcctctcctgcttctttccttcaatcagaacccacatcaatc694
proproserserproalaserpheleuglnsergluprothrserile
100105110
gtacaatcaccaagggctggagctccagctttttcgcccctctcacca742
valglnserproargalaglyalaproalapheserproleuserpro
115120125
aattcccaatccccgacggggccaccatccatctttgctattgggccg790
asnserglnserprothrglyproproserilephealaileglypro
130135140
tatgcacatgagacacaactcgtctcgcctccagtgttctcggccttc838
tyralahisgluthrglnleuvalserproprovalpheseralaphe
145150155160
acaactgaaccatcaactgcccctttcactcctcctccggagtctgtc886
thrthrgluproserthralaprophethrproproprogluserval
165170175
catctgacaaccccttcctctccagaggtcccatatgcaaagcttctg934
hisleuthrthrproserserprogluvalprotyralalysleuleu
180185190
acatcgatcaacaatagcaagaatggtgaagcagggggtgatctccag982
thrserileasnasnserlysasnglyglualaglyglyaspleugln
195200205
tcgtatccaaactacccagacagcccaattgggcgcctgatatctcca1030
sertyrproasntyrproaspserproileglyargleuileserpro
210215220
agctcgggctgttctggcacatcctccccattccctgaccctgagatg1078
serserglycysserglythrserserpropheproaspproglumet
225230235240
caggcttcttcacgcagcgctttacgcttgttcccagttcgtgagccc1126
glnalaserserargseralaleuargleupheprovalargglupro
245250255
cctaagatattggatggcgagggcgttgcgacacagaagttgatacct1174
prolysileleuaspglygluglyvalalathrglnlysleuilepro
260265270
cgccatatgcgcaacggtgggtccctcttggatggccagatctcagca1222
arghismetargasnglyglyserleuleuaspglyglnileserala
275280285
gctgtaccagttgtggacttctctgcccgacttcaacccaacgagcac1270
alavalprovalvalasppheseralaargleuglnproasngluhis
290295300
gcaatggatcaccgggtgtcattcgagttgaccgtcgaagatgtcgcg1318
alametasphisargvalserphegluleuthrvalgluaspvalala
305310315320
cgctgccttgagaagaagactgcaatctccggggattctggcacggca1366
argcysleuglulyslysthralaileserglyaspserglythrala
325330335
tcattccaccttgcaccgaccggcagcggcgaccaccacagagaatcc1414
serphehisleualaprothrglyserglyasphishisarggluser
340345350
aacgaggcaagggcagggctctacgtcgacgaatcataccatgacttg1462
asnglualaargalaglyleutyrvalaspglusertyrhisaspleu
355360365
cccgagaaagcgaggcggtccctgtccctgcgcctggccaaagagttc1510
proglulysalaargargserleuserleuargleualalysgluphe
370375380
aatttcaacaacgtcgacgtcggtagcgtggagccgagcgtgggatcc1558
asnpheasnasnvalaspvalglyservalgluproservalglyser
385390395400
gactggtgggcgaacgagaaagtcgccgggatgacaactgagccaaaa1606
asptrptrpalaasnglulysvalalaglymetthrthrgluprolys
405410415
aagaactggtctttccacccggtggtgcagcctggggtcagctaa1651
lysasntrpserphehisprovalvalglnproglyvalser
420425430
cctttgcactgaccataatgatcttatacagggatgaaggaattagcttcttctgcttct1711
gcttctgcttcagtggtcgattccttagggatgacttgccagcttgttctaaacatgcag1771
ctagtggtaagttctggatttagatagtaataagtaatggggtgtccaccgtctaatatc1831
gtcatgggatactaaaggtttttattctgggagatgtgacaaaccgcaaactctttggtt1891
aggggtcggtcggaactgtcctgttcgtctgttttatcctgataccggtgctgtgttgtg1951
tatgtacataatgaagtagtttttttaaaaaaaagtcaattaaaagggtagaacagtttc2011
cttcgataagttgccagatgctaccaaattgcttgtgattgttccctgcccgttcatgat2071
attgttattacagaatttgtgagagaggaaacttctgggatttttctgaaaaaaaaaaaa2131
<210>13
<211>430
<212>prt
<213>玉米(zeamays)
<400>13
metglnserglyglyaspmetargprovalhisasnservalaspthr
151015
valasnalaalaalavalalailevalthralagluserargthrgln
202530
proproalagluglnargarglystrpalaaspargleuservaltyr
354045
phecyspheglyserglnlysasnglyargargmetargvalasnhis
505560
alaalaleuvalprogluproalaproglnargthraspalaproala
65707580
alagluileproasnhisproproproprovalpheprophevalala
859095
proproserserproalaserpheleuglnsergluprothrserile
100105110
valglnserproargalaglyalaproalapheserproleuserpro
115120125
asnserglnserprothrglyproproserilephealaileglypro
130135140
tyralahisgluthrglnleuvalserproprovalpheseralaphe
145150155160
thrthrgluproserthralaprophethrproproprogluserval
165170175
hisleuthrthrproserserprogluvalprotyralalysleuleu
180185190
thrserileasnasnserlysasnglyglualaglyglyaspleugln
195200205
sertyrproasntyrproaspserproileglyargleuileserpro
210215220
serserglycysserglythrserserpropheproaspproglumet
225230235240
glnalaserserargseralaleuargleupheprovalargglupro
245250255
prolysileleuaspglygluglyvalalathrglnlysleuilepro
260265270
arghismetargasnglyglyserleuleuaspglyglnileserala
275280285
alavalprovalvalasppheseralaargleuglnproasngluhis
290295300
alametasphisargvalserphegluleuthrvalgluaspvalala
305310315320
argcysleuglulyslysthralaileserglyaspserglythrala
325330335
serphehisleualaprothrglyserglyasphishisarggluser
340345350
asnglualaargalaglyleutyrvalaspglusertyrhisaspleu
355360365
proglulysalaargargserleuserleuargleualalysgluphe
370375380
asnpheasnasnvalaspvalglyservalgluproservalglyser
385390395400
asptrptrpalaasnglulysvalalaglymetthrthrgluprolys
405410415
lysasntrpserphehisprovalvalglnproglyvalser
420425430
<210>14
<211>1029
<212>dna
<213>萝卜(raphanussativus)
<220>
<221>cds
<222>(1)..(1029)
<400>14
atgagaggcggcgcgagtggaaacaacgttttggagactataaacgca48
metargglyglyalaserglyasnasnvalleugluthrileasnala
151015
gccgctactgcgttcgcttcctctgatgatcgtgttcatcaccaacct96
alaalathralaphealaserseraspaspargvalhishisglnpro
202530
tccccgattcatagaagaaaacgaatcgggaaagctgctcttgctcct144
serproilehisargarglysargileglylysalaalaleualapro
354045
gaaccggttcctaccgattccacatccaattccggttatcgttcggtt192
gluprovalprothraspserthrserasnserglytyrargserval
505560
atgacggctcttcctttcatagccccaccttcctctccagcttccttc240
metthralaleupropheilealaproproserserproalaserphe
65707580
ttccaatcagaacctccttccgctacacagtcacctgtagggatcctc288
pheglnsergluproproseralathrglnserprovalglyileleu
859095
tcctttagtcctctaccttctaacagccacaacaacaacaacaacagc336
serpheserproleuproserasnserhisasnasnasnasnasnser
100105110
gaagaacgtccttcgatcttcgccatcggaccttacgctcacgaacct384
glugluargproserilephealaileglyprotyralahisglupro
115120125
cagctggtttctcctccggttttctctacttacacaaccgaaccgtct432
glnleuvalserproprovalpheserthrtyrthrthrgluproser
130135140
tcagctccggtcacgccgcctctcgacgagtctttctacttaaccacc480
seralaprovalthrproproleuaspgluserphetyrleuthrthr
145150155160
accacaccgtcttcgcctgaagtccctttcgctcagctctttaactcc528
thrthrproserserprogluvalprophealaglnleupheasnser
165170175
agcagtaactacggtgtcaggtctccggtgtctaactacgagtttcag576
serserasntyrglyvalargserprovalserasntyrgluphegln
180185190
ttttaccaacttcctcccggtagtccactcgctcagcttatctccccc624
phetyrglnleuproproglyserproleualaglnleuileserpro
195200205
agctcggttatgtccggttctggtgcgacttctccgtttcctgacgga672
serservalmetserglyserglyalathrserpropheproaspgly
210215220
ctcgctcagtttcaagtctctgatccaccaaagctgctgagccctggt720
leualaglnpheglnvalseraspproprolysleuleuserprogly
225230235240
aaactgcgttgctccaagtctgttacaactcctaaagagcagaacaag768
lysleuargcysserlysservalthrthrprolysgluglnasnlys
245250255
attgtgagaccgaacaaaccggtttcgttcgatcttgatgcggatcat816
ilevalargproasnlysprovalserpheaspleuaspalaasphis
260265270
ttcattagatgcgttgataagaagctgagaacaacgttccctgaagcg864
pheileargcysvalasplyslysleuargthrthrpheprogluala
275280285
tctgatcaagaagcagctcaacattcctcctccggatccaataaagaa912
seraspglnglualaalaglnhisserserserglyserasnlysglu
290295300
ttcgatttcggcaccaccgatgagatacatttgaccggtgatgatgag960
pheasppheglythrthraspgluilehisleuthrglyaspaspglu
305310315320
catagagattcgaccaagaacagcagcgattggtccttccctgtgatg1008
hisargaspserthrlysasnserserasptrpserpheprovalmet
325330335
caatcaggcacacttagctaa1029
glnserglythrleuser
340
<210>15
<211>342
<212>prt
<213>萝卜(raphanussativus)
<400>15
metargglyglyalaserglyasnasnvalleugluthrileasnala
151015
alaalathralaphealaserseraspaspargvalhishisglnpro
202530
serproilehisargarglysargileglylysalaalaleualapro
354045
gluprovalprothraspserthrserasnserglytyrargserval
505560
metthralaleupropheilealaproproserserproalaserphe
65707580
pheglnsergluproproseralathrglnserprovalglyileleu
859095
serpheserproleuproserasnserhisasnasnasnasnasnser
100105110
glugluargproserilephealaileglyprotyralahisglupro
115120125
glnleuvalserproprovalpheserthrtyrthrthrgluproser
130135140
seralaprovalthrproproleuaspgluserphetyrleuthrthr
145150155160
thrthrproserserprogluvalprophealaglnleupheasnser
165170175
serserasntyrglyvalargserprovalserasntyrgluphegln
180185190
phetyrglnleuproproglyserproleualaglnleuileserpro
195200205
serservalmetserglyserglyalathrserpropheproaspgly
210215220
leualaglnpheglnvalseraspproprolysleuleuserprogly
225230235240
lysleuargcysserlysservalthrthrprolysgluglnasnlys
245250255
ilevalargproasnlysprovalserpheaspleuaspalaasphis
260265270
pheileargcysvalasplyslysleuargthrthrpheprogluala
275280285
seraspglnglualaalaglnhisserserserglyserasnlysglu
290295300
pheasppheglythrthraspgluilehisleuthrglyaspaspglu
305310315320
hisargaspserthrlysasnserserasptrpserpheprovalmet
325330335
glnserglythrleuser
340
<210>16
<211>2350
<212>dna
<213>杨(populustrichocarpa)
<220>
<221>cds
<222>(298)..(1821)
<400>16
ggaaaagagttaatgggattaaaaacaacaaacaagaggataagaaccgtagagaagaga60
gagtgtgcatgtttttcgttttcatgatctgaaaaaacagaacacaactcaacacttctc120
ttctgttctgttctgctgtcttgttttttttttgtttttttttttaaacaaaaatctctt180
cttttttcacttcttttatctaagaaaacagcagccctctttggttttggtatgaattta240
attccctgcatgtgttttatgggtggtgatgtttgctgttgttgttgttaatcaaag297
atgagaagtgtcaataatagcagcatcgaaaccgttaacgccgctgct345
metargservalasnasnserserilegluthrvalasnalaalaala
151015
accgccatcgtctccgccgagtctcgtgtccagccatcctcttcttct393
thralailevalseralagluserargvalglnproserserserser
202530
gttcaaaaaagaaggtggggaggctgctggagtctctactggtgtttt441
valglnlysargargtrpglyglycystrpserleutyrtrpcysphe
354045
ggttctcacggttctcacaagaatagcaagcgaattggtcatgctgtc489
glyserhisglyserhislysasnserlysargileglyhisalaval
505560
cttgttcctgaaccagaagtaccaggagcagtatcctcttcaactgaa537
leuvalprogluprogluvalproglyalavalserserserthrglu
65707580
aatcagactcagtcaactcccattctgctgccctttattgctcctccc585
asnglnthrglnserthrproileleuleupropheilealapropro
859095
tcttctcctgcttctttcctccagtctgatcccccttcctccactcaa633
serserproalaserpheleuglnseraspproproserserthrgln
100105110
tcaccagcaggattgctctctctcacgtccctctcggcgaatgcttat681
serproalaglyleuleuserleuthrserleuseralaasnalatyr
115120125
tccccacgcggacctgcatccatttttgccataggcccttatgcacat729
serproargglyproalaserilephealaileglyprotyralahis
130135140
gaaacccagctagtcacaccacctgtgttttctgctttcaccactgag777
gluthrglnleuvalthrproprovalpheseralaphethrthrglu
145150155160
ccatccactgctccattcactccccctccggagtctgttcaacttact825
proserthralaprophethrproproprogluservalglnleuthr
165170175
acaccttcatctcctgaagtgccatttgctcaactcttgacgtcctct873
thrproserserprogluvalprophealaglnleuleuthrserser
180185190
ctggaacgtgctcgaagaaatagtggccccaatcaaaagttttcgtta921
leugluargalaargargasnserglyproasnglnlyspheserleu
195200205
tctcactatgaatttcagtcatatcatctgtacccaggaagcccaggt969
serhistyrglupheglnsertyrhisleutyrproglyserprogly
210215220
ggtcaaatcatctcaccaggatcagcaatctcgaattcaggtacctct1017
glyglnileileserproglyseralaileserasnserglythrser
225230235240
tctccgttccccgatagacatcctatgcttgagttccgaatgggtgag1065
serpropheproasparghisprometleuglupheargmetglyglu
245250255
gctcccaagctcctgggcttcgaacattttagtactcgtaaatggggt1113
alaprolysleuleuglyphegluhispheserthrarglystrpgly
260265270
tcaagattgggttctggatctttgacaccggatgcgactccagatggc1161
serargleuglyserglyserleuthrproaspalathrproaspgly
275280285
atgggattatccaggctcggttccggtacggtgaccccagatggcatg1209
metglyleuserargleuglyserglythrvalthrproaspglymet
290295300
gggctatcgaggctctgttctggaactgccactccagatggtgcgggc1257
glyleuserargleucysserglythralathrproaspglyalagly
305310315320
ctgcgttcaaggcttggctcaggaactttgacccctgattgcttcgtg1305
leuargserargleuglyserglythrleuthrproaspcyspheval
325330335
cctgcctcgcaaattggtttccttttggagaaccaaatttctgaggtt1353
proalaserglnileglypheleuleugluasnglnilesergluval
340345350
gcatcacttaccaactcagaaaatggatcaaaaaccgaagaaaatgtg1401
alaserleuthrasnsergluasnglyserlysthrglugluasnval
355360365
gttcaccacagagtctcctttgagttgagcggtgaagaggttgcgcgt1449
valhishisargvalserphegluleuserglyglugluvalalaarg
370375380
tgtcttgaaattaagtcagtggcatccactagaactttcccagagtat1497
cysleugluilelysservalalaserthrargthrpheproglutyr
385390395400
ccacaggacaccatgcccgaagacccagtgagaggtgataggttagca1545
proglnaspthrmetprogluaspprovalargglyaspargleuala
405410415
atgaatggtgaacgttgcttacaaaacggggaagcttccagtgaaatg1593
metasnglygluargcysleuglnasnglyglualaserserglumet
420425430
cctgagaaaaattcagaagaaacagaagaggatcacgtctatagaaag1641
proglulysasnserglugluthrglugluasphisvaltyrarglys
435440445
catcgatccatcactcttgggtcaataaaagagtttaactttgataac1689
hisargserilethrleuglyserilelysglupheasnpheaspasn
450455460
tctaaaggagaagtctctgataagcctgctatcagctctgagtggtgg1737
serlysglygluvalserasplysproalaileserserglutrptrp
465470475480
gcaaatgaaacgattgctgggaaggaagctagacctgccaacagctgg1785
alaasngluthrilealaglylysglualaargproalaasnsertrp
485490495
actttctttcctctgctacagccagaggtcagctgacattacaatc1831
thrphepheproleuleuglnprogluvalser
500505
atgaaaaaaagacctctcttccatatttggacagtgaagcaaatatgttgtattgaaagc1891
tagcctgtgagatgcaagtccaaagccagcactgttttggaaactctgattaccatagtc1951
tgcaatcacatgattgaaggatagatttcacagtactattttctcaagtgaccaaaagca2011
caaattcttagaggaagtagtttgtagggatccttggtgtgggcactgatagatcattct2071
tttttttatcttttccttattttagttacacaaaaatagcatttgcatataggtattgta2131
ctctggatattttttttctttcctttttccttttcatgtgtaataataagagaatttggt2191
ttccttcgacaggtgccctcactatagtgattcggtgtctctgtggctgtatatgcccct2251
cctcgtcttgtttctgcaggttctctcgttatagaatcctgatgaattgattatatatat2311
tagaaagataagtttgatattgatctatctacattttta2350
<210>17
<211>507
<212>prt
<213>杨(populustrichocarpa)
<400>17
metargservalasnasnserserilegluthrvalasnalaalaala
151015
thralailevalseralagluserargvalglnproserserserser
202530
valglnlysargargtrpglyglycystrpserleutyrtrpcysphe
354045
glyserhisglyserhislysasnserlysargileglyhisalaval
505560
leuvalprogluprogluvalproglyalavalserserserthrglu
65707580
asnglnthrglnserthrproileleuleupropheilealapropro
859095
serserproalaserpheleuglnseraspproproserserthrgln
100105110
serproalaglyleuleuserleuthrserleuseralaasnalatyr
115120125
serproargglyproalaserilephealaileglyprotyralahis
130135140
gluthrglnleuvalthrproprovalpheseralaphethrthrglu
145150155160
proserthralaprophethrproproprogluservalglnleuthr
165170175
thrproserserprogluvalprophealaglnleuleuthrserser
180185190
leugluargalaargargasnserglyproasnglnlyspheserleu
195200205
serhistyrglupheglnsertyrhisleutyrproglyserprogly
210215220
glyglnileileserproglyseralaileserasnserglythrser
225230235240
serpropheproasparghisprometleuglupheargmetglyglu
245250255
alaprolysleuleuglyphegluhispheserthrarglystrpgly
260265270
serargleuglyserglyserleuthrproaspalathrproaspgly
275280285
metglyleuserargleuglyserglythrvalthrproaspglymet
290295300
glyleuserargleucysserglythralathrproaspglyalagly
305310315320
leuargserargleuglyserglythrleuthrproaspcyspheval
325330335
proalaserglnileglypheleuleugluasnglnilesergluval
340345350
alaserleuthrasnsergluasnglyserlysthrglugluasnval
355360365
valhishisargvalserphegluleuserglyglugluvalalaarg
370375380
cysleugluilelysservalalaserthrargthrpheproglutyr
385390395400
proglnaspthrmetprogluaspprovalargglyaspargleuala
405410415
metasnglygluargcysleuglnasnglyglualaserserglumet
420425430
proglulysasnserglugluthrglugluasphisvaltyrarglys
435440445
hisargserilethrleuglyserilelysglupheasnpheaspasn
450455460
serlysglygluvalserasplysproalaileserserglutrptrp
465470475480
alaasngluthrilealaglylysglualaargproalaasnsertrp
485490495
thrphepheproleuleuglnprogluvalser
500505
<210>18
<211>1523
<212>dna
<213>葡萄(vitisvinifera)
<220>
<221>cds
<222>(177)..(1523)
<400>18
gaatctcgtgtgattttcgttttcccgatcggactcacagctccatatcggaatctcgcc60
ggaaaacttgctgtttcttatggtgaattttaagcacacatagccggaattagaagtttt120
agttgtcggaggaggtttgagttggaaatagggggattttgtgatttataaggaagatg179
met
1
agaagtgtgaataatagcgtagagactataaacgccgcggccactgcg227
argservalasnasnservalgluthrileasnalaalaalathrala
51015
atcgtctcggccgagagtcgagtccagccgactacggttcagaagaga275
ilevalseralagluserargvalglnprothrthrvalglnlysarg
202530
agatggggtagctgcttgagtttatactggtgctttggatctcataga323
argtrpglysercysleuserleutyrtrpcyspheglyserhisarg
354045
cacagcaagcgaataggtcatgctgttcttgttcctgaaccaatggtg371
hisserlysargileglyhisalavalleuvalprogluprometval
50556065
ccaggagctgtcgctcctgcttctgaaaacctgaacctctcaaccagc419
proglyalavalalaproalasergluasnleuasnleuserthrser
707580
attgtactgcctttcattgcacctccctcttctcctgcatctttcctc467
ilevalleupropheilealaproproserserproalaserpheleu
859095
caatcagatcctccgtcctccactcagtcaccagctggatttctatcc515
glnseraspproproserserthrglnserproalaglypheleuser
100105110
ctcactgccctttctgtcaatgcctattccccaagtggtcctgcctcc563
leuthralaleuservalasnalatyrserproserglyproalaser
115120125
atgtttgccattggcccttatgcccatgaaactcaactagtctcacca611
metphealaileglyprotyralahisgluthrglnleuvalserpro
130135140145
cctgtattttctaccttccccacagagccatcgactgctccttttact659
provalpheserthrpheprothrgluproserthralaprophethr
150155160
ccccctccagaatctgtgcaattaactacgccttcatcacctgaagtt707
proproprogluservalglnleuthrthrproserserprogluval
165170175
ccatttgctcagctgctgacatcttcactggaccgctctcgaaggaac755
prophealaglnleuleuthrserserleuaspargserargargasn
180185190
agtggaaccaatcagaagttgtcactgtccaattatgaattccagcct803
serglythrasnglnlysleuserleuserasntyrglupheglnpro
195200205
tatcagctatacccagaaagcccagttggccaccttatatcaccaata851
tyrglnleutyrprogluserprovalglyhisleuileserproile
210215220225
tcaaattctggtacctcttctcctttcccagatagacgcccaattgta899
serasnserglythrserserpropheproaspargargproileval
230235240
gaggctcccaagctcttgggttttgaacatttttccacccgcagatgg947
glualaprolysleuleuglyphegluhispheserthrargargtrp
245250255
ggttcaaggctgggttctggatctttgacaccagatggtgcagggcct995
glyserargleuglyserglyserleuthrproaspglyalaglypro
260265270
gcctcccgagacagtttccttctggagaaccagatctctgaggtggca1043
alaserargaspserpheleuleugluasnglnilesergluvalala
275280285
tcccttgccaactctgagagtggatctcaaaatggtgaaactgtaatc1091
serleualaasnsergluserglyserglnasnglygluthrvalile
290295300305
gatcatagggtctcatttgagttggctggtgaagatgttgcagtttgt1139
asphisargvalserphegluleualaglygluaspvalalavalcys
310315320
gttgaaaagaaaccagtggcatcagctgaaaccgtccaaaacactctt1187
valglulyslysprovalalaseralagluthrvalglnasnthrleu
325330335
caggatatagttgaagaaggcgaaattgaaagagaaagagatgggatt1235
glnaspilevalglugluglygluilegluarggluargaspglyile
340345350
tcagagagtacagaaaattgttgtgagttctgtgtcggagaagctctt1283
sergluserthrgluasncyscysgluphecysvalglyglualaleu
355360365
aaagctgcgtccgagaaagcttcagctgaaggagaggaagagcagtgc1331
lysalaalaserglulysalaseralagluglygluglugluglncys
370375380385
cataaaaagcatcctccgatcagacatggttcaatcaaagagttcaat1379
hislyslyshisproproilearghisglyserilelysglupheasn
390395400
tttgacaacacaaagggagaagtctcagccaagccgaacatcatcggt1427
pheaspasnthrlysglygluvalseralalysproasnileilegly
405410415
tctgagtggtgggtaaacgaaaaggttgtcgggaaaggaacagggccc1475
serglutrptrpvalasnglulysvalvalglylysglythrglypro
420425430
caaaccaactggactttcttcccattgctgcagcccggaatcagttga1523
glnthrasntrpthrphepheproleuleuglnproglyileser
435440445
<210>19
<211>448
<212>prt
<213>葡萄(vitisvinifera)
<400>19
metargservalasnasnservalgluthrileasnalaalaalathr
151015
alailevalseralagluserargvalglnprothrthrvalglnlys
202530
argargtrpglysercysleuserleutyrtrpcyspheglyserhis
354045
arghisserlysargileglyhisalavalleuvalproglupromet
505560
valproglyalavalalaproalasergluasnleuasnleuserthr
65707580
serilevalleupropheilealaproproserserproalaserphe
859095
leuglnseraspproproserserthrglnserproalaglypheleu
100105110
serleuthralaleuservalasnalatyrserproserglyproala
115120125
sermetphealaileglyprotyralahisgluthrglnleuvalser
130135140
proprovalpheserthrpheprothrgluproserthralaprophe
145150155160
thrproproprogluservalglnleuthrthrproserserproglu
165170175
valprophealaglnleuleuthrserserleuaspargserargarg
180185190
asnserglythrasnglnlysleuserleuserasntyrgluphegln
195200205
protyrglnleutyrprogluserprovalglyhisleuileserpro
210215220
ileserasnserglythrserserpropheproaspargargproile
225230235240
valglualaprolysleuleuglyphegluhispheserthrargarg
245250255
trpglyserargleuglyserglyserleuthrproaspglyalagly
260265270
proalaserargaspserpheleuleugluasnglnilesergluval
275280285
alaserleualaasnsergluserglyserglnasnglygluthrval
290295300
ileasphisargvalserphegluleualaglygluaspvalalaval
305310315320
cysvalglulyslysprovalalaseralagluthrvalglnasnthr
325330335
leuglnaspilevalglugluglygluilegluarggluargaspgly
340345350
ilesergluserthrgluasncyscysgluphecysvalglygluala
355360365
leulysalaalaserglulysalaseralagluglyglugluglugln
370375380
cyshislyslyshisproproilearghisglyserilelysgluphe
385390395400
asnpheaspasnthrlysglygluvalseralalysproasnileile
405410415
glyserglutrptrpvalasnglulysvalvalglylysglythrgly
420425430
proglnthrasntrpthrphepheproleuleuglnproglyileser
435440445
<210>20
<211>4092
<212>dna
<213>小立碗藓(physcomitrellapatens)
<220>
<221>cds
<222>(1)..(4092)
<400>20
atggtgctgctcatgaataggaatgcactcggtatccatttcggtgcc48
metvalleuleumetasnargasnalaleuglyilehispheglyala
151015
cgggccagtctggcagtgatggtgatccattggtatttgcggcctggc96
argalaserleualavalmetvalilehistrptyrleuargprogly
202530
gagctaaccccatgctcacccacgaatggcccattatggaaatggatg144
gluleuthrprocysserprothrasnglyproleutrplystrpmet
354045
agaacaatcttaccttttccggtgcctgtagatttgaagagggaggct192
argthrileleupropheprovalprovalaspleulysarggluala
505560
gttctggccggaacggtcgtcgtctacagcgttctaaagatgcccgtg240
valleualaglythrvalvalvaltyrservalleulysmetproval
65707580
ctctggagctcttccctcgacgccgacgccgacgcagcagccactaat288
leutrpserserserleuaspalaaspalaaspalaalaalathrasn
859095
cgacattcatcatcaacgttcgttagagatggttggagggcattaacg336
arghisserserserthrphevalargaspglytrpargalaleuthr
100105110
ggtcgtggccaaggaccgaaattgcggcgggtggagggtatggagact384
glyargglyglnglyprolysleuargargvalgluglymetgluthr
115120125
agggtagtttctgcagaggagcttgttttaccaatggttgccctagct432
argvalvalseralaglugluleuvalleuprometvalalaleuala
130135140
tgggtagtggtacataagttcctgtgggcgctatggaagctatggcgc480
trpvalvalvalhislyspheleutrpalaleutrplysleutrparg
145150155160
tctgccgttgatgacataaacattggaaggacgaaacgtttgtgtcct528
seralavalaspaspileasnileglyargthrlysargleucyspro
165170175
acctcttctcagatggcacgaggtggatgctgctggagtgggccattt576
thrserserglnmetalaargglyglycyscystrpserglyprophe
180185190
tgtcttggctcattatcccgcaagaacaagaagcgaattgttccggca624
cysleuglyserleuserarglysasnlyslysargilevalproala
195200205
accagagtgcacgatggaactgcacagccatctgaccctcagggccag672
thrargvalhisaspglythralaglnproseraspproglnglygln
210215220
tttgccttcctacttgcacccccttcgtctcctgcttcctatgcaaat720
phealapheleuleualaproproserserproalasertyralaasn
225230235240
tctatggcaccttcctctgttcagtcgccctactacccttcatcgtgt768
sermetalaproserservalglnserprotyrtyrprosersercys
245250255
cctgttcctcagggtggtgggtctaggattcctctagaaacacagtct816
provalproglnglyglyglyserargileproleugluthrglnser
260265270
aacatgttcgctgtaggtccgtatgcacatgaaaccgctctagtctct864
asnmetphealavalglyprotyralahisgluthralaleuvalser
275280285
ccaccagttttttctacgttcacaactgctccctccacagctcctttt912
proprovalpheserthrphethrthralaproserthralaprophe
290295300
acgccccctccagagcttgcagcccatttcactacgccttcttcccct960
thrproproprogluleualaalahisphethrthrproserserpro
305310315320
gatgttcctttcgcaaagctgttaggatcttcgttctcagaacagcga1008
aspvalprophealalysleuleuglyserserphesergluglnarg
325330335
actacgaagcgagaagcggagcctccatactcagcctctccattcgct1056
thrthrlysargglualagluproprotyrseralaserpropheala
340345350
tccccagattactatcagcaagatcaccatccgcaggatgacttgcag1104
serproasptyrtyrglnglnasphishisproglnaspaspleugln
355360365
gttgggtatcagttatatccaggaagtccgcttggtcgtttaatttct1152
valglytyrglnleutyrproglyserproleuglyargleuileser
370375380
cctgcaggcaccacgggcgcgtcaactccttttgctgcaggtggcact1200
proalaglythrthrglyalaserthrprophealaalaglyglythr
385390395400
actggtactaatactcctcatgcagagagtgacaatcctactccactg1248
thrglythrasnthrprohisalagluseraspasnprothrproleu
405410415
actgtccttccagcagtggtaagcactttacctaaccttgagcaccag1296
thrvalleuproalavalvalserthrleuproasnleugluhisgln
420425430
gtggctgaagggcttcatcagaggagtattcttgattctcagtgcggg1344
valalagluglyleuhisglnargserileleuaspserglncysgly
435440445
ccaggggaacctctgagtgacagtggtagagagcggcatgggagcttt1392
proglygluproleuseraspserglyarggluarghisglyserphe
450455460
gacagtcactcgcggttcatggtagcaatgatacacgaacgcgatgga1440
aspserhisserargphemetvalalametilehisgluargaspgly
465470475480
tctgattccaactcaaattcgtatggacatgagcggtacagcggaagt1488
seraspserasnserasnsertyrglyhisgluargtyrserglyser
485490495
ttgactgggcttaatgacgtcttggagggacggaacaggtatacgaag1536
leuthrglyleuasnaspvalleugluglyargasnargtyrthrlys
500505510
ttgaagcaggacaagggtcctcgatcctctagtctgagatcccatgag1584
leulysglnasplysglyproargserserserleuargserhisglu
515520525
aaagaggatgatattcaggaagaggacttgcttgaacttcccatgcta1632
lysgluaspaspileglnglugluaspleuleugluleuprometleu
530535540
ttaggcagtgagagttctcatggtggttcgagagcatcacggtctcca1680
leuglysergluserserhisglyglyserargalaserargserpro
545550555560
agtagtagaagtaaagttcagagcaaggcagggagcagagtagggagc1728
serserargserlysvalglnserlysalaglyserargvalglyser
565570575
agggtagaaagtaaggcagctagcagggtagggagtaaggctgggtct1776
argvalgluserlysalaalaserargvalglyserlysalaglyser
580585590
gatgccttgaaagattgggatcacaacgaggataacttattggaatat1824
aspalaleulysasptrpasphisasngluaspasnleuleuglutyr
595600605
gcactaagcttgcttgaaggaactgatggaaaaagacccggaacaatc1872
alaleuserleuleugluglythraspglylysargproglythrile
610615620
gggtgggcgccagtggatacctcgctcccacgagaatctgaaggtccc1920
glytrpalaprovalaspthrserleuproargglusergluglypro
625630635640
attcagtcatcctctaccacgagaaacgagaccgaggagaatgaaaag1968
ileglnserserserthrthrargasngluthrglugluasnglulys
645650655
gatactgtgtataaaagtatcaagaatggtaaaggtagcgacaatgtg2016
aspthrvaltyrlysserilelysasnglylysglyseraspasnval
660665670
ccccagcaagtagtgggggcgggagcaaaggaggtttctgagggaacg2064
proglnglnvalvalglyalaglyalalysgluvalsergluglythr
675680685
attactccggttggacatcatggtagtgcacctgattgctgttcgaga2112
ilethrprovalglyhishisglyseralaproaspcyscysserarg
690695700
tgcgaatcgcttgtctctcagtgtgagcagctatcagtggctttgaag2160
cysgluserleuvalserglncysgluglnleuservalalaleulys
705710715720
gaggctagaaggaaacaacaagagaaagatcgacttgcggaggaaaga2208
glualaargarglysglnglnglulysaspargleualaglugluarg
725730735
gaaaaacaaataaggcaccttacacagctactgcagtcaggggaacag2256
glulysglnilearghisleuthrglnleuleuglnserglyglugln
740745750
ggcagtttcttacaagtcctagatctgttggacgagcacatgccggag2304
glyserpheleuglnvalleuaspleuleuaspgluhismetproglu
755760765
gaatacctgagtcgagcaagcttattctgccctgtttctgaagaattc2352
glutyrleuserargalaserleuphecysprovalserglugluphe
770775780
aacttctccgcagaatctttgctgggttttagtcatgagtttagactt2400
asnpheseralagluserleuleuglypheserhisglupheargleu
785790795800
gccgaatggagcaaactttgtccgaatgatttcgcgctcacattgtgc2448
alaglutrpserlysleucysproasnaspphealaleuthrleucys
805810815
aacgaattacgttgcacatttgctgatgatgagaacgcgttctgttta2496
asngluleuargcysthrphealaaspaspgluasnalaphecysleu
820825830
ggggagtttatgagctacgatcctggttcacaagccaccggcagagtg2544
glygluphemetsertyraspproglyserglnalathrglyargval
835840845
aggaaggtcgcaatgagcgccaactatggcaacttggacattcagaca2592
arglysvalalametseralaasntyrglyasnleuaspileglnthr
850855860
atgctttggtgtcgaagtcttcgaagaacgcatgacgacaatatgaag2640
metleutrpcysargserleuargargthrhisaspaspasnmetlys
865870875880
acggtgaagaaggggatggaggtaaagaggaagagtagattgagtaaa2688
thrvallyslysglymetgluvallysarglysserargleuserlys
885890895
accagcgctcttcaaaagaaaatgcctaactcaaatgtagaggctgct2736
thrseralaleuglnlyslysmetproasnserasnvalglualaala
900905910
aactatcctcgatctgttttgcaaactcgttctcaggagactccctcg2784
asntyrproargservalleuglnthrargserglngluthrproser
915920925
gcgtcacctcgactatcaagaccgaagagtgctcaggtccagcaagct2832
alaserproargleuserargprolysseralaglnvalglnglnala
930935940
aagaagttactgaaaatattacgtctagatcacatgctttctgatggc2880
lyslysleuleulysileleuargleuasphismetleuseraspgly
945950955960
gacgaagaggaagaaacagagaggatgagcaagaagaaagggatcaaa2928
aspglugluglugluthrgluargmetserlyslyslysglyilelys
965970975
gaaaagaagaagcgttgtccgtacaggctgaggtcgacctcgtccaaa2976
glulyslyslysargcysprotyrargleuargserthrserserlys
980985990
atacctctatccagggaaggttctccccgtccagttgagaggagtgta3024
ileproleuserarggluglyserproargprovalgluargserval
99510001005
tctcctttgacatcttggagaatggatgatgccggtatgccagaa3069
serproleuthrsertrpargmetaspaspalaglymetproglu
101010151020
ccactgcgggagaagttgaatatttttaagaaagagcatgagacc3114
proleuargglulysleuasnilephelyslysgluhisgluthr
102510301035
atgtcaagtcaacaaacttcgaaagaaccagtgcagttgaatcaa3159
metserserglnglnthrserlysgluprovalglnleuasngln
104010451050
ggttcggaagaagaagagaattataacgacacatctcatgctacg3204
glyserglugluglugluasntyrasnaspthrserhisalathr
105510601065
cagtggaatcacaaacctttgcgaggacaagcgcggggggaatct3249
glntrpasnhislysproleuargglyglnalaargglygluser
107010751080
tggcaagccgatcacgtaggtctcagtctcaaaaaggagccgcac3294
trpglnalaasphisvalglyleuserleulyslysgluprohis
108510901095
aattttgaactagataagttaaagaatatagtagcaacaatgcag3339
asnphegluleuasplysleulysasnilevalalathrmetgln
110011051110
gaggagcacagacagtgtattcgcagtatcgcggagacccttgaa3384
glugluhisargglncysileargserilealagluthrleuglu
111511201125
catcacagccggcagattgttctcttgacgaaggcaaactcttcc3429
hishisserargglnilevalleuleuthrlysalaasnserser
113011351140
ctgtcgaaccacgcggattccttagagaaagagctcatggtgttg3474
leuserasnhisalaaspserleuglulysgluleumetvalleu
114511501155
cgagctatgcacataaaatccgaaagtctctttaatgctgtaaaa3519
argalamethisilelyssergluserleupheasnalavallys
116011651170
ttggcggatttaagagtaggatgtatcaagaccaccttgttaaat3564
leualaaspleuargvalglycysilelysthrthrleuleuasn
117511801185
gctctcttgttctgccatttgtgcgcagaaacgaaaggcggagtt3609
alaleuleuphecyshisleucysalagluthrlysglyglyval
119011951200
gggcattatcgagggaatagcggtcttggccgaatagacctggcg3654
glyhistyrargglyasnserglyleuglyargileaspleuala
120512101215
agaaacagtgaactcctcgacctgaggttggacgatcttggtgat3699
argasnsergluleuleuaspleuargleuaspaspleuglyasp
122012251230
gagcggcaagcaacaaaggaagatcgaagaaaacaaaggctctcg3744
gluargglnalathrlysgluaspargarglysglnargleuser
123512401245
cctccctcgaatgtgagagcaagacgtggtagggtcacaaaggga3789
proproserasnvalargalaargargglyargvalthrlysgly
125012551260
gcggggccaaggttcgagaaagccttagccaagcaagcaacatgg3834
alaglyproargpheglulysalaleualalysglnalathrtrp
126512701275
cagcagccgatagcacaacttttgaggaacactgcaccgtcatca3879
glnglnproilealaglnleuleuargasnthralaproserser
128012851290
ccgagcagaaaagggcaaaggtcgatgtgctcaatcgcctctgca3924
proserarglysglyglnargsermetcysserilealaserala
129513001305
aggggggagtttctggatcgaacttatcggcgtcaaaggaacccc3969
argglyglupheleuaspargthrtyrargargglnargasnpro
131013151320
gccgaagcttcctccaacactgaatggggagtcaggtggcgtttt4014
alaglualaserserasnthrglutrpglyvalargtrpargphe
132513301335
atccttcgcgccaaaacattccaggtggcgttccatagcaagcat4059
ileleuargalalysthrpheglnvalalaphehisserlyshis
134013451350
cttaaaatttgtgcaaatacatgcaagaattaa4092
leulysilecysalaasnthrcyslysasn
13551360
<210>21
<211>1363
<212>prt
<213>小立碗藓(physcomitellapatens)
<400>21
metvalleuleumetasnargasnalaleuglyilehispheglyala
151015
argalaserleualavalmetvalilehistrptyrleuargprogly
202530
gluleuthrprocysserprothrasnglyproleutrplystrpmet
354045
argthrileleupropheprovalprovalaspleulysarggluala
505560
valleualaglythrvalvalvaltyrservalleulysmetproval
65707580
leutrpserserserleuaspalaaspalaaspalaalaalathrasn
859095
arghisserserserthrphevalargaspglytrpargalaleuthr
100105110
glyargglyglnglyprolysleuargargvalgluglymetgluthr
115120125
argvalvalseralaglugluleuvalleuprometvalalaleuala
130135140
trpvalvalvalhislyspheleutrpalaleutrplysleutrparg
145150155160
seralavalaspaspileasnileglyargthrlysargleucyspro
165170175
thrserserglnmetalaargglyglycyscystrpserglyprophe
180185190
cysleuglyserleuserarglysasnlyslysargilevalproala
195200205
thrargvalhisaspglythralaglnproseraspproglnglygln
210215220
phealapheleuleualaproproserserproalasertyralaasn
225230235240
sermetalaproserservalglnserprotyrtyrprosersercys
245250255
provalproglnglyglyglyserargileproleugluthrglnser
260265270
asnmetphealavalglyprotyralahisgluthralaleuvalser
275280285
proprovalpheserthrphethrthralaproserthralaprophe
290295300
thrproproprogluleualaalahisphethrthrproserserpro
305310315320
aspvalprophealalysleuleuglyserserphesergluglnarg
325330335
thrthrlysargglualagluproprotyrseralaserpropheala
340345350
serproasptyrtyrglnglnasphishisproglnaspaspleugln
355360365
valglytyrglnleutyrproglyserproleuglyargleuileser
370375380
proalaglythrthrglyalaserthrprophealaalaglyglythr
385390395400
thrglythrasnthrprohisalagluseraspasnprothrproleu
405410415
thrvalleuproalavalvalserthrleuproasnleugluhisgln
420425430
valalagluglyleuhisglnargserileleuaspserglncysgly
435440445
proglygluproleuseraspserglyarggluarghisglyserphe
450455460
aspserhisserargphemetvalalametilehisgluargaspgly
465470475480
seraspserasnserasnsertyrglyhisgluargtyrserglyser
485490495
leuthrglyleuasnaspvalleugluglyargasnargtyrthrlys
500505510
leulysglnasplysglyproargserserserleuargserhisglu
515520525
lysgluaspaspileglnglugluaspleuleugluleuprometleu
530535540
leuglysergluserserhisglyglyserargalaserargserpro
545550555560
serserargserlysvalglnserlysalaglyserargvalglyser
565570575
argvalgluserlysalaalaserargvalglyserlysalaglyser
580585590
aspalaleulysasptrpasphisasngluaspasnleuleuglutyr
595600605
alaleuserleuleugluglythraspglylysargproglythrile
610615620
glytrpalaprovalaspthrserleuproargglusergluglypro
625630635640
ileglnserserserthrthrargasngluthrglugluasnglulys
645650655
aspthrvaltyrlysserilelysasnglylysglyseraspasnval
660665670
proglnglnvalvalglyalaglyalalysgluvalsergluglythr
675680685
ilethrprovalglyhishisglyseralaproaspcyscysserarg
690695700
cysgluserleuvalserglncysgluglnleuservalalaleulys
705710715720
glualaargarglysglnglnglulysaspargleualaglugluarg
725730735
glulysglnilearghisleuthrglnleuleuglnserglyglugln
740745750
glyserpheleuglnvalleuaspleuleuaspgluhismetproglu
755760765
glutyrleuserargalaserleuphecysprovalserglugluphe
770775780
asnpheseralagluserleuleuglypheserhisglupheargleu
785790795800
alaglutrpserlysleucysproasnaspphealaleuthrleucys
805810815
asngluleuargcysthrphealaaspaspgluasnalaphecysleu
820825830
glygluphemetsertyraspproglyserglnalathrglyargval
835840845
arglysvalalametseralaasntyrglyasnleuaspileglnthr
850855860
metleutrpcysargserleuargargthrhisaspaspasnmetlys
865870875880
thrvallyslysglymetgluvallysarglysserargleuserlys
885890895
thrseralaleuglnlyslysmetproasnserasnvalglualaala
900905910
asntyrproargservalleuglnthrargserglngluthrproser
915920925
alaserproargleuserargprolysseralaglnvalglnglnala
930935940
lyslysleuleulysileleuargleuasphismetleuseraspgly
945950955960
aspglugluglugluthrgluargmetserlyslyslysglyilelys
965970975
glulyslyslysargcysprotyrargleuargserthrserserlys
980985990
ileproleuserarggluglyserproargprovalgluargserval
99510001005
serproleuthrsertrpargmetaspaspalaglymetproglu
101010151020
proleuargglulysleuasnilephelyslysgluhisgluthr
102510301035
metserserglnglnthrserlysgluprovalglnleuasngln
104010451050
glyserglugluglugluasntyrasnaspthrserhisalathr
105510601065
glntrpasnhislysproleuargglyglnalaargglygluser
107010751080
trpglnalaasphisvalglyleuserleulyslysgluprohis
108510901095
asnphegluleuasplysleulysasnilevalalathrmetgln
110011051110
glugluhisargglncysileargserilealagluthrleuglu
111511201125
hishisserargglnilevalleuleuthrlysalaasnserser
113011351140
leuserasnhisalaaspserleuglulysgluleumetvalleu
114511501155
argalamethisilelyssergluserleupheasnalavallys
116011651170
leualaaspleuargvalglycysilelysthrthrleuleuasn
117511801185
alaleuleuphecyshisleucysalagluthrlysglyglyval
119011951200
glyhistyrargglyasnserglyleuglyargileaspleuala
120512101215
argasnsergluleuleuaspleuargleuaspaspleuglyasp
122012251230
gluargglnalathrlysgluaspargarglysglnargleuser
123512401245
proproserasnvalargalaargargglyargvalthrlysgly
125012551260
alaglyproargpheglulysalaleualalysglnalathrtrp
126512701275
glnglnproilealaglnleuleuargasnthralaproserser
128012851290
proserarglysglyglnargsermetcysserilealaserala
129513001305
argglyglupheleuaspargthrtyrargargglnargasnpro
131013151320
alaglualaserserasnthrglutrpglyvalargtrpargphe
132513301335
ileleuargalalysthrpheglnvalalaphehisserlyshis
134013451350
leulysilecysalaasnthrcyslysasn
13551360
<210>22
<211>1886
<212>dna
<213>拟南芥(arabidopsisthaliana)
<220>
<221>cds
<222>(227)..(1543)
<400>22
aagctggactagataacgaccggtgtgagtgagagagagagagagagaattcgatccatc60
attatacaaaaccttcttcgctttttatttgatttatttatccttttctcatttgatttt120
ctcgggaaaatctctctgcgtagtagatccaagtggtggtttctgagtagtagtaagcaa180
gcaaatctggatgttattgtagtcggtgtgaatctgagttcgaaagatgaggaac235
metargasn
1
gttgttaataacagcgttgagactgttaacgccgccgctaccgccatc283
valvalasnasnservalgluthrvalasnalaalaalathralaile
51015
gtcaccgccgagtctcgagtacagccgtcttcttctcagaagggaaga331
valthralagluserargvalglnproserserserglnlysglyarg
20253035
tggggaaaatgttggagtttatattcatgttttggaactcagaagaac379
trpglylyscystrpserleutyrsercyspheglythrglnlysasn
404550
aataaaaggattggtaatgctgtgcttgtacctgaaccggttacatct427
asnlysargileglyasnalavalleuvalprogluprovalthrser
556065
ggagttccggtagttactgttcaaaactcagctacttcaactactgtt475
glyvalprovalvalthrvalglnasnseralathrserthrthrval
707580
gttcttccctttatagctcctccttcatctccagcttcgtttttgcaa523
valleupropheilealaproproserserproalaserpheleugln
859095
tcggatccttcatcggtttctcactcgcctgttggtccactttctctt571
seraspproserservalserhisserprovalglyproleuserleu
100105110115
actagcaatacattctcgcctaaggagcctcaatctgtctttaccgtt619
thrserasnthrpheserprolysgluproglnservalphethrval
120125130
ggaccttatgctaatgaaactcaaccagtcactcctccggtgttctct667
glyprotyralaasngluthrglnprovalthrproprovalpheser
135140145
gcgtttataactgagccatctactgcaccgtatactccacctcctgaa715
alapheilethrgluproserthralaprotyrthrproproproglu
150155160
tcatcagtccatataactacaccttcttcacctgaagtgccctttgct763
serservalhisilethrthrproserserprogluvalpropheala
165170175
cagttgcttacttcttcgttggagctaactcggagggatagtactagt811
glnleuleuthrserserleugluleuthrargargaspserthrser
180185190195
gggatgaatcaaaagttttcgtcttcgcactatgagtttcggtctaat859
glymetasnglnlyspheserserserhistyrglupheargserasn
200205210
caggtgtgtccggggagtcctggtggtggtaatctaatctctcccggg907
glnvalcysproglyserproglyglyglyasnleuileserprogly
215220225
tcagtgatttcaaactctggtacatcttctccttaccctggtaaatca955
servalileserasnserglythrserserprotyrproglylysser
230235240
cccatggttgagtttcgaataggcgagcctccaaagttcttgggtttt1003
prometvalglupheargileglygluproprolyspheleuglyphe
245250255
gagcactttacagctcgtaaatggggatcgaggttcggttctggatcg1051
gluhisphethralaarglystrpglyserargpheglyserglyser
260265270275
atcacacctgttgggcatggttcaggtttggcttcaggcgctctgaca1099
ilethrprovalglyhisglyserglyleualaserglyalaleuthr
280285290
ccaaatggtccagagatagtatctggaaacttaacacccaacaatacc1147
proasnglyprogluilevalserglyasnleuthrproasnasnthr
295300305
acatggcctcttcaaaatcagatctctgaggtcgcttcactggcaaat1195
thrtrpproleuglnasnglnilesergluvalalaserleualaasn
310315320
tcggatcatggctctgaagtcatggtagcagatcacagagtttcgttt1243
serasphisglysergluvalmetvalalaasphisargvalserphe
325330335
gagttaacaggtgaagacgttgcacgttgtcttgcaagcaagctaaat1291
gluleuthrglygluaspvalalaargcysleualaserlysleuasn
340345350355
cgatcacacgacagaatgaacaacaatgaccggatcgaaacagaggag1339
argserhisaspargmetasnasnasnaspargilegluthrgluglu
360365370
agttcatcaacagacataagaagaaacatagagaaaaggtcaggagac1387
serserserthraspileargargasnileglulysargserglyasp
375380385
agagagaacgaacagcatagaattcagaagctgagttcctcatcgatt1435
arggluasngluglnhisargileglnlysleuserserserserile
390395400
ggatctagcaaagaatttaaattcgacaacacgaaagacgagaatatc1483
glyserserlysgluphelyspheaspasnthrlysaspgluasnile
405410415
gagaaggttgcaggaaacagctggagtttcttcccggggttacgatct1531
glulysvalalaglyasnsertrpserphepheproglyleuargser
420425430435
ggagtcagctaaccaaattaaaatgacatctcttcttcttcttcctcattac1583
glyvalser
caaagacagagatataaaaccctataagtaacaaaccttgaacatggatcgtgaaaaaag1643
gtaagagatttgggttctaacaacttatacacactgaataaataagcttaaagctttgat1703
ttgcagttaacttctgtactcatgtagttggaaactttggaatattcttggatcagctcc1763
tacatctaatcttgttacattgatataaattgtttatttcttgttttactgtttcataca1823
aatatattgtttgaactgagatcgtgaatggtcttattctatatcattactaaaacattt1883
tca1886
<210>23
<211>438
<212>prt
<213>拟南芥(arabidopsisthaliana)
<400>23
metargasnvalvalasnasnservalgluthrvalasnalaalaala
151015
thralailevalthralagluserargvalglnprosersersergln
202530
lysglyargtrpglylyscystrpserleutyrsercyspheglythr
354045
glnlysasnasnlysargileglyasnalavalleuvalproglupro
505560
valthrserglyvalprovalvalthrvalglnasnseralathrser
65707580
thrthrvalvalleupropheilealaproproserserproalaser
859095
pheleuglnseraspproserservalserhisserprovalglypro
100105110
leuserleuthrserasnthrpheserprolysgluproglnserval
115120125
phethrvalglyprotyralaasngluthrglnprovalthrpropro
130135140
valpheseralapheilethrgluproserthralaprotyrthrpro
145150155160
proprogluserservalhisilethrthrproserserprogluval
165170175
prophealaglnleuleuthrserserleugluleuthrargargasp
180185190
serthrserglymetasnglnlyspheserserserhistyrgluphe
195200205
argserasnglnvalcysproglyserproglyglyglyasnleuile
210215220
serproglyservalileserasnserglythrserserprotyrpro
225230235240
glylysserprometvalglupheargileglygluproprolysphe
245250255
leuglyphegluhisphethralaarglystrpglyserargphegly
260265270
serglyserilethrprovalglyhisglyserglyleualasergly
275280285
alaleuthrproasnglyprogluilevalserglyasnleuthrpro
290295300
asnasnthrthrtrpproleuglnasnglnilesergluvalalaser
305310315320
leualaasnserasphisglysergluvalmetvalalaasphisarg
325330335
valserphegluleuthrglygluaspvalalaargcysleualaser
340345350
lysleuasnargserhisaspargmetasnasnasnaspargileglu
355360365
thrglugluserserserthraspileargargasnileglulysarg
370375380
serglyasparggluasngluglnhisargileglnlysleuserser
385390395400
serserileglyserserlysgluphelyspheaspasnthrlysasp
405410415
gluasnileglulysvalalaglyasnsertrpserphepheprogly
420425430
leuargserglyvalser
435
<210>24
<211>1994
<212>dna
<213>拟南芥(arabidopsisthaliana)
<220>
<221>cds
<222>(384)..(1733)
<400>24
gataattttaatattttaacacaataatagcgaataaataatatatacatggaagcgcgt60
gggtccaaaccgtcaaattatgatgacgtggcaaaatctcgtgggaaattgaatgaaaac120
tgtaaaagataacgaccggtgacagagagagagagagaggagaagaagaatagaagaagc180
ggctcatatccataataaaagccatttgctcctttatttattctctcattctttcttctt240
cattattcatttctctcgttattcgtttcgtattttgtttgtgtttatgattttacctcc300
ttagttgctgtgagatcaagaagtggtgaaagaaaagttactaaatctggatgttatttg360
attgtcgtcgttggtcttgagctatgaggagcgttaataatagtagtgtcgac413
metargservalasnasnserservalasp
1510
accgtgaacgccgccgcttccgccatcgtctccgctgagtctagaaca461
thrvalasnalaalaalaseralailevalseralagluserargthr
152025
caaccgtcgtcggttcagaaaaaaaggggaagctggtggagcttgtac509
glnproserservalglnlyslysargglysertrptrpserleutyr
303540
tggtgttttggatccaagaagaacaataaaaggataggccacgcggtg557
trpcyspheglyserlyslysasnasnlysargileglyhisalaval
455055
cttgtacccgaaccagctgcatcaggagctgcggtggctccagtccaa605
leuvalprogluproalaalaserglyalaalavalalaprovalgln
606570
aactcttcgagcaattctacttcaatattcatgccctttatagctcct653
asnserserserasnserthrserilephemetpropheilealapro
75808590
ccttcatctcctgcttcctttctgccatcaggtcctccctctgcgtca701
proserserproalaserpheleuproserglyproproseralaser
95100105
catactcctgatcctggtctactttgttccctaaccgtcaatgaaccg749
histhrproaspproglyleuleucysserleuthrvalasnglupro
110115120
ccttcagcctttactattggaccatacgctcatgagactcaacctgtt797
proseralaphethrileglyprotyralahisgluthrglnproval
125130135
actcctccagtgttctctgctttcacaacggaaccctccaccgcgcca845
thrproprovalpheseralaphethrthrgluproserthralapro
140145150
ttcacgccacctcctgaatcaccttcttcccctgaagtgccttttgct893
phethrproproprogluserproserserprogluvalpropheala
155160165170
cagttacttacatcttcattggaaagggctaggaggaacagtggtggt941
glnleuleuthrserserleugluargalaargargasnserglygly
175180185
ggaatgaatcagaagttttcagctgcacactacgagtttaagtcttgt989
glymetasnglnlyspheseralaalahistyrgluphelyssercys
190195200
caagtgtatcctggaagtccaggtggtaatctaatctctcctggttca1037
glnvaltyrproglyserproglyglyasnleuileserproglyser
205210215
ggtacatcttctccttacccagggaaatgctccatcatcgagtttcgt1085
glythrserserprotyrproglylyscysserileilegluphearg
220225230
atcggcgaacctccaaagtttcttggttttgagcacttcacagcgcgt1133
ileglygluproprolyspheleuglyphegluhisphethralaarg
235240245250
aaatggggatcaagattcggttctggatccatcacacctgctggacaa1181
lystrpglyserargpheglyserglyserilethrproalaglygln
255260265
ggttcaaggttgggttcaggtgctttgactcctgatggctcaaagcta1229
glyserargleuglyserglyalaleuthrproaspglyserlysleu
270275280
acttctggtgtagtgacaccaaatggtgcagagactgttataagaatg1277
thrserglyvalvalthrproasnglyalagluthrvalileargmet
285290295
agttatgggaatctcacaccacttgaaggcagtcttttggatagtcag1325
sertyrglyasnleuthrproleugluglyserleuleuaspsergln
300305310
atctctgaggttgcgtctttagccaattcggaccacgggtcgtcaagg1373
ilesergluvalalaserleualaasnserasphisglyserserarg
315320325330
cataatgatgaagctctcgtggttcctcacagagtttctttcgagttg1421
hisasnaspglualaleuvalvalprohisargvalserphegluleu
335340345
actggtgaagacgttgcacggtgtcttgcaagcaagctaaaccgttcc1469
thrglygluaspvalalaargcysleualaserlysleuasnargser
350355360
ggttcacatgaaaaagcaagcggcgaacatttaagaccaaactgttgt1517
glyserhisglulysalaserglygluhisleuargproasncyscys
365370375
aaaacgtcgggagaaacagagagcgaacagagtcagaagctaagatcg1565
lysthrserglygluthrglusergluglnserglnlysleuargser
380385390
ttttctacaggctctaacaaagaattcaagtttgatagcaccaatgaa1613
pheserthrglyserasnlysgluphelyspheaspserthrasnglu
395400405410
gagatgatagagaaaattcgatcggagtggtgggcgaatgagaaggtc1661
glumetileglulysileargserglutrptrpalaasnglulysval
415420425
gccggaaaaggtgatcacagtccaagaaacagttggactttctttcca1709
alaglylysglyasphisserproargasnsertrpthrphephepro
430435440
gtcttacgctctggacatacttagcacaaagaaatagcttttaccttcttcatt1763
valleuargserglyhisthr
445
acctctaacatgggaagcaaagtcagtgatatggtaagagattggggtctaacaactata1823
catatatatagcttggtattgtaggtgtctttgttctttgtactgttaattatataaaat1883
agttttaacacttaaatggtagacttatcattgacaaaagaaaagaaactgtattctttc1943
ttggttatgtataaacataaaaatcatctctttactatattgtcttacatt1994
<210>25
<211>449
<212>prt
<213>拟南芥(arabidopsisthaliana)
<400>25
metargservalasnasnserservalaspthrvalasnalaalaala
151015
seralailevalseralagluserargthrglnproserservalgln
202530
lyslysargglysertrptrpserleutyrtrpcyspheglyserlys
354045
lysasnasnlysargileglyhisalavalleuvalprogluproala
505560
alaserglyalaalavalalaprovalglnasnserserserasnser
65707580
thrserilephemetpropheilealaproproserserproalaser
859095
pheleuproserglyproproseralaserhisthrproaspprogly
100105110
leuleucysserleuthrvalasngluproproseralaphethrile
115120125
glyprotyralahisgluthrglnprovalthrproprovalpheser
130135140
alaphethrthrgluproserthralaprophethrproproproglu
145150155160
serproserserprogluvalprophealaglnleuleuthrserser
165170175
leugluargalaargargasnserglyglyglymetasnglnlysphe
180185190
seralaalahistyrgluphelyssercysglnvaltyrproglyser
195200205
proglyglyasnleuileserproglyserglythrserserprotyr
210215220
proglylyscysserileileglupheargileglygluproprolys
225230235240
pheleuglyphegluhisphethralaarglystrpglyserargphe
245250255
glyserglyserilethrproalaglyglnglyserargleuglyser
260265270
glyalaleuthrproaspglyserlysleuthrserglyvalvalthr
275280285
proasnglyalagluthrvalileargmetsertyrglyasnleuthr
290295300
proleugluglyserleuleuaspserglnilesergluvalalaser
305310315320
leualaasnserasphisglyserserarghisasnaspglualaleu
325330335
valvalprohisargvalserphegluleuthrglygluaspvalala
340345350
argcysleualaserlysleuasnargserglyserhisglulysala
355360365
serglygluhisleuargproasncyscyslysthrserglygluthr
370375380
glusergluglnserglnlysleuargserpheserthrglyserasn
385390395400
lysgluphelyspheaspserthrasngluglumetileglulysile
405410415
argserglutrptrpalaasnglulysvalalaglylysglyasphis
420425430
serproargasnsertrpthrphepheprovalleuargserglyhis
435440445
thr
<210>26
<211>1947
<212>dna
<213>拟南芥(arabidopsisthaliana)
<220>
<221>cds
<222>(374)..(1669)
<400>26
attctgattaaaatagaaatgctttttccttggtaaggatcatcttttcatgccgattgc60
atcagtgcagatttccttatttactctttctcttctcaaatttccatttttttctttatc120
tctcccactcccactcataaaattagttttgtctttctttccccaaaaatctttacttaa180
aatctttttcacccttttgctttaacttttgtttgctcctcctctggatcgtatcagtaa240
actggaagaagaaattgtgagaaaaaatggagaaaaccaagatgattacgtctatgacct300
gttgaagaaaccctactcagaataagagtaaaactctgtgatttttctgactaacagaga360
gagagacacctaaatgggctcagagcaggatcagaggaaaaggtgggga409
metglysergluglnaspglnarglysargtrpgly
1510
ggttgtttaggagtgttctcttgtttcaagtcacaaaaaggtggaaaa457
glycysleuglyvalphesercysphelysserglnlysglyglylys
152025
agaattgtacctgcttctcgcattcctgaaggtggcaatgtctcagct505
argilevalproalaserargileprogluglyglyasnvalserala
303540
tcacaacctaatggagctcatcaagctggtgtcttaaataatcaagct553
serglnproasnglyalahisglnalaglyvalleuasnasnglnala
45505560
gcaggagggattaatctatcactattggctccaccttcctctcctgct601
alaglyglyileasnleuserleuleualaproproserserproala
657075
tccttcaccaattcagctcttccttcaactactcagtctccaaactgc649
serphethrasnseralaleuproserthrthrglnserproasncys
808590
tacttatctctggctgcaaattcaccgggaggtccttcgtcgagtatg697
tyrleuserleualaalaasnserproglyglyprosersersermet
95100105
tatgccactggaccatatgctcatgaaacccaattagtctcaccgcct745
tyralathrglyprotyralahisgluthrglnleuvalserpropro
110115120
gttttctctactttcaccaccgagccatcgactgctcctttcactcct793
valpheserthrphethrthrgluproserthralaprophethrpro
125130135140
cctccagagcttgcacgtctgactgccccttcttcacctgatgtacct841
proprogluleualaargleuthralaproserserproaspvalpro
145150155
tatgctcgtttcttgacttcctccatggatctcaagaactctggtaaa889
tyralaargpheleuthrsersermetaspleulysasnserglylys
160165170
ggtcattacaatgatcttcaagctacttattctctttatcccggaagt937
glyhistyrasnaspleuglnalathrtyrserleutyrproglyser
175180185
ccagccagtgctcttagatcaccaatctctcgggcttcgggagatggg985
proalaseralaleuargserproileserargalaserglyaspgly
190195200
ttattgtcgcctcaaaatggtaaatgctcaaggagtgattctggcaac1033
leuleuserproglnasnglylyscysserargseraspserglyasn
205210215220
acattcgggtatgacacaaatggagtctcaacacctttgcaggagtca1081
thrpheglytyraspthrasnglyvalserthrproleuglngluser
225230235
aacttcttctgtcctgaaacttttgccaagttttacctggatcacgac1129
asnphephecysprogluthrphealalysphetyrleuasphisasp
240245250
ccttcagttcctcaaaacggtggaagattaagcgtgtcgaaggattca1177
proservalproglnasnglyglyargleuservalserlysaspser
255260265
gatgtgtatcctacaaatggatatggaaacgggaaccagaataggcag1225
aspvaltyrprothrasnglytyrglyasnglyasnglnasnarggln
270275280
aacagaagtcccaagcaagacatggaggagttagaagcttacagggcg1273
asnargserprolysglnaspmetglugluleuglualatyrargala
285290295300
tcctttggttttagtgcagatgaaatcatcacaactagtcagtatgta1321
serpheglypheseralaaspgluileilethrthrserglntyrval
305310315
gagatcactgatgtgatggatgggtcttttaacacatcagcttactct1369
gluilethraspvalmetaspglyserpheasnthrseralatyrser
320325330
ccaagcgatggacaaaagcttctcagaagagaagcaaatttgctgagt1417
proseraspglyglnlysleuleuargargglualaasnleuleuser
335340345
caaacaagccccaaatcagaagccgatcttgattcacaagttgtagac1465
glnthrserprolysserglualaaspleuaspserglnvalvalasp
350355360
ttccaatcaccaaagtcatcaaatagctacaaagatcacaaacaaaga1513
pheglnserprolysserserasnsertyrlysasphislysglnarg
365370375380
aaccggatccatgcggatgaagaggctttattgtcgagagtggggtct1561
asnargilehisalaaspgluglualaleuleuserargvalglyser
385390395
gtaaaaggaagcagaagctatcatatttcaagctctgatgcagaggtc1609
vallysglyserargsertyrhisileserserseraspalagluval
400405410
gagtacagaagaggaagaagcttaagggaaagcagagagaacagacac1657
glutyrargargglyargserleuarggluserarggluasnarghis
415420425
aggaaagcctgatcaagaacaacaacaacaagaagaagaaagcctgatctgt1709
arglysala
430
tcttaaagtcttctaaggtctaaaaagttgatcttgtgatgctaactcttgagataagaa1769
gtctagtttttaagtatatatagatatatataaatataattacagatggttttttagtag1829
aatgatgttatatataagaagagagcaagggtgagaaagtgaatgcttgttttttagttt1889
gacatggcttttttgtaagaccccatcttttatctaattgcaccaatctgtttgagct1947
<210>27
<211>431
<212>prt
<213>拟南芥(arabidopsisthaliana)
<400>27
metglysergluglnaspglnarglysargtrpglyglycysleugly
151015
valphesercysphelysserglnlysglyglylysargilevalpro
202530
alaserargileprogluglyglyasnvalseralaserglnproasn
354045
glyalahisglnalaglyvalleuasnasnglnalaalaglyglyile
505560
asnleuserleuleualaproproserserproalaserphethrasn
65707580
seralaleuproserthrthrglnserproasncystyrleuserleu
859095
alaalaasnserproglyglyprosersersermettyralathrgly
100105110
protyralahisgluthrglnleuvalserproprovalpheserthr
115120125
phethrthrgluproserthralaprophethrproproprogluleu
130135140
alaargleuthralaproserserproaspvalprotyralaargphe
145150155160
leuthrsersermetaspleulysasnserglylysglyhistyrasn
165170175
aspleuglnalathrtyrserleutyrproglyserproalaserala
180185190
leuargserproileserargalaserglyaspglyleuleuserpro
195200205
glnasnglylyscysserargseraspserglyasnthrpheglytyr
210215220
aspthrasnglyvalserthrproleuglngluserasnphephecys
225230235240
progluthrphealalysphetyrleuasphisaspproservalpro
245250255
glnasnglyglyargleuservalserlysaspseraspvaltyrpro
260265270
thrasnglytyrglyasnglyasnglnasnargglnasnargserpro
275280285
lysglnaspmetglugluleuglualatyrargalaserpheglyphe
290295300
seralaaspgluileilethrthrserglntyrvalgluilethrasp
305310315320
valmetaspglyserpheasnthrseralatyrserproseraspgly
325330335
glnlysleuleuargargglualaasnleuleuserglnthrserpro
340345350
lysserglualaaspleuaspserglnvalvalasppheglnserpro
355360365
lysserserasnsertyrlysasphislysglnargasnargilehis
370375380
alaaspgluglualaleuleuserargvalglyservallysglyser
385390395400
argsertyrhisileserserseraspalagluvalglutyrargarg
405410415
glyargserleuarggluserarggluasnarghisarglysala
420425430
<210>28
<211>22
<212>dna
<213>人工序列
<220>
<223>引物
<400>28
ggaagttcatttattcggagag22
<210>29
<211>22
<212>dna
<213>人工序列
<220>
<223>引物
<400>29
ggcaacaggattcaatcttaag22
<210>30
<211>19
<212>dna
<213>人工序列
<220>
<223>引物
<400>30
cgccatccaagctgttctc19
<210>31
<211>20
<212>dna
<213>人工序列
<220>
<223>引物
<400>31
tcacgtccagcaaggtcaag20
<210>32
<211>19
<212>dna
<213>人工序列
<220>
<223>引物
<400>32
cattcgtctctcgggtcca19
<210>33
<211>20
<212>dna
<213>人工序列
<220>
<223>引物
<400>33
tcttcggcgaagctgatcta20
<210>34
<211>20
<212>dna
<213>人工序列
<220>
<223>引物
<400>34
cgagaaaattctcagactca20
<210>35
<211>20
<212>dna
<213>人工序列
<220>
<223>引物
<400>35
aagcagctgcgtttatagta20
<210>36
<211>20
<212>dna
<213>人工序列
<220>
<223>引物
<400>36
ggtggtttctgagtagtagt20
<210>37
<211>19
<212>dna
<213>人工序列
<220>
<223>引物
<400>37
agtctcaacgctgttatta19
<210>38
<211>20
<212>dna
<213>人工序列
<220>
<223>引物
<400>38
gttatttgattgtcgtcgtt20
<210>39
<211>19
<212>dna
<213>人工序列
<220>
<223>引物
<400>39
ttctagactcagcggagac19
<210>40
<211>20
<212>dna
<213>人工序列
<220>
<223>引物
<400>40
ctgttgaagaaaccctactc20
<210>41
<211>18
<212>dna
<213>人工序列
<220>
<223>引物
<400>41
accttttcctctgatcct18
<210>42
<211>24
<212>dna
<213>人工序列
<220>
<223>引物
<400>42
caccatgagaagcggtgctaatgg24
<210>43
<211>21
<212>dna
<213>人工序列
<220>
<223>引物
<400>43
ttagcttagtgtacctgactg21
<210>44
<211>24
<212>dna
<213>人工序列
<220>
<223>引物
<400>44
caccatgcagagtgggagcgagat24
<210>45
<211>19
<212>dna
<213>人工序列
<220>
<223>引物
<400>45
ttagctgacccctggctgt19
<210>46
<211>39
<212>dna
<213>人工序列
<220>
<223>引物
<400>46
aggatttatcacaagtttgtacaaaaaagcaggctccgc39
<210>47
<211>40
<212>dna
<213>人工序列
<220>
<223>引物
<400>47
gtttggtgttactcctgcaggatttatcacaagtttgtac40
<210>48
<211>63
<212>dna
<213>人工序列
<220>
<223>引物
<400>48
caccactttgtacaagaaagctgggtcggcgcgcccacccttttagcttagtgtacctga60
ctg63
<210>49
<211>54
<212>dna
<213>人工序列
<220>
<223>引物
<400>49
ggccgatttggcccctgcaggatttatcaccactttgtacaagaaagctgggtc54
- 还没有人留言评论。精彩留言会获得点赞!