基于VH‑VL域间角的抗体人源化的制作方法
本发明处于抗体人源化领域中。本文中报道了一种用于抗体人源化的方法,其包括把供体残基移植到受体构架上,其中根据所述的人源化的抗体和供体抗体的vh-vl域间角进行受体构架的选择。
背景技术:
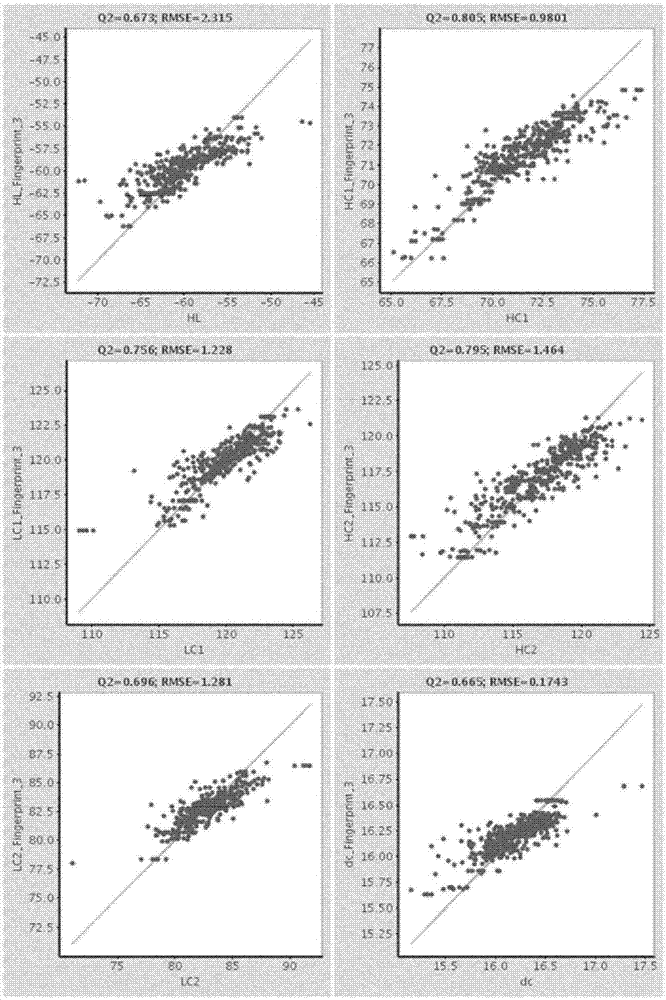
:抗体的抗原结合位点是在重链和轻链可变域vh和vl的界面处形成的,使得vh-vl结构域取向成为影响抗体特异性和亲和性的因素。为了保留供体抗体性质,在抗体工程化和人源化的过程中保存vh-vl结构域取向将会是有利的。预测正确的vh-vl取向已经被认识到是抗体同源性建模中的一个因素。在wo2011/021009中,具有改善的可制造性的变体免疫球蛋白与下列发现相关:修饰免疫球蛋白分子的某些关键位置中的氨基酸序列导致在可制造性方面的改善,特别是导致聚集倾向的减小和/或生产水平的提高。在wo2008/003931中报道了一种用于人源化的抗体的构架筛选的方法,由此通过考虑人受体构架与供体序列的同源性,可能选择最合适的可变区构架,但是更重要地,考虑选择其中特异性残基是专性供体残基的那些可变区构架,即给予加权。因此,已经存在于同源的人构架中的这些加权的(重要的)供体残基越多,所述的人构架越合适,无论总的同源性是否稍微小于具有较少的加权残基匹配的另一个构架。在wo2001/027160(ep1224224)中报道了一种单克隆抗体生产和特异性地同时对单克隆抗体可变区的多个不同结构域进行体外亲和性最优化的方法。通过产生cdr移植的可变区片段的多样库,然后针对结合活性类似于或好于供体的结合活性来筛选所述的库,来完成移植。多样库是通过选择在相应的位置处与供体构架相比不同的受体构架位置并且使得库群体在那些位置中的每一个处包含所有可能的氨基酸残基变化以及在所述的可变区的cdr内的每一个位置处包含所有可能的氨基酸残基变化而产生的。dunbar,j.等人(prot.eng.des.sel.26(2013)611-620)在表征抗体中的vh-vl取向时报道了ab角。bujotzek,a.等人(proteins:structure,function,andbioinformatics83(2015)681-695)报道了vh-vl结构域取向的预测,用于抗体可变域建模。发明概述本文中报道了用于把抗体人源化的基于序列的快速方法,其基于重链和轻链可变域取向、vh-vl域间取向(角)的确定。采用本文中报道的方法,提供了改善的,即更快的、更经济的、更少资源需求的以及更有效的筛选最好的合适的非人抗体的人源化变体。更详细地,根据本文中报道的方法使用基于序列的快速预测子,其预测vh-vl域间取向。用六个绝对ab角参数来描述所述的vh-vl取向,以把vh-vl取向的不同自由度精确地分开。已经发现:采用根据本文中报道的方法,可以实现针对变体(人源化的)抗体的vh-vl取向相对于亲本(非人)抗体的偏差方面,在人源化的抗体的选择方面的改善。这表面关于在亲本(非人)和变体(人源化的)抗体之间vh-vl域间角的相似性方面的改善。与采用不同的方法得到的人源化的抗体比较,根据本文中报道的方法(包括移植方法)正在递送变体(人源化的)抗体的较好的结合性质。可以把其它工程化方法例如构架改组与根据本文中报道的方法组合,导致当一种人构架被交换为另一种人构架以改变抗体的生物物理性能时,所获得的变体抗体的改善的结合。正如根据本文中报道的那样,一个方面是用于筛选来源于亲本抗体fv片段的一种或更多种变体抗体fv片段的方法,其包括下列步骤:-通过把来自亲本抗体fv片段的一个或更多个决定特异性的残基移植/转移到受体抗体fv片段上而产生许多变体抗体fv片段,由此许多变体抗体fv片段中的每一个变体抗体fv片段与其它变体抗体fv片段至少有一个氨基酸残基不同,-基于抗体fv片段的序列指纹,确定亲本fv片段和许多变体抗体fv片段中的每一个变体抗体fv片段的vh-vl取向,-选择与亲本抗体的vh-vl取向比较,在vh-vl取向方面具有最小差别的那些变体抗体fv片段并且从而选择来源于亲本抗体fv片段的一种或更多种变体抗体fv片段,由此所述的一种或更多种变体抗体fv片段和亲本抗体fv片段与相同的抗原结合。在一种实施方式中,所述的方法包括下列步骤:-选择与亲本抗体的vh-vl域间角比较,在vh-vl域间角方面具有最高的(结构上的)相似度的那些变体抗体fv片段并且从而选择来源于亲本抗体fv片段的一种或更多种变体抗体fv片段。一个方面是用于筛选来源于亲本抗体fv片段的一种或更多种变体抗体fv片段的方法,其包括下列步骤:-通过把来自亲本抗体fv片段的一个或更多个决定特异性的残基移植/转移到受体抗体fv片段上而产生许多变体抗体fv片段,由此许多变体抗体fv片段中的每一个变体抗体fv片段与其它变体抗体fv片段至少有一个氨基酸残基不同,-基于抗体fv片段的序列指纹,确定亲本fv片段和许多变体抗体fv片段中的每一个变体抗体fv片段的vh-vl取向,-选择与亲本抗体的vh-vl域间角比较,在vh-vl域间角方面具有最高的(结构上的)相似度的那些变体抗体fv片段并且从而选择来源于亲本抗体fv片段的一种或更多种变体抗体fv片段,由此所述的一种或更多种变体抗体fv片段和亲本抗体fv片段与相同的抗原结合。在一种实施方式中,亲本抗体fv片段是非人抗体fv片段。在一种实施方式中,受体抗体fv片段是人或人源化的抗体fv片段或人抗体fv片段种系氨基酸序列。正如根据本文中报道的那样,一个方面是用于把非人抗体人源化的方法,其包括下列步骤-提供与抗原特异性地结合的非人抗体,-通过把来自非人抗体的一个或更多个决定特异性的残基移植/转移到人或人源化的受体抗体或种系抗体序列上而产生许多变体抗体,由此许多变体抗体中的每一种变体抗体与其它变体抗体至少有一个氨基酸残基不同,-基于抗体fv片段的序列指纹,确定非人抗体的fv片段和许多变体抗体中的每一种变体抗体的fv片段的vh-vl取向,-选择与亲本抗体的vh-vl取向比较,在vh-vl取向方面具有最小差别的那些变体抗体fv片段并且从而选择来源于非人的一种或更多种人源化的抗体,由此所述的一种或更多种人源化的抗体和非人抗体与相同的抗原结合。在一种实施方式中,所述的方法包括下列步骤:-选择与亲本抗体的vh-vl域间角比较,在vh-vl域间角方面具有最高的(结构上的)相似度的那些变体抗体fv片段并且从而选择来源于非人抗体的一种或更多种人源化的抗体。一个方面是用于把非人抗体人源化的方法,其包括下列步骤:-提供与抗原特异性地结合的非人抗体,-通过把来自非人抗体的一个或更多个决定特异性的残基移植/转移到人或人源化的受体抗体或种系抗体序列上而产生许多变体抗体,由此许多变体抗体中的每一种变体抗体与其它变体抗体至少有一个氨基酸残基不同,-基于抗体fv片段的序列指纹,确定非人抗体的fv片段和许多变体抗体中的每一种变体抗体的fv片段的vh-vl取向,-选择与亲本抗体的vh-vl域间角比较,在vh-vl域间角方面具有最高的(结构上的)相似度的那些变体抗体fv片段并且从而选择来源于非人抗体的一种或更多种人源化的抗体,由此所述的一种或更多种人源化的抗体和非人抗体一样与相同的抗原结合。在如本文报道的所有方面的一个实施方式中,所述的序列指纹是一组vh-vl界面残基。在如本文报道的所有方面的一个实施方式中,vh-vl界面残基是氨基酸残基,其侧链原子具有以小于或等于(在至少90%的所有重叠的fv结构中)的距离邻接相反链的原子。在如本文报道的所有方面的一个实施方式中,所述组的vh-vl界面残基包括残基l44、l46、l87、h45、h62(根据chothia指标编号)。在如本文报道的所有方面的一个实施方式中,所述组的vh-vl界面残基包括残基h35、h37、h39、h45、h47、h50、h58、h60、h61、h91、h95、h96、h98、h100x-2、h100x-1、h100x、h101、h102、h103、h105、l32、l34、l36、l38、l43、l44、l46、l49、l50、l55、l87、l89、l91、l95x-1、l95x、l96(根据chothia指标编号)。在如本文报道的所有方面的一个实施方式中,所述组的vh-vl界面残基包括残基h33、h35、h43、h44、h46、h50、h55、h56、h58、h61、h62、h89、h99、l34、l36、l38、l41、l42、l43、l44、l45、l46、l49、l50、l53、l55、l56、l85、l87、l89、l91、l93、l94/l95x-1、l95x、l96、l97、l100(根据chothia指标编号)。在本文中报告的所有方面当中的一种实施方式中,所述组的vh-vl界面残基包括残基h33、h35、h37、h39、h43、h44、h45、h46、h47、h50、h55、h56、h58、h60、h61、h62、h89、h91、h95、h96、h98、h99、h100x-2、h100x-1、h100x、h101、h102、h103、h105、l32、l34、l36、l38、l41、l42、l43、l44、l45、l46、l49、l50、l53、l55、l56、l85、l87、l89、l91、l93、l94/l95x-1、l95x、l96、l97、l100(根据chothia指标编号)。在如本文报道的所有方面的一个实施方式中,所述组的vh-vl界面残基包括残基h35、h37、h39、h45、h47、h50、h58、h60、h61、h91、h95、h96、h98、h100x-2、h100x-1、h100x、h101、h102、h103、h105、l32、l34、l36、l38、l43、l44、l46、l49、l50、l55、l87、l89、l91、l95x-1、l95x、l96、l98(根据chothia指标编号)。在本文中报告的所有方面当中的一种实施方式中,所述组的vh-vl界面残基包括残基h33、h35、h37、h39、h43、h44、h45、h46、h47、h50、h55、h56、h58、h60、h61、h62、h89、h91、h95、h96、h98、h99、h100x-2、h100x-1、h100x、h101、h102、h103、h105、l32、l34、l36、l38、l41、l42、l43、l44、l45、l46、l49、l50、l53、l55、l56、l85、l87、l89、l91、l93、l94/l95x-1、l95x、l96、l97、l98、l100(根据chothia指标编号)。在如本文报道的所有方面的一个实施方式中,所述组的vh-vl界面残基包括残基210、296、610、612、733(根据wolfguy指标编号)。在如本文报道的所有方面的一个实施方式中,所述组的vh-vl界面残基包括残基199、202、204、210、212、251、292、294、295、329、351、352、354、395、396、397、398、399、401、403、597、599、602、604、609、610、612、615、651、698、733、751、753、796、797、798(根据wolfguy指标编号)。在如本文报道的所有方面的一个实施方式中,所述组的vh-vl界面残基包括残基197、199、208、209、211、251、289、290、292、295、296、327、355、599、602、604、607、608、609、610、611、612、615、651、696、698、699、731、733、751、753、755、796、797、798、799、803(根据wolfguy指标编号)。在如本文报道的所有方面的一个实施方式中,所述组的vh-vl界面残基包括残基197、199、202、204、208、209、210、211、212、251、292、294、295、296、327、329、351、352、354、355、395、396、397、398、399、401、403、597、599、602、604、607、608、609、610、611、612、615、651、696、698、699、731、733、751、753、755、796、796、797、798、799、801、803(根据wolfguy指标编号)。在如本文报道的所有方面的一个实施方式中,所述组的vh-vl界面残基包括残基199、202、204、210、212、251、292、294、295、329、351、352、354、395、396、397、398、399、401、403、597、599、602、604、609、610、612、615、651、698、733、751、753、796、797、798、801(根据wolfguy指标编号)。在如本文报道的所有方面的一个实施方式中,所述组的vh-vl界面残基包括残基197、199、202、204、208、209、210、211、212、251、292、294、295、296、327、329、351、352、354、355、395、396、397、398、399、401、403、597、599、602、604、607、608、609、610、611、612、615、651、696、698、699、731、733、751、753、755、796、797、798、799、801、803(根据wolfguy指标编号)。在如本文报道的所有方面的一个实施方式中,关于vh-vl取向,所述的选择/筛选基于顶部80%变体抗体fv片段。在如本文报道的所有方面的一个实施方式中,关于vh-vl取向,所述的选择/筛选是基于顶部20%变体抗体fv片段的。在如本文报道的所有方面的一个实施方式中,关于vh-vl取向,所述的选择是取消选定最差的20%变体抗体fv片段。在如本文报道的所有方面的一个实施方式中,通过计算六个ab角vh-vl取向参数来确定vh-vl取向。在如本文报道的所有方面的一个实施方式中,通过使用随机的森林方法计算所述的ab角vh-vl取向参数来确定vh-vl取向。在如本文报道的所有方面的一个实施方式中,通过对于每一个ab角使用一种随机的森林方法计算所述的ab角vh-vl取向参数来确定vh-vl取向。在如本文报道的所有方面的一个实施方式中,通过使用随机森林模型计算vh和vl的惯常的扭转角、四个弯曲角(每个可变域两个)和中心轴长度(hl、hc1、lc1、hc2、lc2、dc)来确定vh-vl取向。在如本文报道的所有方面的一个实施方式中,仅仅采用复合物抗体结构数据训练所述的随机森林模型。在如本文报道的所有方面的一个实施方式中,最小的差别是在与最高的q2值有关的真实的和预测的角度参数值之间的最小的差别。在如本文报道的所有方面的一个实施方式中,最小的差别是在与最高的q2值有关的亲本抗体角度参数和人源化的变体抗体角度参数值之间的最小的差别。在如本文报道的所有方面的一个实施方式中,最高的结构相似度是最低的平均均方根偏差(rmsd)。在一种实施方式中,rmsd是针对非人或亲本抗体的氨基酸残基的所有cα原子(或羰基原子)与变体抗体的相应cα原子测定的rmsd。一般而言,通过利用vh-vl预测子,相对于结构参比得到了改善。由vh-vl重取向导致的的减小通常转化成更好的rmsd值,特别是相对于构架区。平均起来,对于完整的fv发现了的显著的改善和羰基rmsd的改善。在如本文报道的所有方面的一个实施方式中,把在从或者共有vh或者vl构架上比对,随后在共有fv构架上进行vh-vl重定向的模板结构装配的模型用于测定vh-vl取向。在如本文报道的所有方面的一个实施方式中,把在完全的fv(vh和vl同时)的β-片层核心上比对的模型用于测定vh-vl取向。在如本文报道的所有方面的一个实施方式中,把其中抗体fv片段在共有fv构架上被重取向的模型用于测定vh-vl取向。在如本文报道的所有方面的一个实施方式中,把利用在常见的共有fv构架上比对的模板结构并且vh-vl取向不以任何形式被调整的模型用于测定vh-vl取向。在如本文报道的所有方面的一个实施方式中,把从在或者共有vh或者vl构架上比对,随后在基于相似度选择的vh-vl取向模板结构上进行vh-vl重取向的模板结构装配的模型用于测定vh-vl取向。根据本文中报道的一个方面是用于生产抗体的方法,其包括下列步骤:-根据在本文中报道的方法选择一种或更多种抗体或抗体fv片段,-基于它的结合性质,从所述的一种或更多种抗体或抗体fv片段中选择单一的抗体或抗体fv片段,-把编码vh和vl的核酸克隆到一种或更多种表达载体中,-采用在前一步骤中获得的表达载体转染细胞,-培养所转染的细胞并且从而生产所述的抗体。根据本文中报道的一个方面是用于生产抗体的方法,其包括下列步骤:-选择一种或更多种抗体或抗体fv片段,其包括下列步骤:-通过把来自非人抗体的一个或更多个决定特异性的残基移植/转移到人或人源化的受体抗体或种系抗体序列上而产生许多变体抗体,由此许多变体抗体中的每一种变体抗体与其它变体抗体至少有一个氨基酸残基不同,-通过利用基于由抗体fv片段的残基h33、h35、h37、h39、h43、h44、h45、h46、h47、h50、h55、h56、h58、h60、h61、h62、h89、h91、h95、h96、h98、h99、h100x-2、h100x-1、h100x、h101、h102、h103、h105、l32、l34、l36、l38、l41、l42、l43、l44、l45、l46、l49、l50、l53、l55、l56、l85、l87、l89、l91、l93、l94/i95x-1、i9sx、l96、l97、l100(根据chothia指标编号)组成的一组vh-vl界面残基的随机森林模型,计算vh和vl的惯常的扭转角、四个弯曲角(每个可变域两个)和中心轴长度(hl、hc1、lc1、hc2、lc2、dc),确定非人抗体fv片段和许多变体抗体中的每一种变体抗体的fv片段的vh-vl取向,-选择针对所有对的非人抗体fv片段和变体抗体fv片段的相应的cα原子确定的具有最小的平均均方根偏差(rmsd)的那些变体抗体fv片段,-基于它的结合性质,从所述的一种或更多种抗体中选择单一抗体,-把编码vh和vl的核酸克隆到一种或更多种表达载体中,-采用在前一步骤中获得的表达载体转染细胞,-培养所转染的细胞并且从而生产所述的抗体。根据本文中报道的一个方面是人源化的抗体,其在轻和重链可变域中在氨基酸位置l26-l32、l44、l46、l50-l52、l87、l91-l96、h26-h32、h45、h53-h55、h62和h96-h101(根据chothia指标编号)处包含来自供体非人抗体的氨基酸残基并且在其余位置处包含来自受体人或人源化的抗体或受体人种系氨基酸序列的残基。根据本文中报道的一个方面是人源化的抗体,其在轻和重链可变域中在氨基酸位置h26-h32、h35、h37、h39、h45、h47、h50、h53-h55、h58、h60、h61、h91、h95、h96-h101、h102、h103、h105、l26-l32、l34、l36、l38、l43、l44、l46、l49、l50-l52、l55、l87、l89、l91-l96(根据chothia指标编号)处包含来自供体非人抗体的氨基酸残基并且在其余位置处包含来自受体人或人源化的抗体或受体人种系氨基酸序列的残基。根据本文中报道的一个方面是人源化的抗体,其在轻和重链可变域中在氨基酸位置h26-h32、h33、h35、h43、h44、h46、h50、h53-h55、h56、h58、h61、h62、h89、h96-h101、l26-l32、l34、l36、l38、l41、l42、l43、l44、l45、l46、l49、l50-l52、l53、l55、l56、l85、l87、l89、l91-l96、l97、l100(根据chothia指标编号)处包含来自供体非人抗体的氨基酸残基并且在其余位置处包含来自受体人或人源化的抗体或受体人种系氨基酸序列的残基。根据本文中报道的一个方面是人源化的抗体,其在轻和重链可变域中在氨基酸位置h26-h32、h33、h35、h37、h39、h43、h44、h45、h46、h47、h50、h53-h55、h56、h58、h60、h61、h62、h89、h91、h95、h96-h101、h102、h103、h105、l26-l32、l34、l36、l38、l41、l42、l43、l44、l45、l46、l49、l50-l52、l53、l55、l56、l85、l87、l89、l91-l96、l97、l100(根据chothia指标编号)处包含来自供体非人抗体的氨基酸残基并且在其余位置处包含来自受体人或人源化的抗体或受体人种系氨基酸序列的残基。根据本文中报道的一个方面是人源化的抗体,其在轻和重链可变域中在氨基酸位置h26-h32、h35、h37、h39、h45、h47、h50、h53-h55、h58、h60、h61、h91、h95、h96-h101、h102、h103、h105、l26-l32、l34、l36、l38、l43、l44、l46、l49、l50-l52、l55、l87、l89、l91-l96、l98(根据chothia指标编号)处包含来自供体非人抗体的氨基酸残基并且在其余位置处包含来自受体人或人源化的抗体或受体人种系氨基酸序列的残基。根据本文中报道的一个方面是人源化的抗体,其在轻和重链可变域中在氨基酸位置h26-h32、h33、h35、h37、h39、h43、h44、h45、h46、h47、h50、h53-h55、h56、h58、h60、h61、h62、h89、h91、h95、h96-h101、h102、h103、h105、l26-l32、l34、l36、l38、l41、l42、l43、l44、l45、l46、l49、l50-l52、l53、l55、l56、l85、l87、l89、l91-l96、l97、l98、l100(根据chothia指标编号)处包含来自供体非人抗体的氨基酸残基并且在其余位置处包含来自受体人或人源化的抗体或受体人种系氨基酸序列的残基。附图描述图1分别从具有pdbid1n7m、1dlf和3hzm的晶体结构取得的具有5、10和15个氨基酸长度的三种示例性的cdr-h3环的叠加:a)chothia/kabat编号显示在三种代表性的cdr-h3环中残基97的宽的空间分布;b)wolfguy编号显示残基97的紧凑的空间定位,因为它总是在cdr-h3的末端之前的第三至最后一个残基,根据wolfguy指标命名的397;c)来自cdr的几个氨基酸具有链间接触,特别是位于cdr-h3和cdr-l3的末端的那些(根据wolfguy指标的残基797清楚地与vh定位共定位并且进行接触)。图2针对关于单独的复合物结构的试验数据集的示例性运行,预测的(纵轴)对真实的ab角取向参数(横轴)(把2/3的复合物结构用作训练组,而1/3用作试验组)。完美的预测将依赖于对角线。图3在预测子训练期间关于用筛选频度百分比的六个ab角参数的顶部25个重要的指纹3位置。采用改变的随机选择的训练组(单独的复合物结构)在十次运行上求所述值的平均值。误差线对应于一个标准差。构架和cdr分类遵循wolfguy命名法。图4当使用不受限制的代替受限制的最小化时,在构架(fw)、cdr(cdr)和所有fv残基(all)的羰基rmsd方面的平均变化和在distab角方面的平均变化(显示了三种变体i、ii、iii对1、2、3)。图5在构架(fw)、cdr(cdr)和所有fv残基(all)的羰基rmsd方面的平均变化和在原始模型和重取向模型之间在每种amaii抗体的distab角方面的平均变化。图6在构架(fw)、cdr(cdr)和所有fv残基(all)的羰基rmsd方面的平均变化和在原始模型和重取向的模型之间在每种amaii参与者的distab角方面的平均变化。图7按照它们的平均角度-距离把hc(矩阵的行,左边)和lc(矩阵的列,右边)分类。这些设想被用来剔除“坏的”hc/lc。图8具有不同的hc/lc组合的elisa测定值的矩阵。通过不同的方法取消选择的抗体是带阴影的;1:坏的hc/lc组合;2:被舍弃的全部hc/lc;3:最坏的20%。图9所有三种筛选方法“坏的hc/lc组合”(左边)、“全部hc和lc”(中间)和“最坏的20%”(右边)的elisa测定值的堆叠直方图。直方图条形的浅灰色区域指示被舍弃的抗体。图10按照它们的平均角度-距离把hc(矩阵的行,左边)和lc(矩阵的列,右边)分类。这些设想被用来剔除“坏的”hc/lc。图11具有不同的hc/lc组合的elisa测定值的矩阵。通过不同的方法取消选择的抗体是有阴影的;1:坏的hc/lc组合;2:被舍弃的全部hc/lc;3:最坏的20%。图12所有三种筛选方法““坏的hc/lc组合”(左边)、“全部hc和lc”(中间)和“最坏的20%”(右边)的elisa测定值的堆叠直方图。直方图条形的浅灰色区域指示被舍弃的抗体。图13按照它们的平均角度-距离把hc(矩阵的行,左边)和lc(矩阵的列,右边)分类。这些可视化被用来选择“坏的”hc/lc。图14三幅图,每一幅显示具有不同的hc/lc组合的bl测定值的矩阵;通过不同的方法取消选择的抗体是有阴影的;1:坏的hc/lc组合;2:被舍弃的全部hc/lc;3:最坏的20%。图15所有三种方法“坏的hc/lc组合”(左边)、“全部hc和lc”(中间)和“最坏的20%”(右边)的elisa测定值的堆叠直方图。直方图条形的浅灰色区域指示被取消选择的抗体。图16三幅图,每一幅显示具有不同的hc/lc组合的t1/2测定值的矩阵;通过不同的方法选择的抗体是有阴影的;1:坏的hc/lc组合;2:被舍弃的全部hc/lc;3:最坏的20%。图17所有三种方法“坏的hc/lc组合”(左边)、“全部hc和lc”(中间)和“最坏的20%”(右边)的t1/2测定值的堆叠直方图。直方图条形的浅灰色区域指示被取消选择的抗体。定义wolfguy编号方案所述的wolfguy编号像kabat和chothia定义的并集那样定义cdr区。而且,所述的编号方案基于cdr长度(并且部分地基于序列)注释cdr环尖端,使得cdr位置指标指示cdr残基是否是上行或下行环的一部分。在表1中显示了与已建立的编号方案的比较。表1:使用chothia/kabat(ch-kb)、honegger和wolfguy编号方案给cdr-l3和cdr-h3编号。后者具有从n-末端基底到cdr顶点递增的编号以及从c-末端cdr端开始递减的编号。kabat方案固定最后两个cdr残基并且引入字母以适应cdr长度。与kabat命名法对比,honegger编号不使用字母并且对于vh和vl是常见的。这样设计wolfguy,使得尽可能采用等效的指标来给结构上等效的残基(即在fv结构中在保守的空间定位方面非常相似的残基)编号。在图1中举例说明了这一点。关于wolfguy编号的全长vh和vl序列的例子可以在表2中找到。表2:采用wolfguy、kabat和chothia编号的具有pdbid3pp4(21)的晶体结构的vh(左边)和vl(右边)序列。在wolfguy中,仅仅根据长度给cdr-h1-h3、cdr-l2和cdr-l3编号,同时根据环长度和规范簇关系给cdr-l1编号。通过计算与不同的共有序列的序列相似度来确定规范簇关系。在这里,我们仅仅提供cdr-l1编号的单个例子,因为它对于产生我们的vh-vl取向序列指纹是不重要的。ab角概念(7)当在任何两个基于氨基酸的结构之间进行比较时,一般地使用基于距离的矩阵例如等效原子的均方根偏差(rmsd)。为了表征任何两个三维物体之间的取向,有必要定义:-关于每个物体的参考构架。-用来测定有关的取向参数的轴。-用来描述和定量这些参数的术语。所述的ab角概念是一种方法,其使用五个角度(hl、hc1、lc1、hc2和lc2)和一个距离(dc)在一种一致的并且绝对的意义上完全地表征vh-vl取向。以总体方式把抗体的可变域对vh和vl表示成抗体fv片段。在第一步中,从数据库(例如蛋白质数据库,pdb)中提取抗体结构。把chothia抗体编号(chothia和lesk,1987)应用于每一条抗体链。使成功地编号的链配对而形成fv区。这是通过应用下列约束条件而进行的:重链的h37位cα坐标(在重链可变域第37位的氨基酸残基的α碳原子)必须在轻链的l87位cα坐标的范围之内。非冗余组的抗体是使用cdhit(li,w.和godzik,a.bioinformatics,22(2006)1658-1659),在99%的fv区的构架上应用序列同一性截止而产生的。使用在重和轻结构域中在结构上最保守的残基位置来定义结构域位置。把这些位置表示成vh和vl核心组。这些位置主要地位于构架的β链上并且形成每个结构域的核心。在下面的表3中提供了所述的核心组的位置:使用所述的核心组位置来把参照的构架登记在抗体fv区结构域上。使用cdhit,在结构域中的构架位置上应用80%的序列同一性截止值,将非冗余数据集中的vh结构域聚类。从30个最大的聚类中的每一个随机地选择一个结构。使用mammoth-mult(lupyan,d.等人,bioinf.21(2005)3255-3263)在vh核心组位置上比对这组结构域。从这种比对中提取对应于β-片层界面中的八个在结构上保守的位置h36、h37、h38、h39、h89、h90、h91和h92的cα坐标。通过所产生的240个坐标拟合一个一个平面。对于vl结构域,使用位置l35、l36、l37、l38、l85、l86、l87和l88来拟合所述的平面。上面描述的步骤允许把两个参考构架平面映射在任何fv结构上。因此,可以使得测量vh-vl取向等效于测量所述的两个平面之间的取向。为了完全地并且在绝对意义上做到这一点,需要至少六个参数:距离、扭转角和四个弯曲角。必须针对连接所述的平面的一致地定义的向量来测量这些参数。在下面这种向量被表示为c。为了识别c,把参考构架平面登记在如上所述的非冗余组中的每一个结构上并且在每一个平面上设置网格。因此每个结构具有等效的网格点以及如此等效的vh-vl网格点对。在每个结构中针对每一对网格点测量欧几里德距离。鉴别在它们的分离距离方面具有最小方差的点对。把连接这些点的向量定义为c。使用位于每个平面中并且以对应于c的网格点为中心的向量来完全地定义坐标系。h1是与vh平面的第一主分量平行运行的向量,同时h2与第二主分量平行运行。类似地在vl结构域上定义l1和l2。hl角是在所述的两个结构域之间的扭转角。hc1和lc1弯曲角等效于一个结构域相对于另一个结构域的倾斜样的变化。hc2和lc2弯曲角描述一个结构域相对于另一个结构域的扭曲样的变化。为了描述vh-vl取向,使用六个度量:距离和五个角度。在坐标系中把这些定义如下:-c的长度,dc,-针对c测量的从h1到l1的扭转角hl,-在h1和c之间的弯曲角hc1,-在h2和c之间的弯曲角hc2,-在l1和c之间的弯曲角lc1,以及-在l2和c之间的弯曲角lc2。按照本领域中通常的意义使用术语“vh-vl取向”,正如本领域的技术人员将会理解的那样(参见例如dunbar等人,prot.eng.des.sel.26(2013)611-620;和bujotzek,a.等人,proteins,struct.funct.bioinf.,83(2015)681-695)。它表示vh和vl结构域相对于彼此怎样取向。因此,vh-vl取向是由下列确定的:-c的长度,dc,-针对c测量的从h1到l1的扭转角hl,-在h1和c之间的弯曲角hc1,-在h2和c之间的弯曲角hc2,-在l1和c之间的弯曲角lc1,以及-在l2和c之间的弯曲角lc2,其中通过下列登记参考构架平面:i)比对对应于vh的八个位置h36、h37、h38、h39、h89、h90、h91和h92的cα坐标并且通过它们拟合一个平面,和ii)比对对应于vl的八个位置l35、l36、l37、l38、l85、l86、l87和l88的cα坐标并且通过它们拟合一个平面,iii)在每个平面上设置一个位置,由此每个结构具有等效的网格点和等效的vh-vl网格点对,以及iv)在每个结构中对于每一个网格点对,测量欧几里德距离,由此向量c把在它们的分离距离方面具有最小方差的网格点对连接起来,其中h1是与vh平面的第一主分量平行运行的向量,同时h2是与vh平面的第二主分量平行运行的向量,l1是与vl平面的第一主分量平行运行的向量,l2是与vl平面的第二主分量平行运行的向量,hl角是在所述的两个结构域之间的扭转角,hc1和lc1是等效于一个结构域相对于另一个结构域的倾斜样变化的弯曲角,以及hc2和lc2弯曲角等效于一个结构域相对于另一个结构域的扭曲样的变化。按照chothia指标确定了所述的位置。选择向量c使其在所述的非冗余组的结构上具有最保守的长度。距离dc是这个长度。它具有16.2的平均值以及仅仅0.3的标准差。表4列出了通过随机森林算法鉴定为在确定vh-vl取向的每一个角度尺寸方面重要的顶部10个位置和残基。表4:x代表变量l36va/l38eb/l42ha/l43la/l44fa,b/l45t/l46gb/l49g/l95h(关于更详细的信息,参见参考文献7和bujotzek,a.等人,prot.struct.funct.bioinf.83(2015)681-695,将它们的全部引用并入本文)。更多的定义:“受体人构架”用于本文的目的是包含来源于人免疫球蛋白构架或人共有构架的轻链可变域(vl)构架或重链可变域(vh)构架的氨基酸序列的构架,正如在下面定义的那样。“来源于”人免疫球蛋白构架或人共有构架的受体人构架可以包含其相同的氨基酸序列,或者它可以包含氨基酸序列变化。在一些实施方式中,氨基酸数目的变化是10个或更少、9个或更少、8个或更少、7个或更少、6个或更少、5个或更少、4个或更少、3个或更少或2个或更少。在一些实施方式中,vl受体人构架在序列方面与vl人免疫球蛋白构架序列或人共有构架序列相同。“亲和性”是指在分子(例如抗体)的单个结合位点和它的结合配偶体(例如抗原)之间全部非共价相互作用的强度。正如本文中所用的那样,除非以其它方式指出,“结合亲和性”是指反映结合对(例如抗体和抗原)的成员之间1∶1相互作用的固有结合亲和性。分子x对于它的配偶体y的亲和性一般地可以用解离常数(kd)表示。可以通过包括本文中描述的那些方法在内的本领域中已知的常见方法来测定亲和性。用于测定结合亲和性的具体的举例说明的和示范性的实施方式被描述在下列中。在本文中术语“抗体”以最广泛的意义被使用并且包括不同的抗体结构,包括但不限于单克隆抗体、多克隆抗体、多特异性抗体(例如双特异性抗体)以及抗体片段,只要它们展示所要的抗原结合活性。“抗体片段”是指除完整抗体以外的分子,它包括完整抗体的一部分,其结合完整抗体所结合的抗原。抗体片段的实例包括但是不局限于fv、fab、fab’、fab-sh、f(ab’)2;双抗体;线性抗体;单链抗体分子(例如scfv);和由抗体片段形成的多特异性抗体。术语“嵌合的”抗体是指其中重和/或轻链的一部分源自于特定的来源或物种,同时重和/或轻链的其余部分源自于不同的来源或物种的抗体。抗体的“类别”是指由它的重链占有的恒定域或恒定区的类型。有五个主要的抗体类别:iga、igd、ige、igg和igm,并且这些中的几个可以被进一步分成亚类(同种型),例如igg1、igg2、igg3、igg4、iga1和iga2。对应于不同类别的免疫球蛋白的重链恒定域分别被称为α、β、ε、γ和μ。在本文中使用术语“fc区”来定义包含至少一部分恒定区的免疫球蛋白重链的c端区域。该术语包括天然序列的fc区和变体fc区。在一种实施方式中,人igg重链fc区从cys226或从pro230延伸到重链的羧基端。然而,fc区的c端赖氨酸(lys447)可以存在或可以不存在。在本文中除非另外指定,在fc区或恒定区中氨基酸残基的编号是根据eu编号系统,也被称为eu指标,正如在kabat,e.a.等人,sequencesofproteinsofimmunologicalinterest,第五版,publichealthservice,nationalinstitutesofhealth,bethesda,md(1991),nihpublication91-3242中描述的那样。“构架”或“fr”是指除高变区(hvr)残基以外的可变域残基。可变域的fr一般地由四个fr结构域组成:fri、fr2、fr3和fr4。因此,hvr和fr序列一般地出现在vh(或vl)中的下列序列中:fr1-hi(l1)-fr2-h2(l2)-fr3-h3(l3)-fr4。在本文中术语“全长抗体”、“完整的抗体”和“整个抗体”可被互换地使用,是指具有实质上类似于天然的抗体结构的结构或具有包含在本文中所定义的fc区的重链的抗体。“人抗体”是具有对应于由人或人细胞生产的抗体的氨基酸序列的氨基酸序列的抗体或来源于非人来源利用人抗体全部组成成分或其它人抗体编码序列的抗体。人抗体的这种定义具体地排除了包含结合非人抗原的残基的人源化的抗体。在人免疫球蛋白vl或vh构架序列的选择中,“人共有构架”是代表最通常地存在的氨基酸残基的构架。一般地,对人免疫球蛋白vl或vh序列的选择来自可变域序列的亚群。一般地,所述的序列亚群是正如在kabat,e.a.等人,sequencesofproteinsofimmunologicalinterest,第五版,bethesdamd(1991),nihpublication91-3242,第1-3卷中那样的亚群。在一种实施方式中,对于vl,所述的亚群是正如在kabat等人,上文中那样的亚群kappai。在一种实施方式中,对于vh,所述的亚群是正如在kabat等人,上文中那样的亚群iii。“人源化的”抗体是指包含来自非人hvr的氨基酸残基和来自人fr的氨基酸残基的嵌合抗体。在某些实施方式中,人源化的抗体将会实质上包含全部的至少一个并且典型地两个可变域,其中全部的或实质上全部的hvr(例如cdr)对应于非人抗体的那些,并且全部的或实质上全部的fr对应于人抗体的那些。人源化的抗体任选地可以包含来源于人抗体的至少一部分抗体恒定区。“人源化的形式”的抗体例如非人抗体是指已经经历人源化的抗体。正如本文中所用的那样,术语“高变区”或“hvr”是指在序列中是高变的(″互补决定区″或″cdr″)和/或在结构上形成确定的环(“高变环”)和/或包含接触抗原的残基(“抗原触点”)的抗体可变域的每一个区。一般地,抗体包含六个hvr;三个在vh中(h1、h2、h3),以及三个在vl中(l1、l2、l3)。在本文中hvr包括(a)高变环,其存在于氨基酸残基26-32(l1)、50-52(l2)、91-96(l3)、26-32(h1)、53-55(h2)和96-101(h3)处(chothia,c.和lesk,a.m.,j.mol.biol.196(1987)901-917);(b)cdr,其存在于氨基酸残基24-34(l1)、50-56(l2)、89-97(l3)、31-35b(h1)、50-65(h2)和95-102(h3)处(kabat,e.a.等人,sequencesofproteinsofimmunologicalinterest,第五版,publichealthservice,nationalinstitutesofhealth,bethesda,md(1991),nihpublication91-3242.);(c)抗原触点,其存在于氨基酸残基27c-36(l1)、46-55(l2)、89-96(l3)、30-35b(h1)、47-58(h2)和93-101(h3)处(maccallum等人,j.mol.biol.262:732-745(1996));和(d)(a)、(b)和/或(c)的组合,包括hvr氨基酸残基46-56(l1)、47-56(l2)、48-56(l2)、49-56(l2)、26-35(h1)、26-35b(h1)、49-65(h2)、93-102(h3)和94-102(h3)。除非另有指示,在本文中根据kabat等人,上文来给可变域中的hvr残基以及其它残基(例如fr残基)编号。根据它在本领域中的意义来使用术语“特异性决定残基”。它定义直接地参与抗原的相互作用的抗体的残基(参见例如padlan,e.a.等人,fasebj.9(1995)133-139)。“分离的”抗体是已经被从它的自然环境的组分中分离出来的抗体。在一些实施方式中,抗体被纯化到大于95%或99%纯度,正如通过例如电泳(例如sds-page、等电点聚焦(ief)、毛细管电泳)或层析(例如离子交换或反相hplc)测定的那样。关于用于评估抗体纯度的方法的综述,参见例如flatman,s.等人,j.chromatogr.b848(2007)79-87。正如本文中所用的那样,术语“单克隆抗体”是指从实质上同质的抗体群中获得的抗体,即包含在所述的群的单个抗体是相同的和/或结合相同的表位,除了可能的变体抗体以外,例如包含天然存在的突变或在生产单克隆抗体制剂期间产生的,这样的变体一般地以微小的数量存在。与典型地包括针对不同的决定簇(表位)的不同抗体的多克隆抗体制剂相比,单克隆抗体制剂中的每一种单克隆抗体针对抗原上的单一决定簇。因此,所述的修饰语“单克隆的”指示抗体的特性是从实质上同质的抗体群中获得的,并且不被解释为需要通过任何特定的方法生产抗体。例如,可以通过多种技术来制造根据本发明将要被使用的单克隆抗体,这些技术包括但不限于杂交瘤方法、重组dna方法、噬菌体展示方法和利用包含全部或一部分人免疫球蛋白基因座的转基因动物的方法,这样的方法以及其它示范性的方法用于制造本文中描述的单克隆抗体。术语“可变区”或“可变域”是指参与抗体与抗原结合的抗体重或轻链的结构域。天然抗体的重链和轻链的可变域(分别为vh和vl)一般地具有相似的结构,同时每个结构域包含四个保守性构架区(fr)和三个高变区(hvr)。(参见,例如kindt,t.j.等人,kubyimmunology,第6版,w.h.freemanandco.,n.y.(2007),第91页)。单个vh或vl结构域可以足以赋予抗原结合特异性。而且,可以使用来自结合所述抗原的抗体的vh或vl结构域来分离结合特定抗原的抗体,从而分别地筛选互补的vl或vh结构域的库。参见例如portolano,s.等人,j.immunol.150(1993)880-887;clarkson,t.等人,nature352(1991)624-628)。发明详述在本文中报道了基于序列的快速预测子,其预测vh-vl域间取向。实现了范围从0.67到0.80的q2值。根据六个绝对的ab角参数描述了vh-vl取向以把vh-vl取向的不同自由度精确地分开。在不同的抗体结构上评估了vh-vl取向的影响。已经发现采用在本文中报道的方法可以达到关于变体(人源化的)抗体的vh-vl取向偏差相对于亲本(非人)抗体的改善。通过氨基酸主链的羰基原子的平均均方根偏差(rmsd)显示了这一点。这显示关于在亲本(非人)和变体(人源化的)抗体之间vh-vl域间角的相似度方面的改善。在本文中所报道的方法(包括移植方法)正在递送变体(人源化的)抗体的更好的结合性质。可以把其它工程化方法例如构架改组与在本文中报道的方法组合,当把一种人构架转换为另一种以便改变抗体的生物物理特性时,导致所获得的变体抗体的改善的结合。这导致提供用于从来源于亲本抗体的许多变体抗体中选择更好的人源化抗体的方法。抗体在治疗学和临床诊断学中的用途产生了对抗体结构精确的同源性模型的需要,每当不可得到晶体结构时,该模型确保合理的抗体工程化。因此,已经发展了许多用于计算机辅助的抗体设计(1)的计算方法,在它们之中许多同源性建模方法被通过盲建模研究(8,2)有规则地评估。由于在实验上衍生的抗体结构的数目(结构上的抗体数据库sabdab3把1841个条目算作是2014年5月的),与其它生物分子的同源性模型相比,抗体同源性模型的品质是优良的。所述的两种抗体可变片段(fv)的六种抗原结合环在序列方面是高变的(高变区,hvr)。它们中的五种易于适应正则构象,可以从基于现有的模板结构的序列中预测该正则构象。这一点不适用于在重链可变区上的第三个环hvr-h3。hvr-h3是在序列和长度方面最可变的环,并且典型地是主要的抗原相互作用特异性决定位点。抗体的抗原结合位点在两种fv(重链可变域(vh)和轻链可变域(vl))的界面处形成。每一种可变域包含三个hvr。vh和vl结构域的相对取向增加了抗原结合位点的拓扑学。在它们最近的抗体建模评估研究2(amaii)中,teplyakov等人(2)使用了单个角测度来描述vh-vl取向。利用一组在结构上保守的β-片层核心位置,把在vh-vl倾角相对于参比结构方面的差别计算成通过vl和vh结构域的顺序叠加而实现的坐标变换的球面角系统(ω,φ,κ)中的κ角。narayanan等人(6)使用基于rmsd(均方根偏差)的量度来训练和评估基于能量的vh-vl取向的预测子。chailyan以及同事(5)鉴定了具有类似的vh-vl取向的fv结构簇并且通过测定某些保守性残基的cα叠加rmsd确定了有影响的序列位置。其它研究通过提供一种晶体结构的vh或vl在另一种晶体结构上的重取向所必需的旋转量而增大rmsd值(10-12)。abhinandan和martin(4)定义了vh-vl堆积角,一种用于比较vh-vl取向的绝对量度。vh-vl堆积角是由通过在两个结构域中的每一个中一组高度保守的cα位置的主轴拟合的向量所跨越的扭转角。与相对的rmsd值对比,vh-vl堆积角允许用它在结构空间中的vh-vl取向来描述每一个单个的fv结构。与vh-vl堆积角的定义一起,作者鉴定了一组有影响的位置并且提供了采用神经网络学习的基于序列的vh-vl堆积的预测子。基于过去的观察(其在被认为对vh-vl取向有影响的fv序列位置方面至少有部分不一致)(4,5),dunbar以及同事(7)提示vh-vl取向受多个自由度支配,并且每一个自由度由不同组的有影响的序列位置决定。因此,所述的作者除了惯常的扭转角之外,还确定了四个弯曲角(每个可变域两个)以及vh和vl的中心轴长度,并且利用随机森林模型,针对五个角参数(ab角)中的每一个以及针对vh和vl之间的中心轴长度,鉴定了最有影响的序列位置。在本文中报道了用于在抗体的人源化期间表征和利用vh-vl取向的基于ab角的方法。在本文中报道了针对六个ab角量度中的每一个的基于序列的vh-vl取向的预测子。还报道了在真实的抗体同源性模型中调节vh-vl取向的方法。在本文中报道了用于在结合决定残基从供体抗体转移到受体抗体构架期间表征和利用vh-vl取向的基于ab角的方法。在本文中报道了用于在抗体的部分或全部构架区的交换(构架改组)期间表征和利用vh-vl取向的基于ab角的方法。vh-vl取向预测子表5:用于预测在50次运行上平均化的六个ab角参数的q2和rmse值。手动调整每个随机森林模型的树的数目,以便使q2最大化。在括弧中的数值具体说明标准差。在apo和复合物结构(表5,中心列)的完全数据集上训练随机森林模型一次以及在单独的复合物结构(表格5,右列)上训练随机森林模型一次。尽管训练组被减少了几乎550个结构,但是当单独使用复合物结构时,q2和rmse值改善了。对于hl、lc2和dc,q2值大约是0.68,而hc1、lc1和lc2具有0.75以及以上的q2值(当考虑复合物结构时)。备选地,为了确保在训练组中包括最大多样性的不同取向的指纹,可以使用cd-hit来以100%同一性聚类取向指纹,以及针对每一个聚类,可以把至少一个代表添加到训练组中,直到把可得到的结构分配给训练组为止。可以把其余的用于测试。由于试验组然后将会由也被包括在训练组中的取向指纹组成这样的事实,对于目前的数据组的,所产生q2值例如范围从0.71到0.88,取决于各自的ab角参数,当面对未知的取向指纹时,所述q2值将会夸大预测子的真实能力。在那种情况下,可能发现对于目前的数据集,q2值范围大约为0.54至0.73。图2显示在单独的复合物结构数据集上,关于预测的对比真实的ab角参数的示范性的回归图。例如与由abhinandan和martin(4)报道的相比,相关性得到了改善。不受这种理论的限制,可以把所述的改善归因于用六个ab角参数对vh-vl取向自由度的更精细的描述以及利用wolfguy编号方案减少或甚至避免了hvr残基编号中的模糊度。在图3中描绘了指纹位置作为不同的ab角参数的描述者的重要性排序。基于指纹位置重要性排序的发现,已经发现每一个ab角参数受vh和vl这两者上的大体上不同组的界面位置影响。针对除hc2之外的所有参数,构架位置是最重要的描述符。尽管如此,在每一种情况下,至少两个hvr-h3残基在最重要的描述符之中。已经在原始ab角公布(7)中被排序在顶部10个重要的输入变量之中的位置也被推进到本文中给出的排序中。但是,鉴于dunbar等人(7)发现hc1是排他地由重链上的残基决定的,以及lc1是排他地由轻链上的残基决定的,采用根据在本文中报道的方法确定的hc1和lc1的前十个描述符涉及这两种链上的指纹位置。在本文中排序指纹位置不考虑氨基酸特异性。排序前25的指纹位置也包含由chailyan等人(5)(l41、l42、l43、l44)以及由abhinandan和martin(4)(l41、l44、l46、l87、h33、h45、h60、h62、h91、h105)鉴定的vh-vl取向决定位置组中的许多成员。已经发现l87是hl的顶部描述符,l46是hc1的顶部描述符,h45是lc1的顶部描述符,h62是hc2的顶部描述符,以及l44是lc2的顶部描述符。采用vh-vl重取向的抗体同源性建模mofvab模型关于mofvab(抗体的fv的建模)方法的详细说明已经被bujotzek,a.等人公布(mabs7(2015)838-852)。获得的关于建模变体1的结果(从在或者共有vh或者vl构架上比对,随后在共有fv构架上vh-vl重取向的模板结构装配的模型)被显示在表6中。表6:采用mofvab变体1建立的amaii模型。数值陈述了在β-片层核心上进行逐链比对以后,关于由teplyakov等人(7)所定义的片段的羰基rmsd。为了把由vh-vl取向中的偏差所引起的羰基位移考虑进去,在完全的fv(vh和vl同时)的β-片层核心上比对相同的模型并且重新计算所述的数值。将结果显示在表7中。表7:采用mofvab变体1建立的amaii模型。数值陈述了在完全的fv的β-片层核心上进行比对以后,关于由teplyakov等人(7)所定义的片段的羰基rmsd。最右边的三列具体说明关于基于wolfguy片段定义和kabatcdr定义的构架(fw)、hvr(cdr)和所有fv残基(all)的羰基rmsd。在表6和表7之间的比较揭示了一旦考虑完全的fv结构,rmsd数值是怎么变坏的。对于vl,关于β-片层核心的平均羰基rmsd从增大到并且对于vh,从增大到这种趋势不局限于构架,而是延伸到hvr。例如对于hvr-l3,平均羰基rmsd从增大到而对于hvr-h3,从增大到通过直接地查看六个ab角参数和相对于参比结构的差别,把在vh-vl取向方面的偏差显示在表8中。表8:按照采用mofvab变体1建立的amaii模型的六个ab角参数,在vh-vl取向相对于参比结构的偏差。模型参比δhlδhc1δlc1δhc2δlc2δdcdistae角ab014ma3_b_a1.850.040.332.880.390.273.47ab014ma3_h_l7.722.460.074.510.780.039.31ab024kuz_h_l0.711.571.971.611.100.403.29ab034kq3_h_l1.710.190.952.450.270.103.15ab044kq4_h_l0.591.941.424.950.450.005.55ab054m6m_h_l8.843.903.304.170.320.3511.04ab064m6o_h_l3.731.610.000.540.510.284.14ab074mau_h_l2.602.333.870.382.280.235.71ab084m7k_h_l2.321.613.080.280.090.344.20ab094kmt_h_l2.470.982.083.821.360.055.28ab104m61_b_a0.630.251.154.561.600.205.02ab104m61_d_c0.980.700.315.520.940.295.74ab114m43_h_l2.392.154.382.251.330.356.042.811.521.762.920.880.225.53已经使列在表8中的模型在相同的共有fv构架上重取向并且因此共享基本上相同的大约θcons:=(-59.45,71.65,120.49,117.46,82.77,16.11)的ab角取向。显示了amaii结构固有的在vh-vl取向方面的大的多样性。对于参数hl和hc2,发生的最大的偏差不仅在不同的结构之间,而且也在来自相同的不对称单元的序列相同的结构:4ma3_b_a和4ma3_h_l的参数hl偏差5.87度。这证实vh-vl取向虽然受某些序列特征引导(参见图3),但是也受固有的、非定向的可变性影响。对于未结合形式的(7)结合蛋白的抗体,这是特别显著的。采用建模变体2(从在或者共有vh或者vl构架上比对,随后在基于与预测的ab角参数的相似度选择的vh-vl取向模板结构上进行vh-vl重取向的模板结构装配的模型),利用的对模板结构的相同选择,重建了所有的模型。将结果显示在表9中(数值涉及在完全的fv的β-片层核心上比对的模型-参比对)。表9:采用mofvab变体2建立的amaii模型。针对vh-vl取向优化的变体2模型计算的每个片段的平均羰基rmsd数值显示了与利用一般的vh-vl取向的模型相比,大约的改善(参见表7)。ab角偏差揭示了变体2模型已经移动到更接近参比结构的真实的vh-vl取向(参见表10)。表10:根据采用mofvab变体2建立的amaii模型的六个ab角参数,相对于参比结构在vh-vl取向方面的偏差。在最右边的列中提供了基于预测的ab角参数选择的vh-vl取向模板结构。平均distab角从普通的取向模型的5.53改善到取向优化的变化形式的4.78。在表的最右边列中显示的是基于加权的距离至预测的ab角参数选择的vh-vl取向模板。不是基于指纹相似度而是通过在ab角取向空间方面的相似度来选择vh-vl取向模板的。采用建模变体3,利用在常见的共有fv构架而不是经链的共有结构上比对的模板结构,并且不以任何形式调整vh-vl取向,重建了所有的模型。由于经fv比对了所有的模板结构这样的事实,对于vl,逐链的羰基rmsd(参见表6)从0.37增大到0.43,而对于vh,从0.47增大到0.55(数据没有被显示)。在表11中列出了关于在完全的fv上比对的模型-参比对的羰基rmsd数值。表11:采用mofvab变体3建立的amaii模型。关于变体3的结果不像其它两种变体的结果那样好。尽管在变体3中混合了具有完全无关的vh-vl取向的来自fv结构的模板片段,但是似乎对于模型品质乎没有特别地有害的影响。相应的ab角偏差被显示在表12中。表12:根据采用mofvab变体3建立的amaii模型的六个ab角参数,在vh-vl取向相对于参比结构方面的偏差。模型参比δhlδhc1δlc1δhc2δlc2δdcdistab角ab014ma3_b_a2.990.210.590.890.200.523.23ab014ma3_h_l2.882.290.990.740.590.283.94ab024kuz_h_l3.531.103.382.462.690.336.20ab034kq3_h_l3.630.542.224.811.030.246.53ab044kq4_h_l0.912.090.711.250.730.102.79ab054m6m_h_l9.762.803.220.932.760.0311.04ab064m6o_h_l5.951.511.500.380.920.176.40ab074mau_h_l2.392.452.970.763.110.545.57ab084m7k_h_l2.632.200.182.450.950.414.34ab094kmt_h_l3.250.112.320.550.180.114.04ab104m61_b_a0.920.393.260.410.080.293.45ab104m61_d_c0.570.842.421.370.740.383.08ab114m43_h_l0.941.912.771.421.580.234.103.101.422.041.421.200.284.98变体3模型的平均distab角不像vh-vl取向优化的模型的那样好,但是好于通过变体1产生的具有共有fv取向的模型的平均distab角。不受这种理论限制,取自在常见的共有fv构架上比对的结构的模板片段确实编码一些vh-vl取向信息,不然这些信息将会丢失。上述显示的所有数据涉及在存在对于所有残基的位置限制的情况下被最小化的模型,除了被重塑或位于具有来源于不同模板结构的相邻残基的片段边缘处的那些模型以外。因此,保留了由模板结构和/或vh-vl重取向所赋予的vh-vl取向信息的最大值。为了比较,重复所述的建模方法并且利用相同的力场和隐含的水模型组合(charmm和gbsw),同时省略位置限制,把所有的模型最小化。为了简单起见,当从受限制的转换到完全柔性的最小化时,仅仅总结了在羰基rmsd和distab角方面的平均变化。对于采用mofvab变体1和2建立的模型,相对于参比结构的平均羰基rmsd变得稍微更大了。变体3模型,由于它们的模板结构设置可能最受立体不准确性的影响,受益于经由小的边缘的无限制的能量最小化。所有的三种模型变体在distab角方面相对于参比结构有改善。不受这种理论限制,似乎是无限制的能量最小化诱导模型品质相对于不同的建模变体(力场/隐含的水模型组合)的均等化。原始的amaii模型把通过使它在本文中所报道的vh-vl取向模板上重取向来改善给定的fv同源性模型的方法集成到本领域现有技术的建模软件中。原始的amaii模型是从http://www.3dabmod.com中获得的,采用wolfguy编号给所述的结构注释,为的是便于集成,并且所述的模型重取向在表8中所列的vh-vl取向模板上。在重取向以后每种抗体在羰基rmsd和distab角方面的平均变化(在所有amaii参与者各自的模型上求平均值),被显示在图5中。在十一组抗体模型(由所有通过根据ab02的acc、ccg、jef、joa、mmt、pig和sch提供的结构构成)中的八组的不同fv结构(特别地没有任何后加工)上进行逐链重取向以后,完全的fv主链的羰基rmsd通过vh-vl重取向而得到改善。此外,模型组ab01、ab10和ab11在vh-vl取向和构架rmsd方面改善了。最后,把所述的模型组分开,以便评估本文中所报道的vh-vl重取向方法与采用不同的方法建立的抗体模型在多大程度上一致。在重取向以后羰基rmsd和distab角方面的平均变化(在各自的amaii参与者的所有的模型上求平均值)被显示在图6中。对于所有参与者的模型,相对于参比结构,平均distab角得到了改善。在七种情形中的五种中,特别是关于构架区,由vh-vl重取向导致的distab角的减小转变为更好的rmsd数值。平均起来,发现对于整个fv,有distab角的显著改善和羰基rmsd的小的改善。因此,正如在本文中报道的那样,通过从单一的vh-vl堆积角移动到根据由dunbar等人(7)定义的六个ab角参数的vh-vl取向的更精细描述,可以延伸基于序列特征的vh-vl取向预测概念。针对每一个ab角参数,在迄今已知的fv结构组上训练了随机森林模型。当在仅仅由复合物结构组成的一组上训练时,所述的六个预测子的q2值范围从0.67到0.80。对我们的随机森林模型的顶部描述符的分析揭示了许多以前没有被知晓这样的描述符的hvr-h3残基。在本文中报道抗体编号方案“wolfguy”有助于鉴定这些残基,因为它被设计成使得尽可能采用也(并且特别地)在高变区中的等效的指标来给在结构上等效的残基编号。把没有vh-vl取向预测和调节的两种建模变体(变体1和3)与建模变体相比较,该建模变体根据六个ab角参数预测最可能的vh-vl取向,在抗体模板数据库中自动地搜索最相似地取向的fv结构,并且在进一步加工之前,使原始模型在这种vh-vl取向模板上重取向(变体2)。将会预期:由于vh和vl的改善的预取向,相对于建模hvr-h3环的协同作用效果。而且,基于基于序列的预测子优化vh-vl取向和随后在模板结构上的重取向的计算成本是微不足道的(例如当与合成工作比较时)。本发明已经发现:相对于抗原的亲本非人抗体,在与抗原的相同表位结合的两种(人源化的)抗体变体之间总的vh-vl取向差别与所述抗体各自在抗原结合能力方面的差别相关联。在本文中从fv序列位置(“序列指纹”)的(有意义的)亚组中而不是从完全的fv序列中预测vh-vl取向。基于这样的假定:vh-vl取向由在vh-vl界面上或靠近vh-vl界面的残基决定,已经鉴定了一组界面残基,其中如果在所述的数据库中,例如在rab3d中,在至少90%的所有重叠的fv结构中,一个残基的侧链原子以小于或等于的距离邻接相反链的原子,则把所述残基定义为vh-vl界面的一部分。将结果概括在表29中,该表也基于统计分析(4、5、7)陈述是否已经预先将序列位置连接而使其成为vh-vl取向的决定簇。表29:vh-vl界面残基,其中在rab3d中在至少90%的所有重叠的fv结构中,如果一个残基的侧链原子以小于或等于的距离邻接相反链的原子,则所述残基是所述的界面的一部分。*根据环长度编号+根据chothia等人(13)所定义的vh-vl界面的一部分上述组的界面残基丢失了一些序列位置,所述序列位置已经被列在dunbar等人(7)的vh-vl取向的“顶部10个重要的输入变量”之中。那些序列位置被列在下表30中。表30:被dunbar等人(7)列在vh-vl取向的“顶部10个重要的输入变量”之中的另外的序列位置。*根据环长度编号+根据chothia等人(13)所定义的vh-vl界面的一部分为了选择亲本抗体的合适的(人源化的)变体抗体,根据vh和vl的六个“ab角”取向参数描述了vh-vl取向,所述的取向参数由一个扭转角、四个弯曲角(每个可变域两个)以及中心轴长度组成。已经发现可以使用在vh结构域和vl结构域之间的相对取向(vh-vl取向)来(预先)选择具有最好的结合亲和性的变体抗体。这可应用在一组人源化的抗体内部以及在一组人源化的抗体和亲本非人抗体之间。此外,已经发现每当必须要改变(交换)构架残基或全部构架时,可以基于本文中所报道的方法来评估新的变体与它的抗原的结合。在下面采用具体的抗体来举例说明本发明,这些抗体充当实例而不应该被解释成用来把本发明的保护范围限定于此。在本文中所报道的方法一般地是可应用的方法。所述的序列指纹由54个氨基酸组成,29个在vh区中,以及25个在vl区中。参见下列的表13。表13:序列指纹。*根据环长度编号+根据chothia等人(13)所定义的vh-vl界面的一部分在第一个实例中,根据在本文中所报道的方法评估了与cd81受体胞外域(ecd)的大的胞外环(lel)结合的两种鼠的抗体cd81k04和cd81k13以及其人源化的变体。在第二个实例中,根据在本文中所报道的方法评估了识别来自ptau蛋白(包括s422磷酸化)的肽段的兔抗体以及它的人源化的变体。在第三个实例中,根据在本文中所报道的方法评估了抗hepsin抗体以及它的人源化的变体。在序列比对中,采用灰色背景标记hvr。所使用的hvr定义对应于kabat和chothiacdr定义的并集。采用黑色背景来标记作为用于预测vh-vl取向的序列指纹部分的序列位置。采用‘x’来标记在给定的抗体中没被占据的指纹位置。cd81k04、cd81k13(鼠的)和rb86的原始vh序列:cd81k04、cd81k13(鼠的)和rb86的原始vl序列:cd81k04vh人源化变体(原始的鼠的序列被显示在顶部):cd81k04vl人源化变体(原始的鼠的序列被显示在顶部):cd81k13vh人源化变体(原始的鼠的序列被显示在顶部):cd81k13vl人源化变体(原始的鼠的序列被显示在顶部):rb86vh人源化变体(原始的兔序列被显示在顶部):fr3rftiskas--ttvdlkmtsptaedtgrftisrdnskntlylqmnslraedtarftisrdnskntlylqmnslraedtarftisrdnskntlylqmnslraedtarftisrds--ttlylqmnslraedtarftisrdnskntlylqmnslraedtarftisrdnskntlylqmnslraedtarftisrdnskntlylqmnslraedtarvtmttdtststaymelrslrsddtarvtmtkas--staymelrslrsddtarftisrdnskntlylqmnslraedtarftisrdnskntlylqmnslraedtarftisrdnskntlylqmnslraedtarftisrdnskntlylqmnslraedtarftisrdnskntlylqmnslraedtarftisrdnskntlylqmnslraedtarb86vl人源化变体(原始的兔序列被显示在顶部):已经利用一般的移植原理设计了序列变体。一般而言,发展了移植用来生产人源化的抗体。此外还可以使用移植来获得与其它物种相容的抗体,或仅仅为了交换一种抗体的构架以得到这种抗体或抗体片段的其它生物物理学性质。在人恒定区对应物上克隆人源化的可变区以后,通过把所有的重链质粒与所有的轻链质粒组合来以“矩阵”形式表达所述的抗体。于是第一行和第一列是半人源化的抗体,而第一单元格是呈它的嵌合形式的原始鼠或兔的抗体,以及矩阵的其余部分是完全人源化的抗体。关于抗cd81抗体cd81k04和cd81k13,结合数据是生物化学的(细胞的结合)elisa数据,正如分别地在下面的表14和表15中概括的那样。嵌合形式的cd81k04接近1.15的值,而人源化的变体是效力稍微更小的结合剂。对于一些变体,亲和性下降得更加显著。关于兔抗体rb86,在第一次针对它们的能力的筛选中分析了来自上清液的微纯化物质,以获得缔合参数和解离参数,其没有偏离原始抗体的那些参数太多。针对每一种变体以及针对参比兔抗体,编辑了结合晚期(bl)ru(在缔合阶段结束时在spr/biacore实验中的应答单位),以及解离常数kd[1/s],其可以被转换为抗体在它的靶上的半衰期(t1/2=ln2/kd),分别参见表16和表17。对于一些缔合非常差(在缔合阶段中ru接近于零或是负的)的变体,也没有办法测定kd值;将半衰期数值设定到0。在ab角距离和结合之间的相关性可以使用rv系数或其它系数例如来自protest方法的相关系数来关联所述的矩阵。这些方法本质上评估在两个数据组之间的相关性,其中针对每一种样品,我们不是有一个而是有几个量度,并且因此在某种程度上延伸标准的单变量相关系数。rv系数被用在下列中。在表23中显示了关于来自hc和lc的透视图的三个不同的数据组的rv系数和它的p值。表23:关于全部四个数据组的rv系数和相应的p值。rv系数是从作为样品的hc以及因此作为多变量量度的lc中计算出来的,反之亦然。p值是经由置换检验计算出来的并且指示达到与计算出来的rv系数一样高或比其高的rv系数的概率。关于所述的数据组的更少受限制的观点将会要把每一种mab看作个体。在那种情况下,它使得把这两个矩阵向量化和计算pearson相关性有意义。表24显示关于不同的数据组的相关系数和相应的p值。表24:关于所有的三个数据组的pearson相关系数和相应的p值。计算了角度-距离和结合矩阵的向量化形式的相关性。所述的p值指示根据不具有相关性的零假设达到所计算的相关性的概率。所有的数据组显示相关性。因此,已经发现可以使用仅仅基于角度-距离来舍弃单个抗体的方法来选择人源化的抗体变体。抗体亚组舍弃在下面分析了针对它们的表现的选择抗体亚组的三种方法。这些方法是选择最坏的20%、选择坏的hc/lc组合和选择本文中描述的完整hc或lc。为了评估不同的选择方法的表现,计算了不同的统计数字。第一种统计数字是在保留亚组和舍弃亚组之间中值结合长度的比率。关于这种比率,还利用置换检验计算了p值。此外,使用堆积直方图直观地检查了在所述的两个亚组中结合长度的分布或一个数据组的ic50数值。cd81k04关于这两种使用hc/lc信息的方法,必须选择期望表现得差于其余者的hc/lc。图7描绘关于hc(矩阵的行,左边)和lc(矩阵的列,右边)的平均角距离。把包含hc6、7、8、9、12、13和14以及lc7和9的抗体取消选择了。在图8中显示了通过各自的选择方法除去了哪些抗体以及保留了哪些抗体。表25显示了针对它们的结合长度,比较所述的抗体亚组的结果。计算了在这两个组中的中值结合长度并且形成了这两者的比率。在该表中显示了所有的三种方法连同p值的结果,其指示得到至少这种低的比率的概率。表25:关于这三种不同的抗体舍弃法,计算了在舍弃的抗体和保留的抗体之间中值酶联免疫吸附测定测量值的比率。另外经由置换法计算了p值,其显示达到与所发现的中值比率一样低或更低的值的概率。cd81k04中值比率(取消选择的/选择的)p值舍弃最坏的20%0.22790.0373舍弃坏的hc/lc组合0.206610.0162舍弃全部hc和lc0.3152<0.001不依赖于所述的方法,保留的抗体亚组具有比取消选择的抗体长3-5倍的结合长度。此外,这些结果是显著的(p<0.05)。在图9中显示了关于所述的三种选择方法的elisa测量值的堆积直方图。该直方图证实了中值比率结果。所有的三种方法都舍弃低结合的单克隆抗体。cd81k13对于抗体cd81k13的人源化的变体,进行了与上面所描绘的相同的方法。将结果显示在图10至12以及下表26中。取消选择了包含hc3、4和9以及lc2、5、6、7、10和11的抗体。所有这三种方法导致一个亚组的结合比取消选择亚组中抗体的长1.6-2倍(参见表26)。表26:关于这三种不同的抗体选择方法,计算了在舍弃的抗体和保留的抗体之间中值elisa测量值的比率。另外经由置换法计算了p值,其显示达到与所发现的中值比率一样低或更低的值的概率。图12显示具有较低的结合长度的多数抗体被舍弃了。rb86为抗体rb86的人源化变体进行了与上面描绘的一样的方法。结果被显示在图13至15以及下列表27中。取消选择了包含hc9、10和11以及lc2和8的抗体。关于抗体rb86的变体,在选择/取消选择步骤中使用了spr数据。能够得到关于不同抗体的结合行为的两种不同的量度。基于bl数据,取消选择了不同的抗体(参见图14)。所有三种选择方法都针对“bl”选择平均结合好3-5.5倍的抗体。p值指示结果不是偶然的,而是由于在所述的不同方法中用来选择抗体的有利的方式所致。表27:关于这三种不同的抗体舍弃法,计算了在舍弃的抗体和保留的抗体之间中值bl测量值的比率。另外经由置换法计算了p值,其显示达到与所测定的中值比率一样低或更低的值的概率。这在图15中是带下划线的,其中堆积直方图显示具有低的结合长度的抗体主要地被取消选择了。作为基于t1/2数据的备选方法,取消选择了抗体(参见图16)。正如可以看到的那样,如基于bl数据那样选择了相同的抗体。关于不同的选择方法的中值比率显示:被取消选择的抗体亚组总是差于被保留的亚组(参见表28)。采用“坏的hc/lc组合”方法,仅仅取消选择了少数几种抗体,但是在所述的组中有许多不具有半衰期的抗体。这是为什么关于这种方法的p值不是显著的理由。关于其它方法,p值指示很好地选择了被取消选择的亚组。表28:关于这三种不同的抗体选择方法,计算了在取消选择的抗体和选择抗体之间中值t1/2测量值的比率。另外经由置换法计算了p值,其显示达到与所测定的中值比率一样低或更低的值的概率。在图17中的堆积直方图中也显示了这一点。所有的方法都取消选择了大量的具有很低的半衰期的许多抗体。概述已经发现:当把vh-vl取向(vh-vl角度)预测应用在全部来源于常见的原始亲本抗体的人源化变体上时,良好的人源化的变体更加紧密地遵从亲本抗体的角度参数。所述的被用于舍弃具有次优vh-vl取向的抗体,即舍弃“最坏的20%”或一组全部hc/lc或坏的hc/lc组合的三种方法,产生了类似的抗体亚组。堆积直方图和相关性分析证实:角度-距离是结合行为的良好指标。已经发现通过利用在本文中所报道的选择方法,可以急速地增大这样过滤的人源化矩阵的品质。在一种实施方式中,插入置信度矩阵作为另外的步骤,例如首先选择被取消选择的亚组,然后选择高置信度亚组。在一种实施方式中,使用所有的抗体之间的距离信息来计算抗体聚类并且鉴定距离插入参比的聚类最远的抗体聚类。具体的实施方式1.用于筛选来源于亲本抗体fv片段的一种或更多种变体抗体fv片段的方法,其包括下列步骤:-通过把来自亲本抗体fv片段的一个或更多个决定结合特异性的残基移植/转移到受体抗体fv片段上而产生许多变体抗体fv片段,由此所述的许多变体抗体fv片段的每一种变体抗体fv片段与其它变体抗体fv片段至少有一个氨基酸残基不同,-基于所述的抗体fv片段的序列指纹,确定亲本fv片段和所述的许多变体抗体fv片段中的每一种变体抗体fv片段的vh-vl取向,-选择在vh-vl取向方面与亲本抗体的vh-vl取向比较具有最小差别的那些变体抗体fv片段并且从而选择一种或更多种来源于亲本抗体fv片段的变体抗体fv片段,由此所述的一种或更多种变体抗体fv片段与亲本抗体fv片段与相同的抗原结合。2.用于把非人抗体人源化的方法,其包括下列步骤:-提供与抗原特异性地结合的非人抗体,-通过把来自非人抗体的一个或更多个决定结合特异性的残基移植/转移到人或人源化的受体抗体或种系抗体序列上产生许多变体抗体,由此所述的许多变体抗体中的每一种变体抗体与其它变体抗体至少有一个氨基酸残基不同,-基于所述的抗体fv片段的序列指纹,确定非人抗体fv片段和所述的许多变体抗体中的每一种变体抗体的fv片段的vh-vl取向,-选择在vh-vl取向方面与亲本抗体的vh-vl取向比较具有最小差别的那些变体抗体fv片段并且从而选择来源于非人的一种或更多种人源化的抗体,由此所述的一种或更多种人源化的抗体与非人抗体与相同的抗原结合。3.根据实施方式1所述的方法,其包括下列步骤:-选择在vh-vl域间角方面与亲本抗体的vh-vl域间角比较具有最高的(结构上的)相似度的那些变体抗体fv片段并且从而选择一种或更多种来源于亲本抗体fv片段的变体抗体fv片段。4.根据实施方式2所述的方法,其包括下列步骤:-选择在vh-vl域间角方面与亲本抗体的vh-vl域间角比较具有最高的(结构上的)相似度的那些变体抗体fv片段并且从而选择一种或更多种来源于非人抗体的人源化的抗体。5.根据实施方式1和3中任意一种所述的方法,其中所述的亲本抗体fv片段是非人抗体fv片段。6.根据实施方式1、3和5中任意一种所述的方法,其中所述的受体抗体fv片段是人或人源化的抗体fv片段或人抗体fv片段种系氨基酸序列。7.根据实施方式1至6中任意一种所述的方法,其中所述的序列指纹是一组vh-vl界面残基。8.根据实施方式7所述的方法,其中vh-vl界面残基是氨基酸残基,其侧链原子以小于或等于的距离邻接相反链的原子(在至少90%的所有重叠的fv结构中)。9.根据实施方式7至8中任意一种所述的方法,其中所述组的vh-vl界面残基包括残基l44、l46、l87、h45、h62(根据chothia指标编号)。10.根据实施方式7至9中任意一种所述的方法,其中所述组的vh-vl界面残基包括残基h35、h37、h39、h45、h47、h50、h58、h60、h61、h91、h95、h96、h98、h100x-2、h100x-1、h100x、h101、h102、h103、h105、l32、l34、l36、l38、l43、l44、l46、l49、l50、l55、l87、l89、l91、l95x-1、l95x、l96(根据chothia指标编号)。11.根据实施方式7至9中任意一种所述的方法,其中所述组的vh-vl界面残基包括残基h33、h35、h43、h44、h46、h50、h55、h56、h58、h61、h62、h89、h99、l34、l36、l38、l41、l42、l43、l44、l45、l46、l49、l50、l53、l55、l56、l85、l87、l89、l91、l93、l94/l95x-1、l95x、l96、l97、l100(根据chothia指标编号)。12.根据实施方式7至9中任意一种所述的方法,其中所述组的vh-vl界面残基包括残基h33、h35、h37、h39、h43、h44、h45、h46、h47、h50、h55、h56、h58、h60、h61、h62、h89、h91、h95、h96、h9r、h99、h100x-2、h100x-1、h100x、h101、h102、h103、h105、l32、l34、l36、l38、l41、l42、l43、l44、l45、l46、l49、l50、l53、l55、l56、l85、l87、l89、l91、l93、l94/l95x-1、l95x、l96、l97、l100(根据chothia指标编号)。13.根据实施方式7至9中任意一种所述的方法,其中所述组的vh-vl界面残基包括残基h35、h37、h39、h45、h47、h50、h58、h60、h61、h91、h95、h96、h98、h100x-2、h100x-1、h100x、h101、h102、h103、h105、l32、l34、l36、l38、l43、l44、l46、l49、l50、l55、l87、l89、l91、l95x-1、l95x、l96、l98(根据chothia指标编号)。14.根据实施方式7至9中任意一种所述的方法,其中所述组的vh-vl界面残基包括残基h33、h35、h37、h39、h43、h44、h45、h46、h47、h50、h55、h56、h58、h60、h61、h62、h89、h91、h95、h96、h98、h99、h100x-2、h100x-1、h100x、h101、h102、h103、h105、l32、l34、l36、l38、l41、l42、l43、l44、l45、l46、l49、l50、l53、l55、l56、l85、l87、l89、l91、l93、l94/l95x-1、l95x、l96、l97、l98、l100(根据chothia指标编号)。15.根据实施方式7至8中任意一种所述的方法,其中所述组的vh-vl界面残基包括残基210、296、610、612、733(根据wolfguy指标编号)。16.根据实施方式7至8和15中任意一种所述的方法,其中所述组的vh-vl界面残基包括残基199、202、204、210、212、251、292、294、295、329、351、352、354、395、396、397、398、399、401、403、597、599、602、604、609、610、612、615、651、698、733、751、753、796、797、798(根据wolfguy指标编号)。17.根据实施方式7至8和15至16中任意一种所述的方法,其中所述组的vh-vl界面残基包括残基197、199、208、209、211、251、2r9、290、292、295、296、327、355、599、602、604、607、608、609、610、611、612、615、651、696、698、699、731、733、751、753、755、796、797、798、799、803(根据wolfguy指标编号)。18.根据实施方式7至8和15至17中任意一种所述的方法,其中所述组的vh-vl界面残基包括残基197、199、202、204、208、209、210、211、212、251、292、294、295、296、327、329、351、352、354、355、395、396、397、398、399、401、403、597、599、602、604、607、608、609、610、611、612、615、651、696、698、699、731、733、751、753、755、796、796、797、798、799、801、803(根据wolfguy指标编号)。19.根据实施方式7至8和15至18中任意一种所述的方法,其中所述组的vh-vl界面残基包括残基199、202、204、210、212、251、292、294、295、329、351、352、354、395、396、397、398、399、401、403、597、599、602、604、609、610、612、615、651、698、733、751、753、796、797、798、801(根据wolfguy指标编号)。20.根据实施方式7至8和15至19中任意一种所述的方法,其中所述组的vh-vl界面残基包括残基197、199、202、204、208、209、210、211、212、251、292、294、295、296、327、329、351、352、354、355、395、396、397、398、399、401、403、597、599、602、604、607、608、609、610、611、612、615、651、696、698、699、731、733、751、753、755、796、796、797、798、799、801、803(根据wolfguy指标编号)。21.根据实施方式1至20中任意一种所述的方法,其包括选择预部20%变体抗体fv片段。22.根据实施方式1至21中任意一种所述的方法,其中通过计算六个ab角vh-vl取向参数来确定vh-vl取向。23.根据实施方式1至22中任意一种所述的方法,其中通过利用随机的森林方法计算ab角vh-vl取向参数来确定vh-vl取向。24.根据实施方式1至23中任意一种所述的方法,其中通过利用随机的森林方法为每一个ab角计算ab角vh-vl取向参数来确定vh-vl取向。25.根据实施方式1至24中任意一种所述的方法,其中通过利用随机森林模型计算vh和vl的惯常的扭转角、四个弯曲角(每个可变域两个)和中心轴长度(hl、hc1、lc1、hc2、lc2、dc)来确定vh-vl取向。26.根据实施方式25所述的方法,其中仅仅采用复合物抗体结构数据来训练随机森林模型。27.根据实施方式1至26中任意一种所述的方法,其中所述的最小差别是最高的q2值。28.根据实施方式1至27中任意一种所述的方法,其中最高的结构相似度是最低的平均均方根偏差(rmsd)。29.根据实施方式1至28中任意一种所述的方法,其中使用从在或者共有vh或者vl构架上比对的结构模板装配,随后在共有fv构架上进行vh-vl重取向的模型来确定vh-vl取向。30.根据实施方式1至28中任意一种所述的方法,其中使用在完全的fv(vh和vl同时)的β-片层核心上比对的模型来确定vh-vl取向。31.根据实施方式1至30中任意一种所述的方法,其中使用其中在共有fv构架上使抗体fv片段重取向的模型来确定vh-vl取向。32.根据实施方式1至28和30中任意一种所述的方法,其中使用利用在常见的共有fv构架上比对的模板结构并且不以任何形式调整vh-vl取向的模型来确定vh-vl取向。33.根据实施方式1至28和30中任意一种所述的方法,其中使用从在或者共有vh或者vl构架上比对的模板结构装配,随后在基于相似度选择的vh-vl取向模板结构上进行vh-vl重取向的模型来确定vh-vl取向。34.用于生产抗体的方法,其包括下列步骤:-按照根据实施方式1至33中任意一种所述的方法选择一种或更多种抗体或抗体fv片段,-基于它的结合性质,从所述的一种或更多种抗体或抗体fv片段中选择单一的抗体或抗体fc片段,-把编码vh和vl的核酸克隆到一种或更多种表达载体中,-采用在前一步骤中获得的表达载体转染细胞,-培养所转染的细胞并且从而生产所述的抗体。下列是本发明的方法和组合物的实施例。应该明白:鉴于上面提供的一般说明的情况下,各种其它的实施方式都可以被实施。不要把所述的实施例理解成用来限定本发明。真实的保护范围是在权利要求中给出的。实施例实施例1材料和方法roche抗体数据库3d(rab3d)抗体结构数据库rab3d包含大部分可公开地得到的fv结构。采用内部的“wolfguy”编号方案(参见下一部分)加工和注释fv结构。把所有注释的fv结构叠加在共有fv构架上、叠加在共有vh构架上和叠加在共有vl构架上。利用来自pdb的高分辨结构亚组计算共有结构。注释和重取向的fv结构充当同源性建模的模板仓库。wolfguy编号方案wolfguy编号把cdr区定义为kabat和chothia定义的并集。此外,所述的编号方案基于cdr长度(以及部分地基于序列)注释cdr环尖端,使得cdr位置指标指示cdr残基是否是上升或下行环的一部分。在下面表1中显示了与已确立的编号方案的比较。表1:使用chothia/kabat(ch-kb)、honegger和wolfguy编号方案给cdr-l3和cdr-h3编号。后一种编号方案具有从n端碱基到cdr峰递增的编号以及从c端cdr末端开始递减的编号。kabat方案固定最后两个cdr残基并且引入多个字母以适应cdr长度。与kabat命名法对比,honegger编号不使用字母并且对于vh和vl是常见的。这样设计wolfguy,使得尽可能采用等效的指标来给在结构上等效的残基(即在fv结构中在保守性空间定位方面非常相似的残基)编号。在图1中举例说明了这一点。在下面表2中可以找到wolfguy编号的全长vh和vl序列的实例。表2:采用wolfguy、kabat和chothia编号的具有pdbid3pp4(21)的晶体结构的vh(左边)和vl(右边)序列。在wolfguy中,仅仅依靠长度给cdr-hi-h3、cdr-l2和cdr-l3编号,同时依靠环长度和正则簇成员资格给cdr-l1编号。通过计算与不同的共有序列的序列相似度来确定正则簇成员资格。在这里,我们仅仅提供关于cdr-l1编号的单个实例,因为它对于产生我们的vh-vl取向序列指纹是不重要的。实施例1vh-vl取向指纹选择在本文中从fv序列位置(“序列指纹”)的(有意义的)亚组中而不是从完全的fv序列中预测vh-vl取向。基于这样的假定:vh-vl取向由在vh-vl界面上或靠近vh-vl界面的残基决定,已经鉴定了一组界面残基,其中如果在所述的数据库中,例如在rab3d中,在至少90%的所有重叠的fv结构中,一个残基的侧链原子以小于或等于的距离邻接相反链的原子,则把它定义为vh-vl界面的一部分。将结果概括在表29中,该表也基于统计分析(4、5、7)陈述是否已经预先将序列位置连接而使其成为vh-vl取向的决定簇。表29:vh-vl界面残基,其中在rab3d中在至少90%的所有重叠的fv结构中,如果一个残基的侧链原子以小于或等于的距离邻接相反链的原子,则它是所述的界面的一部分。*根据环长度编号+根据chothia等人(13)所定义的vh-vl界面的一部分上述组的界面残基丢失了一些被dunbar等人(7)列在关于vh-vl取向的“顶部10个重要的输入变量”之中的序列位置。那些序列位置被列在下列表30中。表30:被dunbar等人(7)列在vh-vl取向的“顶部10个重要的输入变量”之中的另外的序列位置。*根据环长度编号+根据chothia等人(13)所定义的vh-vl界面的一部分从这个可能的vh-vl取向决定序列位置集合中,装配了三种序列指纹用于统计学评价:指纹1包含已经被发现是vh-vl界面的一部分的所有的序列位置,正如在表29中所陈述的那样,第801位(l98)鉴于它们高度的序列保守性而被抛弃。指纹2包含所有的列在ab角的“顶部10个重要的输入变量”(7)之中的序列位置,即197、199、208、209、211、251、292、295、296、327、355、599、602、604、607、608、609、610、611、612、615、651、696、698、699、731、733、751、753、755、796、797、798、799、803(h33、h35、h43、h44、h46、h50、h58、h61、h62、h89、h99、l34、l36、l38、l41、l42、l43、l44、l45、l46、l49、l50、l53、l55、l56、l85、l87、l89、l91、l93、l94/l95x-1、l95x、l96、l97、l100)以及第289和290位(h55和h56)。指纹3是指纹1和指纹2的并集。为了评估仅仅基于构架序列预测vh-vl取向可能到什么程度,我们针对所述的三种指纹中的每一种生产了两种减小的变体,即a:仅仅具有处于构架边缘的最外面的cdr残基,和b:没有任何cdr残基。实施例2vh-vl取向预测子训练积累了到2013年10月为止来自rcsbpdb(www.rcsb.org)(24)的可公开地得到的抗体fv晶体结构,并且对于每一种结构,按照本文中描述的那样计算ab角vh-vl取向(参见实施例4)。此外,正如上面举例说明的那样,对于每一种结构产生了vh-vl取向序列指纹(在序列中黑色高亮显示)。所述的序列指纹由54个氨基酸组成,29个在vh区中,以及25个在vl区中。所述的序列指纹也包含属于高变区的残基,取决于环长度,这些残基可能不存在于给定的抗体序列中。在这种情况下,采用‘x’而不是呈单字母密码的规则的氨基酸描述来指示未被占据的序列指纹位置。在已经计算了ab角参数以及序列指纹这两者以后,把数据集(n=2249)分类成复合物结构(n_complex=1468)和apo(n_apo=781)结构。所述的“随机的森林”方法结果是每一个ab角取向参数的统计学上显著最好的预测子,随后是“神经网络”和“决策树”。方法“提高树”在我们的数据集上表现得最不好(没有显示数据)。对于每一个ab角参数,每一个参数进行了50次运行(每一次运行由训练阶段和测试阶段组成),同时在所述的随机森林中从10到100改变决策树的数目,以确定关于试验组的q2值的最佳的树数目。对于每一个单次运行,把输入数据集随机地分成70%训练组和30%试验组。使用accelryspipelinepilot8.5(19)采用组件“学习rp(随机分配)森林模型”以“回归”模式实施随机森林模型。在表31中列出了森林模型的参数设置的列表。表31:关于在accelryspipelinepilot8.5中使用“学习rp(随机分配)森林模型”组件的回归的参数设置。表32显示采用随机地选择的训练和试验组在50次运行上平均化的六个ab角参数的预测值的q2和均方根误差值(rmse)。表32:用于预测在50次运行上平均化的六个ab角参数的q2和rmse值。手动调整每个随机森林模型的树的数目,以便使o2最大化。在括弧中的数值具体说明标准差。在apo和复合物结构(表32,中心列)的完全数据集上训练了随机森林模型一次以及在单独的复合物结构(上面的表32,右边的列)上训练随机森林模型一次。尽管使训练组减少了将近550个结构,但是当考虑单独的复合物结构时,q2和rmse值改善了。对于hl、lc2和dc,q2值是大约0.68,同时hc1、lc1和lc2具有0.75以及以上(当考虑复合物结构时)的q2值。图2显示关于在单独的复合物结构数据集上预测的对比真实的ab角参数的示例性的回归图。进一步评估了确认组的大小(数据集的或者1/3或者1/2)是否对随机的森林预测有影响。关于所有的ab角参数,已经发现:正如将要预期的那样,在r2方面的差别有利于较小的确认组和较大的训练组(没有显示数据)。最后,经过三次重复后,评估了三种指纹和它们的减小的变体对六个不同的ab角参数的预测表现(参见表33)。表33:使用所述的三种序列指纹和它们的仅仅在构架的边缘处具有最外面的cdr残基(a)和没有任何cdr残基(b)的变体,经过三次重复后,预测六个ab角参数hl、hc1、lc1、hc2、lc2和dc的平均r2值。基于界面残基组的指纹1和基于ab角顶部输入变量位置的指纹2同样地好,当组合时,这两种(指纹3)似乎既不赋予另外的预测功效也不损害结果。在所有的三种情形中,所述的两种减小的指纹变体表现更差,这证实单独的构架序列信息不足以确定vh-vl结构域取向。指纹3被选择用于进一步评估以及随机森林模型被选择用于学习。为了把预测组件掺入同源性建模解决方案中,使用具有组件“学习rp(随机分配)森林模型”的accelryspipelinepilot8.5(19),以“回归”模式实施和训练随机森林模型。针对可在rab3d中得到的所有的fv结构(n=2249),计算了ab角vh-vl取向参数以及指纹3,并且把所述的数据集的成员分类到复合物(n=1468)和apo(n=781)结构中。对于每一个ab角参数,每一个进行50次运行,同时从10到100改变所述的随机森林中决策树的数目,以便针对试验组的q2值,确定最佳的树数目。对于每一次单次运行,把输入数据集随机地分成70%试验组和30%训练组。实施例3具有vh-vl取向调节的抗体同源性建模算法用于给抗体的fv区建模的建模软件(mofvab)使用注释的并重取向的结构,例如来自rab3d数据库,作为模板仓库。采用wolfguy为给定的一对重和轻链输入序列注释并且分别地将其简化为vh和vl。然后将vh和vl这两者分成七个功能段,即构架1、cdr1、构架2、cdr2、构架3、cdr3和构架4(参见表10)。与其它同源性建模方法对比,没有依照fv或依照链来选择常见的构架模板,而是基于序列同源性独立地检索/比对/确定每个片段。例如可以从十四个不同的模板结构以及可能甚至更多的模板结构装配单个mofvab模型,因为从不同的模板结构重建cdr环的上行和下行部分也是可行的。按照以下顺序给片段模板命中排序:1)序列相似度(blosum62矩阵得分),2)不完全侧链的数目3)模板结构的分辨率,和4)模板结构对比rab3d共有构架的比对rmsd。任选地,有可能通过从头区段来增大(或甚至整体替换)针对给定片段的可利用的模板选择。在常见的共有构架上比对所有的模板结构,因此所有的模板结构共享相同的坐标系。这样,把模板坐标转移到原始模型文件而没有进一步调整。然后加工原始模型:交换非同源的侧链,重塑不完全的模板侧链,以及通过旋转异构体最优化来消除空间上的冲突。由于独立地选择每一个片段这一事实,每个模型必需的侧链交换的数目是能够控制的。针对charmm力场把加工模型参数化并且使用具有简单变换的generalizedborn(gbsw)隐含的水模型,首先通过最陡下行,然后通过共轭梯度法,使加工模型最小化。为了保留最大量的来自模板结构的构象信息,在所述的最小化期间限制所有的还没有被重塑以及不是位于片段边缘(具有起源于不同模板结构的相邻残基)的残基。基于使用accelrysdiscoverystudio3.5(20)界面的在accelryspipelinepilot8.5(19)中实施的方案,mofvab作为网络服务是可以利用的。比较了三种mofvab建模变体以评估vh-vl结构域调整的影响:变体1:从依照链,即分别地在共有vh构架上和在共有vl构架上比对的模板结构建立模型,并且在模型加工和能量最低化之前,通过在共有fv结构上的逐链比对来调整模型的vh-vl取向。这种变体产生多个模型,其具有与序列无关的一般的、平均的vh-vl取向。变体2:按照上面描述的那样,基于序列指纹预测模型的vh-vl取向,并且根据它的ab角参数在数据库中检索最相似的fv模板。然后通过在所谓的取向模板上逐链比对来调整模型的vh-vl取向。在变体1和2这两者中,通过由dunbar等人(7)定义的35个ab角核心组残基的cα叠加来实现在或者共有fv或者取向模板上的逐链比对。变体3:从在常见的共有fv构架上而不是依照链共有结构比对的模板结构建立模型并且不调整vh-vl取向。为了产生代表性的试验组,使用mofvab来从amaii建立11个抗体fv结构。关于多种物种(兔、小鼠、人),所述的amaii结构是不同的并且主要由结合蛋白的抗体组成,抗dnafaba52(pdbid)除外。所有的amaii参比结构是以未结合的形式被结晶的。在建模的时候,通向除原始的amaii“竞争者”以外的更多种模板结构是可能的,包括许多兔抗体结构在内。因此,不能把在本文中呈现的根据rmsd的建模结果直接地与由原始的盲法建模研究呈现的结果比较。为了至少模拟盲法建模方案,没有使用来自每个链具有大于或等于95%的cdr序列同一性的结构的模板片段,其显而易见地包括amaii的原始晶体结构以及其序列变体在内。使用软件cd-hit(15,16)来进行序列相同的模板结构的鉴定以将其排除在建模之外。实施例4ab角距离计算为了比较在ab角空间方面的相似度,定义了一组ab角参数作为元组。于是在两组ab角参数之间的欧几里德距离是一组预测的ab角参数始于相关联的标准差组当计算具有标准差的一组预测的ab角参数和一组测量的ab角参数θ之间的距离时,通过使用下列加权的距离函数,把预测的不确定性这个因素包括进去:使用加权的距离函数用于在数据库中找到与预测组的ab角参数最佳匹配的取向模板。当distab角和混合角(hl、hc1、lc1、hc2、lc2)与线性(dc)距离度量时,它们不能被解释成在角空间中的实际距离,而是仅仅充当一种抽象的距离量度。为了计算ab角取向参数,以在wolfguy编号的结构上工作的稍微改良版本使用可在http://www.stats,ox.ac.uk/~dunbar/abangle/处得到的由dunbar等人(7)公布的程序代码。实施例5相关性pearson相关系数pearson相关系数度量两个变量x和y之间的线性相关性。将它计算成其中cov(x,y)是x和y之间的协方差以及σ是标准差。使用在r(25)中的标准cor.test方法来计算相关系数和p值,以评估它是否显著地不同于零。rv系数由escoufier引入了rv系数来度量对称方阵(26)之间的相似度。可以容易地把所述的定义延伸到矩形矩阵(27)。对于两个矩阵x和y,可以把rv系数计算成其中s=xxt并且t=yyt。为了计算rv系数的相关联的p值,即它是否是仅仅偶然那么高,在r中使用来自factominer包的coeffrv-方法,其进行置换检验,正如在josse等人(29)中描述的那样。基于与参比抗体的角度距离用来舍弃抗体的方法根据这样的假定:与参比比较,更大的角度距离暗示更坏的抗体结合行为,用来舍弃抗体的许多不同的方法是能够想得到的。i)舍弃最坏的20%在本文中基于它们与参比的角度距离,直接地舍弃某个百分比的抗体。例如在这里我们选择舍弃20%。因此在这种算法中的步骤是1.将角度-距离矩阵排序并且记住指标2.使用最高的20%的角度-距离指标来舍弃最坏的抗体ii)舍弃全部hc/lc在本文中舍弃全部hc或lc并且仅仅产生其它hc/lc组合。为了做到这一点,我们提出下列算法1.计算关于每一个hc/lc的平均角度-距离2.显现这些距离并且选择想要舍弃的hc/lc亚组iii)舍弃坏的hc/lc组合随后的方法的一种变型是将要仅仅舍弃具有“坏的”hc/lc组合的抗体。如果对于个别抗体,角度-距离与抗体的相关性不是如此强,但是在全部hc和lc上被很好地保留,则这种方法可能表现得很好。所述的算法是:1.计算每一个hc/lc的平均角度-距离2.显现这些距离并且选择hc/lc亚组,以便仅仅舍弃这些之间的所有可能的组合。实施例6羰基rmsd为了与amaii一致起见,使用了根据teplyakov等人(2)的β-片层核心和cdr环的羰基rmsd和定义。为了确定给定的片段的羰基rmsd,首先利用β-片层核心的cα原子,采用在accelrysdiscoverystudio3.5(20)中提供的重叠法,把模型叠加在晶体结构上。然后把羰基rmsd计算成给定区段的主链羰基原子相对于晶体结构的偏差。与通常使用的cα或全部主链rmsd比较,所述的羰基rmsd对于在主链构象方面的偏差更敏感。同时基于单独的vh或者vl的β-片层核心的叠加,计算了在amaii中所有的羰基rmsd值,同时在本文中另外基于vh和vl的β-片层核心的叠加,计算了羰基rmsd。在全部fv上叠加导致更坏的rmsd值,因为它把在vh-vl取向方面的缺陷因素包括进去了。当重新计算从http://www.3dabmod.com下载的原始amaii模型的rmsd羰基值时,不是所有的来自原始参比的值都能够被精确地复制,这归因于在叠加算法方面的微小差别或者数值不准确。实施例7采用人源化的抗cd81抗体变体的结合细胞elisa对于结合细胞elisa分析,在37℃和5%co2下,在含有10%fcs的f-12dmem培养基中增殖huh7-rluc-h3(表达cd81的阳性细胞系)和huh7-rluc-l1(阴性对照细胞系)。在第1天,使细胞以大约90%汇合度受胰蛋白酶作用并且以4x105个细胞/ml将其悬浮。在37℃和5%co2下,将2x104个细胞/孔的huh7-rluc-h3和huh7-rluc-l1(阴性对照细胞系)在50μldmem培养基中铺板并且允许其附着于96孔聚-d-赖氨酸板(greiner,目录号655940)24小时。在第2天,以两倍想要的浓度以120μl的最终体积把将要被试验的抗体样品配制在单独的聚丙烯圆底板中。把所有的分析试样稀释在细胞培养基中。把50μl每一种抗体样品(一式两份孔)添加到细胞中,以提供100μl/孔的最终体积并且在4℃下培育持续2小时。在初级培育以后,通过吸出除去样品并且在室温下采用0.05%戊二醛在pbs中的溶液(rochediagnosticsgmbh,mannheim,德国,目录号1666789)固定细胞10分钟。在固定以后,采用200μlpbs/0.05%tween洗涤每一个孔3次。在室温下在往复振荡器上进行次级培育步骤2h,用于检测结合的抗cd81抗体。对于人源化的cd81k抗体,利用过氧化物酶缀合物绵羊抗人iggγ链特异性抗体(thebindingsite,目录号ap004)和山羊抗小鼠igg进行检测,对于js81小鼠阳性对照抗体(bdbiosciences,目录号555675),使用(h+l)-hrp缀合物(biorad,目录号170-6516),这两者都在pbs10%封闭缓冲液中以1∶1000稀释。采用200μlpbs/0.05%tween洗涤每一个孔三次以除去未结合的抗体。使用50μl随时可用的tmb溶液(rochediagnosticsgmbh,mannheim,德国,目录号1432559)检测hrp活性并且在大约7-10分钟以后,采用50μl/孔的1mh2so4停止反应。使用酶联免疫吸附测定tecan读数器在450nm处采用620nm参考波长读取吸光度。实施例8动力学筛选根据schraeml等人(schraeml,m.和m.biehl,methodsmol.biol.901(2012)171-181)在安装有biacorecm5传感器的biacore4000仪器上进行动力学筛选。biacore4000仪器处于v1.1版软件的控制之下。把biacorecm5s系列芯片安装在所述的仪器中并且按照生产商的说明书以水动力方式解决问题并且预先处理。所述仪器的缓冲液是hbs-ep缓冲液(10mmhepes(ph7.4)、150mmnacl、1mmedta、0.05%(w/v)p20)。在所述的传感器表面上准备一种抗体捕获系统。使用nhs/edc化学以10,000ru,以10mm乙酸钠缓冲液(ph5)中30μg/ml把具有人igg-fc特异性的多克隆的山羊抗人抗体(jacksonlab.)固定到所述仪器的流动池1、2、3和4中的斑点1、2、4和5。在每一个流动池中,把抗体捕获到斑点1和5上。使用斑点2和斑点4作为参比斑点。采用1m乙醇胺溶液钝化传感器。以在补充有1mg/mlcmd(羧甲基葡聚糖)的仪器缓冲液中的44nm和70nm之间的浓度应用人源化的抗体衍生物。以30μl/分钟的流速注射抗体持续2分钟。以相对应答单位(ru)测量表面呈现的抗体的捕获水平(cl)。以30μl/分钟的流速,以300nm持续3分钟注射在溶液中的分析物,磷酸化的人ττ蛋白、非磷酸化的人τ蛋白和磷酸化的人ττ突变体蛋白t422s。监测解离持续5分钟。通过以30μl/分钟在所有流动池上注射1分钟的10mm甘氨酸缓冲液ph1.7来再生捕获系统。使用两个报告点:在分析物注射结束之前不久记录的信号(其被表示成结合晚期(bl))和在解离时间结束之前不久记录的信号(稳定性晚期(sl)),来表征动力学筛选表现。此外,按照朗缪尔模型计算了离解速率常数kd(1/s)以及按照公式ln(2)/(60*kd)以分钟计算了抗体/抗原复合物的半衰期。按照公式mr=(结合晚期(ru))/(捕获水平(ru))*(mw(抗体)/(mw(抗原))计算了摩尔比(mr)。在给传感器配置合适数量的抗体配体捕获水平的情况下,每一种抗体应该能够在功能上与在溶液中的至少一种分析物结合,通过mr=1.0的摩尔比来表示它。于是,摩尔比也是分析物结合的化合价方式的指标。对于结合两个分析物的抗体,化合价最大值可以是mr=2,一个分析物伴随每一个fab化合价。在另一种实施方式中,在25℃和37℃下使用相同的实验设置,但是使用每一种分析物在溶液中的多个浓度系列,在0nm(缓冲液)、1.2nm、3.7nm、11.1nm、33.3nm、100nm和300nm下测定了动力学速率。按照生产商的说明书使用biacore评估软件和使用rmax全局的朗缪尔1.1模型,从浓度依赖性的结合行为中计算了动力学数据。参考文献1.kuroda,d.,etal.,proteineng.des.sel.,25(2012)507-521.2.teplyakov,a.,etal.proteins,2014;doi:10.1002/prot.245543.dunbar,j.,etal.,nuc.acidsres.,42(2014)d1140-d1146.4.abhinandan,k.r.andmartin,a.c.r.,proteineng.des.sel.,23(2010)689-697.5.chailyan,a.,etal.,febsj.,278(2011)2858-28666.narayanan,a.,etal.,j.mol.biol.388(2009)941-953.7.dunbar,j.,etal.,proteineng.des.sel.,26(2013)611-620.8.almagro,j.c.,etal.,proteins79(2011)3050-3066.9.jayaram,n.,etal.,proteineng.des.sel.25(2012)523-529.10.banfield,m.j.,etal.,proteins,29(1997)161-171.11.li,y.,etal.,biochem.,39(2000)6296-6309.12.teplyakov,a.,etal.,actacryst.,f67(2011)1165-1167.13.chothia,c.,etal.,j.mol.biol.,278(1998)457-479.14.chothia,c.andlesk,a.m.,j.mol.biol.196(1987)901-917.15.li,w.andgodzik,a.,bioinformatics,22(2006)1658-1659.16.fu,l.,etal.,bioinformatics,28(2012)3150-3152.17.pan,r.,etal.,j.virol.,87(2013)10221-10231.18.kaas,q.,etal.,brieffunct.genom.prot.,6(2007)253-264.19.accelryssoftwareinc.,pipelinepilot,release8.5.0.200,sandiego:accelryssoftwareinc.,201120.accelryssoftwareinc.,discoverystudiomodelingenvironment,release3.5.0.12158,sandiego:accelryssoftwareinc.,2012.21.niederfellner,g.,etal.,blood,118(2011)358-367.22.kaas,q.,etal.,brieffunct.genom.prot.,6(2007)253-264.23.accelryssoftwareinc.,pipelinepilot,release8.5.0.200,sandiego:accelryssoftwareinc.,2011.24.berman,h.m.,etal.,nucl.acidsres.,28(2000)235-242.25.rcoreteam(2013).r:alanguageandenvironmentforstatisticalcomputing.rfoundationforstatisticalcomputing,vienna,austria.urlhttp://www.r-project.org/.26.robert,p.andescoufier,y.,appl.stat.25(1967)257-26527.abdi,h.,(2007).rvcoefficientandcongruencecoefficient,inn.j.salkind(ed):encyclopediaofmeasurementandstatistics.thousandoaks:sage.28.husson,f.,etal.,(2014).factominer:multivariateexploratorydataanalysisanddataminingwithr.rpackageversion1.26.http://cran.r-project.org/package=factominer29.josse,j.,etal.,comp.stat.dataanal.,53(2008)82-91.当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1