一种16SrDNA高通量测序文库的构建方法与流程
本发明涉及高通量测序领域,具体而言,涉及一种适用于16S rDNA高通量测序文库的构建方法。
背景技术:
16S rDNA是编码16S rRNA的DNA序列,存在于所有细菌基因组中,一般由9个保守区和10个可变区组成。保守区在细菌间无显著差异,可用于构建所有生命的统一进化树。可变区在不同细菌中存在一定差异,对16S rDNA可变区进行测序,可将菌群鉴定精细到分类学上属,甚至种的级别,这对研究海洋、土壤、肠道粪便等环境中的微生物构成具有重要指导意义。
目前主要采用Illumina Miseq/Hiseq测序仪对16S rDNA进行测序,但Illumina测序平台的构架决定其读长较短(最长读长为2X300bp),限制其只能对16S rDNA中某一段可变区进行测序。查阅现有文献发现,目前对16S rDNA测序主要有三种方法:测单V区(V3/V4/V6),测双V区(V3-V4或V4-V5),或者测三V区(V1-V3、V5-V7、V7-V9)。根据单V区检测结果发现,V4区在各个水平上的物种鉴定精确度最高,目前16S rDNA V4区测序已广泛应用于微生物的分类鉴定。但双V区比单V区序列读长更长,所含信息量更大,且V3-V4区引物结合特异性好,在细菌和古菌中的覆盖率均高,一次测序可同时检测细菌和古菌的多样性分布,因此,16S rDNA V3-V4区测序是微生物物种鉴定的最佳选择。
目前,基于Illimina测序平台的文库构建方法主要有两步扩增法和一步扩增法。两步扩增法是较早的建库方法,其流程如下:先进行目的片段扩增,扩增产物切胶纯化,末端修复,3’端加A,连接接头,再进行一次PCR扩增,整个建库过程需进行两次PCR。一步扩增法是目前应用较广泛的建库方法,其扩增引物同时包含了接头序列、index序列、测序引物序列和目的片段扩增序列,只需经过一步PCR扩增即可完成文库构建。
上述两种文库构建方法中,两步扩增法因需要进行两次PCR,步骤繁琐,耗时较长,且费用较高。上述一步扩增法根据其所选择目的片段扩增引物的不同,扩增产物在细菌中覆盖率不同,进而最终物种鉴定的精确度也不同,因此选择合适的引物是一步扩增法文库构建的关键步骤。此外,一步扩增法得到的产物,目前多采用琼脂糖凝胶电泳,切胶纯化的方法进行文库纯化,步骤繁琐、耗时长,且胶回收效率较低,PCR产物损失多,可能影响后续上机测序的文库量。
因此,现有的适用于16S rDNA高通量测序文库的构建方法仍需要进行改进和完善:简化步骤、缩短时间、节约成本,或者选择合适引物,改进文库纯化方法以获得高质量的文库,方便后续上机测序。
技术实现要素:
本发明的目的在于克服现有几种建库方法存在的缺点和不足:步骤繁琐、耗时长、费用高,或者扩增序列覆盖率不够高、文库回收效率低等。本发明结合现有几种建库方法的优缺点,提供了一种适用于16S rDNA高通量测序文库的构建方法。
为实现上述目的,本发明采用如下技术方案:
本发明采用一步扩增法来完成16S rDNA高通量测序的文库构建。本发明提供了一种适用于一步扩增法文库构建的引物,该引物包含一条上游引物和一条下游引物。其中,上游引物从5’端到3’ 端分别包括:P5端接头序列、index序列、上游测序引物序列、V3-V4区扩增引物上游序列。其中,下游引物从5’端到3’端分别包括:P7端接头序列、index序列、下游测序引物序列、V3-V4区扩增引物下游序列。其中,index序列由8个碱基序列组成。其中,上游引物1-29位碱基为P5端接头序列,30-37位碱基为index 1序列,38-71位碱基为上游测序引物序列,72-87位碱基为16S rDNA V3-V4区上游扩增引物序列。其中,下游引物1-33位碱基为P7端接头序列,34-41位碱基为index 2序列,42-65位碱基为下游测序引物序列,66-86位碱基为16S rDNA V3-V4区下游扩增引物序列。上下游引物共173bp。
上述一步扩增法所用引物,其中,所述引物的上游引物为下表SEQ ID NO.1-8中任意一条,下游引物为下表SEQ ID NO.9-20中任意一条。
一种16S rDNA高通量测序文库的构建方法,利用所述引物中任意一条上游引物和任意一条下游引物进行一步扩增,即得到16S rDNA测序文库。
经上述一步扩增步骤后,使用Qiagen公司的Agencourt® AMPure® XP Beads对扩增产物进行纯化,得到纯化后的测序文库。
经上述文库纯化步骤后,用Qubit和Agilent 2100进行文库初步定量和质检。
经上述文库质检步骤后,在进行高通量测序之前,采用KAPA Library Quantification Kits进行文库定量。
经上述文库定量步骤后,根据每个样本的数据量要求,进行相应比例的混合。采用Illumina Hiseq 2500/ Miseq测序仪进行测序。
与现有16S rDNA高通量测序建库方法相比较,本发明有以下进步和优势:
1.本发明提供的引物同时包含接头序列、index序列、测序引物序列和目的片段扩增序列,使得整个建库过程只需一步PCR即可完成,与两步扩增法相比,明显缩短建库时间。
2.本发明所提供的引物中所包含的目的片段扩增引物上下游分别是343F和806R,该引物能够扩增出16S rDNA的V3-V4区,与仅扩增V4区相比,扩增片段覆盖率更高,所含信息量更大,并且引物特异性好,灵敏度高。
本发明中,文库纯化所采用的是Qiagen公司的Agencourt® AMPure® XP Beads,只需25分钟即可完成文库纯化步骤,与传统的琼脂糖凝胶电泳切胶纯化(制胶、点样、电泳、纯化至少1.5小时)相比较,简化了操作步骤,节约了文库纯化时间。且本发明对常规的beads纯化法进行了改进,提供了能够使文库纯化达到最佳效果的beads用量及洗脱方法,明显提高了文库回收效率。
附图说明
图1示出了本发明所提供的引物结构示意图。
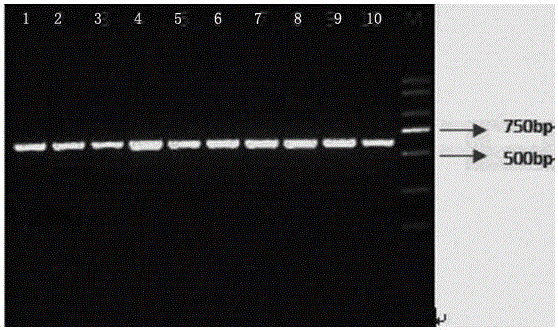
图2示出了本发明具体实施例中10个样本一步扩增法建库完成后电泳检测结果图。
具体实施方式
实施例1
(1)收集10例人肺泡灌洗液样本,每例样本约2ml,室温离心,14000rpm,30min,离心后弃上清,保留沉淀。采用Qiagen公司的QIAamp® UCP Pathogen Mini kit,按照protocol提取基因组DNA。
(2)采用nanodrop 2000检测DNA浓度和质量,选择浓度大于10 ng/ul,OD260/280=1.8-2.0的DNA进行后续文库构建操作,共10个DNA样本。
(3)10例样本PCR扩增所用引物为前述8条上游引物和12条下游引物随机自由组合,具体如下表所示:
(4)采用Takara Premix Ex TaqTM Hot Start Version进行PCR扩增。
PCR反应体系如下:
Premix Ex TaqTM Hot Start Version 25μl
Forward primer(10μM) 1μl
Reverse primer(10μM) 1μl
Template 100 ng
ddH2O 补充至50μl
(5)设定如下反应程序:
98℃ 3 min
98℃ 15 sec
54℃ 30 sec 35cycles
72℃ 45 sec
72℃ 10min
4℃ hold
反应结束后,取出PCR管放置于4℃。
(6)每例样本分别取5μl PCR产物进行琼脂糖凝胶电泳检测,跑胶结果如图2所示,其中根据本发明所用引物扩增出V3-V4区长度约为425bp,再加上上下游引物173bp,扩增产物总长约为600bp。从结果图2中可看出,10例样本扩增产物电泳目的条带大小均在600bp左右,与预期大小相符,且目的条带清晰明亮,无杂带,无拖尾现象,说明本发明所提供引物特异性好,灵敏度高。此外,该10对引物为随机选取的上下游引物自由组合,因此,利用本发明所提供的引物能够有效地对16S rDNA进行一步扩增法建库。
(7)采用Qiagen公司的Agencourt® AMPure® XP Beads进行文库纯化。提前30min将AMPure XP Beads放置于室温,使用之前上下颠倒混合均匀。
(8)向PCR产物中加入9ul AMPure XP Beads,室温静置5min。
(9)将PCR管静置于磁力架上5min,取上清于一新的离心管中,丢弃beads。
(10)向上清中加入6ul AMPure XP Beads,室温静置5min。
(11)将离心管静置于磁力架上5min,弃上清,保留beads。
(12)用80%现配乙醇洗涤两次,空气中干燥5min。
(13)加入25 μl 0.1X TE缓冲液洗脱beads,所得洗脱产物即为纯化好的文库。
(14)将上述步骤得到的纯化好的文库,用Qubit进行初步定量,所有样本浓度均达到后续上机测序要求,且10个样本浓度分别如下表所示:
(15)根据Qubit检测所得文库浓度,将样本稀释10倍,分别取出1μl用Agilent 2100检测文库片段大小,所有样本的文库片段都集中在600bp左右,说明本发明所采用的纯化方式能够有效地纯化出目的片段,去除非特异片段。
(16)经上述文库质检步骤后,在进行高通量测序之前,采用KAPA Library Quantification Kits进行文库定量。
(17)经上述文库定量步骤后,根据每个样本的数据量要求,进行相应比例的混合。采用Illumina Hiseq 2500/ Miseq测序仪进行测序。
(18)对测序所得数据进行生物信息学分析。如下表所示,各样本测序数据 GC 含量均在 50%-56%之间,clean tags 长度在 420bp左右;平均 reads 数为 8 万;数据质控合格率都在 90%以上。该结果进一步说明利用本发明所提供的引物能够有效地对16S rDNA的V3-V4区进行一步扩增法建库,且本发明采用beads法进行文库纯化,根据本发明所提供的beads用量及洗脱方式能够高效地纯化出目的片段,去除杂质及非特异片段,为后续上机测序提供足量合并且格的文库。
以上所述仅为本发明的较佳实施例,凡依本发明申请专利范围所做的均等变化与修饰,皆应属本发明的涵盖范围。
SEQUENCE LISTING
<110> 厦门基源医疗科技有限公司
<120> 一种16S rDNA高通量测序文库的构建方法
<130> 20
<160> 20
<170> PatentIn version 3.3
<210> 1
<211> 85
<212> DNA
<213> 人工序列
<400> 1
aatgatacgg cgaccaccga gatctacact atagcctaca ctctttccct acacgacgct 60
cttccgatct tacggvaggc agcag 85
<210> 2
<211> 85
<212> DNA
<213> 人工序列
<400> 2
aatgatacgg cgaccaccga gatctacaca tagaggcaca ctctttccct acacgacgct 60
cttccgatct tacggvaggc agcag 85
<210> 3
<211> 85
<212> DNA
<213> 人工序列
<400> 3
aatgatacgg cgaccaccga gatctacacc ctatcctaca ctctttccct acacgacgct 60
cttccgatct tacggvaggc agcag 85
<210> 4
<211> 85
<212> DNA
<213> 人工序列
<400> 4
aatgatacgg cgaccaccga gatctacacg gctctgaaca ctctttccct acacgacgct 60
cttccgatct tacggvaggc agcag 85
<210> 5
<211> 85
<212> DNA
<213> 人工序列
<400> 5
aatgatacgg cgaccaccga gatctacaca ggcgaagaca ctctttccct acacgacgct 60
cttccgatct tacggvaggc agcag 85
<210> 6
<211> 85
<212> DNA
<213> 人工序列
<400> 6
aatgatacgg cgaccaccga gatctacact aatcttaaca ctctttccct acacgacgct 60
cttccgatct tacggvaggc agcag 85
<210> 7
<211> 85
<212> DNA
<213> 人工序列
<400> 7
aatgatacgg cgaccaccga gatctacacc aggacgtaca ctctttccct acacgacgct 60
cttccgatct tacggvaggc agcag 85
<210> 8
<211> 85
<212> DNA
<213> 人工序列
<400> 8
aatgatacgg cgaccaccga gatctacacg tactgacaca ctctttccct acacgacgct 60
cttccgatct tacggvaggc agcag 85
<210> 9
<211> 87
<212> DNA
<213> 人工序列
<400> 9
caagcagaag acggcatacg agatcgagta atgtgactgg agttcagacg tgtgctcttc 60
cgatctggac tacvvgggta tctaatc 87
<210> 10
<211> 87
<212> DNA
<213> 人工序列
<400> 10
caagcagaag acggcatacg agattctccg gagtgactgg agttcagacg tgtgctcttc 60
cgatctggac tacvvgggta tctaatc 87
<210> 11
<211> 87
<212> DNA
<213> 人工序列
<400> 11
caagcagaag acggcatacg agataatgag cggtgactgg agttcagacg tgtgctcttc 60
cgatctggac tacvvgggta tctaatc 87
<210> 12
<211> 87
<212> DNA
<213> 人工序列
<400> 12
caagcagaag acggcatacg agatggaatc tcgtgactgg agttcagacg tgtgctcttc 60
cgatctggac tacvvgggta tctaatc 87
<210> 13
<211> 87
<212> DNA
<213> 人工序列
<400> 13
caagcagaag acggcatacg agatttctga atgtgactgg agttcagacg tgtgctcttc 60
cgatctggac tacvvgggta tctaatc 87
<210> 14
<211> 87
<212> DNA
<213> 人工序列
<400> 14
caagcagaag acggcatacg agatacgaat tcgtgactgg agttcagacg tgtgctcttc 60
cgatctggac tacvvgggta tctaatc 87
<210> 15
<211> 87
<212> DNA
<213> 人工序列
<400> 15
caagcagaag acggcatacg agatagcttc aggtgactgg agttcagacg tgtgctcttc 60
cgatctggac tacvvgggta tctaatc 87
<210> 16
<211> 87
<212> DNA
<213> 人工序列
<400> 16
caagcagaag acggcatacg agatagcttc aggtgactgg agttcagacg tgtgctcttc 60
cgatctggac tacvvgggta tctaatc 87
<210> 17
<211> 87
<212> DNA
<213> 人工序列
<400> 17
caagcagaag acggcatacg agatcatagc cggtgactgg agttcagacg tgtgctcttc 60
cgatctggac tacvvgggta tctaatc 87
<210> 18
<211> 87
<212> DNA
<213> 人工序列
<400> 18
caagcagaag acggcatacg agatttcgcg gagtgactgg agttcagacg tgtgctcttc 60
cgatctggac tacvvgggta tctaatc 87
<210> 19
<211> 87
<212> DNA
<213> 人工序列
<400> 19
caagcagaag acggcatacg agatgcgcga gagtgactgg agttcagacg tgtgctcttc 60
cgatctggac tacvvgggta tctaatc 87
<210> 20
<211> 87
<212> DNA
<213> 人工序列
<400> 20
caagcagaag acggcatacg agatctatcg ctgtgactgg agttcagacg tgtgctcttc 60
cgatctggac tacvvgggta tctaatc 87
- 还没有人留言评论。精彩留言会获得点赞!
- 一种构建Hi‑C高通量测序文库的方法与制造工艺
- 桑葚病原菌高通量鉴定及种属分类方法及其应用与制造工艺
- 基于高通量测序技术检测乳腺癌/卵巢癌易感基因的多重PCR特异性引物、试剂盒及方法与制造工艺
- 基于高通量测序检测人EGFR基因变异的质控方法及试剂盒与制造工艺
- 基于高通量测序技术的小麦病原微生物检测方法及其应用与制造工艺
- 一种基于二代高通量测序技术的地中海贫血症的基因检测试剂盒的制造方法与工艺
- 一种基于集群的高通量数据分析方法与制造工艺
- 高通量双折射干涉成像光谱仪装置及其成像方法与制造工艺
- 高通量测序检测人循环肿瘤DNA EGFR基因的捕获探针及试剂盒的制造方法与工艺
- 一种高通量测序检测无创亲子鉴定的方法与制造工艺