产甘草次酸的重组酿酒酵母、其构建方法以及用途与流程
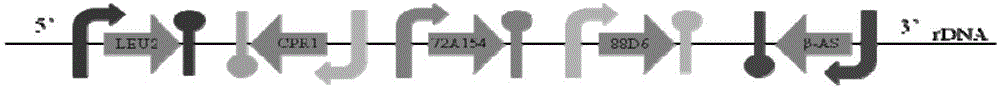
本发明涉及甘草次酸生产的技术领域。具体地,本发明涉及产甘草次酸的重组酿酒酵母、其构建方法以及用途。
背景技术:
甘草(Glycyrrhiza uralensis Fisch)是豆科甘草属多年生草本植物,以根和茎入药,有补脾益气、清热解毒、祛痰止咳、缓急止痛、调和诸药等功效,是我国应用最为广泛的传统中药材。目前,从甘草已分离得到约200余种化合物,主要分为三萜类,黄酮类及多糖类等。其中甘草酸(glycyrrhizic acid)又称甘草甜素(glycyirrhizin),是一种齐墩果烷型五环三萜类皂苷,是甘草最主要的活性成分,占甘草干重的2-8%。现代药理研究证实甘草酸有多种药学活性,抗菌抗炎、调节机体免疫力、抗溃疡等。甘草酸对于多种DNA和RNA病毒如免疫缺陷病毒(HIV),急性呼吸系统冠状病毒(SARS),肝炎病毒等均有明显抑制作用,其中对HIV抑制率高达90%。甘草酸还有降血脂与抗动脉粥样硬化作用,以及防癌、抗癌作用。除了具有上述功效作用,甘草酸的甜度是蔗糖的150倍左右,因此被作为天然甜味剂广泛应用于食品、烟草等领域。
甘草次酸是甘草酸的苷元,药理实验表明甘草酸进入人体后并不能直接被胃肠道吸收,而是首先被人体内肠道菌群首先分解为甘草次酸,被肠粘膜吸收后进入血液发挥作用。因此甘草酸实际是以其苷元的形式,即甘草次酸的形式发挥其药效作用。甘草次酸的研究证实相对甘草酸,甘草次酸具有更加好的药理活性,比如在抗血小板聚集上,甘草次酸的体外实验效果要好于甘草酸。此外,甘草次酸还具备较好的细胞毒性,因此对肿瘤细胞、病毒等都有一定的抑制作用。对甘草次酸的结构中-COOH和-OH官能团进行修饰,得到多种具备活性的衍生物。目前为止,以甘草次酸作为骨架进行修饰得到的衍生物达400多种,其中IC50<30μM的具备细胞毒性的衍生物达128种。除了作为药物之外,甘草次酸因抗炎和美白功效广泛地用于化妆品领域。甘草次酸在甘草的中含量远远低于甘草,约占甘草干重的0.6%-1.6%,不同地区不同种群中差别比较大比如亚洲地区的甘草中甘草次酸的含量一般不超过0.7%。因此,甘草次酸一般是通过甘草酸进行水解脱掉两个葡萄糖醛酸而得到,目前主要方法还是采用化学的酸解方法,另外还有酶解的方法,但是目前为止酶解的方法还处于实验室探索阶段。因其在甘草中的低含量且以甘草酸作为原料进一步水解得来,甘草次酸的价格是甘草酸价格的2-3倍,约为$500-700/kg。
因甘草酸和甘草次酸的用途广泛,国内外对甘草需求量非常大。近两年我国国内每年的需求约6万至7万吨,出口也在持续增长。2007年全球甘草市场交易额约为$42.1million。相对于甘草巨大的需求而言,甘草资源则面临着严重的不足。由于巨大需求下的无序采挖,野生甘草资源遭到严重破坏,且濒临灭绝,目前国内野生甘草储量不足20万吨。野生甘草群落具有极强的防风固沙功能。过度采挖致使野生甘草,带来的植被破坏加重了沙尘暴灾害。2000年中国政府颁布了野生甘草的禁挖令,同时对甘草的出口进行管控。目前的甘草资源主要以种植甘草为主,但是种植甘草存在最主要的问题是其功效成分甘草酸含量低于2%,达不到药典的要求。
甘草次酸结构复杂,目前还无法进行化学合成。从甘草中提取分离,则受限于有限的甘草资源以及其低含量的限制,同时大规模的甘草挖掘会带来生态环境的破坏,引发沙漠化。合成生物学是以工程学理论为依据,设计和合成新的生物元件,或是设计改造已经存在的生物系统。因此,基于合成生物学思想,通过组装药用植物功效成分合成途径,实现在大肠杆菌或者酵母等模式菌株中异源合成药用功效成分为实现中药资源可持续,利用和发展提供了新的发展机遇。美国加州大学Keasling研究团队(Paddon C J,Westfall P J,Pitera D J,et al.High-level semi-synthetic production of the potent antimalarial artemisinin[J].Nature,2013,496(7446):528.)通过在酵母细胞中转入青蒿酸合成途径,并通过代谢调控,使得构建的产青蒿酸的酵母菌株发酵产25g/L青蒿酸,之后经过4步化学催化,成功合成青蒿素,大大推进了青蒿酸的生产。目前这一成果已授权国际制药公司进行工业化生产。除此之外,紫杉醇、丹参酮、人参皂甙、吗啡、长春花碱或其前体成分的合成取得了一定的进展。Stephanopoulos(Ajikumar P K,Xiao W H,Tyo K E J,et al.Isoprenoid pathway optimization for Taxol precursor overproduction in Escherichia coli[J].Science,2010,330(6000):70.)通过模块通路工程化(modular pathway engineering)的策略,将紫杉烯(taxadiene)的合成途径分为两个模块进行调控,一是大肠杆菌上游的MEP途径,使进入萜类物质前体IPP碳流增加,二是异源紫杉烯合成途径的整合入大肠杆菌,通过调控两个模块的代谢流,最终紫杉醇的前体成分紫杉烯的产量达到1g/L。Sarah E.O’Connor团队(Brown S.Clastre M.Courdavault V.et al.2015.De novo production of the plant-derived alkaloid strictosidine in yeast.Proc Natl Acad Sci U S A.112(11):3205-3210..)通过一系列的基因重排,实现了在微生物中合成异胡豆苷这一长春花碱的重要前体,除了一些必要的基因导入菌株外,还对启动子进行优化,增加了5个基因的额外副本tHMGR,MAF1,IDI1,SAM2,ZWF1,删除ERG20,ATF1,OYE2,异胡豆苷产量为0.5mg/L。Dai等(Dai Z,Yi L,Zhang X,et al.Metabolic engineering of Saccharomyces cerevisiae,for production of ginsenosides)将人参来源的达玛二烯合成酶基因(PgDDS)、原人参二醇合成酶基因(CYP716A47)和拟南芥(A.thaliana)来源的At CPR1基因,导入酵母菌株中,并对相关基因tHMG1、ERG20、ERG9、ERG1等进行了系统调控,同时对相关密码子进行优化构建出了产原人参二醇的工程菌株,原人参二醇的产量1.2g/L,较原始出发菌株产量提高了262倍。这是首次人参皂甙重要前体物质原人参二醇在酵母中实现从葡萄糖的合成。
功能基因的挖掘与鉴定是实现天然产物功效成分合成生物学的前题。对于甘草酸生物合成途径的研究,国内外已取得一些进展。其合成途径基本清晰,作为典型的齐墩果烷型三萜化合物,甘草酸的合成由三萜类化合物的共同前体2,3氧化-鲨烯经过环化、羟基化以及糖基化最终形成甘草酸。具体地,2,3氧化鲨烯在β-香树脂醇合酶(β-AS)催化下生成β-香树脂醇,β-AS基因是催化生成甘草酸的重要分支点,在形成甘草酸五环三萜母核骨架上具有重要作用。接着,由一系列的CYP450基因对形成的母核进行羟基化修饰。2008年日本Toshiya Muranaka团队在PNAS(Seki H,Ohyama K,Sawai S,Mizutani M,Ohnishi T,Sudo H,Akashi T,Aoki T,Saito K,Muranaka T.Licorice beta-amyrin 11-oxidase,a cytochrome P450with a key role in the biosynthesis of the triterpene sweetener glycyrrhizin.Proc Natl Acad Sci U S A.2008,105:14204-14209.)发表文章,利用甘草转录组信息成功鉴定了CYP88D6这一基因并在酵母系统中实现成功表达,该基因对β-香树脂醇的C-11进行羟基化和进一步羧基化。2011年该团队在The Plant Cell(Seki H,Sawai S,Ohyama K,Mizutani M,Ohnishi T,Sudo H,Fukushima EO,Akashi T,Aoki T,Saito K,Muranaka T.Triterpene functional genomics in licorice for identification of CYP72A154involved in the biosynthesis of glycyrrhizin.Plant Cell.2011,23:4112-4123.)上发表文章,同样利用甘草转录组进一步鉴定了CYP72A154和CYP72A63这一基因,该基因对β-香树脂醇的C-30催化形成羧酸,形成甘草次酸。至此,甘草次酸合成途径的催化基因全部鉴定。这些工作为甘草次酸的合成打下基础。
利用微生物进行天然产物的生物合成具有多方面的优势,摆脱了对于原料的依赖,减少了土地使用,同时生长周期短、发酵原料低廉且利用率高、发酵产物合成高效单一,副产物少、条件温和、对环境污染少、便于分离。
技术实现要素:
基于甘草次酸的应用市场和巨大需求和当前甘草资源有限的限制,本发明提供了一株产甘草次酸酵母菌株及其构建方法,通过过表达酵母自身10个基因、3个甘草次酸合成途径中外源基因以及酵母自身基因的删除等技术,最终通过酵母发酵即可实现甘草次酸的生产。
一方面,本发明提供了一种产甘草次酸的重组酵母菌株,所述重组酵母菌株过表达酵母基因乙酰乙酰辅酶A硫解酶基因(ERG10)、甲羟戊酸激酶基因(ERG12)、HMG-CoA合酶基因(ERG13)、二磷酸甲羟戊酸脱羧酶基因(ERG19)、法尼基焦磷酸合酶(ERG20)、角鲨烯合酶(ERG9)、鲨烯环氧酶(ERG1)、磷酸甲羟戊酸激酶基因(ERG8)、异戊烯基二磷酸异构酶基因(IDI1)和截短3-羟基-3-甲基戊二酰辅酶A还原酶(tHMG);以及所述重组酵母菌株表达甘草次酸合成途径中的香树精合酶(β-AS)、CYP88D6和CYP72A154基因以及拟南芥的细胞色素P450还原酶(CPR1)基因;同时所述重组酵母菌株删除了GGPP合酶基因(BTS1)。
根据本发明所述的重组酵母菌株,其于2016年10月21日保藏在中国微生物菌种保藏管理委员会普通微生物中心(CGMCC),地址为:北京市朝阳区北辰西路1号院3号,中国科学院微生物研究所;邮编:100101;保藏编号为CGMCC13126;其分类命名为酿酒酵母(Saccharomyces cerevisiae)。
另一方面,本发明提供了一种制备生产甘草次酸的重组酵母菌株的方法,所述方法包括步骤:
a)从甘草植物中获得参与甘草次酸合成途径的β-AS、CYP88D6和CYP72A154基因,从拟南芥中获得CPR1基因片段;
b)将步骤a)中获得的β-AS、CYP88D6、CYP72A154以及CPR1基因片段,构建Ppgk1-β-AS-Tadh1、Ptdh3-CYP88D6-Tcyc1、Padh1-CYP72A154-adh1、Ptdh3-CPR1-Tcyc1基因表达簇,将上述四个基因表达簇整合至酵母菌染色体Cen.pk2-1D的rDNA位点;
c)将步骤b)中的CYP88D6和CYP72A154基因进行密码子优化,获得OPCYP88D6和OPCYP72A154基因,构建Ptdh3-OPCYP88D6-Tcyc1、Padh1-OPCYP72A154-adh1基因簇,连同Ppgk1-β-AS-Tadh1、Ptdh3-CPR1-Tcyc1基因簇,一起整合入酿酒酵母Cen.pk2-1D的rDNA位点;
d)从酵母中PCR扩增获得酵母菌株的ERG9、ERG20、ERG1和tHMG片段,将ERG20和ERG9基因进行融合获得融合片段ERG20+9或者ERG9+20,连同ERG1和tHMG一起,构建其对应的基因表达簇Ptdh3-E20+9-Tcyc1、Ptdh3-E9+20-Tcyc1、Ptef1-ERG1-Tpgk1、Ppgk1-tHMG-Tadh1将上述基因簇整合至步骤c)所构建菌株的染色体delta位点;
e)从酵母中PCR扩增获得酵母菌株的ERG10、ERG8、ERG13,构建其对应的基因簇Padh1-E10-Tadh1、Ptdh3-ERG8-Ttdh3、Padh1-E13-Tadh1,将这三个基因表达簇整合至步骤d)的酵母菌染色体上的trp位点的+314bp位置,使ERG10、ERG8和ERG13基因过表达;和
f)从酵母中PCR扩增获得酵母菌株的ERG12、ERG19和IDI1基因,从甘草中的扩增获得甘草的细胞色素b5的编码基因(CYB5),构建对应的基因表达簇Padh1-ERG12-Tadh1、Ptef2-ERG19-cyc1、Ppgk1-IDI-Tpgk1、Ptdh3-Cyb5-Ttdh3,将这四个基因表达簇整合至步骤e)中酵母菌染色体的BTS1基因位点,获得最终的重组酿酒酵母GA-5。
根据本发明所述的制备生产甘草次酸的重组酵母菌株的方法,其中所述密码子优化的CYP88D6和CYP72A154的序列分别如SEQ ID NO:1和SEQ ID NO:2所示、所述CYB5的编码基因如SEQ ID NO:3所示。
另一方面,本发明提供了根据本发明的重组酵母菌株在生产甘草次酸中的用途。
另一方面,本发明涉及一种生产甘草次酸的方法,所述方法包括在适当的发酵条件下发酵根据本发明所述的产甘草次酸的重组酵母菌株的步骤。
根据本发明所述的生产甘草次酸的方法,所述方法包括在pH=5、在培养温度30℃下,在由50g/L葡萄糖、10g/L胰蛋白胨、20g/L酵母提取物、0.2g/L尿嘧啶、0.04mol/L甲基-β-环糊精组成的培养基中发酵根据本发明所述的产甘草次酸的重组酵母菌株的步骤;待葡萄糖耗尽后,采用乙醇补料发酵,每隔12h流加乙醇使得乙醇的浓度为7g/L。
附图说明
图1A至图1F分别表示实施例3-7中所构建的基因表达簇的整合顺序。其中E10缩写代表基因ERG10,E8代表基因ERG8,E19代表基因ERG19,E13代表基因ERG13,E12缩写代表基因ERG12;72A代表基因CYP72A154,88D6代表基因CYP88D6,OP88D6代表密码子优化的CYP88D6基因,OP72A代表密码子优化的CYP72A154基因。
图2A-图2C,本发明实施例中的质粒构建方法示意图。
图2A,通过无缝连接的方式构建带有启动子-酶切位点-终止子的克隆载体示图puc19L-Ptef1-Tpgk1,puc19L-Ptdh3-Tcyc1,puc19L-Ppgk1-Tadh1,puc19L-Ppgk1-Tpgk1,puc19L-Ptef2-Tcyc1和puc19L-Ptdh3-Ttdh3均通过该方式进行构建;
图2B,通过酶切连接方式进行基因表达簇构建,本专利中的ERG8,ERG12,IDI,ERG10,CYP72A154,OPCYP72A154和Cyb5基因都是通过该方式构建的表达簇;
图2C,通过无缝连接方式构建基因表达簇,本专利中Ptef2-ERG19-cyc,Ptdh3-CYP88D6-Tcyc1,Ptdh3-OPCYP88D6-Tcy,Ppgk1-β-AS-Tadh1,Ppgk1-tHMG-Tadh1,Ptdh3-ERG20+9-Tcyc1,Ptdh3-ERG9+20-Tcyc1,Ptef1-ERG1-Tpgk1,Ptdh3-CPR1-Tcyc1是通过该方式进行基因簇的构建。
图3显示LC-MS色谱图。上图:甘草次酸标准品LC-MS色谱图,横坐标为保留时间,纵坐标为丰度,出峰时间6.72min;下图:工程菌株GA-5发酵样品LC-MS色谱图,出峰时间6.76min。
图4显示LC-MS质谱图,横坐标为M/Z,纵坐标为丰度。上图:甘草次酸标准品在6.72min处峰的LC-MS质谱图;下图:工程菌株GA-5发酵样品在6.76min处峰的LC-MS质谱图。
图5显示工程菌株GA-5发酵产β-香树精的GCMS检测图。上图:GCMS检测发酵样品β-香树精色谱图,出峰时间16.44min处为β-香树精色谱图,横坐标为保留时间,纵坐标为丰度;下图:GCMS检测发酵样品β-香树精质谱图,横坐标为M/Z,纵坐标为丰度。
图6显示工程菌株GA-5发酵产甘草次酸的GCMS检测图。上图:GCMS检测发酵样品甘草次酸色谱图,出峰时间22.5min处为甘草次酸色谱图,横坐标为保留时间,纵坐标为丰度;下图:GCMS检测发酵样品甘草次酸质谱图,横坐标为M/Z,纵坐标为丰度。
图7显示酵母染色体整合方式示意图。
具体实施方式
出于说明本发明各种实施方式的目的给出如下实施例,并非意图以任何方式限制本发明。本领域技术人员将理解,如权利要求的范围所限定的,其中的变化和其它用途包括在本发明精神范围内。下列实施例中所使用的材料、试剂等,如无特殊说明,均可从商业途径得到,实施例中所提到的基因,如无特殊说明,均为基因的完整编码框区。均可以从NCBI获得,所提到的启动子和终止子序列也可以从NCBI下载获得,具体序列起始位置可根据引物表中的引物获悉。
实施例1.甘草次酸合成所需基因以及模块化调控中酵母自身基因元件的获得
以新鲜乌拉尔甘草为材料,采用本领域公知的方法,提取RNA,反转录得到cDNA。根据本领域的常规方法,设计β-AS、CYP88D6、CYP72A154、Cyb5基因的引物,扩增上述片断;以酿酒酵母CEN.PK2-1D的基因组为模板,设计ERG10、ERG12、ERG13、ERG19、ERG20、ERG9、ERG1、ERG8、IDI1、tHMG基因片段的引物,扩增酵母中上述基因片段。
实施例2.甘草次酸合成所需基因以及模块化调控中酵母自身基因元件构成表达簇的获得
具体地表达簇是指启动子+基因片段序列+终止子。进一步表达簇的构建分为两个步骤:首先将酵母常用的组成型启动子和终止子序列扩增出来,这里扩增的启动子包括P-TEF1、P-TEF2、P-TDH3、P-ADH1和P-PGK1,终止子序列包括:T-PGK1、T-CYC1、T-TDH3和T-ADH1,扩增上述启动子和终止子序列所用的引物见表1。将扩增的启动子序列和终止子序列以启动子-酶切位点-终止子的顺序连接到克隆载体puc19L上,构建puc19L-Ptef1-Tpgk1、puc19L-Ptdh3-Tcyc1、puc19L-Ppgk1-Tadh1、puc19L-Ppgk1-Tpgk1、puc19L-Ptef2-Tcyc1、puc19L-Ptdh3-Ttdh3载体,启动子和终止子序列的连接采用无缝连接的方式进行。
将实施例1中扩增的基因通过无缝连接(如图2C所示)将基因片段连接到启动子和终止子中间,构建了相应基因的表达簇,具体为,Ptef2-ERG19-Tcyc1、Ptdh3-ERG9+20-Tcyc1、Ptdh3-ERG20+9-Tcyc1、Ptef1-ERG1-Tpgk1、Ppgk1-tHMG-Tadh1、Ppgk1-β-AS-Tadh1、Ptdh3-CYP88D6-Tcyc1、Ptdh3-CPR1-Tcyc1、ERG8、ERG10、ERG12、ERG13、IDI1这些基因通过酶切连接的方式(如图2B所示)连接到启动子和终止子之间,构成Ptdh3-ERG8-Ttdh3、Padh1-ERG10-Tadh1、Ptdh3-ERG12-Ttdh3、Padh1-ERG13-Tadh1、Padh1-CYP72A154-adh1、Padh1-Cyb5-adh1、Ppgk1-IDI1-Tpgk1、Ptdh3-Cyb5-Ttdh3。
表1
实施例3.在酵母中创建甘草次酸的合成途径
将Ppgk1-β-AS-Tadh1、Ptdh3-CYP88D6-Tcyc1、Padh1-CYP72A154-adh1和Ptdh3-CPR1-Tcyc1这些基因表达簇以同源重组的形式(图7所示)整合到酵母染色体的rDNA位点,PCR检测阳性克隆,检测阳性克隆菌株产甘草次酸的能力,得到产甘草次酸最高的酵母菌株命名GA-1。其中,外源基因表达簇片段整合的顺序如图1A所示。
实施例4.获得产甘草次酸酵母菌株GA-2
对CYP88D6,CYP72A154进行密码子优化,密码子优化的CYP88D6和CYP72A15的序列见表1。连接启动子和终止子形成表达簇Ptdh3-OPCYP88D6-Tcyc1和Padh1-OPCYP72A154-adh1,将Ppgk1-β-AS-Tadh1、Ptdh3-OPCYP88D6-Tcyc1、Padh1-OPCYP72A154-adh1、Ptdh3-CPR1-Tcyc1这些基因表达簇以同源重组形式(如图7所示)整合到酵母染色Cen.pk2-1D体的rDNA位点,PCR检测阳性克隆,检测阳性克隆菌株产甘草次酸的能力,得到产甘草次酸最高的酵母菌株命名GA-1,得到产甘草次酸最高的酵母菌株命名GA-2。其中基因表达簇片段整合的顺序如图1B所示。
表2
实施例5.获得产甘草次酸酵母菌株GA-3
以酵母菌株GA-2为出发菌株,扩增Ptdh3-E9+20-Tcyc1(Ptdh3-E20+9-Tcyc1)、Ptef1-ERG1-Tpgk1、Ppgk1-tHMG-Tadh1的基因表达簇,将这些片段以同源重组形式整合在酿酒酵母GA-2)delta位点,挑选阳性克隆,检测甘草次酸含量,得到产甘草次酸最高的工程菌株酵母GA-3。其中基因表达簇片段整合的顺序如图1C或图1D所示。
实施例6.获得产甘草次酸酵母菌株GA-4
以酿酒酵母GA-3为出发菌株,扩增Ptdh3-ERG8-Ttdh3、Padh1-E13-Tadh1、Padh1-E10-Tadh1的基因表达簇,以his3作为筛选标记,将这些基因表达簇整合入酿酒酵母GA-3染色体的Trp1位点,挑选阳性克隆,检测甘草次酸含量,以甘草次酸含量最高的菌株名为酿酒酵母GA-4。其中基因表达簇片段整合的顺序如图1E所示。
实施例7.获得产甘草次酸酵母菌株GA-5
以酿酒酵母GA-4为出发菌株,扩增Padh1-ERG12-Tadh1、Ptdh3-Cyb5-Ttdh3、Ptef2-ERG19-cyc1、Ppgk1-IDI-Tpgk1的基因表达簇,分别以BTS1的-210-502序列和530-1008的序列为同源序列,以trp1作为筛选标记,共同整合入酿酒酵母GA-4的染色体,在破坏掉BTS1基因编码框的基础上将ERG12、Cyb5、ERG19基因过表达,增强菌株MVA途径的代谢流。挑选阳性克隆,检测甘草次酸含量,以甘草次酸含量最高的菌株名为酿酒酵母GA-5。其中基因表达簇片段整合的顺序如图1F所示。所述GA-5于2016年10月21日保藏在中国微生物菌种保藏管理委员会普通微生物中心,保藏编号为CGMCC13126。
实施例8.酵母菌株GA-5发酵产甘草次酸
通过上述代谢工程优化,获得高产甘草次酸酵母菌株GA-5,进一步对其发酵条件进行优化,具体的发酵条件为pH=5,培养基的组成为葡萄糖:50g/L、胰蛋白胨:10g/L、酵母提取物:20g/L,尿嘧啶:0.2g/L,甲基-β-环糊精:0.04mol/L;待葡萄糖耗尽后,采用乙醇补料发酵策略,每隔12h流加乙醇使得乙醇的浓度为7g/L;培养温度30℃,最终通过GCMS检测,摇瓶发酵产甘草次酸浓度达445mg/L。
实施例9.酵母细胞产甘草次酸及中间代谢物的样品及检测处理方法
样品处理:取4ml的酿酒酵母工程菌株,12000rmp离心10min弃上清,加入无菌水清洗3次,12000rmp离心10min弃上清,加入1ml的甲醇丙酮萃取剂(甲醇:丙酮(v/v)=1:1),超声破碎10min,12000rmp离心10min收集上清液,取100ul加入内衬管进行氮吹蒸干,加入N-甲基-N-三甲基硅三氟乙酰胺,80℃衍生化处理30min。
样品检测:样品定性和定量均采用仪器型号Agilent 7890GC-7000MS/MS气相色谱-三重串联四极杆质谱,进样口温度为300℃;色谱柱为:2根HP-5ms UI 15m×0.25mm×0.25μm,中间以Purged Ultimate Union(PUU)连接;采用3.0mL/min的标准模式进行隔垫吹扫;色谱柱流速:恒流,柱1:1.1mL/min;柱2:1.3mL/min;柱温箱升温程序:80℃保持1min,以20℃/min的速度升至310℃保持17.5min;传输线温度为300℃;反吹设置:柱温箱于310℃反吹7min,辅助气压力50psi,进样口压力2psi;离子源温度为280℃;四级杆Q1,Q2均为150℃;碰撞池气体流速:氦气流量2.25mL/min,氮气流量1.5mL/min;进样体积为2ul,数据采集模式,鲨烯、麦角固醇、羊毛甾醇以及β-香树脂醇采用全扫描scan模式,甘草次酸为MRM模式,具体定性离子如下表所示:
表3
样品中甘草次酸LCMS定性的检测方法:仪器Agilent LCMS 6420,色谱柱为安捷伦C18色谱柱,柱温为25℃,流速为0.2ml/min,流动相为甲醇和水,梯度洗脱条件为,0–0.5min,1%甲醇;0.5–1.0min,1–80%甲醇;1.0–2.0min,80–90%甲醇;2.0–3.5min,90–1%甲醇,进样体积为2ul,对甘草次酸定性质谱采集m/z设置为469.44-355.38。
序列表
<110> 中国中医科学院中药研究所
<120> 产甘草次酸的重组酿酒酵母、其构建方法以及用途
<130> 王彩霞
<160> 22
<170> PatentIn version 3.3
<210> 1
<211> 1482
<212> DNA
<213> 人工序列
<220>
<223> 密码子优化的CYP88D6
<400> 1
atggaagttc attgggtttg tatgtcagct gcaactttgt tggtttgtta catcttcggt 60
tctaagttcg ttagaaattt gaacggttgg tactacgatg ttaagttgag aagaaaggaa 120
catccattac caccaggtga catgggttgg ccattaattg gtgacttgtt gtcttttatt 180
aaggatttct cttcaggtca tccagattct tttattaaca atttggtttt gaagtacggt 240
agatcaggta tctataagac tcatttgttc ggtaacccat ctatcatcgt ttgtgaacca 300
caaatgtgta gaagagtttt gacagatgat gttaacttca agttgggtta cccaaagtct 360
attaaagaat tggctagatg tagaccaatg attgatgttt caaacgcaga acatagattg 420
tttagaagat taatcacatc tccaatcgtt ggtcataaag ctttagcaat gtacttggaa 480
agattggaag aaatcgttat taattcattg gaagaattgt cttcaatgaa gcatccagtt 540
gaattgttga aggaaatgaa gaaagtttct tttaaagcta tcgttcatgt ttttatgggt 600
tcttcaaacc aagatattat taagaaaatt ggttcttctt ttactgattt gtacaacggc 660
atgttctcta tcccaattaa tgttccaggt tttacattcc ataaagcttt ggaagcaaga 720
aagaaattgg ctaagatcgt tcaaccagtt gttgatgaaa gaagattaat gatcgaaaat 780
ggtccacaag aaggttcaca aagaaaggat ttgatcgata tcttgttaga agttaaagat 840
gaaaatggta gaaaattaga agatgaagat atttctgatt tgttaattgg tttgttattt 900
gctggtcatg aatctactgc aacatcattg atgtggtcta tcacttactt aacacaacat 960
ccacatatct tgaagaaagc taaggaagaa caagaagaaa tcactagaac aagattttct 1020
tcacaaaaac aattgtcatt gaaggaaatt aaacaaatgg tttatttgtc tcaagttatt 1080
gatgaaactt tgagatgtgc aaacattgct tttgcaactt ttagagaagc tacagcagat 1140
gttaacatca acggttacat catcccaaaa ggttggagag ttttgatttg ggctagagca 1200
atccatatgg attcagaata ctacccaaat ccagaagagt ttaatccatc tagatgggat 1260
gattacaatg ctaaagcagg tacattttta ccatttggtg ctggttcaag attgtgtcca 1320
ggtgctgatt tggcaaagtt ggaaatctct attttcttgc attacttttt gttaaattac 1380
agattggaaa gaattaatcc agaatgtcat gttacttcat taccagtttc taaaccaaca 1440
gataactgtt tggctaaagt tattaaagtt tcttgtgcat aa 1482
<210> 2
<211> 1572
<212> DNA
<213> 人工序列
<220>
<223> 密码子优化的CYP72A154序列
<400> 2
atggatgctt cttcaactcc aggtgcaatt tgggttgttt tgacagttat tttagctgca 60
attccaattt gggcttgtca tatggttaac actttgtggt tgagaccaaa gagattggaa 120
agacatttga gagcacaagg tttgcatggt gacccataca agttgtcttt ggataactca 180
aagcaaacat acatgttgaa gttgcaacaa gaagctcaat ctaagtcaat cggtttgtct 240
aaggatgatg ctgcaccaag aattttctct ttggcacatc aaactgttca taagtacggt 300
aaaaattctt ttgcttggga aggtactgca ccaaaagtta ttattacaga tccagaacaa 360
attaaagaag tttttaataa gattcaagat ttcccaaagc caaagttgaa cccaattgct 420
aagtacatct caatcggttt gatccaatac gaaggtgaca agtgggctaa gcatagaaag 480
attattaacc cagcattcca tttggaaaag ttgaagggca tgttgccagc tttttctcat 540
tcatgtcatg aaatgatctc taagtggaag ggtttgttgt cttcagatgg tacatgtgaa 600
gttgatgttt ggccattttt gcaaaatttg acttgtgatg ttatctcaag aacagctttt 660
ggttcttcat acgctgaagg tgcaaagatc ttcgaattgt tgaagagaca aggttacgca 720
ttgatgactg ctagatacgc aagaattcca ttatggtggt tgttgccatc tactacaaag 780
agaagaatga aggaaatcga tagaggtatc agagattcat tggaaggtat catcagaaag 840
agagaaaagg ctttgaagtc tggtaaatca acagatgatg atttgttagg tatcttgttg 900
caatctaacc atatcgaaaa taagggtgac gaaaactcta aatcagctgg tatgactaca 960
caagaagtta tggaagagtg taagttgttt tatttggctg gtcaagaaac tacagctgca 1020
ttgttagcat ggactatggt tttgttaggt aaacatccag aatggcaagc tagagcaaag 1080
caagaagttt tgcaaatctt cggtaaccaa aacccaaact tcgaaggttt gggtagattg 1140
aagatcgtta ctatgatctt gtacgaagtt ttgagattgt acccaccagg tatatatttg 1200
acaagagctt tgagaaagga tttgaagttg ggtaatttgt tattgccagc aggtgttcaa 1260
ttttctgttc caatcttgtt gatccatcat gatgaaggta tttggggtaa tgatgctaaa 1320
gaattcaatc cagaaagatt tgcagatggt attgctaagg caacaaaggg tcaagtttgt 1380
tacttcccat ttggttgggg tccaagaatc tgtgttggtc aaaacttcgc tttgttggaa 1440
gcaaagattg ttttgtcttt gttgttgcaa aacttctctt tcgaattatc accaacttac 1500
gctcatgttc caactacagt tttgacatta caaccaaaac atggtgcacc aattattttg 1560
cataaattat aa 1572
<210> 3
<211> 405
<212> DNA
<213> 甘草
<400> 3
atggcttcag atccaaagct tcacactttc gatgaggtgg caaagcacaa ccagaccaaa 60
gattgctggc ttatcatttc tgggaaggtg tatgatgtca ccccttttat ggaggatcat 120
cccggaggtg atgaggtttt gttatctgca acagggaaag atgcaaccaa tgattttgaa 180
gatgtggggc acagtgattc tgctagagaa atgatggaca agtactatat tggtgagatt 240
gattcctcaa ctgtcccact aaagcgtacc tacgttccac ctcagcaaac ccagtacaac 300
cctgacaaga cctcggaatt cgtgatcaag atcttgcagt tcctggtccc tctcctcata 360
ttgggcttag cctttgttgt ccgacactac accaagaatg agtag 405
<210> 4
<211> 1579
<212> DNA
<213> 甘草
<400> 4
tgcagaccaa ttggtgaaaa ctgaagtcac caagaagtct tttactgctc ctgtacaaaa 60
ggcttctaca ccagttttaa ccaataaaac agtcatttct ggatcgaaag tcaaaagttt 120
atcatctgcg caatcgagct catcaggacc ttcatcatct agtgaggaag atgattcccg 180
cgatattgaa agcttggata agaaaatacg tcctttagaa gaattagaag cattattaag 240
tagtggaaat acaaaacaat tgaagaacaa agaggtcgct gccttggtta ttcacggtaa 300
gttacctttg tacgctttgg agaaaaaatt aggtgatact acgagagcgg ttgcggtacg 360
taggaaggct ctttcaattt tggcagaagc tcctgtatta gcatctgatc gtttaccata 420
taaaaattat gactacgacc gcgtatttgg cgcttgttgt gaaaatgtta taggttacat 480
gcctttgccc gttggtgtta taggcccctt ggttatcgat ggtacatctt atcatatacc 540
aatggcaact acagagggtt gtttggtagc ttctgccatg cgtggctgta aggcaatcaa 600
tgctggcggt ggtgcaacaa ctgttttaac taaggatggt atgacaagag gcccagtagt 660
ccgtttccca actttgaaaa gatctggtgc ctgtaagata tggttagact cagaagaggg 720
acaaaacgca attaaaaaag cttttaactc tacatcaaga tttgcacgtc tgcaacatat 780
tcaaacttgt ctagcaggag atttactctt catgagattt agaacaacta ctggtgacgc 840
aatgggtatg aatatgattt ctaaaggtgt cgaatactca ttaaagcaaa tggtagaaga 900
gtatggctgg gaagatatgg aggttgtctc cgtttctggt aactactgta ccgacaaaaa 960
accagctgcc atcaactgga tcgaaggtcg tggtaagagt gtcgtcgcag aagctactat 1020
tcctggtgat gttgtcagaa aagtgttaaa aagtgatgtt tccgcattgg ttgagttgaa 1080
cattgctaag aatttggttg gatctgcaat ggctgggtct gttggtggat ttaacgcaca 1140
tgcagctaat ttagtgacag ctgttttctt ggcattagga caagatcctg cacaaaatgt 1200
tgaaagttcc aactgtataa cattgatgaa agaagtggac ggtgatttga gaatttccgt 1260
atccatgcca tccatcgaag taggtaccat cggtggtggt actgttctag aaccacaagg 1320
tgccatgttg gacttattag gtgtaagagg cccgcatgct accgctcctg gtaccaacgc 1380
acgtcaatta gcaagaatag ttgcctgtgc cgtcttggca ggtgaattat ccttatgtgc 1440
tgccctagca gccggccatt tggttcaaag tcatatgacc cacaacagga aacctgctga 1500
accaacaaaa cctaacaatt tggacgccac tgatataaat cgtttgaaag atgggtccgt 1560
cacctgcatt aaatcctaa 1579
<210> 5
<211> 18
<212> DNA
<213> 人工序列
<220>
<223> P-PGK1-F引物
<400> 5
aaagatgccg atttgggc 18
<210> 6
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> P-PGK1-R引物
<400> 6
gttttatatt tgttgtaaaa agtagataat 30
<210> 7
<211> 21
<212> DNA
<213> 人工序列
<220>
<223> P-TDH3-F引物
<400> 7
gacacaaggc aattgaccca c 21
<210> 8
<211> 25
<212> DNA
<213> 人工序列
<220>
<223> P-TDH3-R引物
<400> 8
tttgtttgtt tatgtgtgtt tattc 25
<210> 9
<211> 29
<212> DNA
<213> 人工序列
<220>
<223> P-TEF2-F引物
<400> 9
gataggtcaa gatcaatgta aacaattac 29
<210> 10
<211> 28
<212> DNA
<213> 人工序列
<220>
<223> P-TEF2-R引物
<400> 10
aaacgtttag ttaattatag ttcgttga 28
<210> 11
<211> 22
<212> DNA
<213> 人工序列
<220>
<223> T-CYC1-F引物
<400> 11
gtttaaacac aggccccttt tc 22
<210> 12
<211> 21
<212> DNA
<213> 人工序列
<220>
<223> T-CYC1-R引物
<400> 12
aaaatatgca catgaggcga a 21
<210> 13
<211> 26
<212> DNA
<213> 人工序列
<220>
<223> T-PGK1-F引物
<400> 13
gaaataaatt gaattgaatt gaaatc 26
<210> 14
<211> 24
<212> DNA
<213> 人工序列
<220>
<223> T-PGK1-R引物
<400> 14
agctttaacg aacgcagaat tttc 24
<210> 15
<211> 21
<212> DNA
<213> 人工序列
<220>
<223> T-ADH1F引物
<400> 15
gcgaatttct tatgatttat g 21
<210> 16
<211> 21
<212> DNA
<213> 人工序列
<220>
<223> T-ADH1R引物
<400> 16
gaatgacgat gaagatagag c 21
<210> 17
<211> 23
<212> DNA
<213> 人工序列
<220>
<223> P-TEF1-F引物
<400> 17
tcaatagtca tacaacagaa agc 23
<210> 18
<211> 25
<212> DNA
<213> 人工序列
<220>
<223> P-TEF1-R引物
<400> 18
tttgtaatta aaacttagat tagat 25
<210> 19
<211> 44
<212> DNA
<213> 人工序列
<220>
<223> P-ADH1-F引物
<400> 19
aaggaaaaaa gcggccgcgt tgtcctctga ggacataaaa taca 44
<210> 20
<211> 34
<212> DNA
<213> 人工序列
<220>
<223> P-ADH1-R引物
<400> 20
ccggaattcc ccatacatcg ggattcctat aata 34
<210> 21
<211> 23
<212> DNA
<213> 人工序列
<220>
<223> T-TDH3-F引物
<400> 21
gtgaatttac tttaaatctt gca 23
<210> 22
<211> 21
<212> DNA
<213> 人工序列
<220>
<223> T-TDH3-R引物
<400> 22
tcctggcgga aaaaattcat t 21
- 还没有人留言评论。精彩留言会获得点赞!