蔓越桔的一种DNA指纹图谱及其获取方法与专用引物与流程
本发明涉及植物DNA指纹图谱技术领域,具体涉及蔓越桔的一种DNA指纹图谱及其获取方法与专用引物。
背景技术:
蔓越桔(Vacciniium macrocarpon)是杜鹃花科(Ericaceae)越橘属(Vaccinium spp.)植物,喜强酸性的湿润土壤,抗寒能力强,集中分布在美国马萨诸塞和新泽西州。蔓越桔是具有很高经济回报率的小浆果,近年来被加工成果汁、果干和果酱等多种产品,供全世界几百万人享用,并且还具有特殊的医疗价值。医学调查表明,蔓越桔制剂成为预防尿道感染的首选保健药物。目前关于蔓越桔保健功能的研究国外主要集中在抗感染、抗氧化、抗肿瘤等方面。蔓越桔是一种生长在矮藤上、小而圆、表皮富于弹性的鲜红果子,也有人称它为小红莓。蔓越桔需要特殊的环境及气候条件栽培,全球的蔓越桔产区较少,产量有限,又因其具有多种保健功能,从而具有“北美红宝石”之美称。蔓越桔与葡萄、蓝莓并称为北美三大水果,对大多数中国人而言尚且陌生,不过,在学术界却早已盛名在外。蔓越桔一直就是北美地区的一种传统健康食品,近20年来,一系列科学研究证实了蔓越桔的各种保健作用。
目前,我国对于蔓越桔的研究还处于初级研究阶段,刚刚起步,但在蔓越桔产业发展方面已经呈现良好的势头,有越来越多的企业家开始投资蔓越桔产业,也有更多的国内科研人员开始注重蔓越桔各方面的研究。据统计,迄今,我国从北美和加拿大地区引进蔓越桔品种有9个(大连大学育种基地-大连森茂现代农业有限公司种质资源圃保存),由于目前我国还没有系统完善的鉴定方法,致使出现同种多名或同名多种现象,品种混乱问题给蔓越桔产业发展带来很大的经济损失。虽然蔓越桔的鉴定可以基于传统的形态学特征,但这些性状易受环境因素的影响,所以对其进行形态学鉴定和分类相当困难。因此,如何快速准确鉴别蔓越桔品种种质资源,已成为当前蔓越桔生产及科研中急需解决的问题。
与形态标记相比,分子标记是建立在基因组基础之上,其能对各发育时期的生物个体、组织、器官甚至细胞进行检测,不受基因表达的影响,具有操作简便、数量丰富及多态性高等特点。实践证明,分子标记可对种质资源进行准确、科学的鉴定和评价,为育种工作者提供直接、可靠的依据。为了充分有效地利用蔓越桔的种质资源,需要建立高效的蔓越桔分子标记技术体系并利用其对蔓越桔种质资源进行鉴定和评价。
技术实现要素:
本发明的目的在于提供蔓越桔的一种DNA指纹图谱及其获取方法与其专用引物,更充分地、有效地利用蔓越桔丰富的种质资源。
为了解决上述技术问题,本发明提供技术方案如下:
一种获取蔓越桔DNA指纹图谱的专用引物,专用引物序列为:
CA112-F-Joe:TCGTCGGCAGCGTCAGATTCCACCCACTTCACAGTTCA,如序列表SEQNO:1所示;
CA112-R:GTTTATTGGGAGGGAATTGGAAAC,如序列表SEQ NO:2所示;
linna-Joe:AGCAGCCGTCGCAGTCTA,如序列表SEQ NO:3所示。
一种获取蔓越桔DNA指纹图谱的方法,包括如下步骤:
步骤A:提取蔓越桔的基因组DNA;
步骤B:以步骤A的基因组DNA为模板,采用权利要求1所述的专用引物进行SSR-PCR扩增;
步骤C:对步骤B中得到的SSR-PCR产物进行琼脂糖凝胶电泳检测,将扩增成功的PCR产物利用毛细管电泳技术(CE)进行分离,即将PCR产物稀释10倍,取1ul,加入甲酰胺,内标Liz500,利用ABI3730xI全自动基因分析仪进行分析;
步骤D:将ABI3730xI的结果导入Genemarker V2.2.0软件进行分析,记录各样品的条带大小情况,并获得相应的峰图,即构建蔓越桔的DNA指纹图谱。
作为本发明的一种优选方式,步骤B中,所述SSR-PCR扩增程序为:包括95℃预变性5min,95℃变性30s,54℃退火15s,72℃延伸30s,其中,变性、退火和延伸三个步骤循环35次,72℃最后一步延伸15min,4℃保存。
作为本发明的一种优选方式,步骤B中,所述SSR-PCR的扩增体系为20μl,包括:2×Phanta buffer 10μl,10uM dNTP 0.4μl,Phanta max super-fidelity DNA polymerase,专用引物CA112-F-Joe 0.3μl,专用引物CA112-R 1.2μl,linna-Joe 1μl,20ng/μl的DNA模板1μl,ddH2O补足。
作为本发明的一种优选方式,所述蔓越桔的基因组DNA来自蔓越桔植株的幼嫩叶片。
一种获取蔓越桔DNA指纹图谱的试剂盒,所述试剂盒包括专用引物、Phanta buffer、dNTP、Phanta max super-fidelity DNA polymerase以及marker,其中专用引物序列见序列表的序列SEQ NO:1~3。
本发明的专用引物可以应用于获取蔓越桔DNA指纹图谱。
利用所述的方法得到的蔓越桔DNA指纹图谱也属于本发明的保护范围。
与现有技术相比,本发明的有益效果为:获得的蔓越桔DNA指纹图谱多态性高、指纹丰富、重复性好,在提高蔓越桔选择育种的准确性和效率,缩短育种周期,以及蔓越桔的分子标记辅助育种中发挥重要作用。
附图说明
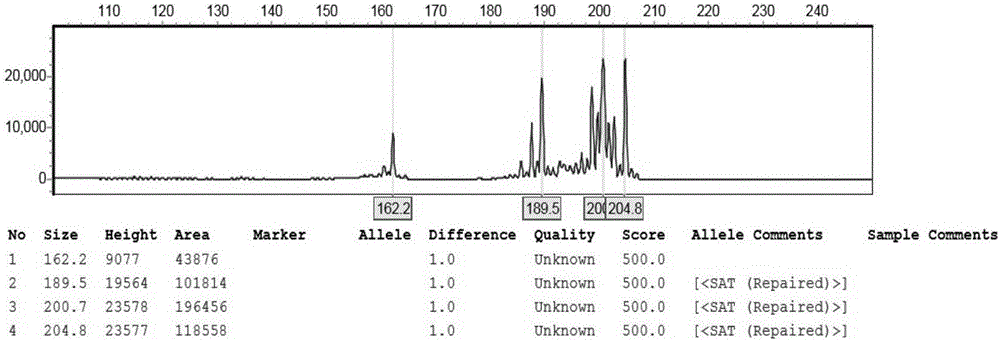
图1为本发明应用所述专用引物SEQ NO:1~3获得的蔓越桔品种C1的DNA指纹图谱。
图2为本发明应用所述专用引物SEQ NO:1~3获得的蔓越桔品种C2的DNA指纹图谱。
图3为本发明应用所述专用引物SEQ NO:1~3获得的蔓越桔品种C3的DNA指纹图谱。
图4为本发明应用所述专用引物SEQ NO:1~3获得的蔓越桔品种C4的DNA指纹图谱。
图5为本发明应用所述专用引物SEQ NO:1~3获得的蔓越桔品种C5的DNA指纹图谱。
图6为本发明应用所述专用引物SEQ NO:1~3获得的蔓越桔品种C6的DNA指纹图谱。
图7为本发明应用所述专用引物SEQ NO:1~3获得的蔓越桔品种C7的DNA指纹图谱。
图8为本发明应用所述专用引物SEQ NO:1~3获得的蔓越桔品种C8的DNA指纹图谱。
图9为本发明应用所述专用引物SEQ NO:1~3获得的蔓越桔品种C9的DNA指纹图谱。
图10为本发明具体实施方式中的9个蔓越桔品种的聚类树状图。
具体实施方式
结合具体实施方式和说明书附图对本发明做进一步说明。
供试材料为蔓越桔的9个品种,即C1:半蓝(Benlear);C2:早黑(Early Black);C3:朝圣者(Pilgrim);C4:克劳利(Crowley);C5:史蒂文(Stevens);C6:博蔓(Bergman);C7:奥斯通(Austoncross Black);C8:森特维尔(Centre Ville);C9:豪斯(Howes),这些材料均来自于大连大学蓝莓科研育种基地大连森茂现代农业有限公司。
一、利用专用引物获取蔓越桔的DNA指纹图谱
操作流程:提取蔓越桔基因组→扩增→琼脂糖凝胶电泳→分离扩增成功的SSR-PCR产物→利用ABI3730xI全自动基因分析仪进行分析→将ABI3730xI的结果导入Genemarker V2.2.0软件进行统计→统计条带大小→获得蔓越桔DNA指纹图谱。
具体方法和过程如下:
1.采用试剂盒(新型植物基因组DNA提取试剂盒DP320-03)提取蔓越桔的基因组DNA
1)处理材料:
取植物新鲜组织100mg或干重组织20mg,加入液氮充分研磨,加入400μl缓冲液LP1和6μl RNase A(10mg/ml),漩涡振荡1min,室温放置10min。
2)加入130缓冲液LP2,充分混匀,漩涡振荡1min。
3)12000rpm(~13400*g)离心5min,将上清移至新的离心管中。
4)加入4.5倍体积的缓冲液LP3(使用前请先检查是否已加入无水乙醇),立即充分振荡混匀15sec,此时可能会出现絮状沉淀。
5)将上一步所得溶液和絮状沉淀都加入一个吸附柱CB3中(吸附柱放入收集管中),12000rpm(~13400*g)离心30sec,倒掉废液,吸附柱CB3放入收集管中。
6)向吸附柱CB3中加入600μl漂洗液PW(使用前请先检查是否已加入无水乙醇)12000rpm(~13400*g)离心30sec,倒掉废液,吸附柱CB3放入收集管中。
注意:如果吸附柱膜呈现绿色,向吸附柱CB3中加入500μl无水乙醇,12000rpm(~13400*g)离心30sec,倒掉废液,吸附柱CB3放入收集管中。
7)重复操作步骤6。
8)将吸附柱CB3放回收集管中,12000rpm(~13400*g)离心2min,倒掉废液。将吸附柱CB3置于室温放置数分钟,以彻底晾干吸附材料中残余的漂洗液。
注意:这一步骤的目的是将吸附柱中残余的漂洗液去除,漂洗液中乙醇的残留会影响后续的酶反应(酶切、PCR等)实验。
9)将吸附柱CB3转入一个干净的离心管中,向吸附膜的中间部位悬空滴加50-200μl洗脱缓冲液TE,室温放置2-5min,12000rpm(~13400*g)离心2min,将溶液收集到离心管中。
注意:为增加基因组DNA的得率,可将离心得到的溶液再加入吸附柱CB3中,室温放置2min,12000rpm(~13400*g)离心2min。洗脱缓冲液体积不应少于50μl,体积过小影响回收效率。洗脱液的pH值对于洗脱效率有很大影响。若用ddH2O做洗脱液应保证其pH值在7.0-8.5范围内。pH值低于7.0会降低洗脱效率;且DNA产物应保存在-20℃,以防DNA降解。
2.SSR-PCR扩增
1)以步骤1中得到的蔓越桔样本的基因组DNA为模板,分别采用专用引物SEQ NO:1~3进行SSR-PCR扩增。
2)所述专用引物的序列为:
①CA112-F-Joe:TCGTCGGCAGCGTCAGATTCCACCCACTTCACAGTTCA;如SEQ IDNo:1所示。(5,端利用FAM标记)
②CA112-R:GTTTATTGGGAGGGAATTGGAAAC;如SEQ ID No:2所示。
③linna-Joe:AGCAGCCGTCGCAGTCTA;如SEQ ID No:3所示。
3)PCR扩增的程序为:包括95℃预变性5min,95℃变性30s,54℃退火15s,72℃延伸30s,其中,变性、退火和延伸三个步骤循环35次,72℃最后一步延伸15min,4℃保存。
4)PCR扩增体系为:20μl,包括:2×Phanta buffer 10μl,10uM dNTP 0.4μl,Phanta maxsuper-fidelity DNA polymerase,专用引物CA112-F-Joe 0.3μl,专用引物CA112-R 1.2μl,linna-Joe 1μl,20ng/μl的DNA模板1μl,ddH2O补足。
3.SSR-PCR产物的琼脂糖电泳检测与分离扩增
1)对步骤中2得到的SSR-PCR扩增产物进行琼脂糖凝胶电泳分离。扩增结束后,扩增产物中加入4μl电泳上样缓冲液(6×Loading Buffer)混匀,取6μl混合样品在2%琼脂糖凝胶中电泳,电泳缓冲液为1×TAE,电泳条件为的120V的电压下电泳1h。
2)将扩增成功的PCR产物利用毛细管电泳技术(CE)进行分离,即将PCR产物稀释10倍,取1ul,加入甲酰胺,内标Liz500,利用ABI3730xI全自动基因分析仪进行分析。
4.条带读取与结果分析
将ABI3730xI的结果导入Genemarker V2.2.0软件进行分析,记录各样品的条带大小情况,并获得相应的峰图,即构建了蔓越桔的DNA指纹图谱,具体结果如图1~图9和表1所示。
表1蔓越桔9个品种的SSR-PCR产物条带大小统计情况
二、蔓越桔DNA指纹图谱的应用
对步骤一获得的蔓越桔指纹图谱进行聚类分析,得到聚类树状图,如图10所示,可以看出,在遗传距离的阈值约为0.62时,可将份蔓越桔品种分成两大组:第一大组包括C1~C8,共8个蔓越桔品种;第二大组只包括C9,1个品种。在遗传距离的阈值约为0.73时,可将第一大组分成两个小组。在遗传距离的阂值约为0.77时,又可进一步划分。
以上内容是结合具体的优选实施方式对本发明所作的进一步详细说明,不能认定本发明的具体实施只局限于这些说明。对于本发明所属技术领域的普通技术人员来说,在不脱离本发明构思的前提下,还可以做出若干简单推演或替换,都应当视为属于本发明的保护范围。
SEQUENCE LISTING
<110> 大连大学
<120> 蔓越桔的一种DNA指纹图谱及其获取方法与专用引物
<130> 1
<160> 3
<170> PatentIn version 3.3
<210> 1
<211> 38
<212> DNA
<213> 人工合成
<400> 1
tcgtcggcag cgtcagattc cacccacttc acagttca 38
<210> 2
<211> 24
<212> DNA
<213> 人工合成
<400> 2
gtttattggg agggaattgg aaac 24
<210> 3
<211> 18
<212> DNA
<213> 人工合成
<400> 3
agcagccgtc gcagtcta 18
- 还没有人留言评论。精彩留言会获得点赞!