与大豆抗疫病基因紧密连锁的分子标记CAPs1516及其应用的制作方法
本发明属于农业生物技术工程和农作物抗病遗传育种领域,具体地说,涉及一种与大豆抗疫病基因rpshc18共分离的分子标记及其应用。
背景技术:
由大豆疫霉(phytophthorasojaekaufmann&gerdemann)引起的大豆疫病是大豆生产中的一种毁灭性病害。该病已在我国黑龙江省、安徽、福建、吉林、新疆等省份发生。大豆疫霉在大豆所有生育期均可以侵染大豆,引起根腐、茎腐、植株矮化、枯萎和死亡,一般田块减产10%-30%,严重地块可达60%-90%,甚至造成绝产。目前全世界每年因大豆疫病造成的经济损失大约为10亿美元(tyler,2007)。
防治大豆疫病最为经济、有效且环境安全的方法是种植抗病品种。目前,国内外共发现了28个大豆抗疫病基因(rps),分布于9个不同的大豆染色体上。rps1、rps7、rps9、rpsyd25、rpsyd29、rpsun1和therpsgeneincultivar‘waseshiroge’被定位在第3号染色体上,其中rps1位点具有5个等位基因分别为rps1a,rps1b,rps1c,rps1d和rps1k(demirbasetal.,2001;wengetal.,2001;gaoetal.,2005;范爱颖等,2009;sugimotoetal.,2011;sunetal.,2010;wuetal.,2011a;linetal.,2013;zhangetal.,2013a)。rps2和rpsun2被鉴定在第16号染色体上(demirbasetal.,2001;linetal.,2013)。rps3、rps8和rpssn10被定位在大豆13号染色体上,并且在rps3位点存在3个等位基因rps3a,rps3b和rps3c(demirbasetal.,2001;sandhuetal.,2005;gordonetal.,2006;yuetal.,2010)。rps4,rps5,rps6,rpsjs,rps12被定位在18号染色体上(demirbasetal.,2001;sandhuetal.,2005;sunetal.,2014;sahooetal.,2017)。而另外5个抗病基因rpszs18、rpsyb30、rpssu、rps10和rps11分别定位在2、19、10、17和7号染色体上(zhuetal.,2007;wuetal.,2011b;zhangetal.,2013b;pingetal.,2015)。大豆疫霉群体结构复杂,容易产生新的毒力型。一般认为大豆抗疫病基因的使用寿命为10-15年,但是新的抗病基因被淘汰的速度正在加快。因此有必要大力挖掘和利用新的抗病基因,并进行抗病基因的累加,培育抗性持久的大豆品种。
随着测序技术的发展,snp和indel等多态性更强的分子标记被开发并且应用到基因的定位中。大豆全基因组测序的完成为大豆基因的精细定位、克隆以及功能标记的开发提供了便利。极端群体混池测序技术(bsa-seq)具有高效,节省时间和人力,并且能够大量的发掘目标区域中的snp等优点,已经应用多种不同性状基因的发掘、定位和功能标记的开发。
技术实现要素:
本发明的目的是提供一个与大豆抗疫病基因rpshc18紧密连锁的分子标记及其应用。
为了实现本发明目的,本研究发明了一个与大豆抗疫病基因rpshc18紧密连锁的分子标记caps1516,所述分子标记位于大豆3号染色体的ssr标记barcsoyssr_03_0269和barcsoyssr_03_0272之间,并且位于rpshc18候选基因内部。
所述分子标记的核苷酸序列如seqidno.1所示,该标记片段大小为541bp。
本发明还提供了用于特异性pcr扩增分子标记caps1516的引物对,包括:
上游引物f:5'-aagtgcaacggactgccttt-3’;
下游引物r:5'-catcagggttgtccaggaatga-3'。
分子标记caps1516与基因rpshc18的遗传距离为0cm。
pcr扩增使用的退火温度为58.4℃,扩增产物大小为541bp,用于特异性切割rpshc18扩增片段的限制性内切酶为bcli。
本发明还提供所述分子标记caps1516在鉴定抗疫病大豆品种中的应用,包括以下步骤:
1)提取待测大豆的基因组dna
2)以待测大豆的基因组dna为模板,利用上述引物f和r,进行pcr扩增反应;
3)检测pcr扩增产物,如果能够扩增出541bp的产物,并且能够被限制性内切酶bcli切割为一个69bp和472bp的两个片段,则判定待测大豆品种含有抗疫病基因rpshc18。
其中,pcr扩增反应使用的扩增体系以10μl计为:大豆基因组dna模板浓度2ng/μl,2×pcrmastermix(tiangen)4μl,引物f和r各0.2μmol/l,用ddh2o补足至10μl。
pcr扩增反应使用的反应条件为:95℃3分钟;94℃45秒,58.4℃45秒,72℃45秒,共40个循环;72℃10分钟。
本发明还提供用于检测抗疫病大豆的pcr试剂盒,所述试剂盒中含有上述用于特异性pcr扩增分子标记caps1516的引物对与限制性内切酶bcli。还包括dntps、taqdna聚合酶、mg2+、pcr反应缓冲液、限制性内切酶标准阳性模板中的一种或多种。
本发明进一步提供所述分子标记caps1516在大豆抗疫病分子育种中的应用。
具体地,本发明的与大豆抗疫病基因rpshc18共分离的分子标记通过如下方法获得:
本研究以抗疫病品种华春18为父本、感病品种吉科豆2号为母本杂交产生的177个f2:3家系为作图群体。采用下胚轴创伤接种法选取不同毒力的大豆疫霉分离物psjs2和psmc1进行群体表型鉴定。选择纯合抗病和感病家系各20个进行极端群体混池测序分析,将该基因定位于大豆3号染色体上767kb的区域,并对候选区域内的基因进行功能注释。
使用ssr标记和indel标记对177家系进行基因型鉴定,构建遗传连锁图谱,将rpshc18进一步精细定位在147kb的区域中,位于indel标记indelhj22和ssr标记barcsoyssr_03_0272之间。根据测序结果,在这个区间中仅有两个nbs-lrr基因含有非同义突变的snp位点。其中一个位于基因rpshc18-nbl2(参考基因组中模型:glyma03g04590)的snp含有限制性酶切位点,被开发成caps标记,并进行群体验证,获得与基因rpshc18共分离的标记。
具体实施方案:
(1)华春18的抗性遗传分析
以抗病品种华春为父本,感病品种吉科豆2号作为母本,杂交产生f1代,f1代自交产生f2代,f2代自交产生的177个f2:3家系的抗性遗传分析、抗病基因鉴定及作图群体。采用下胚轴创伤接种方法,每个家系鉴定:25-30个植株对大豆疫霉分离物psjs2和psmc1的反应进行抗性遗传分析。接种后在24℃,湿度为100%的条件下,保湿48小时,然后传入温室培养,4天后进行病情调查。参照gordon等(2006)方法评价标准,调查f3家系的抗病、分离、感病家系的分离比,研究华春18对大豆疫病的抗性遗传规律。
(2)基因dna的提取和极端群体混池测序
采用tiangen植物基因组提取试剂盒提取2个亲本及177个f2单株的基因组dna。将20个纯和抗病和20个纯和感病的家系分别等量混合进行极端群体混池测序分析,具体分析方法参照(takagietal.2013)。获得华春18中抗疫病基因的候选区域。
(3)利用ssr标记定位华春18的抗病基因
根据混池测序分析的结果,首先采用ssr标记对混池测序分析抗病基因定位结果进行验证。选用目前已经公布的位于混池测序分析获得的候选区域内部及其附近的ssr标记进行亲本间多态性分析,具有多态性ssr标记再用于群体的基因型鉴定。用mapmaker3.0软件分析ssr标记与抗病基因的连锁关系。使用mapdraw软件构建遗传连锁图谱,获得新的定位区间。pcr反应体系为10μl,含有2×taqpcrmastermix(tiangen)4μl,上、下游引物各0.2μmol/l,模板dna浓度为2ng/μl,用ddh2o补足至10μl。
pcr反应扩增程序:95℃预变性3分钟;94℃变性45秒,55℃退火45秒,72℃延伸45秒,共35个循环;72℃10分钟,10℃保存。扩增产物在8-12%聚丙烯酰胺凝胶中进行电泳和分析。
(4)indel标记群体验证及候选基因的发掘
基于遗传连锁图谱结果,获得含有rpshc18的在参考基因组上的物理位置,结合亲本的基因组测序结果,发掘该区间内indel位点。根据indel位点上下游200bp的序列设计引物,开发indel标记,用来鉴定177个家系在ssr定位区间内的重组位置,从而进一步缩小定位区间。在缩小后的定位区间中,查找含有非同义突变snp及indel的基因,并将这些基因作为rpshc18候选的抗疫病基因。
(5)基于rpshc18候选基因的特异性标记的开发及验证
引起非同义突变的snp用于开发成特异性检测rpshc18的caps标记,caps标记的开发方法参照thiel等(2004)的方法,使用premier5.0设计引物用于扩增抗病品种华春18和感病品种吉科豆2号的非同义snp两侧序列。将抗、感亲本的pcr产物进行测序并且发掘各自的酶切位点,通过对比获得华春18特有的酶切位点。再用对应的限制性内切酶bcli切割抗感品种扩增出的片段。能够被切割的说明含有rpshc18。将含有已知抗疫病基因的20个大豆品种按上述方法进行目标区域的pcr扩增,并且酶切,若只有华春18的片段能够被酶切,说明该标记为rpshc18的特异性标记。
大豆疫病是大豆生产过程中的一种毁灭性病害。本发明利用bsa全基因组混池测序性状定位以及ssr和indel分子标记作图方法,对大豆抗疫病基因rpshc18进行精细定位,并发现其共分离和紧密连锁的分子标记。该抗病基因对大豆疫霉具有广谱抗性,可有效控制我国大豆疫病的发生,减轻该病害造成的产量损失。
利用与该基因共分离的caps分子标记caps1516,可有效应用到分子标记辅助育种中,克服常规育种方法所需周期长的缺点;基于pcr技术,在室内对大豆资源进行抗性分析抗疫病基因鉴定;同时为克隆该rpshc18基因奠定基础,这对进一步了解该基因的分子遗传学机理具有重要意义,在大豆生产实践、育种工作和抗病理论研究中具有重要的价值。
本发明的优点在于:
(一)本发明对3号染色体上发现的新的大豆抗疫病基因rpshc18进行了精细定位。该基因对我国大豆疫霉具有广谱抗性,可有效控制我国大豆疫病的发生,减轻该病害造成的产量损失。
(二)本发明提供的与抗疫病基因rpshc18共分离的分子标记caps1516,是在华春18及其抗性稳定的杂交后代家系中获得的分子标记,并进行了有效性验证。可用于抗大豆疫病资源鉴定和抗大豆疫病后代的分子标记辅助育种。
(三)本发明提供的与抗疫病基因rpshc18共分离的分子标记caps1516,为该基因的克隆和测序奠定基础,从而明确该序列信息的结构特征。另外,根据共分离的分子标记和大豆基因组序列信息,进行该基因的单倍型分析,有望克隆获得新的抗疫病基因。
附图说明
图1为本发明实施例1中华春18(左)和吉科豆2号(右)接种大豆疫霉4天后的调查结果,其中,a为接种菌株psmc1的结果,b为接种菌株psjs2的结果。
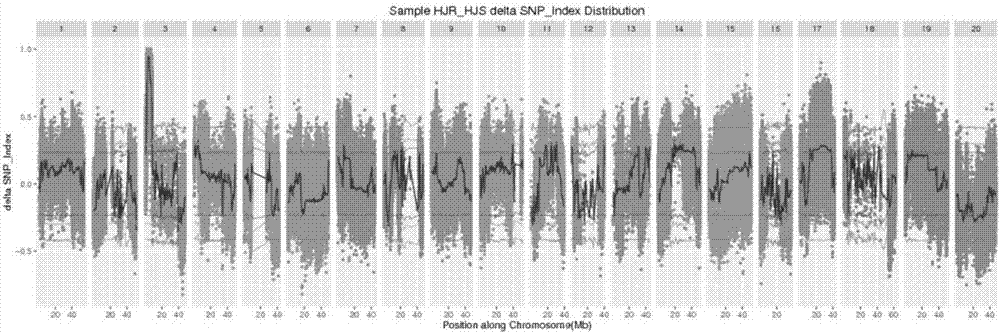
图2为基于极端群体全基因组混池测序分析的delta-snp-index结果。
图3为本发明实施例1中大豆抗疫病基因rpshc18遗传连锁图谱。
图4为本发明caps1516在抗病,杂合和感病家系不同分子带型,以及与含有不同抗疫病基因的材料鉴定后不同带型的比较。
具体实施方式
以下实施例用于说明本发明,但不用来限制本发明的范围。若未特别指明,实施例中所用技术手段为本领域技术人员所熟知的常规手段,所用原料均为市售商品。
实施例1大豆抗疫病基因rpshc18特异性分子标记caps1516的获得
1、华春18的抗性遗传分析
以抗病品种华春为父本,感病品种吉科豆2号作为母本,杂交产生f1代,f1代自交产生f2代,f2代自交产生的188个f2:3家系的抗性遗传分析、抗病基因鉴定及作图群体。采用下胚轴创伤接种方法,每个家系鉴定:25-30个植株对大豆疫霉分离物psjs2和psmc1的反应进行抗性遗传分析。接种后在24℃,湿度为100%的条件下,保湿48小时,然后传入温室培养,4天后进行病情调查(图1)。参照gordon等(2006)方法评价标准,调查f3家系的抗病、分离、感病家系的分离比,研究华春18对大豆疫病的抗性遗传规律。
吉科豆2号与华春18杂交组合衍生的177个f2:3家系对分离物psjs2和psmc1的反应一致,纯和抗病家系为47个,纯和感病家系为50个,杂合型家系为80个。经x2检验符合期望的1:2:1的分离比例。
(2)基因dna的提取和极端群体混池测序
采用tiangen植物基因组提取试剂盒提取2个亲本及177个f2:3家系f2单株的基因组dna。将20个纯和抗病和20个纯和感病的家系分别等量混合进行极端群体混池测序分析,具体分析方法参照(takagietal.2013)。获得华春18中抗疫病基因的候选区域。
通过混池测序分析,抗池、感池以及抗病和感病亲本通过比对共发现了3780800个snp及indel位点。通过计算delta-snp-index的数值来确定抗病基因的候选区域,结果发现位于大豆3号染色体上的碱基位置4152795与4919481之间的767kb的区域平均delta-snp-index大于0.9。因此我们推测这一区域为抗疫病基因的候选区域(图2)。
(3)利用ssr标记定位华春18的抗病基因
根据混池测序分析的结果,首先采用ssr标记对混池测序分析结果进行验证。选用目前已经公布的位于混池测序分析候选区域内部及其附近的ssr标记。用mapmaker3.0软件分析ssr标记与抗病基因的连锁关系。使用mapdraw软件构建遗传连锁图谱,获得新的定位区间。pcr反应体系为10μl,含有2×taqpcrmastermix(tiangen)4μl,上、下游引物各0.2μmol/l,模板dna浓度为2ng/μl,用ddh2o补足至10μl。
pcr反应扩增程序:95℃预变性3分钟;94℃变性45秒,55℃退火45秒,72℃延伸45秒,共35个循环;72℃10分钟,10℃保存。扩增产物在8-12%聚丙烯酰胺凝胶中进行电泳和分析。
在新设计的145对ssr中通过亲本筛选和群体验证。有16对在群体中具有多态性。其中16个标记在群体中均呈现1:2:1的分布。使用mapmaker3.0软件分析表明,这16对多态性ssr引物与rpshc18均连锁。使用mapdraw软件绘制遗传连锁图谱,rpshc18被定位在标记barcsoyssr_03_0254(0.3cm)和barcsoyssr_03_0272(0.5cm)之间,同时与ssr标记barcsoyssr_03_0267和barcsoyssr_03_0269共分离,与rpshc18的遗传距离为0cm(图3a)。
(4)indel标记群体验证及候选基因的发掘
基于遗传连锁图谱结果,获得含有rpshc18的在参考基因组上的物理位置,结合基因组测序结果,发掘该区间的snp以及indel位点,并切根据这些snp和indel位于基因的位置进行注释,包括5’-utr,3’-utr,外显子,基因上游1000bp,基因下游1000bp,内含子,非编码rna或者其他类型。根据indel位点上下游200bp的序列设计引物,共开发了28个indel标记,用来鉴定177个家系在ssr定位区间内的重组位置,从而进一步缩小定位区间。在缩小后的定位区间中,查找含有非同义突变snp及indel的基因,并将这些基因作为候选的抗疫病基因。
通过ssr标记将rpshc18的物理位置定位在3号染色体的barcsoyssr_03_0254(0.3cm)和barcsoyssr_03_0272(0.5cm)之间的295kb的物理距离。基于抗感亲本及混池测序结果,在这一区间中共存在1949个snps及indels位点,但是仅有35个位点为非同义突变位点,分别位于5个不同基因中(表1)。将28个indel标记进行亲本的多态性筛选及亲本验证,11个在亲本及群体上具有多态性。其中3个标记与抗病基因rpshc18共分离(图3b)。indel标记indelhj22与ssr标记barcsoyssr_03_0272之间组成了一个147kb的‘共分离’区域,在这个区域中仅存在两个非同义突变的基因,并且均为抗病基因结构,分别为rpshc18-nbl1(genemodelglyma03g04560)和rpshc18-nbl2(genemodelglyma03g04590)。于是这两个基因被推测为抗病rpshc18的候选基因。
(5)基于rpshc18候选基因特异性标记的开发及验证
引起非同义突变的snp用于开发成特异性检测rpshc18的caps标记,caps标记的开发方法参照thiel等(2004)的方法,使用premier5.0设计引物用于扩增抗病品种华春18和感病品种吉科豆2号的非同义snp两侧序列。将pcr产物进行测序并且发掘各自的酶切位点,通过对比获得华春18特有的酶切位点。再用对应的限制性内切酶切割抗感品种扩增出的片段。能够被切割的说明含有rpshc18。将含有抗疫病基因的20个大豆品种按上述方法进行目标区域的pcr扩增,并且酶切,若只有华春18的片段能够被酶切,说明该标记为rpshc18的特异性标记。
通过对抗病候选基因中的snp位点进行两侧片段的pcr扩增及酶切位点的发掘,仅在候选基因rpshc18-nbl2发现了一个snp位点a→t构成了限制性内切酶位点bcli。用于有效扩增caps标记caps1516的引物对为5'-aagtgcaacggactgccttt-3’/5'-catcagggttgtccaggaatga-3',扩增产物为541bp,通过限制性内切酶切割两小时后,抗病亲本及抗病家系pcr产物能够被切割成两个片段,大小分别为69bp和472bp,而对应的感病亲本及感病家系的pcr产物由于不存在相应的酶切位点而不能被切割,仍然只有541bp的一条带,而杂合家系则产生3条不同带型,分别为69bp、472bp和541bp。将caps标记在20个含有不同抗疫病基因的大豆品种及两个感病对照品种中进行验证发现,仅有华春18对应的条带能够被酶切,说明该标记为rpshc18的特异性标记(图4)。
虽然,上文中已经用一般性说明及具体实施方案对本发明作了详尽的描述,但在本发明基础上,可以对之作一些修改或改进,这对本领域技术人员而言是显而易见的。因此,在不偏离本发明精神的基础上所做的这些修改或改进,均属于本发明要求保护的范围。
参考文献
1.thielt,kotar,grossei,steinn,granera(2004)snp2caps:asnpandindelanalysistoolforcapsmarkerdevelopment.nucleicacidsres32:e5–e5
2.gordonsg,stmartinsk,dorranceae(2006)rps8mapstoaresistancegenerichregiononsoybeanmolecularlinkagegroupf.cropsci46:168–173.
3.takagih,abea,yoshidak,kosugis,natsumes,mitsuokac,uemuraa,utsushih,tamirum,takunos,innanh,canol,kamouns,terauchir(2013)qtl–seq:rapidmappingofquantitativetraitlociinriceby
序列表
<110>中国农业科学院作物科学研究所
<120>与大豆抗疫病基因紧密连锁的分子标记caps1516及其应用
<130>khp171110867.2tq
<160>3
<170>patentinversion3.5
<210>1
<211>541
<212>dna
<213>caps1516
<400>1
aagtgcaacggactgcctttagcagcacagtcgcttggaggcatgttgagaagaaagcat60
gacatcagggattggaataatattctgaatagtgacatttgggaactttctgaaagtgag120
tgtaaagttattccagcactgagacttagttatcattatctccctccacatttaaaacgg180
tgctttgtttattgttcgttgtatccacaagattaccaatttgaaaaaaatgaattaatc240
ttgttgtggatggctgaagatcttttgaggaaaccaagaaaaggtgggactttagaagag300
gttggtcaggagtattttgatgatttggttttgagatcatttttccaacgttcaaataga360
agtagttggtctcatgggaaatggtttgtgatgcatgacctcatgcatgatctagcgaca420
tcactcagtggagatttttattttagatcagaagaacttgggaaagaaacaatgatcaat480
accaagactcgtcatttgtcatttgccaaattcaattcttcattcctggacaaccctgat540
g541
<210>2
<211>20
<212>dna
<213>人工序列
<400>2
aagtgcaacggactgccttt20
<210>3
<211>22
<212>dna
<213>人工序列
<400>3
catcagggttgtccaggaatga22
- 还没有人留言评论。精彩留言会获得点赞!