葡萄糖氧化酶CnGOD19及其改良酶、基因和应用的制作方法
本发明涉及基因工程领域,具体涉及葡萄糖氧化酶cngod19及其改良酶、基因和应用。
背景技术:
葡萄糖氧化酶(god,e.c.1.1.3.4)是由分子量大小为60-90kda的两个相同亚基组成的同型二聚体,为一种黄素依赖型需氧脱氢酶,属于gmc(glucose/methanol/choline)氧化还原酶家族,这一家族的酶广泛应用于食品、化工、医药以及其它生物技术产业,尤为重要的是在医学诊断方面的应用。
目前我国生产的葡萄糖氧化酶工业酶制剂来源少、纯度低且酶学性质极不稳定,长期以来依赖进口,因此寻找进口酶制剂的替代产品具有重要意义。
技术实现要素:
本发明从枝孢菌cladosporiumneopsychrotolernssl-19菌株中挖掘的新来源葡萄糖氧化酶基因,不仅拓展了葡萄糖氧化酶基因的挖掘范围,而且通过对该基因改良设计得到了一种酶学性质较好的葡萄糖氧化酶cngod19a,将进一步推进葡萄糖氧化酶的挖掘和改良工作。
本发明的目的是提供一种新来源且未见报道的葡萄糖氧化酶。
本发明的再一目的是提供上述葡萄糖氧化酶的编码基因。
本发明的再一目的是提供上述葡萄糖氧化酶的改良酶。
本发明的再一目的是提供上述葡萄糖氧化酶改良酶的基因。
本发明的再一目的提供包含上述葡萄糖氧化酶改良酶的编码基因的重组载体。
本发明的再一目的是提供包含上述葡萄糖氧化酶改良酶的编码基因的重组菌株。
本发明的再一目的是提供一种生产新来源葡萄糖氧化酶改良酶的方法。
本发明的再一目的是提供上述葡萄糖氧化酶改良酶的。
本发明中所提供的葡萄糖氧化酶cngod19基因来源于cladosporiumneopsychrotolernssl-19菌株,该基因的cdna序列如下seqidno.1所示。
经比对显示该基因为新基因,该基因所对应的肽链具有葡萄糖氧化酶活性。
根据上述核苷酸序列可以得到所对应的葡萄糖氧化酶cngod19氨基酸序列如下seqidno.2所示。
该序列所对应蛋白质的理论等电点为4.68,理论分子量约为61.58kda,第1-16个氨基酸为信号肽。为了排除该理论信号肽对蛋白后期表达分泌的影响,在构建重组表达载体时,可以去除信号肽所对应的核苷酸序列,而后表达所对应的氨基酸序列如下seqidno.3所示。
通过实验验证上述葡萄糖氧化酶cngod19基因所表达多肽具有葡萄糖氧化酶活性,但是活性较低,设计多种方案对cngod19基因进行改造,最终优化获得有效的改造方案,得到cngod19a基因,其核苷酸序列如下seqidno.4所示。
根据上述核苷酸序列可以得到所对应的葡萄糖氧化酶改良酶cngod19a,其氨基酸序列如下seqidno.5所示。
根据本发明的具体实施方式,在构建重组表达载体时,可以去除信号肽所对应的核苷酸序列,而后表达所对应的氨基酸序列如下seqidno.6所示。
该序列所对应的蛋白质的等电点为4.81,分子量约为64.68kda。通过实验验证上述cngod19a基因所表达的产物具有葡萄糖氧化酶酶活力,改良前酶活力极低,改良后酶活力提高到33.34u/mg,该改良酶的最适反应温度为20℃,在0-50℃之间依然具有60%以上的酶活力,在40℃下处理60min,剩余酶活还有近70%。
附图说明
图1显示本发明的重组葡萄糖氧化酶的最适ph值。
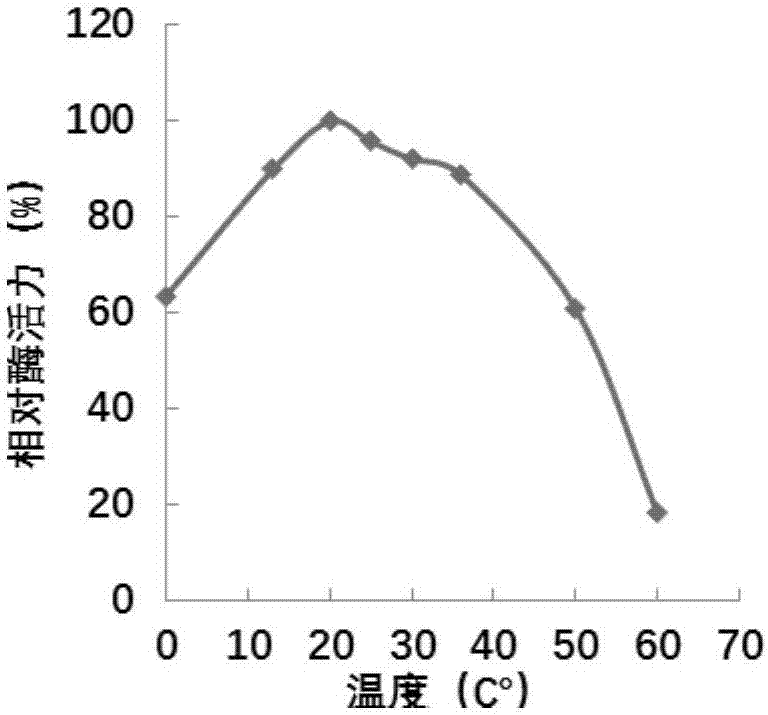
图2显示本发明的葡萄糖氧化酶最适反应温度。
图3显示本发明的葡萄糖氧化酶的ph稳定性。
图4显示本发明的葡萄糖氧化酶热稳定性。
具体实施方式
试验材料和试剂
1、菌株及载体:毕赤酵母(pichiapastorisgs115);毕赤酵母表达载体ppic9及菌株gs115购自于invitrogen公司。
2、酶类及其它生化试剂:内切酶购自takara公司,连接酶购自invitrogen公司,peasy-uniseamlesscloningandassemblykit购自北京全氏金生物技术有限公司,rna提取试剂盒购自普洛麦格生物技术有限公司(promega),质粒提取试剂盒购自于天根生化科技有限公司,核酸回收试剂盒购自于omegabio-tek,其它均为国产试剂(均可从普通生化试剂公司购买得到)。
3、培养基:
(1)产酶培养基(/l):葡萄糖172.11g,玉米浆11.05g,碳酸钙52.29g,牛肉蛋白胨3.5g,(nh4)h2po40.5g,mgso4·7h2o0.125g,feso4·7h2o0.125g。
(2)大肠杆菌lb培养基:1%蛋白胨、0.5%酵母提取物、1%naci,ph7.0。
(3)bmgy培养基:1%酵母提取物,2%蛋白胨,1.34%ynb,0.000049<biotin,1%甘油(v/v)。
(4)bmmy培养基:除以0.5%甲醇代替甘油外,其余成份均与bmgy相同。
说明:以上培养基和实验中用于菌株培养的器材未特殊说明均需灭菌后使用,以下实施例中未作具体说明的分子生物学实验方法,均参照《分子克隆实验指南》(第三版)j.萨姆布鲁克一书中所列的具体方法进行,或者按照试剂盒和产品说明书进行。
实施例1cngod19基因的克隆
(1)提取枝孢菌cladosporiumneopsychrotolernssl-19总rna
首先,将在产酶培养基中培养3天的菌收集到滤纸上并压干,加入已高温灭菌的研钵中,液氮迅速研磨至粉末,然后按照rna提取试剂盒的操作说明提取菌株总rna。
(2)葡萄糖氧化酶cdna的获取
利用oligo(dt)20和反转录酶对该基因进行反转录,然后设计合成引物z19#f和z19#r(见表1),对cngod19的成熟蛋白(去信号肽序列)的编码区进行pcr扩增,获得葡萄糖氧化酶的cdna序列。
表1cngod19克隆引物
实施例2重组菌株gs115/cngod19的构建
(1)重组载体ppic9-cngod19的构建
按照peasy-uniseamlesscloningandassemblykit说明书将ppic9表达载体与经表1引物扩增的目的产物混合进行无缝拼接,构建成酵母表达载体ppic9-cngod19。将ppic9-cngod19质粒转入大肠杆菌感受态trans1-t1,涂布到带有氨苄抗性的lb固体平板上过夜培养,后进行菌落pcr验证,挑选阳性转化子进行dna测序,测序序列正确的转化子用于大量制备重组质粒。用限制性内切酶bglii线性化重组质粒,回收酶切产物,然后电击将其转入酵母gs115感受态细胞,涂布到md平板上30℃培养2-3天,挑取在md平板上生长的转化子进行进一步的表达实验,具体操作请参考毕赤酵母表达操作手册。
(2)gs115/cngod19高产酶转化子的筛选
用灭菌后的牙签从md板上挑取转化子的单菌落,按照顺序先后点到md平板上并编号标记,将md平板置于30℃培养箱中培养1~2天,至菌落长出。按编号从md平板上挑取转化子接种于装有3mlbmgy培养基的培养管中,30℃,220rpm摇床培养48h;将摇床培养48h的菌液3,000×g离心15min,去上清,培养管中再加入1ml含有0.5%甲醇的bmmy培养基,在30℃、220rpm诱导培养;诱导培养48h后,3,000×g离心5min,取上清用于酶活性检测,从中筛选出葡萄糖氧化酶高活性转化子,具体操作请参考毕赤酵母表达操作手册.
酶活检测结果显示,上述构建gs115/cngod19重组菌株表达的cngod19酶具葡萄糖氧化酶活性,但活性较低,因此下面的实施案例中将阐述基因改造后重组菌株gs115/cngod19的构建过程,以及后期cngod19a酶的表达和纯化过程。
实施例3重组菌株gs115/cngod19a的构建
(1)重组载体ppic9-cngod19a的构建
本发明研究发现,cngod19编码基因由于进化导致结构变化,进而影响其表达蛋白结构的变化,最终导致酶活力低。为了解决上述问题,本发明优化多种方案,最终设计了碳末端的区段替换,以氨基酸序列为“yiatlgmmkeelggvvdsrlkvygienvravdasvlpiqlsahlssslygiaekaammikedqgh*”的肽片段替换cngod19中第550位至第615位的肽片段(计数时包含信号肽序列)。
(2)gs115/cngod19a高产酶菌株的构建
参照实施例2过程,将ppic9-cngod19a重组质粒最终转入gs115酵母感受态细胞,并筛选出gs115/cngod19a高产酶菌株。
以同样的方法构建包含信号肽序列的重组载体并转化宿主菌株。
实施例4重组葡萄糖氧化酶在毕赤酵母中的发酵
(1)重组葡萄糖氧化酶在摇瓶水平的大量表达
将筛选出产酶活较高的转化子接种于30mlypd种子培养基中培养48h,后转接到300mlbmgy液体培养基中,30℃,220rpm摇床振荡培养48h,进行菌体富集;4,500rpm离心5min,弃上清,将菌体转接到100ml含有0.5%甲醇的bmmy液体培养基中,30℃,220rpm诱导培养72h。诱导培养期间,每间隔24h补加一次甲醇溶液以补偿甲醇的损失,使甲醇终浓度保持在0.5%左右;诱导培养后12,000×g离心10min,收集上清发酵液,检测酶活性并进行sds-page蛋白电泳分析。
(2)重组葡萄糖氧化酶的纯化
收集摇瓶表达的重组葡萄糖氧化酶上清液,首先利用5kda膜包进行浓缩,此过程中低盐缓冲液置换了发酵液中的培养基,然后用5kda超滤管进一步的浓缩。浓缩到酶液能稀释一定倍数,通过离子交换层析进行纯化。具体地,将cngod19a浓缩液注入到20mmpbs(ph6.5)预先平衡过的hitrapqsepharosexl阴离子柱中,然后用含0-1mol/lnacl的pbs缓冲液进行线性梯度洗脱,检测分步收集的洗脱液酶活性并进行蛋白浓度的测定。
实施例5重组葡萄糖氧化酶部分性质分析
采用分光光度法对本发明的葡萄糖氧化酶进行酶活测定。具体方法如下:在ph6.0,30℃条件下,5ml的反应体系包括2.5ml邻联茴香胺缓冲液,0.3ml18%的葡萄糖溶液,0.1ml90u/ml辣根过氧化物酶,0.1ml适当的稀释酶液,反应3rnin,加入2ml2m硫酸终止反应,冷却后测定od540吸光值。葡萄糖氧化酶活性单位定义:在一定条件下,每分钟能将lμmolβ-d-葡萄糖氧化生成d-葡萄糖酸和过氧化氢的酶量为1个活性单位(u)。
(1)葡萄糖氧化酶cngod19a的最适ph及最适温度
测定实施例4中经纯化的葡萄糖氧化酶样品在不同ph缓冲条件下的酶活力,以确定其最适ph缓冲条件。配制不同ph值所用缓冲液:ph3.0~8.0的柠檬酸-磷酸氢二钠系列缓冲液及ph9.0~l2.0甘氨酸-氢氧化钠系列缓冲液,用不同ph值的缓冲液来配制邻联茴香胺溶液。30℃下测定的最适ph结果(图1)表明:cngod19a的最适ph为7.0,在ph6.0-ph8.0范围内,酶活力能够维持在60%以上。
在ph7.0条件下,测定不同温度(0-60℃)下已纯化葡萄糖氧化酶样品的酶活力。分析实验结果表明,该酶的最适反应温度为20℃,在0-50℃之间依然具有60%以上的酶活力(图2)。
(2)葡萄糖氧化酶cngod19a的ph稳定性及热稳定性
将酶液在不同ph值的缓冲液中于25℃下处理60min,在最适ph下测定酶活性以进行该酶ph稳定性的研究。分析结果表明(图3),该酶在ph6.0-ph9.0之间基本稳定,能够维持80%以上的酶活力,经ph5.0和ph10.0处理后酶活力基本消失。
热稳定性测定为葡萄糖氧化酶样品在不同温度(30℃、40℃、50℃)下处理不同时间后,在30℃下进行酶活性测定。热稳定性实验表明(图4),该葡萄糖氧化酶在30℃下处理60min,酶活基本不变,在40℃下处理60min,剩余酶活还有近70%,但50℃下处理5min后仅剩余约60%的酶活力。
(3)葡萄糖氧化酶cngod19a的金属离子抗性
表2cngod19a纯化后离子抗性实验结果
由表2可以看出,与空白对照组相比,当底物中含有5mm的mn2+、fe3+或co2+离子时,cngod19a酶活性受到抑制,而底物中含有5mm的β-巯基乙醇时,cngod19a酶活性损失严重,其它上述5mm的金属离子和有机试剂对酶活性有较小的促进作用。当底物中含有10mm的cu2+、ni2+或na+离子时,cngod19a酶活性有较小的提升,而底物中含有10mm的β-巯基乙醇时,cngod19a酶活性损失严重,其它上述10mm的金属离子对酶活性有抑制作用。
序列表
<110>中国农业科学院饲料研究所
<120>葡萄糖氧化酶cngod19及其改良酶、基因和应用
<160>6
<170>siposequencelisting1.0
<210>1
<211>3414
<212>dna
<213>枝孢菌(cladosporiumneopsychrotolernssl-19菌株)
<400>1
atggtttacccggctgctattctcacatttctcctcgccactctcgcacaatcattaccc60
taccaaatgggccgacgataagagtgtaaagaggagcggtgagagcgtgttagtaatggg120
cagaagcaacgagcggacaaggcacgcggaatcatcaccgacgccaaccatgtcgccaac180
gtcttcgttgctcgcctgttccgtgcgccttagtagtggctgcggttggtacagcggttg240
aagacctttgactacatcgtctgcggtggcggactgacaggactagtcgtagcgagtcgt300
ttctggaaactgatgtagcagacgccaccgcctgactgtcctgatcagcatcgctcagca360
ctctcagaagaccccgagatatcagtgctagtgatcgagaacggcaacgacgaccacgag420
gagagtcttctggggctctatagtcacgatcactagctcttgccgttgctgctggtgctc480
gaccctcgtgtcaacgatgtcaggacctacggagcagcattcgaatccgagctcgacttc540
ctgggagcacagttgctacagtcctggatgcctcgtcgtaagcttaggctcgagctgaag600
aatcttacatcgacgcctgtcccgtggcagaacgacaccggcctgctcctagttgcgggc660
ttagaatgtagctgcggacagggcaccgtcttgctgtggccggacgaggatcaacgcccg720
aggactctaggcggaagtggcagcatcaacggcgctagctggacgaaaggcgacaagacg780
tcctgagatccgccttcaccgtcgtagttgccgcgatcgacctgctttccgctgttctgc840
cagtatgatttgctaccagtgctgacgggggatgactcttggtctttcgacgccctgaac900
gtcatactaaacgatggtcacgactgccccctactgagaaccagaaagctgcgggacttg960
gagatcatgctcagcatcgaggacttccacgagccaacggaagagcacatcgccaaaggc1020
ctctagtacgagtcgtagctcctgaaggtgctcggttgccttctcgtgtagcggtttccg1080
gcgaacttcacaagcgagttccacggccgggatgggatggtgcaggtttcgtttccgaga1140
cgcttgaagtgttcgctcaaggtgccggccctaccctaccacgtccaaagcaaaggctct1200
ggcatgtttggcggaatccaagttccagcgttggaagcgtccacggccatctggaagggg1260
ccgtacaaaccgccttaggttcaaggtcgcaaccttcgcaggtgccggtagaccttcccc1320
ttggggatagttgcggatttcgcagcgggactcacgaacggcgctacaatcattcctaac1380
aacccctatcaacgcctaaagcgtcgccctgagtgcttgccgcgatgttagtaaggattg1440
atgattgaagacaacgaggatcagaaccgctcttcgcctttcactgtgtatgcaaaggac1500
tactaacttctgttgctcctagtcttggcgagaagcggaaagtgacacatacgtttcctg1560
cagacgcagcagcgggataatttcgtgatcctgacaggtcaccgcgtcacatcgatcaac1620
gtctgcgtcgtcgccctattaaagcactaggactgtccagtggcgcagtgtagctagttg1680
tggcgtccgggcaaagacatggccgccgagggcgtgaacttccaggcatgtcgcagctgc1740
accgcaggcccgtttctgtaccggcggctcccgcacttgaaggtccgtacagcgtcgacg1800
gagaaacacgtcgcgacaacgaagcgcgaagtactgctcgccgcaggctctctacaaagc1860
ctctttgtgcagcgctgttgcttcgcgcttcatgacgagcggcgtccgagagatgtttcg1920
ccacagctgctcgagctgtcaggcgtaggagacccagaagtcctcgccgccgccggcgta1980
ggtgtcgacgagctcgacagtccgcatcctctgggtcttcaggagcggcggcggccgcat2040
cccgtgaagatggcatcccccaacgtcggcaagaacatgcaagaacaaaccaaaaacacc2100
gggcacttctaccgtagggggttgcagccgttcttgtacgttcttgtttggtttttgtgg2160
ctctggttcgaccccgtcagcaccgacttcgacggcaccggccccccaaacgccatcgcc2220
gagaccaagctggggcagtcgtggctgaagctgccgtggccggggggtttgcggtagcgg2280
ttccccaccgtcaaccaactcttcaaaaacaacagcgcatccttgtaccaaagcatcaca2340
aaggggtggcagttggttgagaagtttttgttgtcgcgtaggaacatggtttcgtagtgt2400
tccagcctctggcaatacgccctcgacctcaaagcctctggcctcgtaacaaacgccacc2460
aggtcggagaccgttatgcgggagctggagtttcggagaccggagcattgtttgcggtgg2520
gcaacccacctaatcctctcagcccaagtcaacaacctgtggaaagactccgccggcgcc2580
cgttgggtggattaggagagtcgggttcagttgttggacacctttctgaggcggccgcgg2640
gcagaagtcttcttcgtcacctcgcccgccaccggccaaatcggcatcgacctctggaac2700
cgtcttcagaagaagcagtggagcgggcggtggccggtttagccgtagctggagaccttg2760
ctcatcgtgctgtccaggggttacgtgcacatcacctccaactcctcctgggaccacccg2820
gagtagcacgacaggtccccaatgcacgtgtagtggaggttgaggaggaccctggtgggc2880
gagatcgagccctcgtacttcggccaccccttcgacctcggggtccagctggcagcggcc2940
ctctagctcgggagcatgaagccggtggggaagctggagccccaggtcgaccgtcgccgg3000
cagcaatcgcgcgagatctttcagacggcaccactcgcagctcacgtcgcgtcggagacg3060
gtcgttagcgcgctctagaaagtctgccgtggtgagcgtcgagtgcagcgcagcctctgc3120
ttccctggtcttgaaaccgttcctcaaaacgccactacggaagtctgggaggagtgggtc3180
aagggaccagaactttggcaaggagttttgcggtgatgccttcagaccctcctcacccag3240
aaaggtactttcacttctgtgtggcattatatcgcgacgttggggatgatgaaagaggag3300
tttccatgaaagtgaagacacaccgtaatatagcgctgcaacccctactactttctcctc3360
ctgggggagttgtggatcacagactgagaccccctcaacacctagtgtctgact3414
<210>2
<211>568
<212>prt
<213>枝孢菌(cladosporiumneopsychrotolernssl-19菌株)
<400>2
metvaltyrproalaalaileleuthrpheleuleualathrleuala
151015
glnserleuproglnlysglnargalaasplysalaargglyileile
202530
thraspalaasnhisvalalaasnlysthrpheasptyrilevalcys
354045
glyglyglyleuthrglyleuvalvalalaserargleusergluasp
505560
progluileservalleuvalilegluasnglyasnaspasphisglu
65707580
aspproargvalasnaspvalargthrtyrglyalaalaphegluser
859095
gluleuasppheasnleuthrserthrprovalprotrpglnasnasp
100105110
thrglyleuleuleuvalalaglyargthrleuglyglyserglyser
115120125
ileasnglyalasertrpthrlysglyasplysthrglntyraspleu
130135140
leuprovalleuthrglyaspaspsertrpserpheaspalaleuasn
145150155160
gluilemetleuserilegluaspphehisgluprothrglugluhis
165170175
ilealalysglyalaasnphethrsergluphehisglyargaspgly
180185190
metvalglnvalserpheproargglymetpheglyglyileglnval
195200205
proalaleuglualaserthralailetrplysglyleuglyileval
210215220
alaaspphealaalaglyleuthrasnglyalathrileileproasn
225230235240
metilegluaspasngluaspglnasnargserserprophethrval
245250255
tyralalysaspglnthrglnglnargaspasnphevalileleuthr
260265270
glyhisargvalthrserileasntrpargproglylysaspmetala
275280285
alagluglyvalasnpheglnalacysargsercysglulyshisval
290295300
alathrthrlysarggluvalleuleualaalaglyserleuglnser
305310315320
proglnleuleugluleuserglyvalglyaspprogluvalleuala
325330335
alaalaglyvalprovallysmetalaserproasnvalglylysasn
340345350
metglngluglnthrlysasnthrleutrppheaspprovalserthr
355360365
asppheaspglythrglyproproasnalailealapheprothrval
370375380
asnglnleuphelysasnasnseralaserleutyrglnserilethr
385390395400
serserleutrpglntyralaleuaspleulysalaserglyleuval
405410415
thrasnalathralathrhisleuileleuseralaglnvalasnasn
420425430
leutrplysaspseralaglyalaalagluvalphephevalthrser
435440445
proalathrglyglnileglyileaspleutrpasnleuilevalleu
450455460
serargglytyrvalhisilethrserasnsersertrpasphispro
465470475480
gluilegluprosertyrpheglyhispropheaspleuglyvalgln
485490495
leualaalaalaglnglnserarggluilepheglnthralaproleu
500505510
alaalahisvalalasergluthrpheproglyleugluthrvalpro
515520525
glnasnalathrthrgluvaltrpgluglutrpvallysglythrphe
530535540
thrservaltrphistyrilealathrleuglymetmetlysgluglu
545550555560
leuglygluleutrpilethrasp
565
<210>3
<211>552
<212>prt
<213>枝孢菌(cladosporiumneopsychrotolernssl-19菌株)
<400>3
glnserleuproglnlysglnargalaasplysalaargglyileile
151015
thraspalaasnhisvalalaasnlysthrpheasptyrilevalcys
202530
glyglyglyleuthrglyleuvalvalalaserargleusergluasp
354045
progluileservalleuvalilegluasnglyasnaspasphisglu
505560
aspproargvalasnaspvalargthrtyrglyalaalaphegluser
65707580
gluleuasppheasnleuthrserthrprovalprotrpglnasnasp
859095
thrglyleuleuleuvalalaglyargthrleuglyglyserglyser
100105110
ileasnglyalasertrpthrlysglyasplysthrglntyraspleu
115120125
leuprovalleuthrglyaspaspsertrpserpheaspalaleuasn
130135140
gluilemetleuserilegluaspphehisgluprothrglugluhis
145150155160
ilealalysglyalaasnphethrsergluphehisglyargaspgly
165170175
metvalglnvalserpheproargglymetpheglyglyileglnval
180185190
proalaleuglualaserthralailetrplysglyleuglyileval
195200205
alaaspphealaalaglyleuthrasnglyalathrileileproasn
210215220
metilegluaspasngluaspglnasnargserserprophethrval
225230235240
tyralalysaspglnthrglnglnargaspasnphevalileleuthr
245250255
glyhisargvalthrserileasntrpargproglylysaspmetala
260265270
alagluglyvalasnpheglnalacysargsercysglulyshisval
275280285
alathrthrlysarggluvalleuleualaalaglyserleuglnser
290295300
proglnleuleugluleuserglyvalglyaspprogluvalleuala
305310315320
alaalaglyvalprovallysmetalaserproasnvalglylysasn
325330335
metglngluglnthrlysasnthrleutrppheaspprovalserthr
340345350
asppheaspglythrglyproproasnalailealapheprothrval
355360365
asnglnleuphelysasnasnseralaserleutyrglnserilethr
370375380
serserleutrpglntyralaleuaspleulysalaserglyleuval
385390395400
thrasnalathralathrhisleuileleuseralaglnvalasnasn
405410415
leutrplysaspseralaglyalaalagluvalphephevalthrser
420425430
proalathrglyglnileglyileaspleutrpasnleuilevalleu
435440445
serargglytyrvalhisilethrserasnsersertrpasphispro
450455460
gluilegluprosertyrpheglyhispropheaspleuglyvalgln
465470475480
leualaalaalaglnglnserarggluilepheglnthralaproleu
485490495
alaalahisvalalasergluthrpheproglyleugluthrvalpro
500505510
glnasnalathrthrgluvaltrpgluglutrpvallysglythrphe
515520525
thrservaltrphistyrilealathrleuglymetmetlysgluglu
530535540
leuglygluleutrpilethrasp
545550
<210>4
<211>3690
<212>dna
<213>枝孢菌(cladosporiumneopsychrotolernssl-19菌株)
<400>4
atggtttacccggctgctattctcacatttctcctcgccactctcgcacaatcattaccc60
taccaaatgggccgacgataagagtgtaaagaggagcggtgagagcgtgttagtaatggg120
cagaagcaacgagcggacaaggcacgcggaatcatcaccgacgccaaccatgtcgccaac180
gtcttcgttgctcgcctgttccgtgcgccttagtagtggctgcggttggtacagcggttg240
aagacctttgactacatcgtctgcggtggcggactgacaggactagtcgtagcgagtcgt300
ttctggaaactgatgtagcagacgccaccgcctgactgtcctgatcagcatcgctcagca360
ctctcagaagaccccgagatatcagtgctagtgatcgagaacggcaacgacgaccacgag420
gagagtcttctggggctctatagtcacgatcactagctcttgccgttgctgctggtgctc480
gaccctcgtgtcaacgatgtcaggacctacggagcagcattcgaatccgagctcgacttc540
ctgggagcacagttgctacagtcctggatgcctcgtcgtaagcttaggctcgagctgaag600
aatcttacatcgacgcctgtcccgtggcagaacgacaccggcctgctcctagttgcgggc660
ttagaatgtagctgcggacagggcaccgtcttgctgtggccggacgaggatcaacgcccg720
aggactctaggcggaagtggcagcatcaacggcgctagctggacgaaaggcgacaagacg780
tcctgagatccgccttcaccgtcgtagttgccgcgatcgacctgctttccgctgttctgc840
cagtatgatttgctaccagtgctgacgggggatgactcttggtctttcgacgccctgaac900
gtcatactaaacgatggtcacgactgccccctactgagaaccagaaagctgcgggacttg960
gagatcatgctcagcatcgaggacttccacgagccaacggaagagcacatcgccaaaggc1020
ctctagtacgagtcgtagctcctgaaggtgctcggttgccttctcgtgtagcggtttccg1080
gcgaacttcacaagcgagttccacggccgggatgggatggtgcaggtttcgtttccgaga1140
cgcttgaagtgttcgctcaaggtgccggccctaccctaccacgtccaaagcaaaggctct1200
ggcatgtttggcggaatccaagttccagcgttggaagcgtccacggccatctggaagggg1260
ccgtacaaaccgccttaggttcaaggtcgcaaccttcgcaggtgccggtagaccttcccc1320
ttggggatagttgcggatttcgcagcgggactcacgaacggcgctacaatcattcctaac1380
aacccctatcaacgcctaaagcgtcgccctgagtgcttgccgcgatgttagtaaggattg1440
atgattgaagacaacgaggatcagaaccgctcttcgcctttcactgtgtatgcaaaggac1500
tactaacttctgttgctcctagtcttggcgagaagcggaaagtgacacatacgtttcctg1560
cagacgcagcagcgggataatttcgtgatcctgacaggtcaccgcgtcacatcgatcaac1620
gtctgcgtcgtcgccctattaaagcactaggactgtccagtggcgcagtgtagctagttg1680
tggcgtccgggcaaagacatggccgccgagggcgtgaacttccaggcatgtcgcagctgc1740
accgcaggcccgtttctgtaccggcggctcccgcacttgaaggtccgtacagcgtcgacg1800
gagaaacacgtcgcgacaacgaagcgcgaagtactgctcgccgcaggctctctacaaagc1860
ctctttgtgcagcgctgttgcttcgcgcttcatgacgagcggcgtccgagagatgtttcg1920
ccacagctgctcgagctgtcaggcgtaggagacccagaagtcctcgccgccgccggcgta1980
ggtgtcgacgagctcgacagtccgcatcctctgggtcttcaggagcggcggcggccgcat2040
cccgtgaagatggcatcccccaacgtcggcaagaacatgcaagaacaaaccaaaaacacc2100
gggcacttctaccgtagggggttgcagccgttcttgtacgttcttgtttggtttttgtgg2160
ctctggttcgaccccgtcagcaccgacttcgacggcaccggccccccaaacgccatcgcc2220
gagaccaagctggggcagtcgtggctgaagctgccgtggccggggggtttgcggtagcgg2280
ttccccaccgtcaaccaactcttcaaaaacaacagcgcatccttgtaccaaagcatcaca2340
aaggggtggcagttggttgagaagtttttgttgtcgcgtaggaacatggtttcgtagtgt2400
tccagcctctggcaatacgccctcgacctcaaagcctctggcctcgtaacaaacgccacc2460
aggtcggagaccgttatgcgggagctggagtttcggagaccggagcattgtttgcggtgg2520
gcaacccacctaatcctctcagcccaagtcaacaacctgtggaaagactccgccggcgcc2580
cgttgggtggattaggagagtcgggttcagttgttggacacctttctgaggcggccgcgg2640
gcagaagtcttcttcgtcacctcgcccgccaccggccaaatcggcatcgacctctggaac2700
cgtcttcagaagaagcagtggagcgggcggtggccggtttagccgtagctggagaccttg2760
ctcatcgtgctgtccaggggttacgtgcacatcacctccaactcctcctgggaccacccg2820
gagtagcacgacaggtccccaatgcacgtgtagtggaggttgaggaggaccctggtgggc2880
gagatcgagccctcgtacttcggccaccccttcgacctcggggtccagctggcagcggcc2940
ctctagctcgggagcatgaagccggtggggaagctggagccccaggtcgaccgtcgccgg3000
cagcaatcgcgcgagatctttcagacggcaccactcgcagctcacgtcgcgtcggagacg3060
gtcgttagcgcgctctagaaagtctgccgtggtgagcgtcgagtgcagcgcagcctctgc3120
ttccctggtcttgaaaccgttcctcaaaacgccactacggaagtctgggaggagtgggtc3180
aagggaccagaactttggcaaggagttttgcggtgatgccttcagaccctcctcacccag3240
aaaggtacgttcacatctgtttggcactacatagctacattgggcatgatgaaagaggaa3300
tttccatgcaagtgtagacaaaccgtgatgtatcgatgtaacccgtactactttctcctt3360
ttgggtggtgttgtggacagcaggctgaaggtatatggcattgagaatgtacgagcggtg3420
aacccaccacaacacctgtcgtccgacttccatataccgtaactcttacatgctcgccac3480
gatgctagtgtgctgccgatccagctttcggcgcacttgagctcttcgctgtacggcatt3540
ctacgatcacacgacggctaggtcgaaagccgcgtgaactcgagaagcgacatgccgtaa3600
gcggagaaggctgctatgatgattaaggaagatcagggacattgacgcctcttccgacga3660
tactactaattccttctagtccctgtaact3690
<210>5
<211>614
<212>prt
<213>枝孢菌(cladosporiumneopsychrotolernssl-19菌株)
<400>5
metvaltyrproalaalaileleuthrpheleuleualathrleuala
151015
glnserleuproglnlysglnargalaasplysalaargglyileile
202530
thraspalaasnhisvalalaasnlysthrpheasptyrilevalcys
354045
glyglyglyleuthrglyleuvalvalalaserargleusergluasp
505560
progluileservalleuvalilegluasnglyasnaspasphisglu
65707580
aspproargvalasnaspvalargthrtyrglyalaalaphegluser
859095
gluleuasppheasnleuthrserthrprovalprotrpglnasnasp
100105110
thrglyleuleuleuvalalaglyargthrleuglyglyserglyser
115120125
ileasnglyalasertrpthrlysglyasplysthrglntyraspleu
130135140
leuprovalleuthrglyaspaspsertrpserpheaspalaleuasn
145150155160
gluilemetleuserilegluaspphehisgluprothrglugluhis
165170175
ilealalysglyalaasnphethrsergluphehisglyargaspgly
180185190
metvalglnvalserpheproargglymetpheglyglyileglnval
195200205
proalaleuglualaserthralailetrplysglyleuglyileval
210215220
alaaspphealaalaglyleuthrasnglyalathrileileproasn
225230235240
metilegluaspasngluaspglnasnargserserprophethrval
245250255
tyralalysaspglnthrglnglnargaspasnphevalileleuthr
260265270
glyhisargvalthrserileasntrpargproglylysaspmetala
275280285
alagluglyvalasnpheglnalacysargsercysglulyshisval
290295300
alathrthrlysarggluvalleuleualaalaglyserleuglnser
305310315320
proglnleuleugluleuserglyvalglyaspprogluvalleuala
325330335
alaalaglyvalprovallysmetalaserproasnvalglylysasn
340345350
metglngluglnthrlysasnthrleutrppheaspprovalserthr
355360365
asppheaspglythrglyproproasnalailealapheprothrval
370375380
asnglnleuphelysasnasnseralaserleutyrglnserilethr
385390395400
serserleutrpglntyralaleuaspleulysalaserglyleuval
405410415
thrasnalathralathrhisleuileleuseralaglnvalasnasn
420425430
leutrplysaspseralaglyalaalagluvalphephevalthrser
435440445
proalathrglyglnileglyileaspleutrpasnleuilevalleu
450455460
serargglytyrvalhisilethrserasnsersertrpasphispro
465470475480
gluilegluprosertyrpheglyhispropheaspleuglyvalgln
485490495
leualaalaalaglnglnserarggluilepheglnthralaproleu
500505510
alaalahisvalalasergluthrpheproglyleugluthrvalpro
515520525
glnasnalathrthrgluvaltrpgluglutrpvallysglythrphe
530535540
thrservaltrphistyrilealathrleuglymetmetlysgluglu
545550555560
leuglyglyvalvalaspserargleulysvaltyrglyilegluasn
565570575
valargalavalaspalaservalleuproileglnleuseralahis
580585590
leuserserserleutyrglyilealaglulysalaalametmetile
595600605
lysgluaspglnglyhis
610
<210>6
<211>598
<212>prt
<213>枝孢菌(cladosporiumneopsychrotolernssl-19菌株)
<400>6
glnserleuproglnlysglnargalaasplysalaargglyileile
151015
thraspalaasnhisvalalaasnlysthrpheasptyrilevalcys
202530
glyglyglyleuthrglyleuvalvalalaserargleusergluasp
354045
progluileservalleuvalilegluasnglyasnaspasphisglu
505560
aspproargvalasnaspvalargthrtyrglyalaalaphegluser
65707580
gluleuasppheasnleuthrserthrprovalprotrpglnasnasp
859095
thrglyleuleuleuvalalaglyargthrleuglyglyserglyser
100105110
ileasnglyalasertrpthrlysglyasplysthrglntyraspleu
115120125
leuprovalleuthrglyaspaspsertrpserpheaspalaleuasn
130135140
gluilemetleuserilegluaspphehisgluprothrglugluhis
145150155160
ilealalysglyalaasnphethrsergluphehisglyargaspgly
165170175
metvalglnvalserpheproargglymetpheglyglyileglnval
180185190
proalaleuglualaserthralailetrplysglyleuglyileval
195200205
alaaspphealaalaglyleuthrasnglyalathrileileproasn
210215220
metilegluaspasngluaspglnasnargserserprophethrval
225230235240
tyralalysaspglnthrglnglnargaspasnphevalileleuthr
245250255
glyhisargvalthrserileasntrpargproglylysaspmetala
260265270
alagluglyvalasnpheglnalacysargsercysglulyshisval
275280285
alathrthrlysarggluvalleuleualaalaglyserleuglnser
290295300
proglnleuleugluleuserglyvalglyaspprogluvalleuala
305310315320
alaalaglyvalprovallysmetalaserproasnvalglylysasn
325330335
metglngluglnthrlysasnthrleutrppheaspprovalserthr
340345350
asppheaspglythrglyproproasnalailealapheprothrval
355360365
asnglnleuphelysasnasnseralaserleutyrglnserilethr
370375380
serserleutrpglntyralaleuaspleulysalaserglyleuval
385390395400
thrasnalathralathrhisleuileleuseralaglnvalasnasn
405410415
leutrplysaspseralaglyalaalagluvalphephevalthrser
420425430
proalathrglyglnileglyileaspleutrpasnleuilevalleu
435440445
serargglytyrvalhisilethrserasnsersertrpasphispro
450455460
gluilegluprosertyrpheglyhispropheaspleuglyvalgln
465470475480
leualaalaalaglnglnserarggluilepheglnthralaproleu
485490495
alaalahisvalalasergluthrpheproglyleugluthrvalpro
500505510
glnasnalathrthrgluvaltrpgluglutrpvallysglythrphe
515520525
thrservaltrphistyrilealathrleuglymetmetlysgluglu
530535540
leuglyglyvalvalaspserargleulysvaltyrglyilegluasn
545550555560
valargalavalaspalaservalleuproileglnleuseralahis
565570575
leuserserserleutyrglyilealaglulysalaalametmetile
580585590
lysgluaspglnglyhis
595
- 还没有人留言评论。精彩留言会获得点赞!