从患者肿瘤分离的识别野生型抗原和有效肽模拟表位的T细胞受体的抗原发现的制作方法
交叉引用
本申请要求2017年3月24日提交的美国临时专利申请号62/476,575的权益,所述申请以引用的方式整体并入本文。
背景技术:
t细胞是适应性免疫系统不可或缺的并且提供针对病原体和癌症的保护。它们通过tcr的细胞外识别而起作用,tcr对细胞上的人白细胞抗原(hla)上呈递的短肽具有特异性(birnbaum等人,(2014)cell157,1073-1087)。tcr、肽和hla分子所固有的多样性使鉴定任一个tcr的特异性成为极其复杂的问题。尽管表征t细胞和对其tcr进行测序的能力最近得到了很大改进(han等人,(2014)natbiotechnol32,684-692;stubbington等人,(2016)natmethods13,329-332),但是确定和研究t细胞的抗原特异性的能力没有类似地进展。
每个人个体在他们的体内具有1012个t细胞,具有107至108个独特的t细胞受体。每个t细胞表达独特的t细胞受体(tcr),所述t细胞受体针对与呈递肽的主要组织相容性复合物(mhc)分子结合的能力进行选择。肽-mhc(pmhc)的tcr识别驱动t细胞的发育、存活和效应子功能。虽然tcr配体的亲和力相对较低(1-100μm),但tcr非常敏感,从而仅需要10种激动剂肽来完全活化t细胞。在识别后,信号传导级联允许t细胞执行其免疫功能。
对pmhc的tcr识别的广泛结构研究表明,通过mhc螺旋的顶部与种系编码的tcrcdr1和cdr2环之间的保守性接触驱动,绝大多数研究的tcr-pmhc复合物共有一致的结合取向(参见garcia和adams(2005)cell122,333-336;garcia等人(2009)natimmunol10,143-147;以及rudolph等人(2006)annualreviewofimmunology24,419-466)。这些保守性接触可能已经在适应性免疫系统的整个发展过程中共同演变,并且充当αβtcr谱系的mhc限制的基础(scott-brown等人,2011)。已显示对典型tcr-pmhc相互作用的改变与消除的信号传导相关,并且(当存在于发育中时)与偏斜的tcr谱系相关(adams等人(2011)immunity35(5):681-93;birnbaum等人(2012)immunol.rev.250(1):82-101)。
tcr的另一个重要特征是能够使交叉反应性与特异性平衡。由于独特地识别每种可能的pmhc组合所需的t细胞的数量非常高,并且由于tcr谱系中几乎不存在表征的‘孔’,因此假定tcr交叉反应性的程度高是功能性抗原识别的要求。t细胞谱系能够同时受到mhc限制、交叉反应足以确保可满足所有潜在的抗原激发、但仍具有足够的特异性以避免异常自身免疫仍然是免疫学中待决的紧迫问题。
已经有许多策略用于确定孤儿tcr的特异性(birnbaum等人,(2012)immunolrev250,82-101)。质谱法可提供抗原分离的无偏方法,但限于需要大量细胞数量(通常为107至109)的实验,并且靶标必须仍由正确的hla呈递。传统上,t细胞抗原特异性的大多数研究都涉及凭经验测试候选抗原。例如,抗肿瘤t细胞特异性的研究已正确地假定存在针对新抗原的生产性t细胞应答。此类研究涉及对肿瘤进行测序以鉴定突变,使用表位预测算法来预测免疫原性突变型肽,并测试针对这些突变型肽的t细胞应答(kreiter等人,(2015)nature520,692-696;rajasagi等人,(2014)blood124,453-462;tran等人,(2014)science344,641-645)。其他策略通过使用phla多聚体查询患者中已确立的t细胞特异性(bentzen等人,(2016)natbiotechnol34,1037-1045;newell等人,(2013)natbiotechnol31,623-629)。
用于确定‘孤儿’tcr(即抗原特异性未知的tcr)的特异性的高通量且灵敏的方法可能有助于发现癌症免疫疗法、自身免疫和感染的潜在靶标,并提供对具有极大兴趣的疾病发病机理的机制性洞察。
技术实现要素:
提供了在限定的mhc背景下目标t细胞受体(tcr)的配体的组合物。所述组合物可包含限定的肽或由限定的肽组成,或者可包含编码这种肽的多核苷酸或由编码这种肽的多核苷酸组成。此类肽可以是天然存在的抗原性蛋白的片段;可以是为肿瘤发生期间体细胞突变的对象的新抗原性蛋白的片段,或者可以是抗原性蛋白的合成产生的模拟物。合成肽可充当t细胞受体的高效激动剂。在一些实施方案中,肽或编码序列是选自本文提供的序列,包括但不限于seqidno:1-257中列出的肽序列中的任一者或组合。肽可作为在刺激t细胞中有活性的短抗原性序列提供;或者可以较大蛋白质(例如完整结构域、可溶性蛋白质部分、完整蛋白质等)的形式提供。在一些实施方案中,鉴定了在患者之间共有并提供用于广泛适用疗法的手段的肽抗原。在其他实施方案中,抗原的鉴定提供个人化用药方法。
t细胞受体和同源抗原的鉴定提供免疫疗法的靶标,包括筛选患者t细胞的应答性、用肽或编码此类肽的核酸进行疫苗接种、基于细胞的疗法、基于蛋白质的疗法等。本文公开的肽和方法可用于基于肽抗原特异性对tcr进行分类,这允许鉴定识别跨患者的共有抗原的临床候选tcr。
在一些实施方案中,提供了针对癌症(例如结肠直肠癌)的疫苗接种的方法,所述方法包括施用有效剂量的疫苗组合物,所述组合物可包含本文鉴定的肽;肽的组合,例如2、3、4、5、6、7、8、9、10种或更多种不同的肽;肽和mhc蛋白的至少一部分的复合物;已被刺激以对本文鉴定的抗原肽产生应答的自体或同种异体t细胞;编码本文鉴定的抗原肽的核酸;以及任选的药学上可接受的赋形剂,所述赋形剂可包含疫苗佐剂。肽疫苗接种策略可用于例如用本文提供的合成肽初始引发免疫应答,然后用相应的已知野生型抗原或野生型全蛋白质加强。
通过经由酵母展示筛选肽-mhc文库来鉴定限定的肽,所述酵母展示用于鉴定个别t细胞受体的识别景观。筛选方法可用于多重方法中,以同时筛选多个肽文库,例如同时筛选2、3、4个或更多个文库。多重允许提高抗原发现的效率。每个文库可包含独特的表位标签(例如可被抗体靶向的表位)以允许鉴定;可包含dna条形码;蛋白质条形码等。分别产生利用表位标签的每个文库,并且例如基于来自生长板上初始文库的有限稀释的菌落计数来计算多样性。合并用于文库选择的t细胞受体可进一步使选择多重化,例如肽序列、肽长度、不同mhc或hla等位基因的收集等的多重化。对于选择,可经由抗表位标签染色监测每个条形码、表位标签等以检测肽特异性富集的水平,统计算法和机器学习算法可用于鉴定。
在一些实施方案中,提供了对癌症抗原有反应的t细胞受体的序列。t细胞受体序列可包括但不限于具有任选地与seqidno:259或seqidno:260的β链序列组合的α链的蛋白质,所述α链具有seqidno:258中列出的序列。这些t细胞受体的结合区(cdr)序列可被移植到抗体框架上以提供tcr样抗体。由于t细胞受体具有适应性并且通常从患者至患者独特,因此个别t细胞受体序列可在患者之间不同。尽管存在这些差异,但不同的tcr仍可识别同一靶标。因此,不同的t细胞受体可具有与可结合同一靶标的这些t细胞受体相比的微小序列变异。另外,可修饰t细胞受体以引入将允许与同一抗原结合的氨基酸取代。此类情况包括针对特定靶标的t细胞受体的亲和力成熟或受体修饰,以改进所述t细胞受体对其靶标的特异性。t细胞受体的识别部分可被移植到其他蛋白支架上以用作治疗试剂。由于t细胞受体在某种程度上具有交叉反应性,因此合成肽的列表并不是详尽的。对肽序列的轻微修饰仍可导致t细胞刺激。
在一些实施方案中,从其获得用于筛选的tcr序列的t细胞是从肿瘤部位分离的,并且可包括但不限于肿瘤浸润性t细胞(til)。在其他实施方案中,所述t细胞从对感染(例如细菌、病毒、原生动物等感染)有反应的个体获得。在其他实施方案中,所述t细胞从移植物接受者获得,并且可从移植物部位分离。
附图说明
当结合附图阅读时,从以下详细描述中可最透彻地理解本发明。本专利或申请文件含有至少一幅彩色绘制的附图。要强调的是,根据惯例,附图的各种特征不是按比例的。相反,为清楚起见,各种特征的尺寸任意地放大或缩小。附图中包括以下各图。
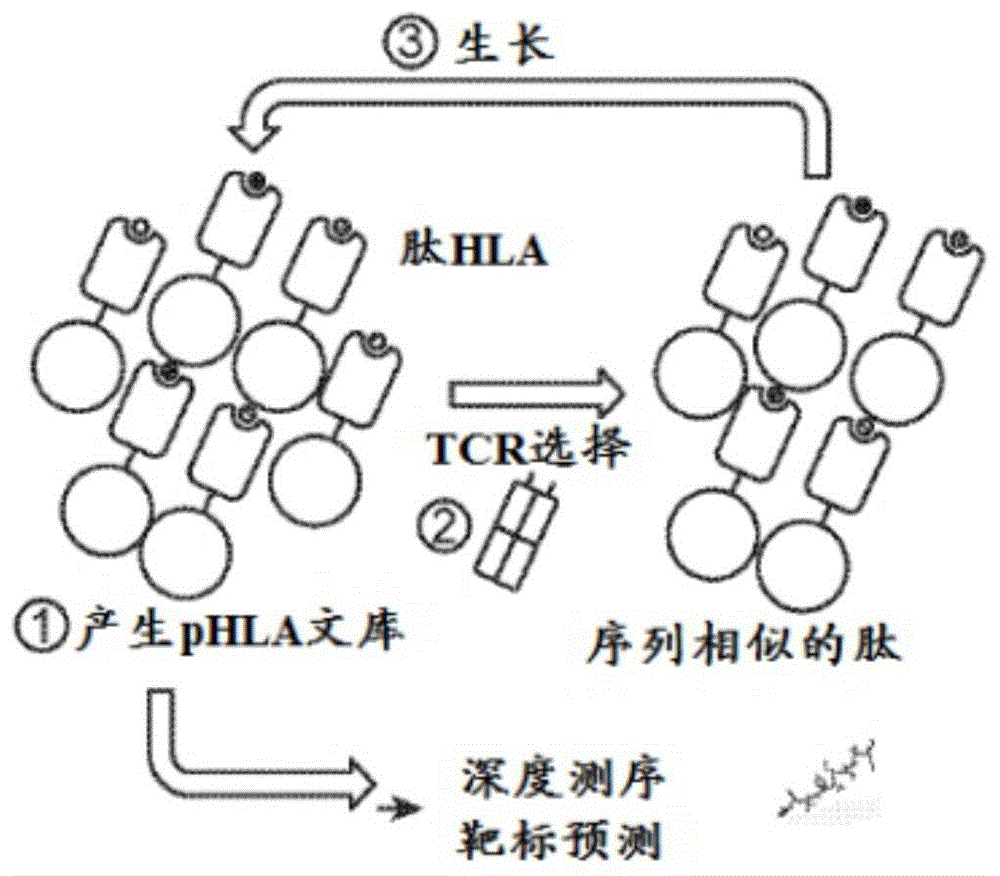
图1.肽-hla-a*02:01酵母展示文库的设计。(a)用于选择phla的酵母展示文库的方法。每种酵母展示经遗传编码的独特的肽。典型的文库含有约108种独特的肽,所述文库可通过目标tcr选择。使酵母在使用珠粒多聚化的tcr的基于亲和力的选择中富集并且生长用于迭代选择轮次。使肽连续富集,并且对所有酵母dna进行深度测序。这些合成的肽序列用于生成对源自人蛋白质组和/或患者特异性外显子组的tcr配体进行预测的模型。(b)所述研究的目标是使用酵母展示选择来破解(de-orphanize)具有未知抗原特异性的tcr。从酵母展示选择由tcr选择的肽产生特定tcr的识别景观,然后将其用于预测孤儿tcr的抗原特异性。可在t细胞刺激测定中验证预测的靶标。(c)所述构建体利用单链设计来展示栓系至表位标签和aga2p的phla-a*02:01复合物,所述复合物与酵母上的天然aga1蛋白结合。每种组分通过gly-ser接头共价连接。引入表位标签以监测文库的表达。(d)用橙色箭头突出显示两个肽锚的mart-1/hla-a*02复合物结构(pdb4l3e)。在肽的p2和pω处的这些肽位置允许肽与hla-a*02结合。(e)示例性8聚体肽文库示出hla-a*02:01文库的锚偏好以及随机化至二十个氨基酸中的任一个的剩余位置(x=20个氨基酸和终止密码子)。密码子使用的核苷酸缩写根据iupac核苷酸代码列出。(f)被设计成捕获由hla-a*02:01呈递的最常见长度肽的多长度文库。使用独特的表位标签将每种肽长度置于构建体中以进行选择监测。所述文库具有由肽长度和文库组成决定的理论核苷酸多样性。功能多样性代表了在文库的有限稀释后基于酵母菌落计数的物理文库的真实容量。
图2.使用dmf5tcr验证hla-a*02:01文库。(a)dmf5tcr对酵母染色,从而在酵母的表面上展示与hla-a*02:01复合的mart-1肽(elagigiltv)(seqidno:264)。链霉抗生物素蛋白-647(sa-647)用于四聚化并荧光标记dmf5tcr。(b)如通过流式细胞术通过抗ha表位标签染色测量的通过dmf5tcr对10聚体长度的hla-a*02:01酵母展示文库的富集。示出四轮选择中的三轮。(c)通过dmf5tcr四聚体对通过dmf5tcr从10聚体选择中测序的高度富集的肽进行染色,并通过流式细胞术进行测量。((c)序列从左至右:seqidno:264、324、286、323、283、285)。(d)根据通过dmf5tcr进行的第3轮10聚体hla-a*02:01文库选择的深度测序,前10种多肽的总测序读数计数的分数。((d)序列从上至下:seqidno:287、326、325、324、286、323、285、322、284、283)。(e)来自第3轮选择的独特的肽落入似乎与野生型mart-1肽序列(seqidno:267)相似的两个主要簇。通过首先计算第3轮选择中存在的所有肽之间的反汉明距离且然后根据得分进行聚类来确定簇。mart-1十聚体结构(pdb:4l3e)与选定的肽进行比对。(f)使用簇1肽的取代矩阵(2014pwm)将mart-1肽预测为在八种其他预测的肽中最可能结合dmf5tcr的肽。((f)序列从上至下:321、320、319、318、317、316、315、314、267)
图3.通过新抗原特异性tcr对hla-a*02:01文库进行盲法验证。(a)具有盲特异性的三种tcr分别根据在多轮选择内的表位标签染色针对特定肽长度富集hla-a*02:01文库。左图指示在已经完成全部4轮选择后的四聚体和表位染色,并且右图指示在整个选择过程中的表位染色。(b)将在第3轮选择中由nki2选择的独特的肽通过肽长度进行解析并通过反汉明距离进行聚类。簇中鉴定的肽的数目以及相应的肽长度在右侧示出。(c)在第3轮通过nki2每10聚体所选肽与来自总计127种新抗原的列表的各10聚体新抗原肽之间计算的最大反汉明距离。((c)序列从上至下:seqidno:501、502、620、503-519。(d)来自所选文库的两种肽lib-1(seqidno:434)和lib-2(seqidno:269)与源自cdk4的10聚体新抗原肽aldphsghfv(seqidno:265)非常类似。与所述新抗原相同的氨基酸以红色标注。(e)由nki2tcr选择的长度10的前5种肽用于刺激经转导以表达tcrnki1或nki2的外周血淋巴细胞,所述tcrnki1或nki2两者均对cdk4新抗原aldphsghfv(seqidno:265)具有特异性。将转导的淋巴细胞与用肽、对照肽或无肽脉冲的jy细胞1:1混合,并且使用流式细胞术评估如通过细胞内抗体染色测量的ifnγ产生。((e)序列从上至下:1)seqidno:269、2)seqidno:427、3)seqidno:423、4)seqidno:420、5)seqidno:417)。
图4.对在两名患有结肠直肠腺癌的hla-a*02患者中鉴定的tcr进行谱分析(a)破解hla-a*02:01文库上患者来源的tcr的研究设计与总结的结果。(b)来自til的独特的成对αβtcr序列的丰度的条形图。*=富集了来自文库的肽的tcr。(c)代表每名患者的肿瘤组织与健康组织之间的个别独特cdr3α或cdr3β链序列的重叠的维恩图(venndiagram)。数字指示维恩图的最近部分中的cdr3序列的量。(d)使用扩增且加条形码的转录物的测序来鉴定转录因子的二进制测量的热图。交替的黑色和白色图指示具有相同受体序列的单一t细胞克隆的边界,其中最丰富的克隆从最左侧开始。左图鉴定具有选自待筛选的患者a的tcr的那些t细胞,并且绿色表示存在转录物。右图鉴定具有选自待筛选的患者b的tcr的那些t细胞,并且蓝色表示存在转录物。白色指示没有检测到的转录物。对tcr1a、2a、3b和4b进行标记。
图5.四种til来源的tcr富集肽的hla-a*02:01文库。(a)从hla-a*02:01文库中选择肽的四种孤儿tcr的tcr序列。示出tcr基因区段可变和连接以及相应的cdr3序列。丰度代表据发现在肿瘤/健康组织中具有确切tcr序列的单个细胞的次数。((a))序列:1acdr3α:(seqidno:472),2acdr3α:(seqidno:261),3bcdr3α:(seqidno:261),4bcdr3α:(seqidno:495),1acdr3β:(seqidno:463),2acdr3β:(seqidno:262),3bcdr3β:(seqidno:263),4bcdr3β:(seqidno:484))(b)从患者a和b分离的两种序列相似tcr的核苷酸序列。未编码的核苷酸以红色突出显示。((b)氨基酸序列:cdr3α2a:(seqidno:261),cdr3α3b:(seqidno:261),cdr3β2a:(seqidno:262),cdr3β3b:(seqidno:263));核苷酸序列:cdr3α2a核苷酸序列:(seqidno:536),cdr3α3b核苷酸序列:(seqidno:537),cdr3β2a核苷酸序列:(seqidno:538),cdr3β3b核苷酸序列(seqidno:539)。(c)如通过流式细胞术测量的通过四种孤儿tcr每轮选择的hla富集和四聚体染色。左图指示在已经完成全部4轮选择后的四聚体和表位染色,并且右图指示在整个选择过程中的表位染色。
图6.通过四种tiltcr对酵母选择的深度测序结果。(a)字词徽标展示对于每种tcr,独特的第3轮所选肽,不考虑深度测序读数计数丰度。氨基酸字母的大小代表其在独特肽中的给定位置处的比例丰度。(b)示出在第3轮选择计入肽富集的肽的每位置的氨基酸组成的热图。较暗的颜色指示在给定位置处的给定氨基酸的丰度更高。锚定残基以黑色勾勒出轮廓。(c)tcr2a和3b在第3轮选择中选择11种肽的重叠集,显示为第3轮中总读数的分数。((c)序列从上至下:seqidno:95、249、54、195、42、191、196、198、200、201、4)。
图7.用预测的人靶标和肽模拟表位活化tiltcr。将tcr逆转录病毒感染到cd8+skw-3细胞中,并且进行分选以获得稳定的tcr(ip26)和cd3(ucht1)共表达。用100μm肽脉冲t2抗原呈递细胞3小时,与t细胞系共孵育18小时,并通过流式细胞术分析cd69表达。(a)将tcr1a、(c)tcr2a、(e)tcr3b和(g)tcr4b在技术三次重复中通过肽刺激针对cd69活化进行测试,并示出标准偏差。代表性实验从生物学三次重复示出。((a)序列从左至右:seqidno:540-555;(c)seqidno:556-574;(e)seqidno:556-574;(g)seqidno:596-619)。(b)、(d)、(f)、(h)每种刺激性肽的剂量反应曲线均显示在右侧,用生物学三次重复的平均值与平均值的标准误差绘制。对于两个实验,均使用普通单向anova计算p值。对于tcr2a和3b,为简单起见,除去17种非刺激性肽。((b)序列从上至下:seqidno:540-543;(d)序列从上至下:556-558、560、562-567;(f)序列从上至下:41、42、193、194、195、257;(h)序列从上至下:596-602、604、608、610、613、615)。
图8.使用dmf5tcr验证hla-α2*01文库。(a)与dmf5tcr野生型肽或肽模拟表位复合的正确折叠的hla-a*02:01的ma2.1抗体染色。直方图示出通过ma2.1抗体、随后第二抗体染色。((a)序列从左至右:seqidno:264、324、286、323、283、285)。(b)在dmf5tcr10聚体选择的第3轮序列的簇2上使用2014pwm算法预测的人多肽的得分。(c)在图8b中鉴定的前10种肽的得分。((c)序列从上至下:seqidno:364、363、362、361、360、359、358、357、356、355)。
图9.患者组织免疫组织化学和tcr谱系测序和表型分析。(a)使用h&e染色、抗cd4/苏木精或抗cd8/苏木精的患者免疫组织化学。所有代表性图像均使用300x放大倍率拍摄。(b)从cys至phe测量的患者cdr3长度。(c)在健康和肿瘤组织中tcr可变α基因的患者分布。(d)在健康和肿瘤组织中tcr可变β基因的患者分布。(e)患者bt细胞的t-sne图,其示出通过转录物测序的转录谱分析(左)和通过流式细胞术的细胞表面标志物(右)。转录物的存在是基于深度测序读数的二进制(1=是,0=否),并且强度与细胞表面标志物的mfi有关。
图10.用于预测人多肽特异性的机器学习算法2017dl的设计。(a)示出从酵母展示文库选择采集数据以训练机器学习模型的过程的示意图,所述模型对来源于来自uniprot数据库或患者特异性外显子组的蛋白质的肽进行评分。利用每种肽的深度测序轮次计数和肽的组成从酵母展示选择数据生成所述模型。使用适应度函数将指数曲线拟合至每种肽以捕获在选择轮次内的富集。(b)用于将指数曲线拟合至由tcr选择的肽的深度测序轮次计数的适应度函数。(c)示例性肽的矩阵表示,其中每个氨基酸表示为独热(one-hot)向量。(d)利用两层卷积神经网络的机器学习算法的体系结构。输入由表示为单热向量的向量的肽序列和从适应度函数确定的肽的适应度得分组成。输出是适应度得分。
图11.根据表达预测的肽靶标的酵母的cd69中值mfi和tcr四聚体染色,skw-3细胞的活化。从图7分析的数据,但使用cd69表达的平均荧光强度而不是(a)、(b)、(c)和(d)的对cd69表达呈阳性的细胞百分比。如图7中将具有tcr(a)1a、(b)2a、(c)3b或(d)4b的skw-3t细胞与肽脉冲的t2抗原呈递细胞共培养。在技术三次重复中从cd3门控的skw-3细胞的抗cd69染色测量平均荧光强度,示出平均值和标准偏差。示出来自生物学三次重复的代表性实验。使用普通单向anova测量p值。将表达tcr(e)1a、(f)2a、(g)3b和(h)4b的文库肽和预测的靶标肽的单链三聚体的酵母用400nmtcr四聚体染色。将四聚体阴性群体仅用链霉抗生物素蛋白-647染色。所有酵母都在表位标签阳性酵母上门控。((a)序列从上至下:seqidno:540-542)。
图12.u2af2定量rna表达和u2af2肽的亲和力测量。(a)使用18s作为管家基因,在患者a和b中相对于健康组织,肿瘤的u2af2转录物表达的定量pcr表达。样品在技术重复四次中进行,示出标准偏差。(b)使用18s作为管家基因,与患者a健康组织中的u2af2rna表达相比,在各种人源性肿瘤中u2af2rna的以2为底的对数定量pcr表达。样品在技术重复四次中进行,示出标准偏差。所示的细胞系以适当的顺序在方法部分中列出。(c)使用18s作为管家基因,与患者b健康组织中的u2af2rna表达相比,在各种人源性肿瘤中u2af2rna的以2为底的对数定量pcr表达。样品在技术重复四次中进行,示出标准偏差。所示的细胞系以适当的顺序在方法部分中列出。(d)使用2倍稀释,在涂有93.6μm至0.365μm范围的mmdffnaqm-hla-a*02:01(seqidno:266)的芯片上流动的递增浓度的tcr2a的表面等离子体共振迹线。为简单起见,将从流动池减法产生的tcr与肽-hla-a*02的缔合之前和之后的峰除去。仅使用标记有浓度的彩色曲线来计算kd。(e)图s4d中标记的各种浓度的tcr处生成的数据点的曲线拟合。
具体实施方式
在进一步描述本发明之前,应理解本发明不限于如下所述的本发明的特定实施方案,因为可进行所述特定实施方案的变化并且所述变化仍然在随附权利要求的范围内。还应该理解所采用的术语仅是用于描述特定实施方案的目的,而并非旨在加以限制。在本说明书和随附的权利要求书中,除非上下文另外明确规定,否则单数形式“一个/种(a/an)”和“所述(the)”包括复数指示物。
在提供值的范围的情况下,应理解本发明内涵盖所述范围的上限与下限之间的各介入值,准确到下限的单位的十分之一(除非上下文另外清楚指示),以及所述陈述范围内的任何其他陈述值或介入值。这些较小范围的上限和下限可独立地包括在更小的范围内,并且也涵盖在本发明内,服从所陈述范围内任何明确排除的极限值。当所述的范围包括限值中的一者或两者时,排除那些所包括的限值中的任一者或两者的范围也包括于本发明中。
除非另外定义,否则本文所用的所有技术术语和科学术语均具有与本发明所属领域中的普通技术人员通常理解的相同的含义。虽然可在本发明的实践或测试中使用与本文所描述的那些方法、装置和材料类似或等效的任何方法、装置和材料,但现在描述说明性方法、装置和材料。
出于描述和公开描述于公布中的本发明的主题组分的目的,本文所提及的所有公布均以引用的方式并入本文,所述组分可与当前描述的发明结合使用。
已经根据本发明人发现或提出的特定实施方案描述了本发明,以包括用于实施本发明的优选模式。本领域普通技术人员将理解,鉴于本公开,在不脱离本发明的预期范围的情况下,可在举例说明的特定实施方案中进行各种修改和改变。例如,由于密码子冗余,可在基础dna序列中进行改变而不影响蛋白质序列。此外,由于生物功能等效性考虑,可在蛋白质结构中进行改变而不影响种类或量的生物作用。所有此类修改意图包括在附随权利要求书的范围内。
筛选方法.通过产生单链多肽的文库发现了抗原序列,所述单链多肽包含:主要组织相容性复合物蛋白的结合结构域;以及各种肽配体。将所述文库引入表达编码的多肽的合适宿主细胞中,所述宿主细胞包括但不限于酵母细胞。使目标tcr多聚化以增强结合,并且用于选择表达与t细胞受体结合的那些单链多肽的宿主细胞。进行迭代选择轮次,即在第一轮中选择的细胞为第二轮提供起始群体等,直到所选群体具有高于背景的信号,进行通常至少三轮且更通常至少四轮选择。使编码来自单链多肽文库的最终选定群体的多核苷酸经受高通量测序。所选组的肽配体在残基(例如接触tcr的残基)处表现出有限的氨基酸选择,所述信息可被输入到算法,所述算法可用于分析公共数据库中符合结合标准的所有肽并且提供一组符合这些标准的肽。
肽配体的长度是约8至约20个氨基酸,通常约8至约18个氨基酸、约8至约16个氨基酸、约8至约14个氨基酸、约8至约12个氨基酸、约10至约14个氨基酸、约10至约12个氨基酸。应理解,完全随机的文库将代表大量可能的组合。在优选的方法中,多样性在将肽锚定至mhc结合结构域的残基处是有限的,所述残基在本文中称为mhc锚定残基。肽中锚定残基的位置由特定mhc结合结构域决定。i类结合结构域可在p2位置和最后一个接触残基处具有锚定残基。ii类结合结构域在p1处并且取决于等位基因在p4、p6或p9中的一个处具有锚定残基。例如,iek的锚定残基是p1{i,l,v}和p9{k};hla-dr15的锚定残基是p1{i,l,v}和p4{f,y}。dr等位基因的锚定残基在p1处共有,在p4、p6、p7和/或p9处具有等位基因特异性锚定残基。
在一些实施方案中,主要组织相容性复合物蛋白的结合结构域是ii类α和β链的可溶性结构域。在一些此类实施方案中,已经使结合结构域经受诱变并且选择增加单链多肽的溶解度而不改变肽结合接触的氨基酸变化。在某些具体实施方案中,结合结构域是包含氨基酸变化集合{m36l,v132m}的hla-dr4α;和包含氨基酸变化集合{h62n,d72e}的hla-dr4β。在某些具体实施方案中,结合结构域是包含氨基酸变化集合{f12s,m23k}的hla-dr15α;和包含氨基酸变化{p11s}的hla-dr15β。在某些具体实施方案中,结合结构域是包含氨基酸变化集合{i8t,f12s,l14t,a56v}的h2iekα和包含氨基酸变化集合{w6s,l8t,l34s}的h2iekβ。
在一些实施方案中,主要组织相容性复合物蛋白的结合结构域包含i类mhc蛋白的α1和α2结构域,所述结构域与β2微球蛋白一起提供于单链中。在一些此类实施方案中,已经使i类蛋白经受诱变并且选择增加单链多肽的溶解度而不改变肽结合接触的氨基酸变化。在某些具体实施方案中,结合结构域是包含氨基酸变化{y84a}的hla-a2α1和α2结构域。在某些具体实施方案中,结合结构域是包含氨基酸变化{m31r}的h2-ldα1和α2结构域。在某些具体实施方案中,结合结构域是包含氨基酸变化{y84a}的hla-b57α1、α2和α3结构域。
肽的序列可通过任何方便的高通量测序方法来确定。可使用聚类算法,例如通过实施例中公开的方法来对序列进行分析。可对肽进行分析以搜索人蛋白质(uniprot)或患者特异性外显子组,以使用滑动窗口对固定长度的肽进行评分。通过确定肽的每个位置的所有氨基酸的频率来制得取代矩阵。对于给定位置处的氨基酸,可设置0.1%频率的截断值以消除噪音。
为了确定肽的统计显著性,对人蛋白质组和外显子组肽集合进行评分。为了计算外显子组肽集合的p值,在人蛋白质组得分的背景下计算百分位数得分。未校正的p值是1-百分位数。bonferroni校正的p值是未校正的p值乘以突变体集合中的肽的数量。
mhc蛋白.主要组织相容性复合物蛋白(也称为人白细胞抗原,hla,或小鼠中的h2基因座)是在细胞表面上表达的赋予这些细胞独特抗原身份的蛋白质分子。mhc/hla抗原是由t细胞和自然杀伤(nk)细胞识别为来源于与免疫效应细胞相同来源的造血重构干细胞(“自身(self)”)或来源于另一种来源的造血重构细胞(“非自身(non-self)”)的靶分子。识别两种主要类别的hla抗原:hlai类和hlaii类。
在本发明的文库和方法中使用的mhc蛋白可来自任何哺乳动物或禽类物种,例如灵长类物种,特别是人;啮齿动物,包括小鼠、大鼠和仓鼠;兔;马科动物、牛科动物、犬科动物、猫科动物;等。特别感兴趣的是人hla蛋白和鼠h-2蛋白。hla蛋白中包括ii类亚基hla-dpα、hla-dpβ、hla-dqα、hla-dqβ、hla-drα和hla-drβ,和i类蛋白hla-a、hla-b、hla-c,以及β2-微球蛋白。鼠h-2亚基中包括i类h-2k、h-2d、h-2l,以及ii类i-aα、i-aβ、i-eα和i-eβ,以及β2-微球蛋白。
mhc结合结构域通常是正常膜结合蛋白的可溶性形式。可溶性形式通过缺失跨膜结构域而源自天然形式。方便地,蛋白质被截短,从而除去细胞质结构域和跨膜结构域两者。在一些实施方案中,主要组织相容性复合物蛋白的结合结构域是ii类α和β链的可溶性结构域。在一些此类实施方案中,已经使结合结构域经受诱变并且选择增加单链多肽的溶解度而不改变肽结合接触的氨基酸变化。
“等位基因”是染色体上的特定基因座处的基因的不同核酸序列之一。一个或多个遗传差异可构成等位基因。hla基因系统的重要方面是其多态性。每种基因mhci类(a、b和c)和mhcii类(dp、dq和dr)存在于不同的等位基因中。hla等位基因的当前命名法由数字指定,如由marsh等人:nomenclatureforfactorsofthehlasystem,2010.tissueantigens75:291-455所描述,所述文献以引用的方式具体地并入本文。关于hla蛋白和核酸序列,参见robinson等人(2011),theimgt/hladatabase.nucleicacidsresearch39增刊1:d1171-6,所述文献以引用的方式具体地并入本文。
使本文公开的各种mhc蛋白和变体上的氨基酸残基的编号与全长多肽一致。边界设置为‘微型’mhc的mhc肽结合结构域的末端(如通过检查晶体结构所判断),如本文以i-ek、h2-ld和hla-dr15所例示的;以及如通过‘全长’mhc的结构和/或序列判断的β2/α2/α3结构域的末端,如本文以hla-a2、-b57和-dr4所例示的。
在一些实施方案中,构建体的mhc部分是seqidno:1-6中的任一者中描绘的mhc部分。本领域技术人员将理解,肽和接头部分可与提供的序列不同。
mhc背景.mhc分子的功能是结合源自病原体的肽片段并将它们展示在细胞表面上,以被适当的t细胞识别。因此,t细胞受体识别可能受到呈递抗原的mhc蛋白的影响。术语mhc背景是指当其通过特定mhc蛋白呈递时,通过给定肽的tcr识别。
ii类hla/mhc.ii类结合结构域通常包含α链的α1和α2结构域以及β链的β1和β2结构域。将包括跨膜结构域的氨基酸中的不超过约10个、通常不超过约5个、优选没有一个。缺失将是使得其不干扰α2或β2结构域结合肽配体的能力。
在一些实施方案中,主要组织相容性复合物蛋白的结合结构域是ii类α和β链的可溶性结构域。在一些此类实施方案中,已经使结合结构域经受诱变并且选择增加单链多肽的溶解度而不改变肽结合接触的氨基酸变化。
在某些具体实施方案中,结合结构域是hla-dr等位基因。hla-dra蛋白可选自但不限于以下的结合结构域:dra*01:01:01:01;dra*01:01:01:02;dra*01:01:01:03;dra*01:01:02;dra*01:02:01;dra*01:02:02;以及dra*01:02:03,所述结合结构域可被修饰以包含氨基酸变化{m36l,v132m};或{f12s,m23k},这取决于是在全长等位基因还是微型等位基因的背景下提供。hla-dra结合结构域可与hla-drb结合结构域中的任一者组合。
在某些此类实施方案中,hla-dra等位基因与hla-drb4等位基因的结合结构域配对。hla-drb4等位基因可选自可公开获得的drb4等位基因。
在其他此类实施方案中,hla-dra等位基因与hla-drb15等位基因的结合结构域配对。hla-drb15等位基因可选自可公开获得的drb15等位基因。
在其他实施方案中,ii类结合结构域是h2蛋白,例如i-aα、i-aβ、i-eα和i-eβ。在一些此类实施方案中,结合结构域是可包含氨基酸变化集合{i8t,f12s,l14t,a56v}的h2iekα;和可包含氨基酸变化集合{w6s,l8t,l34s}的h2iekβ。
i类hla/mhc.对于i类蛋白,结合结构域可包括i类等位基因的α1、α2和α3结构域,包括但不限于hla-a、hla-b、hla-c、h-2k、h-2d、h-2l,所述结构域与β2-微球蛋白组合。将包括跨膜结构域的氨基酸中的不超过约10个、通常不超过约5个、优选没有一个。缺失将是使得其不干扰结构域结合肽配体的能力。
在某些具体实施方案中,结合结构域是hla-a2结合结构域,例如至少包含a2蛋白的α1和α2结构域。已在hla-a2中鉴定了大量等位基因,包括但不限于hla-a*02:01:01:01至hla-a*02:478,所述序列可在例如robinson等人(2011),theimgt/hladatabase.nucleicacidsresearch39增刊1:d1171-6获得。在hla-a2等位基因变体中,hla-a*02:01是最普遍的。结合结构域可包含氨基酸变化{y84a}。
在某些具体实施方案中,结合结构域是hla-b57结合结构域,例如至少包含b57蛋白的α1和α2结构域。hla-b57等位基因可选自可公开获得的b57等位基因。
t细胞受体,是指脊椎动物(例如哺乳动物)的抗原/mhc结合异二聚体蛋白质产物,tcr基因复合物,包括人tcrα、β、γ和δ链。例如,已对人βtcr基因座的完整序列进行了测序,如rowen等人(1996)science272(5269):1755-1762所公开的;已对人αtcr基因座进行了测序和重新测序,例如参见mackelprang等人(2006)humgenet.119(3):255-66;参见ardenimmunogenetics.1995;42(6):455-500中t细胞受体可变基因区段家族的一般分析;所述文献各自针对出版物中提供和引用的序列信息以引用的方式具体地并入本文。
在本发明的方法中用于选择的多聚化t细胞受体是可溶性蛋白质,其包含目标tcr的结合结构域,例如tcrα/β、tcrγ/δ。可溶性蛋白质可以是单链,或者更通常是异二聚体。在一些实施方案中,通过在一种多肽的c末端添加生物素受体肽序列来修饰可溶性tcr。在受体肽处生物素化后,tcr可通过与生物素结合配偶体(例如,抗生物素蛋白、链霉抗生物素蛋白、traptavidin、中性抗生物素蛋白)结合而被多聚化。生物素结合配偶体可包含可检测标记,例如荧光团、质量标记等,或者可与颗粒(例如顺磁性颗粒)结合。结合至tcr的配体的选择可通过流式细胞术、磁性选择等进行,如本领域已知的。
tcr的肽配体是肽抗原,针对其可产生涉及t淋巴细胞抗原特异性应答的免疫应答。此类抗原包括与自身免疫性疾病、感染、诸如麸质等的食物、过敏或组织移植排斥相关的抗原。抗原还包括各种微生物抗原,例如,如在感染、疫苗接种等中发现的,包括但不限于源自病毒、细菌、真菌、原生动物、寄生虫和肿瘤细胞的抗原。肿瘤抗原包括肿瘤特异性抗原,例如免疫球蛋白独特型和t细胞抗原受体;致癌基因,如p21/ras、p53、p210/bcr-abl融合产物;等;发育抗原,如mart-1/melana;mage-1、mage-3;gage家族;端粒酶等;病毒抗原,例如人乳头瘤病毒、爱泼斯坦巴尔病毒(epsteinbarrvirus)等;组织特异性自身抗原,例如酪氨酸酶;gp100;前列腺酸性磷酸酶、前列腺特异性抗原、前列腺特异性膜抗原;甲状腺球蛋白、α-胎蛋白;等;以及自身抗原,例如her-2/neu;癌胚抗原,muc-1等。
在本发明的方法中,产生了多种肽抗原的文库。肽配体的长度是约8至约20个氨基酸,通常约8至约18个氨基酸、约8至约16个氨基酸、约8至约14个氨基酸、约8至约12个氨基酸、约10至约14个氨基酸、约10至约12个氨基酸。应理解,完全随机的文库将代表大量可能的组合。在优选的方法中,多样性在将肽锚定至mhc结合结构域的残基处是有限的,所述残基在本文中称为mhc锚定残基。肽中锚定残基的位置由特定mhc结合结构域决定。如通过结合研究表明,多样性在其他位置,例如在tcr锚处也可能是限制性的。
文库.在本发明的一些实施方案中,提供了多肽的文库或编码此类多肽的核酸的文库,其中多肽结构具有下式:编码p-l1-β-l2-α-l3-t多肽的多核苷酸组合物
其中l1、l2和l3中的每一个是长度为约4至约12个氨基酸的柔性接头,例如包括甘氨酸、丝氨酸、丙氨酸等。
α是i类mhc蛋白或ii类αmhc蛋白的α结构域的可溶性形式;
β是(i)ii类mhc蛋白的β链或(ii)对于i类mhc蛋白,β2微球蛋白的可溶性形式;
t是允许多肽被栓系至细胞表面的结构域,包括但不限于酵母aga2;并且
p是肽配体,通常是如上所述的不同肽配体的文库,其中所述文库中存在至少106、至少107、更通常至少108种不同的肽配体。
可使用组装编码序列的常规方法。为了产生肽配体的多样性,使用本领域中已知的随机化、易错pcr、诱变引物等来产生一组多核苷酸。通常将多核苷酸的文库连接至适合于目标宿主细胞的载体。在各种实施方案中,所述文库以编码p-l1-β-l2-α-l3-t多肽的纯化的多核苷酸组合物的形式提供;以编码可操作地连接至表达载体的p-l1-β-l2-α-l3-t多肽的纯化的多核苷酸组合物的形式提供,其中所述载体可以但不限于适于在酵母细胞中表达;以包含编码p-l1-β-l2-α-l3-t多肽的多核苷酸文库的细胞群体的形式提供,其中所述细胞群体可以是但不限于酵母细胞,并且其中可诱导所述酵母细胞表达多肽文库。
“适合的条件”应具有取决于使用此术语的上下文的含义。即,当与t细胞受体与具有式编码p-l1-β-l2-α-l3-t多肽的多核苷酸组合物的多肽结合使用时,所述术语应意指允许tcr结合至同源肽配体的条件。当此术语与核酸杂交结合使用时,所述术语应意指允许长度为至少15个核苷酸的核酸与具有与其互补的序列的核酸杂交的条件。当与使剂与细胞接触结合使用时,此术语应意指允许能够这样做的剂进入细胞并执行其预期功能的条件。在一个实施方案中,ru本文所用的术语“适合的条件”是指生理条件。
术语“特异性”是指阴性测试结果中为真实阴性测试结果的比例。阴性测试结果包括假阳性和真阴性测试结果。
术语“灵敏度”是指分析方法检测少量分析物的能力。因此,如在此所用,例如,用于检测扩增的dna的更灵敏的方法将能够比不那么灵敏的方法更好地检测少量的此类dna。“灵敏度”是指具有阳性测试结果的预期结果的比例。
如本文所用,术语“可再现性”是指当对相同样品的等分试样重复进行时,分析程序给出相同结果的一般能力。
可在本公开中使用的测序平台包括但不限于:焦磷酸测序、合成测序、单分子测序、第二代测序、纳米孔测序、通过连接测序或通过杂交测序。优选的测序平台是可从illumina(rna-seq)和helicos(digitalgeneexpression或“dge”)商购的那些。“下一代”测序方法包括但不限于由以下商业化的那些:1)454/rochelifesciences,包括但不限于在margulies等人,nature(2005)437:376-380(2005);以及美国专利号7,244,559;7,335,762;7,211,390;7,244,567;7,264,929;7,323,305中描述的方法和设备;2)helicosbiosciencescorporation(cambridge,ma),如美国申请序列号11/167046和美国专利号7501245;7491498;7,276,720;以及美国专利申请公布号us20090061439;us20080087826;us20060286566;us20060024711;us20060024678;us20080213770;和us20080103058中所描述;3)appliedbiosystems(例如solid测序);4)doversystems(例如,polonatorg.007测序);5)illumina,如美国专利号5,750,341;6,306,597;和5,969,119中所描述;以及6)pacificbiosciences,如美国专利号7,462,452;7,476,504;7,405,281;7,170,050;7,462,468;7,476,503;7,315,019;7,302,146;7,313,308;以及美国申请公布号us20090029385;us20090068655;us20090024331;和us20080206764中所描述。所有参考文献均以引用的方式并入本文。此类方法和设备在此通过举例的方式提供并且不意图进行限制。
表达构建体:可将编码本文公开的肽或本文公开的tcr的序列引入表达载体,例如引入待工程化的细胞中,作为疫苗等。可在内源性基因的位点处引入tcr序列,例如使用crispr技术(参见例如,eyquem等人(2017)nature543:113-117;ren等人(2017)protein&cell1-10;ren等人(2017)oncotarget8(10):17002-17011)。
如本文所述,通过将适当的核苷酸变化引入编码序列中来制备氨基酸序列变体。此类变体代表如所述的残基的插入、取代和/或指定的缺失。进行插入、取代和/或指定的缺失的任何组合以得到最终构建体,条件是最终构建体具有如本文所定义的所需生物学活性。
将编码序列的核酸插入载体中以表达和/或整合。许多此类载体是可用的。例如,可通过用编码cas9和sgrna的质粒转染来将crispr/cas9系统直接应用于人细胞。已使用慢病毒和逆转录病毒载体广泛证明了crispr组分的病毒递送。还已经报告了用由非整合病毒,如腺病毒和腺病毒相关病毒(aav)编码的crispr进行基因编辑。较小cas蛋白的最近发现已这种技术与载体(如aav载体)的组合实现并增强,所述载体在它们的安全性概况和效率方面获得了越来越多的成功。
载体组分通常包括但不限于以下中的一者或多者:复制起点、一种或多种标记基因、增强子元件、启动子和转录终止序列。载体包括病毒载体、质粒载体、整合载体等。
序列可作为与异源多肽的融合多肽直接重组产生,所述异源多肽是例如信号序列或在成熟蛋白质或多肽的n端处具有特定裂解位点的其他多肽。一般来说,信号序列可以是载体的组分,或者它可以是插入载体中的编码序列的一部分。优选选择的异源信号序列是被宿主细胞识别并加工(即被信号肽酶裂解)的序列。在哺乳动物细胞表达中,可使用天然信号序列,或其他哺乳动物信号序列可以是适合的,如来自相同或相关物种的分泌多肽的信号序列,以及病毒分泌前导序列,例如单纯疱疹gd信号。
表达载体可含有选择基因,也称为选择性标记。这种基因编码在选择性培养基中生长的转化的宿主细胞的存活或生长所必需的蛋白质。未用含有选择基因的载体转化的宿主细胞在培养基中将不会存活。典型选择基因编码以下蛋白质:(a)赋予对抗生素或其他毒素(例如,氨苄青霉素、新霉素、甲氨蝶呤或四环素)的抗性,(b)弥补营养缺陷不足,或(c)供应不可自复合培养基获得的关键养分。
表达载体将含有被宿主生物体识别并与编码序列可操作连接的启动子。启动子是位于结构基因的起始密码子上游(5')(通常在约100至1000bp内)的非翻译序列,其控制它们所可操作地连接的特定核酸序列的转录和翻译。此类启动子通常分为两类,诱导型和组成型。诱导型启动子是响应于培养条件的某一变化(例如存在或不存在某一养分或温度变化)来引发在它们的控制下自dna的转录水平增加的启动子。许多由多种潜在宿主细胞识别的启动子是熟知的。
可例如通过从以下获得的启动子来控制从哺乳动物宿主细胞中的载体转录:病毒的基因组,所述病毒如多瘤病毒、禽痘病毒、腺病毒(如腺病毒2)、牛乳头瘤病毒、禽肉瘤病毒、巨细胞病毒、逆转录病毒(如鼠干细胞病毒)、丙型肝炎病毒且最优选猿猴病毒40(sv40);异源哺乳动物启动子,例如肌动蛋白启动子、pgk(磷酸甘油酸激酶)或免疫球蛋白启动子;或热激启动子,条件是此类启动子与宿主细胞系统相容。sv40病毒的早期和晚期启动子可方便地以sv40限制性片段的形式获得,所述片段也含有sv40病毒复制起点。
通过高等真核生物转录通常通过将增强子序列插入载体中来增强。增强子是dna的顺式作用元件,长度通常约10至300bp,其作用于启动子以提高其转录。增强子相对不依赖于取向和位置,已在转录单元的5'和3'、在内含子内以及在编码序列自身内发现。现在已知许多来自哺乳动物基因(球蛋白、弹性蛋白酶、白蛋白、α-胎蛋白和胰岛素)的增强子序列。然而,通常,将使用来自真核病毒的增强子。实例包括复制起点的后侧上的sv40增强子、巨细胞病毒早期启动子增强子、复制起点的后侧上的多瘤增强子和腺病毒增强子。增强子可剪接至表达载体中编码序列的5'或3'位置上,但优选位于启动子的5'方向位点上。
用于真核宿主细胞中的表达载体还将含有终止转录和稳定mrna所必需的序列。此类序列通常可从真核或病毒dna或cdna的5’和偶尔3'非翻译区中获得。含有一种或多种以上所列组分的适合载体的构建采用标准技术。
用于在本文中的载体中克隆或表达dna的适合的宿主细胞是上述原核生物、酵母或其他真核生物细胞。有用的哺乳动物宿主细胞系的实例是小鼠l细胞(l-m[tk-],atcc#crl-2648)、由sv40转化的猴肾cv1株系(cos-7,atcccrl1651);人胚肾株系(293细胞或被亚克隆来在悬浮培养物中生长的293细胞);幼小仓鼠肾细胞(bhk,atccccl10);中国仓鼠卵巢细胞/-dhfr(cho);小鼠塞尔托利细胞(sertolicell)(tm4);猴肾细胞(cv1atccccl70);非洲绿猴肾细胞(vero-76,atcccrl-1587);人宫颈癌细胞(hela,atccccl2);犬肾细胞(mdck,atccccl34);水牛大鼠肝细胞(brl3a,atcccrl1442);人肺细胞(w138,atccccl75);人肝细胞(hepg2,hb8065);小鼠乳腺肿瘤(mmt060562,atccccl51);tri细胞;mrc5细胞;fs4细胞;以及人肝细胞瘤株系(hepg2)。
可用上述表达载体转染宿主细胞,包括工程化的t细胞等。细胞可在被调节为适合诱导启动子、选择转化子或扩增编码所需序列的基因的常规营养培养基中培养。哺乳动物宿主细胞可在多种培养基中培养。诸如ham'sf10(sigma)、最低必需培养基((mem),(sigma)、rpmi-1640(sigma)和杜氏改良的伊格尔氏培养基((dmem),sigma)的可商购的培养基适用于培养宿主细胞。必要时任何这些培养基都可补充有激素和/或其他生长因子(如胰岛素、转铁蛋白或表皮生长因子)、盐(如氯化钠、钙、镁和磷酸盐)、缓冲剂(如hepes)、核苷(如腺苷和胸苷)、抗生素、微量元素以及葡萄糖或等效能量来源。还可包括本领域技术人员所已知的适当浓度的任何其他必需补充剂。诸如温度、ph等的培养条件是先前与选择用于表达的宿主细胞一起使用的那些条件,且将为普通熟练技术人员显而易知。
核酸在放置成与另一核酸序列呈功能性关系时是“可操作地连接”的。举例来说,信号序列的dna在其表达为表示多肽的分泌的前蛋白的情况下可操作地连接至多肽的dna;启动子或增强子在其影响编码序列的转录的情况下可操作地连接至所述编码序列;并且核糖体结合位点在其位置促进翻译的情况下可操作地连接至编码序列。一般来说,“可操作地连接”意指所连接的dna序列是连续的,并且在分泌前导序列的情况下,是连续的并且处于阅读框中。然而,增强子不必是连续的。
在本公开的多肽或核酸为“基本上纯”的情况下,它们可以是目标生物分子的至少约60重量%(干重)。例如,所述组合物可以是目标生物分子的至少约75重量%、约80重量%、约85重量%、约90重量%、约95重量%或约99重量%。可通过任何适当的方法测量纯度,例如,柱色谱法、聚丙烯酰胺凝胶电泳或hplc分析。
在本发明的另一个实施方案中,提供了一种含有适用于治疗以上所述的疾患的材料的制品。所述制品包括容器和标签。适合的容器包括例如瓶、小瓶、注射器和试管。所述容器可由多种材料诸如玻璃或塑料形成。容器容纳对治疗所述疾患有效的组合物并且可具有无菌入口(sterileaccessport)(例如,容器可为具有可经皮下注射用注射针刺破的塞子的静脉输液袋或小瓶)。组合物中的活性剂可以是适于将序列引入靶细胞以表达的载体。容器上或与容器相关的标签指示组合物用于治疗所选的疾患。制品可提供有另外的容器,所述容器可容纳例如药学上可接受的缓冲液,如磷酸盐缓冲盐水、林格氏溶液或右旋糖溶液。制品还可包括从商业和用户角度期望的其他材料,包括其他缓冲剂、稀释剂、过滤器、针头、注射器以及带有使用说明的包装插页。
如本文中关于多肽或dna序列使用的术语“序列同一性”是指两个分子之间的亚基序列同一性。当两个分子中的亚基位置均被相同的单体亚基(例如,相同的氨基酸残基或核苷酸)占据时,则所述分子在所述位置处是相同的。两个氨基酸序列或两个核苷酸序列之间的相似性是相同位置的数量的直接函数。通常,将序列进行比对,以便获得最高位匹配。如果需要,可使用公开的技术和可广泛获得的计算机程序,如gcs程序包(devereux等人,nucleicacidsres.12:387,1984)、blastp、blastn、fasta(atschul等人,j.molecularbiol.215:403,1990)来计算同一性。
术语“多肽”、“蛋白质”或“肽”是指氨基酸残基的任何链,而不管其长度或翻译后修饰(例如糖基化或磷酸化)。
本文中的“蛋白质变体”或“变体蛋白质”或“变体多肽”是指由于至少一种氨基酸修饰而不同于野生型蛋白质的蛋白质。亲本多肽可以是天然存在的或野生型(wt)多肽,或者可以是wt多肽的经修饰型式。变体多肽可指多肽本身,包含所述多肽的组合物或编码所述多肽的氨基序列。优选地,与亲本多肽相比,变体多肽具有至少一种氨基酸修饰,例如与亲本相比约1至约10种氨基酸修饰且优选约1至约5种氨基酸修饰。
本文公开的肽可与另外的氨基酸残基侧接,只要所述肽保留其tcr诱导性即可。此类肽可少于约40个氨基酸,例如少于约20个氨基酸,例如少于约15个氨基酸。位于由选自seqidno:3-5、7-9、12、15-19、22、24、27-30、37、67和74的组的氨基酸序列组成的肽侧翼的氨基酸序列没有限制,并且可由任何种类的氨基酸组成,只要它不抑制tcr识别即可。氨基酸序列可通过取代一个或多个氨基酸来进行修饰。本领域技术人员将认识到,改变单个氨基酸或少量氨基酸的对氨基酸序列的单独添加或取代导致原始氨基酸侧链的性质的保守;因此,其被称为“保守性取代”或“保守性修饰”,其中蛋白质的改变产生具有相似功能的蛋白质。
除了肽的上述序列修饰以外,所述肽可进一步与其他物质连接,只要它们保留tcr结合活性即可。可用的物质包括:肽、脂质、糖和糖链、乙酰基、天然和合成聚合物等。肽可含有修饰,如糖基化、侧链氧化或磷酸化;只要所述修饰不破坏如本文所述的肽的生物学活性。可进行这些类型的修饰以赋予另外的功能(例如靶向功能和递送功能)或使多肽稳定。
例如,为了增加多肽的体内稳定性,本领域已知引入特别有用的各种d-氨基酸、氨基酸模拟物或非天然氨基酸;这种概念也可适用于本发明的多肽。多肽的稳定性可以多种方式测定。例如,肽酶和各种生物介质,如人血浆和血清已被用于测试稳定性(参见例如,verhoef等人,eurjdrugmetabpharmacokin11:291-302,1986)。iii.肽的制备
可使用众所周知的技术来制备本文公开的肽。例如,可通过重组dna技术或化学合成来合成制备肽。本文公开的肽可单独合成或作为包含两种或更多种肽(例如,两种或更多种肽或一种肽和一种非肽)的更长的多肽合成。所述肽可被分离,即被纯化以基本上不含其他天然存在的宿主细胞蛋白质及其片段,例如至少约70%、80%或90%纯化的。
如本文所用的“亲本多肽”、“亲本蛋白质”、“前体多肽”、或“前体蛋白质”意指随后要被修饰来产生变体的未被修饰的多肽。亲本多肽可以是野生型(或天然)多肽,或者是野生型多肽的变体或工程化型式。亲本多肽可指多肽本身、包含亲本多肽的组合物、或编码其的氨基酸序列。
术语“接受者”、“个体”、“受试者”、“宿主”和“患者”在本文中可互换使用并且是指需要诊断、治疗或疗法的任何哺乳动物受试者,特别是人。“哺乳动物”出于治疗目的是指分类为哺乳动物的任何动物,包括人、家养动物和农场动物,以及动物园、运动或宠物动物,如狗、马、猫、牛、绵羊、山羊、猪等。优选地,哺乳动物是人。
如本文所用,“治疗有效量”是指足以治疗或控制疾病或病症的治疗剂(例如初免t细胞、肽或多核苷酸疫苗等的输注)的量。治疗有效量可指足以延迟或最小化疾病发作(例如延迟或最小化癌症扩散)的治疗剂的量,或可有效减少或增加来自目标受体的信号传导的量。治疗有效量还可以是指在疾病的治疗或控制中提供治疗益处的治疗剂的量。此外,关于本发明的治疗剂的治疗有效量是指在疾病的治疗或控制中提供治疗益处的单独或与其他疗法组合的治疗剂的量。
如本文所用,术语“给药方案”是指通常相隔时间段来单独向受试者施用的一组单位剂量(通常一个以上)。在一些实施方案中,给定的治疗剂具有可涉及一个或多个剂量的推荐给药方案,。在一些实施方案中,给药方案包括多个剂量,所述多个剂量中的每一个彼此按相同长度的时间段分开;在一些实施方案中,给药方案包括多个剂量和使单独剂量分开的至少两个不同的时间段。在一些实施方案中,给药方案内的所有剂量都具有相同单位给药量。在一些实施方案中,给药方案内的不同剂量具有不同量。在一些实施方案中,给药方案包括以第一给药量施用的第一剂量,继之以一个或多个以不同于所述第一给药量的第二给药量施用的其他剂量。在一些实施方案中,给药方案包括第一给药量的第一剂量,之后是一个或多个与所述第一给药量相同的第二给药量的其他剂量。在一些实施方案中,当在整个相关群体中施用时,给药方案与所需或有益的结果相关(即,是治疗性给药方案)。
如本文所用,术语“癌症”(或“癌性”)或“肿瘤”用于指具有自主生长能力(例如,以迅速增殖的细胞生长为特征的异常状态或状况)的细胞。高度增殖的和赘生性细胞的疾病状态可被归类为病理学的(即,表征或构成疾病状态),或者它们可被归类为非病理学的(例如,偏离正常的但不与疾病状态相关联)。所述术语意指包括所有类型的癌性生长或致癌性过程、转移性组织或恶性转化的细胞、组织或器官,而不考虑组织病理学类型或侵袭的阶段。病理学高度增殖的细胞发生在由恶性肿瘤生长表征的疾病状态中。非病理学高度增殖的细胞的实例包括与伤口修复相关联的细胞增殖。术语“癌症”或“肿瘤”还用于指各种器官系统的恶性肿瘤,包括影响肺、乳腺、甲状腺、淋巴腺和淋巴组织、胃肠器官和泌尿生殖道的那些恶性肿瘤,以及通常被认为包括诸如大多数结肠癌、肾细胞癌、前列腺癌和/或睾丸肿瘤、肺非小细胞癌、小肠癌和食道癌的恶性肿瘤的腺癌。
术语“癌”是本领域公认的并且是指上皮或内分泌组织的恶性肿瘤,包括呼吸系统癌、胃肠系统癌、生殖泌尿系统癌、睾丸癌、乳腺癌、前列腺癌、内分泌系统癌和黑素瘤。“腺癌”是指源自腺组织的癌或其中肿瘤细胞形成可辨认的腺结构的癌。
示例性癌症类型包括但不限于aml、all、cml、肾上腺皮质癌、肛门癌、再生障碍性贫血、胆管癌、膀胱癌、骨癌、骨转移、脑癌、中枢神经系统(cns)癌症、周围性神经系统(pns)癌症、乳腺癌、子宫颈癌、儿童非霍奇金淋巴瘤、结肠和直肠癌、子宫内膜癌、食道癌、尤文氏肿瘤家族(例如,尤文氏肉瘤)、眼癌、胆囊癌、胃肠类癌、胃肠间质瘤、妊娠滋养细胞疾病、霍奇金淋巴瘤、卡波济氏肉瘤、肾癌、喉癌和下咽癌、肝癌、肺癌、肺类癌瘤、非霍奇金淋巴瘤、男性乳腺癌、恶性间皮瘤、多发性骨髓瘤,多发性骨髓瘤、骨髓增生异常综合征、骨髓增生性疾病、鼻腔和鼻窦癌、鼻咽癌、成神经细胞瘤、口腔和口咽癌、骨肉瘤、卵巢癌、胰腺癌、阴茎癌、垂体瘤、前列腺癌、成视网膜细胞瘤、横纹肌肉瘤、唾液腺癌、肉瘤、黑素瘤皮肤癌、非黑素瘤皮肤癌、胃癌、睾丸癌、胸腺癌、甲状腺癌、子宫癌(例如子宫肉瘤)、移行细胞癌、阴道癌、外阴癌、间皮瘤、鳞状细胞癌或表皮样癌、支气管腺瘤、绒毛膜癌、头颈癌、畸胎癌或瓦尔登斯特伦巨球蛋白血症。
方法和组合物
提供了用于在给定mhc背景下准确鉴定由t细胞受体识别的一组肽的组合物和方法;并且提供使用多重方法以同时筛选2、3、4、5个或更多个文库而从这种筛选中获得的抗原。由此鉴定的肽配体(抗原)的长度是约8至约20个氨基酸,通常约8至约18个氨基酸、约8至约16个氨基酸、约8至约14个氨基酸、约8至约12个氨基酸、约10至约14个氨基酸、约10至约12个氨基酸,并且可包括本文中作为seqidno:1-257提供的任何肽。
通过将多聚化tcr与表达所述文库的宿主细胞群体组合在一起来进行与目标tcr结合的肽的选择。用于选择的多聚化t细胞受体是包含目标tcr的结合结构域(例如α/β、tcrγ/δ)的可溶性蛋白,并且可通过任何方便的方法来合成。tcr可以是单链或异二聚体。在一些实施方案中,通过在一种多肽的c末端添加生物素受体肽序列来修饰可溶性tcr。在受体肽处生物素化后,tcr可通过与生物素结合配偶体(例如,抗生物素蛋白、链霉抗生物素蛋白、traptavidin、中性抗生物素蛋白)结合而被多聚化。生物素结合配偶体可包含可检测标记,例如荧光团、质量标记等,或者可与颗粒(例如顺磁性颗粒)结合。结合至tcr的配体的选择可通过流式细胞术、磁性选择等进行,如本领域已知的。
进行多轮选择,直到所选择的群体具有高于背景的信号,进行通常至少三轮且更通常至少四轮选择。在一些实施方案中,使用与磁性试剂(如超顺磁性微粒)偶联(其可被称为“磁化”)的tcr进行初始选择轮次,例如直到存在高于背景的信号。以引用的方式并入本文的molday(美国专利号4,452,773)描述了磁性铁-葡聚糖微粒的制备,并且提供了描述制备适合附着于生物材料的颗粒的各种方式的概述。在美国专利号5,385,707中发现了用于高梯度磁分离(hgms)方法中的磁性颗粒的聚合物涂层的描述。用于制备超顺磁性颗粒的方法在美国专利号4,770,183中进行了描述。微粒的直径将通常小于约100nm,并且直径通常将大于约10nm。用于偶联的确切方法对于本发明的实践并不重要,并且本领域中已知许多替代方案。直接偶联将tcr附着至颗粒。间接偶联可通过若干方法来实现。可将tcr偶联至高亲和力结合系统的一个成员,例如生物素,以及附着至另一成员的颗粒,例如抗生物素蛋白。或者,也可使用识别tcr的物种特异性表位的第二级抗体,例如抗小鼠ig、抗大鼠ig等。间接偶联方法允许使用单个磁偶联实体,例如抗体、抗生物素蛋白等,以及各种分离抗体。
或者,并且在用于最后选择轮次的优选实施方案中,将tcr多聚化为具有可检测标记(例如用于流式细胞术、质谱法等)的试剂。例如,facs分选可用于增加具有与tcr结合的肽配体的细胞的浓度。技术包括荧光活化细胞分类术,其可具有不同程度的复杂性,如多个颜色通道、低角度和钝角光散射检测通道、阻抗通道等。
在最后一轮选择后,从所选择的宿主细胞分离多核苷酸,并且通常通过高通量测序确定所选择的肽配体的序列。本文显示,选择过程使得确定在特定hla背景下被tcr结合的一组肽。这些配体在t细胞活化中的生物活性已得到验证。所选择的配体的集合提供关于与t细胞受体结合所需的氨基酸位置的限制的信息。通常选择多种肽配体,例如达10个、达100个、达500个、达1000个或更多个不同的肽序列。
来自肽配体的这一选定集合的序列数据提供有关肽配体的每个位置处的氨基酸的限制的信息。这可以图形方式显示。所述限制在接触tcr的残基处尤其相关。输入有关肽位置处的氨基酸的限制的数据,以设计用于分析公共数据库的搜索算法。搜索的结果提供了一组肽,所述肽符合在mhc背景下与tcr结合的标准。搜索算法通常体现为可由计算机执行并通过加载到计算机中的软件组件执行的指令程序。
通过这些方法鉴定的肽和t细胞受体可用于疫苗方法、筛选方法中以对患者t细胞群体进行分类,以在体外引发t细胞等。
在一些实施方案中,所述组合物包含在具有至少一种hla等位基因(即hla-a2)的受试者中引发对癌细胞(例如结肠直肠癌细胞)的免疫应答的一种或多种肽。在另一方面,本发明提供了包含编码本文公开的肽的多核苷酸的组合物。在一些实施方案中,所述组合物包含编码本文公开的多种肽的多种(即,两种或更多种)多核苷酸。在一些实施方案中,所述组合物包含编码本文公开的多种肽的多核苷酸。
在相关方面,提供了通过向个体施用肽或编码本文公开的肽的多核苷酸来治疗癌症(例如,减少肿瘤细胞生长、促进肿瘤细胞死亡)的方法。在相关方面,提供了已经用本文公开的肽引发的分离的引发的t细胞。在另一方面,提供了一种抗原呈递细胞,其包含在hla抗原与本文公开的肽之间形成的复合物。在一些实施方案中,分离所述抗原呈递细胞。
术语“疫苗”(也称为免疫原性组合物)是指具有在接种到动物中后诱导抗肿瘤(或抗病原体)免疫的功能的物质。
有待通过药剂治疗的癌症不受限制,并且包括其中在受试者中表达本文所鉴定的肽的相应蛋白质的所有种类的癌症。示例性癌症包括癌症,例如结肠直肠癌。
如果需要,由肽或编码肽的多核苷酸组成的药剂可任选地包含其他治疗性物质作为活性成分,只要所述物质不抑制目标肽的tcr刺激作用即可。例如,制剂可包括抗炎剂、止痛药、化学治疗剂等。除了在药物本身中包括其他治疗性物质外,还可与一种或多种其他药理剂顺序或同时施用药物。药物和药理剂的量取决于例如所使用的药理剂的类型、所治疗的疾病以及施用的时间表和途径。
如果需要,可作为已通过常规配制方法配制的药剂直接施用肽。在此类情况下,除了肽之外,可适当地包括通常用于药物的载体、赋形剂等,而没有特别限制。此类载体的实例是无菌水、生理盐水、磷酸盐缓冲液、培养液等。此外,必要时所述药剂可含有稳定剂、悬浮剂、防腐剂、表面活性剂等。所述药剂可用于治疗和/或预防癌症。
所述肽可组合制备,其包含两种或更多种本文公开的肽,以在体内刺激t细胞。肽可呈混合形式或者可使用标准技术彼此缀合。例如,肽可表达为单一多肽序列。组合中的肽可相同或不同。通过施用所述肽,所述肽以高密度呈递在抗原呈递细胞的hla抗原上,然后刺激对在展示的肽与hla抗原之间形成的复合物特异性反应的t细胞。或者,将肽固定在其细胞表面上的抗原呈递细胞是通过从受试者除去树突细胞而获得,通过肽刺激所述树突细胞,然后通过将肽负载的树突细胞重新施用于受试者来刺激内源性t细胞,并且因此,可增加对靶细胞的攻击性。
包含本文所述的肽作为活性成分的药剂任选地可包含佐剂,以使得将有效地建立细胞免疫,或者它们可与其他活性成分一起施用,并且它们可通过配制成颗粒来施用。佐剂是指当与具有免疫活性的蛋白质一起(或相继)施用时增强针对所述蛋白质的免疫应答的化合物。可应用的佐剂包括文献中描述的那些。示例性佐剂包括磷酸铝、氢氧化铝、明矾、霍乱毒素、沙门氏菌毒素等,但不限于此。
此外,可方便地使用脂质体制剂、其中肽结合至数mcm直径的珠粒的颗粒制剂、以及其中脂质结合至肽的制剂。或者,提供称为外来体的细胞内囊泡,其在它们的表面上呈递在肽与hla抗原之间形成的复合物。与肽类似,外来体可作为疫苗接种。
在一些实施方案中,本文公开的药剂包含引发t淋巴细胞的组分。脂质已被鉴定为能够在体内引发针对病毒抗原的ctl的剂。例如,棕榈酸残基可连接至赖氨酸残基的ε-和α-氨基,且然后连接至本文公开的肽。脂质化的肽然后可直接以胶束或颗粒形式施用,掺入脂质体中,或在佐剂中乳化。作为ctl应答的脂质引发的另一个实例,大肠杆菌脂蛋白,如三棕榈酰基-s-甘油基半胱氨酰丝氨酰-丝氨酸(p3css)当与适当的肽共价连接时可用于引发ctl(参见例如,deres等人,nature342:561,1989)。
施用方法可以是口服、皮内、皮下、静脉内注射等,并且全身性施用或局部施用至靶向部位附近可适用。施用可通过单次施用来进行或者通过多次施用来加强。肽的剂量可根据待治疗的疾病、患者的年龄、体重、施用方法等适当地调节,并且通常是0.001mg至1000mg,例如0.001mg至1000mg、例如0.1mg至10mg,并且可每隔几天至每隔几个月施用一次。本领域技术人员可适当地选择适合的剂量。
本文公开的药剂还可包含以可表达形式编码本文公开的肽的核酸。在本文中,短语“以可表达的形式”是指当引入细胞中时,多核苷酸将在体内表达为具有刺激抗肿瘤免疫的多肽。在一个实施方案中,目标多核苷酸的核酸序列包括在靶细胞中表达多核苷酸所必需的调控元件。可装备多核苷酸以稳定地插入靶细胞的基因组中(关于同源重组盒载体的描述,参见例如,thomaskr&capecchimr,cell51:503-12,1987)。参见例如,wolff等人,science247:1465-8,1990;美国专利号5,580,859;5,589,466;5,804,566;5,739,118;5,736,524;5,679,647;以及wo98/04720。基于dna的递送技术的实例包括“裸dna”、促进的(布比卡因、聚合物、肽介导的)递送、阳离子脂质复合物以及颗粒介导的(“基因枪”)或压力介导的递送(参见例如,美国专利号5,922,687)。
本文公开的肽也可通过病毒或细菌载体表达。表达载体的实例包括减毒的病毒宿主,如痘苗或禽痘。这种方法涉及痘苗病毒的使用,例如,作为表达编码肽的核苷酸序列的载体。在引入宿主后,重组痘苗病毒表达免疫原性肽,并且由此引发免疫应答。在免疫方案中有用的痘苗载体和方法在例如美国专利号4,722,848中进行了描述。另一种载体是bcg(卡介苗)。bcg载体描述于stover等人,nature351:456-60,1991中。多种其他可用于治疗性施用或免疫的载体,例如腺和腺相关病毒载体、逆转录病毒载体、鼠伤寒沙门氏菌载体、去毒的炭疽毒素载体等将是显而易见的。参见例如,shata等人,molmedtoday6:66-71,2000;shedlock等人jleukocbiol68:793-806,2000;hipp等人,invivo14:571-85,2000。
施用方法可以是口服、皮内、皮下、静脉内注射等,并且全身性施用或局部施用至靶向部位附近可适用。施用可通过单次施用来进行或者通过多次施用来加强。在用编码肽的多核苷酸转化的适合载体或的细胞中的多核苷酸的剂量可根据所治疗的疾病、患者的年龄、体重、施用方法等适当地调节,并且通常是0.001mg至1000mg,例如0.001mg至100mg、例如0.1mg至10mg,并且可每隔几天至每隔几个月施用一次。本领域技术人员可适当地选择适合的剂量。
还提供了抗原呈递细胞(apc),所述抗原呈递细胞呈递在hla抗原与其表面上的肽之间形成的复合物。apc是通过接触肽或编码肽的核苷酸获得的,并且可从作为治疗和/或预防的靶标的受试者中备,并且可作为疫苗单独或与包括肽、外来体或细胞毒性t细胞在内的其他药物组合施用。apc不限于任何类型的细胞,并且包括树突细胞(dc)、朗格汉斯细胞、巨噬细胞、b细胞和活化的t细胞,已知它们全部在其细胞表面上呈递蛋白质性抗原以便被淋巴细胞识别。由于dc是apc中具有最强ctl诱导作用的代表性apc,因此dc可用作apc。
例如,可通过从外周血单核细胞诱导树突细胞且然后使其在体外、离体或体内与肽接触(刺激)来获得apc。当将肽施用至受试者时,具有固定在其上的肽的apc在所述受试者体内受到刺激,“诱导apc”包括使细胞与肽或编码肽的核苷酸接触(刺激)以呈递在hla抗原与细胞表面上的肽之间形成的复合物。或者,在将肽固定于apc之后,可将apc作为疫苗施用至受试者。例如,离体施用可包括以下步骤:a:从受试者收集apc;和b:将步骤a的apc与肽接触。可将通过步骤b获得的apc作为疫苗施用至受试者。
此类apc可通过以下方法来制备,所述方法包括以下步骤:将包含编码肽的多核苷酸的基因体外转移至apc。引入的基因可呈dna或rna的形式。对于引入方法,没有特别限制,可使用在此领域中常规进行的各种方法,如脂质转染、电穿孔和磷酸钙方法。
细胞可被工程化以表达在此提供的tcr,或对本文提供的肽抗原作出应答。许多不同的细胞类型适用于工程化,特别是t细胞或nk细胞。在一些实施方案中,用于工程化的细胞是自体的。在一些实施方案中,所述细胞是同种异体的。
针对本文公开的任何肽刺激的t细胞可用作类似于所述肽的疫苗。因此,本发明提供了被任何本发明肽刺激的分离的t细胞。可通过(1)向受试者施用或(2)使受试者来源的apc和cd8阳性细胞或外周血单核白细胞在体外与肽接触来获得此类t细胞。已通过从呈递肽的apc刺激而刺激的t细胞可源自作为治疗和/或预防的靶标的受试者,并且可出于调控效应的目的单独施用或与其他药物(包括肽或外来体)组合施用。所获得的t细胞特异性地作用于呈递肽(例如用于引发的相同肽)的靶细胞。靶细胞可以是内源性表达的细胞,或者用基因转染的细胞,以及由于被这些肽刺激而在细胞表面呈递所述肽的细胞也可成为攻击的靶标。
在一些实施方案中,工程化的细胞是t细胞。术语“t细胞”是指可以cd3和/或t细胞抗原受体的表达为特征的哺乳动物免疫效应细胞,所述细胞可被工程化以表达本文提供的tcr或被刺激以对本文提供的肽作出应答。在一些实施方案中,t细胞是选自天然cd8+t细胞、细胞毒性cd8+t细胞、天然cd4+t细胞、辅助t细胞(例如th1、th2、th9、th11、th22、tfh);调控性t细胞,例如tr1、天然treg、诱导型treg;记忆t细胞,例如中心记忆t细胞、t干细胞记忆细胞(tscm)、效应记忆t细胞、nkt细胞、γδt细胞。在一些实施方案中,工程化的细胞包含免疫细胞的复杂混合物,例如从需要治疗的个体中分离的肿瘤浸润性淋巴细胞(til)。参见例如,yang和rosenberg(2016)advimmunol.130:279-94,“adoptivetcelltherapyforcancer;feldman等人(2015)seminoncol.42(4):626-39“adoptivecelltherapy-tumor-infiltratinglymphocytes,t-cellreceptors,andchimericantigenreceptors”;clinicaltrialnct01174121,“immunotherapyusingtumorinfiltratinglymphocytesforpatientswithmetastaticcancer”;tran等人(2014)science344(6184)641-645,“cancerimmunotherapybasedonmutation-specificcd4+tcellsinapatientwithepithelialcancer”。在一些实施方案中,t细胞在体外与肽接触,即其中然后将t细胞转移至接受者。
出于本发明的目的,效应细胞可包括对靶细胞具有溶细胞活性的自体或同种异体免疫细胞,包括但不限于肿瘤细胞。效应细胞可通过在体外工程化外周血淋巴细胞(pbl)、然后与可增加活化的细胞因子和/或抗原组合一起培养而获得。在培养之前、施用之前或两者,将细胞任选地与不需要的细胞分离。免疫效应细胞对靶细胞的细胞介导的细胞溶解据信由穿透结合的靶细胞的细胞膜的细胞质颗粒的局部定向胞吐作用介导。
与肿瘤细胞反应的细胞毒性t淋巴细胞(ctl)是用于过继性免疫疗法的特异性效应细胞,并且对于通过用本文公开的肽引发进行工程化或工程化以表达本文公开的tcr而引起关注。ctl的诱导和扩增是抗原特异性和mhc限制的。
从受试者收集的t细胞可通过富集所需细胞的技术从细胞的混合物分离,或者可进行工程化和培养而无需分离。适当的溶液可用于分散或悬浮。此类溶液通常将是方便地用胎牛血清或其他天然存在的因子补充、连同低浓度(通常5-25mm)的可接受的缓冲液的平衡盐溶液,例如生理盐水、pbs、汉克氏平衡盐溶液等。合宜的缓冲液包括hepes、磷酸盐缓冲液、乳酸盐缓冲液等。
用于亲和分离的技术可包括使用抗体包被的磁珠进行磁分离、亲和色谱法、与单克隆抗体连接或与单克隆抗体(例如补体和细胞毒素)结合使用的细胞毒性剂,以及用附着至固体基质(例如板)的抗体“淘选”或其他方便的技术。提供精确分离的技术包括荧光活化细胞分类术,其可具有不同程度的复杂性,如多个颜色通道、低角度和钝角光散射检测通道、阻抗通道等。可通过采用与死细胞相关的染料(例如碘化丙啶)来针对死细胞选择细胞。可使用对所选细胞的活力不会过分有害的任何技术。亲和试剂可以是上述细胞表面分子的特异性受体或配体。除抗体试剂外,还可使用肽-mhc抗原和t细胞受体对;肽配体和受体;效应子和受体分子等。
分离的细胞可收集于维持细胞活力的任何适当培养基中,通常在收集管的底部具有血清缓冲垫。各种培养基可商购获得并且可根据细胞的性质加以使用,包括dmem、hbss、dpbs、rpmi、伊斯科夫培养基等,经常补充有胎牛血清(fcs)。
所收集和任选富集的细胞群体可立即用于遗传修饰,或者可在液氮温度下冷冻并储存,解冻并能够重复使用。细胞通常将储存在10%dmso、50%fcs、40%rpmi1640培养基中。
工程化的细胞可通过任何方便的施用途径(通常血管内)在任何生理学上可接受的介质中输注给受试者,尽管它们也可通过其他途径引入,其中细胞可找到适当的生长位点。通常,将施用至少1×106个细胞/kg,至少1×107个细胞/kg、至少1×108个细胞/kg、至少1×109个细胞/kg、至少1×1010个细胞/kg或更通常受收集过程中获得的t细胞的数量限制。
肽和t细胞受体序列也可用于患者样品的筛选测定中,其中分析含有来自个体的样品,例如血液样品、肿瘤活检样品、淋巴结样品、骨髓样品等的t细胞的(i)包含本文鉴定的tcr的t细胞的存在,和/或(ii)对本文所述的肽有应答的t细胞的存在。可根据任何方便的方法来确定t细胞的存在,例如通过测量响应于hla复合物中的或通过apc呈递的肽的存在的增殖等来确定刺激。特定tcr的存在可通过mrna的测序、基因组dna的测序等来确定。对肽有反应或具有目标tcr的t细胞的存在允许患者被分配到可通过疫苗接种、apc转移等治疗的组中。
本文还提供了有形地体现在机器可读介质中的软件产品,所述软件产品包括可操作以使一个或多个数据处理设备执行包括以下的操作的指令:从通过本发明的筛选方法获得的所选肽配体的位置频率生成nx20矩阵,其中n是肽配体文库中氨基酸位置的数目。设定氨基酸频率的截断值,例如小于0.1、小于0.05、小于0.01,并且低于截断值的频率的频率被设置为零。序列数据库,例如一组人类多肽序列;一组病原体多肽序列,一组微生物多肽序列;一组过敏原多肽序列;等通过使用n位置滑动窗口比对并对来自取代矩阵的位置氨基酸频率的乘积计分的算法进行搜索。排除了含有其中频率低于截断值的至少一个氨基酸的比对区段。搜索结果可作为数据文件输出到计算机可读介质中。
可在多种介质中提供肽序列结果和数据库搜索结果,以利于它们的使用。“介质”是指含有本发明的表达谱系信息的制造品。本发明的数据库可被记录在计算机可读介质上,例如可直接通过计算机读取和访问的任何介质。此类介质包括,但不限于:磁存储介质,如软盘、硬盘存储介质和磁带;光学存储介质如cd-rom;电存储介质如ram和rom;以及这些分类如磁存储介质/光学存储介质的混杂体。本领域技术人员可容易地理解,可如何使用任何当前已知的计算机可读介质来创建包括对当前数据库信息的记录的制造品。“记录的”是指使用本领域中已知的任何此类方法在计算机可读介质上存储信息的过程。基于用于访问所存储的信息的方法,可选择任何便利的数据存储结构。各种数据处理器程序和格式可用于存储,例如文字处理文本文件、数据库格式等。
如本文所用,“基于计算机的系统”是指用于分析本发明的信息的硬件装置、软件装置、和数据存储装置。本发明的基于计算机的系统的最小硬件包括中央处理器(cpu)、输入装置、输出装置、以及数据存储装置。本领域的技术人员可易于认识到,当前可用的基于计算机的系统中的任一种适于在本发明中使用。数据存储装置可包括任何制造品,其包括如上所述的本发明的信息的录制品或可访问此制造品的存储器访问装置。
在本发明的基于计算机的系统中,用于输入和输出装置的各种结构格式可用于输入和输出信息。这种呈现为熟练的技术人员提供相似性的评级,并鉴定测试表达谱系中包含的相似度。
搜索算法和序列分析可以硬件或软件或两者的组合来实现。在本发明的一个实施方案中,提供了机器可读存储介质,所述介质包括用机器可读数据编码的数据存储材料,当使用被编程有用于使用所述数据的指令的机器时,所述机器可读数据能够显示本发明的任何数据集和数据比较。在一些实施方案中,本发明在可编程的计算机上执行的计算机程序中实现,所述可编程的计算机包括处理器、数据存储系统(包括易失性和非易失性存储器和/或存储元件)、至少一个输入装置以及至少一个输出装置。将程序代码应用于输入数据以执行上述功能并产生输出信息。将所述输出信息以已知的方式应用于一个或多个输出装置。计算机可以是例如个人计算机、微型计算机或常规设计的工作站。
每个程序可以高水平的程序性或面向对象的编程语言来实现以与计算机系统进行通信。然而,如果需要,所述程序可以汇编语言或机器语言来实现。在任何情况下,所述语言可以是编译或解释语言。每一个这样的计算机程序可被存储在通过一般或特殊目的的可编程计算机可读取的存储介质或装置(例如,rom或磁性软盘)上,以用于在所述存储介质或装置被计算机读取时配置并操作所述计算机,从而执行本文所述的程序。所述系统还可被认为作为配置有计算机程序的计算机可读存储介质而实现,其中这样配置的存储介质使计算机以特定且预定的方式操作,以执行本文所述的功能。
本文还提供了一种经由计算机存储和/或传输通过本文公开的方法收集的序列和其他数据的方法。包括但不限于软件和存储装置的任何计算机或计算机配件都可用来实施本发明。序列或其他数据可由用户直接或间接输入到计算机中。另外,可用于对dna进行测序或分析dna或分析肽结合数据的任何装置都可连接至计算机,以便将数据传输至计算机和/或计算机兼容的存储装置。数据可存储在计算机或适合的存储装置(例如cd)上。数据也可通过本领域众所周知的方法(例如,互联网、地面邮件、航空邮件)从计算机发送至另一台计算机或数据收集点。因此,通过本文描述的方法收集的数据可在任何点或地理位置收集并且被发送至任何其他地理位置。
实验
实施例1
肿瘤浸润性淋巴细胞上表达的孤儿t细胞受体的抗原鉴定
免疫系统可发动针对肿瘤的t细胞应答;然而,对肿瘤浸润性淋巴细胞(til)的抗原特异性了解甚少。鉴于tcr通常对其内源性配体表现出强烈的偏好的最近发现,使用了肽-人白细胞抗原(phla)的酵母展示库来筛选来自人大肠腺癌的til上表达的‘孤儿’t细胞受体(tcr)的抗原。四种til来源的tcr对高度多样的phla-a*02:01文库中呈现的肽表现出强选择。所述tiltcr中的三种对非突变的自身抗原具有特异性,其中两种存在于单独的患者肿瘤中,并且对源自u2af2的非突变的自身抗原具有特异性。这些结果表明,tcr可接近的mhc结合肽的有限识别表面含有足够的结构信息,以能够重建具有未知特异性的病原性tcr的肽靶标的序列。这一发现使得能够容易地鉴定肿瘤抗原。
迄今为止,没有直接的相互作用筛选或组合展示系统用于确定孤儿tcr的抗原特异性。在此,测试了我们的方法,目的是鉴定被源自til的tcr识别的抗原(图1b)。在患有结肠直肠腺癌的两名hla-a2纯合患者中应用了cd8+til的单细胞t细胞表型分析和tcr测序,以从酵母展示文库选择中预测候选抗原靶标(图1b)。在所筛选的tcr中,四种tcr在hla-a*02:01文库中分离了肽靶标。这些tcr中的两种在序列上高度相似,并且对一组重叠的肽具有特异性,从而意味着共有抗原特异性。除了来自uniprot人参考基因组的预测肽外,从文库中分离的合成肽也刺激了相应的目标t细胞受体。令人惊讶的,四种受体中的三种识别出未突变的自身抗原。这充当使用酵母展示文库将t细胞免疫应答及其克隆tcr与直接抗原鉴定方法联系的原理证明。这种方法学可充当鉴定通过免疫应答识别的新型癌症抗原的有力工具。
hla-a*02:01酵母展示文库的设计.hla-a*02:01等位基因高度流行,存在于达50%的多种群体中。由hla-a*02呈递的肽的结合基序已得到充分表征,并且鉴定了许多受限制的临床上相关的tcr。由于这些原因,生成了酵母展示文库用于筛选潜在hla-a*02:01限制性t细胞受体(图1a)。个别酵母表达与hla分子共价连接的随机肽,其使得能够通过dna测序鉴定肽(图1c)。这种phla文库具有n-末端肽文库,所述文库与野生型β-2-微球蛋白(b2m)和具有单点突变y84a的hla-a*02:01重链连接(参见star方法)。为确保肽在结合槽中的正确展示,所述肽文库将p2和pω处的氨基酸使用限制为hla-a*02:01所优选的脂族疏水性残基(图1d-1f)。在其他位置,nnk密码子随机编码所有20种氨基酸,以提供无偏文库。由于hla-a*02:01通常呈递8至11个氨基酸长度的肽,因此使用表位标签生成了多种肽长度的文库以进行多重选择(图1f)。每个文库的理论核苷酸多样性由文库组成和长度决定,但所述文库的功能多样性有限(图1f)。总计,估计在组合文库中代表了8到11个氨基酸范围的大约4亿种独特的肽。
用mart-1特异性dmf5tcr验证文库.为了确定hla-a*02:01复合物是否正确折叠以呈递肽,使用了具有已知特异性的‘代理’tcr。使用了dmf5tcr,其是识别源自与hla-a*02:01结合的mart-1黑素瘤抗原的10个氨基酸序列(eaagigiltv)(seqidno:267)的天然存在的tcr。为了验证hla-a*02:01文库,将具有改进的hla稳定性的10聚体异源肽elagigiltv(seqidno:264)与hla-a*02:01一起展示在酵母上,并通过抗血凝素(ha)抗体和400nm四聚化dmf5tcr两者染色,从而指示蛋白质复合物的表面表达和phla的正确折叠(图2a)。为了确认所述文库可用于鉴定dmf5tcr的抗原,通过macs珠粒多聚化dmf5tcr选择了hla-a*02:0110聚体文库(图1f)(参见star方法,图2b)。通过桑格(sanger)测序对第四轮选择的样品进行测序以鉴定富集的肽,发现其中大多数与mart-110聚体肽高度相关(图2c)。五个序列分别在具有hla-a*02:01的酵母上表达,并且用400nmdmf5tcr四聚体染色以显示tcr特异性结合(图2c)以及用抗hla-a*02染色以显示复合物的构象表达(图8a)。
通过dmf5tcr进行的所有酵母展示选择轮次均进行了深度测序。所述文库到第3轮选择显著会聚至68种独特的肽,其中前10种肽占文库的91.7%(图2d)。最显着的观察结果是,几乎所有选定的肽在p6(p6g)处都具有gly(表1),这与dmf5-mart-1/hla-a*02:01晶体结构一致,从而表明p6g提供柔性以允许cdr3β100f的间隙,p6g氢键键合至所述间隙。深度测序揭示了肽序列的两种主要簇(图2e)。为了阐明这些簇,计算了所有肽之间的反汉明距离,所述距离是用于鉴定两种肽之间的精确氨基酸匹配数量的度量,且然后按得分进行聚类(图2e,表1)。两个主要簇在p4至p6处相异,在29种肽中具有中心‘gig’基序(簇1)以及在32种肽中具有中心‘drg’基序(簇2)。在搜索矩阵中使用了簇1肽以对潜在人多肽靶标进行评分,这是以前用于根据酵母展示选择数据预测人抗原的方法(2014pwm)。然而,由于10聚体文库不允许在文库的p2处的ala,因此将p2a手动包含在搜索矩阵中,从而匹配频率最低的锚-leu为16.67%。根据此分析,预测了来自人蛋白质组的9种肽,其结合dmf5tcr的概率得分各不相同(图2f,表1)。显著地,人mart-1肽最有可能结合预测的9种肽的dmf5tcr(图2f)。使用簇2,预计多个数量级的肽结合tcr(图8b、8c,表1)。然而,dmf5tcr尚未显示任何脱靶毒性,从而表明在所述研究中这种其他‘drg’肽基序可能在癌症患者的免疫应答中没有生理相关性。
用新抗原特异性tcr盲法验证hla-a*02:01文库.为了测试hla-a*02:01文库鉴定具有未知抗原特异性的tcr的抗原的能力,筛选了三种源自黑素瘤患者的tcr,其中所有tcr对新抗原均具有盲特异性。这些抗原已通过肿瘤材料的外显子组测序、通过hla-a*02:01预测新抗原呈递以及使用肽负载的hla-a*02:01多聚体对患者来源的肿瘤浸润性t细胞进行染色而独立地鉴定。重组表达标记为nki1、nki2和nki3的三种tcr,并用于选择含义所有四种肽长度的hla-a*02:01文库。
从第2轮选择开始,仅针对nki2的选择产生了400nm四聚体阳性酵母,从而表明肽-hla-a*02:01文库的强结合(图3a)。对所有选择轮次进行深度测序,且然后基于每个选择轮次的肽长度解析数据(表2)。肽通过第3轮选择会聚并且通过反汉明距离对肽进行聚类(图3b)。nki2的选择结果在9聚体、10聚体和11聚体序列中显示显著相似性。这些肽序列分别在p6、p7和p8处在9聚体、10聚体和11聚体序列中共有保守glu,并且所述肽在9聚体、10聚体和11聚体的p5处共有带正电的残基。nki1和nki3不会产生四聚体阳性的选定酵母(图3a),深度测序也未指示强的肽选择。
作为盲法验证的一部分,预测将由hla-a*02:01呈递的127种新抗原的列表用作nki2tcr的候选配体。与通过nki2选择的10聚体合成肽的列表相比,计算这127种潜在的新抗原肽中的每种的反汉明距离(图3c)。aldphsghfv(seqidno:265)(一种源自细胞周期蛋白依赖性激酶4(cdk4)的肽新抗原)在10个位置中的5个和6个分别与文库肽lib-1和lib-2相同。(图3d)。cdk4被正确鉴定并确认为nki2的新抗原靶标。通过这种盲法验证可能无法明确鉴定nki1和nki3的靶标。nki1对同一cdk4新抗原具有特异性,并且nki3对gcn1l1新抗原alletpslll(seqidno:268)具有特异性。稍后论述缺乏靶标鉴定的原因。
已经确立,从phla文库分离的这些合成肽被nki2特异性地识别。接下来,询问它们是否能够刺激表达nki1或nki2的t细胞。用nki1或nki2转导人外周血淋巴细胞并与负载有通过nki2选择的前5种肽的hla-a*02:01+jy细胞共培养。令人感兴趣的是,所有5种肽都以剂量依赖的方式引发nki2转导的t细胞产生ifnγ(图3f)。此外,最前面选择的肽模拟表位aldsrsehfm(seqidno:269)与cdk4新抗原aldphsghfv本身一样有效地刺激这些细胞。通过nki2进行的第五大选择的肽以剂量依赖性方式刺激nki1受体,从而表明重叠的特异性。
结肠直肠癌患者中肿瘤浸润性淋巴细胞的单细胞表征.最终目标是使用酵母展示平台鉴定源自浸润患者肿瘤的扩增且细胞毒性的t细胞群体的tcr的肽配体(图1b)。用于分析t细胞的单细胞技术提供了对单个t细胞进行单独表型化并以高通量的方式对其配对的αβtcr进行测序的方式。
选择了对于hla-a*02等位基因纯合的患者(图4a)。这提高了从患者分离的t细胞具有限于hla-a*02等位基因的受体的可能性;然而,这并不排除这种tcr可对其他经典或非经典限制性抗原具有特异性的可能性。测定了没有hla-c的两名患者的完整hla基因座(表3)。hla-a*02:01和hla-a*02:06的不同之处仅在于β-折叠底板中的f9y取代,其不太可能影响tcr识别。尽管差异可能等同于phla的tcr多聚体染色的不同模式,但已描述了这些亚等位基因共有可呈递的肽抗原的子集。
两名患者均为患有结肠直肠腺癌的60多岁的男性(图4a)。分析了肿瘤的组织样品中的cd8+和cd4+t细胞的浸润以及通过h&e染色观察到的总体结构(图9a)。对于患者a,在结肠的固有层中发现cd4+和cd8+t细胞,而在肿瘤中较少。对于患者b,cd4+t在结肠组织内并不丰富;然而,存在显著cd8+t细胞浸润到肿瘤中。
从这两名患者,对来自肿瘤部位的数百个cd8+t细胞进行了表型分析并使用来自健康组织的53个配对序列和来自肿瘤组织的709个配对序列进行测序(图4b)。在肿瘤部位多于一次观察到的任何克隆都被视为扩增的克隆。在两种情况下,肿瘤中都存在扩增的tcr克隆,从而表明抗原特异性扩增。最大扩增的tcr克隆分别占患者a中测序群体的12.9%(23/178)和患者b中6.67%(35/526)。肿瘤的这种扩增水平与原发性肝癌和cd4+t细胞浸润性结肠直肠癌中t细胞谱系群体的其他报告一致。由于从健康组织中鉴定出的t细胞很少,因此,如果不共有α或β链,则认为克隆是肿瘤专有的并且不与健康组织共有。对于两名患者,α和β链序列均仅显示肿瘤与健康组织之间的序列的小的重叠(图4c)。这表明大多数tilt细胞克隆由于肿瘤驱动的应答而富集并存在于肿瘤中;然而,由于健康组织的采样有限,不能得出任何tiltcr仅存在于肿瘤中的结论。
从患者测序的t细胞受体表现出典型的cdr3α和cdr3β长度(图9b)。两名患者均具有trav8-3、trav19(图9c)和trbv7-2(图9d)表达的主导。与来自患者a的t细胞不同,通过索引分类对来自患者b的t细胞进行了分析,从而可将细胞表面标记表达与转录物表达配对。当使用t分布随机近邻嵌入(t-sne)基于细胞表面标记和转录谱分离t细胞群体时cd8+和cd4+t细胞群体被分离成主要簇(图9e)。对于患者b,存在显著cd8+t细胞浸润到肿瘤中,并且采样的大多数细胞共表达pd-1和ifnγ,具有其他细胞毒性标记颗粒酶b、穿孔素和tnf-α的异源表达。已经提出pd-1+cd8+t细胞群体是肿瘤反应性群体。
在hla-a*02:01文库上筛选孤儿tcr.基于肿瘤的局部扩增、细胞毒性概况(ifnγ、tnfα、穿孔素、颗粒酶b)并在一些情况下基于常用的tcr链使用情况,选择了二十种候选受体(图4b、4d)。在hla-a*02:01文库上筛选的二十种候选tcr中(表4),四种tcr富集来自文库的肽,源自患者a的tcr1a和2a以及源自患者b的tcr3b和4b(图5a)。令人感兴趣的是,从不同患者分离的两种受体2a和3b表达相同的tcrα链和相似的tcrβ链,它们包含相同长度的cdr3β序列,具有五个保守氨基酸差异以及通过np加成完全产生的中心val残基(图5b)。
在hla-a*02:01文库上筛选每种tcr。四种tcr中的每一种都富集了由酵母表达的hla连接的表位标签,而剩余十六种tcr没有(图5c)。对于tcr1a、2a和3b,四聚体染色的酵母在整个选择轮次中逐渐增加。然而,尽管9聚体表位标签的连续富集,tcr4b仍未使酵母染色(图5c)。剩余十六种tcr缺乏富集的原因很可能是hla限制为替代性hla等位基因,并在讨论中探讨了其他可能性。
对由tcr1a、2a、3b和4b选择的酵母进行深度测序(表4)。对于所有四种tcr,使用通过第3轮选择会聚的序列和独特的肽序列来生成肽基序,以鉴定位置热点(图6a)。为相关肽序列选择了高度相似的tcr2a和3b,其中11种是两者共有的(图6c)。共同肽池的选择表明这些tcr识别相同的抗原。但是,在p6的这两个基序之间观察到显著差异,对于tcr2a,不变asn;而对于tcr3b主要是asn、glu和ser。一般来说,与通过tcr3b选择的66种独特的肽相比,tcr2a展示更大程度的交叉反应性,选择190种具有位置p1、p4和p5的独特肽,从而允许更多的氨基酸取代。tcr1a和4b具有完全不同的基序,分别在第三轮选择中选择了15种和61种独特的肽。
用于测量t细胞受体的交叉反应性的方法是观察在肽的特定位置处耐受的氨基酸的所选择宽度。为此,确定了每个位置处所有氨基酸的比例,考虑了第3轮的肽富集(图6b)。tcr1a和3b的肽基序相对特异性,每个位置的氨基酸偏好更高。相比之下,tcr2a和4b在其特异性上更具交叉反应性,从而除有限的锚残基外还允许沿肽的位置处的简并。尽管2a与3b之间的氨基酸序列非常相似,但tcr在其肽景观的交叉反应性方面展示高对比度。在此方面,phla文库筛选可有效‘测量’tcr的相对交叉反应性,这对于选择用于过继性细胞疗法的tcr可能是重要的,其中可能需要有限的交叉反应性以限制自身反应性。
来自人蛋白质组和患者外显子组的tcr靶标预测。在酵母展示选择中鉴定的肽生成每种tcr的序列的识别景观。如使用2014pwm对dmf5tcr所进行的一样,此信息可用于预测刺激性人抗原的算法中。在将所述算法应用于结肠直肠癌数据时,生成了针对tcr2a的人类预测,但未获得针对tcr1a和tcr3b的预测,并且针对tcr4b的预测有限。这激发了从选择数据中预测人肽的两种另外方法的发展-先前统计方法的改进的变型(2017pwm)和利用两层卷积神经网络的方法(2017dl)(参见star方法)。使用dr15文库来自先前选择的数据来测试2017pwm和2017dl算法在预测肽抗原中的准确性。mbp是使用2017dl的最佳预测,并且对于tcrob1.a12是使用2017pwm的次佳预测,并且对于tcrob1.2f3是两种算法中的次佳预测。
使用uniprot数据库,使用另外两种算法来对来自人蛋白质组的预测肽进行评分。对于tcr2a和3b,存在通过用于两种tcr的多种算法预测的许多肽,从而表明它们共有靶标特异性。总体而言,对于所有四种tcr,这三种算法能够共同地从人蛋白质组进行预测。
因为患者突变可产生被t细胞识别的新抗原,所以进行了外显子组测序和变体调用以鉴定潜在候选者。总计,在患者a中鉴定了762种pass变体并且在患者b中鉴定了4,763种pass变体,对健康组织和肿瘤组织的测序覆盖率为至少30x。通过2017pwm和2017dl算法对外显子组肽进行了评分,但在整个tcr中只有极少数是显著的。一个例外是同一wdr66基因的从内含子至外显子7的21个核苷酸易位,即使在将导致其表达不佳(如果有的话)的亚最佳hla锚的情况下,它也在患者a中产生新抗原肽。这产生了新颖的肽序列eygvsyew(seqidno:270),其与患者a来源的tcr1a的肽基序紧密匹配。总体而言,对四种tcr的预测表明,四种tcr中的三种很可能结合未突变的自身抗原。
合成和预测的人肽的体外靶标验证.选自文库的合成肽和来自人和/或外显子组的预测的人肽均由用于刺激经修饰以表达从患者中鉴定的四种tcr的skw-3cd8+t细胞系的t2细胞呈递。令人感兴趣的是,由tcr1a选择的合成文库肽全部经由cd69活化(图7a,图10a)并以剂量依赖性方式(图7b)有效地刺激t细胞。对于tcr1a,外显子组肽(eygvsyew)(seqidno:270)、锚定修饰的外显子组肽(emgvsyem)(seqidno:271)、人肽预测均未刺激细胞系(图7a)。尽管已经确定了tcr1a的强抗原识别基序,但是还没有能够恢复刺激性内源性抗原,只有模拟表位。
对于三种tcr2a、3b和4b(图7c-7h),能够鉴定刺激性内源性抗原。tcr4b受其选择的合成肽库刺激,并且也受6/19的预测的人肽刺激,这与在酵母选择深度测序分析中观察到的较高程度的交叉反应性相符(图7g、7h,图10d)。令人感兴趣的是,观察到tcr4b被来自两种不同推定驱动基因wdr871310-1318(肽lledldwdv)(seqidno:272)(一种在结肠直肠癌中反复突变的睾丸表达的抗原)和crispld182-90(肽nmeymtwdv)(seqidno:273)(一种在许多癌症中表达的无已知功能的蛋白质)的抗原刺激。富含半胱氨酸的分泌蛋白、抗原4和发病机理相关1蛋白(cap)超家族包括crispld1,并且这些蛋白质一直牵涉于广泛范围的功能中,包括离子通道调控、繁殖、癌症、细胞粘附等。根据外显子组分析,患者b在d143y处在crispld1中具有突变。tcr4b也受到5种其他人抗原的刺激,包括cd74181-189肽tmetidwkv(seqidno:274)、fanci1104-1112肽vleevdwli(seqidno:275)、gemin4771-779肽kleqldwtv(seqidno:276)、pde4a243-251肽tleeldwcl(seqidno:277)或pde4b231-239肽tleeldwcl(seqidno:277)以及klhl7506-514肽nveyydikl(seqidno:278)。真实的体内特异性不能在没有另外肿瘤信息的情况下明确地鉴定。
高度相似的tcr2a和3b对选定的合成肽具有不同的刺激概况(图7c-7f,图10b-10c)。tcr2a细胞受通过tcr2a选择的前五种肽中的四种和通过tcr3b选择的前五种肽中的四种刺激。然而,tcr3b细胞仅受通过其自身tcr选择的前五种肽中的四种刺激,并且不受通过tcr2a选择的任一种刺激。这些结果支持了tcr3b与tcr2a相比具有相对选择性的发现(图6b)。显著地,在从预测测试的26种人肽(表6)中,仅发现单一人肽可刺激带有任一受体的t细胞(图6c、6e)。这种肽是mmdffnaqm(seqidno:279),其源自u2af2174-182(一种参与rna剪接复合物的蛋白质)。u2af2通常在许多人组织中表达,并在许多癌症(包括结肠直肠癌)中过表达,如通过在蛋白质图谱中沉积的抗体染色所确定。实际上,u2af2rna分别在患者a和患者b中在肿瘤组织中相对于健康组织中过表达2.11倍和2.65倍(图11a)。当检查人淋巴瘤、乳腺、结肠和肺肿瘤细胞系时,相对于患者样品,u2af2rna显著过表达(图11b-11c)。u2af2一直牵涉于黑素瘤的肿瘤转移的促进中,并且在慢性髓细胞性白血病、骨髓增生异常综合症和实体瘤(如肺腺癌)中很少突变。u2af1(u2af2的结合配偶体)通常在癌症中突变,并且突变显示rna剪接和外显子跳跃增强,从而导致体外基因失调。在两名患者中,在u2af2或u2af1中均未发现突变。对于与tcr3b相比更具交叉反应性的tcr2a,源自蛋白质txndc11107-115的另一种人肽(seqidno:280)vldfqgql能够刺激受体,所述受体先前并未被描述涉及在癌症中,但在结肠和许多其他组织类型中表达。
通过表面等离子体共振确定tcr2a对由hla-a*02:01展示的肽mmdffnaqm(seqidno:279)的亲和力为110μm,从而鉴定真正的相互作用(图11d-11e)。不能确定对tcr3b的亲和力。这些低亲和力可部分解释表达单链mmdffnaqm-hla-a*02:01(seqidno:281)的酵母的tcr四聚体染色的缺乏(图10f-10g)。在所有研究的tcr中都观察到刺激对比四聚体结合的这些不一致结果(图10e-10h)。相反,mmdffnaqm-hla-a*02:01(seqidno:281)四聚体未能染色表达tcr2a或tcr3b的skw-3细胞。不幸的是,组织样品不能用于通过质谱法确认通过hla-a*02进行的肽呈递。尽管不能确定性地确定靶向源自u2af2的肽的免疫应答,但来自酵母展示筛选、预测算法和体外刺激的证据将这种肽鉴定为可能的靶标。这些结果充当phla文库能够鉴定tcr的抗原特异性的原理证明,已鉴定出两名患者内的共有特异性。phla文库还可正确地区分肽抗原的相对交叉反应性。
从研究中得出的根本令人惊讶的见解是,在对tcr可见的mhc结合肽的小识别核中编码的特异性足以使内源性肽的整个序列重建为特异性未知的tcr。这种发现对鉴定t细胞介导的疾病中的抗原具有重要意义。t细胞在感染性疾病、自身免疫、过敏和癌症中提供了治疗性途径。在大多数这些情况下,由于方法有限,对t细胞特异性的了解很少,尤其是在人中。通过高通量方法直接离体从单个t细胞获得tcr序列使这种情况得以进展,但是仍然面临着确定肽配体的艰巨任务。在此,将单细胞tcr分析方法与酵母展示文库筛选方法的改进版本相结合,以发现人结肠直肠腺癌中的新颖phla特异性。这对于对癌症中t细胞特异性的理解具有广泛意义,并且可应用于其他疾病。
据我们所知,这是使用组合生物学筛选技术进行tcr配体鉴定的第一个实例,其中发现三种tcr对在癌症中具有作用的野生型抗原具有特异性。在所研究的2/2患者中,源自u2af2的单一野生型抗原可能是共有的免疫应答靶标。对于在hla-a*02文库上成功筛选的所有tcr,能够鉴定出刺激这些tcr的多种模拟表位肽,通常比天然肽更有效。类似于新抗原,合成的肽抗原或模拟表位可用作dna、rna或肽疫苗以刺激特定的抗原特异性t细胞,并产生比免疫应答可能耐受的自身抗原更高的免疫原性应答。
从深度测序选择数据预测同源肿瘤抗原的成功取决于改进的且完善的搜索算法和患者组织验证。另外,从给定肿瘤中筛选大量的tcr可能增加将选择数据与同源抗原联系的几率,特别是当与相关的患者数据(包括rna表达和/或洗脱肽的质谱)结合时。
这种方法在免疫治疗中有两个主要应用:1)鉴定孤儿tcr的内源性和模拟表位配体和/或2)作为基于肽抗原特异性对tcr进行分类的方式,这将允许鉴定识别患者中的共有抗原的临床候选tcr。共有的tcr可以是共有相似tcr序列的受体(其可能潜在导致共有的抗原特异性),或者可以是没有任何共有序列、但识别相同抗原的tcr。识别共有抗原的此类tcr在工程化的t细胞或疫苗疗法中特别有用。随着tcr测序继续发展和更多tcr测序数据变得可用,可推断出患者hla的tcr限制,并推断出汇集的tcr序列簇的共同tcr特异性。这使得tcr配体识别能够更有效地针对具有已知hla限制的有影响的tcr。
与其他利用外显子组数据来鉴定可充当t细胞免疫应答的潜在靶点的患者特异性新抗原的方法不同,这种方法是对当前免疫应答的tcr特异性的无偏探询,所述探询依赖于tcr与phla之间的物理相互作用。这种配体鉴定方法在突变负荷低的癌症中可能尤其重要,在所述癌症中,新抗原靶标与野生型抗原相比可能不那么普遍。已经开发出改进酵母菌展示文库用于破解tcr的用途的方法,所述方法可提供用于鉴定临床上重要的tcr和新抗原的方式。已经验证了hla-a*02:01文库作为两名结肠直肠腺癌患者中用于til的破解的工具。主要将野生型抗原鉴定为这些患者免疫应答的靶标,对具有潜在治疗价值的野生型抗原具有共同应答。
star方法
实验模型和受试者细节
人受试者.两名年龄为64和66岁的男性受试者均患有结肠直肠腺癌。斯坦福大学机构审查委员会批准了收集人组织和血液的所有方案。患者样品是从斯坦福医院病理科在患者同意下获得的。两名患者均hla分型为无hla-c,并且因其hla-a*02等位基因表达而特别选择。
原代和细胞系。除非另有说明,否则所有细胞均在37℃、5%co2下生长。
在含有10%胎牛血清(fbs)、2mml-谷氨酰胺(thermofisher)和50u/ml青霉素和链霉素(thermofisher)的rpmi完全培养基(thermofisher)中培养人pbmc。skw-3细胞源自人t细胞白血病,并在含有10%fbs、2mml-谷氨酰胺和50u/ml青霉素和链霉素的rpmi完全培养基中培养。将转导的细胞与额外1ug/ml嘌呤霉素(thermofisher)和20ug/ml博莱霉素(thermofisher)一起培养。t2细胞是用作针对skw-3细胞的抗原呈递细胞的hla-a*02阳性细胞。将它们在含10%fbs、2mml-谷氨酰胺和50u/ml青霉素和链霉素的imdm(thermofisher)中培养。jy细胞是在含有10%fbs、2mm谷氨酰胺和50u/ml青霉素和链霉素的rpmi完全培养基中培养的ebv永生化b细胞系。hek293t细胞在含有10%fbs、2mml-谷氨酰胺、50u/ml青霉素和链霉素的dmem完全培养基(thermofisher)中生长。flyrd18在含10%fbs与2mm谷氨酰胺和50u/ml青霉素和链霉素的dmem完全培养基中生长。
eby100酵母细胞在含有20g右旋糖、6.7gdifco酵母氮源(bdbiosciences)、5gbacto酪蛋白氨基酸(bdbiosciences)、14.7g柠檬酸钠(sigma-aldrich)、4.29g柠檬酸一水合物(sigma-aldrich)/升h2o(ph4.5)的sdcaa中生长或在用半乳糖代替右旋糖的sgcaa中生长。酵母在sdcaa中在30℃或在sgcaa中在20℃生长,以便在大气co2下诱导蛋白质。
highfive细胞在具有最终浓度为10mg/l硫酸庆大霉素(thermofisher)的insectx-press培养基(lonza)中在27℃、大气co2下生长。sf9细胞在含10%fbs和最终浓度10mg/l的硫酸庆大霉素的sf900-iii无血清培养基(thermofisher)中在27℃、大气co2下生长。
酵母展示文库的制备和选择.如先前报告(birnbaum等人,2014),使用化学感受态eby100酵母(atcc)生成酵母展示文库。简言之,使用编码所选密码子集的引物来产生dna编码的肽文库。肽的p2和pω处的锚位置分别具有leu-met和leu-met-val的受限密码子使用,而在所有其他位置允许nnk密码子多样性(图1e,表8)。单独长度的文库编码不同长度的密码子集和使用独特的表位标签进行多重选择的载体:8聚体-v5标签,9聚体-myc标签,10聚体-ha标签,11聚体-vsv标签。为了在酵母上展示肽/hla*a-02:01复合物,用y84a突变对hla*a-02:01的重链进行了修饰,并在s302处将重链截短。这种突变允许接头打开,以在肽的c-末端之间穿过肽结合槽的末端穿至b2m,以产生单链三聚体。跨膜截短的重链连接至与aga2p蛋白连接的表位标签以用于酵母展示。在有限稀释后,通过菌落计数在电穿孔后确定酵母文库的多样性。
将酵母以个别长度文库的10x多样性混合,并在-80℃下于2%甘油和0.67%酵母氮源中冷冻。根据需要将文库在sdcaaph4.5中解冻,传代,在sgcaa中诱导,且随后如前所述(birnbaum等人,2014)使用与链霉抗生物素蛋白包被的磁性macs珠粒(sab)(miltenyi)偶联的生物素化的可溶性tcr进行选择。简言之,在10ml的pbs+0.5%牛血清白蛋白和1mmedta(pbe)中在4℃下用250μlsab阴性选择含有所有四种长度文库的酵母(4x109个细胞)的10x多样性持续1小时。使酵母通过附接至磁台(miltenyi)的ls柱(miltenyi),并洗涤3次。然后将流过液与250μlsab一起在4℃孵育3小时,所述sab与400nm生物素化的tcr一起在4℃预先孵育15分钟。再次,使酵母通过ls柱,并在sdcaa洗涤后将洗脱液在sdcaaph4.5中生长过夜。一旦酵母达到od>2,就将其在含有10%sdcaa的sgcaa中诱导2-3天,然后进行另外的选择。所有后续选择均使用500μl的pbe中的50μlsab或tcr包被的sab完成。在酵母与400nmsa-647于500ulpbe中孵育1小时、随后pbe洗涤且与50μl的抗alexa647微珠(miltenyi)孵育20分钟后,使用阴性选择进行第四轮。在与400nmsa-647tcr四聚体孵育3小时、随后20分钟的抗alexa647微珠20分钟后,进行阳性选择。天然文库和所有选择轮次都按如下所述进行深度测序。诱导后用抗表位染色监测每个轮次,并在4℃下进行400nmtcr四聚体染色3小时。
将从所述选择中分离的个别酵母克隆或用从深度测序中鉴定的重构的肽-hla构建体电穿孔的感受态酵母用标记有sa-647的400nmtcr四聚体或单独sa-647与抗表位标签的组合染色。
phla文库的深度测序.通过miniprep(zymoprepii试剂盒,zymoresearch)每轮选择从5x107酵母中分离dna。通过pcr将个别条形码和随机8聚体序列添加至测序产物的侧翼区中,并扩增25个循环(表8)。这些引物从构建体的信号肽扩增至b2m的中间序列。随后进行额外的pcr扩增,添加illumina芯片引物序列以生成含有illuminap5-truseq读取1-(n8)-条形码-phla-(n8)-truseq读取2-llluminap7的最终产物。通过琼脂糖凝胶纯化对文库进行纯化,通过nanodrop和/或bioanalyzer(agilentgenomics)进行定量,并且对于低多样性文库使用2x150v2试剂盒通过illuminamiseq测序仪进行深度测序。
可溶性tcr的表达.f5tcr的每条链分别在大肠杆菌bl21(de3)中表达,并进行纯化、重折叠和功能验证。对于所有其他tcr,使用sf9细胞分别表达tcr的每条链,以在pacgp67a载体(bdbiosciences)中产生杆状病毒。α链和β链两者均含有对应于tcrvα或tcrvβ的gp67信号肽。两种构建体均利用表达tcrv区的多角体启动子,其中人恒定区在连接肽处被截短以进行可溶性表达,并带有工程化的二硫键(boulter等人,2003)。两条链均表达c末端酸性gcn4拉链-6xhis标签或c末端碱性gcn4拉链-6xhis标签。所有含有酸性拉链的链都含有生物素化受体肽。两条链均在tcr胞外结构域的c末端与gcn4拉链之间包含3c蛋白酶位点。将所述dna与具有cellfectinii(lifetechnologies)的bdbaculogold线性杆状病毒dna(bdbiosciences)一起共转染到sf9细胞中。在2ml培养物中产生病毒。将病毒以在25ml培养物中1:1000的稀释度以1x106个细胞/ml传代,以产生更有效的病毒,然后将其在2ml的highfive(hi5)(thermofisherscientific)细胞中以2x106个细胞/ml共滴定以产生稀释液以获得通过sds-page凝胶和考马斯蓝染色tcrα和β链的1:1表达。对于每个链,共滴定范围从1:1000至1:250。
使用病毒来感染hi5细胞,以获得以2x106个hi5细胞/ml在1-4l体积中的蛋白质表达。感染后2-3天取出细胞,并将上清液处理至100mmtris-hclph8.0、1mmnicl2和5mmcacl2以沉淀污染物。通过离心除去沉淀物,并将上清液在室温下与ni-nta树脂(qiagen)一起孵育3小时。将蛋白质用1xhbsph7.2中的20mm咪唑洗涤,且然后在1xhbsph7.2中的200mm咪唑中洗脱。在30kda过滤器(millipore)中将缓冲液交换至1xhbsph7.2后,在4℃下将蛋白质用bira连接酶、100um生物素、40mmbicineph8.3、10mmatp和10mm乙酸镁生物素化过夜。用3c蛋白酶(10ug/mgtcr)过夜处理用于表面等离子体共振的蛋白质。通过使用aktapurifier(gehealthcare)superdex200柱(gehealthcare)的尺寸排阻色谱法纯化蛋白质。分离级分,在sds-page凝胶上运行以通过链霉抗生物素蛋白转移确认1:1化学计量和生物素化。合并级分并通过nanodrop定量tcr,并且在-80℃下冷冻以储存在ph7.2的1xhbs缓冲液中。
斯坦福大学机构审查委员会批准了收集人组织和血液的所有方案。来自年龄为64岁和66岁的两名男性的患者样品是从斯坦福医院病理科在患者同意下获得的。通过福尔马林固定的石蜡包埋加工一部分肿瘤组织样品以用于免疫组织化学染色。使用抗cd4(克隆1f6,leicabiosystems)、抗cd8(克隆c8/144b,dako)或苏木精/曙红对组织进行染色。新鲜肿瘤和健康样品如先前一样进行加工(han等人,2014)。简言之,将肿瘤组织分开并与pbs中的10mmedta孵育30分钟。制备细胞悬液,并使其通过10-μm尼龙细胞过滤器(bectondickinson),并在含5%fbs的rpmi中用0.5mg/ml的4型胶原酶(worthingtonbiochemical)处理30分钟。用钝头的16号针头和注射器破坏组织。保存一些样品用于抗体染色,以通过针对epcam(克隆9c4,biolegend)和live/dead可固定的死细胞染色试剂盒(invitrogen)染色来分离肿瘤组织,并使用ariaii(bectondickinson)通过facs进行分选,以通过aiiprepdna/rna微型试剂盒(qiagen)进行加工用于dna/rna提取。另外,通过percoll(gehealthcare)梯度离心富集淋巴细胞,并且将细胞冷冻在含有10%二甲基亚砜和40%fbs的rpmi中,或立即用于抗体染色。在染色用于facs之前,使用150ng/mlpma+1μm离子霉素对淋巴细胞进行非特异性预刺激3小时。用pbs+0.05%叠氮化钠+2mmedta+2%fcs洗涤细胞。
将淋巴细胞用以下抗体染色:抗cd4(rpa-t4,biolegend)、抗cd8(okt8,ebiosciences)、抗αβtcr(ip26,biolegend)、抗tim3(f38-2e2,biolegend)、抗cd28(cd28.2,biolegend)、抗cd103(ber-act8,biolegend)、抗ccr7(g043h7,biolegend)、抗lag3(3ds223h,invitrogen)、抗cd38(hit2,biolegend)、抗cd45ro(uchl1,biolegend)以及抗pd1(eh12.2h7,biolegend)。使用live/dead可固定的死细胞染色试剂盒(invitrogen)排除死细胞。使用ariaii(bectondickinson)通过荧光活化细胞分选(facs)将细胞直接分选到一步rt-pcr缓冲液(qiagen)中。通过索引分选对患者b样品进行分析。使用如先前生成的合并引物组(han等人,2014)扩增反应,加条形码并且合并以通过琼脂糖凝胶纯化进行纯化,并使用2x250v2试剂盒通过illuminamiseq进行深度测序。使用定制软件流水线处理数据,并使用vdjfasta调用各个孔的cdr3、tcrα和tcrβ可变、连接和多样性区域。使用基于t细胞转录标记和表型标记的t-sne分析数据,以分离细胞群体。
患者外显子组的测序和变体调用.从肿瘤和健康组织提取的dna用于生成用于外显子组测序的文库。使用nextera(illumina)将来自肿瘤和正常组织的50ngdna制成illumina测序文库。使用rochenimblegenseqcapez3.0(roche)汇集并富集外显子区域的文库。使用nextseq500生成配对末端75bp读数。根据gatk最佳实践确定了肿瘤特异性变体。简言之,使用cutadaptv1.9修整衔接子和低质量碱基。使用bwamem0.7.12将读数与hg19进行比对。使用picard工具v1.119删除重复项,然后使用gatkv3.5和从gatkresourcebundle2.8下载的参考文件进行插入缺失重新排列和碱基重新校准。使用bedtoolsv2.25.0确定中值覆盖范围。最后,使用mutect2确定正常与肿瘤之间的变体。所有试剂盒均遵循制造商的说明书,并且所有流水线种均使用默认软件参数。
使用来自ensembl的grch37组件,所有外显子组变体均用于生成替代编码序列。每个替代编码序列均经过处理,并基于文库肽的长度进行评分。使用2017pwm和2017dl算法对肽进行评分。
开发用于人肽的算法和预测.如先前所进行(birnbaum等人,2014)对深度测序结果进行分析,并进行了修改以并入针对不同肽段长度的文库的去卷积。基于信号肽和gs接头侧翼的氨基酸的数量鉴定不同长度的肽。简言之,使用pandaseq从深度测序结果中确定配对末端读数。使用geneious版本6通过条形码解析配对末端读数,以鉴定选择轮次。在第3轮和第4轮选择中具有少于10个计数并且与所述轮次中的另一序列仅相差仅1个核苷酸序列的所有核苷酸序列被合并至显性序列。任何具有移码或终止密码子的数据均从进一步分析中删除。使用自定义perl脚本和shell命令处理序列。
反汉明距离是从肽的总长度中减去的汉明距离,代表两个肽之间共有氨基酸的数量。使用matlab(mathworksinc.)通过针对来自所选第3轮文库序列中的所有其他肽对每种肽进行迭代来计算它们。产生的输出得分是肽之间匹配氨基酸位置的数目。基于反汉明距离,使用cytoscape对肽进行聚类,并基于肽相似性手动确定截断值。对于dmf5tcr,进行了聚类,并且使用簇来生成取代矩阵以进行预测,而不使用氨基酸频率的截断值。对于nkitcr,反汉明距离足以确定nki2tcr的新抗原特异性。2014pwm模型未从127种新抗原的列表中产生任何预测结果。在算法预测之前,未对四种结肠直肠癌来源的tcr进行聚类。
对于2014pwm和2017pwm,从所有选择的第3轮中生成取代矩阵,并将其用于搜索人蛋白质(uniprot)或患者特异性外显子组,以使用滑动窗口对固定长度的肽进行评分。通过确定肽的每个位置的所有氨基酸的频率来制得取代矩阵。对于使用2014pwm进行的所有预测(针对dmf5tcr进行的那些除外),对于给定位置处的氨基酸设置0.1%频率的截断值,以除去噪声。肽的分数被计算为每个位置处氨基酸频率的乘积。2017pwm不如2014pwm严格,因为它允许预测的肽在文库的选定肽中找不到的位置处并入氨基酸。这防止了丢弃可能尚未选择、但可能是可行的肽溶液的肽序列。
产生深度学习方法2017dl以将肽视为整体实体,而不是像以前的算法那样将肽的每个单独位置视为彼此独立(图12a)。在r中对包括肽序列和轮次计数的测序数据进行预处理,以除去在所有轮次中少于3个计数的任何肽序列。然后通过将每个轮次计数乘以轮次中的计数的平均数量且然后除以给定轮次中的计数的数量来对数据进行标准化。使用适合的适应度得分对源自适应度函数的文库中的每个肽进行评分,所述函数由通过标准化的轮次计数拟合至每种肽的指数曲线表示(图12b)。
接下来,使用每种肽的适应度得分生成模型,并将肽表示为20xl矩阵,其中l是肽序列的长度(图12c)。矩阵的20行与20种可能的氨基酸有关。氨基酸表示为独热向量,其中向量含有单个1,其余为0。将代表肽的矩阵展平至长度20xl的特征向量,以用于训练神经网络。将独热矩阵用作输入,并将适应度得分用作输出。使用来自文库肽的数据,实现了先前描述的利用分别使用10个节点和5个节点的两个隐藏层网络的网络体系结构(图12d)。训练是在lua中用torch软件包进行的。使用这种模型来对来自uniprot数据库(2015年12月18日下载)的给定肽和患者特异性外显子组进行评分,使用转换为独热矩阵进行神经网络输入的从l长度滑动窗口中分离的肽。计算每种肽的p值和经bonferroni校正的p值,从而代表从整个蛋白质组中随机选择适应度得分与所评分的肽一样高或高于所评分的肽的概率。
在共培养测定中测量t细胞活化.使用tcr可变基因和全长、未修饰的恒定区的野生型信号肽,将从自文库中选择肽的结肠直肠癌患者鉴定的四种tcr克隆到呈α-p2a-β构型的基于mscv的载体pmigii中。p2a跳过序列允许tcr的1:1化学计量表达。基于mscv的载体pmigii也用于生成δ-f2a-γ-t2a-ε-p2a-ζ格式的人cd3。包装载体pcl10a用于并入env、gag和pol,以允许人哺乳动物向性和病毒产生。所述载体将嘌呤霉素和博来霉素选择性引入感染的细胞。使用5μgtcr或人cd3dna和3.3μgpcl10adna在人胚肾293t细胞中针对每种tcr和人cd3产生逆转录病毒。使用x-tremegene9dna转染试剂(sigma-aldrich)在无血清dmem中产生病毒。在细胞培养中,使用2%fbsdmem来回收细胞,并在12小时更换培养基。分别在36、40、44和48小时收获2.5ml量的病毒以汇集,用0.45μm注射器过滤器(fischerscientific)过滤,并在-80℃冷冻,或立即用于感染tcr-cd8+skw-3细胞。通过在32℃下以2500rpm旋转2小时,将2mltcr病毒和2ml人cd3病毒与5ug/ml聚丙烯(millipore)一起用于共感染2x106个skw-3细胞。除去病毒,并用培养基和培养的细胞更换。在2-3天后,将转导的skw-3细胞无限期地在20ug博来霉素和1ug嘌呤霉素中培养,以选择tcr和人cd3共表达。然后将细胞针对tcr(ip26,biolegend)和人cd3(ucht1,biolegend)进行共染色,并在sh800细胞分选仪(sonybiotechnologyinc.)上分选。
将转导的skw-3细胞与tap缺陷型t2细胞以2:1的比例以各种肽稀释液共培养。测试了从酵母展示选择分离的前5种合成肽,以及从3种预测算法确定的预测。将肽合成至>70%纯度(genscript)(elimbiopharm),并且重悬于二甲基亚砜中至20mm,并储存在-20℃。使用accuric6(bdbiosciences)通过流式细胞术在18小时测量cd69(fn50,biolegend)以检测早期t细胞活化。通过ucht1染色检测skw-3t细胞,并检查tcr和cd3表达。通过抗体(bb7.2,biolegend)检查t2细胞的hla-a*02表达。使用flowjo版本10(flowjo,llc)分析数据,并通过前向散射和侧向散射以及ucht1+细胞将样品在skw-3细胞上门控,然后分析cd69表达。实验以生物学三次重复和技术三次重复进行。在prism中通过普通单向anova分析计算p值,并用所示的平均值的标准偏差或标准误差绘制实验。
cdk4特异性tcr克隆10(nki1)和17(nki2)来源于黑素瘤患者的til,基本上如所述用负载有预测的新抗原的hla多聚体筛选所述til。将两种tcr的可变部分克隆到编码鼠tcrα和β恒定结构域的逆转录病毒载体中。将flyrd18包装细胞以1.2x106个细胞/孔接种在10cm皿中。在一天后,使用25μlx-tremegenehpdna(sigma-aldrich)用10μg编码cdk4tcr的逆转录病毒载体dna转染细胞。在48小时后,分离逆转录病毒上清液,并转移至重组纤维连接蛋白(retronectin)包被的24孔板中,并以430g离心90分钟。pbmc被活化,并用抗cd3/cd28珠粒(thermofisher)以3:1的珠粒与细胞比率进行选择。在刺激后四十八小时,将t细胞以0.5x106个细胞/ml接种在病毒包被的板上。使用apc标记的cdk4r>lhla-a*02:01四聚体与抗鼠vβtcr-pe标记的抗体(bdbiosciences)的组合,测量转导的t细胞上引入的cdk4tcr的表面表达。使用facscalibur(bectondickinson)分析细胞。用指示浓度的cdk4肽或预测的肽将jy细胞在37℃脉冲1小时,且然后洗涤两次。接下来,在1μl/mlgolgiplug(bdbiosciences)存在下,将0.2x106个tcr转导的t细胞与0.2x106个肽脉冲的jy细胞一起孵育。将未暴露于jy细胞、暴露于未负载的jy细胞以及暴露于负载有无关肽(mart-1)的jy细胞的t细胞用作对照。在37℃、5%co2下孵育5小时后,将细胞洗涤并用percp-cy5.5抗cd8、fitc抗cd3、pe抗鼠vβtcr和apc抗ifnγ标记的抗体染色。
用外源性肽表达重折叠的hla-a*02:01.pet26b载体用于在rosettabl21de3大肠杆菌细胞中分别表达hla-a*02:01(1-275)和β2m(1-100)。将含有分离蛋白的包涵体溶解在8m尿素、40mmtris-hclph8.0、10mmedta和10mmdtt中。对于体外重折叠,将hla-a*02重链、β2m和mmdffnaqm(seqidno:279)肽以1:2:10的摩尔比混合,并稀释到含有0.4ml-精氨酸-hcl、100mmtris-hclph8.0、4mmedta、0.5mm氧化型谷胱甘肽和4mm还原型谷胱甘肽的重折叠缓冲液中。在4℃下72小时后,将蛋白质在10l的10mmtris-hcl中透析,并使用deae纤维素柱通过弱离子交换进行纯化。使用superdex200柱上的尺寸排阻色谱法和5/50monoq柱(gehealthcare)上的离子交换色谱法纯化蛋白质洗脱液。在30kda过滤器(millipore)中将缓冲液交换至1xhbsph7.2后,在4℃下将蛋白质用bira连接酶、100um生物素、40mmbicineph8.3、10mmatp和10mm乙酸镁生物素化过夜,之后在尺寸排阻superdex200柱上运行。
用于测量tcr2a和3b与mmdffnaqm-hla-a*02:01的结合亲和力的表面等离子体共振.使用biacoret100(gehealthcare)生物传感器在25℃下通过表面等离子体共振测量tcr2a和3b与mmdffnaqm-hla-a*02(seqidno:281)的相互作用。将生物素化的mmdffnaqm-hla-α2(seqidno:282)以大约1000个共振单位(ru)固定在链霉抗生物素蛋白包被的biacoresa芯片上。用无关肽-hla-a2固定不同的流动池,以充当空白对照。将不同浓度的2a或3btcr依次流经空白和mmdffnaqm-hla-α2(seqidno:282)。在60s后停止tcr注射,以允许足够的时间使spr信号达到平稳状态。通过使用biacore评价软件将平衡数据与1:1结合模型拟合来获得解离常数(kd)。
用于确定u2af2的相对rna表达的定量pcr.如上所述,先前从肿瘤和健康患者组织提取的rna用于确定u2af2rna表达的相对量。另外,从以下细胞系中提取rna:淋巴瘤:k562,daudi;乳腺:mdamb231;肺:a549、ekvx、hcc78、h358、h441、h1373、h1437、h1650、h1792、h2009、h2126、h3122、lc-2/ad。施用高容量rna至cdna试剂盒(thermofisher)一式三份产生cdna。汇集cdna样品用于使用taqman探针(thermofisher)、taqmanuniversalmastermixii、noung(thermofisher)和quantstudio3实时pcr系统(thermofisher)进行定量和定量实时pcr。u2af2探针(thermofisher,hs00200737_m1)扩增跨越u2af2的外显子的75bp区域。18srna探针(thermofisher,hs99999901_s1)用作管家基因,扩增了187bp区域。计算每种样品的u2af2至18srna的循环阈值,并将其与患者a健康组织或患者b健康组织循环阈值进行比较,以确定相对表达水平。绘制标准偏差。
定量和统计分析.使用skw-3细胞进行t细胞刺激测定.使用flowjo分析数据以门控skw-3细胞和cd3+组以鉴定t细胞。然后使用阴性对照(无肽)在cd69表达上门控t细胞。分析了cd3+组中cd69的中值mfi表达和表达cd69的细胞的百分比。与阴性对照(无肽)相比,使用prism确定了两种分析的单向普通anova。100μm肽刺激在生物学和技术三次重复中完成。仅示出生物学三次重复中的一次。肽滴定实验在生物学三次重复中进行。共同分析所有生物学三次重复。每个图均列出了p值指定的图例。使用sem(n=3;技术重复三次)或sd(n=3,生物学重复三次),并在相应的图例中列出。
2014pwm评分.如(birnbaum等人,2014)中所述进行评分。使用测序读数计数作为肽序列的乘数,从第3轮选择数据生成频率矩阵。将肽的每个位置乘以读数计数以得到给定氨基酸存在的次数。在第3轮中针对每种独特的肽进行此操作,并将每个位置的氨基酸计数除以总读数的数量。然后使用频率矩阵对人蛋白质组的每个n聚体肽进行评分,其中n是来自文库的所选择的肽的长度。通过乘以整个肽中给定氨基酸的频率来进行评分。
2017pwm和2017dl肽评分.在本文中生成了算法。对于两者2017pwm,生成与2014pwm中相同的频率矩阵,除了对于跨所有选择轮次的数据生成额外的频率矩阵,而不仅仅是第3轮。对于第3轮频率矩阵,每个氨基酸每个位置的比率取为所有轮次频率矩阵。对零值实现0.05的伪计数频率,并且取所述比率的log10。此得分被解释为一个位置处的特定氨基酸的富集率。此得分用于通过乘以每个位置的得分来确定来自外显子组或人蛋白质组的给定肽的总体富集。如方法中所述实施2017dl算法。
为了确定肽的统计显著性,对人蛋白质组和外显子组肽集合进行评分。为了计算外显子组肽集合的p值,在人蛋白质组得分的背景下计算百分位数得分。未校正的p值是1-百分位数。bonferroni校正的p值是未校正的p值乘以突变体集合中的肽的数量。
定量pcr分析.在技术重复四次样品中进行定量pcr。通过减去循环阈值来计算u2af2rna相对于18srna(δ循环阈值)的相对表达水平。通过减去参考相对于样品的相对循环阈值(δ循环阈值)来确定相对于健康的倍数变化(δδ循环阈值)。使用下式计算δ循环阈值的标准偏差
其中s=标准偏差,s1=目标样品的标准偏差,并且s2=参考样品的标准偏差。δδ循环阈值标准偏差取自δ循环阈值测试样品的标准偏差。
数据和软件可用性.外显子组测序.数据可在biosample登录samn07350021、samn07350022、samn07350023、samn07350024、samn07350025、samn07350026、samn07350027、samn07350028、samn07350029、samn07350030、samn07350031和samn07350032的短读取存档中获得。
深度测序.数据可在biosample登录samn07977164、samn07977165、samn07977166、samn07977167、samn07977168和samn07977169的读取存档中获得。
表1.dmf5选择数据和人靶标预测.
在通过反汉明距离聚类后,从dmf510聚体文库选择的第3轮深度测序鉴定的序列.使用这些聚类,使用2014pmm在uniprot数据库上进行预测。列出了‘gig’簇的9个预测和‘drg’簇的前10个预测。
表2
表2.根据肽长度的nki2选择数据.
从根据肽长度列出的nki2文库选择的第3轮深度测序鉴定的序列.与图3相关。
表3
患者hla分型结果.
在hla-a*02:01文库上筛选的tcr.基于肿瘤中的克隆性、表型概况、对肿瘤的排他性以及另外通过相关的tcr序列来选择tcr序列。肿瘤和健康标记下方的数字表示从这种组织观察到配对的tcr序列的次数。与图5和6相关。
tcr2a:
tcr包含trav19、traj32、cdr3:(seqidno:261)calsearggatnklif和trbv10-1、trbj1-1、cdr3:(seqidno:262)cassrdtvnteaff
α链(seqidno:258)
β链(seqidno:259)
tcr3b:
tcr包含trav19、traj32、cdr3:(seqidno:261)calsearggatnklif和trbv10-1、trbj2-7、cdr3:(seqidno:263)cassrdfvsneqyf
α与tcr2a相同
β链(seqidno:260)
序列表
<110>小利兰·斯坦福大学托管委员会(theboardoftrustees-lelandstanfordjunioruniversity)
马克m•戴维斯(davis,markm)
凯南克里斯拖弗•加西亚(garcia,kenanchristopher)
马文•吉(gee,marvin)
阿诺德•汉(han,arnold)
<120>从患者肿瘤分离的识别野生型抗原和有效肽模拟表位的t细胞受体的抗原发现
<130>stan-1407wo
<150>us62/476,575
<151>2017-03-24
<160>620
<170>patentinversion3.5
<210>1
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>1
leumetaspmethisasnglyglnleu
15
<210>2
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>2
argleuaspalametasnglyglnleu
15
<210>3
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>3
argmetasptyrasnasnmetglnmet
15
<210>4
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>4
sermetaspthrpheglnglyglnmet
15
<210>5
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>5
glymetasptyrhisasnglyhisleu
15
<210>6
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>6
tyrleuaspphehisasnglyglnleu
15
<210>7
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>7
leumetasptyrthrasnmetglnleu
15
<210>8
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>8
asnleuasptrpalaasnvalglnleu
15
<210>9
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>9
metmetaspleuhisasnglyglnleu
15
<210>10
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>10
lysmetasptyrhisgluglyglnleu
15
<210>11
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>11
thrleuaspglypheasnglyglnmet
15
<210>12
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>12
valmetserhisphegluglyglnleu
15
<210>13
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>13
alametasptyrleuasnalaglnleu
15
<210>14
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>14
glnleuasptrpasnasnmetglnmet
15
<210>15
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>15
argmetglytyrhisasnglyglnleu
15
<210>16
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>16
argmetaspargpheasnglyglnleu
15
<210>17
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>17
alametsertyraspasnmetglnleu
15
<210>18
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>18
valmetthrhisasnasnmetglnleu
15
<210>19
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>19
asnmetsertrpglnasnmetglnleu
15
<210>20
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>20
argmetaspvalasnasnmetglnleu
15
<210>21
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>21
asnleuasptrpasnasnvalglnmet
15
<210>22
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>22
gluleuasptrppheasnserglnleu
15
<210>23
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>23
cysmetaspvalpheasnglyglnleu
15
<210>24
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>24
glymetsertyrserasnmetglnleu
15
<210>25
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>25
sermetthrtrpmetasnglyglnleu
15
<210>26
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>26
sermetaspargpheasnglyglnmet
15
<210>27
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>27
valleuaspglnhisasnglyglnleu
15
<210>28
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>28
hismetasppheasnasnvalglnmet
15
<210>29
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>29
sermetsertrpmetasnglyglnleu
15
<210>30
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>30
metleuasptrpasnasnvalglnleu
15
<210>31
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>31
glumetaspvalhisasnglyglnmet
15
<210>32
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>32
lysmethistrppheasnglyglnleu
15
<210>33
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>33
sermetaspserleuasnglyglnleu
15
<210>34
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>34
valmetthrtyrglnasnglyglnleu
15
<210>35
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>35
valmetasphisleuasnglyglnleu
15
<210>36
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>36
trpmetserasppheglnglyglnleu
15
<210>37
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>37
argleuaspserpheasnglyglnleu
15
<210>38
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>38
sermetaspsertrpasnglyglnmet
15
<210>39
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>39
thrmetasptrphisserglyglnleu
15
<210>40
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>40
lysleuaspiletrpasnglyglnleu
15
<210>41
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>41
thrmetaspphetyrglnglyglnleu
15
<210>42
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>42
lysmetasptyrpheserglyglnleu
15
<210>43
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>43
tyrleuasptyrargasnmetglnleu
15
<210>44
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>44
glumetasphisleuasnmetglnleu
15
<210>45
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>45
hismetaspileasnasnmetglnmet
15
<210>46
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>46
serleuasptrppheasnserglnleu
15
<210>47
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>47
argmetasptrpleuglnalaglnleu
15
<210>48
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>48
pheleuasppheargasnglyglnmet
15
<210>49
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>49
glumetmettrptrpasnglyglnval
15
<210>50
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>50
thrmetglutrppheasnglyhisleu
15
<210>51
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>51
thrmetaspthrleuasnalaglnleu
15
<210>52
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>52
phemetaspserpheasnglyglnmet
15
<210>53
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>53
asnmetmettrppheglnglyglnleu
15
<210>54
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>54
asnmetglyphegluasnmetglnleu
15
<210>55
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>55
asnmetasptyrileasnvalglnleu
15
<210>56
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>56
glumetasptrpserasnleuglnleu
15
<210>57
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>57
leumetglyilehisasnglyglnleu
15
<210>58
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>58
glumetsertrppheserglyglnleu
15
<210>59
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>59
valmetaspleupheglnglyglnmet
15
<210>60
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>60
leuleuaspvalhisasnmetglnleu
15
<210>61
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>61
lysmetasptyrasnasnvalglnmet
15
<210>62
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>62
sermetasptyrasnasnvalglnmet
15
<210>63
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>63
leumetgluasnpheglnglyglnleu
15
<210>64
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>64
argmetserphehisasnglyglnleu
15
<210>65
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>65
sermetmettyrmetasnglyglnleu
15
<210>66
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>66
argmetglutrpglnasnalaglnleu
15
<210>67
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>67
valmetserhisglnasnmetglnleu
15
<210>68
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>68
metmetaspphepheaspglyglnmet
15
<210>69
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>69
ilemetserhisglnasnmetglnleu
15
<210>70
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>70
hismetgluphemetasnmetglnleu
15
<210>71
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>71
asnmetaspthrtyrasnglyglnmet
15
<210>72
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>72
asnleuasptyrthrasnglyglnleu
15
<210>73
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>73
sermetthrtrpgluasnmetglnleu
15
<210>74
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>74
alametthrphehisasnglyglnleu
15
<210>75
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>75
sermetaspphethrasnalaglnmet
15
<210>76
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>76
asnmetserthrargaspgluargmet
15
<210>77
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>77
sermetthrphegluasnmetglnleu
15
<210>78
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>78
glumetasptrptrpasnglyhisleu
15
<210>79
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>79
thrmetaspaspasnasnglyglnleu
15
<210>80
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>80
leumetaspgluasnasnmetglnleu
15
<210>81
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>81
glumetthrasntrpasnglyglnleu
15
<210>82
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>82
tyrmetasptyrhisasnglyhismet
15
<210>83
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>83
lysmetthrtrpasnasnmetglnmet
15
<210>84
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>84
tyrmetthrhisleuasnglyglnleu
15
<210>85
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>85
glumetthrtrpthrasnalaglnmet
15
<210>86
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>86
lysmetasnasnphegluglyglnleu
15
<210>87
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>87
metmetaspleutyrasnglyglnleu
15
<210>88
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>88
valleuaspasnasnasnmetglnleu
15
<210>89
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>89
lysleualatrppheasnglyglnleu
15
<210>90
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>90
asnleuasphisasnasnglyglnmet
15
<210>91
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>91
leumetaspasnserasnmetglnleu
15
<210>92
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>92
asnmetasptyrasnasnvalglnleu
15
<210>93
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>93
argmetasptyrasnasnvalglnmet
15
<210>94
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>94
glumetgluilemetasnmetglnleu
15
<210>95
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>95
tyrmetaspargpheglnglyglnleu
15
<210>96
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>96
tyrmetasnvalphegluglyglnleu
15
<210>97
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>97
leumetaspthrpheasnalaglnmet
15
<210>98
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>98
glymetasptyrhisasnglyglnleu
15
<210>99
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>99
metleuaspleutyrasnglyglnleu
15
<210>100
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>100
argleusertrppheglnglyglnleu
15
<210>101
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>101
valleuasnglypheaspglyglnleu
15
<210>102
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>102
sermetglytrpgluglnleuglnleu
15
<210>103
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>103
sermetthrtrpphethrglyglnleu
15
<210>104
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>104
trpmetaspileserasnmetglnleu
15
<210>105
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>105
thrmetglntrpglnasnalaglnleu
15
<210>106
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>106
sermetthrvalpheasnglyglnleu
15
<210>107
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>107
asnmetaspmethisasnmetglnleu
15
<210>108
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>108
argmetserserpheaspglyglnleu
15
<210>109
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>109
tyrmetserpheaspasnvalglnleu
15
<210>110
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>110
leumetserglypheaspglyglnleu
15
<210>111
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>111
tyrleuasptyrleuasnmetglnleu
15
<210>112
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>112
sermetasptyrasnasnileglnmet
15
<210>113
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>113
glymetaspthrhisasnglyglnleu
15
<210>114
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>114
leumetaspmethisasnglyhisleu
15
<210>115
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>115
serleuasntyrtrpgluglyglnleu
15
<210>116
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>116
alaleuasnhisphegluglyglnleu
15
<210>117
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>117
alametaspasnmetasnglyglnleu
15
<210>118
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>118
argmetglyilepheasnglyglnleu
15
<210>119
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>119
asnleuasptrpserasnalaglnleu
15
<210>120
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>120
argmetasphismetasnglyhisleu
15
<210>121
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>121
metmetserpropheasnglyglnleu
15
<210>122
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>122
thrmetasnsertrpasnglyglnleu
15
<210>123
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>123
sermetasntrpglnasnglyglnleu
15
<210>124
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>124
ilemetgluthrpheasnglyglnmet
15
<210>125
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>125
tyrleuaspasnasnasnmetglnmet
15
<210>126
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>126
glnmetaspleumetlysthrtyrleu
15
<210>127
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>127
glyleuasptrpileasnglyglnleu
15
<210>128
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>128
argleuthrtyrleuasnglyglnleu
15
<210>129
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>129
alametaspasptrpasnglyglnmet
15
<210>130
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>130
asnleuasptrpglnasnmetglnmet
15
<210>131
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>131
thrmetasptyrasnasnalaglnmet
15
<210>132
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>132
thrmetaspgluasnasnmetglnleu
15
<210>133
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>133
trpmetaspaspileasnglyglnleu
15
<210>134
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>134
metleuasptyrmetasnalaglnmet
15
<210>135
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>135
alametasplyshisasnglyglnmet
15
<210>136
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>136
lysmetasptrpargvalvalglnmet
15
<210>137
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>137
argmetasptyrthrasnmetglnleu
15
<210>138
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>138
argmetasphisserasnmetglnmet
15
<210>139
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>139
thrleugluilehisasnglyglnleu
15
<210>140
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>140
leumetaspmethisasnmetglnmet
15
<210>141
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>141
serleuthrtyrpheasnglyglnmet
15
<210>142
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>142
tyrmetaspmethisasnglyglnleu
15
<210>143
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>143
asnmetasparghisasnglyglnmet
15
<210>144
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>144
asnmetaspargasnasnmetglnleu
15
<210>145
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>145
thrleuaspvalhisasnmetglnleu
15
<210>146
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>146
argleuserthrphegluglyglnleu
15
<210>147
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>147
glnmetaspthrmetasnglyglnleu
15
<210>148
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>148
lysmetasptyrhisasnglyhisleu
15
<210>149
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>149
ilemetasptrpserasnvalglnmet
15
<210>150
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>150
lysleuaspalapheasnglyglnmet
15
<210>151
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>151
cysleusergluserleuglntrpval
15
<210>152
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>152
sermetcystyrglnasnmetglnleu
15
<210>153
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>153
leumetthrcysalaglyasnaspmet
15
<210>154
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>154
lysleuaspvalpheasnalaglnleu
15
<210>155
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>155
leumetasptyrasnasnmetglnmet
15
<210>156
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>156
tyrleuaspphehisasnglyhisleu
15
<210>157
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>157
alametaspmethisasnglyglnleu
15
<210>158
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>158
sermetasntyrtyraspglyglnleu
15
<210>159
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>159
tyrmetasptrpserasnserglnmet
15
<210>160
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>160
thrleuasphismetasnalaglnmet
15
<210>161
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>161
hismetasntyrpheaspglyglnmet
15
<210>162
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>162
thrleucystyrasnasnmetglnleu
15
<210>163
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>163
phemetaspasppheserglyglnleu
15
<210>164
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>164
glnleuasptrpasnasnvalglnleu
15
<210>165
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>165
thrleuasppheargasnmetglnleu
15
<210>166
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>166
valleuleuargaspalasertrpmet
15
<210>167
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>167
thrmetglutrppheasnglyglnmet
15
<210>168
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>168
phemetasppheasnserglyglnleu
15
<210>169
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>169
sermetaspmethisasnglyglnleu
15
<210>170
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>170
argleuglnaspileserglyvalmet
15
<210>171
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>171
gluleumetalatrpasnglyglnleu
15
<210>172
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>172
asnleuasptrpasnasnmetglnmet
15
<210>173
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>173
argmetasptyrleuasnalaglnleu
15
<210>174
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>174
phemetaspphehisasnglyglnleu
15
<210>175
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>175
metmetaspleuhisasnglyhisleu
15
<210>176
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>176
leumetaspthrpheglnglyglnmet
15
<210>177
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>177
alametaspphehisasnglyglnleu
15
<210>178
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>178
thrmetasppheserasnileglnleu
15
<210>179
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>179
glymetaspasphisasnmetglnleu
15
<210>180
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>180
lysmethistyrpheasnglyglnmet
15
<210>181
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>181
tyrmetasptyrhisasnglyglnleu
15
<210>182
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>182
argmetasptyrasnasnglyhisleu
15
<210>183
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>183
leumetasptyrhisgluglyglnleu
15
<210>184
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>184
argmetaspargpheasnglyglnmet
15
<210>185
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>185
argmetaspvalasnasnglyglnleu
15
<210>186
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>186
glymetaspthralaasnmetglnleu
15
<210>187
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>187
metleuasptyrmetasnglyglnleu
15
<210>188
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>188
lysmetthrphehisasnalaglnleu
15
<210>189
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>189
phemetasppheasnasnvalglnmet
15
<210>190
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>190
serleuasphispheglnglyhisleu
15
<210>191
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>191
thrmetaspphetyrglnglyglnleu
15
<210>192
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>192
lysmetasptyrpheserglyglnleu
15
<210>193
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>193
sermetasptrppheglnglyglnmet
15
<210>194
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>194
leumetasptyrtrpglnglyglnleu
15
<210>195
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>195
asnmetmettrppheglnglyglnleu
15
<210>196
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>196
lysmethistrppheasnglyglnleu
15
<210>197
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>197
thrmetasptyrtrpglnglyhisleu
15
<210>198
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>198
argmetaspargpheasnglyglnleu
15
<210>199
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>199
sermetaspthrpheglnglyglnmet
15
<210>200
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>200
valmetserhisphegluglyglnleu
15
<210>201
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>201
leumetasptyrthrasnmetglnleu
15
<210>202
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>202
lysmetasptyrhisileglyglnmet
15
<210>203
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>203
valmetasphispheglnalaglnleu
15
<210>204
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>204
asnmetglyphegluasnmetglnleu
15
<210>205
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>205
tyrleuasphislysthrleuargleu
15
<210>206
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>206
thrmetasptyrtrpglnglyglnleu
15
<210>207
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>207
lysmetargmetasnarghislysleu
15
<210>208
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>208
tyrmetaspargpheglnglyglnmet
15
<210>209
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>209
sermetaspphepheasnserglnleu
15
<210>210
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>210
asnmetgluglutyrcysalaleuval
15
<210>211
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>211
sermetaspphetyrglnglyglnleu
15
<210>212
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>212
sermetasptrppheglnglyglnleu
15
<210>213
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>213
asnmetmettrppheglnglyglnmet
15
<210>214
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>214
alamettyrlysleuserglyleumet
15
<210>215
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>215
hismetglutyrargtyralaasnmet
15
<210>216
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>216
leumetasptyrpheserglyglnleu
15
<210>217
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>217
thrmetasptrppheglnglyglnmet
15
<210>218
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>218
phemetservalalalysphevalval
15
<210>219
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>219
argleuasptyrhisasnmetglnleu
15
<210>220
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>220
leumetaspphetyrglnglyglnleu
15
<210>221
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>221
leumetasptyrtrpglnglyhisleu
15
<210>222
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>222
thrmetaspphetyrglnglyglnmet
15
<210>223
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>223
lysmetleuserileaspvalvalmet
15
<210>224
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>224
sermetasptyrpheserglyglnleu
15
<210>225
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>225
lysmetlysasnhishisthrlysval
15
<210>226
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>226
sermetasptyrtrpglnglyglnleu
15
<210>227
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>227
lysleuhisarghislysglnhismet
15
<210>228
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>228
leumetasptrppheglnglyglnmet
15
<210>229
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>229
lysmetthrsertrptrpaspmetleu
15
<210>230
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>230
aspmetasptrppheglnglyglnmet
15
<210>231
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>231
metleutyrgluleuthrgluhisleu
15
<210>232
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>232
sermetasptrppheasnglyglnleu
15
<210>233
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>233
argleuhisargargaspasnleumet
15
<210>234
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>234
aspmetasptyrtrpglnglyglnleu
15
<210>235
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>235
lysmetasptyrthrasnmetglnleu
15
<210>236
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>236
thrmetasptyrtrpglnglyglnmet
15
<210>237
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>237
phemetglyvalsertyrglumetmet
15
<210>238
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>238
leumetasptyrtrpglnglyglnmet
15
<210>239
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>239
sermetaspthrpheglnglyglnleu
15
<210>240
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>240
lysmethisglyhislyshistyrmet
15
<210>241
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>241
lysmethistrppheglnglyglnmet
15
<210>242
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>242
serleuasptyrpheasnserglnleu
15
<210>243
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>243
tyrmetaspargpheglnglyglnleu
15
<210>244
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>244
argmettrpseraspargmetaspleu
15
<210>245
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>245
lysmetasptyrpheasnserglnleu
15
<210>246
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>246
tyrmethisserhisservalleuleu
15
<210>247
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>247
aspmetasptyrpheserglyglnleu
15
<210>248
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>248
sermetasptrppheglnglyhisleu
15
<210>249
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>249
valmetaspleupheglnglyglnmet
15
<210>250
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>250
asnmetglusertrpleusermetmet
15
<210>251
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>251
argmetaspargpheglnglyglnmet
15
<210>252
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>252
sermetgluileserasnleuasnmet
15
<210>253
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>253
aspmetgluargalaleumetasnleu
15
<210>254
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>254
aspmetaspthrpheglnglyglnmet
15
<210>255
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>255
lysmetlyslysasnhisasphismet
15
<210>256
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>256
lysmetargglumetprovallysmet
15
<210>257
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>257
metmetaspphepheasnalaglnmet
15
<210>258
<211>210
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>258
glnlysvalthrglnalaglnthrgluileservalvalglulysglu
151015
aspvalthrleuaspcysvaltyrgluthrargaspthrthrtyrtyr
202530
leuphetrptyrlysglnproproserglygluleuvalpheleuile
354045
argargasnserpheaspgluglnasngluileserglyargtyrser
505560
trpasnpheglnlysserthrserserpheasnphethrilethrala
65707580
serglnvalvalaspseralavaltyrphecysalaleusergluala
859095
argglyglyalathrasnlysleuilepheglythrglythrleuleu
100105110
alavalglnproasnileglnasnproaspproalavaltyrglnleu
115120125
argaspserlysserserasplysservalcysleuphethraspphe
130135140
aspserglnthrasnvalserglnserlysaspseraspvaltyrile
145150155160
thrasplyscysvalleuaspmetargsermetaspphelysserasn
165170175
seralavalalatrpserasnlysseraspphealacysalaasnala
180185190
pheasnasnserileileprogluaspthrphepheproserproglu
195200205
serser
210
<210>259
<211>241
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>259
gluilethrglnserproarghislysilethrgluthrglyarggln
151015
valthrleualacyshisglnthrtrpasnhisasnasnmetphetrp
202530
tyrargglnaspleuglyhisglyleuargleuilehistyrsertyr
354045
glyvalglnaspthrasnlysglygluvalseraspglytyrserval
505560
serargserasnthrgluaspleuproleuthrleugluseralaala
65707580
serserglnthrservaltyrphecysalaserserargaspthrval
859095
asnthrglualaphepheglyglnglythrargleuthrvalvalglu
100105110
aspleulysasnvalpheproprogluvalalavalphegluproser
115120125
glualagluileserhisthrglnlysalathrleuvalcysleuala
130135140
thrglyphetyrproasphisvalgluleusertrptrpvalasngly
145150155160
lysgluvalhisserglyvalcysthraspproglnproleulysglu
165170175
glnproalaleuasnaspserargtyralaleuserserargleuarg
180185190
valseralathrphetrpglnasnproargasnhispheargcysgln
195200205
valglnphetyrglyleusergluasnaspglutrpthrglnasparg
210215220
alalysprovalthrglnilevalseralaglualatrpglyargala
225230235240
asp
<210>260
<211>241
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>260
gluilethrglnserproarghislysilethrgluthrglyarggln
151015
valthrleualacyshisglnthrtrpasnhisasnasnmetphetrp
202530
tyrargglnaspleuglyhisglyleuargleuilehistyrsertyr
354045
glyvalglnaspthrasnlysglygluvalseraspglytyrserval
505560
serargserasnthrgluaspleuproleuthrleugluseralaala
65707580
serserglnthrservaltyrphecysalaserserargasppheval
859095
serasngluglntyrpheglyproglythrargleuthrvalthrglu
100105110
aspleulysasnvalpheproprogluvalalavalphegluproser
115120125
glualagluileserhisthrglnlysalathrleuvalcysleuala
130135140
thrglyphetyrproasphisvalgluleusertrptrpvalasngly
145150155160
lysgluvalhisserglyvalcysthraspproglnproleulysglu
165170175
glnproalaleuasnaspserargtyralaleuserserargleuarg
180185190
valseralathrphetrpglnasnproargasnhispheargcysgln
195200205
valglnphetyrglyleusergluasnaspglutrpthrglnasparg
210215220
alalysprovalthrglnilevalseralaglualatrpglyargala
225230235240
asp
<210>261
<211>16
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>261
cysalaleuserglualaargglyglyalathrasnlysleuilephe
151015
<210>262
<211>14
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>262
cysalaserserargaspthrvalasnthrglualaphephe
1510
<210>263
<211>14
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>263
cysalaserserargaspphevalserasngluglntyrphe
1510
<210>264
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>264
gluleualaglyileglyileleuthrval
1510
<210>265
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>265
alaleuaspprohisserglyhispheval
1510
<210>266
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<220>
<221>misc_feature
<222>(10)..(10)
<223>位置10处的xaa是hla-a*02:01。
<400>266
metmetaspphepheasnalaglnmetxaa
1510
<210>267
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>267
glualaalaglyileglyileleuthrval
1510
<210>268
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>268
alaleuleugluthrproserleuleuleu
1510
<210>269
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>269
alaleuaspserargsergluhisphemet
1510
<210>270
<211>8
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>270
glutyrglyvalsertyrglutrp
15
<210>271
<211>8
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>271
glumetglyvalsertyrglumet
15
<210>272
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>272
leuleugluaspleuasptrpaspval
15
<210>273
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>273
asnmetglutyrmetthrtrpaspval
15
<210>274
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>274
thrmetgluthrileasptrplysval
15
<210>275
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>275
valleuglugluvalasptrpleuile
15
<210>276
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>276
lysleugluglnleuasptrpthrval
15
<210>277
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>277
thrleuglugluleuasptrpcysleu
15
<210>278
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>278
asnvalglutyrtyraspilelysleu
15
<210>279
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>279
metmetaspphepheasnalaglnmet
15
<210>280
<211>8
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>280
valleuasppheglnglyglnleu
15
<210>281
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<220>
<221>misc_feature
<222>(10)..(10)
<223>位置10处的xaa是hla-a*02:01
<400>281
metmetaspphepheasnalaglnmetxaa
1510
<210>282
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<220>
<221>misc_feature
<222>(10)..(10)
<223>位置10处的xaa是hla-a2。
<400>282
metmetaspphepheasnalaglnmetxaa
1510
<210>283
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>283
sermetleuglyileglyilevalproval
1510
<210>284
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>284
sermetalaglyileglyilevalaspval
1510
<210>285
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>285
asnmetglyglyleuglyilemetproval
1510
<210>286
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>286
asnleuserasnleuglyileleuproval
1510
<210>287
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>287
sermetleuglyileglyiletyrproval
1510
<210>288
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>288
thrmetalaglyileglyvalhisvalval
1510
<210>289
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>289
sermetalaglyileglythrleuvalval
1510
<210>290
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>290
sermetserglyleuglyileleupromet
1510
<210>291
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>291
sermetalaglyileglyilevalproval
1510
<210>292
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>292
sermetleuglyileglyilevalaspval
1510
<210>293
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>293
asnmetalaglyileglymetglythrval
1510
<210>294
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>294
sermetleuglyileglyileleuproval
1510
<210>295
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>295
serleuserglyileglyileseralaval
1510
<210>296
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>296
aspleualaglyleuglyleutyrproval
1510
<210>297
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>297
asnmetalaglyileglyileileglnval
1510
<210>298
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>298
asnmetglyglyleuglyileleuproval
1510
<210>299
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>299
sermetalaglyileglyiletyrproval
1510
<210>300
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>300
asnleuserasnleuglyilevalproval
1510
<210>301
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>301
ilemetleuglyileglyileaspthrleu
1510
<210>302
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>302
asnleuserasnleuglyilemetproval
1510
<210>303
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>303
sermetleuglyileglyilevalleuval
1510
<210>304
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>304
sermetalaglyileglyvalhisvalval
1510
<210>305
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>305
asnmetalaglyileglyileleuthrval
1510
<210>306
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>306
metmetalaglyileglyilevalaspval
1510
<210>307
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>307
asnmetglyglyleuglyilevalproval
1510
<210>308
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>308
sermetleuglyilelysilevalproval
1510
<210>309
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>309
gluleuserglyleuglyileglnthrval
1510
<210>310
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>310
sermetleuglyileglyileleupromet
1510
<210>311
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>311
sermetalaglyileglyileleuproval
1510
<210>312
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>312
sermetleuglyileglyilevalproval
1510
<210>313
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>313
glualaalaglyileglyileleuthrval
1510
<210>314
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>314
thrleuglyglyileglyleuvalthrval
1510
<210>315
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>315
ileleuleuglyileglyiletyralaleu
1510
<210>316
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>316
ileleuserglyileglyvalserglnval
1510
<210>317
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>317
ilemetglyasnleuglyleuilealaval
1510
<210>318
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>318
metalaglyasnleuglyileilethrleu
1510
<210>319
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>319
ilemetglyasnleuglyleuilevalleu
1510
<210>320
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>320
ileleualaglyleuglythrserleuleu
1510
<210>321
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>321
gluleuglyglyleulysileserthrleu
1510
<210>322
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>322
metmettrpaspargglymetglyleuleu
1510
<210>323
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>323
ilemetgluaspvalglytrpleuasnval
1510
<210>324
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>324
metmettrpaspargglyleuglymetmet
1510
<210>325
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>325
ileleugluaspargglypheasnglnval
1510
<210>326
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>326
leumetpheaspargglymetserleuleu
1510
<210>327
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>327
leumetleuasppheaspglyserleuleu
1510
<210>328
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>328
ilemetgluaspargglyserleuasnmet
1510
<210>329
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>329
leumetasnaspmetglyphehisileval
1510
<210>330
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>330
ilemetgluaspargglyserglyglumet
1510
<210>331
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>331
leumettrpaspvalglyleuserilemet
1510
<210>332
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>332
sermettrpaspargglythrpheilemet
1510
<210>333
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>333
leumetleuaspargglyserproasnmet
1510
<210>334
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>334
ilemetpheaspargglyileglyilemet
1510
<210>335
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>335
ileleupheaspargglymetasnleumet
1510
<210>336
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>336
metleuleuaspargglyleuserleumet
1510
<210>337
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>337
ilemetgluaspargglyserleuileleu
1510
<210>338
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>338
leumetargasptyrglnleuleuglnval
1510
<210>339
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>339
leumetpheaspargglymetservalleu
1510
<210>340
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>340
leumetgluaspileglyarggluleuval
1510
<210>341
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>341
ileleugluaspargglymetglyleuleu
1510
<210>342
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>342
metmetaspglnpheasnglyleumetmet
1510
<210>343
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>343
ilemettrpaspargasptyrglyvalmet
1510
<210>344
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>344
metmettrpaspargglypheasnglnval
1510
<210>345
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>345
ilemetsermetservalserasntyrleu
1510
<210>346
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>346
alametglyaspglysertyrleuleumet
1510
<210>347
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>347
sermettrpaspargglymetglyleuleu
1510
<210>348
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>348
metmetgluasnargglyserglyalaleu
1510
<210>349
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>349
leumettrpaspserglyleugluleumet
1510
<210>350
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>350
sermettrpaspargglyleuglymetmet
1510
<210>351
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>351
leumettrpaspvalglytrpleuasnval
1510
<210>352
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>352
metmettrpaspargglythrpheilemet
1510
<210>353
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>353
metmettrpaspargglyilevalproval
1510
<210>354
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>354
ileleupheaspargglymetasnleumet
1510
<210>355
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>355
metleutrpaspvalglnserglyglnmet
1510
<210>356
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>356
leuleuleuglnvalglyleuserleuleu
1510
<210>357
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>357
serleugluaspvalvalmetleuasnval
1510
<210>358
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>358
metleugluaspargaspleuphevalmet
1510
<210>359
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>359
metleugluaspmetserleuglyilemet
1510
<210>360
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>360
serleugluasnargglyleusermetleu
1510
<210>361
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>361
ileleuaspaspglyglypheleuleumet
1510
<210>362
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>362
leuleutrpasnpheglyleuleuileval
1510
<210>363
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>363
leuleupheaspileserpheleumetleu
1510
<210>364
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>364
ilemetglyaspargasnargasnleuleu
1510
<210>365
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>365
valmetileserhisgluasnphemet
15
<210>366
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>366
thrmetglnserhisgluvalmetleu
15
<210>367
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>367
thrmetglnserhisgluasnphemet
15
<210>368
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>368
valmetglnserhisgluvalmetleu
15
<210>369
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>369
valmetileserhisgluilepheleu
15
<210>370
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>370
ilemetthrserhisgluvalmetleu
15
<210>371
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>371
ilemetthrserhisgluvalmetmet
15
<210>372
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>372
valmetgluserhisaspvalphemet
15
<210>373
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>373
ilemetasnserhisgluvalmetmet
15
<210>374
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>374
sermetasnserhisgluvalmetmet
15
<210>375
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>375
lysmetasnserhisgluvalmetmet
15
<210>376
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>376
alametglnglyhisglutyrpheleu
15
<210>377
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>377
alametglnglyhisgluilepheleu
15
<210>378
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>378
valleuglnserhisgluvalsermet
15
<210>379
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>379
alametglnserhisgluvalthrleu
15
<210>380
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>380
leumetserglyasptyrglnpheval
15
<210>381
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>381
thrmethisasnhisgluvalmetmet
15
<210>382
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>382
valmethisasnhisgluvalmetmet
15
<210>383
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>383
thrmetthrglyhisgluvalphemet
15
<210>384
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>384
thrmetthrglyhisgluvalpheval
15
<210>385
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>385
valmetglnglyhisgluserpheleu
15
<210>386
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>386
valmetileserhisgluvalmetleu
15
<210>387
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>387
thrmetthrglyhisgluvalmetleu
15
<210>388
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>388
sermetvalglymetgluhissermet
15
<210>389
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>389
alametglnglyhisgluhisphemet
15
<210>390
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>390
valmetgluglyasptyrtrppheleu
15
<210>391
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>391
sermetglnserhisglutrpmetleu
15
<210>392
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>392
tyrmetglnthrhisgluserphemet
15
<210>393
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>393
valmetasnglyaspserglythrpheleu
1510
<210>394
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>394
tyrmetalavalargsergluasnphemet
1510
<210>395
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>395
argmetproasnlysglngluasnpheval
1510
<210>396
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>396
ilemetaspserlyssergluhisphemet
1510
<210>397
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>397
ilemetaspserargglugluvalpheval
1510
<210>398
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>398
ilemetaspserargsergluhisphemet
1510
<210>399
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>399
glymetaspserargalagluvalphemet
1510
<210>400
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>400
alaleuaspserargserglutyrpheleu
1510
<210>401
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>401
lysmetalaasnargaspgluasnpheval
1510
<210>402
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>402
argleuaspglyglnaspthrlysphemet
1510
<210>403
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>403
leumetaspserargsergluhisphemet
1510
<210>404
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>404
ilemetasnserargsergluleupheleu
1510
<210>405
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>405
metmetasnvalargsergluleupheval
1510
<210>406
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>406
thrmetasnvalargsergluleupheval
1510
<210>407
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>407
lysmetasnserargsergluleupheleu
1510
<210>408
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>408
thrmetasnvalargsergluhisphemet
1510
<210>409
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>409
sermetasnserargsergluleupheleu
1510
<210>410
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>410
lysmetasnserargsergluhisphemet
1510
<210>411
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>411
thrmetglnserhisaspalaserpheleu
1510
<210>412
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>412
valmetglnglyhisaspalaserpheleu
1510
<210>413
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>413
lysmetasnserhisserglythrpheleu
1510
<210>414
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>414
lysmetasnglylyssergluaspphemet
1510
<210>415
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>415
aspmetaspasnargleuaspargaspmet
1510
<210>416
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>416
ilemetaspserlyssergluilepheleu
1510
<210>417
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>417
sermetasnserhisserglythrpheleu
1510
<210>418
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>418
sermetasnserargglugluhisphemet
1510
<210>419
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>419
ilemetasnserhisserglythrpheleu
1510
<210>420
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>420
ilemetaspserlyssergluasnpheleu
1510
<210>421
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>421
alametaspserlyssergluasnpheleu
1510
<210>422
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>422
ilemetaspserargalaaspmetpheval
1510
<210>423
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>423
sermetasnserargglugluvalpheval
1510
<210>424
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>424
lysmetasnserargglugluvalpheval
1510
<210>425
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>425
alaleuaspserargsergluhisphemet
1510
<210>426
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>426
alametaspserargsergluhisphemet
1510
<210>427
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>427
alametaspserargalaaspmetpheval
1510
<210>428
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>428
leumetaspserargserglnilepheval
1510
<210>429
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>429
glymetthrserargserasptyrmetval
1510
<210>430
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>430
valmetasnserargsergluhisphemet
1510
<210>431
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>431
valmetasnserargserasptrppheleu
1510
<210>432
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>432
tyrmetasnserhisaspprotyrthrval
1510
<210>433
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>433
argmetaspserargserglnasppheval
1510
<210>434
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>434
argmetglualahisserserhispheval
1510
<210>435
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>435
thrleumetserargseraspleupheleu
1510
<210>436
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>436
ileleuasnserargaspglualametmet
1510
<210>437
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>437
alaleuasnserargaspglualametmet
1510
<210>438
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>438
alaleuaspserargleugluphepheval
1510
<210>439
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>439
valmetaspserargleugluphepheval
1510
<210>440
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>440
alaleuaspserargsergluleupheleu
1510
<210>441
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>441
alamettyrserasnseraspphemetval
1510
<210>442
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>442
valmetaspserargleugluhisphemet
1510
<210>443
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>443
sermetasnserargsergluhisphemet
1510
<210>444
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>444
sermetasnserlyssergluasnpheleu
1510
<210>445
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>445
valleuaspserserserserserpheleu
1510
<210>446
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>446
alaleuaspserargsergluasnpheleu
1510
<210>447
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>447
alaleuaspserlyssergluasnpheleu
1510
<210>448
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>448
alaleuaspserargsergluilepheleu
1510
<210>449
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>449
sermetasnserargalaaspmetpheval
1510
<210>450
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>450
sermettyrserargglnglumetmetval
1510
<210>451
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>451
argmettrpserargsergluaspmetval
1510
<210>452
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>452
valleuargalaargseraspvalpheval
1510
<210>453
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>453
alaleuaspserargglugluvalpheval
1510
<210>454
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>454
sermetasnserargglugluilepheleu
1510
<210>455
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>455
sermetserglyphesergluserpheval
1510
<210>456
<211>11
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>456
ileleuserasnargglyhisgluvalpheval
1510
<210>457
<211>11
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>457
ileleuserasnargglyhisgluasnphemet
1510
<210>458
<211>11
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>458
ileleuserasnargglyhisaspvalphemet
1510
<210>459
<211>11
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>459
ileleuserasnargglyhisgluilepheleu
1510
<210>460
<211>11
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>460
ileleuserasnargglyhisglutyrpheleu
1510
<210>461
<211>11
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>461
cysalaserserleuglyleugluglnphephe
1510
<210>462
<211>14
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>462
cysalaserserleuglyglyglyhisthrglualaphephe
1510
<210>463
<211>13
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>463
cysalaserserleuvalasnglyleuglytyrthrphe
1510
<210>464
<211>15
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>464
cysalathrserargaspargglyglnaspglulysleuphephe
151015
<210>465
<211>15
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>465
cysalaserseralaaspthrglyvalasnglnproglnhisphe
151015
<210>466
<211>14
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>466
cysalaserserargaspthrvalasnthrglualaphephe
1510
<210>467
<211>14
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>467
cysseralaargasptyrglnglyserglnproglnhisphe
1510
<210>468
<211>14
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>468
cysseralaargasptyrglnglyserglnproglnhisphe
1510
<210>469
<211>15
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>469
cysalaserseralaaspthrglyvalasnglnproglnhisphe
151015
<210>470
<211>12
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>470
cysalaglyglyglyglyalaaspglyleuthrphe
1510
<210>471
<211>15
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>471
cysalaleuserglualaglualaalaglyasnlysleuthrphe
151015
<210>472
<211>16
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>472
cysalaleuserglualaglymetaspserasntyrglnleuiletrp
151015
<210>473
<211>18
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>473
cysalametarggluglyargtyrserglyalaglysertyrglnleu
151015
thrphe
<210>474
<211>13
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>474
cysvalvalthrgluthrasnalaglylysserthrphe
1510
<210>475
<211>16
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>475
cysalaleuserglualaargglyglyalathrasnlysleuilephe
151015
<210>476
<211>13
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>476
cysalavalasnserglyasnthrglylysleuilephe
1510
<210>477
<211>15
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>477
cysalavalpropheleutyrasnglnglyglylysleuilephe
151015
<210>478
<211>11
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>478
cysalavalasnasppheasnlysphetyrphe
1510
<210>479
<211>16
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>479
cysalaserserglnglyvalglyglnphelysasnthrglntyrphe
151015
<210>480
<211>17
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>480
cysalaserserleuserglyargglnglyglysertyrgluglntyr
151015
phe
<210>481
<211>15
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>481
cysalaserserserserglyglyleuvalaspthrglntyrphe
151015
<210>482
<211>12
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>482
cysalasermetglyargsertyrglytyrthrphe
1510
<210>483
<211>16
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>483
cysalaserserleugluthrglythralailetyrgluglntyrphe
151015
<210>484
<211>19
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>484
cysalaserserproserglyleualaglyserasnleuglyasnglu
151015
glnphephe
<210>485
<211>14
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>485
cysalaserserargileaspserthraspthrglntyrphe
1510
<210>486
<211>15
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>486
cysalaserserileproargglyserserglnproglnhisphe
151015
<210>487
<211>16
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>487
cysalailelysglyglyaspargglyvalasnthrglualaphephe
151015
<210>488
<211>13
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>488
cysseralaargleualasertyrasngluglnphephe
1510
<210>489
<211>14
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>489
cysalaserserargaspphevalserasngluglntyrphe
1510
<210>490
<211>13
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>490
cysalavalgluthrserasnthrglylysleuilephe
1510
<210>491
<211>12
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>491
cysalaalaserserthrglyasnglnphetyrphe
1510
<210>492
<211>17
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>492
cysalaleuseralaglyalaserglyalaglysertyrglnleuthr
151015
phe
<210>493
<211>15
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>493
cysalaleumetasntyrglyglyalathrasnlysleuilephe
151015
<210>494
<211>12
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>494
cysalaalaaspasnasnasnalaargleumetphe
1510
<210>495
<211>15
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>495
cysalaleuserserargglyserthrleuglyargleutyrphe
151015
<210>496
<211>14
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>496
cysleuvalglygluvalglythralaserlysleuthrphe
1510
<210>497
<211>13
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>497
cysalavalaspserglyglytyrasnlysleuilephe
1510
<210>498
<211>15
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>498
cysalametarggluproasnasnalaglyasnmetleuthrphe
151015
<210>499
<211>15
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>499
cysalavalargargalathraspsertrpglylysleuglnphe
151015
<210>500
<211>16
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>500
cysalaleuserglualaargglyglyalathrasnlysleuilephe
151015
<210>501
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>501
tyrleualaproglnglusertyrglyala
1510
<210>502
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>502
tyralasersertyrileileleualamet
1510
<210>503
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>503
valmetleuglnileileasnilevalleu
1510
<210>504
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>504
valleusertrpleuleulystyrlysile
1510
<210>505
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>505
servalleuasntyrphelysprotyrleu
1510
<210>506
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>506
serleumetthrproasnthrilethrmet
1510
<210>507
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>507
argvalleuserhisaspserilepheile
1510
<210>508
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>508
asnleuasnproasnvalaspproglnval
1510
<210>509
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>509
leuleuglngluglualahisvalproleu
1510
<210>510
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>510
leuiletyrgluleutyrvalsergluleu
1510
<210>511
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>511
lysthrtyrileilephephevalleuval
1510
<210>512
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>512
lysleutyrglyleuasptrpalagluleu
1510
<210>513
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>513
lysleupheglupheleuvaltyrglyval
1510
<210>514
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>514
ilevalalaalaaspleuilemetthrleu
1510
<210>515
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>515
ileglntyrleugluleuasnargleuval
1510
<210>516
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>516
ileglnvaltrpglualaleuleuthrleu
1510
<210>517
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>517
ileleuserglyglyargthrleuglnile
1510
<210>518
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>518
hisvalmetleuglnileileasnileval
1510
<210>519
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>519
hismetmetglypheargthrglngluval
1510
<210>520
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>520
hisiletyrileglyilehismetcysval
1510
<210>521
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>521
glymettyralasersertyrileileleu
1510
<210>522
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>522
glyleuleuprovalleusertrpleuleu
1510
<210>523
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>523
pheasnglnleuiletyrgluleutyrval
1510
<210>524
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>524
phemetthrlysileasnaspleugluval
1510
<210>525
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>525
pheleuvaltyrglyvalargproglymet
1510
<210>526
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>526
pheleuprovalthraspalaserserval
1510
<210>527
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>527
phealaleuleuglngluglualahisval
1510
<210>528
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>528
phealaleuglyasnvalileseralaleu
1510
<210>529
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>529
aspleusertyrthrtrpasnileproval
1510
<210>530
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>530
alavalphetyrthrileleuthrproval
1510
<210>531
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>531
alathrleuasptrpserlysasnalaval
1510
<210>532
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>532
alasermetthrglyilevaltyrserleu
1510
<210>533
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>533
alaleuleugluthrproserleuleuleu
1510
<210>534
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>534
alaleuaspprohisserglyhispheval
1510
<210>535
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>535
alaleualaphethrprovalgluglnval
1510
<210>536
<211>48
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>合成核苷酸
<400>536
tgtgctctgagtgaggcgaggggtggtgctacaaacaagctcatcttt48
<210>537
<211>48
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>合成核苷酸
<400>537
tgtgctctgagtgaggcgcggggcggtgctacaaacaagctcatcttt48
<210>538
<211>42
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>合成核苷酸
<400>538
tgcgccagcagccgggacactgttaatactgaagctttcttt42
<210>539
<211>42
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>合成核苷酸
<400>539
tgcgccagcagtcgggacttcgtgtccaacgagcagtacttc42
<210>540
<211>8
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>540
sermetglyvalthrtyrglumet
15
<210>541
<211>8
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>541
tyrmetglyvalsertyrglumet
15
<210>542
<211>8
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>542
tyrmetglyvalvaltyrglumet
15
<210>543
<211>8
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>543
lysmetglyvalthrtyrglumet
15
<210>544
<211>8
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>544
phemetglyvalthrtyrglumet
15
<210>545
<211>8
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>545
asnmetgluvalthrtyrgluile
15
<210>546
<211>8
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>546
pheilethrvalthrglugluile
15
<210>547
<211>8
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>547
hisileglnvalthrasngluile
15
<210>548
<211>8
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>548
hisleuilevalsertyrgluleu
15
<210>549
<211>8
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>549
hisleuglyvalthrlysgluleu
15
<210>550
<211>8
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>550
argleuglyvalthrtyrpheval
15
<210>551
<211>8
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>551
tyrleuprovalthrtyrhisile
15
<210>552
<211>8
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>552
glyleuglyglnthrtyrgluile
15
<210>553
<211>8
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>553
glutyrglyvalsertyrglutrp
15
<210>554
<211>8
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>554
glutyrglyvalglnasntyrval
15
<210>555
<211>8
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>555
glumetglyvalsertyrglumet
15
<210>556
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>556
leumetaspmethisasnglyglnleu
15
<210>557
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>557
argleuaspalametasnglyglnleu
15
<210>558
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>558
argmetasptyrasnasnmetglnmet
15
<210>559
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>559
sermetaspthrpheglnglyglnmet
15
<210>560
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>560
glymetasptyrhisasnglyhisleu
15
<210>561
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>561
thrmetaspphetyrglnglyglnleu
15
<210>562
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>562
lysmetasptyrpheserglyglnleu
15
<210>563
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>563
sermetasptrppheglnglyglnmet
15
<210>564
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>564
leumetasptyrtrpglnglyglnleu
15
<210>565
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>565
asnmetmettrppheglnglyglnleu
15
<210>566
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>566
valleuaspleupheglnglyglnleu
15
<210>567
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>567
metmetaspphepheasnalaglnmet
15
<210>568
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>568
leuleuasnleuasnasnglyglnleu
15
<210>569
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>569
glnmetasptyrglugluglyglnleu
15
<210>570
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>570
glyleuserserglnasnglyglnleu
15
<210>571
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>571
thrleuhistyrtyrglumethisleu
15
<210>572
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>572
valileasppheleuasnasnglnleu
15
<210>573
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>573
valileaspglnleuasnglyglnleu
15
<210>574
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>574
valvalasppheleulysglyglnleu
15
<210>575
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>575
leumetaspmethisasnglyglnleu
15
<210>576
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>576
argleuaspalametasnglyglnleu
15
<210>577
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>577
argmetasptyrasnasnmetglnmet
15
<210>578
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>578
sermetaspthrpheglnglyglnmet
15
<210>579
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>579
glymetasptyrhisasnglyhisleu
15
<210>580
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>580
thrmetaspphetyrglnglyglnleu
15
<210>581
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>581
lysmetasptyrpheserglyglnleu
15
<210>582
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>582
sermetasptrppheglnglyglnmet
15
<210>583
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>583
leumetasptyrtrpglnglyglnleu
15
<210>584
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>584
asnmetmettrppheglnglyglnleu
15
<210>585
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>585
valleuaspleupheglnglyglnleu
15
<210>586
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>586
metmetaspphepheasnalaglnmet
15
<210>587
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>587
leuleuasnleuasnasnglyglnleu
15
<210>588
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>588
metmetaspphepheasnalaglnmet
15
<210>589
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>589
leuleuasnleuasnasnglyglnleu
15
<210>590
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>590
glnmetasptyrglugluglyglnleu
15
<210>591
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>591
glyleuserserglnasnglyglnleu
15
<210>592
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>592
thrleuhistyrtyrglumethisleu
15
<210>593
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>593
valileasppheleuasnasnglnleu
15
<210>594
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>594
valileaspglnleuasnglyglnleu
15
<210>595
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>595
valvalasppheleulysglyglnleu
15
<210>596
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>596
argmetgluglnvalasptrpthrval
15
<210>597
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>597
lysleugluphemetasptrpargleu
15
<210>598
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>598
trpleuaspasnphegluleucysleu
15
<210>599
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>599
thrleuglutyrmetasptrpleuval
15
<210>600
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>600
glumetmetleupheasptrplysval
15
<210>601
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>601
lysleugluglnleuasptrpthrval
15
<210>602
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>602
thrmetgluthrileasptrplysval
15
<210>603
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>603
aspleugluglnmetgluglnthrval
15
<210>604
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>604
thrleuglugluleuasptrpcysleu
15
<210>605
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>605
thrleugluaspmetalatrpargleu
15
<210>606
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>606
asnvalgluglumetasptrpleuile
15
<210>607
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>607
asnvalgluglumetasptrpmetval
15
<210>608
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>608
leuleugluaspleuasptrpaspval
15
<210>609
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>609
thrleuglualametasnthrthrval
15
<210>610
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>610
valleuglugluvalasptrpleuile
15
<210>611
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>611
trpleugluaspvalglutrpglnval
15
<210>612
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>612
lysmetgluasnpheasplysthrval
15
<210>613
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>613
asnmetglutyrmetthrtrpaspval
15
<210>614
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>614
phevalgluasnvalglutrpargval
15
<210>615
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>615
asnvalglutyrtyraspilelysleu
15
<210>616
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>616
hisleugluglnvalasplysalaval
15
<210>617
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>617
glumetgluglnvalaspalavalval
15
<210>618
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>618
sermetgluglnphethrvalargval
15
<210>619
<211>9
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>619
hismetasnasnvalthrvalthrleu
15
<210>620
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成多肽
<400>620
trpleuileaspmetlysserleuvalmet
1510
- 还没有人留言评论。精彩留言会获得点赞!