生物标志物作为靶标在肺腺癌诊治中的应用的制作方法
本发明属于生物医药领域,涉及生物标志物作为靶标在肺腺癌诊治中的应用,具体的所述生物标志物为CLINT1。
背景技术:
肺癌是全球范围内发病率及死亡率最高的肿瘤之一,严重危害人类健康。肺癌可以分为非小细胞肺癌(non small cell lung cancer,NSCLC)和小细胞肺癌(small cell lung cancer,SCLC)。其中NSCLC约占肺癌总数的80%-85%,其病情发展迅速,转移速度快,复发率较高。大部分病人发现时已发展至中晚期,预后差,5年生存率不足15%。早期接受治疗的NSCLC患者复发率达40%,且70%以上患者被发现时已是局部侵犯或远隔转移而无法手术。
目前,外科手术、化疗、放疗仍是肺癌的主要治疗方式。近年,随着多学科综合治疗(multidisciplinary team,MDT)的发展,肺癌的治疗取得了长足的进步。然而,肺癌5年生存率仍徘徊在15%左右。精准医学模式是当今肿瘤治疗的趋势和方向,其关键是利用有效的生物标志物,进行特异性个体化治疗。因此,寻找肺癌新的有效分子标志物以及潜在治疗靶点就显得尤为重要。
随着人类基因组计划的完成,对肺癌在基因水平有了全新的认识。可以认为,在分子基因水平肺鳞癌和肺腺癌是“两种疾病”。现有资料也提示在肺腺癌中可能更易发现生物标志物,并可能从中获益。
精准医学模式的倡导为肺癌的诊治打开了崭新的思路。精准医学是基于分子生物信息学的医学模式,其关键是利用有效的生物标志物,区分目标人群,从而进行特异性个体化治疗。因此,寻找肺癌新的有效的分子生物标志物和信号通路,进一步阐明肺癌的发生发展机制,为肺癌的个体化精准治疗提供新的理论依据成为当今的研究热点和方向。
技术实现要素:
为了弥补现有技术的不足,本发明的目的在于提供一种与肺腺癌发生发展相关的生物标志物。
为了实现上述目的,本发明采用如下技术方案:
本发明提供了一种CLINT1基因的用途,用于制备预防或治疗肺腺癌的药物组合物。
进一步,所述药物组合物包括CLINT1的下调剂。所述下调剂选自:以CLINT1或其转录本为靶序列、且能够抑制CLINT1基因表达或基因转录的干扰分子,包括:shRNA(小发夹RNA)、小干扰RNA(SiRNA)、dsRNA、微小RNA、反义核酸,或能表达或形成所述shRNA、小干扰RNA、dsRNA、微小RNA、反义核酸的构建物;或特异性与CLINT1编码的蛋白结合的结合分子(如能够抑制CLINT1蛋白活性的抗体或配体)。
进一步,所述的下调剂选自下组序列的siRNA:SEQ ID NO.8、SEQ ID NO.9。
本发明提供了一种用于预防或治疗肺腺癌的CLINT1的下调剂,所述下调剂选自:
核酸抑制物,蛋白抑制剂,蛋白水解酶,蛋白结合分子,其能够在蛋白或基因水平上下调CLINT1基因或其编码的蛋白的表达或活性。
在本发明中,所述CLINT1的下调剂还可用于抑制肺腺癌细胞的侵袭和增殖。
本发明提供了一种用于预防或治疗肺腺癌的药物组合物,所述药物组合物含有:
上面所述的CLINT1的下调剂;和
药学上可接受的载体。
本发明提供了一种CLINT1基因的用途,用于筛选预防或治疗肺腺癌的潜在物质。
本发明提供了一种筛选预防或治疗肺腺癌的潜在物质的方法,所述方法包括:
用候选物质处理表达或含有CLINT1基因或其编码的蛋白的体系;和
检测所述体系中CLINT1基因或其编码的蛋白的表达或活性;
其中,若所述候选物质可降低CLINT1基因的表达或活性,(优选显著降低,如低20%以上,较佳的低50%以上;更佳的低80%以上),则表明该候选物质是预防或治疗肺腺癌的潜在物质。所述体系选自:细胞体系、亚细胞体系、溶液体系、组织体系、器官体系或动物体系。
所述候选物质包括但不限于:针对CLINT1基因或其编码的蛋白或其上游或下游基因或蛋白设计的干扰分子、核酸抑制物、结合分子(如抗体或配体)、小分子化合物等。
在本发明中,所述的方法还包括:对获得的潜在物质进行进一步的细胞实验和/或动物试验,以从候选物质中进一步选择和确定对于预防、缓解或治疗肺腺癌有用的物质。
本发明提供了一种CLINT1基因的用途,用于制备诊断肺腺癌的产品。其中,所述产品包括(但不限于)芯片、制剂或试剂盒。
进一步,所述产品包括检测CLINT1基因表达的试剂。
进一步,所述试剂选自:
特异性识别CLINT1的探针;或
特异性扩增CLINT1的引物;或
特异性结合CLINT1编码的蛋白的抗体或配体。
较佳的,所述的特异性扩增CLINT1基因的引物是引物对,序列如SEQ ID NO.3和SEQ ID NO.4所示。
附图说明
图1是利用QPCR检测CLINT1基因在肺腺癌组织中的表达情况图;
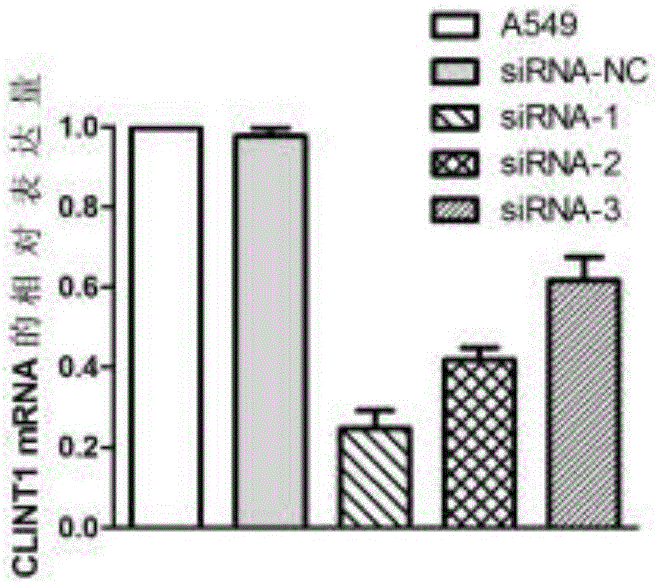
图2是利用QPCR检测CLINT1在肺腺癌细胞中的转染情况图;
图3是用CCK-8法检测CLINT1基因对肺腺癌细胞增殖的影响图;
图4是用流式细胞仪检测CLINT1基因对肺腺癌细胞凋亡的影响图;
图5是利用Transwell小室检测CLINT1对肺腺癌细胞迁移和侵袭的影响图;其中图A是CLINT1对肺腺癌细胞迁移的影响图;图B是CLINT1对肺腺癌细胞侵袭的影响图。
具体的实施方式
本发明经过广泛而深入的研究,通过高通量方法,采用目前覆盖数据库最广的基因芯片,检测肺腺癌标本中基因在肿瘤组织和癌旁组织的表达,发现其中具有明显表达差异的基因片段,探讨其与肺腺癌的发生之间的关系,从而为肺腺癌的早期检测及靶向治疗寻找更好的途径和方法。通过筛选,本发明首次发现了肺腺癌中CLINT1显著性上调。实验证明,通过降低CLINT1的表达水平,能够有效地抑制肺腺癌细胞的生长和侵袭,提示检测CLINT1基因的表达水平可成为肺腺癌早期诊断的辅助诊断指标之一,干扰CLINT1基因表达可成为预防或治疗肺腺癌或肺腺癌转移的新途径。
CLINT1基因
CLINT1是位于人5号染色体长臂3区3带上,一种代表性的人CLINT1基因的核苷酸序列和氨基酸序列如SEQ ID NO.1和SEQ ID NO.2所示。本发明中的CLINT1包括野生型、突变型或其片段。
本发明的人CLINT1核苷酸全长序列或其片段通常可以用PCR扩增法、重组法或人工合成的方法获得。对于PCR扩增法,可根据已公开的有关核苷酸序列,并用市售的cDNA库或按本领域技术人员已知的常规方法所制备的cDNA库作为模板,扩增而得有关序列。当序列较长时,常常需要进行两次或多次PCR扩增,然后再将各次扩增出的片段按正确次序拼接在一起。
本领域技术人员将认识到,本发明的实用性并不局限于对本发明的靶标基因的任何特定变体的基因表达进行定量。如果当核酸或其片段与其它核酸(或其互补链)最佳比对时(具有适当的核苷酸插入或缺失),在至少大约60%的核苷酸碱基、通常至少大约70%、更通常至少大约80%、优选至少大约90%、及更优选至少大约95-98%核苷酸碱基中存在核苷酸序列相同性,则这两个序列是“基本同源的”(或者基本相似的)。
或者,当核酸或其片段与另一核酸(或其互补链)、一条链或其互补序列在选择性杂交条件下杂交时,则其间存在基本同源或(相同性)。当杂交比特异性整体丧失发生更具选择性时,存在杂交选择性。典型地,当在至少大约14个核苷酸的一段序列存在至少大约55%相同性、优选至少大约65%、更优选至少大约75%及最优选至少大约90%相同性时,发生选择性杂交。如本文所述,同源对比的长度可以是较长的序列节段,在某些实施方案中通常为至少大约20个核苷酸,更通常为至少大约24个核苷酸,典型为至少大约28个核苷酸,更典型为至少大约32个核苷酸,及优选至少大约36或更多个核苷酸。
因此,本发明的多核苷酸与SEQ ID NO.1优选具有至少75%、更优选至少85%、更优选至少90%同源性。更优选地,存在至少95%、更优选至少98%同源性。
本发明可以利用本领域内已知的任何方法测定基因表达。本领域技术人员应当理解,测定基因表达的手段不是本发明的重要方面。可以在转录水平上检测生物标志物的表达水平。
下调剂和药物组合物
基于本发明人的发现,本发明提供了一种CLINT1的下调剂的用途,用于制备抑制肺腺癌的药物组合物。如本文所用,所述的CLINT1的下调剂包括但不限于抑制剂、拮抗剂、阻滞剂、阻断剂、核酸抑制物等。
所述的CLINT1基因或蛋白的下调剂是指任何可降低CLINT1蛋白的活性、降低CLINT1基因或蛋白的稳定性、下调CLINT1蛋白的表达、减少CLINT1蛋白有效作用时间、或抑制CLINT1基因的转录和翻译的物质,这些物质均可用于本发明,作为对于下调CLINT1有用的物质,从而可用于预防或治疗肺腺癌。例如,所述的抑制剂是:核酸抑制物,蛋白抑制剂,抗体,配体,蛋白水解酶,蛋白结合分子,只要其能够在蛋白或基因水平上下调CLINT1蛋白或其编码基因的表达。
作为本发明的一种选择方式,所述的CLINT1的下调剂是一种特异性与CLINT1结合的抗体。所述的抗体可以是单克隆抗体或多克隆抗体。可用CLINT1蛋白免疫动物,如家兔,小鼠,大鼠等来生产多克隆抗体;多种佐剂可用于增强免疫反应,包括但不限于弗氏佐剂等。与之相似的,表达CLINT1或其具有抗原性的片段的细胞可用来免疫动物来生产抗体。所述的抗体也可以是单克隆抗体,此类单克隆抗体可以利用杂交瘤技术来制备。抗体的“特异性”是指抗体能结合于CLINT1基因产物或片段。较佳地,指那些能与CLINT1基因产物或片段结合但不识别和结合于其它非相关抗原分子的抗体。本发明的抗体可以通过本领域内技术人员已知的各种技术进行制备。本发明不仅包括完整的单克隆或多克隆抗体,而且还包括具有免疫活性的抗体片段,如Fab’或(Fab)2片段;抗体重链;抗体轻链;遗传工程改造的单链Fv分子;或嵌合抗体。抗CLINT1蛋白的抗体可用于免疫组织化学技术中,检测活检标本中的CLINT1蛋白含量,还可以作为用于预防肝癌转移和侵袭的特异性治疗剂。血样或尿液中的CLINT1蛋白的直接测定可以作为肿瘤的辅助诊断和愈后的观察指标,也可作为肿瘤早期诊断的依据。抗体可以通过ELISA、Western Blot印迹分析,或者与检测基团偶联,通过化学发光、同位素示踪等方法来检测。
作为本发明的一种优选方式,所述CLINT1的下调剂是一种CLINT1特异性的小干扰RNA分子。如本文所用,所述的“小干扰RNA”是指一种短片段双链RNA分子,能够以同源互补序列的mRNA为靶目标降解特定的mRNA,这个过程就是RNA干扰(RNA interference)过程。小干扰RNA可以制备成双链核酸的形式,它含有一个正义链和一个反义链,这两条链仅在杂交的条件下形成双链。一个双链RNA复合物可以由相互分离的正义链和反义链来制备。因此,举例来讲,互补的正义链和反义链是化学合成的,其后可通过退火杂交,产生合成的双链RNA复合物。
在筛选有效的siRNA序列时,本发明人通过大量的比对分析,从而找出最佳的有效片段。本发明人设计合成了多种siRNA序列,并将它们分别通过转染试剂转染肺腺癌细胞系进行验证,选出干扰效果最佳的siRNA,它们分别具有SEQ ID NO.8、SEQ ID NO.9所示的序列,进一步地在细胞水平实验,结果证明对于细胞试验而言抑制效率非常高。
作为本发明的一种可选方式,所述的CLINT1的下调剂也可以是一种“小发夹RNA(Small hairpin RNA,shRNA)”,其是能够形成发夹结构的非编码小RNA分子,小发夹RNA能够通过RNA干扰途径来抑制基因的表达。如上述,shRNA可以由双链DNA模板来表达。双链DNA模板被插进一个载体,例如质粒或病毒载体,然后在体外或体内连接到一个启动子进行表达。shRNA在真核细胞内DICER酶的作用下,可被切割成小干扰RNA分子,从而进入RNAi途径。“shRNA表达载体”是指一些本领域常规用于构建shRNA结构的质粒,通常该质粒上存在“间隔序列”以及位于“间隔序列”两边的多克隆位点或供替换序列,从而人们可以将shRNA(或类似物)相应的DNA序列通过正向和反向的方式插入多克隆位点或替换其上的供替换序列,该DNA序列转录后的RNA可形成shRNA(ShortHairpin)结构。所述的“shRNA表达载体”目前已经完全可以通过商购的途径购买获得,例如一些病毒载体。
本发明的核酸抑制物如siRNA可以化学合成,也可以通过一个重组核酸结构里的表达盒转录成单链RNA之后进行制备。siRNA等核酸抑制物,可通过采用适当的转染试剂被输送到细胞内,或还可采用本领域已知的多种技术被输送到细胞内。
药物组合物
本发明还提供了一种药物组合物,它含有有效量的所述的CLINT1的下调剂,以及药学上可接受的载体。所述的组合物可用于抑制肺腺癌。任何前述的CLINT1的下调剂均可用于组合物的制备。
如本文所用,所述“有效量”是指可对人和/或动物产生功能或活性的且可被人和/或动物所接受的量。下调剂的有效量可随给药的模式和待治疗的疾病的严重程度等而变化。优选的有效量的选择可以由本领域普通技术人员根据各种因素来确定(例如通过临床试验)。所述的因素包括但不限于:所述的CLINT1基因的下调剂的药代动力学参数例如生物利用率、代谢、半衰期等;患者所要治疗的疾病的严重程度、患者的体重、患者的免疫状况、给药的途径等。
所述“药学上可接受的载体”指用于治疗剂给药的载体,包括各种赋形剂和稀释剂。该术语指这样一些药剂载体:它们本身并不是必要的活性成分,且施用后没有过分的毒性。合适的载体是本领域普通技术人员所熟知的。在组合物中药学上可接受的载体可含有液体,如水、盐水、缓冲液。另外,这些载体中还可能存在辅助性的物质,如填充剂、润滑剂、助流剂、润湿剂或乳化剂、pH缓冲物质等。所述的载体中还可以含有细胞(宿主细胞)转染试剂。
本发明可以采用用本领域熟知的多种方法来将所述的下调剂或其编码基因、或其药物组合物给药于哺乳动物。包括但不限于:皮下注射、肌肉注射、经皮给予、局部给予、植入、缓释给予等;优选的,所述给药方式是非肠道给予的。
优选的,可采用基因治疗的手段进行。比如,可直接将CLINT1的下调剂通过诸如注射等方法给药于受试者;或者,可通过一定的途径将携带CLINT1的下调剂的表达单位(比如表达载体或病毒等,或siRNA或shRNA)递送到靶点上,并使之表达活性的CLINT1下调剂,具体情况需视所述的下调剂的类型而定,这些均是本领域技术人员所熟知的。
术语“宿主细胞”可以是原核细胞,如细菌细胞;或是低等真核细胞,如酵母细胞;或是高等真核细胞,如哺乳动物细胞。代表性例子有:大肠杆菌,链霉菌属的细菌细胞;真菌细胞如酵母;植物细胞;果蝇S2或Sf9的昆虫细胞;CHO、COS、或293细胞的动物细胞等。
用重组DNA转化宿主细胞可用本领域技术人员熟知的常规技术进行。当宿主为原核生物如大肠杆菌时,能吸收DNA的感受态细胞可在指数生长期后收获,用CaCl2法处理,所用的步骤在本领域众所周知。另一种方法是使用MgCl2。如果需要,转化也可用电穿孔的方法进行。当宿主是真核生物,可选用如下的DNA转染方法:磷酸钙共沉淀法,常规机械方法如显微注射、电穿孔、脂质体包装等。
本发明中的药物组合物包括CLINT1的下调剂、和/或与所述下调剂配伍的其他药类以及药学上可接受的载体和/或辅料。
本发明的药物组合物还可与其他治疗肺腺癌的药物联用,其他治疗性化合物可以与主要的活性成分同时给药,甚至在同一组合物中同时给药。
本发明的药物组合物还可以以单独的组合物或与主要的活性成分不同的剂量形式单独给予其它治疗性化合物。主要成分的部分剂量可以与其它治疗性化合物同时给药,而其它剂量可以单独给药。在治疗过程中,可以根据症状的严重程度、复发的频率和治疗方案的生理应答,调整本发明药物组合物的剂量。
药物筛选
本发明提供了一种筛选预防或治疗肺腺癌药物的方法,即:
在实验组中,向细胞培养体系中加入待测化合物,并测定CLINT1的表达水平;在对照组中,向同样的培养体系中不加入待测化合物,并测定CLINT1的表达水平;其中,如果实验组中CLINT1的表达水平大于对照组,则说明该候选化合物为CLINT1的下调剂。
在本发明中,所述的方法还包括:对上面步骤获得的候选化合物进一步测试其抑制肺腺癌的效果,若测试化合物对肺腺癌有显著的抑制效果,则说明该化合物为预防或治疗肺腺癌的潜在物质。
芯片、试剂盒
本发明的所述的基因芯片包括:固相载体;以及有序固定在所述固相载体上的寡核苷酸探针,所述的寡核苷酸探针特异性地对应于CLINT1所示的部分或全部序列。
具体地,可根据本发明所述的基因,设计出适合的探针,固定在固相载体上,形成“寡核苷酸阵列”。所述的“寡核苷酸阵列”是指具有可寻址位置(即以区别性的,可访问的地址为特征的位置)的阵列,每个可寻址位置均含有一个与其相连的特征性寡核苷酸。根据需要,可将寡核苷酸阵列分成多个亚阵。
术语“探针”指能与另一分子的特定序列或亚序列或其它部分结合的分子。除非另有指出,术语“探针”通常指能通过互补碱基配对与另一多核苷酸(往往称为“靶多核苷酸”)结合的多核苷酸探针。根据杂交条件的严格性,探针能和与该探针缺乏完全序列互补性的靶多核苷酸结合。探针可作直接或间接的标记,其范围包括引物。杂交方式,包括,但不限于:溶液相、固相、混合相或原位杂交测定法。
本发明中针对CLINT1基因的寡核苷酸探针可以是DNA、RNA、DNA-RNA嵌合体、PNA或其它衍生物。所述探针的长度没有限制,只要完成特异性杂交、与目的核苷酸序列特异性结合,任何长度都可以。所述探针的长度可短至25、20、15、13或10个碱基长度。同样,所述探针的长度可长至60、80、100、150、300个碱基对或更长,甚至整个基因。由于不同的探针长度对杂交效率、信号特异性有不同的影响,所述探针的长度通常至少是14个碱基对,最长一般不超过30个碱基对,与目的核苷酸序列互补的长度以15-25个碱基对最佳。所述探针自身互补序列最好少于4个碱基对,以免影响杂交效率。
本发明中所述固相载体可采用基因芯片领域的各种常用材料,例如包括但不限于塑料制品、微颗粒、膜载体等。所述塑料制品可通过非共价或物理吸附机制与抗体或蛋白抗原相结合,最常用的塑料制品为聚苯乙烯制成的小试管、小珠和微量反应板;所述微颗粒是由高分子单体聚合成的微球或颗粒,其直径多为微米,由于带有能与蛋白质结合的功能团,易与抗体(抗原)形成化学偶联,结合容量大;所述膜载体包括硝酸纤维素膜、玻璃纤维素膜及尼龙膜等微孔滤膜。
所述的CLINT1芯片的制备可采用本领域已知的生物芯片的常规制造方法。例如,如果固相载体采用的是修饰玻片或硅片,探针的5’端含有氨基修饰的聚dT串,可将寡核苷酸探针配制成溶液,然后采用点样仪将其点在修饰玻片或硅片上,排列成预定的序列或阵列,然后通过放置过夜来固定,就可得到本发明的基因芯片。
本发明提供了一种试剂盒,所述试剂盒可用于检测CLINT1基因或蛋白的表达。优选的,所述的制剂或试剂盒中还含有用于标记RNA样品的标记物,以及与所述标记物相对应的底物。此外,所述的试剂盒中还可包括用于提取RNA、PCR、杂交、显色等所需的各种试剂,包括但不限于:抽提液、扩增液、杂交液、酶、对照液、显色液、洗液等。此外,所述的试剂盒中还包括使用说明书和/或芯片图像分析软件。
试剂盒的组分可以以水介质的形式或以冻干的形式来包装。试剂盒中适当的容器通常至少包括一种小瓶、试管、长颈瓶、宝特瓶、针筒或其它容器,其中可放置一种组分,并且优选地,可进行适当地等分。在试剂盒中存在多于一种的组分时,试剂盒中通常也将包含第二、第三或其它附加的容器,其中分离地放置附加的组分。然而,不同组合的组分可被包含在一个小瓶中。本发明的试剂盒通常也将包括一种用于容纳反应物的容器,密封以用于商业销售。这种容器可包括注模或吹模的塑料容器,其中可保留所需的小瓶。
本发明中,术语“样本”以其最广泛的含义使用。在一种含义中,意在包括从任何来源获得的标本或培养物,以及生物和环境样本。生物样本可获自动物(包括人)并涵盖液体、固体、组织和气体。生物样本包括血液制品,诸如血浆、血清等。然而,此类样本不应解释为限制适用于本发明的样本类型。
下面结合附图和实施例对本发明作进一步详细的说明。以下实施例仅用于说明本发明而不用于限制本发明的范围。实施例中未注明具体条件的实验方法,通常按照常规条件,例如Sambrook等人,分子克隆:实验室手册(New York:ColdSpring HarborLaboratory Press,1989)中所述的条件,或按照制造厂商所建议的条件。
实施例1筛选与肺腺癌相关的基因标志物
1、样品收集
各收集8例肺腺癌癌旁组织和肺腺癌组织样本。肺腺癌肿瘤组织标本取材部位为主要肿瘤区域,位于肿瘤肿块中外1/3与正常组织交接处,排除肿瘤中心明显坏死、钙化部分以及肿瘤外围正常肺组织;癌旁正常肺组织标本取自肿瘤边缘5cm以上的部位,肉眼观察无明显变化。上述所有标本的取得均通过组织伦理委员会的同意。
2、RNA样品的制备(利用E.Z.N.A.miRNA kit进行操作)
在研钵中导入液氮,取上述获得的组织放入研钵中,在液氮中剪碎并研磨成粉末,剪碎后投入液氮中并研磨至粉末状,随后转入玻璃匀浆器中;组织匀浆在玻璃匀浆器中加入Trizol试剂,在冰上研磨组织。将匀浆后组织匀浆液转入无RNA酶的EP管中,室温下静置5min。按照试剂盒中的说明书提取分离RNA。具体如下:
1)RNA的分离:
在EP管中加入0.2m1氯仿,盖紧EP管盖,手动剧烈振荡15s,使其充分混匀。室温下孵化5min。然后在4℃下以14000g离心15min。离心后样品分为三层,RNA存在于上层水相中。
2)RNA沉淀
将分离得的水相取450μl移至新的无RNA酶的EP管中,按照1:1的比例加入450μl异丙醇,上下颠倒混匀后在室温下孵化10min,4℃14000g离心10min。
3)RNA洗脱
离心后小心移去上清,加入1ml 75%乙醇(灭酶处理,现用现配并在冰上预冷)冲洗RNA,随后4℃7500g离心5min。
4)RNA再溶解
小心移去洗涤之后的上清,在超净工作台中打开EP管管盖,在室温下放置RNA样本5-10min,晾干。加入无RNA酶处理水20-50μl,55-60℃水浴箱中水浴10min。
5)RNA样品的质量分析
分光光度计检测:
NanoDrop1000分光光度计检测RNA样品,RNA-seq测序的样品要求:OD260/OD280为1.8-2.2。
琼脂糖凝胶电泳检测:
将上述提取的RNA进行琼脂糖凝胶电泳,Agilent Technologies 2100Bioanalyzer检测RNA样品质量,观察28S rRNA和18S rRNA主带明显、无降解、RNA完整性指数合格、浓度达到要求,可以用于芯片的基因表达谱及筛选实验。
3、逆转录和标记
用Low RNA Input Linear Amplification Kit将mRNA逆转录成cDNA,同时用Cy3分别标记实验组和对照组。
4、杂交
基因芯片采用Aglient公司的人全基因组表达谱芯片。按芯片使用说明书的步骤进行。
5、数据分析
利用Agilent GeneSpring软件对芯片结果进行分析,筛选表达量具有显著性差异(标准为该基因在癌与癌旁的表达量相差2倍以上,而且p<0.05)的基因。
6、结果
结果显示,CLINT1在肺腺癌组织中的表达量显著高于癌旁组织中的表达量。
实施例2QPCR测序验证CLINT1基因的差异表达
1、对CLINT1基因差异表达进行大样本QPCR验证。按照实施例1中的样本收集方式选择肺腺癌癌旁组织和肺腺癌组织各50例。
2、RNA提取步骤如实施例1所述。
3、逆转录
1)反应体系:
RNA模板 1μl,随机引物1μl,双蒸水加至12μl,混匀,低转速离心,65℃5min,然后放在冰上冷却。
继续在12μl反应液中加入下列成分:
5×反应缓冲液4μl,RNA酶抑制剂(20U/μl)1μl,10mM dNTP混合液 2μl,AMV反转录酶(200U/μl)1μl;充分混匀并进行离心处理;
2)逆转录反应条件
25℃5min,42℃60min,70℃5min。
3)聚合酶链反应
引物设计:
根据Genebank中CLINT1基因和GAPDH基因的编码序列设计QPCR扩增引物,由博迈德生物公司合成。具体引物序列如下:
CLINT1基因:
正向引物为5’-TCTATGATGAGCACTAAC-3’(SEQ ID NO.3);
反向引物为5’-GGACCATATCTGTATTCT-3’(SEQ ID NO.4)。
GAPDH基因:
正向引物为5’-AATCCCATCACCATCTTCCAG-3’(SEQ ID NO.5);
反向引物为5’-GAGCCCCAGCCTTCTCCAT-3’(SEQ ID NO.6)。
配制PCR反应体系:
2×qPCR混合液 12.5μl,基因引物2.0μl,反转录产物2.5μl,ddH2O 8.0μl。
PCR反应条件:95℃10min,(95℃15s,60℃60s)×40个循环,60℃5min延伸反应。75℃至95℃,每20s升温1℃,绘制溶解曲线。以SYBR Green作为荧光标记物,在Light Cycler荧光定量PCR仪上进行PCR反应,通过融解曲线分析和电泳确定目的条带,ΔΔCT法进行相对定量。
5、统计学方法
实验都是按照重复3次来完成的,结果数据都是以平均值±标准差的方式来表示,采用SPSS18.0统计软件来进行统计分析的,癌与癌旁组织的配对比较采用t检验,认为当P<0.05时具有统计学意义。
6、结果
结果如图1所示,与肺腺癌癌旁组织相比,CLINT1在肺腺癌组织中表达上调,差异具有统计学意义(P<0.05),同芯片检测结果一致。
实施例3CLINT1基因的沉默
1、细胞培养
人肺腺癌细胞株A549,以含10%胎牛血清和1%P/S的RPMI1640培养基在37℃、5%CO2、相对湿度为90%的培养箱中培养。2-3天换液1次,使用0.25%含EDTA的胰蛋白酶常规消化传代。
将培养瓶中的细胞用胰酶进行消化并接种在6孔板中,保证细胞数为2-8×105个/孔,加入细胞培养基。过夜,第二天观察细胞密度,细胞密度为70%以上可进行转染。
2、siRNA的设计
阴性对照siRNA序列(siRNA-NC):
正义链为5’-UUCUCCGAACGUGUCACGU-3’(SEQ ID NO.7)
反义链为5’-ACGUGACACGUUCGGAGAA-3’(SEQ ID NO.8)
siRNA-1:
正义链为5’-UUAUCACUGAAUGGAAAAGCA-3’(SEQ ID NO.9)
反义链为5’-CUUUUCCAUUCAGUGAUAAAU-3’(SEQ ID NO.10)
siRNA-2:
正义链为5’-UCCAAUUCUUUUUGUUGUCUU-3’(SEQ ID NO.11)
反义链为5’-GACAACAAAAAGAAUUGGAGA-3’(SEQ ID NO.12)
siRNA-3:
正义链为5’-AUACUUGUCUUUGUUCUUCUU-3’(SEQ ID NO.13)
反义链为5’-GAAGAACAAAGACAAGUAUGU-3’(SEQ ID NO.14)
3、转染
将实验分为三组:对照组(A549)、阴性对照组(siRNA-NC)和实验组(siRNA1、siRNA2、siRNA-3),其中阴性对照组siRNA与CLINT1基因的序列无同源性,浓度为20nM/孔,同时分别进行转染。
4、QPCR检测CLINT1基因的转录水平
4.1细胞总RNA的提取
1)将6孔板中的细胞培养液倒掉,用PBS冲洗两次,各孔加入1ml Trizol试剂,室温放置5min。
2)加入0.2m1氯仿,剧烈震荡15s,4℃,12000g离心15min。
3)水相转移到新管中,加入4.5m1异丙醇,室温放置10min;4℃,10000g离心10min。
4)倒掉液体,用l ml的75%乙醇洗EP管壁。4℃,7500g,离心5min。
5)倒掉清洗后的75%乙醇,室温晾置5-10min。
6)加入25μl无RNA酶的DEPC水,-70℃保存。
4.2逆转录步骤同实施例2。
4.3QPCR扩增步骤同实施例2。
5、统计学方法
实验都是按照重复3次来完成的,结果数据都是以平均值±标准差的方式来表示,采用SPSS18.0统计软件来进行统计分析的,CLINT1基因实验组与对照组之间的差异采用t检验,认为当P<0.05时具有统计学意义。
6、结果
结果如图2显示,与非转染组与转染siRNA-NC组相比,实验组中的CLINT1的表达水平显著降低,差异具有统计学意义(P<0.05)。
实施例4CLINT1基因对肺腺癌细胞增殖的影响
采用CCK-8实验检测CLINT1基因对肺腺癌细胞增殖能力影响。
1、细胞培养与转染步骤同实施例3,转染后6h换液,放置细胞培养箱过夜。
2、第二天将细胞取出,显微镜下观察细胞生长情况,1ml/孔加入含EDTA的胰酶,进行细胞消化,待消化完成后去除胰酶,加入细胞培养基混匀使细胞悬浮,然后进行细胞计数。
3、将细胞悬液浓度稀释为15000个/ml,之后往96孔板中进行接种,每孔加入细胞悬液200μl,细胞控制在3000个左右,接种8个复孔。设置siRNA-1实验组和siRNA-NC对照组。共铺4块96孔板分别用于24h、48h、72h、96h 4个检测时间点。
4、24h后,将第一块96孔板取出,每孔中加入10μl的CCK-8检测液,将96孔板继续放入细胞培养箱中孵育4h左右,用酶标仪检测各孔在450nm波长处的吸光度值并记录数据。
5、在48h、72h、96h后分别重复步骤4中的操作,最后统计出各时间点的吸光度值,作出生长曲线图。
6、统计学分析
实验都是按照重复3次来完成的,采用SPSS18.0统计软件来进行统计分析,两者之间的差异采用t检验,认为当P<0.05时具有统计学意义。
7、结果
结果如图3所示,与对照相比,实验组在转染siRNA-1后,细胞的增殖明显受到了抑制,差异具有统计学意义(P<0.05)说明CLINT1具有促进细胞增殖的作用。
实施例5CLINT1基因对肺腺癌细胞凋亡的影响
使用流式细胞仪检测CLINT1基因对细胞凋亡的影响。
1、细胞培养步骤同实施例3。
2、细胞转染步骤同实施例3。
3、步骤
1)将3m1 10×上样缓冲液用27m1蒸馏水稀释。
2)收集细胞样本并用预冷的PBS清洗。
3)将细胞加入lml 1×上样缓冲液,300g离心10min,吸出缓冲液。
4)再次加入lml 1×上样缓冲液,将细胞悬液中细胞浓度调整为1×106个/ml。
5)将细胞悬液取出100μl,加入EP管中。
6)将5μl的Annexin V FITC加入EP管中,混匀EP管中的液体,在室温下避光孵育10min。
7)向EP管中加入5μl PI染液,在室温下避光5min。
8)在EP管中加入500μl的PBS溶液,轻轻混匀,1h内上流式细胞仪进行检测。
3、统计学方法
实验都是按照重复3次来完成的,结果数据都是以平均值±标准差的方式来表示,采用SPSS18.0统计软件来进行统计分析的,两者之间的差异采用的t检验,认为当P<0.05时具有统计学意义。
4、结果:
结果如图4所示,实验组与对照组相比,细胞凋亡率显著升高(P<0.05),该结果说明,CLINT1的过表达抑制肺腺癌细胞的凋亡。
实施例6细胞迁移及侵袭实验
1、Transwell小室制备
无菌条件下Matrigel冰浴融化,用PBS进行20倍稀释,以50μl/孔的体积铺在Transwell小室的聚碳酸酯膜上。37℃放置4h,待Matrigel胶聚合成凝胶后取出,轻轻吸出上层析出液。每孔中加入50μl的含BSA的无血清培养液对基底膜进行水化处理,37℃放置30min。
2、配置细胞悬液
细胞撤血清饥饿处理12-24h,对细胞进行消化处理,终止消化后进行离心,去除上层培养液。用PBS对沉淀细胞进行清洗,加入含有BSA的无血清培养基对其进行重悬。调整细胞的密度至5×l05个/ml。
3、细胞接种
取细胞悬液200μl(迁移实验为100μl,侵袭实验为200μl)加入到Transwell小室中。在24孔板下室加入500μl含FBS的1640培养基。把细胞放入细胞培养箱中培养24h。
4、染色
细胞在培养结束后使用DAPI染色。把小室细胞先用PBS漂洗2遍,放入DAPI工作液中室温染色5-20min。用PBS漂洗2遍,放入荧光显微镜下观察并计数。
5、结果
结果如图5所示,在肺腺癌细胞转染干扰RNA之后,与对照组相比,实验组的迁移及侵袭能力均有明显下降,结果说明CLINT1能够促进肺腺癌的迁移及侵袭。
上述实施例的说明只是用于理解本发明的方法及其核心思想。应当指出,对于本领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以对本发明进行若干改进和修饰,这些改进和修饰也将落入本发明权利要求的保护范围内。
SEQUENCE LISTING
<110> 北京泱深生物信息技术有限公司
<120> 生物标志物作为靶标在肺腺癌诊治中的应用
<160> 14
<170> PatentIn version 3.5
<210> 1
<211> 1932
<212> DNA
<213> 人源
<400> 1
atgttgaaca tgtggaaggt gcgcgagctg gtggacaaag ccaccaatgt tgttatgaat 60
tattcagaga tcgagtctaa ggttcgagag gcaacgaacg atgatccttg gggaccttct 120
gggcaactca tgggagagat tgccaaggct acatttatgt atgaacaatt tccagaactt 180
atgaacatgc tttggtcacg aatgttaaaa gacaacaaaa agaattggag aagagtttat 240
aagtcgttgc tgctcctagc ttacctcata aggaatggat cagagcgtgt tgttacaagt 300
gccagagaac acatttatga tttacgatcc ctggaaaatt accactttgt agatgagcat 360
ggtaaggatc aaggtataaa tattcgacag aaggtgaagg aattggttga atttgcccag 420
gatgacgaca ggcttcgtga agagcgaaag aaagcaaaga agaacaaaga caagtatgtt 480
ggggtttcct cagacagtgt tggaggattc agatacagtg aaagatatga tcctgagccc 540
aaatcaaaat gggatgagga gtgggataaa aacaagagtg cttttccatt cagtgataaa 600
ttaggtgagc tgagtgataa aattggaagc acaattgatg acaccatcag caagttccgg 660
aggaaagata gagaagactc tccagaaaga tgcagcgaca gcgatgagga aaagaaagcg 720
agaagaggca gatctcccaa aggtgaattc aaagatgaag aggagactgt gacgacaaag 780
catattcata tcacacaggc cacagagacc accacaacca gacacaagcg cacagcaaat 840
ccttccaaaa ccattgatct tggagcagca gcacattaca caggggacaa agcaagtcca 900
gatcagaatg cttcaaccca cacacctcag tcttcagtta agacttcagt gcctagcagc 960
aagtcatctg gtgaccttgt tgatctgttt gatggcacca gccagtcaac aggaggatca 1020
gctgatttat tcggaggatt tgctgacttt ggctcagctg ctgcatcagg cagtttccct 1080
tcccaagtaa cagcaacaag tgggaatgga gactttggtg actggagtgc cttcaaccaa 1140
gccccatcag gccctgttgc ttccagtggc gagttctttg gcagtgcctc acagccagcg 1200
gtagaacttg ttagtggctc acaatcagct ctaggcccac ctcctgctgc ctcaaattct 1260
tcagacctgt ttgatcttat gggctcgtcc caggcaacca tgacatcttc ccagagtatg 1320
aatttctcta tgatgagcac taacactgtg ggacttggtt tgcctatgtc aagatcacag 1380
cctttgcaaa atgttagcac agtgctgcag aagcctaatc ctctctataa tcagaataca 1440
gatatggtcc agaaatcagt cagcaaaacc ttgccctcta cttggtctga ccccagtgta 1500
aacatcagcc tagacaactt actacctggt atgcagcctt ccaaacccca gcagccatca 1560
ctgaatacaa tgattcagca acagaatatg cagcagccta tgaatgtgat gactcaaagt 1620
tttggagctg tgaacctcag ttctccatcg aacatgcttc ctgtccggcc ccaaactaat 1680
gctttgatag ggggacccat gcctatgagc atgcccaatg tgatgactgg caccatggga 1740
atggcccctc ttggaaatac tccgatgatg aaccagagca tgatgggcat gaacatgaac 1800
atagggatgt ccgctgctgg gatgggcttg acaggcacaa tgggaatggg catgcccaac 1860
atagccatga cttctggaac tgtgcaaccc aagcaagatg cctttgcaaa tttcgccaat 1920
tttagcaaat aa 1932
<210> 2
<211> 643
<212> PRT
<213> 人源
<400> 2
Met Leu Asn Met Trp Lys Val Arg Glu Leu Val Asp Lys Ala Thr Asn
1 5 10 15
Val Val Met Asn Tyr Ser Glu Ile Glu Ser Lys Val Arg Glu Ala Thr
20 25 30
Asn Asp Asp Pro Trp Gly Pro Ser Gly Gln Leu Met Gly Glu Ile Ala
35 40 45
Lys Ala Thr Phe Met Tyr Glu Gln Phe Pro Glu Leu Met Asn Met Leu
50 55 60
Trp Ser Arg Met Leu Lys Asp Asn Lys Lys Asn Trp Arg Arg Val Tyr
65 70 75 80
Lys Ser Leu Leu Leu Leu Ala Tyr Leu Ile Arg Asn Gly Ser Glu Arg
85 90 95
Val Val Thr Ser Ala Arg Glu His Ile Tyr Asp Leu Arg Ser Leu Glu
100 105 110
Asn Tyr His Phe Val Asp Glu His Gly Lys Asp Gln Gly Ile Asn Ile
115 120 125
Arg Gln Lys Val Lys Glu Leu Val Glu Phe Ala Gln Asp Asp Asp Arg
130 135 140
Leu Arg Glu Glu Arg Lys Lys Ala Lys Lys Asn Lys Asp Lys Tyr Val
145 150 155 160
Gly Val Ser Ser Asp Ser Val Gly Gly Phe Arg Tyr Ser Glu Arg Tyr
165 170 175
Asp Pro Glu Pro Lys Ser Lys Trp Asp Glu Glu Trp Asp Lys Asn Lys
180 185 190
Ser Ala Phe Pro Phe Ser Asp Lys Leu Gly Glu Leu Ser Asp Lys Ile
195 200 205
Gly Ser Thr Ile Asp Asp Thr Ile Ser Lys Phe Arg Arg Lys Asp Arg
210 215 220
Glu Asp Ser Pro Glu Arg Cys Ser Asp Ser Asp Glu Glu Lys Lys Ala
225 230 235 240
Arg Arg Gly Arg Ser Pro Lys Gly Glu Phe Lys Asp Glu Glu Glu Thr
245 250 255
Val Thr Thr Lys His Ile His Ile Thr Gln Ala Thr Glu Thr Thr Thr
260 265 270
Thr Arg His Lys Arg Thr Ala Asn Pro Ser Lys Thr Ile Asp Leu Gly
275 280 285
Ala Ala Ala His Tyr Thr Gly Asp Lys Ala Ser Pro Asp Gln Asn Ala
290 295 300
Ser Thr His Thr Pro Gln Ser Ser Val Lys Thr Ser Val Pro Ser Ser
305 310 315 320
Lys Ser Ser Gly Asp Leu Val Asp Leu Phe Asp Gly Thr Ser Gln Ser
325 330 335
Thr Gly Gly Ser Ala Asp Leu Phe Gly Gly Phe Ala Asp Phe Gly Ser
340 345 350
Ala Ala Ala Ser Gly Ser Phe Pro Ser Gln Val Thr Ala Thr Ser Gly
355 360 365
Asn Gly Asp Phe Gly Asp Trp Ser Ala Phe Asn Gln Ala Pro Ser Gly
370 375 380
Pro Val Ala Ser Ser Gly Glu Phe Phe Gly Ser Ala Ser Gln Pro Ala
385 390 395 400
Val Glu Leu Val Ser Gly Ser Gln Ser Ala Leu Gly Pro Pro Pro Ala
405 410 415
Ala Ser Asn Ser Ser Asp Leu Phe Asp Leu Met Gly Ser Ser Gln Ala
420 425 430
Thr Met Thr Ser Ser Gln Ser Met Asn Phe Ser Met Met Ser Thr Asn
435 440 445
Thr Val Gly Leu Gly Leu Pro Met Ser Arg Ser Gln Pro Leu Gln Asn
450 455 460
Val Ser Thr Val Leu Gln Lys Pro Asn Pro Leu Tyr Asn Gln Asn Thr
465 470 475 480
Asp Met Val Gln Lys Ser Val Ser Lys Thr Leu Pro Ser Thr Trp Ser
485 490 495
Asp Pro Ser Val Asn Ile Ser Leu Asp Asn Leu Leu Pro Gly Met Gln
500 505 510
Pro Ser Lys Pro Gln Gln Pro Ser Leu Asn Thr Met Ile Gln Gln Gln
515 520 525
Asn Met Gln Gln Pro Met Asn Val Met Thr Gln Ser Phe Gly Ala Val
530 535 540
Asn Leu Ser Ser Pro Ser Asn Met Leu Pro Val Arg Pro Gln Thr Asn
545 550 555 560
Ala Leu Ile Gly Gly Pro Met Pro Met Ser Met Pro Asn Val Met Thr
565 570 575
Gly Thr Met Gly Met Ala Pro Leu Gly Asn Thr Pro Met Met Asn Gln
580 585 590
Ser Met Met Gly Met Asn Met Asn Ile Gly Met Ser Ala Ala Gly Met
595 600 605
Gly Leu Thr Gly Thr Met Gly Met Gly Met Pro Asn Ile Ala Met Thr
610 615 620
Ser Gly Thr Val Gln Pro Lys Gln Asp Ala Phe Ala Asn Phe Ala Asn
625 630 635 640
Phe Ser Lys
<210> 3
<211> 18
<212> DNA
<213> 人工序列
<400> 3
tctatgatga gcactaac 18
<210> 4
<211> 18
<212> DNA
<213> 人工序列
<400> 4
ggaccatatc tgtattct 18
<210> 5
<211> 21
<212> DNA
<213> 人工序列
<400> 5
aatcccatca ccatcttcca g 21
<210> 6
<211> 19
<212> DNA
<213> 人工序列
<400> 6
gagccccagc cttctccat 19
<210> 7
<211> 19
<212> RNA
<213> 人工序列
<400> 7
uucuccgaac gugucacgu 19
<210> 8
<211> 19
<212> RNA
<213> 人工序列
<400> 8
acgugacacg uucggagaa 19
<210> 9
<211> 21
<212> RNA
<213> 人工序列
<400> 9
uuaucacuga auggaaaagc a 21
<210> 10
<211> 21
<212> RNA
<213> 人工序列
<400> 10
cuuuuccauu cagugauaaa u 21
<210> 11
<211> 21
<212> RNA
<213> 人工序列
<400> 11
uccaauucuu uuuguugucu u 21
<210> 12
<211> 21
<212> RNA
<213> 人工序列
<400> 12
gacaacaaaa agaauuggag a 21
<210> 13
<211> 21
<212> RNA
<213> 人工序列
<400> 13
auacuugucu uuguucuucu u 21
<210> 14
<211> 21
<212> RNA
<213> 人工序列
<400> 14
gaagaacaaa gacaaguaug u 21
- 还没有人留言评论。精彩留言会获得点赞!